HAL Id: tel-02503204
https://tel.archives-ouvertes.fr/tel-02503204
Submitted on 9 Mar 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Étude comparative des peptides antimicrobiens et des
peptides pénétrants
Didier Cheng
To cite this version:
Didier Cheng. Étude comparative des peptides antimicrobiens et des peptides pénétrants. Chimie organique. Sorbonne Université, 2018. Français. �NNT : 2018SORUS294�. �tel-02503204�
1
THESE DE DOCTORAT DE
L’UNIVERSITE PIERRE ET MARIE CURIE
Spécialité Chimie Bio-organiqueEcole doctorale Chimie Moléculaire
Présentée par
Didier Cheng
Pour obtenir le grade de
DOCTEUR de l’UNIVERSITÉ PIERRE ET MARIE CURIE
Sujet de la thèse :Étude comparative des peptides antimicrobiens
et des peptides pénétrants
soutenue le 9 octobre 2018 devant le jury composé de :
Mme Claire Lacombe Directrice de thèse
Mme Isabel Alves Rapporteure
Mme Véronique Vié Rapporteure M. Sébastien Deshayes Examinateur M. Thierry Foulon Examinateur Mme Séverine Zirah Examinatrice
3
Remerciements
La thèse présentée dans ce manuscrit a été réalisée au Laboratoire des Biomolécules (LBM) à l’université de Pierre et Marie Curie, et a été financée par une bourse de l’université Pierre et Marie Curie.
Je tiens tout d'abord à remercier les membres de mon jury, le Dr. Isabel Alves, et le Dr. Véronique Vié qui ont accepté d’évaluer ce travail en qualité de rapporteures. Je remercie également le Dr. Sébastien Deshayes, le Pr. Thierry Foulon et le Dr. Séverine Zirah pour avoir accepté d’être membres du jury en tant qu’examinateurs. Je tiens à tous les remercier pour l’attention qu’ils ont porté à la lecture de mon manuscrit et à ma présentation orale lors de la soutenance de thèse, ainsi que pour la qualité de la discussion que nous avons pu avoir à la suite de cette soutenance.
Je souhaite remercier le Dr Sandrine Sagan pour m’avoir accueilli dans son laboratoire, pour sa gentillesse et pour tout le précieux temps qu’elle a pu m’accorder au cours de ces trois années.
Je ne trouverai sans doute pas de mots ou d’expressions assez forts pour exprimer ma reconnaissance envers Claire Lacombe, qui a été une directrice de thèse exceptionnelle. Je tiens à la remercier de m’avoir offert l’opportunité de faire cette thèse sous son encadrement, et pour sa gentillesse et pour sa patience tout au long de ces trois années. Malgré mes nombreux moments de doutes, elle aura toujours su me guider et me redonner confiance quelque soit la difficulté rencontrée avec un dynamisme extraordianaire que j’espère pouvoir un jour égaler et retransmettre à mon tour.
Je souhaite dire un grand merci à toutes les personnes au laboratoire sans lesquelles cette thèse n’aurait pas vu le jour.
Je remercie Fabienne Burlina pour sa patience et tous ses conseils sur les peptides et leurs synthèses ainsi que pour toutes nos conversations tardives malgré ton emploi du temps très chargé, je n’aurais sans doute pas réussi à obtenir nombreux des peptides dont je parle dans cette thèse sans son aide. Je lui souhaite beaucoup de réussite à elle et à son Equipe 2.
4
De la même manière, je tiens à remercier Astrid Walrant pour sa gentillesse tous ses conseils aussi bien en laboratoire que pour l’organisation de la vie au laboratoire. Je lui souhaite le meilleur à elle et à la petite Judith.
Je remercie Rodrigue Marquant pour sa disponibilité et pour tout le temps qu’il m’a accordé (surtout considérant la quantité incroyable de travail qu’il a au laboratoire !) pour m’avoir montré tous les appareils et nombre de techniques utilisés au laboratoire. Je lui souhaite plein de courage pour la suite et pour les chanceux petits nouveaux arrivants au laboratoire qu’il rencontrera.
Un grand merci à Thierry Drujon, malgré ton habituel humour sarcastique qui n’a jamais manqué à me mettre de bonne humeur, tu as réussi à me transmettre une motivation à travailler avec des bactéries sans pareille, jusqu’à en réaliser des tests antimicrobiens pendant plusieurs heures dans une salle à 40 °C les jours d’été. Quoiqu’il se passe à l’avenir, j’espère qu’il ne changera jamais et que tout marchera pour le mieux pour lui.
Encore à ce jour, je ne crois toujours pas que Françoise Illien est timide malgré notre première conversation. Sans compter ce détail, je crois dur comme fer à tout ce qu’elle m’a dit aussi bien sur les cellules qu’en dehors. Moi, qui ne connaissait pas grand-chose sur les cellules un an avant de la rencontrer, elle m’a appris tant de choses sur ces dernières et sur les techniques les utilisant, et ce toujours avec la bonne humeur qui la caractérise. Pour m’avoir tant aidé, je lui en serais toujours reconnaissant et je lui souhaite une réussite pour l’avenir.
Mis à part ma directrice de thèse, je ne pense pas qu’il y ait eu une autre personne que j’ai autant sollicité : Delphine Ravault. Je m’excuse si je l’ai ennuyé à commander des cultures de cellules toutes les semaines ces dernières années mais, plus que les excuses, je tiens à la remercier pour m’avoir toujours dit « oui » avec son grand sourire à toutes ces demandes. J’espère tout le meilleur pour elle. (PS : Merci éfalement pour les livraisons de café)
Je remercie toute les personnes qui m’ont accueilli et tant aidé à la plate-forme de spectrométrie de masse : Emmanuelle Sachon pour sa gaiété et ses conseils pour que je puisse maintenant réaliser des dépôts sur la plaque que les personnes ne peuvent qualifier de « moches », Gérard Bolbach pour sa patience, sa pédagogie et toutes les solutions qu’il a apporté pour que je puisse enfin voir mes petits peptides internalisés, Gilles Clodic qui a toujours été là pour m’accueillir à la plate-forme et surtout toujours été là pour m’y aider à chaque fois, et enfin Lucrèce Matheron qui a toujours su me guider quand je tirais dans le vide. À eux tous, un grand merci et une excellente continuation.
5
Je tiens à remercier Vincent Humblot du Laboratoire de Réactivité de Surface pour son dynamisme, son précieux temps et son aide pour l’étude structurale des peptides. J’espère sincèrement que cette collaboration apportera un résultat fructueux et que tout se déroulera pour le mieux pour lui de son côté.
Il y a encore tant de permanents que j’aimerais remercier : Dominique Guianvarc’h pour ses conseils pour les présentations orales et à qui je souhaite beaucoup de réussite à Orsay, Sophie Cribier qui est dans doute la personne la plus cultivée que je connaisse, Nicolas Rodriguez avec qui j’ai pu partagé plusieurs de mes meilleures conversations et pizza au long de ces trois années… et encore tant d’autres !
Je manquerai de mots pour exprimer les remerciements que j’exprime pour ceux qui ont partagés avec moi les joies et les difficultés, et qui ont rendu ces trois années extraordinaires et inoubliables : les doctorants du LBM !
Tout d’abord, Sébastien Cardon et Frédéric Thiebaut, vous êtes les meilleurs, mes modèles et réellement les visages de ce bureau 424 dans lequel j’ai passé tant de merveilleux moments. Bien que je ne les ai pas cotoyé aussi longtemps que je l’aurais souhaité mais je tenais quand même à les remercier : Mehdi Amoura, Pierre Soule, Anais Terrien et Lucile Senicourt. Cela aura été court mais j’ai beaucoup apprécié le temps et tous les repas qu’on aura partagé.
Merci à Mathilde Belnou, la meilleure patissière que j’ai connu et celle avec qui j’ai synthétisé mon premier peptide (même si ça a finalement été de juste me placer devant la balance et de confier le reste à Rodrigue), et à Aude Sadet pour ta patience légendaire qui m’avait toujours inspiré.
Un grand merci à ceux qui auront partagé cette aventure de trois ans à mes côtés : Simon Kulifaj toujours franc et dynamique avec qui il est impossible de ne pas passé un bon moment, Claudia Almeida la plus belle doctorante de tous les temps, Benjamin Zagiel avec qui j’ai pris beaucoup de plaisir à organiser la Fête de la Science, et Damien Samson avec qui j’ai toujours pris plaisir pour discuter (en dehors du sujet de la vaisselle à la salle bacterio du moins). Merci à ceux ou plutôt celles qui sont arrivées ensuite : Leïla Bechtella, Bingwei He et Shadai Salazar, merci pour votre aide à la Fête de la Science (dès votre arrivée !) et avec qui j’ai pu partagé beaucoup de bons moment (de grignotages entre autres) au laboratoire.
Je remercie et surtout je souhaite un bon courage aux « petits » nouveaux Pauline Gehan (littéralement) qui m’a appris à faire des bulles avec mes peptides et Alexandre Desert (relatif !)
6
avec qui j’ai pu partager ma passion du sucre sans limite, ainsi qu’aux nouvelles Nathalie Dhayer et Mathilde Le Jeune à qui je souhaite de passer autant de bons moments que j’ai pu passé ces dernières années.
Je souhaite dire un grand merci aux stagiaires qui ont pu partagé un petit moment de ma thèse avec moi et à qui j’espère les avoir faits passé un bon moment au laboratoire malgré tout : Estelle Serre qui a fait un travail incroyable en un petit mois, Bryan Benainous qui a supporté à côté les tests antibactériens dans le « sauna », Célia Boveyron qui était là lorsque mon projet a pris un nouvel élan et Kangou Fofana qui a assuré plusieurs expériences pendant ma rédaction.
Enfin, je souhaite remercier ma famille, mes amis et mes proches pour avoir toujours été à mes côtés même dans les moments les plus difficiles.
Merci à ma grand-mère qui m’a donné la passion des sciences, à ma mère qui m’a poussé dans cette voie quand j’avais perdu espoir, à ma petite sœur qui m’a toujours soutenu et à ma petite rosette qui poursuit mes pas.
J’arrive à la fin de mes remerciements et il y a encore tant de personnes que je n’ai pas encore mentionné alors encore une fois (et pas la dernière) : un grand merci à toutes les personnes extraordinaires que j’ai rencontré lors de ces trois dernières années : membres de l’Équipe 2, membres du Laboratoires des Biomolécules, famille, amis, MERCI !
8
Étude comparative des peptides antimicrobiens et des peptides
pénétrants
Résumé : Les peptides à visée thérapeutique suscitent une attention croissante auprès des
industries pharmaceutiques en raison de leur forte activité biologique. Parmi ces peptides, les peptides antimicrobiens (AMP) peuvent éliminer une grande gamme de microorganismes tels que les bactéries et les champignons pathogènes en perturbant leur membrane tout en ne provoquant qu’une résistance bactérienne limitée contrairement aux antibiotiques couramment utilisés. D’autre part, les peptides vecteurs (CPP) se distinguent par leur capacité à traverser les membranes cellulaires eucaryotes sans les endommager. Cette particularité peut être exploitée pour l’internalisation intracellulaire de molécules bioactives. Malgré leurs différences, les AMP et les CPP sont des peptides membranotropes qui partagent de nombreuses similitudes telles que la petite taille, la présence d’acides aminés chargés positivement et les structures secondaires qu’ils peuvent adopter, telle que la structure en hélice α.
Le projet décrit ici propose de déterminer quels sont les paramètres qui peuvent conférer une activité antimicrobienne ou pénétrante à une séquence peptidique. À cet effet, des séquences inspirées d’un AMP naturel et modifiées par substitution d’acide(s) aminé(s) afin de promouvoir la capacité à pénétrer les membranes eucaryotes ont été établies et synthétisées pour en étudier l’activité antimicrobienne et la pénétration dans les cellules. Les propriétés physico-chimiques de ces peptides (taille, charge globale, hydrophobie …) ont alors été modifiées afin d’étudier l’importance de ces paramètres pour chacune des activités.
Mots clés : Peptides antimicrobiens, peptides pénétrants, peptides riches en tryptophanes,
structure secondaire, extrémité N-terminale, cytotoxicité
Laboratoire des Biomolécules
4, place Jussieu
10
Table des matières
Remerciements ..………..3
Chapitre I – Les peptides membranotropes ...…...31
1. Historique et généralités des peptides membranotropes ... 31
1.1 Les peptides antimicrobiens ... 31
1.1.1. Généralités sur les peptides antimicrobiens ... 31
1.1.2. Les défensines ... 34
1.1.3. Les dermaseptines ... 37
1.2 Les peptides pénétrants ... 39
1.2.1. Généralités sur les peptides pénétrants ... 39
1.2.2. Peptides dérivés de domaines protéiques ... 42
1.2.3. Peptides chimériques ... 43
1.2.4. Peptides synthétiques ou bio-inspirés ... 44
2. Les cibles des peptides membranotropes ... 45
2.1. Les principales cibles des peptides antimicrobiens ... 45
2.1.2 Les bactéries à Gram positif ... 52
2.1.1 Les bactéries à Gram négatif ... 54
2.2. La membrane plasmique des cellules eucaryotes ... 59
3. Modes d’action des peptides membranotropes ... 64
3.1. Modes d’action des peptides antimicrobiens ... 65
3.1.1. Modèle du pore en tonneau ... 66
3.1.2. Modèle du pore mixte ... 67
3.1.3. Modèle du tapis ... 67
3.1.4. Autres mécanismes de perméabilisation membranaire ... 67
3.1.5. Interaction avec des cibles intracellulaires ... 68
3.2. Internalisation des peptides pénétrants ... 69
3.2.1 Interaction des peptides pénétrants avec la membrane plasmique ... 69
3.2.2. Translocation directe ... 71
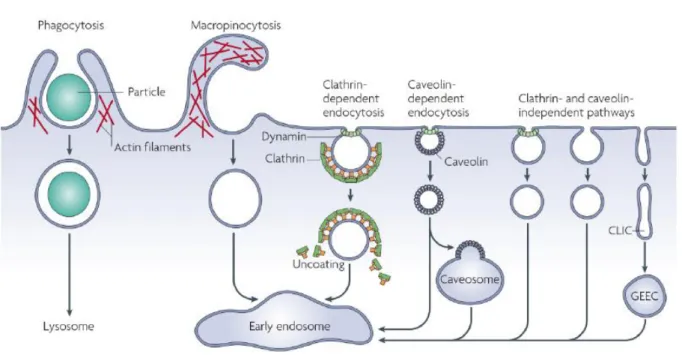
3.2.3. Endocytose ... 73
11
Chapitre II – Prédictions et nouvelles séquences peptidiques : quels effets pour
l'activité antimicrobienne ? ...81
1. Séquence peptidique de départ et établissement de nouvelles séquences peptidiques par prédiction d’activité : passage d’une activité antimicrobienne à une activité pénétrante ? ... 81
2. Taille des peptides et activité antimicrobienne ... 90
2.1. Peptides à dix acides aminés : une taille minimale ? ... 90
2.2. Élongation des peptides : étude de peptides à quinze acides aminés ... 93
2.3. Influence du nombre d’acides aminés : pair ou impair ? ... 96
3. Importance de l’hydrophobie dans l’activité antibactérienne ... 102
3.1. Premières observations sur l’effet de l’hydrophobie ... 102
3.2. Effet de l’incorporation de tryptophanes dans les séquences ... 108
3.3 Comparaison avec une autre série de peptides riches en tryptophanes ... 116
Chapitre III – Expériences sur cellules eucaryotes : les nouveaux peptides
sont-ils pénétrants ? ...123
1. Tests de cytotoxicité des nouveaux peptides ... 123
2. Test hémolytique des peptides hydrophobes ... 126
3. Visualisation de l’internalisation par microscopie de fluorescence ... 127
4. Quantification de l’internalisation par spectrométrie de masse ... 131
Chapitre IV – Relation entre structure et activité : dichroïsme circulaire .….. 135
Discussion, conclusion et perspectives ..………143
Matériels et méthodes ………...153
1. Logiciels de prédictions ... 153
1.1 Prédiction de l’activité antimicrobienne ... 154
1.2 Prédiction de l’activité pénétrante ... 155
1.3 Prédiction de la structure ... 157
12
2.1 Synthèse peptidique sur support solide ... 158
2.2 Test de Kaiser ... 158
2.3 Modifications de l’extrémité N-terminale du peptide ... 161
2.4 Déprotection, clivage à l’acide fluorhydrique et purification ... 162
3. Tests d’hémolyse ... 163
4. Expériences sur cellules eucaryotes ... 163
4.1 Culture cellulaire ... 163
4.2 Test de cytotoxicité ... 164
4.3 Microscopie à fluorescence : test à la streptavidine fluorescente ... 165
4.4 Quantification de l’internalisation par spectrométrie de masse ... 166
5. Tests sur bactéries ... 168
5.1 Souches bactériennes ... 168
5.2 Test antibactérien ... 169
5.3. Suivi cinétique de l’inhibition de croissance bactérienne ... 170
6. Tests avec modèles membranaires ... 171
6.1. Préparation des vésicules uni-lamellaires ... 171
6.2. Dichroïsme circulaire ... 172
1. Spectres de masse ... 175
2. Profils HPLC... 195
3. Tests antimicrobiens ... 215
4. Tests de cytotoxicité sur cellules CHO-K1 ... 230
5. Tests à la streptavidine fluorescente ... 232
13
Table des figures
Figure 1. Schéma représentant l’organisation de la réponse immunitaire innée. ... 32
Figure 2. Répartition des origines des peptides antimicrobiens selon l’Antimicrobial Peptide Database. ... 34
Figure 3. Exemples de séquences de défensines chez les plantes et structure des défensines : crambine et β-purothionine. ... 35
Figure 4. Structure des trois classes de défensines : α-défensine, β-défensine et Ɵ-défensine.. ... 36
Figure 5. Répartition des origines des peptides antimicrobiens chez les vertébrés selon l’Antimicrobial Peptide Database. ... 37
Figure 6. Transport d’une molécule cargaison « cargo » dans les cellules. ... 40
Figure 7. Interactions entre les chaines latérales des peptides membranotropes et les phospholipides des membranes. ... 40
Figure 8. Différents domaines de la protéine Tat. ... 43
Figure 9. Structure générale du peptidoglycane. ... 46
Figure 10. Représentation simplifié des liaisons dans le peptidoglycane. ... 46
Figure 11. Représentation des différentes étapes de la biosynthèse du peptidoglycane et des antibiotiques qui l’inhibe. ... 48
Figure 12. Différences entre les membranes de bactéries à Gram positif et à Gram négatif. ... 49
Figure 13. Structure générale d’un acide phosphatidique. ... 49
Figure 14. Structure générale du phosphatidylglycérol et de la phosphatidyléthanolamine. ... 50
Figure 15. Structure générale de l’alanylphosphatidylglycérol et du lysylphosphatidylglycérol. ... 51
Figure 16. Structure générale des cardiolipines ... 51
14
Figure 18. Structure générale de l’acide téichoïque et de l’acide lipotéichoïque. ... 53
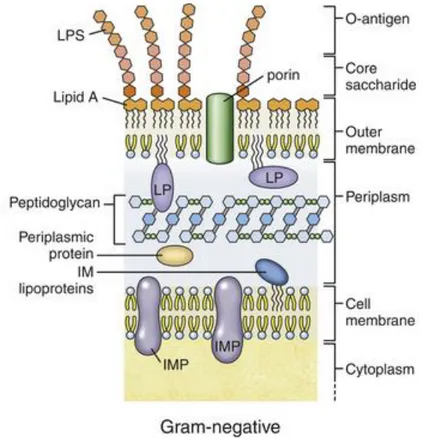
Figure 19. Structure générale d’une membrane de bactérie à Gram négatif.. ... 54
Figure 20. Modèles de transport du lipopolysaccharide à travers la membrane des bactéries à Gram négatif : modèle de l’intermédiaire soluble et du complexe trans-enveloppe. ... 56
Figure 21. Structure générale d’un lipopolysaccharide. ... 57
Figure 22. Modifications possibles du lipide A chez les bactéries à Gram négatif pour lutter contre les molécules antibiotiques et antibactériennes. ... 58
Figure 23. Structure 3D d’une porine en utilisant le logiciel de visualisation PyMol. ... 59
Figure 24. Représentation de la membrane cellulaire. ... 60
Figure 25. Structure du cholestérol. ... 60
Figure 26. Structure générale de la phosphatidylcholine. R1 et R2 représentent les chaines aliphatiques des acides gras estérifiés. ... 61
Figure 27. Structure de la sphingomyéline, sphingolipide possédant une partie polaire, la phosphocholine. ... 61
Figure 28. Structure générale de la phosphatidylsérine. R1 et R2 représentent les chaines aliphatiques des acides gras estérifiés. ... 61
Figure 29. Distribution des lipides dans les membranes de cellules eucaryotes. ... 62
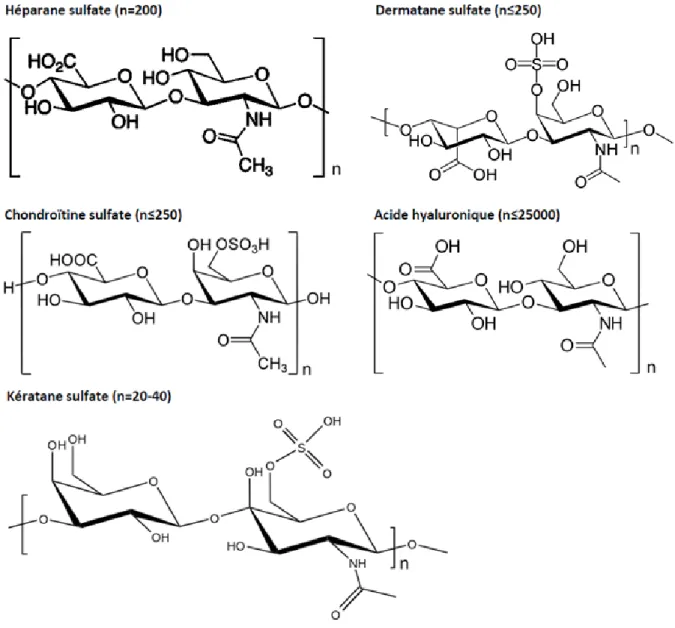
Figure 30. Classification des glycosaminoglycanes selon la nature de leur unité disaccharidique. .. 63
Figure 31. Structure d’un glycosphingolipide : le ganglioside. ... 64
Figure 32. Interaction des peptides antimicrobiens avec les deux types de membranes bactériennes lors de l’insertion des peptides dans les membranes. ... 65
Figure 33. Représentation schématique des différents modèles de perméabilisation membranaire. A : modèle du pore en tonneau ; B : modèle du pore mixte ; C : modèle du tapis ; D : modèle de l’électroporation moléculaire ; E : modèle du radeau. ... 66
15
Figure 34. Les modes d’action des peptides antimicrobiens intracellulaires contre Escherichia coli.
... 69
Figure 35. Quantité internalisée de pénétratine dans les cellules CHO en fonction de la température et de la présence de glycosaminoglycanes. WT, cellule sauvage (wild type) ; GAGdef, cellule déficiente en glycosaminoglycane. ... 70
Figure 36. Différences entre les interactions entre les peptides RL9 et RW9 avec la bicouche. Relation entre les quantités liées aux membranes et les quantités internalisées dans les cellules. 71 Figure 37. Différents modèles de translocation directe. A : formation de micelles inverses ; B : formation de pores transitoires, C : transfert de phase. ... 72
Figure 38. Les différentes voies d’endocytose. ... 74
Figure 39. Mécanisme général de la macropinocytose. ... 75
Figure 40. Différentes étapes de l’endocytose dépendant de la clathrine. ... 76
Figure 41. Interactions des peptides antimicrobiens avec les membranes cellulaires eucaryotes et procaryotes. ... 77
Figure 42. États d’organisation de la membrane en fonction de la température et de la présence de cholestérol. ... 78
Figure 43. Action de peptides antimicrobiens sur une membrane contenant un radeau lipidique (lo) et un domaine en état liquide désordonné (ld). ... 79
Figure 44. Projection en roue hélicoïdale des peptides DA5m-S1-03 et DA5m-S1-04. ... 88
Figure 45. Tests antibactériens de DMS-DA5 sur les bactéries E. coli, P. aeruginosa, K. rhizophila et S. aureus. ... 90
Figure 46. Tests antibactériens des DA5m-S1-01B, DA5m-S1-02B, DA5m-S1-03B et DA5m-S1-04B sur E. coli. ... 91
Figure 47. Tests antibactériens des DA5m-S1-01B, DA5m-S1-02B, DA5m-S1-03B et DA5m-S1-04B sur K. rhizophila. ... 92
16
Figure 48. Représentation de l’importance de la taille du peptide pour l’établissement de pores
dans la membrane bactérienne. ... 93
Figure 49. Tests antibactériens des peptides DA5m-S1-03B et DA5m-S2 sur K. rhizophila. ... 95
Figure 50. Tests antibactériens de DA5m-S2-02N et DA5m-S2-02B sur S. aureus. ... 96
Figure 51. Représentation en ruban des peptides DMS-DA5, DA5m-S2-01 et DA5m-S2-02. ... 97
Figure 52. Représentation de la surface tridimensionnelle des peptides DMS-DA5, DA5m-S2-01 et DA5m-S2-02. ... 98
Figure 53. Représentation en ruban des peptides DA5m-S3-00 « GMWGKIKSTAKEAAKAA » et DA5m-S3-01 « GMWGKIKTAKEAAKAA ». ... 100
Figure 54. Prédictions des structures des peptides DA5m-S3-00 « GMWGKIKSTAKEAAKAA » et DA5m-S3-01 « GMWGKIKTAKEAAKAA » par le logiciel MeDor. ... 100
Figure 55. Tests antibactériens des peptides DA5m-S3 sur E. coli. ... 102
Figure 56. Prédiction par l’algorithme AMPA des régions de la séquence de DMS-DA5 les plus probables de posséder une activité antimicrobienne. ... 104
Figure 57. Tests antibactériens des peptides DA5m-S4 sur P. aeruginosa. ... 106
Figure 58. Tests antibactériens des peptides DA5m-S5-01, DA5m-S5-02 et DA5m-S5-03 sur E. coli. ... 108
Figure 59. Tests antibactériens des peptides DA5m-S6-01 et DA5m-S6-02 sur E. coli. ... 111
Figure 60. Comparaison des concentrations minimales inhibitrices sur les bactéries étudiées du peptide DA5m-S6-02 en fonction de son extrémité N-terminale. ... 114
Figure 61. Test antimicrobien du peptide biotinylé DA5m-S6-02B sur P. aeruginosa en présence de biotine ou non... 116
Figure 62. Comparaison des concentrations minimales inhibitrices sur E. coli des peptides RW en fonction de leur extrémité N-terminale. ... 118
17
Figure 64. Suivi cinétique de l’inhibition de la croissance bactérienne de E. coli par les peptides DA5m-S6-02N et R6W3-1N. ... 122 Figure 65. Tests de cytotoxicité de DMS-DA5 sur les cellules CHO-K1. ... 123 Figure 66. Tests de cytotoxicité des peptides de la série DA5m-S3 sur les cellules CHO-K1. ... 124 Figure 67. Tests de cytotoxicité des peptides R8W1-1, R5W4-1 et R5W4-2 sur les cellules CHO-K1 ... 125
Figure 68. Tests de cytotoxicité des peptides DA5m-S6-01 et DA5m-S6-02 sur les cellules CHO-K1. ... 126
Figure 69. Pouvoir hémolytique des peptides DMS-DA5 et des peptides riches en tryptophanes étudiés sur du sang de rat juvénile. ... 126 Figure 70. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides DA5m-S2-01B et DA5m-S1-02B. ... 128 Figure 71. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides DA5m-S3-01B, DA5m-S3-02B et DA5m-S3-03B. ... 129 Figure 72. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides RW1-1B, RW4-1B, DA5m-S6-01B et DA5m-S6-02B. ... 130 Figure 73. Spectre de masse de l’internalisation de DA5m-S6-02B, DA5m-S6-02D possédant des glycines deutérées a été utilisé en tant qu’étalon interne. ... 131 Figure 74. Quantification de l’internalisation par spectrométrie de masse des peptides DA5m-S6-01B, DA5m-S6-02B, RW9 et pénétratine. ... 132 Figure 75. Spectres de dichroïsme circulaire caractéristiques des principales structures secondaires des peptides. ... 136 Figure 76. Structure des lipides POPG et POPC. ... 137 Figure 77. Spectres de dichroïsme circulaire de DMS-DA5 en présence de LUV (POPC ou POPG) et en solution dans un tampon phosphate. ... 137
18
Figure 78. Spectres de dichroïsme circulaire de DA5m-S2-01N en présence de LUV (POPC ou POPG)
et en solution dans un tampon phosphate. ... 138
Figure 79. Spectres de dichroïsme circulaire de DA5m-S2-02N en présence de LUV (POPC ou POPG) et en solution dans un tampon phosphate. ... 139
Figure 80. Proportion de la structure en hélice α des différents peptides inspirés de DMS-DA5. . 140
Figure 81. Représentation du lien entre les structures de peptides vecteurs, leur interaction avec les membranes et leur mécanisme d’internalisation. ... 141
Figure 82. Comparaison de l’activité antibactérienne sur Escherichia coli de peptides de séquences similaires mais de tailles différentes. ... 144
Figure 83. Schéma du principe de l’expérience en utilisant une bicouche à l’interface d’une émulsion inverse. ... 147
Figure 84. Analyse de l’effet des substitutions d’acides aminés sur l’activité antibactérienne de la bacténécine A2 contre P. aeruginosa. ... 155
Figure 85. Profil binaire de la séquence CIWSRDLISH. ... 156
Figure 86. Principe du test de Kaiser. ... 159
Figure 87. Déamidation de l’asparagine. ... 160
Figure 88. Test de Kaiser avec la proline. ... 160
Figure 89. Spectre RMN 1H de la biotine sulfone dans du D 2O. ... 162
Figure 90. Principe du test de cytotoxicité avec le kit CCK8. ... 164
Figure 91. Principe du test à la streptavidine fluorescente ... 166
Figure 92. Protocole de la quantification de l’internalisation des peptides pénétrants par spectrométrie de masse. ... 168
Figure 93. Plan de plaque d’un test antimicrobien. ... 169
Figure 94. Représentation des dilutions en cascade effectuées pour obtenir les aliquotes à déposer sur milieu gélosé pour le test cinétique de l’inhibition de croissance bactérienne. ... 171
20
Table des tableaux
Tableau 1. Alignement des séquences peptidiques des dermaseptines isolées de Phyllomedusa
sauvagei [51]. ... 38
Tableau 2. Exemple de peptides pénétrants. ... 41 Tableau 3. Prédiction de l’activité antimicrobienne de DMS-DA5 et des peptides issus de sa
séquence en utilisant CAMPR3. ... 82
Tableau 4. Prédiction de l’activité pénétrante de DMS-DA5 et des peptides de dix acides aminés issus de sa séquence en utilisant CellPPD. ... 83 Tableau 5. Prédiction de l’activité pénétrante de la pénétratine, de DMS-DA5 et DA5M-S1-01 en utilisant CPPpred. ... 84 Tableau 6. Prédiction de l’activité pénétrante de DA5m-S1-01 et de ses dérivés présentant une substitution d’une lysine par une arginine en utilisant CPPpred... 85 Tableau 7. Prédiction de l’activité pénétrante de peptides possibles par substitution d’un seul acide aminé dans la séquence de DA5m-S1-01 en utilisant CellPPD... 86 Tableau 8. Prédiction de l’activité pénétrante de DA5m-S1-01 et des dix séquences issues de la substitution d’un seul acide aminé dans sa séquence qui présentent les meilleurs scores CPPpred. ... 87
Tableau 9. Prédiction de l’activité antibactérienne de DA5M-S1-03 et DA5M-S2-01 en utilisant CAMPR3. ... 87
Tableau 10. Liste des souches bactériennes testées. ... 89 Tableau 11. Résultats des tests antibactériens avec les peptides DA5m-S1 sur les quatre souches testées. ... 91 Tableau 12. Prédiction de l’activité antibactérienne de DA5M-S1-03 et DA5M-S2-01 en utilisant CAMPR3. ... 94
Tableau 13. Résultats des tests antibactériens avec les peptides DA5m-S2 sur les quatre souches testées. ... 94
21
Tableau 14. Séquence de plusieurs dermaseptines répertoriées dans la littérature mettant en évidence les motifs conservés. ... 99 Tableau 15. Séquence des peptides DA5m-S3-01, DA5m-S3-02 et DA5m-S3-03 inspirées de
DMS-DA5. ... 101 Tableau 16. Résultats des tests antibactériens avec les peptides DA5m-S3 sur les quatre souches testées. ... 101 Tableau 17. Séquence de quelques dermaseptines possédant des motifs conservés en commun avec DMS-DA5. ... 103 Tableau 18. Séquence des nouveaux peptides DA5m-S4 et DA5m-S5 inspirés de DMS-DA5. ... 105 Tableau 19. Résultats des tests antibactériens avec les peptides DA5m-S4 sur les quatre souches testées. ... 105 Tableau 20. Résultats des tests antibactériens avec les peptides DA5m-S5 sur les quatre souches testées. ... 107 Tableau 21. Séquence des nouveaux peptides DA5M-S6-01 et DA5M-S6-02 inspirés de la séquence de DA5M-S5-05. ... 110 Tableau 22. Concentrations minimales inhibitrices de DA5m-S6-01 et DA5m-S6-02 biotinylés ou non sur les souches E. coli, P. aeruginosa, S. aureus et K. rhizophila. ... 110 Tableau 23. Concentrations minimales inhibitrices de DA5m-S6-01 et DA5m-S6-02 acétylés ou non sur les souches E. coli, P. aeruginosa, S. aureus et K. rhizophila. ... 112 Tableau 24. Concentration minimale inhibitrice de DA5m-S6-01 et DA5m-S6-02 sans modification, avec une chaine polyglycine ou avec une chaine polyglycine biotinylée sur les souches E. coli,
P. aeruginosa, S. aureus et K. rhizophila. ... 113
Tableau 25. Concentrations minimales inhibitrices de DA5m-S6-01 et DA5m-S6-02, avec une chaine polyglycine acétylée ou non et avec une chaine polyglycine biotinylée sur les souches E. coli,
P. aeruginosa, S. aureus et K. rhizophila. ... 114
22
Tableau 27. Concentrations minimales inhibitrices des peptides RW9 sur les souches E. coli et
S. aureus. ... 118
Tableau 28. Concentrations minimales inhibitrices des peptides RW9 acétylés ou non sur les
souches E. coli et S. aureus. ... 119 Tableau 29. Concentrations minimales inhibitrices des peptides RW9 acétylés ou non sur les
souches E. coli et S. aureus. ... 120 Tableau 30. Attribution des structures secondaires selon les caractéristiques du spectre de
23
Table des Annexes
Annexe 1. Spectre de masse de DMS-DA5. ... 175 Annexe 2. Spectre de masse de DA5m-S1-01B. ... 175 Annexe 3. Spectre de masse de DA5m-S1-02B. ... 176 Annexe 4. Spectre de masse de DA5m-S1-03B. ... 176 Annexe 5. Spectre de masse de DA5m-S1-04B. ... 176 Annexe 6. Spectre de masse de DA5m-S2-01N. ... 177 Annexe 7. Spectre de masse de DA5m-S2-01B. ... 177 Annexe 8. Spectre de masse de DA5m-S2-02N. ... 178 Annexe 9. Spectre de masse de DA5m-S2-02B. ... 178 Annexe 10. Spectre de masse de DA5m-S3-01N. ... 179 Annexe 11. Spectre de masse de DA5m-S3-01B. ... 179 Annexe 12. Spectre de masse de DA5m-S3-02N. ... 180 Annexe 13. Spectre de masse de DA5m-S3-02B. ... 180 Annexe 14. Spectre de masse de DA5m-S3-03N. ... 181 Annexe 15. Spectre de masse de DA5m-S3-03B. ... 181 Annexe 16. Spectre de masse de DA5m-S4-01N. ... 182 Annexe 17. Spectre de masse de DA5m-S4-02N. ... 183 Annexe 18. Spectre de masse de DA5m-S4-03N. ... 183 Annexe 19. Spectre de masse de DA5m-S5-01N. ... 184 Annexe 20. Spectre de masse de DA5m-S5-02N. ... 184
24
Annexe 21. Spectre de masse de DA5m-S5-03N. ... 184 Annexe 22. Spectre de masse de DA5m-S5-04N. ... 185 Annexe 23. Spectre de masse de DA5m-S5-01B. ... 185 Annexe 24 . Spectre de masse de DA5m-S5-02B. ... 185 Annexe 25. Spectre de masse de DA5m-S5-03B. ... 186 Annexe 26. Spectre de masse de DA5m-S5-04B. ... 186 Annexe 27. Spectre de masse de DA5m-S5-05B. ... 187 Annexe 28. Spectre de masse de DA5m-S6-01N. ... 187 Annexe 29. Spectre de masse de DA5m-S6-01Ac. ... 188 Annexe 30. Spectre de masse de DA5m-S6-01G. ... 188 Annexe 31. Spectre de masse de DA5m-S6-01Ga. ... 188 Annexe 32. Spectre de masse de DA5m-S6-01B. ... 189 Annexe 33. Spectre de masse de DA5m-S6-02N. ... 189 Annexe 34. Spectre de masse de DA5m-S6-02Ac. ... 189 Annexe 35. Spectre de masse de DA5m-S6-02G. ... 190 Annexe 36. Spectre de masse de DA5m-S6-02Ga. ... 190 Annexe 37. Spectre de masse de DA5m-S6-02B. ... 190 Annexe 38. Spectre de masse de R8W1-1N. ... 191 Annexe 39. Spectre de masse de R8W1-1Ga. ... 191 Annexe 40. Spectre de masse de R7W2-1N. ... 192 Annexe 41. Spectre de masse de R8W1-1Ga. ... 192 Annexe 42. Spectre de masse de R6W3-1N. ... 192
25
Annexe 43. Spectre de masse de R6W3-1Ga. ... 193 Annexe 44. Spectre de masse de R5W4-1N. ... 193 Annexe 45. Spectre de masse de R5W4-1N. ... 193 Annexe 46. Spectre de masse de R5W4-1N. ... 194 Annexe 47. Spectre de masse de R5W4-1N. ... 194 Annexe 48. Profil HPLC de DA5m-S1-01B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 195 Annexe 49. Profil HPLC de DA5m-S1-02B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 195 Annexe 50. Profil HPLC de DA5m-S1-03B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 196 Annexe 51. Profil HPLC de DA5m-S1-04B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 196 Annexe 52. Profil HPLC de DA5m-S2-01N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 197 Annexe 53. Profil HPLC de DA5m-S2-01B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 197 Annexe 54. Profil HPLC de DA5m-S2-02B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 198 Annexe 55. Profil HPLC de DA5m-S3-01N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 198 Annexe 56. Profil HPLC de DA5m-S3-01B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 199 Annexe 57. Profil HPLC de DA5m-S3-02N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 199
26
Annexe 58. Profil HPLC de DA5m-S3-02B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 200 Annexe 59. Profil HPLC de DA5m-S3-03N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 200 Annexe 60. Profil HPLC de DA5m-S3-03B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 201 Annexe 61. Profil HPLC de DA5m-S4-01N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 201 Annexe 62. Profil HPLC de DA5m-S4-02N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 202 Annexe 63. Profil HPLC de DA5m-S4-03N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 202 Annexe 64. Profil HPLC de DA5m-S5-01N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 203 Annexe 65. Profil HPLC de DA5m-S5-01B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 203 Annexe 66. Profil HPLC de DA5m-S5-02N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 204 Annexe 67. Profil HPLC de DA5m-S5-02B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 204 Annexe 68. Profil HPLC de DA5m-S5-04N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 205 Annexe 69. Profil HPLC de DA5m-S5-04B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 205 Annexe 70. Profil HPLC de DA5m-S5-05N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 206
27
Annexe 71. Profil HPLC de DA5m-S6-01N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 206 Annexe 72. Profil HPLC de DA5m-S6-01Ac (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 207 Annexe 73. Profil HPLC de DA5m-S6-01Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 207 Annexe 74. Profil HPLC de DA5m-S6-02N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 208 Annexe 75. Profil HPLC de DA5m-S6-02G (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 208 Annexe 76. Profil HPLC de DA5m-S6-02Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 209 Annexe 77. Profil HPLC de DA5m-S6-02B (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 209 Annexe 78. Profil HPLC de R8W1-1N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 210 Annexe 79. Profil HPLC de R8W1-1Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 210 Annexe 80. Profil HPLC de R7W2-1N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 211 Annexe 81. Profil HPLC de R7W2-1Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 211 Annexe 82. Profil HPLC de R6W3-1N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 212 Annexe 83. Profil HPLC de R6W3-1Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 212
28
Annexe 84. Profil HPLC de R5W4-1N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 213 Annexe 85. Profil HPLC de R5W4-1Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 213 Annexe 86. Profil HPLC de R5W4-2N (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 214 Annexe 87. Profil HPLC de R5W4-2Ga (gradient de 5% à 60% acétonitrile en 10 minutes ; suivi à 220 nm (en noir) et 280 nm (en rouge). ... 214 Annexe 88. Tests antibactériens des DA5m-S1-01B, DA5m-S1-02B, DA5m-S1-03B et DA5m-S1-04B sur P. aeruginosa. ... 215 Annexe 89. Tests antibactériens des DA5m-S1-01B, DA5m-S1-02B, DA5m-S1-03B et DA5m-S1-04B sur S. aureus... 215 Annexe 90. Tests antibactériens des peptides DA5m-S1-03B et DA5m-S2 sur E. coli. ... 216 Annexe 91. Tests antibactériens des peptides DA5m-S1-03B et DA5m-S2 sur P. aeruginosa. ... 216 Annexe 92. Tests antibactériens des peptides DA5m-S1-03B et DA5m-S2 sur S. aureus. ... 217 Annexe 93. Tests antibactériens des peptides DA5m-S3 sur P. aeruginosa. ... 217 Annexe 94. Tests antibactériens des peptides DA5m-S3 sur S. aureus. ... 218 Annexe 95. Tests antibactériens des peptides DA5m-S3 sur K. rhizophila. ... 218 Annexe 96. Tests antibactériens des peptides DA5m-S4 sur E. coli. ... 219 Annexe 97. Tests antibactériens des peptides DA5m-S4 sur S. aureus. ... 219 Annexe 98. Tests antibactériens des peptides DA5m-S4 sur P. aeruginosa. ... 220 Annexe 99. Tests antibactériens des peptides DA5m-S5 sur E. coli. ... 220 Annexe 100. Tests antibactériens des peptides DA5m-S5 sur P. aeruginosa. ... 221 Annexe 101. Tests antibactériens des peptides DA5m-S5 sur S. aureus. ... 221
29
Annexe 102. Tests antibactériens des peptides DA5m-S5 sur K. rhizophila. ... 222 Annexe 103. Tests antibactériens des peptides DA5m-S6-01 sur E. coli. ... 222 Annexe 104. Tests antibactériens des peptides DA5m-S6-01 sur P. aeruginosa... 223 Annexe 105. Tests antibactériens des peptides DA5m-S6-01 sur S. aureus. ... 223 Annexe 106. Tests antibactériens des peptides DA5m-S6-01 sur K. rhizophila. ... 224 Annexe 107. Tests antibactériens des peptides DA5m-S6-02 sur E. coli. ... 224 Annexe 108. Tests antibactériens des peptides DA5m-S6-02 sur P. aeruginosa... 225 Annexe 109. Tests antibactériens des peptides DA5m-S6-02 sur S. aureus. ... 225 Annexe 110. Tests antibactériens des peptides DA5m-S6-01 sur K. rhizophila. ... 226 Annexe 111. Tests antibactériens des peptides R8W1-1 sur E. coli. ... 226 Annexe 112. Tests antibactériens des peptides R8W1-1 sur K. rhizophila. ... 227 Annexe 113. Tests antibactériens des peptides R6W3-1 sur E. coli. ... 227 Annexe 114. Tests antibactériens des peptides R6W3-1 sur S. aureus. ... 228 Annexe 115. Tests antibactériens des peptides R5W4-1 sur E. coli. ... 228 Annexe 116. Tests antibactériens des peptides R5W4-1 sur S. aureus. ... 229 Annexe 117. Tests antibactériens des peptides R5W4-2 sur E. coli. ... 229 Annexe 118. Tests antibactériens des peptides R5W4-2 sur S. aureus. ... 230 Annexe 119. Tests de cytotoxicité des peptides de la série DA5m-S1 sur les cellules CHO-K1. ... 230 Annexe 120. Tests de cytotoxicité des peptides de la série DA5m-S2 sur les cellules CHO-K1. ... 231 Annexe 121. Tests de cytotoxicité des peptides de la série DA5m-S5 sur les cellules CHO-K1. ... 231 Annexe 122. Tests de cytotoxicité des peptides de la série DA5m-S5 sur les cellules CHO-K1. ... 232
30
Annexe 123. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides DA5m-S2-01B et DA5m-S1-02B. ... 232 Annexe 124. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides DA5m-S3-01B, DA5m-S3-02B et DA5m-S3-03B. ... 233 Annexe 125. Résultats des tests de pénétration dans les cellules CHO-K1 utilisant la streptavidine fluorescente des peptides RW1-1B, RW4-1B, DA5m-S6-01B et DA5m-S6-02B. ... 234
31
Chapitre I – Introduction :
Les peptides membranotropes
1. Historique et généralités des peptides membranotropes
Les membranes cellulaires, procaryotes ou eucaryotes, sont principalement de nature hydrophobe ce qui les rend imperméables à un grand nombre de molécules telles que les peptides, protéines et oligonucléotides. Malgré cela, certains peptides, appelés peptides membranotropes ou « membrane-active peptides », interagissent avec ces membranes dans le cadre de leur activité biologique. Parmi ces peptides, deux classes se distinguent par leur important potentiel thérapeutique : les peptides antimicrobiens et les peptides pénétrants. Ces peptides présentent de nombreux points communs, étant tous deux généralement courts (quelques dizaines d’acides aminés en général) et chargés positivement, bien que leur origine et leurs utilisations soient considérablement différentes. Dans le cadre de ce travail, j’ai essayé de déterminer quels sont les paramètres qui peuvent orienter le caractère d’un peptide vers l’activité antimicrobienne ou vers l’activité pénétrante.
1.1 Les peptides antimicrobiens
1.1.1. Généralités sur les peptides antimicrobiens
Tous les organismes vivants, qu’ils appartiennent au règne animal, végétal ou microbien, possèdent des mécanismes de défense afin de lutter contre les agents pathogènes auxquels ils sont régulièrement confrontés. Ces réactions de défense se divisent en deux types : un système immunitaire inné naturel et immédiat, et un système immunitaire adaptif fortement spécifique. Si l’immunité innée ou naturelle est le seul mécanisme de défense présent chez les plantes et les invertébrés, elle est la première ligne de défense chez les vertébrés. Chez ces derniers, il existe également l’immunité adaptative qui se développe plus lentement que la première et par laquelle elle est également stimulée [1]. L’immunité innée dépend en premier lieu de barrières physiques et épithéliales (perméabilité de la peau, mucus des muqueuses, flore commensale …) ainsi que des molécules qu’elle produit en cas
32
d’infection [2]. Parmi les substances sécrétées, un des éléments clés est constitué par les composés capables de neutraliser les micro-organismes pathogènes et notamment les peptides antimicrobiens (PAM), ou AntiMicrobial Peptides (AMP) ou encore Host Defense Peptides (HDP) (Figure 1) [3, 4].
Figure 1. Schéma représentant l’organisation de la réponse immunitaire innée, adaptée de [5].
Les peptides antimicrobiens sont principalement connus pour leur forte activité antibactérienne, en éliminant (peptides bactériolytiques) ou en inhibant totalement la croissance (peptides bactériostatiques) de ces micro-organismes. Ils induisent une résistance limitée de la part des bactéries contrairement aux antibiotiques couramment utilisés, constituant ainsi une alternative prometteuse à ces derniers [6, 7, 8, 9].
Ces peptides sont également actifs contre un grand nombre d’autres micro-organismes, présentant des activités antifongiques et/ou antivirales [10, 11, 12], et certains ont montré des propriétés antitumorales [13, 14]. En plus de leur activité contre les micro-organismes, les peptides antimicrobiens peuvent jouer un rôle dans la réponse inflammatoire et la réponse
33
immunitaire, allant jusqu’à servir de lien entre les réponses immunitaires innée et adaptative (Figure 1) [15].
Les peptides antimicrobiens présentent une grande variété structurale. Ils peuvent généralement être classés en quatre grandes familles selon leur structure : les peptides structurés en hélice α, en feuillets β, présentant une structure mixte en hélices α – feuillets β et les peptides non-structurés, particulièrement riches en un ou plusieurs acides aminés [16, 17, 18, 19]. En plus de cette classification, certains peptides présentent des structures particulières telles que des structures cycliques [20] ou encore en lasso (où ils sont constitués d’une partie cyclique et d’une partie linéaire qui passe à travers le cycle) [21].
Les peptides antimicrobiens sont des peptides de petite taille allant d’une dizaine à quelques dizaines d’acides aminés. La taille des peptides est importante pour la définition de la structure que le peptide peut adopter étant donné qu’un minimum de huit acides aminés est nécessaire pour qu’il puisse adopter une hélice α amphiphile ou présenter un coude β.
Ces peptides sont généralement riches en acides aminés basiques (lysines et arginines) et en acides aminés hydrophobes (leucines, isoleucines, phénylalanines et tryptophanes). Les lysines et arginines sont chargées positivement à pH physiologique et cette charge constitue le facteur d’interaction initial avec les têtes polaires chargées négativement des lipides membranaires procaryotes. D’un autre côté, les acides aminés hydrophobes, qui constituent souvent près de la moitié de la séquence peptidique, interagissent fortement avec les chaines aliphatiques des lipides. Ensemble, cette composition en acides aminés assure des interactions électrostatiques et hydrophobes très favorables avec les membranes bactériennes.
Bien que les premiers peptides antimicrobiens (bactériocines) aient été découverts chez les bactéries vers le milieu des années 20 [22, 23, 24], le premier isolement à partir d’une espèce pluricellulaire fut celui de la cécropine à partir du papillon Hyalophora cecropia en 1981 [25,26]. Depuis cette découverte, de nombreux peptides ont été identifiés chez les plantes, les espèces animales invertébrées et vertébrées, dont l’être humain [7, 18, 27]. À ce jour, près de 2700 peptides antimicrobiens d’origine naturelle dont les séquences peptidiques sont établies (au moins partiellement) ont été recensés sur la base de données « Antimicrobial
34
Peptide Database » (APD), et plus de la moitié de ces peptides (56 %) est retrouvée chez les vertébrés (Figure 2) [28].
Figure 2. Répartition des origines des peptides antimicrobiens selon l’Antimicrobial Peptide Database, d’après les données de [28].
Dans les deux sous-parties suivantes, deux grandes familles de peptides antimicrobiens très étudiées seront présentées. Ces peptides possèdent d’importantes activités biologiques et se différencient par leurs structures qui sont très différentes : les défensines repliées en feuillets β et les dermaseptines principalement en hélice α linéaires.
1.1.2. Les défensines
e nombre très important de peptides antimicrobiens et leurs origines très diverses, certaines familles de peptides sont particulièrement abondantes. Tel est le cas de la famille des défensines qui sont présentes chez de nombreuses espèces pluricellulaires dont les plantes et l’homme. Les défensines sont des peptides possédant une cinquantaine d’acides aminés dont la composition est particulièrement riche en cystéines. Les groupements thiols de ces cystéines forment des ponts disulfures au sein de la structure, généralement entre deux et six ponts intramoléculaires [18].
Chez les plantes, les défensines possèdent des séquences de 45 à 54 acides aminés qui sont assez divergentes mais dans lesquelles sont particulièrement conservées huit cystéines (en jaune sur la figure 3). Ces peptides possèdent souvent des structures mixtes où les parties structurées en hélice α et en feuillets β sont reliées par quatre à cinq ponts disulfures selon le peptide (Figure 3) [29]. Ces peptides permettent aux plantes de se défendre contre les
13%
12%
19% 56%
Origine des peptides
antimicrobiens
Bactéries et micro-organismes Plantes
Invertébrés Vertébrés
35
infections pathogènes ainsi que contre les facteurs environnementaux qui feraient obstacle à leur croissance, tels que la sécheresse ou la présence de métaux lourds dans les sols. Ils sont particulièrement connus pour leur activité antifongique, mais ils peuvent également posséder une activité antibactérienne ou insecticide. Ils sont également capables d’inhiber l’α-amylase et la trypsine, enzymes hydrolysant respectivement les sucres et les protéines, en cas de stress biotique (carence en azote, rayons UV …), ce qui permet d’éviter des complications tels que des réactions inflammatoires [31, 32, 33].
Figure 3. (A) Exemples de séquences de défensines chez les plantes et (B) structure des défensines : crambine (à gauche) et β-purothionine (à droite). Les cystéines conservées et impliquées dans les ponts disulfures sont écrites en jaune ; les structures en hélice α sont représentées en bleu et les feuillets β en rose [29].
En comparaison avec les défensines d’origine végétale, celles des mammifères sont plus courtes et sont constitués de 18 à 45 acides aminés. Ces peptides sont généralement riches en arginines et possèdent six à huit résidus cystéinyles engagés dans des ponts disulfures. Ils sont ainsi classés selon l’alignement des ponts disulfures entre les cystéines : les α-défensines, les β-défensines et les plus rares Ɵ-défensines qui ne sont trouvées que chez
36
certains primates. Les trois types de défensines possèdent toutes une forte activité antibactérienne contre les bactéries à Gram négatif et les bactéries à Gram positif mais elles se différencient par leur structure (Figure 4) [33, 34].
Ainsi, les α-défensines sont caractérisées par une structure composée de feuillets β reliés par trois ponts disulfures au niveau des cystéines 1-6, 2-4 et 3-5. Elles contrôlent positivement la réponse immunitaire par le recrutement de neutrophiles et la régulation de molécules pro-inflammatoires et anti-pro-inflammatoires (Figure 4A) [35].
Les β-défensines sont généralement plus grandes et plus cationiques que les α-défensines mais moins hydrophobes. Comme les défensines de plantes présentées précédemment, les β-défensines se caractérisent par des structures mixtes en hélice α et feuillets β qui présentent trois ponts disulfures intramoléculaires entre les cystéines 1-5, 2-4 et 3-6 (Figure 4B) [36, 37]. Ces peptides sont impliqués dans la production de molécules pro-inflammatoires et la régulation de types cellulaires impliqués dans le processus inflammatoire. En plus de ces activités, les α-défensines et les β-défensines ont montré une activité antivirale, en particulier en provoquant le blocage de la réplication du VIH-1 [38, 39]
La troisième et dernière classe de défensines retrouvée chez les mammifères est représentée par les Ɵ-défensines. Elles sont plus petites que les autres défensines et leur structure est composée de deux feuillets β antiparallèles reliés par trois ponts disulfures. Leur activité est essentiellement antibactérienne, mais elles sont également actives contre les champignons et les virus (Figure 4C) [40, 41].
Figure 4. Structure des trois classes de défensines : (A) α-défensine, (B) β-défensine et (C) Ɵ-défensine. Les hélices α sont représentées en rouge et jaune ; les feuillets β en bleu et les ponts disulfures en jaune [18].
37
1.1.3. Les dermaseptines
Parmi les peptides antimicrobiens originaires des vertébrés, la grande majorité des peptides identifiés est retrouvée exclusivement chez les amphibiens (Figure 5).
Figure 5. Répartition des origines des peptides antimicrobiens chez les vertébrés selon l’Antimicrobial Peptide Database, [28].
La peau des amphibiens est effectivement riche en molécules qui possèdent une activité biologique et qui servent de première ligne de défense contre les pathogènes [42]. Les composants majeurs de cette défense chimique sont constitués de peptides analgésiques, antioxydants, inhibiteurs de protéases et antimicrobiens. Malgré le fait que le mécanisme de défense soit similaire pour toutes ces espèces, les molécules produites pour cette défense, et en particulier les peptides antimicrobiens, sont considérablement différentes et spécifiques à chaque espèce, variant ainsi en taille, charge, hydrophobie et activité. Les peptides produits par une espèce donnée sont toutefois très similaires (Tableau 1), étant des isomères de séquences et de structures conservées mais pouvant posséder des activités biologiques différentes. Il est ainsi possible de classer les peptides en plusieurs familles, selon leur homologie par alignement de séquences, parmi lesquelles peuvent être citées les phylloseptines, dermatoxines, phylloxines, hyposines ou dermaseptines [27, 43]. La famille des dermaseptines est l’une des familles de peptides qui n’est retrouvée que chez les rainettes de la sous-famille des phyllomédusines [44, 45, 46, 47, 48, 49, 50].
8% 14% 62% 4% 7% 2% 3%
Peptides antimicrobiens
chez les vertébrés
Hommes Mammifères Grenouilles Autres amphibiens Poissons Reptiles Oiseaux
38
Tableau 1. Alignement des séquences peptidiques des dermaseptines isolées de
Phyllomedusa sauvagei [51].
Les dermaseptines présentent des séquences d’une trentaine d’acides aminés (généralement entre 24 et 34) dont la composition peut grandement varier d’un peptide à un autre. Malgré cette divergence dans les séquences, les peptides sont tous cationiques du fait de la présence d’un nombre important de lysines. Contrairement aux défensines dont la structure est stabilisée par des ponts disulfures, les dermaseptines possèdent généralement une structure linéaire en hélice α amphiphile en présence de lipides membranaires, grâce à la répartition de leurs acides aminés hydrophobes (alanines et leucines, très présentes chez les dermaseptines) d’un côté, et hydrophiles (lysines) de l’autre [51, 52].
La grande majorité des dermaseptines a été principalement identifiée par leur remarquable activité antibactérienne. Toutefois, de nombreuses dermaseptines se sont révélées posséder des activités biologiques supplémentaires. La dermaseptine DRS-S1 fut le premier peptide antimicrobien qui stimule l’activité microbicide des leucocytes [53]. Depuis, de nombreuses dermaseptines ont révélé des activités immunomodulatrices, en stimulant l’activité du système immunitaire ou en l’inhibant. Par exemple, les dermaseptines DRS-S9 et DRS-DA4 peuvent induire un chimiotactisme, c’est-à-dire un mouvement de certaines cellules en réponse à une stimulation chimique, qui a un effet chémoattracteur envers les leucocytes permettant ainsi leur recrutement [54, 55].
Une dermaseptine étudiée au Laboratoire des Biomolécules est la dermaseptine isolée de la rainette Pachymedusa dacnicolor nommée DMS-DA5 selon la nomenclature proposée par le groupe de Pierre Nicolas : DMS correspondant à dermaseptine, DA pour dacnicolor et 5
39
pour être le cinquième peptide caractérisé chez cette espèce [56]. Cette dermaseptine possède une séquence de 29 acides aminés « GMWGKIKSTAKEAAKAAGKAALNAVSEAL ». En comparaison avec les autres peptides isolés chez Pachymedusa dacnicolor, DMS-DA5 s’est distinguée par son importante activité bactérienne, présentant des concentrations minimales inhibitrices très faibles (concentration la plus faible à laquelle la croissance bactérienne est complètement inhibée) égales à 3,1 µM contre Escherichia coli DH5α, Bacillus subtilis AIA et
Salmonella enterica serovar typhimurium STM14028, et de 25 µM contre Pseudomonas aeruginosa [57].
1.2 Les peptides pénétrants
1.2.1. Généralités sur les peptides pénétrants
Depuis une trentaine d’années, une autre classe de peptides membranotropes a provoqué un intérêt grandissant. Ces peptides sont communément désignés par peptides pénétrants, peptides vecteurs ou Cell-Penetrating Peptides (CPP), et possèdent des appellations alternatives telles que Trojan Peptides ou domaines de transduction des protéines (Protein Transduction Domains ; PTD) [58, 59]. Ils se distinguent par leur capacité à pouvoir entrer dans les cellules sans avoir à se lier à un récepteur spécifique. Cette pénétration peut se réaliser en transportant une molécule dite cargaison (ou « cargo » en anglais) liée de manière covalente ou non à ces peptides, ce qui en fait des outils prometteurs pour le transport de molécules bioactives qui ne pourraient pas traverser la bicouche lipidique seules (Figure 6). En effet, ce transport a pu être réalisé in vitro et in vivo avec des petites et des grosses molécules incluant peptides, protéines (jusqu’à 120 kDa), acides nucléiques peptidiques (molécules similaires à l’ADN mais différenciées par leur structure qui est composée de plusieurs N-(2-aminoéthyl)-glycines liées par des liaisons peptidiques), oligonucléotides et analogues [60, 61, 62, 63, 64]. En comparant aux autres technologies de vectorisation existantes (virus, liposomes, nanoparticules, électroporation …), les peptides pénétrants présentent l’avantage d’être accessibles facilement par synthèse peptidique et de montrer peu ou pas de toxicité.
40
Figure 6. Transport d’une molécule cargaison « cargo » dans les cellules, adaptée de [65].
Les peptides pénétrants présentent généralement dans leur séquence de nombreux acides aminés basiques, notamment des arginines [66, 67], et des acides aminés hydrophobes, en particulier les tryptophanes, qui peuvent jouer un rôle déterminant dans la pénétration dans les cellules [59, 68, 69, 70, 71]. Comme pour les peptides antimicrobiens, cette composition en acides aminés assure ainsi des interactions électrostatiques et hydrophobes avec les phospholipides des membranes cellulaires (Figure 7).
Figure 7. Interactions entre les chaines latérales des peptides membranotropes et les phospholipides des membranes. Les interactions électrostatiques et les interactions hydrophobes sont respectivement indiquées par les flèches blanches et noires [72].
41
Les peptides pénétrants peuvent adopter les mêmes structures secondaires que les peptides antimicrobiens, comme les structures en hélice α et brin β [73, 74], mais il existe aussi certains peptides pénétrants capables de s’internaliser dans les cellules sans être structurés tels que le peptide Tat dérivé de la protéine Tat du virus d’immunodéficience humaine (VIH) [75].
Contrairement aux peptides antimicrobiens, les peptides pénétrants ne sont pas des peptides naturels. En effet, ces peptides sont généralement dérivés de protéines naturelles mais certains d’entre eux sont issus de la fusion de deux séquences naturelles ou encore totalement synthétiques (Tableau 2) [76, 77].
Nom Séquence Origine Référence
Peptides issus de domaines protéiques
Pénétratine RQIKIWFQNRRMKWKK Antennapedia [78]
Tat (48-60) GRKKRRQRRRPPQ Tat du VIH [79]
pVEC LLIILRRRIRKQAHAHSK Cadhérine [80]
VP22 NAKTRRHERRRKLAIER Protéine virale du
VSH [81]
Peptides chimériques
Transportan GWTLNSAGYLLGKINLKALAALAKKIL Galanine /
Mastoparan [82] MPG GALFLGFLGAAGSTMGAWSQPKKKRKV gp41 (VIH) /
Antigène T (SV40) [83]
Pep-1 KETWWETWWTEWSQPKKKRKV Transcriptase (VIH)
/ Antigène T (SV40) [84] Peptides synthétiques Oligo-arginine RRRRRRRR(R)n Dérivé de Tat (48-60) [85] RW16 RRWRRWWRRWWRRWRR Dérivé de la pénétratine [86] RW9 RRWWRRWRR Dérivé de la pénétratine [87]
MAP KLALKLALKALKAALKLA Model Amphipatic
Peptide [88]
42
1.2.2. Peptides dérivés de domaines protéiques
Parmi les nombreux peptides dérivés de domaines protéiques, la pénétratine est l’un des peptides les plus décrits dans la littérature. En 1993, le groupe d’Alain Prochiantz a montré que l’homéodomaine d’Antennapedia, issu d’un gène homéotique de la drosophile, était capable de traverser les membranes cellulaires et d’atteindre les noyaux [78, 89]. L’homéodomaine est un domaine hautement conservé chez les homéoprotéines, facteurs de transcription se liant à l’ADN, impliqués dans les processus morphologiques du développement embryonnaire. Celui d’Antennapedia possède 60 acides aminés structurés en 3 hélices α et un coude β entre les deuxième et troisième hélices. Des expériences de mutations dans la séquence de la troisième hélice ont montré que celle-ci est indispensable pour l’internalisation de l’homéodomaine, étant donné que les mutations (délétions, substitutions d’acides aminés), notamment au niveau des acides aminés hydrophobes, dans cette séquence bloquent totalement l’internalisation dans les cellules [89, 90]. L’étude approfondie de cette troisième hélice a révélé que la séquence minimale nécessaire à l’internalisation est une séquence de 16 acides aminés, correspondants aux positions 43 à 58 de l’homéodomaine, qui est celle de la pénétratine appelée également pAntp : « RQIKIWFQNRRMKWKK » [78, 91]. Depuis cette découverte, d’autres peptides pénétrants inspirés des troisièmes hélices de différents homéodomaines ont pu être établis comme les peptides Engrailed ou Hox provenant des homéoprotéines humaines correspondantes [92].
Un autre peptide pénétrant historique est le peptide Tat (48-60). Ce peptide est dérivé de la protéine Tat du VIH (virus de l’immunodéficience humaine) qui est composée de cinq domaines fonctionnels différents : une région N-terminale riche en acides aminés acides, un domaine riche en cystéines nécessaire à la transactivation, un motif « RKGLGI » conservé chez les VIH-1 et VIH-2, un domaine basique responsable de la localisation nucléaire et un domaine permettant la liaison de la protéine aux protéines transmembranaires (Figure 8).
![Figure 1. Schéma représentant l’organisation de la réponse immunitaire innée, adaptée de [5].](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/33.892.118.781.248.761/figure-schéma-représentant-organisation-réponse-immunitaire-innée-adaptée.webp)
![Figure 5. Répartition des origines des peptides antimicrobiens chez les vertébrés selon l’Antimicrobial Peptide Database, [28]](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/38.892.178.718.237.556/répartition-origines-peptides-antimicrobiens-vertébrés-antimicrobial-peptide-database.webp)
![Figure 6. Transport d’une molécule cargaison « cargo » dans les cellules, adaptée de [65]](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/41.892.280.613.101.470/figure-transport-molécule-cargaison-cargo-cellules-adaptée.webp)
![Figure 20. Modèles de transport du lipopolysaccharide à travers la membrane des bactéries à Gram négatif : modèle de l’intermédiaire soluble (à gauche) et du complexe trans-enveloppe (à droite) [129]](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/57.892.112.783.222.721/modèles-transport-lipopolysaccharide-membrane-bactéries-négatif-intermédiaire-enveloppe.webp)
![Figure 22. Modifications possibles du lipide A chez les bactéries à Gram négatif pour lutter contre les molécules antibiotiques et antibactériennes [141].](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/59.892.109.814.590.1052/figure-modifications-possibles-bactéries-négatif-molécules-antibiotiques-antibactériennes.webp)
![Figure 32. Interaction des peptides antimicrobiens avec les deux types de membranes bactériennes lors de l’insertion des peptides dans les membranes [104]](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/66.892.189.702.503.926/interaction-peptides-antimicrobiens-membranes-bactériennes-insertion-peptides-membranes.webp)
![Figure 37. Différents modèles de translocation directe. A : formation de micelles inverses ; B : formation de pores transitoires, C : transfert de phase [59]](https://thumb-eu.123doks.com/thumbv2/123doknet/14704124.565681/73.892.110.786.186.509/modèles-translocation-formation-micelles-inverses-formation-transitoires-transfert.webp)