Je voudrais remercier tous ceux qui, de près ou de loin, m’ont aidée dans mon travail et m’ont soutenue tout au long de ma thèse. Merci.
RESUME
La famille des facteurs de transcription E2Fs joue un rôle paradoxal dans la régulation du devenir de la cellule, et en particulier dans le développement tumoral. Les facteurs de transcription E2Fs régulent dans le temps, l’expression de nombreux gènes nécessaires à l’entrée en phase S, à la réplication de l’ADN et à la progression du cycle cellulaire. Cependant, il est maintenant connu que l’activation transcriptionnelle par au moins un des membres de la famille, E2F1, peut aussi induire efficacement l’apoptose en réponse aux dommages à l’ADN. Un équilibre approprié entre régulateurs positifs et négatifs apparaît comme essentiel à la modulation de l’activité de tels facteurs, clés du devenir de la cellule en prolifération. Les mécanismes faisant basculer la balance d’un côté ou de l’autre, entre survie et apoptose, sont encore mal compris.
La protéine Api5 est connue dans la littérature pour ses propriétés anti apoptotiques (« Apoptosis inhibitor 5 ») et comme ayant une expression dérégulée et associée à un mauvais pronostic dans différents cancers. Elle a plus récemment été identifiée comme inhibiteur spécifique de l’apoptose dépendante du facteur de transcription E2F1, cependant le mécanisme d’action reste inconnu. Mes travaux de thèse ont porté sur l’étude d’Api5 et de sa fonction dans l’activité pro mitotique et pro apoptotique du facteur de transcription E2F1. Mes résultats mettent en évidence que la protéine Api5 participe à l’expression de gènes sous le contrôle transcriptionnel d’E2F1, nécessaires à la transition G1/S et à la progression du cycle cellulaire. Dans ce sens, nous avons montré que l’inhibition d’Api5 provoque un arrêt des cellules en phase G1 et un retard de croissance. En ce qui concerne l’activité pro apoptotique d’E2F1, il semblerait que la protéine Api5 influence également l’expression de cibles d’E2F1 pro apoptotiques périodiquement exprimés au cours du cycle cellulaire, sans induire l’apoptose, mais pas celle des cibles pro apoptotiques induites par E2F1 en réponse aux dommages à l’ADN. Quant aux efforts portés sur le mécanisme moléculaire mis en jeu, nos observations suggèrent qu’Api5 contribue à l’activité transcriptionnelle d’E2F1 en augmentant sa capacité à se fixer sur ses promoteurs cibles, mais ce de manière indirecte. Plusieurs partenaires d’interaction d’Api5 ont été identifiés. L’étude de leur rôle dans la relation fonctionnelle entre Api5 et E2F1, ainsi que celui des formes nucléaires du FGF2 (liaison directe publiée au laboratoire), est une piste prometteuse pour la suite de mon travail.
A ce stade, le message général qui apparaît est que la protéine Api5, déjà décrite comme inhibiteur de l’activité apoptotique du facteur de transcription E2F1, jouerait parallèlement en faveur d’une régulation positive de l’expression des gènes du cycle cellulaire cibles d’E2F1. Api5, fréquemment dérégulé dans les pathologies cancéreuses, pourrait donc être considéré comme un proto oncogène, statut souvent associé aux régulateurs positifs du cycle. Enfin, de façon intéressante, l’invalidation d’Api5 la souris induit une létalité in utero à des stades embryonnaires très précoces, ce qui révèle son importance dans la prolifération cellulaire et ainsi qu’une absence de redondance fonctionnelle qui pourrait compenser sa perte.
SOMMAIRE
RESUME 1
SOMMAIRE 3
LISTE DES ABRÉVIATIONS 7
PROBLEMATIQUE 9
INTRODUCTION 11
1ÈRE PARTIE: L’INHIBITEUR D’APOPTOSE API5 13
I L’INHIBITEUR D’APOPTOSEAPI5 15
A Généralités 15
1. Le gène API5 15
2. La protéine Api5 17
B Api5, Facteur de Survie 21
1. Mise en évidence d’Api5 en tant que facteur de survie 21
2. Api5 dans la signalisation du FGF2 et de ERa 22
C Api5 et Apoptose 25
1. Api5, régulateur positif de la mort programmée de la cellule chez Oryza sativa 25 2. Identification fonctionnelle d’Api5 comme suppresseur de l’apoptose induite par E2F1 chez la
Drosophile 26
3. Api5 régule la fragmentation de l’ADN en interagissant et en inhibant Acinus dans la cellule
humaine 28
D Api5 dans le Cancer 30
2ÈME PARTIE: LE FACTEUR DE TRANSCRIPTION E2F1 33
II LEFACTEUR DETRANSCRIPTIONE2F1, PROTÉINECLE DUDEVENIR DE LACELLULE 35
A Généralités 35
B La famille E2F chez les eucaryotes superieurs 37
1. Les E2Fs activateurs 37
2. Les E2Fs répresseurs 39
3. Les E2Fs atypiques 40
4. Les cofacteurs DP 41
C Régulation Transcriptionnelle par les E2Fs 42
1. Répression de la transcription 43
2. Activation de la transcription par les facteurs E2Fs 46
3ÈME PARTIE: E2F1 DANS LE CYCLE CELLULAIRE 49
III E2F1 DANS LE CYCLE CELLULAIRE 51
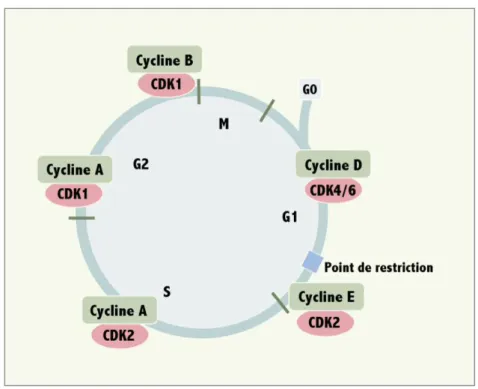
A Le Cycle Cellulaire 51
1. Généralités 51
2. Bases Moléculaires de la Régulation du Cycle Cellulaire 53
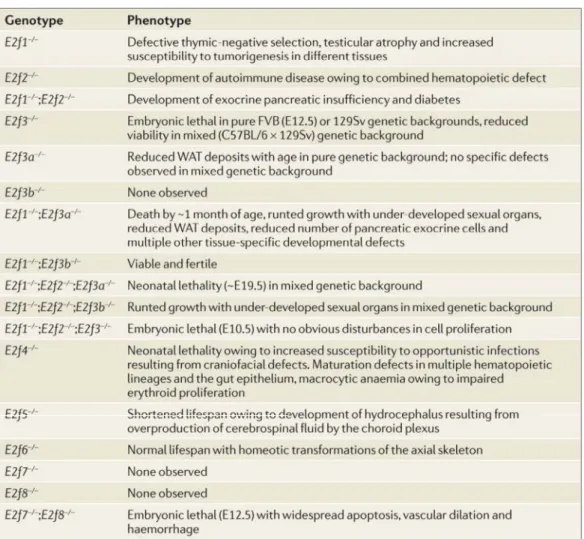
3. Quelques modèles murins du cycle cellulaire 56
B Contrôle du cycle cellulaire par E2F1 57
1. Contrôle de la transition G1/S 58
2. Régulation de la phase S et de la réplication de l’ADN 60
3. Régulation de la mitose 62
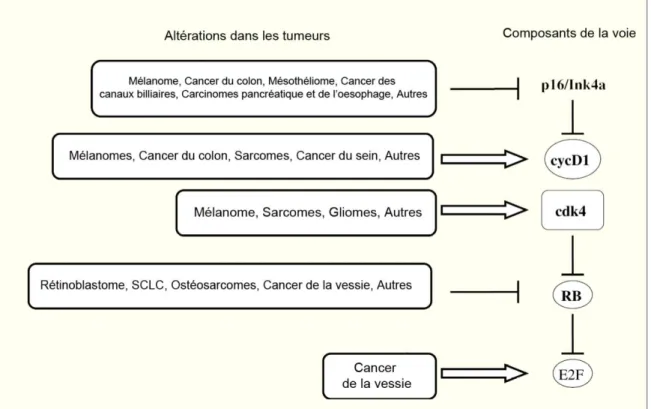
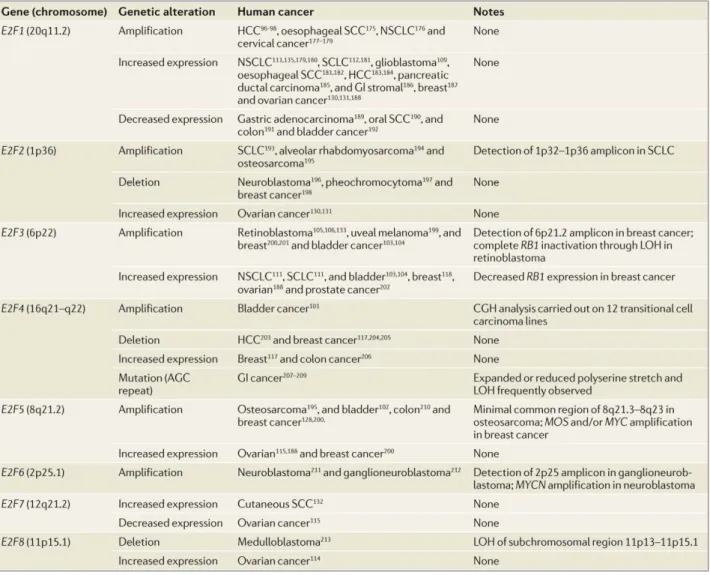
C E2F1 et Cancer 63
1. Protéines régulatrices du cycle cellulaire et cancer : proto oncogènes, oncogènes et suppresseurs
de tumeurs 63
2. La voie E2F/RB dans le cancer 65
3. E2F1 : Oncogène ou Suppresseur de Tumeur 69
4ÈME PARTIE: E2F1 ET APOPTOSE 75
IV INDUCTION DE L’APOPTOSE PAR E2F1 77
A Activation d’E2F1 en Réponse aux Dommages à l’ADN 77
1. Fonction transcriptionnelle d’E2F1 dans les points de contrôle et dans la réparation de l’ADN 77
2. Activation d’E2F1 en réponse à un stress génotoxique 79
B Les Différentes Voies Apoptotiques Régulées par E2F1 82
1. E2F1 active la famille p53 82
2. Induction de la voie apoptotique mitochondriale 84
3. Inhibition des facteurs de survie 86
4. Voies non canoniques de mort cellulaire régulées par E2F1 88
RESULTATS 89
1ÈRE PARTIE: REGULATION DE L’ACTIVITE TRANSCRIPTIONNELLE DU FACTEUR E2F1
PAR API5 91
I REGULATION DE L’ACTIVITE TRANSCRIPTIONNELLE DU FACTEUR E2F1PAR API5 93 A API5 contribue a l’activité transcriptionnelle d’E2F1 au moment de la transition G1/S du cycle
cellulaire 93
Rationnel 93
Objectifs 94
B Effets supplementaires d’Api5 et des hmwFGF2 sur l’activite trancriptionnelle d’E2F1 121
Objectifs 121
Matériel et méthodes 122
1. Effet d’Api5 dans l’expression des cibles pro apoptotiques d’E2F1. 123 2. Effet d’Api5 sur la transcription E2F1 dépendante dans un contexte de stimulation de l’activité
d’E2F1 par un stress génotoxique 125
3. Effet d’Api5 sur l’apoptose des cellules HeLa et H1299 127 4. Rôle du FGF2 dans l’interaction fonctionnelle entre Api5 et E2F1 130
2ÈME PARTIE: RESULTATS COMPLEMENTAIRES 133
II CARACTERISATION D’API5 DANS LA CELLULE ET DANS UN MODELE MURIN 135
A API5 est associe a des complexes protéiques nucleaires 135
Rationnel 135
Objectifs 135
Matériel et méthodes 136
Résultats 136
B Caractérisation de differentes isoformes d’Api5 139
Rationnel 139
Objectifs 139
Matériel et méthodes 140
Résultats 141
C La souris knockout API5 149
Rationnel 149
Objectifs 149
Matériel et méthodes 149
Résultats 151
CONCLUSIONS, DISCUSSIONS, ET PERSPECTIVES 155
I. Rôle de Api5 dans la régulation transcriptionnelle par E2F1. 157 II. Possible boucle de régulation de l’expression entre le FGF2 et le couple Api5/E2F1. 166 III. Api5 appartient à un ou plusieurs complexes protéiques nucléaires. 167 IV. L’épissage alternatif du transcrit Api5 génère 11 isoformes protéiques. 169 V. L’invalidation d’Api5 chez la souris est létale in utero. 169
ANNEXE 195 CONTRIBUTION A D’AUTRES TRAVAUX : ROLES DE SNX6 ET RANBPM DANS LA NUCLEARISATION
DU FGF2 INDUITE PAR LA TRANSLOKINE 197
LISTE DES ABREVIATIONS
aa : acides aminés
AAC 11: antiapoptotic clone 11 ADN : acide déoxyribonucléique ADNc : ADN complémentaire
APC: Anaphase Promoting Complex AIP1/2: Api5 Interacting Protein 1/2 Api5 : Apoptosis inhibitor 5
ARN: acide ribonucléique ARNm: ARN messager ARNr: ARN ribosomique
ATM : Ataxia Telangectasia Mutated ATR : ATMRad3 related
ATP: adenosine tri phosphate Cdk: Cyclin dependent kinases
ChIP : Immunoprécipitation de Chromatine CKI : Cdk inhibitors
E2F: E2 promoter binding factor Era: Estrogen receptor alpha FGF: fibroblast growth factor FGF2: fibroblast growth factor2 FGF2BP: FGF2 binding protein FBR1/2: FGF2 Binding Region 1/2 FGFR: FGF receptor
FIF: FGF2 interaction factor FOXO3 : Forkhead box factor 3 G1/2 : gap 1/2
GFP: green fluorescent protein HMW: High molecular weight Kb: kilobases
KDa: kilo Dalton KO : knockout
LMW: Low molecular weight LZ : leucine zipper
M : mitosis
NFkB : nuclear factor kappa B NLS: nuclear localization sequence
P/CAF : p300/CREB binding protein associated factor pb : paire de base
PCNA : proliferating cell nuclear antigen PBS : Phosphate buffered saline
pRb: protéine du rétinoblastome RBF1/2 : retinoblastoma factor ½ RBL1/2 : Retinoblastoma Like 1/2 RE : Réticulum Endoplasmique
RT PCR: reverse transcription polymerase chain reaction S : synthesis
siRNA: small interfering RNA TBP : TATA Box Binding Protein
TGFb : transforming growth factor beta TNFa : Facteur de nécrose tumorale alpha VHL: Von Hippel Lindau
PROBLEMATIQUE
L’équipe du Professeur Hervé Prats travaille depuis plusieurs années sur les mécanismes de la régulation de l’expression du FGF2, ainsi que sur son rôle dans la cellule, et plus précisément, dans la progression tumorale.
Les grandes isoformes du FGF2 (hmwFGF2) sont des formes nucléaires du facteur de croissance qui ont été impliquées dans la prolifération et la migration cellulaires, événements contribuant à la progression tumorale. Dans le but d’élucider les mécanismes d’action des hmwFGF2, la recherche de complexes comprenant les hmwFGF2 a permis l’identification de l’inhibiteur d’apoptose Api5.
La protéine Api5 a été décrite comme étant surexprimée et associée à un mauvais pronostic dans de nombreuses pathologies cancéreuses. Plus récemment, Api5 a été identifiée comme étant un inhibiteur de la voie apoptotique induite par le facteur de transcription E2F1. A ce jour, aucun mécanisme moléculaire expliquant l’interaction fonctionnelle entre ces deux protéines n’a été proposé.
Dans ce contexte, l’étude de la relation fonctionnelle entre Api5, les hmwFGF2 et E2F1 a été entreprise, afin d’identifier un mécanisme moléculaire reliant ces trois facteurs impliqués dans les processus tumoraux.
1
EREPARTIE
:
I L’INHIBITEUR D’APOPTOSE API5
A
G
ENERALITES
1. Le gène API5
Chez l’Homme, le gène API5 est localisé sur le chromosome 11 (11p11.2) et est constitué de 15 exons. Son expression conduit à la synthèse d’un ARN pré messager qui peut subir plusieurs types d’épissages alternatifs. Il existe également dans le génome humain deux pseudo gènes, API5P1 et API5P2, localisés sur les chromosomes X (Xq24) (Gianfrancesco et al, 1999) et 2 (2q31.2) respectivement.
L’ADN complémentaire (ADNc) codant pour la protéine Api5 a été initialement isolé par Tewari et al. en 1997, lors d’un crible visant à identifier des facteurs de survie à partir d’une banque d’ADNc murine (Tewari et al, 1997). Le produit fut appelé AAC 11 (« Anti Apoptotic Clone 11 »). La protéine humaine homologue, identifiée à partir de la séquence murine, présente 98% d’identité. En parallèle à cette étude, un projet de recherche a été mené dans mon équipe dans le but d’identifier des partenaires d’interaction intracellulaires du FGF2 par un crible double hybride chez la levure. A partir de ce crible, ils ont alors cloné un ADNc humain de séquence (quasi) identique à celle de l’ADNc décrite par Tewari et al. La protéine issue de cet ADNc fut appelée FIF, pour « FGF2 Interacting Factor » (Van den Berghe et al, 2000). Dans la littérature, on retrouve les trois appellations : Api5, AAC 11 et FIF, bien que le nom Api5 (« Apoptosis inhibitor 5 ») soit le plus utilisé.
Dans les travaux de Tewari et al. et Van den Berghe et al., le profil d’expression du gène API5 a été analysé par Northen blot dans de nombreux tissus murins et humains (cœur, placenta, cerveau, poumon, foie, muscles squelettiques, pancréas, rein, rate et testicules) et à différents stades de développement du rat (E15, E19 et D4 après naissance) (Tewari et al, 1997; Van den Berghe et al, 2000). Le gène API5 présente une expression ubiquitaire. Chez
l’Homme, l’ARN messager (ARNm) d’Api5 apparaît relativement plus abondant dans le cœur, le pancréas et le placenta. Chez le rat, le transcrit du gène API5 est autant exprimé pendant le développement fœtal qu’à un stade postnatal précoce.
Ces deux premiers travaux ont permis d’avoir une appréciation de la conservation du gène API5 dans différentes espèces (souris, rat, singe, taureau, homme). Depuis, d’autres études chez la mouche (Drosophila melanogaster) et chez la plante (Oryza sativa) ont permis de compléter ces données. Notamment dans le travail réalisé chez le riz, les auteurs ont construit un arbre phylogénique (Figure 1) (Li et al, 2011; Morris et al, 2006) où l’on peut apprécier la conservation du gène API5 des protistes aux animaux.
A ce jour, il n’a pas été décrit de souris « knockout » pour le gène API5.
2. La protéine Api5
Il existe dans la cellule humaine plusieurs isoformes protéiques d’Api5 issues d’épissages alternatifs. In silico, l’information répertoriée dans les banques de données reflète un épissage alternatif complexe du transcrit du gène d’Api5. Le nombre et la séquence des différentes isoformes sont parfois divergents et de ce fait confus. Il n’est donc pas surprenant que ces différentes isoformes aient aléatoirement été utilisées lors des études.
A l’origine, trois isoformes de 510, 504 et 445 acides aminés (aa) avaient été identifiées à partir d’une banque d’ADNc de lymphocytes humains et des séquences EST retrouvées dans les banques de données (Van den Berghe et al, 2000). A ce jour, sur le site de la NCBI, deux autres isoformes d’Api5 (GeneID: 8539) de 524 et 331aa sont répertoriées ; la forme de 524aa étant considérée comme l’isoforme majeure.
Le clone AAC 11long murin, ainsi que son homologue humain, identifiés par Tewari et al. correspondent à la forme de 504aa. Dans ce travail, les auteurs avaient initialement mis en évidence un ADNc murin codant pour une protéine de 230aa, appelée AAC 11short. C’est cette
protéine qui a été au centre de leurs analyses. Les auteurs ont discuté la possibilité que cette forme de 230aa soit une forme tronquée de AAC 11long (504aa), un artefact apparu au
moment du clonage (Tewari et al, 1997). Cette hypothèse aurait de grandes conséquences sur leur étude puisque dans leurs travaux c’est l’isoforme AAC 11short, et non pas l’isoforme AAC
11long, qui se comporte comme un facteur de survie. Actuellement, l’isoforme AAC 11short
n’apparaît plus dans les banques de données. Par la suite, différents groupes ont travaillé avec des formes d’Api5 chez l’Homme assimilables à la protéine 524aa, alors que les travaux faits chez la Drosophile et le riz ont utilisé des homologues d’Api5 de 536/537aa (Kim et al, 2000; Li et al, 2011; Morris et al, 2006; Rigou et al, 2009).
D’une façon générale, ce sont les derniers exons de l’extrémité 3’ de l’ARN d’Api5 qui sont le siège des épissages alternatifs. Les différentes isoformes protéiques ont souvent la même extrémité N terminale et possèdent grande partie de leur séquence en commun. Dans le cas des formes de grande taille (504, 510 et 524aa chez l’Homme), ces isoformes comportent les mêmes domaines protéiques conservés. A ce jour, aucune différence fonctionnelle n’a été décrite pour ces trois isoformes.
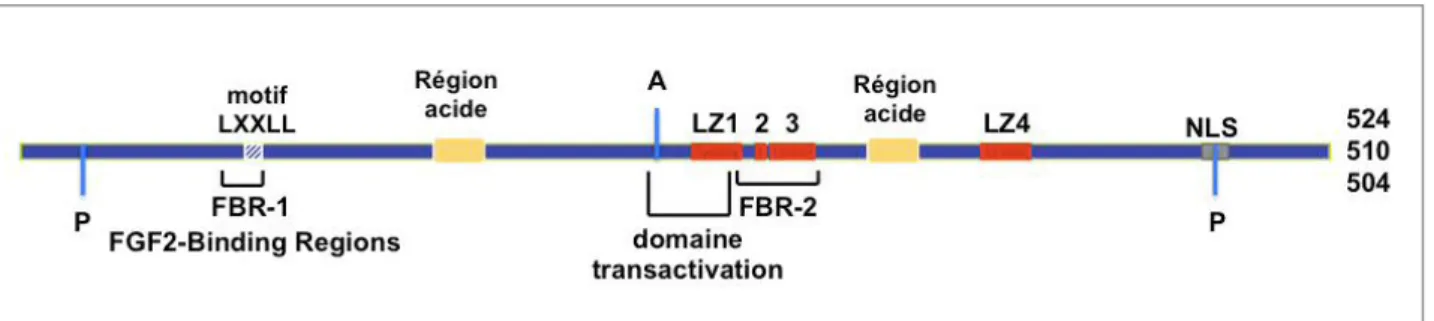
La protéine Api5 apparaît être une protéine structuralement très conservée dans les organismes étudiés. De l’extrémité N terminale à l’extrémité C terminale, la protéine Api5 contient (Figure 2) :
Un motif LXXLL (L102 L106), où L est une leucine et X n’importe quel acide aminé. Ce motif est présent dans de nombreux facteurs de transcription et co facteurs. Il est associé à différents aspects de la régulation transcriptionnelle (positive et négative) (Plevin et al, 2005). Plus particulièrement, il a été décrit comme étant impliqué dans l’interaction entre des co activateurs transcriptionnels et les récepteurs nucléaires (Heery et al, 1997). Le motif LXXLL se trouve au sein de la région FBR1 (« FGF2 Binding Region 1 », résidus 96 à 107), une des deux régions nécessaires à la liaison directe d’Api5 avec les FGF2. Cependant, ce motif n’est pas impliqué dans l’interaction Api5/FGF2 (Van den Berghe et al, 2000).
Deux régions acides (résidus 161 à 190 et 323 à 335). Chez les facteurs de transcription, les domaines acides sont souvent associés avec la fonction transactivatrice. Dans le cas d’Api5, il a été suggéré que ces régions pourraient établir des interactions régulatrices au sein de la protéine, notamment avec le domaine de transactivation (Van den Berghe et al, 2000).
Un domaine de transactivation entre les deux régions acides (résidus 231 à 273). Ce domaine a été mis en évidence en utilisant des constructions avec des formes tronquées d’Api5 (Van den Berghe et al, 2000). Il a été observé que les protéines Api5 tronquées, dont l’extrémité COOH est absente, sont capables d’activer l’expression du gène rapporteur LacZ. Cependant, cette capacité transactivatrice n’est pas retrouvée avec une construction Api5 pleine taille. Ainsi, la région transactivatrice est probablement masquée dans protéine pleine taille. Les auteurs ont émis l’hypothèse qu’une modification post traductionnelle et/ou des changements de la conformation de la protéine pouvaient exposer et activer le domaine de transactivation.
Trois domaines leucine zipper non canoniques, ou autrement dit, trois régions avec des répétitions d’heptamères hydrophobes (259 287, 263 284 et 286 313). L’un de ces domaines contient la deuxième région nécessaire à l’interaction avec le FGF2, appelée FBR2 (résidus 274 à 311) (Van den Berghe et al, 2000).
Un domaine leucine zipper canonique (résidus 370 à 391). Les domaines leucine zipper, pour « fermeture éclair à leucine », sont des motifs structuraux tridimensionnels des protéines. Ils consistent en la répétition de plusieurs résidus leucines à des intervalles
d’approximativement 7 résidus, ce qui forme une hélice alpha amphipathique qui expose une face hydrophobe. Ces motifs fonctionnent comme des domaines de dimérisation. L’interaction débute par contact hydrophobe, ce qui génère des ponts entre les leucines des hélices alpha de deux protéines. Ces domaines sont fréquemment retrouvés au sein des domaines de liaison à l’ADN de différents facteurs de transcription, et sont ainsi impliqués dans la régulation de l’expression de gènes. Dans ce cas, la région hydrophobe est requise pour la liaison à l’ADN.
La dimérisation d’Api5 a été démontrée par Rigou et al. L’oligomérisation d’Api5 fait bien intervenir le domaine leucine zipper et semble avoir un rôle fonctionnel (Rigou et al, 2009).
Une NLS, pour « Nuclear Localisation Sequence », localisée en C terminal (résidus 454 à 474). Cette séquence est similaire à celle retrouvée dans le suppresseur p53. Elle confère une localisation nucléaire à Api5 (Van den Berghe et al, 2000). Certaines isoformes d’Api5, telle que la forme de 445aa (aussi appelée C1), n’ont plus cette séquence en raison d’un épissage alternatif et sont alors cytoplasmiques.
Figure 2. Structures conservées de la protéine Api5.
La protéine Api5 possède dans sa séquence : un motif LXXLL (boîte rayé), deux domaines de liaison au FGF2 (FBR 1/2), deux régions acides (en jaune), un domaine de transactivation, trois domaines « leucine zipper like » (LZ1, 2 et 3 en rouge), un domaine leucine zipper canonique (LZ4 en rouge) et une NLS (en gris). La protéine Api5 peut être phosphorylée (P) et acétylée (A). Les trois isoformes de grande taille (de 524, 510 et 504aa) partagent les mêmes domaines protéiques.
Des analyses récentes de spectrométrie de masse en haut débit ont permis d’identifier des modifications post traductionnelles d’Api5.
Plusieurs travaux montrent que la protéine Api5 peut être phosphorylée au niveau des résidus tyrosine 28 et sérine 464 (Gauci et al, 2009; Kim et al, 2005; Olsen et al, 2006). La
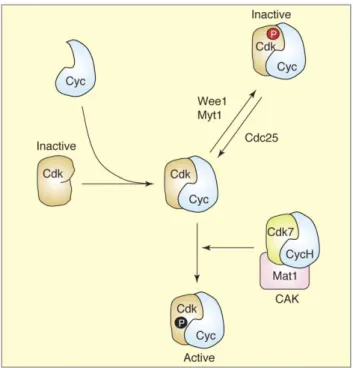
phosphorylation putative de la sérine 464, curieusement au sein de la NLS, est la plus décrite et a été mise en relation avec la régulation du cycle cellulaire. En effet, la séquence entourant cette sérine (SEDTTSGSPPKKSSA) laisse prévoir la reconnaissance et la phosphorylation par la kinase CK1 (S/T X X X S) ainsi que par les kinases Cdk1, Cdk2 et Cdk5 (S/T P X K/R). Ces kinases sont impliquées dans la signalisation et le contrôle de la progression du cycle cellulaire. Leur activité est transitoire car régulée temporellement, en fonction du stade du cycle cellulaire. Ceci laisse supposer le même type de régulation pour la phosphorylationdu résidu S464 d’Api5.
La protéine Api5 a été identifié comme une cible spécifique de la famille des kinases Pim (Peng et al, 2007). Les kinases Pim, qui sont considérées comme des proto oncogènes, ont été impliquées dans la signalisation de la prolifération, la survie et la différenciation cellulaire.
Enfin, une étude de 2009 prédit une acétylation d’Api5 au niveau de la lysine 251 (Choudhary et al, 2009). L’enzyme responsable de cette modification n’a pas été identifiée.
Ces modifications post traductionnelles ont été répertoriées dans la base de données PHOSIDA. On peut y visualiser l’importante conservation de la séquence d’Api5 au niveau des résidus modifiés, dans différentes espèces.
Le nombre de publications cherchant à établir la fonction et les mécanismes moléculaire associés à la protéine Api5 est faible. Dans les chapitres qui suivent, je ferai intentionnellement le détail des sept publications que je considère essentielles à la contextualisation de la fonction d’Api5, ainsi qu’à la compréhension des démarches et des hypothèses que j’ai adoptées lors de mes travaux de recherche. Dans une dernière partie, je ferai la synthèse du statut d’Api5 dans différentes pathologies cancéreuses.
B
A
PI
5, F
ACTEUR DE
S
URVIE
1. Mise en évidence d’Api5 en tant que facteur de survie
En 1997, Tewari et al. ont publié le résultat d’un crible fonctionnel cherchant à identifier de nouveaux gènes codant pour des facteurs de survie (Tewari et al, 1997). Ils ont travaillé dans des cellules murines BALB/3T3, avec une banque d’ADNc murine. Ce crible a permis de mettre en évidence un ADNc murin de 1023 paires de bases (pb) qui code pour une protéine de 230aa, appelé AAC 11short. En utilisant une partie de la séquence AAC 11short
comme sonde pour cribler leur banque murine, ils ont isolé un deuxième ADNc de plus grande taille, AAC 11long, qui code pour une protéine de 504aa. Cette protéine possède un homologue
humain de même taille qui présente 98% d’identité.
De façon intéressante, alors que 95% des cellules BALB/3T3 contrôle meurent au bout de 10 12 jours de privation en sérum, 40% des cellules transfectées avec AAC 11shortrésistent
jusqu’à 15 30 jours dans les mêmes conditions. Certaines cellules transfectées avec AAC 11shortsont viables jusqu’à 4 mois en absence de sérum et sont même capables de proliférer
jusqu’à confluence après addition de sérum. Cette propriété est abolie quand le motif leucine zipper est muté, ce qui démontre un rôle essentiel de ce motif dans l’activité biologique de la protéine.
De façon contrastée, dans cette même expérience, les cellules transfectées avec AAC 11long ont une mortalité deux fois supérieure aux cellules contrôle (100% de mortalité
après 4 jours sans sérum). Le clone AAC 11long contient 274aa additionnelles en N terminal
par rapport à AAC 11short. Les deux formes possèdent le domaine leucine zipper, cependant
seule la petite isoforme semble agir comme un facteur de survie. Les auteurs proposent un rôle pro apoptotique pour AAC 11long, dont l’activité serait régulée par AAC 11short à la
manière d’un dominant négatif.
Par ailleurs, comme j’ai déjà commenté, il a été envisagé que cette forme de 230aa, AAC 11short, soit une forme tronquée de AAC 11long(504aa), un artefact apparu au moment du
clonage. Cette hypothèse aurait de grandes conséquences sur leur étude, puisque dans leurs travaux, seule l’isoforme AAC 11short se comporte comme un facteur de survie. L’amalgame
apoptotiques de AAC 11shorten absence de sérum sont fréquemment attribuées à tort à AAC
11long.
Suite à ce travail, une étude cherchant à identifier des gènes associés au cancer du col de l’utérus humain a mis évidence en une expression différentielle d’Api5 (Kim et al, 2000). L’ARNm d’Api5 (dans le temps assimilé comme AAC 11 de Tewari et al., et associé à une fonction de facteur de survie) se retrouve exprimé dans les métastases des ganglions lymphatiques et dans les lignées cellulaire du cancer du col de l’utérus ; alors qu’il est absent dans les tissus sains et dans les tumeurs primaires de cancer cervical.
Les auteurs ont cloné l’isoforme humaine de 524aa d’Api5. Ils ont établi des lignées stables CUMC 6 de cancer du col de l’utérus surexprimant cette isoforme. La surexpression d’Api5 confère aux cellules une plus grande capacité d’invasion et d’adhésion (deux fois plus que les cellules contrôle). Cette capacité semble mettre en jeu l’activation de l’expression des métalloprotéases MMP 2 et MT1 MMP, retrouvées surexprimées dans les tumeurs invasives. De plus, la surexpression d’Api5 inhibe l’expression de TIMP 1, une protéine inhibitrice des métalloprotéases, considérée comme un suppresseur de tumeurs. Dans un milieu de culture sans sérum, les cellules CUMC 6 surexprimant Api5 survivent deux fois plus longtemps que les cellules contrôle (8 semaines vs. 4 semaines). Ce résultat semble en contradiction avec celui de Tewari et al. Cependant, la différence de résultat obtenu pourrait être expliquée par le modèle cellulaire utilisé mais aussi par l’isoforme d’Api5 utilisée (isoforme de 524aa humaine pour Kim et al. et isoforme de 504aa murine pour Tewari et al.).
2. Api5 dans la signalisation du FGF2 et de ERa
Le FGF2 (« Fibroblast Growth Factor 2 ») fait partie d’une famille de facteurs de croissance qui agissent sur un grand nombre de types cellulaires en stimulant la prolifération et en induisant des changements morphologiques et la différenciation. Plus précisément, le FGF2 est impliqué dans des processus développementaux, la cicatrisation, l’angiogenèse, mais aussi dans la tumorigènese. Chez l’Homme, cinq isoformes de 18, 22, 22.5, 24 et 34 kDa sont synthétisées à partir d’un même ARNm, selon l’utilisation alternative de différents codons d’initiation de la traduction (Cohen Jonathan et al, 1997). Ces isoformes diffèrent uniquement par leur extrémité N terminale. La petite isoforme de 18kDa se localise au niveau du
cytoplasme et de la matrice extracellulaire, alors que l’extension amino terminale confère une localisation nucléaire aux quatre isoformes de haut poids moléculaire (hmwFGF2, pour « High Molecular Weight FGF2 »).
Les hmwFGF2 sont impliquées dans la prolifération cellulaire et dans la tumorigenèse. La plus grande forme de 34kDa a été caractérisée comme étant facteur de survie. Elle est exprimée essentiellement dans des lignées cancéreuses humaines et induit une grande capacité de survie cellulaire aux cellules NIH3T3 en milieu sans sérum (Arnaud et al, 1999). La surexpression des autres formes de haut poids moléculaire conduit à l'immortalisation des cellules endothéliales d'aorte bovine ainsi qu’à une radiorésistance accrue des cellules transformées (Cohen Jonathan et al, 1997). La surexpression de la forme de 24kDa dans les cellules de carcinome de vessie de rat NBTII induit leur dissémination métastatique. De plus, l’injection intraveineuse de cellules exprimant la forme de 24kDa induit la formation de métastases importantes au niveau des poumons de souris nude (Okada Ban et al, 1999). Enfin, la co expression des différentes formes de FGF2 provoque le développement de tumeurs chez la souris (Couderc et al, 1991).
Les mécanismes moléculaires par lesquels les FGF2 endogènes exercent leurs effets intracellulaires restent mal compris. Des données préliminaires montrent que les hmwFGF2 sont associées à des complexes protéiques dans le noyau. Selon le rationnel que les partenaires d’interaction doivent refléter les activités biologiques intracrines des hmwFGF2, mon équipe a réalisé un crible double hybride dans la levure, utilisant le FGF2 comme appât, afin d’identifier des partenaires d’interaction. Ils ont alors identifié la protéine FIF, pour « FGF2 Interacting Factor », maintenant connue sous nom d’Api5 (Van den Berghe et al, 2000). Ce travail a permis de montrer qu’Api5 interagit directement et spécifiquement avec les isoformes de hauts poids moléculaire du FGF2. Cette interaction est dépendante de deux sites de liaison, FBR1 et FBR2, au niveau de la protéine Api5. Par ailleurs, cette étude a permis de cartographier la séquence nucléotidique et protéique d’Api5 et d’étudier son profil d’expression (données utilisées dans les chapitres précédents). Les auteurs ont envisagé une homodimérisation fonctionnelle d’Api5 qui exposerait un domaine de transactivation, masqué dans le monomère. De façon similaire, l’association d’Api5 avec les FGF2 nucléaires pourrait provoquer un changement de conformation d’Api5 et rendre le dimère actif.
Une étude postérieure en collaboration avec mon équipe a démontré l’implication des hmwFGF2 et Api5 dans la voie de signalisation des œstrogènes (Garmy Susini et al, 2004).
Les œstrogènes ont été décrits comme stimulant l’activité angiogénique ainsi que la migration et la prolifération des cellules endothéliales. L’effet angiogénique de ces hormones passe par la signalisation du récepteur aux œstrogènes alpha (ERa), récepteur nuclé ire respons ble de l’ ctiv tion tr nscriptionnelle d’un gr nd nombre de gènes.
L’utilis tion de souris inv lidées pour le gène du FGF2 permit de montrer que les FGF2 endogènes sont essentiels ux effets des œstrogènes sur l migr tion et l prolifér tion des cellules endothéli les. Ces effets ne sont p s dépend nts de l’isoforme du FGF2 de 18kD , m is semblent être contrôlés p r les hmwFGF2. De plus, les uteurs ont observé que les œstrogènes ffectent l’ bond nce des récepteurs ux FGFs, des isoformes du FGF2 et de f çon très intéress nte, celle d’Api5. L’ARNm et l protéine Api5 s’ ccumulent sous l’effet des œstrogènes. L suppression p rtielle d’Api5, en utilis nt des siARNs, ttenue les effets des œstrogènes sur l migr tion des cellules endothéli les FGF2+/+ et bolit complètement les
effets des œstrogènes u niev u des cellules lmwFGF2/ (c’est à dire, n’exprim nt que les
hmwFGF2). L migr tion des cellules endothéli les sous l’effet des œstrogènes p sse donc p r les hmwFGF2 et Api5.
L’étude du rôle d’Api5 d ns l sign lis tion des œstrogènes p ss nt p r le récepteur ERa constitue le projet de recherche du Dr. Céline B sset, médecin n tomop thologiste ré lis nt s thèse u sein de l’équipe. Des résult ts encour ge nts ont étés obtenus. Api5 est fonctionnellement impliquée d ns les ctivités biologiques du récepteur ERa et d ns l régul tion tr nscriptionnelle de ses gènes cibles. Les deux protéines inter gissent directement et sont c p bles de réguler réciproquement leur expression.
C
A
PI
5
ET
A
POPTOSE
1. Api5, régul teur positif de l mort progr mmée de l cellule chez Oryza
sativa
Une étude chinoise très récente, y nt identifié l’homologue d’Api5 chez le riz (Oryza sativa), ccorde à Os Api5 un rôle de régul teur tr nscriptionnel du processus de dégénérescence nécess ire à l form tion des g mètes mâles (Li et l, 2011).
Le t petum est le tissu nourricier d ns l p roi des nthères permett nt l m tur tion des gr ins de pollen. L dégénérescence contrôlée du t petum est essentielle pour le développement des g métophytes mâles. Elle est ssimilée à un processus de mort cellul ire progr mmée. Des déf uts d ns l dégénérescence contrôlée du t petum entr înent l stérilité mâle des pl ntes.
En ré lis nt un crible génétique à l recherche de gènes nécess ires à l form tion des g métophytes mâles, Os Api5 été identifié comme ét nt un régul teur positif de l dégénérescence du t petum. Le contrôle de ce processus développement l p r Os Api5 se f it
u nive u tr nscriptionnel. L protéine Os Api5 inter git directement vec deux ARN hélic ses ATP dépend ntes de l f mille « DEAD box », AIP1 et AIP2 (« Api5 Interacting Protein »). L f mille de protéines « DEAD box » est impliquée d ns les processus mét boliques des ARN (m tur tion des ARNr, ssembl ge des ribosomes, tr nscription/épiss ge/tr nsport des ARNm). Le complexe Os Api5 AIP1/2 est c p ble de se fixer u nive u du promoteur du gène CP1, effecteur de l mort cellul ire progr mmée, et contrôler insi son expression. L li ison du complexe Os Api5 AIP1/2 u nive u du promoteur de CP1 stimule s tr nscription, ce qui conduit à l’initi tion du processus de dégénérescence du t petum.
Les uteurs discutent le f it que leur étude démontre qu’Os Api 5 est un régul teur positif de l mort cellul ire progr mmée ; lors que les tr v ux chez l’Homme, montrent qu’Api5 un rôle inverse, en t nt que f cteur de survie et inhibiteur de l’ poptose. Conscients de ce rôle opposé, ils concluent que l fonction d’Api5 d ns l mort progr mmée de l cellule est l rgement conservée u cours de l’évolution, m lgré les v ri tions d ns son ctivité
2. Identific tion fonctionnelle d’Api5 comme suppresseur de l’ poptose
induite p r E2F1 chez l Drosophile
Les f cteurs de tr nscription de l f mille E2F (« E2 promoter binding factor ») sont des régul teurs import nts de l progression du cycle cellul ire. Ils sont les cibles m jeures de l f mille de l protéine du rétinobl stome (pRb), suppresseurs de tumeurs. L voie de sign lis tion de pRb est fonctionnellement in ctivée d ns l qu si tot lité des cellules tumor les. Cette dérégul tion provoque des nom lies d ns l’expression et l’ ctivité des f cteurs E2Fs, provoqu nt une prolifér tion cellul ire incontrôlée.
En ddition à leur c p cité à réguler le cycle cellul ire, il est m inten nt connu que l’ ctiv tion tr nscriptionnelle d’ u moins un des membres de l f mille, E2F1, peut ussi induire effic cement l’ poptose. L’ ctivité poptotique d’E2F1 limite les conséquences d’une ctivité dérégulée des E2Fs u nive u des tissus. Cepend nt, le processus tumorigène sélectionne non seulement des mut tions de l voie de sign lis tion de pRb, m is ussi des mut tions qui suppriment l’ ctivité poptotique d’E2F1.
D ns le but d’identifier des gènes dont le produit modifie l’ ctivité poptotique d’E2F1, le groupe de Morris et al. mis en pl ce un crible génétique in vivo, en utilis nt l Drosophile comme système modèle (Morris et l, 2006).
En effet, l’import nce de l voie E2F1/Rb est soulignée p r s conserv tion d ns les espèces u cours de l’évolution. L Drosophile possède une version « simplifiée » de l voie E2F/Rb, ce qui f it d’elle un modèle expériment l intéress nt. Chez l Drosophile, l régul tion tr nscriptionnelle p r les E2Fs est simplifiée du f it qu’elle ne possède que deux gènes e2f, cod nt pour un f cteur dE2F1 ctiv teur et un f cteur dE2F2 répresseur (Figure
3). Deux protéines homologues à pRb hum in, RBF1 et RBF2 régulent l’ ctivité des deux
f cteurs dE2Fs.
D ns un premier temps, le crible génétique de ce tr v il consisté à générer de lignées tr nsgéniques de Drosophiles, où une surexpression tissu dépend nte de dE2F1 provoque des phénotypes visibles et mesur bles. Le choix s’est porté sur une lignée de Drosophile où dE2F1 est sous le contrôle du promoteur Actin 88F, spécifiquement ctif pend nt le développement de l mouche. Ces mouches tr nsgéniques présentent des déf uts visibles u nive u des iles, insi que d’ utres phénotypes. Ces phénotypes sont dus à une poptose
import nte et ectopique induite p r dE2F1. Ils peuvent être compensés p r l co expression de RBF1.
Figure 3. La voie E2F/RB chez les drosophiles.
La voie E2F/RB est conservée chez la Drosophile, mais contient moins de membres que la voie E2F/RB chez les mammifères. Le modèle d’interaction est similaire entre les deux groupes : RBF1 est capable de lier les deux dE2Fs, l’activateur et le répresseur ; alors que RBF2 ne lie que le dE2F répresseur. (d’après Dimova & Dyson, 2005)
L deuxième p rtie du crible consisté en l’insertion lé toire d’un élément tr nspos ble P d ns le génome des mouches « Actin 88F/dE2f1 ». L’insertion du tr nsposon peut engendrer des mut tions qui modifient le phénotype des iles des mouches « Actin 88F/dE2f1 », c’est à dire qui modifient l’ poptose induite p r dE2F1. C’est insi que le gène API5 été identifié chez l Drosophile. L’inv lid tion de dAPI5 d ns les mouches « Actin 88F/dE2f1 » ccroit le phénotype induit p r dE2F1, provoqu nt des déf uts m ssifs d ns les
iles.
Afin de confirmer ces résult ts in vitro, les uteurs ont d’ bord tr v illé d ns une lignée cellul ire de Drosophile. Ils ont montré que l’ poptose induite p r l surexpression de dE2F1 ét it ugmentée p r l’inhibition de l’expression de dApi5, de f çon comp r ble à l’inhibition de dRBF1 d ns ce même contexte. De plus, l’inhibition de dApi5 n’ ucune conséquence sur l’ ctiv tion tr ncriptionnelle du promoteur de PCNA, cible connue d’E2F1. Les uteurs émettent l’hypothèse que l’inter ction fonctionnelle entre Api5 et E2F1 se f it en v l de l’ ctivité tr nscriptionnelle d’E2F1. Cepend nt, ils soulignent l nécessité de tester d’ utres cibles promotrices pour conclure.
Ces observ tions ont été ég lement confirmées d ns des lignées cellul ires hum ines, ce qui permet de montrer l conserv tion de l’inter ction fonctionnelle entre E2F1 et Api5 u
cours de l’évolution. Ils ont choisi de tr v iller sur les cellules S os 2, modèle cellul ire où pRb et p53 sont délétés (l’ ctivité E2F1 n’est donc p s régulée et l’ poptose d ns ces cellules ne peut être induite que p r E2F1). D ns ce modèle cellul ire, l’ poptose induite p r l surexpression d’E2F1 est fortement diminuée qu nd Api5 est co surexprimé. L’inhibition de l’ poptose p r Api5 n’ ucune conséquence sur l’induction de l’expression de p14ARF et de l
Cycline E, cibles tr nscriptionnelles d’E2F1. Ce résult t démontre à nouve u que l’ poptose p r Api5 se f it en v l de l’ ctiv tion tr nscriptionnelle p r E2F1.
Enfin, les uteurs testent l c p cité d’Api5 à protéger les cellules d’une poptose induite p r des tr itements chimiques (c mptothécine, vinbl stine, st urosporine, roténone ou f cteur de nécrose tumor le – TNFa) ou p r l surexpression de p53 et p73. De f çon surpren nte, d ns ces contextes pro poptotiques, l’ ctivité d’Api5 n’est p s suffis nte pour bloquer l’ poptose. Ce résult t souligne l spécificité de l’ ctivité d’Api5 d ns l suppression de l’ poptose induite p r l surexpression d’E2F1.
Ce tr v il, élég mment ré lisé p r Morris et al., est à l b se des hypothèses de mon projet de recherche. J’ i cherché à ét blir les méc nismes molécul ires p r lesquels Api5 pourr it contrôler l’ ctivité p r dox le du f cteur E2F1, u nive u de s double fonction pro mitotique et pro poptotique. En p r llèle, j’ i voulu étudier le rôle de l’inter ction directe Api5/hmwFGF2, d ns rel tion fonctionnelle entre Api5 et E2F1.
L synthèse des inform tions bibliogr phiques concern nt l f mille E2F et leurs fonctions biologiques, insi que le rôle d’E2F1 d ns le contrôle du cycle cellul ire et d ns l’induction de l’ poptose, ser pprofondie d ns l suite de l’introduction.
3. Api5 régule l fr gment tion de l’ADN en inter giss nt et en inhib nt
Acinus d ns l cellule hum ine
Le groupe Rigou et al. poursuivi l’étude de l fonction nti poptotique d’Api5 d ns les cellules hum ines (Rigou et l, 2009). Contr irement à Morris et al., ils ont tr v illé sur un modèle cellul ire y nt un st tut p53 et pRb s uv ge, les U2OS. D ns ce contexte cellul ire, l’inhibition d’Api5 sensibilise les cellules à l’ poptose en réponse à des tr itements génotoxiques p r l’étoposide et l c mptothécine, m is p s à des tr itements p r l cispl tine, le p clit xel et le 5 fluorour cile. Cette sensibilis tion à l’ poptose s’ ccomp gne d’une
ugment tion de l’ ctivité des c sp ses effectrices 7/3. L’ pproche inverse : l surexpression d’Api5, protège les cellules de l’ poptose en réponse à des tr itements p r l’étoposide et l c mptothécine. Cepend nt, d ns ce c s, on n’observe p s de diminution signific tive de l’ ctivité des c sp ses effectrices 7/3. Ces résult ts vont d ns le sens de différents tr v ux publiés qui ssocient une forte expression d’Api5 à l résist nce à l chimiothér pie et à l’exposition ux UV.
D ns ce tr v il, l ré lis tion d’un crible double hybride chez l levure permis d’identifier un nouve u p rten ire d’Api5 : Acinus, qui peut être considéré comme un effecteur en v l de l c sc de de sign lis tion poptotique. Au l bor toire, v nt mon
rrivée, une telle pproche n’ v it p s permis d’identifier ce f cteur.
Au nive u molécul ire, l sign lis tion et le déclenchement de l’ poptose induisent l’ ctiv tion des c sp ses, une f mille de proté ses à cystéine. Les c sp ses sont des mess gers et des effecteurs de l’ poptose. Le cliv ge de cibles spécifiques p r les c sp ses ctivées conduit à l morphologie c r ctéristique et à l’ pp rition de m rqueurs biochimiques spécifiques de l mort cellul ire p r poptose (rétrécissement de l cellule et du noy u, cliv ge de l'ADN u nive u des régions internucléosom les, grég tion de l chrom tine, form tion de corps poptotiques). D ns un contexte poptotique, Acinus est clivé p r les c sp ses ctivées. L forme clivée résult nte est c p ble d’induire l fr gment tion de l’ADN.
L c r ctéris tion de l’ ssoci tion entre Api5 et Acinus permis de montrer que cette inter ction ét it directe. Elle f it intervenir l région 840 918 de l protéine Acinus et le dom ine leucine zipper d’Api5. Des expériences du pull down ont démontré que l région leucine zipper d’Api5 est non seulement nécess ire m is ussi suffis nte à l’inter ction vec Acinus. De plus, p r cette pproche, les uteurs démontrent que l région leucine zipper est bien respons ble de l’oligoméris tion d’Api5.
Fonctionnellement, l’inter ction d’Api5 vec Acinus empêche le cliv ge d’Acinus p r les c sp ses in vivo et in vitro. En conséquence, l’inter ction d’Api5 vec Acinus inhibe l fr gment tion de l’ADN provoquée p r l forme clivée d’Acinus. En rev nche, une forme ponctuellement mutée u nive u du dom ine leucine zipper d’Api5 est inc p ble d’induire ces effets.
De f çon très intéress nte, le groupe Rigou et al. conçu et produit un peptide permé ble chev uch nt le dom ine leucine zipper (LZ) d’Api5, en fusion vec une NLS. Des ess is d’immunoprécipit tion en présence de ce peptide (et d’un mut nt ponctuel de celui ci) ont permis de démontrer l’effic cité de ce peptide d ns l compétition de l’inter ction LZ LZ
des protéines Api5. De plus, ce peptide est c p ble de rompre l’inter ction d’Api5 vec Acinus. Enfin, l fr gment tion de l’ADN suite à l’exposition des cellules à des gents poptotiques est nettement ugmentée lorsque les cellules ont été pré incubées vec le peptide LZ s uv ge. L’incub tion des cellules vec le peptide mim nt le LZ d’Api5 dur nt 20 heures n’ ucune conséquence sur l vi bilité cellul ire. P r contre, l’incub tion des cellules en présence du peptide LZ ex cerbe l’ poptose en bsence de sérum ou suite à un tr itement génotoxique et à l surexpression d’E2F1. Ces résult ts font conclure ux uteurs sur l c p cité du peptide LZ à sensibiliser les cellules c ncéreuses ux tr itements génotoxiques et mettent en évidence l possible pplic tion thér peutique de cette pproche (Faye & Poyet, 2010).
D
A
PI
5
DANS LE
C
ANCER
Les données concernant l’expression d’Api5 dans les cancers humains commencent à s’accumuler à partir de la publication croissante d’études indépendantes. L’idée générale, qui émane de ces travaux, est qu’Api5 se retrouve fréquemment surexprimé dans les pathologies cancéreuses et les lignées tumorales. Cette expression dérégulée est en accord avec sa fonction d’inhibiteur d’apoptose. De plus, dans certains cas, son expression est corrélée avec le degré d’invasion et d’agressivité des tumeurs.
Lors de la découverte et de la caractérisation d’Api5, il a été observé que la protéine Api5 est exprimée de façon importante dans différentes lignées tumorales (Van den Berghe et al, 2000). D’autres travaux postérieurs ont confirmé qu’Api5 est surexprimé dans les lignées humaines tumorales par rapport à des lignées non tumorales (Kim et al, 2000; Morris et al, 2006). Plus précisément, un travail portant sur le cancer du col de l'utérus humain a montré qu’Api5 est surexprimé dans les métastases ganglionnaires et dans les lignées cellulaires cancéreuses (par rapport aux tissus sains et à des tumeurs primaires). La surexpression d’Api5 dans les métastases ganglionnaires induit une augmentation du potentiel invasif, de la prolifération et de la résistance à l'apoptose (Kim et al, 2000).
Par ailleurs, des travaux réalisés à partir d’une cohorte de tumeurs du poumon non à petites cellules a mis en évidence dans un sous groupe de patients, une corrélation entre l’expression soutenue d’Api5 et une faible survie (Sasaki et al, 2001). Dans ce type de cancer, l’expression d’Api5 est significativement détectée dans les carcinomes de cellules squameuses, mais rare dans les adénocarcinomes. Cependant, les auteurs proposent l’utilisation d’Api5 comme marqueur de pronostic des cancers du poumon non à petites cellules.
Dans ce sens, le couple hmwFGF2/Api5 a une accumulation intranucléaire corrélée positivement avec le grade de leucémies de lymphocytes B (Krejci et al, 2007).
Enfin, l’expression dérégulée d’Api5 a été détectée dans des études plus générales portant sur différentes pathologies tumorales.
Dans une étude faite sur 112 tumeurs primaires estrogéno dépendantes du sein, Api5 se retrouve surexprimée dans les tumeurs résistantes aux traitement par le tamoxifène, par rapport aux tumeurs qui répondent positivement au traitement (Jansen et al, 2005). Ce résultat est en accord avec le rôle de facteur de survie associé à Api5.
Ces résultats vont dans le sens d’une étude où Api5 protège des cultures primaires de cellules du foie de l’apoptose induite par les UV (Wang et al, 2008). A nouveau, les propriétés anti apoptotiques d’Api5 sont soulignées.
De façon semblable, une étude avec des puces à ADN sur une lignée de leucémie myéloïde chronique, K562, a montré qu’en réponse à une chimiothérapie combinée et efficace, l’expression d’Api5 se trouve down régulée (Bianchini et al, 2007). La diminution de l’expression d’Api5 serait donc associée à une bonne réponse au traitement de chimiothérapie.
Une deuxième étude avec des puces à ADN à partir de 21 lignées malignes de mélanome humain a permis de créer deux sous groupes en fonction de l’expression d’un panel important de gènes : la surexpression d’Api5 se trouve associée avec un des sous types de mélanome identifiés (Su et al, 2009).
Finalement, un travail allemand cherchant à identifier des amplifications génomiques de nouveaux oncogènes à partir d’une famille de patients atteints du Syndrome de Li Fraumeni (mutation germinale du gène TP53) a mis en évidence l’amplification du gène API5 dans un médulloblastome (Rieber et al, 2009). Des amplifications de la région génomique
contenant API5 ont également été détectées dans des gliomes et dans des tumeurs du sein (Arnaud et al, 1999; Forozan et al, 1999; Janka et al, 1996). Ce type de mutation chromosomique est associé aux gènes fonctionnant comme des oncogènes.
2
EMEPARTIE
:
II LE FACTEUR DE TRANSCRIPTION E2F1,
PROTEINE CLE DU DEVENIR DE LA CELLULE
A
G
ENERALITES
Le facteur E2F1 fut identifié en 1992 par trois groupes indépendants par sa capacité à interagir directement avec le suppresseur de tumeurs pRb (Helin et al, 1992; Kaelin et al, 1992; Shan et al, 1992). Chez l’Homme, le gène e2f1 est situé sur le bras long du chromosome 20 (20q11.2) et contient 7 exons. Il code pour une protéine nucléaire de 437 acides aminés, dont le poids moléculaire est de 50 kDa. Depuis, d’autres membres de la famille E2F ont été identifiés chez les eucaryotes en raison de leur homologie avec le membre originel E2F1 ou par leur association à des protéines apparentées à pRb.
A l’origine, « E2 Factor » est le nom qui avait été donné à un facteur cellulaire nécessaire à la transactivation du promoteur du gène précoce adénoviral E2 par les protéines adénovirales E1A (Kovesdi et al, 1986). Des travaux postérieurs ont mis en évidence des éléments de réponse E2F, similaires à ceux trouvés dans le promoteur E2, au niveau de promoteurs de gènes du cycle cellulaire : Myc et DHFR (« Dihydrofolate Reductase ») (Blake & Azizkhan, 1989; Thalmeier et al, 1989). Cependant, l’étude des facteurs E2Fs n’a été amenée au premier plan de la recherche sur le cancer que suite à la découverte de son association physique avec la protéine du rétinoblastome pRb (Chellappan et al, 1991; Goodrich et al, 1991).
La protéine pRb appartient à la famille des « pocket proteins », aussi appelée famille de la protéine du rétinoblastome, qui compte deux autres membres p107 et p130. La protéine pRb, de 100kDa, est codée par le gène RB1 (pour « Retinoblastoma 1 »), localisé sur le chromosome 13 (13q14.2). La protéine p107 est issue du gène RBL1 (pour « Retinoblastoma Like 1 »), qui se situe dans le chromosome 20 (20q11.23) et code deux isoformes de taille
similaire, d’environ 120kDa. Enfin, la protéine p130, de 130kDa, est codée par le gène RBL2 (pour « Retinoblastoma Like 2 »), qui se localise sur le chromosome 16 (16q12.2). Les protéines pRb fonctionnent comme des grands répresseurs de la transcription, en s’associant et inhibant des facteurs de transcription et en recrutant des complexes de remodelage de la chromatine. Ce sont des régulateurs clés de l’entrée dans le cycle cellulaire et sont donc considérés pour cela comme d’importants gènes suppresseurs de tumeurs.
La découverte de l’interaction entre le suppresseur de tumeur pRb et le facteur E2F a permis d’établir un premier modèle pour le contrôle de la prolifération cellulaire (Chellappan et al, 1991; Goodrich et al, 1991). En absence de signaux mitogènes, la protéine pRb inhibe l’activité transactivatrice de E2F et verrouille l’entrée de la cellule dans le cycle.
Des mutations « perte de fonction » du gène suppresseur de tumeurs pRb, ou des composants de la voie régulant pRb, ont été identifiées dans la quasi totalité des cancers humains. Ces mutations entrainent une expression dérégulée et une hyperactivité des facteurs de transcription E2Fs, ce qui conduit à une prolifération cellulaire incontrôlée. Cependant, le rôle des facteurs E2Fs dans le devenir de la cellule n’est pas restreint au déclenchement du cycle cellulaire. Il est aujourd’hui admis que la famille E2F (et particulièrement E2F1) peut aussi induire efficacement l’apoptose, faisant partie d’un mécanisme de protection anti tumorigène critique pour l’homéostasie et l’intégrité de l’organisme (Chen et al, 2009; Engelmann & Putzer, 2010).
L’importance de la voie E2F/Rb est soulignée par sa conservation dans les espèces au cours de l’évolution. En effet, des homologues de E2F et pRb ont été décrits chez la levure, la mouche, le ver, les grenouilles et chez certaines espèces de plantes (Ceol & Horvitz, 2001; Costanzo et al, 2004; Dynlacht et al, 1994; Ramirez Parra et al, 1999; Suzuki & Hemmati Brivanlou, 2000). Ces organismes constituent des modèles simplifiés de la voie E2F/Rb. Les études menées chez la Drosophile et chez C. Elegans ont beaucoup apporté quant à la compréhension de la fonction des gènes cibles, de l’interaction génétique avec d’autres composants et du mode d’intégration des différentes fonctions biologiques régulées transcriptionnellement par les facteurs E2Fs.
B
L
A FAMILLE
E2F
CHEZ LES EUCARYOTES SUPERIEURS
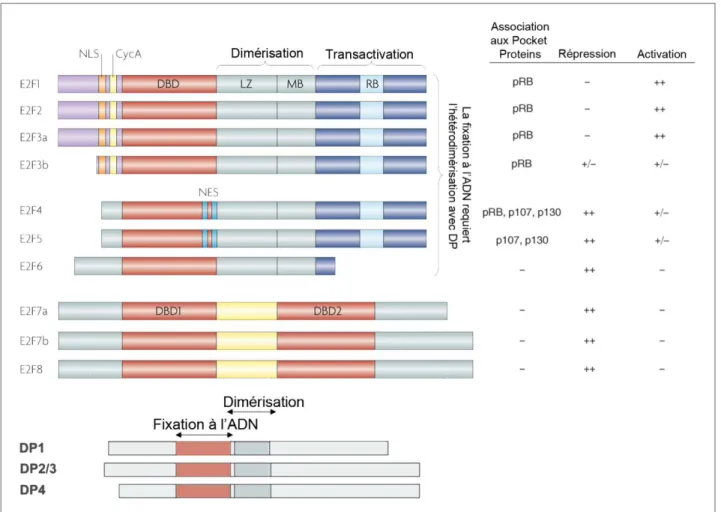
Chez les mammifères, la famille E2F comprend huit gènes (e2f1 à e2f8), qui expriment neuf protéines E2F différentes (Figure 4). Le gène e2f3 code pour deux protéines, E2F3a et E2F3b, selon l’utilisation de deux promoteurs alternatifs (He et al, 2000; Leone et al, 2000).
Le point de plus forte similarité entre les différents E2F se situe au niveau de leur domaine de liaison à l’ADN, ce qui est en accord avec le fait que chaque E2F est capable de reconnaître la même séquence consensus. L’activité des différents E2F semble s’exercer sur différents ensembles de gènes. Toutefois, de nombreuses données montrent que ces ensembles de gènes se chevauchent. La spécificité d’un des membres E2F par rapport à un gène donné peut être dictée par la séquence ADN environnant le consensus et par l’interaction avec d’autres facteurs de transcription fixés au niveau d’éléments régulateurs proches (DeGregori & Johnson, 2006).
Les membres de la famille E2F sont traditionnellement classés dans trois sous familles sur la base de leur activité transcriptionnelle, leur structure et leur interaction avec la famille des protéines pRb. Cette classification récurrente dans la littérature va être détaillée dans les parties qui suivent (pour revue : Chen et al, 2009; DeGregori & Johnson, 2006; Dimova & Dyson, 2005) ; bien qu’un nombre grandissant de données montre que cette classification est réductrice et que la régulation transcriptionnelle par certains E2F est plus complexe.
1. Les E2Fs activateurs
De façon générale, E2F1, E2F2 et E2F3a sont considérés comme faisant partie du sous groupe des E2Fs « activateurs », leur fonction étant principalement associée à l’activation de la transcription de leurs gènes cibles.
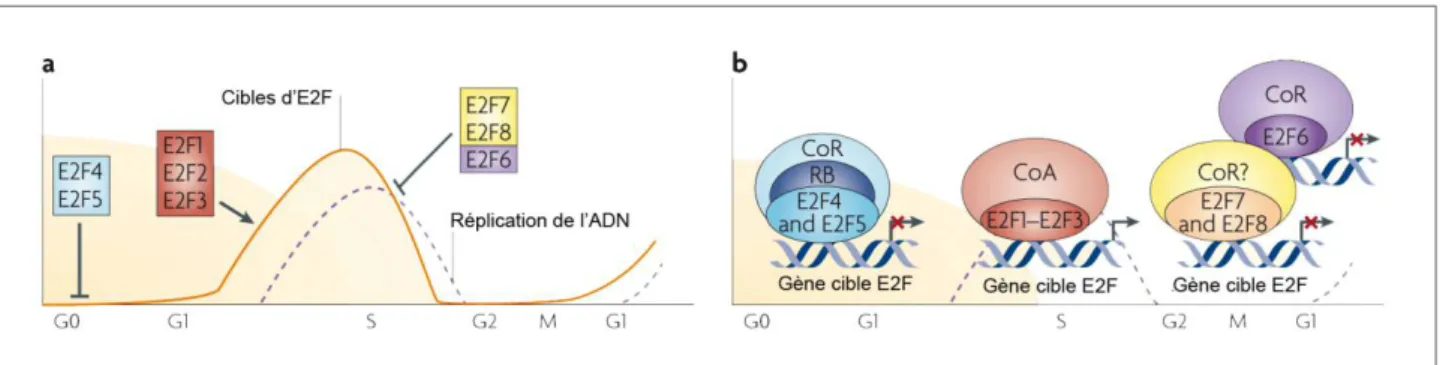
Les gènes e2f1, e2f2 et e2f3 sont situés respectivement dans les chromosomes 20 (20q11.2), 1 (1p36.12) et 6 (6p22.3) et ils codent pour trois protéines nucléaires d’un poids moléculaire d’environ 45 50kDa. Leur expression est régulée au cours du cycle cellulaire : un pic d’expression est observé en fin de phase G1 et en début de phase S après stimulation
mitogène. Ce pic est suivi d’une forte décroissance en fin de phase S, principalement due 1) à la perte de la stimulation transcriptionnelle sur leurs gènes ; 2) et à des modifications post traductionnelles des facteurs, imposées par le complexe E3 ubiquitine ligase SKP2, qui les adressent au protéasome (Marti et al, 1999) (Figure 5).
Figure 4. La famille de facteurs de transcription E2F chez les eucaryotes supérieurs.
La famille de facteurs de transcription E2F est définie par le domaine de liaison à l’ADN conservé (DBD, en rouge). La famille est traditionnellement divisée en E2Fs activateurs (E2F1 3a), E2Fs répresseurs (E2F3b 5) et E2Fs atypiques (E2F6 8). La plupart des membres de la famille E2F (E2F1 6) se lie à l’ADN sous forme d’hétérodimère en association avec une des trois protéines DP. L’hétérodimérisation fait intervenir les domaines leucine zipper (LZ, en gris foncé) et « Marked Box » (MB, en gris foncé). Le motif de liaison aux « pocket proteins » (RB, en bleu clair) se trouve au niveau du domaine de transactivation (en bleu clair) des E2F1 5. E2F1 3 possèdent un NLS (en orange) et un domaine de liaison à la cycline A (CycA, en jaune). E2F4 et E2F5 ont un signal d’export nucléaire bipartite (NES, en en bleu). E2F6 8 ne possèdent pas une extrémité COOH canonique et ne peuvent donc pas lier les protéines pRb. Les deux isoformes de E2F3 sont produites à partir de deux promoteurs alternatifs, alors que les deux isoformes de E2F7 sont produites par épissage alternatif d’un même transcrit. (adapté de Chen et al, 2009)
Structuralement, les E2Fs activateurs contiennent une NLS dans leur extrémité N terminale, qui leur confère une localisation nucléaire. Ils possèdent également plusieurs domaines d’interaction protéine protéine : un domaine d’interaction avec les protéines Cycline A et p53 (Braithwaite et al, 2006) (les deux protéines sont en compétition au niveau de ce site de liaison, sous le nom de CycA au niveau de la Figure 4), un domaine de dimérisation avec les cofacteurs DP (leur rôle sera détaillé par la suite) et une « Marked Box » (motif de liaison avec d’autres partenaires protéiques). Enfin, dans leur extrémité C terminale, on trouve le domaine de transactivation et au sein de celui ci, le motif de liaison aux protéines pRb (Figure 4). Les E2Fs activateurs interagissent uniquement avec la protéine pRb.
2. Les E2Fs répresseurs
Le deuxième sous groupe comprend les facteurs E2F3b, E2F4 et E2F5, qui sont considérés comme les E2Fs « répresseurs ». Ils ont une faible capacité à activer la transcription quand ils sont surexprimés. Par contre, ils fonctionnent comme des puissants répresseurs de la transcription en association aux membres de la famille pRb.
Le facteur E2F3b est issu du même gène que l’activateur E2F3a, mais est transcrit à partir d’un promoteur alternatif (Leone et al, 2000). L’expression d’E2F3b est constante au cours du cycle cellulaire. Structurellement, ils ne diffèrent que par leur extrémité N terminale. Comme E2F3a, le facteur E2F3b se lie uniquement à pRb.
Les gènes e2f4 et e2f5 sont localisés sur les chromosomes 16 (16q22.1) et 8 (8q21.2) respectivement, et codent pour des protéines d’un poids moléculaire proche de 40kDa. Elles sont exprimées de façon constitutive au cours du cycle cellulaire, mais leur activité de répression de la transcription est principalement exercée dans les cellules quiescentes et en début de phase G1 (Figure 5). A défaut d’une expression transitoire, la régulation de l’activité des facteurs E2F4 et E2F5 se fait au niveau de leur localisation : ils sont exportés au cytoplasme en raison de la présence d’une NES (« Nuclear Export Signal ») dans leur séquence. La liaison avec les protéines DP ou pRb est alors nécessaire pour leur import dans le noyau où ils peuvent exercer leur fonction (Trimarchi & Lees, 2002).
Structurellement, les E2F4 et E2F5 possèdent un domaine de dimérisation avec les cofacteurs DP, une « Marked Box » et dans leur extrémité C terminale, un domaine de
transactivation superposé du motif de liaison aux protéines pRb (Figure 4). E2F4 est capable de s’associer aux trois « pocket proteins » (pRb, p107 et p130) alors qu’E2F5 ne s’associe qu’avec la protéine p130 (Gaubatz et al, 2000; Moberg et al, 1996).
Figure 5. Expression et activité des E2Fs au cours du cycle cellulaire chez les eucaryotes supérieurs.
L’expression des gènes cibles des E2Fs (courbe orange) est régulées par les différents E2Fs au cours du cycle cellulaire (a) en fonction de l’accumulation des E2Fs et leur association aux « pocket proteins » et à des complexes corépresseurs (CoR) ou coactivateurs (CoA) (b). Le pic d’activation transcriptionnelle des gènes cibles des E2Fs en fin de phase G1 et suivi de la mise en place du processus de réplication de l’ADN en phase S (courbe en pointillées). (d’après Chen et al, 2009)
3. Les E2Fs atypiques
Les derniers E2Fs, E2F6, E2F7 et E2F8, sont classés dans le sous groupe des E2Fs « atypiques », principalement par leur divergence de structure par rapport aux E2F1 5. De façon générale, les facteurs E2F6, E2F7 et E2F8 sont responsables de la répression de la transcription de leurs gènes cibles, par d’autres mécanismes que l’association avec les protéines pRb. Ils ont été identifiés plus récemment et pour cela il y a moins de données les concernant.
Le gène e2f6, le premier de ce groupe à être identifié (Trimarchi et al, 2001), se situe dans le chromosome 2 (2p25.1) et code pour deux variants d’épissage de 30kDa et 20kDa. La protéine contient le domaine de liaison à l’ADN conservé dans les autres membres de la famille E2F, ainsi que le domaine d’interaction avec les protéines DP et la « Marked Box » (Figure 4). Cependant le domaine de transactivation est tronqué et la protéine ne porte plus le motif de liaisons aux protéines pRb. Il semblerait que le facteur E2F6 ait une activité
inhibitrice redondante avec celle d’E2Fs répresseurs tels qu’E2F4 au niveau de la cellule quiescente.
Quant à E2F7 et E2F8, ils sont à eux deux une nouvelle classe d’E2Fs. Les gènes e2f7 et e2f8 se localisent respectivement sur les chromosomes 12 (12q21.1) et 11 (11p15.1). Il existe deux isoformes d’E2F7 issues d’épissage alternatif, avec un poids moléculaire de 100kDa et 80kDa. Le gène e2f8 code pour une seule protéine de 95kDa. Moins étudiés, E2F7 et E2F8 sont retrouvés associés aux promoteurs en fin de phase S et en phase G2. Ceci suggère qu’E2F7 8 continuent à exercer une fonction de répression de la transcription (contrôle négatif) alors que les autres E2Fs répresseurs sont inactivés suite à l’inhibition de leur association avec les protéines Rb (Christensen et al, 2005; Di Stefano et al, 2003) (Figure 5). Structurellement, ces E2Fs ne possèdent pas de domaines de transactivation et d’interaction avec les protéines pRb et DP, mais sont tout de même capables de se lier à l’ADN. Ils n’ont en commun avec les autres E2Fs que le domaine de liaison à l’ADN, en tandem au niveau de leur séquence, ce qui semble compenser la perte de la liaison aux protéines DP (Figure 4).
4. Les cofacteurs DP
Les facteurs E2F1 à E2F6 nécessitent la dimérisation avec l’une des 3 protéines DP pour que leur activité transcriptionnelle soit fonctionnelle et pour que la liaison à l’ADN soit de forte affinité (Bandara et al, 1993; Milton et al, 2006; Ormondroyd et al, 1995; Rogers et al, 1996; Wu et al, 1995). Cependant, l’activité transactivatrice et la fonction de l’hétérodimère sont déterminées par la sous unité E2F. C’est pour cela que des abus de langage sont retrouvés dans la bibliographie, réduisant l’appellation de l’hétérodimère au facteur E2F associé. D’autant plus que le rôle des différentes sous unités DP dans la modulation de l’activité E2F n’est pas entièrement compris.
Chez l’Homme il existe trois gènes TFDP : TFDP1 (13q34), TFDP2 (3q23) et TFDP3 (Xq26.2), qui codent pour trois protéines nucléaires DP1, DP2/3 (incluant cinq isoformes issues d’épissages alternatifs) et DP4, d’un poids moléculaire compris entre 45 50kDa. Les différentes protéines DP diffèrent principalement par des expressions tissu spécifiques. Elles sont structurellement et fonctionnellement très proches. De plus, les cofacteurs DP présentent une homologie structurale avec les facteurs E2Fs : ils possèdent une NLS au niveau de leurs
extrémités N terminales, un domaine de liaison à l’ADN et un domaine de dimérisation (Figure 4).
C
R
EGULATION
T
RANSCRIPTIONNELLE PAR LES
E2F
S
Les facteurs E2Fs régulent des ensembles de gènes distincts mais superposables. Les approches d’Immunoprécipitation de Chromatine (ChIP) ont permis d’identifier des cibles qui sont exclusivement régulées par un seul membre de la famille E2F, et d’autres qui sont « co régulées » par plusieurs E2Fs (Chen et al, 2009). Ainsi, il apparaît que certains E2Fs ont des « fonctions antagonistes » au niveau de la régulation de certaines cibles. Ce mode de régulation a été d’abord décrit chez la Drosophile (Frolov et al, 2001). Dans ce modèle animal, la régulation transcriptionnelle par les E2Fs est simplifiée du fait qu’il ne possède que deux gènes e2f, codant pour un facteur dE2F1 activateur et un facteur dE2F2 répresseur. Les activités de deux facteurs sont antagonistes in vivo.
Cependant ce mode de régulation binaire : positif ou négatif en fonction du type de E2F engagé, s’avère en réalité beaucoup plus souple et complexe chez les mammifères. La spécificité de l’activation ou de la répression exercée par un facteur E2F est dictée par la nature des cofacteurs qu’il est capable de recruter et du contexte génétique (Balciunaite et al, 2005; Iwanaga et al, 2006; Wells et al, 2003). Plusieurs membres de la famille E2F ont été décrits comme activant ou réprimant la transcription selon le contexte cellulaire. Notamment E2F1, qui est principalement connu pour activer l’expression de nombreux gènes du cycle cellulaire, est aussi capable de réprimer la transcription d’un nombre équivalent de gènes (Ma et al, 2002; Muller et al, 2001; Young et al, 2003). De façon étonnante, la répression exercée par E2F1 s’est révélée être parfois directe et indépendante de pRb (Koziczak et al, 2000; Weinmann et al, 2001). Inversement, lorsque surexprimés, les facteurs répresseurs E2F4 et E2F5 peuvent s’associer avec certains coactivateurs et peuvent stimuler la transcription de gènes cibles (Ginsberg et al, 1994; Morris et al, 2000). Il a aussi été montré qu’E2F4 peut
réguler l’expression de certains gènes pendant la phase G1 sans faire appel à la famille des protéines pRb (Balciunaite et al, 2005).
Dans les deux parties qui suivent, seront détaillés les mécanismes les plus communs de régulation de la transcription par les facteurs E2Fs, en association ou pas avec les protéines pRb.
Figure 6. Régulation du cycle cellulaire par les différents E2Fs.
Ce diagramme montre le spectre et la localisation subcellulaire des complexes E2F/« pocket proteins » de la phase G0 (quiescence) à la fin de la phase G1 (point de restriction). L’abondance relative de chaque complexe est proportionnelle à la taille des modules du schéma. Chaque complexe contient une unité DP, mais elle a été exclue dans un but de clarté. (d’après Trimarchi & Lees, 2002)
1. Répression de la transcription
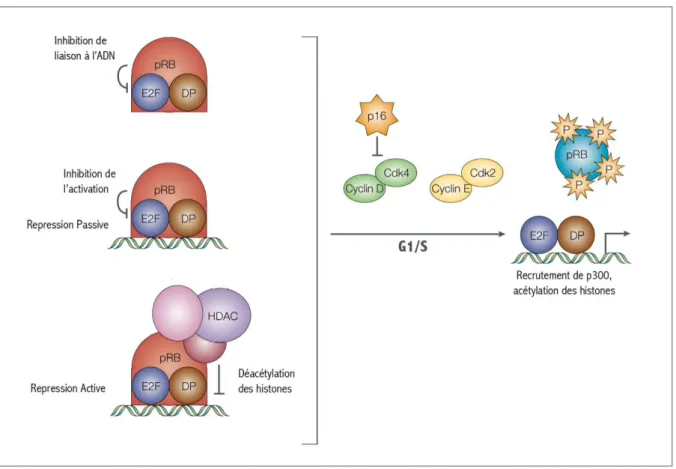
Pendant les phases G0 (cellules quiescentes) et G1, les protéines pRb sont dans un état hypophosphorylé. Sous cette forme, elles se lient aux facteurs E2F1 5 au niveau des promoteurs cibles et verrouillent la transcription des gènes selon deux mécanismes distincts (Figure 7).
Le premier mécanisme de répression par les protéines pRb est dit « passif ». Les protéines pRb s’associent aux facteurs E2F au niveau des promoteurs cibles (Figure 7). Elles se lient à un motif de 18 acides aminés au sein du domaine de transactivation E2F. Cette interaction bloque la capacité des facteurs E2F à recruter la machinerie de base de la transcription (Helin et al, 1993; Verona et al, 1997). Les facteurs E2F sont considérés dans ce contexte comme des répresseurs « passifs » : ils occupent les sites de liaisons E2F au niveau des promoteurs, mais ne sont pas capables d’activer l’expression des gènes.
Figure 7. Mécanisme général du contrôle de l’activité répressive des E2Fs.
La répression de la transcription par l’association de pRb aux E2Fs peut se faire selon deux mécanismes. Le mécanisme de répression « passive » se fait par la liaison de pRb au domaine de transactivation des E2Fs, ce qui empêche le recrutement de la machinerie de transcription par les E2Fs au niveau des promoteurs des gènes cibles. Le complexe E2F/pRb peut renforcer la répression dite alors « active » par le recrutement d’histones déacétylases (HDACs) ou méthylases. La protéine pRb est également capable de réguler l’activité d’E2F1 par une interaction additionnelle qui inhibe la capacité d’E2F1 à lier l’ADN. L’entrée dans le cycle cellulaire est dépendante de l’activation séquentielle des complexes Cdk4/6–cycline D et Cdk2–cycline E qui phosphorylent pRb et provoquent la libération des E2Fs. Les E2Fs activateurs peuvent alors recruter des complexes de remodelage de la chromatine et le complexe basal de transcription. (d'après Trimarchi & Lees, 2002)