HAL Id: tel-03193857
https://tel.archives-ouvertes.fr/tel-03193857
Submitted on 9 Apr 2021HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
dans son transport intracellulaire et/ou sa dégradation :
rôle des lectines EDEMs et la mannosidase du RE
Khadidja Sidelarbi
To cite this version:
Khadidja Sidelarbi. Implication du chaperome de la protéine F508del-CFTR dans son transport intracellulaire et/ou sa dégradation : rôle des lectines EDEMs et la mannosidase du RE. Biochimie, Biologie Moléculaire. Université de Poitiers, 2017. Français. �NNT : 2017POIT2286�. �tel-03193857�
Pour l’obtention du Grade de
DOCTEUR DE L’UNIVERSITE DE POITIERS
(Faculté des Sciences Fondamentales et Appliquées) (Diplôme National - Arrêté du 25 mai 2016)
Ecole Doctorale : Biosanté
Secteur de Recherche : Biomolécules, pharmacologie, thérapeutique
Présentée par :
Khadidja SIDELARBI
************************
Implication du chaperome de la protéine F508del-CFTR dans son transport
intracellulaire et/ou sa dégradation : rôle des lectines EDEMs et de la mannosidase du RE
************************
Directeur de Thèse : Pr. Frédéric BECQ et Dr. Caroline NOREZ ************************
Soutenue le 24 novembre 2017 devant la Commission d’Examen
************************
JURY
Valérie Chappe Professeur associé, Université de Dalhousie, Halifax Rapporteur Thomas Falguières Chargé de recherche INSERM, Saint-Antoine, Paris Rapporteur Christelle Coraux Chargé de recherche INSERM, Université de Reims Examinateur Thierry Berges Professeur des Universités, Université de Poitiers Examinateur Caroline Norez Maître de Conférences, Université de Poitiers Examinateur Frédéric Becq Professeur des Universités, Université de Poitiers Examinateur
1
Sommaire
Sommaire ... 1
Abréviations ... 4
Liste des Figures ... 6
Liste des tableaux ... 9
Introduction ... 10
I. Le contrôle qualité des glycoprotéines ... 10
A. La voie ERAF ... 11
1) Les modifications co- et post-traductionnelles ... 13
2) Les interactions avec les protéines chaperonnes ... 18
B. La voie ERAD ... 33
1) ERMnI et son rôle dans ERAD ... 40
2) EDEMs et leurs rôles dans ERAD ... 43
3) Régulation des régulateurs d’ERAD « ERAD tuning » ... 45
4) Réponse à l’accumulation des protéines mal repliées UPR ... 47
II. Exemple du contrôle qualité : la protéine CFTR vs F508del-CFTR ... 49
A. Du gène à la protéine CFTR ... 49
1) Le gène CFTR ... 49
2) Structure de la protéine CFTR ... 50
3) Les fonctions de la protéine CFTR ... 52
4) Les mutations du CFTR ... 53
B. Biosynthèse et repliement : F508del-CFTR vs WT-CFTR ... 55
C. Le contrôle qualité de CFTR dans le RE et sa dégradation par ERAD ... 58
1) Les principales chaperonnes et lectines ... 58
2) Motif de rétention dans le RE : AFT (Arginine-Framed Tripeptides) ... 61
3) Code de sortie du RE : le code diacide ... 62
2
III. Le système de contrôle qualité du RE comme cible thérapeutique ... 64
Contexte, problématiques et objectifs ... 70
Matériels et méthodes ... 74
I. Matériel cellulaire ... 74
Fiche technique N°1 : ... 74
Lignées cellulaires, culture et entretien ... 74
Fiche technique N°2 : ... 76
Congélation et décongélation des lignées cellulaires ... 76
Fiche technique N°3 : ... 77
Isolement des cellules épithéliales bronchiques des patients CF ... 77
II. Techniques de biologie moléculaire et de biochimie ... 79
Fiche technique N°4 ... 79
Mesure de l’activité du protéasome ... 79
Fiche technique N°5 : ... 80
Transfection de siRNA (small interfering RNA) ... 80
Fiche technique N°6 : ... 82 Western blot ... 82 Fiche technique N°7 : ... 87 Biotinylation de surface ... 87 Fiche technique N°8 : ... 89 Immunomarquage indirect ... 89 Fiche technique N°9 : ... 91
In situ Proximity Ligation Assay (Duolink) ... 91
III. Techniques de physiologie cellulaire ... 93
Fiche technique N°10 : ... 93
Efflux d’iodure ... 93
Fiche technique N°11 : ... 95
Etude des variations du potentiel membranaire avec la sonde oxonol ... 95
Fiche technique N°12 : ... 97
3
Résultats ... 101
I. Les protéines EDEMs comme cibles thérapeutiques dans la mucoviscidose ... 101
A. Contexte ... 101
B. Résultats ... 101
1) Article ... 101
2) Résultats complémentaires ... 133
II. Implication de l’α1,2-mannosidase du RE dans la rétention de F508del-CFTR .. 143
A. Contexte ... 143
B. Résultats ... 144
1) Effet des inhibiteurs des mannosidases sur la fonction de F508del-CFTR ... 144
2) Effet du siRNA d’ERManI sur F508del-CFTR ... 144
3) Comprendre l’effet correcteur sur F508del-CFTR de l’extinction d’ERManI ... 148
Discussion, conclusion et perspectives ... 155
A. Synthèse d’une nouvelle famille d’iminosucres : dérivés multivalents du Deoxymannojirimycin ... 155
B. La famille des protéines EDEMs : de nouvelles cibles thérapeutique pour la mucoviscidose ... 157
C. Etude du trivalent-DMJ comme correcteur de F508del-CFTR ... 159
D. A la recherche d’une autre cible thérapeutique pour la mucoviscidose : α1,2-mannosidase I du RE ... 166
Références bibliographiques ... 171
Annexes ... 204
4
Abréviations
°C: degré celsius
4PBA: sodium-4-phénylbutyrate A: alanine
AAA+: ATPases associated with diverse
cellular activities
AAT: α1-antitrypsin
ABC: ATP binding cassette
Aha1: Activator of Hsp90 ATPase 1 ADP: adénosine diphosphate
AFT: Arginine-Framed Tripeptides AMC: 7-mino-4-methylcoumarin AMPc: adénosine monophosphate
cyclique
ARN: acide ribonucléide Asn: asparagine
ATF6: activating transcription factor-6 ATP: adenosine triphosphate
BAG-1/2: BCL2 associated athanogene 1/2 BCA: bicinchoninique acide
BHK: baby hamster kidney
BiP: binding immunoglobulin protein BSA: bovine serum albumin
Ca2+: ion calcium CF: cystic fibrosis
CFBE: CF branchial epithelial
CFTR: CF transmembrane conductance
regulator
CFTRinh: CFTR inhibiteur
CHIP: C-terminus of Hsp70 interacting
protein
CHO: chinese hamster ovary
CHOP: CCAAT/enhancer-binding protein
homologous protein
Cl-: ion chlorure
COPI/II: coat proteins I/II
CRD: carbohydrate recognition domain Cu2+: ions cuivriques CypB: cyclophilines B Cys: cystéine D: aspartate ddp: différence de potentiel del: deletion DMJ: 1-déoxymannojirimycine
DNAJB12: DnaJ Hsp40 family member
B12
DNJ: deoxynojirimycine DMSO: diméthylsulfoxide
ECL: enhanced chemiluminescence ECLs: extra cellular loops
EGF: epidermal growth factor
EDEM: ER degradation-enhancing
1,2-manosidase-like protein
eIF2α: Eukaryotic Initiation Factor 2α ER: endoplasmic reticulum
ERAD: ER associated degradation ERAF: ER associated folding ERdj: ER-localized DnaJ proteins ERES: ER-exit sites
ERGIC: ER-Golgi intermediate
compartment
ERmanI: ER α1,2-mannosidase I ERQC: ER quality control F: phenylalanine
FKBP: FK506-binding proteins Fluo: florescent
Fsk: forskoline
GH47: glycoside hydrolase family 47 Glc: glucose
GlcNAc: N-Acetylglucosamine Gp78: Glycoprotein 78
Grp: glucose regulated protein Gst: génistéine
GTP: guanosine triphosphate Hdj: human DnaJ
HEK293: human embryonic kidney 293 hERG: human ether-a-go-go-related gene Herp: ER stress-inducible protein
5
HRD1: HMG-CoA reductase degradation
protein 1
HRP: horseradish peroxidase Hsc: heat shock cognate Hsp: heat shock protein
HspBP1: Hsp binding protein 1 Htm1: homologous to mannosidase I I-: ion iodure
ICLs: intracellular loops IRE1: inositol requiring 1 Isc: short circuit current IgG: Immunoglobulin G K+: ion potassium
KCl: chlorure de potassium kDa: kilo Dalton
KO: knockout
LAMP: lysosomal-associated membranes
proteins
LC3: microtubules-associated protein-light
chain 3
Man: mannose
Mns1: α1,2-mannosidase chez la levure Mg: magnésium
MSD: membrane-spanning domain N: asparagine
Na+: ion sodium
NBD: nucleotide-binding domain NEF: nucleotide exchange factors NHK: null hong kong
OS-9: osteosarcoma amplified 9 OST: oligosaccharyl transférase
PAGE: polyacrylamide gel electrophoresis PAF: paraformaldéhyde
PBS: phosphate buffer saline PDI: protein disulfide isomerase
PERK: protein kinase RNA-like ER kinase PPIase: peptidyl-prolylisomérases
PKA: protein kinase A PKC: protein kinase C
PLA: proximity ligation assay QCV: quality control vesicles RD: regulatory domain RE: réticulum endoplasmique
RISC: RNA induced silencing complex RMA1: RING-finger protein with
membrane anchor-1
RNF5: RING finger protein SBD: substrate binding domain SDS: sodium dodecyl sulfate
SEL1L: suppressor/enhancer of
Lin-12-like
SERCA: sarco/ER Ca2+-ATPase
siRNA: small interfering RNA SPP: signal peptide peptidase SRP: signal recognition particule SVF: sérum de veau fœtal
T1/2: demi-vie
TBS: tris buffer saline TG-SDS: tris-glycin-SDS TMD: transmembrane domain Trx: thiorédoxine
UGGT: UDP-Glucose:glycoprotein
glycosyltransferase
UPR: unfolded protein response VIP36: vesicular integral membrane
protein of 36 kDa
VIPL: VIP36-Like wt: wild type (sauvage)
XBP-1: x-box binding protein 1
XTP3-B: XTP3-transactivated gene B
protein
6
Liste des Figures
Figure 1 : Reconnaissance du peptide signal par la SRP. ... 12
Figure 2 : Structure du motif oligosaccharidique N-glycan. ... 13
Figure 3 : Glycosylation d’une protéine naissante. ... 14
Figure 4 : Formation des liaisons disulfure par les protéines PDI. ... 16
Figure 5 : Réaction d’isomérisation peptidyl-prolyl cis-trans catalysée par des PPIases. ... 17
Figure 6 : Interaction des glycoprotéines avec la chaperonne calnexine. ... 19
Figure 7 : Le Cycle de la calnexine/calréticuline. ... 20
Figure 8 : Influence des co-chaperonnes du complexe Hsp70 sur le devenir des protéines. ... 24
Figure 9 : Modes de transport des protéines hors du réticulum endoplasmique. ... 28
Figure 10 : Transport des glycoprotéines dans la voie sécrétoire (Yamamoto, 2014). ... 30
Figure 11 : Représentation schématique de la maturation des oligosaccharides N-liés. ... 31
Figure 12 : Etapes de la dégradation ERAD des glycoprotéines. ... 35
Figure 13 : Structure du motif oligosaccharide N-glycan Glc3Man9GlcNAc2. ... 37
Figure 14 : Les trois principales ligases identifiées chez les mammifères. ... 38
Figure 15 : Représentation schématique des membres de la famille GH47. ... 40
Figure 16 : Modèle de compartimentation du RE dans les cellules de mammifères. ... 42
Figure 17 : Le processus ERAD tuning. ... 46
Figure 18 : Représentation des trois voies de l’UPR. ... 48
Figure 19 : Du gène à la protéine CFTR. ... 50
Figure 20 : Représentation schématique de la structure de CFTR. ... 51
Figure 21 : Régulation des canaux ioniques par la protéine CFTR. ... 52
Figure 22 : Classification des mutations du CFTR et leurs conséquences sur la protéine CFTR. ... 54
Figure 23 : Localisation de la protéine WT- et F508del-CFTR. ... 55
Figure 24 : Biosynthèse de WT- et F508del-CFTR et assemblage des domaines. ... 56
Figure 25 : Rôle de Hsp70/90 et leur partenaires dans le contrôle de qualité de CFTR. ... 59
Figure 26 : Adaptation de la capacité des voies ERAF et ERAD pour corriger les défauts impliqués dans les pathologies conformationnelles. ... 65
Figure 27 : Représentation schématique des principales étapes de la biosynthèse de F508del-CFTR et les sites d’action du Miglustat et des inhibiteurs de pompes Serca... 71
7
Figure 29 : Principe de la transfection de siRNA. ... 80
Figure 30 : Principe de la technique du western blot. ... 82
Figure 31 : Assemblage du sandwich de western blot. ... 85
Figure 32 : Principe de la biotinylation de surface. ... 87
Figure 33 : Principe d’immunomarquage indirect. ... 89
Figure 34 : Principe de la technique de Duolink. ... 91
Figure 35 : Exemple de représentation graphique des résultats d’efflux d’iodures. ... 94
Figure 36 : Structure de la sonde oxonol [DiSBAC2(3)]. ... 95
Figure 37 : Exemple de représentation graphique de la fluorescence de la sonde oxonol. ... 96
Figure 38 : Principe de la technique de chambre de Ussing. ... 97
Figure 39 : Chambre de Ussing. ... 99
Figure 40 : Implication de RMA1/RNF5 et CHIP dans la dégradation de F508del-CFTR. .. 133
Figure 41 :Effet du trivalent-DMJ sur l’interaction de F508del-CFTR avec RNF5/RMA1 ou CHIP. ... 134
Figure 42 : Effet du trivalent-DMJ sur l’activité du protéasome 20S intracellulaire. ... 135
Figure 43 : Effet du trivalent-DMJ sur l’activité du protéasome 20S purifié. ... 137
Figure 44 : Effet du DMJ et ses dérivés multivalent sur l’activité du protéasome 20S purifié. ... 138
Figure 45 : Effet de la tunicamycine sur l’expression protéique de BiP/GRP78 et eif2α. ... 140
Figure 46 : Effet du trivalent-DMJ sur l’expression de la protéine BiP/GRP78 et eIF2α. .... 141
Figure 47 : Efficacité du potentiateur VX-770 sur F508del-CFTR corrigé par le trivalent-DMJ. ... 142
Figure 48 : Effet des inhibiteurs de mannosidase sur l’activité de F508del-CFTR. ... 144
Figure 49 : Diminution de l’expression protéique d’ERManI ou de Golgi-Man par siRNA. 145 Figure 50 : Effet du siRNA ERManI et Golgi-ManII sur l’activité de F508del-CFTR. ... 146
Figure 51 : Effet de siRNA ERManI sur la maturation de F508del-CFTR. ... 147
Figure 52 : Effet du siRNA ERManI sur l’expression protéique des EDEMs. ... 149
Figure 53 : Effet du siRNA ERManI sur l’interaction de F508del-CFTR avec les EDEMs. 150 Figure 54 : Effet du siRNA ERManI sur l’expression protéique de CHIP et RNF5/RMA1. 151 Figure 55 : Effet du siRNA ERManI sur l’interaction de F508del-CFTR avec CHIP ou RNF5/RMA1. ... 152
Figure 56 : Effet du siRNA ERManI sur l’activité du protéasome 20S intracellulaire. ... 153
Figure 57 : Hypothèses de la restauration de F508del-CFTR à la membrane cellulaire sous sa forme immature par le trivalent-DMJ. ... 161
8 Figure 58 : Principales protéines impliquées dans le repliement et/ou la dégradation de CFTR.
... 163
Figure 59 : Modèle d’ubiquitinylation séquentielle de F508del-CFTR par des complexes contenant RMA1/RNF5 et Gp78... 164
Figure 60 : Les trois principales ligases identifiées chez les mammifères. ... 165
Figure 61 : Devenir des glycoprotéines correctement- ou mal-repliées. ... 167
9
Liste des tableaux
Tableau 1 : Liste non exhaustive des principales protéines du ERQC. ... 11
Tableau 2 : Composition du milieu de culture pour les lignées cellulaires Hela WT- et F508del- CFTR. ... 74
Tableau 3 : Composition du milieu de culture pour la lignée cellulaire CFBE41o-. ... 75
Tableau 4 : Composition du milieu de décontamination pour échantillons CF. ... 77
Tableau 5 : Composition du milieu de culture primaire CF. ... 78
Tableau 6 : Composition du tampon de lyse cellulaire. ... 82
Tableau 7 : Composition des gels d’électrophorèse des protéines. ... 84
Tableau 8 : Composition des tampons 4X Lower et Upper. ... 84
Tableau 9 : Composition du tampon de transfert. ... 84
Tableau 10 : Récapitulatif des anticorps utilisés en western blot. ... 85
Tableau 11 : Récapitulatif des anticorps utilisés en western blot. ... 86
Tableau 12 : Récapitulatif de l’ensemble des anticorps primaires utilisés. ... 90
Tableau 13 : Récapitulatif de l’ensemble des anticorps secondaires utilisés. ... 90
Tableau 14 : Récapitulatif de l’ensemble des anticorps utilisés en Duolink. ... 92
Tableau 15 : Composition du milieu d’efflux utilisé. ... 93
Tableau 16 : Composition de la solution Krebs sans Cl-... 96
10
Introduction
Dans toutes les cellules vivantes, des programmes génétiques différents contrôlent toutes les étapes de la naissance, de la vie et de la mort des protéines. Avant de pouvoir exercer sa fonction biologique, une protéine va subir un ensemble de processus biochimiques complexes entre sa naissance (au moment de la lecture de l’ARN) et sa localisation définitive le temps qu’elle soit fonctionnelle.
Nous allons examiner dans cette introduction quelques-uns des mécanismes et déterminants moléculaires généraux avant d’aborder les particularités de notre protéine d’intérêt, CFTR (Cystic Fibrosis Transmembrane conductance Regulator).
I. Le contrôle qualité des glycoprotéines
Le contrôle qualité du réticulum endoplasmique (RE), ERQC (Endoplasmic Reticulum Quality Control) est un mécanisme de surveillance de la biosynthèse et du repliement des protéines dans le RE. Ce processus est composé de deux voies : la voie ERAF (ER Associated Folding) qui assiste les protéines au cours de leur repliement et s’assure que seules les protéines correctement repliées puissent quitter le RE, et la voie ERAD (ER Associated Degradation) qui séquestre les protéines mal repliées et/ou mutées dans le RE puis les dirige vers la dégradation (Araki and Nagata, 2011).
Ce système est composé de diverses protéines chaperonnes et enzymes dont les principaux acteurs ainsi que leur fonction sont présentés dans le tableau 1.
11
Familles Principaux membres Fonctions
α-glucosidase I et II Clivage des résidus glucoses permettant l’entrée des glycoprotéines dans le cycle calnexine ou calréticuline Enzymes de
glycosylation
UGGT (UDP–glucose glycoprotein:
glucosyltransferase)
Reconnaissance des glycoprotéines mal repliées et re-glycosylation afin de permettre leur entrée dans un nouveau cycle de repliement calnexin ou calréticuline ER α-1,2 mannosidase Clivage du résidu mannose permettant la sortie des
glycoprotéines du cycle calnexine ou calréticuline Malectine Empêche l’agrégation des polypeptides naissants et
recrutement de l’α-glucosidases II Calnexine, Calréticuline Repliement des glycoprotéines Lectines
EDEM1 à 3 Reconnaissance des glycoprotéines mal repliées, clivage des résidus mannoses afin de les diriger vers ERAD OS-9, XTP3-B Reconnaissance des glycoprotéines mal repliées et
adressage vers le complexe de rétrotranslocation Hsp70, BiP/grp78,
Hsp90, Grp94 Empêche l’agrégation des protéines et aide au repliement Hsp40 : ERdj1 à 7 Co-chaperonnes qui régulent BiP
Chaperonnes
Hsp40 : DNAJB12 Co-chaperonne qui dirige les protéines mal repliées vers ERAD
CHIP (carboxy terminus of Hsc70 interacting protein)
E3 ubiquitine ligase, co-chaperonne qui se lie à Hsc70 ou Hsp70 et dirige les protéines mal repliées vers ERAD Oxydo-
réductases
PDI (protéine disulfure
isomérase), ERp57 Formation et isomérisation des ponts disulfures PPIase Cyclophilines Co-chaperonnes qui facilitent le repliement tardif des
substrats de Hsp90
Tableau 1 : Liste non exhaustive des principales protéines du ERQC.
A. La voie ERAF
Chez les eucaryotes, les protéines solubles ou membranaires de la voie de sécrétion sont synthétisées au niveau du RE. Ces protéines doivent alors être transloquées à travers la membrane de ce compartiment, et dans le cas des protéines transmembranaires, s’intégrer dans la bicouche lipidique. Pour cela, les peptides naissants destinés à cette voie de sécrétion présentent une courte séquence hautement hydrophobe qui assure leur adressage vers la membrane du RE. Selon le type de la protéine, soit cette séquence est appelée peptide signal, lorsqu’elle est située dans l’extrémité N-terminal (cas des protéines sécrétées et certaines protéines transmembranaires de type I), soit elle correspond au premier segment transmembranaire de la protéine (McCaffrey and Braakman, 2016).
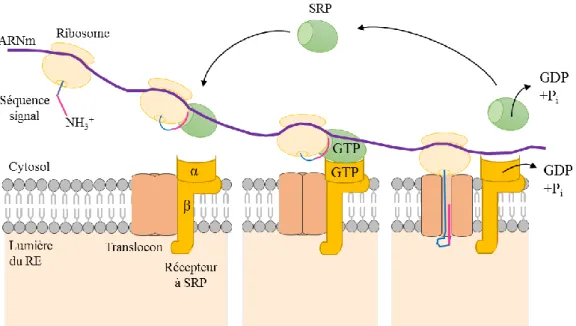
12 Dès sa sortie du ribosome, le peptide signal est reconnu par une particule ribonucléoprotéique SRP (signal-recognition particle) qui après sa fixation provoque un arrêt temporaire de la traduction en bloquant l’accès aux facteurs d’élongation (Figure 1).
Figure 1 : Reconnaissance du peptide signal par la SRP.
Le peptide signal (rose) de la séquence peptidique (bleu) est reconnu par la SRP. Cette dernière interrompt la traduction et favorise la fixation du ribosome au translocon (Sec61). Cela, permet la dissociation de la SRP de son récepteur, le relâchement du peptide signal, son entré dans le translocon et enfin la reprise de la translocation de manière co-traductionnelle (Modifié d’après http://biochemprotein.blogspot.fr).
La SRP est une GTPase qui possède une faible affinité intrinsèque pour le GTP. Son récepteur est à la fois ancré dans la membrane du RE et lié au translocon, un complexe hétérotrimérique de protéines Sec61 (Figure 1).
La fixation de la SRP au complexe « ribosome-chaîne polypeptidique naissante » augmente son affinité pour le GTP ce qui favorise sa fixation à son récepteur. Le ribosome interagit alors avec le translocon Sec61 et permet l’hydrolyse du GTP de la SRP et de son récepteur. Cette hydrolyse provoque la dissociation de la SRP de son récepteur, le relâchement du peptide signal et la reprise de la translocation qui s’effectue de manière co-traductionnelle.
Le peptide signal est clivé après son intégration dans la membrane du RE par l’enzyme signal peptidase et est ensuite dégradé par la signal peptide peptidase (SPP). Dans le cas du signal d’ancrage, la séquence ferait partie de la protéine et permettrait son ancrage dans la bicouche lipidique (Ng and Walter, 1994; McCaffrey and Braakman, 2016).
13
1) Les modifications co- et post-traductionnelles
Dans la lumière du RE, la protéine naissante intègre un processus de repliement lui permettant d’acquérir une structure tridimensionnelle précise (forme native), indispensable à sa transition du RE vers l’appareil de Golgi et à sa fonction. Ce processus commence alors que la chaine polypeptidique est toujours en cours de translocation et de traduction. Il consiste en des modifications post traductionnelles telles que la N-glycosylation et la formation de ponts disulfures sur la protéine cliente.
a) N-glycosylation
Dans les cellules eucaryotes, la grande majorité des polypeptides naissants qui entrent dans la lumière du RE sont glycosylés sur leur résidu asparagine (Asn) contenu dans la séquence consensus -Asparagine-X-Sérine/Thréonine-, où X correspond à n’importe quel acide aminé autre que la proline (Caramelo and Parodi, 2015). Elle se caractérise par le transfert en bloc sur le résidu Asparagine, d’un groupement oligosaccharidique appelé N-glycan. Ce groupement est constitué de 2 résidus N-acetylglucosamine (GlcNAc), 9 mannoses (Man) et 3 glucoses (Glc) (Glc3Man9GlcNAc2) réparti en trois branches A, B et C (Figure 2).
Figure 2 : Structure du motif oligosaccharidique N-glycan.
L’oligosaccharide préformé est composé de 3 glucoses, 9 mannoses et 2 N-acetylglucosamines. Les branches oligosaccharidiques sont représentées par A, B et C. Les enzymes qui modifient ce motif sont listées sur la figure et leur action est représentée. Le type de liaison glycosidique est représenté en couleur (D'après Tannous et al., 2015).
14 Le N-glycan est préalablement assemblé au niveau d’un dolichol-phosphorylé (-PP-dolichol) ancré dans la membrane du RE (Schwarz and Aebi, 2011; Lannoo and Van Damme, 2015). Il est ensuite transféré sur le résidu Asparagine par l’oligosaccharyl transférase (OST), un complexe protéique multimérique situé dans la membrane du RE et associé au ribosome et au translocon sec61 (Harada et al., 2009) (Figure 3).
Figure 3 : Glycosylation d’une protéine naissante.
Le motif oligosaccharidique N-glycan Glc3Man9GlcNAc2 est préassemblé sur un dolichol-phosphorylé ancré dans la membrane du RE. Au cours de sa translocation dans le RE via un canal de translocation, le polypeptide néosynthétisé glycosylé par le complexe OST (oligosaccharyl transférase). Ce complexe transfert le motif N-glycan sur le résidu asparagine (Asn) contenu dans la séquence consensus -Asn-X-Sérine/Thréonine-, où X correspond à n’importe quel acide aminé autre que la proline (Modifié d'après Mazola et al., 2011).
Immédiatement après son transfert sur le polypeptide, le N-glycan est modifié par différentes enzymes glycoside hydrolases (α-glucosidases I et II) et par une glucosyl transférase, l’UGGT (UDP-Glucose:glycoprotein glycosyltransferase) dans une cascade de réactions générant différentes structures qui servent de ligand spécifique aux protéines chaperonnes du RE. Ainsi l’état de glycosylation d’une protéine peut nous renseigner sur son stade de repliement par le contrôle de qualité.
Les N-glycans jouent un rôle majeur à la fois direct et indirect dans les processus de repliement et de contrôle qualité des protéines qui les portent. En effet, de part leur nature encombrante et hydrophile, ces motifs augmentent la solubilité intrinsèque des polypeptides naissants auxquels ils sont attachés et préviennent leur agrégation. De plus, leur fixation aux polypeptides pourrait affecter la structure secondaire locale de la protéine en stabilisant la conformation des résidus proximaux au site de glycosylation. L’analyse de la structure
15 secondaire autour de ce dernier a soulevé, en effet, la possibilité que les glycans puissent agir comme marqueurs des points où des changements se produisent dans la structure secondaire (Petrescu et al., 2004). De plus, il a été montré que l’interaction du N-glycan avec la chaîne polypeptidique facilite l’acquisition de structures secondaires particulières telles que les feuillets β (O’Connor and Imperiali, 1996; Petrescu et al., 2004; Vagin et al., 2008). D’autre part, il a été observé que des acides aminés aromatiques éloignés dans la séquence, deviennent proches aux sites de glycosylation dans la structure tertiaire. Ceci suggère que les N-glycans liés pourraient avoir un rôle encore plus direct dans le repliement, en agissant comme des sites de nucléation pour des parties éloignées de la chaîne protéique riche en acides aminés aromatiques. Cette hypothèse est soutenue par des expériences in vitro indiquant que les N-glycans favorisent le repliement basé sur les interactions hydrophobes glycan-protéine (Nishimura et al., 1998; Jitsuhara et al., 2002). D’autres études comparant des glycoprotéines et leur homologues non glycosylés ont montré que la présence du N-glycan augmentait la stabilité et la résistance à la protéolyse (Beggah et al., 1997; Imperiali and O’Connor, 1999; Kundra and Kornfeld, 1999; Crothers et al., 2004). Une résistance probablement due à la restriction d’accès des sites de clivage à la protéase (Vagin et al., 2008). Enfin, les N-glycans favorisent également le repliement et le contrôle qualité de manière indirecte en recrutant de manière spécifique les chaperonnes et les enzymes impliquées dans le repliement et la dégradation des glycoprotéines (Slominska-Wojewodzka and Sandvig, 2015).
b) Formation et réarrangement des ponts disulfures
La formation des ponts disulfures entre deux résidus de cystéine est essentielle pour le repliement correct et la maturation de beaucoup de protéines de sécrétion. En effet, il s’agit des liaisons covalentes qui stabilisent les structures des protéines et garantissent le maintien de leur conformation tridimensionnelle fonctionnelle. Ce processus, appelé repliement oxydatif, prend place dans le RE et est catalysé par les protéines de la famille PDI (Protein Disulfide-Isomerase).
Les PDIs sont des protéines localisées principalement dans le RE et sont essentielles pour le maintien de son environnement oxydatif et son homéostasie. Elles sont dotées d’une activité oxydoréductase/isomérase et interviennent de manière co- et post-traductionnelle pour catalyser la formation de ponts disulfures (oxydation des groupements thiols des cystéine), leur coupure (réduction) et/ou le réarrangement des liaisons incorrectement établies entre deux cystéines proches (isomérisation) (Wallis and Freedman, 2013) (Figure 4).
16 Figure 4 : Formation des liaisons disulfure par les protéines PDI.
Mécanismes d’oxydation, de réduction et d’isomérisation des liaisons disulfure catalysées par les protéines PDI (D'après Tannous et al., 2015).
En plus de leur activité oxydoréductase/isomérase, la majorité des membres de PDI présentent une fonction chaperonne et jouent un rôle crucial dans l’ERQC. Elles peuvent en effet se lier sur des protéines nouvellement synthétisées pour contribuer à leur repliement, ou bien sur des protéines mal repliées pour faciliter leur dégradation (Ushioda et al., 2008; Lee et al., 2010; Parakh and Atkin, 2015). Ainsi, certains membres de la famille PDI sont des protéines multifonctionnelles, dont leur fonction comme oxydoréductase ou chaperonne est dépendante du substrat (Kozlov et al., 2010a). A noter, l’activité chaperonne du PDI semble être indépendante de son activité enzymatique (Fu and Zhu, 2010).
Plus de 20 membres ont été découverts chez les mammifères. La nécessité d’un si grand nombre de membres de la famille PDI demeure une question ouverte. Cependant, plusieurs membres, les plus abondants ont des spécificités de substrat distinctes (Jessop et al., 2009). Bien que ces membres présentent des séquences en acides aminés et des fonctionnalités diverses, ils présentent tous au moins un site catalytique qui contient un domaine homologue à la thiorédoxine (Trx) (Edman et al., 1985; Appenzeller-Herzog and Ellgaard, 2008; Kojima et al., 2013). PDI est la première oxydoréductase découverte et la plus abondante dans le RE. Ubiquitaire et très fortement exprimée dans les cellules sécrétoires, PDI a la particularité d’assurer les diverses fonctions de la famille : oxydase, réductase, isomérase et chaperonne (Määttänen et al., 2010; Irvine et al., 2014; Okumura et al., 2015).
La protéine ERp57 présente une similarité de séquence significative avec PDI (Oliver et al., 1997). Cependant, au lieu du domaine assurant l’activité de chaperon pour PDI, ERp57 présente une région de liaison pour les domaines P de calnexine et calréticuline, chaperonnes impliquées dans le repliement et le contrôle qualité des glycoprotéines (Zapun et al., 1998; Frickel et al., 2002; Russell et al., 2004; Kozlov et al., 2006). Un rôle spécifique et essentiel dans l’isomérisation de liaisons disulfure non-natives des glycoprotéines néo-synthétisées lui a était attribué (Oliver et al., 1997; Jessop et al., 2007) ; cependant, ERp57 ne semble pas avoir
17 des propriétés intrinsèques de lectine (Zapun et al., 1998). Ces observations suggèrent qu’ERp57 fonctionne de concert avec la calnexine et la calréticuline pour moduler spécifiquement le repliement des glycoprotéines (Oliver et al., 1997; Zapun et al., 1998; Oliver et al., 1999).
Il a été observé qu’ERp57 peut s’associer à des glycoprotéines qui ne présentent pas de résidu de cystéines et ne nécessitent donc pas d’isomérisation de disulfure intramoléculaire (Elliott et al., 1997; Oliver et al., 1997). Le rôle d’ERp57 ne semble donc pas se limiter à la catalyse de la formation et de l’isomérisation de liaisons disulfure. Mais comme PDI, ERp57 peut avoir un rôle plus vaste en tant que chaperonne moléculaire. Son extinction in vivo est létal chez des souris KO (knockout) ERp57 ; en revanche, elle est tolérée in vitro sur des cellules en culture, suggérant que certaines glycoprotéines peuvent engager d’autres assistants de repliement afin d’acquérir leur conformation native. Comme par exemple, ERp72 qui semble pouvoir remplacer ERp57 pour assurer une maturation efficace des glycoprotéines du virus forestier Semliki (Soldà et al., 2006).
c) Les liaisons peptidiques
Les liaisons peptidiques, reliant n’importe quel acide aminé avec un résidu proline peuvent adopter une conformation cis ou trans, avec une faible préférence pour la conformation trans (Fischer and Aumüller, 2003). Les peptidyl-prolylisomérases (PPIases) sont des protéines ubiquitaires capables de catalyser l’isomérisation des acides aminés prolines des protéines de conformation trans en cis et inversement (Figure 5). Cela permet le changement d’orientation de la chaine peptidique dans l’espace.
Figure 5 : Réaction d’isomérisation peptidyl-prolyl cis-trans catalysée par des PPIases. (Tannous et al., 2015)
La correction de l’orientation des liaisons proline a lieu tardivement pendant le processus de repliement et intervient lorsque la protéine a atteint une conformation quasi-complète. L’isomérisation de liaisons peptidyl-prolyle mal placées peut-être une étape limitante pour la vitesse du repliement des protéines (Kiefhaber et al., 1990).
18 La famille PPIase des mammifères comprend trois sous-groupes, parvulines, cyclophilines (Cyps) et protéines de liaison FK506 (FKBPs) (Göthel and Marahiel, 1999; Fischer and Aumüller, 2003; Stocki et al., 2014). Le RE des mammifères contient 6 membres de FKBP et 2 membres de Cyps. La cyclophiline B est la PPIase la plus caractérisée (Bernasconi et al., 2010; Bergeron et al., 2011; Jansen et al., 2012; Stocki et al., 2014). Elle a été identifiée dans un complexe protéique incluant les chaperonnes calnexine et calréticuline. L’absence de site de fixation chaperonne-like sur cette PPIase suggère son interaction avec ses substrats via d’autres chaperonnes. Cette hypothèse a été confirmée par des études de cristallographie identifiant un site d’interaction de CypB à l’extrémité du domaine P de calnexine et calréticuline (Kozlov et al., 2010b). Ces données supportent le rôle actif de CypB dans le cycle calnexine ou calréticuline en favorisant l’orientation correcte des liaisons peptidyl-prolyl dans les protéines en cours de repliement (Kozlov et al., 2010a).
2) Les interactions avec les protéines chaperonnes
a) Les lectines calnexine/calréticuline et les glycosidases
La calnexine et son homologue soluble, la calréticuline, sont deux lectines chaperonnes responsables du repliement et du contrôle qualité des glycoprotéines dans le RE (Fliegel et al., 1989; Wada et al., 1991; Michalak et al., 1992; Hebert et al., 1996). La calnexine est une protéine transmembranaire de type I, de 90 kDa qui interagit avec les glycoprotéines solubles et transmembranaires ; tandis que la calréticuline interagit préférentiellement avec les glycoprotéines solubles (Hebert et al., 1997). Ces deux lectines partagent une organisation générale similaire. Chacune possède dans sa partie N-terminale un domaine globulaire de liaison aux glucides, permettant la liaison spécifique des glycoprotéines monoglucosylées de type Glc1Man9GlcNAc2 (Kozlov et al., 2010c; Schrag et al., 2001). À partir de ce domaine
s’étend une longue boucle en forme de bras appelé domaine P pour sa richesse en résidus de proline. Ce domaine a pour rôle de recruter l’oxydoreductase ERp57 (Frickel et al., 2002; Kozlov et al., 2006) et la PPIase CypB (Kozlov et al., 2010b). Le domaine globulaire des deux lectines contient également des sites de liaison pour les ions Ca2+ et l’ATP, deux cofacteurs régulateurs importants pour la stabilisation des lectines chaperonnes. Le Ca2+ régule également la liaison des lectines aux substrats (pour revue Lamriben et al., 2016).
Bien que réticulaires, ces deux chaperonnes ont également été localisées hors du RE où elles joueraient d’autres fonctions. La calréticuline a été localisée dans le cytoplasme, à la surface
19 externe de la cellule et la matrice extracellulaire ; et la calnexine à la surface cellulaire de nombreuses cellules (Michalak et al., 2009; Wang et al., 2012; Lamriben et al., 2016).
i. Cycle de la calnexine/calréticuline
La forme tri-glucosylée Glc3Man9GlcNAc2 du N-glycan des protéines nouvellement
synthétisées a une demi-vie de quelques secondes. Immédiatement après son transfert sur le résidu asparagine de la chaine polypeptidique naissante, son glucose terminal est clivé avec une t1/2 < 2 min par l’action de l’α-glucosidase I, une enzyme faisant partie du complexe translocon
avec l’OST (Helenius and Aebi, 2004; Dejgaard et al., 2010; Roth and Zuber, 2017).
Le carbohydrate di-glucosylé Glc2Man9GlcNAc2 ainsi généré recrute et s’associe avec la
malectine, une lectine transmembranaire de type I du RE (Figure 6). Le rôle de cette protéine induite par le stress du RE serait de prévenir l’agrégation des polypeptides naissants durant la période de synthèse précoce et de recruter l’α-glucosidase II (Schallus et al., 2008; Galli et al., 2011).
Figure 6 : Interaction des glycoprotéines avec la chaperonne calnexine.
OST : oligosaccharyl transférase, BiP : chaperonne réticulaire, PDI et ERp57 : oxydoréductases, SPC : signal peptide peptidase, GlcI et GlcII : α-glucosidases I et II, Points et traits rouges : liaisons disulfures (D'après Braakman and Hebert, 2013).
L’α-glucosidase II ainsi recrutée clive le deuxième glucose avec une t1/2 ~ 5 min et génère un
carbohydrate mono-glucosylé Glc1Man9GlcNAc2 qui devient ainsi un substrat pour la calnexine
ou la calréticuline (Roth and Zuber, 2017). La calnexine ou calréticuline interagit avec ce N-glycan au niveau de son domaine globulaire, tandis que sa chaîne polypeptidique forme des liaisons disulfures mixtes transitoires avec ERp57. Ces interactions aident au repliement de la glycoprotéine d’une part en réduisant son espace de repliement et en conséquence le nombre de conformations potentielles, et d’autre part, en prévenant son agrégation et en facilitant l’isomérisation des ponts disulfures grâce à l’ERp57 (Jessop et al., 2007; Gidalevitz et al., 2013;
20 Lamriben et al., 2016). Ce dernier semble jouer un rôle supplémentaire dans la stabilisation de l’ensemble de ces interactions.
À la fin de son repliement, la glycoprotéine doit être relâchée de la calnexine ou la calréticuline. L’α-glucosidase II clive alors le dernier glucose du N-glycan et génère le motif Man9GlcNAc2,
avec une t1/2 ~ 20 min (Roth and Zuber, 2017). Le motif généré n’est ainsi plus reconnu par les
deux chaperonnes. Cependant, il pourra être reglucosylé par l’enzyme UGGT afin de restaurer son affinité pour ces lectines, on parle alors de « cycle calnexine/calréticuline » (Figure 7).
Figure 7 : Le Cycle de la calnexine/calréticuline.
(1) Assemblage de l’oligosaccharide Glc3Man9GlcNAc2,(2) Glycosylation de la protéine par l’OST, (3) Clivage de glucose par les α-glucosidases I et II, (4) Liaison de la glycoprotéine à la calnexine ou calréticuline, (5 et 8) Clivage du dernier glucose par l’α-glucosidase II, (6) Re-glucosylation des protéines n’ayant pas atteint leur conformation native par UGGT et (7) leur réentrée dans le cycle calnexine ou calréticuline pour un nouveaux cycle de repliement, (9 et 10) Les protéines correctement repliées sont démannosylées en Man8GlcNAc2 par les mannosidases du RE puis dirigées vers l’appareil de Golgi, (11) Les protéines définitivement mal repliées sont extensivement démannosylées par les mannosidases du RE puis conduites vers la dégradation hors du RE (D'après Lamriben et al., 2016).
21 L’UGGT apparait comme étant un senseur de repliement crucial dans le RE extrêmement sensible, qui peut reglucosyler les glycoprotéines mal repliées pour un nouveau cycle de repliement et ignorer les glycoprotéines natives et fortement mal conformées. Cependant, comment cette spécificité est accomplie reste encore mal comprise.
ii. Distinction des protéines correctement repliées des mal repliées
Une des questions importantes dans l’ERQC est de savoir comment ce processus distingue les glycoprotéines définitivement mal repliées de celles ayant acquis leur conformation native et surtout des intermédiaires de repliement. Il a été proposé que la présence de résidus d’acides aminés hydrophobes exposés sur la protéine mal repliée serait le motif de distinction entre les différentes formes de repliement. Ces motifs seraient reconnus par un « senseurs de repliement », l’enzyme UGGT (Lamriben et al., 2016) ou BiP, une protéine de la famille des Hsp70 (Heat Shock Protein at 70 kDa) (Flynn et al., 1991). En effet, tant qu’une protéine présente des séquences hydrophobes exposées, elle restera retenue dans le RE afin de subir d’autres cycles de repliement et d’atteindre la bonne conformation. A la fin de chaque cycle, soit la protéine est correctement repliée et pourra donc quitter le RE pour transiter vers l’appareil de Golgi, soit elle est reconnue comme définitivement mal repliée et sera alors envoyée vers le système de dégradation, ou bien, cette protéine présente toujours un repliement intermédiaire, elle subira dans ce cas-là, une autre tentative de repliement. Le système basé sur BiP est utilisé principalement pour les protéines non glycosylées ou les glycoprotéines dans lesquelles le N-glycan se produit relativement tard (Otero et al., 2010). Ainsi, nous détaillerons ici uniquement le deuxième système, propre aux glycoprotéines.
Le système de contrôle de qualité des glycoprotéines est basé non seulement sur la reconnaissance des séquences hydrophobes exposées sur la surface des glycoprotéines par l’enzyme UGGT, mais également sur la structure de leur chaine glycanique, d’où le nom de « système basé sur le code glycan ». En effet, comme nous l’avons abordé dans la partie précédente, au cours de son processus de repliement, la glycoprotéine sous sa forme mono glucosylée Glc1Man9GlcNAc2 interagit avec la chaperonne calnexine/calréticuline et subit un
cycle de repliement « cycle de calnexine/calréticuline ». Suite au clivage de son résidu glucose par la glucosidaseII, la glycoprotéine sous la forme Man9GlcNAc2 n’est plus reconnue par la
calnexine. A cet instant, elle va être « scannée » par l’enzyme UGGT qui reconnaît et interagit préférentiellement avec les régions protéiques hydrophobes exposées à la surface des glycoprotéines n’ayant pas atteint leur conformation native. Ainsi, trois cas de figures sont
22 possibles (Braakman and Hebert, 2013) : (1) la glycoprotéine n’expose pas de séquences hydrophobes, elle est donc correctement repliée et pourra quitter le RE et transiter vers l’appareil de Golgi (Geva and Schuldiner, 2014a), (2) la glycoprotéine expose des séquences hydrophobes, elle est donc mal repliée et sera reglucosylée par l’UGGT. Elle reforme alors un complexe avec calnexine-ERp57 afin de subir un nouveau cycle de repliement (Hebert et al., 1995), ou (3) la glycoprotéine présente toujours des séquences hydrophobes avec un repliement anormal, elle sera donc extensivement démannosylée par les mannosidases du RE afin de fournir un signal pour sa prise en charge par le système de dégradation (Helenius and Aebi, 2004).
b) Les protéines de choc thermique (Hsp)
Le système Hsp (Heat Shock Protein) est un système chaperon majeur dans le RE en plus du complexe calnexine/calréticuline (Otero et al., 2010; Balchin et al., 2016). Localisées dans le cytoplasme, le noyau ou même dans le RE, les Hsps sont des chaperonnes inductibles par le stress (Shastry et al., 2002; McClung et al., 2008). Elles sont recrutées pour aider à la maturation des protéines non glycosylées ou des domaines sur des protéines glycosylées (Beckmann et al., 1990). Certains d’entre eux identifient les protéines immatures, aberrantes ou favorables à l’agrégation de par la présence de segments hydrophobes exposés sur ces protéines (Glover and Lindquist, 1998; Ben-Zvi et al., 2004; Li and Srivastava, 2004). Ce système est également impliqué dans la transduction du signal, la régulation du cycle cellulaire, la différenciation et la mort cellulaire programmée (pour revue Jacob et al., 2017). Leur liaison est régulée par des cofacteurs adénine-nucléotidiques et cofacteurs spécialisés. Malgré ces similitudes, leur gamme de substrats et leurs rôles sont divers. Ici, nous allons nous intéresser principalement aux chaperonnes et co-chaperonnes les plus pertinentes pour notre étude.
i. Hsp70 et Hsp40
La famille des Hsp70 regroupe des protéines d’environ 70 kDa inductibles par un choc thermique. Les chaperonnes de ce système se lient à des séquences enrichis en résidus hydrophobes, typiquement exposés par des protéines mal repliées (Rüdiger et al., 1997; Swain et al., 2007; Qi et al., 2013). Leur rôle dans le repliement des protéines néo-synthétisées se caractérise par la prévention de l’agrégation, l’aide au repliement et la solubilisation des protéines agrégées.
23 Les membres de cette famille partagent une séquence composée de deux domaines : un domaine ATPase, nommé NBD (Nucteotide Binding Domain) localisé en N-terminal et un domaine de liaison au substrat SBD (Substrate Binding Domain) localisé en C-terminal. Ce dernier domaine contient une région hydrophobe appropriée en forme de poche à laquelle se fixe la protéine à replier (Mayer and Bukau, 2005). Cette poche est sous le contrôle d’un couvercle étendu, ATP-dépendant, qui peut s’ouvrir et se fermer en emprisonnant le substrat dans la poche (Mayer et al., 2000). Suite à la fixation de l’ATP au domaine NBD, le couvercle s’ouvre, permettant la liaison du substrat au domaine SBD avec une faible affinité. Cette liaison de substrat stimule l’activité ATPase de la chaperonne et par conséquent l’hydrolyse d’ATP (Flynn et al., 1989). Hsp70 lié à l’ADP acquiert ainsi une forte affinité pour son substrat et le couvercle se ferme, isolant la partie de substrat fixé au SBD. Cette liaison avec une haute affinité protège les substrats d’un repliement prématuré et de l’agrégation. Cependant, l’échange de l’ADP par un ATP résulte en l’ouverture du couvercle et par conséquent la libération de la protéine qui peut alors poursuivre son repliement.
Ce cycle de Hsp70 est régulé par des protéines de la famille Hsp40 et des facteurs d’échange de nucléotides NEF (nucleotide exchange factors). Les co-chaperonnes Hsp40 (DnaJ chez les bactéries), connue aussi sous le nom de protéines à domaine-J contiennent une région, appelée domaine J requise pour leur liaison avec les différents membres de Hsp70. Leur liaison stimule et accélère fortement l’hydrolyse de l’ATP, générant l’état fermé de Hsp70 piégeant le substrat (Kityk et al., 2012; Nunes et al., 2015). La liaison ultérieure des NEF au NBD quant à elle, facilite l’échange ADP en ATP, ouvrant le SBD et permettant la libération du substrat pour le repliement ou le transfert vers les chaperonnes en aval ou vers la machinerie de dégradation (Sharma et al., 2010; Braakman and Hebert, 2013).
Parmi les membres de la famille Hsp70, on retrouve la protéine GRP78 (Glucose Regulated Protein), connue sous le nom de BiP pour Binding Immunoglobulin Protein. Il s’agit d’une protéine soluble réticulaire de 78 kDa qui se lie à la plupart des chaines peptidiques naissantes qui transitent dans le RE. BiP est l’une des chaperonnes du RE les plus abondantes et possède de multiples rôles allant du repliement productif à une participation dans le processus ERAD. Il a été proposé que BiP serait un senseur de repliement qui distingue entre les différentes formes de repliement des protéines non glycosylées ou des glycoprotéines dans lesquelles le N-glycan se produit tardivement (Flynn et al., 1991; Otero et al., 2010).
24 L’activité ATPase de BiP, mais aussi sa localisation et ses diverses fonctions, nécessitent l’assistance des co-chaperonnes de la famille Hsp40 connues sous le nom de ERdj 1 à 7 (ER-localized DnaJ proteins) (Braakman and Hebert, 2013). Certains membres d’ERdj jouent un rôle dans le processus de repliement (Shen and Hendershot, 2005; Otero et al., 2010), alors que d’autres s’associent au protéines mal repliées et aident dans l’accélération de leur dégradation (Dong et al., 2008; Lai et al., 2012). Il a été démontré par exemple qu’ERdj5 interagit avec EDEM (ER-degradation enhancing alpha-mannosidase like protein), protéine de type α-mannosidase impliquée dans la dégradation des protéines mal repliées (Ushioda et al., 2008). De même, ERdj4 et ERdj5 surexprimés interagissent avec p97, une composante de la machinerie ERAD (Dong et al., 2008).
Les membres de Hsp70 cytoplasmiques participent aussi au bon repliement des protéines, mais des domaines exposés dans la face cytosolique du RE. Il a été démontré que Hsp70 en association avec Hsp40 et d’autres co-chaperonnes (telles que Hdj1/2, CHIP (C-terminus of Hsp70 interacting protein), HspBP1 (Heat Shock Protein Binding 1)) interviennent en complexe sur des domaines cytoplasmiques des protéines du RE. En fonction du complexe formé, la protéine sera envoyée soit vers la voie de sécrétion soit vers la voie de dégradation (Figure 8).
Figure 8 : Influence des co-chaperonnes du complexe Hsp70 sur le devenir des protéines. CHIP : C-terminus of Hsp70 interacting protein. Hdj : Human DnaJ2. Hop : Hsp-organising protein
Hsp : Heat Shock Protein. (Modifié d'après Goldberg, 2003).
Hdj1 et Hdj2 semblent faciliter le repliement des protéines (Meacham, 1999; Farinha et al., 2002). HspBP1 quant à elle, régule négativement la co-chaperonne CHIP qui augmente l’activité du système ERAD (Connell et al., 2001; Meacham et al., 2001a; Alberti et al., 2004). CHIP est une E3 ligase qui interagit avec une famille d’enzymes de conjugaison d’ubiquitine E2 (E2 ubiquitin conjugating enzymes) et se lie sur Hsp70 ou sa forme constitutivement exprimée, Hsc70 (heat shock cognate) mais également sur Hsp90 (Cyr et al., 2002). Même si
25 les deux chaperonnes lient CHIP de la même affinité, elles présentent des effets opposés sur l’ubiquitinylation de protéines. En effet, il a été proposé que Hsp70 favorise l’ubiquitinylation et la dégradation de substrats, tandis que Hsp90 les inhibe (Peng et al., 2009).
ii. Hsp90
La famille des Hsp90 regroupe des protéines d’environ 90 kDa et qui sont présentes dans la plupart des compartiments cellulaires tels que le cytosol, le RE et les mitochondries. Elles jouent un rôle dans la prévention de l’agrégation des protéines néo-synthétisées et facilitent leur repliement ou leur dégradation. Cette famille de protéine joue également un rôle essentiel dans de nombreux autres processus cellulaires, y compris la réponse de la cellule au stress, le contrôle du cycle cellulaire, la survie cellulaire et d’autres voies de signalisation (Pour revue Jackson, 2012; Mayer and Le Breton, 2015; Schopf et al., 2017).
Les membres de HSP90 fonctionnent comme des homodimères et la dimérisation est essentielle pour leur fonction in vivo (Wayne and Bolon, 2007). Un monomère de HSP90 se compose de trois domaines hautement conservés : un domaine N-terminal NTD, un domaine intermédiaire MD et un domaine C-terminal CTD. Le domaine NTD permet la fixation de l’adénine, du calcium, des co-chaperonnes et régule l’hydrolyse d’ATP (Prodromou et al., 1997). Le domaine MD possède une large boucle hydrophobe qui contrôle la fixation de l’ATP et permet la liaison de substrats. Ces 2 domaines NTD et MD sont connectés par un connecteur long, flexible et chargé qui module les contacts NTD-MD et affecte la fonction de Hsp90 (Hainzl et al., 2009; Tsutsumi et al., 2012; Jahn et al., 2014). Le domaine CTD quant à lui, est responsable de la dimérisation (Harris et al., 2004; Cunningham et al., 2008). Il contient également un motif C-terminal important pour l’interaction avec certaines co-chaperonnes (Buchner, 1999). En l’absence d’ATP, Hsp90 favorise une conformation ouverte. En revanche, lors de la liaison d’ATP, un segment de couvercle de NTD se ferme sur le nucléotide lié, conduisant à une dimérisation de NTD. Après l’hydrolyse de l’ATP et la libération de nucléotides, Hsp90 revient à son état ouvert (Shiau et al., 2006; Hellenkamp et al., 2017). Hsp90 coopère avec divers co-chaperonnes qui régulent son activité ATPase et recrutent les substrats (Li et al., 2012; Röhl et al., 2013) . Ces facteurs agissent en séquentiel le long du cycle Hsp90 et, dans certains cas, forment des complexes mixtes avec la chaperonne (Li et al., 2011). Parmi ces co-chaperonnes, Hop (Hsp-organising protein) et Cdc37 qui stabilisent la conformation ouverte du dimère Hsp90, inhibent l’hydrolyse d’ATP et facilitent la liaison avec le substrat. Hop facilite le transfert de substrat de Hsp70 vers Hsp90, alors que Cdc37
26 fonctionne comme un adaptateur pour les substrats kinase. Une autre co-chaperonne, Aha1 (Activator of Hsp90 ATPase protein 1) se lie asymétriquement au MD et NTD du dimère Hsp90, facilitant la transition vers la conformation fermée et accélérant ainsi l’hydrolyse d’ATP (Panaretou et al., 2002; Meyer et al., 2004; Koulov et al., 2010; Retzlaff et al., 2010). Quant à la co-chaperonne P23, elle agit vers la fin du cycle et facilite la maturation du substrat en stabilisant l’état fermé des domaines N de Hsp90 et en inhibant l’hydrolyse de l’ATP (Prodromou, 2000; Ali et al., 2006; Li et al., 2012). Hsp90 coopère avec une gamme d’autres cofacteurs contenant des domaines TPR (Tetratricopeptide). Certains de ces facteurs contiennent également des domaines peptidyl-prolyl-isomérase (PPIase) et participent au repliement du substrat sur Hsp90 (Taipale et al., 2012). D’autres facteurs contiennent un domaine avec une activité ubiquitine ligase comme CHIP, qui se lie aux deux chaperonnes Hsc70 et Hsp90 à travers son domaine Tetratricopeptide (TPR) (Pratt et al., 2010).
Le mécanisme d’interaction de Hsp90 avec ses substrats et comment son cycle ATPase est couplé à leur maturation n’est pas encore bien compris. Il a été cependant postulé que l’hydrolyse de l’ATP de Hsp90 régulerait le transfert du substrat de Hsp70 vers Hsp90, probablement grâce à un couplage des cycles d’ATP des deux chaperonnes (Kirschke et al., 2014). Des études avaient démontré en effet que Hsp70 et Hsp90 agiraient ensemble mais en séquentiel pour prévenir l’agrégation des chaines polypeptidiques en cours de repliement et que Hsp90 agirait en aval en se liant sur les intermédiaires de repliement tardifs (Kosano et al., 1998). De plus, le rôle de Hsp40, une co-chaperonne de Hsp70 pour l’activité de Hsp90 a été démontré in vivo par des études de génétiques dans la levure montrant que des mutations dans Ydj1 (yeast dnaJ) compromettent la capacité de Hsp90 à chaperonner les récepteurs de stéroïdes ou la protéine kinase pp60v-src (Caplan et al., 1995; Kimura et al., 1995). Sa nécessité pour l’activité de Hsp90 a également été démontrée par des études in vitro sur des complexes de Hsp90 avec le récepteur progestérone (PR) (Kosano et al., 1998; Hernández et al., 2002) ou glucocorticoïde (GR) (Dittmar et al., 1998; Kirschke et al., 2014). Il a été constaté que cette co-chaperonne se lie beaucoup plus facilement à PR dans sa forme native que sous sa forme dénaturée, suggérant que sa liaison serait la première étape pour la voie de chaperon HSP90 pour PR (Hernández et al., 2002).
Parmi les membres de la famille Hsp90, Grp94 (glucose-regulated protein 94), fortement exprimée dans la lumière du RE (Marzec et al., 2012). Il présente de nombreuses similitudes avec les Hsp90 cytosoliques. Cependant, Grp94 lie le calcium dans le RE, possède un ensemble distinct de substrats à plusieurs domaines, tous liées avec des disulfures et ne
27 présente pas de motif Tetratricopeptide (TPR) (Soldano et al., 2003; Dollins et al., 2007; Frey et al., 2007; Immormino et al., 2009; Marzec et al., 2012). Les co-chaperonnes qui pourraient moduler son activité n’ont pas encore été identifiés. Il a été démontré que GRP94 est souvent associée à BiP. L’inhibition par voie pharmacologique ou moléculaire de l’une des deux chaperonnes induit une up-régulation de l’autre chaperonne. GRP94 agirait après BiP en se liant sur les intermédiaires de repliement tardifs. Cette distinction semble être due à des indices structurels différents auxquels la GRP94 est sensible (Gidalevitz et al., 2013). Il a été observé que le knockdown de GRP94 stabilise le substrat classique d’ERAD, α-1-antitrypsin NHK (Null Hong Kong), suggérant que tout comme BiP, cette chaperonne jouerait un rôle dans l’adressage de cette protéine mal repliée vers la voie de dégradation ERAD (Christianson et al., 2008). De plus, une interaction de GRP94 avec OS-9 (OsteoSarcoma amplified 9), une protéine qui reconnait les glycoprotéines mal repliées et les envoie vers la voie ERAD a été observée (Christianson et al., 2008; Braakman and Hebert, 2013).
Tout comme Hsp70 cytosolique, Hsp90 cytosolique interagit avec les domaines cytoplasmiques de différentes protéines insérées dans la membrane du RE, dont la protéine CFTR (Cystic Fibrosis Transmembrane conductance Regulator), et influence leur stabilité.
c) Les chaperonnes de transport hors le RE
La sortie des protéines correctement repliées du RE se produit au niveau de sites spécifiques de la membrane du RE, appelés sites de sortie du RE (ERES pour ER-exit sites), qui sont séparés spatialement des régions où la translocation active a lieu. Au niveau de ces sites, les protéines correctement repliées sont empaquetées dans des vésicules pour leur adressage vers un compartiment situé entre le RE et l’appareil de Golgi appelé ERGIC (ER-Golgi Intermediate Compartment) selon un transport antérograde (Appenzeller-Herzog, 2006; McCaffrey and Braakman, 2016). Ces vésicules de transport sont appelées vésicules COPII (COatomer protein II), en référence à leur complexe de revêtement spécifique qui entraîne l’assemblage et le bourgeonnement de la vésicule. Les vésicules COPII sont composées d’une enveloppe externe comprenant entre autre les protéines Sec13 et Sec31 qui favorisent la polymérisation de revêtement (Coat) ainsi que le processus de bourgeonnement. Elles sont également composées d’une enveloppe interne comprenant les protéines Sec23 et Sec24 qui trient les protéines néosynthétisées par absorption sélective (Lederkremer et al., 2001; Miller et al., 2002, 2003; D’Arcangelo et al., 2013). Les protéines correctement repliées sont en effet
28 sélectivement concentrées par liaison directe ou indirecte à la sous-unité Sec24 afin de permettre leur exportation. Les protéines résidentes du RE et les protéines mal/non repliées ne sont pas reconnues par ce système et ne vont donc pas sortir du RE (Geva and Schuldiner, 2014a).
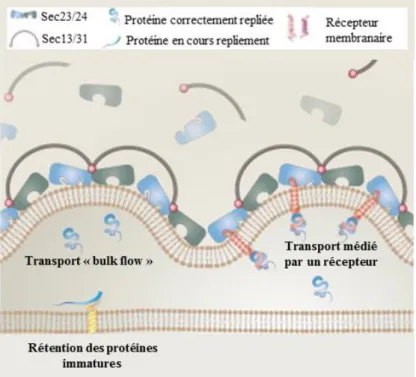
L’empaquetage des protéines dans les vésicules COPII suivrait deux modèles (Figure 9) :
Figure 9 : Modes de transport des protéines hors du réticulum endoplasmique. (Geva and Schuldiner, 2014b)
Le transport « bulk flow » : pour ce mode de transport, les protéines sont incorporées de manière non sélective dans les vésicules COPII. Il est proposé pour ce modèle que les protéines quittent le RE en suivant le fluide de la membrane des vésicules formées sans l’intervention de récepteurs. Ce flux de protéines hors du RE est complètement contrôlé par les chaperonnes et autres composants de l’ERQC qui retiennent les protéines mal/non repliées telles que les chaperonnes calnexine et calréticuline. Ces interactions réduiraient en effet la mobilité des protéines dont la mise en conformation n’est pas terminée, leur accès aux vésicules COPII et ainsi leur sortie du RE. Les protéines ayant atteint leur conformation native, quant à elles, sont libérées de ces interactions et peuvent ainsi pénétrer dans les vésicules COPII d’une manière non sélective et quitter le RE (Balch and Gist Farquhar, 1995; Geva and Schuldiner, 2014b).
29 Le transport médié par un récepteur membranaire : ce mode de transport implique l’interaction des glycoprotéines avec un récepteur membranaire présent aux sites de sorties du RE et capable d’interagir avec le manteau de la vésicule (Balch et al., 1994). Parmi ces récepteurs sont retrouvés ERGIC53, VIP36 (36 kDa Vesicular Integral membrane Protein) et VIPL (VIP36-Like) (Yamamoto, 2014). Ces récepteurs sont classés comme lectines type-L car ils possèdent un domaine homologue de CRD (Carbohydrate Recognition Domain) (Kamiya et al., 2008). Des études antérieures ont révélé qu’ERGIC53 et VIP36 présentent des spécificités et affinités aux substrats différentes malgré des similitudes structurelles dans leur CRD (46% de similitude), suggérant leurs rôles biologiques distincts dans le trafic intracellulaire.
La lectine ERGIC53 est localisée principalement dans le compartiment ERGIC. Ce récepteur qui forme des homodimères et des homohexamères, possède un grand domaine lectine luminale dans la partie N-terminale qui aurait pour rôle de lier les protéines glycosylées, et un domaine dans sa partie cytosolique qui permet la liaison avec les molécules COPII. Constitutivement recyclé entre le RE et l’ERGIC, ERGIC53 facilite le transport du RE vers l’ERGIC de nombreux substrats présentant un motif N-glycan à haute teneur en mannose, de manière dépendante du Ca2+ et du pH (Kamiya et al., 2008; Satoh et al., 2014a; Yamamoto,
2014) (Figure 10). ERGIC-53 semble lier efficacement le mannose de ses substrats à un pH de 7.4 minimum (Kamiya et al., 2005). Il présente une affinité de liaison plus faible et une spécificité plus large que VIP36 et VIPL et peut se lier aux glycans avec une branche A déglucosylée ou monoglucosylée (Kamiya et al., 2008; Satoh et al., 2014b). Il a été révélé que l’expression d’ERGIC53 avec d’autres chaperonnes du RE est surexprimée dans des conditions de stress du RE (Qin et al., 2012).
30 Figure 10 : Transport des glycoprotéines dans la voie sécrétoire (Yamamoto, 2014). Les protéines ayant acquis leur structure correctement repliée dans le cycle de calnexine/calréticuline sont transportées vers le compartiment ERGIC via le récepteur ERGIC53 dans des vésicule COPII. Une fois dans le compartiment ERGIC, les glycoprotéines sont libérées puis transportées vers l’appareil de Golgi, et ERGIC53 est recyclé vers le RE via des vésicules de type COPI. Dans le cis-Golgi, les glycoprotéines sont soit clivées en Man5 avant la reglucosylation par les glycosyltransférases de Golgi, soit marquées avec le signal lysosomale Man-6-P par l’action séquentielle de phosphotransférase (PT) et phosphodiesterase (PD). Certaines glycoprotéines échappent aux clivages, mais elles peuvent être reconnues par VIP36 dans le trans-Golgi et recyclées au cis-Golgi pour une autre tentative de clivage ou bien recyclées au RE pour les transmettre à la chaperonne BiP. Les protéines transportant des résidus Man-6-P sont reconnues par les MPR (mannose 6-phosphate receptors) dans le trans-Golgi et transportées vers les endosomes via des vésicules revêtues de clathrin. Les glycoprotéines correctement repliées sont transportées vers leur localisation physiologique. Ce processus peut être médiés par des lectines qui sont cependant inconnues (Modifié d'après Hauri et al., 2000; Yamamoto, 2014).
31 Une fois dans l’appareil de Golgi, la glycoprotéine est démannosylée par l’α-mannosidase I de Golgi formant la structure Man5GlcNAc2, qui est un substrat pour
N-acetylglucosaminyltransferases (GnT-I), une enzyme clé dans le développement d’organismes multicellulaires. L’addition du premier résidu GlcNAc par GnT-I génère GlcNAcMan5GlcNAc2, substrat pour GnT-III ou α-mannosidase II. La GnT-III oriente la voie
vers des structures hybrides, alors que l’α-mannosidase II clive deux résidus mannose et génère GlcNAcMan3GlcNAc2. Ce dernier peut être encore modifié par une série de GnTs (GnT-II, IV,
V et VI) qui génèrent différentes branches de complexes N-glycans (Vasconcelos-dos-Santos et al., 2015) (Figure 11).
Figure 11 : Représentation schématique de la maturation des oligosaccharides N-liés. GNT-I à V : N-acetylglucosaminyltransferases. FucT : Fucosyltransferase. α-Mann I/II : α-Mannosidase I. DMJ : 1-deoxymannojirimycin. LacNac : N-Acetyl-D-lactosamine. Lex : LewisX. SLex : Sialyl LewisX. Ley : Lewisy (D'après Vasconcelos-dos-Santos et al., 2015).
Les protéines mal repliées qui « accidentellement » se retrouvent transportées par les vésicules COPII jusqu’à l’appareil de Golgi sont recyclées vers le RE selon un transport rétrograde par des vésicules de type COPI (Hsu et al., 1991; Teasdale and Jackson, 1996). Cela est assuré par des molécules de capture, tels que les récepteurs de protéines ou le complexe COPI lui-même qui servent de médiateur à la récupération sélective de protéines mal/non repliées. Ainsi, le compartiment ERGIC est considéré comme un point de contrôle laissant transiter vers l’appareil de Golgi uniquement les protéines correctement repliées (Kukushkin et al., 2011).