© Abderrahim Benmoussa, 2019
Diversité des vésicules extracellulaires dans le lait
bovin et leurs activités dans les maladies
inflammatoires de l'intestin
Thèse
Abderrahim Benmoussa
Doctorat en biologie cellulaire et moléculaire
Philosophiæ doctor (Ph. D.)
Diversité des vésicules extracellulaires dans le lait
bovin et leurs activités dans les maladies
inflammatoires de l’intestin
Thèse
Abderrahim Benmoussa
Sous la direction de :
iii
RÉSUMÉ
Les vésicules extracellulaires (VE) sont des « fragments » de cellules activement libérés dans tous les fluides biologiques. Elles transitent dans la circulation corporelle et transmettent leur contenu bioactif à d’autres cellules. Le lait est le fluide qui contient le plus de VE et celles-ci encapsulent plusieurs éléments bioactifs qui ont des effets anticancéreux, anti-inflammatoire et diminuent notamment les symptômes de la polyarthrite rhumatoïde in vivo. Durant ma thèse j’ai exploré la diversité des VE présentes dans le lait bovin commercial et j’ai étudié leurs activités biologiques dans le cadre des maladies inflammatoires chroniques de l’intestin (MICI).
Les résultats que j’ai obtenus démontrent l’existence de plusieurs populations de VE dans le lait bovin commercial que j’ai pu discriminer grâce à l’utilisation du citrate de sodium pour leur isolation. J’ai découvert qu’elles étaient capables de survivre lors de la digestion in vitro durant laquelle elles protègent leur contenu bioactif, notamment des microARN. Après avoir décrit en détail les différents microARN et protéines encapsulées dans ces VE, j’ai pu trouver des marqueurs spécifiques pour certains sous-ensembles de VE du lait. J’ai aussi montré le transfert de miR-223 bovin à des cellules humaines in vitro et son activité biologique sur l’expression d’un gène rapporteur. J’ai alors exploré l’activité biologique des VE du lait dans un modèle murin de colite induite par le Dextran sulfate sodium (DSS). La prise orale de VE du lait a diminué les symptômes de la maladie, restauré en partie le microbiote intestinal et rétabli la barrière digestive et les niveaux de mucines. J’ai aussi découvert que différentes populations de VE du lait ont différents effets sur l’inflammation du côlon, notamment en modulant le niveau de certains microARN impliqués dans le développement des MICI.
Le lait contient donc différentes VE avec des activités biologiques différentes capables de moduler l’inflammation et le développement de pathologies digestives. L’étude des mécanismes qui sous-tendent leur bioactivité pourrait impacter la prise en charge des maladies inflammatoires, permettrait une amélioration des formulations de lait pour les nouveau-nés et serait d’importance pour la santé publique et le traitement industriel du lait commercial.
iv
ABSTRACT
Extracellular vesicles (EVs) are cellular “fragments” actively released in all biological fluids. They are transported through body circulation and transmit their bioactive content to remote recipient cells. Milk is the biological fluid most enriched in EVs and these encapsulate several bioactive elements with anti-cancer and anti-inflammatory effects and reduce the symptoms of rheumatoid arthritis in vivo. During my thesis, I explored the diversity of EVs present in commercial bovine milk and studied their biological activities in the context of inflammatory bowel diseases (IBD).
The results I obtained demonstrate the existence of several EV subsets in commercial bovine milk that I could discriminate using sodium citrate for their isolation. I found these EVs can survive during in-vitro digestion and protect their bioactive content, including microRNAs. After detailing the different microRNAs and proteins encapsulated in these EVs, I found specific markers for certain populations of milk EVs. I also reported the transfer of vesicular bovine miR-223 to human cells in vitro and its biological activity on the expression of a reporter gene. I then explored the biological activities of milk EVs in a mouse model of DSS-induced colitis. The oral intake of milk EVs decreased the symptoms of the disease, restored part of the intestinal microbiota, restored the intestinal barrier and replenished mucin levels. Also, different populations of milk EVs differentially modulated inflammation in the colon, notably by regulating the level of certain IBD-associated microRNAs.
Milk therefore contains different EV subsets with different biological activities capable of modulating inflammation and the development of digestive pathologies. Studying the mechanisms underlying their bioactivity could impact the management of inflammatory diseases, improve milk formulations for newborns, and be of importance to public health and industrial milk processing.
v
TABLE DES MATIÈRES
Résumé _______________________________________________________________ iii Abstract _______________________________________________________________ iv Table des matières _______________________________________________________ v Liste de Figures et illustrations ____________________________________________ xi Liste des tableaux ______________________________________________________ xiv Liste des abréviations ____________________________________________________ xv Remerciements _______________________________________________________ xviii Avant-propos __________________________________________________________ xx INTRODUCTION ______________________________________________________ 1 1.1. Aspects généraux ________________________________________________ 1 1.2. Les microARN __________________________________________________ 2 1.2.1. Définition et généralités _______________________________________________________ 2 1.2.2. Historique __________________________________________________________________ 2
1.2.3. Biogenèse des microARN ______________________________________________________ 4
1.2.4. Fonction et activité des microARN _______________________________________________ 9
1.2.5. Modulation de la biogenèse et de l’activité des microARN ____________________________ 17
1.2.6. MicroARN extracellulaires ____________________________________________________ 20
1.3. Les vésicules extracellulaires ______________________________________ 23
1.3.1. Introduction, historique et définitions ____________________________________________ 23
1.3.2. Classification des vésicules extracellulaires_________________________________________ 24
1.3.3. Biogenèse des VEs __________________________________________________________ 26
1.3.4. Isolation des vésicules extracellulaires ____________________________________________ 36
1.3.5. Caractérisation des vésicules extracellulaires _______________________________________ 39
1.3.6. Internalisation des vésicules extracellulaires ________________________________________ 44
1.4. Les vésicules extracellulaires du lait ________________________________ 50
1.4.1. Le lait : un aliment fonctionnel _________________________________________________ 50
1.4.2. Les vésicules extracellulaires : des éléments bioactifs du lait ____________________________ 50
1.5. Le système digestif : un équilibre fragile_____________________________ 55
1.5.1. La digestion humaine ________________________________________________________ 55
1.5.2. Le tractus digestif : une barrière protectrice complexe ________________________________ 57
1.6. Les maladies inflammatoires de l’intestin ____________________________ 62
1.6.1. Introduction _______________________________________________________________ 62
1.6.2. Économie, épidémiologie et statistiques __________________________________________ 62
1.6.3. La maladie de Crohn _________________________________________________________ 62
1.6.4. La colite ulcéreuse ___________________________________________________________ 67
1.6.5. Traitement des maladies inflammatoires de l’intestin _________________________________ 69
1.6.6. Modèles expérimentaux ______________________________________________________ 70
CHAPITRE II : RAISONNEMENT, HYPOTHÈSES, ET OBJECTIFS _________ 72
2.1. Raisonnements _________________________________________________ 72
2.1.1. Données préliminaires et littérature ______________________________________________ 72
2.1.2. Limites des précédentes études _________________________________________________ 73
2.2. Hypothèse globale ______________________________________________ 73
2.3. Objectifs ______________________________________________________ 74
2.4. Importance et implications du projet _______________________________ 74
CHAPITRE III. COMMERCIAL DAIRY COW MILK MICRORNAS RESIST
DIGESTION UNDER SIMULATED GASTROINTESTINAL TRACT
CONDITIONS ________________________________________________________ 75
3.1. Avant-propos ___________________________________________________ 75
vi
3.3. Abstract _______________________________________________________ 77
3.4. Introduction ___________________________________________________ 78
3.5. Materials and Methods ___________________________________________ 79
3.5.1. Dairy milk samples __________________________________________________________ 79
3.5.2. TNO GI in vitro model (TIM-1) ________________________________________________ 79
3.5.3. Dairy milk exosome isolation and enrichment ______________________________________ 80
3.5.4. Particle size measurements ____________________________________________________ 81
3.5.5. MicroRNA detection by quantitative PCR (qPCR) __________________________________ 81
3.5.6. Western blot analysis ________________________________________________________ 82
3.5.7. Statistical analysis ___________________________________________________________ 82
3.6. Results ________________________________________________________ 83
3.6.1. Dairy milk microRNAs withstand digestion under simulated GI tract conditions ___________ 83
3.6.2. Dairy milk microRNAs bta-miR-223 and bta-miR-125b that resist digestion are associated with exosomes ________________________________________________________________________ 84
3.6.3. ___ Large amounts of non-digested commercial dairy milk microRNAs are associated with exosomes ________________________________________________________________________________ 86
3.6.4. Most commercial dairy milk microRNAs bta-miR-223 and bta-miR-125b are associated with 200-nm particles sedimenting at a centrifugation speed lower than for exosomes ______________________ 87
3.7. Discussion _____________________________________________________ 88
3.8. Conclusion and declarations ______________________________________ 90
3.8.1. Contribution of the authors ___________________________________________________ 91
3.8.2. Acknowledgements __________________________________________________________ 91
3.8.3. Disclosure of conflict of interests _______________________________________________ 91
3.9. References _____________________________________________________ 92
3.10. Figures _______________________________________________________ 96
3.11. Supplementary material _________________________________________ 104
3.11.1. Supplementary tables _______________________________________________________ 104
3.11.2. Supplementary figures _______________________________________________________ 105
CHAPITRE IV : A SUBSET OF EXTRACELLULAR VESICLES CARRIES THE BULK OF MICRORNAS IN COMMERCIAL DAIRY COW MILK _____________ 111
4.1. Avant-propos __________________________________________________ 111
4.2. Résumé ______________________________________________________ 112
4.3. Abstract ______________________________________________________ 113
4.4. Introduction __________________________________________________ 114
4.5. Material and methods ___________________________________________ 115
4.5.1. Dairy milk samples _________________________________________________________ 115
4.5.2. Dairy milk EV isolation and enrichment _________________________________________ 115
4.5.3. Particle hydrodynamic size measurements ________________________________________ 116
4.5.4. MicroRNA detection by reverse transcription and quantitative PCR (RT-qPCR) ___________ 116
4.5.5. Double stranded RNA labeling and DiR membrane staining __________________________ 117
4.5.6. Protein extraction and measurements ___________________________________________ 117
4.5.7. Western blot analysis _______________________________________________________ 118
4.5.8. Transmission electron microscopy (TEM) _______________________________________ 119
4.5.9. LC-MS/MS_______________________________________________________________ 119
4.5.10. EV labeling and HS-FCM analysis _____________________________________________ 121
4.5.11. Proteinase K and RNase protection assays _______________________________________ 121
4.5.12. Statistical analysis __________________________________________________________ 122
4.6. Results _______________________________________________________ 122
4.6.1. The bulk of milk microRNAs sediment at low ultracentrifugation speeds ________________ 122
4.6.2. The bulk of milk microRNAs cofractionate with a specific subset of EV-associated proteins _ 123
4.6.3. Identification of XDH as a marker of a new microRNA-enriched EV subset in commercial dairy cow milk ________________________________________________________________________ 123
vii
4.6.5. The microRNA-enriched milk EVs have a hydrodynamic diameter of ~200 nm and have a phospholipidic membrane ___________________________________________________________ 127
4.6.6. Cytoplasmic esterase activity supports the presence of cytoplasm and EVs in IDG fractions F7 and F8 ________________________________________________________________________ 128
4.6.7. Transmission electron microscopy (TEM) analysis revealed that milk microRNA-enriched EVs are ~100 nm in diameter _______________________________________________________________ 129
4.6.8. MicroRNAs found in IDG fractions F7 and F8 resist proteinase K and RNAse A digestion, and are likely contained within a distinct subset of EVs ________________________________________ 130
4.7. Discussion ____________________________________________________ 131
4.8. Conclusion and declarations _____________________________________ 136
4.8.1. Contribution of the authors __________________________________________________ 137
4.8.2. Acknowledgements _________________________________________________________ 137
4.8.3. Disclosure of conflict of interests ______________________________________________ 137
4.9. Abbreviations _________________________________________________ 137
4.10. References ____________________________________________________ 138
4.11. Figures ______________________________________________________ 143
4.12. Supplementary figures __________________________________________ 157
CHAPITRE V. COMPLEXITY OF THE MICRORNA TRANSCRIPTOME OF COW’S MILK AND MILK-DERIVED EXTRACELLULAR VESICLES ISOLATED BY DIFFERENTIAL ULTRACENTRIFUGATION ________________________ 162
5.1. Avant-propos __________________________________________________ 162
5.2. Résumé ______________________________________________________ 163
5.3. Abstract ______________________________________________________ 164
5.4. Introduction __________________________________________________ 165
5.5. Material and methods ___________________________________________ 167
5.5.1. Dairy milk samples _________________________________________________________ 167
5.5.2. Sedimentation of dairy milk extracellular vesicles (EVs) by differential ultracentrifugation ____ 167
5.5.3. RNA isolation and sequencing ________________________________________________ 167
5.5.4. Cell experimentations _______________________________________________________ 168
5.5.5. Molecular biology analysis in cell experiments _____________________________________ 169
5.5.6. Statistical analysis __________________________________________________________ 169
5.5.7. Illustrations _______________________________________________________________ 170
5.6. Results _______________________________________________________ 170
5.6.1. Commercial cow’s milk contains different small RNAs, including numerous microRNAs ___ 170
5.6.2. Milk EV subsets separated by differential ultracentrifugation have different small RNA content 171
5.6.3. Different EV subsets contain different microRNAs ________________________________ 172
5.6.4. Different EV subsets contain different isomiRs ___________________________________ 173
5.6.5. Milk EVs enrich human cultured HeLa cells in functional miR-223 _____________________ 174
5.7. Discussion ____________________________________________________ 175
5.8. Conclusion and declarations _____________________________________ 177
5.8.1. Contribution of the authors __________________________________________________ 177
5.8.2. Disclosure of conflict of interests ______________________________________________ 177
5.9. References ____________________________________________________ 178
5.10. Figures ______________________________________________________ 182
5.11. Supplementary material_________________________________________ 190
CHAPITRE VI. IDENTIFICATION OF PROTEIN MARKERS FOR
EXTRACELLULAR VESICLE (EV) SUBSETS IN COW’S MILK ______________ 194
6.1. Avant-propos __________________________________________________ 194
6.2. Résumé ______________________________________________________ 195
6.3. Abstract ______________________________________________________ 196
6.4. Graphical Abstract _____________________________________________ 197
viii
6.6. Introduction __________________________________________________ 198
6.7. Material and methods ___________________________________________ 200
6.7.1. Dairy milk samples _________________________________________________________ 200
6.7.2. Sedimentation of dairy milk extracellular vesicles (EVs) by differential ultracentrifugation ____ 200
6.7.3. Trizol LS protein isolation and resuspension ______________________________________ 200
6.7.4. Sample preparation for liquid chromatography-tandem mass spectrometry (LC-MS/MS) ____ 201
6.7.5. LC-MS/MS_______________________________________________________________ 201
6.7.6. Database searching and Label Free Quantification _________________________________ 202
6.7.7. Statistical analysis __________________________________________________________ 202
6.7.8. Cellular component analysis __________________________________________________ 203
6.7.9. Pathway analysis ___________________________________________________________ 203
6.7.10. Figures and illustrations _____________________________________________________ 203
6.8. Results _______________________________________________________ 203
6.8.1. Milk 35K and 100K pellets have similar protein content, but specific protein markers ______ 203
6.8.2. Can usual EV markers be used to classify commercial dairy cow milk EVs? ______________ 205
6.8.3. Unraveling specific protein markers for the 35K and 100K pellets, and their associated EVs _ 207
6.8.4. Molecular function analysis unveils functional role of 100K milk EVs in translation regulation, galactose and protein metabolism _____________________________________________________ 208
6.8.5. Milk EVs and associated proteins may impact metabolism, translation and immunity modulating pathways upon internalization ________________________________________________________ 208
6.9. Discussion ____________________________________________________ 209
6.10. Conclusion and declarations _____________________________________ 213
6.10.1. Acknowledgments and fundings _______________________________________________ 213
6.10.2. Disclosure of conflict of interests ______________________________________________ 213
6.11. References ____________________________________________________ 213
6.12. Figures ______________________________________________________ 218
CHAPITRE VII. TWO SUBSETS OF EXTRACELLULAR VESICLES FROM COW’S MILK MODULATE SYMPTOMS AND INFLAMMATION IN EXPERIMENTAL COLITIS _____________________________________________________________ 227
7.1. Avant-propos __________________________________________________ 227
7.2. Résumé ______________________________________________________ 228
7.3. Abstract ______________________________________________________ 229
7.4. Introduction __________________________________________________ 229
7.5. Material and methods ___________________________________________ 230
7.5.1. Dairy milk samples _________________________________________________________ 230
7.5.2. Sedimentation of dairy milk extracellular vesicles (EVs) by differential ultracentrifugation ____ 231
7.5.3. Mouse experiments _________________________________________________________ 231
7.5.4. Molecular biology analyses ___________________________________________________ 234
7.5.5. Statistical Analysis __________________________________________________________ 235
7.5.6. Figures and illustrations _____________________________________________________ 236
7.6. Results _______________________________________________________ 236
7.6.1. Milk EVs from P35K and, to a certain extent, P100K, improve recovery of DSS-treated mice 236
7.6.2. Milk EVs alleviate colon dysbiosis in colitic mice __________________________________ 237
7.6.3. Milk EVs restore normal colon histologic architecture and extracellular matrix secretion ____ 238
7.6.4. Milk EVs restore gut barrier after two days of feeding_______________________________ 239
7.6.5. Milk EVs modulate inflammation in DSS-induced colitis ____________________________ 240
7.6.6. P100K EVs down regulate colitis-associated microRNAs ____________________________ 241
7.6.7. P100K EVs increased expression of anti-inflammatory A20 and normalized the levels of COX-2 and ZO-1 _______________________________________________________________________ 242
7.7. Discussion ____________________________________________________ 242
7.8. Conclusion and declarations _____________________________________ 247
7.8.1. Acknowledgements and funding _______________________________________________ 247
7.8.2. Authors’ contributions ______________________________________________________ 248
ix
7.8.4. Competing interests ________________________________________________________ 248
7.8.5. Data availability statement____________________________________________________ 248
7.8.6. Author biography __________________________________________________________ 248 7.9. References ____________________________________________________ 249 7.10. Figures ______________________________________________________ 254 7.11. Supplementary Materials ________________________________________ 263 DISCUSSION ________________________________________________________ 269 8.1. Mise en contexte _______________________________________________ 269
8.2. Résumé des découvertes et discussion _____________________________ 269
8.2.1. Le citrate de sodium, une découverte fondamentale ________________________________ 270
8.2.2. Bioaccessibilité des VE du lait _________________________________________________ 271
8.2.3. Diversité des VE du lait _____________________________________________________ 272
8.2.4. Diversité et répartition des microARN dans les VE du lait ___________________________ 274
8.2.5. Contenu protéique et marqueurs des VE du lait ___________________________________ 276
8.2.6. Activité des VE du lait dans un modèle de colite induite au Dextran Sodium Sulfate ________ 277
8.2.7. Perspectives ______________________________________________________________ 290 CONCLUSIONS ______________________________________________________ 292 RÉFÉRENCES BIBLIOGRAPHIQUES __________________________________ 294 ANNEXE 1. MISEV2018. _______________________________________________ 320 1.1. Avant-propos __________________________________________________ 320 1.2. Résumé ______________________________________________________ 321 1.3. Abstract ______________________________________________________ 322 1.4. Introduction __________________________________________________ 323
1.5. Note on applicability of MISEV2018: species, cells, sample types, and
experimental conditions _________________________________________________ 325
1.6. Nomenclature _________________________________________________ 325
1.7. Collection and pre-processing: pre-analytical variables ________________ 326
1.7.1. Cell culture conditioned media ________________________________________________ 326
1.7.2. Biological fluids ___________________________________________________________ 328
1.7.3. Tissue ___________________________________________________________________ 329
1.7.4. Storage __________________________________________________________________ 330
1.8. EV separation and concentration: how MISEV2014 evolves in 2018 ______ 331
1.9. EV characterization: how MISEV2014 evolves in 2018 _________________ 332
1.9.1. Quantification of EVs _______________________________________________________ 333
1.9.2. Characterization of EVs by their protein composition _______________________________ 336
1.9.3. Single vesicle analysis _______________________________________________________ 341
1.9.4. New recommendation: determine the topology of EV-associated components ____________ 343
1.10. Functional studies: how MISEV2014 evolves in 2018 __________________ 344
1.10.1. Determine the specific versus common functions of different types of EVs ______________ 345
1.10.2. How to normalize amount of EVs used for comparative functional studies ______________ 346
1.10.3. Demonstrate that the activity is observed in the absence of direct cell-cell contact _________ 347
1.10.4. Demonstrate that the activity is predominantly associated with EVs rather than with soluble mediators _______________________________________________________________________ 348
1.10.5. Demonstrate the specific association of the activity with EVs rather than with co-isolated components _____________________________________________________________________ 349
1.10.6. Determine whether a function is specific to exosomes, as compared with other small EVs ___ 350
1.10.7. How to attribute particular effects mediated by EVs to specific EV components __________ 353
1.10.8. Consider whether an EV-dependent function is specific to a given EV source ____________ 354
1.11. General considerations __________________________________________ 354
1.11.1. Reporting requirements _____________________________________________________ 354
1.11.2. Exceptions to compliance with MISEV guidelines _________________________________ 355
1.12. Conclusions ___________________________________________________ 355
x 1.14. Acknowledgments _____________________________________________ 361 1.15. Disclosure statement ___________________________________________ 361 1.16. Funding ______________________________________________________ 361 1.17. Authors’ contributions __________________________________________ 361 1.18. ORCID ______________________________________________________ 361 1.19. References ____________________________________________________ 362 1.20. Tables _______________________________________________________ 379
ANNEXE 2 : MILK MICRORNAS IN HEALTH AND DISEASE _____________ 390
2.1. Avant-propos __________________________________________________ 390
2.2. Résumé ______________________________________________________ 391
2.3. Abstract ______________________________________________________ 392
2.4. List of abbreviations ____________________________________________ 393
2.5. Introduction __________________________________________________ 394
2.6. MicroRNAs: small molecules, but big players _______________________ 394
2.7. Circulating microRNAs: the paradigm shift _________________________ 395
2.8. MicroRNAs in milk ____________________________________________ 396
2.8.1. Abundant microRNA in human breast milk ______________________________________ 397
2.8.2. MicroRNAs found in milk from other species_____________________________________ 398
2.8.3. Cellular origin of milk microRNAs _____________________________________________ 399
2.9. Milk microRNAs in health and disease _____________________________ 400
2.9.1. Milk microRNAs as biomarkers _______________________________________________ 400
2.9.2. Limits for bioactivity of milk microRNAs ________________________________________ 402
2.9.3. Milk extracellular vesicles: functional carriers of microRNAs _________________________ 403
2.9.4. Evidence for the bioavailability of milk extracellular vesicles __________________________ 404
2.9.5. The bioactivity of milk extracellular vesicles ______________________________________ 404
2.9.6. Milk microRNA stability and bioaccessibility______________________________________ 406
2.9.7. Transfer and function of milk microRNAs _______________________________________ 408
2.10. Limitations and future perspectives _______________________________ 415
2.11. Conclusion and statements ______________________________________ 417
2.12. Contribution of the authors ______________________________________ 418
2.13. Acknowledgements ____________________________________________ 418
2.14. Disclosure of conflict of interests __________________________________ 418
2.15. References ____________________________________________________ 418
2.16. Figures ______________________________________________________ 431
2.17. Tableaux _____________________________________________________ 433
ANNEXE 3 : PRE-TREATING MILK WITH SODIUM CITRATE AS A KEY STEP TO ISOLATE MILK EXTRACELLULAR VESICLES _______________________ 435
3.1. Avant-propos __________________________________________________ 435
3.2. Résumé ______________________________________________________ 435
3.3. Abstract ______________________________________________________ 435
3.4. Letter to the editor _____________________________________________ 436
3.5. References ____________________________________________________ 439
xi
LISTE DE FIGURES ET ILLUSTRATIONS
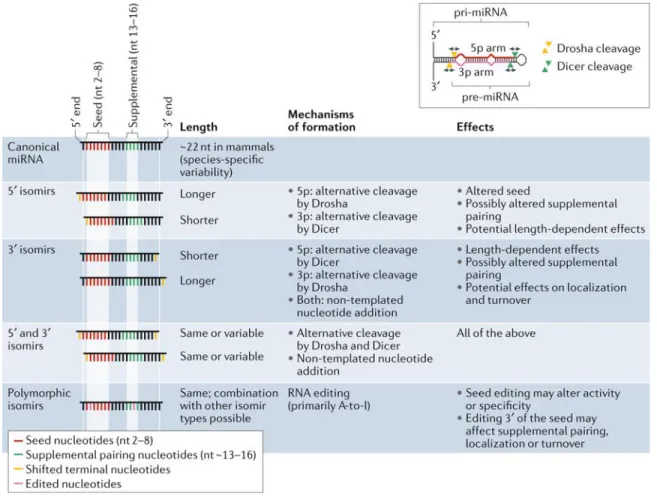
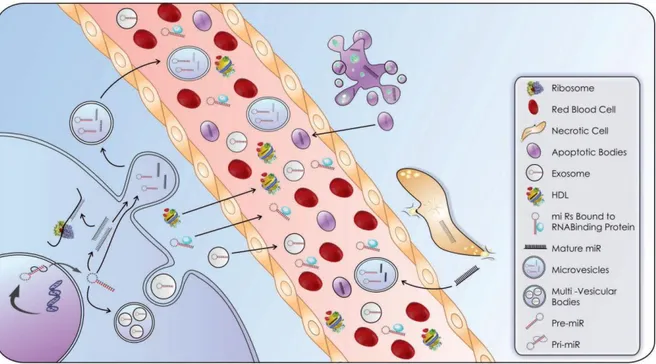
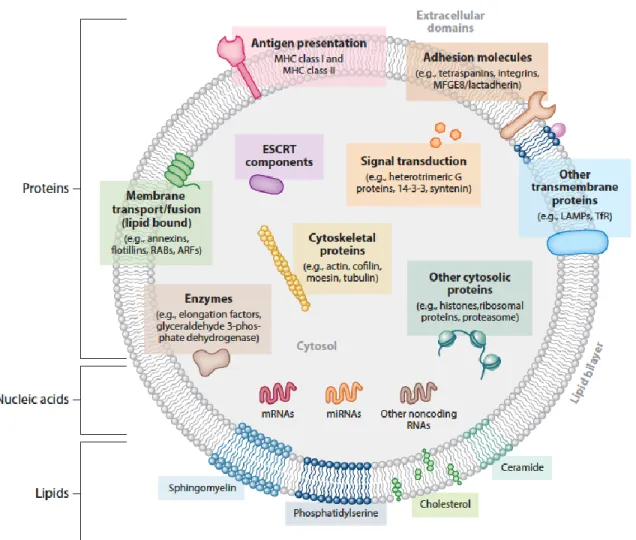
Chapitre IFigure 1.1. Biogenèse et fonction d’un microARN typique. __________________________ 6 Figure 1.2. Voies canoniques et alternatives de biogenèse des microARN. ______________ 8 Figure 1.3. Mécanisme d’action des microARN. _________________________________ 12 Figure 1.4. Les différents types d’isomiRs et leur activité biologique. __________________ 16 Figure 1.5. Les microARN extracellulaires sont contenus et protégés par différentes particules incluant des complexes RNP et des corps vésiculaires. _____________________________ 22 Figure 1.6. Différentes vésicules peuvent être libérées par les cellules de mammifères._____ 25 Figure 1.7. Biogenèse des exosomes. _________________________________________ 29 Figure 1.8. Biogenèse des microvésicules. ______________________________________ 32 Figure 1.9. Aspect général d’une vésicule extracellulaire. ___________________________ 42 Figure 1.10. Fixation et internalisation des vésicules extracellulaires (VE). ______________ 46 Figure 1.11. Rang d’abondance des microARN du lait. ____________________________ 52 Figure 1.12. Anatomie et composants de la barrière intestinale. ______________________ 59 Figure 1.13. Étiologie et composantes immunitaires des MICI. ______________________ 66
Chapitre III
Figure 3. 1. Absolute quantification and distribution of dairy milk microRNAs bta-miR-223 and bta-miR-125b in the gastrointestinal (GI) tract during digestion under simulated conditions. 97 Figure 3. 2. Dairy milk microRNAs bta-miR-223 and bta-miR-125b that resist digestion are associated with exosomes. __________________________________________________ 98 Figure 3. 3. The majority of digested dairy milk microRNAs bta-miR-223 and bta-miR-125b is associated with 200-nm particles sedimenting at a centrifugation speed lower than for exosomes. _____________________________________________________________________ 100 Figure 3. 4. Large amounts of non-digested commercial dairy milk microRNAs are associated with exosomes. _________________________________________________________ 101 Figure 3. 5. Most commercial dairy milk microRNAs bta-miR-223 and bta-miR-125b are associated with 200-nm particles sedimenting at a centrifugation speed lower than for exosomes. ____________________________________________________________ 103 Supplementary Figure 3. 1. Picture of the TNO gastrointestinal (GI) in vitro model-1 (TIM-1) simulating the digestion of dairy milk in healthy adults. _________________________ 106 Supplementary Figure 3. 2. Plan of the experimentations performed on digested (left) and non-digested (right) commercial dairy milk. ____________________________________ 107 Supplementary Figure 3. 3. The TIM-1 effluent sample obtained after 30 min of digestion contains several bovine microRNAs that withstand digestion. ______________________ 108 Supplementary Figure 3. 4. Fractions F5 to F7 derived from Iodixanol density gradient (IDG) of commercial dairy milk are the most enriched in microRNAs bta-miR-223 ___________ 109 Supplementary Figure 3. 5. TIM-1 effluent sample obtained after 30 min of digestion contains several bovine microRNAs that withstand digestion and sediment at lower speed than exosomes. _____________________________________________________________ 110
xii Chapitre IV
Figure 4. 1. The bulk of milk microRNA pellet a low ultracentrifugation speeds and their distribution does not correspond to EV-associated proteins profiles. _________________ 144 Figure 4. 2. Identification of XDH as a protein marker associated to the bulk of microRNAs in commercial milk. ________________________________________________________ 145 Figure 4. 3. Cow milk microRNAs are contained within 12,000 g and 35,000 g EVs of discrete density. _____________________________________________________________ 148 Figure 4. 4. The composition protein of the 12,000 g and 35,000 g pellets is compatible with the existence of multiple EV subsets in milk pellets. _____________________________ 149 Figure 4. 5. The presence of EVs in low-speed ultracentrifugation pellets of IDG fractions is supported by detection of particles with hydrodynamic diameter of ~200 nm and of phospholipids. _________________________________________________________ 150 Figure 4. 6. High-sensitivity flow cytometry analysis of fractionated IDG revealed the peculiar enrichment of EVs in the 12,000 g pellet. _____________________________________ 152 Figure 4. 7. Transmission electron microscopy (TEM) analysis revealed that milk microRNA-enriched EVs are ~100 nm in diameter. ______________________________________ 153 Figure 4. 8. Proteinase K and RNAse A sensitivity of the microRNA-enriched 12,000 g and 35,000 g EVs and associated microRNAs. _____________________________________ 155 Figure 4. 9. Schematic illustration of the new EV subset carrying the bulk of microRNAs in commercial dairy cow milk. ________________________________________________ 156 Supplementary Figure 4. 1. RNA in commercial milks. __________________________ 158 Supplementary Figure 4. 2. Estimation of MP enrichment by using the dynamic light scattering system attenuator position (ATTN). _________________________________________ 159 Supplementary Figure 4. 3. Milk EVs found in 12,000 and 35,000g pellets resist proteinase K digestion. _____________________________________________________________ 161 Chapitre V
Figure 5. 1. Small RNA and microRNA profile of commercial cow’s milk. ____________ 183 Figure 5. 2. Small RNA profiles of milk EV subsets. ____________________________ 185 Figure 5. 3. MicroRNA distribution in milk EV subsets. __________________________ 187 Figure 5. 4. MicroRNA isomer (isomiR) distribution in milk EV subsets. _____________ 188 Figure 5. 5. Milk EVs enrich human cultured HeLa cells in functional miR-223. ________ 189 Supplementary Figure 5. 1. Small RNAs sequencing comparing milk microRNAs with other microRNAs datasets._____________________________________________________ 191 Supplementary Figure 5. 2. Bta-miR-30a and its isomiRs may have different mRNA targets.
_____________________________________________________________ 193 Chapitre VI
Graphical Abstract 6. 1. Bovine milk Contains two EVs subsets with different protein markers _____________________________________________________________ 197 Figure 6. 1. Quantitative proteomic identification of milk proteins that can be sedimented by differential ultracentrifugation. _____________________________________________ 219 Figure 6. 2. Comparative enrichment of canonical EV protein markers in the 35K and 100K pellets. _____________________________________________________________ 221
xiii
Figure 6. 3. Identification of the proteins differentially enriched between 35K and 100K pellets. _____________________________________________________________ 222 Figure 6. 4. The predicted molecular function of the proteins associated to the milk pellets.
_____________________________________________________________ 223 Chapitre VII
Figure 7. 1. Milk EVs prevent weight loss and modulate disease activity during experimental colitis. _____________________________________________________________ 255 Figure 7. 2. Milk EVs help restore colon length and manage colitis-induced colonic dysbiosis.
____________________________________________________________ 256 Figure 7. 3. Milk EVs help restore colon histologic integrity, manage inflammation and regulate extracellular matrix (ECM) production. _______________________________________ 257 Figure 7. 4. Milk EVs restore colon permeability in vivo. __________________________ 259 Figure 7. 5. Milk EVs modulate cytokine production in the colon. __________________ 260 Figure 7. 6. P100K EVs downregulated colitis-associated microRNAs, including miR-125b, leading to higher levels of the anti-inflammatory A20 and restored expression levels of COX2 and Zo-1. _____________________________________________________________ 261 Figure 7. 7. Putative and confirmed mechanisms through which milk EVs modulate inflammation. __________________________________________________________ 262 Supplementary Figure 7. 1. Schematic representation of the experimental procedures. __ 263 Supplementary Figure 7. 2. Supplementary Figure S2. Setup of the mouse DSS-induced colitis model. _____________________________________________________________ 265 Supplementary Figure 7. 3. Ingestion of DSS and P35K EVs influence colonic bacterial levels after 4 and 14 days, respectively. ____________________________________________ 267 Annexe 2
Annexe 2. Figure 1. The rank of milk microRNAs, as determined by small RNA sequencing studies. _____________________________________________________________ 431 Annexe 2. Figure 2. Wordle representation of the top 10 targets known to be regulated by the most expressed milk microRNAs. ___________________________________________ 432 Annexe 3
Annexe 3. Figure 1. Addition of sodium citrate to commercial cow’s milk prevents casein gel formation upon ultracentrifugation. __________________________________________ 442
xiv
LISTE DES TABLEAUX
Chapitre III
Supplementary table 3. 1. Parameters of the TNO gastrointestinal (GI) in vitro model-1 (TIM-1) simulating the GI transit associated with the digestion of a liquid meal in healthy adults. 104 Chapitre VI
Table 6. 1. Pathways involving the proteins common to both 35K and 100K pellets. ____ 224 Table 6. 2. Pathways involving 35K proteins. __________________________________ 225 Table 6. 3. Pathways involving 100K proteins. _________________________________ 226 Chapitre VII
Supplementary table 7. 1. RT-qPCR primers used to quantitate bacterial strains. _______ 268 Annexe 1
Annexe 1. Table 1. Considerations for EV separation/enrichment. _________________ 379 Annexe 1. Table 2. Steps of EV characterization. _______________________________ 381 Annexe 1. Table 3. Protein content-based EV characterization. ____________________ 383 Annexe 1. Table 4. EV-associated and EV-excluded biological activities. _____________ 386 Annexe 1. Text Box 1. Example of a change from MISEV2014 as a paradigm for considering negative markers. _______________________________________________________ 387 Annexe 1. Text Box 2. Example of a possible change from MISEV2018 in the future as markers of non-EV co-isolated components. _________________________________________ 388 Annexe 1. Text Box 3. Aside on uptake. _____________________________________ 389 Annexe 2
Annexe 2. Table 1. The top 10 microRNAs in human, cow and goat milk, whey, fat, and/or extracellular vesicles (EVs). ________________________________________________ 433
xv
LISTE DES ABRÉVIATIONS
3’UTR, 3’ Untranslated Region ACTN1, Actin 1
ADARs, Adenosine Deaminases Acting On RNA AGO, Argonaute
ALIX, ALG-2 Interacting Protein X AMP, Antimicrobial Peptides ANOVA, Analysis Of Variance
ARF6, Ras-Related GTPase ADP-Ribosylation Factor 6 ARNdb, ARN Double Brin
ARNsb, ARN Simple-Brin ARNt, ARN de transfert
ARRDC1, Arrestin Domain-Containing Protein-1 ATTN, Attenuator Position
BTA, Bos Taurus
BTN1A1, Butyrophilin Subfamily 1 Member A1 C3B, Complement Component 3
C8B, Complement C8 Beta Chain CCR4, Carbon Catabolite Repression 4
CD, Cluster De Differentiation/ Cluster Domain (p. ex.
CD9)
CDH1, E-Cadherin
CeRNA, Competing endogenous RNA CLDN8, Claudin-8
CMVs, Corps Multivésiculaires CNS, Central Nervous System COX-2, Cyclo-Oxygénase-2 CRTAP, Cartilage-Associated Protein
CSDC2, Cold-Shock Domain-Containing Protein 2 CSN1, S1 Alpha-S1-Casein
CYC1, Cytochrome C
DCP1-DCP2, mRNA-Decapping Enzymes 1 Et 2 DCTN2, Dynactin Subunit 2
DGCR8, Digeorge Syndrome Critical Region Gene 8 DiR, Carbocyanine Dioc18
DLS, Dynamic Light Scattering DNA, Deoxyribonucleic Acid DNMT, DNA Methyl Transferase DSS, Dextran Sulfate Sodium ECC, Enterochromaffin Cell ECL, Enhanced Chemiluminescence EDTA, Ethylenediaminetetraacetic Acid EHD4, EH Domain Containing 4 EiR, Eukaryotic Initiation Factor
EmPAI, Exponentially Modified Protein Abundance Index
ENS, Enteric Nervous System EP, Extracellular Particle
EPS15, Epidermal Growth Factor Receptor Substrate 15 ESCRT, Endosomal Sorting Complexes Required For Transport
ESI MS/MS, Electrospray Mass Spectrometry
EV, Extracellular Vesicle F7/F8, Fraction 7/8 FASN, Fatty Acid Synthase FcRn, Neonatal Fc Receptor FDR, False Discovery Rate FN1, Fibronectin 1 Foxp3, Forkhead Box P3
FPLC/ HPLC, Fast Protein/High Perfomance Liquid Chromatography
FRQS, Fond De Recherche Québec Santé FSC, Forward Scatter
GALT, Gut-Associated Lymphoid Tissue
GC/MS, Gaz Chromatography/Mass Spectrometry GDI2, Rab GDP Dissociation Inhibitor Beta GGLs, Globule gras du Lait
GI, Gastrointestinal GLRX3, Glutaredoxin-3 GO, GeneOntology
HCD, Higher Energy Collision-Induced Dissociation HDAC, Histone Deacetylases
HD-EVs, High Density EVs HDL, High Density Lipoproteins
HnRNP, Heterogeneous Nuclear Ribonucleoprotein HRP, Horseradish Peroxidase
HSC70, Heat Shock Cognate 70
HS-FCM, High Sensitivity Flow Cytometer HSP, Heat Shock Protein
IBD, Inflammatory Bowel Diseases ICAMs, Intercellular Adhesion Molecules IDG, Iodixanol Density Gradient IDH, Isocitrate Dehydrogenase IEC, Intestinal Epithelial Cell
IGF-1R, Insulin-Like Growth Factor 1 (IGF-1) Receptor IRSC, Instituts De Recherche En Santé Du Canada IS, Immune System
ISC, Intestinal Stem Cell
ISEV, International Society For Extracellular Vesicles ITGAV, Integrin Alpha-V
JEV, Journal Of Extracellular Vesicles KDa, KiloDalton
KO, Knock Out
LAMP2, Lysosome-Associated Membrane Protein 2 LC-MS/MS, Liquid Chromatography Tandem-Mass Spectrometry
LD-EVs, Low Density EVs LFQ, Label Free Quantification LncRNAs, Long Non-Coding RNA LPS, Lipopolysaccharide
xvi L-Th, Lymphocytes T Helper
MAN2A, Mannosidase 2 Alpha MDP, Muramyl-Dipeptides MFG, Milk Fat Globule
MFG-E8, Milk-Fat Globule Membrane Element 8 MFGM, MFG Membranes
MICI, Maladies Inflammatoires Chroniques de
L’intestin
MiRNP, MicroRNA Ribonucleoprotein Complex MISEV2018, Minimal Information For Studies Of Extracellular Vesicles 2018
MP, Microparticle
MPO, Myélopéroxydase mRNA, Messenger RNA
mTOR, Mammalian Target Of Rapamycin MVB, Multivesicular Bodies
MVEs, Multivesicular Elements MVP, Major Vault Protein
NCAM2, Neural Cell Adhesion Molecule 2
NFκB, Nuclear Factor Kappa-Light-Chain-Enhancer Of Activated B Cells
NKT, Natural Killer T-Lymphocytes NLR, NOD-Like Receptors
NLRP6, NOD-Like Receptor Family Pyrin Domain Containing 6
NOT1, Negative On TATA-Less 1 NTA, Nanoparticle Tracking Analysis OMVs, Outer Membrane Vesicles PACT, Protein Activator Of PKR
PAEP, Progestagen-Associated Endometrial Protein Analog
PAGE, Polyacrylamide Gel Electrophoresis PAZ, Piwi-Argonaute-Zwille
P-Bodies, Processing Bodies PBS, Phosphate Buffered Saline
PCC, Phosphate de Calcium Colloidal PCNA, Proliferating Cell Nuclear Antigen PHGDH, Phosphoglycerate Dehydrogenase PI3K, Phosphoinositide 3-Kinase
PIGR, Polymeric Immunoglobulin Receptor PLA2, Phospholipase Sécrétoire A2 PLIN2, Perilipin
PLOD3, Procollagen-Lysine 2-Oxoglutarate 5-Dioxygenase 3
PMSF, Phenylmethylsulfonyl Fluoride PMTP, photomultiplier Tube
Pol II, ARN Polymérase De Type II
PPAR-γ, Peroxisome Proliferator-Activated Receptor Gamma
PRKAR2B, Protein Kinase CAMP-Dependent Type II Regulatory Subunit Beta
PRR, Pattern Recognition Receptor VE, Vésicules Extracellulaires
VILs, Vésicules Intraluminales PVDF, Polyvinylidene Fluoride qPCR, Quantitative PCR Rab, Ras-Related Proteins
RanGTP, Ran (Ras-Related Nuclear) Protein - Guanosine Triphosphate
REG3G, Regenerating Islet-Derived Protein 3 Gamma RhoB, Rho Gtpase B
RI, Refractive Index
RISC, RNA-Induced Silencing Complex RNA, Ribonucleic Acid
RNAse, Ribonuclease RPS, Resistive Pulse Sensing
RT-qPCR, Reverse Transcription and Quantitative Polymerase Chain Reaction
SCFA, Short Chain Fatty Acids SD, Standard Deviation SDS, Sodium Dodecyl Sulfate SEM, Standard error of the mean SERPIN, Serine Protease Inhibitor sIgA, Secretory IgA
SN, Supernatant
SNARE, Soluble NSF Attachment Protein Receptors SoPs, Standard Operating Procedures
SPR, Plasmon Resonance SYT, Synaptotagmin
TAR, Trans-Activation Response TBS, Tris Buffered Saline
TDMD, Target RNA–Directed miRNA Degradation TEM, Transmission Electron Microscopy
TGA, Transcriptional Gene Activation TGF-ß, Tumor Growth Factor ß TGI, Tractus Gastro-Intestinal TGS, Transcriptional Gene Silencing
TIM-1, TNO “Toegepast Natuurwetenschappelijk Onderzoek” Intestinal Model
TLR, Toll-Like Receptor
TNFAIP3, Tumor Necrosis Factor, Alpha-Induced Protein 3 (A20)
TReg, T-Regulatory Lymphocyte TSG-101, Tumor Susceptibility Gene-101 TSPAN4, Tetraspanin-4
TUT, Terminal Uridyl Transferases UHT, Ultra-Haute Temperature UPR, Unfolded Protein Response UTR, Untranslated Region
XDH, Xanthine Dehydrogenase/Oxidase XPO5, Exportin 5
ZO-1, Zonua occludens 1 UV, Ultraviolet
xvii
« Songez que toutes les merveilles, objets de vos études,
expriment l’œuvre de plusieurs générations ; une œuvre collective exigeant de tous un effort enthousiaste, et une peine certaine.
Tout cela, dans vos mains, devient un héritage. Vous le recevez, vous le respectez, vous l’accroissez et, plus tard ;
vous le transmettez fidèlement à votre descendance. Nous sommes ainsi des mortels immortels parce que nous créons, ensemble, des œuvres qui nous survivent. »
Albert Einstein
xviii
REMERCIEMENTS
Tout voyage commence par de petits pas. Des sommets à franchir, des vallées à découvrir, des merveilles à voir et des déceptions à subir. Pour moi, ce voyage a commencé lorsque j’ai quitté mon pays de naissance, l’Algérie, puis s’est poursuivi lorsque j’ai quitté mon pays d’adoption, la France, qui m’a vu grandir, pour enfin venir explorer le Nouveau Monde. Bien que ce ne soit pas une finalité en soit, une étape de ce voyage se termine avec ce doctorat. Presque 5 années, intenses et formatrices. 5 années de souffrances et de bonheurs, de fierté et d’angoisses ; 5 années d’une vie loin de ma famille, loin de ma conjointe, mais loin d’être seul. Les succès que j’ai pu connaitre, durant ce temps, et ma résilience face l’adversité, je les dois aux nombreuses personnes qui ont été là pour moi.
Tout d’abord, je voudrai remercier mon directeur de recherche Patrick Provost pour m’avoir accepté au sein de son équipe, et pour son support tout au long de mon doctorat. Merci de m’avoir encouragé à travers les moments difficiles, les moments de doute, merci pour tous ces moments d’échanges personnels et professionnels, merci pour tous tes efforts pour m’aider à développer mon esprit critique et de synthèse, pour m’aider à ne pas me perdre dans les multiples projets. Merci pour ton mentorat, au travers duquel j’ai pu apprendre tant sur le métier de chercheur, et sans lequel je n’en serais pas là aujourd’hui. Surtout merci pour ton engagement pour tes étudiants, et merci pour tout ce que tu m’as appris. Tout cela n’a pas de prix pour moi, mais revêt la plus grande des valeurs.
Je tiens aussi à remercier particulièrement la Dre Caroline Gilbert qui m’a tant appris et transmit durant ce doctorat. Aucun de ces projets n’aurait abouti sans tes conseils, ton expertise et ton excellence lors de nos collaborations. Je remercie, dans la foulée, toutes les étudiantes et tous les étudiants de Caroline, qui m’ont transmis leurs connaissances et m’ont formé pour l’étude des vésicules extracellulaires. Je pense notamment à mon collègue, et ami, Julien Vitry, à Audrey Hubert, Caroline Subra, Alma Posvandzic et Myriam Vaillancourt entre autres. Votre enseignement a été indispensable au développement de mes compétences. Je remercie aussi les nombreux collaborateurs/trices avec qui j’ai travaillé durant ce doctorat. Je pense aux Drs Éric Boilard, Ismail Fliss, Jean Sevigny, Arnaud Droit, Iwona Rudkowska et tous les membres de leurs équipes dont Sylvie Bourassa, Clarisse Gotti, Patricia Savard, Charles Joly Beauparlant, et Céline Bourdon, entre autres. Un remerciement particulier pour Mabrouka Salem pour sa gentillesse, et son excellence, et pour tout ce qu’elle m’a appris sur les maladies inflammatoires de l’intestin. À tous, votre excellence et vos conseils sont les socles sur lesquels j’ai pu bâtir cette thèse et sans lesquelles rien de cela n’aurait été possible.
Un grand merci à Geneviève Pepin pour m’avoir encadré et accueilli au sein du laboratoire, pour m’avoir fait sentir à ma place, et pour avoir encourager mes idées et cadrer mes pensées. Merci aussi à Benoit Laffont pour tout ce que tu m’a transmis et pour ta bonne humeur et ton énergie pour le développement de mes projets. Toute ma reconnaissance à Natalie Majeau avec qui j’ai eu le plaisir de travailler pendant quelque temps. Ta participation à mon projet de recherche a été plus que décisive pour l’avancée de mes travaux ! J’ai aussi une pensée émue pour feue Véronique Dorval qui nous a quittés prématurément. Yes, she was a « drummer », qui a lutté jusqu’au bout et son passage parmi nous restera gravé dans nos mémoires.
Je ne saurai oublier Jonathan Laugier, alias « Pepito ». Un collègue, un camarade de sport et de jeux, « engraineur professionnel » et surtout un ami sincère et un mec au grand cœur. Ton chemin t’aura mené vers d’autres horizons que la recherche, mais je suis certain que tu te destines
xix
à de grandes choses. Je pense aussi à ma collègue et amie Marine Lambert. On en aura passé des choses ensemble. Des bons moments, des moments difficiles, on en aura vécu des péripéties, mais on s’en est sorti et on a grandi ensemble. Merci pour tout Marine, je n’aurai pas assez de lignes pour t’exprimer ma gratitude ici. Il en est de même pour mon camarade et ami Idrissa Diallo. Merci pour ta bonne humeur quotidienne, même dans l’adversité, merci pour ton engagement et toute l’aide que tu as pu donner pour ces travaux. C’est avec des personnes comme toi que le monde va bouger ; ne lâche pas, l’ami ; you will get through and thrive ! Merci aussi à tous les étudiants, et toutes les étudiantes, que j’ai pu encadrer : Chris Lee, Sophia Ly, Christine Shan et Sara Michel. Je n’ai aucun doute, au vu de votre excellence, que vous irez loin.
Mes plus chaleureux remerciements à toutes les personnes que j’ai pu côtoyer, de près ou de loin, avec qui j’ai pris quelques verres ou participé à quelques aventures en nature. Je pense, sans ordre particulier, à Isabelle Allaeys, Manu, Audrée, Romu, Tania, Halima, Anthony « le Breton », Yasmine, Anne, Jean, Imène, Nicolas, Sofiane, Étienne et Étienne, Maria, et tous les autres que je n’ai pas cités, mais auxquels je suis tout aussi reconnaissants. Les moments passés avec vous n’ont pas été assez nombreux, mais ont toujours été un plaisir. C’est votre bonne humeur quotidienne qui construit les bons souvenirs de ce doctorat.
Je pense aussi à feu Alexandre Parks partit bien trop jeune et à qui je dédie cette thèse. On pense à toi et on fera avancer la recherche dans ton souvenir.
Un immense merci mes camarades, et les amis, avec qui je vis au quotidien, au laboratoire comme à la maison. Je pense évidemment à Geneviève, Stéphane, Camille (bibi et guigui aussi) et Yann. Je n’aurai aucun mot pour votre exprimer ma gratitude pour tous ces bons moments passés ensembles. Vous êtes ma seconde famille ici. Une pensée spéciale pour Yann avec qui nous sommes en collocation depuis maintenant 4 ans. Camarade, collègue, ami, colocataire, la liste est longue. Merci pour tous ces moments, ces échanges, cette vie au quotidien. On a on a survécu aux hivers et aux rudes épreuves du doc, on a traversé bien des tempêtes, mais on en sort grandi. Merci d’être toi et merci pour ton inestimable apport à la poésie du monde. Fredo, Bastien, Remy, Vincent, Justine, Jonathan, Hichem et Calixte, je pense à vous qui m’aviez vu grandir depuis si longtemps et avec qui j’ai avancé tout le long du chemin. Votre amitié fait partie des choses les plus précieuses dans ma vie.
Aussi, je ne serais pas là aujourd’hui sans mon incroyable famille. Merci à mon grand-père et mes oncles et tantes, les scientifiques de la famille, qui m’ont élevé lorsque mes parents étaient loin, et tentaient de construire une nouvelle vie loin de la misère. Vous m’avez inculqué l’excellence et transmis votre abnégation, le gout des sciences et entretenu ma flamme, ma curiosité. Merci surtout à mes parents, qui ont tout quitté pour nous offrir une meilleure vie. Qui ont tant sacrifié pour éduquer leurs trois enfants ; eux qui n’avaient pas eu la chance d’avoir une éducation et merci à mon frère et ma sœur d’avoir toujours été là pour moi.
Enfin, les mots me manquent pour toi mon amour. Annie, on a été séparés alors même que notre histoire commençait à peine, lorsque je suis parti démarrer cette étape de ma vie, ce doctorat. Tu m’as soutenu depuis le début et nous avons résister 5 années durant à la distance, nous avons eu nos hauts et nos bas, mais sommes toujours là aujourd’hui. Nous avons grandi, ensemble, nous avons voyagé et vu tant de merveilles, nous avons vécu tant de choses… J’ai passé les plus belles années de ma vie avec toi et une nouvelle étape de notre vie commence bientôt. Mon futur, proche comme lointain, je le vois avec toi. Côtes-à-côtes, ou que le vent nous emporte. Je t’aime.
xx
AVANT-PROPOS
À mon arrivée au laboratoire du Dr Provost, l’équipe travaillait principalement sur le devenir des microARN contenus dans les microparticules (MP) de plaquettes sanguines. Les travaux des Drs Benoit Laffont et Aurélie Corduan, deux anciens étudiants du laboratoire, ont notamment montré l’internalisation et la fonction de microARN plaquettaires au sein de cellules endothéliales en culture [1] et l’effet des MP sur l’activité des macrophages [2]. En réalité les MPs sont des vésicules extracellulaires (VE) dérivées de la membrane plasmique lorsque les plaquettes sont activées. Après avoir étudié l’effet des inhibiteurs de pathogènes sur les microARN des VE de plaquettes dans les concentrés plaquettaires[3], l’équipe a soulevé la possibilité de leur transmission interindividuelle dans le cadre de transfusion sanguine ou de concentré de plaquettes. Cette idée de transfert interindividuel a suggéré la possibilité de transfert interespèce de microARN. L’équipe a alors développé, avec l’aide du Dr Arnaud Droit, un pipeline bio-informatique pour étudier les microARN de plaquettes humaines, et déterminer s’ils contenaient des microARN d’autres espèces. Cette approche a permis de révéler quelques microARN exogènes, mais en très faibles quantités. De plus, ce genre d’approche est grandement limité par l’existence d’isomères de microARN dont la séquence est très proche des microARN d’autres espèces. Cette approche a donc été abandonnée au profit de l’étude d’un autre fluide biologique, largement consommé dans le monde : le lait. L’équipe a pu quantifier par RT-qPCR quelques microARN dans le lait commercial, ce qui a donc ouvert la voie à l’étude des microARN du lait, dans notre laboratoire.
C’est dans ce cadre que mon projet de doctorat a commencé en 2014. À partir de ces quelques données préliminaires limitées, il m’a fallu transposer les technologies du laboratoire au lait, et développer des protocoles reproductibles, pour étudier les microARN qu’il contient. J’ai pu alors étudier leur bioaccessibilité, définir leur association avec des VE, élucider la diversité des VE du lait et trouver des marqueurs spécifiques pour chacune de ces VEs. L’étude de leur contenu biologique (ARN et protéines) m’a, finalement, amené à explorer leurs effets dans le cadre des maladies inflammatoires de l’intestin. Ce travail a mené à la production de 5 manuscrits de recherche originale, 1 revue extensive de la littérature et une lettre à l’éditeur détaillant une de nos découvertes d’importance pour le domaine des VEs. Les approches utilisées pour ces travaux ont été reprises, en partie, dans le papier de position de l’international society for extracellular
vesicles (ISEV), document majeur de référence pour l’étude des VE et pour lequel j’ai contribué
en tant que coauteur. Recherches originales
Le manuscrit présenté au chapitre III intitulé « Commercial dairy cow milk microRNAs resist
digestion under simulated gastrointestinal tract conditions » a été publié au Journal of Nutrition en octobre
2016. Il traite de la biodisponibilité des microARN du lait et de leur association avec des VEs. J’en suis l’auteur principal et il est coécrit avec Chan Ho Chris Lee, Benoit Laffont, Patricia Savard, Jonathan Laugier, Eric Boilard, Caroline Gilbert, Ismail Fliss et Patrick Provost. J’ai effectué les expériences, l’analyse, l’interprétation des résultats et la révision de ce manuscrit, en collaboration avec les coauteurs. Ce projet de recherche a été supporté par les Instituts de recherche en santé du Canada (IRSC) [IG1-134171 et MOP-137081 via l’institut de Génétique, et PJT-165806]. Des modifications mineures du format ont été effectuées dans le manuscrit et la numérotation des figures a été adaptée à la facture de cette thèse.
xxi
Le manuscrit présenté au chapitre IV intitulé « A subset of extracellular vesicles carries the bulk
of microRNAs in commercial dairy cow milk » a été publié dans le Journal of Extracellular Vesicles (JEV)
en novembre 2017. C’est le premier manuscrit rapportant l’existence de plusieurs populations de VE dans le lait et détaillant leur contenu. J’en suis l’auteur principal et il est coécrit avec Sophia Ly, Christine Shan, Jonathan Laugier, Eric Boilard, Caroline Gilbert et Patrick Provost. J’ai effectué les expériences, l’analyse, l’interprétation des résultats et la révision de ce manuscrit, en collaboration avec les coauteurs. Le Dr Richard Janvier a été remercié pour son aide pour la microscopie. Ce projet de recherche a été supporté par les Instituts de recherche en santé du Canada (IRSC) [IG1-134171 et MOP-137081 via l’institut de Génétique, et PJT-165806]. Des modifications mineures du format ont été effectuées dans le manuscrit et la numérotation des figures a été adaptée à la facture de cette thèse.
Le manuscrit présenté au chapitre V intitulé « Complexity of the microRNA transcriptome of
cow’s milk and milk-derived extracellular vesicles isolated by differential ultracentrifugation» a été soumis au Journal of Dairy Science en avril 2019. Il décrit la diversité des petits ARNs dans le lait commercial
et les différentes VE du lait. J’en suis l’auteur principal et il est coécrit avec Charles-Joly Beauparlant, Jonathan Laugier, Marine Lambert, Arnaud Droit et Patrick Provost. J’ai effectué les expériences, l’analyse, l’interprétation des résultats et la révision de ce manuscrit, en collaboration avec les coauteurs. Ce projet de recherche a été supporté par les Instituts de recherche en santé du Canada (IRSC) [IG1-134171 et MOP-137081 via l’institut de Génétique, et PJT-165806] et j’ai reçu une bourse du Fonds de recherche Québec Santé (FRQS) pour ce projet (No. 262093). Des modifications mineures du format ont été effectuées dans le manuscrit et la numérotation des figures a été adaptée à la facture de cette thèse.
Le manuscrit présenté au chapitre VI intitulé « Identification of protein markers for extracellular
vesicle (EV) subsets in cow’s milk » a été publié dans le Journal of Proteomics en février 2019. Il rapporte
l’existence de marqueurs protéiques spécifiques à certaines populations de VE et prédit leurs activités biologiques. J’en suis l’auteur principal et il est coécrit avec Clarisse Gotti, Sylvie Bourassa, Caroline Gilbert et Patrick Provost. J’ai effectué les expériences, l’analyse, l’interprétation des résultats et la révision de ce manuscrit, en collaboration avec les coauteurs. Ce projet de recherche a été supporté par les Instituts de recherche en santé du Canada (IRSC) [IG1-134171 et MOP-137081 via l’institut de Génétique, et PJT-165806]. Des modifications mineures du format ont été effectuées dans le manuscrit et la numérotation des figures a été adaptée à la facture de cette thèse.
Le manuscrit présenté au chapitre VII intitulé « Two subsets of extracellular vesicles from cow’s
milk modulate symptoms and inflammation in experimental colitis » a été soumis au journal Nature Scientific Reports en avril 2019. Il décrit comment deux populations de VE du lait peuvent avoir des effets
marqués, et parfois différents, sur l’inflammation et les symptômes de la colite. J’en suis l’auteur principal et il est coécrit avec Idrissa Diallo, Mabrouka Salem, Sara Michel, Caroline Gilbert, Jean Sevignyet Patrick Provost. J’ai effectué les expériences, l’analyse, l’interprétation des résultats et la révision de ce manuscrit, en collaboration avec les coauteurs. Ce projet de recherche a été supporté par les Instituts de recherche en santé du Canada (IRSC) [IG1-134171 et MOP-137081 via l’institut de Génétique, et PJT-165806] et j’ai reçu une bourse du Fonds de recherche Québec Santé (FRQS) pour ce projet (No. 262093). Les expérimentations animales, l’hébergement et les soins ont été approuvés par le Comité de protection des animaux de l’Université Laval en concordance avec les recommandations du Conseil canadien de Protection des Animaux (protocole 2016024-1). Des modifications mineures du format ont été effectuées dans le manuscrit et la numérotation des figures a été adaptée à la facture de cette thèse.