Valorisation de la flore nordique du Québec : de
l'extraction à la découverte de composés bioactifs
Thèse
Claudia Carpentier
Doctorat en chimie
Philosophiæ doctor (Ph. D.)
Valorisation de la flore nordique du Québec : de
l’extraction à la découverte de composés bioactifs
Thèse
Claudia Carpentier
Sous la direction de :
Normand Voyer
Résumé
L’orientation principale des travaux de recherches qui sont présentés dans cette thèse est de valoriser les espèces nordiques de la région du Nunavik en démontrant la diversité structurale et la spécificité biochimique des métabolites qu’elles biosynthétisent. Nous croyons que la flore nordique du Québec est une source inestimable de composés bioactifs en raison des stress environnementaux auxquels les espèces sont soumisses. En fait, c’est cette pression évolutive qui mène à la diversification structurale des métabolites spécialisés afin que les espèces soient mieux adaptées à leur environnement. La dynamique des écosystèmes du Grand Nord du Québec est toutefois appelée à changer drastiquement avec les changements climatiques. L’expansion de l’arbuste Betula glandulosa depuis les années 1990 est d’ailleurs un problème préoccupant puisque la densification menace la biodiversité en altérant tant l’environnement biotique qu’abiotique des écosystèmes nordiques. Il est ainsi pertinent de caractériser le métabolome nordique de la flore du Québec via l’investigation phytochimique de ces espèces qui sont en continuelle évolution.
La présente thèse rapporte les investigations phytochimiques du lichen Stereocaulon paschale qui sont axées vers l’identification de composés anti-inflammatoires. En plus de démontrer le potentiel d’inhibition de l’acide lobarique et de pseudodepsidones envers plusieurs cibles pro-inflammatoires, une relation structure-activité et un mécanisme d’action ont été proposés en réalisant des simulations d’arrimage moléculaire. L’étude de la composition phytochimique des feuilles de l’arbuste Betula glandulosa et l’évaluation du potentiel d’inhibition des métabolites vis-à-vis l’élastase sont également rapportées. L’étude de cette espèce a été un défi de taille en termes d’extraction et de purification en raison de son profil en métabolites très complexe. L’approche des réseaux moléculaires a ainsi été employée pour accélérer le rythme de la découverte des composés inédits et/ou bioactifs. L’étude détaillée du profil en métabolites et la localisation des composés d’intérêts qui ont été respectivement dérépliqués et ciblés par l’utilisation des réseaux moléculaires, sont ainsi présentées.
Abstract
The main orientation of this thesis is to enhance species in subarctic ecosystem in Northern Quebec by demonstrating the high chemical diversity and biochemical specificity of the secondary metabolites they biosynthesize. We believe that northern flora of Quebec is of great interest for the discovery of novel or bioactive secondary metabolites due to extreme growing conditions in the Nunavik region. In fact, it is this evolutionary pressure that leads to the adaptation of living organisms in their specific ecosystem and the chemical diversification of natural products in nature. The dynamics of the ecosystems of Northern Quebec are, however, about to change drastically with the warming trend that is observed since the 1990s in Nunavik. The expansion and extreme densification of the dwarf birch (Betula glandulosa) in lichen-dominated ecosystems threatens biodiversity by altering biotic and abiotic environment of northern ecosystems. Hence, it is important to characterize the metabolome of northern flora of Quebec by carrying out phytochemical investigations on species that are continuously evolving.
In this thesis, the phytochemical investigations of the lichen Stereocaulon paschale focused on the discovery of anti-inflammatory secondary metabolites. Herein we report the potential of lobaric acid and pseudodepsidones to reduce the expression of several pro-inflammatory targets. In addition, docking simulation experiments provided insight on the structure-activity relationship and the mechanism of action. The phytochemical study of the leaf of the shrub Betula glandulosa and the potential of the metabolites to exhibits significant elastase inhibition are also reported in the thesis. The very high complexity of the crude extracts of this species was very challenging in term of extraction and purification. The identification of novel bioactive NPs from such complex matrices remain a significant challenge in drug discovery programs. A molecular networking approach was thus used to explore the chemical diversity and accelerate the speed of the discovery of novel and bioactive NPs. By using this advanced mass spectrometry approach of dereplication, the thorough metabolite profiling of the crude extract was performed with the objective of targeting compounds of interest.
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des figures ... viii
Liste des tableaux ... xii
Liste des schémas ... xiii
Liste des abréviations ... xiv
Remerciements ... xviii
Introduction ... 1
Métabolites spécialisés : bioactivité et chimiodiversité ... 1
La recherche en PNs appliquée en pharmaceutique ... 4
Intérêts de la recherche en PNs pour le développement de médicaments ... 4
L’inflammation et les maladies chroniques ... 9
Potentiel anti-inflammatoire des métabolites spécialisés ... 12
La recherche en PNs appliquée en cosméceutique ... 16
Le vieillissement de la peau : mécanisme et prévention ... 17
Les produits naturels : source d’actifs cosméceutiques ... 21
Le Grand Nord du Québec : une pharmacie naturelle pour la découverte de composés bioactifs ... 26
Objectifs et structure de la thèse ... 29
Chapitre 1 : Identification de métabolites anti-inflammatoires issus du lichen Stereocaulon paschale ... 32
1.1 Investigations phytochimiques antérieures du lichen Stereocaulon paschale ... 32
1.1.1 Extraction, purification et identification de métabolites lichéniques bioactifs... 32
1.1.2 Similarités structurelles et considérations biosynthétiques ... 35
1.2 Études des propriétés anti-inflammatoires des métabolites issus de S. paschale ... 37
1.2.1 Évaluation de l’activité anti-inflammatoire via des macrophages stimulés au LPS ... 37
1.2.2 Évaluation de la liaison envers PPAR-γ ... 45
1.2.3 Modélisation moléculaire de l’interaction avec le récepteur PPAR-γ ... 47
1.3 Perspectives et travaux en cours ... 51
1.3.1 Développement d’agents anti-inflammatoires synthétiques ... 51
1.3.1 Méthodologie synthétique ... 52
Chapitre 2 : Investigations phytochimiques de Betula glandulosa : de l’extraction à l’isolement ... 63
2.1 Généralités sur l’espèce Betula glandulosa... 63
2.1.1 Classification et morphologie végétale ... 63
2.1.2 Distribution, habitat et climat ... 64
2.1.3 Métabolites spécialisés et voies biosynthétiques ... 65
2.1.4 Propriétés biologiques des métabolites spécialisés ... 73
2.2 Investigations phytochimiques des feuilles de Betula glandulosa ... 76
2.2.1 Échantillonnage et identification ... 76
2.2.2 Extraction de la biomasse par macération ... 76
2.2.3 Profilage des métabolites par HPLC-UV-ELSD et HPLC-MS ... 78
2.2.4 Isolement et identification des métabolites spécialisés ... 83
2.3 Investigations bioguidées : inhibition de l’élastase ... 108
2.4 Perspectives ... 111
Chapitre 3 : La spectrométrie de masse : un outil pour l’identification de PNs ... 113
3.1 Principes fondamentaux et état de l’art ... 113
3.1.1 Stratégies de déréplication et d’annotation ... 113
3.1.2 Profilage des métabolites par LC-HRMS et LC-MS/MS ... 116
3.1.3 Outils informatiques de déréplication: un aperçu ... 120
3.2 Les réseaux moléculaires ... 124
3.2.1 Principes fondamentaux... 124
3.2.2 Méthode pour la génération des réseaux moléculaires (workflow) ... 129
3.2.3 Processus d’annotation des réseaux moléculaires et outils bio-informatiques ... 138
3.3 L’identification de polyphénols par spectrométrie de masse tandem ... 150
3.3.1 Patron de fragmentation caractéristique des flavonoïdes ... 150
3.3.2 Patrons de fragmentation caractéristiques des tannins hydrolysables... 155
3.3.3 Patrons de fragmentation caractéristiques des phénylpropanoïdes ... 157
3.4 Perspectives ... 160
Chapitre 4 : Déréplication des métabolites et localisation d’actifs cosméceutiques des feuilles de B. glandulosa ... 161
4.1 Objectifs et analyse critique de la méthodologie ... 161
4.2 Interprétation des réseaux moléculaires de l’extrait méthanol des feuilles de Betula glandulosa ... 165
4.3 Enrichissement du réseau moléculaire ... 181
4.3.2 Fractionnement des fractions XAD sur gel de silice ... 200
4.4 Localisation des inhibiteurs de l’élastase... 227
4.5 Perspectives ... 233
Conclusion ... 235
Retour sur les objectifs de la thèse ... 235
Travaux en cours et perspectives ... 237
Développement d’une méthodologie pour l’extraction et la purification ciblées d’actifs cosméceutiques ... 237
Étude comparative des métabolites issus des feuilles de B. glandulosa, B. papyrifera et B. alleghaniensis par une approche de réseaux moléculaires ... 240
Valorisation des tiges de Betula glandulosa : extraction et purification d’agents chimiopréventifs ... 242
Approches bio-informatiques pour la valorisation de la flore boréale du Québec ... 244
Avancées et remarques finales ... 246
Partie expérimentale ... 247
Partie expérimentale générale ... 247
Partie expérimentale chapitre 1 ... 249
Partie expérimentale chapitre 2 ... 256
Partie expérimentale chapitre 4 ... 261
Bibliographie ... 265
Annexes ... 280
Annexes Chapitre 2 ... 280
Annexes Chapitre 3 ... 366
Annexes Chapitre 4 ... 368
Liste des figures
Figure 1 Facteurs biotiques et abiotiques des écosystèmes ... 1
Figure 2 Cristaux d’acide usnique à la surface de l’hyphe du champignon ... 2
Figure 3 Inclusion des PNs dans les criblages HTS (A) par rapport à l’impact des PNs dans le développement de médicaments entre 1981 et 2019 (B). ... 5
Figure 4 Domaines de recherches impliqués lors du développement d’un médicament issus de PNs ... 9
Figure 5 Activation canonique de la voie du facteur de transcription NF-ĸB ... 10
Figure 6 Polyphénols aux propriétés anti-inflammatoires ... 13
Figure 7 Processus des voies biochimiques menant à la dégradation de la peau ... 19
Figure 8 Polyphénols aux propriétés préventives vis-à-vis la dégradation de la peau ... 23
Figure 9 Écosystème et flore nordique de la région du Nunavik ... 26
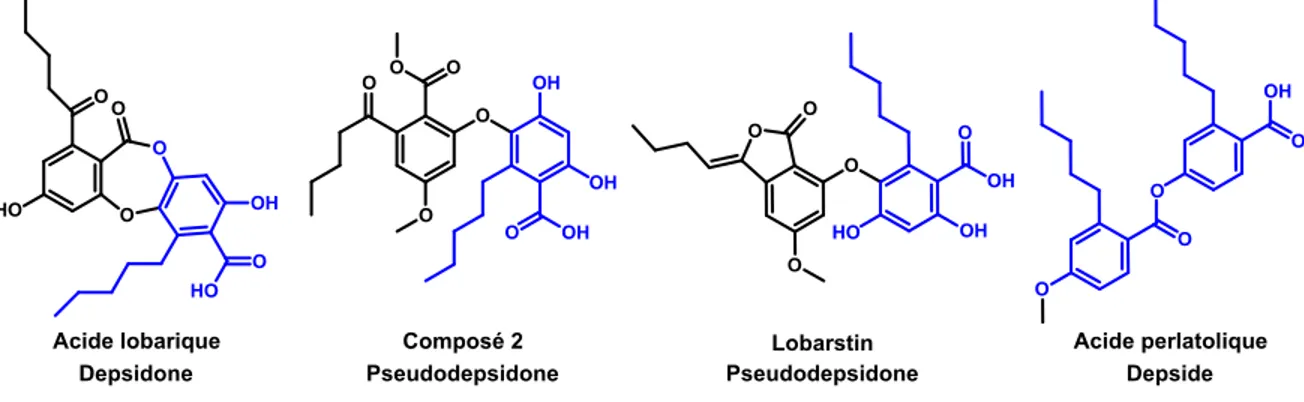
Figure 10 Métabolites identifiés dans le lichen Stereocaulon paschale ... 33
Figure 11 Inhibition de la sécrétion des cytokines en présence des composés 1, 2, 7 et 9 : A. sécrétion IL-6, B. sécrétion IL-8. C. Sécrétion IL-1β ... 43
Figure 12 Inhibition de la sécrétion d’IL-1β, du TNF-α et de l’activation de NF-ĸB par les composés 1 et 2 .... 44
Figure 13 Structures des agonistes naturels de PPAR-γ et du rosiglitazone ... 46
Figure 14 Capacité de liaison à PPAR-γ via un essai de liaison compétitif par TR-FRET : CON. control négatif ROSI. Rosiglitazone... 47
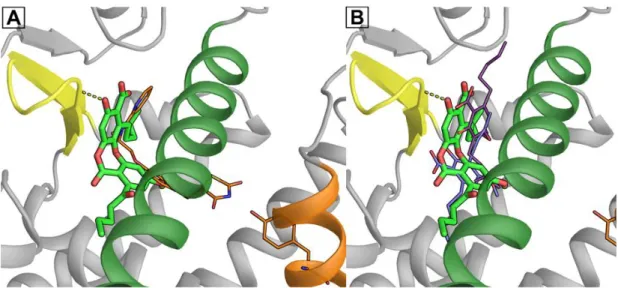
Figure 15 Résultats d’arrimage moléculaire avec le LBD de PPAR-γ pour l’acide lobarique en comparaison au rosiglitazone (A) et à l’amorfrutin 2 (B). ... 48
Figure 16 Résultats d’arrimage moléculaire avec le LBD de PPAR-γ pour les pseudodepsidones ... 49
Figure 17 Métabolites lichéniques ayant un motif d’acide olivetolique représenté en bleu ... 51
Figure 18 Capacité de liaison à PPAR-γ des composés monoaromatiques 14 et 15 en comparaison aux métabolites lichéniques de S.paschale: CON. control négatif ROSI. rosiglitazone ... 58
Figure 19 Évaluation de la toxicité cellulaire des composés monoaromatiques 14 et 15 ... 58
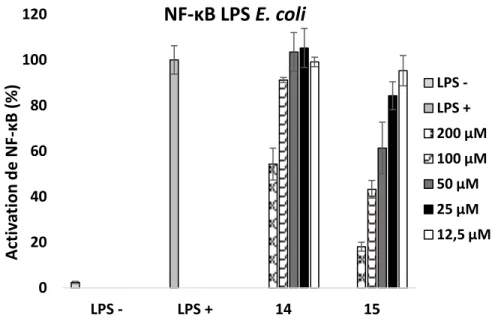
Figure 20 Inhibition de l’activation de NF-ĸB par les composés monoaromatiques 14 et 15 ... 59
Figure 21 Inhibition de la sécrétion du TNF-α par les composés monoaromatiques 14 et 15 ... 60
Figure 22 Activité inhibitrice des composés monoaromatiques 14 et 15 vis-à-vis la sécrétion de l’interleukine IL-1β induit par le LPS E. coli ... 61
Figure 23 Activité inhibitrice des composés monoaromatiques 14 et 15 vis-à-vis la sécrétion de l’interleukine IL-8 induit par le LPS E. coli ... 61
Figure 24 Activité inhibitrice des composés monoaromatiques 14 et 15 vis-à-vis la sécrétion de l’interleukine IL-6 induit par le LPS E. coli ... 62
Figure 25 Photographie du bouleau glanduleux A. tiges et chatons fructifères, B. buisson de bouleau glanduleux à Kuujjurapik, C. feuilles ... 64
Figure 26 Distribution des espèces du genre Betula ... 64
Figure 27 Habitats du bouleau glanduleux à Kuujjurapik ... 65
Figure 28 Métabolites spécialisés identifiés des feuilles des espèces de la famille Betulaceae ... 66
Figure 29 Polyphénols de type flavonoïdes et tannins hydrolysables ayant des propriétés anti-cancéreuses, antioxydantes, anti-inflammatoires, anti-tyrosinase, anti-collagénase et anti-hyaluronidase ... 74
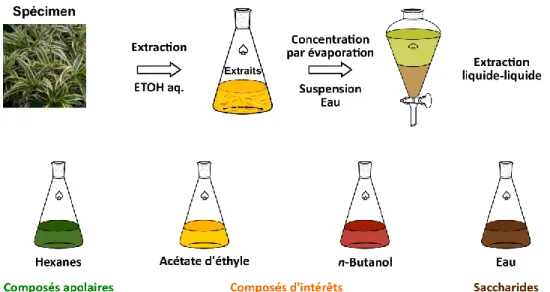
Figure 30 Préparation des extraits bruts par macération avec gradient de polarité ... 77
Figure 31 Chromatogrammes des extraits hexanes, dichlorométhane et méthanol des feuilles de B. glandulosa ... 79
Figure 32 Chromatogramme optimisé de l’extrait méthanol de B. glandulosa et spectre d’absorption UV des
composés majoritaires de type gallotannins, ellagitannins et flavonoïdes ... 79
Figure 33 Fractionnement de l’extrait MeOH de B. glandulosa par MPLC-PDA ... 81
Figure 34 Optimisation des gradients d’élution des fractions mixtes 1-6 par HPLC-PDA de l’extrait MeOH de B. glandulosa ... 81
Figure 35 Fractionnement d’un extrait par un processus d’extraction liquide-liquide ... 83
Figure 36 Processus de fractionnement/purification sur une résine polymérique ... 84
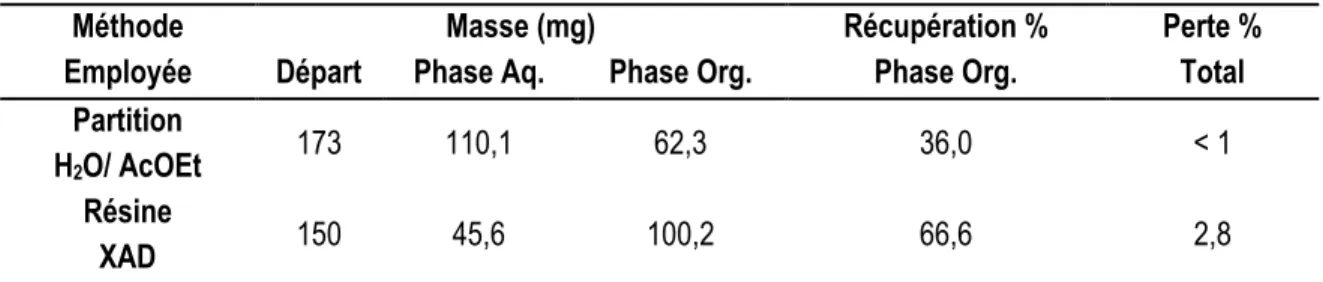
Figure 37 Profil HPLC-ELSD de l’extrait MeOH après partition H2O/AcOEt et traitement sur XAD-16 ... 85
Figure 38 Fractionnement de l’extrait MeOH sur résine XAD-16 avec gradient d’élution ... 87
Figure 39 Complémentarité des systèmes de solvants pour la purification des flavonoïdes ... 88
Figure 40 Méthodologie pour la purification des flavonoïdes par HPLC semi-préparative ... 89
Figure 41 Fragmentation des flavonoïdes glycosylés ionisés dans la source ... 89
Figure 42 Structures des flavonoïdes glycosylsés identifiées (* identification partielle) ... 92
Figure 43 Détermination de la configuration anomérique d’un hexose selon l’équation de Karplus ... 93
Figure 44 Procédure d’extraction des polyphénols et tannins hydrolysable sur résine polyamide utilisée ... 97
Figure 45 Analyses HPLC-ESI-TOF-HRMS du traitement sur résine polyamide de la faction XAD1 4-5 ... 99
Figure 46 Arbre de fractionnement et de purification détaillé de la fraction XAD1 4-5 ... 100
Figure 47 Composés issus de XAD1 4-5 et XAD2 8 identifiés par RMN et spectrométrie de masse ... 103
Figure 48 Processus de purification de XAD1 4-5 et XAD2 8 ... 104
Figure 49 Structures des composés putativement identifiés par RMN et dérépliqués par HRMS ... 105
Figure 50 Tentative de purification de la pédunculagine/casuariine issue de la fraction XAD1 2-3 ... 106
Figure 51 Assignation partielle des protons de la tellimagrandine I par RMN 1H ... 107
Figure 52 Inhibition de l’élastase par les fractions 5C, 3G, 4H, 1L, XAD1 2-3 et XAD1 4-5 ... 110
Figure 53 Articles et citations par années au sujet des procédures de déréplication ... 114
Figure 54 Schéma d’un appareil MS/MS de type LTQ-Orbitrap : Transfer Multipole (Quadripôle LTQ), HCD Cell (Cellule de collision). ... 118
Figure 55 Schématisation de l’approche MetFusion qui combine l’outil MetFrag (gauche) avec la recherche dans les librairies spectrales (droite) ... 121
Figure 56 Schématisation de l’outil FingerID : Prédiction de fragments via la conversion du spectre MS/MS en vecteur qui est basée sur 3 types d’informations spectrales (Noir : pics, Rouge : fragmentation primaire et Bleu : fragmentation secondaire) ... 122
Figure 57 Génération de réseaux moléculaires via l’alignement spectral ... 125
Figure 58 Détermination du Δ m/z pour une paire de spectre MS/MS ... 126
Figure 59 Visualisation d’un réseau moléculaire dans la plateforme GNPS ... 127
Figure 60 Annotation des réseaux moléculaires via l’outil de visualisation Cystoscape ... 128
Figure 61 Étapes du traitement des données MS/MS brutes dans le logiciel MZmine (étapes 6-8 optionnelles) ... 131
Figure 62 Construction du chromatogramme à partir des ions détectés... 132
Figure 63 Déconvolution du chromatogramme pour éliminer les pics mal résolus et larges ... 133
Figure 64 Exemple d’un chromatogramme des ions déconvolués ... 134
Figure 65 Alignement, filtration et remplissage des ions pour les différents échantillons analysés ... 135
Figure 66 Exemples de type d’annotations des réseaux moléculaires ... 139
Figure 68 Schématisation du processus de déréplication par réseaux moléculaires (MN) et annotation in silico
des spectres MS/MS (ISDB) ... 142
Figure 69 Visualisation dans Cytoscape des nœuds dérépliqués via la librairie ISDB ... 143
Figure 70 Système automatisé de reclassement des structures prédites par l’outil ISDB en fonction de l’alignement spectral et d’un score taxonomique ... 145
Figure 71 Visualisation dans Cytoscape de la classe de composés chimiques des réseaux moléculaires via l’outil d’annotation ClassyFire ... 146
Figure 72 Classification de taxonomie chimique pour le digalloylglucopyranose via l’outil ClassyFire ... 147
Figure 73 Schématisation du processus de déréplication des réseaux moléculaires par l’outil MolNetEnhancer ... 149
Figure 74 Nomenclature des fragments issus de la fragmentation MS/MS des flavonoïdes glycosylés ... 150
Figure 75 Clivages homolytique et hétérolytique de l’ion pseudomoléculaire de la quercétine-3-O-rhamnose ... 151
Figure 76 Variation des fragments pour les spectres MS/MS de la lutéoline et du kaempférol ... 153
Figure 77 Clivages fréquents et caractéristiques des flavones et flavonols par CID ... 153
Figure 78 Fragments caractéristiques aux gallotannins et ellagitannins ... 156
Figure 79 Schématisation du système de reclassement des annotations en fonction de filtres orthogonaux 163 Figure 80 Visualisation des classes de composés des réseaux moléculaires de l’extrait BGF MeOH ... 166
Figure 81 Structures des flavonoïdes dérépliqués du réseau MN1 de l’extrait brut BGF MeOH ... 168
Figure 82 Structures des ellagitannins dérépliqués du réseau MN2 de l’extrait brut BGF MeOH... 170
Figure 83 Structures des gallotannins dérépliqués du réseau MN3 de l’extrait brut BGF MeOH ... 171
Figure 84 Structures des tannins de l’extrait BGF MeOH dérépliqués des réseaux MN10, MN12 et d’une paire de nœuds ... 173
Figure 85 Structures des phénylpropanoïdes dérépliqués du réseau MN4 de l’extrait BGF MeOH ... 175
Figure 86 Structures des phénylpropanoïdes dérépliqués du réseau MN5 de l’extrait BGF MeOH ... 176
Figure 87 Structures des phénylpropanoïdes dérépliqués des réseaux MN6 et MN7 de l’extrait BGF MeOH 177 Figure 88 Structures des diarylheptanoïdes dérépliqués du réseau MN11 de l’extrait BGF MeOH ... 178
Figure 89 Structures potentielles de diarylheptanoïdes pour l’ion 309,11 m/z ... 179
Figure 90 Annotations putatives des classes de composés chimiques pour les paires de nœuds de l’extrait BGF MeOH ... 180
Figure 91 Processus de génération des fractions XAD2 4-12 issues de l’extrait brut MeOH des feuilles de Betula glandulosa ... 182
Figure 92 Visualisation des classes de composés des réseaux moléculaires des fractions XAD2 4-12 ... 183
Figure 93 Déréplication du réseau MN6 des fractions XAD2 4-12 : gallotannins et dérivés ... 186
Figure 94 Déréplication du réseau MN8 des fractions XAD2 4-12 et patron isotopique du nœud 299 ... 187
Figure 95 Patron isotopique des ions [M-2H]2- de la pédunculagine du réseau MN16 des fractions XAD2 4-12 ... 188
Figure 96 Prédiction ISDB de l’ion 461,07 m/z du réseau MN20 des fractions XAD2 4-12 ... 189
Figure 97 Déréplication du réseau MN3 des fractions XAD2 4-12 : phénylpropanoïdes et dérivés... 190
Figure 98 Visualisation du réseau MN2 des fractions XAD2 4-12 : phénylpropanoïdes et dérivés ... 192
Figure 99 Structures des phénylpropanoïdes dérépliqués du réseau MN2 des fractions XAD2 4-12 ... 194
Figure 100 Déréplication des réseaux MN11, MN14 et MN18 des fractions XAD2 4-12 : phénylpropanoïdes et dérivés ... 195
Figure 102 Déréplication du réseau MN7 des fractions XAD2 4-12 : flavonoïdes et dérivés ... 198
Figure 103 Déréplication du réseau MN19 des fractions XAD2 4-12 : diaryheptanoïdes ... 199
Figure 104 Visualisation de l’annotation ClassyFire pour les réseaux moléculaires issus des 23 sous-fractions de XAD1 4-5 et XAD2 8 ... 202
Figure 105 Visualisation du réseau MN1 issus des sous-fractions de XAD1 4-5 et XAD2 8 sous la forme de sous-réseaux (A-D) ... 203
Figure 106 Déréplication du sous-réseau A du réseau MN1 : phénylpropanoïdes et dérivés ... 205
Figure 107 Visualisation du sous-réseau B du réseau MN1 ... 207
Figure 108 Structures dérépliquées du sous-réseau B du réseau MN1 ... 209
Figure 109 Déréplication du sous-réseau C du réseau MN1 : phénylpropanoïdes et dérivés ... 210
Figure 110 Déréplication du sous-réseau D du réseau MN1 : phénylbutanoïdes, phénylpropanoïques et dérivés ... 211
Figure 111 Déréplication du réseau MN7 des sous-fractions XAD1 4-5 et XAD2 4-12 : phénylbutanoïdes et dérivés ... 213
Figure 112 Déréplication réseau MN4 des sous-fractions XAD1 4-5 et XAD2 4-12 : phénylpropanoïdes et dérivés ... 214
Figure 113 Visualisation du réseau MN10 des sous-fractions XAD1 4-5 et XAD2 4-12 ... 215
Figure 114 Structures potentielles de type terpénoïdes glycosylés du réseau MN10 ... 216
Figure 115 Structures potentielles de type diglucosides du réseau MN10 ... 217
Figure 116 Structures potentielles de types phénylpropanoïdes diglycosylés du réseau MN10 ... 218
Figure 117 Structures potentielles de l’ion ayant un rapport m/z de 443,16 du réseau MN10 ... 219
Figure 118 Visualisation du réseau MN2 des sous-fractions XAD1 4-5 et XAD2 4-14 : gallotannins et dérivés ... 220
Figure 119 Structures dérépliquées de types gallotannins et dérivés du réseau MN2 ... 222
Figure 120 Visualisation du réseau MN11 et déréplication des nœuds enrichis : gallotannis et ellagitannins 223 Figure 121 Visualisation et déréplication des réseaux MN6, MN12, MN18 et MN27 : gallotannis et ellagitannins ... 224
Figure 122 Déréplication du réseau MN36 des sous-fractions XAD1 4-5 et XAD2 4-12 : diarylheptanoïdes et dérivés ... 225
Figure 123 Déréplication des réseaux MN37 et MN44 des sous-fractions XAD1 4-5 et XAD2 4-12 : diarylheptanoïdes et dérivés ... 226
Figure 124 Évaluation de l’activité inhibitrice des fractions XAD2 4-14 et sous-fractions envers l’élastase ... 227
Figure 125 Annotation des activités inhibitrices des fractions XAD2 4-12 vis-à-vis l’élastase ... 228
Figure 126 Annotation des réseaux moléculaires des sous-fractions de XAD1 4-5 et XAD2 4-12 en fonction de l’activité inhibitrice envers l’élastase ... 230
Figure 127 Visualisation et annotation des réseaux moléculaires issus des extraits des feuilles de B. glandulosa, B. papyrifera et B. alleghaniensis ... 241
Figure 128 Identification putative de triterpènes issus de l’extrait BGT DCM ... 243
Liste des tableaux
Tableau 1 Propriétés anti-inflammatoires des métabolites polyphénoliques ... 14
Tableau 2 Formulation topiques à base de produits naturels de la compagnie Lucas Meyer Cosmetics ... 21
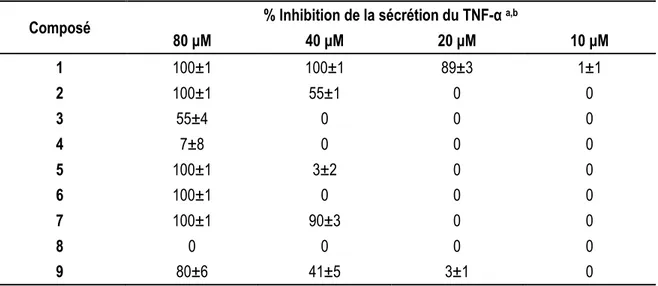
Tableau 3 Effet des métabolites issus de S. paschale envers l’activation de NF-кB induite par LPS ... 38
Tableau 4 Effet des métabolites issus de S. paschale envers la sécrétion du TNF-α induite par LPS ... 39
Tableau 5 Évaluation de la cytotoxicité des métabolites issus de S. paschale envers les macrophages ... 40
Tableau 6 Classification du bouleau glanduleux ... 63
Tableau 7 Types et nombre de métabolites rapportés chez la famille des Betulaceae ... 67
Tableau 8 Résultats de déréplication par HPLC-UV-ESI-TOF-HRMS des fractions 1-6 ... 82
Tableau 9 Rendement de la partition et du traitement sur résine polymérique de l’extrait MeOH ... 86
Tableau 10 Caractérisation des composés purs et semi-purifiés par HPLC-MS et RMN ... 90
Tableau 11 Analyse des fractions semi-purifiées par HPLC-ESI-TOF-HRMS ... 91
Tableau 12 Comparaison des déplacements chimiques (1H et 13C) des flavonoïdes glycosylés isolés ... 94
Tableau 13 Tannins hydrolysables identifiés dans les feuilles de Betula pubescens ... 96
Tableau 14 Caractérisation RMN et ESI-TOF-HRMS des composés purifiés issus du fractionnement test de XAD1 4-5 ... 101
Tableau 15 Caractérisation RMN et ESI-TOF-HRMS des tannins hydrolysables purifiés issus des fractions XAD1 4-5 et XAD2 8 ... 102
Tableau 16 Paramètres à optimiser pour la génération des réseaux moléculaires via la plateforme GNPS .. 137
Tableau 17 Génération des ions pseudomoléculaires et des fragments lors de l’ionisation et la collision dissociation induite de flavonoïdes glycosylés ... 152
Tableau 18 Fragments observés dans les spectres MS/MS des dérivés phénylpropanoïdes glycosylés ... 159
Tableau 19 Sélectivité des classes de composés en fonction du gradient d’élution ... 184
Tableau 20 Comparaison de l’enrichissement en métabolites en fonction des procédures de fractionnement et de la distribution des métabolites en fonction des classes de composés ... 201
Liste des schémas
Schéma 1 Proposition d’une relation biosynthétique entre les pseudodepsidones 2-7 et l’acide lobarique (1) via
les similarités structurales ... 35
Schéma 2 Voie de synthèse potentielle de l’acide perlatolique (16) à partir de l’acide olivetolique (14) ... 53
Schéma 3 Voie synthétique permettant d’accéder aux composés monoaromatiques 14 et 15 ... 54
Schéma 4 Voie synthétique potentielle de la pseudodepsidone 2 ... 56
Schéma 5 Voie biosynthétique de l’acide gallique, l’acide shikimique, l’acide protocatéchique, l’acide quinique et l’acide 3-déhydroquinique ... 68
Schéma 6 Biosynthèse des ellagitannins via un couplage oxydatif des gallotannins ... 69
Schéma 7 Biosynthèse des précurseurs des acides aminés aromatiques ... 70
Schéma 8 Voie biosynthétique des phénylpropanoïdes et des dérivés glycosylés ... 71
Schéma 9 Voie biosynthétique des flavanones et des stilbènes ... 72
Schéma 10 Voie biosynthétique des diarylheptanoïdes ... 73
Schéma 11 Fragmentation du cycle C d’un flavonoïdes par rétro-Diels-Alder ... 154
Schéma 12 Fragmentation de type rétro-Diels-Alder d’un dihydroxyflavonol menant aux ions 1,2A- et 1,2B-.... 155
Schéma 13 Fragmentation du galloylglucopyranose par spectrométrie MS/MS ... 157
Schéma 14 Fragmentation MS/MS caractéristiques des phénylpropanoïdes ... 158
Schéma 15 Procédure d’extraction et de purification globale des phénylpropanoïdes, flavonoïdes et tannins ... 238
Liste des abréviations
ABTS : L’acide(2,2’-azino-bis-(3-éthylbenzothiazoline-6-sulfonique ACN : Acétonitrile
AIBN : Azobisisobutyronitrile
AINS : Anti-inflammatoire non stéroïdien AMX : Système de spin d’un cycle aromatique aq. : Aqueux
AND : Acide désoxyribonucléique ARN : Acide ribonucléique
BNICE: Biochemical Network Integrated Computaional Explorer
C4 : Silice fonctionnalisée butyl C8 : Silice fonctionnalisée octyl C18 : Silice fonctionnalisée octadécyl
CAS : Numéro CAS d’une substance chimique CCM : Chromatographie sur couche mince CFM-ID : Competitive Fragmentation Modeling-ID CID : Collision induite par dissociation
CMI : Concentration minimale inhibitrice CoA : Coenzyme A
Col. : Colonne Conc. : Concentration COX : Cyclooxygénase
CRC : Numéro CRC d’une substance chimique c-Rel : Sous-unité facteur de transcription CPR : P450 réductase
CV : Nombre de volume de colonne δ : Déplacement chimique d : Doublets (spectre RMN) Da : Dalton DAHP : Acide-3-désoxy-D-arabino-heptulosonique-7-phosphate DCM : Dichlorométhane
DDA: Data depentdent acquisition DIPEA : Diisopropyléthylamine DIOL : Silice fonctionnalisée DIOL DMF : Diméthylformamide DMSO : Diméthylsulfoxyde
DNP : Dictionary of Natural Products DOS : Synthèse orientée vers la diversité DPPH : Radical 2,2-diphényl-1-picryl-hydrazyl ECD : Dichroïsme circulaire électronique
EGCG : Gallate d’épigallocatéchine EIC : Extracted-ion chromatogram
ELISA : Enzyme-linked immunosorbent assays ELSD : Détecteur évaporatif à diffusion de la lumière
ECM : Matrice extracellulaire du derme ESI : Mode ionisation par électronébulisation eq. : Équivalent de réactif ou composé F3H : Flavanone-3-hydroxylase FA : Acide formique
FBDD : Fragment-based drug discovery FLS : Flavonol synthase
FMO : P450 flavonoïde monooxygénase FR : Fraction
FRAP : Ferric reducing-antioxidant power FRET : Transfert d’énergie par résonance de fluorescence
Galloy- : Unité galloyle
GNPS : Global Natural Products Social Molecular Networking GPP : Géranyl-pyrophosphate GPx : Glutathionne peroxydase 1H-1H : Couplage proton-proton 1H-13C : Couplage proton-carbone H2O2 : Peroxyde d’hydrogène
HaCAT : Lignée cellulaire de kératinocytes Hex : Hexanes
HHDP : Hexahydroxydiphénique HLB : Résine polymérique hydrophilique HPLC : Chromatographie liquide à haute performance
HRMS : Spectrométrie de masse haute résolution HTS : High-throughput screening
IC50 :Concentration inhibitrice médiane
IL-6, -8 et -1β : Interleukines-6, -8, 1β % inhb. : % inhibition
IĸB : Protéine inhibitrice du NF-ĸB IKK-α et -β : Enzyme IĸB kinase InChi : Identifiant chimique international Inj. : Injection
iNOS : Oxyde nitrique synthase IPP : Isopentényl-pyrophosphate
IR : Spectroscopie infrarouge ISDB : In silico Database
ISDB-MN : In silico Database-Molecular Networking
IT-MS : Spectrométrie de masse à trappe d’ions J : Constante de couplage en hertz
LBD : Domaine de liaison du ligand
LC-MS/MS : Chromatographie liquide couplée à la spectrométrie de masse en tandem
5-LO : 5-Lipoxygenase
5- et 12- LOX : Lipoxygénases-5 et -12 LPS : Lipopolysaccharide bactérien LTQ : Trappe ionique quadripolaire m/z : Rapport masse sur charge
MAPK: Mitogen-activated protein kinases MF : Formule moléculaire
MINES : Metabolic In silico Network Expansions MMPs : Métalloprotéinases
MN : Réseau moléculaire
MOM : Méthyle chlorométhyle éther mPGES-1 : Prostaglandine E synthase-1 MPLC : Chromatographie liquide à moyenne pression
MS : Spectrométrie de masse
MS1 : Spectre MS de niveau 1 (Full scan) MS2 : Spectre MS/MS de niveau 2
MS/MS : Spectrométrie de masse en tandem MTT : Bromure de 3-(4,5-diméthylthiazol-2-yl)-2,5-diphényl tétrazolium
Na : Sodium
NAD : Nicotinamide adénine dinucléotide NADPH : Nicotinamide adénine dinucléotide phosphate
NAP : Network Annotation Propagation NCI : US National Cancer Institute
NF-κB : Facteur de transcription nucléaire (nuclear factor-kappa B)
NHTP : Nonahydroxyterphenoyl NO· : Oxyde nitrique radicalaire
NPNPD : Programme de découverte de produits naturels de l’institut NCI
[O] : Réaction d’oxydation org. : Organique
OTBDMS : Éther tert-butyldiméthylsilyle
ORD : Dispersion rotatoire optique
p50, p52, p65 : Sous-unité facteur de transcription PAL : Phénylalanine ammonia-lyase
PDA : Détecteur à barrette de diodes PEP : Phosphoénolpyruvate
PGE2 : Prostaglandine E synthase-2 PKS : Polycétide-synthase
PLP : Phosphate de pyridoxal PNs : Produits naturels
PPAR-γ : Proliférateurs de peroxysomes de type gamma
PTP1B : Tyrosine-protein phosphatase non-receptor type 1
Q-MS : Spectromètre de masse à quadridole Q-TOF : Spectromètre de masse à temps de vol quadripolaire
ReIA : Sous-unité facteur de transcription RMN : Spectroscopie de résonance magnétique nucléaire
RMN 1H : Étude RMN 1D du proton
RMN 13C : Étude RMN 1D du carbone
RMN 13C-DEPTQ : Étude RMN de type 1D du
carbone par Distorsionless Enhancement by Polarization Transfert
RMN COSY : Étude RMN de type COrrelation SpectroscopY
RNM HMBC : Étude RMN de type Heteronuclear
Multiple Bond Coherence
RMN HSQC : Étude RMN de type Heteronuclear
Single-Quantum Correlation
RMN NOESY: Étude RMN de type Nuclear
Overhauser Effect SpectroscopY
ROS : Dérivés réactifs de l’oxygène SAR : Étude de relation structure-activité S-Enz : Liaison souffre-enzyme
SCoA : Liaison souffre coenzyme A
SMILES : Simplified Molecular Input Line Entry Specification
SOD : Superoxyde dismutase SPE : Extraction en phase solide t.a. : Température ambiante TAL : Tyrosine ammonia-lyase TBDMS : tert-Butyldiméthylsilyle
THF : Tétrahydrofurane
TLR : Récepteurs transmembranaires de type Toll TMEDA : Tétraméthyléthylènediamine
TNF-α : Facteur de nécrose tumorale alpha TOF : Spectrométrie de masse à temps de vol TIC : Chromatogramme total des ions
TR : Temps de rétention
UHPLC : Chromatographie liquide ultra performance
UV : Détecteur UV-visible
UV-A : Rayonnements ultraviolets (400-315 nm) UV-B : Rayonnements ultraviolets (315-280 nm) UV-VIS : Rayonnements ultraviolets et visibles VCAM-1 : Vasopressin-activated Ca2+-mobilizing
‘’ Qu’est-ce donc qu’une mauvaise herbe,
sinon une plante dont on n’a pas encore
découvert les vertus.’’ Ralph Waldo Emerson
Remerciements
Lors de mon parcours académique au département de chimie de l’Université Laval, j’ai eu le plaisir de rencontrer et de côtoyer de nombreuses personnes qui ont contribué à la réalisation de cette thèse. Je tiens d’abord à remercier mon directeur de recherche Normand Voyer de m’avoir fait confiance pendant toutes ces années. Je suis consciente qu’il n’a pas dû être facile de me superviser avec tous les projets que je voulais entreprendre. Merci de m’avoir encouragé à voyager et à profiter pleinement des études de cycles supérieurs. Je n’oublierais jamais tous les bons moments que j’ai passés en compagnie de mes collègues du groupe Voyer. Plusieurs d’entre vous sont devenus des amis proches : Christopher, Sébastien, Pierre-Alexandre, Jean-Daniel, Nicolas, Corinne, Meggan et Jean-Christophe. Merci d’avoir été là pour discuter de science, mais aussi pour l’ambiance unique qui régnait au laboratoire, au bureau et au Cactus sur l’Avenue Myrand. Une mention spéciale à François Otis qui a été d’une précieuse aide que ce soit pour la relecture de nombreux documents, la réparation des appareils ou la supervision au laboratoire.
Il ne fait aucun doute que mes stages de formation dans le laboratoire du professeur Jean-Luc Wolfender ont été parmi mes plus belles expériences scientifiques. Durant ces séjours, j’ai eu la chance d’échanger avec des étudiants et chercheurs spécialisés en chimie des produits naturels, ce qui a été essentiel pour la poursuite de mes travaux de doctorat. C’est pourquoi je tiens à remercier Jean-Luc Wolfender et Emerson Ferreira Queiroz de m’avoir accueillie dans leurs laboratoires en plus de m’avoir supervisée lors de ces stages. Je voudrais également remercier Adriano Rutz et Pierre-Marie Allard de m’avoir appris les rudiments de l’approche des réseaux moléculaires. Je voudrais également remercier Daniel Grenier, Jabrane Azelmat, Xavier Bardeau et Stéphane Boudreau qui ont été collaborateurs essentiels à la réalisation des travaux de recherche qui sont présentés dans cette thèse.
Je souhaiterais également remercier François Béland et Hugo St-Laurent qui m’ont permis de réaliser mon doctorat en collaboration avec la compagnie SiliCycle. Je remercie aussi les chercheurs du laboratoire R&D qui m’ont accueilli au sein de l’équipe et qui m’ont prodigué de nombreux conseils sur la chromatographie. Je pense particulièrement à Vincent Bédard, Xavier Pigeon et Denis Boudriau. Une mention spéciale à Michel Morin et David Dubé que j’ai eu le plaisir de côtoyer au laboratoire, vous savez vraiment mettre de l’ambiance !
Finalement, j’aimerais remercier mes parents et ma famille qui m’ont supportée tout au long de mon parcours académique et qui m’ont encouragée à entreprendre des études supérieures. Je voudrais remercier ma mère de m’avoir fait découvrir la science et la recherche en laboratoire dès un très jeune âge. Je suis persuadée que les nombreuses visites dans ton laboratoire ont influencé mon choix de carrière. Je tiens également à remercier mon père de m’avoir fait aimer les études et de m’avoir transmis le plaisir d’apprendre. J’aimerais aussi
témoigner ma reconnaissance à Étienne pour m’avoir accompagné tout au long de nos études en chimie. Tu as été le meilleur des partenaires tant au laboratoire qu’à la maison. Je tiens à te remercier comme je suis persuadée que tu as fait de moi, la scientifique que je suis devenue. Chaque soir, nous pouvions passer des heures à échanger sur nos projets respectifs, ce qui a souvent mené à de judicieux conseils. J’ai bien hâte de voir ce que la vie nous réserve et quels sont les nouveaux défis que nous allons relever ensemble.
Introduction
Métabolites spécialisés : bioactivité et chimiodiversité
Les organismes vivants tels que les végétaux, les champignons, les algues et les bactéries produisent des métabolites spécialisés en réponse aux différents stimulus externes qui sont présents dans leur environnement.1,2 Ces composés naturels sont communément appelés métabolites spécialisés puisqu’ils ne
sont pas essentiels à la croissance et au développement de l’organisme, mais ils sont nécessaires à sa survie.3,4
Ces composés sont en fait des métabolites de défense qui sont biosynthétisés en réponse à des facteurs biotiques, abiotiques et relatifs aux humains auxquels l’organisme est soumis dans son environnement (Figure 1).1 Par exemple, plusieurs plantes ou organismes vivants secrètent dans le cortex des tiges et à la surface des
feuilles des résines, des gommes ou d’autres substances végétales comme du mucilage.3 La composition
chimique de ces substances regorge de métabolites de défense tels que des terpènes qui agissent en tant qu’insecticide.3
Figure 1 Facteurs biotiques et abiotiques des écosystèmes
Les composés phénoliques tels que les flavonoïdes font aussi partie des mécanismes de défense des organismes vivants.1 En fait, ces composés sont majoritairement distribués dans les cellules de l’épiderme des
feuilles et des tiges puisqu’ils protègent les cellules des rayons UV-B.1 Ils auraient également un rôle antioxydant
en captant les dérivés réactifs de l’oxygène (ROS).1 Selon l’intensité lumineuse de l’environnement, les végétaux
et organismes peuvent accumuler de grandes quantités de composés photoprotecteurs. Ce mécanisme de protection est d’ailleurs l’un des principaux chez les organismes lichéniques.5 En fait, une diversité de
métabolites photoprotecteurs (depsidones, depsides, anthraquinones, dibenzofuranes, etc.) sont sécrétés sous forme cristalline à la surface de l’hyphe du thalle pour protéger le partenaire photosynthétique (algue) des
radiations UV.6,5 Chaque type de composé a un spectre d’absorption des rayons UV distincts, ce qui permet
d’assurer une protection sur une large gamme de radiation UV.7,5 Les métabolites lichéniques qui sont
biosynthétisés à la surface du thalle peuvent également avoir des propriétés cytotoxiques, antimicrobiennes et antifongiques pour se protéger des pathogènes et des insectes.7 L’acide usnique est un exemple de métabolite
de défense/protection qui peut être sécrété en grande quantité à la surface du champignon lichénique (Figure 2).7 Les tannins hydrolysables sont aussi d’excellents exemples de produits naturels (PNs) qui jouent un rôle
dans les mécanismes de défense et de protection des plantes vis-à-vis les herbivores.1 Ces composés de masse
moléculaire élevée (600-3000 g/mol) ont la capacité d’induire la génération de ROS en plus de faire précipiter les protéines, d’où leurs propriétés cytotoxiques.8,9 De plus, ces métabolites repoussent également les
mammifères herbivores en raison de leur goût très astringent.10 La teneur en métabolites de défense est très
variable dans le temps et selon les stress environnementaux auxquels les espèces sont soumises.10 Au Québec
par exemple, la composition en métabolites est très variable entre les différentes saisons. Il est généralement recommandé de récolter les spécimens durant l’été, après la floraison puisque la diversité chimique est plus élevée. Généralement, le pourcentage massique par rapport à la masse sèche d’une plante en métabolites spécialisés est entre 1-10 %.3
Carpentier, C. Mémoire, Université Laval (2016)7
Figure 2 Cristaux d’acide usnique à la surface de l’hyphe du champignon
A : surface de l’hyphe du champignon lichénisé, B : structure moléculaire de l’acide usnique et C : cristaux d’acide usnique. Les propriétés défensives des PNs sont souvent étroitement liées à leurs propriétés biologiques.4 Par exemple,
les propriétés photoprotectives des composés phénoliques sont très intéressantes du point de vue de la cosméceutique pour le développement de formulations topiques qui préviennent le vieillissement de la peau.6,11
Les propriétés antibactériennes ou antifongiques des métabolites spécialisés sont également à l’origine du développement de traitements contre les infections bactériennes ou fongiques.12,13 La recherche en chimie des
PNs a d’ailleurs comme objectif d’étudier les organismes vivants soit par rapport aux rôles écologiques des métabolites de défense ou aux propriétés bioactives des métabolites spécialisés.4 Les organismes vivants sont
ainsi une source inestimable de composés bioactifs dont la diversité chimique a évoluée à travers le temps pour mieux s’adapter à leur environnement.4,2,14 Cette pression évolutive a ainsi favorisé la diversité structurale des
métabolites spécialisés qui sont biosynthétisés au sein d’un même genre et/ou espèce.4 Selon Pichersky et
Lewinsohn, le nombre total de métabolites spécialisés qui se trouve dans le règne des plantes dépasse de loin la capacité du génome d’une plante à encoder les enzymes nécessaires à la biosynthèse de ces métabolites.4,2
En évoluant une espèce ou une lignée végétale développe la capacité de produire des métabolites spécialisés inédits, tout comme elle perd la capacité d’encoder des enzymes spécifiques à la biosynthèse de certains composés.4,2 Il est ainsi commun que le profil en métabolites spécialisés d’une même espèce qui est dans deux
environnements distincts, comporte plusieurs différences. Ce concept de diversification des métabolites biosynthétisés fait référence à la chimiodiversité, soit la diversité chimique des organismes vivants.4,15 La
diversité structurale des métabolites est généralement visualisée dans un espace chimique en fonction de descripteurs moléculaires tels que la masse moléculaire, la lipophilicité, les groupements fonctionnels et la topologie moléculaire.4,15 Selon des études, la chimiodiversité des plantes surpasse celle de toutes les
molécules organiques qui ont été obtenus par une approche de chimie combinatoire à haut débit.15,16 C’est
d’ailleurs pour cette raison qu’il y a un intérêt renouvelé pour la recherche en PNs pour l’identification de molécules ‘’têtes de séries’’ dans l’industrie pharmaceutique.4,15
La recherche en PNs appliquée en pharmaceutique
Intérêts de la recherche en PNs pour le développement de médicaments
Les plantes et organismes vivants sont utilisés depuis des lunes (millénaires) en médecine traditionnelle pour traiter une vaste gamme de problèmes de santé.17 Les humains ont ainsi continué à s’inspirer de la nature pour
le développement de médicaments.17 Le succès des PNs dans le domaine de la pharmaceutique est associé à
leur diversité structurale et à leur spécificité biochimique.18 Les PNs sont d’ailleurs considérés comme des
structures privilégiées pour le développement de médicaments aux mécanismes d’action inédits puisqu’ils ont des caractéristiques structurales qui favorisent l’interaction avec des cibles biologiques spécifiques telles que des protéines et des récepteurs membranaires.4,18 En fait, c’est la pression évolutive qui favorise la biosynthèse
de métabolites dont la structure est adaptée pour faire face à divers stimulus externes tels que des pathogènes.18
Lors de ce processus, il est fréquent que les organismes vivants produisent plusieurs analogues d’un métabolite bioactif.18,19 L’organisme est ainsi en mesure d’évaluer la relation structure-activité des métabolites
biosynthétisés, ce qui lui permet de produire une deuxième génération de métabolites qui sont mieux adaptés.18
Dans les années 1970 à 1980 la recherche pharmaceutique était principalement axée sur l’investigation des PNs comme source de composés ‘’têtes de séries’’, ce qui a eu pour effet qu’entre 1981 et 2002, près de 49% des 877 nouveaux médicaments qui ont été approuvés étaient des PNs, des dérivés semi-synthétiques ou les pharmacophores étaient des PNs.18 Malgré l’immense succès des PNs dans le domaine de la pharmaceutique,
les programmes de recherche ont délaissé les PNs dans les années 1990 jusqu’au début dans années 2000.4,18
La principale raison de ce délaissement est associée à l’arrivée des programmes de criblages à haut débit (high-throughput screening, HTS) qui ont grandement accéléré le rythme auquel une chimiothèque pouvait être testée.4,18,19 Le développement de la chimie combinatoire qui permet de générer rapidement des chimiothèques
de composés ayant une grande diversité structurale est aussi une autre raison.18,19 Les avancées dans le
domaine de la biologie moléculaire et cellulaire ont aussi permis de réduire le temps nécessaire pour l’évaluation des activités biologiques d’une chimiothèque, en plus d’augmenter le nombre de cibles biologiques, ce qui a grandement favorisé les criblages à haut débit.18,19 Il y a eu également un déclin de la recherche dans le domaine
des maladies infectieuses, où les PNs étaient une source importante pour le développement de médicaments.18
Dans ce contexte, les programmes de recherche traditionnels en PNs ne sont pas adaptés et compétitifs face aux chimiothèques de composés synthétiques qui permettent le criblage à haut débit.19,20,21 En fait, les
procédures de fractionnement bioguidé qui sont traditionnellement utilisées en isolation de PNs et en pharmacognosie ont mené à la découverte de plusieurs médicaments tel que l’artémisinine (1972, Artemisia annua, anti-malaria),22 le paclitaxel (1971, Taxus brevifolia, antitumoral),23 la vincristine (1965, Catharanthus
rapamycine (1975, Streptomyces hygroscopicus, immunosuppresseur).26,27,4,15,18 Les nombreuses étapes de
purification, l’élucidation de la structure et l’évaluation des propriétés biologiques du composé purifié font toutefois en sorte que le processus est lent, couteux et mène souvent à l’isolation de composés bioactifs connus.4,15,18 C’est pourquoi la chimie des PNs traditionnelle est considérée non compatible avec les
programmes de criblages à haut débit, ce qui explique qu’un très petit pourcentage de produits naturels ait été inclus dans ce type de programmes par les pharmaceutiques (Figure 3).19
Wilson, B.A.P et al. Nat. Prod. Rep (2020) 19 et Newman, D.J. et al. J. Nat. Prod. (2020) 28
Figure 3 Inclusion des PNs dans les criblages HTS (A) par rapport à l’impact des PNs dans le développement de médicaments entre 1981 et 2019 (B).
A : publications publiées sur les criblages HTS au total incluant les chimiothèques synthétiques ainsi que les PNs (gris) et les publications publiées sur le criblage HTS des PNs (bleu) et B : distribution des nouveaux médicaments approuvés entre 1981 et 2019 selon leur classe : macromolécule biologique (bleu), vaccin (mauve), molécule synthétique (jaune-orange), molécule synthétique dont le pharmacophore est un produit naturel (orange), produit naturel (vert foncé), dérivé d’un produit naturel (vert), molécule synthétique mimant un produit naturel (beige-jaune et pêche) et botanique/formulation (vert pâle). Il est toutefois intéressant de noter que malgré ce délaissement, les PNs demeurent encore aujourd’hui une source d’inspiration importante pour le développement de médicaments. Entre 1981 et 2014, plus de 60% des petites molécules qui ont été approuvées comme traitement contre le cancer ont été développées à partir de la structure d’un produit naturel.14 Durant cette période, moins de 1 % des PNs ont été inclus dans les criblages à
haut débit, ce qui démontre le potentiel bioactif des produits naturels.14 À titre de comparaison, la chimie
combinatoire qui a été largement utilisée dans les programmes de recherches, a seulement mené à l’approbation de 3 nouveaux médicaments entre 1981 et 2019, soit le Sorafenib (Nexavar, Bayer, 2005) un composé antitumoral pour le traitement du cancer du rein, l’Ataluren (Translarna, PTC Therapeutics, 2014) un traitement pour la dystrophie musculaire et le Vemurafenib (Zelboraf, ROCHE, 2011) un traitement des mélanomes de stade avancé.28 Dans cette même période (1981-2019), 42 % des 1881 nouveaux médicaments
(Figure 3).28 Un des principaux problèmes qui explique pourquoi les chimiothèques générées par chimie
combinatoire ont connu peu de succès dans les programmes de criblages à haut débit est qu’elles ont été désignées pour avoir une diversité structurale maximale plutôt que de maximiser les propriétés structurales importantes qui favorise l’interaction avec des cibles biologiques.18 Des études computationnelles ont d’ailleurs
permis de mettre en lumière que les PNs ont plusieurs propriétés (masse moléculaire, nombre de centre chiraux, flexibilité moléculaire, distribution des atomes lourds, règles de Lipinski) qui les rendent prompt aux développements de médicaments, en plus d’occuper un espace chimique qui est très similaire à celui des médicaments.4,18 De plus, la diversité chimique des PNs qui est décrite dans un espace multidimensionnel
(masse moléculaire, lipophilie, topologie, etc.), est plus grande que celle de tous les composés qui ont été synthétisés par une approche de chimie combinatoire.4,15
À ce jour, plus de 250 000 PNs ont été rapportés dans la base de données Dictionary of natural products (DNP).14,4 Cette dernière répertorie principalement les métabolites qui ont été isolés chez le règne des plantes,
ce qui implique que l’espace chimique des PNs n’est seulement que partiellement investigué.4 À lui seul,
l’espace chimique des organismes microbiens est environ 4 fois plus grand que celui des plantes.4 De plus, il
est estimé qu’environ seulement 10 % des métabolites spécialisés qui sont issus des organismes végétaux, marins et microbiens ont été investigués dans les programmes pharmaceutiques pour la découverte de médicaments.14 Devant cet immense potentiel de diversité chimique et de spécificité biochimique, un regain
d’intérêt envers la recherche en chimie des PNs pour le développement de médicament a été observé durant la dernière décennie.18,17 Le défi principal est toutefois de tirer profit de cette diversité chimique tout en étant
compatible avec les programmes modernes de découverte de médicaments qui sont principalement basés sur le criblage à haut débit.14 Afin d’accélérer le rythme de la découverte de PNs bioactifs, des avancées
technologiques importantes ont été réalisées dans le domaine de la spectrométrie de masse pour le profilage des métabolites dans des matrices complexes.15,17,20,21 Un des défis principaux de la recherche en produits
naturels est que les extraits de plantes ou d’autres organismes vivants sont des matrices très complexes, qui peuvent contenir plusieurs centaines voire des milliers de métabolites dont la teneur est très variable.4 Des
systèmes performant de spectrométrie de masse en tandem couplé à la chromatographie qui permettent de détecter les métabolites sur une large gamme dynamique et de générer des spectres riches en informations, ont ainsi été développés pour identifier les métabolites.4,19 Des avancés computationnelles qui permettent
d’interpréter automatiquement les spectres MS/MS, ont également vue le jour.19 Ces avancées technologiques
permettent d’identifier les métabolites qui ont précédemment été investigués sans avoir à les isoler (procédures de déréplication), ce qui permet grandement d’accéléré le rythme de la découverte de composés inédits.20,21
Les procédures de déréplication par spectrométrie MS/MS sont maintenant un outil courant dans les programmes modernes de recherche en PNs, mais les équipes de recherche doivent également adapter leurs
méthodologies en termes d’extraction et de purification afin de fournir des chimiothèques de PNs qui sont compatibles avec le criblage à haut débit.14,19 Le US National Cancer Institute (NCI) est d’ailleurs un pionnier
dans ce domaine.14,19 Durant les dernières années, le laboratoire de Barry R. O’Keefe a concentré ses efforts
à développer une chimiothèque d’extraits de PNs fractionnés qui est accessible à toute la communauté scientifique sous la forme de plaques 384 puits pour le criblage d’activité biologique.14,19 À ce jour, le programme
de découverte de produits naturels (NPNPD) de l’institut NCI détient une des plus grandes et diverses collections d’extrait de produits naturels (>125 000) qui provient de partout dans le monde.14,19 Ils ont ainsi décidé de
stimuler la recherche en PNs pour le développement de médicament en développant une chimiothèque qui est compatibles avec le criblage à haut débit.14,19 Pour ce faire, ils ont dû développer une plateforme automatisée
de fractionnement SPE qui implémente des phases stationnaires (diol, C4, C8, C18 et HLB) et des phases mobiles aux sélectivités complémentaires afin de produire des fractions semi-purifiées qui contiennent une distribution uniforme des métabolites.14,19 Les fractions ainsi générées doivent également être concentrées en
métabolites minoritaires, en composés bioactifs et contenir du matériel en quantité suffisantes pour les étapes subséquentes de déréplication (HPLC-MS/MS), d’isolation des composés actifs et d’élucidation de la structure.14
Les méthodologies de fractionnement employées doivent aussi conserver la diversité chimique de l’extrait tout en étant peu couteuses et rapides.14,19 Il a été démontré que les chimiothèques d’extraits pré-fractionnés ont
généralement une activité biologique accrue en comparaison aux chimiothèques d’extraits bruts (extractothèque).19 En fait, les extraits sont des matrices très complexes qui peuvent contenir plusieurs
composés qui interfèrent avec la méthode de détection des essais biologiques.19 Par exemple, il est possible
que des composés absorbent, fluorescent ou dévient la lumière parce qu’ils sont insolubles.19 De plus, les
métabolites bioactifs inédits sont souvent en très faible quantité, il est ainsi avantageux de les concentrer par fractionnement.19 Finalement, même si les extractothèques sont plus rapides et moins couteuses initialement,
des efforts de purification devront être effectués pour isoler le composé bioactif et l’identifier.19 Il est ainsi
avantageux de développer des méthodologies de fractionnements automatisés qui permettent de générer des fractions peu complexes. Dans le cas de la chimiothèque du NCI, les fractions contenaient entre 2-28 composés, les composés inédits dans les fractions actives peuvent ainsi être ciblés par HPLC-MS/MS, puis isolés en une étape supplémentaire de HPLC préparative.19
Malgré les efforts et les investissements des pharmaceutiques pour augmenter l’espace chimique et le potentiel bioactif des chimiothèques en titrant profit des PNs, seulement 20 % des squelettes (scaffolds) de PNs sont représentés dans les chimiothèques synthétiques commerciales.14 Les chimiothèques préfractionnées d’extraits
de PNs qui sont générées par des plateformes automatisées sont ainsi une approche viable qui favorise l’inclusion de PNs dans les programmes de criblage à haut débit.14,19 Pour favoriser la découverte de molécules
bioactives inédites à un rythme qui soutient les programmes de recherche, les procédures de déréplications sont essentielles lors de la génération des fractions afin d’identifier leurs profils en métabolites. En plus des
chimiothèques d’extraits préfractionnés, il est possible de tirer profit de la diversité chimique des PNs en développant des approches synthétiques de criblage qui s’inspirent du squelette des PNs pour la génération de chimiothèque.18,19 Par exemple, la conception de médicaments par la méthode des fragments, qui est connu
sous le nom de fragment-based drug discovery (FBDD),19 est une approche où des fragments de molécules qui
ont des propriétés physico-chimiques prompts au développement de médicaments, sont utilisés pour le criblage à haut débit.29 Les squelettes des PNs peuvent également être inclus dans les approches de synthèse orientés
divergentes, soit le diversity-oriented synthesis (DOS).19,30 Cette stratégie synthétique permet de générer
rapidement une chimiothèque qui présente une grande diversité structurale en effectuant des réactions divergentes à partir d’un motif structural au potentiel bioactif élevé.30 Il est aussi possible de tirer profit de la
diversité chimique et du potentiel des PNs à interagir avec des cibles biologiques en gérant des chimiothèques par des approches de chimie combinatoire.18 Lors de cette stratégie, le design de la chimiothèque est effectué
en élaborant des modifications structurales à partir du squelette d’un produit naturel bioactif afin d’améliorer sa spécificité biochimique envers une cible.
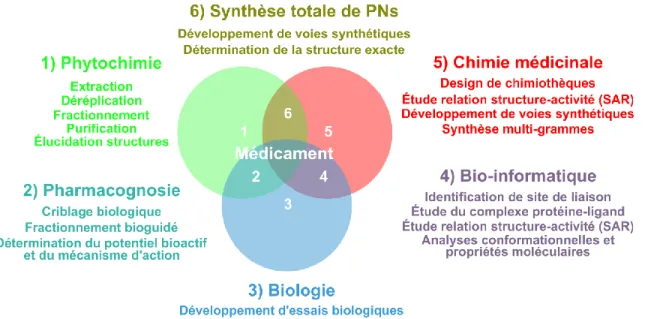
L’investigation phytochimique des organismes vivants qui proviennent de différents écosystèmes reste à ce jour une source inestimable de squelettes uniques de PNs bioactifs. Pour que ce domaine soit en mesure de soutenir le développement de médicaments, des approches modernes de recherche en chimie des PNs sont toutefois primordiales autant pour l’extraction/purification des PNs, que pour dérépliquer les métabolites connus dans les extraits et fractions, que pour le criblage des propriétés biologiques, que pour l’étude des mécanismes d’action, que pour le design des analogues et que pour le développement d’approches synthétiques. La phytochimie, la pharmacognosie, la bio-informatique et la chimie médicinale sont des domaines de recherche qui sont étroitement reliés dans le processus de développement d’un médicament issu d’un produit naturel (Figure 4).18
De façon générale, la plante ou l’organisme est d’abord extrait, puis purifié pour générer des fractions qui seront parallèlement profilées par HPLC-MS/MS pour identifier les métabolites (phytochimie) et criblés vis-à-vis différentes cibles biologiques (pharmacognosie).18 Les composés inédits dans les fractions actives sont ensuite
purifiés à l’échelle du milligramme pour élucider leur structure (phytochimie) et évaluer leur potentiel bioactif (pharmacognosie).18 Le design d’analogues est ensuite réalisé en utilisant des approches de modélisation
moléculaire (bio-informatique).18 Des approches synthétiques sont ensuite développées pour générer une
chimiothèque de composés qui dont le potentiel bioactif sera évalué envers une ou plusieurs cibles (chimie médicinale et pharmacognosie).18 Des voies synthétiques sont alors développées pour accéder à une plus
grande quantité des composés bioactifs (1-100 g), ce qui est nécessaire pour l’étude du mécanisme d’action et les étapes subséquentes en lien avec le développement du médicament (chimie médicinale, pharmacognosie et chimie des procédés).18 Les avancées technologiques en métabolomique, génomique, microbiologie,
bio-informatique/chimie computationnelle et synthèse chimique sont ainsi aussi primordiales pour tirer profit de la diversité chimique des PNs pour le développement de médicaments.
Figure 4 Domaines de recherches impliqués lors du développement d’un médicament issus de PNs
L’inflammation et les maladies chroniques
Le processus d’inflammation est un mécanisme de défense qui joue un rôle essentiel dans le système immunitaire.31 En réponse à un stimulus tel qu’une bactérie, un virus, une réaction allergique ou une
lésion tissulaire, les macrophages et les monocytes induisent le processus d’inflammation en secrétant des cytokines pro-inflammatoires.31,32,33 Ces médiateurs de l’inflammation sont des protéines solubles qui activent
des voies de signalisation en interagissant avec des récepteurs membranaires. Par exemple, lorsqu’une cytokine ou une protéine bactérienne/virale se lie au récepteur de type Toll (TLR4) la voie de signalisation du facteur de transcription nucléaire kappa-B (NF-κB) peut être activité.31,34 Le NF-κB joue un rôle clé dans les
réponses immunitaires et inflammatoires en induisant l’expression de plus de 150 gènes qui sont associés à divers processus cellulaires dont la sécrétion de cytokines pro-inflammatoires.31,35,36 Une dérégulation du
processus d’inflammation, soit la sécrétion excessive et incontrôlée des médiateurs pro-inflammatoires, peut mener au développement de maladies inflammatoires chroniques telles que l’athérosclérose, l’asthme, le psoriasis, l’arthrite et les maladies parodontales (parodontite et gingivite).32 Le développement de médicaments
anti-inflammatoires occupe ainsi une grande importance dans le domaine de la pharmaceutique. En effet, entre 1981 et 2019, les anti-inflammatoires sont en 5e position en termes de nombre de médicaments approuvés avec
3,3 %, derrière les médicaments qui servent à traiter le diabète (3,9 %), l’hypertension (5,1 %), les cancers (25 %) et les maladies infectieuses (25 %).28 Il est ainsi intéressant de spécifier que la sécrétion excessive et
non contrôlée des médiateurs pro-inflammatoires mène aussi au développement de troubles chroniques tels que le diabète et les maladies cardiovasculaires, qui font également partie du top 5 des médicaments les plus
développés entre 1981 et 2019.28 De plus, l’inflammation chronique est aussi directement liée à la
carcinogenèse, soit un processus qui est impliqué dans l’initiation, la promotion et la progression des cancers. La régulation de la sécrétion de cytokines et chimiokines par l’inhibition des processus inflammatoires peut ainsi servir de traitement chimiopréventif.37,38
Cardinal, S. Thèse, Université Laval (2016)39
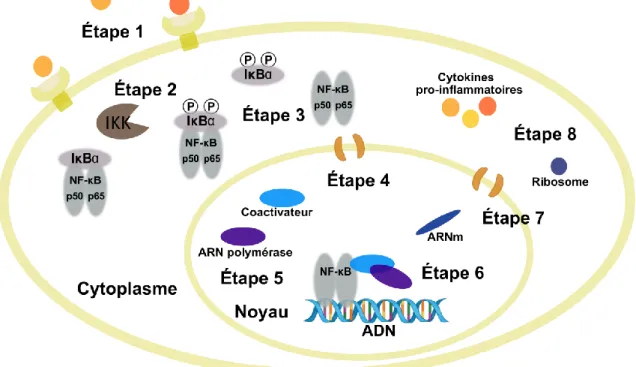
Figure 5 Activation canonique de la voie du facteur de transcription NF-ĸB
Une cible importante des médicaments anti-inflammatoires est le facteur NF-κB, car son inhibition peut atténuer la sécrétion des médiateurs pro-inflammatoires. Le NF-κB est une protéine qui appartient à une famille de facteurs de transcription qui sont formés de sous-unités (p50, p52, p65 (RelA), c-Rel et ReIB).35,40 La forme la
plus commune du NF-κB est l’hétérodimère p50:p65. Ce dernier est inactivé dans le cytoplasme par des protéines inhibitrices de type IκB qui masque son site de liaison.40,41,34 Le mécanisme d’activation du NF-κB par
lequel le complexe NF-κB/IκB sera dissocié pour mener à la transcription des gènes dans le noyau, est un processus biochimique complexe (activation canonique) qui peut être résumé en 8 étapes : (1) activation des récepteurs transmembranaires, (2) formation du complexe enzymatique IKK, (3) dégradation de la protéine inhibitrice IκBα, (4) translocation de NF-κB dans le noyau cellulaire, (5) recrutement de l’ARN polymérase, (6) transcription assistée par NF-κB, (7) migration de l’ARNm maturé vers le cytoplasme et (8) traduction du brin d’ARNm en protéine (Figure 5).39,41,36,40,42 Selon Bremner et Heinrich, les étapes 2 et 3 de l’activation canonique