HAL Id: tel-00361317
https://tel.archives-ouvertes.fr/tel-00361317
Submitted on 13 Feb 2009
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of
sci-entific research documents, whether they are
pub-lished or not. The documents may come from
teaching and research institutions in France or
abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est
destinée au dépôt et à la diffusion de documents
scientifiques de niveau recherche, publiés ou non,
émanant des établissements d’enseignement et de
recherche français ou étrangers, des laboratoires
publics ou privés.
Segmentation des tissus et structures sur les IRM
cérébrales : agents markoviens locaux coopératifs et
formulation bayésienne.
Benoît Scherrer
To cite this version:
Benoît Scherrer. Segmentation des tissus et structures sur les IRM cérébrales : agents markoviens
locaux coopératifs et formulation bayésienne.. Modélisation et simulation. Institut National
Poly-technique de Grenoble - INPG, 2008. Français. �tel-00361317�
INSTITUT NATIONAL POLYTECHNIQUE DE GRENOBLE
Noattribu´e par la biblioth`eque
TH `ESE
pour obtenir le grade de
DOCTEUR DE L’Institut Polytechnique de Grenoble Sp´ecialit´e : « Math´ematiques Appliqu´ees et Informatique »
pr´epar´ee aux laboratoires
TIMC(Technique de l’Ing´enierie M´edicale et de la Complexit´e),
GIN(Grenoble Institut des Neurosciences, INSERM), et LIG(Laboratoire d’Informatique de Grenoble), dans le cadre de l’ ´Ecole Doctorale « Math´ematiques, Sciences et Technologies de
l’Information, Informatique » pr´epar´ee et soutenue publiquement par
Benoit Scherrer le 12 d´ecembre 2008
Segmentation des tissus et structures
sur les IRM c´
er´
ebrales : agents
markoviens locaux coop´
eratifs et
formulation bay´
esienne.
sous la direction de Catherine Garbay et Michel Dojat.
JURY
Pr Christian Jutten Pr´esident
Pr Dr Jean-Philippe Thiran Rapporteur
Dr Christian Barillot Rapporteur
Dr Gr´egoire Malandain Examinateur
Dr Catherine Garbay Directeur de th`ese
Dr Michel Dojat Directeur de th`ese
Titre : Segmentation des tissus et structures sur les IRM c´er´ebrales : agents markoviens locaux coop´eratifs et formulation bay´esienne.
R´esum´e : La segmentation des IRM c´er´ebrales est une ´etape cruciale pour de nombreuses applications, tant dans le domaine clinique que pour les neurosciences. Elle est rendu difficile par les art´efacts inh´erents `a ce type d’image, leur faible contraste et les importantes variations individuelles qui limitent l’introduction de connaissances a priori. Dans cette th`ese nous proposons une nouvelle approche de segmentation des IRM c´er´ebrales dont l’originalit´e r´eside (1) dans le couplage de la segmentation des tissus, de la segmentation des structures et de l’int´egration de connaissances anatomiques et (2) la volont´e de prendre en compte la localit´e de l’information.
La localit´e est mod´elis´ee via un cadre multi-agents : des agents sont distribu´es dans le volume et r´ealisent une segmentation markovienne locale. Dans une premi`ere approche (LOCUS, Local Cooperative Unified Segmentation) nous proposons des m´ecanismes intuitifs de coop´eration et de couplage pour assurer la coh´erence des mod`eles locaux. Les structures sont segment´ees via l’int´egration de contraintes de locali-sation floue d´ecrites par des relations spatiales entre structures. Dans une seconde approche (LOCUSB,
LOCUS in a Bayesian framework) nous consid´erons l’introduction d’un atlas statistique des structures. Nous reformulons le probl`eme dans un cadre bay´esien nous permettant une formalisation statistique du couplage et de la coop´eration. Segmentation des tissus, r´egularisation des mod`eles locaux, segmentation des structures et recalage local affine de l’atlas sont alors r´ealis´es de mani`ere coupl´ee dans un cadre EM, chacune des ´etapes s’am´eliorant mutuellement.
L’´evaluation sur des images simul´ees et r´eelles montrent les performances de l’approche et en particulier sa robustesse aux art´efacts pour de faibles temps de calculs. Les mod`eles markoviens locaux distribu´es et coop´eratifs apparaissent alors comme une approche prometteuse pour la segmentation d’images m´edicales.
Mots-cl´es : Segmentation, Champ de Markov, Multi-agents, Estimation EM distribu´ee, IRM c´er´ebrale
Title : MR brain scan tissus and structures segmentation: local cooperative Markovian agents and Bayesian formulation.
Abstract : Accurate magnetic resonance brain scan segmentation is critical in a number of clinical and neuroscience applications. This task is challenging due to artifacts, low contrast between tissues and inter-individual variability that inhibit the introduction of a priori knowledge. In this thesis, we propose a new MR brain scan segmentation approach. Unique features of this approach include (1) the coupling of tissue segmentation, structure segmentation and prior knowledge construction, and (2) the consideration of local image properties.
Locality is modeled through a multi-agent framework : agents are distributed into the volume and perform a local Markovian segmentation. As an initial approach (LOCUS, Local Cooperative Unified Segmentation), intuitive cooperation and coupling mechanisms are proposed to ensure the consistency of local models. Structures are segmented via the introduction of spatial localization constraints based on fuzzy spatial relations between structures. In a second approach, (LOCUSB, LOCUS in a Bayesian framework) we consider the introduction of a statistical atlas to describe structures. The problem is reformulated in a Bayesian framework, allowing a statistical formalization of coupling and cooperation. Tissue segmentation, local model regularization, structure segmentation and local affine atlas registration are then coupled in an EM framework and mutually improve.
The evaluation on simulated and real images shows good results, and in particular, a robustness to non-uniformity and noise with low computational cost. Local distributed and cooperative MRF models then appear as a powerful and promising approach for medical image segmentation.
Keywords : Segmentation, Markov Random Field, Multi-agents system, Distributed EM esti-mation, MR Brain scan
Remerciements
Cette th`ese est le fruit d’un travail de trois ans r´ealis´e en collaboration avec des laboratoires de recherche de diff´erents domaines. Je suis bien incapable d’´enum´erer ici toutes les personnes que je souhaiterais remercier et j’esp`ere que celles que j’aurais oubli´e ne m’en tiendront pas rigueur.
Je tiens `a remercier les membres de mon jury pour l’accueil qu’ils ont su r´eserver `a mon travail et pour les efforts consacr´es `a la lecture de ce manuscrit. Merci `a mes deux rapporteurs, Christian Barillot et Jean-Philippe Thiran, pour l’attention port´ee `a mon rapport de pr´esoutenance malgr´e un emploi du temps charg´e. Merci ´egalement `a Gr´egoire Malandain pour avoir corrig´e un bon nombre de coquilles, et `a Christian Jutten de m’avoir fait l’honneur de pr´esider ce jury.
Je remercie tout particuli`erement et sans r´eserve mes co-directeurs de th`ese Catherine Garbay et Michel Dojat, pour leur soutien sans relˆache, leur disponibilit´e, leur ´ecoute et leur rigueur. Merci de m’avoir encourag´e `a prendre du recul sur mon travail, et de m’avoir initi´e au fonctionnement de la recherche scientifique. De mˆeme, un immense merci `a Florence Forbes de l’´equipe MISTIS `a l’INRIA pour son influence consid´erable sur ma compr´ehension des champs de Markov. Merci pour sa pr´esence `a toutes nos r´eunions hebdomadaires, pour tous les ´echanges que nous avons pu avoir, pour son dynamisme et son enthousiasme. Travailler dans cet environnement pluridisciplinaire encadr´e par deux puis trois chercheurs au sens humain incroyable a ´et´e un vrai plaisir. Merci `a eux pour tous ces moments partag´es. Je garderai particuli`erement en m´emoire les longues marches dans Alger, Amsterdam ou New York avec eux.
Je tiens aussi `a remercier Christoph Segebarth de m’avoir accueilli dans l’´equipe Neuro-imagerie Fonctionnelle et M´etabolique du Grenoble Institut des Neurosciences, ainsi que Yves Demazeau pour l’accueil dans l’´equipe MAGMA du Laboratoire d’Informatique de Grenoble. Merci `a toutes les personnes que j’ai eu l’occasion de cˆotoyer dans ces laboratoires, en particulier Flor Vasseur, Senan Doyle et Emilie Chipon avec qui j’ai eu de tr`es bons moments. J’exprime aussi ma gratitude aux gestionnaires des ´equipes dans lesquelles j’ai travaill´e, Zilora Zouaoui, Marie-Claude Zanoni, Barta Angles et C´eline Fontant, pour leur gentillesse alors que j’ai rarement ´et´e dans les temps pour les formalit´es administratives.
Un grand merci aussi `a tous mes amis de Grenoble, qui m’ont permis de passer des ann´ees inoubliables dans cette ville avec beaucoup de souvenirs : ski, waterslide, kayak, VTT, randonn´ees, refuges, sauna, golf, ... Merci `a mes diff´erents colocataires, aussi bien de l’avenue Foch, que de l’ile Verte. Merci aussi `a Quentin et Audrey pour l’appartement dans la maison `a Eybens, ainsi qu’au Chalet Crew et aux m´emorables repas du lundi. En particulier, merci `a Antoine Vermeille, mon plus vieil ami que j’ai retrouv´e en venant habiter `a nouveau `a Grenoble.
Enfin, je remercie bien sˆur ceux qui ont fait de moi ce que je suis, mes fr`eres et sœurs Matthieu, Vincent et Estelle, et mes parents Claire et Paul, qui ont toujours su me soutenir et me donner beaucoup d’amour, et `a qui je d´edie cette th`ese.
Th`
ese pr´
epar´
ee au sein des laboratoires
• GIN - Grenoble Institut des Neurosciences, INSERM Chemin Fortun´e Ferrini,
Universit´e Joseph Fourier - Site Sant´e 38706 La Tronche Cedex
• LIG - Laboratoire d’Informatique de Grenoble Maison Jean Kuntzmann
110 avenue de la Chimie
Domaine Universitaire de Saint-Martin-d’H`eres BP 53 - 38041 Grenoble cedex 9
• TIMC - Technique de l’Ing´enierie M´edicale et de la Complexit´e Facult´e de M´edecine
38706 La Tronche cedex
Collaboration
• INRIA - Equipe MISTIS Inovall´ee, 655 avenue de l’Europe Montbonnot
Table des mati`eres
Couverture i
R´esum´e i
Remerciements iii
Table des mati`eres vii
Table des figures xi
Notations xiii
Pr´eface 1
1 Introduction 3
1.1 Motivations . . . 3
1.2 Approches propos´ees . . . 6
1.3 Principales contributions. . . 7
1.4 Organisation du manuscrit. . . 8
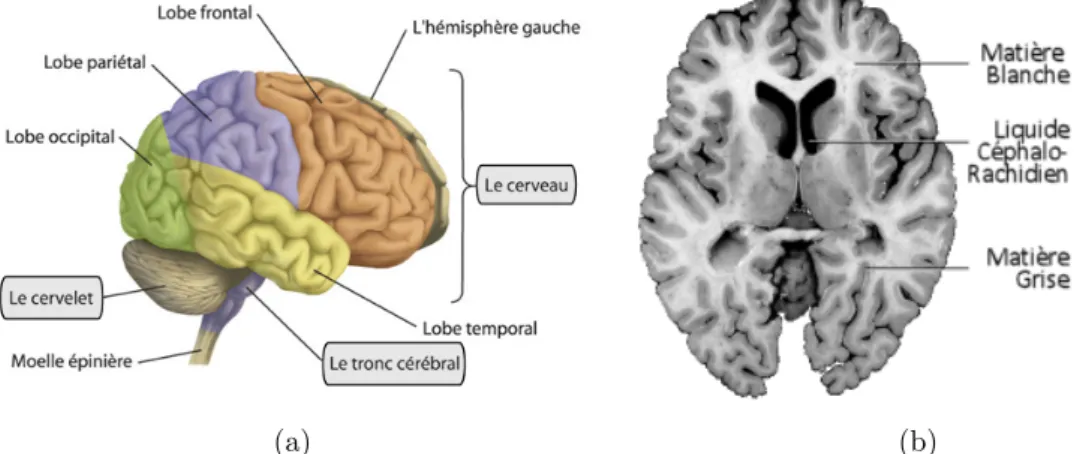
2 Imagerie C´er´ebrale 11 2.1 El´ements d’anatomie c´er´ebrale . . . 11
2.1.1 L’enc´ephale, centre du syst`eme nerveux central . . . 11
2.1.2 Les principales substances pr´esentes dans l’enc´ephale . . . 12
2.1.3 Quelques structures c´er´ebrales d’int´erˆet . . . 13
2.2 Observer le cerveau avec l’imagerie m´edicale. . . 14
2.3 Imagerie par R´esonance Magn´etique . . . 15
2.3.1 Principe physique de la r´esonance magn´etique nucl´eaire (RMN). . . 15
2.3.2 Application `a l’imagerie m´edicale . . . 16
2.3.3 Imperfections de l’acquisition . . . 18
2.4 Interpr´etation automatique des IRM c´er´ebrales . . . 21
2.4.1 Probl´ematique . . . 21
2.4.2 La segmentation : un maillon crucial dans de nombreuses applications . . . 22
2.4.3 Evaluation de la segmentation . . . 24
3 Segmentation des IRM c´er´ebrales 25 3.1 Diff´erentes familles d’approches de la segmentation . . . 26
3.1.1 Segmentation orient´ee contours . . . 26
3.1.2 Segmentation orient´ee r´egions. . . 28
3.1.3 Approches « hybrides » combinant segmentation orient´ee r´egions et contours 31 3.1.4 Extraction de l’enc´ephale . . . 31
3.2 La classification probabiliste des tissus, g´en´eralement consid´er´ee de mani`ere globale 32 3.2.1 Mod´elisation statistique de la segmentation d’image . . . 33
3.2.2 Estimation des param`etres du mod`ele . . . 34
3.2.3 Robustesse au bruit . . . 35
3.2.4 Volume partiel . . . 37
3.2.5 Correction d’inhomog´en´eit´e d’intensit´e . . . 39
3.3.1 Principe, forces et difficult´es. . . 41
3.3.2 Une approche encore peu explor´ee . . . 42
3.3.3 Conclusion . . . 43
3.4 Le paradigme multi-agents pour l’interpr´etation d’images . . . 43
3.4.1 Pr´esentation . . . 43
3.4.2 Formalisme du cadre multi-agents . . . 44
3.4.3 Un cadre adapt´e pour la segmentation locale . . . 46
3.5 La segmentation des structures, consid´er´ee comme un traitement s´epar´e . . . 46
3.5.1 Introduction d’un atlas `a recaler . . . 47
3.5.2 Introduction d’une description floue de l’anatomie c´er´ebrale.. . . 51
3.5.3 Conclusion . . . 52
4 Segmentation coop´erative des tissus et structures dans un cadre multi-agents 53 4.1 Mod´elisation markovienne de la segmentation . . . 54
4.1.1 Voisinage et clique . . . 54
4.1.2 Champ de Markov . . . 55
4.1.3 Segmentation markovienne . . . 55
4.1.4 Estimation des param`etres du mod`ele markovien . . . 59
4.2 Agent markovien local . . . 60
4.3 Segmentation locale et coop´erative des tissus . . . 61
4.3.1 D´efinition hi´erarchique des agents . . . 61
4.3.2 Coop´eration entre mod`eles locaux . . . 62
4.3.3 Coordination entre les agents . . . 63
4.3.4 Algorithme DILEM . . . 65
4.4 Segmentation locale et coop´erative des tissus et structures . . . 65
4.4.1 Agents markoviens locaux sp´ecialis´es structure (AML-S) . . . 65
4.4.2 Int´egration d’a priori anatomique . . . 67
4.4.3 Coop´eration entre mod`eles locaux . . . 67
4.4.4 Coordination entre les agents . . . 69
4.5 Synth`ese . . . 70
5 Segmentation des tissus, structures et recalage : formulation bay´esienne jointe 71 5.1 Motivations . . . 71
5.2 Analyse bay´esienne d’un probl`eme `a donn´ees manquantes . . . 72
5.2.1 Interpr´etation fonctionnelle de EM . . . 73
5.2.2 Estimation d’un mod`ele `a donn´ees manquantes coupl´ees . . . 75
5.3 Mod´elisation de LOCUSB . . . . 76
5.3.1 Mod´elisation markovienne et d´ecomposition en distributions conditionnelles 77 5.3.2 Formulation des interactions . . . 78
5.4 Estimation du mod`ele . . . 81
5.4.1 Etape E : mise `a jour de qS et qT . . . 81
5.4.2 Etape M : mise `a jour des param`etres . . . 82
5.5 Recalage hi´erarchique affine local . . . 85
5.6 Mod´elisation dans le cadre multi-agent . . . 87
6 Impl´ementation 89 6.1 Plate-forme multi-agents g´en´erique . . . 89
6.1.1 Gestionnaire du syst`eme . . . 90
6.1.2 Mod`ele g´en´erique d’agent `a base de comportements . . . 90
6.1.3 Mod`ele g´en´erique de groupe . . . 91
6.1.4 Descripteur g´en´erique d’´ev`enements et interface utilisateur . . . 91
6.1.5 Impl´ementation . . . 92
6.2 Segmentation markovienne g´en´erique . . . 92
6.3 Impl´ementation de LOCUS . . . 93
6.4 Impl´ementation de LOCUSB . . . . 97
7 Evaluation 99 7.1 Donn´ees utilis´ees . . . 99
7.1.1 Evaluation quantitative . . . 100
7.1.2 Evaluation qualitative sur images r´eelles . . . 102
7.2 Algorithmes de segmentation consid´er´es . . . 102
7.3 Algorithmes d’estimation des champs de Markov . . . 103
7.4 Evaluation de LOCUS . . . 103
7.4.1 Evaluation de LOCUS-T . . . 103
7.4.2 Comportements int´eressants de l’approche locale . . . 107
7.4.3 Evaluation de LOCUS-TS . . . 110
7.4.4 Simulation d’image . . . 113
7.5 Evaluation de LOCUSB . . . . 114
7.5.1 Evaluation de LOCUSB-T . . . 114
7.5.2 Evaluation quantitative de LOCUSB-TS et LOCUSB-TSR . . . 114
7.5.3 Evaluation qualitative de LOCUSB-TS et LOCUSB-TSR sur images r´eelles. 118 7.5.4 Evaluation sur une image pathologique. . . 121
7.6 Conclusion . . . 123
8 Synth`ese, Conclusion et Perspectives 125 8.1 Approche LOCUS. . . 127
8.2 Approche LOCUSB . . . 130
8.3 Perspectives . . . 131
8.3.1 Synth`ese. . . 133
A Annexes 135 A.1 Publications scientifiques. . . 135
A.1.1 Approche LOCUS . . . 135
A.1.2 Approche LOCUSB . . . . 136
A.2 Estimation des param`etres d’un champ de Markov . . . 137
A.2.1 Algorithme ICM non supervis´e . . . 137
A.2.2 Algorithme EM . . . 138
A.3 Description floue de l’anatomie via des relations spatiales . . . 142
A.3.1 Traduction des relations spatiales en paysages flous . . . 142
A.3.2 Application `a l’anatomie c´er´ebrale : description de huit structures sous-corticales . . . 144
A.4 Interpolation par krigeage . . . 146
A.4.1 Interpolation mono-dimensionnelle . . . 146
A.4.2 Extension au cas multi-dimensionnel . . . 147
A.4.3 Conclusion . . . 147
A.5 Conversion 8 bits . . . 148
A.6 Comparaison des modalit´es d’imagerie c´er´ebrale. . . 150
A.6.2 Imagerie fonctionnelle . . . 151
Bibliographie 153
Table des figures
1 Atlas de Vesale, neurones color´es et phr´enologie de F.J. Gall. . . 1
1.1 Acquisition IRM d’un cerveau et segmentation . . . 4
2.1 Description de l’enc´ephale et illustration des trois mati`eres principales qui la compose 12 2.2 Quelques structure anatomiques d’int´erˆet . . . 13
2.3 Diff´erentes modalit´es d’imagerie appliqu´ees au cerveau . . . 14
2.4 Le principe de r´esonance magn´etique nucl´eaire. . . 16
2.5 Diff´erentes images acquises par IRM . . . 17
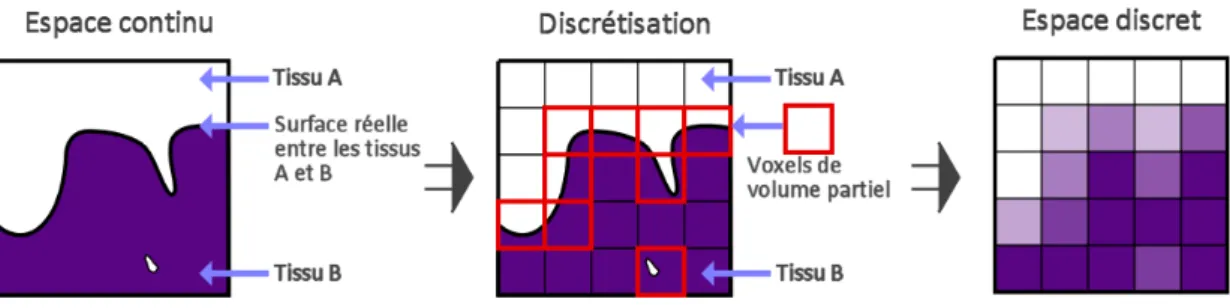
2.6 Art´efact de volume partiel dˆu `a la discr´etisation de l’espace. . . 19
2.7 Art´efact d’inhomog´en´eit´e d’intensit´e . . . 20
2.8 Int´erˆet port´e `a segmentation d’images m´edicales depuis les ann´ees 1980 . . . 22
2.9 Illustration de la segmentation . . . 22
2.10 Diff´erents niveaux d’interpr´etation d’une IRM c´er´ebrale. . . 23
3.1 Approches duales de la segmentation : mod`eles orient´es r´egions ou contours . . . . 26
3.2 Repr´esentation 3D d’une image 2D . . . 27
3.3 Segmentation de structures sous-corticales par mod`ele d´eformable explicite . . . . 28
3.4 Principe de l’extraction de l’enc´ephale . . . 31
3.5 Extraction de l’enc´ephale par BET . . . 32
3.6 Mod´elisation d’une image comme un ensemble de sites organis´es sur une grille. . . 32
3.7 La segmentation vue comme un probl`eme `a donn´ees manquantes. . . 34
3.8 Atlas statistique des tissus . . . 36
3.9 Mod´elisation du volume partiel par Van Leemput et al. (2003) . . . 38
3.10 La majorit´e des approches estiment leurs param`etres de mani`ere globale.. . . 39
3.11 Avantage d’une mod´elisation locale . . . 42
3.12 Les diff´erentes strat´egies de coop´eration . . . 45
3.13 Les distributions d’intensit´es des structures se recouvrent partiellement. . . 47
3.14 Atlas d’´etiquettes . . . 48
3.15 Atlas statistique orient´e r´egions de structures sous-corticales. . . 48
3.16 Les relations spatiales exprim´ees par des paysages flous 3D . . . 51
4.1 Repr´esentation sur une grille r´eguli`ere de diff´erents voisinages . . . 54
4.2 Repr´esentation de diff´erentes cliques 2-D . . . 55
4.3 Sch´ema classique de l’estimation globale par EM . . . 59
4.4 Sch´emas d’un agent markovien local . . . 60
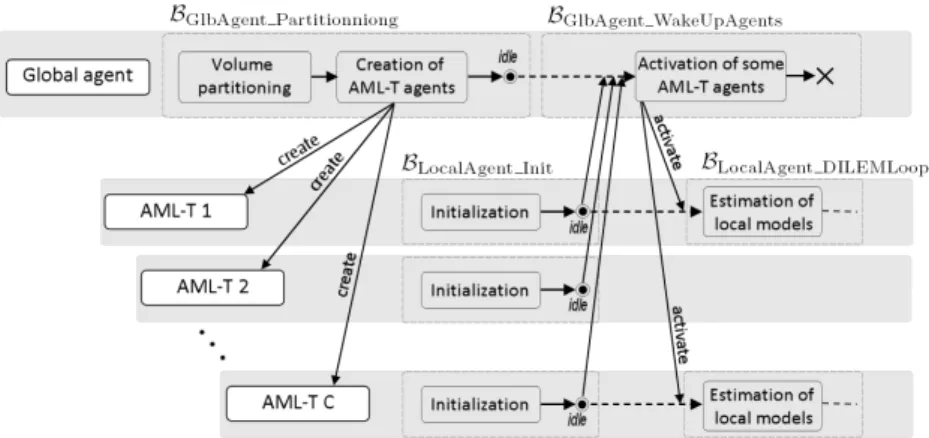
4.5 Partitionnement du volume en sous-volumes cubiques r´eguliers et syst`eme de voisinage 62 4.6 Interactions entre l’agent global et les AML-T `a l’initialisation de la segmentation. 64 4.7 Diagramme d’´etat d’un AML-T et algorithme DILEM . . . 64
4.8 Illustration des groupes d’accointances entre agents tissus et agents structures . . . 66
4.9 Entropie d’une distribution d’intensit´e . . . 68
4.10 Sch´ema synth´etique de LOCUS . . . 70
6.1 Gestionnaire du syst`eme . . . 90
6.2 Descripteur d’´ev`enements et interface utilisateur . . . 91
6.3 Sch´ema synth´etique de l’impl´ementation de la plate-forme multi-agent g´en´erique. . 92
6.4 Conception modulaire pour l’estimation des champs de Markov . . . 93
6.6 Conception modulaire de l’agent global. . . 95
6.7 Conception modulaire d’un AML-T, et 26-voisinage . . . 95
6.8 Interface graphique de la premi`ere approche . . . 96
7.1 Simulateur d’image BrainWeb et ´evaluation . . . 100
7.2 Base de donn´ees IBSR . . . 101
7.3 Evaluation des algorithmes d’estimation des champs de Markov - image 2-D . . . . 104
7.4 Evaluation des algorithmes d’estimation des champs de Markov sur BrainWeb. . . 104
7.5 Evaluation de LOCUS-T et comparaison `a SPM5 et FAST sur huit fantˆomes BrainWeb105 7.6 Evaluation de LOCUS-T sur les deux bases de donn´ees IBSR . . . 105
7.7 Evaluation qualitative de la robustesse aux inhomog´en´eit´es sur une image 3T r´eelle 106 7.8 Evaluation sur une acquisition r´ealis´ee avec une antenne de surface . . . 106
7.9 Evaluation de la robustesse au param`etre de taille des sous-volumes . . . 107
7.10 Illustration de l’activit´e des agents dans le syst`eme . . . 108
7.11 Comportements des agents selon leur position dans le volume . . . 109
7.12 Evaluation de LOCUS-TS sur BrainWeb . . . 110
7.13 La mati`ere grise de certaines structures est sous-segment´ee sur la r´ef´erence de Brain-Web . . . 111
7.14 Apport qualitatif du lien entre segmentation des tissus et des structures . . . 111
7.15 Evaluation qualitative de LOCUS-TS sur des images r´eelles . . . 112
7.16 Simulation d’images `a partir des param`etres du mod`ele de segmentation . . . 113
7.17 Evaluation de LOCUSB-TS et LOCUSB-TSR sur BrainWeb . . . . 115
7.18 Illustration du couplage de la segmentation des structures avec le recalage . . . 116
7.19 Segmentation d’une image BrainWeb avec LOCUSB-TS . . . . 116
7.20 Evaluation de LOCUSB-TS et LOCUSB-TSR sur IBSR v2 . . . . 117
7.21 Evaluation et comparaison de LOCUSB-T, LOCUSB-TS et LOCUSB-TSR sur une image r´eelle 3 Tesla. . . 118
7.22 Reconstruction 3-D des structures segment´ees avec LOCUSB-TSR. . . . 119
7.23 Evaluation de LOCUSB-TSR sur une image r´eelle 3 Tesla . . . . 119
7.24 Evolution de la segmentation et du recalage sur une image r´eelle. . . 120
7.25 Evolution de la segmentation sans recalage coupl´e . . . 120
7.26 Evolution du recalage et de la segmentation pour un recalage artificiellement perturb´e.121 7.27 Evaluation sur une image pathologique. . . 122
7.28 Segmentation des structures sur une image pathologique . . . 122
A.1 Illustration de la relation spatiale « `a gauche de ». . . 142
A.2 Construction du paysage flou d’une relation spatiale de distance. . . 143
A.3 Construction du paysage flou d’une relation spatiale d’orientation. . . 144
A.4 Construction du paysage flou d’une relation spatiale de sym´etrie en trois ´etapes. . 144
A.5 Conversion 8 bits : destruction d’information . . . 148
A.6 Conversion 8 bits : apparition de pics. . . 149
Notations
Notations math´
ematiques
R Ensemble des r´eels.RK Ensemble des vecteurs r´eels `a K dimensions. a(k) K-i`eme composante d’un vecteur a.
ta Transpos´ee d’un vecteur a.
ha, bi Produit scalaire de deux vecteurs a et b, ´egal `ata b =P
ka(k)b(k). E Esp´erance d’une variable al´eatoire.
Uc Fonction potentielle associ´ee `a la clique c. Ui Potentiel sur les singletons.
Uij Potentiel sur les paires de sites voisins.
Notations en lien avec la mod´
elisation multi-agents
AML Agent Markovien Local.AML-T Agent Markovien sp´ecialis´e Tissu. Ac R´ef´erence d’un AML-T.
VT
c Sous-volume d´efinissant le territoire de l’agent markovien sp´ecialis´e tissu. AML-S Agent Markovien sp´ecialis´e Structure.
Al R´ef´erence d’un AML-S.
VlS Sous-volume d´efinissant le territoire de l’agent markovien sp´ecialis´e structure.
Notations en lien avec l’approche bayesienne coupl´
ee
ek Vecteur unitaire de RK repr´esentant la classe de tissu (parmi K tissus).e0l Vecteur unitaire de RL+1 repr´esentant la classe de structure (parmi L structures et le fond). ψ Param`etres des mod`eles d’intensit´es des tissus.
R Param`etres de recalage.
ζS Atlas statistique des structures.
ρ(R, i) Fonction associant la position d’un voxel i de l’espace de l’image `a l’espace de l’atlas. fS Champ d´ecrivant la connaissance a priori sur les structures issue de l’atlas.
Pr´eface
L
e cerveau fascine. Il a pourtant ´et´e longtemps consid´er´e comme un organe d’importance mineure dans le corps humain. Les ´egyptiens ne se pr´eoccupaient par exemple pas de le conserver lors des momifications. Alcm´eon (500 av. JC), disciple de Pythagore, fut le premier auteur occidental `a affirmer que ”ce qui gouverne si`ege dans le cerveau”, ouvrant une p´eriode o`u diff´erentes hypoth`eses s’affront`erent. Ses conceptions furent reprises par Hippocrate (460-370 av. JC) et partag´ees en partie par Platon (427-348/347 av. JC). Elles furent contest´ees par Aristote (384-332 av. JC), qui situait plutˆot le si`ege des pens´ees et des ´emotions dans le cœur. Le cerveau n’´etait alors qu’une machine thermique, destin´ee `a refroidir le sang surchauff´e par les ´emotions ressenties par le cœur. Ce n’est que quelques si`ecles plus tard que Galien (131-201) mit en ´evidence le parcours de l’influx nerveux depuis le cerveau et ´etudia l’influence des nerfs sur le mouvement des muscles. Pendant plus de mille ans les connaissances sur le cerveau n’´evolu`erent gu`ere, par manque d’outils d’exploration efficace d’une part, et par doctrine religieuse qui interdisait toute ´etude anatomique humaine d’autre part, sous peine d’excommunication.C’est durant la Renaissance (seizi`eme si`ecle) que d´ebuta la r´evolution anatomique, avec en parti-culier Vesale (1514-1564), ouvrant la voie `a la dissection humaine et `a l’anatomie descriptive. Il fut `a l’origine de la premi`ere description rigoureuse illustr´ee du corps humain, et du cerveau en particulier (voir Figure1). Franz Joseph Gall (1758-1828), avec la phr´enologie, fut le premier `a attribuer une localisation aux diff´erentes fonctions du cerveau, en supposant que le crˆane ´etait le reflet de la surface c´er´ebrale. Cette id´ee fut tr`es controvers´ee jusqu’aux exp´eriences de Paul Broca (1824-1880), qui la confirma partiellement. Il d´ecrivit le cas d’un de ses patients comprenant le langage mais ne pouvant pas parler. A la mort de son patient, il pu mettre en relation cet handicap avec une petite l´esion dans une r´egion sp´ecifique du cerveau (mais non du crˆane). Les d´ecouvertes de la cellule par Th´eodor Schwann (1840), puis des cellules du syst`eme nerveux, les neurones, par Golgi (1843-1926) et Cajal (1852-1934) grˆace `a une nouvelle technique de coloration, furent fonda-mentales pour les ´etudes sur la forme, les propri´et´es, les fonctions et les connexions des neurones. Korbinian Brodmann (1868-1918) proposa de partitionner le cortex en 52 aires en se basant sur des caract´eristiques cytoarchitectoniques des neurones. Certaines de ces aires furent plus tard associ´ees `
a des fonctions cognitives bien d´efinies (aires 41 et 42 : cortex auditif primaire ; aire 17 : cortex visuel primaire ; etc...) .
(a) (b) (c)
Fig. 1 – Atlas de Vesale (image a) (source : De numani corporis fabrica), neurones color´es par la m´ethode de Golgi (image b) (source : Hubel) et phr´enologie de F.J. Gall.
Depuis le d´ebut du vingti`eme si`ecle, toutes les grandes r´evolutions scientifiques et techniques, de l’´electronique `a la m´ecanique quantique, de la g´en´etique `a l’informatique, ont trouv´e application dans l’´etude du cerveau.
Particuli`erement, la d´ecouverte en 1895 des rayons X par Wilhelm R¨ontgen permit la mise au point au cours des deux d´ecennies suivantes de la radiographie m´edicale. Il n’´etait alors plus n´ecessaire d’avoir recours `a la dissection pour explorer l’int´erieur du corps. Au d´ebut des ann´ees 1970 les pre-miers ordinateurs assez puissants pour r´ealiser la reconstruction 3D de l’image ont permis la mise au point du scanner (Hounsfield, 1970). L’utilisation d’ultrasons (´echographie) s’est d´evelopp´ee (Wild, 1952), `a l’origine pour le diagnostic de tumeurs du cerveau. Puis l’imagerie par r´esonance magn´etique nucl´eaire pour l’´etude de l’anatomie c´er´ebrale (IRM, 1973, Paul Lauterbur et Peter Mansfield) et l’IRM du tenseur de diffusion (IRMd, ann´ees 1990) pour la mise en ´evidence les faisceaux de fibres connectant les diff´erentes aires c´er´ebrales. En parall`ele `a ces techniques d’ob-servation de l’anatomie c´er´ebrale se sont d´evelopp´ees des techniques pour l’exploration de l’acti-vit´e c´er´ebrale : l’´electro-enc´ephalographie (H. Berger, 1929), la magn´eto-enc´ephalographie (Cohen, ann´ees 1970), la TEP (ann´ees 1970) puis l’IRM fonctionnelle (Seiji Ogawa et John Belliveau, 1991). Ces m´ethodes d’imagerie c´er´ebrale permettent d’observer des aspects compl´ementaires de l’anato-mie ou de l’activit´e c´er´ebrale. Accompagn´ees par l’´evolution de l’informatique, elles ont entraˆın´e une v´eritable r´evolution dans la connaissance du fonctionnement du cerveau ; d’une part `a des fins de diagnostics et de traitements m´edicaux, d’autre part en bouleversant le rapport entre psychologie et neurosciences, entraˆınant l’´emergence d’une nouvelle discipline : les neurosciences cognitives.
Les nombreux d´eveloppements en physique et en informatique ont permis l’observation de plus en plus fine de diff´erents aspects du cerveau. La puissance de calcul aujourd’hui disponible doit maintenant en aider l’interpr´etation. Parmi les outils d’interpr´etation, la segmentation des IRM c´er´ebrales anatomiques vise `a localiser les diff´erents tissus et structures anatomiques qui com-posent l’enc´ephale. Elle est au cœur de nombreuses applications, tant dans le domaine clinique (planification d’op´erations neurochirurgicales, suivi de l’´evolution du volume des structures, ...) que des neurosciences (quantification de la variabilit´e inter-individuelle, support pour la projection des donn´ees IRMf, ...) La segmentation met en jeu diverses ´etapes de traitements g´en´eralement consid´er´es de mani`ere globale et s´equentielle : correction des art´efacts de l’image, recalage d’un atlas connu a priori, etc... De mˆeme, les buts, c’est-`a-dire la segmentation des tissus et des struc-tures, sont g´en´eralement consid´er´es comme des probl`emes globaux s´epar´es. L’objet de cette th`ese est de proposer une nouvelle approche de segmentation des IRM c´er´ebrales avec (1) la volont´e de coupler les diff´erents traitements et de coupler les diff´erents buts et (2) la volont´e de ne pas mod´eliser globalement le probl`eme mais de prendre en compte la localit´e de l’information.
1
Introduction
L
’imagerie par r´esonance magn´etique (IRM) a connu un v´eritable essor ces derni`eres ann´ees. Si son coˆut tr`es ´elev´e a au d´ebut ´et´e un handicap, ses nombreux atouts l’ont progressivement aid´ee `a se d´emocratiser. Les champs d’exploration offerts sont larges : l’IRM anatomique est aujourd’hui la m´ethode non invasive la plus adapt´ee pour observer avec une r´esolution fine les tissus biologiques, et donc en particulier les tissus c´er´ebraux. Les d´eveloppements r´ecents permettent aujourd’hui avec le mˆeme imageur d’explorer diff´erents aspects du cerveau : l’activit´e c´er´ebrale avec l’IRM fonction-nelle, ou la connectivit´e des aires c´er´ebrales avec l’IRM de diffusion. Cette modalit´e d’imagerie est ainsi devenu un outil de plus en plus central dans la m´edecine du cerveau ou dans la recherche en neurosciences cognitives.1.1 Motivations
Aujourd’hui, la masse d’information produite par l’imagerie m´edicale a consid´erablement aug-ment´ee. Si les acquisitions m´edicales ont d’abord ´et´e 2D elles sont aujourd’hui majoritairement 3D. Ainsi, l’ensemble des examens IRM r´ealis´es pour la pr´eparation d’op´erations neurochirurgi-cales ou pour l’´etude de pathologies c´er´ebrales (accident vasculaire c´er´ebral, tumeur, scl´erose en plaque...) peut repr´esenter plusieurs centaines de m´ega-octets (100Mo = plus de 0.8 milliards de bits 0 ou 1). Une ´etude en neurosciences cognitives r´ealis´ee `a partir d’une vingtaine de patients en IRM et IRMf peut repr´esenter quant `a elle plusieurs dizaines de giga-octets (1Go = 1024Mo). L’interpr´etation automatique des IRM c´er´ebrales est donc devenu un enjeu majeur. Les m´edecins et les chercheurs en sciences cognitives ont besoin d’outils fiables pour les assister dans leur prise de d´ecision et dans l’interpr´etation de la masse d’information cr´e´ee.
Une acquisition IRM produit un volume 3D de donn´ees dont chaque ´el´ement est appel´e voxel (vo-lume element). Interpr´eter l’image1consiste `a en produire une description symbolique, c’est-`a-dire
`
a reconnaˆıtre et `a d´ecrire les diff´erentes entit´es qui la compose. Parmi les outils d’interpr´etation, la segmentation (ou ´etiquetage) est un maillon crucial dans de nombreuses applications et ana-lyses quantitatives. Le but est, `a partir d’une ou plusieurs images, d’attribuer `a chaque voxel une ´etiquette pour en donner une description. Dans le cadre de la segmentation d’IRM c´er´ebrales, on consid`ere g´en´eralement diff´erents niveaux de description (voir Figure1.1) :
– la segmentation en tissus, qui vise `a d´ecrire la composition de chaque voxel parmi les trois mati`eres principales du cerveau : la mati`ere blanche (MB), la mati`ere grise (MG) et le liquide c´ephalo-rachidien (LCR). Elle est notamment mise en œuvre pour la reconstruction 3D du cortex
1
dans ce manuscrit le terme « image » fait r´ef´erence `a une acquisition, qui peut ˆetre 2D pour une coupe ou 3D pour un volume.
(a) (b) (c) (d)
Fig. 1.1 – Acquisition IRM (image a), segmentation en tissus (image b), segmentation en structures (image c) et segmentation d’une l´esion (image d).
(´etudes en neurosciences cognitives), pour des ´etudes sur la variation de volume de mati`ere grise, ou en planning d’op´erations de chirurgie c´er´ebrale.
– la segmentation en structures, qui d´ecrit l’appartenance de chaque voxel `a une structure c´er´ebrale connue : thalamus, putamen, syst`eme ventriculaire, etc. Elle permet d’en ´etudier le volume lors de pathologies d´eg´en´eratives (maladies de Huntington, de Parkinson, de Alzheimer), de dissocier mati`ere grise du cortex et mati`ere grise des structures sous-corticales, etc.
– la segmentation de l´esions c´er´ebrales (tumeurs, scl´erose en plaque, accidents vasculaires c´er´ e-braux) dans le cadre clinique, qui vise `a ´etudier leur localisation, leur volume, etc.
La segmentation est donc un outil central tant dans le domaine clinique que dans celui des neu-rosciences. Les images IRM souffrent cependant d’un grand nombre d’art´efacts qui rendent leur segmentation automatique difficile : le bruit dans les donn´ees, dˆu `a la mesure physique qui ne peut ˆetre qu’imparfaite ; les variations d’intensit´e au sein d’un mˆeme tissu ou d’une mˆeme structure, dont la source est `a la fois mat´erielle et biologique ; l’effet de volume partiel, du `a la discr´etisation de l’espace, etc.
Ainsi, la segmentation manuelle sur chaque coupe, tr`es coˆuteuse en temps humain (une segmen-tation manuelle de quelques structures sur un seul volume IRM peut prendre une demi-journ´ee), a longtemps ´et´e utilis´ee. Le contraste ´etant faible, certaines fronti`eres entre r´egions sont diffici-lement d´elimitables. la segmentation manuelle introduit alors une variation intra- et inter-expert non n´egligeable : (1) elle est peu reproductible, mˆeme par le mˆeme expert et (2) diff´erents experts proposent des versions diff´erentes selon leur exp´erience. Aujourd’hui, des outils semi-automatiques sont largement utilis´es (particuli`erement pour la segmentation des structures ou des l´esions), mais requi`erent toujours l’intervention humaine.
Pour parvenir `a une segmentation automatique pr´ecise et robuste, il est n´ecessaire de prendre en compte les art´efacts avec des mod`eles math´ematiques adapt´es. Par exemple, un int´erˆet particu-lier a ´et´e port´e r´ecemment sur la mod´elisation statistique avec champ de Markov (Held et al.,
1997;Van Leemput et al.,1999b;Zhang et al.,2001). Cette mod´elisation permet d’introduire une d´ependance spatiale entre ´etiquettes voisines, r´egularisant l’´etiquetage et am´eliorant la robustesse au bruit. De plus, le cadre statistique permet `a tout moment d’avoir une mesure de l’incertitude pour appr´ecier la confiance dans le r´esultat. Si l’id´ee intuitive est simple, certains d´eveloppements math´ematiques formels sont n´ecessaires pour sa mise en œuvre de mani`ere rigoureuse.
Pour la segmentation des tissus, l’approche param´etrique classique est de mod´eliser chaque classe de tissu par un mod`ele d’intensit´e, g´en´eralement gaussien. Il s’agit alors d’estimer les param`etres
de ce mod`ele, `a savoir la moyenne et la variance, pour chaque classe. Pour la segmentation des structures, la seule information d’intensit´e n’est pas suffisante pour l’´etiquetage : par exemple, des structures grises telles les noyaux caud´es, l’hippocampe ou le thalamus pr´esentent toutes une intensit´e grise tr`es proche. Pour leur segmentation, il est n´ecessaire d’apporter de l’information a priori, qui g´en´eralement introduit une connaissance sur leur localisation, voire leur forme. Les m´ethodes classiques utilisent un atlas connu construit au pr´ealable et recal´e sur l’image `a segmenter. La qualit´e de la segmentation est alors principalement d´ependante de la qualit´e du recalage effectu´e en pr´etraitement. R´ecemment l’introduction d’une description floue de l’anatomie c´er´ebrale a ´et´e propos´ee (Barra et Boire,2001;Colliot et al.,2006). Chaque structure est d´ecrite par ses relations spatiales de distance, d’orientation ou de sym´etrie relativement aux autres structures. Par exemple « le noyau caud´e gauche est situ´e `a moins de 5mm de la corne frontale gauche ». Ces relations, fournies par des anatomistes et consid´er´ees comme stables dans l’anatomie humaine, permettent de construire une information possibiliste de localisation de chaque structure
Dans la litt´erature, la majorit´e des approches consid`erent :
(1) La segmentation des tissus et la segmentation des structures comme des tˆaches successives rela-tivement ind´ependantes. Pourtant, une structure est compos´ee d’un tissu connu. L’information tissu est donc li´ee `a la structure, et la connaissance de la localisation des structures fournit une connaissance pertinente quant `a l’intensit´e locale des tissus. Elle informe en particulier sur les variations d’intensit´es dues aux propri´et´es biologiques des tissus.
(2) Une mod´elisation globale de l’intensit´e des tissus. Chaque tissu est d´ecrit par un mˆeme mod`ele gaussien sur tout le volume malgr´e l’art´efact d’inhomog´en´eit´e des intensit´es. Ces approches glo-bales n´ecessitent alors la mod´elisation et l’estimation de mod`eles sp´ecifiques de non-uniformit´e (mod`eles de champ de biais, ou « bias field » en anglais), introduisant des hypoth`eses et une charge calculatoire suppl´ementaires.
(3) Le recalage de l’atlas sur l’image `a segmenter comme un processus global, dont la transformation estim´ee satisfait le volume entier, et pas n´ecessairement des propri´et´es locales. Le recalage se base alors sur une unique transformation ´elastique pour assurer une mise en coh´erence fine des images, induisant d’importants temps de calcul et le risque d’atteindre un grand nombre d’optima locaux.
(4) Le recalage de l’atlas comme un pr´etraitement `a la segmentation et non comme un probl`eme li´e. La connaissance de la segmentation des tissus et des structures peut cependant participer `
a l’am´elioration du recalage.
Les approches classiques ne profitent ainsi pas du couplage entre segmentation des tissus, seg-mentation des structures et recalage. Ce couplage est pourtant pertinent pour raffiner de mani`ere it´erative chaque traitement. Elles ne prennent non plus pas assez en compte la localit´e et le contexte de l’information, pr´ef´erant des mod´elisations globales.
Certaines approches de recalage affine locale ont ´et´e propos´ees (Pohl et al.,2006;Commowick et al.,
2008), dans lesquelles un ensemble de transformations affines locales sont estim´ees et repr´esentent mieux les d´eformations locales. Quelques approches locales de segmentation des tissus ont aussi ´et´e propos´ees. L’id´ee est d’estimer des mod`eles d’intensit´es locaux `a diff´erents endroits du volume. Ces mod`eles refl`etent beaucoup mieux les propri´et´es locales de l’image. En particulier, ils prennent en compte sans mod´elisation explicite les variations d’intensit´es des tissus. La difficult´e majeure est toutefois d’assurer la coh´erence globale des mod`eles locaux. Dans les approches actuelles, la localit´e est mise en œuvre (1) soit en pr´etraitement afin d’estimer un champ de biais (Shattuck et al.,2001), (2) soit en estimant des mod`eles d’intensit´es sur des territoires qui se recouvrent pour
assurer la coh´erence des mod`eles locaux. Si cette redondance peut ˆetre mise en œuvre avec une mod´elisation simple de la segmentation (Zhu et Jiang,2003), elle introduit une charge calculatoire beaucoup trop importante lorsqu’une mod´elisation markovienne (Rajapakse et al., 1997) robuste au bruit est consid´er´ee.
Ces approches montrent cependant l’int´erˆet de la localit´e et motivent la recherche dans cette direc-tion. L’approche r´ecente deRichard et al.(2007) d´emontre l’int´erˆet d’une architecture multi-agents pour assurer la r´egularisation des mod`eles locaux de fa¸con efficace. L’approche r´ecente de Pohl et al.(2006) couplant segmentation des structures et recalage d’atlas illustre par ailleurs l’int´erˆet de coupler les diff´erents traitements n´ecessaires `a la segmentation. L’objet de cette th`ese s’inscrit dans cet esprit : nous proposons deux approches markoviennes locales de segmentation des IRM c´er´ebrales avec la volont´e (1) de coupler les mod`eles de segmentations des tissus et des structures, (2) de profiter du caract`ere local de l’information et (3) d’int´egrer dans une approche unifi´ee les ´etapes suivantes de la segmentation : recalage, prise en compte du bruit, des inhomog´en´eit´es, et introduction de connaissances anatomiques.
1.2 Approches propos´
ees
LOCUS : Segmentation locale coop´erative des tissus et structures dans un cadre multi-agents
Le caract`ere distribu´e du processus de segmentation locale engendre des besoins relatifs `a la colla-boration entre processus, `a la coordination des traitements, `a la communication et `a la gestion des conflits. Le cadre multi-agents est un formalisme adapt´e qui propose des solutions en termes de repr´esentation d’entit´es autonomes (les agents), de m´ecanismes de coop´erations et de coordination.
Dans une premi`ere approche nous proposons donc une mod´elisation multi-agents de la segmentation markovienne locale et coop´erative des tissus et structures. Nous d´efinissons la notion g´en´erale d’agent coop´eratif markovien charg´e d’estimer les param`etres d’un champ de Markov local. Ces agents sont situ´es spatialement dans le volume et se voient attribuer (1) un territoire local de segmentation et (2) un ensemble d’accointances repr´esentant les entit´es avec lesquelles ils coop`erent. Des agents sp´ecialis´es tissus sont distribu´es dans le volume et segmentent leur territoire en tissus (LCR, MG, MB). Les territoires consid´er´es sont disjoints de mani`ere `a assurer l’efficacit´e de l’es-timation des champs de Markov ; la coh´erence des mod`eles locaux est assur´ee par des m´ecanismes de coop´eration entre agents voisins. Des m´ecanismes de coordination permettent d’ordonner l’es-timation locale de mani`ere asynchrone afin de consid´erer d’abord les zones les plus « sures ». Des agents sp´ecialis´es structure sont aussi distribu´es et int`egrent des contraintes de localisation floue des structures d´ecrites par des relations spatiales. Cette connaissance n’est pas statique mais mise `
a jour par des m´ecanismes de coop´eration entre agents structures. La segmentation des structures n’est pas consid´er´ee comme une tˆache isol´ee : l’information structure est int´egr´ee dans le mod`ele des tissus via des m´ecanismes de coop´eration, tandis que la segmentation des structures s’appuie sur l’estimation affin´ee des mod`eles d’intensit´es des tissus. Cette premi`ere approche, LOCUS (LOcal Cooperative Unified Segmentation), fournit des r´esultats comparables aux principaux outils de segmentation utilis´es en neuroscience avec des temps de calcul comp´etitifs.
LOCUSB : Segmentation des tissus, structures et recalage : formulation bay´esienne jointe
Le cadre multi-agents permet de mod´eliser de mani`ere ´el´egante la complexit´e des interactions entre processus locaux. Cependant, certains m´ecanismes de coop´eration dans la premi`ere approche restent « ad-hoc » et introduisent des param`etres dont la sensibilit´e est difficile `a ´evaluer. Ils
souffrent par ailleurs de l’absence d’un cadre th´eorique formel assurant la convergence des mod`eles math´ematiques.
Dans la seconde approche nous proposons d’exprimer la coop´eration entre la segmentation des tissus et des structures via une mod´elisation probabiliste jointe traduisant le couplage des deux mod`eles. Pour la segmentation des structures, nous int´egrons la connaissance d’un atlas statistique a priori d´ecrivant la distribution spatiale de chaque structure. La mise en correspondance de l’atlas sur l’image `a segmenter est consid´er´ee de mani`ere locale via l’estimation d’une transformation affine propre `a chaque structure. De plus, plutˆot que de consid´erer le recalage de l’atlas comme un pr´etraitement, celui-ci est incorpor´e dans le mod`ele de segmentation et raffin´e it´erativement. Nous formulons alors le probl`eme g´en´eral de la segmentation coop´erative via une unique distribu-tion jointe liant ´etiquettes des tissus et des structures, param`etres locaux d’intensit´e et param`etres de recalage. Nous d´efinissons un champ de Markov sur cette distribution jointe pour introduire des d´ependances dans le mod`ele. Notre approche est alors fond´ee sur la d´ecomposition de la distribu-tion jointe en distribudistribu-tions condidistribu-tionnelles, permettant d’identifier et de sp´ecifier naturellement les diff´erents types d’interactions : des interactions (1) pour traduire la corr´elation spatiale entre les ´etiquettes, (2) pour r´egulariser les mod`eles d’intensit´e locaux, (3) pour traduire le couplage tissus-structures et (4) pour guider le recalage d’atlas. De plus, le cadre statistique dans lequel nous nous pla¸cons offre des outils permettant de d´ecrire de mani`ere rigoureuse les ´etapes n´ecessaires `a l’estimation d’un tel mod`ele. Segmentation des tissus, r´egularisation des mod`eles locaux, segmen-tation des structures et recalage de l’atlas sont alors r´ealis´es de mani`ere coupl´ee dans un cadre Expectation Maximization (EM), chacune des ´etapes s’am´eliorant mutuellement.
Cette approche, LOCUSB (LOcal Cooperative Unified Segmentation in a Bayesian framework), est elle aussi impl´ement´ee dans un paradigme multi-agents. Elle permet de combiner les avantages d’une mod´elisation statistique aux fondements solides avec ceux d’une impl´ementation distribu´ee efficace.
1.3 Principales contributions
Les principales contributions de cette th`ese sont donc de :
(1) Casser la logique traditionnelle du calcul (estimation globale sur tout le volume) en distribuant des processus d’estimation locale autonomes, qui refl`etent beaucoup mieux les caract´eristiques locales de l’image.
(2) Assurer la coh´erence des mod`eles locaux non pas par une redondance d’information mais par une nouvelle modalit´e de r´egulation entre mod`eles voisins. La premi`ere approche propose une r´egulation ad-hoc qui fournit de bons r´esultats. La seconde fournit une modalit´e de r´egulation statistiquement fond´ee via la d´efinition d’un champ de Markov sur les mod`eles d’intensit´es locaux.
(3) Orienter l’estimation locale d’abord sur les zones les plus riches et les plus sures en information en mettant en œuvre des m´ecanismes de d´esynchronisation et de coordination entre processus. (4) Consid´erer diff´erentes sources d’information a priori pour segmenter les structures sous-corti-cales : le recalage d’un atlas ou la description de l’anatomie c´er´ebrale par un ensemble de relations spatiales floues entre structures.
(5) Casser le cloisonnement segmentation des tissus / construction de connaissances anatomiques / segmentation des structures. On ne les consid`ere plus comme des tˆaches successives mais on les consid`ere dans leur globalit´e, comme des tˆaches collaboratives. Cette fusion d’informations permet `a chaque processus de se renforcer et de s’am´eliorer mutuellement.
(6) Proposer une mod´elisation statistique coupl´ee de la segmentation des tissus, des structures, de la correction des mod`eles locaux et du recalage local affine dans un cadre EM via la d´efinition de distributions conditionnelles qui permettre d’identifier naturellement les interactions.
1.4 Organisation du manuscrit
Le Chapitre II introduit les termes et concepts essentiels pour appr´ehender l’analyse des images IRM c´er´ebrales. Nous pr´esentons quelques notions ´el´ementaires d’anatomie c´er´ebrale (Section2.1, page 11), donnons un rapide aper¸cu des diff´erentes modalit´es d’imagerie m´edicale (Section 2.2, page 14) et nous nous focalisons sur le principe de l’imagerie par r´esonance magn´etique (IRM) (Section2.3, page15). Nous d´etaillons alors la description des imperfections de ce type d’imagerie, `
a l’origine d’artefacts dans l’image (Section 2.3.3, page 18). Nous pr´esentons ensuite la notion g´en´erale d’interpr´etation des IRM c´er´ebrales et d´ecrivons en particulier la segmentation. Nous mettons en ´evidence ses nombreuses applications dans le cadre clinique et celui des neurosciences (Section2.4, page21), soulignant l’int´erˆet d’une segmentation aussi robuste et fiable que possible.
Le Chapitre III vise `a ´etablir un ´etat de l’art des directions retenues dans la litt´erature pour la seg-mentation des IRM c´er´ebrales. Nous d´ecrivons bri`evement les principales familles d’approches pour la segmentation, en distinguant notamment celles orient´ees contours, celles orient´ees r´egion (Sec-tion3.1, page26) et certaines m´ethodes « hybrides ». (Section3.1.3, page31). Pour la segmentation des tissus sur les IRM c´er´ebrales nous nous focalisons ensuite sur une pr´esentation d´etaill´ee des approches de classification probabiliste et exposons diff´erentes mod´elisation des artefacts dans ce cadre (Section3.2, page32). La Section3.3(page41) pr´esente le concept de segmentation locale de l’image, encore peu exploit´ee dans la litt´erature, et pointe les limites des approches locales actuelles. Nous introduisons ensuite le paradigme multi-agents (Section3.4, page43), offrant un formalisme adapt´e pour la mod´elisation du caract`ere distribu´e de la segmentation locale. Enfin, nous d´ecrivons et comparons deux types d’approches pour la segmentation des structures sous-corticales (Section
3.5, page46) : des approches reposant sur l’introduction d’un atlas a priori connu des structures, et des approches reposant sur une description g´en´erale de l’anatomie c´er´ebrale via la d´efinition de relations spatiales floues entre structures.
Dans le Chapitre IV nous d´etaillons notre premi`ere approche LOCUS fond´ee sur une mod´elisation multi-agents. Nous commen¸cons par introduire le cadre g´en´eral de la segmentation markovienne (Section4.1, page54) et proposons la d´efinition d’un agent markovien local charg´e d’estimer de mani`ere coop´erative les param`etres d’un champ de Markov local (Section 4.2, page 60). Nous montrons ensuite comment ce type d’agent peut ˆetre sp´ecialis´e pour segmenter `a la fois les tissus et les structures sur les IRM c´er´ebrales (Sections4.3, page61et4.4, page61).
Le Chapitre V d´ecrit la seconde approche LOCUSB fond´ee sur la formulation jointe du probl`eme. Nous introduisons le probl`eme g´en´eral d’un probl`eme `a donn´ees manquantes coupl´ees dans un cadre bay´esien et d´ecrivons sa r´esolution via une vision fonctionnelle de l’algorithme EM (Section 5.2, page72). Nous montrons ensuite que la d´efinition d’un champ de Markov sur la distribution jointe conditionnelle des param`etres et des donn´ees manquantes permet d’identifier les diff´erents types d’interactions au sein du mod`ele (Section 5.3, page76). Nous sp´ecifions ces interactions (Section
5.3.2, page78) et d´etaillons les diff´erentes ´etapes n´ecessaires `a l’estimation du mod`ele (Section5.4, page81). Nous pr´esentons alors bri`evement comment ce type d’approche peut aussi ˆetre mod´elis´e dans un cadre multi-agents (Section5.6, page87).
L’impl´ementation des deux approches est d´etaill´ee dans le Chapitre VI, qui souligne le souci mo-dulaire avec lequel l’impl´ementation a ´et´e r´ealis´ee. L’´evaluation des approches est pr´esent´ee dans le chapitre VII. Elle ne vise pas seulement `a ´etablir les performances pures des approches, mais aussi `
a illustrer certains comportements int´eressants des entit´es locales plong´ees dans le syst`eme multi-agents. Le Chapitre VIII donne une conclusion g´en´erale et esquisse des perspectives int´eressantes `
2
Imagerie C´er´ebrale
Sommaire
2.1 El´ements d’anatomie c´er´ebrale . . . 11 2.1.1 L’enc´ephale, centre du syst`eme nerveux central . . . 11 2.1.2 Les principales substances pr´esentes dans l’enc´ephale. . . 12 2.1.3 Quelques structures c´er´ebrales d’int´erˆet . . . 13 2.2 Observer le cerveau avec l’imagerie m´edicale . . . 14 2.3 Imagerie par R´esonance Magn´etique . . . 15 2.3.1 Principe physique de la r´esonance magn´etique nucl´eaire (RMN). . . 15 2.3.2 Application `a l’imagerie m´edicale . . . 16 2.3.3 Imperfections de l’acquisition . . . 18 2.4 Interpr´etation automatique des IRM c´er´ebrales . . . 21 2.4.1 Probl´ematique . . . 21 2.4.2 La segmentation : un maillon crucial dans de nombreuses applications . 22 2.4.3 Evaluation de la segmentation . . . 24
2.1 El´
ements d’anatomie c´
er´
ebrale
Cette premi`ere section pr´esente quelques notions ´el´ementaires d’anatomie c´er´ebrale. Elle d´efinit les termes et concepts essentiels qui permettent de mieux comprendre ce que l’on observe avec l’imagerie c´er´ebrale.
2.1.1
L’enc´
ephale, centre du syst`
eme nerveux central
L’enc´ephale (du grec en, dans, et k´ephalˆe, tˆete ; qui est plac´e dans la tˆete ) est la partie du syst`eme nerveux contenue dans la boˆıte crˆanienne et reposant sur la base du crˆane. Il comprend le cerveau, le cervelet et le tronc c´er´ebral (voir Figure2.1a) :
– Le cerveau (du latin cerebrum) occupe la majeure partie de l’enc´ephale et est divis´e en 2 h´emisph`eres c´er´ebraux : l’h´emisph`ere droit et l’h´emisph`ere gauche. La surface du cerveau pr´esente de nombreux replis, avec un grand nombre de circonvolutions limit´ees par des sillons. Certains sillons plus profonds, les scissures, permettent de diviser chaque h´emisph`ere en 4 lobes : le lobe frontal, le lobe pari´etal, le lobe temporal et le lobe occipital. Chaque lobe comprend des zones bien pr´ecises impliqu´ees dans des fonctions sp´ecifiques (motricit´e, sensibilit´e etc.). – Le cervelet (du latin cerebellum, diminutif de cerebrum) est situ´e en parall`ele sur deux grandes
(a) (b)
Fig. 2.1 – L’image (a) illustre l’enc´ephale, compos´ee du cerveau, du cervelet et du tronc c´er´ebral. L’image (b) montre une coupe histologique d’un cerveau mettant en ´evidence les trois mati`eres principales de l’enc´ephale.
l’analyse ; et celles qui partent du cortex et descendent vers les muscles pour les faire se contracter. Le cervelet est en partie impliqu´e dans la coordination des mouvements du corps.
– Le tronc c´er´ebral repr´esente un carrefour majeur du syst`eme nerveux central entre le cerveau et le cervelet. Il est responsable en particulier de la r´egulation de la respiration et du rythme cardiaque. C’est ´egalement un centre de passage des voies motrices et sensitives, ainsi qu’un centre de contrˆole de la douleur.
Le poids moyen de l’enc´ephale est de 1400 `a 1800 grammes. Il est envelopp´e et prot´eg´e par des membranes appel´ees m´eninges : la dure-m`ere (la plus externe), l’arachno¨ıde, et la pie-m`ere (la plus interne).
2.1.2
Les principales substances pr´
esentes dans l’enc´
ephale
Outre la pr´esence d’art`eres et de veines c´er´ebrales, de tissus servant de parois, ou de nombreuses petites structures telles des glandes, l’enc´ephale contient principalement trois substances (voir Figure2.1.b) :
Le liquide c´ephalo-rachidien (LCR) (ou liquide c´er´ebro-spinal, LCS) est le liquide dans le-quel baignent le cerveau et le cervelet. D’un volume moyen de 150ml, il est absorb´e par le syst`eme veineux c´er´ebral et continuellement renouvel´e. Ses principaux rˆoles sont :
– la protection m´ecanique du syst`eme nerveux central contre les chocs par amortissement des mouvements,
– la protection contre les infections, car il contient les m´ediateurs de l’immunit´e humorale et cellulaire,
– le transport des hormones entre les diff´erentes r´egions ´eloign´ees du cerveau.
La mati`ere grise (MG) (ou substance grise) correspond aux corps cellulaires des neurones avec leur dense r´eseau de dendrites. Elle se r´epartit en :
– une couche superficielle d’une ´epaisseur de quelques millim`etres recouvrant le cerveau et le cervelet : le cortex.
– en noyaux profonds : les noyaux gris centraux.
La mati`ere blanche (MB) (ou substance blanche) correspond `a la gaine de my´eline qui recouvre les axones des neurones pour en acc´el´erer la conduction. Les axones my´elinis´es s’assemblent en faisceaux pour ´etablir des connexions entre groupes de neurones.
(a) (b)
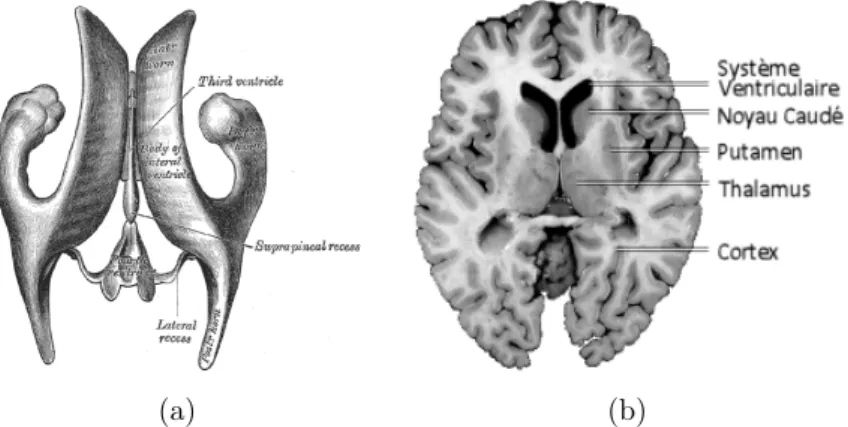
Fig. 2.2 – L’image (a) montre les quatre ventricules (source : 20th U.S. edition of Gray’s Anatomy of the Human Body). L’image (b) pointe quelques structures c´er´ebrales sur une coupe histologique.
2.1.3
Quelques structures c´
er´
ebrales d’int´
erˆ
et
Les trois substances pr´ec´edemment introduites se regroupent en r´egions spatialement stables dans l’anatomie humaine : les structures c´er´ebrales. Si leur rˆole fonctionnel exact n’est pas encore parfai-tement connu, de multiples exp´eriences ont n´eanmoins permis de comprendre en partie leur impli-cation dans diff´erents m´ecanismes neurologiques. Diff´erents niveaux de description des structures c´er´ebrales peuvent ˆetre consid´er´es. Par exemple, le cortex peut ˆetre d´ecrit comme une structure `
a part enti`ere. On peut aussi consid´erer ses diff´erents lobes, ou mˆeme les 52 aires d´ecrites par K. Brodmann. Cette section d´ecrit bri`evement le cortex ainsi que quelques structures c´er´ebrales qui seront ´etiquet´ees dans les approches de segmentation propos´ees. Leur implication fonctionnelle est aussi abord´ee, motivant l’int´erˆet d’algorithmes de segmentation robustes pour en am´eliorer l’´etude et le diagnostic clinique.
Le cortex (du latin cortex : ´ecorce) est le mince manteau superficiel de mati`ere grise recouvrant le cerveau (cortex c´er´ebral) et le cervelet (cortex c´er´ebelleux). Il comporte de multiples re-plis, parmi lesquels on diff´erencie les sillons (la partie profonde) et les circonvolutions (les replis en surface). Sa surface externe repr´esente environ 2200cm2dont les deux tiers sont en-fouis dans les sillons. Le cortex est constitu´e de plusieurs couches de neurones. Son ´epaisseur moyenne est de 3mm, avec des variations allant de 1.5mm `a 4.5mm selon les r´egions corti-cales. Elles rassemblent environ 75% des 100 milliards de neurones de l’enc´ephale. Le cortex humain abrite, en plus des aires motrices, visuelles et sensorielles comme le cortex des autres mammif`eres, des r´egions propres au langage, `a l’imagination, au raisonnement...
Les structures sous-corticales sont enfouies dans l’espace prot´eg´e par le cortex. Parmi elles, on distingue notamment :
Le syst`eme ventriculaire est la structure centrale dans laquelle circule le liquide c´ ephalo-rachi-dien. On distingue quatre cavit´es communicantes, appel´ees « ventricules » : deux ventricules lat´eraux dans les h´emisph`eres c´er´ebraux, un ventricule dans le dienc´ephale et un quatri`eme situ´e entre le cervelet et le bulbe. Le syst`eme ventriculaire est en particulier le si`ege de la synth`ese du LCR, au niveau des plexus choro¨ıdes, `a raison d’environ 600 ml/jour.
Le thalamus est constitu´e de 2 gros noyaux gris sym´etriques. Il relaie les informations visuelles, auditives, gustatives et tactiles vers le cortex et d´etermine lesquelles vont atteindre la cons-cience. Il participe aux ´echanges moteurs entre le cortex, les ganglions de la base et le cervelet. Il est aussi impliqu´e dans la douleur et l’attention.
Les ganglions de la base sont constitu´es de multiples structures de mati`ere grise, parmi les-quelles le pallidum, le striatum form´e par le noyau caud´e et le putamen, etc. Ils sont
tra-(a) (b) (c) (d)
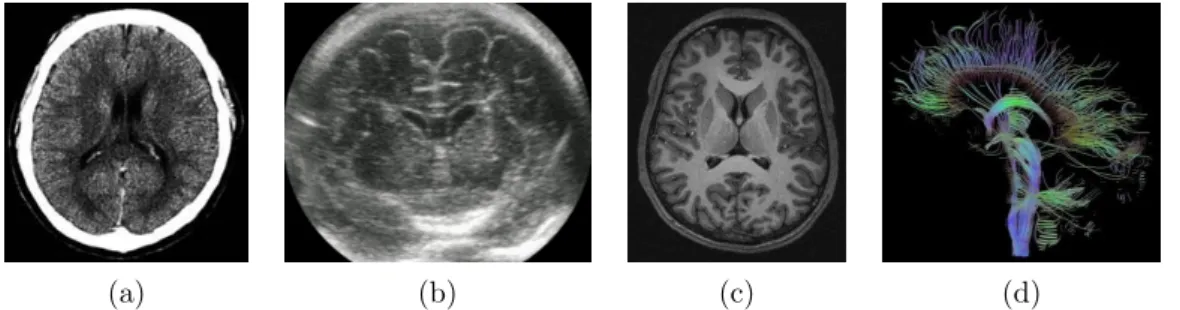
Fig. 2.3 – Diff´erentes modalit´es d’imagerie appliqu´ees au cerveau : le scanner X (image a), l’ul-trasonographie (image b), l’IRM anatomique (image c) et le r´esultat du traitement d’une IRM du tenseur de diffusion (image d).
vers´es par des axones my´elinis´es. Ces faisceaux de mati`ere blanche forment des stries (visibles sur la Figure 2.2a) qui se d´emarquent de la mati`ere grise des noyaux qu’ils traversent. Ils forment avec le cortex c´er´ebral et le thalamus un circuit striato-thalamo-cortical qui joue un rˆole fondamental dans la motricit´e volontaire mais aussi dans de nombreuses autres fonctions cognitives comme l’apprentissage, la m´emoire ou les ´emotions.
L’hippocampe est une partie ancienne du cortex apparue avec les premiers mammif`eres. C’est la porte d’entr´ee des informations `a m´emoriser. L’hippocampe relaie ces informations au cortex pour leur stockage `a long terme et les r´ecup`ere lors de la rem´emoration. Ses nombreuses connexions avec l’ensemble des aires sensorielles du cortex permettent d’encoder tout le contexte associ´e `a un ´ev`enement.
L’amygdale, en forme d’amande, est situ´ee pr`es de l’hippocampe et joue un rˆole dans l’appren-tissage, la m´emoire et surtout dans la r´egulation des ´emotions. Elle est particuli`erement impliqu´ee dans les r´eactions de peur et les r´eactions agressives.
2.2 Observer le cerveau avec l’imagerie m´
edicale
L’imagerie m´edicale a r´evolutionn´e les connaissances sur le cerveau car elle en permet l’observa-tion in vivo. Diverses modalit´es ont ´et´e mises au point durant le si`ecle dernier. Chacune d’entre elles se base sur des propri´et´es physiques diff´erentes et met en œuvre un dispositif d’acquisi-tion sp´ecifique. Les images obtenues selon chacune des modalit´es ont des caract´eristiques propres (r´esolution, contraste, art´efacts, acquisition 2D, 3D ou 3D+t, ...) et fournissent des observations compl´ementaires. Les modalit´es d’imagerie m´edicales sont g´en´eralement regroup´ees selon deux familles :
L’imagerie anatomique (ou structurelle) pour observer l’anatomie c´er´ebrale. Les modalit´es d’im-agerie anatomique peuvent se baser sur des propri´et´es acoustiques (ultrasonographie), des propri´et´es d’att´enuation des rayons X (scanner-X = tomodensitom´etrie), des propri´et´es op-tiques (imagerie optique) ou des propri´et´es magn´etiques (IRM). Le scanner-X est davantage adapt´e pour l’observation des tissus durs, alors que l’IRM est adapt´e pour les tissus mous. L’ultrasonographie fournit des images de faible qualit´e mais est suffisante pour un grand nombre d’applications. Elle est largement utilis´ee car tr`es bon march´e et sans danger connu. L’imagerie fonctionnelle permet d’´etudier l’activit´e c´er´ebrale, et donc de mettre en ´evidence des m´ecanismes du fonctionnement c´er´ebral. Certaines modalit´es sont invasives et reposent sur le suivi d’un traceur radioactif (TEP, TEMP), n´ecessitant des installations parfois lourdes. D’autres reposent sur l’observation non invasive des signaux ´electriques sur la surface du cer-veau (´electro-enc´ephalogramme) ou des champs magn´etiques induits par l’activit´e ´electrique
des neurones (magn´eto-enc´ephalographie). L’IRM fonctionnelle repose quant `a elle sur une mesure indirecte de l’activit´e c´er´ebrale (effet BOLD) : elle met en ´evidence l’afflux sanguin transporteur d’oxyg`ene n´ecessaire `a l’activit´e des neurones.
L’IRM anatomique est la modalit´e non invasive qui permet la meilleure caract´erisation des tissus. C’est donc la modalit´e la plus adapt´ee pour observer les tissus et les structures du cerveau. Elle offre une r´esolution fine (≤ 1mm)1pour des temps d’acquisition de 1min `a 20min. Les travaux pr´esent´es
dans cette th`ese se focalisent sur la segmentation d’IRM c´er´ebrales anatomiques. Certaines id´ees pourraient cependant ˆetre reprises pour d’autres modalit´es d’imagerie.
La section suivante pr´esente le principe physique de l’IRM anatomique, permettant de mieux comprendre les art´efacts rencontr´es sur ces images.
2.3 Imagerie par R´
esonance Magn´
etique
L’imagerie par r´esonance magn´etique est une technique d’imagerie m´edicale bas´ee sur le ph´enom`ene de r´esonance magn´etique nucl´eaire (RMN, terme cr´e´e en 1930 par Isidor Isaac Rabi ; prix Nobel de Physique en 1944). Ce ph´enom`ene fut observ´e pour la premi`ere fois en 1945 de fa¸con simultan´ee par deux ´equipes am´ericaines, l’´equipe de Felix Bloch `a Stanford et celle de Edward Mills Purcell `
a Harvard, qui publi`erent leurs r´esultats en 1946 (Bloch et al., 1946; Purcell et al., 1946) (prix Nobel de physique conjoint en 1952). Destin´ee `a l’origine `a analyser la structure mol´eculaire, la r´esonance magn´etique nucl´eaire permet l’analyse du comportement des protons soumis d’une part `
a un champ magn´etique, et d’autre part `a une impulsion de radiofr´equence.Damadian(1971) posa les premi`eres pierres de l’imagerie par RMN en observant des diff´erences de param`etres RMN (les temps de relaxation) entre tissus sains et tissus pathologiques. La premi`ere image par RMN fut publi´ee en 1973 par Paul Christian Lauterbur (Lauterbur,1973). De mani`ere ind´ependante, Peter Mansfield proposa une technique semblable utilisant des gradients de champ magn´etique pour s´eparer spatialement les signaux RMN (Mansfield et Grannell,1973). Leurs travaux fondamentaux pour l’´emergence de l’imagerie par r´esonance magn´etique leur ont valu le prix Nobel de m´edecine en 2003.
La section suivante pr´esente le principe physique de r´esonance magn´etique nucl´eaire pour l’´etude du comportement des protons. Ensuite, une description succincte de son application en imagerie m´edicale est propos´ee, abordant les concepts d’encodage spatial, de formation de l’image et de s´equence d’acquisition. Enfin, une attention particuli`ere est port´ee sur les art´efacts rencontr´es en IRM, dont la connaissance est indispensable pour proposer des traitements automatiques adapt´es.
2.3.1
Principe physique de la r´
esonance magn´
etique nucl´
eaire (RMN)
Le ph´enom`ene de r´esonance magn´etique nucl´eaire repose sur les propri´et´es magn´etiques des noyaux des atomes. Un noyau ayant un nombre impair de protons poss`ede en effet un moment magn´etique, appel´e spin nucl´eaire (Otto Stern et Walter Gerlach, 1922). En l’absence de champ magn´etique ext´erieur, ces spins sont orient´es de fa¸con al´eatoire dans l’espace (Figure 2.4-a). La r´esultante magn´etique des spins des noyaux est nulle.
Champ polarisant B0 : Plac´es dans un champ magn´etique constant B0 (qui d´efinit arbitrai-rement la direction z de l’espace), les spins s’alignent suivant B0 selon deux orientations : l’une
1
interessante par exemple pour le calcul du volume des structures ou l’´etude du cortex, d’une ´epaisseur de 1.5mm `a 4.5mm.