© Clément Fage, 2019
Étude de la résistance des virus influenza B
contemporains aux inhibiteurs de la neuraminidase et
son impact sur le fitness viral
Thèse
Clément Fage
Doctorat en microbiologie-immunologie
Philosophiæ doctor (Ph. D.)
I
Résumé :
Les virus influenza ont toujours eu un impact considérable sur l’humanité. Les virus influenza B (IB) ont longtemps été négligés et sous-estimés par rapport aux virus influenza A (IA). Ils ne représentent que 10 à 20% des infections annuelles par les virus influenza, mais peuvent devenir majoritaire certaines années et entrainer des symptômes similaires à ceux des virus IA. Les inhibiteurs de la neuraminidase (INA) sont les principaux traitements disponibles contre les virus influenza. Cependant, à la suite de mutations, certains virus influenza ont développé des mécanismes de résistance limitant dangereusement les options thérapeutiques. Actuellement, très peu de souches résistantes sont isolées en clinique car le fitness de ces virus résistants, c’est-à-dire leur capacité à se répliquer et se transmettre, est altéré. Or, il a été montré que des virus IA peuvent améliorer leur fitness et propager le phénotype de résistance. L’objectif de ce projet de thèse est de mieux comprendre la résistance des virus influenza B et son impact sur le fitness viral afin de mieux prévenir l’émergence de souches résistantes. Nous avons, dans un premier temps, étudié la résistance des virus influenza A et B au peramivir, le plus récent des INA. Nous avons confirmé que cette molécule est hautement active contre les souches saisonnières A et B. De plus, elle conserve une activité antivirale in vitro contre certaines souches virales ayant une sensibilité réduite ou hautement réduite contre d’autres INA (l’oseltamivir et le zanamivir). Dans un second temps, nous avons étudié le fitness de virus influenza B contemporains résistants in vitro et chez la souris. Nous avons montré que certains virus influenza B, ayant un phénotype de résistance croisée, peuvent maintenir un fitness similaire à celui du virus sauvage in vitro et in vivo. De plus, nous avons pu décrire et analyser un nouveau mécanisme de résistance chez les virus influenza B observé chez un cas clinique. Ces résultats soulignent la dangerosité de notre dépendance aux INA et nous encouragent à faire évoluer les stratégies thérapeutiques.
II
Sommaire
Résumé : ... I Sommaire ... II Liste des tableaux : ... VI Liste des figures : ... VII Liste des abbréviations :... IX Remerciements ... XI Avant-propos ... XIII
Introduction ... 1
Section I : Les virus influenza ... 1
1. Historique ... 1
2. Classification des virus influenza et leurs réservoirs naturels ... 2
2.1. Alphainfluenzavirus (influenza A) ... 3
2.2. Betainfluenzavirus (influenza B) ... 3
2.3. Gammainfluenzavirus (influenza C) ... 3
2.4. Deltainfluenzavirus (influenza D) ... 4
3. Les virus influenza A et B, structure et morphologies ... 5
3.1. Structure des particules virales ... 5
3.2. Morphologies des particules virales ... 6
3.3. Structure et organisation du génome viral ... 7
3.3.1. Influenza A ... 8
3.3.2. Influenza B ... 10
4. Les protéines virales et leurs fonctions... 11
4.1. Les glycoprotéines de surface ... 11
4.1.1. Hémagglutinine (HA) ... 11
a- Mécanismed’attachement ... 12
b- Rôle de HA dans le spectre d’hôte ... 15
c- Clivage de HA et pathogenèse ... 15
4.1.2. Neuraminidase (NA) ... 16
a- Structure du site actif ... 19
b- Fonction de la NA dans le cycle viral ... 20
III
4.2.1. M1 ... 21
4.2.2. M2/BM2 ... 21
4.3. Les protéines virales composant le complexe RNP ... 22
4.4. Les protéines non structurales NS1 et NEP ... 23
4.5. Protéines accessoires ... 24
5. Cycle de réplication virale ... 25
5.1. Fixation et internalisation ... 25
5.2. Échappement de l’endosome ... 25
5.3. Transcription et réplication des ARN viraux dans le noyau ... 26
5.4. Assemblage et bourgeonnement ... 26
6. Mécanismes de l’évolution virale ... 27
6.1. Mutations ponctuelles et formation de quasi-espèces ... 27
6.2. Réassortiment génétique ... 28
6.3. Évolution différente entre les IA et IB ... 29
7. Variation antigénique ... 29
7.1. Dérive antigénique (antigenic drift) ... 30
7.2. Cassure antigénique (antigenic shift) ... 31
Section II : Aspects immunologiques, cliniques et épidémiologiques des virus influenza 33 1. Réponse immunitaire dirigée contre les virus influenza ... 33
1.1. Réponse immunitaire innée ... 33
1.1.1. Détection virale et induction des interférons ... 33
1.1.2. Mise en place des mécanismes intracellulaires antiviraux ... 35
1.2. Réponse immunitaire adaptative ... 36
1.2.1. Réponse humorale ... 36
1.2.2. Réponse cytotoxique ... 37
2. Tableau clinique et épidémiologie ... 37
2.1. Mode de transmission interhumaine des virus influenza ... 37
2.2. Manifestations cliniques ... 38
2.3. Pathogenèse ... 39
2.4. Impact des virus Influenza sur l’Homme ... 40
2.4.1. Impact des pandémies et exemple de la souche H1N1 de 2009 ... 40
2.4.2. Impact des épidémies saisonnières ... 43
IV
a- Zoonoses aviaires ... 44
b- Zoonoses porcines ... 46
2.5. Diagnostic en laboratoire ... 46
Section III : Traitement et contrôle des infections ... 49
1. Les vaccins, première ligne de défense ... 49
1.1. Les vaccins inactivés ... 49
1.2. Vaccins vivants atténués... 50
1.3. Vaccin universel ... 51
2. Les antiviraux, deuxième ligne de défense ... 52
2.1. Les adamantanes ... 53
2.1.1. Indication clinique ... 53
2.1.2. Mécanisme d’action ... 53
2.1.3. Mécanisme de résistance ... 53
2.2. Les inhibiteurs de la neuraminidase ... 55
2.2.1. Indication clinique ... 55
2.2.2. Mode d’action des INA ... 58
2.2.3. Mécanisme de résistance ... 62
2.2.4. Impact de la résistance sur le fitness viral ... 67
2.2.5. État de la résistance dans le monde ... 73
2.3. Les inhibiteurs de la polymérase virale ... 76
2.3.1. Favipiravir ... 76
2.3.2. Baloxavir-marboxil ... 77
2.4. Combinaison thérapeutique ... 78
2.4.1. Association de molécules antivirales ... 78
2.4.2. Association molécules antivirales / immunomodulateurs ... 78
Chapitre I : Hypothèses et objectifs ... 80
1. Hypothèses ... 80
2. Objectifs ... 81
Chapitre II : Caractérisation de la sensibilité au peramivir de virus influenza A et B sélectionnés avec plusieurs inhibiteurs de la neuraminidase. ... 82
1. Résumé ... 82
V
3. Contribution des auteurs ... 84
4. Article 1 ... 85
Chapitre III : Caractérisation in vitro et in vivo de la sensibilité, du fitness et de la virulence de virus influenza B contemporains porteurs de mutations de résistance croisée aux inhibiteurs de la neuraminidase. ... 102
1. Résumé ... 102
2. Abstract ... 103
3. Contribution des auteurs ... 104
4. Article 2 ... 105
Chapitre IV : Influenza B avec une sensibilité réduite détecté dans un isolat clinique, Canada. ... 127
1. Résumé ... 127
2. Abstract ... 128
3. Contribution des auteurs ... 129
4. Article 3 ... 130
Chapitre V : Discussion ... 139
1. Création d’outils et adaptation des techniques pour l’étude de la sensibilité et du fitness des IB ... 140
2. Étude de la sensibilité de souches influenza aux INA ... 141
3. Recherche de mutations permissives/compensatrices ... 144
4. Étude du fitness des souches IB contemporaines ... 145
5. Étude de nouveaux mécanismes de résistance chez les IB ... 148
Conclusions et perspectives ... 150
Bibliographie ... 152
Annexe A : Substitutions en acide aminés dans la neuraminidase des virus influenza associées à une réduction de la sensibilité aux INA. ... 186
VI
Liste des tableaux :
Tableau 1 : Fonctions connues des protéines accessoires codées par les virus influenza A et
B ... 24
Tableau 2 : Principales protéines effectrices de la résistance antivirale médiée par les
interférons chez l’Homme ... 35
Tableau 3 : Principaux signes cliniques observés au cours de la grippe et leurs valeurs
diagnostiques. ... 39
Tableau 4 : Résumé des différents tests diagnostiques approuvés par la FDA, leurs
avantages et leurs inconvénients... 48
Tableau 5 : Dosages et durée recommandés par la FDA pour le traitement ou la
VII
Liste des figures :
Figure 1 : Spectre d’hôte des virus influenza A, B, C et D ... 4
Figure 2 : Structure des virus influenza A (gauche) et B (droite) sous leur forme sphérique. ... 5
Figure 3 : Observation des formes sphérique et bacilliforme des virus influenza ... 6
Figure 4 : Observation de la forme filamenteuse des virus influenza ... 7
Figure 5 : Structure d’un segment génomique viral et de son promoteur ... 8
Figure 6 : Organisation des segments génomiques des IA ... 9
Figure 7 : Organisation des segments génomiques des IB ... 10
Figure 8 : Structure tridimensionnelle de HA des virus influenza A et B ... 12
Figure 9 : Structure et interaction du RBS des virus influenza avec un récepteur cellulaire humain ... 13
Figure 10 : Les différentes formes d’acides sialiques pouvant se lier à HA ... 14
Figure 11 : Les différents modes de clivage et d’activation de HA0 des virus IA et IB ... 16
Figure 12 : Vue du dessus de la structure de la neuraminidase N9 sous forme tétramérique d’un virus influenza A ... 17
Figure 13 : Évolution et phylogénie de la neuraminidase des virus influenza A et B par analyse de séquences. ... 18
Figure 14 : Structure tridimensionnelle du site actif de la neuraminidase N9 complexé à un acide sialique. ... 19
Figure 15 : Cycle de réplication des virus influenza ... 25
Figure 16 : Illustration des mécanismes de dérive ou de cassure antigénique ... 30
Figure 17 : Historique de l’émergence des pandémies grippales depuis le début du XXe siècle. ... 32
Figure 18 : Reconnaissance des virus influenza et induction de la réponse IFN-I et III entrainant la mise en place de mécanismes antiviraux ... 34
Figure 19 : Émergence de la souche pandémique H1N1 de 2009 suite à plusieurs réassortiments génétiques ... 42
Figure 20 : Exemple d’hémagglutinine chimérique cH8/1 pour l’élaboration d’un vaccin universel ... 52
Figure 21 : Fixation d’une molécule de rimantadine (A) et d’amantadine (B) au niveau du canal ionique de la protéine virale M2 ... 54
Figure 22 : Structure chimique des inhibiteurs de la neuraminidase ... 59
VIII
Figure 24 : Modélisation du mécanisme moléculaire de la résistance induite par la
substitution H274Y chez les virus H1N1 ... 64
Figure 25 : Positionnement des résidus E-105 et G-141 sur deux monomères de la
neuraminidase d’un virus IB... 65
Figure 26 : Réseau d’interaction entre les résidus I-427, G401, S-400 et R-368 chez la
neuraminidase N1. ... 65
Figure 27: Critères définissant la susceptibilité des souches influenza A et B pour les
inhibiteurs de la neuraminidase. ... 67
Figure 28 : Production de virus influenza recombinants par génétique inverse avec un
système de 8 ou 12 plasmides ... 69
Figure 29 : Schéma représentant la balance HA/NA ... 71 Figure 30 : Évolution de l’émergence de souches résistantes à l’oseltamivir de 2012 à 2017
dans le monde ... 73
Figure 31 : Évolution de l’émergence de souches résistantes au zanamivir de 2012-2017
dans le monde. ... 74
Figure 32 : Évolution de l’émergence de souches résistantes au peramivir de 2012-2017
dans le monde ... 75
Figure 33 : Évolution de l’émergence de souches résistantes au laninamivir de 2012-2017
IX
Liste des abbréviations :
a.a : acide aminé
A(H1N1)pdm09 : virus H1N1 pandémique de 2009
ADCC : « Antibody-dependent cell-mediated cytotoxicity » AIV : influenza A aviaire
ARNdb : acide ribonucléique double brins ARNm : acide ribonucléique messager ARNsb : acide ribonucléique simple brin ARNv : acide ribonucléique viral
BCR : récepteur cellulaire des lymphocytes B C-term : extrémité carboxy-terminale d’une protéine CDC : « Center Disease Control and prevention » CI50 : concentration inhibitrice médiane
CTL : lymphocyte T cytotoxique
CMH-I : complexe majeur d’histo-incompatibilité de type I Da : Dalton
DANA : 2,3-didehydro analogue de l’acide N-acetyl-neuraminidique DCs : Cellules dendritiques
ddPCR : « Droplets Digital PCR » EC50 : concentration effectrice médiane FDA : « Food and Drugs Administration » HA : Hémagglutinine
HEF :
HEK-293T : cellule rénale embryonnaire humaine – 293T HPAI : influenza A aviaire hautement pathogène
IA : influenza A IB : influenza B IC : influenza C ID : influenza D Il : interleukine IFN : interféron Ig : immunoglobuline
INA : inhibiteur(s) de le neuraminidase ISG : « Interferon stimulated gene »
LAIV : vaccin contenant une souche influenza vivante atténuée LB : lymphocyte B
LRT : partie basse de l’arbre respiratoire LT : lymphocyte T
MDCK : « Madin-Darby Canine Kidney cells » mg : milligramme
ml : millilitre
N-term : extrémité aminoterminale d’une protéine NA : neuraminidase
NES : séquence exportatrice du noyau ng : nanogramme
NHBE : cellules épithéliales bronchiques humaine NLS : séquence de localisation nucléaire
NK : cellule « natural Killer » ORF : cadre de lecture
X
pA : séquence poly-A
PAMPs : signal associé aux pathogénes pfu : plage de lyse
PIPS : « Phylogenetic Inference of Protein Stability » prCMV : promoteur du cytomégalovirus
RBS : site de liaison de l’hémagglutinine
RIDT : test rapide de dépistage des virus influenza RLR : « RIG-Like-Receptors »
RNP : complexe ribonucléoprotéique viral
RT-PCR : « Reverse Transcriptase – Polymerase Chain Reaction » Rz : ribozyme
Sial-2,3 : acide sialique -2,3 Sial-2,6 : acide sialique -2,6
snRNA : petit acide ribonucléique nucléaire snoRNA : petit acide ribonucléique nucléolaire TCR : récepteur cellulaire des lymphocytes T Thf : lymphocyte T folliculaire
TLR : « Toll Like Receptors » TNF : « Tumor Necrosis Factor »
tPolI : séquence de terminaison de la polymérase 1 URT : partie haute de l’arbre respiratoire
OMS : Organisation Mondiale de la Santé WT : sauvage
XI
Remerciements
Ce doctorat a été long et difficile, mais formateur. Il m’a appris à penser, à ouvrir mon esprit et surtout que pour chaque problème, il y’a une solution. Je tiens à remercier toutes les personnes que j’ai pu croiser durant cette épreuve et que je n’aurai pas citées (vous êtes si nombreux). Je remercie également les docteur(e)s Mariette Ducatez, Caroline Duchaine et Gary Kobinger du temps qu’ils m’ont accordé pour la lecture de ce travail.
Je tiens à remercier tout particulièrement Guy de m’avoir offert l’opportunité de travailler dans son laboratoire et permis de concrétiser un rêve. Ce travail, je le dois aussi à Yacine qui, chaque jour, m’orientait dans le brouillard qu’est cette épreuve du doctorat par sa patience, sa ténacité, nos « brainstorming » et ses blagues. Merci à lui d’avoir été un mentor.
Un doctorat sans travail d’équipe, ça n’existe pas. J’ai eu la chance d’en avoir une merveilleuse ; merci aux membres de l’équipe GB : Andrés, Émilien, Chantal, Coraline, Daniela, Jocelyn, Julia, Julie, Mariana, Karima, Liva, Christine, Eve, Marie-Hélène, Martin, Miguel, Nathalie, Olus, Rafik, Véronique, William, Xavier, Zeineb pour leurs encouragements, leur aide et surtout de m’avoir supporté durant plus de 4 ans pour certains. Je remercie également Minnie, Didou et Chuck d’avoir permis un flot sans fin de plaisanteries qui ont bien embêté Julie et Sophia.
Ce doctorat, je le dois également à mes anciens collègues des laboratoires de Tours et de Toulouse qui m’ont formé et aidé dans mes objectifs.
Un doctorat ou une vie universitaire sans amis, c’est impossible. Ils ont été là dans les hauts et les bas, la joie et la tristesse. Merci à mes camarades de galère pour leurs soutiens : Anaïs, Bruno, Carlos, Flora, Jessica, Joan (et sa sœur), Joey, Julia, Julien, Karima, Kéké, LoLo Gil, Malika et M.C. Merci également à mes amis intemporels de France, qui même avec la distance, ont toujours eu une pensée (ou une moquerie) pour me remonter le moral
XII
dans les froids (trop froid) hivers québécois. Merci à Arthur, Claire, Fred, Hélène, Mychou, Tristan et PV et tant d’autres d’avoir partagé cette aventure avec moi.
La séparation familiale, la distance, le froid, les échecs, la solitude et bien d’autres paramètres ont fait que ce projet de thèse n’a pas été simple pour moi. Mais depuis qu’elle est entrée dans ma vie, Sophia a facilité beaucoup de choses. Merci de m’avoir sorti la tête de l’eau quand je me noyais dans la masse de travail, d’avoir été là quand j’avais le blues, mais aussi d’avoir partagé des moments inoubliables et qui continueront bien après cette thèse.
Enfin, merci à ma famille. Merci à mes parents et à mon frère qui m’ont soutenu bien plus qu’ils ne l’auraient dû dans ces années. Merci à eux d’avoir fait de moi ce que je suis maintenant, d’avoir été patient avec moi et de m’avoir soutenu contre vents et marées. Merci également à mes grands-parents qui, malheureusement, n’ont pas pu voir leur petit-fils devenir docteur.
XIII
Avant-propos
Dans ce manuscrit, je vais commencer par vous présenter une revue de la littérature sur les virus influenza axée sur la microbiologie, l’immunologie, l’épidémiologie et les aspects cliniques (section I et II de l’introduction). Puis, dans un second temps, je vous présenterai les principales connaissances sur la sensibilité des virus influenza aux inhibiteurs de la neuraminidase et leur fitness (section III de l’introduction). Les résultats de ce projet de thèse sont présentés sous la forme de trois articles (chapitre IV à VI) publiés dans des revues internationales et consultables sur PubMed. Vous pourrez trouver un résumé ainsi qu’un avant-propos spécifique pour chaque article en début de chapitre.
Durant ce projet de thèse, j’ai également travaillé sur un projet secondaire ayant pour objectif de déterminer l’efficacité d’une combinaison entre l’oseltamivir et une molécule immunomodulatrice. Ce projet s’éloignant quelque peu de mon objectif initial d’étudier la résistance et la fitness des virus influenza B, il n’a donc pas été inclus dans ce manuscrit, mais il me semblait important de le mentionner.
« The combination of oseltamivir with azithromycin does not show additional benefits over oseltamivir monotherapy in mice infected with influenza A(H1N1)pdm2009 virus »
Fage C, Pizzorno A, Rhéaume C, Abed Y, Boivin G.
Publié dans la revue « Journal of Medical Virology », 2017 Dec;89(12):2239-2243. DOI: 10.1002/jmv.24911
1
Introduction
Section I : Les virus influenza
1. Historique
uand on parle de la grippe, le monde se souvient du fléau qu’a été la grippe espagnole de 1918-1919 qui a fait plus de morts que la 1ère guerre mondiale. Néanmoins, cette pathologie infectieuse était déjà parmi nous depuis l’Antiquité. En 2400 avant J.C, Hippocrate avait décrit une maladie nommée la toux de Périnthe, dont les signes cliniques ressemblaient à ceux d’une grippe moderne. Son apparition serait due selon lui au vent frais du nord durant l’hiver [1, 2]. Cette description est considérée comme la première observation d’une épidémie grippale chez l’Homme. Toutefois, il est difficile d’en avoir la certitude du fait que la grippe partage également un certain nombre de symptômes avec d’autres maladies comme la diphtérie, les pneumonies bactériennes ou encore la typhoïde. Les premières descriptions détaillées de pandémie grippale, faisant consensus chez les spécialistes, ont été faites en 1580. Elle a débuté en Asie durant l’été puis s’est disséminée en Asie mineure, Afrique du nord puis en Europe ou elle a sévi durant 6 mois faisant environs 8000 morts à Rome et décimant certaines villes espagnoles [3]. Durant le XXe siècle, plusieurs pandémies ont été répertoriées : en 1918 avec la terrible « Grippe espagnole » (H1N1) qui aura tué entre 50 et 100 millions de personnes à travers le monde selon des estimations actuelles [4]. En 1957 avec la souche asiatique H2N2, en 1968 avec la souche H3N2 apparaissant à Hong Kong et finalement, la première pandémie du second millénaire commença en 2009 par l’apparition d’un virus H1N1 d’origine porcine au Mexique. Bien que créant une grande inquiétude dans la communauté scientifique, la pandémie de 2009 eut une gravité moindre. Néanmoins, en un an elle causa 12 000 morts et infecta entre 43 et 89 millions de personnes rien qu’aux États-Unis (É.-U) [5].
Il aura fallu attendre de nombreuses découvertes médicales et technologiques pour qu’en 1933, Wilson Smith, Sir Christopher Andrewes et Sir Patrick Laidlaw puissent enfin
2
isoler d’un patient et caractériser pour la première fois le virus influenza humain de type A, agent causal des épidémies et pandémies de grippe [6]. Puis en 1940, Thomas Francis Jr. fût le premier à isoler d’un jeune garçon, nommé Lee, le virus influenza de type B qu’il baptisa B/Lee/1940. Il remarqua que les sera de lapins infectés par divers virus de type A ne protégeaient pas les lapins infectés avec le virus B/Lee/1940 et en conclu donc qu’il y a bien deux types différents de virus [7]. Grâce à ces découvertes et de nombreuses autres recherches, les scientifiques ont pu mettre au point les premiers vaccins (1945) et les premiers traitements antiviraux (1966) afin de contrôler les infections grippales.
Malgré l’avancée importante de nos connaissances sur les virus influenza, ils représentent toujours un coût humain important. On estime que chaque année, dans le monde, un milliard de personnes sont infectées par ces virus causant ainsi 3 à 5 millions de cas d’infection sévère et 650 000 morts [8]. En plus du coût humain vient s’ajouter un coût économique non négligeable. Une étude a estimé qu’en moyenne par saison, les virus influenza couteraient 87 milliards de dollars aux É.-U. si l’on additionne les frais médicaux divers et jours d’absentéisme [8].
2. Classification des virus influenza et leurs réservoirs naturels
Les virus influenza font partie de la famille des Orthomyxoviridae (du grec ortho, droit ; myxo, mucus) regroupant 7 genres, Alpha, -Béta, -Gamma et Deltainfluenzavirus ainsi que les Isavirus, Thogotovirus et Quaranjavirus. Les virus influenza de type A, B et C sont les agents étiologiques des grippes humaines et sont les seuls Orthomyxovirus pathogènes de l’Homme [9-11]. Dans la nomenclature actuelle, on nomme les virus en fonction de leur type, du lieu où la souche a été isolée, du numéro de souche et de l’année où elle a été isolée. Par convention, l’origine humaine d’un isolat n’est pas précisée dans la nomenclature (ex: B/Yamagata/16/1988) [12].
3
2.1. Alphainfluenzavirus (influenza A)
Les virus influenza A (IA) composent l’unique espèce du genre Alphainfluenzavirus. Ils ont pour réservoir naturel les oiseaux palmipèdes, mais peuvent aussi infecter l’Homme et un large spectre de mammifères incluant des animaux domestiques (chien, chat, chevaux, porc) (figure 1) [13]. Les virus influenza A possèdent des glycoprotéines de surface, hémagglutinine (HA) et neuraminidase (NA), ayant des propriétés antigéniques différentes et pouvant être sous-typées. Dans la nomenclature, on indique alors les sous-types de HA et NA entre deux crochets (ex: A/Chicken/Vietnam/C58/14[H5N1]) [12]. Actuellement, il a été dénombré 18 sous-types de HA et 11 sous-types de NA ce qui mathématiquement fait 198 combinaisons possibles, mais toutes n’ont pas été observées dans la nature [14].
2.2. Betainfluenzavirus (influenza B)
Les virus influenza B (IB) composent l’unique espèce du genre Betainfluenzavirus. Ils ont pour principal réservoir l’Homme, néanmoins il a été observé qu’ils peuvent naturellement infecter à de rares occasions plusieurs espèces de mammifères comme les porcs domestiques, les phoques, les chiens et chevaux (figure 1) [15, 16]. Contrairement aux virus de type A, les IB ne peuvent pas être sous-typés par leurs HA ou leurs NA. Toutefois, depuis 1983 on peut distinguer deux lignées génétiquement et antigéniquement différentes représentées par les souches B/Victoria/2/87 et B/Yamagata/16/88 [17, 18].
2.3. Gammainfluenzavirus (influenza C)
Les virus influenza C (IC) composent l’unique espèce du genre Gammainfluenzavirus. Ils ont été isolés en 1947 de patients présentant des symptômes grippaux, mais dont les virus isolés n’étaient pas sensibles aux sera dirigés contre IA et IB [19]. Les IC ont pour réservoir l’Homme, mais peuvent aussi infecter le porc, le dromadaire et le chien (figure 1) [16, 20]. Contrairement aux IA et IB, les IC ne possèdent pas de HA mais une l’Hemaglutinin Esterase Function (HEF) et ne peuvent donc pas être sous-typés. Néanmoins, par séquençage de cette HEF, on peut distinguer 6 lignées différentes représentées par les virus C/Taylor/47, C/Kanagawa/1/76, C/Yamagata/26/81, C/Aichi/1/81, C/Sao Paulo/378/82, and C/Mississippi/80 [21].
4
2.4. Deltainfluenzavirus (influenza D)
Les virus influenza D (ID) composent l’unique espèce du genre Deltaainfluenzavirus. Ils ont été isolés en 2011 aux É.-U. d’un porc présentant des symptômes grippaux [10]. Les ID ont d’abord été considérés comme des virus influenza C-like jusqu’à ce que des études montrent que les séquences en acides aminés de ces virus ne possèdent globalement que 50% d’identité avec les IC. De plus, les sera dirigés contre les IC ne réagissent pas contre ces virus IC-like ce qui fait d’eux un genre de virus à part entière [22]. Partant de ce constat, le comité international de la taxonomie virale (ICTV) a décidé de les classer dans un genre à part en 2017 [11]. Ces virus ont pour réservoir naturel la vache, mais peuvent infecter plusieurs mammifères, dont l’Homme, comme le porc, la chèvre ou encore le cheval [16]. Des études sérologiques ont montré qu’une partie de la population humaine (les pourcentages diffèrent selon les études) possède des anticorps dirigés contre les ID [10, 23]. Toutefois, il est à noter qu’aucune détection directe n’a pour le moment été faite chez l’Homme [24].
5
3. Les virus influenza A et B, structure et morphologies
Ce projet thèse se concentre essentiellement sur l’étude des virus influenza de type A et B comme nous le verrons par la suite. Dans l’ensemble de ce manuscrit, nous ne traiterons que de ces deux types qui représentent la majorité des cas d’infections cliniques de l’Homme par le genre influenzavirus.
3.1. Structure des particules virales
Les virus influenza A et B sont enveloppés, c’est-à-dire qu’ils possèdent une membrane lipidique issue de la cellule hôte dans laquelle on retrouve principalement les glycoprotéines de surface HA, NA ainsi que la protéine de matrice 2 (M2 pour les IA et BM2 pour IB, figure 2). En plus de ces 3 protéines, les IB possèdent également une protéine en plus que les IA, la protéine NB. Cette membrane recouvre une capside composée de la protéine de la matrice 1 (M1). La capside renferme le génome viral qui est composé de 8 segments d’ARN de polarité négative, simples brins complexés aux nucléoprotéines (NP). Chaque segment génomique est associé à un complexe polymérase composé des protéines PA (Polymerase Acid), PB1 (Polymerase Basic 1) et PB2 (Polymerase Basic 2) le tout formant le complexe Ribo-Nucléo-Protéique (RNP) [12]. La Nuclear Export Protein (NEP) ainsi que la protéine Non structurale 1 (NS1) ont longtemps été considérés comme des protéines non structurales jusqu’à ce que des études aient démontré leur présence dans les virions [25, 26]. En général, les virus influenza sont composés d’environ 1% d’ARN, 5 à 8% de sucres, 20% de lipides et 70% de protéines. Toutefois ces chiffres sont susceptibles de varier avec l’évolution des techniques et surtout avec le fait que les IA comme les IB ont une structure pléomorphique [12].
Figure 2 : Structure des virus influenza A (gauche) et B (droite) sous leur forme sphérique. Tiré de
6
3.2. Morphologies des particules virales
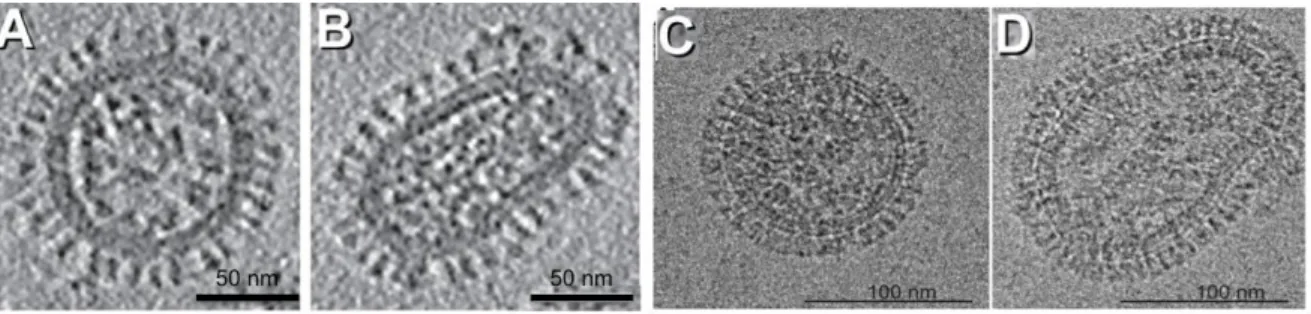
La forme des virus IA et IB est un caractère déterminé principalement par la protéine M1. Le rôle d’autres protéines comme la NA ou HA en association avec M1 a également été démontré [28]. Les particules peuvent être sphériques (la forme majoritaire 65-75%), bacilliformes ou filamenteuses. Des études ont montré que des mutations sur le gène codant la protéine M1 peuvent entrainer des modifications de morphologie observable grâce à la cryomicroscopie [29-31]. Les IA et IB sphériques ont un diamètre de 100 à 130 nm de diamètre en moyenne (figure 3A et 3C). Les virions ayant morphologie bacilliforme ont une forme allongée d’une longueur variable n’excédant pas 250 nm (figure 3B et 3D) [31].
Figure 3 : Observation des formes sphérique et bacilliforme des virus influenza. Les virus
A/H3N2/X-31 (A et B) et B/Lee/40 (C et D) sont observés par cryomicroscopie électronique. Photos Tirées de [32, 33].
La morphologie filamenteuse a également été observée pour les IA et IB [31]. Cette structure, dont la taille varie de 250 nm à 300 nm, peut être suffisamment grande pour qu’elle puisse être observée par microscopie confocale (figure 4A). La cryomicroscopie électronique montre une structure étendue recouverte de glycoprotéine HA tandis que la NA forme un groupement au pôle proximal à l’opposé du pôle contenant le génome viral (figure 4B) [28, 31]. Le rôle des différentes morphologies dans la pathogenèse reste encore mal connu et est toujours sujet à débat. Toutefois, il a été montré que la forme sphérique confère un avantage sélectif au virus influenza lors de leur culture sur cellules eucaryotes ou sur œufs embryonnés. Inversement, la forme filamenteuse est sélectionnée au dépens des autres lors d’infections in vivo [34].
7
Figure 4 : Observation de la forme filamenteuse des virus influenza. Observation
par microscopie confocale (A) ainsi que par cryomicroscopie électronique (B) de particules filamenteuses virales de virus A/Udorn/72(H3N2). Tiré de [35].
3.3. Structure et organisation du génome viral
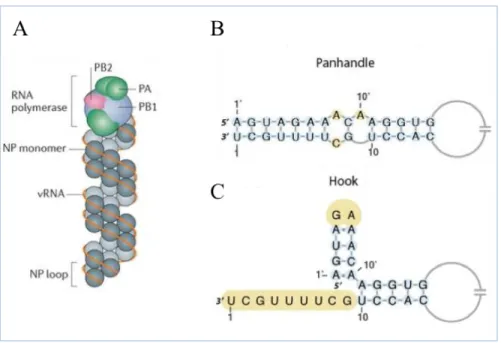
Le génome des IA et IB est composé de 8 segments d’ARN qui ont été classés en fonction de leur taille respective du plus grand (segment 1) au plus petit (segment 8) [12]. Ils ont une polarité dite négative, car ils ne codent pas directement pour une protéine, mais servent de support à la transcription d’ARN messagers (ARNm) [36]. Les extrémités 3’ et 5’ non codantes de ces segments sont composées d’une partie hautement conservée, spécifique du type viral. Elles permettent le repliement de l’ARN sur lui-même afin de former le promoteur interagissant avec la polymérase virale ainsi que le signal d’empaquetage du segment dans le nouveau virion (figure 5A) [37]. La structure de ce promoteur est encore matière à débat, mais il a été démontré par cristallographie que ce dernier peut prendre plusieurs formes. Lorsque l’ARN n’est pas complexé à la polymérase virale alors le promoteur aurait une structure dite « panhandle » (manche, figure 5B) et une fois associé à celle-ci il aurait une structure dite « hook » (crochet, figure 5C) [38]. De manière générale, l’organisation génomique des IA et IB est similaire avec néanmoins quelques différences notables qui seront présentées par la suite (figure 6 et 7).
A
8
Figure 5 : Structure d’un segment génomique viral et de son promoteur. Structure de la
particule vRNP composée de la polymérase virale (complexe PB2, PB1 et PA), de monomère NP ainsi que du génome ARN simple brin de polarité négative (A). Structures proposées du promoteur viral lorsque le génome est libre (B) ou associé à la polymérase virale (C). Les bases surlignées montrent le mésappariement ou les bases libres. Tiré de [37, 39].
3.3.1. Influenza A
Les IA possèdent un génome d’une longueur totale d’environ 13 600 nucléotides codant pour 17 protéines. Ces virus ont la particularité de pouvoir augmenter leur capacité de codage grâce à diverses stratégies comme l’épissage, l’utilisation de cadre de lecture alternatif ou alors, plus récemment découvert, le saut de cadre de lecture (figure 6) [12].
L’épissage alternatif est un mécanisme post-transcriptionnel qui permet, par coupure et ligation, la production d’un nouvel ARNm à partir d’un premier ARNm. Les virus influenza ont détourné ce procédé employé par les cellules eucaryotes afin de pouvoir produire à partir de leur segment génomique une plus grande diversité d’ARNm. Ainsi, par ce procédé, le segment 1 transcrit un ARNm codant pour la protéine PB2 et suite à l’épissage de cet ARNm, PB2-S1 [40]. Le segment 7 code pour une protéine M1, mais également pour M2 et M42 par épissage alternatif de l’ARNm de M1 [41]. Le segment 8 transcrit un ARNm codant pour NS1 et suite à l’épissage de cet ARNm, NS2 [42].
9
Une autre stratégie très employée par les virus influenza est l’utilisation de codon alternatif d’initiation de la traduction et le changement de cadre de lecture. Un ARNm peut être constitué de plusieurs codons d’initiation ou d’arrêt de la traduction. Un ribosome pourra alors débuter la traduction en plusieurs endroits de l’ARNm produisant ainsi différentes protéines à partir du même ARNm. Ainsi à partir de l’ARNm des segments 2 et 3 (PB1 et PA) les IA peuvent produire plusieurs protéines (figure 6) [42]. Il a été récemment montré que le segment 3 code pour une protéine PA-X issue de deux cadres de lecture différents grâce à un saut de cadre de lecture au cours de la traduction [43].
Figure 6 : Organisation des segments génomiques des IA. Le
virus A/Puerto Rico/1934(H1N1 a été pris pour exemple. Les segments ARN sont montrés en polarité positive avec leur Open Reading Frame (ORF, les boites bleu et verte correspondent respectivement au cadre de lecture 1 et 2). La position des codons d’initiation ou d’arrêt de la traduction est indiquée pour chaque ORF par un chiffre noir au-dessus des boites, les épissages des ARN (segments PB2, M et NS) sont indiqués par des lignes en V et le saut de cadre de lecture du segment PA est indiqué par une ligne rouge.
10
3.3.2. Influenza B
Les IB n’ont pas une organisation génomique aussi complexe que celle des IA. Ils possèdent un génome dont la longueur totale est d’environ 14 200 nucléotides et ne codent que pour 11 protéines. Contrairement aux IA, les segments PB2, PB1 et PA ne produisent qu’une protéine chacun et le segment M n’a pas de site d’épissage, mais contient deux cadres de lecture qui se succèdent, codant pour les protéines M1 et BM2 [12]. De plus, le segment NA des IB possède un site alternatif d’initiation de la traduction lui permettant de produire une protéine supplémentaire, NB, que les IA ne possèdent pas [44] (figure 7).
Figure 7 : Organisation des segments génomiques des IB. Le virus B/Brisbane/60/2008 a été pris
11
4. Les protéines virales et leurs fonctions
4.1. Les glycoprotéines de surface
4.1.1. Hémagglutinine (HA)
HA est une glycoprotéine intégrale de type I codée par le segment génomique 4, recouvrant 80 à 90% de la surface du virus et ayant un poids moléculaire d’approximativement 60 kDa dans sa forme non glycosylée. Toutefois, sa taille en acides aminés (a.a) ainsi que le nombre de sites de glycosylation peuvent varier en fonction du sous-type et du type viral [12]. Sa structure homotrimérique a été pour la première fois décrite en 1981 [45]. Les monomères la composant sont liés entre eux de manière non covalente et possèdent une partie carboxy-terminale (C-term) insérée dans la membrane virale et une partie aminoterminale (N-term) globulaire hydrophile projetée à l’extérieur du virus (figure 8A) [12]. Les monomères de HA sont synthétisés par les ribosomes au niveau du réticulum endoplasmique puis, relâchés dans le lumen sous la forme de précurseurs polypeptidiques inactifs HA0. Ces polypeptides vont ensuite subir plusieurs étapes de
maturation post-traductionnelle, de clivage protéolytique, de glycosylation et d’acétylation au niveau de l’appareil de Golgi. Suite à cette maturation, chaque monomère va être composé de 2 sous-unités protéiques HA1 et HA2 reliées entre elles par des ponts disulfures
(figure 8A) [46]. La sous-unité HA1 compose la partie globulaire externe au virus, elle
porte les sites de glycosylation ainsi que le site d’attachement « Receptor-Binding Site » (site de liaison au recepteurs, RBS) et représente le principal déterminant antigénique de HA. La sous-unité HA2 porte le peptide de fusion ainsi que le domaine transmembranaire
ancrant HA dans la bicouche lipidique virale.
Bien qu’évoluant séparément et ne possédant que 25% d’identité dans leur séquence en a.a, les HA des IA et IB ont une structure très proche comme l’ont démontré des études cristallographiques (figure 8B) [47, 48]. Par ailleurs, des études utilisant des virus chimériques IA et IB ont également montré que la HA des IB peut remplacer la HA des IA sans altérer la réplication virale démontrant ainsi une fonction hautement conservée entre ces deux types viraux [12, 49, 50].
12
Figure 8 : Structure tridimensionnelle de HA des virus influenza A et B. Observation de HA
sous forme monomérique et trimérique avec les sous unités HA1 (bleu) et HA2 (violet) ainsi que la localisation du RBS et du peptide de fusion (A). Comparaison de la structure tridimensionnelle du sous domaine d’un monomère de HA des virus influenza A et B (B). Tiré de [47, 51].
a- Mécanisme d’attachement
En 1941, George K. Hirst fut le premier à observer que les virus influenza ont la capacité d’agglutiner les hématies grâce à un élément qui fut alors nommé « hémagglutinine » [52]. Cette observation et ce nom illustrent bien la première fonction de HA qui est de permettre l’attachement des virus influenza à la surface des cellules hôtes. Cette action est rendue possible grâce au site d’attachement viral, RBS, qui va se lier spécifiquement à des récepteurs cellulaires contenant des acides sialiques.
- Structure du RBS des virus influenza
Du fait de sa fonction hautement conservée, les IA et IB ont gardé un site d’attachement structuralement proche. Le RBS est une cavité dont la base est composée par de 4 a.a hautement conservés : Y98, W153, H183 et Y195 (numérotation H3) pour les IA et F95, W158, H191 et 202Y (numérotation B) pour les IB [46, 47]. Comme nous pouvons le
I Influenza B
Influenza A
I I
B A
13
constater sur la figure 9, il est formé par une hélice (190-helix) au sommet ainsi que de 2 boucles (130/140 et 220/240 pour les IA/IB respectivement). Ces structures, ainsi que les a.a qui les composent, vont permettre de définir le mode d’interaction de HA avec son substrat, les acides sialiques. Ainsi, les différences en a.a entre les RBS des IA et IB vont entrainer des différences dans son affinité pour son substrat. Des études cristallographiques ont pu montrer que le RBS des IA a une plus forte affinité pour les acides sialiques humains que le RBS de IB [48]. Ceci est dû au nombre plus important de liaisons hydrogène que peut établir le RBS de IA avec son substrat. Comme le montrent les modèles informatiques de la figure 9, le RBS de H3 peut établir 10 liaisons hydrogène avec un acide sialique humain alors que celui des IB ne peut en faire que 8. Ceci est dû à la phénylalanine en position 95 qui empêche des liaisons de se faire et modifie l’orientation de W158 [48].
Figure 9 : Structure et interaction du RBS des virus influenza avec un récepteur cellulaire humain. La structure des RBS des virus A/Aichi/2/68(H3N2) (A) et B/HongKong/8/73 (B) est
représentée en gris avec les a.a effectuant des liaisons hydrogène (pointillés bleus) entre le RBS et un acide sialique simulant un récepteur humain (vert/rouge). Tiré de [48]
14
- Spécificité de récepteurs
Les cellules eucaryotes sont recouvertes par le glycocalix, une couche composée de glycolipides, glycoprotéines et protéoglycanes. Il existe plusieurs types de glycanes liés à des protéines de surface (N- et O-glycan) de taille variable dont certains sont terminés par un galactose lié à un acide sialique par une liaison -2,3 (sial-2,3) ou -2,6 (sial-2,6) (figure 10) [53]. Les virus influenza, en fonction de la structure du RBS et des sites de glycosylation de HA, peuvent se lier de manière préférentielle au sial-2,3 ou -2,6 [54]. Ainsi, il a été montré que les virus ayant une glycoprotéine de surface H1 se fixe préférentiellement au sial-2,6 mais que des substitutions, E190D et G225D, augmentent leur affinité pour les sial-2,3 [55]. De même, pour des virus ayant une glycoprotéine de surface H3, les résidus 226Q et 228G au niveau de HA favorise la fixation du virus au sial-2,3 alors que des résidus 226L et 228S permettent une meilleure fixation au sial-2,6 [56-58].
Contrairement aux IA, la spécificité de récepteurs des IB reste peu étudiée et est encore matière à débats. En compilant des données de cristallographie issues de différentes études, un modèle a néanmoins été proposé. Le RBS des IB aurait une structure lui permettant de se lier avec la même affinité au sial-2,3 et -2,6. Ainsi les IB ne feraient pas de distinction entre ces deux récepteurs [59, 60]. Néanmoins, un site de glycosylation au niveau des résidus 194/196 du RBS entrainerait un encombrement stérique [61], limitant l’interaction entre l’hélice- 190 et les sial-2,3 [59]. Il a été montré que des substitutions permettant d’éliminer ces sites de glycosylation augmentent grandement l’affinité de HA des IB pour les sial-2,3 tout en conservant une affinité similaire pour les sial-2,6 [61, 62].
Figure 10 : Les différentes formes d’acides sialiques pouvant se lier à HA. L’acide N-acétylneuraminique
est un acide sialique qui peut être relié à un autre sucre, ici un galactose, par une liaison covalente entre les carbones en position 2 et 3 (A, -2,3) ou entre les carbones 2 et 6 (B, -2,6). Tiré de [53].
A
15
b- Rôle de HA dans le spectre d’hôte
Par sa capacité d’interagir avec les sial-2,3 ou -2,6, HA va permettre de définir en partie le spectre d’hôte des virus influenza. En effet, les souches IA d’origine aviaire ont une préférence naturelle pour les sial-2,3 alors que les souches IA humaines ont une préférence pour les sial-2,6 [54]. Or, la distribution de ces récepteurs au niveau tissulaire varie en fonction des hôtes. Les oiseaux expriment essentiellement des sial-2,3 au niveau des cellules épithéliales composant leurs tractus respiratoire et digestif alors que l’Homme exprime majoritairement des sial-2,6 au niveau de son tractus respiratoire (fosses nasales, pharynx, trachée, bronche et alvéole) [63, 64]. Toutefois, il a également été montré qu’au niveau de la partie basse du tractus respiratoire humain, certaines cellules non ciliées à l’intersection entre les bronches et les alvéoles, ainsi que les pneumocytes de type II expriment également des sial-2,3 [64]. Ainsi les virus aviaires, ayant une faible affinité pour les sial-2,6, pourront difficilement infecter l’Homme du fait du manque de récepteurs sial-2,3 dans la partie haute de son arbre respiratoire. Ceci explique pourquoi les infections humaines par des souches aviaires comme A/H5N1 sont rares, mais possible suite à des contacts prolongés avec des animaux infectés [65-67]. Toutefois, ceci n’explique pas pourquoi les IB sont limités à un nombre restreint d’hôtes puisque, s’ils sont libérés de l’encombrement stérique dû au site de glycosylation, ils peuvent se fixer aussi bien au sial-2,3 ou -2,6. Ceci est dû au fait que la HA n’est pas la seule protéine à définir le spectre d’hôte, de nombreuses études ont montré que d’autres protéines virales comme NS1 [68] ou la polymérase virale [69] ou NA sont importantes dans la définition de ce paramètre [60].
c- Clivage de HA et pathogenèse
Le clivage de HA0 par des protéases de l’hôte est essentiel dans le cycle de vie des
virus influenza. Il permet le changement conformationnel indispensable au second rôle de HA, la fusion de l’enveloppe virale avec la membrane endosomale [12]. Trois groupes de protéases sont responsables de ce clivage et leur disponibilité au niveau des tissus de l’hôte va déterminer en grande partie le tropisme tissulaire des virus. Un premier groupe de protéases à sérine ubiquitaire de la famille des subtilisines, les furines, vont cliver HA au niveau de l’appareil de Golgi. Un second groupe de protéase à sérine transmembranaire, TMPRSS2 et HAT, vont cliver HA au niveau de la membrane de la cellule hôte [70]. Le
16
dernier groupe comprend des protéases solubles dans le milieu extracellulaire comme les protéases Clara, plasmine, miniplasmine ou encore des protéases bactériennes [71] (figure 11). Les enzymes des deux derniers groupes ont toutes pour cible un site de clivage situé sur HA0, composé d’une séquence en a.a hautement conservée (R-X-K*R329-R pour les IA
et K-L-L-K-E-R361*G pour les IB, * étant le site de coupure) [72, 73]. Les protéases ubiquitaires « furine » ont pour site de clivage une séquence contenant de multiples a.a basiques (R-R-R-K-K*R329-R) présents uniquement chez certaines souches hautement pathogènes d’IA aviaire (HPAI). Grâce à ce site, les HPAI vont pouvoir causer des infections systémiques chez les oiseaux du fait de la grande biodisponibilité des furines dans différents tissus de l’hôte [12, 54].
Figure 11 : Les différents modes de clivage et d’activation de HA0 des virus IA et IB. Tiré de
[71].
4.1.2. Neuraminidase (NA)
La NA est la deuxième glycoprotéine de surface des virus IA et IB, elle recouvre 20% de la surface virale. C’est une protéine membranaire de type intégrale II codée par le
17
segment génomique 6 et a une taille de 454 à 466 a.a (environs 60 kDa) dépendamment du type viral [12]. Sa structure a été pour la première fois caractérisée par Peter Colman en 1983 [74]. Dans sa forme active, la NA est un homotétramère où les monomères sont reliés entre eux par plusieurs ponts disulfures. Elle est produite au niveau du réticulum endoplasmique où elle va être oligomérisée et glycosylée [75, 76]. Elle possède trois domaines distincts : un ectodomaine, un domaine transmembranaire et une extrémité N-term formant la queue cytoplasmique (figure 12) [77]. L’ectodomaine est constitué d’une tête globulaire où est localisée une cavité centrale correspondant au site actif de l’enzyme lui permettant de cliver les acides sialiques terminaux [78]. La tête globulaire est ensuite suivie d’une queue contenant un domaine transmembranaire permettant d’ancrer l’enzyme dans l’enveloppe virale [78]. De par sa fonction essentielle dans le cycle viral (que nous décrirons bientôt), la structure de la NA est hautement conservée entre les types viraux A et B [12, 79]. Des études ont montré qu’un virus IA chimérique possédant une NA d’un virus IB a une réplication virale non altérée démontrant ainsi une fonction hautement conservée entre ces deux types viraux [50].
Figure 12 : Vue du dessus de la structure de la neuraminidase N9 sous forme tétramérique d’un virus influenza A. Pour chaque monomère, la couleur change du bleu (N-term) au rouge
18
Au cours du temps, suite à des adaptations successives à de nouveaux hôtes et de la pression de sélection qui en résulte, la NA des IA a évolué en 11 sous-types différents. Les analyses phylogénétiques ont montré qu’après avoir divergé en deux types A et B, les gènes de la NA du type A peuvent être classés en deux groupes : Le groupe I comprenant N1, N4, N5 et N8 et le groupe II comprenant N2, N3, N6, N7 et N9 ayant tous les deux un large spectre d’hôtes (figure 13) [80, 81]. Seuls les types B et les sous-types N1 (H1N1 pandémique de 2009 et H1N1 saisonnier avant 2009) et N2 (H3N2 humain/porcin) de type A sont actuellement pathogènes pour l’Homme. Les sous-types N10 et N11 n’appartiennent pas à ces deux groupes car il s’agit de « NA-like ». Elles ont de grandes divergences avec les autres NA dans leurs séquences en a.a mais également dans leurs sites actifs perdant ainsi leur fonction de sialidase [82]. Comme nous pouvons le voir sur la figure 13, les IB n’ont pas la même diversité dans leur NA du fait d’une dynamique évolutive plus faible et d’un spectre d’hôte plus restreint dont nous parlerons plus en détail dans la section I-7.
Figure 13 : Évolution et phylogénie de la neuraminidase des virus influenza A et B par analyse de séquences. L’origine des différents hôtes pour chaque souche est représentée par un code
couleur (vert, humaine ; bleu, porcine ; rouge, aviaire ; violet, équine). L’apparition des différents clades est symbolisée par un carré suivant l’axe horizontal des années. Tiré de [80].
19
a- Structure du site actif
La cavité formant le site actif de chaque monomère est composée d’a.a hautement conversé chez les IA comme les IB. Le site catalytique est composé de huit résidus (R118, D151, R152, R224, E276, R292, R371 et Y406 ; numérotation N2 [83]) qui vont entrer en contact avec le substrat et de onze autres résidus structuraux (E119, R156, W178, S179, D/N198, I222, H274, E277, N294 et E425) qui vont soutenir et stabiliser la conformation du site actif [74, 84]. Toute mutation entrainant une substitution d’un de ces résidus peut entrainer une baisse ou une perte d’activité enzymatique pouvant conduire à une perte de fonction. Ce site actif étant hautement conservé et essentiel pour le virus, il représente donc une cible de choix pour des molécules thérapeutiques comme les inhibiteurs de la NA (INA) que nous développerons dans la section III.
Une autre cavité proche du site actif a récemment été découverte par des études cristallographiques [85]. Cette cavité, nommée « 150-cavity », formée par les boucles 150 et 430 aurait une conformation ouverte pour les virus ayant une NA du groupe I (à l’exception du virus H1N1 pandémique de 2009) et fermée pour les NA du groupe II et la NA des IB [86, 87]. Des études ont montré que cette cavité pourrait être une nouvelle cible pour de potentielles molécules inhibitrices de la NA. Néanmoins, ce sujet reste encore à l’étude du fait d’une efficacité moindre par rapport aux molécules thérapeutiques actuellement commercialisées [88, 89].
Figure 14 : Structure tridimensionnelle du site actif de la neuraminidase N9 complexé à un acide sialique. Les résidus
formant le site catalytique sont colorés en bleu et les résidus soutiens du site actif en violet (numérotation N2). L’acide sialique est coloré en vert. Tiré de [90].
20
b- Fonction de la NA dans le cycle viral
- Échappement du mucus et empêchement de l’agrégation virale
Lors de l’infection, les virus influenza infectent les voies haute et basses de l’arbre respiratoire protégées par une couche de mucus riche en acides sialiques pouvant lier la HA et ainsi séquestrer les particules virales [91, 92]. Le premier rôle de la NA sera donc de permettre aux particules virales de se libérer de ce piège tout en accédant aux cellules hôtes [92]. De plus, l’enveloppe virale étant d’origine cellulaire, celle-ci contient également des glycoprotéines cellulaires et des acides sialiques pouvant lier les HA. Le second rôle de la NA sera de cliver les acides sialiques de l’enveloppe virale afin d’empêcher l’agrégation virale médiée par la HA [93].
- Favoriser l’attachement et l’endocytose du virus
Il a été récemment démontré que la NA, en association avec HA, favorise l’attachement et l’internalisation du virus. Ce mécanisme nommé « rolling » (roulement) permet par un processus d’association/dissociation entre HA et les récepteurs cellulaires le roulage du virus à la surface de la cellule hôte jusqu’à ce que la particule virale soit fixée puis endocytée [94, 95]. En l’absence de ce processus de roulage, l’internalisation virale est réduite de manière significative sans pour autant stopper l’infection [94].
- Favoriser la survie de la cellule hôte
Les protéines virales vont interagir avec les nombreuses protéines cellulaires afin de permettre la production de nouveaux virions [12]. Une étude a montré que la NA des IA interagit avec une protéine chaperonne cellulaire, Hsp90, connue pour stabiliser et protéger ses protéines-cibles de la dégradation par le protéasome ainsi que de favoriser la survie cellulaire [91, 96]. Grâce à cette interaction, la NA va échapper à la dégradation par le protéasome et augmenter la survie cellulaire lors de l’infection favorisant par la même occasion la production de nouveau virus [91].
21
-
Libérer les nouveaux virions lors du bourgeonnement
C’est la fonction principale et la mieux connue de la NA. Les particules virales nouvellement formées au niveau de la membrane cellulaire vont, lors du bourgeonnement, rester ancrées à celle-ci par un lien entre la HA et des récepteurs cellulaires (contenant des Sial) présents à proximité du site de libération. Afin de libérer les nouveaux virions, la fonction de sialidase de la NA est essentielle pour cliver ce lien au niveau des Sial terminaux [12]. Une perte ou une inhibition d’activité sialidase de la NA entraine une baisse significative de la réplication virale suite à l’impossibilité des virions à être libérés [97].
4.2. Les protéines de la matrice
4.2.1. M1
La protéine de la Matrice 1 (M1) est codée par le segment génomique 7 et à un poids moléculaire d’environ 28 kDa [98]. C’est la protéine la plus abondante des virus influenza, elle se situe sous l’enveloppe virale et a la particularité de pouvoir s’auto-oligomériser afin de former la capside virale [12, 31, 99]. Du fait de sa position, elle va pouvoir interagir avec de nombreux composés viraux, ce qui en fait un élément central dans l’assemblage des nouveaux virions et leur bourgeonnement [98]. En effet, il a été montré que M1 peut interagir avec les RNP permettant leur transport vers la membrane cellulaire [100, 101]. De plus, M1 peut également interagir avec les queues cytoplasmiques des glycoprotéines de surface HA et NA [102] ainsi qu’avec M2 (ou BM2 pour les IB) [103] faisant ainsi le lien entre l’enveloppe virale et l’intérieur de la capside. Une inhibition de ces interactions entraine une déficience dans l’assemblage des virus [104, 105].
4.2.2. M2/BM2
La protéine de la Matrice 2 (M2, ou BM2 pour les IB) est également codée par le segment génomique 7. Elle est composée de 97 a.a chez les IA et de 109 a.a pour les IB. C’est une protéine transmembranaire de type intégrale III avec un côté N-term projeté à l’extérieur de l’enveloppe virale et un coté C-term orienté vers l’intérieur du virus [106, 107]. La protéine M2 est un canal à proton sous forme d’un homotétramère ayant un rôle essentiel dans la libération des particules RNP dans le cytoplasme [12]. Lors de
22
l’endocytose des virus influenza, l’acidification de l’endosome va activer l’ouverture du canal et permettre le transfert des protons à l’intérieur de la capside afin d’abaisser le pH. Cela va entrainer la dépolymérisation des protéines M1 entrainant la destruction de la capside [108]. Les protéines M2 et BM2 ont une faible homologie de séquence à l’exception des résidus H37/19 (position pour IA/IB respectivement) et W41/23 (position pour IA/IB respectivement) [109]. Ces a.a hautement conservés sont essentiels pour le contrôle de la conductance des protonsau travers du canal et de leur sélectivité en ions [107]. Le résidu histidine va être responsable de l’activation pH-dépendante du canal tandis que le résidu tryptophane va faire office de barrière aux protons [110, 111].
4.3. Les protéines virales composant le complexe RNP
La structure de l’ARN polymérase ARN-dépendante des virus IA et IB est composée de 3 protéines : PB2, PB1 et PA [38, 112]. Ces protéines une fois produites dans le cytoplasme de la cellule hôte vont migrer vers le noyau et s’associer entre elles afin de constituer une nouvelle polymérase. En s’associant avec un segment génomique, lui-même recouvert de protéine NP, il y aura alors formation du complexe RNP qui aura pour rôle la transcription mais également la réplication des ARN viraux [113].
PB2 est codée par le segment 1 et a un poids moléculaire de 85 kDa. Elle a pour
fonction principale de capter les coiffes 5’ méthylés des ARN nucléaires non codants comme les snRNA («small nuclear RNA ») ou snoRNA (« small nucleolar RNA ») [114]. Une fois l’ARN nucléaire clivé, PB2 va orienter la coiffe dans le site actif de la polymérase afin de pouvoir initier la transcription des ARNm viraux. En plus de cette activité, PB2 pourrait avoir potentiellement une fonction hélicase permettant de séparer les brins d’ARN lors de l’élongation [112].
PB1 est codée par le segment génomique 2, elle a un poids moléculaire de 85 kDa.
Cette protéine est la sous-unité centrale de la polymérase et interagit avec chaque autre protéine composant le complexe RNP. Avec son domaine N-term, PB2 forme le site actif de la polymérase et va donc permettre l’intégration des acides nucléiques durant la transcription ou la réplication des ARN viraux [39].
23
PA est codée par le segment 3 et a un poids moléculaire de 82 kDa. De précédentes
études avaient conféré par erreur à PA le rôle de PB1 dans l’intégration des acides nucléiques. Il a été montré qu’en réalité PA fait partie de la famille des endonucléases PD-(D/E)XK [115]. Elle va se positionner sur l’ARN cellulaire capté par PB2 et le clivé à 15 nucléotides après la coiffe afin de permettre à PB2 de l’insérer la dans le site actif de la polymérase [39].
NP est codée par le segment 5 et a un poids moléculaire de 56 et 61 kDa pour les IA et
IB, respectivement. Elle a une structure trimérique pour les IA et tétramérique pour les IB [116, 117]. Malgré cette différence structurale, NP conserve une fonction similaire chez les IA et IB. Bien que n’ayant aucun rôle direct dans l’activité polymérase, NP n’en reste pas moins un partenaire essentiel dans l’architecture du complexe RNP. En recouvrant et protégeant les huit segments d’ARNv (ARN viraux), elle va jouer un rôle clé dans le transport des RNP et dans la régulation des mécanismes de réplication et de transcription de l’ARNv [118, 119].
4.4. Les protéines non structurales NS1 et NEP
NS1 est codée par le segment 8 et a un poids moléculaire de 25 et 32 kDa pour les IA
et IB, respectivement. Bien que nommée protéine non structurale, une étude a pu démontrer par spectrométrie de masse que NS1 est bien présente en faible quantité dans le virion, probablement intégrée lors du bourgeonnement viral [26]. NS1 sous sa forme active est un homodimère où chaque monomère est composé d’un domaine RNA-binding en N-term et d’un domaine effecteur en C-term pouvant interagir avec un large panel de protéines et d’ARN aussi bien cellulaires que viraux [120, 121]. Ces domaines vont ainsi lui conférer une multitude de fonctions, la principale étant l’inhibition de la réponse interféron de type I (IFN-I). De plus, NS1 est également connu pour inhiber directement la mise en place de mécanismes antiviraux médiés par la réponse IFN-I (mécanismes que nous verrons plus en détail dans la section II) [122].
24
NEP pour « Nuclear Export Protein » (également appelée NS2) a un poids moléculaire
de 14 kDa et est la seconde protéine codée par le segment 8 obtenu après un épissage (voir section I-4) [42]. Comme pour NS1, NEP était considérée comme une protéine non structurale jusqu’à ce que des études aient démontré qu’elle est également présente dans le virion [26]. NEP est présente aussi bien dans le cytoplasme de la cellule hôte que dans son noyau. La présence d’une séquence signal d’exportation nucléaire (NES) dans son domaine N-term va permettre l’exportation des complexes RNP hors du noyau en association avec les protéines virales M1 et NP [119]. De plus, NEP permet l’accumulation d’ARNv dans le noyau ce qui pourrait permettre la régulation de la transition entre la transcription et la réplication du génome virale [123].
4.5. Protéines accessoires
Ces protéines ont été identifiées chez les virus IA ou IB mais ne semblent pas être indispensables pour la réplication virale. En revanche, la perte de certaines protéines pourrait impacter la virulence in vitro ou in vivo (tableau 1).
Type viral Protéines Fonctions connues Références
A
PB2-S1 Inhibition de la voie de signalisation RIG-I permettant la
synthèse des IFN-I [40]
PB1-N40 Très peu connues à ce jour [124]
PA-X Activité endonucléase permettant l’inhibition de l’expression des gènes cellulaires
Participe à l’inhibition de la réponse inflammatoire
[125, 126]
PA-N155
Très peu connues à ce jour [127]
PA-N182
M42 Protéine M2-like pouvant compenser la perte de M2 [41]
B NB Présente dans la membrane virale
Peu connues à ce jour [128]
25
5. Cycle de réplication virale
Figure 15: Cycle de réplication des virus influenza. Tiré de [39].
5.1. Fixation et internalisation
Comme nous l’avons précédemment vu, HA est la glycoprotéine de surface du virus influenza responsable de la fixation du virus à la surface de la cellule hôte. Une fois fixé, le virus va ensuite être internalisé dans la cellule par un mécanisme d’endocytose. Traditionnellement, le virus influenza empreinte ce mécanisme au niveau des puits de clathrine, toutefois il peut emprunter d’autres voies alternatives comme l’endocytose non médiée par la clathrine ou la cavéoline [129].
5.2. Échappement de l’endosome
Afin de pouvoir se libérer de l’endosome, les virus influenza ont besoin d’un pH acide permettant la fusion de leurs enveloppes avec la membrane endosomale. Ce mécanisme est dépendant de HA qui doit avoir été préalablement clivée en deux sous-unités HA1 et HA2
par des protéases cellulaires (voir figure 11). L’acidification de l’endosome va permettre à la HA mature de modifier sa conformation et de présenter ses peptides de fusion (présent