Étude du contexte chromatinien de la transcription des
gènes codant les ARN ribosomiques chez la souris
Thèse
Jean-Clement Mars
Doctorat en biologie cellulaire et moléculaire
Philosophiæ doctor (Ph. D.)
Étude du contexte chromatinien de la transcription des
gènes codant les ARN ribosomiques chez la souris
Thèse
Jean-Clément Mars
Sous la direction de :
Résumé
Au centre du ribosome, lui-même centre de synthèse de toutes les protéines, se trouve quatre molécules d’ARN, nommées les ARNs ribosomiques. Ces ARNs sont synthétisés dans le nucléole des cellules eucaryotes à partir des gènes répétés en tandem, les gènes d’ARN ribosomiques. Ces gènes nécessitent une régulation très stricte de la transcription des ARN ribosomiques pour permettre un cycle cellulaire contrôlé et surtout contrôlable. En effet, un débalancement de la transcription des ARN ribosomiques a été observée dans de nombreuses maladies comme par exemple le cancer. En conséquence, l’étude de la régulation de la synthèse des ARN ribosomiques est d’une importance cruciale dans la compréhension et subséquemment le traitement de ces maladies.
En raison du nombre de répétitions des gènes d’ARN ribosomiques, souvent évaluées aux alentours de 175 par génome haploïde pour les mammifères, l’étude in vivo de leur régulation est difficile par les techniques de mutagénèse. L’approche que j’ai développée durant cette thèse de doctorat est celle de l’immuno-précipitation de chromatine suivie de séquençage à haut débit (ChIP-Seq) en combinaison avec des lignées cellulaires conditionnelles pour des facteurs essentiels de la transcription ribosomique. Les gènes d’ARN ribosomique possèdent la particularité d’être transcrits grâce à une machinerie entièrement dédiée à sa transcription. Ces facteurs généraux de transcription agissent de concert pour effectuer efficacement leur rôle. Dans l’organisme modèle utilisé ici, à savoir Mus musculus, l’ARN polymérase I transcrit ces gènes avec l’aide, lors de l’initiation, des facteurs UBF, Rrn3/TIF-1A et SL-1/TIF-1B. Le facteur de terminaison TTF-1 permet de terminer la transcription de l’ARN précurseur, et joue plusieurs rôles dans la régulation de l’état des gènes.
Dans un premier temps, nous avons été amenés à développer, en complément du séquençage à haut débit, une approche de déconvolution des données pour permettre d’améliorer l’interprétation subséquente. Cette approche a été validée par l’amélioration notamment des profils d’immuno-précipitation de chromatine obtenus avec l’ARN polymérase I qui montrent une distribution homogène le long des gènes d’ARNr comme montrée par la technique de Miller
Spread. Cette amélioration des données a également pu mettre en avant un double rôle d’UBF en fonction soit de sa complémentarité avec SL-1 soit de son affinité pour les séquences riches en GC de l’ADN. Par la suite nous avons pu mettre en évidence la localisation des régions régulatrices et, en amont de ces régions, d’une barrière nucléosomique permettant de créer et maintenir une zone sans nucléosomes le long des gènes d’ARN ribosomiques. Cette barrière possède deux particularités. La première est d’être identifiée par les marques épigénétiques associées habituellement à l’activation de la transcription comme H3K4me3, H2A.Z ou encore l’acétylation de H2A.Z. La deuxième particularité est d’être indépendante de la présence d’UBF qui est elle-même indépendante de la transcription. Cependant, ce travail n’a pas détecté la présence des marques épigénétiques de l’activation de la transcription le long des gènes d’ARNr remettant en question certaines hypothèses sur la régulation de la transcription ribosomique.
Finalement, les études en parallèle dans les cellules souche embryonnaires (mESC) et fibroblastes embryonnaires (MEF) a permis d’identifier 3 catégories de gènes ribosomiques dans une même cellule. Une première forme nucléosomale ou l’ADN est méthylé et hétérochromatique, une deuxième nucléosomale mais non méthylée et finalement une forme transcrite. Une comparaison quantitative de la synthèse d'ARNr dans les mESCs et les MEFs a montré que le nombre de gènes actifs n'est pas un facteur significatif dans la régulation de la synthèse d’ARNr. Dans des cellules souches embryonnaires, tous les gènes sont transcrits en même temps avec une faible efficacité. Dans des cellules différenciées, une faible portion des gènes est transcrite mais très efficacement, cette efficacité étant relié au nombre de polymérase en cours de transcription. Les différents profils d'interaction de Rrn3 et de PolI près du site d'initiation de la transcription suggèrent que cette différence est due à une régulation de l'initiation.
Abstract
At the heart of the ribosome, itself the center of synthesis of all proteins, are four RNA molecules, called ribosomal RNAs. These RNAs are synthesized in the nucleolus of eukaryotic cells from the tandemly repeated genes, the ribosomal RNA genes. A very strict regulation of the transcription of ribosomal RNA needs to be made to allow a controlled and above all a controllable cell cycle. Indeed, the imbalance of ribosomal RNA synthesis has been observed in many diseases such as cancer. Consequently, the study of transcriptional regulation of ribosomal RNAs is of crucial importance in the understanding and subsequent treatment of these diseases.
Due to the number of repeats of ribosomal RNA genes, evaluated at around 175 per haploid genome for mammals, the in vivo study of their regulation by mutagenesis techniques is difficult. The approach I developed during this doctoral thesis is that of chromatin immunoprecipitation followed by high throughput sequencing (ChIP-Seq) applied to cell lines conditional for the basal transcription factors. The ribosomal RNA genes have the particularity of being transcribed thanks to an entirely dedicated transcriptional machinery. In mice, these general transcription factors act in concert to perform their role effectively. In the model organism used here, namely Mus musculus, RNA polymerase I transcribes the genes with the help, during initiation, of the factors UBF, Rrn3 / TIF-1A and SL-1 / TIF-1B. The TTF-1 termination factor makes it possible to terminate transcription of precursor ribosomal RNA, and also plays roles in gene regulation.
In addition to high-throughput sequencing, we have developed a deconvolution approach to improve the interpretation of ChIP-Seq data. This approach has been validated by the improvement in particular of immunoprecipitation profiles obtained for RNA polymerase I that confirm the electron microscopy images of Miller spread type. This improvement of the data could also highlight a dual role of UBF depending on its complementarity with SL-1 and its affinity for GC-rich sequences of DNA. Subsequently we have been able to highlight the upstream localization of the regulatory regions and of a
nucleosome barrier allowing to create and maintain a zone without nucleosomes along the rRNA genes. This barrier has two peculiarities, the first is that it contains the epigenetic marks usually associated with the activation of transcription such as H3K4me3, H2A.Z or the acetylation of H2A.Z. The second particularity is that it is independent of the presence of UBF, which is itself independent of transcription. Challenging certain assumptions about the regulation of ribosomal transcription, our work did not detect the presence of epigenetic markers of transcriptional activation throughout the rRNA gene body.
Finally, parallel studies in embryonic stem cells (ESC) and embryonic fibroblasts (MEFs) made it possible to identify 3 categories of ribosomal genes in the same cell. First, a heterochromatic DNA methylated and nucleosomal form, a nucleosomal but non-DNA methylated form, and finally a transcribed form. A quantitative comparison of rRNA synthesis in ESCs and MEFs has shown that the number of active genes is not a significant factor in the regulation of rRNA synthesis. In embryonic cells, all genes are transcribed at the same time with low efficiency. In differentiated cells, a small portion of the genes are transcribed but very efficiently, this efficiency being related to the number of polymerase being transcribed. The different interaction profiles of Rrn3 and PolI near the transcription initiation site suggest that this difference is due to the regulation of initiation.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières... vii
Liste des figures ... xi
Liste des tableaux ... xiii
Abréviations ... xiv
Remerciements ... xvi
Avant propos ... xviii
Introduction ... 1
Chapitre 1 :
Revue de littérature ... 5
1.1. Le nucléole ...5 1.1.1. Découverte ... 5 1.1.2. Rôles ... 7 1.2. L'ADN ribosomique... 10 1.2.1. Organisation ... 11 1.2.2. Transcription ... 13 1.3. Épigénétique et chromatine ... 24 1.3.1. Présentation de la chromatine ... 24 1.3.2. Le nucléosome ... 26 1.3.3. Méthylation de l'ADN ... 31
1.3.4. Épigénétique de l'ADN ribosomique ... 32
1.4. Méthodes d'études de l'ADN ribosomiques ... 36
1.4.1. Utilisation des techniques du séquençage à haut débit ... 36
1.4.2. Présentation des modèles d'études cellulaire ... 39
1.4.3. Traitements ... 41
1.5. Objectifs de la thèse ... 42
Chapitre 2 :
A deconvolution protocol for ChIP-seq reveals analogous enhancer
structures on the mouse and human ribosomal RNA genes ... 44
2.1. Résumé... 45
2.2. Abstract ... 46
2.3. Introduction ... 47
2.5. Results ... 51
2.6. Discussion ... 68
2.7. Acknowledgments ... 70
2.8. Conflict of interest ... 70
2.9. References ... 71
Chapitre 3 :
A unique Enhancer Boundary Complex on the Mouse Ribosomal RNA
genes persists After loss of Rrn3 and UBF, and the inactivation of RNA Polymerase I
transcription ... 76
3.1. Résumé... 77
3.2. Abstract ... 78
3.3. Introduction ... 79
3.4. Results ... 81
3.4.1. A High-resolution map of basal factors across the mouse rDNA. ... 81
3.4.2. UBF binding precisely delimits to the functional rDNA unit. ... 84
3.4.3. A stalled RPI transcription complex lies near the upstream UBF boundary. ... 85
3.4.4. Rrn3-TIF1A is an essential factor in mouse. ... 85
3.4.5. RPI transcription is not required to maintain actively poised rDNA... 86
3.4.6. UBF is essential for the recruitment of the RPI transcription machinery. ... 88
3.4.7. UBF determines psoralen accessibility and nucleosome exclusion. ... 91
3.4.8. Rrn3-loss causes partial rDNA chromatin collapse but does not disrupt the nucleolus. .... 93
3.4.9. UBF may replace core histones on the active rDNA. ... 95
3.4.10. The domain of UBF recruitment is delineated by a unique upstream Enhancer Boundary. 98 3.4.11. The Enhancer Boundary is maintained in the absence of UBF. ... 98
3.4.12. Cohesin is also recruited to the Enhancer boundary... 101
3.5. Discussion ... 102
3.6. Materials and Methods ... 108
3.6.1. Isolation and culturing of MEFs. ... 108
3.6.2. Embryo collection and genotyping... 108
3.6.3. Inactivation of Ubf or of Rrn3 in cell culture, and analysis of genotype, rRNA synthesis and proteins. 109 3.6.4. Antibodies for Western Blot, Immunofluorescence and ChIP ... 110
3.6.5. Chromatin immunoprecipitation (ChIP). ... 110
3.6.6. Analysis of ChIP samples by massively parallel sequencing... 111
3.6.7. DNase-Seq ... 111
3.6.8. Analysis of massively parallel sequence data. ... 111
3.6.9. Psoralen crosslinking accessibility. ... 112
3.6.10. MNase digestion of chromatin ... 112
3.7. Ethics Statement ... 114
3.8. Acknowledgments ... 114
3.9. Competing Interests ... 114
3.10. Réferences ... 114
Chapitre 4 :
Caractérisation de la transcription ribosomique dans des cellules
embryonnaires de souris. ... 132
4.1. Introduction: ... 133
4.2. Matériel et méthodes ... 134
4.2.1. Culture cellulaire ... 134
4.2.2. Immuno-Précipitation de Chromatine ... 135
4.2.3. Analyse du séquençage haut débit ... 135
4.2.4. Digestion à la DNaseI... 135
4.2.5. Digestion à la MNase ... 135
4.2.6. Séquençage au bisulfite... 136
4.2.7. Anticorps pour Western-Blot et ChIP ... 136
4.2.8. Marquage métabolique et calcul de l’élongation ... 136
4.2.9. Comptages ... 136
4.3. Résultats... 137
4.3.1. Les gènes ribosomiques dans les Cellules ES sont d'avantage sous conformation actives que dans des cellules fibroblastiques ... 137
4.3.2. La barrière nucléosomique est présente sur les gènes actifs ... 143
4.3.3. La localisation des Facteurs Généraux de Transcriptions (FGTs) au SpPr suggère un rôle de cette zone dans la détermination de l'état d'activité des gènes codant l'ARNr ... 146
4.3.4. Caractérisation des différences de transcription de l'ADNr entre ES et les cellules de type fibroblaste153 4.4. Discussion ... 157
Conclusion ... 161
A) UBF stabilise la liaison de SL-1 au niveau du promoteur des gènes d'ARNr ... 162
B) UBF remplace les nucléosomes sur les gènes d’ARNr ... 163
C) La barrière nucléosomique permet de maintenir ouverts les gènes ribosomiques ... 164
D) UBF stabilise les zones sans nucléosomes ... 165
E) Il n'y a pas de site spécifique de modification des histones au niveau du promoteur principal. ... 167
F) La perte de la transcription ribosomique augmente la modification de la barrière nucléosomique ... 169
G) Mise en évidence d’une régulation différente de l’initiation de la transcription ribosomique ... 169
H) Le nombre de gènes d’ARNr transcrits est corrélé à la différentiation cellulaire ... 170 I) La transcription des gènes ribosomiques permet l’ouverture de la chromatine ... 172
Bibliographie ... 174
Liste des figures
Figure 1-1 : Image de microscopie électronique du noyau d’une cellule de pancréas de
rat. ________________________________________________________________________________ 2
Figure 1-2 : Section histologique de cancer du sein chez deux patientes. ______________ 6 Figure 1-3 : Nucléole d’une cellule P815 de souris. ___________________________________ 7 Figure 1-4 : Représentation de la chaine de production des ribosomes chez la souris. __ 8 Figure 1-5 : Régulation de P53 par l’ubiquitine-ligase MDM2 ________________________ 10 Figure 1-6 : Localisation chromosomique des RONs chez l’humain et la souris. _______ 11 Figure 1-7 : Structure du gène codant les ARN ribosomiques chez la souris. __________ 13 Figure 1-8 : Image originale de microscopie électronique de la technique de Miller spread
sur des ovocytes de Triturus viridescens. ___________________________________________ 15
Figure 1-9 : Schéma de la structure de SL-1 chez la souris. _________________________ 17 Figure 1-10 : Structure schématique d’UBF. ________________________________________ 18 Figure 1-11 : Structure schématique de l’enhancesome et modèle hypothétique du
promoteur principal. _______________________________________________________________ 19
Figure 1-12 : MDM2 contrôle les fonctions ribosomiques de TTF-1. ___________________ 21 Figure 1-13 : Modèle de la transcription ribosomique in vitro. ________________________ 23 Figure 1-14 : Régulation de la transcription ribosomique au niveau des FGTs. ________ 24 Figure 1-15 : Image de microscopie électronique à transmission d'une cellule HeLa. __ 25 Figure 1-16 : Représentation des différents niveaux de compaction de l'ADN. ________ 26 Figure 1-17 : Représentation d'un nucléosome et des modifications associées aux
histones canoniques. ______________________________________________________________ 28
Figure 1-18 : Modèle de la régulation épigénétique de la transcription ribosomique. ___ 34 Figure 1-19 : Modèle de l’incorporation au psoralène. _______________________________ 35 Figure 1-20 : Schéma du processus de séquençage Illumina. ________________________ 37 Figure 2-1 : Improved mapping with ChIP-Seq deconvolution. ________________________ 55 Figure 2-2 : ChIP-Seq deconvolution maps are highly reproducible. __________________ 57 Figure 2-3 : Preferential positioning of UBF across the mouse rDNA. _________________ 59 Figure 2-4 : Mapping of preinitiation complexes at the Spacer and 47S Promoters of
MEFs.). ___________________________________________________________________________ 61
Figure 2-5 : Direct comparison of interaction proles of the TAF1B, -C and TBP
components of SL1, and of UBF and RPI at the Spacer and 47S Promoter regions in MEFs (MTAB-5893). _____________________________________________________________________ 62
Figure 2-6 : Identification of a Spacer Promoter in the human rDNA.. _________________ 64 Figure 2-7 : Fine mapping of TBP complexes and potential enhancer repeat suggest
functional parallels between the human and mouse rDNA. e. _________________________ 67
Figure 3-1 : High resolution ChIP-Seq maps of RPI, the RPI basal factors and TTF1 across
the mouse rDNA repeat unit of Ubf+/+/Rrn3+/+p53-/- control MEFs.. ____________________ 83
Figure 3-2 : Conditional deletion of the Rrn3 gene inhibits RPI transcription but does not
affect pre-initiation complex formation.. _____________________________________________ 88
Figure 3-3 : Conditional deletion of the UBF gene not only ablates RPI transcription but
also prevents preinitiation complex formation.. ______________________________________ 91
Figure 3-4 : UBF determines the activated state of the rDNA chromatin. 4. ___________ 93 Figure 3-5 : Comparative nucleolar structures before and after UBF or Rrn3 gene
Figure 3-6 : The UBF binding domain is delineated by an Enhancer Boundary Complex.
__________________________________________________________________________________ 97
Figure 3-7 : The Enhancer Boundary Complex exists independently of UBF and rDNA
activity. __________________________________________________________________________ 100
Figure 3-8 : Cohesin subunits and chromatin remodelers also map to the Enhancer
Boundary. _______________________________________________________________________ 102
Figure 3-9 : Diagrammatic summary of factor binding and chromatin structure across the
active mouse rDNA repeat (Wild Type) and after deletion of Rrn3 (Rrn3-KO) or UBF (UBF-KO). _____________________________________________________________________________ 103
Figure 3-10 : Figure S1. GC content of the rDNA and UBF binding. ____________ 125 Figure 3-11 : Figure S2. Deletion of the Rrn3-Tif1a gene arrests mouse
development during early cleavage divisions. __________________________________ 126 Figure 3-12 : Figure S3. Functional analysis of UBF and Rrn3 inactivation in conditional MEFs. ______________________________________________________________ 129 Figure 3-13 : Figure S4. TTF1 may bind at low level throughout the 47S gene body during active transcription. ______________________________________________ 129 Figure 3-14 : Figure S5. UBF determines the activated state of the rDNA
chromatin.. _____________________________________________________________________ 130 Figure 3-15 : Figure S6. H3K9ac, H3K36me3 and H3K27ac histone modifications also map to the Enhancer Boundary Complex.. ________________________________ 131 Figure 4-1 : Alignement du séquençage après traitement au bisulfite sur le gène de l'ARN
ribosomique.. ____________________________________________________________________ 138
Figure 4-2 : Les cellules ES montrent une forte incorporation au psoralène
dépendamment de la présence d’UBF. _____________________________________________ 140
Figure 4-3 : L’accessibilité de l’ADN dans les cellules ES est dépendant de la présence
d’UBF est de l’absence de nucléosomes A Digestion à la MNase de l’ADN après induction de la déplétion d’UBF dans des cellules ES. ________________________________________ 142
Figure 4-4 : Profil de liaison des histones H3 et H2A.Z. ____________________________ 144 Figure 4-5 : ChIP-seq de H3K4me3, H3, H2A.Z, H2A.Zac et CTCF dans des cellules ES
UBFfl/fl ERCre+/+ avant et 72h après induction de la déplétion d’UBF avec le 4HOT. 145
Figure 4-6 : ChIP-seq des protéines UBF (bleu), TAF68 (jaune), TTF-1 (rouge), PolI (vert
foncé) et Rrn3 (vert clair) dans des cellules E14. ____________________________________ 147
Figure 4-7 : Comparaison du ChIP-seq de PolI et de Rrn3 entre des cellules mESs et des
MEFs. ___________________________________________________________________________ 149
Figure 4-8 : ChIP-seq de PolI ou Rrn3 dans des cellules NIH3T3 en présence de sérum
ou en présence de PD98057 50µM. ________________________________________________ 150
Figure 4-9 : Effet du CX-5461 sur la transcription et la localisation des FGTs dans des
cellules NIH3T3. _________________________________________________________________ 151
Figure 4-10 : ChIP-seq de PolI et Rrn3 dans des conditions contrôles. ou traité avec
10uM de CX-5461. _______________________________________________________________ 152
Figure 4-11 : Mesure de la durée et de la quantité d’ARN par type cellulaire. ________ 154 Figure 4-12 : Expérience de marquage métabolique par de l'uridine marqué au 3H. __ 155
Figure 4-13 : Modèle de la transcription ribosomique dans les cellules ES et les cellules
Fibroblastiques. __________________________________________________________________ 159
Figure 5-1 : Pourcentage en GC des pics de DNase-seq en fonction de la présence d’UBF.
_________________________________________________________________________________ 165
Liste des tableaux
Tableau 1 : Pourcentage de méthylation moyen des sites CpG de l'ADNr au niveau des
séquences inter-géniques (SIG) ou géniques.________________________________________ 139
Tableau 2 : Quantification des deux bandes de migration après traitement au psoralène
après induction de la Cre-recombinase dans des cellules ES. ________________________ 141
Tableau 3 : Mesure des ratios des signaux maximums des ChIP seq d'UBF et TAF68
entre le promoteur principal (Prom) et le Spacer Promotes (SpPr). _____________________ 147
Tableau 4 : compilation des résultats de marquage métabolique, cycle cellulaire, quantité
Abréviations
ADN : Acide-Deoxyribo-Nucléique ARF :
ADP-Ribosylation Factors
ARN : Acide-Ribo-Nucléique
ARNr : Acide-Ribo-Nucléique ribosomique Cas9 : Caspase 9
CBP : CREB-binding protein CF : Centre Fibrillaire
CFD : Composante Fibrillaire Dense ChIP : Chromatin Immuno-Precipitation
ChIP-seq : Chromatin Immuno-Precipitation sequencing CG : Composante Granulaire
CpG : Cytosine-phosphate-Guanine CPI : Complexe de Pré-Initiation
CRISPR : Clustered Regularly Interspaced Short Palindromic Repeats CTCF : CCCTC-binding factor
Ctrl : Contrôle
DMEM : Dubelcco’s Modified Eagle Medium DNaseI : Deoxyribonucleicacid Nucelase I EGF : Epidermal Growth Factor
ERCre : Estrogen Receptor Cre
ERK : Extracellular signal-Regulated Kinase ESC : Embryonic Stem Cell
FGTs : Facteurs Generaux de la Transcription HAT : Histone Acetyl Transferase
HMG : High Mobility Group
Hmo1 : High mobility group protein-1 HMT : Histone Methyl Transferase HP1 : Heterochromatin Protein 1 LoxP : Locus of X-over P1
MAPkinase : Mitogen Activated Protein kinase MDM2 : murine double minute 2
MEF : Mouse Embryonic Fibroblast mESCs : mouse ESCs
MNase: Micrococcal Nuclease
mTOR : mammalian Target Of Rapamycin NoRC : Nucleolar Remodelling Complex P53 : Protein 53
pARN : promoteur ARN
PolI/RPI : RNA Polymerase I/ RPI Prom : promoteur principal
qPCR : quantitative-Polymerisation Chain Reaction RMN : Rayonnement Magnétique Nucléaire
RON : Régions Organisatrices des Nucléoles RP : Ribosomal-Protein
RPA : RNA Polymerase I-Associated Factor RSN : Réponse au Stress Nucléolaire
SIGs : Séquences Intergéniques
SIT : Site d’Initiation de la Transcription SL-1 : SeLectivity factor-1
snoRNP : small nucleolar Ribo-Nucleo-Protein SpPr : Spacer Promoter
TAD : Trans-Activating-Domain TAF : TBP Associated Factor TBP : TATA box Binding Protein
TIF1A : Transcription Initiation Factor-1A Tsp : Terminateur Spacer Promoteur TTF-1 : Transcription Termination Factor-1 UAF : Upstream Activating Factor
UBF : Upstream Binding Factor
UBTF : Upstream Binding Transcription Factor UCE : Upstream Core Element
WGBS : Whole Genome Bisulfite Sequencing ZDN : Zones Déplétées en Nucléosomes
Remerciements
Ce n’est pas vraiment mon truc de remercier des gens surtout que si je devais être parfaitement honnête il y a beaucoup plus de gens qui ont participé directement ou non à ce travail de thèse. Je me contenterai d’un hommage aux personnes impliqués directement dans les années de laboratoire que j’ai passé à et au Québec.
En premier lieu je tiens à remercier mon directeur de recherche, le Dr Thomas Moss de sa présence et des très nombreuses discussions scientifiques ou non que l’on a pu avoir au cours de ces 5 ans de travail passionnant. Il a clairement pris la place de mentor et a été un merveilleux pédagogue pour réussir à faire rentrer un peu de rigueur scientifique dans ma tête de rêveur.
Ensuite chacun des membres du laboratoires à mon éternelle reconnaissance pour l’aide, les conseils et les bons moments passés ensemble. À ce titre, je tiens à remercier Mike et Victor, les piliers du labo (à défaut des comptoirs). Plus que juste des professionnels, tout le temps que l’on a passé devant les mots croisés, les bières et les croissants ont été riches (en discussion politico-phylosophico-scientifico-religieuse entre autres) et extrêmement plaisants (voir rigolos, amusants, divertissants… et pleins d’autres synonymes bizarres que l’on a dû chercher sur internet). Chelsea bien entendu qui fut une complice et une amie précieuse. Fascinante de passion et d’envie d’apprendre un tas de choses, toujours d’attaque pour finir les journées de travail par des pichets de bières dans les bars de Québec. Mais aussi Nourdine, Prakash et Joe qui m’ont très bien accueilli au laboratoire et intégré dans leur environnement. Merci à Nourdine en particulier qui m’ayant enseigné le ChIP a permis indirectement la réalisation de la majorité de mon projet de recherche.
La première personne étrangère au laboratoire a qui vont mes remerciements sera Alice qui est responsable de ma venue au Québec, une complice et amie depuis plusieurs riches années (surtout en conneries mais c’est ça qu’est bon comme on dit). Ensuite je voudrais remercier Pierre qui m’a accueilli chez lui les premières semaines de mon arrivée, c’est toujours marrant de voir quelqu’un qu’on connait de l’autre côté d’un océan, mais c’est encore mieux
quand c’est un ami (P.S. J’ai toujours des photos dossiers qui trainent). Finalement j’aimerai remercier tous les gens du centre et en dehors qui m’ont accompagné sur cette fin de diplomation (C’est pas trop tôt).
Un grand merci à tous les amis, passés et présent qui sont passés dans ma vie et ont fait en sorte que ce séjour à Québec soit peuplé de merveilleux souvenirs. La liste serait trop longue mais merci à vous tous.
Pour refermer cette courte partie, je tenais bien entendu à remercier ma famille qui me supporte depuis toujours. En particulier mes parents et mes frangins qui ont eu à me supporter depuis ma naissance. Je leur suis éternellement reconnaissant d’avoir su me porter ou j’en suis maintenant. Et ce même si ils préfèreraient que je sois de l’autre côté de l’océan.
Avant propos
Cette thèse présente 4 chapitres suivis d’une conclusion essayant de mettre en avant les découvertes faites au cours de ce travail de doctorat.
Le chapitre premier est une revue de littérature présentant les différents thèmes de biologie pertinents dans le cadre de cette étude. Dans un premier temps, j’ai choisi de présenter le nucléole qui est le lieu d’intérêt de mon étude. Dans un deuxième temps, ce sont les ARN ribosomiques ainsi que leurs gènes qui font l’objet d’une présentation étant donné la place centrale de la régulation de leur production dans ma thèse. La troisième partie est une introduction à l’épigénétique qui joue un rôle important dans la modulation de la transcription des gènes d’ARNr. Dans une quatrième partie, j’ai voulu présenter certains modèles cellulaires utilisés dans les éxpèriences. Les objectifs de la thèse sont présentés à la fin du chapitre.
Le chapitre numéro 2 a été publié sous forme d’article dans la revue G3 : Genes | genomes | Genetics. Les lignées cellulaires conditionnelles pour UBF ont été générées par Michel G. Tremblay. Marianne Sabourin-Félix a réalisé l’intégralité des analyses de données ainsi que participé à l’élaboration du protocole de normalisation. Le Dr. Tom Moss a rédigé le manuscrit et créé les figures, il a également participé à l’élaboration du protocole de normalisation ainsi qu’à l’identification du Spacer Promoter humain ainsi que de la région de répétition enhancer. J’ai pour ma part réalisé la totalité des expériences de ChIP-seq et de DNaseI-ChIP-seq. J’ai également participé à l’élaboration du protocole de normalisation et à la rédaction du manuscrit. Tous les auteurs ont participé aux corrections du manuscrit. Cette publication est reproduite avec l’autorisation de tous les coauteurs.
Le troisième chapitre a été publié sous forme d’article dans la revue Plos Genetics en juillet 2017. Les lignées cellulaires conditionnelles pour UBF ont été générées par Michel G. Tremblay. Victor stefanovsky a réalisé les expériences de digestion à la MNase, marquages métaboliques, incorporation de psoralène et certains western-Blot. Le Dr. Chelsea Herdman a généré les lignées cellulaires conditionnelles pour Rrn3 ainsi que réalisait la caractérisation de la déplétion
cellulaire de Rrn3, réalisées la caractérisation du phénotype de déplétion embryonnaire de Rrn3. Elle a également généré les premières données de ChIP-seq pour la déplétion de Rrn3. Marianne Sabourin-Félix a réalisé l’intégralité des analyses de données bio-informatiques. Helen Lindsay ainsi que le Dr. Mark D.Robinson ont fourni la plateforme de stockage des données et ont aidé à l’analyse des données de séquençage à haut débit. Le Dr. Tom Moss a rédigé le manuscrit et créé les figures ainsi que supervisé tous les travaux du laboratoire. J’ai pour ma part réalisé la majorité des expériences de ChIP-seq et de DNaseI-seq ainsi que les caractérisations de la déplétion d’UBF. J’ai également participé aux différentes étapes d’élaboration du manuscrit. Tous les auteurs ont participé aux corrections du manuscrit. Cette publication est reproduite avec l’autorisation de tous les coauteurs.
Finalement le chapitre 4 présente les travaux que j’ai réalisé durant la fin de mon travail de doctorat, il porte sur la différence des modèles cellulaires concernant la transcription des ARNs ribosomiques. Le séquençage au bisulfite ainsi que les éxpériences de digestion à la MNase ont été réalisées par Victor Stefanovsky, l’alignement des séquençages a été réalisé par Marianne Sabourin-Félix.
Introduction
Le ribosome est au cœur de la vie cellulaire de par son rôle central dans la synthèse des protéines. Sa découverte remonte aux années 1950 après que la microscopie électronique ait rendu possible la visualisation du réticulum endoplasmique granuleux (figure 1). Ces petits granules qui, par la suite, furent identifiés comme l'usine de création de toutes les protéines du monde vivant : le ribosome. Il est intéressant de noter que l’observation de cette macromolécule a été permise en raison de sa taille imposante pour une machinerie cellulaire. En effet, chez les eucaryotes, le ribosome est un complexe de 4.2MDa (250-300 Å), ce qui en fait une des plus grosse machinerie moléculaire cellulaire avec le pore nucléaire. En contrepartie, sa taille a posé problème pour permettre d'en étudier précisément la structure par des techniques de biophysiques précises comme la cristallographie ou rayonnement magnétique nucléaire. Ce n’est que récemment, dans les années 2010 que finalement sa structure a pu être résolue pour le ribosome humain (Khatter et al. 2015).
Le ribosome est une macromolécule dite nucléo-protéique. Il est composé à la fois de protéines (environ 80) appelées les ribo-protéines (RP) ainsi que d'acides-ribo-nucléiques (ARN) dits ARN ribosomiques (ARNr) qui représentent environ 60% de la masse du ribosome. Fait non anodin dans le vivant ce sont les ARNr qui possèdent la fonction de catalyse enzymatique, et ici de catalyse de la liaison peptidique (Huttenhofer and Noller 1992). À titre anecdotique, mais non moins passionnant, ce fait est un argument fondateur de la théorie du monde de l'ARN (Review in Vázquez-Salazar and Lazcano 2018), l'une des deux théories découlant de la "soupe originelle" sur l'apparition de la vie sur terre. Cet argument a été mis en place suite à l'observation des propriétés de l'ARNr. Ce dernier possède à la fois des qualités de l'ADN par son encodage simple et sa stabilité, mais aussi des protéines, notamment avec l'arrangement en 3 dimensions ou encore la catalyse enzymatique. Cette théorie introduit le fait qu'à l'instar de la cellule, les ribosomes, et plus particulièrement les ARNr, sont au centre de la vie elle-même.
Figure 0-1 : Image de microscopie électronique du réticulum endoplasmique
granuleu ainsi que du noyau d’une cellule de pancréas de rat. n le noyau, nm1 la membrane nucléaire, nm2 une membrane du réticulum endoplasmique rugueux, g et x des structures granulaires denses correspondant au ribosomes. z un granule de proenzyme. (Adapté de : Palade 1955)
Outre cette mise en exergue de l'importance de ce composant cellulaire, il existe d'autres raisons de s'y intéresser consciencieusement. L'une d'entre elle est que le ribosome est la macromolécule la plus présente dans les cellules. Son rôle ainsi que la quantité nécessaire aux différents processus cellulaires suggèrent que la régulation de sa production est une étape limitante de la majorité des mécanismes cellulaires fondamentaux comme la division, la différentiation ou encore la croissance cellulaire (Campbell and White 2014; Tom Moss 2004; C Mayer and Grummt 2006). À titre d'illustration, pour certaines levures en phase de croissance, la production des ribosomes représente à elle seule 60% de la transcription totale d'une cellule (Warner 1999).
cellu-Même si ces constatations placent l'étude du ribosome et de sa production comme primordiale dans la compréhension de la biologie de manière générale mais également au niveau de la biologie médicale plus particulièrement. En effet, étant donné le rôle central de la production des protéines dans la biologie, le mécanisme de fabrication des ribosomes est également l'un des mécanismes le plus souvent dérégulé dans de nombreuses pathologies. C'est le cas pour la majorité des cancers connus ou encore des maladies métaboliques (Núñez Villacís et al. 2018). Les efforts de la médecine pour lutter contre ces fléaux ont permis de découvrir de nouvelles molécules efficaces dans leur traitement. Certaines d'entre elles sont dirigées directement contre la synthèse des ribosomes et en particulier contre la production des ARNr (Bywater et al. 2012). D'autres molécules, initialement suspectées d’agir via les dommages à l’ADN, impactent la production des ribosomes en premier lieu (Hamdane et al. 2015; Jordan and Carmo-Fonseca 1998). Dans tous les cas, ces molécules permettent d'étudier la transcription ribosomique et augmentent l'arsenal chimique de la médecine.
En tant que mécanisme essentiel à la vie, c’est tout naturel que la transcription ribosomique soit sujette à un haut degré de conservation à travers l’évolution et donc que son étude soit facilitée par l'utilisation de très nombreux modèles différents. Cependant, malgré les similitudes entre organismes, certaines spécificités importantes existent. Les eucaryotes ont alloué à la transcription ribosomique une machinerie spécialisée se singularisant ainsi du monde procaryote. Les résultats de cette thèse de doctorat portent sur la production des ARNr chez la souris. Étant donné la conservation de ce mécanisme central de la vie cellulaire au cours de l’évolution, certains résultats de cette étude sont interprétables au niveau des eucaryotes en général. En revanche, étant donné les facteurs étudiés, la majorité des résultats sont spécifiques aux métazoaires voire aux mammifères. La machinerie moléculaire utilisée par la souris dans la transcription ribosomique comprend entre autre l’ARN polymérase de type I (PolI, RNA Polymerase I ; RPI), ainsi qu'une série de facteurs essentiels nommés Facteurs Généraux de la Transcription (FGTs) comprenant les protéines UBF (Upstream Binding Factor; ou Upstream Binding Transcription Factor ; UBTF), Rrn3 (TIF1A), TTF-1, ou encore le complexe SL-1 (SeLectivity factor I) composé lui-même de TBP (TATA-box Binding Protein) et de,
au moins, 3 protéines associées TAF48/TAF1A, TAF68/TAF1B et TAF95/TAF1C (TBP Associated Factor). Un 4ième facteur TAF41/TAF1D a également été identifié
par la suite comme potentiel membre de SL-1. Chose remarquable, la production des ARNr est réalisée au niveau d’un sous-compartiment nucléaire spécialisé, le nucléole. Il fut le premier non délimité par une membrane qui a été identifié historiquement.
Chapitre 1 : Revue de littérature
1.1. Le nucléole
1.1.1.
Découverte
Découvert dans les années 1830, le nucléole fut l'un des premiers marqueurs des cellules cancéreuses découverts (Revue dans : Montanaro, Treré, and Derenzini 2008). En effet, des changements au niveau de la taille ainsi que la forme de ce sous-compartiment nucléaire ont été, dans un premier temps, reliés aux cellules malignes (figure 1.2). Il peut se présenter sous forme d'un unique compartiment en particulier dans la levure ce qui permet une observation facile du volume ainsi que de la forme. En revanche, dans les cellules mammifères, il existe plusieurs nucléoles par cellule. Dans ce cas de figure, les déformations et les hypertrophies peuvent être dues à des augmentations du volume nucléolaire mais également à une augmentation du nombre de nucléoles.
Par la suite, le caractère diagnostique du nucléole a dû être abandonné étant donné son manque de spécificité (Derenzini et al. 1998). En effet, l’observation est désormais reliée à un phénomène beaucoup plus "normal" de l'état des cellules. Étant donné sa fonction de synthèse des ribosomes, le nucléole et sa taille reflètent l'état métabolique de la cellule et non un état malin particulier. Une conséquence logique sera que plus une cellule va avoir besoin de produire des protéines plus elle va devoir produire de ribosomes et plus le nucléole sera gros ; il en découle qu’une diminution de moitié du cycle cellulaire entrainera, dans le même temps, une multiplication par un facteur 4 de la quantité de ribosomes nécessaire. En conséquence, tous les phénomènes de dérégulation du cycle cellulaire ainsi que les changements du métabolisme cellulaire auront un effet direct sur la taille du nucléole. Il y a donc un lien physique entre la production des ARNr et la taille du nucléole, le cycle cellulaire et le métabolisme.
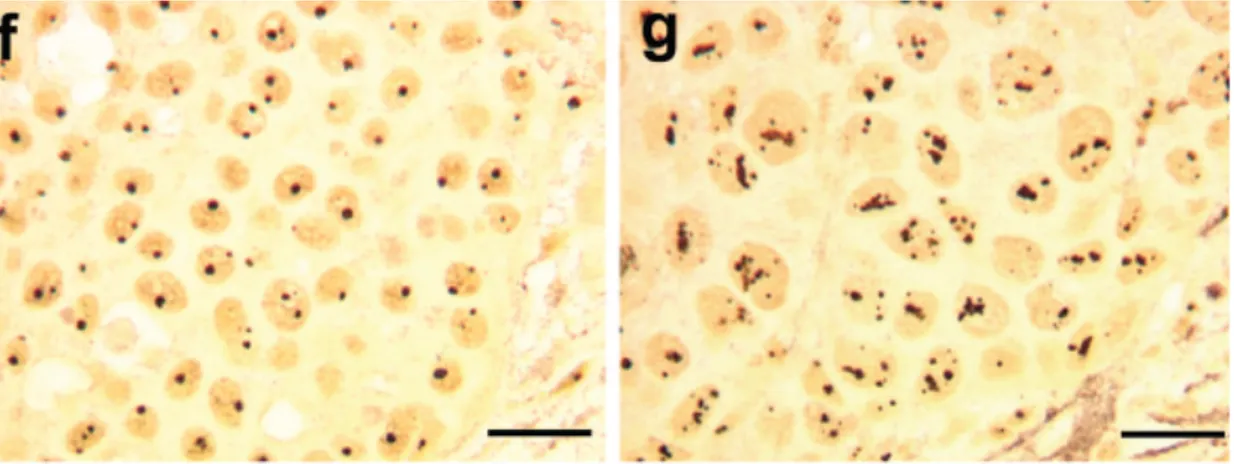
Figure 1-1 : Section histologique de cancer du sein chez deux patientes. Coloration
à l'argent, permet d'observer les AgNOR (Argent Nucleolar Organiser Regions). F. Patiente avec un bon pronostic vital. G. Patiente avec un mauvais pronostic. (bar d'échelle: 20µm) (Adapté avec autorisation de : Montanaro, Treré, and Derenzini 2008)
Malgré les variations, la structure du nucléole est relativement stable en fonction des différentes cellules. Il est habituellement sous-divisé en 3 compartiments en fonction de leur densité lors des observations de microscopie électronique (figure 1.3). Il existe le centre fibrillaire (CF), la composante fibrillaire dense (CFD) et la composante Granulaire (CG). Cette séparation en plusieurs compartiments différents est supportée par la localisation de différents marqueurs comme l’ARNr, l’ADN, UBF ou encore PolI.
Récemment, certains chercheurs ont commencé à étudier le phénomène de compartimentation indépendant des membranes reliées à certains organites particuliers comme le nucléole ou les granules de stress (Mitrea and Kriwacki 2016). Ces hypothèses font intervenir la séparation de phases liquide-liquide et permettent d’expliquer comment ce genre de compartiment peut se former et se maintenir. Sans entrer dans les détails, la formation de ces compartiments est reliée directement à leurs rôles. Pour le nucléole, la transcription des ARNr induit la formation du nucléole à cause entre autres de la production des ARNr eux-mêmes. Cette production d’ARN permet de recruter et d’assurer la présence des protéines de maturation des ARNr qui in vitro ont été impliquées dans la formation de compartiments non-membranaires (Mitrea et al. 2018).
Figure 1-2 : Nucléole d’une cellule P815 de souris. La CFD (flèches) apparait
comme une structure dense aux électrons entourant le centre fibrillaire. La majorité du nucléole est composé de la composante granulaire (astérisque). Bar d’échelle, 0.5µm (Adapté de : Biggiogera et al. 2001)
1.1.2.
Rôles
1.1.2.1. La production des ribosomes
Le nucléole est une structure nucléaire qui a été associé à la synthèse des ribosomes chez les eucaryotes dans les années 1960 (review: Pederson, 2011). Ce sous-compartiment nucléaire est le lieu d'assemblage des deux sous-unités des ribosomes, et également le lieu de synthèse des ARNr ou tout du moins de 3 des 4 ARNr : les ARN 5.8S, 18S et 28S. Chez la plupart des eucaryotes, le gène codant l'ARNr 5S est transcrit séparément dans le noyau par l'ARN polymérase de type III. La production des ARNr est un phénomène extrêmement central pour une cellule en phase de croissance.
Le CF est composé en partie d'ADN, notamment des gènes codant les ARNr, des protéines structurales comme UBF ou encore des protéines associées à la transcription ribosomique comme PolI. Ce centre nucléolaire est peu dense aux électrons et il est associé généralement à la production des ARNr. C'est plus précisément à l'interface entre le CF et la CFD que se trouve la synthèse des ARNr
(Junéra et al. 1995). La différence de densité aux électrons après coloration entre les deux compartiments au niveau de la figure 1.3 est due à la présence des ARNr, majoritairement absent du CF et très présent au niveau du CFD. Le dernier des compartiments est la CG. il possède lui aussi des ARNr mais très peu d'ADN et pas de transcription ribosomique. C'est d’un côté le centre de maturation des ARNr mais également là où les protéines ribosomales ainsi que les ARNr sont assemblés pour former les deux sous-unités ribosomales.
Figure 1-3 : Représentation de la chaine de production des ribosomes chez la
souris. La flèche rouge représente la transcription ribosomique (rouge pour l'ARN 18S, noir pour le 5.8S et le 28S), la flèche verte représente la transcription de l'ARNr 5S (vert clair) et la flèche jaune représente la transcription des ARNm (jaune) des protéines ribosomiques. Les flèches blanches représentent les processus d’import/export des différents composants et la chaine d’assemblage simplifiée.
En tant que mécanisme central de la vie cellulaire, il est également intéressant de noter que la production des ribosomes fait intervenir la totalité des types de polymérases mais également les ribosomes eux-mêmes. La figure 1.4 résume cette observation chez la souris. L’ARN polymérase III produit l'ARNr 5S dans le noyau des cellules de mammifères qui sera acheminé vers le nucléole
pour entrer dans la composition de la grande sous-unité ribosomique. L’ARN polymérase II transcrit les ARN messagers (ARNm) des protéines en général et donc également celui des protéines ribosomiques (RP) qui seront traduits dans le cytoplasme par des ribosomes avant de rejoindre le nucléole pour s'associer aux ARNr et former les deux sous-unités ribosomiques.
1.1.2.2. Contrôle du cycle cellulaire
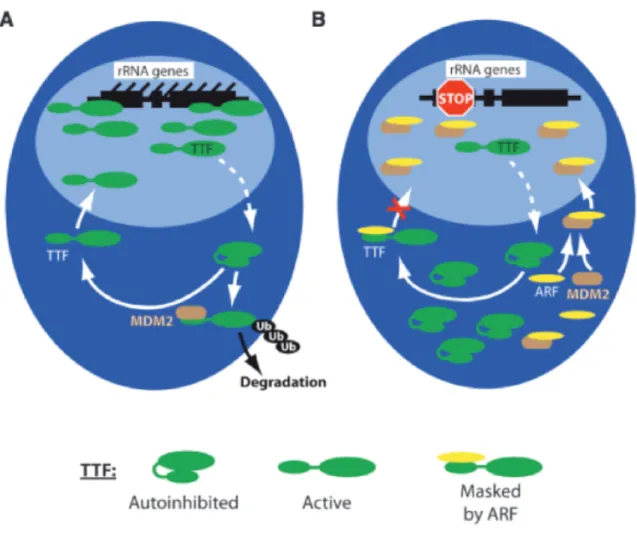
De nombreux éléments sont nécessaires afin de contrôler le cycle cellulaire comme l'accessibilité aux nutriments, les voies de la croissance cellulaire ou encore l'environnement physico-chimique. L'intégration de ces différents stimuli externe permet de contrôler finement la division cellulaire. Ce phénomène est central dans la compréhension de l'organisation du vivant en général puisque les organismes doivent pouvoir s'adapter à des variations de disponibilité des nutriments, des variations de température, de pH etc. La production des ribosomes est reliée directement aux différentes voies de signalisation qui impactent sur la division cellulaire. À titre d'exemple, en réponse aux stress, il y a accumulation de la protéine suppressive de tumeur P53 entraine la répression de la transcription ribosomique (Golomb, Volarevic, and Oren 2014; Núñez Villacís et al. 2018). Ce phénomène est connu sous le nom de réponse au stress nucléolaire (RSN). Il peut être causé autant par l'accumulation des protéines ribosomales que par l'inhibition de l'ubiquitinylation de P53 par MDM2 (figure 1.5).
Plus généralement, au début des années 2000, des études ont montré que le nucléole était un lieu de localisation de nombreuses protéines qui ne sont pas toutes impliquées dans la formation du ribosome. Ces études ont donné lieu à l'élaboration d'une banque de données de au moins 4500 protéines identifiées dans le nucléole humain (Scherl 2002; Andersen et al. 2002; Ahmad et al. 2009). On parle désormais pour le nucléole de centre censeur de stress de la cellule.
Figure 1-4 : Régulation de P53 par l’ubiquitine-ligase MDM2 A. P53 est régulé
négativement en condition normale par MDM2 qui entraine son export nucléaire et sa dégradation cytoplasmique. B. L'accumulation des protéines ribosomiques permet l'accumulation de P53 en séquestrant MDM2 entrainant l'arrêt du cycle cellulaire ou la mort cellulaire. (Adapté avec autorisation de : Núñez Villacís et al. 2018)
1.2. L'ADN ribosomique
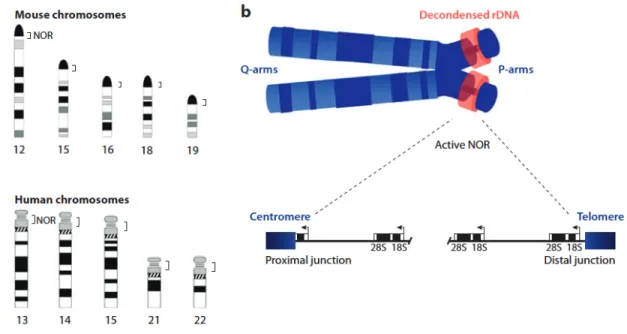
Le gène codant les ARNr est un gène répété dans tout le règne du vivant. Chez les eucaryotes, on dénombre environ 100 copies par génome haploïde pour la levure et environ 200 chez la souris ou l'humain (Hernandez-Verdun, Roussel, and Gébrane-Younès 2002). Ces dénombrements sont sujets à controverses à cause de variabilités potentielles ainsi que du peu de précision des mesures (Gibbons et al. 2014). Chez les mammifères, ils sont regroupés sur les bras courts des chromosomes acrocentriques (figure 1.6.). Les gènes ribosomiques sont situés au niveau de locus spécifiques aussi appelés régions organisatrices des nucléoles (RONs) (Kurihara et al. 1994). La population des gènes codant les ARNr est plus simplement nommée l'ADN ribosomique (ADNr). Cette nomination concerne tous les gènes ribosomiques peu importe leur localisation, elle regroupe les gènes d’ARNr peu importe leur niveau d’expression.
Figure 1-5 : Localisation chromosomique des RONs chez l’humain et la souris. A.
Représentation des chromosomes acrocentriques chez la souris (haut) et chez l'humain (bas). B. Représentation du locus ribosomique sur un chromosome acrocentrique. Les unités de transcriptions sont orientées dans le même sens et séparées l’une de l’autre par une séquence inter-génique (Adapté avec autorisation de : McStay and Grummt 2008)
L'ADNr est divisé conventionnellement en différentes catégories suivant l'état transcriptionnel des gènes d'ARNr. On distingue les gènes constitutivement réprimés (hétérochromatiniens), les gènes potentiellement transcrits et les gènes transcrits (euchromatiniens). Les gènes constitutivement réprimés sont suspectés être méthylés sur les sites CpG (Cytosine phosphate Guanine) des régions transcrites et régulatrices (Santoro and Grummt 2001; Christine Mayer et al. 2006). Les deux autres populations diffèrent de par leur activité mais il reste difficile de les distinguer à l'heure actuelle. Ces différents aspects sont développés dans la partie 3 de l’introduction (Épigénétique).
1.2.1.
Organisation
1.2.1.1. Régions d’organisation des nucléoles
Les RONs chez la souris sont situés sur les bras courts des chromosomes 12, 15, 16, 18 et 19. Au sein de ces RONs, les gènes codants les ARNr possèdent une organisation spatiale particulière. Ils s’organisent à la suite les uns des autres dans la même orientation. Le site d’initiation de la transcription orienté vers les télomères et les régions terminatrices vers le centromère du chromosome.
Chaque gène est séparé du suivant par des zones nommées Séquences InterGéniques (SIG). Ces SIG sont des zones répétées de l’ADN comportant les séquences de régulation de la transcription que sont le promoteur et le terminateur et également des sites permettant de réguler la réplication de l’ADNr (Gerber et al. 1997; Putter and Grummt 2002).
1.2.1.2. Le gène d’ARN ribosomique
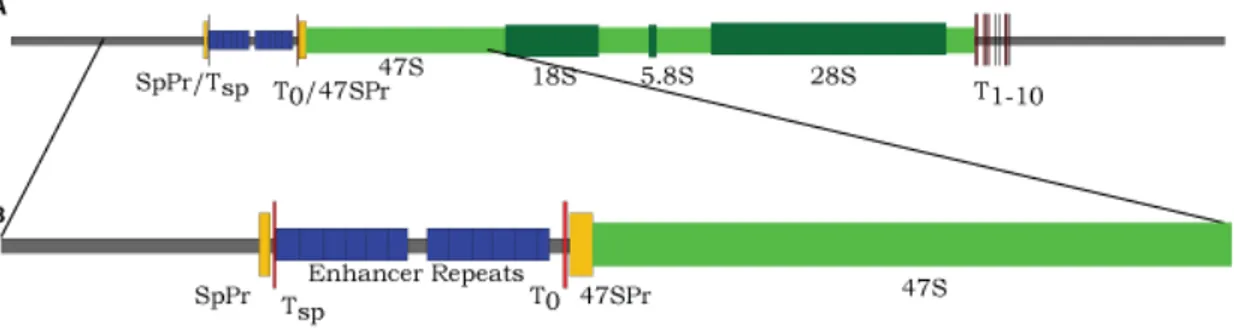
Le gène codant les ARNr est organisé classiquement avec une région régulatrice, une région transcrite comprenant des régions de types introns et 3 exons correspondant aux ARN 18S, 5.8S puis 28S organisés l'un à la suite de l'autre et séparés par des séquences transcrites internes (STI), cette région transcrite mesure 13403 paires de bases (pb) et elle est représentée en vert sur la figure 1.7. La partie finale du gène est une région appelée terminatrice (t1-10 sur
la figure 1.7), elle est constituée d'un site répété 10 fois chez la souris appelé Sal-box. La région régulatrice en amont est dite promotrice. Elle comprend une région majeure que j'appellerai le promoteur 47S ou promoteur principal et qui est le promoteur minimal nécessaire à l'initiation de la transcription. Il est situé juste en amont du site d'initiation de la transcription (SIT) et est lui-même composé de deux éléments distincts, le promoteur principal (core Promoter) et l'UCE ou UBE (Upstream Core Element/ Upstream Binding Element).
Juste en amont du promoteur se trouve un site de type Sal-box identique à la répétition identifiée au niveau du terminateur, il est appelé T0. La région en
bleu sur la figure 1.7 est composée d'environ 1770pb. Elle est nommée enhancer repeats et est composée de 12 répétitions. L’enhancer repeat a été impliquée dans l'augmentation de la transcription chez la grenouille Xenopus laevis, chez la souris et chez la drosophile (Tom Moss 1983; Paalman, Henderson, and Sollner-Webb 1995; Grimaldi, Fiorentini, and Di Nocera 1990). Finalement cette région se situe en amont d'un élément appelé Spacer Promoter (SpPr) qui a été identifié comme important dans la stimulation de la transcription en complément de l'enhancer repeats (De Winter and Moss 1986, 1987). La région du SpPr a également été montrée comme active transcriptionellement. De cette région est issue un petit ARN, le pARN (promoteur ARN) nommé ainsi en raison de sa complémentarité avec la séquence promotrice (Christine Mayer et al. 2006). Il
permet le remodelage de la chromatine par le complexe NoRC (Nucleolar Remodelling Complex) qui induit entre autre la propagation de la méthylation de l'ADNr à travers les cycles cellulaires (Santoro and Grummt 2001; Christine Mayer et al. 2006; Savić et al. 2014). Cette région est séparée de l'enhancer repeats par un site de type Sal-box appelé Tsp (Terminator Spacer Promoteur). Ensemble ils permettent une production efficace des ARNr et constituent donc une région importante dans la régulation de la transcription.
Figure 1-6 : Structure du gène codant les ARN ribosomiques chez la souris. En
jaune les régions promotrices (Spacer Promoter et promoteur principal), en rouge les sites Sal-box, en bleu l’enhancer repeats, en vert la région transcrite et en vert sombre les régions codantes des 3 différents ARN ribosomiques. A. Schéma global du gène. B. Agrandissement de la région promotrice.
1.2.2.
Transcription
Il existe 4 différents ARNr nommés en fonction de leur coefficient de sédimentation dans des conditions standardisées (S: Svedberg) 5S, 5.8S, 18S et 28S. Trois d'entre eux sont synthétisés par l'ARN polymérase de type I à partir de l'ADNr. L'ARNr 5S est produit en dehors du nucléole à partir d'un gène spécifique transcrit par PolIII pour être acheminé par la suite vers la composante granulaire ou il entrera dans la composition du ribosome. Les ARNr 5.8S, 18S et 28S sont synthétisés à partir d'un seul gène sous forme d'un précurseur 47S. Par la suite ce précurseur est maturé par différents facteurs comme les snoRNP (small nucleolar Ribo-Nucleo-Protein), des complexes nucléolaires permettant la modification du précurseur des ARNr. Finalement les ARN 5S, 5.8S et 28S rentreront dans la composition de la grande sous-unité ribosomale (le complexe 60S) en complexe avec 49 protéines ribosomales. L'ARN 18S rentrera dans la composition de la petite sous-unité ribosomale (40S) avec 33 protéines
ribosomales. Les deux sous-unités agiront de concert lors de la traduction des ARNm sous la forme du ribosome (80S).
1.2.2.1. Les Facteurs Généraux de la Transcription
Dans les mammifères, il existe trois composés essentiels fonctionnant de concert avec l'ARN polymérase de type I pour produire les ARNr: SL-1 ou également TIF1B, UBF, Rrn3 aussi nommé TIF1A. Tous ces facteurs ont été identifiés en tant que facteur d'initiation de la transcription ribosomique. En plus de ces facteurs, il existe un facteur impliqué dans la terminaison de la transcription appelé TTF-1. La majorité des connaissances à propos de ces facteurs concernant leurs mécanismes d’action sont connus grâce à des expériences in vitro. Transposer ces données dans un contexte in cellulo a été la motivation majeure de mon travail de doctorat. Tous ces facteurs sont considérés ou démontrés comme essentiels grâce aux lignées déplétées précédemment pour une sous-unité essentielle de PolI (Rpa194), pour UBF et pour Rrn3. Tous ces modèles murins ont montré le même arrêt développemental très tôt dans l’embryogénèse (stade 3.5 chez la souris) mettant ainsi en exergue l’aspect absolument fondamental de ces facteurs.
1.2.2.1.1. L'ARN polymérase I:
RPI ou encore PolI est une ARN polymérase dépendante de l’ADN spécifique de la transcription des ARNr chez les eucaryotes. La PolI est un complexe multi-protéiques de 14 sous-unités dont 12 sont communes avec les ARN polymérases de type II et III, les deux sous-unités restantes étant spécifiques à PolI. Cette enzyme est spécifique au nucléole, elle existe sous au moins deux formes. La première est une forme majoritaire appelée PolIα (90%) et est incompétente dans l'initiation de la transcription. En revanche, elle est suspectée de représenter en partie la population d’ARN polymérases en cours d'élongation.
Figure 1-7 : Image originale de microscopie électronique de la technique de Miller
spread sur des ovocytes de Triturus viridescens (Adapté de : O. L. Miller and Beatty 1969). S désigne la séquence inter-génique et M la partie transcrite.
La deuxième forme nommée β correspond à la forme compétente pour l'initiation est se distingue entre autre par sa liaison a un facteur de transcription: Rrn3 (voir partie suivante pour plus de détails). Cette forme représente, suivant les estimations, de 1% à 10% de la PolI isolée des nucléoles (Revue: Russell and Zomerdijk 2005). Les structures de PolIα (Engel et al. 2013) et plus récemment celle de PolIβ (Engel, Plitzko, and Cramer 2016) en complexe avec Rrn3 ont été décrite. La structure résolue est celle d'un dimère de PolI et il a été suggéré que c'est cette forme, libre dans le nucléoplasme, qui serait la forme "inactive" de PolI alors que celle liée à Rrn3 serait la forme compétente pour l’initiation (Woods et al. 2015). Les observations précédentes suggèrent que la forme en cour d’élongation représenterait une forme à part.
La distribution des PolI sur les gènes codant les ARNr est connue depuis longtemps grâce à la technique de Miller Spread (figure 1.8). La structure visible en "arbre de noël" représente le gène en train d'être transcrit, le tronc étant l'ADNr avec les PolI accrochées et chaque branche est un ARN en cours de transcription et de maturation (les petites boules sont des facteurs impliqués dans la
maturation des ARNr). Il existerait donc probablement au moins 3 formes de PolI: une forme dimérique non active, une deuxième compétente pour l'initiation qui est monomérique et liée à Rrn3 et enfin une troisième forme liée à l'ADN ribosomique et à l'ARN ribosomique en cours d'élongation. Dans le contexte de la transcription, ceux sont les deuxième et troisième formes que j’ai davantage étudiées étant donné les techniques employées.
1.2.2.1.2. Rrn3/TIF1A:
Rrn3 ou TIF1A est un facteur qui a été identifié lié à PolI sous sa forme β d'abord dans la levure comme un facteur essentiel de la transcription ribosomique (Milkereit and Tschochner 1998). Grâce à la résolution des structures cristallographiques chez la levure de PolI et de PolI-Rrn3, une idée plus précise du rôle de Rrn3 a émergé. Dans la levure, il semblerait que Rrn3 empêche la dimérisation de PolI déplaçant ainsi l’équilibre entre les dimères de PolI stockée sous forme inactives et une forme liée à Rrn3 compétente pour l’initiation de la transcription (Engel, Plitzko, and Cramer 2016; Pilsl et al. 2016). Il a également été montré que la phosphorylation de Rrn3 était directement impliquée dans la régulation de la transcription des gènes codant les ARN ribosomiques en partie en régulant l'équilibre entre les deux formes de PolI non liées aux gènes ribosomiques (Cavanaugh et al. 2002; Cavanaugh, Evans, and Rothblum 2007). La phosphorylation de Rrn3 est également suspectée de réguler les interactions entre SL-1 est Rrn3. Il est également possible que Rrn3 puisse interagir directement avec l'ADN au niveau du promoteur des gènes ribosomiques (Stepanchick et al. 2013). Les données semblent suggérer que Rrn3 est nécessaire uniquement au recrutement de PolI active mais que son absence n'impacte pas le recrutement de PolI au niveau du promoteur de l'ADNr (Schnapp et al. 1993). Plus récemment Rrn3 a été identifié, avec PolI ainsi que les autres FGTs, associé au SpPr des gènes codant les ARNr suggérant que la présence du Complexe de Pré-Initiation (CPI) à cet endroit-là est également fonctionnel en matière de transcription (Hamdane et al. 2014).
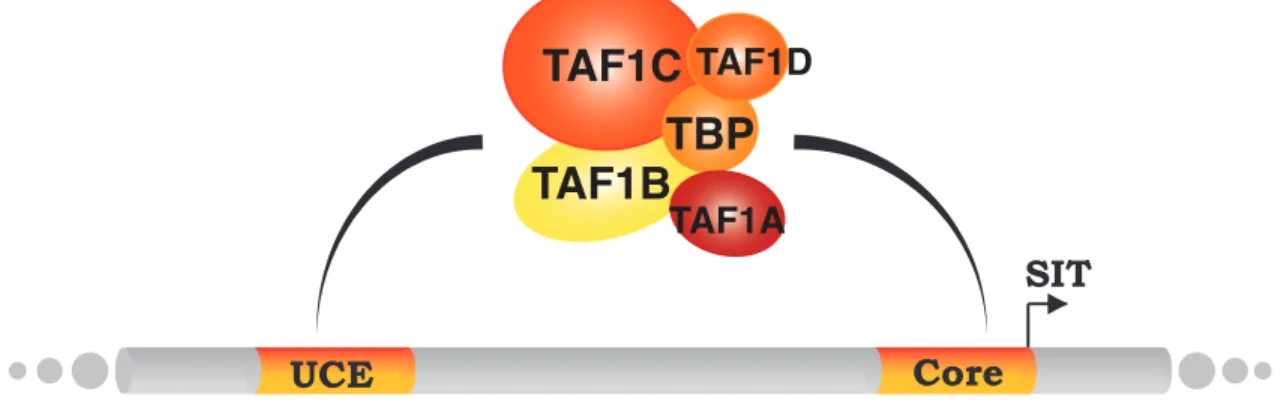
1.2.2.1.3. SeLectivity factor 1 (SL-1)
Chacune des différentes polymérases possède des facteurs propres qui lui sont spécifiques. Ils sont également appelés facteur de sélectivité parce qu'ils
permettent la formation des différents complexes de pré-initiation au niveau des sites spécifiques à chacune d'entre elles. Le facteur de sélectivité de PolI est nommé SL-1 (ou TIF-1B). C'est un complexe multi-protéique composé de TBP (Tata-box Binding protein) et de, au moins, 4 différents TAF (TBP Associated Factor (41, 48, 68, 95) également nommées respectivement TAF1A, TAF1B, TAF1C) (Comai, Tanese, and Tjian 1992; Gorski et al. 2007). Pour PolII, TBP permet la spécificité de séquence au niveau de certains promoteurs et les TAFs apportent la spécificité vis-à-vis de l’enzyme. L'ARN polymérase de type III possède différents types de promoteurs également et les TAFs apportent la spécificité du complexe. Ici, malgré le caractère essentiel de TBP, il n’est pas défini que c’est la protéine TBP qui permet l’interaction spécifique avec la séquence promotrice. En revanche, le facteur TAF48 permet le recrutement de PolI en interagissant avec Rrn3 et donc permettrait le recrutement des PolI actives (G. Miller et al. 2001).
Figure 1-8 : Schéma de la structure de SL-1 chez la souris. SL-1 est composé de
TBP et de au moins 4 facteurs associés. Il reconnait à la fois la séquence UCE (Upstream Core Element) et le Core promoteur en amont du SIT.
Il existe un débat sur l’interaction de SL-1 avec le promoteur des gènes codant les ARNr. En effet, des études in vitro ont montré que SL-1 seul permet d’avoir une transcription active de ces gènes. Cependant, un autre facteur a été montré important dans la formation d’une structure particulière au niveau du promoteur principal sur laquelle je reviendrai dans la partie suivante. Le modèle en figure 1.9 présente le modèle de fixation de SL-1 qui reconnait autant l'élément UCE que le Core promoteur. En réalité, 2 modèles de reconnaissance éxistent. Soit 2 complexes SL-1 reconnaissent indépendamment l’UCE et le Core element,
UCE
Core
SIT
TAF1B
TBP
TAF1C
TAF1A
TAF1D
soit une structure de l’ADN permet de rapprocher les deux éléments et un seul complexe suffit à reconnaître les 2 éléments.
La fixation de SL-1 ainsi que son interaction avec Rrn3/PolI sont régulées par différentes voies de signalisation comme la voie mTOR ou MAP kinase mais également par des kinases spécifiques du cycle cellulaire (Heix et al. 1998)
1.2.2.1.4. Upstream Binding (transcription) Factor (UBF/ UBTF)):
Le facteur UBF a également été montré in vitro comme important dans la fixation de SL-1 (Learned et al. 1986; Friedrich et al. 2005; Tuan, Zhai, and Comai 1999). UBF est une protéine nucléaire qui a été impliquée dans la régulation de la transcription des ARNr. Elle est codée par un gène unique dont la délétion entraîne une létalité embryonnaire très tôt au cours du développement (Hamdane et al. 2014).
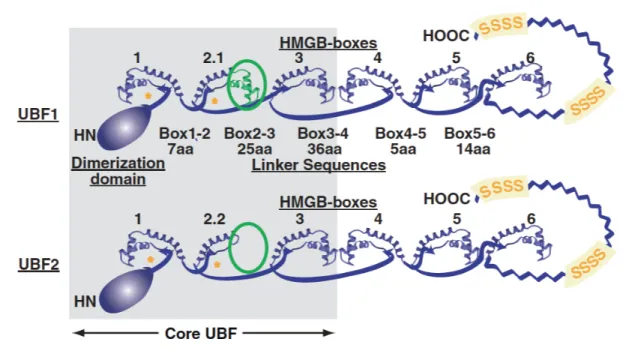
Figure 1-9 : Structure schématique d’UBF de souris. Les variants 1 et 2 d'UBF ne
diffèrent que partiellement par la présence dans UBF-1 d’une région suplémentaire dans la 2ième boite HMG. UBF est composé d’un domaine de dimérisation, de 6
domaines HMG-box reliées par des portions non structurées. En C-terminal, UBF possède un domaine acide (indiqué par les S des Sérines). Adapté avec autorisation de : Victor Y. Stefanovsky and Moss 2008)
UBF est présent dans les mammifères sous forme de deux variants notés UBF-1 ou UBF-2 (voir Figure 1.10). UBF-1 est une protéine composée de 6 domaines de type HMG-boxes (High Mobility Group), d'une région N-terminale de type Homéo domain-like permettant la dimérisation d'UBF (homo ou hétéro) et en C-terminal d'une région riche en acide aminé acides. UBF-2 est issu du même gène qu’UBF-1, c’est un variant d’épissage auquel il manque une série de 30 acides aminés dans la deuxième boîte HMG. Cette différence n’impacte pas sa capacité de dimérisation ni ses fonctions in vitro cependant il a été démontré qu’UBF-2 pourrait avoir un rôle différent d’UBF-1 en servant de facteur de transcription pour certains gènes transcrits par PolII en particulier des gènes codant pour des histones (Sanij et al. 2015). Les boites HMG sont reliées entre elles par un morceau d’ADN non structuré. UBF est capable de courber l'ADN grâce à ces boites HMG de manière non spécifique le long d’une séquence d’ADN (Copenhaver et al. 1994).
Figure 1-10 : Structure schématique de l’enhancesome et modèle hypothétique du
promoteur principal. À gauche, la structure déterminée par microscopie électronique et à droite le repliement possible du promoteur principal par deux enhancesomes induit par la fixation d’UBF. UBF est en bleu et l’ADN est en rouge, les astérisques (*) indiquent les sites de phosphorylation par ERK. À droite le modèle hypothétique proposé de rapprochement de l’UCE (jaune et noir) et du Core Promoteur (jaune) grâce à la juxtaposition de deux dimères d’UBF (Adapté avec autorisation de : T. Moss et al. 2007).