Université de Sherbrooke
EFFETS DES RADIATIONS GAMMA ET DES ELECTRONS DE
BASSE ENERGIE SUR LA FONCTIONNALITE DE L’ADN
Par
SALOUA SAHBANI
Programme en Sciences des Radiations et Imagerie Biomédicale
Thèse présentée à la Faculté de Médecine et des Sciences de la Santé en vue de l’obtention du diplôme de philosophiae doctor (Ph.D.) en sciences des radiations et imagerie biomédicale, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4
Sherbrooke, Québec, Canada
August 2014
Membres du jury:
Professor Darel Hunting
Encadreur, Sciences des Radiations et Imagerie BiomédicaleProfessor Leon Sanche
Encadreur, Sciences des Radiations et Imagerie BiomédicaleProfessor Brigitte Guerin
Présidente, Sciences des Radiations et Imagerie BiomédicaleProfessor Jean Cadet
Évaluateur externe à l’Université, CEA, Grenoble, FranceProfessor Gylain Boissonneault
Évaluateur externe au département, Département de Biochimie, Université de SherbrookeProfessor Jean Paul Jay-Gerin
Évaluateur interne au département, Sciences des Radiations et Imagerie BiomédicaleDEDICACE
A mes parents Salah et Jannette
pour leur tendresse et leur sacrifice
A mon mari Salah pour
son encouragement
A mes enfants Rania et Rabai
A mes frères et sœurs Sami, Bachar, Samira et
ma plus belle sœur Amel
pour leur affection
A toute la famille et mes amis
RÉSUMÉ
EFFETS DES RADIATIONS GAMMA ET DES ÉLECTRONS DE BASSE
ÉNERGIE SUR LA FONCTIONNALITE DE L’ADN
Par Saloua Sahbani
Programme en sciences des radiations et imagerie biomédicale
Thèse présentée à la Faculté de Médecine et des Sciences de la Santé en vue de l’obtention du diplôme de philosophiae doctor (Ph.D.) en sciences des radiations et imagerie biomédicale,
Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4
Il est généralement admis que les cassures double-brin (CDB) de l’ADN sont parmi les lésions les plus toxiques induites par les radiations ionisantes (RI). Les CDBs non ou mal réparées peuvent conduire à une instabilité génomique et à la mort cellulaire. La chimioradiothérapie concomitante est l’une des modalités la plus efficace pour le traitement de certains cancers surtout en stade avancé. Le rendement des CDBs a augmenté quand l’ADN a été irradié en présence de cisplatine avec des électrons de basse énergie (EBEs). Notre étude a pour objectif de réévaluer la contribution des CDBs et d’autres lésions induites par les RI dans la létalité cellulaire. L'effet des RI sur la fonctionnalité de l’ADN plasmidique modifié ou non de façon covalente par le cisplatine a été étudié par mesure de l'efficacité de transformation du plasmide dans E. coli. Les complexes cisplatine-ADN ont été préparés de telle sorte qu’il y avait en moyenne deux adduits de cisplatine par plasmide tel que mesuré par ICP-MS. Nos échantillons ont été irradiés en solution avec des doses croissantes de rayonnements gamma (137Cs). La présence de cisplatine a augmenté la formation des CDBs par un facteur de 2.6 par comparaison avec l'ADN non modifié. Malgré cette augmentation, le rendement des CDBs reste très faible et ne peut pas expliquer la perte de fonctionnalité observée. Alors que, les dommages multiples localisés (LMDS) (non-DSB cluster damage) donnant naissance à des CDBs sous l’action des enzymes de réparation la formamidopyrimidine [fapy]-DNA
glycosylase (Fpg) et l’endonuclease III (Nth) où leur rendement a été augmenté d’un facteur de
2.1 lorsque l’ADN a été irradié en présence de cisplatine, ont pu expliquer la perte de fonctionnalité observée. Ces résultats suggèrent que le cisplatine peut agir, non seulement comme un agent chimiothérapeutique, mais aussi comme un radiosensibilisateur efficace par addition d’autres lésions à l’ADN. Aussi, pour la première fois nous avons pu évaluer l’effet des EBEs sur la létalité cellulaire. Des films d'ADN ont été préparés en utilisant la méthode d’adsorption douce sur un substrat de graphite pyrolytique, en présence de 1,3- diaminopropane (Dap2+) et ont été irradiées avec des EBEs 10 eV. Nous avons pu conclure, qu’en plus des CSBs, CDBs et des dommages de base, les EBEs sont capables aussi d’induire des LMDS (non-DSB cluster damage) et induire la perte de fonctionnalité de l’ADN. Le rendement des CDBs est très faible d’où ils n’ont pas pu expliquer la perte de fonctionnalité de plasmide observée, après irradiation avec les EBEs. Le rendement très faible des LMDS (non-DSB
cluster damage) ne peut pas expliquer la perte de fonctionnalité de l’ADN. Il semble que les
EBEs sont capables d’induire des dommages très proches les uns des autres et qui ne peuvent pas être révélés par les enzymes de réparation Fpg et Nth. Plus les dommages sont proches les uns des autres, plus leur réparation est difficile, car une de ces lésions peut inhiber la réparation de l’autre la plus proche.
Mots-clés: Rayonnements gamma, électrons de basse énergie, cisplatine, fonctionnalité de l’ADN, cassure double-brin, LMDS (Non-DSB cluster damage), radiosensibilisation.
IV
ABSTRACT
EFFECT OF GAMMA RADIATION AND LOW ENERGY ELECTRON
ON THE DNA FUNCTIONALITY
By Saloua Sahbani
Program of Radiation Sciences and Biomedical Imaging
Thesis submitted to the Faculty of Medicine and Health Sciences for the degree of Doctorate of Philosophy (PhD) in Radiation Sciences and Biomedical Imaging, Faculty of Medicine and
Health Sciences, Université de Sherbrooke, Sherbrooke, Québec, Canada
It is generally accepted that DNA double-strand breaks (DSB) are among the most toxic lesions induced by ionizing radiation (IR). Unrepaired or misrepaired DSB can lead to genomic instability and cell death. It is known that concomitant chemoradiation therapy is one of the most preferred methods for the treatment of certain cancers especially in advanced stage. The yield of DSBs was increased when DNA was irradiated with low energy electron (LEEs). The aims of our study was to reassess the contribution of DSBs and other lesions induced by indirect and direct effect of IR in cell lethality. The effect of IR on the DNA functionality of the plasmid modified covalently with cisplatin was studied by measuring the transformation efficiency of the plasmid in E. coli. Cisplatin-DNA complexes were prepared such that there was an average of two cisplatin adducts per plasmid as measured by ICP-MS. Aqueous solutions of the samples were irradiated with 137Cs -rays at various doses. Gel electrophoresis analysis shows that cisplatin enhances, by a factor of 2.6, the formation of DSB by -rays relative to those in unmodified DNA. Despite this increase, the yield of DSBs is very low and cannot explain the loss of functionality observed after transformation with plasmids modified with cisplatin. While locally multiple damaged sites (LMDS) revealed by repair enzymes Fpg (Formamidopyrimidine [fapy]-DNA glycosylase) and Nth (Endonuclease III) as DSB
(non-DSB cluster damage), where their yield was increased by a factor of 2.1 when DNA was
irradiated in the presence of cisplatin were able to explain the observed loss of DNA functionality. These results suggest that cisplatin may act not only as a chemotherapeutic agent, but also as an effective radiosensitizer by addition of other DNA lesions.
For the first time, we could also evaluate the effect of low energy electrons (LEEs) on DNA functionality. Highly ordered DNA films were prepared on pyrolytic graphite by molecular self-assembly using 1,3-diaminopropane ions (Dap2+) to bind together the plasmids and irradiated with LEE (10 eV). We concluded that in addition to CSBs, DSBs and base damage, LEEs induced the formation of non-DSB cluster damage and also induced the loss of DNA functionality under LEE irradiation. The yields of DSBs and of non-DSB cluster damage are too low and so one unable to explain the loss of DNA functionality. It seems that LEEs are able to induce a high complex damage that cannot be revealed by repair enzymes Fpg and Nth. The high complex damage is difficult to repair possibly because the repair of one lesion, may inhibit the repair of another.
Keywords: Gamma radiation, Low energy Electron, cisplatin, DNA functionality, Double strand break, LMDS (Non-DSB cluster damage), Radiosensitization.
TABLE DES MATIÈRES
LISTE DES TABLEAUX VII
LISTE DES FIGURES VIII
LISTE DES ABBRÉVIATIONS XV
I. INTRODUCTION 1
I.1. Effets chimiques et biologiques des radiations ionisantes 1 I.1.1 Effets chimiques des radiations ionisantes 1
I.1.1.1. Effets indirects des radiations ionisantes: la radiolyse de l’eau
1 I.1.1.2. Effets directs des radiations ionisantes 3 I.1.2. Effets biologiques des radiations ionisantes: les dommages
à l’ADN
3
I.1.2.1. Les dommages de bases 4
I.1.2.2. Les cassures simple et double-brin de l’ADN 4 I.1.2.3. Les pontages de l’ADN 6 I.1.2.4. Les dommages multiples localisés 8 I.2. Les dommages à l’ADN induit par les electrons de basse énergie 12 I.3. Réparation des cassures double-brin 13 I.3.1. Voie de réparation par recombinaison homologue (RH) 13 I.3.1.1. Chez les eucaryotes 13 I.3.1.2. Chez les procaryotes: Echerichia coli 14 I.3.2. Voie de réparation par jonction des extrimités non-homologues 16
I.3.2.1. Chez les eucaryotes 16 I.3.2.2. Chez les procaryotes: Echerichia coli 17
I.4. Chimioradiothérapie 19
VI
II.1. Premier article
The relative contributions of DNA strand breaks, base damage and clustered lesions to the loss of DNA functionality induced by ionizing radiation.
23
II.2. Deuxième Article
Non-DSB clustered DNA lesions induced by ionizing radiation are largely responsible for the loss of plasmid DNA functionality in the presence of cisplatin.
57
II.3. Troisième article
Cell death induced by low-energy (10 eV) electrons measured by the loss of plasmid DNA functionality
87
III. DISCUSSION
III.1. Les cassures double-brin de l’ADN ne sont pas responsables de la
perte de fonctionnalité de l’ADN 118
III.2. Rôle des cassures double-brin de l’ADN ainsi que les dommages multiples localisés (non-DSB cluster damage) dans la perte de fonctionnalité de l’ADN induite par les électrons de basse énergie (10 eV)
120
IV. CONCLUSION 123
REMERCIEMENTS 126
LISTE DES TABLEAUX Chapitre I : Introduction
Table 1 Réparation de dommages multiples localiés de l’ADN: lesions en
Tandem
10 Chapitre II: Premier Article
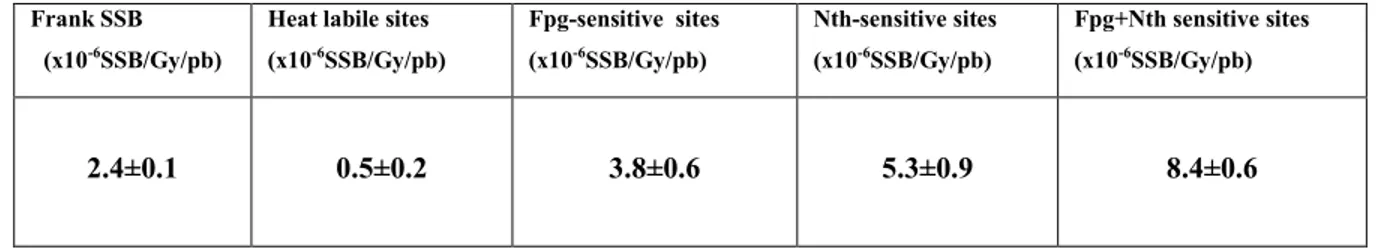
Table 1 Yields of SSB induced in pGEM-3-Zf (-) plasmid DNA by gamma-irradiation followed by incubation with Nth and Fpg (Yields of SSB/Gy/bp).
33
Table 2 Yields of DSB induced in pGEM-3-Zf(-) plasmid DNA by gamma-irradiation followed by incubation with Nth and Fpg (Yields of DSB/Gy/bp).
34
Chapitre II: Deuxième Article
Table 1 Yields of SSB induced in pGEM-3-Zf (-) plasmid DNA unmodified or modified by cisplatin by gamma-irradiation followed by incubation with Nth and Fpg (Yields of SSB/Gy/bp).
63
Table 2 Yields of DSB induced in pGEM-3-Zf (-) plasmid DNA unmodified or modified by cisplatin by gamma-irradiation followed by incubation with Nth and Fpg (Yields of DSB/Gy/bp).
65
Chapitre II: Troisième Article
Table 1 The yield for SSB induction in pGEM-3-Zf (-) plasmid DNA by 10 eV-electrons irradiation followed by incubation with Nth and Fpg (x10-16 cm2/ plasmid).
96
Table 2 The yield for DSB induction in pGEM-3-Zf (-) plasmid DNA by 10 eV-electrons irradiation followed by incubation with Nth and Fpg (x10-16 cm2/ plasmid).
LISTE DES FIGURES Chaptire I
Figure I.1 Formation de pontage interbrin (5) entre le radical 5-(2′-deoxyuridinyl) methyl (1) et le 2′-deoxyadenosine qui se trouve sur le brin opposé.
7
Figure I.2 Les symboles ouverts et solides sont les rendements quantiques mesurés (événements/ électron incident) pour l'induction respectivement des CSBs (a) et CDBs (b) de l’ADN.
13
Figure I.3 Réparation des cassures double-brin par recombinaison homologue chez les eucaryotes.
14
Figure I.4 Réparation des cassures double-brin par recombinaison homologue chez les procaryotes (E.coli).
15
Figure I.5 Réparation des cassures double-brin par la ligature d’extrémité non-homologue (JENH) chez les eucaryotes.
17
Figure I.6 Réparation des cassures double-brin par la ligature d’extrémité non-homologue (JENH) chez les procaryotes (A) et par la ligature d’extrémité non-homologue alternative (JENH-A) chez E. coli (B).
18
IX
Figure 1 Agarose gel electrophoresis of gamma-irradiated extracellular plasmid pGEM-3Zf(–). Panel a: Laser scan of agarose gel after electrophoresis to separate the different forms of the plasmid. The DNA standards are in the left lane “S”. Panel b: The loss of supercoiled DNA ( ) and the formation of circular ( ) and linear (▴) DNA as a function of irradiation dose (Gy).
31
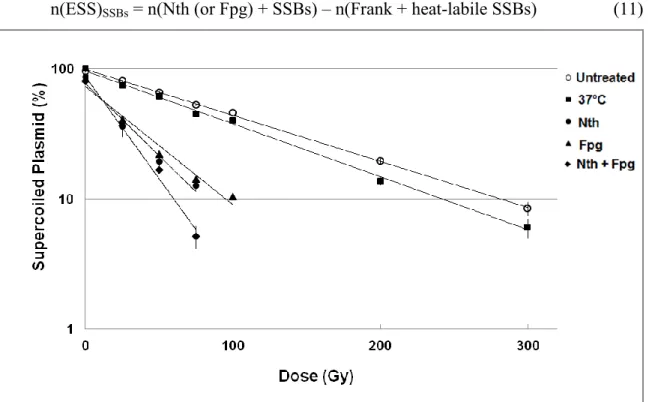
Figure 2 Loss of Supercoiled pGEM-3Zf (-) DNA (10 ng/µl) versus dose for DNA irradiated in the presence of phosphate buffer (10 mM) with γ-rays. After irradiation DNA was untreated (open circles) or incubated at 37°C (closed squares) or incubated with Nth (closed circle) or Fpg (closed triangles) or both enzymes Nth and Fpg (closed diamonds).
32
Figure 3 Dose response plots showing the fractions of linear DNA. After irradiation DNA was untreated (solid lines) or incubated at 37°C (closed squares) or incubated with Nth (closed circle) or Fpg (closed triangles) or both enzymes Nth and Fpg (closed diamonds).
X
Figure 4 Impact of gamma-irradiation on transformation efficiency. The continuous line is calculated with equation 10. The best fit to the data is when the ratio of ρC /ρS = 3, ρL = 0, n = 3 and βR = (60 ± 2) x 10-4/Gy/plasmid. The values for βS and βD, and the value for ρ, were determined by gel electrophoresis from the data presented in Fig. 1b. The data are the mean of three independent experiments.
35
Figure 5
Figure 6
Impact of DNA form on transformation efficiency. SC: supercoiled DNA form; C: circular DNA form generated by digesting supercoiled plasmid with Nb.BtsI, and L: linear DNA form generated by digesting supercoiled plasmid with EcoRI. The data are the mean of three independent experiments.
Biological activity of plasmid pGEM-3Zf (-) following gamma-irradiation. The “survival function” (dashed curve) of the plasmid is calculated using equati on 10. The dotted line represents the variation in the “entry function” as a function of radiation dose (equation 10). The behaviour is due to the change in the number of plasmids with different entry coefficients. For comparison, the formation of linear plasmid as a function of dose (taken from Figure 1b) is also shown (straight line). Inset shows the functionality of plasmid in the logarithmic scale.
36
37
XI
Figure 1 Fig. 1A: Effect of gamma-irradiation on transformation efficiency of plasmid pGEM-3Zf (-) DNA unmodified or modified by cisplatin. The curves for Unmodified DNA (continuous line) and modified DNA by cisplatin (dashed line) were calculated with equation 1. The best fit to the data is when the ratio of ρC /ρS = 3,
ρL = 0, n = 3 and βR equal to (60 ± 2) x 10-4 per Gy per plasmid
and (90 ± 5) x 10-4 per Gy per plasmid respectively for unmodified or DNA modified by cisplatin. The values for βS and
βD, and the value for ρ, were determined by gel electrophoresis
from the data presented in Fig. 2B. The data are the mean of three independent experiments.
56
Fig. 1B: Biological activity of plasmid pGEM-3Zf (-) DNA unmodified or modified by cisplatin following gamma-irradiation. Fig.1B1 represents the “survival function” (DNA unmodified (continuous line) and modified with cisplatin (dashed curve)) of the plasmid calculated using equation 1. Fig. 1B2 represents the variation in the “entry function” as a function of radiation dose (equation 1) for both DNA unmodified (continuous curve) or modified with cisplatin (dashed curve). The behaviour is due to the change in the number of plasmids with different entry coefficients. The inset shows the functionality of the plasmid using tradional logarithmic scale.
XII
Figure 2 Agarose gel electrophoresis of gamma-irradiated extracellular plasmid pGEM-3Zf (-) unmodified or modified by cisplatin. (A) Laser scan of agarose gel following electrophoresis to separate the different forms of the plasmid. The DNA standards are in the left lane (S). (B) Exposure-response curves for the loss of supercoiled DNA and the formation of circular and linear DNA unmodified (continuous line) or modified by cisplatin (dashes line).
59
Figure 3 Fig. 3A: Loss of Supercoiled pGEM-3Zf (-) DNA unmodified or modified by cisplatin versus dose for DNA irradiated in the presence of phosphate buffer (10 mM) with γ-rays. After irradiation DNA was untreated and incubated at 37°C (1) or incubated with Nth (2) or Fpg (3) or both enzymes Nth and Fpg (4). Fig. 3b: Dose response plots showing the fractions of linear DNA unmodified or modified by cisplatin. After irradiation DNA incubated at 37°C (1) or incubated with Nth (2) or Fpg (3) or both enzymes Nth and Fpg (4) at 37°C.
60
Figure 4 Summary of Poisson distribution of cluster damage and loss of plasmid functionality with increasing dose of gamma radiation in DNA unmodified and modified by cisplatin. Fig. 4A and 4B represent the number of plasmids having respectively 2 or more and 3 or more separate DNA cluster damage.
64
XIII
Figure 1 Impact of 10 eV-electrons irradiation on transformation efficiency. The continuous line is calculated with equation 1. The best fit to the data is when the ratio of ρC /ρS = 3, ρL = 0, n = 3 and
βR = (1.2 ± 0,1) x 10-14 cm2 per plasmid. The data are the mean of
three independent experiments.
91
Figure 2 Biological activity of plasmid pGEM-3Zf (-) following 10 eV-electrons irradiation. The “survival function” (dashed curve) of the plasmid is calculated using equation 1. The dotted line represents the variation in the “entry function” as a function of electron fluence (equation 1). The behaviour is due to the change in the number of plasmids with different entry coefficients. For comparison, the formation of linear plasmid as a function of electron fluence (taken from Figure 3b) is also shown (straight line). The Inset shows the functionality of plasmid DNA on a logarithmic scale.
92
Figure 3 Agarose gel electrophoresis of 10 eV-electrons irradiated extracellular plasmid pGEM-3Zf (-).The loss of supercoiled DNA () and the formation of circular () and linear () DNA as a function of electron fluence.
94
Figure 4 Loss of Supercoiled pGEM-3Zf (-) DNA versus electron fluence. After irradiation DNA was untreated (closed circles) or incubated at 37°C (closed triangles) or incubated with Nth (Asterix) or Fpg (solid lines) or both enzymes Nth and Fpg (plus).
XIV
Figure 5 Dose response plots showing the fractions of linear DNA. After irradiation DNA was untreated (close circles) or incubated at 37°C (closed triangles) or incubated with Nth (Asterix) or Fpg (solid lines) or both enzymes Nth and Fpg (plus).
LISTE D’ ABBRÉVIATIONS
γ Gamma
8-oxo-G 8-oxo-guanine
ADN Acide désoxyribonucléique
AP Site abasique
BER Réparation d’excision de base
137Ba Baryum 137 C Circulaire 60Co Cobalt 60 137Cs Césium 137 CSB Cassure simple-brin CDB Cassure double-brin D Dose Dap2+ 1,3- diaminopropane dT Thymidine e-aq Électron aqueux e- Électron
EBEs Électrons de basse énergie
E. coli Echerichia coli
EDTA Ethylène Diamine Tétra acétique
Eq Equation
ESs Électrons secondaires
eV électronvolt Fe2+ Fer ferreux Fe3+ Fer ferrique Fpg Formamidopyrimidine N glycosylase Gy Gray H Atome d’hydrogène .H Radical d’hydrogène
XVI
H2O Eau
H2O.+ Eau cationique
H2O* Eau excitée
H2O2 Peroxyde d’hydrogène
hOGG1 The human 8-oxoguanine DNA N-glycosylase 1 JENH Jonction des extrémités non-homologues
JENH-A Jonction des extrémités non-homologue-Alternatives
keV Kiloélectron-volt
L Linéaire
LB Luria-Bertani
LMDS Locally multiply damaged site
MeV Mégaélectron-volt MS Spectrométrie de Masse mC Méthyl cytocine ng Nanogramme 60Ni Nickel 60 nm Nanomètre Nth Endonucléase III .OH Radical hydroxyle OH- Ion hydroxyde
Phase G2 Phase Gap (= Intervalle)
Phase M Phase de Mitose
Phase S Phase de Synthèse
Pb Paire de base
RI Radiation ionisante
RH Recombinaison homologue
S Surenroulée
TE Éfficacité de Transformation
TEL Transfert d’énergie linéique
INTRODUCTION I. INTRODUCTION
I. Introduction
I.1. Effets chimiques et biologiques des radiations ionisantes I.1.1. Effets chimiques des radiations ionisantes
Les rayonnements ionisants déposent suffisamment d’énergie dans la matière qu’ils pénètrent pour créer une ionisation. Il existe des rayonnements directement ionisants où les particules chargées (électrons et protons) constituent le rayonnement ionisant en lui-même et les rayonnements indirectement ionisants où le rayonnement incident, constitué de particules non chargées (photons et neutrons) qui produisent des ionisations par l’intermédiaire des particules qu’elles mettent en mouvement.
I.1.1.1. Effet indirect des radiations ionisantes: la radiolyse de l’eau Au cours de ma thèse j’ai utilisé comme source de dommages à l’ADN les rayonnements γ. Les rayonnements γ sont constitués de photons caractérisés par leur énergie inversement proportionnelle à leur longueur d’onde. Le rayonnement γ provient de désintégrations nucléaires et son énergie est caractéristique de cette réaction (la désintégration β du 137Cs en 137Ba est accompagnée d’une émission de γ de 660 keV et celle du 60Co en 60Ni est accompagnée d’une double émission de γ de 1.17 et 1.33 MeV). En traversant la matière, les photons déposent de l’énergie d’une manière qui varie avec la profondeur.
Lors de l’interaction entre un photon de haute énergie et la matière, quatre types d’interaction peuvent être rencontrés dont leur répartition dépend de l’énergie des photons et aussi du matériau traversé (Attix, 1991).
-Pour des énergies inférieures à 50 keV, l’effet photoélectrique domine. Il est lié à l’absorption de l’énergie d’un photon incident par un électron du cortège électronique d’un atome. Ce dernier se trouve ainsi éjecté et le phénomène peut s’accompagner de la production d’électrons Auger.
L’effet Compton est dominant lorsque les énergies sont comprises entre 50 keV et 20 MeV.
Il résulte d’une collision élastique du photon avec un électron des couches périphériques de l’atome cible et conduit à l’éjection de ce dernier qui est doté d’une énergie dont le photon
diffusé sera privé. Un autre phénomène se produit lorsque les énergies sont supérieures à 1.02 MeV, c’est la production de paires, le photon ainsi interagit avec le noyau des atomes créant ainsi l’émission d’une paire de particules électron-positron (β+,β-).
L’eau joue un rôle très important dans les phénomènes d’irradiation indirecte, il représente plus que 70% de la masse des organismes vivants. Suivant leur énergie, les radiations ionisantes interagissent avec l’eau par des phénomènes d’excitation électronique et d’ionisation.
La dissociation des molécules d’eau ionisées donne naissance à une molécule d’eau cationique et un électron (1).
La réaction (1) est suivie par la formation d’un radical hydroxyle(.OH) car l’eau cationique est un acide fort qui perd rapidement un proton au profit de l’eau environnante (2).
Les électrons arrachés (1) sont capables de parcourir certaines distances car ils sont doués d’une énergie cinétique initiale qu’ils vont perdre par collision. Ces électrons ralentis sont piégés par les molécules d’eau donnant naissance aux électrons hydratés e-aq (3).
Alors que, les molécules d’eau excitées peuvent soit se dissocier et donner naissance à deux radicaux .OH et .H (4), ou ils peuvent perdre leur énergie d’excitation et donc retourner alors à leur état d’énergie fondamentale.
En plus de la formation de radicaux très réactifs à la suite de la radiolyse de l’eau comme les radicaux hydroxyles, les radicaux ainsi formés par ionisation ou excitation peuvent se recombiner et donner naissance à l’eau oxygénée (H2O2) qui pourra participer à la formation
de radicaux hydroxyles à l’intérieur de la cellule en présence de Fe2+ (5) ou Cu+ (Réaction
de Fenton).
INTRODUCTION I.1.1.2. Effet direct des radiations ionisantes
Prenons l’exemple d’un rayonnement qui est constitué d’électrons qui se déplacent à grande vitesse et qui peuvent être produits par des accélérateurs. En traversant un milieu donné, ces électrons perdent progressivement leurs énergies cinétiques par interaction avec le cortège électronique ou le noyau des atomes qui constituent ce milieu. Les électrons du milieu sont mis en mouvement à cause du transfert de l’énergie. Ce transfert d’énergie peut être suivi par ionisation des molécules du milieu quand l’énergie échangée est supérieure à l’énergie de liaison de l’électron. Ces molécules ionisées par les électrons libérés peuvent créer à leur tour des ionisations en cascade. En effet, les interactions entre l’électron incident et les électrons de la cible peuvent être soit inélastiques ou élastiques.
I.1.2. Effets biologiques des radiations ionisantes: les dommages à l’ADN L’irradiation ionisante peut induire des lésions létales aux cellules et aussi des lésions sublétales ; ces dernières pourront se transformer en lésions létales par accumulation. L’ADN est considéré comme la cible critique des radiations ionisantes (Bernhard & Close, 2003). Les radicaux libres générés par la radiolyse de l’eau à la suite d’une irradiation par des rayonnements γ de l’ADN en solution aqueuse diluée sont capables d’induire plusieurs dommages à l’ADN (von Sonntag, 1987), comme les cassures simple et double brin de l’ADN, les dommages de base: oxydation ou perte (sites abasiques), les pontages intra- ou interbrins ADN-ADN ou les pontages ADN-protéines (Ward, 1988). Les radicaux hydroxyles jouent un rôle important dans l’induction de ces dommages surtout quand l’irradiation est effectuée en milieu oxygéné (Breen & Murphy, 1995a). Les doubles liaisons des bases jouent un rôle crucial dans la réaction des radicaux hydroxyles avec les bases de l’ADN puisque 80% de ces radicaux réagissent avec l’ADN en s’additionnant sur ces doubles liaisons et 20% seulement vont arracher un atome d’hydrogène des sucres induisant ainsi la formation d’autres radicaux (Bernhard & Close, 2003).
Certains paramètres influent le nombre et la distribution des dommages induits par les RI à l’ADN comme le transfert d’énergie linéique (TEL) du rayonnement ainsi que la forme de l’ADN et d’autres comme la température et surtout la présence de radioprotecteurs ou radiosensibilisateurs (Teoule, 1987, Frankenberg-Schwager, 1990).
I.1.2.1. Les dommages aux bases
Les RI sont capables d’induire des dommages isolés similaires aux lésions induites par le métabolisme oxydatif, tel que des lésions de bases puriques et pyrimidiques par arrachement d’un atome d’hydrogène (.H) ou surtout par addition sur les carbones des liaisons π, mais ils peuvent induire même la perte de base et donc la formation de site abasique (AP) (Kuwabara, 1991, Breen & Murphy, 1995b). Les méthodes chromatographiques ainsi que la spectrométrie de masse (MS) sont utilisées pour détecter et identifier les dommages radioinduits aux bases. Les oxydations de bases sont les lésions les plus fréquentes et les plus variées (Cadet et al., 1997a, Spotheim-Maurizot et al., 2008). L’oxydation des bases pyrimidiques proviennent principalement de l’attaque des radicaux
.OH des cycles aromatiques surtout en position C5 et C6 (von Sonntag, 2006, Cadet et al.,
2010). De plus, les bases puriques peuvent être oxydées par les radicaux .OH par des attaques sur des atomes C4, C5 et C8 dont le produit le plus fréquent est le 8-oxo-7,8 dihydroguanine (8-oxo-G) (Cadet et al., 1997b, Douki & Cadet, 1999, Cadet et al., 2002). Les conséquences majeures des lésions aux bases sont les mutations surtout la 8-oxo-G (Shibutani et al., 1991, Moriya et al., 1991, Moriya, 1993). Des attaques aussi avec les radicaux .OH sur les atomes du sucre en position C1’, C2’ et C5’ou sur la base peuvent également conduire à la perte de la base et donc la formation de sites AP par la rupture de la liaison N-glycosidique sans que le squelette phosphodiester soit brisé en milieu neutre. Ces dommages sont appelés aussi «sites alcali-labiles», car quand le milieu est alcalin, un traitement avec pipéridine des coupures peut être observé (Chung et al., 1992, Cadet et al., 1997a).
I.1.2.2. Les cassures simple et double- brin de l’ADN
L’abstraction d’un atome H du sucre par .OH peut induire la coupure de la liaison phosphodiester entre le phosphate et le 2-désoxyribose (Miaskiewicz & Osman, 1994, Colson & Sevilla, 1995, Pogozelski & Tullius, 1998). La CSB résulte d’une coupure d’un des deux brins d’ADN. Les atomes hydrogène du 2-désoxyribose situés en position C4’ et C5’ sont les plus accessibles aux attaques radicalaires et la formation de coupures est plus probable à ces positions (von Sonntag, 1987, Sy et al., 1997, Pogozelski & Tullius, 1998, Aydogan et al., 2002, Evans et al., 2004). Une cellule irradiée avec un rayonnement ionisant
INTRODUCTION de faible TEL pourra avoir jusqu'à 1000 CSB Gy-1 et 2000 bases endommagées Gy-1 (Goodhead, 1994a).
Une CDB est une rupture de deux chaines d’ADN à des sites distants de moins de 10 paires de base (pb) (Vogin, 2011). Un seul radical .OH peut induire une CDB par attaque de 2-désoxyribose et transfert du radical sur le deuxième brin (Aslam Siddiqi & Bothe, 1987). Aussi, les CDBs peuvent être induites par des attaques par plusieurs radicaux dans des zones rapprochées (Ward, 1985).
Les CDBs sont considérées comme étant les plus toxiques et si elles sont mal ou non réparées, elles peuvent être létales (Ward, 1988, Goodhead et al., 1993, Rydberg et al., 1994, Little, 2000, Trevor et al., 2001, Little, 2002). Les CDBs peuvent être à l’origine d’une perte des chromosomes, des mutations et des réarrangements chromosomiques et donc d’instabilité génomique (Iliakis, 1991a, Dikomey et al., 1998a, Hoeijmakers, 2001, Elliott & Jasin, 2002, Varga & Aplan, 2005), de l’arrêt de cycle cellulaire ainsi que de l’apoptose (Rich et al., 2000, Norbury & Hickson, 2001, Jackson, 2002). Les CDBs radioinduites ne sont pas facilement réparables (extrimités modifiées). Après irradiation, ces CDBs jouent donc un rôle déterminant dans la survie cellulaire (Dikomey et al., 1998b, Iliakis, 1991b). Diverses méthodes sont utilisées pour détecter et quantifier les CDBs, tel que l’électrophorèse en champ pulsé (Blocher & Kunhi, 1990), les techniques d’élution neutre sur filtre (Kohn & Grimek Ewig, 1973).
Les radiations ionisantes, et plus spécifiquement les radicaux .OH formés par la radiolyse de l’eau, peuvent générer des structures chimiques complexes au niveau des bouts de l’ADN libérés par la cassure (Rass et al., 2012) empêchant ainsi les enzymes de réparation et plus particulièrement les enzymes de ligation de terminer la dernière étape de réparation et donc la légation de deux extrémités par l’ADN-Ligase (Rass et al., 2012). La correction de ces bouts constitue ainsi une étape cruciale et très peu d’enzymes sont connus aujourd’hui pour jouer ce rôle. La radiosensibilité cellulaire a été corrélée avec le taux de CDBs non réparées. Une seule CDB est suffisante pour induire la létalité chez les bactéries déficientes en systèmes de réparation des CDBs (Freifelder, 1965). En plus, Kemp et ces collaborateurs ont montré que les organismes comme les cellules de rongeurs ou les levures déficientes en système de réparation des CDBs sont les plus radiosensibles (Kemp et al., 1984).
I.1.2.3. Les pontages de l’ADN
Les RI peuvent induire trois types de pontages: intra- et interbrins ADN-ADN et ADN-protéines. Les radicaux .OH peuvent être responsables des pontages ADN-ADN (von Sonntag, 1987, Cadet et al., 1997c) et aussi des pontages ADN-protéines (Cadet et al., 1997c). En effet, l’attaque radicalaire d’une des bases de l’ADN ainsi qu’un acide aminé de protéines qui se trouve proche de l’ADN peuvent induire la formation d’une liaison covalente entre les radicaux intermédiaires ainsi formés à partir de la base et de l’acide aminé (Cadet et al., 1997c).
a- Les pontages intra- et interbrins de l’ADN
Les pontages intrabrins, surtout les pontages entre les bases adjacentes (lésion en tandem), peuvent être induits par les rayonnements γ (Bellon et al., 2002, Gu & Wang, 2004, Zhang & Wang, 2005). Des pontages intrabrins entre la base purique guanine (G) et 5-methylcytosine (mC) ont été observés après exposition aux rayonnements γ avec un rendement 10 fois plus élevé lorsque l’ADN est irradié en absence plutôt qu’en présence d’oxygène (Zhang & Wang, 2005). En milieu anaérobique, le pontage intrabrins mC^G est 10 fois plus faible que la lésion G^mC (Zhang & Wang, 2005). Il est connu que les pontages interbrins sont induits à des fréquences très faibles par les RI (Hong & Greenberg, 2005, Ding & Greenberg, 2007). Ces dommages sont très toxiques, car ils peuvent inhiber la réplication et la transcription en cas de non réparation (von Sonntag., 2006). Ding et Greenberg, (2007) ont montré que le radical 5-(2′-deoxyuridinyl) methyl qui est formé par l’abstraction d’un atome d’hydrogène du groupement méthyl de thymidine (dT) joue un rôle très important dans la formation des pontages interbrins (Figure 1) (Hong & Greenberg, 2005, Ding & Greenberg, 2007). Price et al., ont pu montrer que des pontages interbrins peuvent être formés entre des résidus adénine et des sites abasiques avec un rendement assez remarquable (15-70%) (Price et al., 2014).
INTRODUCTION
Figure 1: Formation de pontage interbrin (5) entre le radical 5-(2′-deoxyuridinyl) méthyle (1) et le 2′-désoxyadénosine qui se trouve sur le brin opposé (Ding & Greenberg, 2007). Figure reproduite avec la permission d’American Chemical Society Publications.
b- Les pontages ADN-protéine
Des liaisons covalentes entre l’ADN et les protéines nucléaires peuvent être induites par les RI (Oleinick et al., 1990). Les acides aminés: tyrosine et lysine jouent un rôle déterminant dans la formation de ces liaisons. L’irradiation de cellules de mammifères avec des doses faibles de RI peut provoquer la formation de pontages ADN-protéines avec une fréquence quatre fois plus élevée que les CDBs (Barker et al., 2005). Des pontages entre la tyrosine et la cytosine, la tyrosine et la thymine et la lysine et la thymine ont été mis en évidence (Margolis et al., 1988, Dizdaroglu et al., 1989, Gajewski & Dizdaroglu, 1990, Weir Lipton et al., 1996).
I.1.2.4. Les dommages multiples localisés
En plus des lésions simples comme les CSBs et les dommages aux bases isolées purines et pyrimidine, les RI sont capables d’induire aussi des dommages multiples localisés (LMDS= Locally multiply damaged site). En 1987, John Ward est le premier qui a introduit le concept de dommages multiples (Ward et al., 1987). Ward définit les LMDS comme deux ou plusieurs lésions formées sur un ou deux pas d’hélice de l’ADN le long de la trajectoire d’une seule particule ionisante (Ward, 1994, Goodhead, 1994b). Il y a différents types de LMDS: les dommages multiples incluant des CDBs (Schema 1) et d’autres n’incluant pas de CDBs (Schema 2) (Eccles et al., 2011). Pour ces deux types de dommages, il y a les dommages multiples simples ou complexes et on parle de lésions simples lorsqu’il y a seulement deux lésions situées sur les deux brins d’ADN et de lésions complexes lorsque plusieurs dommages (<2) sont situés sur les deux brins d’ADN (Nikjoo et al., 2001, Datta et al., 2005). On parle de lésion en tandem lorsque les deux bases oxydées par exemple sont adjacentes (Eccles et al., 2011). L’un des mécanismes réactionnels proposé pour ce dernier type de dommage est le suivant, un radical attaque une base, il peut induire un dommage simple et peut former aussi un radical intermédiaire (radical peroxyle) qui est capable d’attaquer une autre base adjacente (In et al., 2007).
INTRODUCTION
Schémas 1 et 2: les lésions multiples simples et complexes.
Il est difficile de caractériser les dommages multiples en irradiant des cellules à cause de la complexité des dommages induits à l’ADN par les RI et de la variété de protéines qui sont impliquées dans l’initiation de la réparation (Sage & Harrison, 2011).
Il est aussi difficile de déterminer comment chaque lésion est réparée et si la réparation d’une lésion est prévenue et de montrer comment elle a été inhibée (Sage &
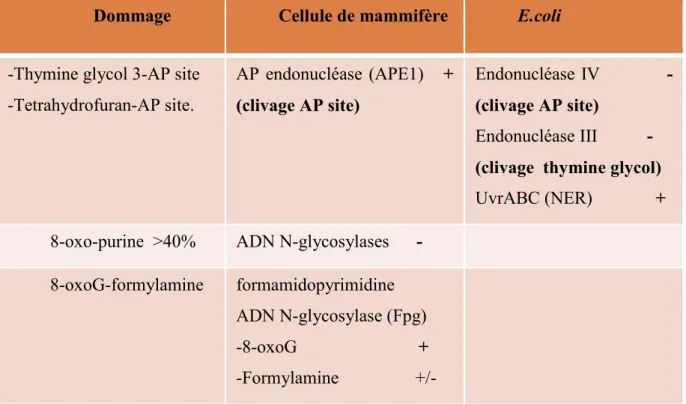
Harrison, 2011). Pour surmonter toutes ces difficultés, les chercheurs ont utilisé des oligonucluéotides ou des plasmides sur lesquelles ont été synthétisées des lésions simples ou complexes en utilisant des enzymes de réparation d’origine mammifères ou bactériennes (Sutherland et al., 2003, Barnes & Lindahl, 2004). Différentes méthodes ont été utilisées pour la détection des dommages multiples localisés, comme par exemple la chromatographie liquide couplée à la spectrométrie de masse pour détecter les guanines adjacentes oxydées et les dérivés formamide des pyrimidine dans l’ADN de thymus de veau irradié avec rayonnement X (Box et al., 1995, Douki et al., 2002), l’électrophorèse sur gel d’agarose combiné avec un traitement enzymatique (Georgakilas et al., 2004) et aussi le test des Comètes qui a été adapté pour mesurer les dommages multiples oxydatifs (Blaisdell & Wallace, 2001). En utilisant des bactéries E. coli, des études in vitro ont démontré que les lésions en tandem sont difficiles à réparer et une de ces deux lésions peut inhiber la réparation d’une autre (Imoto et al., 2008). En effet, la présence de thymine glycol 3 ou tetrahydrofurane en tandem avec un site abasique (AP) peut inhiber l'enzyme endonucléase IV- AP de couper au niveau de site AP et l’endonucléase III de couper au niveau de thymine glycol chez E. coli. Par contre, l’endonucléase AP de cellules des mammifères est capable de couper au niveau de site AP (Tableau 1) (Imoto et al., 2008).
Tableau 1: Réparation de dommages multiples localisés de l’ADN: lésion en Tandem.
Dommage Cellule de mammifère E.coli
-Thymine glycol 3-AP site -Tetrahydrofuran-AP site. AP endonucléase (APE1) + (clivage AP site) Endonucléase IV -(clivage AP site) Endonucléase III - (clivage thymine glycol) UvrABC (NER) + 8-oxo-purine >40% ADN N-glycosylases -
8-oxoG-formylamine formamidopyrimidine ADN N-glycosylase (Fpg) -8-oxoG + -Formylamine +/-
INTRODUCTION Une partie des 8-oxo-purines forment des lésions en tandem et plus que 40 % de ces dommages ne peuvent pas être réparés par la N-glycosylases. Par exemple, une 8-oxo-G qui se trouve en tandem avec une formylamine; la 8-oxo-G ne peut être réparée par la formamidopyrimidine N glycosylase (Fpg) et la formylamine est plus ou moins réparée par cette enzyme (Imoto et al., 2008).
Lorsque la 8-oxo-G se trouve en tandem avec la lésion formylamine, ≈ 70% de ces 8-oxo-G ne vont être pas réparées mais elles vont être présentes pendant la réplication de l’ADN (Box et al., 2000). Aussi, ≈ 45% de la formylamine vont être introduit pendant la réplication par insertion d’une adenine en face de cette lésion. Il est connu que la formylamine est très mutagène quand elle est formée à partir de l’oxydation d’une cytosine (Box et al., 2000). L’insertion de lésion en tandem pendant la réplication diminue de 17% lorsqu’une adenine est insérée en face d’une formylamine et une cytosine en face d’une 8-oxo-G (Box et al., 2000).
Chez les cellules déficientes en système de réparation par jonction des extrémités non-homologue (JENH), ≈ 10% de lésions multiples n’incluant pas de CDBs induites par l’irradiation γ sont transformées en CDBs après 30 min (Gulston et al., 2004). L’augmentation de létalité, mutagénicité et les CDBs au niveau de cellules irradiées peuvent être à l’origine de l’hyper-expression des enzymes de réparation comme hNTH1 (human endonuclease III) et hOGG1 (human 8-oxoguanine DNA N-glycosylase 1) (Yang et al., 2004). L’hyper-expression de hOGG1 est à l’origine de l’augmentation de mutation Hprt (Paul et al., 2006).
La létalité et la mutagénicité des dommages complexes peuvent aussi être expliquées par l’inhibition de la réparation d’une lésion par la présence d’une autre (lésion en tandem) (Lomax et al., 2004). Comme par exemple la réparation d’un site AP ou une CSB peut être difficile ou même inhibée par la présence d’une base endommagée qui se trouve à 5 pb de ces deux lésions. Encore, la nature de la lésion de base peut influer la réparation de ces deux lésions. Comme par exemple, la 8-oxo-G et le glycol de thymine sont responsables de la réduction 2 à 8 fois de l’efficacité de la réparation des CSBs qui se trouvent sur le brin opposé de l’ADN (Lomax et al., 2004). Aussi, le résidu glycol de thymine inhibe l’activité de polymérase. Quand les lésions complexes incluent une base oxydée et un site AP sur un brin et une CSB sur le brin opposé, elles se trouvent en orientation négative ; ces deux
lésions sont ainsi initiées davantage avec le système de réparation d’excision de base (BER) short patch, alors que si ces lésions complexes sont en orientation positive, la réparation de site AP/CSB est initiée par les deux voies «long» et «short» patch BER (Lomax et al., 2004).
I. 2. Les dommages à l’ADN induit par les électrons de basse énergie
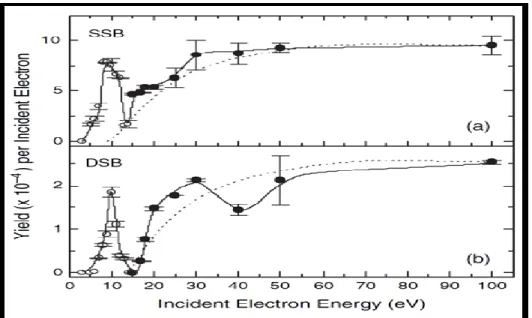
L’interaction des RI avec un milieu biologique, comme les cellules, produit des électrons secondaires (ESs), dont les plus abondants sont des électrons de basse énergie (EBEs). La plupart des électrons secondaires qui sont créés le long de la trace (4 x 104/MeV) ont une énergie inférieure à 30 eV (Kaplan & Miterev, 1985, Pimblott & LaVerne, 2007, Scifoni et al., 2010). Plusieurs études ont pu démontrer que les EBEs sont capables d’induire des dommages à l’ADN, surtout des CSBs et CDBs (Figure 2) (Barrios et al., 2002, Huels et al., 2003, Pimblott & LaVerne, 2007, Ptasinska & Sanche, 2007, Sanche, 2009,). En plus de l’induction des cassures, les EBEs peuvent aussi générer des dommages aux bases de l’ADN (Abdoul-Carime et al., 2001, Pan et al., 2003, Abdoul-Carime & Sanche, 2004) et même la perte de base et donc la formation de site AP (Sanche, 2009). Les CSBs peuvent être induites par des EBEs ayant des énergies de 0-10 eV, tandis que les CDBs peuvent être induites par des EBEs ayant des énergies de 5-10 eV. Ces énergies sont plus basses que celle du seuil d'ionisation de l'ADN (Figure 2). Ces résultats sont expliqués par un attachement de l'électron incident sur les différents composants de l'ADN (phosphate, sucre, base ou H2O) qui conduit à la formation d'un état d'anion moléculaire transitoire de courte
durée de vie, ce qu’on appelle résonance (Schulz, 1973). Malgré les grands progrès dans la compréhension de l’effet de RI sur la matière biologique, le rôle des ESs dans la létalité cellulaire est encore mal connu. La question qui a été posée au début de ce projet est celle-ci, est-ce que ces ESs, et plus particulièrement les EBEs, sont capables d’induire la létalité cellulaire comme les radicaux hydroxyles générés par l’effet indirect de l’RI de la radiolyse de l’eau? Si elles sont capables d’induire la létalité cellulaire, quels sont les mécanismes responsables de cette létalité?
INTRODUCTION
Figure 2: Les symboles ouverts et solides sont les rendements quantiques mesurés (événements/ électron incident) pour l'induction respectivement des CSBs (a) et CDBs (b) de l’ADN (Huels et al., 2003). Figure reproduite avec la permission d’American Chemical
Society Publications.
I.3. Réparation des cassures double-brin
I.3.1. Voie de réparation par recombinaison homologue (RH) I.3.1.1. Chez les eucaryotes
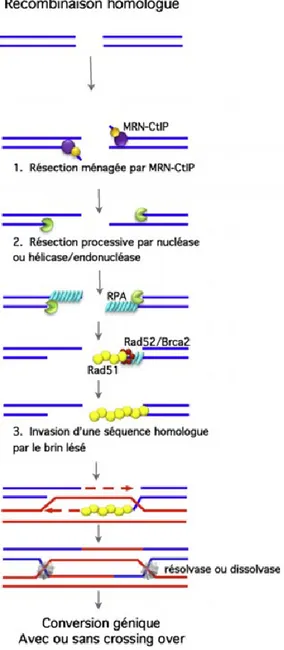
Lors de la réparation d’une CDB, la séquence de l’ADN endommagée peut être re-synthétisée à partir de la chromatide sœur ; ce mécanisme est appelé recombinaison homologue (RH). Il dépend de la phase du cycle cellulaire puisqu’il peut se dérouler uniquement pendant les phases S et G2. Cette voie de réparation est dite fidèle. Un complexe appelé MRN (Mre11-Rad50-NBS1) est responsable de la reconnaissance de la CDB (Huertas, 2010) (Figure 3). La résection des extrémités de la CDB de 5’ vers 3’ est effectuée par l’enzyme Mre11 (Huertas, 2010). La protection des extrémités 3’ sortantes de la dégradation par les exonucléases est assurée par les protéines RPA et Rad52. Rad52 joue un rôle très important dans le recrutement des différents partenaires tel que Rad51, acteur principal de la RH (Figure 3). Une étape cruciale de la RH est la recherche des séquences homologues sur les chromatides sœurs appariées, cette étape est assurée par la protéine Rad51, sous le contrôle de Brca1 et Brca2 (Bouwman et al., 2010, Bunting et al., 2010). Ensuite, des polymérases synthétisent la séquence d’ADN lésée en prenant comme matrice
la séquence homologue intacte sur la chromatide sœur. La dernière étape est la ligature de deux extrémités franches neoformées (Figure 3).
Figure 3 : Réparation des cassures double-brin par recombinaison homologue chez les eucaryotes (Rass et al., 2012). Figure reproduite avec la permission de
Cancer/Radiothérapie.
I.3.1.2. Chez les procaryotes: Escherichia coli
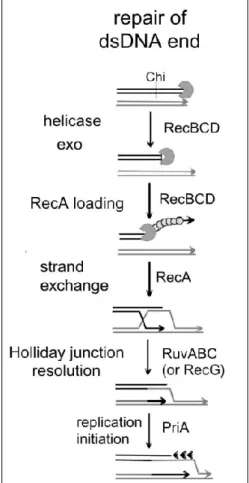
En 1960, Clark et al. ont mis en évidence la première recombinase: RecA chez E.
INTRODUCTION formation d’un filament nucléoprotéique suivie par la recherche de la séquence homologue et enfin l’échange de brins. Chez E.coli, l’enzyme RecBCD effectue l’ouverture de la double hélice au niveau de la CDB et la destruction d’un brin d’ADN pour la formation d’extrémités simple brin (Figure 4). Il peut aussi aider RecA à lier les extrémités simple brin. En effet, RecA joue le même rôle que RAD51 chez les eucaryotes. RecBCD joue le même rôle que l’ensemble Mer11 chez les eucaryotes (Wyman et al., 2004). Le RecA forme le filament nucléoprotéique ou filament présynaptique (Figure 4). Ce dernier est responsable de la recherche de la séquence homologue dans le génome. Une fois les deux séquences homologues alignées, les brins homologues peuvent être échangés lors de la synapse.
Figure 4 : Réparation des cassures double-brin par recombinaison homologue chez les procaryotes (E.coli) (Rocha et al., 2005). Figure reproduite avec la permission de PLoS
I.3.2. Voie de réparation par jonction des extrémités non-homologue (JENH) I.3.2.1. Chez les eucaryotes
Contrairement au mécanisme de réparation des CDBs par RH, le JENH peut intervenir tout au long du cycle cellulaire. Le complexe DNA-PK se compose de l’hétérodimère Ku70/Ku80 et de la protéine DNA-PKcs (DNA-dependent protein kinase, catalytic subunit) responsable de la reconnaissance de la lésion (Figure 5). Artemis est une nucléase recrutée par le complexe DNA-PK qui provoque la résection des oligonucléotides simple-brin situés aux extrémités de la brèche double-brin. L’ADN ligase IV associée aux protéines XRCC4 et XLF effectue la relégation des extrémités résultantes (Figure 5). Le mécanisme de réparation JENH est généralement infidèle, car l'étape de résection effectuée par Artemis implique une perte d’information génétique. Deriano et al., en 2005, ont démontré que les cellules déficientes en KU70/80 ou ADN-PK sont sensibles à la formation des CDBs induites par les RI et aussi aux agents utilisés en chimiothérapie (Deriano et al., 2005). C’est ce qui a motivé les chercheurs à développer des agents capables de cibler ADN-PK et certains d’entre eux sont testés aujourd’hui en clinique (Bolderson et al., 2009, Vogin, 2011).
INTRODUCTION
Figure 5 : Réparation des cassures double-brin par la ligature d’extrémité non-homologue (JENH) chez les eucaryotes (Rass et al., 2012). Figure reproduiteavec la permission de Cancer/Radiothérapie.
I.3.2.2. Chez les procaryotes : Escherichia coli
Les bactéries aussi ont un système de réparation par JENH (Weller et al., 2002). Weller et al., ont montré que les bactéries ont un hétérodimère Ku analogue à celui identifié chez les eucaryotes et qu’il est capable de recruter une ADN ligase à l’extrémité de l’ADN et donc de stimuler la légation (Weller et al., 2002). L’E. coli est incapable de réparer les CDB par le système de réparation JENH, car elle est dépourvue des protéines Ku et aussi de l’ADN ligase D qui sont indispensables pour ce type de réparation. En 2009, Chayot et ses collaborateurs ont mis en évidence qu’E. coli est capable de réparer les CDBs de l’ADN même en l’absence de deux systèmes de réparation par RH et JENH, grâce au système de réparation de ligature d’extrémités non homologues alternative (JENH-A) (Figure 6)
(Chayot et al., 2010). Ce système a été aussi mis en évidence chez la levure, les cellules de hamster ou de souris ou in vitro dans les extraits acellulaires ou d’œufs de xénope (Liang & Jasin, 1996, Boulton & Jackson, 1996, Kabotyanski et al., 1998, Göttlich et al., 1998, Feldmann et al., 2000). Une nucléase ou une hélicase initie le JENH-A en générant des extrémités d’ADN simple-brin (Figure 6). Au contraire de JENH où la protéine Ku et la ligase D sont présentes pour protéger les extrémités de l’ADN coupées, le JENH-A repose sur la dégradation des extrémités par le complexe nucléase/hélicase RecBCD (Chayot et al., 2010). L’étape suivante est la recherche de microhomologies de part et d’autre de la cassure, dont l’hybridation permettrait la ligature de l’ADN (Chayot et al., 2010, Rass et al., 2012). Des délétions peuvent être détectées lors de l’initiation de JENH-A, ce qui nous amène à conclure que ce mécanisme de réparation est mutagène et n’est pas conservatif (Rass et al., 2012).
Figure 6: Réparation des cassures double-brin par la ligature d’extrémité non-homologue (JENH) chez les procaryotes (A) et par la ligature d’extrémité non-non-homologue alternative (JENH-A) chez E. coli (B) (Chayot et al., 2010). Figure reproduite avec la permission de Proceedings of the National Academy of Sciences.
INTRODUCTION I.4. Chimioradiothérapie
La chimioradiothérapie est devenue le traitement le plus utilisé pour traiter de nombreux cancers tel que le cancer pancréatique ou du sein. Des résultats encourageants en clinique ont été observés lors de l’administration concomitante de deux modalités. En effet, il a été démontré que cette modalité de traitement induit une augmentation de la destruction des cellules tumorales. Une amélioration du contrôle locorégional de tumeurs, la préservation de l’organe et une amélioration de la survie des patients ont été constatées (Samant et al., 1999, Peters III et al., 2000, Candelaria et al., 2006, Salama et al., 2007, Boscolo-Rizzo et al., 2011). Ces résultats peuvent être la conséquence des modifications des lésions radio-induites ainsi que de leur réparation ou d’un spectre d’action différent en fonction des phases des cycles cellulaires (Hennequin et al., 2009). Les substances chimiothérapeutiques peuvent transformer une lésion facilement réparable en une lésion irréparable (Dolling et al., 1999). Prenant l’exemple de cisplatine qui est un complexe à base de platine, cette substance est capable de se fixer surtout sur des résidus guanine en formant des ponts intra- ou interbrins. Le cisplatine peut inhiber la réparation d’une CDB radioinduite (Begg, 1990, Yang et al., 1995). Une interaction supra-additive aussi a été démontrée lors de traitement concomitant de l’étoposide qui est un poison de topo-isomérase II-α et les RI surtout au cours du bloc en phase G2 du cycle cellulaire radio-induits (Giocanti N, et al., 2000). Le 5-fluorouracile a la capacité de radiosensibiliser les cellules tumorales en empêchant la réparation des lésions radio-induites, par la diminution du pool de dTTP (Bruso et al., 1990). La gemcitabine est un analogue pyrimidique connu pour sa large activité surtout contre les tumeurs solides et il a radiosensibilisé les cellules tumorales par inhibition de la ribonucléotide réductase (Shewach et al., 1994, Lawrence et al., 1996). Il est reconnu que la phase S du cycle cellulaire est la plus radiorésistante et au contraire la phase G2/M est la plus radiosensible (Sinclair & Morton, 2012). Une augmentation de la létalité cellulaire a été observée lors du traitement concomitant avec des agents cytotoxiques spécifiques de la phase S (la camptothécine et ses dérivés; poisons des topo-isomérases I) et de l’irradiation ionisante (Hennequin, et al., 1994). Le paclitaxel et le docétaxel sont deux substances capables de changer la dynamique des microtubules (Blagosklonny & Fojo, 1999). Ils ont pu bloquer la progression de la mitose à forte dose (Hennequin, 2004) et donc synchroniser le cycle cellulaire dans la phase la plus
radiosensible (Choy et al., 1993, Hennequin, 2004, Pawlik & Keyomarsi, 2004). Au niveau tissulaire, l’irradiation peut augmenter la captation cellulaire des agents cytotoxiques, probablement par augmentation de débit sanguin (5-fluorouracile) (Davis et al., 1995). Aussi, la diminution du volume tumoral après un traitement avec l’une des modalités peut améliorer la vascularisation et donc radiosensibiliser de plus en plus les cellules à cause de la réoxygénation des tissus (Milas et al., 1995, Mason et al., 1999).
Plusieurs études ont donc montré l’efficacité de l’utilisation concomitante avec la chimiothérapie et la radiothérapie dans le traitement de nombreux cancers mais les mécanismes spécifiques responsables des effets synergiques observés restent encore inconnus.
20 OBJECTIFS
Objectifs:
Les cassures double-brin (CDBs) de l’ADN sont considérées comme les lésions radioinduites les plus toxiques. Plusieurs études ont montré l’effet létal de ces lésions en cas de non réparation. L’hyperfocalisation sur ces lésions (CDBs) a peut-être retardé la mise en évidence d’autres lésions radioinduites aussi toxiques et létales que les CDBs, mais qui pourraient être induites avec des fréquences plus élevées.
Le premier objectif de cette thèse est donc de réévaluer la contribution des CDBs et d’autres lésions radioinduites dans la létalité cellulaire en utilisant un simple modèle, soit l’ADN (pGEM-3Zf(-)) irradié en solution avec des doses croissantes de rayonnements γ puis transformé à l’intérieur d’E. coli. La fonctionnalité des plasmides, qui représente la proportion de plasmides qui sont encore capables de se répliquer et d'exprimer le gène de résistance à l'ampicilline, pourra ensuite être évaluée indirectement par une mesure directe de l’efficacité de transformation (l'efficacité de transformation peut être séparé en deux composantes. La première composante, dite « fonction d'entrée», contient les probabilités ρS ρC, ρL et que l'ADN dont les formes topologiques: surenroulée, circulaire ou linéaire, respectivement, traversent la membrane cytoplasmique. Ces probabilités sont supposées ne dépendent que de la conformation de l'ADN, mais pas de la dose de rayonnement. La deuxième composante, dite «fonction de survie», représente la proportion de plasmides qui sont encore capables de se répliquer et d'exprimer le gène de résistance à l'ampicilline) en fonctionéon Sanche de la dose d’irradiation. Cette étude nécessitera l’utilisation de modèles mathématiques pour déterminer la fonctionnalité de l’ADN en fonction de la dose d’irradiation et la comparer avec les rendements des différents dommages tel que les CSBs, CDBs, les dommages aux bases puriques et pyrimidiques et les dommages multiples localisés (LMDS = non-DSB
cluster damage). Ces derniers pourront être révélés sous forme de CDBs par l’utilisation
d’enzymes de réparation tel que Fpg (Formamidopyrimidine [fapy]-DNA glycosylase) et Nth (Endonuclease III).
La chimioradiothérapie concomitante est l’une des modalités les plus utilisées pour le traitement de certains cancers surtout en stade avancé. Cependant, la contribution des mécanismes spécifiques responsables de l'efficacité de cette combinaison n'a pas encore
été déterminée. Le rendement des CDBs a augmenté quand l’ADN a été irradié en présence de cisplatine, qui est un complexe à base de platine. La question qu’on a posée est celle-ci, est-ce que cette augmentation de rendement de CDBs pourra expliquer la létalité cellulaire observée après un traitement combiné: RI et cisplatine?
La réponse à cette question été notre deuxième objectif pour évaluer la contribution des CDBs et d’autres lésions radioinduites dans la létalité cellulaire en utilisant le même modèle, soit l’ADN (pGEM-3Zf(-)), irradié en solution avec des doses croissantes de rayonnements γ puis transformé à l’intérieur d’E. coli. Mais cette fois-ci, l’ADN a été modifié avec du cisplatine (deux adduits de cisplatine par plasmide en moyenne).
Notre troisième objectif a été d’évaluer l’effet direct des RI dans la létalité cellulaire. Nous désirons utiliser les électrons de basse énergie (EBEs) comme un moyen d’irradiation. En effet, l’interaction des EBEs (EBE de 0-20 eV) avec l’ADN peut provoquer des dommages biologiques significatifs, sous forme de cassures simples et double brins, à des énergies en-dessous du seuil d’ionisation de l’ADN, qui est de 8 à 10 eV. Par contre, le rôle joué par les EBEs dans la létalité cellulaire est encore inconnu. C’est donc la première fois qu’on testera le rôle des EBEs dans la perte de fonctionnalité de l’ADN. C’est, pour cette raison, l’un de nos ultimes objectifs.
Des films d'ADN (pGEM-3Zf(-)) seront préparés en utilisant la méthode d’adsorption douce sur un substrat de graphite pyrolytique, en présence de 1,3- diaminopropane (Dap2+) et seront irradiés avec une source d’électrons de 10 eV, dans des conditions d’hypervide pendant des temps variables. En effet, l’uniformité des films joue un rôle important dans la réussite de cette expérience, plus le film est uniforme plus nos électrons intéragissent avec l’ADN et donc plus on aura des plasmides attaqués par ces électrons. Pour cette raison, nous désirons bien irradier notre ADN en présence de Dap2+, afin d’obtenir des films les plus uniformes possible.
RÉSULTATS: PREMIER ARTICLE
Article 1:
The relative contributions of DNA strand breaks, base damage and
clustered lesions to the loss of DNA functionality induced by ionizing
radiation.
Kouass Sahbani S. , Girouard S., Cloutier P., Sanche L., Hunting J.D. Article publié dans : Radiation Research2014, 181(1):99-110.
Avant-propos:
Ma contribution à ce travail été par la réalisation de la totalité des travaux expérimentaux, la rédaction et la soumission de l’article. Dr. Pierre Cloutier nous a aidé à analyser nos résultas expérimentaux à l’aide des modèles mathématique.
Résumé:
Les cassures doubles brins (CDBs) sont généralement connues comme les lésions toxiques les plus fréquemment induites par le rayonnement ionisant. Afin de déterminer l’effet de CDBs et d’autres types de lésions créées par l'effet indirect de la radiation ionisante sur la fonctionnalité (‘‘viabilité’’) du plasmide, nous avons utilisé un modèle simple, soit E. coli transformée avec un plasmide (pGEM-3Zf (-)) préalablement irradié en solution aqueuse avec des rayonnements gamma (137Cs). Comme prévu, nous avons constaté que l'efficacité de transformation diminue avec l'augmentation de la dose de rayonnement, mais cette diminution ne peut pas être expliquée par la formation des CDBs. Par exemple, pour une dose de 500 Gy, l'efficacité de transformation relative a diminué de 100% à 53%, alors que seulement 5.7% des plasmides contiennent des CDBs. Malgré le fait que les CDBs sont clairement toxiques, leur nombre pour une dose donnée, est insuffisant pour expliquer la perte de la viabilité du plasmide. La perte de la ‘‘viabilité’’ du plasmide peut être expliquée par une lésion (s) formée à une fréquence environ 8 fois plus grande que celle des CDBs. Ces lésions peuvent être des sites multiples localement endommagés (LMDS) (incluant un pontage inter-brins), de sorte que l'information est
perdue sur les deux brins d’ADN. Elles semblent être réparables, car la courbe de survie présente un épaulement assez remarquable à des doses de rayonnements ionisants faibles. Le rendement des LMDS (non-DSB cluster damage), tel que révélé par les enzymes de réparation Fpg et Nth, est 31 fois plus élevé que celui des CDBs franches. De plus, à l’aide de modèles mathématiques, nous avons estimé qu’il faut au moins trois lésions toxiques pour inactiver les plasmides irradiés.
RÉSULTATS: PREMIER ARTICLE
ABSTRACT
The majority of studies on lethal radiobiological damage have focused on double-strand breaks (DSBs), a type of clustered DNA damage and the evaluation of their toxicity, while other types of clustered DNA damage have received much less attention. The main purpose of this study is to evaluate the contribution of different lesions induced by ionizing radiation to the loss of plasmid DNA functionality. We employed a simple model system comprising E. coli transformed with an irradiated plasmid [pGEM-3Zf (–)] to determine the effect of DSBs and other lesions including base damage and clustered lesions on the functionality (“viability”) of the plasmid. The yields of γ-radiation-induced single-strand breaks (SSBs) and DSBs were measured by gel electrophoresis. We found that the transformation efficiency decreases with radiation dose, but this decrease cannot be explained by the formation of DSBs. For example, at doses of 500 and 700 Gy, the relative transformation efficiency falls from 100% to 53% and 26%, respectively, while only 5.7% and 9.1% of the plasmids contain a DSB. In addition, it is also unlikely that randomly distributed base lesions could explain the loss of functionality of the plasmid, since cells can repair them efficiently. However, clustered lesions other than DSBs, which are difficult to repair and result in the loss of information on both DNA strands, have the potential to induce the loss of plasmid functionality. We therefore measured the yields of γ-radiation-induced base lesions and cluster damage, which are respectively converted into SSBs and DSBs by the base excision repair enzymes endonuclease III (Nth) and
formamidopyrimidine-DNA glycosylase (Fpg). Our data demonstrate that the yield of cluster damage (i.e., lesions that yield DSBs following digestion) is 31 times higher than that of frank DSBs. This finding suggests that frank DSBs make a relatively minor contribution to the loss of DNA functionality induced by ionizing radiation, while other toxic lesions formed at a much higher frequencies than DSBs must be responsible for the loss of plasmid functionality. These lesions may be clustered lesions/locally multiply damaged sites (LMDS), including base damage, SSBs and/or intrastrand and interstrand crosslinks, leading to the loss of vital information in the DNA. Using a mathematical model, we estimate that at least three toxic lesions are required for the inactivation of plasmid functionality, in part because even these complex lesions can be repaired.