HAL Id: tel-01505073
https://tel.archives-ouvertes.fr/tel-01505073
Submitted on 11 Apr 2017HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Métabolisme secondaire de Streptomyces ambofaciens :
exploration génomique et étude du groupe de gènes
dirigeant la synthèse du sphydrofurane
Drago Haas
To cite this version:
Drago Haas. Métabolisme secondaire de Streptomyces ambofaciens : exploration génomique et étude du groupe de gènes dirigeant la synthèse du sphydrofurane. Biochimie, Biologie Moléculaire. Univer-sité Paris Sud - Paris XI, 2015. Français. �NNT : 2015PA112052�. �tel-01505073�
1
UNIVERSITÉ PARIS-SUD
ÉCOLE DOCTORALE 426 : GÈNES GÉNOMES CELLULES
Laboratoire : Institut de Biologie Intégrative de la Cellule
THÈSE DE DOCTORAT
SCIENCES DE LA VIE ET DE LA SANTÉ
par
Drago HAAS
METABOLISME SECONDAIRE DE STREPTOMYCES AMBOFACIENS :
EXPLORATION GENOMIQUE ET ETUDE DU GROUPE DE GENES
DIRIGEANT LA SYNTHESE DU SPHYDROFURANE
Date de soutenance : 10/04/2015Composition du jury :
Directeur de thèse : Sylvie LAUTRU Chargée de recherche (I2BC, Orsay) Rapporteurs : Christophe CORRE Research fellow (University of Warwick)
Soizic PRADO Professeur (Muséum National d’Histoire Naturelle)
Examinateurs : Bertrand AIGLE Professeur (Université de Lorraine, Nancy)
Marc-Henri LEBRUN Directeur de recherche (INRA, Grignon)
3
Remerciements
« A vaincre sans péril, on triomphe quand même » Adapté de « Le Cid » de Pierre Corneille1637 Acte 2 , Scène 2
Je souhaiterais tout d’abord remercier le Docteur Sylvie Lautru, pour m’avoir fait découvrir le monde merveilleux (au sens où Lewis Carol l’entendait…) des Streptomyces et pour avoir encadré, tout au long, mon travail de thèse (et de Master 2) tout en acceptant mes défauts. Merci de m’avoir permis de réaliser une thèse dans de si bonnes conditions (avec de la lumière, de la nourriture et parfois même des week ends). Je voudrais également remercier le Docteur/Directeur-de-département/Conseiller-aux-thèses Jean-Luc Pernodet pour m’avoir accueilli au sein de son équipe Microbiologie Moléculaire des Actinomycètes, pour m’avoir coaché durant ces 3,5232877 années de thèse et pour avoir toujours trouvé du temps pour discuter, malgré son emploi du temps parfois un peu chargé.
Merci également à nos collaborateurs : les Docteurs Jamal Ouazzani, Géraldine Le Goff et le reste de l’équipe de l’ICSN, pour m’avoir accueilli au sein de l’unité, pour m’avoir initié à la chimie et pour leur grande part de travail dans la caractérisation de molécules réfractaires à la purification. Je remercie également le Professeur Olivier Lespinet et le Docteur Matthieu Barba pour le développement de l’outil Break Viewer ainsi que les Docteurs Claude Gerbaud et Annabelle Thibessard et le Professeur Pierre Leblond pour leur participation à l’assemblage des génomes de S. ambofaciens. Je tiens également à remercier les membres de mon jury pour avoir accepté ces fonctions : merci aux Professeurs Soizic Prado, Bertrand Aigle et Nicolas Bayan, ainsi qu’aux Docteurs Christophe Corre et Marc-Henri Lebrun, j’espère que ce manuscrit leur sera agréable à lire et instructif.
Je voudrais aussi remercier mes collègues de l’enseignement : tout d’abord le Docteur Karine Blondeau, qui a accepté de m’engager, quand bien même je m’étais monumentalement planté à son examen en M1, et qui m’a permis de communiquer mon enthousiasme pour Streptomyces et le métabolisme secondaire à ses étudiants. Merci aussi aux Docteurs Florence
4
Constantinesco, Pascale Servant et Cécile Labarre qui m’ont permis de m’investir dans l’univers (impitoyable) de l’enseignement et de survivre à mes TP.
Enfin, un petit mot de remerciement pour le comité GGC (maintenant SDSV). Merci au Professeur /Directeur Pierre Capy pour m’avoir « encouragé » à être représentant doctorant et pour m’avoir permis d’assister aux concours doctoraux en étant du coté le moins stressant. Merci aussi à Sandrine Le Bihan et à Rozenn Le Guyader qui rendent les démarches administratives surmontables (notamment grâce à tous ces liens qui permettent d’arriver directement au bon endroit sur le site de l’UP-Sud). Merci également à mes homologues représentants doctorants : Docteur Cyril Denby-Wilkes, Jean-Baptiste Briand, Charlotte Urien et Selma Machaa (et désolé pour ceux qui seront devenus Docteur entre l’écriture des remerciements et la publication de ma thèse…)
”When you do things right, people won’t be sure you’ve done anything at all” Bender (Futurama)
Merci à l’amicale des thésards dépressifs pour m’avoir permis de vivre ces trois années (à une vache près, ce n’est pas une science exacte…) pas si mal que ça. Merci à Cécile Pereira (thésarde) et Marc Descrimes (post-doc honorifique) pour leur soutien, leur humour et pour m’avoir nourri un jour par semaine. Merci à Geoffrey « John » Hutinet (thésard Docteur) pour sensiblement les mêmes choses (mais le mardi au lieu du jeudi). Merci à Moano (non-thésard) pour ta bonne humeur et pour m’avoir permis de parler d’autre chose que de ma thèse. Merci à Kévin Verstraete (thésard détendu (oxymore)) pour ta sympathie et parce que tu fabriques des sabres. Merci également à Julien Recoquillay (thésard Docteur à l’étranger (Tours)) pour m’avoir initié aux mondes du JdR. A tous, merci notamment pour : « qu’est-ce qu’une table ? », les nombreuses parties de jeux de société (que Marc a gagnées), les chaînes de jeux de mots interminables (les chats, l’électricité entre autres…), les blagues « comme ta thèse », le whisky-rhum-porto etc…
Je tiens également à remercier tous mes collègues et amis. Tout d’abord (et par ordre alphabétique), les « djeuns » : Aaron Millan, Audrey Vingadassalon, Florence Lorieux, Jerzy (prononcé Yèjeux, mais écrit Jerzy) Witwinowski, Laura Guidici, Maxime Vergne,
Pierre-5
Alexandre Mariel, Priscilla Pierre-Elies, Soren Helmark, Soumaya Najah Ben Youssef, Thierry Dulermo et Tony Ruzzini. Merci pour beaucoup de choses : Le soutien, les discussions pas du tout scientifiques, les soirées bowling, les batailles d’eau près des armoires électriques dans des endroits approuvés par Hygiène & Sécurité... Merci à vous également toutes ces « petites » farces : le papier alu, les cotillons (qui fondent dans l’eau le glycérol), les 600 verres pour n’en citer que quelques-unes.
Je remercie également les « un petit peu moins djeuns » (je vais me faire tuer pour ça…) : Emmanuelle Darbon, pour avoir râlé pour deux, Alain Raynal, pour savoir beaucoup de choses, notamment sur la science magie de la PCR, Cécile Martel, pour les problèmes d’HPLC partagés, Sylvain Pendino, pour les milieux ; pour les nombreux milieux, et Michel Guérineau pour ses nombreuses anecdotes sur quand la Recherche n’était pas une succursale de l’ANPE. Encor (oui « encor » sans « e » car c’est accepté par l’Académie Française) merci à Corinne, Sylvine, Marie-Hélène, Catherine et Marie-Joëlle pour avoir donné vie à cette aile MMA-MES.
Je voudrais aussi remercier tous les gens de « l’autre aile » qui m’ont accepté dans leur cafétéria malgré mes origines douteuses… Merci en particulier à Olivier Bugaud et Sandra Blanchet d’avoir accepté de perdre quelques heures de productivité en ma compagnie. Enfin, je me dois de remercier les personnes du service administratif (en particulier Muriel Decraene), dont la qualité de travail réfute activement un certain sketch de Coluche.
« A Lannister always pays his debts » La majorité des Lannister (Game of Throne)
Je me sens également obligé de remercier mes amis de plus longue date, qui ont aussi joué un rôle dans le maintien de ma santé mentale (à un seuil acceptable). Merci à Arthur, Guillaume, Max, Mathieu, Romain, Pierre et Marc. Pour les soirées nuits de jeux de sociétés de plus de 8 heures (sans compter les 2 heures d’explication des règles), pour avoir enrichi ma culture (Mon Petit Poney, Adventure Time, et bien d’autres…) et pour m’avoir invité à toutes ces sorties sur Paris où je ne suis jamais venu. Je voudrais aussi remercier « les autres » que je ne vois plus si souvent (peut être par ce qu’au lieu de faire des études vous avez décidé
6
d’avoir une vie de famille et/ou un vrai boulot…), notamment Christo, Flo et Thomas pour tous les trucs complètement stupides qu’on a pu faire ensemble.
Je me dois également d’adresser ma reconnaissance à mes parents, qui ont accepté sans broncher de me laisser vivre à leurs dépens, tel un rabouin sédentaire et photophobe, pendant toutes ces années. Merci aussi au reste de ma famille (trois personnes à tout casser…) d’avoir cru en moi (ou, du moins, d’avoir fait semblant…), en particulier mon grand-père, Jean Rettagliati, qui aurait sans doute apprécié de voir l’accomplissement de ce travail.
Pour finir sur une touche d’humour (…), je souhaiterais remercier un certain nombre de choses qui n’ont pas forcément besoin d’être remerciées. Je commencerai par Streptomyces, sans qui cette thèse n’aurait pas eu lieu (et sans qui beaucoup de gens seraient morts de maladies infectieuses, mais c’est secondaire…), merci d’avoir créé le sphydrofurane, cette molécule pour laquelle la seule fonction connue aura été de m’avoir occupé trois ans (un peu plus en fait…). Merci également à tous les gens qui auraient dû être remerciés plus haut (voir beaucoup plus haut) et que j’ai complètement oublié (merci, et désolé). Enfin, si jamais cette thèse venait (par accident) à être publiée, voir vendue, Ami Lecteur, merci à toi !
7
Table des Matières
Remerciements 3
Abréviations 11
Table des Figures 13
Etat de l’Art 17
1. Le métabolisme secondaire 19 1.1. Définition et généralités 19 1.1.1. Métabolisme secondaire chez les plantes 19 1.1.2. Métabolisme secondaire chez les microorganismes 20 1.2. Activités des métabolites secondaires 21 1.2.1. Applications 21 1.2.1.1. Antibiotiques 21 1.2.1.2. Antitumoraux 23 1.2.1.3. Immunosuppresseurs 23 1.2.1.4. Antifongiques et antiparasitaires 24 1.2.1.5. Inhibiteurs d’enzymes 25 1.2.2. Rôles biologiques 26 1.2.2.1. Elimination de compétiteurs 26 1.2.2.2. Dose et affinité : au-delà d’un rôle 27 1.2.3. Evolution du métabolisme secondaire 30 1.2.3.1. Acquisition de clusters par transfert horizontal 30 1.2.3.2. Vers de nouveaux clusters de biosynthèse 31 1.3. Les principales familles du métabolisme secondaire et leurs enzymes 33 1.3.1. Les NRPS et peptides non ribosomaux 33 1.3.1.1. Organisation 33 1.3.1.2. Mode d’assemblage 34 1.3.1.3. Diversité 35 1.3.2. Les PKS et polycétides 36 1.3.2.1. Mécanisme d’assemblage 36 1.3.2.2. Les différents types de PKS 37 1.3.3. Autres familles 39 1.3.3.1. Les terpènes 40 1.3.3.2. Les aminoglycosides 41 2. Les Streptomyces et leur métabolisme secondaire 42 2.1. Présentation du genre Streptomyces 42 2.1.1. La classe des Actinobactéries 42 2.1.2. Ecologie des Streptomyces 43 2.1.3. Cycle de vie 45 2.1.3.1. Germination 46 2.1.3.2. Croissance végétative 47 2.1.3.3. Croissance aérienne 47
8
2.1.3.4. Sporulation 49 2.2. Génome des Streptomyces 49 2.2.1. Organisation du chromosome 49 2.2.2. Biais en G/C 50 2.3. Richesse du métabolisme secondaire des Streptomyces 51 2.3.1. Age d’or de la découverte d’antibiotiques 51 2.3.2. Ere post-génomique, redécouverte du métabolisme secondaire 52 3. Recherche de nouveaux métabolites secondaires 53 3.1. Genome mining 53 3.1.1. Prédiction du métabolisme secondaire par bioinformatique 53 3.1.2. Inactivation de gènes et recherche phénotypique 55 3.1.3. Expression hétérologue 56 3.1.4. Approche génomisotopique 57 3.2. Activation de clusters du métabolisme secondaire par des systèmes de régulation
57
3.2.1. Les régulateurs globaux 57 3.2.2. Les régulateurs spécifiques d’un cluster de gènes 60 3.2.3. Les molécules signal 61 3.3. Expression de clusters cryptiques par des approches empiriques 63 3.3.1. La méthode OSMAC 63 3.3.2. Ribosome Engineering 64 4. Métabolisme secondaire de Streptomyces ambofaciens 66 4.1. Présentation de Streptomyces ambofaciens 66 4.2. Métabolites connus 66 4.2.1. La spiramycine 66 4.2.2. La congocidine 68 4.2.3. Les kinamycines 69 4.2.4. Les stambomycines 70 4.2.5. Autres métabolites 71 4.3. Recherche et caractérisation de nouveaux métabolites 72 4.3.1. Clusters putatifs du métabolisme secondaire 72 4.3.2. Ilots génomiques 73 5. Objectifs du travail de thèse 74
Chapitre I : Exploration du métabolisme secondaire de Streptomyces ambofaciens
ATCC23877 : comparaison des outils de recherche antiSMASH 2.0. et Break Viewer 77
1. Introduction 79
2. Matériel et méthodes 80 2.1. Extraction de l’ADN génomique 80 2.2. Séquençage des génomes de S. ambofaciens ATCC23877 81 2.3. Recherche de ruptures de synténie entre S. ambofaciens et S. coelicolor 81
3. Résultats 82
3.1. Assemblage du génome de S. ambofaciens ATCC23877 82 3.2. Recherche de gènes du métabolisme secondaire par approche bioinformatique
83 3.3. Etude des clusters putatifs du métabolisme secondaire 85 3.3.1. Clusters putatifs de type sidérophore-like 85
9
3.3.2. Cluster putatif de type CDA-like 88 3.3.3. Cluster putatif PKS de type I 90 3.3.4. Clusters putatif de biosynthèse de peptides ribosomiques 92 3.3.5. Cluster de type terpène 94 3.3.6. Clusters butyrolactones 96 3.3.7. Recherche de clusters par similitude de séquence : bilan 96
3.4. Comparaison des génomes de S. ambofaciens et S.coelicolor : recherche d’ilots
génomiques 97
3.4.1. Organisation du chromosome et synténie chez les Streptomyces 97 3.4.2. Identification d'ilots génomiques chez S. ambofaciens ATCC23877 par
comparaison avec le génome de S. coelicolor 98 3.4.3. Avantages et inconvénients des deux stratégies de recherche 102
4. Conclusion 104
Chapitre II : Etude de l’ilot génomique RS3-SAM/SCO : lien avec la production de
métabolites secondaires 105
1. Introduction 107
2. Matériel et méthodes 108
2.1. Souches, plasmides et conditions de culture 108 2.2. Préparation et manipulation de l’ADN 108 2.3. Recherche de ruptures de synténie entre S. ambofaciens et S. coelicolor 108 2.4. Expression hétérologue de l’ilot RS3-SAM/SCO chez S. coelicolor M1154 108
2.5. Analyses HPLC 109
3. Résultats 111
3.1. Choix de l’ilot génomique étudié 111 3.2. Etude de l’ilot RS3-SAM/SCO chez S. ambofaciens 113
4. Conclusions 115
Chapitre III : Isolation and characterization of the gene cluster directing the biosynthesis of sphydrofuran in Streptomyces ambofaciens ATCC23877 117
Chapitre IV : Comparaison des profils du métabolisme secondaire de plusieurs espèces
proches de Streptomyces 147
1. Introduction 149
2. Matériel et méthodes 150
2.1. Extraction de l’ADN génomique 150 2.2. Séquençage des génomes des souches DSM40697 et M1013 150 2.3. Construction des arbres phylogénétiques 150 2.4. Recherche de clusters du métabolisme secondaire 151
3. Résultats 151
3.1. Assemblage des génomes des souches DSM40697 et M1013 151 3.2. Phylogénie des souches étudiées 151 3.3. Clusters du métabolisme secondaire des souches étudiées 153
3.3.1. Profils du métabolisme secondaire des souches ATCC23877 et
DSM40697 154
3.3.2. Profil du métabolisme secondaire de S. coelicolor A3(2) 156 3.3.3. Profil du métabolisme secondaire de Streptomyces sp. M1013 157
10
3.3.4. Clusters conservés et clusters spécifiques 159 3.4. Existence d’un « core secondary metabolome » chez les Streptomyces 161 3.4.1. Clusters conservés chez Streptomyces collinus Tü365 161 3.4.2. Clusters conservés chez Streptomyces griseus NBRC 13350 162
4. Conclusion 164
Conclusion Générale et Perspectives 165
11
Abréviations
A : Adenylation
ACV : L-aminoadipyl-L-cysteinyl-D-valine ACP : Acyl Carrier Protein
ADN : Acide Désoxyribonucléique
alp : Alpomycine/Kinamycine (gènes)
AMP : Adenosine Mono-Phosphate
antiSMASH : antibiotics and Secondary Metabolite Analysis SHell ARN : Acide Ribonucléique
ARO : Aromatase AT : Acyltransferase
ATCC : American Type Culture Collection ATP : Adenosine Tri-Phosphate
C : Condensation
CDA : Calcium Dependent Antibiotic CDPS: CycloDiPeptide Synthase
cgc : Congocidine (genes)
CLF : Chain Length Factor
cll: CDA-like Lipopeptide
CSR : Cluster Situated Regulator CYC : Cyclase
DEBS : 6-DeoxyErythronolide B Synthase
DEDL : Détecteur Evaporatif à Détection de Lumière DH : Dehydratase
DMAPP : Diméthylallyl Pyrophosphate E : Epimerase
ER : Enoyl-Reductase FAS : Fatty Acid Synthase
GABR : γ-butyrolactone receptor GBL : γ-butyrolactone
G+C : Guanine + Cytosine Glc-NAc : N-acetylglucosamine GPP : Geranyl Diphosphate
HMG-CoA : 3-hydroxy-3-methylglutaryl-coenzyme A HPLC : High Pressure Liquid Chromatography
HRESIMS : High Resolution ElectroSpray Ionisation Mass Spectrometry HTH : Helix-Turn-Helix
12
Kb : Kilobase
KR : Keto-Reductase KS ; Keto-Synthase
LAL : Large ATP-binding regulators of the LuxR family Mb : Mégabase
MiGenIs : Mining Genomic Islands MMF : Methylenomycin Furan MT : Methyltransferase NRP : Non-Ribosomal Peptide
NRPS : Non-Ribosomal Peptide Synthetase NRRL : Northern Regional Research Laboratory ORF : Open Reading Frame
OSC : Orsay Strain Collection
OSMAC : One Strain MAny Compounds PCP : Peptidyl Carrier Protein
pH : potentiel Hydrogène PKS : Polyketide Synthase
ppGpp : Guanosine Tétraphosphate PPTase : Phosphopantetheinyltransferase pSAM2 : Streptomyces ambofaciens plamid 2
RiPP : Ribosomally synthetized and Post-translationally modified Peptide RMN : Résonance Magnétique Nucléaire
ROS : Reactive Oxygen Species
RT-PCR : Reverse Transcriptase Polymerase Chain Reaction
RS1-SAM/SCO : Rupture de Synténie N°1 du classement ; ilot génomique chez SAM par
rapport à SCO
SAM : Streptomyces ambofaciens
SAML/SAMR : gene de S. ambofaciens bras gauche/droit SARP : Streptomyces Antibiotics Regulatory Protein SCO: Streptomyces coelicolor
SFM: Soya Floor Manitol
spf : Sphydrofurane srm : Spiramycine
TAP : Telomere Associated Protein Te : Thioesterase
TIR : Terminal Inverted Repeats TLC : Thin Layer Chromatography TP : Terminal Protein
13
Table des Figures
Figures
Etat de l’Art1) Structures de l’artémisinine et de la morphine 20 2) Structures de différents antibiotiques 22 3) Structures de différents anticancéreux 23 4) Structures de différents immunosuppresseurs 24 5) Structures de la nystatine et de la spinosyne 25 6) Structures d’inhibiteurs d’enzymes 25 7) Effet de la concentration d’antibiotiques sur la transcription 29 8) Différents groupements post-assemblage d’un squelette NRP 32 9) Chaîne d’assemblage classique d’une NRPS 34 10) Fonctionnement des domaines A, PCP et C d’une NRPS 35 11) Principaux substrats utilisés par les PKS 36 12) Synthèse de l’érythromycine par des PKS de type I 38 13) Schéma d’assemblage des PKS II et III 39 14) Structures et mode d’assemblage des terpènes linéaires C10 et C15 41 15) Structures de la kanamycine et de la streptomycine 42 16) Phylogénie des Actinobactéries 43 17) Exemples de modes de vie symbiotiques et parasites chez des Streptomyces 45 18) Cycle de développement des Streptomyces 46 19) Structure et organisation du mycélium végétatif 47 20) Production d’antibiotiques et sénescence au sein d’une colonie de Streptomyces 48 21) Organisation du chromosome de Streptomyces 50 22) Principaux antibiotiques découverts au XXème siècle 52
23) Exemple de résultat obtenu avec antiSMASH 55 24) Stratégie de comparaison de profils sauvage/mutant 56 25) Principe de l’expression hétérologue 57 26) Exemple de réseaux de régulation des métabolismes primaire et secondaire 59 27) Schéma de la réponse stringente bactérienne 62 28) Cascade de régulation impliquant le facteur-A chez S. griseus 63 29) Principe de la méthode OSMAC 64 30) Effet de la délétion de rsmG sur la production d’actinorhodine chez S. coelicolor 65 31) Cluster de biosynthèse et structure de la spiramycine 67 32) Cluster de biosynthèse et structure de la congocidine 68 33) Cluster de biosynthèse et structure de la kinamycine 69 34) Cluster de biosynthèse et structure de la stambomycine 71 35) Localisation des clusters du métabolisme secondaire chez S. ambofaciens 72 36) Ilots génomique et métabolisme secondaire chez les Salinispora 74
14
Chapitre I Figures
1) Clusters putatifs identifiés sur le chromosome de S. ambofaciens 84 2) Clusters sidérophore-like N°13 et 18 et pics HPLC liés au cluster N°18 87 3) Organisation des gènes et des NRPS du cluster cll 89 4) Organisation du cluster putatif N°27 de PKS de type I 91 5) Organisation des clusters bactériocines putatifs N°16 et 22 94 6) Organisation du cluster terpène putatif N°21 95 7) Synténie observée entre S. ambofaciens et différentes espèces de Streptomyces 98 8) Comparaison de la synténie entre S. ambofaciens et S. coelicolor avec Break Viewer 99 9) Comparaison du cluster cgc tel que détecté par antiSMASH 2.0. et Break Viewer 103
Chapitre II
1) Représentation de la zone de rupture de synténie RS3 112 2) Chromatogrammes HPLC de OSC4 et OSC416 (pics liés à l’ilot RS3-SAM/SCO) 114 3) Chromatogrammes HPLC de SPFSH001 et M1154 (pics liés à l’ilot RS3-SAM/SCO) 115
Chapitre III
1) Kinetics of the M1-M3 metabolites production over 5 days culture 122 2) Structures of the sphydrofuran molecule two forms 123 3) Comparison of the spf gene cluster and several homologous clusters 124 4) HPLC chromatograms of sphydrofuran M2 and M3 forms 129 5) Hypothetical biosynthesis pathway of the sphydrofuran molecule 130 S1) NMR spectra of sphydrofuran 133 S2) HRESIMS spectra of sphydrofuran and M3 134 S3) Structures of the methylenomycin A and syringolide molecules 134 S4) Phylogeny of several species containing the spf gene cluster 135 S5) HPLC chromatograms of sphydrofuran (Δspf01-04 and 15-16) 135 S6) HPLC chromatograms of sphydrofuran (Δspf06-10 and 13-14) 137
Chapitre IV
1) Arbres phylogénétiques de différentes espèces de Streptomyces 153 2) Représentation et comparaison des clusters de S. ambofaciens ATCC23877 et DSM40697
154 3) Représentation et comparaison des clusters de S. ambofaciens et S. coelicolor 156 4) Exemple de cluster très conservé : le cluster desferrioxamine 160 5) Digramme de Venn des clusters spécifiques et conservés chez plusieurs Strepotmyces
15
Tableaux
Chapitre I1) Souches utilisées dans cette étude 82 2) Clusters de gènes identifiés par antiSMASH 2.0. chez S. ambofaciens ATCC23877 84 3) Annotation des gènes du cluster putatif NIS-like N°18 86 4) Annotation des gènes du cluster putatif cll 90 5) Annotation des gènes du cluster putatif PKS de type I N°27 92 6) Annotation des gènes du cluster putatif terpène N°21 95 7) Comparaison de la synténie entre S. ambofaciens et S. coelicolor avec Break Viewer 98 8) Classement des ruptures de synténie entre S. ambofaciens et S. coelicolor 100 9) Comparaison des outils antiSMASH 2.0. et Break Viewer 103
Chapitre II
1) Souches et plasmides utilisés dans l’étude 109 2) Oligonucléotides utilisés dans l’étude 110 3) Classement des ruptures de synténie entre SAM/SCO 111
Chapitre III
1) NMR spectroscopic data 123 2) In silico analysis of the spf cluster genes 125 3) Production of the three metabolites in wild type and mutant contexts 128 S1) Strains and plasmids used in this study 138 S2) Primers used in this study 141
Chapitre IV
1) Liste des clusters antiSMASH 2.0. identifiés chez S. ambofaciens ATCC23877 et DSM40697 155 2) Liste des clusters antiSMASH 2.0. identifiés chez S. coelicolor 157 3) Liste des clusters antiSMASH 2.0. identifiés chez Streptomyces sp.M1013 158 4) Comparaison des clusters conservés chez plusieurs espèces de Streptomyces 162
17
19
1. Le métabolisme secondaire
1.1. Définition et généralitésLe métabolisme secondaire est défini comme l’ensemble des voies permettant la synthèse de petites molécules, non essentielles mais pouvant procurer un avantage sélectif dans certaines conditions. Ces molécules sont appelées métabolites secondaires. Le métabolisme secondaire est généralement opposé au métabolisme primaire qui regroupe l’ensemble des voies cataboliques et anaboliques indispensables à la survie et à la reproduction de la cellule. Cela dit, il est difficile de poser une définition bien tranchée séparant métabolisme primaire et secondaire, cette séparation n’existant dans les faits que pour des conditions de laboratoire bien définies. Par exemple, les sidérophores, petites molécules utilisées pour capturer le fer dans l'environnement, sont considérés comme des métabolites secondaires, bien que ces molécules soient souvent indispensables à la survie des organismes producteurs dans leur environnement naturel.
Il existe une grande variété de métabolites secondaires pouvant avoir des structures chimiques diverses et complexes et de très nombreuses activités biologiques. On peut citer la capacité à tuer ou inhiber la croissance des bactéries (on parle alors d’antibiotiques), des champignons (antifongiques) ou d’autres organismes eucaryotes, tels des protistes ou des helminthes (antiparasitaires). D’autres composés peuvent jouer un rôle dans la régulation du métabolisme de l’organisme. Certaines molécules peuvent également servir à la communication entre cellules (quorum sensing). La plupart des métabolites secondaires sont produits par les plantes, les bactéries et les champignons. Ils sont souvent synthétisés après la phase de croissance végétative (dans le cas de microorganismes) quand des stress de différentes natures commencent à s’exercer.
1.1.1. Métabolites secondaires chez les plantes
L’utilisation de plantes en médecine remonte à plus de 2600 ans av. J.-C. avec par exemple l’usage d’huiles de cèdre et de cyprès par les Mésopotamiens. Aujourd’hui encore, de nombreux peuples utilisent des essences de plantes dans ce que l’on appelle la médecine traditionnelle. Les molécules liées aux propriétés thérapeutiques de ces plantes sont la plupart
20
du temps des métabolites secondaires. On sait par exemple aujourd’hui que les propriétés analgésiques du pavot somnifère (Papaver somniferum) sont dues à un alcaloïde: la morphine (Figure 1). De même pour le Quinhaosu (Artemisia annua), qui tient ses propriétés antipaludiques de la lactone sesquiterpène artémisinine (Cragg et al., 1999). Le nombre total de métabolites secondaires produits par des plantes est estimé entre 500 000 et 600 000 produits, principalement des alcaloïdes, des flavonoïdes, des terpènes, des stéroïdes et des carbohydrates (Berdy, 2005).
Figure 1 : A) Artemisia annua et la structure de l’antipaludique artémisinine. B) Papaver somniferum et la structure de l’analgésique morphine
1.1.2. Métabolites secondaires chez les microorganismes
Si l’utilisation de plantes en médecine remonte à plusieurs millénaires, l’usage des microorganismes en thérapeutique est relativement récent. L’exploitation de ces organismes pour la recherche de métabolites secondaires est amorcée par Fleming en 1928. La découverte de la pénicilline chez le champignon Penicillium notatum, suivie de la découverte de la streptomycine par Waksman en 1944 chez Streptomyces griseus annonce l’âge d’or de la découverte d’antibiotiques (Woodruff, 2014).
Au sein d’une très large diversité, certains genres bactériens et fongiques se démarquent par la richesse de leur métabolisme secondaire. Ainsi les champignons filamenteux des genres Penicillium et Aspergillus (Sanchez et al., 2012) constituent d’excellents modèles pour la recherche de nouveaux métabolites secondaires. Chez les
21
bactéries, les actinomycètes sont particulièrement prolifiques pour la production de métabolites secondaires. On peut notamment citer les genres Amycolatopsis, Micromonospora, Salinospora ou plus spécialement les Streptomyces (discutés partie 2) chez qui la majorité des composés utilisés par l’homme ont été isolés.
1.2. Activités des métabolites secondaires
1.2.1. Applications des métabolites secondaires par l’Homme
L’étude du métabolisme secondaire ces dernières décennies a conduit à la découverte de nombreuses molécules, le plus souvent dans une optique d’utilisation thérapeutique. Cette partie propose d’expliciter les différents usages possibles des métabolites secondaires. Etant donnée la part de molécules utilisées issues de Streptomyces, la grande majorité des exemples cités sont des composés synthétisés par ces bactéries.
1.2.1.1. Les antibiotiques
Du grec « anti » (contre) et « bios » (vie), le terme antibiotique désigne toute molécule capable de tuer ou d’inhiber la croissance des bactéries, et ce de façon sélective. Cette terminologie ne s’applique pas si l’effet est dirigé contre un organisme eucaryote. Les antibiotiques permettent de lutter activement contre de nombreuses pathologies autrefois souvent fatales (ex : tuberculose), et ont contribué de façon non négligeable à l’allongement de l’espérance de vie. De nombreuses familles d’antibiotiques ont été identifiées. On peut citer les β-lactames, les cyclines, les macrolides ou encore les aminoglycosides par exemple (Figure 2). La plupart de ces composés agissent sur une des trois cibles suivantes : la synthèse du peptidoglycane, la réplication de l’ADN ou la synthèse des protéines.
22
23
1.2.1.2. Agents antitumoraux
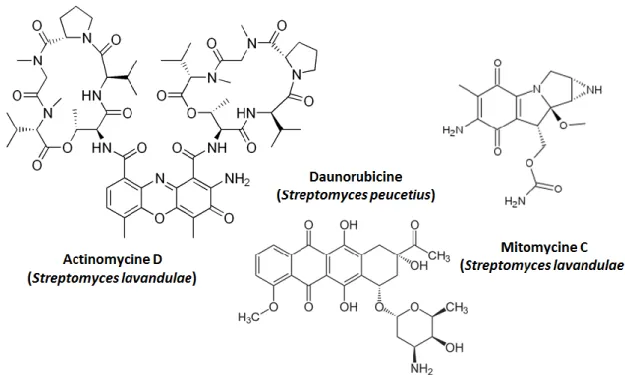
Le cancer constitue l’une des pathologies humaines responsables d’un grand nombre de décès chaque année. Parmi les métabolites secondaires, certaines molécules se révèlent capables d’inhiber la multiplication des cellules cancéreuses. On peut citer l’actinomycine D, les anthracyclines (daunorubicine, doxorubicine), la bléomycine ou encore la mitomycine C (Figure 3). Ces composés agissent en se fixant sur l’ADN. L’actinomycine D, par exemple, se fixe au niveau du complexe d’initiation de la transcription et empêche l’élongation par l’ARN polymérase (Sobell, 1985).
Figure 3 : Quelques structures de produits naturels ayant des propriétés anticancéreuses
1.2.1.3. Immunosupresseurs
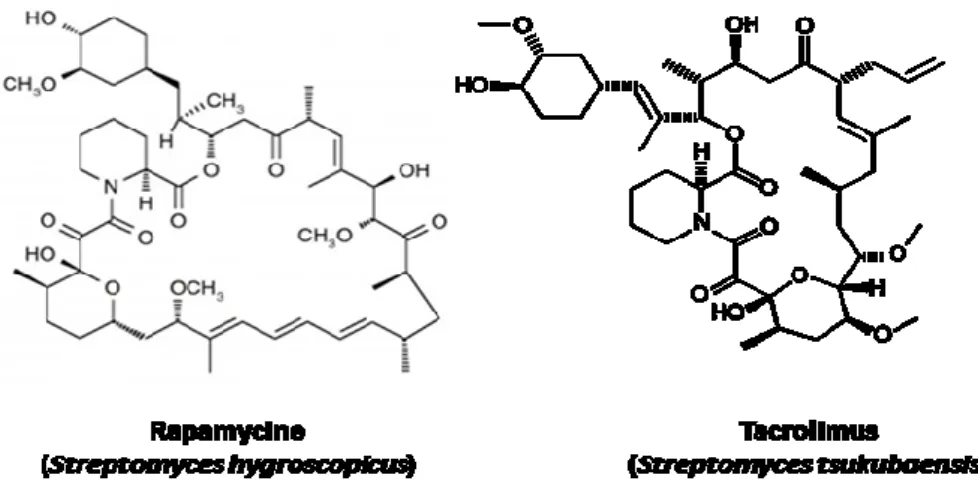
Certains métabolites sont capables d’agir sur le système immunitaire de façon à inhiber celui-ci. Une telle activité se révèle très utile pour le traitement de maladies auto-immunes ou pour empêcher le rejet d’organes greffés. Par exemple, la rapamycine issue de Streptomyces hygroscopicus est utilisée pour ses propriétés immunosuppressives et antiprolifératives lors de transplantations de reins (Figure 4). Le tacrolimus (FK506 ou fujimycine) isolé chez S. tsukubaensis, est utilisé à très faible dose pour la transplantation de foies depuis 1994 (Demain & Sanchez, 2009).
24
Figure 4 : Quelques structures de produits naturels ayant des propriétés immunosuppressives
1.2.1.4. Antifongiques et antiparasitaires
Le mot antifongique réfère à un composé capable de tuer ou d’inhiber la croissance d’un champignon. De nombreux antifongiques sont utilisés en médecine. La nystatine produite par Streptomyces noursei est par exemple utilisée pour le traitement de différentes infections par des levures, notamment des Candida (Fjaervik and Zotchev, 2005). Le terme antiparasitaire désigne plus globalement un composé permettant de lutter contre différents types d’organismes eucaryotes (protistes, insectes, helminthes). Ces métabolites présentent des utilités en médecine humaine et vétérinaire, mais aussi en agriculture. L’usage de métabolites insecticides en agriculture permet de diminuer fortement les pertes de rendement. Ils sont aussi moins toxiques que les insecticides de synthèse. Par exemple les spinosynes (Figure 5), produites par Saccharopolyspora spinosa, sont très spécifiques et ne présentent aucun risque pour les animaux (Kirst, 2010 ; Waldron et al., 2001). L’avermectine produite par Streptomyces avermitilis est active contre certaines espèces d’arthropodes et de nématodes. Ce composé est en revanche non toxique pour les mammifères et ne possède pas de propriété antibiotique (donc pas d’effet sur la flore intestinale) (Burg et al., 1979). L’avermectine est utilisée chez l’Homme pour le traitement de l'onchocercose (cécité provoquée par le nématode Onchocerca volvulus) et en médecine vétérinaire pour la prévention et le traitement de diverses infections (Goa et al., 1991).
25
Figure 5 : Exemples de structure de molécules aux propriétés antiparasitaires (spinosyne) et antifongiques (nystatine)
1.2.1.5. Inhibiteurs d’enzymes
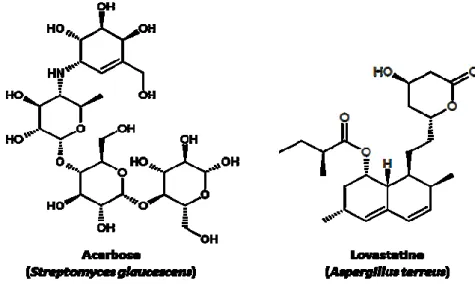
Certains métabolites secondaires sont capables d’agir sur le niveau d’efficacité d’enzymes du métabolisme primaire et de diminuer celui-ci. Ces propriétés ont donné lieu à de nombreuses applications notamment en médecine. La lovastatine (isolée chez Aspergillus terreus) est capable d’inhiber l’action de la réductase d’HMG-CoA, une enzyme permettant la synthèse du mévalonate, précurseur du cholestérol (Figure 6). Les propriétés de la lovastatine sont utilisées pour le traitement de maladies cardiovasculaire (Newman et al., 2000). D’autres métabolites sont utilisés pour le traitement du diabète de type 2. C’est notamment le cas de l’acarbose (produit par Streptomyces glaucescens). Cette molécule ralentit l’hydrolyse de polysaccharides issus de l’alimentation. En conséquence, le glucose est moins rapidement assimilé par l’intestin ce qui fait chuter son taux sanguin (Tsunosue et al., 2010).
26
1.2.2. Rôles biologiques des métabolites secondaires
Il est acquis en évolution qu’un caractère n’est sélectionné que s’il apporte un avantage sélectif à l’organisme, c’est-à-dire qu’il augmente ses chances de survie et/ou reproduction par rapport à ces compétiteurs. Dans une optique plus neutraliste, il est également accepté que si le caractère n’a pas d’effet délétère, il peut être fixé par dérive génétique (Kimura, 1991). Les gènes du métabolisme secondaire sont la plupart du temps organisés en cluster pouvant occuper plusieurs dizaines de kilobases de génome. C’est par exemple le cas de l’érythromycine qui, chez Saccharopolyspora erythrea, occupe 60 kb pour l’assemblage du macrolide, les modifications post-assemblage et le système de résistance à cette molécule. Un tel poids au sein du génome doit être contrebalancé par des effets bénéfiques sans quoi ce cluster aurait été effacé par la sélection naturelle.
S’il est évident qu’une molécule aux propriétés anticancéreuses est utilisée par l’Homme car anticancéreuse, la raison pour laquelle cette même molécule a été sélectionnée par l’évolution reste souvent obscure. Poser la question du rôle biologique des métabolites secondaires revient à se poser la question de l’avantage sélectif apporté par un métabolite produit à une concentration inconnue dans des conditions de vie inconnues. C’est un domaine où la spéculation est facile et la vérification quasi impossible (Davies, 2013).
1.2.2.1. Métabolites antimicrobiens ayant un rôle antimicrobien ?
Certains biomes, tels les sols, accueillent une grande mixité de genres et d’espèces de microorganismes devant lutter pour l’accès aux ressources nutritives. Il est généralement accepté que les molécules antibiotiques joueraient un rôle d’armes biologiques destinées à éliminer les organismes compétiteurs. Cette supposition, bien que logique d’un point de vue évolutif, n’en reste pas moins une hypothèse qui n’exclut pas d’autres possibilités. Cela dit, le rôle d’antibiotique est pour certains métabolites fortement probable, comme illustré avec les deux exemples qui suivent (Challis & Hopwood, 2003).
Le premier exemple est celui de la production des « antibiotiques » céphamycines et de l’acide clavulanique par Streptomyces clavuligerus. L’acide clavulanique est un inhibiteur de β-lactamases (pouvant hydrolyser les antibiotiques β-lactames). De façon intéressante, plusieurs espèces identifiées comme produisant les céphamycines sont aussi capables de produire l’acide clavulanique (S. jumonjinensis, S. katsurahamanus). Au contraire, à ce jour, aucun exemple d’espèce produisant l’acide clavulanique mais pas de céphamycine n’a été
27
rapporté(Jensen et al., 1999). Second fait intéressant, les clusters de biosynthèse de l’acide clavulanique et de la céphamycine sont, chez les espèces précédemment citées, adjacents sur le chromosome (Ward et al., 1993), suggérant qu’ils pourraient être acquis et/ou transférés simultanément par transfert horizontal. Ces faits semblent donc corroborer l’hypothèse du rôle antibactérien des céphamycines. Dans une optique de course à l’armement, ces espèces auraient donc été sélectionnées, car capables de produire un antibiotique efficace contre des souches sensibles mais également contre un panel de souches « résistantes » à cet antibiotique.
Le second exemple concerne les streptogramines. Les streptogramines sont une paire de molécules (A et B) agissant de façon synergique. Ces molécules ont été isolées chez des Streptomyces, notamment les pristinamycines I et II chez Streptomyces pristinaespiralis. Prise séparément, chaque molécule possède des propriétés bactériostatiques. Ensemble, leur activité devient bactéricide. La fixation des streptogramines A sur le ribosome augmente d’un facteur 40 l’affinité des streptogramines B pour un second site du ribosome, rendant sa fixation irréversible (Di Giambattista et al., 1989). Aujourd’hui, aucune souche capable de produire uniquement une streptogramine A ou B n’a encore été isolée. Chez S. pristinaespiralis, les gènes de biosynthèse des pristinamycines sont organisés en un seul méga-cluster de 200 kb (Bamas-Jacques et al., 1999). Ces différentes observations laissent fortement penser que les streptogramines auraient coévolué de façon à renforcer mutuellement leur activité antibiotique.
1.2.2.2. Dose et affinité : au-delà d’un rôle biologique
La recherche de métabolites secondaires ayant souvent été conduite dans une optique pharmaceutique, les molécules étudiées sont la plupart du temps associées à des activités biologiques utiles pour l’homme. De plus, ces molécules sont généralement testées dans des conditions et à des concentrations très éloignées de ce qui peut se passer dans une cellule. Cette partie propose de regarder le métabolite non pas comme ayant un « rôle » au sein de la cellule, mais comme ayant des propriétés physico-chimiques conduisant à différentes activités globalement bénéfiques pour l’organisme. Deux points seront particulièrement discutés : l’effet de la dose et l’affinité entre une enzyme et sa cible.
28
« Tout est poison et rien n’est sans poison, seule la dose permet à quelque chose de ne pas être poison ». Ce principe énoncé par Paracelse est à la base de la toxicologie et s’applique aussi bien à l’Homme qu’aux producteurs de métabolites secondaires. De nombreux composés révèlent des activités biologiques insoupçonnées lorsqu’ils sont présents à très faible concentration. Il a été montré que certains « antibiotiques » pouvaient avoir, à des concentrations sub-inhibitrices, un effet sur le niveau de transcription de certains gènes. C’est par exemple le cas des antibiotiques érythromycine et rifampicine qui, à concentrations sub-inhibitrices entraînent la transcription de gènes sous le contrôle de luxI (Goh et al., 2002) (Figure 7). L’exemple inverse a également été observé : la N-(3-oxodo-decanoyl)-L-homoserine lactone, impliquée dans le quorum-sensing chez Pseudomonas aeruginosa, a été montrée comme possédant également des propriétés antimicrobiennes contre certaines bactéries Gram-négatives ainsi que la capacité à fixer le fer (Kaufmann et al., 2005). Ces métabolites pourraient donc avoir été sélectionnées car possédant plusieurs effets biologiques en fonction de leur concentration dans l’environnement. Cette caractéristique est qualifiée d’hormésie (Yim et al., 2007)
29
Figure 7 : Effet de la concentration de deux antibiotiques sur la croissance bactérienne et sur la transcription (bas de la bandelette : concentration faible ; haut : concentration forte). L’effet antibiotique de l’érythromycine est visualisé en A) et celui de la rifampicine en C). L’intensité lumineuse observée due à l’activation du promoteur luxI par l’érythomycine est observée en B) et celui de la rifampycine en D). La couleur correspond à l’intensité du signal (échelle correspondante est représentée à droite de (B et D) (Goh
et al., 2002).
Pour posséder une activité, il est nécessaire qu’une enzyme puisse interagir avec un ligand (interaction protéine/ligand). Ce ligand doit nécessairement posséder une structure permettant sa fixation précise dans le site actif de la protéine. Cela doit pouvoir se faire à des concentrations physiologiques d’enzyme et de substrat. Il est donc rare qu’une mutation isolée mène à un composé pouvant se fixer efficacement à faible concentration sur une cible. Considérant ces faits, Firn et Jones (2009) proposent une hypothèse : en produisant une grande variété de molécules, les chances qu’un mutant capable de produire une molécule bénéfique apparaisse augmentent. Des espèces produisant une diversité de métabolites secondaires seraient donc sélectionnées par l’évolution même si certains de ces métabolites n’ont pas de « rôle » à proprement parler. Autrement dit, ces clusters de gènes, apparemment
30
sans fonction, auraient pour « rôle » d’évoluer afin de permettre l’apparition d’individus possédant un « kit » de gènes permettant la synthèse d’un composé augmentant sa « fitness ». Ce trait ayant un avantage sélectif, il serait alors fixé au sein de l’espèce.
1.2.3. Evolution du métabolisme secondaire
Comment se transmettent les gènes du métabolisme secondaire ? Comment l’évolution génère-t-elle de nouvelles molécules ? Poser la question de l’évolution du métabolisme secondaire soulève plusieurs points à la fois intéressants et complexes. En effet, il ne s’agit pas ici de comprendre comment évolue un gène, mais un groupe de gènes fonctionnant de façon collective. Les clusters de gènes du métabolisme secondaire sont également considérés comme constituant une des familles d’éléments génétiques parmi les plus diversifiés au sein du monde vivant et évoluant rapidement (Fischbach et al., 2008). Cette partie s’intéressera, d’une part, à la manière dont se propagent ces clusters, et, d’autre part, aux changements menant à l’apparition de nouveaux métabolites.
1.2.3.1. Acquisition de clusters par transfert horizontal
Il est aujourd’hui admis que les clusters de gènes de biosynthèse se propagent par transfert vertical, mais également par transfert horizontal (Fischbach et al., 2007). Plusieurs exemples de clusters acquis par transferts horizontaux ont été reportés chez des bactéries. On peut citer le cas du cluster de biosynthèse du sidérophore yersiniabactine, initialement identifié chez l’agent pathogène de la peste Yersinia pestis (Pelludat et al., 1998), et retrouvé chez de nombreuses espèces, parfois phylogénétiquement éloignées. Ce cluster a notamment été retrouvé chez le symbionte de nématode Photorhabdus luminescens (Duchaud et al., 2003), le pathogène de plantes Pseudomonas syringae, E. coli (Bultreys et al., 2006) et la bactérie marine Salinispora tropica (Udwary et al., 2007). Certaines observations suggèrent que certains clusters auraient pu passer du règne eucaryote au règne procaryote (ou l’inverse), comme illustré avec le cas de β-lactames, retrouvées chez des champignons et des bactéries (Liras et al., 1998). Les clusters du métabolisme secondaires peuvent être transférés d’un organisme à l’autre via un échange de plasmide (ex : méthylènomycine (Wright and Hopwood, 1976)) et/ou par l’insertion de fragments d’ADN aux niveaux de « points chauds » d’intégration. Il a été montré que la présence de gènes d’ARN de transfert (ARNt) pouvait indiquer la présence de tels loci d’intégration (Reiter et al., 1989). En effet, les gènes d’ARNt
31
contiennent fréquemment le site attB, point d’ancrage pour la recombinaison (Hudson et al., 2014). Cette propagation efficace fait que les clusters du métabolisme secondaire se retrouvent chez de nombreuses espèces, évoluant parfois dans des environnements où des pressions évolutives différentes vont s’exercer, menant à l’apparition de nouvelles molécules.
1.2.3.2. Vers de nouveaux clusters de biosynthèse
L’évolution est la conséquence de modifications qui se sont avérées bénéfiques. L’apparition de nouveaux métabolites est donc le résultat de substitutions, de délétions et/ou d’insertions dans la séquence d’ADN des clusters de biosynthèse, conduisant à la synthèse d’une molécule globalement meilleure pour la « fitness » de l’organisme. Ces modifications peuvent ne toucher qu’un seul gène comme plusieurs.
Dans le cas des peptides synthétisés par voie ribosomale, une simple mutation dans le gène responsable de la synthèse du squelette peptidique peut mener à l’apparition de nouveaux métabolites. C’est ce qui a notamment été observé dans le cas du cluster de biosynthèse des patellamides, produits par plusieurs espèces de Prochloron, où une région « hypervariable » est observée dans un gène du cluster quand les six autres sont quasiment identiques (Donia et al., 2006). Les mutations ponctuelles peuvent également affecter la structure de métabolites assemblés par des complexes enzymatiques tels que les PKS et les NRPS. Dans le cas du polycétide ascomycine (FK520), deux modules possédant les domaines DH+KR sont impliqués dans la biosynthèse du métabolite. Alors que le premier domaine DH est fonctionnel, une mutation a rendu le domaine DH présent dans le module suivant inactif, menant à l’incorporation de substrats modifiés de façon différente dans la chaîne d’assemblage (Wu et al., 2000).
Les clusters de biosynthèse peuvent également subir des modifications plus importantes faisant intervenir l’apparition de nouveaux gènes ou, au contraire, la délétion de gènes déjà présents. Ces gènes peuvent être liés à des modifications post-assemblage du squelette de la molécule. Par exemple, dans le cas des antibiotiques de la famille des téicoplanines, où la structure du peptide est conservée, les groupements postérieurement ajoutés sont différents (Figure 8) (Pootoolal et al., 2002). On peut également citer le cas des aminoglycosides. Ces clusters conservent les gènes de biosynthèse de la partie 2-deoxystreptamine alors que les gènes responsables de la synthèse des sucres branchés diffèrent (Flatt and Mahmud, 2007).
32
Un autre aspect à prendre en compte dans l’évolution du métabolisme secondaire est la duplication des clusters au sein du génome. Ces évènements sont associés à une divergence fonctionnelle. En effet, le fait de posséder deux copies d’une voie métabolique autorise l’accumulation de mutations dans une des copies (Fischbach et al., 2008). La duplication liée à la divergence est notamment pensée comme pouvant être à l’origine de la diversité des PKS et des NRPS. Une étude phylogénétique des PKS de Streptomyces a montré que ces PKS ont dérivé d’un module ancestral par évènements de duplication (Jenke-Kodama et al., 2006). Les mêmes résultats ont également été observés pour les PKS de champignons (Kroken et al., 2003). On peut illustrer ces observations avec l’exemple des clusters congocidine et distamycine chez Streptomyces ambofaciens et Streptomyces netropsis (Juguet et al., 2009 ; Hao et al., 2014). Alors que S. ambofaciens possède un unique cluster congocidine (cluster 1), S. netropsis possède une copie « dégénérée » de ce cluster (cluster 2). Il a récemment été montré que, chez cette espèce, la quasi-totalité des gènes essentiels à la synthèse de la congocidine sont présents dans le cluster 1 (un seul gène essentiel situé dans le cluster 2), tandis que la distamycine, ainsi qu’un composé hybride nommé disgocidine, nécessitaient la présence des deux clusters pour être synthétisés (Vingadassalon et al., 2014).
Figure 8 : Différents groupements post-assemblages des molécules de teicoplanine et A47934 possédant le même squelette NRP (Pootoolal et al., 2002).
33
1.3. Les principales familles du métabolisme secondaire et leurs enzymes
Il est possible de classer les métabolites secondaires de plusieurs façons. La partie 1.2.1. présente ces derniers en fonction de leurs activités biologiques et de leurs intérêts pour l’Homme, mais il est également possible de classer ces molécules sur des bases structurales et de modes d’assemblage. En étudiant ces familles de molécules, ainsi que les enzymes impliquées dans leur assemblage, il est parfois possible de prédire des éléments de leur voie de biosynthèse. Il est également possible de prédire la localisation et la fonction des gènes impliqués dans cette biosynthèse. Ces prédictions sont particulièrement utiles pour la recherche de nouveaux métabolites secondaire (Cf partie 3.1.). Cette partie se concentre sur la description de familles majeures du métabolisme secondaire. Il ne s’agit pas ici de donner une liste exhaustive de ces familles, mais plutôt d’offrir un regard sur les mécanismes d’assemblage et la diversité des molécules synthétisables par quelques familles relativement bien étudiées comme les peptides non ribosomaux ou les polycétides.
1.3.1. Les NRPS et peptides non ribosomaux 1.3.1.1. Organisation des NRPS
Les peptides non ribosomaux (non ribosomal peptide : NRP) constituent l’une des principales familles de métabolites secondaires. Ces molécules sont assemblées par des méga-complexes enzymatiques appelés synthétases de peptides non ribosomaux (non ribosomal peptide synthetase : NRPS). Les NRPS sont constituées d'un ou plusieurs modules qui ont pour rôle l’incorporation d’un acide aminé dans la chaîne d’assemblage du peptide. Chacun de ces modules est lui-même subdivisé en domaines. Tous les modules contiennent obligatoirement le domaine d’adénylation (A), le domaine porteur de peptide (peptidyl carrier protein : PCP) et le domaine de condensation (C), à l’exception du module d’initiation qui lui ne contient pas de domaine C. Il existe d’autres domaines, accessoires, qui ne sont pas nécessairement présents mais qui permettent d’apporter des modifications au peptide en cours de synthèse. Ce sont, par exemple, les domaines méthyltransferase (MT) ou épimérase (E). Enfin, le dernier module contient un domaine thioestérase (Te) capable de libérer le peptide (Figure 9) (Finking and Marahiel, 2004).
34
Figure 9 : Chaîne d’assemblage classique d’une NRPS
1.3.1.2. Mode d’assemblage
Le recrutement d’un acide aminé se fait de façon spécifique au niveau du site actif du domaine A. Ce dernier est capable de recruter les 20 acides aminés protéinogéniques mais également d’autres acides aminés (ex : ornithine, β-alanine). L’étude du domaine A-phénylalanine de la gramicidine S a permis de déchiffrer le code de reconnaissance des acides aminés par les domaines A (Stachelhaus et al., 1999). Une fois l’acide aminé recruté, celui-ci est converti en aminoacyl-AMP (consommation d’une molécule d’ATP). Le domaine PCP sur lequel vient se fixer le substrat est préalablement modifié par une enzyme phosphopantétheinyltransferase (PPTase). Celle-ci ajoute un groupement phosphopantétheinyl sur un résidu sérine du module PCP. Cette action permet l’attachement par liaison thioester de l’aminoacyl adénylé par le domaine A sur le domaine PCP ainsi activé (Figure 10) (Lambalot et al., 1996). Le domaine C catalyse la réaction de condensation par liaison peptidique. Cette réaction va de l’acide aminé situé sur le domaine PCP du module en amont vers le PCP situé sur le même module que le domaine C, les facteurs contrôlant cette direction de l’élongation sont encore très mal connus (Fischbach and Walsh, 2006). L’élongation de la chaîne peptidique est unidirectionnelle et se termine au niveau du domaine Te par hydrolyse de la liaison thioester au niveau du dernier domaine PCP. Il existe cela dit des exceptions à ces règles. C’est par exemple le cas de la NRPS impliquée dans la synthèse de la coelichéline, dont un module fonctionne de façon itérative (Lautru et al. 2005).
35
1.3.1.3. Diversité des NRPS
De nombreux métabolites secondaires sont assemblés par des NRPS. On peut notamment citer le groupe ACV (L-aminoadipyl-L-cysteinyl-D-valine), tripeptide impliqué dans la synthèse des antibiotiques de la famille des β-lactames. Les antibiotiques vancomycine (glycopeptide) et daptomycine (lipopeptide) font également partie de la famille des NRP. On peut également citer des molécules ayant d’autres activités, comme la cyclosporine (immunosuppresseur) ou encore la bléomycine (antitumeur).
Cette diversité des NRP s’explique par le nombre de substrats pouvant être sélectionnés par les domaines A, estimé à 500 acides aminés différents. Ces substrats peuvent être modifiés au cours de la chaîne d’élongation (en cis) par exemple par des domaines E ou MT. A ces modifications, s’ajoutent d’autres types de modifications (en trans), par exemple l’ajout de sucres sur la vancomycine (glycosylation) ou encore l’ajout de chaînes d’acides gras sur la daptomycine. Afin de répertorier ces molécules, une banque de données a été créée : NORINE qui compte plus de 1000 NRP (Caboche et al., 2008).
Figure 10 : A) Activation du domaine PCP par la phosphopantétheinyl transférase. B) Recrutement d’un substrat par le domaine A et fixation sur le domaine PCP. C) Réaction de condensation entre les substrats des modules n-1 et n par le domaine de condensation C (adapté de « Antibiotics » Walsh 2003)
36
1.3.2. Les Polycétides synthases
Les polycétides synthases (PolyKetide Synthase : PKS) sont responsables de la synthèse des polycétides, une des grandes familles de métabolites secondaires. Ces métabolites présentent des activités biologiques variées : antibiotique (érythromycine), antifongique (nystatine), antiparasitaire (avermectine) ou encore antitumorale (daunorubicine). Les polycétides sont assemblés à partir de petits acides carboxyliques par les PKS qui peuvent fonctionner de façon modulaire (PKS de type I) ou de façon itérative (PKS de type II et III). Le système d’assemblage des PKS est souvent comparé à celui des synthases d’acide gras (Fatty Acid Synthase : FAS) avec lequel il existe de nombreuses analogies.
1.3.2.1. Mécanisme d’assemblage
Les PKS assemblent de petits acides carboxyliques en une longue chaîne acyle grâce à un nombre défini de cycles d’élongation incorporant chacun 2 à 3 atomes de carbone. Le choix du substrat d’initiation de la chaîne d’assemblage est diversifié, cela peut être de l’acétyl-, du malonyl-, du malonamyl-, du propionyl- ou du butyryl-CoA par exemple. Le choix du substrat d’élongation est en revanche plus réduit, généralement le malonyl- ou méthylmalonyl-CoA (Figure 11).
37
Comme pour les NRPS, les PKS fonctionnent grâce à des domaines ayant chacun un rôle défini. Le substrat est sélectionné par le domaine Acyltransférase (AT) et fixé sur un domaine porteur d’acyle (Acyl Carrier Protein : ACP) préalablement activé grâce à l’ajout d’un bras phosphopantéthéinyl par une phosphopantéthéinyltransférase (PPTase) (Lambalot et al., 1996). La condensation entre les substrats n et n+1, se fait par l’intermédiaire du domaine
céto-synthase (Keto-Synthase : KS) qui, par une décarboxylation, génère un carbone nucléophile permettant la liaison carbone-carbone (C-C) entre les deux substrats. La terminaison de la chaîne se fait grâce au domaine Te (Thioestérase) qui permet la libération de la molécule, par hydrolyse de la liaison thioester, et dans certains cas la cyclisation du polycétide (Weissman, 2009). Tous les domaines précédemment cités sont nécessaires à la synthèse du polycétide. D’autres domaines, accessoires, permettent d’apporter une plus grande diversité de structure. Ces domaines sont les domaines cétoréductase (KetoReductase : KR), déhydratase (DH) et énoylréductase (ER). Le domaine KR seul permet de transformer un groupement β-cétone en groupement alcool. Les domaines KR+DH permettent la déshydratation de l’acyle tout en laissant une liaison Cα=Cβ insaturée. L’action des trois domaines KR, DH et ER laisse cette fois-ci une liaison Cα-Cβ saturée. A ces modifications peuvent s’ajouter d’autres types de modifications post-assemblage, notamment des glycosylations, des réductions et/ou des oxydations.
1.3.2.2. Les différents types de PKS
Comme expliqué précédemment, il existe trois classes de PKS (I, II et III) Les PKS de type I fonctionnent selon un mode analogue aux NRPS. Celles-ci sont organisées sous forme de méga-complexes enzymatiques constitués d'un ou plusieurs modules, chacun impliqué dans l’incorporation d’un acyle dans le polycétide. Dans le cas d’un assemblage faisant intervenir plusieurs complexes enzymatiques, l’ordre d’intervention est déterminé par des séquences spécifiques situées en C-terminal et en N-terminal des complexes, appelés « docking domains » (dd). L’extrémité C-terminale de la PKS n s’accroche spécifiquement à l’extrémité N-terminale du complexe PKS n+1 (Buchholz et al., 2009) (Wu et al., 2002). Chaque module est au moins constitué des domaines AT, PCP et KS à l’exception du module d’initiation qui ne comporte pas de domaine KS et du module de terminaison qui s’accompagne d’un domaine Te nécessaire à la libération du polycétide. Chaque domaine n’est en principe utilisé qu’une seule fois, bien qu’il existe des contre-exemples où un module peut intégrer plusieurs substrats à la chaîne d’acyle. C’est le cas de l’aureothine produite par Streptomyces thioluteus, qui est assemblée par une chaîne PKS de quatre modules exécutant
38
cinq extensions du polycétide (He and Hertweck, 2003). Dans ce cas, on parle de PKS de type I itératives, elles sont néanmoins peu présentes chez les bactéries.
L’exemple le plus cité est celui de l’érythromycine A chez Saccharopolyspora erythraea. Cet antibiotique, dérivé hydroxylé, glycosylé et méthylé du 6-déoxyérythronolide B (6dEB), est assemblé par une PKS constituée de trois chaîne polypeptidiques/sous-unités : DEBS1, 2 et 3 (6-DeoxyErythronolide B Synthase) (Cortes et al., 1990). Deux de ces enzymes (DEBS 2 et 3) sont responsables de l’incorporation de deux acides carboxyliques et une (DEBS 1), responsable de l’incorporation de trois acides carboxyliques (Figure 12).
Figure 12 : Synthèse de l’érythromycine A par des PKS de type I. Les modifications post-PKS sont représentées en rouge sur la molécule finale (adapté de Staunton & Weissman, 2001)
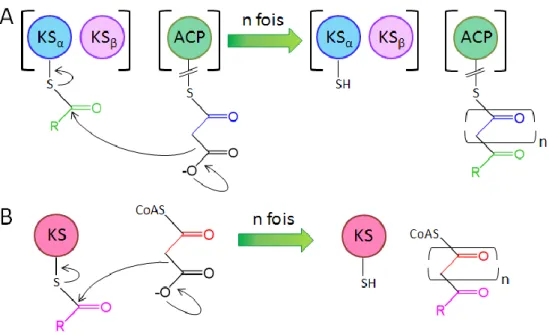
Les PKS de type II sont dites itératives. Chaque enzyme possède une fonction unique, elles se regroupent pour former un complexe multi-enzymatique qui assure la synthèse du polycétide. Contrairement aux PKS de type I, les domaines sont utilisés plusieurs fois pour intégrer les substrats dans la chaîne d’élongation. Une PKS de type II « minimale » est constituée d’un domaine ACP unique et de deux domaines KS (KSα et KSβ) (Figure 13A). Le domaine KSα assure les réactions de condensation entre les différents substrats. Le domaine KSβ est lui impliqué dans le contrôle du nombre de cycle d’élongation. Il est également
39
appelé CLF (Chain Length Factor). A ce complexe minimal peuvent s’ajouter d’autres enzymes pouvant modifier la structure de la chaîne. Ces enzymes sont les cétoréductase (KetoReductase: KR), les cyclases (CYC) et les aromatases (ARO). A ces modifications peuvent s’ajouter des oxydations, des hydroxylations et des méthylations une fois la chaîne d’assemblage terminée (Rix et al., 2002). Le substrat d’initiation choisi est la plupart du temps l’acétyl-CoA, bien que d’autres choix soient possibles (propionyl-CoA dans le cas de la daunorubicine). La seule unité d’élongation connue pour les PKS de type II est le malonyl-CoA, apporté du métabolisme primaire par une enzyme MAT (Malonyl-CoA:ACP AcylTransferase). Les polycétides générés par les PKS de type II sont en général aromatiques (famille des tétracyclines) et moins complexes que ceux synthétisés par les PKS de type I.
Le dernier type de PKS, le type III est sans doute le moins bien connu. Cette classe est également appelée chalcone synthase en raison du fait qu’il s’agit de la première enzyme de ce type qui fut caractérisée. La majorité des PKS de type III connues sont synthétisées par les plantes bien qu’il existe des exemples au sein du règne bactérien (1,3,6,8-tetrahydroxynaphtalene chez Streptomyces griseus (Funa et al., 1999)). Les produits des PKS de type III sont la plupart du temps des molécules aromatiques monocycliques ou bicycliques (Yu et al., 2012). Ces enzymes diffèrent des PKS de type I et II par l’absence de domaine ACP. L’incorporation d’unité d’élongation se fait directement sur l’acyl-CoA par un unique module KS (Figure 13B) (Austin and Noel, 2003).
Figure 13 : A) Schéma d'assemblage des PKS de type II. B) Schéma d'assemblage des PKS de type III (Weissman 2009)
40
1.3.3. Autres familles du métabolisme secondaire
Outre les polycétides et les NRP (et les hybrides polycétide-NRP), il existe de nombreuses familles de métabolites secondaires. On peut citer les composés C-P (ex: fosfomycine) (Rogers and Birnbaum, 1974), qui comme leur nom l’indique, contiennent des liaisons carbone-phosphate. Les lantibiotiques (ex : nisine) (Buchman et al ., 1988), qui sont des peptides ribosomaux utilisant des acides aminés reliés par des liaisons thio-esters (lanthionines et méthyllantionines) ainsi que des acides aminés déshydratés (Asaduzzaman and Sonomoto, 2009). Les cyclodipeptides sont, quant à eux, de petites molécules cycliques constituées de deux acides aminés qui, dans certains cas, ne sont pas assemblés par un système NRPS (ex : albonoursine) (Gondry et al., 2009 ; Lautru et al., 2002). Contrairement au NRP et au polycétides, certaines familles de métabolites secondaires sont relativement peu caractérisées et/ou avec peu de molécules décrites. Cette partie propose de s’intéresser à deux familles de métabolites bien caractérisées et dont les mécanismes d’assemblage diffèrent de ceux explicités précédemment : les terpènes et les aminoglycosides.
1.3.3.1. Les terpènes
Les terpènes sont une large famille de métabolites secondaires possédant des structures variées. Bien qu’identifiées principalement chez les plantes, ces molécules sont également présentes chez les bactéries, en particulier les Actinobactéries comme les Streptomyces (Cane and Ikeda, 2012). Ces métabolites peuvent avoir différentes activités biologiques. Certaines de ces molécules ont des propriétés odorantes, comme la géosmine qui donne aux Streptomyces une odeur de « terre humide ». D’autres peuvent posséder des propriétés antibiotiques comme le pentalenolactone ou le terpène glycoside phénalinolactone (Figure 14) (Binz et al., 2008).
Les terpènes sont assemblés à partir d’unités d’isoprène (5 carbones) reliées les unes aux autres selon un mode tête-queue. On distingue les hémiterpènes (C5), les
monoterpènes (C10) les sesquiterpènes (C15), les diterpènes (C20), les sesterterpènes (C25),
les triterpènes (C30) et les tétraterpènes (C40). La synthèse d’un monoterpène se fait à
partir d’isopentenyl pyrophosphate (IPP) et de son isomère le diméthylallyl pyrophosphate (DMAPP) pour donner une molécule de géranyl diphosphate (GPP). L’ajout successif de monomères d’IPP permet d’obtenir une chaîne allant jusqu’à 25 carbones (Figure 14). Des chaînes plus longues peuvent être obtenues par la dimérisation de chaînes de 15 ou 20