THÈSE
Pour l'obtention du grade de
DOCTEUR DE L'UNIVERSITÉ DE POITIERS UFR des sciences fondamentales et appliquées Ecologie et biologie des interactions - EBI (Poitiers)
(Diplôme National - Arrêté du 7 août 2006)
École doctorale : Sciences pour l'environnement - Gay Lussac (La Rochelle) Secteur de recherche : Aspects moléculaires et cellulaires de la biologie
Présentée par :
Vincent Delafont
Diversité et implication des amibes libres dans la survie
et la persistance des mycobactéries non tuberculeuses
au sein d'un réseau d'eau potable
Directeur(s) de Thèse :
Yann Héchard, Didier Bouchon, Laurent Moulin Soutenue le 21 octobre 2015 devant le jury Jury :
Président Emmanuelle Cambau Professeur, APHP Lariboisière, Paris Rapporteur Thierry Soldati Professeur, Université de Genève, Suisse
Rapporteur Benoit Cournoyer Directeur de recherche CNRS, Université de Lyon 1 Membre Yann Héchard Professeur des Universités, Université de Poitiers Membre Didier Bouchon Professeur des Universités, Université de Poitiers
Membre Laurent Moulin Docteur, Eau de Paris
Membre Vincent Thomas Docteur, BIOASTER, Paris
Pour citer cette thèse :
Vincent Delafont. Diversité et implication des amibes libres dans la survie et la persistance des mycobactéries non tuberculeuses au sein d'un réseau d'eau potable [En ligne]. Thèse Aspects moléculaires et cellulaires de la biologie. Poitiers : Université de Poitiers, 2015. Disponible sur Internet <http://theses.univ-poitiers.fr>
THESE
Pour l’obtention du grade de
DOCTEUR DE L’UNIVERSITÉ DE POITIERS
FACULTÉ DES SCIENCES FONDAMENTALES ET APPLIQUÉES(Diplôme National – Arrêté du 7 août 2006)
Ecole Doctorale : Science pour l’environnement Gay Lussac Spécialité : Aspects Moléculaires et Cellulaires de la Biologie
D
IVERSITE ET IMPLICATION DES AMIBES LIBRES
DANS LA SURVIE ET LA PERSISTANCE DES
MYCOBACTERIES NON TUBERCULEUSES AU SEIN
D
’
UN RESEAU D
’
EAU POTABLE
Préparée au sein des laboratoires Ecologie et Biologie des Interactions (équipe Microbiologie de l’Eau) Poitiers, France, et de la Direction de la Recherche et Développement pour la Qualité d l’Eau (Equipe R&D Biologie) Ivry/Seine, France
Présentée par :
Vincent DELAFONT
Directeurs de Thèse : Yann Héchard, Professeur, Université de Poitiers Laurent Moulin, Docteur, Eau de Paris
Didier Bouchon, Professeur, Université de Poitiers
Soutenue le 21 octobre 2015, devant la commission d’examen
JURY
Rapporteurs : Professeur Thierry Soldati (Université de Genève)
Professeur Benoit Cournoyer (Université Claude Bernard, Lyon 1) Examinateurs : Professeur Yann Héchard (Université de Poitiers)
Professeur Didier Bouchon (Université de Poitiers) Docteur Laurent Moulin (Eau de Paris)
Professeur Emmanuelle Cambau (CNR-MyRMA, APHP) Docteur Vincent Thomas (BIOASTER)
THESE
Pour l’obtention du grade de
DOCTEUR DE L’UNIVERSITÉ DE POITIERS
FACULTÉ DES SCIENCES FONDAMENTALES ET APPLIQUÉES(Diplôme National – Arrêté du 7 août 2006)
Ecole Doctorale : Science pour l’environnement Gay Lussac Spécialité : Aspects Moléculaires et Cellulaires de la Biologie
D
IVERSITE ET IMPLICATION DES AMIBES LIBRES
DANS LA SURVIE ET LA PERSISTANCE DES
MYCOBACTERIES NON TUBERCULEUSES AU SEIN
D
’
UN RESEAU D
’
EAU POTABLE
Préparée au sein des laboratoires Ecologie et Biologie des Interactions (équipe Microbiologie de l’Eau) Poitiers, France, et de la Direction de la Recherche et Développement pour la Qualité d l’Eau (Equipe R&D Biologie) Ivry/Seine, France
Présentée par :
Vincent DELAFONT
Directeurs de Thèse : Yann Héchard, Professeur, Université de Poitiers Laurent Moulin, Docteur, Eau de Paris
Didier Bouchon, Professeur, Université de Poitiers
Soutenue le 21 octobre 2015, devant la commission d’examen
JURY
Rapporteurs : Professeur Thierry Soldati (Université de Genève)
Professeur Benoit Cournoyer (Université Claude Bernard, Lyon 1) Examinateurs : Professeur Yann Héchard (Université de Poitiers)
Professeur Didier Bouchon (Université de Poitiers) Docteur Laurent Moulin (Eau de Paris)
Professeur Emmanuelle Cambau (CNR-MyRMA, APHP) Docteur Vincent Thomas (BIOASTER)
"Imagination is more important than knowledge.
Knowledge is limited. Imagination encircles the world."
- Albert Einstein
Remerciements
Cette étude a été effectuée au sein des équipes Microbiologie de l’Eau, de l’UMR CNRS 7267 Ecologie et Biologie des Interactions dirigée par Didier Bouchon, et R&D Biologie de la Direction de la Recherche, du Développement et de la Qualité de l’Eau d’Eau de Paris dirigée par Michel Joyeux. Je leur adresse mes plus sincères remerciements pour m’avoir donné l’opportunité de réaliser ces travaux au sein de leurs laboratoires. Merci également à
Yann Héchard et Laurent Moulin qui m’ont accueilli eu sein de leurs équipes.
J’adresse mes remerciements à Thierry Soldati, Professeur dirigeant son laboratoire au département des Sciences Biochimiques, à l’Université de Genève, ainsi qu’à Benoit
Cournoyer, Directeur de Recherche CNRS au sein de l’équipe de recherche Bactéries
Pathogènes Opportunistes et Environnement, à l’Université Lyon 1, pour m’avoir fait l’honneur d’accepter de rapporter ce travail. Veuillez trouver par ces mots l’expression de ma plus sincère gratitude.
Je remercie également très chaleureusement Emmanuelle Cambau, Professeur et Praticienne Hospitalière, Chef du service de Bactériologie-Hygiène de l’hôpital Lariboisière, à l’Assistance Publique des Hôpitaux de Paris, ainsi que Vincent Thomas, responsable de l’unité Biocollections au sein de Bioaster, d’avoir accepté d’évaluer ce travail. Votre présence parmi le jury de thèse représente un véritable honneur.
Les mots me manquent véritablement pour remercier mon directeur de thèse Yann
Héchard, pour cette fabuleuse expérience qu’a été ce travail de thèse. Merci d’avoir cru en
moi dès le début, depuis la Licence. Merci de m’avoir enseigné, encadré, conseillé, aiguillé tout au long de ce travail et plus encore. Merci également pour toutes les discussions (scientifiques ou non), les moments en dehors de travail, le soutient lorsqu’il y en avait beaucoup, et ton implication tout au long de cette aventure. Je comprends avec le recul que l’utilisation de ces mots n’est pas anodine, tant ils reflètent non pas seulement ton impact sur ma vie professionnelle, mais également sur ma personne. Yann, j’ai trouvé en toi un mentor, je sors grandi de cette expérience grâce à toi, et je te serai éternellement reconnaissant pour tout cela.
Je ne sais comment remercier Laurent Moulin, pour m’avoir encadré au cours de ce travail à Eau de Paris. Merci de m’avoir transmis ta passion pour la Recherche, et de m’avoir fait découvrir le monde de l’eau potable. Au delà même du cercle professionnel, tu m’a
passage (de presque 2 ans) à Paris. Nos discussions, autour des tables de réunions, ou d’un troquet parisien, m’ont enrichies sur le plan scientifique, mais également sur le plan culturel, tant ton savoir parait inépuisable ! En plus de tes grandes qualités d’Homme de Science, la générosité dont tu as fait preuve à mon égard est un fait pour lequel je te serai toujours reconnaissant.
Merci enfin à Didier Bouchon, pour m’avoir co-encadré tout au long de la thèse. Merci pour ta pédagogie, tes compétences qui m’ont permis de véritablement développer ma passion pour ce travail de thèse. Je me sens privilégié du temps que tu as pu m’accorder tout au long de la thèse, et honoré d’avoir appris tant de choses auprès de toi.
Mes plus sincères remerciements vont également aux personnes avec qui j’ai eu la chance de collaborer, en particulier Emmanuelle Cambau, Faïza Mougari et Laurent Raskine, au CNR-MyRMA. Merci à vous de m’avoir accueilli, aidé et tant appris sur les mycobactéries. Vos connaissances et compétences n’ont d’égales que votre gentillesse ; grâce à vous j’allais toujours à l’hôpital avec le sourire (!), et ce travail n’aurait pu être tel qu’il est sans votre aide.
Merci également à Laurence Guidicelli et Sylvie Dubrou, du Laboratoire d’Hygiène de la Ville de Paris, qui m’ont permis de récupérer ces nombreuses cultures de mycobactéries au cours de la campagne de prélèvement.
J’exprime mes remerciements aux membres de la DRDQE à Eau de Paris, avec qui j’ai pu travailler et passer de bons moments, plus particulièrement à Sébastien, Sophie, Héberte et Marion de l’équipe R&D Biologie. Votre accueil au sein de l’équipe a été formidable, et même si Sébastien me faisait peur au début, j’ai plus qu’apprécié les bons moments partagés au labo. Merci à Jean, Guillaume, Alexis et Xavier, les voisins de la R&D chimie des matériaux, pour leur bonne humeur et les parties de rigolade ; il faut dire, c’est facile avec le rire communicatif de Xavier !
Je souhaite remercier tout particulièrement Carole et sa bonne humeur à toute épreuve, sans qui j’aurais été perdu dans les méandres administratifs ! Avec toi, tout paraissait si simple ! Un grand merci également à Anne et à tout le département prélèvement, sans qui ces travaux n’auraient tout simplement pas pu être accomplis. Merci à Gwenaelle pour les discussions quand nous nous retrouvions lors des pauses !
Merci à Benoit, Adélaïde et Camille, avec qui j’ai passé des moments exceptionnels. Les passages au Bootlagers, ou à manger des flammekueches à volo au FLAM’s, en tentant de répondre aux questions bizarre de Benoit… Merci à vous de m’avoir accueilli à Paris, surtout quand je préviens que j’arrive la veille ! Grâce à vous j’ai passé de super moments.
Je remercie également l’ensemble des membres de l’équipe Microbiologie de l’Eau, qui m’ont accueilli tout au long de la thèse, mais aussi assez régulièrement depuis la Licence. Merci aux « tenanciers » du labo, Yann et Jean-Marc. Jean-Marc, tu m’impressionneras toujours par ton don divin à nous affubler de magnifiques sobriquets. Merci également pour ton écoute, ta bonne humeur communicative et ta pédagogie, avec toi la biochimie devient magique ! Et je persiste à dire que le Transperceneige c’est pas mal ! Merci du fond du cœur à Emilie, ma collègue de bureau (a.k.a. chasseuse de coquilles, ou WAR MACHINE). Je pense que notre consommation de café les matins en discutant de tout et de rien doit maintenant pouvoir se compter en mètres cubes. Je n’aurais pu rêver de meilleure collègue/amie pour partager ce bureau et tous ces moments de joie, de stress, de craquage total. Peu importe où nous finirons, on se retrouvera ! Je remercie particulièrement Ascel, mon premier collègue de bureau, et mon instructeur de biologie cellulaire. Tu as véritablement influé sur mon travail de thèse, en m’apprenant tant de choses et en me donnant tous ces conseils ; tu es mon directeur de thèse officieux (chut !). Au delà du travail, nous avons quand même vécu des choses assez inoubliables à Vienne, et par deux fois ! Je pense que notre choix d’hôtel nous a permis de vivre une expérience unique qui nous a… changé !
Merci à Julien, pour toujours être disponible quand j’ai eu besoin de conseils, ou tout simplement de décompresser. Je garderai d’excellents souvenirs de nos conversations, qui malheureusement, sont restées incomprises (ou mal interprétées dirons-nous) au labo… Merci aux membres du labo basés au CHU, Marie-Hélène, Christine, Estelle, Marion,
Elodie et Damien, pour les collaborations et les bons moments partagés. Un labo ne serait
pas un vrai labo sans ses stagiaires, et je dois admettre avoir fait face à des cuvées exceptionnelles ces dernières années. Un immense merci à Ségolène et Antoine, pour ces quelques mois qui me font l’impression de vous connaître depuis des années ! Même si vous vous retrouvez au fin fond de nul part (ce sera d’ailleurs un peu la cas quand même Ségo), on se retrouvera aussi. Merci à tous les collègues du labo, Luce, Laurence
Christophe, Dominique, Stéphanie, Clémence, Willy grâce à qui j’ai pu vivre une
expérience de thèse absolument géniale.
Au delà de l’équipe, je souhaite remercier plusieurs membres de l’équipe Ecologie, Evolution Symbioses qui ont un rôle important au cours de ces travaux. Tout d’abord, merci encore à Didier, pour toute l’aide apportée au cours ce cette thèse. Un grand merci à
Bouziane, pour m’avoir fait bénéficié de ces talents de bioinformaticien. Merci à Jessica,
qui a essuyé les plâtres des manips de pyroséquençage, nous permettant ainsi de tirer profit de son expérience. Merci Joanne pour ton incroyable expérience en microscopie, et tes précieux conseils en FISH notamment.
Je finirai par remercier toutes les personnes qui m’ont entouré au cours de cette aventure, amis et famille. Merci à ma mère et ma sœur, qui sont mes soutiens inconditionnels.
Maman, Sandrine, vous êtes mes modèles, si un jour je pouvais envisager d’égaler vos
accomplissements, d’être une personne aussi bonne, alors j’aurai réussi ma vie, c’est certain. Alain et Lydie, merci pour tout le soutient, votre gentillesse envers moi, et tous ces moments exceptionnels passés en votre présence, je ne pouvais rêver de meilleure famille que vous. J’adresse mes remerciements les plus sincères à Daniel et Eliane, qui m’ont ouvert leur porte et tant donné au cours de mes nombreux séjours à Paris. Je vous suis reconnaissant que toute l’aide que vous m’avez apportée. Trouvez en ces mots toute mon affection et ma gratitude.
Merci à Louis, mon vieux frère viking, dont l’amitié, solide telle un roc, est mon point de repère.
Enfin, Marlene, merci. Sans toi, je ne serais pas la moitié de ce que je suis à présent. Tu es la raison pour laquelle j’ai fait ce travail, tout autant que la personne qui m’a donné les moyens de le faire. Tu as cru en moi, tu m’as soutenu, tu as été là, tout simplement, et sans cela je n’aurais pas pu en arriver là. Je t’aime, pour toutes ces raisons et plus encore.
Sommaire
Liste des abréviations ________________________________________________________________ 1 Liste des figures ______________________________________________________________________ 3 Liste des tableaux ____________________________________________________________________ 5
Introduction générale _________________________________________________________________ 6 Etude Bibliographique ________________________________________________________________ 8
I. Le réseau d’eau potable de Paris ____________________________________________________ 9 I.1. Les sources d’approvisionnement ______________________________________________ 10 I.2. Les filières de potabilisation _____________________________________________________ 11
I.2.1. Traitement des eaux de surface _____________________________________________________ 11 I.2.1. Traitement des eaux souterraines __________________________________________________ 14
I.3. Distribution de l’eau potable ___________________________________________________ 15 I.4. Ecologie des réseaux d’eau potable ____________________________________________ 16
I.4.1. Les biofilms dans les réseaux d’eau potable _________________________________________ 17 I.4.2. Vers la caractérisation du microbiome des réseaux d’eau potable ___________________ 18
II. Les amibes libres dans l’eau _______________________________________________________ 19 II.1. Taxonomie ____________________________________________________________________ 21 II.2. Morphologie __________________________________________________________________ 23
II.2.1. Le trophozoïte _____________________________________________________________________ 23 II.2.2. Le kyste ___________________________________________________________________________ 25 II.2.3. La forme flagellée _________________________________________________________________ 27 II.2.4. La forme sociale __________________________________________________________________ 27
II.3. Nutrition ______________________________________________________________________ 28
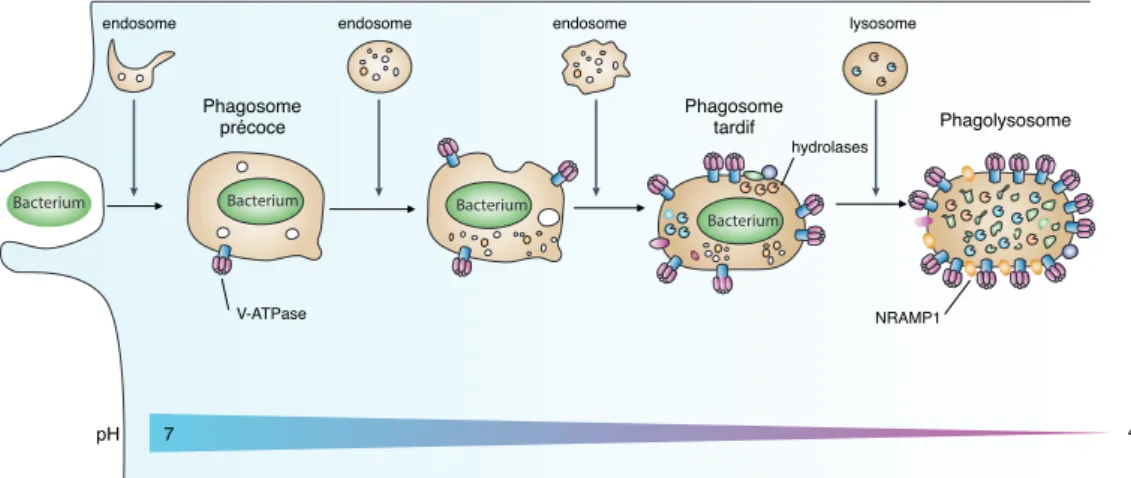
II.3.1. Sources nutritives _________________________________________________________________ 28 II.3.2. La phagocytose ___________________________________________________________________ 29 II.3.2.1 La reconnaissance ____________________________________________________________ 29 II.3.2.2 L’internalisation _______________________________________________________________ 29 II.3.2.3 Le phagosome ________________________________________________________________ 30
II.4. Pouvoir pathogène ____________________________________________________________ 31 II.5. Ecologie des amibes dans les eaux douces destinées à la consommation ______ 32
II.5.1. Les amibes libres dans les sources d’eau douce ____________________________________ 32 II.5.2. Les amibes dans les réseaux d’eau artificiels ______________________________________ 33
III. Les mycobactéries ________________________________________________________________ 35 III.1. Taxonomie ____________________________________________________________________ 35
III.1.2. Les mycobactéries non tuberculeuses ____________________________________________ 37
III.2. Morphologie __________________________________________________________________ 39
III.2.1. La paroi des mycobactéries _______________________________________________________ 40
III.3. Physiologie ___________________________________________________________________ 41
III.3.1. Croissance _______________________________________________________________________ 41
III.4. Génomique ___________________________________________________________________ 42
III.4.1. L’opéron rrn chez les mycobactéries ______________________________________________ 43 III.4.2. Les gènes PE/PPE uniques au genre Mycobacterium ______________________________ 43 III.4.3. Les systèmes de sécrétion de type 7 ______________________________________________ 44
III.5. Pouvoir pathogène des mycobactéries non tuberculeuses _____________________ 45
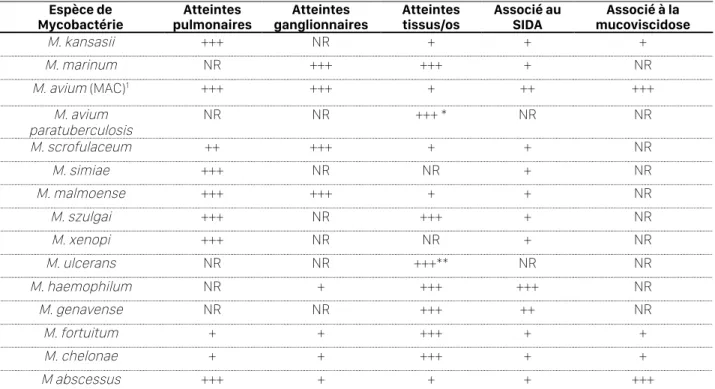
III.5.1. Atteintes pulmonaires ____________________________________________________________ 46 III.5.2. Atteintes ganglionnaires __________________________________________________________ 47 III.5.3. Atteintes cutanées _______________________________________________________________ 48 III.5.4. Les mycobactérioses à MNT chez les patients atteints d’immunodéficience et de mucoviscidose _________________________________________________________________________ 48
III.6. Ecologie des mycobactéries non tuberculeuses dans les réseaux d’eau ________ 48
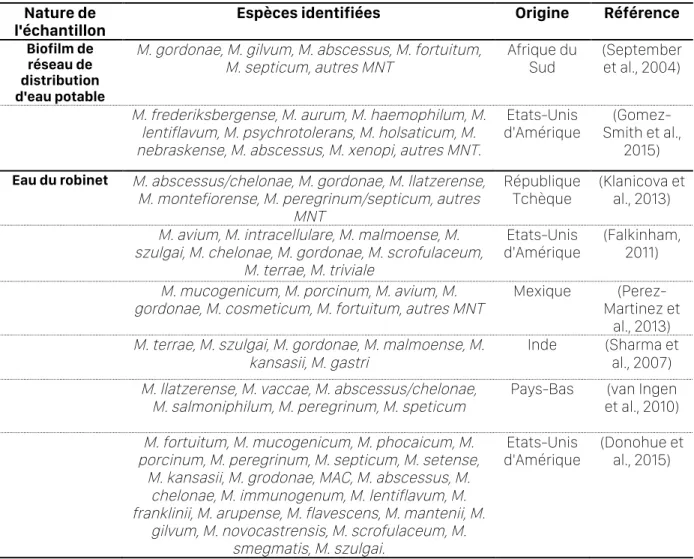
III.6.1. Occurrence des mycobactéries non tuberculeuses dans les réseaux d’eau potable _ 49 III.6.2. Résistance aux traitements de désinfection _______________________________________ 52
IV. Les interactions entre amibes libres et bactéries __________________________________ 53 IV.1. Les relations endosymbiotiques entre amibes et bactéries _____________________ 54
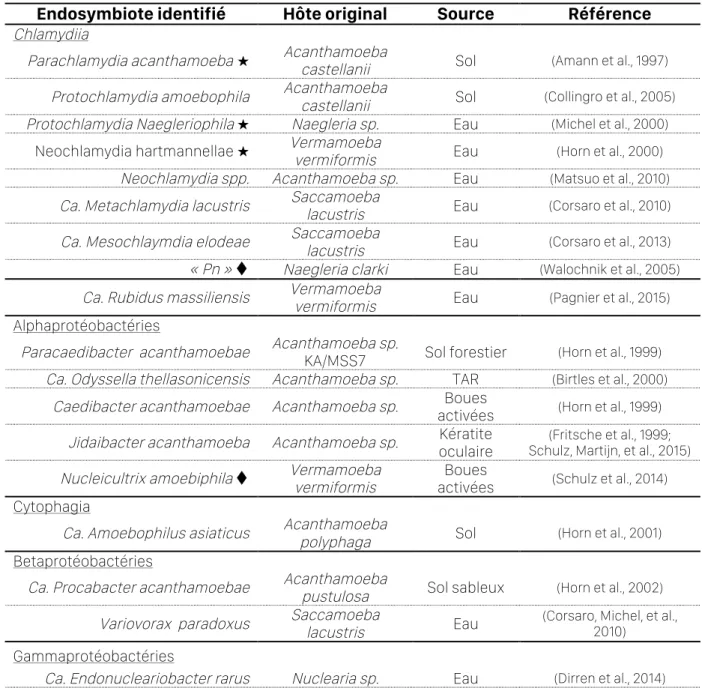
IV.1.1. Les endosymbiotes d’amibes libres ________________________________________________ 54 IV.1.1.1 Diversité des endosymbiotes d’amibes _________________________________________ 54 IV.1.1.2 Nature des interactions endosymbiotiques ____________________________________ 56 IV.1.1.3 Rôle des amibes libres dans l’écologie de la matière noire microbienne ? _______ 58
IV.2. Les bactéries résistantes aux amibes _________________________________________ 59
IV.2.1. Diversité des ARB _________________________________________________________________ 59
IV.3. Mécanismes de survie à la prédation amibienne _______________________________ 61
IV.3.1. Evitement de la phagocytose _____________________________________________________ 61 IV.3.2. La résistance à la phagocytose ___________________________________________________ 62 IV.3.2.1 Legionella pneumophila ______________________________________________________ 62 IV.3.2.2 Les mycobactéries non tuberculeuses ________________________________________ 64
Mycobacterium avium __________________________________________________________ 65 (1) Mycobacterium marinum _______________________________________________________ 66 (2) Mycobacterium abscessus _____________________________________________________ 68 (3)
IV.4. Les amibes comme lieu d’échange génétique _________________________________ 69
Matériel et Méthodes ________________________________________________________________ 73
I. Campagne d’échantillonnage du réseau d’eau potable de Paris _____________________ 74 I.1. Echantillonnage ________________________________________________________________ 74 I.2. Traitement des échantillons ____________________________________________________ 75 II. Microorganismes __________________________________________________________________ 76 II.1. Souches de microorganismes _________________________________________________ 76 II.2. Culture d’amibes libres ________________________________________________________ 77
II.2.1. Culture des échantillons d’amibes environnementaux ______________________________ 77 II.2.2. Cultures d’amibes liquides en milieu monoxénique liquide _________________________ 77 II.2.3. Cultures d’amibes axéniques ______________________________________________________ 77
II.3. Culture bactérienne ___________________________________________________________ 78
II.3.1. Mycobactéries non tuberculeuses _________________________________________________ 78 II.3.1.1 Isolement des mycobactéries de l’eau potable __________________________________ 78 II.3.1.2 Entretien des souches en laboratoire __________________________________________ 78 II.3.2. Culture d’Escherichia coli _________________________________________________________ 78
II.4. Culture des levures ____________________________________________________________ 79 II.5. Co-cultures d’amibes et bactéries _____________________________________________ 79
II.5.1. Infection des amibes par les mycobactéries ________________________________________ 79 II.5.1.1 Internalisation des mycobactéries par les amibes _______________________________ 79 II.5.1.2 Evaluation de la persistance des mycobactéries au sein des amibes ____________ 80 II.5.1.3 Induction de la voie autophagique, et inhibition de la maturation des phagosomes chez les amibes infectées ____________________________________________________________ 80 II.5.1.4 Test de résistance en goutte ___________________________________________________ 80
II.6. Numération des microorganismes _____________________________________________ 81
II.6.1. Numération des bactéries _________________________________________________________ 81 II.6.1.1 Numération par densité optique ________________________________________________ 81 II.6.1.2 Numération par unité formant colonies (UFC) ___________________________________ 81 II.6.2. Numération des amibes ___________________________________________________________ 81 II.6.2.1 Comptage direct des cellules ambiennes _______________________________________ 81 II.6.2.2 Comptage différentiel des trophozoïtes et des kystes amibiens ________________ 81
II.7. Coloration des mycobactéries selon Ziehl-Neelsen _____________________________ 82 III. Méthode de biologie moléculaire __________________________________________________ 83 III.1. Extraction d’acides nucléiques ________________________________________________ 83
III.1.1. Extraction d’ADN __________________________________________________________________ 83 III.1.2. Extraction d’ARN __________________________________________________________________ 83 III.1.3. Transcription inverse ______________________________________________________________ 84
III.2. Polymerase Chain Reaction : PCR _____________________________________________ 84 III.3. PCR quantitative (PCRq) ______________________________________________________ 85
III.3.1. Réalisation de la gamme étalon ___________________________________________________ 86 III.3.2. Amplification _____________________________________________________________________ 86
III.4. Purification des produits de PCR ______________________________________________ 88 III.5. Séquençage par la méthode dérivée de Sanger ________________________________ 88 III.6. Pyroséquençage ______________________________________________________________ 89
III.6.1. Principe __________________________________________________________________________ 89 III.6.2. Protocoles _______________________________________________________________________ 90 III.6.2.1 Séquençage des génomes de mycobactéries environnementales ______________ 90 III.6.2.2 Metabarcoding _______________________________________________________________ 90
III.7. Identification des bactéries par spectrométrie de masse MALDI-TOF ___________ 91 IV. Méthodes d’analyses bioinformatiques ____________________________________________ 92 IV.1. Analyses génomiques _________________________________________________________ 92 IV.2. Analyses de diversité _________________________________________________________ 92
IV.2.1. Quantitative Insights Into Microbial Ecology (QIIME) ________________________________ 92 IV.2.2. Analyses statistiques _____________________________________________________________ 93
IV.3. Analyses phylogénétiques ____________________________________________________ 93 V. Techniques de microscopie _______________________________________________________ 94 V.1. Hybridation fluorescente in situ (FISH) _________________________________________ 94 V.2. Microscopie électronique à transmission ______________________________________ 94
Résultats et Discussion _____________________________________________________________ 95
I. Etude de la diversité des amibes libres et de leur microbiome bactérien au sein du réseau d’eau potable ________________________________________________________________ 96
I.1. Microbiome des amibes libres isolées d’un réseau d’eau potable ________________ 96
I.1.1. Publication 1 : Microbiome of free-living amoebae isolated from drinking water _______ 97 I.1.2. Résultats complémentaires _______________________________________________________ 106 I.1.2.1 Occurrence des amibes libres dans les échantillons d’eau potable ______________ 106 I.1.2.2 Evaluation des charges bactériennes du microbiome des amibes libres. ________ 107
I.2. Impact des conditions environnementales sur le diversité des amibes libres et leur microbiome bactérien, au sein du réseau d’eau potable ___________________________ 109
I.2.1. Publication 2 : Environmental factors shaping free-living amoebae population and their microbiome in drinking water network __________________________________________________ 109 I.2.2. Résultats complémentaires _______________________________________________________ 127 I.2.2.1 Occurrence des amibes par point de prélèvement ______________________________ 127
I.3. Rôle des amibes libres dans l’écologie de la matière noire microbienne ________ 128
I.3.1. Publication 3 : Shedding light on microbial dark matter: a TM6 bacterium as natural endosymbiont of a free-living amoeba __________________________________________________ 128
II. Implication des amibes libres dans la survie et la persistance des mycobactéries non tuberculeuses dans un réseau d’eau potable ________________________________________ 145
II.1. Relation entre amibes libres et mycobactéries non tuberculeuses (MNT) au sein du réseau d’eau potable _____________________________________________________________ 145
II.1.1. Publication 4 : First evidence of amoebae – mycobacteria association in drinking water network. _______________________________________________________________________________ 145 II.1.2. Résultats complémentaires _______________________________________________________ 159 II.1.2.1 Identification des mycobactéries colonisant le réseau d’eau potable. ___________ 159
II.2. Caractérisation des stratégies de survie des mycobactéries non tuberculeuses à la prédation amibienne. _____________________________________________________________ 161
II.2.1. Publication 5 : Mycobacterium llatzerense, a waterborne mycobacterium, that resists phagocytosis by Acanthamoeba castellanii ______________________________________________ 161 II.2.2. Résultats complémentaires ______________________________________________________ 187 II.2.2.1 Caractérisation de la résistance de M. chelonae à la prédation amibienne ______ 187 Résistance à la phagocytose ___________________________________________________ 187 (1)
Persistance et croissance intracellulaire _______________________________________ 188 (2)
Criblage génomique des facteurs de résistance ________________________________ 189 (3)
II.3. Discussion ___________________________________________________________________ 192
Conclusion __________________________________________________________________________ 196 Bibliographie _______________________________________________________________________ 200 Annexes ____________________________________________________________________________ 221
I. Implication des amibes libres dans les infections liées au soins ____________________ 222 II. Analyse par pyroséquençage de la diversité bactérienne colonisant les units de soin dentaire ___________________________________________________________________________ 233
Liste des abréviations
AG : arabinogalactane AM : arabinomannane
ARNr : acide ribonucléique ribosomique ATP : adénosine triphosphate
BLAST : basic local alignment search tool CCD : charge-coupled device
CFP-10 : culture filtrate protein 10 kDa CHU : centre hospitalo-universitaire CWD : cell wall deficient
DO : densité optique
dot/icm : defect in organelle trafficking/intracellular replication EAG : encéphalite amibienne primitive
emPCR : emulsion polymerase chain reaction ESAT-6 : early secreted antigenic target 6 kDa ESX : early secretory antigenic target system FISH : fluorecent in situ hybridization
GC : guanine et cytosine gen. nov. : genus novum
iNOS : inducible nitric oxyde synthase LCV : Legionella containing vacuole LLAP : Legionella like amoebal pathogen LOS : lipooligosaccharides
MAC : Mycobacterium avium complex
MALDI-TOF : matrix-assisted laser desorption ionisation – time of flight MAMPS : microbes associated molecular patterns
MAP : méningite amibienne primitive MBP : mannose binding protein mce : mammalian cell entry operon MID : multiplex identifier
MNT : mycobactéries non tuberculeuses MOI : multiplicity of infection
NAM : d’acide muramique N-acétylé
NRAMP1 : natural resistance associated macrophage protein 1 OTU : operational taxonomic unit
PAS : Page’s amoeba saline
PE/PPE : proline glutamine/proline proline glutamine PG : peptidoglycane
PI3K : phosphoinositide 3-kinase PLC : phospholipase C
PPi : pyrophosphate ppm : partie par million
PRR : pathogen recognition receptor PTP : picotiter plate
PYG : proteose peptone, yeast extract, glucose medium QIIME : quantitative insights into microbial ecology RAST : rapid annotation using subsystem technology SDS : sodium dodécyl sulfate
SIDA : syndrome d’immunodéficience acquise spp. : species pluralis
T4SS : type 4 secretion system T7SS : type 7 secretion system UDI : unité de distribution UFC : unité formant colonie v/v : volume/volume
Liste des figures
Figure 1 : Les différentes sources d’eau alimentant le réseau d’eau potable de Paris. _____ 10 Figure 2 : Principe de fonctionnement des filières de potabilisation des eaux de surface
mises en place dans les usines d’Orly traitant l’eau de Seine, et Joinville traitant l’eau de Marne. _________________________________________________________________________ 13 Figure 3 : Principe de fonctionnement des filières de potabilisation des eaux souterraines
mise en place au sein des usines de l’Hay-Les-Roses, Saint-Cloud, Sorques et Longueville. _______________________________________________________________________ 14 Figure 4 : Carte des unités de distribution (UDI) de l’eau potable à Paris. _________________ 15 Figure 5 : Aperçu global d’un réseau d’eau potable théorique. ___________________________ 16 Figure 6 : Biofilm microbien se développant sur une canalisation métallique d’un réseau
d’eau industriel. ___________________________________________________________________ 17 Figure 7 : Distribution, interactions et diversité des microorganismes colonisant un réseau
d’eau potable. _____________________________________________________________________ 18 Figure 8 : Phylogénie simplifiée des amibes. ____________________________________________ 22 Figure 9 : Structure d’un trophozoïte d’Acanthamoeba castellanii ATCC 30234 observée par
microscopie électronique à transmission. __________________________________________ 23 Figure 10 : Morphotypes des Amoebozoa en fonction des principaux clades déterminés par
comparaison de séquences du gène codant l’ARNr 18S. ____________________________ 24 Figure 11 : Caractérisation de la morphologie des kystes d’Acanthamoeba castellanii. (A, B)
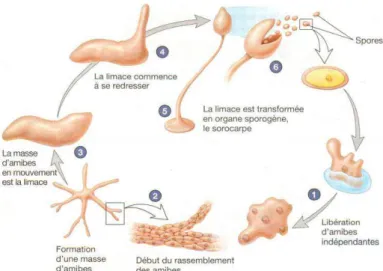
et de Vermamoeba vermiformis (C, D), par microscopie électronique à balayage et à transmission. ______________________________________________________________________ 26 Figure 12 : Formes flagellées de Tetramitus (A), Naegleria (B), et Willaertia (C). ____________ 27 Figure 13 : Cycle de vie de Dictyostelium discoideum. ___________________________________ 28 Figure 14 : Modification membranaires impliquées dans l’internalisation des
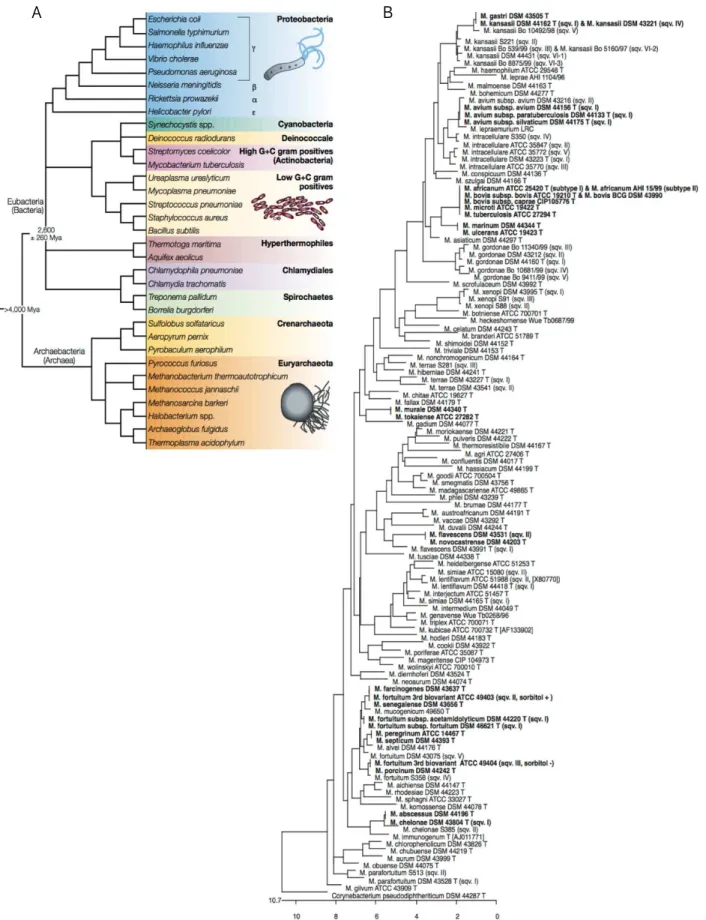
microorganismes par les amibes libres. ____________________________________________ 30 Figure 15 : Les principales étapes de la phagocytose. ____________________________________ 31 Figure 16 : Phylogénie simplifiée des modèles de référence du règne bactérien. _________ 36 Figure 17 : Classification des mycobactéries selon les homologies de séquences des
marqueurs phylogénétiques rpoB (A) ou hsp65 (B). _________________________________ 39 Figure 18 : Comparaison de deux souches de Mycobacterium abscessus, présentant des
morphotypes distincts lisses (S) et rugueux (R). ____________________________________ 40 Figure 19 : Composition de la paroi des mycobactéries. __________________________________ 40 Figure 20 : Taille (A) et composition en bases GC (B) des différents génomes de
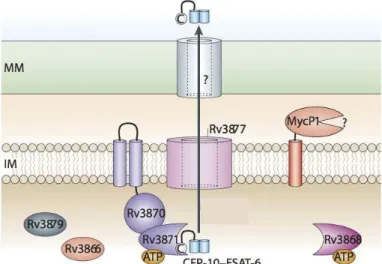
Figure 21 : Famille des protéines PE et PPE. _____________________________________________ 44 Figure 22 : Modèle prédictif du système de sécrétion de type 7 chez Mycobacterium spp. 45 Figure 23 : Foyers de prolifération des endosymbiotes d’amibes libres. __________________ 57 Figure 24 : Stratégie d’évitement de la phagocytose de B. thurigiensis par Acanthamoeba
polyphaga. ________________________________________________________________________ 62 Figure 25 : Cycle de vie de L. pneumophila au sein de phagocytes professionnels tels que
les macrophages et les amibes. ____________________________________________________ 63 Figure 26 : Trophozoïtes d’Acanthamoeba polyphaga à l’issue d’une infection par M. avium
à 48 heures de co-incubation. _____________________________________________________ 65 Figure 27 : Visualisation de la capacité de M. marinum à polymériser l’actine de l’hôte. ___ 67 Figure 28 : Quantification des gènes issus de transferts horizontaux inter-règnes chez des
phagocytes professionnels unicellulaires. __________________________________________ 69 Figure 29 : Répartition géographique des points de prélèvements effectués sur les
réservoirs (points libellés) et le réseau de distribution du réseau d’eau potable de Paris. ___________________________________________________________________________________ 74 Figure 30 : Limite du réseau de distribution publique d’eau potable de Paris. _____________ 75 Figure 31 : Principe de la technique de pyroséquençage. ________________________________ 90 Figure 32 : Greffage des adaptateurs sur des produits de PCR. __________________________ 91 Figure 33 : Occurrence des amibes libres sur l’ensemble des sites analysés lors de la
campagne de prélèvement d’eau potable. _________________________________________ 106 Figure 34 : Ratio du nombre de bactéries (en équivalent génomes) quantifiés par amibes. __________________________________________________________________________________ 108 Figure 35 : Evaluation de l’occurrence des amibes libres pour chaque site de prélèvement
de fin de réseau. __________________________________________________________________ 127 Figure 36 : Identification des espèces de MNT isolées du réseau d’eau potable de Paris. 159 Figure 37 : Localisation de M. chelonae au sein de A. castellanii à 4h (A), 24h (B) et 72h (C)
post infection, par microscopie électronique à transmission. ______________________ 187 Figure 38 : Potentiel infectieux de M. chelonae en comparaison avec celui de M. llatzerense
(A), et croissance en présence d’A. castellanii (B). __________________________________ 188 Figure 39 : Survie de M. chelonae suite à l’activation de la machinerie autophagique par
traitement à la rapamycine (A), et suite au blocage de la maturation du phagosome par traitement au NH4Cl (B). ___________________________________________________________ 189
Liste des tableaux
Tableau 1 : Identification des amibes libres à différentes étapes du circuit de potabilisation de l’eau. ___________________________________________________________________________ 34 Tableau 2 : Pouvoir pathogène des principales espèces de mycobactéries non
tuberculeuses impliquées dans les pathologies humaines. _________________________ 47 Tableau 3 : Diversité des mycobactéries non tuberculeuses à différents points des réseaux
d’eau potable, selon la littérature. __________________________________________________ 50 Tableau 4 : Diversité des endosymbiotes bactériens isolés naturellement au sein des
amibes libres dans l’environnement. _______________________________________________ 55 Tableau 5 : Genres bactériens comprenant au moins une espèce considérée comme ARB. ___________________________________________________________________________________ 60 Tableau 6 : Mycobactéries non tuberculeuses décrites comme résistantes aux amibes,
ainsi que les hôtes amibiens associés. _____________________________________________ 64 Tableau 7 : Souches de microorganismes utilisés au cours des travaux. _________________ 76 Tableau 8 : Amorces utilisées au cours de cette étude pour les expériences de PCR en point
final. ______________________________________________________________________________ 85 Tableau 9 : Amorces et sondes nucléotidiques utilisées au cours de cette étude pour les
expériences de qPCR. _____________________________________________________________ 87 Tableau 10 : Caractéristiques générales du génome de la souche environnementale M.
chelonae EDP_6, ainsi que des autres assemblages génomiques de cette espèce disponible publiquement. _________________________________________________________ 189 Tableau 11 : Facteurs de virulence impliqués dans le blocage de la maturation du
L’équipe Microbiologie de l’Eau (MDE) de l’unité Ecologie et Biologie des Interactions (UMR CNRS 7267) cherche à comprendre et maitriser le développement de microorganismes pathogènes dans l’eau, comme par exemple la bactérie pathogène Legionella pneumophila, dont l’écologie est en lien étroit avec les amibes libres. Les amibes libres sont des microorganismes ubiquitaires des environnements hydriques, et sont naturellement présentes dans les réseaux d’eau potable. Les amibes libres ne sont que rarement incriminées directement dans les pathologies humaines. En revanche, elles sont considérées comme un véritable « cheval de Troie », favorisant la protection, le transport et l’implantation de bactéries pathogènes dans l’eau. Ce caractère protecteur permet notamment à des bactéries potentiellement pathogènes comme L. pneumophila et Mycobacterium spp. de survivre à des conditions environnementales qui leurs sont d’ordinaire défavorables.
Dans le contexte des réseaux d’eau potable, les amibes libres pourraient faciliter l’implantation de ces bactéries potentiellement pathogènes. Le risque de santé publique associé à la présence d’amibes libres dans le réseau est ainsi légitimement questionné. L’état des connaissances actuelles sur la diversité et l’écologie des amibes libres dans les réseaux d’eau potable, de même que sur leur véritable implication dans la survie et la persistance des diverses bactéries résistantes, était encore fragmentaire au début de ce travail de thèse. Parmi les bactéries résistantes aux amibes, les mycobactéries représentent un genre fréquemment retrouvé au sein des réseaux d’eau potable. Les mycobactéries non tuberculeuses sont considérées comme un problème de santé publique émergent lié à l’eau, impliquant des pathologies contraignantes et coûteuses à soigner dont la prévalence augmente sans cesse depuis les dernières décennies. Leur forte présence au sein des réseaux témoigne d’une capacité particulièrement efficace de colonisation et de persistance de cet environnement. Ajouté à ces constats, des études menées sur plusieurs espèces de mycobactéries ont pu mettre en évidence in vitro la résistance de ces dernières aux amibes libres classiquement isolées dans des réseaux d’eau potable.
Dans le cadre de cette problématique, Eau de Paris, le service public gérant la préparation et la distribution de l’eau potable pour la ville de Paris, s’intéresse à la compréhension du rôle probable des amibes comme vecteur facilitant l’implantation et la dispersion de microorganismes potentiellement pathogènes.
Ce travail de thèse, effectué en collaboration avec l’UMR CNRS 7267 Ecologie et Biologie des Intéractions et Eau de Paris, a pour but d’étudier la diversité et l’implication des amibes dans la survie et la persistance des mycobactéries non tuberculeuses au sein d’un réseau d’eau potable.
I. Le réseau d’eau potable de Paris
La ville de Paris comptabilise plus de 2'220'000 habitants intra-muros, auxquels s’ajoutent environ un million de personnes venant à Paris chaque jour pour y travailler. Pour répondre aux besoins en eau de l’ensemble de la population parisienne, entre 450'000 et 600'000 m3 d’eau potable sont distribués chaque jour via le réseau d’eau
potable de Paris. Ce réseau fût initialement conçu entre 1854 et 1860 par Eugène Belgrand (1810-1878), sous l’impulsion de préfet de l’époque, Georges Eugène Haussmann. La construction du réseau d’eau potable fût concomitante à celle du réseau d’évacuation des eaux usées. En cela, le réseau d’eau potable de Paris est unique car 90% des canalisations sont en galerie souterraine et visitables; les réseaux d’eau conçus classiquement étant enterrés. Cette particularité favorise l’accès à tous les points du réseau, permettant une réponse rapide en cas d’avarie ou de fuite. De ce fait, le réseau d’eau potable de Paris bénéficie d’un rendement de 91,4% (taux représentant le volume d’eau arrivé au consommateur par rapport au volume d’eau injecté dans le réseau) parmi les meilleurs au monde.
Le réseau d’eau potable de Paris représente actuellement 1900 km de canalisations, 470 km d’aqueducs, disposant de 6 usines de traitements, ainsi que de 5 réservoirs de stockages situés aux portes de Paris.
I.1. Les sources d’approvisionnement
Le réseau d’eau potable de Paris est alimenté par de multiples sources (Figure 1). La construction des aqueducs de Voulzie, du Loing, de la Vanne et de l’Avre a permis d’avoir accès à 102 points de captages, éloignées parfois de plus de 100 km de Paris.
Figure 1 : Les différentes sources d’eau alimentant le réseau d’eau potable de Paris. Les sources de Verneuil-sur-Avre, Dreux, Nemours, Sens, Longueville et Provins approvisionnent le réseau en eau d’origine souterraine. Ces eaux sont acheminées par le réseau d’aqueducs au sein des usines de Saint-Cloud, l’Hay-les-Roses, Sorques et Longueville pour la potabilisation. Les eaux de surface, issues des rivières Seine et Marne, sont puisées à proximité de Paris, puis traitées au sein des usines d’Orly et Joinville. L’usine d’Ivry, encore indiquée sur ce plan, a été mise en arrêt en 2009. (Source : document interne Eau de Paris).
L’eau approvisionnant ces aqueducs est exclusivement d’origine souterraine, et représente environ 50% de l’alimentation en eau du réseau. L’eau de surface, puisée dans les rivières de la Seine et de la Marne, représente l’autre moitié de l’eau alimentant le réseau. Les eaux de surface sont acheminées au sein des usines d’Orly et Joinville, tandis que les eaux d’origine souterraine sont acheminées au sein des usines de l’Hay-les-roses, Saint-Cloud, Sorques et Longueville.
I.2. Les filières de potabilisation
Les traitements visant à potabiliser l’eau ont la nécessité d’être adaptés à l’origine de l’eau. En effet, des eaux d’origine souterraine vont différer, tant sur les paramètres physico-chimiques que sur les paramètres microbiologiques, d’eaux de surface puisées dans des rivières. De manière générale, les eaux de surface sont plus chargées en microorganismes que les eaux souterraines. En revanche les eaux issues de sources souterraines sont plus concentrées en sels minéraux et en molécules polluant les sols qu‘elles traversent, tels que les nitrates ou de potentiels produits phytosanitaires. Pour cela, les différentes usines de potabilisation emploient des filières adaptées au type d’eau les approvisionnant.
I.2.1. Traitement des eaux de surface
Les eaux de surface, provenant des rivières Seine et Marne, sont exclusivement traitées dans les usines d’Orly et Joinville respectivement (Figure 2). Malgré quelques différences dans les processus de potabilisation entre ces deux usines, il est possible de distinguer plusieurs étapes communes pour le traitement des eaux de surface.
Suite au pompage de l’eau, un dégrillage est effectué (1) : cette étape correspond à une
filtration grossière permettant d’exclure les macroéléments potentiellement présents dans les eaux de rivière. Suite à cela, les usines d’Orly et de Joinville appliquent un traitement de pré-ozonation à faible concentration (2), visant principalement à oxyder
les molécules organiques. L’eau va subir ensuite une étape de coagulation-floculation sur filtres dégrossisseurs (3). Cette étape consiste à faire coaguler les particules
colloïdales (particules fines d’un diamètre compris entre 1 nm et 1 µm), grâce à l’ajout de sels métalliques. Ces matières colloïdales vont ensuite être rassemblées pour former un floc, grâce à l’ajout d’un polymère favorisant les phénomènes d’agglomération. Cette étape permet donc d’agglomérer toutes les particules colloïdales, de façon à obtenir des amas suffisamment lourds pour qu’ils décantent naturellement. Au sein de l’usine d’Orly l’étape suivante consiste à décanter les flocs, permettant de récolter l’eau en surface. Les
récupérées ensuite. Au sein de l’usine de Joinville, les flocs sont récupérés par flotation, grâce à l’ajout d’air formant des bulles, faisant ainsi remonter les flocs à la surface des bassins. Cette différence de méthodologie est directement liée à la source d’eau, car certains paramètres, notamment la présence de microorganismes comme des algues et cyanobactéries, interfèrent fortement avec le processus de décantation.
L’eau va ensuite être filtrée dans des bassins comportant un lit de sable (4). Cette étape
va permettre de clarifier l’eau. En fonction du temps, cette étape de clarification est dite « sur filtre à sable » (l’eau met environ 8h pour traverser le lit de sable à l’usine d’Orly), ou « biologique » (l’eau met environ 24h pour traverser le lit de sable à l’usine de Joinville)
(5). Cette filtration lente favorise l’activité biologique contribuant à la clarification de
l’eau. Un traitement à l’ozone est appliqué en sortie des bassins de filtration lente (6),
ayant pour but d’inactiver les microorganismes. Il est à noter que cette étape de traitement doit être extrêmement bien maitrisée, car une ozonation trop importante favorise la formation de bromates, dont la concentration dans l’eau potable est réglementée, ne devant pas dépasser 10 µg/L. Une dernière étape de filtration sur charbon actif est ensuite appliquée (7). Le charbon actif est un composé carboné
hydrophobe, ayant la capacité de retenir de nombreuses molécules organiques, parmi lesquelles les pesticides ou les résidus médicamenteux. La filtration sur charbon actif vise à purifier chimiquement l’eau destinée à la consommation. Finalement, l’eau va subir une désinfection aux rayonnements ultraviolets (8), à raison de 40 mJ/cm2 (Hijnen
et al., 2006), puis par chloration (9), permettant de compléter l’étape d’ozonation en
inactivant les microorganismes. Une chloration à 0,3 ppm (partie par million) de chlore actif est utilisée pour un temps de stockage court, avant envoi dans les réservoirs d’eau potable localisés à Belleville, Ménilmontant et les Lilas (Figure 1).
Figure 2 : Principe de fonctionnement des filières de potabilisation des eaux de surface mises en place dans les usines d’Orly traitant l’eau de Seine, et Joinville traitant l’eau de Marne. Les principales étapes de la potabilisation des eaux de surfaces consistent en des étapes de filtrations (étapes 1, 3, 4, 5) et des étapes de traitements (étapes 2, 6, 7, 8, 9). (Source : www.eaudeparis.fr/les-metiers/produire/#traiter_eaux_rivieres).
I.2.1. Traitement des eaux souterraines
Les eaux souterraines subissent un traitement très différent des eaux de surface. En effet, ces eaux sont issues des précipitations infiltrant les sols, formant une nappe lorsque ces infiltrations rencontrent une couche tellurique imperméable. Ce processus d’infiltration représente un premier traitement naturel de clarification de l’eau, qui présente alors moins de particules colloïdes et de microorganismes. Cependant, ce même processus peut impliquer la présence de molécules chimiques polluant les sols, telles que des nitrates, pesticides ou leurs produits de dégradation (Gustafson, 1989; Spalding and Exner, 1993; Stuart et al., 2011). Pour cela, la filière de potabilisation est adaptée en conséquence, au sein des usines de l’Hay-Les-Roses, Saint-Cloud, Sorques et Longueville (Figure 3).
Figure 3 : Principe de fonctionnement des filières de potabilisation des eaux souterraines mises en place au sein des usines de l’Hay-Les-Roses, Saint-Cloud, Sorques et Longueville, alimentées par les différents aqueducs du réseau. La filière de potabilisation diffère de celle mise en place pour les eaux de surface. L’eau souterraine ne nécessite pas de dégrillage ni de filtration sur sable, car son infiltration au travers de sols représente une étape de filtration naturelle. L’eau acheminée en usine est donc uniquement traitée avec du charbon actif et une étape finale de chloration. Une seule étape d’ultrafiltration est appliquée, afin de clarifier l’eau et filtrer bactéries et autres microorganismes. (Source : document interne Eau de Paris).
15
Les eaux souterraines acheminées par aqueducs sont dans un premier temps stockées en bassins, avant de subir un traitement par filtration sur charbons actifs. Cette étape va principalement permettre d’éliminer les contaminations en molécules organiques, comprenant les pesticides. Suite à cela, l’eau va subir une mise en pression, afin de pouvoir passer au travers des modules d’ultrafiltration. Ces modules sont composés d’un réseau de fibres d’une porosité de 10 nm en moyenne, et retiennent donc théoriquement les eucaryotes, bactéries ainsi qu’une grande partie des virus. L’eau ultrafiltrée est finalement chlorée, puis stockée en réservoirs d’eau potable, situés à Montsouris, Saint-Cloud et l’Hay-Les-Roses (Figure 1).
I.3. Distribution de l’eau potable
Les réservoirs de stockage, répartis aux portes de Paris, peuvent contenir 1'087'000 m3
d’eau potable, correspondant à environ deux jours de consommation pour la ville de Paris. Ces réservoirs alimentent ainsi continuellement le réseau de distribution de Paris. Ce réseau est composé de 1900 km de canalisations, dont 70% sont sur la rive droite de la Seine, et 30% sur la rive gauche. Les différents quartiers sont alimentés par les différents réservoirs, selon les unités de distribution (Figure 4).
Figure 4 : Carte des unités de distribution (UDI) de l’eau potable à Paris.
n
UDI Sud-Ouest alimenté par les eaux de la Vanne traitées à l’Hay-Les-Roses et les eaux de la Seine traitées à Orly.n
UDI Nord-Ouest alimenté par les eaux de l’Avre traitées parl’usine de Saint-Cloud.
n
UDI Est alimenté par les eaux de la Seine et de la Marne, traitées par les usines d’Orly et de Joinville.n
UDI centre alimenté par les eaux du Loing et de la Voulzie, traitées par les usines de Sorques et Longueville. (Source :www.eaudeparis.fr/la-qualite-de-leau-a-paris/).
La qualité de l'eau à Paris
Pour connaître la qualité de votre eau, sélectionnez votre secteur d'habitation sur la carte ci-dessous. Cela vous permet de télécharger le dernier rapport correspondant.
CARTE DES UNITÉS DE DISTRIBUTION DE L'EAU À PARIS
L'EAU DE PARIS EST SÛRE
L’eau de Paris respecte les 56 paramètres de potabilité définis par le Code de la santé publique dans le cadre des normes européennes qui forment le standard de qualité sanitaire le plus élevé au monde. Elle ne présente aucun risque pour la santé d’une personne qui en consommerait toute sa vie.
Chaque année, plus d’un million de mesures de qualité sont réalisées pour assurer la surveillance sanitaire et garantir une qualité irréprochable de l’eau fournie au robinet des Parisiens. L’eau de Paris est soumise à plusieurs types de contrôles :
329
I.4. Ecologie des réseaux d’eau potable
Les réseaux d’eau potable, bien qu’étant des structures artificielles, représentent un environnement à part entière. Malgré l’utilisation de traitements biocides, cet écosystème oligotrophe est colonisé par une riche communauté de microorganismes. L’analyse de microorganismes présents dans les réseaux d’eau potable a longtemps été restreinte à la détection d’indicateurs de contamination fécale, telles que les bactéries Escherichia coli et Enterococcus faecalis, permettant de témoigner d’une eau biologiquement propre à la consommation (Fewtrell et al., 2001; Tallon et al., 2005). Le suivi de protozoaires parasites particulièrement résistants aux traitements de potabilisation, tels que Cryptosporidium parvum et Giardia intestinalis a également été implémenté (LeChevallier et al., 1991; Teunis et al., 1997). Le suivi des charges bactériennes par dénombrement sur milieu gélosé R2A a longtemps représenté le seul indicateur de la colonisation microbienne des réseaux d’eau potable (WHO | Heterotrophic plate counts and drinking-water safety; Reasoner and Geldreich, 1985). Au cours des dernières décennies, les approches globales visant à définir avec précision les communautés microbiennes colonisant les réseaux d’eau potable, et non plus uniquement les indicateurs de contamination, ce sont multipliées (pour revue ; Berry et al., 2006; Douterelo et al., 2014). Ces analyses ont permis d’observer la complexité de ces communautés, à tous les points du réseau de distribution d’eau potable (Figure 5 ; Proctor and Hammes, 2015).
Figure 5 : Aperçu global d’un réseau d’eau potable théorique, depuis le puisage de la source d’eau, le traitement, la distribution, jusqu’à l’acheminement au sein des habitations. Les textes encadrés soulignent les facteurs majeurs influant sur la diversité et la composition des communautés microbiennes. Figure tirée de (Proctor and Hammes, 2015).
source water treatment distribution household
reservoirs biofiltration (= growth) oxidation (= disinfection) Ground water Surface water Sea water Reclaimed water no treatment membrane filtration UV disinfection disinfection residuals • indigenous communities • anthropogenic contamination • seasonal variation • sudden events
source-to-network seeding (= no treatment or treatment breakthrough)
source-to-biofilter seeding biofilter-to-network seeding network-to-household seeding
final treatment step: elimination of viable microbes
• filter-specific microbiome
• functionally relevant
• pollutant biodegradation
• bio-augmentation potential
• planktonic, biofilm, & particle-associated cells
• widely varied pipe materials
• warm temperatures
• hot & cold water distribution
• disinfection residual loss
• long stagnation times
• potential for growth and community structural changes
chlorination
•planktonic, biofilm and particle-associated cells
• pipes materials: cast iron, cement,
PVC, etc...
• seasonal temperature fluctuations •varied residence times
•contamination through pipe breakage
microbiome continuum
I.4.1. Les biofilms dans les réseaux d’eau potable
Le biofilm désigne un assemblage de microorganismes adhérents à une surface immergée, enchâssé dans une matrice (Costerton et al., 1995) (Figure 6) . Dans le cadre d’un réseau d’eau potable, les biofilms vont se former à la surface des canalisations. La matrice extracellulaire qui entoure les microorganismes représente environ 90% de la masse sèche des biofilms, et est constituée de molécules organiques (exopolymères polysaccharidiques, protéines, lipides, ADN…) et inorganiques (oxydes, molécules dérivées de la corrosion du matériau supportant le biofilm…) (Flemming and Wingender, 2010). Les microorganismes colonisant le biofilm sont qualifiés de sessiles, par opposition aux microorganismes planctoniques, circulant librement. Ces biofilms bactériens représentent la forme de vie majoritaire dans ces environnements (Remis et al., 2010).
Figure 6 : Biofilm microbien se développant sur une canalisation métallique d’un réseau d’eau industriel, visualisé par microscopie électronique à balayage. D’après (Donlan and Costerton, 2002). Barre d’échelle : 1 µm.
Les communautés microbiennes, notamment sous forme de biofilms, peuvent être impliquées dans les phénomènes néfastes de biofouling (accumulation non désirée de microorganismes sur les surfaces immergées) et de biocorrosion (détérioration accélérée des canalisations dûe à la présence de microorganismes) à divers endroits du réseau d’eau (Flemming, 2002; Beech and Sunner, 2004). Outre l’implication des microorganismes dans la compromission de l’intégrité du réseau, les communautés microbiennes sous forme de biofilms peuvent représenter une niche de persistance pour des bactéries, virus et eucaryotes potentiellement pathogènes pour l’Homme, entrainant une véritable problématique de santé publique (Payment and Robertson, 2004; Wingender and Flemming, 2011).
Alors que les problématiques posées par la colonisation microbienne des réseaux d’eau ont été largement abordées dans la littérature, des propositions alternatives ont récemment émergé quant au statut et au bénéfice associé à la présence de
proposées, consistant à une introduction contrôlée de microorganismes bénéfiques, inhibant ainsi la croissance et le développement d’autres microorganismes pathogènes (Wang, Edwards, et al., 2013). Ce genre de traitements reste cependant exploratoire, et nécessite encore des travaux de recherche conséquents afin de démontrer la faisabilité et l’efficacité de ce type d’approche dans le cadre d’un réseau d’eau potable.
I.4.2. Vers la caractérisation du microbiome des réseaux d’eau
potable
A l’image du microbiome intestinal, les canalisations d’un réseau d’eau potable sont colonisées par un microbiome riche, soumis aux variations des traitements et substrats propres aux réseaux (Figure 7). Pour souligner la richesse de cet environnement, il a pu être identifié jusqu’à 48 phyla bactériens différents dans la composition du microbiome des réseaux d’eau potable, dominé par les Protéobactéries, et comportant de nombreuses bactéries non identifiées, affiliées à des phyla tels que TM6, TM7, OP3, OP11, OD1, GN2, ZB2, SM2F11 et WS3 (Proctor and Hammes, 2015). Cette diversité contient naturellement de nombreux colonisateurs bactériens classiques des réseaux, mais également des pathogènes potentiels comme énoncé précédemment. Au-delà de la diversité bactérienne, les réseaux d’eau potable peuvent abriter divers microorganismes eucaryotes tels que des amibes libres et autres protozoaires, des champignons et des nématodes, ainsi que des virus (Buse et al., 2013; Ashbolt, 2015a, 2015b). Très peu d’études sont en revanche disponibles concernant la diversité virale et eucaryotique des réseaux d’eau potable, qui, à l’image des amibes libres, sont considérées comme un « biome ignoré » (Neglected Biome) (Figure 7).
Figure 7 : Distribution, interactions et diversité des microorganismes colonisant un réseau d’eau potable. D’après (Proctor and Hammes, 2015).
biofouling (excessive EPS formation) planktonic cells (103 – 105 cells/mL) biofilm cells (104 – 108 cells/cm2) detachment biodegradation (synthetic polymer pipes) bio-corrosion
(stainless steel; concrete) sediments / loose deposits
(altered environment) community shifts, growth
transport attachment
substrate leaching (e.g. flexible tubing)
biofilm inhibition (e.g., copper pipes)
re-suspension
disinfectant residuals
hydraulic influences
unwanted consequences of biofilm deposition
pipe material influences
neglected biome predation, protection, DNA exchange free DNA viruses invertebrates protozoa
II. Les amibes libres dans l’eau
Le terme amibe (du grec amoibê, signifiant «changeant») désigne un ensemble de microorganismes eucaryotes unicellulaires hétérotrophes, se déplaçant par projection cytoplasmique. La première description des amibes dans l’environnement est datée de 1755, décrivant un organisme adoptant de nombreuses formes, et ainsi dénommé «Der Kleine Proteus» (Rosenhof and Kleemann, 1755). Le terme « Amibe » a été employé pour la première fois au cours du 19e siècle, représentant la première classification
taxonomique de ces microorganismes. Le genre Amiba est ainsi décrit comme «un corps membraneux, tellement diffluent et contractile tour à tour, que l’animal n’a de forme que celle qu’il veut se donner» (Vincent, 1826). Les amibes actuelles descendent d’un forme de vie datant d’environ 2 milliards d’années, représentant très probablement une des premières émergences de la vie eucaryote (Stanley, 1973). Ces microorganismes, comme de nombreux autres unicellulaires eucaryotes, se multiplient principalement de façon asexuée, par fission binaire (Byers, 1979). Certaines amibes font exception, leur cycle de reproduction incluant une phase de méiose suivie d’une fusion nucléaire (Lahr et al., 2011). Du fait de la caractérisation très ancienne de ces microorganismes, basée sur des critères exclusivement morphologiques, le terme amibe rassemble en réalité un groupe polyphylétique. Ainsi, plus de 5000 espèces d’amibes sont actuellement décrites, et on estime la présence d’environ 25000 espèces amibiennes différentes, réparties au sein de plusieurs super-règnes, arborant une grande diversité de morphologie et de mode de vie (Adl et al., 2007).
Les amibes parasitaires, par définition, nécessitent un hôte pour leur développement et leur multiplication. Elles sont retrouvées au sein du super-groupe des Amoebozoa, et sont connues pour coloniser principalement le système digestif de l’Homme et autres mammifères. Les amibes parasitaires sont affiliées aux genres Entamoeba, Endolimax, Pseudolimax, Iodamoeba, et Dientamoeba.
Par contraste, les amibes libres sont des microorganismes pouvant proliférer sans la nécessité de coloniser un hôte. Celles-ci sont largement répandues dans les milieux naturels et anthropiques, notamment les écosystèmes microbiens présents dans les sols et l’eau, ainsi que dans l’air (Rodriguez-Zaragoza et al., 1993; Rodríguez-Zaragoza, 1994; Loret and Greub, 2010; Conza et al., 2013). Dans les environnements hydriques, les amibes libres sont isolées à partir de l’eau circulante ainsi que des biofilms recouvrant les surfaces immergées (Rodríguez-Zaragoza, 1994). Deux catégories sont distinguables au sein des amibes libres : les amibes nues, et les amibes à thèque. Les amibes à thèque sont facilement reconnaissables, utilisant une coque de nature minérale ou organique (le
test ou la thèque) qui joue un rôle protecteur. Les amibes nues, par opposition, sont dépourvues de thèque. Au cours de cette introduction, l’intérêt se portera principalement sur les amibes libres nues, qui sont celles principalement représentées dans les réseaux d’eau potable.