HAL Id: tel-00964872
https://tel.archives-ouvertes.fr/tel-00964872
Submitted on 24 Mar 2014HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Internalisation cellulaire et activité biologique de PNA
bloqueurs stériques de la traduction, conjugués au
peptide (R/W)9
Céline Cordier
To cite this version:
Céline Cordier. Internalisation cellulaire et activité biologique de PNA bloqueurs stériques de la tra-duction, conjugués au peptide (R/W)9. Médecine humaine et pathologie. Université René Descartes - Paris V, 2014. Français. �NNT : 2014PA05P601�. �tel-00964872�
!
!
THESE DE DOCTORAT DE LÕUNIVERSITE PARIS DESCARTES Ð PARIS V
Ecole Doctorale MŽdicament, Toxicologie, Chimie, Environnement
Discipline : Biologie cellulaire et molŽculaire
PrŽsentŽe par
CŽline CORDIER
Pour obtenir le grade de Docteur de lÕUniversitŽ Paris Descartes
Internalisation cellulaire et activitŽ biologique de PNA
bloqueurs stŽriques de la traduction, conjuguŽs au peptide
(R/W)9
Soutenue le 23 janvier 2014 devant le jury composŽ de :
Pr. Claude Malvy UniversitŽ Paris Sud, Paris Rapporteur Dr. Sandrine Sagan UniversitŽ Pierre et Marie Curie, Paris Rapporteur Dr. Virginie Escriou UniversitŽ Paris Descartes, Paris Examinateur Dr. Jean-Jacques ToulmŽ INSERM, Bordeaux Examinateur Pr. Michel Vidal UniversitŽ Paris Descartes, Paris Examinateur Pr. Tula Saison-Behmoaras MusŽum National dÕHistoire Naturelle, Directrice de th•se
Paris
Th•se rŽalisŽe au sein du laboratoire de RŽgulation et Dynamique des GŽnomes du MusŽum National dÕHistoire Naturelle, INSERM U565, CNRS UMR 7196
!
!
! !
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
!
Remerciements
Je remercie vivement le Docteur Sandrine Sagan et le Professeur Claude Malvy d!avoir accept" d!#tre rapporteurs de cette th$se. Merci "galement au Professeur Michel Vidal et aux Docteurs Virginie Escriou et Jean-Jacques Toulm" d!avoir accept" d!#tre membres du jury et d!"valuer ce travail.
Je remercie l!"cole doctorale M"dicament, Toxicologie, Chimie et Environnement %MTCE& et l!universit" Paris Descartes pour avoir financ" cette th$se.
Je remercie le Docteur Carine Giovannangeli et le Professeur Jean-Fran'ois Riou pour m!avoir donn" la chance de travailler dans leur unit". Je tiens ( remercier ma directrice de th$se, le Professeur Tula Saison-Behmoaras, pour sa disponibilit" et pour m!avoir toujours apport" son soutien et sa confiance.
Un grand merci ( toutes les personnes avec qui nous avons eu l!occasion de collaborer sur ce sujet : les Docteurs Fabienne Burlina et G"rard Chassaing pour les CPP, les Docteurs St"phanie Bonneau et Christine Vevert-Bizet pour les aspects PCI, et le Docteur Alain Joliot et Edmond Dupont du Coll$ge de France pour les exp"riences de microcopie confocale.
Je remercie les membres de l!"quipe avec qui j!ai travaill", les Docteurs Carine Giovannangeli et Fanny Marlin pour notre travail commun sur l!internalisation cellulaire des PNA et pour nos nombreuses discussions ( ce sujet, le Docteur Fatima Boutimah et le Docteur Sabine Lecosnier pour son travail sur la partie IGF1R et l!exp"rience qu!elle m!a transmise. Merci "galement au Docteur Jean-Christophe Fran'ois pour ses comp"tences et son investissement dans le projet sur IGF1R.
Je tiens "galement ( remercier les diff"rents "tudiants qui m!ont assist"e sur ce projet : Arlette Le Grand de Mercey, Myriam Rahhab, Mathilde Bourdeloux, Florian Dupuy et Elisabeth Met. J!ai beaucoup appr"ci" travailler avec vous tous et je vous souhaite une bonne continuation dans vos "tudes ou emplois.
Mes remerciements se tournent ensuite vers plusieurs personnes du laboratoire qui m!ont particuli$rement aid"e au cours de ma th$se et avec qui j!ai appr"ci" pouvoir interagir : Fran'ois Loll pour son aide pr"cieuse au microscope, Lo)c Perrouault pour #tre toujours pr#t ( apporter son aide pour tous genres de gels et pour ses conseils pratiques, Patrizia Alberti pour les exp"riences de spectroscopie d!absorption et pour nos discussions int"ressantes qui les accompagnaient, Charlotte Boix pour tous ses conseils sur la culture cellulaire ou autre, Anne De Cian pour son aide pour tout type de clonage ou PCR et pour ses conseils pour la r"daction de ma th$se, Lo)c Ponger pour ses r"ponses ( mes questions de bioinformatique, Sacha Boutorine pour m!avoir initi"e ( la
!
!
chromatographie HPLC et Julien Cochennec pour sa disponibilit" pour nous les *d!jeun!s! et son aide pour r"soudre tous mes probl$mes informatiques.
Je remercie tous les membres de l!unit" pour leur sympathie et pour la bonne ambiance qui r$gne au laboratoire. Une pens"e pour tous les anciens doctorants que j!ai eu la chance de conna+tre : Julien, Kahina, Elodie, Tiphanie, Fatima, Alex, Pierre, Fanny, Sabine, Christine, C"line M, Asta, Najah, LayalÉ Merci ( Fara, Corinne et Patricia du secr"tariat pour leur patience et leur gentillesse. Des remerciements sp"ciaux pour Charlotte et Fran'ois avec qui j!ai partag" mon bureau
%le bureau des *k!, on se comprendÉ& avec grand plaisir. Merci pour votre humour %sp"ciale d"dicace
( Frankois& et votre bonne humeur quotidienne. Je remercie bien s,r les doctorants encore pr"sents
au laboratoire : Jean, Armelle, Vivien, Karine, Marion, Hind, Lorena. Merci pour votre enthousiasme et votre bonne humeur et bon courage pour la fin de votre th$se. Une pens"e particuli$re pour Jean, qui soutiendra sa th$se quelques semaines apr$s moi. Merci "galement aux post-doctorants, notamment ( X"nia, merci pour ta gentillesse, je te souhaite de trouver ta voie et ne doute pas que ta culture scientifique te permettra de t!"panouir dans un nouveau projet. J!adresse ensuite mes remerciements aux personnes qui m!ont le plus soutenue au quotidien au labo, dans les bons moments comme dans les moments difficiles. Merci Fanny pour nos midis au badminton ou au stretching, nos heures partag"es dans le L2 et pour m!avoir transmis une partie de ton talent d!imitatrice. Merci Hind pour ta sinc"rit", pour notre humour commun %la cr$me br,l"e& et tes bricks d!anniversaire. Merci Charlotte pour tes petites attentions qui m!ont souvent touch"e. J!ai "t" heureuse de vous c-toyer, au labo ou ailleurs et je ne compte pas nos fous rires et les bons souvenirs que je garderai, et ceux ( venir ;&
Enfin, je remercie tr$s affectueusement mes amis et ma famille, chez qui je puise mon souffle et mes forces. Merci de m!avoir support"e dans des moments parfois difficiles. Merci ( Aur"lie, mon ex-voisine de quartier, pour nos s"ances sportives et nos discussions sur nos avenirs professionnels. Merci ( Sana et Bibi pour votre soutien, bon courage pour votre derni$re ann"e de th$se. Merci ( Laurent, mon bisontin pr"f"r", mais j!esp$re que tu ne comptes pas t!exiler encore apr$s ta th$se ;& Merci ( Mathilde et Audrey pour votre amiti", votre confiance et votre patience. L!UPMC restera toujours un lieu particulier pour nous, emprunt de tant de souvenirs. Merci ( ma meilleure amie Lorena d!#tre pr"sente ( chaque instant et de me redonner un peu de confiance en moi quand cela me fait d"faut. Nos petits d"jeuners matinaux et tous ces moments pass"s ensemble au labo vont beaucoup me manquer. Je ne peux trouver de mots suffisants pour te remercier ( la hauteur de ton soutien. Merci ( ma s.ur et ( mon fr$re. Merci ( mes grands parents. Merci ( mes parents d!avoir toujours cru en moi et de m!avoir donn" les moyens de r"ussir, je leur d"die cette th$se.
!
!
AbrŽviations
ADN Acide DŽsoxyribonuclŽique
AGO Argonaute
AHX Acide 6-aminohexano•que
ARNi ARN interfŽrence
ARN Acide RibonuclŽique
ATP AdŽnosine Triphosphate
CFTR Cystic Fibrosis Transmembrane Conductance Regulator
CHO Chinese Hamster Ovary
CMV CytomŽgalovirus
CPP Cell-penetrating Peptides
DMD Duchenne Muscular Dystrophy
DMEM Dulbecco's Modified Eagle Medium
DOG 2-DŽoxy-D-Glucose
DOPE 1,2-Dioleoyl-sn-Glycero-3-PhosphoŽthanolamine
DOSPA
2,3-Dioleyloxy-N-[2(Sperminecarboxamido)ethyl]-N,N-dimŽthyl-1-Propanaminium Trifluoroacetate
DOTMA N-[1-(2, 3-dioleyloxy)propyl]-N,N,N-trimethylammonium chloride
dsARN Double Stranded ARN
EMSA Electrophoretic Mobility Shift Assay
ESE Exonic Splicing Enhancers
ESS Exonic Splicing Silencers
GTP Guanosine Triphosphate
GAG Glycosaminoglycanes
HCV Hepatitis C Virus
HDL High Density Lipoprotein
HeLa Henrietta Lacks
HRP Horseradish Peroxydase
HS HŽparane Sulfate
HSPG HŽparane Sulfate ProtŽoglycanes
IGF-1 Insulin-like Growth Factor-I
IRES Internal Ribosome Entry Site
IR Insulin Receptor
ISE Intronic Splicing Enhancers
!
!
LDH Lactates DŽshydrogŽnases
LDL Low-Density Lipoprotein
LNA Locked Nucleic Acid
MALDI-TOF MS Matrix-assisted Laser Desorption/Ionization Time-Of-Flight Mass
Spectrometer
MAPK Mitogen-activated Protein Kinase
miARN Micro ARN
2Õ-MOE 2Õ-O-MŽthoxyethyl
NLS Nuclear Localisation Signal
nt NuclŽotide
2Õ-OMe 2Õ-O-MŽthyl
pb Paire de Bases
PCI Photochemical Internalization
PCR Polymerase Chain Reaction
PEG PolyEthyl•ne Glycol
PEI PolyŽthylenimine
PKA ProtŽine Kinase A
PKC ProtŽine Kinase C
PKR ProtŽine Kinase R
PNA Peptide Nucleic Acid
PO Phosphodiester
PRR Pattern Recognition Receptor
PS Phosphorothioate
PSMA Prostate-Specific Membrane Antigene
RFP Red Fluorescent Protein
RISC RNA-Induced Silencing Complex
RNase RibonuclŽase
ROS Reactive Oxygen Species
RPMI Roswell Park Memorial Institute Medium
SDS-PAGE Sodium Dodecyl Sulfate Polyacrylamide Gel Electrophoresis
shARN Short Hairpin ARN
siARN Small Interfering ARN
SLO Streptolysine O
SVF SŽrum de Veau FÏtal
SV40 Simian Vacuolating virus 40
TAMRA Carboxytetramethylrhodamine
!
!
Tm Melting temperature
TPPS2a TŽtraphŽnylporphine disulfonate
TRBP TAR RNA Binding Protein
TR Texas Red
UR! ! ! ! Untranslated Region
!
!
! ""!
Table des mati•res
Table des mati•res ... 11!
Introduction ... 19!
Chapitre I : La stratŽgie antisens ... 20!
I. 1. LÕARN interfŽrence ... 21!
1. 1. ARN interfŽrents ... 22!
a) miARN ... 22!
b) siARN et shARN ... 24!
1. 2. MŽcanismes de rŽpression post-transcriptionnelle conduite par les miARN ... 24!
1. 3. DŽfis ... 28!
1. 4. DŽveloppement clinique ... 30!
I. 2. Les oligonuclŽotides antisens ... 31!
2. 1. Modifications chimiques et mŽcanismes associŽs ... 31!
a) 1•re gŽnŽration ... 33! b) 2•me gŽnŽration ... 34! c) 3•me gŽnŽration ... 36! 2. 2. Les applications ... 41! a) Inhibition de la traduction ... 41! b) Modulation de lÕŽpissage ... 44!
c) Blocage des miARN ... 51!
I. 3. Conclusion : utilisation et dŽfis de la stratŽgie antisens ... 52!
Chapitre II : Internalisation cellulaire des acides nuclŽiques ... 53!
II. 1. NŽcessitŽ de la vectorisation ... 53!
1.1. Des premiers essais ˆ la recherche de transporteurs pour les acides nuclŽiques ... 53!
1.2. Les barri•res biologiques extracellulaires ... 54!
1.3. Internalisation cellulaire ... 55!
a) MŽcanismes dÕinternalisation cellulaire ... 55!
b) StratŽgies dÕŽchappement endosomal ... 58!
c) Le noyau : une barri•re supplŽmentaire ? ... 61!
II. 2. StratŽgies non virales dÕinternalisation des acides nuclŽiques ... 61!
2.1. MŽthodes physiques ... 62!
2.2. MŽthodes photochimiques ... 64!
! ! "#! a) Nanotransporteurs ... 66! i) Lipoplexes et polyplexes ... 66! ii) Nanoparticules ... 70! b) Aptam•res ... 71!
c) Petites molŽcules organiques et peptides ... 73!
d) Cell Penetrating Peptides (CPP) ... 75!
i) Classification ... 76!
ii) MŽcanismes dÕinternalisation ... 77!
iii) Interactions avec la membrane plasmique ... 80!
iv) StratŽgie covalente / non covalente ... 83!
v) Conclusions ... 87!
Chapitre III : Ciblage du rŽcepteur ˆ lÕInsulin-like Growth Factor 1 ... 89!
III. 1. Le rŽcepteur ˆ lÕIGF1 ... 90!
3. 1. Structure et fonction ... 90!
a) IGFIR et lÕaxe IGF ... 90!
b) Voies de signalisation intracellulaires ... 91!
c) Fonctions biologiques ... 93!
d) IGF1R et IR ... 94!
3. 2. Implication dans le cancer ... 95!
3. 3. StratŽgies de ciblage ... 96!
a) Anticorps monoclonaux ... 96!
b) Inhibiteurs de tyrosines kinases ... 97!
c) Inhibition de lÕexpression de lÕIGF1R par stratŽgie antisens ... 98!
d) ProtŽines tronquŽes dominants nŽgatifs de lÕIGF1R ... 99!
e) Autres approches ... 101!
III. 2. Inhibition de lÕŽlongation de la traduction de lÕIGF1R pour gŽnŽrer une protŽine tronquŽe dominant nŽgatif ... 102!
Chapitre IV : Projet de th•se ... 103!
RŽsultats ... 105!
Chapitre I : Inhibition de la traduction par des oligonuclŽotides bloqueurs stŽriques ... 107!
I. 1. Comparaison de diffŽrentes modifications chimiques de bloqueurs stŽriques in vitro et in cellulo ... 107!
1. 1. Syst•me dÕŽtude : la sŽquence Polypurine Tract (PPT) de lÕintŽgrase du VIH . 108! 1. 2. ActivitŽ biologique dÕoligonuclŽotides dirigŽs contre la sŽquence PPT ... 109!
!
! "$!
b) Inhibition de lÕinitiation de la traduction dans les cellules HeLa ... 111!
1. 3. Etudes des complexes formŽs in vitro avec lÕARN complŽmentaire ... 114!
1. 4. Conclusions ... 119!
I. 2. Inhibition de lÕŽlongation de la traduction in cellulo ... 119!
I. 3. Optimisation du PNA anti-IGF1R ... 124!
3.1. Le PNA 959 et ses variants de taille ... 124!
3. 2. ActivitŽ in vitro ... 125!
a) CaractŽrisation des complexes formŽs entre les PNA et lÕARN ... 125!
b) EfficacitŽ pour arr•ter lÕŽlongation de la traduction ... 130!
3. 3. ActivitŽ in cellulo ... 132!
3. 4. Conclusions ... 135!
I. 4. Discussion ... 135!
Chapitre II! : Internalisation cellulaire des PNA ... 141!
II. 1. Internalisation des PNA dirigŽs contre la sŽquence PPT dans les cellules HeLa 1002 par le CPP (R/W)9 ... 142!
1. 1. ConjuguŽs PNA-CPP ... 142!
1. 2. Etude de lÕinternalisation du PNA AS2 par les peptides (R/W)9 et (K/W)9 ... 143!
a) Internalisation libre ... 143!
b) Internalisation assistŽe par la chloroquine et la PCI ... 144!
Article : Cell Delivery of Antisense Peptide Nucleic Acids (PNAs) by the Small Arginine-Rich Cell-Penetrating Peptide (R/W)9 ... 144!
c) Internalisation Photochimique (PCI) : Žtudes complŽmentaires ... 147!
1. 3. ConjuguŽs CPP-AS1 ... 149!
1. 4. Etudes complŽmentaires ... 155!
a) Profils de marquage dans les cellules HeLa non permŽabilisŽes vs permŽabilisŽes ... 155!
b) Comparaison de lÕefficacitŽ de diffŽrents agents lysosomotropiques ... 157!
c) Observation des conjuguŽs en microscopie confocale ... 159!
II. 2. Internalisation du PNA C-13 dirigŽ contre lÕARNm de lÕIGF1R dans les cellules DU145 par le CPP (R/W)9 ... 162!
II. 3. Discussion ... 164!
Conclusion gŽnŽrale et perspectives ... 172!
MatŽriel et MŽthodes ... 176!
!
! "%!
!
! "&!
Sommaire des Figures
Figure 1: MŽcanismes impliquŽs dans la voie des ARN interfŽrents (miARN, shARN et siARN) dans les
cellules de mammif•res ... 23!
Figure 2 : MŽcanismes suggŽrŽs pour la rŽpression post-transcriptionnelle conduite par les miARN dans les cellules animales ... 27!
Figure 3 : PRR (Pattern Recognition Receptors) impliquŽs dans la reconnaissance des siARN et rŽponse immunitaire innŽe associŽe ... 30!
Figure 4 : MŽcanismes dÕaction des oligonuclŽotides antisens. ... 32!
Figure 5 : Structures chimiques des oligonuclŽotides antisens ... 32!
Figure 6 : Structures chimiques comparŽes des PNA et de l'ADN ... 38!
Figure 7 : Fixation des PNA sur une sŽquence dÕADN double-brin homopurique par un mŽcanisme dÕinvasion de brin ... 40!
Figure 8 : MŽcanismes de correction dÕŽpissage par les oligonuclŽotides antisens bloqueurs stŽriques ... 45!
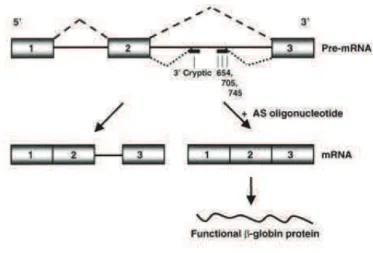
Figure 9 : Correction dÕŽpissage de lÕARN pre-messager de la !-globine ... 46!
Figure 10 : R™les des sŽquences exoniques ESS/ESE (Exonic Splicing Silencer/ Exonic Splicing Enhancer) et des sŽquences introniques ISS/ISE (Intronic Splicing Silencer/Intronic Splicing Enhancer) ... 48!
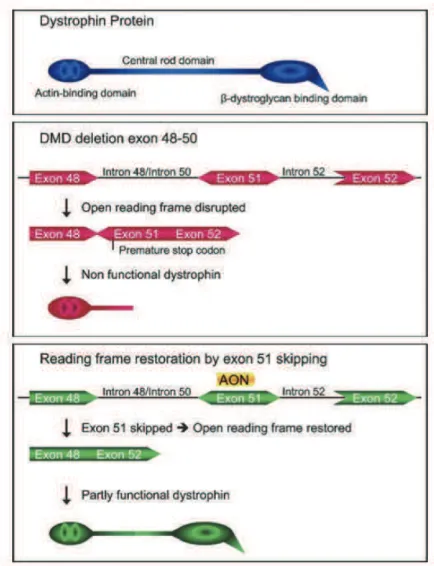
Figure 11 : Approche de saut dÕexon dans le cadre de la Dystrophie Musculaire de Duchenne (DMD) ... 49!
Figure 12 : Voies dÕendocytose ... 57!
Figure 13 : MŽcanismes dÕŽchappement endosomal ... 60!
Figure 14 : MŽcanisme proposŽ pour la libŽration de Tat-Cre dans le cytoplasme, assistŽe par un co-traitement avec un conjuguŽ Tat-HA2 ... 60!
Figure 15 : Principe de la Photochemical Internalization (PCI) ... 64!
Figure 16 : Structures chimiques des photosensibilisateurs tŽtraphŽnylporphine disulfonate (TPPS2a) et phthalocyanine dÕaluminium disulfonate (AlPcS2a) ... 65!
Figure 17 : Composition et structure dÕun liposome de type SNALP (Stabilized Nucleic Acid Lipid Particle) ... 68!
Figure 18 : Composition des nanoparticules CALAA-01. ... 69!
Figure 19 : Structure de la chim•re entre lÕaptam•re anti PSMA et un siARN dirigŽ contre le g•ne de survie cellulaire Plk1 ... 71!
Figure 20 : Structures schŽmatiques des chim•res constituŽes de l'aptam•re anti-gp120 et d'un siARN ... 72!
Figure 21 : Structures chimiques de petites molŽcules organiques utilisŽes pour la vectorisation des acides nuclŽiques ... 74!
Figure 22 : Principaux mŽcanismes de translocation directe des CPP dans les cellules ... 79!
Figure 23 : Structure des protŽoglycanes membranaires (HSPG) ... 81!
Figure 24 : RŽduction de la rŽpulsion de charge par les lysines et les arginines ... 82!
Figure 25 : MŽcanismes proposŽs pour lÕinternalisation de R9 dans les cellules CHO ... 83!
!
! "'!
Figure 27 : Structure de l'IGF1R. ... 91!
Figure 28 : Signalisation cellulaire associŽe ˆ lÕIGF1R. ... 93!
Figure 29 : ProtŽines tronquŽes dominants nŽgatifs de lÕIGF1R dŽcrites dans la littŽrature. ... 101!
Figure 30 : Structure chimique des PMO, LNA et PNA. ... 108!
Figure 31 : ReprŽsentation schŽmatique de lÕARNm de lÕintŽgrase de HIV-1 et position de la sŽquence PPT. ... 108!
Figure 32 : Inhibition de lÕŽlongation de la traduction de lÕintŽgrase in vitro par les PNA 13mer AS1 et AS2 ... 109!
Figure 33 : Inhibition de la traduction de lÕintŽgrase in vitro par les PNA 13-mer (AS1 et AS2), PNA-18 mer (PNAPNA-18) et LNA PNA-18-mer (LNAPNA-18). ... 111!
Figure 34 : Construction intŽgrŽe stablement dans les cellules HeLa 1002 ... 111!
Figure 35 : Fixation des PNA AS1 et AS2 sur la sŽquence PPT mutŽe contenue dans la construction des cellules HeLa 1002. ... 112!
Figure 36 : ActivitŽ lucifŽrase et expression de la GFP dans les cellules HeLa permŽabilisŽes. ... 113!
Figure 37 : Inhibition de la traduction par les PNA 13-mer (AS1, AS2 et scr2), PNA-18 mer (PNA18), LNA 18-mer (LNA18) et PMO 18-mer (PMO18). ... 114!
Figure 38 : Retards de migration sur gel de polyacrylamide des complexes formŽs entre lÕARN 19-mer et les PNA AS1 et AS2 ... 115!
Figure 39 : Retard de migration sur gel dÕacrylamide 15% des complexes formŽs entre lÕARN sauvage 19 mer radiomarquŽ (2 nM) et le PNA18, le LNA 18 ou le PMO 18. ... 116!
Figure 40 : ReprŽsentation schŽmatique des ARN PPT WT (ARN WT) et mutŽ (ARN mut) et des complexes formŽs avec le PNA AS1. ... 117!
Figure 41 : Retards de migration sur gel de polyacrylamide 15%, des complexes formŽs entre lÕARN 19 mer radiomarquŽ (2 nM), dŽnaturŽ par prŽchauffage 10 min ˆ 90¡C, et des quantitŽs croissantes de PNA AS1k (gauche) et AS2k (droite), en tampon Tris 50 mM. ... 118!
Figure 42 : SŽquences PPT sauvage et synthŽtique. ... 121!
Figure 43 : Expression de lÕintŽgrase synthŽtique dans les cellules humaines DU145 transfectŽes avec le plasmide pVax-INsyn. ... 122!
Figure 44 : Traductions in vitro de lÕintŽgrase synthŽtique dans du lysat de rŽticulocyte de lapin, en prŽsence ou en absence des PNA AS1 et AS2. ... 123!
Figure 45 : Retards de migration sur gel de polyacrylamide 15% (EMSA) des PNA C-16 ˆ C-13 (A) et N-16 ˆ N-13 (B). ... 127!
Figure 46 : SŽparation sur gel natif de polyacrylamide 15% (gel retard), des complexes formŽs entre lÕARN 959 17-mer radiomarquŽ et le PNA indiquŽ, ˆ 100 ou 300 nM. ... 127!
Figure 47 : Analyses spectroscopiques du PNA 959 et des PNA 13- et 14-mer. ... 129!
Figure 48 : ReprŽsentation schŽmatique de la fixation des anticorps (Ac) H60 et C20 sur lÕIGF1R. 130! Figure 49 : Inhibition de la traduction in vitro et formation de la protŽine tronquŽe, en prŽsence des diffŽrents PNA variants de taille. ... 132!
Figure 50 : Inhibition de lÕexpression de la sous-unitŽ ! de lÕIGF1R par les PNA N-13 et C-13 dans les cellules humaines DU145. ... 134!
Figure 51 : Inhibition de lÕexpression de la sous-unitŽ ! de lÕIGF1R par les PNA C-11 et C-12 dans les cellules humaines DU145. ... 134!
!
! "(!
Figure 53 : PCI observŽe pour le conjuguŽ (R/W)9-scr2-TAMRA dans les cellules HeLa 1002. ... 148!
Figure 54 : ActivitŽ lucifŽrase obtenue pour les conjuguŽs (R/W)9-PNA-TAMRA apr•s PCI. ... 149!
Figure 55 : Inhibition de lÕactivitŽ lucifŽrase par les PNA et conjuguŽs PNA-CPP dans les cellules HeLa 1002 permŽabilisŽes avec la SLO. ... 150!
Figure 56 : Internalisation des PNA AS1 et AS2 conjuguŽs au CPP (R/W)9 dans les cellules HeLa 1002. ... 151!
Figure 57 : Internalisation des PNA AS1 et AS2 conjuguŽs au CPP (R/W)9 dans 4 lignŽes cellulaires humaines. ... 151!
Figure 58 : DŽpendance ŽnergŽtique de lÕinternalisation du conjuguŽ (R/W)9-AS1-TR dans les cellules HeLa 1002. ... 153!
Figure 59 : Comparaison de lÕinternalisation du conjuguŽ (R/W)9-AS1-TR dans les cellules CHOK1 et dans les cellules CHO745. ... 154!
Figure 60 : Effet du co-traitement des cellules HeLa 1002 avec les diffŽrents conjuguŽs CPP-PNA-TR et la chloroquine (CQ). ... 155!
Figure 61 : Internalisation cellulaire des conjuguŽs (R/W)9-AS2-TR et (R/W)9-AS2-TR dans les cellules HeLa 1002. ... 156!
Figure 62 : PermŽabilisation par la SLO des cellules HeLa 1002 en prŽsence des conjuguŽs (R/W)9-AS2-TR et (K/W)9-(R/W)9-AS2-TR. ... 157!
Figure 63 : Internalisation cellulaire du conjuguŽ (R/W)9-AS1-TR dans les cellules HeLa 1002, en prŽsence de diffŽrents agents lysosomotropiques. ... 158!
Figure 64 : ActivitŽs lucifŽrase apr•s internalisation libre du conjuguŽ (R/W)9-AS2 en prŽsence de CaCl2. ... 159!
Figure 65 : Observation en microscopie confocale, de cellules HeLa 1002 co-traitŽes pendant 4h avec le conjuguŽ (R/W)9-AS1-TR et la chloroquine (CQ). ... 160!
Figure 66 : Effets de la concentration du conjuguŽ (R/W)9-AS1-TR sur lÕinternalisation dans les cellules HeLa 1002. ... 161!
Figure 67 : Expression dÕIGF1R-! dans les cellules DU145 ŽlectroporŽes en prŽsence des PNA indiquŽs ou du conjuguŽ (R/W)9-C13. ... 162!
Figure 68 : Internalisation du conjuguŽ (R/W)9-AS2-TR dans les cellules DU145 en absence (NT) ou en prŽsence de 150 µM de chloroquine (CQ). ... 163!
Figure 69 : Expression dÕIGF1R-! dans les cellules DU145 traitŽes avec le conjuguŽ (R/W)9-C13. 164! Figure 70 : Syst•me rapporteur dÕactivitŽ de correction dÕŽpissage par les oligonuclŽotides antisens dans le noyau. ... 169!
Figure 71 : Structure chimique dÕun GPNA (Tg)10 ... 172!
Figure 72 : Structure chimique des conjuguŽs PNA-acide cholique ... 173!
!
! ")!
Sommaire des Tableaux
Tableau 1 : Description des principaux CPP dŽcrits dans la littŽrature. ... 76!
Tableau 2 : SŽquences des oligonuclŽotides 13-mer et 18-mer dirigŽs contre la sŽquence PPT. ... 110!
Tableau 3 : RŽcapitulatif des rŽsultats reprŽsentŽs sur les Figures 38 et 39. ... 117!
Tableau 4 : SŽquence des PNA raccourcis en C-terminal (PNA N-16 ˆ N-13) ou en N-terminal (PNA C-16 ˆ C-11) par rapport au PNA 959. ... 125!
Tableau 5 : Valeurs des IC50 (nM) correspondants ˆ la fixation des diffŽrents PNA variants de taille du
959 sur lÕARN 959 17-mer. ... 128!
Tableau 6 : Conditions de PCI rŽalisŽes dans nos Žtudes avec les conjuguŽs AS2 et (R/W)9-AS2-TAMRA. ... 149!
Tableau 7 : SŽquences des PNA dirigŽs contre la cible PPT. ... 176!
Tableau 8 : SŽquences des PNA dirigŽs contre lÕIGF1R. ... 176!
Tableau 9 : Composition en bases des PNA contr™les (scramble) utilisŽs dans les Žtudes avec les PNA ciblant lÕIGF1R. ... 177!
Tableau 10 : SŽquences et caractŽristiques peptidiques des CPP (R/W)9 et (K/W)9. ... 177!
Tableau 11 : CaractŽristiques et conditions de culture des lignŽes cellulaires utilisŽes dans ce travail. ... 178!
! ! "*!
Introduction
! ! !Chapitre I
La stratŽgie antisense
Chapitre II
Internalisation cellulaire des acides nuclŽiques
Chapitre III
Ciblage du rŽcepteur ˆ lÕIGF1
Chapitre IV
Projet de th•se
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #>!
Chapitre I : La stratŽgie antisens
La plupart des mŽdicaments actuellement sur le marchŽ sont des petites molŽcules qui ciblent des protŽines, principalement des enzymes ou des rŽcepteurs cellulaires (Landry & Gies 2008). La stratŽgie antisens consiste en lÕutilisation dÕoligonuclŽotides antisens ou dÕARN interfŽrents qui ciblent les ARN (ARN messagers, ARN prŽ-messagers ou miARN) et sÕy fixent par complŽmentaritŽ de bases, de mani•re sŽquence spŽcifique, permettant ainsi de moduler lÕexpression dÕun g•ne. Cette stratŽgie est nŽe de la dŽcouverte, en 1978, par Zamecnik et Stephenson du potentiel dÕun oligonuclŽotide long de 13 bases et complŽmentaire ˆ une sŽquence de lÕARN du virus du sarcome de Rous ˆ inhiber la traduction des protŽines virales en sÕhybridant ˆ lÕARN viral. Ceci a pour consŽquence lÕinhibition de la rŽplication virale dans des fibroblastes embryonnaires de poulet (Zamecnik & Stephenson 1978; Stephenson & Zamecnik 1978).
Un oligonuclŽotide antisens comporte en gŽnŽral de 13 ˆ 25 bases, enti•rement complŽmentaires ˆ lÕARN cible, et sÕhybride ˆ ce dernier par liaisons Watson-Crick. Les mŽcanismes dÕactions inhŽrents ˆ cette interaction impliquent un clivage catalytique ou blocage stŽrique et sont dŽpendants de la structure chimique de lÕoligonuclŽotide. Cette approche prometteuse, permettant en thŽorie de cibler nÕimporte quel g•ne, pourrait donc constituer un outil prŽcieux en recherche pour des Žtudes fonctionnelles. De plus, elle serait en mesure dÕapporter une solution thŽrapeutique ˆ tous types de maladies impliquant un dŽfaut de rŽgulation de lÕexpression gŽnique (cancers, maladies auto-immunes, maladies inflammatoires, infections virales ou bactŽriennesÉ). LÕenthousiasme ainsi soulevŽ fut malheureusement confrontŽ ˆ certains obstacles, notamment ˆ des probl•mes de toxicitŽ et de stabilitŽ des oligonuclŽotides ADN dans les milieux biologiques. NŽanmoins, la conception dÕune vingtaine de modifications chimiques introduites dans la structure de lÕADN ou de lÕARN a permis dÕamŽliorer nettement leurs propriŽtŽs. De plus, la comprŽhension des rŽgulations post-transcriptionnelles de lÕexpression des g•nes fut bouleversŽe par la dŽcouverte de la voie de lÕARN interfŽrence (ARNi) ˆ la fin des annŽes 1990, et de lÕutilisation des small interfering RNA (siRNA) pour moduler lÕexpression gŽnique (Fire et al. 1998; Elbashir et al. 2001).
Dans cette partie seront prŽsentŽes les diffŽrentes chimies dÕoligonuclŽotides antisens, ainsi que les siRNA, et les mŽcanismes qui leur sont associŽs. Il existe trois principales applications des oligonuclŽotides antisens : inhibition de lÕinitiation ou de lÕŽlongation de la
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #"!
traduction (ciblage de lÕARN messager), modulation de lÕŽpissage des ARN prŽ-messager et inhibition de la fonction de certains ARN non codants, notamment les micro-ARN (miARN). Celles-ci seront explicitŽes et illustrŽes par les principaux exemples connus dans la littŽrature.
I. 1. LÕARN interfŽrence
Le concept dÕARN interfŽrence fut introduit en 1998 par Fire et Mello, qui montr•rent que lÕintroduction de faibles quantitŽs de longs ARN exog•nes double-brins (>299 pb) chez le nŽmatode C.elegans provoquait lÕinhibition des g•nes cibles correspondants (Fire et al. 1998). Dans les annŽes qui suivirent, des Žtudes chez la drosophile montr•rent que les ARN double-brins rŽgulent lÕexpression des g•nes en mŽdiant spŽcifiquement un clivage endonuclŽolytique des ARN messagers correspondants (Zamore et al. 2000; Hammond et al. 2000). LÕidentification de lÕenzyme Argonaute 2 (AGO2) en tant que nuclŽase impliquŽe dans ce clivage fut rŽalisŽe peu de temps apr•s (Hammond et al. 2001).
Dans les cellules de mammif•res, il a ŽtŽ montrŽ que les ARN double-brins de plus de 30 pb dŽclenchent une rŽponse immune innŽe antivirale en activant la protŽine kinase R (PKR), qui induit alors une inhibition non spŽcifique de la traduction, ainsi que la destruction des ARNm (Williams 1997). La m•me annŽe, deux Žquipes ont montrŽ (Elbashir et al. 2001; Caplen et al. 2001) pour la premi•re fois que des duplexes synthŽtiques dÕARN double-brins de 21 nuclŽotides, appelŽs small interfering ARN (siARN), induisent une inhibition spŽcifique de la traduction de g•nes cibles endog•nes ou exog•nes, apr•s transfection dans des cellules de mammif•res, Žvitant la rŽponse non spŽcifique induite par la PKR. Dans ces deux Žtudes, lÕinterfŽrence provoquŽe par les siARN Žtait plus importante que lÕinhibition dŽclenchŽe par les ARN simple-brins de m•me sŽquence. Elbashir dŽfinit donc lÕARN interfŽrence comme Žtant un processus de rŽgulation de lÕexpression gŽnique post-transcriptionnelle, sŽquence spŽcifique et initiŽ par des ARN double-brins complŽmentaires de la sŽquence du g•ne cible. Ce processus est en outre conservŽ au sein du vivant, puisquÕil a ŽtŽ retrouvŽ chez les plantes, les champignons et les animaux.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;! ! ##!
1. 1. ARN interfŽrents
a) miARN
La voie de lÕARN interfŽrant est une voie cellulaire endog•ne (Figure 1) qui est utilisŽe pour la rŽgulation de lÕexpression gŽnique initiŽe par de petits ARN non codants endog•nes, appelŽs microARN (miARN), qui ont un r™le important dans diffŽrents processus biologiques, tels que le dŽveloppement, la prolifŽration cellulaire, lÕhŽmatopo••se et lÕapoptose (Rana 2007).
Chez les mammif•res, les prŽcurseurs primaires de ces ARN sont gŽnŽralement transcrits par lÕARN polymŽrase II, ˆ partir de rŽgions intergŽniques ou dÕADN intronique, et sont appelŽs primary miARN (pri-miARN). Ces molŽcules sont maturŽes dans le noyau par la RNase de type III Drosha qui les transforme en prŽcurseurs de micro-ARN (pre-miARN), comportant une structure en boucle hairpin, une extrŽmitŽ 5Õ phosphate et deux nuclŽotides sortants en 3Õ. Ces prŽcurseurs sont ensuite exportŽs activement dans le cytoplasme via Ran-GTP et le rŽcepteur nuclŽaire exportin-5, o• ils sont substrats de lÕendoribonuclŽase Dicer. Dicer, souvent associŽe ˆ la protŽine de liaison ˆ lÕARN double-brin, TRBP (TAR RNA Binding Protein), clive la boucle, aboutissant au miARN mature, constituŽ de 21 nuclŽotides dont deux sont 3Õ sortants. La particularitŽ des miARN est quÕils comportent une complŽmentaritŽ imparfaite pour leur cible ARN. Ces miARN sont ensuite reconnus et pris en charge par un complexe multi-protŽique appelŽ RISC (RNA-Induced Silencing Complex). Un des deux brins de lÕARN, appelŽ Ôbrin guideÕ, est sŽlectionnŽ dans le complexe RISC, tandis que lÕautre brin, appelŽ Ôbrin passagerÕ est clivŽ et ŽliminŽ du complexe RISC. Le brin guide correspond au brin antisens, qui est partiellement complŽmentaire ˆ la sŽquence de lÕARNm cible. Le brin guide, complexŽ au RISC, reconna”t alors lÕARNm cible imparfaitement complŽmentaire, se fixe dans la rŽgion 3Õ UTR (Untranslated Region) de celui-ci et inhibe sa traduction.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #$!
!
Figure 1: MŽcanismes impliquŽs dans la voie des ARN interfŽrents (miARN, shARN et siARN) dans les cellules de mammif•res (dÕapr•s Burnett et Rossi 2012).
A. Les miARN sont transcrits sous forme de primary miARN (pri-miARN) qui sont ensuite clivŽs par Drosha et DGCR8 pour former les prŽcurseurs de miARN de 70 nuclŽotides (pre-miARN). Ces prŽcurseurs sont exportŽs dans le cytoplasme par une protŽine de liaison ˆ lÕARN double-brin : lÕexportine-5, o• ils sont maturŽs par le complexe Dicer/TRBP en miARN dÕenviron 22 nuclŽotides. Ces miARN matures sont alors pris en charge par le complexe RISC et par la protŽine AGO, au sein duquel le brin passager est ŽliminŽ et le brin guide est sŽlectionnŽ, permettant sa fixation sur lÕARNm cible avec une complŽmentaritŽ imparfaite. LÕinhibition de la traduction rŽsulte alors de diffŽrents mŽcanismes possibles, tels quÕun stockage dans les P-bodies ou un clivage de lÕARNm. B. Les shARN sont transcrits par lÕARN polymŽrase III ˆ partir de lÕADN et maturŽs par Dicer, avant dÕ•tre pris en charge par le complexe RISC de la m•me mani•re que les miARN. A la diffŽrence des miARN, le brin guide prŽsente une complŽmentaritŽ parfaire pour lÕARNm cible et le transcrit est clivŽ par lÕenzyme AGO2. C. Les siARN, introduits de mani•re exog•ne dans la cellule, utilisent ce m•me mŽcanisme dÕinhibition de la traduction de leur cible.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #%!
b) siARN et shARN
La voie de lÕARNi peut aussi •tre utilisŽe par les siARN synthŽtiques introduits dans la cellule. Ces petits ARN interfŽrents double-brins (dont la taille est comprise entre 19 et 21 pb), sont pris en charge par le complexe RISC, au sein duquel le brin sens (passager) est ŽliminŽ, tandis que le brin antisens (guide) se lie ˆ sa cible parfaitement complŽmentaire sur lÕARNm. Pour la sŽlection des brins, des donnŽes thermodynamiques indiquent que la protŽine AGO2 dŽtermine le brin guide comme Žtant celui comportant lÕextrŽmitŽ 5Õ la moins stable (principalement dž ˆ un fort contenu en A/T) (Schwarz et al. 2003). Du fait de la complŽmentaritŽ parfaite entre le brin guide et la sŽquence cible sur le messager, lÕendonuclŽase AGO2 au sein du RISC clive lÕARNm entre les nuclŽotides 10 et 11 (comptŽs ˆ partir de lÕextrŽmitŽ 5Õ du brin antisens). Le complexe RISC associŽ au brin antisens est recyclŽ et peut ˆ nouveau dŽclencher plusieurs Žv•nements de clivage catalytique.
Alternativement, les siARN peuvent •tre produits ˆ partir de longs ARN double-brins exog•nes appelŽs dsARN (double stranded ARN) ou de shARN (short hairpin ARN), qui sont des courts duplexes intramolŽculaires dÕARN. Ces ARN seront clivŽs par lÕenzyme Dicer, qui produira des duplexes siARN, de 20 ˆ 25 nuclŽotides de longueur, comportant une extrŽmitŽ 5Õ-monophosphate et une extrŽmitŽ 3Õ avec deux nuclŽotides sortants. Ces param•tres (longueur et nature des extrŽmitŽs) sont cruciaux pour la prise en charge par le complexe RISC. LÕavantage dÕexprimer les siARN ˆ partir de dsARN ou de shARN rŽside dans la possibilitŽ dÕexprimer ces ARN stablement, ˆ partir dÕun vecteur plasmidique ou viral. En effet, ˆ la diffŽrence des siARN, dont lÕeffet apr•s transfection est transitoire (inhibition de la cible jusquÕˆ cinq ˆ sept jours), les shARN stablement intŽgrŽs dans la cellule peuvent induire une inhibition ˆ long terme.
1. 2. MŽcanismes de rŽpression post-transcriptionnelle
conduite par les miARN
Des centaines de miARN sont aujourdÕhui connus, chacun Žtant prŽsumŽ rŽguler lÕexpression de centaines de cibles (Behm-Ansmant et al. 2006). Bien que les mŽcanismes prŽcis de lÕinhibition post-transcriptionnelle gouvernŽe par les miARN soient encore controversŽs, il semblerait quÕil sÕagisse dÕun blocage stŽrique dans la majoritŽ des cas, sans destruction de lÕARNm (Deleavey & Damha 2012; Filipowicz et al. 2008). La Figure 2
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #&!
rŽcapitule les diffŽrents mŽcanismes suggŽrŽs pour la rŽpression post-transcriptionnelle de lÕexpression des g•nes dans les cellules animales.
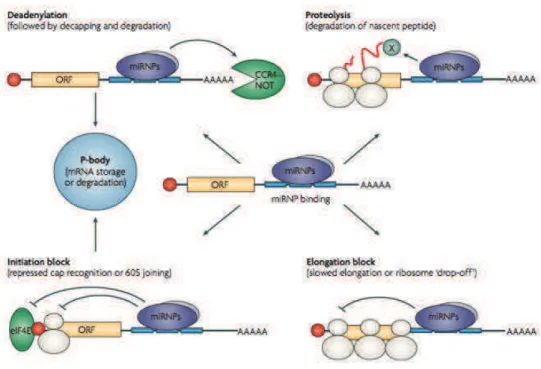
Dans le cas dÕune rŽpression stŽrique de la traduction, de nombreux exemples sont en faveur dÕun blocage de lÕŽtape dÕinitiation de la traduction. Certaines Žtudes dans des cellules HeLa utilisant des ARN messagers rapporteurs exprimant la lucifŽrase et comportant plusieurs sites de fixation pour un miARN naturel (let-7 ; Pillai et al. 2005) ou synthŽtique (CXCR4 ; Humphreys et al. 2005) dans leur 3ÕUTR, ont montrŽ lÕimportance de la coiffe 7-mŽthylguanosine pour la rŽpression induite par les miARN. En effet, lÕintroduction dÕune sŽquence virale IRES (Internal Ribosomal Entry Site) en 5ÕUTR de lÕARNm, permettant lÕassociation des ribosomes avec lÕARNm indŽpendamment de la coiffe, abolit lÕactivitŽ rŽpressive des miARN, mettant en Žvidence la nŽcessitŽ de la 7-mŽthylguanosine pour cette rŽpression traductionnelle. Plus prŽcisŽment, ces Žtudes sugg•rent une inhibition due ˆ une interfŽrence avec le recrutement du facteur dÕinitiation de la traduction eIF4E sur la 7-mŽthylguanosine, ou avec lÕinteraction entre les facteurs eIF4E et eIF4G. En effet, ces facteurs ont un r™le important dans la reconnaissance de lÕARNm par la sous-unitŽ ribosomale 40S, ainsi que dans la circularisation de lÕARNm, qui stimule lÕinitiation de la traduction. De plus, certaines Žtudes (Bhattacharyya et al. 2006; Pillai et al. 2005) ont relevŽ que lÕARNm rŽprimŽ, ainsi que le miARN et la protŽine AGO2, colocalisent dans les processing bodies (P-bodies), qui sont des structures cytoplasmiques impliquŽes dans le catabolisme des ARNm (dŽadŽnylation, decapping et dŽgradation), mais qui pourraient Žgalement avoir un r™le dans le stockage de ces ARN. Cette relocalisation dans les P-bodies serait plut™t une consŽquence quÕune cause de la rŽpression (Pillai et al. 2005; Eulalio et al. 2007), et apporte potentiellement un caract•re rŽversible ˆ lÕinhibition provoquŽe par les miARN. Un mŽcanisme alternatif de lÕaction des miARN identifie le facteur dÕinitiation eIF6 comme cible potentielle de la machinerie miARN (Chendrimada et al. 2007). Ce facteur est essentiel pour la biogen•se de la sous-unitŽ ribosomale 60S, et jouerait aussi un r™le dans la rŽgulation de lÕassociation de cette-unitŽ ˆ la sous-unitŽ 40S lors de lÕinitiation de la traduction. En interagissant avec ce facteur, les protŽines AGO pourraient rŽprimer la traduction en emp•chant la sous-unitŽ 60S de sÕassocier ˆ la sous-unitŽ 40S, interfŽrant ainsi avec lÕinitiation de la traduction.
La rŽpression engendrŽe par les miARN pourrait Žgalement se rŽaliser ˆ des Žtapes de la traduction postŽrieures ˆ lÕinitiation (Petersen et al. 2006; Olsen & Ambros 1999). Il a ŽtŽ
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #'!
montrŽ par des expŽriences de fractionnement des polysomes (ensemble des ribosomes sur un ARNm) par ultracentrifugation en gradient de sucrose, que lÕARNm rŽprimŽ par le miARN CXCR4 est associŽ ˆ des polysomes actifs (Petersen et al. 2006). De plus, dans cet exemple, la traduction initiŽe par une sŽquence IRES est Žgalement rŽprimŽe par le miARN, et il semblerait que le miARN induise une terminaison prŽmaturŽe de la traduction. Dans lÕensemble, les rŽsultats seraient en faveur dÕun mod•le dit de Ôribosome drop offÕ, dans lequel les ribosomes seraient dissociŽs prŽmaturŽment de lÕARNm rŽprimŽ. Les mŽcanismes fins de la rŽpression traductionnelle engendrŽe par les miARN sont donc encore mal compris, et pourraient •tre variables selon le syst•me considŽrŽ (organisme, type cellulaire, niveaux dÕexpressions, cible ARNm, miARNÉ).
Cependant, les mod•les de rŽgulation de lÕinitiation et de lÕŽlongation de la traduction ne sont pas forcŽment mutuellement exclusifs. Par exemple, il est envisageable que lÕŽtape dÕinitiation soit toujours inhibŽe, mais que les ribosomes puissent sÕaccumuler quand lÕŽtape dÕŽlongation est Žgalement inhibŽe, masquant ainsi la dŽtection de lÕeffet sur lÕinitiation (Filipowicz et al. 2008).
Une hypoth•se supplŽmentaire parfois rapportŽe est un effet rŽpressif rŽsultant dÕune dŽgradation de la protŽine successivement ˆ la traduction. Cependant, cette possibilitŽ a ŽtŽ plusieurs fois envisagŽe et aucune protŽolyse ou protŽase associŽe nÕa pu •tre mise en Žvidence. De plus, des inhibiteurs du protŽasome sont sans effet sur la rŽpression (Pillai et al. 2005).
Un dernier mŽcanisme envisagŽ concerne une dŽgradation de lÕARNm. En effet, selon le mod•le gŽnŽral, le degrŽ de complŽmentaritŽ entre le miARN et sa cible gouverne le mode de rŽpression : le miARN induira un clivage si lÕARNm contient une complŽmentaritŽ suffisante (mŽcanisme semblable ˆ celui des siARN), tandis quÕil rŽprimera la traduction sans dŽgradation de lÕARNm si celui-ci ne contient plus de mŽsappariements (Bartel 2004). Cependant, certaines Žtudes dŽmontrent une dŽstabilisation de lÕARNm cible, dans des cas de complŽmentaritŽ imparfaite. Une Žtude in vivo chez le nŽmatode C.elegans, montre que le miARN endog•ne let-7 induit une inhibition dÕun facteur trois de lÕARNm cible lin-41. Ce m•me miARN induit Žgalement lÕinhibition des ARNm lin-14 et lin-28 qui comportent Žgalement des sites de fixation ˆ ce miARN dans leur 3ÕUTR (Bagga et al. 2005). De plus, certaines donnŽes (Sen & Blau 2005; Liu et al. 2005) rapportant une colocalisation des miARN et de lÕARNm rŽprimŽ avec les protŽines Argonautes (AGO1-AGO4), les enzymes dÕexcision de la coiffe DCP1 et DCP2 et lÕexonuclŽase 5Õ! 3Õ Xrn1 au sein des P-bodies, confortent lÕhypoth•se que certains miARN pourraient induire la dŽgradation de leur cible
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #(!
dans ces centres spŽcialisŽs. En effet, si le mŽcanisme de clivage dÕun miARN parfaitement complŽmentaire ˆ sa cible est semblable ˆ celui utilisŽ par les siARN (clivage par Ago2 de la liaison phosphodiester entre les nuclŽotides en position 10 et 11), la dŽgradation induite par les miARN serait consŽcutive ˆ une dŽadŽnylation et une excision de la coiffe. Notamment, une Žtude chez la drosophile montre que la protŽine GW182, requise pour lÕintŽgritŽ des P-bodies, interagit avec les protŽines AGO et initie la dŽgradation du transcrit via une dŽadŽnylation par le complexe CCR4-NOT et une excision de la coiffe via le complexe DCP1-DCP2 (Behm-Ansmant et al. 2006). On ignore encore si cette dŽgradation de lÕARNm se dŽroule de mani•re concomitante ˆ la rŽpression traductionnelle stŽrique, ou si elle la prŽc•de ou la succ•de. Les mod•les supposant une inhibition de la circularisation de lÕARNm lors de la traduction pourraient favoriser une dŽgradation simultanŽe, la circularisation rendant la queue poly A moins vulnŽrable aux nuclŽases.
!
Figure 2 : MŽcanismes suggŽrŽs pour la rŽpression post-transcriptionnelle conduite par les miARN dans les cellules animales (dÕapr•s Filipowicz et al. 2008).
Le brin guide des miARN, associŽ au complexe RISC (miRNP, pour micro-ribonuclŽoprotŽines), peut rŽprimer la traduction de sa cible par diffŽrents mŽcanismes, tels quÕune dŽadŽnylation par le complexe CCR4-NOT, ou un clivage protŽolytique de la cha”ne peptidique en cours de synth•se par une protŽase (X). Un blocage stŽrique a Žgalement ŽtŽ suggŽrŽ, au niveau de lÕinitiation de la traduction (interfŽrence avec la reconnaissance de la coiffe par le facteur dÕinitiation eIF4E, ou avec lÕassociation des sous-unitŽs ribosomales) ou de lÕŽlongation (ralentissement de la vitesse dÕŽlongation ou dissociation prŽmaturŽe des ribosomes). Une relocalisation dans les P-bodies successive ˆ la rŽpression de lÕinitiation de la traduction ou ˆ la dŽadŽnylation est suggŽrŽe, impliquant une dŽgradation ou un stockage de lÕARNm. Cercle rouge : coiffe 7-mŽthylguanosine.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #)!
1. 3. DŽfis
Le potentiel thŽrapeutique de ces ARNi, mis en Žvidence par toutes les Žtudes in vitro et in vivo, serait donc immense. Cependant, de nombreux points restent ˆ amŽliorer pour parvenir au dŽveloppement thŽrapeutique de ces technologies.
Tout dÕabord, les acides nuclŽiques, et plus particuli•rement lÕARN, sont rapidement dŽgradŽs dans les cellules. Les siARN ont donc une durŽe de vie limitŽe du fait de leur sensibilitŽ aux nuclŽases. En effet, la demi-vie des siARN nus dans le sŽrum varie de quelques minutes ˆ une heure (Behlke 2006). Le clivage, notamment par les enzymes de la famille des RNAses A, se produit plus frŽquemment dans les rŽgions riches en pyrimidines (Turner et al. 2007). Pour augmenter la rŽsistance aux nuclŽases, lÕintroduction de nuclŽotides 2Õ-O-mŽthyl et de liaisons internuclŽotides phosphorothioates (voir Figure 5) ˆ lÕextrŽmitŽ 3Õ du brin antisens est possible. Cependant, les modifications chimiques envisageables pour le brin antisens sont limitŽes, afin de conserver la fonctionnalitŽ du siARN. Concernant le brin sens, un plus grand nombre de nuclŽotides modifiŽs est tolŽrŽ (possibilitŽ de substituer plus de nuclŽotides par des 2Õ-O-mŽthyl). Les modifications de chimie 2Õ-fluoro, seules ou en combinaison avec des purines 2Õ-O-mŽthyl, stabilisent Žgalement les siARN. Des nuclŽotides LNA peuvent Žgalement •tre incorporŽs dans les siARN, mais leur position est plus critique que pour les 2Õ-O-mŽthyl et 2Õ-fluoro.
De plus, des Žtudes de transcriptome (Jackson et al. 2003) ont rŽvŽlŽ un effet Ôoff-targetÕ des siARN, cÕest-ˆ-dire lÕinhibition de lÕexpression de g•nes autres que le g•ne cible. Il a ŽtŽ montrŽ que la plupart de ces effets non spŽcifiques rŽsultent dÕune homologie avec une rŽgion prŽcise du siARN appelŽe rŽgion ÔseedÕ. Cette rŽgion, prŽsente dans les siARN et les miARN, est composŽe des nuclŽotides 2 ˆ 8 en partant de lÕextrŽmitŽ 5Õ du brin antisens et est une rŽgion dans laquelle la complŽmentaritŽ de sŽquence pour la cible est parfaite, assurant la spŽcificitŽ de ciblage. Chez les miARN, le reste de la sŽquence nÕest que partiellement complŽmentaire. Il suffirait donc que cette sŽquence ÔseedÕ corresponde ˆ dÕautres g•nes pour que le siARN puisse exercer un effet semblable ˆ celui dÕun miARN, sur une cible non spŽcifique. Des approches bioinformatiques prŽdictives sont donc ˆ considŽrer lors du design des siARN pour Žvaluer les possibles effets Ôoff-targetÕ.
Enfin, les ARN double-brins Žtant reconnus par la cellule comme la signature dÕune infection virale, les siARN peuvent dŽclencher une rŽponse immune innŽe conduisant ˆ des
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! #*!
effets secondaires indŽsirables. En effet, la reconnaissance des siARN via des rŽcepteurs appelŽs ÔPattern Recognition ReceptorsÕ (PRR) gŽn•re une rŽponse inflammatoire et antivirale (Whitehead et al. 2011). Parmi ces rŽcepteurs, certains Toll-Like Receptors (TLR) sont impliquŽs dans la dŽtection des siARN : TLR3 (localisŽ dans les membranes plasmique et endosomale, reconna”t lÕARN double-brin), TLR7 (localisŽ dans la membrane des endosomes, reconna”t lÕARN simple-brin) et TLR8 (localisŽ dans la membrane des endosomes, reconna”t lÕARN simple-brin).
Parmi les PRR, deux protŽines cytoplasmiques jouent Žgalement un r™le dans cette reconnaissance des siARN : la protŽine kinase PKR et la protŽine RIG-I (Retinoic Acid-Inducible Gene-1). La PKR rŽagit de mani•re non spŽcifique avec des ARN double-brins de 11 pb de longueur, et induit une rŽponse interfŽron et une inhibition gŽnŽrale de la traduction. La rŽponse PKR provoquŽe par les siARN reste cependant modŽrŽe, par comparaison ˆ celle rŽsultant de plus longs ARN double-brins. La protŽine RIG-I reconna”t les ARN simple ou double-brins 5Õ triphosphate et non coiffŽs. LÕactivation de ces diffŽrentes protŽines PRR, conduit ˆ une transduction de signaux intracellulaires aboutissant ˆ lÕactivation de facteurs de transcription et ˆ lÕexpression dÕinterfŽrons de type I (IFN-! et IFN-") et de cytokines pro-inflammatoires (Figure 3).
Il est ˆ noter Žgalement que TLR7 et TLR8 reconnaissent les ARN simple-brins de mani•re dŽpendante de la sŽquence. Certains motifs de sŽquence sont donc particuli•rement immunostimulateurs, comme la sŽquence UGUGUU (Judge et al. 2005) ou la sŽquence GUCCUUCAA (Hornung et al. 2005). LÕintroduction de certaines modifications chimiques des nuclŽotides, comme les 2Õ-dŽoxy, 2Õ-O-mŽthyl ou LNA (voir Figure 5), peut rŽduire lÕactivation de la rŽponse immune, de mani•re dŽpendante de la position de ces modifications dans la sŽquence.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $>!
!
Figure 3 : PRR (Pattern Recognition Receptors) impliquŽs dans la reconnaissance des siARN et rŽponse immunitaire innŽe associŽe (dÕapr•s Whitehead et al. 2011).
Les protŽines cytoplasmiques PKR et RIG-I, ainsi que certains TLR (Toll-Like Receptors) reconnaissent les siARN et induisent une rŽponse inflammatoire en stimulant la transcription dÕinterfŽrons de type I et de cytokines inflammatoires via activation dÕune cascade de signalisation spŽcifique.
Enfin, le dernier point limitant pour un dŽveloppement clinique des siARN est leur internalisation cellulaire. En effet, les siARN, qui sont gros (environ 13 kDa) et nŽgativement chargŽs, traversent difficilement la membrane plasmique lipophile. Il est donc nŽcessaire dÕutiliser un syst•me transporteur pour internaliser, voire vectoriser (cÕest ˆ dire cibler certains types cellulaires) les siARN. Bien sžr, la nature de ce transporteur influence les diffŽrents points ŽvoquŽs dans cette partie (stabilitŽ, off-target et immunostimulation). Les diffŽrentes stratŽgies pour augmenter lÕinternalisation cellulaire des siARN seront abordŽes dans le second chapitre de cette introduction.
1. 4. DŽveloppement clinique
Plus de dix ans apr•s la dŽcouverte de lÕARNi, une vingtaine de siARN ou shARN sont en dŽveloppement clinique (phases I ou II), mais aucun nÕa encore ŽtŽ approuvŽ sur le marchŽ (Burnett & Rossi 2012). Des progr•s ont ŽtŽ apportŽs pour optimiser la distribution
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $"!
aux tissus cibles, lÕinternalisation cellulaire et lÕefficacitŽ, dans des limites acceptables de toxicitŽ. Parmi les premiers candidats ˆ entrer en essais cliniques, beaucoup Žtaient des siARN sans aucune modification chimique, et pr•s de la moitiŽ des essais cliniques utilisent des siARN nus, non vectorisŽs (Watts & Corey 2012). Le plus avancŽ dÕentre eux, le Bevasiranib (Opko Health), un siARN dirigŽ contre le VEGF pour le traitement de la dŽgŽnŽrescence maculaire liŽe ˆ lÕ‰ge, a ŽtŽ stoppŽ en phase III en 2009 pour des raisons de manque dÕefficacitŽ. Le composŽ le plus avancŽ en cours de dŽveloppement clinique (phase IIb) est un siARN non modifiŽ (ALN-RSV01 ; Alnylam/Cubist), dirigŽ contre la nuclŽocapside du virus RSV (Respiratory syncytial Virus) pour traiter les infections des patients ayant re•u une transplantation pulmonaire (injection intranasale ou par inhalation).
I. 2. Les oligonuclŽotides antisens
!
2. 1. Modifications chimiques et mŽcanismes associŽs
!LÕutilisation des oligonuclŽotides antisens ADN phosphodiester (PO) est limitŽe principalement par leur dŽgradation rapide par les nuclŽases dans les milieux biologiques (sang ou milieu intracellulaire). Un oligonuclŽotide non modifiŽ est dŽgradŽ en quelques heures dans 10% de SŽrum de Veau FÏtal (SVF) (Stein et al. 2008) et sera donc probablement compl•tement dŽgradŽ avant dÕatteindre sa destination. Afin dÕaugmenter la stabilitŽ, des oligonuclŽotides modifiŽs chimiquement ont ŽtŽ dŽveloppŽs, nÕŽtant pas reconnus comme substrats pour les nuclŽases et possŽdant donc une plus grande rŽsistance ˆ la dŽgradation nuclŽolytique. La nature de ces modifications affecte le mode dÕaction des oligonuclŽotides, qui fait intervenir deux mŽcanismes : le clivage catalytique du transcrit ou un blocage stŽrique de la fonction des ARN (Figure 4). Les diffŽrentes gŽnŽrations de modification des oligonuclŽotides seront dŽcrites dans cette partie, ainsi que les limites et le dŽveloppement clinique qui leur sont associŽs.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $#!
!
Figure 4 : MŽcanismes dÕaction des oligonuclŽotides antisens.
A) Clivage de lÕARNm par la RNase H au sein de lÕhŽtŽroduplexe oligonuclŽotide/ARN. B) Blocage stŽrique de lÕinitiation ou de lÕŽlongation de la traduction.
!
!
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $$!
a) 1
•regŽnŽration
La premi•re gŽnŽration dÕoligonuclŽotides modifiŽs chimiquement est composŽe principalement des Phosphorothioates (PS), dans lesquels un atome dÕoxyg•ne du groupement phosphate (non engagŽ dans la liaison phosphodiester) a ŽtŽ remplacŽ par un atome de soufre (Figure 5). Cette modification augmente drastiquement la stabilitŽ de lÕoligonuclŽotide dans les cellules en culture et in vivo, la demi-vie dÕun PS Žtant approximativement de neuf-dix heures dans le sŽrum humain contre environ une heure pour un oligonuclŽotide non modifiŽ (Kurreck et al. 2002; Kurreck 2003). Les PS ont ŽtŽ synthŽtisŽs pour la premi•re fois en 1969 par Eckstein et ses coll•gues (De Clercq et al. 1969) et plusieurs molŽcules PS ont progressŽ dans des essais cliniques depuis, certaines Žtant aujourdÕhui en phase III. Seulement une, le Fomivirsen ou Vitravene¨ (ISIS/Novartis), a obtenu lÕautorisation de mise sur le marchŽ (en 1998), pour le traitement des rŽtinites induites par le cytomŽgalovirus (CMV), par injection intraoculaire (http://www.isispharm.com). LÕexpŽrience des essais prŽ-cliniques et cliniques utilisant les PS a permis de mettre en Žvidence certains inconvŽnients relatifs ˆ lÕutilisation de ces oligonuclŽotides. Tout dÕabord, leur fixation non spŽcifique sur des protŽines cellulaires, notamment avec des protŽines qui interagissent avec des poly-anions tels que lÕhŽparine (laminine, fibronectine, bFGF (basic fibroblast growth factor), VEGF (vascular endothelial growth factorÉ) engendre des effets secondaires toxiques (Guvakova et al. 1995; Dias & Stein 2002). De m•me, les PS peuvent interagir avec certains rŽcepteurs cellulaires, tels que les rŽcepteurs ˆ lÕEGF (Epidermal Growth Factor) ou au VEGF (Vascular Endothelial Growth Factor) et perturber leur activation (Rockwell et al. 1997). Il a aussi ŽtŽ montrŽ que les PS peuvent inhiber les ADN polymŽrases humaines et les RNase H1 et H2 (ˆ fortes concentrations) (Gao et al. 1991). Leur fixation non spŽcifique ˆ des protŽines plasmatiques (hŽparine, thrombine ou albumine) pourrait •tre bŽnŽfique dÕun point de vue pharmacocinŽtique puisquÕelle leur conf•rerait une demi-vie dans le sŽrum augmentŽe, une protection vis-ˆ-vis de la filtration rŽnale, ainsi quÕune pŽnŽtration facilitŽe dans les tissus (Kurreck 2003; Bennett & Swayze 2010). Enfin, lÕaffinitŽ des PS pour lÕARN est lŽg•rement diminuŽe, en comparaison de celle de leurs analogues PO (rŽduction de la tempŽrature de demi-association, ou Tm, de 0,5¡C par nuclŽotide pour lÕassociation dÕun PS ˆ son ARN complŽmentaire, en comparaison au PO) (Crooke 1999). Ceci est compensŽ en partie par une lŽg•re augmentation de la spŽcificitŽ dÕhybridation des PS sur lÕARN.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $%!
Les oligonuclŽotides non modifiŽs (PO) et PS agissent sur lÕexpression post-transcriptionnelle des g•nes, en recrutant la RNase H, enzyme constitutive et nuclŽaire, connue pour son r™le dans la rŽplication de lÕADN (dŽgradation des amorces dÕARN). Cette enzyme clive le brin dÕARN au sein dÕun duplexe ARN/ADN (Stein & Hausen 1969). Chez les mammif•res, la RNase H spŽcifique recrutŽe aux duplexes OligonuclŽotides/ARN est la RNase H1 (Wu et al. 2004). Cette enzyme se lie ˆ lÕhŽtŽroduplexe ARN/ADN et clive lÕARN, en prŽsence de cations divalents (Mg2+, Mn2+), ˆ une position variable entre le septi•me et le dixi•me nuclŽotide ˆ partir de lÕextrŽmitŽ 5Õ de lÕARN. Les oligonuclŽotides PO et PS dirigŽs contre nÕimporte quelle rŽgion de lÕARNm (5Õ ou 3Õ UTR ou rŽgion codante) peuvent ainsi provoquer le clivage puis la dŽgradation du transcrit et inhiber lÕexpression de la protŽine.
b) 2
•megŽnŽration
Dans le but dÕamŽliorer lÕaffinitŽ pour lÕARN, la rŽsistance aux nuclŽases et les param•tres pharmacocinŽtiques des oligonuclŽotides, dÕautres modifications ont ŽtŽ introduites. Les olignonuclŽotides dits de seconde gŽnŽration comportent une modification (alkylation) en 2Õ du sucre et sont appelŽs 2Õ-O-mŽthyl (2Õ-OMe ; comportent un groupe methyl) et 2Õ-O-mŽthoxyethyl (2Õ-MOE ; comportent un groupement methoxyethyl) (Figure 5). Ces modifications conf•rent une bonne rŽsistance aux nuclŽases (demi-vie dÕenviron 12 heures), une toxicitŽ rŽduite par rapport aux PS et une affinitŽ pour lÕARN augmentŽe en comparaison aux PO et PS isosŽquentiels. La Tm des 2Õ-OMe est augmentŽe de moins dÕ1¡C par modification par rapport aux PO (Kurreck et al. 2002), contre une augmentation de 2¡C par modification 2Õ-MOE (Bennett & Swayze 2010).
Ces oligonuclŽotides de seconde gŽnŽration, en revanche, ne peuvent activer la RNase H. En effet, les Žtudes mŽcanistiques et structurales sur la RNase H ont montrŽ lÕimportance de trois param•tres pour permettre la reconnaissance et le clivage de lÕARN cible (Zamaratski et al. 2001). Tout dÕabord, lÕaccessibilitŽ du groupement 2Õ-OH de lÕARN est cruciale pour le clivage. Toute modification ˆ proximitŽ du petit sillon doit donc •tre ŽvitŽe pour prŽserver lÕaccessibilitŽ de ce groupe 2Õ-OH. De plus, la largeur du petit sillon de lÕhŽlice formŽe par le duplexe OligonuclŽotide/ARN est cruciale pour la reconnaissance par la RNase H. Celle-ci doit •tre suffisamment large pour sÕaccommoder au site de clivage de lÕenzyme, ce qui pourrait expliquer en partie la spŽcificitŽ de lÕenzyme pour les duplexes ARN/ADN.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $&!
Structurellement, les duplexes ADN/ADN sont de forme B, dont la largeur du petit sillon varie entre 3 et 7,4 •. LÕhŽlice formŽe par lÕhŽtŽroduplexe ARN/ADN comporte un petit sillon beaucoup plus large (entre 8,9 et 10,5 •), plus proche de celui adoptŽ par les duplexes ARN/ARN (de 9,3 ˆ 10,8 • : forme A). La discrimination vis-ˆ-vis de la forme A pourrait dŽpendre de la rigiditŽ des deux brins dans les duplexes ARN/ARN (Mangos et al. 2003). En effet, la flexibilitŽ de lÕoligonuclŽotide doit permettre une adaptation conformationnelle nŽcessaire au clivage par lÕenzyme. Ainsi, les modifications en 2Õ du sucre des oligonuclŽotides de seconde gŽnŽration ne sont pas compatibles avec un clivage par la RNase H.
Selon leur cible, ces oligonuclŽotides inhibent par blocage stŽrique le mŽtabolisme de divers ARN : inhibition de la traduction des ARNm (Figure 4B), interfŽrence avec lÕŽpissage des prŽ-ARNm, ou blocage de la fonction des miARN. Ce mŽcanisme dÕaction a ŽtŽ montrŽ pour la premi•re fois en 1997 (Baker et al. 1997), avec un oligonuclŽotide 2Õ-MOE dirigŽ contre la rŽgion de la coiffe en 5Õ du transcrit de ICAM-1 (Intercellular Adhesion Molecule 1), qui bloque la formation du complexe 80S dÕinitiation de la traduction, sans interfŽrer avec lÕŽpissage ou le transport nuclŽo-cytoplasmique des transcrits.
Alternativement, des oligonuclŽotides antisens dits ÔgapmersÕ peuvent •tre utilisŽs pour induire un clivage de lÕARNm par la RNase H. Ces oligonuclŽotides comportent des modifications de 2•me ou 3•me gŽnŽration ˆ leurs extrŽmitŽs, de mani•re ˆ augmenter la stabilitŽ vis-ˆ-vis des nuclŽases ainsi que lÕaffinitŽ pour lÕARN, et une fen•tre de 6-8 nuclŽotides PO ou PS contigus minimum (Kurreck et al. 2002), de mani•re ˆ recruter la RNase H.
Une quinzaine dÕoligonuclŽotides 2Õ-OMe ou 2Õ-MOE, dont la grande majoritŽ sont des gapmers, sont actuellement en essais cliniques (principalement en phase I ou II). Un gapmer 2Õ-MOE/PS, le Mipomersen (Genzyme/ISIS), a re•u lÕautorisation de mise sur le marchŽ en janvier 2013 (http://www.kynamro.com). Cet oligonuclŽotide cible lÕARNm de lÕapolipoprotŽine B100 (APOB100) et est administrŽ en injections sous-cutanŽes aux patients atteints de formes familiales dÕhypercholestŽrolŽmie. Il sÕagit du premier oligonuclŽotide antisens dŽlivrŽ en systŽmique ˆ atteindre le marchŽ.
! +,-./012-3/,! 45673-.8!+!9!:6!;-.6-<=38!6,-3;8,;!
! $'!
c) 3
•megŽnŽration
Afin dÕamŽliorer les propriŽtŽs antisens des oligonuclŽotides, un grand nombre dÕanalogues de lÕARN ou de lÕADN ont ŽtŽ dŽveloppŽs et constituent la troisi•me gŽnŽration dÕoligonuclŽotides antisens modifiŽs. Les modifications chimiques concernent les liaisons phosphodiester, le sucre ou les nuclŽotides. Seules certaines de ces modifications, parmi les plus prometteuses, seront dŽcrites ici.
Les Locked Nucleic Acids (LNA) :
Les LNA, initialement synthŽtisŽs en 1998 par Imanishi et Wengel (Ers et al. 1998; Koshkin et al. 1998), sont des ribonuclŽotides contenant un pont mŽthyl•ne reliant lÕoxyg•ne 2Õ du ribose avec le carbone en 4Õ (Figure 5), qui leur impose une conformation C3Õ-endo et par consŽquent une flexibilitŽ restreinte. Ces molŽcules poss•dent des propriŽtŽs intŽressantes pour leur rŽsistance ŽlevŽe aux nuclŽases, mais surtout pour leur affinitŽ dÕhybridation exceptionnelle vis-ˆ-vis de lÕADN et de lÕARN. Cette augmentation dÕaffinitŽ est plus importante vis-ˆ-vis de lÕARN, et la Tm est classiquement augmentŽe de 5 ˆ 7¡C par base LNA (Stein et al. 2008). La plus grande augmentation de Tm est observŽe lorsque les nuclŽotides LNA sont introduits dans un oligonuclŽotide PS, qui conf•re une ŽlŽvation de la
Tm de 9 ˆ 10¡C par modification. LÕintroduction de nuclŽotides LNA dans un oligonuclŽotide
conduit ˆ un changement conformationnel du duplexe vers une hŽlice de type A. De plus, les modifications LNA induisent un changement conformationnel des dŽoxynuclŽotides avoisinants, ce qui explique que lÕinsertion de seulement quelques nuclŽotides LNA suffit ˆ confŽrer une tr•s haute affinitŽ ˆ lÕoligonuclŽotide entier.
Les modifications LNA peuvent •tre introduites sous deux formes dans un oligonuclŽotide, aboutissant ˆ un oligonuclŽotide dit ÔmixmerÕ, ou ˆ un oligonuclŽotide dit ÔgapmerÕ. Les mixmers LNA sont des oligonuclŽotides dans lesquels des nuclŽotides LNA sont dispersŽs tout le long de la sŽquence, ˆ raison dÕun LNA tous les deux ou trois nuclŽotides. Tout comme la grande majoritŽ des oligonuclŽotides comportant des modifications en 2Õ du ribose, les LNA nÕactivent pas la RNase H et peuvent agir selon un mŽcanisme de blocage stŽrique, sur la traduction (Fluiter et al. 2003) ou lÕŽpissage (Aartsma-Rus et al. 2004), ou en interfŽrant avec la fonction dÕun miARN (Lanford et al. 2010). Les oligonuclŽotides LNA gapmers sont constituŽs dÕun segment central dÕADN ou de PS et de deux segments de LNA aux extrŽmitŽs de lÕoligonuclŽotide. Une sŽquence centrale de 7-8