HAL Id: tel-01827019
https://tel.archives-ouvertes.fr/tel-01827019
Submitted on 1 Jul 2018HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
nucléaire de régulation post-transcriptionnelle par les
micro-ARN
Nedra Tekaya Hamouda
To cite this version:
Nedra Tekaya Hamouda. Identification et étude d’un nouveau mécanisme nucléaire de régulation post-transcriptionnelle par les micro-ARN. Sciences agricoles. Université Nice Sophia Antipolis, 2016. Français. �NNT : 2016NICE4007�. �tel-01827019�
Identification et
Etude d’un Nouveau
Mécanisme Nucléaire de Régulation
Post-Transcriptionnelle par les micro-ARN
Ecole Doctorale des Sciences de la Vie et de la Santé de Nice (ED 85) Centre Méditerranéen de Médecine Moléculaire de Nice
Thèse de Doctorat
Pour l’obtention du grade de Docteur en Sciences de la Vie et de la Santé Spécialité : Interactions Moléculaires et Cellulaires
Présentée par
Madame Nedra HAMOUDA-TEKAYA
Thèse soutenue le
31 Mars 2016
Au Centre Méditerranéen de Médecine Moléculaire de Nice
Devant un jury de thèse composé de :
Dr. Jean-François TANTI Président du Jury
Dr. Sébastien FRIBOURG Rapporteur
Dr. Annick HAREL-BELLAN Rapporteur
Dr. Stéphane AUDEBERT Examinateur
Dr. Pascal BARBRY Examinateur
Les travaux de doctorat présentés dans ce manuscrit
ont été réalisés de
Janvier 2013
à
Février 2016
dans le
Centre Méditérranéen de Médecine Moléculaire de
Nice au sein de l’équipe
«
Control of Gene Expression
»
sous la direction du
Je tiens à exprimer mon profond respect à tous les membres composant mon jury de thèse et à leur adresser mes remerciements de
m’avoir honorée de faire partie de ce jury.
Je souhaite remercier le Dr. Jean-François TANTI d’avoir accepté de présider mon jury de thèse et pour ses précieux conseils.
Mes sincères remerciements s’adressent aussi
au Dr. Annick HAREL-BELLAN et au Dr. Sébastien FRIBOURG qui ont consacré de leur temps pour évaluer ce manuscrit. Enfin, je remercie chaleureusement le Dr. Pascal BARBRY et le Dr. Stéphane AUDEBERT d’avoir accepté d’examiner mon travail de
Je souhaite ensuite remercier tous les membres de l’équipe 10, en particulier :
Michele, je te remercie de m’avoir accueillie dans ta jeune équipe il y a trois années qui ne cesse de s’agrandir et d’évoluer. Tu m’avais prévenu depuis le premier jour que ça n’allait pas être facile au quotidien… et tu avais raison! Je tiens à te remercier pour tout ce que tu m’as appris, pour les discussions scientifiques enrichissantes (surtout ton fameux « new layers of conplexity » !), pour m’avoir transmis ton esprit d’analyse critique et ta rigueur et enfin de m’avoir permis de vivre l’expérience de l’encadrement pendant ma thèse… Merci pour tout !
Emy, j’aurais aimé t’écrire ces mots en italien mais il voudrait mieux que je ne le fasse pas , tu connais mon vocabulaire particulier… Plus sérieusement, tu as été comme une grande sœur pour moi, merci pour tes conseils et tes petites attentions. Merci aussi pour tout ce que tu m’as appris à la paillasse. Je te souhaite le meilleur pour la suite de ta carrière et aussi pour les trois bouts de choux.
Zoheir, mon co-thésard et colocataire de bureau et de paillasse. Je te remercie d’avoir supporté mon « ralage » quotidien (intensif ces derniers mois) pour absolument tout (hhh) ! Nos petits délires vont me manquer notamment sur l’actualité ciné ou musicale (accompagné de « one O six point five riviera Radio ») ainsi que les sushis préparés par tes soins de temps à autre . Je te souhaite de finir ta thèse en beauté et un bel avenir avec ta future épouse Carole (vivement Bali).
Silvia, I am super glad that I have met you and worked with you. You are such talented scientist. I wish you the best for your career but also for you and your family (huge kiss to princess Sofi ^^).
Anaïs C, tu n’as pas cessé de m’encourager tout le long, je te remercie pour tout.
Anthony P et Elisiane KL, j’étais ravie de vous avoir rencontré et encadré pendant vos masters respectifs. J’espère que je ne vous ai pas trop traumatisés :) je vous souhaite un avenir radieux.
Jeannine C, je te remercie pour ton aide dans toutes les démarches administratives et surtout pour toutes les commandes hyper urgentes (toutes quoi) que tu as due passer pour moi :).
Un grand merci à toute l’équipe 5 : Mohamed B, Claire M, Carole B et Katia, pour vos encouragements et votre sympathie.
Rachel B, je te remercie énormément de m’avoir accueillie dans ton bureau pendant ces mois d’écriture. Merci aussi pour tes conseils, pour les divers kits de survie que tu as mis à ma disposition pour les moments de stress ou juste pour accompagner un café . Je n’oublierai surtout pas de te dire combien ton aide surtout pour le manuscrit m’a été précieuse. Un GRAND merci <3.
Rihab et Amal, qu’est ce qu’on a rigolé ! Merci les filles pour ces beaux moments qui ont égayé la vie au labo.
Ma petite Wafa, (tu remarqueras que j’ai évité de dire Wifi, oups !) nos pauses café m’ont vraiment manqués ces derniers mois ! Je te remercie d’avoir été là pour moi surtout merci pour ton amitié. Je te souhaite le meilleur pour ta thèse (et après la thèse) et un bonheur infini avec ta petite famille qui s’est agrandie.
Ma Lilouch, j’espère de tout mon cœur que tu iras au bout de ce chemin, avec excellence, 2016 c’est NOTRE année. Tu m’avais vachement manqué cette dernière année. Merci pour ton amitié et pour ta grande générosité, tu es une fille en OR!
Milouch, je te remercie pour ta gentillesse, tes encouragements quotidiens et ta bonne humeur. Je vous adore les filles !
(bientôt à trois). Merci au plus gentil et sympa de tous, Patrick M par contre je ne te félicite pas pour les moments où tu n’as pas manqué de me rappeler « qu’est ce que tu fais en blouse, tu n’as pas une thèse à écrire ?! » (hhh). Anne D, Orane V, Maria, Elsa G et Greg et Laurent; merci à tous sans exception pour votre aide, votre bonne humeur et votre sympathie. Merci aussi pour le dépannage de produits par-ci par-là, toujours avec une grande générosité. You are the best guys ;)
Mon équipe de cœur, Philippe, Stephy, Débo, Béa, GiGi. J’ai passé beaucoup de moments avec vous depuis mon master 1. Vous étiez toujours à mon écoute et votre soutien m’était précieux. Je tiens ici à remercier du fond du cœur Béa et Philippe, vous m’avez fait confiance pour entamer une thèse avec vous il y a déjà quatre ans …
Mon équipe adoptive par alliance, l’équipe 2. Vous m’avez un peu trop vu trainée dans vos labos, je ne sais pas pourquoi (hhh). Sandrine M, Pascal C, et Nathan merci pour votre sympathie et vos encouragements. Ma petite Alix, merci pour ta gentillesse et ta bonté je te souhaite aussi le meilleur pour ta thèse et pour la suite. Arnaud, je suis d’accord avec Amine quand il dit « la carrétude » d’Arnaud est inégalable, je te remercie infiniment pour tous tes conseils et surtout pour ta lecture et tes remarques pertinentes, comme toujours. Fredo, je n’ai pas oublié le lapin que tu m’as posé pour le master 2 (hhh). Tu es une personne formidable, tu sais à quel point je t’apprécie et je tiens à te le dire ici. Merci pour tous les « pas clair » et « mal dit » (hhhhh) qui m’ont beaucoup aidé aussi. Guigou, toujours là pour nous faire rire avec tes blagues plus drôles les unes que les autres, merci pour ton aide technique notamment pour le Facs et le test de prolif. Enfin, Patrick, merci pour ta grande sympathie, ton immense modestie et ta disponibilité. Je tiens tout particulièrement à te remercier pour tes conseils ainsi que ton soutien dans toutes mes démarches tout le long de ma thèse.
Fred B, tu es et tu seras toujours le rêve de tout étudiant et post-doc ;). Merci pour ta grande sympathie, ta gentillesse sans faille et pour tes précieux conseils. Merci d’avoir partagé avec nous des souvenirs exceptionnels de vacances ;).
Chris, je te croisais à l’IPMC mais ici je t’ai connu et beaucoup apprécié. Ton attitude zen et posée ainsi que ta vision philosophique des choses m’ont toujours impressionnées. Merci pour tous tes conseils et d’avoir été là quand j’en avais besoin !
Miss zObba, beaucoup de beaux souvenirs depuis la licence (surtout notre passion pour CG hhhh). Je te souhaite beaucoup de succès et de bonheur avec PL pour votre nouvelle vie à Oxford .
Elodie, quelle belle rencontre ! Avec ta joie et ta bonne humeur on ne peut que t’adorer :), je suis sure que tu vas cartonner pour ta thèse et dans ta future carrière. Merci pour tes petits mots d’encouragements et de soutien au quotidien (vivement le 29 Mai hhh).
Diogo, le portugais préféré des tunisiens :-p merci d’être toujours là pour nous faire rire avec tes anecdotes et tes blagues, tu vas nous manquer ! pfff qu’est ce que je raconte je pars aussi lol.
To the columbian beauty, Camila notre séjour à Barcelone n’aurait pas été le même sans tes conseils et tes bons plans ^^ merci pour ta gentillesse... I wish you the best!
Lazaro, (alias szzzszzzz hhhhh) tu m’as transmis beaucoup de bonnes ondes positives surtout le matin dans le 22 en arrivant au C3M! Merci pour ta gentillesse et je te souhaite de réaliser tout ce que tu désires dans la vie…
wish you the best my friend !
Un énorme MERCI à Fabienne M, Laura M, Dominique S, Carole S, Virginie M, Stephan C, Bastien V, Jérome, Mireille C, Karine D, Jean P, Nadia H, Ali, GMDD, Nadia, Zoulikha, Ruxanda, Jean François P, Didier M et Véro I pour votre amitié et votre sympathie (et au passage le crosslinker de l’équipe 4 pour les longues heures passer à irradier les cellules sans jamais lâcher).
Mon ancienne chef de master, Véronique B à l’IPMC. Je tiens à te remercier pour tout ce que tu m’as appris mais aussi de m’avoir redonnée confiance en moi.
A mes amis qui m’ont soutenu durant ma thèse :
Anaïs, ma « coupine », ma petite rousse préférée et Toni, pleins de merveilleux souvenirs ensemble (nos mariages respectifs, la naissance des petits loulous…). J’espère qu’on partagera ensemble pleins d’autres beaux moments! Je vous souhaite à tous les deux, Cléo et Mattéo tout le bonheur du monde.
Anouar, je te remercie pour tous les moments passés ensemble depuis le master à l’IPMC et pour ceux à venir. Une belle carrière t’attend. Beaucoup de bonheur pour toi et Lenka.
Steph P, Chokran beaucoup pour tous les fous rires et les déconnades, je t’adore <3.
Charly, alias le Nez, tu m’impressionneras toujours, reste comme tu es ;).
Clém et Maeva, merci pour tout ce que vous êtes : gentilles, à l’écoute et de bons conseils, je vous souhaite le meilleur aussi.
Selma, Mansour, Fériel, Ophélie « la Cassut’ » et Coline (« Claudine »), je vous remercie tous pour les moments passés ensemble et pour votre amitié.
Yousra. Plus qu’une amie, même à des centaines de km tu es présente et tu m’encourages sans cesse, je t’adore ma Yoss.
Omar, on était le quatuor de choc avec Amine et Sophie, ya hasra ! Les années ISSBAT me manquent tellement. Je te souhaite tout le bonheur du monde et une belle carrière de végétaliste comme tu l’as toujours souhaité !
Un merci tout particulier plein d’amour à ma belle famille pour leur soutien et encouragements. Je pense surtout à ma deuxième famille : Tata Fouzia, Hinda, Tristan, Kais, Magali, Karim, tata Souad, ainsi qu’à tata Rafika, tonton Habib et omi Aicha…
Je ne pouvais pas ne pas dédier quelques mots à ma deuxième maman Feiza… tu m’avais dit des choses très importantes il y a très longtemps que je n’ai jamais oublié… merci <3
A mon mari,
Mon chéri et mon être le plus cher, Amine. Malheureusement je n’ai que quelques lignes pour t’exprimer ma gratitude pour tout ce que tu m’apportes. Merci d’être toujours à mes côtés pour me soutenir (et me supporter surtout) dans toutes les épreuves et me pousser vers l’avant surtout dans les durs moments (je n’étais pas facile parfois, je le sais). Je tiens à t’exprimer ici combien t’avoir à mes côtés me rassure et procure de la force. Je t’aime.
A mes sœurs et frères,
Mouna, ma sœur, ma deuxième maman merci pour tes douces paroles et d’être là pour moi <3 allez, c’est toi la prochaine et n’oublie pas « une thèse réussie est une thèse… » (tu connais la suite ;)). Latifa, ton soutien à ta manière tellement particulière est inégalable et essentiel pour moi, merci pour tout ma Lattoufti. A Sofiane et Azaiez, merci d’être les grands frères que je n’ai jamais eu! Mes petits neveux d’amour Yassou, Sassou, Rayouna et Malouka, que je n’ai pas vu grandir à mon plus grand regret, je vous souhaite beaucoup de réussite et de bonheur. Je vous aime tous.
Le meilleur pour la fin, Papa et Maman.
Les mots ne sont pas assez puissants pour vous exprimer à quel point je vous aime et combien je vous suis reconnaissante pour tout ce que vous avez fait et continuez à faire pour moi. C’est grâce à vous que je suis là aujourd’hui, vous m’avez toujours soutenu dans mes choix. C’est en vous que j’ai puisé ma force pendant les moments difficiles. Je vous dédie donc ce travail et cette distinction. J’espère que vous êtes fiers de moi. Je vous aime tellement fort.
1
LISTE DES ABREVIATIONS ... 4
LISTE DES FIGURES ET TABLEAU ... 8
PREAMBULE ... 10
INTRODUCTION ... 12
I- Généralités ... 13
II- Découverte des micro-ARN ... 15
III- Caractéristiques et nomenclature des micro-ARN ... 16
IV- Biogenèse des micro-ARN ... 17
IV-1- Arrangement génomique des micro-ARN ... 17
IV-2-Transcription en micro-ARN primaires ... 18
IV-3-Clivage des micro-ARN primaires en précurseurs micro-ARN ... 20
IV-3-1- Régulation de l’activité du microprocesseur ... 22
IV-3-2- Modèles murins d'étude de la fonction de Dgcr8 et Drosha ... 25
IV-3-3- Maturation des mirtrons – processus indépendant du microprocesseur ... 26
IV-4-Export nucléaire des pre-micro-ARN ... 27
IV-5-Clivage des pre-micro-ARN en micro-ARN matures ... 27
IV-5-1-Régulation de l’activité de DICER ... 28
IV-5-2- Modèles murins d'étude de la fonction de Dicer ... 30
IV-6- Formation du complexe miRISC (miRNA-induced silencing complex) ... 31
IV-6-1- Les protéines AGO et l’incorporation du duplex miARN-miARN* ... 31
IV-6-2- Régulation post-traductionnelle des protéines AGO ... 32
IV-7- Sélection du brin mature ... 33
V- Modes d’action de la machinerie miARN ... 34
V-1- Notion de région « Seed » ... 34
V-2- Les protéines GW182 et les Processing-bodies ... 35
V-3- Par déadénylation puis dégradation de l’ARNm ... 35
V-4- Par inhibition de l’initiation à la traduction ... 36
V-5- Par inhibition de la traduction après initiation ... 36
V-6- Stimulation de la traduction ... 38
V-7- Cas particulier de ciblage ... 38
V-8- Effet de la présence de plusieurs sites de miARN... 38
VI- Localisation subcellulaire de la machinerie miARN ... 40
VI-1- Cytoplasme ... 40
VI-1-1- Le Réticulum Endoplasmique Rugueux : Site de maturation du RISC et de clivage de la cible ... 40
VI-1-2- Les endosomes: Site d’assemblage du RISC ... 43
VI-1-3- Les exosomes : Export des miARN vers le milieu extracellulaire ... 44
VI-1-4- La mitochondrie ... 45
VI-1-5- Les autophagosomes : lieu de dégradation du complexe RISC ... 47
VI-2- Le Noyau ... 49
VI-2-1- Rôle dans la régulation transcriptionnelle ... 49
2
VI-2-3- Transport des miARN vers le noyau ... 54
VII- Eléments régulateurs du ciblage par les miARN ... 56
VII-1- Complémentarité entre le miARN et sa séquence cible ... 56
VII-1-1- Sites de liaison canoniques ... 56
VII-1-2- Sites de liaison non-canoniques ... 57
VII-1-3- Effets des sites non-canoniques sur l’expression génique ... 58
VII-1-4- Au-delà du site de liaison ... 59
VII-2- Eléments liés au microenvironnement génétique des sites de liaison des miARN ... 60
VII-2-1- L'énergie libre de la liaison miARN/ARNm ... 61
VII-2-2- L'accessibilité du site cible du miARN ... 61
VII-2-3- Présence d’éléments riches en AU aux alentours du site cible ... 62
VII-2-4- Position du site du miARN au sein de l’ARNm... 62
VII-2-5- Les protéines de liaison à l’ARN ... 62
VII-3- Prédiction bioinformatique du site de liaison du miARN ... 64
VII-4- Caractérisation des cibles de miARN par des outils biologiques ... 66
VII-4-1- Analyse de l’expression génique ... 66
VII-4-2- Approches biochimiques ... 66
VII-4-2-1- PAR-CLIP ... 67
VII-4-2-2- iCLIP... 67
VII-4-2-3- CLASH ... 68
VIII-Importance des protéines de liaison à l’ARN (RBP) dans le ciblage des miARN ... 69
VIII-1- HuR ... 69 VIII-2- DND1 ... 71 VIII-3- PUM1 ... 72 VIII-4- PTB ... 73 VIII-5- CRD-BP ... 74 VIII-6- RBM38 ... 75
VIII-7- Cas particuliers ... 75
VIII-7-1- ROQUIN... 75
VIII-7-2- MOV10/FMRP ... 76
IX- Sfpq : une protéine multifonctionnelle ... 78
IX-1-Généralités ... 78
IX-2- Structure et domaines de SFPQ ... 78
IX-3- Fonctions liées à SFPQ ... 80
IX-3-1- Epissage ... 80
IX-3-2- Processing du 3' ... 81
IX-3-3- La rétention nucléaire ... 81
IX-3-4- Traduction ... 81
IX-3-5- Transcription ... 82
IX-3-6- Réparation suite aux dommages à l’ADN ... 83
IX-3-7- Infection virale ... 83
IX-4- Régulation post-traductionnelle ... 84
RESULTATS ... 86
DISCUSSION & PERSPECTIVES ... 141
3
II- Importance de Sfpq dans le complexe ... 144
III- Mécanisme(s) régulateur(s) de la machinerie miARN ... 145
IV- Rôle des paraspeckles ? ... 146
V- Impact général de Sfpq sur la liasion d’Ago2 aux transcrits cibles ... 148
CONCLUSION... 150
RÉFÉRENCES BIBLIOGRAPHIQUES ... 152
4
Liste des
5
Aa: Acide Aminé
ADAR: Adenosine DeAminase RNA-specific ADN: Acide DésoxyriboNucléique
ADNc: Acide DésoxyriboNucléique
complémentaire
ADP: Adénosine DiPhosphate AGO: ARGONAUTE
AGO.ca: chromatin-associated AGO
AKT3: V-Akt Murine Thymoma Viral Oncogene
Homolog 3
ALCL: Anaplastic Large Cell Lymphomas ALK: Anaplastic Lymphoma Kinase AMP1: Altered Meristem Program 1 ARN: Acide RiboNucléique
ARNm: ARN messager ARNt: ARN de transfert ARNr: ARN ribosomal ARE: AU Rich Element
ATG: AuTophaGy-related proteins ATM: Ataxia-Telangiectasia Mutated BRCA1: BReast Cancer 1 protein CARM1: Coactivator-associated Arginine
Methyltransferase 1
CDE: Constitutive Decay Element CDS: Coding Sequence
CD45: Cluster of Differentiation 45 ChIP: Chromatin ImmunoPrecipitation
CLASH: Cross-Linking ligation And Sequencing
of Hybrids
CLIP: CrossLink ImmunoPrecipitation COX-2: CycloOXygenase 2
CRAC: UV CRoss-linking and Analysis of cDNA CRD-BP: Coding Region Determinant-Binding
Protein
CRP: C-Reactive Protein
COX-1: CycloOXygenase 1
DCP1-2: mRNA DeCapping enzyme DDR: DNA Damage Response DDX: Dead-box
DGCR8: DiGeorge syndrome Critical Region gene
8
DBHS: Drosophila-Behavior Human Splicing DMPK: Dystrophia Myotonica-Protein Kinase DND1: Dead-End 1
DSB: Double Strand Break
dsRBD: double-stranded RNA Binding Domain eIF: eukaryotic Initiation Factors
EF1A: Elongation Factor 1A
EGFR: Epidermal Growth Factor Receptor Elav: Embryonic lethal abnormal vision ERBB-2: Erb-B2 Receptor Tyrosine Kinase 2 ESCRT: Endosomal Sorting Complex Required for
Transport
EXP5: Exportin 5
FMR1: Fragile X mental Retardation protein 1 FSTL1: Follistatin 1
Gly: Glycine
GDP: Guanosine DiPhosphate GTP: Guanosine Triphosphate HCC: HepatoCellular Carcinoma HDAC: Histone Deacetylases
HITS-CLIP: High-Throughput Sequencing of
RNA isolated by CrossLinking
IimmunoPrecipitation
HIV: Human Immunodeficiency Virus
hnRNPA1: heterogeneous nuclear RiboNucleoProtein A1
HP1: Heterochromatin Protein 1
HSP90: Heat Shock Protein 90
6
IFN: InterFeron
IgE: Immunoglobuline de classe E IL: Interleukine
IPO8: Importin 8
IRALU: Inverted Repeats ALU elements IRES: internal ribosome entry segment kDa: kilo-Dalton
KMT: Lysine MethylTransferase KO: Knock-Out
KSRP: KH-type Splicing Regulatory Protein LAST2: LArge Tumor Suppressor homolog 2 LH: Luteinizing Hormone
lnRNA: long non-coding RNA
MAPKAPK2: MAPK-activated protein kinase 2 MEF: Mouse Embryonic Fibroblasts
miARN: micro-ARN MID: MIDDLE domain
miRISC: miRNA-induced silencing complex MITF: MIcrophthalmia-associated Transcription
Factor
MitomiARN: ARN mitochondrial MNK: MAP kinase interacting kinase mTOR: mammalian Target Of Rapamycin MVB: MultiVesicular Bodies mP 7 P G: 7-methylguanosine NCL: Nucleolin
NDP52: Nuclear Dot Protein 52 kDa
ND1: NADH-ubiquinone oxidoreductase chain 1 NEAT1: Nuclear Enriched Abundant Transcript 1 NES: Nuclear Export Signal
NLS: Nuclear Localization Signal nm: nanomètre
NONO: Non-POU Domain Containing,
Octamer-Binding
nt: nucléotide
PAR-CLIP:
PhotoActivatable-Ribonucleoside-Enhanced Crosslinking and ImmunoPrecipitation
Pb: paire de bases
PMA: Phorbol 12-Myristate 13-Acetate PNPASE: PolyNucleotide PhosphorylASE PSPC1: ParaSpeckle Protein Component 1 RAD51D: DNA repair protein RAD51 homolog 4 R-SBE: RNA Smad-Binding Element
RhoB: Ras homolog B
RPL18: 60S Ribosomal Protéin L18
PABPC: Cytoplasmic Poly(A)-Binding Protein PAZ: PIWI-Argonaute-ZWILLE
piRNA: Piwi interacting RNA
PIWI: P-element-Induced WImpy testes pre-miARN: miARN précurseur pri-miARN: miARN primaire
PTB: Polypyrimidine Tract-Binding protein PSF: PTB-associated Splicing Factor PUF: PUmilio Fem-3-binding factor PUM: PUMilio
Pum-HD: Pumilio-Homoly Domain P-bodies: Processing bodies RAN: RAs-related Nuclear protein RIIID: RNase III domain
RBM38: RNA Binding Motif protein 38 RBP: RNA Binding Protein
RER: Reticulum Endoplasmique Rugueux PER1: PERiod protein 1
RPS6: Ribosomal Protein S6
7
RT-PCR: Reverse Transcription – Polymerase Chain Reaction
SA-TGS: Senescence-Associated Transcriptional
Gene Silencing
SDS-PAGE: Sodium Dodecyl Sulfate– PolyAcrylamide Gel Electrophoresis
SFPQ: Splicing Factor Proline-Glutamine Rich shRNA: short-hairpin RNA
SILAC: Stable Isotope Labeling by Amino acids in
Cell culture
SINE: Short INterspersed Elements siRNA: small interfering RNA SM: Spectrométrie de Masse SMN2: Survival of Motor Neuron 2 snRNP: small nuclear RiboNucleoProtein snoRNA: small nucleolar RNA
STAT6: Signal Transducers and Activators of
Transcription 6
TDP-43: Tar DNA-Binding Protein-43 TGF: Transforming Growth Factor
TGS: Transcriptional Gene Silencing
TNF: Tumor Necrosis Factor alpha
TNRC6A-B-C: TriNucleotide Repeat-Containing
gene 6A-B-C protein
TOP2A: Topoisomerase II alpha
TRAP150: Tyroid Receptor-Associated Protein
Complex 150 kDa
TRBP: TAR RNA-Binding Protein Trp: Tryptophane
tRNA: transfer RNA
TSH: Thyroid-Stimulating Hormone
Tyr: Tryrosine URR: U Rich Region UTR: UnTranslated Region UV: Ultraviolet
WT: Wild-Type XPO1: Exportin 1 XPO5: Exportin 5 XRN: ExoRibonuclease
Zc3h12a: Zinc-finger CCCH-type containing 12A 4PH: prolyl 4-hydroxylase de type I
8
Liste des Figures
9
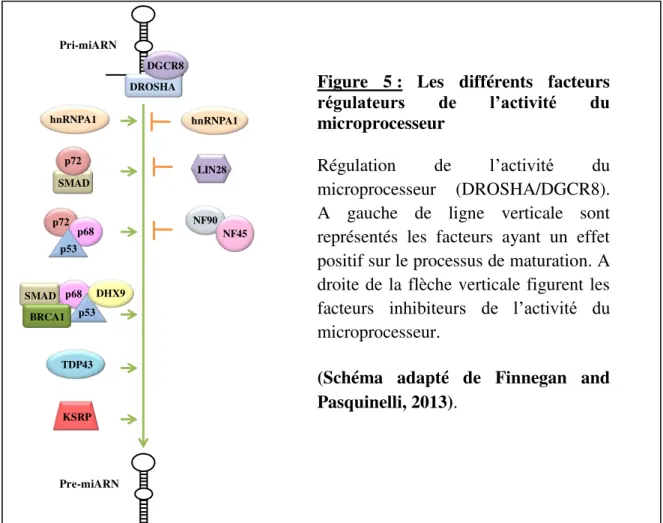
Figure 1 : L'implication des micro-ARN dans les principaux processus biologiques ____________________ 14 Figure 2 : Différentes localisations génomiques du micro-ARN ____________________________________ 17 Figure 3 : Biogenèse des microARN _________________________________________________________ 20 Figure 4 : Structures de miARN primaire et du miARN précurseur _________________________________ 21 Figure 5 : Les différents facteurs régulateurs de l’activité du microprocesseur _________________________ 25
Figure 6 : Les différents facteurs régulateurs de l’activité de DICER ________________________________ 29
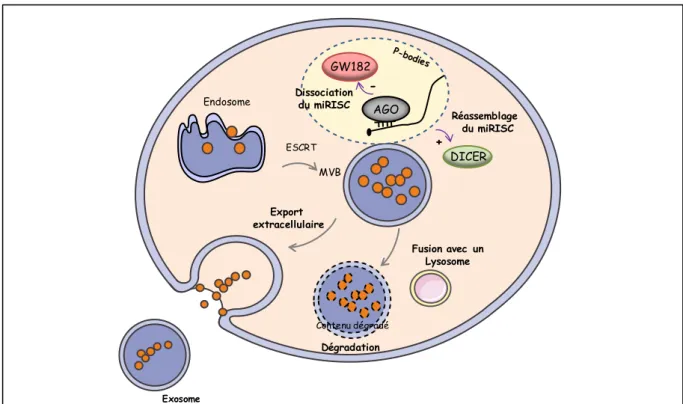
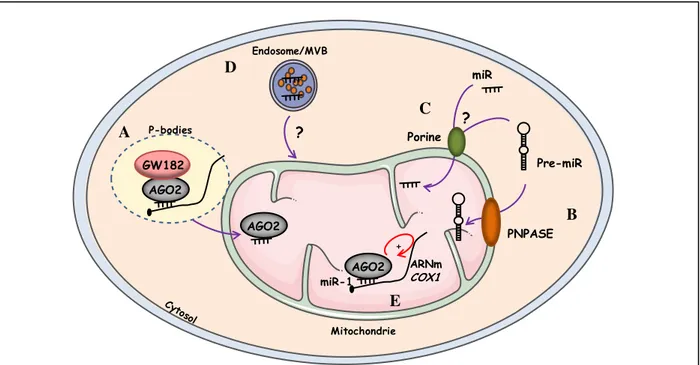
Figure 7 : Organisation des domaines de la protéine humaine AGO2 ________________________________ 32 Figure 8: Interaction miARN/ARNm _________________________________________________________ 34 Figure 9: Représentation schématique des différents mécanismes de ciblage par les miARN _____________ 37 Figure 10: Synergie entre deux sites de miARN proches. _________________________________________ 39 Figure 11: Schéma récapitulatif des principaux rôles des miARN au niveau du réticulum endoplasmique
rugueux ________________________________________________________________________________ 42
Figure 12: Schéma représentatif du rôle des corps multivésiculaires (MVB) dans désassemblage/assemblage du
RISC __________________________________________________________________________________ 44
Figure 13: Schéma représentatif des voies de transports des miARN vers la mitchondrie ________________ 46 Figure 14: Schéma représentatif du rôle de l’autophagie dans la dégradation de composants du RISC : AGO2 et DICER_________________________________________________________________________________ 48
Figure 15: Schéma du modèle de la fonction d’AGOβ/miARN dans le processus de Senescence-Associated-Transcriptional Gene Silencing ______________________________________________________________ 51
Figure 16: Influence des miARN sur l’épissage alternatif suite aux modifications des histones. ___________ 53
Figure 17: Schéma des différentes voies proposées de transport des composants de la machinerie des miARN du
cytoplasme vers le noyau __________________________________________________________________ 55
Figure 18: Les différents types de sites de miARN canoniques _____________________________________ 56 Figure 19: Principaux types de sites de miARN non-canoniques ___________________________________ 58 Figure 20: Position et effet d’un appariement additionnel du γ’ du miARN avec sa séquence cible ________ 60
Figure 21: Principaux facteurs extrinsèques à la séquence seed importants pour la liaison du miARN/ARNm 63 Figure 22: Représentation schématique des étapes clées de la HITS-CLIP. ___________________________ 68 Figure 23: Schéma général du mode d’action des RBP régulatrices de l’activité des miARN _____________ 77
Figure 24: Représentation schématique de la structure primaire de la protéine SFPQ humaine mettant en
évidence les différents domaines protéiques ____________________________________________________ 80
Figure 25 : Schéma mettant en évidence les fonctions de SFPQ dans la cellule ________________________ 84 Figure 26: Principe des HITS-CLIP de SFPQ et d’AGO2 dans les cellules P19 _______________________ 149
Tableau 1 : Tableau récapitulatif des 10 programmes bioinformatiques les plus populaires de prédiction de
10
Préambule
11 Ce travail de thèse a porté sur l’étude de la régulation du ciblage des micro-ARN par les protéines de liaison à l’ARN. Ces protéines de liaison à l’ARN sont des facteurs multifonctionnels qui touchent à plusieurs niveaux (stabilité, transcription, traduction, dégradation) les ARN messagers présents dans la cellule. Le lien entre l’activité des micro-ARN et des protéines de liaison à l’ARN est complexe. Toutefois quelques exemples ont démontré que certaines de ces protéines régulent directement l’activité des micro-ARN.
Pour aborder notre hypothèse de travail, il est nécessaire dans un premier temps de connaitre la biogenèse, le mécanisme de ciblage, la distribution cellulaire ainsi que les acteurs principaux des voies des micro-ARN qui feront l’objet de la première partie de l’introduction. Puis, nous consacrons un chapitre pour décrire la distribution ainsi que les rôles des micro-ARN dans les différents compartiments cellulaires. Ensuite, nous présenterons tous les paramètres régulateurs du ciblage des micro-ARN intrinsèques et extrinsèques au duplex formé par le micro-ARN et sa séquence cible. Enfin, nous dresserons l’état des connaissances des protéines décrites comme étant importantes dans la voie des micro-ARN. Un chapitre descriptif sera dédié en fin de l’introduction à notre protéine d’intérêt, qui représente une partie importante pour une meilleure compréhension de la partie discussion et perspectives.
Les résultats obtenus lors de ces trois années de travail seront présentés sous forme d’un article en préparation pour être soumis dont je suis co-auteur (plus de détails dans le chapitre Résultats).
Un dernier chapitre du manuscrit sera consacré à la conclusion, la discussion et aux perspectives.
12
Introduction
13
I- Généralités
Différentes classes de petits ARN ont été identifiées tels que les miARN (micro-ARN), les siARN (small interfering RNA), les piARN (piwi-interacting RNA) et ceux dérivés du processing des snoARN (small nucleolar RNA), des tARN(transfer RNA), des répétitions Alu et des ARN-Y (Kim et al., 2009b; Repetto et al., 2015). Leurs particularités communes sont de ne pas coder pour une protéine, d’être stables en milieu intra et extracellulaire et de posséder un groupement phosphate à leur extrémité 5’. Cependant, ils possèdent des tailles, une localisation cellulaire, une biogenèse et des fonctions diverses. Dans ce manuscrit nous nous intéresserons à la classe des micro-ARN. D’après leur profil d’expression ainsi que leur conservation, ces petites molécules jouent un rôle important dans de nombreux processus cellulaires tels que le développement (Bernstein et al., 2003), la prolifération cellulaire, l’apoptose (Brennecke et al., 2003), le métabolisme (Poy et al., 2004), la cardiogenèse (Zhao et al., 2005), les fonctions neuronales (Conaco et al., 2006; Schratt et al., 2006), l’immunité (Taganov et al., 2006), etc…. Par conséquent, une dérégulation de l’expression de ces micro-ARN altère ces processus biologiques critiques pour les organismes et entraîne le développement de diverses pathologies comme le cancer du sein (Muluhngwi and Klinge, 2015), le cancer du poumon (Ebrahimi and Sadroddiny, 2015) ou encore le cancer du foie (Mao and Wang, 2015), les maladies infectieuses virales par le HIV (Human Immunodeficiency Virus) (Roşca et al., 2014), le virus de l’influenza (Głobińska et al., β014), les maladies auto-immunes (la sclérose en plaques (Zare-Shahabadi et al., 2013), le lupus érythémateux systémique (Qu and Shen, 2015), les maladies neurodégénératives (Parkinson et Alzheimer (Qiu et al., 2014)), etc… (Figure 1). Sans doute, les microARN représentent un vaste champ à explorer afin de mieux comprendre les processus biologiques et les pathologies qui leurs sont associées.
14
Figure 1 : L'implication des micro-ARN dans les principaux processus biologiques
En violet, quelques exemples des processus biologiques dans lesquels les micro-ARN sont essentiels. En orange, quelques exemples de pathologies dans lesquelles des micro-ARN sont dérégulés.
Développement
Les micro-ARN
Prolifération
cellulaire
Apoptose
Métabolisme
Différenciation
Vieillissement
Programme
Epigénétique
Cancers
Maladies
Infectieuses
Maladies
Immunitaires
Maladies
Métaboliques
Maladies
Dégénératives
15
II- Découverte des micro-ARN
La mise en évidence des miARN, est vraisemblablement l’une des plus importantes découvertes dans le domaine biomédical des vingt dernières années. L’identification du premier miARN date des années 1980 où les post-doctorants Victor Ambros et Gary Ruvkun étudiaient comment les gènes Lin-4 et Lin-14P
1
P
régulaient le développement du nématode
Caenorhabditis elegans et ceci au sein du prestigieux Massachusetts Institute of Technology.
Ils voulaient comprendre d’une part pourquoi les mutations de Lin-4 empêchaient la larve de
C.elegans de se transformer en un ver adulte complètement formé et d’autre part, pourquoi
celles qui touchent Lin-14 provoquaient une maturation précoce de la larve (Ambros and Horvitz, 1984). Ambros a fini par montrer en 1989 que Lin-4 agit comme un répresseur de l’activité de 14 (Ambros, 1989). Cependant, le mécanisme par lequel 4 réprime
Lin-14 n’avait pas été élucidé. Ce n’est qu’en 1991, que le groupe dirigé par Ruvkun découvre
que les mutations touchant la région γ’ non-traduite (γ’ untranslated region : γ’UTR) du gène de Lin-14 entraînaient un excès de production de la protéine Lin-14 dont l’ARN messager (ARNm) est ciblé par Lin-4 (Wightman et al., 1991). Deux années plus tard, l’équipe d’Ambros réussit à cloner Lin-4 et découvre que le gène Lin-4 ne contient aucune phase ouverte de lecture et que son produit final n’est pas une protéine standard mais deux petites molécules d’ARN non codantes dont la taille est de 22 et 61 nucléotides (nt) (Lee et al., 1993). De plus, ils ont également montré que la région γ’UTR (γ’ UnTranslated Region) de
Lin-14 possède des séquences avec une complémentarité partielle avec celle de Lin-4 leur
permettant ainsi de conclure que Lin-4 était responsable de l’inhibition de Lin-14 (Lee et al., 1993). Depuis, ces découvertes ont valu à Ambros et Ruvkun l’obtention de nombreuses distinctions dans le monde de la recherche scientifique (récemment Ambros a reçu le prix « Breakthrough Prize 2015 »). L’idée que des petites molécules d’ARN peuvent réguler l’expression génique au sein d’autres organismes n’a pas été immédiatement étudiée. En 1999, David Baulcombe publie une étude décrivant l’implication d’une classe d’ARN dans des processus de répression affectant les virus, les transposons et l’expression génique chez les plantes (Baulcombe, 1999). Cette dernière a été suivie par la découverte du miARN let-7
1
Dans ce manuscrit nous avons choisi d’annoter les protéines en lettres majuscule et les gènes/transcrits en lettres majuscules et italiques. Toutefois lorsqu'une espèce donnée est mentionnée nous adopterons la nomenclature et les symboles officiels HGNC (HUGO Gene Nomenclature Committee) pour l’homme : symbole des gènes ou des transcrit en majuscules et en italiques et le symbole des protéines en majuscules. Pour les murins, selon MGI : Les gènes ou les transcrits seront en minuscules et en italiques et les protéines en minuscules sauf la première lettre. Pour les autres espèces nous suivrons la même nomenclature que pour les murins.
16 par le groupe de Ruvkun chez C. elegans, qui est conservé au cours de l’évolution chez plusieurs espèces y compris chez l’homme (Reinhart et al., 2000). La découverte du mécanisme d’action de l’interférence à l’ARN en 1998 par Andrew Fire et Craig Mello a été récompensée par le prix Nobel de Médecine et Physiologie en 2006 (Fire et al., 1998).
Au cours de ces quinze dernières années, plus de 35000 miARN matures ont été mis en évidence chez 223 espèces différentes parmi lesquel β588 miARN chez l’homme (selon miRBase version 21, Juin 2014 ; 23Twww.mirbase.org23T) (Ha and Kim, 2014).
III- Caractéristiques et nomenclature des micro-ARN
Les miARN ont été donc définis de manière générale comme étant des petits ARN non codants dont la taille varie entre 21 à 24 nt. Ils jouent un rôle de régulateurs post-transcriptionnels en utilisant leur complémentarité de séquence avec l’ARNm cible afin d’induire sa répression (Pasquinelli, 2012).
La nomenclature des miARN ne suit pas une règle particulière (Ha and Kim, 2014); les miARN trouvés lors d’études développementales ont porté un nom lié aux phénotypes auxquels ils sont associés (lin-4, let-7a et lsy-6 chez C.elegans). En revanche, la plupart des miARN identifiés par clonage ou séquençage ont reçu une nomenclature numérique (par exemple : l’homologue de lin-4 chez d’autres espèces est le miR-125). Les gènes codants pour des miARN partageant une grande identité de séquence sont appelés miARN “soeurs” (“miRNA sisters”) et sont indiqués par des lettres suffixes comme miR-125a et miR-125b (Ha and Kim, 2014; Wahdan-Alaswad and Liu, 2013). Si des miARN matures partagent la même séquence mais sont issus de loci différents, on les distingue alors par l’ajout d’un chiffre en suffixe tels que miR-125b-1 et miR-125b-2 (Ha and Kim, 2014). Enfin, le précurseur du miARN qui donne naissance au miARN mature peut générer deux formes différentes selon qu’elle soit issue de l’extrémité 5’ (miR-125a-5p) ou de l’extrémité γ’ (miR-125a-3p). Toutefois, un seul brin est souvent sélectionné comme étant le brin fonctionnel, appelé aussi le brin guide comparé au brin restant désigné comme brin passager par une étoile (miARN*). Une famille de miARN regroupe l’ensemble des miARN (famille des miARN let-7) possèdant la même séquence entre le nt 2 au nt 7. Cette séquence représente la région « seed » nécessaire à la liaison du miARN à l’ARNm cible (Ha and Kim, 2014).
17
IV- Biogenèse des micro-ARN
IV-1- Arrangement génomique des micro-ARN
Certains miARN sont intégrés dans un gène codant pour une protéine ou dans des transcrits de longs ARN non codants au niveau d’un exon (miARN exoniques) ou d’un intron (miARN introniques) (Figure 2-A et 2-B). D’autres miARN sont issus de gènes ayant leurs propres promoteurs et éléments régulateurs (miARN géniques) (Figure 2-C). Souvent, de multiples miARN sont arrangés en cluster exonique ou intronique dans le génome entraînant la formation d’un unique transcrit qui sera clivé en plusieurs miARN fonctionnels (Figure 2-D et 2-E). Enfin, des miARN peuvent être générés à partir d’introns épissés, à l’origine de leur nomenclature « mirtrons » (Figure 2-F) (Finnegan and Pasquinelli, 2013; Treiber et al., 2012).
Figure 2 : Différentes localisations génomiques du micro-ARN
Les miARN peuvent être localisés dans un exon (A) ou un intron (B) de transcrits codants ou non-codants. Certains gènes de miARN peuvent être intergéniques, produits sans épissage (C). Les miARN groupés en cluster peuvent se situer au niveau intronique (D) ou exonique (E) d’un transcrit codant ou non-codant. Les miARN issus d’intron par épissage (sans l’intervention du microprocesseur) sont appelés mirtrons (F). La couleur violette représente les régions de l’ADN non transcrites, le vert représente les exons espacés par des introns. La flèche indique le site de début de la transcription.
(Schéma adapté de Finnegan and Pasquinelli, 2013).
A. miARN exonique
B. miARN intronique
C. miARN gènique
D. Cluster de miARN intronique
E. Cluster de miARN exonique
18
IV-2-Transcription en micro-ARN primaires
La majorité des miARN sont transcrits par l’ARN polymérase II (ARN Pol II) à partir du transcrit du gène hôte ou à partir d’une unité transcriptionnelle indépendante. L’ARN naissant, appelé miARN primaire (pri-miARN) reçoit alors une coiffe méthylée en 5’ (5′ 7-methylguanosine, m7G) et une queue polyadénylée en γ’ similaires à d’autres transcrits de l’ARN Pol II (Finnegan and Pasquinelli, 2013; Sarnow et al., 2006). Plus rarement, lorsque la séquence du miARN est proche de répétition Alu, la transcription par l’ARN polymérase III (ARN Pol III) est réalisée (Borchert et al., 2006).
La première étape de la biogenèse des miARN est la formation d’une longue molécule d’ARN : pri-miARN qui contient une structure en tige-boucle (stem-loop) (Figure 3)2. Un pri-miARN typique est formé donc d’une région double brin (tige) longue de 33-35 nt présentant des mésappariements, de bras d’ARN simples brins de part et d’autre de la structure en tige double brin et d’une boucle terminale simple brin (Figure 4-A) (Ha and Kim, 2014).
2
Nous avons choisi de représenter la biogenèse des miARN de manière complète avec une vue d'ensemble dans la figure 3. Cette figure est détaillée sur plusieurs chapitres (I-4-2 jusqu'à I-4-7).
20
IV-3-Clivage des ARN primaires en précurseurs
micro-ARN
Le pri-miARN subit plusieurs étapes de maturation pour générer un miARN mature. Il est clivé une première fois par le microprocesseur pour former le précurseur miARN aussi noté pre-miARN (Figure 3). Le microprocesseur, présent dans le noyau, est un complexe protéique formé par l’enzyme RNase III : DROSHA et la protéine DGCR8 (DiGeorge syndrome Critical Region gene 8, aussi appelé PASHA) (Figure 3-A). La protéine DGCR8 reconnait, grâce à ses domaines de liaison à l’ARN double brin (dsRBD, double-stranded RNA binding domain) la tige précisément à la jonction simple brin/double brin. Cette reconnaissance facilite la liaison de DROSHA, grâce à son dsRBD, à la tige du pri-miARN (Figure 4-B). Cette fixation se fait de telle sorte que son centre catalytique constitué des deux domaines RNase IIIa et IIIb (RIIIDa et b), se positionne et clive le duplex d’ARN à 11 paires de bases (pb) après la jonction (Figure 4-A et 4-B) (Han et al., 2006; Kim et al., 2009b). La protéine DGCR8 a été décrite comme essentielle dans le processus de clivage par DROSHA car elle mesure la distance entre les fragments simples brins et ceux doubles brins pour la détermination du site de clivage. Elle serait aussi requise pour le déroulement de la structure en hélice de la loupe du pri-miARN, afin de rendre accessible le site de clivage de DROSHA
Figure 3 : Biogenèse des microARN
La voie de biogenèse standard d’un miARN débute par la transcription du miARN primaire (pri-miARN) contenant une coiffe en 5’ (7-methylguanosine, m7G) et une queue poly(A) en γ’. Le pri-miARN, de structure en tige-boucle, est clivé dans le noyau par l’endonucléase DROSHA avec l’aide de la protéine de liaison double brin DGCR8. Après clivage, le miARN précurseur (pre-miARN), dépourvu de ses extrémités simples brins est exporté du noyau vers le cytoplasme via l’Exportin 5. Le clivage cytoplasmique par l’endonucléase DICER aidée par son partenaire TRBP (transactivation-response RNA-Binding Protein ; Loqs, chez la Drosophile) libère un duplex miARN-miARN*. Les protéines AGO, chaperonnées par le complexe HSC70-HSP90, prennent en charge le duplex miARN-miARN* (pre-RISC : pre-RNA-Induced Silencing Complex) pour la sélection du miARN mature (RISC mature ou miRISC). Le miRISC cible différentes régions de l’ARNm cible. La voie alternative de biogenèse (à droite du schéma) concerne les mirtrons qui sont des fragments épissés à partir de transcrits. Dans le noyau, les mirtrons sont déroulés puis clivés par des nucléases (autrement que par le microprocesseur) pour générer un pre-miARN qui est transloqué vers le cytoplasme via l’EXPORTIN 5. Une voie alternative est observée pour le pre-miR-451 qui échappe au clivage par DICER dans le cytoplasme et qui est directement pris en charge par une protéine AGO se chargeant de sa maturation en un miARN simple brin.
21 (Han et al., 2006). Cette étape donne lieu à une petite molécule d’ARN d’environ 65 nt toujours sous forme de tige-boucle qui représente le pre-miARN (Figures 3 et 4) (Ameres and Zamore, 2013; Ha and Kim, 2014; Han et al., 2006; Kim et al., 2009b).
A
B
Figure 4 : Structures de miARN primaire et du miARN précurseur
A. Schéma du pri-miARN avec les sites clés de reconnaissance et de clivage par le
microprocesseur. Le complexe microprocesseur (DROSHA et DGCR8) reconnait la queue simple brin, la tige d’environ γ5nt et la boucle terminale du pri-miARN. Il reconnait une séquence située entre les onze derniers nucléotides de la tige et la partie supérieure du miARN primaire qui sera clivée par DROSHA. L’enzyme DROSHA est formée par deux domaines catalytiques RNase III (RIIID) en tandem qui dimérisent pour former un centre de clivage ou de processing pour incorporer la structure double brin du miARN primaire. Un domaine se charge de cliver le brin en γ’ alors que le deuxième est spécifique du brin en 5’.
B. Les structures primaires des protéines humaines DROSHA, DGCR8 et de DICER mettant
en évidence leurs différents domaines. C. Schéma de mise en évidence des sites importants du pre-miARN reconnu par DICER. Le domaine PAZ de DICER se fixe aux extrémités γ’ et 5’ du pre-miARN. La tige est contenue le long de la protéine de telle sorte que les deux domaines catalytiques RIIIDa et b clivent 22 nt après les extrémités du pre-miARN.
Illustrations issues de (Ha and Kim, 2014; Kim et al., 2009). A
B
22
IV-3-1- Régulation de l’activité du microprocesseur
L’activité du microprocesseur peut être modulée par plusieurs facteurs. En effet, il a été montré que des facteurs (environ 20 protéines différentes) (Gregory et al., 2004) peuvent s’associer à DROSHA/DGCR8 et réguler positivement ou négativement la maturation des pri-miARN selon la combinaison des complexes protéiques formés (Figure 5).
Ainsi, les ARN hélicases p68 et p72 (aussi appelées, respectivement DDX5 et DDX17, DEAD-box) co-précipitent avec DROSHA et ont été montrées comme importantes dans le processus de sélection et de maturation de certains pri-miARNs spécifiques (Fukuda et al., 2007)3. Ces deux hélicase p68 et p72 peuvent aussi fonctionner comme des intermédiaires pour l’association d’autres protéines régulatrices du microprocesseur telles que les protéines p53 et SMAD (Finnegan and Pasquinelli, 2013). La protéine p53 est à la fois un facteur de transcription mais aussi une protéine majeure dans le processus de suppression de tumeur induite en réponse à un dommage à l’ADN (Suzuki et al., 2009). Afin de contrecarrer le stress que subit la cellule, p53 peut aussi agir sur l’expression des miARN au niveau transcriptionnel mais aussi au niveau post-transcriptionnel en se liant au microprocesseur afin de favoriser la maturation de certains miARN tels que miR-16-1, miR-143 et miR-145 (Corney et al., 2007; Suzuki et al., 2009).
Les protéines SMAD, faisant partie de la cascade de signalisation du TGF-(Transforming Growth Factor ), peuvent aussi réguler la biogenèse des miARN de manière dépendante de p68. Ces protéines se lient grâce à des éléments de liaison à l’ARN (R-SBE, RNA Smad-Binding Element) aux tiges de certains pri-miARN régulés par la voie du TGF- . Ceci permet donc le recrutement du microprocesseur, y compris p68, vers certains pri-miARN pour favoriser spécifiquement leur maturation (Davis et al., 2008, 2010).
La régulation de la biogenèse des miARN a été aussi considérée comme étant une voie supplémentaire de lutte contre la cancérisation de la cellule. En effet, BRCA1 (BReast Cancer 1 protein), connue comme facteur suppresseur de tumeur, promouvoit la biogenèse des miARN en s’associant avec le complexe microprocesseur. Parmi les miARN induits par cette voie nous pouvons citer let-7, miR-16, miR-145 et miR34a (Kawai and Amano, 2012) qui ont été donc considérés comme étant des miARN suppresseurs de tumeurs. Ces données ont montré que BRCA1 régule la biogenèse des miARN en formant un large complexe avec Smadγ/p5γ/p68/DHX9 pour favoriser positivement l’activité de DROSHA/DGCR8 sur
3
Cet article a été retiré du journal Nat. Cell Biol en β014 en raison d’une manipulation des figures, les éditeurs réclament tout de même la validité des résultats et des conclusions présentés par les auteurs de l’article (Fukuda et al., 2014).
23 certains miARNs renforçant ainsi les mécanismes de lutte de la cellule contre le stress (Kawai and Amano, 2012).
Un exemple particulier de protéines régulatrices associées au microprocesseur est celui de la protéine de liaison à l’ARN hnRNPA1 (heterogeneous nuclear RiboNucleoProtein A1). Sa particularité découle de son double rôle dans la régulation de la biogenèse à l’étape de pri-miARN. HnRNPA1 se lie directement et spécifiquement au pri-miARN membre de la famille miR-17-92, à savoir le miR-18a, afin d’induire une modification structurale de ce dernier qui favorise le clivage par DROSHA (Michlewski et al., 2008). Contrairement à l’activité de hnRNPA1 sur le miR-18a, il a été montré par le même groupe deux années plus tard que cette protéine peut aussi fixer le pri-let-7a-1. Cependant, son effet est opposé au précédent car elle entraîne le blocage du clivage par DROSHA médié par KSRP (KH-type Splicing Regulatory Protein) (Michlewski and Cáceres, 2010). En effet, la protéine de régulation de l’épissage KSRP se lie préférentiellement à une région riche en G au niveau de la boucle terminale de certains pri-miARN (tel que le pri-let-7a) et stabilise leur interaction avec le microprocesseur, particulièrement DROSHA (Trabucchi et al., 2009). De plus, suite au clivage par DROSHA, KSRP resterait liée à la boucle du pre-miARN afin de faciliter l’étape suivante du processus de biogenèse des miARN qui est effectuée dans le cytoplasme par la protéine DICER (Trabucchi et al., 2009). Notre équipe a montré qu’au cours de l’organogenèse, une boucle de régulation négative se crée entre let-7b/c et KSRP. Cette boucle de régulation va induire la maturation de let-7b/c qui en retour réprime KSRP en ciblant sa région γ’UTR. De manière générale, ce mécanisme servirait à contrôler la prolifération cellulaire en régulant la maturation de let-7 mais aussi à contrôler la différenciation cellulaire lors de l’organogenèse (Repetto et al., 2012).
Les dommages à l’ADN peuvent aussi affecter la maturation des pri-miARN toujours
via KSRP. La phosphorylation de KSRP par la kinase ATM (Ataxia-Telangiectasia Mutated),
facteur essentiel de la voie de réponse aux dommages à l’ADN, favorise son interaction avec les pri-miARN et par conséquent l’augmentation de son activité sur la maturation. Cette étude a révélé pour la première fois le lien qui existe entre les dommages à l’ADN et la voie de biogenèse des miARN (Zhang et al., 2011).
Comme KSRP, la protéine TDP-43 (Tar DNA-Binding Protein-43) intervient dans plusieurs étapes de maturation du miARN (par exemple : miR-132-3p et miR-212) (Magill et al., 2010). Tout d’abord dans le noyau, TDP-43 se lie à la fois à DROSHA et à la boucle d’une sous-population de pri-miARN. Ensuite dans le cytoplasme, TDP-43 reste liée aux
pre-24 miARN pour faciliter le clivage par DICER (étape suivante de la maturation des miARN) (Kawahara and Mieda-Sato, 2012).
Cette première étape de maturation des miARN est aussi sujette à une régulation négative à travers la liaison de facteurs au pri-miARN afin de prévenir le clivage par le microprocesseur (Figure 5). La protéine de liaison à l’ARN LIN-28 est un parfait exemple de ce blocage. LIN-28 se fixe au pri-let-7 afin de bloquer l’accès de DROSHA à son site de clivage (Newman et al., 2008). Il existe une boucle régulatrice entre let-7 et LIN-28 qui représente sa cible directe. L’expression de LIN-28 est restreinte aux phases précoces du développement. En effet, elle est importante pour le maintien de l’état indifférencié des cellules souches. Par ailleurs, let-7 est requis pour la différenciation cellulaire (Roush and Slack, 2008) et sa forme mature est indétectable dans les cellules souches à cause du blocage exercé par LIN-28. (Roush and Slack, 2008). Au cours de la différenciation des cellules, la diminution du niveau d’expression de la protéine LIN-28 entraîne la libération du pri-let-7 lié par KSRP pour finir son processus de maturation (Newman et al., 2008; Trabucchi et al., 2009, 2010). Chez les mammifères, il existe deux paralogues de LIN-28 : LIN-28A (aussi nommée LIN-28) et LIN-28B. La forme LIN-28B est nucléaire et se lie au pri-let-7 afin de le séquestrer dans le nucléole bloquant ainsi l’accès du microprocesseur au pri-let-7. Par contre, LIN-28A est essentiellement localisée dans le cytoplasme où elle inhibe la deuxième étape de maturation des miARN (Piskounova et al., 2011).
Il a été rapporté que le complexe nucléaire formé par NF90-NF45 (Nuclear Factor 90 ou 45) joue aussi un rôle inhibiteur sur la maturation du pri-miARN en pre-miARN. En se liant à la structure double brin du pri-miR-21, NF90 et NF45 inhibent in vitro son clivage en bloquant l’accès du microprocesseur. In vivo, ce complexe présente une meilleure affinité vis-à-vis du pri-let-7a que le pri-miR-21 (Sakamoto et al., 2009).
25
IV-3-2- Modèles murins d'étude de la fonction de Dgcr8 et Drosha
L’invalidation de Dgcr8 (Knock-Out, KO) chez la souris provoque une mort embryonnaire au stade E6.5 par apoptose, indiquant un rôle essentiel des miARN dans le développement embryonnaire précoce (Wang et al., 2007). L’importance de Dgcr8 pour l’auto-renouvellement des cellules souches a été montrée par l’utilisation de cellules souches embryonnaires (ES) murines invalidées pour Dgcr8. Sur le plan moléculaire, ces cellules présentent une perte massive de pre-miARN et de miARN matures alors que la forme primaire des miARN ne semble pas être affectée, confirmant le rôle primordial de Dgcr8 dans la maturation des pri-miARN. Phénotypiquement, l’altération de la différenciation en absence de la protéine Dgcr8 montre que les miARN sont impliqués dans le développement embryonnaire (Wang et al., 2007).
Le même phénotype de létalité a été observé aussi chez les souris KO pour Drosha (Park et al., 2010; Wu et al., 2012). Afin de contrecarrer la létalité précoce associée au souris KO et d’étudier sa fonction dans différents tissus, plusieurs groupes ont crée des modèles murins
DGCR8 DROSHA hnRNPA1 p72 SMAD p72 p68 p53 p68 p53 SMAD DHX9 BRCA1 TDP43 KSRP Pri-miARN Pre-miARN hnRNPA1 LIN28 NF90 NF45
Figure 5 : Les différents facteurs régulateurs de l’activité du microprocesseur
Régulation de l’activité du microprocesseur (DROSHA/DGCR8). A gauche de ligne verticale sont représentés les facteurs ayant un effet positif sur le processus de maturation. A droite de la flèche verticale figurent les facteurs inhibiteurs de l’activité du microprocesseur.
(Schéma adapté de Finnegan and Pasquinelli, 2013).
26 conditionnels inductibles grâce au système Cre-Loxp. Par exemple, le KO conditionnel de
Drosha a été réalisé spécifiquement dans les spermatogonies par la stratégie Cre-loxp (Wu et
al., 2012). Dans ce cas, l’absence de la protéine Drosha diminue drastiquement de 80% l'expression des miARN totaux dans ces cellules par rapport aux cellules normales. La morphologie des testicules déficients pour Drosha montre une taille réduite de 50% à l'âge de 6 semaines par rapport aux testicules de souris de phénotype sauvage (WT, Wild Type). De même, sur le plan histologique, les testicules KO présentent (i) une déplétion sévère en cellules germinales, (ii) une altération de l'épithélium séminifère contenant peu ou pas de spermatides, (iii) de nombreuses vacuoles et des cellules multinuclées géantes indiquant un défaut/arrêt de la spermatogenèse et (iv) un épididyme dépourvu de spermatozoïdes. Ces altérations entraînent l’infertilité des mâles due à la déficience de la spermatogenèse (Wu et al., 2012). D’autre part, il a été montré que le KO conditionnel de Drosha dans les lymphocytes T entraîne une forte diminution du nombre de cellules T (CD4+/CD8+) à l'âge de 6 semaines comparées à des souris WT du même âge (Chong et al., 2008). Des souris déficientes âgées de 4 mois présentent des symptômes inflammatoires spontanés caractérisés par une forte sécrétion d'IFN (InterFeron ) et d'Interleukine 17A (IL-17A) dans la rate et dans les ganglions lymphatiques accompagnés d'une réduction de la masse musculaire et du tissu adipeux viscéral. Par ailleurs, un taux de mortalité de 50 % a été observé chez ces souris déficientes âgées de 6 mois (Chong et al., 2008).
Ces exemples d’études in vivo démontrent l’importance de ces facteurs du microprocesseur: Dgcr8 et Drosha pour la maturation de miARN et in fine pour le maintien des fonctions vitales.
IV-3-3- Maturation des mirtrons – processus indépendant du microprocesseur
La formation des miARN contenus dans les introns ne nécessite pas l’activité du microprocesseur mais dépend plutôt des événements d’épissage de leur gène hôte (Figure 2, voie alternative de biogenèse) (Ruby et al., 2007). Suite à l’épissage, un débranchement de la forme lasso de l’intron se produit pour faire apparaître une structure en tige-boucle de type pre-miARN qui rejoint la voie canonique de maturation du miARN (Okamura et al., 2007; Ruby et al., 2007). Cependant, une étude a montré que tous les miARN introniques ne sont pas dépendants de l’épissage du gène hôte. Elle montre ainsi que la biogenèse des miARN introniques miR-1225 et miR-1228, présents chez l’homme, est indépendante de l’épissage du transcrit hôte et de la participation de DGCR8 (Havens et al., 2012). En revanche, ces miARN
27 nommés simtrons (pour Splicing Independant mirtrons) nécessitent DROSHA pour générer le précurseur. Cette étude a donc mis en évidence pour la première fois l’importance de DROSHA dans la biogenèse des mirtrons sans que DGCR8 ne soit impliquée (Havens et al., 2012).
IV-4-Export nucléaire des pre-micro-ARN
Le pre-miARN doit à son tour subir une maturation afin de produire le miARN fonctionnel. Pour cela, il doit être transloqué vers le cytoplasme où a lieu la deuxième étape de clivage (Figure 3). Le transport des pre-miARN se fait grâce la protéine EXPORTIN 5 (EXP5, codé par le gène XPO5) couplée au cofacteur nucléaire RAN-GTP (RAs-related Nuclear protein – Guanosine TriPhosphate). L’EXP5 s’associe à la région double brin du précurseur en reconnaissant la structure double brin ainsi que le bras simple brin en γ’ du précurseur (long de 1-8 nt) (Zeng and Cullen, 2004). Le guidage du pre-miARN vers le cytoplasme se fait à travers les pores nucléaires. Le complexe formé par EXP5/RAN-GTP/pre-miARN se dissocie suite à l’hydrolyse de la molécule de GTP en GDP (Guanosine DiPhosphate) libérant ainsi le pre-miARN pour la suite du processus de maturation (Ha and Kim, 2014; Zeng and Cullen, 2004).
IV-5-Clivage des pre-micro-ARN en micro-ARN matures
Une fois dans le cytoplasme, le pre-miARN doit subir un deuxième clivage par une enzyme RNase III cytoplasmique appelée DICER, pour générer la forme mature du miARN dont la taille est comprise entre 21-24 nt. L’enzyme DICER se caractérise par la présence d’un domaine hélicase, d’un domaine PAZ (PIWI-Argonaute-ZWILLE), d’un domaine de liaison à l’ARN double brin et de deux domaines RIIIDa et b (Figure 4-B). Le domaine PAZ sert à reconnaitre le petit bras de 2 nt à la base de la tige du pre-miARN alors que les deux domaines RIIID se dimérisent et forment un centre catalytique similaire à celui de DROSHA, qui entoure la tige du pre-miARN. Quant au domaine hélicase, il facilite la reconnaissance du miARN en interagissant avec la boucle terminale (Kim et al., 2009b). Une fois que le pre-miARN est proprement chargé dans la protéine DICER, chaque domaine RIIID coupe un des deux brins de la tige libérant ainsi la boucle du reste du précurseur qui devient donc une molécule d’ARN double brin avec des extrémités γ’ sortantes de β nt de chaque côté (Figure 4-C) (Finnegan and Pasquinelli, 2013; Ha and Kim, 2014; MacRae et al., 2007). Cette forme appelée aussi duplex miARN de taille comprise entre 21-24 pb, est composée d’un brin
28 miARN « guide » ou mature et d’un brin « passager » ou miARN* (Finnegan and Pasquinelli, 2013).
Il existe une voie de maturation indépendante de DICER particulière au miR-451. Ce processus a été identifié chez les souris exprimant une protéine Ago2 catalytiquement inactive (mutée au niveau de son domaine PIWI). Ces souris meurent rapidement après la naissance. La mort précoce des souriceaux est accompagnée d’une forte anémie due à la perte du miR-451 qui est important pour le bon déroulement de l’érythropoïèse (Cheloufi et al., 2010). Par contre, dans le cytoplasme, le miR-451 ne suit pas la voie canonique de maturation car DICER n’intervient pas dans son clivage. En effet, Ago2 a été montré comme étant catalytiquement primordiale pour le clivage du pre-miR-451 en miARN mature grâce à son centre catalytique (Figure 3) (Cheloufi et al., 2010).
IV-5-1-Régulation de l’activité de DICER
La maturation du pre-miARN peut être modulée en affectant le niveau d’expression ainsi que l’activité de la protéine DICER (Figure 4-C). L’ARNm de DICER peut être lui-même ciblé par les miARN. Ainsi trois sites conservés de let-7 sont présents au niveau de la séquence codante de l’ARNm (Forman et al., 2008). L’altération de l’activité de DICER survient lorsque d’autres facteurs se lient à cette protéine. Parmi ces facteurs, la protéine TRBP (HIV-1 TAR RNA-Binding Protein) appelée aussi TARBP2 est une protéine de liaison à l’ARN capable, par interaction directe avec DICER, d’augmenter son efficacité de clivage. Certaines études ont montré que l’interaction entre ces deux protéines stabilise DICER (Chendrimada et al., 2005; Koscianska et al., 2011), alors que d’autres indiquent que l’absence de TRBP induit une diminution des niveaux de miARN mature et/ou des précurseurs miARN (Chakravarthy et al., 2010; Haase et al., 2005).
Une deuxième protéine PACT, aussi appelée Loqs chez la drosophile, capable de se lier à l’ARN double brin, a été décrite comme étant un partenaire direct de DICER facilitant le clivage des pre-miARN. La déplétion de PACT dans des cellules de mammifères entraîne aussi une réduction de l’expression de miARN (Chakravarthy et al., 2010; Haase et al., 2005; Koscianska et al., 2011). Malgré la redondance des rôles de TRBP et PACT, il a été suggéré que les deux protéines aident de manière synergique le double clivage de DICER par chacun de ses deux domaines RIIID (Koscianska et al., 2011).
Récemment, RBM3 a été aussi impliquée dans la stimulation du processus de maturation du pre-miARN. RBM3 se lie aux pre-miARN et facilite l’association de ce dernier avec DICER
29 (Pilotte et al., 2011). Toutefois, RBM3 ne serait importante pour la biogenèse des miARN uniquement en conditions de stress (Finnegan and Pasquinelli, 2013).
D’un autre coté, une répression de l’étape de conversion du pre-miARN en miARN mature peut être due à l’endonucléase Zc3h12a (Zinc-finger CCCH-type containing 12A) qui dégrade le pre-miARN au niveau de la boucle terminale (Suzuki et al., 2011).
Comme pour DROSHA, LIN-28 joue également un rôle dans la régulation de l’activité de DICER lors de la maturation du pre-let-7 en let-7 fonctionnel. La protéine LIN-28 se fixe sur la boucle du pre-miARN et recrute le facteur TUT4/7 (RNA-uridylyltransferase) qui ajoute des uridines (U) à l’extrêmité γ’ du pre-let-7 le rendant susceptibles à une dégradation rapide par l’exonucléase DIS3L2 (Heo et al., 2008, 2009; Piskounova et al., 2011; Ustianenko et al., 2013). La capacité de LIN-28 à réguler plusieurs étapes de la biogenèse de let-7 souligne les rôles opposés de ces deux facteurs : LIN-28 versus let-7. LIN-28 est responsable de la pluripotence cellulaire alors que let-7 est un facteur crucial de la différenciation cellulaire (Viswanathan and Daley, 2010).
D’autre part, il est à noter que l’activité de DICER peut être régulée par une modification post-traductionnelle de la protéine. La phosphorylation de DICER sur deux résidus conservés au niveau de ses domaines de liaison à l’ARN entraîne l’inhibition de la fonction de la protéine aussi bien chez les vers, que chez les souris ou chez l’homme. Par exemple chez C. elegans, il a été montré que Dicer est inhibée lorsqu’elle est phosphorylée par la kinase ERK durant la méiose permettant ainsi d’établir la transition ovocyte-embryon (Drake et al., 2014).
Figure 6 : Les différents facteurs régulateurs de l’activité de DICER
Régulation de l’activité de DICER. A gauche de la ligne verticale sont représentés les facteurs ayant un effet positif sur le processus de maturation. A droite de la flèche verticale figurent les facteurs inhibiteurs de l’activité de DICER
(Schéma adapté de Finnegan and Pasquinelli, 2013). TUT4 DICER Pre-miARN LIN28 PACT TRBP RBM3 MCPIP1 miARN double brin