HAL Id: tel-02178722
https://tel.archives-ouvertes.fr/tel-02178722
Submitted on 10 Jul 2019
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Annotation des ARN longs non-codants chez la poule et
les espèces d’élevage : Focus sur les ARNlnc régulateurs
du métabolisme des lipides
Kevin Muret
To cite this version:
Kevin Muret. Annotation des ARN longs non-codants chez la poule et les espèces d’élevage : Focus sur les ARNlnc régulateurs du métabolisme des lipides. Génétique animale. Agrocampus Ouest, 2018. Français. �NNT : 2018NSARC139�. �tel-02178722�
T
HESE DE DOCTORAT DE
AGROCAMPUS
OUEST
COMUE UNIVERSITE BRETAGNE LOIRE
ECOLE DOCTORALE N° 600
Ecole doctorale Ecologie, Géosciences, Agronomie et Alimentation
Spécialité : Génétique, génomique et bio-informatique
Annotation des ARN longs non-codants
chez la poule et les espèces d’élevage
Focus sur les ARNlnc régulateurs du métabolisme des lipides Thèse présentée et soutenue à Rennes, le jeudi 20 décembre 2018 (AO, Amphi. Moule)
Unité de recherche : Physiologie, environnement, et génétique pour l’animal et les systèmes d’élevage INRA / AGROCAMPUS OUEST – Equipe Génétique et Génomique
Thèse N° : C-139_2018-32
Par
Kévin MURET
Rapporteurs avant soutenance :
Mark COCK
Directeur de recherche, CNRS, UMR LBI2M
Laboratoire de Biologie Intégrative des Modèles Marins
Christophe HITTE
Ingénieur de recherche, CNRS, UMR IGDR Institut de Génétique et Développement de Rennes
Composition du Jury :
Maria MANZANARES-DAULEUX, Présidente
Professeure, AGROCAMPUS OUEST, UMR IGEPP
Mark COCK, Rapporteur
Directeur de recherche, CNRS, UMR LBI2M
Christophe HITTE, Rapporteur
Ingénieur de recherche, CNRS, UMR IGDR
Cédric COULOUARN, Examinateur
Chargé de recherche, INSERM, UMR NuMeCan
Sandrine LAGARRIGUE, Directrice de thèse
i
iii
« Le génome est un recueil de nouvelles
rédigé il y a plusieurs milliards d'années,
vous pourriez l'avoir sur votre table de chevet,
comme les contes des Mille et Une Nuits,
et y lire une histoire différente chaque soir. »
— Eric Lander —
Fondateur et directeur du Broad
Institute of MIT and Harvard
Leader du projet Génome Humain
Ce manuscrit raconte l’une de ces histoires génomiques. Il
n’a pas la prétention de finir sur l’une de vos tables de chevet,
mais il aura, je l’espère, le mérite de titiller votre curiosité
sur ce que sont les ARN longs non-codants …
v
Remerciements
En premier lieu, je tiens à remercier ma directrice de thèse, Sandrine LAGARRIGUE, qui a su, pendant ces 4 années me transmettre sa passion de la génétique. C’était un réel plaisir de travailler avec elle, toujours pleine d’énergie, pleine d’idées et surtout, toujours très encourageante. Les compliments n’ont jamais manqué, et il faut le dire, le moral et la satisfaction du travail accompli passe très souvent par les compliments et les encouragements. Je tiens également à la remercier pour la tolérance qu’elle a pu avoir à mon égard tout au long de ces années. Un grand merci pour tous tes retours et nos échanges sur les travaux, les écrits, les oraux.
Ensuite, mes remerciements iront à l’équipe. Colette DESERT, Morgane BOUTIN, Laetitia LAGOUTTE et Françoise BOISSEL qui ont travaillé sur la validation expérimentale de mes résultats informatiques. J’espère vous laisser de quoi alimenter une belle aventure avec DHCR24-DT qui fût le sujet de nombreuses discussions, réunions et doutes. Dans cette même dynamique, je remercie Frédéric JEHL qui, malgré son début de thèse il y a un an, est présent chez nous depuis toujours : étudiant, stagiaire, bénévole, ingénieur puis thésard. Il a travaillé à chacun de ces postes sur des thématiques liées à ma thèse et je l’en remercie.
Je remercie également les membres de mon comité de thèse : mon tuteur Thomas DERRIEN de l’Institut de Génétique et Développement de Rennes (équipe du chien) qui a pris son rôle très au sérieux ainsi que nos collègues Dominique ROCHA de l’INRA, unité Génétique Animale et Biologie Intégrative (équipe Génétique et Génomique Bovine), Denis TAGU de l’Institut de Génétique, Environnement et Protection des Plantes (équipe Ecologie et Génétique des Insectes) et Yann AUDIC de l’IGDR (équipe Expression Génétique et Développement). Je remercie bien évidemment les membres du jury qui ont bien accepté les missions de rapporteurs et d’examinateurs de mes travaux de recherche : Mark COCK de la Station Biologique de Roscoff (équipe Génétique des Algues), Christophe HITTE de l’IGDR (équipe du chien), rapporteurs de mes travaux ainsi que Cédric COULOUARN de l’unité Nutrition, Métabolismes et Cancer (équipe Signalisation TGFβ, Homéostasie du Glutathion & Thérapies Innovantes dans le Cancer) et Maria MANZANARES-DAULEUX de l’IGEPP (équipe Résistance et Adaptation).
vi Pour l’aspect ambiance et énergie, je remercie Sophie ALLAIS et Frédéric LECERF qui ont animé ces trois années de thèse en musique, en cris ou en bataille de Nerf. Je remercie les autres membres de l’équipe précités ainsi que nos nouveaux collègues du bâtiment 15 qui ont partagés mes repas et mes pauses, mes hauts et mes bas, mon caractère, mon entêtement, mes rires, mes anecdotes improbables et même mon régime puis mes écarts.
vii Je remercie mes financeurs, la Région Bretagne et le département de Génétique Animale de l’INRA qui m’ont permis d’effectuer ces 3 années au sein du laboratoire.
Je remercie nos collègues des chantiers d’abattage, Frédérique PITEL (mon premier contact à l’INRA qui m’a permis d’obtenir un entretien avec Sandrine), Sophie LEROUX et Tatiana ZERJAL avec qui j’ai partagé d’agréables moments de … dissections à Tours.
Un grand merci également à nos collègues du projet FR-AgENCODE et tout particulièrement à Sylvain FOISSAC, Christophe KLOPP et Sarah DJEBALI qui ont suivi mes travaux sur les ARNlnc pendant ces 4 années et avec qui j’ai pu avoir des échanges très intéressants.
Un remerciement à tous les stagiaires que j’ai pu côtoyer et avec qui j’ai travaillé. Victor YTHIER et Nicolas FONTRODONA que j’ai formé et co-encadré avec Sandrine et Julien ROBERT que j’ai co-encadré avec Laetitia.
Un énorme merci à Pint of Science France de m’avoir fait confiance pour l’organisation du festival sur la ville de Rennes ainsi qu’à toute notre équipe d’organisation avec qui nous avons fait un superbe travail sur ces 3 années. Un remerciement particulier à Julien MORICEAU, Sandie CABON, Marie GUIONIE, Manon AUFFRET et Marion GUILLAUMIN qui ont fait partie du noyau dur de l’équipe pour la communication, le management des équipes, la recherche de financements et l’organisation du festival d’une manière générale.
Et enfin je remercie ma famille pour leur soutien moral et financier, ainsi que mes amis avec qui j’ai passé la quasi-totalité de mes week-ends et qui me faisaient oublier le temps de quelques heures que j’étais en doctorat, ainsi que le reste…
ix
Communications sur les travaux de thèse
Articles :
- Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Désert C., Boutin M., Jehl F., Acloque H., Giuffra E., Djebali S., Foissac S., Derrien T., Lagarrigue S. Long noncoding RNA repertoire in chicken liver and adipose tissue. Genetics Selection Evolution. 2017;49. - Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Désert C., Boutin M., Jehl
F., Acloque H., Giuffra E., Djebali S., Foissac S., Derrien T., Lagarrigue S. Une nouvelle facette du génome de poule : les gènes « longs non-codants ». 12ème Journées de la Recherche Avicole.
2017.
- Foissac S., Djebali S., Munyard K., Villa-Vialaneix N., Rau A., Muret K., Esquerré D., Zytnicki M., Derrien T., Bardou P., Blanc F., Cabau C., Crisci E., Dhorne-Pollet S., Drouet F., Gonzales I., Goubil A., Lacroix-Lamandé S., Laurent F., Marthey S., Marti-Marimon M., Momal Leisenring R., Mompart F., Quéré P., Robelin D., San Cristobal M., Tosser-Klopp G., Vincent-Naulleau S., Fabre S., Pinard-Van der Laan M., Klopp C., Tixier-Boichard M., Acloque H., Lagarrigue S., Giuffra E. Livestock genome annotation : transcriptome and chromatin structure profiling in cattle, goat, chicken and pig. Soumis à Genome Biology en juillet 2018.
Communications orales :
- Muret K., Lagarrigue S. Identification chez la poule de gènes régulateurs du métabolisme des lipides par des approches intégrées de (1) cartographie génétique (2) analyse de données d’expression (RNA-seq) – Focus particulier sur les ARN longs non-codants. A la Journée des Doctorants, 19/04/2016, St-Gilles, France.
- Muret K., Lagarrigue S. Long noncoding RNA catalog in liver and adipose tissue in chicken. Au NGS-Barcelona, 04-06/04/2016, Barcelone, Espagne.
- Muret K., Lagarrigue S. Long noncoding RNA repertoires in liver and two T lymphocyte cell types in four livestock species – FAANG pilot project « FR-AgENCODE ». A la 36ème
International Society for Animal Genetics Conference, 16-21/07/2017, Dublin, Irlande.
Posters :
- Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Acloque H., Giuffra E., Foissac S., Djebali S., Derrien T., Lagarrigue S. Long noncoding RNA repertoire in chicken liver and adipose tissue. Au GO-FAANG Workshop, 07-08/10/2015, Washington, USA. (p. 249)
- Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Désert C., Boutin M., Acloque H., Giuffra E., Foissac S., Djebali S., Derrien T., Lagarrigue S. Long noncoding RNA
x
repertoire in chicken liver and adipose tissue. A la Plant & Animal Genome Conference XXIV, 09-13/01/2016, San Diego, USA. (p. 250)
- Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Jehl F., Désert C., Boutin M., Acloque H., Giuffra E., Foissac S., Djebali S., Derrien T., Lagarrigue S. Long noncoding RNA repertoire liver and adipose tissue in chicken. Au 19ème Séminaire des thésards du département de Génétique Animale, 16-17/03/2016, Castanet-Tolosan, France. (p. 251) - Muret K., Klopp C., Wucher V., Esquerré D., Legeai F., Lecerf F., Désert C., Boutin M., Jehl
F., Acloque H., Giuffra E., Djebali S., Foissac S., Derrien T., Lagarrigue S. Une nouvelle facette du génome de poule : les gènes « longs non-codants ». Aux 12ème Journées de la Recherche
Avicole, 05-06/04/207, Tours, France. (p. 252)
- Muret K., Djebali S., Derrien T., Cabau C., Klopp C., Esquerré D., Munyard K., Tosser-Klopp G., Acloque H., Giuffra E., Foissac S., Lagarrigue S. Long noncoding RNA repertoires in liver and two T lymphocyte cell types in four livestock species – FAANG pilot project « FR-AgENCODE ». A la 36ème International Society for Animal Genetics Conference,
16-21/07/2017, Dublin, Irelande.(p. 253)
- Muret K., Klopp C., Esquerré D., Boutin M., Désert C., Lagarrigue S. RNA-seq data of liver and adipose tissue highlight lncRNAs as candidates for the lipid metabolism regulation in broilers. A la Plant & Animal Genome Conference XXVI, 13-17/01/2018, San Diego, USA. (p. 254)
Clip vidéo vulgarisé :
- Muret K., Jehl F., Boutin M., Herry F., Désert C., Lagoutte L., Robert J., Allais S., Lecerf F., Lagarrigue S. La Thèse de Kévinou. A la Journée des Doctorants, 16/04/2018, St-Gilles, France.
xi
Table des matières
Remerciements ... v
Communications sur les travaux de thèse ... ix
Table des matières ... xi
Liste des figures ... xvii
Liste des tableaux ... xxiii
Introduction ... 1
1 – Contexte et objectifs de la thèse ... 2
1.1 – Contexte lié au premier objectif de la thèse : annotation des ARN longs non-codants (ARNlnc) chez la poule ... 2
1.1.1 – Des gènes codant des protéines aux ARNlnc... 2
1.1.2 – Premier objectif de ma thèse : annotation des ARNlnc chez les espèces d’élevage dont la poule ... 6
1.2 – Contexte lié au second objectif de la thèse : ARNlnc et lipides ... 8
1.2.1 – Importance des lipides ... 8
1.2.2 – Second objectif de ma thèse : identifier des ARNlnc régulateurs du métabolisme des lipides ... 9
2 – L’espèce poule ... 10
2.1 – De la poule sauvage aux lignées commerciales ... 10
2.2 – Importance économique du poulet de chair ... 12
2.3 – Importance de la poule en recherche ... 13
3 – Les ARN longs non-codants ... 17
3.1 – Le monde des ARNlnc ... 17
3.1.1 – Qu’est-ce qu’un ARNlnc ? ... 17
3.1.2 – Qui du gène codant ou non-codant est arrivé en premier ? ... 18
3.1.3 – Les ARNlnc : entre ARN codant et bruit transcriptionnel ... 19
xii
3.1.5 – Classification des ARNlnc ... 24
3.1.6 – Nomenclature des ARNlnc ... 27
3.2 – Les interactions des ARNlnc avec les autres molécules ... 29
3.2.1 – Interactions ARN-ARN : les doubles hélices d’ARN ... 29
3.2.2 – Les duplex ARN-ADN : cas des boucles ribonucléiques ... 29
3.2.3 – Les triplex nucléotidiques ... 30
3.2.4 – Les multiplex nucléotidiques ... 31
3.2.5 – Interactions ARN-protéines ... 32
3.2.6 – Les ARNlnc, de « super-régulateurs » ... 34
3.3 – Les différents mécanismes d’action des ARNlnc ... 34
3.3.1 – ARNlnc et structure de la cellule ... 34
3.3.2 – ARNlnc, intégrité et structure du génome ... 35
3.3.3 – Rôle des ARNlnc sur la transcription des gènes ... 37
3.3.4 – Rôle des ARNlnc sur la maturation des ARN ... 38
3.3.5 – Rôle des ARNlnc sur la demi-vie des ARN ... 41
3.3.6 – Rôle des ARNlnc sur la traduction ... 42
3.3.7 – Rôle des ARNlnc sur le transport des protéines ... 43
3.3.8 – Rôle des ARNlnc sur la signalisation cellulaire ... 43
3.4 – Les ARNlnc hôtes de petites ORF et de petits ARN ... 44
3.4.1 – Des ARN longs non-codants codants... 44
3.4.2 – Les ARN, de formidables hôtes ... 45
Résultats – Partie I – Annotation des ARNlnc de la poule et des espèces d’élevage ... 47
1 – Annotation des ARNlnc du foie et du tissu adipeux chez la poule ... 48
1.1 – Résumé ... 48
1.2 – Article 1 ... 52
xiii 2 – Annotation des ARNlnc des lymphocytes T (CD4+, CD8+) et du foie chez 4 espèces
d’élevage : le bovin, la chèvre, le porc et la poule ... 79
2.1 – Résumé ... 79
2.2 – Article ... 84
3 – Mise à jour du catalogue d’ARNlnc chez la poule en apportant de nouvelles données RNA-seq et en combinant plusieurs bases de données ... 126
3.1 – Modélisation des ARNlnc à partir de 364 expériences RNA-seq ... 127
3.1.1 – Présentation des deux stratégies de modélisation ... 128
3.1.2 – Variation importante du nombre de transcrits d’une stratégie à l’autre ... 129
3.1.3 – Variation du nombre de transcrits monoexoniques d’une stratégie à l’autre ... 131
3.1.4 – Phénomène de fusion de gènes ... 132
3.1.5 – Bilan des deux stratégies ... 133
3.2 – Vers un fichier d’annotation combinant différentes sources d’ARNlnc ... 135
3.2.1 – ALDB ... 136
3.2.2 – NCBI ... 136
3.2.3 – NONCODE ... 136
3.2.4 – FR-AgENCODE ... 137
3.2.5 – Méthode d’agrégation des bases de données ... 137
3.3 – Fichier d’annotation final .GTF_FINAL ... 138
Résultats – Partie II – ARNlnc et lipides ... 141
1 – Les ARNlnc connus comme étant impliqués dans le métabolisme des lipides... 142
1.1 - Résumé ... 142
1.2 – Article (en cours de soumission) ... 143
2 – Les ARNlnc connus comme étant impliqués dans l’adipogenèse ... 179
2.1 – Introduction ... 179
2.2 – Matériels et méthodes ... 180
Méthode bibliographique ... 180
xiv
Analyse de l’expression des gènes conservés chez la poule et l’Homme ... 180
2.3 – Résultats ... 181
Une analyse bibliographique approfondie sur les ARNlnc permet de doubler le répertoire des ARNlnc impliqués dans l’adipogenèse ... 181
Les mécanismes d’action sont décrits pour ⅓ des gènes longs non-codants ... 185
20% des ARNlnc sont conservés de l’Homme/souris à la poule ... 187
Conservation de ces ARNlnc au sein du règne animal, de l’Homme au poisson-zèbre . 189 2.4 – Discussion et conclusion ... 190
3 – De nouveaux ARNlnc potentiellement régulateurs du métabolisme des lipides ... 193
3.1 – Introduction ... 193
3.2 – Matériels et méthodes ... 194
Préparation des échantillons ... 194
Traitements informatiques des données RNA-seq ... 195
Contrôle des échantillons ... 195
Analyse des expressions différentielles ... 196
Analyse des termes GO enrichis ... 197
Construction des groupes de gènes DE co-localisés ... 197
3.3 – Résultats ... 197
Expression des gènes codant pour des protéines dans les deux tissus : foie et tissu adipeux ... 197
Les gènes codant des protéines différentiellement exprimés entre lignées grasse et maigre dans le foie et le tissu adipeux sont enrichis en gènes cholestérogéniques et lipogéniques ... 198
Les gènes codant des protéines et différentiellement exprimés entre régimes le sont essentiellement dans le foie et sont des gènes cholestérogéniques et lipogéniques ... 200
Expression des gènes longs non-codants dans les deux tissus : foie et tissu adipeux... 201
xv Les ARNlnc sont DE entre régimes uniquement dans le foie comme pour les gènes codants
... 204
Co-localisation dans le genome de gènes différentiellement exprimés ... 206
3.4 – Discussion et conclusion ... 208
Discussion et perspectives ... 211
1 – RNA-seq et annotations ... 212
1.1 – La modélisation des gènes ... 212
1.2 – Annotation fonctionnelle des ARNlnc ... 216
2 – RNA-seq et variants ... 218
2.1 – Recherche de variants causaux ... 218
2.2 – Recherche de cis-régulation : expression allèle spécifique (ASE) ... 221
3 – RNA-seq et lipides ... 222
3.1 – Lipides et synténie ... 222
3.2 – Cas particuliers des ARNlnc DHCR24-DT et FASN-DT ... 223
Bibliographie ... 227
Annexes ... 249
Poster – GO-FAANG Workshop – 7-8 octobre 2015 – Washington ... 249
Poster – International Plant & Animal Genome XXIV – 9-13 janvier 2016 – San Diego . 250 Poster – 19ème Séminaire des thésards – 16-17 mars 2016 – Castanet-Tolosan ... 251
Poster – 12ème journée de la Recherche Avicole et Palmipèdes à Foie Gras – 5-6 avril 2017 – Tours ... 252
Poster – 36th International Society for Animal Genetics Conference – 16-21 juillet 2017 – Dublin ... 253 Poster – International Plant & Animal Genome XXVI – 13-17 janvier 2018 – San Diego254
xvii
Liste des figures
FIGURE 1–RESULTATS SYNTHETISES DE KAPRANOV ET COLLEGUES [6] SUR LEUR EXPERIENCE DE TILING ARRAY SUR LES CHROMOSOMES
21 ET 22.LES CHIFFRES EN BAS FONT REFERENCE AUX CHIFFRES (1),(2) ET (3) DU TEXTE. ... 3
FIGURE 2–ETUDE BIBLIOMETRIQUE DES ARNLNC,ARNMI ET ARNCIRC DE 2007 A 2018. ... 5 FIGURE 3–IMPORTATION DES POULES ASIATIQUES EN EUROPE AU TRAVERS DES CONQUETES ... 11 FIGURE 4–CONSOMMATION FRANÇAISE DE VIANDE.A.TOUTES VIANDES PAR HABITANT ENTRE 1994 ET 2017.LES BOVINS
CORRESPONDENT AUX GROS BOVINS ET AUX VEAUX.LES VOLAILLES CORRESPONDENT AU POULET, A LA DINDE, AU CANARD ET A LA PINTADE. KGEC : KILOGRAMME EQUIVALENT-CARCASSE B. PRODUCTION AVICOLE EN 2017.C.CONSOMMATION DE POULETS ENTIERS, DECOUPES OU TRANSFORMES EN FRANCE ENTRE 2000 ET 2017.DONNEES ISSUES DE FRANCEAGRIMER. ... 13 FIGURE 5–TAILLE ET REPETITIVITE DES GENOMES DES ESPECES EUCARYOTES.LA REPETITIVITE DES GENOMES A ETE EVALUEE PAR LE %
DE 31-MERS NON UNIQUES.FIGURE EXTRAITE DE JIAO ET SCHNEEBERGER [40]. ... 14 FIGURE 6-CARYOTYPE DE L'HOMME (EN HAUT) ET DU POULET (EN BAS).POUR AVOIR UN ORDRE D'IDEE DE LA TAILLE DES
CHROMOSOMES, LE CHROMOSOME 22 HUMAIN FAIT A PEU PRES LA MEME TAILLE QUE LE CHROMOSOME 5 DE LA POULE
(>50MB). ... 15 FIGURE 7–SCHEMA SUR L’EVOLUTION DES GENES.A.PHENOMENES DE DUPLICATION DE GENES, PSEUDOGENEISATION ET NEO
-FONCTIONNALISATION.IL EST A NOTER QUE LES REMANIEMENTS PAR ELEMENTS TRANSPOSABLES, TRANSFERTS DE GENES HORIZONTAUX ET REARRANGEMENTS CHROMOSOMIQUES N’ONT PAS ETE NOTES ICI.INSPIRE DE CASANE ET LAURENTI [67].B. EXEMPLE DE RETRO-TRANSPOSITIONS DE L’ARNCIRC CIRCRFWD2 A 5 EXONS (A, B, C, D ET E) PROCESSE (I.E. MATURE, SANS INTRON) DANS LE GENOME DE LA SOURIS.6 COPIES DE CET ARN MATURE ONT ETE TROUVEES SUR DES CHROMOSOMES DIFFERENTS.INSPIRE DE DONG ET COLLEGUES [68]. ... 19
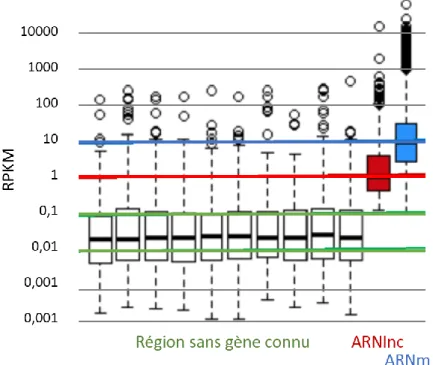
FIGURE 8–EXPRESSION DES ARNLNC ET DES ARNM VS. DE 10 REGIONS SANS GENE CONNU.RPKM :LECTURES NORMALISEES D’UN ECHANTILLON EXPRIMEES PAR KILOBASES ET PAR MILLION DE LECTURES DANS L’ECHANTILLON.FIGURE TRADUITE DE MURET ET COLLEGUES [23]. ... 20 FIGURE 9–EVOLUTION DU NOMBRE DE PUBLICATIONS SELON L’OUTIL DE PREDICTION DES ARNLNC UTILISE.LES RESULTATS POUR
2018 ONT ETE RAMENES A 12 MOIS. ... 23 FIGURE 10–LES DIFFERENTES CLASSES PAR RAPPORT AU GENE CODANT PROCHE ET AUX ELEMENTS DE REGULATION.A.LES ARNLNC
INTERGENIQUES ET GENIQUES.B.LES ARNLNC ASSOCIES AUX PROMOTEURS, ENHANCERS ET EXTREMITES UTR. ... 25
FIGURE 11–CLASSIFICATION SELON LEUR MECANISME D’ACTION ... 26 FIGURE 12-ARBRE DE DECISION POUR NOMMER LES ARNLNC SELON LA NOMENCLATURE DU HGNC.DECISION A PRENDRE
UNIQUEMENT LORSQUE L’ARNLNC N’A PAS DE NOM ILLUSTRANT SA FONCTION.LORSQUE PLUSIEURS CHOIX SONT POSSIBLES DUS A L’EXISTENCE DE PLUSIEURS TRANSCRITS, PRIVILEGIER LES TRACES VERTS AUX ROUGES.* :GENE DEFINI PAR LA
CONCATENATION DE L’ENSEMBLE DES ISOFORMES.** :SI L’ARNLNC CHEVAUCHE PLUSIEURS GENES CODANTS, CHOISIR LE NOM DE GENE CODANT LE PLUS EN 5’ DE L’ARNLNC.PETITE EXCEPTION POUR LES CLUSTERS DE GENES OU GENE PEUT
CORRESPONDRE AU CLUSTER(E.G.HOXD-AS1, L’ARNLNC CHEVAUCHANT PLUSIEURS GENES DU CLUSTER HOXD).SCHEMA DE WRIGHT [87] COMPLETE ET MIS-A-JOUR. ... 28 FIGURE 13–PHOTOGRAPHIE EN MICROSCOPIE ELECTRONIQUE (A GAUCHE) ET SCHEMA ASSOCIE MONTRANT LA FORMATION DE
xviii
CONTACT ENTRE LES DEUX MOLECULES.* : REGIONS DOUBLE BRIN AMBIGUËS.PHOTOGRAPHIE ISSUE DE UMLAUF ET COLLEGUES
[96] ET SCHEMA REVISE. ... 30 FIGURE 14–MODELES DE STRUCTURES DE TRIPLEX ET G-QUADRUPLEX.A. TRIPLEX D’ARN CREANT UN PSEUDO-NŒUD [PDB:437D]
[104]B. TRIPLEX D’ARN(ROUGE) AVEC DE L’ADN(BLEU/BEIGE)[PDB:1D3X][105]C. MODELE DE G-QUADRUPLEX D’ADN[PDB:4U5M][106]D. MODELE DE LA STRUCTURE EN G-QUADRUPLEX DE L’ARNM MYC(ORANGE) RECONNU PAR LA PROTEINE RHAU(HELICASE ; GRIS)[PDB:5VHE][107]. ... 31 FIGURE 15–LA PROTEINE PUMILIO1(GRISE) COMPLEXEE AVEC UN ARN(BLEU)[PDB:1M8Y].LES LIAISONS HYDROGENES SONT
SYMBOLISEES PAR DES POINTILLES.FIGURE EXTRAITE DE AUWELER ET COLLEGUES [108]. ... 33 FIGURE 16–INTERACTION DES ARNLNC NEAT1 ET VIM-AS1 AVEC LEUR CIBLE PROTEIQUE ET ADN.A.L’ARNLNC NEAT1RNA
CO-LOCALISE AVEC LES PROTEINES DU PARASPECKLE DANS LE NUCLEOPLASME DES CELLULES HELA.A GAUCHE, L’ARNLNC
NEAT1(EN VERT) LOCALISE EN PATCH.AU MILIEU, LA PROTEINE PSP1(EN ROUGE), MARQUEUR DES PARASPECKLE ETANT PRESENTE DANS TOUT LE NUCLEOPLASME AVEC QUELQUES ZONES PLUS DENSES EN PROTEINE (FLECHES).A DROITE, LE CHEVAUCHEMENT DES 2 SIGNAUX (JAUNE), CO-LOCALISATION PARFAITE ENTRE LES ZONES DENSES EN PSP1 ET LES PATCHS D’ARNLNC NEAT1 DELIMITANT LES PARASPECKLES NUCLEOPLASMIQUES.PHOTOS ISSUES DE CLEMSON ET COLLEGUES [116]. B. A GAUCHE, ACTIVATION DE CELLULES DE MUSCLE LISSE PAR 5-HT.EN VERT, LA PROTEINE VIM STRUCTUREE EN FILAMENT. PHOTO ISSUE DE LI ET COLLEGUES [117].A DROITE, EN ROUGE, LE PRE-ARNLNC VIM-AS1(EN ROUGE) CO-LOCALISE AVEC LE PRE-ARNM VIM(EN VERT) AU NIVEAU DE LEUR SITE INITIAL DE TRANSCRIPTION (FLECHE).LES PETITS POINTS EN DEHORS DU SITE INITIAL REPRESENTENT LE PRE-ARNM VIM DANS LE NOYAU DE LA CELLULE.PHOTO ISSUE DE BOQUE-SASTRE ET COLLÈGUES
[95]. ... 35 FIGURE 17–ROLE DES ARNLNC TERRA ET NORAD DANS LA PROTECTION DES CHROMOSOMES ET LA REGULATION DU CYCLE
CELLULAIRE.A.L’ARNLNC TERRA CO-LOCALISE AVEC LES EXTREMITES DES CHROMOSOMES.(1)DETECTION DE
G-QUADRUPLEX DE L’ARNLNC TERRA(VERT) EN FIN DE METAPHASE.(2)L’ADN CHROMOSOMIQUE (ROUGE).(3)LA
SUPERPOSITION DES DEUX PHOTOS PERMET D’OBSERVER LA CO-LOCALISATION DES G-QUADRUPLEX D’ARNLNC TERRA SUR LES EXTREMITES DES CHROMOSOMES.PHOTOS ISSUES DE XU ET COLLEGUES [120].B.MODELE D’ACTION DE NORAD SUR LA REPLICATION ET LE CYCLE CELLULAIRE.LA DESTRUCTURATION DU COMPLEXE RIBOPROTEIQUE LORSQUE NORAD N’EST PAS TRANSCRIT INDUIT UNE DEREGULATION DE LA REPLICATION DE L’ADN ET DU CYCLE CELLULAIRE.INSPIRE DE MUNSCHAUER
[121]. ... 36 FIGURE 18–ROLE DES ARNLNC XIST ET FIRRE.A.L’ARNXIST DETECTE PAR RNA-FISH(RNAFLUORESCENCE IN SITU
HYBRIDIZATION) ET LOCALISE SUR LE CHROMOSOME X INACTIVE.PHOTO EXTRAITE DE NG ET COLLEGUES [122].B.L’ARNLNC
FIRRE AGISSANT COMME INTERMEDIAIRE ENTRE LES CHROMOSOMES 2,9,15 ET 17 VIA LA RIBONUCLEOPROTEINE HNRNPU
AFIN DE LES METTRE EN CONTACT.FIGURE EXTRAITE DE MARCHESE ET COLLEGUES [123]. ... 37 FIGURE 19–ROLE DE L’ARNLNC FENDRR EN CIS ET EN TRANS (HAUT) ET ROLE DE L’ARNLNC ANRIL SUR LA TRANSCRIPTION DU LOCUS
INK4A-INK4B EN CIS VIA LE RECRUTEMENT DE PRC1 ET PRC2(BAS). ... 38
FIGURE 20–SCHEMA DES MECANISMES D’ACTION DES ARNLNC SAF,ZEB2OS, UC.283+A ET UC.372.A.ROLE DE SAF SUR L’EXPRESSION DU GENE FAS IMPLIQUE DANS LA SURVIE DES CELLULES TUMORALES.B.ROLE DE ZEB2OS SUR L’EXPRESSION DU GENE ZEB2 IMPLIQUE DANS LA TRANSITION EPITHELIO-MESENCHYMATEUSE.SCHEMAS ADAPTES DE ROMERO-BARRIOS ET COLLEGUES [131].C.ROLE D’UC.283+A ET UC.372 DANS LE PROCESSUS DE MATURATION DES ARNMI PAR CAMOUFLAGE DES SITES DE CLIVAGE DE DROSHA ET DICER. ... 40
xix
FIGURE 21–MODELE D’ACTION DES ARNLNC NRON(A) ET LNC_ASNR(B) DANS LE TRANSPORT PROTEIQUE ET DE L’ARNLNC
LINK-A(C) DANS LA SIGNALISATION TUMORALE. ... 44 FIGURE 22-INTERSECTION ENTRE LES ARNLNC QUE NOUS AVONS IDENTIFIES ET CEUX DES BASES DE DONNEES D'ENSEMBL ET DU NCBI ... 51 FIGURE 23–LES PICS ATAC-SEQ SONT ENRICHIS DANS LES REGIONS TSS PAR RAPPORT AUX TTS.EN BLEU LES GENES CODANT POUR
DES PROTEINES, EN ORANGE LES GENES LONGS NON-CODANTS.L’AXE DES ABSCISSES REPRESENTE LA POSITION DU PIC ATAC-SEQ PAR RAPPORT AU GENE : EN NOIR LES REGIONS AUTOUR DU GENE EN KB ET EN ROUGE LA POSITION DANS LE GENE EN %. . 82 FIGURE 24-SCHEMA DES DEUX STRATEGIES DE MODELISATION DES GENES PAR CUFFLINKS.A.PAR ECHANTILLON (SPE).B.PAR
TISSU (SPT) ... 129 FIGURE 25–IMPACT DU NOMBRE D'ECHANTILLONS REGROUPES DANS LA METHODE SPE SUR LE NOMBRE DE TRANSCRITS TOTAUX.EN
ROUGE, DIFFERENTS NOMBRES D’ECHANTILLONS ANALYSES PARMI LES 364 ECHANTILLONS DE POULETS DE LA PRESENTE ANALYSE ; EN BLEU,4 ET 12 ECHANTILLONS ANALYSES ISSUS DU PROJET FR-AGENCODE[24]. ... 130 FIGURE 26–DONNEES RNA-SEQ SIMULEES VIA FLUXSIMULATOR [195].FLUXSIMULATOR A GENERE DES LECTURES EN COHERENCE
AVEC LES MODELES MAUVES.CUFFLINKS A ENSUITE MODELISE AB INITIO LES MODELES ROUGE (IL REPLACE SIMPLEMENT LES MODELES DE REFERENCE) ET A CREE UN TRANSCRIT ARTEFACTUEL ORANGE. ... 131 FIGURE 27–LA SPT FAVORISE L'APPARITION DE TRANSCRITS MONOEXONIQUES ... 132
FIGURE 28–LA SPE FAVORISE L'APPARITION DE TRANSCRITS FUSIONNANT DES GENES CODANTS CONNUS.VERT : DEUX GENES (1) ET
(2) ET LEURS TRANSCRITS CONNUS DE ENSEMBL.BLEU : TRANSCRITS MODELISES PAR SPE AGREGEANT LES 2 LOCUS GENIQUES DE ENSEMBL EN UN SEUL LOCUS GENIQUE (1+2).MARRON : TRANSCRITS MODELISES PAR SPT CONDUISANT A 3 LOCI
GENIQUES, LES 2 CONNUS DE ENSEMBL ET UN NOUVEAU. ... 133
FIGURE 29–IMPACT DU NOMBRE D'ECHANTILLONS REGROUPES DANS LA METHODE SPE SUR LE POURCENTAGE DE GENES CODANTS CONNUS (ENSEMBLE V86.5) FUSIONNES.EN ROUGE, NOS 364 ECHANTILLONS DE POULETS ; EN BLEU, LES 12 ECHANTILLONS DE POULETS DU PROJET FR-AGENCODE. ... 133 FIGURE 30-INFLUENCE DE LA TAILLE DU GENE SUR L'EXPRESSION.A.FORMULES DE CALCUL DU RPKM, NORMALISATION TRES UTILISEE
PAR LA COMMUNAUTE SCIENTIFIQUE ET SA VARIANTE LE TPM, DE PLUS EN PLUS UTILISE PAR LA COMMUNAUTE SCIENTIFIQUE
(OU LA SOMME DES LECTURES POUR TOUS LES GENES (N) EST EGALEMENT NORMALISEE PAR LA TAILLE DES TRANSCRITS).B. INFLUENCE DE LA TAILLE DU GENE (TAILLEG EN ABSCISSE) SUR SON EXPRESSION EN COMPTAGE DE LECTURE BRUT (LECTUREG EN ORDONNEE) POUR UN TPM DE 0,1 ET DES LIBRAIRIES VARIABLES DE 48M(SANG DES POULES COMMERCIALES) A 132M DE LECTURES (TISSU ADIPEUX DES POULETS DE CHAIR INRA). ... 135 FIGURE 31–CONSERVATION DES ARNLNC AU SEIN DES VERTEBRES.A.ARBRE PHYLOGENETIQUE DES 8 ESPECES ETUDIEES.B.CINQ
EXEMPLES PARMI LES 12ARNLNC CONSERVES ENTRE LA SOURIS ET LA POULE.LES GENES CODANT POUR DES PROTEINES SONT EN BLEU, LES ARNLNC EN JAUNE ET LES ARNMI EN MAUVE. ... 190 FIGURE 32–ANALYSES EN COMPOSANTES PRINCIPALES DES FOIES (A ET B) ET TISSUS ADIPEUX (C).A&C.ACP CORRESPONDANT AUX
16 TISSUS DISPONIBLES.B.ACP DE 15 FOIES SUITE AU RETRAIT DU FOIE EXTREME MIS EN EVIDENCE EN A.EN VARIABLES :
EXPRESSION DES GENES EXPRIMES DANS LE TISSU, EN DONNEES BRUTES LOG-NORMALISEES DES GENES EXPRIMES.UN GENE EST CONSIDERE COMME EXPRIME DANS UN TISSU SI 60% DES ECHANTILLONS ONT UN TPM≥0,1.● :LIGNEE MAIGRE, REGIME HAUT EN LIPIDES ;● : LIGNEE MAIGRE, REGIME BAS EN LIPIDES ;● : LIGNEE GRASSE, REGIME HAUT EN LIPIDES ;● :LIGNEE GRASSE, REGIME BAS EN LIPIDES... 196
xx
FIGURE 33-EXPRESSION DES GENES CODANT POUR DES PROTEINES DANS LE FOIE ET LE TISSU ADIPEUX.A.NOMBRE DE GENES EXPRIMES DANS LES DEUX TISSUS.B.NOMBRE DE GENES DIFFERENTIELLEMENT EXPRIMES ENTRE LES LIGNEES GRASSE ET MAIGRE DANS LES DEUX TISSUS.F :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE FOIE ET SOUS-EXPRIMES DANS LE TISSU ADIPEUX.TA :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE TISSU ADIPEUX ET SOUS-EXPRIMES DANS LE FOIE.C. RATIOS D’EXPRESSION DES GENES DIFFERENTIELLEMENT EXPRIMES DANS LES DEUX TISSUS.● : LES GENES AYANT DES RATIOS D’EXPRESSION SIMILAIRES ;● : LES GENES AYANT DES RATIOS D’EXPRESSION ANTAGONISTES. ... 199 FIGURE 34–NOMBRE DE GENES DIFFERENTIELLEMENT EXPRIMES ENTRE LES REGIMES HAUT ET BAS EN GRAISSES/FIBRES DANS LE FOIE
ET LE TISSU ADIPEUX.F :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE FOIE ET SOUS-EXPRIMES DANS LE TISSU ADIPEUX.TA :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE TISSU ADIPEUX ET SOUS-EXPRIMES DANS LE FOIE.● : LES GENES AYANT DES RATIOS D’EXPRESSION SIMILAIRES ;● : LES GENES AYANT DES RATIOS D’EXPRESSION ANTAGONISTES. ... 200 FIGURE 35–EXPRESSION DES GENES A ARNLNC DANS LE FOIE ET LE TISSU ADIPEUX.A.NOMBRE DE GENES EXPRIMES DANS LES DEUX
TISSUS.B.NOMBRE DE GENES DIFFERENTIELLEMENT EXPRIMES ENTRE LES LIGNEES GRASSE ET MAIGRE DANS LES DEUX TISSUS. F :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE FOIE ET SOUS-EXPRIMES DANS LE TISSU ADIPEUX.TA :GENES SUREXPRIMES DANS LA LIGNEE GRASSE DANS LE TISSU ADIPEUX ET SOUS-EXPRIMES DANS LE FOIE.C.RATIOS D’EXPRESSION DES GENES DIFFERENTIELLEMENT EXPRIMES DANS LES DEUX TISSUS.● : LES GENES AYANT DES RATIOS D’EXPRESSION SIMILAIRES ;● :
LES GENES AYANT DES RATIOS D’EXPRESSION ANTAGONISTES.D.EXPRESSION DES GENES A ARNLNC ET CODANT POUR DES PROTEINES (COULEURS PALES).LES EXPRESSIONS SONT EN LECTURES MOYENNES PAR GENES... 202 FIGURE 36–COUPLES DE GENES « ARNLNC – CODANT PROCHE » DIFFERENTIELLEMENT EXPRIMES (A) CORRELES EN EXPRESSION (B)
ET DIVERGENT PROCHE OU ANTISENS (C).A.COUPLES DE GENES DE ENTRE LIGNEES.B.COUPLES DE GENES DE PRESENTANT DES CORRELATIONS D’EXPRESSION SIGNIFICATIVEMENT ELEVEES (SPEARMAN ; P-VALUE ≤ 5×10-2).C.COUPLES ISSUS DE (B) AYANT UN ARNLNC GENIQUE (G ;*ANTISENS ET SENS INTRONIQUES, ANTISENS EXONIQUES) OU DIVERGENT PROCHE (<2KB) (D).HORIZONTALE HAUTE :SUREXPRIMES DANS LA LIGNEE GRASSE ; HORIZONTALE BASSE : SOUS-EXPRIMES DANS LA LIGNEE GRASSE ; HORIZONTALE MEDIANE : COUPLES DE GENES AYANT DES RATIOS D’EXPRESSION ANTAGONISTES ENTRE LES DEUX GENES.D.EXEMPLES DE COUPLES ISSUS DE (C).PARTIE HAUTE :3 COUPLES DIVERGENTS PROCHES PUIS 2 COUPLES ANTISENS D’INTRON ($) ET 1 COUPLE SENS D’INTRON ($$) ; PARTIE BASSE :6 COUPLES ANTISENS D’EXONS.LES EXPRESSIONS SONT EN
TPM LOG-TRANSFORMES, L’AXE DES ABSCISSES REPRESENTE L’ARNLNC ET LES ORDONNEES LES GENES CODANTS.LES CORRELATIONS SONT INDIQUEES SUR CHAQUE PLOT ET LA P-VALUE ASSOCIEE :** :<1% ;***<0,1%.● : LIGNEE MAIGRE ; ● :LIGNEE GRASSE. ... 203 FIGURE 37–NOMBRE DE GENES DIFFERENTIELLEMENT EXPRIMES ENTRE LES DEUX REGIMES DANS LES DEUX TISSUS. ... 205 FIGURE 38–COUPLES DE GENES « ARNLNC – CODANT PROCHE » DIFFERENTIELLEMENT EXPRIMES (A) CORRELES EN EXPRESSION (B)
ET DIVERGENTS PROCHES OU ANTISENS (C).A.COUPLES DE GENES DE ENTRE REGIMES.B.COUPLES DE GENES DE PRESENTANT DES CORRELATIONS D’EXPRESSION SIGNIFICATIVEMENT ELEVEES (SPEARMAN ; P-VALUE ≤ 5×10-2).C.COUPLES ISSUS DE (B)
AYANT UN ARNLNC GENIQUE (G ;*ANTISENS ET SENS INTRONIQUES, ANTISENS EXONIQUES) OU DIVERGENT PROCHE (<2KB)
(D).HORIZONTALE HAUTE :SUREXPRIMES DANS LA LIGNEE GRASSE ; HORIZONTALE BASSE : SOUS-EXPRIMES DANS LA LIGNEE GRASSE ; HORIZONTALE MEDIANE : COUPLES DE GENES AYANT DES RATIOS D’EXPRESSION ANTAGONISTES ENTRE LES DEUX GENES.D.EXEMPLES DE COUPLES ISSUS DE (C).LES EXPRESSIONS SONT EN TPM LOG-TRANSFORMES, L’AXE DES ABSCISSES REPRESENTE L’ARNLNC ET LES ORDONNEES LES GENES CODANTS.LES CORRELATIONS SONT INDIQUEES SUR CHAQUE PLOT ET LA
xxi
FIGURE 39–GROUPE DE GENES CODANTS CO-LOCALISES ET DE ENTRE LIGNEES DANS LE FOIE.A. SCHEMATISATION DU LOCUS DE 5 GENES SUR LE CHROMOSOME 3(104943000-104962000).LES GENES EN DIVERGENCE SONT DISTANTS DE MOINS DE 1KB. B. CORRELATIONS D’EXPRESSION DES GENES DU LOCUS PAR RAPPORT A ABHD1.C.SCHEMATISATION DU LOCUS DE 4 GENES SUR LE CHROMOSOME 6(16566000-16645000).D.CORRELATIONS D’EXPRESSION DES GENES DU LOCUS PAR RAPPORT A
XLOC_026220.$ :GENES CODANTS OU LONGS NON-CODANTS (¤) NOUVELLEMENT MODELISES.LES GENES VERTS SONT PRIS
EN REFERENCE POUR LES PLOTS.LES CORRELATIONS DE SPEARMAN SONT INDIQUEES AVEC LEUR P-VALUE ASSOCIEE :** :<1% ; *** :<0,1% ;NS : NON SIGNIFICATIF. ... 207 FIGURE 40-CAPTURE D'ECRAN D'IGV MONTRANT LA FUSION DE DEUX GENES OIT3 ET MCU.EN BLEU, LES MODELES DE GENES ; EN
VERT, LES MODELES CONNUS SOUS ENSEMBL ;EN ROUGE, LA STRATEGIE PAR ECHANTILLON GENERANT 2 PROBLEMES
IMPORTANTS : DES MODELES BIEN TROP NOMBREUX ET LA FUSION DES DEUX GENES... 213 FIGURE 41–DEUX EXEMPLES DE POTENTIELLES FUSIONS DE GENES NOUVEAUX.A.&B.LES LOCI POTENTIELS DES GENES SONT
ENTOURES EN ROUGE ET LES TRANSCRITS QUI FORMENT LA FUSION SONT POINTES PAR LA FLECHE ROUGE. ... 214 FIGURE 42-MODELES ENSEMBL DU GENE SCD(DROITE) ET DE SON ARNLNC PRESUME (GAUCHE).LA PREMIERE LIGNE REPRESENTE
LA COUVERTURE EN LECTURES.LA LIGNE DU MILIEU REPRESENTE L’EMPILEMENT DES LECTURES (GRISES) ET LES INTRONS (BLEU CIEL).LA LIGNE DU BAS REPRESENTE LES MODELES SOUS ENSEMBL. ... 215 FIGURE 43–TESTS DE FEELNC AVEC DIFFERENTS JEUX D’ENTRAINEMENT.LES DIFFERENTS GRAPHES SONT NUMEROTES DANS
L’ENCADRE :1.DOUBLE COURBE ROC DE LA PUBLICATION MURET ET COLLEGUES CHEZ LA POULE SOUS GALGAL4 AVEC UNE SENSIBILITE ET SPECIFICITE DES ARNLNC FIXEES A 97% ; ENTRAINEMENT DES ARNM :ARNM D’ENSEMBL V.85 ;
ENTRAINEMENT DES ARNLNC :ARNLNC DE NONCODE.2-5.TESTS LORS DE LA PARUTION DE LA NOUVELLE ANNOTATION D’ENSEMBL V.87 SOUS GALGAL5 CHEZ LA POULE ; ENTRAINEMENT DES ARNM : LES ARNM D’ENSEMBL V.87 ; ENTRAINEMENT DES ARNLNC :ARNLNC D’ENSEMBL V.87(2),ARNLNC DE NONCODE(3), REGIONS INTERGENIQUES (4),ARNM EN MODE
SHUFFLE (5).6.TEST AVEC UN SOUS-SET D’ARNM AVEC UN WIKIGENE NAME ; ENTRAINEMENT DES ARNM : LES ARNM AVEC
WIKIGENE NAME D’ENSEMBL V.87 ; ENTRAINEMENT DES ARNLNC : LES ARNM AVEC WIKIGENE NAME EN MODE SHUFFLE. 7-9.MEME METHODE QUE (6) CHEZ LE BOVIN (7), LE PORC (8) ET LA CHEVRE (9).EN ROUGE, LA SENSIBILITE ET EN BLEU LA SPECIFICITE DE LA METHODE POUR LES ARNLNC. ... 217 FIGURE 44-EFFET DU SNP EN POSITION 17:4978358 SUR LA PROTEINE SLC27A4 D'APRES VEP[295].LE GENE EST REPRESENTE
EN HAUT, LES TRAITS VERTICAUX FONT REFERENCE AUX POSITIONS DES CODONS CODANTS LES SITES IMPORTANTS DE LA PROTEINE.LA PROTEINE EST REPRESENTEE EN DESSOUS, LES SITES IMPORTANTS SONT EN COULEUR.LA FLECHE BLEUE POINTE LE
SNP ETUDIE QUI TRANSFORME UN CODON CODANT L’ARGININE EN CODON STOP.POUR INFORMATION, LE GENE EST LOCALISE SUR LE BRIN NEGATIF. ... 220 FIGURE 45-OBSERVATION DES SNP CHEZ 4 INDIVIDUS.LES POLYMORPHISMES SONT MIS EN EVIDENCE PAR DES COULEURS.
L'ACCOLADE MONTRE LE TRIPLET ETUDIE ICI.LA MUTATION CODON STOP IDENTIFIEE PAR VEP EST LE CHANGEMENT D’UNE GUANINE PAR UNE ADENINE EST INDIQUEE PAR UN ASTERISQUE (*).LA MUTATION QUI RESTITUE UNE PROTEINE COMPLETE EST INDIQUE PAR LE SIGNE ($).LES INDIVIDUS (1) ET (2) SONT ISSUS DES DONNEES RNA-SEQ DE FOIES DE 2 POULETS DE LA LIGNEE
MAIGRE ET (3) ET (4), DE LA LIGNEE GRASSE... 220
FIGURE 46–RESULTATS DES ALIGNEMENTS (PAR BLAST) DE LA SEQUENCE HUMAINE DE DHCR24-DT SUR LES ESPECES SOURIS, PORC,
VACHE, CHEVRE ET CHIEN.DHCR24 EST REPRESENTE PAR L’ENCHAINEMENT DES EXONS ROUGES OU JAUNES.DHCR24-DT HUMAIN EST BLEU ENTOURE DE MAUVE.LES RESULTATS DES ALIGNEMENTS SONT ORANGE ENTOURES DE MAUVE. ... 224
xxiii
Liste des tableaux
TABLEAU 1–ANNOTATIONS D’ENSEMBL V.83(DECEMBRE 2015) DE L’HOMME, DE 3 ESPECES MODELES SOURIS, POISSON-ZEBRE,
DROSOPHILE ET DE 3 ESPECES D’ELEVAGE POULE, BOVIN ET PORC. ... 6
TABLEAU 2 –BIBLIOGRAPHIE COMPLETE CONCERNANT LE ROLE D’EPONGE DE TUG1 ... 42
TABLEAU 3–NOMBRE DE TRANSCRITS DANS LA BASE DE DONNEES DE REFERENCE OU NOUVELLEMENT MODELISES. ... 81 TABLEAU 4-AVANTAGES ET INCONVENIENTS DES DIFFERENTES STRATEGIES DE MODELISATION DES GENES ET TRANSCRITS.* PEU
REPRODUCTIBLE : PEUT ETRE DU BRUIT COMME UNE ISOFORME RARE. ... 134 TABLEAU 5-RECAPITULATIF DU FICHIER D'ANNOTATION FINAL ... 139 TABLEAU 6–LES 58ARNLNC IMPLIQUES DANS L’ADIPOGENESE. ... 182 TABLEAU 7–LE MECANISME D’ACTION DES 19ARNLNC ... 186
1
Introduction
2
1 – Contexte et objectifs de la thèse
1.1 – Contexte lié au premier objectif de la thèse : annotation des ARN longs
non-codants (ARNlnc) chez la poule
1.1.1 – Des gènes codant des protéines aux ARNlnc
Sydney Brenner (Cambridge, UK), François Jacob (Paris, FR) et Matt Meselson (Pasadena, US) publient en 1960 un article dans Nature [1] sur la découverte d’un intermédiaire entre les gènes et la synthèse protéique qu’ils nomment ARN messager. De cette découverte est née la notion de région transcrite. La notion inverse de région “non-transcrite” – et inutile – semble communément usitée à Cambridge au début des années 60 sous le terme de “junk DNA” (échanges entre Dan Graur, professeur à l’université de Houston, et Sydney Brenner1). Susumo
Ohno, à qui on attribue la paternité de l’expression, la popularisa en 1972 dans son article “So much "junk" DNA in our genome” [2]. Malgré ce très contesté terme péjoratif, Sydney Brenner a tenu à nuancer dans les années 90, ce “junk DNA” : “There is the rubbish we keep, which is junk, and the rubbish we throw away, which is garbage”2 [3]. L’anisomorphie, correspondance imparfaite, entre les langues a fait naître des termes souvent inadaptés pour traduire le “junk DNA”, comme le métonyme “ADN poubelle” en français3 [4]. On ne retrouve ainsi
aucune nuance pour cet “ADN poubelle” que l’on devrait plutôt appeler “ADN bric-à-brac4”, illustrant ainsi l’idée d’ensemble hétérogène regroupant résidus de gènes, régions non-codantes ou régions intergéniques dont la fonction n’est actuellement pas identifiée. Cela englobe aussi l’idée que ce bric-à-brac puisse être utile : une fonction insoupçonnée d’un locus donné ou un réarrangement chromosomique néo-fonctionnalisant un locus. Susumo Ohno proposait en 1972 que ces régions d’ADN poubelle (99% du génome) soient des vestiges de gènes pseudogénisés et jouent un rôle d’écarteur génique ironisant sur “l’importance de ne rien faire” [2]. Brosius et Gould, biologistes de l’évolution, proposent en 1992 [5] une nomenclature nuançant
1 Tumblr de Dan Graur alias Judge Starling
2 Proposition de traduction : Il y a les déchets que nous gardons, qui sont du bric-à-brac, et les déchets que nous
jetons, qui sont des ordures.
3 En français, on utilise plus rarement la notion d’ADN déchet qui serait pourtant plus adaptée. Dans les autres
langues européennes, on retrouve la même métonymie inadaptée. En espagnol, ADN basura qui se traduit clairement comme de l’ADN poubelle et son synonyme plus adapté et pourtant moins usité d’ADN chatarra. En allemand, on retrouve les termes de Müll-DNA et de Schrott-DNA qui signifient ADN déchet et ADN déchet avec une notion de recyclage. Malgré la forte proximité du second terme allemand avec la réelle définition du junk
DNA, c’est le terme Müll-DNA qui est le plus retrouvé. Les termes italiens DNA spazzatura et portugais ADN lixo
assez inadaptés font plutôt références à de l’ADN ordure, qui ne serait pas valorisable.
3 positivement le “junk DNA” reprenant l’idée d’ADN bric-à-brac avec des potentialités5 : le nuon étant une séquence d’acides nucléiques définie, le potonuon étant une séquence d’acides nucléiques ayant la possibilité de devenir fonctionnelle.
En 2002, Kapranov et collègues [6] présentent des travaux ayant pour but d’analyser la part du génome exprimé en prenant pour exemples les chromosomes les plus petits du génome humain (les chromosomes 21 et 22 d’environ 50Mb). Ils élaborent une puce à ADN en créant 1 million de sondes espacés de 35nt sur l’intégralité de ces chromosomes (=tiling array). Sur ces 2 chromosomes, on sait qu’il y avait 770 gènes codants connus6. Sur ces 1 million de sondes,
2,6% des sondes hybridées l’étaient sur des régions géniques, montrant ainsi que le reste des sondes hybridées avait détecté un signal en dehors des modèles connus. Les raisons proposées quant à la non-observation préalable de ces entités géniques sont (1) que les modèles connus étaient à l’époque incomplets (i.e. pas tous connus et/ou pas full-length), (2) que le niveau d’expression des ARN inconnus était trop faible et (3) que les séquences de nombreux clones d’ADNc des bases de données publiques avaient été éliminés lors de filtres sur leur capacité à coder des protéines7 (Figure 1, partie basse). Ils concluent sur le fait que les transcriptomes sont
encore loin d’avoir livré tous leurs secrets et qu’il existe certainement encore de nombreux transcrits inconnus à caractériser.
Figure 1 – Résultats synthétisés de Kapranov et collègues [6] sur leur expérience de tiling array sur les chromosomes 21 et 22. Les chiffres en bas font référence aux chiffres (1), (2) et
(3) du texte.
5 Un nuon est défini comme étant une séquence d’acides nucléiques qui est identifiable par un quelconque critère :
exon, SINE (i.e. élément transposable) ou encore un élément de réponse. Les dérivés de nuons sont : potonuon (séquence nucléique avec la potentialité de devenir fonctionnelle), potogene (idem pour un gène ≈ pseudogène), xaptonuon (potonuon ayant acquis une nouvelle fonction) et naptonuon (potonuon qui n’a jamais acquis de fonction).
6 Pour information, aujourd’hui le même ordre de grandeur de gènes codants se trouve sur ces chromosomes : 724
gènes codants
7 Pour information, aujourd’hui 910 ARNlnc et 527 pseudogènes se trouvent sur ces chromosomes, ce qui montre
4 D’autres expériences de tiling array de plus grande ampleur (50M et 70M de sondes) ont montré également que l’annotation des gènes humains était encore fortement incomplète et qu’elle était fortement lacunaire en ARNnc [7, 8]. C’est par ces observations que naît en 2003-2004 le projet ENCODE [9] (ENCyclopedia Of DNA Elements) qui souhaite identifier tous les nuons possédant une fonction biologique. En 2005, le consortium FANTOM et deux autres groupes de recherche se sont alliés pour publier un article majeur [10] sur la transcription des génomes humain et murin montrant qu’il existerait autant – voire plus – de gènes non-codant pour des protéines que de gènes codant. En 2006, Willingham et Gingeras [11] résument ces dernières années de course à l’amélioration des annotations du génome humain par : “transcription is widespread throughout the genome and many-fold higher than existing genome annotations would predict”. Ce “junk DNA” n’est finalement pas si déchet que cela mais cette transcription généralisée n’est-elle pas que du bruit transcriptionnel ? C’est à cette question que Ponjavic et collègues en 2007 [12] tentent de répondre en argumentant en faveur d’une transcription fonctionnelle puisque ces transcrits sont enrichis en séquences soumises à une sélection purifiante conduisant à la conservation de l’intégrité des promoteurs et des jonctions d’épissage (et dans une moindre mesure, la séquence primaire). La même année Kapranov et collègues publient un article [13], résumé par Ponjavic et Ponting [14], qui définit pour la première fois la taille des futurs ARNlnc (i.e. ≥200nt) appelés ici, lRNAs (long RNAs). Ils créent également la classification des PALR (Promoter Associated Long RNAs) correspondant à de longs gènes non-codants antisens du promoteur et de l’extrémité 5’ d’un gène codant. Quelques mois plus tard, le consortium du projet ENCODE publie un article montrant que 5% du génome humain a été identifié comme étant sous contrainte évolutive : il est composé de 40% d’ARN codants, 20% de régions non-codantes identifiées et 40% de régions inconnues [15]. Ils expliquent également que leur annotation des ARNm semble quasiment complète mais pas celle des ARNnc.
Avec l’émergence, fin des années 2000, des technologies de séquençage haut débit [16], le séquençage des génomes (DNA-seq) et transcriptomes (RNA-seq) est devenu facilement accessible et à coûts réduits. A la suite du projet ENCODE, le sous-projet GENCODE est fortement impulsé par une subvention de l’Institut Américain de Recherche sur le Génome Humain (NHGRI). La première version accessible de GENCODE humain qui recense 47553 gènes (https://www.gencodegenes.org/) date de juillet 2009. Ce premier catalogue chez l’Homme répertorie 6496 loci de type ARNlnc. Une première publication de 2010 de Ørom et collègues sur un sous-set de 3019 ARNlnc montre l’importance des ARNlnc comme régulateurs
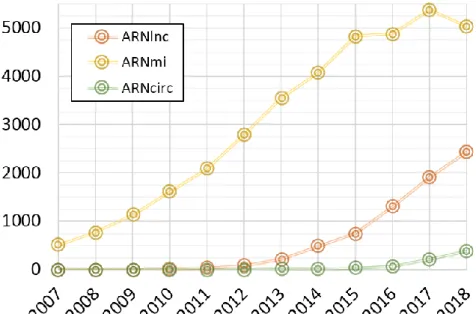
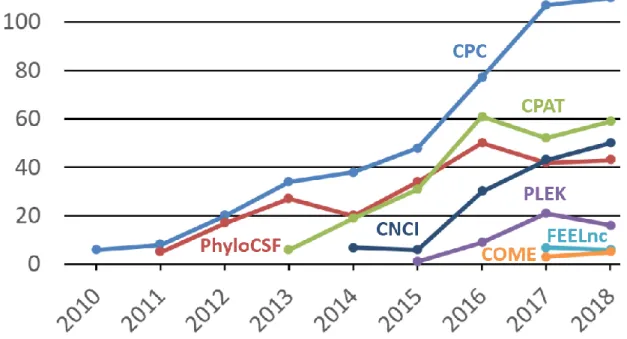
5 de l’expression génique [17, 18]. Ce rôle maintenant bien admis de régulateurs de l’expression génique (cf. 3.3 – Les différents mécanismes d’action des ARNlnc, p. 34) leur donne ainsi une importance majeure dans le fonctionnement du vivant. En effet, la régulation de l’expression génique est un levier de la différenciation tissulaire, de l’homéostasie des êtres vivants face aux conditions environnementales toujours changeantes, et également une cause majeure des différences phénotypiques entre les individus. A partir de cette date, les publications sur les ARNlnc commencent à augmenter progressivement comme indiqué en Figure 2. En 2012, Derrien et collègues publieront l’article majeur du sous-projet GENCODE sur les ARNlnc humain [19, 20], suivi en 2015 de l’annotation des ARNlnc chez la souris [21]. A partir de 2014-2015, on assiste à une course effrénée pour décrire les ARNlnc fortement impulsée par la Chine et les Etats-Unis [22] qui, contrairement aux ARNmi, n’a pas encore atteint son maximum d’effervescence (Figure 2).
Figure 2 – Etude bibliométrique8 des ARNlnc, ARNmi et ARNcirc de 2007 à 2018.
Pour conclure, en 2015, Francis Collins, directeur du NIH, s’est exprimé9 sur le “junk DNA” et
les récentes découvertes sur l’épigénome impliqué dans les processus de régulation : « I would say, in terms of junk DNA, we don't use that term any more 'cause I think it was pretty much a case of hubris to imagine that we could dispense with any part of the genome as if we knew enough to say it wasn't functional. There will be parts of the genome that are just, you know, random collections of repeats, like ALU's, but most of the genome that we used to think was
8 Recherche sur PubMed: ((Journal Article[Publication Type] OR Review[Publication Type]) AND
"lncrna"[MeSH Terms]) AND (("2007/01/01"[PDAT] : "2018/12/31"[PDAT]) AND English[lang])
6 there for spacer turns out to be doing stuff and most of that stuff is about regulation and that's where the epigenome gets involved, and is teaching us a lot. ».
1.1.2 – Premier objectif de ma thèse : annotation des ARNlnc chez les espèces d’élevage dont la poule
Au démarrage de ma thèse, fin 2015, l’annotation des gènes humains comprenait environ 20000 gènes codant pour des protéines et 15000 gènes à ARNlnc. Les statistiques des espèces modèles comme la souris, le poisson-zèbre et la drosophile mentionnaient entre 14000 et 26000 gènes codant pour des protéines et seulement 2300 à 6800 gènes à ARNlnc soit 2 à 6 fois moins que chez l’Homme (Tableau 1). Ces derniers chiffres montrent, à cette époque, un certain retard de l’annotation chez les espèces modèles par rapport à l’Homme, retard toujours présent aujourd’hui avec 9602 (+40%) ARNlnc chez la souris et 3278 (+20%) chez le poisson-zèbre dans la v.93.6 d’Ensembl de juillet 2018, soit des nombres bien inférieurs aux 15000 gènes ARNlnc humains. Pour les espèces d’élevage, cet effort d’annotation des génomes au niveau des gènes à ARNlnc n’avait pas encore été amorcé puisque 0, 0 et 135 ARNlnc étaient recensés fin 2015 dans la base de données Ensembl (version 83 de décembre 2015) chez la poule, le bovin et le porc respectivement (Tableau 1). Dans ce cadre, le premier
objectif de ma thèse était d’établir un premier catalogue d’ARNlnc chez la poule grâce à des
données de RNA-seq. Les données de RNA-seq correspondent à un très grand nombre (~100 millions) de courtes séquences (~100nt) appelés lectures, et correspondant à des fragments de transcrits du tissu et de l’individu analysé.
Tableau 1 – Annotations d’Ensembl v.83 (décembre 2015) de l’Homme, de 3 espèces modèles souris, poisson-zèbre, drosophile et de 3 espèces d’élevage poule, bovin et porc.
Gènes codant pour des protéines Pseudogènes Gènes à petits ARN non-codants Gènes à ARN longs non-codants Homme 20313 14453 7703 14896 Souris 22533 9349 5530 6830 Poisson-zèbre 25642 293 3172 2741 Drosophile 13918 257 1018 2366 Poule 15508 42 1408 0 Bovin 19994 797 3650 0 Porc 21630 568 2804 135
7 Très brièvement, à partir de données RNA-seq de foies et tissus adipeux de poulets de chair, nous avons modélisé, sur le génome de référence Galgal4, de nouvelles entités géniques, desquelles nous avons établi un premier catalogue de 1670 ARNlnc de nos deux tissus exprimés de façon reproductible. Ce premier catalogue a été publié en janvier 2017 dans la revue Genetics Selection Evolution [23] (Résultats – Partie I – 1 – Annotation des ARNlnc du foie et du tissu adipeux chez la poule, p. 48). Sur la même période (octobre 2016), un nouvel assemblage du génome de la poule Gallus_gallus-5.0 (alias Galgal5) ainsi qu’une nouvelle annotation sont parus dans les bases de données publiques d’Ensembl (v.86.5) et du NCBI (v.103). Cette nouvelle annotation apportait environ 8000 ARNlnc. Nous avons fait une comparaison de notre catalogue par rapport aux deux ensembles d’ARNlnc des bases de données publiques. Ces travaux ont été présentés sous la forme d’un poster (p. 252) et d’un article vulgarisé rédigé en français (Résultats – Partie I – 1.3 – , p. 73) lors des 12ème Journées de la Recherche Avicole.
Mes connaissances et mon expertise sur les ARNlnc m’ont également permis d’intégrer le projet pilote INRA FR-AgENCODE qui vise à améliorer l’annotation des génomes des espèces d’élevage dans le cadre d’un projet international plus large appelé FAANG (Functional Annotation of Animal Genome). J’ai ainsi pu participer à l’annotation des ARNlnc chez le bovin, le porc, la chèvre et la poule à partir de données RNA-seq de foie et de deux types cellulaires (lymphocytes T CD4+ et T CD8+). Les expressions de ces gènes connus ou nouvellement identifiés ont alors été intégrées à d’autres analyses de données de type Hi-C et ATAC-seq et ont fait l’objet d’une publication soumise en juillet 2018 dans Genome Biology [24] (Résultats – Partie I – 2 – Annotation des ARNlnc des lymphocytes T (CD4+, CD8+) et du foie chez 4 espèces d’élevage : le bovin, la chèvre, le porc et la poule, p.79).
Début 2017, en collaboration avec nos collègues de la plateforme de bio-informatique de Toulouse, nous avons relancé nos pipelines d’alignement de lectures et modélisation de gènes en apportant une nouvelle série de 364 expériences RNA-seq issues d’échantillons de foie, tissu adipeux, sang et hypothalamus de poules commerciales. Nous avons ainsi modélisé 38612 ARNlnc nouveaux par rapport à l’annotation d’Ensembl pris en référence pour l’alignement des lectures et la modélisation des gènes faisant un total de 44583 ARNlnc. En binôme avec Frédéric Jehl, autre doctorant du laboratoire, nous avons ajouté à ce catalogue les modèles géniques de type long non-codant ne chevauchant pas les modèles précédents et provenant d’autres sources publiques. Il s’agit de la base de données publique du NCBI ainsi que les bases de données spécialisées de NONCODE [25] et ALDB [26] qui proposent des
8 catalogues indépendants d’ARNlnc. On y a ajouté, de la même manière, les ARNlnc du sous-projet FR-AgENCODE [24] dont il a été parlé ci-dessus. Cet effort de regroupement des sources précitées a permis de former un nouveau catalogue de 59486 ARNlnc. La constitution de ce nouveau catalogue (Résultats – Partie I – 3 – Mise à jour du catalogue d’ARNlnc chez la poule en apportant de nouvelles données RNA-seq et en combinant plusieurs bases de données, p.126) sera intégré dans un article plus large qui est en préparation.
1.2 – Contexte lié au second objectif de la thèse : ARNlnc et lipides
1.2.1 – Importance des lipides
Les lipides ont un rôle essentiel dans les espèces animales puisqu’ils composent les membranes, agissent comme molécules de signalisation et sont une importante source d’énergie. La dérégulation des processus d’homéostasie lipidique est donc responsable de dyslipidémies et de nombreuses maladies d’une importance majeure en santé humaine (e.g. obésité, diabète, maladie du « foie gras », maladies cardiovasculaires). D’un point de vue agronomique, ce métabolisme est lié à des caractères économiquement importants. Chez le porc, le bœuf, le mouton ou les poissons, les acides gras peuvent être stockés sous forme de graisses intramusculaires et contribuent ainsi positivement à la qualité nutritionnelle et organoleptique de la viande [27–29]. En revanche, le gras de carcasse localisé majoritairement dans la cavité abdominale (cas du poulet) ou encore sous la peau (cas du canard ou du porc), n’est pas valorisé par l’industrie agroalimentaire et représente une perte économique importante pour toutes les espèces d’élevage.
Le site majeur de synthèse des lipides, plus précisément de la synthèse de novo des acides gras, peut différer d’une espèce animale à l’autre. Elle se fait principalement dans le foie chez l’Homme, les rongeurs, les oiseaux [30–32] et de nombreux poissons [33], et dans le tissu adipeux chez le porc ou la vache [34]. La synthèse du cholestérol chez les mammifères, les oiseaux et les poissons se fait de façon plus hétérogène mais avec une prédominance également dans le foie [31, 35, 36]. Le stockage des acides gras en triglycérides, principale réserve énergétique des animaux constituée en période d’excès et mobilisée en période de privation énergétique, se fait essentiellement dans le tissu adipeux des animaux au sein de cellules spécialisées, les adipocytes.
9 1.2.2 – Second objectif de ma thèse : identifier des ARNlnc régulateurs du métabolisme des lipides
Ce métabolisme des lipides étant donc très important et fortement régulé au niveau des transcrits, mon deuxième objectif de thèse est d’identifier de nouveaux gènes régulateurs de ce métabolisme avec un focus particulier sur les ARNlnc qui sont encore aujourd’hui méconnus. Cet objectif entre dans une problématique scientifique plus large de l’équipe qui est de mieux comprendre les causes génétiques des caractères complexes en lien avec le métabolisme des lipides (adiposité corporelle, efficacité alimentaire, …) que les sélectionneurs cherchent à contrôler dans les populations commerciales.
Dans ce contexte, j’ai effectué une étude approfondie de la littérature afin de recenser tous les ARNlnc décrits comme étant impliqués dans le métabolisme des lipides ; cette étude a été faite chez toutes les espèces animales dont l’Homme. La majorité de ces ARNlnc étant décrits chez les espèces les plus travaillées que sont l’Homme et la souris, j’ai souhaité identifier les orthologues chez la poule afin d’apporter de la connaissance chez cette espèce. J’ai donc mené une étude sur la conservation par synténie de ces ARNlnc chez la poule et l’ai étendue jusqu’aux poissons. Cette revue et ces résultats ont fait l’objet d’un article écrit et prêt à être soumis à la revue Scientific Reports (Résultats – Partie II – 1 – Les ARNlnc connus comme étant impliqués dans le métabolisme des lipides, p.142).
J’ai également effectué ce travail sur les ARNlnc impliqués dans l’adipogenèse, processus de différenciation des préadipocytes en adipocytes lié au stockage des lipides et à la formation du tissu adipeux. Cette revue qui fera ultérieurement l’objet d’une publication est présentée en français, sous la forme d’un article (Résultats – Partie II – 2 – Les ARNlnc connus comme étant impliqués dans l’adipogenèse, p.179).
Ayant à ma disposition des données de RNA-seq de foie et de tissu adipeux issues de deux lignées de poulets de chair, divergentes pour le poids de gras abdominal (appelées. lignée maigre et lignée grasse), j’ai également recherché les gènes différentiellement exprimés (DE) entre ces deux lignées. J’ai pu mettre en évidence des exemples intéressants d’ARNlnc DE génomiquement proches de gènes emblématiques du métabolisme des lipides. Par exemple, deux gènes longs non-codants nouvellement modélisés par nos travaux qui sont à proximité du gène DHCR24, codant une enzyme de la synthèse du cholestérol, ou encore le gène FASN, codant une enzyme clé de la synthèse des acides gras. Cette analyse est présentée sous la forme
10 d’un article en français et sera soumis ultérieurement (Résultats – Partie II – 3 – De nouveaux ARNlnc potentiellement régulateurs du métabolisme des lipides, p.193)
***
Les deux objectifs de thèse étant en lien avec l’espèce poule et les ARNlnc, j’ai souhaité avant de présenter les différents articles et résultats, apporter quelques informations sur l’espèce poule et terminer par un état de l’art sur les ARNlnc, ces entités géniques qui sont au cœur de ma thèse.
2 – L’espèce poule
2.1 – De la poule sauvage aux lignées commerciales
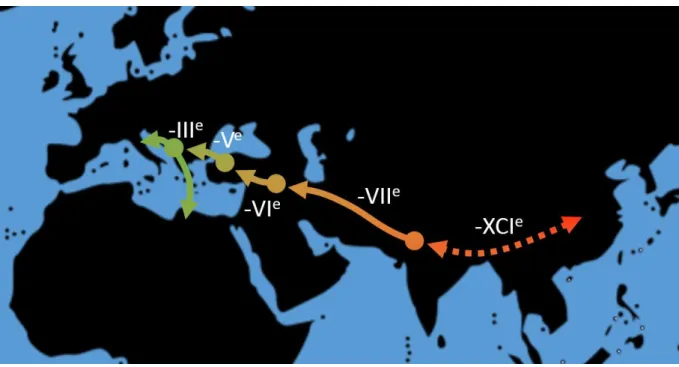
Les premières traces de poule remontent à la fin du Néolithique (4000 avant J-C.) dans la province du Henan en Chine [37] où vivait le peuple des Yangshao. De 1200 avant J-C. au IIIème siècle, de la dynastie Shang aux Trois Royaumes chinois, l’élevage des poules semble avoir un but sacrificiel, de nombreux ossements étant retrouvés dans les tombeaux royaux [37]. Les écrits en Europe et en Afrique liés à la poule de ces époques sont inexistants. Les Perses ont très certainement découvert cet animal lors des conquêtes perses (VIIème au VIème siècle avant J-C.), royaume qui s’étendait de l’Inde à l’Asie Mineure (Figure 3, 2ème flèche). La poule semble atteindre la Lydie (i.e. actuelle Turquie) sous l’ère de Crésus, roi lydien (VIème siècle avant J-C.) qui entretenait de bonnes relations avec la Grèce (Figure 3, 3ème flèche). Aristophane poète grec du Vème siècle témoigne de l’arrivée de la poule en Grèce puisqu’il mentionne la poule comme étant « l’oiseau persique » [38] (Figure 3, 4ème flèche). La poule a ensuite été
disséminée sur le pourtour du bassin méditerranéen lors des conquêtes romaines d’Alexandre le Grand (IVème siècle avant J-C.) puisqu’on retrouve les premières représentations de la poule
sur les monuments égyptiens lors de la période hellénistique (IIIème siècle avant J-C.) (Figure
3, 5ème flèche, bas). Columelle et Cicéron, respectivement agronome et orateur romains sous la
dynastie julio-claudienne (Ier siècle), témoignent de la présence de poules dans la région de Rome (Figure 3, 5ème flèche, haut). Il se pourrait même que le principe même de la castration
des jeunes coqs en vue d’en faire de bons chapons bien gras soit une invention romaine [39]. Dans le livre VIII de « De L’Economie Rurale » [39], Columelle écrit : « Le revenu qui provient de ces oiseaux de basse-cour n'est pas à dédaigner, si on les soigne convenablement, comme faisaient la plupart des Grecs […]. Ces peuples recherchaient surtout, en raison de leur grande espèce et de leur courage dans les combats, les races de Tanagra et de Rhodes […]. Nous
11 pensons, nous, que les poules doivent constituer un revenu pour le père de famille industrieux, et non pas pour un instructeur d'oiseaux batailleurs, dont souvent tout le patrimoine, gage de la joute, lui est ravi par la victoire d'un athlète emplumé. » car en effet, l’élevage avait également vocation à produire des coqs de combat, des poules longtemps élevées pour leurs œufs et coqs ornementaux pour les expositions voire des animaux de compagnie pour les poules naines. Avec la révolution des transports au XIXème siècle, la poule 九斤黄 Jiujinhuang,
littéralement « Jaune de 9 kati » (i.e. 4,5 kilos) réputée pour sa viande a été exportée en Europe et en Amérique [37]. Fort de son succès, elle sera en partie à l’origine des poules Rhodes Island des Etats-Unis, initialement créées comme poules de chair et de ponte [37].
Figure 3 – Importation des poules asiatiques en Europe au travers des conquêtes
Au XIXème siècle, la révolution industrielle transforme vivement un monde rural, artisanal et agricole en une société urbaine et industrielle. De l’élevage des poules et poulets dans les fermes pour l’alimentation familiale est né l’élevage intensif pour subvenir à une alimentation de la population mondiale en perpétuelle croissance. L’amélioration génétique par sélection a permis l’apparition de lignées spécialisées dans la ponte (e.g. Rhodes Island, Leghorn) ou la chair (e.g. Cornish, White Plymouth Rock). L’amélioration génétique et donc la spécialisation des races s’est accélérée ces 50 dernières années grâce à des méthodes de génétique quantitative efficaces basées sur les performances des individus apparentés et plus récemment, durant les années 2010, par la sélection sur le génome.
12
2.2 – Importance économique du poulet de chair
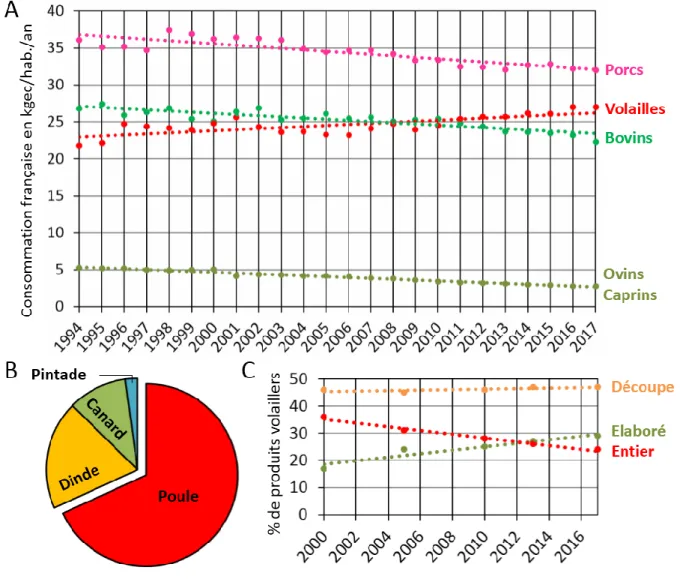
A l’inverse de nombreuses espèces d’élevage10, la consommation du poulet de chair n’est
soumise à aucune contrainte ethnique ou religieuse. Du poulet tikka masala indien, au poulet Marengo en France en passant par le plof caucasien, le galinhada brésilien, le poulet Yassa sénégalais ou les yakitoris japonais, on retrouve le poulet dans toutes les cultures du monde. En 2017, la consommation mondiale de viande volaillère par rapport aux autres viandes est de 41% (1ère place) avec des disparités géographiques allant de 35% en Asie à cause des peurs liées à la grippe aviaire jusque 50 à 63% respectivement dans les pays d’Amérique du Nord et du Moyen-Orient/Maghreb (Données issues des « Perspectives agricoles de l’OCDE et de la FAO 2018-2027 »). Entre 1961 et 2016, la production mondiale de volailles a explosé passant de 9 à 120 millions de tonnes et les estimations pour 2030 prévoient des augmentations de +271% en Asie du Sud (Données FAO). Sa consommation nationale comme dans le reste du monde est en progression constante depuis plus de 20 ans au détriment des viandes bovine, porcine, ovine et caprine (Figure 4A). Selon FranceAgriMer, en 2017, la filière avicole française représentait 1,81 million de tonnes de volailles produites dont 68% de poule (Figure 4B) et 15 milliards d’œufs. Malgré un reclassement entre 2013 et 2017 de la 1ère à la 3ème place des plus grands
pays européens producteurs de volailles, la France se démarque des autres pays avec une exportation importante dans les pays du Moyen-Orient et en Afrique Subsaharienne. Avec l’essor de la malbouffe, depuis 2013, la proportion des volailles entières consommées a laissé place aux produits volaillers transformés (e.g. nuggets, cordons bleus). La consommation de volailles découpées (e.g. filet de poulet) reste malgré tout constante depuis 5 ans (Figure 4C). La poule est donc d’une grande importance en alimentation humaine. Les efforts de sélection ont jusqu’à présent permis une très forte amélioration des caractères de production comme la croissance et l’efficacité alimentaire. Par exemple un poulet de chair commercial atteint 2 kg en 6 semaines contre 1 kg en 20 semaines pour le poulet sauvage ancestral (MOOC – l’Aviculture, une filière d’avenir). Aujourd’hui, la tendance est toujours de produire plus d’œufs ou de viandes, en élevant les animaux à moindre coût11, résistants aux maladies12, et qu’ils
10 Les juifs ont des interdits alimentaires comprenant le porc, le lapin, l’autruche ou les fruits de mer. Pour les
musulmans, il s’agit du porc ou de l’âne. La vache est vénérée par les hindouistes.
11 Rendement et efficience de l’animal, adaptation de l’alimentation
12 Etudes des maladies aviaires : maladie de Newcastle, maladie de Gumboro, maladie de Marek, le coryza ou les
13 conservent leur efficience malgré des conditions changeantes13 et pour les poules pondeuses qu’elles soient utilisées plus longtemps.
Figure 4 – Consommation française de viande. A. Toutes viandes par habitant entre 1994 et
2017. Les bovins correspondent aux gros bovins et aux veaux. Les volailles correspondent au poulet, à la dinde, au canard et à la pintade. kgec : kilogramme équivalent-carcasse
B. Production avicole en 2017. C. Consommation de poulets entiers, découpés ou transformés
en France entre 2000 et 2017. Données issues de FranceAgriMer.
2.3 – Importance de la poule en recherche
La poule a l’intérêt d’être un animal de petite taille, peu coûteux et ayant un temps de génération court, 6 à 8 mois suivant les races. Son génome est plus petit que celui de l’Homme – 1 milliard contre 3 milliards de bases (Figure 5) – et est composé de 38 chromosomes autosomes et 2 chromosomes sexuels Z et W.
13 Adaptation à des changements de régimes pour des raisons économiques et géographiques, à des changements
![Figure 1 – Résultats synthétisés de Kapranov et collègues [6] sur leur expérience de tiling array sur les chromosomes 21 et 22](https://thumb-eu.123doks.com/thumbv2/123doknet/14546925.725402/29.892.149.748.658.865/figure-résultats-synthétisés-kapranov-collègues-expérience-tiling-chromosomes.webp)