Ministère de l'Enseignement Supérieur et de La Recherche Scientifique Université des Frères MENTOURI Constantine
Faculté des Sciences de la Nature et de la Vie N° d’ordre : 38/D3C/2016
N° de série : 03/Bioch/2016
THESE
En vue de l’obtention du diplôme de
DOCTORAT 3
èmeCYCLE
Spécialité : Biochimie et Biotechnologies
Présentée par
Melle MEDOURI Asma
Intitulé
Soutenue le 03 juillet 2016
Devant le jury composé de
Président : A. BOULAHROUF Prof. Université des Frères Mentouri, Constantine Directeur de thèse : D. KHELIFI Prof. Université des Frères Mentouri, Constantine Examinateurs : M.A. HAMIDECHI Prof. Université des Frères Mentouri, Constantine A. CHIKHI Prof. Université des Frères Mentouri, Constantine M. HAFSI Prof. Université Ferhat Abbas, Sétif
Diversité génétique de l'espèce Aegilops geniculata Roth en Algérie
par approches morphologique, biochimique et moléculaire
A
Mes parents qui ont fait de moi ce que je suis, mon père
Mohammed Cherif et ma mère Chafia. Que Dieu vous garde et
m’aide pour vous rendre heureux et fiers de moi.
Mon petit trésor, ma nièce Balsam
Mon petit amour, mon cher frère Ahmed Mouaad
Mon très cher frère El Moundir
Ma moitié et ma très chère sœur Nawel
Mes très chers grands parents Djaber et Zelikha
Tous ceux qui me sont chers
La thèse, quelle aventure à la fois unique, passionnante et éprouvante ! Pas moins de quatre années se sont écoulées depuis mes premiers pas au laboratoire de Génétique Biochimie et Biotechnologies Végétales et la fin de la rédaction de ce document. Je ne pourrais dire si ces années se sont écoulées vite ou lentement, mais une chose est sûre, elles auront été remplies de moments et de rencontres inoubliables. Au terme de ce travail je voudrais remercier toutes les personnes qui m’ont apporté leur aide et qui ont contribué à l’élaboration de ce travail, et tout particulièrement certaines d’entre elles. A commencer par l’initiateur et le soutien de cette thèse Mr. KHELIFI Douadi, directeur du laboratoire de Biochimie Générique et Biotechnologies Végétales. Il a passé ces dernières années à m’encadrer, avec la bonne dose de patience et de courage. Vous m’avez fait confiance pour défendre un sujet de thèse mûri depuis des années qui vous tenait à cœur. Je vous remercie pour m’avoir guidée, encouragée, et conseillée tout au long de ces années. Pour cette confiance, la disponibilité, l’enthousiasme, le dynamisme, les remarques pertinentes et l’autonomie que vous m’avez accordée, je vous remercie très sincèrement.
J’adresse mes vifs remerciements aux membres du jury pour l’honneur qui m’ont fait en acceptant d'examiner ce travail. Je remercie le Pr. BOULAHROUF A. qui me fait l’honneur de présider ce jury. Merci à messieurs les professeurs HAMIDECHI M.A., CHIKHI A. et HAFSI M., examinateurs de cette thèse. Qu’ils soient assurés de ma sincère reconnaissance.
Je tiens à adresser ma profonde reconnaissance à :
Ma très chère amie et collègue Imane MEDOUKALI, sans qui la prospection et la collecte n’auront pu être jamais faites. Elle a toujours été là pour moi. Même quand ça n’allait pas, elle m’a toujours dit de ne pas baisser les bras.
Sarah BECHKRI qui est devenue comme une grande sœur pour moi. Elle m’a apporté chaque fois le support moral et les conseils scientifiques. Je la remercie aussi pour sa relecture intéressante des différentes parties de la thèse.
Mohammed CHEKARA BOUZIANI qui m’a continûment soutenu, pour la générosité, pour son aide inestimable surtout dans la partie moléculaire.
Inès BELLIL pour m’avoir accompagné au début de ce travail, pour ses conseils et pour l’intérêt qu’elle a donné à ce sujet. Je tiens à la remercier tout particulièrement pour son aide tout au long de la rédaction, la correction et la révision des articles.
La merveilleuse équipe de Biochimie génétique qui d’une façon ou d’une autre a contribué à la réalisation de cette thèse. Je remercie très spécialement les doctorantes Souheir BENSARI et Rayène OUALBANI qui par leur humour ont su créer une ambiance agréable.
Tout le personnel du laboratoire de Biochimie Génétique et Biotechnologies Végétales, en particulier Mr. BELBEKRI Mohamed Nadir, BOULDJED Rima, DJEGHAR Radia, MOUALLEF Adra, BENAMARA Meriem et BOUCHEMAL Karima.
Mes cousines Aïcha BENYAHIA, Samia et Safia MEDOURI et mon amie Ikram HANTOUT pour leur aide et leur disponibilité chaque fois que j’en ai eu besoin.
Cela va de soi, je remercie avec grande émotion mes parents qui ont toujours su m’offrir leur soutien, leur compréhension, leurs encouragements, leur patience et affection. Merci à ma famille entière pour son irremplaçable et inconditionnel soutien. Elle m’a toujours encouragée à aller de l’avant dans la vie malgré la difficulté. Merci d’avoir été là pour écarter les doutes, soigner les blessures et partager les joies. Cette thèse est aussi la vôtre.
ABREVIATIONS
Ae. Aegilops
4-VP 4-vinylpyridine
AFLP Amplified fragment length polymorphism A-PAGE Acid polyacrylamide gel electrophoresis
APS Ammonium persulfate
CAPS Cleaved amplified polymorphic sequence
CM Cysteine motif
DTT Dithiothréitol
FPM Faible poids moléculaire HMW High molecular weight HPM Haut poids moléculaire
ICARDA International center for agricultural research in the dry areas
LMW Low molecular weight
MAS Marker assisted selection NORs Nucleolus organizer regions
pb Paire de base
PCA Principal component analysis PCR Polymerase chain reaction
RAPD Random amplified polymorphic DNA RFLP Restriction fragment length polymorphism SCAR Sequence-characterized amplified region
SDS-PAGE Sodium dodecyl sulfate polyacrylamide gel electrophoresis SG Sous unités de gluténines
SNP Single nucleotide polymorphism SSR Simple sequence repeat
T. Triticum
TCA Trichloroacetic acid
Glycine Gly G Proline Pro P Alanine Ala A Valine Val V Leucine Leu L Isoleucine Ile I Méthionine Met M Cystéine Cys C Phénylalanine Phe F Tyrosine Tyr Y Tryptophane Trp W Histidine His H Lysine Lys K Arginine Arg R Glutamine Gln Q Asparagine Asn N
Acide glutamique Glu E Acide aspartique Asp D
Serine Ser S
LISTE DES TABLEAUX
Partie I : Revue bibliographique Tableau 1 Aperçu des importantes classifications des AegilopsTableau 2 Sections et espèces d’Aegilops avec leurs formules génomiques
Tableau 3 Les principaux gènes de résistance aux stress biotiques identifiés chez les
Aegilops et transférés chez le blé
Tableau 4 Les espèces d’Aegilops considérées comme source potentielle de résistance au stress abiotique
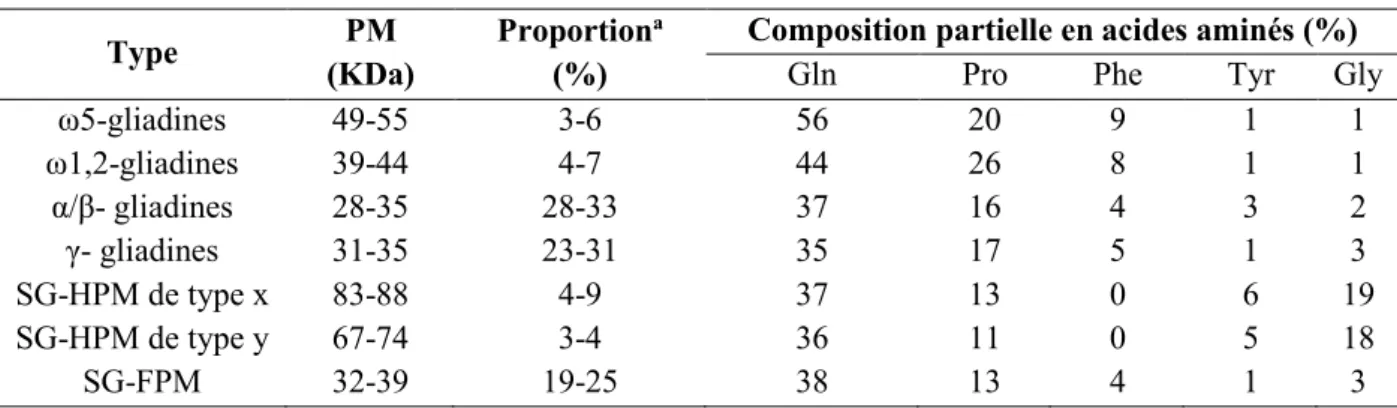
Tableau 5 Autres traits exploitables à partir des espèces d’Aegilops Tableau 6 Caractérisation des protéines du gluten
Tableau 7 Les différents systèmes de classification des SG-FPM
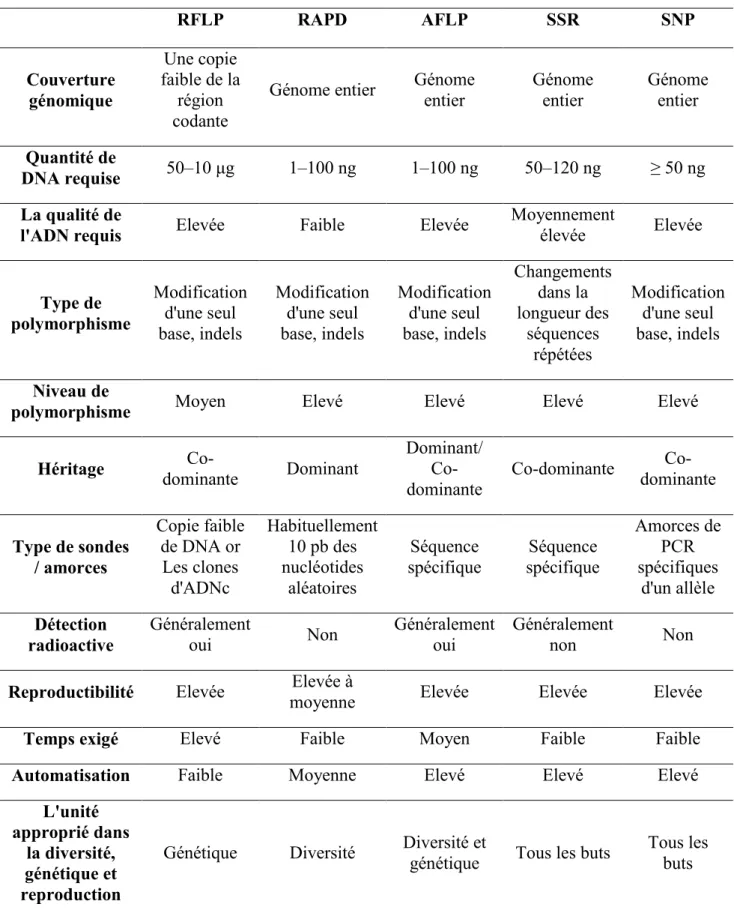
Tableau 8 Comparaison entre les cinqs marqueurs d’AND principalement utilisés pour les plantes
Tableau 9 Les principaux marqueurs moléculaires liés aux gènes de résistance à la rouille chez les Aegilops
Tableau 10 Synthèse des principaux travaux de la diversité génétique et la phylogénie des
Aegilops
Partie II : Résultats Chapitre 1
Tableau 1 Origin and allelic composition at the Glu-1 and Glu-3 loci in diploid accessions Tableau 2 Allele frequencies at HMW and LMW glutenin subunits loci in Ae. comosa and
Ae. umbellulata
Tableau 3 Gliadin patterns and diversity indexes within diploid species Chapitre 2
Tableau 1 Sampling sites and main bioclimatic parameters
Tableau S1 Allelic composition at Glu-M1 and Glu-U1 loci found in Ae. geniculata accessions collected in Algeria
Tableau S2 Allele frequencies at HMW glutenin subunits and genetic index diversity at the
Glu-M1 and Glu-U1 loci in Ae. geniculata accessions collected in Algeria
Chapitre 3
Tableau 1 Allelic composition of HMW and B-LMW glutenin subunits of Ae. geniculata accessions
Tableau 2 Allele frequencies at HMW glutenin subunits and genetic index diversity at the Glu-M1 and Glu-U1 loci in Aegilops geniculata accessions
Chapitre 4
Tableau 1 Locations and ecological parameters of different Ae. geniculata accessions used in this study
Tableau 2 Number of gliadin bands, patterns, and the genetic diversity in gliadins for the accessions of Ae. geniculate
Chapitre 5 Tableau 1 List and coordinates of accessions used
LISTE DES FIGURES
Partie I : Revue bibliographique Figure 1 Spéciation par allopolyploïdisationFigure 2 Les espèces des genres Triticum et Aegilops constituent un exemple classique de spéciation par polyploïdie
Figure 3 Aperçu sur l’évolution du blé et les évènements ayant abouti à sa formation Figure 4 Morphologie d’Ae. geniculata et de ses progéniteurs diploïdes
Figure 5 Aire de distribution d’Ae. geniculata Figure 6 Schéma d'un grain de blé en coupe
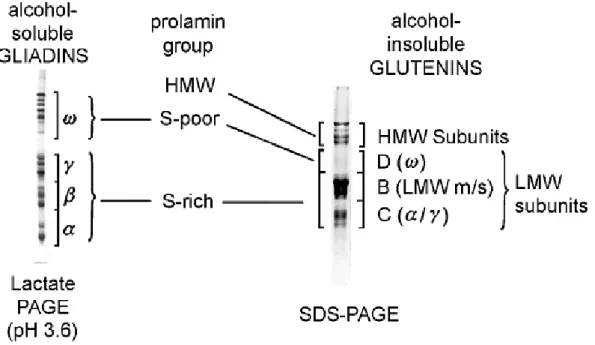
Figure 7 Classification et nomenclature des protéines de gluten séparées par SDS-PAGE et Acid-PAGE
Figure 8 Electrophorèse bidimensionnelle (IEF et SDS-PAGE) des sous unités de gluténines
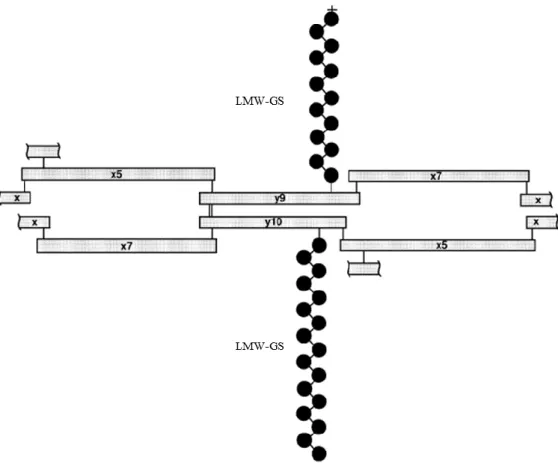
Figure 9 Schémas représentatifs de la structure des SG-HPM et de leurs ponts disulfures Figure 10 Model théorique de la structure du polymère des gluténines
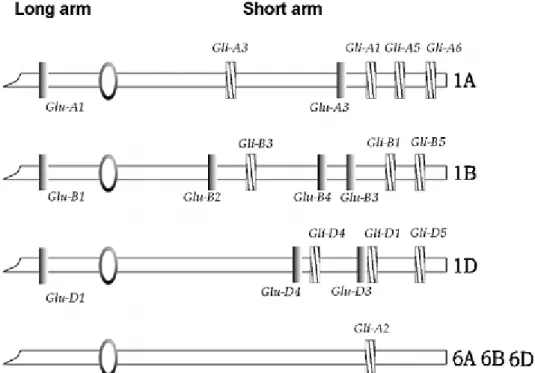
Figure 11 Localisation chromosomique des loci codants les gliadines et les gluténines chez le blé tendre
Partie II : Résultats Chapitre 1
Figure 1 SDS-PAGE patterns of HMW- and LMW-GS in some Ae. comosa and Ae.
umbellulata accessions
Figure 2 Acid-PAGE gliadins patterns of Ae. comosa accessions analyzed Figure 3 Acid-PAGE gliadins patterns of Ae. umbellulata accessions analyzed
Chapitre 2
Figure 1 Distribution of the studied Aegilops geniculata Roth accessions in the different eco-geographical regions of Northern Algeria
Figure 2 Principal component analysis of Aegilops geniculata accssions based on a fifteen morphological traits
Figure 3 SDS-PAGE patterns of high molecular weight glutenin subunits in Algerian
Aegilops geniculata
Figure S1 Schematic representation of the mobility on SDS-PAGE of the different HMW glutenin subunits alleles encoded at Glu-M1 and Glu-U1 loci found in Ae.
geniculata accessions studied
Figure S2 Dendrogramm based on HMW glutenin subunits polymorphism, showing the relationships among accessions of Ae. geniculata Roth sampled in Algeria
Chapitre 3
Figure 1 SDS-PAGE patterns of HMW and LMW in some accessions of Ae. geniculata collected in Algeria
Figure 2 Diagrammatic representation of HMW-GS patterns found in Ae. geniculata from Algeria
Ae. geniculata accessions
Figure 4 Schematic representation of the mobility on SDS-PAGE of the different alleles encoded at Glu-1 and Glu-3 loci found in Ae. geniculata accessions studied
Chapitre 4
Figure 1 Variations detected using Acid-PAGE for some representative samples of Ae.
geniculata
Figure 2 Cluster analysis of Ae. geniculata accessions from Algeria based on gliadin polymorphism using Ward’s method
Figure 3 The relative loading of each studied variable on the principal components of 36 Algerian accessions of Ae. geniculata
Chapitre 5
Figure 1 RAPD amplification patterns of some Ae. geniculata genotypes
Figure 2 Dendrogram based on RAPD data using UPGMA cluster analysis of the distance matrix among 34 Ae. geniculata accessions
Figure 3 Dendrogram based on fifteen morphological traits using UPGMA cluster analysis of the distance matrix among 34 Ae. geniculata accessions
SOMMAIRE
RemerciementsListe des tableaux, des figures et des abréviations Sommaire
Introduction générale………….………... 1
Partie I : Revue bibliographique I- Le genre Aegilops………... 3
I-1- Présentation………. 3
I-2- Historique de la classification………...………... 3
I-3- Génétique des Aegilops………... 4
I-3-1- La polyploïdie………... 4
I-3-2- Constitution génomique des espèces d’Aegilops………... 5
I-3-3- Évolution des génomes des espèces polyploïdes d’Aegilops……… 6
I-4- Aire de répartition des Aegilops………... 7
I-5- Intérêts des Aegilops……… 8
I-5-1- Contribution dans la constitution des génomes des blés………... 8
Aperçu sur les espèces sauvages et cultivées du blé……….. 8
Origine des blés tétraploïdes……….. 9
Origine des blés héxaploïdes……….. 9
I-5-2- Amélioration des blés cultivés……….. 10
Amélioration de la résistance et ou la tolérance aux stress biotique et abiotque……….. 10
Amélioration de la qualité technologique du blé………... 10
II- Présentation de l’espèce Aegilops geniculata……….. 12
II-1- Description morphologique………... 12
II-2- Le génome d’Aegilops geniculata……… 12
II-3- Distribution géographique……… 13
III-Caractéristiques biochimiques et génétiques des protéines du grain de blé et de ses apparentés………... 14
III-1- Le grain d’Aegilops……… 14
III-2- Les différentes fractions protéiques du grain………... 14
III-2-1- Protéines cytoplasmiques ou métaboliquement actives…………... 15
III-2-2- Protéines de réserve………... 15
- Les prolamines……….. 15
A- Les gliadines………... 16
B- Les gluténines……….. 17
Les sous unités gluténines de faible poids moléculaire... 17
Les sous unités gluténines de haut poids moléculaire... 18
III-3- Déterminisme génétique et localisation chromosomique des prolamines……….. 19
III-3-1- Les gliadines……….. 19
III-3-2- Les sous unités gluténines de haut poids moléculaire………... 20
III-3-3- Les sous unités gluténines de faible poids moléculaire …... 20
IV- Les marqueurs génétiques………... 22
IV-1- Définition d’un marqueur génétique……….... 22
IV-2- Propriétés recherchées d’un marqueur génétique………... 23
IV-3- Types de marqueurs génétiques……… 24
IV-3-1- Les marqueurs traditionnels……… 24
• Les marqueurs morphologiques... 24
• Les marqueurs cytologiques………. 24
• Les marqueurs biochimiques……… 25
IV-3-2- Les marqueurs moléculaires (marqueurs à ADN) ………... 25
• Les marqueurs de type RFLP………... 25
• Les marqueurs de type PCR………. 26
- Les marqueurs RAPD ………... 26
IV-4- Intérêt des marqueurs moléculaires dans l’amélioration des blés cultivés à travers les Aegilops………... 27
IV-5- Synthèse des travaux de diversité génétique des Aegilops et leurs relations au genre Triticum………. 28
Références bibliographiques………... 29
Partie II : Résultats Chapitre 1 : Evaluation de la variation génétique de deux espèces diploïdes d’Aegilops en termes de gluténines et gliadines………... 44
Chapitre 2 : Diversité génétique et morphologique d’Aegilops geniculata Roth dans le Nord Algérien………... 61
Chapitre 3 :Diversité génétique des sous unités gluténines de haut et faible poids moléculaire d’Aegilops geniculata en Algérie………... 77
Chapitre 4 :Evaluation de la diversité génétique des gliadines d’Aegilops geniculata prospectée en Algérie………. 85
Chapitre 5 :Diversité génétique d’Aegilops geniculata en Algérie révélée par les marqueurs morphologiques et moléculaires... 93
Partie III : Synthèse générale et perspectives 1- Variabilité biochimique des diploïdes……….. 111
2- Diversité morphologique, biochimique et moléculaire d’Ae. geniculata…... 111
3- Corrélation avec les facteurs climatiques….………... 113
4- Perspectives………... 113 Annexes
“For the sake of future generations, we MUST collect and study wild and weedy relatives of our cultivated plants as well as the domesticated races. These sources of germplasm have been dangerously neglected in the past, but the future may not be so tolerant. In the plant breeding programs of tomorrow we cannot afford to ignore any source of useable genes.”
Introduction générale
1
Indéniablement, les merveilles de l'amélioration ont eu comme conséquence l'érosion de la diversité génétique de beaucoup de récoltes, y compris le blé, à cause de l’utilisation des mêmes génotypes parentaux et l’introduction des techniques modernes pour les besoins de la sélection. Cette érosion a rendu les cultures du blé de plus en plus vulnérables à de nouveaux stress biotiques et abiotiques. L’insuffisance de la variabilité des blés cultivés peut être résolue par l’exploitation des formes sauvages apparentées qui constituent des ressources importantes de gènes utiles pour l’amélioration de la qualité des produits finis et l’adaptation des plantes cultivées aux contraintes environnementales.
L’amélioration d’une espèce cultivée repose en grande partie sur la gestion et l’exploitation de la variabilité génétique qu’elle présente. Elle consiste à créer une variabilité génétique nouvelle, puis sélectionner et fixer parmi cette diversité les génotypes intéressants.
Les espèces apparentées au blé qui appartiennent à la tribu des Triticées et en particulier les espèces du genre Aegilops, ont servi comme un réservoir génétique précieux dans les perspectives de l’amélioration technologique des blés cultivés. En effet, elles sont des donneurs potentiels de gènes à intérêt agronomique tel que : la tolérance au stress abiotique, la résistance aux maladies et le polymorphisme des protéines de réserve (gluténines et gliadines).
A cet égard, les études de diversité génétique ont été accentuées sur les ancêtres diploïdes du blé (donneurs du génome B : espèces de la section Sitopsis et donneur du génome D : Aegilops
tauschii). Néanmoins, ces ancêtres, comme la majorité des espèces diploïdes d’Aegilops
présentent une aire de répartition limitée (van Slageren 1994). Par conséquent, l’utilisation d’espèces polyploïdes d’Aegilops largement distribuées est fortement préconisée.
L’espèce allotétraploïde Aegilops geniculata Roth est l’une des espèces les plus répandues du genre (van Slageren 1994), montrant ainsi une vaste capacité d’adaptation aux contraintes de l’environnement. En outre, cette espèce a montré son pouvoir de résistance aux maladies et aux insectes (Gill et al. 1985 ; Dimov et al. 1993) et à la salinité (Farooq et al. 1996), ce qui fait d’elle une source potentielle de gènes de résistance à ces stress. En Algérie, Ae. geniculata se trouve dans des groupes morphologiques suivant une large gamme de conditions climatiques et géographiques, allant des plaines côtières au nord jusqu’aux hauts plateaux steppiques au sud, incluant les montagnes de l’Atlas Tellien.
La mise en valeur de l’apport des ressources phytogénétiques dans l’amélioration des blés cultivés ne peut être réalisé qu’après une analyse du polymorphisme de ces ressources. L’évaluation ayant pour objectif de décrire la diversité génétique fait souvent appel à des marqueurs morphologiques, biochimiques (protéines de réserve et isozymes) ou moléculaires
2
(marqueurs à ADN). Les récents développements des biotechnologies offre la possibilité d’établir de nouvelles approches pour améliorer les stratégies de sélection.
Ce travail s’inscrit dans ce contexte, il a pour objectif la valorisation de cette ressource phytogénétique apparentée au blé et l’appréciation de sa diversité génétique par l’analyse du polymorphisme des marqueurs morphologiques, biochimiques (sous unités gluténines de haut et de faible poids moléculaire et gliadines) et moléculaires (RAPD). L’étude est réalisée sur différentes accessions de l’espèce Ae. geniculata collectées dans plusieurs régions écogéographiques à travers le nord Algérien. Ensuite, l’association de quelques paramètres climatiques et altitudinales avec la diversité génétique des accessions analysées par les trois approches (morphologique, biochimique et moléculaire) sera évaluée.
Le manuscrit de thèse est structuré en trois parties. La première partie dresse une brève présentation du genre Aegilops et de l’espèce d’étude (Ae. geniculata) ainsi que leurs relations phylogénétiques avec le blé et leurs intérêts dans l’amélioration des blés cultivés, tout en donnant des généralités sur les caractéristiques biochimiques et génétiques du grain d’Aegilops. Les types de marqueurs génétiques utilisés dans l’analyse du polymorphisme des plantes et leur intérêt dans la sélection du blé à travers les Aegilops sont aussi abordés. La deuxième partie quant à elle, présente les résultats qui sont structurés en 5 chapitres sous forme d’articles. Et enfin, une synthèse générale des résultats obtenus et les perspectives dégagés de cette étude sont présentés dans la troisième partie.
PREMIERE PARTIE
3
I- Le genre Aegilops I-1- Présentation
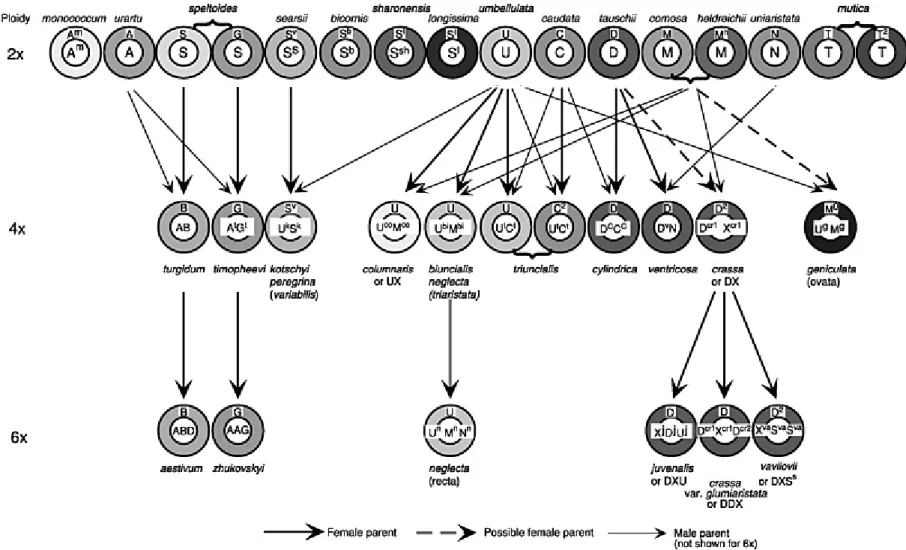
Le mot Aegilops est dérivé de deux mots grecs, Aegos = chèvre, ôps = œil, car les Aegilops ont été utilisés pour la première fois par les agriculteurs de la Grèce antique, pour leurs propriétés curatives. Selon Dodoeus (1644, cité par van Slageren 1994), ils permettent de guérir une maladie oculaire des chèvres. Le genre Aegilops appartient à la tribu Triticeae de la famille Gramineae (Poaceae). Cette tribu comprend six grands genres dont les plus importants et cultivés sont : Triticum (blé), Hordeum (orge) et Secale (seigle). Aegilops prend une position intermédiaire entre le genre Triticum et Agropyrum (Zhukovsky 1928 dans Raskina et al. 2004).
I-2- Historique de la classification
Les Aegilops sont classés par Emberger et Chadefaud (1960) comme suit : Règne : Végétal
Embranchement : Spermaphytes Sous embranchement : Angiospermes Classe : Monocotylédones
Ordre : Glumiflorales Famille : Gramineae Tribu : Triticeae
Sous - tribu : Tritinaceae Genre : Aegilops
Linné fut l’un des premiers à décrire le genre Aegilops en 1737 (Raskina et al. 2004). Dans son ouvrage Species Plantarum (1753), il a décrit ce genre en proposant une classification en cinq espèces : Ae. ovata L., Ae. caudata L., Ae. triuncialis L., Ae. squarrosa L. et une cinquième espèce Ae. incurva L. considérée depuis comme espèce appartenant au genre
Parapholis. Plus tard, plusieurs auteurs ont classé le genre en groupes et sous-groupes, où le
nombre d’espèces passe à 11 regroupées en six sous-groupes par Jaubert et Spach 1851. Longtemps après et en se basant sur les études des caractères morphologiques et la distribution géographique des espèces du genre Aegilops, Zhukovsky (1928) et Eig (1929) ont divisé le genre Aegilops en 20 espèces classées dans 9 sections. Vers les années 40, les études de cytogénétique ont permis de proposer de nouvelles classifications. En effet, Kihara (1940, 1954) proposa une classification des Aegilops basée sur une analyse des génomes et donna des formules pour les différents génomes. Stebbins (1956), par contre, a proposé de fusionner
Tableau 1 : Aperçu sur les importantes classifications des Aegilops
Génome van Slageren (1994) Kimber et Sears (1987) Whitcombe (1983) Hammer (1980) Kihara (1954) Zhukovsky (1928) Section Aegilops L. Section Polyeides Zhuk. Section Aegilops Section Polyeides Zhuk.
U Ae. umbellulata Zhuk. T. umbellulatum (Zhuk.)
Bowden Ae. umbellulata Zhuk. Ae. umbellulata Zhuk. Ae. umbellulata Zhuk. Ae. umbellulata Zhuk.
UM Ae. columnaris Zhuk. T. columnare (Zhuk.)
Morris & Sears Ae. columnaris Zhuk. Ae. columnaris Zhuk. Ae. columnaris Zhuk Ae. columnaris Zhuk.
UM Ae. biuncialis Vis. T. macrochaetum (Shuttl.
& Huet) Richter Ae. lorentii Hochst Ae. lorentii Hochst. Ae. biuncialis Vis. Ae. biuncialis Vis.
UM Ae. geniculata Roth T. ovatum (L.) Raspail Ae. ovata L. Ae. geniculata Roth Ae. ovata L. Ae. ovata L.
UM / UMN Ae. neglecta Req. ex
Bertol. (4x et 6x) T. triaristatum (Willd.) Godr. & Gren. (4x et 6x) Ae. triaristata Willd. (4x et 6x) Ae. neglecta Req. ex Bertol. ssp. neglecta (4x) ssp. recta (Zhuk.) Hammer (6x)
Ae. triaristata Willd. (4x
et 6x) Ae. triuncialis L.
UC Ae. triuncialis
var. triuncialis
var. persica (Boiss.) Eig
T. triunciale (L.) Raspail Ae. triuncialis L. Ae. triuncialis L.
ssp. triuncialis
ssp. persica (Boiss.) Zhuk.
Ae. triuncialis L. Ae. triuncialis L.
US Ae. kotschyi Boiss. T. kotschyi (Boiss.)
Bowden Ae. kotschyi Boiss Ae. kotschyi Boiss. Ae. kotschyi Boiss.
US Ae. peregrina (Hack. in J.
Fraser) Marie & Weiller var. peregrina
var. brachyathera (Boiss) Marie & Weiller
T. kotschyi (Boiss.)
Bowden Ae. peregrina (Hackel) Maire et Weiller Ae. peregrina (Hackel) Maire et Weiller Ae. variabilis Eig
Section Comopyrum
(Jaub. & Spach) Zhuk. (Jaub. & Spach) Zhuk. Section Comopyrum Section Comopyrum (Jaub. & Spach) Zhuk. emend. Senjan.-Korcz.
Section Comopyrum (Jaub. & Spach)
Zhuk. M Ae. comosa Sm. in Sibth.
& Sm. var. comosa
var. subventricosa Boiss.
T. comosum (Sibth. &
Sm.) Richter Ae. comosa Sibth. & Sm. Ae. comosa Sibth. & Sm. ssp. comosa ssp. heldreichii (Boiss.) Eig
Ae. comosa Sibth. & Sm. Ae. comosa Sibth.
et Sm.
N Ae. uniaristata Vis. T. uniaristatum (Vis.)
Richter Ae. uniaristata Vis. Ae. uniaristata Vis. Ae. uniaristata Vis. Ae. uniaristata Vis.
Section Cylindropyrum
(Jaub. & Spach) Zhuk. (Jaub. & Spach) Zhuk. Section Cylindropyrum (Jaub. & Spach) Zhuk. Section Cylindropyrum emend Kihara
Section Cylindropyrum (Jaub. & Spach) Zhuk. C Ae. caudata L. T. dichasians (Zhuk.)
Bowden Ae. caudata L. Ae. markgrafii (Greuter) Hammer Ae. caudata L. Ae. caudata L.
S Ae. speltoides Tausch
var. speltoides
var. ligustica (Savign.) Fiori
T. speltoides (Tausch)
Gren. ex Richter Ae. speltoides Tausch Ae. ligustica (Savign.)
Coss.
Ae. speltoides Tausch ssp. speltoides
ssp. ligustica (Savign.) Zhuk
Ae. speltoides Tausch Ae. speltoides
Tausch
Sb Ae. bicornis (Forssk.).
Jaub. & Spach var. bicornis var. anathera Eig
T. bicorne Forssk Ae. bicornis (Forssk.)
Jaub. & Spach Ae. bicornis (Forssk.) Jaub. & Spach Ae. bicornis (Forssk.) Jaub. & Spach Ae. bicornis (Forssk.) Jaub. et Spach
Sl Ae. longissima
Schweinf. & Muschl. T. longissimum (Schweinf. & Muschl.) Bowden
Ae. longissima
Schweinf. & Muschl. Ae. longissima Schweinf. & Muschl. emend. Eig s. l. ssp. longissima
ssp. sharonensis (Eig) Hammer
Ae. longissima
Schweinf. et Muschl. Ae. longissima (Schweinf. Et Muschl.) Eig
Ss Ae. searsii Feldman &
Kislev ex Hammer T. searsii (Feldman & Kislev) Feldman, comb. nov.
T. searsii Feldman &
Kislev Ae. searsii Feldman & Kislev ex Hammer
Ssh Ae. sharonensis Eig Ae. sharonensis Eig Ae. sharonensis Eig
Section Vertebrata Zhuk.
emend. Kihara Section Vertebrata Zhuk. Section Vertebrata Zhuk. emend. Kihara Section Vertebrata Zhuk. D Ae. tauschii Coss. T. tauschii (Coss.)
Schmalh. Ae. squarrosa L. Ae. tauschii Coss. Ae. squarrosa L. Ae. squarrosa L.
DMU Ae. juvenalis (Thell).Eig T. juvenale Thell. Ae. juvenalis (Thell).
Eig Ae. juvenalis (Thell). Eig Ae. juvenalis (Thell). Eig Ae. turcomanica Roshev.
DM/DU Ae. turcomanica Roshev.
DMS Ae. vavilovii (Zhuk.)
Chennav. T. syriacum Bowden Ae. vavilovii (Zhuk.) Chennav
DM/
DDM Ae. crassa Boiss. (4x et 6x) T. crassum (Boiss.) Aitch. & Hensl. (4x et 6x)
Ae. crassa Boiss. (4x
et 6x) Ae. crassa Boiss. (4x et 6x) Ae. crassa Boiss. (4x et 6x) Ae. crassa Boiss. (4x and 6x)
DN Ae. ventricosa Tausch T. ventricosum Ces. Ae. ventricosa Tausch Ae. ventricosa Tausch Ae. ventricosa Tausch Ae. ventricosa
Tausch
Sous genre Amblyopyrum
(Jaub. & Spach) Eig* Section Amblyopyrum (Jaub. & Spach) Zhuk. Sous genre Amblyopyrum Jaub. et Spach Section Amblyopyrum (Jaub. & Spach) Zhuk.
Amblyopyrum muticum
(Boiss.) Eig
T. tripsacoides
(Jaub. & Spach) Bowden Ae. mutica Boiss. Ae. mutica Boiss. Ae. mutica Boiss. Ae. mutica Boiss.
T var. muticum
var. loliaceum (Jaub. & Spach) Eig
var. mutica
var. loliacea (Jaub. & Spach) Eig
Revue bibliographique
4
barrières génétiques entre les deux (possibilité d’établir des croisements entre les espèces des deux genres). En 1959, Bowden a soutenu cette proposition de classification qui a suscité un débat qui dure jusqu’à nos jours. Cette révision taxonomique a été soutenue et expliquée par Chennaveeraiah (1960). Toutefois, Bor (1968) l’a controversée. En 1980, Hammer publia une révision complète du genre Aegilops en le divisant en trois sous genres répartis en quatre sections, 21 espèces et 105 taxons (Hammer 1980a, b). Löve (1982-1984) donna dans la tribu des Triticeae, un statut générique à chaque génome ou constitution génomique. Whitcombe (1983) classa Triticum et Aegilops dans deux groupes morphologiques distincts. Par conséquent, cette classification a été suivie par beaucoup de scientifiques et de nouveaux noms ont été donnés aux espèces diploïdes et polyploïdes (Kimber et Sears 1983 ; Kimber et Feldman 1987). La classification la plus récente est la révision de van Slageren (1994) basée sur des caractères morphologiques. van Slageren a fait des prospections et des collectes entre les années 1988 et 1994, et a examiné 20.000 échantillons d’herbier qui représentent selon lui 75-85% des Aegilops disponibles. Cette classification rejoint celle de Mac key (1966), et regroupe deux genres, le genre Aegilops divisé en cinq sections (Aegilops, Cylindropyrum,
Vertebrata, Comopyrum et Sitopsis), représenté par 22 espèces et 5 variétés, et le genre Amblyopyrum comprenant une seule espèce (au total 23 espèces, dont 11 diploïdes et 12
polyploïdes).
Depuis la découverte des Aegilops par Linné jusqu’à aujourd’hui, plusieurs classifications contradictoires ont été proposées. En effet, les avis entre botanistes et généticiens sont divergents. Le problème majeur réside dans le fait de classer les Aegilops avec les Triticum ou les traiter séparément et vice versa, ce qui a conduit à une grande variété de nomenclature (Tableau 1).
I-3- Génétique des Aegilops I-3-1- La polyploïdie
La polyploïdie, ou l’assortiment de plusieurs jeux complets de chromosomes dans un noyau, est un facteur important dans l’évolution des génomes des eucaryotes (polyploïdisation), et constitue un mécanisme de diversification et de création de variabilité génétique. Le terme de polyploïdie désigne les individus présentant plus de deux jeux haploïdes (2n) de chromosomes (tétraploïde : 2n= 4x, hexaploïde : 2n=6x etc.). Elle est fréquente chez les plantes, particulièrement les angiospermes. La polyploïdie peut avoir deux formes :
Revue bibliographique
5
Une autopolyploïdie : résultant de l’addition d’un ou de plusieurs jeux de chromosomes identiques au jeu haploïde de l’espèce, autrement dit ; les gamètes viennent de la même espèce voire du même individu (autofécondation).
Une allopolyploïdie : plus fréquente, générée par la combinaison de deux jeux de chromosomes d’espèces différentes suite à un croisement interspécifique c’est-à-dire les gamètes viennent d’individus d’espèces différentes mais suffisamment proches pour s’hybrider. Quand les deux espèces originales sont connues, le terme amphiploïdie est préféré (Klug et al. 2006).
Dans le cas de l’allopolyploïdie, l’individu hybride ayant une copie (un lot) de chromosomes de chaque espèce est en général stérile, puisque les chromosomes ne s’apparient pas correctement au cours de la méiose. Cependant de tels individus peuvent devenir fertiles à la suite d’un doublement du stock chromosomique suite à une anomalie (méiose anormale). Par conséquent, l’individu aura deux copies de chaque assortiment chromosomique, l’appariement au cours de la méiose serait possible et la reproduction sexuée redevient possible (gamètes haploïdes viables) (Figure 1). L'hybride résultant des gamètes de ce type serait directement tétraploïde et fertile (Raven et al. 2011).
I-3-2- Constitution génomique des espèces d’Aegilops
Les genres Triticum et Aegilops appartiennent à un complexe d’espèces sauvages et domestiquées, dont les membres allopolyploïdes ont évolué par spéciation des hybrides (Kimber et Sears 1987). Les études cytogénétiques de Kihara (1937) et Sears (1941), sur plusieurs espèces d’Aegilops ont démontré que leur nombre chromosomique de base (x=7) est le même que celui du blé avec les mêmes niveaux de ploïdie. En plus, les sept groupes d’homologie définis chez le blé sont homologues à ceux définis chez le genre Aegilops.
Aegilops regroupe 11 espèces diploïdes (2n =2x = 14) et 12 polyploïdes (tétraploïdes : 2n= 4x= 28 ; hexaploïdes : 2n= 6x= 42). Ae. crassa et Ae. neglecta présentent les deux types de
ploïdie (tétraploïdes et hexaploïdes) (van Slageren 1994). Le genre est divisé en cinq sections ; les deux sections Comopyrum et Sitopsis renferment des espèces diploïdes uniquement et les trois sections Aegilops, Cylindropyrum et Vertebrata regroupent des espèces diploïdes et polyploïdes.
La section Comopyrum (Jaub. & Spach) Zhuk. regroupe les espèces diploïdes portant le
génome M ou le génome N : Ae. comosa (M) et Ae. uniaristata (N).
La section Sitopsis (Jaub. & Spach) Zhuk. regroupe les espèces diploïdes portant le génome S : Ae. bicornis (Sb), Ae. longissima (Sl), Ae. sharonensis (Ssh), Ae. searsii (Ss) et Ae.
Section Espèces diploïdes G C Espèces tétraploïdes G C Espèces héxaploïdies G C Aegilops L. Ae. umbellulata U U Ae. biuncialis UM U
Ae. columnaris UM U2 Ae. geniculata MU M0 Ae. kotschyi SU S2 Ae. neglecta ssp. neglecta UM U Ae. peregrina
Ae. triuncialis UC, SU CU
S2
C,U Ae. neglecta ssp. recta UMN U
Comopyrum (Jaub. et
Spach) Zhuk. Ae. comosa Ae. uniaristata M N M N Cylindropyron (Jaub.
et Spach) Zhuk. Ae. markgrafii C C
Sitopsis (Jaub. et
Spach) Zhuk Ae. bicornis S
b Sb Ae. longissima Sl Sl Ae. sharonensis Ssh Sl Ae. searsii Ss Ss Ae. speltoides S S Vertebrata Zhuk. emend. Kihara Sous genre Amblyopyrum Ae. tauschii Ae. mutica D T D nd Ae. crassa ssp. crassa (4x) DM DN D2 D
Ae. crassa ssp. crassa
(6x) DDM D2
Ae. ventricosa Ae. vavilovii DMS D2
Ae. juvenalis DMU D2
Revue bibliographique
6
La section Aegilops L. comprend les espèces portant le génome U et les combinaisons d’autres génomes avec le génome U : Ae. umbellulata (U), Ae. biuncialis (UM), Ae.
columnaris (UM), Ae. geniculata (MU), Ae. kotschyi (SU), Ae. neglecta (UM, UMN), Ae. peregrina (SU) et Ae. triuncialis (UC).
La section Cylindropyrum (Jaub. & Spach) Zhuk. renferme les espèces avec le génome
C et les génomes DC : Ae. markgrafii (C) et Ae. cylindrica (DC).
La section Vertebrata Zhuk. emend. Kihara : regroupe les espèces avec le génome D et les combinaisons d’autres génomes avec le génome D : Ae. tauschii (D), Ae. crassa (DM, DDM), Ae. ventricosa (ND), Ae. vavilovii (DMS) et Ae. juvenalis (DMU).
Selon la dernière clé de van Slageren (1994), le sous genre Amblyopyrum (Jaub. & Spach) Eig est inclus dans le genre Aegilops et est représenté par une seule espèce diploïde : Ae.
mutica (génome T). Le tableau 2 représente les espèces de chaque section et leurs formules
génomiques.
I-3-3- Évolution des génomes des espèces polyploïdes d’Aegilops
Les espèces polyploïdes du genre Triticum et Aegilops constituent un exemple classique de l’évolution par amphiploïdie. Des études sur les hybrides suggèrent que l’hybridation interspécifique a joué un rôle significatif dans le processus de divergence intraspécifique des espèces polyploïdes du genre Aegilops (Zohary et Feldman 1962).
Toutes les espèces diploïdes du genre Aegilops possèdent des génomes bien distincts qui peuvent être facilement différenciés sur la base des caractères morphologiques de la plante, contrairement aux espèces polyploïdes où les différences morphologiques ne sont pas claires entre les espèces (Zohary et Feldman 1962 ; Zohary 1966).
Les recherches entreprises sur l’origine des espèces polyploïdes du genre Aegilops ont montré que certaines espèces polyploïdes ont des génomes très similaires à leurs géniteurs (parents) diploïdes, alors que ces génomes sont modifiés dans les autres espèces polyploïdes (Kihara 1954). Ce même auteur a suggéré que les espèces diploïdes donneuses sont soit inexistantes (ont disparu), soit sont encore présentes et leurs génomes ont été modifiés par réarrangement durant l’évolution pour donner les génomes des espèces polyploïdes. Selon une hypothèse alternative (Zohary et Feldman 1962 ; Kimber et Feldman 1987), le degré de modification du génome parental dans les espèces polyploïdes est différent. Le phénomène de spéciation par polyploïdisation a été donc accompagné de substitutions chromosomiques entières et/ou partielles. En règle générale, l’un des génomes est très proche voir identique au génome parental, appelé « génome pivot » ou génome invariable, alors que le deuxième (ou le troisième) est modifié « génome différentiel » ou variable. Autrement dit ; le génome pivot
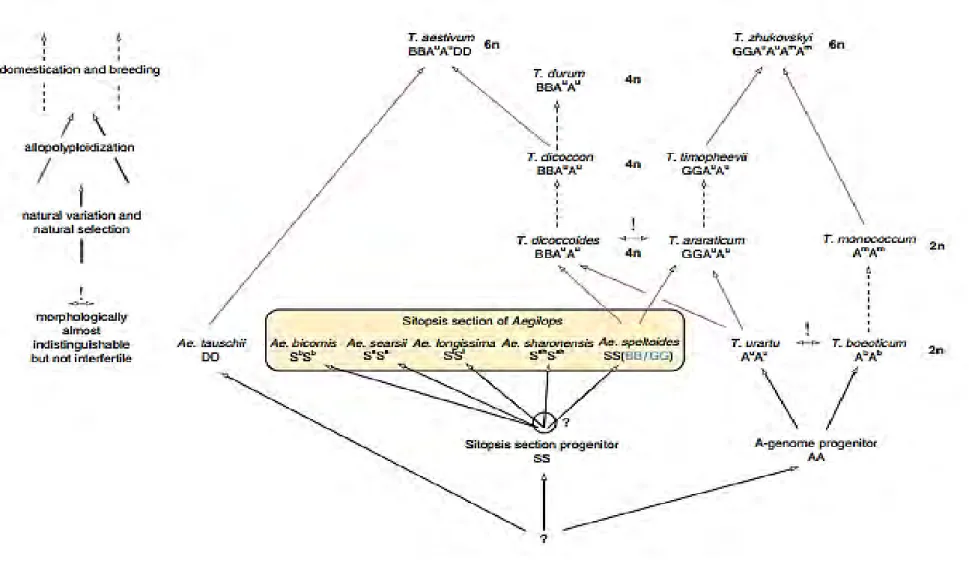
Figure 2 : Les espèces des genres Triticum et Aegilops constituent un exemple classique de spéciation par polyploïdie. Les polyploïdes constituent les trois clusters : A, D et U (Gill et al. 2006)
Revue bibliographique
7
joue le rôle d’un tampon génétique qui facilite l’hybridation et assure la fertilité dans l’hybride résultant, alors que la recombinaison aura lieu entre les génomes variables. Trois génomes “pivot” ont été identifiés dans le complexe Triticum/Aegilops (A, U et D). Ceci a permis la classification de toutes les espèces polyploïdes du complexe en trois clusters. Chaque cluster renferme des espèces polyploïdes ayant un génome pivot (A, U ou D) et un deuxième (ou bien un troisième) génome variable (M, N, C et S). Toutes les espèces diploïdes et polyploïdes du blé sont inclues dans le cluster « génome A ». Le cluster « génome-D » regroupe le diploïde Ae. tauschii Coss. et les cinq espèces polyploïdes contenant le génome D. Le cluster « génome-U » inclut l’espèce diploïde Ae. umbellulata Zhuk. et sept espèces polyploïdes portant ce génome (Figure 2) (Morris et Sears 1967 ; Kimber et Feldman 1987).
I-4- Aire de répartition des Aegilops
La zone d’extension naturelle du genre Aegilops est localisée entre 10° à l’ouest et 82° à l’est, et entre 24° et 47° au nord (Kilian et al. 2011). Selon Hammer (1980a), le genre Aegilops est originaire de la Transcaucasie et l’espèce la plus primitive Ae. speltoides a été identifiée au centre de la région. Kimber et Feldmen (1987) rapportent que tous les diploïdes (à l’exception d’Ae. tauschii) ont une aire de distribution limitée alors que les tétra- et hexa- ploïdies ont un potentiel d’adaptation plus grand et par conséquent ont une large distribution. Ces mêmes auteurs mentionnent que les Aegilops se développent en association avec d’autres graminées dans des environnements dégradés (pâturages, bords des routes et des champs cultivés, sur les formations d’arbrisseaux et les formations herbacées, etc.). D’après van Slageren (1994), les 22 espèces d’Aegilops sont distribuées principalement dans le bassin méditerranéen et au centre et au sud-ouest de l’Asie. La distribution altitudinale du genre est comprise entre 400 et 2700 m mais elle varie énormément entre les espèces (Hodgkin et al. 1992 ; van Slageren 1994). Les espèces se répartissent également dans les régions centrales de l’Europe et au nord du Sahara en Afrique. Kimber et Feldmen (1987) rapportent que la région du croissant fertile est considérée comme le centre primitif de la variabilité des espèces du genre Aegilops et Triticum, allant de la Palestine, le Liban, la Syrie, le sud-est de la Turquie et le nord de l’Irak vers le ouest de l’Iran. La région du nord-ouest de la Jordanie, la Palestine, le Liban, l’nord-ouest de la Syrie, l’Irak et la Turquie renferme plus de neuf espèces d’Aegilops. Deux zones ont été trouvées à avoir 12 à 14 espèces d’Aegilops : (1) l’ouest de la Syrie et le nord est du Liban et (2) le nord de l’Irak (van Slageren 1994 ; Maxted et al. 2008). Jusqu’à maintenant, ce centre préserve son potentiel de spéciation (Zhucovsky 1928 dans Raskina et al. 2004). Ces mêmes auteurs mentionnent que les zones de répartition des espèces Ae. longissima, Ae. searsii et Ae. sharonensis chevauchent la partie
8
sud-ouest de la zone de répartition d’Ae. speltoides. Il semblerait que ces espèces substituent
Ae. speltoides dans le sud où l’environnement climatique et édaphique est particulier. Ces
mêmes auteurs proposent Ae. speltoides comme espèce ancestrale de la section Sitopsis. Le diploïde Ae. longissima est considéré comme une espèce endémique à la Palestine et le sud du Liban.
Selon la prospection réalisée par l'ICARDA (International center for agricultural research in the dry areas) entre 1989 et 1990, les espèces représentatives du genre Aegilops en Algérie sont les espèces allotétraploïdes suivantes : les plus dominantes sont Ae. geniculata Roth (MU) et Ae. ventricosa Tausch (DN), puis Ae. triuncialis L. (UC) et d’un degré moindre Ae.
biuncialis (UM). Des populations mixtes d’Ae. neglecta (UM, UMN) avec Ae. geniculata ont été trouvées en Algérie (Bandou et al. 2009 ; Kilian et al. 2011).
I-5- Intérêts des Aegilops
I-5-1- Contribution dans la constitution des génomes du blé
L’étroite relation entre les deux genres (Aegilops et Triticum) a été établie depuis longtemps puis a confirmé que les Aegilops sont les ancêtres du blé. Les espèces du genre
Triticum comme celles d’Aegilops existent comme des blés diploïdes, tétraploïdes ou
hexaploïdes avec un nombre chromosomique de base n=7. Ensemble, le genre Triticum et
Aegilops regroupent 13 espèces diploïdes et 18 espèces polyploïdes. Les espèces diploïdes
contiennent huit génomes distincts : le génome A (A (Au) et Ab/Am) pour le genre Triticum et sept génomes D, S (S, Ss, Sb, Sl, Ssh), M, C, U, N et T pour le genre Aegilops (Figure 2). Les deux génomes B et G trouvés dans les espèces polyploïdes du blé sont nommés ainsi (différemment des génomes de base) parce que leurs donneurs diploïdes ne sont pas encore bien élucidés (Golovnina et al. 2007).
Aperçu sur les espèces sauvages et cultivées du blé
Les espèces diploïdes du blé sont : l’engrain sauvage T. urartu (AA ou AuAu) et T. monococcum avec ses deux formes ; sauvage (ssp. Aegilopoides, syn. T. boeticum, genome AbAb) et domestiquée (T. monococcum ssp. monococcum, genome AmAm). Les espèces polyploïdes du blé résultent des évènements d’hybridation interspécifiques suivis par le doublement chromosomique spontané. Il existe deux espèces tétraploïdes du blé : l’amidonnier T. turgidum (AABB) et T. timopheevii (AAGG) avec leurs sous espèces sauvages et cultivées. Les espèces hexaploïdes du blé sont T. eastivum (AABBDD) et T.
zhukovskyi (AmAmAAGG) qui sont toutes les deux des espèces domestiquées. Les études génétiques ont révélé qu’il existe deux lignées évolutives polyploïdes du blé : la première étant composée de T. turgidum et T. aestivum et la deuxième renferme T. timopheevii et T.
9
zhukovskyi. La culture moderne du blé est essentiellement basée sur deux espèces
allopolyploïdes : le blé dur allotétraploïde (2n=4x=28, BBAA) et le blé tendre allohexaploïde (2n=6x=42, BBAADD. La formation de ces espèces du blé (la domestication du blé) a impliqué de nombreux évènements de polyploïdisation entre plusieurs espèces des genres
Triticum et Aegilops (Figure 3).
Origine des blés tétraploïdes
L’amidonnier sauvage T. turgidum ssp. dicoccoides (2n=4x=28, BBAA) s’est formée il y a environ 0,5 million d’années par un événement d’allopolyploïdisation entre l’espèce diploïde T. urartu (2n=2x=14, AuAu), progénitrice du génome A (Nishikawa 1983 ; Dvorák et al. 1993 ; Huang et al. 2002) et une espèce de la section Sitopsis, progénitrice du génome B qui reste à identifier (Feldman et al. 1995 ; Blake et al. 1999 ; Feldman 2001 ; Huang et al. 2002 ; Dvorák et al. 2006). L’espèce donneuse la plus proche dans cette section est Ae. speltoides (2n=2x=14, SS) (Feldman et al. 1995 ; Bálint et al. 2000 ; Feldman 2001, Provan et al. 2004). De même, l’hybridation entre l’espèce diploïde sauvage T. urartu (progéniteur du génome A) avec une autre espèce diploïde sauvage progénitrice du génome G, qui demeure inconnue, a donné l’espèce tétraploïde T. timopheevii ssp. armeniacum (AAGG) (Huang et al. 2002). Récemment, les analyses génétiques et moléculaires ont montré que le génome S d’Ae. speltoides est presque identique au génome G de l'espèce tétraploïde
T. timopheevii (Dvorák et al. 1998 ; Huang et al. 2002 ; Golovnina et al. 2007 ; Kilian et al.
2009). L’engrain T. monococcum (2n=2x=14, AmAm) et l’amidonnier T. turgidum spp.
dicoccum (2n=4x=28, BBAA) sont les premières espèces domestiquées du blé qui viennent
respectivement de l’engrain sauvage T. boeoticum (2n=2x=14, AmAm) et de l’amidonnier sauvage T. turgidum ssp. dicoccoides (BBAA). Le blé dur actuellement cultivé est T.
turgidum ssp. durum. Son origine et sa relation avec les premières espèces domestiquées
tétraploïdes du blé (T. turgidum spp. dicoccum) restent mal caractérisées (Charles 2010).
Origine des blés hexaploïdes
Les formes domestiquées du blé (T. monococcum et T. turgidum spp. dicoccum) disséminaient progressivement à travers l’Asie, l’Europe et l’Afrique. L’amidonnier (BBAA) sera ainsi mis en contact avec l’espèce diploïde Ae. tauschii (Coss.) (syn. Ae. squarrosa,
2n=2x=14, DD), donneur du génome D, dans la région allant de l’Arménie à la côte sud-ouest
de la mer Caspienne (Feldman et al. 1995 ; Dvorák et al. 1998 ; Huang et al. 2002). Leur hybridation et doublement chromosomique ont donné le blé tendre allohexaploïde actuellement cultivé T. aestivum ssp. aestivum (2n=6x=42, BBAADD) (Kihara 1944 ; Feldman et al. 1995 ; Nesbitt et Samuel 1996). Le blé hexaploïde cultivé T. zhukovskyi
Tableau 3 : Les principaux gènes de résistance aux stress biotiques identifiés chez les
Aegilops et transférés chez le blé
Gène (s) de Résistance Espèce donneuse
Maladie
Lr 9
Ae. umbellulata Rouille brune (Puccinia
recondita) Ae. speltoides Lr 28, Lr35, Lr36, Lr47, Lr 51 Lr 21, Lr 22, Lr 32, Lr39, Lr40, Lr 41, Lr 42, Lr 43 Ae.tauschii
Lr 37 Ae.ventricosa
Plusieurs gènes de résistance
Ae.geniculata Gènes (Lr Tr) Ae.triuncialis Lr 57 Ae.geniculata Sr 32, Sr 39 Ae .speltoides Rouille noire
(Puccinia graminis) Ae.comosa Sr34
Sr38 Ae .ventricosa Sr 33 Ae.tauschii Yr 8 Ae.comosa Rouille jaune
(Puccinia stiiformis) Ae.tauschii Yr28
Yr 17 Ae.ventricosa Yr 40 Ae.geniculata Pm 12 Ae.speltoides Oïdium
(Erysiphe graminis) Ae.longissima Pm13
Pm32 Ae. speltoides Pm 19, Pm 35 Ae.tauschii Pm 29 Ae .geniculata Pch 1 Ae.ventricosa Piétin verse (Tapesia yallundae)
Parasite Espèce donneuse Gène(s) de Résistance Nématodes à kystes
( Heterodera avenae) Ae. ventricosa Ae. tauschii Cre2,Cre5, Cre6 Cre 3, Cre4 Ae. triuncialis Cre 7
Nématodes à galles
(Meloidogyne naasi) Ae. peregrina Ae. tauschii Rkn-mnl Cécidonsyie
(Mayetiola destructor) Ae. tauschii H22,H23,H24,H13,H26,
Ae. triuncialis H30 Ae .ventricosa H27 Punaise du blé
(Schizaphis graminum) Ae. tauschii Gb 3, Gb4, Gb7, Gb a, Gbba
Gbca, Gbda, Gbxa, Gbza
Ae. speltoides Gba Gbx Gb5
10
(AmAmAAGG) s’est formé de l’hybridation induite entre le tétraploïde T. timopheevii (AAGG) et l’espèce diploïde T. monococcum (AmAm) (Dvorák et al. 1993).
I-5-2- Amélioration des blés cultivés
Le blé est considéré comme la céréale la plus importante économiquement et historiquement. Indéniablement, les merveilles de l'amélioration ont eu comme conséquence l'érosion de la diversité génétique du blé à cause de l’usage fréquent des mêmes génotypes parentaux. L’existence chez les espèces sauvages apparentées au blé d’un important réservoir de gènes utiles est évidente si l’on prend en compte l’adaptation de ces diverses espèces à des environnements très différents. Les Triticées sauvages constituent donc des ressources importantes de variabilité génétique. Elles sont porteuses de nombreux gènes à fort potentiel économique qui interviennent dans des caractères tels que la résistance aux maladies, la tolérance au stress abiotique et la qualité technologique. Les espèces du genre Aegilops constituent une bonne partie du génome du blé, puisqu’ils sont impliqués dans l’évolution des blés polyploïdes. En effet, leurs génomes présentent une grande affinité avec ceux des blés et donc peuvent être efficacement exploités dans les opérations de transfert de gènes (Schneider at al. 2008).
Amélioration de la résistance et/ou la tolérance aux stress biotique et abiotique
Beaucoup d’Aegilops sont connues porteuses de nombreux gènes de résistance aux bioagresseurs les plus néfastes aux cultures et pour lesquels les moyens de lutte actuels ne donnent pas satisfaction, soit par manque d’efficacité, soit parce qu’ils sont préjudiciables à l’environnement (Srivastata et Damania 1989). Il s’agit principalement des rouilles, de l’oïdium, du piétin verse, des fusarioses, des pucerons et des virus qu’ils transmettent et des nématodes. Les principaux gènes de résistance aux maladies et aux insectes (Gill et al. 1983, 1985, 1987 ; Raupp et al. 2001 ; Zaharieva et al. 2001) à la rouille et l’oïdium (Damania et Pecetti 1990 ; Dimov et al. 1993) transférés chez les Aegilops sont présentés dans le tableau 3. De plus, les Aegilops portent des gènes de haute tolérance aux différents stress abiotiques (Tableau 4) en particulier la tolérance à la sécheresse et à la salinité (Molnár et al. 2004 ; Damania et al. 1992 ; Farooq et al. 1989, 2001 ; Colmer et al. 2006).
Amélioration de la qualité technologique du blé
Branlard et Autran (1986) définissent la notion de qualité comme étant un caractère polygénique qui est à la fois dépendant des variétés, des conditions de culture et de l’interaction génotype × milieu. La qualité du grain de blé recouvre un grand nombre de critères : la disponibilité du produit, des critères physiques, technologiques et sanitaires (Gatel
Tableau 4 : Les espèces d’Aegilops considérées comme source potentielle de résistance aux stress abiotiques
Stress abiotique Espèce Déficit hydrique
Ae. columnaris, Ae. geniculata, Ae. kotschyi, Ae. longissima, Ae. peregrina, Ae. sharonensis, Ae. tauschii, Ae. triuncialis, Ae. umbellulata
Froid Ae. cylindrica, Ae. neglecta, Ae. tauschii, Ae. geniculata Ae. triuncialis, Ae. umbellulata
Stress salin Ae. tauschii, Ae. comosa, Ae, umbellulata,Ae. geniculata, Ae.
Trait exploitable Espèces
donneuses Gène étudié/position chromosomique Aconitase Ae. longissima Aco-S (6S)
Ae. umbellulata Beta-Amy-U1
Tolérance à
l’aluminium Ae. uniaristata Endopeptidase Ae. bicornis
Ae. longissima Ep-S (7S)
Ae. umbellulata Ep-U1 (1U)
Pourcentage
protéine du grain Ae. longissima Protéine de dureté
du grain Ae. bicornis, longissima, searsii, sharonensis, speltoides
Pina et PinbS1
, Sbl, Sl1, Ss, Ssh Protéine de
tendreté du grain Ae. tauschii GspD1 Poids du grain Ae. longissima
Tolérance à la chaleur (Phase végétative) Ae. longissima, searsii, speltoides HPM, FPM et
gliadines Ae. tauschii, longissima, umbellulata
Glu-D1, Glu-Sl,
Glu-U, Gli-D, Gli-Sl,
Gli-U
Régions
organisatrices de nucléole
Ae. umbellulata Nor-U1 (1U)
Ae. umbellulata Nor-U3 (5U)
Ae. speltoides Nor-2 (6S)
Peroxydase Ae. longissima Per-S (2S)
Inhibiteur de
subtilisine Ae. longissima Ae. umbellulata Si-S (1S) Si-U2 (1U)
Tolérance à la
toxicité au Mn Ae. speltoides Tolérance a la
déficience en Zinc Aegilops species with U-genome
Inhibiteur de
Revue bibliographique
11
et al. 1999). Le plus important des critères technologiques est la teneur en protéines, qui est corrélée à l’aptitude à la panification. Cette aptitude est déterminée par des tests rhéologiques comme : la force boulangère liée à l’élasticité fournie par les gluténines, l’indice de Harberg déterminée par l’activité amylasique, la viscosimétrie approuvée par la teneur en arabinoxylanes (pentosanes) et la dureté/tendreté du grain chez les blés hexaploïdes etc. (Gatel et al. 1999). Les analyses de la qualité technologique relatives aux protéines de réserve ont montré le rôle de l’ancêtre diploïde Ae. tauschii dans l’amélioration de cette dernière (Hsam et Kieffer 2001 ; Weiser et al. 2003). D’autres études ont été consacrées à l’analyse des gluténines notamment chez les espèces diploïdes donatrices du génome D (Ae. tauschii) et du génome B (espèces de la section Sitopsis) (Cox et Gill 1992). Les études sur les relations phylogénétiques et évolutives du complexe Aegilops/Triticum suggèrent certains degrés d’homologie du génome D avec les génomes U et M (Badaeva et al. 1996 ; Wang et al. 2011). Par conséquent, les études s’étendent de plus en plus aux autres espèces diploïdes et polyploïdes du genre Aegilops. Selon Harlan (1992), les espèces d’Aegilops ayant un génome différent du génome D sont considérées comme un pool génétique secondaire pour le blé, et le transfert de gènes depuis ces espèces nécessite des mises au point dans les manipulations cytogénétiques pour améliorer la recombinaison entre chromosomes homéologues. Plusieurs gènes à intérêt agronomique visant à améliorer la qualité technologique ont pu être transférés depuis les espèces d’Aegilops au blé par hybridation interspécifique. Le tableau 5 englobe les traits liés à la qualité technologique transférés à partir des espèces du genre Aegilops au blé.
*
Les ancêtres du blé (espèces diploïdes) présentent une aire de répartition limitée (van Slageren 1994). Par conséquent, l’utilisation d’espèces largement distribuées est fortement préconisée pour l’amélioration du blé. Parmi les 22 espèces du genre Aegilops, Ae. geniculata Roth (MU) semble particulièrement intéressante dans la résistance aux maladies et aux insectes (Gill et al. 1985 ; Dimov et al. 1993 ; Kuraparthy et al. 2007) et à la salinité (Farooq et al. 1996). Ceci fait d’elle une source potentielle de gènes de résistance à ces stress (Tableau 3). Par ailleurs, c’est l’une des espèces les plus répandues du genre (van Slageren 1994) montrant ainsi une vaste capacité d’adaptation aux contraintes de l’environnement (Tableau 4). Ainsi, une meilleure compréhension du système d’adaptation d’Ae. geniculata permettra une meilleure utilisation dans les programmes d’amélioration du blé (Zaharieva et al. 2001).
(1)
(2)
(3)
Figure 4 : Morphologie d’Ae. geniculata (1) et de ses géniteurs diploïdes : Ae. umbellulata (2) et Ae. comosa (3) (Kilian et al. 2011)
Revue bibliographique
12
II- Présentation de l’espèce Aegilops geniculata
Ae. geniculata Roth (syn. Ae. ovata auct. non L.) est une espèce annuelle autogame
allotétraploïde (2n = 4x = 28) appartenant à la section Aegilops L. (Figure 4.1) avec une formule génomique (MU) (van Slageren 1994). Ses progéniteurs diploïdes sont respectivement, Ae. umbellulata Zhuk. (2n = 2x = 14, UU) de la même section (Figure 4.2) et Ae. comosa Sm.in Sibth. & Sm. (2n = 2x = 14, MM) de la section Comopyrum (Jaub. & Spach) Zhuk. (Figure 4.3).
II-1- Description morphologique
La première clé qui permet de déterminer les espèces du genre et quelques espèces des taxons infraspécifiques est celle de Hammer (1980b) initialement publiée en allemand, et suivie par la version anglaise (Hammer 1982). La dernière clé de van Slageren (1994) contient quelques races infraspécifiques seulement. Nielsen (1981) a ajouté le caractère « longueur du rachis de l’épillet fertile inferieur » pour la distinction entre quelques espèces : ex., Ae.
triuncialis (3–6 mm) et Ae. kotschyi (1–2 mm), Ae. geniculata (4-8 mm) et Ae. umbellulata
(1–3 mm) (Kilian et al. 2011). Selon Hammer (1980b) et van Slageren (1994), l’espèces Ae.
geniculata est présentée comme suit :
La plante adulte est cespiteuse et velue, elle atteint 30 à 40 cm de hauteur (entre 10 et 30 cm sans arêtes). Les feuilles sont glabres ou poilues. La surface de la gaine cylindrique est toujours hérissée. La semence est généralement dispersée avec l’inflorescence entière ou avec un épillet et une partie du rachis. La feuille est plus ou moins poilue. La ligule membraneuse est dentée et les oreillettes sont courtes. L’épi est ovale et court (de 1.2- 1.8 cm sans arêtes), non fragile, d'un vert pâle ou glauque, se rétrécissant généralement après 1 à 3 cm (Figure 4.1) et se désarticulant à maturité. L’épi contient de 2 à 4 épillets dont un ou deux petits supérieurs stériles, deux gros épillets inferieurs fertiles et un ou deux épillets rudimentaires à la base de l'épi. Les épillets sont imbriqués, ovoïdes, brusquement renflés, ventrus au milieu. Les glumes sont enflées au milieu (subventricoses) munis de 4 à 5 arêtes sétacées et scabres de 1.5 à 2.5 cm de longueur, celles-ci sont plus nombreuses aux épillets inférieurs. Les arêtes du lemme (3 à 5 longues arêtes) sont presque aussi longues que celles de la glume. La sous espèce geniculata a des épis plus relâchés avec généralement trois épillets, alors que la sous espèce gibberosa Zhuk. possède des épis plus durs et pas plus de 3 épillets par épi. Des variétés botaniques de cette espèce ont été décrites par Hammer (1980b).
II-2- Le génome d’Aegilops geniculata
L’espèce tétraploïde Ae. geniculata appartient au cluster du génome U. Dans ce cluster, le génome U peut être combiné avec le génome M, c’est le cas des espèces Ae. geniculata Roth.,
Revue bibliographique
13
Ae. biuncialis Vis., Ae. columnaris Zhuk., Ae. neglecta Req. Ex Bertol.. Le génome U de ces
espèces est homologue voire similaire au génome parental d’Ae. umbellulata (génome pivot), alors que le deuxième génome est modifié (génome variable) en comparaison avec le génome de l’espèce parentale (Chennaveeraiah 1960 ; Kimber et Abu Bakar 1981 ; Kimber et Zhao 1983 ; Kimber et Feldman 1987 ; Kimber et Yen 1989). Néanmoins, les deux génomes d’Ae.
triuncialis (UM) ont été trouvés très similaires à ceux de leurs espèces ancestrales (Kihara
1954 ; Chennaveeraiah 1960 ; Kimber et Yen 1989 ; Zhang et Dvorák 1992 ; Dvorák et Dubcovsky 1996). En se basant sur les analyses cytogénétiques (Badaeva et al. 2004), la comparaison d’Ae. geniculata avec ses progéniteurs diploïdes (Ae. umbellulata et Ae. comosa) révèle des différences entre les chromosomes des génomes Mg et Ug par rapport aux génomes parentaux (M et U). Cela montre que les deux génomes M et U ont été modifiés (Kimber et Abu-Bakar 1981 ; Talbert et al. 1993 ; Badaeva et al. 2004). Ces mêmes auteurs rapportent que plusieurs types d’altérations chromosomiques ont survenu durant la spéciation : inactivation des NORs (Nucleolus organizer region) majeurs situés sur les chromosomes du génome M, redistribution des sites de l’ADNr 5S et la perte de quelques loci mineurs d’ADNr 18S – 26S d’Ae. geniculata.
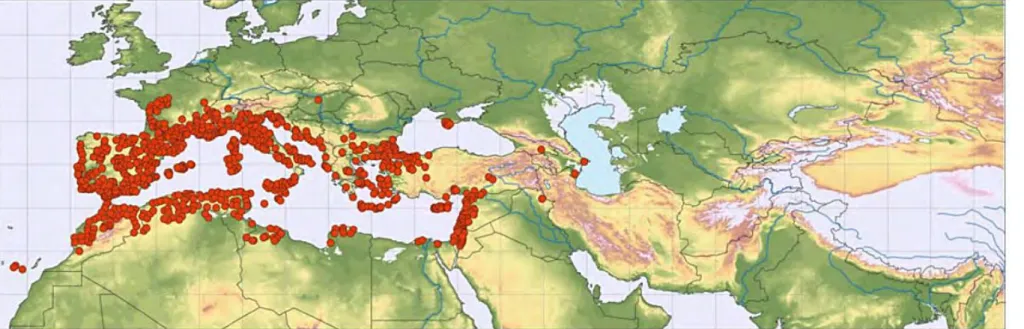
II-3- Distribution géographique
Selon van Slageren (1994), l’espèce est fréquente en Europe du sud, en Égée et au nord d’Afrique, moins fréquente en Turquie et en Transcaucasie, introduite dans les îles Canaries et très rare dans certaines parties du nord-est de l’Europe et l’Europe centrale (Figure 5). En Algérie, Ae. geniculata se trouve, dans une large gamme de conditions climatiques. Cette espèce tétraploïde se répartit sous des conditions écogéographiques différentes, allant des plaines côtières au nord jusqu’aux hauts plateaux steppiques au sud, incluant les montagnes de l’Atlas Tellien. Ae. geniculata peut former des populations mixtes avec Ae. neglacta