—————
Chaˆınes de caract`eres : ´editer une base
bibliographique
J. Lobry & D. Chessel
—————
permet toutes les manipulations sur les chaˆınes de caract`eres. La fiche introduit aux recherches et substitutions avec des expres-sions r´eguli`eres. L’illustration porte sur une bibliographie au format BibTex.
Table des mati`
eres
1 Introduction 2
2 Recherche de chaˆınes de charact`eres 4
2.1 Identifier les d´ebuts de fiche . . . 4 2.2 Trouver une chaˆıne de caract`eres . . . 4 2.3 Rechercher les num´eros manquants . . . 4
3 Substituer une chaine de caract`eres `a une autre 6
3.1 Rechercher un auteur . . . 6 3.2 La distribution du nombre d’articles par auteur . . . 6 3.3 La distribution du nombre d’auteurs par article . . . 6
4 Pour faire des citations dans LATEX 6
1
Introduction
T´el´echarger le fichier http://pbil.univ-lyon1.fr/R/donnees/pco.bib. Ou-vrir ce fichier dans un ´editeur de texte :
pco001,
Author = {Udalova, I.A. and Mott, R. and Field, D. and Kwiatkowski, D.}, Title = {Quantitative prediction of NF-[Kappa;]B DNA-protein interactions}, Journal = {Proceedings of the National Academy of Sciences of the United States of America}, Volume = {99}, Number = {12}, Pages = {8167-8172}, Year = {2002} } ...
Il contient des r´ef´erences bibliographiques au format BibTex :
http://www.ecst.csuchico.edu/~jacobsd/bib/formats/bibtex.html De nombreux outils manipulent ce type de fichier, par exemple le d´ej`a ancien BibEdit sous Windows :
http://www.iui.se/staff/jonasb/bibedit/
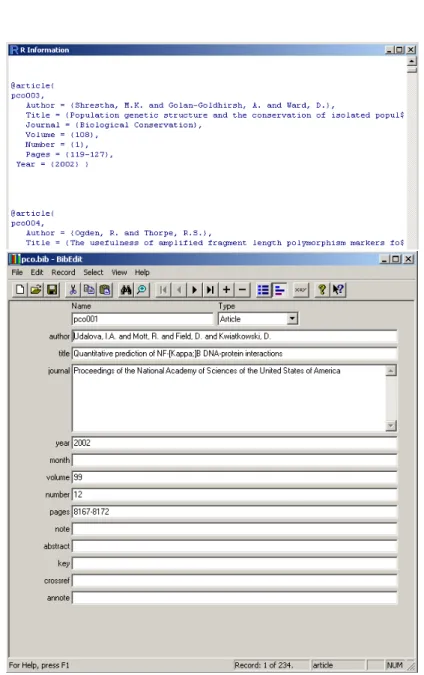
Le logiciel indique qu’il y a 234 r´ef´erences dans le fichier (figure 1). Pour placer le contenu int´egral du fichier texte en m´emoire dans un vecteur de chaˆınes de caract`eres : rm(list = ls()) download.file("http://pbil.univ-lyon1.fr/R/donnees/pco.bib", "pco.bib", mode = "wb") tmp <- readLines("pco.bib") tmp[1:10] [1] "" [2] "@article{" [3] "pco001,"
[4] " Author = {Udalova, I.A. and Mott, R. and Field, D. and Kwiatkowski, D.}," [5] " Title = {Quantitative prediction of NF-[Kappa;]B DNA-protein interactions},"
[6] " Journal = {Proceedings of the National Academy of Sciences of the United States of America}," [7] " Volume = {99}," [8] " Number = {12}," [9] " Pages = {8167-8172}," [10] " Year = {2002} }" nlines <- length(tmp) length(tmp) is.vector(tmp) mode(tmp)
Fig. 1 – Le fichier pco.bib vu par la commande show.file et la premi`ere fiche vue dans le logiciel BibTex qui indique qu’il y a 234 r´ef´erences.
2
Recherche de chaˆınes de charact`
eres
2.1
Identifier les d´
ebuts de fiche
Chaque fiche bibliographique commence par le caract`ere @. Identifier les nu-m´eros de lignes qui commencent par ce caract`ere. Noter la fonction substr (sous-chaˆıne / sub-string).
isEntryLine <- function(line) {
substr(x = line, start = 1, stop = 1) == "@" }
entryLines <- (1:nlines)[sapply(tmp, isEntryLine)] + 1 entryLines[1:20]
[1] 3 16 29 42 55 68 81 94 107 120 133 146 159 172 185 198 211 224 237 250
R´ecup´erer les entr´ees :
entries <- sub(",", "", tmp[entryLines]) entries[1:30]
[1] "pco001" "pco002" "pco003" "pco004" "pco005" "pco006" "pco007" "pco008" "pco009" [10] "pco010" "pco011" "pco012" "pco013" "pco014" "pco015" "pco016" "pco017" "pco018" [19] "pco019" "pco020" "pco021" "pco022" "pco023" "pco024" "pco025" "pco026" "pco027" [28] "pco028" "pco029" "pco030"
Normalement ces entr´ees sont des clefs d’identification et doivent ˆetre uniques. Est-ce le cas ?
any(duplicated(entries)) which(duplicated(entries))
2.2
Trouver une chaˆıne de caract`
eres
Sinon ´editer les r´ef´erences qui ont une entr´ee multiple. Utiliser la fonction grep : grep("pco152", tmp) grep("pco154", tmp) grep("pco354", tmp) tmp[grep("pco154", tmp)] tmp[1976:1990] length(grep("Pages", tmp)) length(grep("@", tmp)) all(entryLines == grep("@", tmp) + 1)
Nous pouvons donc dire qu’il y a 234 entr´ees dans la base bibliographique, mais qu’il y a une ´etiquette pr´esente deux fois. Il va falloir s’en d´ebarasser.
2.3
Rechercher les num´
eros manquants
Si il y a une clef utilis´ee deux fois, l’utilisation d’un codage naturel a laiss´e des clefs non utilis´ee. Pour chercher une chaˆıne de caract`eres, on peut utiliser cette chaˆıne exactement, mais rapidement se pose la question des jokers. C’est le domaine des expressions r´eguli`eres. On ne fera pas le tour de la question ici, mais on indiquera juste que la fonction grep prend en premier argument des expressions r´eguli`eres, c’est `a dire des chaˆınes de caract`eres `a jokers qui permettent de chercher ce qui commence par, finit par, contient ceci ou cela.
cle contient un vecteur de chaˆınes de caract`eres :
[1] "pco001," "pco002," "pco003," "pco004," "pco005," "pco006," [7] "pco007," "pco008," "pco009," "pco010," "pco011," "pco012," [13] "pco013," "pco014," "pco015," "pco016," "pco017," "pco018," ...
Rep´erer quelques commandes utiles dans cette liste.
cle[grep("pco118",cle)] donne les composantes qui contiennent pco118 ; cle[grep("pco1.1",cle)] donne les composantes qui contiennent pco11 suivi
d’un caract`ere unique quelconque ;
cle[grep("^pco00",cle)] donne les composantes qui commencent par pco00 ; cle[grep("18,$",cle)] donne les composantes qui finissent par 18 ;
cle[grep("pco2*,",cle)] donne les composantes qui contiennent pco suivi d’une chaˆıne de 2 de longueur quelconque (´eventuellement nulle), suivi de , ;
cle[grep("2+,",cle)] donne les composantes qui contiennent une chaˆıne de 2 de longueur au moins 1 ;
cle[grep("pco0.+0",cle)] donne les composantes qui contiennent pco suivie d’une chaˆıne non vide quelconque, suivie de 0 ;
cle[grep("[7-9]",cle[1 :99])] donne, parmi les 99 premi`eres, les compo-santes qui contiennent les caract`eres de 7 `a 9 .
Rechercher les num´eros manquants :
w1 <- paste("pco00", 1:9, sep = "") w2 <- paste("pco0", 10:99, sep = "") w3 <- paste("pco", 100:235, sep = "") w <- c(w1, w2, w3)
which(sapply(w, function(x) (length(grep(x, cle)) == 0)))
pco152 pco155 152 155
which(sapply(as.character(1:235), function(x) (length(grep(x, cle)) == 0)))
152 155 152 155
3
Substituer une chaine de caract`
eres `
a une autre
Le 152 n’est pas utilis´e, le 154 l’est deux fois. On r´etablit l’unicit´e avec :tmp[num[153]] <- "pco152," num <- grep("@", tmp) + 1 clefs <- tmp[num]
any(duplicated(clefs))
[1] FALSE
Question : pourquoi ne fallait-il pas utiliser la fonction tmp <- sub("pco154","pco152",tmp) ?
3.1
Rechercher un auteur
Retrouver tous les Smith :
w <- tmp[grep("Smith", tmp)] w <- sub(".*\\{", "", w) w <- sub("\\}.*", "", w) w <- unlist(strsplit(w, " and ")) w <- w[grep("Smith", w)] w
[1] "Smith, D.R." "Smith, R." "Smith-Ramirez, C." [4] "Smith-Ramirez, C." "Russell-Smith, J. J." "Smith, R.W."
3.2
La distribution du nombre d’articles par auteur
Donner la distribution du nombre d’articles par auteur :
w <- tmp[grep("Author", tmp)] w <- sub(".*\\{", "", w) w <- sub("\\}.*", "", w) w <- unlist(strsplit(w, " and ")) table(table(w)) 1 2 3 4 6 7 716 54 10 2 1 1
Quels sont ceux qui ont le plus grand nombre d’articles dans cette bibliographie ?
3.3
La distribution du nombre d’auteurs par article
Combien d’auteurs par articles ?
4
Pour faire des citations dans L
ATEX
Un dernier exercice, pour les amateurs de LATEX : citer les 25 premiers articles de cette bibliographie :
pco001 : [25] pco002 : [22] pco003 : [21] pco004 : [16] pco005 : [15] pco006 : [14] pco007 : [13] pco008 : [12] pco009 : [11] pco010 : [10] pco011 : [9] pco012 : [8] pco013 : [7] pco014 : [6] pco015 : [5] pco016 : [4] pco017 : [3] pco018 : [2] pco019 : [1] pco020 : [30] pco021 : [29] pco022 : [28] pco023 : [27] pco024 : [26] pco025 : [24] pco026 : [23] pco027 : [20] pco028 : [19] pco029 : [18] pco030 : [17]
On retiendra de ces exercices les fonctions d’entr´ee-sortie sur des fichiers textes (writeLines readLines), le travail sur les vecteurs de chaˆınes de carac-t`eres (longueur par nchar, substitution par sub, recherche de sous-chaˆınes par grep et la pr´esence des expressions r´eguli`eres.
R´
ef´
erences
[1] M. Ahmad. Assessment of genomic diversity among wheat genotypes as de-termined by simple sequence repeats. Genome / National Research Council Canada = Genome / Conseil National de Recherches Canada, 45(4) :646– 651, 2002.
[2] P. Ajmone-Marsan, R. Negrini, E. Milanesi, R. Bozzi, I.J. Nijman, J.B. Buntjer, A. Valentini, and J.A. Lenstra. Genetic distances within and across cattle breeds as indicated by biallelic aflp markers. Animal Genetics, 33(4) :280–286, 2002.
[3] G. Birmeta, H. Nybom, and E. Bekele. Rapd analysis of genetic diversity among clones of the ethiopian crop plant ensete ventricosum. Euphytica, 124(3) :315–325, 2002.
[4] Daniel Borcard and Pierre Legendre. All-scale spatial analysis of ecological data by means of principal coordinates of neighbour matrices. Ecological Modelling, 153(1-2) :51–68, 2002.
[5] M.C.J. Bottini, A. De Bustos, N. Jouve, and L. Poggio. Aflp characte-rization of natural populations of berberis (berberidaceae) in patagonia, argentina. Plant Systematics and Evolution, 231(1-4) :133–142, 2002. [6] C.K. Boyce and A.H. Knoll. Evolution of developmental potential and the
multiple independent origins of leaves in paleozoic vascular plants. Paleo-biology, 28(1) :70–100, 2002.
[7] J.B. Buntjer, M. Otsen, I.J. Nijman, M.T.R. Kuiper, and J.A. Lenstra. Phy-logeny of bovine species based on aflp fingerprinting. Heredity, 88(1) :46–51, 2002.
[8] K.L. Burgher, A.R. Jamieson, and X. Lu. Genetic relationships among lowbush blueberry genotypes as determined by randomly amplified poly-morphic dna analysis. Journal of the American Society for Horticultural Science, 127(1) :98–103, 2002.
[9] K.Y. Ho, J.C. Yang, and J.Y. Hsiao. An assessment of genetic diversity and documentation of hybridization of casuarina grown in taiwan using rapd markers. International Journal of Plant Sciences, 163(5) :831–836,
[10] S.Y. Hwang, Y.T. Tseng, and H.F. Lo. Application of simple sequence repeats in determining the genetic relationships of cultivars used in sweet potato polycross breeding in taiwan. Scientia Horticulturae, 93(3-4) :215– 224, 2002.
[11] S. Jarraud, C. Mougel, J. Thioulouse, G. Lina, H. Meugnier, F. Forey, X. Nesme, J. Etienne, and F. Vandenesch. Relationships between staphy-lococcus aureus genetic background, virulence factors, agr groups (alleles), and human disease. Infection and Immunity, 70(2) :631–641, 2002. [12] G. Khoo, K.F. Lim, D.K.Y. Gan, F. Chen, W.-K. Chan, T.M. Lim, and
V.P.E. Phang. Genetic diversity within and among feral populations and domesticated strains of the guppy (poecilia reticulata) in singapore. Marine Biotechnology, 4(4) :367–378, 2002.
[13] A.A. Linos, P.J. Bebeli, and P.J. Kaltsikes. Cultivar identification in upland cotton using rapd markers. Australian Journal of Agricultural Research, 53(6) :637–642, 2002.
[14] F. Liu, G.-L. Sun, B. Salomon, and R. Von Bothmer. Characterization of genetic diversity in core collection accessions of wild barley, hordeum vulgare ssp. spontaneum. Hereditas, 136(1) :67–73, 2002.
[15] G. Nieto Feliner, J. Fuertes Aguilar, and J.A. Rossello. Reticulation or di-vergence : The origin of a rare serpentine endemic assessed with chloroplast, nuclear and rapd markers. Plant Systematics and Evolution, 231(1-4) :19– 38, 2002.
[16] R. Ogden and R.S. Thorpe. The usefulness of amplified fragment length polymorphism markers for taxon discrimination across graduated fine evo-lutionary levels in caribbean anolis lizards. Molecular Ecology, 11(3) :437– 445, 2002.
[17] M.C. Sawkins, B.L. Maass, B.C. Pengelly, H.J. Newbury, B.V. Ford-Lloyd, N. Maxted, and R. Smith. Geographical patterns of genetic variation in two species of stylosanthes sw. using amplified fragment length polymorphism. Molecular Ecology, 10(8) :1947–1958, 2001.
[18] G. Schmalisch, M. Schmidt, and B. Foitzik. Novel technique to average breathing loops for infant respiratory function testing. Medical and Biolo-gical Engineering and Computing, 39(6) :688–693, 2001.
[19] F. Sebastiani, R. Meiswinkel, L.M. Gomulski, C.R. Guglielmino, P.S. Mel-lor, A.R. Malacrida, and G. Gasperi. Molecular differentiation of the old world culicoides imicola species complex (diptera, ceratopogonidae), infer-red using random amplified polymorphic dna markers. Molecular Ecology, 10(7) :1773–1786, 2001.
[21] M.K. Shrestha, A. Golan-Goldhirsh, and D. Ward. Population genetic structure and the conservation of isolated populations of acacia raddiana in the negev desert. Biological Conservation, 108(1) :119–127, 2002. [22] J.-C. Svenning and F. Skov. Mesoscale distribution of understorey plants
in temperate forest (kalo, denmark) : The importance of environment and dispersal. Plant Ecology, 160(2) :169–185, 2002.
[23] P.W. Sweeney and R.A. Price. A multivariate morphological analysis of the cardamine concatenata alliance (brassicaceae). Brittonia, 53(1) :82–95, 2001.
[24] L. Triest. Hybridization in staminate and pistillate salix alba and s. fragilis (salicaceae) : Morphology versus rapds. Plant Systematics and Evolution, 226(3-4) :143–154, 2001.
[25] I.A. Udalova, R. Mott, D. Field, and D. Kwiatkowski. Quantitative predic-tion of nf-[kappa ;]b dna-protein interacpredic-tions. Proceedings of the Napredic-tional Academy of Sciences of the United States of America, 99(12) :8167–8172, 2002.
[26] Jean K. Whelan, Lorraine Eglinton, Mahlon C. Kennicutt, II, and Yaorong Qian. Short-time-scale (year) variations of petroleum fluids from the u.s. gulf coast. Geochimica et Cosmochimica Acta, 65(20) :3529–3555, 2001. [27] J.K. Whelan, L. Eglinton, M.C. Kennicutt II, and Y. Qian. Short-time-scale
(year) variations of petroleum fluids from the u.s. gulf coast. Geochimica et Cosmochimica Acta, 65(20) :3529–3555, 2001.
[28] C.D. Wilkinson and D.R. Edds. Spatial pattern and environmental corre-lates of a midwestern stream fish community : Including spatial autocor-relation as a factor in community analyses. American Midland Naturalist, 146(2) :271–289, 2001.
[29] B. Yacoubi Loveslati, A. Sanchez-Mazas, H. Ennafaa, R. Marrakchi, J.-M. Dugoujon, M.-P. Lefranc, and A. Ben Ammar Elgaaied. A study of gm allotypes and immunoglobulin heavy gamma ighg genes in berbers, arabs and sub-saharan africans from jerba island, tunisia. European Journal of Immunogenetics, 28(5) :531–538, 2001.
[30] S. Zhou, D.R. Smith, and G.R. Stanosz. Differentiation of botryosphaeria species and related anamorphic fungi using inter simple or short sequence repeat (issr) fingerprinting. Mycological Research, 105(8) :919–926, 2001.