Rôle du microbiote intestinal dans l’altération de
l’homéostasie immunitaire par la phospholipase A2 de
groupe IIA
Mémoire
Etienne Doré
Maîtrise en microbiologie-immunologie - avec mémoire
Maître ès sciences (M. Sc.)
Rôle du microbiote intestinal dans l’altération de
l’homéostasie immunitaire par la phospholipase A
2de groupe IIA
Mémoire
Etienne Doré
Sous la direction de :
Résumé
La flore intestinale, composée de l’ensemble des microorganismes retrouvés dans le tractus digestif, joue un rôle essentiel dans le développement et l’homéostasie du système immunitaire. Son altération est associée à de multiples maladies inflammatoires ayant des répercussions non seulement à l’intestin, mais bien dans l’ensemble de l’organisme. Les causes de cette dérégulation restent toutefois méconnues. Parmi les multiples facteurs susceptibles de moduler sa composition, on retrouve plusieurs enzymes bactéricides produites à l’intestin. L’une de ces protéines, la phospholipase A2-IIA sécrétée (sPLA2-IIA), est fortement surexprimée en condition inflammatoire.
Nous avons émis l’hypothèse que la surexpression de la sPLA2-IIA à l’intestin au cours de maladies inflammatoires pourrait altérer la composition de la flore intestinale, contribuant ainsi à la physiopathologie de ces maladies.
L’utilisation de souris surexprimant la sPLA2-IIA humaine (sPLA2-IIATGN) nous a permis d’observer l’apparition spontanée d’un désordre immunitaire encore jamais caractérisé dans ce modèle. En nous intéressant à l’impact de cette enzyme sur la flore intestinale, nous avons pu identifier plusieurs altérations dans le microbiome de ces souris. Nos résultats suggèrent également que l’environnement, et plus spécifiquement le microbiote intestinal, joue un important rôle dans le développement de ce désordre immunitaire.
Nos résultats suggèrent que la modulation de l’inflammation systémique par la sPLA2-IIA est dépendante de la flore intestinale.
Abstract
The mammalian digestive tract harbors trillions of microorganisms that collectively form the intestinal microbiota. This flora has been shown to play a prominent role in the development and homeostasis of the immune system. As such, its alteration was associated with a wide array of inflammatory diseases with intestinal and systemic afflictions. However, the cause of this unbalance remains poorly understood. While the intestinal flora may be regulated by a large number of environmental factors, multiple endogenous intestinal enzymes have been shown or proposed to play an important part in the shaping of its composition. One of those enzymes, the secreted phospholipase A2-IIA (sPLA2-IIA), possesses great bactericidal properties and is overexpressed during multiple inflammatory disorders.
We hypothesized that the overexpression of sPLA2-IIA in the intestine during inflammatory processes could alter the composition of the intestinal microbiota, thereby contributing to the pathophysiology of those diseases.
To verify this hypothesis, we used transgenic mice overexpressing the human sPLA2 -IIA (sPLA2-IIATGN) and observed a yet uncharacterized spontaneous immune disorder in this model. We aimed to evaluate the impact of sPLA2-IIA on the intestinal flora in this model. We identified multiple alterations in the microbiome of sPLA2-IIATGN mice. Our results also suggest that the environment, and more specifically the intestinal microbiota, play a prominent role in the development of this immune disorder.
Our results suggest that the modulation of systemic inflammation by sPLA2-IIA is dependent upon the intestinal flora.
Table des matières
Résumé ... ii
Abstract ... iii
Table des matières ... iv
Liste des figures ... viii
Introduction ... viii
Results ... viii
Annexe A ... viii
Liste des tableaux ... ix
Results ... ix
Liste des abréviations, sigles, acronymes ... x
Remerciements ... xvi
Avant-propos ... xviii
Introduction ... 1
1.
Le système immunitaire... 1
1.1.
Barrières physiques et chimiques... 1
1.2.
Système immunitaire innée ... 2
1.2.1. Macrophages ... 3 1.2.2. Cellules dendritiques ... 3 1.2.3. Neutrophiles ... 4 1.2.4. Éosinophiles ... 4 1.2.5. Basophiles ... 5 1.2.6. Mastocytes ... 5 1.2.7. Système du complément ... 5
1.3.
Système immunitaire adaptatif ... 6
1.3.1. Les organes lymphoïdes ... 6
1.3.1.1. Organes lymphoïdes primaires ... 6
1.3.1.2. Organes lymphoïdes secondaires ... 7
1.3.1.2.1. Lymphocytes T auxiliaires CD4+ (Th) ... 7
1.3.1.2.2. Lymphocytes T cytotoxiques CD8+ (Tc) ... 9
1.3.1.2.3. Lymphocytes B et plasmocytes ... 9
1.3.2. Immunité à médiation humorale ... 10
1.3.2.1. Les anticorps ... 10 1.3.2.1.1. Immunoglobuline de type D ... 10 1.3.2.1.2. Immunoglobuline de type M ... 10 1.3.2.1.3. Immunoglobuline de type G ... 11 1.3.2.1.4. Immunoglobuline de type E ... 11 1.3.2.1.5. Immunoglobuline de type A ... 11
2.1.
Principaux rôles ... 11
2.1.1. Métabolisme... 12
2.1.2. Barrière contre l’implantation des pathogènes ... 12
2.1.3. Développement et homéostasie immunitaire ... 13
2.2.
Dysbiose ... 15
2.2.1. Pathogènes au cours de maladies inflammatoires chroniques ... 16
2.3.
Facteurs régulant la composition bactérienne ... 17
2.3.1. Facteurs exogènes ... 18
2.3.2. Facteurs endogènes ... 18
3.
Phospholipases A
2... 19
3.1.
Phospholipases A
2sécrétées ... 20
3.2.
Phospholipase A
2sécrétée de groupe IIA ... 21
3.2.1. Distribution tissulaire ... 22
3.2.2. Structure ... 22
3.2.3. Activité non-catalytique ... 23
3.2.4. Substrats de préférence ... 24
3.2.4.1. Substrats endogènes ... 24
3.2.4.1.1. Activité sur les cellules apoptotiques ... 25
3.2.4.1.2. Activité sur les vésicules extracellulaires ... 26
3.2.4.1.3. Activité sur les mitochondries extracellulaires ... 27
3.2.4.2. Substrats exogènes ... 27
3.2.4.2.1. Bactéries à gram négatif ... 27
3.2.4.2.2. Bactéries à gram positif ... 28
3.2.4.2.3. Phase de croissance bactérienne ... 30
3.2.5. sPLA2-IIA dans la défense contre les pathogènes ... 30
3.2.5.1. Synergie avec la sPLA2-IIA dans l’élimination de bactéries ... 31
3.2.5.2. Mécanisme de résistance contre la sPLA2-IIA ... 31
3.2.5.2.1. Restriction de l’accès à la membrane ... 32
3.2.5.2.2. Suppression de l’expression de la sPLA2-IIA ... 33
3.2.5.3. Induction de l’expression de la sPLA2-IIA par des pathogènes ... 35
4.
Problématique, hypothèses et objectifs de recherche ... 38
4.1.
Problématique ... 38
4.2.
Hypothèse ... 39
4.3.
Objectifs généraux et spécifiques de la recherche ... 39
1
stChapter: Methods ... 41
1.1.
Mice... 41
1.2.
Cell isolation ... 41
1.3.
Flow Cytometry ... 41
1.4.
Complete blood count ... 42
1.5.
Cytokine dosage ... 42
1.6.
Immunoglobulin dosage ... 42
1.7.1. Swelling score ... 43
1.7.2. Mass score ... 43
1.8.
Whole-genome shotgun sequencing ... 43
1.7.1. Bioinformatic analysis ... 43
1.9.
Microbiota depletion ... 44
1.10.
Microbiota depletion assessment ... 44
1.11.
Intestinal lipid profiling ... 45
1.12.
sPLA
2-IIA activity and concentration assessment ... 46
1.12.1. Lysis of intestinal samples ... 46
1.12.2. Enzymatic activity ... 46
1.12.3. Time-resolved florescence immunoassay ... 46
2
ndChapter: Results ... 48
2.1.
sPLA
2-IIA-induced immune disorder ... 48
2.1.1. Characterization of immune cell populations ... 49
2.1.2. Circulating immunoglobulins and cytokines in sPLA2-IIATGN mice 51
2.2.
Environmental impact on the immune disorder ... 52
2.2.1. Mouse housing conditions ... 52
2.2.2. Microbiota composition assessment ... 55
2.2.3. Role of the microbiota in the immune disorder ... 58
2.3.
Role of cPLA2-α and 12-LO ... 60
2.4.
Eicosanoids released in the intestine ... 61
2.5.
Implication of IL-17A in the immune disorder ... 62
2.6.
sPLA
2-IIA concentration... 63
3
rdChapter: Discussion ... 65
Conclusion ... 71
Bibliographie ... 72
Annexe A : Rôles de la phospholipase A
2sécrétée de groupe IIA
dans l’inflammation et la défense de l’hôte ... 92
Résumé en français : ... 92
Abstract ... 94
1.
Introduction ... 94
2.
sPLA2-IIA and cell membrane hydrolysis in inflammation ... 95
3.
sPLA2-IIA-interacting proteins ... 98
4.
sPLA2-IIA substrates in the extracellular milieu ... 99
5.
sPLA2-IIA and in vivo models of inflammation ... 101
6.
sPLA2-IIA as a biomarker ... 103
7.
Bacterial membrane as a substrate for sPLA2-IIA ... 104
7.1. Gram-negative bacteria ... 104
7.3. Bacterial growth phase ... 106
8.
sPLA2-IIA in host defense ... 107
9.
Resistance to sPLA2-IIA ... 108
9.1. Increased bacterial cell wall density ... 108
9.2. Sortase A-attached LPXTG proteins ... 109
9.3. Downregulation of sPLA2-IIA expression ... 110
10.
Induction of sPLA2-IIA expression by pathogens ... 113
10.1. P. aeruginosa-mediated sPLA2-IIA expression during cystic fibrosis ... 113
10.2. Porphyromonas gingivalis-mediated sPLA2-IIA expression ... 114
11.
Concluding remarks ... 114
Transparency document ... 115
Acknowledgements ... 115
Conflicts of interests ... 116
Liste des figures
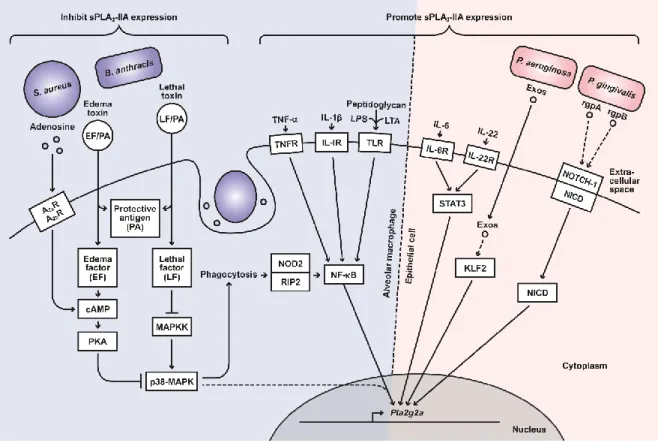
Introduction
Figure 1.1 : Représentation simplifiée de l’hématopoïèse. Figure 1.2 : Structure d’un ganglion lymphatique.
Figure 1.3 : Le microbiote intestinal protège l’organisme contre les pathogènes. Figure 1.4 : Induction d’une réponse Th par des bactéries de la flore intestinale. Figure 1.5 : Représentation de l’activité catalytique des phospholipases.
Figure 1.6 : Représentation structurelle des différentes sPLA2s retrouvées chez les mammifères.
Figure 1.7 : Substrats endogènes de la sPLA2-IIA.
Figure 1.8 : Capacité de la sPLA2-IIA à traverser la paroi bactérienne.
Figure 1.9 : Voies de signalisation régulant l’expression du gène Pla2g2a codant pour la
sPLA2-IIA.
Results
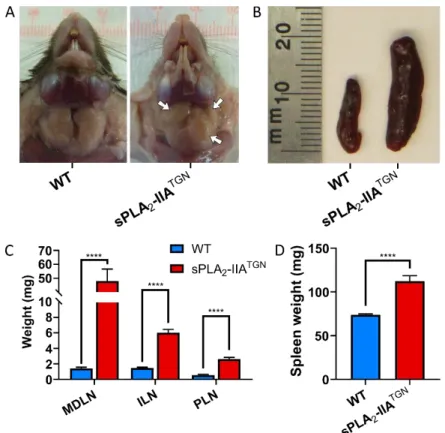
Figure 2.1: Expansion of lymph nodes and the spleen in sPLA2-IIATGN mice.
Figure 2.2: Characterization of immune cell populations from MDLN, bone marrow and
spleen of sPLA2-IIATGN mice.
Figure 2.3: Blood cells in WT and sPLA2-IIATGN mice.
Figure 2.4: Concentrations of circulating antibodies and cytokines.
Figure 2.5: The housing environment impacts the severity of the immune disorder. Figure 2.6: sPLA2-IIA is present and active in the intestine of sPLA2-IIATGN mice.
Figure 2.7: The microbiota of sPLA2-IIATGN mice is altered compared to WT mice.
Figure 2.8: Assessment of the immune disorder following the depletion of the microbiota. Figure 2.9: cPLA2-α and 12-lipoxygenase are not involved in the sPLA2-IIA-mediated immune disorder.
Figure 2.10: Modulation of intestinal lipid mediators in sPLA2-IIATGN mice.
Figure 2.11: The suppression of IL-17A expression has no impact on the immune disorder. Figure 2.12: Serum and intestinal sPLA2-IIA concentration.
Annexe A
Fig. 1. Endogenous substrates of sPLA2-IIA.
Fig. 2. Ability of group IIA sPLA2 to penetrate the bacterial cell wall. Fig. 3. Group IIA sPLA2 expression pathway.
Liste des tableaux
Results
Table 2.1: Bacterial exclusion list for the SPF and Elite SPF+ animal facilities at CHU de
Liste des abréviations, sigles, acronymes
12-HETE Acide 12(S)-hydroxyeicosatetraénoïque
(12(S)-hydroxyeicosatetraenoic acid) 1- et 2-AG 1- et 2-arachidonylglycérol (1- and 2-arachidonoylglycerol) AA Acide arachidonique (Arachidonic acid) ABHD Hydrolase α/β (α/β hydrolase) ABX Antibiotiques (Antibiotics) ACD Anticoagulant-citrate-dextrose
(Anticoagulant citrate dextrose)
AIEC Souche adhérente et invasive d’Escherichia coli (Adherent-invasive Escherichia coli)
AEA Anandamide ou N-arachidonoyléthanolamide
(Anandamide or N-arachidonoylethanolamide)
AMP Peptide antimicrobien
(Antimicrobial peptide)
BCR Récepteur des cellules B
(B cell receptor)
BPI Protéine bactéricide/augmentant la perméabilité
(Bactericidal/permeability-increasing protein)
cAMP Adénosine monophosphate cyclique
(Cyclic adenosine monophosphate)
CAMP Peptide antimicrobien cationique
(Cationic antimicrobial peptide)
CBA Cytometric bead array
CCL2 Ligand des chimiokines 2
(Chemokin ligand 2)
CMH-II Complexe majeur d’histocompatibilité de classe II (Major histocompatibility complexe class II)
COX Cyclooxygénase
(Cyclooxygenase)
cPLA2 Phospholipase A2 cytosolique (Cytosolic phospholipase A2)
cPLA2- Phospholipase A2 cytosolique de groupe IVA (Group IVA cytosolic phospholipase A2)
DAMP Motif moléculaire associé aux dommages
(Damage-associated molecular pattern)
DHA Acide docosahexaénoïque
(Docosahexaenoic acid)
DPA Acide docosapentaénoïque
(Docosapentaenoic acid)
DPM Désintégrations par minutes
(Disintegration per minutes)
ELISA Dosage d'immunoabsorption par enzyme liée
(Enzyme linked immunosorbent assay)
EPA Acide eicosapentaénoïque
(eicosapentaenoic acid) EV Vésicule extracellulaire (Extracellular vesicle) GPI Glycosylphosphatidylinositol (Glycophosphatidylinositol) HS Héparane sulfate (Heparan sulfate)
IBD Maladies inflammatoires de l’intestin
(Inflammatory Bowel Disease)
IFN-γ Interféron-γ (Interféron-γ) Ig Immunoglobuline (Immunoglobulin) IL Interleukine (Interleukin)
ILN Ganglion inguinal
(Inguinal lymph node)
iPLA2 Phospholipase A2 indépendante du calcium (Calcium-independent phospholipase A2)
IκBα Inhibiteur α de NF-κB
(NF-κB inhibitor α)
KLF2 Facteur de transcription 2 de type Krüppel (Krüppel-like factor 2)
LA Acide linoléique
LDL Lipoprotéine à faible densité (Low density lipoprotein)
LO Lipoxygénase
(Lipoxygenase)
LPLA2 Phospholipase A2 lysosomale
(Lysosomal phospholipase A2)
LPS Lipopolysaccharide
(Lipopolysaccharide)
LTA Acide lipotéichoïque
(Lipoteichoic acid)
LTB4 Leucotriène B4
(Leukotriene B4)
MAPK Protéine kinase activée par des agents mitogènes (Mitogen-activated protein kinase)
MAPKK Kinase de protéines kinases activées par des agents mitogènes (Mitogen-activated protein kinase kinase)
MCP-1 Facteur chimiotactique des monocytes-1
(Monocyte chemoattractant protein 1)
MDLN Ganglion mandibulaire
(Mandibular lymph node)
m-pIgR Récepteur polymérique des immunoglobulines murines
(Mouse polymeric immunoglobulin receptor)
mtDNA ADN mitochondrial
(Mitochondrial DNA)
NADPH Nicotinamide adénine dinucléotide phosphate
(Nicotinamide adenine dinucleotide phosphate)
NF-κB Facteur de transcription nucléaire κB
(Nuclear factor κB)
NICD Domaine intracellulaire de Notch
(Notch intracellular domain)
NLR Récepteur de type NOD
(NOD-like receptor)
NOD Domaine de liaison des nucléotides et d’oligomérisation (Nucleotide-binding oligomerization domain)
NOTCH-1 Homologue 1 de Notch
(Notch homolog 1)
NOX NADPH oxydase
OEA N-Oléoyléthanolamine
(N-Oleoylethanolamine)
PAF-AH Acétyle-hydrolases des facteurs d’activation plaquettaires (Platelet-activating factor acetylhydrolase)
PAMP Motif moléculaire associé aux pathogènes
(Pathogen-associated molecular pattern)
PC Phosphatidylcholine (Phosphatidylcholine) PE Phosphatidyléthanolamine (Phosphatidylethanolamine) PFA Paraformaldéhyde (Paraformaldehyde) PG Phosphatidylglycérol (Phosphatidylglycerol) PGB2 Prostaglandine B2 (Prostaglandin B2) PGE2 Prostaglandine E2 (Prostaglandin E2) PLA2 Phospholipase A2 (Phospholipase A2)
PLAAT Phospholipase A acyltransférase
(Phospholipase A and acyltransferase)
PLN Ganglion poplité
(Popliteal lymph node)
PRR Récepteur de motif moléculaire
(Pattern recognition receptor)
PSA Polysaccharide A
(Polysaccharide A)
qPCR Réaction quantitative de polymérisation en chaîne (Quantitative polymerase chain reaction)
rgpA et rgpB Protéases à cystéine hydrolysant l’arginine A et B (Arginine-gingipains A and B)
RIP2 Sérine/thréonine kinase interagissant avec un récepteur 2 (Receptor-interacting protein kinase-2)
ROS Espèce réactive de l’oxygène
(Reactive oxygen species)
SCFA Acide gras à courte chaîne
SFB Bactérie filamenteuse segmentée (Segmented filamentous bacteria)
SPF Exempt de pathogène spécifique
(Specific pathogen-free) sPLA2 Phospholipase A2 sécrétée
(Secreted phospholipase A2)
sPLA2-IIA Phospholipase A2 sécrétée de groupe IIA (Groupe IIA secreted phospholipase A2)
sPLA2-IIATGN Souris transgénique pour la phospholipase A2-IIA sécrétée humaine (Transgenic human secreted phospholipase A2-IIA mice)
SrtA Sortase A
(Sortase A)
STAT3 Transducteur de signal et activateur de la transcription-3 (Signal transducer and activator of transcription-3)
T3SS Système de sécrétion de type 3
(Type 3 secretion system)
TA Acide téichoïque
(Teichoic acid)
Tc Lymphocyte T cytotoxique
(Cytotoxic T cell)
TCR Récepteur des cellules T
(T cell receptor)
TFH Lymphocyte T auxiliaire folliculaire
(T follicular helper cell)
TGF-β Facteur de croissance transformant β
(Transforming growth factor β)
Th Lymphocytes T auxiliaire
(T helper cell)
TLR Récepteur de type Toll
(Toll-like receptor)
TNF Facteur de nécrose tumorale
(Tumor necrosis factor)
Treg Lymphocyte T régulateur
(Regulatory T cell)
TRFIA Spectrofluorimétrie à résolution temporelle (Time-resolved fluoroimmunoassay)
TXB2 Thromboxane B2
WT Souche de souris C57BL/6J sauvage (Wild-type C57BL/6J mice)
Remerciements
Le travail qui suit est le fruit de plusieurs années de travail et n’aurait pas été possible sans la collaboration et le soutien d’un grand nombre d’individus. J’aimerais en tout premier lieu remercier mon directeur de recherche, le Dr Eric Boilard, pour m’avoir donné il y a cela déjà plus de 3 ans l’opportunité de réaliser un stage dans son équipe, puis de poursuivre ma formation à la maîtrise sous sa direction. Ce dernier a su m’encadrer, m’encourager et me pousser tout au long de mon parcours, m’incitant ainsi à mettre tous les efforts nécessaires pour réussir. Je remercie également le Dr Boilard pour toutes les opportunités qu’il m’a présentées, que ce soit la possibilité d’écrire une revue de littérature, de réaliser un stage à l’étranger, et même de présenter mon projet à l’occasion d’un congrès international. Ces expériences furent extrêmement enrichissantes et n’auraient pas été possibles sans son important soutien.
Tout bon chef s’entoure d’une bonne équipe et l’équipe Boilard n’y fait pas exception. À mon arrivée, je n’avais aucune expérience de recherche. C’est de la main de notre technicienne hors pair, Tania Lévesque, que j’ai pu apprendre tous les rudiments du travail en laboratoire. Son aide dans la réalisation des multiples expériences nécessitant des modèles animaux fut également grandement appréciée. Merci au Dre Emmanuelle Rollet-Labelle pour son encadrement au début de mon projet, ainsi que pour son aide dans l’embellissement de mes demandes de bourses. Je remercie les Dres Isabelle Allaeys et Natalie Cloutier pour leur aide technique et leurs conseils essentiels au bon déroulement de nombreux protocoles. Finalement, j’aimerais remercier tous les étudiant et autres membres de mon équipe que j’ai pu côtoyer et qui m’ont soutenu tout au long de mon cheminement, dont Nicolas, Geneviève, Yann, Imène, Audrée, Anne, Anne-Claire, Florian, Jenifer, Sara, Carolanne et Chanel.
Je ne remercie toutefois pas les alpagas pour leur omniprésence qui me hante jour et nuit.
J’aimerais ensuite remercier le Dr Gérard Lambeau pour avoir accepté de me superviser dans le cadre d’un stage dans son laboratoire. Je suis tout particulièrement reconnaissant des multiples périodes où ce dernier a pris le temps de s’assoir à mes côtés afin de partager avec moi son expertise en rapport aux différentes techniques utilisées pendant mon stage. Ma compréhension et mon projet dans son ensemble bénéficièrent grandement de ces enseignements. Je remercie également toute son équipe pour l’accueil chaleureux qu’ils
m’ont présenté à mon arrivée, avec une mention spéciale pour Dre Christine Payre et Dre Agnès Petit-Paitel pour l’aide technique apportée dans la mise au point et l’exécution des expériences réalisées.
Je remercie tous nos collaborateurs pour leur contribution indispensable à la réussite de ce projet. Merci aux Dr Arnaud Droit, Charles Joly-Beauparlant et à Antoine Bodein pour leur travail essentiel dans l’analyse des résultats de séquençage. Merci également au Dr Nicolas Flamand et à Cyril Martin pour l’analyse du profil des médiateurs lipidiques de l’intestin. Merci aux Drs Jacques Corbeil et Frédéric Raymond pour les conseils et la réalisation du séquençage. Je remercie ensuite tous les individus dont les suggestions et conseils permirent de faire progresser mon projet, dont les Drs Jonathan Schertzer, Abderrahim Benmoussa et Joanie Baillargeon. J’aimerais également remercier tous les individus que j’ai pu côtoyer au quotidien, qui mettent de la vie dans le laboratoire et qui ont rendu cette expérience si exceptionnelle.
En concluant ces remerciements, j’aimerais remercier toute ma famille, qui m’a toujours soutenu et encouragé dans la poursuite de mes études. C’est à leur appui constant que je dois ma persévérance, qui m’a menée à la toute fin de ce parcours. Je tire également cette qualité de mes grands-parents, qui nous ont si souvent répété « Quand on veut, on peut » et « C’est difficile, mais t’es capable ». Bien que vous nous ayez quittés il y a quelques mois seulement, je tiens à vous remercier des importantes qualités et valeurs que vous avez pu nous transmettre.
Avant-propos
Le mémoire qui suit contient des résultats qui n’ont pas encore été publiés. En effet, les multiples mois nécessaires au développement du désordre immunitaire étudié dans le cadre de ce projet de maîtrise ont considérablement allongé la réalisation de ce projet. Étant donné le potentiel des résultats obtenus, mon directeur et moi-même avons pris la décision stratégique de cumuler des données supplémentaires dans un modèle murin d’arthrite dans le cadre de mes études doctorales. Les résultats retrouvés dans ce mémoire, en plus de résultats additionnels obtenus dans le cadre de mes études doctorales, seront ainsi utilisés pour la rédaction d’un article de plus grande envergure que nous prévoyons soumettre en cette année 2020. Afin de directement pouvoir utiliser ces résultats pour la rédaction de cet article, les méthodes, les résultats et la discussion de ce mémoire ont été rédigés en anglais. Mon directeur m’a encadré lors de l’écriture, mais je demeure l’auteur de ce manuscrit.
Dans le cadre de ma maîtrise, j’ai également rédigé une revue en tant que premier auteur en collaboration avec mon directeur de recherche. Cette revue porte sur les rôles de la phospholipase A2-IIA dans l’inflammation et la protection de l’hôte,1 un sujet se rapprochant grandement de mon projet de recherche (voir annexe A). Ayant par moi-même écrit les sections traitant des propriétés antimicrobiennes de cette enzyme, je me suis basé sur ces passages pour la rédaction de certaines sections de l’introduction du mémoire qui suit. Les informations retrouvées dans ces passages, quoiqu’adaptées et mises à jour, apparaissent principalement dans les sections 3.2.4.2 et 3.2.5.
Cette revue a été commandée par le journal Biochimica et Biophysica Acta et fait partie d’une thématique spéciale nommée « Novel functions of phospholipase A2 ». Dore, E. & Boilard, E. Roles of secreted phospholipase A2 group IIA in inflammation and host defense. Biochimica et Biophysica Acta - Molecular and Cell Biology of Lipids vol. 1864 789–802 (2019).
Introduction
1. Le système immunitaire
Afin de se défendre contre la multitude de pathogènes présents dans son environnement, le corps humain est muni d’un système immunitaire chargé de combattre ces microorganismes. Ce système est constitué de différents organes, tissus, cellules et molécules dont les fonctions sont de prévenir les infections et maintenir l’homéostasie immunitaire. Il comporte plusieurs lignes de défense qui seront discutées plus en détail dans le cadre de cette introduction de mémoire.
1.1. Barrières physiques et chimiques
Le premier et principal obstacle prévenant l’entrée de microorganismes au sein du corps humain est composé des différentes barrières physiques et chimiques à sa surface. Les tissus épithéliaux, localisés à l’interface avec le milieu externe du corps, sont souvent combinés à différents mécanismes de sécrétion afin de prévenir l’implantation et l’infiltration de pathogènes.2 La peau est par exemple constituée de multiples couches de cellules épaisses et serrées qui bloquent l’entrée des microorganismes.3 Sa surface est également peu propice à la croissance de différents pathogènes en raison de son pH acide causé par diverses sécrétions produites notamment par les glandes sébacées.4,5 Cette protection est accentuée par la libération de peptides antimicrobiens (antimicrobial peptides, AMP) comme la cathélicidine et les β-défensines par certains kératinocytes, les principales cellules de la peau.6–8
Dans les muqueuses, des cellules épithéliales glandulaires produisent un épais mucus qui contribue à la défense de ces tissus. En effet, ce mucus constitue une importante barrière prévenant l’implantation des pathogènes, et sa constante élimination permet simultanément l’élimination de ces microorganismes.2,3,9 Tout comme au niveau de la peau, de nombreux AMPs sont libérés dans le mucus et limitent la prolifération bactérienne.7 Si des pathogènes parviennent tout de même à traverser cette première barrière, les cellules épithéliales sont liées entre elles par des jonctions serrées qui préviennent l’entrée de ces pathogènes dans l’épithélium.2,9
1.2. Système immunitaire innée
Dans l’éventualité où des pathogènes parviennent à traverser le tissu épithélial, le système immunitaire inné sera le prochain en rang pour combattre ces envahisseurs. Ce système aura pour fonction de détecter les intrus et mobiliser ses « troupes » afin de rapidement les éliminer. Pour y arriver, le système immunitaire inné est composé de différentes cellules effectrices jouant des fonctions complémentaires afin de répondre rapidement et efficacement à toute éventualité.
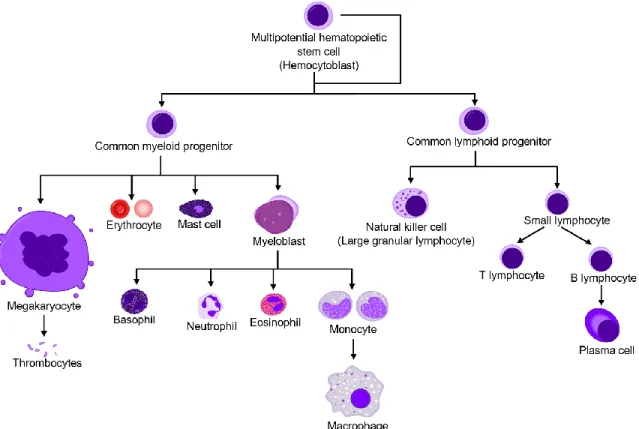
Figure 1.1 : Représentation simplifiée de l’hématopoïèse. La majorité des cellules en
circulation sont générées dans la moelle osseuse à partir d’une cellule souche hématopoïétique. Ces progéniteurs peuvent se différencier pour générer deux lignées distinctes : la lignée myéloïde et la lignée lymphoïde. Le progéniteur de la lignée myéloïde est responsable de générer la majorité des cellules présentent dans le sang. Les globules rouges, ou érythrocytes, et les plaquettes sanguines, c’est-à-dire les thrombocytes, sont d’ailleurs issus de cette lignée. De nombreuses cellules participant à l’immunité innée, comme les mastocytes, les basophiles, les neutrophiles, les éosinophiles et les monocytes/macrophages, proviennent également de cette lignée. Quant à elles, les cellules de la lignée lymphoïde contribuent principalement à l’immunité adaptative (voir section 1.3). Comme leur nom l’indique, les différents lymphocytes descendent de cette lignée. Figure par A. Rad et M. Häggström, utilisée avec permission.10
1.2.1. Macrophages
Les macrophages sont des cellules retrouvées dans l’ensemble des tissus du corps humain, et dont le rôle principal dans l’immunité est l’élimination active des pathogènes. Ces cellules immunitaires ont différentes origines, la plupart étant générées au cours du développement embryonnaire, tandis que d’autres proviennent de la différentiation des monocytes circulant à la suite de leur migration dans les tissus.11–13 Afin d’identifier les pathogènes à éliminer, les macrophages possèdent des récepteurs de motifs moléculaires (Pattern recognition receptor, PRR) tels les récepteurs de type Toll (Toll-like receptor, TLR) pouvant reconnaître des motifs moléculaires associés aux pathogènes (Pathogen-associated
molecular pattern, PAMP). Ainsi, la reconnaissances de PAMPs, comme les
lipopolysaccharides (LPS) et le peptidoglycane, active les macrophages, entrainant la phagocytose de ces pathogènes et la libération de divers composés pro-inflammatoires et antimicrobiens.14,15 À la surface des macrophages sont également retrouvés des récepteurs reconnaissant des protéines du complément ou des anticorps, permettant d’amplifier les fonctions de ces leucocytes.2 L’importance de ces protéines sera respectivement discutée plus en détails dans les sections 1.2.7 et 1.3.2 de cette introduction.
1.2.2. Cellules dendritiques
Les cellules dendritiques tirent leur nom de leurs nombreuses extensions cellulaires qui rappellent les dendrites des neurones. Tout comme les macrophages, les cellules dendritiques possèdent des PRRs permettant de reconnaître les pathogènes et induire leur phagocytose. En digérant ces microorganismes, les cellules dendritiques isolent des antigènes qu’ils présentent alors à leur surface sur des complexes majeurs d’histocompatibilité de classe II (CMH-II). Les cellules dendritiques migrent ensuite vers les organes lymphoïdes secondaires (voir section 1.3.1.2) afin de présenter ces antigènes aux lymphocytes T, initiant ainsi la réponse immunitaire adaptative.2,15,16 Cette réponse sera décrite dans la section 1.3.2 de cette introduction. Bien que les cellules dendritiques soient spécialisées dans la présentation antigénique, d’autres cellules comme les macrophages peuvent également migrer dans les organes lymphoïdes secondaires afin d’exercer cette fonction.11,17
1.2.3. Neutrophiles
Les neutrophiles, éosinophiles et basophiles sont collectivement nommés « granulocytes » en raison des granules retrouvés dans leur cytoplasme.2,18 Ces granules contiennent divers composés jouant des rôles variés dans la réponse immunitaire. Chez les neutrophiles, ces granules contiennent différentes enzymes aidant à la dégradation des pathogènes ingérés par phagocytose, ou pouvant être libérées par dégranulation afin d’éliminer ces pathogènes. Parmi ces enzymes sont retrouvées des péroxydases, lysozyme, défensines, lactoferrine, collagénase et autres.3,19 Originaires de la moelle osseuse, les neutrophiles sont les cellules immunitaires les plus abondantes dans la circulation sanguine. Contrairement aux macrophages, ils ne sont généralement pas retrouvés dans les tissus sains.2,3 Afin d’éliminer les pathogènes envahissant, les neutrophiles migrent hors de la circulation sanguine en réponse à diverses chimiokines libérées par les cellules résidentes des tissus.20,21 Ils sont généralement les premiers répondants au site du dommage. Après avoir infiltré le tissu, les neutrophiles éliminent les pathogènes envahissants et contribuent à la réponse immunitaire via plusieurs mécanismes incluant la phagocytose, la libération de cytokines et chimiokines, la dégranulation, la libération d’espèces réactive de l’oxygène (Reactive oxygen species, ROS) et la NETose, c’est-à-dire la libération d’une matrice constituée d’ADN et de protéines granulaires qui emprisonne et élimine les bactéries.21–23 Pour compenser la migration des neutrophiles hors de la circulation au cours des processus inflammatoires, la production de ces granulocytes dans la moelle osseuse peut être augmentée.24
1.2.4. Éosinophiles
Les éosinophiles jouent un rôle similaire aux neutrophiles. Présents en circulation, ils sont recrutés dans les tissus, principalement les muqueuses, pour exercer leur fonction phagocytaire.25 Cependant, ces cellules s’attaquent plutôt aux parasites tels les protozoaires et helminthes, dont la taille relativement grande les protège de la phagocytose par les macrophages et neutrophiles.26,27 Pour exercer leur fonction antiparasitaire, les éosinophiles libèrent des médiateurs cytotoxiques présents dans leurs granules (dégranulation) afin d’efficacement neutraliser ces envahisseurs.28
1.2.5. Basophiles
Les basophiles sont les leucocytes les moins abondants en circulation. Contrairement aux autres granulocytes, les basophiles ne sont pas des phagocytes.2,3 En effet, ces cellules contiennent des granules contenant des composés jouant notamment un rôle dans la vasodilatation associée à l’inflammation et les allergies, tels l’histamine, l’héparine et des eicosanoïdes comme des prostaglandines et des leucotriènes. Ces cellules sont particulièrement susceptibles à l’activation par les immunoglobulines de type E (voir section 1.3.2.4).29–32
1.2.6. Mastocytes
Les mastocytes, comme les granulocytes, sont issus de la moelle osseuse et contiennent des granules. Toutefois, ces cellules migrent dans les tissus où ils complètent leur maturation.30,33 L’activation de ces cellules via leurs PRRs entraine leur dégranulation, libérant ainsi des médiateurs similaires à ceux retrouvés dans les granules des basophiles.34,35 Ils participent également à l’élimination des pathogènes par phagocytose et via la libération de ROS et d’AMPs. Il a également été démontré que les mastocytes peuvent libérer une matrice d’ADN selon un mécanisme similaire à la NETose des neutrophiles.33–35
1.2.7. Système du complément
Le système du complément est constitué de plus de 30 protéines présentes en circulation et jouant différents rôles dans l’immunité.36 Ce système contribue notamment à l’immunité innée grâce à un processus nommé « opsonisation ». Ce processus est médié par les opsonines du complément, qui recouvrent les organismes pathogènes afin de les marquer pour la phagocytose. Un second rôle associé au complément est le recrutement de phagocytes au site de leur activation afin d’éliminer les pathogènes rencontrés.36–38 Enfin, d’autres molécules du complément participent activement à l’élimination des agents pathogènes. Ces molécules forment le complexe d’attaque membranaire, un complexe qui crée un canal dans la membrane cellulaire des pathogènes rencontrés. L’accumulation de pores à la surface de ces microorganismes entraine la mort de ces derniers. Enfin, le système du complément contribue à l’immunité adaptative, notamment par l’amplification de la réponse aux anticorps.36–38
1.3. Système immunitaire adaptatif
Contrairement au système immunitaire inné, le système immunitaire adaptatif est programmé pour reconnaître des antigènes spécifiques à la surface des pathogènes. Cela permet une réponse ciblée permettant l’élimination de pathogènes spécifiques. Ce système comporte ainsi des cellules « mémoires » qui sont générées suite à la reconnaissance d’un antigène et permettent l’induction d’une réponse cytotoxique (voir section 1.3.1.2.2) ou humorale (voir section 1.3.2) rapide lorsque cet antigène est de nouveau rencontré.2
1.3.1. Les organes lymphoïdes
1.3.1.1.
Organes lymphoïdes primaires
Les organes lymphoïdes primaires sont les sites de production des lymphocytes, c’est-à-dire des cellules effectrices de la réponse adaptative, et où la compétence et tolérance de ces cellules sont mises à l’épreuve. On retrouve deux organes lymphoïdes primaires : la moelle osseuse et le thymus.39,40
La moelle osseuse est le site où les cellules souches lymphoïdes responsables de la production des lymphocytes sont localisées. Ces dernières se différentient notamment en lymphocytes T et B immatures (Figure 1.1), dont la maturation a lieu dans différents organes. En effet, alors que les lymphocytes T immatures sont relâchés dans la circulation pour rejoindre le thymus, les lymphocytes B poursuivent leur maturation dans la moelle osseuse.40,41
Dans leur organe respectif, les lymphocytes subissent différentes étapes de réarrangement de leur récepteur caractéristique : le récepteur des cellules T (T cell receptor, TCR) ou le récepteur des cellules B (B cell receptor, BCR). Ces étapes combinées à la sélection positive et négative dans ces organes permettent l’élimination des lymphocytes incapables de reconnaître les antigènes, ainsi que des cellules reconnaissant les antigènes du soi. Les lymphocytes T acquièrent également un co-récepteur CD4 ou CD8 permettant la reconnaissance du CMH-II ou CMH-I respectivement, ce qui définit alors les fonctions de ce type cellulaire. Au terme des étapes de sélection, les lymphocytes matures mais toujours naïfs n’ayant pas été éliminés migrent hors de la moelle osseuse et du thymus vers la périphérie, en direction des organes lymphoïdes secondaires.39,40,42–46
1.3.1.2.
Organes lymphoïdes secondaires
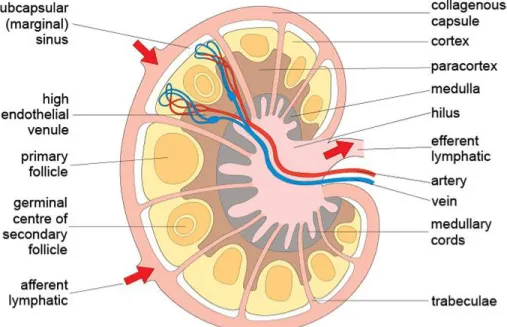
La rate et les ganglions lymphatiques sont les principaux organes lymphoïdes secondaires. Dans ces derniers, les cellules dendritiques et macrophages présentent les antigènes rencontrés sur leurs CMH-II et -I aux lymphocytes T CD4+ et CD8+ naïfs, respectivement.16 Cette présentation combinée à un signal de co-stimulation additionnel entraine l’activation de ces cellules, leur permettant de proliférer et de se différencier en cellules effectrices ou mémoires. Cette présentation et différenciation se produit dans la zone « T » des ganglions, c’est-à-dire le paracortex (Figure 1.2).39,40
Figure 1.2 : Structure d’un ganglion lymphatique. Traductions : Sinus sous-capsulaire
(subcapsular sinus), veinule à endothélium épais (high endothelial venule), follicule primaire (primary follicle), centre germinatif du follicule secondaire (germinal centre of secondary
follicle), vaisseau lymphatique afférent (afferent lymphatic), capsule de collagène
(collagenous capsule), cortex (cortex), paracortex (paracortex), médulla (medulla), hile (hilus), vaisseau lymphatique efférent (efferent lymphatic), cordons médullaires (medullary
cords), trabécule ou travée fibreuse (trabeculae). Figure tirée de « Histology, microscopy,
anatomy and disease », OpenLearn.47
1.3.1.2.1. Lymphocytes T auxiliaires CD4+ (Th)
Les lymphocytes T CD4+, aussi nommés T auxiliaires (T helper, Th), sont d’importants médiateurs de l’inflammation. Ils n’agissent pas directement sur les pathogènes,
mais induisent ou amplifient plutôt la réponse d’autres cellules effectrices. Selon l’environnement cytokinique, ces cellules se différencient pour adopter un sous-type associé au type de réponse généré. Pour produire des cellules Th1, de l’interféron-γ (IFN-γ) et interleukine (IL)-12 sont nécessaires, tandis qu’il faut de l’IL-4 pour les Th2, le facteur de croissance transformant (Transforming growth factor, TGF)-β, IL-6 et IL-23 pour les Th17, l’IL-6 et d’autres stimuli pour les T auxiliaires folliculaires (TFH) et de l’IL-2 et l’absence d’IL-6 pour les T régulateurs (Treg).2,48,49
Les lymphocytes Th1 contribuent principalement à la défense de l’organisme contre les pathogènes intracellulaires. Ces cellules sont principalement caractérisées par la production d’IFN-γ, mais produisent également d’autres cytokines tels l’IL-2 et le facteur de nécrose tumorale (tumor necrosis factor, TNF)-β. Elles participent notamment à l’activation des lymphocytes T cytotoxiques (Tc) (voir section 1.3.1.2.2) et amplifient l’activité des macrophages.40,50
Les lymphocytes Th2 jouent quant à eux un important rôle dans la défense contre les parasites, entre autres par la sécrétion d’IL-4, d’IL-5 et d’IL-13. Elles promeuvent l’activation des éosinophiles et mastocytes, ainsi que la production d’immunoglobulines de type E (IgE) par les plasmocytes, les cellules productrices d’anticorps (voir section 1.3.2.1.4).40,49,51
Les lymphocytes Th17 participent notamment à la défense contre les pathogènes extracellulaires au niveau de la peau et des muqueuses par la libération d’IL-17A et d’IL-22. Ces sécrétions permettent le recrutement des neutrophiles et induisent la libération d’AMPs par différentes cellules épithéliales.40,49,52,53
Les lymphocytes TFH ne quittent pas les ganglions suite à leur activation et migrent plutôt vers le cortex (Figure 1.2), où ils peuvent rencontrer les lymphocytes B. Dans cette zone, ils contribuent également au développement des centres germinatifs, d’importants sites de prolifération et de mutation somatique pour les lymphocytes B. Les lymphocytes TFH participent à l’activation des lymphocytes B et contribuent notamment à leur prolifération et différenciation en plasmocytes (voir section 1.3.1.2.3) par la sécrétion d’IL-21.48,49,54,55
Les lymphocytes Treg jouent quant à eux un rôle immunosuppresseur. Ces derniers sécrètent principalement de l’IL-10, qui inhibe la réponse inflammatoire par les cellules Th.
Les lymphocytes Treg sont particulièrement importants pour le maintien de la tolérance au soi en inhibant les lymphocytes auto-réactifs dans la périphérie. Ils jouent également un grand rôle dans la tolérance aux microorganismes mutualistes notamment au niveau de l’intestin.56– 58
1.3.1.2.2. Lymphocytes T cytotoxiques CD8+ (Tc)
La principale fonction des lymphocytes T CD8+, ou T cytotoxiques (Tc), est l’élimination des cellules infectées par des pathogènes intracellulaires. Ces cellules sont activées suite à la reconnaissance d’un antigène présenté sur le CMH-1 d’une cellule présentatrice d’antigènes (généralement une cellule dendritique ou un macrophage). Contrairement au CMH-II qui est presque exclusivement exprimé par les cellules présentatrices d’antigènes, le CMH-I est retrouvé à la surface de toutes les cellules du corps. Sur leur CMH-I, les cellules présentent différents antigènes qu’ils ont rencontrés, qu’ils soient des antigènes du soi ou du non-soi. Si un lymphocyte Tc activé rencontre une cellule présentant à sa surface l’antigène qu’il reconnait, le Tc entrainera l’élimination de cette cellule en créant des pores dans sa membrane par la libération de perforine et en la forçant à entrer en apoptose, c’est-à-dire en mort cellulaire programmée.15,40,59,60
1.3.1.2.3. Lymphocytes B et plasmocytes
L’activation des lymphocytes B se produit généralement dans le cortex des organes lymphoïdes secondaires (Figure 1.2). Pour leur activation, ces cellules doivent en premier lieu reconnaître un antigène via leur BCR. Comme les lymphocytes T, elles nécessitent un second signal pour leur activation. Bien que dans certaines situations, les cellules B peuvent être activées suite à la reconnaissance d’un composé bactérien comme second signal, ces cellules nécessitent généralement la coopération des lymphocytes TFH.3,61–63 Pour ce faire, la reconnaissance d’un antigène par le BCR entraine l’internalisation de cet antigène et sa dégradation. Les peptides issus de cette dégradation sont présentés à la surface des lymphocytes B sur leur CMH-II. La reconnaissance de ces peptides par un lymphocytes TFH entraine l’activation de la cellule B, stimulant sa prolifération.40,62,63 Certains lymphocytes B se différencieront alors en plasmoblastes, les précurseurs des plasmocytes capables de générer des anticorps mais pouvant toujours proliférer, et migreront dans les cordons médullaires des ganglions (Figure 1.2).62,63 D’autres migreront plutôt dans les centres
germinatifs64 pour proliférer et se différencier en plasmoblastes, puis en plasmocytes, ou en cellules mémoires avec l’aide des TFH.62,64,65 Les plasmocytes issus de cette différenciation migreront finalement vers les cordons médullaires ou quitteront les ganglions pour rejoindre la moelle osseuse.65,66
1.3.2. Immunité à médiation humorale
La réponse immunitaire à médiation humorale repose sur la reconnaissance d’antigènes par les lymphocytes B, leur différenciation en plasmoblastes ou plasmocytes et la libération d’anticorps pour contribuer à l’élimination des pathogènes rencontrés.40,67
1.3.2.1.
Les anticorps
En se différenciant en plasmoblastes et plasmocytes, les lymphocytes B acquièrent la capacité de produire une forme sécrétée et soluble de leur BCR, alors nommé anticorps ou immunoglobuline (Ig). Ces glycoprotéines sont constituées de chaînes légères responsables de leur affinité pour un antigène et d’une chaîne lourde pouvant être reconnue par différentes cellules. Cette chaîne lourde, qui décide du type de l’anticorps, lui confère ainsi différents rôle dans l’immunité adaptative.17,67,68 Certaines cytokines notamment produites par les lymphocytes TFH peuvent induire la recombinaison du gène codant pour cette chaîne lourde, modifiant ainsi le type d’anticorps généré par les plasmocytes.69,70
1.3.2.1.1. Immunoglobuline de type D
Les IgD sont coexprimés avec les IgM à la surface des lymphocytes B naïfs. Toutefois, suite à l’activation et la différenciation des lymphocytes B en plasmoblastes et plasmocytes, cet anticorps est très peu libéré. La sécrétion de cet anticorps ne semble pas avoir de rôle marquant dans la réponse immunitaire. 2,3,69
1.3.2.1.2. Immunoglobuline de type M
Les IgM sont les premiers anticorps produits suivant la stimulation antigénique. On les retrouve principalement dans le sang sous la forme de pentamères. Ils sont particulièrement efficaces pour activer le système du complément et participent également à l’opsonisation des antigènes.2,3,69
1.3.2.1.3. Immunoglobuline de type G
Les IgG sont, et de loin, les anticorps les plus abondants en circulation chez l’humain. On en retrouve plusieurs sous-groupes aux fonctions variées, mais de façon générale, ces anticorps jouent un important rôle dans la neutralisation et l’opsonisation des agents pathogènes, ainsi que dans l’activation du complément.69,71
1.3.2.1.4. Immunoglobuline de type E
Les IgE sont peu abondantes en circulation. Ces immunoglobulines fixent efficacement les mastocytes, induisant la libération de médiateurs qui entrainent de fortes réactions comme la toux, les éternuements et les vomissements. Leur production est induite par l’IL-4 et inhibée par l’IFN-γ.69,72
1.3.2.1.5. Immunoglobuline de type A
En général, les IgA sont les deuxièmes plus abondants anticorps en circulation. On les retrouve toutefois principalement au niveau des muqueuses, où leur sécrétion joue un important rôle dans la défense contre les pathogènes. La production d’IgA par les plasmocytes est induite par le TGF-β et l’IL-21, et augmentée par l’IL-5.2,69,73,74
2. Flore bactérienne intestinale
Le corps humain est constamment en contact avec des billions de microorganismes qui compétitionnent pour occuper l’espace à sa surface. Toutefois, plutôt que de systématiquement éliminer l’ensemble de ces colonisateurs microscopiques, le corps humain entretient une relation mutualiste avec ces microorganismes, en particulier au niveau de la muqueuse intestinale.58,75 En effet, c’est à la surface de ce tissu que se retrouve la plus importante concentration de microorganismes, soit plus de 100 fois plus qu’à la surface de tout autre tissu humain.76
2.1. Principaux rôles
La proximité de la flore microbienne intestinale, aussi nommée « microbiote » intestinal, avec l’épithélium de l’intestin lui permet de jouer d’importants rôles au niveau du métabolisme, de la protection contre les pathogènes et de l’homéostasie du système immunitaire humain.75
2.1.1. Métabolisme
Le microbiote intestinal est parfaitement positionné pour métaboliser certains aliments indigestes par la machinerie enzymatique humaine. En effet, certaines bactéries retrouvées dans l’intestin peuvent fermenter des composés comme des carbohydrates et polysaccharides complexes afin de libérer et d’utiliser l’énergie contenue dans ces polymères.77,78 Cette activité entraine la libération de sous-produits tels des monosaccharides et acides gras à courte chaîne (short-chain fatty acids, SCFA) comme le butyrate, le propionate et l’acétate, qui peuvent être absorbés et utilisés comme source d’énergie par le corps humain.77–80 L’un de ces SCFAs, le butyrate, fait également le sujet de nombreuses études en raison de ses propriétés anti-inflammatoires.81–86 Il a notamment été démontré chez la souris que le butyrate régule la différenciation des lymphocytes T, réduisant le ratio Th17/Treg dans la rate via l’inhibition de l’histone désacétylase 8.86 Le butyrate peut également inhiber l’activation du facteur de transcription nucléaire κB (nuclear factor κB, NF-κB), un facteur de transcription notamment impliqué dans l’induction de l’expression de plusieurs cytokines pro-inflammatoires incluant l’IL-1β, le TNF-α et l’IL-6. Le potentiel clinique du butyrate a été évalué au cours de certaines maladies inflammatoires incluant la maladie de Crohn, pendant laquelle la régulation de NF-κB par le butyrate semble réduire la réponse inflammatoire localisée à l’intestin des patients.87 Des évidences suggèrent également que la supplémentation de butyrate peut réduire le phénotype inflammatoire des monocytes circulants chez les patients atteints du syndrome métabolique.88 Ainsi, en contribuant au métabolisme du corps humain, la flore intestinale participerait à la régulation de la réponse inflammatoire chez l’homme.
2.1.2. Barrière contre l’implantation des pathogènes
Outre son rôle dans le métabolisme, le microbiote intestinal contribue également à divers mécanismes de résistance prévenant l’implantation de pathogènes. Localisé à la surface de la muqueuse intestinale, ce microbiote protège le corps humain en compétitionnant avec ces pathogènes pour l’espace et les nutriments (Figure 1.3).58,89–91 Ces microorganismes peuvent également combattre les bactéries délétères par la libération de composés telles les bactériocines, des toxines ciblant les souches pathogènes de leur espèce ou d’une espèce semblable.92,93 Un autre mécanisme employé par la flore microbienne afin d’éliminer les
pathogènes est la stimulation du système immunitaire afin d’induire une réponse contre ces envahisseurs. Cette flore peut en effet promouvoir la libération d’AMPs et d’IgA au niveau de la muqueuse intestinale par les cellules de Paneth et les plasmocytes, respectivement, permettant la neutralisation des pathogènes envahissant (Figure 1.3).94–97
Figure 1.3 : Le microbiote intestinal protège l’organisme contre les pathogènes. La flore
intestinale compétitionne avec les pathogènes pour l’espace et les nutriments retrouvés à la surface de la muqueuse intestinale. Ces organismes mutualistes et commensaux peuvent également promouvoir la libération de peptides antimicrobiens (AMPs) et d’IgA, permettant la neutralisation de ces pathogènes. Figure tirée de Belkaid & Hand, 2014.90
2.1.3. Développement et homéostasie immunitaire
L’utilisation de souris axéniques, c’est-à-dire de souris dépourvues de tout microorganisme dès leur naissance, a permis de mettre en lumière l’importante contribution du microbiote intestinal dans le développement et l’homéostasie du système immunitaire. En effet, l’absence de flore chez ces animaux provoque des déficiences tant au niveau de l’immunité adaptative que de l’immunité innée. À l’intestin, l’absence de microorganisme entraine une réduction de la taille et de la quantité des plaques de Peyer, qui sont des organes lymphoïdes secondaires associés à l’intestin, ainsi que des ganglions mésentériques.98,99 En
accord avec ces observations, une plus faible quantité de plasmocytes sont retrouvés dans ces organes, engendrant une production réduite d’IgA.73,100 De plus, plusieurs populations lymphocytaires sont moins abondantes dans la lamina propria de l’intestin,98 la couche de tissu conjonctif localisée sous l’épithélium intestinal. L’absence de microbiote a également été associée à une diminution de la sécrétion de certains AMPs à l’intestin.101,102
Bien que l’intestin soit particulièrement affecté par l’absence de flore chez les souris axéniques, ce dernier n’est pas le seul organe touché. Plusieurs organes lymphoïdes en sont également affectés, incluant d’autres ganglions périphériques, mais également la rate et la moelle osseuse.103,104 Il a par exemple été démontré que la flore intestinale stimule la myélopoïèse, c’est-à-dire la production des cellules de la lignée myéloïde (Figure 1.1). En effet, comparativement aux souris hébergées dans des animaleries exemptes de pathogènes spécifiques (Specific pathogen-free, SPF), le standard dans le milieu de la recherche, les souris axéniques présentent une importante réduction de différents leucocytes, incluant les neutrophiles, les monocytes et les macrophages, dans la rate et la moelle osseuse.105 Cette réduction de la myélopoïèse chez les souris axénique serait l’une des causes pour lesquelles ces animaux sont plus susceptible à développer divers infections, comme celles causées par
Listeria monocytogenes et Staphylococcus aureus.105,106
Considérant l’importance de la présentation antigénique dans l’induction d’une réponse immunitaire adaptative, il n’est pas surprenant que ce système soit fortement dérégulé en l’absence de microorganisme. Par exemple, les lymphocytes Th17, qui jouent un important rôle dans la défense de l’hôte au niveau des muqueuses, sont indétectables dans la lamina propria intestinale de souris axéniques.107 Pour remédier à cette déficience, la colonisation avec certaines bactéries comme la bactérie filamenteuse segmentée (SFB) permet d’induire la prolifération de cette population lymphocytaire, augmentant de ce fait la quantité intestinale de lymphocytes Th17 (Figure 1.4).108,109 La fonction des lymphocytes Treg est également grandement altérée en l’absence de flore et, comme les Th17, leur abondance en est réduite à l’intestin.110,111 Ces lymphocytes jouent un rôle majeur dans la tolérance immunitaire et aux microorganismes mutualistes, ce qui en fait d’importants agents dans le maintien de l’homéostasie immunitaire.57 Toutefois, chez les souris axéniques, ces cellules voient leurs propriétés anti-inflammatoires grandement diminuées.112 Enfin, un débalancement entre les lymphocytes Th1 et Th2 en faveur de ces derniers a été rapporté en
environnement stérile. Un rôle de la flore dans la résolution de ce dérèglement a été décrit, et impliquerait notamment la présentation du polysaccharide A (PSA) de Bacteroides
fragilis, un symbiote retrouvé dans l’intestin humain, aux lymphocytes Th par les cellules
dendritiques (Figure 1.4).113 Le PSA semble également favoriser la prolifération des lymphocytes Treg, donc d’une réponse anti-inflammatoire, au détriment des Th17 via l’activation du TLR2 à la surface des lymphocytes Th.114
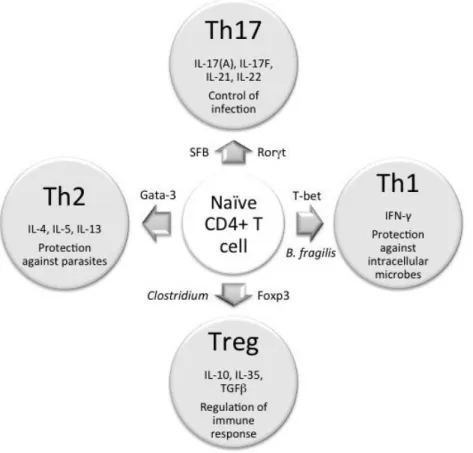
Figure 1.4 : Induction d’une réponse Th par des bactéries de la flore intestinale.
Différentes bactéries peuvent contribuer à la différenciation des lymphocytes Th (CD4+) naïfs en un sous-type spécifique. Chaque sous-type de lymphocytes Th requiert l’expression d’un facteur de transcription distinct et sécrète des cytokines spécifiques lui permettant d’exercer ses fonctions caractéristiques. Figure tirée de Wu & Wu, 2012.115
2.2. Dysbiose
Dans l’ensemble, le microbiote est essentiel au bon développement du système immunitaire et joue un grand rôle dans le maintien de son homéostasie. La flore intestinale constitue toutefois un écosystème complexe dont l’altération peut contribuer à de
nombreuses maladies inflammatoires.116 Le terme « dysbiose » a été associé à ce débalancement, caractérisé par une altération de la composition de la flore normale, par exemple par l’expansion de populations bactériennes nuisibles souvent de la famille des
Enterobacteriaceae, et impliquant généralement une réduction de la diversité
microbienne.117,118 Bien que les conséquences d’une dysbiose varient d’une condition à l’autre, une telle dérégulation peut entrainer l’élimination de bactéries ayant des fonctions protectrices et favoriser la croissance de pathogènes, contribuant au développement ou à l’amplification d’une réponse inflammatoire chronique.116
2.2.1. Pathogènes au cours de maladies inflammatoires chroniques
Les maladies inflammatoires de l’intestin (IBD), dont les plus connues sont la maladie de Crohn et la colite ulcéreuse, sont des maladies systémiques principalement caractérisées par une importante inflammation qui, selon la maladie, affecte des régions distinctes du tractus digestif.119 Ces maladies sont généralement associées à une importante réduction de la diversité microbienne à l’intestin. Des évidences suggèrent que cette diversité diminuée serait en partie causée par une perte de tolérance des lymphocytes T face aux bactéries mutualistes.95,120,121 L’élimination de ces bactéries, importantes dans le maintien de l’homéostasie intestinale, pourrait contribuer à l’inflammation retrouvée dans ces maladies. Supportant cette hypothèse, plusieurs évidences démontrent une réduction de bactéries productrices de butyrate, ainsi que de symbiotes promouvant la prolifération de cellules exprimant l’IL-10 chez les patients atteints d’IBD.122–124 La prolifération de pathogènes pourrait également contribuer aux processus inflammatoires retrouvés chez ces patients. En effet, contrairement aux souris colonisées avec le microbiote d’un sujet sain, il a été démontré qu’un modèle de souris transgénique axénique propice au développement d’IBD colonisé par le microbiote de patients atteints de maladie de Crohn développe une colite.125 De plus, la croissance de différentes espèces d’Enterobacteriaceae a été observée chez les patients atteints d’IBD, incluant la souche d’Escherichia coli adhérente et invasive (AIEC).126 Cette souche pathogène a notamment été démontrée comme pouvant infiltrer l’épithélium intestinal, se répliquer dans les macrophages et induire une réponse Th17 et Tc chez la souris, pouvant contribuer à l’inflammation retrouvée chez les patients atteints d’IBD.127–131 Malgré
ces évidences, nous ne savons toujours pas si la dysbiose observée chez les patients atteints d’IBD serait plutôt une cause ou une conséquence dans le développement de cette maladie.
Une quantité croissante d’évidences pointent vers un rôle important du microbiote intestinal dans la pathogenèse de la polyarthrite rhumatoïde, une maladie inflammatoire systémique et chronique affectant principalement les articulations.132 En effet, l’apparition d’une dysbiose a été observée chez les patients atteints de cette maladie, et des associations ont été démontrées entre la présence de pathogènes spécifiques tels Prevotella copri et
Porphyromonas gingivalis et son développement.111,133–138 Des études ont également dévoilé que SFB peut amplifier la sévérité de l’arthrite dans un modèle murin. Il a été décrit que cette bactérie lie l’épithélium de l’intestin et induit la sécrétion de sérum amyloïde A par les entérocytes, menant ultimement au recrutement et à la prolifération des lymphocytes Th17.109,139–141 L’IL-17A libérée par ces cellules contribuerait à amplifier la sévérité de l’arthrite dans ce modèle. Bien que SFB soit peu répandu chez l’humain, d’autres études ont permis d’identifier différentes bactéries retrouvées chez l’homme et ayant une activité similaire. Ces recherches ont identifié Bifidobacterium adolescentis, une bactérie notamment retrouvée dans certains suppléments de probiotiques, comme pouvant induire une réponse Th17 similaire à celle de SFB chez les souris arthritiques.142 Il semblerait ainsi que l’induction de l’immunité par cette bactérie pourrait avoir des effets bénéfiques chez certains individus, mais pourrait également amplifier l’inflammation retrouvée au cours des maladies inflammatoires, du moins dans un modèle murin.
2.3. Facteurs régulant la composition bactérienne
Une panoplie de facteurs peut favoriser la prolifération de certaines espèces bactériennes plutôt que d’autres, ce qui complique grandement la tâche d’identifier les causes des dysbioses. Parmi ces derniers, on retrouve plusieurs facteurs exogènes telles la diète, la consommation d’antibiotiques ou les infections. Différents facteurs endogènes contribuent toutefois à maintenir une composition équilibrée par la libération de différents composés tels les IgA et les AMPs.117
2.3.1. Facteurs exogènes
Comme le métabolisme des différentes bactéries qui constituent la flore intestinale varie d’une espèce à l’autre, la consommation de quantités variables de protéines, de gras et de carbohydrates affecte grandement l’abondance de ces microorganismes.143 Par exemple, la consommation d’une diète riche en gras a été associée à une réduction de la diversité de la flore intestinale chez la souris.144 La consommation de gras saturés issus du lait favoriserait également la prolifération de Bilophila wadsworthia, une bactérie ayant la capacité d’induire une réponse Th1 et ainsi contribuer au développement de colites dans un modèle de souris propice au développement d’IBD.116,145
L’administration d’antibiotiques représente un autre facteur pouvant altérer la composition de la flore intestinale. En effet, en plus des pathogènes, de nombreuses espèces bactériennes mutualistes peuvent être affectées par la consommation d’antibiotiques à large spectre administrés pour combattre ou prévenir les infections. Parfois, cette altération de la flore peut même être mesurée jusqu’à plusieurs années suivant l’administration passagère des antibiotiques.146–150
2.3.2. Facteurs endogènes
Malgré le caractère hautement variable de la flore intestinale, sa composition est généralement plus similaire au sein d’une même espèce. Cette observation témoigne de l’importance des facteurs endogènes pour le maintien d’un microbiote équilibré.151
Comme précédemment décrit, les IgA sont des anticorps jouant un important rôle dans la défense de l’organisme au niveau des muqueuses. Relâchés à la surface de l’épithélium intestinal, ils préviennent l’implantation de pathogènes à ce niveau, favorisant une composition bénéfique de la flore intestinale. En effet, leur capacité à cibler des bactéries ou composants bactériens spécifiques leur permet notamment d’éliminer ces pathogènes sans s’attaquer aux bactéries mutualistes colonisant cet environnement, permettant le développement d’une flore diversifiée et équilibrée.116,152–156
L’activité bactéricide des AMPs au niveau des muqueuses contribue également à moduler la composition de la flore intestinale.157 Chez les mammifères, les défensines sont probablement les AMPs les plus abondants à l’intestin.158 Libérées par les cellules de Paneth,