Segmentation robuste et automatique du cartilage du
genou sur des images d'IRM
Mémoire
Ali Hajiabadi
Maîtrise en génie électrique
Maître ès sciences (M.Sc.)
Québec, Canada
Segmentation robuste et automatique du cartilage du
genou sur des images d'IRM
Mémoire
Ali Hajiabadi
Sous la direction de :
iii
Résumé
L’objectif de cette recherche est de développer un algorithme de segmentation du cartilage du genou à partir des images IRM en façon automatique et robuste. Cette recherche est réalisée en collaboration avec Laboratoire BodyCad. Le but final de cette étude est de permettre à l’entreprise de générer des prothèses du genou personnalisées. L’algorithme développé dans cette étude sera utilisé pour planification des chirurgies médicales de genou, donc la précision et la fiabilité de l’algorithme sont des points critiques.
Afin de mieux comprendre les algorithmes existants de segmentation médicale, nous avons fait une évaluation de la littérature de ce domaine. Nous présentons une revue brève des recherches touchant le domaine de la segmentation des images médicales.
L’algorithme proposé dans cette étude est implémenté en deux versions : la version primaire et la version étendue. Dans la version primaire, la segmentation est effectuée basée sur les informations extraites à partir d’une image IRM, tandis que la version étendue de notre algorithme extrait les informations de deux images IRM simultanément. Les résultats obtenus en utilisant trois ensembles distincts d’images IRM se présentent et se comparent dans la section de conclusion. La comparaison montre que l’algorithme a la capacité de produire les résultats de grande qualité.
iv
Abstract
The objective of this research is to develop a robust automatic algorithm for segmentation of the knee cartilage from MRI images. This research is carried out in collaboration with BodyCad Laboratories. The ultimate goal of this study is to allow the company to generate personalized knee prostheses. The algorithm developed in this study will be used in medical knee surgery planning, so the precision and reliability of the algorithm are of crucial importance.
In order to achieve a better understanding of the existing algorithms in the field of medical segmentations, we performed a profound review of the literature in this field. We present a brief review of these studies in the literature review section.
The algorithm proposed in this study is implemented in two versions: the primary version and the extended version. In the primary version, segmentation is performed based on information extracted from an MRI image, while the extended version of our algorithm extracts information from two MRI images simultaneously. The results obtained using three distinct sets of MRI images are presented in the results section and a comparison is drawn in the conclusion section of this document. This comparison shows that the algorithm has the ability to produce high quality results.
v
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières... v
Liste des tableaux ... vii
Liste des figures ... vii
Remerciements ... ix 1 Introduction ... 1 1.1 Objectif ... 1 1.2 Problématique ... 2 1.3 Notre approche ... 2 2 Introduction à l’IRM ... 4
2.1 Historique de l’imagerie par résonance magnétique (IRM) ... 4
2.2 Le concept d’imagerie par résonance magnétique ... 5
2.2.1 Résonance magnétique ... 6
2.2.2 Bobines de shim ... 7
2.2.3 Champ magnétique secondaire et bobines RF ... 8
2.2.4 Solénoïdes supraconducteurs ... 8
2.2.5 Signal RM ... 9
2.2.6 Codage spatial ... 9
2.2.7 Les bobines de gradient ... 11
2.2.8 La structure d’une machine d’IRM ... 12
2.2.9 Caractéristiques du signal RM ... 14
2.2.10 Des images pondérées T1 et T2 ... 15
2.2.11 Les paramètres TE, TR et la séquence d'écho de spin (SE) ... 15
3 Revue de littérature ... 16
4 Méthodologie ... 25
4.1 Vue d'ensemble de notre méthode ... 25
4.2 Étude de cas (ensembles de données) ... 32
4.3 Structure des données ... 33
4.4 Segmentation osseuse ... 35
vi
4.6 Sélection de caractéristiques ... 38
4.6.1 La position du pixel dans l'image ... 39
4.6.2 L'intensité du pixel ... 40
4.6.3 Les dérivées secondes ... 40
4.6.4 Tenseur de structure ... 40
4.6.5 La matrice hessienne ... 41
4.6.6 Distance de la surface osseuse ... 42
4.6.7 Intensité des pixels dans l'image RM auxiliaire ... 42
4.6.8 Résumé de la liste des caractéristiques ... 43
4.7 Fonction d'extraction de caractéristiques ... 43
4.8 Normalisation de caractéristiques et pondération... 45
4.9 Optimisation (sélection de poids) ... 51
5 Résultats et discussion ... 58
5.1 Paramètres d'évaluation de chevauchement ... 59
5.1.1 Coefficient de similarité des données (DSC) ... 59
5.1.2 Intersection Over Union (IOU) ... 59
5.1.3 Précision, sensibilité et spécificité ... 60
5.2 Résultats en utilisant l’ensemble SKI10 ... 60
5.3 Utilisation de l’ensemble BodyCad et version préliminaire de l'algorithme ... 75
5.4 Utilisation de l’ensemble BodyCad et la version étendue de l'algorithme ... 81
5.5 Comparaison et discussion ... 82
vii
Liste des tableaux
Tableau 1 – Résumé de la liste des caractéristiques utilisées dans notre algorithme ... 43
Tableau 2 – DSC des résultats obtenus en utilisant trois différentes approches de normalisation ... 50
Tableau 3 – Séquences de poids de test utilisées pour l'optimisation ... 51
Tableau 4 – Définitions des opérandes utilisés dans les équations 10 à 14 ... 58
Tableau 5 – Résultats obtenus à l'aide de l’ensemble de test SKI10, sans optimisation ... 61
Tableau 6 – Résultats obtenus à l'aide de l’ensemble de test SKI10, avec optimisation ... 61
Tableau 7 – Résultats obtenus à l'aide de l’ensemble d’entraînement SKI10, sans optimisation ... 62
Tableau 8 – Résultats obtenus à l'aide de l’ensemble d’entraînement SKI10, avec optimisation ... 62
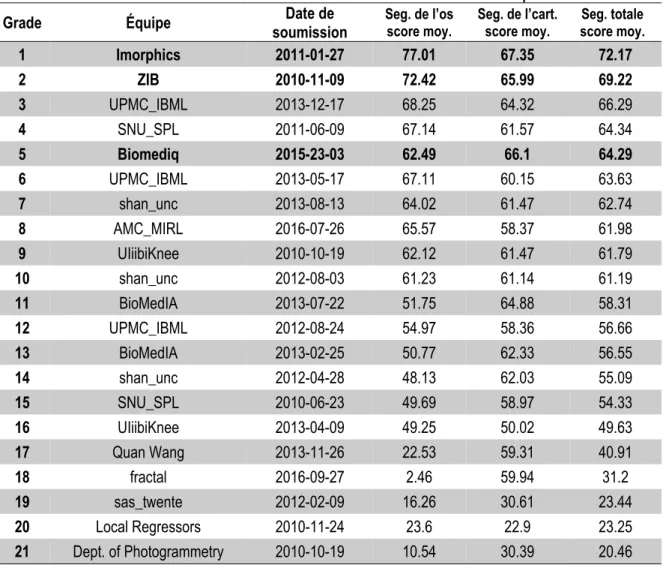
Tableau 9 – Tableau des résultats tirés du site Web SKI10 ... 63
Tableau 10 – Comparaison de notre algorithme avec trois algorithmes supérieurs du défi SKI10 ... 64
Tableau 11 – Résultats obtenus à l'aide de l'ensemble de test de BodyCAD, en utilisant une image IRM ... 75
Tableau 12 – Résultats obtenus à l'aide de l'ensemble de test BodyCAD, avec deux images IRM ... 81
Tableau 13 – Résultats obtenus en utilisant quatre images de l’ensemble SKI10 ... 82
Liste des figures
Figure 1 – Image d’un doigt humain ... 5Figure 2 – La première image du corps entier (la poitrine de Minkoff) ... 5
Figure 3 – Proton sous l'influence du champ statique B0 ... 7
Figure 4 – Concept des gradients de champ magnétique ... 10
Figure 5 – Maxwell paire de bobines générant un gradient dans le long de l’axe Z ... 12
Figure 6 – Bobines de gradient dans une machine d’IRM clinique ... 13
Figure 7 – Schéma écorché d’un scanographe RM ... 13
Figure 8 – Séquence d’écho de spin et ses TR et TE correspondants ... 15
Figure 9 – Organigramme du processus d'entraînement ... 27
Figure 10 – Organigramme du processus d'entraînement – 2e partie ... 28
Figure 11 – Capture d’écran de l’outil de segmentation conçu pour notre étude ... 29
Figure 12 – Organigramme du processus de segmentation ... 31
Figure 13 – Structure standard pour les ensembles utilisés dans notre étude ... 34
Figure 14 – Exemple de ROI pour la segmentation du cartilage du fémur ... 38
Figure 15 – Exemple de tenseur de structure, calculé au bord du cartilage ... 41
Figure 16 – Carte de distances des os fémoraux (a) Tranche IRM (b) La carte de distances ... 44
Figure 17 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Intensité dans l'image principale » — Normalisées en utilisant la valeur moyenne ... 46
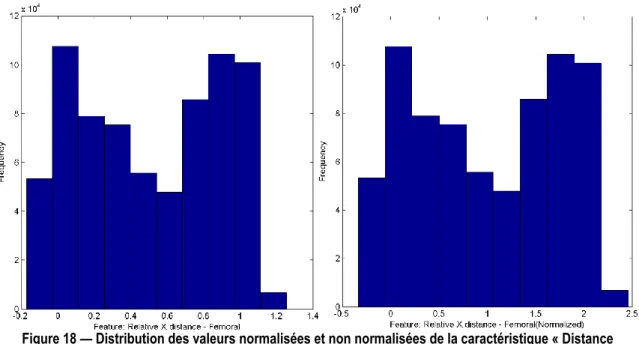
Figure 18 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Distance horizontale relative » — Normalisées en utilisant la valeur moyenne ... 47
Figure 19 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Intensité dans l’image principale » — Normalisées en utilisant la valeur maximale ... 48
Figure 20 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Distance horizontale relative » — Normalisées en utilisant la valeur maximale ... 48
viii
Figure 21 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Intensité dans
l'image principale » — Normalisée en utilisant 𝝋𝒊 = 𝟏𝝁𝒊 + 𝟐 ∙ 𝝈𝒊 ... 49
Figure 22 – Distribution des valeurs normalisées et non normalisées de la caractéristique « Distance horizontale relative » — Normalisée en utilisant 𝝋𝒊 = 𝟏𝝁𝒊 + 𝟐 ∙ 𝝈𝒊 ... 50
Figure 23 – Organigramme de sélection de poids (optimisation) ... 53
Figure 24 – Organigramme de sélection de poids (optimisation) – 2e partie ... 54
Figure 25 – Courbes de DSC par poids générées par la fonction d'optimisation - Caractéristiques 1 à 3 ... 55
Figure 26 – Courbes de DSC par poids générées par la fonction d'optimisation - Caractéristiques 4 à 7 ... 56
Figure 27 – Courbes de DSC par poids générées par la fonction d'optimisation - Caractéristiques 8 à 10 ... 57
Figure 28 – Diagramme de Venn décrivant les paramètres de chevauchement de segmentation ... 59
Figure 29 – Distribution du DSC des segmentations fémorales dans l’ensemble de test SKI10 (optimisé) ... 65
Figure 30 – Distribution du DSC des segmentations tibiales dans l’ensemble de test SKI10 (optimisé) ... 65
Figure 31 – Distribution de l’IOU des segmentations fémorales dans l’ensemble de test SKI10 (optimisée) ... 66
Figure 32 – Distribution de l’IOU des segmentations tibiales dans l’ensemble de test SKI10 (optimisée) ... 66
Figure 33 – Distribution de la précision des segmentations fémorales dans l’ensemble de test SKI10 ... 67
Figure 34 – Distribution de la précision des segmentations tibiales dans l’ensemble de test SKI10 ... 67
Figure 35 – Distribution de la sensibilité des segmentations fémorales dans l’ensemble de test SKI10 ... 68
Figure 36 – Distribution de la sensibilité des segmentations tibiales dans l’ensemble de test SKI10 ... 68
Figure 37 – Distribution de la spécificité des segmentations fémorales dans l’ensemble de test SKI10 ... 69
Figure 38 – Distribution de la spécificité des segmentations tibiales dans l’ensemble de test SKI10 ... 69
Figure 39 – Distribution du DSC des segmentations fémorales dans l’ensemble d’entraînement SKI10 ... 70
Figure 40 – Distribution du DSC des segmentations tibiales dans l’ensemble d’entraînement SKI10 ... 70
Figure 41 – Distribution de l’IOU des segmentations fémorales dans l’ensemble d’entraînement SKI10 ... 71
Figure 42 – Distribution de l’IOU des segmentations tibiales dans l’ensemble d’entraînement SKI10 ... 71
Figure 43 – Distribution de la précision des segmentations fémorales dans l’ensemble d’entraînement SKI10 ... 72
Figure 44 – Distribution de la précision des segmentations tibiales dans l’ensemble d’entraînement SKI10 ... 72
Figure 45 – Distribution de la sensibilité des segmentations fémorales dans l’ensemble d’entraînement SKI10 ... 73
Figure 46 – Distribution de la sensibilité des segmentations tibiales dans l’ensemble d’entraînement SKI10 ... 73
Figure 47 – Distribution de la spécificité des segmentations fémorales dans l’ensemble d’entraînement SKI10 ... 74
Figure 48 – Distribution de la spécificité des segmentations tibiales dans l’ensemble d’entraînement SKI10 ... 74
Figure 49 – Distribution du DSC des segmentations fémorals dans l’ensemble de test BodyCAD ... 76
Figure 50 – Distribution du DSC des segmentations tibiales dans l’ensemble de test BodyCAD ... 76
Figure 51 – Distribution de l’IOU des segmentations fémorals dans l’ensemble de test BodyCAD ... 77
Figure 52 – Distribution de l’IOU des segmentations tibiales dans l’ensemble de test BodyCAD ... 77
Figure 53 – Distribution de la précision des segmentations fémorals dans l’ensemble de test BodyCAD ... 78
Figure 54 – Distribution de la précision des segmentations tibiales dans l’ensemble de test BodyCAD ... 78
Figure 55 – Distribution de la sensibilité des segmentations fémorals dans l’ensemble de test BodyCAD ... 79
Figure 56 – Distribution de la sensibilité des segmentations tibiales dans l’ensemble de test BodyCAD ... 79
Figure 57 – Distribution de la spécificité des segmentations fémorals dans l’ensemble de test BodyCAD ... 80
ix
Remerciements
J'adresse mes remerciements aux personnes qui m'ont aidé à réaliser ce mémoire.
J’aimerais d’abord remercier Robert Bergevin. En tant que mon directeur de recherche, il m’a guidé dans mon travail avec des conseils et des explications exhaustives. Je remercie aussi Geoffroy Rivet-Sabourin, Directeur de recherche de l’entreprise Bodycad, pour sa collaboration en me fournissant des données et des équipements pour effectuer cette recherche.
J’aimerais exprimer ma gratitude à mes amis Félix et Quentin qui m'aidaient à la correction de ce mémoire.
Je tiens à remercier mes très chers parents et mon cher frère, Kamran qui ont été toujours là pour moi. Je remercie ma chère belle-soeur, Parisa, et mon cher beau-frère, Kaveh, pour m’avoir supporté.
Enfin, je souhaite particulièrement remercier ma femme, Shiva, qui était toujours à mes côtés pour son support également pour sa précieuse aide à la relecture et à la correction de mon mémoire.
1
1 Introduction
L'imagerie médicale est devenue l'un des examens les plus couramment utilisés par les médecins pour diagnostiquer une grande variété de maladies. Parmi toutes les méthodes d'imageries médicales, l'imagerie par résonance magnétique (IRM) pourrait être considérée comme la plus populaire en raison de sa fiabilité, de ses coûts relativement faibles, de sa nature non invasive et de sa souplesse d'utilisation sur différentes parties du corps, ce qui fait de cette méthode un outil de diagnostic pratique pour les médecins. La croissance d'utilisation de cette méthode ainsi que l’augmentation de la puissance informatique des ordinateurs et des processeurs au cours des dernières décennies ont encouragé les chercheurs à mener de nombreuses études dans le domaine du traitement numérique d’images captées par résonance magnétique.
Les objectifs visés par la plupart des études effectuées dans ce domaine sont de diagnostiquer une maladie, d’évaluer l’extension de son avancement ou de prédire le danger futur d’une maladie pour le patient. Dans ce document, nous examinerons et résumerons plusieurs de ces études. Nous expliquerons également comment fonctionne une machine d’imagerie par résonance magnétique, puis nous présenterons une petite terminologie de l’imagerie par RM. Nous continuerons en décrivant notre méthodologie et, finalement, nous présenterons nos résultats et conclurons en comparant nos résultats avec des études similaires dans ce domaine.
1.1 Objectif
L’objectif de cette étude est de développer un algorithme automatique et robuste de segmentation du cartilage du genou à partir des images IRM. Cette recherche est réalisée en collaboration avec BodyCad Inc., un pionnier dans les restaurations orthopédiques personnalisées. Cette entreprise, fondée en avril 2011, s’est engagée à fournir une éducation, une formation et un service de pointe, y compris l’accès aux techniciens de Bodycad PREP (processus d’évaluation de la restauration personnalisée) pour assurer la réussite chirurgicale. Incorporée en 2014, la société Bodycad USA a soumis ses premières soumissions réglementaires pour Santé Canada et la Food and Drug Administration (FDA) pour Bodycad Unicompartmental Knee System en 2015 ainsi que sa première procédure BodyCad OnCall. L’algorithme que nous avons développé dans cette étude sera utilisé par BodyCad Laboratories pour générer des modèles 3D précis des cartilages de genou à utiliser pour des chirurgies de genou personnalisées sur des patients souffrant d’arthrose (OA).
BodyCad est en possession d’un grand ensemble de données d’images IRM et CT Scan 3D, soit environ 250 images, obtenues sur quelques années à partir d’examens d’imagerie médicale effectués sur près de 100 patients. Les sujets d’examen appartiennent à un groupe diversifié concernant leur âge et leur sexe, certains souffraient d’OA au moment de l’examen alors que la plupart étaient en bonne santé. En plus de la base de données d’images, plusieurs fonctions Matlab ont également été développées par l’entreprise avant ce projet,
2
c’est le cas, par exemple, pour un algorithme de segmentation osseuse entièrement automatisé que nous utilisons dans notre algorithme de segmentation du cartilage.
1.2 Problématique
Comme mentionné précédemment, le résultat de l’algorithme sera utilisé dans des chirurgies, donc la précision et la fiabilité de l’algorithme développé sont des points critiques. Étant donné que la forme, la taille et presque toutes les caractéristiques du cartilage du genou diffèrent d’une personne à l’autre, l’algorithme devra être assez robuste pour produire des résultats fiables dans la plupart des cas. Il convient de mentionner que les caractéristiques physiques et chimiques du cartilage du genou dépendent de l’âge, du sexe, de la structure osseuse et du style de vie du patient. L’algorithme n’a accès à aucune de ces informations, les seules données accessibles sont des images IRM prises par les scanographes IRM 3T, donc l’algorithme se base uniquement sur les caractéristiques visuelles du genou et du cartilage dans les images IRM.
Le but final de cette étude est de permettre à l’entreprise de générer des modèles 3D précis du cartilage du genou afin de produire des prothèses personnalisées. Ce modèle 3D devra être généré pour le cartilage de chaque os de genou séparément, donc l’algorithme devra considérer le cartilage de chaque os comme un objet séparé. Les prothèses sur mesure seront plus tard utilisées dans la chirurgie du genou afin de restaurer une partie du genou des patients souffrant d’OA.
1.3 Notre approche
L’algorithme proposé dans cette étude est implémenté en trois parties : renseignement, optimisation et test (ou segmentation).
Renseignement : La partie « renseignement » consiste en une étape d’extraction de la caractéristique suivie de la production d’un modèle kNN (k Nearest Neighbor). Un grand nombre de vecteurs de caractéristiques sont extraits à partir des pixels de plusieurs images RM, qui sont segmentées manuellement. Puisque chaque vecteur de caractéristiques contient neuf composantes, un système de coordonnées 9-dimensionnel est formé comme un modèle kNN.
Optimisation : Afin d’attribuer des poids aux composantes du modèle kNN le plus efficacement possible, un algorithme itératif de sélection de poids, qui consiste en une boucle de « train-verify-adjust », est développé.
Segmentation : Notre algorithme de segmentation passe par les images IRM tranche par tranche en segmentant une tranche à la fois. La partie principale de l’algorithme de segmentation consiste en deux classifieurs binaires de kNN qui itèrent à travers les pixels de la région d’intérêt (ROI). Un de ces classifieurs,
3
en tant que classifieur à deux classes, décide si le pixel en question appartient au cartilage tibial tandis que le deuxième décide si le pixel appartient au cartilage fémoral.
4
2 Introduction à l’IRM
2.1 Historique de l’imagerie par résonance magnétique (IRM)
Ces dernières années, l’imagerie par résonance magnétique du genou est devenue le dernier cri de la modalité d’imagerie pour évaluer des troubles liés aux genoux. La diminution des coûts et la nature non envahissante des examens IRM la rendent largement acceptée pour l’évaluation du ménisque et des ligaments mais également de la moelle osseuse, des blessures des tissus mous ou des anomalies (Bolog, Andreisek, & Ulbrich, 2015).Dans ce chapitre, nous présentons aux lecteurs le concept, les principes de base, l’historique et l’évolution des systèmes d’imagerie par résonance magnétique. Bien que la première image de RM ait été acquise dans des tubes de fantôme expérimentaux il y a presque 40 ans, il est quelque peu difficile de définir exactement le début de l’imagerie par résonance magnétique (Elmaoğlu & Çelik, 2012).
En 1972, Raymond Damadian (Downstate Medical Centre, Brooklyn) a mesuré les temps de relaxation de T1 et T2 d’un tissu normal et d'une tumeur de rat en utilisant un appareil de résonance magnétique nucléaire (RMN) et a montré que la tumeur avait des temps de relaxation plus longs que celui du tissu normal. On peut considérer ces études comme les premières études cliniquement pertinentes de RMN.
En 1973, Paul Lauterbur, de l’Université de l’Illinois — Urbana-Champaign, a eu la brillante idée d’utiliser des gradients magnétiques dans trois plans (Gx, Gy et Gz) pour exciter l’emplacement de tranche choisi (le codage
spatial). Plus tard, Lauterbur a utilisé une technique de projection arrière utilisée pour la tomographie, a calculée
et a reconstruit la toute première image de RM. Les toutes premières images reconstruites de RM des « tubes à essai » ont l’air simples, mais elles ont marqué le début de l’IRM d’aujourd’hui.
Peter Mansfield, de Nottingham, a amélioré la technique IRM avec des études qu’il a faites en 1973, 1975 et 1977. Plus tard, il a développé l’utilisation de gradients dans le champ magnétique et a créé des modèles mathématiques en 1977 avec Andrew A. Maudsey. Il l’a utilisé cettes modèles pour prendre l’image d’un doigt humain (présentée à la figure 1) et, en 1978, celle d’une poitrine (montrée à la figure 2). Les temps requis pour produire ces images étaient respectivement de 23 minutes pour le doigt et de 5 heures pour la poitrine (“Invention of MRI,” n.d.). Mansfield a été le premier à utiliser la technique fondamentale qu’aujourd’hui nous appelons EPI (Echo-Planar Imaging).
En 1974, Jim Hutchison, Bill Edelstein et leurs collègues à l'Université d'Aberdeen, en Angleterre, ont développé le premier prototype de l'équipement de RM. Ces scientifiques ont reflété un rat en utilisant la technique d'écho de spin (Elmaoğlu & Çelik, 2012).
5
Figure 1 - Image d’un doigt humain, par Mansfield et Maudsley en 1977, tirée de (“Invention of MRI,” n.d.)
Figure 2 – La première image du corps entier (la poitrine de Minkoff), juillet 1978, tirée de (“Invention of MRI,” n.d.)
2.2
Le concept d’imagerie par résonance magnétique
L’atome le plus abondant dans le corps humain est l’atome d’hydrogène et celui-ci a le même numéro atomique que la masse atomique, qui est égale à un. Les atomes peuvent avoir le même numéro atomique en ayant des masses atomiques différentes (un nombre différent de neutrons) et ceux-ci sont appelés des isotopes.
Les protons dans le noyau tournent continuellement autour d’un axe et créent leur propre champ magnétique qui est orienté dans l’axe de rotation. Cette magnétisation peut être représentée par un vecteur appelé vecteur magnétique. Selon le théorème de Larmor, un moment magnétique (p. ex. un proton en spin) placé dans un champ magnétique avec un angle différent de l’angle du champ magnétique sent une force qui tente de l’aligner
6
le long de la direction du champ magnétique. Puisque le moment magnétique a une quantité de mouvement angulaire, la force amènera le moment magnétique à se balancer (Acremann et al., 2000).
Dans le domaine de la résonance magnétique, « le spin » a une définition spécifique : si un noyau a un nombre pair comme son numéro atomique et masse atomique, ce noyau n’a aucun spin, c.-à-d.qu’il est insensible à la résonance et ne peut donc pas être détecté par l’imagerie RM, tels l’argon et le cerium (Elmaoğlu & Çelik, 2012). Puisque le noyau d’hydrogène (1H) est présent dans 99,98 % des tissus du corps humain, c’est le noyau de
choix dans la grande majorité des examens RM d’aujourd’hui. Le phosphore (31P), le sodium (23Na) et le carbone
(13C) sont aussi reflétés avec IRM pour un certain nombre d’applications et de recherches.
2.2.1 Résonance magnétique
Quand le proton est placé dans l’influence d’un champ magnétique externe, son axe de rotation tournoie en s’accordant à la direction du champ magnétique. Cette interaction du vecteur magnétique du proton avec le champ magnétique crée une résonance magnétique.
La fréquence angulaire du mouvement du proton est déterminée par l'équation de Larmor (équation 1), l'équation la plus essentielle de la résonance magnétique dans laquelle la fréquence de précession (rotation), 𝑓 équivaut à une constante multipliée par la force du champ magnétique principal (Pooley, 2005).
𝑓 = 𝛾𝐵0 (1)
La constante 𝛾 est appelée le rapport gyromagnétique qui est une caractéristique de chaque type de noyaux. Pour le proton d’hydrogène, le rapport gyromagnétique est égal à 42,6 MHz/T (mégahertz par Tesla). 𝐵0 est
l’intensité du champ magnétique statique principal appliquée dans l’unité de Tesla et 𝑓 est la fréquence angulaire des protons placés dans le champ magnétique avec l’unité de MHz (le mégahertz).
Parce que nous venons de présenter une référence à une direction, il est important de discuter du système de coordonnées qui nous orientera pour la discussion ultérieure. La direction parallèle au champ magnétique principal est la direction longitudinale, qui peut aussi être appelée la direction Z. Le plan perpendiculaire à cette direction est appelé le plan transversal ou le plan x-y (Pooley, 2005).
La somme de tous les vecteurs magnétiques individuels des protons en spin produit un vecteur magnétique combiné appelé la magnétisation totale de ce groupe de protons. Puisque cette magnétisation est produite par le champ statique B0, nous montrons ce paramètre avec M0. Cette magnétisation, comme le champ
7
Figure 3 – Proton sous l'influence du champ statique B0, adopté de (Elmaoğlu & Çelik, 2012)
Avant l’application du champ magnétique principal, les protons avaient des spins aléatoires, neutralisant le vecteur magnétique de l’un et l’autre, c.-a-d. que leur magnétisation totale était zéro. Même après l’application du champ magnétique, les composantes X et Y de la magnétisation totale sont négligeables. Par conséquent, appliquer un champ magnétique momentané et perpendiculaire au champ existant balancerait facilement les axes de rotation vers le plan x-y, réduisant la composante Z de la magnétisation totale significativement. Afin de faire une résonance avec les protons en spin et ne pas fausser le modèle de précession des protons, ce champ magnétique devrait avoir la même fréquence que les protons en spin (voir l’équation 1).
Le champ magnétique secondaire appliqué s’appelle 𝐵1. Le signal généré par ce champ est appelé « l'impulsion
RF1 » et l’inclinaison causée par ce champ magnétique est appelée l'angle de nutation2. L'angle de nutationpeut
être ajusté en modifiant la durée de l'impulsion RF et l’intensité du champ B1.
La coupe du champ secondaire fait en sorte que l’axe de rotation des protons commence à se plier à nouveau sur sa direction originelle. Ayant une bobine installée perpendiculairement à cette variation du flux magnétique, l’appareil RM détecte et reçoit le courant induit qui s’appelle le « Signal de RM ».
2.2.2 Bobines de shim
La qualité du signal reçu exige une bonne homogénéité dans le champ principal 𝐵𝟎. Quand un objet est placé
dans le champ principal, il crée des effets de susceptibilité locaux, perturbant le champ magnétique local, si bien que ces artéfacts de susceptibilité doivent être corrigés. Les bobines de shim, aussi connues comme des bobines de correction, sont utilisées pour ajuster ou « shimmer » des inhomogénéités du champ magnétique 𝐵𝟎 et sont très importantes pour la qualité du signal reçu (Jacobs, Ibrahim, & Ouwerkerk, 2007).
1 En anglais : RF Pulse 2 En anglais : Flip Angle
8
Il y a deux familles de bobines de shim, soit passives et actives. Les bobines passives sont des plats de métal installés dans le tunnel de la machine RMou à la surface de l’aimant. Les bobines de shim actives fournissent plus de rajustements au champ autour de l’objet d’intérêt. La majorité des systèmes cliniques et des systèmes de recherche utilisent autant des bobines passives qu’actives pour obtenir des images de grande qualité.
Le champ magnétique secondaire est produit en passant un signal CA (courant alternatif) par une bobine perpendiculaire à l'axe Z. L'amplitude de ce signal change la force du champ produit et, comme mentionné précédemment, sa fréquence est calculée en utilisant l'équation 1.
2.2.3 Champ magnétique secondaire et bobines RF
Avant d’appliquer le champ magnétique principal, les protons avaient des spins aléatoires, donc leurs vecteurs magnétiques s’annulaient l’un l’autre, par conséquent l’aimantation totale est nulle. Même après avoir appliqué le champ magnétique, l’aimantation totale a des composants X et Y négligeables. Donc, l’application d’un champ magnétique momentané perpendiculaire au champ existant balance les vecteurs de rotation vers le plan x-y plus facilement, ce qui réduit significativement la composante Z de l’aimantation totale. Afin d’obtenir une résonance avec les protons en rotation et de ne pas distordre le rythme de précession des protons, ce champ magnétique devrait avoir exactement la même fréquence que les protons en rotation.
Le champ magnétique secondaire est produit en faisant passer un signal en courant alternatif par une bobine perpendiculaire à l’axe Z. L’amplitude de ce signal modifie l’intensité du champ généré et, comme déjà mentionné, sa fréquence est calculée à l’aide de l’équation 1.
Le champ magnétique secondaire s’appelle 𝐵1, le signal CA que crée ce champ s’appelle impulsion RF (ou RF Séquence) et l’inclinaison causée par ce champ magnétique s’appelle « angle de flip ». L’angle de flip peut être réglé en modifiant la durée de l’impulsion RF et l’intensité du champ 𝐵1. La bobine qui génère l’impulsion RF
s’appelle « bobine RF » et agit également comme récepteur du signal RM3. Plusieurs bobines RF sont incluses
dans l’ensemble de scanographe d’imagerie RM clinique : il y a une grande bobine RF qui est utilisée pour les balayages corporels avec plusieurs bobines plus petites conçues pour balayer des parties du corps, par ex. le genou, la tête, le bassin, etc.
2.2.4 Solénoïdes supraconducteurs
Les aimants utilisés pour produire le champ statique principal dans les scanographes RM sont des solénoïdes supraconducteurs, c.-à-d. que les conducteurs utilisés pour ces solénoïdes sont refroidis à hauteur de 4 °K. Un
9
fort courant électrique peut donc circuler à travers eux pour produire des champs magnétiques de plusieurs teslas sans dissipation d’énergie majeure.
Quant au système de refroidissement, desrafraîchissants cryogéniques (habituellement de l’hélium liquide) sont utilisés dans des scanographes RM et ils sont généralement dispendieux. Il est aussi nécessaire d’entretenir le système de refroidissement à intervalle de quelques mois en remplissant à nouveau les réservoirs d’hélium liquide de la machine. La plupart des systèmes RM cliniques utilisent des aimants supraconducteurs pour produire un champ statique principal de 1,5 T à 3,0 T. Les machines RM de recherche (clinique ou expérimentale) peuvent avoir des intensités de champ de 4,0 T à 9,4 T (Jacobs et al., 2007).
2.2.5 Signal RM
Comme mentionné ci-dessus, le tunnel principal de la machine de RM contient un solénoïde supraconducteur énorme qui produit un champ magnétique statique très fort. Le patient est placé au centre de ce tunnel. Ensuite, la machine produit un champ magnétique secondaire de courte durée pour produire un angle de flip spécifique. En coupant ce champ secondaire, le vecteur de rotation des protons commence à se replier vers sa direction originelle. Ayant une bobine installée perpendiculairement au plan transversal, la machine MR capte le courant induit par ces mouvements, appelé le signal RM.
2.2.6 Codage spatial
Avec le processus décrit jusqu’ici, la machine de RM capte le signal RM produit par tous les protons en spin du corps en même temps. Ce n’est cependant pas exactement ce que nous cherchons dans l’imagerie médicale. Nous avons besoin d’informations spatiales (les coordonnées X, Y et Z) que nous obtenons également du signal. Pour ce faire, les concepteurs de machines d’IRM ont divisé l’espace à l’intérieur du tunnel principal de la machine en petits voxels. La machine ajuste légèrement la fréquence de résonance des protons d’hydrogène situés à l’intérieur de ces voxels pour distinguer le signal RM généré par chacun d’eux. En mettant ces signaux RM dans une matrice 3D concernant l’emplacement spatial du voxel correspondant, la machine produit l’image de RM.
Dans le domaine des télécommunications, la modulation (p. ex. la modulation de fréquence) est utilisée afin de faire la différence entre les données transmises par un seul média. Le même concept peut être utilisé ici, si nous pouvons faire une petite modification dans la fréquence de la précession de tous les protons à l’intérieur des quelques voxels. Ainsi, le champ magnétique secondaire résonne seulement avec ces protons, créant le signal RM produit par la précession d’entre eux seulement. Ceci est exactement ce que les machines de RM font. Des bobines spéciales installées dans les machines de RM produisent des gradients de champ magnétique. Ces
10
gradients magnétiques ajustent la force du champ magnétique principal le long d’un axe. On montre le concept d’un gradient magnétique appelé codage spatial à la figure 4.
Figure 4 – Concept des gradients de champ magnétique, adapté de (HarounExplainIt, 2013)
Comme indiqué à la figure 4, le gradient de champ magnétique change la fréquence de la précession des protons en fonction de leur emplacement. Par conséquent, seuls certains protons (ceux placés sur la ligne pointillée dans la figure) sont excités par le champ magnétique principal (𝐵0), résonnant avec le champ
secondaire et produisant le signal RM. La valeur de 15 mT montrée dans la figure est un exemple, les pentes des gradients magnétiques étant typiquement de 1 % à 3 % de l’intensité du champ statique principal et ayant une unité de milli Tesla par mètre (mT/m) (Elmaoğlu & Çelik, 2012).
En déplaçant ce gradient le long d’un axe, nous concentrons l’imagerie sur un plan de l’espace intérieur du tunnel du scanographe, c’est-à-dire le plan contenant la ligne pointillée à la figure 4. Dans quelques sources de littérature, le déplacement du gradient, qui change le plan d’imagerie le long de l’axe Z, est appelé la « Sélection
de tranche » ou « Sélection de coupe »4 (Jacobs et al., 2007). L’épaisseur de la tranche choisie dépend de la
largeur de la bande de l’impulsion RF et de la pente du gradient, donc de l’inclinaison de la ligne rouge (figure 4). En général, les gradients avec les plus grandes inclinaisons donneront des tranches plus minces (Jacobs et al., 2007).
11
En plus de la sélection de tranche, soit la coordonnée en Z, nous devrions aussi avoir les coordonnées de X et de Y des voxels étant parcourus. À ces fins, deux autres encodeurs spatiaux sont utilisés dans les machines d’IRM. Un d’entre eux produit un gradient magnétique le long de X, produisant un spectre linéaire dans la fréquence de résonance des protons le long de l’axe de X, tandis que l’autre produit un gradient de phase le long de l’axe de Y. Pour résumer, après la sélection de tranche, le signal RM pris par la bobine RF est produit par un groupe de protons qui appartiennent à une tranche spécifique et sont spatialement encodés par leur fréquence et leur phase unique. En contenant les composants de la fréquence et de la phase, le signal RM peut être transformé dans le domaine fréquentiel (utilisant FFT par exemple) afin d’obtenir une image 3D de RM.
Il importe de mentionner ici que, contrairement à la plupart des cas de traitement du signal dans lesquels le composant de phase dans le domaine fréquentiel est rejeté parce qu’il ne contient pas d’informations utiles (Smith et al., 1997), le composant de phase est très important et pourrait nous fournir les informations spatiales des voxels le long de l’axe Y.
La résolution d’image RM est directement proportionnelle à la transformation utilisée pour convertir le signal RM à son représentant dans le domaine fréquentiel. Si la transformée de Fourier discrète (TFD) a été utilisée pour transformer le signal RM au domaine fréquentiel, l’image aurait la même résolution que le TFD.
2.2.7 Les bobines de gradient
Pour un système clinique standard de RM, les gradients magnétiques sont produits en utilisant deux bobines dans lesquelles les courants circulent dans des directions opposées l’une à l’autre (appelées paire de Maxwell). Le champ magnétique au centre d’une bobine s’ajoute au champ 𝐵0, tandis que le champ magnétique au centre de l’autre se soustrait de 𝐵0, créant ainsi un gradient dans le champ 𝐵0 (Currie, Hoggard, Craven, Hadjivassiliou, & Wilkinson, 2013). Un exemple de ce concept est illustré à la figure 5.
12
Figure 5 – Maxwell paire de bobines générant un gradient dans le long de l’axe Z, pris de (Currie et al., 2013)
Les bobines de gradient des axes X et Y ont des formes différentes, la figure 6 montre la forme et le positionnement de toutes les bobines de gradient dans une machine d’IRM clinique. Les bobines de gradient sont la cause de la plupart des bruits qu’une machine d’IRM fait lorsqu’elle fonctionne.
2.2.8
La structure d’une machine d’IRM
La figure 7 présente un schéma écorché d’un scanographe de RM. Dans cette figure, l’aimant est un aimant supraconducteur qui produit le champ statique principal et les bobines de gradient produisent les gradients magnétiques nécessaires pour : la sélection de tranche (le long de Z), le codage de la phase (le long d’Y) et le codage de la fréquence (le long de X). Finalement, la bobine radiofréquence produit l’impulsion RF (la séquence RF) et capte le signal RM.
13
Figure 6 – Bobines de gradient dans une machine d’IRM clinique, prise de (“MRI,” n.d.)
Figure 7 – Schéma écorché d’un scanographe RM, adapté de (“MRI,” n.d.)
14
2.2.9 Caractéristiques du signal RM
Pendant l’examen IRM, le signal RF est activé et éteint fréquemment, ce qui provoque l’alternance au champ magnétique créé par des protons (aimantation transversale). Ces altérations du champ magnétique induisent un signal électrique dans la bobine RF qui s’appelle le signal RM. Des radiologues appellent le signal capté (avant le traitement) les données brutes ou k-space (Elmaoğlu & Çelik, 2012). Comme décrit précédemment, nous devons effectuer une transformée de Fourier rapide (FFT) sur les données brutes de chaque coup afin d’obtenir le signal généré par les protons dans chaque voxel.
Lorsque l’impulsion RF est activée, deux processus se produisent en même temps. D’une part, l’amplitude du vecteur d’aimantation transversale (sur le plan x-y) augmente, entraînant une diminution de sa projection sur l’axe Z; d’autre part, lorsque l’impulsion RF est désactivée, les protons commencent à se détendre, ce processus s’appelle la relaxation du signal RM. L’étape de relaxation consiste en deux diminutions graduelles : la diminution de l’aimantation transversale vers zéro, qui s’appelle « relaxation T2 », et le retour de la force du champ magnétique principal vers sa valeur originale, appelé « relaxation T1 ».
Les diminutions mentionnées ci-dessus se produisent progressivement sur une courte période. Lorsque l’impulsion RF est éteinte, les protons commencent à distribuer leur énergie à leur environnement afin de revenir à leur état stationnaire. La vitesse à laquelle les protons perdent leur énergie est proportionnelle au taux de tumbling de la molécule auquel ils appartiennent, soit le taux de mouvement moléculaire (Currie et al., 2013). Étant donné que le taux de change de l’énergie entre les protons et leur environnement est proportionnel à la différence entre leur niveau d’énergie, la courbe de relaxation est une exponentielle qui, théoriquement, ne rencontrera jamais sa valeur cible, ce qui fait que l’examen IRM prendra trop de temps si nous attendons que tous les protons perdent complètement leur énergie induite. Par conséquent, la constante de temps de la courbe est utilisée comme représentant de la vitesse à laquelle la courbe s’approche de sa valeur cible, et cette vitesse représente le taux de tumbling du tissu auquel les protons appartiennent. Par exemple, l’eau libre et l’hydrogène lié ont des temps de relaxation T1 relativement longs (~ 4 secondes), mais T1 pour l’eau structurée (l’eau liée par d’autres tissus) est beaucoup moindre (~ 400 à 800 ms) (Hahn, 1950).
Il existe également une courbe lorsque la force du champ magnétique principal augmente à sa valeur originale, la constante de temps de cette courbe ascendante s’appelle T1, tandis que T2 est la constante de temps de la courbe de décroissance de la magnétisation transversale. Pour les tissus vivants, T2 est beaucoup plus court que T1.
15
2.2.10 Des images pondérées T1 et T2
Ce qui aide les radiologues à distinguer les tissus adjacents dans une image IRM, c’est le contraste entre ces tissus dans l’image. Dans les images IRM pondérées en T1, la valeur T1 gère la luminosité des pixels, d’où le contraste. De même, dans les images pondérées en T2, c’est T2 qui contrôle ces paramètres.
2.2.11 Les paramètres TE
5, TR
6et la séquence d'écho de spin (SE)
Une fois que le patient est placé dans le tunnel du scanographe d’IRM, l’appareil active et désactive l’impulsion radiofréquence. Cette impulsion modulée induit le signal RM, composé des courbes ascendantes et descendantes mentionnées dans la dernière section, dans la bobine RF. Comme expliqué précédemment, la largeur d’impulsion du signal RF ajuste l’angle de flip des protons en rotation.
Différentes séquences RF peuvent être générées en mélangeant différents modèles d’angles de flip. Par exemple, l’une des séquences les plus élémentaires utilisée dans l’imagerie par IRM consiste en une impulsion de 90° suivie de plusieurs impulsions de 180°. Cette séquence (figure 8), appelée la séquence « écho de spin », est largement utilisée dans l’imagerie IRM pour obtenir des images pondérées T1 et T2.
Figure 8 – Séquence d’écho de spin et ses TR et TE correspondants
À la Figure 8, les impulsions noires représentent les impulsions RF envoyées par la bobine RF, tandis que les impulsions rouges représentent le signal capté par elle. Comme on peut le voir, TE est le délai entre la première impulsion du flip 90 degrés et le premier pic du signal capté, et TR est la période de la séquence RF complète, c.-à-d., l’intervalle entre deux impulsions du flip 90 degrés ultérieures.
5 Temps d’écho (en anglais : Echo Time)
16
3 Revue de littérature
Dans la littérature existante, la segmentation de l’IRM, en particulier la segmentation du cartilage du genou, est discutée de manière exhaustive. Une courte revue des écrits touchant de près le domaine de la segmentation des images médicales est présentée dans cette section.
Étant donné que l’objectif de notre étude est de produire un modèle 3D précis (ou un nuage de points) du cartilage du genou, nous avons fait une évaluation approfondie de la littérature de ce domaine pour mieux comprendre les algorithmes existants de segmentation du cartilage du genou ainsi que leurs avantages et lacunes. Dans ce chapitre, nous explorons quelques études en lien avec notre travail et décrivons leurs méthodologies, leurs capacités et leurs inconvénients.
Obtenir des modèles 3D à partir d’images de résonance magnétique est possible grâce à deux approches : (1) par segmentation directe de la partie en question en utilisant des méthodes de segmentation tridimensionnelle ou (2) par segmentation de la partie tranche par tranche dans les images IRM qui produit un nuage de points 3D.
Il y a un large éventail de solutions proposées par les chercheurs à cause de la variété des paramètres dans la problématique. La séquence d’IRM à utiliser, l’étendue de l’autonomie de l’algorithme, la précision requise, etc. ne sont que quelques exemples de tels paramètres.La caractéristique évaluée du cartilage est un autre paramètre, par exemple certaines recherches se concentrent sur la mesure de l’épaisseur du cartilage (Haubner et al., 1997; S. Koo, Gold, & Andriacchi, 2005; Lösch, Eckstein, Haubner, & Englmeier, 1997) alors que certaines attirent l’attention sur l’affirmation de la morphologie du cartilage du genou en général (Cohen et al., 1999; F. Eckstein et al., 2002; F. Eckstein, Cicuttini, Raynauld, Waterton, & Peterfy, 2006; Hohe, Ateshian, Reiser, Englmeier, & Eckstein, 2002).
Plusieurs algorithmes sont suggérés pour comparer les caractéristiques du cartilage du genou entre différentes personnes (Carballido-Gamio et al., 2008; Li et al., 2007), et il existe également une vaste variété d’études qui portent sur le suivi des changements des montants des caractéristiques pour un patient sur une durée spécifique (F. Eckstein et al., 2007, 2009; Gandy et al., 2002; Kauffmann et al., 2003).
En plus des études orientées sur le mesurage, il y a aussi des recherches ayant pour but de générer des modèles 3D du cartilage.Généralement, ces méthodes sont classifiées en deux catégories : supervisées et non supervisées. La majorité des méthodes supervisées nécessite une interaction de l’utilisateur afin de générer tout résultat (Grau, Mewes, Alcaniz, Kikinis, & Warfield, 2004; Warfield, Kaus, Jolesz, & Kikinis, 2000). Cette interaction de l’utilisateur varie de la sélection des quelques points-graines pour dessiner une ROI (région
17
d’intérêt) jusqu’à l’ajustement des paramètres d’une transformation affine sur une segmentation générée automatiquement afin de la recaler parfaitement à l’image. Il y a aussi des méthodes automatiques dans le sens où l’intervention de l’opérateur peut être nécessaire pour compléter le processus. Dans ce cas, le résultat doit être indépendant de l’opérateur pour que l’algorithme soit considéré automatique (Clarke et al., 1995).
Cohen et coll. (1999) évaluent la précision tridimensionnelle de l’imagerie par résonance magnétique (IRM) à mesurer la topographie des surfaces articulaires et l’épaisseur des cartilages sur des genoux cadavériques. Ils examinent différentes séquences d’IRM en passant en revue des études existantes. Selon leur revue, Hayes et ses collègues (1990) montrent que les images pondérées T1 sont capables de l’identification de toutes les lésions du cartilage du genou, à l’exception du ramollissement précoce du tissu cartilagineux sans changement à son contour.
Une autre étude révèle que la détection de petites lésions n’est pas fiable, puisque les techniques d’imagerie pondérée T1 produisent des artefacts de changement chimique et un effet de volume partiel (Hodler, Trudell, Pathria, & Resnick, 1992). Bien que les images pondérées T2 se préconisent également pour détecter le cartilage, des tests supplémentaires ne présentent aucun avantage (Recht et al., 1993).
L’étude de cas de Cohen et coll. (1999) porte sur six genoux cadavériques fraîchement congelés et les genoux de quatre bénévoles, imagés avec une acquisition tridimensionnelle « Spoiled Gradient-Recalled » (SPGR) avec suppression de graisse en utilisant une machine d’IRM 1,5 T avec une résolution de 0,47 x 0,47 x 1,0 mm. Ils évaluent leur méthode en comparant leurs résultats avec la méthode stéréophotogrammétrique (SPG) étalonnée (Ateshian, 1993; Ateshian, Kwak, Soslowsky, & Mow, 1994; Kwak et al., 1997; Soslowsky et al., 1992).
Deux segmentations sont effectuées pour extraire des mesures topographiques de l’IRM, soit une segmentation manuelle et une semi-automatique. Après l’examen IRM, les six genoux cadavériques sont disséqués et leurs surfaces articulaires sont quantifiées en utilisant des méthodes stéréophotogrammétriques, employées par Ateshian et d’autres auparavant (Ateshian, Rosenwasser, & Mow, 1992; Ateshian, Soslowsky, & Mow, 1991). En dernier lieu, les mesures de surface de l’IRM sont comparées numériquement avec les mesures SPG.
Pour les six genoux cadavériques, les précisions moyennes des mesures de la surface du cartilage et de l’os sous-chondral sont de 0,22 mm et 0,14 mm respectivement, et les mesures de l’épaisseur montrent une précision moyenne de 0,31 mm. L’étude constate que tandis que la plupart des erreurs peuvent être attribuées à erreur aléatoire de la mesure, la précision est quelque peu affectée par des erreurs systématiques.
18
Cependant, Cohen et al. (1999) démontrent que l’IRM clinique est capable de fournir des mesures précises; ils confirment aussi que la séquence SPGR avec suppression de graisse peut être utilisée pour quantifier la topographie, l’épaisseur, les zones de contact et les courbures de surface du cartilage du genou.
Une approche computationnelle a été développée par Stammberger et al. (1999) pour quantifier la distribution de l’épaisseur du cartilage articulaire en trois dimensions par la résonance magnétique (MR), indépendamment du plan d’imagerie et pour tester la reproductibilité de la méthode au sein du vivant. Dans cette recherche, les auteurs ont l’intention de surmonter les limitations des travaux antérieurs (Felix Eckstein et al., 1996; Haubner et al., 1997; Lösch et al., 1997; Schnier et al., 1997) en développant une nouvelle technique computationnelle basée sur la segmentation de la surface du cartilage séparément de l’interface os-cartilage en utilisant la carte de distances euclidienne en 3D.
La précision de la méthode est évaluée par six ensembles MR répliqués du cartilage articulaire du genou de huit bénévoles. Leurs résultats montrent un excellent chevauchement avec la solution analytique. Les valeurs d’épaisseur maximale sont identiques (entre 0,03 et 0,08 mm) et la différence de valeurs d’épaisseur moyenne est d’environ 0,1 voxel (dans la plage de 0,11 à 0,18 mm). Les auteurs affirment que leur travail est le premier à signaler une haute reproductibilité de la quantification de l’épaisseur du cartilage du genou au sein du vivant avec l’IRM en 3D, indépendamment de la position de la section originale et de l’orientation d’imagerie (Stammberger et al., 1999).
Une méthode non supervisée populaire est proposée par Pakin et al. (2002) pour générer une carte d’épaisseurs 3D du cartilage articulaire. La méthode consiste en deux parties principales : l’extraction du cartilage et le calcul de la carte d’épaisseurs. La segmentation initiale pour l’extraction du cartilage est obtenue en utilisant l’algorithme proposé dans (Tamez-Pena, 1999) basé sur la croissance de région. Les régions produites au cours de ce processus sont ensuite étiquetées comme cartilage ou non-cartilage en utilisant une procédure de vote binaire (Pakin, Gaborski, Barski, Foos, & Parker, 2001) basée sur des connaissances anatomiques préalables établissant que le cartilage et les tissus osseux devraient être proches les uns des autres.
Après l’extraction du cartilage, le cartilage fémoral se sépare du cartilage tibial par détection de l’interface entre eux en utilisant un modèle déformable proposé par Kass, Witkin et Terzopoulos (1988). La plupart des algorithmes de calcul d’épaisseur ont l’inconvénient de travailler sur la grille discrète qui limite le résultat à n’obtenir que certaines valeurs discrètes. Ceci est problématique, spécialement pour un objet mince comme le cartilage. Cette méthode a l’avantage de fournir des résultats avec une précision de sous-pixel. L’algorithme a été testé sur une seule image IRM et les auteurs ont vérifié le résultat en le comparant à une segmentation effectuée par un radiologue. La comparaison donne l’exactitude, la sensibilité et la spécificité à 98,87 %, 66,22 % et 99,56 % respectivement (Pakin et al., 2002).
19
Dodin et al. (2010) suggèrent un nouvel algorithme de segmentation automatique pour quantifier le volume du cartilage d’un genou humain à partir de l’IRM. Dans l’approche proposée, les interfaces os-cartilage pour le fémur, le tibia et la patella sont obtenues, comme une étape de préparation avant l’algorithme principal, en utilisant une procédure indépendante de l’algorithme de segmentation (Lorenz & von Berg, 2005).L’algorithme commence par le redimensionnement du voisinage de la surface osseuse le long de l’interface os-cartilage, ce qui donne une image rectangulaire, c.-à-d. les pixels ayant la même distance de la surface d’os appartiennent à la même ligne dans l’image obtenue. Ensuite, une étape d’analyse de texture est effectuée sur l’image afin que le cartilage soit discriminé en tant qu’un tissu brillant et homogène. Ce processus d’exclusion des tissus mous, développé et évalué par Stachowiak, Stachowiak et Podsiadlo (2006), permet de détecter les frontières externes du cartilage. Par la suite, une technique de prise de décision basée sur Bayesian permet de séparer automatiquement le cartilage du liquide synovial.
Ils valident leur algorithme par (1) une analyse de volume du cartilage et ses changements au fil du temps sur neufs patients atteints d’arthrose du genou en comparant avec les résultats obtenus d’un système de calcul du volume semi-automatique validé (Kauffmann et al., 2003; Raynauld et al., 2003) et par (2) une procédure test-retest sur cinq patients atteints d’arthrose du genou. Les médianes du coefficient de Dice des résultats du premier test étaient de 0,85, 0,84, 0,84 pour le fémur, le tibia et la totalité du genou respectivement. Le test-retest a révélé une excellente erreur de mesure de -0,3 ± 1,6 % pour la totalité du genou et de 0,14 ± 1,7 % pour le fémur (Dodin et al., 2010).
Malgré des résultats excellents, l’intégrité de la segmentation du cartilage dépend entièrement de la précision de la détection de l’interface os-cartilage. Si les auteurs voulaient régénérer le modèle 3D du cartilage après la segmentation, ils auraient dû redimensionner l’image segmentée du système de coordonnées paramétrique de la surface de l’os au système de coordonnées original d’IRM. Cette étape occasionnerait une énorme erreur dans le modèle généré si la frontière entre l’os et le cartilage n’était pas segmentée précisément.
Kornaat et coll. (2005) dans leur étude comparent un scannographe IRM de 1,5 T avec un de 3,0 T concernant le rapport signal sur bruit (SNR), le rapport contraste sur bruit (CNR) et les mesures d’épaisseur du cartilage articulaire en utilisant une séquence SPGR en 3D et deux séquences « Steady-State Free Precession » (SSFP) en 3D. Dans cette étude, les deux genoux de cinq bénévoles (quatre hommes, une femme, âgés entre 26-38 ans) ont été examinés afin de créer un ensemble de validation en utilisant une machine de RM à 1,5 T et une autre à 3,0 T. Chaque examen a consisté en une séquence sagittale 3D-SPGR, une séquence sagittale 3D-SSFP supprimée de graisse (FS-SSFP) et une séquence sagittale Dixon SSFP. Les séquences 3D-SPGR permettent une excellente représentation de la morphologie du cartilage. Pour une analyse quantitative,
20
l’efficacité du SNR et du CNR du cartilage a été comparée ainsi que les mesures moyennes de l’épaisseur du cartilage.
Les auteurs concluent qu’en utilisant les séquences FS-3D-SPGR, malgré des temps de balayage raisonnables (environ 5 à 6 minutes), une couverture tridimensionnelle avec SNR élevé est réalisable. Ces séquences RM sont également avantageuses pour la mesure du volume, cependant, leur principal inconvénient se situe au niveau du faible contraste entre le cartilage et le liquide synovial.
Le contraste produit par la séquence SSFP supprimée en graisse peut être utile pour l’imagerie du cartilage. Il produit un signal hyperintense pour le liquide synovial, ce qui l’amène à apparaître visuellement de façon brillante dans les images tout en préservant la luminosité du cartilage. L’efficacité du SNR et la vitesse des techniques SSFP ce qui fait de cettes techniques très attrayantes pour l’imagerie morphologique ordinaire du cartilage (Kornaat et al., 2005). La principale lacune de ces techniques SSFP se situe au niveau de la sensibilité aux artefacts hors de la résonance (Gold, McCauley, Gray, & Disler, 2003).
Selon Kornaat et coll. (2005), en géneral, l’imagerie du cartilage articulaire du genou avec un scanographe RM à 3,0 T montre des efficacités améliorées du SNR et du CNR comparativement à un scanographe à 1,5 T. Par ailleurs, avec un scanographe 1,5 T, les techniques basées sur SSFP présentent la plus forte augmentation de l’efficacité du SNR et CNR. En utilisant les séquences indiquées ci-dessus, il existe une différence négligeable entre les mesures de l’épaisseur du cartilage effectuées sur les images captées par les deux différents scanographes.
Pour une séquence 3D-SPGR, l’efficacité du SNR du cartilage à 3,0 T augmente par rapport à celle à 1,5 T par un facteur de 1,83 (plage : de 1,40 à 2,09). Par rapport à 3D-SPGR, l’efficacité du SNR de FS-SSFP augmente par un facteur de 2,13 (plage : de 1,81 à 2,39) et pour Dixon SSFP, par un facteur de 2,39 (plage : de 1,95 à 2,99). Pour 3D-SPGR à 3,0 T, le CNR entre le cartilage et les tissus alentours augmente par rapport à celui à 1,5 T par un facteur de 2,12 (plage : de 1,75 à 2,47), pour FS-SSFP par un facteur de 2,11 (plage : de 1,58 à 2,80) et pour Dixon SSFP, par un facteur de 2,39 (plage : de 2,09 à 2,83). À l’encontre de ce qui vient d’être dit, l’épaisseur moyenne des régions porteuses du cartilage est identique pour les deux intensités de champ et pour toutes les séquences (P> 0,05). La moyenne de l’épaisseur mesurée dans tous les genoux est de 2,28 mm.
Les résultats montrent également que l’angle de flip optimal pour avoir le meilleur rapport signal sur bruit du cartilage en utilisant les séquences FS-SSFP et Dixon SSFP est de 30° pour les scanographes IRM à 1,5 T et à 3,0 T. Par ailleurs, pour les séquences 3D-SPGR, l’angle de flip optimal est de 12° pour les machines à 1,5 T et 10° à 3,0 T (Kornaat et al., 2005).
21
Grau et coll. dans ses études de (2004) développent une méthode de ligne de partage des eaux améliorée, ajustée pour être utilisée dans les algorithmes semi-automatiques de segmentation MR. Ils mettent en place des informations préalables dans le calcul de la transformation conventionnelle de ligne de partage des eaux afin qu’il soit plus approprié aux fins de segmentation des images médicales.
La ligne de partage des eaux transforme à des propriétés intéressantes qui la rendent utile pour de nombreuses applications de segmentation d’images différentes (Beucher & Meyer, 1992) : la méthode est simple et intuitive, cela peut être parallélisé et produit toujours une division complète de l’image (Roerdink & Meijster, 2000). Cependant, lorsque la méthode de la ligne de partage des eaux est appliquée à l’analyse des images médicales, elle présente des inconvénients importants tels que la sursegmentation, la sensibilité au bruit et la mauvaise détection de structures minces (telles que les cartilages).
Dans leurs études, Grau et coll. testent leur approche sur deux types d'applications : la délinéation du cartilage du genou et la segmentation complète de la matière blanche et de la matière grise du cerveau (Grau et al., 2004). Pour la segmentation du genou, ils utilisent des images d’IRM acquises en utilisant une séquence SPGR 3D saturée de graisse, avec une résolution élevée de 0,23 × 0,23 × 1,5 mm. Pour chaque image IRM, environ 50 marques par classe (le cartilage, l'os et d'autres tissus) sont choisies manuellement.
Leur ensemble de test contient 40 images prises sur quatre sujets ; ils font cinq examens RM sur un des patients, puis segmentent chaque image cinq fois, à la main. En utilisant ces segmentations comme réalité de terrain, ils atteignent un coefficient de Dice (DSC7) de 0,8745 pour leur pire cas et de 0,9119 pour leur meilleur.
Bien que, par rapport à d’autres algorithmes semi-automatiques, leurs résultats ne présentent pas d’amélioration significative, leur méthode offre quelques avantages. Les lignes de partage des eaux correspondent toujours aux contours les plus importants entre les marqueurs, même s’il n’y a pas de frontière forte entre les marqueurs, la transforme de ligne de partage des eaux8 détecte toujours un contour dans la zone. Ce contour est situé sur
les pixels avec un contraste plus élevé, par conséquent, et contrairement aux méthodes de minimisation de l’énergie qui pourraient produire des minimums locaux et donc des résultats erronés à cause du bruit, cette technique n’est pas affectée par des bords à contraste plus faible.
Une approche intégrée et automatique de la segmentation du genou est proposée par J. Fripp et coll. (2010) et implique un algorithme de segmentation pour tous les os utilisant un modèle de forme actif tridimensionnel, une extraction de l’interface os-cartilage (BCI) et une segmentation du cartilage du BCI utilisant un modèle déformable. Les auteurs suggèrent une hiérarchie de segmentation dans laquelle les objets plus difficiles à
7 En anglais : Dice similarity coeficient 8 En anglais : Watershed
22
segmenter sont segmentés selon les informations, les pointes d’initialisation et les contraintes fournies par les objets plus faciles à segmenter, qui eux sont segmentés en premier. Dans l’articulation du genou, il existe une forte relation spatiale entre les os et les cartilages. Cet algorithme bénéficie de cette relation pour exécuter la segmentation de cartilage du BCI (l’interface os-cartilage). Jurgen Fripp et coll. ont également développé un algorithme pour extraire le BCI des images RM (Jurgen Fripp, Crozier, Warfield, & Ourselin, 2007).
L’exactitude de ce schéma est validée expérimentalement en utilisant une validation croisée « leave-one-out » sur un ensemble d’images RM de « Spoiled Gradient-Recalled » (SPGR) avec suppression de graisse. Les auteurs utilisent un ensemble contenant 20 images RM anonymes, segmentées manuellement et captées de volontaires sains qui n’étaient pas connus pour avoir de l’OA ou des douleurs au genou.
J. Fripp et coll. comparent leurs résultats à trois approches de pointe, soit à une classification des tissus (Folkesson, Dam, Olsen, Pettersen, & Christiansen, 2007), à un algorithme semi-automatique modifié (Grau et al., 2004) et à une déformation de la forme libre à base de B-spline (Rueckert et al., 1999). Chacune de ces trois approches est formée et validée contre un ensemble hétérogène d’images RM de FS-SPGR segmentées manuellement par des radiologues (J. Fripp et al., 2010). Ils obtiennent des coefficients de Dice (DSC) moyens de 0,83, 0,83, 0,85 pour patella, tibia et fémur respectivement, tandis qu’en utilisant le classifieur de tissus, les DSC sont de 0,82, 0,81, 0,86 et de 0,73, 0,79, 0,76 avec l’enregistrement non rigide (méthode basée sur B-spline).
La moyenne de DSC obtenue pour la totalité d’un genou montre une petite amélioration par rapport à la méthode de la classification des tissus (Folkesson et al., 2007), tandis qu’elle est légèrement inférieure à la méthode de ligne de partage des eaux (Grau et al., 2004) malgré l’utilisation du même ensemble de données. La principale et peut-être la seule limitation de cette méthode est qu’elle n’a été validée que sur des genoux non pathologiques et des changements peuvent être exigés pour s’adapter aux sujets avec OA.
Saadatmand-Tarzjan (2015) propose une nouvelle méthode de contour actif adaptable pour différentes applications de segmentation d’images RM médicales appelée « self-affine snake ». Les contours actifs sont des silhouettes déformables qui se déforment continuellement dans le domaine de l’imagerie afin de minimiser les énergies internes et externes. L’énergie externe déplace les frontières du contour vers l’objet d’intérêt dans l’image et l’énergie interne lisse les courbes du contour. Cette méthode intègre la transformée en ondelettes, le contour actif paramétrique (le « snake ») et le système de cartographie autoaffine (Ida & Sambonsugi, 2000) pour détecter les limites faibles.
L’auteur évalue sa méthode par cinq ensembles d’expériences en utilisant des images synthétiques et médicales. Les images RM ont été segmentées manuellement par deux experts en radiologie comme la réalité
23
de terrain pour l’évaluation. En ce qui concerne le processus d’évaluation, après la convergence des contours, le résidu (l’erreur) de chaque composante du contour est calculé comme la distance euclidienne minimale à la limite désirée. Ensuite, la qualité des résultats est mesurée en calculant l’erreur maximale (𝜓𝑚𝑎𝑥), moyenne (𝜓𝑎𝑣𝑟) et l’écart-type de tous les composants du contour (𝜎𝜓). L’auteur compare ses résultats à ceux d’autres algorithmes basés sur le contour : méthode de ballon (Xu, Pham, & Prince, 2000), Gradient Vector Flow (GVF), GVF généralisé (Xu & Prince, 1998) et Active Contours Without Edges (ACWE) (Chan & Vese, 2001).
Leurs résultats montrent une petite amélioration par rapport aux autres méthodes basées sur le contour discuté dans l’article avec une moyenne de l’erreur de 𝜓𝑎𝑣𝑟= 1,08 , 𝜎𝜓 = 0,45, évaluée par rapport aux images RM
segmentées par le premier radiologue, et de 𝜓𝑎𝑣𝑟= 0,94 , 𝜎𝜓 = 0,30, basée sur les images segmentées par le deuxième radiologue. Cependant, la méthode proposée n’a jamais été testée sur le cartilage du genou ni, d’ailleurs, sur tout autre composant mince.
Zhang et coll. (2011) présentent une autre solution pour la segmentation précise et automatique du cartilage à l’aide d’images RM à contraste multiple, basée sur la classification des pixels. Les méthodes de la classification des pixels précédents de leurs, s’appuyaient sur la classification par des classifieurs tels que SVM9
(US20090306496 A1, 2009) ou kNN (Folkesson et al., 2007) sans considérer l’information spatiale. Zhang et coll. incorporent des dépendances spatiales dans la classification en adoptant les caractéristiques basées sur la structure locale de l’image ainsi que les informations géométriques. Ils mirent également en place une optimisation convexe sur leur méthode basée sur SVM et sur les champs aléatoires discriminatifs10. Bien que
l’emploi de la combinaison de SVM et de DRF était proposé pour la localisation du cancer de la prostate (Artan et al., 2010) ainsi que pour l’extraction des tumeurs cérébrales (Lee et al., 2005) avant, n’avait pas été spécialisé dans la segmentation du cartilage.
Zhang et coll. valident leur algorithme sur un ensemble d’images RM à contraste multiple. Ils utilisent un scénario d'entraînement sujet-spécifique où le classifieur est entraîné sur un sous-ensemble de données du sujet et ensuite testé sur un autre sous-ensemble ou sur tout l'ensemble. Cette méthode de validation est utilisée dans plusieurs études sur la segmentation d’images médicales (Chen & Metaxas, 2003; García & Moreno, 2004; Kaus et al., 2001; J. Zhang, Ma, Er, & Chong, 2004). Dans cette recherche, une tranche sur cinq est utilisée comme données de l'entraînement et toutes autres tranches en tant qu’ensemble de test. Le DSC de 0,913 ± 0,090 est réalisé à l'aide d'intensités normalisées d'images RM à contraste multiple, de structure locale et d'informations géométriques. Bien que l’utilisation d’un scénario sujet-spécifique augmente la qualité des
9 Support Vector Machines
24
résultats légèrement, une comparaison entre leurs résultats et d’autres résultats utilisant des scénarios sujets-spécifiques montre que l'utilisation des images RM à contraste multiple améliore considérablement le résultat.