Université de Sherbrooke
La régulation de l’expression génique peut passer par un mécanisme de terminaison prémature de la transcription dépendant de la RNase III chez
Saccharomyces cerevisiae
Par
Francis Malenfant
Département de microbiologie et infectiologie
Mémoire présenté à la Faculté de médecine et des sciences de la santé en vue de l’obtention du grade de maitre en sciences (M.Sc) en microbiologie et
infectiologie
Mai, 2017
Membres du jury d’évaluation
Sherif Abou Elela, département de microbiologie et infectiologie Benoit Chabot, département de microbiologie et infectiologie
La régulation de l’expression génique peut passer par un mécanisme de terminaison prémature de la transcription dépendant de la RNase III chez Saccharomyces cerevisiae
Par
Francis Malenfant
Département de microbiologie et infectiologie
Mémoire présenté à la Faculté de médecine et des sciences de la santé en vue de l’obtention du grade de maitre en sciences (M.Sc) en microbiologie et infectiologie,
Faculté de médecine et des sciences de la santé, Université de Sherbrooke,
Résumé
L’expression des gènes est un ensemble hautement régulé de mécanismes ayant pour objectifs de synthétiser les protéines fonctionnelles dont la cellule a besoin à partir des codes inscrits dans l’ADN. Pour contrôler la quantité de signal utilisé et pour que ce message puisse physiquement traverser la cellule, celle-ci utilise la transcription des ARN messagers comme intermédiaire. Pour assurer la qualité de ce signal et pour contrôler son niveau d’expression, plusieurs mécanismes de dégradation des ARNm se coordonnent dans les organismes en fonction de leurs spécificités propres. La littérature a depuis longtemps démontré les liens entre les machineries de synthèse des ARNm et celles de leur dégradation en identifiant comment ceux-ci travaillent ensemble pour assurer une bonne régulation génique. Un de ces mécanismes induit une terminaison dans la région non-codante en 3’ de certains gènes à partir d’un clivage par la ribonucléase III. Dans ce mémoire, nous voulons démontrer qu’un mécanisme similaire dépendant de la ribonucléase III peut induire une terminaison prémature de la transcription à l’intérieur même de séquences codantes. Ce mécanisme semble être indépendant du promoteur et du terminateur des gènes, préférant réguler sa sélectivité à partir de la structure liée au clivage de l’ARNm. Plusieurs séquences et structures sous forme de tige-boucles d’ARNm peuvent être reconnues par la ribonucléase III. Cependant, il existe des différences fonctionnelles entre les différentes tige-boucles et toutes n’induisent pas un mécanisme de terminaison prématurée. Comme ce type de mécanisme doit être inductible et/ou permissible afin de ne pas empêcher complètement l’expression des gènes cibles, nous pouvons potentiellement faire affaire à un nouveau modèle de régulation génique.
Mots clés : Terminaison de la transcription, terminaison prémature, RNase III, Saccharomyces cerevisiae, Poly-A indépendant, modèle torpille.
Gene expression can be regulated with a premature termination mechanism targeting ongoing transcription dependent on the RNase III in Saccharomyces cerevisiae
By
Francis Malenfant
Microbiology and Infectiology Department
Master thesis presented to the Faculté de médecine et des sciences de la santé for the obtention of the title of Master of Sciences (M.Sc) in microbiology and infectiology,
Faculté de médecine et des sciences de la santé, Université de Sherbrooke,
Summary
Gene expression is a highly regulated coordination of processes with the objective of producing functional proteins from the DNA code of the cell. To control the amount of proteins produced, cells use messenger RNAs as an intermediate to permit genomic information to move across the cell. To assure the quality of this signal and to control the level of gene expression, many RNA degradation mechanisms coordinate together according to their own specificities. Scientific literature has demonstrated long ago the existing interactions between RNA synthesis and RNA degradation pathways and how they closely work together to achieve viable gene expression regulation. One of those mechanisms induces transcription termination in the 3’ untranscribed region of coding genes initiated with a RNA cleavage from a ribonuclease III. In this master thesis, we show that a similar RNase III dependent mechanism can induce premature transcription termination inside the coding sequence. This mechanism seems promotor and terminator independent and depends mostly on the sequence coding for a stem-loop structure in the mRNA. Different sequences can induce a stem-loop structure recognizable by RNase III. However, there are some functional differences between stem-loop structures and not all of them can induce premature transcription termination. Since this mechanism must not happen every time and somehow must be inducible to permit gene expression when needed, this could possibly lead to a new gene regulation model.
Key Words : Transcription termination, premature termination, RNase III, Saccharomyces
Table des matières
Résumé III
Summary IV
Table des matières V
Liste des tableaux VII
Liste des figures VIII
Liste des abréviations IX
Introduction 1
1. Introduction générale et présentation du laboratoire 1 2. L’expression des gènes est un ensemble de mécanismes hautement régulés 2 2.1. Mécanismes généraux de l’expression génique 2 2.2. Bref historique de la recherche sur l’expression génique 5 2.3. La structure de la chromatine et ses rôles dans la régulation génique 6 3. La régulation génique se produit par la gestion de la stabilité des ARN et par leurs
mécanismes de dégradation 7
3.1. L’importance de la dégradation des ARN et les rôles des mécanismes de
surveillance dans le contrôle de la qualité 7
3.2. Le rôle de la coiffe et la protection de l’extrémité 5’ des ARNm 9 3.3. Le rôle de la queue poly-A et la protection de l’extrémité 3’ de l’ARNm 10 3.4. Le rôle des ribonucléases est essentiel dans le bon déroulement de la
dégradation spécifique des ARN 11
3.5. Rnt1p, la ribonucléase de la famille III chez la levure 14 4. La transcription est un ensemble coordonné de mécanismes essentiels dans la
régulation de l’expression génique 17
4.1 L’initiation de la transcription 18
4.2 L’élongation de la transcription 20
4.3 La terminaison de la transcription 22
4.3.1 Mécanisme de terminaison poly-A dépendante : Rat1p dépendante 22 4.3.2 Mécanisme de terminaison poly-A indépendante : Rat1p indépendant
(Courts ARN non-codants) 25
4.3.3 Mécanisme de terminaison poly-A indépendante : Rnt1p dépendant
(Long ARN non-codants) 28
Présentation de l’article 31 Avant-propos 31 Résumé de l’article: 32 Introduction 33 Results 36 Discussion 52 Experimental Procedures 57 References: 70 Discussion 75
6. La structure de reconnaissance de Rnt1p sur l’ARNm est l’élément le plus
déterminant de la présence d’un mécanisme la terminaison prémature. 75 7. Le promoteur et le terminateur ne semblent pas avoir un rôle aussi critique que la
tige-boucle reconnue par Rnt1p. 77
8. Le recrutement de Rnt1p à la chromatine est dépendant de l’ARNm 80 9. Les caractéristiques des substrats menant à une terminaison prémature sont
toujours inconnues. 81
10. Comment un mécanisme de terminaison prémature de la transcription serait
bénéfique pour la cellule. 82
11. Les faiblesses soulevées lors de la soumission de l’article 84 12. Où peut mener ce projet de recherche et quelles sont les prochaines étapes ? 86
Conclusion 87
Remerciements 88
Liste des références 89
Liste des tableaux
Article
Table S1 : Summary of the features and transcription patterns of genes carrying internal Rnt1p cleavage signals (IRCS)……….. p.64 Table S2 : Summary of the strains used in this study……… p.66 Table S3 : Summary of the primers used for QPCR in this study……… p.67 Table S4 : Summary of the primers used for transformation in this study………... p.69
Liste des figures
Introduction
Figure 1 : Schématisation simplifiée des grandes étapes de l’expression génique… p.4 Figure 2 : Les différentes voies de dégradation des ARNm eucaryotes ………..…. p.13 Figure 3 : Groupements fonctionnels de la famille des RNase III……… p.15 Figure 4 : Terminaison Poly-A dépendante et Rat1p dépendante……… p.23 Figure 5 : Le recrutement cotranscriptionnel de Rat1p induit le
décrochage de l’ARN polymérase après le clivage...………. P.24 Figure 6 : Le recrutement des facteurs de terminaison de la voie
Sen1-Nrd1 se fait aussi de manière cotranscriptionnelle………... P.26 Figure 7 : La dégradation par l’exosome dans la voie Nab3-Sen1……… p.27 Figure 8 : Mécanisme de terminaison Rnt1p-Rat1p dépendant………. p.29 Article
Figure 1 : Cleavage within mRNA coding sequence triggers
premature transcription termination……….. p.37 Figure 2 : Rnt1p associates with chromatin of certain genes to induce
premature transcription termination and reduces RNA abundance…….. p.39 Figure 3 : RNA cleavage by Rnt1p is required for premature transcription
termination………. P.42 Figure 4 : Rnt1p uses components of the “torpedo” and not the Nrd1-Nab3-Sen1
mechanism of transcription termination……… p.45 Figure 5 : Promoter and terminator sequences are not responsible for
Rnt1p-dependent premature transcription termination……….. p.48 Figure 6 : Rnt1p induces transcription termination in a substrate-dependent
manner………... P.50 Figure 7 : Model describing the mechanism of Rnt1p dependent premature
transcription termination……… p.55 Figure S1 : Replacement of the DGR2 stem-loop inhibit PTT……….. p.59 Figure S2 : DGR2 iRCS is sufficient to induce Rnt1p dependent PTT………. p.60 Figure S3 : Cleavage within the coding sequence of MIG2 does not induce
Rat1p dependent premature transcription termination……… p.61 Figure S4 : The sequence surrounding the DGR2 stem-loop isn’t required for
the induction of premature transcription termination……….. P.62 Figure S5 : Predicted secondary structures of Rnt1p cleavage sites used in
this study………. p.63 Annexe
Figure 9 : Distribution de l’ARN polymérase II sur le gène MIG2 avec ou sans
Liste des abréviations
ADN : Acide désoxyribonucléique ARN : Acide ribonucléique
ARNnc : ARN non-codant ARNm : ARN messager ARNr : ARN ribosomal
CstF : Facteurs de stimulation du clivage
CPSF : Facteurs spécifiques au clivage et à la polyadénylation RNase : Ribonucléase
Introduction
1. Introduction générale et présentation du laboratoire
Ce mémoire présente l‘aboutissement d’un projet de recherche effectué dans le but de mieux comprendre le rôle et les impacts de la Ribonucléase III Rnt1p sur les mécanismes de la transcription et de la dégradation des ARNm chez les cellules eucaryotes. Vous retrouverez la proposition d’un mécanisme de terminaison prémature de la transcription présente sur un sous-ensemble de gènes. Ce projet fut fait sous la supervision du professeur Sherif Abou Elela au sein de l’Université de Sherbrooke dans le département de microbiologie et infectiologie.
Le laboratoire du professeur Abou Elela possède une approche multidisciplinaire en génétique, biologie moléculaire et en biochimie afin de mieux comprendre les mécanismes sous-jacents à la biologie du cancer. Ainsi, le laboratoire contient quatre axes majeurs, soit la compréhension des mécanismes de maturation et de dégradation des ARN, l’étude fonctionnelle de l’épissage dans des cellules humaines cancéreuses, la recherche sur les fonctions des introns à l’intérieur du génome ainsi que l’étude des propriétés différentielles des séquences ohnologues dans la fonction des ribosomes.
Ce projet fait ainsi partie d’une série de publications fait par le laboratoire pour mieux comprendre les mécanismes de maturation et de dégradation des ARN, plus spécifiquement auprès de son expertise dans l’étude des impacts biologiques des ribonucléases de la famille III. Dans le cadre du travail effectué pour ce mémoire, l’organisme modèle utilisé fut la levure Saccharomyces cerevisiae. La présentation des mécanismes dans l’introduction de ce mémoire restera le plus général possible pour discuter des concepts biologiques mais lorsque des précisions devront avoir lieu, celles-ci se feront chez les eucaryotes.
2. L’expression des gènes est un ensemble de mécanismes hautement régulés
2.1. Mécanismes généraux de l’expression génique
Les cellules de tous les organismes vivants possèdent un code génétique. Ce code, dans la presque totalité des organismes, certains virus faisant exception, est fait à partir de séquences spécifiques d’acides désoxyribonucléiques (ADN). L’ADN se présente sous la forme de très longues séquences continues nommées chromosomes. Certaines sections de l’ADN contiennent les codes nécessaires à la production de protéines fonctionnelles particulières, ces régions sont des gènes codants. Les séquences présentes entre les différents gènes codants sont eux appelés séquences non-codantes et ont elles aussi certaines fonctions dans la régulation de l’expression des gènes (Alberts B et al, 2002). La principale distinction entre ces deux types de région génique est donc la finalité souhaité, soit la synthèse d’une protéine fonctionnelle ou non. Dans le cadre de ce mémoire, le mécanisme étudié a été fait chez des séquences codantes pour des protéines.
Pour offrir un niveau de contrôle accru, la cellule synthétisera, à partir de nucléotides, une copie des séquences codantes de l’ADN à l’aide d’une enzyme, l’ARN polymérase II (Lodish H et al, 2012). Cette copie des séquences codantes se présente sous la forme d’un acide ribonucléique messager (ARNm). Un ARNm fraichement transcrit subira de nombreuses étapes de maturation pour, dans une seconde étape, se rendre dans le cytoplasme afin d’y rencontrer les ribosomes et initier la synthèse de protéines. Évidemment, ce portrait simpliste de l’expression génique chez les eucaryotes se complexifie rapidement lorsque l’on décortique chacun des événements impliqués. En effet, si l’ARNm est considéré sans défaut, que sa maturation est complète, que sa structure coiffe et sa queue poly-A sont bien installés, que tous ces sous-ensembles sont reconnus par les bonnes protéines d’export, tout en n’étant pas reconnus par les ribonucléases, alors l’ARNm pourra être exporté hors du noyau pour être traduits (Alberts B et al, 2002). Dans le cadre de ce mémoire, nous discuterons plus en détail de ces divers mécanismes pour mieux comprendre la machinerie de transcription dans la cellule et ses possibles relations avec la machinerie de dégradation des ARNm.
Chacune des étapes impliquées dans l’expression des gènes est hautement régulée par plusieurs mécanismes et plusieurs d’entre eux restent encore partiellement inconnus ou encore mal compris. Leur étude reste cependant très importante, car il s’agit de mécanismes essentiels à la vie et des défauts dans ceux-ci peuvent induire un nombre incalculable de pathologies (Cooper TA et al, 2009). Le regard de ce mémoire sur la régulation génique sera simplifié pour se limiter aux mécanismes impliqués dans la synthèse de l’ARNm, soit la transcription et la maturation standard des ARNm. Nous discuterons très peu des mécanismes de maturation tels que l’épissage, du transport cellulaire et de sa coordination, de la traduction ou même de la régulation apportée par certains ARN non-codants. Il faut cependant comprendre qu’il s’agit d’un sous ensemble particulièrement complexe d’un nombre élevé d’acteurs pouvant prendre plusieurs formes différentes. Au-delà des ARNm, certaines séquences de l’ADN peuvent avoir différentes fonctions et former d’autres produits comme les ARN non-codants (ARNnc). Comme mentionné précédemment, ces ARN n’ont pas comme fonction de mener à la synthèse d’une protéine fonctionnelle mais elles peuvent avoir d’importantes fonctions de régulation. Cette catégorie contient entre autre les petits ARN, les ARN ribosomaux, les ARN de transfert et même chez l’humain des ARN interférents (Nishikawa, 2017).
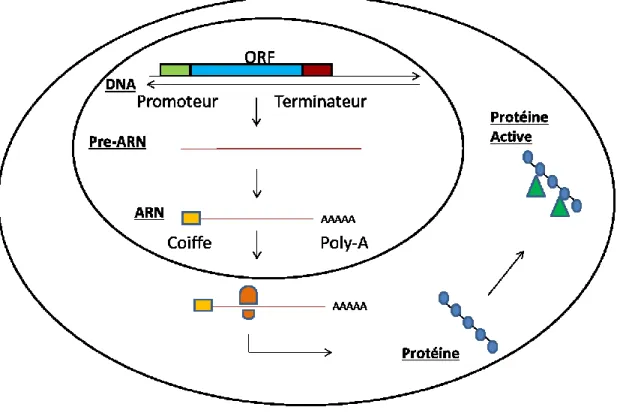
Figure 1: Schématisation simplifiée des grandes étapes de l’expression génique
De l’ADN, la cellule synthétisera des pré-ARNm qui subiront plusieurs étapes de maturation. Ces ARN seront modifiés cotranscriptionnellement pour ajouter une coiffe et une queue poly-A. L’extrémité 5’ de l’ARNm sera protégée par une coiffe installée dès le début de la transcription et l’extrémité 3’ sera protégée par une queue poly-A installée lors de la terminaison. Une fois mature, l’ARNm sera reconnu par un ensemble de protéines et pourra sortir du noyau pour se rendre au cytoplasme. Cet ARNm pourra y être reconnu par le ribosome et permettra l’assemblage d’acides aminés en protéines fonctionnelles.
2.2. Bref historique de la recherche sur l’expression génique
La littérature semble attribuer la découverte de la régulation génique à une étude sur l’activité de l’ARN polymérase faite dans les noyaux de foie chez le rat seulement 6 ans après que Watson et Crick aient déterminé la structure de l’ADN (Weiss S et Gladstone L, 1959). Provenant d’un organisme où il était difficile d’extraire la polymérase, les scientifiques de l’époque ont réussi à utiliser des bactéries comme organismes modèles. Cet avancement aura mené au prix Nobel de médecine en 1965, bien qu’on ne retrouve pas encore les termes transcription ou polymérase dans les publications liées au prix (Nobel Foundation, 1973). Ce changement de méthodologie mena à plusieurs autres découvertes peu de temps après. Ce n’est que cinq ans plus tard que les chercheurs de l’époque découvrirent que les cellules eucaryotes possédaient trois polymérases différentes contrairement aux cellules procaryotes (Roeder RG et Rutter WJ, 1969). La recherche se développa à grande vitesse et en 1995, trois grandes questions finirent par être posées chez les chercheurs du domaine. Ceux-ci voulaient comprendre comment les éléments activateurs et répresseurs pouvaient affecter la machinerie de transcription, comment les activités de ces éléments étaient coordonnées et régulées et enfin, ils voulaient comprendre comment les voies régulatrices pouvaient coordonner la croissance des cellules en fonction de signaux externes (Struhl K, 1995). Aujourd’hui, de nombreux facteurs impliqués dans la régulation des gènes furent étudiés mais beaucoup de découvertes restent encore à faire. Ce mémoire sera un aperçu des connaissances actuelles sur les mécanismes généraux de la régulation des gènes se concluant par la proposition d’un nouveau mécanisme cotranscriptionnel initié par un membre de la famille des ribonucléases III.
2.3. La structure de la chromatine et ses rôles dans la régulation génique
À proprement parler, il existe plusieurs étapes de régulation avant même que la transcription commence à initier la synthèse d’un brin d’ARNm. L’expression des gènes implique des complexes d’histones et de protéines chaperonnes ayant comme fonction de structurer l’ADN pour la protéger (Harteis S et Schneider S, 2014). Ces interactions sont très importantes car elles dictent s’il sera possible aux facteurs de transcription et aux enzymes de reconnaitre et lier la région d’ADN codante pour qu’elle soit transcrite. Ainsi, cette structure en réalité tridimentionnelle et très complexe doit être flexible afin de permettre l’accès des facteurs impliqués dans la transcription (Harteis S et Schneider S, 2014).
L’ADN eucaryote est maintenu dans une structure dynamique qui la protège tout en permettant de minimiser son espace dans la cellule. À la base structurale, on retrouve le nucléosome, formé d’histones associées à 147 paires de bases d’ADN liées en double-hélice, considéré comme l’unité de base de la chromatine (Horn JP et al, 2002). La chromatine permet de réguler l’accessibilité des facteurs liés à la transcription mais aussi à ceux liés à la recombinaison, à la réparation et à la réplication de l’ADN (Venkatesh et al, 2015). Ainsi, pour que la bonne région soit disponible à recruter les facteurs de transcription et l’ARN polymérase, il faut ouvrir la structure tertiaire de l’ADN. Cela se fait principalement par des mécanismes d’acétylation des histones (Kurdistani SK et Grunstein M, 2003). Une fois la région génique disponible, des facteurs de transcription activateurs et/ou inhibiteurs pourront aller reconnaitre des séquences spécifiques dans la région en 5’ de la séquence codante et entamer l’initiation de la transcription.
3. La régulation génique se produit par la gestion de la stabilité des ARN et par leurs mécanismes de dégradation
3.1. L’importance de la dégradation des ARN et les rôles des mécanismes de surveillance dans le contrôle de la qualité
Dans le cadre de ce mémoire, l’emphase sera mise sur la dégradation des ARNm et ses rôles dans la régulation des mécanismes de transcription. Il est cependant important de mentionner que tous les ARN, que ce soit les ARN de transfert, ARN ribosomaux et même les ARN non-codants subissent des mécanismes de dégradation hautement régulés. Certains de ceux-ci sont généraux alors que d’autres sont beaucoup plus minutieusement contrôlés. L’ensemble de ces mécanismes sont étudiés depuis plus de 20 ans mais il reste encore beaucoup de questions en suspens (Parker R, 2012).
Comme mentionné brièvement, afin de produire des protéines, la cellule utilise l’ARNm comme intermédiaire. Si l’on peut dire que l’expression génique est la capacité de la cellule à activer toute la mécanique qui aura comme finalité la création de protéines, nous pourrions dire que la régulation de l’expression génique est la gestion entre l’activation et la désactivation de ce signal. En effet, une protéine utile dans une situation particulière peut devenir nuisible si la situation vient à changer. La cellule doit, en réalité, être capable d’ajuster les niveaux d’expression de ses gènes en fonction de son environnement et de ses stimuli. Pour exprimer cette flexibilité, la cellule peut utiliser plusieurs moyens. Ainsi, la transcription des gènes peut être arrêtée avec l’aide de répresseurs, ou par le retrait des activateurs dans les séquences précédents le gène. À la fin de la chaine d’expression, les protéines peuvent être dégradées pour arrêter toute activité. Mais comme l’expression des gènes est au final un ensemble de mécanismes travaillant d’un commun accord, la diminution de l’expression des gènes utilise aussi de nombreux mécanismes de dégradation des ARNm. Chez la levure Saccharomyces cerevisiae, la dégradation générale des ARNm se produit à l’aide de deux mécanismes commençant tous les deux par la déadénylation de la queue poly-A en 3’ de l’ARNm (Doma MK et Parker R, 2007). Après cette première étape initiatrice de la dégradation, le premier mécanisme impliquera la dégradation de l’ARNm dans l’orientation 3’-5’ alors que le deuxième consistera à l’enlèvement de la
structure coiffe de l’ARNm et de sa dégradation dans l’orientation 5’-3’ (Parker R, 2012). À l’aide de ceux-ci, une fois que l’ARNm a rempli son rôle et permis la traduction d’une ou plusieurs protéines, celui-ci sera dégradé pour ne pas conserver la machinerie de traduction dans un perpétuel état activé. C’est ce qui est le plus communément appelé turnover de l’ARNm.
La dégradation des ARN se produit de nombreuses manières et répond, en réalité, à plusieurs fonctions spécifiques autres que la dégradation généralisée. Au-delà de supprimer le signal d’expression pour répondre au besoin d’arrêt de production de protéines, la dégradation des ARNm a un rôle très important dans le contrôle de la qualité de son signal. Lorsqu’un ARNm possède des défauts de synthèse ou de maturation qui le rend non-fonctionnel, la cellule est capable de reconnaitre ces défauts et de dégrader l’ARNm avant que celui puisse être traduit en protéines aberrantes et causer des dommages, ce sont les mécanismes du Nonsense Mediated Decay, du No-Go Decay et du Non-Stop Decay (Atkinson GC et al, 2008). Dans le noyau de la levure, les ribonucléases Rat1p, Rrp6p et l’exosome sont généralement impliqués dans la dégradation des ARNm alors que dans le cytoplasme, la cellule utilise les ribonucléases Xrn1p et l’exosome (Doma MK et Parker R, 2007). Nous entrerons plus en détail dans leur fonctionnement à la section 3.4. De plus, nous regarderons les rôles et fonctionnalités de la ribonucléase Rnt1p qui est un acteur essentiel du mécanisme proposé dans ce mémoire. Mais avant de voir plus en détail les mécanismes de dégradation des ARNm et leur impacts sur la machinerie de transcription, il faut comprendre comment la cellule protège et surveille ses ARNm.
3.2. Le rôle de la coiffe et la protection de l’extrémité 5’ des ARNm
L’installation d’une coiffe à l’extrémité 5’ de l’ARNm est l’une des phases de maturation les plus importantes et répandues à travers le génome. Cette coiffe est essentielle pour traduire efficacement les gènes et pour assurer la viabilité de la cellule. En effet, il a été démontré que la coiffe, plus précisément la méthylation de sa guanosine, était essentielle à la traduction de la plupart des ARNm dans les organismes eucaryotes (Cowling VH, 2010). En réalité, la coiffe a des rôles très importants dans la transcription, l’épissage, dans l’export des ARNm au cytoplasme et même dans la polyadénylation de l’extrémité 3’ de l’ARN (Cowling VH, 2010).
La coiffe est une structure particulière qui contient une 7-méthylguanosine liée d’une manière inversée par une liaison 5’-5’ triphosphate à l’extrémité 5’ de l’ARNm (Alberts B et al, 2002). De plus, plusieurs ARNm chez les eucaryotes possèdent une modification additionnelle, soit une méthylation du groupe 2’-OH sur le second ribose de l’ARNm (Alberts B et al, 2002). La fonction première de la coiffe est de protéger l’extrémité 5’-phosphate de l’ARNm, mais celle-ci est impliquée dans d’autres mécanismes très importants comme l’export nucléaire et dans l’initiation de la traduction (Lewis JD et Izaurralde E, 1997). Le mécanisme d’installation de la coiffe est mis en place très rapidement au début de la transcription pour assurer la stabilité de l’ARNm produit. En effet, après que la transcription de l’ARNm ait atteint ses premiers 25 à 30 nucléotides, la machinerie responsable de l’installation de la coiffe a déjà commencé son travail sur l’extrémité de l’ARNm (Lodish H et al, 2012). Sans trop entrer dans les détails, la synthèse de la coiffe est une coordination entre trois enzymes, soit la triphosphatase Cet1p, la méthyltransférase Abd1p et la guanylyltransférase Ceg1p (Shuman S, 2002). Le groupement guanylate de la coiffe protègera l’ARNm pour qu’il ne soit pas dégradé par une exoribonucléase 5’-3’ alors que le groupe méthyle participera à la reconnaissance de l’ARNm par le mécanisme de traduction (Shuman S, 2002). Chez la levure, les ribonucléases ayant les fonctions les plus importantes dans la dégradation de l’extrémité 5’ des ARNm sont Rat1p dans le noyau et Xrn1p dans le cytoplasme.
3.3. Le rôle de la queue poly-A et la protection de l’extrémité 3’ de l’ARNm
Nous discuterons dans la section 4.3 des différents mécanismes de terminaison de la transcription chez Saccharomyces cerevisiae. Mais pour bien comprendre leur raison d’être, il est important de comprendre à quoi ressemblera l’ARNm à la toute fin de la transcription. Chez les ARNm codants, la dernière étape de la terminaison traditionnelle se complète par l’addition d’une queue poly-A qui va, un peu comme la coiffe à l’autre extrémité, protéger l’extrémité 3’-OH de l’ARNm (Alberts B et al, 2002). De plus, comme la coiffe, la queue poly-A aura des fonctions supplémentaires importantes dans la régulation post-transcriptionnelle de son ARNm. La queue poly-A consiste en une accumulation de 100 à 250 nucléotides d’adénine à l’extrémité 3’ de l’ARNm, ajoutés un après l’autre à l’aide de l’enzyme poly-A polymérase (Lodish H et al, 2012). Contrairement à l’ARN polymérase, la poly-A polymérase ne nécessite aucun modèle sur lequel copier sa séquence, voilà pourquoi aucune séquence équivalente d’adénine n’est encodée à la fin de des gènes (Balbo PB et Bohm A, 2007). La synthèse de la queue poly-A est dépendante de la machinerie de terminaison de la transcription poly-A dépendante, celle-ci sera décrite en détail dans la section 4.3.1 de ce mémoire.
En plus de protéger l’extrémité 3’ des ribonucléases, la queue poly-A a plusieurs autres fonctions assurant la bonne expression des gènes. Comme mentionné précédemment, la coiffe devra être reconnue pour initier la traduction aux ribosomes. Cependant, pour éviter de traduire un ARNm endommagé qui aurait pu être brisé ou coupé par une ribonucléase, la queue poly-A devra elle aussi être reconnue par la machinerie de traduction avant de pouvoir initier la synthèse de protéines (Alberts B et al, 2002). Si le ribosome peut reconnaitre les deux extrémités de l’ARNm et leur bonne maturation, celui-ci met les chances de son côté à ce que le messager soit en bon état. De plus, la queue poly-A possède un rôle très important dans l’export nucléaire en agissant comme une plateforme servant à recruter les facteurs nécessaires à la liaison de l’ARNm avec le récepteur d’export TAP (Fuke H et Ohno M, 2008). Chez la levure, les ribonucléases les plus importantes pour dégrader l’extrémité 3’ des ARNm sont Rrp6p dans le noyau et l’exosome, présent simultanément dans le noyau et le cytoplasme.
3.4. Le rôle des ribonucléases est essentiel dans le bon déroulement de la dégradation spécifique des ARN
Pour dégrader l’ARN, la cellule emploie un grand nombre de ribonucléases, chacune avec ses spécificités et fonctions. Celles-ci sont donc aptes à reconnaitre leurs propres substrats chez l’ARN. Il existe trois familles générales d’activité chez les RNases, les exoribonucléases qui hydrolysent les ARN à partir de leur extrémité 5’, les endoribonucléases qui sont capables de dégrader l’ARN à partir de l’intérieur de leur séquence et enfin, les exoribonucléases qui dégradent les ARN à partir de leur extrémité terminale 3’ (Houseley J et Tollervey D, 2009).
Au travers de toutes ses options, la levure utilise dans ces mécanismes généraux de dégradation deux exoribonucléases 5’-3’, la RNase nucléaire Rat1p (équivalente à la ribonucléase XRN2) et la RNase cytoplasmique Xrn1p. Ces deux enzymes reconnaissent spécifiquement une extrémité 5’ non protégée chez l’ARNm, plus précisément une extrémité 5’monophosphate (Nagarajan VK et al, 2013). Faisant partie de la même famille de ribonucléases, ces enzymes sont très conservées entre les espèces eucaryotes (Nagarajan VK et al, 2013). Comme tous les l’ARNm sont susceptibles d’avoir une extrémité 5’-phosphate libre, ces RNases peuvent avoir des interactions considérées comme très générales et peuvent affecter tous les ARNm. Cependant, Rat1p possède un rôle très spécifique dans la terminaison poly-A dépendante de la transcription, comme il sera expliqué en détail à la section 4.3.1. Rat1p est une ribonucléase essentielle à la survie de la cellule, alors que Xrn1p est en réalité considéré comme non essentielle. Cependant, les cellules ayant un défaut dans leur gène XRN1 possèdent un phénotype très malade, deviennent très sensibles à plusieurs facteurs de stress et ont une vie beaucoup plus courte que les cellules sauvages (Giaever G et al, 2002).
De l’autre extrémité de l’ARNm, l’exoribonucléase Rrp6p est capable de dégrader l’extrémité 3’-OH dans le noyau. Cette enzyme possède des fonctions dans la maturation et dans les mécanismes de surveillance de l’ARNm (Alberts B et al, 2002). De plus, Rrp6p possède dans son domaine C-terminal deux domaines de liaison, un à l’exosome et un autre à l’ARN (Wasmuth EV et Lima CD, 2017). En réalité, les deux groupes de dégradation des
ARNm en 3’-5’ sont de très proches partenaires. De son côté, le complexe de l’exosome est un rassemblement de 11 exoribonucléases combinées avec d’autres facteurs, comme une RNA hélicase, qui viennent ensemble coordonner la dégradation de l’extrémité 3’ de l’ARNm dans le cytoplasme et le noyau (Lodish H et al, 2012). Parmi cet assemblage, six sous-unités de l’exosome lient les ribonucléases Rrp6p et Rrp44 (Wasmuth EV et Lima CD, 2017). Au total, 31 gènes sont impliqués dans sa synthèse (Giaever G et al, 2002).
RRP6 est un gène non essentiel qui, lors de son inactivation, cause une accumulation de
plusieurs ARNm, ARNr, petits ARN nucléaires et petits ARN nucléolaires ayant des défauts de maturation (Giaever G et al, 2002). Son implication très globale dans tous ces types d’ARN est un signe de son importance dans la maturation et dans les processus dégradation généralisés de l’ARNm.
Enfin, il existe une autre approche à la dégradation des ARNm, soit la dégradation par clivage endonucléotidique. Rnt1p, la ribonucléase de la famille III de la levure, utilise cette approche pour dégrader l’ARNm. En effet, celle-ci va reconnaitre une structure tige-boucle particulière sur l’ARNm et va pouvoir couper celle-ci de l’intérieur (Lamontagne B et al, 2004). Comme le mécanisme de régulation proposé dans ce mémoire est complètement dépendant de cette ribonucléase, celle-ci sera vue spécifiquement dans la prochaine section.
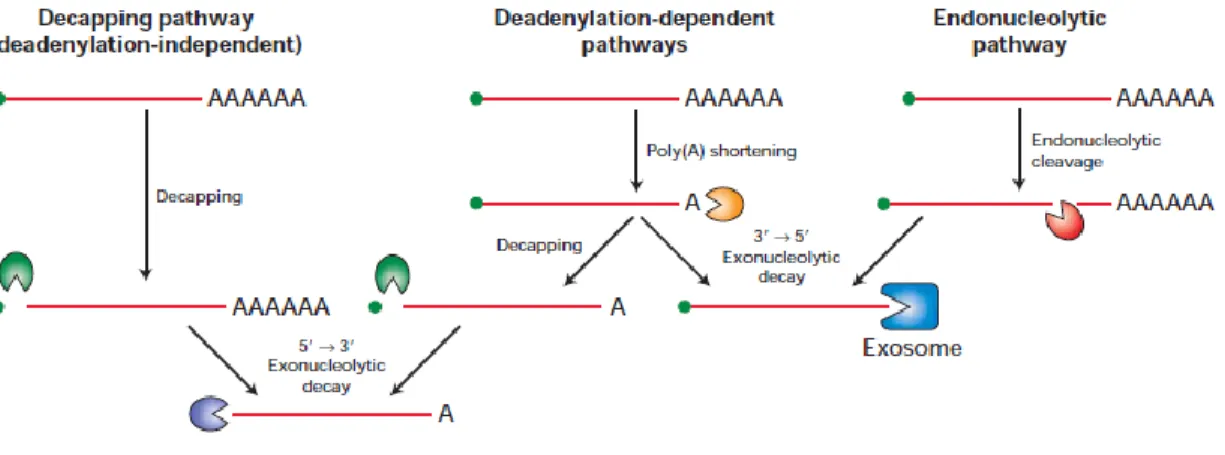
Figure 2 : Les différentes voies de dégradation des ARNm eucaryotes
La voie la plus utilisée pour dégrader les ARNm est la voie dépendante de la déadénylation de la queue poly-A. Une fois raccourci, l’ARNm pourra être dégradé par les ribonucléases Xrn1p et Rat1p en 5’ après l’abolition de la coiffe. Autrement, l’ARNm sera dégradé en 3’ par Rrp6p et l’exosome car la queue poly-A ne sera plus présente pour protéger l’ARNm. Autrement, il existe deux autres approches pour dégrader les ARNm. La cellule peut passer outre la phase de déadénylation de l’extrémité 3’ et directement enlever la coiffe avec des enzymes, ce qui ouvre la voie à la dégradation par Xrn1p et Rat1p. Autrement, des endoribonucléases peuvent venir couper l’ARNm de l’intérieur. Ce clivage va ainsi créer deux extrémités non protégées dans l’ARNm, une extrémité 5’-phosphate qui pourra être reconnue par les exoribonuclase 5’-3’ ainsi qu’une extrémité 3’ non protégée qui pourra être dégradée par les exoribonucléases 3’-5’ dans une deuxième étape.
3.5. Rnt1p, la ribonucléase de la famille III chez la levure
Les ribonucléases de la famille III sont des endoribonucléases aptes à reconnaitre une structure tige-boucle d’ARN double-brin dont une majorité est constituée d’une boucle AGNN. (Gagnon J et al, 2015) Cette famille d’enzymes est autant présente dans les bactéries que dans les cellules eucaryotes. Cependant, elle se présente sous différentes formes et structures en fonction de la complexité de l’organisme. Chez Saccharomyces
cerevisiae, Rnt1p possède deux domaines fonctionnels importants, un domaine de liaison à
l’ARN double-brin (dsRBD) et un domaine catalytique (NuclD) afin de faire le clivage de l’ARN (Blaszczyk J et al, 2001). Le domaine catalytique de Rnt1p est caractérisé par la présence d’un motif très conservé de 9 acides aminés qu’on retrouve à l’intérieur de toutes les classes des ribonucléases III (Filippov V. et al, 2000). Contrairement à la ribonucléase III de la bactérie, Rnt1p possède un domaine N-terminal supplémentaire qui est utilisé pour influencer les interactions intermoléculaires de l’enzyme (Lebars I et al, 2001).
Rnt1p se localise spécifiquement dans le noyau de la levure et ne se retrouve pas dans le cytoplasme (Ge D et al, 2005). Les rôles de Rnt1p sont multiples et affectent un large spectre de fonctions dans la cellule, passant de la régulation de l’expression génique jusqu’à la régulation du cycle cellulaire (Catala M et al, 2004). En pourcentage de travail, la plus grande tâche de Rnt1p est d’assurer la terminaison de la transcription des ARN ribosomaux transcrits par l’ARN polymérase I, plus précisément dans la région en 3’ de l’ARNr 35S (Catala M et al, 2008). Pour procéder à ce mécanisme, il a été identifié que Rnt1p est capable de se lier à l’ARN polymérase I et peut être coimmunoprécipité avec elle (Catala M et al, 2008). De plus, il a été démontré que Rnt1p était aussi apte à venir couper plusieurs petits ARN non-codants, certains ARNm liés dans la régulation du glucose et d’autres ARNm impliqués dans la régulation du cycle cellulaire et dans la réponse au stress de la membrane cellulaire (Lavoie M et al, 2012. Catala M et al, 2004).
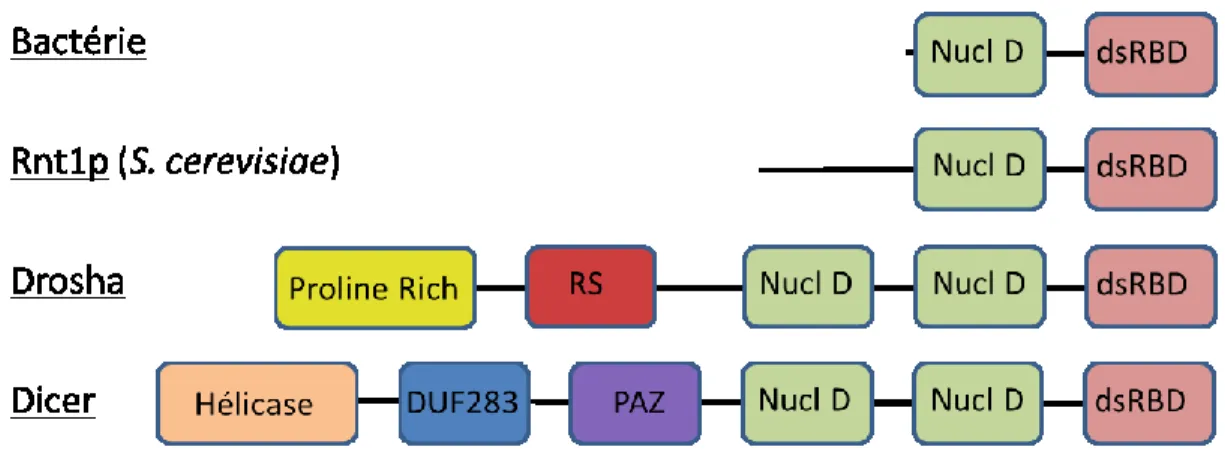
Figure 3 : Groupements fonctionnels de la famille des RNase III
Il existe 4 classes de ribonucléases III. La première classe correspond à la version présente dans les bactéries. La classe II comprends Rnt1p chez
Saccharomyces cerevisiae et Pac1p dans Saccharomyces Pombe. Un domaine
N-terminal sur les RNases de classe II les distingue des RNases bactériennes. Dans les eucaryotes supérieurs, Dicer et Drosha se partagent le rôle de ribonucléase III et possèdent plusieurs domaines fonctionnels supplémentaires.
RNT1, comme la plupart des autres ribonucléases présentées dans ce mémoire, n’est
pas à proprement parler essentielle à la survie des cellules. Cependant, une cellule avec une inactivation de RNT1 développe de nombreux problèmes de croissance combinés à une thermosensibilité à 370C (Catala M et al, 2004). De plus, une inactivation du gène induit plusieurs défauts de maturation des ARN ribosomaux et autres ARN non-codants (Lamontagne B et Abou Elela S, 2001). Lors d’une suppression du gène de RNT1, plus de 450 gènes ont vu leur niveau d’expression augmenter d’au moins deux fois à partir de profils d’expression par puces à ADN (Ge D et al, 2005). Ceci est en quelque sorte un signe avant-coureur démontrant la difficulté à identifier tous les substrats réels de Rnt1p. En effet, pour valider si un ARNm est un substrat, un clivage in-vitro peut être accompli. Cependant, ce clivage forcé n’est pas synonyme de ce qui se passe réellement dans la cellule et ne tient pas compte de tous les éléments régulateurs entrant en jeu lors d’un clivage, que celui-ci soit fait durant la transcription ou de manière post-transcriptionnelle. Enfin, un défaut en ARNr peut créer plusieurs effets indirects sur le phénotype observé.
La ribonucléase Rnt1p reconnait comme substrat une structure particulière d’ARN double brin composée d’une tétraboucle contenant une séquence de nucléotides NGNN. Autrement dit, l’élément le plus important dans la formation de la boucle d’ARN est qu’il y ait une guanine à la seconde position de celle-ci. Lorsque Rnt1p est apte à reconnaitre la boucle, l’enzyme peut venir se lier à l’ARN et venir cliver la tige. Cette coupure aura lieu à 16 et 14 nucléotides de chaque côté de la tétraboucle pour former un brin d’ARN de 34 nucléotides de long (Lamontagne et al, 2001). Ce mode de reconnaissance est le plus commun chez les substrats de Rnt1p. Il existe cependant quelques exceptions à ce mécanisme de reconnaissance et certains clivages non canoniques créent des ARN plus longs que 34 nucléotides.
Lors de l’analyse de la position des sites de clivage de Rnt1p, certains sites furent identifiés dans la région en 3’UTR de certains gènes et un mécanisme de terminaison Rnt1p dépendant fut identifié; il sera présenté à la section 4.3.3 (Ghazal G et al, 2009). Cependant, lorsque les sites de clivages de Rnt1p furent recherchés au travers du génome, 254 349 sites potentiels furent identifiés bioinformatiquement chez la levure, dont 6321 avaient un score aussi élevé que des gènes préalablement validés comme substrats dans la littérature (Gagnon J et al, 2015). Ainsi, en plus de ces fonctions importantes dans la maturation des ARN ribosomaux, Rnt1p pourrait affecter théoriquement la stabilité d’un nombre très important de gènes chez Saccharomyces cerevisiae. Cependant, la majorité des sites de clivage potentiels se retrouvent à l’intérieur de séquences codantes et non dans les régions en 3’ de celles-ci. Cette constatation fut le point interrogatoire initial de ce projet.
4. La transcription est un ensemble coordonné de mécanismes essentiels dans la régulation de l’expression génique
Une des différences notables chez les eucaryotes, comparativement aux organismes bactériens, est la présence de trois polymérases différentes. Celles-ci possèdent plusieurs sous-unités fonctionnelles similaires, mais elles transcrivent des ensembles différents de gènes (Alberts B et al, 2002). Chez les eucaryotes, l’ARN polymérase I transcrit les ARNr, sauf l’ARNr 5S qui est transcrit par la polymérase III (Lodish H et al, 2012). Ces ARNr participent donc à l’assemblage des ribosomes, unités fonctionnelles au cytoplasme pouvant traduire les ARNm en protéines. L’ARN polymérase III, de son côté, a la responsabilité de l’ARNr 5S, des ARNt et de plusieurs autres petits ARN ayant comme fonctions la maintenance de la cellule (Dieci G et al, 2007). Enfin, la polymérase II est responsable de l’ensemble de la transcription des gènes codants. Bien qu’en volume, les ARNr représentent près de 80% des ARN totaux extraits, les ARNm représentent la plus grande diversité d’ARN (Zhao W et al, 2014). Ces différences s’expriment dans l’ensemble des mécanismes de régulation de la transcription qui ont toutes comme point central leur polymérase.
Le travail effectué dans le cadre de ce mémoire cherche à mieux comprendre les mécanismes de terminaison de la transcription et ceux de la régulation cotrancriptionnelle de l’expression des gènes. Pour ce faire, il faut étudier les liens entre les mécanismes de dégradation des ARN présentés à la section 3 de ce mémoire avec les mécanismes de synthèse des ARN présentés ici. En effet, plusieurs mécanismes de maturation et de terminaison utilisent les ribonucléases comme outils. Ainsi, pour décrocher l’ARNm de son unité de synthèse et lui permettre de quitter le noyau pour le cytoplasme, il faut que celui-ci soit coupé de manière contrôlée. Comme vous le constaterez dans la section 4 de ce mémoire, la plupart des mécanismes de régulation se produisent simultanément à la transcription et la terminaison en fait partie. Elle est enclenchée en cours de transcription et est, au final, l’étape qui lui permet de prendre fin. Ainsi, ce mémoire cherche à mieux comprendre comment les mécanismes liés à terminaison de la transcription, de par l’action des ribonucléases, peuvent être régulés en cours de transcription.
4.1 L’initiation de la transcription
Cette étape est reconnue comme particulièrement importante, car la grande majorité des étapes génétiques enclenchées ne pourront pas être annulées une fois lancée (Lodish H et al, 2012). Bien que ce mémoire propose une nouvelle méthode d’interruption de la transcription sur un sous-ensemble de gènes, il reste que la régulation en amont est la manière la plus facile pour la cellule de contrôler l’expression de la totalité de génome. En effet, il est actuellement conçu que la cellule doit compléter la synthèse de tous les brins d’ARNm en cours de production pour que, dans une seconde étape, elle puisse enclencher les mécanismes qui les dégraderont comme il a été présenté à la section 3.4.
Ainsi, une fois l’hélice d’ADN devenu disponible comme discuté à la section 2.3, les facteurs activateurs pourront venir interagir. Le positionnement génomique où le recrutement des facteurs aura lieu est très important. La machinerie de transcription doit pouvoir identifier où elle commencera et où elle devra terminer pour que l’ARNm produit puisse former, à la toute fin, la bonne protéine, donc la bonne séquence avec le bon cadre de lecture (Barbosa C et al, 2013). Pour reconnaitre la bonne région dans l’organisme
Saccharomyces cerevisiae, des facteurs de transcription activateurs ou répresseurs
reconnaîtront des séquences spécifiques activatrices ou répressives en amont du gène pour identifier où la transcription commencera (Hahn S et Young ET, 2011). Pour qu’un gène soit transcrit par l’ARN polymérase II au-delà d’un niveau basal, il faut qu’un gène possède une ou plusieurs régions activatrices, souvent situées à plusieurs milliers de bases avant le site d’initiation de la transcription (Hahn S et Young ET, 2011). Chez la levure, 169 gènes sont reconnus pour avoir une fonction de facteurs de transcription et 257 séquences spécifiques de liaisons ont été identifiées pour un total de 12 346 associations possibles et distinctes (Teixeira et al, 2006). Une fois liés à l’ADN, les facteurs activateurs vont participer à l’ouverture de la chromatine et permettre la liaison de l’ARN polymérase II au promoteur. Celle-ci se retrouve initialement dans un complexe de pré-initiation composé de plusieurs autres facteurs de transcription (Lodish H et al, 2012). Chez la levure, le complexe de pré-initiation est recruté dans la région TATA riche pour que l’ARN polymérase II puisse identifier le site d’initiation de la transcription (Lubliner et al, 2015)
.Après plusieurs courtes tentatives de départ, l’ARN polymérase II quittera finalement la région promotrice pour aller transcrire l’ADN du gène en ARNm. (Lubliner et al, 2015)
Ainsi, l’initiation de la transcription peut être résumée à trois grandes étapes. L’assemblage des complexes de préinitiation où les facteurs de transcription se joignent à l’ARN polymérase au promoteur, l’initiation où il y aura la formation des premiers liens phosphodiester entre les nucléotides et enfin le départ du complexe transcriptionnel du promoteur pour aller parcourir le gène. (Otero G et al, 1999) La région promotrice a de grands impacts sur la force d’adhésion de la polymérase et de nombreux facteurs sont impliqués dans la régulation du mécanisme. (Lodish H et al, 2012) Ainsi, le promoteur d’un gène peut avoir une grande incidence sur les événements qui auront lieu plus tard dans la transcription. Dans le cadre de ce projet, l’impact du promoteur a été analysé, car nous avions émis l’hypothèse que celui-ci avait une importante implication dans le mécanisme qui sera proposé dans l’article présenté.
4.2 L’élongation de la transcription
Une fois que tous les facteurs sont en place et que la phase d’initiation s’est terminée, la production d’une chaine d’ARNm peut avoir lieu. Pour permettre le passage de la phase d’initiation à la phase d’élongation, le CTD de la plus grosse sous-unité de l’ARN polymérase II doit devenir hyperphosphorylé à l’aide de la TFIIH kinase. (Otero G et al, 1999) Avec l’ouverture de la double hélice d’ADN, un des deux brins peut maintenant servir de modèle pour la synthèse d’un ARNm. Ainsi, avec l’aide de la polymérase, un ribonucléotide complémentaire à la séquence d’ADN peut se lier de manière covalente à la chaine croissante d’ARN par une réaction de catalyse enzymatique. (Alberts B et al, 2002) Cet ARN ne restera pas lié très longtemps à son modèle d’ADN. En effet, lorsque l’ARN polymérase se déplace sur le long de l’ADN, l’ancienne section du simple brin d’ARN se libère de son interaction avec l’ADN et devient disponible pour interagir avec d’autres facteurs. (Lodish H et al, 2012) C’est par ce principe que l’ARNm pourra déjà commencer ses processus de maturation, débutant par l’installation d’une coiffe sur son extrémité 5’-phosphate. L’élongation est de plus facilitée par un nombre de facteurs ciblant la chromatine et/ou l’ARN polymérase II pour empêcher les pauses et permettre le déplacement fluide de celle-ci le long de l’ADN. (Reines D et al, 1999)
Lors du processus d’élongation, l’avancée de la polymérase sur le gène entrainera des réactions chez l’enzyme qui seront très utiles pour la prochaine étape de la transcription. En effet, plus celle-ci avancera en cours de transcription, plus la sérine 2 de son domaine c-terminal deviendra phosphorylée jusqu’à atteindre l’extrémité 3’ du gène codant. (Heo DH et al, 2013) Comme il sera mentionné plus tard, il s’agit d’un des principaux signaux de la terminaison de la transcription, la dernière étape de la synthèse de l’ARNm. Plusieurs liens importants sont à faire entre l’élongation de la transcription et la régulation de la stabilité des ARNm. En réalité, les processus de maturation sont très bien coordonnés chronologiquement et se font simultanément à la synthèse de l'ARNm. En effet, l’ajout de la coiffe, la synthèse de la queue poly-A, le recrutement de la machinerie d’épissage et l’édition de l’ARNm se font tous cotranscriptionnellement. (Bentley DL, 2014) Dans le cadre de ce projet, il semble que Rnt1p est recruté en cours de transcription pour pouvoir reconnaitre le site de clivage qui sera présent sur l’ARNm. De plus, notre RNase III aura
une collaboration importante avec l’exoribonucléase Rat1p dans le mécanisme de terminaison. Il serait donc logique de penser que les deux ribonucléases se coordonnent en cours d’élongation pour induire le mécanisme de la terminaison. En effet, il a été démontré que XRN2, l’équivalent de RAT1, pouvait s’associer cotranscriptionnellement et induire la dégradation de certains transcrits. (Davidson L et al, 2012)
4.3 La terminaison de la transcription
Chez les eucaryotes, la terminaison des séquences responsables de la transcription des ARNr par la polymérase I nécessite la présence de protéines liant l’ADN. (Lodish H, 2012) Dans le cas des séquences dépendantes de l’ARN polymérase III, la terminaison se produit par la polymérisation de résidus uraciles et la formation d’un hybride ADN-ARN. (Lodish H, 2012) Chaque polymérase possède en réalité sa propre série de mécanismes distinguant la terminaison de ses transcrits. Dans ce mémoire, une nouvelle application d’un mécanisme de terminaison de l’ARN polymérase II est proposée.
Une fois le gène parcouru dans sa totalité par l’ARN polymérase II, l’enzyme doit pouvoir arrêter pour relâcher le brin d’ARNm précédemment synthétisé. La terminaison aura un rôle majeur dans la mise en place de la queue poly-A en 3’ des ARNm. Dans la levure, plusieurs mécanismes de terminaison sont connus et varient en fonction de l’ARN en question. En effet, comme les transcrits qui proviennent des différentes polymérases utilisent des mécanismes de terminaison différents, différents ARN transcrits par la polymérase II peuvent posséder des mécanismes de terminaison qui leur sont particuliers. En effet, les ARNm n’utiliseront pas forcément les mêmes mécanismes de terminaison que les ARNnc. Enfin, il a même été démontré qu’il existe certains mécanismes présents afin d’assurer une terminaison de secours ou failsafe (Lemay JF et Bachand F, 2015). Il est pertinent d’entrer dans le détail des mécanismes généraux afin de bien mettre en contexte le projet de recherche supporté par ce mémoire.
4.3.1 Mécanisme de terminaison poly-A dépendante : Rat1p dépendante
Ce premier modèle de terminaison est le plus commun au niveau des ARNm. Ce mécanisme de terminaison commence par le recrutement de plusieurs facteurs de maturation des ARNm auprès du domaine C-terminal de l’ARN polymérase II. Parmi cet ensemble de facteurs se retrouvent les protéines liées à la polyadénylation qui reconnaissent la sérine 2 phosphorylée du domaine C-terminal de la polymérase (Kim M, 2004). Plus la polymérase parcours le gène lors de la phase d’élongation, plus la phosphorylation de la
sérine 2 augmente, ce qui en fait un important signal de terminaison de la transcription (Kim M, 2004). Parmi les différents facteurs impliqués dans ce mécanisme se retrouvent la ribonucléase Rat1p et les complexes protéiques qui vont reconnaitre la région en 3’ de l’ARNm. L’ARN polymérase II va transcrire l’ARNm au-delà du site de polyadénylation présent en 3’ de la séquence codante et va transcrire sur son ARNm les sites de reconnaissance pour ses protéines de terminaison. Deux sous-unités protéiques, CstF (cleavage stimulation factors) et CPSF (cleavage and polyadenylation specificity factor) vont ensuite reconnaitre ces dites séquences génomiques spécifiques dans cette région et se lier sur l’ARNm (Alberts B et al, 2002). Celles-ci vont entrainer avec elles plusieurs autres protéines impliquées dans la terminaison pour leur permettre de localiser l’extrémité 3’ de la séquence codante. Après cette étape de reconnaissance, l’endonucléase YSH1 va reconnaitre et cliver l’ARNm pour le libérer de la polymérase (Schaughency P et al, 2014). Cela va libérer une extrémité 3’ qui sera reconnue par l’ARNm polyA polymérase Pap1p (Kim M et al, 2006). Pap1p pourra ajouter une chaine d’approximativement 200 adénines à la nouvelle région exposée en 3’ sans avoir besoin d’un modèle d’ADN, contrairement à l’ARN polymérase (Albert B et al, 2002).
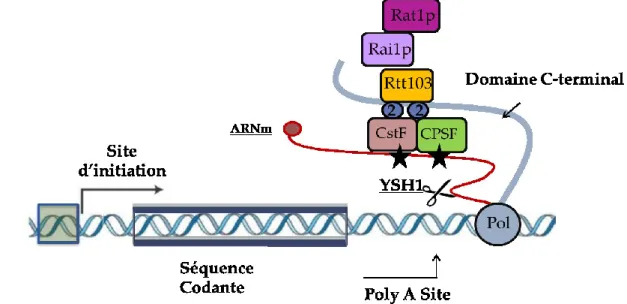
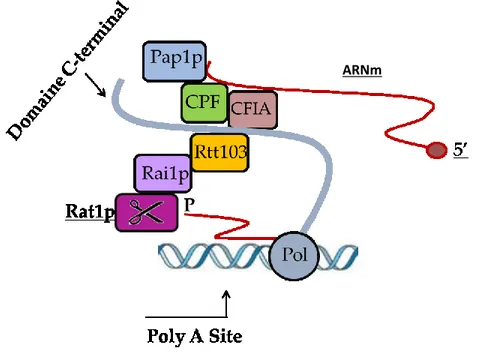
Figure 4 : Terminaison Poly-A dépendante et Rat1p dépendante
Après avoir quitté la région codante, la polymérase continuera toujours la transcription de l’ADN. Les facteurs CstF et CPSF vont reconnaitre la région des séquences spécifiques sur l’ARNm et s’y lier. L’endonucléase YSH1 reconnaitra la région d’ARNm à couper et la libérera de la polymérase.
Une fois la queue poly-A mise en place, certaines protéines pourront se lier sur elle à la fin de l’ARNm afin de guide celle-ci dans son passage vers le cytoplasme jusqu’aux ribosomes (Albert B et al, 2002). Voici le mécanisme au niveau de l’ARNm.
Du côté de l’ADN, après le clivage, il faut mentionner que la polymérase restera encore attachée à l’ADN et continuera son processus de transcription sur une région de plusieurs centaines de nucléotides après la région codante (Alberts B et al, 2002). L’extrémité d’ARNm encore attachée à l’ARN polymérase exposera une extrémité non protégée en 5’ qui pourra être reconnue par la ribonucléase Rat1p. Même si le mécanisme pour décrocher l’ARN polymérase reste peu connu, la littérature semble suggérer que lorsque Rat1p aura dégradé l’ARNm excédent et sera parvenu à rattraper la polymérase, la déstabilisation du complexe entrainera le relâchement de la polymérase de l’ADN (Kim M, 2004). Ce mécanisme porte le nom de modèle torpille.
Figure 5: Le recrutement cotranscriptionnel de Rat1p induit le décrochage de l’ARN polymérase après le clivage.
Une fois l’ARNm clivé, la liaison des protéines de reconnaissance permettra le positionnement de Rat1p et Pap1p. Rat1p dégradera le brin d’ARNm toujours produit par l’ARN polymérase II alors de Pap1p ajoutera des nucléotides A à l’extrémité 3’ de l’ARNm codant pour le gène initialement transcrit.
4.3.2 Mécanisme de terminaison poly-A indépendante : Rat1p indépendant (Courts ARN non-codants)
La majorité du temps, lorsque nous parlons d’expression génique, il s’agit de l’expression de séquences codantes. Cependant, certaines séquences d’ADN doivent être exprimées pour remplir des fonctions importantes, même si elles ne codent pas pour des protéines (Cech TR et Steitz JA, 2014). Dans le cas des petits ARN non-codants, ceux-ci possèdent un mécanisme de terminaison de leur extrémité 3’ différent du mécanisme présenté précédemment. L’ARN polymérase II de Saccharomyces cerevisiae participe à la transcription de certains ARN non-codants comme les petits ARN nucléaires, les petits ARN nucléolaires et certains transcrits cryptiques instables (Lemay et al, 2016). Ce choix de mécanisme est indépendant du clivage de l’ARNm et de la présence de l’exoribonucléase Rat1p (Vasiljeva L et al, 2008). Le mécanisme de terminaison poly-A indépendant nécessite la présence de Nrd1p et Nab3p, des protéines aptes à lier l’ARN, en plus de l’hélicase Sen1p (Porrua O et Libri D, 2013). Dans le cas présent, la protéine Nrd1p interagit avec la sérine 5 du domaine C-terminal de l’ARN polymérase II (Heo DH et al, 2013). Contrairement à la sérine 2 qui devient de plus en plus phosphorylée plus la transcription avance, la sérine 5 se déphosphoryle tout au long de la transcription (Heo DH et al, 2013). Pour que la sérine 5 soit un bon signal de terminaison, il faut donc que la séquence transcrite soit courte, autrement le recrutement des protéines liées au mécanisme poly-A dépendant sera fortement avantagé. Il semble donc que la longueur du gène à terminer soit un des éléments les plus importants afin de déterminer quel mécanisme de terminaison la cellule doit choisir (Porrua O et Libri D, 2013). L’hélicase Sen1p serait, quant à elle, complètement indépendante du domaine c-terminal de la polymérase (Porrua O et Libri D, 2013).
Nrd1p et Nab3p vont initier ce modèle de terminaison en reconnaissant des motifs spécifiques sur l’ARN et se lier à ceux-ci. Sen1p va interagir auprès de la polymérase pour la faire quitter l’ADN et arrêter la transcription (Vasiljeva L et al, 2008). Le mécanisme derrière la capacité de Sen1p à arrêter la transcription est encore mal connu mais l’hypothèse actuelle est que cette protéine participerait à défaire les hybrides RNA-DNA, plus précisément les R loops, formés lors de la transcription près de la polymérase et finirait par déstabiliser celle-ci (Porrua O et Libri D, 2013). Avec l’arrêt de l’ARN polymérase II, l’ARN formé exposera une extrémité 3’ non protégée qui sera reconnu par le complexe Tramp et l’exosome (LaCava, 2005).
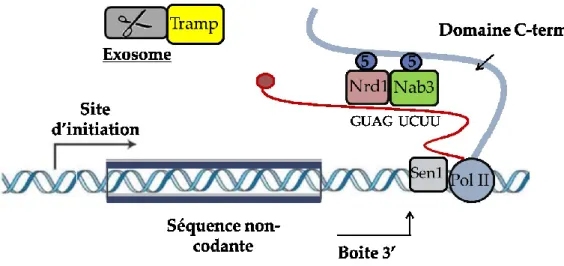
Figure 6 : Le recrutement des facteurs de terminaison de la voie Sen1-Nrd1 se fait aussi de manière cotranscriptionnelle.
Les deux protéines Nrd1p et Nab3p liés aux domaines C-terminal de la polymérase vont reconnaitre des séquences spécifiques à la fin du transcrit non-codant. L’hélicase Sen1p va déstabiliser la polymérase et la forcer à se décrocher de l’ADN, arrêtant simultanément le processus de transcription.
Le complexe Tramp est reconnu pour sa forte capacité à activer l’exosome et est composé d’une hélicase ATP-dépendante nommée Mtr4p, de la poly-A polymérase Trf4p et la protéine capable de lier l’ARN Air2p (LaCava et al, 2005). De son côté, l’exosome chez Saccharomyces cerevisiae est un assemblage de différentes protéines auxquelles s’associeront les ribonucléases Rrp6p et Dis3p (Chanfreau GF et Tamanoi F, 2012). Les deux reconnaitront ensemble l’extrémité 3’ de l’ARN non-codant et commenceront à dégrader celle-ci jusqu’à atteindre le point de liaison formé par Nrd1p et Nab3p. À ce moment, la poly-A polymérase Tfr4, aidée de Tfr5, pourra synthétiser la queue poly-A sur l’ARN (Jamonnak N et al, 2011).
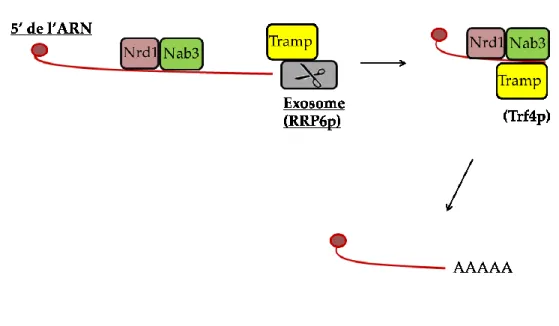
Figure 7 : La dégradation par l’exosome dans la voie Nab3-Sen1
Le complexe Tramp et l’exosome vont reconnaitre l’extrémité 3’ non protégée de l’ARN et dégrader celui-ci jusqu’à rejoindre le complexe formé par Nrd1p et Nab3p. Ces deux protéines vont être le signal d’arrêter la dégradation. À ce moment, Trf4p commencera à synthétiser la queue poly-A.
4.3.3 Mécanisme de terminaison poly-A indépendante : Rnt1p dépendant (Long ARN non-codants)
L’implication de l’exoribonucléase Rat1p avait été démontrée dans la maturation des ARN transcrits par l’ARN polymérase II. Lors de recherches sur la terminaison de certains transcrits produits par l’ARN polymérase I, il a été découvert que la nucléase Rat1p était aussi essentielle au mécanisme de terminaison des ARN ribosomaux (El Hage A et al, 2008). Or, la maturation des ARN ribosomaux, plus précisément l’extrémité 3’ du pré-ARN 35S, nécessite initialement un clivage par l’endoribonucléase Rnt1p pour se produire. (Kawauchi J et al, 2008) Ainsi, la démonstration du partenariat entre Rnt1p et Rat1p dans la terminaison des ARN ribosomaux fut faite.
Cependant, certains gènes transcrits par l’ARN polymérase II possédaient eux aussi un signal de clivage de Rnt1p dans leur région en 3’, ce qui laissait sous-entendre la possibilité d’un rôle similaire avec la polymérase II. Ainsi, lors de l’étude des gènes NPL3 et GPI17, physiquement localisés très près l’un de l’autre et transcrits par la polymérase II chez la levure, il a été observé qu’une suppression de RNT1 causait l’apparition de transcrits polycistroniques entre les deux gènes (Ghazal G et al, 2009). Dans cette étude, il a été démontré qu’entre les deux gènes existe un site de clivage de Rnt1p et celui-ci participe à la terminaison de NPL3 (Ghazal G et al, 2009). Ce mécanisme de terminaison fonctionne très similairement à la terminaison de la polymérase I. En formant une structure en tige-boucle NGNN reconnue par Rnt1p, l’endonucléase coupe l’ARNm toujours en cours de transcription et produit deux extrémités non protégées. Il a été démontré que Rrp6p était impliqué en partie dans la dégradation des extrémités 3’ libres alors que Rat1p dégradait les ARNm avec l’extrémité 5’ libre (Ghada Ghazal et al, 2009). Le rôle de Rnt1p dans la terminaison de certains transcrits par la polymérase II s’est donc plus concrétisé. Cependant, il a été déterminé qu’un clivage de Rnt1p ne menait pas toujours à une terminaison de la transcription et que des éléments supplémentaires comme un promoteur particulier ou bien des séquences précises devaient être impliquées pour coordonner tous les acteurs de la terminaison (Meaux S et al, 2011). Cela restait cependant à déterminer.
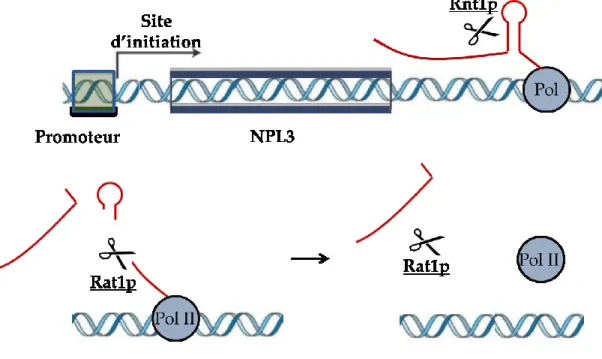
Figure 8 : Mécanisme de terminaison Rnt1p-Rat1p dépendant
La région entre les gènes NPL3 et GPI17 contient les séquences nécessaires afin de permettre la formation d’une structure tige-boucle NGNN dans l’ARNm correspondant. L’endoribonucléase Rnt1p est apte à reconnaitre cette structure pour venir initier un clivage de l’ARNm. Au moment de l’étude, il avait été démontré que la longueur du transcrit observé augmentait en l’absence des ribonucléases Rat1p (inactivation thermique) et Rrp6p, ce qui laissait présager la présence d’un modèle similaire au modèle torpille.
5. Problématiques de recherche
Avant ce projet de recherche, de nombreuses fonctions de la ribonucléase Rnt1p étaient connues dans la maturation de plusieurs ARN non-codants (Ghazal G et al, 2005). Rnt1p était aussi reconnue pour sa capacité à couper un certain ensemble d’ARNm et participer à la régulation d’un sous-ensemble de gènes (Lavoie M et al, 2012). Ces différentes fonctions ne semblaient donc pas à priori se rejoindre, jusqu’à ce que la capacité de Rnt1p à cliver certains ARNm dans leur région en 3’ soit faite.
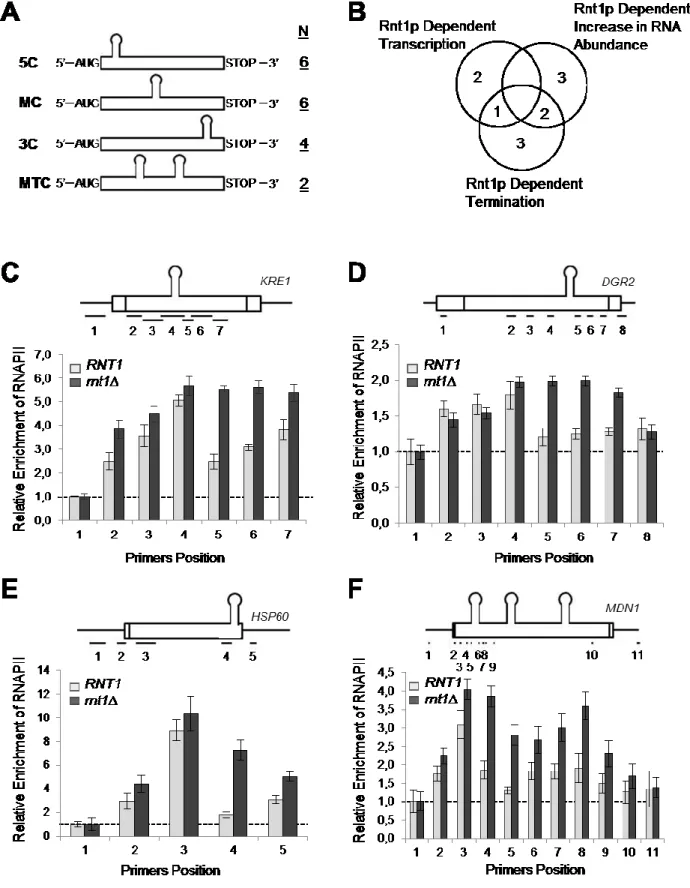
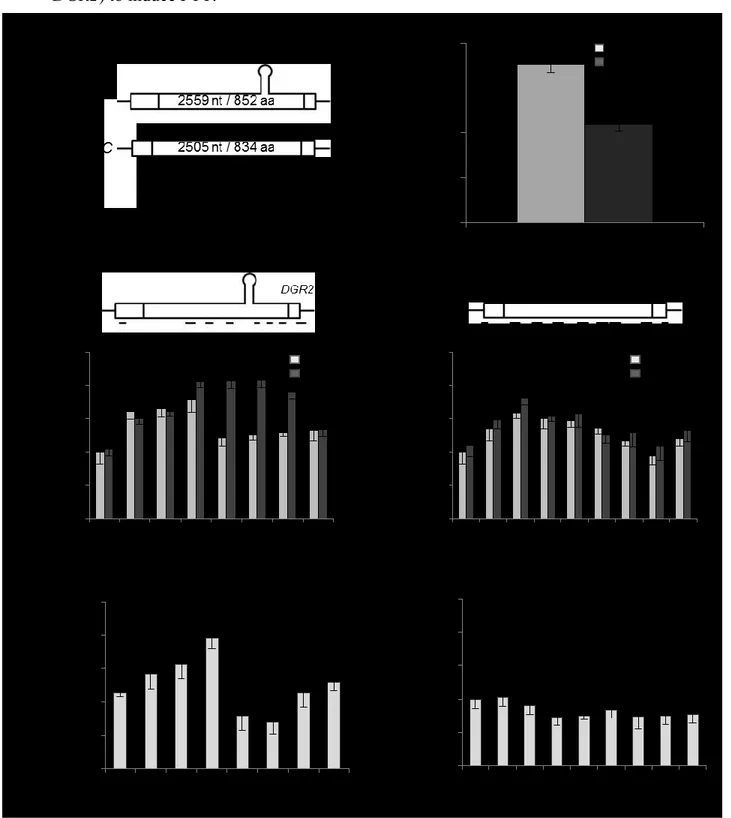
Avec l’étude des sites de clivages de Rnt1p, une question se pose. Si un site de clivage de Rnt1p présent en 3’ d’un ARNm peut induire une terminaison, est-ce qu’un site de clivage à l’intérieur même d’une séquence codante peut induire ce même mécanisme ? Cette question est très importante car lorsqu’on regarde les prédictions de positionnement des sites de clivage de Rnt1p près des séquences codantes, il est possible de remarquer que la majorité des sites de clivage sont à l’intérieur de gènes (Gagnon J et al, 2015). Ainsi, si ce qui a été observé à la fin des gènes ne représente qu’une faible proportion des clivages de Rnt1p, y aurait-il un plus grand rôle dans la régulation même des gènes ?
De plus, il a été démontré qu’un clivage d’ARN par Rnt1p n’induit pas forcément un événement de terminaison (Meaux S et al, 2011). Si une terminaison de la transcription existe à l’intérieur de gènes, par quel mécanisme celle-ci se produit-elle et quels sont les éléments distinguant un événement de terminaison prémature à un simple clivage de l’ARNm par Rnt1p ? Ces questions ont donc mené à la recherche d’un événement de terminaison de la transcription au travers de différents gènes. Une fois un modèle identifié, plusieurs expériences furent faites pour définir les différents éléments nécessaires afin de différencier un événement de terminaison d’un clivage standard. Vous retrouverez les détails dans l’article suivant.
Présentation de l’article
La dégradation cotranscriptionnelle des ARNm peuvent induire un mécanisme de terminaison prémature de la transcription
Avant-propos
Co-transcriptional RNA Degradation Triggers Premature Transcription Termination Francis Malenfant, Mathieu Catala, Mélodie Berthoumieux et Sherif Abou Elela
Article soumis dans Nucleic Acid Research
Mars 2017
Contribution : Les expériences effectuées dans le cadre de ce mémoire et la majorité des figures présentées dans cet article ont été faites par Francis Malenfant. Les co-auteurs ont aidé dans la préparation et la transformation des souches de levure, dans le cheminement du raisonnement scientifique du projet et dans la révision de l’article. De plus, Francis Malenfant a rédigé la section « matériel et méthodes ». Mathieu Catala a travaillé sur les tables supplémentaires, sur la figure S5 et a participé à la rédaction de la figure 1. Mélodie Berthoumieux a travaillé sur la figure 7. Le professeur Sherif Abou Elela a rédigé le reste du manuscrit initial auquel tous les auteurs ont apportés leur contribution lors de la révision.