THESE
THESE
En vue de l'obtention du
DOCTORAT DE L’UNIVERSITÉ DE TOULOUSE
DOCTORAT DE L’UNIVERSITÉ DE TOULOUSE
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Cancérologie
JURY
Pr Bettina COUDERC Présidente du jury Dr Jean-Luc TEILLAUD Rapporteur
Dr Christophe DE ROMEUF Rapporteur
Pr Hervé PRATS Directeur de thèse Dr Philippe MONDON Co-directeur de thèse
Ecole doctorale : Biologie - Santé - Biotechnologies de Toulouse Unité de recherche : INSERM - I2MR- U858
Directeur(s) de Thèse : Pr Hervé PRATS / Dr Philippe MONDON
Présentée et soutenue par Stéphanie FALLOT Le 24 Septembre 2009
Titre : Elaboration de vecteurs polycistroniques à stœchiométrie variable
Je dédie cette thèse
A mes parents,
A mon frère Ben,
A toute ma famille,
en particulier Yves, Karine, Erwan, Corentin et mon Papy à qui je pense très
fort,
Et enfin à ma grand-mère Lucette avec qui j’aurais aimé partager cela et qui
aurait sûrement été fière de moi.
Je souhaiterais remercier Hervé Prats et Philippe Mondon, mes deux co-directeurs de thèse, pour m’avoir permis de réaliser cette thèse à l’interface entre deux environnements, qui n’a pas
toujours été facile à mener à bien mais qui m’a été profitable sur de nombreux plans. Je tiens également à remercier les membres de mon jury, Madame Bettina Couderc, et Messieurs Christophe de Romeuf et Jean-Luc Teillaud qui m’ont fait l’honneur d’accepter de
juger mon travail et de participer à ma soutenance.
C’est également pour moi l’occasion de remercier toutes les personnes que j’ai pu croiser au cours de ces années passées à Millegen et au laboratoire et qui m’ont beaucoup apporté tant sur le plan
scientifique que sur le plan humain.
Christian, je sais que ça n’a pas toujours été évident pour toi de m’encadrer mais je me dois de reconnaître que tu m’as beaucoup appris.
Toutes les personnes de Millegen pour leur accueil, leur aide et leur soutien: Hakim, Khalil, Christine (merci pour ta disponibilité et ta grande gentillesse), Fabien, Nathalie, Céline, Anne, Laurence, Yann, Olivier, Marie-Julie,…merci et bonne route à vous tous, sans oublier Patrick,
David et Frédéric qui sont maintenant partis vers d’autres horizons.
Merci également à tous ceux qui m’ont accompagnée au quotidien dans ce cadre idyllique qu’est le troisième étage du L3, je sais que je n’ai pas toujours été une collègue gaie, enjouée et souriante mais il paraît qu’on ne se refait pas alors je vous remercie tous d’avoir fait avec et de
m’avoir aidée, soutenue ou tout simplement fait rire :
Les anciens du labo qui m’ont montré la voie et qui m’ont accompagnée à divers moments de la thèse: Zeïneb (tu sais tout ce que je te souhaite pour la suite), Yoyo (mon australo-portuguais que je n’ai pas eu le temps d’aller voir dans l’hémisphère sud) , Jason (see you in
Messanges…), Raouia, Sylvain, Amande, Sara (ma coloc-éclair), Sophie, Monique, Olivier (mon apprenti médecin préféré) et tous ceux qui y sont encore : Guillaume (le roi de la mécanique, Scooty te remercie aussi), Hugo, Audrey M., Marina, Céline, Tania, Eric, Elodie,
Hortense, Pierre (alias Dr Gourdy), Victorine, Loïc, Aurélie, Piou, Henrik et Odile. Merci à tous pour votre gentillesse et votre patience et surtout bon courage à tous ceux qui termineront
bientôt leur thèse et qui voudraient à cet instant être à ma place : COURAGE à vous tous, nul doute que vous allez tous y parvenir !
Une pensée bien particulière pour Cathy (merci pour TOUT) mais aussi pour Nadéra, Audrey R. et Dudu. Merci à vous pour votre amitié, pour les bons moments passés au labo et hors du labo. Un grand MERCI également pour avoir été là, près de moi, dans les moments difficiles,
je ne vous l’ai jamais dit mais vous m’avez beaucoup aidé et je ne l’oublie pas. Merci aussi à mes amis de Toulouse et d’ailleurs qui m’ont permis de garder le cap et de passer de supers moments dans l’ « autre vie » : Cyril et toute la famille Viaud, Sylvie et Stéphane mes basquo-bourguino-béarno-landais préférés, Manu le banquier parisien, Mathieu
et Tamara, Julie, Delphine, Jess, Caï, Dédé et les autres…
RESUME
Les outils permettant la co-expression de plusieurs protéines dans une même cellule se sont fortement développés depuis plusieurs années, et sont désormais indispensables pour de nombreuses applications tant en recherche fondamentale que dans le cadre d’expérimentations de thérapie génique. Plusieurs systèmes ont été envisagés pour cela, utilisant des stratégies variées allant de l’utilisation de plusieurs vecteurs indépendants à celle de vecteurs polycistroniques dans lesquels l’expression du second cistron est rendue possible grâce à l’insertion de séquences intercistroniques particulières. C’est le cas, par exemple, des sites d’entrée interne des ribosomes (IRES) ou du peptide 2A issu du génome du virus FMDV (Foot-and-Mouth Disease Virus).
Au cours de ma thèse, j’ai eu à développer un vecteur permettant la co-expression de deux polypeptides qui soit applicable, entre autres, à l’expression d’anticorps recombinants. La production d’anticorps recombinants à visée thérapeutique se fait classiquement dans des cellules de mammifères, dans lesquelles l’expression simultanée des deux chaînes est requise. Le ratio intracellulaire entre les deux chaînes a un fort impact sur la productivité d’anticorps recombinants. Le rapport optimum varie selon plusieurs critères, comme la séquence de l’anticorps, le type cellulaire utilisé, les conditions de production (stable ou transitoire)… Les systèmes évoqués précédemment pour la co-expression de deux protéines ne permettent pas de moduler ce ratio de façon reproductible et étendue. Afin de générer un outil permettant de faire varier la stœchiométrie entre les deux protéines, facilement adaptable à la production du plus grand nombre d’anticorps quelles que soient les conditions choisies, un nouveau type de vecteur bicistronique a été conçu. Ces vecteurs, basés sur l’épissage alternatif, permettent l’expression de deux protéines à partir d’un seul ARN pré-messager possédant un site donneur d’épissage et deux sites accepteurs alternatifs placés en amont de chacun des deux cistrons. Le ratio entre les deux protéines est ainsi adapté en mutant les différents sites accepteurs d’épissage, ce qui a pour conséquence de modifier leur fréquence d’utilisation.
Le système a été construit avec deux gènes rapporteurs, ce qui a permis de démontrer que le ratio pouvait facilement être modulé et permettre aux quantités relatives des deux protéines de varier d’un rapport 10:1 à 1:10. Le vecteur a ensuite été validé pour l’expression de plusieurs anticorps recombinants, dans différents types cellulaires et dans des conditions de transfection stable et transitoire. Dans tous ces cas, différents mutants des sites d’épissage produisant divers ratios entre la chaîne lourde et la chaîne légère ont été testés et il a été confirmé que le rendement de production d’anticorps entiers sécrétés par les cellules varie en fonction des mutants et donc en fonction du ratio intracellulaire entre les deux chaînes.
Le vecteur à épissage alternatif développé au cours de ma thèse est donc applicable à la production d’anticorps recombinants fonctionnels dans les cellules de mammifères et pourra également être utilisé pour de très nombreuses applications comme la co-expression de deux
SOMMAIRE
RÉSUMÉ………...1
SOMMAIRE………. 3
LISTE DES ABRÉVIATIONS………7
INTRODUCTION……….9
I - L’ÉPISSAGE : MÉCANISMES ET RÉGULATION………..11
A) Introduction : transcription et maturation des ARN pré-messagers……….13
B) L’épissage : mécanisme et régulation………14
1) Les sites d’épissage………...14
2) Mécanisme général de l’épissage……….17
3) Régulation de l’épissage………...18
a) Les séquences exoniques et les facteurs associés……….19
b) Les séquences introniques de régulation………..…23
C) L’épissage alternatif………....24
1) Mécanisme et caractéristiques………..24
2) Régulation de l’épissage alternatif………27
3) Epissage alternatif et pathologies……….30
D) Couplage entre l’épissage et les autres étapes de la vie d’un ARN messager……32
II - LES ANTICORPS : GÉNÉRALITÉS………..35
A) Structure, assemblage et sécrétion des anticorps……….37
B) Différents formats d’anticorps recombinants………..41
C) Chimérisation et humanisation des anticorps………..42
E) Applications thérapeutiques des anticorps monoclonaux recombinants:
de 1975 à aujourd’hui……….47
III - PRODUCTION D’ANTICORPS RECOMBINANTS………..51
A) Les différents hôtes et systèmes pour la production d’anticorps recombinants...53
1) Les systèmes procaryotes……….54
2) Les levures et champignons filamenteux………..55
3) Les systèmes baculovirus / cellules d’insectes……….55
4) Les plantes transgéniques……….56
5) Les animaux transgéniques………...56
6) Les cellules de mammifères………..57
B) Profil de glycosylation et systèmes d’expression………..57
C) Les différents modes d’expression dans les cellules de mammifères………..59
1) L’expression stable………...59
a) La transcription des transgènes……….60
b) Le nombre de copies du transgène………62
c) La traduction et la sécrétion des protéines d’intérêt……….64
d) Survie, prolifération et apoptose des cellules………...65
2) L’expression transitoire………66
a) Choix des vecteurs et de la méthode de transfection………68
b) Choix des cellules hôtes : les HEK, un outil de choix………..69
D) Influence du ratio entre les chaînes lourdes et légères sur la production d’anticorps recombinants………...71
E) Vecteurs utilisés pour la production d’anticorps recombinants……….77
1) Co-transfection de deux plasmides différents………...79
2) Vecteurs à plusieurs promoteurs………...79
3) Vecteurs à IRES………81
4) Vecteurs comportant des peptides « auto-clivables »………...83
RÉSULTATS………...89
RATIONNEL………91
I - DÉVELOPPEMENT DU VECTEUR À ÉPISSAGE ALTERNATIF………97
Article : Alternative-splicing-based bicistronic vectors for ratio-controlled protein expression and application to recombinant antibody production………...99
II - RÉSULTATS COMPLÉMENTAIRES……….….119
A) Utilisation des vecteurs pour l’expression d’un autre anticorps murin………...121
1) Rationnel……….121
2) Matériel et méthodes………...122
3) Résultats………..124
a) Etude des sites cryptiques d’épissage……….124
b) Modulation du ratio entre les deux chaînes de l’anticorps……….128
c) Analyse des quantités d’anticorps sécrétés dans les surnageants de culture………..129
B) Mise au point d’outils pour la recherche de sites cryptiques d’épissage……….133
1) Rationnel………133
2) Matériel et méthodes………..135
3) Résultats……….136
C) Autres types de vecteurs envisagés………..137
1) Rationnel……….137
2) Matériel et méthodes………...140
3) Résultats………..142
D) Utilisation des vecteurs pour la production d’anticorps recombinants à moyenne échelle………..144
CONCLUSIONS, DISCUSSIONS ET PERSPECTIVES……….149
BIBLIOGRAPHIE………163
ANNEXES……….……….179
LISTE DES ABRÉVIATIONS
5’ SS / 3’SS : 5’/ 3’ Splice Site AAV: Adeno-Associated Virus
ADCC : Antibody-Dependent Cellular Cytotoxicity
ADN : Acide Désoxyribonucléique ADNc: ADN complémentaire ARN : Acide Ribonucléique ARNm : ARN messager ARNt : ARN de transfert
ASF/SF2 : Alternative Splicing Factor/Splicing Factor 2
Asn : Asparagine
BAC : Bacterial Artificial Chromosome BHK : Baby Hamster Kidney
BiP : immunoglobulin Binding Protein CBP : Cap Binding Protein
CDC : Complement-Dependent Cytotoxicity CDR : Complementarity Determining Region CELF : CUGBP and ETR-Like Factors CHO : Chinese Hamster Ovary
CMV: Cytomegalovirus CTD : C-Terminal Domain DHFR : Dihydrofolate Reductase
DMEM : Dulbecco's Modified Eagle Medium DO : Densité Optique
Domaine CH : domaine constant de la chaîne lourde d’un anticorps (Constant Heavy) Domaine CL : domaine constant de la chaîne légère d’un anticorps (Constant Light) Domaine VH : domaine variable de la chaîne lourde d’un anticorps (Variable Heavy) Domaine VL : domaine variable de la chaîne
E. coli : Escherichia coli EF1α : Elongation Factor 1 α
EGFR : Epidermal Growth Factor Receptor eIF : Eukaryotic translation Initiation Factor EJC : Exon Junction Complex
ELISA : Enzyme-Linked Immunosorbent Assay EMCV : Encephalomyocarditis virus
EMEM : Eagle’s Minimal Essential Medium ESE : Exonic Splicing Enhancer
ESS : Exonic Splicing Silencer EST : Expressed Sequence Tag Fab : Fragment antigen binding Fc : Fragment crystallizable FCS : Fœtal Calf Serum
FDA : Food and Drug Administration FMDV : Foot-and Mouth Disease Virus GFP : Green Fluorescent Protein GS : Glutamine Synthetase
HAMA : Human Anti-Mouse Antibodies HAT : Histone Acetyltransferase
HC : Heavy Chain
HDAC : Histone Déacétylase
HeLa : lignée cellulaire cancéreuse issue d’un prélèvement de métastase effectué sur une patiente atteinte d’un cancer du col de l’utérus et décédée en 1951, Henrietta Lacks.
HEK-293 : Human Embryonic Kidney 293 HIV-1 : Human Immunodeficiency Virus type 1 hnRNP : heterogenous nuclear
RiboNucleoProtein
HRP : Horseradish Peroxidase IgG : Immunoglobuline G
ISE : Intronic Splicing Enhancer ISS : Intronic Splicing Silencer IRES : Internal Ribosome Entry Site kb: kilobase
kDa : kilo Dalton LC : Light Chain
LCR : Locus Control Region Luc : Luciferase
MSX : Méthionine Sulphoximine MTX : Méthotrexate
NMD : Nonsense Mediated Decay NOVA-1 : Neuro-Oncological Ventral Antigen-1
nt : nucleotides NT : Non traduite
ORF : Open Reading Frame PAP : poly(A) polymérase PABP : poly(A) Binding Protein pb : paire de base
PBS : Phosphate Buffered Saline PCR: Polymerase Chain Reaction PDI : Protein Disulfide Isomerase PEI: Polyethylène Imine
Pré-ARNm: ARN pré-messager
PTB : Polypyrimidine Tract Binding protein RE: Reticulum Endoplasmique
RRM : RNA Recognition Motif RT : Reverse Transcription
S/MAR : Scaffold/Matrix-Associated Region SA : Site Accepteur d’épissage
SC35 : Spliceosomal Component 35 scFv : single chain variable Fragment SCID: Severe Combined ImmunoDeficiency SD : Site Donneur d’épissage
SDS : Sodium Dodecyl Sulfate SF1 : Splicing Factor 1
snRNP : small nuclear Ribonucleoprotein Particle
SR : Serine arginine Rich
SRP: Signal Recognition Particle STAR: STabilising and Anti-Repressor SV40: Simian Virus 40
TGE : Transient Gene Expression TMB : Tetramethylbenzidine TNFα : Tumor Necrosis Factor α
U2AF : U2 small nuclear ribonucleoprotein Auxiliary Factor
UCOE : Ubiquitous Chromatin Opening Element
UTR : Untranslated Region
I
ERE
PARTIE
L’EPISSAGE : MECANISMES ET
REGULATION
I – L’épissage : mécanismes et régulation
A) Introduction : transcription et maturation des ARN pré-messagers :
La transcription est l’étape au cours de laquelle divers types d’ARN, parmi lesquels les ARN messagers, sont synthétisés à partir d’une matrice d’ADN. La liaison de facteurs de transcription au niveau de certaines régions du promoteur (boîte TATA) permet le recrutement de la machinerie d’initiation incluant les enzymes qui vont permettre la synthèse du brin d’ARN, les polymérases. L’élongation de l’ARN se poursuit jusqu’à ce que la polymérase ne rencontre un site de terminaison (signal de polyadénylation AATAAA chez les eucaryotes) et ne se détache de l’ARN. Celui-ci peut ensuite être exporté dans le cytoplasme afin d’être traduit.
Dans le noyau des cellules eucaryotes, la synthèse des ARN messagers est assurée par la polymérase II (Pol II). Les ARN pré-messagers (pré-ARNm) subissent plusieurs modifications co-transcriptionnelles permettant leur maturation, après laquelle on parle d’ARN messager (ARNm), avant l’étape de transport vers le cytoplasme. Ces modifications mettent en jeu un grand nombre de protéines qui vont réaliser l’ajout d’une coiffe à l’extrémité 5’ des ARNm, l’épissage des introns, le clivage à l’extrémité 3’ et la synthèse d’une queue de polyadénosines (queue poly[A]).
La coiffe est constituée d’un résidu guanosine méthylé en position N7, relié au premier nucléotide trouvé en 5’ du pré-ARNm, via une liaison 5’-5’ triphosphate. Cette coiffe est indispensable à plusieurs titres. En effet, il a été montré qu’elle augmente la demi-vie des ARN messagers en les protégeant des exonucléases qui agissent dans la direction 5’-3’. Elle facilite l’excision des introns grâce au complexe CBP20-CBP80 (Cap Binding Protein 20 et 80) qui se lie à sa structure, permet le recrutement du complexe d’épissage et participe au transport des ARN matures vers le cytoplasme. Enfin, la coiffe interagit, via le groupement méthyle du résidu guanosine, avec le facteur eIF4E (eukaryotic translation Initiation Factor-4E), appartenant au complexe eIF4F impliqué dans le recrutement des ribosomes lors des phases précoces d’initiation de la traduction (Proudfoot et al., 2002).
Certaines séquences encadrent le site de clivage composé des deux nucléotides CA (hexanucléotide AAUAAA 10 à 30 nucléotides en amont et motif riche en GU 20 à 40 nucléotides en aval) et permettent la fixation de facteurs qui vont couper l’ARN alors que la PAP (Poly(A) Polymérase) ajoute ensuite des résidus adénosines (environ 250) en 3’ du produit 5’ de clivage (Proudfoot et al., 2002). Cette queue poly(A) protège les ARN messagers de la dégradation en 3’ par des nucléases et participe elle aussi à leur transport vers le cytoplasme. Elle intervient également lors de la traduction car la PABP (poly(A) Binding Protein) se fixe à cette queue et interagit avec le complexe d’initiation de la traduction, via une protéine d’échafaudage eIF4G, permettant la circularisation de l’ARNm en cours de traduction.
La maturation des ARN pré-messagers nécessite enfin une étape dite d’épissage qui permet l’élimination de séquences non-codantes entrelacées entre les régions codantes (ou exons). Ces séquences non-codantes appelées introns (pour INTervening RegiON, terme inventé en 1978 par Walter Gilbert ; Tonegawa et al., 1978) ont été mises en évidence pour la première fois en 1976, dans l’ARN messager de l’adénovirus de type 2 par Phillip Allen Sharp et Richard J. Roberts, ce qui leur a valu le prix Nobel de physiologie ou médecine en 1993 (Berget et al., 1977 ; Chow et al., 1977).
Le mécanisme d’épissage, qui nous intéresse plus particulièrement dans le cadre de ce projet de thèse, va maintenant être présenté plus en détails.
B) L’épissage, mécanisme et régulation :
1) Les sites d’épissage :
Chez les mammifères, les ARN pré-messagers issus de la transcription sont classiquement composés de plusieurs exons, dont la longueur est relativement courte, qui sont interrompus par des introns de taille beaucoup plus importante, en moyenne 10 à 100 fois plus long. Afin de générer des ARN matures prêts à être traduits, ces exons doivent être identifiés et assemblés les uns à la suite des autres de façon précise et efficace. Ce mécanisme constitue une étape clé dans le contrôle de l’expression génique car elle détermine la nature de la phase codante du transcrit mature (Black, 2003).
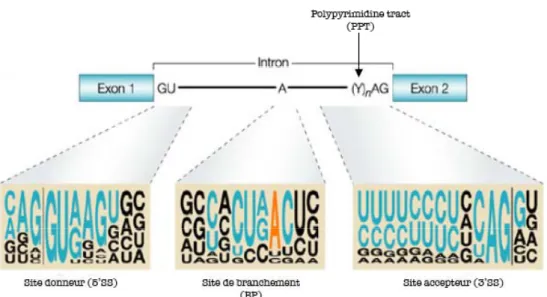
L’épissage d’un pré-ARNm est dirigé par des éléments de séquence en cis indispensables au recrutement de la machinerie d’épissage (cf. figure 1).
Le dinucléotide GU est situé à la jonction exon-intron, et constitue la borne 5’ de l’intron, on parle de site d’épissage en 5’ (site donneur d’épissage ou 5’SS, 5’ Splice Site). Le site 3’ d’épissage (site accepteur ou 3’SS, 3’ Splice Site), situé à la borne 3’ de l’intron, comporte trois éléments de séquence : le site de branchement (A), suivi d’une séquence riche en pyrimidines (Yn) de composition et de longueur variable (entre 10 et 40 nucléotides) et du
dinucléotide terminal AG.
Le site donneur est trouvé le plus souvent plusieurs centaines voire milliers de bases en amont du site de branchement et le site accepteur est situé immédiatement en aval de ce site et de la région riche en pyrimidines. Ces sites sont très conservés entre les mammifères mais ils ne suffisent toutefois pas à la reconnaissance, par la machinerie d’épissage, des bornes d’un intron.
En effet, il est clair que les bases situées dans l’environnement direct de ces sites jouent également un rôle important (Stephens & Schneider, 1992). C’est pourquoi des séquences dites « consensus » ont été définies grâce à l’alignement de nombreuses séquences introniques eucaryotes ou plus particulièrement humaines. De façon simplifiée ces séquences consensus pourraient être représentées ainsi :
- site donneur AG | GURAGU - site de branchement YNCURAY - site accepteur YAG | RNNN
avec les séquences introniques représentées en italique, les nucléotides invariables soulignés, les autres bases étant celles retrouvées le plus fréquemment aux diverses positions des sites d’épissage (R = purine, Y = pyrimidine, N = nucléotide aléatoire) (Parent & Bisaillon, 2006).
Toutefois, cette représentation ne rend pas compte de la fréquence de chaque nucléotide aux différentes positions. C’est pourquoi on peut trouver un autre type de représentation de ces séquences consensus, illustré dans la figure 1, et sur laquelle la taille des nucléotides est directement proportionnelle à leur fréquence, calculée par alignement de séquences d’ARN pré-messagers.
Figure 1: Les séquences consensus des sites d’épissage, adapté de Cartegni et al., 2002.
Pour chacun des sites, la taille d’un nucléotide à une position donnée est proportionnelle à la fréquence de ce nucléotide obtenue lors de l’alignement des séquences conservées de 1683 introns humains. Les nucléotides appartenant au consensus classique sont en bleu, excepté la base A du site de branchement qui est représentée en
orange. Les lignes verticales représentent les frontières exon-intron.
Différents algorithmes, aujourd’hui disponibles sur Internet, ont été créés par plusieurs groupes, en se basant sur ces séquences consensus, et permettent d’identifier les potentiels sites d’épissage dans une séquence donnée (Houdayer & Stoppa-Lyonnet, 2005). Parmi eux on peut citer « Splice Site Prediction », « MaxEntScan », « GeneSplicer » et « Splice Site Finder » (Reese et al., 1997; Pertea et al., 2001; Yeo & Burge, 2004).
Il faut noter ici qu’il existe des variants à la forme standard des introns décrits ci-dessus et dits « GT-AG » qui sont très largement majoritaires. Le variant majeur correspond aux introns GC-AG et représente 0,5% des sites donneurs annotés (Burset et al., 2001). Ceux-ci sont reconnus par les mêmes facteurs d’épissage que les introns classiques (cf. paragraphe suivant). Il existe également une autre classe d’introns, rare, et caractérisée par les dinucléotides terminaux AT-AC (Jackson, 1991). Ces introns sont épissés par un complexe dit « mineur » ou « U-12 spliceosome » qui contient des snRNP (small nuclear Ribonucleoprotein Particles) spécifiques peu abondantes, comme U11 et U12.
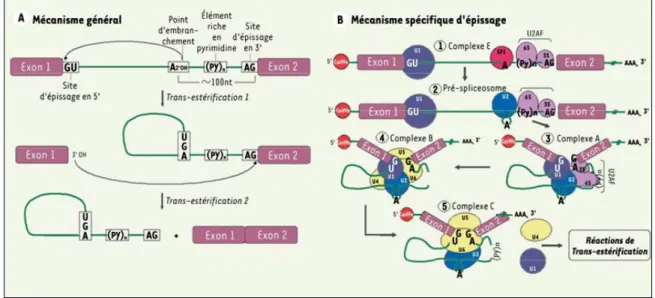
2) Mécanisme général de l’épissage :
L’épissage des introns est un processus qui se produit en deux étapes de trans-estérification (cf figure 2A). La liaison 3’-5’ phosphodiester du résidu guanosine présent au niveau du site donneur subit une attaque nucléophile par le résidu adénosine hautement conservé trouvé au site de branchement. Il y a alors formation d’une interaction covalente entre ces deux nucléotides permettant à l’intron d’adopter la forme d’un lasso. L’extrémité 3’ libre de l’exon est soumise à la seconde trans-estérification par l’attaque nucléophile de la liaison 3’-5’ phosphodiester du résidu guanosine présent au niveau du site accepteur. Il s’ensuit le détachement de l’intron et la liaison entre les deux exons adjacents (Black, 2003).
Les deux étapes de trans-estérification sont réalisées suite à la fixation sur les séquences en cis d’un complexe catalytique macromoléculaire nommé « spliceosome ». Parmi les facteurs impliqués, on trouve cinq complexes de ribonucléoprotéines nucléaires (snRNP) et un grand nombre d’autres facteurs, jusqu’à plusieurs centaines, dont la protéine SF1 (Splicing Factor 1) et le dimère U2AF (U2small nuclear ribonucleoprotein Auxiliary Factor) constitué de deux sous-unités nommées U2AF-65 et -35.
Chacune des snRNP est constituée d’un petit ARN nucléaire, dont la séquence est complémentaire d’une séquence particulière des ARN pré-messagers, associé à plusieurs protéines pour former les complexes snRNP U1, U2, U4-U6 et U5. Par exemple, la séquence 5’-CAG/GUAAGU-3’ est complémentaire aux neuf nucléotides situés à l’extrémité 5’ de l’ARN de la snRNP U1. Les sites donneurs d’épissage sont constitués de nucléotides plus ou moins identiques à cette séquence. La vaste majorité d’entre eux varient sur deux ou trois positions, les positions les plus conservées étant le dinucléotide GU et la guanosine en position +5 (par rapport à la jonction exon/intron) présents dans 85% des séquences analysées (Shapiro & Senapathy, 1987). Certaines études montrent qu’un nombre minimal de bases complémentaires est nécessaire pour la reconnaissance par U1, les bases -1 et +5 étant très importantes, en plus du dinucléotide GU (Carmel et al., 2004).
Figure 2: Mécanismes de l’épissage, d’après Parent & Bisaillon, 2006.
La formation du spliceosome sous forme active doit préalablement se réaliser pour permettre l’excision intronique. Pour cela un assemblage en cinq étapes est nécessaire. Il débute par la formation du complexe E (pour Early) où il y a appariement de U1 au site donneur, de SF1 au résidu adénosine du site de branchement, de U2AF-65 à la zone de polypyrimidines et de U2AF-35 au site accepteur (cf. figure 2B). Par la suite, le pré-spliceosome est formé par la délocalisation de SF1 par U2 au site de branchement en laissant le résidu adénosine disponible pour la première attaque nucléophile. Le complexe A apparaît grâce à la réorganisation du pré-spliceosome via des interactions entre U1, U2 et U2AF contribuant ainsi au rapprochement physique des exons. L’arrivée du tri-snRNP U5/U4-U6 au niveau des séquences consensus entraîne la formation du complexe B et permet de libérer le site de branchement et le site accepteur. Finalement, le complexe catalytique, ou complexe C, est formé par l’association de U6 et U2 au niveau du site donneur déclenchant le départ de U1 et U4. Ce complexe catalytique réalise enfin les réactions de trans-estérification permettant la ligation exonique.
3) Régulation de l’épissage :
Le spliceosome a donc une double fonction : reconnaître et sélectionner les sites d’épissage mais aussi catalyser la ligation des exons. L’efficacité avec laquelle il va agir est déterminée par une balance entre différents critères comme la force des sites d’épissage (c'est-à-dire leur conformité par rapport aux séquences consensus), la taille des exons et la présence de séquences auxiliaires.
En effet, la reconnaissance des sites d’épissage est nécessaire mais pas suffisante pour déterminer l’assemblage du spliceosome et permettre l’épissage qui en découle. D’autres séquences en cis et facteurs protéiques en trans sont indispensables pour conduire à la définition de l’exon (exon definition, Berget, 1995) et activer l’utilisation de ces sites par la machinerie d’épissage.
Ces séquences auxiliaires présentent toutes des caractéristiques communes : elles sont courtes (moins de 10 nucléotides), variables en terme de séquence, individuellement faibles et souvent présentes en plusieurs copies. La plupart d’entre elles sont conservées entre les espèces mais contiennent des motifs dégénérés ce qui les rend difficiles à identifier. Elles peuvent être exoniques ou introniques et quand elles sont dans les introns, elles peuvent se situer en amont, en aval ou de chaque côté de l’exon qui est régulé. Enfin, on peut les classer selon qu’elles sont activatrices ou inhibitrices (Ladd & Cooper, 2002).
a) Les séquences exoniques et les facteurs associés :
La grande majorité des exons contient des séquences régulatrices de l’épissage, qu’elles soient activatrices (ESE pour Exonic Splicing Enhancer) ou inhibitrices (ESS pour Exonic Splicing Silencer) (Cartegni et al., 2002; Black, 2003).
- Les ESE et les protéines SR :
Les ESE sont des éléments qui sont très variés en termes de séquence et qui constituent des sites de fixation spécifiques de protéines régulatrices de l’épissage, parmi lesquelles les protéines SR (Serine/arginine Rich).
Ces protéines sont des facteurs d’épissage essentiels et multifonctionnels, requis à différentes étapes de l’assemblage du spliceosome (Graveley, 2000). Elles forment la famille de protéines régulatrices de l’épissage la plus étudiée et bien que chacune ait une fonction spécifique, elles présentent toutes une structure commune. Elles sont ainsi caractérisées par la présence d’un ou deux motifs reconnaissant l’ARN (RRM pour RNA Recognition Motifs) et d’un domaine carboxy-terminal de longueur variable riche en dipeptide sérine/arginine, le domaine RS. Les motifs RRM permettent la fixation spécifique à certaines séquences des ARN pré-messagers alors que le domaine RS est impliqué dans les interactions protéine-protéine. Ce domaine peut ainsi faciliter la définition de l’exon en recrutant directement ou
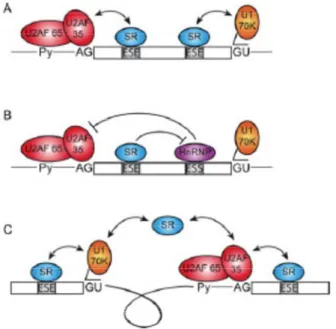
Cette famille regroupe entre autres les protéines SRp20, SRp30c, SRp40, SRp55, SRp70, ASF/SF2 (Alternative Splicing Factor/Splicing Factor 2) et SC35 (Spliceosomal Component 35). Ces facteurs peuvent agir sur la sélection des sites d’épissage selon différents modèles (cf. figure 3) (Graveley, 2000).
Ainsi, les protéines SR fixées aux ESE pourraient recruter et stabiliser les interactions entre la snRNP U1 et le site donneur d’épissage et entre U2AF-35 et le site accepteur au cours du processus de définition de l’exon (cf. figure 3A). Le domaine RS stabilise également les interactions entre le snRNA du complexe U2 et le site de branchement (Shen & Green, 2006). Un autre mode d’action consiste à antagoniser l’action d’un facteur inhibiteur fixé sur une autre séquence régulatrice (ESS, cf. paragraphe suivant) qui lui-même inhibe l’interaction entre U2AF et le site accepteur (cf. figure 3B). Les protéines SR peuvent aussi former un réseau d’interactions protéine-protéine visant à juxtaposer les sites donneurs et accepteurs durant l’assemblage du spliceosome comme cela a été montré pour ASF/SF2 et SC35 qui sont capables d’interagir avec U1 et U2AF-35 (cf. figure 3C) (Wu & Maniatis, 1993).
Figure 3: Représentation schématique des modes d’action des protéines SR, d’après Long & Caceres, 2009.
Les protéines SR pourraient aussi faciliter le recrutement du tri-snRNP U5/U4-U6 au pre-spliceosome (Long & Caceres, 2009). Notons que les protéines SR sont également impliquées dans l’épissage des introns de type U12 en facilitant l’interaction entre U11 et le site donneur ainsi qu’entre U12 et le site de branchement (Hastings & Krainer, 2001).
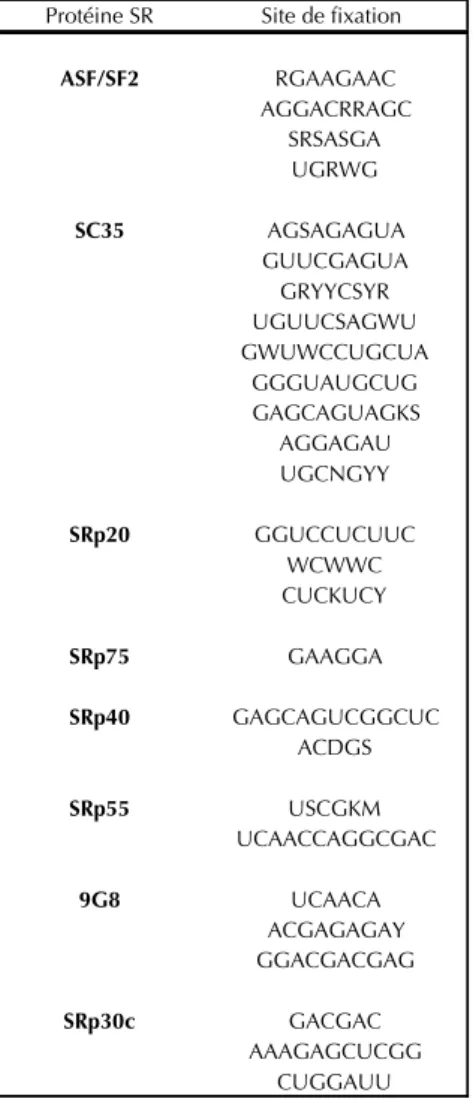
Il n’existe pas de séquence consensus commune à tous les ESE. Toutefois, différentes approches ont permis de proposer plusieurs motifs susceptibles de fixer les différentes protéines de la famille SR et d’autres protéines régulatrices (cf. tableau 1) (Cartegni et al., 2002; Matlin et al., 2005; Long & Caceres, 2009). La compilation de ces différentes données a permis à différents outils de recherche d’ESE de voir le jour comme « ESE finder » et « RESCUE-ESE » (Fairbrother et al., 2002; Cartegni et al., 2003).
Protéine SR Site de fixation
ASF/SF2 RGAAGAAC AGGACRRAGC SRSASGA UGRWG SC35 AGSAGAGUA GUUCGAGUA GRYYCSYR UGUUCSAGWU GWUWCCUGCUA GGGUAUGCUG GAGCAGUAGKS AGGAGAU UGCNGYY SRp20 GGUCCUCUUC WCWWC CUCKUCY SRp75 GAAGGA SRp40 GAGCAGUCGGCUC ACDGS SRp55 USCGKM UCAACCAGGCGAC 9G8 UCAACA ACGAGAGAY GGACGACGAG SRp30c GACGAC AAAGAGCUCGG CUGGAUU
Tableau 1: Exemples de séquences d’ARN identifiées comme sites de fixation de certaines protéines SR; adapté de Long & Caceres, 2009.
N = n’importe quel nucléotide ; R = purine ; Y = pyrimidine ; D = A, G ou U ; K = U ou G ; M = A ou C ; S = G ou C et W = A ou U.
- Les ESS et les hnRNP :
Les éléments exoniques répresseurs de l’épissage, les ESS, sont moins bien caractérisés que les ESE mais on peut penser qu’ils sont aussi fréquents. Ils interagissent avec des protéines régulant négativement l’épissage, appartenant le plus souvent à la famille des hnRNP (heterogenous nuclear RiboNucleoProtein) (Black, 2003). Comme les protéines SR, les hnRNP sont constituées d’un ou plusieurs domaines capables de se lier aux ARN immatures et un domaine auxiliaire impliqué dans des interactions protéine-protéine (Krecic & Swanson, 1999). Ces protéines très abondantes sont distribuées dans tout le noyau, à l’inverse des protéines SR qui, elles, co-localisent préférentiellement avec différents autres facteurs d’épissage dans des structures particulières du noyau, les nuclear speckles (Lamond & Spector, 2003).
Les hnRNP peuvent fonctionner en compétition directe avec les protéines SR sur certains éléments ESE et ESS qui se chevauchent comme c’est le cas entre hnRNPA1 et SC35 lors de l’épissage du messager tat de HIV-1. La fixation de hnRNPA1 sur un ESS particulier prévient la fixation de SC35 sur un ESE chevauchant et empêche donc le recrutement de U2AF sur un site accepteur faible proche (House & Lynch, 2008).
Les hnRNP peuvent aussi directement réprimer la fixation de U1 ou U2 sur un exon en se fixant sur un ESS ou un ISS (Intronic Splicing Silencer, cf. paragraphe suivant) situé proche de l’exon, causant ainsi un encombrement stérique rendant impossible l’accès des composants du spliceosome aux sites d’épissage environnants.
L’un des membres les plus étudié de la famille, hnRNPA1 est capable de réprimer l’épissage de différentes façons en fonction des transcrits :
- en bloquant des étapes initiales d’assemblage du spliceosome grâce à la fixation de plusieurs molécules tout au long de l’ARN au niveau de séquences spécifiques ou non (processus appelé « multimérisation coopérative »)
- en antagonisant l’effet de certaines séquences régulatrices environnantes
- en créant une boucle dans le pré-ARNm au niveau d’un exon (par fixation de plusieurs molécules qui vont dimériser) le rendant ainsi indisponible pour la machinerie d’épissage (Black, 2003).
La protéine hnRNPI ou PTB (Polypyrimidine Tract Binding protein) appartient à la famille des hnRNP mais elle a la capacité de se fixer directement à la région riche en pyrimidine des introns. Elle entre ainsi en compétition avec U2AF, exerçant donc un effet inhibiteur sur l’épissage (Caceres & Kornblihtt, 2002).
Notons, par ailleurs, que certains membres des hnRNP, tout comme certaines protéines SR, peuvent rester fixés aux ARN après épissage. En plus de leur rôle lors de l’épissage elles auraient donc également un rôle sur le transport, la localisation, la traduction des ARN matures ou encore lors du processus de NMD (Nonsense Mediated Decay) (Krecic & Swanson, 1999; Long & Caceres, 2009).
b) Les séquences introniques de régulation :
D’autres séquences régulatrices de l’épissage sont présentes dans les introns. Les sites de fixation pour les protéines impliquées dans cette régulation sont souvent situés dans la région riche en pyrimidines, dans les zones adjacentes au site de branchement ou au site donneur. Toutefois ces éléments peuvent également agir à distance alors qu’ils se trouvent à des centaines de nucléotides de l’exon qu’ils régulent. Ce sont les ISE (Intronic Splicing Enhancers) et les ISS (Intronic Splicing Silencers). Comme leurs homologues exoniques, ces éléments sont souvent regroupés dans certaines zones où les deux types de séquences sont proches et influent l’une sur l’autre (Black, 2003).
Plusieurs éléments sont connus pour agir en tant qu’ISE mais les protéines qui les reconnaissent sont moins bien caractérisées que pour les ESE. Cependant, il est connu que certains sont dépendants des protéines SR et d’autres sont reconnus par des membres de la famille CELF (CUGBP and ETR-Like Factors), une autre famille de protéines ayant des rôles multiples dans le métabolisme des ARN (Ladd et al., 2001; Barreau et al., 2006). Les séquences et les positions de ces ISE sont, une fois encore, assez variables mais certains motifs ont pu être fréquemment observés. Ainsi, certains sites donneurs sont fortement activés lorsqu’une séquence riche en uridine est située immédiatement en aval. Un autre élément commun est l’hexanucléotide UGCAUG qui a la propriété d’activer fortement l’épissage, en particulier lorsqu’elle est répétée (Black, 2003).
Il a également été montré que certaines protéines portant un domaine RS, comme les protéines SR, et donc appelées protéines « SR-like », sont capables de se lier à ces ISS, et entraînent l’inhibition de l’épissage en antagonisant l’action de certaines protéines SR (Cowper et al., 2001). A l’opposé, certaines hnRNP peuvent fonctionner comme des activateurs de l’épissage (Smith & Valcarcel, 2000).
Qu’elles soient exoniques ou introniques toutes ces séquences présentent des partenaires et des modes d’action proches voire identiques. La régulation de l’épissage résulte d’une compétition entre les phénomènes activateurs et répresseurs dont les acteurs sont nombreux et non encore tous identifiés. L’épissage est donc influencé à la fois par la force des sites situés aux jonctions exon-intron et par l’ensemble des effets positifs et négatifs des différents éléments situés dans les exons et les introns environnants. La complexité et la subtilité de cette modulation prend tout son sens quand on aborde la notion d’épissage alternatif.
C) L’épissage alternatif :
1) Mécanisme et caractéristiques :
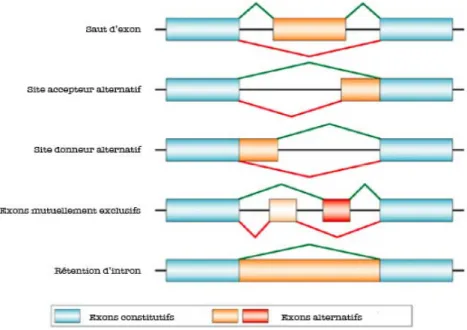
L’épissage alternatif est un processus naturel au cours duquel plusieurs ARN messagers différents peuvent être générés à partir d’un même pré-ARNm en utilisant diverses combinaisons d’exons, générant ainsi des isoformes protéiques variées. Ceci peut se faire par le biais de différents mécanismes (cf. figure 4).
Certains exons sont dits « constitutifs », cela signifie qu’ils sont toujours inclus dans les ARN messagers matures. D’autres exons sont « régulés » et sont donc parfois inclus dans leur totalité et parfois éliminés (cas des exons mutuellement exclusifs). Un autre mécanisme permet d’allonger ou de raccourcir certains exons, ou d’inclure une partie ou la totalité d’un intron grâce à l’utilisation de sites donneurs et/ou accepteurs d’épissage distincts des sites constitutifs, ce sont des sites d’épissage alternatifs.
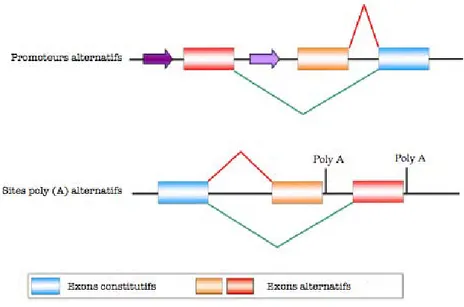
Enfin deux autres mécanismes, l’un dépendant de l’utilisation de promoteurs alternatifs et l’autre de sites de polyadénylation alternatifs, peuvent aussi conduire à l’utilisation de sites alternatifs d’épissage (cf. figure 5).
Figure 5: Autres types de mécanismes d’épissage alternatif.
On estime que pour les trois quarts des gènes de mammifères il existe des possibilités alternatives, parfois nombreuses d’épissage de chaque transcrit primaire. Cela peut expliquer en partie l’écart entre le nombre de gènes (entre 31000 et 39000) et celui des protéines qui est environ dix fois supérieur (Johnson et al., 2003). L’épissage alternatif est donc un niveau essentiel de la régulation qualitative de l’expression des gènes puisqu’il permet à une même séquence d’ADN de produire des protéines différentes selon le tissu ou les conditions d’environnement. Il est considéré comme étant la source la plus importante de la diversité protéique chez les vertébrés (Graveley, 2001).
Certaines caractéristiques propres aux exons alternativement épissés ont pu être mises en évidence lors d’études comparatives avec les exons constitutifs dans le cas particulier de
La première est que les sites d’épissage bordant ces exons alternatifs sont moins forts que les sites d’épissage constitutifs (c'est-à-dire plus éloignés de la séquence consensus) et donc plus sensibles à la régulation (Garg & Green, 2007). Une conséquence directe est que le nombre de séquences régulatrices est plus important dans les exons alternatifs (Itoh et al., 2004).
Il semble également que les exons alternatifs soient souvent entourés de régions introniques plus longues et beaucoup plus conservées que les exons constitutifs, ce qui a été utilisé pour la prédiction des évènements d’épissage alternatif (Sorek & Ast, 2003). Ces régions seraient impliquées dans la régulation de l’épissage, soit parce qu’elles comportent de multiples séquences régulatrices, qu’elles sont des régions structurées de l’ARN ou parce qu’elles sont le siège d’interaction avec la machinerie transcriptionnelle. Nous verrons plus loin que tous ces éléments sont susceptibles de réguler l’épissage alternatif.
Les exons alternativement épissés sont également plus courts, présentent un degré de conservation plus important entre les exons orthologues et leur longueur est plus souvent un multiple de trois (conservation du cadre de lecture) que celle des exons constitutifs (Kim et al., 2008). Les introns pouvant être inclus dans l’ARNm lors du processus de rétention d’intron sont, quant à eux, significativement plus courts que les autres classes d’introns (Zheng et al., 2005) et possèdent eux aussi des sites d’épissage plus faibles. Ce phénomène est retrouvé sur les ARNm de gènes dont les introns sont petits et qui comprennent une densité d’ESS et de l’ISE particulier « GGG » plus faible (Kim et al., 2008).
La prédiction des évènements d’épissage alternatif, basée sur l’identification et la localisation des sites d’épissages et des séquences régulatrices potentielles, reste un challenge important et différentes approches utilisant à la fois les données expérimentales et des outils informatiques ont été développées (Holste & Ohler, 2008).
Les diverses méthodes utilisées pour la détection des variants d’épissage chez différents eucaryotes (analyse des banques d’EST, d’ARNm, séquences génomiques, microarrays…) ont contribué à alimenter plusieurs bases de données sur l’épissage comme « SpliceNest », « ProSplicer », « Splice DB », « Altextron » ou « Alternative Splice Site Predictor » (Burset et al., 2001; Johnson et al., 2003; Gupta et al., 2004; Philipps et al., 2004; Wang & Marin, 2006). Celles-ci peuvent être utilisées pour prédire la présence de sites alternatifs d’épissage.
2) Régulation de l’épissage alternatif :
Il existe assez peu d’exemples pour lesquels les mécanismes d’épissage alternatif ont été précisément élucidés. Le choix de sites alternatifs d’épissage est effectué lors des étapes précoces d’assemblage du spliceosome.
La séquence même de ces sites a une influence sur leur utilisation par la machinerie d’épissage. Ainsi, les sites alternatifs peuvent avoir une séquence relativement différente des sites consensus et la variabilité dans la séquence du site donneur peut avoir, par exemple, une influence importante sur les mécanismes de régulation de l’épissage (Sorek et al., 2004).
De même, la « force » de la région riche en pyrimidines, qui peut être plus ou moins longue et parsemée de purines, peut modifier la sélection du site 3’ d’épissage en favorisant l’utilisation d’un autre site de branchement (Coolidge et al., 1997). Enfin les facteurs ASF/SF2 et hnRNPA1 peuvent être impliqués dans le choix entre deux sites donneurs de même force, un proximal et un distal, interagissant avec un même site accepteur (Caceres et al., 1994) ou bien encore dans le choix de sites accepteurs alternatifs en compétition pour interagir avec un site donneur commun (Bai et al., 1999). Cette observation s’étend également à d’autres facteurs.
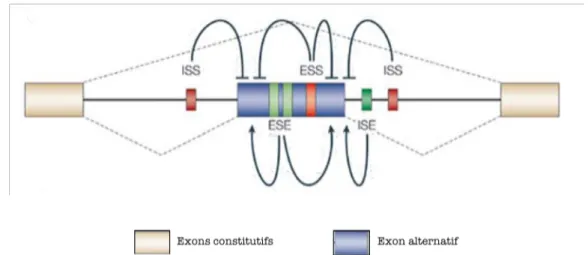
La capacité de la machinerie d’épissage à reconnaître ces sites alternatifs en compétition avec les sites constitutifs dépend de la force des sites en question mais la balance peut être déséquilibrée par la fixation différentielle de divers facteurs protéiques sur les séquences régulatrices évoquées ci-avant (ESE, ESS, ISE, ISS) (cf figure 6).
Figure 6: Représentation schématique de l’influence des séquences régulatrices en cis sur l’épissage alternatif dans le cas du saut d’exon adapté de Matlin et al., 2005. ESE = Exonic Splicing Enhancer; ESS = Exonic Splicing Silencer; ISE = Intronic Splicing Enhancer; ISS = Intronic Splicing Silencer. Les séquences
Par exemple, les protéines SR sont impliquées dans les interactions entre les exons dans les ARN constitutivement épissés comme dans ceux alternativement épissés (Graveley et al., 1999; Graveley, 2000) et préviennent le saut d’exon en se fixant aux ESE. Des études menées sur les séquences des exons constitutivement ou alternativement épissés ont permis de montrer que les ESE et les facteurs régulateurs impliqués dans les deux mécanismes d’épissage sont globalement les mêmes.
Il semblerait toutefois que certains motifs ESE soient retrouvés plus spécifiquement dans certains types d’exons. C’est le cas, par exemple, de la séquence activatrice CACC qui a pu être mise en évidence dans des exons constitutifs mais pas dans les exons alternatifs. A l’inverse, la séquence GAAGAA semble être plus spécifique des exons alternatifs (Kechris et al., 2008).
Une autre différence apparaît quand on étudie plus particulièrement les différents modes d’épissage alternatif (saut d’exon, sites alternatifs, rétention d’intron…) et les séquences régulatrices associées. Différents hexamères régulateurs sont sur- ou sous-représentés selon les cas, indiquant que les différents modes d’épissage alternatif auraient des préférences pour certains ESE ou ESS (Zheng et al., 2005).
L’organisation des séquences régulatrices à l’intérieur des séquences des ARN pré-messagers, les ratios relatifs entre les différentes protéines de régulation et la compétition qui s’exerce entre elles déterminent quels sites seront utilisés (Goldstrohm et al., 2001; Caceres & Kornblihtt, 2002).
En plus des protéines ubiquitaires impliquées généralement dans le processus d’épissage alternatif, un certain nombre de protéines spécifiques de certains tissus ont pu être mises en évidence. C’est le cas, chez les mammifères du facteur NOVA-1 (Neuro-Oncological Ventral Antigen-1) qui régule l’épissage alternatif dans les neurones (Dredge et al., 2001). Le ratio entre certains facteurs activateurs (protéines SR comme ASF/SF2) et répresseurs (hnRNP) peut varier en fonction des tissus ou au cours des différentes étapes du développement. Cela influence fortement l’épissage alternatif et dicte l’expression de certaines isoformes protéiques particulières dans ces différentes conditions (Caceres et al., 1994; Kamma et al., 1995; Smith & Valcarcel, 2000; Yeo et al., 2004).
D’autres paramètres peuvent également apporter un degré supplémentaire à la complexité de cette régulation.
C’est le cas, par exemple, de la structure secondaire de l’ARN qui peut aussi avoir un rôle sur l’épissage des ARN pré-messagers, en « séquestrant » certains éléments de séquence ou, au contraire, en favorisant leur rapprochement (Buratti & Baralle, 2004).
En outre, certaines modifications des différents facteurs régulateurs, comme la phosphorylation peuvent jouer un rôle dans la régulation de l’épissage alternatif. En effet, il a été montré que la localisation sub-cellulaire des protéines SR ainsi que leur activité sont régulées par leur phosphorylation. Le domaine RS est hautement phosphorylé sur les résidus sérine et les cycles de phosphorylation/déphosphorylation sont requis pour l’épissage des ARN pré-messagers et influencent le choix des sites d’épissage (Graveley, 2000; Smith & Valcarcel, 2000; Long & Caceres, 2009). De plus, l’état de phosphorylation peut lui-même être modulé en fonction de l’environnement cellulaire et de divers stimuli extérieurs (Stamm, 2002).
Enfin, l’épissage alternatif est aussi régulé par un processus impliquant la machinerie de transcription. L’épissage des ARN pré-messagers est un processus co-transcriptionnel, tout comme les autres modifications évoquées au début de cette section, et les différents acteurs de ces machineries interagissent de plusieurs manières. Ainsi, le domaine CTD (C-Terminal Domain) de la RNA PolII peut recruter des facteurs d’épissage, faciliter l’assemblage du spliceosome en rapprochant les exons consécutifs et influe sur la distribution nucléaire des différents composants des machineries de transcription et d’épissage (Maniatis & Reed, 2002). D’autres éléments comme la structure du promoteur, la vitesse d’élongation et certains activateurs transcriptionnels ont également un rôle dans la régulation de l’épissage alternatif (Kornblihtt et al., 2004).
3) Épissage alternatif et pathologies :
Comme évoqué précédemment, l’épissage alternatif est un moyen naturel de générer des isoformes protéiques variées qui peuvent avoir des rôles spécifiques nécessaires dans certains tissus, au cours de certaines étapes développement ou en fonction de certaines conditions physiologiques. Toutefois, certaines mutations peuvent entraîner des évènements aberrants d’épissage alternatif conduisant à plusieurs pathologies.
Le fait que les sites donneurs et accepteurs d’épissage se réduisent à quelques nucléotides et leur nature dégénérée impliquent que, statistiquement, on peut retrouver de tels sites assez fréquemment dans les séquences des ARN pré-messagers.
Nous avons vu que la force de ces sites, c'est-à-dire leur identité par rapport aux séquences consensus, et de nombreuses autres séquences régulatrices influent sur leur reconnaissance et leur utilisation par la machinerie d’épissage, permettant ainsi la régulation de l’épissage constitutif et de l’épissage alternatif.
Toutefois, certains sites, qui ne sont ni des sites constitutifs ni des sites alternatifs d’épissage, ont des séquences proches de ces derniers et sont donc intrinsèquement fonctionnels, ce sont des sites dits « cryptiques ». Nous verrons plus tard dans ce manuscrit que l’existence de ces sites cryptiques d’épissage a eu un impact important sur mon travail de thèse.
Dans des conditions « classiques », ces sites ne sont pas reconnus ni utilisés par les différents facteurs d’épissage, parce que leur force est moindre comparée aux sites constitutifs et qu’ils sont potentiellement inhibés par différentes séquences régulatrices proches (Domenjoud et al., 1991). Toutefois, lorsque les sites authentiques subissent une mutation, certains sites cryptiques qui sont dans l’environnement proche du site muté, peuvent alors accéder à la compétence et être utilisés en lieu et place du site naturel.
Cela a été mis en évidence, par exemple, dans un cas où la mutation d’un site donneur d’épissage de l’intron 1 du gène humain de la β–globine, mutation associée à un phénotype de thalassémie, active l’utilisation de trois sites cryptiques situés en amont et en aval du site naturel (Treisman et al., 1983). Certains de ces sites cryptiques subissent parfois eux-mêmes une mutation qui, en augmentant la complémentarité entre l’ARN de la snRNP U1 et ce site, peut favoriser l’utilisation du site muté au détriment du site naturel environnant (Nelson & Green, 1990).
Enfin, certaines mutations peuvent interférer avec la fixation de protéines SR perturbant ainsi l’épissage, comme cela a pu être observé sur l’ARNm codant pour le gène humain smn2 (survival of motor neuron 2). Dans cet exemple, une mutation silencieuse située dans un ESE affecte le site de fixation de la protéine ASF/SF2 et conduit à l’exclusion d’un exon (Cartegni & Krainer, 2002). Certaines mutations, jusqu’alors considérées comme de simples polymorphismes car n’affectant pas la séquence codante, peuvent donc avoir un retentissement profond sur la structure des protéines par le biais d’altérations de l’épissage.
Des données indiquent que 70% à 88% des événements d’épissage alternatif changeraient la nature de la protéine, et seulement 19% conduiraient à la production de protéines tronquées (Modrek & Lee, 2002). Cela résulte en grande partie du fait que l’épissage alternatif est souvent associé à l’insertion de codons stop prématurés (situés en amont de la position occupée par le codon stop habituellement utilisé), ce qui peut être à l’origine de la production de protéines tronquées, mais surtout du mécanisme de NMD.
Ce mécanisme est en effet très souvent observé si le codon stop est situé à plus de 50-55 nucléotides en amont de la dernière jonction exon-exon. Il conduit à diminuer considérablement la stabilité des transcrits correspondants, donnant ainsi rarement lieu à la synthèse d’une protéine abondante (Hentze & Kulozik, 1999). Ce mécanisme est interprété comme une surveillance physiologique destinée à prévenir la production de protéines tronquées potentiellement délétères produites suite à un épissage alternatif « aberrant » lié à une mutation. (N.B. : dans des conditions « normales » le couplage épissage alternatif / NMD peut aussi être considéré comme un moyen naturel de réguler l’expression d’une protéine.)
En revanche, quand l’épissage alternatif induit un changement dans la séquence polypeptidique, les propriétés des protéines comme la fixation du ligand, l’activité enzymatique ou la localisation peuvent être affectées. Cela implique que l’épissage alternatif, en contrepartie de son rôle physiologique créateur de diversité protéique, peut aussi produire des transcrits aberrants aux conséquences pathologiques très variées (Faustino & Cooper, 2003). C’est pourquoi diverses approches impliquant des modifications des voies d’épissage alternatif ont été envisagées dans le cadre de thérapies contre certaines maladies génétiques (Garcia-Blanco et al., 2004; Corcos & Solier, 2005; Jeanteur & Tazi, 2005).
D) Couplage entre l’épissage et les autres étapes de la vie d’un ARN messager :
Au cours des différentes étapes de l’expression génique, l’ARN pré-messager est transcrit, puis il subit différentes étapes de maturation avant que l’ARN mature puisse être exporté vers le cytoplasme pour être traduit. Chacune de ces étapes est réalisée par différentes machineries à multiples composants mais il est maintenant clair que celles-ci sont couplées de manière physique et fonctionnelle et ce, de façon réciproque (Proudfoot et al., 2002).
Par exemple, le domaine CTD de la RNA PolII est capable d’interagir avec des facteurs impliqués dans le coiffage, l’épissage et la polyadénylation des pré-messagers (Maniatis & Reed, 2002). Ces trois étapes de maturation sont aussi couplées entre elles à différents niveaux, ce qui permet d’en augmenter la vitesse, l’efficacité et la fidélité. Ces mécanismes sont aussi couplés aux étapes suivant la maturation, à savoir l’export des ARN dans le cytoplasme et la traduction.
L’épissage semble, en particulier, impliqué dans l’export par l’intermédiaire des complexes EJC (Exon Junction Complex). Ces complexes consistent en un assemblage de facteurs protéiques qui sont déposés par le spliceosome, de manière indépendante de la séquence, entre 20 et 24 nucléotides en amont des jonctions exons/exons formées après épissage (Le Hir et al., 2000). Ce complexe contient notamment la protéine UAP56, un facteur du spliceosome, capable de recruter le complexe (Aly/Tap/p15) qui peut s’associer aux pores nucléaires lors du passage vers le cytoplasme (Reed, 2003).
Les complexes EJC sont donc « une plateforme » sur laquelle viennent se fixer différents facteurs, dont certains sont impliqués dans l’export des ARN alors que d’autres jouent un rôle dans le processus du NMD ou encore sur la localisation cytoplasmique des ARN (Le Hir et al., 2001). Le dépôt de ces complexes EJC n’est pas indispensable à l’export des ARN mais leur confère un avantage sélectif pour cela (Luo & Reed, 1999; Valencia et al., 2008).
Certaines études suggèrent en outre que les composants de la machinerie d’export peuvent être recrutés sur l’ARN grâce à un couplage avec les machineries de transcription ou de polyadénylation.
Une dernière possibilité est la présence, sur la molécule d’ARNm, d’éléments de séquence spécifiques capables de recruter certains facteurs impliqués dans l’export (Reed & Hurt, 2002). Cela permet d’expliquer que certains messagers viraux ou cellulaires, ne contenant pas d’introns, sont exportés correctement.
De plus, l’épissage influe également sur la traduction. En effet, il a été montré que les ARN épissés sont plus efficacement traduits (Nott et al., 2003). Ceci est corrélé au fait qu’on retrouve un plus grand nombre de ces ARN couplés aux polysomes, et les complexes EJC sont, une fois encore, impliqués dans ce phénomène (Nott et al., 2004).
II
ÈME
PARTIE
II – Les anticorps : généralités
A) Structure, assemblage et sécrétion des anticorps: (Janeway et al., 2003)
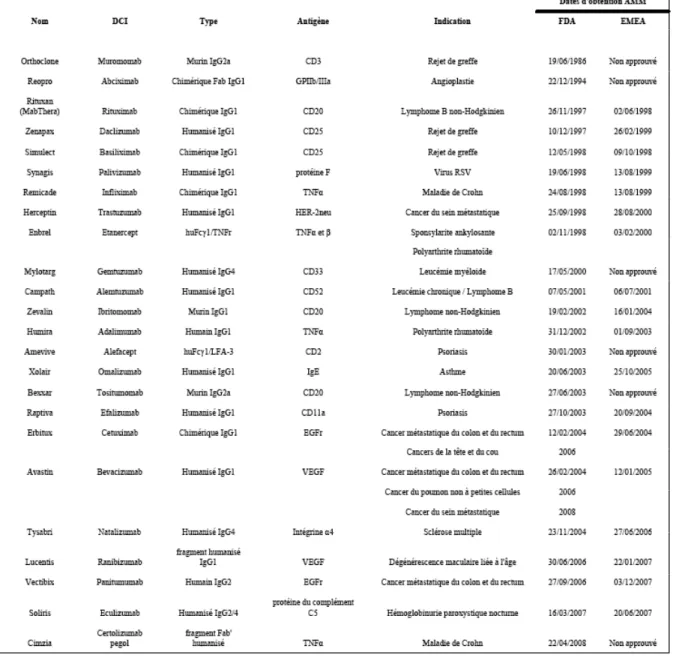
Chez l’homme on distingue cinq classes d’immunoglobulines ou Ig : les Ig A, D, E, G et M. C’est la structure de la molécule d’immunoglobuline de classe G (IgG), d’un poids moléculaire de 150 kDa, qui a été le mieux étudiée (Baty & Chames, 2006). Précisons également que c’est la classe d’immunoglobulines qui nous intéresse plus particulièrement car tous les anticorps recombinants thérapeutiques utilisés à l’heure actuelle en font partie et c’est pourquoi la majeure partie des paragraphes suivants y sont consacrés.
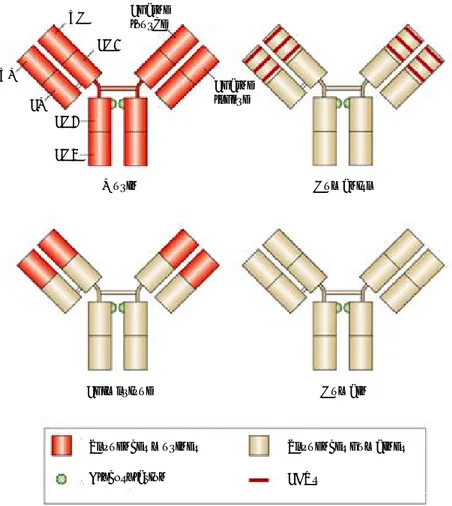
Les IgG constituent les anticorps « conventionnels » protecteurs circulant dans le sérum. Ils se répartissent en quatre sous-classes IgG1, IgG2, IgG3 et IgG4 comportant respectivement des chaînes lourdes γ1, γ2, γ3 et γ4 et dont les taux sériques sont différents (Jefferis, 2009). Les différences structurales entre ces sous-classes incluent le nombre et la position des ponts disulfure dans la région hinge (zone charnière entre les domaines VH-CH1 et CH2-CH3, cf. figure 7) et la position des ponts disulfure qui relient les chaînes lourdes et légères.
Il est important de noter que chacune de ces sous-classes possède des fonctions effectrices qui lui sont propres. Quelle que soit la sous-classe d’IgG, les chaînes légères sont de type kappa ou de type lambda (Cκ à 60% ou Cλ à 40% chez l’homme). La reconnaissance de l’antigène se fait par une interaction directe entre le site de combinaison de l’anticorps et une très petite portion de l’antigène natif que l’on appelle épitope.
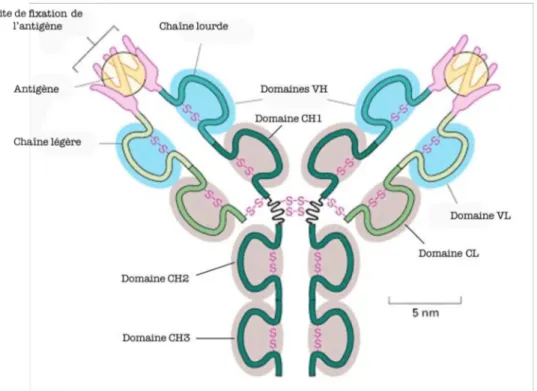
Les IgG sont les immunoglobulines majoritaires (75 à 80% des anticorps circulants) et sont constituées de deux chaînes lourdes identiques (HC pour Heavy Chain) d’environ 50 kDa et de deux chaînes légères identiques (LC pour Light Chain) d’environ 25 kDa. Chacune des chaînes est organisée en domaines d’environ 110 acides aminés, deux pour les chaînes légères (appelés VL et CL) et quatre pour les chaînes lourdes (VH, CH1, CH2 et CH3) (cf. figure 7).
Figure 7: Représentation schématique de la structure d’un anticorps; source inconnue.
Les parties variables sont représentées sur fond bleu, les parties constantes sur fond brun. VH=partie variable de la chaîne lourde; VL=partie variable de la chaîne légère; CH=partie constante de la chaîne lourde; CL=partie
constante de la chaîne légère.
Les domaines VL et VH sont appelés domaines variables, ce sont ceux qui lient l’antigène. Chaque domaine variable possède trois boucles hypervariables appelées CDR (Complementarity Determining Region) qui se caractérisent par une très grande variabilité en acides aminés d’un anticorps à l’autre (5 à 10 acides aminés par CDR). Ces régions constituant le site de combinaison antigénique représentent 5% de la partie variable et sont adjacentes à des zones présentant moins de variabilité et constituant l’armature (charpente ou framework = FR) du segment variable. Les domaines CL, CH1, CH2 et CH3 sont appelés domaines constants.
Chacune des chaînes lourde et légère consiste donc en une série de domaines alignés en tandem qui se replient de façon indépendante l’une de l’autre en deux feuillets β stabilisés par un pont disulfure intra-chaîne constituant une structure sphérique dont la partie interne est hydrophobe. La chaîne légère est reliée à la chaîne lourde par un pont disulfure localisé aux extrémités C-terminales des domaines CL et CH1. Les chaînes lourdes sont reliées entre elles par deux ponts disulfure localisés au niveau des acides aminés 226 et 229 situés entre les domaines CH1 et CH2 dans la région charnière (appelée hinge).
Cette zone, qui est plus ou moins longue selon le sous-type d’IgG, est riche en résidus proline et cystéine formant une région flexible dans les deux plans de l’espace.
La structure quaternaire mature d’un anticorps (HC2LC2) est formée par l’assemblage
de deux chaînes lourdes identiques attachées à deux chaînes légères identiques par un pont disulfure inter-chaine. Cet assemblage a lieu lors du transit dans la lumière du réticulum endoplasmique (RE) et est suivi du passage de l’anticorps natif à travers l’appareil de Golgi pour aboutir ensuite à l’obtention de sa structure tridimensionnelle finale et sa sécrétion dans le milieu.
On peut cliver une IgG par la papaïne et obtenir trois fragments d’environ 50kDa, dont deux sont identiques et comportent chacun un site de liaison pour l’antigène ; ce sont les fragments Fab pour « antigen binding » ; le troisième fragment possédant les fonctions dites effectrices comme la fixation au complément ou aux récepteurs Fc ; c’est le fragment Fc pour « cristallisable ». La pepsine coupe, quant à elle, la molécule d’IgG à la base des régions charnières, permettant ainsi d’obtenir le fragment F(ab’)2, le fragment Fc étant dégradé (cf.
figure 8).
Figure 8: Formats d’anticorps utilisés pour les applications thérapeutiques, d’après Peterson et al., 2006.
Chaque ovale représente un domaine particulier de l’immunoglobuline. Les ponts disulfures sont représentés en rouge et le polypeptide de liaison du scFVest représenté en bleu. F(ab’)2 = dimeric antigen binding fragment,