ÉTUDE DE L’ÉPISSAGE ALTERNATIF DES UGT2B
Thèse
Vincent Ménard
Doctorat en pharmacie
Philosophiae Doctor (Ph.D.)
Québec, Canada
© VINCENT MÉNARD, 2013
iii
RÉSUMÉ
Les UDP-glucuronosyltransférases (UGT) sont des enzymes impliquées dans le métabolisme de plusieurs molécules endogènes et exogènes, entraînant leur inactivation et leur élimination subséquente via la bile et l‘urine. Étant donné l‘impact potentiel de l‘épissage alternatif sur la variabilité de la glucuronidation chez l‘humain, nous avons étudié ce processus chez deux membres de la famille des UGT2B, soit UGT2B4 et UGT2B7. Dans le cas d‘UGT2B4 nous avons détecté 3 nouveaux exons et 11 nouveaux transcrits principalement exprimés au foie. Les nouvelles protéines dérivées de ces ARNm n‘ont pas d‘activité de conjugaison, mais trois d‘entre elles ayant une extrémité C-terminale alternative (i2, i3 et i5) modulent la capacité de glucuronidation de l‘enzyme active UGT2B4_i1. Dans le cas d‘UGT2B7 nous avons découvert 6 nouveaux exons retrouvés dans 22 transcrits pleines-longueurs codant pour 7 protéines UGT2B7. Tous les ARNm détectés l‘ont été au niveau du rein, mais nous avons observé une grande spécificité tissulaire dans la transcription: les ARNm contenant l‘exon 1 classique contrôlés par un promoteur proximal sont exprimés dans les tissus du tractus gastro-intestinal et le foie, alors que les transcrits caractérisés par la présence d‘exons 1 alternatifs et distaux sont exprimés en périphérie. De plus, un contrôle de la spécificité d‘expression des promoteurs UGT2B7 semble exister au rein, le promoteur associé à l‘enzyme active étant absent dans les tissus foetaux et néoplasiques. Les 6 protéines UGT2B7 alternatives sont enzymatiquement inactives, mais deux d‘entre elles ayant une extrémité C-terminale tronquée (UGT2B7_i2 et i4) modulent négativement la glucuronidation par l‘enzyme i1. Cette régulation a été observée en surexpression mais également lors d‘expérimentations de répression des isoformes endogènes d‘i2 et i4 par interférence à l'ARN entraînant une augmentation de la conjugaison. Des expériences de co-immunoprécipitation et d‘immunofluorescence supportent que cette répression survienne via l'interaction directe des protéines i2 et i4 avec i1 dans la membrane du réticulum, formant des complexes inactifs. Nos résultats démontrent que des évènements d'épissage alternatif des gènes UGT2B humains influenceraient la voie de glucuronidation, ce qui pourrait constituer un nouveau mécanisme d'autorégulation et influencer le métabolisme de substances endogènes et la réponse à des traitements pharmacologiques.
v
ABSTRACT
UDP-glucuronosyltransferases (UGT) are enzymes involved in the metabolism of several endogenous and exogenous molecules in the human body via inactivation and further elimination through bile and urine. Because of the potential impact of alternative splicing on the variability of the glucuronidation pathway, we studied this process in two UGT2B family members, UGT2B4 and
2B7. For UGT2B4, we detected 3 novel exons and 11 transcripts mainly expressed in the liver. The
novel proteins derived from those mRNAs are enzymatically inactive, but three of them bearing alternative C-terminal ends (UGT2B4_i2, i3 and i5) have the ability to modulate glucuronidation efficiencies of the UGT2B4_i1 enzyme. For UGT2B7 we discovered 6 novel exons in 22 full-length transcripts coding for seven UGT2B7 proteins. All mRNAs were detected at least in the kidney, but a complex tissue-specificity of the transcription of UGT2B7 was observed: mRNAs containing the classical exon 1 are mainly expressed in gastrointestinal tract and liver, whereas transcripts characterized by the presence of alternative exon 1s (derived from the action of the distal promoter 1a) can be found in peripheral tissues. Fine-tuning of the specificity of UGT2B7 expression seems to exist at least in the kidney, this organ having a modulated UGT2B7 expression profile depending if the tissue is embryonic, adult or neoplastic. All six novel UGT2B7 proteins are enzymatically inactive, but two of them with truncated C-terminal ends (UGT2B7_i2 and i4) modulate the glucuronidation activity of the i1 enzyme. This regulation has been observed in overexpressing cellular models, but also in RNA interference experiments where repression of i2 and i4 increased glucuronidation by UGT2B7. Co-immuoprecipitation as well as immunofluorescence experiments support that this repression is related to the interaction of i2 and i4 with the i1 enzyme in the ER membrane, this process forming inactive complexes. The results presented here show that alternative splicing in UGT2B genes in human influences the glucuronidation process, a feature that could constitute an auto-regulation mechanism and could modify the susceptibility to adverse effect and response related to the treatment of several pathologies. Also, this process could influence elimination capacity of endogenous substances like steroid hormones.
vii
TABLE DES MATIÈRES
RÉSUMÉ ... iii
ABSTRACT ... v
TABLE DES MATIÈRES ... vii
LISTE DES TABLEAUX ... xi
LISTE DES FIGURES ... xiii
LISTE DES ABRÉVIATIONS ... xv
REMERCIEMENTS ... xxiii
AVANT-PROPOS ... xxix
CHAPITRE I : INTRODUCTION ... 1
1. La voie de glucuronidation ... 2
1.1 Présentation générale et importance physiologique ... 2
1.2 Structure des UGT ... 3
1.3 La superfamille des UGT ... 5
1.3.1 Nomenclature et classification ... 5
1.3.2 Description des familles d’UDP-glucuronosyltransférases ... 5
1.3.2.1 Les UGT1 ... 5
1.3.2.2 Les UGT2 ... 7
1.3.2.3 Les UGT3 ... 9
1.3.2.4 UGT8A1 ... 9
1.4 Distribution tissulaire des UGT et activité ... 10
1.5 Pathologies associées à la voie de la glucuronidation ... 12
2. Les UGT2B : présentation et analyse de la littérature ... 17
2.1 Structure génomique ... 17
2.2 Structure des enzymes UGT2B ... 17
2.3 Présentation en détail de chacun des membres de la famille UGT2B... 21
2.3.1 UGT2B4 ... 22 2.3.2 UGT2B7 ... 23 2.3.3 UGT2B10 ... 30 2.3.4 UGT2B11 ... 31 2.3.5 UGT2B15 ... 32 2.3.6 UGT2B17 ... 35 2.3.7 UGT2B28 ... 38
3. Mécanismes à l’origine de la variabilité de la glucuronidation ... 39
3.1 Importance physiologique de la variabilité de la glucuronidation ... 40
3.2 Modulation de la transcription et de l’activité ... 42
3.2.1 Modulation de la glucuronidation par des composés endogènes ... 42
3.2.2 Modulation de la glucuronidation par des composés exogènes ... 42
3.3 Les polymorphismes génétiques : importance clinique ... 43
3.4 L’épigénétique ... 44
3.5 Modifications post-transcriptionnelles de l’ARN ... 46
3.5.1 Épissage et épissage alternatif ... 46
3.5.2 Modifications en 5’ du transcrit d’ARN ... 49
viii
3.6 Modifications post-traductionnelles ... 52
3.6.1 Repliement ... 52
3.6.2 Clivage du peptide-signal ... 54
3.6.3 Modifications chimiques des protéines ... 55
3.6.3.1 Glycosylation ... 55
3.6.3.2 Phosphorylation ... 56
3.6.3.3 Autres modifications ... 57
3.7 Interactions protéines-protéines ... 58
4. L’ARN : fonction, édition et contrôle ... 61
4.1 Contrôle de l’expression... 62
4.1.1 Structure de la chromatine ... 62
4.1.2 Modifications chimiques des promoteurs ... 63
4.1.3 Initiation de la transcription ... 64
4.1.4 Transport ... 66
4.1.5 Stabilité ... 66
4.2 Fonction de l’ARN ... 67
4.2.1 Les ARN messagers ... 67
4.2.2 Les ARN de transfert ... 68
4.2.3 Les microARN ... 69
4.2.4 Autres fonctions ... 70
4.3 L’épissage alternatif : mécanisme et découvertes récentes ... 70
4.3.1 Présentation du mécanisme ... 70
4.3.2 Régulation du mécanisme ... 72
4.3.3 Importance physiologique de l’épissage alternatif ... 73
5. Objectifs, hypothèses de recherche, méthodologie et importance des articles dans la démarche scientifique ... 74
5.1 La présence de nouveaux exons au loci UGT2B4 et UGT2B7 pourrait entraîner la production de transcrits alternatifs { l’ARNm classique encodant l’enzyme. ... 77
5.2 L’expression des transcrits alternatifs pourrait être spécifique aux tissus. ... 77
5.3 Les protéines dérivées des produits d’épissage des gènes UGT2B4 et UGT2B7 pourraient avoir un effet modulateur sur l’activité de glucuronidation cellulaire in vitro. ... 78
5.4 Malgré leurs structures différentes, les nouvelles protéines résident en partie dans les mêmes organites que l’enzyme classique. ... 79
5.5 Le rôle biologique des variants UGT2B7 pourrait être de moduler l’activité de glucuronidation cellulaire lorsque co-exprimés avec l’enzyme active. ... 80
5.6 Suite { cette découverte, nous avons alors testé l’hypothèse d’un rôle modulateur des formes variantes d’épissage, et ce dans un contexte d’expression endogène. ... 81
5.7 La répression observée pourrait être causée par l’interaction des isoformes variantes avec l’enzyme active. ... 81
CHAPITRE II : IDENTIFICATION DE NOUVELLES ISOFORMES D’ÉPISSAGE DU
LOCUS UGT2B4 ... 83
CHAPITRE III : IDENTIFICATION DE NOUVELLES ISOFORMES D’ÉPISSAGE DU
LOCUS UGT2B7 ... 119
CHAPITRE IV : QUANTIFICATION DE L’EXPRESSION DES NOUVEAUX
ÉVÈNEMENTS D’ÉPISSAGE D’UGT2B7 ... 151
CHAPITRE V : CARACTÉRISATION DE DEUX NOUVELLES ISOFORMES UGT2B7
MODULATRICES DE L’ACTIVITÉ DE GLUCURONIDATION ... 183
ix
CHAPITRE VI : REVUE DE LA DIVERSITÉ GÉNOMIQUE DES UGT ET DE SON
IMPACT SUR L’ACTIVITÉ DE GLUCURONIDATION ... 219
DISCUSSION ET CONCLUSION ... 265
Discussion ... 266
Conclusion ... 291
BIBLIOGRAPHIE ... 293
ANNEXE I : TABLEAUX DE RÉFÉRENCE ... 369
ANNEXE II : CARACTÉRISATION DES GRANDES DÉLÉTIONS DU LOCUS UGT2B . 387
ANNEXE III : ANALYSE DES HAPLOTYPES DU GÈNE UGT2B7 DANS PLUSIEURS
POPULATIONS... 421
xi
LISTE DES TABLEAUX
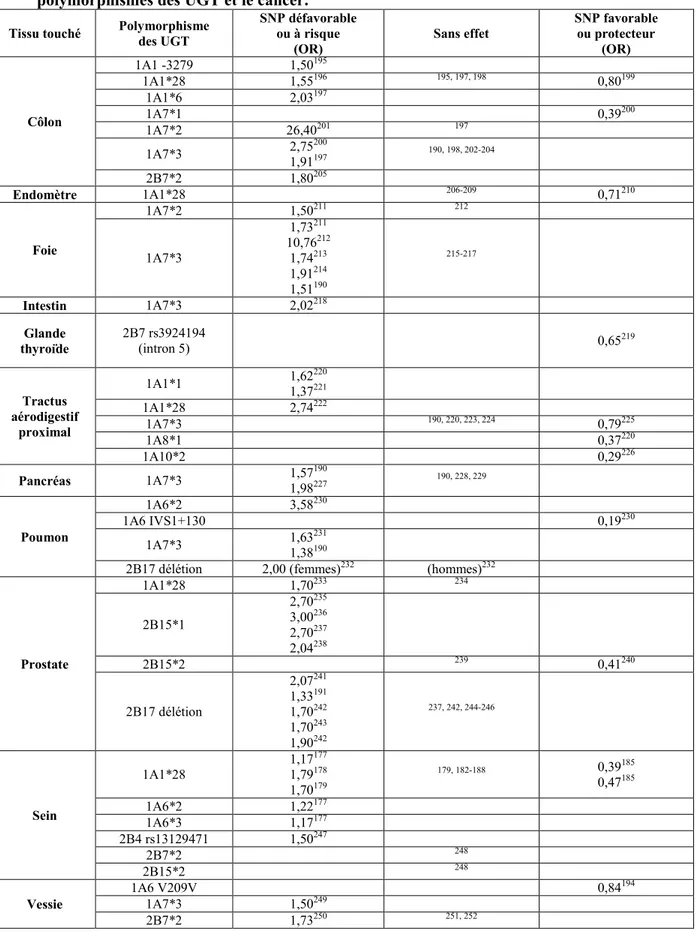
Tableau 1 : Études cas-témoins ayant testé des associations potentielles entre les polymorphismes des UGT et le cancer.
Tableau 2 : Interactions protéine-protéine des UDP-glucuronosyltransférases rapportées dans la littérature et méthode de détection.
Tableau 3 : Caractéristiques spécifiques à chacun des transcrits UGT2B rapportés jusqu‘à présent.
Tableau A1 : Expression des UGT dans le tractus gastro-intestinal, le rein et la vessie. Tableau A2 : Expression des UGT dans le tractus aéro-digestif supérieur.
Tableau A3 : Expression des UGT dans les tissus hormo-sensibles. Tableau A4 : Expression des UGT dans les autres tissus.
Tableau A5 : Substrats endogènes métabolisés par UGT2B7 rapportés dans la littérature et dont les données relatives aux cinétiques enzymatiques ont été décrites.
Tableau A6 : Substrats exogènes métabolisés par UGT2B7 rapportés dans la littérature et dont les données relatives aux cinétiques enzymatiques ont été décrites.
Tableau A7: Substrats d‘UGT2B7 dont les cinétiques n‘ont pas été décrites.
Tableau A8 : Substances inhibitrices de l‘activité médiée par l‘isoforme UGT2B7 surexprimée dans des cellules humaines.
Tableau A9: Analyse de la littérature concernant l‘impact du polymorphisme UGT2B7*2 dans le cadre d‘expériences in vitro et/ou ex vivo.
xiii
LISTE DES FIGURES
Figure 1 : Voie de la glucuronidation
Figure 2: Structure des UGT et représentation graphique de leur localisation transmembranaire au niveau du réticulum endoplasmique.
Figure 3: Arbre phylogénétique de la superfamille des UGT humains. Figure 4: Représentation du locus UGT1.
Figure 5 : Niveaux d‘expression relatifs des UGT dans les tissus humains. Figure 6: Structure du locus UGT2 (4q13) et des gènes le composant. Figure 7 : Cristallographie de la partie C-terminale de UGT2B7. Figure 8 : Motif de dimérisation des UGT2B.
Figure 9 : Importance de UGT2B7 dans le métabolisme de la morphine et ses dérivés. Figure 10 : Distribution de l‘excrétion de testostérone-glucuronide.
Figure 11: Formation des différents produits d‘épissage. Figure 12 : Structure moléculaire de la coiffe en 5‘ des ARN. Figure 13 : Description des types de polyadénylation alternative.
Figure 14 : Fréquence estimée des différentes modifications post-transcriptionnelles du protéome humain.
Figure 15 : Assemblage du complexe de pré-initiation de la transcription. Figure 16 : Évènements d‘épissage alternatif.
Figure 17 : Séquences génomiques des jonctions intron/exon des exons 4 et 5.
Figure 18 : Arbre phylogénétique des UGT1 et des UGT2 incluant leurs variants d‘épissage. Figure 19 : Utilisation d‘anticorps ciblant l‘exon 1 de UGT2B4 et UGT2B7 dans des lignées
cellulaires et dans certains tissus humains.
Figure 20 : Représentation des différents types d‘interférence transcriptionnelle liée à la présence de plusieurs promoteurs au sein d‘un même gène.
xv
LISTE DES ABRÉVIATIONS
3-alpha-diol androstanediol
4-MU 4-methylumbelliferone
4-OHE1 4-hydroxyestrone
aa Acide aminé
ADN Acide désoxyribonucléique
ADNc ADN complémentaire
ADT androstérone
AR Récepteur des androgènes
ARE « AU-rich element » ou élément riche en adénine/uracile
ARN Acide ribonucléique
ARNm ARN messager
ARNr ARN ribosomal
ARNt ARN de transfert
ATP Adénosine triphosphate
AZT Zidovudine ou azidothymidine
BPS « Branch-point sequence »
BSA « Bovine serum albumin » ou albumine de sérum bovin CAR « Constitutive androstane receptor » ou récepteur constitutif de l‘androstane
CBC « cap binding complex » ou complexe de fixation à la coiffe
CDCA
Acide chénodésoxycholique
CDK « cyclin-dependent kinase » ou kinase dépendante de la cycline
CEC « capping enzyme complex » ou complexe enzymatique d‘ajout de la coiffe circRNA « circular RNA » ou ARN circulaire
CNV « Copy number variation » ou variation du nombre de copies géniques
COOH groupement carboxyl
CpG Cytosine – lien phosphate – guanidine
CPSF « Cleavage and Polyadenylation Specificity Factor » ou facteur de spécificité pour le clivage et la polyadénylation
CRD « Coding Region instability Determinant » ou déterminant d‘instabilité des régions codantes
CRD-BP « CRD-Binding Protein » ou protéine se fixant aux CRD CstF « cleavage-stimulating factor » ou facteur stimulant le clivage CTD « carboxy-terminal domain » ou domaine carboxy-terminal
CTLA « Cytotoxic T-Lymphocyte Antigen » ou antigène du lymphocyte T cytotoxique
CYP Cytochrome P450
DCP « decapping complex » ou complexe d‘enlèvement de la coiffe DE-APA « different exons alternative polyadenylation » ou polyadénylation
alternative impliquant différents exons
DHEA Déhydroépiandrostérone
DHT Dihydrotestostérone
DNMT « DNA methyltransferase » ou méthyltransférase de l‘ADN
dL décilitre
EJC « Exon junction complex » ou complexe de jonctions d‘exons
xvi
ESE séquences « exonic splicing enhancers » ESS séquences « exonic splicing silencers »
Ex. Exon
FDA Food and Drug Administration (USA)
FGF « Fibroblastic growth factor » ou facteur de croissance des fibroblastes
Fig. Figure
FRET « Fluorescence resonance energy transfer » ou transfert d'énergie par résonance de fluorescence
FXR « Farnesoid X receptor » ou récepteur farnesoid X
G Groupement glucuronide G6PD Glucose-6-phosphate déshydrogénase GAG Glycosaminoglycans GMP Guanosine mono-phosphate GST Glutathione-S-transférase GTP Guanosine triphosphate
GTPase Enzymes hydrolysant le Guanosine triphosphate
HDCA Acide hyodésoxycholique
HETE acide hydroxyeicosatétraénoïque
HEK « Human embryonic kidney » ou rein humain embryonnaire HNF « Hepatic nuclear factor » ou facteur nucléaire hépatique
hnRNP « heterogeneous nuclear ribonucleoprotein » ou ribonucléoprotéine nucléaire hétérogène
HODE acide hydroxyoctadécadiénoïque
HSP « Heat shock protein » ou protéine de choc thermique
i Isoforme
Il Interleukine
Int. Intron
IRES « Internal Ribosome Entry Sites » ou site d‘entrée interne du ribosome ISE séquences « intronic splicing enhancers »
ISS séquences « intronic splicing silencers »
kDa Kilodalton
Kb Kilobases
Ki Constante d‘inhibition
km Affinité
LCA acide lithocholique
LNCaP « Lymph node carcinoma of the prostate »
M-3-G Morphine-3-glucuronide
M-6-G Morphine-6-glucuronide
MBD « methyl-CpG-binding domain » ou domaine de fixation méthyl-CpG
mg milligramme
miARN microARN
MPA Acide mycophénolique
MRA « Micro-Anchoring Region » ou région d‘arrimage
NAT N-acétyltransférase
NCBI « National Center for Biotechnology Information » (USA)
nm nanomètre
nM nanomolaire
NMD « Nonsense Mediated Decay » ou dégradation des ARN non-sens NNAL 4-(méthylnitrosamino)-1-(3-pyridyl)-1-butanol
xvii
NPC « Nuclear Pore Complexe » ou complexe de pore nucléaireOH Groupement hydroxyl
OR « Odds ratio » ou rapport des chances ou rapport des cotes
PA polyadénylation alternative
PABP « Poly-A Binding Protein » ou protéine se fixant au poly-A
PAP polyadénylate polymérase
PCR « Polymerase chain reaction » ou réaction en chaîne de la polymérase piRNA « Piwi-interacting RNA », ARN interagissant avec les protéines Piwi
PKC protéine kinase C
PMA phorbol 12-myristate 13-acétate
Pol Polymérase
RE Réticulum endoplasmique
RISC « RNA-induced silencing complex », complexe d‘inhibition par les ARN
RNase Ribonucléase
RNP Ribonucloprotéine
ROS « reactive oxygen species » ou dérivé réactif de l‘oxygène
RT-PCR « Reverse-transcription PCR » ou transcription inverse couplée au PCR RXR « Retinoid X receptor » ou récepteur rétinoïde X
SAHA acide suberoylanilide hydroxamique
SAM S-adénosylméthionine
SE-APA « same exon alternative polyadenylation » ou polyadénylation alternative impliquant un seul exon
shRNA « short hairspin RNA », petit ARN en épingle
SLDE « Stem-Loop Destabilizing Element » ou élément déstabilisateur d‘épingle
SN-38 irinotécan
snARN « Small nuclear RNA », petit ARN nucléaire
snRNP « Small nuclear ribonucleoprotein » ou petite ribonucléoprotéine nucléaire snoARN « Small nucleolar RNA » ou petit ARN nucléolaire
SNP « Single nucleotide polymorphisme » ou polymorphisme ponctuel SR « Serine/arginine rich » ou élément riche en sérine/arginine SRF « serum response factor » ou facteur de réponse au sérum
SRP « signal recognition particle » ou particule de reconnaissance du signal SWI/SNF « Switch/Sucrose nonfermentable »
TAF « TBP-Associated Factor » ou facteur associé à TBP
TBHQ t-butylhydroquinone
TBP « TATA-Binding Protein » ou protéine de fixation à la boîte TATA TCPOBOP 1,4-bis[2-(3,5-dichloropyridyloxy)] benzène
TF « Transcription factor » ou facteur de transcription
TK Tyrosine kinase
TTNPB 4-(E-2-[5,6,7,8-tétrahydro-5,5,8,8-tétraméthyl-2-naphthalenyl]-1-propényl) benzoic acid
U2AF « U2 auxiliary factor » ou facteur auxiliaire à U2 UDP-GlcA Uridine diphosphate acide glucuronique
UGT
UDP-glucuronosyltransférasesUDP Uridine diphosphate
USF « Upstream stimulatory factor » ou facteur de stimulation en amont UTR « Untranslated region » ou région non-traduite
v Variant
xix
"…, il n'est pas de vraie création sans secret." Albert Camus, Le mythe de Sisyphexxi
À mon grand-papa Louis et à mon fils Éliotxxiii
REMERCIEMENTS
Même si j‘ai adoré les années passées dans le laboratoire du Dr Guillemette, je dois tout d‘abord avouer que le travail de laboratoire et les activités liées aux études supérieures trainent avec eux des périodes de stress et de découragement (comme n‘importe quel travail ou occupation, en fait…). Je veux remercier les gens qui m‘ont appuyé, qui m‘ont changé les idées, qui m‘ont appris, qui m‘ont fait réaliser des choses que je n‘avais pas vues, qui m‘ont poussé à me dépasser, qui ont cru en moi et qui m‘ont accepté dans leur vie, malgré mes défauts et mes absences. Sans vous tous, rien de ce qu‘humblement je présente ici n‘aurait été réalisable, et je vous remercie d‘être là, avec moi. Dans les prochains paragraphes je serai un peu plus spécifique, mais je tenais tout d‘abord à souligner le fait que je me trouve chanceux d‘être aussi bien entouré…
Il y a plus de sept ans maintenant, ma directrice de recherche Dr Chantal Guillemette m‘a fait l‘immense cadeau de m‘accepter au sein de son équipe de recherche. J‘étais alors loin de me douter à quel point ce passage allait changer ma vie… Chantal, le temps passé à te côtoyer et tes conseils m‘ont fait grandir, m‘ont permis de me dépasser et m‘ont aidé à accomplir des choses que je ne me croyais pas capable de faire. Pour moi, tu es un exemple de succès (tant au niveau professionnel que familial) et de dévouement (à tes recherches et à tes étudiants). Je considère être une bien meilleure personne grâce à toi, et ta présence dans ma vie m‘a également permis de rencontrer des gens incroyables… En plus de tout ce que tu as fait pour moi, je crois sincèrement que je ne pourrai jamais te remercier assez de m‘avoir engagé au printemps 2006!
En plus de Chantal, je considère devoir mon intérêt pour la recherche scientifique à trois personnes, soit les Dr Éric Lévesque, Hugo Girard et Julie Fradette. Éric, ta passion contagieuse pour la science a été pour moi une source de motivation inépuisable durant mes études. Tu m‘as toujours fait confiance, et cette attitude m‘a poussé à tout faire pour m‘améliorer. Pour tout ça et bien d‘autres choses, travailler à tes côtés restera un de mes meilleurs souvenirs de laboratoire. Hugo, merci de m‘avoir tout montré et merci d‘avoir été un partenaire de discussion (et de squash…) aussi polyvalent! On ne s‘ennuie jamais avec toi… Quant à toi Julie, tu as été la première à me présenter la « vraie » science, celle qui existe lorsqu‘on ferme les livres d‘université et qu‘on ouvre les portes d‘un laboratoire. Tu as réussi à m‘accrocher, à me prouver que j‘étais au bon endroit et à me convaincre que je détenais les aptitudes essentielles pour faire des études aux cycles supérieurs.
xxiv
Merci aussi de m‘avoir fait confiance avec tes échantillons inestimables… Je suis honoré d‘avoir pu contribuer aux premières années de ton laboratoire maintenant florissant.
Durant mes années dans le laboratoire du Dr Guillemette, j‘ai eu la chance de rencontrer des dizaines d‘étudiants et d‘employés de son équipe qui sont tous devenus des amis très proches. Parmi ceux-ci je pense entre autres à Judith, Isabelle L., Isabelle G., Mélanie, Johanie, Joannie, Jelena, Marie-Odile, Jean-Philippe et Sarah-Ann, des gens incroyables que j‘ai pu côtoyer au labo, mais également dans plusieurs autres congrès, party ou évènements… Je vous aime tous! (même si pour JP je ne rappelle plus trop pourquoi…). Je voudrais aussi souhaiter bon succès à la nouvelle troupe française du laboCG (Alan, Anaïs et Guillaume), des gens supers qui accompliront beaucoup lors des prochaines années. Bien sûr, je m‘en voudrais d‘oublier nos petits cousins de laboratoire du Dr Olivier Barbier, qui ont toujours su m‘aider quand j‘en avais le plus besoin (parfois même sans qu‘ils le sachent…) Je pense plus spécifiquement à Laurent (notre français dans la lune préféré), Jocelyn (un grand joueur de golf, dans tous les sens du mot) et Mélanie (que j‘ai peu vue depuis 2006, et que j‘aurais beaucoup de difficulté à reconnaitre dans la rue sans sa bedaine de femme enceinte). Un petit merci particulier à Kim, une maman extraordinaire et une confidente encore plus extraordinaire (c‘est peu dire…) qui est prête à tout pour nous, ses amis. Merci d‘avoir été là quand ça comptait. Enfin, il est impossible pour moi de passer sous silence l‘importance de Lyne dans le cadre de mes sept dernières années… J‘ai eu tellement de plaisir à jaser avec toi et à te faire rire! Tu as toujours été là pour moi, et ta présence au laboratoire a été très réconfortante pour moi lorsque je doutais de mes capacités. Merci d‘avoir mis du soleil dans chacune de mes journées de travail, Lyne!
Dans les dernières années, mon passage au laboratoire m‘a aussi permis de rencontrer deux gars tout simplement incroyables qui ont dû surmonter un nombre incalculable d‘embuches pour parvenir à leurs rêves; Dr Martin Perreault et « futur Dr» Pierre Collin. Martin, sous ton attitude de gros côlon se cache un père affectueux, un scientifique passionné et un joueur de hockey moyen (mais avec beaucoup de cœur). Je n‘aurais jamais pensé rencontrer un gars comme toi dans un laboratoire, mais je me rends maintenant compte que la recherche scientifique serait en bien meilleure posture si plus de ses participants étaient comme toi… Continue à avancer, ton talent te permettra d‘avoir une incroyable carrière. Pierre, ta persévérance, ton courage et ton intelligence font de toi un modèle pour les gens qui, comme moi, l‘ont un peu ‗tout cuit dans le bec‘… Ta force de caractère t‘a permis d‘avoir beaucoup de succès, et tu peux maintenant avoir confiance en ce que
xxv
tu es capable d‘accomplir. Bien sûr, comment ne pas parler de l‘histoire de la clé pétée dans ta porte d‘appartement? On sera très vieux et c‘est certain qu‘on en rira encore…Mon implication au sein de la Faculté de Pharmacie de l‘Université Laval m‘a permis de rencontrer une foule de professeurs et d‘employés soucieux du bien-être et du succès des étudiants. Tout d‘abord, je souhaiterais souligner le travail acharné du Dr Thérèse Di Paolo et sa dévotion envers tous les étudiants. Votre travail n‘est vraiment pas facile, mais vous réussissez toujours à prioriser les étudiants dans chacune de vos décisions. Chacun de nous à la Faculté peut être fier d‘être représenté par une aussi grande dame. J‘ai eu la chance de siéger sur plusieurs comités en compagnie de plusieurs professeurs dévoués dont le Dr Jean Lefebvre, un doyen qui a fait de la réussite des étudiants sa priorité. Je voudrais également noter la disponibilité et la qualité du travail des différentes agentes administratives de la Faculté que j‘ai pu rencontrer, soit principalement Sylvie Lacasse, Jessica Jean, Lysanne Tanguay et Lucie Fréchette.
J‘ai la certitude que les différents résultats présentés dans ce document n‘auraient pu être obtenus sans l‘apport inestimable de Mario et Olivier. Olivier, tu as été un stagiaire et un étudiant à la maîtrise remarquable, et ton apport à la réalisation des expérimentations et à l‘analyse des résultats nous a permis de démarrer le projet sur des bases solides. Il y a un petit peu de ton travail dans chacun des résultats présentés ici, et je tiens à t‘en remercier. Ça aurait été impossible d‘être épaulé par un meilleur « wingman » que toi Oli! Quant à toi Mario, je dois dire que c‘est difficile de trouver les bons mots pour te remercier assez pour tout ce que tu as fait pour moi. En plus de me sortir du trouble à de nombreuses reprises au labo, c‘est grâce toi (et à ta fête de 40 ans) que j‘ai pu rencontrer Marie-Eve, la femme de ma vie et la mère de mon fils. Ton éternel jeunesse et ton attitude m‘ont toujours permis de dédramatiser mes petits problèmes, et nos conversations sans queue ni tête (souvent) ou sérieuses (des fois) resteront pour moi dans les plus beaux souvenirs de mes sept dernières années. Je suis heureux que ta copine Julie et toi soyez dans ma vie, et j‘espère vous y voir longtemps…
J‘étais presque en train d‘oublier Étienne… mais comment oublier Étienne? J‘ai eu la chance de commencer mon doctorat au même moment que toi, et je suis bien content de t‘avoir eu à mes côtés, même si des fois tu me tapes tellement sur les nerfs! En fait, tu me gosses principalement parce que c‘est impossible d‘être aussi bon que toi en sciences, et ça c‘est vraiment dérangeant… Pourquoi fallait-il que je fasse mon doc en même temps qu‘une machine comme toi? J‘avais l‘air de quoi moi, à côté? Sérieusement, merci beaucoup de m‘avoir poussé à être meilleur, et ce juste en
xxvi
étant toi-même. Nicos et toi m‘offrez la chance d‘une vie, celle de m‘associer à des gagnants pour créer NEV/Conseils, quelque chose qui peut nous mener plus loin qu‘on peut même l‘imaginer. Même à Montréal (ou ailleurs…), Christine et toi allez toujours garder une place importante dans ma vie et dans celle de ma petite famille. Merci à vous deux d‘être là!
Je voudrais remercier mes amis de l‘extérieur de laboratoire qui m‘ont toujours attendu (c‘est le cas de le dire) et qui m‘ont toujours soutenu durant mes périodes de non-disponibilité! Je pense plus particulièrement à Alex, un ami comme on en rencontre peu dans le cours d‘une vie, quelqu‘un de tellement important qu‘il est plus de la famille qu‘un ami. Olivier et Marie-Hélène sont aussi de cette catégorie de gens avec qui j‘ai grandi et qui sont devenus une partie essentielle de mon existence. Merci à vous trois de me soutenir quand ça va mal, et de rendre tout meilleur quand ca va bien. Je voudrais aussi souligner l‘importance dans tout ce processus de mes amis du bacc, et un peu plus spécifiquement Mélissa, Marie-Ève, Rachel et Dario. Sans oublier mes partenaires de musique, Louis-Pierre et Rémy, qui m‘ont permis de me changer les idées lors de nos nombreux jams en tant que Sosbambi. D‘ailleurs, je me trouve bien chanceux qu‘on existe encore et je souhaite continuer avec vous autres encore longtemps!
En cette fin d‘une grande étape de ma vie, mes pensées vont beaucoup à ma famille, des gens sur qui j‘ai toujours pu compter et que j‘aime plus que tout. Francine et Jean-François, vous m‘avez fait l‘immense honneur de m‘accepter dans le clan des Gagnon/Patry à bras ouverts, et j‘ai peu de mots pour vous exprimer à quel point je vous en suis reconnaissant. Dès le jour un vous m‘avez fait confiance, et votre attitude envers moi contribue à faire grandir le bonheur que j‘ai de vivre aux côtés de votre fille. Isabelle, ta persévérance et ton attitude fonceuse malgré les embuches font de toi un modèle à suivre. Je suis chanceux d‘avoir une belle-sœur comme toi… Je vous aime les Patry!
Jo et Philippe, vous avez comblé mon cœur de frère lorsque vous nous avez offert, à Marie-Eve et moi, d‘être la marraine et le parrain de votre belle Juliette. J‘ai l‘impression que les liens qui nous unissent sont de plus en plus forts, et je voudrais vous remercier de la belle place que vous nous offrez dans votre vie. Votre réussite et votre bonheur me rendent fier et me poussent à m‘améliorer tant au plan professionnel que familial. Juliette est pour nous une source de joie inépuisable, et je voudrais vous remercier tous les trois d‘être avec nous dans tout ce que nous sommes en train de vivre.
xxvii
Papa et maman, votre confiance tranquille en mes capacités m‘a toujours permis de me dépasser et d‘accomplir des choses que je ne me croyais pas capable de faire. Le fait de vous savoir derrière moi me permet de toujours regarder en avant, ce qui m‘a permis de mener à bien tous mes projets sans m‘interroger sur la suite des choses. Dans les moments les plus difficiles vous avez toujours été encore plus présents pour moi, et pour tout ça il n‘existe pas de mot assez fort pour vous remercier. J‘ai vraiment les meilleurs parents du monde, et chaque jour je réalise la chance que j‘ai de vous avoir dans ma vie. Je vous aime tellement…Le 12 novembre dernier, ma copine Marie-Eve m‘a fait le plus beau cadeau : elle a fait de moi le père de notre beau petit Éliot. Marie, mes remerciements pour toi seront simples : je te remercie d‘être toi. Je te remercie d‘être à mon écoute, de tout faire pour me rendre heureux, de m‘attendre, de m‘appuyer quand j‘ai trop de choses à faire, de me faire découvrir notre nouvelle vie de famille, de créer quelque chose de beau autour de moi, de faire en sorte que je sois une meilleure personne, de me comprendre… Ta présence avec moi me rend plus fort, et ensemble je sais qu‘on peut tout faire. Tu m‘a fait l‘honneur de vouloir devenir la mère de mes enfants, et construire notre vie ensemble me rend heureux. Il y a beaucoup de toi dans tout ce qui est écrit ici, et l‘amour que j‘ai pour toi m‘a permis de trouver un sens à toutes ces choses. Je t‘aime plus que tout.
J‘aurais beaucoup aimé que mon grand-papa Louis puisse lire cette thèse, mais j‘ai en même temps très hâte que mon fils Éliot le fasse.
xxix
AVANT-PROPOS
Cette thèse, intitulée « Étude de l‘épissage alternatif des UGT2B », est présentée à la Faculté des études supérieures et post-doctorales de l‘Université Laval pour l‘obtention du grade de
Philosophiae Doctor et est rédigée sous forme d‘insertion d‘articles. Cette première section sert
principalement à définir ma contribution et celle des différents co-auteurs des articles insérés. Le premier article traite de l‘épissage alternatif du gène UGT2B4, les trois suivants de l‘épissage alternatif du gène UGT2B7 et le dernier porte sur l‘étude de certains haplotypes de ce dernier gène.
Le premier article auquel j‘ai contribué dans le cadre de mon doctorat en tant que deuxième auteur est intitulé « Extensive splicing of transcripts encoding the bile acid-conjugating enzyme UGT2B4 modulates glucuronidation », un article publié dans le journal scientifique
Pharmacogenetics and Genomics (facteur d‘impact=3.485) en mars 2010. L‘auteur principal de cet article est le Dr Éric Lévesque et les autres collaborateurs ont été Isabelle Laverdière, Dr Judith Bellemare, Dr Olivier Barbier, Dr Hugo Girard et l‘auteur sénior, Dre Guillemette. Isabelle Laverdière a contribué aux expériences de PCR en temps réel. La Dr Judith Bellemare a assisté dans les différentes expériences d‘immunofluorescence. Le Dr Olivier Barbier a révisé l‘article. Le Dr Hugo Girard a participé activement à la recherche des différentes banques de données publiques et à l‘élaboration des différentes stratégies d‘amplification par RT-PCR. La Dr Chantal Guillemette a conceptualisé l‘étude, en a assuré la supervision et le financement, a interprété les données et a contribué à l‘écriture et la révision de l‘article. Le Dr Éric Lévesque a quant à lui participé à la conceptualisation de l‘étude, à l‘interprétation des résultats, au choix et à la supervision d‘expériences et à l‘écriture de l‘article. Pour ma part, j‘ai effectué la grande majorité des expériences de laboratoire en plus de participer à l‘interprétation des résultats, de concevoir les figures et de contribuer à l‘écriture de l‘article. Dans l‘ensemble, j‘estime ma contribution à la réalisation des travaux contenus dans ce premier article à 40%.
Le deuxième article intitulé « Transcriptional diversity at the UGT2B7 locus is dictated by extensive pre-mRNA splicing mechanisms that give rise to multiple mRNA splice variants » a été publié en octobre 2011 dans le journal Pharmacogenetics and Genomics (facteur d‘impact= 3.485). Je suis l‘auteur principal de cet article et les co-auteurs sont Olivier Eap, Joannie Roberge, Mario Harvey, Dr Éric Lévesque, et l‘auteur sénior, Dre Guillemette. Le travail d‘Olivier Eap a été de mettre au point des méthodes de qPCR utilisées dans cet article, en plus d‘aider aux différents clonages. Joannie Roberge a quant à elle contribué à des qPCR suite à la diplomation d‘O.E.. Mario
xxx
Harvey a participé à la supervision et l‘interprétation d‘expériences de laboratoire et a contribué à l‘écriture. Les Dr Chantal Guillemette et Éric Lévesque ont conceptualisé et supervisé la mise en place de l‘étude, en plus de participer à la supervision des expériences, à l‘analyse des résultats et à la rédaction de l‘article. Pour ma part, j‘ai contribué à la mise en place des outils d‘analyse (stratégie RT-PCR, clonage des transcrits, transfection de clones) en plus d‘effectuer la plupart des expériences. Avec les résultats obtenus, j‘ai pu participer à l‘écriture de l‘article, en plus de concevoir et réaliser toutes les figures présentes dans l‘article publié. J‘estime à 50% ma contribution à la réalisation des travaux présentés dans cet article scientifique.
Je suis le premier auteur du troisième article de cette thèse qui est intitulé « Expression of UGT2B7 Is Driven by Two Mutually Exclusive Promoters and Alternative Splicing in Human Tissues: Changes from Prenatal Life to Adulthood and in Kidney Cancer » et qui a été accepté en septembre 2013 pour publication dans Pharmacogenetics and Genomics (facteur d‘impact= 3.485). Les autres auteurs de cet article sont le Dr Éric Lévesque, Sylvia Chen, Olivier Eap, la Dr Melanie Joy, la Dr Lena Ekström, le Dr Anders Rane, et l‘auteur sénior, Dre Guillemette. Dans le cadre de ce projet, le Dr Éric Lévesque a contribué à l‘analyse des résultats, l‘interprétation et l‘écriture de l‘article. Sylvia Chen a participé à certaines des expériences de PCR en temps réel tandis qu‘Olivier Eap a contribué, tel que mentionné dans le travail précédent, à la mise en place de stratégies d‘amplification. Dr Melanie Joy a gracieusement fourni les différents échantillons de reins adultes normaux et tumoraux, tandis que les Dr Lena Ekström et Anders Rane ont fait de même pour tous les tissus foetaux; il est également à noter que ces trois derniers collaborateurs ont également participé à la lecture critique de l‘article. En plus de fournir le financement pour ce projet, la Dr Chantal Guillemette a conceptualisé et supervisé l‘étude et a participé à l‘analyse des résultats et à l‘écriture de l‘article. Pour ma part, j‘ai effectué la majorité des expériences de laboratoire décrites dans cet article, ai contribué à l‘analyse des résultats et à la rédaction de l‘article en plus de concevoir et réaliser toutes les figures qui y sont présentées. Je considère que le pourcentage de ma contribution à la réalisation du produit final est de 70%.
Le quatrième article présenté dans cette thèse s‘intitule « Modulation of the UGT2B7 Enzyme Activity by C-terminally Truncated Proteins Derived from Alternative Splicing » et a été accepté en octobre 2013 pour publication dans le journal Drug Metabolism and Disposition (facteur d‘impact=3.891). Je suis premier auteur de cet article. Les co-auteurs sont Pierre Collin, Guillaume Margaillan et l‘auteur sénior, Dre Guillemette. Dans le cadre de ce projet, Guillaume Margaillan a principalement été impliqué dans la validation par PCR en temps réel des ARNm pour les candidats
xxxi
ayant été identifiés par l‘analyse globale de l‘expression génique. Pierre Collin a quant à lui contribué aux expériences de co-immunoprécipitation. Dr Chantal Guillemette a financé, supervisé et conceptualisé l‘étude, a contribué à l‘analyse et à l‘interprétation des résultats et à la rédaction de l‘article. Quant à moi, j‘ai participé à l‘élaboration des outils moléculaires nécessaires, j‘ai effectué la majorité des expériences, j‘ai analysé les résultats, conçu et réalisé les figures et contribué à l‘écriture de l‘article. Je considère que ma participation à la réalisation du produit final se situe à environ 60%.Le cinquième et dernier manuscrit présenté dans cette thèse est un article de revue intitulé « UGT genomic diversity: beyond gene duplication » qui a été publié en février 2010 dans la revue Drug
Metabolism Review (facteur d‘impact=5.538). Je suis le cinquième auteur de cet article qui a été
écrit par Dr Chantal Guillemette, avec l‘aide du Dr Éric Lévesque, de Mario Harvey et du Dr Judith Bellemare. La Dr Guillemette a conceptualisé cet article et a écrit la revue. Dr Éric Lévesque, Mario Harvey et Dr Judith Bellemare ont participé à la lecture critique de l‘article, en plus de contribuer à générer certaines des figures. Dans mon cas, j‘ai produit la majorité des figures du manuscrit. Je considère que le pourcentage de ma contribution à la réalisation du produit final est de 10%.
Le premier manuscrit non discuté dans cette thèse mais présenté en annexe s‘intitule « Copy-number variations (CNVs) of the human sex steroid metabolizing genes UGT2B17 and UGT2B28 and their associations with a UGT2B15 functional polymorphism » publié en septembre 2009 dans le journal Human Mutation (facteur d‘impact=5.213). Je suis le premier auteur de cet article que j‘ai produit avec la précieuse collaboration des co-auteurs Olivier Eap, Mario Harvey, Dr Chantal Guillemette et Dr Éric Lévesque. Olivier Eap a effectué les amplifications par PCR, tandis que Mario Harvey a participé à la supervision d‘expériences et à la lecture critique du manuscrit. Dr Chantal Guillemette a financé, conceptualisé et supervisé l‘étude, a participé à l‘analyse des résultats ainsi qu‘à l‘écriture du manuscrit. Dr Éric Lévesque a contribué à la conceptualisation, à la supervision des travaux, à l‘interprétation des résultats et à l‘écriture de l‘article. Pour ma part, j‘ai réalisé la majeure partie des travaux de laboratoire, j‘ai élaboré la majorité des méthodes expérimentales, réalisé les figures, analysé les résultats et participé à l‘écriture de l‘article. Je considère avoir contribué à 60% du produit final. Cet article est présenté à l‘Annexe II.
Le deuxième article présenté en annexe s‘intitule « Worldwide variation in human drug-metabolism enzyme genes CYP2B6 and UGT2B7: implications for HIV/AIDS treatment » et a
xxxii
été publié en avril 2012 dans le journal Pharmacogenomics (facteur d‘impact=3.857). Je suis le deuxième auteur de cet article, l‘auteure principale de ce manuscrit étant la Dr Jing Li. Les autres collaborateurs ont été dans ce cas-ci Dr Rebekah L. Benish, Dr Richard J. Jurevic, Dr Chantal Guillemette, Dr Mark Stoneking, Dr Peter A. Zimmerman et Dr Rajeev K. Mehlotra. La Dr Li a effectué la majorité des manipulations de laboratoire (consistant principalement dans des amplifications PCR), a compilé les résultats et a participé à la rédaction de l‘article. Dr Benish a participé à la compilation des données. Les Dr Stoneking, Zimmerman, Jurevic et Guillemette ont contribué à la mise en place du projet et à la révision critique de l‘article. Le Dr Mehlotra a conceptualisé, coordonné et supervisé les travaux, a analysé les résultats et a écrit l‘article. Dans mon cas, j‘ai analysé les données génotypiques et j‘ai contribué à la détermination des différentes structures haplotypiques exposées dans l‘article. Je considère que le pourcentage de ma contribution à cet article est de 15%. Ce manuscrit est présenté ici à l‘Annexe III.
Cette thèse comprend une introduction où j‘approfondis les différents thèmes étudiés dans le cadre de mes travaux de doctorat. Suivant cette introduction se retrouve une section décrivant l‘hypothèse de recherche de mes travaux, ainsi que les différents objectifs s‘y rattachant. Cinq articles rédigés en anglais ont été par la suite insérés dans des chapitres distincts, chacun précédés d'un résumé en français. Les articles insérés dans cette thèse ont tous la structure standard d‘une publication scientifique, soit le résumé, l‘introduction, la section « matériel et méthodes », les résultats, la discussion, la bibliographie, les tableaux et les figures. Le corps principal de la thèse se termine avec une section de discussion des résultats obtenus, une conclusion ainsi qu‘une ouverture sur les perspectives rattachées à mes recherches. Il est également à noter qu‘une liste des références citées dans l'introduction et la discussion se retrouve après la conclusion. En annexe ont été ajoutés des tableaux de résumé de la littérature en lien avec l‘introduction ainsi que deux articles supplémentaires auxquels j‘ai contribué mais qui ne sont pas traités dans les sections principales.
Je vous remercie de l‘intérêt que vous portez à ma thèse et vous souhaite bonne lecture.
Vincent Ménard
1. La voie de glucuronidation
1.1 Présentation générale et importance physiologique
La glucuronidation est la plus importante réaction de phase II et est impliquée dans le métabolisme des médicaments ainsi que de plusieurs autres substances endogènes et exogènes : hormones stéroïdiennes, bilirubine, éléments chimiques carcinogènes et autres molécules possiblement toxiques 1. La réaction de glucuronidation consiste en l‘ajout enzymatique d‘un acide glucuronique provenant du co-substrat l‘uridine diphosphate acide glucuronique (UDP-GlcA) sur un aglycone (i.e. un groupement fonctionnalisé) d‘une molécule-cible. Le mécanisme chimique de la glucuronidation consiste en une attaque nucléophile du groupement accepteur du substrat sur le carbone en position C1 de l‘UDP-GlcA, ce qui entraîne la production d‘un UDP et d‘un produit β-D-glucuronide 2. Ce mécanisme est sommairement représenté dans la figure 1. Ce transfert augmente la polarité du substrat, ce qui favorise donc son excrétion par la bile et l‘urine 3, 4. Cet ajout provoque aussi un encombrement stérique diminuant l‘affinité de la plupart des composés glucuronidés pour leur récepteur spécifique 3, 5, 6.
La majorité des molécules glucuronidées sont inactivées par ce processus métabolique, mais il existe quelques exceptions où l‘ajout d‘un acide glucuronique augmente l‘effet pharmacologique : par exemple, citons la morphine-6-glucuronide qui a un pouvoir analgésique 20 fois plus puissant que la morphine non conjuguée, le dérivé conjugué de l‘ézétimibe qui se fixe plus efficacement à la paroi intestinale et inhibe plus fortement l‘absorption du cholestérol que sa contrepartie non-conjuguée ainsi que l‘hydroxytamoxifène glucuronidé en position N qui détient une affinité pour le récepteur aux estrogènes similaire à sa molécule-mère 7, 89. Malgré ces quelques exceptions, la fonction biologique principale de la glucuronidation réside dans l‘inactivation de substrats et la détoxification de l‘organisme de molécules endogènes ou exogènes. Les enzymes de la famille des UDP-glucuronosyltransférases (UGT) catalysent la réaction de glucuronidation 10. Ces protéines sont des enzymes transmembranaires situées au réticulum endoplasmique (RE) et ayant leur site actif dans la lumière de cet organite. Le foie constitue la niche principale de la glucuronidation, où les concentrations du co-substrat l‘UDP-GlcA sont 15 fois plus élevées que dans les tissus extra-hépatiques. Cette réaction a cependant pu être détectée dans la grande majorité des tissus 11-14.
1.2 Structure des UGT
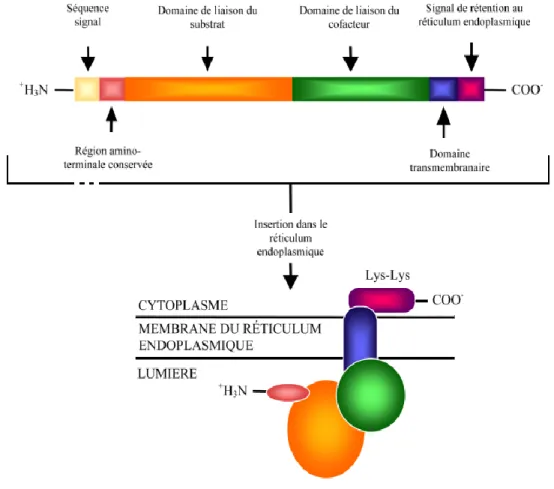
Les UGT sont des enzymes composées de 523 à 541 acides aminés (59 à 62 kDa) 4, 15. Leur région N-terminale a été rapportée comme étant principalement responsable de la reconnaissance du substrat, mais détient également à son extrémité un peptide signal essentiel pour le positionnement au RE. Cette petite région est clivée après l‘insertion et ne fait pas partie de la protéine active 16. Cependant, il a été démontré qu‘au moins UGT1A10 ne possède pas ce peptide signal de positionnement, mais est tout de même retrouvé au niveau du RE dans la cellule, ce qui laisse croire qu‘une autre séquence peptidique interne pourrait jouer le même rôle 17-20. Une courte séquence de 19 à 26 acides aminés fait protubérance du côté cytoplasmique et serait possiblement impliquée dans des interactions avec des protéines cytosoliques 19, 21, 22.
Figure 1 : Voie de la glucuronidation : l‘enzyme UDP-glucuronosyltransférase catalyse la substitution nucléophile de type 2 (SN2) entre le C1 de l‘acide glucuronique (provenant du co-substrat l‘uridine diphosphate acide glucuronique, UDP-GlcA) et un groupement aglycone (-OH, -COOH, -SH, -NH2) de divers substrats lipophiles. Le substrat ainsi glucuronidé est rendu plus hydrophile (polaire) et est plus facilement excrété par l‘urine ou la bile 3, 23.
De plus, la région C-terminale de tous les UGT est responsable de la reconnaissance du co-substrat (UDP-GlcA) et contient un domaine transmembranaire. La présence du domaine transmembranaire en 5‘ divise la protéine en deux parties inégales, la plus grosse partie de la protéine étant dans la lumière du RE (95% de la protéine, 500 premiers acides aminés, contenant le site catalytique) et une plus petite portion située au niveau du cytoplasme (25 derniers acides aminés). Les 17 acides aminés hydrophobes du domaine transmembranaire traversent la bicouche de lipide du réticulum endoplasmique 24, 25, mais il semble que cette région ne soit pas la seule à interagir avec cette membrane : en effet, certains auteurs avancent qu‘une certaine portion de la partie luminale de la protéine serait également enfouie au sein de la bicouche 26-29. La figure 2 représente graphiquement la structure générale des UGT humains.
Figure 2: Structure des UGT et représentation graphique de leur localisation transmembranaire au niveau du réticulum endoplasmique. Il est à noter que la séquence-signal (en jaune) est clivée, donc ne se retrouve pas dans la protéine mature. Le domaine transmembranaire est principalement composé d‘une suite d‘acides aminés hydrophobes (tels la valine et la lysine) et est essentiel à la localisation subcellulaire au niveau du RE. Les UGT décrits jusqu‘à maintenant ont tous cette structure générale 30-32.
1.3 La superfamille des UGT
1.3.1 Nomenclature et classification
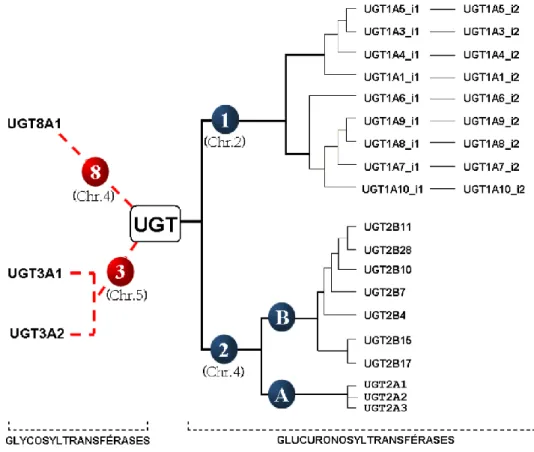
La nomenclature des UDP-glucuronosyltransférases a évolué en fonction de la découverte successive des isoformes ainsi que de la résolution parallèle de l'organisation génomique des loci UGT. Dans la nomenclature actuelle, on regroupe sous le terme « UGT » non seulement les UDP-GlucuronosylTransférases (responsables du transfert d‘un acide glucuronique sur une molécule lipophile), mais aussi tous les membres de la superfamille des UDP-GlycosylTransférases (responsables du transfert d‘un sucre quelconque sur une molécule lipophile). À ce jour, plus de 125 UGT ont été identifiés dans différentes espèces de mammifères, nommées et classifiés sur une base de divergence évolutive 33, 34. Chez l‘homme, on a répertorié 30 protéines UGT distribué dans 4 familles distinctes (UGT1, UGT2, UGT3 et UGT8) et dans 5 sous-familles (UGT1A, UGT2A, UGT2B, UGT3A, UGT8A). La nomenclature actuelle exige que chaque famille partage un minimum de 30% d‘identité de séquence en acides aminés avec les autres familles, alors qu‘à l‘intérieur d‘une même famille ce type d‘homologie doit être supérieur à 50% 33, 34. L‘arbre phylogénétique des différents UGT humains est présenté à la figure 3.
1.3.2 Description des familles d’UDP-glucuronosyltransférases
1.3.2.1
Les UGT1
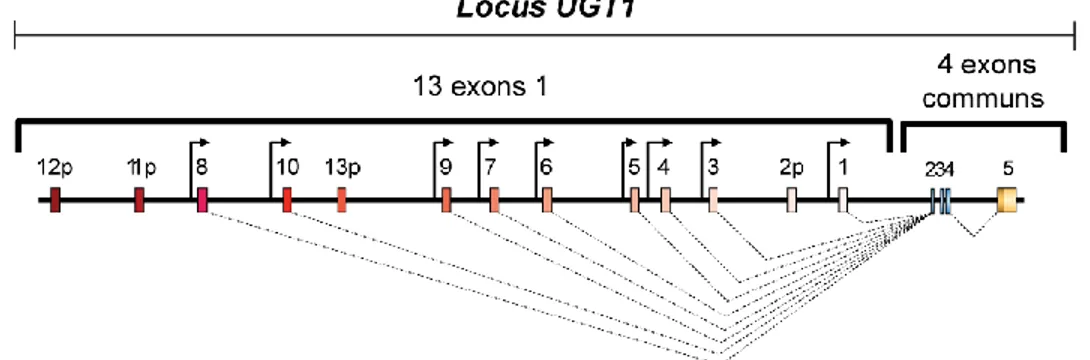
Le gène humain encodant les protéines UGT1 est long de 198 kilobases a été précisément localisé dans la région q37 du chromosome 2 (2q37) 35. La famille UGT1 comporte 9 enzymes uniques, toutes codées par ce gène complexe contenant 17 exons. À l‘origine, la structure particulière du locus UGT1 a été déterminée comme étant constituée d‘une série de 13 exons 1 uniques en 5‘ du gène et de 4 exons communs à tous les transcrits UGT1 en 3‘. Ce locus permet une génération d‘ARNm par partage d‘exons (« exon sharing ») : seulement un des treize exons 1 est choisi et est regroupé à tous les exons 2 à 5 communs (figure 4)36. Chaque transcrit est donc composé de cinq exons, le premier (l‘exon 1) étant unique à chaque isoforme, alors que les quatre exons terminaux sont identiques pour tous. Au niveau génique, chaque exon 1 possède une séquence promotrice unique permettant une spécificité d‘expression propre à chaque UGT1. Au niveau structural, l‘exon 1 code pour le domaine de liaison au substrat, alors que les exons communs 2 à 5 codent pour le domaine de liaison au co-facteur ainsi que pour le domaine transmembranaire.
Figure 3: Arbre phylogénétique de la superfamille des UGT humains. Entre elles, les glucuronosyltransférases des familles UGT1 et UGT2 partagent environ 50% d‘homologie dans leurs chaînes d‘acides aminés. À l‘intérieur des familles 1 et 2, les différentes isoformes partagent au moins 66% et 59% d‘homologie, respectivement. Les UGT1A_i2 sont à 98% identiques aux UGT1A_i1 (étant donné qu‘ils comportent une région différente en 3‘) et sont enzymatiquement inactifs; leurs caractéristiques sont expliquées en détail dans la section 3.5.1 32-34.
Les enzymes de la famille UGT1 démontrent une affinité pour un grand nombre de substances, comme par exemple la bilirubine, les hormones stéroïdiennes (principalement les estrogènes) 37, 38, les acides biliaires 39, les hormones thyroïdiennes 31 ainsi que plusieurs médicaments 40. De plus, les molécules contenant des amines primaires, secondaires et tertiaires 41, 42, les opioïdes 43, les flavonoïdes 44, 45 et les benzopyrènes 46-48 sont pour la plupart glucuronidés par certains UGT1. UGT1A1, l‘enzyme la plus étudiée de tous les UGT1, peut conjuguer divers composés endogènes comme l‘acide rétinoïque 49 et les estrogènes 38, mais également des composés exogènes comme les médicaments irinotécan 50 et ézétimibe 51. UGT1A1 est également la seule isoenzyme à métaboliser la bilirubine 52 : d‘ailleurs, des variants de sa région génique sont associés à l‘hyperbilirubinémie (souvent appelée syndrome de Gilbert et caractérisée par une jaunisse)53, à la maladie de Crigler-Najjar (hyperbilirubinémie sévère menant souvent à des dommages
cérébraux)54, à la susceptibilité à certains cancers estrogéno-dépendants 55, 56 et à d‘autres caractéristiques physiologiques 57-59.
Figure 4: Représentation du locus UGT1. Lors du processus de transcription, un des 13 exons 1 du locus est joint aux 4 exons communs (2, 3, 4 et 5), ce qui donne une possibilité d‘expression de 13 transcrits UGT1 distincts. Les exons 1 nommés 12p, 11p, 13p et 2p sont considérés comme étant des pseudogènes car ils contiennent tous au moins un codon d‘arrêt entraînant une traduction prématurée 60.
Récemment, notre laboratoire a mis au jour la présence d‘un exon commun supplémentaire, l‘exon 5b. Cet exon alternatif peut se retrouver au sein de transcrits UGT1 après l‘exon 4 et il précède ou remplace l‘exon 5 classique (renommé exon 5a). L‘épissage alternatif de cet exon produit deux nouvelles classes de transcrits, les UGT1A_v2 et v3, tous les deux menant à un seul type de nouvelles protéines tronquées en C-terminal (les UGT1A_i2) qui modulent négativement l‘activité des enzymes UGT1A (renommées UGT1A_i1) 61-66. Ce mécanisme moléculaire est exposé de façon plus exhaustive dans la section 3.5.1.
1.3.2.2
Les UGT2
Il existe 2 sous-familles UGT2 chez l‘humain, UGT2A et UGT2B, qui sont toutes deux codées sur le chromosome 4q13 67. Un total de trois membres de la sous-famille 2A ont été découverts chez l‘homme, soit UGT2A1, UGT2A2 et UGT2A3. Ces 3 enzymes partagent environ 60% d‘identité avec les UGT2B et sont connues comme étant principalement actives au niveau de l‘épithélium olfactif 68-70. La fonction biologique principale proposée aux UGT2A est la terminaison du signal odorant dûe à la glucuronidation de composés phénoliques 71.
UGT2A1 et UGT2A2 sont exprimés à partir du même locus et, comme les UGT1A, sont caractérisés par des exons 1 uniques et par un partage d‘exons communs (exons 2 à 6)72. Les deux exons 1 comprennent des promoteurs distincts, ce qui leur confère une spécificité d‘expression.
Ces deux enzymes sont les seuls UGT exprimés de façon majeure le long des voies respiratoires 72-74quoiqu‘ils aient également été détectés au côlon, au rein et au sein, mais pas dans le foie 72. Il a été remarqué que UGT2A1 avait des niveaux d‘expression environ deux fois plus élevés que UGT2A2 dans les tissus où les deux sont exprimés70 75. UGT2A1 est considéré comme l‘UGT le plus efficace pour la conjugaison de l‘indométacine, un médicament anti-inflammatoire 76. Cette enzyme métabolise également les androgènes (la testostérone, l‘épitestostérone 77, l‘androstérone, l‘étiocholanolone et les 5-alpha- et 5-bêta-androstanediol 78), les estrogènes (l‘estradiol 79 et l‘estriol 75), le 1-naphtol, le scopolétol, le 4-méthylumbelliférone (4-MU) 75, certains composés phénoliques75, plusieurs composés carcinogéniques du tabac74, l‘acide hyodésoxycholique (HDCA)75 et le diclofénac 76. L‘activité de UGT2A2 est beaucoup plus faible, mais cette enzyme est aussi capable conjuguer la testostérone, l‘épitestostérone 77, l‘androstérone, l‘étiocholanolone, les 5-alpha- et 5-bêta-androstanediol 78, l‘estradiol 75, 79, l‘acide hyodésoxycholique 75 le 1-naphtol, certains composés phénoliques 75, le 4-méthylumbelliférone 75 et plusieurs composés carcinogéniques du tabac 72. Il a récemment été démontré qu‘un transcrit variant sans exon 3 existait pour UGT2A1 et 2A2 (les ARNm UGT2A1Δexon3 et UGT2A2Δexon3, menant respectivement à la présence des protéines UGT2A1_i2 et UGT2A2_i2) 72, 80. Un peu comme dans le cas des formes i2 du locus UGT1A, ces protéines diminuent la vitesse maximale de l‘activité de glucuronidation des formes classiques (UGT2A_i1) sans modifier l‘affinité pour le substrat. Les formes courtes ont été détectées dans les mêmes tissus que les i1, mais ne sont très présentes qu‘au niveau du côlon 72, 80. UGT2A3 est un des UGT qui détient le plus large spectre de distribution tissulaire, étant exprimé au niveau du foie, de l‘estomac, du pancréas, du côlon, du rein, du tissu adipeux, de la trachée, du larynx, des amygdales, du poumon, du testicule et du tissu nasal 70, 72, 81. Pour l‘instant, très peu de substrats ont été associés à UGT2A3, soit le 1-OH-pyrène, le 1-naphtol 72 et certains acides biliaires (l‘acide hyodésoxycholique, l‘acide chénodésoxycholique, l‘acide désoxycholique et l‘acide ursodésoxycholique 75 81). Cette enzyme a une capacité de glucuronidation très faible sur les composés testés jusqu‘à maintenant.
La sous-famille UGT2B compte quant à elle sept enzymes fonctionnelles chez l‘homme, soit UGT2B4 48, UGT2B7 82, 83, UGT2B10 84, UGT2B11 85, UGT2B15 86, 87, UGT2B17 88 et UGT2B28 89. Les principaux substrats métabolisés par les membres de la sous-famille UGT2B sont les acides biliaires 90, les hormones stéroïdiennes 89, 91, les leucotriènes 92 et plusieurs substances exogènes comprenant des médicaments tels la morphine 8, 93, la zidovudine 94 et le
tacrolimus 95. Étant donné que mes travaux de doctorat ont principalement porté sur les UGT2B, la section 2.3 de cette introduction leur est consacrée.
1.3.2.3
Les UGT3
Les UGT3 (UGT3A1 et 3A2) ont approximativement 30% d‘homologie avec les autres familles UGT et sont codés sur le chromosome 5p13.2. Ils sont constitués de 7 exons et sont composés d‘un total de 523 acides aminés 60. UGT3A1 est spécialisé dans le transfert du N-acétylglucosamine sur certains substrats, tels les acides biliaires (acide lithocholique et l‘acide désoxycholique), les flavonoïdes (biochanine A, 2‘/4‘OH-flavanone, formononétine, chrysine et génistéine), l‘estradiol, la testostérone, le 4-nitrophénol, le 1-naphtol, le 4-méthylumbelliférone et l‘ibuprofène; cependant, son substrat principal semble être l‘acide ursodésoxycholique 96, 97. Comme le métabolite majeur de cet acide dans l‘urine est sa forme N-acétylglucosaminidée 98, l‘importance de UGT3A1 dans le métabolisme de cette molécule utilisée pour contrer les cholestases pourrait être majeure 99. UGT3A1 a été principalement retrouvé dans le foie ainsi que le rein, mais il semble également exprimé au niveau de l‘estomac, du duodénum, du côlon et des testicules96. Malgré une forte homologie de séquence 97, UGT3A2 ne transfère pas le N-acétylglucosamine comme UGT3A1, mais se spécialise plutôt dans l‘utilisation de l‘UDP-glucose et de l‘UDP-xylose comme co-substrats. Il a été remarqué que UGT3A2 était impliqué dans le métabolisme de plusieurs flavonoïdes (naringénine, génistéine, chrysine, isorhamnétine, biochanine A, acacétine, diadzéine et formononétine), certains estrogènes (estradiol, estrone et estriol), l‘acide mycophénolique et plusieurs autres substrats classiques des UGT (le 4-nitrophénol, le 1-naphtol, le 4-méthylumbelliférone, le 7-hydrocoumarin et le 1-OH-pyrène) 71. Le transcrit d‘UGT3A2 a été détecté dans les tissus périphériques (testicule, thymus, prostate, rate) et le rein.
1.3.2.4
UGT8A1
Il n‘existe qu‘un seul UGT8A humain, UGT8A1, une enzyme encodée par un gène à 5 exons sur le chromosome 4q26 produisant une protéine de 541 acides aminés. L‘activité médiée par UGT8A1 est très différente des autres UGT, cette enzyme catalysant le transfert du galactose sur la céramide, une étape cruciale dans la biosynthèse des glycosphingolipides, des cérébrosides et des sulfatides 96. L‘activité de UGT8A1 a été principalement notée dans les cellules nerveuses, plus particulièrement au niveau de la fabrication des gaines de myéline 100. Le rôle des galactocéramides que UGT8A1 contribue à produire est peu connu, mais il a été suggéré que ces
molécules galactocéramides seraient essentielles à la fonction et à la stabilité de cette gaine 101, 102. En lien avec son rôle, le transcrit d‘UGT8A1 a été détecté principalement au cerveau 103, mais son activité et/ou son expression ont également été notées au rein 103, 104, au foie 103, 105, au testicule, au pancréas, dans la peau, dans les os 103 et dans l‘intestin 103, 106.
1.4 Distribution tissulaire des UGT et activité
Les UGT sont principalement exprimés au foie et au rein, mais ils ont également été détectés dans un grand nombre d‘autres tissus. Les tableaux A1 à A4 de l‘Annexe I constituent un résumé de la littérature concernant la détection des différents transcrits UGT dans les tissus humains. Dans certains cas, les résultats publiés sont divergents, ce qui est représenté par le symbole « ~ » dans les tableaux. Ces différences pourraient être expliquées par une présence très faible de l‘UGT en question ou une expression polymorphique de celui-ci 107, 108; également, une méthode de détection inadéquate ou non spécifique pourrait affecter les résultats obtenus 109.
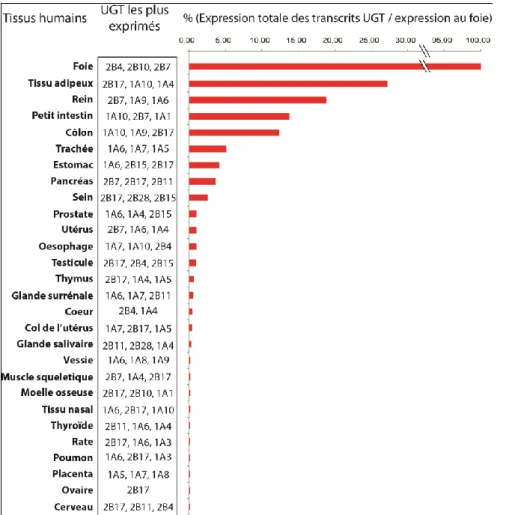
La forte homologie des UGT couplée à l‘absence d‘anticorps très spécifique a pour l‘instant freiné l‘avancement des connaissances concernant l‘expression comparative des UGT. Ce problème majeur a été contourné récemment par l‘avènement de la quantification par PCR en temps réel qui rend possible la comparaison des niveaux de transcription des différents UGT dans un même tissu et/ou d‘une même enzyme dans des tissus différents. Cette méthode n‘est pas sans limite, car comme elle dépend de l‘amplification d‘un petit fragment d‘ADN complémentaire (de moins de 200 paires de bases 110), il est souvent plus complexe (voire impossible) de distinguer les variants d‘épissage d‘un UGT. De plus, la comparaison de l‘expression entre des transcrits peut se faire uniquement si l‘efficacité d‘amplification des fragments est la même, ou si les résultats ont été rapportés sur une courbe d‘efficacité 111. Malheureusement, certains auteurs négligent ces éléments mais comparent tout de même les résultats entre eux 112, ce qui peut constituer une importante source de divergences. Quoi qu‘il en soit, deux études ont été récemment menées dans le but de quantifier par PCR en temps réel l‘expression des UGT dans plusieurs tissus humains 70, 113. La figure 5 résume les niveaux de transcrits UGT retrouvés dans les tissus analysés, en plus d‘énumérer les UGT les plus exprimés dans ces matrices.
La présence du tissu adipeux au deuxième rang de cette liste peut sembler surprenante, et selon l‘auteur ce résultat devrait être interprété avec prudence et validé subséquemment, étant donné