T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Microbiologie et Génétique
Présentée et soutenue par
Laetitia ATTAIECH
Le 11 Décembre 2009Titre :
Déterminants de stabilité et de maturation de l'ADN internalisé lors de la transformation génétique naturelle
chez Streptococcus pneumoniae
JURY
Suzanne SOMMER Rapporteur Pablo RADICELLA Rapporteur Vincent MEJEAN Examinateur Bernard MARTIN Directeur de Thèse Jean-Pierre CLAVERYS Directeur de Thèse
Ecole doctorale : Biologie Santé Biotechnologie
Unité de recherche : Laboratoire de Microbiologie et Génétique Moléculaire, CNRS, UMR5100 Directeur(s) de Thèse : Bernard MARTIN et Jean-Pierre CLAVERYS
D
ETERMINANTS DE STABILITE
ET DE MATURATION
DE L
'ADN
INTERNALISE
LORS DE LA
TRANSFORMATION GENETIQUE NATURELLE
CHEZ
2
Remerciements
Je remercie Suzanne Sommer et Pablo Radicella d’avoir accepté d’être rapporteurs de cette thèse malgré un emploi du temps chargé et un timing vraiment serré en cette fin d’année, ainsi que Vincent Méjean pour son rôle d’examinateur.
Merci à mon équipe qui m’a supportée (et pas que dans le sens de « soutenir » !) depuis si longtemps. A Bernard et Jean-Pierre, mes 2 chefs, pour le temps qu’ils m’ont accordé, parce qu’ils ont toujours pris le temps de discuter avec moi, même de mes hypothèses les plus saugrenues (et j’en ai !!), et aussi pour avoir supporté que je ne sois pas vraiment « du matin » et que je sois parfois un danger ambulant (heureusement, je ne fait mal qu’à moi-même…) ! A Marc, qui a été mon premier chef dans l’équipe lors de mon stage de Maîtrise et avec qui j’ai adoré faire de la recherche (la preuve : je suis restée !). A Isa pour les manip’ en communs et les nombreux conseils techniques, à Nathalie que je n’ai pas arrêté de harceler de questions depuis qu’elle a intégré l’équipe, à Chantal pour un peu tout (les souches, les plasmides, les milieux, les ragots partagés et autres nombreux services… !). A tous ceux qui sont passés au labo (les anciens partis et les nouveaux arrivés il y a peu) et qui ont contribué à la bonne humeur générale : Anne-Lise, Audrey, Calum, Christophe, Mathieu (2), Nicolas, Sébastien, Vincent… et je dois certainement en oublier, mais bon, une fin de thèse, c’est usant ! Un merci global à tous pour les encouragements, la disponibilité, l’ambiance, les gâteaux et autres bons moments… ça va vraiment me manquer…
Un merci généralisé à tous ceux de l’étage et même du bâtiment que j’ai eu l’occasion de croiser ça et là et dont l’accueil a toujours été chaleureux.
Un merci particulier à Philippe Liauzun et Patrick Rouimi, deux de mes anciens maîtres de stage, qui ont gardé le contact avec moi et qui ont contribué à ce que j’aime la recherche, ainsi qu’à mes profs de BTS qui m’ont redonné l’envie d’apprendre.
Merci à mes amis, les scientifiques et les non-scientifiques (que j’aime autant, comme quoi, ça n’a rien à voir !!!) d’être tout simplement là quand il faut. Dans le 1er groupe, Hélène et Magali (vous aviez raison, c’est crevant sur la fin !), Max, Stéphanie et Thomas (rigolez pas, c’est bientôt votre tour !!!) ; dans le 2ième groupe, Marion (depuis siiiiiiiii longtemps), David, Sandra-Julie ; et Carole que je ne sais pas dans quel groupe ranger et qui ne sait pas ce qu’elle a raté en abandonnant la bio !
Merci aussi à ma famille qui ne comprend absolument pas ce que je fabrique avec des pipettes mais qui me soutient quand même !
3
Résumé
Streptococcus pneumoniae est une bactérie commensale de l’homme colonisant le
rhinopharynx. Ce pathogène majeur de l’homme (~1,6 millions de morts par an dans le monde) pose un problème de santé publique du fait de sa variabilité antigénique, qui limite l’efficacité des vaccins, et de sa capacité à acquérir des gènes de résistance aux antibiotiques. Ces 2 phénomènes sont permis par une propriété naturelle de S. pneumoniae : la transformation génétique. Cette bactérie peut en effet développer un état physiologique transitoire régulé génétiquement (la compétence), lors duquel elle peut internaliser et intégrer dans son chromosome par recombinaison homologue de l’ADN exogène.
Au début de ma thèse, j’ai terminé des travaux, initiés en Maitrise qui ont permis de montrer que la compétence (et donc la transformation) pouvait être induite par des antibiotiques couramment utilisés en thérapie (Prudhomme, Attaiech et al., Science, 2006, 313(5783):89-92). Ainsi, l’utilisation mal contrôlée d’antibiotiques peut augmenter le pouvoir pathogène de S. pneumoniae en favorisant les transferts génétiques.
Mon travail s’est ensuite concentré sur le devenir de l’ADN transformant après son internalisation. J’ai étudié les déterminants de stabilité de cet ADN, i.e. les protéines le stabilisant ou le protégeant d’attaques nucléolytiques et j’ai tenté d’identifier une/des nucléase(s) que l’on sait agir sur cet ADN. En effet, il avait été montré qu’en l’absence de RecA ou DprA, protéines exprimées lors de la compétence, l’ADN simple-brin internalisé (ADNsb) est dégradé dès son entrée dans la cellule, suggérant que ces deux protéines le protègent de l’action de nucléases endogènes. Dans le but d’identifier la (les) nucléase(s) responsable(s) de cette dégradation de l’ADNsb entrant, j’ai analysé plusieurs mutants par différentes méthodes (génétiques et biochimiques).
Je n’ai pas réussi à identifier cette nucléase mais parmi les candidats testés, j’ai pu montrer que Pms et CoiA influent sur l’efficacité du système général de réparation des mésappariements (Hex) et j’ai étudié RadC dont l'expression est induite lors de la compétence. Nous avons montré que cette protéine n’était pas la nucléase recherchée et n’était pas non plus impliquée ni dans la recombinaison ni dans la réparation de l’ADN. Ces résultats nous ont conduit à proposer une révision des annotations de la famille RadC dans la base de protéines pfam (Attaiech, et al., J. Bact., 2008, 190(16):5729-32).
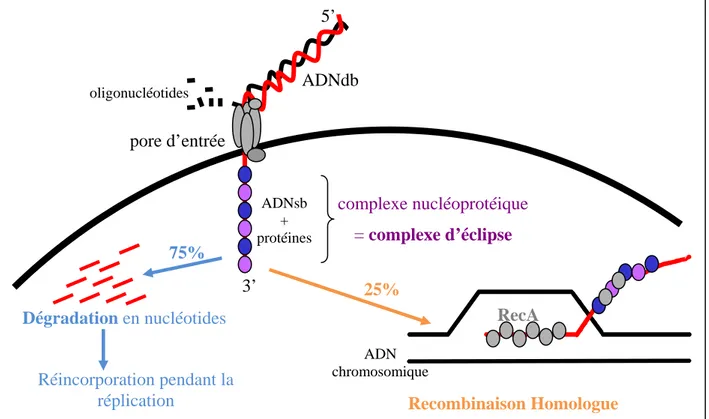
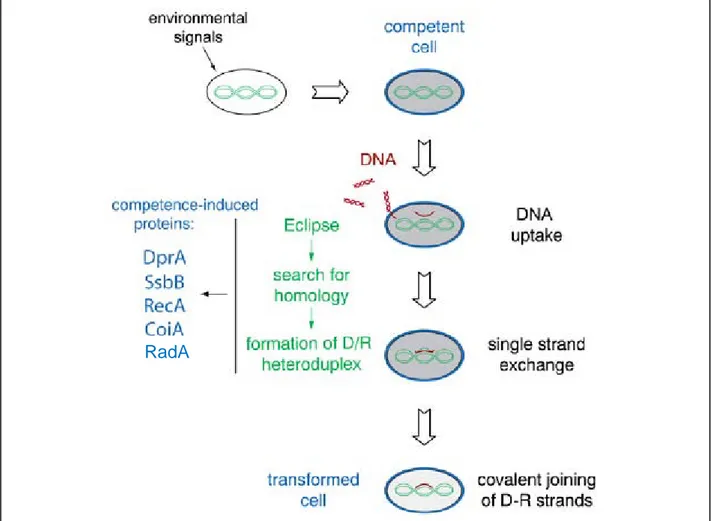
On sait, depuis une trentaine d’années, que l’ADNsb internalisé est pris en charge par une (des) protéine(s) pour former un complexe résistant à l’action de nucléases in vitro, le « complexe d’éclipse ». Des expériences de fractionnement et de purification nous ont permis de proposer que la protéine majeure du complexe d’éclipse est SsbB, une protéine affine de l’ADNsb dont l'expression est induite en compétence (Morrison, Mortier-Barrière et al., J. Bact., 2007, 189(17):6497-500). Toutefois, l'absence de SsbB, à la différence de celle de DprA ou RecA ne provoque pas de dégradation majeure de l'ADNsb internalisé. Afin de détecter la présence éventuelle de ces protéines dans le complexe d’éclipse et de mieux cerner le rôle de SsbB, j’ai poursuivi la caractérisation de ce complexe. Il semble que RecA et DprA ont une forte affinité pour le support utilisé lors de la purification, elles pourraient donc former un nucléocomplexe avec l’ADNsb mais ne plus pouvoir être éluées de ce support. Ces résultats montrent que la nature même de ce que l’on nomme « complexe d’éclipse » et la composition protéique de ce nucléocomplexe reste à élucider (manuscrit en préparation).
4
Summary
During my doctorate, I studied different aspects of genetic transformation in the human pathogen Streptococcus pneumoniae. This bacterium can naturally develop a transient physiological state, named competence, in which it is able to internalize and integrate exogenous DNA into its chromosome by homologous recombination.
At the beginning of my thesis, I completed some experiments on the subject I worked on during my Masters Degree project. We showed for the first time that antibiotic treatment can trigger a response enabling bacteria to undergo competence and uptake DNA and integrate it into their genome. (Prudhomme, Attaiech, et al., Science, 2006, 313(5783):89-92).
Then, my work concentrates on the fate of DNA after internalization during the process of transformation. In particular, I studied parameters influencing the stability of internalized DNA, i.e. proteins stabilizing/protecting it from nucleases as well as potential nucleases that could degrade it.
It is already known that during genetic transformation of S. pneumoniae, exogenous double-stranded DNA (dsDNA) is cut and one of the two strands is transported into competent cells (3’-end first). Single stranded internalised DNA (ssDNA) is then complexed with protein(s), this nucleocomplex has been known as the Eclipse Complex since 1977. It has been also showed that internalized ssDNA requires protection against endogenous DNases and that RecA and DprA, two competence proteins, are essential for this protection.
I have analysed, by various methods, several mutants, with the aim of identifying the nuclease responsible for the rapid degradation of ssDNA in the absence of DprA or RecA. So far, I have been unable to identify this nuclease. However, work on some of these mutants has produced interesting information regarding the contribution of CoiA and Pms to ssDNA processing. I also carried out experiments on the radC gene which encoded a protein originally annotated as [a DNA repair protein (that) plays a role in repair of DNA damage
after UV and X-ray irradiation in prokaryotes...] (pfam04002). I properly inactivated this
gene and showed that radC- cells remain fully transformable with various DNA substrates and are not affected in repair mechanisms. There is thus no evidence of a role of RadC in connection with repair and/or recombination in any prokaryote (Attaiech, et al., Journal of Bacteriology, 2008, 190(16):5729-32).
As part of my thesis studies, I aimed to identify more precisely the component(s) of the EC. By fractionation and purification experiments, we showed that the major protein component is the competence-induced protein SsbB, a paralogue of the essential ssDNA-binding protein SSB (Morrison, Mortier-Barrière et al., Journal of Bacteriology, 2007, 189(17):6497-500). As RecA and DprA are essential the protection of internalized DNA, their absence in the EC is puzzling. Trying to understand this apparent paradox, I have done further biochemical experiments on the characteristics of EC and show that DprA and RecA have a strong affinity for the support used during purification of EC. This could explain the only detection of SsbB in the EC and imply that the real nature of the EC has to be discovered (Attaiech et al., in prep).
5
Sommaire
A.
Introduction ... 10
A.1.
Présentation de Streptococcus pneumoniae ... 11
A.2.
La transformation génétique naturelle ... 13
A.2.1. Historique ... 13
A.2.2. Un phénomène répandu ... 13
A.2.3. Rôle de la transformation génétique naturelle ... 14
A.2.4. L’état de « compétence » ... 16
A.3.
La compétence pour la transformation génétique chez Streptococcus
pneumoniae ... 17
A.3.1. La compétence et sa régulation ... 17
A.3.1.1. Conditions d’induction de la compétence ... 17
A.3.1.2. Le CSP et l’opéron comAB ... 18
A.3.1.3. L’opéron comCDE ... 19
A.3.1.4. La boucle auto-catalytique ... 21
A.3.2. Le développement de la compétence ... 22
A.3.2.1. Les gènes précoces ... 23
A.3.2.2. Les gènes tardifs ... 25
A.3.2.3. Les gènes retardés ... 27
A.3.2.4. La sortie de l’état de compétence... 27
A.3.3. L’initiation de l’état de compétence ... 29
A.3.3.1. L’expression basale ... 29
A.3.3.2. Les mutants dérégulés ... 30
A.3.4. La compétence et la réponse au stress ... 31
A.4.
Les mécanismes de la transformation génétique ... 34
A.4.1. L’entrée d’ADN exogène ... 34
A.4.1.1. Fixation de l’ADN exogène ... 34
A.4.1.1.1. Passage du peptidoglycane : rôle des protéines ComGs... 35
A.4.1.1.2. La fixation de l’ADNdb par ComEA ... 37
A.4.1.2. Dégradation d’un brin et entrée concomitante de l’autre brin ... 37
A.4.1.3. L’internalisation de l’ADNsb ... 38
A.4.1.3.1. Passage de la membrane : rôle de ComEC ... 38
A.4.1.3.2. Force d’internalisation ... 39
6
A.4.2.1. Le complexe d’éclipse ... 41
A.4.2.1.1. Nature du complexe d’éclipse ... 42
A.4.2.1.2. La composante protéique du complexe d’éclipse ... 44
A.4.2.2. La recombinaison homologue ... 46
A.4.2.2.1. Les protéines habituellement impliquées dans la recombinaison homologue .. 46
A.4.2.2.2. Les protéines nécessaires spécifiquement pour la recombinaison homologue lors de la transformation ... 50
A.4.2.3. Le système Hex ... 52
A.4.2.4. La transformation plasmidique ... 57
B.
Résultats ... 59
B.1.
Etude de la dégradation de l’ADN internalisé ... 60
B.1.1. Existence d’une nucléase antagonisée par DprA et RecA ... 60
B.1.2. Rôle du domaine carboxy-terminal de ComEC ... 61
B.1.2.1. Approches génétiques permettant la caractérisation des fonctions de la protéine ComEC ... 62
B.1.2.1.1. Capacité de transformation ... 63
B.1.2.1.2. Stabilité de l’ADNsb internalisé dans une souche dprA- et effet des variants de ComEC ... 67
B.1.2.1.3. Conclusion sur la fonction de ComEC par l’approche génétique ... 69
B.1.2.2. Caractérisation biochimique du domaine carboxy-terminal de la protéine ComEC ... 69
B.1.2.2.1. Mise en évidence d’une activité DNase ... 71
B.1.2.2.2. Mise en évidence d’une capacité de liaison à l’ADN ... 74
B.1.2.2.3. Conclusions sur l’analyse de ComEC Cter par les approches biochimiques .. 75
B.1.2.3. Conclusion sur la fonction du domaine Cter de ComEC ... 75
B.1.3. Tests d’autres candidats nucléases ... 77
B.1.3.1. Les différents candidats ... 78
B.1.3.2. Etude de la stabilité de l’ADNsb internalisé en contexte triple-mutant ... 79
B.1.4. Conclusion sur la recherche de nucléase ... 80
B.2.
Etude de la protection de l’ADN internalisé (le complexe d’éclipse) .. 82
B.2.1. Identification de la composante protéique du complexe d’éclipse ... 83
B.2.2. Revisiting the Eclipse Complex (in prep) ... 89
B.3.
Etude de la maturation de l’ADNsb internalisé ... 112
B.3.1. coiA, pms et le système Hex ... 112
B.3.1.1. Rôle de Pms dans la transformation génétique naturelle ... 113
7
B.3.1.1.2. Effet de la mutation pms- sur l’efficacité du système Hex ... 113
B.3.1.1.3. Conclusion sur le rôle de Pms ... 115
B.3.1.2. Rôle de CoiA dans la transformation génétique naturelle ... 116
B.3.1.2.1. Capacité de transformation d’un mutant coiA- ... 116
B.3.1.2.2. Efficacité du système Hex en contexte coiA- ... 119
B.3.1.2.3. Conclusion sur le rôle de CoiA ... 120
B.3.2. RadC est-elle impliquée dans la maturation de l’ADN ? ... 122
C.
Discussion, Conclusion ... 135
D.
Matériel et Méthodes ... 143
D.1.
Souches, plasmides et utilisés ... 144
D.2.
Conditions de Culture et Transformation ... 149
D.3.
Test de conversion génique ... 149
D.4.
Analyse statistique des résultats ... 149
D.5.
Détermination de l’expression du gène rapporteur ssbB::luc ... 150
D.6.
Mutagénèse mariner ... 151
D.7.
Construction des mutants comEC
D511N, comEC
H562Aet comEC
N650A... 152
D.8.
Synthèse de fragment d’ADNdb ... 153
D.9.
Suivi du devenir de l’ADN internalisé pendant la transformation de S.
pneumoniae ... 153
D.10.
Production et purification de protéines ... 154
D.11.
Détection d’une activité DNase in situ ... 156
D.12.
Dégradation d’oligonucléotides ... 156
D.13.
Retard sur gel ... 157
E.
Annexes ... 158
E.1.
Publication : « Antibiotic stress induces genetic transformability in the
human pathogen Streptococcus pneumoniae. » ... 159
8
Table des illustrations
Figure 1 : Développement de la compétence chez S. pneumoniae. ... 18
Figure 2 : Séquence protéique du produit du gène comC. ... 19
Figure 3 : Transduction du signal au sein d’un système à deux composants classique. ... 20
Figure 4 : La boucle auto-catalytique d'induction de la compétence. ... 22
Figure 5 : Les gènes précoces et leurs boites ComE. ... 24
Figure 6 : Les gènes tardifs et leurs boites cin. ... 26
Figure 7 : Les gènes retardés. ... 27
Figure 8 : Les inducteurs du système SOS. ... 32
Figure 9 : Liaison et entrée de l’ADN pendant la transformation chez S. pneumoniae. ... 35
Figure 10 : Modèle de la topologie adoptée par ComEC chez B. subtilis. ... 39
Figure 11 : Représentation schématique du devenir de l'ADN internalisé lors de la transformation. ... 40
Figure 12 : Processing de l’ADN internalisé. ... 42
Figure 14 : Modèle général de la recombinaison homologue. ... 49
Figure 15 : Principales étapes dans le processus de transformation génétique chez S. pneumoniae et protéines impliquées. ... 52
Figure 16 : Efficacité du système Hex. ... 54
Figure 17 : Modèle d'action du système Hex. ... 55
Figure 13 : Installation d’un plasmide réplicatif monomérique chez S. pneumoniae. ... 58
Figure 18 : Alignement d’une partie de la séquence de ComEC de S. pneumoniae et de ses homologues chez d’autres bactéries naturellement compétentes. ... 63
Figure 19 : Capacité de transformation d’un mutant dprA- et effet suppresseur de la mutation comECD511N. ... 65
Figure 20 : Stabilité de l’ADNsb internalisé. ... 68
Figure 21 : Séquence de ComEC de S. pneumoniae. ... 70
Figure 22 : Révélation d’une activité DNase sur gel. ... 72
Figure 23 : Révélation d’une activité DNase par dégradation d’oligonucléotides. ... 74
Figure 24 : Retard sur gel. ... 74
Figure 25 : Modèle du mécanisme d’entrée de l’ADN transformant et de son devenir après internalisation chez S. pneumoniae. ... 77
Figure 26 : Stabilité de l’ADNsb internalisé. ... 80
9 Figure 28 : Efficacité du système Hex lors de la transformation génétique en contexte pms-. ... 114
Figure 29 : Effet de la mutation pms- sur la conversion génique. ... 115
Figure 30 : Rôle potentiel de Pms lors de la reconnaissance de mésappariements chez S.
pneumoniae. ... 116
Figure 31 : Capacité de transformation d’un mutant coiA-. ... 117
Figure 32 : Capacité de transformation des mutants coiA-, ssbB- et combinaison de ces mutations. ... 118
Figure 33 : Efficacité du système Hex lors de la transformation génétique en contexte coiA- et/ou ssbB-. ... 119
Figure 34 : Rôle potentiel de CoiA lors de la transformation génétique chez S. pneumoniae. ... 121
Figure 35 : Modèle de la maturation de l’ADNsb internalisé durant la transformation génétique naturelle chez S. pneumoniae. ... 142
Figure 36 : Ecart-types. ... 150
Figure 37 : Représentation schématique d’un mini-transposon et de son mode d’insertion. . 151
10
A.
11
A.1. Présentation de Streptococcus pneumoniae
Streptococcus pneumoniae, aussi appelée pneumocoque, est une bactérie capsulée à
Gram positif, commensale de l’homme, isolée en 1881 simultanément par Louis Pasteur en France et George Sternberg aux Etats-Unis (Pasteur 1881)(Sternberg 1881).
30 à 50% des enfants de moins de 6 ans et 4 à 12% des adultes sont porteurs sains de pneumocoques localisés au niveau du rhinopharynx dans un état ''latent'' (Lynch & Zhanel 2009). Cependant, cette bactérie peut devenir invasive lorsque les conditions deviennent favorables. Elle pose ainsi un grave problème de santé publique, y compris dans les pays industrialisés. Pathogène majeur du système respiratoire, elle est la cause principale des pneumonies. Elle est également responsable d’otites, de méningites et de surinfections au cours de leucémies lymphocytaires chroniques. Les jeunes enfants, les personnes âgées et les personnes immunodéprimées (notamment par des infections virales comme le VIH) se trouvent particulièrement exposés aux infections par S. pneumoniae (Lynch & Zhanel 2009). Les bactériémies et méningites bactériennes causées par S. pneumoniae sont une cause importante de morbidité sévère et de mortalité. En 2007, l’Organisation Mondiale de la Santé estimait à 1,6 millions le nombre de morts par an par pneumococcies dont 1 million d’enfants (World Health Organization 2007).
En France, S. pneumoniae est à l’origine d’une dizaine de milliers d’infections invasives par an. Depuis 1987, un réseau de laboratoires hospitaliers (Epibac) surveille l’incidence des infections invasives bactériennes. Les données obtenues contribuent à l’évaluation des mesures de prévention, notamment vaccinales, mises en place au niveau national (Georges et al. 2005).
S. pneumoniae est naturellement sensible à de nombreux antibiotiques, mais, depuis la
description de la première souche de pneumocoque de sensibilité diminuée à la pénicilline G (PSDP) en 1967 en Australie, de nombreuses études montrent une augmentation de la résistance aux antibiotiques et particulièrement aux β-lactames. Les données publiées en France montrent une augmentation continue des PSDP (55,4 % en 2001), particulièrement chez l’enfant où le taux atteint 71 %, mais ceci avec une variation importante selon les régions (Maugein et al. 2006).
La lutte contre S. pneumoniae passe par l’emploi soit d’antibiotiques, soit de vaccins. Ces deux stratégies se heurtent à la grande plasticité génétique de cette bactérie. Cette
12 variabilité génétique est directement reliée à la transformation génétique naturelle. Ce phénomène, découvert chez le pneumocoque, permet aux cellules de capturer l’ADN présent dans l’environnement, puis de l’intégrer dans le chromosome par recombinaison homologue.
La transformation génétique est très probablement responsable de l’apparition de nouveaux gènes de résistance aux antibiotiques ainsi que de l’accumulation d’isolats cliniques présentant des résistances multiples aux antibiotiques. Par exemple, la résistance aux β-lactames passe par une modification de leurs cibles que sont les PBPs (Penicillin-Binding Proteins). Celles-ci sont impliquées dans la synthèse du peptidoglycane et leurs activités sont bloquées par les β-lactames (Tomasz 1986). Chez S. pneumoniae, les gènes codant les PBPs résistantes aux β-lactames sont dits « mosaïques », ce sont en fait des combinaisons de gènes formées par transfert horizontal entre différents streptocoques (Dowson et al. 1989)(Hakenbeck et al. 2001).
De plus, en ce qui concerne la mise au point d’un vaccin, la diversité capsulaire de S.
pneumoniae est problématique. En effet, la plupart des souches de S. pneumoniae sont
capsulées. La capsule, enveloppe principalement constituée de polysaccharides, joue notamment un rôle central dans la virulence du pneumocoque en le protégeant de la phagocytose par les macrophages (Paton et al. 1993). Elle est également un des principaux déterminants antigéniques cibles du système immunitaire, raison pour laquelle elle est utilisée dans la préparation de vaccins. Cependant, on dénombre plus de 90 sérotypes capsulaires, ce qui rend difficile la mise au point d’un vaccin universel (Henrichsen 1995). Cette diversité capsulaire est aussi vraisemblablement due à la capacité naturelle de transformation génétique de S. pneumoniae (Claverys et al. 2000).
Ainsi S. pneumoniae pose un problème de santé publique majeur à l’échelle mondiale. Ceci justifie l’intérêt de nombreuses équipes de recherche et de laboratoires pharmaceutiques pour la recherche sur les phénomènes de virulence et de transformation génétique. Le séquençage de différentes souches de pneumocoque a favorisé et aidé les recherches génétiques dans ces domaines. Les séquences de deux souches virulentes (types capsulaires 4 et 19F) ainsi que de la souche R6, avirulente de type 2 ont été publiées dès 2001 (Tettelin et al. 2001)(Dopazo et al. 2001)(Hoskins et al. 2001). Désormais, le séquençage de nombreuses souches de pneumocoques a été réalisé, ce qui simplifie encore les recherches par approches globales et les études génétiques (e.g. (Hiller et al. 2007)).
13
A.2. La transformation génétique naturelle
A.2.1.
Historique
La capacité à acquérir de nouveaux caractères par transformation génétique naturelle a été démontrée dès 1928 chez S. pneumoniae par Frederick Griffith. Ce bactériologiste anglais a démontré qu’il pouvait y avoir un transfert de caractère entre deux types de pneumocoques (Griffith 1928). Il disposait alors de deux souches, l’une, virulente chez la souris et formant sur boites des colonies d’aspect lisse (nommée S, pour Smooth) ; et l’autre, avirulente, formant des colonies d’aspect rugueux (nommée R, pour Rough). L’observation de Griffith fut la suivante : une injection d’un mélange de bactéries R (non virulentes) et de bactéries S tuées par la chaleur à une souris induit la mort de celle-ci par septicémie et on retrouve dans son sang des bactéries S vivantes. Il en conclut que le caractère morphologique « S » porté par les bactéries S mortes avait été transmis aux bactéries R. Il appela ce phénomène « la transformation » et en déduit l’existence d’un « principe transformant » dans la suspension de bactéries tuées.
Il a fallu attendre 1944 pour que cette expérience soit reprise et que le « principe transformant » soit identifié comme étant l’ADN (Avery, MacLeod, & McCarty 1944). Avery, MacLeod et McCarthy ont testé les différents composants présents dans une suspension de bactéries S tuées pour leur capacité à transformer des bactéries R en S. Leurs résultats ont permis de montrer que l’ADN permettait le transfert de caractères d’une bactérie à l’autre et qu’il était donc le support de la transformation et de l’hérédité.
A.2.2.
Un phénomène répandu
S. pneumoniae est la première bactérie pour laquelle la transformation génétique
naturelle a été démontrée. Mais depuis, ce phénomène a pu être mis en évidence dans de nombreuses autres espèces.
Une revue, parue en 1994, dénombrait une quarantaine d’espèces naturellement transformables (Lorenz & Wackernagel 1994). Ces espèces, réparties dans tous les taxons (même chez les archaebactéries), peuvent être à Gram positif ou négatif et colonisent tout types de niches écologiques. Ainsi, l’autre bactérie modèle pour la transformation génétique naturelle chez les Gram + est une bactérie du sol, Bacillus subtilis. Parmi les bactéries du sol et de l’eau naturellement transformables, on trouve aussi par exemple la cyanobactérie
14
Anacystis nidulans (renommée Synechococcus PCC 6301), ainsi que Vibrio cholerae
(pathogène humain extrêmement virulent). La bactérie Ralstonia solanacearum, pathogène végétal, est aussi naturellement transformable. De nombreuses bactéries commensales ou pathogènes des animaux et de l’homme présentent également la capacité d’intégrer de l’ADN exogène (e.g. Mycobacterium smegmatis, Haemophilus influenzae, Helicobacter pilori,
Neisseria gonorrhoeae).
Le nombre d’espèces qualifiées de naturellement transformables augmente sans arrêt. Il s’agit en fait de trouver les conditions dans lesquelles une espèce en particulier pourra être transformée. Les progrès du séquençage ont permis une meilleure connaissance des génomes et les annotations font apparaitre la conservation des gènes nécessaires au processus de transformation génétique chez de très nombreuses espèces, il est donc tentant de penser que la capacité de transformation d’autres bactéries pourra être observée et démontrée (Claverys & Martin 2003).
A.2.3.
Rôle de la transformation génétique naturelle
Au contraire des autres mécanismes permettant les transferts horizontaux (conjugaison et transduction), la transformation naturelle est un mécanisme actif, pour lequel les protéines impliquées sont directement codées par le génome de la bactérie.
Deux hypothèses sont avancées pour expliquer l’apparition et surtout la conservation de ce mécanisme dans le monde bactérien : l’hypothèse nutritionnelle et l’hypothèse « génomique ». La première propose que l’ADN internalisé est utilisé comme substrat pour augmenter le pool de nucléotides dans la cellule alors que la deuxième donne à l’ADN entrant des rôles plus « nobles » (réparation de lésions du chromosome et acquisition de diversité génétique) (Redfield 2001)(Finkel & Kolter 2001)(Szöllosi, Derényi, & Vellai 2006) (Claverys, Prudhomme, & Martin 2006)(Johnsborg, Eldholm, & Håvarstein 2007).
L’hypothèse nutritionnelle est la plus critiquable. En effet, internaliser de l’ADN par la transformation génétique parait un processus très couteux du point de vue énergétique. Le déclenchement de la compétence (état physiologique dans lequel la bactérie est transformable,
cf. paragraphe suivant A.2.4) induit l’expression de nombreux gènes et la mise en place de
structures multiprotéiques complexes alors qu’il serait plus simple, par exemple, de sécréter une nucléase et d’importer directement les nucléotides libres. Par exemple, V. cholerae sécrète la nucléase Dns, ce qui lui permet effectivement de disposer d’une source de nucléotides, mais la production de cette DNase est justement inhibée lors du déclenchement de la compétence (Blokesch & Schoolnik 2008). De plus, chez S. pneumoniae, la compétence
15 est un phénomène très transitoire qui permet l’internalisation de seulement 200kb d’ADN exogène simple-brin (alors que l’autre brin est dégradé, et les nucléotides ainsi créés libérés dans le milieu de culture)(Lacks 1962)(Lacks, Greenberg, & Neuberger 1974)(Méjean & Claverys 1993). Pourquoi déclencher un processus aussi complexe pour un gain aussi limité ? Un autre point important est que, chez H. influenzae et N. gonorrhoeae par exemple, l’ADN internalisé doit contenir une séquence spécifique qui témoigne de son homologie avec le chromosome des bactéries transformées (Deich & Smith 1980)(Goodman & Scocca 1988). La aussi, il semble étrange d’imposer une telle limite, si l’ADN internalisé doit être catabolisé.
L’hypothèse « génomique » recouvre deux idées : l’ADN internalisé pourrait servir de matrice pour la réparation de dommages chromosomiques et/ou à l’acquisition de nouveaux gènes.
L’idée que l’ADN internalisé serait utilisé pour permettre une réparation du chromosome a été notamment proposée chez B. subtilis (Michod, Wojciechowski, & Hoelzer 1988). En effet, dans cette bactérie, quelques uns des gènes potentiellement impliqués dans la réparation des lésions de l’ADN sont induits dans les cellules compétentes (e.g. DinB, DinC et RecA) (Love, Lyle, & Yasbin 1985)(Lovett, Love, & Yasbin 1989)(Berka et al. 2002)(Claverys et al. 2006). Cette théorie présente toutefois plusieurs points faibles. Par exemple, il a été montré que la mitomycine C et les UV, des agents endommageant l’ADN, n’induisent pas la compétence chez B. subtilis (Redfield 1993). De plus, chez H. influenzae, après irradiation des cellules aux UV, la transformation par un fragment d’ADN obtenu par PCR permet le même gain de survie que la transformation par des chromosomes entiers (Mongold 1992). Ainsi, il semble que la compétence puisse être un bon moyen de lutter contre des dommages à l’ADN mais plus par l’intermédiaire des protéines qu’elle induit que par l’ADN potentiellement internalisé.
Finalement, l’hypothèse la plus tentante est donc que la transformation génétique soit apparue et ait perduré tout au long de l’évolution car elle permet un puissant brassage génétique. De nombreuses preuves de transferts horizontaux, vraisemblablement par transformation génétique, entre espèces proches ont pu être mises en évidence chez différentes bactéries transformables comme H. influenzae, N. meningitis, H. pylori et S.
pneumoniae (Kroll et al. 1998)(Suerbaum et al. 1998)(Muñoz et al. 1992)(Laible, Spratt, &
Hakenbeck 1991). Comme dit précédemment (cf. paragraphe A.1), chez S. pneumoniae, la résistance aux β-lactames est due à la variabilité des PBPs car certains des gènes codant ces protéines sont formés de différents blocs de nucléotides provenant de différents streptocoques par transfert horizontal (Dowson et al. 1989)(Hakenbeck et al. 2001)(Chi et al. 2007). Il
16 semble même que chez S. pneumoniae et les autres streptocoques apparentés (Streptococcus
mitis et Streptococcus oralis), la compétence ait évolué pour favoriser la diversité génétique.
En effet, lors de la compétence, le phénomène du fratricide/sobrinicide permet aux bactéries compétentes de lyser des bactéries non-compétentes de la même espèce/d’espèces proches (Guiral, Mitchell, et al. 2005)(Johnsborg et al. 2008). Ce mécanisme permet aux streptocoques compétents d'accéder à un pool de gènes commun qui est beaucoup plus important que leur propre génome. Cette propriété représente ainsi un avantage considérable pour ces bactéries pour echapper aux pressions de sélection auxquelles elles sont soumises lors de la vaccination et de traitements par antibiotiques.
A.2.4.
L’état de « compétence »
La capacité de transformation nécessite l’expression de nombreuses protéines permettant la liaison et l’internalisation d’ADN exogène ainsi que la prise en charge de l’ADN internalisé. On appelle « compétence » l’état physiologique dans lequel les cellules expriment de telles protéines. Dans la plupart des espèces bactériennes, la compétence est un phénomène transitoire très finement régulé. Les seules exceptions connues sont N.
gonorrhoeae et Neisseria meningitidis qui sont transformables tout au long de leur croissance
en laboratoire car elles expriment les protéines de compétence de façon constitutive (Sparling 1966)(Johnsborg et al. 2007).
Hormis ces deux espèces, les bactéries expriment les protéines nécessaires à la transformation génétique à un moment donné de leur croissance. Par exemple, des bactéries appartenant aux genres Deinococcus, Acinetobacter, Synechococcus et Chlorobium sont compétentes tout au long de la phase exponentielle de croissance alors que B. subtilis déclenche la compétence à la transition phase exponentielle/phase stationnaire (Moseley & Setlow 1968) (Essich, Stevens, & Porter 1990)(Palmen et al. 1993)(Dubnau 1991) (Johnsborg et al. 2007). S. pneumoniae n’est compétente que durant une quarantaine de minutes en début de phase exponentielle de croissance (Pakula & Walczak 1963)(Tomasz & Hotchkiss 1964)(voir paragraphe A.3 pour une description plus détaillée de la régulation de la compétence chez S. pneumoniae).
Le moment du déclenchement et la durée de la compétence ne sont pas les seuls paramètres variables entre les différentes espèces. Par exemple, la proportion de cellules compétentes au sein d’une population varie de 5 à 10% chez B. subtilis à environ 100% chez
S. pneumoniae (Singh & Pitale 1968)(Javor & Tomasz 1968). De plus, les conditions
physico-17 chimiques et nutritionnels du milieu de culture peuvent influer sur le développement de la compétence.
A.3. La compétence pour la transformation génétique chez
Streptococcus pneumoniae
Comme signalé dans les paragraphes précédents, la compétence est un phénomène complexe, génétiquement régulé et pour lequel chaque espèce bactérienne présente des spécificités. Cette partie de l’introduction sera donc consacrée à la compétence chez S.
pneumoniae, l’organisme sur lequel j’ai effectué tous mes travaux de thèse.
A.3.1.
La compétence et sa régulation
A.3.1.1. Conditions d’induction de la compétence
In vitro, dans un milieu bien défini, une culture de S. pneumoniae peut déclencher la
compétence spontanément de façon coordonnée pour toute la population (Pakula & Walczak 1963)(Tomasz & Hotchkiss 1964). Cette état physiologique dure une quarantaine de minutes et a lieu en début de phase exponentielle de croissance.
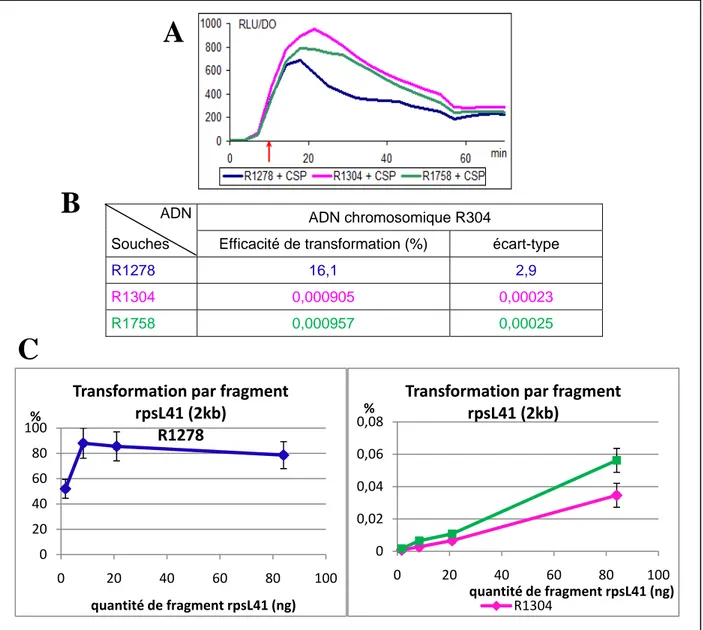
Si l’on prélève des aliquots de culture à différents temps et qu’on les teste pour leur capacité à transformer, la courbe représentant le pourcentage de transformants au cours du temps montre un déclenchement et un arrêt très rapide de la capacité de transformation. On parle ainsi de « pic de compétence » (cf. Figure 1).
La synchronisation de la population est en fait due à la sécrétion d’un petit peptide, nommé CSP (pour Competence Stimulating Peptide) qui diffuse dans le milieu et permet le déclenchement de la compétence.
18
Figure 1 : Développement de la compétence chez S. pneumoniae.
Une culture d’une souche de S. pneumoniae sensible à la streptomycine (SmS) est réalisée en milieu C+Y, et la croissance est suivie par la lecture de la DO à 492nm (carrés roses). A différents temps, des aliquots sont prélevés et mis en contact avec de l’ADN donneur portant un marqueur de résistance à la streptomycine (SmR). La numération des bactéries totales et des bactéries SmR permet de déterminer le pourcentage de transformation (triangles bleus)
A.3.1.2. Le CSP et l’opéron comAB
Dès les années 60, des travaux montraient qu’un facteur protéique, nommé CF (pour Competence Factor), était présent dans le surnageant de cultures compétentes, et que ce surnageant pouvait être utilisé pour induire la compétence d’une autre culture (Pakula & Walczak 1963)(Tomasz & Hotchkiss 1964).
Pendant longtemps, aucune technique de purification n’a permis d’isoler le CF. L’identification du CF et du gène le codant a été permise grâce à différentes découvertes. En 1984, un mutant ne devenant pas compétent spontanément, ne produisant pas de CF mais restant capable d’y répondre a été isolé (Morrison et al. 1984). Dans cette souche le locus muté contient 2 gènes : comA et comB (Chandler & Morrison 1987)(Chandler & Morrison 1988). La séquence de comA a montré que son produit était une protéine de la famille des transporteurs ABC (Hui & Morrison 1991). Cette famille de protéines couple l’hydrolyse de l’ATP avec l’import ou l’export de divers composants (ions, sucres, antibiotiques, peptides, protéines, acides aminés, etc…) (pour revue voir (Davidson et al. 2008)). Les ABC-transporteurs dédiés aux bactériocines permettent le clivage d’un pré-peptide dit « à motif double Glycine (GG) » et son export dans le milieu extérieur (Håvarstein, Diep, & Nes 1995). L’homologie entre comAB et d’autres loci codant des systèmes ABC dédiés à l’export de bactériocines, a permis de penser que ComAB pourrait, de même, cliver et exporter un petit peptide (Hui, Zhou, & Morrison 1995). Cependant, le locus codant la/les bactériocine(s) est habituellement proche de l’opéron codant le système d’export dédié (Stoddard et al. 1992), ce
19 qui n’est pas le cas pour comAB (Hui et al. 1995). Grâce à l’adaptation d’un protocole utilisé pour la purification de bactériocines, un peptide de 17 acides aminés a été purifié dans le surnageant de cellules compétentes. Ce peptide, nommé CSP, a été séquencé et cela a permis, par génétique inverse, de trouver le gène comC codant le pré-CSP (Håvarstein, Coomaraswamy, & Morrison 1995). Il s’est aussi avéré que le CSP n’était pas modifié post-traductionnellement puisque un peptide synthétique est aussi actif que le CSP purifié du surnageant de cultures compétentes (Håvarstein et al. 1995).
Le fait de pouvoir synthétiser de façon chimique le CSP a grandement facilité son utilisation. Ainsi grâce à la possibilité de déclencher la compétence et donc la transformation dans 100% des cellules par la simple adjonction d’un petit peptide synthétique, S. pneumoniae a pu être utilisé comme outil génétique puissant.
A.3.1.3. L’opéron comCDE
Le gène comC code pour le pré-CSP, un peptide de 41 acides aminés (cf. Figure 2) (Pestova, Håvarstein, & Morrison 1996). Les 22 premiers acides aminés (Nter) sont suivis d’un motif double glycine spécifique des peptides clivés et exportés par les transporteurs du type ABC. On trouve ensuite les 17 acides aminés qui composent le CSP mature. Ces observations ont confirmé l’hypothèse selon laquelle le produit du gène comC serait exporté et maturé sous la forme du CSP par les protéines ComA/ComB.
Figure 2 : Séquence protéique du produit du gène comC.
L’étude du locus du gène comC a permis d’identifier deux autres gènes nommés comD et comE. Ces deux nouveaux gènes codent respectivement pour une histidine kinase et un régulateur de réponse (Pestova et al. 1996). Ces 3 gènes sont organisés en opéron (comCDE) (Alloing et al. 1998)
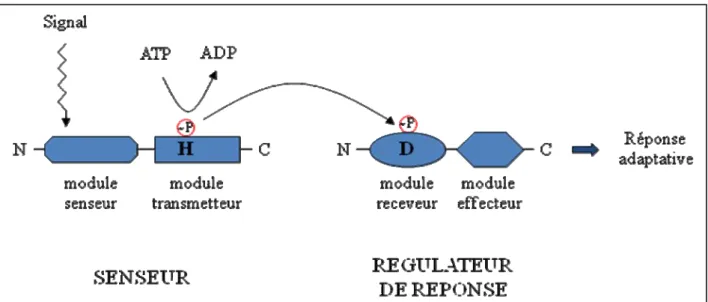
comD et comE forment ce que l’on appelle un système à deux-composants (ou TCS,
20 régulateur transcriptionnel (cf. Figure 3). Dans un TCS, le récepteur est une protéine kinase, généralement membranaire, qui en présence de son signal, s’autophosphoryle sur une histidine (H), puis transmet le phosphate sur un résidu aspartate (D) du régulateur de réponse (cf. Figure 3). Celui-ci peut alors moduler la transcription de gènes cibles par action directe sur leurs promoteurs (pour revue, voir (Parkinson & Kofoid 1992) ; (Stock, Robinson, & Goudreau 2000) ; (Hoch 2000)). Les TCS sont très répandus dans le monde bactérien et permettent la perception de l’environnement et le développement de réponse(s) adaptée(s). Il a donc été supposé qu’un tel système pouvait jouer un rôle dans la détection et la réponse au CSP. Le fait que des mutants perte de fonction des gènes comD et comE ne peuvent plus répondre au CSP confirme cette hypothèse (Pestova et al. 1996).
Figure 3 : Transduction du signal au sein d’un système à deux composants classique.
Aucune expérience n’a pu mettre en évidence d’interaction directe entre ComD et le CSP, mais de nombreux arguments permettent de postuler que ComD est bien le récepteur du CSP. Chez S. pneumoniae, il existe une variabilité importante du locus comCDE, mais on peut tout de même dissocier principalement 2 types d’allèles pour le gène comD (comD1 et
comD2) et pour le gène comC (comC1 et comC2) (Pozzi et al. 1996)(Ramirez, Morrison, &
Tomasz 1997)(Håvarstein, Hakenbeck, & Gaustad 1997)(Whatmore, Barcus, & Dowson 1999)(Johnsborg, Godager, & Nes 2004). Récemment, des expériences ont effectivement pu démontrer que le modèle de reconnaissance CSP/ComD, déjà illustré chez Streptococcus
gordonii en 1996 (Håvarstein et al. 1996), est conservé chez S. pneumoniae (Johnsborg et al.
2006). Il a été montré que le CSP1 ne peut activer efficacement la compétence que d’une souche possédant l’allèle comD1 (Johnsborg et al. 2006). Les prédictions de structure pour les protéines ComD définissent 7 segments transmembranaires. ComD1 et ComD2 diffèrent par
21 des changements d’acides aminés dans la partie N-terminale de la protéine, prédite comme formant une boucle extra-cytoplasmique dont on pense qu’elle permet la formation du site de reconnaissance de CSP (Johnsborg et al. 2004).
En tant que régulateur de réponse du TCS ComD/E, ComE doit être activé (phosphorylé) par ComD et agir sur la transcription de gène cible. Effectivement, ComE se fixe en amont des opérons comCDE, comAB, et d’un gène codant pour un ABC-transporteur de fonction inconnue, sur une séquence nucléotidique constituée de deux segments de 9 paires de bases imparfaitement répétées et espacés de 12 nucléotides (boite ComE ou ComE-box) (Ween, Gaustad, & Håvarstein 1999). Cependant, l’état de phosphorylation de ComE lors de sa fixation n’est pas connu. En revanche, il a été montré que le CSP induit effectivement la transcription des opérons comCDE et comAB, il semble donc bien que ComE soit le régulateur de réponse responsable de l’induction de la compétence (Pestova et al. 1996)(Alloing et al. 1998).
A.3.1.4. La boucle auto-catalytique
Le déclenchement et la synchronisation de la compétence chez S. pneumoniae fait donc intervenir deux opérons : comCDE et comAB (cf. Figure 4).
Le gène comC code pour le précurseur du CSP (pré-CSP) qui est clivé, puis libéré dans le milieu sous forme de CSP mature, grâce au système d’export ComAB. Après interaction avec le CSP, le senseur ComD s’autophosphoryle puis transfère son phosphate au régulateur de réponse ComE. Celui-ci peut alors activer la transcription des 2 opérons comAB et comCDE en se fixant sur les ComE-box situées en amont de leurs promoteurs. Il y a ainsi mise en place d’une boucle auto-catalytique qui permet l’augmentation de la concentration en CSP dans le milieu et donc le basculement en compétence de toute la population bactérienne.
(Pour une analyse plus détaillée des phénomènes qui influent sur la mise en place de cette boucle catalytique, voir paragraphe A.3.3)
22
ComD
ComE
comCDE
(ComC) pre-CSPComAB
CSPcomAB
ComE
~P
Figure 4 : La boucle auto-catalytique d'induction de la compétence.
A.3.2.
Le développement de la compétence
En plus des deux opérons détaillés précédemment, de nombreux gènes sont induits lors du développement de la compétence, c’est ce que l’on nomme le « régulon compétence ».
Historiquement, deux classes de gènes avaient été décrites : les gènes précoces dont l’expression était induite rapidement après l’ajout de CSP, et les gènes tardifs dont l’expression était retardée.
En effet, l’un des premiers gènes étudiés du fait de son rôle essentiel lors de la recombinaison homologue a été recA (Martin et al. 1995)(Pearce et al. 1995). Chez, S.
pneumoniae, il fait partie de l’opéron cinA-recA-dinF-lytA (Mortier-Barrière et al. 1998) où lytA code pour l’autolysine majeure de S. pneumoniae (López et al. 1997) et où cinA et dinF
sont de fonction inconnue. La comparaison des cinétiques d’expression de recA et de
comCDE après ajout de CSP a permis de révéler un retard d’expression pour recA (Alloing et
al. 1998). C’est pourquoi comCDE a été nommé opéron précoce et cinA-recA-dinF-lytA opéron tardif.
Plusieurs études transcriptomiques ont confirmé l’existence de ces deux classes de gènes et ont permis d’identifier une nouvelle classe, les gènes retardés, dont l’expression est encore plus tardive (Peterson et al. 2000)(Rimini et al. 2000)(Dagkessamanskaia et al. 2004)(Peterson et al. 2004). Parmi les 105 et 124 gènes respectivement révélés comme induits en compétence par les deux dernières analyses, 91 gènes sont communs aux deux études (~80% de recouvrement, ~5% du génome de S. pneumoniae). En revanche, les gènes trouvés
23 comme réprimés durant la compétence diffèrent d’une étude à l’autre, c’est pourquoi ils ne seront pas détaillés par la suite.
Les 91 gènes induits sont répartis en 17 gènes précoces, 60 gènes tardifs et 14 gènes retardés (Guiral, Dagkessamanskaia, et al. 2005).
A.3.2.1. Les gènes précoces
La transcription des gènes précoces est induite directement après l’ajout de CSP, atteint son maximum ~5 min plus tard puis s’éteint rapidemment. L’expression de ces gènes dépend de la fixation du régulateur de réponse ComE sur les boites ComE présentes en amont de leurs promoteurs. Le transcriptome réalisé en 2004 par Peterson et ses collaborateurs a identifié 13 opérons comme « précoces » (soit 24 gènes, cf. Figure 5) et parmi ceux-ci, 10 présentaient une boite ComE, ce qui a permis de définir une séquence consensus (cf. Figure 5) (Peterson et al. 2004).
Parmi ces gènes précoces, on retrouve comAB et comCDE impliqués dans la boucle auto-catalytique décrite précédemment, des gènes codant pour des protéines de fonctions prédites diverses (e.g. SP1717-1716 code un ABC-transporteur), mais également des gènes de fonction inconnue.
Deux autres gènes précoces sont extrêmement importants pour le développement de la compétence puisqu’ils sont nécessaires à l’expression des gènes tardifs. Il s’agit de comX (qui est présent en 2 copies, comX1 et comX2, chez S. pneumoniae) et comW.
24
Figure 5 : Les gènes précoces et leurs boites ComE.
(adapté de (Peterson et al. 2004)) Les numéros des gènes proviennent de la séquence de la souche virulente de type 4 (TIGR4, (Tettelin et al. 2001)). A : Les gènes requis pour la transformation chromosomique sont représentés en rouge, les gènes non requis pour le développement de la compétence sont en vert, les gènes essentiels ou non analysés sont en gris. Les flèches noires signalent la présence d’un promoteur et les traits verticaux noirs celle d’un terminateur. DR signifie Direct Repeat et signale la présence d’une boite ComE dont la séquence est donnée en B. B : Alignement de séquences des boites ComE putatives trouvées en amont des gènes précoces et séquence consensus (surlignée en jaune).
comX1 et comX2 codent la protéine ComX ou σX, un facteur sigma alternatif qui, en se liant au core de l’ADN polymérase, permet la liaison du complexe transcriptionnel en amont des gènes tardifs par la reconnaissance d’une séquence spécifique nommée boite cin ou boite
com (cin-box ou com-box) (Campbell, Choi, & Masure 1998)(Pestova & Morrison
1998)(Claverys & Martin 1998)(Lee & Morrison 1999).
Des expériences réalisées en 2003 ont montré que l’expression de ComX seul ne permet pas d’induire la transcription des gènes tardifs de compétence sauf si on le sur-exprime. Cela a conduit les auteurs à proposer que ComX subissait une régulation post-transcriptionnelle (Luo, Li, & Morrison 2003). Dans la même équipe, l’année suivante, les expériences de mutagénèse ciblée réalisées après l’étude du transcriptome ont montré que
comW était essentiel au développement de la compétence (Peterson et al. 2004). Les auteurs
ont alors plus précisément étudié ce gène et ont montré qu’il code une protéine de 80 acides
A
S P0014
25 aminés, ComW, qui, exprimée en même temps que ComX, permet d’induire la compétence indépendamment de la boucle auto-catalytique (Luo, Li, & Morrison 2004). Des études complémentaires ont permis de démontrer que ComW active ComX en stabilisant son interaction avec l’ARN polymérase et en le protégeant de la dégradation par les protéases Clp (Sung & Morrison 2005)(Piotrowski, Luo, & Morrison 2009).
A.3.2.2. Les gènes tardifs
ComX et ComW permettent la transcription de tous les gènes tardifs de compétence. La transcription de ces gènes débute 5 min après l’ajout du CSP, atteint son maximum entre 10 et 15 min puis diminue. Le transcriptome réalisé dans l’équipe de Morrison en 2004 a révélé 23 opérons tardifs (81 gènes) dont 19 présentent une cin-box (cf. Figure 6) (Peterson et al. 2004).
Parmi les gènes tardifs identifiés, on peut définir différentes classes en fonction du rôle qu’ils jouent dans la physiologie bactérienne.
De nombreux gènes sont directement impliqués dans le mécanisme de la transformation génétique lui-même, depuis l’entrée de l’ADN exogène jusqu’à son intégration par recombinaison homologue dans le chromosome de la bactérie (voir paragraphe A.4 pour plus de détails).
Certains gènes tardifs semblent être impliqués dans la virulence de S. pneumoniae. En effet, il avait été montré que des mutants ne pouvant pas devenir compétents présentaient une atténuation de la virulence dans la souris mais le lien entre compétence et virulence n’était pas compris (Lau et al. 2001)(Bartilson et al. 2001). En 2005, la découverte du phénomène de fratricide a permis de penser que la diminution de la virulence vient de la perte de cette propriété (cf. paragraphe A.2.3) (Guiral, Mitchell, et al. 2005). Dans cette étude, il est montré que le fratricide est dépendant de différentes protéines codées par des gènes tardifs comme CibAB une bactériocine à deux peptides ainsi que CibC son facteur d’immunité, CbpD une amidase potentielle et LytA l’autolysine majeure de S. pneumoniae. Le lysozyme LytC est également impliqué dans ce mécanisme, mais est produit de façon constitutive (García et al. 1999). Ainsi, lors du fratricide, la lyse des cellules libère de nombreux facteurs de virulence. Par exemple, la pneumolysine est une protéine cytoplasmique de la famille des cytolysines dépendantes du cholestérol (comme la listériolysine de Listeria monocytogenes) qui peut former des pores dans les membranes cellulaires. Les acides téichoïques et lipotéichoïques induisent une forte réaction inflammatoire (Kengatharan et al. 1998). Enfin, l’ADN chromosomique bactérien peut être à l’origine de chocs septiques, de production de cytokines
26 induisant une réponse inflammatoire et d’une stimulation de production de lymphocytes (Krieg et al. 1995)(Sparwasser et al. 1997).
Figure 6 : Les gènes tardifs et leurs boites cin.
(adapté de (Peterson et al. 2004)) Les numéros des gènes proviennent de la séquence de la souche virulente de type 4 (TIGR4, (Tettelin et al. 2001)). A : Le code couleur est le même que pour la Figure 5, les cin-box sont notées C. B : Alignement de séquences des cin-box putatives trouvées en amont des gènes tardifs et séquence consensus (surlignée en jaune).
Parmi les gènes tardifs de compétence identifiés par les différentes études transcriptomiques, un certains nombre sont encore de fonction inconnue. On peut donc penser que, outre le fratricide, la virulence et la transformation génétique, le régulon compétence pourrait englober d’autres fonctions.
A
27
A.3.2.3. Les gènes retardés
Les 14 autres gènes induits à la compétence ont une cinétique dite retardée. Leur expression augmente 10 à 15 min après l’ajout du CSP et n’est pas directement dépendante de l’expression de ComX (cf. Figure 7) (Peterson et al. 2004). Ils appartiennent aux régulons de réponse au stress CiaR, HrcA et CtsR (Dagkessamanskaia et al. 2004)(Peterson et al. 2004).
Ces régulons de stress contrôlent la synthèse des protéines chaperonnes assurant le contrôle de la conformation des protéines néo-synthétisées (système GroES/GroEL, chaperonnes DnaJ/DnaK/GrpE, pour revue voir (Hartl & Hayer-Hartl 2002)). Ils contrôlent aussi la synthèse de protéases assurant la dégradation des protéines mal conformées (protéases Clp, pour revue voir (Gottesman & Maurizi 1992) ; HtrA, une sérine protéase de surface, pour revue voir (Pallen & Wren 1997)). Les mécanismes de régulation de ces gènes retardés ne sont pas encore compris. L’induction de ces gènes doit vraisemblablement répondre à un stress interne dû à la différenciation des cellules dans leur état de compétence. En effet, la synthèse de protéines chaperonnes et de protéases pourraient être nécessaires pour gérer l’abondance de nouvelles protéines synthétisées lors de l’induction de la compétence (Peterson et al. 2004). Cette hypothèse est étayée par le fait que des cultures compétentes mutantes pour le gène ciaR sont très affectées en croissance (Dagkessamanskaia et al. 2004).
Figure 7 : Les gènes retardés.
(tiré de (Peterson et al. 2004)) Les numéros des gènes proviennent de la séquence de la souche virulente de type 4 (TIGR4, (Tettelin et al. 2001)). Le code couleur est le même que pour la Figure 5.
A.3.2.4. La sortie de l’état de compétence
Si le régulon compétence est aujourd’hui bien défini grâce, notamment, aux différentes études transcriptomiques, les mécanismes qui permettent l’arrêt de la compétence sont encore mal compris.
Par exemple, il a été constaté que les cellules activées étaient incapables de répondre au CSP pendant environ 1,5 génération après l’induction. Il a été proposé que les cellules étaient « aveugles au CSP » (Morrison 1997), suggérant l’existence d’un phénomène actif de fermeture de la compétence.
28 L’arrêt de la compétence nécessite, d’une part l’arrêt de la transcription des gènes tardifs et d’autre part, l’arrêt de celle des gènes précoces.
La fin de la transcription des gènes tardifs peut s’expliquer par la faible durée de vie du facteur sigma alternatif ComX (Luo & Morrison 2003). Cependant, la raison de l’instabilité de ComX reste encore à définir. Il a également été observé une induction de la transcription du gène rpoD (codant pour le facteur sigma de ménage) de manière simultanée avec les gènes tardifs (Peterson et al. 2000)(Dagkessamanskaia et al. 2004)(Peterson et al. 2004). Il est proposé que RpoD puisse entrer en compétition avec ComX pour la liaison à l’ARN polymérase, ce qui serait à l’origine de l’extinction de la transcription des gènes tardifs (Claverys & Håvarstein 2002).
L’arrêt de la transcription des gènes précoces et l’incapacité des cellules activées à répondre au CSP ne peuvent pas être expliqués par la dégradation du régulateur de réponse ComE. En effet, ComE est stable pendant au moins de 80 min après le début de l’activation (Ween et al. 1999). De plus, lors de l’analyse du transcriptome réalisée dans une souche
comX-, il a été observé que la fermeture de la transcription des gènes précoces a bien lieu, même s’il reste un niveau résiduel de transcription d’environ un cinquième du niveau maximum d’induction (Peterson et al. 2000). Donc, un gène tardif joue peut-être un rôle minime dans l’arrêt de transcription des gènes précoces, mais la majeure partie de l’inhibition est réalisée par le produit d’un gène précoce. Une hypothèse proposée pour expliquer ces observations est que l’activité de ComE pourrait être régulée par la variation de son niveau de phosphorylation ou par sa multimérisation (Alloing et al. 1998)(Martin et al. 2000). Cette hypothèse est appuyée par le comportement d’un mutant ponctuel de ComE (ComER120S) dont la fermeture de la compétence est perturbée. La mutation R120S affecte un résidu localisé à la limite du domaine receveur et de la région charnière de ce régulateur de réponse. Cette mutation pourrait donc déstabiliser la protéine et ainsi affecter son état de phosphorylation (Martin et al., 2000). La protéine mutante ComER120S pourrait alors être plus stable sous sa forme phosphorylée. Elle pourrait ainsi continuer d’activer la transcription des gènes précoces et empêcher une fermeture normale de la compétence. Il a aussi été proposé que cette mutation affecterait la capacité de dimérisation de ComE ce qui provoquerait une stabilité plus grande et/ou une activité de régulateur transcriptionnel plus efficace. Toutefois, l’état de phosphorylation et d’oligomérisation de ComE au cours de la compétence et les mécanismes de régulation de cet état restent pour l’instant inconnus.
29
A.3.3.
L’initiation de l’état de compétence
Comme expliqué précédemment (paragraphe A.3.1), le déclenchement de la compétence nécessite la mise en place d’une boucle autocalytique faisant intervenir les opérons comAB et comCDE. Ces opérons étant sous le contrôle de ComE, la question qui se pose est : comment s’effectue le démarrage de la boucle autocatalytique ?
A.3.3.1. L’expression basale
Pour répondre à cette question, 2 modèles ont été proposés.
Selon le premier modèle, le CSP s’accumulerait de façon passive grâce à une fuite transcriptionnelle du gène codant l’ARNtArg qui se situe directement en amont de comCDE (Pestova et al. 1996). Le CSP serait alors un peptide rendant compte d’une mesure de la densité cellulaire dans la culture, permettant ainsi un mécanisme de quorum-sensing (Morrison 1997). Un autre modèle postule que le CSP serait une phéromone ou alarmone dont la production pourrait être temporairement augmentée en réponse aux changements de conditions environnementales (i.e. modification des paramètres nutritionnels, physiques ou chimiques du milieu) (Claverys et al. 2000)(Martin et al. 2000).
En ce qui concerne ce dernier modèle, il est connu depuis longtemps que certains aspects du métabolisme ou de l'environnement influent sur l'initiation de la compétence. Notamment, le milieu doit contenir du CaCl2, de la BSA (Bovine Serum Albumin) et une concentration élevée en phosphate (pour revue, voir (Håvarstein & Morrison 1999)). De plus, un faible pH initial est inhibiteur de la compétence (Chen & Morrison 1987). Néanmoins, le rôle exact de ces différents facteurs n’a jamais été bien défini.
Enfin, l’étude d’un mutant obl a permis de montrer qu’une carence en acides aminés influe sur le déclenchement de la compétence (Alloing et al. 1998). Ce mutant est en fait muté pour les gènes amiA, aliA et aliB, codant trois protéines du système Obl (Oligopeptide-Binding Lipoproteins), un système de transport d’oligopeptides et d’acides aminés (Alloing, de Philip, & Claverys 1994). Comparativement à une souche sauvage, la compétence se déclenche de façon spontanée à une densité optique 50 fois plus faible dans le mutant obl (Alloing et al. 1998). Ce phénotype de déclenchement précoce de la compétence a pu être reproduit dans un mutant possédant deux copies du gène comC dans son génome. Cela a conduit les auteurs à émettre l’hypothèse qu’une augmentation de l’expression de comC était peut-être à l’origine du phénotype du mutant obl (Alloing et al. 1998).
30 Ces travaux tendent à montrer qu’il existe effectivement une régulation de l’expression basale des opérons comCDE et comAB et donc que la compétence n’est pas un réel mécanisme de quorum-sensing.
Pour tenter de confirmer cette hypothèse, une recherche génétique de mutants dérégulant l’expression de l’opéron comCDE a été réalisée dans l’équipe de Jean-Pierre Claverys (Martin et al. 2000).
A.3.3.2. Les mutants dérégulés
Deux approches visant à obtenir des mutants dits cup (pour competence up) ont été réalisées au laboratoire : une PCR mutagène a été effectuée sur le locus comCDE ; et une mutagénèse in vitro par insertion aléatoire de mini-transposons mariner dans le génome a été effectuée (Martin et al. 2000). Une souche possède un phénotype cup, si elle induit la compétence dans des conditions d’inhibition pour une souche sauvage. Un crible de sélection a permis d’identifier 8 mutations ponctuelles situées dans le locus comCDE et 52 insertions
mariner indépendantes dans divers loci au sein du génome, aboutissant à des phénotypes cup
(Martin et al. 2000).
L’étude des différents mutants ponctuels a permis de montrer que des modifications de la quantité de protéines ComCDE produites, de leur capacité à interagir et à être actifs pouvaient avoir des répercutions importantes sur la cinétique et l’intensité de développement de la compétence (Martin et al. 2000).
Parmi les insertions mariner induisant des phénotypes cup, on trouve majoritairement des insertions localisées dans deux loci, comAB et ciaRH. Par exemple, 2 insertions cup ont été localisées en amont de comAB. Celles-ci sont co-transcrites avec comAB et provoquent ainsi une augmentation de la transcription de cet opéron. Ceci indique que lors de l’initiation du développement de la compétence, l’export du CSP est limitant et souligne l’importance de
comAB dans la régulation de la compétence.
Différentes insertions mariner sont situées dans l’opéron ciaRH. Celui-ci code un TCS dont CiaR et CiaH sont respectivement le régulateur de réponse et le senseur. On ne connait pas les signaux activateurs de CiaRH mais on pense généralement que ce système permet l’intégration de stress dans la paroi et/ou la membrane cellulaire. De nombreuses études ont montré une interconnexion entre les régulons compétence et CiaRH mais l’origine de ce lien reste incompris. Par exemple, il avait déjà été montré qu’une souche ciaR- développait la compétence dans des conditions non permissives pour une souche sauvage (Giammarinaro, Sicard, & Gasc 1999). De plus, le fait que l’inactivation de ciaRH provoque la surexpression
31 de comCDE a permis de proposer que CiaR régulait négativement cet opéron (Echenique, Chapuy-Regaud, & Trombe 2000). L’étude par analyse transcriptomique du régulon CiaR a confirmé cette hypothèse (Sebert et al. 2002)(Mascher et al. 2003). Enfin, le régulon CiaR est induit lors de la compétence, et des cellules ciaR- ont un problème de viabilité si elles deviennent compétentes (Dagkessamanskaia et al. 2004).
Enfin, d’autres insertions mariner induisant un phénotype cup ont été retrouvées dans des gènes conduisant à des altérations des pools d’acides aminés ou de nucléotides, à des modifications de la paroi ou de la membrane, etc… (Marc Prudhomme, communication personnelle)(Claverys & Håvarstein 2002).
Ainsi, différentes mutations pouvant être considérées comme induisant ou reproduisant des signaux de stress induisent une régulation positive de l’opéron comCDE (Claverys & Håvarstein 2002). Il a donc été suggéré que, chez S. pneumoniae, la compétence pourrait permettre une réponse adaptative à des changements environnementaux et que le CSP pourrait agir comme une alarmone (Martin et al. 2000)(Claverys & Håvarstein 2002).
A.3.4.
La compétence et la réponse au stress
Contre les agressions de l’environnement, les bactéries ont développé de multiples systèmes de réparation. Un des mieux caractérisé est le système SOS qui permet la réparation des lésions présentes sur l’ADN par trois mécanismes : élimination des lésions par excision, réparation par recombinaison post-réplicative ou synthèse translésionnelle (mutagenèse SOS) (Radman 1975)(pour revue, voir (Walker 1995) ; (Janion 2008)). Il a été découvert chez E.
coli mais est conservé chez de nombreuses espèces bactériennes (Erill, Campoy, & Barbé
2007).
Chez E. coli, la réponse SOS est dépendante de la protéine RecA. A l’état basal, le répresseur LexA empêche la transcription des gènes de la réponse SOS. Mais, lors de dommages sur l’ADN, de l’ADN simple-brin (ADNsb) se forme. La protéine RecA polymérise sur cet ADNsb, formant un nucléo-filament, elle est alors notée RecA*. RecA* catalyse alors l’auto-clivage du répresseur LexA et les gènes du régulon din (pour damage inductible) peuvent être exprimés. Les produits de ces gènes permettent une réparation des dommages (pour revue voir (Janion 2008)).
Bien que le système SOS soit associé à la réponse directe aux dommages à l’ADN, il est apparu plus récemment qu’il permettait une réponse intégrée pour un grand nombre de stress. En effet, les carences métaboliques, les stress environnementaux (stress oxydatif,