THÉSE
en vue de l'obtention du
DOCTORAT DE L'UNIVERSITÉ DE TOULOUSE délivré par l'Université Toulouse III - Paul Sabatier
Ecole doctorale : science de la matière Spécialité : Chimie-Biologie-Santé présentée et soutenue par
Gabrielle CHATAIGNÉ
le 23 novembre 2007Titre :
Détermination structurale des lipopolysaccharides
de surface chez Sinorhizobium.
Directeur de thèse : Dr. Véréna POINSOT
JURY
M. F. COUDERC Professeur à l'Université Paul Sabatier de Toulouse Président M. J.-C. MICHALSKI Directeur de recherche Université des Sciences et Technologies de Lille Rapporteur Mme M.-C. RALET Chargée de recherche à l'unité BIA de l'INRA de Nantes Rapporteur M. G. PUZO Directeur de recherche à l'Université Paul Sabatier de Toulouse Examinateur V. POINSOT Chargée de recherche à l'université Paul Sabatier (IMRCP) Directeur de thèse
Recherches effectuées au
Laboratoire des Interactions Moléculaires et Réactivités Chimiques et Photochimiques 118 route de Narbonne 31062 Toulouse CEDEX 9
2 UNIVERSITE PAUL SABATIER
Thèse d'Université, spécialité Chimie-Biologie-Santé Date de soutenance
Nom du candidat CHATAIGNÉ Gabrielle
Titre en Français Détermination structurale des liposaccharides de surface de Sinorhizobium. Résumé en Français
Pour survivre dans des sols pauvres en nutriments, les plantes de la famille des légumineuses sont capables d'entrer en symbiose avec des bactéries de la famille des rhizobiaceae. Les polysaccharides de surface de ces bactéries jouent un rôle crucial dans l'établissement du processus d'infection. Sinorhizobium meliloti 1021 porte à sa surface trois types de polysaccharide, les lipopolysaccharides (LPS), les polysaccharides capsulaires (KPS) et les exopolysaccharides (EPS). Ses KPS sont des polymères de Kdo de petite taille rattachés à une ancre lipidique. Nous avons étudié la structure des KPS d'un mutant de la souche Sm1021 déficient en exopolysaccharides et produisant des KPS de haute masse molaire. Son activité symbiotique a été déterminée. Les KPS et les LPS possèdent des propriétés physico-chimiques communes (même taille, même charge et même balance hydrophobe-hydrophile). Pour analyser les constituants de ce mélange sans purification préalable lourde et en utilisant un minimum d'échantillon, nous avons mis au point un couplage entre une chromatographie liquide à haute performance échangeuse d'anion (HPAEC), efficace dans la séparation des oligosaccharides, et un spectromètre de masse (ESI-Q-ToF). Nous avons ainsi pu déterminer la structure des KPS de haute masse molaire synthétisés par le mutant mais aussi démontrer que la présence d'une ancre lipidique est, sur les KPS, très commune chez les Sinorhizobia. Nous avons aussi recueilli de nouvelles données concernant la caractérisation structurale de l'oligosaccharide de cœur des LPS de Sm1021 et des lipides A de deux mutants AcpXL et LpxXL.
Liste des mots clés : KPS, LPS, Sinorhizobium, HPAEC-PAD-MS, ESI-Q-ToF Nom et adresse du ou des laboratoires
Laboratoire des Interactions Moléculaires et Réactivités Chimiques et Photochimiques 118 route de Narbonne 31062 Toulouse CEDEX 9
3 Titre en Anglais: Structural determination of surface liposaccharides of Sinorhizobium
Abstract
Soil bacteria belonging to the rhizobia are able - when the rhizosphere is starved in nitrogen - to infect legume roots and to establish a nitrogen-fixing symbiosis. Surface polysaccharide appeared to be essential to the symbiosis establishing. S. meliloti produces cell-surface polysaccharides including lipopolysaccharides (LPS), capsular polysaccharides (KPS) and exopolysaccharides (EPS). Sinorhizobium meliloti strain 1021 produces a low-molecular-mass capsular polysaccharide that is a Kdopolymer harboring a phospholipid anchor. Exopolysaccharide-deficient mutant of Sm1021 affected in the KPS size (increased of polymerization level) has been studied. we performed a structural analysis of the K polysaccharides using NMR, ESI-MS and GC-MS. LPS and KPS were impossible to separate (same size, charge and hydrophilic/lipophilic balance). To determine the complexity oligosaccharide composition of biological samples and to use the smallest possible quantity of the surface polysaccharide extract, we decided to develope on-line coupling of High performance anion exchange chromatography (HPAEC), well established technique for determining underivatized carbohydrates, and mass spectrometry. In this thesis are presented new data concerning the structure of LPS core oligasaccharide and lipidA of the Sinorhizobium meliloti strain 2011 and mutant AcpXL and LpxXL.
Key words : KPS, LPS, Sinorhizobium, HPAEC-PAD-MS, ESI-Q-ToF Nom et adresse du ou des laboratoires
Laboratoire des Interactions Moléculaires et Réactivités Chimiques et Photochimiques 118 route de Narbonne 31062 Toulouse CEDEX 9
4
Remerciements...
Les travaux présentés dans ce manuscrit de thèse ont été réalisés au laboratoire des Interactions
Moléculaires et Réactivité Chimique et Photochimique (IMRCP) à l’Université Paul Sabatier de
Toulouse. Je tiens à remercier les deux directrices qui se sont succédé : Isabelle Rico-Lattes et
Monique Mauzac pour m’avoir accueillie dans ce laboratoire.
je tiens à exprimer ma profonde reconnaissance à Monsieur François Couderc pour avoir accepté de
présider à ce jury de thèse mais aussi pour sa présence et ses conseils tout au long de ces trois années.
je remercie sincèrement Madame Marie-Christine Ralet, Monsieur Jean-claude Mishalsky d'avoir
accepté de consacrer du temps, en tant que rapporteur pour juger et aider à l'amélioration de ce
travail. Merci aussi à Monsieur Germain Puzo d'avoir participé à ce jury et pour ses conseils.
Je voudrais adresser toute ma reconnaissance à ma directrice de thèse, le Dr Véréna Poinsot. Merci
de m’avoir accueillie dans ton équipe et de m'avoir soutenue dans les périodes tempétueuses pour
moi, merci de m’avoir enseigné la pratique des sciences et la rigueur de la démarche expérimentale, de
m’avoir transmise ta capacité d’enthousiasme et ta force, merci de m’avoir offert cette grande liberté
de travail et de m’avoir accordée ta confiance. J’ai énormément appris de toi et je garderai
longtemps le souvenir de ton enseignement et de ton humanité.
Merci encore à celles qui n’ont été que de passage mais avec qui j’ai passé de réels bons moments au
coin d’une paillasse, d’une hotte vrombissante ou au détour d’un café, et plus particulièrement à
Delphine, que j’ai appris à connaître tout au long de ces années et qui m'a apportée sa bonne
humeur et ce quelque soit les circonstances. Pour tous nos bons moments, et les moins bons.
Enfin, merci à tous ceux qui partagent ma vie et qui m’accompagnent avec tant d’amour et de
tendresse. Merci à ma famille, à mes amis, vous mériteriez des lignes, des chapitres, des livres
entiers, il y a tant d’histoire, de rencontres, d’émotions partagées à raconter, mais vous connaissez
mes difficultés d'écriture alors merci de me laisser simplement vous serrer dans mes bras.
5 aa, acide aminé
Ac, acétyl
Ac2O, anhydride acétique
ASRS, suppresseur autorégénérant d’anions ATP, adenosine triphosphate
BuOH, hydroxybutyryl
Cn:x, n représente le nombre de carbone de la chaine carbonée de l’acide gras x représente le nombre d’insaturation
CID, collisions avec un gaz rare induisant des dissociations DHB, acide 2,5-dihydroxybenzoique
DNase, désoxyribonucléase DO, densité optique
DOC, acide désoxycholique
E., Escherichia
EDTA, acide éthylènediaminetétraacetique EPS, exopolysaccharides
EPS-, déficience en exopolysaccharide ESI+, mode de détection /ionisation positif ESI-, mode de détection /ionisation négatif
ESI/MS, ionisation électrospray couple à un spectromètre de masse EI, impact électronique
FAO, Organisation des Nations Unies pour l'alimentation et l'agriculture
Fix-, phénotype dans lequel les symbiosomes fixent l’azote, si positif la plante est verte, sinon elle est jaune
FT-ICR, résonance cyclotronique d'ions et à transformée de Fourier Gal, galactose
GalA, acide galacturonique
GC/MS, chromatographie en phase gazeuse couplé à un spectromètre de masse Glc, Glucose
GlcA, acide glucuronique GlcN, glucosamine Hex, hexose
HexA, acide uronique
HFBA, anhydride heptafluorbutyrique
HMBC, corrélation multiple liaison hétéronucléaire HMW, haute masse molaire
HPAEC, chromatographie haute performance échangeuse d’anion HSQC, corrélation simple quantum hétéronucléaire
Kdo, acide 2-ceto,3-désoxy-manno-octulosonique Kdx, acide 2-céto-3-désoxy-ulosonique
KPS, polysaccharides capsulaires
KPS-, déficient en polysaccharide capsulaire LA, lipides A
LCO, lipooligomère de chitine LMW, faible masse molaire LOD, limite de détection LPS, lipopolysaccharide
LysoPI, lysophosphatidylinositol
MALDI, ionisation désorption sur matrice assistée par laser Man, mannose
6 MMW, masse molaire moyenne
MS/MS, fragmentation d'un ion déterminé MW, masse moléculaire
MWCO, Limite de séparation du poids moléculaire
N., Neisseria
NAD(P), le P entre parenthèse signifie que le donneur d'électron est soit le NADH,H+ ou le NADPH,H+
NAD, nicotinamide adénine dinucléotide
NADP, nicotinamide adénine dinucléotide phosphate NANA, acide N-acétyl neuraminique
BuOH, hydroxy-butyryl
PAD, détection à ampérométrie pulsée
PAGE, électrophorèse sur gel de polyacrylamide PC, phosphatidylcholine
PE, phosphatidyléthanolamine PI, phosphatidylinositol
PMAA, acétate d’alditol partiellement méthylé Pse, acide pseudaminique
Q-ToF, quadripôle- temps de vol
R., Rhizobium
rARN, acide ribonucléique ribosomial Rha, rhamnose
r-LPS, "rough" lipopolysaccharides RMN, résonnance magnétique nucléaire RNase, ribonucléase A
S., Sinorhizobium
SEC, chromatographie exclusion stérique Sf, Sinorhizobium fredii
SIM, appel d'ion unique
s-LPS, "smooth" lipopolysaccharides Sm, Sinorhizobium meliloti.
TEMED, Tétramethyl-èthyl éthylènediamine TFA, acide trifluoroacétique
TIC, appel d'ions totaux
TLC, chromatographie sur couche mince TRIS, tris(hydrxyméthyl)aminométhane UDP, 1-phosphate uridyl
YEM, milieu aux extraits de levure
3OHCn, acide gras portant un hydroxyle en position 3 de sa chaîne carbonée n représente le nombre de carbone
7
I Introduction.
... 111 Généralités ... 13
2 La formation du nodule ... 15
3 Les signaux symbiotiques ... 16
3.1 Facteurs Nod et reconnaissance entre les deux partenaires (étapes 1,2,3)... 16
3.2 Rôle symbiotique des polysaccharides de surface (Etape 6)... 17
3.2.1 Les exopolysaccharides (EPS) ... 18
3.2.2 Les polysaccharides capsulaires (KPS)... 19
3.2.3 Les lipopolysaccharides (LPS)... 20
4 Biosynthèse des polysaccharides de surface... 22
4.1 Les exopolysaccharides... 22
4.2 Les lipopolysaccharides... 23
4.3 Les polysaccharides capsulaires... 27
5 Analyse des oligosaccharides par spectrométrie de masse. ... 31
5.1 Les sources et modes d’ionisation... 31
5.1.1 Bombardement par des atomes rapides (FAB) ... 31
5.1.2 Desorption/Ionisation Assistée par Laser (MALDI)... 32
5.1.3 Ionisation ElectroSpray (ESI) ... 33
5.1.4 Désorption/ionisation par electrospray (DESI) ... 34
5.2 Mode de fragmentation des oligosaccharides...Erreur ! Signet non défini. 6 Mise en perspective des travaux de thèse... 38
II-Résultats
. ... 39Chapitre 1 : Caractérisation des KPS de haute masse
molaire, actifs dans la symbiose fixatrice d'azote : rôle de
rkpZ1 et rkpZ2 chez Sm1021
.
... 41CONTEXTE ... 411
RESULTATS ... 49
1 Choix de la mutation et effet du plasmide pMW23 sur la biosynthèse des KPS... 49
2 Analyse par spectrométrie de masse de l’extrait brut ... 522
3 Analyse de la composition saccharidique des KPS en fonction de leur taille ... 600
4- Détermination du mode d’enchaînement... 644
5- Mise en évidence de la présence d’une ancre lipidique ... 6767
6- Structure de l'ancre lipidique du KPS ... 700
7- Activité symbiotique ... 833
8- Des paralogues de rkpZ dans la souche Sm1021 ... 8486
BILAN ... 955
Chapitre 2: Analyse des polysaccharides bactériens par
couplage HPAEC-PAD-MS
...99
CONTEXTE ... 99
RESULTATS ... 1055
1 Couplage ESI-Q-ToF /HPAEC-PAD en ligne ... 1055
1.1 Choix du gradient... 1066
8
1.3 Effet du dessaleur sur la résolution chromatographique... 1111
1.4 Choix du mode d'ionisation en MS... 1122
1.5 Sensibilité respective des détecteurs PAD et MS... 114
2 Analyse des polysaccharides contenus dans les extraits bactériens... 116
2.1 Sm1021... 118
2.2 S. fredii HH103... 1211
2.3 Sm41 exoB (AK631)... 1222
2.4 S sp. NGR234... 12424
BILAN ... 1277
Chapitre 3 : Analyse structurale du cœur des LPS de
Sm1021
.
...
1311CONTEXTE ... 1311
RESULTATS ... 13939
1 Détermination de la masse molaire du cœur ... 13939
1.1 Masse et structure du lipide A... 13939
1.1.1 Sm1021... 139
1.1.2 AcpXL... 1411
1.1.3 LpxXL ... 1411
1.2 Masse molaire des LPS... 1433
2 Analyse structurale de l’oligosaccharide de cœur... 1466
2.1 Composition saccharidique... 1466
2.1.1 Analyse en GC-MS ... 1466
2.1.2 Analyse en HPAEC-PAD... 1511
2.2 Analyse du mode de jonction : méthode de Hakomori... 1566
2.3 Analyse du cœur entier par HPAEC-PAD... 1611
BILAN ... 1655
III Conclusions et perspectives
... 1677IV Matériels et Méthodes
. ... 17551 Cultures bactériennes ... 1777
2 Test de Gram ... 17878
3 Purification des Polysaccharides... 17979
3.1Extraction au phénol à chaud... 17979
3.2 Digestion enzymatique... 1800
3.2.1 RNase, DNase, Protéinase K... 1800
3.2.2 Phospholipases ... 1800
4 Purifications des KPS et des LPS... 1811
4.1 Par chromatographie d’exclusion stérique (S300)... 1811
4.2 Par chromatographie d’affinité (polymyxine B)... 1822
5 Caractérisations... 1822
5.1 Gel DOC-PAGE... 1822
5.2 Analyse par spectrométrie de masse des polysaccharides... 1855
5.2.1 ESI-MS des extraits bruts et lipides A ... 1855
5.2.2 MALDI-ToF des extraits bruts et des lipides A... 1855
5.3 Analyse des ancres lipidiques par TLC préparative... 1866
9
5.3.2 Coloration à l’acide sulfurique... 1866
5.3.3 Coloration à l’anthrone... 186
5.3.4 Coloration à la primuline... 1877
5.3.5 Coloration à l'acide phosphomolybdique ... 187
5.3.6 Coloration à l'azure A... 187
5.3.7 Coloration de Dragendorff ... 187
5.3.8 Coloration à la ninhydrine... 187
5.3.9 Élution des composés adsorbés sur silice... 187
6 Analyses structurales ... 18888 6.1 Hydrolyse douce... 18888 6.2 Hydrolyse forte... 18888 6.3 Analyse de l’ancre... 189 6.3.1 Saponification... 18989 6.3.2 Acétylation ... 18989
6.4 Etude par HPAEC... 189
6.4.1 HPAEC-PAD ... 1911
6.4.2 HPAEC-PAD-MS ... 1911
6.5 Etude par GC-MS... 1922
6.5.1 Dérivation par l’acide heptafluorobutyrique ... 1933
6.5.2 Méthylation/acétylation : méthode Hakomori... 1933
6.6 Etude par RMN... 1944
11
13 1 Généralités
Il est aujourd’hui commun de dire que les symbioses sont sources de diversité et d’adaptation. De nombreux exemples de symbioses existent dans le monde du vivant : elles prennent des formes aussi diversifiées que les Lichens (union d’une algue et d’un champignon), la microflore intestinale chez l'homme ou encore l'acacia comigera, qui survit grâce aux colonies de fourmis. Dans le monde végétal, elles permettent, le plus souvent, la survie de la plante dans un environnement défavorable qu’il soit sec ou carencé en nutriments. Elles permettent ainsi l’implantation de nombreuses espèces dans des milieux hostiles en enrichissant les sols.
Nos travaux concernent une association symbiotique particulière: la symbiose fixatrice d’azote. L'azote, le phosphore, le calcium, et plus généralement les minéraux indispensables à la croissance des végétaux sont prélevés dans le sol. Après la récolte, les éléments qui ont servi à la croissance des plantes, doivent être restitués au sol, afin d'en conserver la fertilité. Dans le système d’agriculture intensive que nous connaissons, la restitution se fait sous forme d’engrais d'origine chimique (azote/phosphore/potasse), parfois appliqués en excès. Cette pratique est une source de nuisances pour la santé humaine et l’environnement. Elle pollue les eaux de surface et des nappes phréatiques, par lessivage des engrais, mais engendre aussi une forte consommation d’énergie et une émission de gaz à effet de serre pour la production des engrais azotés. Ces engrais purement minéraux amènent par ailleurs, un déséquilibre croissant de la structure et de la composition des sols.
Parmi les éléments nutritifs essentiels, l’azote est très important puisqu’il est nécessaire à la fois aux biosynthèses des acides aminés et au métabolisme des plantes. Bien que l'azote représente 80% de l’air sous forme N2, sa forme combinée (NH4OH, NO3-) est
plus rare. Seul l'azote combiné sous forme de nitrate ou d’ammoniaque est assimilable par la plante. Les sources naturelles d’azote combiné sont les éclairs (Dickerson et al., 1984), la décomposition d’organismes vivants, les cyanobactéries présentes dans l’environnement proche des plantes (Burris et al., 1974) et enfin, la plus importante des sources, les bactéries de type Rhizobia qui entrent en symbiose avec les légumineuses. Le tonnage en ammoniaque produit par cette dernière est proche de celui produit par l'industrie chimique, approximativement 107 millions de tonnes (http://www.fertilizer.org). Cette symbiose est de
14 type mutualiste (E. Toby Kiers et al., 2004): la plante apporte les nutriments carbonés et un environnement pauvre en oxygène et sans stress hydrique, tandis que la bactérie apporte l’ammoniaque. La réduction de l’azote atmosphérique n’est rendu possible que par la mise en commun du potentiel des deux partenaires. La bactérie sous forme de bactéroïde réduit l'azote atmosphérique grâce à une enzyme, la nitrogénase (Figure 1). Cette transformation demande un apport énergétique très important, puisque 1) 16 ATP sont nécessaires pour réaliser chaque réduction et 2) la formation d'un nouvel organe racinaire dans lequel la pression de l’oxygène est maintenue à un niveau assez bas dans l’environnement de l’enzyme, est indispensable à la réduction de l’azote.
Figure 1: Transformation de l’azote en ammoniaque par le complexe réductase/nitrogénase via la ferrédoxine.
La symbiose légumineuse/rhizobium est le résultat d'une interaction hautement spécifique entre la plante et la bactérie. L'établissement et le fonctionnement de la symbiose sont sous le contrôle génétique des deux partenaires. La reconnaissance entre les deux symbiotes est rendue possible par des échanges de signaux moléculaires. Ainsi, des composés de types flavonoïdes sont exsudés par les racines de la plante et vont induire la transcription des gènes de la nodulation nod et nol qui codent pour la production de composés de lipooligomère de chitine (LCO), les facteurs Nod. Ces composés sont libérés dans la rhizosphère et perçus en retour par le végétal. Seule une structure très précise de LCO modulée par la présence ou non de certains substituants est reconnue par la plante hôte (Broughton et al., 2000). Ces facteurs Nod, une fois reconnus, vont avoir une activité « à distance » au niveau de la plante.
Une fois ce stade précoce passé, d’autres composés saccharidiques provenant de la bactérie entrent en jeu. Ces polysaccharides d’origine bactérienne sont soit sécrétés dans le milieu extérieur soit restent au contact de la bactérie.
15 Une communication complexe entre les deux symbiotes s’effectue ainsi tout au long du processus d’infection. La compréhension, puis la maîtrise des processus permettant la mise en place d’une telle symbiose, constituent un grand pas vers le développement d’une agriculture moins polluante. La prédiction du département européen pour l'agriculture indique que si aujourd'hui les engrais n'apportent que 43% des nutriments nécessaires à la plante, dans un avenir proche, ce pourcentage sera de 84%, étant donné le modèle agricole actuel (FAO, communiqué de juin 2003). Une utilisation de la symbiose fixatrice d'azote peut diminuer l’apport d’engrais azoté chimique nécessaire aux cultures tout en structurant les sols en réalisant des cycles légumineuses/céréales performants. L'optimisation de la symbiose permettrait aussi une culture du soja et des autres légumineuses plus raisonnée.
2 La formation du nodule
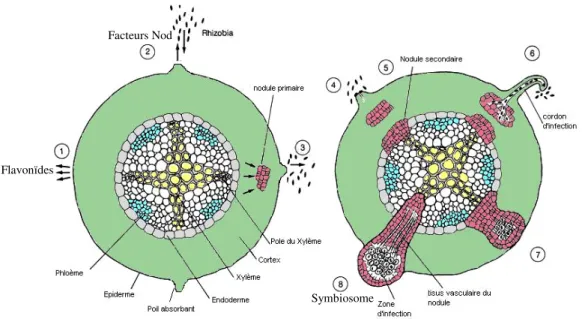
Figure 2: Etapes de la formation d’un nodule dans une racine latérale
Les étapes de la mise en place de la symbiose peuvent être énumérées dans un ordre chronologique : (1) la racine exsude des flavonoïdes ; (2) les Rhizobia, en réponse secrètent des facteurs Nod ; (3) sous l’action des facteurs Nod, les cellules du cortex se mettent en mitose pour former le primordium nodulaire dans le méristème ; (4) la bactérie s’attache à l’épiderme du poil absorbant et le poil (par croissance polaire) se recourbe sur lui-même ; (5) les cellules du péricycle près des pôles du xylème se mettent en mitose ; (6) il y a formation du cordon d’infection amenant la bactérie au nodule primaire ; (7) les deux masses de cellules fondent en un bloc unique tandis que le cordon d'infection continue à se développer ; (8) le nodule se prolonge, le raccordement vasculaire avec le stèle de la racine est mis en place. Les
Facteurs Nod
Symbiosome Flavonïdes
16 bactéries sont acheminées dans les cellules du nodule et se différencient en bactéroïdes. Le nodule et les bactéroïdes forment le symbiosome.
3 Les signaux symbiotiques
3.1 Facteurs Nod et reconnaissance entre les deux partenaires (étapes 1,2,3 de la Figure 2).
La plante étant par définition immobile, elle doit attendre que les Rhizobia entrent dans sa rhizosphère. Toutefois la mobilité de la bactérie et le chemotactisme ne jouent qu’un rôle restreint dans la reconnaissance (Fellay et al., 1995). Dans ce contexte il apparaît difficile que les deux partenaires se rencontrent. C'est sans compter sur le fait que la plante exsude dans son proche environnement une grande diversité de nutriments qui, par leur nature (source de carbone, d’azote, etc...), attirent à elle des microorganismes symbiotiques et pathogènes (Barbour et al., 1991). Pour réaliser une sélection dans cette flore, la plante sécrète en continu des flavonoïdes. Lorsqu’un Rhizobium compatible pénètre dans sa rhizosphère, la concentration en flavonoïde augmente. La bactérie reconnaît ce composé grâce à une protéine NodD, il se forme un complexe qui active une séquence de gène (nod, nol et
noe). Ces gènes sont responsables de la biosynthèse des facteurs Nod (Schlaman et al., 1992).
Il s’ensuit un processus impliquant les facteurs Nod mais aussi les polysaccharides de la surface des bactéries.
Les facteurs Nod sont des lipooligosaccharides composés de 3 à 5 β1-4N-Acétylglucosamines portant sur leur sucre non réducteur un groupement acyle et sur leurs deux extrémités des substituants divers (fucose, sulfate, acétyle, carbanoyle) dépendant de la souche bactérienne (Figure 3). Ces lippochitooligomères sont sécrétés dans le milieu externe et vont déclencher une série de modifications chez la plante. Ils sont impliqués dans les stades les plus précoces de la reconnaissance entre les deux partenaires et induisent les modifications du poil absorbant, débutant ainsi l'organogenèse du nodule.
17
Figure 3: Schéma structural d'un facteur Nod.
Si les facteurs Nod sont le point de départ d’une communication indispensable à la mise en place spécifique de la symbiose fixatrice d’azote, ils ne sont pourtant pas impliqués dans les étapes plus tardives. En effet, lorsque des racines sont mises en présence des seuls facteurs Nod, les organes symbiotiques ne se forment pas ou sont incomplets. Seul le recourbement du poil absorbant est visible. Il existe à la surface des bactéries d’autres composés dont le rôle est de succéder aux facteurs Nod : les polysaccharides de surface.
De nombreux articles de revue sur le rôle de ces polysaccharides ont été réalisés au cours des dernières années, dont les plus importantes ont été écrits par Fraysse et al. en 2004 ou encore par Niehaus et al. en 1998. L’exposé succinct du rôle de ces polysaccharides de surface qui suit repose sur ces revues.
3.2 Rôle symbiotique des polysaccharides de surface (Etape 6)
Les bactéries de la famille des Rhizobia sont de type α-proteobacter, et possèdent donc une physiologie classique de Gram négatif (Figure 4). Ainsi, leur paroi est composée de trois parties : une bicouche phospholipidique, la frontière entre le milieu externe et le cytoplasme, puis une couche de peptidoglycanes séparée par un espace périplasmique de la membrane externe. Cette membrane externe asymétrique est constituée sur sa face interne de
18 phospholipides et de lipoprotéines et, sur sa couche externe, de lipopolysaccharides. Des ions Ca2+ jouent un rôle primordial dans la cohésion de cette membrane externe. Au voisinage proche de la membrane externe, une couche supplémentaire composée de polysaccharides capsulaires entoure la bactérie. Son rôle est de protéger le microorganisme contre les perturbations abiotiques telles que les stress hydriques et salins. Une troisième catégorie de polysaccharides se trouve encore plus en surface des bactéries : les exopolysaccharides. Ils sont continuellement secrétés dans la rhizosphère ou dans le canal d’infection, mais leur production est stoppée une fois le nodule atteint. Dans les conditions de stress hydrique, ces polysaccharides transforment leur environnement en gel afin de maintenir une couronne humide autour de la bactérie.
Figure 4: Schéma de la surface d’une bactérie de type Gram négatif.
3.2.1 Les exopolysaccharides (EPS)
Ce sont les plus étudiés de tous les polysaccharides de surface. Un intérêt particulier à été porté aux succinoglycanes présents à la fois chez Sinorhizobium meliloti et
Agrobacterium. Leur structure est constituée d’unités répétitives (8 à 12 mères), composées
19 des pyruvates, des succinates ou des acétates. Ils peuvent être linéaires ou ramifiés. Les sucres sont reliés entre eux par une grande variété de liaisons (alpha ou béta, 1-2, 1-3 etc…) (Figure 5). Certaines bactéries telles que S. meliloti sont capables de produire un autre type d’EPS appelés EPS II qui sont composés d’une unité répétitive disaccharidique de type hexose. L’absence des exopolysaccharides bactériens provoque chez la plante la production excessive de composés phénoliques qui colorent en brun les racines et provoquent une apoptose des cellules infectées, ainsi que la mort des bactéries. De plus, le cordon d'infection ne progresse pas et des aberrations (poches) apparaissent. Ainsi leur rôle dans la symbiose fixatrice d’azote se situe dans l’inhibition des réactions de défenses des plantes et dans la progression du cordon d’infection.
Figure 5 : Schéma de la structure de différents exopolysaccharides en fonction de la souche qui les produits (Skorupska, 2006).
3.2.2 Les polysaccharides capsulaires (KPS)
Au contact de la surface de la bactérie se trouvent les polysaccharides capsulaires. Ceux de la famille des Rhizobia sont composés de sucres appartenant à la famille de l’acide 2-céto-3-désoxy-ulosonique (Kdo), notés Kdx. Les premières structures élucidées ont été celles
20 des KPS des souches S. fredii USDA 205 et S. meliloti 41 (Reuhs et al., 1993). Ces polysaccharides ont une structure analogue à celle de l’antigène KR5 de E. coli, c’est pour cela
que les initiales KPS leur ont été attribuées. Un motif consensus a pu être établi : il s’agit d’un polymère de disaccharide composé d’un Kdx et d’un hexose ou d’un acide uronique (Tableau 1).
Tableau 1:Composition des unités répétitive des KPS en fonction de la souche (Reuhs et al., 1998).
Ces polymères peuvent atteindre 30 kDa (pour Sm41) mais leur masse molaire est en moyenne de 7 à 12 kDa. Ils sont en général facilement identifiables sur un gel de polyacrylamide car ils se colorent au bleu d’alcian (colorant cationique formant une paire d’ions avec les Kdx). La nature particulière des Kdx les rend sensibles à l’hydrolyse acide. C’est cette propriété chimique qui induit une grande disparité dans la taille observée des KPS, qui n’est pas obligatoirement représentative des formes synthétisées in vivo.
Leur rôle dans la symbiose fixatrice d’azote est à ce jour inconnu. Peu d’études ont été réalisées pour comprendre leurs modes d'action. Ils pourraient avoir un simple un rôle passif de protection lors des changements brusques d’environnement (passage du sol à la plante) et faces aux défenses de la plante (antibiotiques de nature peptidique) et au choc alcalin.
3.2.3 Les lipopolysaccharides (LPS)
Beaucoup de structures de LPS de Rhizobia sont partiellement décrites mais relativement peu sont complètes à ce jour. Les LPS sont composés de trois parties : i) le lipide A qui permet d’ancrer la molécule dans la bicouche phospholipidique, ii) un oligosaccharide
21 de cœur, iii) un antigène O. Les LPS se distinguent en deux classes suivant leurs tailles. Leur nom provient de la forme qu’ils donnent aux colonies bactériennes. Lorsque les colonies présentent un contour irrégulier, ils sont appelés r-LPS : r pour « rough ». Lorsque les colonies présentent des contours réguliers les LPS sont dits s-LPS, s pour « smooth ». Les r-LPS ne sont constitués que d'un lipide A et d'un oligosaccharide de cœur. Les s-r-LPS sont quant à eux composés d'un lipide A, d'un oligosaccharide de cœur et des unités répétitives formant l’antigène de type O (Figure 6).
Figure 6: Schéma structural des LPS.
Une analyse sur gel d'acrylamide permet de les identifier. La coloration en jaune, après traitement au nitrate d’argent, est caractéristique de ces composés en partie lipidiques. Sur un gel de polyacrylamide, les r-LPS, de petites tailles, migrent le plus rapidement que les s-LPS.
Leur rôle dans la symbiose fixatrice d’azote réside dans l’inhibition des réactions de défenses de la plante dans les étapes plus tardives de l’infection, lorsque les EPS ne sont plus synthétisés, en empêchant la mise en place du choc oxydant (Scheidle et al., 2005). Ils ont par ailleurs un rôle dans la pénétration des bactéries dans les cellules nodulaires, lorsqu’elles sont libérées du cordon d’infection. Les composés sont aussi des agents de cohésion membranaire. Pour cette raison, les LPS semblent nécessaires à la survie de la bactérie lors de sa transformation en bactéroïde (changement de forme et de taille).
monosaccharide
s-LPS
22 4 Biosynthèse des polysaccharides de surface
4.1 Les exopolysaccharides
Les gènes impliqués dans la biosynthèse des exopolysaccharides (exo/exs ou pss) forment un grand groupe de gènes situé sur le megaplasmide symbiotique pSymB (Finan et
al., 2001). Les protéines synthétisées par ce groupe de gènes sont de quatre types : les
enzymes impliquées dans la biosynthèse des précurseurs de sucre, les transférases responsables de l'assemblage de l'unité répétitive, les enzymes responsables des substitutions non-sacharidiques et enfin celles permettant la polymérisation des unités répétitives entre elles et l'export des EPS.
La biosynthèse des EPS représente un processus multi-étape et dépend d'un complexe protéique localisé à la fois sur les membranes interne et externe. Elle débute par la formation d'un précurseur, nucléotide de sucre diphosphorylé (par exemple UDP-glucose), transféré sur un accepteur le undecaprenydiphosphate (UndP). L'unité répétitive est formée dans le cytoplasme et sera ensuite transférée vers le périplasme dans lequel se déroule la polymérisation. Le polysaccharide est acheminé vers la membrane externe par des transporteurs de type ABC (Glucksmann et al., 1993).
Prenons plus en détail la biosynthèse des EPS I de Sinorhizobium meliloti qui sont les EPS actifs dans la symbiose (Figure 7). Tout d'abord la biosynthèse débute par la formation d'un nucléotide de sucre. exoC code pour une phosphoglucomutase qui transforme le glucose-6-phosphate en glucose-1-phosphate qui est le précurseur de UDP-glucose (Uttaro et al., 1995). Intervient ensuite le gène exoB qui code pour une UDP-glucose-4-épimérase qui convertit UDP-glucose en UDP-galactose (Canter Cremers et al., 1990). La mutation des gènes exoB ou exoC induit la non-production des EPS, mais affecte aussi la biosynthèse d'autres polysaccharides tels que les LPS et les glycanes cycliques. L'assemblage de l'unité répétitive des EPS est initié par ExoY qui est une galactosyltransférase (Müller et al., 1993).
exoF est le gène codant pour la protéine permettant la liaison entre le galactose et
l'undecaprenyl-diphosphate (Reuber et al., 1993). ExoA, ExoL, ExoM, ExoO, ExoU et ExoW sont les glycosyltransférases qui vont ajouter séquentiellement les sucres de l'unité répétitive tandis que ExoZ, ExoH, et ExoV vont introduire les motifs non-saccharidiques (Skorupska et
23
al., 2006). Les protéines ExoP, ExoQ, ExoT et ExsA sont responsables de la polymérisation
et de l'acheminement vers le milieu externe des EPS entiers.
Figure 7 Schéma représentant la biosynthèse de l'unité répétitive des EPS I de S. meliloti. En orange et en bleu sont indiquées les protéines impliquées dans le transfert du précurseur (galactose) sur le transporteur lipidique (undecaprenol-diphosphate : PP-prenol), en vert sont indiquées les protéines impliquées dans les modifications non-saccharidiques, en jaunes les protéines
permettant la polymérisation et l'export des EPS. (Skorupska et al., 2006)
4.2 Les lipopolysaccharides
Price et ses collaborateurs en 1994 ont démontré que R. leguminosarum et R. etli possédés une activité enzymatique se recoupant avec celle des enzymes de E. coli impliquées dans la synthèse du précurseur .
Ainsi, sur le précurseur UDP-N-acetylglucosamine deux acides gras sont liés en position 2 et 3 par LpxACD. LpxH substitue la position 1 de l'entité obtenue précédemment. LpxB va permettre la liaison entre une glucosamine diacylée et la glucosamine diacylé et phosphatée. La position 4 de la glucosamine non réductrice est alors substituée par un phosphate et KdtA ajoute enfin les deux Kdo sur la position 6 de celle-ci (Figure 8) (Raetz et
al., 2002).
C'est à ce stade qu'intervient la substitution alcoxy-acyl, par un acide gras normal chez lez bactéries usuelles mais à très longue chaîne chez Rhizobiaceae ce qui représente la plus
24 grande spécificité de leurs lipides A . L’enzyme responsable de cette modification est LpxXL qui associé à un donneur d'acide gras AcpXL permet de modifier par une chaîne grasse en C28 ou C30 l'hydroxyle de l’acide gras porté par l’azote de la glucasomine.
Figure 8 schéma de la biosynthèse du Kdo2-lipideIVA de R.Leg. O UDP NH HO HO OH O O UDP NH O HO OH O O HO O UD P NH2 O HO OH O HO O UDP NH O HO OH O HO O OH O O NH O HO OH O HO O OH P O O O O O NH O HO O HO O OH P O O O O O NH O OH O HO O OH O P O O O O O NH O H O O HO O OH P O O O O O N H O OH O HO O OH HO O OH OH OH OH O O P O O cyti di ne CMP-Kdo CMP-Kdo CMP CMP O O OH OH OH OH O O O O NH O HO O HO O OH P O O O O O NH O O O HO O OH O P O O O O OH OH OH O O O O OH OH OH OH O O O O NH O HO O HO O OH P O O O O O NH O O O HO O O P O O O O OH OH OH O O O O O OM e O
3OHC16-ACP acétate 3OHC18-ACP
UDP ATP ADP LpxA LpxC LpxD LpxH LpxB KdtA KdtA AcpXL 27OHC28-ACP LpxXL Kdo2-lipideIVA
25 Une fois l'ancre lipidique du LPS synthétisée, la biosynthèse de l'oligosaccharide de cœur peut débuter. Une seule étude a été réalisée pour déterminé les enzymes responsables de la biosynthèse de l’oligosaccharide de cœur pour la famille des Rhizobiceae. Cette étude porte sur la souche R. leguminosarum. Pour cette souche, le Kdo2-lipideIVA sert alors d’accepteur
pour la biosynthèse du cœur. Un mannose est fixé sur le Kdo interne grâce à la protéines LpcC. Des glycosyltransférases (LpcA et LpcB) permettent ensuite l’élongation de l’oligosaccharide de cœur (Kadrmas et al., 1998) (Figure 3).
Figure 9 schéma de la biosynthèse de l'oligosaccharide de cœur de R. Leg
Une fois l'oligosaccharide de cœur synthétisé l'entité cœur lipide A est alors transféré vers le périplasme. Chez R. leg, le lipide A va alors perdre les deux phosphates (en 1 et 4') grâce aux enzymes LpxE et LpxF. Après cette déphosphorylation, LpxQ va oxyder la position 1 pour donner le gluconate. La position 4', le mannose ainsi le Kdo externe du Kdo2-lipide IVA que du cœur vont être substitué par les acides galacturoniques par les enzymes RgtABCD
(Kanjilal-Kolar et al., 2006 a et b).
Pour la souche Sinorhizobium meliloti dont le lipide A diffère de R. leg et dont le cœur reste indéterminé, a la présence d’un homologue de lpcC nommé lpsB pu être démontrer. L’analyse des séquences de gènes suivant cet homologue ont démontré une forte homologie avec des glycosyltransférases et ont été nommés lpsCDE (Lagares et al., 2001).
Lipide A Kdo(interne) Kdo(externe) Man Gal Kdo GalA GalA GalA Antigène O LpcC RgtC LpcA LpcB RgtA et RgtB KdtA Lipide A Kdo(interne) Kdo(externe) Man Gal Kdo GalA GalA GalA Antigène O LpcC RgtC LpcA LpcB RgtA et RgtB KdtA
26 Contrairement à l’oligosaccharide de cœur, la partie antigène O est synthétisée séparément. Mais comme pour les autres constituants du LPS ils s'agit d'un processus séquentiel qui se déroule en trois étapes i) synthèse de l'unité répétitive ii) polymérisation de la chaîne iii) transport à travers les membranes plasmiques et liaison au complexe lipide cœur. Il existe peu d'études sur la synthèse de la partie antigène O chez Rhizobia. Tandis que leur biosynthèse chez les entérobactéries et en particulier E. coli, sont bien connues (Raetz et al., 2002).
Le début de la synthèse de l'unité répétitive se fait sur un lipide porteur l'undecaprenylphosphate (UndP) déjà utilisé dans la biosynthèse du peptidoglycane et des exopolysaccharides. Cette unité répétitive est synthétisée à la surface interne de la membrane cytoplasmique.
Le mode de polymérisation et sa localisation dépendent ensuite du mode de transfert de la partie antigène O vers le périplasme. Dans le cas de E. coli, il existe deux mécanismes principaux.
Le mécanisme dépendant de Wzy induit le transport des unités "UndP-unité répétitive" vers la face périplasmique de la membrane cytoplasmique. Ce transfert est contrôlé par une flipase (Wzx). Une fois dans le périplasme les unités répétitives vont pouvoir polymériser. Pour cela une unité va être ajoutée à un complexe "UndP-unité répétitive". Ce dimère va ensuite être attaché à un complexe "UndP-unité répétitive" et ainsi de suite jusqu'à atteindre la taille adéquate. Ceci implique que au cours de la polymérisation (l'unité répétitive)n est
détaché du lipide porteur pour être lié à un complexe "UndP-unité répétitive" pour obtenir un nouveau complexe UndP-(unité répétitive)n+1. Wzy et WaaL permettrons ensuite la liaison
entre l'antigène O et l'unité lipide A-cœur qui sera ensuite acheminé à la surface de la bactérie (Figure 10A).
Le deuxième mécanisme implique des transporteurs de type ABC. Dans ce cas l'élongation de la chaîne se situe dans le cytoplasme. La synthèse de l'antigène O ne nécessite dans ce cas que 1 UndP puisque les sucres des unités répétitives sont ajoutés séquentiellement jusqu'à atteindre la taille adéquate. l'antigène O est ensuite transféré vers le périplasme via un complexe de transport de type ABC (ATP Binding Cassette ) (Figure 10B).
En 2001, Lerouge et ses collaborateurs ont mis en évidence la présence d'un homologue des transporteurs de type ABC. La mutation du gène implique la formation
27 uniquement de r-LPS. R. etli utiliserai donc le mécanisme de biosynthèse impliquant les transporteurs de type ABC.
Figure 10 schéma de la biosynthèse de l'antigène O A) biosynthèse impliquant l'enzyme Wzy : -a- formation de l'unité répétitive -b- transfert vers l'espace périplasmique -c- polymérisation B) biosynthèse impliquant les transporteur de type ABC
: -d- synthése de la totalité de l'antigène O -e- transfert vers l'espace périplasmique (Szalo et al., 2006)
4.3 Les polysaccharides capsulaires
Il existe, là aussi, peu d’études sur le mécanisme de la biosynthèse des polysaccharides capsulaires chez les Rhizobiaceae. Elles sont surtout basées sur la comparaison des gènes de
Sinorhizobium avec ceux d’autres bactéries pathogènes (Kiss et al., 2001). Il existe deux
mécanismes de biosynthèse et d’assemblage commun à de nombreuses bactéries pathogènes. Ces deux mécanismes de biosynthèse sont présents chez E.coli. Or, E.coli a été scindé en quatre groupes présentant des propriétés physiques et chimiques communes (Tableau 2). Ainsi les KPS des groupes 1 et 4 ont un mécanisme de biosynthèse impliquant Wzy et possèdent une ancre lipidique composée par un lipide A. Les groupes 2 et 3 impliquent les transporteurs de type ABC et ont pour ancre lipidique un glycérophospholipide.
Tableau 2 récapitulatif des spécificités de chaque groupe de KPS de E. coli (Roberts, 1996)
Groupe Caractéristique
1 et 4 2 et 3
Thermostabilité oui Non
Structure de la partie lipidique Lipide A- noyau α-glycerophosphate Système de polymérisation Dépendant de Wzy Transporteur ABC Similitude avec la capsule de Klebsiella, Erwinia Neisseria, Haemophilus
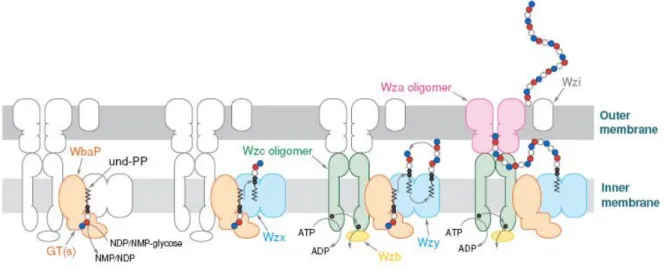
28 Pour les groupes 1 et 4, le mode de biosynthèse de ces KPS est un processus dépendant de Wzy, similairement à celui décrit pour la biosynthèse des antigènes O. Les unités répétitives sont assemblées sur un récepteur lipidique (l'undecaprenyl-phosphate) à l’interface entre la membrane cytoplasmique et le cytoplasme au moyen de l’enzyme WbaP. Ces unités sont ensuite acheminées à travers la membrane interne par Wzx . Ces unités sont ensuite accrochées à l’ancre lipidique des KPS (Lipide A-cœur) grâce à Wzy. La polymérisation est ensuite réalisée dans l’espace périplasmique et nécessite la transphosphorylation de Wzc et la déphosphorylation de Wzb. Une fois les KPS synthétisés, ils sont acheminés vers la surface de la bactérie via Wza (Figure 11) (Whitfield, 2006).
Figure 11 schéma du processus de biosynthèse et de transfert impliquant l'enzyme Wzy (groupe 1 et 4 de E.coli) (Whitfield, 2006)
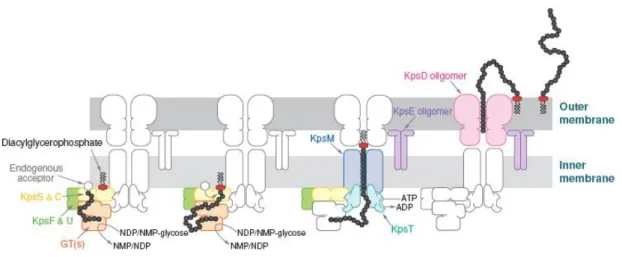
Pour les groupes 2 et 3, le processus de biosynthèse implique les transporteurs de type ABC. Le polysaccharide est dans ce cas synthétisé en totalité dans le cytoplasme sur un accepteur endogène inconnu. Le polysaccharide est ensuite transféré à l’ancre lipidique (glycérophosphate pour E.coli) dans le cytoplasme. Il n’a pas été déterminé si ce transfert était initié avant ou après la fin de la polymérisation. Le lipoKPS est ensuite exporté vers l’espace périplasmique grâce à aux protéines KpsC et KpsT. D’autres protéines contenues dans le groupe de gènes comprenant KpsS et KpsC ont été démontrées comme indispensables. Les enzymes KpsF et KpsU participent à la biosynthèse mais leur rôle demeure inconnu. Le transport à travers le périplasme et la membrane externe est réalisée par les protéines KpsE et KpsD (Figure 12) (Whitfield, 2006).
29
Figure 12 : schéma du processus de biosynthèse et de transfert impliquant un transporteur de type ABC (groupes 2 et 3 de
E.coli) (Whitfield, 2006)
Des analogies de structure entre les polysaccharides capsulaires des Rhizobia et les KPS du groupe II de E. coli ont été trouvées. Les premières structure des KPS de rhizobia ont été rapportées pour Sinorhizobium fredii USDA205 et Sinorhizobium meliloti Rm41 par Reuhs et ses collaborateurs (1993). Ces dernières années, des progrès significatifs ont été accomplis dans l'analyse moléculaire et génétique de la biosynthèse des KPS de
Sinorhizobium meliloti 41. Ils se composent d'unités répétitives dissaccharidiques composées
d'acide glucuronique et pseudaminique substitué par butyrates et d'acétates (Reuhs et al., 1993, 1998) (fig. 1b).
Trois groupes de gènes sont impliqués dans la production de ces KPS (Kereszt et
al., 1998 ; Putnoky et al., 1990).
La région chromosomique rkp-1 (ancien fix-23) semble déterminer la biosynthèse d'un porteur lipidique spécifique, nécessaire à la biosynthèse des KPS (Kiss et al., 1997 ; Petrovics et al., 1993). Des résultats récents, on mis en valeur l'importance des gènes
rkpABCDEF du groupe de gène rkp-1. Quand ils ont été identifiés dans Sm41, ces six gènes
ont été annotés comme gènes codant pour une acide gras synthase (Petrovics et al.,1993). D'ailleurs, la fonction biochimique de RkpF comme protéine donneur d'acyle a été démontrée (Epple et al.,1998). Cependant, les données actuellement disponibles permettent une meilleure prévision de la fonction du produit des gènes codés par rkpABCDEF qui ressemblent à une protéine multidomaines codant pour une polyketide synthase de type I.
La région chromosomique rkp-2 (codant pour lpsL et rkpK) est impliquée dans le métabolisme des nucléotides de sucre. rkp-1 et rkp-2 sont en plus indispensables à la
30 production de LPS. Seul le gène rkpK est nécessaire à la seule synthèse de KPS (Kereszt et
al., 1998).
Le dernier groupe de gène rkp-3 située sur le mégaplasmide de pSymB comprend 10 gènes. Six de ces gènes, rkpLMNOPQ, sont impliqués dans la synthèse des précurseurs spécifiques des sucres et déterminent la composition en saccharide des KPS (Kiss et al., 2001).
Sinorhizobium meliloti 1021 contient également des groupes de gènes homologues
de rkp-1 et rkp-2, mais le groupe de gènes rkp-3 ne possède que peu de similitudes celui de Sm41 (Kiss et al., 2001). Toutefois les gènes rkpRSTZ, qui sont peut-être impliqués dans l'exportation et la polymérisation des KPS, sont des orthologues de ceux de Sm41. Par contre il n'y a aucun homologue aux gènes biosynthétiques rkpLMNOPQ. Trois paralogues putatifs du gène de rkpZ de Rm41 sont présents dans cette région du génome Sm1021, mais seulement un de ces derniers semble coder pour une protéine dans son intégralité. Précédemment, aucun homologue du gène rkpZ (autrefois noté lpsZ) n'avait pu être détecté par hybridations du type Southern dans Sm1021 (Williams et al., 1990).
rkpABCDEF codant pour une protéine multidomaine, il n'est pas surprenant, dans Sm1021,
que ces gènes aient été réunis en un seul gène appelée rkpA, qui code pour un long polypeptide de 2.504 acides aminés. La biosynthèse du lipide par l'intermédiaire des polyketides-synthases est fondamentalement différente de la voie de synthèse des acides gras. Bien que les polyketide synthases utilisent les mêmes réactions de base que les acide-gras-synthases, les cycles de condensation et d'élongation sont souvent abrégés, produisant des chaînes grasses fortement fonctionnalisées pouvant porter des cétones, des hydroxyles ou des doubles liaisons (Wallis et al., 2002). Jusqu'ici, le paradigme de l'analogie de la biosynthèse des KPS de rhizobia avec les entérobactéries a dominé. Par conséquent, il a semblé embarrassant que S. meliloti utilise un lipide dérivé de polyketide synthase pour la production des KPS. L'analyse du génome d'autres procaryotes a pourtant fait apparaître clairement que l'utilisation des lipides dérivés de polyketides synthases, dans la production de KPS, n'est pas un dispositif singulier de S. meliloti. Un orthologue de rkpA de Sm1021 a été identifié dans
Burkholderia mallei (DeShazer et al.2001) et Bordetella bronchiseptica (Parkhill et al.,
2003). Dans ces deux organismes, le gène orthologue est appelé wcbR et est situé dans le groupe de gènes codant pour les KPS (DeShazer et al., 2001 ; Parkhill et al., 2003). Depuis l'identification des gènes de rkpABCDEF dans Sm41 (Petrovics et al., 1993), il a été supposé qu'un porteur ou du moins une ancre lipidique était exigé pour la biosynthèse des KPS. Cependant, aucune partie lipidique n'a été détectée dans les KPS de grande taille de Sm41
31 (Reuhs et al., 1993). Des recherches récentes sur les KPS de petite taille de Sm1021 ont indiqué la présence d'une ancre lipidique attachée aux polyKdo (Fraysse et al., 2005).
5 Analyse des oligosaccharides par spectrométrie de masse.
Les polysaccharides possèdent un intérêt biologique tout particulier, puisqu'ils interviennent dans de nombreux processus biologiques (reconnaissance, adhésion, différenciation cellulaire etc...). La compréhension de la relation liant leur structure et leur activité est donc un domaine important d’investigation. La spectrométrie de masse est un outil de choix pour l'analyse structurale des sucres. Elle offre des résultats précis, une polyvalence analytique et une sensibilité élevée pour des composés de nature et de compositions diverses. Il existe quelques revues récentes concernant l’analyse des sucres par spectrométrie de masse, desquelles est tiré ce paragraphe (Zaia, 2004 ; Harvey, 1999 et 2006 ; Morelle et al., 2005). Nous présenterons dans la suite l’intérêt que présentent différentes techniques d’ionisation ou d’analyses spectrometriques de masse pour l’étude des saccharides.
5.1 Les sources et modes d’ionisation
5.1.1 Bombardement par des atomes rapides (FAB)
Cette méthode peut s’avérer être utile, bien qu’elle n’ait été commercialisée que sur un période courte (10 ans), laissant la place belle à l’électrospray. Cette méthode utilise un faisceau d'atomes neutres (Argon) de grande énergie cinétique pour l'ionisation de substances non volatiles, ionisées ou non, présentes sous forme de solution dans une matrice de glycérol ou thioglycérol. L'ionisation s'effectue à température ambiante et fournit en abondance des ions pseudo-moléculaires ([M+H]+ ou [M+Na]+en mode positif ou [M-H]- en mode négatif). L’analyse à partir d'une source d'ions de type FAB nécessite souvent une dérivation dont les plus utilisées sont la perméthylation et le peracétylation en raison de l'accroissement de la sensibilité présentée par ces dérivés qui est de 10 à 50 fois meilleure que celle d'échantillons natifs. De plus l'analyse en FAB-MS des oligosaccharides perméthylés et peracétylés produit des données concernant la séquence saccharidique, qui ne sont pas accessibles avec les échantillons natifs. Les spectres de FAB-MS sont caractérisés par les ions moléculaires abondants [M+H]+ et [M+Na]+ fournissant des informations sur leur composition. En outre, pendant l'ionisation, l'énergie interne forte donnée aux molécules, produit la fragmentation
32 des liaisons labiles. En fait, les fragments produit sont des ions terminaux non-réducteurs issus de la rupture intracyclique (ions type A) ou terminaux réducteurs de la jonction osidique (ions de type Y). Quand les sucres aminés sont présents, la fragmentation de type Y se produit du côté non-réducteur en formant un ion B, principalement dans les composés peracetylés et exclusivement pour ceux perméthylés.
Souvent associées à des analyseurs de type magnétique, les sources FAB produisent des spectres permettant de déduire grâce à la valeur précise des m/z des ions moléculaires ainsi qu’à celles des masse perdues, des informations quant à la structure de ces oligosaccharides et cela à des concentrations très faibles, de l'ordre du picomolaires.
5.1.2 Ionisation par Desorption Assistée par Laser (MALDI)
L’ionisation de type MALDI a été développée au milieu aux années 1980 pour l'analyse des molécules ayant de grands poids moléculaires (protéines intactes jusqu’à 200kDa). Cependant, il est devenu évident que beaucoup d'autres types de composés, y compris des oligosaccharides, pourraient être analysés par cette technique à des concentrations extrêmement faibles (femtomoles -picomoles). L’ionisation MALDI est 10 à 100 fois plus sensible que la FAB pour l'analyse des glycoprotéines et des dérivés de N-glycanes. Dans une expérience MALDI, l'échantillon est mélangé à une matrice de faible masse moléculaire (par exemple l’acide 2,5-dihydroxybenzoique) absorbant dans l'ultraviolet à la longueur d'onde du laser (337 nm pour le laser à azote habituellement utilisé). Le mélange est alors cristallisé par évaporation du solvant (la technique "de gouttelette sèche") et l'ionisation est effectuée par un laser pulsé. La matrice absorbe l'énergie de l'impulsion de laser qui est transférée à l'échantillon, par des mécanismes qui jusqu'à ce jour sont inconnus. Le transfert d'énergie à la matrice a comme conséquence la désorption de la matrice et de l'analyte de la surface de la cible sur laquelle ils avaient été déposés. Les ions ainsi formés sont présents dans la phase gazeuse, ils peuvent être électrostatiquement extraits, puis accélérés vers un analyseur de masse à temps de vol. Il existe un grand nombre de matrices dont le choix dépend du type d'oligosaccharide à étudier (l'acide 2,5-hydroxybenzoïque pour les oligosaccharides neutres et la 6-Aza-2-thiothymine ou la 2,4,6-trihydroxyacetophenone pour les oligosaccharides acides (Harvey, 1999)) .
33 Une ionisation de type MALDI permet la formation prédominante d'ions moléculaires monochargés et produit très peu d'ions fragments. Habituellement, les oligosaccharides donnent des espèces de type [M+Na]+ comme ion principal. Cet ion est souvent accompagné d'un plus faible [M+K]+. La MALDI-MS est donc la technique prépondérante pour examiner les ions moléculaires particulièrement quand une sensibilité élevée est exigée. Elle est particulièrement adaptée aux mélanges oligosaccharidiques.
L'inconvénient majeur de cette technique pour l’analyse des sucres réside dans l'analyse des sucres acides de type acide 2céto-3-desoxy ulosonique et uronique, qui possèdent une faible ionisation, car il se forme à la fois des ions de type [M+Na]+ et [M-H]- ainsi qu’une multitude de pics liés à des adduits de sodium ou potassium et à une dégradation aisée. Pour les acides ulosoniques, l'énergie d'ionisation produit une fragmentation au niveau de l'hydroxyle anomérique des acides ulosonique ou une décarboxylation. Les acides uroniques semblent quant à eux stable dans les conditions d'analyse.
5.1.3 Ionisation par “ElectroSpray” (ESI)
La spectrométrie de masse à ionisation Electrospray est un outil puissant pour l'analyse de molécules de grandes tailles et non-volatiles, notamment celles d'intérêt biologique, et a contribué aux progrès en sciences de la vie. L'ESI a révolutionné l'analyse des biopolymères labiles, avec des applications s'étendant de l'analyse des ADN, ARN, oligonucléotides, protéines, glycoprotéines, sucres, lipides, glycolipides, et lipopolysaccharides. Dans ce mode d'ionisation, une solution contenant les analytes est injectée à un débit de quelques microlitres/min par un capillaire en métal, tenu au potentiel élevé. L'effet du champ électrique élevé sur la solution produit la formation d'une nébulisation (spray) dont les gouttelettes sont fortement chargées. Le gaz, la chaleur, ou l’association des deux, sont appliqués aux gouttelettes fortement chargées, faisant évaporer le solvant. La taille des gouttelettes diminue . Lorsque les gouttelettes atteignent la taille critique pour laquelle les forces coulombiennes de surface surpassent les forces de tensions de surface, les gouttelettes explosent en de plus petites gouttelettes. Ce processus continue jusqu'à ce que soit atteint le point où un ion désorbe d'une gouttelette et se retrouve en phase gazeuse (le solvant est complètement éliminé). Le mécanisme exact de la formation des ions est en discussion, et il est probable que différents mécanismes s'appliquent en fonction de l'analyte et du solvant. Les composés ainsi libérés peuvent alors porter plusieurs charges. La distribution de charge est proportionnelle au
34 nombre de groupements ionisables présents dans la molécule. Les ions sont accélérés vers un analyseur de masse par une série de lentilles. L’electrospray possède deux intérêts majeurs pour la caractérisation de biopolymères : une ionisation efficace et la capacité d'obtenir des ions multichargés. La première permet de produire des faisceaux d'ions intenses tandis que la seconde permet de transmettre des composés de masse molaire élevée. Le procédé d'ionisation dans ESI-MS est très doux, ayant pour résultat peu ou pas de fragmentation. La fragmentation peut être induite par la manipulation de la tension des lentilles de la source d'ion, appelées cônes, ou par collision des ions dans une région choisie du spectromètre de masse : le quadridôle de collision situé entre deux analyseurs de masse. La dernière approche est habituellement préférable, puisqu'elle permet la sélection d'un ion précurseur par le premier analyseur et un meilleur contrôle de la fragmentation dans la chambre de collision. Pour cette raison, beaucoup d'analyseurs couplés à l'ESI sont des analyseurs dit en tandem qui permettent la détection des ions fragments produits par la collision (dissociation induite par collision (CID) des ions moléculaires. Cette analyse est appelée expérience MS-MS : l'ion parent est choisi par le premier analyseur et les ions fragments sont étudiés par le deuxième analyseur. Bien que les instruments triple-quadripôles soient le plus largement répandus, l’ESI peut également être couplé avec des analyseurs « ion-trap », temps-de-vol (ToF)et des analyseurs de masse hybrides. Avec des analyseurs « ion-trap », les analyses multi-étapes (MSn) sont possibles et sont très utiles pour l’analyse des oligosaccharides car elles permettent l'analyse des ramifications osidiques. La technique Q-TOF (spectromètre hybride entre un quadripôle et un temps de vol) est également compatible avec l'ESI et a montré des données analytiques extrêmement sensibles.
5.1.4 Ionisation par Désorption par electrospray (DESI)
Ces dernières années, la recherche s’est concentrée sur le développement de nouvelles techniques d’ionisation. L’analyse de solides à pression atmosphérique est facilement mise en application sur les sources d’ionisation existantes (ESI/APCI). Récemment, l'ionisation Par désorption par electrospray (DESI) a été mise au point par une société américaine (Prosolia). En DESI, des gouttelettes de solvant chargées sont accélérées par un gaz (azote) à haute pression vers une surface sur laquelle les analytes ont été déposés. Après impact, des gouttelettes secondaires contenant l’analyte sont formées et envoyées vers l’analyseur de masse. L'analyse des espèces de haute masse (>20kDa) est une de ses limitations. Ceci peut
35 être partiellement expliqué par l'incapacité du jet de solvant à dissoudre des protéine (ou toute autre molécule) de grande taille de la surface de la cible. De ce fait un problème de désorption est créé et diminue la sensibilité globale de l'analyse pour des espèces de haute masse molaire. En raison de sa sensibilité élevée pour de plus petites molécules, la DESI semble particulièrement adaptée à l'étude des oligosaccharides (Bereman et al., 2007). La comparaison des spectres de masse du même échantillon analysé par MALDI-FTICR et DESI-FTICR sont presque identiques et ce, malgré les différents mécanismes d’ionisation (la DESI étant réalisée à pression atmosphérique tandis que la MALDI est exécutée sous vide). Bereman et ses collaborateurs ont observé plus de crêtes dans le spectre de masse obtenu en DESI dues à des formes protonées et sodées des glycanes, tandis que dans le spectre obtenu en mode d’ionisation MALDI, seuls les adduits au sodium sont présents. La similitude entre les deux spectres démontre le potentiel de DESI-FT-ICR-MS pour les études d’oligosaccharides dans des échantillons complexes.
5.2 Mode de fragmentations des oligosaccharides
La nature des monosaccharides, les ramifications, et éventuellement les modes de liaisons peuvent être déterminés grâce à la fragmentation. La dissociation induite par Collision (CID) des glycoconjugés a comme conséquence l'observation des ions qui correspondent à la coupure de la partie oligosaccharide de la molécule. Typiquement, ces ions sont produits avec une plus grande abondance pour cette partie que ceux produits pour la partie non-oligosaccharide des glycoconjugés. La nomenclature utilisée en spectrométrie de masse pour la fragmentation des oligosaccharides est montrée sur le Figure 13. Les ions fragments qui contiennent la partie non-réductrice sont marqués avec les lettres majuscules du début de l'alphabet (A, B, C), et ceux qui contiennent l'extrémité réductrice de l'oligosaccharide ou de la partie non-oligosaccharide sont marqués avec des lettres de la fin de l'alphabet (X, Y, Z). Les ions produis à partir de la rupture des résidus successifs sont marqués : Am, Bm, et Cm, pour l'extrémité non-réductrice et le Xn, Yn, et Zn, pour l’extrémité réductrice. Notez qu’Y0 et Z0 se rapportent à la fragmentation de la partie non saccharidique. Les ions de type A et X sont produits par la rupture intracyclique, et sont marqués en indiquant les liaisons impliquées dans la coupure en partant de la liaison hémicétal intracyclique (0) et en augmentant l’indice dans le sens des aiguilles d’une montre. La figure 13 montre l’exemple de deux ions fragments obtenus par ce type de coupure (0,2A1 et1,5X1).
36
Figure 13 nomenclature des fragments obtenus en MSn (Zaia, 2004)
Pour la spectrométrie de masse en tandem des oligosaccharides, il y a plusieurs possibilités concernant l'état de l'ion précurseur, dont le choix influencera nettement le modèle de fragmentation, et ainsi l'information structurale produite. Pour les oligosaccharides natifs, les possibilités incluent la présence d’ions protonés [M+nH]n+, les ions déprotonés [M-nH]n- et les adduits sodium[M+Na]+. Ils sont monochargés en MALDI et multichargés en ESI. Les principes de la fragmentation des oligosaccharides pour la MSn ont été étudiés avec l'ionisation par bombardement rapide d'atome (FAB). Lorsque l’ionisation est de faible énergie, on observe plus particulièrement les fragments de type Y et B. Lorsque l’énergie est plus grande tous les fragments sont observés. La fragmentation impliquant une charge à distance exige plus d'énergie que la fragmentation induite par une charge, et le degré avec lequel la fragmentation se produit est proportionnel à la taille de cation impliqué. L’utilisation de grandes énergies en CID augmente la teneur en information des spectres de masse mais rend l’analyse des spectres plus délicate.
En plus de l’importance de l’énergie de collision pour l’obtention de fragments, la nature des sucres et la linéarité des molécules influencent la production des fragments. Pour les oligosaccharides protonés et cationisés, la présence des résidus de HexNAc place une charge sur l'azote de l'amide. La proximité de cette charge de l'oxygène anomérique prédispose à la rupture au niveau de l’HexNAc. Un modèle semblable est observé pour des oligosaccharides deprotonés en mode négatif. La présence de sucres particuliers tels les acides 2-céto-3-désoxy-octuolosonique produit la présence prépondérante en négatif et en positif de fragments obtenu après scission de la liaison glycosidique les impliquant. Les résultats obtenus en MALDI-FTMS sur des oligosaccharides alcalino-cationisés ont montré une corrélation entre le degré de ramification des oligosaccharides et le degré de fragmentation. En effet plus un oligosaccharide est ramifié moins il produit de fragments (Zaia, 2004).
37 L’analyse par spectrométrie de masse des oligosaccharides a subit au cours de ces dernières années de grands progrès. Malgré cela l’analyse des oligosaccharides ramifiés reste difficile. De plus il est toujours nécessaire de coupler cette méthode analytique performante avec d’autre méthodes afin de déterminer la composition saccharidique mais aussi de déterminer le mode de liaison.
Dans le cadre de ce travail de thèse, l'instrument dont nous disposons au sein du groupe spectrométrie de masse, fluorescence et bioanalytique du laboratoire des IMRCP est un ESi-Q-Tof Ultima commercialisé par la société Waters. Cet instrument, au vu de la littérature est bien adapté à notre problématique et devrait nous avancer dans l'analyse structurale des polysaccharides de surface de rhizobia. Néanmoins, comme il sera signalé au cours de ce travail, nous avons eu recours à des analyses complémentaires en MALDI-Tof (Brücker Daltonic, Billerica, MA, USA) et FT-ICR (Thermo Fisher, San Jose, CA). Ce dernier appareil étant localisé à l'Université de Borstel (Allemagne), il n'a pu être utilisé sur tout son panel d'applications.