HAL Id: tel-02614615
https://tel.archives-ouvertes.fr/tel-02614615
Submitted on 21 May 2020
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Laura Belot
To cite this version:
Laura Belot. Etude structurale et fonctionnelle de la glycoprotéine des Rhabdovirus. Biochimie [q-bio.BM]. Université Paris Saclay (COmUE), 2019. Français. �NNT : 2019SACLS522�. �tel-02614615�
de la glycoprotéine des
Rhabdovirus
Thèse de doctorat de l'Université Paris-Saclay
Préparée à l’Université Paris-Sud
École doctorale n°577 :
Structure et dynamique des systèmes vivants (SDSV)
Spécialité de doctorat: Sciences de la vie et de la santé
Thèse présentée et soutenue à Gif-Sur-Yvette, le 16 décembre 2019, par
Laura BELOT
Composition du Jury :
Mr Denis GERLIER
Directeur de Recherche, Centre international de Recherche en Infectiologie (CIRI) (UMR 5308)
Rapporteur
Mr Pierre-Yves Lozach
Directeur de Recherche, Institut National de la Recherche Agronomique (INRA) (UMR 754)
Rapporteur
Mme Marie-Christine Vaney
Chargée de Recherche, Institut Pasteur (Unité de Virologie structurale) Examinatrice
Mr Philippe Minard
Professeur, Institut de biologie Intégrative de la cellule (I2BC) (Département Biochimie, Biophysique et Biologie Structurale)
Président du jury Mr Yves Gaudin
Directeur de Recherche, Institut de biologie Intégrative de la cellule (I2BC) (CNRS–UMR 9198)
Directeur de thèse Mme Aurélie Albertini
Chargée de Recherche, Institut de biologie Intégrative de la cellule (I2BC) (CNRS–UMR 9198)
1 En premier lieu je souhaite remercier mon co-directeur de thèse Yves Gaudin pour m’avoir accueillie dans son département et plus particulièrement dans l’équipe Rhabdovirus. Je le remercie pour l’expérience enrichissante, et pleine d’intérêt, qu’il m’a fait vivre durant ces années. Il m’a fait découvrir le monde de la recherche et m’en a fait une vocation. Je lui suis extrêmement reconnaissante d’avoir toujours été à l’écoute, bienveillant, compréhensif et généreux de conseils avisés ainsi que pour la pertinence de ses critiques.
Je souhaite témoigner à Aurélie Albertini, ma co-directrice de thèse, toute ma gratitude pour sa patience, la grande confiance qu’elle m’a accordée, et le savoir-faire qu’elle m’a transmis. Travailler à ses côtés m’a permis cette émulation scientifique quotidiennequi a été l’un des piliers de ma motivation depuis mon premier stage dans cette équipe, durant ma première année de Master. Aurélie m’a accompagnée durant mes deux stages de Master, puis pour ces 3 années de thèse. Au fur et à mesure, nous avons développé une connivence qui m’a été très précieuse et pour laquelle je la remercie chaleureusement.
Je tiens également à remercier une nouvelle fois, et conjointement, mes deux co-directeurs de thèse Aurélie Albertini et Yves Gaudin, pour le temps qu’ils ont consacré à la relecture de mon manuscrit de thèse.
Je remercie (non sans une certaine émotion) l’ensemble des personnes de cette équipe, sans qui cette expérience n’aurait pas été aussi épanouissante.
Je remercie vivement Hélène Raux, pour l’intérêt qu’elle a porté à mes travaux tout au long de ma thèse, ainsi que pour les précieux conseils techniques et généraux, qu’elle a su partager avec moi.
De la même façon je remercie Danielle Blondel pour les nombreuses réponses qu’elle a su m’apporter, et pas seulement aux questions relatives à la thèse ou au futur post-doctorat. Je remercie Cécile Lagaudrière-Gesbert qui m’a fait découvrir l’équipe et sans qui je n’aurais pas vécu cette expérience.
Je remercie Nathalie Scrima pour son encadrement lors de mes premiers clonages et sa grande disponibilité tout le long de ma thèse.
Je remercie Ahmet pour sa gentillesse.
Je remercie Christine Maheu pour la quantité de virus qu’elle m’a fournie, en plus de sa bonne humeur.
Tous ont su m’intégrer, avec gentillesse et jovialité, y compris les anciens que je n’oublie pas, qui étaient là avant que l’aventure de ma thèse ne commence: Nolwenn Poccardi, Julie Takissian, Zoé Lama. Je remercie également avec nostalgie Jovan Nikolic pour sa bienveillance, sa grande gentillesse, son soutien et les chasses aux Ferrero. Je remercie Abbas Abou Hamdam et je remercie chaleureusement Alice Decombe pour le soleil qu’elle a su m’apporter durant ses 6 mois de stage au sein de ce laboratoire.
Je remercie également très chaleureusement Malika Ould Ali pour sa sympathie, son écoute, et pour m’avoir formée à la microscopie électronique. Je la remercie pour la confiance qu’elle m’a accordée sur les appareils, ainsi que pour le temps qu’elle a consacré à regarder nos petits objets en cryo-microcopie électronique.
En somme, je remercie l’ensemble de l’équipe pour l’accueil chaleureux et professionnel qu’elle m’a réservé et la contribution de chacun à la réalisation de ma thèse dans les meilleures conditions. Je remercie également les autres membres du département de
2
une pensée particulière pour Stéphane Roche pour les très nombreuses discussions que l’on a eues autour des cristaux que j’ai obtenus, pour ses encouragements et sa bonne humeur perpétuelle.
Je vous remercie tous, très sincèrement, de m’avoir permis de profiter pleinement de cette expérience enrichissante. Tant au plan professionnel que personnel.
Je tiens à remercier les membres de mon jury de thèse, les Dr. Denis Gerlier et Pierre-Yves Lozach d’avoir accepté d’être rapporteurs de ma thèse, ainsi que le Dr. Marie-Christine Vaney et le Pr. Philippe Minard qui ont accepté d’en être les examinateurs. Je profite de ces remerciements pour en adresser un particulier au Pr. Philippe Minard, l’enseignant qui a éveillé en moi l’attrait pour la biochimie et qui m’a amenée à suivre le Master dont il est co-responsable.
Je remercie mes tuteurs Bernard Delmas et Julie Ménétrey pour leur accompagnement durant ma thèse ainsi que leur très grande gentillesse.
Je voudrais remercier également :
Magali Aumont-Nicaise, pour les mesures d’ITC mais surtout pour sa patience et sa grande amabilité ; Christophe Velours, pour l’UCA et le SEC-MALS, toujours réalisés avec le sourire ; Stéphane Plancqueel pour les cribles de cristallogenèse, les fous rires et surtout sa très grande disponibilité.
Pierre Legrand, avec qui nous collaborons, pour sa disponibilité et le savoir-faire qu’il a su partager avec gentillesse et pédagogie ainsi que l’aide qu’il nous a apportée.
Enfin je souhaiterais remercier affectueusement les personnes sans qui je ne pourrais pas être celle que je suis, qui m’ont soutenue sans relâche, et ont également rendu possible cette thèse. Je commencerai de toute évidence par mon père et ma mère, qui ont toujours cru en moi et m’ont fait confiance. Je remercie ma famille et mes amis. Je remercie Maëlle, de m’avoir tant exprimé sa fierté. Je terminerai en témoignant mon affection à François qui m’a accompagnée tout au long de cette expérience et surtout de m’avoir soutenu pour les derniers moments de cette thèse.
3
Remerciements ... 1
Table des figures ... 8
Liste des abréviations ... 10
INTRODUCTION ... 11
1. Introduction générale ... 11
1.1. La famille des Rhabdoviridae ... 11
1.2. Le VSV ... 12
1.2.1. Etiologie de la stomatite vésiculeuse ... 12
1.2.2. Physiopathologie de la stomatite vésiculeuse ... 13
1.3. Le virus de la rage ... 14
1.3.1. Étiologie de la rage ... 14
1.3.2. Physiopathologie de la rage ... 15
1.3.3. Traitement ... 15
1.4. Structure de la particule virale des Rhabdovirus ... 16
1.5. Le cycle viral ... 17
1.5.1. Entrée virale ... 17
1.5.2. Synthèse des composants viraux de VSV et de RABV ... 18
1.5.2.a. La transcription virale ... 18
1.5.2.b. La traduction des messagers viraux ... 19
1.5.2.c. La réplication virale... 19
1.5.3. Assemblages et bourgeonnement ... 20
1.6. Les protéines virales et leur implication dans l’infection ... 22
1.6.3. La ribonucléoprotéine ... 22
1.6.3.a. La nucléoprotéine ... 22
1.6.3.b. La phosphoprotéine ... 24
1.6.3.c. L’ARN polymérase ARN-dépendante ... 26
1.6.4. Les protéines associées à l’enveloppe virale ... 28
1.6.4.a. La protéine de matrice ... 28
2. La glycoprotéine et l’entrée des Rhabdovirus ... 31
2.1. Généralités sur G ... 31
2.2. Structure de la glycoprotéine des vésiculovirus... 32
2.3. Attachement et récepteurs... 34
2.3.1. Les facteurs d’attachement ... 35
2.3.2. Les récepteurs ... 35
4
2.3.2.b. Les récepteurs de RABV ... 37
(i) Le récepteur nicotinique de l’acétylcholine ... 37
(ii) La molécule d’adhésion des cellules neuronales ... 38
(iii) Le récepteur murin du facteur de croissance nerveux de faible affinité ... 38
(iv) Le récepteur métabotropique du glutamate 2 ... 39
2.3.2.c. Les récepteurs d’autres Rhabdovirus ... 39
2.4. Voie d’entrée des Rhabdovirus dans la cellule-hôte ... 40
2.4.1. Entrée par endocytose médiée par la clathrine ... 40
2.4.2. Entrée dépendante de l’actine ... 40
2.4.3. Le transport des Rhabdovirus après internalisation ... 41
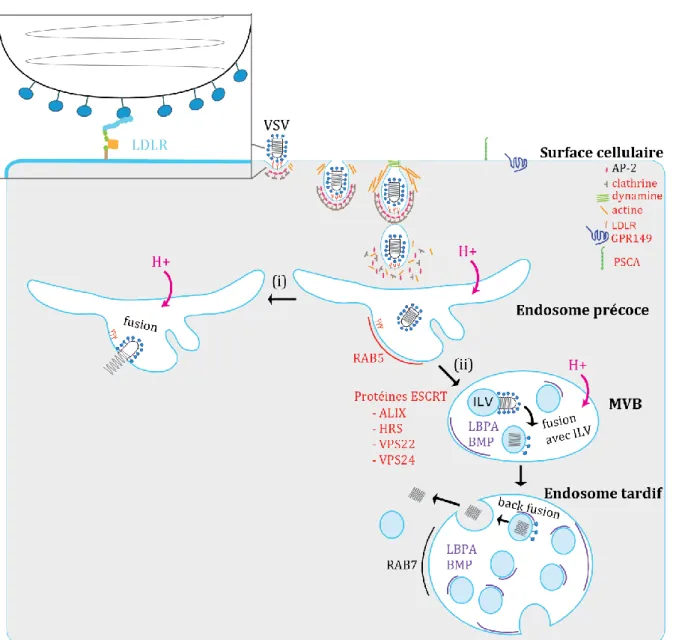
2.4.3.a. Le transport du VSV dans la voie endosomale ... 41
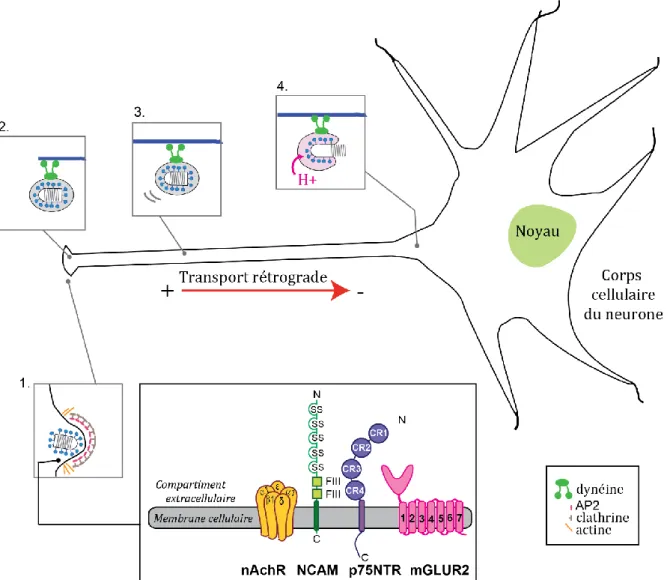
2.4.3.b. Le transport de RABV dans l’axone... 42
2.5. La fusion membranaire ... 44
2.5.1. Considérations générales ... 44
2.5.2. Propriétés de fusion des Rhabdovirus ... 46
2.5.3. Les différentes classes de protéines de fusion virales ... 46
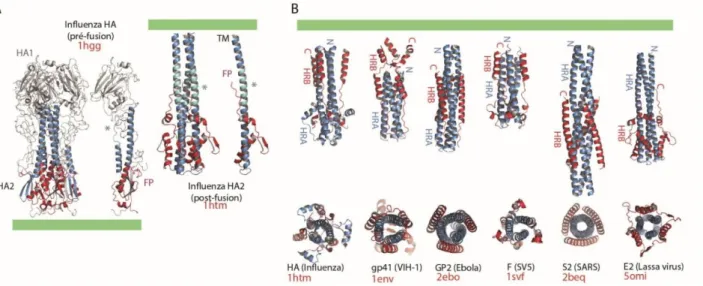
2.5.3.a. Les protéines de fusion de classe I ... 47
(i) Influenza HA, le prototype des glycoprotéines de fusion de classe I ... 47
(ii) Caractéristiques communes aux protéines de fusion de classe I ... 48
2.5.3.b. Les protéines de fusion de classe II ... 49
(i) TBEV E, le prototype des glycoprotéines de fusion de classe II ... 49
(ii) Caractéristiques communes aux protéines de fusion de classe II ... 50
2.5.3.c. Les protéines de fusion de classe III ... 51
(i) VSV G, le prototype des glycoprotéines de fusion de classe III ... 51
(ii) Caractéristiques communes aux protéines de fusion de classe III ... 51
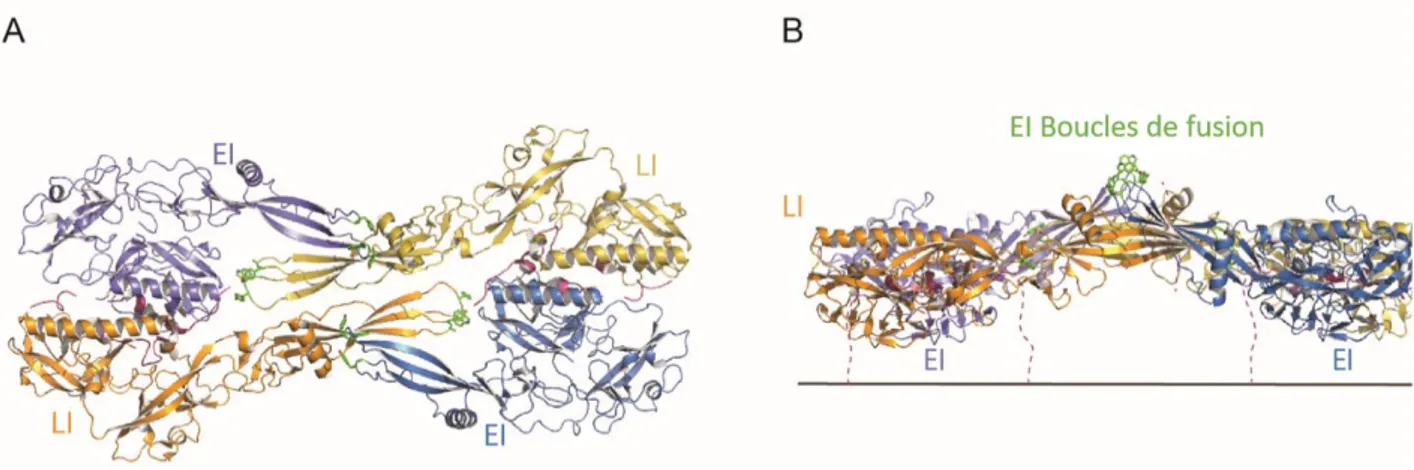
2.5.4. Mécanisme de la fusion membranaire induite par les Rhabdovirus ... 52
2.5.4.a. Une coopérativité nécessaire ... 52
2.5.4.b. Piégeage d’intermédiaires durant le processus de fusion ... 52
2.5.4.c. Fonctionnement de la machinerie de fusion des Rhabdovirus ... 53
(i) Interaction des boucles de fusion avec la membrane cible ... 53
(ii) Interrupteurs sensibles au pH ... 53
(iii) Intermédiaires structuraux durant la transition structurale de G ... 54
3. Antigénicité des Rhabdovirus ... 58
3.1. Anticorps et réponse immunitaire ... 58
5
3.2.1.b. Le modèle d’étude viral ... 58
(i) Sélection de mutants d’échappement ... 58
(ii) Utilisation de mutants de délétion, de chimères ou bien pontuels ... 59
(iii) Alignement de séquences ... 59
(iv) Génération de fragments chevauchants ... 59
3.2.2. Les sites antigéniques de RABV G ... 59
3.2.3. Les sites antigéniques de VSV G ... 62
3.2.5. Les anticorps et les traitements alternatifs de la rage ... 62
4. Les objectifs de la thèse ... 65
4.1. Les bases moléculaires de l’interaction entre VSV G et son récepteur ... 65
4.2. L’antigénicité de VSV G ... 65
4.3. Structure de la G de Lyssavirus ... 65
MATERIEL ET METHODES ... 67
1. Biologie moléculaire ... 67
1.1. Souches bactériennes ... 67
1.2. Milieux de cultures bactériens ... 67
1.3. Clonage ... 67
2. Biologie cellulaire ... 68
2.1. Souches cellulaires ... 68
2.2. Virus et souches ... 69
2.3. Infection des cellules ... 69
2.4. Production et purification de virus ... 69
2.5. Neutralisation de l’infection virale par les Fab ... 70
2.6. Interaction entre VSV G et les Fab ... 70
3. Biochimie ... 70
3.1. Production et purification des protéines ... 70
3.2. SEC MALS ... 73
4. Biologie structurale ... 73
4.1. Cristallogenèse... 73
4.2. Microscopie et cryo-EM ... 73
4.2.1. Microscopie électronique en coloration négative de VSV ou de différentes G ... 73
4.2.2. Cryo-EM de VSV Gth-Fab et GPVfl ... 74
RESULTATS ... 77
1. Les bases moléculaires de l’interaction entre VSV G et son récepteur ... 77
6
2. Caractérisation structurale de la glycoprotéine de VSV en complexe avec un anticorps
neutralisant ... 96
2.1. Contexte ... 96
2.2. Objectifs ... 96
2.3. Résultats ... 97
2.3.1. Neutralisation de l’infection virale par le Fab ... 97
2.3.2. Etude de l’interaction entre VSV G et le Fab ... 97
2.3.2.a. Interaction entre Gth et le Fab 8G5F11 ... 97
2.3.2.b. Interaction entre VSV G et le Fab 8G5F11, à la surface des particules virales . 98 2.3.3. Isolation et caractérisation du complexe G/Fab ... 99
2.3.4. Etude structurales du complexe Gth-Fab... 101
2.3.4.a. Cristallisation du complexe Gth-Fab ... 101
2.3.4.b. Etude du complexe Gth-Fab par microscopie électronique... 103
2.3.4.c. Etude du complexe Gth-Fab par cryo-EM ... 105
2.4. Conclusion ... 106
3. Structure de la G des Lyssavirus ... 107
3.1. Contexte ... 107
3.2. Objectifs ... 108
3.3. Résultats ... 108
3.3.1. Etude de l’ectodomaine de GPV ... 108
3.3.1.a. Obtention de l’ectodomaine soluble de GPV ... 108
3.3.1.b. Etude structurale de l’ectodomaine de GPV ... 109
(i) Cristallogenèse de l’ectodomaine de GPV... 109
(ii) Observation par microscopie électronique de l’ectodomaine de GPV ... 109
3.3.2. Etude de la forme complète de GPV ... 110
3.3.2.a. Obtention de la forme entière de GPV à partir des particules virales ... 110
3.3.2.b. Etude structurale de GPVfl ... 111
(i) Observation par microscopie électronique de GPVfl ... 111
3.3.3. Etude structurale de l’ectodomaine de la glycoprotéine d’autres Lyssavirus ... 112
3.3.3.a. Obtention des ectodomaines des glycoprotéines des virus MOK et WBC ... 113
3.3.3.b. Etude structurale la glycoprotéine des virus MOK et WCB ... 113
(i) Cristallogenèse de l’ectodomaine de GMOK ... 113
(ii) Etude par microcopie électronique de l’ectodomaine de GMOK ... 114
(iii) Etude par microcopie électronique de l’ectodomaine de GWCBm ... 115
7
1.1. Les récepteurs du VSV ...117
1.2. VSV G, un outil en biotechnologie ...118
2. Caractérisation structurale de VSV G en complexe avec un anticorps neutralisant ...119
2.1. Caractérisation de l’interaction entre VSV G et le Fab 8G5F11 ...119
2.2. Mécanisme de neutralisation des Fab ...121
2.3. Structure du complexe G-Fab 8G5F11 ...121
3. Structure de la G des Lyssavirus ...121
3.1. L’étude de l’ectodomaine de G des Lyssavirus ...122
3.2. L’étude de la forme complète de GPV ...122
RÉFÉRENCES BIBLIOGRAPHIQUES ... 123
ANNEXES ... 141
Images de microscopie électronique ...141
8
Figure 2 : Distribution mondiale de la stomatite vésiculeuse et troubles physiques induits par
l’infection. ... 13
Figure 3. Distribution du risque d’être en contact avec le virus de la rage en 2013 ... 14
Figure 4. Cliché de Rhabdovirus par EM après coloration négative. ... 16
Figure 5. Organisation de la particule virale et génome des Rhabdovirus. ... 17
Figure 6. Cycle viral des Rhabdovirus. ... 17
Figure 7.Schéma du génome viral ... 18
Figure 8. Schéma de la réplication et transcription virale ... 19
Figure 9.Schéma de la transcription et de la réplication du virus de la rage. ... 20
Figure 10.Modèle d'assemblage et de bourgeonnement des Rhabdovirus. ... 21
Figure 11. Structure de la N de RABV. ... 23
Figure 12. La phosphoprotéine des Rhabdovirus. ... 26
Figure 13. Structure de la polymérase de VSV. ... 27
Figure 14. Structure de la protéine de matrice de VSV. ... 29
Figure 15. Structures de la glycoprotéine de VSV et de CHAV. ... 33
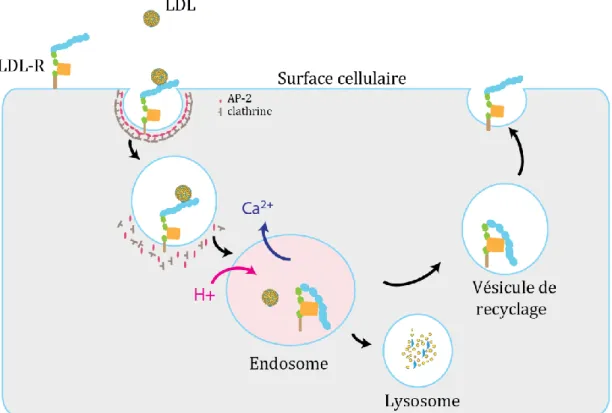
Figure 16. Cycle du LDL Récepteur dans l'homéostasie du cholestérol ... 36
Figure 17. Structure cristallographique de la forme endosomale du LDLR et schéma du LDLR sous sa forme endosomale à pH acide et extracellulaire à pH neutre. ... 37
Figure 18. Les récepteurs proposés pour RABV. ... 37
Figure 19. Voies proposées pour l'entrée du VSV dans la cellule hôte. ... 42
Figure 20.Mécanisme proposé pour l'entrée de RABV et son transport rétrograde dans les neurones. ... 44
Figure 21. Schéma du processus de fusion membranaire par l'intermédiaire d'un intermédiaire hémifusion. ... 45
Figure 22. Protéines de fusion de classe I. ... 48
Figure 23. Protéines de fusion de classe II. ... 50
Figure 24. Les éléments interrupteurs sensibles au pH. ... 54
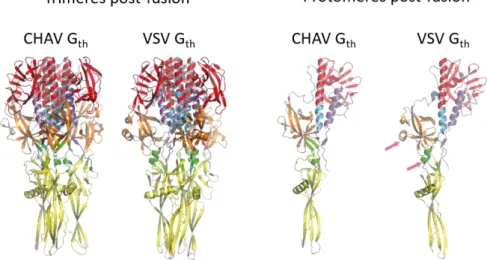
Figure 25.Structure de la G de CHAV dans l'unité asymétrique du cristal. ... 55
Figure 26.Voie plausible du changement de conformation des Vésiculovirus G. ... 56
Figure 27. Dimère de G de CHAV. ... 57
Figure 28. Sites antigéniques de RABV G. ... 61
Figure 29. Sites antigéniques de VSV G. ... 63
Figure 30. Différences structurales entre les G de VSV et CHAV sous conformation post-fusion. ... 64
Figure 31. Les domaines CR du LDLR. ... 76
9 Figure 35. Morphologie de G à la surface des particules virales à pH 8 et à pH 6, en absence et
en présence des Fab 8G5F11. ...100
Figure 36. Purification de Gth, des Fab 8G5F11 et du complexe Gth-Fab 8G5F11 ...102
Figure 37. Galerie des cristaux du complexe Gth-Fab. ...103
Figure 38. Morphologie de Gth en solution à pH 8 et à pH 6, en absence et en présence des Fab 8G5F11, visualisés par microscopie électronique en coloration négative. ...104
Figure 39. Observations de Gth à pH 6, en absence et en présence des Fab, par cryo-EM. ....106
Figure 40. Arbre phylogénétiques des Lyssavirus. ...107
Figure 41. Purification de GPV440m. ...108
Figure 42. Galerie des cristaux de GPV440m-streptag. ...109
Figure 43. Morphologie de GPV440m en coloration négative à pH 8 et pH 6. ...110
Figure 44. Purification de GPVfl. ...111
Figure 45. Morphologie de GPVfl à pH 6, après coloration négative à l’acétate d’uranyle. ....111
Figure 46. Images de cryo-EM de GPVfl en présence de détergent à pH 6. ...112
Figure 47. Purification de GMOKm et GWCBm. ...113
Figure 48.Galerie des cristaux de GMOKm-streptag. ...114
Figure 49. Morphologie de GMOKm et GMOKwt à pH 6. ...114
Figure 50. Morphologie de GWCBm à pH 8 et à pH 6 en microscopie électronique. ...115
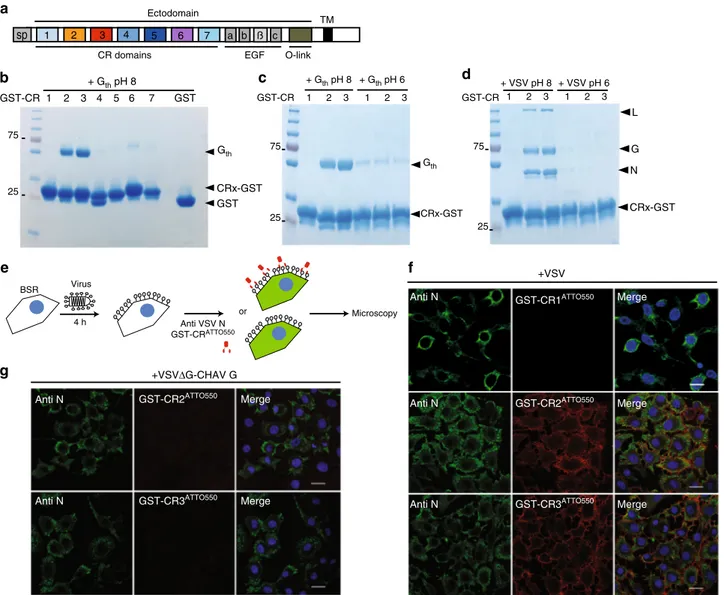
Figure 51. Interaction HRV2VP1-V3, VSV G-CR2 et VSV G-CR3. ...118
Figure 52. Schéma illustrant le ciblage de cellules d’intérêt avec VSV Gnano*. ...119
Figure 53. Localisation de l'épitope reconnu par l'anticorps 8G5F11. ...120
Figure 54. Variation de l'épaisseur de la couche de G à la surface des particules virales en présence des Fab. ...120
10
ARNm ARN messager
BEFV Le virus de la fièvre éphémère bovine CHAV Virus Chandipura
CMC Concentration micellaire critique CME Endocytose médiée par la clathrine CN Corps de Negri
DAG Diacylglycérol
DNTS Segment N-terminal désordonné DUVV Virus Duvenhage
ESCRT Complexe de sortie endosomale requis pour le transport Fab Fragment de liaison à l’antigène
FC Fragment cristallin G Glycoprotéine
GPV Glycoprotéine du virus de la rage souche PV GNS Glycoprotéine non structurale
ILV Vésicule intraluminale
HA Hemmaglutinine du virus de la grippe IRF Facteur régulateur de l’interféron kDa kiloDalton
L ARN polymérase ARN-dépendant LPC Lysophosphatidylcholine
M Protéine de Matrice MOKV Virus Mokola
MVB Corps multivésiculaires N Nucléoprotéine
NC Nucléocapside
NES Signal d’export nucléaire P Phopshoprotéine
PC Phosphatidylcholine ou tampon phosphate-citrate PE Phosphatidyléthanolamine
PKC Proteine kinase C
PML Protéine de leucémie promyélocytaire PS Phosphatidylsérine
PV Virus de la rage souche Pasteur Virus RABV Virus de la rage
RNP Ribonucléoprotéine Rpm Rotation par minute
RVPK Phosphoprotéine kinase du virus de la rage SFV Virus de la forêt de Semliki
SNC Système nerveux central
STAT 1 Le transducteur de signal et l'activateur de transcription 1 VHSV Virus de la septicémie hémorragique virale
VSV Virus de la stomatite vésiculeuse WCBV Virus de la chauve-souris ouest africaine
11
1. Introduction générale
1.1. La famille des Rhabdoviridae
La famille des Rhabdoviridae appartient à l'ordre des Mononegavirales, et contient 18 genres différents et une espèce non attribuée (Virus Moussa) (Walker et al., 2018).
Les Rhabdovirus sont répandus parmi une grande diversité d’organismes. On les retrouve chez les vertébrés (mammifères, oiseaux, reptiles, poissons), les invertébrés (insectes, arachnides, crustacés) et les plantes (Figure 1 et Tableau 1). Plusieurs membres des Rhabdoviridae sont associés à des pathologies importantes chez l’homme et le bétail (Dietzgen et al., 2017).
Figure 1. Taxonomie de l’ordre des Mononegavirales.
Les génotypes indiqués en gras correspondent au prototype de chaque famille virale. Les encarts rouges indiquent la phylogénie des souches virales étudiées durant la thèse.
Les deux genres les plus étudiés chez les Rhabdovirus sont le genre Lyssavirus, dont le prototype est le virus de la rage (RABV) et le genre Vésiculovirus, pour qui le prototype est le virus de la stomatite vésiculeuse (VSV). Les Lyssavirus n'infectent que les mammifères chez qui ils causent des encéphalites mortelles. Ce genre est subdivisé en 3 phylogroupes différents, basés essentiellement sur la distance génétique et l’antigénicité de chaque groupe d’isolat.
12
*sont transmis par les arthropodes
Tableau 1. Tableau représentant les différents genres de la famille Rhabdoviridae et leurs hôtes respectifs.
1.2. Le VSV
1.2.1. Etiologie de la stomatite vésiculeuse
Le VSV est un Vésiculovirus responsable de la stomatite vésiculeuse. Cette maladie n’est pas une maladie émergente puisque des cas datant du début des années 1800 ont été répertoriés aux Etats-Unis (Hanson, 1952). Cette maladie s’est fait connaitre plus tard lorsqu’elle a handicapé 4000 chevaux durant la Guerre civile américaine en 1862.
La stomatite vésiculeuse est endémique dans certaines régions au Sud du Mexique, en Amérique centrale, au Venezuela, en Colombie, en Equateur et au Pérou. Des foyers ont été rapportés au Nord du Mexique et dans l’Ouest des Etats-Unis, notamment en 2014 au Texas et au Colorado. Des cas sporadiques ont été rapportés en Argentine et au Brésil également. La contagion hors de ces pays a ensuite été causée lors de l’envoi de chevaux contaminés depuis les Etats-Unis vers l’Afrique du Sud (à partir des années 1880) puis en Europe au cours de la première Guerre Mondiale, elle a historiquement été décrite en France en 1915 et 1917. Aujourd’hui, elle est principalement confinée au continent américain (Figure 2,A).
Genre Hôte
Almendravirus Les moustiques
Cytorhabdovirus Les plantes et les insectes vecteurs
Curiovirus Les moustiques, les moucherons et phlébotomes, vertébrés*
Dichorhavirus Les plantes
Ephemerovirus Le bétail* et les moustiques
Hapavirus Les moustiques, les moucherons, les lézards, les oiseaux* et les mammifères*
Ledantevirus Les moustiques, les tiques, les puces, les mammifères* dont l’Homme*
Lyssavirus Les mammifères dont l’Homme
Novirhabdovirus Les poissons
Nucleorhabdovirus Les plantes et les sauterelles
Perhabdovirus Les poissons
Sigmavirus Les drosophiles
Sprivivirus Les poissons*
Spipuvirus Les moucherons, les lézards
Tibrovirus Le bétail*, les buffles*
Tupavirus Les oiseaux
Varicosavirus Les plantes
13 Figure 2 : Distribution mondiale de la stomatite vésiculeuse et troubles physiques induits par l’infection.
A. La carte (Kumar et al, 2018) montre l’incidence mondiale de la maladie, les pays comportant des
preuves sérologiques et récurrentes d’une infection sont représentés en rouge. B. Présence de vésicules sur la langue, les babines et les sabots d’un cheval, due à une infection par le VSV.
1.2.2. Physiopathologie de la stomatite vésiculeuse
Chez les bovins, les chevaux et les porcs, la stomatite vésiculeuse est caractérisée par une apparition de vésicules sur la langue, les babines, les muqueuses buccales, les sabots, les tétines et le prépuce. Ces lésions sont douloureuses et s’accompagnent de fièvre. Les animaux retrouvent leur état normal en deux à trois semaines. La maladie n’est pas facilement reconnaissable car elle ne peut être distinguée de la fièvre aphteuse sur ses signes cliniques, donc en attendant le diagnostic, les animaux sont isolés.
Bien que VSV ne soit pas mortel, il engendre un taux de morbidité élevé du fait de sa contagiosité, ce qui a des conséquences économiques puisque l’infection peut s’étendre à un élevage entier. Comme il n’y a pas de traitement, une règle européenne exige l’abattage des animaux diagnostiqués, afin de limiter la contagion, qui est toujours possible malgré l’isolement. En effet, le virus peut infecter des animaux par transmission directe par voie transcutanée et transmucosale, via la salive, et après rupture des vésicules induisant la libération du liquide vésiculaire chargé en particules virales ; mais aussi par voie indirecte par transmission vectorielle par les insectes. Cette transmission vectorielle peut se faire par un insecte piqueur, et dans ce cas le confinement d’un animal infecté ne suffit pas à éviter la transmission aux animaux sains. La transmission peut également se faire lors de l’ingestion d’insectes infectés présents dans les pâturages (Drolet et al., 2009).
La stomatite vésiculeuse est une zoonose, c’est-à-dire une maladie infectieuse des animaux vertébrés transmissible à l'être humain. L’infection humaine dans les zones endémiques apparait de façon commune et est directement liée au contact avec les animaux contaminés. Elle peut être aussi détectée chez les travailleurs dans les laboratoires étudiant
14
le VSV. De fait, avant que ne soient instaurés les procédures et équipements modernes de sécurité biologique, les personnes infectées étaient des personnes manipulant des animaux infectés par le VSV ou le virus lui même. La maladie peut apparaitre 20-30 h après l'exposition, dure de 3 à 6 jours et n'est pas associée à d’autres complications que les lésions vésiculaires qui peuvent être présentes dans le pharynx, la muqueuse buccale ou la langue. VSV peut tout de même induire une immunodépression chez la personne porteuse qui peut subir des épisodes fébriles. (Letchworth et al., 1999)
1.3. Le virus de la rage
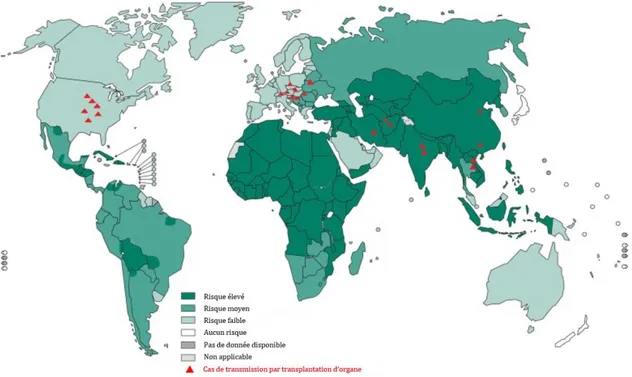
1.3.1. Étiologie de la rageL’origine du mot rage provient du mot latin « rabere » : être fou. La rage est l'une des plus anciennes maladies, avec des cas décrits remontant à 2000 ans avant notre ère. Les personnes étaient tellement effrayées par la rage que, après avoir été mordues par un animal potentiellement enragé, beaucoup se suicidaient. Aujourd’hui, la rage est présente sur tous les continents à l'exception de l'Antarctique. Les chercheurs estiment qu'environ 59 000 décès humains sont imputables à la rage chaque année, les pays les moins développés étant davantage touchés : la plupart des cas sont signalés en Afrique(entre 21 000 et 25 000) et en Asie (environ 35 000)(Broban et al., 2018; Hampson et al., 2015) (Figure 3).
Le virus de la rage a un large réservoir d’hôtes chez les mammifères parmi les ordres Carnivora (en particulier les chiens, les chats, les animaux sauvages tels que le renard, les loups, les ratons laveurs, les chacals, les moufettes, les coyotes) et Chiroptera (les chauves-souris). Dans les pays développés, les animaux domestiques ne sont responsables que d’environ 10% des cas de transmission du virus de la rage, tandis que les animaux sauvages (surtout les chauves-souris) sont responsables de la majorité des cas. Tout mammifère peut être porteur de la rage. La transmission peut se faire par la morsure d’un animal infecté, mais aussi par le contact de la salive de l’animal enragé avec une peau lésée ou des muqueuses. Les autres voies d'infection comprennent l'inhalation du virus sous forme d'aérosol, l'ingestion, la voie transplacentaire et même la transplantation d'organes (Lu et al., 2018).
15
1.3.2. Physiopathologie de la rage
La rage est une zoonose généralement transmise à l'homme par morsure d’un animal infecté. Dans un premier temps, le virus reste localisé au niveau du site d’infection (Murphy and Bauer, 1974), il se multiplie au niveau des tissus périphériques et des tissus musculaires. Ce myotropisme précoce rend le traitement post-exposition possible. Le virus se déplace ensuite dans le système nerveux périphérique pour finalement envahir le système nerveux central. Au sein des neurones, le virus se déplace de façon rétrograde avec une vitesse de déplacement d’environ 3 mm/h (Tsiang, 1979) et dépend pour cela des microtubules. Le virus retourne ensuite au système nerveux périphérique, en particulier dans les zones très innervées, comme par exemple les glandes salivaires, et ainsi l’animal atteint peut à son tour transmettre le virus.
Après inoculation, la rage comporte cinq stades :
(i) L’incubation qui est la période définie entre l’inoculation et l'apparition des premiers symptômes. Elle peut durer de quelques jours à plusieurs années, en fonction de de la charge virale reçue, de la localisation et de la gravité de la plaie lors de l’infection.
(ii) Le prodrome survient à la fin de l’incubation et comprend des symptômes semblables à ceux causés par une infection virale (fièvre, malaise, maux de tête) (iii) La maladie neurologique aiguë correspond à l'apparition des symptômes
neurologiques. On distingue ainsi la maladie neurologique encéphalitique appellée "rage furieuse" (représentant environ 85% des cas) et la maladie neurologique paralytique appelée "rage muette".
La rage furieuse démarre sur une anxiété se traduisant par une hydrophobie qui produit des spasmes musculaires pharyngiens intenses à la vue, au contact ou au son de l'eau. L’état du malade se dégrade avec l’apparition de tachycardie, augmentation du rythme respiratoire, fièvre, hypersalivation, hyperactivité et enfin un délire franc.
Au contraire la rage paralytique engendre une paralysie flasque ascendante et des troubles sensoriels (Warrell, 1976).
Une forme plus rare de la maladie est associée à des convulsions et à des symptômes moteurs et sensoriels plus profonds, pouvant être corrélés au fait que chez certains patients atteints, la consommation d'oxygène cérébral est considérablement réduite, ce qui induit une atteinte cérébrale irréversible.
(iv) Le coma peut débuter dix jours après l’apparition des symptômes de la maladie neurologique aigue, développer des apnées prolongées et avoir une paralysie flasque.
(v) La mort survient rapidement après le coma. Les causes peuvent résulter d’une insuffisance cardiopulmonaire ou être liées à la gravité de l’encéphalite.
1.3.3. Traitement
Lorsque les premiers symptômes de la rage apparaissent, il n'y a pas de traitement efficace et l’issue est forcément fatale. Les travaux de Pasteur ainsi que ceux des prédécesseurs oubliés, Toussaint et Galtier, puis le travail de Roux visant à atténuer le vaccin vivant de Pasteur (Laurioz, 2005), ont permis de développer les premiers vaccins antirabiques afin de prévenir l’apparition de la maladie, cela concerne actuellement les animaux domestiques mais également les personnes exposées (personnel de laboratoire,
16
pays à risque). Des campagnes de vaccination orale des animaux sauvages ont également été réalisées utilisant des appâts piégés par des virus vivants atténués.
Après la morsure d’un animal enragé, il est possible d’éviter la maladie par l’utilisation d’un vaccin antirabique en prophylaxie ou l’injection d’immunoglobulines antirabiques. Ce traitement est possible avant l’apparition des symptomes. Aucune prophylaxie n'est possible une fois que le virus a atteint le système nerveux central. Dans ce cas, le pronostic est très mauvais. Parfois, les soins intensifs ont produit des résultats prometteurs : il est possible de prévenir les complications potentiellement mortelles (Warrell, 1976) mais la gravité de l'encéphalite constitue le dernier obstacle à la survie.
1.4. Structure de la particule virale des Rhabdovirus
Les Rhabdovirus sont des virus enveloppés qui possèdent un génome non segmenté constitué d'une molécule d'ARN simple brin de polarité négative, qui n’est pas traduisible en protéine car est incapable de recruter des ribosomes et est donc non infectieux isolément. De façon très générale, en microscopie électronique, le virion a une forme caractéristique en obus avec une base plate et une extrémité arrondie ( Figure 4 A). Les virions du genre Nucleorhabdovirus comme par exemple le virus de la mosaïque du maïs, apparaissent quant-à-eux bacilliformes et ont leurs deux extrémités arrondies ( Figure 4 B).
Figure 4. Cliché de Rhabdovirus par EM après coloration négative.
A. VSV. B. Virus de la mosaïque du maïs.
Les barres d’échelle représentet 100 nm.
Au sein d’un même genre, les virus ont en général la même organisation génomique, particulièrement en ce qui concerne le nombre et les emplacements des gènes accessoires. Le génome, de taille allant de 10,8 à 16,1 kb (Walker et al., 2015), contient jusqu'à une douzaine de gènes, dont cinq seulement sont communs à tous les membres de la famille. Ces gènes communs codent, par ordre d’apparition dans le génome (Flamand and Delagneau, 1978; Tordo et al., 1986a) pour la nucléoprotéine (N), la phosphoprotéine (P), la protéine de matrice (M), la glycoprotéine (G) et l’ARN-polymérase ARN-dépendante (L) (Figure 5). Le génome est associé à N, L et P pour former la ribonucléoprotéine (RNP), qui est enroulée en une structure hélicoïdale à l'intérieur de la particule virale et qui est séparée de la membrane virale par la protéine M. M tapisse l’intérieur de la membrane virale et établit un lien entre celle-ci et la RNP. A la surface du virion on retrouve une unique protéine transmembranaire : la glycoprotéine virale (G) (Figure 5).
17
Figure 5. Organisation de la particule virale et génome des Rhabdovirus. Adapté de ViralZone,2017, Swiss Institute of Bioinformatics.
1.5. Le cycle viral
1.5.1. Entrée viraleLe cycle viral des Rhabdovirus est initié par la liaison de G aux récepteurs cellulaires. La particule virale pénètre ensuite dans la cellule par voie endocytique, puis l’acidification de l’endosome dans lequel le virus a été internalisé induit un changement conformationnel de G depuis une conformation pré-fusion (ou native) vers une conformation post-fusion (ou finale). Ce changement conformationnel catalyse la fusion des membranes virale et endosomale. La fusion permet la libération de la RNP virale dans le cytoplasme de la cellule pour les étapes ultérieures du cycle (Figure 6).
Les étapes du cycle viral médiées par G seront abordées en détail dans la partie 2 de l’introduction.
Figure 6. Cycle viral des Rhabdovirus.
18
1.5.2. Synthèse des composants viraux de VSV et de RABV
1.5.2.a. La transcription virale
Le génome viral arrivant dans le cytoplasme est toujours associé à la nucléoprotéine N. A son extrémité 3’, il débute par une séquence, codant pour un ARN leader non codant (Conzelmann, 1998; Tordo et al., 1986b), qui régule la transition de la transcription vers la réplication (Figure 7). Le génome code ensuite pour les gènes des cinq protéines virales dans l’ordre N,P,M,G,L (Flamand and Delagneau, 1978). Les gènes viraux sont bordés par des régions intergéniques contenant des séquences conservées représentant les signaux d’initiation et de terminaison/polyadénation de la transcription (Hinzman et al., 2008; Tordo et al., 1986a) (Finke 2000) (Figure 7). A l’extrémité 5’ se trouve une autre région régulatrice dite trailer (Figure 7). La région leader en 3’ du génome et trailer en 3’ de l’antigénome contiennent les promoteurs de réplication pour la synthèse de l’antigénome et du génome respectivement. (Conzelmann and Schnell, 1994; Finke and Conzelmann, 1997; Schnell et al., 1994)
La RNP, appartenant au virus entrant, une fois libérée dans le cytoplasme après la fusion, (Figure 6, étape 3), permet une première étape de transcription qui ne dépend que des constituants viraux : le complexe N-ARN sert de matrice à la polymérase virale L et son co-facteur P. Cette première étape est appelée transcription primaire. Elle se fait selon un modèle « stop-start » (Testa, 1980), la polymérase qui possède un site d’entrée unique à l’extrémité 3’ du génome (Emerson, 1982) se fixe sur le promoteur transcriptionnel localisé dans la région leader qu’elle transcrit jusqu’à rencontrer le « gene start » du premier gène et démarrer la transcription de celui-ci ; elle le coiffe et le transcrit jusqu’à rencontrer le site de poly-adénylation sur laquelle elle bégaie pour ajouter la queue polyA. Puis la transcription s’interrompt sur les 3 nucléotides de la région intergénique avant de se réinitier pour transcrire le gène aval (Figure 7) (Barr et al., 1997; Stillman and Whitt, 1999 ; Abraham et al., 1975; Ivanov et al., 2011; Ogino and Banerjee, 2007).
Figure 7.Schéma du génome viral
L’efficacité de la ré-initiation de la transcription au niveau du gène suivant n’est pas de 100% (Finke et al., 2000). Comme la polymérase n’a qu’un site d’entrée sur le génome, cela entraine une transcription décroissante des gènes viraux en fonction de leur position dans le génome (Iverson and Rose, 1981) et donc un gradient de transcrits en fonction de leur ordre d’apparition des gènes dans le génome. Ainsi la quantité d’ARNm de N est supérieure à celle de P, elle-même supérieure à celle de M et ainsi de suite jusqu’à celle de l’ARNm de L qui sera le moins abondant (Figure 8).
19 Figure 8. Schéma de la réplication et transcription virale
1.5.2.b. La traduction des messagers viraux
Durant l’infection virale, la machinerie de traduction de l’hôte est détournée, ce qui provoque une baisse de traduction des ARNm de l’hôte au profit des ARNm viraux. Il semble également que la voie d’initiation de la traduction des ARNm de VSV et de RABV soit différente de celle des ARNm cellulaires (Lee et al., 2013). La protéine RPL40, appartenant à la sous-unité 60S du ribosome, semble être spécifique de cette voie de traduction propre. Il a été montré que la traduction des ARNm de VSV est dépendante de RPL40 qui est aussi requise pour la multiplication de RABV.
La présence d’un peptide signal au début du gène de la glycoprotéine permet sa translocation co-traductionnelle dans le réticulum endoplasmique (Figure 6).
1.5.2.c. La réplication virale
Lorsque la quantité de protéines virales, en particulier de nucléoprotéines, synthétisées est suffisante, L change son activité de transcriptase pour une activité réplicase (Figure 8,9). L associée à P copie alors le génome négatif en un brin antigénomique positif qui à son tour servira de matrice à la synthèse de nouveaux génomes viraux par ce même complexe L-P. Le génome et l’antigénome sont encapsidés, de façon concomitante à leur synthèse, par la nucléoprotéine. L’association de N à l’ARN viral est régulée par P qui joue le rôle de chaperon pour la protéine N (Peluso and Moyer, 1988). La P empêche la N soluble, non associée à l’ARN (N0) d’interagir avec les ARN cellulaires en formant le complexe N0-P. La différence de force
des promoteurs génomique et antigénomique se traduit par des proportions différentes de génomes et d'antigénomes dans les cellules infectées. Ainsi, il y a 50 fois plus de génomes qui sont synthétisés que d’antigénomes. Les génomes neo-synthétisés pourront être ensuite incorporés dans de nouveaux virions lors des étapes d’assemblage ou servir de matrice pour la réplication ou la transcription secondaire.
Le mécanisme faisant passer de la transcription à la réplication de la polymérase virale est encore mal compris. Il semble être lié à l’encapsidation des ARN leader par le complexe N0-P,
(Blumberg and Kolakofsky, 1983; Blumberg et al., 1981). La transition de la transcription vers la réplication implique donc qu’il y a suffisamment de N à avoir été synthétisée. La protéine M de RABV est également proposée comme régulateur de cette transition puisqu’elle inhibe la transcription virale tout en stimulant la réplication (Finke, 2003; Finke and Conzelmann, 2003).
20
Figure 9.Schéma de la transcription et de la réplication du virus de la rage.
D’après Albertini et al., 2011. Pendant la transcription et la réplication, la sous-unité catalytique de la polymérase L est associée à un dimère de P. La liaison de L-P à une sous-unité de N dans la nucléocapside déclenche probablement une libération locale d'ARN permettant l'accès de la polymérase virale à l'ARN viral. Au cours de la transcription (A), l'ARNm est coiffé et méthylé à son extrémité 5’ naissante. Pendant la réplication (B), l'ARN nouvellement synthétisé est instantanément encapsidé par les complexes N0-P
entrants. L'extension du processus de réplication conduit à la production d'un nouveau complexe N-ARN génomique ou antigénomomique encapsidé.
La transcription et la réplication ainsi que l’assemblage des RNP se font dans des usines virales qui concentrent les protéines et ARN viraux, et des facteurs cellulaires. Dans les cellules infectées par le virus de la rage, les usines virales sont appelées corps de Negri (CN), et ont des propriétés d’organites liquides (Lahaye et al., 2009; Nikolic et al., 2016, 2017). Ces usines sont très dynamiques : des RNPs en sont éjectées et importées via un transport dépendant des microtubules. P effectue la navette entre le cytoplasme et les CN dans lesquels elle est concentrée, permettant le recrutement de protéines cellulaires au sein des CN (Nikolic et al., 2017).
Des corps d'inclusion avec des propriétés d’organites liquides sont aussi formés dans le cytoplasme des cellules infectées par VSV et servent eux aussi de sites de réplication du VSV (Heinrich et al., 2018).
1.5.3. Assemblages et bourgeonnement
La membrane virale est composée à 50% de protéines et 50% de lipides. Sa composition lipidique est très proche de la membrane dont elle provient, cependant elle est enrichie en cholestérol. En effet les virions bourgeonnent à des endroits particuliers de la membrane cellulaire. Il s’agit de zones où se trouvent les radeaux lipidiques enrichis en cholestérol ainsi que les protéines virales qui seront associées à la membrane des virions en devenir (Brown and Lyles, 2003). La protéine de matrice M et la glycoprotéine G y sont présentes et permettent l’assemblage des nouveaux virus au site de bourgeonnement.
21 Les virions sont directement assemblés au niveau du site de bourgeonnement. Le bourgeonnement pour VSV a lieu très majoritairement à la membrane et implique le détournement de la machinerie cellulaire ESCRT (endosomal sorting complex required for transport) (Chen and Lamb, 2008) (Figure 10). La machinerie ESCRT est responsable de la formation des corps multivésiculaires (MVB de l’anglais multivesicular bodies) et le bourgeonnement de vesicule au sein des MVB est topologiquement équivalent au bourgeonnement des virions dans le milieu extracellulaire.
Dans le cas de la rage, le bourgeonnement s’effectue à la fois à la membrane plasmique et au niveau des corps de Negri lorsque ceux-ci s’entourent d’une double membrane aux temps tardifs de l’infection. Il dépend aussi du complexe ESCRT.
L’inhibition de VPS4, une ATPase qui dissocie le complexe ESCRT à la fin de la réaction de bourgeonnement, conduit à l’inhibition de l’étape finale chez VSV, c’est-à-dire la fission membranaire conduisant au relargage du virus dans le milieu (Taylor et al., 2007).
Pour RABV, la glycoprotéine semble faciliter le processus de sortie des virions et lorsqu’elle est absente, l’efficacité de bourgeonnement est réduite (Mebatsion et al., 1996). M est indispensable car en son absence, il n’y a plus de bourgeonnement (Mebatsion et al., 1999). Pour VSV, G s’accumule au niveau de la membrane en recrutant et concentrant la protéine M au site de bourgeonnement (Schnell, 1998). Par ailleurs, lorsque M est exprimée en absence des autres composants viraux, on observe le bourgeonnement de vésicules. (Justice et al., 1995). De plus, il apparaît que les RNP cytoplasmiques doivent subir une étape d’amorçage qui leur permet de pénétrer dans la voie d’assemblage. Un tel événement d’amorçage pourrait être dû à l’association de petites quantités de M dans une conformation distincte (protéine M initiatrice sur Figure 10) aux RNP. Ce complexe RNP-M initiatrice interagirait avec les protéines M associées à la membrane plasmique déclenchant la condensation des RNP (Jayakar et al., 2004).
Figure 10.Modèle d'assemblage et de bourgeonnement des Rhabdovirus.
Adapté de Jayakar et al, 2004.1. Les RNP qui ont lié une petite quantité de M initiatrice, s’associent au
feuillet interne de la membrane plasmique via une interaction avec la M membranaire. 2. L’interaction des RNPs avec les protéines M liées à la membrane déclenche la condensation de la RNP, ce qui entraîne la formation de sites de bourgeonnement. Ces sites se forment préférentiellement au niveau des régions de la membrane plasmique qui contiennent des concentrations localement élevées de G. Au niveau de ces sites, la protéine recrute la machinerie ESCRT. 3. La condensation de la RNP au niveau du site de bourgeonnement continue en interagissant avec les protéines M liées à la membrane, ce qui entraine la protrusion du virion en formation. 4. La machinerie ESCRT, permet l'étape finale du bourgeonnement des virions, i.e. la fission membranaire.
22
1.6. Les protéines virales et leur implication dans l’infection
Le génome des Rhabdovirus code le plus souvent pour 5 protéines structurales, c’est le cas de VSV et de RABV. Nous l’avons vu, la quantité de protéines produites dans la cellule infectée dépend directement de la place du gène dans le génome. Cette quantité relative dans la cellule n’est pas retrouvée dans le virion néosynthétisé. Les Rhabdovirus ont donc peu de protéines permettant d’assurer l’ensemble des fonctions nécessaires au bon déroulement du cycle et au détournement des machineries cellulaires. Le virus doit également échapper au système immunitaire et certaines protéines virales ont pour rôle de neutraliser les défenses cellulaires. Ces cinq protéines virales sont par conséquent multifonctionnelles. Ce sont ces différents rôles et les mécanismes employés qui vont être décrits de façon succincte dans la partie suivante.
1.6.3. La ribonucléoprotéine
La RNP regroupe la nucléoprotéine N, la phosphoprotéine P et la polymérase L, en un complexe liant l’ARN viral, et permettant les fonctions de transcription et réplication du génome viral.
1.6.3.a. La nucléoprotéine
Fonction
La protéine N encapside les ARN génomiques et antigénomiques formant ainsi la nucléocapside (NC), ce qui les protège des nucléases cellulaires et empêche également l’activation de la réponse immunitaire innée (en évitant la formation d’ARN double brin). La polymérase sous forme du complexe P-L n’est pas processive sur l’ARN nu au-delà de 18 nucléotides. La processivité nécessite à la fois que la matrice d’ARN soit recouverte d’un homopolymère continu de la protéine N sous forme d’une nucléocapside hélicoïdale et la fixation à la nucléocapside du complexe P-L via la P (Morin et al., 2017).
Structure
La protéine N présente une forte affinité pour l’ARN indépendamment de l’origine, de la longueur ou de la séquence de celui-ci. Elle peut donc s'assembler avec les ARN cellulaires ou génomiques en formant des structures en forme d'anneaux fermés contenant une dizaine de sous-unités, ou en longs filaments hélicoïdaux, en fonction de la taille de l’ARN avec lequel elle interagit (Iseni et al., 1998).
La structure à basse résolution des complexes N-ARN a d’abord montré que l’ARN était localisé à l’intérieur de l’anneau, protégeant ainsi le squelette phosphodiester (Chen et al., 2004; Iseni et al., 2000). Les analyses cristallographiques aux rayons X des complexes N-ARN de RABV et de VSV ont ensuite apporté plus de détails. Les anneaux de N ont révélé que la protéine est composée de deux domaines N-terminal (NTD) et C-terminal (CTD), formant entre eux un sillon contenant des résidus basiques accueillant l’ARN (Figure 11).
La transcription et la réplication d’un mini-réplicon de VSV sont abolies ou diminuées par des mutations de ces résidus basiques (Rainsford et al., 2010). Chaque sous unité de N lie neuf nucléotides. (Albertini et al., 2006; Green, 2006; Iseni et al., 1998; Schoehn et al., 2001). Dans les complexes N-ARN, chaque N est associée aux N voisines. Le NTD d’un protomère N s'étend sur N+1 et le CTD s'étend sur N-1. Cette disposition conduit à l’interaction du NTD du protomère N-1 avec le CTD du protomère N+1 au contact du protomère N (Figure 11)
23 La boucle C-terminale de deux N adjacentes dans le complexe VSV N-ARN fournit un site de liaison pour le domaine C-terminal de la phosphoprotéine P (Green and Luo, 2009). Un mode similaire de liaison du domaine C-terminal de la protéine RABV P à la matrice N-ARN a été proposé à partir d'études de modélisation moléculaire (Ribeiro et al., 2009).
Interactome
N existe sous une forme libre (N0), non liée à l’ARN, uniquement lorsqu’elle est associée à un
dimère de P (Masters and Banerjee, 1988) (Mavrakis et al., 2003). Ce complexe constitue la réserve de N libres, nécessaire à la synthèse de nouvelles nucléocapsides lors de la réplication. Dans ce complexe, le segment N-terminal de P occupe le sillon de liaison à l’ARN de N, empêchant à la fois l’auto-assemblage de N et l’interaction non spécifique avec l’ARN cellulaire. Ce mécanisme assure un approvisionnement constant de N pour l’encapsidation du génome viral (Leyrat et al., 2011; Ruigrok et al., 2011).
Figure 11. Structure de la N de RABV.
D’après Albertini et al., 2006.
A. Structure cristallographique
du complexe N-ARN (PDB : 2GTT). À gauche, le diagramme en ruban de la structure de l'anneau constitué de 11 sous-unités, vu de dessous. Chaque
protomère N est coloré
différemment. L'ARN est
représenté par la ligne noire. À droite, une vue de l'intérieur de l’anneau, le NTD est en haut et le CTD en bas. La flèche du haut
indique que les NTDs
n'interagissent pas les uns avec les autres ; la flèche du bas pointe vers l'interface étendue entre les CTDs.
B. Représentation du potentiel
électrostatique de la surface d’un monomère de la N montrant la cavité basique liant l’ARN.
C. Représentation sous forme
de ruban du monomère de N. Le NTD de la protéine est en vert et le CTD en jaune.
D. Représentation en surface de
la structure de l’anneau N-ARN, vu de côté montrant que les deux régions charnières stabilisent la polymérisation de N. Le NTD du protomère N s'étend sur le protomère N+1 et le CTD s'étend sur le protomère N-1. Cette disposition conduit à l’interaction du NTD du protomère N-1 avec le CTD du protomère N+1 au contact du protomère N.
24
1.6.3.b. La phosphoprotéine
Fonction
La phosprotéine P est la protéine la moins conservée chez les Rhabdovirus. Elle joue plusieurs rôles au cours du cycle de vie du virus grâce à des interactions directes avec diverses protéines virales. Elle est le co-facteur de la polymérase L et, en se liant à N, elle bloque sa capacité à encapsider l’ARN. P est par ailleurs indispensable à la création des usines virales et pourrait avoir un rôle dans le transport axonal rétrograde.
Enfin, à travers des interactions avec des protéines cellulaires, P est connue pour neutraliser les fonctions de divers facteurs cellulaires impliqués dans les réponses antivirales.
Il existe un mécanisme de « leaky-scanning » lors de la synthèse de la protéine P des Rhabdovirus, qui donne lieu à la synthèse de 4 sous-produits nommés P2 – P5 chez RABV (Chenik et al., 1995), et 2 sous-produits C et C’ chez VSV. Lors de la synthèse protéique le premier AUG de l’ARNm de P se trouve dans un environnement peu favorable et donc certains ribosomes vont démarrer aux AUG suivants. Les sous-produits RABV P proviennent d’AUG initiateurs qui sont dans le même cadre de lecture que celui de P et donc correspondent à des P écourtées en N-terminal. Ce n’est pas le cas pour VSV, C et C’ sont traduits dans un autre cadre de lecture. Pour RABV ces sous-produits n’ont pas tous la même localisation que P car la partie N-terminale de la P contient un signal d’export nucléaire (NES). Les sous-produits P3, P4 et P5 en sont dépourvus et sont donc localisés dans le noyau. Les sous-produits de P sont connus pour neutraliser les fonctions de divers facteurs cellulaires impliqués dans les réponses antivirales, tels que IRF-3, IRF-7, STAT1 et PML (Rieder and Conzelmann, 2011). Structure
La P des Rhabdovirus est un dimère en solution (Gerard et al., 2007). Le protomère de P possède une organisation modulaire (Figure 12).
RABV P contient 3 domaines structuraux séparés les uns des autres par deux domaines intrinsèquement désordonnés (Gerard et al., 2009) (Figure 12). Les domaines structuraux comprennent un domaine N-terminal d’interaction avec N0, un domaine central de
dimérisation, et un domaine C-terminal d’interaction avec la N associée à l’ARN.
Les structures du domaine central et du domaine C-terminal de RABV P sont connues. Le domaine de dimérisation consiste en deux hélices formant une épingle à cheveux. La cohésion du dimère est assurée principalement par des interactions hydrophobes (Ivanov et al., 2010). La structure cristallographique du domaine C-terminal décrit un domaine en forme de demie poire structuré en hélice (Mavrakis et al., 2004).
Les domaines de dimérisation, C-terminal et le domaine intrinsèquement désordonné les reliant, sont requis pour la transition de phase liquide-liquide (Nikolic et al., 2017) et donc pour la formation des usines virales.
VSV P suit la même organisation que RABV P. La région N-terminale est très dynamique en solution avec l’apparition transitoire de formes compactes par l’intermédiaire d’hélices permettant à P d’interagir avec N0 (Leyrat et al., 2011).
Le domaine de dimérisation de VSV P est constitué de deux paires de brins formant chacun un motif en épingle à cheveux. Au centre du domaine se trouve une hélice . Dans le dimère cette hélice du premier protomère s’associe de façon parallèle par interactions hydrophophes à l’hélice de l’autre protomère, permettant la dimérisation de la protéine. Les interactions entre les brins fournissent une force supplémetaire pour stabiliser le dimère (Ding et al., 2006). Ce domaine de dimérisation présente donc une organisation différente de celui de la P du RABV.
25 La région C-terminale forme un domaine compact formant un prisme triangulaire de structure semblable à la région C-terminale de la P rabique (Ribeiro et al., 2008).
Interactome
Les domaines N-terminal (résidus 4 à 40) et C-terminal (résidus 186 à 297) de RABV P interagissent respectivement avec la protéine N0 (Mavrakis et al., 2003, 2006) et le complexe
N-ARN (Mavrakis et al., 2004; Ribeiro et al., 2009). La structure d’une forme soluble de la N avec un fragment de P de VSV a été résolue (Leyrat et al., 2011). En solution, le complexe N0
-P est formée d’une molécule de N pour un dimère de -P. Le site de liaison N-terminal de -P sur N0 est localisé au niveau du sillon de liaison à l'ARN de la protéine N0 et au niveau du bras de
N qui lie la sous-unité voisine dans son processus d’auto-assemblage (Leyrat et al., 2011). Cette liaison empêche donc simultanément l’interaction de N avec l’ARN et l’interaction entre sous-unités conduisant à la formation de la nucléocapside.
Le domaine C-terminal de P lie la N associée à l’ARN et permet donc de faire le pont entre la polymérase L et la matrice N-ARN (Mavrakis et al., 2004). Sur la nucléocapside, le domaine d’interaction est situé au niveau de la zone de contact entre 2 molécules adjacentes de N, ce qui explique pourquoi le domaine C-terminal de P ne peut pas interagir avec une forme libre de N (Mavrakis et al., 2006).
Il a été suggéré que deux régions indépendantes (résidus 1 à 19 et 40 à 100) de RABV P se lient à la partie C-terminale de L (Castel et al., 2009; Chenik et al., 1998), tandis que les résidus 11 à 50 sont responsables de la stimulation de l’activité de L (Morin et al., 2017). Les résidus 65 à 175, qui comprennent le domaine de dimérisation, sont indispensables pour l’activité de transcription (Jacob et al., 2001).
Par ailleurs, P interagit avec de nombreuses protéines cellulaires.
Elle interagit avec la chaine légère de la dynéine LC8 (Jacob et al., 2001; Poisson et al., 2001; Raux et al., 2000). L’interaction est nécessaire pour une transcription efficace du génome plutôt que pour le transport axonal rétrograde des nucléopcapsides dans les cellules neuronales comme cela avait été initialement proposé (Mebatsion, 2001; Tan et al., 2007). La kinase d'adhésion focale cellulaire (FAK) interagit également avec le domaine de dimérisation central de la protéine RABV P. Elle régule positivement l'activité de synthèse des ARNm et génomiques du RABV est est d’ailleurs associée aux corps de négri (Fouquet et al., 2015).
Pour le RABV, P est la protéine qui va interférer avec la réponse immunitaire innée de l’hôte. Ainsi, la région C-terminale fixe STAT1, un facteur de transcription des gènes induits par l’interféron (Brzozka et al., 2006; Rieder et al., 2011; Vidy et al., 2005) et la protéine PML (Promyelocytic Leukaemia Protein). P est ainsi capable d’interagir avec les corps PML et de les réorganiser, mais le rôle biologique de cette interaction reste encore inconnu. La synthèse de cette protéine est induite par l’interféron. Cette protéine fait partie des corps nucléaires qui portent son nom (corps PML) et aurait un rôle dans la défense cellulaire. Des cellules qui sont dépourvues de PML peuvent produire 20 fois plus de virus que les cellules produisant PML (Blondel et al., 2002).
RABV P est donc un antagoniste multifonctionnel de l’interféron pouvant interférer avec plusieurs processus de signalisation impliquant cette cytokine et ce, dans différents sub-compartiments cellulaires. Pour VSV, le sous-produit C’ de P permettrait d’augmenter l’activité transcriptionnelle de la polymérase(Richardson and Peluso, 1996).
26
Figure 12. La phosphoprotéine des Rhabdovirus.
D’après Albertini et al, 2011, Ivanov et al, 2011 et Leyrat et al, 2012.
A. Schéma de l’organisation de la phosphoprotéine des Rhabdovirus. Les domaines structurés sont
représentés (le domaine N-terminal, le domaine de dimérisation et le domaine C-terminal). DID1 et DID2 représentent les domaines intrinsèquement désordonnés reliant les domaines structurés.
B. Structures des domaines structurés de la P de VSV (en haut) et celle du virus de la Rage (en bas),
réprésentés en ruban, le domaine de dimérisation (PDB: 2FQM pour VSV, 3L32 pour RABV) est en orange et le domaine C-terminal (PDB: 2K47 pour VSV, 1VYI pour RABV) est en rouge, et les régions désordonnées sont représentées par des lignes pointillées. Les partenaires d’interaction de P sont indiqués au niveau duquel ils interagissent.
C. Complexe N-ARN-P. Le diagramme en ruban de RABV P montre le positionnement de la partie
C-terminale de P à la surface du complexe N-ARN. Trois protomères de N adjacents sont représentés en bleu et le domaine C-terminal de P en orange.
1.6.3.c. L’ARN polymérase ARN-dépendante
Fonction
L’ARN-polymérase ARN-dépendant doit son acronyme L du fait de sa grande taille, L pour « Large ». En effet elle a un poids moléculaire de près de 240 kDa chez VSV et RABV. C’est une protéine constituée de plusieurs domaines, catalysant la réplication et la transcription ainsi que le coiffage, la méthylation en 5’ et la polyadénylation en 3’ des ARNm qu’elle synthétise. Structure
La structure de VSV L a été obtenue par cryo-microscopie électronique (cryo-EM) (Figure 13) (Liang et al., 2015).
Cette structure contient aussi un peptide de P (résidus 35 à 106) qui a permis de stabiliser L dans une conformation donnée. Les polymérases des Mononegavirales sont globalement conservées. Celles des Rhabdovirus sont plutôt conservées au niveau de la séquence en acides aminés (Bujnicki and Rychlewski; Ogino and Banerjee, 2011; Poch et al., 1990), suggérant des similitudes fonctionnelles et au niveau de l’organisation en domaines (Ogino and Green, 2019). Au niveau N-terminal se trouve le domaine portant l’activité polymérase, suivi du domaine responsable de l’ajout de la coiffe, d’un domaine connecteur le reliant au domaine responsable de la méthylation de la coiffe qui se trouve juste en amont du domaine C-terminal
27 (Figure 13). Le domaine polymérase possède la structure classique en doigts, paume et pouce de ce type d’enzyme (Figure 13,C). Le sous-domaine de la paume de RABV L possède deux résidus aspartate (D618 et D729) conservés universellement dans d’autres polymérases, qui pourraient servir de résidus catalytiques pour la polymérisation nucléotidique (Genna et al., 2016; Steitz, 1998), et en effet le résidu D729 est nécessaire pour son activité de synthèse d'ARN in vitro (Ogino et al., 2018) et in cellula (Schnell and Conzelmann, 1995).
L’ajout de la coiffe se fait grâce à une activité polyribonucléotidyltransférase, sa méthylation se fait par une activité méthyltransférase.
Figure 13. Structure de la polymérase de VSV.
D’après Liang et al, 2015.
A. Organisation en domaines de VSV L. Le domaine de l’ARN polymérase ARN dépendante (RdRp) est en
cyan, le domaine de coiffage (Cap) en vert, le domaine connecteur (CD) en jaune, la méthyltransférase (MT) en orange, le domaine C-terminal (CTD) en rouge. Les régions conservées au sein des polymérases des Rhabdovirus sont étiquetées CR I – VI. Les astérisques indiquent la position des résidus du site actif.
B. Structure de VSV L représentée en diagramme en ruban avec le même code couleur qu’en A (PDB :
5A22).
C. Domaine RdRp. Les résidus 35 à 865 sont représentés sous forme de diagramme en ruban. Le
sous-domaine paume est en rouge, les doigts en bleus, le pouce en vert et la région N-terminale en gris.
Interactome
Le peptide N-terminal de P, à travers son interaction avec L a permis de stabiliser une conformation de VSV L (Liang et al., 2015). En effet le repliement fonctionnel de la L nécessite à la fois son association/stabilisation par la P et d’être transitoirement chaperonnée par le complexe HSP90 (Bloyet et al., 2016). VSV L interagit aussi avec le domaine C-terminal de P mais son site de liaison sur la polymérase n’est pas connu (Castel et al., 2009; Chenik et al., 1998; Nakagawa et al., 2017). Cette interaction stimule l'initiation et l’élongation de la transcription médiée par VSV L (Morin et al., 2017; Ogino et al., 2018).
28
Les résidus 1079 à 1083, appartenant au domaine responsable du coiffage des ARNm, de RABV L interagissent avec la protéine cellulaire LC8 (Light Chain 8). LC8 régule positivement la transcription en se liant à la fois à P et à L (Bauer et al., 2015; Jacob et al., 2000; Raux et al., 2000).
1.6.4. Les protéines associées à l’enveloppe virale
La membrane virale est associée à deux protéines : la protéine de matrice M localisée sur sa face interne qui joue un rôle lors de l’assemblage et du bourgeonnement et la glycoprotéine G qui joue un rôle essentiel lors des étapes précoces du cycle viral. Le glycoprotéine G, faisant l’objet de ma thèse, sera décrite dans la deuxième partie de cette introduction.
1.6.4.a. La protéine de matrice
Fonction
La protéine de matrice M est une protéine membranaire extrinsèque, associée à la face interne de la membrane virale, via une interaction avec les têtes hydrophiles de lipides chargés négativement, tels que la phosphatidylsérine.
Elle est principalement impliquée dans les étapes tardives du cycle viral, en permettant l’assemblage des particules virales et leur bourgeonnement. Elle permet la compaction de la RNP sous sa forme hélicoïdale retrouvée à l’intérieur des particules virales, avant le bourgeonnement (Newcomb et al., 1982). Cette étape de compaction est rendue possible par les capacités d’auto-assemblage de la M (Gaudin et al., 1995a, 1997).
M possède plusieurs motifs L-domain (late budding domain) ou domaine « tardif ». Ils permettent l’interaction avec des partenaires cellulaires, impliqués dans la formation des MVB. Le premier L-domain de M est un motif PPPY dans sa partie N-terminale, il permettrait l’interaction avec le motif WW de l’ubiquitine ligase Nedd4 (Harty et al., 1999, 2001) et le recrutement de la machinerie ESCRT dans des étapes précoces (Figure 10). L’ubiquitinylation qui en résulte entraine un adressage aux MVB. Chez VSV, des mutations qui alterent le motif PPPY de M entrainent son incapacité d’induire le bourgeonnement (Jayakar et al., 2000). Un second motif PSAP est présent sur M, il est impliqué dans l’interaction avec TSG101, composant de ESCRT, qui est impliquée dans les évènements tardifs de bourgeonnement chez de nombreux virus enveloppés (Bieniasz, 2006) ; en revanche, à lui seul, ce motif ne confère pas une activité de bourgeonnement efficace dans tous les types cellulaires (Irie et al., 2004; Obiang et al., 2012).
M intervient également au niveau de la régulation de la réplication et de la transcription du génome viral (Connor et al., 2006; Finke and Conzelmann, 2003), mais aussi au niveau de la transcription et traduction cellulaire (Whitlow et al., 2006, 2008). Pour VSV, M interfère en effet avec le transport des ARNm cellulaires du noyau vers le cytoplasme (Faria et al., 2005) et module également la redistribution des facteurs d'épissages grâce à des sous-produits de M produits eux-aussi par un mécanisme de leaky-scanning (Jayakar et al., 2000; Redondo et al., 2015).
M participe à l’invasion neuronale de RABV (Faber et al., 2004). Chez VSV, M inhibe l'activation de NF-KB en ciblant un événement en amont d'IKK dans la voie canonique d’induction des interferons (Varble et al., 2016). Elle contribue ainsi à limiter les réponses antivirales de l’hôte.
Chez RABV et VSV, M a aussi un rôle dans l’induction de l’apoptose (Gaddy and Lyles, 2005; Larrous et al., 2010). M cible partiellement les mitochondries et induit une apoptose