HAL Id: tel-03190840
https://tel.archives-ouvertes.fr/tel-03190840
Submitted on 6 Apr 2021
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Caractérisation de l’holobionte des isopodes par des
approches omiques : exemple du processus de
dégradation de la lignocellulose
Marius Bredon
To cite this version:
Marius Bredon. Caractérisation de l’holobionte des isopodes par des approches omiques : exemple du processus de dégradation de la lignocellulose. Biologie végétale. Université de Poitiers, 2019. Français. �NNT : 2019POIT2318�. �tel-03190840�
THÈSE
Pour l’obtention du Grade de
DOCTEUR DE L’UNIVERSITE DE POITIERS
Faculté des Sciences Fondamentales et Appliquées
(Diplôme National - Arrêté du 25 mai 2016)
Ecole Doctorale Chimie Ecologie Géosciences Agrosciences « Théodore Monod » Secteur de Recherche Biologie de l'environnement, des populations, écologie
Présentée par :
Marius BREDON
************************Caractérisation de l’holobionte des isopodes par des
approches omiques : exemple du processus de
dégradation de la lignocellulose
************************ Directeurs de Thèse :
Didier BOUCHON, Bouziane MOUMEN
Soutenue le 12 décembre 2019 devant la Commission d’Examen ************************
JURY
Jean-Christophe SIMON Directeur de Recherche, Université de Rennes Rapporteur
Roland MARMEISSE Directeur de Recherche, Université de Lyon Rapporteur
Géraldine DUBREUIL Maître de conférences, Université de Tours Examinateur
Fabrice VAVRE Directeur de Recherche, Université de Lyon Examinateur
Didier BOUCHON Professeur, Université de Poitiers Examinateur
Remerciements
Ces trois années de thèse signent la fin d’une belle aventure, et probablement le début d’une nouvelle bien plus longue… Je vais essayer de par ces quelques lignes, de tous vous remercier, vous qui m’avez aidé, accompagné et soutenu tout le long de ma thèse.
Je tiens tout d’abord à remercier les membres de mon jury d’avoir accepté d’évaluer ces travaux. Un grand merci à Roland Marmeisse et Jean-Christophe Simon pour avoir accepté d’être rapporteurs. Je remercie particulièrement Fabrice Vavre et Jean-Christophe Simon pour leur aide précieuse lors de mon comité de suivi de thèse, et des échanges qu’on a pu avoir pendant les congrès. Enfin merci à Géraldine Dubreuil, que je suis très content de retrouver après ces années passées à Tours.
Bien sûr, ces remerciements ne pourraient pas se faire sans vous remercier, Didier et Bouziane. Vous avez su me donner la liberté de faire de ce projet, mon projet de thèse. Vous avez toujours été là pour m’aider, m’épauler et me conseiller, et ce malgré des emplois du temps parfois compliqués. Je me souviendrai toujours de cet appel téléphonique d’un certain « Didier Bouchon » (à ce moment-là je n’avais aucun souvenir de qui était cette personne !), pour un entretien assez inattendu à cause de mails qui ne sont jamais arrivés … Je ne vous remercierai jamais assez pour tout ça, en particulier pour avoir su me pousser toujours plus loin même quand je n’en avais pas la motivation.
Je remercie ensuite toute l’équipe EES pour leur aide au quotidien. Je commencerai par l’équipe technique, car sans elle il est certain qu’une grande partie de ces travaux de thèse n’aurait pas pu exister. Merci Maryline pour ton aide et tes réponses à mes questions sur tout et n’importe quoi concernant le cloporte. Merci Carine pour ton expertise en biologie moléculaire. Un grand merci à Tiffany qui s’est toujours montrée disponible quand j’en avais besoin, et pour ces bons moments passés au labo (et pour
m’avoir dépanné de quelques corvées…). Merci à Alexandra sans qui toutes ces manips sur les cloportes ne pourraient exister. Merci Hélène pour ton aide sur les TPs, et en général pour les manips. Merci Christelle pour tout l’aspect administratif, toujours géré d’une main de maître. Je remercie également tous les membres du labo, autant pour leur aide pour les enseignements que pour les discussions que nous avons pu avoir ensemble. Je remercie particulièrement Joanne pour son aide précieuse, autant au niveau des enseignements qu’au niveau scientifique.
Merci également aux étudiants qui ont fait leur stage avec moi. Baptiste pour commencer, ton évolution entre le début et la fin du stage m’a tellement impressionnée ! Lucas, enfin Mr. Catastrophe, je dois dire que tu m’as vendu du rêve dès ton premier jour de stage, RIP la poignée… Je te remercie énormément pour ton aide au projet Koh-Lanta, semé d’embûches, mais on y est arrivé ! Et bien sûr Elisabeth, grande fan de FF (vive les Chocobo !), fervente défenseuse de la Play’, mais que tu sois d’accord ou non, les rois ça reste Nintendo… Et puis n’oublions pas le stage, les manips de biomol (encore des contaminations !), et cette fameuse aventure à Paris dont on évitera tout commentaire sur mon sens de l’orientation. Je profite de cet aparté sur Paris pour remercier toute l’équipe d’Eau de Paris qui nous a aidé dans ce projet, avec un é-n-o-r-m-e remerciement à Ciriac pour toute ton aide, sincèrement merci.
J’en arrive à l’équipe avec qui j’ai partagé l’espace d’un bureau pendant trois ans… Je commencerai par mon acolyte de thèse, Clémentine. Même moment, même galère, je crois que ça résume bien ces trois années. Merci pour ce soutien mutuel, j’espère pouvoir réentendre ce fameux accent créole un jour. Et puis : on va boire un coup ? Ensuite merci Sylvine pour tout ton soutien et ces grandes discussions qu’on a pu avoir le midi. J’espère qu’on finira par conquérir le monde comme prévu… J’en viens à Thomas, ou plutôt Maitre Jedi, qui m’a beaucoup aidé au début de ma thèse et par la suite. Conseiller professionnel et personnel ! Bien évidement je ne t’oublie pas non plus Camarade (Jérémy pour les non-initiés). Tu es un exemple de persévérance et de force de travail, j’espère vraiment que tu vas réussir ce que tu as entrepris. Charlotte, mon (ancienne) voisine de derrière, merci pour tous ces fous rires que tu nous as offert. Cybèle, la gentillesse incarnée, j’espère qu’on pourra refaire des séances de spiritualisme. Sans oublier Salamèche, notre magnifique et très vif gecko, et les 835 (environ) phasmes du bureau.
Enfin, Big up aux Deux-Sévriens, Junior (Thomas) et Elie. Je regrette que vous ne soyez pas arrivés plus tôt, mais dans tous les cas vous avez bien égayé ces derniers mois de thèse. Et bonne chance aussi à la dernière arrivée Cécilia, que ta thèse te soit fructueuse ! Ah, et j’ai failli oublier la mention spéciale, seuls les vrais sauront.
Egalement merci aux « bioinfos ». Tout d’abord Amine, notamment pour ton pot de thèse, aaah ce couscous… (Comment ça les thésards ne pensent qu’à manger ?). Alex, le grand fan de Doom, de MisterMv et de Benzaie. Tu as bon goût, et pas que pour ces sujets là… Et Cyril bien sûr, qui m’a toujours aidé en bioinfo, même le week-end. Un grand merci à vous trois.
Je terminerai ces lignes en remerciant ceux qui me sont proches. Micka, mon ami de toujours, avec qui je partage toutes mes passions. Merci du fond du cœur à ma famille : Papa, Maman, Jules et Lucien. De même pour toi Mathilde, merci d’avoir toujours été à mes côtés durant ces belles années, et pour toutes celles à venir. C’est impossible de tout résumer en quelques lignes, juste, merci à tous.
Sommaire
Introduction générale ... 15
1. Les interactions hôte-microbiote : vers le concept de l’holobionte ... 15
1.1 Un monde de microbes ... 15
1.2 La domestication du microbiote ... 17
1.3 De la symbiose à l’holobionte ... 20
1.4 Vers une compréhension plus approfondie de l’holobionte ... 23
2. Les isopodes, de très bons exemples d’holobiontes ... 27
2.1 Le système digestif des isopodes, un facteur clef de leur succès colonisateur ... 28
2.2 Le cortège microbien des isopodes ... 30
A) Généralités sur le microbiote des isopodes ... 30
B) Une association historique : Wolbachia ... 31
C) D’autres microbes d’intérêt ... 34
D) Le virome des isopodes ... 37
2.3 Le microbiote digestif ... 39
3. La décomposition de la lignocellulose ... 42
3.1 Le processus de dégradation de la lignocellulose ... 42
A) Les lignases ... 44
B) Les cellulases ... 44
C) Les hémicellulases ... 45
D) D’autres systèmes ingénieux pour dégrader la lignocellulose ... 46
3.2 La dégradation de la lignocellulose dans le monde vivant ... 48
A) Des stratégies diverses pour dégrader la lignocellulose ... 48
B) Les isopodes, des acteurs particuliers dans la décomposition de la lignocellulose ... 49
4. Objectifs de la thèse ... 52
Chapitre 1. Le cloporte commun
Armadillidium vulgare
, un
décomposeur efficace de la lignocellulose ... 55
1. La dégradation de la lignocellulose au niveau de l’holobionte d’un isopode
terrestre : le fruit d’un travail d’équipe ... 56
1.1 Avant-Propos ... 56
1.2 Publication : Lignocellulose degradation at the holobiont level: teamwork in a keystone soil invertebrate ... 58
1.3 Perspectives et conclusions ... 77
2. Interactions multiples au sein du digestome d’Armadillidium vulgare ... 80
2.1 Introduction ... 80
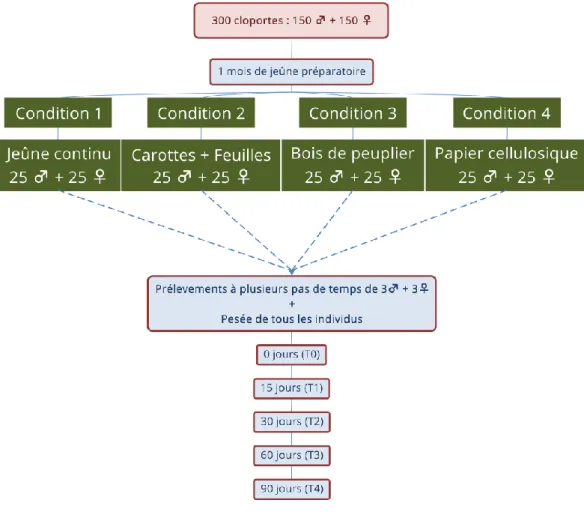
2.2 Modèle biologique et Méthodes ... 82
Modèle biologique et schéma expérimental ... 82
Extraction des acides nucléiques... 84
Quantification de la charge bactérienne totale ... 84
Estimation du niveau d’expression de CAZymes de l’hôte ... 85
Analyses statistiques ... 86
Diversité des microbiotes ... 86
Analyse des données de metagénomique ciblée ... 87
2.3 Résultats ... 89
Variations de la masse des cloportes selon les régimes alimentaires ... 89
Expression des CAZymes de l’hôte en fonction du régime alimentaire... 90
La charge bactérienne dans les caeca et le tube digestif... 93
Diversité des microbiotes en fonction des régimes alimentaires et du temps ... 93
2.4 Discussion ... 101
2.5 Conclusion ... 106
Chapitre 2. L’évolution de la dégradation de la lignocellulose au
sein des isopodes... 109
1. La dégradation de la lignocellulose chez les isopodes : nouveaux éclairages sur l’adaptation à la vie terrestre ... 110
1.1 Avant-Propos ... 110
1.2 Publication : Lignocellulose degradation in isopods: new insights into the adaptation to terrestrial life ... 112
1.3 Perspectives et conclusions ... 114
2. Les isopodes, des modèles prometteurs pour étudier la dégradation de la lignocellulose ... 128
2.1 Avant-Propos ... 128
2.2 Publication : Isopod holobionts as new models for lignocellulose degradation ... 130
Chapitre 3. La diversité des microbiotes des isopodes ... 161
1. Caractérisation du microbiote des isopodes ... 162
1.1 Introduction ... 162
1.2 Matériels et Méthodes ... 165
Banques métagénomique... 165
Profils taxonomiques des métagénomes ... 166
Détermination de MAGs par une approche de « binning » ... 166
Reconstitution de génomes bactériens à partir des métagénomes ... 166
1.3 Résultats ... 169
Composition des microbiotes... 169
Reconstitution de MAGs par une approche de binning ... 175
Assemblage de génomes bactériens ... 178
1.4 Discussion ... 183
1.5 Conclusion ... 188
2. La composante virale du microbiote des isopodes ... 189
2.1 Introduction ... 189
2.2 Matériels et Méthodes ... 192
Stratégie d’identification des virus au sein des métagénomes ... 192
Tri des faux-positifs ... 194
Assignation taxonomique des contigs viraux ... 195
Quantification des virus... 196
2.3 Résultats ... 197
Prédiction des séquences virales ... 197
Assignation taxonomique des contigs viraux ... 198
Quantification des séquences virales dans les métagénomes ... 202
2.4 Discussion ... 205
2.5 Conclusion ... 208
Synthèse générale ... 211
1.Le rôle du microbiote dans la nutrition des isopodes terrestres ... 211
2.Le microbiote, un acteur important dans la conquête des écosystèmes terrestres par les isopodes ... 213
3.Qui sont les réels acteurs de la dégradation de la lignocellulose chez les isopodes ? ... 214
4.La sélection du microbiote digestif est sous la dépendance de la dégradation de la lignocellulose ... 215
5.Des biontes méconnus chez les isopodes ... 216
6. Qu’en est-il de Wolbachia ? ... 217
Références ... 219
Annexes ... 263
15
Introduction ge ne rale
1. Les interactions hôte-microbiote : vers le concept de
l’holobionte
1.1 Un monde de microbes
Tout organisme pluricellulaire vit en association avec des microbes. Ces microbes façonnent les êtres vivants, et ce depuis très longtemps. Déjà il y a 470 Ma, les plantes ne sont pas sorties des eaux seules. Les premiers fossiles de plantes terrestres montrent qu’elles étaient au départ dépourvues de racines, ce qui ne les a pas empêchées d’accéder à l’air libre et d’exploiter les ressources terrestres. Des fossiles issus de la flore de Rhynie en Ecosse, datés de 400Ma, montrent des plantes (des genres Rhynia et Aglaophyton) pourvues de tiges rampantes et d’autres dressées (Iwatsuki and Raven, 1997; Strullu-Derrien et al., 2018). Néanmoins, aucune de ces plantes ne possède des feuilles ni des racines. En 1921, il a été observé que les tiges rampantes contenaient des hyphes de champignons appartenant probablement aux Gloméromycètes (Kidston and Lang, 1921; Strullu-Derrien et al., 2018). Plus tard il s’est avéré que des cellules observées dans ces fossiles contenaient des structures appelées « arbuscules », témoignant d’une association entre des champignons et des plantes (Remy et al., 1994). Ainsi l’interaction avec des champignons aurait permis à ces plantes d’exploiter les ressources hydrominérales du milieu et de coloniser les terres émergées. Les deux partenaires obtiennent des bénéfices mutuels de ce système : le champignon accède à des nutriments récoltés par la plante, et la plante reçoit en échange des ressources environnementales rares et difficiles d’accès tel que l’azote. Cette association étroite et durable entre deux organismes est appelée symbiose. Le plus grand des deux organismes impliqués est généralement défini comme étant l’hôte, et sert
Introduction générale
16 au moins de support à l’autre partenaire qui est plus petit et défini comme étant le symbiote. La notion de symbiose a été généralisée par Anton De Bary en 1879 sous la définition de « la vie ensemble d’organismes de noms différents » (De Bary, 1879). Les interactions entre hôte et symbiote se répartissent le long d’un gradient du mutualisme au parasitisme. Le bénéfice de cette association peut être aussi unidirectionnel et sans impact pour l’hôte (i.e. commensalisme).
Après les premières observations de micro-organismes par Leeuwenhoek dans la seconde moitié du 17ème siècle, l’Homme s’est rapidement rendu compte de leur richesse
et de leur présence dans tous les environnements. Cependant, les recherches sur les associations symbiotiques se sont longtemps intéressées à l’interaction d’un hôte avec un symbiote en particulier, omettant tout le reste des communautés microbiennes. Cette vision binaire a longtemps limité notre compréhension de la diversité du microbiote (c’est-à-dire l’ensemble des micro-organismes vivant dans un environnement donné, incluant les bactéries, les virus, les archées, les champignons et divers autres micro-eucaryotes). Il est aujourd’hui admis que le microbiote influe sur le développement des animaux, sur leur santé et sur leur évolution (McFall-Ngai et al., 2013; Peterson and Scharf, 2018). Une vision holistique est donc nécessaire pour comprendre la biologie des organismes, en particulier celle des métazoaires. Dans des conditions environnementales changeantes, le microbiote peut évoluer rapidement et influencer l’adaptation et l’évolution de l’hôte. De ce fait, les microbiotes et leurs effets sur le phénotype de l’hôte sont des facteurs majeurs à prendre en compte dans la biologie des organismes hôtes. La « révolution du microbiote » (Blaser, 2014) de ces dix dernières années a permis de mettre en évidence l’influence du microbiote sur presque tous les aspects de la biologie des organismes eucaryotes. Un grand nombre d’études montre en effet l’impact du microbiote sur la valeur adaptative de son hôte. Le microbiote est connu notamment pour protéger l’hôte contre certains pathogènes. On peut citer l’exemple des espèces du genre
Corynebacterium qui protègent l’humain des infections dues au staphylocoque doré
(Staphylococcus aureus) (Ramsey et al., 2016). De même, la crevette Palaemon macrodactylus est protégée contre l’infection d’un champignon (Lagenidium callinectes) par des protéobactéries du genre Alteromonas qui produisent du 2,3-indolinedione à la surface de l’embryon (Gil-Turnes et al., 1989). Le microbiote agit aussi sur la valeur adaptative de son hôte en lui
17 fournissant des nutriments essentiels à son développement. C’est le cas par exemple de la bactérie Wigglesworthia glossinidia qui fournit des vitamines essentielles à la mouche tsé-tsé (Glossina sp.) (Akman et al., 2002; Pais et al., 2008). Le microbiote peut également stocker des graisses et être un facteur d’obésité chez l’humain (Castaner et al., 2018; Koren et al., 2012), tout comme il est connu pour influencer le comportement tel que l’anxiété et l’humeur chez l’humain (Cryan and Dinan, 2012; Ezenwa et al., 2012; Heijtz et al., 2011; Mohajeri et al., 2018). Enfin, il peut affecter l’immunité de son hôte en interagissant avec le système immunitaire inné (Thaiss et al., 2016; Van der Ent et al., 2009; Weiss et al., 2012), réguler le développement et la croissance d’animaux (Sjögren et al., 2012) et de plantes (Martínez-Hidalgo and Hirsch, 2017).
1.2 La domestication du microbiote
La transmission du microbiote
Afin d’étudier l’histoire évolutive des interactions entre un hôte et son microbiote, il est important de comprendre comment se maintiennent les associations hôtes-microbiote au cours des générations. Il existe deux principaux modes de transmission du microbiote : la transmission verticale et la transmission horizontale (Bright and Bulgheresi, 2010). Le mode de transmission le plus répandu est celui de la transmission verticale (Russell, 2019). De cette façon le microbiote est directement transmis des parents vers la génération suivante. Beaucoup de plantes transfèrent leur microbiote de manière verticale, grâce notamment à la reproduction végétative ou via leurs graines (Fell, 1993). On retrouve également ce mode de transmission chez les animaux, où certains symbiotes sont transmis par les ovocytes (Stouthamer et al., 1999) ou par le biais du lait maternel chez les mammifères (Fernández et al., 2013).
Dans les cas où les hôtes sont en association avec des symbiotes bénéfiques, la théorie de la coévolution prédit alors que leur mode de transmission devrait tendre vers une transmission verticale afin de pérenniser l’association (Ewald, 1987; Sachs et al., 2011; Yamamura, 1993). Cette théorie se vérifie souvent dans des systèmes simples où les hôtes
Introduction générale
18 sont dépendants de leurs symbiotes. Cependant, dans des systèmes plus complexes où la valeur adaptative des hôtes est dépendante de tout un cortège microbien, le microbiote n’est pas systématiquement transmis de façon verticale (Björk et al., 2019). Le microbiote peut alors être acquis de manière horizontale. Dans ce cas, il n’y a pas de transfert direct des parents vers la nouvelle génération ; l’individu acquiert ses microbes au cours de son développement par contact avec son environnement. Ce mode de transmission se distingue aussi par la succession de phases asymbiotiques et symbiotiques durant la vie de l’organisme hôte (Bright and Bulgheresi, 2010), et il implique principalement des bactéries environnementales libres du sol ou de milieux aquatiques. Il n’est pas rare que certains animaux acquièrent leur microbiote avec les deux modes de transmission (Russell, 2019). Les éponges par exemple reçoivent des microbes de leurs parents et de l’environnement (Björk et al., 2019). Il existe également des comportements qui intègrent les deux modes de transmission. C’est le cas par exemple de la coprophagie, puisqu’elle fait intervenir des microbes à la fois de l’hôte et de l’environnement (Brune and Dietrich, 2015; Osawa et al., 1993).
Symbioses obligatoires et facultatives
La fréquence de chaque microbe varie dans le temps et dans l’espace en fonction des conditions environnementales et des effets de sélection de l’hôte. Au cours de sa vie, l’hôte peut sélectionner un microbe si ce dernier augmente sa valeur adaptative. Une fois sélectionné par l’hôte, le micro-organisme pourra être transmis à la génération suivante, ce qui favorisera l’établissement d’une association durable (Herre et al., 1999). Ainsi, cette domestication du microbiote se fait selon un continuum entre micro-organismes issus de l’environnement et symbiotes (Sachs et al., 2011; Toft and Andersson, 2010). On distingue alors deux types de symbioses : les symbioses obligatoires (ou primaires) et les symbioses facultatives (ou secondaires). Les symbiotes obligatoires sont indispensables à la survie de l’hôte et sont le plus souvent la résultante de longues histoires évolutives entre l’hôte et son symbiote. A contrario, la survie de l’hôte ne dépend pas des symbiotes facultatifs, mais ces derniers peuvent conférer un avantage adaptatif à l’hôte dans des conditions environnementales changeantes.
19
La symbiose, facteur de modifications importantes de l’hôte…
Les interactions entre un hôte et ses symbiotes ont entraîné des modifications importantes dans la physiologie de certains hôtes. Beaucoup d'entre eux possèdent des structures spécialisées où se logent leurs microbes, tels que des organes distincts ou des cellules spécialisées. C’est le cas des riftias (Riftia pachyptila), des vers tubicoles géants extrêmophiles fixés au sol des fonds abyssaux dont le tube digestif a disparu (Rimskaya-Korsakova et al., 2017). Ces vers possèdent un organe appelé trophosome abritant une grande quantité de bactéries. Ces bactéries nourrissent les riftias en oxydant, en présence d’oxygène, l’hydrogène sulfuré (H2S) qui s’échappe des cheminées hydrothermales, ce qui
leur permet grâce à l’énergie libérée de fabriquer des sucres à partir du CO2. L’hôte peut ainsi profiter des sucres produits par les bactéries et s’en nourrir. Bien sûr, le bénéfice de cette symbiose mutualiste est à double sens, puisque les bactéries profitent ici de leur hôte qui concentre les ressources d’un milieu pauvre.
…et également pour le symbiote
A l’inverse, certaines associations ont entrainé des modifications importantes chez des bactéries, allant jusqu’à impacter leur génome. Buchnera aphidicola est un des symbiotes les plus étudiés ; c’est une bactérie vivant en symbiose avec des pucerons. Là encore, à l’image des riftias, l’hôte possède des cellules spécialisées appelées bactériocytes qui abritent les bactéries. Elles sont transmises maternellement à chaque génération, et synthétisent des acides aminés essentiels au développement de leur hôte depuis plus de 160 millions d’années (Peccoud et al., 2009; Perez-Brocal et al., 2006). Cette symbiose mutualiste a conduit à une réduction du génome du symbiote, entrainant l’inactivation voire la perte de certains gènes (Chong et al., 2019; van Ham et al., 2003). Les pertes peuvent être importantes : par exemple chez certaines souches de B. aphidicola, seuls 354 gènes ont été identifiés, alors qu’une bactérie libre possède en moyenne entre 2000 et 4000 gènes. Le symbiote a perdu des gènes habituellement reconnus comme étant essentiels au bon fonctionnement de l’organisme, tout en conservant les gènes nécessaires à la pérennité de l’association. Ce type d’association aboutit à une dépendance stricte entre l’hôte et le symbiote, et elle a été conceptualisée sous le nom de théorie endosymbiotique (Margulis and Fester, 1991). Selon cette hypothèse, les symbioses sont des moteurs majeurs de l’évolution à l’origine de l’émergence des cellules des organismes
Introduction générale
20 pluricellulaires. L’exemple des mitochondries illustre très bien cette théorie. Ce sont des organites présents chez la plupart des cellules eucaryotes, elles possèdent leur propre génome et présentent beaucoup d’homologies dans leur structure avec les bactéries (Nass, 1963). Les mitochondries seraient le fruit d’une endosymbiose entre une bactérie et un eucaryote, vieille d’il y a environ 2 milliards d’années (Sagan, 1967). La bactérie concernée, une α-protéobactérie de l'ordre des Rickettsiales (Gray, 1998), aurait perdu un grand nombre de gènes et en aurait transféré vers son hôte (i.e. transfert horizontal), rendant ainsi la bactérie complètement dépendante de son hôte. Les modifications sont telles que la bactérie a fini par s’intégrer au sein même de la cellule de l’eucaryote hôte en tant qu’organite fonctionnel. Elle produit l’énergie nécessaire au fonctionnement de la cellule, et elle aurait ainsi contribué à la naissance de la cellule eucaryote telle qu’elle est aujourd’hui.
1.3 De la symbiose à l’holobionte
Ces exemples parmi tant d’autres démontrent l’étroite relation que peut avoir un hôte avec son microbiote. L’acquisition de nouveaux microbes peut avoir des effets comparables, voire supérieurs à l’apparition d’un nouveau gène chez l’hôte. Ainsi l’expression du microbiome (i.e. l’ensemble des gènes du microbiote) va affecter son environnement, qui n’est autre que l’hôte. Le phénotype de chaque organisme ne se limite donc plus à l’organisme en lui-même, mais s’étend aux autres organismes et à son environnement. Cela reprend le concept de « phénotype étendu » soutenu par Richard Dawkins (Dawkins, 1984) qui stipule que les organismes sont capables de s’adjoindre divers éléments du milieu, qu’ils soient abiotiques (e.g. minéraux) ou biotiques (e.g. microbes), pour enrichir leur phénotype. L’hôte et son microbiote ne peuvent donc plus être considérés comme étant des entités isolées, mais comme un ensemble évolutif où il existe de nombreuses interactions. Le concept de l’holobionte répond à cette problématique en définissant l’hôte et l’ensemble de ses micro-organismes comme une unité propre soumise à la sélection et l’évolution (Rosenberg et al., 2007; Theis et al., 2016).
21
Superorganisme, Holobionte ou les deux ?
Le terme de « superorganisme » a également été proposé pour définir l’assemblage d’un organisme eucaryote et de son microbiote (Segre and Salafsky, 2016; Sleator, 2010). Néanmoins, l’emploi du terme « superorganisme » a plus de signification quand il est associé aux insectes sociaux, comme décrit par Wheeler en 1928 (Wheeler, 1928). Les insectes sociaux se caractérisent par des associations d’individus d’une même espèce, où chaque individu appartient à une caste remplissant des tâches spécifiques pour la colonie (e.g. protection, fourragement, reproduction…). Ainsi, la colonie forme un superorganisme composé d’une multitude d’individus œuvrant au fonctionnement de cette entité. Elle est donc un assemblage d’holobiontes, eux-mêmes étant des assemblages de « biontes », unités de base de l’holobionte. De ce fait, le terme « d’holobionte » est préférable à celui de « superorganisme » pour définir un hôte et l’ensemble de ses micro-organismes (Gordon et al., 2013).
Le concept d’holobionte fut introduit pour la première fois en 1991 par Lynn Margulis dans son livre « Symbiosis as a Source of Evolutionary Innovation: Speciation and
Morphogenesis » (Margulis and Fester, 1991). Elle a défini l’holobionte comme étant une
entité biologique comprenant un hôte et son symbiote associé. Sa définition s’est ensuite étendue à l’ensemble des micro-organismes de l’hôte. Ce concept a été largement utilisé pour les écosystèmes coralliens (Rosenberg et al., 2007; Thompson et al., 2015), et est aujourd’hui appliqué, dans divers contextes, à de nombreux autres organismes (métazoaires essentiellement) (Figure 1). Récemment, Vannier et al. (2019) ont élargi l’holobionte au concept de meta-holobionte. Un meta-holobionte est un réseau d’holobiontes qui s’échangent des ressources et transfèrent leur microbiote à travers les générations, impactant ainsi la valeur sélective des holobiontes et plus généralement celle des méta-holobiontes. Ce concept présuppose l’existence de passerelles physiques entre les individus pour faciliter les échanges et créer ainsi des réseaux d’interactions. Introduit à l’origine pour les plantes produisant des ramets (i.e. clones), les auteurs discutent de la viabilité du concept chez les insectes sociaux. Effectivement certains insectes sociaux tels que les termites, s’échangent des ressources et leur microbiote par le biais de la trophallaxie. Le microbiote est ainsi transmis de génération en génération et impacte la
Introduction générale
22 valeur adaptative de l’ensemble de la colonie. Néanmoins la validité de ce concept dans ce contexte pose question du fait de la complexité sociale d’une colonie.
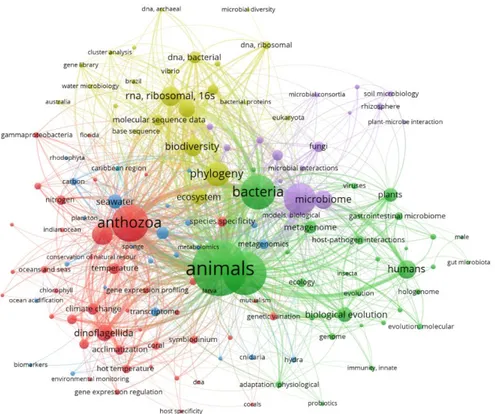
Figure 1. Principaux termes associés à l’holobionte dans la littérature actuelle. Le diagramme a été généré par VOSviewer à partir d’une recherche bibliographique sur PubMed utilisant le terme « holobiont » en incluant tous les domaines scientifiques.
L’hologénome
Les associations au sein de l’holobionte peuvent être transitoires ou stables (Hester
et al., 2016). La perte ou l’acquisition d’un nouveau bionte, ainsi que l’évolution d’un ou
plusieurs biontes (e.g. mutations, transferts horizontaux de gènes…) sont autant de facteurs qui jouent un rôle fondamental dans l’adaptation et l’évolution de l’holobionte. Ainsi chaque bionte évolue à la suite de sélections qui s’opèrent au niveau de l’holobionte et de son génome. De ce fait, le concept de l’hologénome a été introduit en 2007 pour définir l’ensemble des génomes présents dans l’holobionte, soit la réunion des génomes du microbiote et de l’hôte (Rosenberg and Zilber-Rosenberg, 2018; Rosenberg et al., 2007; Zilber-Rosenberg and Rosenberg, 2008). De la même façon que l’holobionte,
23 l’hologénome est considéré comme une unité de sélection indépendante. Bien que le concept de l’holobionte soit aujourd’hui communément accepté par la communauté scientifique, le concept de l’hologénome est quant à lui sujet à de nombreux débats. Le fait de considérer l’hologénome comme une unité de sélection pose question, car bien que de nombreux symbiotes et hôtes aient coévolué ensemble, d’autres symbiotes ne sont que transitoires et n’ont pas co-évolué avec l’holobionte (Moran and Sloan, 2015; Suárez, 2018). Dans ce cas, l’hologénome ne peut être considéré comme une unité de sélection, il doit donc être considéré avec prudence.
1.4 Vers une compréhension plus approfondie de l’holobionte
La naissance des technologies de séquençage et de la métagénomique
L’étude des microbes qui nous entourent s’est longtemps faite au travers des microbes cultivables (Lagier et al., 2015). Cependant, seules environ 19,000 espèces de procaryotes sont cultivables alors qu’on estime à plus de 10 millions leur nombre total (Parte, 2018). Beaucoup de microbes sont difficilement cultivables, voire incultivables, du fait de la nécessité de conditions spécifiques propres à leur habitat naturel. L’essor des technologies de biologie moléculaire, et en particulier l’arrivée du séquençage Sanger en 1977, a permis d’accéder aux premières séquences d’ADN de procaryotes sous la forme de courtes séquences appelées lectures. En combinaison avec des marqueurs génétiques permettant de classer taxonomiquement les espèces tels que les gènes codant l’ARN ribosomique (Pace et al., 1986), il était désormais possible d’identifier des espèces microbiennes sans passer par l’étape de culture.
L’avènement et les progrès conséquents en matière de séquençage à haut débit (aussi appelé NGS pour « Nouvelles Générations de Séquençage ») ont accéléré notre compréhension des holobiontes. Nous sommes aujourd’hui capables de séquencer tout l’ADN et l’ARN d’un environnement donné et ainsi d’en décrypter les espèces présentes et leurs fonctions biologiques associées. Le perfectionnement des techniques de séquençage et de leurs méthodes d’analyse, ainsi que la baisse de leur coût ont permis de
Introduction générale
24 rendre ces technologies de plus en plus accessibles. Ainsi durant ces 15 dernières années, on observe un nombre croissant d’études ayant utilisé des approches de métagénomique (Figure 2) ; approche qui consiste à séquencer directement l’ensemble des ADNs présents dans un milieu donné (Handelsman et al., 1998).
Figure 2. Evolution de 2002 à 2018 du nombre de publications scientifiques faisant mention de la métagénomique dans le texte selon PubMed.
La métagénomique ciblée
Les premières études de métagénomique ont employé des méthodes dites « ciblées » ou metabarcoding. Comme son nom l’indique, la métagénomique ciblée vise une région spécifique du génome, dans le but de caractériser taxonomiquement un échantillon. Les gènes des ARNs ribosomiques (ARNr) 16S et 18S sont les gènes marqueurs les plus utilisés, car le séquençage de leurs régions conservées permet une approche rapide de la composition taxonomique d’un échantillon. La première exploration du microbiote intestinal humain utilisant cette méthode (Eckburg, 2005), a ainsi permis d’identifier 13 355 séquences géniques d’ARN 16S appartenant à des procaryotes, regroupées en 395 OTUs (i.e. Unités Taxonomiques Opérationnelles : séquences regroupées sur la base de leur similarité). Néanmoins cette méthode présente plusieurs limitations, notamment au niveau du choix des amorces pour amplifier les gènes codant l’ARNr. En effet, le choix de la région amplifiée détermine les OTUs potentiellement détectables, ce qui ne permet pas d’avoir l’entièreté des communautés
0 500 1000 1500 2000 2002 2003 2004 2005 2006 2007 2008 2009 2010 2011 2012 2013 2014 2015 2016 2017 2018
25 microbiennes présentes dans l’échantillon ni les virus qui ne possèdent pas d’ARNr. L’assignation taxonomique des séquences se fait grâce à une comparaison avec des banques de séquences annotées, cette méthode repose donc sur des bases de données connues. De même, sans une profondeur de séquençage importante cette méthode ne permet pas de détecter les espèces présentes en faible densité. Enfin, l’utilisation d’un marqueur spécifique tel que le l’ADNr 16S limite l’investigation aux espèces présentes dans l’échantillon sans en comprendre leurs fonctions biologiques.
La métagénomique plein-génome
L’arrivée de la métagénomique plein-génome (dite shotgun ou Whole Genome
Sequencing en anglais) a permis de dépasser les limites de la métagénomique ciblée. Cette
méthode consiste à séquencer la totalité de l’ADN d’un environnement donné sous forme de lectures de 25 à 300 nucléotides. Un métagénome, qui correspond à l’ensemble des ADNs présents dans un milieu, permet d’identifier et de caractériser non seulement les bactéries présentes dans un échantillon, mais aussi les archées, les virus et autres composants du microbiote. Au-delà de la composition taxonomique, la métagénomique plein-génome permet aussi d’avoir une idée des fonctions biologiques codées dans les génomes des espèces présentes. Mais cette méthode se heurte encore à certaines limitations technologiques, que ce soit au niveau de la puissance de calcul nécessaire à l’analyse de ces gigas voire téraoctets de données, qu’au niveau des algorithmes permettant l’assemblage des lectures en séquences plus longues (i.e. contigs) et de leur analyse. La présence notamment d’espèces proches dans les métagénomes complique l’assemblage des lectures en contigs. De la même manière que pour la métagénomique ciblée, la compréhension d’un métagénome se fait en grande partie grâce à la comparaison des séquences obtenues avec celles des banques de séquences accessibles publiquement. On retrouve encore un nombre considérable de séquences inconnues, qui n’ont aucune affiliation taxonomique ni de fonction connue. L’arrivée des méthodes de séquençage de longues lectures, telles que celles développées par PacBio et Oxford Nanopore, ouvre de nouvelles perspectives qui aideraient à dépasser les limitations actuelles de la métagénomique plein-génome. L’accès à des lectures de séquençage plus longues pourrait faciliter l’assemblage des métagénomes et permettre ainsi une identification plus précise des espèces et des fonctions présentes dans les métagénomes.
Introduction générale
26 Mais ces deux approches que sont la métagénomique ciblée et plein-génome ne sont pas exclusives. Cette dernière, malgré des avantages non négligeables, n’a pas supplanté la métagénomique ciblée, elle l’a au contraire enrichie. La métagénomique ciblée est une approche rapide et peu coûteuse pour obtenir la méta-taxonomie d’un milieu, et elle permet avec l’utilisation de marqueurs spécifiques d’identifier des organismes rares, là où la métagénomique plein-génome peine à extraire les séquences de ces organismes de ses giga-octets de données. Beaucoup d’études ont utilisé la métagénomique ciblée afin d’obtenir une première image du milieu considéré, ce qui est le cas par exemple du projet TARA Océans (Bork et al., 2015). Il en est de même pour les travaux de cette thèse, qui font suite à des études faites en utilisant la métagénomique ciblée sur le microbiote d’isopodes (Dittmer et al., 2016). Elle se propose ici d’aller plus loin dans l’exploration de l’holobionte étudié avec, entre autres, la métagénomique plein-génome.
27
2. Les isopodes, de très bons exemples d’holobiontes
Les isopodes forment un groupe remarquable de crustacés puisqu’il s’agit du seul groupe qui compte des espèces marines, dulcicoles et exclusivement terrestres. On dénombre actuellement plus de 10,000 espèces d’isopodes réparties dans 8 sous-ordres (Poore and Bruce, 2012; Schmidt, 2008) (Figure 3). D’origine marine, les isopodes ont aujourd’hui colonisé avec succès tous les écosystèmes terrestres et aquatiques. Comme de nombreux arthropodes, les isopodes sont associés à des micro-organismes, que ce soit de manière durable ou transitoire (Bouchon et al., 2016; Dittmer et al., 2016). Ils constituent ainsi un modèle de choix pour étudier les interactions au sein de l’holobionte. Leur histoire évolutive et leur richesse taxonomique en font par ailleurs d’excellents modèles sous bien des aspects de la biologie.
Figure 3. Hypothèses phylogénétiques sur l’organisation des sous-ordres d’isopodes. Les trois phylogénies ont été construites à partir de caractères morphologiques. La relation de parenté de ces sous-ordres est méconnue. De même la monophylie de certains sous-ordres n’est pas encore clairement établie (adapté de Schmidt, 2008).
Introduction générale
28
2.1 Le système digestif des isopodes, un facteur clef de leur succès
colonisateur
Parmi les différents sous-ordres constituant les isopodes, celui des Oniscidea est le seul constitué d’espèces exclusivement terrestres et semi-terrestres. Depuis leur sortie des eaux, il y a environ 300 millions d’années (période du Permo-carbonifère) (Lins et al., 2017), les isopodes terrestres (aussi appelés cloportes) ont développé divers traits morphologiques, écologiques et comportementaux (détaillés dans Hornung, 2011), qui leur ont permis de coloniser tous les écosystèmes terrestres jusqu’aux déserts. La colonisation des sols terrestres s’est accompagnée de nouveaux défis pour les cloportes, ils ont notamment dû s’adapter à un nouveau régime alimentaire composé en grande partie de lignocellulose riche en lignine, plus complexe à digérer. Les isopodes terrestres sont devenus des espèces clefs dans les écosystèmes, ils contribuent de manière directe à la décomposition de la matière organique et au recyclage des nutriments grâce à leur digestion efficace de la litière du sol (Abd El-Wakeil, 2015; Hassall et al., 1987; Hättenschwiler et al., 2005; Jambu et al., 1988; Kozlovskaja and Striganova, 1977; Mocquard et al., 1987; Zimmer et al., 2005), et de manière indirecte par le biais de leurs fèces qui favorisent l’activité et le développement des communautés microbiennes du sol (Hassall, 1977; Jia et al., 2015; Špaldoňová and Frouz, 2014). Certains isopodes aquatiques sont également connus pour être des décomposeurs efficaces des parois végétales. De nombreux isopodes marins se nourrissent de lignocellulose et de ses dérivés contenus dans le bois immergé et les algues (Besser et al., 2018; Han et al., 2018; Zimmer et al., 2002). La plupart des isopodes d’eau douce se nourrissent aussi de la lignocellulose qu’ils trouvent dans les débris de végétaux terrestres (Graça et al., 1993; Zimmer and Bartholmé, 2003). Le développement du système digestif des isopodes terrestres et aquatiques a donc contribué à faire d’eux des décomposeurs efficaces, et il pourrait être est un des facteurs clef de leur succès colonisateur.
Le système digestif des isopodes terrestres et aquatiques se compose d’un long tube allant de l’orifice buccal à l’anus, et d’un hépatopancréas constitué de deux à trois paires de caeca (Figure 4). La région antérieure du tube digestif comprend un court tube
29 appelé œsophage et un proventricule, suivie d’un long tube se terminant par le rectum et l’anus (Goodrich, 1939; Holdich and Ratcliffe, 1970; Schmitz and Schultz, 1969; Zimmer, 2002). Le proventricule prévient la régurgitation de la nourriture dans l’œsophage. Chez les isopodes terrestres, il sert aussi à la mastication et au broyage de la nourriture déjà pré-broyée par les pièces buccales. Chez les espèces marines, le proventricule joue un rôle dans la sécrétion d’enzymes digestives (Štrus et al., 1995) alors que chez les espèces terrestres seul l’hépatopancréas est connu pour sécréter des enzymes digestives. Le proventricule filtre la nourriture en séparant les liquides des solides : ces derniers sont envoyés vers la partie centrale du tube digestif (l’intestin) alors que les premiers sont envoyés vers l’hépatopancréas. L’hépatopancréas rejette ensuite des fluides chargés en enzymes digestives vers l’intestin pour y dégrader les particules de nourriture présentes (Hassall and Jennings, 1975).
Figure 4. Schéma du système digestif d’un isopode terrestre (Oniscidea).
Une fois la nourriture dégradée, les fluides sont réinjectés dans les caeca où les nutriments seront absorbés et stockés. On y distingue deux types de cellules : les cellules B et les cellules S. Ces cellules jouent un rôle dans l’absorption de la nourriture, et également dans la sécrétion d’enzymes digestives pour les cellules B (Clifford and Witkus, 1971). Elles servent aussi au stockage de certains nutriments tels que le glycogène et les lipides (Alikhan, 1972a; Jones et al., 1969; Steeves, 1963), ainsi que le cuivre, nécessaire à l’hémocyanine, stocké dans les cellules S (Alikhan, 1972b; Dallinger and Prosi, 1988; Wieser, 1968). Enfin les différents déchets issus de la digestion ainsi que les composants
Introduction générale
30 non assimilés se retrouvent dans la région postérieure du tube digestif. Cette région est construite de façon à limiter les pertes prématurées de nourriture et d’eau du tube digestif, ce qui permet in fine le compactage de la matière fécale avant son éjection par l’anus (Hames and Hopkin, 1989).
2.2 Le cortège microbien des isopodes
A) Généralités sur le microbiote des isopodes
Les isopodes abritent de nombreux micro-organismes dans chacun de leurs tissus, incluant de nombreuses bactéries, des archées, des virus, et des micro-eucaryotes (Bouchon et al., 2016). Leur microbiote contribue à leur métabolisme et à leur phénotype, allant jusqu’à déterminer leur sexe (Rousset et al., 1992). Il est composé essentiellement de bactéries issues de l’environnement (Dittmer et al., 2016; Wenzel et al., 2018), et il est dépourvu de symbiotes primaires. Cependant, plusieurs symbiotes secondaires sont connus chez les isopodes, telle que la bactérie féminisante Wolbachia spp., le pathogène
Rickettsiella spp. ou encore les bactéries Candidatus Hepatincola porcellionum et Candidatus
Hepatoplasma crinochetorum. Chaque espèce d’isopode arbore des communautés bactériennes différentes, qui peuvent aussi varier entre les individus d’une même espèce en fonction de l’environnement, du sexe et de la saison, et au sein même des individus en fonction des tissus (Bouchon et al., 2016; Dittmer and Bouchon, 2018; Dittmer et al., 2016; Horváthová et al., 2019; Wenzel et al., 2018).
Le mode de transmission du microbiote est assez mal connu chez les isopodes. Alors que certains symbiotes seraient transmis directement dans les œufs ou par le liquide contenu dans le marsupium (i.e. la poche incubatrice de la mère), la plupart des microbes seraient acquis au cours de la vie de l’isopode de manière horizontale via l’environnement (Bouchon et al., 2016; Horváthová et al., 2015). Il a notamment été montré que pour se nourrir, les isopodes terrestres privilégient les feuilles colonisées par des bactéries ou plus généralement des biofilms (Edwards, 1974; Horváthová et al., 2016; Ihnen and Zimmer, 2008). Ce comportement favoriserait l’acquisition de nouveaux microbes qui fourniraient
31 des enzymes digestives pour aider les cloportes à digérer les différents composés des parois végétales. De même, les cloportes pourraient se nourrir des bactéries elles-mêmes pour y trouver des nutriments rares tel que l’azote (Coughtrey et al., 1980; Gunnarsson and Tunlid, 1986; Ullrich et al., 1991; Zimmer and Topp, 1998a). De plus, les isopodes terrestres consomment régulièrement leurs propres fèces et celles de leurs congénères (Gunnarsson and Tunlid, 1986; Hassall and Rushton, 1982; Kautz et al., 2002). Ce comportement de coprophagie pourrait également jouer un rôle déterminant dans le transfert de certaines bactéries à la génération suivante, aboutissant ainsi à une transmission environnementale stable équivalente à une transmission verticale comme c’est le cas chez certains termites et myriapodes (Bignell et al., 2011).
Finalement, peu d’études ont caractérisé le microbiote d’isopodes dans leur ensemble. La plupart des études se sont intéressées soit à un symbionte en particulier (Wolbachia notamment), soit au microbiote digestif. On peut néanmoins citer Wenzel et al. (2018) qui ont utilisé la métagénomique ciblée pour étudier les variations des communautés bactériennes de l’isopode marin Jaera albifrons en fonction des saisons. De la même façon, Dittmer et al. (2016, 2018) ont étudié les variations des communautés bactériennes de l’isopode terrestre Armadillidium vulgare en fonction de l’origine de l’hôte, des tissus et de la présence ou non du symbiote Wolbachia. La part d’ombre persistant sur le microbiote des isopodes reste donc importante, tant au niveau des communautés qui le composent, que de ses fonctions biologiques et de son rôle au sein de l’holobionte.
B) Une association historique : Wolbachia
Wolbachia est un des endosymbiotes les plus répandus sur Terre qui infecte de
nombreux arthropodes et nématodes (LePage and Bordenstein, 2013; Makepeace and Gill, 2016). Les isopodes ne faisant pas exception, on estime à 60% la proportion d’espèces d’isopodes terrestres infectées par Wolbachia (Bouchon et al., 2008). Elle est également présente chez des espèces marines et dulcicoles tel que Sphaeroma hookeri et
Asellus aquaticus (Bouchon et al., 1998). Historiquement, Wolbachia a été découverte chez le
moustique Culex pipiens en 1929 par Hertig et Wolbach (1929). Elle a ensuite été observée chez les isopodes pour la première fois en 1973 chez le cloporte commun Armadillidium
Introduction générale
32
vulgare (Martin et al., 1973), puis elle fut réellement identifiée en tant que Wolbachia qu’en
1992 par Rousset et al. (1992). L’association Wolbachia-arthropodes est un cas très étudié d’endosymbiose, en raison des multiples interactions qu’elle a pu établir avec de nombreux hôtes. Chez les isopodes terrestres, beaucoup d’études se sont notamment intéressées à l’impact de Wolbachia sur la physiologie et la génétique de l’hôte (Bouchon et
al., 2008).
Cette alphaprotéobactérie (ordre des Rickettsiales) est considérée comme un parasite de la reproduction (Stouthamer et al., 1999) puisqu’elle induit différents phénotypes sexuels ayant pour conséquence de favoriser les femelles infectées dans une population (Figure 5). Cela favorise sa transmission, car Wolbachia se transmet principalement par voie ovocytaire. Elle est présente majoritairement dans les cellules de la lignée germinale femelle, mais également dans les autres tissus tels que les hémocytes, les tissus nerveux et digestifs (Dittmer et al., 2014). De façon remarquable, Wolbachia est à l’origine d’un nouveau chromosome sexuel chez A. vulgare en transférant une partie de son génome dans le génome du cloporte hôte (Leclercq et al., 2016; Legrand et al., 1984).
Wolbachia est donc un puissant facteur du déterminisme du sexe chez les isopodes, allant
jusqu’à modifier le fonctionnement même des chromosomes sexuels du cloporte.
Mais Wolbachia n’est pas qu’un parasite de la reproduction, elle est aussi connue pour avoir des effets bénéfiques sur son hôte. C’est le cas par exemple chez le nématode
Brugia malayi où Wolbachia est nécessaire à sa fertilité et son développement larvaire (Taylor et al., 2005). Chez les insectes, Wolbachia fournit de la vitamine B à la punaise de lit (Cimex lectularius) (Hosokawa et al., 2010; Jiggins, 2016; Nikoh et al., 2014), protège son hôte
drosophile contre les infections bactériennes et virales (Gupta et al., 2017; Hedges et al., 2008; Teixeira et al., 2008), et elle est nécessaire à l’ovogenèse de la guêpe parasitoïde
Asobara tabida (Dedeine et al., 2001). Concernant les isopodes, le seul bénéfice connu de Wolbachia est une meilleure protection de l’hôte contre les agents pathogènes
(Braquart-Varnier et al., 2015). Wolbachia constitue donc un bon exemple de la transition d’un état parasitaire vers l’établissement d’une symbiose mutualiste avec son hôte (Weeks et al., 2007).
33
Figure 5. Les différents phénotypes induits par Wolbachia. Wolbachia est connue pour induire quatre phénotypes étendus différents, tous favorisant les femelles porteuses de Wolbachia dans une population. Elle peut induire (1) la féminisation où un mâle génétique se développe en femelle, (2) la parthénogenèse thélytoque qui est une forme de parthénogenèse aboutissant à une descendance uniquement composée de femelles, (3) le male killing où la présence de Wolbachia a pour effet la mort ciblée des embryons mâles, et (4) l’incompatibilité cytoplasmique, ou isolement reproducteur post-zygotique, qui empêche les mâles infectés de se reproduire avec des femelles non infectées. Chez les isopodes deux de ces quatre phénotypes sont connus : la féminisation et l’incompatibilité cytoplasmique (adapté de Werren et al., 2008).
En protégeant son hôte contre certains virus et bactéries, Wolbachia agit directement sur la composition du microbiote de son hôte. Sa présence ne se limitant pas qu’aux cellules germinales, elle peut ainsi impacter l’entièreté du microbiote. La caractérisation du microbiote intestinal de Drosophila melanogaster a permis de mettre en évidence l’effet de Wolbachia sur la composition du microbiote tout au long du développement de la drosophile (Simhadri et al., 2017). Comparativement aux drosophiles asymbiotiques, la présence de Wolbachia altère l’abondance ainsi que la présence ou l’absence de certains microbes. Cependant, les auteurs n’ont pas observé d’interactions directes entre Wolbachia et les bactéries intestinales, l’impact de Wolbachia serait alors bien plus complexe qu’une interaction directe. Plus récemment, Dittmer et Bouchon (2018) ont aussi mis en évidence l’influence de Wolbachia sur la structure du microbiote d’A.
vulgare. Non seulement elle affecte la présence ou l’absence de certaines bactéries, mais elle
modifie aussi l’abondance de l’ensemble des communautés microbiennes du cloporte. Ainsi, les effets sur le phénotype de l’hôte ne sont pas seulement la résultante d’une dualité de Wolbachia avec son hôte, mais bien le résultat d’une interaction à trois niveaux
Introduction générale
34 intégrant l’endosymbiote, l’hôte, et le microbiote associé. Cet exemple démontre une fois de plus l’importance d’avoir une approche intégrative incluant l’ensemble de l’holobionte ainsi que son environnement, pour comprendre les associations hôtes-microbiotes (Brinker et al., 2019) (Figure 6).
Figure 6. L’interaction tripartite et ses effets sur le phénotype de l’hôte. Wolbachia interagit avec le microbiote de l’hôte de manière directe par compétition pour les ressources et l’espace, et probablement de manière indirecte en provoquant une réponse immunitaire accrue de la part de l’hôte. L’origine de l’hôte, son sexe, son mode de nutrition, les facteurs environnementaux, mais aussi les bactéries issues de l’environnement sont autant de facteurs qui vont interagir avec ce triptyque Wolbachia, Hôte et Microbiote (Dittmer and Bouchon, 2018). Le phénotype de l’hôte n’est pas seulement dépendant de son interaction avec Wolbachia, mais bien le résultat de tout un complexe multifactoriel (adapté de Brinker et al., 2019).
C) D’autres microbes d’intérêt
Rickettsiella, des bactéries pas si pathogènes
Les bactéries du genre Rickettsiella pourraient constituer un autre exemple de la transition d’un état parasitaire vers une symbiose mutualiste chez les isopodes. Ces bactéries appartiennent aux Gammaproteobacteria, elles sont connues en tant que
35 pathogènes chez un grand nombre d’arthropodes (Bouchon et al., 2011). Chez les isopodes, elles ont été identifiées chez plusieurs espèces d’isopodes terrestres et dulcicoles (Cordaux et al., 2007; Dittmer et al., 2016; Kleespies et al., 2014; Wang et al., 2007). Les individus infectés par Rickettsiella développent une maladie symptomatique ou « blue
disease », facilement identifiable dans les stades avancés de l’infection de par la coloration
bleutée de la face ventrale des individus infectés (Vago et al., 1970). La maladie se traduit aussi par un développement ralenti de l’hôte, et provoque dans la plupart des cas la mort de l’individu. Néanmoins, la virulence de la bactérie n’est pas aussi forte que pourrait l’être celle d’un virus ou un champignon. De même, des porteurs sains de bactéries du genre
Rickettsiella ont été identifiés dans plusieurs populations. Effectivement plusieurs souches
de Rickettsiella sont connues chez les isopodes (Kleespies et al., 2014), et certaines de ces souches pourraient ne pas être pathogènes. C’est notamment le cas pour le puceron du pois Acyrthosiphon pisum, où des bactéries du genre Rickettsiella protègent leur hôte contre les prédateurs en modifiant sa couleur (Tsuchida et al., 2010, 2014). Rickettsiella pourrait alors constituer un nouvel exemple de symbiote mutualiste chez certains isopodes.
Le parasite Rhabdochlamydia
La bactérie Rhabdochlamydia porcellionis est également un des pathogènes reconnus chez les cloportes. Ce parasite intracellulaire, proche des Chlamydiae, a été découvert dans l’hépatopancréas du cloporte rugueux Porcellio scaber (Drobne et al., 1999; Kostanjsek, 2004; Shay et al., 1985). Il présente un cycle développemental complexe découpé en trois phases : (1) une phase sous forme de corps élémentaires infectieux qui (2) une fois rentrés dans la cellule infectée, se développent en corps réticulés capables de se diviser et (3) de se différencier en corps intermédiaires. Ces corps intermédiaires vont finalement se différencier en corps élémentaires infectieux, pour être enfin libérés de la cellule grâce à l’éclatement de sa vacuole. Le cycle peut ainsi recommencer jusqu’à la mort de l’individu infecté. Il reste néanmoins beaucoup d’aspects de la biologie de cette bactérie à élucider, tant au niveau des voies de transmissions qu’au niveau de son évolution chez les isopodes (Bouchon et al., 2016).
Introduction générale
36 Candidatus Hepatincola et Candidatus Hepatoplasma, deux bactéries hépatopancréatiques
L’hépatopancréas des cloportes a longtemps suscité un fort intérêt pour la recherche de bactéries symbiotiques. Il est supposé que des bactéries symbiotiques de l’hépatopancréas produisent les enzymes nécessaires à la dégradation de composés lignocellulosiques, le composant majeur des parois végétales qui constitue la nourriture principale de beaucoup d’espèces d’isopodes terrestres (Zimmer, 2006). En plus de la bactérie Rhabdochlamydia porcellionis détaillée plus haut, deux bactéries hépatopancréatiques extracellulaires ont été observées chez plusieurs espèces d’isopodes terrestres : Candidatus Hepatincola porcellionum et Candidatus Hepatoplasma crinochetorum (Dittmer et al., 2016; Eberl, 2010; Fraune and Zimmer, 2008; Wang et al., 2007). La première appartient aux Rickettsiales alors que la seconde fait partie des Mollicutes (Wang et al., 2004a, 2004b). Ces deux bactéries semblent être des symbiotes facultatifs du fait de la présence d’individus non infectés dans les populations hôtes, et semblent exclusives puisqu’aucun individu bi-infecté n’a été identifié (Fraune and Zimmer, 2008; Wang et al., 2007). Fraune et Zimmer (2008) ont observé qu’en présence de Candidatus Hepatoplasma, la survie des individus infectés augmente dans des milieux pauvres en termes de qualité nutritive, notamment au niveau cellulosique. C’est pourquoi il a d’abord été supposé que cette bactérie aide à la production d’enzymes dégradant la cellulose. Néanmoins, l’analyse de son génome (Leclercq et al., 2014) montre que ce dernier ne possède pas de gènes codant des enzymes connues pour participer à la dégradation de la cellulose, et plus largement de la lignocellulose (Bredon et al., 2018; Zimmer, 2006). Des analyses de cophylogénie montrent que Candidatus Hepatoplasma a une histoire évolutive très ancienne avec les isopodes terrestres (Fraune and Zimmer, 2008). A contrario, Candidatus Hepatincola n’aurait colonisé l’hépatopancréas des isopodes terrestres que très récemment (Wang et al., 2007), et il a été observé qu’elle réduit la longévité des individus infectés (Fraune and Zimmer, 2008). Fraune et Zimmer (2008) ont donc émis l’hypothèse que Candidatus Hepatincola aurait envahi et pris la place de Candidatus Hepatoplasma dans certaines populations grâce à un mimétisme moléculaire. Il reste une grande part d’ombre quant au rôle et l’origine de ces deux bactéries. L’acquisition du génome de Candidatus Hepatincola et la comparaison de différentes souches de ces deux bactéries pourraient aider à comprendre leur histoire évolutive avec les isopodes.
37 D) Le virome des isopodes
Les virus sont présents dans l’ensemble des écosystèmes terrestres et aquatiques (Danovaro et al., 2008; Zablocki et al., 2014). Il existe une grande variété de virus infectant les trois domaines du vivant : les bactéries, les archées et les eucaryotes (Koonin and Dolja, 2013). Ils constituent ainsi une part importante de l’holobionte ; ils peuvent impacter la physiologie de leur hôte tout comme ils peuvent altérer la composition du microbiote dans son ensemble. Le virome d’un environnement englobe l’ensemble des virus qui y sont présents, incluant les virus d’eucaryotes et de procaryotes. Il est constitué d’une part importante de virus infectant les bactéries ou phages. Ces derniers peuvent avoir un fort impact sur la composition de l’holobionte en exerçant une pression sélective vis-à-vis des bactéries (Letarov and Kulikov, 2009; Reyes et al., 2012; Rodriguez-Valera et
al., 2009), et en affectant la virulence de certaines bactéries pathogènes (Boyd, 2012).
Les arthropodes sont les vecteurs de nombreux virus pathogènes touchant un grand nombre de plantes et d’animaux (Li et al., 2015; Nault, 1997; Rosario et al., 2018; Shi
et al., 2016). De nombreux virus sont également connus pour leurs effets létaux sur les
arthropodes, en particulier chez les crustacés (Bateman and Stentiford, 2017; Grandjean et al., 2019). Ces dernières années, beaucoup d’études ont révélé la richesse des communautés de virus chez les crustacés (Bistolas et al., 2017; Dunlap et al., 2013; Edwards and Rohwer, 2005; Gudenkauf and Hewson, 2016; Hewson et al., 2013; Rosario
et al., 2015), et pourtant, seule une infime partie du virome des isopodes a été caractérisée.
On connait à ce jour quatre familles de virus chez les isopodes : la famille des Cruciviridae, des Picornaviridae, des Iridoviridae et des Reoviridae (Bistolas et al., 2017; Johnson, 1983; Juchault et al., 1991; Loiseau et al., 2017). Ces deux dernières familles sont les plus impressionnantes du fait de leurs effets sur la physiologie de leur hôte.
Les iridovirus, les virus du cloporte bleu
Des trois familles de virus connues chez les isopodes, il en est une qui se distingue des autres par la couleur bleutée qu’elle confère aux cloportes infectés. Les Iridoviridae IIVs (pour « Invertebrate Iridescent Viruses ») de l’ordre des Megavirales, sont des virus à ADN qui infectent un large spectre d’invertébrés, allant des nématodes aux insectes, en passant par
Introduction générale
38 les mollusques, les annélides et bien sûr les isopodes (Williams, 2008). Les iridovirus caractérisés chez le cloporte commun A. vulgare appartiennent à l’espèce IIV31. Ils sont facilement détectables puisque les individus infectés renvoient une couleur iridescente bleue violette du fait de l’accumulation de virions, rangés de façon à réfléchir la lumière bleue ou violette dans le cytoplasme des cellules infectées (Federici, 1980; Loiseau et al., 2017; Smith and Williams, 1957). Les individus infectés présentent de plus des troubles au niveau du comportement alimentaire, mais aussi au niveau du comportement fouisseur et ils sont moins réactifs face à certains stimuli comme la lumière ou l’eau (Loiseau et al., 2017; Lupetti et al., 2013; Wijnhoven and Berg, 1999). L’infection est rapide, systémique (tous les tissus sont infectés sauf, curieusement, l’hépatopancréas) et les individus infectés meurent sept à huit semaines après le début de l’infection (Federici, 1984; Lupetti et al., 2013). Cependant, le mode de transmission de ce virus reste inconnu, il est supposé que l’infection pourrait se faire par simple contact, ou bien à cause des comportements du cannibalisme ou de coprophagie connus chez les cloportes (Cole and Morris, 1980).
L’OMV, le virus masculinisant
Il existe chez les cloportes un autre virus modifiant de manière impressionnante des caractères physiologiques. Il se nomme OMV (« Oniscoidea Masculinizing Virus ») ; c’est un virus proche de la famille des Reoviridae qui a été identifié chez Porcellio dilatatus, et potentiellement chez Armadillidium vulgare (Juchault et al., 1991). Ce virus en apparence inoffensif pour son hôte et peu infectieux, induit une masculinisation partielle des femelles génétiques. Les femelles infectées présentent la particularité d’être intersexuées, c’est-à-dire qu’elles présentent à la fois des caractères sexuels mâles et femelles. Le virus agit sur le cytoplasme des cellules épithéliales en mimant l’action de l’hormone mâle. De ce fait, seuls les organes sexuels externes sont touchés, les autres organes sexuels tels que les ovaires ne sont pas affectés. En général les femelles infectées sont asymétriques, présentant d’un côté un stylet copulateur mâle et de l’autre un orifice sexuel femelle. Malheureusement, ce virus n’a pas été observé depuis la publication de Juchault et al. (1991). Sa faible tolérance thermique (une température excédant les 27° degrés lui est létale), ainsi que son faible niveau d’infection pourraient être des facteurs limitants pour son succès invasif.