HAL Id: hal-02916800
https://hal.inrae.fr/hal-02916800
Submitted on 18 Aug 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Isolement et caractérisation de bactéries appartenant au
groupe des Bdellovibrio et organismes apparentés
(BALOs) et de leurs proies dans un grand lac Resp:
Stéphan Jacquet (INRAE CARRTEL)
Gaetan Pavard
To cite this version:
Gaetan Pavard. Isolement et caractérisation de bactéries appartenant au groupe des Bdellovibrio et organismes apparentés (BALOs) et de leurs proies dans un grand lac Resp: Stéphan Jacquet (INRAE CARRTEL). Sciences de l’environnement. 2019. �hal-02916800�
UE Stage
M2 Microbiologie fondamentale
2018/2019
Gaëtan PAVARD
Encadrants : Stéphan JACQUET, Jade EZZEDINE
INRA CARRTEL / Directeur : Jean GUILLARD
Email :
stephan.jacquet@inra.fr
Tel : +33 4 50 26 78 12
INRA – UMR CARRTEL 75 bis, avenue de Corzent
74203 THONON-LES-BAINS
Isolement et caractérisation de bactéries appartenant au
groupe des Bdellovibrio et organismes apparentés
Remerciements
J
e tiens à remercier Jean GUILLARD, directeur de l’UMR CARRTEL, de m’avoir permis d’effectuer mon stage au sein de l’établissement qu’il dirige et de m’avoir laissé profiter des locaux pour me loger.J
e remercie infiniment mes deux maîtres de stage, Stéphan JACQUET et son doctorant Jade EZZEDINE, pour leur encadrement et leur suivi permanent afin que mon stage se déroule dans les meilleures conditions. J’ai beaucoup appris de leur expertise et, grâce à eux, les 6 mois de stage en laboratoire ont été très enrichissants et sont passés à une vitesse folle.J
e remercie particulièrement Cécile CHARDON et Louis JACAS, techniciens de recherche, pour leurs précieux conseils concernant les différentes parties techniques, et leur aide pour m’adapter à ce nouvel environnement de travail. Un grand merci aussi à Marine GARDILLON, stagiaire BTS arrivée au mois de mai, de m’avoir épaulé dans la réalisation des dernières manipulations de mon stage.J
e souhaite aussi remercier Séverine RUFFIER qui s’est occupée de toute la partie administrative de mon stage.P
our finir, je remercie tout le personnel du CARRTEL, en particulier les doctorants et post-doctorants pour la bonne ambiance qu’ils maintiennent quotidiennement au sein du laboratoire. Ainsi que tous les stagiaires avec qui j’ai passé d’agréables moments au château et en dehors.Liste des abréviations
ALBD – Annecy Léman Bourget Dranse BALOs – Bdellovibrio and Like Organisms
CIRM – Centre International de Ressources Microbiennes DO – Densité Optique
LB – Liquid Broth
LPS – Lipopolysaccharides PBS – Phosphate Buffered Saline p/v – poids/volume
qPCR – quantitative Polymerase Chain Reaction rpm – rotations par minute
TCC – Thonon Culture Collection TSB – Tryptic Soil Broth
TBE LB NB TSB TAE HM Fur
Liste des figures et tableaux
Figure 1 : Photographies obtenues en microscopie de B. bacteriovorus 109J
Figure 2 : Cycle de prédation et de reproduction endobiotique de B. bacteriovorus HD100
Figure 3 : Arbre phylogénétique simplifié des BALOs
Figure 4 : Exemple d’abondances de différentes familles de BALOs dans 3 grands lacs péri-alpins
Figure 5 : Schéma expérimental de l’obtention de proies à partir de la TCC et de l’échantillon nommé ALBD
Figure 6 : Schéma expérimental du prélèvement de l’échantillon d’eau du Léman et de son enrichissement
Figure 7 : Schéma expérimental de la technique d’isolement des BALOs par centrifugation et filtration
Figure 8 : Marqueur de masse moléculaire FastGene® 100 bp DNA Ladder RTU
Figure 9 : Photographies de souches de bactéries proies potentielles ALBD et TCC après coloration de Gram
Figure 10 : Photographies de cultures bactériennes au cours de la phase d’enrichissement
Figure 11 : Photographies de gels d’électrophorèse avec les produits PCR utilisant les primers universels
Figure 12 : Photographie de plaques de prédation de B. bacteriovorus HD100
Tableau 1 : Souches bactériennes utilisées au cours de cette étude
Tableau 2 : Composition des milieux et tampons utilisés pour la croissance des bactéries
Tableau 3 : Microalgues sélectionnées pour l’isolement des proies potentielles à partir de la TCC
Tableau 4 : Détail des amorces nucléotidiques utilisées
Table des matières
1. Introduction ... 1
2. Matériels et Méthodes ... 5
2.1 Matériels ... 5
2.1.1 Souches bactériennes et conditions de culture utilisées... 5
2.1.2 Milieux de cultures ... 5
2.1.3 Réactifs et solutions utilisées ... 5
2.1.4 Matériel d’observation microscopique et autres matériels du laboratoire ... 5
2.2 Méthodes ... 7
2.2.1 Obtention des proies à partir de la TCC et ALBD ... 7
2.2.2 Préparation des proies ... 7
2.2.3 Culture liquide des BALOs caractérisés ... 9
2.2.4 Stockage des souches bactériennes ... 9
2.2.5 Isolement des BALOs des grands lacs périalpins ... 9
2.2.5.1 Prélèvement des eaux et enrichissement des échantillons ... 9
2.2.5.2 Isolement de BALOs sur boite en double couche d’agar ... 11
2.2.6 Caractérisation des souches bactériennes ... 11
2.2.6.1 Extraction d’ADN génomique ... 11
2.2.6.2 Dosage de l’ADN ... 11
2.2.6.3 Protocole PCR ... 13
2.2.6.4 Purification de produits PCR ... 13
2.2.6.5 Clonage et ligation des inserts dans le vecteur ... 13
2.2.6.6 Transformation bactérienne ... 13
2.2.6.7 Electrophorèse en gel d’agarose ... 13
2.2.6.8 Séquençage et analyse des séquences ... 13
3. Résultats ... 15
3.1 Isolements et caractérisation des proies potentielles de TCC et ALBD ... 15
3.2.1 Enrichissement de la proportion de BALOs dans l’échantillon ... 15
3.2.2 Isolement par centrifugation différentielle et double-agar ... 15
3.3 Caractérisation génétique des souches bactériennes ... 17
3.3.1 PCR et clonage dans le vecteur plasmidique ... 17
3.3.3 Séquençage des produits PCR ... 17
4. Discussion et Perspectives ... 19
5. Conclusions ... 24
6. Références ... 25
7. Annexes ... 28
Protocole coloration de Gram ... 28
Protocole d’extraction d’ADN génomique GeneElute optimisé par le laboratoire ... 28
Protocole PCR ... 29
Figure 1 : (A) Photographie obtenue en microscopie électronique de Bdellovibrio bacteriovorus 109J1. (B) et (C) Photographies révélant la prédation de B. bacteriovorus HD100 obtenues en microscopie aux ions d’hélium2.
Figure 2 : Cycle de prédation et de reproduction endobiotique de B. bacteriovorus HD1003. Les étapes 1 et 2 correspondent à la phase d’attaque du prédateur. La reconnaissance avec la proie s’effectue grâce aux oligosaccharides associés aux lipopolysaccharides (LPS). Lorsqu’il y a contact entre le contenu cytoplasmique de la proie et l’enveloppe cellulaire du prédateur, la phase de croissance et de reproduction est enclenchée. En (4) il y a formation du bdelloplaste. Le prédateur croît en forme de filament dans le périplasme (étapes 5 à 7). Quand le cytoplasme de la proie est consommé, il y a séparation des cellules (étape 8 et 9). La progéniture se voit alors dotée de flagelle, et libère des enzymes hydrolytiques pour digérer le peptidoglycane et lyser la proie (étape 10).
1
1. Introduction
La majorité des microorganismes présents sur notre planète sont non-cultivables en laboratoire4. Les conditions de croissance permettant de les cultiver sont en effet difficiles à reproduire ou ne sont pas connues. C’est le cas pour de nombreuses bactéries dont l’incroyable diversité a surtout été mise en évidence grâce à l’avènement et l’utilisation des outils issus de la biologie moléculaire : PCR, clonage-séquençage, métagénomique, séquençage à haut débit5,6 permettant d’éviter la mise en culture. Les différentes interactions entre ces microorganismes et leur environnement, ainsi que leurs rôles fonctionnels, restent souvent à découvrir et tenter d’isoler certains d’entre eux peut alors présenter un véritable intérêt scientifique. C’est le cas pour un groupe de bactéries, les Bdellovibrio et organismes apparentés ou BALOs (acronyme anglo-saxon pour
Bdellovibrio And Like Organisms) qui constituent un groupe fonctionnel de bactéries prédatrices obligatoires
d’autres bactéries7 dont on connait encore peu le rôle dans de nombreux environnements.
Le groupe des BALOs est composé de bactéries Gram négatif, prédatrices obligatoires d’autres bactéries également Gram négatif. Les BALOs sont des bactéries type vibrio de petite taille (mesurant entre 0,7 et 1,5 µm x 0,5 µm), planctoniques et très mobiles grâce à leur unique flagelle polaire qui permet un déplacement à plus de 50 µm/s8 (Figure 1). La reconnaissance suivie de l’attachement du prédateur sur sa proie, qui constitue la phase d’attaque, serait liée à la composition de la membrane cellulaire de la proie, en particulier les oligosaccharides
associés au LPS (Lipopolysaccharides - composant majeur de la
membrane externe des bactéries Gram négatif)
de la proie. Les BALOs ne peuvent ni répliquer leur ADN, ni entamer de division cellulaire au cours de cette phase (Figure 2). Après l’interaction du prédateur avec sa proie, les BALOs passent par une phase transitoire avant d’entrer en phase de croissance et de réplication9. L’ouverture de la membrane externe de la proie se produit grâce à la libération locale d’enzymes hydrolytiques du prédateur. Dès lors, il existe deux stratégies différentes possibles pour la suite du cycle de reproduction du prédateur : le cycle de vie épibiotique et le cycle de vie endobiotique10.Le cycle de vie épibiotique, retrouvé par exemple chez Micavibrio aeruginosavorus ou Bdellovibrio
exovorus, consiste pour le prédateur à s’ancrer sur la paroi de sa proie, à la digérer de l’extérieur et à se diviser
de façon binaire11. Le cycle de vie endobiotique ou périplasmique est très différent et consiste, pour le prédateur, à pénétrer à l’intérieur de l’hôte et former dans l’espace périplasmique un bdelloplaste, véritable habitat protecteur contre l’attaque des bactériophages et des conditions physico-chimiques environnantes. Le développement et la réplication du prédateur est rendu possible par la digestion de la bactérie proie hôte par l’action de diverses enzymes type protéases, hydrolases et peptidases. Dans le bdelloplaste, la cellule prédatrice se développe sous la forme d’un filament, qui va, par la suite, se segmenter en 2 à 7 progénitures de même taille. Lorsque la cellule proie est réduite à la forme d’un protoplaste et que celui-ci est entièrement consommé, les cellules nouvellement formées vont développer leur flagelle. Des enzymes hydrolytiques vont permettre de dégrader le peptidoglycane pour libérer ces nouveaux prédateurs12. Ce cycle de vie se retrouve typiquement chez B. bacteriovorus.
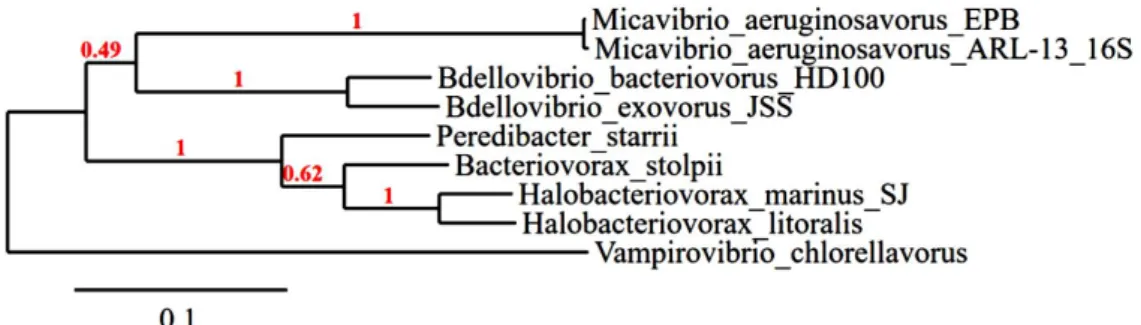
2 Figure 3 : Arbre phylogénétique simplifié des BALOs13. Arbre basé sur l’analyse de séquences du gène de la sous-unité ribosomique 16S des BALOs.
Figure 4 : Abondances et distribution de différentes familles de BALOs dans trois grands lacs péri-alpins7. Dynamique des abondances pour trois familles de BALOs en nombre de copies du gène 16S par mL (déterminé par qPCR), sur des profondeurs différentes. Les cercles correspondent aux eaux de surface (entre 2 et 3 m de profondeur),les carrés plein aux eaux plus profondes (entre 45 et 50 m de profondeur).
3 Les BALOs forment un groupe fonctionnel, composé de deux groupes polyphylétiques distincts. L’un de ces deux groupes est composé de cinq familles insérées dans la classe des Oligoflexia : les
Bdellovibrionaceae, les Bacteriovoracaceae, les Peredibacteraceae, les Pseudobacterovoracaceae et les Halobacteriovorax. Le genre Micavibrio constitue le deuxième groupe, qui est inséré dans la classe des
alpha-protéobactéries (Figure 3). Les BALOs résident partout au sein de la biosphère (océans, mers, littoraux, estuaires…), et sous diverses formes (planctonique ou en biofilm)14,15. Il apparait qu’ils ne forment pas de populations majeures dans les milieux naturels ; leur abondance a en effet été rapportée en moyenne comprise entre 0,2 et 0,5% de la communauté bactérienne totale9,16. Toutefois, leur rôle fonctionnel peut être important et, de par une capacité de croissance et de division importante, ils peuvent constituer des régulateurs significatifs de la biomasse et de la diversité bactérienne, à l’instar des protistes flagellés, des ciliés et des bactériophages. Dès lors, leur abondance peut augmenter d’un facteur mille en quelques heures17.
Les BALOs ont été récemment découverts dans les grands lacs péri-alpins, avec, pour certaines familles (i.e. les Peredibacteraceae), des abondances pouvant être importantes7. Cette étude a aussi révélé différentes distributions des familles Bdellovibrionaceae, Bacteriovoracaceae, et Peredibacteraceae au sein de la colonne d’eau pouvant correspondre à des niches écologiques préférentielles pour chacune (Figure 4). Cette étude préliminaire a permis à Stéphan Jacquet d’élaborer le projet C-BALO (thèse de Jade Ezzedine, 2017-2020) visant à décrypter plus en détail la diversité de ce groupe d’organismes (par séquençage à haut débit), déterminer leurs abondances (par qPCR) et commencer à apprécier leur rôle fonctionnel pour les lacs d’Annecy et du Léman. Ce projet de MASTER entre dans le cadre du projet C-BALO, avec comme objectif premier de tenter d’isoler et caractériser de nouveaux BALOs (et leurs proies) provenant d’un de ces lacs (le Léman) pour, dans un second temps, permettre l’étude des relations proies-prédateurs dans différentes conditions expérimentales. Les questions auxquelles nous avons tenté de répondre ont été les suivantes :
Est-il possible d’isoler, maintenir en culture et caractériser des bactéries susceptibles d’être des proies pour différents BALOs lacustres ?
Comment peut-on isoler des BALOs d’un grand lacs péri-alpin en conditions de laboratoire et en utilisant les proies précédemment isolées ?
Quels sont les spectres de prédation de ces prédateurs ? Peuvent-ils prédater d’autres types de proies (typiquement venant d’autres milieux, conservés dans des banques d’organismes, etc.) ?
4 Tableau 1 : Souches bactériennes utilisées au cours de cette étude
Tableau 2 : Composition des milieux et tampons utilisés pour la croissance des bactéries
Nom Composition par litre
LB Tryptone 10 g, Extrait de levure 5 g, NaCl 10 g TSB Peptone 20 g, Glucose 2,5 g, NaCl 5 g, K2HPO4 2,5 g
NB Peptone 15g, Extrait de levure 3 g, Glucose 1 g, NaCl 6 g
Tampon HM HEPES 6 g, 15 g agar, 6 mL CaCl2 (à 0,5 M), 3,33 mL MgCl2 (à 0,6 M) HM plates Tampon HM supplémenté de 15 g d'agar
HM top Tampon HM supplémenté de 5 g d'agar (0,5%) ou 7 g (0,7%) Tampon PBS NaCl 8 g, KCl 0,2 g, Na2HPO4 1,44 g, KH2PO4 0,24 g
Fur Tryptone 10 g, Extrait de levure 5 g, NaCl 2,5 g, Tyrosine 1,0 g
Souche Description
Proie référencée
Escherichia coli ML35 Souche envoyée par E. Jurkevitch
Pseudomonas aeruginosa (souche inconnue) Souche envoyée par E. Jurkevitch
Citrobacter freundii ATCC 8090 Souche type commandée au CIRM INRA de Tours
Escherichia coli ATCC 10536 Souche commandée au CIRM INRA de Tours
Hafnia alvei ATCC 13337 Souche type commandée au CIRM INRA de Tours
Pseudomonas fluorescens ATCC 13525 Souche type commandée au CIRM INRA de Tours
Pseudomonas putida ATCC 12633 Souche type commandée au CIRM INRA de Tours
Aeromonas salmonicida salmonicida SA28 Souche bactérienne responsable de la furonculose
des salmonidés, isolée par le laboratoire
Synechococcus sp TCC793 Souche picocyanobactérienne isolée du lac du
Bourget par le laboratoire
BALO
Bdellovibrio bacteriovorus HD100 Souche envoyée par E. Jurkevitch
Bdellovibrio bacteriovorus 109J Souche envoyée par E. Jurkevitch
5
2. Matériels et Méthodes
2.1 Matériels
2.1.1 Souches bactériennes et conditions de culture utilisées
Les différentes souches bactériennes utilisées lors de ce stage sont représentées dans le Tableau 1. Les proies Escherichia coli ML35 et Pseudomonas aeruginosa ont été fournies par le laboratoire d’E. Jurkevitch de l’Université Hébraïque de Jérusalem, afin de nourrir les BALOs envoyés par ce même laboratoire. E. coli ML35 a été utilisée comme proie pour les souches de Bdellovibrio bacteriovorus HD100 et B. bacteriovorus 109J, alors que P. aeruginosa a été utilisée pour nourrir Micavibrio aeruginosavorus.
Une variété de souches bactériennes Gram négatif lyophilisées a été commandée au Centre International de Ressources Microbiennes (CIRM) de l’INRA de Tours. Dès réception, ces souches ont été remises en culture dans un milieu LB liquide incubé à 37°C sauf pour les souches de Pseudomonas incubées à 30°C.
D’autres bactéries proies potentielles ont été sélectionnées depuis la collection algale du laboratoire d’accueil (la TCC pour Thonon Culture Collection) et d’un mélange d’eau des lacs Léman, du Bourget, d’Annecy et de l’embouchure de la Dranse (source notée ALBD par la suite). L’isolement de ces bactéries est détaillé plus loin (cf.2.2.1).
Les cultures de BALOs en milieu liquide ont été réalisées dans du tampon HM et incubées à 25°C sous agitation à 200 rotations par minute (rpm) dans une chambre thermostatée.
2.1.2 Milieux de cultures
Différents milieux de culture liquide et solide ont été testés (NB, TSB, LB, HM, et Fur) (Tableau 2). L’agar (EuroBio) a été ajouté à raison de 15 g.L-1 afin d’obtenir des milieux gélosés pour des cultures en boite de Petri. Pour réaliser le top agar HM (voir plus bas), deux concentrations d’agar ont été testées : 5 et 7 g.L-1.
2.1.3 Réactifs et solutions utilisées
Pour réaliser la coloration de Gram sur les souches isolées de la TCC et ALBD, un kit de coloration (Sigma-Aldrich®) comprenant une solution de cristal violet, une solution d’iodine, une solution de décoloration et une solution de safranine, a été utilisé.
Une solution de glycérol à 80% stérile a été préparée à partir de glycérol pur puis autoclavée pour stocker les proies et prédateurs à -80°C.
2.1.4 Matériel d’observation microscopique et autres matériels du laboratoire
L’observation des bactéries a pu être réalisée en utilisant un microscope de la marque ZEISS (modèle AX10). Les observations ont été réalisées avec l’objectif × 100 sous huile à immersion.
6 Tableau 3 : Microalgues sélectionnées pour l’isolement des proies potentielles à partir de la TCC
Classe Famille Genre espèce Origine de
l'échantillon
TCC
Chlorophycées Ulotrichaceae Ulothrix sp. Lac Léman TCC1a
Cyanophycées Phormidiaceae Planktothrix rubescens Lac Léman TCC13 Cyanophycées Synechococcaceae Synechococcus rhodobaktron Lac Léman TCC33 Zygophycées Desmidiaceae Staurastrum sebaldii Lac Léman TCC106 Chlorophycées Oocystaceae Mychonastes homosphaera Lac Léman TCC108b Chlorophycées Scenedesmaceae Scenedesmus serratus Lac Léman TCC110 Chlorophycées Chlamydomonadaceae Chlamydomonas intermedia Lac Léman TCC113 Chlorophycées Scenedesmaceae Scenedesmus acutus Lac Léman TCC116 Chlorophycées Hydrodictyaceae Pediastrum duplex Lac Léman TCC120 Chlorophycées Dictyosphaerioideae Botryococcus protuberans Lac Léman TCC123
Chlorophycées Volvocaceae Eudorina elegans Lac Léman TCC125
Chlorophycées Coelastraceae Coelastrum reticulatum Lac Léman TCC129
Chlorophycées Oocystaceae Mychonastes sp. Lac Léman TCC136-1
Diatomophycées Bacillariaceae Nitzschia palea Lac Léman TCC139-3 Chlorophycées Prasiolaceae Stichococcus bacillaris Lac Léman TCC145-7 Chlorophycées Chlorellaceae Chlorella vulgaris Lac Léman TCC213 Chlorophycées Chlorellaceae Monoraphidium contortum Lac Léman TCC223 Diatomophycées Fragilariaceae Fragilaria crotonensis Lac Léman TCC365 Diatomophycées Fragilariaceae Fragilaria perminuta Lac Léman TCC743 Chlorophycées Scenedesmaceae Scenedesmus costatus Lac Léman TCC744 Diatomophycées Achnanthidiaceae Achnanthidium minutissimum Lac Léman TCC746 Diatomophycées Fragilariaceae Fragilaria perminuta Lac Léman TCC747 Diatomophycées Achnanthidiaceae Achnanthidium minutissimum Lac Léman TCC748 Diatomophycées Fragilariaceae Fragilaria perminuta Lac Léman TCC749 Diatomophycées Achnanthidiaceae Achnanthidium straubianum Lac Léman TCC833
Tableau 4 : Détail des amorces nucléotidiques utilisées
Nom de l'amorce Séquence nucléotidique Références
M13 Forward (-20) GTAAAACGACGGCCAG TOPO® TA Cloning® Kit
M13 Reverse CAGGAAACAGCTATGAC TOPO® TA Cloning® Kit
515 F GTGCCAGCMGCCGCGGTAA 18
7 Le microscope a été configuré en mode « contraste de phase » pour l’observation des cultures de BALOs. Pour les observations des tests de la coloration de Gram, l’appareil a été utilisé en mode « lumière transmise ».
Une caméra de la marque ZEISS reliée au microscope et le logiciel AxioVisio ont été utilisés afin de réaliser des captures d’images et vidéos.
2.2 Méthodes
2.2.1 Obtention des proies à partir de la TCC et ALBD
Préalablement à la tentative d’obtention de BALOs, des bactéries Gram négatif pouvant potentiellement être (utilisées comme) des proies ont été sélectionnées et/ou isolées de différentes matrices, via différentes stratégies. Un premier lot de bactéries a ainsi pu être obtenu à partir d’un mélange d’eaux provenant des lacs d’Annecy, du Léman, du Bourget et de l’embouchure de la Dranse (échantillon nommé ALBD) prélevés pendant les mois de décembre 2018 et janvier 2019. Un mélange unique de l’ensemble a été réalisé dans un erlenmeyer en prenant 100 mL de chaque eau. Un second lot a été obtenu à partir des bactéries présentes dans des cultures non-axéniques de microalgues d’eau douce, provenant de la TCC. Il a été constitué à partir de 25 cultures d’algues différentes (balayant un panel assez large en termes de diversité phytoplanctonique) isolées à partir d’échantillons provenant du Léman (Tableau 3). A partir des flacons contenant ces algues, 3 mL de culture de chaque espèce ont été prélevés puis mélangés ensemble dans un flacon unique contenant un mélange de milieux nutritifs adapté à ces algues.
Ces deux sources potentielles de proies ont alors été filtrées à travers un filtre GF/F (Whatman) pour ne conserver que les bactéries. Le filtrat a été ensuite centrifugé afin de culoter les cellules. Le culot cellulaire a été repris avec 3 mL de tampon PBS, séparé en trois aliquots, chacun servant d’inoculum afin d’ensemencer 3 Erlenmeyers contenant 30 mL de milieu LB, NB ou TSB. Après incubation, la concentration cellulaire de ces cultures a été estimée par cytométrie en flux. Les cultures ont alors été diluées de 10 en 10 jusqu’à 10-7, pour réaliser des étalements sur gélose en boite de Petri, et afin d’obtenir des colonies distinctes (Figure 5).
2.2.2 Préparation des proies
Les souches bactériennes servant de proies (souches caractérisées ou proies isolées) ont alors été préparées à partir d’une colonie isolée sur gélose en boite de Petri. 50 mL de milieu LB ont été ensemencés dans un Erlenmeyer et incubés à 25°C sous agitation à 200 rpm pendant une nuit. Puis, la culture a été centrifugée à 5000 g à 4°C pendant 10 minutes. Le surnageant éliminé, le culot a été récupéré et lavé avec 1 mL via une centrifugation avec les précédents paramètres. Puis, le culot a alors été repris dans quelques millilitre de tampon HM (afin d’obtenir une concentration d’environ 1010 cellules par mL), utilisé immédiatement ou stocké à 4°C. La concentration cellulaire a été obtenue par lecture de densité optique (DO) au spectrophotomètre à 600 nm.
8 Figure 5 : Schéma expérimental de l’obtention de proies à partir de la TCC et de ALBD. En (A) les mélanges d’eaux ALBD (Annecy Léman Bourget Dranse) et les cultures des 25 algues mélangées de la TCC (Thonon Culture Collection) ont d’abord été filtrés avec un filtre GF/F afin d’éliminer les grosses particules. Les filtrats ont ensuite été centrifugés pour culoter les bactéries. Le culot a été repris par du tampon PBS puis cette suspension a été utilisée comme inoculum. Trois Erlenmeyer possédant soit 30 mL de milieu LB, NB ou TSB ont ensuite été incubés sous agitation pendant deux jours. Les concentrations cellulaires ont été mesurées par cytométrie en flux après fixation des cellules au glutaraldéhyde (0,5%) et coloration au SYBR Green I (1/10000). Les concentrations étant identiques dans les 6 croissances, le milieu LB a été choisi arbitrairement comme milieu de croissance pour la suite. En (B) les croissances en milieu LB liquide ALBD et TCC ont été diluées puis étalées sur milieu LB gélosé en boite de Petri. Les boites possédant des colonies bien distinctes ont été sélectionnées.
Croissance en milieu LB des échantillons ALBD et TCC
1 mL de culture est dilué en série de 10 en 10 dans du PBS 1X jusqu’à 10-7
10-5 10-6
10-7
Étalement de 100 µL par dilution sur les milieux gélosés LB
Incubation 24h à 25 C puis observation des colonies distinctes
B.
Échantillon d’eaux mélangées ALBD Échantillon mixte de la TCC - 50 mL des échantillons sont filtrés sur filtre GF/F- Centrifugation 20 minutes à 12000 g - Culot repris avec 3 mL de PBS 1X
1 mL par Erlenmeyer ayant 30 mL de milieu liquide
LB NB TSB LB NB TSB
Incubation 48h à 25 C sous agitation 150 rpm
Détermination de la concentration par cytométrie en flux
9
2.2.3 Culture liquide des BALOs caractérisés
En complément du travail d’isolement des proies potentielles et des BALOs présents dans les lacs péri-alpins, une collaboration avec le Professeur E. Jurkevitch de l’Université Hébraïque de Jérusalem nous a permis d’obtenir 3 souches de BALOs et leurs proies correspondantes (cf §2.1.1.). Ces BALOs ont permis de tester nos protocoles de croissance proies-prédateurs en milieu liquide et solide. Ces prédateurs ont aussi été utilisés comme témoins positifs pour tester des primers dessinés par Jade Ezzedine dans le cadre de son travail de thèse, visant à déterminer la diversité (par séquençage à haut débit) et l’abondance relative (par qPCR) des différents groupes de BALOs possiblement retrouvés dans les grands lacs périalpins. 500 µL des solutions de prédateurs envoyés par le laboratoire d’E. Jurkevitch, ont été inoculés en Erlenmeyer avec 30 mL de tampon HM supplémentés par 2 mL de solution de proies puis incubés à 25°C. Lorsqu’il a été décidé de prolonger plusieurs jours la croissance des BALOs, des proies fraîches ont été réinoculées toutes les 48 heures.
2.2.4 Stockage des souches bactériennes
Afin de sauvegarder les différentes souches bactériennes sur une longue durée, des stocks glycérolés ont été réalisés. Une solution de glycérol à 80% stérile a été utilisée à raison de 20% final après dilution avec la croissance bactérienne réalisée pendant la nuit. Cette suspension a ensuite été placée dans des cryotubes puis stockés dans un congélateur à - 80°C.
2.2.5 Isolement des BALOs des grands lacs périalpins
2.2.5.1 Prélèvement des eaux et enrichissement des échantillons
La première tentative d’isolement de BALOs provenant de grands lacs péri-alpins a été réalisée à partir d’un échantillon du Léman pris devant la Station INRA d’Hydrobiologie Lacustre de Thonon. Ce prélèvement effectué le 02 avril 2019 a consisté en un mélange d’eau de surface, de sédiment et de biofilms fixés sur des pierres. Laissé au repos à 4 C pendant une nuit afin de faire sédimenter les grosses particules, l’échantillon a alors été filtré à travers un tamis de maille 200 µm, puis à travers des filtres de porosité variable : GF/F et PC 5 µm et 2 µm. Pour finir, deux groupes d’échantillons, <5 et <2 µm ont été testés pour tenter d’isoler un prédateur BALO au moyen de la technique d’enrichissement. Le même jour, faisant suite à la filtration, les deux filtrats précités ont été traités comme suit : 100 mL de filtrat (répétés trois fois) ont été placés dans un Erlenmeyer stérile de 500 mL auxquels ont été rajoutés 5 mL d’une solution de proies concentrée. Un Erlenmeyer témoin (eau stérile) a aussi été inoculé avec 5 mL de la solution de proies. La totalité des Erlenmeyers a été placée en chambre chaude à 25°C sous agitation à 200 rpm et auscultée quotidiennement (Figure 6). La préparation de la suspension concentrée de proies a été réalisée de la façon suivante. Les souches bactériennes suivantes ont été cultivées en milieu LB liquide les jours précédents. Les croissances d’E. coli ML35, P. aeruginosa, les cinq souches provenant du CIRM et les dix souches TCC et ALBD ont été préparées en suivant le protocole de préparation des proies (cf.§2.2.2.). Un mélange (mix) des différentes préparations de proies a été préparé en prenant 5 mL de chacune des solutions préparées précédemment : E. coli (ML35),
10 Figure 6 : Schéma expérimental du prélèvement de l’échantillon d’eau du lac Léman et de son enrichissement. L’échantillon d’eau a été prélevé au port de la station INRA. Le mélange d’eaux de surface, de sédiments et de biofilms a été filtré à travers une série de filtre pour n’obtenir que les microorganismes inférieurs à 2 µm. 100 mL d’eau filtrée ont été répartis dans trois Erlenmeyer, et une solution de proies concentrée a été additionnée à raison de 5 mL par Erlenmeyer. Un enrichissement témoin a été réalisé en utilisant de l’eau stérile. Ces enrichissements ont été incubés à 25 C sous agitation jusqu’à l’observation d’une baisse de la turbidité par rapport à l’Erlenmeyer témoin. Une observation microscopique à contraste de phase permet ensuite de vérifier la présence de BALOs.
11
P.aeruginosa, E. coli (CIRM), P. fluorescens, P. putida, C. freundii, H. alvei, TCC (culture des 5 souches
isolées ensemble) et ALBD (culture des 5 souches isolées ensemble).
2.2.5.2 Isolement de BALOs sur boite en double couche d’agar
A la suite de l’étape d’enrichissement, un abaissement de la turbidité conjugué à l’observation de la présence de cellules très motiles de taille inférieure à 2 µm de longueur (microscopie à contraste de phase), l’isolement sur boite a été lancé. 50 mL des cultures d’enrichissement ont été centrifugés une première fois à 500 × g pendant 5 minutes à 4°C, le culot a été éliminé. Le surnageant a été de nouveau centrifugé à 27 000 × g pendant 20 minutes à 4°C, afin de récupérer toutes les cellules restées en suspension dans le précédent surnageant. Le culot a été repris avec 3 mL de tampon HM, puis filtré à travers un filtre ayant des pores de 1,2 µm. Ce filtrat contenant les possibles prédateurs a été utilisé pour réaliser une gamme de dilution de 10 en 10 jusqu’à 10-4 dans un volume final de 5 mL de tampon HM. 100 µL de chaque dilution ont été ajoutés à 4 mL de HM top en surfusion (0,5% ou 0,7 %), supplémenté avec 300 µL d’une solution de proies appropriées (solution préparée au préalable cf.§2.2.2). Après l’avoir mixé rapidement au Vortex, le HM top a été coulé sur une boite de Petri contenant 20 mL de HM plate solidifié. Une fois le HM top solidifié, les boites ont été parafilmées et incubées à 25°C (Figure 7). Après 5 à 8 jours d’incubation, la présence de halos transparents de quelques millimètres (ressemblant à des plages de lyse) témoignant de la consommation des proies du HM top, a été recherchée. Ces plaques de prédation ont alors été prélevées grâce à un cône de pipette P1000 ayant été au préalable découpé. Le morceau de gélose a ensuite été resuspendu dans 0,5 mL de tampon HM. La présence de BALOs dans la gélose a été confirmée par observation au microscope à contraste de phase. Si cela est avéré, la solution est filtrée à travers un filtre de 0,45 µm. Cette l’étape d’isolement sur boite doit être répétées trois fois afin d’obtenir une souche « purifiée » de BALOs.
2.2.6 Caractérisation des souches bactériennes
La caractérisation (i.e. l’identification) des différentes souches de BALOs et de proies ont été réalisées via la technique de clonage séquençage du gène de l’ARN 16S.
2.2.6.1 Extraction d’ADN génomique
Les extractions d’ADN génomiques ont été réalisées à partir des bactéries en conditions de croissance en milieu liquide. Un protocole d’extraction modifié GenElute a été utilisé (cf. Annexes) ; les culots d’ADN génomiques ont été repris dans 20 µL de solution de Tris-EDTA. Les extractions d’ADN génomiques de BALOs ont impliqué de filtrer les cultures à travers un filtre 0,45 µm.
2.2.6.2 Dosage de l’ADN
Les extractions et purifications d’ADN ont été quantifiées (à partir d’un volume de 2 µl) en utilisant le Nanodrop X1000 du fabricant Denovix, et en vérifiant la qualité du produit obtenu au moyen des ratios 260/230 et 260/280.
12 Figure 7 : Schéma expérimental de la technique d’isolement des BALOs par centrifugation et filtration. En (A) le schéma présente la technique d’isolement par centrifugation différentielle. La solution d’enrichissement a été centrifugée une première fois à 500 × g de façon à éliminer les microorganismes de grande taille. Les microorganismes de petites tailles ont été culotés par centrifugation du surnageant à une vitesse de 27000 × g. Le culot repris a été filtré pour isoler les microorganismes inférieurs à 1,2 µm. Le schéma (B) présente la technique d’isolement des BALOs par double-agar. Le filtrat précédent a été dilué en série, et il a été additionné à 4 mL de HM top en surfusion contenant la solution de proie. Ce mélange a été déposé sur une gélose HM plate solide, et laissé à solidifier. Après incubation, la présence de disques clairs dans la couche supérieur de gélose peut présager de la présence de BALOs.
13
2.2.6.3 Protocole PCR
Les protocoles PCR et programmes utilisés avec le thermocycleur TProfessional de la marque Biometra™ pour la caractérisation des souches bactériennes sont détaillés dans les annexes. Les amorces utilisées (M13 ou 16S universelles) sont détaillées dans le Tableau 4.
2.2.6.4 Purification de produits PCR
Les produits PCR utilisés pour le clonage ont été purifiés en utilisant le kit de purification illustra™ de la marque GE Healthcare. Le protocole de purification utilisé est celui indiqué par le fabricant, auquel est rajouté 10 µL d’acétate de sodium à 3,0 M (pH 5,2). L’élution finale a été réalisée avec 15 µL de tampon Tris-HCl à 10 mM (pH 8,0).
2.2.6.5 Clonage et ligation des inserts dans le vecteur
Le kit utilisé pour réaliser le clonage des produits PCR ARN 16S utilise la technologie TOPO® TA Cloning® (Invitrogen). Le vecteur utilisé, le plasmide pCR™2.1 TOPO®, est également présenté dans les annexes. La quantité de vecteur utilisée pour le clonage était de 10 ng. La masse d’inserts utilisée est déterminée par la formule suivante :
𝑚𝑎𝑠𝑠𝑒 𝑑𝑒 𝑣𝑒𝑐𝑡𝑒𝑢𝑟 (𝑛𝑔) × 𝑡𝑎𝑖𝑙𝑙𝑒 𝑑𝑢 𝑣𝑒𝑐𝑡𝑒𝑢𝑟 (𝑘𝑏)
𝑡𝑎𝑖𝑙𝑙𝑒 𝑑𝑒 𝑙′𝑖𝑛𝑠𝑒𝑟𝑡 (𝑘𝑏) × 𝑟𝑎𝑡𝑖𝑜 𝑛𝑏 𝑖𝑛𝑠𝑒𝑟𝑡/𝑣𝑒𝑐𝑡𝑒𝑢𝑟 = 𝑚𝑎𝑠𝑠𝑒 𝑑′𝑖𝑛𝑠𝑒𝑟𝑡 (𝑛𝑔)
Le protocole appliqué est celui fourni par le fabricant du kit. Le ratio du nombre d’inserts par rapport au nombre de vecteurs utilisé était de 3. Le volume final de réaction du clonage variait de 6 à 9 µL.
2.2.6.6 Transformation bactérienne
2 µL du produit de ligation ont été utilisés pour réaliser la transformation. Ce volume a été ajouté à 50 µL de solution d’E. coli compétentes, fournies avec le kit de clonage. Les plasmides ont été incorporés aux cellules grâce à un choc thermique de 40 secondes à 42°C puis un passage dans la glace. Les bactéries ont été étalées sur un gélose LB contenant 100 mg/L d’ampicilline, et une solution de X-Gal / IPTG. Après incubation, les colonies blanches (positives) ont été sélectionnées, réensemencées en milieu LB liquide avec ampicilline et revérifiées par PCR.
2.2.6.7 Electrophorèse en gel d’agarose
Les électrophorèses ont été réalisées en gel d’agarose à 1,5% (p/v) dans du tampon TBE 0,5X. La taille des amplicons a été obtenue par comparaison avec un marqueur de masse moléculaire FastGene® 100 bp DNA Ladder RTU (Figure 8).
2.2.6.8 Séquençage et analyse des séquences
Les parties d’ADN à séquencer ont été envoyées à la société GATC-Eurofins, sous forme des produits PCR déposés dans une plaque 96 puits. L’enchaînement des bases nucléotidiques est déterminé par la technique de séquençage Sanger. Le séquençage demandé est un séquençage de type single-end, autrement dit un séquençage unidirectionnel 5’ – 3’. Au moment du dépôt de ce manuscrit, nous n’avons pas pu récupérer
14 Figure 8 : Marqueur de masse moléculaire FastGene® 100 bp DNA Ladder RTU.
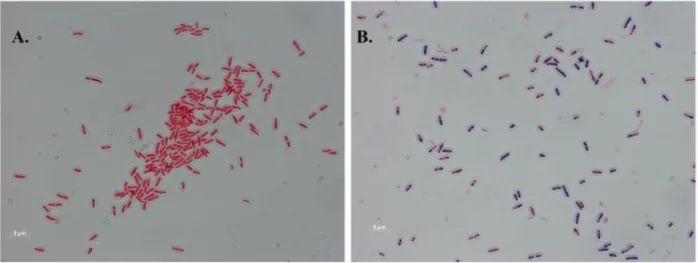
Figure 9 : Exemples de photographies des souches de bactéries proies potentielles ALBD et TCC après coloration de Gram. Deux exemples de photographies obtenues par microscopie optique au grossissement ×1000 sous huile à immersion. (A) souche bactérienne appelée A2, en référence à la deuxième colonie isolée depuis l’échantillon ALBD. Cette souche possède une coloration Gram négatif (bacilles roses). (B) souche de bacilles à coloration Gram positif (bacilles violets) nommée T3, en référence à la troisième colonie sélectionnée à partir de l’échantillon TCC.
Tableau 5 : Résultats de la coloration de Gram des souches de proies potentielles ALBD et TTC. Les noms donnés à ces souches font référence à la colonie isolée à partir des échantillons ALBD ou TCC.
Echantillon ALBD Echantillon TCC
A1 Gram négatif T1 Gram négatif
A2 Gram négatif T2 Gram négatif
A3 Gram négatif T3 Gram positif
A4 Gram négatif T4 Gram négatif
15 l’analyse de nos séquences par le prestataire, ce dernier ayant subi à la fin du mois de mai une cyber-attaque massive (largement repris par les médias). Dans l’avenir, nous espérons donc récupérer nos séquences afin de les contrôler (vérification du chromatogramme de séquençage via le logiciel SnapGene viewer), nettoyer (de la partie plasmidique et des erreurs de séquençage des extrémités 5’ et 3’) puis comparer (« blaster ») à celles disponibles sur la base de données NCBI-nucléotide (pour assignation).
3. Résultats
3.1 Isolements et caractérisation des proies potentielles de TCC et ALBD
Isoler et caractériser des proies potentielles a été une étape essentielle et relativement chronophage au cours de ce projet. Après avoir fait pousser dans un premier les bactéries dans différents milieux de culture, nous avons choisi l’un d’eux pour la suite, le milieu LB (non montré). Dès lors nous avons pu obtenir de nombreuses colonies distinctes et dix souches de bactéries proies potentielles différentes ont été sélectionnées (Figure 5). Neufs se sont révélés être des bactéries Gram négatif et une seule était Gram positif (Figure 9 et Tableau 5). Ces bactéries issues des différents écosystèmes aquatiques locaux et de la TCC sont venues compléter la sélection de 5 souches bactériennes de référence acquise via le CIRM et celles reçues du collaborateur Israélien (Tableau 1).
3.2 Isolements de BALOs du Léman
3.2.1 Enrichissement de la proportion de BALOs dans l’échantillon
La méthode dite d’enrichissement a été choisie afin d’augmenter les chances d’isoler un prédateur conjointement au fait qu’il n’y avait pas, à ce niveau, d’aspect quantitatif à considérer. Les Erlenmeyer incubés à 25°C ont été observés quotidiennement. Dès la diminution de la turbidité constatée, des observations microscopiques en contraste de phase ont permis de détecter la présence possible des bactéries prédatrices (Figure 10). Pour rappel, deux types d’enrichissements ont été réalisés. Pour le premier, la solution de proies ajoutées dans l’Erlenmeyer avec l’échantillon d’eau du port filtrée était composée de l’ensemble des proies sélectionnées toute mélangées. Le deuxième enrichissement a été opéré en discriminant chaque proie potentielle.
L’évidence de la présence de BALOs a donc été suggérée dans un premier temps par l’éclaircissement des flacons de culture d’enrichissement, puis, dans un second temps, par l’observation sous microscope de cellules de petites tailles très motiles, comme cela peut être observé sur le montage vidéo réalisé (https://vimeo.com/340615225). Cette observation a été faite pour la culture en milieu liquide du prédateur de la proie A4, obtenu avec le deuxième enrichissement.
3.2.2 Isolement par centrifugation différentielle et double-agar
Une fois la présence de BALOs suspectée ou avérée, l’isolement à proprement parlé a été lancé. Le premier essai d’enrichissement avec les différentes proies mélangées n’a rien révélé ; aucune plaque de prédation n’ayant pu clairement être identifiée à la suite de la période d’incubation. Après incubation pendant
16 Figure 10 : Photographies des Erlenmeyers au cours de la phase d’enrichissement. Les photographies (A) et (B) montre respectivement l’Erlenmeyer témoin au moment du lancement de l’enrichissement, et après 6 jours d’incubation. Même configuration avec les photographies (B) et (D) mais avec l’échantillon d’eau prélevé au port. En comparaison avec le test témoin, on observe une nette diminution de la turbidité dans l’échantillon d’eau du port (D) après 6 jours d’incubation.
Figure 11 : Photographies de gels d’électrophorèse d’agarose avec les produits PCR utilisant les primers universels. Les photographies (A) et (B) présentent les résultats des migrations de produits PCR par électrophorèse sur gel d’agarose. Il s’agît de PCRs utilisant comme matrice les ADN génomiques extraits, avec amplification par des primers universels 515F et 909R. Les échantillons nommés “Proie XX” signifient que l’extraction a été réalisée sur une culture pure de la proie. Les échantillons nommés “Prédateur XX” utilisent l’ADN génomique extrait à partir d’une croissance liquide de BALOs qui a été filtrée à 0,45 µm pour ne récupérer que les prédateurs.
17 7 jours à 25°C, le second essai a, quant à lui, été plus fructueux ; certaines boites présentant des plaques de prédation. Cela concernait P. putida, P. florescens, les cinq souches provenant de l’échantillon ALBD (A1, A2, A3, A4 et A5), et la boite avec la proie T3. Toutes les plaques de prédation ont été prélevées, remises en milieu liquide et filtrés à travers 0,45 µm. Ce filtrat a alors été utilisé pour (i) inoculer de nouvelles boites en double-agar, et (ii) réensemencer une culture liquide de BALOs dans du tampon HM, afin de réaliser l’extraction d’ADN génomique de ces prédateurs afin de les identifier avec leurs proies.
3.3 Caractérisation génétique des souches bactériennes
3.3.1 PCR et clonage dans le vecteur plasmidique
Le clonage séquençage a très bien fonctionné avec les différents échantillons A3, A4, A5, T1 et T3 et 10 colonies blanches pour chaque traitement ont été récupérées, vérifiées par PCR avec le couple d’amorces M13 et par électrophorèse avant leur envoi au séquençage.
3.3.3 Séquençage des produits PCR
Ce résultat était très attendu pour confirmer notre succès à isoler un ou plusieurs BALOs du Léman et surtout pour le(s) identifier. Malheureusement, le prestataire EuroFins Genomics ayant subi une cyber-attaque majeure au même moment où nous avons envoyé nos produits PCR à séquencer, aucun résultat n’a pu être obtenu et mis en avant au moment du dépôt de ce manuscrit.
18 Figure 12 : Photographie de plaques de prédation obtenues avec B. bacteriovorus HD100. Cette photographie présente une boite de Petri où a été coulé un HM top contenant la souche B. bacteriovorus HD100 et sa proie E. coli ML23. Après 5 jours d’incubation à 25 C, on distingue des disques clairs à travers la couche de proie. Il s’agît de plaques de prédation, causées par la consommation des proies.
19
4. Discussion et Perspectives
Si ces dernières années, les nouvelles technologies de séquençage ont fortement accéléré les recherches en écologie microbienne et les approches « omiques » permis d'aborder les organismes d’un écosystème en les considérant, non plus comme des entités isolées, mais comme un ensemble d'éléments agissant au sein d’un réseau complexe d'interactions, il n’en reste pas moins que pour comprendre leur fonctionnement, les approches culturales demeurent souvent indispensables. Complémentaires des approches moléculaires, elles peuvent notamment permettre (i) de confirmer ou d’interroger les données récoltées via des approches globales, (ii) d’effectuer des tests de sensibilité (ici sur les relations proie-prédateur), (iii) tester et déterminer diverses conditions de croissance, etc ...
Ce projet de Master a consisté à tenter d’isoler, pour la première fois, des bactéries prédatrices obligatoires d’autres bactéries (et de leurs proies) au sein d’un écosystème lacustre, le Léman. Les BALOs ont clairement été étudiés majoritairement dans les milieux marins et terrestres17,20 , si bien que les lacunes de connaissance pour les eaux douces restent, à ce jour, importantes. Des travaux antérieurs effectués au sein du CARRTEL ont toutefois révélé la présence de BALOs au sein des grands lacs péri-alpins, à savoir les lacs Léman, d’Annecy et du Bourget7. Notre objectif final étant d’en apprendre davantage sur les interactions entre ces bactéries et leur importance fonctionnelle au sein des grands lacs alpins, il a donc fallu s’intéresser à la fois aux proies et aux prédateurs. Ce projet a donc été intéressant à plus d’un titre. Au-delà de la découverte et la caractérisation possibles de nouveaux prédateurs bactériens (les seuls connus en milieu lacustre à ce jour), ce sujet a aussi permis de prendre conscience d’un certain nombre de difficultés méthodologiques et tenter de s’adapter.
Premièrement, le postulat de base pour isoler ces prédateurs bactériens nécessitait de posséder un panel de bactéries susceptibles d’être consommées comme proie. Le premier choix s’est porté sur l’utilisation de bactéries déjà caractérisées, soit que le laboratoire avait en sa possession, ou d’en commander depuis des collections disponibles. Parmi les souches disponibles au laboratoire, A. salmonicida salmonicida SA28 et
Synechococcus sp TCC793 ont été sélectionnées. La première provient d’un isolement réalisé sur un omble
chevalier souffrant de la furonculose, issus de la pisciculture du laboratoire. C’est une infection bactérienne souvent retrouvée dans les piscicultures de production, causant de larges pertes pour les éleveurs. La possibilité de trouver un moyen de lutte contre ces pathogènes via l’utilisation d’un microorganisme de biocontrôle, nous a poussé à tester cette souche comme une proie potentielle. De plus, elle possède une paroi de type Gram
négatif. Tout comme Synechococcus sp TCC793, qui a longuement été étudiée par le laboratoire dans le passé.
Nous souhaitons savoir si cette souche pouvait être prédatée par curiosité scientifique.
Pour compléter la liste des bactéries caractérisées pouvant potentiellement être utilisées comme proie, nous avons choisi de commander cinq autres souches. C’est cinq souches ont été choisies en fonction de deux critères : (i) être à coloration de Gram négatif, (ii) être de classe de risque de niveau 1. Le coût a aussi été pris en compte dans le nombre sélectionné, puis elles ont été commandées depuis le CIRM de Tours (Tableau 1).
21 Pour parfaire notre panel de proies, il a été décidé d’isoler des bactéries provenant des mêmes écosystèmes où nous allions tenter d’isoler des BALOs. Un premier choix s’est tourné vers l’utilisation des échantillons d’eaux ALBD, car il s’agît directement des lacs concernés par cette étude. Le deuxième choix a été d’utiliser la TCC, propriété du laboratoire (https://www6.inra.fr/carrtel-collection). La sélection faite sur une partie des microalgues isolées depuis le lac Léman, avait pour but de recueillir les bactéries encore présentes au moment de leur échantillonnage. Car la TCC est composée de cultures non-axéniques (cf. Tableau 1 et §2.2.1). Le nombre de 10 proies potentielles sélectionnées après l’isolement depuis la TCC et ALBD, s’est fait arbitrairement afin de ne pas surcharger le panel de proies. Tout en gardant à l’esprit que ces 10 proies potentielles isolées, peuvent provenir de contaminations via les manipulations depuis le prélèvement et/ou les repiquages au laboratoire, n’étant pas stérilement réalisés.
Initialement prévu, nous devions tenter d’isoler un ou plusieurs BALOs depuis les lacs péri-alpins du Bourget, du Léman et d’Annecy. Mais il a été décidé de se focaliser seulement sur le lac Léman. La principale raison est que le lac est à proximité directe du laboratoire car nous avons un accès au port, en bas de la station. Une autre raison est que le traitement des échantillons d’un seul lac engageait suffisamment de travail, et qu’il aurait été techniquement difficile de s’occuper d’échantillons de plusieurs lacs simultanément.
Cette proximité directe avec le lac Léman a été d’autant plus appréciable qu’il nous a fallu effectuer deux prélèvements pour réaliser nos deux enrichissements, comme précisé précédemment. Le premier avec un mélange de toutes les bactéries proies potentielles, et un deuxième avec chaque potentielles proies séparées. Le choix de réaliser un deuxième enrichissement a été décidé de façon à discriminer plus facilement et plus rapidement les proies ayant un meilleur potentiel. Et par la même occasion, éviter des manipulations supplémentaires avec des bactéries non-consommées par les prédateurs isolés.
L’isolement des prédateurs lancé, nous avions émis plusieurs hypothèses concernant les futurs prédateurs isolés. Premièrement nous nous attendions à probablement isoler un prédateur non-halophile. Ce qui exclut la possibilité de trouver un Halobacteriovorax (environnement salin), ainsi qu’un
Pseudobacteriovorax retrouvé autour de coraux21. L’hypothèse qui nous semble être la plus plausible une fois les résultats de séquençage obtenus et analysé, est de retrouvé un BALOs faisant parti des Peredicbacter. Car comme précisé dans l’étude menée par Paix et al.(2019)7 la niche écologique préférentielle de ce genre de BALOs est en surface, là où nous avons réalisé nos prélèvements.
Malheureusement, il a été difficile d’obtenir des plages de prédation. Pourtant, nous avons testé différentes approches. Par exemple, nous avons testé deux types de milieu HM top, l’un à 0,7% d’agar, l’autre à 0,5%, supposé faciliter les rencontres entre les proies et les prédateurs. Pour augmenter encore plus cette probabilité, il a été décidé d’additionner une solution de proies au HM top en surfusion à une haute concentration (environ 1010 cellules/mL), permettant aussi d’améliorer les contrastes visuels pour observer les zones de prédation vs les zones sans prédateurs. Peu de plaques visibles ont été observées, les seules pouvant être décrites comme tels étant celles obtenues avec la souche B. bacteriovorus HD100 (Figure 12). Clairement, la raison de l’absence de grandes plaques de prédation pour les BALOs et proies isolées du lac reste à creuser.
23 Les plaques obtenues sont restées de très petite taille et susceptibles d’être plus représentatives de plages de lyse (due à l’action lytique de virus lytiques). Les plages de lyse causées par des bactériophages apparaissent plus rapidement que celles des prédateurs bactériens, et ne dépassent pas quelques millimètres. De plus, elles ne s’étendent pas contrairement à celles des BALOs qui peuvent s’élargir pendant une semaine22. Il serait donc possible de les annoter pour ne pas les confondre. Parmi les protocoles à tester à l’avenir pour augmenter les chances d’obtenir et observer des prédateurs, l’utilisation d’un gradient de Ficoll serait surement une solution à envisager car elle permet d’éviter une filtration réduisant la quantité de prédateur à isoler14. Il est aussi probable que les prédateurs nouvellement isolés ne forment pas des plaques de prédation semblables à ce que l’on peut retrouver classiquement chez B. bacteriovorus HD100 (cf. Figure 12). La taille et la forme de ces plaques lytiques dépendent de l’espèce qui les produits22.
Un des résultats présentés paraît surprenant, il s’agît de la suspicion d’un prédateur capable d’utiliser comme proie la souche T3. Or, cette dernière présente la caractéristique d’être une bactérie à coloration de
Gram positif, mais il a précédemment été indiqué que les BALOs prédatent uniquement des bactéries Gram négatif. Par ailleurs, il a déjà été rapporté qu’en présence exclusive de bactéries à coloration de Gram positif, B. bacteriovorus devient après quelques heures un prédateur épibiotique alors qu’à l’origine il favorise un
mode de prédation endobiotique23. La caractérisation par clonage - séquençage pourrait trancher sur cette possibilité pour ce probable prédateur. Malheureusement, les produits PCR obtenus grâce aux amorces universelles étaient trop peu concentrés après l’étape de purification pour pouvoir les utiliser dans le clonage. Les expériences de coculture impliquant les potentiels prédateurs isolés du lac Léman et la souche d’A.
salmonicida salmonicida SA28 du laboratoire, n’ont pas révélé de prédation envers cette souche. Il est fort à
parier qu’elle puisse posséder une couche de S layers à sa surface. Il a précédemment été montré que ce type de structure protéique protège ces bactéries contre la prédation de BALOs. C’est notamment le cas pour A.
salmonicida A449 qui est protégé grâce à ce réseau de protéines de surface des attaques de B. bacteriovorus
109J24.
Malgré tout, de probables plaques de prédation ont été observées avec l’utilisation de P. putida comme bactéries proies. Cela abonde dans le sens que les prédateurs isolés pourraient être faire parti des Peredibacter, car il est mentionné que Peredibacter starrii est capable de prédater P. putida25.
L’observation de cellules de petites tailles très motiles (inférieures à 2 µm de longueur) en microscopie a quand même permis de révéler la présence de BALOs et la possibilité d’en isoler du Léman. Une coloration de Gram aurait pu être faite à cette étape, fournissant un argument supplémentaire si ces bactéries se révélaient être Gram négatif. In fine, l’identification sera obtenue grâce à la caractérisation moléculaire.
In fine, ce projet a permis d’isoler et caractériser de nombreuses bactéries dont certaines susceptibles
d’appartenir au groupe fonctionnel des BALOs. L’étape d’après sera d’étudier finement les interactions proies-prédateurs sous différentes conditions environnementales et mieux appréhender le rôle potentiel ou avéré de ces prédateurs. Une chance unique va aussi consister à profiter du déploiement de mésocosmes au lac au cours de l’été 2019 pour analyser à des échelles de temps inégalés certaines de ces interactions.
24
5. Conclusions
L’objectif principal de l’étude était d’obtenir un ou plusieurs BALOs depuis un grand lac alpin. Pour cela une partie très chronophage du stage s’est attardée sur l’obtention d’une dizaine de souches bactériennes pouvant être utilisées par la suite comme proies pour tenter d’isoler ces prédateurs bactériens.
Plusieurs indicateurs permettent de dire qu’au moins un prédateur a pu être isolé. Le fait d’avoir observé des diminutions de DO lors des enrichissements, avec l’observation en microscopie de bactéries très motiles et de petites tailles, conjugué à la présence de plaques de prédation nous laisse à penser que l’objectif est en bon route pour être atteint.
Pour valider cette hypothèse, la caractérisation moléculaire par le clonage séquençage est indispensable. Et cela constitue un travail qui reste encore à accomplir durant la fin de ce stage. Ce stage qui se voit prolongé jusqu’à la durée maximale légale, afin d’aller au plus loin dans les analyses.
Les analyses des spectres de prédation des BALOs n’ont pas encore été réalisés, mais le seront d’ici la fin du stage.
L’analyse du séquençage me permettra de me former à l’utilisation d’outils en bio-informatique. Cela complètera la formation suivie au cours de ce stage, sur le metabarcoding avec l’utilisation de Mothur.
Pour finir, l’encadrement de Marine GARDILLON sur des techniques microbiologiques fut une expérience très enrichissante.
25
6. Références
1. Davidov, Y., Friedjung, A. & Jurkevitch, E. Structure analysis of a soil community of predatory bacteria using culture-dependent and culture-independent methods reveals a hitherto undetected diversity of Bdellovibrio-and-like organisms. Environ. Microbiol. 8, 1667–1673 (2006).
2. Said, N., Chatzinotas, A. & Schmidt, M. Have an Ion on It: The Life‐Cycle of Bdellovibrio bacteriovorus Viewed by Helium‐Ion Microscopy. Adv. Biosyst. 3, 1800250 (2019).
3. Negus, D. et al. Predator Versus Pathogen: How Does Predatory Bdellovibrio bacteriovorus Interface with the Challenges of Killing Gram-Negative Pathogens in a Host Setting? Annu. Rev. Microbiol. 71, 441– 457 (2017).
4. Staley, J. T. & Konopka, A. Measurement of in Situ Activities of Nonphotosynthetic Microorganisms in Aquatic and Terrestrial Habitats. Annu. Rev. Microbiol. 39, 321–346 (1985).
5. Huber, J. A. et al. Microbial Population Structures in the Deep Marine Biosphere. Science 318, 97–100 (2007).
6. Heidelberg, K. B., Gilbert, J. A. & Joint, I. Marine genomics: at the interface of marine microbial ecology and biodiscovery. Microb. Biotechnol. 3, 531–543 (2010).
7. Paix, B., Ezzedine, J. A. & Jacquet, S. Diversity, Dynamics, and Distribution of Bdellovibrio and Like Organisms in Perialpine Lakes. Appl. Environ. Microbiol. 85, e02494-18 (2019).
8. Jashnsaz, H. et al. Hydrodynamic Hunters. Biophys. J. 112, 1282–1289 (2017).
9. Rotem, O., Pasternak, Z. & Jurkevitch, E. Bdellovibrio and Like Organisms. in The Prokaryotes:
Deltaproteobacteria and Epsilonproteobacteria (eds. Rosenberg, E., DeLong, E. F., Lory, S.,
Stackebrandt, E. & Thompson, F.) 3–17 (Springer Berlin Heidelberg, 2014). doi:10.1007/978-3-642-39044-9_379
10. Chanyi, R. M., Ward, C., Pechey, A. & Koval, S. F. To invade or not to invade: two approaches to a prokaryotic predatory life cycle. Can. J. Microbiol. 59, 273–279 (2013).
11. Chanyi, R. M., Koval, S. F. & Brooke, J. S. Stenotrophomonas maltophilia biofilm reduction by Bdellovibrio exovorus. Environ. Microbiol. Rep. 8, 343–351 (2016).
12. Rendulic, S. et al. A Predator Unmasked: Life Cycle of Bdellovibrio bacteriovorus from a Genomic Perspective. Science 303, 689–692 (2004).
26 13. Ezzedine, J. A. & Jacquet, S. Bactéries prédatrices : zoom sur les Bdellovibrio et organismes apparentés
(BALOs). 25 (2019).
14. Jurkevitch, E. Isolation and Classification of Bdellovibrio and Like Organisms. Curr. Protoc. Microbiol. 26, 7B.1.1-7B.1.20 (2012).
15. Markelova, N. Y. & Gariev, I. A. Predatory bacteria Bdellovibrio: survival strategy. Process Biochem. 40, 1089–1094 (2005).
16. Chen, H., Athar, R., Zheng, G. & Williams, H. N. Prey bacteria shape the community structure of their predators. ISME J. 5, 1314–1322 (2011).
17. Henry N. Williams, N. L. Data report: exploring the presence of Bdellovibrio and like organisms in deep-sea sediment by culture-independent and culture-dependent methods. doi:10.14379/iodp.proc.349.202.2018
18. Reysenbach, A. L. uise & Pace, N. R. Reliable amplification of hyperthermophilic Archaeal 16S rRNA genes by the polymerase chain reaction. Robb, F T, Place, A R Archaea (1995). Available at: https://eurekamag.com/research/033/177/033177965.php. (Accessed: 5th June 2019)
19. Brunk, C. F. & Eis, N. Quantitative Measure of Small-Subunit rRNA Gene Sequences of the Kingdom Korarchaeota. Appl. Environ. Microbiol. 64, 5064–5066 (1998).
20. Sutton, D. C. & Besant, P. J. Ecology and characteristics of bdellovibrios from three tropical marine habitats. Mar. Biol. 119, 313–320 (1994).
21. McCauley, E. P., Haltli, B. & Kerr, R. G. Description of Pseudobacteriovorax antillogorgiicola gen. nov., sp. nov., a bacterium isolated from the gorgonian octocoral Antillogorgia elisabethae, belonging to the family Pseudobacteriovoracaceae fam. nov., within the order Bdellovibrionales. Int. J. Syst. Evol.
Microbiol. 65, 522–530 (2015).
22. Jurkevitch, E. The Genus Bdellovibrio. in The Prokaryotes: Volume 7: Proteobacteria: Delta, Epsilon
Subclass (eds. Dworkin, M., Falkow, S., Rosenberg, E., Schleifer, K.-H. & Stackebrandt, E.) 12–30
(Springer New York, 2006). doi:10.1007/0-387-30747-8_2
23. Iebba, V. et al. Bdellovibrio bacteriovorus directly attacks Pseudomonas aeruginosa and Staphylococcus aureus Cystic fibrosis isolates. Front. Microbiol. 5, (2014).
24. Koval, S. F. & Hynes, S. H. Effect of paracrystalline protein surface layers on predation by Bdellovibrio bacteriovorus. J. Bacteriol. 173, 2244–2249 (1991).
27 25. Peredibacter starrii (Seidler et al.) Davidov and Jurkevitch ATCC ®. Available at:
http://www.lgcstandards-atcc.org/products/all/15145.aspx?geo_country=fr#generalinformation. (Accessed: 6th June 2019)
28
7. Annexes
Protocole coloration de Gram
1. Réaliser un frotti sur une lame porte objet 2. Fixer les cellules à l’alcool
3. Une fois sèche, imbiber la lame avec la solution de cristal violet pendant 60 secondes 4. Rincer la lame à l’eau distillée
5. Déposer la solution de lugol et patienter 60 secondes 6. Rince à l’eau distillée
7. Décolorer la lame sous un jet de la solution de décoloration pendant environ 10 secondes 8. Stopper la décoloration en rinçant la lame à l’eau distillée
9. Faire la contre coloration en déposant la solution de safranine sur la lame pendant 60
10. Rincer à l’eau distillée. Sécher la lame délicatement avec du papier absorbant. Lire l’échantillon sous un microscope optique au grossissement × 1000 sous huile à immersion
Protocole d’extraction d’ADN génomique GeneElute optimisé par le laboratoire
Centrifuger les croissances liquides pour récupérer uniquement les cellules. 1. Ajouter 350µL de TE et 400µL de tampon de lyse
2. Agiter horizontalement pendant 10 minutes
3. Faire un choc thermique : 15 min à -80°C puis 2 min à 55°C
4. Vortexer et centrifuger le tube rapidement avec une centrifugeuse de paillasse 5. Ajouter 25 µl de SDS 20% et 25µl de protéinase K ( 20mg/ml)
6. Incuber 1h à 37°C sous agitation à 200 t/min 7. Incuber 20min à 55°C
8.Centrifuger 6min à 13000rpm à 4°C. Puis transférer le surnageant dans un tube de 2mL.
9.Ajouter 0.1 volume d’acétate de sodium à 3M (pH=5.2). Et ajouter 1µL de solution GenElute. Laisser 10min à température ambiante
10. Ajouter 2.5 volume d’éthanol absolu froid ou 1 volume d’isopropanol froid. Homogénéiser les tubes par retourenements
11. Centrifuger 10min à 12000g à 4°C, et éliminer le surnageant
12. Laver le culot avec 300µL d’étanol 80%. Puis centrifuger 10min à 12000g à 4 C, et éliminer le surnageant 13. Evaporer le reste de l’alcool 20 min au SpeedVac
14. Resuspendre dans 20µL de tampon TE
29
Protocole PCR
Préparation de la solution de réaction :
Programme utilisé pour les PCR :
Réactifs Unités Concentrations
Initiales Concentrations finales Volume pour 1 échantillon (µL) Eau / / / 18,3 Tampon U 10 1 2,5 MgCl2 Mm 50 2 1 BSA mg/mL 10 0,4 1 dNTP mM 100 0,4 0,1 515F µM 10 0,2 0,5 909R µM 10 0,2 0,5 BioTaq U/µL - U 5 0,5 0,1 ADN matrice ng/µL / 25 1
Températures (°C) Temps (seconde) Cycles
95 120 (2min) 94 30 58 30 72 30 72 300 (5min) 30
30