Université des frères Mentouri Constantine Faculté des sciences de la Nature et de Vie
Département de Microbiologie
N° de série : 11/D3L/2015 N° d’ordre : 02/microbio/2015
Thèse de Doctorat 3ème cycle LMD
En Biotechnologies microbiennes, Génomes et Environnement
Thème :
Application des outils biomoléculaires 16S, MALDI-TOF et
métagénomique pour la détermination des variantes
taxonomiques bactériennes telluriques
Présentée par :
Kaoutar KHELILI
Soutenue le: 31 mai 2015
Devant le jury :
Président : Mr. BOULAHROUF Abderrahmane Professeur. Université Constantine 1 Rapporteur : Mr. HAMIDECHI Mohamed Abdelhafid Professeur. Université Constantine 1
Examinateurs : Mr. ARHAB Rabah Professeur. Université Oum El
Bouaghi.
Mr. BOUDEMAGH Allaou Eddine Maître de Conférences A. Université Constantine 1.
Mr. DJEBRI Belgacem Maître de Conférences A. Université Tébessa.
Afin d’être reconnaissante envers ceux qui m’ont appuyé et encouragé à effectuer ce travail
de recherche, je dédie cette thèse :
À ma très chère maman « Zahia » et à mon très cher papa « Abdelkader » qui ont fait de moi
ce que je suis.
À ma chère sœur « Khaoula » et mes deux chers frères « Loqman » et « Aymene » qui n’ont
pas cessé de me combler par leur amour et leur tendresse.
À mon conseiller « cher époux Abdelghani » pour son soutien moral, et pour tous les
sentiments d’affection et d’amour.
À mon petit cœur « mon fils Kynane » qui m’a donné un coup de pouce pour compléter ma
thèse.
À mes chères amies « Zeinouba », « Amoura », « Hinoucha », « Asouma » et « Ouideda » pour
leur gentillesse et leur encouragement.
À tous les membres de ma famille.
Remerciements
J’aimerai, en premier lieu, remercier mon dieu « Allah » qui m’a donné la volonté et le
courage pour la réalisation de se travail.
Avant tout, je remercie les membres du jury, M
rBoulahrouf Abderrahmane, M
rArhab
Rabah, M
rBOUDEMAGH Allaou Eddine et M
rDjebri Belgacem pour avoir accepté de
juger mon travail et pour la discussion qu’ils ont apportée sur ce projet.
Un projet de thèse ne se réalise jamais seul, c’est pourquoi je souhaiterai ici remercier toutes
les personnes qui ont participé à ces cinq années d’aventure scientifique et humaine.
Je souhaiterai tout d’abord remercier M
rHamidechi Mohamed Abdelhafid, mon directeur de
thèse, qui a cru en mes capacités pour relever le défi de ce projet. Je le remercie aussi pour
m’avoir transmis son goût pour la recherche et pour m’avoir fait profiter de son
expérience dans le domaine.
Au sein des deux laboratoires, de microbiologie et de biochimie, situés au sein de la faculté
des sciences de la nature et de la vie de l’université Constantine 1, je souhaiterai remercier
particulièrement toutes les techniciennes et tous les techniciens pour leur aide et leur
sympathie durant mes travaux pratiques. Ainsi que le staff administratif de notre
département.
J’aimerai bien remercier M
meSoltani Mounira, chef de service à l’ANRH (Constantine), de
m’avoir effectué les analyses physicochimiques de mes échantillons de sol. De plus, je
souhaiterai lui dire merci aussi pour sa gentillesse et sa générosité.
Durant mon premier stage pratique, j’ai eu l’occasion de travailler avec M
rMichel
Drancourt du Laboratoire de microbiologie, hygiène, épidémiologie hospitalisation à
l’hôpital de la Timone (Marseille, France). A son côté j’ai appris à utiliser le MALDI-TOF
MS, la PCR et le séquençage de l’ADNr 16S. Je le remercie pour son accueil dans son
laboratoire, et pour les discussions enrichissantes que nous avons eu sur le projet. Cela m’a
permis d’explorer les données selon de nouvelles perspectives et de développer mon
esprit critique sur les méthodes moléculaires que nous utilisons.
Je voudrai dire un grand merci à M
rEmmanuel Lemichez du Centre Méditerranéen de
Médecine Moléculaire (Nice, France) de m’avoir appris beaucoup de connaissance dans le
domaine de la biologie moléculaire durant mon deuxième stage. Merci à toute l’équipe de
recherche (Daniel, Emilie, Patrick et Anne) de m’avoir apporté une vision différente au
projet et pour les discussions très intéressantes que nous avons eu. Cela m’a permis
de données : les librairies et les séquences de gènes d’ARNr 16S. Merci à lui de
m’avoir fait partager son expérience dans le domaine de la métagénomique.
Enfin, en dehors du laboratoire, au cours de ces cinq années, j’ai eu la chance de rencontrer
d’autres jeunes chercheurs en biologie, qui sont des gens formidables, auprès des quels
j’ai non seulement développé mon expérience du management et de la gestion de projet, mais
j’ai aussi passé de très bons moments de détente post-réunion. Je tiens à remercier
particulièrement : Soumya, Sarah, Sofiane, Mehdi, Narjesse, Rita, Mamadou et Fatima.
Liste des figures i
Liste des tableaux iv
Liste des abréviations et des acronymes v
Introduction générale 1
Synthèse bibliographique
1. Objet sol : une matrice vivante complexe, hétérogène mais structurée 4
1.1. Fraction minérale 5
1.2. Matière organique 6
1.3. Structure du sol 6
1.4. Eau et l’air 7
2. Le sol : un réservoir de la diversité bactérienne 8 2.1. Facteurs influençant la structure des communautés bactériennes telluriques 9
2.1.1. Facteurs abiotiques 10
2.1.2. Facteurs biotiques 11
3. Caractérisation des bactéries telluriques 12 3.1. Approche phénotypique, le fondement de la microbiologie du sol 12
3.2. Approche moléculaire 13
3.2.1. Ere protéomique et l'identification bactérienne 14
3.2.1.1. Spectrométrie de masse 14
3.2.1.2. MALDI-TOF MS en microbiologie : une histoire récente 15 4. Etude de la diversité bactérienne tellurique: 19
4.1. Diversité bactérienne dans le sol 19
4.2. Paramètres influençant la diversité bactérienne dans le sol 20
4.3. Approche métagénomique 23
4.3.1. Définition 23
4.3.2. Contexte et origine 23
4.3.3. La notion d'espèce : de la microbiologie à la microbiologie de l'environnement
25
4.3.4. Contraintes de la définition d'une OTU en métagénomique 26
4.3.5. Méthodes et applications 27
4.3.6. Défis 30
4.3.6.4. Séquençage à haut débit : la nouvelle technologie " pyroséquençage 454 "
35 4.3.6.5. Traitement des données et analyse de la diversité 40
Matériels et méthodes
Partie 1 : Isolement et caractérisation phénotypique et moléculaire des bactéries telluriques
42
1. Échantillonnage de sol 42
2. Isolement et purification des souches 42
2.1. Réalisation des dilutions 42
2.2 Ensemencement et incubation 42
3. Identification phénotypique des isolats 42
4. Identification moléculaire des isolats 43
4.1. Identification protéomique par MALDI-TOF MS 43
4.1.1. Préparation de la matrice 43
4.1.2. Dépôt des isolats 44
4.1.3. Introduction de la cible MALDI dans le spectromètre de masse Microflex LT
44
4.1.4. Lancement de l’acquisition MALDI-TOF MS et interrogation de la base de données
46
4.2. Identification génotypique (amplification et séquençage de l’ADNr 16S) 48
Partie 2 : Caractérisation de la diversité bactérienne par analyse de l’ADN extrait des sols (approche métagénomique)
50
1. Prélèvement des échantillons de sol 50
2. Analyses physicochimiques des sols 50
3. Approche métagénomique 50
3.1. Extraction des acides nucléiques 50
3.2. Amplification d’ADNr 16S et pyroséquençage 454 50
3.3. Traitement et analyse des données 52
Résultats et discussion
Partie 1 : Le MALDI-TOF MS fournie un outil utile pour l’identification rapide des bactéries telluriques
1.1. Approche phénotypique 53
1.2. Approche moléculaire 57
1.2.1. Identification protéomique (MALDI-TOF MS) 57 1.2.2. Identification génotypique (amplification et séquençage de l’ADNr 16S) 65
Partie 2 : L’approche métagénomique : accès à la diversité bactérienne tellurique et l’influence des caractéristiques physicochimiques
70
1. Caractérisation des propriétés physicochimiques des trois types de sol 70
1.1. Couleur 70
1.2. Répartition granulométrique 70
1.3. Teneur en calcaire (CaCO3) 71
1.4. Teneur en matière organique 71
1.5. pH 72
2. Mesure de la diversité bactérienne tellurique 72 2.1 Assignation des séquences d’ADNr 16S des bactéries 72
2.2. Mesure de la diversité bactérienne 79
2.2.1. Courbe de raréfaction 79
2.2.2. Indices de la diversité bactérienne 81
2.2.2.1. Indice de Shannon-Weaver 81
2.2.2.2. Inverse de l’indice de Simpson 82
2.2.2.3. Indice Chao1 83
3. Effet des caractéristiques physicochimiques sur la diversité bactérienne 83
Conclusion générale et perspectives 85
Références bibliographiques 87
i
Liste des figures :
Figure 1 : Le positionnement et la complexité externe et interne du sol (Gobat et al., 2003). Figure 2: Proportion des principaux composants du sol en volume (White, 2006).
Figure 3 : Schéma d’identification par MALDI-TOF à partir des colonies.
Figure 4 : Relation entre le pH des sols et la diversité bactérienne, en utilisant la diversité
phylogénétique (A) et le nombre de phylotypes (B) défini à 97% de similarité. La courbe pleine représente la courbe de tendance. Les indices de diversité ont été calculés en utilisant 1200 séquences par sol (Lauber et al., 2009).
Figure 5 : Contribution des séquences d’ARNr 16S bactériens aux phylums les mieux
représentés dans le sol. Les données ont été compilées à partir de 21 banques (2920 clones) de clones d’ARNr 16S obtenus par amplification a partir d’ADN du sol. La barre horizontale représente le pourcentage moyen de séquences attribuées au phylum, le rectangle représente l’ecart type et les barres verticales représentent les valeurs maximales et minimales (North et
al., 2004).
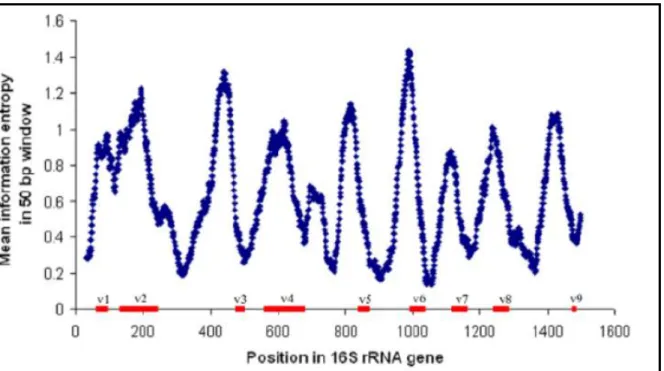
Figure 6 : Représentation schématique de la variabilité le long de l'ADNr.
La variabilité est exprimée par l'entropie de Shannon en fonction des régions hypervariables (en rouge) sur les gènes codant pour l'ARNr 16S (Andersson et al., 2008).
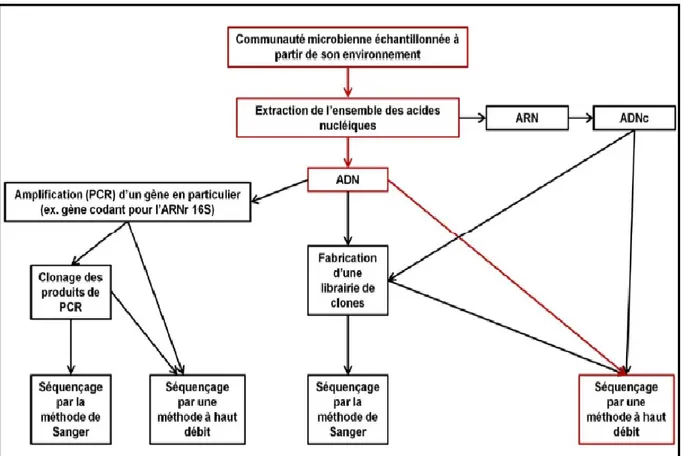
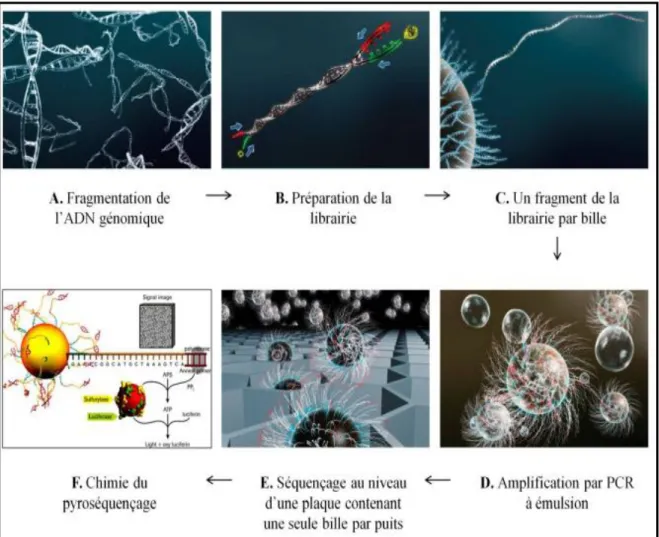
Figure 7 : Schématisation de plusieurs méthodologies courantes dans une analyse
métagénomique (figure adaptée d'Ederer, 2011). Le chemin constitué de cadres et de flèches rouges symbolise la méthode métagénomique utilisée dans les différentes analyses présentées dans notre thèse.
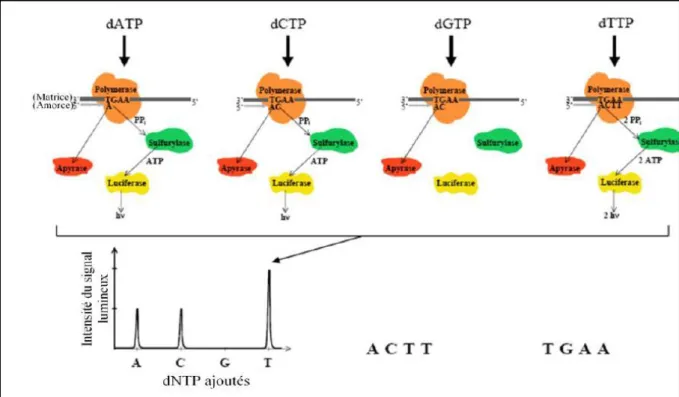
Figure 8 : Etapes du pyroséquençage (Ahmadian et al., 2006).
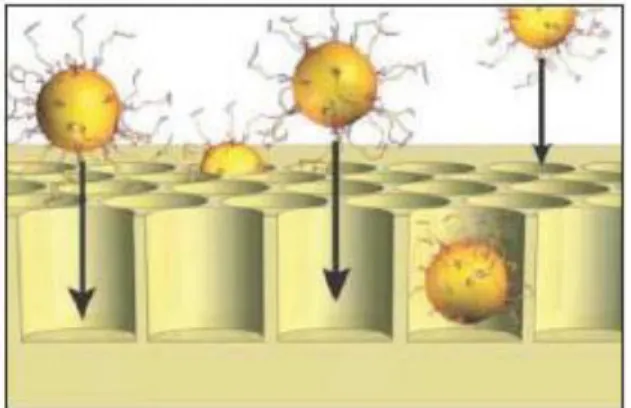
Figure 9 : Représentation du dépôt d’une microbille dans un puits d’une plaque en fibre
optique contenant 1,4 millions de puits (Shendure et Ji, 2008).
Figure 10 : Principe du pyroséquençage 454 (figure adaptée de 454 Life sciences, ©
1996-2012 Roche Diagnostics Corporation, www.454.com).
Figure 11 : Cible MALDI type Microflex, modèle " MSP 96 " (11 colonnes et 8 lignes). Figure 12 : Trappe d’insertion en position ouverte sans (a) et avec (b) cible.
ii Figure 13 : Introduction de la cible Microflex LT.
Figure 14 : Interface du logiciel MALDI BioTyper TM 2.0 (Bruker Daltonics).
Figure 15 : Interface du logiciel d’acquisition FlexControl.
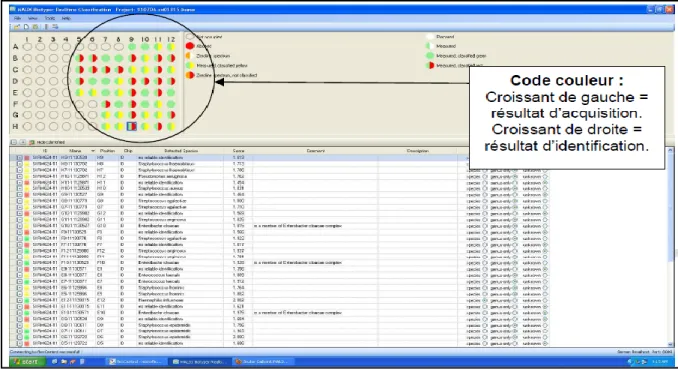
Figure 16 : Code couleur des analyses et identifications BioTyper TM 2.0 (Bruker Daltonics).
Figure 17 : Constitution des amorces fusionnées utilisées pour le pyroséquençage. Figure 18 : Spectre de masse de l’isolat S1.
Figure 19 : Spectre de masse de l’isolat S2. Figure 20 : Spectre de masse de l’isolat S3. Figure 21 : Spectre de masse de l’isolat S4. Figure 22 : Spectre de masse de l’isolat S5. Figure 23 : Spectre de masse de l’isolat S6. Figure 24 : Spectre de masse de l’isolat S7. Figure 25 : Spectre de masse de l’isolat S8. Figure 26 : Spectre de masse de l’isolat S9.
Figure 27 : Dendrogramme des neuf isolats bactériens réalisé par le logiciel MALDI
BioTyper TM 2.0 (Bruker Daltonics).
Figure 28 : Produits d’amplification PCR du gène d’ARNr 16S des neuf isolats bactériens,
sur gel d’agarose à 1,5 %. M) marqueur moléculaire 1 kpb.
Figure 29 : Arbre phylogénétique construit avec la méthode Neighbor-joining montrant les
liens phylogénétiques entre 9 séquences basées sur les gènes d’ARNr 16S des isolats bactériens. Les nombres figurant au niveau des nœuds indiquent les taux de Bootstrap (1000 réplicats), la barre 0.02 indique le nombre de substitution par position de nucléotide. Les numéros d’accession sont indiqués entre parenthèse.
Figure 30 : Histogramme montrant les pourcentages des séquences identifiées au niveau du
iii Figure 31: Carte phylogénétique ("heatmap") des phyla identifiés dans les trois sols.
Figure 32: Histogramme montrant les pourcentages des séquences identifiées au niveau de
famille dans les trois échantillons de sol.
Figure 33: Carte phylogénétique ("heatmap") des familles identifiées dans les trois sols. Figure 34: Histogramme montrant les pourcentages des séquences identifiées au niveau du
genre dans les trois échantillons de sol.
Figure 35 : Carte phylogénétique ("heatmap") des genres identifiés dans les trois sols.
Figure 36: Courbes de raréfaction (nombre d’OTU identifiées par rapport au nombre de
séquences analysées) pour les trois échantillons de sol.
Figure 37: Histogramme des nombres des OTUs identifiées dans les trois échantillons de sol. Figure 38: Histogramme représentant les valeurs de l’indice de diversité "
Shannon-Wiener ".
Figure 39: Histogramme représentant les valeurs de " l’inverse de l’indice de diversité de
Simpson ".
iv
Liste des tableaux :
Tableau 1 : Score de concordance des spectres obtenus à partir d’une bactérie d’intérêt avec
ceux de la base de données Biotyper, reflétant le degré de confiance à accorder à l’identification.
Tableau 2 : Amorces utilisées pour l’amplification et le séquençage du gène de l’ARNr
16S (Adékambi et Drancourt, 2004).
Tableau 3 : Liste des séquences code à barres (Hamady et al., 2008).
Tableau 4: Aspect macroscopique des colonies bactériennes isolées sur gélose au sang. (Les
photos sont réalisées au laboratoire de cultures spéciales à la faculté de médecine de Marseille).
Tableau 5: Résultats de l’identification présomptive des isolats bactériens. Tableau 6: Identification des isolats bactériens par MALDI-TOF MS. Tableau 7 : Confrontation et correspondance biomoléculaire avec GenBank. Tableau 8: La répartition granulométrique des trois échantillons de sol. Tableau 9 : Norme d'interprétation du taux du calcaire du sol (Baize, 1988). Tableau 10 : Echelle d'interprétation du pH (Gagnard et al., 1988).
v
Liste des abréviations et des acronymes :
ACE : Abundance based coverage estimator.
ANRH : Agence Nationale des Ressources Hydrauliques.
bcPCR : Barcode Polymerase Chain Reaction. BLAST : Basic Local Alignment Search Tool. BLASTn : BLAST Nucleotide.
BTS : Bruker Bacterial Test Standard. CCD : Charge-Coupled Device. DHB : Acide 2,5-dihydroxybenzoïque. dNTP : déxoxyribonucléotides-triphosphate. EMBL : European Molecular Biology Laboratory.
EMP : Earth Microbiome Project.
emPCR : Emulsion Polymerase Chain Reaction. GOS : Global Ocean Sampling.
gyrB : gyraseB gene.
HMP : Human Microbiome Project.
HPLC : High-performance liquid chromatography.
MALDI-TOF MS: Matrix Assisted Laser Desorption/Ionization Time-Of-Flight Mass
Spectrometry.
MID : Multiplex Identifier.
MLST : Multilocus sequence typing.
MSP: Main Spectra Projection.
NCBI : National Center for Biotechnology Information.
NGS : Next Generation Sequencing. OTU : Operational Taxonomic Unit. PTP : PicoTiterplate.
vi RDP : Ribosomal Database Project.
SARAMIS : Spectral ARchive And Microbial Identification System. SBS : sequencing by synthesis.
TFA : Trifluoroacetic Acid.
1
L’identification des bactéries est une priorité absolue surtout lorsqu’il s’agit de germes pathogènes ou à intérêt industriel. La description phénotypique permet, certes, de "cadrer" le germe, mais est-elle vraiment suffisante ? Tous les microbiologistes diront que non. L’identification au niveau taxonomique de l’espèce a fait couler beaucoup d’encre et soulevé plus d’un débat, à cause des différentes méthodes développées jusque-là par plusieurs microbiologistes.
D’un point de vue écologique, le sol est l’un des plus importants réservoirs de diversité microbienne et surtout l’un des derniers remparts pour cette biodiversité (Swift, 1998) résultant de la complexité des assemblages taxonomiques et fonctionnels des communautés microbiennes et des interactions entre eux (Coleman et Whitman, 2005). Ceci est d’autant plus vrai pour les communautés bactériennes qui sont encore considérées, sur la base d’un postulat proposé par Beijerinck (1913) : "tout est partout" (everything is everywhere), comme des organismes ubiquistes présentant une forte redondance fonctionnelle. A ce titre, les communautés bactériennes telluriques apparaissent encore de façon récurrente comme une "boîte noire" fonctionnelle génératrice de flux dont l’intensité dépend de plusieurs facteurs biotiques et abiotiques.
Malgré que ce compartiment fondamental de la biosphère constitue une mosaïque d’habitats avec pour chacun des populations bactériennes propres, notre niveau de connaissance reste limité. Il en résulte un manque de prise en compte des communautés bactérienne en terme de diversité et de distribution spatiale à petite comme à grande échelle (Ranjard et al., 2010). Ce qui peut être expliquer par le retard méthodologique sur la caractérisation des bactéries du sol in situ qui s’explique aussi par les caractéristiques propres du modèle bactérien (i) petite taille des organismes, (ii) accessibilité plus ou moins importante des populations au sein d’une matrice hétérogène et structurée comme le sol, (iii) difficulté à résoudre une information constituée de 100 000 à 1 000 000 d’espèces différentes par gramme de sol (Curtis et Sloan, 2005 ; Maron et al., 2007). Toutefois, depuis une vingtaine d’années, les importantes avancées en biologie moléculaire ont permis de mettre au point des techniques qui permettent d’appréhender et de résoudre la diversité des communautés bactériennes du sol in situ et sans a priori (Ranjard et al., 2000).
Jusqu’au début des années 2000, au sein des laboratoires de microbiologie du sol, l’identification bactérienne reposait essentiellement sur l’analyse de caractères phénotypiques dont l’interprétation nécessitait une expertise. Pour la plupart des cas, le résultat n’était
2
obtenu qu’au bout de 18 heures. En cas d’échec, l’identification était acquise par des analyses de biologie moléculaire comme celles des séquences du gène de l’ARNr 16S. Depuis près de dix ans, des automates utilisant la spectrométrie de masse de type MALDI-TOF (Matrix Assisted Laser Desorption/Ionization Time-Of-Flight) (Mellmann et Müthing, 2013) ont révolutionné l’identification bactérienne dans le domaine clinique. À partir de colonies, des résultats d’identification fiables sont acquis en moins de cinq minutes avec une facilité d’utilisation technique. Ces atouts ont largement contribué à son succès. Dès lors qu’un laboratoire s’équipe avec ce type d’automate, l’essentiel des identifications est assuré par cette nouvelle approche. Mais malheureusement, cette technique est très peu exploiter dans le domaine de la microbiologie du sol (Uhlik et al., 2011 ; Koubek et al., 2012) d’où la nécessité d’autres travaux de recherche. Pour cette raison, nous avons choisi de se focaliser, dans la première partie de nos études expérimentales, sur l’applicabilité du MALDI-TOF MS couplé à l’algorithme Biotyper dans ce domaine assez complexe.
Cependant, cette technique est dépendante des conditions de culture. En effet, l’étude des populations par isolement de bactéries sur milieux de culture ne permet la mise en évidence que d’une très faible proportion des espèces bactériennes présentes (Nocker et al., 2007) car seulement 0,1 % à 1 % des bactéries telluriques peuvent être cultivées (Torsvik et
al., 1998 ; Kirk et al., 2004). Ces limites imposées par la culture in vitro ont permis le
développement d’approches s’en affranchissant. L’arrivée de la métagénomique a restreint l’étude des bactéries à leur ADN directement extrait de l’environnement (McHardy et Rigoutsos, 2007). L’ADN alors collecté à partir d’échantillons environnementaux (le métagénome) peut être analysé par des techniques permettant de caractériser la structure d’une communauté bactérienne complexe (Ramette, 2009). Ces méthodes nécessitent souvent une étape d’amplification. Récemment les nouvelles techniques de séquençage d’ADN ont permis d’obtenir des gigabases de données de séquences venant de bactéries non cultivées (Roesch et al., 2007), permettant une variété d’applications génétiques.
Par rapport à l’absence d’un organigramme microbiologique consensus pour l’identification formelle des bactéries, nous proposons, dans cette thèse, d’étudier la diversité bactérienne du sol d’El-Meridj (Constantine, Algérie) en mettant en exergue l’importance des techniques moléculaires actuelles dans l’identification bactérienne ; à savoir :
- Evaluer la capacité du MALDI-TOF MS, par rapport aux gènes d’ADNr 16S en tant que référence, à identifier les différentes variantes taxonomiques bactériennes telluriques en confrontant leurs résultats sous forme de dendrogrammes.
3
- Mesurer la diversité bactérienne tellurique par la technique de métagénomique et étudier l’effet des caractéristiques physicochimiques du sol sur cette biodiversité.
4
1. Objet sol : une matrice vivante complexe, hétérogène mais structurée
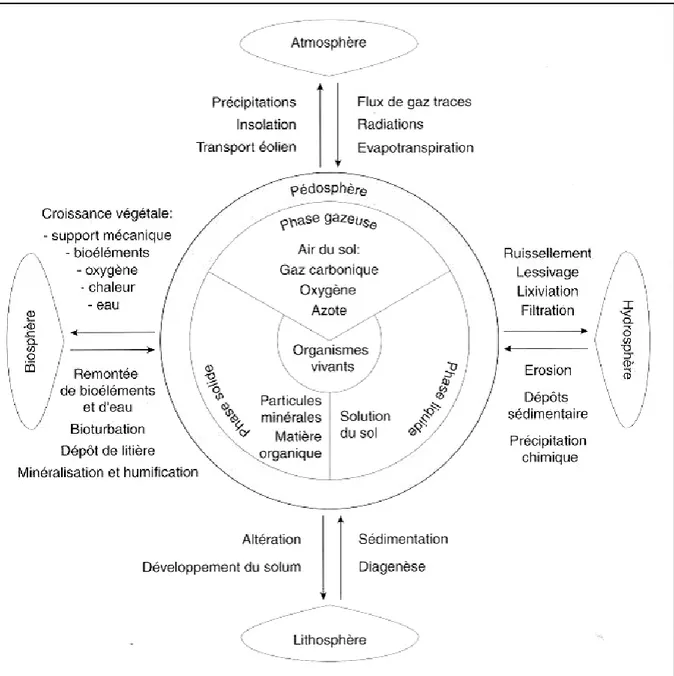
Le sol est considéré comme l’un des environnements les plus complexes de la biosphère et est à ce titre un réservoir majeur de la diversité microbienne. Il est à l’interface entre la lithosphère, l’atmosphère et l’hydrosphère (Alexander, 1977) et sert de support à une partie de la biosphère (Figure 1 ; Gobat, 2003).
Figure 1 : Le positionnement et la complexité externe et interne du sol (Gobat et al., 2003).
Il est constitué de cinq composants majeurs : fraction minérale, matière organique, eau, air et organismes vivants (Figure 2 ; Alexander, 1977). Les matières organiques et minérales s’organisent de manière à créer des vides alors occupés par l’air et l’eau (les pores).
5
Figure 2: Proportion des principaux composants du sol en volume (White, 2006). 1.1. Fraction minérale
Elle dépend du type de roche mère à partir duquel le sol s’est formé. La fraction minérale des sols résulte de l’action de deux principaux phénomènes (Best, 1995). La désagrégation physique due aux alternances chaud/froid, le gel, le vent ou l’eau fractionnant la roche en morceaux de taille de plus en plus faible, les particules générées conservant la composition minéralogique initiale (Gobat, 2003). L’altération due à l’eau, associée ou non à l’oxygène, au gaz carbonique CO2 ou à des acides organiques (Gobat, 2003) provoque une transformation des minéraux primaires avec formation de minéraux secondaires (comme l’argile) dont l’ensemble constitue le complexe d’altération (Duchaufour, 1977).
Cette fraction minérale est constituée de particules de différentes tailles, classées généralement selon leur diamètre : la fraction grossière (>2mm) et la terre fine (<2mm) (sables, limons et argiles). La proportion relative des éléments de la terre fine détermine la texture d’un sol conditionnant directement la structure d’un sol. Les argiles sont le résultat de l’altération des roches par hydrolyse des minéraux silicatés (Gobat, 2003). Les minéraux argileux formés sont des phyllosilicates, constitués de feuillets organisés en couches. Certaines argiles sont capables d’expansion suite à l’hydratation des couches interfoliaires (Gobat, 2003). Une autre composante de la réactivité des particules argileuses est leur surface spécifique. Plus leur surface disponible est élevée, plus leur réactivité sera prédominante
6
(Alexander, 1977). L’adsorption de molécules et ions à la surface des argiles augmente avec leur surface disponible par unité de masse (White, 2006). Par ailleurs les phyllosilicates argileux sont des colloïdes possédant d’autres propriétés qui vont influencer la structure, la porosité ou la capacité d’échange ionique du sol. En effet les argiles sont chargées électro-négativement, sont hydrophiles, mais aussi lipophiles présentant un pouvoir de dispersion et de floculation. Ces propriétés varient suivant les structures minéralogiques et les surfaces développées respectives, régissent la formation d’agrégats, cimentent les particules et favorisent l’habitat microbien (Gobat, 2003).
1.2. Matière organique
Par ses propriétés physicochimiques, la matière organique interagit avec les particules de sol et participe ainsi à sa structuration. La matière organique fraiche provient des déchets des organismes, de leurs sécrétions ou des tissus morts. Mais elle provient majoritairement de débris végétaux (cellulose, hémicellulose, lignine, tanins) (Chenu, 1993). Ces composés organiques humifiés sont séparés en trois fractions suivant leur solubilité : les acides fulviques très solubles à tout pH, les acides humiques insolubles à pH acide, et l’humine insoluble. Les acides fulviques sont électronégatifs et ont la capacité de complexer les cations. Les acides humiques forment des complexes avec les argiles, les minéraux, et ions métalliques ainsi qu’avec les composés organiques hydrophobes (Parent et Velegol, 2004). Des composés lipidiques (acides gras, cires, résines) et azotés (ADN, chitine, muréline, urée, amines, protéines sont également présents dans le sol. Ces composés provenant des bactéries, plantes ou animaux peuvent se fixer à des composés humifiés (Gobat, 2003) ou à des colloïdes minéraux. Le sol contient également des êtres vivants appartenant à chacun des grands groupes du vivant : Bacteria, Archaea et Eucarya. Nous allons nous intéresser plus particulièrement aux bactéries, même si les autres groupes participent également à la structuration du sol interagissant donc avec les bactéries.
1.3. Structure du sol
Les agrégats du sol sont des assemblages opérationnels hétérogènes de particules organiques et minérales qui se distinguent par la taille : les macros (>250μm) et les micros (<250μm) (Tisdall, 1982). Les macro-agrégats sont formés par des associations temporaires de micro-agrégats, de minéraux et de particules de matière organique principalement par enchevêtrement des hyphes fongiques et des racines de plantes (Oades, 1984). Les
micro-7
agrégats typiquement formés par des processus microbiens à l’intérieur de macro-agrégats sont largement dépendants de la persistance des agents liants organiques pour la stabilité structurelle (Tisdall, 1982). Des perturbations physiques du sol résultent généralement en une diminution des macro-agrégats, qui deviennent alors des blocs de construction pour le prochain cycle de formation des macro-agrégats (Oades, 1984 ; Six, 1999 ; Six, 2000). Le sol peut donc présenter de nombreux niveaux d’organisation, chaque niveau ayant une stabilité variable. La structure du sol représente une mosaïque de microenvironnements qui diffèrent par leurs propriétés physiques, chimiques et biologiques. Le volume des espaces des sols dépend plus de la taille, de la forme et de la stabilité des agrégats que des particules minérales elles mêmes (Ranjard et Richaume, 2001).
1.4. L’eau et l’air
L’eau circule et est retenue dans le sol par le réseau des pores. Ces pores sont occupés par un réseau aqueux discontinu, sauf quand le sol est saturé, portant des substances inorganiques ou organiques dissoutes et comprennent une phase gazeuse. L’eau gravitationnelle circule librement dans les macro-pores de diamètre supérieur à 10μm, constitués entre les agrégats et à l’intérieur des macro-agrégats. Une partie de l’eau est fortement retenue par capillarité à l’intérieur des micropores (<10μm) (Robert, 1992) constitués à l’intérieur des micro-agrégats. La disponibilité de l’eau résulte de la combinaison des potentiels matriciel et osmotique. La rétention et potentiellement l’immobilisation de l’eau sont plus importantes dans les microporosités dues à un potentiel matriciel plus élevé que dans les macroporosités, maintenant les microporosités dans un état plus humide que les macroporosités quand le sol est soumis à de fortes pluies. L’eau du sol est très importante dans l’écologie des micro-organismes du sol car la disponibilité des nutriments et l’intégrité des membranes bactériennes nécessitent que la solution du sol soit largement disponible et circulante (Ranjard et Richaume, 2001).
La diffusion des gaz, comme la circulation de l’eau, dépendent de la distribution de l’espace poreux. La diffusion de gaz est faible dans les micropores (Baver, 1961). Le faible renouvellement de l’atmosphère dans ces microenvironnements combiné avec la consommation biologique de l’oxygène, peuvent conduire à un développement rapide et à la persistance de conditions anaérobies. La survie des bactéries dépend donc de leur capacité pour une respiration alternée, remplaçant l’oxygène par un autre accepteur final d’électrons. Il a été montré par exemple que les zones anaérobies à l’intérieur d’un agrégat artificiel sont
8
préférentiellement colonisées par des populations bactériennes capables d’utiliser le nitrate comme accepteur final d’électrons (Philippot et al.,, 1996). La proportion relative de liquide et de gaz affectant les phénomènes aérobies et anaérobies, dépend de la saturation des sols et de la circulation à l’intérieur du réseau des pores (Robert, 1992).
2. Le sol : un réservoir de la diversité bactérienne
L’importance de la variabilité spatiale en microbiologie du sol a depuis longtemps été reconnue et la distribution spatiale des microorganismes a été décrite à des échelles allant de quelques millimètres à plusieurs mètres (Grundmann et Debouzie, 2000 ; Nunan et al., 2002). Des différences de populations bactériennes sont associées à des tailles différentes de particules de sol ou des fractions d’agrégats, incluant les populations associées avec les sous-fractions des micro-agrégats. Mummey et al. (2004) ont clairement démontré une stratification spatiale suivant la localisation à l’intérieur et à l’extérieur des micro-agrégats. Le fractionnement des micro-agrégats du sol par des irradiations UV permet de comparer les structures de communautés bactériennes au niveau de la micro-échelle (Mummey, 2004). A micro-échelle, les organisations structurelles du sol fournissent par conséquent différents habitats dans lesquels les bactéries indigènes sont distribuées de manière hétérogène (Ranjard et Richaume, 2001). Les bactéries sont localisées soit dans les pores du sol qui représentent jusqu’à 50% du volume total du sol, soit enlisées dans l’argile (Foster, 1988 ; Grundmann, 2004). La distribution quantitative hétérogène des bactéries dans le sol résulte en une plus grande abondance des bactéries dans les micropores (Ranjard et Richaume, 2001). Le diamètre maximum des pores les plus fréquemment colonisés par les bactéries est estimé entre 2.5 μm et 9 μm pour respectivement les textures fines et grossières du sol (Hattori, 1988 ; Postma, 1989). 80% des bactéries sont préférentiellement localisées dans la partie interne des micro et macro porosités, quel que soit le type de sol (Hattori, 1988 ; Ranjard, 1997). Les bactéries localisées dans les macropores sont sujettes à des alternances plus intenses entre sécheresses et inondations. Par conséquent, leur survie dépend de leurs capacités à s’adapter aux fluctuations des conditions d’humidité. La dessiccation d’un sol conduit à une nette diminution du nombre total de bactéries localisées dans la partie extérieure par rapport à celle intérieure (Hattori, 1988). La microporosité à l’intérieur de micro-agrégats stables est préférentiellement colonisée, ce qui suggère une combinaison de conditions favorables pour la vie bactérienne dans ces environnements. Les micro-agrégats sont les habitats les plus favorables pour les bactéries dans la plupart des types de sol. En effet, les régions intérieures des micro-agrégats peuvent constituer un habitat relativement
9
stable et isolé pour les microorganismes, caractérisé par une pression de prédations faible, une disponibilité d’eau relativement stable et de forts gradients de diffusion résultant en une faible disponibilité de nutriments et d’oxygène (Hattori, 1988 ; Ranjard, 1997). Ces micro-agrégats protègent donc les bactéries qui les colonisent de la dessiccation et d’autres substances solubles toxiques exogènes. Il est aussi probable que les microorganismes habitant l’intérieur des micro-agrégats contribuent peu à l’ensemble des activités microbiennes mesurables. Ces microorganismes peuvent cependant jouer un rôle clé dans le fonctionnement de l’écosystème dans des environnements anoxiques et en aidant à la formation et à la stabilisation des agrégats (Mummey, 2004). Il est également probable que, à cause de leur isolement, les organismes occupant ces microhabitats soient beaucoup moins disponibles pour leur isolement in vitro, leur extraction ou celle de leur ADN que les microorganismes colonisant la surface des agrégats (Hattori, 1988). Mummey et al. ont montré que ces cellules peuvent être classées comme des ultra microbactéries (un volume de 0.1μm3), qui utilisent préférentiellement des sucres comme substrats de croissance. Une petite taille et un métabolisme simple peuvent être un avantage pour la vie à l’intérieur des microagrégats. Hassink et al. ont montré une corrélation positive entre la taille bactérienne et le volume des pores (Hassink, 1993).
La localisation des bactéries à l’intérieur de la matrice sol est un facteur clé affectant leur survie, leur activité et leurs fonctions écologiques. Déterminer la structure de la communauté bactérienne dans l’espace qui concerne la structure du sol est d’abord fondamental pour élucider les fonctions écologiques. Définir les populations associées avec ces niches est important pour comprendre comment des perturbations influencent la diversité microbienne des sols, leur niveau d’activité et les fonctions susceptibles d’être réalisées.
2.1. Facteurs influençant la structure des communautés bactériennes telluriques
Beaucoup de paramètres biotiques et abiotiques interviennent pour modeler la structure des communautés bactériennes des sols tant en termes de diversité que d’effectifs (Hassink, 1993 ; Borneman et al., 1996 ; Borneman et Triplett, 1997). La dynamique bactérienne est aussi très importante, l’écosystème sol étant soumis en permanence à des perturbations de tous ordres qui vont avoir de profondes répercussions sur la structure des communautés. Il n’est pas encore possible aujourd’hui de prendre en compte tous les paramètres influant sur la composition de la microflore tellurique et surtout leurs interactions qui confèrent une myriade
10
de conditions différentes (Tarlera et al., 2008). Un certain nombre d’études ont toutefois permis d’identifier les facteurs présentant une influence marquée sur la structure des communautés bactériennes.
2.1.1. Facteurs abiotiques
Les structures des communautés bactériennes sont variables et dépendent de nombreux facteurs (Noguez, 2005). En effet, nous savons que la structure des communautés bactériennes ainsi que sa composition peuvent varier considérablement suivant l’espace (Martiny et al., 2006 ; Ramette et Tiedje, 2007). Plusieurs études suggèrent même que les caractéristiques des sols (Girvan et al., 2003 ; Singh, 2006), qui conditionnent la distribution spatiale (Nunan et al., 2005 ; Ritz, 2004), sont les facteurs les plus importants dans le modelage de la structure des communautés bactériennes. Par exemple le pH influence fortement la composition des Acidobacteria et des Actinobacteria. Le pH est l’un des forts prédicateurs de la composition des communautés bactériennes et de la diversité, mais suivant le type de sol d’autres facteurs peuvent agir sur la structure des communautés comme la disponibilité des nutriments, la solubilité des métaux (Muller et al., 2002), le contenu en carbone (Asuming-Brempong, 2008) et en azote (Fierer et al., 2007), l’humidité des sols, la salinité (Rajendhran et Gunasekaran, 2008), les variations climatiques (Lauber, 2009).
Au cours du temps, le sol subit des changements, qui augmentent la complexité globale de l'environnement sol (Nunan et al., 2001 ; Zhou et al., 2002). Tarlera et al. (2008) ont montré que le développement de l’écosystème sol favorisait le développement de communautés bactériennes distinctes. Les changements d’utilisation des terres, particulièrement la conversion des forêts en pâturage ou en champs cultivés, est un événement fréquent et un facteur affectant la biodiversité et le fonctionnement des écosystèmes terrestres (Sala et al., 2000). Les effets de l’utilisation des terres sur l’altération des propriétés physiques et chimiques des sols ont été beaucoup étudiés. En effet, des changements d’utilisation des terres (au niveau de la composition des espèces de plantes et des pratiques de gestion utilisées) peuvent avoir des impacts significatifs et de long terme sur le carbone du sol, le contenu en nutriments, la texture du sol et le pH (Murty, 2002). La variabilité des facteurs édaphiques à travers différentes utilisations des terres peut avoir un effet significatif sur la structure des communautés bactériennes (Lauber et al., 2008).
Les communautés sont dominées par 5 groupes majeurs Acidobacteria, Actinobacteria,
11
les communautés bactériennes dépend de sa durée et de sa spécificité. Après une perturbation transitoire, le système peut retrouver son état, tandis qu’une perturbation permanente résulte en un nouvel état altéré (Rykiel, 1985). Les perturbations avec un mode spécifique d’action altèrent seulement quelques groupes d’organismes (exemple : le groupe des Proteobacteria réagit de manière sensible au changement des niveaux de carbone dans le sol (Asuming-Brempong, 2008)) tandis que celles qui agissent non spécifiquement affectent un grand nombre de bactéries (Muller et al., 2002).
2.1.2. Facteurs biotiques
Pour comprendre les changements de structure des communautés bactériennes il faut prendre en compte différents paramètres incluant le groupe taxonomique en question (Lauber
et al., 2008), le niveau de résolution phylogénétique avec lequel sont étudiées les
communautés (exemple : Pseudomonas) (Cho et Tiedje, 2000). Il a été montré que les plantes peuvent avoir un impact identique ou même supérieur à celui des facteurs abiotiques, tel que le climat, dans le contrôle des fonctions des écosystèmes terrestres mais aussi au niveau de la structure des communautés bactériennes du sol (Verville et al., 1998). La présence de plantes résulte en une augmentation significative de l’humidité du sol, de la biomasse et de la respiration microbienne (Singh et al., 2009). La présence de plantes joue sur la croissance des communautés bactériennes des sols et influe aussi sur les propriétés abiotiques du sol. La présence de plantes provoque des changements profonds au sein de la communauté bactérienne du sol. Il a été observé que beaucoup de plantes sélectionnent des groupes de micro-organismes spécifiques via l’exsudation de composés dans la rhizosphère, qui représente une association positive où les plantes fournissent le carbone pour la croissance microbienne et les microorganismes en retour fournissent des éléments majeurs tels que l’azote et le phosphore, ainsi qu’une protection contre les attaques des pathogènes et parasites (Singh et al., 2004).
Des changements significatifs des structures des communautés bactériennes dans le sol ont été montrés lors de changements du couvert végétal. Ces changements induits par la végétation ont été observés aussi bien au niveau des groupes universels bactériens (Grayston
et al., 1998 ; Nusslein et Tiedje, 1999 ; Tiedje et al., 1999) qu’au niveau des groupes
fonctionnels (Singh et al., 2007).
Bien que les communautés bactériennes et fongiques aient été largement étudiées indépendamment l’une de l’autre dans de nombreux habitats, quelques études ont examiné
12
simultanément ces deux groupes de microorganismes (Costa et al., 2006 ; Singh et al., 2007), sans toutefois tenter de comprendre les interactions entre ces deux communautés qui demeurent très difficiles à prendre en compte, bactéries et champignons vivant en étroite relation dans le sol. Les interactions champignons bactéries sont très certainement de tous types, positive négative ou neutre. Singh et al (2009) ont observé une forte corrélation entre les communautés bactériennes et fongiques indépendamment des facteurs abiotiques du sol (humidité, C et N) à l’échelle du champ (Singh et al., 2009). Des résultats précédents avaient déjà montré qu’un champignon Glomus mossae (Murty et al., 2002) avait un impact direct sur la structure de la communauté bactérienne au niveau des racines de plantes (Artursson et
al., 2005). Singh et al. (2008) avaient montré aussi que la communauté fongique influence la
structure de la communauté bactérienne au niveau du rhizoplan des graminées.
3. Caractérisation des bactéries telluriques
3.1. Approche phénotypique, le fondement de la microbiologie du sol
Les méthodes phénotypiques comprennent toutes les techniques ne faisant pas appel aux acides nucléiques et reposants sur la détermination de caractéristiques morphologiques, physiologiques et biochimiques des bactéries via des techniques standardisées. Les tests sont basés soit sur les critères classiques ou traditionnels utilisés dans les schémas d’identification pratiqués dans la plupart des laboratoires de microbiologie, soit sur des tests spécialisés réservés à certains laboratoires (Denis et al., 2007). L’aspect des colonies observées sur la boîte de culture permet dans certains cas d’orienter l’identification vers un groupe de bactéries. La taille, la forme, la couleur (opaque, translucide, pigmentée…), l’état de la surface (lisse ou rugueuse), l’odeur éventuelle ainsi que la présence et le type d’hémolyse sur gélose au sang constituent des critères orientatifs d’identification. L’examen microscopique fournit les renseignements concernant la morphologie des bactéries (cocci, bacilles, spiralées, à bords parallèles ou non, à extrémités arrondies ou effilées …) ainsi que leur taille. Un examen à l’état frais entre lame et lamelle indiquera la présence et le type de mobilité. La coloration de Gram ou de Ziehl-Neelsen permettra de les classer en bactéries à Gram positif ou à Gram négatif ou acido alcoolorésistantes ainsi que d’observer des arrangements particuliers (diplocoques, palissades, chaînettes, cordes…), la présence ou non de flagelles, et la localisation d’une éventuelle endospore.
13
3.2. Approche moléculaire
Les techniques de biologie moléculaire ont bouleversé l'identification des bactéries, et ont mis en lumière les insuffisances et les erreurs d'identifications phénotypiques qui étaient jusque là seules disponibles (Ronsin, 2005 ; Denis et al., 2007).
L'identification moléculaire des bactéries repose essentiellement sur l'analyse de la séquence du gène ARNr 16S. La séquence obtenue au laboratoire est comparée via le réseau internet, à l'aide de logiciel spécialisé avec les banques électroniques de séquences comme " Nucleotide " du portail NCBI (appelée GenBank), ou EMBL en Europe.
L'identification bactérienne va reposer sur le pourcentage de similarité des séquences. Nous admettons généralement, qu'une similarité de 99% est identifiante au niveau de l'espèce. Une similarité de 97% identifiante au niveau du genre et qu'en dessous de 97%, il y a une possibilité de nouvelle espèce non décrite dans les banques de données. Par ailleurs, certains logiciels permettent de positionner la séquence obtenue au laboratoire parmi l'ensemble des séquences ARNr 16S disponibles sous forme d'arbres phylogénétiques (Sanger et al., 1977 ; Thomas, 1980 ; Smith et al., 1986 ; Swerdlow et al., 1991 ; Zhang et
al., 2003). Cette possibilité est particulièrement intéressante lorsque nous déterminons au
laboratoire une séquence ARNr 16S de similarité inférieure à 97%; cela permet alors d'avoir une idée du groupe bactérien dans lequel se situe la bactérie nouvellement identifiée (Staden, 1979).
L'identification moléculaire des bactéries est utile dans plusieurs cas de figure : elle est utile pour les bactéries fastidieuses de croissance lente, parmi lesquelles se trouvent évidemment les bactéries intracellulaires, qui sont souvent des bactéries d'identification difficile exprimant peu de caractères phénotypiques, mais également le cas de certains genres bactériens cultivant en milieu axénique comme les mycobactéries. Elle est également utile lorsque l'identification phénotypique n'est pas performante (cas des Acinetobacter, exprimant très peu de caractères phénotypiques) ou dans le cas ou il existe une discordance entre cette identification phénotypique et la sensibilité aux antibiotiques par exemple. Elle est enfin utile pour toutes les souches bactériennes qui sont isolées dans une circonstance inhabituelle pour laquelle il convient absolument de confirmer l'identification.
14
3.2.1. Ere protéomique et l'identification bactérienne 3.2.1.1. Spectrométrie de masse
a. Historique : Apparue au début du 20ème siècle, la spectrométrie de masse a apporté des éléments clés dans la compréhension du noyau atomique. Elle est issue de la découverte par Euge Goldstein en 1886, des rayons canaux dus aux ions positifs et de leur analyse par un champ magnétique, par Wilhelm Wien en 1898.
En 1912, Joseph John Thomson obtient des spectres de masses de plusieurs composés gazeux : N2, O2, CO, CO2, etc. Il met en évidence les ions négatifs et les ions multichargés. L'année suivante, il découvre les isotopes A (nombre de masse) égale à 20 et A égale à 22 du néon (Hoffmann et al., 1994). Les innovations qui suivent tentent principalement d'augmenter la sensibilité du dispositif.
En 1918, Dempster construit un spectromètre à focalisation en direction. L'année suivante, Francis William Aston introduit le tri des ions en fonction de leur vitesse, ce qui lui permet de déterminer les abondances isotopiques du néon et de mettre en évidence les écarts entre les masses atomiques réelles et les nombres entiers.
En 1932, Bainbridge, en associant au dispositif un filtre de vitesse de Wien, vérifie expérimentalement l'équivalence entre masse et énergie. Il propose alors la double focalisation en direction et en vitesse, principe qui sera mis en œuvre par Jordan, Mattauch et Herzog.
En 1936, Penning propose de combiner un champ électrique et un champ magnétique pour piéger les ions dans un très faible volume : c'est le piège de Penning.
En 1940, Nier perfectionne la source d'ions à impact électronique. Ensuite, en 1948, Cameron invente le spectromètre de masse à temps de vol. Puis, entre 1953 et 1960, Paul et Steinwedel mettent au point l'analyseur quadripolaire, puis le piège ionique quadripolaire qui diffère de celui de Penning par l'absence de champ magnétique. Le piège quadripolaire aura un tel retentissement en physique fondamentale que le prix Nobel sera attribué en 1989 à Paul et à Dehmelt.
Pendant les années 1960, Smith met au point le spectromètre de masse à radiofréquence.
15
Pour ce qui est des applications, depuis 1950, les techniques d'ionisation se diversifient de façon à s'adapter à la diversité des échantillons à analyser, particulièrement pour la chimie organique. Cette technique d'analyse bénéficie alors du couplage avec un chromatographe en phase gazeuse puis liquide. Les progrès de l'informatique ont permis l'automatisation des réglages de l'appareil ainsi que la mise en œuvre des analyseurs à quadripôle et des pièges ioniques dans de nombreuses applications de la spectrométrie de masse (Hoffmann et al., 1994 ; Constantin, 1996).
b. Principe de fonctionnement : La spectrométrie de masse est une technique
d'analyse de la matière en fonction de la masse de ses constituants : particules subatomiques, atomes, molécules, macromolécules biologiques ou non, agrégats, etc. Elle offre trois fonctions principales : la résolution des constituants atomiques ou moléculaires, la mesure de leur abondance relative et la mesure précise de leurs masses atomiques ou moléculaires. Contrairement à ce qui se passe à l'échelle macroscopique, où la gravitation sert à déterminer la masse des objets, à l'échelle atomique, c'est la masse inertielle qui est déterminée à travers un processus dynamique (Andrade et al., 2008). La matière à analyser est injectée, à l'aide d'un système d'introduction, dans le vide poussé de l'appareil où la source d'ions la transforme en particules électriquement chargées "les ions". Ces derniers sont analysés en masse, à l'aide d'un système analyseur qui met en jeu des combinaisons de champs électromagnétiques. Les ions transmis par le dispositif sont détectés, comptés, voire identifiés, à l'aide d'un détecteur. Un ordinateur pilote l'appareil, effectue l'acquisition des données réduites sous forme de spectres de masse qu'il permet d'analyser. Les performances d'un spectromètre de masse se caractérisent par sa limite en masse, la masse la plus élevée qu'il est capable d'analyser, son pouvoir de résolution, son aptitude à séparer des ions de masses voisines et sa sensibilité. La plus petite quantité de matière qu'il peut détecter (< 10-15 g) (De Lara et al., 2008). Lorsqu'il s'agit de mesurer la masse atomique ou moléculaire, il est caractérisé par sa justesse.
3.2.1.2. MALDI-TOF MS en microbiologie : une histoire récente
Depuis sa mise au point au début des années 1990, la spectrométrie de masse de type MALDI-TOF (Matrix Assisted Laser Desorption/Ionization Time-Of-Flight) s’est développée dans différents domaines de la recherche fondamentale (Hemmersbach, 2008 ; Toyo'oka, 2008). Grâce à une ionisation douce, il est devenu possible de détecter de larges fragments et/ou complexes protéiques, ouvrant ainsi de nouvelles perspectives d’étude impossible jusqu’alors (Karas et Hillenkamp, 1988). Rapidement, les microbiologistes se
16
sont intéressés à cette technique et les premières identifications bactériennes à partir de cellules intactes, sans utilisation de protocoles complexes de préparation des échantillons, ont été obtenus avec succès (Claydon et al., 1996 ; Krishnamurthy et Ross, 1996). Depuis ces premiers travaux, l’ensemble des publications concernant l’identification des bactéries, permet d’affirmer que les empreintes spectrales des principales espèces cultivables identifiées en routine ont été obtenues. Seng et al. ont récemment publié une revue exhaustive sur l’ensemble des bactéries identifiables par cette approches (Seng et al., 2010).
L’identification par cette technique repose sur le fait que le spectre obtenu varie d’un genre à l’autre, d’une espèce à l’autre voire même d’une sous-espèce à l’autre, permettant ainsi de discriminer les bactéries entre elles. La réalisation des banques de données doit donc sélectionner parmi l’ensemble des pics, ceux qui sont spécifiques de la souche de référence étudiée. En effet, parmi les peptides détectés, il a été démontré que certains pics sont spécifiques du genre et de l’espèce et qu’ils sont fréquemment retrouvés à condition que les principaux critères culturaux soient maîtrisés et comparables d’une acquisition à l’autre. Malgré l’efficacité de cette technique, le développement et la généralisation de cette technique au sein des laboratoires n’ont pas connu un succès immédiat.
En effet, identifier un nombre restreint d’espèces et si possible le plus éloignées taxonomiquement parlant comme c’est le cas dans la plupart des premières études réalisées est une chose, une utilisation en routine nécessitant des identifications d’espèce souvent proches les unes des autres en est une autre.
Le développement des banques de données permettant l’identification en routine repose donc sur les différents points évoqués. Plusieurs stratégies existent actuellement, mais elles ont toutes pour objectifs de sélectionner les pics spécifiques d’une espèce donnée. Emonet et
al. ont récemment publié une revue détaillant les principes des trois principales banques de
données (MALDI Biotyper Bruker, SARAMIS Anagnostec BioMérieux, ANDROMAS) permettant l’identification par MALDI-TOF-MS (Emonet et al., 2010).
L’identification de la bactérie étudiée repose sur la comparaison du spectre obtenu à ceux de références contenus dans la base de données. Comme pour les analyses des séquences nucléotidiques par BLAST ou FASTA, la plus forte concordance (« matches ») est retenue et les résultats sont rendus avec un coefficient de similarité.
Les progrès réalisés dans le domaine de la génomique avec l’augmentation du nombre de génomes séquencés disponibles ont permis d’identifier certains pics au sein des empreintes
17
spectrales. Il est maintenant établi que la grande majorité des pics détectés entre 2 et 20 kDa correspond aux protéines ribosomales et à des protéines des gènes de ménage (« house keeping » gene) ce qui explique en partie le caractère spécifique et constant des spectres par espèce malgré les variations rencontrées lors de différentes acquisitions. Ces données laissent envisager le développement de stratégies permettant, au même titre que le MLST, de typer les bactéries et de réaliser des comparaisons phylogénétiques. Parmi les autres protéines identifiées, on retrouve des protéines de choc thermique (cold shock proteins), des DNA binding proteins ou encore des RNA chaperones (Ryzhov et Fenselau, 2001 ; Jones et al., 2003 ; Dieckmann et al., 2008).
Afin d’obtenir des identifications fiables, il est important de maîtriser les différents paramètres expérimentaux qui influencent la qualité des spectres. Les milieux de culture, la concentration en NaCl, la nature des tampons utilisés influencent la qualité des spectres. Une fois la majorité de ces paramètres standardisés, la reproductibilité et la précision des identifications sont parfaitement fiables au sein d’un laboratoire et d’un laboratoire à l’autre (Valentine et al., 2005 ; Wunschel et al., 2005). Ces données soulignent l’importance lors de la réalisation des bases de données, d’avoir à disposition un large panel d’espèces différentes représentatif des principales espèces rencontrées en routine avec si possible le plus grand nombre de souches d’une même espèce. Il est ensuite nécessaire de faire varier pour chaque souche les conditions expérimentales afin de prendre en compte les variations inhérentes aux conditions expérimentales.
Le schéma général de l’identification par MALDI-TOF MS est illustré dans la figure 2 et un protocole détaillé des différentes étapes peut être retrouvé dans la publication de Freiwald et Sauer (Freiwald et Sauer, 2009). Les bactéries à identifier sont obtenus à partir des colonies poussant sur des milieux de culture solides ou à partir de cultures en milieu liquide. Cette approche simplifie au maximum la préparation de l’échantillon à analyser, on parle alors d’acquisition sur bactéries intactes. Cette méthode de préparation de l’échantillon est simple et très rapide pouvant être réalisé par du personnel non spécialisé en spectrométrie de masse. Les bactéries sont déposées directement sur une plaque métallique support (figure 3). Aucune étape complexe de purification de l’échantillon n’est nécessaire.
18
Figure 3 : Schéma d’identification par MALDI-TOF à partir des colonies.
L’obtention d’un spectre de qualité dépend essentiellement de la qualité du dépôt effectué. Peu de matériel est nécessaire pour obtenir une empreinte spectrale de qualité et bien souvent les difficultés d’identification rencontrées sont liées à de mauvais dépôts avec excès de matériel déposé. Une fois l’échantillon séché sur la plaque cible, il est recouvert par la matrice dont la nature est adaptée au type d’analyse que l’on souhaite réaliser. Le rôle de la matrice est d’absorber l’énergie provenant du laser ce qui provoque la vaporisation de l’échantillon avec formation d’ions. Ces ions de masse et de charges différentes sont soumis à un champ électrique. Ils « volent » ensuite jusqu’à un détecteur situé à l’extrémité du tube de vol et la distance parcourue en un temps donné (Time-Of-Flight) est fonction du rapport de leur masse sur la charge (m/z). Les informations essentielles utilisées pour l’identification sont donc contenues dans une liste de pics contenant les rapports m/z et les intensités relatives de chaque pic, l’ensemble de ces données caractérisant l’empreinte spectrale de l’échantillon. Les matrices les plus courantes pour ce type d’analyse sont l’acide 2,5-dihydroxybenzoïque (acide gentisique ou DHB), l’acide trans-3,5-diméthyloxy-4-hydroxycinnamique (acide sinapinique ou SA) et l’acide α-cyano-4-hydroxy-cinnamique (α-CHCA). Elles permettent entre autre l’analyse de substrats peptidiques, glycopeptidiques et lipidiques. Actuellement, le dépôt en « goutte épaisse » utilisant l’ α-CHCA sur l’échantillon sec est largement utilisé pour les identifications de routine. En effet, la cristallisation obtenue est homogène contrairement à la DHB, permettant une utilisation automatique des acquisitions par le spectromètre avec une excellente qualité de spectres.
D’autres techniques de dépôt existent : les préparations en « couches minces » pour lesquelles l’échantillon est déposé sur une couche de matrice préalablement déposée sur la plaque cible pour former de larges films minces polycristallins, les préparations en "sandwich" où l’échantillon est déposé su r un film de matrice avant d’être lui-même
19
recouvert par une dernière couche de matrice. Après évaporation des solvants, la matrice co-cristallise avec l’échantillon et l’ensemble peut être analysé.
Des protocoles plus complexes ont été proposés afin d’augmenter la qualité des empreintes spectrales mais dans la grande majorité des cas, ils ne sont pas nécessaires pour une utilisation de routine (Liu et al., 2007). En revanche, pour certains micro-organismes comme les levures ou les mycobactéries, certains auteurs préconisent de réaliser auparavant une étape d’extraction (Amiri-Eliasi et Fenselau, 2001).
4. Etude de la diversité bactérienne tellurique
Le sol est un environnement hétérogène complexe qui contient une grande variété de communautés et d’espèces bactériennes (Daniel, 2005).
De ce fait, accéder à la diversité bactérienne de manière exhaustive constitue un des défis majeurs de ces dernières décennies en microbiologie du sol et les techniques qui y sont dédiées n’ont cesse d’évoluer (Fierer et Lennon, 2011).
Avant le développement des techniques moléculaires, l’identification et la classification taxonomique de bactéries nécessitaient leur isolement, leur mise en culture et leur caractérisation sur la base de critères morphologiques, physiologiques, métaboliques, biochimiques et écologiques. Ces critères de classification ont rapidement trouvé leurs limites du fait de la diversité phénotypique et physiologique des bactéries mais aussi de par la simplicité de la classification engendrée.
Palliant ce problème, le développement d’approches moléculaires a révolutionné le monde de la microbiologie du sol en permettant d’affiner la classification bactérienne et en augmentant de manière significative le nombre d’espèces considérées, par rapport aux méthodes basées sur les seules bactéries cultivables.
4.1. Diversité bactérienne dans le sol
Les bactéries sont de loin les plus abondants microorganismes du sol à la fois en termes de biomasse et de diversité taxonomique (Buckley et Schmidt, 2002). En 1990, par observation microscopique en utilisant l’acridine orange (fluorochrome qui se fixe sur les acides nucléiques), Torsvik et al., (1990) ont estimé qu’un gramme de sol forestier naturel contient près de 1,5.1010 bactéries. Plus tard, Torsvik et al., (1998) ont extrapolé des données
20
issues d’hybridation ADN-ADN et estimé qu’un gramme de sol forestier se compose de près de 6000 génomes bactériens. Dans cette même étude, les auteurs soulignent la limite des approches culturales qui, dans ce même gramme de sol révèlent la présence de 35 génomes bactériens (chiffre établi à partir de la taille du génome d’E. coli : 4,1.106 pb).
Plus récemment, l’étude de métagénomes de sols contrastés en termes de caractéristiques physicochimiques et géographiques a permis d’estimer entre 2000 et 10000 le nombre d’espèces bactériennes (et donc de génomes) par gramme de sol (Roesch et al., 2007).
D’une manière générale, nous considérons aujourd’hui qu’un gramme de sol héberge, en fonction de ses caractéristiques physicochimiques, plusieurs milliers d’espèces et que l’abondance de ces espèces peut varier de 108
à 1011 cellules par gramme de sol (Curtis et al., 2002, Roesch et al., 2007).
4.2. Paramètres influençant la diversité bactérienne dans le sol
La diversité bactérienne est exprimée par le nombre d’espèces différentes ainsi que par leur abondance relative dans la microflore du sol (Kennedy et Smith, 1995).
La diversité des espèces dans un écosystème local est considérée comme la diversité alpha. La variation de la diversité alpha des écosystèmes dans un même environnement est considérée comme la diversité bêta, et, lorsqu'elle est mesurable, la diversité gamma représente la richesse en espèces à l'échelle régionale et mondiale. La diversité gamma est sensible principalement à des phénomènes qui ont un impact environnemental à l'échelle mondiale (par exemple des changements majeurs sur le climat), par opposition aux impacts a l’échelle locale. La diversité bactérienne peut donc s’observer a différentes échelles, révélant d’une part la complexité des communautés bactériennes dans le sol mais aussi la conservation de certaines caractéristiques.
Le pH apparait ainsi comme un paramètre qui influence de manière majeure la diversité des communautés bactériennes dans les sols. L’analyse de 88 échantillons de sol a permis de montrer une corrélation entre la diversité des bactéries et le pH de ces sols (Figure 4) (Lauber
et al., 2009). L’analyse de la structure des communautés bactériennes montre que
l’abondance relative de certains phylums bactériens comme par exemple les Actinobacteria, les Bacteroidetes et les Acidobacteria, est corrélée au pH.
21
Figure 4 : Relation entre le pH des sols et la diversité bactérienne, en utilisant la diversité
phylogénétique (A) et le nombre de phylotypes (B) défini à 97% de similarité. La courbe pleine représente la courbe de tendance. Les indices de diversité ont été calculés en utilisant 1200 séquences par sol (Lauber et al., 2009).
Cette corrélation entre pH et diversité s’explique par le fait que le pH est directement corrélé à plusieurs paramètres du sol comme la disponibilité des nutriments, la solubilité des cations métalliques, la teneur en carbone organique, l’humidité et la salinité. Une seconde explication à cette corrélation est que le pH impose directement une contrainte physiologique sur les bactéries du sol.
22
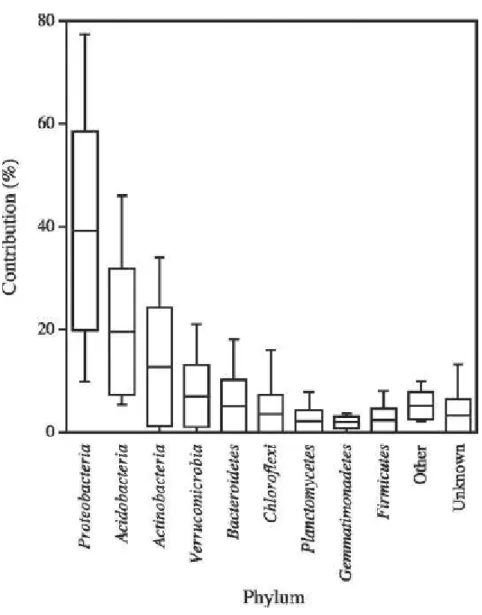
Outre le pH, la structure du sol, la teneur en matière organique et la végétation influencent la diversité et l’abondance des bactéries. L’étude metagénomique des sols montre que quelle que soit la méthode d'analyse, certains phylums bactériens prédominent. C’est en tout cas le constat fait par Janssen et son équipe en 2006, qui se sont intéressés à l’analyse de différentes banques de séquences d’ADNr 16S provenant de sols différents (prairies, forêts, sols arides, agricoles…). Le résultat de cette étude montre que malgré les différences entre sols, certains phylums sont systématiquement dominants (Figure 5).
Figure 5 : Contribution des séquences d’ARNr 16S bactériens aux phylums les mieux
représentés dans le sol. Les données ont été compilées à partir de 21 banques (2920 clones) de clones d’ARNr 16S obtenus par amplification a partir d’ADN du sol. La barre horizontale représente le pourcentage moyen de séquences attribuées au phylum, le rectangle représente l’ecart type et les barres verticales représentent les valeurs maximales et minimales (North et