HAL Id: tel-01382365
https://tel.archives-ouvertes.fr/tel-01382365
Submitted on 17 Oct 2016
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
étude protéomique
Severine Clavier
To cite this version:
Severine Clavier. Effet moléculaire du peptide vecteur (R/W)9 sur le phénotype de cellules modèles du sarcome d’Ewing : étude protéomique. Chimie analytique. Université Pierre et Marie Curie - Paris VI, 2014. Français. �NNT : 2014PA066308�. �tel-01382365�
- 1 -
THESE DE DOCTORAT DE
L’UNIVER“ITE PIERRE ET MARIE CURIE
Spécialité Chimie Analytique
Ecole Doctorale Chimie Moléculaire
Présentée par
Séverine CLAVIER
Pour obtenir le grade de
DOCTEUR de L’UNIVER“ITE PIERRE ET MARIE CURIE
Effet moléculaire du peptide vecteur (R/W)
9sur le phénotype de
ellules od les du sa o e d’E i g : Etude protéomique
Soutenance le 16 octobre 2014
Devant le jury composé de :
Dr. Joëlle VINH
, SMBP, ESPCI RapportricePr. Guillaume VAN DER REST
, LCP, Université Paris Sud RapporteurPr. Tula SAISON-BEHMOARAS
, Régulation et dynamique des génomes, MNHN ExaminatricePr. Richard COLE
, IPCM-LCSOB, UPMC ExaminateurDr. Jacques GHYSDAEL
, Signalisation, Neurobiologie et Cancer, Institut Curie ExaminateurDr. Emmanuelle SACHON
, LBM, UPMC Directrice de thèseDr. Gérard BOLBACH,
LBM,
UPMC Directeur de thèse- 3 -
Remerciements
Pour commencer je souhaite vivement remercier Joëlle Vinh et Guillaume van der Rest qui o t a ept d’ t e les appo teu s de e t a ail de th se, ainsi que Richard Cole, Tula Saison-Behmoaras et Jacques Ghysdaël pour leur participation à mon jury de soutenance en tant
u’e a i ateu s.
Merci à Laurence Sabatier, qui à Strasbourg lors des enseignements de la filière chimie analytique puis lo s de o stage M , ’a fait d ou i le passio a t do ai e de la ioa al se.
Au cours de ces t ois elles a es, o euses so t les pe so es a e les uelles j’ai eu la ha e de travailler au sein de la fac de Jussieu et que je souhaite chaleureusement remercier.
Tout d’a o d, Ma u et G a d, es di e teu s de th se, u o-encadrement qui a idéalement fonctionné avec des avis, des conseils toujours très complémentaires. Un grand merci à Gérard de ’a oi , d s u’u e o asio se p se tait, fait fi ie de so e p ie e e spe t o t ie de asse. J’ai eau oup app i os o euses dis ussions scientifiques ou non qui me permettait de p e d e du e ul, de fai e olue o poi t de ue pou la alisatio d’e p ie es et aussi pou des hoi de ie. Me i i fi i e t à Ma u, a o pli e au ou s de es t ois a es. C’ tait formidable de t a aille a e toi et e ’ai pas sa s u pi e e t au œu ue je ’app te à tou e ette page. Tu t’es toujou s o t e t s dispo i le et e toutes i o sta es p te à ’aide à faire face aux difficultés rencontrées dans la réalisation de mon projet de thèse. Merci pour ton dynamisme et ton optimisme à toute épreuve ! Je suis sûre que cette amitié forte que nous avons développée au cours de ces trois belles années persistera très très longtemps.
Merci Sandrine pour ton accueil au sein du LBM2, tes précieux conseils scientifiques et ton implication régulière dans mon projet de thèse et cela malgré tes nombreuses responsabilités. Merci pou l’ e gie ue tu d pe ses au uotidie pou ai te i des o es o ditio s de t a ail et la très agréable ambiance du laboratoire.
Merci Gilles pour ton incroyable gentillesse et bienveillance ! C’ tait supe de t’a oi su la plateforme. Tu es une personne sur laquelle on peut compter et je te souhaite le meilleur pour le futur.
Merci Claire, Dominique, Karine et Nicolas pour votre accueil toujours chaleureux dans la pièce qui fut jus u’à uel ues jou s de cela votre bureau au Bât F. Pendant les migrations des gels ou les i u atio s su illes, et … je e o pte pas le o e de fois où je suis e ue « squatter » chez ous et j’ai app i ue e soit auta t d’o asio s de dis ute et d’ ha ge su os p ojets de e he he ou su le tie d’e seig a t-chercheur. Merci Sophie pour les coups de pouce, les infos utiles…et le ho olat ;-). Merci Jésus pour ta disponibilité et les discussions scientifiques.
Me i à l’ uipe de o deu i e u eau d’a ueil lo s de es a ips au ât F et du café de l’ap e . A Fa ie e pou ses o seils d’e pe te e i te alisation de CPPs et à Françoise pour toutes les techniques de bio hi ie u’elle ’a t a s ise sa s ou lie l’app e tissage de la culture cellulaire !!
- 4 -
thèse : Lu e, Ast id, Ch i e et Di it a. C’ tait g ial de partager des verres avec vous à l’I ita le et de pou oi o ti ue à ha ge a e ous pa ail ap s ot e d pa t du la o Est-ce que vous auriez accès à la publi X par hasard ?? ;- . Me tio sp iale pou Lu e ui ’a, en quelques sortes, ouvert la porte du LBM (Viens donc faire un tour chez nous si tu veux faire de la bioanalyse ! et ui a o ti u à ’off i so aide et ses o seils depuis les Pa s-Bas. Je suis contente que tu sois parvenue à revenir à Jussieu !
Un grand merci aussi à la team de gars ayant succédé à cette team de filles : Tom, Pierre et Mehdi. C’ tait ai e t houette de se oise au ât F ou de pa tage des o e ts de a ip ! Merci Tom d’a oi toujou s eill à e p e i , lo s ue, « exilée » au bât A, je risquais de manquer le départ vers la cantine, les bords de seine ou la rue Linnée. Et merci aux thésards LBM3, Shahid (« Beers tonight Dude ? » , les A aïs, Lu ie, Lu ille ai si u’au do to a ts et postdo to a ts Besse , Jea , Adrian, Ba ie, Que ti de l’IPCM-CSOB voisin pour les bons moments partagés !
Merci à nos voisins du LBM3 et enseignants LC351, Olivier, Ludovic, Emeric et Lucie.
J’ai o e t app i l’a ia e du ème étage : M e a e u e d o diso s…dat e o peut
travailler dans la bonne humeur ! Vive les productions cinématographiques du LBM2 et en particulier la « Danse de la salle de culture ».
Merci Rodrigue pour la synthèse et la purification de ce fameux peptide (R/W)9 sous ses différentes
versions, photoactivable, deutérée, de o figu atio D… C’ tait parfait et sans cela je ne serais pas allée bien loin !
Me i à Elia e, Nathalie, “t pha e et ai te a t Ka i e pou la gestio ad i ist ati e du LBM. J’ai apprécié votre disponibilité et votre efficacité.
Me i à tous es oll gues d’e seig e e ts et e particulier à ceux du module LC351 pour les coups de mains, les échanges de créneaux de cours. Merci à Laurent Gaillon pour la formation aux TPs et TDs d’HPLC et à Ri ha d Cole pou ses e pli atio s su e tai s e os de spe t o t ie de asse. Merci encore u e fois à Ma u ui ’a gale e t fou i u e aide p ieuse pou la p pa atio des enseignements !
Cot Bât A, je souhaite e e ie Ch istophe, Taha ai si ue les e es de l’ uipe BIOSIPE. Merci « tonton »Thie d’a oi t o tuteu de th se ! Merci Ali pour ta confiance lors des ateliers du NV601. Merci à Sonia et Mouna pour leur sympathie, les ateliers microbalance et speedvac auraient été nettement moins agréables sans vous !
Me i aussi au e es de l’IPCM, Jea -Philippe Goddard et Cyril Olli ie ai si u’à leu s tudia ts ui ’o t pe is d’utilise le s st e Ra o et.
Au-delà des personnes rencontrées à Jussieu, je souhaite remercier mes collaborateurs et soutiens extérieurs.
Xiu ia Du et Ada Ba te de l’U i e sit de Cha lotte e Ca oli e du No d ui ous o t d’a o d aid s pour les données de cross-li ki g hi i ue et ui ous o t e suite sui i pou la ise au poi t d’u logiciel pour le photocross-linking. Merci pour leur patience dans le débugage du logiciel et pour les sympathiques discussions Skype.
- 5 -
les d pa ages et les o seils. Je te suis e t e e t e o aissa te pou l’aide ue tu ’as apportée tout au long de ma thèse. Merci Jacques !
Me i eau oup au ioi fo ati ie s de l’I stitut Cu ie, Pat i k Poullet et Guillau e A as pou ’a oi pe is d’utilise M P oM“. Me i Pat i k d’a oi po du à es i o a les e ails et d’a oi toujou s t e esu e de gle les p o lèmes informatiques.
Me i à la houette uipe du “MBP de l’E“PCI et e pa ti ulie à es a is “hake et “ophie pou leurs dépannages Nano ! (Un bout de capillaire de 20µm à me prêter ? une idée pour le passeur d’ ha tillo s ??)
Me i à l’E ole Do to ale I i ui ’a att i u e o fi a e e t de th se. Me i e pa ti ulie à Ge ai pou so i estisse e t da s la ie de l’ED a e les jou es de e t e et la supe fo atio BioNoBio. Malheu euse e t, les elles hoses e du e t pas toujou s…
Je souhaite également remercier la Société Française de Spectrométrie de Masse (SFSM) qui a financé ma participation à deux congrès (à Orléans en 2012 et en Suisse en 2014) au cours de ma th se et ui ’a pe is de ’e gage da s u e elle a e tu e asso iati e ia le bureau du CJSM. Me i A a di e de ’a oi do l’e ie de ’i esti da s le u eau et de ’a oi o t e la oie et e i au e es a tifs de l’asso iatio faisa t pa tie du u eau ou o pou les supe s moments partagés lors des congrès ou des RCJSMs !!
Merci à Martine, ma prof de danse du 5ième et à ses danseuses. Ces cours géniaux vont tellement me a ue …
Fi ale e t last BUT ot least… je oud ais e e ie tous eu ui o t sui i et pa ti ip à ette aventure parisienne : mes fantastiques amis de lycées, Déborah, Maé, Kana, Steven, Jacqueline, Ramzi, Pauline et les supers pièces rapportées ;-) Sylvain et Matthieu, mais aussi Goulven et Kristen, Emilie et Loic, et puis aussi les supe s de l’ECPM, Ca ille, Ca o, “hake , “ophie, Pauli e, Ma o et leurs +1). Sans oublier Nadège, Thomas, Mathilde et Yoann. “i j’ai pass de si elles a es ’est aussi grâce à vous !!
Me i i fi i e t à es pa e ts ui ’o t toujou s soute ue, o fo t e, o seill e… d ag e ! Merci pour votre amour et merci à ma soeurette et mamie adorées ! Merci aussi à ma géniale belle famille
- 7 -
Publications :
Clavier S, Du X, Sagan S, Bolbach G, Sachon E.« An integrated cross-linking-MS approach to
investigate cell penetrating peptides interacting partners. EuPA Open Proteomics, Vol 3, 2014 June
Matheron L, Clavier S, Diebate O, Karoyan P, Bolbach G, Guianvarc'h D, Sachon E « Improving the
selectivity of the phosphoric acid B-elimination on a biotinylated phosphopeptide » J Am Soc Mass
Spectrom. 2012 Nov 23(11):1981-90
Christine C, Koubemba M, Shakir S, Clavier S, Ehret-Sabatier L, Saupe F, Orend G, Charbonnière LJ.
Synthesis of an activated phosphonated bifunctional chelate with potential for PET imaging and radiotherapy. Org Biomol Chem. 2012 Dec 14;10(46):9183-90
Communications :
Orales :
U de sta di g the molecular basis of (R/W)9 CPP effe t o tu o al EF ells phe otype
(SMAP 2014, Lyon,Congrès national).
Etudes des partenaires intracellulaires du peptide vecteur (R/W)9 par pontage covalent et
spectrométrie de masse Re o t es du Clu des Jeu es “pe t o t istes de Masse, Pau,
RCJSM 2013).
Chemical Cross-linking and Mass Spectrometry for the study of (R/W)9 Cell Penetrating
Peptide interactions with its partner proteins JF“M , O l a s, Co g s atio al
Posters :
Mole ula effe t of R/W9Cell Pe et ati g Peptide o E i g’s sa o a odel ells
phe otype : P oteo i study (IMSC 2014, Genève, Congrès international). Séverine Clavier,
Jacques Ghysdaël, Sandrine Sagan, Gérard Bolbach and Emmanuelle Sachon
Understanding (R/W)9 Cell Penetrating Peptide effect on tumoral cells: Association of
targeted and differential approaches Eupa , “t Malo, Co g s eu op e . Séverine
Clavier, Rodrigue Marquant, Xiuxia Du, Sandrine Sagan, Gérard Bolbach and Emmanuelle
- 9 - ABP actin binding protein
ACN Acétonitrile
ADN/ARN acide déoxyribonucléique/ acide ribonucléique AP-MS affinity purification-mass spectrometry
ATP/ADP Adenosine triphosphate/ Adenosine diphosphate BCA BiCinchoninic acid Assay
BN-PAGE Blue native polyacrylamide gel electrophoresis BSA Bovin serum albumin
CHCA α-Cyano-4-hydroxycinnamic acid CHO Chinese Hamster Ovary
CID collision induced dissociation
CPP Cell Penetrating Peptide ou peptide vecteur DAPI 4',6'-diamidino-2-phénylindole
DAVID Database for Annotation Vizualization and Integrated Discovery DDA Data Dependent Acquisition
DIA Data Independent Acquisition DMEM Dulbecco's Modified Eagle's Medium DTT Dithiothréitol
ECD Electron capture dissociation ESI electrospray ionisation ETD electron transfert dissociation
FA formic acid
FASP Filter aided sample preparation FBS Fœtal o i se u
FDR false discovery rate FITC fluorescein isothiocyanate
FT-ICR fourier transform-ion cyclotron resonance GAG Glycosaminoglycanes
GO Gene ontology
GTP Guanosine triphosphate HBSS Hank's Balanced Salt Solution HCl Hydrochloric acid
HDX hydrogen-deuterium exchange
HPLC High Performance Liquid chromatography HRP Horseradish peroxidase
IAA Iodoacetamide
IEF isoelectrofocalisation
ITC isothermal titration calorimetry LTQ Linear trap quadrupole
MALDI matrix assisted laser desorption/ionisation
MS mass spectrometry
NCS Newborn calf serum
- 10 - PBS Phosphate buffered saline
PSMs Peptide-to-spectrum matches PTM Post-translational modification RMN résonance magnétique nucléaire ROS Reactive oxygen species
SAINT Significance Analysis of Interactome
SDS-PAGE Sodium dodecyl sulfate- polyacrylamide gel electrophoresis SILAC Stable Isotope Labelling with Amino acids in Cell culture SPE Solid phase extraction
SPR surface plasmon resonance
STRING Search Tool for the Retrieval of Interacting Genes/Proteins SWATH Sequential Window Acquisition of all Theoritical spectra TFA trifluoroacetic acid
TOF Time of Flight
UV ultraviolet
- 11 -
Remerciements ___________________________________________________________ 3
Liste des publications et communications ______________________________________ 7
Liste des abbréviations et acronymes _________________________________________ 9
Liste des figures et tableaux ________________________________________________ 15
Préface ________________________________________________________________ 21
-Chapitre 1 : Des interactions protéine-
p ot i e au ph ot pe cellulai e, l’e e ple du
c tos uelette d’acti e ____________________________________________________ 23
-Au sujet des interactions protéine-protéine ___________________________________ - 24 - I.
1. Les diff e ts t pes d’i te a tio s p ot i e-protéine ___________________________________ - 24 - 2. Les do ai es d’i te a tio s des p ot i es ___________________________________________ 24 -3. Régulation et effets biologiques des interactions protéine-protéine _______________________ - 25 - 4. Les seau d’i te a tio s des p ot i es _____________________________________________ - 26 -
Le tos uelette d’a ti e : illust atio de la o ple it des seau d’i te a tio s ___ - 27 - II.
1. L’a ti e : fi he d’ide tit [19] ______________________________________________________ 27 -2. Pol isatio de l’a ti e _________________________________________________________ 28 -3. Les p ot i es de liaiso de l’a ti e et leu s ôles _______________________________________ - 29 - 4. Le rôle du cytosquelette dans la réponse de la cellule aux contraintes mécaniques externes ___ - 33 -
Cytosquelette et oncogenèse _____________________________________________ - 34 - III.
1. Généralités [72] _________________________________________________________________ - 34 - 2. “a o e d’E i g : Comment la protéine EWS-FLI1 agit-elle sur le cytosquelette ?____________ - 35 -
Chapitre 2
: M thodes pou l’ tude des i te actio s p ot i e-protéine : de la découverte de
ouveau pa te ai es à l’i t g atio da s les seau d’i te actio s ______________ 39
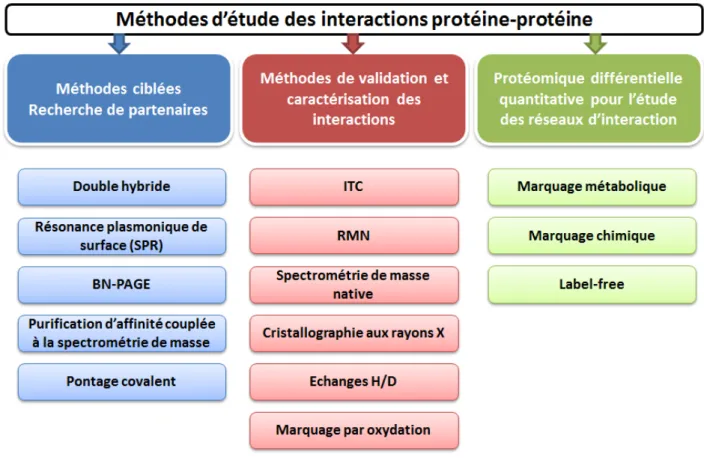
-M thodes i l es de d ou e te de pa te ai es d’i te a tio ____________________ - 41 - I.
1. Systèmes ellulai es d’e p essio de dou le h ide ___________________________________ 41 -2. Résonance plasmonique de surface (Surface plasmon resonance, SPR) _____________________ - 42 - 3. Blue Native PAGE ________________________________________________________________ - 45 - 4. M thodes de pu ifi atio d’affi it couplées à la spectrométrie de masse (APMS) ___________ 45 -5. Pontage o ale t ou C oss-li ki g oupl à la spe t o t ie de asse ___________________ 55 Méthodes de validation et de caractérisation __________________________________ - 59 -
II.
1. Calorimétrie de titrage isotherme (ITC) ______________________________________________ - 59 - 2. Résonance magnétique nucléaire (RMN) _____________________________________________ - 61 - 3. Cristallographie aux rayons X ______________________________________________________ - 63 - 4. Spectrométrie de masse native _____________________________________________________ - 63 - 5. L’ ha ge h d og e/deut iu HDX ______________________________________________ 65 -6. Le marquage par oxydation (OFP) ___________________________________________________ - 67 -
App o hes de p ot o i ue diff e tielle ua titati e pou l’ tude des seau III.
d’i te a tio s _______________________________________________________________ - 68 -
1. Présentation des principales méthodes de quantification en protéomique différentielle _______ - 68 - 2. I t t pou les tudes d’i te a tio s p ot ineprotéine ________________________________ 73
-Co pl e ta it des diff e ts outils d’ tude des i te a tio s p ot i e-protéine _ - 74 - IV.
Perspectives : Vers une étude dynamique possible des interactions protéine-protéine,
V.
exemple de la technique AP-SWATH _____________________________________________ - 76 -
Chapitre 3 : Les peptides vecteurs ___________________________________________ 79
-Présentation générale des peptides vecteurs :_________________________________ - 79 - I.
- 12 -
M a is e d’i te alisatio et outils d’ tudes ________________________________ - 81 - II.
1. Description des méca is es p opos s pou l’i te alisatio des CPPs _____________________ - 81 - 2. M thodes d’ tudes de la apa it d’i te alisatio des CPPs da s les ellules _______________ 86
-Seulement des peptides vecteurs ? ________________________________________ - 88 - III.
1. Les do ai es d’appli atio s des CPPs _______________________________________________ 88 -2. L’i flue e des argos sur le devenir intracellulaire des conjugués ________________________ - 88 - 3. Des peptides vecteurs avec une activité biologique intrinsèque ___________________________ - 89 -
Présentation du projet de recherche _________________________________________ 91
-Co te te de l’ tude ______________________________________________________ - 91 - I.
Objectif et structure de ce projet de thèse ____________________________________ - 93 -
II.
Chapitre 4 : Cross-linking chimique couplé à la spectrométrie de masse pour étudier
l’i te actio des CPPs R/W
9et (R/W)
16avec l’acti e. __________________________ 95
-O je tifs de l’a ti le _____________________________________________________ - 95 - I.
Principaux résultats ______________________________________________________ - 95 -
II.
1. Compositions des complexes ______________________________________________________ - 95 - 2. )o es d’i te a tio ______________________________________________________________ 96
-Chapitre 5
: Ve s la captu e i cellulo de pa te ai es d’i te actio du peptide vecteu
(R/W)
9par photocrosslinking et spectrométrie de masse ________________________ 97
-Mise au poi t et alidatio iologi ue d’u e e sio photoa ti a le du CPP R/W9 :
I.
photo(R/W)9 ________________________________________________________________ - 97 - 1. Design et synthèse de photo(R/W)9 _________________________________________________ - 97 - 2. Validation biologique de photo(R/W)9 _______________________________________________ - 99 - Validation de la faisabilité de la réaction de photocross-linking in vitro et mise au point
II.
d’outils ___________________________________________________________________ - 106 -
1. Réaction de photocross-linking in vitro _____________________________________________ - 106 - 2. Analyses des espèces photocross-linkées par spectrométrie de masse MALDI-TOF/TOF et nanoLC-ESI-LTQ-Orbitrap. ___________________________________________________________________ - 108 -
Expériences de photocross-linking in cellulo ________________________________ - 126 - III.
1. E aluatio de la toto i it de l’i adiatio _________________________________________ - 126 - 2. Expériences de photocross-linking in cellulo _________________________________________ - 126 -
Chapitre 6 : Pull down et photocrosslinking __________________________________ 139
-P se tatio de l’e p ie e de pull-down associée au photocross-linking _________ - 139 - I.
Résultats obtenus _______________________________________________________ - 141 -
II.
1. Electrophorèse sur gel 1D-SDS-PAGE _______________________________________________ - 141 - 2. Résultats des analyses de spectrométrie de masse haut débit. __________________________ - 141 -
Interprétation des données : Quelles sont les « vraies » interactions? ___________ - 142 - III.
1. G atio des listes de pa te ai es d’i te a tio pote tiels ___________________________ 142 -2. I te p tatio a e les outils d’e i hisse e t de te es de gene ontology _______________ - 144 - 3. E a e a uel des listes de pa te ai es d’i te a tio pote tiels ________________________ 145
-Signification biologique des résultats obtenus ______________________________ - 146 - IV.
1. Une interaction avec la protéine EWS ______________________________________________ - 146 - 2. Possi ilit d’u e i te a tio a e EW“-FLI1 __________________________________________ - 147 -
- 13 -
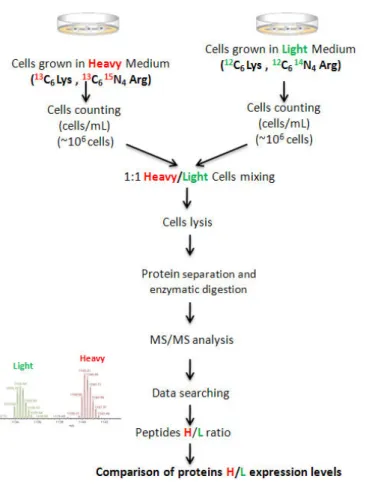
Mise e œu e de la te h i ue “ILAC. _______________________________________ - 151 - I.
1. V ifi atio du tau d’i o po atio des a ides a i s a u s. ________________________ 151 -2. Détermination des seuils de significativité pour la variation des ratios H/L _________________ - 153 - Comparaison des protéomes des cellules EF et 3T3. ____________________________ - 155 -
II.
1. Protéines significativement surexprimées dans les cellules EF par rapport aux cellules 3T3. ___ - 157 - 2. Protéines significativement sous-exprimées dans les cellules EF par rapport aux cellules 3T3. _ - 159 -
Effet du peptide vecteur photo(R/W)9 sur le protéome des cellules EF. __________ - 162 -
III.
Conclusion _____________________________________________________________ 167
Matériel et Méthodes ____________________________________________________ 171
-Matériel _______________________________________________________________ - 171 - I. 1. Origine du matériel _____________________________________________________________ - 171 - 2. Composition de tampons et solutions couramment utilisés : ____________________________ - 171 -Te h i ues d’a alyses et culture cellulaire ___________________________________ - 172 - II.
1. Spectrométrie de masse MALDI-TOF _______________________________________________ - 172 - 2. Couplage NanoLC-ESI-MS/MS _____________________________________________________ - 172 - 3. Culture cellulaire _______________________________________________________________ - 173 -
Protocoles ___________________________________________________________ - 173 - III.
1. Evaluation de la cytotoxicité de peptides vecteurs ____________________________________ - 174 - 2. Evaluation de la quantité de peptide internalisé ______________________________________ - 175 - 3. Observation du cytosquelette d’a ti e des ellules pa i u ofluo es e e_______________ 176 -4. Etude in vitro de la réaction de photo-crosslinking ____________________________________ - 176 - 5. Expérience de photocross-linking avec la stratégie N°1_________________________________ - 177 - 6. R alisatio d’u este lot pou d te te les esp es ioti l es ______________________ 179 -7. Expérience de photocross-linking avec la stratégie N°2_________________________________ - 179 - 8. Expérience de pull-down + photocross-linking ________________________________________ - 180 - 9. Expériences de protéomique différentielle avec la technique SILAC (Stable Isotope Labelling with Amino acids in cell Culture) ___________________________________________________________ - 181 -
Références bibliographiques ______________________________________________ 185
Annexes _______________________________________________________________ 205
-Annexe instrumentale : Spectrométrie de masse ______________________________ - 207 - I.
1. Spectromètres de masse : Généralités ______________________________________________ - 207 - 2. Représentations schématiques des spectromètres de masse utilisés ______________________ - 212 - Annexes diverses _______________________________________________________ - 215 -
- 15 -
Figure 1 : Représentation schématique de la structure cristallog aphi ue d’u o o e
d’a ti e………..- 28 -
Figu e : “ h a de la pol isatio de l’a ti e. ... - 28 -
Figure 3 : Représentatio s h ati ue de la d a i ue de pol isatio de l’a ti e………... - 30 -
Figure 4 : Les t ois a a ge e ts de l’a ti e ... - 32 -
Figure 5 : Représentation schématique de l'obtention de la protéine de fusion EWS-FLI1 par translocation chromosomale. ... - 36 -
Figure 6 : Frise chronologique récapitulant les évolutions de la recherche sur les interactions protéine-protéine. ... - 39 -
Figure 7 : Schéma récapitulant les différentes méthodes d'étude d'interactions protéine-protéine qui seront abordées ... - 40 -
Figure 8 : Exemple de stratégie utilisée pour détecter une interaction protéine-protéine avec un s st e dou le h ide ta lissa t l’a ti it de la p ot i e GAL . ... - 41 -
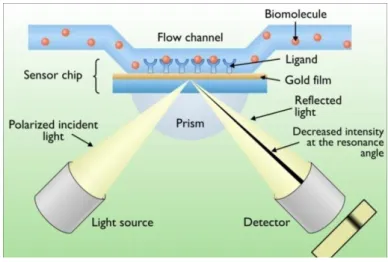
Figure 9 : Schéma de principe de la résonance plasmonique de surface (SPR) ... - 43 -
Figure 10 : Représentation schémati ue d’u e esu e de “PR ... - 44 -
Figure 11 : Des iptio s h ati ue des tapes u i e selles d’u e e p ie e d’i u op ipitatio couplée à la spectrométrie de masse [113]: ... - 46 -
Figure 12 : O te tio d’u e p ot i e e o i a te po teuse d’u e ti uette ... - 48 -
Figure 13 : “ h a de l’e p ie e de TAP-tag initiale . ... - 49 -
Figure 14 : Réaction de cross-linking chimique. ... - 55 -
Figure 15 : Structure du formaldéhyde PFA et du glutaraldéhyde GA. ... - 56 -
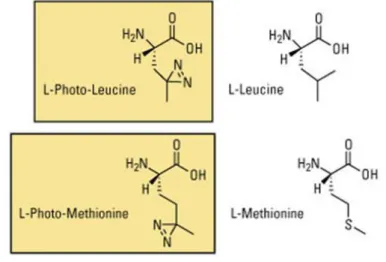
Figure 16 : Structure chimique des acides aminés photoactivables leucine et méthionine et de leurs équivalents naturels. ... - 58 -
Figure 17 : Configuration classi ue d’u alo i t e de titrage isotherme . ... - 60 -
Figure 18 : A Do es utes e t aites d’u e e p ie e d’ITC. B Isothe e de liaiso ep se ta t l’ ha ge de haleu e fo tio du ratio molaire ligand/protéine. ... - 60 -
Figure 19 : Exemple de spectres 1H-15N H“QC de l’i te leuki -2 (IL-2) en présence (bleu) et en l’a se e a o d’u e petite ol ule i hi it i e de IL-2 ... - 62 -
Figure 20 : Spectres de masse obtenus en infusion avec une source électrospray et permettant de sui e l’h d ol se de l’he asa ha ide de N-acétylglycosamine (NAG6) par le lyzozyme de la d’œuf de poule (HEWL). ... - 64 -
Figure 21 : Graphiques donnant le taux de deutération de différents domaines de la protéine apo-oglo i e pou u e e p ie e d’HDX-MS ... - 67 -
Figure 22: Schéma des protocoles de spectrométrie de masse quantitative indiquant à quelle étape les deu o ditio s d’u e e p ie e so t asse l es et do à uel i eau l’i t odu tio d’e eu s systé ati ues da s la ua tifi atio li es à la a ipulatio d’ ha tillo est a t e. Les e ta gles bleus et jaunes correspondent aux deux conditio s e p i e tales à o pa e ………- 69 -
Figure 23 : Exemple de protocole pouvant être utilisé pour réaliser la quantification relative de protéines par la technique SILAC. ... - 70 -
Figure 24 : Image d'un gel 2D réalisée avec la technique DiGE ... - 71 -
- 16 -
Figure 27 : Formation de liaisons hydrogènes divalentes entre les groupements guanidinium des chaînes latérales des arginines et des groupements phosphates, sulfates et carboxylates . ... - 82 - Figure 28 : Les différentes voies d'entrée par endocytose dans la cellule ... - 84 - Figure 29 : Modèle proposé pour expliquer la translocation directe des CPPs par formation de micelles inverses ... - 85 - Figure 30 : Schéma des différentes étapes de translocation directe par formation de pores. ... - 86 - Figure 31 : Analyse quantitative des effets de différents peptides vecteurs sur la morphologie des cellules 3T3 et EF ... - 92 - Figure 32: Structure développée du peptide photo(R/W)9 mettant en évidence les fonctions de ses
différentes parties constitutives. ... - 98 - Figure 33 : Signal obtenu en spectrométrie de masse MALDI-TOF pour un mélange 1/1 des versions deutérées et non deutérées du peptide photo(R/W)9. ... - 98 -
Figure 34 : Mécanisme de transformation du WST-8 (composé légèrement jaune) en WST-8 formazan (composé orange) en présence de cellules viables . ... - 99 - Figure 35 : Pou e tage de ia ilit des ellules EF à l’issue de h, h et h d’i u atio a e le peptide photo(R/W)9 pour des concentrations comprises entre 1 et 30 µM ... - 100 -
Figure 36 : Les différentes étapes du protocole de quantification de l'internalisation d'un peptide vecteur (CPP). ... - 102 - Figure 37 : Résultats obtenus pour les expériences de quantification de l'internalisation des peptides photo(R/W)9 et (R/W)9 réf dans les cellules EF à 37 °C pour des concentrations extracellulaires en
peptides de 5 et 7,5 µM et une période d'incubation de 75 min. ... - 103 - Figure 38 : I ages de i os opie de fluo es e e alis es pou o fi e l’effet du peptide photo(R/W)9su le e odelage du tos uelette d’a ti e da s les cellules EF... - 105 -
Figure 39 : Représentation schématique du protocole utilisé pour étudier les peptides cross-linkés issus de la réaction de photocross-linking du peptide photo(R/W)9 avec la BSA. ... - 106 -
Figure 40 : Mécanisme de la réaction de photocross-linking entre une sonde de type benzophénone et une protéine. ... - 107 - Figure 41 : Schéma du montage de photoirradiation UV avec une lampe à tungstène. ... - 107 - Figure 42 : Spectre MALDI-TOF (gamme de m/z 1000-3000) obtenu pour le milieu réactionnel de photocross-linking entre photo(R/W)9et la B“A à l’issue de la digestio t psi ue et de la pu ifi atio
d’affi it . ... - 108 - Figure 43 : Spectre de fragmentation MALDI-TOF/TOF du doublet de peptides photocross-linkés 2803-2813. ... - 110 - Figure 44 : Représentation schématique de la structure potentielle de l'ion du peptide photocross-linké correspondant au doublet 2803-2813. ... - 111 - Figure 45 : Peptides correspondant à la protéine BSA (P02769) identifiés lors de la recherche avec Mas ot de l’a al se NanoLC-ESI-MS/MS du milieu de photocross-linking photo(R/W)9-BSA. ... - 113 -
Figure 46 : Liste de candidats obtenus avec Xlink-Identifier pour le peptide photocross-linké (M+H)+
m/z 2803.. ... - 114 - Figure 47 : Schéma des différents fragments b et y (fragmentation CID) pouvant être obtenus à partir d'un ion précurseur composé de deux peptides liés covalemment par photocross-linking.. ... - 115 - Figure 48 : Exemple de fichier candidat Xlink-Identifier 2.0 répertoriant les pics identifiés dans un spectre MS/MS donné. ... - 116 -
- 17 -
réaction de photocross-linking. ... - 117 - Figure 50 : Représentation des sites de fragmentation d'un peptide selon la nomenclature de Biemann... - 119 - Figure 51 : Production du fluoranthène radical anion dans le volume d'ion. ... - 119 - Figure 52: Récapitulatif des ions observés en CID basse énergie (ESI-LTQ/orbitrap) pour l'ion photocross-linké de masse moléculaire 2803-2813 chargé 4+. ... - 121 - Figure 53 : Récapitulatif des ions observés en ETD (avec énergie supplémentaire de 20 %) (ESI-LTQ/orbitrap) pour l'ion photocross-linké de masse moléculaire 2803-2813 chargé 4+.. ... - 122 - Figure 54 : Récapitulatif des ions observés en CID haute énergie (MALDI-TOF/TOF) pour l'ion photocross-linké de (M+H)+ 2803-2813 chargé 1+.. ... - 123 -
Figu e : “t at gie i itiale N° e isag e pou l’ide tifi atio des pa te ai es d’i te a tio (intracellulaires et/ou membranaires) du peptide (R/W)9 par photocross-linking.. ... - 127 -
Figure 56: Gel 1D SDS-PAGE coloré au bleu de Coomassie pou l’e périence de photocrosslinking in
cellulo avec le peptide photo(R/W)9. ... - 128 -
Figure 57 : Spectre MALDI-TOF o te u pou l’ luat o espo da t au ilieu a tio el ν dig et purifié sur billes.. ... - 129 - Figure 58 : Image de 1) gel 1D SDS-PAGE coloré au bleu de Coomassie et de 2) western-blot réalisé avec de la streptavidine HRP pour une expérience de photocrosslinking sur cellules entières.. ... - 131 - Figu e : I age d’u este lot alisé avec de la streptavidine HRP pour des milieux de photocrosslinking sur cellules dialysés ou purifiés sur billes . ... - 132 - Figure 60 : Spectre MALDI-TOF d’u digest de l sat ellulai e ellules photoi adi es e p se e de photo(R/W)9) après purification sur billes de streptavidine.. ... - 134 -
Figure 61 : Schéma de la stratégie "adaptée" (N°2) de photocross-linking in cellulo. ... - 136 -
Figure 62 : Gel 1D SDS-PAGE coloré au bleu de Coomassie et Western blot avec de la streptavidine HRP pou l’e p ie e de photo oss-linking sur membranes. ... - 137 - Figu e : Gel d’ le t opho se D “D“-PAGE coloré au bleu de Coomassie pour les milieux de pull-down + photocross-linking (1), au pull-pull-down simple (2) et au contrôle négatif (3). ... - 141 - Figure 64 : Présentation des séquences en acides aminés des protéines EWS, FLI1 et EWS-FLI1 et de leu deg d’ho ologie. ... - 147 - Figure 65 : Schéma de la perturbation de l'interaction de la protéine EWS-FLI a e l’ARN h li ase conduisant à l'inhibition de l'activité de transcription du complexe. ... - 148 - Figure 66 : Spectres MALDI-TOF pour la gamme de m/z 1100- o t a t le tau d’i o po atio des acides aminés marqués 13C
6 Lys et 15N4 Arg seuls et en mélange 1-1 des cellules (L) et (H).. . - 152 -
Figu e : Figu e illust a t la ifi atio du tau d’i o po atio des a ides a inés marqués dans les protéines ... - 153 - Figu e : G aphi ue de t pe ol a o plot o te u pou deu pli ats iologi ues de l’e p ie e contrôle EF (H) / EF (L).. ... - 154 - Figure 69 : Graphique de type volcano plot pour la comparaison du protéome des cellules EF (H) et des cellules 3T3 (L).. ... - 156 - Figu e : R seau d’i te a tio s p ot i e-protéine généré sur le logiciel STRING à partir de la liste de protéines surexprimées dans les cellules EF par rapport aux cellules 3T3.. ... - 157 - Figu e : Nœud du seau d'i te a tio o espondant au processus de glycolyse ... - 158 -
- 18 -
des cellules EF (L) incubées avec le peptide photo(R/W)9 (18 h, 5 µM).. ... - 163 -
Figure 73 : Modifications du métabolisme caractéristiques de la transformation tumorale ... 164
-Tableau 1 : Etiquettes communément utilisées en purificatio d’affi it . ... - 47 - Tableau 2 : Exemple de détergents classiquement utilisés en biochimie et propriétés associées. .. - 53 - Tableau 3 : Trois groupements photoactivables communément utilisés : la benzophénone (Bp), le phényle azide (Pa) et le trifluorométhyl phényl diazirine (TFMFD). ... - 57 - Tableau 4 : Avantages et inconvénients des différentes techniques d'étude des interactions protéine-protéine. ... - 75 - Tableau 5 : Origines et séquences des CPPs les plus étudiés (adapté de [207]) ... - 80 - Tableau 6 : Exemples de fluorophores communément utilisés en immunofluorescence. ... - 104 - Tableau 7 : Attribution des doublets observés dans le spectre MALDI-TOF présenté (Figure 42)..- 109 - Tableau 8 : Attribution des fragments du spectre MALDI-TOF/TOF présenté Figure 43.. ... - 111 - Tableau 9 : Tableau récapitulant les observations et comparaisons réalisées pour les modes de fragmentations CID, haute et basse énergie, et ETD des ions photocross-linkés 2803-2813. ... - 124 - Ta leau : P se tatio des diff e ts ha tillo s de l’e p ie e de pull-down associée au photocross-linking ... - 140 - Tableau 11 : Nombres moyens de peptides et de protéines identifiés pour les différentes pistes pour les deux réplicats biologiques.. ... - 142 - Tableau 12 : Extrait du tableau de résultats obtenu avec le logiciel SAINT pour l'expérience de Pull-down simple.. ... - 146 - Ta leau : R apitulatif des p ot i es de liaiso s de l’a ti e figu a t da s la liste des p ot i es significativement sous-exprimées dans les cellules tumorales EF par rapport aux cellules non-tumorales 3T3... - 161 -
Figures
de l’a e e i st u e tale : Spectrométrie de masse
Figure annexe 1: Structure d’u spe t o t e de asse. ... - 207 - Figure annexe 2 : “ h a de p i ipe de la p odu tio d’io s pa MALDI ... - 208 - Figure annexe 3 : “ h a du p o essus d’io isatio pa le t o ulisatio . ... - 209 - Figure annexe 4 : Principe de la séparation des ions et de la mesure de leur rapport m/z avec un analyseur à temps de vol (TOF). ... - 210 - Figure annexe 5 : Schéma de la réflexion électrostatique des ions dans un analyseur TOF en mode réflecteur ... - 210 - Figure annexe 6 : A = Schéma du LTQ B = Détection des ions piégés ... - 211 - Figure annexe 7 : Vue e oupe de l’a al seu O it ap. Les io s so t i t oduits da s l’a al seu au niveau de la flèche rouge. ... - 211 - Figure annexe 8 : Représentation schématique du spectromètre de masse MALDI-TOF/TOF 4700 Proteomics Analyzer ... - 212 - Figure annexe 9 : Schéma du spectromètre de masse LTQ-Orbitrap XL (Thermo Scientific) ... - 213 -
- 19 -
masse)
Annexe II 1: Recherches des peptides photocross-linkés (R/W)9-BSA avec Mascot sur P.D 1.3 .... - 215 -
Annexe II 2: Spectres de fragmentation CID (basse et haute énergie) et ETD des ions de (M+H)+
2803-2813 ... - 217 -
Annexe II 3 : COMPARAISON A : Pull-down + photocross-linking versus contrôle négatif ... - 222 - Annexe II 4 : COMPARAISON B : Pull-down versus contrôle négatif ... - 226 - Annexe II 5: Attributions des spectres MS/MS aux peptides [269-304] et [373-386] de la protéine. .... -
229 -
Annexe II 6: Liste de protéines sur- ou sousexprimées dans les cellules EF (H) par rapport aux cellules
3T3 ... - 230 -
Annexe II 7: Réseau d’i te a tio s des p ot i es sous-exprimées... - 234 -
Annexe II 8 : Volcano plots obtenus pour les expériences de protéomique différentielle avec le
- 21 -
Les peptides vecteurs ou CPPs, courtes séquences peptidiques (< 30 acides aminés), ont la capacité de traverser la membrane plasmique des cellules avec une toxicité très limitée. Depuis leur découverte il y a aujou d’hui ie tôt t ois d e ies, ils o t sus it eau oup d’i t t da s la communauté scientifique du fait de leur aptitude à transporter avec eux des molécules, appelées cargos, de tailles parfois très importantes par rapport à celles des CPPs. Alors considérés comme des molécules inertes, les CPPs ouvraient des perspectives très intéressantes en termes de vectorisation. Cepe da t, depuis uel ues a es, de plus e plus d’ tudes e ette t e ause l’a se e d’a ti it biologique des CPPs et le terme de bioportide a même été créé pour désigner les CPPs ayant un effet
biologique intrinsèque
[1]
. Parmi eux, se trouve le peptide (R/W)9 de séquence RRWWRRWRR,peptide vecteur largement étudié au Laboratoire des BioMolécules (UMR 7203) dans lequel j'ai réalisé mes travaux de thèse. Il a été montré au cours de précédents travaux
[2]
, que ce peptide était capable de modifier le phénotype de cellules tumorales de type EF, modèle cellulaire du sarcome d’E i g, a a t is pa u tos uelette d’a ti e t s d so ga is . Au out de uel ues heu es d’i u atio a e le peptide e teu R/W 9, u ha ge e t de o phologie est o se ai si u’uretour des fibres de stress asse lage de i ofila e ts d’a ti e e fais eau . Ce ha ge e t de morphologie, li à u e odelage du tos uelette d’a ti e, s’a o pag e d’u e di i utio de la itesse de ig atio et d’u e di i utio de la apa it à oît e e i d pe da e d’a age, toutes deux étant caractéristiques des cellules cancéreuses.
De manière générale, il est nécessaire de comprendre comment ces CPPs peuvent affecter la d a i ue des ellules de a i e à pou oi u jou les utilise e li i ue sa s ai d e d’effets se o dai es li s à leu a ti it iologi ue i t i s ue plutôt u’à elle de la ol ule e to is e. Dans le cas du peptide (R/W)9, ette tude est d’auta t plus intéressante du fait de son activité de
e odelage du tos uelette d’a ti e o se su les ellules tu o ales EF. E effet, l’ tude du ode d’a tio du peptide R/W9est sus epti le d’appo te des l e ts qui pourront faire évoluer
la compréhension et le t aite e t du sa o e d’E i g, a e t s ag essif des os et des tissus conjonctifs, touchant majoritairement les adolescents et ayant un très mauvais pronostic en raison de son fort potentiel métastasique.
L’o jet de e t a ail de th se a ai si t d’a a e da s la compréhension de l'effet moléculaire
observé du peptide vecteur (R/W)9 sur le phénotype de cellules tumorales EF. Le te e d’« effet moléculaire » fait f e e à l’e se le des i te a tio s p ot i e-protéine ou peptide-protéine pote tielle e t i pli u es da s le a is e d’a tio du peptide R/W9.
Le sujet des interactions protéine-protéine se t ou e pa o s ue t au œu de ette tude et j’ai ainsi hoisi d’ o sa e u e pa t i po ta te de l’introduction. Dans un premier chapitre, les interactions protéine-p ot i e, i pli u es da s tous les a is es de la ie d’u e ellule, seront p se t es. L’e e ple des i te a tio s p ot i e-protéine dans la régulation de la dynamique de l’a ti e se a t ait e d tail de a i e à pou oi i te p te os sultats et dis ute pa la suite l’effet du CPP R/W9 sur le remodelage du cytosquelette. Dans le deuxième chapitre, différentes
- 22 -
consacré aux CPPs, en particulier à leur(s) mode(s) d’i te alisatio et à leur devenir intracellulaire. La partie résultats sera, elle, structurée en quatre hapit es. Tout d’a o d, un chapitre concernant le travail réalisé in vitro pou a a t ise l’i te a tion du CPP (R/W)9 a e l’a ti e pa oss-linking
chimique et spectrométrie de masse. Puis, les trois chapitres suivants correspondent aux différentes stratégies développées pour identifier des interactions peptide-protéine ou protéine-protéine in
cellulo ou da s des l sats ellulai es sus epti les d’e pli ue l’effet du CPP R/W 9. Il s’agit de
stratégies ciblées développées pour la aptu e des pa te ai es d’i te a tio du peptide,
(photocross-linking in cellulo ( hapit e et pu ifi atio d’affi it ouplée à la spectrométrie de asse hapit e et d’u e stratégie indirecte correspondant à une approche de protéomique diff e tielle a a t pou o je tif d’ alue l’i pa t du peptide R/W9su l’e p essio des p ot i es des cellules EF pour en déduire so ode d’a tio Chapit e .
- 23 -
au phénotype cellulaire,
l’e e ple du
tos uelette d’a ti e
La p ot o i ue est la dis ipli e ui tudie l’e se le des protéines et leurs rôles dans une cellule, un organite, un tissu, un organe ou un organisme à un moment donné et sous des conditions données. Dans un premier temps, la p ot o i ue s’est atta h e à l’ide tifi atio glo ale des protéines extraites d'une culture cellulaire, d'un tissu ou d'un fluide biologique et à la détermination de leur localisation dans les compartiments cellulaires, de leurs éventuelles modifications post-traductionnelles ainsi que de leur quantité. Aujou d’hui, elle s’o ie te de plus e plus e s la compréhension du rôle de ces protéines de façon intégrée dans la cellule et vers l’ aluatio de l’i ide e des odifi atio s de leu e p essio au i eau d’u o ga is e.
Jus u’au d ut des a es , ota e t e aiso du a ue d’outils app op i s, les fonctions des protéines ont été étudiées de manière indépendante. Cependant, les fonctions cellulaires étant rarement attribuées à une seule molécule, il est apparu nécessaire de prendre en compte les interactions existant entre différentes protéines organisées en modules fonctionnels [3] pour comprendre le ôle d’u e p ot i e e pa ti ulie . De plus, la plupa t des p ot i es so t i pli u es dans plusieurs processus cellulaires distincts et en association avec différentes protéines. La complexité des interactions est également augmentée en raison de la diversité spatiale et temporelle des protéines. En effet, une même protéine peut faire partie de complexes protéiques différents en fonction de sa localisation subcellulaire ou du stade du cycle cellulaire. Les modifications post-traductionnelles peuvent égale e t i flue su la apa it d’u e p ot i e à ta li u e i te a tio . Finalement les interactions protéine-protéine varient énormément en termes de nature, de force et de durée.
Ai si la o p he sio de la fo tio ellulai e d’u e p ot ine peut être une tâche extrêmement lou de e aiso de l’e t ao di ai e o ple it des i te a tio s protéine-molécule de façon générale, et plus particulièrement protéine-protéine da s les uelles elle est sus epti le d’ t e impliquée.
Dans ce premier chapitre, nous allons commencer par poser quelques bases pour la compréhension de la « sociologie moléculaire de la cellule » [4]. Ensuite, et compte tenu du sujet développé, nous ous i t esse o s à u des seau d’i te a tio s p ot ine-protéine les plus étudiés, à savoir le cytos uelette d’a ti e et ses p otéines de liaison (ABPs pour actin binding proteins). Finalement ous i t odui o s le o te te iologi ue de l’ tude, le sa o e d’E i g, esse tielle e t a a t is pa l’e p essio d’u e p ot i e de fusion EWS-FLI1 induisant d’i po tantes modifications du phénotype des cellules en particulier au niveau de leur cytosquelette.
- 24 -
1.
Les diff e ts t pes d’i te a tio s p ot i e-protéine
Ho is uel ues a es e e ples tel ue l’atta he e t o ale t de l’u i uiti e ou des p ot i es SUMO aux protéines de signalisation [5], les protéines se lient les unes aux autres très majoritairement de manière non-covalente. Ces liaisons non- o ale tes, do t l’ e gie est o p ise entre 0 et 40 kJ.mol-1, résultent de la combinaison de liaisons hydrophobes, de liaisons hydrogènes,
d’i te a tio s de Van der Waals et/ou de liaisons ioniques, plus fortes, au niveau de domaines spécifiques de chacu e des deu p ot i es. Ces do ai es d’i te a tio s peu e t t e li it s à de petites po hes ou s’ te d e su de plus g a des su fa es et peuvent ainsi impliquer de quelques sidus à plusieu s e tai es d’a ides a i s. Il est important de comprendre la nature des interactions protéine-protéine car les différents types de liaison vont être plus ou moins sensibles au ha ge e ts de l’e i o e e t ellulai e, que ce soit la composition ionique du milieu, le pH ou d’aut es pa a t es hi i ues.
On distingue généralement les interactions protéiques stables des interactions transitoires.
Les interactions stables so t elles i pli u es da s l’asso iatio de p ot i es ui peu e t t e purifiées sous forme de complexes constitués de plusieurs sous-unités de même nature ou de natures différentes. C’est le as pa e e ple des p ot i es du i oso e ui ’e iste t da s les cellules que sous forme associées.
Ce sont les interactions transitoires ui so t aise la le e t à l’o igi e de la ajo it des fonctions cellulai es. Co e leu o l’i di ue, les interactions transitoires sont limitées dans le te ps et o ditio es pa diff e ts l e ts tels u’u e odifi atio post-traductionnelle, un ha ge e t o fo atio el ou l’a i e da s u o pa ti e t ellulai e pa ticulier. Par exemple, e so t des i te a tio s t a sitoi es ui so t à l’o igi e de la t a s issio de signaux intracellulaires telle ue la liaiso d’u liga d à u epteu oupl à la p ot i e G. Les complexes résultant d’i te a tio s t a sitoi es so t eau oup plus o pli u s à tudie a l’ide tification des protéines et/ou des o ditio s essai es à l’o se ation de ces complexes sont des prérequis à leur étude. Il faud ait pou ela alle e s l’e hausti it da s l’ide tifi atio des l e ts d’un système, ce qui constitue un des défis parmi les plus importants.
2.
Les do ai es d’i te a tio s des p ot i es
Les interactions protéine-protéine epose t su l’e iste e de do ai es d’i te a tio définis au sein des p ot i es. Ces do ai es d’i te a tio peuvent permettre de définir la localisation de la protéine dans la cellule, permettre de reconnaître les modifications post-traductionnelles des protéines pa te ai es, t e à l’o igi e de la fo atio d’u o ple e ultip ot i ue ou e o e o t ôle la co fo atio , l’a ti it ou la sp ifi it pou u su st at d’u e z e.
T pi ue e t, u do ai e d’i te a tio d’u e p ot i e e o ait u élément central déterminant
ainsi que des résidus adjacents contigus ou non qui permettent des contacts supplémentaires et
a lio e t la sp ifi it de l’i te a tio . Pa e e ple, le do ai e “H de la t osi e ki ase cytoplasmique (Csk) reconnaît un motif central PXXP (P : proline, X : ’i po te uel a ide a i de l’e t it de la PEP t osi e phosphatase ui adopte alors une hélice polyproline de type II typique des sites de liaison aux domaines SH3. Ensuite, le domaine SH3 Csk est également en interaction avec
- 25 - spécificité de l’asso iatio in vivo [6].
Cependant, dans certains cas, l’affi it d’u seul do ai e d’u e p ot i e pou u otif peptidi ue peut être suffisante pour obtenir une interaction spécifique.
Les do ai es d’i te a tio so t a a t is s pa u e s ue e o se sus d’a ides a i s. De nombreux domaines sont présents en plusieurs centaines de copies dans le protéome et sont utilisés pour réguler différentes fonctions cellulaires. Certains domaines ont des fonctions bien spécifiques comme le domaine SH2 qui est dédié à la signalisation des tyrosines kinases [7]. Au contraire, d’aut es do ai es so t o u s à u g a d o e de p ot i es et impliqués dans des fonctions ellulai es a i es. C’est le as du do ai e “H ui gule à la fois la t a sdu tio de sig au , le t afi de p ot i es et de si ules, l’o ga isatio du tos uelette ou e o e la iog se d’o ga elles. La cellule fait ainsi appel à un ense le de do ai es d’i te a tio qui peuvent être combinés de diff e tes a i es pou pe ett e l’a tio de ses s st es de gulatio [8].
C’est su la ase des o aissa es a uises au fil des a es o e a t es do ai es d’i te a tio u’a t d elopp e la ase de do es Pfa Protein families database) qui regroupe une large olle tio d’alig e e t de s ue es ainsi que des annotations fonctionnelles et des références bibliographiques de chaque famille de protéines. Elle a pour but de permettre de déterminer la fo tio de ou elles p ot i es e se asa t su l’e iste e da s leu s ue e de do ai es caractéristiques d’u e fa ille pa ti uli e de p ot i es [9]. La fonction prévue par cette approche bioinformatique doit ensuite être confirmée par des expériences in vitro mais constitue un point de départ intéressant.
3. Régulation et effets biologiques des interactions protéine-protéine
Beaucoup des protéines impliquées dans les voies de signalisation ont une activité enzymatique et ont la capacité de modifier leur substrat de manière post-traductionnelle [10]. La phosphorylation d’u e p ot i e est la odifi atio post-traductionnelle la plus courante et la mieux comprise pour la gulatio d’i te a tio s p ot i e-p ot i e ais d’aut es odifi atio s o e l’a t latio et l’h d o latio so t aise la le e t gale e t i pli u es da s la odulatio de es interactions. Les modifications post-traductionnelles peuvent affecter les interactions protéine-protéine selon deux mécanismes. Premièrement, les modifications post-traductionnelles changent
les p op i t s hi i ues de l’a ide a i odifi e t aî a t la atio ou la dispa itio d’u e liaison chimique avec la protéine partenaire. Deuxièmement, les modifications post-traductionnelles peuvent affecter le repliement de la protéine et dans le même temps la structure secondaire des do ai es d’i te a tio . Ainsi les modifications post-traductionnelles peuvent soit permettre d’e pose u ou eau do ai e à la su fa e d’u e p ot i e ou, au contraire, rendre un domaine d’i te action inaccessible pour la liaison avec une protéine partenaire [11]. Les protéines 14-3-3 qui sont capables de reconnaître les acides aminés phosphorylés de séquences particulières sont un bon e e ple de la gulatio d’i te a tio s protéine-protéine par les modifications post-traductionnelles [12].
Phizicky E. M. et Fields “. o t d fi is i at go ies d’effets iologi ues liés aux interactions protéine-protéine [13]. (1) Dans le cas de protéines allostériques, ces interactions peuvent modifier les p op i t s i ti ues de la p ot i e ai si ue leu l’affinité pour un substrat [14]. (2) Un certain
- 26 -
i te diai e di e te e t d’u e enzyme à une autre sans libération en solution (substrate
channeling). (3) Certaines interactions protéine-protéine peuvent donner naissance à de nouveaux
sites de fixation. (4) Des interactions protéine-protéine o duise t à l’i a ti atio d’u e des deu p ot i es, la p ot i e pa te ai e est alo s ualifi e d’i hi iteu . (5) Des interactions protéine-protéine peu e t odifie la sp ifi it d’u e p ot i e pou so su st at.
4.
Les seau d’i te a tio s des p otéines
Les interactions protéine-protéine ne doivent pas être envisagées comme des événements isolés. Ces interactions doivent au contraire être replacées au sein de réseaux denses et interconnectés. En effet, l’a ti atio d’u e as ade de sig alisatio eposant sur un complexe protéique implique également bien souvent des effecteurs placés à la fois en amont et en aval de cette voie de sig alisatio . Il est i po ta t gale e t de ga de à l’esp it u’u e e p ot i e peut t e impliquée dans différentes interactions protéine-protéine.
La cartographie systématique des interactions protéine-p ot i e ou a tog aphie de l’i te a to e
a été initiée pour des organismes modèles avec des processus biologiques définis [15], [16] avant d’ t e te due à l’ helle du p ot o e. “i l’étude des interactions protéine-protéine peut permettre de co p e d e le ôle d’u e p ot i e ; la o p he sio de l’o ga isatio glo ale d’u seau d’i te a tio peut, elle, pe ett e d’a lio e la o aissa e de a is es o ple es e iologie fo da e tale et d’e pli ue le d eloppe e t de diff e tes pathologies. Ainsi, par exemples, un réseau à 186 interactions i pli u es da s la aladie d’Hu ti gto a t ta li [17] tandis que 755 interactions ont été rassemblées dans la voie de signalisation de TGF-β (transforming growth factor-β)[18].
De nombreuses approches ayant pou ut d’ tudie les fo tio s d’u e p ot i e epose t par conséquent su l’ide tifi atio de pa te ai es d’i te a tio de a i e à pouvoir replacer la protéine tudi e da s u seau d’i te a tio s i pli u da s la gulatio d’u p o essus o u. Nous aborderons ces différentes approches dans le chapitre 2.
- 27 -
réseau
d’i te a tio s
Le tos uelette i te e des ellules eu a otes est o pos de i ofila e ts d’a ti e, de microtubules et de filaments intermédiaires. Le cytosquelette est un ensemble dynamique qui joue un rôle crucial dans l’a ti it ellulai e en contrôlant notamment la morphologie de la cellule, sa motilité et sa capacité à se diviser. Il est également fortement impliqué dans le transport intracellulaire. Nous allons nous intéresser tout particulièrement à la régulation de la dynamique des microfilame ts d’a ti e.
1.
L’a ti e : fi he d’ide tit [19]
L’a ti e est u e p ot i e u i uitai e da s les ellules eu a otes, t s o se e d’u o ga is e à u autre, et qui présente trois isoformes principales α-,β-,γ-. C’est da s les ellules us ulai es ue la o e t atio totale d’a ti e est la plus fo te ≈ % de la quantité totale de protéines), tandis u’elle ep se te e o e e environ 5 % des protéines dans les autres types de cellules où elle est impli u e da s la o phologie ellulai e, le t a spo t d’o ga elles, la gulatio du t a spo t des ions ou la réponse à des signaux extracellulaires transmis par les récepteurs.
L’a ti e se t ou e ajo itai e e t da s le toplas e des ellules. L’a ti e onomérique (Actine G pou a ti e glo ulai e s’asse le da s les ellules pou fo e des fila e ts d’a ti e A ti e F) de structure légèrement hélicoïdale de 5 à 9 nm de diamètre (Figure 2).
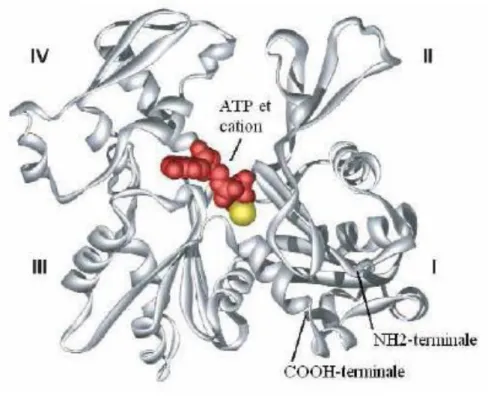
La détermination de la structure atomi ue de l’a ti e G (complexée avec la ADNase I [20] est à la base de la o p he sio a tuelle de l’a ti e. L’a ti e G est constituée de 375 acides aminés et a une masse moléculaire de 43 kDa. Elle est repliée de manière à former deux grands domaines qui eux-mêmes comprennent deux sous-domaines (les sous-domaines sont numérotés de I à IV selon la nomenclature de Kabsch[20]).
Dans cette structure de l’a ti e ep se t e figu e , on note la présence de deux lobes constitués chacun de deux sous-domaines. Le premier sous-domaine (sous-domaine I) est constitué par les segments non-consécutifs 1-32, 70-144 et 338-372 et contient donc les deux extrémités de la protéine (NH2-terminale et COOH-terminale). Le sous-domaine II qui complète le premier lobe est
formé par le segment 33- . L’aut e lo e o po te les sous-domaines III, constitué des segments 145-180 et 230-337, et IV qui englobe la partie centrale de la protéine entre les résidus 181 et 269. Au coeur de ce globule, dans le sillon formé entre les deux domaines, se trouve un site de o ple atio d’u u l otide ATP ou ADP asso i à u atio di ale t Mg2+ ou Ca2+) [21]–[23].
Cette asso iatio de l’a ti e a e es ofa teu s esse tiels pe et la stabilisation de la protéine et limite donc sa dénaturation.
- 28 -
Figure 1 : Représentation schématique de la st u tu e istallog aphi ue d’u o o e d’a ti e. Les 4
sous-do ai es I à IV so t ep se t s e g is, l’ATP en rouge et le cation divalent en jaune. (Figu e d’ap s
[24])
2.
Pol
isatio de l’a ti e
In vitro, en présence de 100 M de KCl, d’ATP et de MgCl2, les o o es d’a ti e G purifiés et
associés à une molécule d’ATP pol ise t pou fo e des fila e ts d’a ti e F. Ce processus se déroule en trois étapes théoriques schématisées dans la figure 2 : la u l atio , l’ lo gatio et l’ tat stationnaire [25].
Figure 2 : Schéma de la pol isatio de l’a ti e (Figu e d’ap s [24]).
Au ou s de la u l atio , les o o es d’a ti e s’ag ge t e di es puis e t i es i sta les jus u’à fo e des o au de pol isatio , les nuclei qui sont les premiers oligomères et dont la disso iatio est oi s fa o a le ue l’asso iatio a e d’aut es o o es. Ap s la fo atio de ces nuclei i te ie t l’ tape d’ lo gatio au ou s de la uelle des o o es d’a ti e so t additio s au i eau des deu e t it s du fila e t. Lo s de l’i o po atio d’u o o e d’a ti e-ATP dans le polymère, le u l otide est h d ol s pou do e de l’ADP a e li atio d’un phosphate inorganique (Pi) [26]. L’h d ol se du u l otide s’a o pag e de a a ge e ts conformationnels qui permettent la différenciation entre les actines présentes aux deux extrémités
- 29 -
pas les es et la itesse de pol isatio de l’e t it + dite a e est à 10 fois plus apide ue elle à l’e t it -) dite pointue [27].
De faço g ale, ette tape oit la pa t de l’a ti e F aug e te alo s ue la o e t atio e monomère diminue [28]. Lorsque celle-ci atteint la concentration critique de polymérisation de l’a tine (Cc), le régime devient alors équilibré. On observe simultanément une polymérisation d’a ti e à l’e t it a e et u e d pol isatio au i eau de l’e t it poi tue. L’ tat d’ uili e se a a t ise do pa u e galisatio des itesses d’asse lage à l’e t it + et de d sasse lage à l’e t it - . Pou ou le e ph o e d’ ha ge de o o es ou
treadmiling, le nucléotide ADP est remplacé par un ATP afin de restaurer le monomère polymérisable
actine-ATP.
In vivo
In vivo, l’ tude de la d a i ue du tos uelette d’a ti e de ie t eau oup plus o ple e. La
polymérisation des o o es d’a ti e G ne repose plus seulement sur la concentration en cofacteurs et sur le ratio actine G/actine F mais également, comme nous allons le voir dans la suite, sur un nombre très impo ta t d’i te a tio s p ot i e-protéine qui sont régulées en fonction des contraintes auxquelles les cellules sont soumises.
3. Les protéines de liaison
de l’a ti e et leu s ôles
La localisation, la longueur, la polarisation des fila e ts d’a ti e ai si ue l’aspe t te po el de l’asso iatio des o o es d’a ti e pou fo e es fila e ts so t gul s pa u g a d o e de p ot i es de liaiso de l’a ti e actin binding proteins ou ABPs) qui sont, elles, régulées par les cascades de signalisation initiées à la surface de la cellule. Les filaments sont à leurs tours organisés de manière tridimensionnelle grâce à un autre groupe de protéines de liaison qui peuvent s’associer à ces différents filaments, les séquestrer, donner naissance à des faisceaux ou encore les ancrer à la membrane.
L’a ti e est u e des p ot i es, si e ’est la p ot i e, i pli u e da s le plus g a d nombre d’i te a tio s p ot i e-protéine da s la ellule. Ai si, il a t o t jus u’à p se t ue plus de p ot i es o tie e t u do ai e de liaiso à l’a ti e et so t sus epti les d’i flue e so tat de polymérisation [29]. Il est ainsi hors de question de dresser la liste de tous ses partenaires et de leur action. Nous allons simplement essayer de définir les rôles principaux et de donner des exemples significatifs de protéines impliquées dans ces fonctions.
La régulation de la polymérisation des microfilaments
a.
Régulation de la concentration en actine G libre
Da s les ellules, u e g a de ua tit d’a ti e est ai te ue sous forme monomérique (actine G) par des protéines dites de séquestration [30]. La s uest atio de l’a ti e G pa es p ot i es pe et de o se e u sto k i po ta t d’a ti e G da s la ellule tout e ai tenant la o e t atio d’a ti e G libre basse dans la cellule lorsque la polymérisation des microfilaments a attei t l’ tat statio naire. Cependant, suite à une stimulation appropriée, la polymérisation de
- 30 - monomères séquestrés.
Il apparaît que deux p ot i es de liaiso de l’a ti e G sont impliquées dans la séquestration de l’a ti e : La thymosine 4, une petite protéine de 5 kDa, ui a t ide tifi e o e l’age t de s uest atio p i ipal de l’a ti e [31] et la profiline, une protéine de masse moléculaire comprise entre 12 et 15 kDa découverte en 1977 [32] et o ue pou fo e u o ple e : a e l’a ti e G. Nucléation et régulation de la nucléation
Le complexe Arp2/3 est un complexe de sept sous unités dont les protéines Arp2 et 3 (Actin related
protein) et cinq autres protéines nommées p41, p34, p2, p20 et p16 [33]. Ce complexe induit la
nucléation de nouveaux filaments après stimulation par les protéines de la famille WASP
(Wiskott-Aldrich Syndrom Protein)/Scar [34] qui possèdent un domaine WA dans leur partie C-terminale. La
région "verproline homology", dite W, recrute deux monomères d'actine et la séquence acide, dite A, active le complexe Arp2/3.
Le complexe Arp 2/3 est le point de départ du pontage de filaments nouvellement formés et organise les fila e ts d’a ti e e u seau a a t u e st u tu e e Y [35] comme schématisé dans la figure 3.
Figure 3 : Représentation schématique de la dynamique de pol isatio de l’a ti e. L’a ti e-ATP est
ep se t e e la , l’a ti e ADP-P e o a ge, l’a ti e-ADP en rouge et la profiline en noir Figu e d’ap s
![Figure 5 : Représentation schématique de l'obtention de la protéine de fusion EWS-FLI1 par translocation chromosomale Figu e d’ap s [87])](https://thumb-eu.123doks.com/thumbv2/123doknet/14719045.569552/37.892.213.683.414.724/figure-représentation-schématique-obtention-protéine-fusion-translocation-chromosomale.webp)

![Figure 11 : Des iptio s h ati ue des tapes u i e selles d’u e e p ie e d’i u op ipitatio couplée à la spectrométrie de masse [113]:](https://thumb-eu.123doks.com/thumbv2/123doknet/14719045.569552/47.892.156.737.657.1091/figure-iptio-tapes-selles-ipitatio-couplée-spectrométrie-masse.webp)
![Tableau 1 : Etiquettes communément utilisées en pu ifi atio d’affi it (Figure d’ap s [114])](https://thumb-eu.123doks.com/thumbv2/123doknet/14719045.569552/48.892.135.756.741.1047/tableau-etiquettes-communément-utilisées-ifi-atio-affi-figure.webp)