RÉSISTANCE AUX -LACTAMINES À LARGE
SPECTRE CHEZ LES BACTÉRIES
À GRAM NÉGATIF
Épidémiologie et diagnostic
Mémoire
MARILYSE VALLÉE Maîtrise en microbiologie-immunologie Maître ès sciences (M.Sc.) Québec, Canadaβ
Résumé
La résistance des bactéries à Gram négatif aux céphalosporines de troisième génération (C3G) et aux carbapénèmes constitue une menace de santé publique majeure à l’échelle mondiale. Ces β-lactamines à large spectre d’action sont spécifiquement utilisées dans le traitement des infections résistantes potentiellement mortelles causées par des bactéries multirésistantes. Le principal mécanisme de résistance aux C3G et aux carbapénèmes est l’acquisition de gènes encodant respectivement des β-lactamases à spectre étendu (BLSE) et des carbapénémases. Plusieurs études ont répertorié les principales β-lactamases responsables des épidémies dans de nombreux pays à savoir les types TEM, SHV et CTX-M pour les bactéries productrices de BLSE, et plus récemment les types ICTX-MP, KPC, NDCTX-M, OXA-48 et VIM pour les bactéries productrices de carbapénémases.
L’Organisation mondiale de la Santé a recommandé des mesures contrôle et prévention et de contrôle afin de réduire le risque de transmission des bactéries à Gram négatif productrices de BLSE ou de carbapénémases. Ces stratégies incluent l’identification des bactéries productrices de BLSE et de carbapénémases par des méthodes diagnostiques rapides de même que la mise en place d’une surveillance épidémiologique des principaux gènes de résistance encodant à ces enzymes.
Ce mémoire de maîtrise se présente en 2 volets. D’une part, le développement d’un essai PCR en temps réel pour la détection rapide et sensible des gènes encodant les principales carbapénémases. D’autre part, la réalisation d’une étude épidémiologique dans deux hôpitaux de la ville de Québec sur les principaux gènes de BLSE chez des souches cliniques résistantes aux céphalosporines de 3e génération. Ces résultats démontrent que ces approches moléculaires pourraient être utilisées dans les laboratoires de microbiologie clinique afin de faciliter la détection et la surveillance des bactéries productrices de BLSE et de carbapénémases.
Abstract
The resistance of Gram-negative bacteria against third-generation cephalosporins (3GC) and carbapenems is a major public health threat worldwide. These antibiotics are used specifically in the treatment of life-threatening infections caused by multidrug-resistant bacteria. The acquisition of genes encoding for extended-spectrum β-lactamases (ESBL) and carbapenemases is the main mechanism of resistance to 3GC and carbapenems, respectively. Several studies have identified the main β-lactamases responsible for epidemics in many countries namely TEM, SHV, and CTX-M for ESBL-producing bacteria, and more recently IMP, KPC, NDM, OXA-48-type, and VIM for bacteria producing carbapenemases.
The World Health Organization has recommended preventives measures to control and reduce the risk of transmission of Gram-negative bacteria producers of ESBL and carbapenemases. These strategies include the identification of ESBL and carbapenemases with rapid diagnostic methods as well as the establishment of epidemiological surveillance of major resistance genes for these enzymes.
This master’s thesis is presented in two parts. On one hand, the development of a real-time PCR assay for rapid and sensitive detection of key genes encoding carbapenemases. On the other hand, an epidemiological study in two hospitals in Quebec City of genes coding for ESBL in clinical strains resistant to third generation cephalosporins. These results demonstrate that these molecular approaches could be used in clinical microbiology laboratories to facilitate the detection and monitoring of ESBL- and carbapenemases-producing bacteria.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux ... xi
Liste des figures ... xiii
Liste des abréviations ... xvv
Dédicace ... xix
Avant-Propos ... xxi
Chapitre 1 Introduction générale ... 1
1. Historique ... 3
Ère pré-antibiotique ... 3
Ère antibiotique ... 4
Impacts globaux ... 5
Infections causées par les bactéries à Gram négatif ... 7
Antibiotiques ... 8
1.5.1 Mode d’action ... 10
1.5.2 β-lactamines ... 11
1.5.2.1 Céphalosporines de 3ième génération ... 14
1.5.2.2 Carbapénèmes ... 14
1.5.2.3 Mode d’action des β-lactamines ... 15
Résistance des bactéries aux antibiotiques ... 17
1.6.1 Résistance naturelle ... 18
1.6.2 La résistance acquise ... 19
1.6.2.1 Structures d'ADN qui assurent la transmission de la résistance aux antibiotiques ... 19
1.6.2.1.1 Plasmides ... 19
1.6.3 Facteurs responsables de l’augmentation de la résistance aux antibiotiques 20
1.6.4 Résistance aux β-lactamines chez les bactéries à Gram négatif ... 23
1.6.4.1 Les β-lactamases ... 24
1.6.4.1.1 Mode d’action ... 24
Classification des β-lactamases ... 25
1.7.1 β-lactamases à spectre étendu ... 29
1.7.1.1.1 TEM ... 30 1.7.1.1.2 SHV ... 30 1.7.1.1.3 CTX-M ... 30 1.7.1.2 Carbapénémases ... 32 1.7.1.2.1 Carbapénémases de classe A ... 33 1.7.1.2.2 Carbapénémases de classe B ... 34 1.7.1.2.3 Carbapénémases de classe D ... 36
Méthode de détermination de la sensibilité à un antibiotique ... 37
1.8.1 Méthodes phénotypiques ... 37
1.8.1.1 La concentration minimale inhibitrice (CMI) ... 38
1.8.1.2 Méthodes phénotypiques pour la détection de la production de BLSE ... 39
1.8.1.2.1 Test de double synergie ... 39
1.8.1.2.2 Bandelettes Etest® BLSE ... 40
1.8.1.2.3 Méthode des disques combinés ... 41
1.8.1.2.4 Méthodes automatisées ... 42
1.8.1.3 Méthodes phénotypiques pour la détection de la production de carbapénémases ... 42
1.8.1.3.1 Test de Hodge modifié ... 43
1.8.1.4 Limitations associées aux méthodes phénotypiques ... 44
1.8.2 Méthodes génotypiques ... 45
1.8.2.1 Réaction de polymérisation en chaîne (PCR) ... 45
1.8.2.1.1 PCR en temps réel ... 45
1.8.2.1.1.1 PCR multiplex en temps réel ... 46
1.8.2.1.1.2 Tests PCR pour la détection des carbapénémases ... 46
Problématique ... 51
Objectifs de recherche ... 52
2.2.1 Objectifs spécifiques ... 52
Chapitre 3 Développement d’un essai PCR multiplex en temps réel pour la détection rapide et sensible des gènes codant pour les carbapénémases ... 55
Abstract ... 61
Introduction ... 62
Materials and methods ... 64
3.3.1 Bacterial strains ... 64
3.3.2 Phenotypic confirmation tests ... 64
3.3.3 DNA extraction and purification from bacterial strains ... 65
3.3.4 Multiplex primer and probe design ... 65
3.3.5 Multiplex PCR for the detection of blaKPC, blaNDM, blaOXA-48-like blaIMP, and blaVIM ... 66
3.3.6 Internal control (IC) ... 66
3.3.7 Validation of the multiplex PCR assay ... 67
3.3.8 Limit of detection (LOD) ... 67
3.3.9 Analytical performance of the MHT and RDS test ... 68
Results ... 68
3.4.1 Analytical performance of the multiplex PCR assay ... 68
3.4.2 Analytical performance of the MHT and RDS test ... 69
Discussion ... 70
Conclusions ... 74
Acknowledgments ... 74
Contributions ... 74
References ... 75
Chapitre 4 Épidémiologie moléculaire des β-lactamases à spectre étendu chez les bactéries à Gram négatif dans deux hôpitaux de la ville de Québec ... 85
Abstract ... 91
Introduction ... 92
4.3.3 Molecular characterization of resistance genes ... 93
4.3.4 PCR detection of the Escherichia coli clonal complex O25b-ST131. ... 93
Detection of ISEcp1 ... 94 4.3.5 ... 94 Results ... 94 Conclusions ... 98 References ... 99 Acknowledgments ... 99
Chapitre 5 Conclusions et perspectives ... 101
Liste des tableaux
Tableau 1. Les postulats de Koch. ... 4
Tableau 2. Micro-organismes producteurs d'antibiotiques. ... 9
Tableau 3. Mécanismes d’action des principaux agents antimicrobiens. ... 10
Tableau 4. Spectre d’activité des principales classes de β-lactamines. ... 13
Tableau 5. Mécanismes de résistance aux antibiotiques. ... 18
Tableau 6. Facteurs contribuant à l'augmentation de la résistance aux antibiotiques. ... 21
Tableau 7. Phénotypes de résistance aux β-lactamines associés à la production des principales BLSE de types SHV, TEM et CTX-M. ... 29
Tableau 8. Phénotypes de résistance aux β-lactamines associés à la production des principales carbapénémases de types IMP, KPC, NDM, OXA-48 type et VIM. ... 33
Tableau 9. Exemples de tests moléculaires rapides visant la détection de gènes de résistance associés à la production de carbapénémases chez les bactéries à Gram négatif...…47
Liste des figures
Figure 1. Inhibition de la croissance d'une culture de Staphylococcus aureus par la
moisissure Penicillium notatum………..5
Figure 2. Structure du noyau β-lactame………...……11
Figure 3. Structures générales des principales classes de β-lactamines………...…………12
Figure 4. Structure de la paroi cellulaire des bactéries à Gram négatif…………...………15
Figure 5. Représentation schématique de la structure du peptidoglycane d’une bactérie à Gram négatif………...………...…………17
Figure 6. Réaction d’inactivation d’une β-lactamine par l’action d’une β-lactamase……..25
Figure 7. Schéma révisée de la classification d’Ambler………...…..26
Figure 8. Répartition mondiale des différents variants de l’enzyme CTX-M………...…..31
Figure 9. Distribution géographique des bactéries productrices de NDM-1……….…...…36
Figure 10. Test de double synergie………...…...….40
Figure 11. Bandelettes Etest® BLSE……….….41
Figure 12. Méthode des disques combinés ……….………42
Figure 13. Test de Hodge modifié……….……..………43
Liste des abréviations
ABPA Acide aminophénylboronique ADN Acide désoxyribonucléique ATCC American Type Culture Collection ATP Adénosine triphosphate BSA Bovine serum albumin
BLSE β-lactamase à spectre étendu
BPC Bactéries productrices de carbapénémases
Caz Ceftazidime
CDC Centers for Disease Control and Prevention (Atlanta, GA)
°C Degré Celcius
CHUL Centre hospitalier de l'Université Laval CLSI Clinical Laboratory Standard Institute
Cla Acide clavulanique
CMI Concentration minimale inhibitrice
Ctx Céfotaxime
CTX-M Céfotaximase-Munich D-Ala-D-Ala D-alanyl-D-alanine DPA Acide dipicolinique EDTA Ethylenediaminetetraacetic acid
EUCAST European Committee for Antimicrobial Susceptibility Testing
FN Faux négatif
FP Faux positif
GES Guiana extended-spectrum I Intermédiaire
IMI Imipénème
IMP Imipénémase
IUCPQ Institut de Cardiologie et de Pneumologie de Québec KCl Chlorure de potassium
MBLs Métallo-β-lactamases MgCl2 Chlorure de magnésium
MIC Minimal inhibitory concentration
min Minute
mL Millilitre
Na+ Ion sodium
NAG N-acétyl-glucosamine
NAM N-acétyl-muramique
NaCl Chlorure de sodium
NCBI National Center for Biotechnology Information NDM New-Deli metallo-β-lactamase
nt Nucléotide
LSPQ Laboratoire de santé publique du Québec
MALDI-TOF Matrix-assisted laser desorption/ionisation-time-of-flight mass spectrometry (Spectromètre de masse couplant une source
d’ionisation laser assistée par une matrice et un analyseur à temps de vol)
MLST Multilocus sequence typing NMC Non-metallo-carbapenemase
OMS Organisation mondiale de la Santé
Pb Paire de bases
PCR Polymerase chain reaction PBP Penicillin-binding protein
R Résistant
RUCLANAP Rapid Universal Cell Lysis and Nucleic Acids Preparation PLP Protéine liant la pénicilline
S Sensible
SHV Sulfhydryl variable SME Serratia marcescens enzyme
TEM Temohiera
USI Unités de soins intensifs UFC Unité formatrice de colonies
μm Micromètre ou micron µM Micromolaire
μg Microgramme
VIM Verona integron-encoded VN Vrai négatif
VP Vrai positif
VPP Valeur prédictive positive VPN Valeur prédictive négative
Avant-propos
Je tiens à communiquer mes plus sincères remerciements à tous ceux qui ont contribué d'une quelconque manière à la réalisation de ce projet, de ce défi personnel. Ces trois dernières années ont été enrichissantes à plusieurs égards, et j'en ressors professionnellement changée et humainement grandie. Ce mémoire est issu d'énormément d'efforts, mais est également empreint de belles journées, de souvenirs impérissables et de beaucoup de rires.
D'abord, j'aimerais souligner ma profonde reconnaissance à mon directeur de recherche, Dr. Michel G. Bergeron. Votre vision et votre passion ont su nourrir mon intérêt afin de pleinement m’investir dans vos recherches. Mon admiration envers vos accomplissements est absolue. Mille mercis de m’avoir offert cette opportunité.
Évidemment, je tiens à remercier profondément ma co-directrice, Dre Ann Huletsky, une femme qui m’a inspirée quotidiennement. J'ai énormément de respect non seulement pour sa carrière professionnelle extraordinaire, mais également pour ses qualités et ses valeurs personnelles. Grâce à elle, j’ai pu approfondir mes connaissances scientifiques, développer un regard critique sur mes recherches et présenter mes travaux de façon claire. Cette formation m’aura certainement permis d’accroître ma confiance en moi et aura assurément établi des bases solides pour ma carrière professionnelle. Merci de m'avoir montré à me surpasser en recherchant toujours l’excellence ainsi que de m'avoir permis de me faire une place dans ce monde scientifique. Je suis reconnaissante de l’énergie et du temps que tu as voués à mon encadrement. J’ai eu beaucoup de plaisir à travailler et j’ai grandement apprécié apprendre de toi.
Nul projet ne serait possible à réaliser sans le soutien de Dominique Boudreau. Ton expérience et tes conseils judicieux ont apporté beaucoup à mon apprentissage. Bien que ta présence contribue à résoudre les différents problèmes qui ne manquent pas de faire surface lors de la réalisation d'un tel projet, c’est ta personnalité qui te distingue avant tout. Tu es
Je désire également remercier Dr. Maurice Boissinot pour son soutien scientifique et sa rigueur qui m’ont permis de développer mon potentiel scientifique. La richesse de l'équipe de diagnostic moléculaire repose sur la diversité des gens et la complémentarité des connaissances. Je tiens également à remercier mes autres collègues, actuels et ancients, Marie-Claude Hélie, Lucile Belley-Montfort, Isabelle Martineau, Catherine Ouellette, Johanne Frenette, Martine Bastien, Ève Bérubé, Luc Bissonnette, Gale Stewart, Martin Gagnon, Jean-Luc Simard, Marthe Bernier, Hélène Morin, Éric Martel, Richard Giroux et Amin Ouameur. Évidemment, une équipe n'est jamais complète sans les autres étudiants. Laurie, merci de ton enthousiasme contagieux, Rana de ton positivisme et Karel de ton écoute. Les liens d'amitiés qui nous unissent sauront toujours franchir les frontières qui nous séparent. Merci Fred pour les midis divertissants remplis de «frederies». J'ai fait de belles rencontres, j’ai eu de belles opportunités, et pour ces raisons, je me sens tellement enrichie aujourd'hui que je recommencerais sans hésiter.
Finalement, je tiens à remercier ma famille pour leur support moral et affectif de même que pour leurs encouragements soutenus dans tous les projets que j'ai entrepris depuis mon tout jeune âge. Merci de m'avoir permis de partager de nombreux moments heureux avec vous et de m'avoir épaulée lors d’épreuves plus difficiles. Mes parents m'ont transmis de belles valeurs, dont la persévérance qui m’a grandement aidée à me rendre jusqu’ici. À travers votre fierté et votre amour, j’ai souvent trouvé la motivation à poursuivre et à me surpasser. Il est difficile de décrire tout ce que vous avez pu m’apporter, mais sachez que j’en suis pleinement reconnaissante. Je remercie aussi Émilie, une grande sœur de qui j'ai tant à apprendre. Tu es un exemple de force et de dévouement. Ayant été mon premier professeur, tu as assurément eu une incidence dans mes accomplissements. Merci à Marc-Antoine de la rendre aussi heureuse. Un merci à ma grande amie Justine qui a été particulièrement présente pour discuter et rire durant ces années. Tes conseils et ta franchise m’ont grandement fait cheminer. Je pense également à Joanny, Catherine, Anne et Stéphanie pour leur amitié sincère et tous ces moments inoubliables.
Chapitre 1
1. Historique
Ère pré-antibiotique
À travers l’histoire, les maladies infectieuses ont été la principale cause de mortalité au niveau mondial. De grandes pandémies ainsi que de multiples épidémies locales, répertoriées depuis l’Antiquité, ont entraîné l’effondrement de plusieurs civilisations (Kardos & Demain, 2013). À cette époque, il était admis que ces phénomènes étaient relatifs aux miasmes, une émanation provenant de la matière organique en décomposition dont la propagation était influencée par différents facteurs environnementaux (Koplan et al, 1999). Ce n’est qu’au 19e siècle, qu’une série d’observations ont ciblé la présence de «germe» en tant qu’agent responsable de ces épidémies. La corrélation entre la présence de microorganismes et le développement de maladies infectieuses a toutefois été formellement établie en 1869 par les travaux du chimiste et physicien français Louis Pasteur (1822-1895). Ses observations relatives à la pébrine lui ont permis de démontrer qu’un protozoaire parasite était l’agent responsable du développement de la maladie chez le verre à soie et que cette dernière pouvait être contrôlée par le biais de chenilles provenant d’œufs pondus par des papillons sains (Prescott et al. 2010). La théorie des germes liant directement les microorganismes aux maladies infectieuses a ainsi entraîné une évolution importante au plan de la santé humaine. Le médecin allemand Robert Koch (1843-1910) a formalisé cette découverte en définissant quatre principes unissant les microorganismes à certaines maladies, connus sous le nom de postulats de Koch. Ces derniers sont brièvement présentés dans le Tableau 1. Dès lors, les recherches dans le domaine médical se sont dirigées vers la découverte des agents causaux des différentes maladies et enfin vers le développement de méthodes permettant d’éliminer ou de contrôler leur présence.
Tableau 1. Les postulats de Koch. Tableau adapté de Prescott et al. (2010).
1. Le micro-organisme doit être invariablement présent lors de la maladie mais absent des organismes sains.
2. Le micro-organisme, qui semble être responsable de la maladie, doit pouvoir être isolé et cultivé en culture pure. 3. La même maladie doit se développer lorsque le micro-
organisme isolé est inoculé à un organisme sain. 4. Le même micro-organisme doit être à nouveau isolé de
l'hôte malade.
Ère antibiotique
Le concept de l’antibiothérapie a été établi par les recherches d’Alexander Fleming (1881-1955) en 1928. En étudiant les propriétés antimicrobiennes des lysosomes, Fleming a remarqué un phénomène inhabituel sur une ancienne plaque de culture de Staphylococcus aureus (Fleming, 1929). Un champignon contaminant, Penicillium notatum, avait inhibé la croissance bactérienne des staphylocoques en libérant un agent antibactérien dans la zone adjacente à sa croissance (Figure 1). Il a ainsi attribué le nom «pénicilline» à ce composé inhibiteur en raison de sa provenance. Suite à cette découverte, Fleming a décrit les conditions nécessaires à la croissance du champignon et a tenté de retrouver cette même production antimicrobienne chez d’autres micro-organismes. Malheureusement, il n'a pu démontrer la valeur thérapeutique de cette nouvelle substance en raison de l’instabilité de la molécule et de la difficulté d'extraction de la forme active (Zaffiri et al, 2012). Ce sont les travaux d’Ernst Chain et d’Howard Florey, en 1940, qui ont mené à l’utilisation thérapeutique de la pénicilline. Ils ont développé une technique afin d'extraire une quantité suffisante de l’agent antimicrobien actif à partir d’une culture microbienne. En effet, ils ont réussi à préparer une formule stable de la pénicilline et démontrer l’efficacité de son activité antibactérienne, en absence de toxicité chez la souris. La pénicilline a ainsi été le premier agent thérapeutique naturel capable de détruire les bactéries in vivo, sans être éliminé dans le
corps, en plus d’être non toxique pour les humains (Chain et al, 1940). En 1945, le prix Nobel de physiologie et médecine a été décerné à Chain, Fleming et Florey pour leur découverte de la pénicilline ainsi que la mise en évidence de son effet curatif envers plusieurs maladies infectieuses (Ligon, 2004). Cette découverte a été la première étape vers le développement de tous les antibiotiques connus à ce jour (Kardos & Demain, 2011) et de ce fait, a mené à la naissance de l’ère antibiotique.
Figure 1. Inhibition de la croissance d'une culture de Staphylococcus aureus par la moisissure Penicillium notatum. Figure adaptée de Prescott et al. (2010).
Impacts globaux
Les bénéfices les plus significatifs au plan de la santé humaine suite à l’introduction des agents antimicrobiens ont été une diminution massive des décès relatifs aux maladies infectieuses. En effet, le taux de mortalité globale est passé de près de 33% à environ 4% entre la fin du 19ème et du 20ème siècle, subséquemment à l’utilisation des antibiotiques (Kardos & Demain, 2011). Pour ces mêmes raisons, l'espérance de vie a bondi de huit ans entre 1944 et 1972 (Kardar, 2005).
L’arrivée d'antibiotiques efficaces a également eu un impact significatif sur l'ensemble de la médecine, permettant, entre autres, la réalisation d'opérations complexes avec un minimum de risque d'infections, l’exécution de diverses procédures invasives et de transplantations d'organes, de même que l’utilisation de chimiothérapies
Colonies normales de Staphylococcus aureus Colonies de Penicillium notatum Zone d’inhibition de croissance de Staphylococcus aureus
notamment permis de réduire le taux d’infections résultant d’une intervention chirurgicale de 40% à 2% (Wallace et al, 2000) et a diminué de 75% la transmission de maladie néonatale lors d’accouchement lorsque combiné avec une antibioprophylaxie intrapartum (CDC, 1996; Davies et al, 2001). Aucun de ces progrès de la médecine n’aurait été possible en absence d’agents antimicrobiens efficaces.
Le succès de l'antibiothérapie a rapidement convaincu de nombreux experts médicaux qu’ils avaient enfin identifié le remède miracle dans le traitement des maladies infectieuses qui affligeaient la population mondiale depuis des milliers d’années. En effet, un sentiment d’optimisme général dans la communauté de l’époque s’est reflété dans la pratique médicale, marqué par une absence de recommandations quant à l’utilisation des antibiotiques.
Toutefois, certains scientifiques craignaient les conséquences négatives potentielles qui pouvaient découler de cette utilisation non-contrôlée. En effet, certaines bactéries ont présenté rapidement leur capacité à détruire l’antibiotique par dégradation enzymatique (Abraham & Chain, 1940). De plus, Alexander Fleming lui-même, avait également mis en garde l’utilisation non contrôlée des antibiotiques:
"The greatest possibility of evil in self-medication is the use of too small doses so that instead of clearing up infection the microbes are educated to resist penicillin and a host of penicillin-fast organisms is bred out which can be passed to other individuals and from them to others until they reach someone who gets a septicaemia or pneumonia which penicillin cannot save."
(New York Times June 26, 1945) Seulement sept décennies plus tard, il semble que l’utilisation excessive et peu contrôlée des antibiotiques a mené à un déclin prémédité de ce remède miracle. Ce mémoire, abordera plus précisément la difficulté thérapeutique actuelle et grandissante des infections causées par des bactéries à Gram négatif ayant acquis une résistance, envers les β-lactamines, la famille d’antibiotiques la plus utilisée en pratique clinique, par la
Infections causées par les bactéries à Gram négatif
Les bactéries à Gram négatif constituent une large catégorie de micro-organismes qui sont incapables de retenir une coloration de cristal violet en raison de la structure distincte de leur paroi cellulaire. Parmi ceux-ci, les entérobactéries de même que les bactéries non-fermentaires forment deux groupes largement responsables de plusieurs maladies infectieuses. Les bactéries regroupées dans la famille des Enterobacteriaceae se retrouvent principalement dans le sol, l'eau, et certaines (incluant Citrobacter sp., Enterobacter sp., Escherichia coli, Klebsiella sp., Proteus sp. et Serratia marcescens) sont des constituants naturels de la flore intestinale de l’homme. Ces micro-organismes sont d’importants pathogènes en milieux hospitaliers (Laurent et al, 2008 ; Piednoir et al, 2011) et communautaires (Arslan et al, 2005 ; Chong et al, 2011 ; Song et al, 2011). Ces derniers se propagent facilement entre humains par le biais des mains, de l’eau et de la nourriture contaminée de même que de sources environnementales (NIH-NIAID, 2012). Les bactéries non-fermentaires sont également répandues dans l’environnement et sont responsables d’infections dites opportunistes. Les principales bactéries à Gram négatif causant des infections sont Escherichia coli et Klebsiella pneumoniae parmi les entérobactéries ainsi que Pseudomonas aeruginosa et Acinetobacter baumannii parmi les bactéries non-fermentaires.
Les infections causées par des bactéries à Gram négatif constituent une préoccupation majeure dans les établissements de soins de santé ainsi que dans la communauté en général. Dans certains cas, les bactéries peuvent pénétrer dans l'organisme par le biais de cathéters, de ventilateurs respiratoires, ou directement dans une plaie. L’entrée de ces micro-organismes peut conduire à une infections respiratoire, une infection des voies urinaires, du site opératoire ou à une septicémie. Ces types d'infections affectent majoritairement les personnes immunosupprimées et les personnes âgées, représentant généralement la clientèle des établissements de soins de santé. De récentes études ont montré que 30 % des infections nosocomiales étaient causées par des bactéries à Gram négatif (NIH, 2009) et que ces dernières prédominaient dans les cas de pneumonies
prévalence de ces mêmes infections a augmenté à près de 70% dans les unités de soins intensifs (USI) (Gaynes et al, 2005).
Les différents agents antimicrobiens développés avec le temps représentent des outils thérapeutiques de choix pour le contrôle des infections causées par ces micro-organismes. Malheureusement, l'incidence de la résistance aux antibiotiques chez les bactéries à Gram négatif a augmenté à un rythme alarmant au niveau mondial au cours des 20 dernières années (Cantòn et al, 2012 ; El Salabi et al, 2013 ; Nordmann et al, 2011 ; Woodford et al, 2011) (Voir section 1.6.4).
Antibiotiques
Un antibiotique (du grec anti, contre et bios, la vie) est une substance chimique naturelle ou synthétique, ayant un mode d’action spécifique contre les bactéries. La majorité des antibiotiques sont des molécules naturelles, produites essentiellement par des bactéries ou certains champignons afin d’éliminer les micro-organismes sensibles (effet bactéricide) ou d’inhiber leur croissance (effet bactériostatique) (Tableau 2) (Kohanski et al, 2010). De plus, il existe des antibiotiques semi-synthétiques, qui sont en fait des antibiotiques naturels modifiés par l’addition de groupements chimiques dans le but de les rendre moins sensibles à l’inactivation par les micro-organismes (Prescott et al, 2010). Les antibiotiques n’ont toutefois aucune action sur les virus ou les champignons. Dans ces cas-ci, on parle plutôt respectivement d’antiviraux et d’antifongiques.
Tableau 2. Micro-organismes producteurs d'antibiotiques. Tableau adapté de Prescott et al. (2010) et Walsh (2003).
Micro-organismes Antibiotiques Bactéries Streptomyces sp. Amphotéricine B Carbapénèmes Chloramphénicol Érythromycine Kanamycine Néomycine Nystatine Rifampicine Streptomycine Tétracyclines Vancomycine Micromonospora sp. Gentamicine Bacillus sp. Bacitracine Mycètes Penicillium sp. Polymyxines Griséofulvine Pénicilline Cephalosporium sp. Céphalosporines
Les antibiotiques sont également classés en fonction de leur champ d’efficacité. D’une part, on retrouve ceux ayant un spectre d’action étroit, c’est-à-dire possédant une efficacité restreinte à une variété de micro-organismes. D’autre part, il y a ceux caractérisés par un large spectre d’action, capable de détruire différents types de micro-organismes. Cette caractéristique donne un avantage significatif à ce type d’antibiotique lorsque l’identification du pathogène responsable de l’infection est inconnue.
sur la flore normale, en perturbant l’équilibre des micro-organismes présents. Les conséquences de cette utilisation seront discutées dans la section 1.6.
1.5.1
Mode d’action
Les mécanismes d’action des agents antimicrobiens se divisent en fonction de la cible de l’antibiotique. Ils agissent notamment en inhibant la synthèse de la paroi cellulaire (peptidoglycane ou membrane externe), des acides nucléiques, des protéines ou sur l’inhibition de voies métaboliques. Le Tableau 3 présente les principaux antibiotiques utilisés en fonction de leur mécanisme d’action. À ce jour, des milliers de molécules d’antibiotiques naturelles ou synthétiques ont été développés. Toutefois, la toxicité de certaines molécules les empêche d’être utilisées en médecine humaine. En somme, une centaine d’antibiotiques sont administrés dans un contexte clinique afin de maîtriser les maladies infectieuses causées par des agents bactériens.
Tableau 3. Mécanismes d’action des principaux agents antimicrobiens. Tableau adapté de Prescott et al. (2010)
Mécanismes d'action Agents antimicrobiens Inhibition de la synthèse de la paroi cellulaire β-lactamines
Vancomycine
Éthionamide et isoniazide Inhibition de la synthèse protéique Aminoglygosides ou aminosides
Tétracyclines Chloramphénicol Macrolides Inhibition de la synthèse des acides nucléiques Rifampicine
Quinolones Inhibition des voies métaboliques Sulfamides
1.5.2
β-lactamines
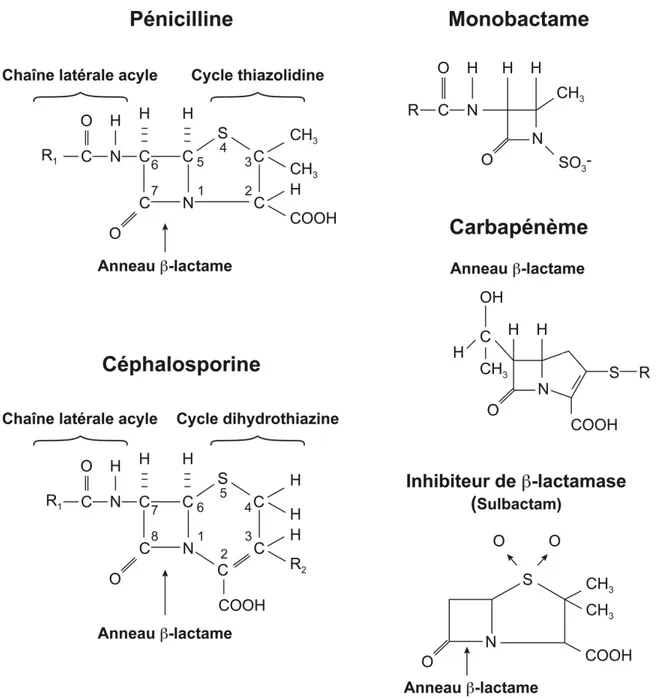
Les β-lactamines, représentent la famille d’antibiotiques la plus utilisée en antibioprophylaxie et en antibiothérapie. L’importance de leur utilisation résulte de leur large spectre d’action, leur faible toxicité, leur efficacité thérapeutique ainsi que le faible coût de certaines molécules (Georgopapadakou, 1993). La résistance bactérienne croissante à cette famille d’antibiotiques sera l’objet de ce mémoire. Les β-lactamines regroupent les pénicillines, les monobactames, les inhibiteurs de β-lactamases, les céphalosporines (1ère à 4ème génération) et les carbapénèmes. Elles se caractérisent par la
présence d’un élément structural commun, soit le noyau β-lactame, nécessaire à l’activité antibactérienne de l’antibiotique (Figure 2).
Figure 2. Structure du noyau β-lactame. Le noyau β-lactame est constitué de trois atomes de carbone et un d’azote. En raison de sa conformation, le cycle β-lactame est fortement tendu. En effet, la liaison entre le groupe carbonyle et l'atome d'azote est instable et rend donc la molécule réactive (Kong, 2010).
La classification des β-lactamines dépend de la chaîne latérale additionnelle ajoutée au noyau β-lactame, induisant essentiellement des différences dans la biodisponibilité de l’antibiotique ainsi qu’une extension du spectre d'activité à l'égard des bactéries à Gram négatif (Mascaretti, 2003). La Figure 3 présente la structure générale des 5 classes de β-lactamines.
Figure 3. Structures générales des principales classes de β-lactamines. Figure adaptée de Wingard et al. (1991).
Le développement des différentes β-lactamines s’est fait en fonction d’une meilleure couverture contre les bactéries à Gram négatif, tel que le démontre le Tableau 4.
Tableau 4. Spectre d’activité des principales classes de β-lactamines. Tableau adapté de Mascaretti (2003).
β-lactamines Spectre d'activité
Pénicillines Actives contre les coques et les bacilles à Gram positif ainsi que les coques à Gram négatif
Monobactames Actives uniquement contre les bacilles à Gram négatif, y compris Pseudomonas aeruginosa
Inhibiteurs de β-lactamases
Actifs contre la majorité des β-lactamases à l'exception des céphalosporinases et des métallo-β-lactamases.
Céphalosporines 1ère génération (C1G)
Actives contre les coques à Gram postif et quelques bactéries à Gram négatif
Céphalosporines 2ème génération (C2G)
Actives contre les coques à Gram postif et ont une action augmentée contre les bactéries à Gram négatif par rapport aux C1G
Céphalosporines 3ème génération (C3G)
Actives contre les bactéries Gram négatif, y compris contre
Pseudomonas aeruginosa. Cette couverture plus large des bactéries à Gram négatif diminue la couverture pour les bactéries à Gram positif Céphalosporines
4ème génération (C4G)
Actives contre certaines bactéries à Gram négatif, y compris Pseudomonas aeruginosa. Elles sont stables à l'action des céphalosporinases.
Carbapénèmes Actives contre toutes les bactéries à Gram négatif, y compris Pseudomonas aeruginosa. Elles sont stables à la plupart des β-lactamases, y compris les BLSE.
Les céphalosporines de 3ème génération (C3G) et les carbapénèmes font partie des β-lactamines ayant le plus large spectre d’action à l’égard des bactéries à Gram négatif. Leur utilisation est spécifique à certaines situations cliniques particulières. En effet, les C3G sont utilisées dans le traitement des infections nosocomiales graves et les carbapénèmes sont utilisées en thérapie empirique ou comme traitement contre des infections causées par des bactéries déjà résistantes aux C3G. Ils représentent donc un traitement de dernière ligne. La résistance bactérienne aux C3G et aux carbapénèmes
limite les possibilités de traitements antimicrobiens ou mène à une impasse thérapeutique. La résistance à ces antibiotiques sera étudiée dans le cadre de ce mémoire.
1.5.2.1 Céphalosporines de 3ième génération
L'introduction de six molécules de céphalosporines de troisième génération (C3G) au début des années 1980 a révolutionné la pratique clinique dans la lutte contre la résistance bactérienne. Les trois principales molécules utilisées sont la céfotaxime, la ceftazidime et le ceftriaxone. Ces agents, comparativement aux céphalosporines de 1ère et 2ème génération, sont caractérisés par une plus grande stabilité contre les enzymes qui inactivent spécifiquement les β-lactamines, soit les β-lactamases (présentées à la section 1.7). Généralement, la sensibilité des bactéries à Gram négatif à l’égard des différentes C3G est très similaire. Exceptionnellement, la ceftazidime a une activité cliniquement significative contre Pseudomonas aeruginosa. Non seulement les C3G sont caractérisées par leur efficacité contre les infections nosocomiales graves, mais ils ont également l'avantage majeur d’avoir peu d'effets néphrotoxiques par rapport aux autres antibiotiques tels les aminosides et les polymyxines (Paterson, 2005).
1.5.2.2 Carbapénèmes
Les carbapénèmes jouent, quant à eux, un rôle crucial dans l’arsenal antibiotique. Parmi les centaines de β-lactamines, les carbapénèmes possèdent le plus large spectre d'activité et la plus grande activité antimicrobienne contre les bactéries à Gram positif et Gram négatif (Papp-Wallace et al, 2011). Ainsi, les carbapénèmes sont devenues des outils essentiels dans le traitement des infections nosocomiales sévères ou acquises dans la communauté (Patel et al, 2013). Les carbapénèmes sont utilisées lors d’un traitement empirique ou dans le traitement d’infections par des bactéries à Gram négatif résistantes aux C3G. Les carbapénèmes sont les seules β-lactamines dont l'efficacité est prouvée dans les infections graves dues aux bactéries productrices de β-lactamases à spectre étendu (BLSE), majoritairement responsables de la résistance aux C3G (Hawkey & Livermore, 2012). Il a été suggéré que cette activité soit due aux effets combinés d’une meilleure pénétration de la paroi bactérienne par une voie d’absorption impliquant la porine OprD, une bonne stabilité à l’hydrolyse de la plupart des β-lactamases et une forte
liaison aux PLPs (Mascaretti, 2003). Cette classe d’antibiotiques comprend le doripénème, l’ertapénème, l’imipénème et le méropénème.
1.5.2.3 Mode d’action des β-lactamines
Le mode d’action des β-lactamines se situe au niveau de l’inhibition de la synthèse de la paroi cellulaire. La composante majeure de cette structure conservée chez toutes cellules bactériennes est le peptidoglycane, élément essentiel pour la stabilisation des membranes cellulaires contre les pressions osmotiques internes élevées. Son intégrité, essentielle à la survie cellulaire, doit donc être maintenue au cours de la croissance et de la division cellulaire. En effet, l’endommagement du peptidoglycane conduit à la lyse bactérienne. Chez les bactéries à Gram négatif, la paroi cellulaire est plus fine que celle des bactéries à Gram positif et présente une structure plus complexe (Figure 4). En effet, les bactéries à Gram négatif possèdent une enveloppe cellulaire constituée d’une membrane cytoplasmique, d'un périplasme et d'une membrane externe. Le peptidoglycane, d’une épaisseur d’environ 5 nm, se situe dans l’espace périplasmique et constitue l’interface entre le milieu extracellulaire et le cytoplasme bactérien.
Figure 4. Schéma représentant la paroi cellulaire des bactéries à Gram négatif. Les bactéries à Gram négatif possèdent une couche plus fine de peptidoglycane entourée d’une membrane externe sur laquelle sont fixés des lipopolysaccharides. Le
peptidoglycane est localisé dans l’espace périplasmique, entre les membranes cytoplasmique et externe. Des porines assurant le passage de petites molécules hydrophiles sont présentes dans la membrane externe.
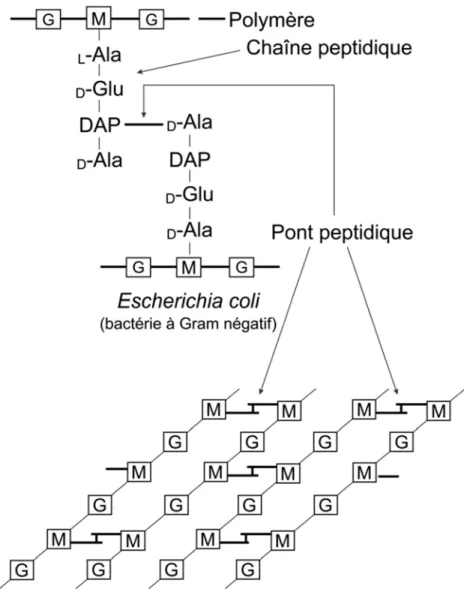
Le peptidoglycane est un polymère composé d’une alternance d’unités d’acide N-acétyl-muramique (NAM) et de N-acétyl-glucosamine (NAG). Ces composés sont reliés entre eux par de courtes chaînes peptidiques constitués, entre autres, d’un dipeptide D-alanine-D-alanine. L’assemblage final de ces unités, processus appelé transpeptidation, est catalysé par des enzymes appelées protéines liant la pénicilline (PLPs), qui sont impliquées dans la biosynthèse de la paroi bactérienne (Figure 5). Leur activité DD-transpeptidase permet la formation des liens peptidiques croisés entre les chaînes de polysaccharides formant le peptidoglycane. Cette catalyse se déroule en deux étapes et débute par l’acylation de l’enzyme à un premier peptide. Lors de la seconde étape, un deuxième peptide est lié au premier par condensation peptidique, libérant ainsi l’enzyme. Les antibiotiques composant la famille des β-lactamines inhibent cette étape finale de la synthèse de la paroi cellulaire à cause de leur homologie structurale avec le substrat des PLPs, soit le D-alanine-D-alanine. Ainsi, le peptidoglycane formé est non fonctionnel et il en résulte une mort cellulaire.
Figure 5. Représentation schématique de la structure du peptidoglycane d’une bactérie à Gram négatif (Brock et al., 1994). La partie supérieure de la figure illustre les liens entre les unités structurales au sein du peptidoglycane d’une espèce de bactéries à Gram négatif (Escherichia coli). L’ensemble des unités sont assemblées suite à un processus catalysé par les PLPs se terminant par le clivage du résidu D-alanine terminal. Abréviations: G : N-acétylglucosamine, M : acide N-acétylmuramique, Ala : alanine, Gly : glycine, Glu : acide glutamique.
Résistance des bactéries aux antibiotiques
La résistance bactérienne se définit par la capacité d’un micro-organisme à se développer en présence d’un agent antimicrobien, dont l’action empêche ou ralentit normalement sa
croissance (NIH, 2009). Ce phénomène de résistance résulte initialement d’un processus normal de sélection naturelle et est à l’origine de la coexistence de bactéries et champignons producteurs d’agents antimicrobiens avec d’autres micro-organismes dans un même environnement. Des gènes responsables de la résistance ont été retrouvés, entre autres, dans différents sites éloignés tel que le sol de l’Alaska, dans les profondeurs de l’océan Pacifique et même dans des sédiments de pergélisol datant de 30 000 ans (Allen et al, 2009; D’Costa et al, 2011; Toth et al, 2010).
La résistance bactérienne envers divers agents antimicrobiens se regroupe selon quatre mécanismes distincts (Tableau 5).
Tableau 5. Mécanismes de résistance aux antibiotiques. Tableau adapté de Carle S. (2009).
Mécanismes Conséquences
Diminution de la perméabilité
membranaire Empêche l'antibiotique d'atteindre sa cible Altération des sites de liaison à
l'antibiotique
Diminution de l’affinité de l’antibiotique pour son site d’action
Pompes à efflux Antibiotique éjecté de la cellule par transport actif
Inactivation enzymatique Production d’une enzyme qui inactive ou détruit l’antibiotique
La résistance aux agents antibactériens peut être une propriété intrinsèque à la bactérie. Dans ce cas précis, on parle de résistance naturelle. Au contraire, elle peut également être acquise soit par mutation de gènes chromosomiques préexistants ou par acquisition de gènes.
1.6.1
Résistance naturelle
La résistance naturelle résulte de la présence d’un ou plusieurs gènes chromosomiques communs à toutes les bactéries d’une même espèce. Ainsi, pour chaque classe d’antibiotiques, il existe des espèces bactériennes pour lesquelles certains antibiotiques
sont inactifs soit en raison de l’absence ou de l’inaccessibilité à la cible, d’une faible affinité pour celle-ci ou de la présence d’enzymes inactivant ces antibiotiques. Par exemple, les β-lactamines sont inactives sur les mycoplasmes par l’absence de paroi cellulaire, le site d’action de cette famille d’antibiotiques. La résistance naturelle est stable et transmise à la descendance lors de la division cellulaire (transmission verticale). Toutefois, elle n’est généralement pas transférable d’une bactérie à l’autre (transmission horizontale). De ce fait, la résistance naturelle ne représente pas une problématique majeure au niveau épidémiologique.
1.6.2
La résistance acquise
La résistance acquise à un antibiotique n’est présente que chez certaines souches d’une espèce donnée. Cette résistance résulte d’une modification génétique par mutation ou de l’acquisition de matériel génétique étranger. Il existe différentes structures d’ADN par lesquels les gènes de résistance aux antibiotiques peuvent être mobilisés horizontalement à savoir les plasmides, les éléments génétiques transposables et les intégrons.
1.6.2.1 Structures d'ADN qui assurent la transmission de la résistance aux antibiotiques
1.6.2.1.1 Plasmides
Les plasmides sont des molécules d'ADN extrachromosomiques capables de réplication autonome, et pouvant conférer une résistance aux principales classes d'antibiotiques (Carattoli, 2009). Une même bactérie peut porter un ou plusieurs plasmides selon leur compatibilité mutuelle et chacun d’eux peut contenir un ou plusieurs gènes de résistance (Harbottle et al., 2006). Les plasmides peuvent également se transmettre à une souche sensible principalement par conjugaison mais aussi par transformation ou transduction. Les plasmides, en fonction de leur efficacité de conjugaison, vont permettre le transfert horizontal de ces composantes à d’autres bactéries de différentes espèces, genres et familles (Thomas & Nielsen, 2005). Les plasmides peuvent acquérir d’autres gènes de résistance par l’intermédiaire d’éléments transposables et d’intégrons par exemple. La dissémination dans les milieux hospitaliers ou dans la communauté de la résistance aux
β-lactamines chez les bactéries à Gram négatif est largement attribuée à ce type de transmission.
1.6.2.1.2 Éléments génétiques transposables
Les éléments transposables sont des séquences d’ADN ayant la capacité de se déplacer et de s’insérer à différents endroits dans un chromosome ou un plasmide par un processus de transposition. Il existe différentes types d’éléments transposables dont les séquences d’insertion (SI) et les transposons. Les SI sont de courtes séquences génétiques de 800 à 2000 pb qui codent uniquement pour la transposition. Les transposons codent également pour les déterminants de la transposition ainsi que pour d’autres fonctions, notamment celle de conférer la résistance aux antibiotiques (El Salabi et al., 2013).
1.6.2.1.3 Intégrons
L'ampleur de la diffusion de la résistance est attribuable en majeure partie par le déplacement des gènes de résistance par le biais d’intégrons (Dominques et al. 2012). Les intégrons sont des éléments génétiques mobiles avec une structure spécifique composée de deux segments conservés encadrant une région centrale appelée « cassette » (Dominques et al. 2012 ; Recchia & Hall, 1997). Les intégrons constituent un système de capture et d’expression de gènes, dont les gènes de résistance, sous forme de cassettes. Les cassettes sont des éléments mobiles capables d’être intégrés ou excisés par recombinaison spécifique. Les intégrons peuvent être associés à des SI, des transposons ou des plasmides pouvant leur conférer une mobilité (Dominques et al. 2012). Les intégrons de classe 1 sont dominants parmi les quatre classes associées à la résistance aux antibiotiques, et les plasmides sont des vecteurs importants pour leur transmission entre les bactéries dans l'environnement hospitalier (Espedido et al., 2005).
1.6.3
Facteurs responsables de l’augmentation de la résistance aux
antibiotiques
Tel que mentionné, la résistance aux antibiotiques est un phénomène naturel relié à l’évolution des espèces bactériennes. Toutefois, une variété de facteurs
environnementaux contribue également à l'émergence et la propagation de la résistance. Ceux-ci sont résumés dans le Tableau 6.
Tableau 6. Facteurs contribuant à l'augmentation de la résistance aux antibiotiques. Tableau adapté de Carle S. (2009) et Knobler et al. (2003).
Facteurs contribuant à l'augmentation de la résistance aux antibiotiques
1. Pression sélective exercée par l'utilisation d'antibiotiques 2. Sur-utilisation ou utilisation innapropriée des antibiotiques 3. Utilisation massive des antibiotiques en milieu hospitalier 4. Dissémination internationale de clones épidémiques résistants 5. Utilisation des antibiotiques dans l'industrie agroalimentaire
D’abord, les bactéries se caractérisent par une capacité d’adaptation à leur environnement, notamment par leur reproduction rapide, la fréquence de mutation génétique aléatoire et leur capacité à s’échanger du matériel génétique entre elles. L’utilisation d’antibiotiques exerce une pression de sélection sur les populations bactériennes en favorisant la survie des bactéries présentant une ou des mutations conférant une résistance aux antibiotiques ou ayant acquis un ou des gènes de résistance à ces antibiotiques par l’entremise d’éléments génétiques mobiles. Celles-ci continuent à se reproduire, en transmettant à leur descendance leurs gènes de résistance, produisant rapidement une génération de bactéries majoritairement résistantes (Hamilton-Miller, 2004; Walsh, 2003). Des bactéries dites compétentes sont aussi capables d’intégrer de l’ADN exogène présent dans le milieu extérieur et ainsi acquérir potentiellement des gènes de résistance aux antibiotiques provenant d’autres espèces bactériennes. Elles peuvent ensuite transmettre à leur descendance leurs gènes de résistance lors de la réplication produisant ainsi une population de bactéries résistantes à l’agent antibactérien utilisé.
En second lieu, la surutilisation ou l’utilisation inappropriée des antibiotiques constitue un facteur important dans l’augmentation de la résistance. Dans les pays développés, les pratiques médicales inappropriées sont souvent favorisées par l'incertitude du diagnostic, le manque d'opportunité pour le suivi des patients, le manque de connaissances sur les traitements optimaux ou suite à la demande du patient (Knobler et al, 2003). Les outils diagnostiques actuels reposent sur une identification phénotypique appuyée principalement sur la culture du micro-organisme. Le temps nécessaire pour l’identification ainsi que la détermination de la sensibilité de la souche envers différents antibiotiques varie entre 48 et 72 heures. Les cliniciens vont donc fréquemment prendre des décisions relatives au traitement sans connaître l’agent étiologique qui causent l’infection. En absence d’informations, un agent antibactérien à large spectre est souvent prescrit dans le cas où un antibiotique plus spécifique ou même aucun antibiotique, aurait représenté un meilleur choix. En effet, environ 25% des prescriptions d’antibiotiques sont données dans le traitement d’infections d’origine virale (Leekha et al, 2013). Dans le cas des pays en voie de développement, ces problèmes surviennent généralement en raison d’agents antimicrobiens disponibles sans prescription médicale (Knobler et al, 2003). Certains comportements des patients jouent également un rôle dans l’émergence de la résistance, notamment l’automédication et le non-respect des traitements recommandés. Le non-respect survient lorsque les patients oublient de prendre leurs médicaments, interrompent le traitement prématurément lorsque leur état s’améliore ou dans le cas où ils ne peuvent se permettre un traitement complet. Quant à l'automédication, elle s’avère presque toujours soit inutile, inadéquate ou avec un dosage inapproprié. Dans certains pays, les problèmes de non-conformité et d’automédication sont amplifiés en raison de la mauvaise synthèse d'antimicrobiens ou l’expiration de la durée de vie efficace de ceux-ci (Knobler et al, 2003).
Diverses pratiques courantes dans les hôpitaux contribuent aussi au problème de résistance aux antibiotiques. Les hôpitaux sont, en effet, des milieux particulièrement fertiles pour la propagation des microbes résistants. On y retrouve un grand nombre de patients, dont plusieurs avec un système immunitaire affaibli, en contact étroit avec les autres patients, et traités avec une thérapie antimicrobienne intensive et prolongée. La
transmission des micro-organismes résistants aux antimicrobiens chez les patients peut être aéroportée, à partir d'une source ponctuelle, tel du matériel contaminé, ou par contact direct ou indirect avec un environnement contaminé ou les mains contaminées du personnel (Larson, 2007). Certaines clones transfèrent plus facilement leurs éléments génétiques mobiles et portent aussi souvent des facteurs de virulence qui facilite leur diffusion épidémique et contribue à l’augmentation de la résistance à grande échelle (Johnson et al, 2009).
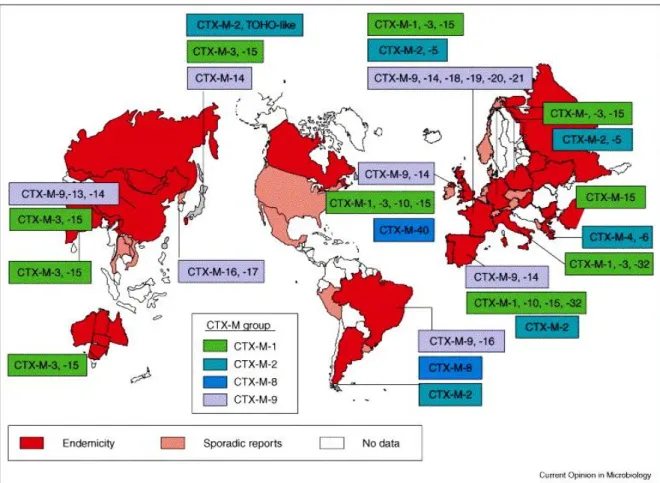
La fréquence de voyages internationaux, le commerce et même le tourisme médical ont contribué à la dissémination de la résistance aux antimicrobiens en permettant aux microbes résistants de se répandre dans le monde avec facilité (Hill, 2001). Plusieurs pays sont endémiques pour certains micro-organismes résistants et sont une source centrale comme il sera abordé dans la section 1.7.1.
Finalement, en dehors d'un usage médical, l'utilisation de divers agents antimicrobiens chez les animaux, que ce soit pour usage thérapeutique ou comme facteur favorisant la croissance, peut conduire au développement de micro-organismes résistants aux agents antimicrobiens qui sont ensuite transmis à l'homme, le plus souvent par le biais des produits alimentaires (Vidaver, 2002).
1.6.4
Résistance aux β-lactamines chez les bactéries à Gram négatif
L‘ensemble des facteurs précédemment présentés a induit une expansion du phénomène de résistance aux antibiotiques au point où nous faisons actuellement face à un problème répandu de multirésistance aux antibiotiques. Cette problématique est d’autant plus menaçant considérant le nombre très limité de nouveaux agents antimicrobiens qui sont en développement (Boucher et al., 2009). Il y a quelques années l’importance de la résistance étaient principalement concentrée chez les bactéries à Gram positif tel que pour SARM (Staphylococcus aureus résistant à la méthicilline) et ERV (Entérocoques résistants à la vancomycine) (CDC, 2103). Toutefois, la résistance aux bactéries à Gram négatif a pris de l’importance les dernières années. En effet, l’Infectious Diseases Society of America a récemment relevé six bactéries regroupées sous le nom « ESKAPE » dontEnterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa et les espèces d’Enterobacter qui sont responsables des deux-tiers des infections acquises en milieu hospitalier et dont les deux-tiers sont des bactéries à Gram négatif (Boucher et al., 2009). De fait, l’OMS identifie ce phénomène comme une menace de santé publique majeure (WHO, 2009-2010). Les antibiotiques de la classe des β-lactamines, qui représentent à eux seuls 60% des prescriptions d’antibiotiques notamment utilisées dans le traitement des infections causées par les bactéries à Gram négatif (voir section 1.5.2) n’échappent pas à cette réalité, Le taux de mortalité associé à une infection bactérienne par une souche à Gram négatif résistante aux β-lactamines est de 2 à 4 fois plus élevé que celui associé à une infection causée par une souche sensible (ReAct, 2012). La résistance aux β-lactamines chez les bactéries à Gram négatif peut être causée par plusieurs mécanismes décrits de manière générale dans le Tableau 5 mais est majoritairement causée par un mécanisme d’inactivation enzymatique, soit par l’hydrolyse du noyau β-lactame par l’action d’une β-lactamase (Bush et Jacoby, 2010). Au cours des 70 dernières années, l'utilisation des β-lactamines, incluant les nouvelles β-lactamines comme les C3G et les carbapénèmes, ayant un spectre d’action de plus en plus large envers les bactéries à Gram négatif, a sélectionné des enzymes de type β-lactamases, chacune plus puissante que la précédente.
1.6.4.1 Les β-lactamases
Le terme β-lactamase se réfère aux enzymes produites par les micro-organismes capables d’hydrolyser le noyau β-lactame. À ce jour, plus de 1300 β-lactamases ont été identifiées (Bush, 2013). Chez les bactéries à Gram négatif, les gènes codant pour ces enzymes sont soit d’origine chromosomique ou plasmidique.
1.6.4.1.1 Mode d’action
Les β-lactamases sont des enzymes bactériennes qui agissent en hydrolysant la liaison amide du cycle β-lactame des antibiotiques de la classe des β-lactamines formant ainsi un acyl-enzyme, qui sera dégradé en acide inactif (Figure 6). Cette action est irréversible, ce qui entraîne donc la perte complète de l’activité antibactérienne de l’antibiotique. Les β-lactamases sont sécrétées dans l’espace périplasmique chez les bactéries à Gram négatif.
Cette localisation particulière permet aux enzymes d’agir directement sur les β-lactamines, avant que celles-ci n’aient le temps de rencontrer leurs cibles, les PLPs, situées dans la membrane cytoplasmique. Les β-lactamases ont une vitesse d’hydrolyse très efficace, soit de 1000 cycles β-lactame par seconde, comparativement aux PLPs ayant une capacité d’hydrolyse d’un cycle β-lactame par heure (Ghuysen, 1990; Massova & Mobashery, 1998; Suvorov et al, 2007).
Figure 6. Réaction d’inactivation d’une β-lactamine par l’action d’une β-lactamase. L’ouverture du cycle à quatre membres rompt la fonction amide imitant la chaîne peptidique (D-alanine-D-alanine) formant les liaisons dans le peptidoglycane.
Classification des β-lactamases
Les β-lactamases peuvent être divisées selon 2 classifications soit celle d’Ambler, basée sur la structure primaire des enzymes (Ambler, 1980) ou celle élaborée par Bush-Jacoby-Meideros qui repose sur les caractéristiques fonctionnelles des enzymes (Bush et al, 1995 ; Bush et Jacoby, 2010, http://www.lahey.org/Studies/). Les quatre classes moléculaires (A, B, C et D) possèdent leurs propres caractéristiques relativement à la séquence primaire de chaque β-lactamase. En contrepartie, les classes fonctionnelles particulières à une enzyme présente l’information utile au clinicien pour la prescription d’une antibiothérapie efficace selon leur profil de substrat et d’inhibiteurs.
La Figure 7 présente les quatre principales classes moléculaires de β-lactamases (A-D), qui sont divisées en groupes fonctionnels.
Figure 7. Schéma des classes moléculaires et fonctionnelles des principales β-lactamases. Adapté de Bush. (2013). Les classes A, C et D présentent une homologie structurale suffisante pour affirmer qu’ils descendent d'un ancêtre commun. Les MLBs hydrolysent le cycle β-lactame par un processus enzymatique qui est nettement différent de celui des β-lactamases à sérine active. Les MBLs sont donc regroupées séparément (classe B). Les groupes fonctionnels se distinguent par un profil de substrats spécifiques à l’enzym et possèdent un profil d’inhibition particulier. AC : Acide clavulanique Cb : Carbapénèmes; Cf : Céphalosporines de 1ère et 2ème génération; EDTA :
β-lactamases
Sérine
Site actif Métallo (Zn)
C A D B Classe moléculaire Principaux sous-groupes fonctionnels 1 2 2d 3 Groupe fonctionnelle 1 1e 2a 2b 2be 2br 2f 2de 2df 3a 3b Enzymes ou familles d’enzymes représentatives Profil d’inhibition -- AC EDTA -- -- -- + + + -- +/- +/- +/- -- ---- ---- -- -- -- -- + + AmpC CMY GC1 PC1 TEM-1SHV-1 CTX-M BLSEs (TEM, SHV) CphA IMP, VIM, NDM OXA-23 OXA-48 OXA-11 OXA-15 KPC SME IRT SHV-10 Substrats connus Cf Cf Cse Pn PnCf Cse, MPn, Cf Pn Pn, Cf, Cb, Cse, M Pn, Cse, M Pn Cb Pn, Cf Cb, Cb
Ethylenediaminetetraacetic acid; Cse : Céphalosporines à spectre étendu; M : Monobactames; Pn : Pénicillines.
Les enzymes de la classe A, C (β-lactamases AmpC) et D (β-lactamases OXA) agissent par un mécanisme de sérine active. Les enzymes de la classe B, également appelées métallo-β-lactamases (MBLs), contiennent un ion zinc (Zn2+) dans leur site actif pour catalyser l’hydrolyse du cycle β-lactame. Les trois classes de β-lactamases à sérine active ont évolué indépendamment des MBLs. Au niveau moléculaire, les MLBs sont un groupe hétérogène de protéines, classées en trois sous-groupes soit B1, B2 et B3, selon les similitudes de séquence et la spécificité des substrats (Galleni et al. 2001) (Garau et al. 2004). La caractéristique structurale particulière des sous-classes B1 et B3, est la présence de deux ions Zn2+ impliqués dans l’hydrolyse des β-lactamines, tandis que la sous-classe B2 utilise un seul ion Zn2+ pour l'activité enzymatique maximale (Valladares et al., 1997). La sous-classe B2 a un spectre étroit de substrat limité aux carbapénèmes (Valladares et al., 2000), tandis que les sous-classes B1 et B3 ont des spectres de substrat plus larges, où la sous-classe B3 montre une activité préférentielle pour les céphalosporines (Bush & Fisher, 2011; Thomson, 2010). La sous-classe B1 contient pour sa part les enzymes IMP, VIM et NDM, codés par des gènes localisés sur des éléments génétiques mobiles. Ces derniers sont majoritairement responsables de la diffusion des MBLs. Une différence structurale majeure de l’enzyme NDM-1 a nécessité la division de la sous-classe B1 en sous-groupes B1a et B1b. La β-lactamase NDM-1 est classée dans le groupe B1b tandis que les β-lactamases IMP et VIM sont classées dans le sous-groupe B1a.
La classe moléculaire C est associée à deux sous-groupes fonctionnels, 1 et 1e, qui ont la capacité d’hydrolyser efficacement les céphalosporines de première et deuxième génération et sont peu affectés par les inhibiteurs de β-lactamases (Bush & Jacoby, 2010). Les classes moléculaires A et D regroupent la classe fonctionnelle 2, constitué de plusieurs sous-groupes qui englobent l'hydrolyse globale de toutes les β-lactamines. Parmi ces sous-groupes, les sous-groupes 2be et 2f sont composés d’enzymes responsables d’une importante diffusion de la résistance aux β-lactamines à l’échelle mondiale. Le sous-groupe 2be comprend les enzymes capables d’hydrolyser les
pénicillines et l’ensemble des céphalosporines (de la 1ère à la 4ème génération). Elles sont inhibées par l’acide clavulanique mais sont insensibles à l’EDTA. Ce profil de résistance correspond aux BLSE, majoritairement représentés par les enzymes de type CTX-M et certains de type SHV et TEM. Le sous-groupe 2f comprend les enzymes capables d’hydrolyser les pénicillines, l’ensemble des céphalosporines (de la 1ère à la 4ème génération) et les carbapénèmes. Elles sont inhibées par l’acide clavulanique mais présentent des niveaux variables de sensibilité à l’EDTA. L’enzyme de ce sous-groupe d’une importance au niveau épidémiologique est KPC. La classe moléculaire D est composé de deux sous-groupes fonctionnels soit 2de et 2df. Le sous-groupe 2de comprend les enzymes capables d’hydrolyser les pénicillines et les céphalosporines à large spectre (3ème et 4ème génération) tandis que les enzymes du sous-groupe 2df hydrolysent les pénicillines et les carbapénèmes. Dans les deux cas, elles sont inhibées par l’acide clavulanique mais présentent des niveaux variables de sensibilité à l’EDTA. Bien que ces β-lactamases soient souvent sous-estimées, elles provoquent une résistance sérieuse chez les bactéries telles qu’Acinetobacter baumannii et Pseudomonas aeruginosa (Walther-Rasmussen & Høiby, 2006).
La classe moléculaire B est composé de 2 sous-groupes fonctionnels soit 3a et 3b. Le sous-groupe 3a est caractérisé par une résistance aux pénicillines, à l’ensemble des céphalosporines (de 1ère à la 4ème génération) et aux carbapénèmes. Ce profil de résistance correspond aux principales carbapénémases du type IMP, NDM et VIM. Les enzymes du sous-groupe 3b hydrolysent, pour leur part, les carbapénèmes. Dans les deux cas, les enzymes de ces sous-groupes sont insensibles à l’acide clavulanique mais sont inhibées par l’EDTA (Bush & Jacoby, 2010).
Relativement à l’importance épidémiologique de certaines enzymes à l’échelle mondiale de même que leur importance clinique, mon projet de maîtrise porte principalement sur les BLSE et les carbapénémases.
1.7.1
β-lactamases à spectre étendu
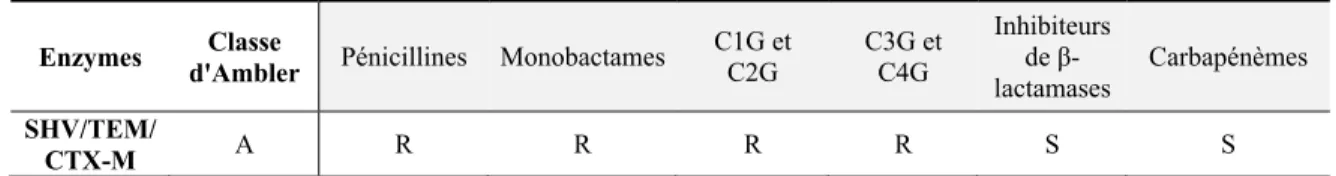
Les infections causées par des bactéries productrices de BLSE sont associées à un taux d’échec thérapeutique et de mortalité élevé (Oteo et al, 2010; Tumbarello et al, 2006). Les BLSE sont des enzymes à large spectre d’action capables d’hydrolyser les pénicillines, toutes les générations de céphalosporines ainsi que les monobactames et sont inhibés par l’acide clavulanique. En contrepartie, les BLSE n’ont aucune action sur la céphamycine (β-lactamines différant des céphalosporines par la présence d’un groupement 7α-methoxy) et les carbapénèmes. Le Tableau 7 présente le profil de résistance aux β-lactamines conféré par les principales BLSE. Les méthodes phénotypiques de détection de production de BLSE sont majoritairement basées sur la résistance aux C3G et de la sensibilité aux inhibiteurs de β-lactamases (Section 1.9.1.2). Tableau 7. Phénotypes de résistance aux β-lactamines associés à la production des principales BLSE de types SHV, TEM et CTX-M.
Tableau adapté de Livermore et al. (2012).
Des bactéries productrices de β-lactamases de types TEM-1, TEM-2 et SHV-1 caractérisées par leur capacité à hydrolyser les pénicillines et les céphalosporines de première et deuxième génération ont été identifiées au début des années 1980. Afin de traiter les infections causées par ces bactéries résistantes, les céphalosporines de 3e génération ont été introduites en pratique clinique (Grall et al, 2011). Toutefois, subséquemment à l’utilisation des C3G, une première bactérie productrice de BLSE de l’espèce Klebsiella pneumoniae a rapidement été identifiée en Allemagne (Knothe et al, 1983). Les premières BLSE répertoriées étaient issues de substitutions d'acides aminés dans le site actif des enzymes TEM-1, TEM-2 et SHV-1. Jusqu’à la fin des années 1990, la majorité des BLSE de types SHV et TEM étaient responsables d’infections
Profil de résistance aux β-lactamines conféré par les BLSE
Enzymes d'Ambler Classe Pénicillines Monobactames C1G et C2G C3G et C4G Inhibiteurs de β-lactamases
Carbapénèmes
SHV/TEM/