T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Microbiologie
"microorganismes, du génome aux interactions avec l'hôte"
JURY
François-Xavier Barre, Directeur de recherche, CNRS, Gif-sur-Yvette Marie-Agnès Petit, Chargée de recherche, INRA, Jouy-en Josas
Jean-Yves Bouet, Chargé de recherche, CNRS, Toulouse
David Lane, Directeur de recherche, CNRS, Toulouse (membre invité) Claude Gutierrez, Professeur, Université Paul Sabatier de Toulouse
Ecole doctorale : Ecole doctorale Biologie-Santé-Biotechnologies de Toulouse Unité de recherche : UMR5100
Directeur(s) de Thèse : Jean-Yves Bouet,David Lane Rapporteurs : François-Xavier Barre,Marie-Agnès Petit Présentée et soutenue par Jean-Philippe Castaing
Le 18 septembre 2009
Titre : La ségrégation du plasmide F d'Escherichia coli : étude du rôle de la fixation de l'ATPase SopA à l'ADN
Doctorat de l’Université de Toulouse délivré par
l’Université Toulouse III - Paul Sabatier
U.F.R. Sciences de la Vie et de la Terre
THÈSE
Pour obtenir le grade de
DOCTEUR DE L’UNIVERSITÉ TOULOUSE III
Discipline : Microbiologie
« Microorganismes, du génome aux interactions avec l’hôte »
Présentée et soutenue par Jean-phillipe CASTAING Le 18 SEPTEMBRE 2009 à 14h00
La ségrégation du plasmide F d’Escherichia coli :
Étude du rôle de la fixation de l’ATPase SopA à l’ADN
JURY
François-Xavier Barre, Chargé de recherche, CNRS, Gif-sur-Yvette Marie-Agnès Petit, Chargée de recherche, INRA, Jouy-en-Josas Jean-Yves Bouet, Chargé de recherche, CNRS, Toulouse David Lane, Directeur de recherche, CNRS, Toulouse
Claude Gutierrez, Professeur, Université Toulouse Paul Sabatier
Recherches effectuées au Laboratoire de Microbiologie et Génétique Moléculaires UMR 5100, CNRS
Bâtiment IBCG 118 route de Narbone 31 062 Toulouse cedex 4, France
Liste des abréviations
ABREVIATIONS
ADN Acide Deoxy-ribo-Nucléique ADP Adenosine Diphosphate ARN Acide Ribo-Nucléique
ARNm ARN messager
ARNt ARN de transfer
ATP Adenosine Triphosphate CFP Cyan Fluorescent Protein
CHUB Condensed nucleoïd, Hypertrophic, Undividing Bacteria ctRNA s coutertranscribed RNAs
Da Dalton
DLS Dynamic Light Scattering
dNTP désoxyribonucléotides Tri-phosphate
DO Densité Optique
DRS Discriminator recognition sequence
DTT Dithiothreitol
EDTA Acide éthylène-diamine-tétraacétique FISH Fluorescent In Situ Hybridation
FROS Fluorescent Repressor-Operator System FPLC Fast Protein Liquid Chromatography
GDP Guanosine Diphosphate
GFP Green Fluorescent Protein GTP Guanosine Triphosphate
HTH Helix Turn Helix
IHF Integration Host Factor
IPTG Isopropyl β-D-1-thiogalactopyranoside
kb kilobase
Kd Constante de dissociation
kDa Kilodalton
KOPS FtsK Orienting Polar Sequence
LB Luria-Bertani
mol molécule
NAPs Nucleoïd Associated Proteins oriC origine de réplication
PAGE polyacrylamide Gel Electrophoresis
pb paires de bases
PCR Polymerase Chain Reaction PLR Polar Localization Region
PNPase polynucléotide phosphorylase
pRNA ARN primer
RHH Ribbon-Helix-Helix
SDS-PAGE SulfateDodécylique Sodium- PolyAcrylamide Gel Electrophoresis SMC Structural Maintenance of Chromosome
SPR Surface Plasmon Resonance
TA Toxine-Antitoxine
TopoIV Topoisomérase IV
UV Ultra Violet
Xgal 5-bromo-4-chloro-3-indolyl-beta-D-galactopyranoside YFP Yellow Fluorescent Protein
Liste des figures et tableaux
LISTE DES FIGURES ET TABLEAUX
Introduction
Chapitre II
Figure 1 : Exemple de plasmides dont la réplication est contrôlée par des itérons. 12 Figure 2 : Exemple de plasmides dont la réplication est contrôlée par des ARN antisens. 13 Figure 3 : Contrôle de la réplication du plasmide F : mécanisme de titration de RepE et de
«handcuffing ». 15
Figure 4 : Modèle topologique de la recombinaison au site cer. 17
Figure 5 : Modèle de recombinaison spécifique de site Cre-loxP. 17
Figure 6 : Représentation schématique du système d’addiction des plasmides, exemple du
système ccd du plasmide F. 18
Chapitre III
Figure 7 : Classification des systèmes de partition. 19
Figure 8 : Localisation intracellulaire du plasmide mini-F par hybridation in situ (FISH). 21 Figure 9 : Suivi dans le temps de la localisation intracellulaire du plasmide mini-P1. 22
Figure 10 : Filaments de ParM et d’actine F. 24
Figure 11 Le modèle de partition du plasmide R1 par le mécanisme de « pushing ». 25 Figure 12 : Représentation schématique de l’interaction entre ParR-parC et les deux
extrémités d’un filament de ParM. 25
Figure 13 : Le modèle de la polymérisation de ParM en « marche d’escalier ». 26
Figure 14 :Diversité des régions centromériques plasmidiques. 27
Figure 15 :Les modèles d’incompatibilité de partition. 28
Figure 16 : Région cristallisée (142-334) de ParB portant les domaines de liaison à l’ADN. 30 Figure 17 : Structure de dimère de ParB (142-334) se fixant à l’ADN. 30 Figure 18 :Représentation schématique de la protéine SopB du plasmide F. 31 Figure 19 : Effet hypothétique de la fixation des protéines SopB sur la topologie de l’ADN. 31 Figure 20 : Modèle de l’étalement des protéines de type ParB autour du site centromérique. 33
Liste des figures et tableaux Figure 21 : Visualisation par microscopie électronique d’événements d’appariement. 33 Figure 22 : Stimulation de la formation de dimères par des protéines SopB. 34 Figure 23 :Les motifs ATPases de type Walker de SopA du plasmide F et ParA du plasmide
P1. 35
Figure 24 : Les régions promotrices des opérons sop du plasmide F et par du plasmide P1. 36 Figure 25 : Modifications structurales de ParA de P1, induites par la fixation d’ADP. 37 Figure 26 : Visualisation par microscopie à fluorescence de l’oscillation de ParA-GFP du
plasmide pB171. 39
Figure 27 : Visualisation par microscopie à fluorescence de l’oscillation de SopA-GFP du
plasmide F. 39
Figure 28 : Dynamique des protéines SopA et SopB au sein de la cellule. 40 Figure 29 : Modèle de partition du plasmide F par « réaction-diffusion ». 40 Figure 30 : Dynamique des protéines SopA et SopB au sein de la cellule. 40
Figure 31 : Modèle de partition du plasmide F par « balayage ». 40
Figure 32 : Modèle de l’oscillation des protéines Min. 41
Figure 33 : Polymérisation in vitro de MinD. 41
Figure 34 : Polymérisation in vitro des ATPases plasmidiques de type Ib. 41
Figure 35 : Polymérisation in vitro de SopA du plasmide F. 42
Figure 36 : Modèle de la régulation de la polymérisation de SopA. 44
Chapitre IV
Figure 37 : Structure schématique des protéines SMC. 47
Figure 38 : Représentation schématique des macrodomaines du chromosome d’Escherichia
coli. 49
Figure 39 : Distribution asymétrique des chromosomes d’Escherichia coli dans une cellule
sur le point de se diviser. 50
Figure 40 : Les étapes clés du cycle de sporulation de Bacillus subtilis. 51
Figure 41 :Ségrégation du chromosome dans la préspore. 52
Figure 42 : Modèle de la régulation de l’entrée en sporulation. 52
Figure 43 : Les étapes du cycle cellulaire de Streptomyces coelicolor. 53 Figure 44 : Les étapes du cycle cellulaire de Caulobacter crescentus. 53 Figure 45 : Positionnement des séquences parS sur le chromosome de B. subtilis. 54
Liste des figures et tableaux Figure 46 : Modèle de la coopération entre les systèmes de partition et les protéines SMC. 56 Figure 47 : Modèle de ségrégation des chromosomes lors de la phase de sporulation de
Streptomyces coelicolor. 58
Figure 48 : Modèle de ségrégation des chromosomes de Caulobacter crescentus. 59
Figure 49 : Modèle de ségrégation du chromosome I Vibrio cholerae. 59
Figure 50 : Structure de Soj de Thermus thermophilus. 60
Figure 51 : Interaction entre les systèmes de partition et de la division cellulaire chez C.
crescentus. 62
Figure 52 : Interaction entre les systèmes de partition et de la division cellulaire chez B.
subtilis. 63
Figure 53 : Division et ségrégation de la protéine MreB. 66
Figure 54 : Rôle de la protéine MreB dans l’activation de la protéine TopoIV. 67
Résultats
Figure 55 : Représentation schématique et mutagenèse de la protéine SopA. 73 Figure 56 : Expression des protéines SopA produites en contexte mini-F. 73 Figure 57 : Capacité de répression et de fixation aux séquences promotrices sopOP des
mutants SopA. 74
Figure 58 : Fractions d’ADN non spécifique fixées en fonction de la concentration de la
protéine SopA. 74
Figure 59 : Suivi de la polymérisation de SopA par Dynamic light scattering. 74 Figure 60 : Test de stabilité du plasmide F porteur des mutations de sopA. 74 Figure 61 : Identification de candidats supplémentaires impliqués dans la reconnaissance de
l’ADN par SopA. 78
Figure 62 : Propriétés in vivo des mutants SopA. 78
Figure 63 : Capacité de fixation aux séquences promotrices sopOP des mutants de SopA. 79 Figure 64 : Fractions d’ADN non spécifique fixées en fonction de la concentration de SopA.
. 79
Figure 65 : Suivi de la polymérisation des mutants de SopA par Dynamic light scattering. 80 Figure 66 : Les motifs ATPases de type Walker des ATPases de type I. 83
Liste des figures et tableaux
Figure 68 : Hydrolyse de l’ATP par les protéines SopA. 85
Figure 69 : Fixation de l’ATP par les protéines SopA. 86
Figure 70 : Capacité de fixation à l’ADN spécifique des protéines SopA. 87 Figure 71 : Fractions d’ADN non spécifique fixées en fonction de la concentration de SopA.
87 Figure 72 : Analyse de l’oligomérisation des protéines SopA par cross link. 88 Figure 73 : Suivi de la polymérisation des mutants de SopA par Dynamic light scattering.
88
Discussion
Figure 74 : Représentation schématique du mécanisme de réaction-diffusion / balayage. 93
Figure 75 : « vagues » des protéines Min in vitro. 94
Matériel et Méthodes
Tableau 1 : Tableau des plasmides et des souches utilisées. 97
Tableau 2 : Tableau des oligonucléotides utilisés pour l’introduction de mutation ponctuelle
dans SopA. 97
Figure 76 : Exemple de l’analyse des étapes de purification de la protéine SopA sauvage. 99
Sommaire
SOMMAIRE
INTRODUCTION... 5
I. LE PLASMIDE COMME MODELE D’ETUDE DE LA PARTITION
DES GENOMES BACTERIENS... 5
I.1 Définitions générales des génomes bactériens ... 5
I.2 La géométrie ... 6
I.3 La taille... 7
I.4 Présence de gènes essentiels... 7
I.5 La réplication et le contrôle du nombre de copie ... 8
I.6 Les plasmides à bas nombre de copies comme modèles d’étude de la partition. ... 9
II.
LES FACTEURS INFLUENÇANT LA SEGREGATION ET LE
MAINTIEN DES PLASMIDES... 11
II.2 Contrôle du nombre de copie ... 12
II.2.1 La réplication de type θ... 12
II.2.2 Contrôle par ARN antisens ... 13
A. Le modèle du plasmide ColEI : ciblage des amorces ... 13
B. Le modèle du plasmide R1 : ciblage de l’ARNm de RepA. ... 14
II.2.3 Contrôle par les itérons ... 15
II.2.4 Réflexion sur le contrôle du nombre de copies ... 16
II.3 Contrôle du nombre d’unité de ségrégation ... 16
II.4 Le système plasmidique d’addiction... 17
III.
LES SYSTEMES ACTIFS DE PARTITION PLASMIDIQUES ... 19
III.1 Identification et organisation générale des systèmes de partition ... 19
III.2 Localisation et dynamique intracellulaires des plasmides... 21
III.3 Le système parRMC du plasmide R1, un système de type mitotique ... 24
III.4 Les sytèmes parABS plasmidique de type I... 26
III.4.1 Les séquences centromériques parS ... 27
A. parS du plasmide P1 ... 27
B. sopC du plasmide F... 28
C. L’incompatibilité de partition ... 28
Sommaire
A. ParB de P1... 30
B. SopB du plasmide F ... 31
C. Topologie du complexe de partition ... 31
D. L’étalement des protéines ParB ou « spreading »... 32
E. L’appariement des plasmides... 33
III.4.3 L’ATPase ParA au centre du processus de partition... 35
A. La famille des ATPases de type I... 35
B. L’autorégulation de l’opéron ... 36
C. ParA interagit avec le complexe de partition ... 38
D. Le comportement dynamique des protéines ParA de type I ... 38
E. La capacité d’auto-assemblage des ATPases de type I ... 41
IV.
LA SEGREGATION DES CHROMOSOMES BACTERIENS... 45
IV.1 Organisation du chromosome bactérien ... 45
IV.1.1 De la compaction de l’ADN à la formation du nucléoïde... 46
IV.1.2 Rôle des protéines SMC dans la condensation et la ségrégation du chromosome47 IV.2 Dynamique du chromosome bactérien ... 48
IV.2.1 L’organisation en macrodomaines ... 48
IV.2.2 La dynamique du réplisome et du chromosome bactérien ... 49
IV.3 Les systèmes de partition parABS chromosomique... 51
IV.3.1 Les bactéries modèles... 51
A. Bacillus subtilis ... 51
B. Streptomyces coelicolor ... 52
C. Caulobacter crescentus... 53
D. Les bactéries multi-chromosomiques : Vibrio cholerae et Burkholderia cenocepacia (Bcc).. ... 53
IV.3.2 Des sytèmes de partition proches des sytèmes plasmidiques... 54
A. Une séquence parS universelle ? ... 54
B. La formation du complexe de partition... 55
C. La localisation des régions origines ... 56
D. Les ATPases chromosomiques ... 57
IV.3.3 Les partenaires spécifiques des systèmes de partition chromosomiques ... 61
A. Interactions avec les systèmes de division cellulaire ... 61
B. Interactions avec les systèmes de contrôle de la réplication ... 63
IV.4 Les acteurs secondaires de la ségrégation des chromosomes ... 64
IV.4.1 Les protéines de la translocation du chromosome ... 64
A. SpoIIIE de B. subtilis ... 64
B. FtsK d’E.coli ... 65
IV.4.2 MreB... 66
IV.5 Conclusion : la ségrégation du chromosome est multifactorielle... 68
RESULTATS ... 70
V.
ETUDE DE L’IMPLICATION DU DOMAINE
HELICE-TOUR-HELICE DE SOPA DANS LA FIXATION DE SOPA A L’ADN NON
SPECIFIQUE... 72
Sommaire
V.1 Résultats ... 72
V.1.1 Etude par délétion... 72
V.1.2 Etude par mutagenèse dirigée. ... 73
V.2 Discussion et perspectives ... 75
VI.
RECHERCHE DU DOMAINE DE FIXATION DE SOPA A
L’ADN NON SPECIFIQUE. ... 76
VI.2 Article. ... 76
VI.3 Discussion supplémentaire ... 77
VI.4 Recherches de résidus supplémentaires impliqués dans la reconnaissance de l’ADN non spécifique. ... 77
VI.4.1 Résultats ... 78
A. Analyse des propriétés in vivo :... 78
B. Capacité de fixation à l’ADN in vitro ... 79
C. Capacité de polymérisation... 79
VI.4.2 Discussions et perspectives ... 80
VII.
ANALYSES DES MUTANTS ATPASES DE SOPA ... 83
VII.2 Introduction ... 83
VII.3 Résultats ... 84
VII.3.1 Propriétés in vivo du mutant V125A ... 84
VII.3.2 Capacités d’hydrolyse de l’ATP in vitro... 85
VII.3.3 Capacité de fixation de l’ATP ... 86
VII.3.4 Propriétés de fixation à l’ADN... 87
VII.3.5 Capacité de dimérisation et d’oligomérisation... 88
VII.3.6 Capacités de polymérisation... 88
VII.4 Discussion et perspectives ... 89
DISCUSSION GENERALE ... 93
MATERIELS ET METHODES ... 97
I. SOUCHES ET PLASMIDES UTILISES... 97
II.
MUTAGENESE ... 97
III.
ADN UTILISES ... 97
Sommaire
V.
TEST DE STABILITE ... 99
VI.
TEST DE L’ACTIVITE DE REPRESSION ... 99
VII.
EXPERIENCE DE RETARD SUR GEL... 100
VIII.
EXPERIENCE DE DOUBLE-FILTRATION ... 100
IX.
TEST DE FIXATION DE L’ATP... 101
X.
TEST DE L’ACTIVITE ATPASE ... 101
XI.
TEST DE DIMERISATION... 102
XII.
DYNAMIC LIGHT SCATERRING ... 102
XIII.
MICROSCOPIE A FLUORESCENCE... 102
Le plasmide comme model d’étude de la partition des génomes bactériens La transmission du patrimoine génétique de tout organisme nécessite deux processus majeurs : la réplication fidèle du matériel génétique puis la ségrégation avant la division cellulaire. Chez les eucaryotes, l’étape fondamentale de ségrégation est appelée mitose. Celle-ci se caractérise par la mise en place d’un fuseau mitotique constitué de microtubules qui s’allongent à partir des centrosomes et s’ancrent au niveau des kinetochores des chromosomes. Les microtubules se rétractent permettant la séparation des chromosomes appariés au centre de la cellule [pour revue, (Kline-Smith and Walczak, 2004)]. Le mécanisme de ségrégation chez les procaryotes, appelé partition, est encore mal connu. Afin de mieux appréhender ce processus biologique au centre de mon travail de thèse, il est important de définir certaines notions concernant les génomes bactériens et de définir pourquoi le plasmide est présenté comme un modèle biologique particulièrement bien adapté à étudier de la partition.
I. Le plasmide comme modèle d’étude de la
partition des génomes bactériens
I.1
Définitions générales des génomes bactériens
Le génome est défini à l'origine par les généticiens comme l'ensemble des déterminants héréditaires propre à une espèce donnée. Les génomes des êtres vivants possèdent des formes et des structures différentes mais ils ont un point commun : l’ADN, dont la séquence des nucléotides est la source de l’information génétique. Celle-ci est transmise de façon fidèle aux cours de générations. Pour cela, le matériel génétique de la cellule mère doit être dupliqué par réplication pour ensuite être réparti équitablement dans les cellules filles. Chaque structure génétique dont la réplication est autonome est nommée « réplicon ». Cette définition du réplicon, encore valable aujourd’hui, fut proposée en 1963 par Jacob, Brenner et Cuzin (Jacob and Brenner, 1963). Cette unité de réplication contient une séquence d’ADN appelée « réplicateur », maintenant appelée « origine de réplication », ainsi qu’un gène codant pour une protéine initiant la réplication au réplicateur.
Deux types de réplicons sont retrouvés au sein de la cellule bactérienne : le chromosome et les éléments extrachromosomaux, appelés plasmides. Les premières études sur les réplicons
Le plasmide comme model d’étude de la partition des génomes bactériens bactériens ont été menées sur Escherichia coli et Bacillus subtilis. Avant l’apparition des techniques de séquençage, les génomes de ces bactéries faisaient office de modèles génétiques chez les procaryotes et leurs études ont permis de définir simplement les deux types de réplicons.
Le chromosome bactérien se définit comme une molécule d’ADN de grande taille (de l’ordre du Mpb) portant les gènes nécessaires à sa transmission ainsi que les gènes essentiels à la survie de la cellule, dans les conditions naturelles de croissance.
Les plasmides sont considérés comme des réplicons de plus petite taille (de l’ordre de plusieurs dizaines de kpb), non essentiels, mais porteurs de gènes conférant un avantage sélectif et adaptatif à leur hôte.
Ces paradigmes définissant les réplicons bactériens sont appropriés pour la majorité des bactéries. Cependant, l’avènement récent des techniques de séquençage a permis de découvrir de nouvelles architectures des génomes nécessitant la reconsidération des définitions initiales. Les paragraphes suivants ont pour but de décrire les critères majeurs différenciant chromosomes et plasmides mais aussi de nuancer ces différences aux vues des récentes découvertes apportées par le séquençage des génomes.
I.2
La géométrie
Le modèle de Jacob et Wollman, en 1956, présentait le chromosome comme une molécule d’ADN circulaire, de grande taille et, en 1963, Cairns confirmait cette hypothèse en montrant la forme circulaire du chromosome d’Escherichia coli par autoradiographie (Cairns, 1963; Jacob and Wollman, 1956). Bien que la majorité des bactéries possèdent un chromosome circulaire, certaines possèdent des chromosomes linéaires dont les premiers ont été découverts chez les spirochètes Borrelia (Davidson et al., 1992) et les actinomycètes
Streptomyces (Lin et al., 1993). La majorité des plasmides présentent une forme circulaire. Il
existe cependant des plasmides linéaires, majoritairement présents chez les bactéries dont le chromosome est aussi linéaire [pour revue, (Meinhardt et al., 1997)]. Par conséquent, la géométrie ne permet pas de différencier chromosomes et plasmides. Le concept d’une molécule d’ADN circulaire étant partagé par la majorité des réplicons bactériens, la description des mécanismes de réplication et de partition se basera sur les modèles des réplicons circulaires.
Le plasmide comme model d’étude de la partition des génomes bactériens
I.3
La taille
Les deux bactéries modèles Escherichia coli et Bacillus subtilis possèdent un chromosome de 4,6 et 4,2 Mpb, respectivement. Le séquençage des génomes bactériens a mis en évidence la grande variabilité de taille des chromosomes bactériens. Le plus petit chromosome connu est celui de Carsonella ruddii composé de 160 kpb (Nakabachi et al., 2006) et le plus important est celui de Sorangium cellulosum avec 13 Mpb (Schneiker et al., 2007).
Au sein de la bactérie, les plasmides sont généralement de taille inférieure au chromosome. En réalité, la gamme de taille chez les plasmides est extrêmement large. Le plasmide pRKU1 de Thermotoga petrophila RKU1, bactérie anaérobie stricte et hétérotrophe, ne fait que 850 pb (Nesbo et al., 2006) alors que Ralstonia solanacearum GMI100, bactérie du sol pathogène de plante, possède un mégaplasmide de 2.09 Mpb (Salanoubat et al., 2002). Aux vues de cette forte variabilité, la taille n’apparaît plus comme un critère fort de différenciation entre les chromosomes et les plasmides.
En revanche, la taille du génome peut refléter le mode de vie des bactéries. Les bactéries possédant de petits chromosomes sont souvent des bactéries symbiotiques comme Carsonella
ruddii, endosymbiote d’insectes, ou des parasites obligatoires comme Mycoplasma genitalium
chez l’Homme (Fraser et al., 1995). Les bactéries à grands génomes sont des bactéries « généralistes », avec des métabolismes complexes comme Sorangium cellulosum ou des bactéries possédant parfois des étapes de développement complexe comme la sporulation ou la formation de mycélium. De la même manière, les analyses statistiques de Slater montrent qu’il existerait une corrélation entre la taille des plasmides et les pressions écologiques que subissent les bactéries hôtes. Plus un plasmide est grand, plus il devient indispensable à la survie de son hôte dans les conditions naturelles de croissance (Slater et al., 2008).
I.4
Présence de gènes essentiels
Le chromosome a longtemps été considéré comme l’unique détenteur des gènes essentiels à la survie de la bactérie, contrairement au plasmide qui, par définition, ne possède aucun gène essentiel, mais des gènes d’adaptation aux conditions de croissance de son hôte. L’analyse du génome de R. Sphaeroides a montré qu’il existait des gènes de ménage sur les
Le plasmide comme model d’étude de la partition des génomes bactériens deux types de réplicons. En particulier, deux opérons ribosomiques sont localisés sur le petit réplicon de 900 kpb et un sur le grand réplicon de 3Mpb. Le réplicon de 900 kpb a donc été appelé chromosome secondaire. (Suwanto and Kaplan, 1989).
La bactérie symbiotique Sinorhizobium meliloti 1021 possède un chromosome (3,65 Mpb) et 2 mégaplasmides pSymA (1.35 Mb) et pSymB (1.68 Mb) possédant des gènes essentiels à leur réplication (Galibert et al., 2001; Sobral et al., 1991). Cependant, pSymB possède l’unique copie d’un gène essentiel codant pour l’ARNt de l’arginine. pSymB a aussi perdu la majorité de ses gènes de transferts, et n’est donc plus mobilisable, caractéristique majeure d’un plasmide. pSymA possède les gènes de nodulation et de fixation de l’azote, indispensables à la symbiose de Sinorhizobium meliloti avec son hôte (Barnett et al., 2001). Même si certains méga plasmides ne sont pas essentiels dans des conditions de laboratoire, leur présence constante dans l’hôte naturel suggère qu’ils le sont dans l’environnement de l’organisme hôte (Slater et al., 2008).
I.5
La réplication et le contrôle du nombre de copie
Le nombre de copies d’un réplicon est l’une des caractéristiques essentielles différenciant chromosomes et plasmides. La régulation drastique de l’initiation de la réplication permet à chaque réplicon de conserver le même nombre de copie au cours des générations. Le modèle du réplicon de Jacob est applicable aussi bien aux chromosomes qu’aux plasmides. En effet, leur réplication est initiée au niveau d’un site unique et spécifique à chaque réplicon, appelé origine de réplication (oriC). Chaque réplicon possède une protéine responsable de l’initiation de la réplication (DnaA pour les chromosomes et Rep pour les plasmides). Le contrôle de l’initiation repose sur une réduction de la disponibilité et/ou de l’activité de la protéine initiatrice et de la région d’origine. Ces mécanismes ne seront détaillés que pour les plasmides, dans le chapitre suivant [pour les chromosomes : pour revue, (Boye et
al., 2000)]. Il existe toutefois une minorité de plasmides dont l’initiation de la réplication ne
nécessite pas de protéine initiatrice (voir chapitre II.1.2.1. le cas du plasmide ColEI).
Pour la majorité des chromosomes bactériens, ce contrôle strict limite au nombre de une le nombre d’initiation de la réplication à une origine donnée durant le cycle cellulaire. En général, le nombre de copie d’un chromosome entier dans la cellule est ainsi limité à deux, en fin de cycle cellulaire.
Le plasmide comme model d’étude de la partition des génomes bactériens La réplication des plasmides est initiée indépendamment de celle du chromosome bactérien. Elle n’est donc pas soumise au même contrôle. Tous les plasmides naturels « mesurent » leur propre nombre de copies et ajustent en conséquence leur fréquence de réplication (Nordstrom and Austin, 1993).
Les plasmides peuvent alors être différenciés en fonction de leur nombre de copies par équivalent génome (nombre de copies par rapport au nombre de chromosome) :
- les plasmides à haut nombre de copies, comme ColE1 sont présents à 20 copies par équivalent génome.
- les plasmides à bas nombre de copies, comme le plasmide F d’Escherichia coli sont présents moins de 5 copies par équivalent génome.
I.6
Les plasmides à bas nombre de copies comme modèles
d’étude de la partition.
Le maintien d’un réplicon dans une population bactérienne est un problème de nature différente suivant son nombre de copie. En effet, la diffusion passive des plasmides dans le cytoplasme engendre une ségrégation aléatoire lors de la division cellulaire. La probabilité d’apparition d’une cellule sans plasmide par division cellulaire suit la formule suivante : P0 = 2 1-n, où n est le nombre de copies du plasmide avant la division cellulaire. Ce mode de ségrégation est suffisant au maintien de plasmides à haut nombre de copies comme par exemple le plasmide ColE1. En effet, la perte d’un plasmide présent à environ 20 copies dans la cellule est prédite une fois toutes les 5.105 divisions. Dans ces conditions, la transmission correcte du plasmide est tributaire de la fréquence de réplication. En revanche, un plasmide présent à 5 copies par cellule va être perdu une fois toute les 16 générations et un plasmide présent à 2 copies par cellule, toutes les 2 générations. Cette diffusion passive ne suffirait pas au maintien des plasmides à bas nombre de copie, tel que le plasmide F.
Le facteur F, ou facteur de fertilité, est une séquence d’ADN contenant toute l’information génétique nécessaire à son propre transfert par conjugaison et à son maintien chez E. coli. C’est un épisome, c’est-à-dire qu’il peut être intégré au chromosome, ou excisé sous une forme plasmidique circulaire d’une taille d’environ 100 kpb (Scaife, 1966). Le plasmide F est présent à 1,8 copies par équivalent génome chez E. coli (Gordon et al., 2004) mais il se maintient de manière autonome dans la bactérie grâce à un système de partition actif. Le
Le plasmide comme model d’étude de la partition des génomes bactériens découpage par digestion enzymatique de ce plasmide a révélé qu’un fragment (f5) était toujours capable de se répliquer de manière autonome et était maintenu chez E. coli (Timmis
et al., 1975). Ce petit plasmide, appelé mini-F, est « curable », non essentiel et contient tous
les éléments nécessaires à sa réplication et à sa partition. De plus, son nombre de copies dans la cellule est proche du nombre de copies du chromosome, faisant de lui un outil idéal concernant l’analyse génétique des mécanismes généraux de partition.
Les facteurs influençant la ségrégation et le maintien des plasmides
II. Les facteurs influençant la ségrégation et le
maintien des plasmides
La ségrégation de l’ADN permet une transmission conservative du matériel génétique présent au sein de la cellule. Elle empêche les divisions inéquitables aboutissant à la naissance de cellules anucléées ou sans plasmide. En absence de sélection, les plasmides sont capables de se maintenir durant la croissance de leur hôte. Cette stabilité est dépendante de plusieurs mécanismes dont les acteurs majeurs sont codés par les plasmides.
Tout d’abord, une régulation drastique de l’initiation de la réplication est nécessaire au contrôle du nombre de copies de réplicons qui devront être ségrégés. Dans la majeure partie des cas cette régulation se fait via la protéine initiatrice Rep.
Ensuite, l’étape de réplication peut être interrompue par des événements de cassures de l’ADN pris en charge par les systèmes dédiés à la recombinaison homologue. Celle-ci peut avoir lieu entre deux copies du plasmide, et créer un dimère qui se comportera comme une seule unité de ségrégation lors de la division cellulaire. Cette dimérisation constitue un facteur d’instabilité des plasmides. Le contrôle du nombre d’unités de ségrégation dépend de la présence de systèmes de recombinaison spécifique catalysant la résolution des dimères et codés par les plasmides.
Lorsque le nombre d’unité de ségrégation est faible ou modéré, les plasmides sont ségrégés avant la division cellulaire grâce aux systèmes actifs de partition, nommés par, qui sont au centre de mes recherches. Ces systèmes seront décrits dans le chapitre suivant. Les systèmes de partition et de régulation du nombre de copies maximisent la distribution équitable de l’information génétique durant le cycle cellulaire. En cas « d’accident » de transmission suite à la défaillance d’un des systèmes de maintien, un dernier mécanisme peut être activé : le système d’addiction. Ce système cause la mort des cellules n’ayant pas hérité d’une copie de plasmide durant la division cellulaire.
Tous ces processus vont être décrits dans ce chapitre, mais c’est leur synergie qui permet aux plasmides de se maintenir de manière efficace au sein d’une population bactérienne.
Boites DnaA AT-riche
Promoteur repE
ori2 incC
Plasmide F
Figure 1 : Exemple de plasmides dont la réplication est contrôlée par des itérons.
Pointes de flèches bleues : itérons ; flèches noires : fixation des protéines initiatrices ; flèches en pointillés : ARNm. Plasmide P1 Boites DnaA AT-riche repA ori Promoteur incA
Les facteurs influençant la ségrégation et le maintien des plasmides
II.2 Contrôle du nombre de copie
Les plasmides initient leur réplication indépendamment du chromosome. Cependant leur réplication doit être coordonnée à la croissance de l’hôte. En effet, un taux de réplication insuffisant diminuerait la probabilité d’être présent dans les deux cellules issues d’une division cellulaire. A l’inverse, un taux de réplication trop élevé est délétère pour l’hôte. Le plasmide utilise la machinerie de réplication de l’hôte, ce qui peut interférer avec la réplication de son propre chromosome et ralentir sa croissance. Dans une population bactérienne, la présence d’un plasmide possédant un taux de réplication trop élevé sera « contre » sélectionné.
Afin de définir et de maintenir leur nombre de copies, les plasmides utilisent des boucles de régulation négative. Les stratégies développées par les plasmides dépendent de deux catégories d’inhibiteurs de l’initiation de la réplication : (i) l’ARN antisens s’hybridant avec une région complémentaire d’un ARN essentiel à l’initiation de la réplication (ColE1, R1) (ii) les sites de fixation de types itérons permettant la fixation des protéines initiatrices (plasmide F, plasmide P1). Ces modes de régulations ont été principalement étudiés chez les plasmides possédant un mode de réplication de type θ. Les deux autres modèles sont la réplication par cercle roulant et la réplication par déplacement de brin [pour revue (del Solar et al., 1998)]. Seule l’initiation de la réplication de type θ sera brièvement décrite ici afin de mieux appréhender les mécanismes de régulation.
II.2.1 La réplication de type θ
L’initiation de la réplication de type θ est proche du modèle de la réplication bi directionnelle du chromosome. La première étape est l’ouverture de la région origine, ori. Dans la majeure partie des cas, celle-ci est catalysée par une protéine Rep codée par le plasmide et la protéine initiatrice de l’hôte, DnaA. Dans ces plasmides (plasmide F, prophage P1) l’organisation générale de la région origine est semblable à celle des chromosomes. Ces plasmides possèdent dans la région ori, des séquences répétées, spécifiquement reconnues par la protéine Rep, appelés itérons (figure 1). La région ori contient une zone riche en AT favorisant l’ouverture de l’ADN.
Figure 2 : Exemple de plasmides dont la réplication est contrôlée par des ARN antisens.
A : région origine du plasmide ColEI
RNA I inhibe la maturation de RNA II nécessaire à l’initiation de la réplication B : région origine du plasmide R1
La protéine initiatrice RepA peut être traduite à partir de 2 transcrits (pointillés bleus : ARNm de repA ; pointillés bleus et rouges : ARNm de copB et repA)
Il existe deux voies de régulation de la synthèse de RepA : répression du promoteur PrepApar CopB. L’ARN copA inhibe la traduction de ARNm de repA.
(D’après Nordström, 2006) Protéine ARN ARN ADN + copA copT copA
copB tap repA oriR1
PcopB P repA PcopA
-ori RNA II RNA IA
B
-
+Les facteurs influençant la ségrégation et le maintien des plasmides La protéine Rep, sous sa forme monomérique, se fixe aux itérons présents dans ori, et DnaA se fixe sur les « boîtes DnaA » formant ainsi un complexe d’initiation nucléoprotéique. Ceci permet le recrutement séquentiel du réplisome dont la principale hélicase, DnaB, catalyse alors l’ouverture des brins d’ADN.
La deuxième étape est la synthèse d’un ARN primer, pRNA, par l’ARN polymérase ou des primases de l’hôte ou du plasmide. L’ADN polymérase III de l’hôte va permettre ensuite l’élongation par extension des amorces ARN. Dans la majeure partie des cas, la réplication de type θ se fait de manière bi-directionnelle mais asymétrique. Deux fourches de réplication sont mises en place mais une des deux va répliquer la quasi-totalité du plasmide, alors que la seconde va être arrêtée à moins d’un kb de l’origine. La réplication se termine lorsque la fourche la plus active arrive au niveau de la seconde (Kuzminov et al., 1997). Après la fin de la réplication, les plasmides frères sont décaténés grâce à la topoisomérase IV de l’hôte.
II.2.2 Contrôle par ARN antisens
Ces ARN antisens sont complémentaires d’une région en 5’ d’un transcrit cible essentiel à l’initiation de la réplication (pRNA ou mRNA de rep) et sont appelés “countertranscribed RNAs” (ctRNAs). Ce système de régulation concerne principalement les plasmides à haut nombre de copie comme par exemple ColEI, p15A. Le plasmide R1 à bas nombre de copie fait office d’exception dans ce cas, la régulation de sa réplication sera donc détaillée.
A. Le modèle du plasmide ColEI : ciblage des amorces
La réplication de type θ du petit plasmide à haut nombre de copie, ColE1, est indépendante d’une protéine initiatrice codée par le plasmide. Le contrôle de sa réplication se fait via un ctRNA ciblant la maturation de pRNA (figure 2A). L’initiation de la réplication nécessite un transcrit, RNA II, produit par l’ARN polymérase et maturé par la RNAse H. Cette maturation est requise car elle génère une extrémité 3’-OH sur laquelle l’ADN polymérase I démarre la synthèse de l’ADN. Cette maturation est inhibée en présence d’un ARN de 108 nucléotides, l’antisens RNA I, complémentaire à la région 5’ de RNA II. La formation de l’hybride entre RNA II et RNA I altère la structure secondaire de RNA II. En
Les facteurs influençant la ségrégation et le maintien des plasmides conséquence, l’extrémité 3’ du primer ne peut plus reconnaître l’ADN. La maturation par la RNAse H ne peut plus se faire, bloquant ainsi l’initiation de la réplication. RNA I est un ARN instable (demi-vie d’une minute) qui est synthétisé sous le contrôle d’un promoteur constitutif. C’est donc au niveau post-transcriptionnel que se situe l’essentiel de la régulation de la quantité de RNA I. Sa dégradation est initiée par un clivage par la RNAseE, suivi d’une prise en charge par la poly(A) polymérase. Cette enzyme ajoute des résidus adénosyl en 3’ du produit de clivage de la RNAaseE, ce qui a pour effet d’activer la voie de dégradation dépendante de l’activité 3’-5’ exoribonucléase de la polynucléotide phosphorylase PNPase (Lin-Chao and Cohen, 1991; Xu and Cohen, 1995). La concentration de RNA I est en partie dépendante du nombre de copie du plasmide et fait donc office de senseur et de régulateur de la réplication (Tomizawa and Itoh, 1981).
B. Le modèle du plasmide R1 : ciblage de l’ARNm de RepA.
La synthèse de la protéine d’initiation peut aussi être ciblée par un ARN antisens. Le contrôle du nombre de copie du plasmide R1 se fait suivant ce modèle, via les produits des gènes copB et copA (figure 2B). Deux promoteurs, PcopB et PrepA sont présents en amont de
repA. PrepA est presque totalement réprimé par le répresseur CopB, codé par la partie 5’ du long transcrit initié à PcopB. L’ARN transcrit constitutivement à partir du gène copA, copA RNA, est complémentaire à une région « leader », nommé copT de l’ARNm de RepA. La fixation de l’ARN copA à copT inhibe la synthèse de RepA en empêchant la traduction du peptide leader, Tap qui est couplée à la traduction de RepA (Blomberg et al., 1992). La force de l’inhibition est dépendante de la stabilité du complexe copA-copT. Ce complexe est la cible de la RNAse III (Blomberg et al., 1990). L’ensemble de ces mécanismes permet d’ajuster finement la fréquence d’initiation de la réplication du plasmide R1 [pour revue, (Nordstrom, 2006)].
Figure 3 : Contrôle de la réplication du plasmide F : mécanisme de titration de RepE et de « handcuffing ».
A : RepE est capable de dimériser (RepE2). Le dimère peut se fixer au répétition inversées (IR) afin d’inhiber la transcription de repE. Des chaperonnes convertissent une partie du pool de RepE2 en monomère RepEf se fixant sur les itérons de la région ori (1 à 4) et dans la région incC (5 à 8).
B : Deux formes possibles de « handcuffing » en cis, en équilibre avec la forme sans « handcuffing » du dessus.
(D’après Morrison and Chattoraj, 2004)
repE incC ori2 IR [RepE] RepE2 RepEf 5 6 7 8 1 2 3 4
…
A
B
Les facteurs influençant la ségrégation et le maintien des plasmides
II.2.3 Contrôle par les itérons
Les itérons sont retrouvés dans les origines de réplication où ils permettent le recrutement des protéines initiatrices Rep. Certains plasmides à bas nombre de copies (plasmide F, prophage P1) possèdent, en dehors de la région origine, des itérons jouant un rôle majeur dans la régulation de la réplication. Différents modèles ont été proposés.
En premier lieu, le rôle des itérons a été décrit à l’origine pour le plasmide F, en se basant sur l’observation suivante : la réplication d’un plasmide F est inhibée si les itérons de l’origine sont clonés sur un plasmide compatible. Le modèle soutient que la protéine Rep est titrée par les itérons apportés en trans, limitant ainsi la fréquence d’initiation. C’est le modèle de titration de la protéine Rep (Tsutsui et al., 1983). Le locus d’itérons supplémentaires chez le plasmide F nommé incC (incA chez le plasmide P1) serait responsable de cette titration. L’analyse détaillée de l’origine du plasmide F a révélé la présence en amont du gène repE, en plus des 4 itérons dans ori, de deux motifs de 8pb en séquences inversées répétées présente dans les itérons permettant la fixation d’un dimère de RepE (figure 1). Celle-ci inhibe la transcription du promoteur responsable de l’autorégulation de la protéine initiatrice (Masson and Ray, 1986).
Ces deux modèles de titration et d’autorégulation sont paradoxaux. Cependant, Chattoraj propose que les protéines RepA fixées aux itérons de incA de P1 soient toujours capables de réprimer le promoteur de RepA (Chattoraj et al., 1988). Ce modèle se base sur la capacité des protéines RepA de se fixer à deux itérons simultanément, entraînant la formation de boucle d’ADN au sein de la même molécule (cis). Par conséquent, deux plasmides frères peuvent aussi s’apparier (en trans) via des interactions Rep-Rep, causant un encombrement stérique des 2 origines. C’est le modèle de “handcuffing” (McEachern et al., 1989). Dans ce cas, c’est la concentration en itérons, plus que le niveau d’expression de Rep, qui détermine la fréquence de réplication. Pour le plasmide F, il a été démontré que le contrôle du nombre de copies était compatible avec un couplage des mécanismes de titration et de « handcuffing » (Morrison and Chattoraj, 2004) (figure 3). La probabilité d’initier un cycle de réplication chez le plasmide F est de plus en plus importante au fur et à mesure que la cellule se développe et devient maximale avant que la division cellulaire ait lieu (Helmstetter et al., 1997). Cette observation implique qu’une fois initiée, la réplication est inhibée de manière drastique par le système des itérons pour n’avoir lieu qu’une fois par cycle.
Les facteurs influençant la ségrégation et le maintien des plasmides
II.2.4 Réflexion sur le contrôle du nombre de copies
La régulation de la disponibilité et/ou de la synthèse d’une protéine initiatrice fait intervenir différents facteurs suivant les plasmides étudiés : titration et auto-répression ou régulation de son promoteur par une autre protéine et ciblage de l’ARNm. Ce type de régulation est plus complexe et plus fin que les mécanismes ciblant directement les amorces ARN nécessaire à l’élongation. Une régulation drastique est particulièrement adaptée pour les plasmides à bas nombre de copie ce qui pourrait expliquer que ces plasmides possèdent un mode de régulation par itérons, comme le plasmide F, le prophage P1. Le plasmide R1 ne possède pas d’itérons mais la synthèse de sa protéine initiatrice est finement régulée.
II.3 Contrôle du nombre d’unité de ségrégation
La duplication de l’ADN peut parfois être interrompue par le blocage d’une fourche de réplication ou par des « accidents » de réplication induisant des cassures d’ADN. Un événement de recombinaison homologue entre deux copies d’une même molécule peut aboutir à la création d’un dimère de chromosome ou de plasmide qui se comportera comme une seule unité de ségrégation au moment de la division cellulaire. La diminution du nombre d’unité de ségrégation augmente le risque d’apparition de cellules sans plasmide, constituant un facteur majeur d’instabilité des plasmides (Summers and Sherratt, 1984).
En absence de sélection, le dérivé du plasmide à haut nombre de copie ColEI, pBR322, présente un taux de perte plus important dans une souche permettant la recombinaison homologue que dans une souche déficiente pour ce type de mécanisme. Summers et ses collaborateurs proposèrent que la formation de dimère soit à l’origine de cette observation. C’est le modèle de « catastrophe des dimères » (Summers et al., 1993). Ce modèle se base sur le choix aléatoire des origines initiant la réplication. Le niveau de réplication est fonction du nombre d’origines présentes dans la cellule. Par conséquent, un dimère, formé de novo par recombinaison homologue, possède deux fois plus de chance d’être répliqué qu’un monomère. Ceci aboutit à l’accumulation de multimères dans la cellule. Cette accumulation favoriserait la répartition hétérogène des dimères et serait la cause de l’apparition de cellules ne contenant que des multimères (Boe and Tolker-Nielsen, 1997).
Dimère de ColEI Synapse Jonction de holliday 2 monomères de ColEI intercaténés 1 2 3
Figure 4 : Modèle topologique de la recombinaison au site cer .
Triangle : sites cer. Traits gras : séquences accessoires reconnues par les protéines ArgR et PepA (non représentées). 1 : formation de la synapse grâce aux protéines accessoires. Les sites cer sont en contact étroit. 2 : échange de brin par XerC formant une jonction de holliday transitoire. 3 : deuxième échange de brin par XerD permettant la résolution du dimère.
(D’après Alen et al., 1997)
Figure 5 : Modèle de recombinaison spécifique de site Cre-loxP.
Les protéines Cre (blanc et noir) se fixent sur les sites loxP pour former une synapse de recombinaison (1). Deux des quatre recombinases clivent le substrat d’ADN (trait épais) grâce à leur tyrosine (Y) conservée, formant une liaison covalente 3’-phosphotyrosine (2). Il y a libération d’un groupement 5’-hydroxyl. Ce groupement peut “attaquer” la liaison phosphotyrosine adjacente et reformer le substrat original où il peut attaquer l’autre 3’-phosphotyrosine pour former un échange de brin (intermédiaire de jonction de Holliday) (3). L’isomérisation de cette jonction, par la migration de branche (4), désactive la première paire de Cre et active la seconde qui va à son tour cliver et échanger la seconde paire des brins d’ADN (5), aboutissant au produit de recombinaison (6).
(D’après Van Duyne, 2001).
Y Y Y Y Y Y Y Y Y Y Y Y Y Y Y Y OH OH OH OH 5’ 5’ 3’ 5’ 3’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ (1) (2) (3) (6) (5) (4)
Les facteurs influençant la ségrégation et le maintien des plasmides Les plasmides possèdent des systèmes de recombinaison spécifique catalysant la résolution des dimères. La présence d’un site de recombinaison spécifique plasmidique est commune à tous les plasmides. Certains nécessitent la résolvase de l’hôte alors que les autres possèdent un gène codant pour leur propre résolvase.
Les plasmides de type ColEI portent un site de recombinaison nommé cer, reconnu par le système de résolution des dimères chromosomiques Xer, permettant la conversion des dimères de plasmide en monomère (Summers and Sherratt, 1984). Les deux recombinases XerC et XerD catalysent la réaction d’échange de brin à cer. Des protéines accessoires sont nécessaires à la formation d’un complexe synaptique qui sera responsable de la directionnalité de la réaction (Alen et al., 1997). En effet, la formation du complexe nucléoprotéique n’est efficace que lorsque les sites cer sont présents sur la même molécule (figure 4).
Il est intéressant de noter que le système de résolution Xer des dimères de chromosome, agissant aux sites dif (séquence de 28pb dans la région terminus du chromosome), joue un rôle essentiel durant la ségrégation du chromosome. Ce point sera discuté dans les mécanismes de partition du chromosome.
Les plasmides à bas nombre de copies possèdent leur propre résolvase. Le modèle le mieux connu est le système Cre/loxP, nécessaire à la stabilité du plasmide P1 (Austin et al., 1981). Cre est une résolvase à tyrosine permettant l’échange de brin au niveau de deux sites loxP (figure 5). Le plasmide F possède le même type de système. In vivo, la présence du gène resD et du site rfsF favorise la conversion des dimères en monomère mais le mécanisme sous-jacent est encore mal connu (Disque-Kochem and Eichenlaub, 1993; Lane et al., 1986).
II.4 Le système plasmidique d’addiction
Les systèmes actifs de partition et les stratégies de régulation du nombre de copies ont pour but de minimiser les risques de perte de matériel génétique lors de la division cellulaire. Cependant, des « accidents » de ségrégation peuvent avoir lieu. Certains plasmides (dont F, P1 et R1) possèdent des systèmes de types « addictifs » permettant de tuer les cellules ayant perdu le plasmide au cours de la division cellulaire. Ce processus est effectué par les systèmes toxine-antitoxine (TA) codés par une cassette des deux gènes co-transcrits et le plus souvent soumis à un mécanisme d’autorégulation. La toxine est une protéine extrêmement stable et séquestrée par l’antitoxine. Cette dernière est beaucoup plus sensible à la dégradation par les
Figure 6 : Représentation schématique du système d’addiction des plasmides, exemple du système ccd du plasmide F.
Rouge : toxine CcdB ; bleu : antitoxine CcdA ; bleu hachuré : antitoxine dégradée ; vert : protéase de l’hôte ; violet : DNA gyrase exerçant son activité sur l’ADN (marron) ; marron clair : ADN dont la superhélicité est altérée.
A droite : lorsque le plasmide est présent, la toxine est inactivée par l’antitoxine.
A gauche : lorsque le plasmide est perdu, le système ccd est responsable de la mort de la cellule (pointillés).
Les facteurs influençant la ségrégation et le maintien des plasmides protéases de l’hôte (Clp et Lon). Ainsi, dans une cellule ayant perdu le plasmide, l’antitoxine est plus rapidement dégradée que la toxine, laquelle est alors libre et peut cibler un mécanisme essentiel à la survie de la cellule (figure 6) [pour revue, (Hayes, 2003)].
Un des systèmes les plus étudiés est le système ccd du plasmide F. Ce système n’est pas indispensable au maintien du plasmide car un plasmide F délété de ccd est stable (Hiraga et
al., 1985). Ce n’est qu’en cas de dysfonctionnement des systèmes de réplication et de partition
que ce système a un impact majeur sur le maintien du plasmide dans une population bactérienne en inhibant la croissance des cellules qui n’héritent pas du plasmide (Jaffe et al., 1985).
Lorsque le plasmide F est présent dans la cellule, l’antitoxine CcdA interagit avec la toxine CcdB (Salmon et al., 1994). Lorsque le plasmide est perdu, la protéase Lon dégrade CcdA, plus instable que CcdB (Van Melderen et al., 1994). CcdB intéragit avec la DNA gyrase et inhibe ainsi son activité (figure 6). En effet, des mutations dans gyrA, gène codant pour la sous unité A de la gyrase, confèrent la résistance à l’effet cytotoxique de CcdB (Maki et al., 1992). CcdB piègerait l’ADN gyrase après la fixation de cette dernière sur l’ADN. In vitro, l’ADN gyrase incubée avec CcdB clive l’ADN (Bernard et al., 1993). En plus d’altérer la superhélicité de l’ADN, la formation de ce complexe sur l’ADN entraîne le blocage de la réplication et de la transcription.
D’autres systèmes d’addiction utilisent un ARN antisens en tant qu’antitoxine. Celui-ci contrôle la synthèse de la protéine toxique. L’ARN antisens est instable, alors que l’ARNm codant pour la toxine est très stable. Ce système a été largement détaillé pour le plasmide R1 (système hok/sok) mais ne sera pas décrit ici [pour revue, (Gerdes and Wagner, 2007)].
Figure 7 : Classification des systèmes de partition.
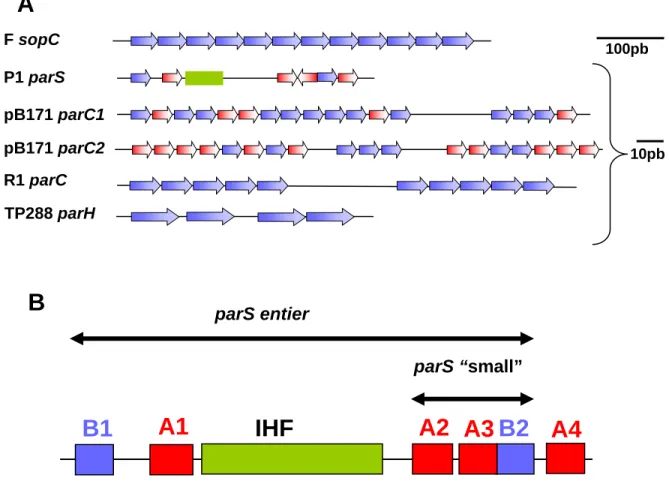
Rouge : gène codant pour une ATPase ; bleu : gène codant pour une protéine de fixation aux séquences centromériques (traits épais). * : parS et sopC ont une localisation similaire mais leur organisation est différente. Les loci de partition de type Ib regroupent aussi ceux présents sur les chromosomes bactériens.
(D’après Schumacher, 2008) Repression de la transcription Liaison au centromère parS* sopC
la P1, F parA, sopA parB, sopB
Type II par loci
Ib TP228 parF parG Repression de la transcription Liaison au centromère R1
Type I par loci
parC parR Repression de la transcription parM Liaison au centromère par1 par2 parC2
parR parM P2 parA parB
Double : type Ib et II
pB171
parC1
Les systèmes actifs de partition plasmidiques
III. Les systèmes actifs de partition plasmidiques
III.1 Identification et organisation générale des systèmes de
partition
Le maintien des plasmides à bas nombre de copies est dépendant des systèmes actifs de partition. Les premières analyses génétiques ont été réalisées sur le plasmide mini-F, dérivé du plasmide F, capable de se maintenir chez E.coli (Timmis et al., 1975). L’analyse par délétion ou par l’insertion d’un transposon Tn3 a permis de définir une région essentielle à la stabilité située en dehors de la région essentielle à la réplication (Bergquist et al., 1981). Cette région est capable de stabiliser des plasmides instables porteurs de l’origine de réplication de
E.coli sans augmenter leur nombre de copies (Ogura et al., 1980; Ogura and Hiraga, 1983).
La poursuite de l’analyse de cette région de partition par délétions successives et par des expériences de transcomplémentation a mis en évidence deux gènes, agissant en trans nommés sopA et sopB (stability of plasmid) et une région agissant en cis nommée sopC (figure 7). Le séquençage nucléotidique a confirmé la présence de deux cadres de lecture
sopA, sopB codant pour deux protéines de 388 et 323 acides aminés, respectivement. Les deux
gènes sont organisés en opéron et sont cotranscrits à partir d’un promoteur unique Psop situé en amont de sopA. L’expression des gènes est réprimée par la fixation de SopA au promoteur. Cette fixation est stimulée par SopB (Mori et al., 1989). Enfin, le site sopC est composé de 12 répétitions imparfaites de 43 pb sur lesquelles se fixent la protéine SopB, formant un complexe nucléoprotéique, nommé complexe de partition (Mori et al., 1986; Mori et al., 1989). Alors que la délétion du site sopC rend un mini-F instable, une seule copie du motif de 43 pb suffit à assurer la stabilité du plasmide (Biek and Shi, 1994; Lane et al., 1987). Ce site
sopC a alors été considéré comme une séquence centromérique procaryote.
Le même type d’organisation du locus de partition nommés par a aussi été découvert sur le plasmide P1 (Abeles et al., 1985; Austin and Abeles, 1983). Les protéines ParA et ParB codées par le locus par de P1 sont homologues aux protéines SopA et SopB du plasmide F. En revanche, le site agissant en cis, nommé parS possède une organisation différente de celle de sopC (figure 7 et figure 14). Un plasmide portant parS est stabilisé par l’apport en trans des protéines ParA et ParB (Martin et al., 1987).
Les systèmes actifs de partition plasmidiques L’analyse des séquences protéiques de SopA et ParA a permis de découvrir que ces protéines appartenaient à une même famille d’ATPase présentant des motifs « Walker » conservés (Motallebi-Veshareh et al., 1990; Walker et al., 1982). Cette découverte a initié le classement des systèmes de partition en fonction des homologies des ATPases qu’ils codent. Les systèmes sop du plasmide F et par du plasmide P1 sont donc les modèles d’études des loci de partition de type I et seront largement détaillés au cours de ce chapitre. Le système de type I peut être subdivisé en deux sous groupes, Ia et Ib, sur la base des caractéristiques propres des protéines qu’il code (figure 7). La différence majeure est la présence d’un domaine HTH (helix turn helix) de liaison à l’ADN en N terminal des ATPase de type Ia, dont SopA et ParA font partie (Radnedge et al., 1998; Ravin et al., 2003). Par l’intermédiaire de ce domaine, ces protéines se fixent sur les séquences opératrices en amont de l’opéron (Davis et al., 1992; Mori et al., 1989).
Les ATPases de type Ib sont donc de plus petite taille. Elles regroupent les ATPases de certains plasmides à bas nombre de copies tels que les plasmides TP228 et pB171 mais aussi les ATPases des systèmes de partition mis en évidence sur la majorité des chromosomes bactériens. Ces dernières feront l’objet d’une description détaillée dans le chapitre suivant. Dans le cas des systèmes de type Ib, c’est la protéine de fixation au centromère (ParG de TP228 ou ParB de pB171) qui joue le rôle de régulateur transcriptionnel en se fixant sur les séquences centromériques positionnées en amont des opérateurs (Carmelo et al., 2005; Ringgaard et al., 2007). Les autres propriétés des ATPases de type Ib sont très proches de celle des ATPase de type Ia et seront donc décrites en parallèle.
Un deuxième groupe majeur a été caractérisé. Il regroupe les systèmes de partition dont l’ATPase est de type actine (Bork et al., 1992). Le système du plasmide R1 fait office de modèle. Il est constitué de deux gènes, parM et parR et d’une séquence centromérique parC (figure 7) (Gerdes and Molin, 1986). La protéine ParR se fixe sur parC et la protéine ParM est l’ATPase du système. Un réplicon portant le locus parC est stabilisé par l’apport en trans de ParM et ParR (Dam and Gerdes, 1994). Bien que minoritaires au sein des réplicons bactériens, ces systèmes sont mieux caractérisés que les systèmes par de type I, notamment au niveau des mécanismes moléculaires sous tendant leur rôle dans la stabilité des plasmides qui le portent.
Récemment une troisième famille a été découverte. Elle comprend des homologues de la tubuline, codés par de larges plasmides dans différentes variétés de Bacillus. Les uniques représentants sont TubZ et RepX. TubZ est nécessaire à la stabilité du plasmide pBtoxis de
Figure 8 : Localisation intracellulaire du plasmide mini-F par hybridation in situ (FISH). A : mini-F sopABC+.
B : mini-F sopABC-.
Le plasmide F est révélé grâce à une sonde portant le fluorochrome Cy3. Les nucléoïdes sont marqués au DAPI. Dans les deux cas, les images sont combinées avec le contraste de phase.
En dessous de chaque série d’images, se trouve la représentation schématique correspondante (sopABC + ou -). Point noir : focus (plasmide). Région blanche : nucléoïde. Région grisée : espace cytosolique. Échelle : 1µm.
(Extrait de Niki and Hiraga, 1997)
A
B
Les systèmes actifs de partition plasmidiques
cereus (Larsen et al., 2007; Tinsley and Khan, 2006). Ces deux protéines sont capables de
polymériser in vitro et in vivo de manière GTP dépendante et pourraient être les « moteurs » de la ségrégation des plasmides qui les codent, suggérant l’existence d’un système mitotique primitif (Akhtar et al., 2009; Chen and Erickson, 2008; Larsen et al., 2007). Cependant, leur rôle dans la partition n’est pas encore clairement défini et ne sera donc pas débattu ici.
Depuis une dizaine d’années, les données issues de la visualisation spécifique et de la localisation des molécules d’ADN dans la cellule, par microscopie à fluorescence, sont venues compléter les connaissances sur la partition acquises par les approches génétiques. En effet, les réplicons sont positionnés dans la cellule et suivent une chorégraphie reproduite de cycle en cycle, et ceci de manière dépendante du système de partition.
III.2 Localisation et dynamique intracellulaires des
plasmides
Les premiers travaux de localisation du plasmide F ont été effectués grâce à la technique de FISH (Fluorescent In Situ Hybridation) (Niki and Hiraga, 1997). Cette méthode consiste à révéler la localisation subcellulaire d’une séquence spécifique d’ADN, par hybridation d’une sonde ADN couplée à un fluorochrome. Cette technique permet la localisation simultanée de plusieurs loci au sein d’une même cellule. Elle nécessite la fixation des cellules et limite donc l’analyse à une visualisation statique des loci d’intérêt.
Niki et Hiraga ont évalué la distribution statistique du plasmide mini-F suivant la taille de la cellule. Dans les jeunes cellules, de petite taille, le focus de fluorescence est localisé au centre de la cellule. Lorsque la cellule présente deux foci, ceux-ci sont localisés aux positions ¼ et ¾ de la cellule, correspondant au futur centre des cellules filles, quelque soit la taille de la cellule. Cette dernière observation suggère que la migration des plasmides n’est pas couplée à l’élongation de la cellule. De plus la fréquence de cellules avec deux foci très proches est très faible, suggérant qu’une fois répliqués les plasmides sont directement déplacés vers les positions ¼ et ¾ de la cellule. Ils y sont maintenus jusqu’à la fin de la cytokinèse (figure 8A). En absence du locus sop, les plasmides sont localisés de manière aléatoire dans le cytoplasme, mais en dehors des régions occupées par le nucléoïde bactérien (figure 8B). Ces résultats ont mis en évidence que le système sop est responsable du positionnement des plasmides dans la cellule (Niki and Hiraga, 1997).
Figure 9 : Suivi dans le temps de la localisation intracellulaire du plasmide mini-P1. Le plasmide mini-P1 est visualisé grâce à une fusion GFP-ParB se fixant sur des séquences
parS insérées sur le plasmide. Les flèches montrent les petits foci pouvant se dissocier et
s’associer au focus central. (Extrait de Li and Austin, 2002)