ANALYSE SPATIOTEMPORELLE DES ENZYMES
DE DÉMÉTHYLATION DE L’ADN ET DES
HISTONES DANS L’EMBRYON BOVIN
Mémoire
FLORENCE PAGÉ-LARIVIÈRE
Maîtrise en biologie cellulaire et moléculaire Maître ès sciences (M.Sc.)
Québec, Canada
iii
Résumé
Chez les mammifères, le passage d’une génération à une autre nécessite la reprogrammation du génome. Cette reprogrammation nécessite la déméthylation de l’ADN parternel et maternel ainsi que celle des lysines des histones suite à la fécondation. Des familles d’enzymes ont récemment été associées à ces processus : les déaminases, notamment Aicda (activation-induced cytosine deaminase), et les Tet (Ten-eleven translocation) désoxygénase, Tet1, Tet2 et Tet3. Plusieurs déméthylases des lysines des histones (KDM) ont été identifiées au cours des dernières années mais très peu d’informations sont disponibles à leur sujet, particulièrement en ce qui a trait à l’embryon bovin. Notre étude s’est attardée à dresser un profil d’expression spatiotemporel de ces familles d’enzymes lors des différents stades embryonnaires précoces chez la vache. Nous suggérons que Tet3 participe activement à la déméthylation de l’ADN, possiblement assisté par Tet2, mais sans Tet1 ni Aicda. Nous montrons également que KDM3A, KDM4A, KDM4C et KDM5B sont présents à des stades et à des endroits précis de l’embryon suggérant ainsi un rôle dans certains processus clés du développement embryonnaire. Ces informations ouvrent la voie à de nouvelles recherches afin de comprendre les modifications de l’épigénome et de réduire les anomalies épigénétiques rencontrées chez les animaux issus de certains protocoles de reproduction assistée.
v
Abstract
In mammals, the transition from one generation to the next requires genomic reprogramming. Such epigenetic change is mediated by paternal and maternal DNA demethylation as well as histone lysines demethylation after fertilization, which is a poorly understood process. Some family of enzymes have recently been associated to those process: the deaminases, like Aicda (activation-induced cytosine deaminase), and Tet (Ten-eleven translocation) dioxygenases, Tet1, Tet2 and Tet3. Many lysine specific histone demethylases (KDM) have been identified in the past few years but little is known about their roles in mammalian embryo. The objective of this study was to develop of a spatiotemporal expression profile of those proteins at different preimplantation stage of bovine embryo. We suggest an active participation of Tet3 in DNA methylation, possibly supported by Tet2 but without Tet1 or Aicda. We also demonstrate the presence and specific localization of KDM3A, KDM4A, KDM4C and KDM5B which may suggest a role during the different embryonic stages. This information opens up the possibilities for further research in order to reduce epigenetic abnormalities associated to assisted reproduction technologies.
Table des matières
RÉSUMÉ ... III ABSTRACT ... V LISTE DES TABLEAUX ... IX LISTE DES FIGURES ... XI LISTE DES ABRÉVIATIONS ... XIII AVANT-PROPOS ... XV CHAPITRE 1 ... 1 1.1 Introduction générale ... 1 CHAPITRE 2 ... 3 REVUE DE LITTÉRATURE ... 3 2. 1 Mise en contexte ... 3 2.1.1 Considérations biologiques ... 5 2.2. Épigénétique ... 7 2.2.1. La chromatine, généralités : ... 8 2.2.2. Hétérochromatine ... 10 2.2.3. Euchromatine ... 11 2.2.4. Gènes à empreinte ... 11
2.2.5. Les marques épigénétiques sont sujettes aux changements ... 12
2.3. Méthylation de l’ADN ... 13
2.3.1. Méthyltransférases ... 16
2.3.2. La déméthylation de l’ADN ... 16
2.3.3. Reprogrammation de l’épigénome ... 17
2.3.3.1. Cellules somatiques vs germinales ... 17
2.3.3.2. Reprogrammation lors de la gamétogenèse ... 19
2.3.3.3. Reprogrammation lors de la fécondation ... 20
2.3.3.4. Conséquences d’une mauvaise reprogrammation ... 21
2.3.4. Déméhylation passive ... 22
viii 2.4. Les histones ... 26 2.4.1 L’acétylation ... 28 2.4.2 La méthylation ... 31 2.4.3 Déméthylation ... 33 2.4.3.1 KDM1 ... 34 2.4.3.2 KDM2 ... 36 2.4.3.3 KDM3 ... 37 2.4.3.4 KDM4 ... 38 2.4.3.5 KDM5 ... 39 2.3.4.6 KDM6 ... 41 CHAPITRE 3 ... 43
3.1 De l’importance de comprendre ces enzymes ... 43
3.2 Objectifs ... 45
3.3 Résumé ... 47
3.3. Article ... 49
RNA extraction and reverse transcription ... 55
Real-time PCR ... 56 Statistical analysis... 57 Immunoblotting ... 57 Immunofluorescence ... 58 3.4. Conclusion ... 77 3.5 Bibliographie ... 79
ix
Liste des tableaux
CHAPITRE 2
Tableau 1. Les membres des différentes classes de HAT ... 30 Tableau 2. Les membres des différentes classes de HDAC. ... 30
CHAPITRE 3
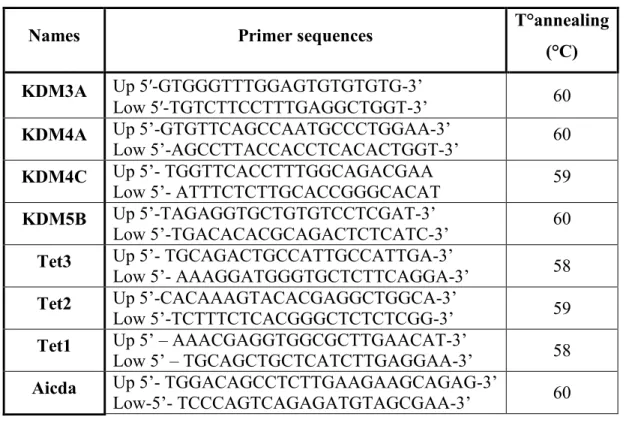
Tableau 1. Information on genes studied.. ... 52 Tableau 2. List of primers used in real-time PCR experiments ... 56 Tableau 3. Pearson determination coefficients of relative mRNA abundance across embryo
xi
Liste des figures
CHAPITRE 2
Figure 1. Quantification relative et origine des ARNm d’un embryon. ... 6
Figure 2. Structure du nucléosome.. ... 9
Figure 3. Cycle de modification de l’épigénome ... 19
Figure 4. Modifications des cytosines et enzymes associées. ... 26
Figure 5 Principales méthylations des lysines situées sur les queues N-terminales des histones H3 et H4.. ... 33
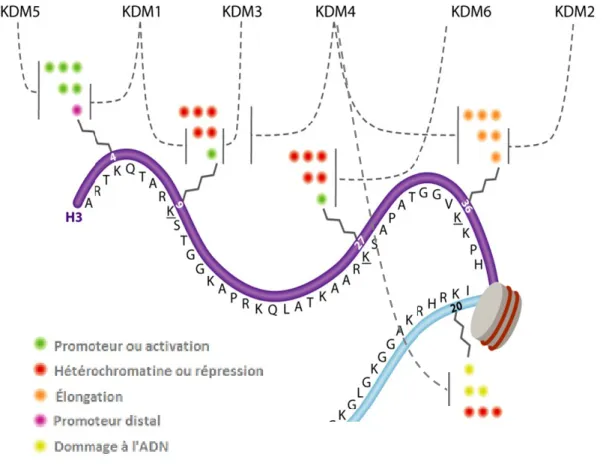
Figure 6. Les familles de déméthylases des lysines des histones associées à chacune des principales méthylations.. ... 42
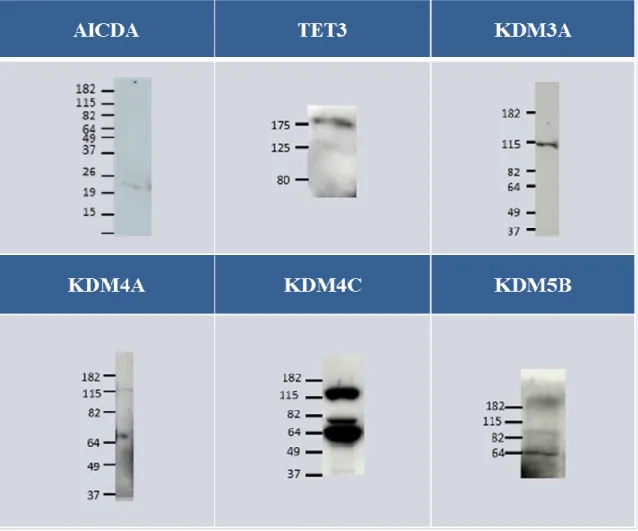
CHAPITRE 3 Figure 1 Immunoblot of AICDA, Tet3, KDM3A, KDM4A KDM4C and KDM5B. ... 59
Figure. 2 Relative abundance of Tet3 transcripts through early developmental stages. ... 61
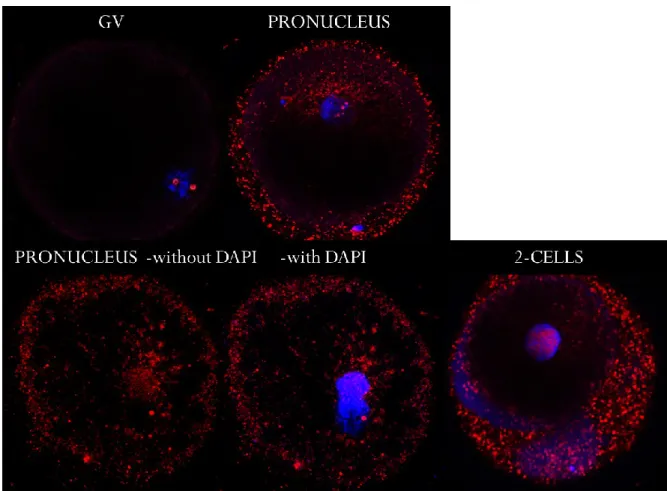
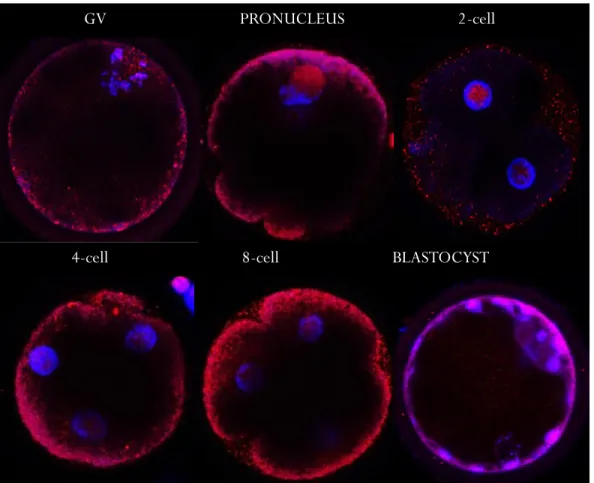
Figure. 3 Immunofluorescence of Tet3 and DNA at different embryo stages………..61
Figure 4 Immunofluorescence of 5hmC and DNA at different embryo stages. ... 62
Figure. 5 Relative abundance of KDM3 transcripts through early developmental stages. .. 63
Figure 6. Immunofluorescence of KDM3A and DNA at different embryo stages. ... 64
Figure. 7. Relative abundance of KDM4A transcript through embryo development. ... 65
Figure 8. Immunofluorescence of KDM4A and DNA at different embryo stages. ... 66
Figure. 9. Relative abundance of KDM4C transcripts through early developmental stages 67 Figure. 10. Immunofluorescence of KDM4C and DNA at different embryo stages. ... 68
Figure. 11. Immunofluorescence of KDM5B and DNA at different embryo stages. ... 69
xiii
Liste des abréviations
2n : diploïde 20G : 2-oxoglutarate 5caC : 5-carboxylcytosine 5fC : 5-formylcytosine 5hmC : 5-hydroxyméthylcytosine 5mC : 5-méthylcytosine Acétyl-CoA : acétyl-coenzyme A ADN : acide désoxyribonucléique
Aicda : activation-induced cytosine deaminase ARNm : acide ribonucléique messager
ATP : adénosine triphosphate CGP : cellule germinale primordiale CpG : cytosine-phosphate-guanine
CXXC : domaine contenant 8 cystéines consécutives Dnmt : DNA methyl transferase
FAD : flavine adénine dinucléotide
FBXL : F-box/leucine rich repeat, F-box vient de la cycline F, première protéine où ce motif a été identifié
FIV : fécondation in vitro GP : globule polaire H1/2/3/4 : histone 1/2/3/4 HAT : histone acétyltransférase HDAC : histone déacétylase HOX : homeobox
HP1 : hétérochromatine protéine IAP : intracisternal A-particule IGF2 : insulin-like growth factor 2
IGF2R : insulin-like growth factor 2 receptor Jarid : jumonji AT-rich domain
Jhdm : jumonji histone déméthylase Jmjd : jumonji domain
K.O. : knock-out
KDM : lysine déméthylase KMT : lysine méthyltransférase LSD : lysine-spécifique déméthylase MBD : methyl binding protein MCI : masse cellulaire interne
MECP2 : methyl CpG binding protein 2 miARN : micro ARN
xiv
NAD+ : nicotinamide adénine dinucléotide
NANOG : nommé d’après le mythe celte irlandais Tír na nÓg qui, en gaélique, désigne « la Terre de l'éternelle jeunesse »
OCT4 : octamer-binding transcription factor 4 PcG : polycomb group
PHD : plant homeobox domain
PRMT : protéin arginine N-methyltransférase REB : réparation par échange de base
RMT : arginine méthyltransférase SAM : S-adénosine methionine TDG : thymine DNA glycosylase TE : transfert d’embryon
Tet : ten-eleven translocation
TI-RCPq : Transcriptase inverse suivi d'une réaction en chaîne de polymérase quantitative TrxG : trithorax group
xv
Avant-propos
Il m’importe d’abord de remercier mon directeur de recherche, Monsieur Marc-André Sirard, pour l’humanité dont il a fait preuve. Il m’a témoigné confiance et respect, m’a guidée sans me presser en ne m’encadrant que de balises discrètes. Le temps venu, il m’a également appuyée financièrement et a ainsi permis l’achèvement de mon projet de maîtrise.
J’aimerais remercier Isabelle Dufort pour l’apprentissage qu’elle m’a offert des techniques de biologie moléculaire, principalement en ce qui a trait aux amplifications en chaîne par polymérase. Je remercie Isabelle Laflamme pour l’enseignement en biologie cellulaire et en fécondation in vitro de même que pour son amitié. Merci Isabelle pour ces conversations improvisées et ô combien appréciées. Je remercie les étudiants du laboratoire de Marc-André Sirard et de Claude Robert pour leurs conseils, leur présence, leur amitié. Plus spécifiquement, je remercie Maëlla Gohin, stagiaire postdoctorale, Sara-Myriam Scantland, étudiante au doctorat, Audrey Bunel, étudiante au doctorat, Anne-Laure Nivet, étudiante au doctorat et Gaël Cagnone, étudiant au doctorat, pour leurs conseils judicieux en matière d’immunobuvardage, d’amplification en chaîne et de présentation d’affiche.
Finalement, je remercie le Fonds de recherche du Québec – Nature et technologies pour le support financier pendant mes études de maîtrise.
xvii
Voilà, j’y suis. À mes parents adorés, à ma sœur bienveillante,
xviii
1
CHAPITRE 1
1.1 Introduction générale
L’ovule est une cellule très complexe. Il contient une multitude de molécules, chacune étant finement régulée, mais le vaste bagage de connaissance que nous possédons à son sujet ne suffit toujours pas à saisir la subtilité des mécanismes qui s’y déroulent. Ainsi, nous sommes encore incapables de déterminer ce qui fait la compétence d’un ovocyte et le rend apte au développement embryonnaire, non plus sommes-nous capables d’expliquer les processus moléculaires en jeu suite à la fécondation et pendant les divisions cellulaires subséquentes. Au cours des premiers stades embryonnaires, la transcription est arrêtée. L’ovule pallie alors ce manque en formant pendant sa croissance une réserve d’ARNm afin de répondre aux besoins de l’embryon. Cette réserve s’épuise graduellement jusqu’à ce que le génome embryonnaire soit activité et que l’embryon puisse à son tour produire ses propres ARNm. La dépendance absolue de l’embryon face à la réserve ovocytaire implique que le moindre dysfonctionnement de l’ovocyte ou la moindre perturbation dans la maturation a des répercussions majeures sur le développement de l’embryon. Aussi, les nombreuses étapes faisant suite à la fécondation sont critiques au devenir de l’embryon, si bien que la qualité de l’ovocyte, bien qu’essentielle, ne suffit pas à prémunir contre l’avortement spontané. En effet, d’autres facteurs, notamment les environnements nutritionnel, chimique et thermique dans lesquels évolue l’embryon, doivent être en tout temps optimaux, la moindre perturbation entraînant souvent l’arrêt du développement à un stade plus ou moins avancé. Depuis quelques années déjà, plusieurs techniques de reproduction assistée ont fait leur apparition dans l’univers agricole et plus particulièrement dans celui de l’industrie laitière. La fécondation in vitro (FIV) compte parmi ces techniques et est largement utilisée à travers le monde, tant chez les animaux que chez l’humain, avec toutefois des taux de rendement relativement faibles. L’une des explications à ce tiède succès est l’impossibilité de savoir si l’ovule ponctionné est compétent au développement
2
embryonnaire ou si l’embryon qui se développe in vitro a le potentiel de former un fœtus viable. Considérant le prix élevé des protocoles FIV et des autres techniques de reproduction assistée, il est pertinent pour de nombreuses industries de mettre à jour des outils permettant de différencier les « bons » des « mauvais » ovules. Une seconde explication tient dans le fait qu’un vase de pétri n’est pas un utérus et que, dans ces circonstances, les perturbations environnementales sont nettement supérieures dans les protocoles in vitro que dans les in vivo. Ces perturbations ont, croyons-nous, le potentiel de nuire au bon développement d’un embryon en en modifiant le métabolisme et le profil d’expression des gènes. Des recherches ont été menées afin de déterminer pourquoi et comment la reproduction assistée pouvait affecter le développement d’un embryon. Les conclusions pointent toutes dans la même direction : l’épigénétique.
3
CHAPITRE 2
REVUE DE LITTÉRATURE
2.
1 Mise en contexte
La croissance de l’industrie laitière à travers le monde au cours des dernières décennies a eu comme résultante la sélection de vaches offrant en abondance un lait de grande qualité. Force est toutefois de constater que cette sélection n’est pas sans conséquence et qu’ainsi, la production laitière accrue s’est soldée par une réduction drastique de la fertilité des vaches. Plusieurs auteurs suggèrent un étranglement au niveau métabolique opposant la production de lait et la gestation comme cause à cette diminution. D’autres, principalement au Brésil où l’industrie laitière est en pleine croissance, regardent du côté du stress thermique subit par les vaches en période estivale. Devant ce constat qui met en péril la pérennité des cheptels et de leurs rendements supérieurs, les éleveurs ont de plus en plus recours à différentes techniques de reproduction assistée. La première utilisée a été la synchronisation de l’ovulation qui permet de contrôler la très courte période d’oestrus (c.-à-d. ovulation), période dont les signes comportementaux et phénotypiques sont difficilement perceptibles. La seconde, nommée superovulation, permet la génération de plusieurs ovules en un même cycle qui sont par la suite récupérés par voie naturelle avant ou après la fécondation. Les développements scientifiques dans le domaine de la reproduction ont ensuite permis l’élaboration de techniques plus pointues présentant chacune des avantages et des inconvénients. La plus courante à travers le monde est le transfert d’embryon (TE). Cette technique se pratique suite à une superovulation et à une insémination artificielle standard et consiste en un prélèvement intra-utérin des embryons pour les transférer ensuite dans l’utérus de vaches receveuses « hormonalement » synchronisées à la donneuse. Ceci permet d’augmenter dans le cheptel la prévalence de traits phénotypiques recherchés en n’utilisant que les embryons issus de vaches hautement performantes. Les embryons peuvent également être congelés et cette innovation a permis d’une part le transfert
4
ultérieur d’embryons à des vaches receveuses, principalement lorsque la quantité de receveuses est moindre que la quantité d’embryons transférables, et d’autre part permis de faciliter et de réduire les coûts d’import et d’export des animaux d’élevage. Toutefois, les frais associés à cette technique sont considérables, d’abord à cause des hormones utilisées pour la superovulation, puis à cause des frais d’expertise médicale et technique inhérents aux protocoles de transfert. De plus, environ 20% des vaches ne répondent pas aux traitements hormonaux, entraînant alors des frais inutiles et compromettant la transmission de leur génome.
Plus récemment, les freins techniques ralentissant l’expansion de la fécondation in vitro (FIV) ont été levés, si bien que cette technique connaissait entre 2008 et 2009 une croissance de 12.7% à travers le monde, comparativement au transfert d’embryons qui lui chutait de 5,6% (IETS, 2011). Cette technique est utile non seulement pour traiter des problèmes d’infertilité, mais également pour mener différentes recherches en science fondamentale, pour sélectionner le sexe de l’embryon, pour améliorer la qualité de la viande et la production laitière de même que pour prémunir les espèces aux populations précaires d’une extinction définitive. Pour les éleveurs, les avantages de la FIV sont majeurs en comparaison avec le transfert d’embryons. Premièrement, elle ne requiert pas nécessairement de superovulation, ce qui évite les injections quotidiennes onéreuses de FSH (folliculostimulating hormone) pendant les 5-6 jours précédant l’ovulation et, parallèlement, la perturbation endocrinienne de ces vaches. En utilisant la stimulation modérée sur 3 jours suivie d’une période de 54 heures sans hormones, une compagnie québécoise de fécondation in vitro obtient des taux de succès inégalés (60 à 80 %) de par le monde (Nivet et al., 2012). Certaine vaches produisent ainsi 100 % d’embryons transférables de sexe connu (insémination in vitro avec semence sexée). La FIV permet également de pallier au problème posé par les 20% de vaches qui ne répondent pas au traitement de superovulation.
La FIV sans la stimulation hormonale donne des taux de succès relativement décevants en comparaison avec d’autres méthodes de reproduction assistée (32% (IETS) versus 60% pour l’insémination artificielle (Martinez et al., 2012). De nombreux ovocytes présentent une absence de développement embryonnaire ou un arrêt précoce, d’autres mènent à la
5 formation d’un fœtus au phénotype ou au métabolisme anormal (Constant et al., 2006; Hori et al., 2010; Martinez et al., 2012). Chez la femme, la technique est fréquemment utilisée pour traiter les problèmes d’infertilité. En fécondation in vitro humaine, les contraintes financières conduisent parfois à l’implantation de plusieurs embryons dans l’utérus dans l’espoir d’en voir un s’implanter et se développer jusqu’à terme. Toutefois, si plusieurs protocoles de FIV échouent, d’autres fonctionnent et produisent parfois des grossesses multiples. Celles-ci sont à la fois dangereuses pour la mère et risquées pour le fœtus. On observe alors des enfants ayant un temps de gestation raccourci, un plus faible poids à la naissance et des complications postnatales fréquentes. En 2011, le gouvernement du Québec annonçait la mise sur pied d’un programme universel de traitement de l’infertilité humaine incluant notamment des protocoles de FIV, ceci afin de réduire le nombre d’embryons transférés et, ultimement, le nombre de grossesses multiples. De près de 2000 protocoles de FIV en 2009, le nombre a plus que doublé en 2012 avec des chiffres estimés à 4400, au Québec uniquement, montrant ainsi la popularité de la FIV subventionnée chez l’humain. Parallèlement, le taux de grossesses multiples est passé de 30% à 4,5%, démontrant tristement le pouvoir de l’argent sur les pratiques médicales.
2.1.1
Considérations biologiques
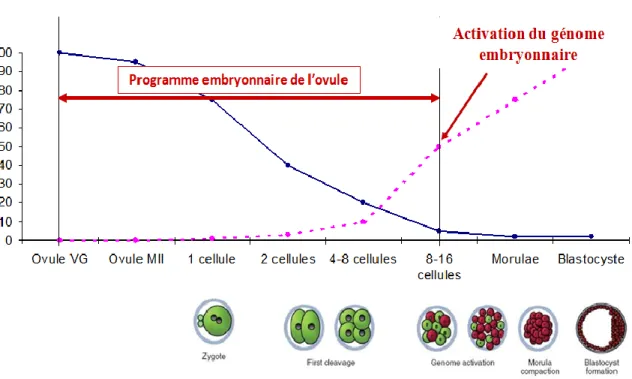
Il est connu que les ovocytes contenus dans les ovaires sont maintenus latents au stade prophase de la méiose I de la naissance à l’ovulation. Chez la plupart des mammifères, une stimulation hormonale par la FSH a lieu une fois par cycle sexuel (2 ou 3 chez le bovin) et amène plusieurs follicules à emprunter la voie du développement, stimulant se faisant l’ovocyte qu’ils contiennent. Cette maturation transforme le follicule qui prend alors de l’expansion et se gonfle de liquide folliculaire. L’ovocyte baigne dans ce liquide, entouré par ses cellules nourricières et protectrices nommées cumulus, et entreprend lui aussi une phase de mûrissement durant laquelle il entrepose une grande quantité d’ARNm. Cette réserve servira à combler les besoins protéiques du jeune embryon pendant les premiers stades de développement alors que la transcription embryonnaire ne sera pas encore activée. Chez le bovin, l’activation du génome embryonnaire se produit entre les stades 8- et 16-cellules, moment à partir duquel l’embryon s’affranchit de la contribution maternelle.
6
La fin de la maturation ovocytaire s’accompagne de l’expulsion d’un premier globule polaire (GP). La méiose est ensuite à nouveau arrêtée, cette fois au stade métaphase II, et ne s’achève que suite au signal induit par la fécondation.
Figure 1. Quantification relative et origine des ARNm d’un embryon. Les ARNm maternels (bleu) diminuent progressivement avant l’activation du génome embryonnaire, moment à partir duquel les ARNm embryonnaires (rose) croissent rapidement. (Sirard, M-A)
Lors d’un protocole de FIV humaine, les follicules sont ponctionnés dans l’ovaire et les ovules sont récoltés au stade de métaphase II. La fécondation de même que le développement précoce se font ensuite en laboratoire dans des milieux de culture synthétiques mimant le contexte tubaire/utérin. À ces étapes s’ajoute, chez la vache, la maturation in vitro de l’ovule durant 24 heures dans un milieu exempt des facteurs folliculaires inhibiteurs (Fair, 2003).
Une autre technique a été mise au point ces dernières années : le clonage. Elle consiste en l’énucléation d’un ovocyte suivi par le transfert d’un noyau 2n souvent extrait d’une cellule
7 somatique. La perspective de produire des animaux par cette méthode a initialement soulevé de grands espoirs, entre autres parce qu’elle faisait miroiter la possibilité de créer des cheptels au sexe et au phénotype prédéfinis, mais ceux-ci ont rapidement été déçus suite aux nombreux échecs rencontrés. Par exemple, en plus des maigres taux de succès (moins de 4% chez la vache) (Wilmut et al., 2002), on retrouve chez le bovin et la chèvre le syndrome du gros veau fœtal (Young et al., 1998) qui se caractérise entre autres par une hypertrophie de certains organes et un poids trop élevé à la naissance obligeant le recours à une césarienne. Également, on observe fréquemment une mort précoce, divers troubles développementaux et des dysfonctions placentaires (Wilmut et al., 2002). Parallèlement, des observations faites sur des embryons, des fœtus et des rejetons issus de la FIV animale ont montré que les manipulations extra-utérines avaient des impacts notables sur l’expression des gènes et, souvent, sur les phénotypes qui leurs étaient associés (Hori et al., 2010; Su et al., 2011; Wrenzycki et al., 2004). Devant ces ratés, les pistes de réponses aux nombreuses interrogations soulevées se sont traduites en autant de projets de recherche, si bien que nous possédons maintenant une nouvelle vision des mécanismes physiologiques et moléculaires s’opérant dans les gamètes et dans le jeune embryon, une vision beaucoup plus fine permise par l’émergence de l’épigénétique.
2.2.
Épigénétique
L’ovule fécondé est à la base de tout animal : Omne vivum ex ovo comme l’écrivait William Harvey. Le zygote se divise et chacune des cellules qui en découlent se divise à son tour, toujours en copiant l’ADN de la cellule mère. Il advient donc que l’entièreté des cellules d’un animal contient précisément le même génome. Comment expliquer alors qu’une si grande diversité de types cellulaires puisse exister au sein d’un même individu? La réponse réside dans le contrôle de l’expression des gènes, domaine de la biologie qu’on nomme épigénétique.
8
« L’épigénétique ressemble un peu à la façon dont on organise ses papiers à la maison : on garde à portée de la main ceux que l’on utilise régulièrement, mais on range les vieux bulletins scolaires dans des boîtes que l’on met au grenier. » Peter Becker
Plus précisément, il s’agit de signaux cellulaires, temporaires ou permanents, directement apposés sur l’ADN pour les uns et ajoutés aux histones pour les autres afin d’induire ou d’inhiber la transcription des gènes. Ces modifications épigénétiques sont reconnues par diverses protéines et permettent de discerner parmi tous les gènes l’information pertinente à transcrire à un moment précis et pour une cellule donnée (Jenuwein and Allis, 2001). Ces marques sont regroupées en deux catégories distinctes qui agissent en synergie : des méthylations sur le 5e carbone des cytosines (5mC) et des modifications post-traductionnelles sur les histones, soit des méthylations, des acétylations, des sumoylations, des phosphorylations, des ubiquitinations et des ADP-ribosylations. En aucun cas la séquence nucléotidique n’est modifiée, seul l’environnement chromatinien est affecté. Ces signaux sont hautement régulés et permettent le recrutement de protéines spécifiques. Ils peuvent également altérer l’affinité de liaison d’autres protéines. Ainsi, une cellule épithéliale aura des marques épigénétiques différentes de celles d’une cellule musculaire et ce sont précisément ces marques qui définissent le profil d’expression génique d’un type de cellule. Les marques épigénétiques permettent donc la différenciation cellulaire.
2.2.1. La chromatine, généralités :
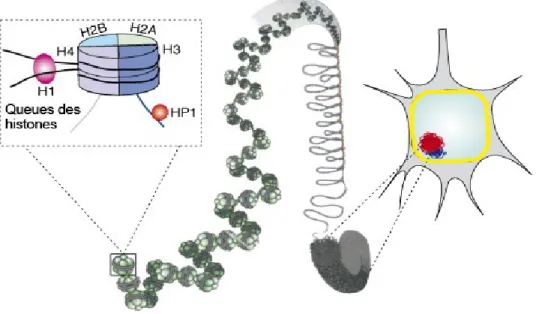
La chromatine est constituée de nucléotides qui permettent l’encodage des protéines sous la forme de gènes. La longueur de l’ADN oblige une compaction extrême en plusieurs niveaux afin que ce double brin soit contenu tout entier dans chaque noyau. L’unité fondamentale qui se répète sur la chromatine est le nucléosome. Il constitue le premier niveau de compaction chez les eucaryotes et est formé de l’octamère d’histones, un agencement de 4 paires de monomères (H2A, H2B, H3, H4) ayant des charges nettes respectives de +15, +19, +20 et +16 et qui s’ancrent fortement sur l’ADN chargé négativement (Turner, 2001). Le nucléosome comprend 147 paires de bases enroulées en une super hélice de 1.7 tour autour d’un octamère d’histones (Luger et al., 1997). Cette
9 structure organisationnelle de la chromatine porte le nom de collier de perles, ou fibre de 11nm (Luger et al., 1997). Chaque monomère contient une queue N-terminale riche en résidus lysine et arginine. Les niveaux subséquents de compaction sont dus à l’accolement des nucléosomes entre eux grâce à la queue N-terminale de chacune des histones qui est chargée positivement et qui peut ainsi interagir avec le corps chargé négativement des histones adjacentes. Également, un 5e monomère d’histone, H1, intervient pour lier entre eux les autres monomères et accoler les nucléosomes les uns aux autres. On parle alors de la fibre de 30nm (Robinson and Rhodes, 2006). Les étapes de compactions suivantes mènent à la formation des chromosomes. Dans le spermatozoïde toutefois, les molécules de compactions de l’ADN ne sont majoritairement pas des histones, mais bien des protamines. Celles-ci sont de petites protéines riches en arginines qui permettent la compaction extrême de l’ADN dans la tête du spermatozoïde mature. Elles sont retirées suite à la fécondation et remplacées par des histones (Ecklund and Levine, 1975).
Figure 2. Structure du nucléosome. La chromatine (ligne noire) est enroulée autour d’un nucléosome constitué de deux copies de H2A, H2B, H3 et H4. En rose l’histone H1 et en orange la protétine Hétérochromatine 1 (HP1) qui reconnaît le motif H3K9me3. Image adaptée de Takizawa (Takizawa and Meshorer, 2008).
10
2.2.2.
HétérochromatineIl existe deux types d’hétérochromatine, soit la constitutive et la facultative (Brown, 1966). La constitutive est la même dans toutes les cellules d’un organisme et se retrouve dans certains territoires chromatiniens spécifiques tels que les télomères et les régions péricentriques et périnucléolaires (Yasmineh and Yunis, 1970). Ces régions contiennent peu de gènes et sont principalement formées de séquences répétées ainsi que d’éléments transposables (Pimpinelli and Ripoll, 1986). À l’opposé, l’hétérochromatine facultative est variable d’une cellule à l’autre et tend à évoluer dans le temps. Elle est dépendante du développement embryonnaire et fœtal (Brown, 1966) et permet l’expression génique différentielle d’une cellule à l’autre de même qu’à des moments variés au sein d’une même cellule. Les deux types d’hétérochromatines portent des marques épigénétiques qui lui sont propres et sont constituées d’un assemblage de macromolécules qui permettent la séquestration des séquences régulatrices de la transcription en les rendant inaccessibles à la machinerie cellulaire transcriptionnelle (Tartof et al., 1989). Un exemple classique d’hétérochromatine facultative est l’inactivation d’un des chromosomes X chez la femelle (Barr and Carr, 1962). Le X éteint est hautement enrichi en marques épigénétiques associées à la répression, telles que H3K27me3 et H3K9me2 (Heard et al., 1997) et est relocalisé en périphérie du noyau, associé à la lamine.
La famille HP1, Heterochromatine protein 1, doit son nom à sa localisation dans les télomères et les régions péricentromériques de même qu’à son rôle dans l’établissement et le maintien du haut degré de condensation de l’ADN dans ces régions non transcrites. Elles sont ubiquitaires et très conservées de la drosophile au mammifère et les fonctions que nous lui connaissons ne cessent de se multiplier. Dans l’hétérochromatine, HP1-α et β ont un domaine de reconnaissance de la lysine 9 méthylée portée par l’histone H3. Cette marque épigénétique de l’histone est associée à l’inhibition de la transcription et, en s’y ancrant, HP1α/β renforcent l’inhibition en plus de recruter une méthyltransférase de l’histone H3K9 afin de propager l’inhibition. Une nouvelle fonction de HP1 vient toutefois d’être révélée avec le membre HP1-γ. Celui-ci est présent non seulement dans l’hétérochromatine mais également dans l’euchromatine, c’est-à-dire près de certains gènes actifs. Les HP1 peuvent, à la manière des histones, être modifiées post-traductionnellement afin de faire varier leur
11 localisation et leur fonction. Il a ainsi été montré que HP1-γ, lorsque modifié sur la sérine 83 (Ser83), avait une localisation euchromatinienne et une fonction d’élongation de la transcription (Lomberk et al., 2006).
2.2.3.
EuchromatineLes régions contenant les gènes exprimés forment l’euchromatine. Il s’agit de régions considérées « ouvertes » et qui sont moins condensées que l’hétérochromatine afin de permettre l’accès aux facteurs et à la machinerie de transcription. Des marques épigénétiques différentes de celles de l’hétérochromatine sont alors présentes sur les histones et l’ADN afin d’en signaler l’état d’ouverture. Par exemple, les cellules souches embryonnaires ont des marques épigénétiques induisant la transcription des gènes spécifiques à la pluripotence, alors que les cellules somatiques ont des marques de répression au niveau de ces gènes. Ainsi, différents types cellulaires peuvent être définis selon leur profil épigénétique (Wu and Zhang, 2010).
2.2.4.
Gènes à empreinte
Un gène à empreinte est un gène dont les deux allèles sont différemment exprimés et qui ne répond donc pas aux principes de la génétique mendélienne. Normalement, les allèles paternel et maternel sont également exprimés et leur régulation est similaire. Dans le cas des gènes à empreinte, un seul des deux allèles est transcrit alors que l’autre est fermé à la transcription d’une façon définitive. On dira que leur expression n’est pas aléatoire mais plutôt dépendante de l’origine (paternelle ou maternelle). Ainsi, pour certains gènes, seul l’allèle maternel sera exprimé (ex. : H19, insulin-like growth factor-2 receptor (IGF2R)) alors que pour d’autres (ex. : IGF2), seul l’allèle paternel sera transcrit. Les gènes soumis à un tel contrôle servent principalement au développement embryonnaire et environ 80% d’entre eux se trouvent regroupés dans l’ADN (cluster). Considérant que les quelque cent gènes soumis à l’empreinte s’expriment selon un allèle parental défini, ils semblent
12
constituer le principal frein à la parthénogenèse (Kono et al., 2004). Aussi, puisqu’aucune compensation par l’allèle complémentaire n’est possible, un défaut d’ordre génétique ou épigénétique dans l’allèle s’exprimant peut résulter en l’absence d’une protéine et, possiblement, en une maladie liée à l’empreinte. Certaines sont bien connues, notamment les syndromes de Beckwith-Wiedemann, de Silver-Russell, de Prader-Willi et d’Angelman (Ideraabdullah et al., 2007; Zeschnigk et al., 2008).
2.2.5.
Les marques épigénétiques sont sujettes aux changements
Contrairement à ce qu’on a longtemps cru, les marques épigénétiques sont sujettes à de nombreux changements, tant additifs que suppressifs. Dans un contexte de cellule somatique, l’épigénome est relativement stable. Toutefois, des évènements de reprogrammation de l’épigénome ont lieu dans les cellules germinales au moment de la gamétogenèse de même que dans le zygote (De La Fuente et al., 2004). Deux groupes de protéines sont considérés comme étant de fins régulateurs de l’expression des gènes pendant le développement embryonnaire : Polycomb Group (PcG) et Trithorax Group (TrxG). Ces complexes synergiques associent plusieurs enzymes de modification de l’épigénome et agissent à des moments précis afin d’activer ou de réprimer des gènes (Sha and Boyer, 2008). Ces phénomènes de reprogrammation seront étudiés en détail dans une prochaine section.
Autrement, lors du développement embryonnaire, les cellules totipotentes se divisent puis, une fois le stade de morula atteint, elles subissent une première étape de différenciation. Certaines cellules prendront la voie du trophectoderme, futur tissu extraembryonnaire auquel le placenta appartient, alors que d’autres formeront la masse cellulaire interne (MCI), futur embryon. Au fil des mitoses, la spécialisation des cellules s’affinera et, se faisant, le patron épigénétique se modifiera.
Considérant que les étiquettes épigénétiques sont portées à évoluer, il est nécessaire que l’épigénome soit modulable, dynamique et flexible (Wu and Zhang, 2010). Aussi, puisque
13 certaines marques doivent être conservées pour la vie entière de la cellule alors que d’autres doivent être modifiées rapidement, il importe d’avoir un système de distinction entre ces modes de régulation à courte et longue durée (Wu and Zhang, 2010). Par exemple, les gènes à empreinte de même que les transposons doivent être éteints pendant tout le développement et toute la vie de l’organisme. Ces séquences de chromatine sont donc « fermées » grâce à des méthylations sur l’ADN en des sites spécifiques qui induisent le recrutement de protéines de fermeture de la chromatine et par des marques sur les histones qui inhibent durablement l’expression des gènes. À l’opposé, les gènes devant être exprimés à de nombreuses reprises sont rendus silencieux temporairement par des modifications post-traductionnelles sur les histones qui permettent la transcription (Wu and Zhang, 2010). Ainsi, le profil épigénétique global d’une cellule somatique reste toujours sensiblement le même, avec d’un côté l’hétérochromatine constitutive et facultative et, d’un autre, l’euchromatine qui subit des modifications mineures constantes.
L’épigénétique peut également être modifiée au cours de la vie d’une cellule due à des facteurs externes telle l’exposition à certains agents chimiques ou à des rayons solaires, le stress chronique et l’alimentation. Aussi, si les cancers étaient jusqu’à présent grandement expliqués par des mutations dans l’ADN, il est maintenant convenu que de nombreux cancers sont issus d’un dérèglement d’ordre épigénétique (Pedersen and Helin, 2010) (Catchpole et al., 2011; Hagelkruys et al., 2011; Metzeler et al., 2011) (Yang et al., 2012). Par exemple, des gènes suppresseurs de tumeur peuvent être inhibés ou, à l’inverse, des oncogènes peuvent être activés. Le fin contrôle de l’épigénétique est donc crucial.
2.3.
Méthylation de l’ADN
La méthylation de l’ADN est une marque épigénétique qui joue un rôle clé dans le contrôle de l’expression génique et la protection de la séquence codante. Elle permet la régulation de la transcription des gènes, la modification de la structure de la chromatine, la régulation du
14
développement embryonnaire et le marquage des gènes sujets à l’empreinte parentale. Aussi, elle prévient les dommages à l’ADN et les réarrangements chromosomiques indus grâce au niveau de compaction accru qu’elle induit (Robertson, 2005) et prévient l’expression des transposons (Yoder et al., 1997).
Les patrons de méthylation diffèrent d’un tissu à l’autre, reflétant les fonctions variées et l’expression différentielle des gènes selon le tissu. Le groupement méthyle provient de la S-adénosine méthionine (SAM) et est ajouté par une méthyltransférase sur le 5e carbone d’une cytosine (5mC) normalement adjacente à une guanine, un arrangement dinucléotidique nommé CpG. Il subvient toutefois quelques méthylations sur les cytosines séparées d’une guanine par une adénine, une thymine ou une autre cytosine dans un arrangement de type CHG, ou alors sans qu’il n’y ait de guanine, un CHH (Ramsahoye et al., 2000). Ces sites de méthylation sont rares et se retrouvent principalement dans les cellules ES (Ramsahoye et al., 2000). Selon Ehrlich (Ehrlich et al., 1982), les dimères C-G ne sont pas distribués uniformément dans l’ADN des vertébrés : la probabilité de rencontrer le couple de nucléotides CpG est de 75 à 80% inférieure à ce qui pourrait être attendu si les lois du hasard étaient respectées. Chez le bovin, ce pourcentage se chiffre à 76.6% (Elsik et al., 2009). Aussi, toujours selon Ehrlich, entre 70 et 80% des CpG des cellules somatiques sont méthylés.
Certaines régions de l’ADN sont enrichies en CpG et portent le nom d’îlots CpG. Les îlots CpG sont des régions d’ADN de plus de 200 paires de bases ayant un contenu en C-G supérieur à 50%. Cooper et Gerber-Huber ont démontré en 1987 (Gardiner-Garden and Frommer, 1987) que ces îlots sont abondants dans les régions promotrices de nombreux gènes, notamment ceux de pluripotence et de gardien cellulaire (housekeeping). On estime que 60 à 70% des gènes ont des îlots CpG dans leur promoteur proximal. Ces îlots se retrouvent également dans le corps de certains gènes et pourraient être impliqués dans l’épissage alternatif en entrainant une pause forcée de l’ARN polymérase II (Malousi et al., 2008; Shukla et al., 2011). Les méthylations portées sur les cytosines des CpG agissent tels des signaux de reconnaissance pour les protéines de la famille des Methyl Binding Protein (MBP). Dans cette famille, on retrouve MBD4 qui joue un rôle dans la réparation de
15 l’ADN, mais aussi MECP2 (Methyl-CpG binding protein 2) et MBD1-3 qui sont associés à des complexes protéiques comportant entre autres des histones déacétylases et plusieurs autres protéines de modification de la conformation de la chromatine (Jones et al., 1998) (Ballestar and Wolffe, 2001). Ces complexes agissent en compactant l’ADN davantage et en inhibant la transcription (Ballestar and Wolffe, 2001). Les sites promoteurs deviennent donc inaccessibles à la machinerie de transcription.
La méthylation de l’ADN est globalement associée à la répression de la transcription, mais certaines nuances s’imposent. Ainsi, l’effet d’une méthylation est davantage lié à son positionnement dans un gène qu’à une signification répressive intrinsèque. Dans les gamètes, les patrons de méthylations varient grandement selon qu’il s’agisse d’un ovocyte ou d’un spermatozoïde. Dans le premier, 40% des CpG sont méthylés et ces méthylations sont regroupées aux îlots CpG de certains promoteurs tels que ceux des gènes Kcnq1 et
Rlim (Kobayashi et al., 2012). Dans le second, l’ADN est hyperméthylé avec environ 89%
des CpG touchés, mais ces méthylations sont absentes de la plupart des îlots CpG (Kobayashi et al., 2012). Différents promoteurs et corps de gènes ont été analysés sous l’angle de la position de leurs méthylations et corrélés à leur niveau d’expression. Il ressort de cela que, dans les gamètes mâles et femelles, la méthylation du corps du gène est fortement liée à la transcription (Spearman's ρ = 0.14–0.16, p<1×10−9 et ρ>0.5, p<1×10−9, respectivement). L’hypothèse veut que la méthylation du corps du gène permette de protéger la séquence nucléotidique de la dégradation. Toutefois, dans l’ovocyte, les gènes les plus fortement exprimés ont parfois un promoteur hyperméthylé (Kobayashi et al., 2012) alors que dans le spermatozoïde, la méthylation du promoteur est associée à un faible niveau de transcription. Les sites d’initiation de la transcription sont néanmoins hypométhylés pour tous les promoteurs (Kobayashi et al., 2012).
En somme, il est de plus en plus mis de l’avant que les méthylations portées par l’ADN ont un rôle relatif à leur positionnement et que, si d’une manière générale, elles sont associées à l’inactivité transcriptionnelle, elles constituent souvent un prérequis à l’expression d’un gène.
16
2.3.1.
Méthyltransférases
Les méthyltransférases sont connues et étudiées depuis longtemps déjà et différentes classes ont été établies afin de discerner leurs rôles respectifs. La classe des méthyltransférases de maintenance a été découverte par Bestor qui, en 1988 (Bestor, 1988), a caractérisé Dnmt1. Elle agit en phase S du cycle cellulaire menant à la mitose en ajoutant des groupements méthyles sur le brin néosynthétisé afin que le patron de méthylation présent sur l’ADN parental soit transmis aux cellules filles. Au niveau embryonnaire, le maintien des méthylations est nécessaire afin que les transposons et les gènes soumis à empreinte restent réprimés d’une mitose à l’autre et que les gènes uniquement requis dans les tout premiers stades s’éteignent aux stades subséquents. La seconde classe de méthyltransférases inclut les Dnmt qui font la méthylation de novo, c’est-à-dire sans qu’un patron préexistant ne les guide. Il s’agit de Dnmt3a, Dnmt3b et Dnmt3L, des méthyltransférases qui sont impliquées principalement dans l’établissement d’un nouveau patron de méthylation suite à la reprogrammation de la gamétogenèse et de la fécondation. Dnmt3a et Dnmt3L sont requises pour l’établissement des empreintes sur les gènes à réprimer dans les lignées germinales (Bourc'his et al., 2001; Kaneda et al., 2004).
2.3.2.
La déméthylation de l’ADN
Les méthylations sur l’ADN ont longtemps été perçues comme étant stables et indélogeables chez les mammifères. Néanmoins, bien que ces marques épigénétiques sur l’ADN soient plus durables que celles apposées sur les histones, il n’en demeure pas moins que des mécanismes sont présents dans les cellules afin de permettre le retrait des méthylations sur la chromatine. Deux vagues de déméthylation majeures ont été identifiées dans certaines cellules de mammifère. L’une a lieu dans les cellules germinales primordiales et débute dès la migration vers les crêtes génitales, alors que la seconde se produit suite à la fécondation de l’ovocyte par le spermatozoïde.
17 La question du processus de déméthylation demeure un grand mystère. Initialement, plusieurs proposaient qu’il s’agisse d’un mécanisme passif, mais d’autres observaient parallèlement des évidences de processus actifs, c’est-à-dire sans réplication de l’ADN pour diluer les méthylations. Dès 1982, une étude montrait l’existence d’une déméthylation in
vitro en absence de division cellulaire, suggérant alors un mécanisme actif. En 1993, Jost et
al montrait qu’une enzyme maintenant connue sous le nom de TDG (Thymine DNA glycosylase) permettait la déméthylation active dans des embryons de poule, sans toutefois pouvoir expliquer le mécanisme moléculaire impliqué (Jost, 1993). Devant quelques évidences d’un processus actif mais en absence de toute preuve mécanistique, une autre équipe a observé la déméthylation de l’ADN post-fécondation afin de statuer sur la passivité ou l’activité du processus. Il a ainsi été constaté dans des cellules embryonnaires souches (ES) de souris que les gènes de pluripotence OCT4 et NANOG voyaient les CpG situés sur leur promoteur se déméthyler en absence de toute division cellulaire (Morgan et al., 2005). Cette observation tendait donc vers la thèse de la déméthylation active de l’ADN. Nous savons aujourd’hui qu’il se produit, suite à la fécondation, une déméthylation à la fois active et passive des pronoyaux.
2.3.3.
Reprogrammation de l’épigénome
Deux évènements majeurs de reprogrammation ont lieu dans l’existence d’un organisme. Le premier concerne les cellules germinales au moment de la gamétogenèse, le second se produit tout juste après la fécondation.
2.3.3.1. Cellules somatiques vs germinales
Deux grandes classes de cellules existent chez un animal : les cellules somatiques et les cellules germinales.
18
Les cellules somatiques sont des cellules diploïdes qui forment la quasi-totalité d’un organisme. Elles ont divers degrés de spécialisation, allant de la cellule souche à la cellule parfaitement différenciée. Aucune d’elle n’a toutefois le potentiel de reformer un nouvel individu, car des étapes de différenciation ont déjà été franchies. Ainsi, quoiqu’il advienne à l’ADN d’une cellule somatique, aucune modification ne saura être transmise à la génération suivante puisque les cellules somatiques ne sont pas des cellules sexuelles. Chez les animaux, ces cellules sont normalement en phase G0 qui correspond à un stade stable de non-division. Lorsqu’elles se répliquent, les cellules diploïdes suivent un cycle cellulaire en 4 phases, soit la phase de croissance G1, la phase de synthèse de l’ADN S, la phase de vérification/prémitose G2 et la phase mitotique M. Cette dernière phase se divise en 4 sous-phases, soit la prophase, la métaphase, l’anaphase et la télophase. Par opposition aux cellules somatiques, les cellules germinales sont celles-là mêmes qui forment les gamètes, rendant alors leur ADN très vulnérable aux modifications de nature génétique et épigénétique. Ces cellules sont dites pluripotentes, c’est-à-dire qu’elles ont le potentiel de former presque tous les types cellulaires. Elles apparaissent dès les premiers stades embryonnaires au niveau de l’épiblaste du blastocyste et, progressivement, elles migrent vers les crêtes génitales. Elles s’y divisent abondamment par mitose pour augmenter le nombre potentiel de gamètes et les cellules filles ainsi produites portent le nom de gonocytes ou cellules germinales. Chez le mâle, la division des cellules germinales est continue pendant la vie adulte (Roosen-Runge, 1962). À l’opposé, la femelle nait en possédant déjà l’entièreté de ses réserves de cellules reproductrices. Celles-ci seront ensuite soumises à la méiose et formeront finalement des gamètes après maturation et différenciation cellulaire (Green and Zuckerman, 1951).
19 Figure 3. Cycle de modification de l’épigénome. Les empreintes maternelles (rouge) et paternelles (bleu) persistent malgré la vague de déméthylation de l’ADN qui suit la fécondation et elles sont présentes dans les cellules différenciées. Parmi celles-ci, quelques cellules germinales primordiales (CGP) subissent une deuxième vague de déméthylation de l’ADN qui efface cette fois les gènes à empreinte. De nouvelles marques sont ensuite établies pendant la gamétogenèse. Adaptée de Li, , 2011(Li and Sasaki, 2011)
20
Lorsque les cellules germinales primordiales (CGP) entament leur migration vers les crêtes génitales, leur chromatine est empreinte d’une multitude de marques épigénétiques, notamment celles inactivant certains gènes à empreinte. Après leur migration vers les crêtes génitales, les CGP entament une reprogrammation qui affecte tout leur génome : presque toutes les marques épigénétiques sont retirées, même celles du chromosome X inactivé et des gènes à empreinte, et il ne subsiste alors que des méthylations partielles sur certains transposons nommés IAPs pour intracisternal A-particles (Lane et al., 2003; Popp et al., 2010). Cette reprogrammation permet d’effacer toute trace de différenciation cellulaire et d’éviter le transfert intergénérationnel (Surani 2001) de modification de la chromatine. Une fois cette reprogrammation achevée et suite à l’enclenchement de la gamétogenèse, un nouveau profil épigénétique se met en place afin de donner aux cellules l’épigénome propre aux gamètes qui devra lui aussi être effacé une fois la fécondation accomplie.
2.3.3.3.
Reprogrammation lors de la fécondation
Lors de la fécondation, l’épigénome de type gamète n’est plus approprié et doit être retiré. L’ovule, qui possède la capacité à reprogrammer l’ADN mâle et le sien, agit activement sur le génome mâle et passivement sur le sien afin d’en retirer les méthylations. L’ADN paternel est alors rapidement déméthylé tandis que le maternel, initialement moins méthylé, se déméthyle progressivement au fil des divisions cellulaires. Cette reprogrammation à la fois active et passive permet d’atteindre, tout juste avant l’activation du génome embryonnaire au stade 8-16-cellules, une déméthylation relative du génome. La reméthylation se fait alors au rythme des différenciations cellulaires et des besoins de l’embryon afin qu’un patron épigénétique propre au nouvel individu s’inscrive. Il est toutefois à noter que certaines régions du génome ne sont pas déméthylées lors de la fécondation. Par exemple, l’hétérochromatine entourant les centromères reste méthylée de même que les IAP et les gènes à empreinte. Notre compréhension actuelle de la régulation génique par la méthylation soutient que ces séquences demeurent méthylées pour augmenter la stabilité des chromosomes, pour empêcher l’expression des rétrotransposons
21 et pour maintenir l’empreinte parentale, respectivement. Il reste cependant beaucoup de travail à faire afin de comprendre comment la méthylation fonctionne.
2.3.3.4.
Conséquences d’une mauvaise reprogrammation
Les conséquences d’une mauvaise reprogrammation ne se résument pas aux syndromes énumérés dans la section portant sur les gènes à empreinte, elles sont nombreuses et souvent lourdes de conséquences. Une constatation flagrante de reprogrammation erronée s’observe lors de tentative de clonage. Puisqu’il s’agit d’un processus forcé plutôt que naturel, les embryons issus du clonage par transfert de noyau présentent de nombreuses anomalies épigénétiques telles que des altérations dans l’épigénome des gènes à empreinte qui sont impliqués dans la croissance fœtale et placentaire (Yang et al., 2005). Des recherches ont été menées sur ces marques épigénétiques anormales et ont démontré que la reprogrammation lors du clonage se faisait selon un processus aléatoire impossible à prévoir et où les gènes déméthylés ne le sont pas toujours complètement ou adéquatement (Su et al., 2011; Wrenzycki et al., 2004). Les embryons clonés meurent dans la majorité des cas et très souvent à cause d’anomalies épigénétiques, démontrant l’incapacité de l’ovocyte à reprogrammer correctement le noyau reçu. On comprend donc que les clones n’ont d’identique qu’un génome alors que leur épigénome varie grandement (de Montera et al., 2010). Bien que les problèmes développementaux soient très marqués dans les cas de clonage, plusieurs perturbations surviennent également lors de FIV. En effet, on rencontre à l’occasion des syndromes de gros veau foetal suite à des protocoles de FIV standard (McEvoy et al., 2000). De ce fait, on constate que le développement précoce dans un contexte in vitro induit suffisamment de perturbations environnementales pour altérer le processus normal de reprogrammation d’un zygote (Dean et al., 1998; Stojanov and O'Neill, 2001), particulièrement au niveau des gènes à empreinte. Ces modifications dans l’ovocyte ou l’embryon perdurent pendant les stades fœtaux et certaines études suggèrent qu’il pourrait y avoir un lien entre ces « épimutations » et l’émergence de cancer dans l’organisme mature (Delaval et al., 2006; Niemitz and Feinberg, 2004). On a également montré que l’alimentation de la mère joue un rôle prépondérant dans le profil épigénétique de son fœtus. En effet, une diète hypocalorique imposée à une rate prégestante ou en début
22
de gestation produit des blastocystes pourvus d’un nombre de cellules réduit tant dans la MCI que dans le trophectoderme. Les rejetons présentent une réduction du poids à la naissance, du taux de croissance postnatal et du ratio poids des organes/poids corporel en plus d’avoir un comportement significativement plus anxieux (Mahsoudi et al., 2007). Il appert donc essentiel de comprendre les causes et conséquences de telles anomalies épigénétiques afin d'augmenter l’efficacité du clonage et de la fécondation in vitro de même d’améliorer la qualité des embryons fécondés in vitro.
2.3.4.
Déméhylation passive
La déméthylation passive de l’ADN maternel se fait par séquestration dans le cytoplasme de Dnmt1. En n’ayant plus accès à l’ADN en réplication, le brin hémiméthylé ne transfère pas son patron de méthylation au brin néosynthétisé. Le niveau de méthylation diminue donc à chaque réplication de l’ADN jusqu’à l’activation du génome embryonnaire au stade 8 cellules chez le bovin. À ce moment, Dnmt1 se relocalise au noyau et permet de copier le patron de méthylation induit par Dnmt3 lors des mitoses subséquentes (Bestor, 2000). Il en découle que les cellules filles pourraient avoir des épigénomes différents selon le brin reçu.
2.3.5.
Déméthylation active
Chez les plantes, la déméthylation active est possible entre autres grâce à DEMETER1 (Zhu, 2009). DEMETER1 est une enzyme à double fonction : dans un premier temps, elle reconnaît la cytosine méthylée et y hydrolyse le lien N-glycosydique unissant le squelette de désoxyribose phosphate et la base méthylée. Puis, dans un second temps, elle agit à titre de lyase en créant un site apyrimidique pouvant être réparé par une DNA polymérase. Chez les animaux, aucune glycosylase capable de reconnaître les cytosines méthylées n’a été
23 découverte, ce qui suggère que des enzymes doivent altérer la cytosine ou le groupement méthyl avant qu’une glycosylase puisse détecter la base à retirer.
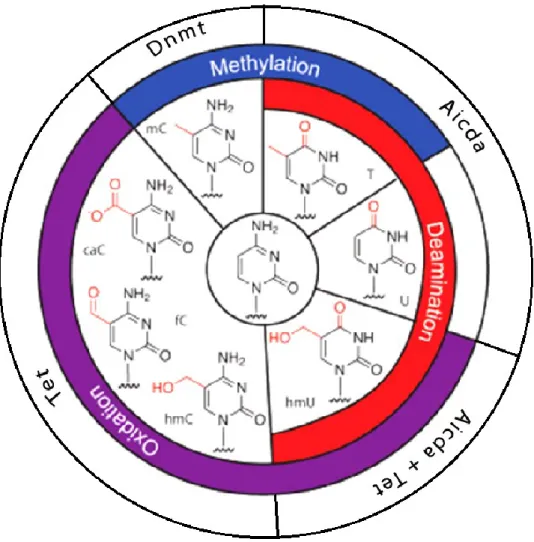
Activation-induced cytidine deaminase (Aicda) est une de ces enzymes qui agissent en modifiant la nature de la cytosine méthylée. Il s’agit d’une déaminase connue depuis longtemps pour son rôle dans l’hypermutation somatique et le changement de classe des immunoglobulines au sein des lymphocytes B immatures (Neuberger et al., 2003). Sa présence a néanmoins été révélée chez la souris dans l’ovocyte, les tissus embryonnaires et les cellules germinales primordiales (Morgan et al., 2004) de même que dans l’ovule du poisson zèbre (Rai et al., 2008), ce qui sous-entend une deuxième fonction pour cette enzyme. Au niveau du génome, le gène Aicda fait partie d’un groupe de gènes de pluripotence tels Stella et Nanog, gènes qui s’expriment exclusivement dans les zygotes et les CGP. Un séquençage au bisulfite a mis en évidence que les niveaux de méthylation de l’ADN des CGP mâles et femelles de souris déficientes en Aicda étaient respectivement 4% et 13% plus élevés que chez des CGP normaux, ce qui montre que Aicda contribue à la déméthylation des cellules germinales (Popp et al., 2010). L’activité de Aicda se fait en retirant le groupement NH2 d’une cytosine méthylée et en y substituant un atome d’oxygène. La cytosine prend alors la forme d’une thymine et, ce faisant, fait naître un mésappariement G : T. La base mésappariée est alors reconnue puis retirée par une glycosylase (Thymine DNA glycosylase chez la souris (TDG) (Morgan et al., 2004) et Methyl binding protein 4 (MBD4) chez le poisson-zèbre (Rai et al., 2008) qui brise le double lien carbone unissant la base au corps phosphaté de l’ADN. Une lyase du système de réparation de l’ADN par excision de base (REB) est ensuite recrutée pour retirer la pyrimidine, puis le trou est comblé par une ADN polymérase. Par contre, les souris déficientes en Aicda ne présentent pas de phénotype particulier, hormis des difficultés au niveau de l’hypermutation somatique dans les lymphocytes B immatures, et demeurent fertiles. La contribution de Aicda ne serait donc pas essentielle au développement embryonnaire et fœtal (Muramatsu et al., 2000).
Néanmoins, si Aicda n’est pas essentielle au développement embryonnaire des souris, il a dernièrement été démontré que TDG l’est. Des souris homozygotes TDG-/- présentaient de
24
graves troubles du développement et mourraient avant la 2e semaine de gestation. L’enzyme de réparation de l’ADN a donc un rôle crucial dans la survie embryonnaire. Récemment, un « cinquième nucléotide » a été découvert dans des cellules de Purkinje, la 5-hydrométhylcytosine (5hmC). Lors de sa première observation, ce nucléotide altéré constituait 0.6% des nucléotides du génome et l’équipe à la base de la découverte a suggéré qu’il pouvait s’agir d’une voie alternative de déméthylation de l’ADN (Kriaucionis and Heintz, 2009). Quelques mois plus tard, un autre groupe publiait qu’une protéine identifiée au début des années 2000 en association avec la méthyltransférase de l’histone H3K4, MLL (mixed-lineage leukemia) (Ono et al., 2002), dans des cellules de leucémie myéloïde aigüe possédait l’activité enzymatique dioxygénase et transformait les 5mC en 5hmC (Tahiliani et al., 2009). Cette enzyme, Ten-eleven translocation 1 (Tet), fait partie d’une famille de dioxygénases constituée de trois membres – Tet1-3 – aux fonctions similaires mais à l’expression tissulaire différentielle (Ito, D'Alessio et al . 2010). Elles sont toutes relativement ubiquitaires, mais Tet1 est nettement plus abondante dans les cellules ES alors qu’elle est absente de l’embryon préimplantatoire, et Tet3 est abondante dans le très jeune embryon ainsi que dans les cellules hématopoïétiques. Aussi, la famille Tet est souvent surexprimée ou mutée dans les cellules cancéreuses (Mercher et al., 2012; Metzeler et al., 2011; Yang et al., 2012). Ces enzymes portent toutes un domaine en doigts de zinc CXXC de liaison aux CpG non méthylés et un domaine dioxygénase dépendant de Fe(II)- et 2-oxoglutarate (2OG).
L’hydroxylation des 5mC n’est pas reconnue par les protéines possédant un domaine d’ancrage spécifique à cette modification telles que les familles MBD et Dnmt. Il est ainsi suggéré que l’hydroxylation rende silencieuse la méthylation, impliquant alors que certains gènes réprimés par des méthylations sur leurs promoteurs puissent être transcrits (Gu et al., 2011). Aussi, en considérant que des protéines de structure de la chromatine sont recrutées sur les CpG méthylés, il est plausible que l’hydroxylation puisse empêcher ce processus et contribuer à une configuration différente de la chromatine (Thalhammer et al., 2011). Aussi, dans les cellules en prolifération, on suggère que l’hydroxylation des 5mC facilite la déméthylation passive lors de la réplication en contrant la liaison de Dnmt1 et la méthylation conséquente sur le brin néosynthétisé (Salvaing et al., 2012; Tahiliani et al.,
25 2009). La fonction pourrait s’appliquer également aux pronoyaux mâle et femelle. Le niveau de 5hmC et de 5mC a été analysé dans des zygotes et des embryons de souris avec comme résultat une corrélation inverse entre les deux marques épigénétiques : le nombre de 5hmC augmentait rapidement dans les pronoyaux paternels de souris alors que celui des 5mC diminuait au même rythme (Gu et al., 2011). Les pronoyaux femelles ne subissaient pas de changement à cet égard (Gu et al., 2011). Conformément à ces résultats, la localisation par immunofluorescence a montré que Tet3 était dans le pronoyau mâle des souris pour être ensuite confiné au cytoplasme. L’importance de Tet3 a finalement été démontrée par l’impossibilité à générer des souris homozygotes mutantes pour Tet3, celles-ci mourant à l’état d’embryon.
L’hydroxylation de la 5mC n’est toutefois pas suffisante à la déméthylation active. Considérant que la glycosylase TDG est inapte à reconnaître l’hydroxylation, une étape intermédiaire est requise entre les deux activités enzymatiques. La solution vient de la famille Tet elle-même qui peut agir sur les 5mC autant que les 5hmC. L’enzyme catalyse ainsi la transformation de 5hmC en 5-formylcytosine (5fC) et en 5-carboxylcytosine (5caC) (Ito et al., 2011), deux sous-produits reconnus par TDG. De plus, la très grande rapidité avec laquelle TDG agit sur eux incite à croire en une nouvelle explication concrète du processus de déméthylation de l’ADN chez les mammifères (Maiti and Drohat 2011).
26
Figure 4. Modifications des cytosines et enzymes associées. Dnmt ajoute un groupement méthyle à une cytosine pour former une 5-méthyl-cytosine (mC). Aicda déamine la cytosine en formant une thymine (T) ou une uridine (U). Tet oxyde de une à trois fois le groupement méthyle pour former une hydroxyméthylcytosine (hmC), une formylcytosine (fC) ou une carboxycytosine (caC). L’action successive de Aicda et Tet forme une hydroxyuridine. Adaptée de Nabel et al . (Nabel et al., 2012)
27 Les nucléosomes sont formés de deux copies des histones H2A, H2B, H3 et H4 et forme un octamère autour duquel l’ADN s’enroule. Cette structure est conservée chez tous les eucaryotes et est maintenant considérée comme étant un déterminant crucial, à tous les niveaux, des gènes et de leurs fonctions (Kornberg and Lorch, 1999). En effet, bien que les histones soient extrêmement conservées, elles sont sujettes à un nombre impressionnant de manipulations et de modifications post-transcriptionnelles accomplies par des enzymes, ce qui leur confère une capacité quasi infinie de variabilité (Turner, 2001). Les modifications des histones, les variants d’histones et le positionnement des nucléosomes travaillent en synergie avec la méthylation de l’ADN pour réguler l’expression des gènes et modifier l’accessibilité des régions promotrices à la machinerie de transcription (Jenuwein and Allis, 2001).
En modulant le degré de compaction de l’ADN, les histones occupent ou libèrent les promoteurs et jouent ainsi un rôle de gardien de la transcription. Des études de cristallographie ont permis de mettre en évidence la queue N-terminale des histones qui est en quelque sorte un appendice au complexe nucléosomal (Luger et al., 1997) et qui permet des interactions internucléosomales et protéiques. Des enzymes ont pour fonction de modifier post-traductionnellement les queues N-terminales des histones afin de signaler l’état de la chromatine, d’autre encore agissent en déplaçant ou démantelant temporairement les nucléosomes. Les modifications des histones semblent se faire selon un ordre précis où certaines modifications sont préalables à d’autres, créant ainsi le code des histones (Strahl and Allis, 2000). Ces modifications permettent une communication complexe et dynamique au sein du noyau et impliquent des interactions avec la chromatine et les protéines qui y sont associées, des protéines de régulation, des molécules non-protéiques, des composants du cytosquelette nucléaire et des ions.
Au grand étonnement de plusieurs, il a été observé dans des embryons et des cellules ES que plusieurs gènes portent des marques épigénétiques bivalentes, c’est-à-dire que leur signification est opposée, l’une activant la transcription et l’autre la freinant. Plusieurs des gènes impliqués dans le développement embryonnaire et la différenciation cellulaire sont touchés par ce chevauchement de signaux. On constate qu’ils sont maintenus dans un état
28
actif et qu’ils sont exprimés, quoique faiblement et à des moments précis (Zhao et al., 2007).
2.4.1 L’acétylation
Certaines lysines des queues N-terminales des histones sont très conservées et servent de sites pour des modifications post-traductionnelles. Des groupements acétyles peuvent ainsi influencer d’une manière électrostatique les interactions entre les histones et l’ADN de par la charge nette négative qu’ils portent. Ces groupements sont ajoutés par des enzymes de la famille histones acétyltransférase (HAT) et viennent former la ε-N-acetyl lysine (Kleff et al., 1995). Le groupement acétyle provient d’un cofacteur, l’acétyl-coenzyme A (acétyl-CoA) (Marmorstein, 2001). Ces marques épigénétiques sont temporaires et peuvent être retirées par des histones déacetylases (HDAC) (Kleff et al., 1995).
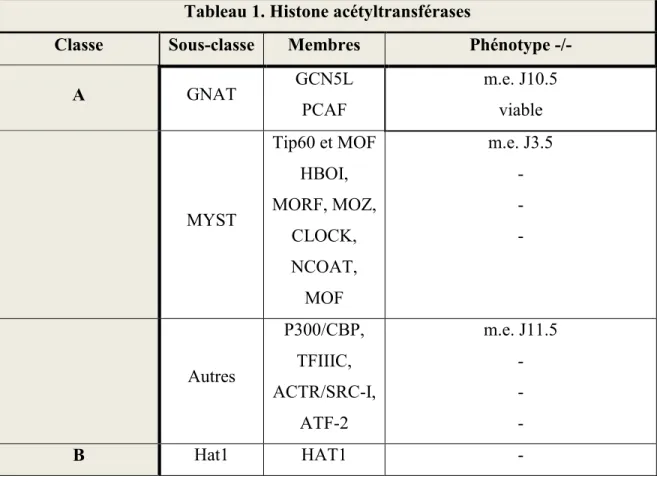
Les HAT se divisent en deux grandes classes chez les mammifères : la classe A dont les membres agissent sur les histones après l’assemblage du nucléosome et la classe B dont les membres sont localisés dans le cytoplasme et acétylent les histones avant leur inclusion dans la chromatine. La classe A se subdivise en trois sous-classes : les MYST (MOZ, Ybf2/Sas3, Sas2 et TIP60), les GNAT (Gcn5 N-Acetyltransférase related) et les « Autres » dont p300/CBP et ATF-2 font partie. On trouve souvent les HAT liées à des complexes multimériques. Cette association a des sous-unités de liaison à l’ADN ou d’interaction protéine-protéine et guide les HAT vers leurs sites d’action.
29 Tableau 1. Histone acétyltransférases
Classe Sous-classe Membres Phénotype -/-
A GNAT GCN5L PCAF m.e. J10.5 viable MYST Tip60 et MOF HBOI, MORF, MOZ, CLOCK, NCOAT, MOF m.e. J3.5 - - - Autres P300/CBP, TFIIIC, ACTR/SRC-I, ATF-2 m.e. J11.5 - - - B Hat1 HAT1 -
Tableau 1. Les membres des différentes classes de HAT chez les mammifères et le phénotype associé à la déplétion homozygote du gène. m.e. J: mort embryonnaire au jour X postfécondation. NAD+ : nicotinamide adénine dinucléotide (Lemoine and Younes, 2010; Voss and Thomas, 2009).