T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Microbiologie
JURY
Jackie PLUMBRIDGE, Directeur de Recherche, CNRS, Paris, Rapporteur Christelle BRETON, Professeur, CERMAV, Grenoble, Rapporteur Charles MANCEAU, Ingénieur de Recherche, INRA, Angers, Rapporteur Valérie VERDIER, Directeur de Recherche, IRD, Montpellier, Examinateur Bernard MARTIN, Professeur de l’Université Paul Sabatier, Toulouse, Examinateur Emmanuelle LAUBER ? Charg2e de Recherche, CNRS, Toulouse, Co-Directeur de thèse
Ecole doctorale : Sciences Ecologiques, Vétérinaires, Agronomiques et Bioingénieries Unité de recherche : Laboratoire des Interactions Plantes-Microorganismes
Directeur de Thèse : Matthieu ARLAT
Présentée et soutenue par Alice BOULANGER Le 15 mai 2009
Titre : Analyse d'un nouveau système CUT impliqué dans l'acquisition et l'utilisation du N-acétylglucosamine par Xanthomonas campestris pathovar campestris.
Doctorat de l’Université de Toulouse délivré par
l’Université Toulouse III - Paul Sabatier
U.F.R. Sciences de la Vie et de la Terre
THÈSE
Pour obtenir le grade de
DOCTEUR DE L’UNIVERSITÉ TOULOUSE III
Discipline : Microbiologie
« Microorganismes, du génome aux interactions avec l’hôte »
Présentée et soutenue par
Alice BOULANGER
Le 15 MAI 2009 à 14h00
Analyse d’un nouveau système CUT impliqué dans
l’acquisition et l’utilisation du N-acétylglucosamine par
Xanthomonas campestris pathovar campestris.
JURY
Jackie PLUMBRIDGE, Directeur de Recherche, CNRS, Paris, Rapporteur Christelle BRETON, Professeur, CERMAV, Grenoble, Rapporteur
Charles MANCEAU, Ingénieur de Recherche, INRA, Angers, Rapporteur Valérie VERDIER, Directeur de Recherche, IRD, Montpellier, Examinateur
Bernard MARTIN, Professeur de l’Université Paul Sabatier, Toulouse, Examinateur Emmanuelle LAUBER, Chargée de Recherche, CNRS, Toulouse, Co-Directeur de thèse
Recherches effectuées au Laboratoire des Interactions Plantes-Microorganismes, UMR CNRS / INRA 2594 / 441
24, Chemin de Borde Rouge BP 52627 31 326 Castanet Tolosan cedex, France
Je souhaite remercier chacun des membres du jury d’avoir accepté d’examiner ce
travail et d’être venu assister à la soutenance de cette thèse.
Merci à :
Matthieu pour m’avoir acceptée au sein de son équipe. Tu m’as appris les bases de
la phytopathologie et la patience (enfin pour la patience tu as essayé !!). Tu m’as
toujours laissé la possibilité de m’exprimer. Merci également pour les blagues
carambar et les sessions bizutages.
Manue, la femme qui décroche plus vite que son ombre. Tu m’as soutenue toutes
ces années, merci pour toute l’aide que tu m’as apportée et tout ce que tu m’as
enseigné. J’ai tenu le coup grâce à toi. Je te remercie également pour les bons
moments passés ensemble, ta patience, les places de cinéma et le super fromage de
chèvre.
Martine et Claudine pour vous être occupées de moi comme de vraies mères.
Martine, merci pour tous les coups de mains que tu n’hésites pas à donner à tous
moments et pour ta gentillesse. J’ai beaucoup apprécié les discussions sur les
péripéties du monde des microbiologistes toulousains. Claudine, merci pour tout le
travail que tu effectues chaque jour et qui nous facilite grandement la vie que ce
soit au sein du laboratoire ou de l’équipe. Les décorations, le chocolat, le muguet et
toutes les petites attentions dont tu nous gâtes tous rendent nos journées plus
belles.
Guitoune et Matcha avec qui j’ai eu de nombreux fous rires. Vive le tableau des
boulettes (bien que vous soyez des tricheurs), les batailles de glace (là aussi deux
contre une c’est de la triche !) et les discussions croustillantes (no comment).
Guitoune, je conserve la boulette d’or, et peut être qu’un jour je te la remettrai. Il
va falloir que tu t’entraînes dur pour arriver à mon niveau et la mériter.
Servane, ma morue. Nous avons passé de très bons moments en ta compagnie.
« vendredi c’est permis » m’ont fait beaucoup de bien et tu m’as ouvert la voie
dans bien des domaines.
Damien pour m’avoir initié aux cultures de plantes et pour ses imitations
inoubliables.
Vincent avec qui je me suis beaucoup amusé pendant les manips.
Laurent pour ton aide apportée lors de la préparation de ma soutenance.
Pauline et Endrick qui sont la nouvelle génération de fous ayant envie de passer
un doctorat. Je vous souhaite beaucoup de réussite et un avenir radieux.
A tous les étudiants qui ont passé un moment avec nous et qui ont participé à la
bonne humeur de l’équipe.
Alice, avec qui je partage de nombreux points communs. Je suis heureuse d’avoir
trouvé une nouvelle amie avec qui partager les joies et les difficultés de la vie. Tu
m’as aidé à passer les moments difficiles de fin de thèse et je t’en remercie.
Solène, Sandra, Lisa, Marie et Céline pour les soirées filles et votre amitié. J’espère
qu’on aura l’occasion de remettre ça rapidement. On va avoir encore pleins
d’heureux événements à fêter.
A toutes les personnes qui animent la vie du labo, qui donnent de leur temps et de
leur énergie pour faciliter le travail de chacun.
Sans les amis et la famille, la vie serait trop dure. Je remercie ma maman qui me
soutien et m’encourage depuis toujours et à qui je dois tant. Sans toi tout ceci
n’aurait pas été possible. Merci à tous mes amis, à Anne, Candice, Nanou et
Zarha. Vous avez toujours été là pour me soutenir et me remonter le moral et
faire la fête quand j’en avais besoin. Enfin, je remercie Jean-philippe qui partage
ma vie. Merci pour tout le bonheur que tu m’apportes chaque jour. Ce n’est pas
tous les jours facile de supporter les états d’âme d’une thèsarde en détresse mais tu
as toujours su me redonner le sourire.
Il était une fois, dans un laboratoire lointain, très lointain, une jolie thésarde prénommée Alice. Alice était en train de finir sa thèse, et avait donc eu beaucoup de bons résultats, seulement,
on aurait dit que le destin l'avait frappé lors de sa naissance et la fit déesse de la boulette. Elle ne pouvait s'empêcher de commettre des boulettes, c'était inné, de temps à autres, ce qu'elle touchait se
transformait en petites ou grosses conneries, si vous me passez l'expression mes vaillants lecteurs. Je vais donc vous conter l'histoire de la boulette du plasmide pVO.
C'était un jour triste du mois de novembre de l'année 2008, et le valeureux guillaume était en train de faire des mutants pVO.
Les fragments avaient déjà été amplifiés et séquencés.
Il en était donc arrivé à la terrible étape de la ligation dans le plasmide pVO. Mais guillaume le bon s'était déjà affranchi de biens de ligation par le passé, et n'appréhendait aucunement cette étape. Seulement, il n'avait plus en sa possession de pVO.
Il parti donc en quête de ce plasmide.
Il réussi à en trouver auprès de la belle Alice, qui, remplie de générosité (j'en rajoute, c'est pour le conte), s'empressa de lui offrir un aliquot.
Il lui demanda si le plasmide était vide et alice confirma avec célérité qu'il ne contenait effectivement pas de fragments en son sein.
(C'est très important pour la suite de l'histoire).
Guillaume le vaillant, accompagné de Martine la grande firent donc leurs ligations, sûr de ce plasmide, et passèrrent à l'étape de la digestion afin de vérifier si leurs fringants fragments étaient en place au sein de leur bien aimé plasmide (car j'avais fait une PCR de vérif avec oligos sur pVO et j'amplifiais un fragment
de 300 environ donc c'était bon pour moi).
Je vous laisse donc imaginer la stupeur de guillaume lorsqu'il vit que ses enzymes ne coupaient pas. Martine tenta donc de couper avec une enzyme, puis l'autre, avec une purif entre les deux,... Rien! Guillaume fît alors une PCR avec les oligos de construction, et, à sa grande surprise, ce fût du grand
n'importe quoi!
Il décida donc de retraverser toutes ses terribles étapes afin d'obtenir le grâle, un mutant pVO. Mais les semaines passèrent (2 quand même) et les ligations ne marchaient toujours pas.
Guillaume le bienheureux ne comprenait pas jusqu'au jour où Martine, imaginant une quelconque baleine sous un gravillon, fit séquencer les pVO
et vit que les séquences correspondaient au gène 0150.
Guillaume le magnifique demanda alors à Alice si, par le plus grand des hasards, il y aurait pu y avoir quelque chose dans le plasmide.
Et effectivement, Alice avait passé à Guillaume une miniprep de pVO avec un fragment dedans, tout en lui disant qu'il était vide. La malédiction avait encore frappée!!
La déesse de la boulette avait accouchée de sa plus belle réalisation en trois ans de thèse. Guillaume digéra donc un autre pVO (sans rien dedans),
fît les ligation, les transformation et eût beaucoup de bons et beaux transformants. Guy.
Il était une fois, dans un laboratoire lointain, très lointain, une jolie thésarde prénommée Alice. Alice était en train de finir sa thèse, et avait donc eu beaucoup de bons résultats, seulement,
on aurait dit que le destin l'avait frappé lors de sa naissance et la fit déesse de la boulette. Elle ne pouvait s'empêcher de commettre des boulettes, c'était inné, de temps à autres, ce qu'elle touchait se
transformait en petites ou grosses conneries, si vous me passez l'expression mes vaillants lecteurs. Je vais donc vous conter l'histoire de la boulette du plasmide pVO.
C'était un jour triste du mois de novembre de l'année 2008, et le valeureux guillaume était en train de faire des mutants pVO.
Les fragments avaient déjà été amplifiés et séquencés.
Il en était donc arrivé à la terrible étape de la ligation dans le plasmide pVO. Mais guillaume le bon s'était déjà affranchi de biens de ligation par le passé, et n'appréhendait aucunement cette étape. Seulement, il n'avait plus en sa possession de pVO.
Il parti donc en quête de ce plasmide.
Il réussi à en trouver auprès de la belle Alice, qui, remplie de générosité (j'en rajoute, c'est pour le conte), s'empressa de lui offrir un aliquot.
Il lui demanda si le plasmide était vide et alice confirma avec célérité qu'il ne contenait effectivement pas de fragments en son sein.
(C'est très important pour la suite de l'histoire).
Guillaume le vaillant, accompagné de Martine la grande firent donc leurs ligations, sûr de ce plasmide, et passèrrent à l'étape de la digestion afin de vérifier si leurs fringants fragments étaient en place au sein de leur bien aimé plasmide (car j'avais fait une PCR de vérif avec oligos sur pVO et j'amplifiais un fragment
de 300 environ donc c'était bon pour moi).
Je vous laisse donc imaginer la stupeur de guillaume lorsqu'il vit que ses enzymes ne coupaient pas. Martine tenta donc de couper avec une enzyme, puis l'autre, avec une purif entre les deux,... Rien! Guillaume fît alors une PCR avec les oligos de construction, et, à sa grande surprise, ce fût du grand
n'importe quoi!
Il décida donc de retraverser toutes ses terribles étapes afin d'obtenir le grâle, un mutant pVO. Mais les semaines passèrent (2 quand même) et les ligations ne marchaient toujours pas.
Guillaume le bienheureux ne comprenait pas jusqu'au jour où Martine, imaginant une quelconque baleine sous un gravillon, fit séquencer les pVO
et vit que les séquences correspondaient au gène 0150.
Guillaume le magnifique demanda alors à Alice si, par le plus grand des hasards, il y aurait pu y avoir quelque chose dans le plasmide.
Et effectivement, Alice avait passé à Guillaume une miniprep de pVO avec un fragment dedans, tout en lui disant qu'il était vide. La malédiction avait encore frappée!!
La déesse de la boulette avait accouchée de sa plus belle réalisation en trois ans de thèse. Guillaume digéra donc un autre pVO (sans rien dedans),
fît les ligation, les transformation et eût beaucoup de bons et beaux transformants. Guy.
Liste des abréviations
ABREVIATIONS
ABC ATP Binding Cassette ADN Acide Deoxy-ribo-Nucléique ADNc ADN complémentaire
AHL N-acyl homoserine lactone
AMPc Adénosine Monophosphate cyclique anhMurNAc acide N-acétylmuramique anhydre
ARN Acide Ribo-Nucléique
Asn Asparagine
ATP Adenosine Triphosphate
Avr Avirulence
CASA Casamino Acid
CAZy Carbohydrate Active Enzymes CBP Chitin-Binding Protein
CC Coiled-coil
CDS Coding DNA Sequence CFU Colony Forming Unit
CHS Chitine Synthase
CLP cAMP receptor Like Protein
COS ChitoOligoSaccharides
CRP cAMP Receptor Protein
CUT Carbohydrate Utilization containing TBDR
Da Dalton
Dap acide Diaminopimélique
DATDH 2,4-diacetamido-2,4,6-trideoxyhexose
DF Diffusible Factor
DMSO Diméthylsulfoxyde
dNTP désoxyribonucléotides Tri-phosphate
DO Densité Optique
DSF Diffusible Signal Factor
DTT Dithiothreitol
ECF Extracellular Cytoplasmic Function EDTA Acide éthylène-diamine-tétraacétique
EI Enzyme I
EII Enzyme II
EPS Exopolysaccharides
ET Ethylène
ETI Effector-Triggered Immunity
Fru Fructose
FUR Ferric Uptake Regulator
GAG Glycosaminoglycane Gal Galactose GalNAc N-acétylgalactosamine GlcNAc N-acétylglucosamine GMP Guanosine Monophosphate GPI Glycophosphatidylinositol GRP Glycine Rich Protein
HAMP Host-Associated Molecular Pattern
Hop Hrp outer protein
HPLC Chromatographie en phase liquide à haute performance HPr Histidine containing protein
HPRP HydroxyProline Rich protein HPt Histidine Phosphotransférase HR Hypersensitive Response
Hrc Hrp conserved
Hrp Hypersensitive response and pathogenicity
HSP Heat-Shock Protein
IPTG Isopropyl β-D-1-thiogalactopyranoside ISR Induced Systemic Resistance
JA Jasmonic Acid
kb kilobase
Kd Constante de dissociation
kDa Kilodalton
KDG 2-keto-3-deoxygluconate LAR Localized Acquired Resistance
Lpp Lipoprotéines
LPS Lipopolysaccharides LRR Leucine rich repeat
LZ Leucine zipper
MAMP Microbes-Associated Molecular Pattern
Man Mannose
MIMP Microbes-Induced Molecular Pattern MCP Methyl accepting Chemotaxis Protein MFS Major Facilitator Superfamily
MS Mass Spectrometry
MurNAc acide N-acétylmuramique
NB / NBS Nucleotide Binding Site
NCBI National Center for Biotechnology Information NeuAc Acide Neuraminique
NLS Nuclear Localization Signal NMR Nuclear Magnetic Resonance
NO Nitric Oxyde
NRP NonRibosomal Peptide
OGT O-GlcNAc Transférase
OMP Outer Membrane Protein ORF Open Reading Frame OTase OligosaccharylTransférase
PAGE polyacrylamide Gel Electrophoresis PAMP Pathogen Associated Molecular Pattern PNPG Paranitrophényl-β-D-galactoside
pb paires de bases
PBP Penicillin-Binding Protein PCD Programmed Cell Death PCR Polymerase Chain Reaction
PEP phosphoénolpyruvate
PG Peptidoglycane
Pgl Protein glycosylation PIP Plant Inducible Promoter
Liste des abréviations PME Pectine Méthyl Estérase
Pop Pseudomonas outer protein
PR Pathogenesis-Related PRP Proline Rich Protein
PRR Pattern Recognition Receptor
PTI PAMP-triggered immunity
PTS Phosphotransferase System pv. pathovar QS Quorum Sensing R Résistance RE Réticulum Endoplasmique RLK Receptor-Like Kinase RLP Receptor-Like Protein rpm rotation per minute
ROK Repressor Open reading frame Kinase ROS Reactive Oxygen Species
Rpf Regulation of pathogenicity factors
RT Reverse Transcription
SA Salicylic Acid
SAR Systemic Acquired Resistance
SDS-PAGE SulfateDodécylique Sodium- PolyAcrylamide Gel Electrophoresis
Ser Sérine
SNP Single Nucleotide Polymorphism
sp. Species
TBDT TonB-Dependent Transporter TCA TriCarboxylic Acid
TCT Trachéal CytoToxine
Thr Thréonine
TIR Toll / Interleukine1 Receptor domain Trh Transcriptional regulator for hrp T3SE Type Three Secretion Effector T3SS Type Three Secretion System UDP Uridine Diphosphate
UV Ultra Violet
Xcc Xanthomonas campestris pv. campestris
Xgal 5-bromo-4-chloro-3-indolyl-beta-D-galactopyranoside Xgluc 5-bromo-4-chloro-3-indolyl-beta-D-glucuronic acid Xop Xanthomonas outer protein
LISTE DES FIGURES
Introduction générale
Chapitre 1
Figure 1 : Quelques exemples de Brassicacées cultivées et la plante modèle
Arabidopsis thaliana
8 Figure 2 : Représentation schématique du cycle de vie de Xanthomonas campestris
pv. campestris (Xcc) lors d’une interaction avec la plante hôte.
9
Figure 3 : Symptômes de la nervure noire sur les feuilles de choux. 9
Chapitre 2
Figure 4 : Signalisation de la réponse basale et cibles de certains effecteurs de type III
connus
11
Figure 5 : Représentation de la théorie “Gène pour Gène” de Flor (1971). 12
Figure 6 : Différentes classes de Protéines de Résistance. 13
Figure 7 : Le modèle en zig zag illustrant la production quantitative de l’équipement
nécessaire à la défense immunitaire de la plante.
15
Chapitre 3
Figure 8 : Représentation schématique de trois types de mobilité bactérienne. 17
Figure 9 : Modèle du système d’acquisition du fer des bactéries Gram– et de sa régulation.
19
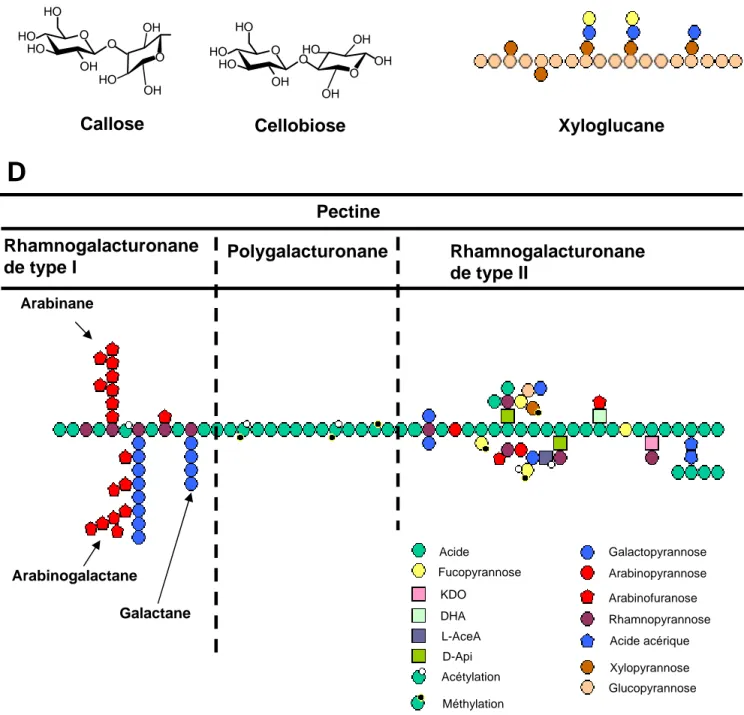
Figure 10 : La paroi végétale. 20
Figure 11 : Représentation schématique de différents polymères de sucres composant
la paroi primaire végétale.
21 Figure 12 : Modèle de fonctionnement d’un locus CUT basé sur le locus sux de
Xanthomonas campestris pv. campestris.
24 Figure 13 : La translocation de groupe par le système PTS, mode de transport actif de
carbohydrates à travers la membrane interne des bactéries à Gram négatif.
24
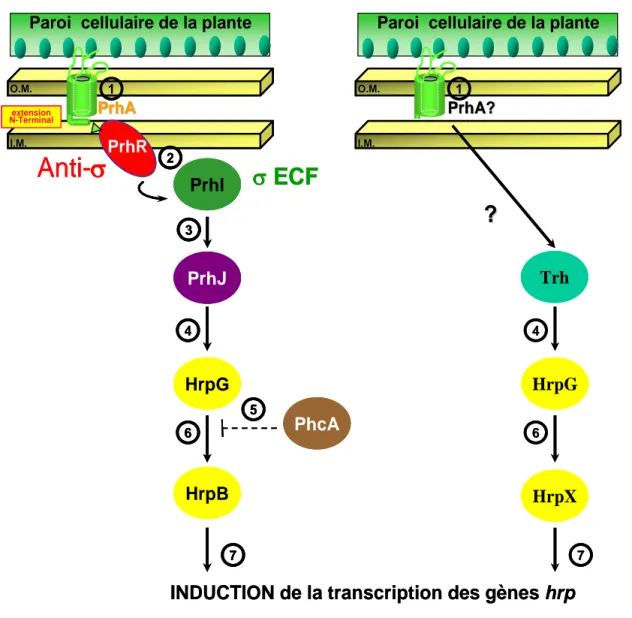
Figure 14 : Représentation schématique du système Hrp. 25
Figure 15 : Modéles de régulation des gènes du système Hrp chez la bactérie
Ralstonia solanacearum et Xanthomonas.
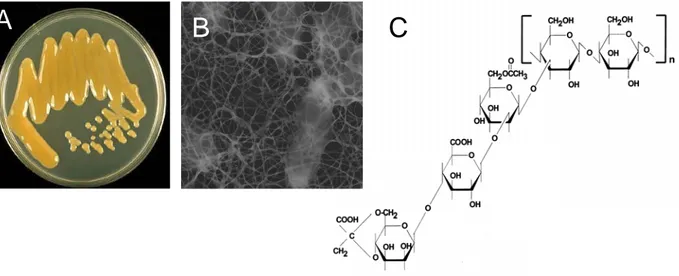
28 Figure 16 : Le xanthane, un exopolysaccharide produit par Xanthomonas campestris
pv. campestris.
29 Figure 17 : Voie de synthèse de Xanthane chez Xanthomonas campestris pv.
campestris (Xcc).
29 Figure 18 : Structures des molécules signales des deux systèmes de quorum sensing
de Xanthomonas campestris pv. campestris.
30 Figure 19 : Modèle de la transduction du signal DSF chez Xanthomonas campestris
pv. campestris.
30
Figure 20 : Représentation schématique du quorum sensing chez Xcc. 31
Figure 21 : Illustration des interactions entre Xanthomonas campestris pv. campestris
et une plante-hôte.
Liste des figures
Etude de l’acquisition et de l’utilisation du GlcNAc
Chapitre 2
Figure 22 : Le peptidoglycane 35
Figure 23 : Voie de biosynthèse du peptydoglycane (PG). 36
Figure 24 : Représentation schématique de l’activité d’enzymes clivant le
peptidoglycane.
37
Figure 25 : Recyclage du peptidoglycane chez Escherichia coli. 38
Figure 26 : Représentation schématique des parois bactériennes. 39
Figure 27 : Structure du lipopolysaccharide (LPS) de E. coli O111:B4. 39
Figure 28 : Structure de la chitine. 40
Figure 29 : Structure schématique de glycosaminoglycanes (GAGs) et de
protéoglycanes.
41 Figure 30 : Structure de l’ancre GPI de l’érythrocyte acétylcholinestérase humaine. 41
Figure 31 : Exemple d’interactions impliquant des carbohydrates de surface. 42
Figure 32 : Relation dynamique entre phosphorylation et modification par le
O-GlcNAc d’une protéine.
44 Figure 33 : Structure du N-glycane précurseur greffé sur la protéine eucaryote en
cours de synthèse.
45 Figure 34 : Représentation simplifiée des premières étapes de N-glycosylation dans le
réticulum endoplasmique (RE).
45 Figure 35 : Structure des principaux N-glycanes rencontrés chez les mammifères. 45
Figure 36 : Structure des principaux N-glycanes rencontrés chez les plantes. 46
Figure 37 : Localisation majoritaire des différents types de N-glycanes dans la cellule
végétale.
46 Figure 38 : Synthèse des différents types de N-glycanes dans le réticulum
endoplasmique (RE) et l’appareil de Golgi des cellules végétales.
46 Figure 39 : Exemples de sucres rares constituant les glycanes des glycoprotéines des
Archées et des bactéries.
52 Figure 40 : Modèle de N- et O-glycosylation chez les archées et les bactéries. 53
Figure 41 : La N-glycosylation chez Campylobacter jejuni. 54
Chapitre 3
Figure 42 : Modèle de la voie de dégradation de la chitine chez Saccharophagus
degradans (anciennement nommé Microbulbifer degradans).
56 Figure 43 : Représentation schématique du catabolisme du GlcNAc chez les bactéries. 59 Figure 44 : Séquences consensus reconnues par les régulateurs transcriptionnels
NagC, NagR et NagQ.
63
Résultats
Figure R1. Xanthomonas campestris pv. campestris (Xcc) utilizes
N-acetylglucosamine and chitobiose as a carbon source.
73 Figure R2. The Xanthomonas campestris pv. campestris N-acetylglucosamine CUT
system and proposal model for N-glycans degradation.
Figure R3. Glycoside hydrolases encoded by the Xanthomonas campestris pv.
campestris glycan cluster are functional and controlled by NagR.
77 Figure R4. Xanthomonas campestris pv. campestris Nag proteins are involved in the
utilization of N-acetylglucosamine.
79 Figure R5. Xanthomonas campestris pv. campestris NagK-II enzymes are involved in
the phosphorylation of N-acetylglucosamine.
80 Figure R6. Xanthomonas campestris pv. campestris nagA mutants are sensitive to
N-acetylglucosamine, in vitro.
81 Figure R7. N-acetylglucosamine and N-acetylglucosamine-6P are the signalling
molecules of NagR and NagQ respectively.
83 Figure R8. Xanthomonas campestris pv. campestris nagA and nagB-II mutants are
affected in pathogenicity.
84 Figure R9. Xanthomonas campestris pv. campestris Nix glycoside hydrolases and the
TonB-dependent transporter NixD play a central role in sensitivity to N-acetylglucosamine in planta.
86
Figure R10. Schematic representation of the N-acetylglucosamine utilization pathway
in Gram negative bacteria.
88 Figure S1. Growth of Xanthomonas campestris pv. campestris wild-type strain in the
presence of various carbon sources at 10 mM.
73 Figure S2. Conservation of genes belonging to the N-acetylglucosamine (GlcNAc)
utilization pathway among completely sequenced Xanthomonadaceae.
81 Figure S3. Conservation of genes belonging to GlcNAc and glycan clusters in
Xanthomonas campestris pv. campestris ATCC33913 and Novosphingobium aromaticivorans (Saro_DSM 12444).
91
Figure S4. Location of mutations introduced in genes belonging to the Xanthomonas
campestris pv. campestris N-acetylglucosamine utilization pathway and regions cloned
in expression plasmids.
94
Discussion
Figure D1. Représentation schématique du réseau de régulation du système GlcNAc
de Xanthomonas campestris pv. campestris.
112 Figure D2. Utilisation du phénotype du mutant ΔnagA pour suivre l’expression d’un
gène in planta.
113
Matériel et Méthodes
Figure M1. Principe de la construction des mutants par insertion du plasmide
pVO155.
116
Figure M2. Délétion d’un gène par la stratégie Cre/Lox. 117
Figure M3. Délétion d’un gène par la stratégie SacB. 117
Figure M4. Carte génétique du plasmide pCZ1034. 118
Figure M5. Index de la maladie développée par Xcc, après infection par « piercing »
de l’écotype Sf-2 d’A. thaliana ou de feuilles de choux Bartolo d’après Meyer et al. (2005).
119
Figure M6. Représentation schématique de la méthode de suivi de la croissance
bactérienne in planta.
Liste des tableaux
LISTE DES TABLEAUX
Interactions Plantes-Microorganismes
Tableau 1. Liste des bactéries phytopathogènes dont le génome est entièrement
séquencé et disponible, ainsi que les maladies provoquées par ces organismes.
4
Introduction générale
Chapitre 1
Tableau 2. Caractéristiques générales et comparaison des 3 génomes de Xanthomonas
campestris pv. campestris (Xcc) séquencés.
7
Chapitre 2
Tableau 3. Propriétés des différentes familles de protéines PR. 13
Chapitre 3
Tableau 4. Enzymes de dégradation de la paroi végétale de Xanthomonas campestris
pv. campestris, source da Silva et al., (2002).
22 Tableau 5. Effecteurs de type III connus chez la souche 85-10 de Xanthomonas
axonopodis pv. vesicatoria.
27
Etude de l’acquisition et de l’utilisation du GlcNAc
chez Xcc
Chapitre 2
Tableau 6. Enzymes impliquées dans le recyclage du peptidoglycane d’Escherichia
coli.
37 Tableau 7. Diversité des O-glycanes de type mucine rencontrés chez l’homme et leur
localisation tissulaire.
43
Chapitre 3
Tableau 8. Occurrence et caractéristiques des gènes impliqués dans l’utilisation de la
chitine et du GlcNAc.
61
Résultats
Table R1. Relative expression ratios of genes belonging to the N-acetylglucosamine
utilization pathway.
75
Table R2. [14C]N-acetylglucosamine transport rates related to Xanthomonas
campestris pv. campestris wild-type strain (Xcc568).
Table S1. Putative function of enzymes encoded by the Xanthomonas campestris pv.
campestris glycan cluster.
74 Table S2. Inhibition of [14C] N-acetylglucosamine uptake by various carbohydrates in
Xanthomonas campestris pv. campestris wild-type strain.
79 Table S3. List of plasmids and Xanthomonas campestris pv. campestris strains used or
generated in this study.
95
Matériel et Méthodes
Sommaire
1
SOMMAIRE
LES INTERACTIONS PLANTES-MICROORGANISMES ... 4
INTRODUCTION GENERALE ... 6
CHAPITRE 1. NOTRE MODÈLE D’ÉTUDE, LA BACTÉRIE PHYTOPATHOGÈNE XANTHOMONAS CAMPESTRIS PV. CAMPESTRIS
... 6
1.1. LE GENRE XANTHOMONAS... 6
1.2. LE GENOME DE XCC... 7
1.3. LES PLANTES HOTES DE XCC :LES BRASSICACEES... 8
1.4. LE CYCLE DE VIE DE XCC... 8
CHAPITRE 2. LES SYSTÈMES DE DÉFENSE DES PLANTES
... 10
2.1 LA RECONNAISSANCE DU « NON SOI » ET L’INDUCTION DE L’IMMUNITE INNEE... 10
2.1.2 Les éliciteurs généraux et la réponse basale ... 10
2.1.3 Les éliciteurs race spécifique Avr reconnus par les protéines de résistance R et la réponse hypersensible ... 12
2.2 LA REPONSE SYSTEMIQUE ACQUISE... 14
2.3 LA COEVOLUTION PLANTES/PATHOGENES, UN VERITABLE PING PONG... 15
CHAPITRE 3. LES DÉTERMINANTS DU POUVOIR PATHOGÈNE BACTÉRIEN
... 16
3.1 L’ADHESION... 16
3.2 LA MOBILITE ET LE CHIMIOTACTISME... 17
3.3 LES SYSTEMES D’ACQUISITION DU FER... 18
3.4 LA DEGRADATION DE LA PAROI VEGETALE ET L’ACQUISITION DES SUCRES... 20
3.4.1 Composition de la paroi primaire ... 20
3.4.2 La biodégradation de la paroi végétale par les bactéries ... 21
A. Dégradation de la pectine... 22
B. Dégradation de la cellulose ... 22
C. Dégradation de l’hémicellulose... 22
D. Dégradation des protéines ... 23
E. Le transport ... 23
F. La régulation ... 24
3.5 LE SYSTEME HRP ET SES EFFECTEURS... 25
3.5.1 La structure du T3SS ... 25
3.5.2 Les effecteurs de type III (T3SE) ... 26
3.5.3 Organisation et régulation des gènes du système Hrp... 27
3.6 LA PRODUCTION D’EPS... 28
3.7 LE QUORUM SENSING... 30
3.8 LES PHYTOTOXINES... 32
ETUDE DE L’ACQUISITION ET DE L’UTILISATION DU
N-ACETYLGLUCOSAMINE ... 34
CHAPITRE 1. INTRODUCTION AU TRAVAIL DE THÈSE
... 34
CHAPITRE 2. LE N-ACÉTYLGLUCOSAMINE DANS LE MONDE DU VIVANT
... 35
2.1 LE GlCNAC, UN ELEMENT DE STRUCTURE... 35
2.1.1 La paroi bactérienne ... 35
A. Le peptidoglycane ... 35
B. Le recyclage du peptidoglycane... 36
C. La paroi des bactéries à Gram+... 39
D. La paroi des bactéries à Gram-... 39
2.1.2 La chitine... 40
2.2 LE GlCNAC ET LES PROTEINES EUCARYOTES : LA GLYCOSYLATION... 41
2.2.1 Les protéines O-glycosylées ... 42
A. Les O-glycanes de type mucine ... 43
B. Les O-GlcNAc... 43
2.2.2 Les protéines N-glycosylées ... 44
2.2.3 Rôles des O- et des N-glycanes ... 47
2.3 LA GLYCOSYLATION CHEZ LES BACTERIES... 52
2.3.1 La O-glycosylation ... 52
2.3.2 La N-glycosylation ... 54
CHAPITRE 3. LE GlcNAc COMME SOURCE D’ÉNERGIE
... 56
3.1 LA DEGRADATION DE LA CHITINE... 56
3.2 LA DEGRADATION DES GLYCANES... 58
3.3 LE CATABOLISME DU GlCNAC... 59
3.3.1 Le transport du GlcNAc ... 60
3.3.2 L’étape de phosphorylation du GlcNAc ... 60
3.3.3 La déacétylation du GlcNAc (par NagA) ... 61
3.3.4 La déamination et l’isomérisation... 61
3.3.5 La régulation ... 62
CHAPITRE 4. LE GlcNAc ET LE POUVOIR PATHOGÈNE
... 65
RESULTATS ... 67
DISCUSSION GENERALE ... 107
MATERIELS ET METHODES ... 115
1 SOUCHES ET PLASMIDES... 115
2 MILIEUX ET RÉACTIFS... 115
3 BIOLOGIE MOLÉCULAIRE... 115
3.1. PREPARATION D’ADN GENOMIQUE... 115
3.2. CONJUGAISON TRIPARENTALE... 116
3.3. CONSTRUCTION DE MUTANTS D’INSERTION ET DE FUSIONS TRANSCRIPTIONNELLES116 3.4. CONSTRUCTION DES MUTANTS DE DELETION... 117
3.4.1 Par la méthode Cre/Lox ... 117
3.4.2 Par la méthode « SacB » ... 117
3.5. COMPLEMENTATION... 118
3.6. RT-PCRQUANTITATIVE (QRT-PCR) ... 118
4 TEST DU POUVOIR PATHOGÈNE DES DIFFÉRENTS MUTANTS DE XCC
119
4.1. INOCULATION PAR « PIERCING » SUR ARABIDOPSIS THALIANA SF2 ET CHOUX BARTOLO ... 119Sommaire
3 4.3. CROISSANCE BACTERIENNE IN PLANTA SUR ARABIDOPSIS THALIANA SF2 (IGC ;
INTERNAL GROWTH CURVE) ... 120
5 TEST DE STABILITÉ DES INSERTIONS pVO IN PLANTA
... 121
6 TESTS PHÉNOTYPIQUES
... 121
6.1. TEST DU ROLE DU GlCNAC COMME SOURCE D’AZOTE... 121
6.2. EFFET TOXIQUE DU GlCNAC ET DE LA PHASE STATIONNAIRE DE CROISSANCE SUR LA CROISSANCE DE MUTANTS... 121
7 DOSAGES D’ACTIVITÉS ENZYMATIQUES
... 122
7.1. DOSAGES DE L’ACTIVITE β-GLUCURONIDASE... 122
7.2. AUTRES ACTIVITES ENZYMATIQUES... 123
8 TESTS DE TRANSPORT DE [14C]GlcNAc
... 123
9 TESTS DE PHOSPHORYLATION DU GlcNAc
... 124
10 ANALYSES IN SILICO
... 125
D'après http://cpgr.plantbiology.msu.edu/cgi-bin/warehouse/cpgr_warehouse.cgi?group=Bacteria&status=Finished
Groupe Espèce Maladie
Taille du génome (MégaBases)
Gram -
α-proteobactéries Agrobacterium tumefaciens str. C58 galle du collet (crown gall) 5.67
Burkholderia cenocepacia AU 1054 pourriture aigre (sour skin) de l'oignon - pathogène sur
patients atteints de fibrose kystique 7.25
Burkholderia cenocepacia HI2424 pourriture aigre (sour skin) de l'oignon - pathogène sur
patients atteints de fibrose kystique 8.09
Burkholderia cenocepacia J2315 pourriture aigre (sour skin) de l'oignon - pathogène sur
patients atteints de fibrose kystique 8.07
Burkholderia cenocepacia MC0-3 pourriture aigre (sour skin) de l'oignon - pathogène sur
patients atteints de fibrose kystique 7,9
Ralstonia solanacearum GMI1000 flétrissement bactérien (bacterial wilt) 5.81
Erwinia carotovora subsp. atroseptica SCRI1043 pourriture molle (soft rot) et jambe noire (blackleg) de la
pomme de terre 5.06
Pseudomonas aeruginosa PA7 pourriture molle (soft rot) 6,6
Pseudomonas aeruginosa PAO1 pourriture molle (soft rot) - pathogènes sur patients atteints
de fibrose kystique 6.26
Pseudomonas aeruginosa UCBPP-PA14 pourriture molle (soft rot) - pathogènes sur patients atteints
de fibrose kystique 6.53
Pseudomonas syringae pv. phaseolicola 1448A graisse à halo du haricot (halo blight of bean) 6.11
Pseudomonas syringae pv. syringae B728a graisse du haricot (brown spot) 6.09
Pseudomonas syringae pv. tomato str. DC3000 moucheture bactérienne (bacterial speck) de la tomate 6.54
Xanthomonas axonopodis pv. citri str. 306 chancre des agrumes (citrus canker) 5.27
Xanthomonas campestris pv. campestris str. 8004 pourriture noire (black rot) des crucifères 5.15
Xanthomonas campestris pv. campestris str. ATCC 33913 pourriture noire (black rot) des crucifères 5.08
Xanthomonas campestris pv. campestris str. B100 pourriture noire (black rot) des crucifères 5.08
Xanthomonas axonopodis pv. vesicatoria str. 85-10 tache bactérienne (bacterial spot) de la tomate et du poivron 5.42
Xanthomonas oryzae pv. oryzae KACC10331 rouille des feuilles (bacterial blight) du riz 4.94
Xanthomonas oryzae pv. oryzae MAFF 311018 rouille des feuilles (bacterial blight) du riz 4.94
Xanthomonas oryzae pv. oryzae PXO99A rouille des feuilles (bacterial blight) du riz 5,24
Xylella fastidiosa 9a5c chlorose variégée des agrumes (citrus variegated chlorosis) 2.73
Xylella fastidiosa M12 chlorose variégée des agrumes (citrus variegated chlorosis) 2,48
Xylella fastidiosa M23 chlorose variégée des agrumes (citrus variegated chlorosis) 2,5
Xylella fastidiosa Temecula1 maladie de Pierce (Pierce's disease) du raisin 2.52
Clavibacter michiganensis subsp. sepedonicus pourriture annulaire (ring rot) de la pomme de terre 3.38
Leifsonia xyli subsp. xyli str. CTCB07 rabougrissement des repousses (ratoon stunting) de la
canne à sucre 2.58
Streptomyces scabies 87-22 gale commune (scab) de la pomme de terre 10.15 Gram + Actinobactéries Gram - β-proteobactéries Gram - γ-proteobactéries
Les interactions plantes-micoorganismes
4
1
Les interactions Plantes-Microorganismes
Au cours de sa vie, la plante va cohabiter et interagir avec de nombreux microorganismes tels que les virus, les mollicutes (mycoplasmes et spiroplasmes), les champignons, les bactéries et les nématodes. On peut définir trois types d’interactions, les interactions de type symbiotique, de type saprophytique et les interactions de type pathogénique.
Aujourd’hui, le terme symbiose est souvent associé au mutualisme, défini par l’association de deux organismes hétérospécifiques à bénéfice mutuel. Cependant, les interactions symbiotiques incluent le commensalisme (l’association n’est bénéfique que pour l’un des deux partenaires, le second n’est pas affectés par cette interaction), l’amensalisme (un des acteurs inhibe le développement de l’autre), et le parasitisme (l’un des acteurs se développe au détriment de l’autre). L’organisme le plus grand est appelé hôte, alors que le plus petit est nommé symbiote ou symbionte.
Les microorganismes saprophytes quant à eux, se nourrissent par absorption de matières organiques mortes, inertes ou en décomposition. Certaines bactéries ont développé une relation étroite avec la plante, leur permettant d’échapper à la compétition avec d’autres microorganismes. Ces bactéries vivent en saprophytes au niveau de la rhizosphère ou de la phyllosphère sans développer de relation mutualiste ou parasite avec la plante.
Tout organisme parasite provoquant une maladie est dit pathogène. Le pouvoir pathogène d’un organisme correspond à la capacité de cet organisme à développer une maladie sur son hôte, tandis que la virulence d’un organisme correspond à une notion quantitative de l’intensité du pouvoir pathogène. Un organisme pathogène peut avoir des souches plus ou moins virulentes.
Pour induire une maladie, une bactérie pathogène doit être capable d’adhérer, de coloniser et d’envahir l’hôte, de se multiplier et par conséquent d’adapter son métabolisme à l’environnement de l’hôte, d’échapper aux systèmes de défense de l’hôte et de résister aux différents stress rencontrés et enfin de survivre entre deux cycles infectieux. Tous les éléments bactériens permettant la mise en place de ces différentes étapes sont définis comme facteurs de virulence. Il existe de nombreuses espèces de bactéries phytopathogènes (infectant les végétaux) regroupées majoritairement dans trois classes de protéobactéries, les α-, les β- et les γ-protéobactéries. Le génome de certaines de ces bactéries a été séquencé (Tableau 1).
Dans certains cas, la distinction entre un organisme saprophyte, symbiotique ou pathogène n’est pas toujours évidente. Certaines bactéries peuvent être saprophytes puis pathogènes au cours de différentes étapes de leur cycle de vie. La bactérie Burkholderia
pseumallei, par exemple, est une bactérie saprophyte du sol également responsable de la
mélioïdose, maladie humaine de l’Asie du sud-est (Thaïlande, Myanmar, Singapour, Malaisie, Laos, Cambodge, Vietnam) (Kaestli et al., 2007).
Dans l’introduction générale, après une brève présentation de notre modèle d’étude, je vous présenterai les systèmes de défenses mis en place par les plantes puis je vous parlerais des facteurs de virulence impliqués dans le développement de la maladie en prenant pour exemple notre modèle d’étude. Dans une seconde partie, je développerai les différents aspects du rôle du N-acetylglucosamine (GlcNAc) dans le monde du vivant. Enfin, je discuterai les résultats obtenus au cours de ma thèse permettant la mise en évidence d’un système particulier, dédié à l’acquisition et à l’utilisation du GlcNAc au cours de la vie in planta de
1
Chapitre 1. Notre modèle d’étude, la bactérie
phytopathogène Xanthomonas campestris pv.
campestris
L’agriculture génère des conditions favorables au développement de maladies voire d’épidémies par la culture d’une même espèce de plante sur de grandes surfaces. Malgré les progrès effectués ces derniers siècles en pratiques phytosanitaires, le développement du commerce international, l’accroissement des volumes de production et la politique de réduction de la diversité des espèces cultivées favorisent l’émergence d’épidémies. L’élargissement de nos connaissances en phytopathologie est donc d’une importance économique et écologique majeure afin d’établir des plans de luttes durables et efficaces contre ces épidémies. Pour cela, des études approfondies des mécanismes de défense des plantes d’une part et de mise en place de cycles infectieux d’autre part, sont indispensables à une meilleure compréhension des interactions entre les plantes et leurs parasites.
C’est dans ce contexte que nous avons choisi d’étudier les déterminants de l’adaptation à son milieu et de la mise en place du pouvoir pathogène de la bactérie phytopathogène Xanthomonas campestris pv campestris (Xcc). La pourriture noire (black rot) ou autrement appelée nervure noire causée par Xcc est la maladie des Brassicacées (anciennement nommées Crucifères) la plus préjudiciable de par le monde (Williams, 1980). Toutes les Brassicacées maraîchères sont sensibles à cette maladie, sauf certains cultivars de radis et de chou frisé, qui sont moins facilement infectés.
1.1. Le genre Xanthomonas
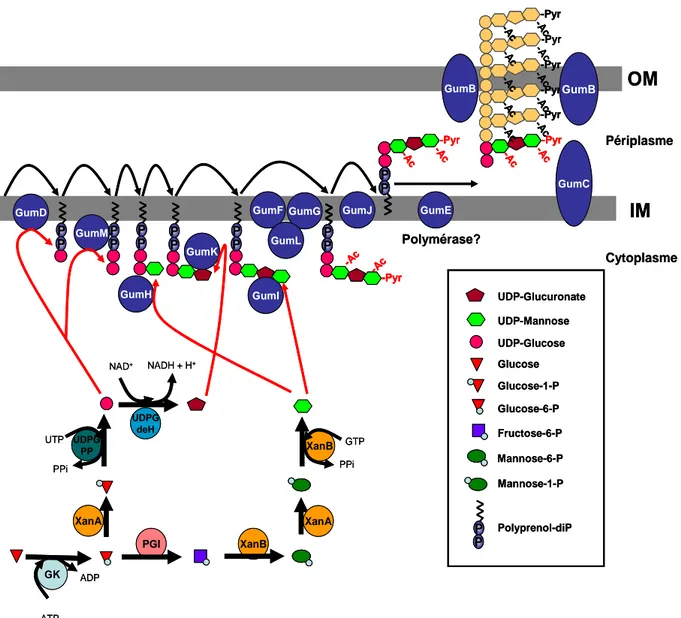
Les bactéries du genre Xanthomonas sont des bactéries à Gram négatif faisant partie de la classe des γ-protéobactéries. Ces bactéries sont aérobies strictes et ont pour particularité de produire des caroténoïdes appelés xanthomonadines qui pigmentent les colonies en jaune. D’autre part Xcc est très étudiée pour sa capacité à produire un exopolysaccharide particulier, le xanthane qui donne un aspect mucoïde aux colonies (voir paragraphe 3.6), très utilisé comme agent épaississant dans les industries cosmétiques, agroalimentaires anisi que dans le bâtiment.
Xcc 8004 Xcc ATCC33913 a Xcc B100
Caractéristiques générales
Taille du génome (pb) 5 148 708 5 076 187 5 079 002
Pourcentage en GC 64.94% 65.00% 65.00%
Nombre total de séquences codantes (CDS) 4273 (87)b 4181 4471
Nombre de CDS avec fonction assignée 2671 (1)b 2708 2878 Nombre de CDS codant de putatives protéines conservées 1523 (27)b 1276 1323
Nombre de CDS codant des protéines hypothétiques 79 (59)b 198 270
Différences de séquences dans les CDS
Nombre de gènes parfaitement identiques 3467c 3408 Nombre de gènes identiques, de même taille, avec SNP 498 500c
Nombre de gènes identiques avec insertions ou délétions 200 211c Nombre de gènes spécifiques de la souche 108 62
Taille moyenne des CDS (pb) 1023 1030
Nombre de séquences d'insertion (IS) 115 109
c Traduit l'existence de duplications de CDSs.
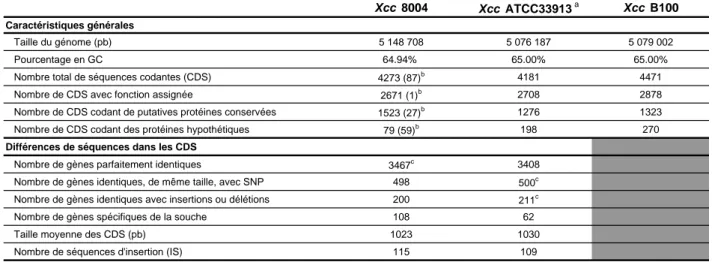
Tableau 2 : Caractéristiques générales et comparaison des 3 génomes de Xanthomonas campestris pv. campestris (Xcc ) séquencés.
a
Données de da Silva et al . (2002) et GenBank (accession n° AE008922).
CDS, Coding DNA Sequences ; SNP, Single Length Polymorphism ; IS, Séquences d'insertion. (D'après Qian et al. , 2005 ; Vorholter et al. , 2008).
b
Les nombres entre parenthèses indiquent le nombre de CDS possédant une protéine identique chez Xcc ATCC33913 mais non annotés par da Silva et al. (2002).
Les membres du genre Xanthomonas infectent 124 espèces de plantes monocotylédones et 268 espèces de plantes dicotylédones (Leyns et al., 1984). Ce genre regroupait dans les années 90, plus de 170 espèces dont bon nombre sont des espèces phytopathogènes subdivisées en plus de 140 pathovars selon leurs plantes hôtes (http://www.ncbi.nlm.nih.gov/Taxonomy). Des analyses d’hybridation ADN-ADN (Vauterin
et al., 1995) ont ensuite permis de reclasser ces bactéries, menant à la description de 20
espèces. L’espèce Xanthomonas campestris comporte six pathovars : aberrans, armoraciae,
barbareae, campestris, incanae et raphani, reclassés par la suite en trois pathovars, campestris, raphani, incanae par des analyses de pouvoir pathogène (Fargier and Manceau,
2007).
1.2. Le génome de Xcc
Le séquençage de trois souches de Xcc, la souche ATCC33913 séquencée au Brésil (da Silva et al., 2002), la souche 8004 séquencée en Chine (Qian et al., 2005) et la souche B100 séquencée en Allemagne à l’université de Bielefeld (Vorholter et al., 2008), a facilité l’étude des bases moléculaires de l’interaction de la bactérie avec la plante. Les trois génomes sont dépourvus de plasmide, présentent un fort pourcentage en GC (65%) et sont riches en éléments transposables (Tableau 2). Le degré de conservation et la colinéarité entre les deux génomes des souches ATCC33913 et 8004 sont en général bien respectés, bien que plusieurs réarrangements aient été détectés entre les chromosomes de ces deux souches. Cependant, ces deux souches présentent de légères différences dans leur pouvoir pathogène sur différentes plantes. La souche ATCC33913 serait légèrement moins agressive sur certains cultivars de chou et de radis (Qian et al., 2005). La souche 8004 présente 92 séquences codantes putatives (CDS) supplémentaires par rapport à la souche ATCC33913, ainsi qu’un nombre plus élevé de gènes « souche spécifiques » (108 contre 62). Des SNP (Single Nucleotide Polymorphism) sont observés dans 12% des CDS, et des réarrangements génomiques significatifs (translocations, inversions, insertions et délétions) différencient les deux souches (Qian et al., 2005).
Figure 1 : Quelques exemples de Brassicacées cultivées et la plante modèle
Arabidopsis thaliana.
A. Chou frisé (http://www.flickr.com/photos/shutterblade/1457241213/)
B. Fleurs d’Arabidopsis thaliana (http://www.flickr.com/photos/jakedude/2470384257/) C. Chou rouge (http://www.flickr.com/photos/mcorreia/1027409405/)
D. Choux-fleurs (http://www.flickr.com/photos/22901003@N00/2084825155/) E. Choux raves (http://www.flickr.com/photos/a_rud_beth/2395348561/)
F. Choux de Bruxelles (http://www.flickr.com/photos/89223198@N00/2670656349/)
A
B
C
E
F
1.3. Les plantes hôtes de Xcc : Les Brassicacées
Xcc est capable d’infecter de nombreuses Brassicacées telles que le chou-fleur, le
brocoli, le rutabaga, le radis, le navet, le chou de Bruxelles, la moutarde, le canola, et de façon intéressante la plante modèle Arabidopsis thaliana dont le génome est séquencé (Bent et al., 1992; Lummerzheim et al., 1993; Meyer et al., 2005; Parker et al., 1993; Simpson and Johnson, 1990). Ceci facilite les études d’interaction entre Xcc et son hôte.
La famille des Brassicacées, anciennement Crucifères, consiste en une importante famille de plantes dicotylédones. Leurs fleurs sont composées de quatre pétales disposés en forme de croix. Elle comprend plus de 3000 espèces réparties en 350 genres. Ce sont principalement des plantes herbacées présentes dans le monde entier mais principalement localisées dans les régions tempérées de l’hémisphère nord. Les Brassicacées colonisent presque tous les habitats : sables et rochers maritimes, bords de ruisseaux, pelouses, cultures, prairies de montagnes, etc. Parmi les plantes appartenant à cette famille, on trouve de nombreuses plantes cultivées pour la production d’huile, la consommation humaine (choux, brocolis, navets…) et animale, ainsi que des plantes d’ornement (Fig. 1).
Une des caractéristiques intéressante des Brassicacées est la production de métabolites secondaires, les glycosinolates, qui ont un rôle dans la défense contre les insectes. Ces composés sont stockés dans la plante et libérés lors d’une attaque de phytophages. Le glycosinolate est hydrolysé par la myrosinase en molécule toxique, l’isothiocyanate. Ce dernier va être ingéré par les phytophages (Pfalz et al., 2007).
1.4. Le cycle de vie de Xcc
Xcc est une bactérie épiphyte vivant à la surface des feuilles. Lorsque les conditions
environnementales sont favorables, correspondant à une température comprise entre 25 et 30°C et un taux d’humidité élevé, elle va développer un cycle infectieux sur sa plante hôte. La première étape d’infection de Xcc (Fig. 2) correspond à l’entrée de la bactérie dans les tissus de la feuille. L’entrée d’une bactérie dans les tissus de la plante peut s’effectuer grâce à des blessures, à l’aide d’insectes qui vont servir de vecteur ou encore par pénétration via des ouvertures naturelles. En effet, les bactéries de la phyllosphère ne peuvent avoir accès directement aux tissus internes des feuilles, ceux-ci étant protégés par la cuticule et l’épiderme. Plusieurs structures naturelles vont pouvoir permettre l’entrée de ces bactéries. Le
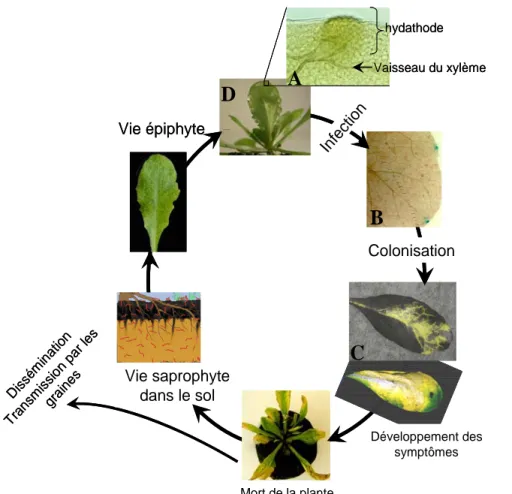
Figure 2 : Représentation schématique du cycle de vie de Xanthomonas campestris pv.
campestris (Xcc) lors d’une interaction avec la plante hôte.
Après une phase de vie épiphyte, Xcc va, dans certaines conditions, pénétrer à l’intérieur des tissus végétaux par les hydathodes. La bactérie va ensuite pouvoir envahir les vaisseaux du xylème et induire les symptômes de la maladie. Enfin, la bactérie va survivre dans le sol et sur les débris végétaux après l’infection. La dissémination des bactéries s’effectue par le vent, la pluie, les insectes, mais aussi et surtout par le transport et l’utilisation de graines contaminées.
A. Vue en microscopie d’un hydathode d’Arabidopsis thaliana (Photo de Jacques Vasse, LIPM)
B. Xcc exprimant le gène uidA codant la glucuronidase, constitutivement (D’après Hugouvieux
et al., 1998). On voit ici les bactéries au niveau des hydathodes
C. Xcc exprimant le gène de la luciférase (Lux) constitutivement (D’après Meyer et al., 2005). Cette photo illustre la colonisation des vaisseaux du xylème par les bactéries.
D. Gouttes de guttation présentes à la marge d’une feuille d’Arabidopsis thaliana. (D’après Blanvillain 2007). Dis sém inat ion Tran smis sion par l es grai nes Vie épiphyte Infe ctio n Colonisation Vie saprophyte dans le sol
B
C
A
hydathode Vaisseau du xylème Mort de la plante Développement des symptômesD
Dis sém inat ion Tran smis sion par l es grai nes Vie épiphyte Infe ctio n Colonisation Vie saprophyte dans le solB
C
A
hydathode Vaisseau du xylème Mort de la plante Développement des symptômesD
Figure 3 : Symptômes de la nervure noire sur les feuilles de choux.
Zones chlorotiques en forme de « V » produites par l’infection du chou par la bactérie
Xanthomonas campestris pv. campestris (Xcc)
site d’entrée naturel de Xcc est un stomate aquifère localisé à la marge des feuilles, l’hydatode (Hugouvieux et al., 1998). Les hydathodes sont localisés à l’aboutissement des vaisseaux du xylème et sont impliqués dans les phénomènes de transpiration de la plante ou phénomène de guttation, lorsque la pression racinaire due à l’humidité élevée est trop forte. Les bactéries vivant à la surface des feuilles vont se retrouver dans les gouttes de guttation est être absorbées en même temps que ces gouttes lorsque l’humidité et donc la pression racinaire va diminuer. Xcc peut dans certains cas, entrer dans les tissus via des blessures ou par les stomates.
Une fois localisée dans les vaisseaux du xylème, Xcc va pouvoir commencer la colonisation de la plante et provoquer l'apparition de lésions jaunes en forme de « V » devenant brunes et nécrosées au fil de leur progression vers la base des feuilles (Fig. 2 et 3). La production de xanthane et la croissance bactérienne vont boucher les vaisseaux entraînant le flétrissement et la nécrose des feuilles qui vont se dessécher complètement pour finir par tomber (Onsando, 1992; Williams, 1980).
Xcc est capable de survivre pendant plusieurs années en saprophyte sur des débris
végétaux et dans le sol, et se transmet à la graine lors de la production des semences. Les jeunes plants cultivés à partir de ces graines contaminées sont souvent infectés de façon systémique (Shaad et al., 1980; Shultz and Gabrielson, 1986). Les semences infectées et l’eau de pluie sont la source majeure de dissémination de la bactérie.
Pour éviter une épidémie, il faut sélectionner des semences saines, alterner les cultures avec des plantes qui ne sont pas des Brassicacées ou des écotypes résistants, travailler dans les champs seulement quand le feuillage est sec, éviter l’arrosage par aspersion et irriguer au niveau des pieds et utiliser des pesticides pour éviter la dissémination via les insectes (http://vegetablemdonline.ppath.cornell.edu/factsheets/Crucifers_BR.htm).
Introduction générale
10
2
Chapitre 2. Les systèmes de défense des plantes
En général, l’infection d’une plante par un pathogène reste une exception. Aux cours de leur coévolution, les plantes ont développé des stratégies élaborées leur permettant de se défendre efficacement contre les agents pathogènes. Il existe trois niveaux de défense de la plante. Tout d’abord on trouve les barrières physiques : le microorganisme ne peut tout simplement pas entrer dans la plante. Cette fonction est assurée entre autres par les cires cuticulaires ou la paroi végétale. Si ces défenses ne suffisent pas, la plante peut détecter la présence de l’agent pathogène afin d’initier la mise en place de systèmes de défense locales et/ou systémiques.
2.1 La reconnaissance du « non soi » et l’induction de
l’immunité innée
La reconnaissance du microorganisme pathogène par la plante et l'induction des mécanismes de défense sont initiées par la perception de molécules appelées éliciteurs ou effecteurs. Les éliciteurs sont des signaux microbiens classiquement regroupés en deux catégories, les éliciteurs généraux qui provoquent des réactions sur tous les cultivars d’une espèce végétale donnée et les éliciteurs race-spécifiques qui n’ont d’effet que sur des cultivars particuliers et qui sont codés par des gènes dits d’avirulence. Ces systèmes de « chimioréception » permettent à la plante de percevoir la présence du « non soi » (Boller, 1995). La reconnaissance du pathogène via des récepteurs spécifiques va entraîner une reprogrammation génétique des cellules de l’hôte et induire une réponse de défense généralement suffisante pour stopper la croissance du pathogène (Chisholm et al., 2006; Dangl and Jones, 2001; Nurnberger et al., 2004; Zipfel and Felix, 2005).
2.1.2 Les éliciteurs généraux et la réponse basale
Les éliciteurs généraux aussi appelés PAMPs pour « Pathogen-Associated Molecular Patterns » sont généralement des constituants stables et héréditaires, caractéristiques de l’agent pathogène, souvent structuraux et de surface. On les appelle également MAMPs pour « Microbes-Associated Molecular Patterns ». Ce terme comprend également les molécules
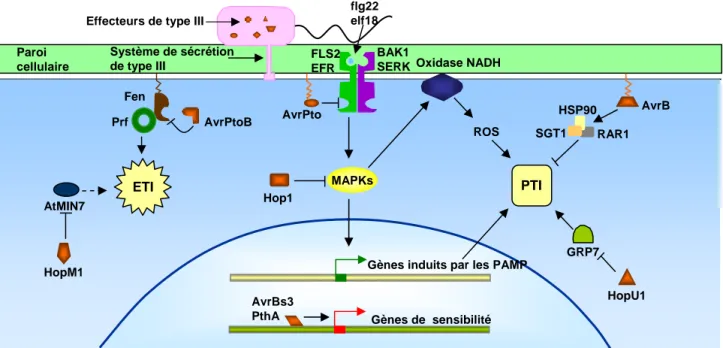
Figure 4 : Signalisation de la réponse basale et cibles de certains effecteurs de type III connus.
FLS2 et EFR sont des exemples de PRRs de surface. Le récepteur SERK est similaire à BAK1 et pourrait se dimériser avec les PRRs au moment de la stimulation par le ligand. Les protéines RAR1, GRP7, et AtMIN7 régulent les réponses de défense au niveau de la paroi végétale via des mécanismes inconnus. RAR1 réprime le dépôt de callose induit par flg22, alors que GRP7 induit le dépôt de callose lors de la reconnaissance des PAMPs. AtMIN7 pourrait jouer un rôle dans la voie ETI (ligne en pointillé).
L’effecteur AvrPto inhibe la transduction du signal par FLS2 et EFR pour bloquer la voie PTI. L’effecteur HopAI1 inactive la signalisation MAP Kinase inhibant le voie PTI. HopU1 inhibe la voie PTI en modifiant la protéine GRP7. AvrB augmente l’activité de RAR1 pour inhiber la voie PTI. L’effecteur AvrPtoB induit l’ubiquitination et la dégradation de la protéine Fen et empêche l’initiation de la voie ETI via la protéine Prf (Protéine NBS-LRR). HopM1 déstabilise AtMIN7 pour inhiber les défenses au niveau de la paroi végétale. Les effecteurs de la famille AvrBs3/PthA se lient directement aux séquences promotrices des gènes de plantes et activent la transcription de certains gènes.
Les effecteurs de type III sont en orange.
PAMP, Pathogen-Associated Molecular Patterns ; ETI, effector-triggered immunity ; PTI, PAMP-triggered immunity ; NBS-LRR, Nucleotide Binding Site with Leucine Rich Repeat domain.
(D’après Zhou and Chai, 2008).
MAPKs HopM1 AtMIN7 Paroi cellulaire ETI PTI Prf Fen AvrPtoB AvrPto Hop1
Gènes induits par les PAMP
Gènes de sensibilité AvrBs3 PthA ROS GRP7 HopU1 AvrB RAR1 HSP90 SGT1 Oxidase NADH Système de sécrétion de type III BAK1 SERK FLS2 EFR
Introduction générale
11 reconnues provenant de microorganismes non pathogènes. Cette notion de PAMPs est également valable chez les animaux, ces molécules vont alors induire les systèmes de défense de la réponse innée. Ces éliciteurs peuvent être de nature très variée. Il existe des PAMPs de natures peptidiques ou protéiques, comme les peptidoglycanes (Erbs et al., 2008), la flagelline (Felix et al., 1999; Sun et al., 2006) ou le facteur d’élongation EF-Tu (Zipfel et al., 2006) qui peuvent être reconnus par la plante et induire les réponses de défense des plantes. La flagelline par exemple, est un puissant éliciteur. Un peptide de seulement 22 acides aminés (fgl22), situé au niveau N-terminal de la flagelline, peut être reconnu par le récepteur FLS2 et induire une réponse de défense chez Arabidopsis (Gomez-Gomez and Boller, 2002; Zipfel et
al., 2004). On trouve également des PAMPs de nature oligosaccharidique ou lipidique comme
les lipopolysaccharides (LPS) qui sont des constituants de la surface bactérienne. Les PAMPs peuvent aussi être produits à partir des constituants de la plante suite à une activité microbienne. On les appelle alors HAMPs pour « Host-Associated Molecular Patterns » (de Lorenzo, 2007) ou MIMPS pour « Microbe-Induced Molecular Patterns » (Bent and Mackey, 2007; Mackey and McFall, 2006). C’est le cas des oligosaccharides provenant de la dégradation de la paroi végétale par les enzymes bactériennes (voir chapitre 3.4) comme les oligogalacturonates ou les glucanes (de Lorenzo, 2007).
Les plantes reconnaissent ces substances avec une très grande sensibilité (Felix et al., 1999) [pour revue (Zipfel, 2008)]. Les récepteurs FLS2 reconnaissant la flagelline et EFR reconnaissant EF-Tu, sont des protéines transmembranaires possédant un domaine extracellulaire de type LRR (Leucine Rich Repeat) et un domaine intracellulaire de type Kinase. On appelle ce type de récepteurs aux PAMPs des RLK pour « Receptors-Like Kinase » ou PRR pour « Pattern Recognition Receptor ». La reconnaissance des PAMPs par ces récepteurs induit l’immunité basale équivalente à l’immunité innée des mammifères (Nurnberger et al., 2004; Zipfel and Felix, 2005). Cette reconnaissance est nommée voie PTI pour “PAMP-triggered immunity” [(Chisholm et al., 2006) Fig. 4]. Au cours d’un premier niveau de défense, la voie PTI va être induite via une cascade de signalisation de type MAP Kinase et une reprogrammation transcriptionnelle. Cette reprogrammation effectuée grâce à des facteurs de transcription de type WRKY, va mener à l’induction de la production d’espèces réactives de l’oxygène (ROS) telles que les ions super oxydes (O2-) et l’H2O2
(Delledonne et al., 2002; Torres et al., 2006) et d’oxyde nitrique (NO). Elle ménera également à un flux rapide d’ions à travers la membrane plasmique, une alcalinisation du milieu et un renforcement de la paroi par lignification et production de callose (polymère de D-glucose) ainsi qu’une augmentation de la synthèse de protéines de parois riches en glycine (GRP), en
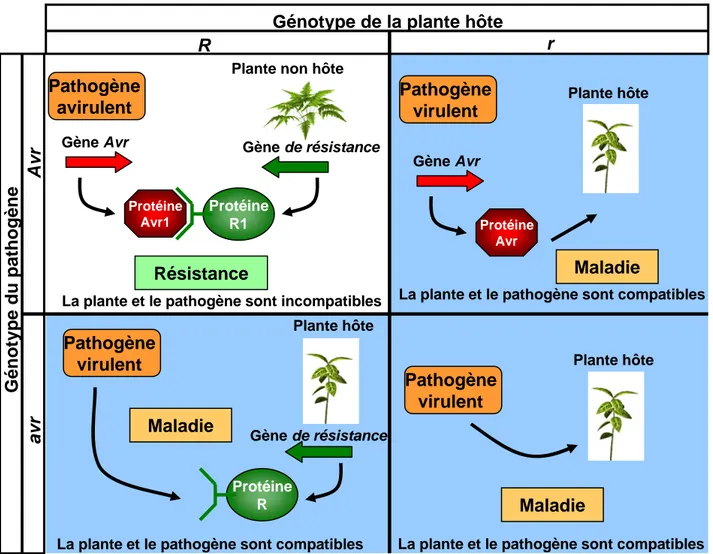
Figure 5 : Représentation de la théorie “Gène pour Gène” de Flor (1971).
La reconnaissance d’une protéine dite d’avirulence de la bactérie par le produit d’un gène de résistance de la plante mène à une interaction incompatible. On dit alors que la plante est non hôte et que la bactérie est avirulente. Lorsque l’un des deux composants est absent, la relation devient compatible et la bactérie est capable de provoquer la maladie.
Pathogène avirulent
Gène Avr
Protéine Avr1
Plante non hôte
Gène de résistance Protéine R1 Résistance Plante hôte Pathogène virulent Gène Avr Protéine Avr Maladie Gène de résistance Protéine R Pathogène virulent Maladie R r Génot y p e du pathogène avr Avr
La plante et le pathogène sont incompatibles
Plante hôte
Pathogène virulent
Maladie
La plante et le pathogène sont compatibles La plante et le pathogène sont compatibles
La plante et le pathogène sont compatibles Plante hôte
Introduction générale
12 proline (PRP) ou en hydroxyproline (HPRP) et de subérine (Chisholm et al., 2006; Hardham
et al., 2007; Maor and Shirasu, 2005). Cependant, ces mesures ne sont pas toujours suffisantes
pour stopper la progression du pathogène, et d’autres systèmes de résistance doivent être mis en place.
2.1.3 Les éliciteurs race spécifique Avr reconnus par les
protéines de résistance R et la réponse hypersensible
Le mécanisme moléculaire qui conduit à l'induction de la résistance est classiquement expliqué par le modèle de H.H. Flor appelé modèle "gène pour gène" (Flor, 1971) (Fig. 5). Pour que la plante soit résistante à un agent pathogène spécifique, une paire de gènes complémentaires de la plante (gène de résistance R) et de l'agent pathogène (gène d'avirulence
avr) doit exister. L'interaction directe ou indirecte entre les produits de ces deux gènes conduit
à la résistance. La plante est dite non hôte ou hôte résistant, l’interaction est dite incompatible et le pathogène est avirulent. L'absence ou l'altération de l'un ou l'autre des gènes conduit au développement de la maladie. La plante devient alors hôte, l’interaction compatible et le pathogène virulent (Fig. 5).
La présence ou non de gènes de résistance correspondant au gène d’avirulence va déterminer le spectre d’hôte de l’agent pathogène. Chez les bactéries phytopathogènes, de nombreux facteurs d'avirulence (plus de 40) ont majoritairement été isolés chez Pseudomonas
syringae et Xanthomonas (Gopalan et al., 1996). Parmi ces gènes d’avirulence, on trouve des
gènes codant des effecteurs de type III qui sont des protéines transloquées par les bactéries dans la cellule eucaryote grâce à un système de sécrétion de type III (voir chapitre 3.5). Certains de ces effecteurs vont agir sur les voies de signalisation de défense pour les inhiber (Fig. 4). En effet, certains effecteurs de type III des bactéries vont pouvoir bloquer la voie PTI. Dans certains cas, ces effecteurs peuvent également trahir la présence du pathogène et induire d’autres systèmes de défense. La plante va alors reconnaître ces effecteurs grâce à des protéines de résistance (Dangl and Jones, 2001; Van der Biezen and Jones, 1998) et initier la voie ETI (Effector-Triggered Immunity) qui correspond à une réponse PTI beaucoup plus forte et qui mène systématiquement à une mort cellulaire programmée (PCD, Programmed Cell Death ; ou apoptose) nommée réponse hypersensible (HR) (Fig. 4). La HR va permettre de confiner l’agent pathogène dans la zone d’infection (Lam et al., 2001). Elle a lieu dans les heures qui suivent l’infection et peut être initiée par la reconnaissance de la plupart des microorganismes phytopathogènes. Les cellules qui meurent émettent alors des molécules
(D'après Sels et al. , 2008)
Famille Membre type Taille
typique (kDa)
Propriété Cible microbienne proposée
Référence d'origine
PR-1 Tobacco PR-1a 15 Antifongique Inconnue (Antoniw et al , 1980)
PR-2 Tobacco PR-2 30 β-1,3-Glucanase β-1,3-Glucane (Antoniw et al , 1980)
PR-3 Tobacco P, Q 25–30 Chitinase (class I, II, IV, V, VI, Chitine (Van Loon, 1982) PR-4 Tobacco "R" 15–20 Chitinase de classe I, II Chitine (Van Loon, 1982)
PR-5 Tobacco S 25 "Thaumatin-like" Membrane (Van Loon, 1982)
PR-6 Tomato Inhibitor I 8 Inhibiteur de protéase a (Green et Ryan, 1972)
PR-7 Tomato P69 75 Endoprotéase a (Vera et al , 1988)
PR-8 Cucumber chitinase 28 Chitinase de classe III Chitine (Métraux et al , 1988)
PR-9 Tobacco "lignin-forming" 35 Péroxidase a (Lagrimini et al , 1987)
PR-10 Parsley "PR1" 17 ‘Ribonuclease-like’ a (Somssich et al , 1986)
PR-11 Tobacco "class V" 40 Chitinase de class I Chitine (Melchers et al , 1994)
PR-12 Radish Rs-AFP3 5 Défensine Membrane (Terras et al , 1995)
PR-13 Arabidopsis THI2.1 5 Thionine Membrane (Epple et al , 1995)
PR-14 Barley LTP4 9 Protéine de transfer de lipides Membrane (Garcia-Olmedo et al , 1995)
PR-15 Barley OxOa (germin) 20 Oxalate oxidase a (Zhang et al , 1995)
PR-16 Barley OxOLP 20 ‘Oxalate oxidase-like’ a (Wei et al , 1998)
PR-17 Tabac PRp27 27 Inconnue a (Okushima et al , 2000)
Le tableau a été adapté de http://www.bio.uu.nl/wfytopath/PR-families.htm a Pas d'activité antimicrobienne identifiée in vitro