Étude de la régulation génique par les microARN
dans les cellules germinales
Thèse
Alexandra Dallaire
Doctorat en biologie cellulaire et moléculaire
Philosophiae doctor (Ph. D.)
Québec, Canada
Étude de la régulation génique par les microARN
dans les cellules germinales
Thèse
Alexandra Dallaire
Sous la direction de :
Résumé
La régulation post-transcriptionnelle joue un rôle essentiel dans le contrôle du développement des animaux et des maladies. Parmi les mécanismes de régulation post-transcriptionnelle identifiés à ce jour, les microARNs jouent un rôle central. Ils constituent une large classe de courts ARNs régulateurs qui ont été découverts chez le nématode C. elegans et ont été répertoriés chez tous les métazoaires. Les microARN sont transcrits par l'ARN polymérase II sous forme d'un long transcrit primaire et vont subir deux étapes de clivage successives pour former le microARN mature. Le microARN mature est pris en charge par une protéine Argonaute pour former un complexe effecteur, le miRISC. Les microARN s’associent avec la région 3' non traduite de leurs ARNm cibles pour réprimer leur traduction et/ou initier leur dégradation. L'orchestration dynamique de ces processus n'est pas bien comprise, mais on pense actuellement qu'elle dépend en grande partie d'un partenaire clé des Argonautes, GW182. Les cellules germinales sont la source d’ARNm maternels dont la régulation est essentielle aux premières étapes de l’embryogénèse. Le maintien de la stabilité de ces transcrits est assuré par des mécanismes post-transcriptionnels. Les microARNs sont abondants dans la lignée germinale et donc pourraient potentiellement participer à cette régulation. C’est ce qui nous amène à étudier la fonction des microARNs dans les cellules germinales. L’objectif de mon doctorat a été de comparer les mécanismes moléculaires utilisés par les microARN dans les cellules germinales et somatiques afin de mieux comprendre comment ils régulent l’expression des gènes. Grâce à une combinaison d’analyses protéomiques et de rapporteurs de l’activité des microARN in vivo, nous avons pu disséquer la composition et la fonction des miRISC germinaux et somatiques. Nous avons découvert un mécanisme de répression indépendant de GW182 qui réprime et stabilise les cibles de microARN. De façon intéressante, nous démontrons que des sites de liaison des miARN sont suffisants pour localiser un transcrit germinal à la région périnucléaire. Finalement, nous identifions GLH-1, une composante des granules P germinaux, comme étant un cofacteur de la fonction des microARN germinaux. Collectivement, mes travaux de doctorat nous ont permis de contribuer à améliorer les connaissances des mécanismes de la régulation des gènes par les microARN.
Abstract
Post-transcriptional regulation plays a key role in controlling animal development and diseases. Among all post-transcriptional regulatory mechanisms identified to date, microRNAs play a central role. They constitute a large class of short non-coding RNAs that were discovered in C. elegans and have been reported in all metazoans. MicroRNAs are transcribed by RNA polymerase II as a long primary transcript that will undergo two successive cleavage steps to form the mature microRNA. The mature microRNA is the loaded onto an Argonaute protein to form an effector complex, the miRISC. MicroRNAs associate with the 3' untranslated region of their target mRNAs to repress their translation and/or initiate their degradation. The dynamic orchestration of these processes is not well understood, but at the moment is thought to be largely dependent on a key Argonaute partner, GW182. In developmental contexts such as oogenesis and early embryogenesis, the expression of maternally supplied mRNAs is tightly controlled. In oocytes, translational repression rather than irreversible mRNA decay, is preferred to accumulate and maintain a pool of maternal mRNAs whose timely expression is critical later in development. MicroRNAs are abundant in the germ line and therefore could potentially participate in this regulation. This leads us to study the function of microRNAs in germ cells. The aim of my PhD was to compare the molecular mechanisms used by microRNAs in germline and somatic cells to better understand how they regulate gene expression. Using a combination of proteomic analyzes and in vivo microRNA activity reporters, we were able to dissect the composition and function of germline and somatic miRISCs. We uncover a GW182-independent silencing mechanism used by germline miRNAs to both repress and unexpectedly stabilize their target mRNA. Interestingly, miRNA binding sites are sufficient to localize a germline reporter transcript to the perinuclear region. Finally, we identify GLH-1, a germline P granule component, as an miRISC cofactor involved in the repression of germline microRNA targets. Collectively, my doctoral work helps us gain new insights about the mechanisms used by microRNAs to regulate gene expression.
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des tableaux ... ix
Liste des abréviations ... x
Remerciements ... xiii
Avant-propos ... xv
Chapitre 1. Introduction ... 1
1.1 La régulation post-transcriptionnelle de l’expression des gènes ... 1
1.1.1 La maturation de l’ARNm ... 1
1.1.2 L’export de l’ARNm au cytoplasme ... 4
1.1.3 Le contrôle de la stabilité et de la traduction par le 5’UTR ... 7
1.1.4 Contrôle de la stabilité et de la traduction par les régions codantes ... 11
1.1.5 Le contrôle de la stabilité et de la traduction par le 3’UTR ... 12
1.1.6 Interactions 5’UTR-3’UTR ... 13
1.1.7 La localisation subcellulaire des ARNm et des protéines ... 15
1.2 Les microARN ... 17
1.2.1 La biogénèse des microARN ... 17
1.2.2 Les mécanismes de régulation des ARNm par les microARN ... 19
1.2.3 Les modes de régulation spécifiques au contexte ... 22
1.3 Régulation post-transcriptionnelle dans la lignée germinale ... 24
1.3.1 Introduction à la lignée germinale de Caenorhabditis elegans ... 24
1.3.2 Mécanismes de régulation par des protéines ciblant des séquences spécifiques . 26 1.3.3 Mécanismes de régulation par des protéines liant l’ARN de façon non-spécifique ... 28
1.3.4 Mécanismes de régulation par des courts ARN non-codants germinaux ... 32
1.4 La fonction des microARN dans la lignée germinale des animaux ... 32
1.4.1 Les courts ARN germinaux ... 32
1.4.2 Les microARN germinaux ... 32
1.4.3 Phénotypes germinaux causés par la perte de fonction de gènes de la voie des microARN ... 34
1.4.4 Les mécanismes utilisés par les miARN dans la lignée germinale ... 42
1.4.5 La transition maternelle-zygotique ... 44
1.5 Problématique et Objectifs ... 47
Chapitre 2. Somatic and germline microRNAs form distinct silencing complexes to regulate their target mRNAs differently ... 49
2.3 Introduction ... 52
2.4 Results ... 54
2.4.1 Germline miRNAs use a specialized silencing mechanism ... 54
2.4.2 Different miRISC composition in germline and somatic tissues ... 58
2.4.3 Tissue specific miRISC co-factors contribute to miRNA function ... 61
2.4.4 Germline miRNA targets are preferentially localized to the nuclear periphery .. 70
2.4.5 The loss of GLH-1 lead to an increase in cytoplasmic germline miRNA targets 74 2.5 Discussion ... 79
2.5.1 GW-dependency and microRNA mechanisms ... 79
2.5.2 GLH-1 is involved in the regulation of miRNA targets ... 80
2.5.3 What confers specificity for the protection of maternal mRNA? ... 81
2.5.4 miRNA function: from the germline to the embryo ... 82
2.6 Materials and methods ... 85
2.7 Resource Tables ... 90
Chapitre 3. Discussion et Perspectives ... 95
3.1 Le miRISC et son contexte post-transcriptionnel ... 96
3.2 Fonctions potentielles des autres interacteurs du miRISC ... 105
3.3 Conclusions ... 112
Bibliographie ... 113
Liste des figures
Figure 1.1 - Les étapes de maturation de l’ARNm et son export au cytoplasme. ... 5
Figure 1.2 - Facteurs affectant la stabilité et/ou la traduction des ARNm chez les eucaryotes. ... 8
Figure 1.3 - Modèle illustrant le couplage des régions 5’ et 3’ lors de la régulation de la traduction. ... 15
Figure 1.4 - Voie de biogénèse des miARN. ... 18
Figure 1.5 : Mécanismes de régulation par les miARN. ... 20
Figure 1.6 - La lignée germinale de C. elegans. ... 25
Figure 1.7 : Rôles connus de composantes des granules P dans la formation de granules. . 29
Figure 1.8 - La perte des gènes nécessaires à la voie microARN entraîne des défauts de la lignée germinale femelle chez l'animal. ... 35
Figure 2.1 - Tissue-specific activity of miR-228. ... 56
Figure 2.2 - Germline activity of miR-35. ... 57
Figure 2.3 - Comparative proteomic analysis of germline and somatic miRISCs. ... 60
Figure 2.4 - Efficiency of miR-35 and miR-241 pull-downs and co-immunoprecipitation of GLH-1::HA with FLAG::ALG-2. ... 61
Figure 2.5 - RNAi of factors required for germline miRNA function. ... 63
Figure 2.6 - RNAi knock-down efficiency in the germline miRNA reporter. ... 64
Figure 2.7 - Biochemical interaction between GLH-1 and ALG-2 ... 65
Figure 2.8 - Effect of glh-1 RNAi on miRNA and Argonaute levels. ... 66
Figure 2.9 - RNAi of factors required for somatic miRNA function. ... 69
Figure 2.10 - RNAi knock-down efficiency in somatic mutated and sensor reporters. ... 69
Figure 2.11 - Whole worm single molecule mRNA FISH analysis of gfp mRNA. ... 71
Figure 2.12 - GLH-1 and miRNAs cooperate in the localization of miRNA targets. ... 73
Figure 2.13 - Analysis of PGL-1::RFP and gfp mRNA localization upon knock-down of P granule factors. ... 75
Figure 2.14 - Effect of P granule factors knock-down on gfp mRNA localization and expression. ... 77
Figure 2.15 - Model for the retention of miRISC-targeted transcripts to the nuclear periphery by the P granule component GLH-1. ... 84
Figure 3.1 - Détection d’ALG-1, ALG-2, NTL-1 et CCF-1 à miR-35 et miR-241 purifiés par 2’O-methyl. ... 98
Figure 3.2 - Modèle intégrant le rôle du complexe GLH-1-miRISC dans la régulation post-transcriptionnelle et la répression de l’expression de transcrits somatiques dans les cellules germinales. ... 104 Figure 3.3 - ZBP-1 détecté par un immunobuvardage de type western. ... 106 Figure 3.4 - Interaction biochimique entre ALG-2 et ZBP-1. ... 107
Liste des tableaux
Tableau 1.1 – Voies de dégradation des ARNm. ... 10 Tableau 1.2 : Protéines sans spécificité de séquence impliquées dans la régulation de la traduction et la stabilité des ARNm germinaux. ... 30 Table 2.1: Key Resource Table ... 92 Table 2.2: Oligonucleotides used in this study ... 94 Tableau 3.1 – Protéines détectées par analyses de spectrométrie de masse des complexes associés à miR-35, miR-241 et miR-228, leurs orthologues et liens avec la voie de
Liste des abréviations
2’-O-me: 2’-O-methyl3’UTR : 3’ UnTranslated Region 4EBP: 4E-Binding Protein 5’UTR: 5’ UnTranslated Region
ADAR: Adenosine Deaminase Acting on RNA ADN: Acide DésoxyriboNucléique
AGO: ArGoNaute
ARN : Acide RiboNucléique ARNm : ARN Messager ARNnc : ARN Non-Codant CAF1: CCR4 Associated Factor 1 CBC: Cap Binding Complex
CCR4: Carbon Catabolite Repression 4 CDS: CoDing Sequence
CPEB: Cytoplasmic Polyadenylation Element Binding CPSF: Cleavage and Polyadenylation Specificity Factor CSTF: Cleavage Stimulatory Factor
DCP: DeCapping Protein DCPS: DeCaPping Scavenger DTC: Distal Tip Cell
endo-siARN: siARN endogène GFP: Green Fluorescent Protein HSP: Heat Shock Protein IP: ImmunoPrecipitation
IRES: Internal Ribosome Entry Site KH: K-Homology
METTL: METhylTransferase-Like miARN : microARN
miRISC: miRNA-Induced Silencing Complex NOT: Negative On Tata less
PABPC: Poly(A) Binding Protein Cytoplasmic PABPN: Poly(A) Binding Protein Nuclear PAN: Poly(A) Nuclease
PARN: Poly(A)-specific RiboNuclease PAP: poly(A) Polymerase
piARN: Piwi Interacting ARN
PIWI: P element-Induced Wimpy testes PUF: PUmilio-FBF
Pré-ARNm : ARN pré-messager pri-miARN: miARN primaire m7G : N-7-MéthylGuanosine m6A : N6-MethylAdenosine
RISC: RNA-Induced Silencing Complex RNAi: RNA interference
RNP: RiboNucleoProtein siARN: small interfering RNA SL1: Selective Factor 1
TOE: Transition Oocyte-Embryon TMZ: Transition Maternelle-Zygotique WT: Wild Type
Curiosity did not kill the cat. Boredom killed the cat.
Remerciements
Merci à mes parents pour être qui ils sont, et pour m’avoir laissée être moi-même. Ça prend beaucoup d’amour et de confiance pour faire ça. Merci à ma mère, Nicole Dubé, pour la leçon suivante : si quelque chose n’existe pas, invente-le et demande à ton père de le faire. Merci à mon père, Martin Dallaire, de m’avoir appris à faire tout ce qu’il sait faire. Y compris démonter un alternateur à -30℃.
Merci Olivier Pouliot d’être qui tu es. Tu es la personne que je connais qui a le plus d’ambition, tout en sachant toujours ce qui ne vaut pas la peine d’être fait. Ces deux qualités te mèneront loin, et la deuxième me permettrait de sauver beaucoup d’énergie si je suivais toujours tes conseils.
Merci Phil St-Cyr pour tout ton amour et pour avoir partagé ma vie pendant les trois dernières années. Tu es la personne la plus humaine et empathique que je connaisse. Merci de m’avoir appris à penser comme toi.
Merci à mes ami(e)s Catherine Hamel, Alexandra Rodrigue, Émilie Savard, Catherine Gilbert, Julien Charest, Hm et Marie-Andrée pour avoir partagé avec moi les choses douces et pas douces de la vie. Avec vous je me sens à ma place, et ça c’est quelque chose qui n’a pas de prix.
Merci aux membres du laboratoire passés et présents. Merci pour toutes les discussions, les répétitions de présentations, les relectures, les critiques. Merci d’avoir partagé vos souches quand je perdais les miennes, et d’avoir écouté la musique que je mettais. Merci aux gens du centre de recherche, étudiants et chercheurs, pour m’avoir fait réfléchir.
Merci à Martin de m’avoir guidée à peu près de la même façon que mes parents. Ça n’a certainement pas été facile, mais les choses les plus faciles à faire ne valent souvent pas la peine d’être faites. Je suis très fière d’être une F1 de ta lignée.
Finalement, je tiens à remercier les membres du jury, Dr Jacques Côté, Dr Thomas Duchaîne et Dr Rachid Mazroui, pour avoir accepté d’évaluer ma thèse de doctorat.
Avant-propos
Le premier chapitre de cette thèse est une revue de la littérature permettant d’introduire mon sujet de doctorat. Dans ce chapitre se trouve une partie consacrée aux fonctions des miARN dans la lignée germinale des animaux. Ce sujet a fait l’objet d’une publication dans la revue Developmental Biology en 2016 (Annexe 4). Le texte et les figures pertinentes à cette thèse ont été traduites en français et mises à jour pour former la section 1.4 de cette thèse.
Le corps du chapitre 2 présente les résultats des expériences qui ont été menées dans le cadre de mon projet de recherche et porte sur l’article accepté dans le journal Developmental Cell en 2018 intitulé : Somatic and germline microRNAs form distinct silencing complexes to regulate their target mRNAs differently. Cette publication
représente l’aboutissement des travaux que j’ai réalisés durant ma maîtrise et mon doctorat au laboratoire du Dr Martin Simard. Le chapitre 2 est structuré sous forme d’article. L’ordre et la numérotation des figures et des tableaux des articles a été modifiée pour une intégration adéquate dans le manuscrit. Sur cet article, mon statut est celui du premier auteur. La version publiée de cet article est présentée à l’Annexe 5.
En 2012, juste avant le début de ma maîtrise, Irfan Bukhari publiait dans le journal Cell Research l’article synthétisant ses travaux de doctorat intitulé : The microRNA pathway controls germ cell proliferation and differentiation in C. elegans. Les résultats présentés dans cet article indiquent que les gènes des Argonautes associées à la voie des miARN, alg-1 et alg-2, sont impliqués dans la régulation de la biogénèse des cellules germinales chez le nématode. Ces travaux sont à la base de mon projet de maîtrise, qui visait à améliorer la compréhension moléculaire du fonctionnement des miARN germinaux. Mes deux années de maîtrise ont été consacrées à la création d’outils permettant d’évaluer directement in vivo la contribution de miARNs (famille miR-35-42) à la régulation d’un ARNm-cible. Ceci nous a permis de confirmer que les miARN sont actifs dans la lignée germinale. J’ai aussi utilisé une technique de purification de courts ARN, développée au laboratoire par le Dr Guillaume Jannot. Cette technique nous a permis de comparer les complexes protéiques
associés à des miARN germinaux et somatiques, ce qui a mené à l’identification de facteurs potentiellement impliqués spécifiquement dans la fonction des miARN germinaux.
Durant mes travaux de doctorat, j’ai donc utilisé ces outils pour déterminer lesquels des facteurs identifiés participent réellement à la fonction des miARN. J’ai aussi développé de nouveaux rapporteurs in vivo permettant de comparer l’effet d’un seul miARN (miR-228) sur l’expression d’une cible somatique ou germinale. Ceci a été essentiel pour confirmer l’implication différentielle des co-facteurs somatiques et germinaux du miRISC présentés dans cet article. J’ai eu la chance de travailler avec Pierre-Marc Frédérick, un stagiaire d’été devenu étudiant à la maîtrise, qui m’a aidé à produire certains résultats, dont ceux présentés dans ce chapitre à la Figure 2.4B. Toutes les autres expériences réalisées dans le cadre de ce manuscrit ont été conçues par moi, avec la collaboration essentielle du Dr Martin Simard. J’ai écrit le manuscrit et préparé toutes les figures, sous la supervision active du Dr Martin Simard.
Finalement les annexes 1, 2 et 3 présentent les articles auxquels j’ai participé durant ma maîtrise et le début de mon doctorat, intitulés Down regulation of miR-124 in both Werner syndrome DNA helicase mutant mice and mutant Caenorhabditis elegans wrn-1 reveals the importance of this microRNA in accelerated aging (Annexe 1), A New Role for the GARP Complex in MicroRNA-Mediated Gene Regulation (Annexe 2), et Expression profile of Caenorhabditis elegans mutant for the Werner syndrome gene ortholog reveals the impact of vitamin C on development to increase life span (Annexe 3).
Chapitre 1. Introduction
1.1 La régulation post-transcriptionnelle de l’expression des gènes
L’un des phénomènes les plus fascinants de la biologie est la capacité de générer un organisme complexe à partir d’une seule cellule, l’oocyte fertilisé. Durant le développement, chaque cellule contient normalement exactement la même information génétique et pourtant, différents types cellulaires sont générés. L’ADN, qui est répliqué à chaque division cellulaire, est transcrit en ARN. L’ARN est quant à lui traduit en protéines possédant des activités enzymatiques et biochimiques spécifiques. Les caractéristiques physiologiques et structurales propres à chaque type cellulaire sont donc conférées par l’assortiment des gènes qui y sont exprimés. Cependant, il ne suffit pas de transcrire des gènes spécifiques à un type cellulaire donné pour assurer l’adoption d’un destin cellulaire approprié. La maturation des transcrits produits, leur stabilité, leur taux de traduction et de dégradation, ainsi que leur localisation, doivent être finement régulés afin de coordonner les évènements développementaux et cellulaires de façon spatio-temporelle. Nous savons maintenant qu’une portion des ARN produits par une cellule ne sont pas traduits en protéines. Ces ARN sont appelés ARN non-codants (ARNnc). Depuis les années 1980, la découverte d’une incroyable variété de types d’ARNnc et des fonctions qui leurs sont associées a révolutionné le dogme central de la biologie moléculaire défini par Francis Crick (Crick 1958). L’ARN, qui était autrefois considéré comme un simple support temporaire de l’information génétique (d’où le terme ARN messager ; ARNm), représente maintenant une partie intégrante et essentielle à la régulation du fonctionnement biologique des cellules (Cech and Steitz 2014). Par ailleurs, on constate que les ARNnc sont impliqués dans la plupart des maladies humaines, incluant le cancer (Esteller 2011). La régulation post-transcriptionnelle est donc un processus fondamental qui ajoute une complexité essentielle aux fonctions biochimiques encodées par le génome.
1.1.1 La maturation de l’ARNm
Les ARNm sont transcrits au noyau par l’ARN polymérase II. L’ARN pré-messager (pré-ARNm) ainsi formé est immature et doit être extensivement modifié avant de pouvoir être exporté au cytoplasme pour être traduit. Les modifications apportées incluent le coiffage,
l’épissage et la polyadénylation, qui visent notamment à préciser la fonction finale de l’ARNm mature, permettre sa traduction, et lui conférer la stabilité nécessaire pour résister au métabolisme cytoplasmique. Tous ces processus sont couplés à la dynamique transcriptionnelle et à ses différentes étapes (Figure 1.1) (Hocine et al. 2010). La première étape de maturation du pré-ARNm naissant est le coiffage, qui est effectué très tôt après le début de la transcription, soit après l’addition des premiers 25-30 nucléotides (Moteki and Price 2002). La coiffe, composée d’une guanosine méthylée (m7G), protège l’extrémité 5’ de l’ARN de la dégradation par les exonucléases 5’->3’ (Ramanathan et al. 2016). Elle est reconnue par le complexe liant la coiffe (cap-binding complex, CBC), qui sert entre autres à assurer l’export de l’ARN mature par le port nucléaire (Ramanathan et al. 2016). Une fois au cytoplasme, cette structure permettra l’interaction avec le complexe d’initiation de la traduction. Dans la majorité des cas, la présence d’une coiffe en 5’ est nécessaire à la traduction des ARNm, mais il existe aussi un mécanisme alternatif d’initiation de la traduction (Komar and Hatzoglou 2011).
Les pré-ARNm des eucaryotes sont discontinus : ils contiennent des segments codants, les exons, et des segments non-codants, qui incluent la région 5’ non-traduite (5’UTR), la région 3’ non-traduite (3’UTR), et les introns. L’épissage permet d’éliminer de façon précise les introns afin de connecter ensemble les exons qui dirigeront la synthèse protéique. Souvent, une forme alternative d’épissage peut survenir, menant à une diversité de combinaisons d’exons et parfois l’inclusion d’introns, ce qui altère la forme mature de l’ARNm produit (Lee and Rio 2015). Par conséquent, la séquence traduite ne sera pas la même et la fonction de la protéine produite peut être altérée.
L’épissage alternatif représente une façon remarquable d’augmenter la diversité du protéome fonctionnel des organismes possédant un nombre limité de gènes. Il est donc étonnant de constater qu’une grande proportion des évènements d’épissage alternatif affectent les régions 5’ et 3’ non-codantes des gènes (Mockenhaupt and Makeyev 2015). Ces régions contiennent des séquences régulatrices et des éléments structuraux importants pour l’efficacité de la traduction, la stabilité et la localisation des ARNm. Ces éléments régulateurs peuvent, non seulement par leur présence, mais aussi par leur distance par
rapport aux codons d’initiation et de terminaison, altérer drastiquement l’expression du gène concerné (Mockenhaupt and Makeyev 2015). Un autre mécanisme de maturation des ARN existe : l’édition d’adénosine en inosine (Nishikura 2016). Sur les pré-ARNm, ces modifications sont retrouvées particulièrement dans des régions qui forment des structures secondaires double-brin, et des études suggèrent qu’elles seraient ajoutées de façon co-transcriptionnelle et en coordination avec l’épissage alternatif (Laurencikiene et al. 2006, Rodriguez et al. 2012). Si cette modification se trouve dans un segment codant d’ARN, elle sera interprétée par la machinerie de traduction comme une guanosine, ce qui peut avoir pour effet de modifier la séquence des acides aminés correspondants. L’ARN peut aussi être méthylé en position N6 des adénosines (m6A). Cette modification réfère à une méthylation de la base adénosine en position azote 6, qui est déposée co-transcriptionnellement (au noyau) par des méthyl-transférases d’adénosine (METTL) (Liu et al. 2014, Ke et al. 2017, Slobodin et al. 2017). Contrairement à l’édition d’adénosine en inosine, la m6A n’affecte pas la séquence d’acide aminés produits. Cependant, cette modification peut affecter l’épissage de certains pré-ARNm (au noyau) (Xiao et al. 2016), et, si elle est présente sur l’ARNm mature, peut affecter sa stabilité (Wang et al. 2014, Du et al. 2016) et/ou sa traduction (Meyer et al. 2015).
La dernière étape de la maturation du pré-ARNm concerne son extrémité 3’. À cette étape, plusieurs processus sont inter-reliés : la terminaison de la transcription, la présence du complexe d’épissage au dernier intron du gène, et la présence du complexe de clivage/polyadénylation à l’exon terminal du gène. Le couplage entre l’épissage et le clivage/polyadénylation qui définit l'exon terminal entraîne également la stimulation mutuelle de ces deux réactions (Niwa et al. 1990, Niwa and Berget 1991, Antoniou et al. 1998, Li et al. 2001), et la reconnaissance du site d'épissage 3’ est également couplée à la transcription (Dye and Proudfoot 1999). Une fois que l’exon terminal est défini, un clivage se produit près du site de polyadénylation, (habituellement l’hexamère AAUAAA), et la séquence de l’extrémité 3’ de l’ARNm est déterminée de façon irréversible. Le complexe CPSF (cleavage and polyadenylation specific factor) coopère avec le complexe de stimulation au clivage (CSTF), la poly(A) polymérase (PAP) et l’extrémité carboxyterminale de l’ARN polymérase II pour former le multi-complexe actif qui clive le
pré-ARNm et stimule sa polyadénylation (Colgan and Manley 1997). Ainsi, la fin d’un transcrit mature est normalement caractérisée par une suite d’adénosines d’une longueur variable, et qui est un déterminant majeur de la stabilité et la traductibilité du transcrit (Jalkanen et al. 2014). À la suite de l’élongation de la queue poly(A), une protéine de liaison à la queue poly(A) de nucléaire (PABPN) lie la queue poly(A) sous forme de multimère. Les fonctions des PABP de type nucléaire sont moins bien connues que celles des PABP de type cytoplasmique (PABPC), mais tout comme ces dernières, elles jouent un rôle dans la détermination de la longueur de la queue poly(A) (Mangus et al. 2003).
Chez les animaux, une grande proportion de gènes possèdent plus d’un site de polyadénylation : environ 75% chez les mammifères (Derti et al. 2012) et environ 35% chez le nématode (Mangone et al. 2010, Jan et al. 2011, Haenni et al. 2012). Les sites de polydénylation alternatifs peuvent se situer soit en amont du site de terminaison (dans un intron ou un exon), ou en aval, dans la région 3’ non-codante (Tian and Manley 2013). L’utilisation d’un site de polyadénylation alternatif peut donc avoir un effet sur la protéine produite, ou altérer la séquence de la région 3’ non-codante, ce qui peut affecter la régulation dépendante des éléments qui s’y trouvent.
1.1.2 L’export de l’ARNm au cytoplasme
L’ARNm coiffé, épissé, polyadénylé et parfois édité, est maintenant mature et prêt à être exporté au cytoplasme. Le recrutement de facteurs d’export se fait co-transcriptionnellement, au niveau du pré-ARNm naissant, et durant sa maturation (Delaleau and Borden 2015). L’ARNm mature acquiert ainsi un complexe d’export qui est principalement associé à l’extrémité 5’ et à la coiffe de l’ARNm. Ensemble, ils forment une particule ribonucléoprotéique (RNP) apte à se localiser à la périphérie du pore nucléaire. L’interaction avec des protéines présentes sur le côté cytoplasmique du pore nucléaire initie la translocation du RNP au cytoplasme. Certaines protéines nucléaires demeurent confinées au noyau (Nakielny and Dreyfuss 1996), tandis que d’autre accompagnent l’ARNm au cytoplasme avant d’être remplacées des protéines cytoplasmiques (Pinol-Roma and
suivent l’ARNm mature et engagent le premier cycle de traduction, dit pionnier (Maquat et al. 2010). Ensuite, le complexe liant la coiffe est remplacé par le facteur d’initiation de la traduction E (eIF4E) et la machinerie de traduction générale. De façon similaire, les PABPN sont remplacées par les PABP de type cytoplasmique (PABPC), bien connues pour favoriser la stabilité des transcrits au cytoplasme (Baer and Kornberg 1980, Deo et al. 1999).
Figure 1.1 - Les étapes de maturation de l’ARNm et son export au cytoplasme.
(A) Une coiffe est ajoutée au pré-ARNm naissant transcrit par l’ARN polymérase II.
La coiffe est liée par le complexe de liaison de la coiffe (CBC, vert foncé). (B) Le pré-ARNm est épissé. (C) Certaines adénosines (ici représentée par un ‘A’), généralement présentes dans des structures double-brin, peuvent être transformées en inosines par les protéines ADAR. (D) Certaines adénosines peuvent être méthylées en position 6 par les protéines METTL. (E) L’ARNm est clivé en 3’ et polyadénylé. Le CBC s’associe au pore nucléaire pour initier l’export de l’ARNm. (F) L’ARNm mature est exporté au cytoplasme. (G) Lors du premier cycle de traduction, les protéines nucléaires CBC et PABPN sont remplacées par leurs contreparties eiF4E et PABPC. La formation du complexe d’initiation de la traduction requiert eiF4G, eiF4A, eiF3 et la sous-unité ribosomale 40S.
L’une des caractéristiques importantes de l’initiation de la traduction est l’interaction entre les extrémités 5’ et 3’ des ARNm. Ceci est dépendant du facteur d’initiation de la traduction G (eIF4G), qui lie la coiffe et la queue poly(A) indirectement, via son interaction avec eIF4E et PABPC respectivement (Sachs and Varani 2000), ce qui mène à la circularisation de l’ARNm (Wells et al. 1998, Sachs and Varani 2000). Étant donné que cette circularisation est importante pour l’efficacité de la traduction (Gallie 1991), ce sont à la fois les particularités du 5’UTR, du 3’UTR, et l’interaction entre ces derniers, qui déterminent la stabilité et la traductibilité d’un transcrit donné.
La dégradation d’un ARNm peut être initiée par trois évènements distincts : un clivage endonucléolytique, un décoiffage, ou une déadénylation menant à la perte de PABPC (Roy and Jacobson 2013). Tous ces évènements mènent à une perte de la forme circulaire de l’ARNm, ce qui convertit l’ARNm d’une forme ‘traductible’ à une forme ‘non- ou peu traductible’. Inversement, une réduction du taux d’initiation de la traduction promeut également la dégradation des ARNm (Chan et al. 2018). Ainsi, il est difficile de déterminer si un facteur donné affecte la traduction ou déstabilise les ARNm puisque les deux processus sont souvent interdépendants. Pour certains facteurs, on englobe donc les deux mécanismes jusqu’à ce qu’une preuve directe de causalité soit faite. Dans les sections 1.1.3, 1.1.4 et 1.1.5, les éléments régulateurs présents dans les régions 5’, les régions codantes
(CDS) et les régions 3’ de l’ARNm seront décrits individuellement. Ensuite, les interactions régulatrices possibles entre les éléments en 5’ et en 3’ de l’ARNm seront résumées dans la section 1.1.6. La Figure 1.2 illustre les éléments régulateurs présentés aux sections 1.1.3, 1.1.4, 1.1.5 et 1.1.6.
1.1.3 Le contrôle de la stabilité et de la traduction par le 5’UTR
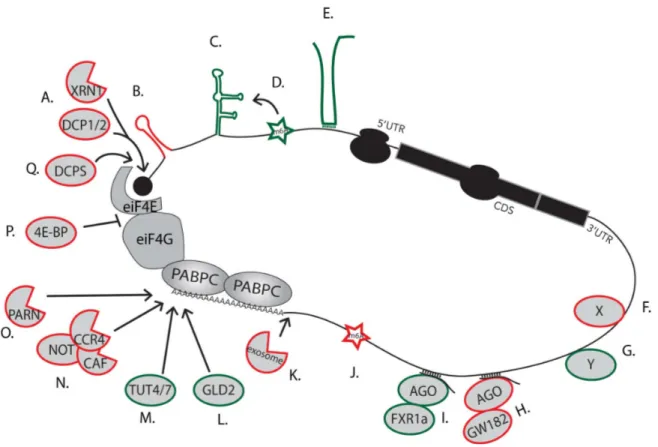
Les mécanismes de régulation des ARNm les plus répandus sont associés au statut traductionnel et aux caractéristiques intrinsèques en principe communes à tous les ARNm. La reconnaissance initiale de la coiffe par eIF4E, l’assemblage et la liaison du complexe d’initiation 43S, l’étape de balayage et de reconnaissance du codon d’initiation sont des étapes limitantes qui peuvent être régulées à plusieurs niveaux (Kozak 1991, Sonenberg and Dever 2003, Gebauer and Hentze 2004). Par exemple, la famille des protéines liant eIF4E, les ‘’4E-binding proteins’’ ou 4EBP, lorsque phosphorylé, lie eIF4E, ce qui déplace eIF4G. L’interaction entre eIF4E et eIF4G est essentielle, donc la présence de 4EBP inhibe l’association du 43S avec l’ARNm, et donc l’initiation de la traduction (Figure 1.2).
Figure 1.2 - Facteurs affectant la stabilité et/ou la traduction des ARNm chez les eucaryotes.
Les facteurs ayant un effet positif sur la stabilité et/ou la traduction de l’ARNm sont en vert et ceux ayant un effet négatif sont en rouge. Certains facteurs peuvent avoir un effet positif ou négatif, tout dépendant de leur localisation sur l’ARNm. Les flèches indiquent une contribution catalytique et les flèches à bout fermé indiquent un antagonisme. (A) DCP1/2 hydrolyse la coiffe des ARNm déadénylés et XRN1 dégrade l’ARNm de façon exoribonucléolytique (5’ à 3’). (B) Une structure secondaire peut masquer ou empêcher le balayage nécessaire à la reconnaissance du codon d’initiation, ce qui empêche l’initiation de la traduction. (C) IRES. Un site interne d’entrée du ribosome promouvoit la traduction de façon indépendante de la coiffe. (D) Un site de méthylation m6A dans la région 5’ non-traduite promeut l’initiation la traduction independante de la coiffe, via un IRES. (E) Un long ARN non-codant peut promouvoir la traduction. (F) Une protéine liant l’ARN peut bloquer la traduction ou stimuler la dégradation de l’ARNm. (G) Une protéine liant l’ARN
peut stimuler la traduction ou bloquer la dégradation de l’ARNm. (H) Le miRISC bloque la traduction et initiation la dégradation de l’ARNm. (I) Le miRISC alternatif incluant FXR1a mais pas GW182 stimule la traduction. (J) Un site de méthyltion m6A dans la région 3’ non-traduite accélère la déadénylation et la dégradation de l’ARNm. (K) Le complexe exosome cytoplasmique cible les ARNm déadénylés et les dégrade (3’ à 5’). (L) La poly(A) polymérase GLD2 allonge la queue poly(A), rendant l’ARNm plus résistant à la dégradation. (M) TUT4/7 urydilent les ARNm, ce qui les destine à la dégradation. (N) Le complexe CCR4/NOT/CAF est un complexe de déadénylation majeur responsable de l’initiation de la dégradation d’ARNm. (O) PARN est une autre déadénylase qui cible les ARNm. (P) Les protéines de la famille 4E-BP lient eIF4E et l’empêchent de s’associer à eIF4G, ce qui bloque l’initiation de la traduction (Q) Le ‘decapping scavenger’ DCPS dégrade la coiffe résiduelle après la dégradation d’un ARNm par la voie 3’à5’.
Les ARNm des eukaryotes portent en général une coiffe m7G. L’hydrolyse de la coiffe est faite par le complexe de décoiffage DCP1/DCP2, ce qui inhibe l’initiation de la traduction et engage rapidement l’ARNm dans la voie de dégradation 5’à3’, médiée par les exoribonucléases XRN. La famille des XRN inclue XRN1 et XRN2, des enzymes cytoplasmiques et nucléaires, respectivement (Nagarajan et al. 2013). Tout ARNm dont minimalement l’une des extrémités 5’ et 3’ est décoiffée ou déadénylée est susceptible d’être un substrat pour une exoribonucléase XRN ou l’exosome. Lorsqu’un ARN est dégradé par la voie 3’à5’, l’hydrolyse de la coiffe résiduelle est effectuée par DCPS (Wang and Kiledjian 2001). De façon similaire, un clivage par une endonucléase crée des extrémités 5’ et 3’ non protégées qui peuvent être ciblées par les mêmes mécanismes de dégradation. Ainsi, une diminution de la stabilité de l’ARNm est souvent associée à une perte de la coiffe. Ceci peut être appliqué soit en masse, soit à des ARNm spécifiques. Le décoiffage est souvent associé à la dégradation d’ARNm ciblés spécifiquement via des motifs présents dans la séquence de l’ARN (p. ex. les miARN, Staufen). Certains activateurs du décoiffage tels que Dhh1/DDX6 sont d’ailleurs recrutés par des facteurs spécifiques en plus d’être impliqués dans les voies générales de dégradation des ARNm (Section 1.1.6, Figure 1.3) (Coller and Parker 2005, Nissan et al. 2010).
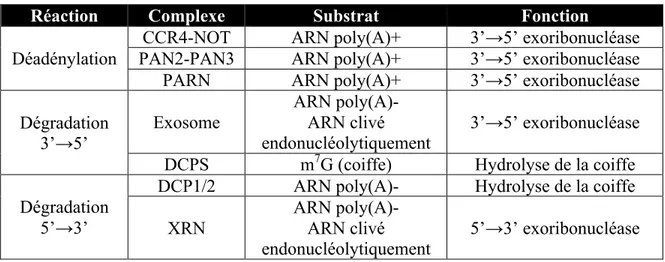
Tableau 1.1 – Voies de dégradation des ARNm.
Les complexes ou protéines responsables de la déadénylation et des deux voies de dégradation sont présentées avec leurs substrats et les fonctions qui leur sont associées.
Ainsi, tous les ARNm peuvent en principe être régulés par les mécanismes décrits ci-haut. Certains ARNm peuvent aussi être contrôlés de façon spécifique, soit par des caractéristiques propres à leurs séquences (p. ex. des structures secondaires ou des modifications), soit par des sites de liaison pour des protéines ou des ARN régulateurs (Leppek et al. 2018).
Par exemple, l’initiation de la traduction peut se faire indépendamment de la coiffe. Dans ce cas, une structure secondaire complexe de l’ARNm, nommée IRES, est nécessaire pour diriger le recrutement des complexes ribosomaux dans la région 5’ non-codante, souvent très près du codon d’initiation (Thompson 2012). Les éléments IRES promeuvent la traduction, mais dans d’autres cas, des structures stables présentes dans la région 5’ non-traduite ou près du codon d’initiation répriment la traduction chez les bactéries et les eucaryotes (Kozak 1989, de Smit and van Duin 1990, Gray and Hentze 1994, Bugaut and Balasubramanian 2012). Des structures secondaires ou tertiaires telles que des tige-boucles et des pseudonoeuds peuvent se former, majoritairement dans la région 5’ non-codante,
Réaction Complexe Substrat Fonction
Déadénylation PAN2-PAN3 CCR4-NOT ARN poly(A)+ ARN poly(A)+ 3’→5’ exoribonucléase 3’→5’ exoribonucléase PARN ARN poly(A)+ 3’→5’ exoribonucléase Dégradation
3’→5’ Exosome
ARN poly(A)- ARN clivé
endonucléolytiquement 3’→5’ exoribonucléase DCPS m7G (coiffe) Hydrolyse de la coiffe Dégradation
5’→3’
DCP1/2 ARN poly(A)- Hydrolyse de la coiffe XRN ARN poly(A)- ARN clivé
peuvent ralentir ou bloquer le balayage par les ribosomes, et donc diminuer l’efficacité de traduction (Kozak 1989, Qu et al. 2011).
Au contraire, certains motifs de liaison spécifiques ou des modifications de l’ARN peuvent favoriser la traduction de certains ARNm ciblés. En condition de stress, par exemple, des sites de méthylation m6A peuvent recruter directement le complexe d’initiation, ce qui permet d’initier la traduction de façon indépendante de la coiffe (Meyer et al. 2015). De façon similaire, certains motifs de liaison permettent de recruter de longs ARN non-codants capables de promouvoir la traduction de leurs cibles (Carrieri et al. 2012, Schein et al. 2016).
Des membres de la famille des courts ARN non-codants, les microARN (miARN), ciblent majoritairement le 3’UTR des ARNm, mais certains exemples démontrent qu’ils peuvent aussi affecter l’expression des gènes en ciblant la région codante ou le 5’UTR (Lytle et al. 2007, Duursma et al. 2008). Dans la majorité des cas observés jusqu’à maintenant, les miARN répriment la traduction et/ou déstabilisent l’ARNm ciblé, mais certaines fonctions non-conventionnelles commencent à émerger (Dragomir et al. 2018). La voie des miARN sera présentée plus en détail à la section 1.2.
1.1.4 Contrôle de la stabilité et de la traduction par les régions codantes
Certaines caractéristiques des régions codantes affectent la traduction (et par conséquent la stabilité) des ARNm, possiblement au niveau de l’élongation. Par exemple, malgré l’activité hélicase du ribosome, des structures secondaires stables présentes dans la région codante des ARNm diminuent l’efficacité de traduction in vitro (Wen et al. 2008, Qu et al. 2011) et in vivo (Burkhardt et al. 2017).
Un aspect important de la région codante a récemment intéressé la communauté scientifique : l’utilisation de codons optimaux. Certains codons sont plus efficacement traduits que les autres, et sont associés à des espèces d’ARN de transfert plus abondants (Presnyak et al. 2015, Bazzini et al. 2016). Les ARNm ayant une séquence enrichie en
codons optimaux sont significativement plus stables et ont un taux de traduction plus élevé. Les ARNm ayant plutôt des codons non-optimaux sont plus facilement dégradés, et ce d’une façon dépendante du complexe CCR4-NOT. Cette idée avait initialement été proposée en 2008 par Drummond et Wilke, dont l’hypothèse était que les codons optimaux seraient sélectionnés évolutivement afin de ralentir la traduction des régions protéiques qui sont les plus sensibles à un mauvais repliement (au niveau de la structure) (Drummond and Wilke 2008, Zhou et al. 2009). Cette idée intéressante n’a toujours pas été testée directement. Bizarrement, l’utilisation des codons non-optimaux est associée à une terminaison précoce de la transcription, et à une répression transcriptionnelle via la triméthylation en lysine 9 de l’histone H3 (Zhou et al. 2016, Zhou et al. 2018). Ainsi, l’identité des codons est une caractéristique qui peut affecter l’expression des gènes au niveau transcriptionnel, via un mécanisme encore inconnu, et au niveau post-transcriptionnel.
1.1.5 Le contrôle de la stabilité et de la traduction par le 3’UTR
Plusieurs facteurs du 3’UTR contribuent à sa stabilité et à l’efficacité de sa traduction. En général, la longueur du 3’UTR d’un ARNm (Tanguay and Gallie 1996, Mishima and Tomari 2016) et de sa queue poly(A) (Eckmann et al. 2011) corrèlent avec sa stabilité et son efficacité de traduction.
Paradoxalement, plus un 3’UTR est long, plus il a de chances de contenir des éléments régulateurs. Cependant, sa longueur est régulée par la présence de sites de clivages et de polyadénylation alternatifs qui entraînent un raccourcissement du 3’UTR, sans affecter la fonction de la protéine produite (Mayr and Bartel 2009, Yeh and Yong 2016). Puisque le 3’UTR contient souvent des régions régulatrices importantes pour la stabilité, la traduction et la localisation subcellulaire de l’ARNm, l’utilisation de sites de polyadénylation
alternatifs représente un niveau de contrôle post-transcriptionnel dont l’effet biologique peut être drastique (Berkovits and Mayr 2015, Mitra et al. 2015, Blazie et al. 2017, Flamand et al. 2017). La longueur de la queue poly(A) est quant à elle régulée négativement par les complexes de déadénylation cytoplasmiques CCR4-NOT, CAF1, PARN, ou au noyau par l’exosome. Elle peut aussi être régulée négativement par des urydilases (TUT4/7) (Lim et al. 2014, Morgan et al. 2017), ou positivement par des poly(A) polymérases comme GLD2 (Nousch et al. 2014).
Des protéines et des courts ARN non-codants peuvent détourner les facteurs mentionnés ci-hauts et ainsi cibler leur effet sur des ARNm spécifiques. Par exemple, les miARN recrutent le complexe CCR4-NOT à leurs cibles et peuvent aussi induire leur urydilation, ce qui les engage dans une voie de dégradation (Lim et al. 2014, Xu et al. 2016). De façon similaire, les protéines SMAUG recrutent le complexe CCR4-NOT à leurs cibles (Loh et al. 2013). Les protéines Argonautes ne sont pas les seules à utiliser un ARN comme intermédiaire pour interagir avec leur cible. STAUFEN, une protéine dirigeant la dégradation des ARNm, peut utiliser un long ARN non-codant pour cibler des ARNm spécifiques (Gong and Maquat 2011).
Finalement, il existe des sites de méthylation m6A dans le 3’UTR des gènes et près du codon d’arrêt, et leur présence corrèle avec le choix de sites de polyadénylation alternatifs (Meyer et al. 2012, Ke et al. 2015, Yue et al. 2018). D’ailleurs, la méthylation m6A peut être reconnue par YTHDF2, qui recrute directement le complexe CCR4-NOT et affecte la stabilité des transcrits (Du et al. 2016). Ainsi, comme illustré à la Figure 1.2, l’effet de cette modification sur la stabilité et la traduction d’un ARNm dépend de l’endroit où elle est déposée et de la présence d’autre éléments régulateurs comme un IRES.
1.1.6 Interactions 5’UTR-3’UTR
Lors de l’initiation de la traduction, l’interaction entre eIF4G et PABPC permet de rejoindre les extrémités 5’ et 3’ de l’ARNm, ce qui le pseudo-circularise. Ainsi, les
régulateurs de la traduction ciblant l’une des extrémités de l’ARNm peuvent affecter directement ou indirectement l’autre extrémité. De multiples interactions protéiques existent entre les facteurs liant le 5’ et le 3’, indiquant en général un couplage entre leurs activités (Figure 1.3).
Chez la levure, la drosophile et les mammifères, le complexe CCR4-NOT est associé à des activateurs du décoiffage tel que Dhh1 ou ses orthologues Me31B/DDX6/RCK1/p54 (Haas et al. 2010, Ozgur et al. 2010, Chen et al. 2014, Mathys et al. 2014), qui sont impliqués dans la répression de la traduction et la fonction des miARN (Minshall et al. 2001, Nakamura et al. 2001, Minshall et al. 2009, Scheckel et al. 2012, Rouya et al. 2014). DDX6 interagit à la fois avec le complexe CCR4-NOT (Rouya et al. 2014) et avec le complexe de décoiffage (Fenger-Gron et al. 2005, Tritschler et al. 2009, Mugler et al. 2016). Cependant, plusieurs études démontrent que DDX6 peut bloquer la traduction indépendamment de l’activation du décoiffage (Carroll et al. 2011, Sweet et al. 2012). Son implication dans la régulation de la traduction et la stabilité des ARNm suggèrent que DDX6 pourrait coupler ces deux mécanismes, mais son rôle exact n’est pas très bien compris (Coller and Parker 2005, Presnyak et al. 2015).
Un autre niveau de couplage entre la régulation en 5’ et en 3’ est l’interaction entre la déadénylation, le décoiffage et la dégradation exonucléolytique 5’à3’. Les ARNm déadénylés sont un substrat pour l’enzyme de décoiffage DCP2 et son co-activateur DCP1, qui interagit directement avec l’enzyme XRN1 (Braun et al. 2012). Ceci indique que la déadénylation par le complexe CCR4-NOT est coordonnée avec la répression de la traduction et la stabilité des ARNm.
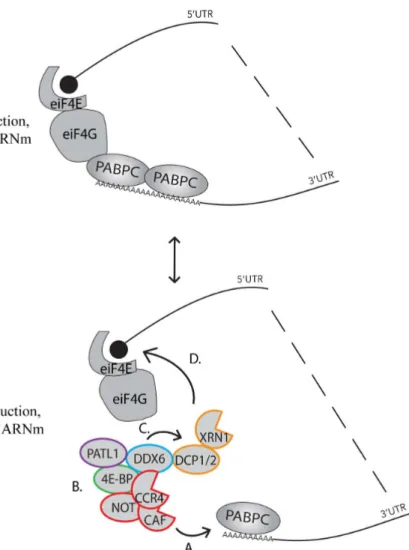
Figure 1.3 - Modèle illustrant le couplage des régions 5’ et 3’ lors de la régulation de la traduction.
Haut : Circularisation de l’ARN Durant l’initation de la traduction. eIF4G interagit
avec eIF4E (en 5’) et PABPC (en 3’). Bas : (A) CCR4-NOT déadényle l’ARNm, ce qui déplace PABPC. (B) 4E-BP favorise la perte d’interaction entre eIF4G et PABPC. C) DDX6 active DCP1/2. (D) L’ARNm sera décoiffé par DCP2 et son co-activateur DCP1, et l’interaction directe entre ce dernier et XRN1 stimule la dégradation exonucléolytique 5’à3’.
1.1.7 La localisation subcellulaire des ARNm et des protéines
De nombreuses protéines impliquées dans la dégradation et la répression de la traduction des ARNm telles que CCR4-NOT, DCP1, DCP2, PATL1 et DDX6, sont enrichies dans des foyers cytoplasmiques. Il existe différents types de foyers cytoplasmiques : les corps P
(processing bodies, ou P bodies), les granules de stress (Brengues et al. 2005, Parker and Sheth 2007, Kedersha and Anderson 2009) et les granules germinaux, qui seront abordés à la section 1.4. Les protéines GW182 et Argonautes liées à la voie des miARN forment aussi des granules cytoplasmiques (Eystathioy et al. 2003, Parker and Sheth 2007). Ainsi, les processus de régulation traductionnelle, qu’ils fassent partie d’une voie générale ou qu’ils soient ciblés à certains transcrits, sont concentrés dans des domaines cytoplasmiques. Parallèlement, les ARNm transitent dans ces domaines suivant leur état traductionnel.
Les granules de stress et les corps P sont des structures dynamiques (Andrei et al. 2005, Kedersha et al. 2005). Il est important de noter que la présence d’un ARNm dans ce type de granule ne le destine pas nécessairement à être indéfiniment réprimé et/ou dégradé. En effet, il a été démontré que des ARNm peuvent sortir des corps P ou des granules de stress sans être décoiffés, pour être ensuite recrutés à la machinerie de traduction et ré-exprimés (Brengues et al. 2005, Bhattacharyya et al. 2006, Takahara and Maeda 2012). Ainsi, malgré la proximité de tous les facteurs permettant de coupler la répression de la traduction et la dégradation des ARNm, il semble que les deux voies puissent aussi être régulées indépendamment, du moins pour certains transcrits.
1.2 Les microARN
1.2.1 La biogénèse des microARN
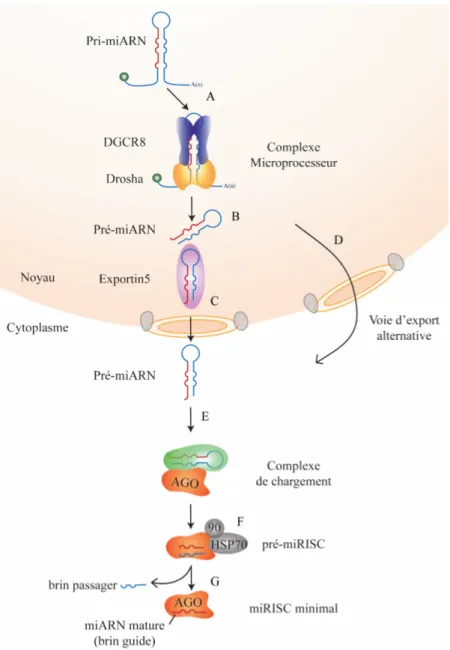
La majorité des gènes de miARN sont transcrits par l’ARN polymérase II, produisant des long miARN primaires (pri-miARN) (Daugaard and Hansen 2017). Les pri-miARN sont coiffés, polyadénylés et ont une structure en forme de tige-boucle, avec des extrémités 5’ et 3’ simple-brin. Certains pri-miARN sont polycistroniques; ils contiennent plusieurs miARN différents. Le pri-miARN est soumis à une première étape de clivage endonucléolytique effectuée au noyau par le complexe Microprocesseur, constitué de la ribonucléase de type III Drosha et de son co-facteur DGCR8. Ce clivage élimine les longues extrémités simple-brin du pri-miARN, formant un produit appelé précurseur de miARN (pré-miARN) qui conserve la structure caractéristique en forme de tige-boucle. Le pré-miARN (environ 70 nucléotides) est exporté au cytoplasme par l’Exportin-5 (Okada et al. 2009) ou une autre voie d’export (Kim et al. 2016), et est ensuite clivé en fragments d’environ 21 nucléotides par Dicer, une autre RNAse de type III (Zhang et al. 2004, Lau et al. 2012). À cette étape, le duplexe est transféré de Dicer à l’Argonaute (AGO), et ce duo forme le complexe de chargement. Les protéines HSP70/90 aident la prise en charge du duplexe de miARN par une protéine AGO. AGO clive le brin passager du duplex (Bouasker & Simard, 2012; Diederichs & Haber, 2007; Matranga, Tomari, Shin, Bartel, & Zamore, 2005; Rand, Petersen, Du, & Wang, 2005) et incorpore seulement le brin possédant l’appariement de base le moins stable en 5’ (le brin guide) et élimine l’autre brin, appelé brin passager, qui sera dégradé (Nakanishi 2016). Le complexe pre-miRISC correspond à cette étape de séparation du duplexe. La prise en charge d’un miARN mature par une Argonaute forme un complexe appelé miRISC minimal (minimal microRNA-Induced Silencing Complex). Ce complexe peut reconnaître des ARNm-cibles grâce à une complémentarité de bases imparfaite entre le miARN et une séquence habituellement présente dans la région 3’ non-traduite de sa cible. Le miRISC minimal représente une plateforme qui recrute des complexes protéiques effecteurs impliqués dans la régulation de la traduction et de la stabilité des ARNm, formant ainsi un miRISC actif.
Figure 1.4 - Voie de biogénèse des miARN.
(A) Le miARN primaire est clivé par le complexe Microprocessur pour former le (B)
précurseur de miARN. (C) Le pré-miARN est exporté au cytoplasme par l’Exportin5 ou (D) par une voie alternative. (E) Le pré-miARN s’associe à Dicer former le complexe de chargement. Dicer clive le pré-miARN, ce qui forme un duplexe d’environ 21 nucléotides. (F) HSP70/90 aide au chargement du duplexe dans l’AGO, formant le pré-miRISC. À cette étape, AGO clive le brin passager, qui sera détruit, et conserve le brin guide du miARN. (G) Le miRISC minimal, composé d’un miARN mature et d’une Argonaute est ainsi formé et prêt à cibler des ARNm.
1.2.2 Les mécanismes de régulation des ARNm par les microARN
Les protéines Argonautes sont des plateformes de recrutement des facteurs qui effectuent les mécanismes répresseurs dirigés par les miARN (Figure 1.5). Les principaux acteurs de ces mécanismes sont les protéines GW182, qui peuvent interagir avec plusieurs Argonautes via leur motifs GW (Elkayam et al. 2017). GW182 recrute CCR4/NOT et peut lier PABP directement, ce qui facilite l’interaction entre le miRISC et l’ARNm-cible. Il est communément accepté que c’est grâce à l’interaction entre GW182 et CCR4-NOT que le miRISC peut réprimer ses cibles (Chekulaeva et al. 2009, Eulalio et al. 2009, Lazzaretti et al. 2009, Huntzinger et al. 2010). Cependant, il existe d’autres mécanismes pouvant affecter négativement l’expression des cibles des miARN, et il n’existe toujours pas de consensus quant à leur nature, leur orchestration temporelle et leurs prévalences respectives.
L’observation d’un maintien de l’association des cibles de miARN aux polysomes lors de leur répression suggère que les miARN bloquent la traduction après l’initiation, possiblement au niveau de l’élongation (Olsen and Ambros 1999, Seggerson et al. 2002, Maroney et al. 2006, Petersen et al. 2006). D’ailleurs, les miARN sont capables de réprimer des rapporteurs dont la traduction est initiée par un IRES, indiquant que leur activité ne cible pas nécessairement les facteurs canoniques d’initiation de la traduction (Petersen et al. 2006). Curieusement, des groupes utilisant les mêmes types de rapporteurs (avec un IRES) ont observé que ces derniers échappaient à la régulation par les miARN (Humphreys et al. 2005, Pillai et al. 2005). De plus, il a été observé que la répression par les miARN peut être renversée in vitro en ajoutant un excès d’eIF4F, ce qui suggère aussi un rôle au niveau de l’initiation de la traduction (Mathonnet et al. 2007). À cause de ces contradictions, la nature de la répression traductionnelle exercée par les miARN n’a pas encore été résolue, et il demeure possible qu’elle puisse être affectée à plusieurs niveaux.
Figure 1.5 : Mécanismes de régulation par les miARN.
(A) Répression de la traduction au niveau de l’initiation et/ou de l’élongation. (B)
Déadénylation par le complexe CCR4-NOT, recruté par le miRISC via GW182. (C) Décoiffage par DCP1/2 et dégradation 5’à3’ par XRN1.
Il a été démontré que les miARN initient la déadénylation de leurs cibles (Wu et al. 2006, Wakiyama et al. 2007, Eulalio et al. 2009), ce qui requiert aussi GW182 et le complexe CCR4-NOT. La déadénylation des cibles de miARN les rend susceptibles au décoiffage et à la dégradation exonucléolytique. DDX6, qui interagit avec CCR4-NOT, pourrait être un médiateur important dans l’activation du décoiffage par DCP1/2 et la dégradation subséquente par XRN1 (Rouya et al. 2014). Ainsi, l’effet déstabilisateur des miARN sur leurs cibles, qui se fait via la déadénylation, est bien établi.
Cependant, une question demeure : la dégradation des cibles est-elle un mécanisme indépendant de la répression, ou est-elle une conséquence de la répression traductionnelle ? La connexion intime entre les deux mécanismes complexifie l’analyse des résultats. Certains auteurs prétendent que la dégradation à elle seule explique la diminution de la traduction causée par les miARN (Guo et al. 2010, Eichhorn et al. 2014). Cependant, deux
études de la cinétique de l’expression des cibles de miARN suggèrent fortement que la répression de la traduction précède la dégradation, du moins en cellules S2 et chez les embryons de poisson-zèbre (Bazzini et al. 2012, Djuranovic et al. 2012, Subtelny et al. 2014). De plus, il a été démontré que la dégradation initiée par les miARN se fait en majeure partie durant la traduction et en association avec les polysomes, ce qui suggère aussi que l’inhibition de la traduction précède la dégradation (Tat et al. 2016). Ainsi, la cinétique développementale représente un atout majeur pour évaluer les subtilités des mécanismes mis en œuvre par les miARN, et potentiellement par d’autres régulateurs. Pour le moment, les conclusions tirées ici doivent être considérées spécifiquement à l’intérieur du contexte de l’embryogénèse, mais malgré tout, ces études pionnières sont un exemple à suivre dans le futur.
Comme mentionné à la section 1.2.7, plusieurs protéines impliquées dans la fonction des miARN forment des foci cytoplasmiques et colocalisent avec des marqueurs des corps P comme CCR4-NOT, DCP1, DCP2, et XRN1. À cause de cette colocalisation, les corps P ont été proposé pour être des sites de répression et de dégradation des ARNm (Parker and Sheth 2007). Ceci suggère que la séquestration des cibles de miARN à l’intérieur de ces foci serait un mécanisme pour assurer leur répression. Cependant, il a été démontré que la dissociation de ces granules n’affecte pas la répression par les miARN (Eulalio et al. 2007). Au contraire, l’intégrité des corps P dépend de la fonctionnalité de la voie des miARN. Donc, il semble que la formation de foci soit une conséquence de la répression des cibles, et non pas un mécanisme de répression.
Finalement, les miARN semblent utiliser plusieurs mécanismes pour bloquer la traduction et déstabiliser leurs cibles, mais leurs contributions respectives sont toujours disputées. Il est important de noter que plusieurs mécanismes peuvent potentiellement fonctionner simultanément, plus particulièrement in vivo, où la régulation de l’expression des gènes est dynamique et complexe.
1.2.3 Les modes de régulation spécifiques au contexte
L’étude des fonctions des miARN dans des contextes cellulaires et développementaux spécifiques améliore notre compréhension de leurs modes de régulation et surtout, fournit des exemples intéressants où certains miRISCs utilisent des mécanismes différents.
Par exemple, dans les cellules quiescentes et les oocytes de xénope, les miARN peuvent stimuler la traduction (Vasudevan et al. 2007, Vasudevan et al. 2007, Vasudevan et al. 2008, Mortensen et al. 2011, Truesdell et al. 2012, Bukhari et al. 2016). Durant la phase G0 du cycle cellulaire, l’expression de TNRC6 (GW182) est réduite et les ARNm ont une très courte queue poly(A). La même chose est observée dans les oocytes, où les ARNm maternels ont aussi une courte queue poly(A) qui empêche temporairement leur traduction sans qu’ils soient dégradés, jusqu’à ce qu’une poly(A) polymérase permette l’activation de leur traduction (Radford et al. 2008, Reyes and Ross 2016). Les résultats du groupe de Vasudevan suggèrent qu’un mécanisme d’activation de la traduction miARN-dépendant existe, et que ce dernier serait dépendant d’un miRISC alternatif incluant FXR1 mais indépendant de GW182, de la queue poly(A) et de PABPC.
Durant l’embryogénèse précoce, les miARN semblent fonctionner différemment. Chez le poisson-zèbre, la traduction des cibles de miARN est réprimée, et précède leur dégradation (Bazzini et al. 2012, Subtelny et al. 2014), ce qui suggère que les deux mécanismes sont découplés pendant un certain temps. Il est intéressant de constater que la fonction des miARN durant l’embryogénèse de C. elegans est indépendante des protéines GW182 (Jannot et al. 2016), ce qui fournit un autre exemple supportant l’existence de miRISC alternatif. De plus, chez les embryons de nématodes, la répression de la traduction des cibles de miARN est dépendante des PABPC, mais indépendante de la queue poly(A) (Flamand et al. 2016). Ensemble, ces études indiquent que la fonction des miARN peut être modulée au cours du développement, et est probablement dépendante de la composition du miRISC et de son environnement post-transcriptionnel.
Les granules ou foci cytoplasmiques sont une caractéristique importante de l’environnement post-transcriptionnel cellulaire. On peut, en examinant la colocalisation des composantes des miRISC et leurs cibles, des complexes de décoiffage, de déadénylation et de dégradation, raisonnablement supposer l’existence d’interactions biochimiques fonctionnelles entre eux. Des études de colocalisation in vivo démontrent l’existence de différents types de granules dans les embryons précoces de la drosophile : 1) Des corps P, où des facteurs de déadénylation, de décoiffage et les Argonautes (AGO1, AGO2) colocalisent, et 2) des corps GW (GW182-bodies, GW-bodies), qui ne colocalisent significativement avec aucune des composantes des corps P (Patel et al. 2016). Ceci suggère que les corps GW, contrairement aux corps P, ne sont pas des sites de dégradation des ARNm. Cette étude contredit ce qui a été observé dans des cellules en culture (Eystathioy et al. 2013, Rewhinkel et al. 2005, Behm-Ansmant et al. 2006, Schneider et al. 2006), souligne la diversité de la composition des foci cytoplasmiques in vivo, et suggère que le contexte développemental module la régulation des ARNm de façon spatiale.
1.3 Régulation post-transcriptionnelle dans la lignée germinale
Pour beaucoup d’espèces, les premières étapes du développement embryonnaire se font en l’absence de transcription et dépendent du contrôle spatio-temporel des ARNm maternels. La lignée germinale des animaux produit, en plus des ARNm nécessaires au cycle cellulaire et l’oogénèse, des ARNm maternels qui sont transmis à l’embryon. Dans la lignée germinale, et plus particulièrement dans celle de C. elegans (Merritt et al. 2008), la régulation post-transcriptionnelle est un mécanisme prévalent. Dans cette section, plusieurs mécanismes de régulation des ARNm germinaux décrits chez C. elegans seront présentés afin d’illustrer les principes généraux conservés chez les autres organismes modèles.
1.3.1 Introduction à la lignée germinale de Caenorhabditis elegans
Avant même d’être choisis comme modèle d’étude par Sydney Brenner au milieu des années 1960, le nématode Caenorhabditis elegans était étudié par les zoologistes pour répondre à des questions fondamentales. Durant la période pré-Brenner, on étudiait exclusivement les modes de reproduction, la méiose, la fertilisation, et l’hérédité du nématode. Déjà, sa transparence était mise à profit. Les premières publications scientifiques évoquant C. elegans sont le fruit du travail d’Ellsworth Dougherty et Victor Nigon, qui ont comparé sa fertilité à celle de C. briggsae, celle du premier étant deux fois supérieure à celle du deuxième (254 descendants par animal pour C. elegans ; 134 pour C. briggsae, à 16°C) (Nigon and Dougherty 1949). Par la suite, Nigon choisira d’utiliser C. elegans pour décrire les diverses structures nucléaires observables durant l’ovogénèse (Nigon and Brun 1955). Durant les années soixante, une époque où les femmes avaient un accès plus que limité à l’éducation universitaire, le premier article publié par la revue Science portant sur C. elegans aura comme première auteure une femme, Dr. Hélène V. Fatt (Fatt and Dougherty 1963). Notez qu’à cette époque, le concept de facteur d’impact en était à ses tout débuts, et la revue Science était beaucoup moins glamour qu’aujourd’hui. Malgré tout, les travaux d’Hélène V. Fatt, réalisés durant sa maîtrise et son doctorat, nous auront aidés à comprendre les premières variations génétiques associées à la fertilité de C. elegans en réponse à la température.
Durant les années 70, les gonades de C. elegans sont à la mode, et les premières descriptions détaillées du développement de son système reproducteur voient le jour (Figure 1.6) (Hirsh et al. 1976, Ward and Carrel 1979). Durant la même période, la cartographie du lignage cellulaire du nématode, faite en observant au microscope toutes les divisions, migrations et apoptoses cellulaires, de l’embryogénèse à l’adulte, a eu un impact profond sur la science (Sulston and Horvitz 1977, Kimble and Hirsh 1979, Sulston et al. 1983). La définition du lignage des cellules neuronales et la découverte de la mort cellulaire programmée ont pavé la voie à plusieurs avenues de recherche qui sont toujours poursuivies aujourd’hui par la communauté scientifique.
Figure 1.6 - La lignée germinale de C. elegans.
Représentation schématique de l’un des deux bras identiques des gonades de C. elegans. La région la plus distale de la gonade contient des cellules souches germinales qui prolifèrent continuellement par mitose (zone colorée en rose). Ces cellules sont entourées d’une cellule distale somatique (distal tip cell ; DTC) qui sert de plateforme signalétique. Les cellules mitotiques s’éloignent du DTC et passent par une zone transition (colorée en violet) vers la méiose (colorée en bleu). Jusqu’à la fin
de la zone pachytène, la gonade est un syncytium : les noyaux partagent un cytoplasme unique. Dans la zone diplotène, une membrane cytoplasmique se forme autour des noyaux, ce qui forme des oocytes dont les volumes nucléaire et cytoplasmique augmentent rapidement. La maturation des oocytes a lieu dans la zone proximale. Les oocytes matures traversent la spermatèque un par un pour être fertilisés.
Aux sections 1.1 et 1.2, nous avons vu qu’il existe un aspect global à la régulation de la traduction et de la stabilité des transcrits, mais que ces processus généraux peuvent aussi être ciblés à des transcrits spécifiques (en étant recrutés par des protéines ou des ARN). Ce principe s’applique aussi à la lignée germinale, où une dimension spatio-temporelle s’ajoute à la régulation d’ARNm spécifiques. Dans les cellules germinales, un niveau important de régulation qui s’apparente à la localisation subcellulaire (introduit à la section 1.1.7) existe, et sera présenté à la section 1.3.3. La majorité des mécanismes décrits sont essentiels pour la prolifération et/ou la différenciation des cellules germinales.
1.3.2 Mécanismes de régulation par des protéines ciblant des séquences spécifiques
Certaines protéines lient et régulent les ARNm via des séquences spécifiques. C’est le cas des familles PUF (Pumilio et FBF). Les protéines PUF lient l’ARN via le motif UGUANAUA et UGURNNAUA (R=purine, N=n’importe quelle base) (White et al. 2001, Bernstein et al. 2005). Chez la drosophile et les cellules humaines, l’inhibition de la traduction et l’initiation de la dégradation des ARNm par les PUF se fait, du moins en partie, en recrutant le complexe CCR4-NOT (Van Etten et al. 2012, Bhandari et al. 2014). Dans les oocytes de xénope, PUM lie directement la coiffe des ARNm, ce qui empêche l’accès à eIF4E et bloque l’initiation de la traduction (Padmanabhan and Richter 2006, Cao et al. 2010).
Chez C. elegans et la drosophile, les protéines de la famille NHL NCL-1 et Brat utilisent le motif UUGUU pour bloquer la traduction de certains transcrits (Sonoda and Wharton 2001,
Laver et al. 2015, West et al. 2018). Le mécanisme utilisé par Brat est inconnu, mais pourrait dépendre de ses interacteurs, les protéines PUF et Nanos.
Les protéines à domaine KH, un domaine librement associé à la liaison d’ADN simple brin, d’ARN et/ou de protéines, participent à la régulation de l’ARN. Chez C. elegans, les protéines GLD-1, GLD-3 et MEX-3 font partie de cette famille. GLD-1 lie le 3’UTR ou le 5’UTR des ARN via le motif UACUUAC (Ryder et al. 2004, Wright et al. 2011), et bloque leur traduction tout en les stabilisant (Scheckel et al. 2012, West et al. 2018). La stabilisation des ARNm par GLD-1 les protège entre autre contre la voie de dégradation des ARNm non-sens, et permet leur accumulation dans les oocytes (Lee and Schedl 2004). Le mécanisme utilisé par GLD-1 est toujours inconnu, mais son interaction avec des composantes des granules P, des granules germinales s’apparentant aux corps P (Gallo et al. 2008, Updike and Strome 2010), suggère que sa fonction passe par la protection des ARNm dans des domaines cytoplasmiques inaccessibles aux machineries de traduction et de dégradation.
Le motif de polyadénylation AAUAAA et ses dérivés reconnus par CPSF peuvent être considérés comme une séquence affectant la traduction et la stabilité des transcrits parce qu’en plus de permettre la polyadénylation nucléaire et l’activation de la traduction, ils déterminent la longueur du 3’UTR, et donc l’identité des séquences régulatrices utilisées. Le motif de polyadénylation cytoplasmique UUUUUAUU (et dérivés) est bifonctionnel ; il maintient les ARNm maternels dormants dans les oocytes immatures, puis active leur traduction durant leur maturation. Ce motif est reconnu par les protéines CPEB, qui peuvent inhiber la traduction indirectement en recrutant un complexe protéique inhibiteur (Stebbins-Boaz et al. 1999), et recruter des poly(A) polymérases activatrices conservées comme GLD2. Dans la lignée germinale de la drosophile, du xénope et du nématode, les poly(A) polymérases, en conjonction avec CPEB, promeuvent la traduction des ARNm (Rouhana et al. 2005, Benoit et al. 2008, Radford et al. 2008, Kim et al. 2009, Nousch et al. 2017). Il est important de noter que les poly(A) polymérases adénylent aussi les miARN dans les oocytes, ce qui les déstabilise et peut contribuer indirectement à affecter la répression de leurs cibles (Lee et al. 2014).