T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Microbiologie Cellulaire
JURY
Dr Catherine JESSUS (Rapporteur) Pr Philippe SANSONETTI (Rapporteur) Pr Bernard DUCOMMUN (Examinateur)
Dr Olivier MARCHES (Examinateur) Pr Eric OSWALD (Directeur de thèse) Dr Frédéric TAIEB (Co-directeur de thèse)
Ecole doctorale : Biologie-Santé-Biotechnologies de Toulouse Unité de recherche : UMR1225 INRA-ENVT
Directeur(s) de Thèse : Eric OSWALD et Frédéric TAIEB Rapporteurs : Catherine JESSUS et Philippe SANSONETTI
Présentée et soutenue par SAMBA LOUAKA Ascel Regis Le 28 Octobre 2009
Titre : Détermination de la voie de signalisation cellulaire eucaryote détournée par la protéine bactérienne Cif
2
Remerciements
Mes remerciements vont à tous les membres du jury pour avoir accepté de juger mon travail de thèse. Docteur Catherine JESSUS et Professeur Philippe SANSONETTI, soyez remerciés pour votre investissement en tant que rapporteur. Je tiens à remercier le professeur Bernard DUCOMMUN pour sa disponibilité et son écoute. Merci également au Docteur Olivier MARCHES pour avoir accepté d’examiner mon travail.
Toute mon affection à l’équipe de Pathogénie Moléculaire et Cellulaire des Infections à
Escherichia coli. Je tiens à exprimer ma profonde reconnaissance à Eric OSWALD qui dirige
cette équipe et qui m’a accueilli dans son laboratoire. Merci pour l’attention portée à mon travail et pour le soutien tant sur le plan personnel que professionnel. J’exprime toute ma gratitude à Frédéric TAIEB, pour sa confiance, son aide et sa disponibilité qui m’ont permis d’arriver là où je suis. Je lui souhaite beaucoup de bonheur avec sa grande famille. Je n’oublierai jamais ce que m’ont apporté mes co-directeurs de thèse. Je leur souhaite beaucoup de réussite dans leurs entreprises.
A Michèle, ma seconde mère. Que puis-je t’offrir pour te remercier ? Ton nom aura toujours une place particulière dans mon cœur. Le bébé a grandi Claude. Merci d’avoir usé tes oreilles à écouter les plaintes du petit Régis. Sans oublier Marie, Marie-Paule, Daniel, Monique, Hubert et Claude. Merci pour l’accueil et la gentillesse. Ma profonde admiration et mon grand respect à Jean-Phi. Merci pour la science partagée, c’est un plaisir. Maiwenn, merci pour tout on n’oublie pas les stats.
A la salle des cadres jetables (stagiaires, thésards et post-doc). Pendant quatre ans, j’ai passé les deux tiers de ma vie dans cette salle. Aux formidables occupants, présents et partis, Greg, Gabriel, Delphine, Ingrid (alias Ingrit), Olivier, Rika, Meredith, Lucile, Olga, Ali, Estelle, Bastien et Mehdi. Contemplez le bonheur autour de vous, je vous souhaite de le vivre. Dans quelques années, j’espère vous voir au CONGO, mon très beau pays natal. Gab, Ingrit et Greg, je vous transmets mes sentiments les meilleurs pour vos petites familles. Je remercie les secrétaires (Laurence, Corinne, Michèle et mes chaleureuses embrassades à Marie). Je n’oublie pas Cécile et Christian. Merci pour votre gentillesse.
A ma chère Emilie, merci d’avoir été toujours là. A toute la famille BOLOKO qui m’a accueilli comme un enfant perdu. Mes pensées vont aussi aux SITA qui m’ont assisté et soutenu.
Je ne pourrai terminer sans penser à ma chère Laurence. Merci pour ton soutien, ta présence et ton amour. Je pense également à mon père, à Laurina, Marietta et Price. Leur absence sera comblée par mes sentiments pour eux.
Femme noire, femme africaine,
ô toi, ma mère, merci ; merci pour tout ce que tu fais pour moi, ton fils,
si loin, si près de toi !
(CAMARA LAYE, L’enfant noir)
4
Table des matières
Remerciements 2
Table des matières 4
Liste des abréviations 8
Liste des figures 11
Avant propos 12
Publications 13
1
èrepartie : Synthèse bibliographique
14
I - Introduction générale sur la bactérie Escherichia coli 15
II- Cif est un effecteur des Escherichia coli entéropathogènes (EPEC) 17
II.1 Les EPEC 17
II.2 Le système de sécrétion de type III et l’induction des lésions d’A/E 18 II.3 Cif est un effecteur bactérien, non codé par le LEE, qui modifie la
physiologie de la cellule hôte 20
II.3.1 Découverte de Cif 20
II.3.2 Cif est un effecteur du SST3 21
II.3.3 Les effets de Cif sur les cellules eucaryotes 22
III. Les protéines Cifs constituent une large famille d’effecteurs
de bactéries pathogènes et/ou symbiotiques 23 III.1 Les espèces bactériennes portant une protéine hétérologue de Cif 23
III.2 Les protéines hétérologues de Cif 24
III.3 Les protéines Cifs sont des membres divergents de la superfamille
des cystéines protéases et acétyl-transférases
26 III.4 Les protéines Cifs bloquent la transition G2/M indépendamment
des modifications du cytosquelette 28
III.4.1 Les protéines Cifs modulent le cycle cellulaire et le
III.4.2 L’arrêt du cycle cellulaire provoqué par les protéines
Cifs ne résulte pas des modifications du cytosquelette 28
IV- Cif est une Cyclomoduline 30
IV.1 Le cycle cellulaire eucaryote 30
IV.1.1 Introduction 30
IV.1.2 Les complexes CDK/Cyclines 30
IV.1.3 Régulation du cycle cellulaire par les complexes
CDK/Cyclines 31
IV.1.4 Le complexe CDK1/Cycline B1 ou l’ inducteur universel
de mitoses 32
IV.1.5 Les inhibiteurs des complexes CDK/Cyclines 32
IV.1.6 Les points de Contrôle 33
IV.1.7 La dérégulation des points de Contrôle 35
IV.2 La mort cellulaire 35
IV.2.1 Description de quelques mécanismes de mort cellulaire 36
IV.2.2 L’apoptose 37
IV.2.3 Liens entre les différents types de mort cellulaire 39 IV.3 Les bactéries sont des micro-organismes pirates 40
IV.3.1 Les Cyclomodulines 41
IV.3.2 Les cyclomodulines forment une famille qui continue de
s’agrandir 42
IV.3.3 Le rôle des cyclomodulines 42
V. Revue bibliographique 44
2
èmepartie : Résultats
56I – Objectifs de thèse 57
II- Article 1 : Escherichia coli cyclomodulin Cif induces G2 arrest of the host cell cycle without activation of the DNA-damage
6 III- Article 2: Bacterial cyclomodulin Cif blocks the host cell
cycle by stabilizing the cyclin-dependent kinase inhibitors p21waf1
and p27kip1 72
IV- Article 3: The EPEC effector Cif induces delayed apoptosis
in epithelial cells 87
3ème partie: Discussion et perspectives
110I- Bilan des résultats 111
II- Le mode d’action de Cif 111
II.1 Le processus d’ubiquitinylation et son détournement
Par les bactéries 111
II.2 La stabilisation des CKIs par Cif 112
II.2.1 Le Mécanisme 112
II.2.2 La cible de Cif 113
II.3 Les conséquences de l’inhibition de la dégradation des
protéines induite par Cif 114
II.3.1 La re-réplication de l’ADN 114
II.3.2 Cif et les autres effecteurs 115
II.3.3 Pistes restant à explorer 115
III- Le rôle de Cif 116
III.1 Les lésions d’A/E et la mort de l’hôte 116 III.2 La colonisation de l’épithélium intestinal 117
III.2.1 L’effet anti-prolifératif de Cif 117
III.2.2 Le rôle des CKIs 117
III.2.3 Le cytosquelette 118
III.2.4 Cif et la symbiose 119
IV- Conclusion 121
IV.1 Originalité du mode d’action de Cif 121
IV.2 Cif, un outil pour explorer le cycle cellulaire 121 IV.3 Une étude du rôle de Cif in vivo s’impose 122
4
èmepartie : Références bibliographiques
1235ème partie : Annexes
140Article : Cycle Inhibiting Factors (CIFs) are a growing family of functional cyclomodulins present in invertebrate and mammal
8
Liste des abréviations
Les termes en anglais sont entourés de guillemets
A/E : Attachement/effacement
ADNc : Acide Désoxyribonucléique complémentaire ADNr : Acide Désoxyribonucléique ribosomique APC : « Anaphase promoting complex »
Arp2/3 : « Actin-related protein 2/3 » ATM : « Ataxia telangiectasia mutated » ATP : Adénosine triphosphate
ATR : « ATM and Rad3 related »
AvrPphB : « Avirulence Pseudomonas syringae pv. phaseolicola B » BFP : « Bundle-forming pilus »
BRCA1 : « Breast cancer associated gene 1 » BrdU : Bromo-désoxy-Uridine
CAD : « Caspase-activated deoxyribonuclease » CagA : « Cytotoxin-associated antigen A » Cdc25 : « Cell division cycle 25»
CDK : « Cyclin-dependent Kinase » CDT : « Cytolethal distending toxin »
Cdt1 : « Chromatin licensing and DNA replication factor 1 » Chk : « Checkpoint kinase »
Cif : « Cycle inhibiting factor » Cip1 : « CDK-interacting protein-1 » CKI : Inhibiteurs de CDK
CNF : « Cytotoxic necrotizing factor » CRL : « Cullin-ring ubiquitin ligases » DAEC : E. coli à adhésion diffuse
DDB1 : « DNA damage-binding protein 1 » DNT : « Dermonecrotic toxin »
Eae : Intimine (EPEC attachement/effacement) EAF : Facteur d’adhésion des EPEC
EAggEC : E. coli entéroaggrégatives EHEC : E. coli entérohémorragiques EIEC : E. coli entéroinvasives EPEC : E. coli entéropathogéniques ERO : Espèces réactives de l’oxygène EspJ : EPEC secreted protein J
ETEC : E. coli entérotoxigéniques
ExPEC : E. coli pathogène extra-intestinale FADD : « Fas-associated death domain » FAS : « Fluorescence actin staining »
FIP : « Fusobacterial immunosuppressive protein » IAP : « Inhibitor of apoptosis protein
IEC-6 : « Intestinal epithelial cells-6 » INK4 : Inhibiteurs de CDK4
IpaB : « Invasion plasmid antigen B »
IVOC : « in vitro human intestinal organ culture » FRET : Fluorescence Resonance Energy Transfert Kip : « Kinase inhibitor protein »
LEE : Locus d’effacement des entérocytes Mad2: « Mitotic arrest deficient 2 »
Map : « mitochondrial associated protein »
MAPKK : « Mitogen-activated protein kinase kinase » Nck : « Non-catalytic region of tyrosine kinase »
N-WASP : « neural Wiskott-Aldrich-syndrome protein » PAI : « Pathogenicity island »
PARP : « Poly(ADP-ribose) polymerase » PCNA : « Proliferating cell nuclear antigen » PMT : « Pasteurella multocida toxin » Rb : Rétinoblastome
SCF : « Skp1/Cullin1/F-box » SiRNA : « Small interfering RNA »
SST : signal de sécrétion et de translocation SST3 : Système de sécrétion de type III Tir : récepteur transloqué de l’intimine
10 TNFR : Tumor necrosis factor receptor
TRADD : « TNFR-associated death domain » VacA : « Vacuolating cytotoxin »
Waf1 : « Wild-type p53 activated fragment-1 »
Liste des figures
Figure 1 : Potentiel génomique d’E. coli 15
Figure 2 : Contribution des éléments génétiques mobiles dans l’évolution
d’une E.coli commensale vers un caractère pathogène 16 Figure 3 : Les lésions d’attachement et d’effacement induites par les EPEC 17 Figure 4 : Représentation schématique de l’appareil de sécrétion de type III
des EPEC 18
Figure 5 : La protéine Cif modifie la physiologie des cellules eucaryotes 22 Figure 6 : Alignement et comparaison de séquences entre CifEc, CifYp, CifBp,
CifPl, CifPa et GOS_5485515 25
Figure 7 : Structures cristallisées des protéines Cifs provenant de E. coli
(forme tronquée), B. pseudomallei et P. luminescens 26 Figure 8A : Les principales phases du cycle cellulaire des eucaryotes supérieurs 30
Figure 8B : Régulation de la transition G1/S 33
Figure 9A : Vue générale et simplifiée des différentes issues possibles après
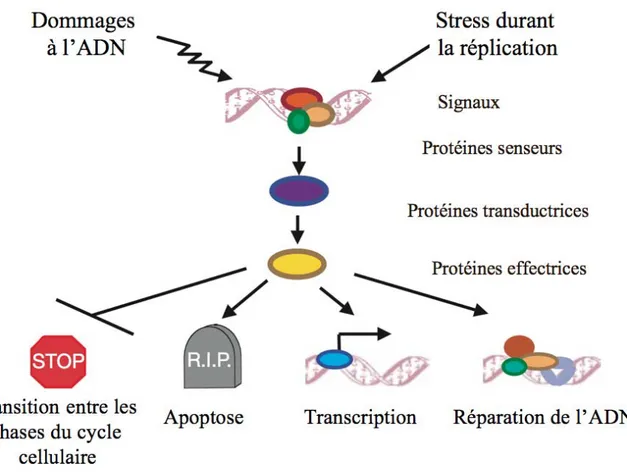
l’activation d’un point de contrôle en réponse aux dommages à l’ADN 34 Figure 9B : Le point de contrôle du cycle cellulaire à la transition G1/S 35
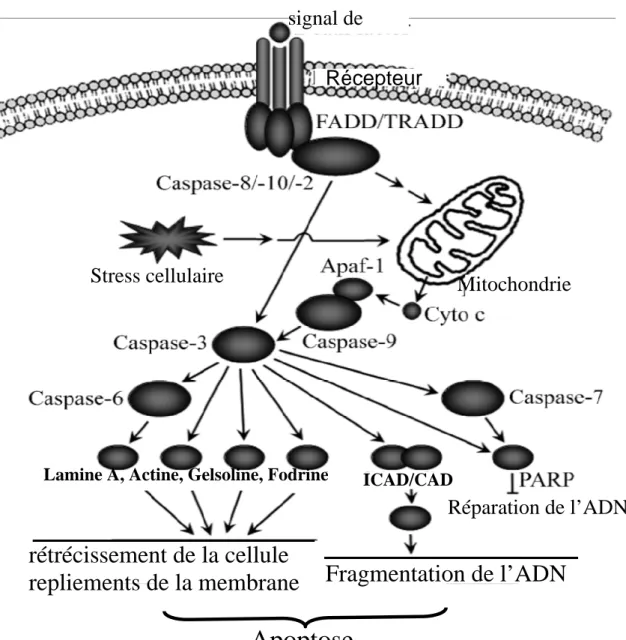
Figure 10 : Schéma du mécanisme d’apoptose avec quelques substrats
des caspases exécutrices 38
Figure 11 : Les cyclomodulines et le cycle cellulaire 41 Figure 12 : Les protéines bactériennes qui interfèrent avec le système
d’ubiquitinylation 112
Figure 13 : Mode d'action hypothétique de Cif sur les cellules HeLa 115
12
Avant propos
Cette thèse a été réalisée dans l’équipe de « Pathogénie Moléculaire et Cellulaire des infections à Escherichia coli », faisant partie de l’Unité Mixte de Recherche (UMR) 1225 INRA-ENVT. Notre laboratoire a développé une approche multidisciplinaire comprenant la microbiologie et la biologie cellulaire pour étudier les interactions entre les souches pathogènes d’E. coli et les cellules eucaryotes hôtes. Ainsi, suite aux nombreuses études portant sur les toxines bactériennes, l’équipe a défini le concept de « Cyclomoduline » qui est une famille de toxines et d’effecteurs bactériens affectant le cycle cellulaire eucaryote.
Les travaux présentés ici sont consacrés à l’étude d’une cyclomoduline appelée Cif pour Cycle inhibiting factor, capable de bloquer la prolifération des cellules infectées.
A l’inverse des cyclomodulines étudiées au laboratoire comme CDT ou la Colibactine, qui provoquent des cassures doubles brins de l’ADN, à mon arrivée au laboratoire, les mécanismes impliqués dans l’arrêt du cycle cellulaire associé à Cif n’étaient pas identifiés. Mon travail de thèse a donc consisté à mettre en lumière la signalisation cellulaire utilisée par la toxine Cif pour modifier la physiologie des cellules eucaryotes.
Nous avons choisi de présenter cette thèse sur articles. Nous avons fait précéder ces articles par une synthèse bibliographique. Celle-ci fait l’état des connaissances concernant la protéine Cif, les organismes portant le gène cif, ainsi qu’un rappel du cycle cellulaire eucaryote. La synthèse bibliographique se conclut par une revue sur la protéine Cif que nous venons de publier très récemment. Après la présentation des articles, je discuterai les différents résultats et exposerai différentes hypothèses quant au rôle de Cif dans la pathogénie des bactéries.
Publications
-
Taieb, F., Nougayrede, J.P., Watrin, C., Samba-Louaka, A. and Oswald, E. (2006)Escherichia coli cyclomodulin Cif induces G2 arrest of the host cell cycle without
activation of the DNA-damage checkpoint-signalling pathway. Cell Microbiol. 8: 1910-1921.
-
Samba-Louaka, A., Nougayrede, J.P., Watrin, C., Jubelin, G., Oswald, E. and Taieb, F. (2008) Bacterial cyclomodulin Cif blocks the host cell cycle by stabilizing the cyclin-dependent kinase inhibitors p21 and p27. Cell Microbiol. 10: 2496-2508.-
Jubelin, G., Chavez, C.V., Taieb, F., Banfield, M.J., Samba-Louaka, A., Nobe, R., etal (2009) Cycle inhibiting factors (CIFs) are a growing family of functional
cyclomodulins present in invertebrate and mammal bacterial pathogens. PLoS ONE. 4: e4855.
-
Samba-Louaka, A., Taieb, F., Nougayrède, J.-P. and Oswald, E. (2009) Cif type III effector protein: a smart hijacker of the host cell cycle. future microbiology. Vol 4 (7), 867-877.-
Samba-Louaka, A., Nougayrède, J-P., Watrin, C., Oswald, E. and Taieb, F. (2009) The EPEC effector Cif induces delayed apoptosis in epithelial cells. Infection and14
I-
Introduction générale sur la bactérie Escherichia coli
C’est en 1885 que le pédiatre allemand Theodor Escherich décrivit la morphologie et les propriétés d’une bactérie “Bacterium coli commune” qu’il isola des selles de nourrissons (Escherich, 1885). Nommée plus tard “Escherichia coli”, en hommage à son découvreur, E.
coli est un bacille à Gram négatif de la famille des Entérobactéries. Elle colonise le tractus
intestinal des animaux à sang chaud et constitue l’espèce bactérienne dominante de la microflore anaérobie facultative de l’intestin. Chez l’homme, la colonisation du tractus intestinal par E. coli se passe quelques heures après la naissance et sa niche écologique est la surface luminale du colon. E. coli fait ainsi partie du microbiote intestinal, véritable écosystème composé de plus de 5600 taxons différents (classification basée sur le pyroséquençage des ADNr 16s) (Dethlefsen et al., 2008). Si nous hébergeons ces populations bactériennes, en échange (symbiose), celles-ci nous assurent des fonctions indispensables, comme la modulation permanente du système immunitaire, la régulation de la prise de poids, la dégradation des aliments non digestibles, la synthèse de certaines vitamines et la protection vis-à-vis la plupart des infections pathogènes. De plus, le microbiote participe au développement, à la différenciation et à l’homéostasie des muqueuses. L’implication possible du microbiote est maintenant clairement proposée dans l’étiologie du cancer sporadique du côlon (Macdonald and Monteleone, 2005; Hooper et al., 2002; Berg, 1996).
Dans les conditions physiologiques normales, E. coli est donc une bactérie commensale. Cependant, la nature inoffensive d’E. coli peut être remise en cause dans de nombreuses situations comme en présence de brèches dans l’épithélium intestinal ou en cas d’immuno-dépression. De plus, certaines souches d’E. coli peuvent occuper des niches écologiques autres que l’intestin. C’est ainsi que certaines bactéries E. coli peuvent survivre dans le tractus urinaire. Ces souches se sont dotées d’un système d’adhésion efficace constitué de nombreux pili ainsi que d’un système de captation du fer important constitué de sidérophores, crucial pour survivre dans cet environnement (Wiles et al., 2008). La capacité de s’adapter à de nouvelles niches écologiques est donc conférée par l’acquisition de gènes spécifiques. Il est édifiant de constater que si la taille du génome de la souche K12 (isolat MG1655 ; souche de laboratoire) d’origine intestinale est de 4,64 millions de paires de bases (Blattner et al., 1997), celle de la souche CFT073, provenant du tractus urinaire, est de 5,23 millions de paires de bases (Welch et al., 2002). D’autre part, une récente étude sur 20 souches d’E. coli a montré
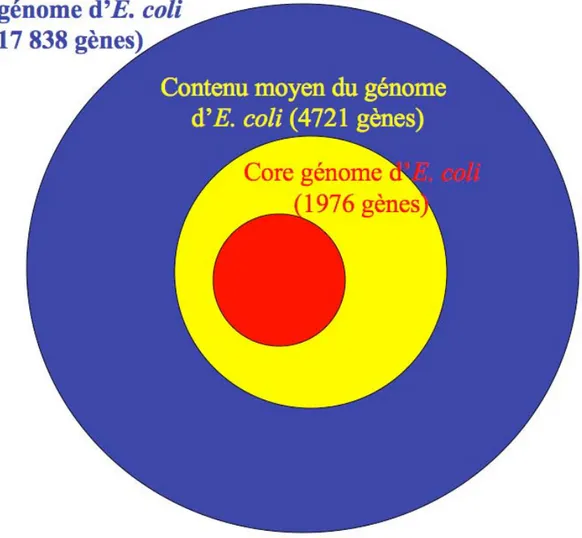
Figure 1 : Potentiel génomique d’E. coli. Diagramme représentant le pan-génome (violet), le contenu moyen d’un génome (jaune) ainsi que le core génome (rouge) pour les souches d’E. coli séquencées (Touchon et al., 2009). Figure d’après Hendrickson, 2009.
que le core génome (ensemble de gènes communs) n’est composé que d’environ 2000 gènes (représentant 40% du génome de chaque bactérie) sur les 18000 gènes du pan-génome (totalité des gènes des souches utilisées) (Touchon et al., 2009; Welch et al., 2002). Cette différence entre le core génome et le pan-génome souligne la prodigieuse plasticité du génome de cette espèce qui, loin d’être confinée dans un rôle uniquement commensal, peut lui conférer un pouvoir pathogène (figure 1). Cette pathogénicité s’explique par la capacité d’E.
coli à acquérir par transfert horizontal des gènes codant pour des facteurs dits de virulence et à
les transmettre à la descendance. Ces gènes sont portés par des éléments génétiques mobiles comme des transposons, des plasmides ou des bactériophages (figure 2) (Kaper et al., 2004). Les bactéries peuvent également faire acquisition d’îlots génomiques pouvant aller jusqu’à 200 kb et portant plusieurs gènes de virulence appelés îlot de pathogénicité (Hacker et al., 1997).
Les E. coli pathogènes sont impliquées dans les infections intestinales et extra-intestinales (figure 1). Étant donné la diversité des souches pathogènes rencontrées, celles-ci ont été classées en pathovars, groupement de souches pathogènes d’une même espèce classée en fonction de symptômes et de caractéristiques pathogéniques propres qui dépendent des différents facteurs de virulence exprimés par la bactérie. On distingue des pathovars intestinaux (responsables d’infections intestinales telles que les diarrhées) et extra-intestinaux (à l’origine d’infections extra-intestinales telles que les infections urinaires et les méningites). Les principaux pathovars intestinaux sont les ETEC (E. coli entérotoxigéniques) à l’origine de diarrhées aqueuses aiguës du voyageur ou « turista » ; les EAggEC (E. coli entéroaggrégatives) qui provoquent des diarrhées persistantes avec présence de mucus; les EIEC (E. coli entéroinvasives) qui provoquent des diarrhées aqueuses et des symptômes de dysenterie (fièvre, douleurs abdominales, diarrhée avec du sang et du mucus); les EHEC (E.
coli entérohémorragiques) qui sont responsables de diarrhées et de colites hémorragiques avec
de graves complications pouvant déboucher sur un syndrome hémolytique et urémique (anémie, diminution du taux de plaquettes sanguines, insuffisance rénale) et les EPEC (E. coli entéropathogéniques) à l’origine de diarrhées aqueuses aiguës ou persistantes (Nataro and Kaper, 1998; Giron et al., 1991b).
Dans le chapitre suivant, nous nous intéresserons au pathovar des EPEC dans lequel la protéine Cif a été identifiée pour la première fois.
Figure 2 : Contribution des éléments génétiques mobiles dans l’évolution d’une E. coli commensale vers un caractère pathogène. De nombreux facteurs de virulence sont codés par les éléments génétiques mobiles tels que des transposons (l’enterotoxine ST) ; des plasmides (l’enterotoxine LT), des bactériophages (la toxine Shiga) ou des îlots de pathogénicité (PAI, pathogenicity island) comme le LEE (locus d’effacement des entérocytes). Figure de Kaper et al., 2004.
II- Cif est un effecteur des Escherichia coli entéropathogènes
(EPEC)
II.1 Les EPEC
Les EPEC furent les premières souches d’E. coli pathogènes à être incriminées comme étant la cause des diarrhées infantiles dans les années 1940 et 1950 (Chen and Frankel, 2005). Elles provoquent des diarrhées aiguës accompagnées de fièvres, de malaises et de vomissements (Levine, 1987). De nos jours, les diarrhées à EPEC sévissent principalement dans les pays en voie de développement et ces pathogènes posent un véritable problème de santé publique car le taux de mortalité chez les jeunes enfants peut s’élever jusqu’à 30% (Senerwa et al., 1989). Par ailleurs, l’Organisation Mondiale de la Santé rapporte que la survenue de maladies diarrhéiques chez l’enfant peut avoir des conséquences à long terme se reportant chez l’adulte par un affaiblissement de la santé et de la productivité.
La caractéristique principale des EPEC est leur capacité à induire des lésions histopathologiques intestinales dites « lésions d’attachement et d’effacement ou A/E» (Knutton et al., 1987; Staley et al., 1969). Ces lésions se caractérisent par un attachement des bactéries sur la membrane des entérocyes et par une destruction des microvillosités (effacement de la bordure en brosse) (figure 3). Les lésions A/E sont associées à un réarrangement du cytosquelette de la cellule infectée. En effet, le réseau d’actine polymérisée est réorganisé pour permettre la formation d’un piédestal (structure pouvant s’allonger jusqu’à 10 m ressemblant à un pseudopode) (Knutton et al., 1989). La présence du piédestal assure un attachement très intime de la bactérie à la cellule hôte (figure 3).
L’interaction des EPEC avec les cellules en culture donne lieu à deux phénotypes d’adhésion. Les bactéries peuvent occuper toute la surface cellulaire (adhésion diffuse) ou se restreindre à quelques zones (adhésion localisée) (Scaletsky et al., 1984). Cette différence du profil d’adhésion dépend de la présence du plasmide EAF. Ce dernier code pour un pili, le BFP (bundle-forming pilus), qui participe à l’adhésion des EPEC à la cellule épithéliale et permet la formation de micro-colonies bactériennes (Giron et al., 1991a). Ces micro-colonies sont à l’origine de l’adhésion localisée. Les EPEC portant le plasmide EAF sont des EPEC « typiques » et celles ne le portant pas sont dites « atypiques ».
Figure 3 : Les lésions d’attachement et d’effacement induites par les EPEC. Ces photos de microscopie électronique illustrent les points caractéristiques de cette lésion, à savoir l’attachement intime des bactéries sur les cellules (A) grâce à la destruction localisée des microvillosités (B et C) et la formation d’un piédestal (D) Photos d’après (Frankel et al., 1998) (A-C) et (Rosenshine et al., 1996) (D).
D
A B
C EPEC
II.2 Le système de sécrétion de type III et l’induction des lésions d’A/E
Les gènes nécessaires à l’établissement des lésions A/E se trouvent sur un îlot génomique de pathogénicité de 35 kb appelé LEE (Locus d’Effacement des Entérocytes) (McDaniel and Kaper, 1997; McDaniel et al., 1995). Ainsi, les deux pathovars de E. coli (EPEC et EHEC) ou les souches de Citrobacter rodentium (pathogène murin) capables d’induire les lésions d’A/E ont acquis un LEE (Deng et al., 2001). Le LEE code pour approximativement 41 protéines qui permettent l’assemblage du système de sécrétion de type III (SST3) (figure 4), l’injection des protéines impliquées dans l’attachement intime de la bactérie, l’effacement des microvillosités et la dérégulation des fonctions des cellules cibles (Garmendia et al., 2005). Le SST3 est un appareil qui permet aux bactéries Gram négatives de sécréter et d’injecter des protéines directement dans le cytosol des cellules eucaryotes (Hueck, 1998). Ainsi, les EPEC se servent très efficacement et très habilement du SST3 pour, entre autres, mettre en place leur attachement à la cellule eucaryote. Pour cela, le LEE code pour les protéines Tir (récepteur transloqué de l’intimine) et intimine (ou eae, EPEC attachement/effacement). La protéine Tir est injectée, via le SST3, dans le cytosol de la cellule cible. Il est fascinant de constater que Tir s’insère ensuite dans la membrane plasmique et sert de récepteur à l’intimine (une adhésine des EPEC) (Kenny and Finlay, 1997). L’interaction entre Tir et intimine est essentielle à l’établissement d’un piédestal (Kenny and Finlay, 1997; Rosenshine
et al., 1996). En effet, le domaine intra-cellulaire de la protéine Tir est phosphorylé sur un
résidu tyrosine (Y474) (Rosenshine et al., 1992). Cette phosphorylation permet de fixer la protéine adaptatrice Nck qui est nécessaire au recrutement des protéines N-WASP (neural Wiskott-Aldrich-syndrome protein) et Arp2/3 (Actin-related protein) qui participent à la nucléation et à la polymérisation de l’actine (Gruenheid et al., 2001; Kalman et al., 1999). De plus, de nombreuses protéines cytosquelettiques sont recrutées au niveau du domaine intra-cellulaire de Tir permettant l’émission du piédestal (Goosney et al., 2001).
Les piédestaux concentrent de nombreux filaments d’actine. In vitro, les piédestaux peuvent être révélés par la phalloidine couplée à la fluoresceine. Cette caractéristique est utilisée dans un test appelé FAS (Fluorescence actin staining) pour révéler la présence d’un piédestal sur une cellule eucaryote (Knutton et al., 1989).
Outre les protéines Tir et intimine, d’autres effecteurs codés par le LEE participent à l’établissement des lésions d’A/E. Nous entendons par le terme « effecteur », toutes
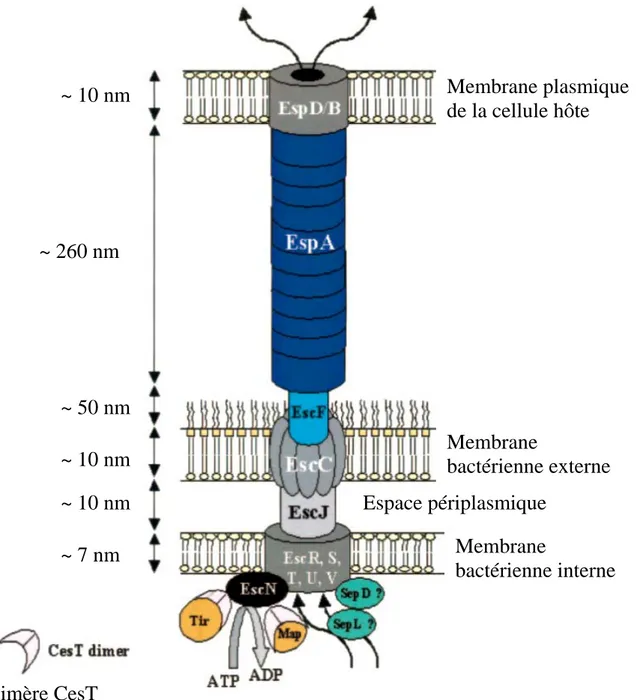
Figure 4 : Représentation schématique de l’appareil de sécrétion de type III des EPEC. Le corps basal du SST3 est composé d’une sécrétine (protéine qui forme un large canal dans la membrane externe) EscC, des protéines de membrane interne EscR, EscS, EscT, EscU et EscV et la lipoprotéine EscJ qui connecte les structures en anneau de la membrane interne et externe. EscF constitue la structure ressemblant à une aiguille alors que les sous-unités EspA polymérisent pour former le filament EspA. Les protéines EspB et EspD forment le pore de translocation dans la membrane plasmique de la cellule hôte connectant la bactérie et la cellule eucaryote. L’ATPase cytoplasmique EscN fournit l’énergie au système en réalisant l’hydrolyse des molécules d’ATP en ADP. SepD et SepL sont des composés cytoplasmiques du SST3. Les effecteurs Tir et Map se fixent aux protéines chaperons CesT qui se lient à EscN et assurent la translocation des deux effecteurs par le SST3 (Garmendia et al., 2005).
Membrane
bactérienne externe
Membrane
bactérienne interne
Espace périplasmique
Dimère CesT
~ 10 nm
~ 260 nm
~ 50 nm
~ 10 nm
~ 10 nm
~ 7 nm
Membrane plasmique
de la cellule hôte
molécules bactériennes, codées ou non par le LEE, introduites à l’intérieur des cellules eucaryotes par le SST3. Chez les EHEC par exemple, la protéine Tir transloquée dans la cellule eucaryote ne peut être phosphorylée sur le résidu Y474 (DeVinney et al., 1999). La protéine Tir des EHEC ne peut donc pas interagir avec la protéine adaptatrice eucaryote Nck pour recruter les protéines N-WASP. Pour y remédier, les EHEC utilisent un effecteur codé par un prophage, la protéine TccP (Tir-cytoskeleton coupling protein) ou EspFu qui s’associe à Tir et lie les protéines N-WASP. C’est ainsi qu’indépendamment de la protéine Nck, les EHEC polymérisent l’actine et initient la formation du piédestal (Campellone et al., 2004; Garmendia et al., 2004). Certaines souches d’EPEC utilisent les deux systèmes (Nck et Tccp) pour remodeler le réseau d’actine (Whale et al., 2006). Deux autres effecteurs, Map (mitochondrial associated protein) et EspH (EPEC secreted protein H) sont impliqués dans la balance entre l’apparition de filopodes et l’émergence du piédestal. Au début de l’interaction entre la bactérie et la cellule eucaryote, Map induit la formation de structures ressemblant à des filopodes sur le site de l’infection. Une fois l’interaction Tir-intimine réalisée, Tir réprime l’effet de Map et permet la mise en place du piédestal (Kenny et al., 2002). De même l’effecteur EspH réprime la formation de filopodes, permettant ainsi l’émergence du piédestal sur des cellules HeLa (Tu et al., 2003). Toutefois, les lésions d’A/E ne sont pas les seuls effets provoqués par les EPEC. En effet, ces pathogènes peuvent injecter plusieurs dizaines d’effecteurs par le SST3 pour moduler des fonctions cellulaires eucaryotes aussi diverses que la perméabilité des jonctions serrées, le potentiel membranaire mitochondrial, la régulation des microtubules, les fonctions de l’appareil de Golgi ou l’apoptose (Garmendia et al., 2005). Parmi ces différents effecteurs, on trouve la protéine Cif à laquelle s’est intéressé notre laboratoire.
20
II.3 Cif est un effecteur bactérien, non codé par le LEE, qui modifie la
physiologie de la cellule hôte
II.3.1 Découverte de Cif
Si la première observation d’un phénotype appelé effet cytopathique associé à certaines souches d’EPEC remonte à 1997 (De Rycke et al., 1997), ce n’est qu’en 2003 que le laboratoire a identifié et caractérisé le premier effecteur des EPEC non codé par le LEE responsable de ce phénotype. Il s’agit d’une protéine de 282 acides aminés appelée Cif (cycle inhibiting factor) (Marches et al., 2003). Le gène cif est localisé sur un prophage de type lambda qui est inductible. Ce prophage se trouve dans la plupart des cas sur le chromosome au niveau des sites d’insertion (att) des phages lambda entre les gènes ybhC et ybhB
(Loukiadis et al., 2008; Marches et al., 2003). Il existe une stricte association entre le gène cif
et celui de l’intimine (eae) ; ce qui évoque une co-sélection positive du prophage portant le gène cif avec le LEE (Iguchi et al., 2009; Loukiadis et al., 2008; Asakura et al., 2007; Marches et al., 2003). Par ailleurs, la congruence des arbres phylogénétiques entre un gène du prophage portant cif et un gène de ménage des EPEC suggère que le prophage portant le gène
cif aurait été acquis tôt dans l’évolution des EPEC (Loukiadis et al., 2008).
Le gène cif n’est présent que dans deux pathovars d’E. coli ; les EPEC et les EHEC. Dans une étude récente, le pourcentage de souches pathogènes d’EPEC et d’EHEC portant le gène cif s’élevait à 71%. Toutefois, parmi les souches d’EPEC portant cif, seules 34% induisent un phénotype associé à Cif (Loukiadis et al., 2008). En effet, dans la majorité des souches y compris dans la souche E2348/69 (souche humaine de référence des EPEC), le gène cif est muté et sous forme de pseudogène (Loukiadis et al., 2008; Marches et al., 2003). Dans les EHEC, seul le pseudogène de cif a été identifié et aucun phénotype lié à Cif n’a été mis en évidence dans ces souches. Le fond génétique des bactéries doit certainement influencer l’acquisition, le maintien ou l’expression du gène cif.
Au laboratoire, nous travaillons avec deux souches d’EPEC qui expriment une protéine Cif fonctionnelle ; une souche isolée du lapin) appelée E22 (O103 :H2), responsable de diarrhées profuses et aqueuses associées à une forte morbidité et mortalité chez les lapereaux (Leroy et
al., 1994; Pohl et al., 1993) et une souche pathogène humaine appelée B171-8 (O111 : H-) qui
a donné des diarrhées très sévères au cours d’une étude sur des volontaires humains (Bieber et
nucléotide qui provoque la substitution d’un acide aminé (P8L) sans que cela n’affecte l’activité de la protéine (Taieb et al., 2006). Bien qu’étant des EPEC, ces deux souches sont très différentes au niveau génétique. En effet, ces souches ont été séquencées et la souche B171-8 est une EPEC typique alors que la souche E22 est atypique. Par ailleurs, la souche B171-8 code pour au moins 24 effecteurs alors que la souche E22 code pour au moins 40 effecteurs (Iguchi et al., 2009; Rasko et al., 2008).
II.3.2 Cif est un effecteur du SST3
Dans le chapitre précédent, j’indique que la présence du gène cif dans les EPEC est associée à celle du LEE. Il est intéressant de noter que toute mutation des composants du SST3 abolit la capacité des souches bactériennes à induire les lésions d’A/E et le phénotype provoqué par la protéine Cif (décrit plus loin) (Nougayrede et al., 2001; Nougayrede et al., 1999; De Rycke et al., 1997). Ce résultat s’explique par le fait que la protéine Cif est injectée dans la cellule cible à travers le SST3 (Marches et al., 2003). L’injection des effecteurs du SST3 est un processus régulé et coordonné voire hiérarchisé, non seulement par leur concentration intra-bacterienne et par l’attachement des bactéries à la cellule hôte, mais aussi grâce à l’interaction de certains effecteurs avec une protéine chaperon (Mills et al., 2008; Wang et al., 2008a; Woestyn et al., 1996; Menard et al., 1994). Ainsi, il existe des protéines chaperons pour les effecteurs d’EPEC comme Tir (Creasey et al., 2003; Abe et al., 1999) et Map (Creasey et al., 2003). Cependant, concernant la protéine Cif, aucune protéine chaperon n’a été mise en évidence pour permettre sa stabilité, sa sécrétion et sa translocation par le SST3 (Charpentier and Oswald, 2004).
Pour étudier la translocation de Cif dans les cellules eucaryotes, le laboratoire a mis au point un test utilisant un système rapporteur dans lequel une fusion traductionnelle est réalisée entre la protéine d’intérêt et la protéine TEM-1 -lactamase. En présence d’un substrat, CCF2/AM, qui émet une fluorescence verte lorsqu’il est intact ou bleue lorsqu’il est clivé par la -lactamase, il est possible de suivre la translocation cellulaire d’une protéine.(Charpentier and Oswald, 2004). Cette technique a permis de déterminer la séquence d’acides aminés permettant l’adressage et la translocation de Cif via le SST3, le signal de sécrétion et de translocation (SST), qui est composé des 20 premiers acides aminés. Par ailleurs, ce SST peut
22 être échangé avec le SST d’autres effecteurs comme celui de Tir sans que cela n’affecte son activité (Charpentier and Oswald, 2004). A l’inverse, le SST de Cif peut être fusionné à des protéines pour permettre leur injection au travers de la seringue moléculaire. Le SST de Cif, par exemple, a été fusionné à la sous-unité active de la toxine CDT (CdtB). Pour être introduit dans la cellule, CdtB nécessite les sous-unités A et C. Le fait que la fusion de la sous-unité B et du SST de Cif provoque des effets décrit pour CDT montre que la protéine de fusion a bien été injectée dans la cellule (Taieb et al., 2006). De même, le SST de Cif peut également servir de contrôle positif dans des expériences visant à vérifier la translocation d’un nouvel effecteur par le SST3 (Dahan et al., 2005). Comme tous les effecteurs du SST3, Cif est donc une protéine modulaire avec un domaine N-terminal nécessaire à la translocation par le SST3 et un domaine C-terminal portant l’activité de la protéine.
II.3.3 Les effets de Cif sur les cellules eucaryotes
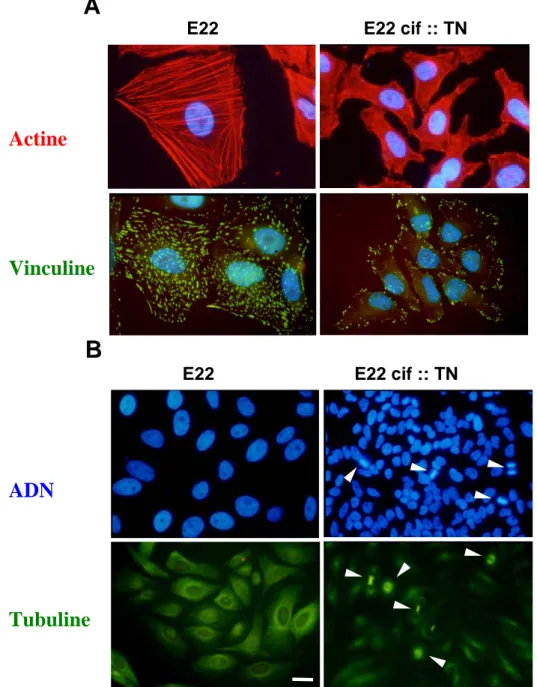
Le laboratoire a développé un test d’interaction qui consiste à mettre les bactéries et les cellules eucaryotes en contact pendant un temps court (de 30 min à 4 h). Les bactéries sont éliminées par rinçage puis par un traitement antibiotique. Cette technique permet de suivre un phénotype à long terme (jusqu’à 72 h typiquement). Ainsi, il fut observé, trois jours après l’interaction bactéries-cellules, que l’infection des cellules HeLa avec certaines souches d’EPEC altère le cytosquelette des cellules eucaryotes. Cela se caractérise par l’apparition de nombreuses fibres de stress, de plaques d’adhésions focale et par l’augmentation de la taille du noyau et des cellules d’environ 2,5 fois (figure 5A) (Marches et al., 2003; Nougayrede et
al., 2001; De Rycke et al., 1997). D’autre part, on remarque l’absence de figure de mitose et
un arrêt de la prolifération des cellules eucaryotes (figure 5B) (Marches et al., 2003; Nougayrede et al., 2001). Ces effets sur les cellules HeLa sont nommés « effets cytopathiques ». Ces derniers ne sont pas observés dans les souches mutées pour le SST3 ou délétées pour le gène cif. L’induction des effets cytopathiques résulte de la translocation de Cif dans la cellule infectée (Marches et al., 2003; Nougayrede et al., 2001). Purifiée puis lipofectée dans les cellules HeLa, Cif est capable d’induire ces effets cytopathiques démontrant que son action ne nécessite pas de co-facteurs bactériens (Taieb et al., 2006). Les mêmes effets sont obtenus suite à la transfection du gène cif dans les cellules HeLa confirmant que Cif est suffisant pour modifier la physiologie des cellules cibles (Jubelin et al., 2009).
Actine
Vinculine
ADN
Tubuline
Figure 5: La protéine Cif modifie la physiologie des cellules eucaryotes. (A) A gauche, les cellules ont été infectées avec une EPEC E22 qui produit Cif. Les cellules et les noyaux (ADN coloré au DAPI en bleu) augmentent en taille. Les fibres de stress (actine en rouge) et les plaques d’adhésion focales se multiplient. Les plaques d’adhésion focales sont de larges complexes protéiques par lesquelles la cellule est connectée à la matrice extra-cellulaire. La vinculine (en vert) fait partie de ces complexes protéiques. A droite, les cellules infectées avec la souche mutée pour le gène cif (E22 cif ::TN) présente un phénotype semblable à des cellules non infectées. (B) A l’inverse des cellules infectées avec la souche mutée pour le gène cif, les cellules en contact avec l’EPEC E22 ne présentent aucune figure de mitose
A
B
E22 E22 cif :: TN
23
III- Les protéines Cifs constituent une large famille
d’effecteurs de bactéries pathogènes et/ou symbiotiques
III.1 Les espèces bactériennes portant une protéine hétérologue de Cif
La protéine Cif a été initialement identifiée et caractérisée chez les EPEC. Si elle ne possède aucune homologie avec des protéines de fonctions connues, Cif présente des similarités de séquence avec des protéines codées par les génomes de quatre autres espèces bactériennes Yersinia pseudotuberculosis, Photorhabdus luminescens et Photorhabdus
asymbiotica et Burkholderia pseudomallei (Jubelin et al., 2009; Yao et al., 2009; Marches et al., 2003).
Y. pseudotuberculosis est une bactérie qui est répandue dans l’environnement (le sol par
exemple) qui affecte l’homme, et les animaux (Wren, 2003; Naktin and Beavis, 1999; Attwood et al., 1987). Après l’ingestion de la bactérie via l’eau ou les aliments, la bactérie se retrouve dans l’intestin grêle, traverse l’épithélium intestinal à l’intérieur des cellules M des plaques de Peyer pour migrer vers les nœuds lymphatiques mésentériques. La bactérie peut être présente également dans le foie et la rate. La multiplication du pathogène est suivie d’une inflammation qui donne lieu à des symptômes associés à une gastro-entérite, des crampes abdominales, des fièvres, des nausées et des vomissements (Wren, 2003).
P. luminescens et P. asymbiotica sont deux bactéries qui se distinguent de B. pseudomallei et
de Y. pseudotuberculosis par le fait qu’elles peuvent être symbiotiques et pathogènes. En effet, la bactérie P. luminescens colonise l’intestin de nématodes juvéniles entomopathogènes (pathogènes pour l’insecte). La bactérie et le nématode sont en symbiose. Le nématode pénètre dans des larves d’insectes et relargue P. luminescens. Cette dernière y prolifère et tue l’insecte. Le cadavre de l’insecte permet la nutrition, la croissance et la prolifération des nématodes. La bactérie s’associe de nouveau avec des nématodes juvéniles et le cycle recommence (Ciche and Ensign, 2003; Forst et al., 1997). Des travaux en cours démontrent que la protéine hétérologue de Cif chez P. luminescens est produite par la bactérie à l’intérieur de son hôte (nématode) (Chavez et al., communication personnelle). Quant à P. asymbiotica, elle entre également en symbiose avec un nématode (Gerrard et al., 2006). Cependant, P.
asymbiotica est un pathogène humain qui provoque des abcès localisés au niveau des tissus.
Les infections à P. asymbiotica ont été décrites en Amérique du Nord et en Australie. (Weissfeld et al., 2005). La transmission de la bactérie du nématode à l’homme reste à établir.
Les espèces bactériennes portant une protéine hétérologue de Cif et citées ci-dessus appartiennent à la classe des -protéobactéries et à la famille des Enterobacteriaceae. Toutefois, une autre protéine hétérologue de Cif a également été trouvée chez B.
pseudomallei. Cette bactérie de la classe des -protéobactéries (famille des Burkholderiaceae)
est présente dans le sol (boue et sédiments) et dans les eaux (eaux stagnantes des rizières, des berges). Bien que la température optimale de croissance soit comprise entre 37 et 39°C, B.
pseudomallei peut vivre en dehors des régions tropicales et résiste bien au froid. Elle est donc
présente à l’état endémique dans le sud-est asiatique, le nord de l’Australie, l’Inde, l’Iran, l’Afrique et l’Amérique. B. pseudomallei est l’agent causal de la mélioïdose. Cette maladie endémique du Sud-est asiatique et du nord de l’Australie touche les hommes et les animaux. La mélioïdose entraîne de nombreux symptômes allant de la séroconversion asymptomatique à des symptômes cliniques apparents (pneumonie aiguë et chronique, infections du foie, de la rate et du rein, septicémie pouvant entraîner un choc septique). Les deux principaux modes de contamination sont l’infection de plaies souillées par de la terre et l’inhalation de poussières contaminées. (http://www.bacterio.cict.fr/bacdico/bb/pseudomallei.html) (Cheng and Currie, 2005).
III.2 Les protéines hétérologues de Cif
Cif partage jusqu’à 72% de similarité de séquence avec la protéine Ypk1971 présente dans la souche YPIII de Yersinia pseudotuberculosis. Le pourcentage de similarité entre CifEc
et les protéines des trois autres espèces bactériennes se situe aux alentours de 40 et 50% (tableau 1). Pour distinguer Cif de ses hétérologues, la protéine Cif identifiée chez les EPEC sera nommée CifEc. Les protéines Ypk1971 (Y. pseudotuberculosis), Bpss1385 (B.
pseudomallei), Plu2515 (P. luminescens) et Pha4011 (P. asymbiotica) seront nommées
respectivement CifYp, CifBp, CifPl et CifPa. Il faut noter également que CifEc partage une
similarité de séquence avec une hypothétique protéine tronquée obtenue par traduction d’un fragment d’ADN isolé dans la mer des Sargasses (GOS5485515) (tableau 1).
25 Tableau 1 : Pourcentage d’identité et de similarité des différents hétérologues de Cif (Jubelin
et al., 2009).
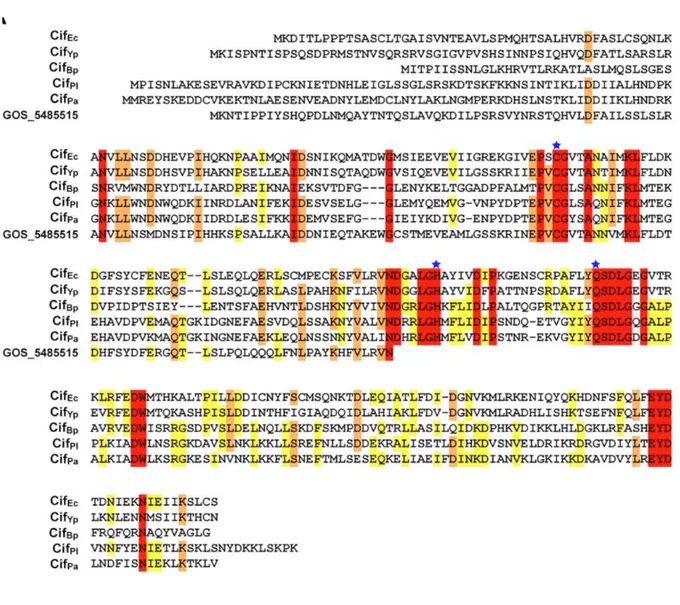
La figure 6 représente les séquences d’acides aminés alignées des différents Cifs. Ces dernières possèdent plusieurs résidus conservés après la région N-terminale. L’absence de conservation de séquence dans la région N-terminale est en accord avec l’hypothétique fonction de cette région qui servirait de séquence de translocation pour le SST3 des différentes espèces bactériennes. A ce jour, aucune donnée n’indique que CifYp, CifBp,CifPl et
CifPa sont des effecteurs du SST3, mais il est frappant de remarquer que toutes les espèces
portant un hétérologue de Cif, possèdent au moins un SST3 (Davis and Mecsas, 2007; Brugirard-Ricaud et al., 2004; Winstanley et al., 1999). A l’instar de CifEc, le domaine
N-terminal de CifBp n’est pas nécessaire à l’activité de la protéine (Yao et al., 2009). De plus, le
fait que CifBp soit capable d’être injectée dans une cellule eucaryote à travers le SST3 d’une
EPEC suggère que la translocation des différents Cifs via le SST3 des bactéries respectives est envisageable (Jubelin et al., 2009). L’absence de similarité de la région N-terminale (SST) n’est pas surprenante puisqu’il n’existe pas de séquence consensus d’adressage au SST3 y compris au sein d’une même bactérie (Ramamurthi and Schneewind, 2003). De récentes publications ont présenté des méthodes de prédiction pour identifier les SST des effecteurs (Arnold et al., 2009; Samudrala et al., 2009). Ces programmes prennent en compte de multiples facteurs comme les propriétés physico-chimiques des séquences en N-terminale, la composition en acides aminés ainsi que la fréquence de chaque résidu et le contenu G+C. La nature de cette séquence SST consensus fait encore débat. Selon le programme (http://www.chlamydiaedb.org), seuls CifEc et CifBp seraient des effecteurs sécrétés par le
SST3.
Identit (%) Similarit (%)
CifEc E. coli -
-CifYp (Ypk1971) Y. pseudotuberculosis 56 72
CifBp (Bpss1385) B. pseudomallei 26 45
CifPl (Plu2515) P. luminescens 23 42
CifPa (Pha4011) P. asymbiotica 26 46
GOS5485515 n/a 51 66
Par rapport ˆ Cif de E. coli
Figure 6 : Alignement et comparaison de séquences entre CifEc, CifYp, CifBp,CifPl, CifPa
et GOS_5485515 (protéine potentielle tronquée identifiée dans le métagénome isolé dans la mer des Sargasses). Les résidus fortement conservés sont surlignés en rouge et les acides aminés conservés à plus de 60 et 80% sont respectivement surlignés en jaune et en orange. Les étoiles bleues indiquent les résidus C, H et Q de la triade catalytique (chapitre suivant)
26
III.3 Les protéines Cifs sont des membres divergents de la
superfamille des cystéines protéases et acétyl-transférases
Récemment, les protéines CifEc, CifBp et CifPl ont été cristallisées. Malgré le faible
pourcentage d’identité entre les différentes Cifs, celles-ci présentent une structure semblable qui peut être divisée en deux parties ; une extension N-terminale composée d’hélices et un noyau glomérulaire composé de feuillets et en C-terminal (figure 7). A l’inverse des deux autres Cifs, la structure cristallisée de CifEc est partielle (acides aminés 100-282). Toutes les
protéines Cifs peuvent présenter des formes dimériques. Cependant, en condition de salinité physiologique ces molécules sont sous forme monomérique. Toutes les molécules Cifs sont fonctionnelles. En effet, tous les hétérologues de Cif lipofectés sur les cellules HeLa reproduisent les effets cytopathiques caractéristiques de CifEc (activation des fibres de stress et
arrêt du cycle cellulaire) (Jubelin et al., 2009). La conservation d’une protéine fonctionnelle à travers des espèces bactériennes phylogénétiquement très éloignées et pathogènes pour des hôtes allant des mammifères aux insectes est sans doute unique et remarquable.
Au niveau structural, la protéine la plus proche des molécules Cifs est AvrPphB de
Pseudomonas syringea (Hsu et al., 2008). CifEc partage également une homologie structurale
avec un autre effecteur bactérien, la N-acétyltransférase arylamine de Salmonella enterica. La protéine AvrPphB appartient à la famille des cystéines protéases dont l’activité dépend d’un site catalytique composé des résidus cystéine, histidine et aspartate/asparagine (Shao et al., 2002). L’alignement structural du noyau glomérulaire de CifEc avec d’autres membres de la
famille des cystéines protéases et des acétyltransférases révèle que le site actif de CifEc est
composé de la Cystéine 109, de l’Histidine 165 et de la Glutamine 185 (Hsu et al., 2008). Les différents hétérologues de CifEc présentent une triade catalytique identique. Chez CifBp, la
triade est composée des résidus Cys 156, His 211 et Gln 231 tandis que chez CifPl on retrouve
les résidus Cys 123, His 181 et Gln 200 (figure 6) (Crow et al., 2009; Yao et al., 2009). La présence des résidus Glutamines à la place des résidus Aspartate ou Asparagine, normalement présents et conservés dans les autres enzymes, est nécessaire pour avoir une bonne superposition de la triade catalytique des Cifs avec celle des autres membres de la superfamille des cystéines protéases, acétyltransférases et transglutaminases. Cette différence de composition de la triade catalytique suggère que Cif est un membre divergent de cette superfamille enzymatique. Comme Chez AvrPphB, le résidu Cys se trouve sur une hélice
Figure 7: Structures cristallisées des protéines Cifs de E. coli (forme tronquée), B.
pseudomallei et P. luminescens. La partie supérieure montre des diagrammes en ruban. Les
hélices et les feuillets sont respectivement colorés en bleu et rouge. Les acides aminés de la triade catalytique sont sous forme de bâtonnets. Le site catalytique est agrandi dans le carré gauche. La partie inférieure représente les surfaces des différents monomères de Cif. Le site catalytique est obstrué par une boucle en violet et le résidu cystéine du site catalytique est en vert. Le résidu aspartate qui fournit la charge négative près de la cystéine active est en bleu ciel (Samba-Louaka et al., 2009).
27 en N-terminale et les résidus His et Gln sur un feuillet (figure 7) (Hsu et al., 2008). L’activité de Cif dépend de cette triade. En effet, la mutation des résidus de la triade catalytique inhibe (pour les résidus Cys et His) ou réduit (pour le résidu Gln) les effets de CifEc (Hsu et al., 2008). Par ailleurs, le résidu Asp (187 chez CifEc), qui est conservé dans tous
les Cifs, confère une charge négative près du site catalytique (figures 6 et 7). Cette charge négative est essentielle à l’activité des Cifs puisque sa mutation entraîne une inactivation de Cif (Yao et al., 2009).
A ce jour, l’activité de Cif reste à déterminer. En effet, malgré une homologie avec les cystéines protéases de type papaïne, aucune activité protéolytique in vitro n’a été associée aux différents Cifs (Yao et al., 2009; Hsu et al., 2008). Le site catalytique des protéines Cifs est partiellement obstrué par une boucle qui n’est pas présente sur les cystéines protéases (figure 7). Cette obstruction partielle ainsi que la charge négative près du site catalytique pourraient conférer une certaine spécificité pour le substrat. Cependant, la délétion de cette boucle ne permet pas le clivage de la caséine, substrat classique des cystéines protéases (Hsu et al., 2008). Plus surprenant encore, Yao et al. ont montré que l’inhibiteur E64, qui se fixe irréversiblement sur la cystéine du site catalytique des cystéines protéases, se fixe spécifiquement sur la cystéine de la triade catalytique de Cif (Yao et al., 2009; Hsu et al., 2008). Or le traîtement de Cif par E-64 ou son utilisation sous forme diffusible (E64-d) sur les cellules infectées n’empêche pas les effets de Cif (Hsu et al., 2008) alors que la mutation de cette même cystéine abolit l’activité de Cif. Ce résultat paradoxal n’a pas encore pu être éclairci. Par ailleurs, il existe des enzymes portant à la fois une activité cystéine protéase et acétyltransférase. C’est le cas de l’effecteur YopJ de Yersinia qui déubiquitine (phénomène détaillé dans la discussion) IB grâce à son activité cystéine protéase et acétyle une MAPK (mitogen-activated protein kinase) Kinase 6 (MAPKK6) (Mukherjee et al., 2006; Zhou et al., 2005). On ne peut exclure que Cif possède une activité acétyltransférase ou transglutaminase.
III.4 Les protéines Cifs bloquent la transition G
2/M indépendamment
des modifications du cytosquelette
III.4.1 Les protéines Cifs modulent le cycle cellulaire et le cytosquelette des
celluleshôtes
Les cellules qui ont été exposées à CifEc se caractérisent par une apparition de nombreuses
fibres de stress, de plaques d’adhésion focale et un arrêt de la prolifération cellulaire qui se manifeste par une absence de mitose (Marches et al., 2003). Ces cellules sont arrêtées en phase G2 (Taieb et al., 2006; Marches et al., 2003; Nougayrede et al., 2001). L’inhibition de
l’entrée en mitose se caractérise au niveau moléculaire par une accumulation de la forme phosphorylée (sur Tyr 15) et inactive de l’inducteur de mitose CDK1, et au niveau cellulaire par une majorité de cellules avec 4N quantité d’ADN (Taieb et al., 2006; Marches et al., 2003; Nougayrede et al., 2001). Par ailleurs, 72 h après l’infection, des cellules HeLa avec 8N quantité d’ADN sont observées en présence de CifEc (Nougayrede et al., 2001). Le chapitre
suivant évoquera plus en détail le rôle de la protéine CDK1 ainsi que la notion de quantité d’ADN.
La translocation ou l’expression des différents hétérologues de Cif dans les cellules HeLa provoque des effets identiques à ceux induits par CifEc. On assiste à une présence importante
de fibres de stress ainsi qu’un arrêt du cycle cellulaire à la transition G2/M. Ces deux effets
dépendent de la triade catalytique des différents Cifs (Jubelin et al., 2009). Ainsi, les protéines Cifs forment une famille d’effecteurs bactériens capables d’arrêter la prolifération cellulaire qui s’étend des bactéries pathogènes aux symbiontes de nématodes (Jubelin et al., 2009).
III.4.2 L’arrêt du cycle cellulaire provoqué par les protéines Cifs ne résulte pas des modifications du cytosquelette
L’arrêt de la prolifération cellulaire induit par les protéines Cifs n’est pas la conséquence des modifications visibles du cytosquelette. D’abord, chronologiquement, Cif bloque la prolifération des cellules dans les 24 h alors qu’il faut attendre 3 jours pour observer les modifications du cytosquelette. Ensuite, l’apparition des fibres de stress et des plaques d’adhésion focale résulte de l’activation des protéines eucaryotes Rho (Ridley and Hall,
29 1992). L’inhibition des protéines RhoA, B et C par la toxine DC3B (l’exoenzyme C3 produite par Clostridium botulinum fusionnée au fragment B de la toxine diphtérique) ou par l’exotoxine EDIN (epidermal cell differentiation inhibitor produite par Staphylococcus
aureus) ne suffisent pas à lever l’arrêt de la prolifération cellulaire induite par Cif
(Nougayrede et al., 2001). Enfin, les modifications du cytosquelette observées sont restreintes aux cellules HeLa car les IEC-6 (cellules intestinales non-transformées) ou les Caco-2 (cellules de colon transformées issues d’un carcinome, non différenciées), ne montrent pas d’induction de fibres de stress ou de plaques d’adhésion focale (Taieb et al., 2006).
A l’inverse des effets sur le cytosquelette, toutes les lignées cellulaires testées (Caco-2, HeLa, HCT116, 293T) ou les cellules IEC-6 infectées avec une EPEC qui exprime Cif présentent le même phénotype; elles ne prolifèrent pas (Yao et al., 2009; Samba-Louaka et al., 2008; Taieb
et al., 2006; Marches et al., 2003; Nougayrede et al., 2001). La capacité de Cif à arrêter le
cycle cellulaire eucaryote constitue le phénotype majeur des protéines Cifs. En d’autres termes, « le cycle cellulaire des cellules eucaryotes exposées à Cif est bloqué ». Cette propriété permet de définir les protéines Cifs comme faisant partie d’une famille de molécules bactériennes appelée « Cyclomodulines ».
IV- Cif est une Cyclomoduline
Avant d’évoquer la notion de cyclomoduline, je commencerai par faire un rappel de quelques notions fondamentales du cycle et de la mort des cellules eucaryotes.
IV.1 Le cycle cellulaire eucaryote
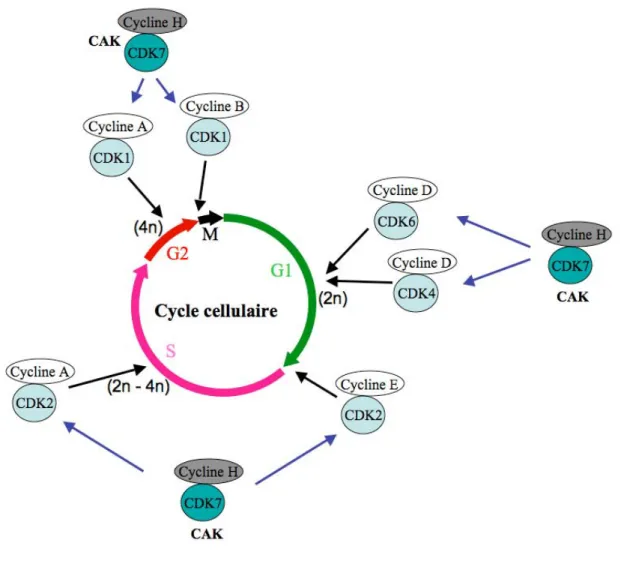
IV.1.1 Introduction
Le cycle cellulaire est un processus complexe et très régulé qui permet la duplication d’une cellule « mère » en deux cellules « filles » identiques à la cellule mère. Lorsqu’elles ne se divisent pas, les cellules sont dites quiescentes ou en phase G0. En présence de signaux
mitogènes, les cellules eucaryotes débutent un cycle cellulaire qui comprend quatre phases essentielles: la phase G1 pendant laquelle les cellules passent le point de restriction qui est un
point de non-retour à partir duquel la progression dans le cycle ne dépend plus de signaux mitogènes ; la phase S pendant laquelle s’opère la duplication du génome ; la phase G2 qui
prépare les cellules à la mitose et la phase M (ou mitose) caractérisée par la répartition du contenu cellulaire et du matériel génétique dans deux cellules filles. Il est possible de déterminer les phases dans lesquelles se trouvent les cellules en analysant leur contenu en ADN par cytométrie en flux. Les cellules en phase G1 possèdent 2n quantité d’ADN alors que
celles en phase G2 et au début de la mitose disposent de 4n quantité d’ADN (figure 8A).
IV.1.2 Les complexes CDK/Cyclines
Les protéines essentielles à la progression du cycle cellulaire sont les kinases CDKs (Cyclin-dependent Kinases) (figure 8A). Trois niveaux de régulation de ces protéines ont été décrits : l’association avec une sous-unité activatrice, la cycline ; un profil de phosphorylation faisant intervenir des phosphorylations activatrices et inhibitrices et enfin la fixation d’inhibiteurs des CDKs, les protéines CKIs. Chez les eucaryotes supérieurs, les CDKs sont actives uniquement sous forme de complexe avec des unités régulatrices spécifiques dont le
Figure 8A: Les principales phases du cycle cellulaire des eucaryotes supérieurs. La progression à l’intérieur ou entre les phases est régulée par l’activité des kinases CDKs. Trois niveaux de régulation des CDKs ont été décrits : l’association à la cycline, un profil de phosphorylation faisant intervenir des phosphorylations activatrices et inhibitrices et enfin la fixation d’inhibiteurs des CDKs. La figure ci-dessus représente deux niveaux d’activation des CDKs : la fixation aux cyclines et la phosphorylation par la CAK. Ce schéma est inspiré de (Malumbres and Barbacid, 2005).
niveau varie en fonction des différentes phases du cycle cellulaire; les Cyclines (Grana and Reddy, 1995; Gautier et al., 1990; Draetta et al., 1989; Meijer et al., 1989). Les cyclines D sont exprimées lors des phases G1 et sont dégradées en phase S par le protéasome après leur
ubiquitinylation (Diehl et al., 1997). Les cyclines qui interviennent dans la mitose (A et B) s’accumulent durant les phases G1, S et G2 et sont dégradées en mitose (King et al., 1995;
Glotzer et al., 1991). Il existe de nombreuses CDKs mais seules 4 (CDK1, 2, 4 et 6) ont un rôle clairement défini dans la régulation du cycle cellulaire.
IV.1.3 Régulation du cycle cellulaire par les complexes CDK/Cyclines
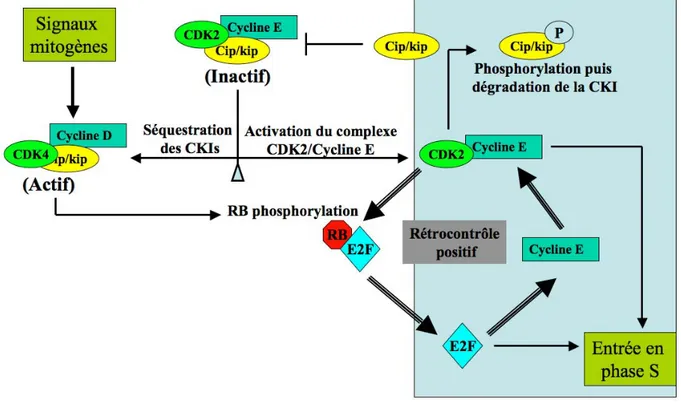
La CDK4 et la CDK6 se fixent aux cyclines D1, D2 et D3 pour assurer le déroulement de la phase G1 (Sherr, 1993). Schématiquement, les signaux mitogènes induisent la synthèse des
cyclines de type D qui s’associent aux CDK4 et 6 et permettent leur translocation dans le noyau. Les complexes CDK4,6/Cyclines D phosphorylent des protéines telles que le rétinoblastome (Rb), les protéines p107 et p130. Ces trois protéines, appelées « pocket proteins », s’associent et inhibent l’activité des facteurs de transcription de la famille E2F. La phosphorylation des complexes Rb/E2F conduit à la libération des facteurs E2F (Dyson, 1998; Nevins, 1998). Parmi les gènes régulés par les facteurs E2F, on trouve la cycline E qui s’associe avec la CDK2.
Le complexe CDK2/Cycline E formé phosphoryle des sites additionnels de Rb (rétrocontrôle positif) pour permettre la libération complète des facteurs E2F qui induisent la transcription d’autres gènes nécessaires à la phase S telle que la cycline A (Pagano et al., 1992; Zindy et
al., 1992; Girard et al., 1991). Le complexe CDK2/Cycline E assure donc la transition G1/S
(Strausfeld et al., 1996; Ohtsubo et al., 1995) tandis que le complexe CDK2-cycline A est nécessaire pour la réplication (Pagano et al., 1992).
La kinase CDK1 peut s’associer aux cyclines A et B et phosphoryle plus de 70 protéines dont celles intervenant dans sa propre régulation, dans la réplication de l’ADN ou dans la mitose (avec l’assemblage du fuseau mitotique ou même la séparation d’organites telle que le centrosome) (Malumbres and Barbacid, 2005; Ubersax et al., 2003). Dans le chapitre qui suit, j’évoquerai la transition G2/M qui est assurée par le complexe CDK1/Cycline B1 (Arellano
32
IV.1.4 Le complexe CDK1/Cycline B1 ou l’inducteur universel de mitoses
Durant la phase G2 les cellules synthétisent les cyclines B qui forment des complexes avec
CDK1. Cette dernière subit une phosphorylation sur la thréonine 161 par le complexe CAK (composé de la CDK7/Cycline H). Cette phosphorylation induit un changement de conformation qui facilite la fixation des cyclines (Larochelle et al., 2007; Sutton and Freiman, 1997; Jeffrey et al., 1995). Les complexes CDK1/Cycline B1 sont maintenus sous une forme inactive via les phosphorylations de la tyrosine 15 et de la thréonine 14 de CDK1 par les kinases Wee1 d’une part et Myt1 d’autre part (Booher et al., 1997; Liu et al., 1997; Parker et
al., 1992; Parker and Piwnica-Worms, 1992). Lorsque la cellule est prête à entrer en phase M,
les résidus Tyr15 et Thr14 sont déphosphorylés par les phosphatases Cdc25s ce qui rend le complexe actif (Draetta and Eckstein, 1997). Dans les cellules de mammifères, il existe trois isoformes de phosphatases Cdc25s : A, B et C. Toutes les isoformes jouent un rôle essentiel et surtout coopèrent pour assurer la déphosphorylation des CDKs ainsi que les transitions G1/S
et G2/M (Boutros et al., 2006). Une fois activé, le complexe CDK1/Cycline B1 peut interagir
et phosphoryler ses nombreux substrats (tels que les lamines, Wee1, Cdc25, les condensines, la kinésine Eg5 nécessaire à l’assemblage du fuseau mitotique, etc.) pour assurer la transition entre la phase G2 et la mitose (Blangy et al., 1995; Hoffmann et al., 1993; Courvalin et al.,
1992; Heald and McKeon, 1990).
La protéine CDK1 peut exécuter tous les évènements nécessaires à la progression du cycle. En effet, il existe des phénomènes de compensation entre les molécules de CDK et également entre les cyclines. Les cellules peuvent proliférer en absence de la cycline E, des CDK2, 4 et 6. De plus, la kinase CDK1 est capable de se fixer aux Cyclines D et E et de phosphoryler le Rb. Toutefois, l’activité de la protéine CDK1 ne peut être compensée (Santamaria et al., 2007; Geng et al., 2003).
IV.1.5 Les inhibiteurs des complexes CDK/Cyclines
Le troisième niveau de régulation des complexes CDK/Cyclines consiste à fixer des inhibiteurs de CDK (CKI). Deux familles de CKI ont été identifiées ; les protéines INK4 (inhibiteurs de CDK4) et les protéines Cip/kip. La famille des INK4 comprend les protéines