HAL Id: dumas-01671410
https://dumas.ccsd.cnrs.fr/dumas-01671410
Submitted on 22 Dec 2017HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Mise en place d’un modèle dynamique de biofilm
pluri-espèces
Hinarii Chonsui
To cite this version:
Hinarii Chonsui. Mise en place d’un modèle dynamique de biofilm pluri-espèces. Chirurgie. 2017. �dumas-01671410�
1
Université de Bordeaux
U.F.R. D'ODONTOLOGIE
Année 2017 N°85
Thèse pour l’obtention du
DIPLOME d’ETAT de DOCTEUR EN CHIRURGIE DENTAIRE
Présentée et soutenue publiquement
Par Hinarii CHONSUI
Née le 23 janvier 1990 à Tahiti
Le 4 décembre 2017
Mise en place d’un modèle dynamique
de biofilm pluri-espèces
Directeur de thèse
Docteur Darrène NGUYEN
Jury
Président Mme V. DUPUIS Professeur des Universités
Directeur Mme D. NGUYEN Assistante Hospitalo-Universitaire
Rapporteur Mme C. BADET Maître de Conférences des Universités
4
Remerciements
A notre Présidente de thèse
Madame le Professeur Véronique DUPUIS Professeur de Universités – Praticien Hospitalier
Sous-section Prothèses 58-01
Je vous remercie de l'honneur que vous me faites en acceptant de présider mon jury de thèse. Veuillez recevoir ici toute ma gratitude et ma reconnaissance.
A notre Directrice de thèse
Madame le Docteur Darrène NGUYEN Assistante Hospitalo-Universitaire
Sous-section Biologie orale 57-01
J'exprime ici toute ma gratitude pour la confiance que tu m'as accordée et pour m'avoir donné l'occasion de travailler sur ce sujet très intéressant. Cela a été une expérience très enrichissante.
Je tiens à te remercier pour ta patience pour toutes mes sollicitations, pour ton aide et tes conseils, qui m’ont permis de mener à bien ce travail.
Reçois ici le témoignage de ma reconnaissance pour le soutien que tu m'as apporté tout au long de ce chemin, pour ta positivité constante et pour ta bonne humeur.
5
A notre Rapporteur de thèse
Madame le Docteur Cécile BADET
Maître de Conférences des Universités – Praticien Hospitalier
Sous-section Biologie orale 57-01
Je tenais très à cœur de vous avoir parmi les membres du jury de ma thèse; aussi, c'est avec toute ma gratitude et mon profond respect que je vous remercie d'avoir accepté d'en être le rapporteur.
Je vous remercie infiniment pour votre aide et vos conseils bienveillants, pour votre disponibilité, votre patience et le temps passé pour les lectures, re-lectures et corrections apportées à la rédaction de ma thèse, ainsi qu’à sa préparation.
Je vous remercie également pour votre présence et votre bonne humeur, en clinique comme au laboratoire.
Veuillez trouver ici le témoignage de ma plus vive reconnaissance.
A notre Assesseur
Monsieur le Docteur Johan SAMOT
Maître de Conférences des Universités – Praticien Hospitalier
Sous-section Biologie orale 57-01
Je vous remercie infiniment de l'honneur que vous me faites en acceptant de faire partie des membres du jury de ma thèse et je vous en suis très reconnaissante. Je vous remercie également pour votre aide et votre gentillesse lors de mes vacations hospitalières.
6
Je dédie cette thèse à mes parents, à mon frère et à toute ma famille du fenua. Merci
pour tout votre Amour et votre soutien, même à 20 000 km !
Mes remerciements vont également :
Aux enseignants qui m’ont appris le métier avec patience et pédagogie, en particulier les
Docteurs N. Thébaud, A. Naveau et J-F. Lasserre.
A Martine, pour son aide et sa collaboration au laboratoire.
A Jennyfer et Samantha, avec qui j’ai partagé des moments importants et une grande amitié. Remerciements particuliers à Jennyfer, ma « messagère personnelle » ;-p
7
Table des matières
Introduction ... 11
Partie 1 - Etat des connaissances ... 12
I. Ecosystème buccal et biofilm dentaire ... 12
A- L’écosystème buccal 1 ... 12 1- Facteurs physico-chimiques ... 12 a- Température... 12 b- Humidité... 13 c- pH ... 13 d- Potentiel d’oxydo-réduction... 13
e- Pression partielle en gaz ... 14
2- Facteurs nutritionnels ... 14
B- Le biofilm dentaire 12 ... 14
1- Composition du biofilm ... 14
2- Formation du biofilm ... 14
a- Adhésion réversible des bactéries aux surfaces dentaires ... 15
b- Adhésion irréversible aux surfaces dentaires ... 15
c- Maturation du biofilm dentaire ... 15
d- Détachement cellulaire ... 16
C- Interactions bactériennes 34 ... 16
1- Co-agrégation ... 16
2- Interactions métaboliques ... 17
3- Sécrétion de bactériocines et autres métabolites inhibiteurs ... 17
4- Communication inter-cellulaire ... 18
a- Quorum-sensing ... 18
b- Competence-Stimulating Protein (CSP) ... 18
8
II- Les modèles expérimentaux de biofilms buccaux 5 ... 20
A- Eléments constitutifs d’un modèle de biofilm buccal ... 20
1- L’inoculum ... 20
a- Bactéries à l’état planctonique ... 20
b- Biofilms mono-espèces ... 21
c- Biofilms pluri-espèces ... 21
2- Pré-conditionnement du support du biofilm ... 22
a- Salive humaine ... 22 b- Salives artificielles ... 22 3- Milieux de développement ... 22 a- Milieux spécifiques ... 23 b- Salives... 23 4- Substrats ... 23
a- Les tissus dentaires et leur analogue ... 24
b- Le verre ... 25
B- Modèles statiques ... 26
1- Milieux gélosés ... 26
2- Plaques multi-puits et variantes ... 27
a- Plaques multi-puits ... 27
b- Le Calgary Biofilm Device ... 27
Partie 2 – Etude expérimentale ... 30
I- Matériels et méthodes ... 30
A- Souches bactériennes et conditions de culture ... 30
B- Mise en place du biofilm ... 31
1- Montage du dispositif ... 31
2- Mise en place du biofilm ... 32
C- Recueil du biofilm ... 32
9 1- Protocole 1 ... 33 a- Spectrométrie MALDI-TOF... 33 b- PCR et électrophorèse ... 33 2- Protocole 2 ... 35 3- Protocole 3 ... 36 PCR quantitative (qPCR) ... 36
4- Recherches pour identifier F. nucleatum ... 37
a- Identification de F. nucleatum sur le biofilm prélevé dans le milieu ... 38
b- Isolement de F. nucleatum sur géloses Trypticase Glucose Yeast (TGY) vert brillant ... 38
c- Co-cultures ... 38
d- qPCR sur les colonies recueillies sur les géloses Columbia au sang ... 38
II- Résultats... 39
A- Protocole 1 ... 39
Disque ... 39
Milieu ... 39
B- Protocole 2 ... 40
1- Biofilms avec S.oralis, S. mutans, A. Viscosus, P. gingivalis et F. nucleatum ... 40
2- Biofilms avec S. mutans, A. viscosus, P. gingivalis, F. nucleatum et FUM enrichi en glucose ... 41
3- Isolement de F. nucleatum sur géloses TGY vert brillant ... 41
C- Protocole 3 ... 42
1- Biofilms avec S. mutans, A. viscosus, P. gingivalis, F. nucleatum et FUM enrichi en glucose ... 42
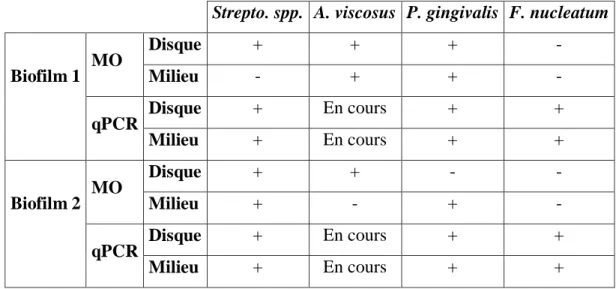
2- Biofilms avec S. salivarius, S. oralis, A. viscosus, P. gingivalis et F. nucleatum ... 43
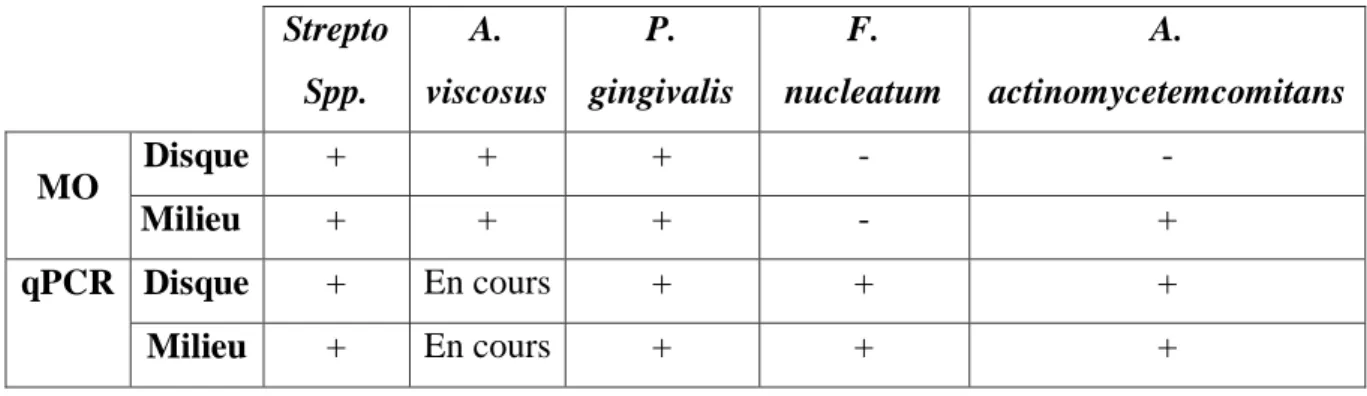
3- Biofilms avec S. oralis, S. mutans, A. viscosus, P. gingivalis, F. nucleatum et A. actinomycetemcomitans ... 44
4- Biofilms avec S. oralis, S. mutans, A. viscosus, P. gingivalis, F. nucleatum et T. forsythia... 44
5- Biofilms avec S. oralis, S. mutans, A. viscosus, P. gingivalis, F. nucleatum, A. actinomycetemcomitans et T. forsythia ... 45
10 6- Mesures du pH pour les biofilms avec S. mutans, A. viscosus, P. gingivalis, F. nucleatum et les
biofilms avec S.salivarius, S. oralis, A. viscosus, P. gingivalis et F. nucleatum ... 45
7- Identification de F. nucleatum sur le biofilm prélevé dans le milieu ... 46
8- Co-cultures ... 46
9- qPCR sur les colonies recueillies sur les géloses Columbia au sang ... 46
Partie 3 – Discussion ... 47
Conclusion ... 51
Bibliographie ... 52
Annexes ... 56
Annexe 1: Composition du bouillon TS/gélose TS/gélose TS + Sang ... 56
Annexe 2 : Composition du bouillon WCA/gélose WCA ... 56
Annexe 3 : Composition du bouillon ANA ... 57
Annexe 4 : Composition du bouillon BHI + cystéine ... 57
Annexe 5 : Composition du FUM/FUM enrichi/gélose FUM ... 58
Annexe 6 : Composition de la gélose Fuso ... 58
Annexe 7 : Composition de la gélose Mitis salivarius ... 59
Annexe 8 : Composition de la gélose TGY vert brillant ... 59
Annexe 9 : Composition de la gélose Schaedler + 5% sang de mouton ... 60
Annexe 10 : Composition de la gélose FAA + Vancomycine + 5% sang de mouton ... 60
Annexe 11 : Protocole d’extraction d’ADN (kit GenElute™ Bacterial Genomic DNA Kit Protocol)... 61
Préparations préalables ... 61
Préparation des bactéries ... 61
Préparation des bactéries à gram – ... 61
Préparation des bactéries à gram + ... 62
Isolation de l’ADN des bactéries à Gram + et Gram – ... 62
11
Introduction
Le biofilm dentaire fait partie d’un écosystème complexe qui comporte de nombreuses espèces bactériennes.
Son étude est très importante car il est à l’origine de pathologies telles que les maladies carieuse et parodontales. Ces dernières peuvent apparaître lorsqu’un déséquilibre survient au sein de l’écosystème buccal.
Le développement de modèles expérimentaux est devenu essentiel face à la difficulté d'étudier les biofilms buccaux in vivo. En effet, cela implique, entre autres, des considérations éthiques et des contraintes de reproductibilité des protocoles. De nombreux modèles ont ainsi été développés dans le but de simuler l'environnement buccal. Ils vont du plus simple, avec des modèles statiques, au plus complexe pour les modèles dynamiques.
Un protocole de modèle de biofilm statique reproductible, basé sur le modèle de Guggenheim
et al., a déjà été développé au sein du laboratoire. Nous avons ainsi voulu le faire évoluer en
modèle dynamique, avec un flux continu d’apport de milieu de culture et d’évacuation des déchets, dans le but de se rapprocher des conditions in vivo. Nous avons également fait évoluer la composition du biofilm avec un plus grand nombre d’espèces bactériennes.
Pour être validé, ce modèle doit être fiable, avec un protocole reproductible de mise en culture, de recueil et d'étude de ce biofilm.
Une fois validé, ce modèle pourra servir de support à l'évaluation de propriétés antibactériennes potentielles de molécules. La modification et/ou la maîtrise du pH et de la température seront également des objectifs à atteindre par la suite.
La première partie de ce travail présentera l’état des connaissances sur l’écosystème buccal et le biofilm dentaire, ainsi que les biofilms statiques qui existent. La deuxième partie exposera le protocole et les résultats de notre étude expérimentale. Nous finirons enfin sur une troisième partie de discussion.
12
Partie 1 - Etat des connaissances
I. Ecosystème buccal et biofilm dentaireA- L’écosystème buccal 1
Un écosystème est un ensemble composé par une communauté biotique (organismes vivants) et d’un environnement abiotique (éléments physiques et biochimiques), entre lesquels s’établissent des interactions.
La cavité buccale représente un des écosystèmes les plus complexes de l’organisme, à l’intérieur duquel cohabitent une grande variété de micro-organismes (bactéries, levures, protozoaires et virus).
Lorsque le système est en équilibre, les relations entre les micro-organismes ainsi que leurs interrelations avec l’environnement sont stables. Toute modification de l’environnement entrainera un déséquilibre de l’écosystème buccal, avec un développement plus important de certaines espèces au détriment des autres espèces, dont la population diminuera ou disparaîtra, ainsi que l’apparition de métabolites. Toutes ces modifications peuvent alors être à l’origine de pathologies bucco-dentaires telles que la carie dentaire et les parodontopathies.
La composition de la flore buccale dépend des déterminants écologiques :
1- Facteurs physico-chimiques a- Température
Chaque espèce bactérienne présente des températures optimales de développement. Celles de la plupart des bactéries buccales se situent entre 20°C et 45°C. La température moyenne de la cavité buccale est de 37°C et est relativement constante. Les bactéries sont toutefois soumises à de fortes variations de température lors des repas (boissons chaudes, glace…). Elles doivent alors pouvoir s’adapter à ces changements de températures extrêmes.
13
b- Humidité
La cavité buccale est un milieu humide car elle baigne continuellement dans la salive. Le fluide gingival, dont la production est inconstante, contribue également à l’humidité du milieu buccal. Ces deux liquides sont constitués essentiellement d’eau.
Le débit salivaire moyen au repos est de 0.35mL/mn. Une diminution de ce débit entraînera une modification de la flore buccale, favorisant l’apparition de pathologies.
c- pH
Le pH optimal de croissance de la plupart des bactéries buccales se situe entre 6 et 7,8.
Le pouvoir tampon de la salive permet de stabiliser le pH intra-buccal à des valeurs proches de la neutralité.
Cependant, en présence d’hydrates de carbone (apportés par l’alimentation), les bactéries acidogènes produisent des acides, ce qui fait chuter le pH en-dessous de 5,5. Seules les bactéries aciduriques, capables de supporter un pH acide, pourront donc survivre.
d- Potentiel d’oxydo-réduction
Au cours d’une réaction d’oxydo-réduction, un produit, l’oxydant, est réduit en captant un électron, et l’autre, le réducteur, est oxydé en émettant un électron. Le potentiel d’oxydo-réduction ou potentiel redox (Eh), exprimé en mV, indique le degré d’oxydation et de d’oxydo-réduction.
Le potentiel redox du milieu buccal est influencé par la présence ou l’absence d’oxygène et influe sur le développement de la flore. La présence d’oxygène entraîne une oxydation du milieu. Un environnement à Eh positif (ex : salive, surface de l’émail) est propice aux bactéries aérobies tandis qu’un milieu à Eh négatif (ex : plaque de 7 jours, poches parodontales) est propice aux bactéries anaérobies. La consommation de l’oxygène par les bactéries entraîne également une diminution de l’Eh. Ceci explique ainsi, en partie, le changement de la flore buccale au cours de la formation du biofilm ainsi que lors du passage d’un biofilm supra-gingival à un biofilm sous-supra-gingival.
14
e- Pression partielle en gaz
Les concentrations en oxygène et en dioxyde de carbone dans la salive ont aussi une influence sur le développement bactérien. On retrouve une concentration en oxygène plus faible chez les personnes présentant une susceptibilité à la carie élevée que chez celles qui sont résistantes à la carie. Quant au CO2, il intervient dans la stabilité du pH salivaire.
2- Facteurs nutritionnels
Pour se développer, les bactéries ont également besoin de nutriments. Ces besoins sont spécifiques à chaque espèce. L’apport des nutriments peut être exogène (hydrates de carbone et protéines issus de l’alimentation) ou endogène (hydrates de carbone, protéines, acides aminés, vitamines, issus de la salive et du fluide gingival). Il peut également provenir des produits de dégradation de nutriments par certains micro-organismes.
B- Le biofilm dentaire 12
1- Composition du biofilm
Le biofilm dentaire, encore appelé « plaque dentaire », est composé d’une fraction cellulaire, qui représente 70% de la masse, et d’une fraction acellulaire, qui représente 30% de la masse. La matrice, fraction acellulaire, comprend 80% d’eau et 20% de phase solide. Cette dernière est constituée de protéines, de polysaccharides, de lipides, d’oligoéléments et d’éléments minéraux. Ces éléments proviennent essentiellement des bactéries et de leur activité. La matrice permet la protection, la nutrition et le développement des bactéries.
2- Formation du biofilm
Les bactéries peuvent se trouver sous la forme planctonique ou sous la forme de biofilm. Cette dernière représente cependant le mode de vie privilégié des bactéries. En effet, la vie en biofilm offre plusieurs avantages aux micro-organismes, favorisant donc leur croissance. Ainsi, d’une part, cette structure est plus stable, et d’autre part, l’organisation en biofilm permet des échanges métaboliques entre les bactéries. Cette structure constitue également un environnement protecteur face aux différents facteurs environnementaux tels que les variations de pH, les agents antimicrobiens…
La forme planctonique ne constitue donc qu’une étape transitoire permettant le passage d’un biofilm à un autre.
15
a- Adhésion réversible des bactéries aux surfaces dentaires
Rapidement après le nettoyage des dents, une fine pellicule, appelée Pellicule Acquise Exogène (PAE), se forme à leur surface. Celle-ci est composée de protéines et de glycoprotéines provenant de la salive et, dans une moindre mesure, du fluide gingival et des bactéries elles-mêmes.
Quelques espèces bactériennes peuvent alors s’y attacher. Ce sont les « bactéries pionnières », principalement des Streptocoques (Streptococcus mitis, Streptococcus gordonii, Streptococcus
oralis), et certaines « colonisatrices précoces » tels qu’Actinomyces naeslundi.
Au départ, l'attachement des bactéries à la PAE est réversible et résulte d’interactions physico-chimiques non spécifiques : force hydrodynamique, forces électrostatiques et forces de Van Der Waals.
b- Adhésion irréversible aux surfaces dentaires
Les bactéries peuvent présenter à leur surface différents éléments, tels que les flagelles, qui leur permettent de se rapprocher, et les fimbriae, qui leur permettent de se fixer aux surfaces dentaires. Ces médiateurs bactériens de l’adhérence portent des molécules appelées adhésines, qui se lient aux récepteurs de la PAE par des interactions de type ligand-récepteur. L’adhésion bactérienne devient alors irréversible. On peut également retrouver des interactions de type enzyme-substrat, comme c’est le cas avec S. mutans. Cette bactérie présente à sa surface des enzymes, de type glycosyl-transférase (GTF), qui synthétisent des glucanes. Ces derniers adhèrent à la surface dentaire et leur interaction avec les GTF permet ainsi à la bactérie de se fixer à la PAE.
c- Maturation du biofilm dentaire
Les bactéries pionnières vont continuer à se multiplier et leur métabolisme entraînera la modification du milieu environnemental. L’épaisseur de la matrice extracellulaire va ainsi augmenter et les gradients d’oxygène, de substrats et de pH se modifier.
Les nouvelles conditions environnementales seront alors propices au développement de bactéries anaérobies appelées "colonisatrices tardives", telles que Fusobacterium nucleatum,
16
Par des processus de co-adhésion et co-aggrégation, ces colonisateurs secondaires vont se lier aux récepteurs des bactéries pionnières par l'intermédiaire des adhésines présentes à leur surface. Ils participent ainsi à la diversification du biofilm dentaire.
d- Détachement cellulaire
L’augmentation du nombre de bactéries et la dégradation enzymatique entraînent un appauvrissement nutritionnel au sein de la structure, ce qui favorise le détachement des bactéries. Ces dernières vont alors se disperser et coloniser d’autres sites, constituant des réservoirs de bactéries pathogènes.
C- Interactions bactériennes 34
La composition bactérienne d’un biofilm est déterminée par les relations qui existent entre les bactéries.
1- Co-agrégation
La co-adhésion entre une bactérie libre et une bactérie déjà fixée ainsi que la co-agrégation entre deux bactéries sont des mécanismes importants qui sont impliqués dans le développement d’un biofilm. En effet, comme nous l’avons vu précédemment, seules quelques espèces, les bactéries pionnières, ont la capacité de s’attacher à la PAE. Les bactéries secondaires vont alors se lier à ces dernières grâce à des récepteurs présents à leur surface. Ces relations ne se font pas au hasard mais entre des bactéries spécifiques. Des combinaisons plus complexes, mettant en relation plusieurs espèces, sont également possibles, ce qui permet à des bactéries qui ne peuvent pas co-agréger de se retrouver liées. Fusobacterium nucleatum est ainsi une bactérie importante jouant un rôle de « pont » entre certaines espèces primaires et d’autres qui sont tardives.
17
2- Interactions métaboliques
La co-agrégation contribue aux interactions métaboliques entre les espèces bactériennes en permettant leur proximité. Les interrelations métaboliques peuvent se faire par différents moyens :
- Un organisme peut utiliser comme source d’énergie un métabolite sécrété par un autre organisme ou un substrat dégradé par l’activité enzymatique d’un autre organisme.
- La vitamine K a un effet stimulateur important mais n’est pas synthétisé par les humains. Les bactéries auxotrophes à cette vitamine l’obtiennent donc grâce à des bactéries présentes dans le biofilm dentaire qui la produisent.
- La consommation de l’oxygène environnemental par les espèces aérobies procure des conditions anaérobies, indispensables à la survie des espèces anaérobies strictes.
- Certaines espèces bactériennes aident à la survie d’autres espèces en diminuant l’acidité du milieu, offrant ainsi un milieu plus favorable aux bactéries sensibles aux milieux acides.
3- Sécrétion de bactériocines et autres métabolites inhibiteurs
Les bactériocines sont des substances protéiques bactéricides produites par des bactéries pour inhiber la croissance de souches ou d’espèces étroitement apparentées.
Parmi toutes les bactéries buccales, les streptocoques sont celles qui ont la plus grande capacité à produire des bactériocines. S. mutans produit par exemple deux sortes de bactériocines, appelées mutacines :
- les lantibiotiques, synthétisés par de nombreuses espèces bactériennes à Gram positif, possèdent un spectre d’inhibition sur les bactéries à Gram positif qui est relativement large.
- les non-lantibiotiques ont un spectre d’action efficace uniquement sur S. sanguinis.
La production de bactériocines par les bactéries et leur sensibilité à ces substances contribue ainsi à la sélection bactérienne.
La production de peroxyde d’hydrogène et d’acides gras à courtes chaînes améliore également la compétitivité des bactéries qui les produisent. La production d’acide lactique, par exemple, diminue le pH au sein du biofilm, ce qui défavorise les bactéries qui sont moins aciduriques.
18
4- Communication intercellulaire
a- Quorum-sensing
Le quorum-sensing (QS) est un mécanisme de régulation de gènes induit par des molécules signal auto-générées, appelées auto-inducteurs, lorsqu’un certain seuil de la densité bactérienne est atteint. Ce système de communication contrôle diverses fonctions telles que la virulence, la tolérance à un milieu acide et la formation de biofilms.
Ce système de communication est très spécifique et précis. En effet, chaque bactérie peut produire différentes molécules signal qui vont réguler cette espèce en particulier sans affecter les autres espèces. Différentes molécules signal peuvent aussi agir les unes sur les autres, établissant ainsi un réseau tridimensionnel de systèmes de signalisations à plusieurs niveaux et de grande échelle.
On distingue trois types de systèmes de QS :
- celui utilisant des oligopeptides comme molécule signal est présent chez les bactéries à Gram+
- celui utilisant une acyl-homoserine lactone comme molécule signal est présent chez les bactéries à Gram-
- celui utilisant AI-2 comme molécule signal se retrouve chez beaucoup de bactéries à Gram+ et à Gram-
b- Competence-Stimulating Protein (CSP)
Les CSP sont de petits peptides de 14 à 23 acides aminés, produits par de nombreuses espèces de streptocoques buccaux. Ils sont impliqués dans la production de bactériocines, la virulence, la formation de biofilms et augmentent la compétence génétique, ce qui permet ensuite aux bactéries de recevoir de l’ADN exogène et de changer leurs informations génétiques.
Les CSP peuvent entraîner la production d’alarmones, molécules signal intracellulaires, en présence de facteurs environnementaux rudes. Ils peuvent également transmettre des messages complexes qui peuvent entraîner par exemple le suicide cellulaire face à des conditions de stress.
Les CSP sont très espèces-spécifiques, ce qui veut dire qu’un CSP produit par une bactérie interfère rarement avec l’activité d’un CSP produit par une bactérie différente.
19
5- Echanges génétiques
Les échanges d’ADN entre bactéries peuvent se faire de 3 façons :
- par conjugaison : une bactérie, le donneur, transfère un plasmide à une autre bactérie, le receveur. Un plasmide est un ADN extra-chromosomique circulaire à double-brin. Sa réplication est indépendante de celle de l’ADN chromosomique.
- par transduction : le transfert d’ADN se fait par l’intermédiaire de bactériophages. - par transformation : l’ADN situé à l’extérieur de la cellule est fragmenté puis importé à l’intérieur de la cellule bactérienne.
L’ADN remplace ensuite une partie de l’ADN original par un phénomène de recombinaison.
Ces échanges de gènes jouent un rôle important dans la résistance des bactéries aux antibiotiques. En effet, une bactérie sensible aux antibiotiques pourra ainsi acquérir des propriétés de résistance. La résistance aux antibiotiques est d’ailleurs un des avantages considérables de la vie en biofilm par rapport à l’état planctonique.
20
II- Les modèles expérimentaux de biofilms buccaux 5
Plusieurs caractéristiques sont à prendre en compte dans la conception d’un modèle de biofilm, notamment l'inoculum, le milieu de développement, la nature du substrat et son pré-conditionnement, et les conditions d'apport des nutriments.
A- Eléments constitutifs d’un modèle de biofilm buccal 1- L’inoculum
a- Bactéries à l’état planctonique
Les études sur bactéries à l’état planctonique sont surtout effectuées pour comparer leur adaptation face à des facteurs nocifs (choc acide, antibiotiques…) avec celle de bactéries organisées en biofilm.
En effet, les chercheurs se sont rendu compte que les biofilms constituaient la forme de vie principale des bactéries, notamment celles qui sont pathogènes, car, comme nous l’avons vu précédemment, cette structure facilite les interactions entre les bactéries et forme une barrière de protection, augmentant ainsi leur chance de survie face à des conditions environnementales hostiles 3,4.
Zhu et al. 6 ont ainsi évalué l’impact d’une période de famine sur la résistance à l’acidité de S.
mutans à l’état planctonique ou organisé en biofilm. Les résultats ont montré qu’un épisode de
famine entraînait une protection contre un choc acide, à la fois pour les biofilms et les cellules planctoniques, et que ce choc acide soit provoqué par un apport externe d’acide lactique ou par la consommation de saccharose (choc acide endogène). En ce qui concerne la formation de biofilms, celle-ci ne semble protéger les bactéries que d’un choc acide exogène et pas d’un choc acide endogène.
Sedlacek et al. se sont, eux, intéressés à la résistance à des antibiotiques de bactéries liées à la plaque sous-gingivale 7. Ils ont ainsi montré que la plupart des espèces bactériennes étaient beaucoup plus résistantes au sein de biofilms qu’en cultures planctoniques. De plus, cette résistance semblait augmenter avec la maturation du biofilm.
Suite à de telles constatations, les recherches sur les biofilms sont alors devenues plus répandues.
21
b- Biofilms mono-espèces
Les modèles de biofilms mono-espèces permettent l'étude des activités physiologiques d'espèces bactériennes spécifiques et d’évaluer l’effet de substances sur des bactéries spécifiques. K. Cogo et al. ont par exemple cultivé des biofilms mono-espèces de S. gordonii,
P. gingivalis et F. nucleatum afin d’étudier l’effet de la nicotine, de la cotinine et de la caféine
sur la viabilité de ces bactéries ainsi que leur capacité à métaboliser ces substances 8.
Cependant, ils ne reflètent pas la situation naturelle car les espèces bactériennes présentes dans la cavité buccale interagissent entre elles et forment des communautés multi-espèces 3,4.
c- Biofilms pluri-espèces
Les modèles pluri-espèces permettent ainsi de se rapprocher de la situation in vivo. Ils peuvent aller du plus simple, avec deux espèces, à des plus complexes contenant un nombre d’espèces plus important. Ces modèles rendent possible l’étude des interactions qui existent entre les différentes espèces bactériennes présentes et de leurs effets sur le développement des biofilms. S. Periasamy et al. ont par exemple étudié les interactions mutualistes entre A.
actinomycetemcomitans, F. nucleatum et Veillonella 9.
L’extrapolation des résultats à l’environnement buccal naturel doit cependant être considérée avec précaution. En effet, la composition du biofilm dentaire est beaucoup plus riche et se diversifie au cours du développement de la plaque dentaire 1; les interactions bactériennes sont donc plus complexes.
Afin de mimer les conditions naturelles, certains auteurs ont alors utilisé la salive comme inoculum. C’est le cas par exemple de J. Pratten et al. qui ont étudié la susceptibilité antimicrobienne et la composition du microcosme dentaire enrichi en saccharose 10.
Ce type d’inoculum présente néanmoins de nombreux inconvénients tels que la difficulté à standardiser l’inoculum pour la reproductibilité des expériences et la difficulté à modifier la composition à des fins expérimentales.
Des auteurs ont également proposé des inocula avec des compositions définies. Un des exemples les plus utilisés est appelé le « Marsh Consortium » et est composé de 9 bactéries. Ce consortium est reproductible et stable dans le temps 1112.
22
2- Pré-conditionnement du support du biofilm
Il consiste en la formation de la PAE, étape préalable à la formation du biofilm. Pour cela, deux types de salives peuvent être employées.
a- Salive humaine
Certains auteurs ont recours à de la salive collectée chez des volontaires sains au moins 1,5 heure après avoir mangé, bu ou s’être brossé les dents. Les échantillons de salives sont ensuite rassemblés et centrifugés puis le surnageant est pasteurisé puis centrifugé à nouveau et conservé à -20°C en attendant leur utilisation 131415
L’emploi de salive humaine présente cependant plusieurs inconvénients tels que la difficulté à recueillir de grandes quantités de salive et la variabilité de la composition. D’autres auteurs ont donc mis au point des salives artificielles.
b- Salives artificielles
On distingue des salives complexes, contenant des composants indéfinis, et des salives chimiquement définies, dont la composition est rigoureusement connue.
Parmi les salives complexes, la Basal Medium Mucin (BMM) 16 contient de l’extrait de levure,
des peptones et de la mucine.
La composition des salives complexes étant approximative, pour les expériences visant à étudier les effets des conditions environnementales et nutritionnelles sur la plaque dentaire, il est préférable d’utiliser des salives définies car elles permettent de faire des modifications de composants spécifiques.
Parmi celles-ci, la Defined Medium Mucin (DMM) est composé d’ions, de mucine, d’acides aminés, de vitamines et de facteurs de croissance à des concentrations similaires à celles de la salive 17. Cette salive artificielle a été développée à partir d’une autre salive chimiquement définie, mise au point par Shellis 18.
3- Milieux de développement
Pour permettre au biofilm de se développer, l’environnement doit être favorable aux espèces bactériennes présentes et leur fournir les nutriments nécessaires.
23
a- Milieux spécifiques
Afin de simuler l’environnement buccal, différents milieux de culture existent selon les bactéries à cultiver. Ils peuvent être achetés prêts à l’emploi pour certains, ou préparés à partir de poudres commercialisées.
Pour les modèles de biofilms mono-espèces, on choisira un milieu connu pour favoriser la croissance de l’espèce concernée.
Un milieu de culture décrit par Wilkins et Chalgren peut par exemple être utilisé pour le développement de bactéries anaérobies 19.
Pour les modèles de biofilms pluri-espèces, le milieu de croissance choisi doit permettre le développement de toutes les espèces présentes.
Pour ceux-là, le Fluid Universal Medium dans sa version modifiée (mFUM) a beaucoup été utilisé car il permet la croissance d’espèces variées 1314.
b- Salives
Pour se rapprocher des conditions in vivo, de la salive humaine peut aussi être utilisée, seule 9 ou en complément d’un autre milieu de culture 13.
De la même façon que pour le pré-conditionnement des substrats, pour pallier les inconvénients de la salive humaine, une salive artificielle peut également servir de milieu de croissance 1610.
La composition de la salive artificielle a un impact sur le développement du biofilm. J. Pratten
et al. ont par exemple comparé une salive de base (formulée par Russell et Coulter), une salive
hybride (constituée de composants empruntés à la salive précédente et à la salive de Shellis), une salive modifiée (avec plus de mucine que la salive de base et sans lab-lemco) et une salive complète (avec lab-lemco et plus de mucine) et montré que S. Sanguinis se développait peu dans les deux premières et que la dernière était un bon compromis car permettait une meilleure croissance des bactéries, tout en étant proche de la composition de la salive humaine 20.
4- Substrats
Différents substrats peuvent être utilisés comme support pour le développement d’un biofilm. Ils incluent les tissus dentaires (émail, dentine) et leur analogue (hydroxyapatite) mais également des matériaux inertes tels que verre.
24
a- Les tissus dentaires et leur analogue
L’émail et la dentine sont employés comme substrats dans les modèles de biofilms destinés à la cariologie. Cavalcanti et al. ont par exemple développé un modèle de biofilm comprenant trois espèces bactériennes (S. mutans, S. gordonii, A. naeslundii), destiné à l’étude du potentiel cariogène de glucides alimentaires et à l’évaluation des effets du fluor sur des substrats dentaires
21. Les recherches sur les moyens de prévention de la carie 2221 ou l’évaluation des propriétés
antibactériennes de divers agents 2010 sont nombreuses.
La dentine est également employée dans les études relatives à l’endodontie 2324. Enterococcus
faecalis est la cible principale de ses études car cette bactérie est très souvent retrouvée dans les
échecs de traitements endodontiques 2526.
L’émail comme la dentine peuvent être d’origine humaine 2223, ce qui permet de se rapprocher des conditions réelles. Cependant, de nombreux inconvénients inhérents à l’utilisation de dents humaines existent 27 : manque de disponibilité, variations qualitatives, contrôle de la provenance et de l’âge difficile, surface faible pouvant représenter une limite pour certaines études. Ainsi, on a souvent recours à des substrats d’origine animale, principalement bovine 20
10 21 24. Les résultats doivent cependant être interprétés avec prudence car des différences de
propriétés physiques, morphologiques et de composition chimique existent et des conclusions contradictoires ont été trouvées quant à savoir si les dents d’origine bovine pouvaient représenter un substitut adapté à celles d’origine humaine 28
L’hydroxyapatite représente aussi une alternative synthétique permettant de simuler les tissus dentaires. Ce substrat est très utilisé car il a l’avantage de ne pas nécessiter la recherche de dents extraites et permet d’établir un protocole reproductible et fiable.
Ce substrat a été très utilisé, notamment dans le modèle de biofilm mis au point par Guggenheim
et al., qui a prouvé son utilité pour l’étude d’agents antibactériens 1314.
Le choix de la nature du substrat est important car en fonction des objectifs de l’étude, elle pourra influer sur ses résultats.
Par exemple, pour l’évaluation des effets d’agents antimicrobiens, J. Pratten et al. ont démontré que les résultats dépendaient fortement de la nature du substrat.
25
Ces auteurs ont comparé l’efficacité de 3 bains de bouche contenant du triclosan, de la chlorhexidine (CHX) ou du chlorure de cétylpyridinium (CPC), sur disques d’hydroxyapatite (HAP) et sur émail bovin. Les résultats sur ce dernier ont montré que la CHX était la plus efficace pour prévenir la formation de plaque. Ces résultats étaient similaires à ceux d’une autre étude réalisée in vivo, tandis que sur les disques d’HAP, le triclosan était le plus efficace. Il serait donc préférable d’utiliser l’émail bovin pour estimer l’efficacité probable d’agents anti-plaque in vivo 20.
b- Le verre
Le verre, sous différentes formes, peut servir de support au développement de biofilms. Periasamy et al. ont ainsi étudié les interactions mutualistes entre A. actinomycetemcomitans,
F. nucleatum et Veillonella, cultivés sur des lames de verres 9. Cheng et al. ont évalué les effets d’extrait de Galla Chinensis sur la croissance et le métabolisme de biofilms en microcosme développés sur des disques de verres 29.
Divers modèles de biofilms expérimentaux ont ainsi été développés, chacun intégrant tout ou partie de ces éléments.
26
B- Modèles statiques 3031
Dans les modèles statiques, aussi appelés systèmes fermés, aucun élément nutritif n’est ajouté, ni les déchets produits (bactéries, produits bactériens, substances nutritives en excès) ne sont éliminés en cours d’expérience.
1- Milieux gélosés
Ce modèle de biofilm représente le plus simple modèle. Il consiste à faire croître des bactéries sur un milieu gélosé, contenant des substances nutritives permettant la croissance bactérienne. Bien que ce modèle soit assez simpliste, il peut être utilisé pour évaluer l’inhibition bactérienne de souches bactériennes ou d’agents antibactériens. Différentes méthodes sont décrites. Les plus employées sont :
- La méthode de diffusion sur disque : l’inoculum sur lequel on souhaite tester l’agent antibactérien est étalé sur un milieu gélosé adapté pour la croissance de l’espèce concernée, puis un disque de papier filtre imprégné du produit à tester est placé sur la gélose. Cette dernière est ensuite incubée dans des conditions appropriées (T°, atmosphère…) à l’espèce bactérienne. Après la période d’incubation, la zone d’inhibition autour du disque est mesurée 32.
- La méthode de diffusion dans des puits : un certain volume d’inoculum est étalé sur un milieu gélosé adapté. Un puits de 6 à 8mm de diamètre est ensuite réalisé dans la gélose puis une certaine quantité de l’agent à tester, à une concentration souhaitée, est ajoutée dans le puits. La gélose est ensuite incubée dans des conditions appropriées. La zone d’inhibition est ensuite observée après la période d’incubation 33.
- La méthode d’inhibition bactérienne en gélose superposée : la souche à tester (souche test) est déposée en spot sur un milieu gélosé adapté et incubée dans des conditions appropriées afin d’obtenir des colonies macroscopiquement visibles. La souche indicatrice (souche sur laquelle l’action inhibitrice de la souche test est testée) est ajustée à une certaine densité optique puis une gélose molle, préalablement ensemencée avec la souche indicatrice, est coulée sur la première gélose. La zone d’inhibition est ensuite mesurée après une période d’incubation 34.
Les résultats doivent cependant être interprétés avec prudence si on utilise des agents antibactériens cationiques car des interactions avec la gélose anionique peuvent se produire et ainsi influer sur leur efficacité.
27
La quantité de nutriments étant restreinte, le développement de biofilms sur gélose est donc variable et limité dans le temps, c’est-à-dire jusqu’à ce que toutes les ressources aient été consommées par les microorganismes.
Il est aussi à noter que, les biofilms développés sur gélose consommant les nutriments à partir du substrat, leur croissance est différente de celle s’effectuant sur tissus durs.
2- Plaques multi-puits et variantes
a- Plaques multi-puits
Une plaque multi-puits (ou de micro-titrage) est constituée d’un plateau, généralement en polystyrène, avec plusieurs puits. Le principe consiste à remplir les puits d’un milieu de culture stérile puis à inoculer les souches bactériennes, qui vont alors former des biofilms.
Dans la méthode de base, la formation des biofilms se fait sur la surface des puits en polystyrène puis, afin d’exploiter le biofilm, le milieu de culture est retiré et les puits rincés pour éliminer les cellules planctoniques. L’adhérence du biofilm, sa formation, ainsi que l’effet d’agents antibactériens peuvent ainsi être étudiés 35.
D’autres substrats, milieux de culture et inoculum peuvent également être employés.
Guggenheim et al. ont par exemple développé un modèle comportant 24 puits et dans lequel le substrat est un disque d’HAP, le milieu de culture composé de salive + mFUM et l’inoculum consiste en 5 espèces bactériennes (A. naeslundii, Veillonella dispar, F. nucleatum, S. sobrinus et S. oralis). Avant de pouvoir être exploité, le biofilm est alors recueilli. Pour cela, le disque d’HAP est rincé et vortexé dans du sérum physiologique afin d’éliminer les cellules planctoniques, puis le biofilm est recueilli (par grattage manuel et/ou à l’aide d’ultra-sons) 13.
b- Le Calgary Biofilm Device
Dans les modèles de plaques multi-puits, les cellules du biofilm ne résultent pas uniquement du processus de développement mais proviennent également de la sédimentation de cellules au fond du puits qui sont ensuite enrobées par des substances polymériques extracellulaires. Cela constitue donc un biais pour la quantification de la biomasse.
Le Calgary Biofilm Device (CBD), variante de la plaque multi-puits et commercialisé sous le nom de Minimal Biofilm Eradication Concentration (MBEC) Assay System, a ainsi été développé par Ceri et al. 36 pour pallier cet inconvénient.
28
Il permet, de plus, de réduire le nombre de bactéries à l’état planctonique qui pourraient persister après les lavages effectués comme c’est le cas avec les plaques de micro-titrage.
Ce modèle est constitué d’un couvercle avec 96 picots placé sur un récipient avec des rigoles, dans lesquelles sont plongés les picots. Le dispositif est placé sur une table oscillante et les cannelures guident le milieu de culture entre les picots. Ceci permet le développement de biofilms similaires sur les picots, qui peuvent ensuite être démontés individuellement ou simultanément.
Le couvercle est également conçu de façon à pouvoir être ajusté sur une plaque à 96 puits.
L’efficacité d’agents antibactériens peut ainsi être évaluée. Pour se faire, le couvercle sur lequel ont été cultivés les biofilms est transféré sur un plateau à 96 puits, dans lesquels auront été ajoutées les différentes concentrations des solutions à tester. Après la période d’exposition les cellules viables sont alors quantifiées 37.
Le modèle Amsterdam Active Attachment Model développé par Exterkate et al. 38 est une
amélioration du Calgary Biofilm Device dans lequel le couvercle, en acier inoxydable et adapté à une plaque à 24 puits, présente 24 « clamps » permettant de fixer différents types de substrats.
Le principal avantage de cette catégorie de modèle est qu’ils sont appropriés à des études nécessitant un haut débit car ils permettent la formation de plusieurs biofilms en même temps, indépendants les uns des autres, ce qui permet d’utiliser différents milieux de culture, substrats, inoculum, et de tester différentes substances simultanément et de façon indépendante.
Bien que ces modèles soient statiques, le milieu de culture peut toutefois être renouvelé, mais de façon discontinue.
Les modèles statiques ont l’avantage d’être simples et faciles à utiliser, ils sont généralement peu coûteux et ne nécessitent, la plupart du temps, pas d’équipement sophistiqué. Ils sont cependant loin de représenter la réalité. L’absence de flux entrant et sortant entraînant un épuisement des ressources nutritives et une accumulation des produits métaboliques, l’environnement se modifie mais ce changement n’est pas comparable à celui qui se produit au cours du développement d’un biofilm dans des conditions naturelles.
29
Pour cela, les modèles dynamiques sont plus indiqués car ils rendent possible l’apport continu de milieu de culture et l’élimination des déchets, simulant ainsi les conditions in vivo. De plus, ils permettent également la simulation des conditions environnementales complexes rencontrées dans la cavité buccale, et auxquelles sont soumises les bactéries ; le pH, la température, les conditions atmosphériques (atmosphère anaérobie, pression) et le débit du flux peuvent ainsi être contrôlés.
30
Partie 2 – Etude expérimentale
I- Matériels et méthodesNous sommes partis d’un modèle de biofilm statique qui avait été validé au sein du laboratoire
39. Ce modèle était lui-même basé sur celui de Guggenheim et al. 13, décrit précédemment.
Le modèle statique du laboratoire comptait cinq espèces (A. viscosus, F. nucleatum, S. oralis,
V. dispar et S. sobrinus), le dispositif était une plaque 24 puits en polystyrène, des disques
d’hydoxyapatite servaient de supports au biofilm et le milieu de culture était constitué de salive et de mFUM, renouvelés au bout de 24 heures par de la nouvelle salive et du mFUM enrichi (mFUMe) avec 0.15% de glucose et 0.15% de saccharose.
A- Souches bactériennes et conditions de culture
Au départ, l'inoculum était constitué des espèces bactériennes suivantes :
- S. oralis ATCC 20627 - S. mutans ATCC 25175 - A. viscosus ATCC 15987 - F. nucleatum ATCC 10953 - P. gingivalis ATCC 33277
Puis, au fur et à mesure de l'expérimentation, la composition de celui-ci a évolué pour finir avec un biofilm contenant sept espèces ; les cinq espèces de base + Aggregatibacter
actinomycetemcomitans ATCC 33384 et Tannerella forsythia ATCC 43037.
Ces bactéries ont été mises en culture dans leur milieu spécifique et incubées à 37°C : - bouillon Trypticase/Soja (TS) pour S. oralis, S. mutans et A. viscosus
- bouillon Wilkins Chalgren Anaerobe (WCA) pour P. gingivalis et F. nucleatum - bouillon Brain-Heart Infusion (BHI) + cystéine pour A. actinomycetemcomitans - bouillon ANA pour T. forsythia
300µL de sérum de cheval ont également été ajoutés au milieu pour A. viscosus, P. gingivalis,
31
48 heures avant la mise en place du biofilm, chaque espèce bactérienne est mise en culture dans du mFUM, additionné de 300µL de sérum de cheval pour A. viscosus, P. gingivalis, F.
nucleatum, A. actinomycetemcomitans et T. forsythia, et maintenue à 37°C.
Une vérification de la pureté des cultures est régulièrement réalisée, de même que celle des pré-cultures avant la mise en place d’un biofilm, grâce à une observation au microscope optique (MO) au grossissement 100, après coloration de Gram.
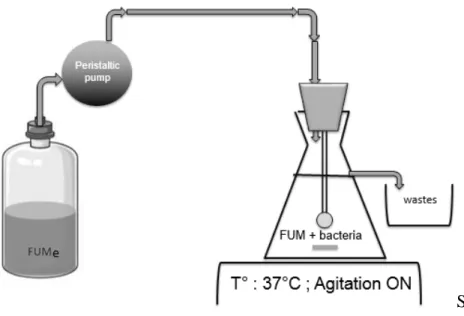
B- Mise en place du biofilm 1- Montage du dispositif
Le dispositif est composé d’un Erlenmeyer fermé à l’aide d’un bouchon présentant 2 orifices. Une burette et un fil orthodontique en acier au bout duquel est suspendu un disque d’hydroxyapatite (HAP) sont insérés dans un des orifices. La burette permet l’incorporation de sérum de cheval et des souches (et éventuellement d’autres produits) dans l’Erlenmeyer. Le disque d’HAP sert de support pour la formation du biofilm et baigne dans le milieu de culture. Dans le deuxième orifice est mis en place un tuyau relié à un flacon contenant du mFUM enrichi. L’Erlenmeyer comporte également une voie de sortie pour l’évacuation des déchets.
Préalablement à la mise en place du biofilm, un aimant est placé dans l’Erlenmeyer et 500mL de mFUM y sont versés. Pour que le dispositif soit étanche, l’interface bouchon/Erlenmeyer ainsi que les orifices sont hermétiquement fermés. Les entrées à l’extrémité des tuyaux et de la burette sont également hermétiquement fermées. Le dispositif est alors mis à l’autoclave à 120°C pendant 18 minutes.
32
Source : Dr D. NGUYEN
Figure 1 : Représentation schématique du dispositif
2- Mise en place du biofilm
La densité optique de chaque espèce est ajustée de telle sorte que DO550nm = 1 ± 0.05.
35mL de sérum de cheval sont ajoutés au mFUM dans l’Erlenmeyer par l’intermédiaire de la burette. De la même façon, les bactéries sont incorporées à part égale au milieu de culture. L’Erlenmeyer est placé sur un agitateur magnétique à plaque chauffante, réglée sur 65°C de façon à obtenir un milieu à 37°C. L’agitateur magnétique est réglé à 90tr/mn.
Le dispositif est raccordé à une pompe péristaltique qui est activée environ 18 heures après la mise en culture et réglée à un débit de 6mL/mn. Celle-ci permettra l’apport continu de mFUMe avec 0.15% de glucose + 0.15% de saccharose.
C- Recueil du biofilm
Au bout de 72 heures de culture, le disque d’HAP est récupéré et mis dans un tube en plastique contenant 1ml d’eau distillée stérile. 1mL de milieu est également prélevé au fond de l’Erlenmeyer à l’aide d’une pipette graduée stérile, de façon à recueillir les bactéries qui s’y sont développées. Les deux tubes sont ensuite placés dans une cuve à ultra-sons pendant 1 mn puis vortexés pendant 1 mn pour détacher les cellules des biofilms et les mettre en suspension. Le pH du milieu est mesuré à l’aide d’une électrode pH en verre reliée à un pHmètre.
33
D- Etude du biofilm
La composition bactérienne du biofilm est analysée pour vérifier que l'on retrouve bien les bactéries de départ.
Plusieurs techniques d’identification bactérienne ont été employées et trois protocoles ont été mis en place :
1- Protocole 1
Des dilutions à 10-2 et 10-4 sont réalisées à partir des suspensions bactériennes issues du disque et du milieu de culture.
50μL de chaque solution diluée à 10-4 sont ensuite étalés en nappes sur six géloses différentes : gélose Columbia au sang, Gélose TS + sang, gélose Fuso, gélose Mitis Salivarius, gélose WCA, gélose FUM.
Après 5 jours de croissance à 37°C en anaérobiose, les différentes colonies qui se sont développées sur les géloses sont caractérisées puis colorées par une coloration de Gram, et observées au microscope optique (MO).
Suite à cette première identification de présomption, on sélectionne, dans la mesure du possible, au moins une colonie qui pourrait correspondre à chaque espèce du biofilm, et ceci, pour les deux échantillons de biofilms recueillis (celui issu du disque et celui du milieu). Les colonies sélectionnées sont ensuite identifiées par spectrométrie MALDI-TOF et par électrophorèse.
a- Spectrométrie MALDI-TOF
Chaque colonie sélectionnée est remise en culture sur gélose Columbia au sang de façon à obtenir des colonies bien individualisées. Après 48 heures d’incubation à 37°C en anaérobiose, les géloses sont envoyées au Laboratoire de Microbiologie du CHU de Bordeaux. Une ou des colonies bien isolées sont alors sélectionnées puis identifiées par spectrométrie MALDI-TOF.
b- PCR et électrophorèse
Après 96 heures de croissance, sur gélose Columbia au sang, à 37°C en anaérobiose, les colonies sont recueillies à l’aide d’un écouvillon stérile puis mises en suspension dans un tube contenant 1mL d’eau distillée stérile. L’extraction d’ADN est alors réalisée à l’aide du kit GenElute™ Bacterial Genomic DNA Kit Protocol.
34
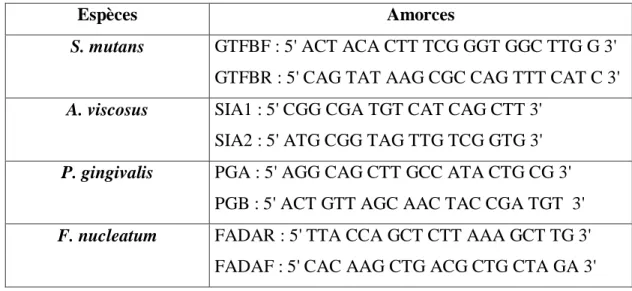
On effectue ensuite une amplification génomique par PCR, avec des amorces et selon un programme spécifique à chaque espèce présumée (cf. tableaux 1 et 2). L’ADN amplifié est mise en évidence par électrophorèse sur gel d’agarose.
Tableau 1 : Amorces de chaque espèce
Espèces S. mutans A. viscosus P. gingivalis F. nucleatum
Nombre de cycles 30 32 30 30 Dénaturation initiale 94°C 2mn 94°C 4mn Dénaturation 95°C 30s 96°C 15s 94°C 30s 94°C 30s Hybridation 59°C 30s 55°C 30s 55°C 30s 55,8°C 30S Elongation 72°C 1mn 72°C 1mn 72°C 30s 72°C 40s Elongation finale 72°C 10mn 72°C 10mn 72°C 10mn 72°C 6mn
Tableau 2 : Programmes PCR de chaque espèce
Si l’extraction d’ADN n’est pas effectuée immédiatement après le recueil des colonies, les tubes sont conservés à -80°C. De même, si l’amplification génomique n’est pas réalisée juste après l’extraction d’ADN, les tubes sont conservés à -20°C.
Espèces Amorces
S. mutans GTFBF : 5' ACT ACA CTT TCG GGT GGC TTG G 3'
GTFBR : 5' CAG TAT AAG CGC CAG TTT CAT C 3'
A. viscosus SIA1 : 5' CGG CGA TGT CAT CAG CTT 3'
SIA2 : 5' ATG CGG TAG TTG TCG GTG 3'
P. gingivalis PGA : 5' AGG CAG CTT GCC ATA CTG CG 3'
PGB : 5' ACT GTT AGC AAC TAC CGA TGT 3'
F. nucleatum FADAR : 5' TTA CCA GCT CTT AAA GCT TG 3'
35
2- Protocole 2
Pour ce protocole, la composition bactérienne était la même que celle du protocole 1 pour certains biofilms, et pour d’autres, seuls S. mutans, A. viscosus, P. gingivalis et F. nucleatum ont été intégrés et le FUM enrichi l’était uniquement en glucose, sans saccharose.
Les méthodes d’identification bactérienne utilisées ont été la coloration de Gram et/ou la spectrométrie de masse MALDI-TOF.
L’analyse par spectrométrie de masse MALDI-TOF a été réalisée sur deux biofilms. Le protocole a été légèrement différent pour chacun d’eux.
Pour le premier, différentes colonies isolées ont été sélectionnées sur les géloses qui ont servi à la coloration de Gram. Elles ont ensuite été identifiées directement par spectrométrie MALDI-TOF. Ce protocole n’ayant pas fonctionné (l’appareil ne détectait pas les bactéries), un deuxième protocole a donc été testé pour le deuxième biofilm.
Pour ce dernier, après avoir sélectionné trois colonies isolées différentes sur la gélose « Disque 10-2 » (gélose sur laquelle la dilution à 10-2 correspondant au biofilm issu du disque a été étalée)
qui a servi à la coloration de Gram, celles-ci sont mises en suspension dans 100µL d’eau distillée stérile puis 50µL sont étalés en nappes sur des géloses Columbia au sang. Après 72 heures à 37°C en anaérobiose, une colonie isolée sur chaque gélose est alors sélectionnée puis une extraction des protéines est effectuée avant de réaliser l’identification par spectrométrie MALDI-TOF.
36
3- Protocole 3
Pour ce protocole, nous avons mis en place des biofilms avec uniquement S. mutans, A.
viscosus, P. gingivalis et F. nucleatum, dans un milieu FUM enrichi uniquement avec du
glucose, sans saccharose.
Nous avons également mis en place des biofilms dont l’inoculum était composé de S. salivarius,
S. oralis, A. viscosus, P. gingivalis et F. nucleatum et le FUMe était un FUMe habituel.
Enfin, pour les derniers biofilms mis en place, nous sommes revenus à la composition bactérienne de départ et nous avons commencé à intégrer les espèces A. actinomycetemcomitans et T. forsythia. En ce qui concerne les milieux de culture, nous avons également étalé ces biofilms sur des géloses Fastidious Anaerobe Agar (FAA) + Vancomycine + 5% de sang de mouton défibriné et sur des géloses Schaedler + 5% de sang de mouton défibriné, en plus des étalements habituels sur géloses Columbia au sang.
Les méthodes d’identification bactérienne utilisées ont été la coloration de Gram et la PCR quantitative.
Après 5 jours de croissance à 37°C en anaérobiose, les différentes colonies qui se sont développées sur les géloses sont caractérisées puis colorées à la coloration de Gram et observées au MO au grossissement 100.
PCR quantitative (qPCR)
La méthode de qPCR nécessite d’abord de constituer une gamme étalon pour chaque espèce bactérienne utilisée pour le biofilm. Celles-ci serviront ensuite de références pour l’analyse de tous les échantillons de biofilms qui seront recueillis.
Pour réaliser ces gammes étalons, pour chacune des espèces bactériennes, des dilutions sérielles de 10-1 à 10-6 sont effectuées en triplicata à partir de suspensions non diluées (ND) de chaque bactérie. Ces dernières constituent ainsi, avec les dilutions, les échantillons standards.
2mL de chaque échantillon standard sont ensuite congelés à -80°C en attendant l’extraction d’ADN puis l’établissement des gammes étalons.
Parallèlement, pour chaque souche, 50µL de chaque échantillon standard sont étalés en nappes, de façon indépendante, sur des géloses Columbia au sang. Après 96 heures de croissance à 37°C en anaérobiose, on procède alors au comptage des Unités Formant Colonies (UFC). Ce dénombrement est ensuite mis en relation avec les gammes étalons et permettra ainsi de quantifier le nombre de bactéries présentes, pour chaque espèce, dans les échantillons des biofilms qui seront recueillis.
37
Suite à ces expérimentations préliminaires, pour chaque biofilm mis en place, nous procédons comme suit :
2mL de milieu sont prélevés au fond de l’Erlenmeyer, de façon à recueillir le biofilm qui s’y est développé, puis sont mis dans un tube en plastique stérile. Ceci est réalisé en triplicata. Ces trois tubes, ainsi que le reste des suspensions bactériennes du biofilm recueilli sur le disque, sont congelés à -80°C en attendant l’extraction d’ADN. Suite à cette dernière, les tubes sont conservés à -20°C en attendant que l’identification par qPCR se fasse à l’Institut des Sciences de la Vigne et du Vin (ISVV).
Figure 3 : Schéma du protocole 3
4- Recherches pour identifier F. nucleatum
En raison des difficultés rencontrées pour retrouver F. nucleatum sur nos biofilms, plusieurs manipulations supplémentaires ponctuelles ont été entreprises afin d’essayer de situer à quelle étape du protocole F. nucleatum n’est plus présent :
38
a- Identification de F. nucleatum sur le biofilm prélevé dans le milieu
Lors du recueil du biofilm, les bactéries qui s’étaient développées dans le milieu environnant ont été prélevées et observées au MO après coloration de Gram.
b- Isolement de F. nucleatum sur géloses Trypticase Glucose Yeast (TGY) vert brillant
Trois biofilms ont été étalés sur des géloses TGY vert brillant pour essayer d’isoler F.
nucleatum 1. Pour cela, le même protocole d’étalement que celui décrit précédemment pour les géloses Columbia au sang, FAA et Schaedler, a été appliqué. De même, ces étalements ont également été suivis d’une caractérisation des colonies, d’une coloration de Gram, ainsi que de l’observation au MO X100.
c- Co-cultures
Afin de voir s’il existe des inhibitions ou compétitions entre F. nucleatum et chacune des quatre autres espèces bactériennes composant le biofilm (S. oralis, S. mutans, A. viscosus et P.
gingivalis), nous avons effectué des co-cultures entre ces bactéries, que nous avons ensuite
étalées sur des géloses Columbia au sang pour vérifier par la suite la présence de F. nucleatum. Pour ce faire, la densité optique de chaque espèce est ajustée de telle sorte à avoir DO550nm = 1
± 0.05, puis 500µL des espèces correspondantes sont mis en culture dans un tube contenant 15mL de FUM additionnés de 300µL de sérum de cheval. Les tubes sont incubés à 37°C en anaérobiose pendant 72 heures, puis 50µL de chaque co-culture sont alors étalés en nappes sur des géloses Columbia au sang. Après 48 heures de croissance à 37°C en anaérobiose, les colonies sont caractérisées puis identifiées par une observation au MO X100 après coloration de Gram.
d- qPCR sur les colonies recueillies sur les géloses Columbia au sang
Afin de confirmer ou d’infirmer l’hypothèse selon laquelle l’absence de F. nucleatum serait un problème d’identification au niveau des géloses, nous avons employé la méthode d’identification par qPCR sur l’ensemble des colonies recueillies sur les géloses Columbia au sang sur lesquelles ont été étalés les biofilms. En effet, soit après étalement sur géloses Columbia au sang, F. nucleatum ne survivrait pas suite aux compétitions avec les autres bactéries, soit il serait présent mais nous n’arrivons pas à le détecter à l’œil nu.
Pour cette étape, l’ensemble des colonies sélectionnées après coloration de Gram a été recueillies à l’aide d’un écouvillon stérile puis mise en suspension dans un tube contenant 1.5mL d’eau distillée stérile. Les tubes sont ensuite conservés à -80°C en attendant l’extraction d’ADN puis à -20°C avant l’identification par qPCR.
39
II- Résultats
A- Protocole 1 Disque
Strepto. spp. A. viscosus P. gingivalis F. nucleatum
Biofilm 1 MO + + + + MALDI-TOF - - - - PCR S. mutans + - - Biofilm 2 MO - + + -
MALDI-TOF n.a. - - n.a.
PCR n.a. - n.a. n.a.
Biofilm 3
MO + + + -
MALDI-TOF S. mutans - - n.a.
PCR S. mutans - - n.a. n.a.: non applicable
Tableau 3: Identifications bactériennes sur les biofilms recueillis sur le disque
Milieu
Strepto. spp. A. viscosus P. gingivalis F. nucleatum
MO + + + +
Biofilm 1 MALDI-TOF - - - -
PCR S. mutans + - -
MO + + + -
Biofilm 2 MALDI-TOF - n.a. n.a. n.a.
PCR n.a. n.a. n.a. n.a.
MO - + + -
Biofilm 3 MALDI-TOF n.a. - - n.a.
PCR n.a. - - n.a.
n.a. : non applicable
Tableau 4 : Identifications bactériennes sur les biofilms recueillis dans le milieu environnant
Pour ce protocole, F. nucleatum n’a été détecté sur aucun des biofilms, sauf sur le premier, mais uniquement sur la base de l’observation au MO.
40
Les résultats de la spectrométrie MALDI-TOF ont été négatifs pour la majorité des cas. Ils ont de plus révélé la présence de contaminations. Seul S. mutans a pu être détecté avec cette technique.
Les résultats de la PCR + électrophorèse ont été positifs pour tous les essais réalisés sur S.
mutans et seulement un test réalisé sur A. viscosus. Ils ont été négatifs pour tous les protocoles
effectués sur P. gingivalis et F. nucleatum.
A. viscosus et P. gingivalis ont été retrouvés à la fois sur le disque et dans le milieu tandis que
les streptocoques n’ont été retrouvés que dans le milieu pour le biofilm 2 et uniquement sur le disque pour le biofilm 3.
Sans différencier les espèces retrouvées sur le disque et celles retrouvées dans le milieu, toutes les bactéries n’ont été retrouvées sur le même biofilm que sur le biofilm 1.
B- Protocole 2
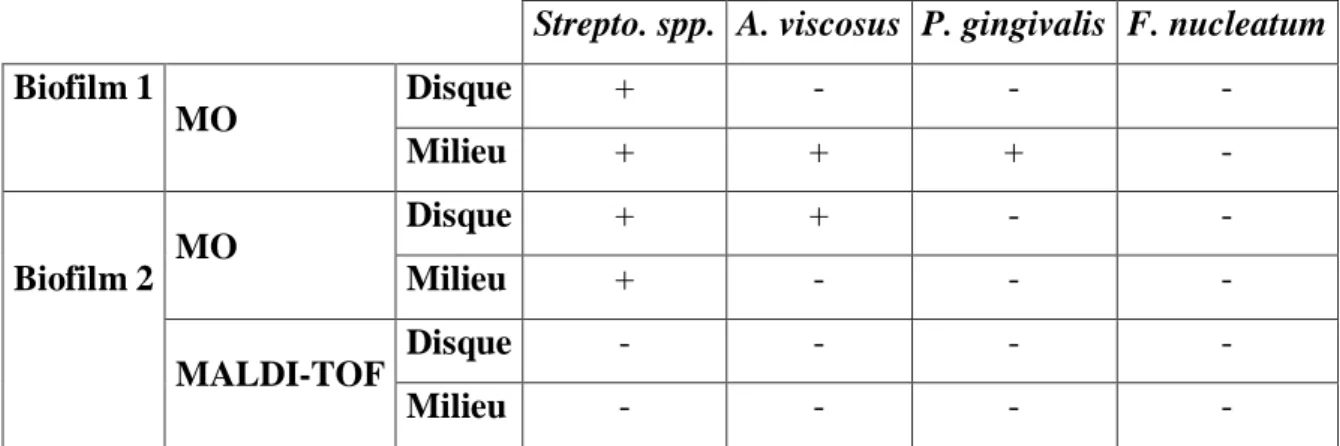
1- Biofilms avec S. oralis, S. mutans, A. viscosus, P. gingivalis et F.
nucleatum
Strepto. spp. A. viscosus P. gingivalis F. nucleatum
Biofilm 1 MO Disque + - - - Milieu + + + - MO Disque + + - - Biofilm 2 Milieu + - - - MALDI-TOF Disque - - - - Milieu - - - -
Tableau 5 : Identifications bactériennes sur les biofilms avec S. oralis, S. mutans, A. viscosus,
P. gingivalis et F. nucleatum
41
2- Biofilms avec S. mutans, A. Viscosus, P. gingivalis, F. nucleatum et FUM enrichi en glucose
S. mutans A. viscosus P gingivalis F. nucleatum
MO Disque + + + - Biofilm 1 Milieu - - + - MALDI-TOF Disque + + - - Biofilm 2 MO Disque + + + - Milieu + - + -
Tableau 6 : Identifications bactériennes sur les biofilms avec S. mutans, A. viscosus, P.
gingivalis, F. nucleatum et FUM enrichi en glucose
F. nucleatum n’a été retrouvé sur aucun des biofilms.
Les trois autres espèces ont été retrouvées sur tous les biofilms.
3- Isolement de F. nucleatum sur géloses TGY vert brillant
Strepto. spp. A. viscosus P. gingivalis F. nucleatum
Biofilm 1 Disque - + - -
Milieu + + + -
Biofilm 2 Disque - - - -
Milieu - - - -
Tableau 8 : Identifications bactériennes par observation au MO des colonies qui se sont développées sur les géloses TGY vert brillant après étalement des biofilms recueillis
Les géloses TGY vert brillant n’ont pas permis d’isoler F. nucleatum. Aucune colonie ne s’est développée sur les géloses du dernier biofilm.
42
C- Protocole 3
1- Biofilms avec S. mutans, A. viscosus, P. gingivalis, F. nucleatum et FUM enrichi en glucose
S. mutans A. viscosus P. gingivalis F. nucleatum
MO Disque + - + - Biofilm 1 Milieu + - + - qPCR Disque + En cours + + Milieu + En cours + + MO Disque - - + - Biofilm 2 Milieu - - + - qPCR Disque + En cours + + Milieu + En cours + + MO Disque - + + - Biofilm 3 Milieu - + + - qPCR Disque + En cours + + Milieu + En cours + + MO Disque - - + - Biofilm 4 Milieu - + + - qPCR Disque + En cours + + Milieu + En cours + +
Tableau 9 : Identifications bactériennes sur les biofilms avec S. mutans, A. viscosus, P.
gingivalis, F. nucleatum et FUM enrichi en glucose
Les résultats sont identiques dans l’ensemble entre le disque et le milieu pour chaque biofilm. Sur la base de l’identification phénotypique par MO, F. nucleatum n’a été retrouvé sur aucun des biofilms alors qu’il a été identifié par la qPCR.
Seul P. gingivalis a été identifié sur tous les biofilms, aussi bien par la méthode microscopique que par la méthode de biologie moléculaire (qPCR).