Étude de la régulation transcriptionnelle au locus
ENPP2.
Thèse
Déborah Argaud
Doctorat en médecine moléculaire
Philosophiæ doctor (Ph. D.)
Étude de la régulation transcriptionnelle
au locus ENPP2
Thèse
Déborah Argaud
Sous la direction de :
R
ÉSUMÉPour maintenir leurs intégrités, les cellules doivent continuellement moduler leurs programmes transcriptionnels selon les signaux environnementaux qu’elles perçoivent. Le contrôle de l’expression des gènes est un processus biologique complexe qui repose sur l’accessibilité de la chromatine aux facteurs de transcription et à l’ARN polymérase II (Pol II). Pour assurer un certain niveau d’accessibilité, la position des nucléosomes est finement régulée par des complexes protéiques. Certains utilisent l’hydrolyse de l’ATP pour altérer les liens histones-ADN tandis que d’autres mettent en jeux des modifications post-traductionnelles sur la queue N-terminale des histones. Les régions régulatrices distales, de type enhancer, capable de former des boucles chromatiniennes et de réguler l’activité d’un promoteur cible, à un moment précis, jouent également un rôle primordial dans l’établissement de programmes transcriptionnels spécifiques.
Bien souvent, en situation pathologique, l’altération de ces mécanismes moléculaires et/ou épigénétiques va conduire à l’expression aberrante du programme transcriptionnel normal. Ainsi, une meilleure compréhension du processus de transcription et des mécanismes sous-jacents pourrait aboutir au développement de nouvelles avenues thérapeutiques. Dans ce cadre, l’objectif général de cette thèse est d’étudier la régulation de la transcription au locus
ENPP2. ENPP2 est un gène impliqué dans le cancer, les pathologies immunes et les maladies
cardiovasculaires. Si sa surexpression est clairement établie en situation pathologique, les mécanismes moléculaires sous-jacents restent à préciser.
Dans un premier temps, dans les cellules HEK293T, nous avons montré que le promoteur du gène ENPP2 possède des caractéristiques épigénétiques très particulières, correspondantes à un promoteur bivalent. A l’état basal, la présence de marques d’histones activatrices H3K4me3 et répressives H3K27me3 a été détectée dans la région promotrice d’ENPP2, ainsi que le recrutement de l’ARN Polymérase II, bien que le gène soit non transcrit. Sous l’effet d’une stimulation au LPS, ENPP2 est induit via une signalisation NF-кB et la réduction de la marque H3K27me3 par l’histone déméthylase UTX/KDM6A. Simultanément, l’histone déméthylase JMJD3/KDM6B est recrutée au site d’initiation de la transcription (TSS), et dans le corps du gène contrôlant ainsi l’expression d’ENPP2 indépendamment de son activité enzymatique. Une analyse de l’interactome de JMJD3 par spectrométrie de masse a permis d’identifier l’ARN hélicase DDX21, connue pour limiter la formation des R-loops, comme un nouveau partenaire protéique de JMJD3. Nous proposons ici que la résolution des R-loops par DDX21, au locus ENPP2 favorise la synthèse de transcrits naissants.
Dans un second temps, nous présentons des données suggérant qu’un enhancer distal participe à la régulation transcriptionnelle d’ENPP2 via la formation d’une boucle chromatinienne permettant l’enrichissement du complexe JMJD3-DDX21 au TSS d’ENPP2 et sa progression le long du corps du gène.
A
BSTRACTIn order to define and maintain cellular functions, eukaryotic cells continuously adapt their transcriptional programs to environmental signals. The control of gene expression is a complex biological process, which depends on the chromatin accessibility to transcription factors and RNA polymerase II. To ensure a certain level of accessibility, the position of nucleosomes is finely regulated by protein complexes. Some hydrolyse ATP to alter histone-DNA bounds, while others add or remove post-translational modifications on N-terminal histone tails. Distal regulatory elements, such as enhancers, which are able to form physical chromatin interactions with a target promoter and control its transcriptional activity, play a fundamental role in establishing specific transcriptional programs.
The alteration of these molecular and/or epigenetic mechanisms are common in human diseases and lead to the aberrant expression of transcriptional programs. Thus, a better understanding of the transcription process could lead to the development of new therapeutics. In this context, the general objective of this thesis is to study the transcriptional regulation of the ENPP2 locus. ENPP2 is a gene involved in cancer, immune and cardiovascular diseases. Although its overexpression is clearly established in various pathological situations, the underlying molecular mechanisms remain elusive.
Here, we show in HEK293T cells that ENPP2 has all the characteristics of a bivalent promoter, marked by an enrichment of H3K4me3 and H3K27me3 and associated with promoter-proximal stalling of RNA polymerase II. Upon LPS stimulation, ENPP2 is induced through NF-κB signaling and the reduction of the H3K27me3 mark by the histone demethylase UTX/KDM6A. Simultaneously, the histone demethylase JMJD3/KDM6B is recruited to the TSS, along the gene body and controls the expression of ENPP2, independently of its enzymatic activity. Mass spectrometry analysis revealed a novel interaction for JMJD3 with DDX21, an RNA helicase that unwinds R-loops created by hybridization between nascent RNA transcript and DNA template. Here, we propose that R-loops unwinding by DDX21 at the
ENPP2 locus favors the synthesis of nascent transcripts. In a second step, we present data
suggesting that a distal enhancer participates in the transcriptional regulation of ENPP2 via the formation of a chromatin loop, allowing the enrichment of the JMJD3-DDX21 complex at ENPP2 TSS and its progression through the gene body.
T
ABLE DES MATIERESRÉSUMÉ ... III ABSTRACT ... V LISTE DES FIGURES ... X LISTE DES ABRÉVIATIONS ... XII REMERCIEMENTS ... XV AVANT-PROPOS ... XVI
INTRODUCTION ... 1
1. DES SÉQUENCES RÉGULATRICES AU CŒUR DE L’ADN ... 2
1.1. Les promoteurs ... 2
a) Les principales caractéristiques des promoteurs ... 2
b) État de la chromatine au niveau des promoteurs ... 4
1.2. Les enhancers ... 4
a) La découverte des enhancers ... 4
b) Les caractéristiques fonctionnelles des enhancers ... 5
Les super-enhancers, une sous population d’enhancers: ... 7
La formation des biomolecular condensates ... 7
c) La communication enhancer-promoteur ... 8
La formation des boucles enhancer-promoteur est affectée par l’organisation 3D du génome ... 9
Plusieurs protéines structurales participent à la formation des boucles enhancer-promoteur ... 10
2. LE PROCESSUS DE TRANSCRIPTION ... 13
2.1. Composition et structure de la Pol II ... 13
2.2. Le cycle de transcription ... 13
a) L’initiation de la transcription : l’assemblage du PIC ... 15
Le modèle séquentiel... 15
Le modèle de l'holoenzyme ... 16
b) L’élongation de la transcription ... 16
c) La terminaison de la transcription ... 17
d) La phosphorylation du CTD au cours de la transcription ... 17
2.3. L’expression des gènes est finement régulée par le phénomène de pause transcriptionnelle ... 19
a) Mise en évidence des gènes en pause ... 19
b) Les mécanismes de maintien de la pause et de relâche ... 21
c) Les fonctions biologiques de la pause transcriptionnelle ... 22
2.4. La formation des R-loops : un processus co-transcriptionnel ... 23
a) Les différents modèles de formation des R-loops... 24
c) La distribution génomique des R-loops et leurs fonctions ... 25
d) Les mécanismes de surveillance des R-loops ... 27
Les enzymes RNase H ... 27
Les ARN/ADN hélicases ... 28
o La famille des DEAD box proteins (DDX) ... 28
Les topoisomérases ... 29
Des protéines impliquées dans la biogenèse des ARNs ... 29
3. LE CONTRÔLE DE LA TRANSCRIPTION EN CONDITIONS INFLAMMATOIRES ... 30
3.1. La transduction des signaux inflammatoires ... 30
3.2. NF-ΚB, un facteur de transcription contrôlant la réponse inflammatoire ... 32
a) Les membres de la famille NF-κB ... 32
b) Les voies de signalisation NF-κB ... 33
c) Les modifications post-traductionnelles de NF-κB ... 35
La phosphorylation et l’acétylation de p65 ... 35
L’hypothèse des « codes-barres » NF-κB ... 36
3.3. Transcription et remodelage de la chromatine ... 36
a) Les acteurs de la transcription coopèrent avec des complexes de remodelage de la chromatine ... 37
b) Les modifications post-traductionnelles des histones ... 38
La tri-méthylation de la lysine 27 de l’histone H3 (H3K27me3) ... 38
Rôle fonctionnel de JMJD3 dans la transcription ... 39
3.4. La cinétique d’activation des gènes ... 40
a) Les primary response genes (PRGs) ... 40
b) Les secondary response genes (SRGs) ... 40
4. ENPP2 : UN GÈNE INFLAMMATOIRE ... 42
4.1. ENPP2 : une lysophospholipase D ... 42
a) La synthèse du LPA ... 42
b) Les voies de signalisation du LPA ... 42
c) La dégradation du LPA ... 43
4.2. Le rôle physiopathologique d’ENPP2 ... 44
a) Au cours du développement embryonnaire et à l’âge adulte ... 44
b) Durant l’inflammation ... 44
c) Une perturbation de la signalisation ENPP2/LPA dans les pathologies humaines ... 44
Les cancers ... 44
Les maladies cardiovasculaires ... 45
Obésité diabète ... 47
4.3. Les mécanismes moléculaires impliqués dans la régulation d’ENPP2 ... 47
a) La régulation transcriptionnelle d’ENPP2 ... 47
b) La régulation de l’activité enzymatique ... 48
CHAPITRE 1 : PROBLÉMATIQUE ET OBJECTIFS ... 49
OBJECTIF GÉNÉRAL ET APPROCHE EXPÉRIMENTALE : ... 49
Objectif 1 : ... 49
Objectif 2 : ... 49
Objectif 3 : ... 50
CHAPITRE 2: ENHANCER-MEDIATED ENRICHMENT OF INTERACTING JMJD3-DDX21 TO ENPP2 LOCUS PREVENTS R-LOOP FORMATION AND PROMOTES TRANSCRIPTION. ... 51
1. RÉSUMÉ ... 52
2. ABSTRACT ... 53
3. INTRODUCTION ... 54
4. MATERIALSANDMETHODS ... 55
4.1. Real-time polymerase chain reaction ... 55
4.2. Cell transfection with siRNA and plasmids ... 56
4.3. Luciferase reporter assay ... 56
4.4. Chromatin immunoprecipitation (ChIP) and detection of R-loops ... 57
4.5. ChIP-ReChIP ... 57
4.6. Co-immunoprecipitation ... 58
4.7. Western blot ... 58
4.8. Purification and identification of JMJD3-associated proteins ... 58
4.9. Capture nascent RNAs ... 59
4.10. Promoter capture Hi-C (PCHi-C) ... 59
4.11. CRISPR/Cas9 editing ... 60
4.12. Chromosome Conformation Capture (3C) ... 60
4.13. Statistical analysis ... 61
5. RESULTS ... 61
5.1. NF-κB response elements mediate the expression of ENPP2 ... 61
5.2. ENPP2 promoter has bivalent histone marks ... 62
5.3. JMJD3 and DDX21 are recruited to the promoter and gene body of ENPP2 ... 63
5.5. R-loops resolution at ENPP2 locus promotes the synthesis of nascent transcript ... 65
5.6. A distant-acting enhancer contacts the promoter region of ENPP2 and controls its expression 65 5.7. Enhancer-mediated enrichment of JMJD3-DDX21 at ENPP2 ... 67
6. DISCUSSION... 67 7. FIGURES ... 70 8. ACKNOWLEDGEMENT ... 78 9. FUNDING ... 78 10. SUPPLEMENTARYINFORMATIONS ... 78 11. REFERENCES ... 89 DISCUSSION ET CONCLUSION... 93
1. L’ORGANISATION DE LA TRANSCRIPTION DANS LE NOYAU ... 93
1.1. Des compartiments hiérarchisés et fonctionnels ... 93
1.2. Comment sont délivrés les régulateurs transcriptionnels au site de transcription? ... 94
2. LA RÉGULATION DU LOCUS ENPP2 ... 94
2.1. L’induction d’ENPP2 par une voie NF-κB /JMJD3 ... 94
a) Comment JMJD3 régule l’expression d’ENPP2 indépendamment de son activité enzymatique ? ... 95
b) Zoom sur l’interactome de JMJD3 ... 97
2.2. La répression transcriptionnelle et les R-loops ... 98
a) Comment les R-loops interfèrent avec le processus de transcription ? ... 98
b) Le rôle fonctionnel des R-loops est-il dépendant du contexte génomique ? ... 99
2.3. Un enhancer distal régule l’expression d’ENPP2 ... 99
a) Par quel processus les enhancers sont sélectionnés avant d’être activés? ... 100
b) L’interaction enhancer-promoteur est-elle déjà présente avant l’activation du gène ENPP2 ? ... 100
2.4. Implication clinique et perspective ... 101
3. IDENTIFICATION D’UNE SIGNATURE MOLÉCULAIRE À L’ÉCHELLE « CELLULE UNIQUE » ... 102
4. CONCLUSION... 103
L
ISTE DESF
IGURESIntroduction
Figure 1.1: Les éléments régulateurs composant le core promoter. ... 3
Figure 1.2: La formation des biomolecular condensates. ... 8
Figure 1.3: Techniques utilisées pour étudier les interactions enhancer-promoteur. ... 10
Figure 1.4: Le modèle de loop extrusion. ... 12
Figure 1.5: La Pol II est l'élément central dans la transcription... 14
Figure 1.6: Le code du CTD au cours de la transcription. ... 18
Figure 1.7: La structure des R-loops. ... 23
Figure 1.8: Les R-loops régulent la transcription en modulant l’architecture de la chromatine. ... 26
Figure 1.9: Les voies de signalisation des récepteurs TLRs. ... 31
Figure 1.10: Les membres de la famille NF-κB. ... 33
Figure 1.11: Les voies de signalisation NF-κB. ... 34
Figure 1.12: La sous-unité p65 est la cible de multiples modifications post-traductionnelles... 35
Figure 1.13: Modèle de l'enhanceosome au locus IFN-β... 37
Figure 1.14: L’organisation de la chromatine au niveau des PRGs et SRGs. ... 41
Figure 1.15: ENPP2, une lysophospholipase D. ... 43
Chapitre 2
Figure 2.1: NF-κB response elements mediate the expression of ENPP2. ... 70Figure 2.2: ENPP2 promoter has bivalent histone marks. ... 71
Figure 2.3: JMJD3 and DDX21 are recruited to the promoter and gene body of ENPP2. ... 72
Figure 2.4: The complex formed by JMJD3 and DDX21 is required for R-loops resolution at ENPP2 locus. ... 73
Figure 2.5: R-loops resolution at ENPP2 locus promotes the synthesis of nascent transcript. ... 74
Figure 2.6: A distant-acting enhancer contacts the promoter region of ENPP2 and controls its expression. ... 75
Figure 2.7: Enhancer-mediated enrichment of JMJD3–DDX21 at ENPP2. ... 76
Supplementary figure S1: Level of p65 overexpression in HEK 293T. ... 79
Supplementary figure S2: Efficiency of EZH2 siRNA in HEK 293T cells. ... 80
Supplementary figure S3: Efficiency of UTX siRNAs in HEK 293T cells. ... 81
Supplementary figure S4: Efficiency of JMJD3 siRNAs in HEK 293T cells. ... 82
Supplementary figure S5: Overexpression of MYC-JMJD3. ... 83
Supplementary figure S6: ChIP-qPCR assays showing crosstalk between UTX and JMJD3 ... 84
Supplementary figure S7: JMJD3 and DDX21 interaction. ... 85
Supplementary figure S8: DDX21 overexpression in HEK-293T cells... 86
Supplementary figure S9: CRISPR/Cas9-mediated enhancer deletion in HEK 293T cells ... 87
L
ISTE DES ABRÉVIATIONS3'UTR three prime untranslated transcribed region
AGO1/2 Argonaute 1/2
ANK motifs ankyrines
AP1 Activator protein 1
ARNm ARN messager
ASO oligonucléotide anti-sens
ATX autotaxine
BAFF-R BAFF receptor
BER réparation de l'ADN par excision de base BRD4 bromodomain-containing protein 4
BRE TFIIB recognition element
BREd TFIIB recognition element downstream BREu TFIIB recognition element upstream brUTP 5’-bromouridine 5’-triphosphate
C cytosine
CBP CREB-binding protein
CBX ChromoBox protein
cdk9 cyclin-dependant kinase 9
ChIP immunoprécipitation de la chromatine CREB cAMP-responsive-element-binding protein
CTD domaine carboxy-terminal
CUT transcriptions cryptiques instables DAMPs Danger-associated molecular pattern
DCE downstream core element
DIP DNA-RNA immunoprecipiation
Dnase I Déoxyribonucléase I
DPE Downstream Promoter Element
DRIVE-sequencing DNA-RNA in vitro enrichment DSIF DRB sensitivity inducing factor
ENPPs Ecto-Nucléotide Pyrophosphatase/Phosphodiesterase eQTL expression Quantitative Trait Loci
eRNA enhancer RNA
GRO-seq Global-Run-On-Sequencing
H2AK119ub1 ubiquitination de la lysine 119 de l’histone H2A H3K27Ac acétylation de la lysine 27 de l’histone H3 H3K27me3 tri-méthylation de la lysine 27 de l'histone H3 H3K4me1 mono-méthylation de la lysine 4 de l’histone H3 H3K4me3 tri-méthylation de la lysine 4 de l'histone H3 H3K9Ac et H3K14Ac acétylation des lysines 9 et 14 de l’histone H3
H3R3me2 di-méthylation de l’arginine de l’histone 3
HDAC histone déacétylase
HEV high endothélial venule
HEXIM1 hexamethylene bisacetamide inducible 1
HPH Human Polyhomeotic Homolog
IDR domaine protéique intrinsèquement désordonnés
IFN β interféron β
IKK Inhibitor of kappa B Kinase
IL-8 interleukine-8
Inr élément initiateur
IRF IFN-regulatory factors
IκB Inhibitor kappa B
JMJD3 Jumonji Domain-Containing Protein 3
kb kilo bases
KRAP1 KRAB-interacting protein 1
LDTFs lineage-determining transcription factors
LPA acide lysophosphatidique
LPC lysophosphatidylcholine
LPP lipides phosphate phosphohydrolases
LPS Lipopolysaccharide
LTβR lymphotoxin β receptor
MG monoacylglycérol
Myd88 myeloid differentiation factor 88
NELF Negative Elongation Factor
NET-seq native elongation transcript sequencing
NF1 nuclear factor 1
NF-κB Nuclear-Factor κB
NLS signal de localisation nucléaire
nt nucléotide
PAMPs Pathogen-associated molecular pattern
pb paire de base
PCGF PolyComb Group Factor
PIC Complexe de pré-initiation de la transcription
Pol II ARN Polymérase II
poly(A) polyadnénylation
PRC2 Polycomb Repressive Complex 2
PRDs positive regulatory domains
PRGs primary response genes
PRR Pattern Recognition Receptor
RHD Rel Homology Domain
RNP ribonucléoprotéine
SDTFs signal-dependant transcription factor
SEC super elongation complex
Sen1 l’ADN/ARN hélicase Senataxin
Ser sérine
Shh sonic hedgehog
shRNAs short hairpin RNAs
siRNA ARN inhérents
SMC structural maintenance of chromosomes
SMD domaine somatomedin
SRGs secondary response genes
SRPK2 protéine kinase spécifique des sérine/arginine STARR-seq self-transcribing active regulatory region sequencing
SV40 virus simien 40
TAD domaine d'associations topologiques TAD1 et TAD2 domaine de transactivation 1 et 2
TAFs TBP-associated factors
TBP TATA-binding protein
Thr thréonine
TLRs Toll-like receptors
TOP1 topoisomérase I
TOP3B topoisomérase IIIB
TPR répétition tétratricopeptide TSS Site d'initiation de la transcription
Tyr tyrosine
U uracile
UTX Ubiquitously transcribed tetratricopeptide repeat X chromosome VICs cellules interstitielles de valves
R
EMERCIEMENTSCe doctorat aura été une expérience unique tant sur le plan scientifique qu’humain. Aussi, je souhaite remercier comme il se doit, chacune de ces personnes, sans qui cette thèse n’aurait jamais abouti.
En premier lieu, je tiens à remercier mon directeur de thèse, Dr Patrick Mathieu pour la confiance qu’il m’a accordée dans la gestion quotidienne de mon projet de recherche. Sa disponibilité et son accompagnement tout au long de mon doctorat m’ont permis de pousser un peu plus loin mon raisonnement. Je remercie également le Dre Marie-Chloé Boulanger pour les nombreuses discussions sur les détails techniques des expériences, et pour ses suggestions toujours avisées. Votre aide fut précieuse.
J’adresse également mes remerciements à tous les étudiants de l’équipe du Dr Patrick Mathieu qui ont contribué à la bonne ambiance générale. Plus que des collègues, Ghada, Romain et Nicolas m’ont apporté un soutien inestimable et ont rendu mes journées de travail plus agréables. Je vous souhaite beaucoup de réussite dans vos projets futurs. Je n’oublie pas, bien sûr, les étudiants exilés du 4e
étage pour leur assistance aussi bien matérielle que morale.
Un grand merci également à mes parents, qui malgré la distance m’ont toujours soutenu. Merci également à mes frères, Quentin et Tonin à qui je souhaite de s’épanouir pleinement dans leurs projets futurs. Enfin, mais non des moindres, tous mes remerciements à Félix, pour sa joie de vivre et son soutien sans faille au quotidien. Merci pour les relectures de mon manuscrit et merci de supporter les bêtises de Mme Apache.
A
VANT-P
ROPOSLe présent document résume mes travaux de recherche, réalisés durant mes études doctorales, sous la supervision du Dr Patrick Mathieu, au sein de l’Institut Universitaire de Cardiologie et de Pneumologie de Québec. Cette thèse avait pour objectif de mettre en lumière les mécanismes moléculaires participant à la régulation transcriptionnelle d’ENPP2. De nombreuses évidences rapportent que le gène ENPP2, qui code pour une enzyme lysophospholipase D, est dérégulé dans le cancer, les pathologies immunes et les maladies cardiovasculaires. Toutefois, les mécanismes sous-jacents étaient jusque-là que très peu décrits.
Ce manuscrit débute par une revue de la littérature afin de remettre en contexte les objectifs de mon doctorat, énoncés dans le chapitre 1. Les hypothèses de travail sont également spécifiées dans le chapitre 1. S’en suit le chapitre 2 qui comprend mes travaux de recherche ayant conduit à une publication, intitulée : « Enhancer-mediated enrichment of
interacting JMJD3-DDX21 to ENPP2 locus prevents R-loop formation and promotes transcription » dans le journal Nucleic Acids Research, en juin 2019. Dans cette étude
fondamentale dont je suis l’auteure principale, nous avons montré qu’ENPP2 est induit en réponse à une stimulation inflammatoire de nature Lipopolysaccharide (LPS) dans les cellules HEK293T via une signalisation NF-κB/JMJD3. De manière intéressante, JMJD3 contrôle le processus de transcription d’ENPP2 indépendamment de son activité enzymatique en recrutant une ARN hélicase, DDX21, nécessaire à la résolution des R-loops dans la région promotrice d’ENPP2 et dans le corps du gène. Enfin, une conclusion et une discussion des points soulevés dans les parties précédentes clôtureront ce manuscrit.
Marie-Chloé Boulanger, Arnaud Chignon, Ghada Mkannez et le Dr Patrick Mathieu ont grandement participé à l’avancement de ce projet de recherche. Tous les co-auteurs ont lu et approuvé la version finale du manuscrit. Pour ma part, j’ai réalisé l’ensemble des expériences, réalisé l’analyse statistique des données et rédigé le manuscrit avant de le soumettre. Marie-Chloé Boulanger a aidé au design d’expériences, en particulier les clonages et la délétion de l’enhancer par la technique de CRISPR-Cas9. Arnaud Chignon a mis au point et réalisé l’expérience de Chromosome Conformation Capture (3C) sur les cellules HEK293T. Ghada Mkannez a contribué à ce projet en réalisant des mesures d’expression d’ENPP2 par q-PCR. Chacune de ces étapes a été supervisée et guidée par le Dr Patrick Mathieu.
INTRODUCTION
Le corps humain est un environnement hétérogène, constitué d’environ 1018
cellules. Pour assurer leurs fonctions, chacune de ces cellules instaure un programme transcriptionnel qui lui est propre et qui définit son identité cellulaire. Lorsque des changements environnementaux s’opèrent, les cellules réagissent différemment et mettent en place une réponse transcriptionnelle transitoire, nécessaire au rétablissement de l’état d’équilibre. Une telle adaptation repose sur des systèmes intégrés capables de détecter ces variations ponctuelles et de stimuler les voies de signalisations adéquates. La transduction de signal permet ensuite le recrutement de la machinerie transcriptionnelle au niveau d’éléments régulateurs, localisés au cœur de l’ADN. À ce moment précis débute le processus de transcription qui permet la synthèse du transcrit naissant. Plusieurs évidences suggèrent qu’au cours de ce processus biologique complexe, les cellules mettent en place divers mécanismes de régulation pour s’assurer du bon déroulement de la transcription. À titre d’exemple, des ARN hélicases et des facteurs d’élongation positifs sont recrutés pour limiter les évènements de pauses transcriptionnelles. Les topoisomérases, quant à elles, réduisent les contraintes imposées à l’ADN au fur et à mesure que la Pol II avance, prévenant ainsi l’apparition de cassures d’ADN. Enfin, le dépôt de marques d’histones H3K36me3 dans le corps du gène inhibe la transcription cryptique.
Bien souvent, en situation pathologique, l’altération de ces mécanismes moléculaires va conduire à l’expression aberrante du programme transcriptionnel normal. Ainsi, une meilleure compréhension du processus de transcription et des mécanismes sous-jacents pourrait aboutir au développement de nouvelles avenues thérapeutiques. Dans ce cadre, l’objectif général de cette thèse est d’étudier la régulation de la transcription au locus ENPP2.
ENPP2 est un gène impliqué dans le cancer, les pathologies immunes et les maladies
cardiovasculaires. Si sa surexpression est clairement établie en situation pathologique, les mécanismes moléculaires sous-jacents restent à préciser.
Le présent document résume mes travaux de recherche, réalisés durant mes études doctorales, sous la supervision du Dr Patrick Mathieu, au sein de l’Institut Universitaire de Cardiologie et de Pneumologie de Québec. Dans l’introduction, nous exposerons en détail les principaux mécanismes intervenant dans le processus de transcription avant d’évoquer son lien avec l’organisation du génome. S’en suivra une énumération des différents objectifs de notre projet de recherche ainsi que les hypothèses de travail dans un chapitre 1. Les résultats
obtenus durant ces quatre années de doctorat sont relatés dans le chapitre 2. Enfin, en guise de conclusion, la dernière partie clôturera ce manuscrit et reprendra les points clés.
1.
Des séquences régulatrices au cœur de l’ADN
La transcription repose sur une interaction dynamique entre des éléments régulateurs agissants en cis et des facteurs protéiques agissant en trans. Les éléments cis-régulateurs sont définis comme des séquences d’ADN non codantes, auxquelles s’associe la machinerie transcriptionnelle. Parmi ces éléments régulateurs, on distingue les promoteurs et les éléments distaux de nature enhancers. À l’inverse, les facteurs en trans regroupent les facteurs de transcription, les complexes de remodelage de la chromatine, les facteurs généraux de la transcription incluant la Pol II [1].
1.1. Les promoteurs
a) Les principales caractéristiques des promoteurs
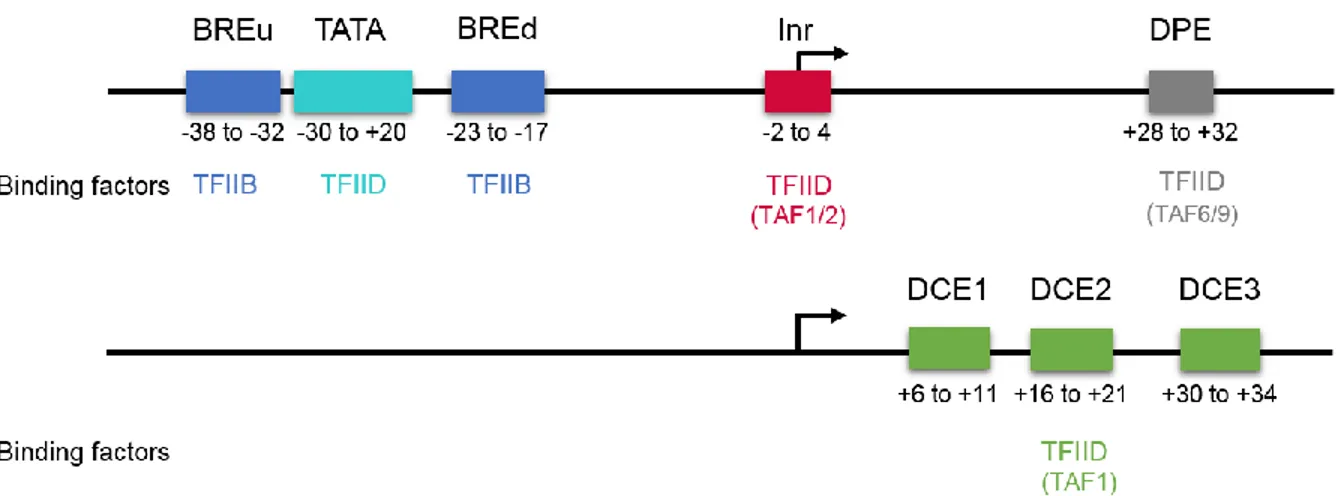
Les promoteurs sont des séquences régulatrices constituées de deux grandes régions : le « core promoter » en anglais et le promoteur proximal. Le core promoter est un segment d’ADN de 50-100 paires de bases (pb) autour du TSS, et constitue une plateforme pour l’assemblage du complexe de pré-initiation (PIC) [2]. Cet assemblage définit notamment la position du site d’initiation de la transcription (TSS), ainsi que la direction de la transcription en plus de contrôler la transcription basale des gènes. Des analyses structurelles à l’échelle du génome ont identifié une série d’éléments associés au core promoter (Figure 1.1). Parmi ces éléments, la boîte TATA et l’élément Initiator (Inr) sont les plus caractérisés. La boîte TATA est localisée à la position -30 à -20 pb en amont du TSS et sert de site d’ancrage pour le facteur de transcription général TFIID. [3]. Le complexe TFIID est composé de la TATA-binding protein (TBP) et des protéines TBP-associated factors (TAFs) [4, 5]. Chez les eucaryotes, 24% des promoteurs possèdent une boîte TATA [6]. Généralement, les promoteurs eucaryotes dépourvus de boîte TATA contiennent un élément Inr [7, 8]. L’élément Inr est une séquence d’ADN riche en pyrimidine, chevauchant le TSS et également impliqué dans l’assemblage du PIC, bien que la nature précise de l’interaction reste peu décrite.
D’autres éléments peuvent être associés au core promoter et moduler l’activité de la boîte TATA et de l’élément Inr. Parmi eux, l’élément DPE (Dowstream Promoter Element) est majoritairement retrouvé dans les promoteurs sans boîte TATA [8]. Le DPE va agir comme un site de recrutement pour les composants de TFIID en combinaison avec l’élément Inr.
Également en aval du TSS, le motif DCE (downstream core element) est défini par une séquence reconnut par les composants du complexe TFIID (TAF6/9). À l’inverse de ces motifs, l’élément BRE (TFIIB recognition element), comme son nom l’indique, permet le recrutement de TFIIB [9, 10]. Il s’agit d’une séquence de 7pb localisée de part et d’autre de la boîte TATA. On parle du motif BRE upstream (BREu) en positon 38 à 32 et BRE downstream (BREd) en -23 à -17. De manière intéressante le BRE peut activer la transcription ou au contraire agir comme un répresseur [8].
Figure 1.1: Les éléments régulateurs composant le core promoter. Les promoteurs des gènes humains sont constitués d’une combinaison variable d’éléments régulateurs. Le DCE est représenté séparément uniquement pour faciliter son illustration. BREu: upstream TFIIB recognition element; TATA: TATA box; BREd: downstream TFIIB recognition element; Inr: élément Initiator; DPE: downstream promoter element; DCE: downstream core element.
Les propriétés structurales des promoteurs humains ne sont pas sans lien avec leurs activités transcriptionnelles. Bien que la boîte TATA et l’élément Inr peuvent être retrouvés seuls ou en combinaison, ils semblent contrôler des familles de gènes distinctes. Les promoteurs minimaux des gènes tissus spécifiques sont caractérisés par la présence d’une boîte TATA qui initie la transcription au niveau d’un nucléotide précis et sont référencés comme des « sharp or focused promoter ». À l’inverse, les promoteurs des gènes, dont l’expression est ubiquitaire possèdent un élément Inr associé à de multiple TSS, dispersés sur des régions allant jusqu’à 150pb [11, 12]. On parle alors de « broad promoter ». Initialement, la proportion de GC et la fréquence des dinucléotides CpG étaient utilisées pour distinguer les promoteurs des gènes tissu-spécifiques de ceux des gènes ubiquitaires. Les premiers étant pauvres en GC et les seconds riches en GC et caractérisés par la présence d’ilots CpG. Une
troisième catégorie de promoteur, également enrichit en îlots CpG et positifs pour la tri-méthylation de la lysine 27 de l’histone H3 (H3K27me3) a été proposée [13]. Ces promoteurs contrôleraient des gènes impliqués dans le développement ou la différenciation cellulaire.
Le promoteur proximal, quant à lui, est défini comme étant la région localisée immédiatement en amont du promoteur cœur. Cette portion du promoteur contient de multiples sites de fixation pour des facteurs de transcription tels que la boîte CAAT et la boîte GC [8, 14]. La boite CAAT permet le recrutement des facteurs de transcription NF1 (nuclear factor 1) ou CBP (CREB-binding protein), tandis que la boîte GC facilite la liaison du facteur de transcription ubiquitaire sp1.
b) État de la chromatine au niveau des promoteurs
L’état de la chromatine au niveau des éléments régulateurs joue un rôle primordial dans le processus de transcription. En agissant comme une barrière physique, les nucléosomes limitent l’accessibilité de l’ADN aux facteurs de transcriptions et influencent le recrutement de la machinerie transcriptionnelle. De ce fait, les modifications post-traductionnelles des histones peuvent aussi renseigner quant à l’activité des promoteurs. Les promoteurs transcriptionnellement actifs sont enrichis en tri-méthylation de la lysine 4 de l’histone H3 (H3K4me3) autour du TSS, tandis que les gènes réprimés sont caractérisés par la présence de la marque H3K27me3 [15]. Les promoteurs bivalents sont définis par la présence de ces deux marques activatrices et répressives.
1.2. Les enhancers
a) La découverte des enhancers
Au début des années 1980, un autre type d’éléments régulateurs distaux contrôlant l’expression des gènes en coopérant avec le promoteur ont été caractérisés [16–18]. Il s’agit des enhancers. Les enhancers ont d’abord été décrits comme des séquences d’ADN non codantes capables d’activer la transcription d’un locus dans des essais gènes rapporteurs [19]. Dans ces expériences, les auteurs montrent qu’une région de 72 pb du génome du virus simien 40 (SV40) augmente la transcription d’un gène humain, celui de la β-globine [19].
b) Les caractéristiques fonctionnelles des enhancers
L’identification des enhancers constitue un défi important en biologie moléculaire puisqu’aucune séquence consensus ne permet de les localiser dans le génome et leur action sur la transcription se fait indépendamment de leur distance et de leur orientation. Néanmoins, l’activité des enhancers corrèle avec certaines propriétés de la chromatine.
Initialement, la conservation intra-espèces de séquences non-codantes a été utilisée pour prédire l’activité enhancer [20, 21]. Par la suite, les enhancers sont caractérisés par une faible densité en nucléosomes, qui se traduit par une hypersensibilité de l’ADN à la digestion par des nucléases à ADN, tel que la Déoxyribonucléase I (Dnase I) [22]. Les nucléosomes localisés de part et d’autre des régions enhancers sont enrichis en marques d’histones comme la mono-méthylation de la lysine 4 de l’histone H3 (H3K4me1) [23, 24] et l’acétylation de la lysine 27 de l’histone H3 (H3K27Ac) [25]. Plusieurs évidences suggèrent que la marque H3K4me1 pourrait faciliter le recrutement du complexe cohésine, impliquée dans les interactions enhancers-promoteurs [26, 27]. Cette marque H3K4me1 est également reconnue par des protéines lectrices possédant des chromodomaines tels que CHD1 [28, 29], CHD7 [30], ou TIP60 [31]. De manière intéressante, les protéines CHD1 et CHD7 jouent un rôle important dans le remodelage de la chromatine, tandis que le complexe TIP60/p400 catalyse le dépôt de variants d’histones comme H2A.Z et H3.3 [32, 33]. L’incorporation de ces variants d’histones contribue à la plasticité des nucléosomes, en les rendant hyper mobiles [32, 33].
Les enhancers contiennent également de nombreux motifs ADN qui agissent comme des sites de fixation pour des facteurs de transcription. Une fois fixés aux enhancers, ces facteurs de transcription recrutent des co-activateurs transcriptionnels comme l’histone acétyle transférase p300 [34] ou le complexe Mediator [35]. La combinaison de ces régulateurs transcriptionnels détermine l’activité des enhancers [18] et ultimement contribue à l’assemblage de la machinerie de transcription [36].
En 2010, la découverte des ARNs associés aux enhancers (eRNAs) apporte un vent de renouveau dans la biologie des enhancers en offrant une nouvelle stratégie permettant de localiser et de prédire les enhancers actifs. Des études à l’échelle du génome mettent en évidence le recrutement de la Pol II au niveau d’enhancers actifs et son implication dans la transcription d’une nouvelle classe d’ARN non codants, les eRNAs [37, 38]. La grande majorité des eRNAs est définie comme étant courts (avec une médiane de 346 nucléotide (nt)), non-polyadenylée, non-épissée et faiblement exprimée [39]. Le rôle fonctionnel de ces eRNA est partiellement compris. Toutefois, de nombreuses études supportent le fait que ces eRNAs sont transcrits à partir d’enhancers actifs et que leur expression est étroitement corrélée avec la
transcription des gènes à proximité [37, 38, 40]. En effet, l’utilisation de short hairpin RNAs (shRNAs) [41], d’ARN interférents (siRNA) [42] ou encore d’oligonucléotide anti-sens (ASO) [43] confirme qu’un knockdown de ces eRNAs est associé à une réduction du niveau d’expression de gènes à proximité, suggérant ainsi que les eRNAs sont importants pour la fonction enhancer. Les mécanismes moléculaires sous-jacents sont divers et semblent dépendants du contexte génomique. Certaines évidences supportent un rôle des enhancers dans l’établissement et/ou le maintien des boucles chromatiniennes enhancer-promoteur [38, 44, 45], tandis que d’autres suggèrent que les eRNA agissent en favorisant le recrutement de facteurs de transcription aux enhancers [46, 47], ou même en induisant la relâche de la pause transcriptionnelle [41, 48]. Le phénomène de pause transcriptionnelle est détaillé dans l’introduction; partie 2.4.
Si l’utilisation de cette signature épigénétique, qui semble propre aux enhancers, a permis d’estimer leur nombre dans le génome humain à 1 million [49], elle ne garantit pas leur fonctionnement en tant que tel [50]. En effet, bien que les marques d’histones activatrices et la présence d’eRNAs soient associées avec l’activité des enhancers, leur détection ne permet pas de mesurer l’activité transcriptionnelle des enhancers [18, 51]. Notons également que cette signature épigénétique n’est pas exclusive aux enhancers actifs et peut conduire à la détection de faux positifs [18]. Des essais gènes rapporteurs sont alors nécessaires pour s’assurer de l’activité intrinsèque de ces séquences ADN. En parallèle, le système d’édition du génome CRISPR-Cas9, est utilisé pour moduler l’activité de ces potentiels enhancers et étudier leur effet sur un locus d’intérêt [50].
Aussi, plus récemment le développement d’essais fonctionnels, comme le
self-transcribing active regulatory region sequencing (STARR-seq) permet d’identifier et de
quantifier l’activité de plusieurs milliers de régions enhancers en parallèle, en interrogeant l’intégralité du génome humain [52]. Brièvement, des fragments d’ADN aléatoires sont insérés en aval d’un promoteur minimal, dans le three prime untranslated transcribed region (3’UTR) d’un gène rapporteur (tel que GFP) [53]. Chaque construction plasmidique contient en général une séquence unique appelée code-barre qui peut être transcrite et qui permet d’associer un transcrit à un candidat enhancer. La librairie d’ADN est ensuite transfectée dans une population cellulaire, et un RNA-sequencing (RNA-seq) et DNA-sequencing (DNA-seq) sont ensuite réalisés. Le rapport ARN/ADN est utilisé pour estimer l’abondance des transcrits du gène et en déduire l'activité de la séquence enhancer correspondante [53]. Si le développement de ces approches expérimentales renseigne sur l’activité intrinsèque d’une séquence ADN, elles ne permettent pas d’analyser l’activité des enhancers dans leur contexte génomique natif. Par la suite, des criblages génétiques basés sur le système d’édition du
génome CRISPR-Cas9 offrent l’opportunité d’identifier des enhancers dans leur contexte génomique d’origine et d’associer fonctionnellement les enhancers à leurs gènes cibles [54].
Les super-enhancers, une sous population d’enhancers:
Grâce à l’avènement du séquençage au débit, l’identification et la prédiction d’enhancers ont connu un réel engouement au cours de la dernière décennie. En 2013, une sous-population d’enhancers est identifiée [55, 56]. Il s’agit des super-enhancers, définis comme un regroupement d’enhancers sur une distance de 12.5 kilo bases (kb), qui pourraient à eux seuls déterminer l’identité cellulaire [56]. Correspondant à environ 3% des enhancers totaux, ces super-enhancers ont été définis pour la première fois dans les cellules souches embryonnaires de souris. Ils présentent une forte densité en facteurs de transcription importants pour l’identité cellulaire (Sox2, Nanog, Oct4 pour la pluripotence des cellules souches) et un fort enrichissement pour le complexe Mediator [56, 57]. La densité en facteur de transcription ou co-activateur au niveau des super-enhancer est approximativement 10 fois supérieure à celle d’un enhancer canonique [56, 57].
La formation des biomolecular condensates
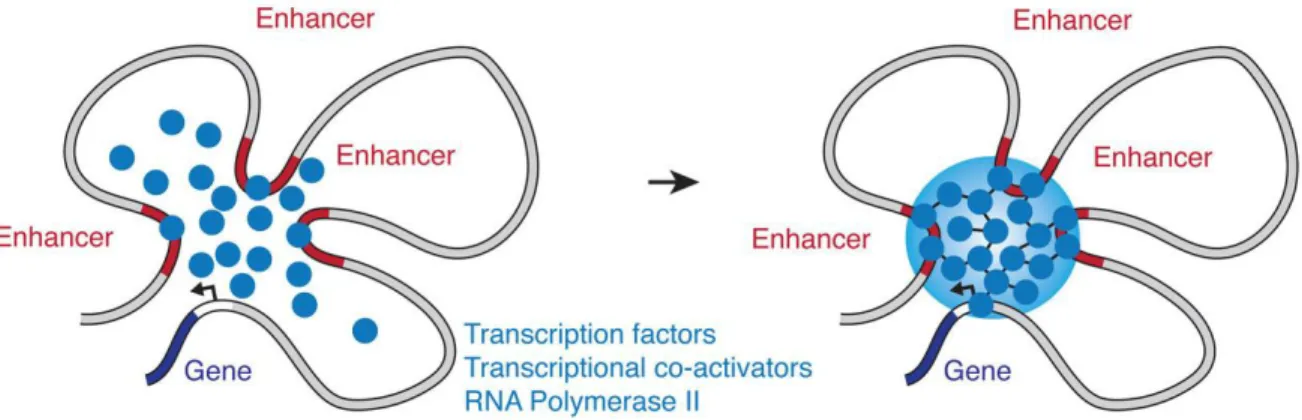
Une large fraction des régulateurs transcriptionnels localisés au niveau des enhancers et super-enhancers sont sujets à des modifications post-traductionnelles. Une fois modifiées, ces molécules multivalentes obéissent au principe physico-chimique de séparation de phase liquide-liquide et sont responsables de la formation de clusters dynamiques appelés
biomolecular condensates (Figure 1.2) [58].
Au sein de ces structures qui se manifestent comme des gouttelettes liquides, des interactions ADN-facteur de transcription, ainsi que les interactions faibles entre protéines présentant des régions intrinsèquement désordonnées se mettent en place [59–61]. D’un point de vue fonctionnel, ces compartiments contribuent à l’assemblage d’enhancer [62] ou de super-enhancer actifs [60] et pourraient participer à la communication enhancer-promoteur [63].
Figure 1.2: La formation des biomolecular condensates. D’après https://www.hniszlab.org/. La forte densité de facteurs de transcription, co-activateurs et de Pol II favorise la formation de
biomolecular condensates. Ces structures jouent un rôle important dans l’activité des
enhancers.
c) La communication enhancer-promoteur
Les enhancers agissent sur la transcription des gènes indépendamment de leur distance. En effet, ces éléments cis-régulateurs fonctionnent lorsqu’ils sont situés à proximité ou au contraire à très grande distance (entre des centaines et des milliers de kb) de leurs gènes cibles [64]. S’il est largement accepté que le fonctionnement des enhancers implique une réorganisation de la chromatine, les mécanismes de communications enhancer-promoteur sont encore que partiellement compris.
Plusieurs modèles non exclusifs ont été proposés au cours des dernières années. Le
linking model, implique que le recrutement de complexes multi-protéiques au niveau des
enhancers induit une modification de la structure chromatinienne, qui se propage ensuite le long de l'ADN jusqu’à à ce qu’un promoteur compétent en matière de transcription soit finalement rencontré et inclus dans cette configuration « ouverte » [65]. En parallèle, le recrutement en cascade des facteurs de transcription se poursuit et ultimement conduit à l’enrôlement de la machinerie transcriptionnelle [66, 67]. Ce modèle est proposé lorsque les enhancers sont localisés à proximité de leurs gènes cibles.
Le tracking model est similaire au linking model. Ici, des activateurs transcriptionnels, incluant la Pol II sont recrutés au niveau des enhancers et diffusent le long de la fibre de chromatine (de manière unidirectionnelle) vers un promoteur cible, parfois sans même quitter la séquence enhancer [67–69]. Il en résulte la formation progressive d’une boucle chromatinienne permettant un rapprochement physique de l’enhancer et du promoteur cible.
De manière intéressante, lorsque le gène est induit, des transcrits unidirectionnels sont détectés au niveau des séquences génomiques localisées entre l’enhancer et le promoteur, ce qui pourrait refléter le tracking de la Pol II [39, 70].
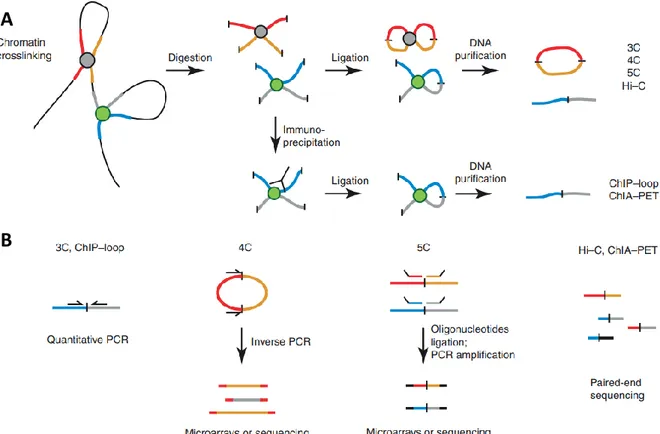
Le modèle le plus populaire aujourd’hui est sans aucun doute le looping model, qui propose que les enhancers activent la transcription d’un ou plusieurs loci en interagissant avec le promoteur de leurs gènes cibles via la formation de boucle chromatinienne [71]. Le développement des expériences de Chromosome Conformation Capture (3C) et des techniques dérivées (4C, 5C, HI-C, ChIA-PET) a permis de mettre en lumière le rôle fonctionnel de ces interactions physiques enhancer-promoteur au cours de la transcription (Figure 1.3). Ainsi, en augmentant la concentration locale de régulateurs transcriptionnels au niveau des promoteurs, les enhancers via la formation de boucles chromatiniennes peuvent stimuler l’initiation de la transcription [72], ou même favoriser la transition entre l’étape d’initiation et l’élongation [41, 73, 74]. Notons également que la mobilisation des enhancers dans des transcription hubs, tels que des biomolecular condensates est proposée comme un mécanisme pouvant influencer l’activité de ces éléments régulateurs [75]. Au sein de ces structures, la promiscuité enhancer-promoteur est exacerbée par l’agrégation du complexe Médiator, de la Pol II qui agissent comme des ponts moléculaires permettant la communication enhancer-promoteur [75].
La formation des boucles enhancer-promoteur est affectée par l’organisation 3D du génome L’organisation 3D du génome favorise les interactions enhancer-promoteur en sculptant des compartiments hiérarchiques. Ces boucles chromatiniennes, dessinées entre enhancer-promoteur, s’intègrent dans des domaines topologiques plus larges, appelés
topologically associated domains (TAD). Ces TADs de l’ordre de 1-5Mb fonctionnent comme
des micro-environnements, favorisant les interactions au sein d’un même TAD, tout en limitant celles avec le reste du génome. L’organisation structurale de ces domaines repose sur des frontières, enrichies en protéines insulatrices telles que CTCF ou architecturale comme le complexe cohésine [77, 78]. Ainsi, le confinement du génome sous forme de TAD influence la transcription à plusieurs niveaux [79]. 1) En réduisant la distance entre les éléments régulateurs [80]. 2) En limitant la propagation de l’euchromatine ou de l’hétérochromatine à un TAD voisin [81]. 3) En isolant un promoteur de l’action d’un enhancer localisé dans un TAD voisin [82, 83]. La perte des frontières entre TADs conduit à des changements transcriptionnels et a des interactions enhancer-promoteur inappropriées [77, 78].
Figure 1.3: Techniques utilisées pour étudier les interactions enhancer-promoteur. D’après [76] (A) Approche expérimentale du 3C. Les interactions ADN-protéines sont figées par une étape de crosslink, et la chromatine est fragmentée par digestion enzymatique. Par la suite, une étape de ligation est réalisée et l’abondance des produits de ligation est détectée par PCR. Plusieurs techniques dérivent du 3C dont le ChIA-PET, le 4C et 5C et le HiC. (B) Différentes approches de biologie moléculaire sont utilisées pour détecter des interactions entre 2 locus (3C), entre un locus d’intérêt et le reste du génome (4C), et pour cartographier des interactions multiples entre librairies (5C) ou à l’échelledu génome (HI-C).
Plusieurs protéines structurales participent à la formation des boucles enhancer-promoteur Récemment, le modèle de loop extrusion propose que la cohésine et CTCF organisent la structure du génome au sein des TADs et façonnent les boucles chromatiniennes [84]. Le complexe cohésine, constitué de 4 sous unités : deux sous unités SMC (structural
maintenance of chromosomes), SMC1 et SMC3, une sous unité kléisine RAD21, et l’antigène
stromal (SA) forme une structure en anneau autour de l’ADN. Une fois recruté, le complexe cohésine agirait comme un moteur d’extrusion en glissant le long de la chromatine jusqu’à la rencontre des sites CTCF (Figure 1.4A). L’orientation convergente des sites CTCF semble être un critère déterminant dans la formation des boucles chromatiniennes [85, 86]. En effet,
A
l’inversion de motifs CTCF à un locus précis, par la technique CRISPR-Cas9, déstabilise les interactions à longues distances et conduit à des changements transcriptionnels [87, 88]. Selon le modèle de loop extrusion, l’extension des boucles est finement contrôlée par le facteur de libération WAPL qui prévient la formation de boucles entre des sites CTCF mal orientés et assure le déchargement du complexe cohésine sur l’ADN [89, 90].
En dépit des connaissances actuelles, il reste à préciser si les boucles chromatiniennes qui se forment entre deux motifs CTCF et celles qui se forment entre enhancer-promoteur résultent de mécanismes divergents [91]. La coexistence de deux complexes cohésine avec une composition en sous-unités distinctes pourrait expliquer une certaine spécificité dans la formation de ces boucles, par le modèle d’extrusion. Par exemple, le complexe cohésine contenant SA1 contrôlerait l’organisation globale des TADs, tandis que les contacts enhancer-promoteur impliquent presque exclusivement les complexes cohésine contenant SA2 [91]. Le recrutement différentiel de ces complexes cohésine est encore mal compris. Une implication du facteur de charge NIBPL, enrichi au niveau des enhancers-promoteurs actifs, a été proposée [72]. Dans ce contexte, la cohésine n’agirait pas comme une protéine stabilisatrice des boucles, mais plus comme un acteur permettant à un enhancer de scanner l’ADN et de faciliter sa rencontre avec un promoteur au détriment d’un autre, via le mécanisme de loop extrusion [84] (Figure 1.4B).
La rétention de la cohésine au niveau de sites dépourvus de CTCF, en particulier des enhancers, pourrait également expliquer les accélérations transcriptionnelles ou phénomènes de burst transcriptional. La transcription est un phénomène épisodique, constituée de courte période à haute intensité, où la transcription se fait en « rafale ». Chacune de ces périodes est entrecoupée par des phases durant lesquelles le promoteur est réfractaire à la transcription. De cette façon, l’activité des promoteurs oscille entre un état « on/off » afin d’adapter la disponibilité d’une protéine aux besoins physiologiques des cellules. Lorsque le promoteur est en position « on », « une rafale » (ou burst) de transcription a lieu [93–95]. Plusieurs évidences suggèrent que les enhancers vont jouer un rôle déterminant dans le niveau d’expression d’un ARN messager (ARNm) d’intérêt en modulant l’amplitude et la fréquence de ces accélérations [96]. Outre CTCF, et la cohésine, d’autres protéines jouent un rôle dans la formation de ces boucles chromatiniennes. Par exemple le facteur de transcription ubiquitaire yin and yang 1 (YY1) interagit avec CTCF et stabiliserait la formation de ces boucles [97]. La perte de YY1 perturbe la fonction des enhancers ainsi que l’expression des gènes [97, 98]. Le complexe Médiator, constitué de 30 sous unités et impliqué dans l’assemblage du PIC, participe également à la communication enhancer-promoteur en interagissant avec la cohésine [72, 99,
Figure 1.4: Le modèle de loop extrusion. D’après [92]. (A) Le complexe cohésine est chargée sur la chromatine par le facteur de recrutement NIPBL. Une fois fixé, le complexe cohésine glisse tel un moteur d’extrusion jusqu’à rencontrer les sites CTCF, qui agissent comme une barrière physique et bloque la progression de la cohésine. (B) Les boucles chromatiniennes formées entre des sites CTCF forment des domaines permettant à un enhancer de scanner l’ADN et de faciliter sa rencontre avec un promoteur via le mécanisme de
2. Le processus de transcription
2.1. Composition et structure de la Pol II
La Pol II est l’élément central de la transcription. Cette enzyme est un assemblage de 12 sous unités d’un poids moléculaire de 550kDa qui lui confère sa capacité à fixer l’ADN et l’ARN, mais aussi à incorporer les nucléotides triphosphates dans la chaîne d’ARN naissante. Les sous-unités protéiques sont nommées Rbp1 à 12, en fonction de leur poids moléculaire et peuvent être classées en trois catégories : 1) les 3 plus grosses sous-unités : Rbp1, Rbp2, et Rbp3 forment le cœur de l’enzyme et sont très conservées au cours de l’évolution ; 2) les sous-unités spécifiques de la Pol II (Rbp4, 7,9 et 11) ; et 3) les sous-unités présentes dans les 3 Polymérases (Rbp5, 6,8, 10alpha, 10beta).
La plus grosse sous-unité, Rbp1, possède à son extrémité C-terminale un domaine carboxy-terminal (CTD) remarquable. Ce domaine est constitué de la répétition d’un même motif Tyrosine1(Tyr1)-Sérine2(Ser2)-Proline3(Pro3)-Thréonine(Thr4)-Ser5-Pro6-Ser7 (Figure 1.5). Si la séquence peptidique est extrêmement conservée au cours de l'évolution, le nombre de répétitions varie d’une espèce à l’autre. On retrouve 26 répétitions chez la levure S.cerevisiae contre 52 chez les mammifères [101, 102]. L’importance du motif réside dans le fait que parmi les 7 acides aminés, 5 peuvent faire l’objet d’une phosphorylation [103]. Bien que le CTD joue un rôle important dans la progression du cycle de la transcription et dans le couplage de la transcription à d’autres processus co-transcriptionnels, il n’est pas indispensable à la fonction catalytique de la Pol II [104].
2.2. Le cycle de transcription
Chez les eucaryotes, la transcription des gènes par la Pol II s’articule autour de trois étapes qui font partie intégrante du cycle de transcription. L’initiation concerne la synthèse des premiers nucléotides et précède la phase d’élongation qui implique l’allongement de la chaîne d’ARN. Enfin, la terminaison regroupe les processus de maturation du transcrit naissant, incluant le clivage endoribonucléolytique et la polyadénylation de l’extrémité 3’. Chacune de ces étapes sont hautement régulée par différents états de phosphorylation du domaine CTD de la Pol II [105].
Figure 1.5: La Pol II est l'élément central dans la transcription. D’après [106]. La Pol II est un assemblage de 12 sous unités d’un poids moléculaire de 550kDa. La plus grosse sous-unité possède à son extrémité C-terminale un domaine carboxy-terminal (CTD) constitué de la répétition d’un même motif qui comprend 7 acides aminés. CTD: domaine carboxy-terminal; Y: tyrosine; S: sérine; P: proline ; T: thréonine.
a) L’initiation de la transcription : l’assemblage du PIC
La Pol II seule n’est pas capable de reconnaitre un core promoter et d’initier la transcription de manière spécifique, sans la présence d’un certain nombre de facteurs auxiliaires. De ce fait, l’intervention des facteurs généraux de la transcription TFIIA, TFIIB, TFIID, TFIIE, TFIIF et TFIIH est indispensable et constitue, avec la Pol II, la machinerie transcriptionnelle de base qui va se lier en amont du core promoter (boite TATA et TSS).
Parmi tous les facteurs généraux de la transcription, TFIID est l’un des plus complexes, car il régule les différentes étapes liées au démarrage de la transcription. En effet, un constituant de TFIID, appelé TBP va lier spécifiquement la boîte TATA située à environ 30pb du TSS et déclencher l’assemblage du PIC [107]. À l’heure actuelle, il existe deux modèles d’assemblage du PIC : le modèle séquentiel et le modèle de l’holoenzyme [108–110]. Quel que soit le modèle envisagé, la fixation de TFIID sur le promoteur semble être l’étape préalable nécessaire à l’assemblage du PIC.
Le modèle séquentiel
D’après le modèle séquentiel, les facteurs généraux de la transcription et la Pol II s’assemblent au niveau du core promoter de manière successive, les uns à la suite des autres [108]. En se fixant au promoteur après la reconnaissance de la boîte TATA, TFIID va induire une courbure de l’ADN d’environ 90° de part et d’autre de la boîte TATA, permettant ainsi le rapprochement des séquences reconnues par des facteurs généraux de transcription [111, 112]. Ainsi, les facteurs TFIIB et TFIIA peuvent se fixer afin de stabiliser le complexe TFIID/TBP avec l’ADN et recrutent à leur tour TFIIF associé à la Pol II. TFIIF, serait essentiel au positionnement correct de la Pol II sur le +1, en limitant les liaisons non spécifiques de celle-ci avec l’ADN [113]. L’assemblage du PIC s’achève avec la liaison de TFIIE sur la Pol II, et l’arrivée de l’hélicase TFIIH. TFIIE agit en synergie avec TFIIH et permet l’ouverture des deux brins d’ADN en -9 et +2, et la formation de la bulle de transcription [114, 115]. Ensuite, TFIIH phosphoryle le CTD de la Pol II. À noter que seul le CTD non phosphorylé permet l’intégration de la Pol II dans le PIC. Le modèle séquentiel est basé sur des observations faites lors d’une reconstitution de la réaction de transcription in vitro, dans laquelle les facteurs sont ajoutés successivement. La position des facteurs est déterminée par la technique de migration sur gel et d’empreinte à la DNase [108, 116, 117].
Le modèle de l'holoenzyme
La purification de très gros complexes protéiques appelés holoenzymes, au sein duquel tous les facteurs seraient pré-assemblés et viendraient se fixer ensemble au promoteur, a permis de spéculer sur l’existence d’un second modèle : le modèle de l’holoenzyme. L’holoenzyme humaine a été purifiée in vivo à partir des nucléoplasmes des cellules HeLa et contient la Pol II et un certain nombre de facteurs généraux de la transcription, dont TFIIF, TFIIE et TFFIIH [109, 110]. Dans ce macro-complexe, la présence de co-activateurs transcriptionnels a également été détectée, notamment le complexe de remodelage de la chromatine SWI/SNF, CBP, ainsi que des membres du complexe Médiator [109, 110].
b) L’élongation de la transcription
Après l’ouverture de l’ADN au niveau des positions -9 et +2, l’incorporation des premiers nucléotides peut débuter et la formation de la première liaison phosphodiester aboutit. À cette étape la synthèse des transcrits naissants (entre 2 et 10 nucléotides) est très souvent abortive. La Pol II n’est pas stable et peine à produire des transcrits supérieurs à 10 nucléotides. L’étape suivante, appelée clairance du promoteur permet à la Pol II d’échapper au promoteur et d’avancer la bulle de transcription jusqu’à la position +11 [118]. La région comprise entre les positions -9 et -2 reprend alors sa conformation double brin, empêchant ainsi la Pol II de revenir sur ses pas. Cette étape marque le début de la phase d’élongation productive. Le remaniement du PIC semble être l’événement clé. TFIID reste fixé au promoteur tandis que TFIIE, TFIIH et TFIIB se décrochent du PIC [119]. La Pol II associée à TFIIF génère de courts transcrits avant de faire une pause à environ 50pb en aval du TSS [120]. Deux facteurs d’élongation négatifs, DSIF (DRB sensitivity inducing factor) et NELF (Negative Elongation Factor) s’associent à la Pol II et bloquent sa progression. La levée de la pause et l’entrée dans une phase de transcription plus efficace nécessitent le recrutement du complexe d’élongation P-TEFb composé des kinases Cdk9 et CyclinT1 [121]. P-TEFb va alors phosphoryler DSIF, NELF et la Ser2 de la Pol II, permettant de dissocier NELF de la Pol II. Finalement, la Pol II poursuit la polymérisation du transcrit naissant jusqu’à atteindre les sites de terminaisons à l’extrémité 3’ du gène.
c) La terminaison de la transcription
La phase de terminaison de la transcription se produit lorsque la Pol II et l’ARN naissant se dissocient de la matrice ADN. Chez les eucaryotes, pour la grande majorité des gènes codant les protéines, la terminaison de la transcription est étroitement couplée à des évènements de maturation du transcrit naissant. En effet, la transcription du signal poly(A) à l’extrémité 3’ des gènes induit la pause de la Pol II ainsi que le clivage endoribonucléolytique du transcrit naissant. Dans un second temps, la section du transcrit naissant en amont du site de clivage est polyadénylé (ajout d’environ 200 nt chez les mammifères) tandis que le reste du produit de clivage est dégradé.
Trois complexes protéiques participent à ces évènements de maturation. Il s’agit de
cleavage and polyadenylation specificity factor (CPSF), cleavage stimulatory factor (CstF) et poly(A) polymerase [122]. La réaction de clivage endoribonucléolytique est initiée lorsque la
Pol II transcrit le signal pol(A), et marque une pause. À ce moment précis, CPSF et CstF qui sont associé à la Pol II, reconnaissent deux éléments du site poly(A) sur le transcrit naissant, et entraine le clivage entre ces deux éléments [123–125]. Rappelons que la plupart des précurseurs d’ARNm possèdent un signal poly(A) extrêmement conservé, incluant une séquence 5’-AAUAAA-3’ suivie par une section riche en guanine/uridine [126]. Une fois le transcrit clivé, la Pol II continue à transcrire. Il est couramment admis que le clivage des transcrits naissants à lieu avant a dissociation de la Pol II de la matrice d'ADN, qui se produit environ 1500 nt en aval du signal de poly(A) [127, 128].
Les détails biochimiques de la réaction de terminaison restent à préciser. Toutefois, deux lignes de pensées prédominent [129]. Selon le modèle allostérique, lorsque la Pol II transcrit le signal poly(A), un changement de conformation est induit. Le recrutement des facteurs de terminaisons entraine le départ des facteurs d’élongation ce qui conduit à la dissociation de la Pol II de l’ADN matrice [130, 131]. Enfin, le modèle torpedo propose que le clivage endoribonucléolytique du transcrit naissant fournit un site d’entré pour l’exoribonucléase 5’-3’ Xrn2 qui va alors dégrader la portion de l’ARN naissant toujours attaché à la Pol II, et permettre le déplacement de la Pol II [132, 133].
d) La phosphorylation du CTD au cours de la transcription
La transcription est hautement régulée par des états de phosphorylation du domaine CTD de la Pol II. On parle du code du CTD [105, 106] (Figure 1.6). Parmi les 5 acides aminés du CTD pouvant être phosphorylé (Tyr1, Ser2, Thr4, Ser5 et Ser7), tous ont fait l’objet d’étude
levure et dans des cellules mammifères [134, 135]. Le profil de phosphorylation est similaire entre les espèces, excepté pour la Tyr1 qui semble suivre le profil de la Ser2 chez la levure, tandis que chez l’homme, cette phosphorylation semble restreinte autour du TSS et associée à la pause transcriptionnelle [136] (Figure 1.6A). De manière générale, ces évènements de phosphorylations successives du CTD dictent le passage entre les différentes étapes de la transcription, mais aussi les interactions entre les acteurs de la machinerie transcriptionnelle (Figure 1.6B).
Figure 1.6: Le code du CTD au cours de la transcription. D’après [106]. (A) Le profil de phosphorylation des acides aminés du CTD est similaire entre les espèces, excepté pour la Tyr1. (B) Chez les eucaryotes, la transcription s’articule autour de plusieurs étapes : l’initiation, l’élongation et la terminaison. Étape 1 : Le PIC incluant le complexe Médiator s’assemble au promoteur lorsque le CTD est non phosphorylé. La phosphorylation de la Ser5 entraine un remaniement du PIC et permet à la Pol II d’échapper au promoteur. Étape 2 : La Pol II s’associe aux facteurs d’élongation négatifs DSIF, NELF et entre en pause. Le CTD est phosphorylé sur la Ser5 et la Ser7. L’arrivée du facteur d’élongation positive P-TEFb va permettre la relâche de la pause en phosphorylant DSIF, NELF et la Ser2. Étape 3 : Durant la phase d’élongation, la phosphorylation de la Ser2 augmente tout comme la phosphorylation de la Thr4 (étape4) et semble requise pour le recrutement des facteurs impliqués dans la terminaison de la transcription. CTD : domaine carboxy-terminal; Y: tyrosine; S: sérine; P: proline ; T: thréonine.
Seul le CTD non phosphorylé permet l’intégration de la Pol II dans le PIC [137]. En effet, le complexe Médiator qui possède une forte affinité pour le CTD non phosphorylé facilite les interactions entre les différents constituants de la machinerie transcriptionnelle de base au niveau du promoteur. Ensuite, durant l’étape d’initiation, la Ser5 est phosphorylée par TFIIH, induisant ainsi la dislocation du complexe Médiator et la libération de la Pol II du promoteur. La phosphorylation de la Ser5 duCTD ne persiste que sur les 100 premières bases, jusqu’à l’arrêt de la Pol II [135]. Cette pause survient lorsque la Pol II est associée aux facteurs d’élongation négatifs DSIF et NELF et que le CTD est phosphorylé sur la Ser5 et la Ser7. Au moment de la relâche de la pause, la phosphorylation de la Ser5 se dissipe progressivement et est remplacée par une phosphorylation sur la Ser2 déposée par le complexe P-TEFb [121]. Cette marque augmente au fur et à mesure de la progression de la Pol II le long du corps du gène, tout comme la phosphorylation de la Thr4 et semble requise pour le recrutement des facteurs impliqués dans la terminaison de la transcription (facteurs de clivage et de polyadénylation) [138, 139].
2.3. L’expression des gènes est finement régulée par le phénomène de pause transcriptionnelle
L’étape d’initiation de la transcription a longtemps été considérée comme la plus importante dans le contrôle de l’expression des gènes. Puis, au milieu des années 1980, cette vision conventionnelle est remise en question avec la découverte de gènes maintenus dans un état de pause transcriptionnelle.
a) Mise en évidence des gènes en pause
Ce phénomène de pause transcriptionnelle est décrit comme un arrêt temporaire de l’élongation, après la synthèse d’environ 25 à 50 nucléotides. La Pol II reste stablement associée au transcrit en cours de synthèse et attend l’intégration de signaux d’activation pour entrer dans une phase d’élongation productive. Chez la Drosophile, en absence d’induction, les promoteurs proximaux des gènes inductibles en réponse au choc thermique et notamment celui de hsp70 sont les premiers gènes en pause à être caractérisés [140, 141]. La levée de la pause et l’entrée dans une phase d’élongation productive seront induites par un choc thermique. Par la suite, d’autres promoteurs en état de pause sont identifiés chez la Drosophile, et chez les mammifères comme les oncogènes humains c-myc, c-myb et c-fos [142–144].
![Figure 1.4: Le modèle de loop extrusion. D’après [92]. (A) Le complexe cohésine est chargée sur la chromatine par le facteur de recrutement NIPBL](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/28.918.140.799.134.566/figure-modèle-extrusion-complexe-cohésine-chargée-chromatine-recrutement.webp)
![Figure 1.5: La Pol II est l'élément central dans la transcription. D’après [106]. La Pol II est un assemblage de 12 sous unités d’un poids moléculaire de 550kDa](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/30.918.165.806.111.939/figure-élément-central-transcription-assemblage-unités-poids-moléculaire.webp)
![Figure 1.6: Le code du CTD au cours de la transcription. D’après [106]. (A) Le profil de phosphorylation des acides aminés du CTD est similaire entre les espèces, excepté pour la Tyr 1](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/34.918.136.792.338.704/figure-transcription-profil-phosphorylation-aminés-similaire-espèces-excepté.webp)
![Figure 1.7: La structure des R-loops. D’après [197]. Les R-loops sont des structures hybrides ARN/ADN formées au cours du processus de transcription](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/39.918.190.732.777.950/figure-structure-loops-structures-hybrides-formées-processus-transcription.webp)
![Figure 1.9: Les voies de signalisation des récepteurs TLRs. D’après [227]. Les récepteurs TLRs sont activés via la reconnaissance de différents ligands](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/47.918.129.790.231.819/figure-voies-signalisation-récepteurs-récepteurs-activés-reconnaissance-ligands.webp)
![Figure 1.10: Les membres de la famille NF-κB. D’après [234]. La famille des facteurs de transcription NF-κB est constituée de cinq membres : p50, p52, p65 (RelA), c-Rel et Rel B, qui partagent à l’extrémité N-terminale le domaine d’homologie](https://thumb-eu.123doks.com/thumbv2/123doknet/3119131.88617/49.918.247.672.113.329/facteurs-transcription-constituée-partagent-extrémité-terminale-domaine-homologie.webp)