DOCTEUR DE L'UNIVERSITÉ DE POITIERS UFR des sciences fondamentales et appliquées
Laboratoire Signalisation et transports ioniques membranaires - STIM (Poitiers) (Diplôme National - Arrêté du 25 mai 2016)
École doctorale : Biologie-santé - Bio-santé (Limoges)
Secteur de recherche : Aspects moléculaires et cellulaires de la biologie
Présentée par :
Hélène Cabanas
Rôle de la signalisation calcique dans la leucémie
myéloïde chronique
Directeur(s) de Thèse : Nicolas Bourmeyster, Nadine Déliot Soutenue le 05 décembre 2016 devant le jury
Jury :
Président Jean-Marc Gombert Professeur des Universités, Université de Poitiers Rapporteur Christophe Vandier Professeur des Universités, Université de Tours Rapporteur Laurence Bresson-Bépoldin Chargée de recherche CNRS, Université de Bordeaux Membre Nicolas Bourmeyster Maître de conférences, Université de Poitiers
Membre Nadine Déliot Maître de conférences, Université de Poitiers
Membre Fabrice Gouilleux Directeur de recherche CNRS, Université de Tours
Pour citer cette thèse :
Hélène Cabanas. Rôle de la signalisation calcique dans la leucémie myéloïde chronique [En ligne]. Thèse Aspects moléculaires et cellulaires de la biologie. Poitiers : Université de Poitiers, 2016. Disponible sur Internet
THESE
Pou l o te tio du G ade de
DOCTEUR DE L’UNIVERSITE DE POITIERS
(Faculté des Sciences Fondamentales et Appliquées)(Diplôme National – Arrêté du 7 Août 2006) Ecole Doctorale : BioSanté
Secteur de Recherche : Aspects Moléculaires et Cellulaires de la Biologie
Présentée par :
Hélène CABANAS
*********************
Rôle de la signalisation calcique dans la
Leucémie Myéloïde Chronique
*********************
Directeur de thèse : Dr. Nicolas BOURMEYSTERCo-directeur de thèse : Dr. Nadine DELIOT
*********************
Soutenue le 5 Décembre 2016 De a t la Co issio d E a e~
JURY
~
Dr. BRESSON-BEPOLDIN Laurence CR-HDR, Institut Bergonié, Bordeaux Rapporteur
Pr. VANDIER Christophe PU, Université de Tours Rapporteur Pr. GOMBERT Jean-Marc PU, Université de Poitiers Examinateur
Dr. GOUILLEUX Fabrice DR, Université de Tours Examinateur Dr. BOURMEYSTER Nicolas MCU-HDR, Université de Poitiers Examinateur Dr. DELIOT Nadine MCU, Université de Poitiers Examinateur
THESE
Pou l o te tio du G ade de
DOCTEUR DE L’UNIVERSITE DE POITIERS
(Faculté des Sciences Fondamentales et Appliquées)(Diplôme National – Arrêté du 7 Août 2006) Ecole Doctorale : BioSanté
Secteur de Recherche : Aspects Moléculaires et Cellulaires de la Biologie
Présentée par :
Hélène CABANAS
*********************
Rôle de la signalisation calcique dans la
Leucémie Myéloïde Chronique
*********************
Directeur de thèse : Dr. Nicolas BOURMEYSTERCo-directeur de thèse : Dr. Nadine DELIOT
*********************
Soutenue le 5 Décembre 2016 De a t la Co issio d E a e~
JURY
~
Dr. BRESSON-BEPOLDIN Laurence CR-HDR, Institut Bergonié, Bordeaux Rapporteur
Pr. VANDIER Christophe PU, Université de Tours Rapporteur Pr. GOMBERT Jean-Marc PU, Université de Poitiers Examinateur
Dr. GOUILLEUX Fabrice DR, Université de Tours Examinateur Dr. BOURMEYSTER Nicolas MCU-HDR, Université de Poitiers Examinateur Dr. DELIOT Nadine MCU, Université de Poitiers Examinateur
C est a e u e e tai e otio et eau oup de si it ue je oud ais e e ie toutes les personnes ayant soutenu et apprécié mon travail.
J ai e ai a a t tout ad esse es si es e e ie e ts à es deu directeurs de thèse : le Dr. Nicolas Bourmeyster et le Dr. Nadine Déliot ui o t a ueilli et permis de travailler sur ce sujet de th se i he et passio a t. Ils o t su e guide , e courager, me conseiller. Je les remercie pour leur confiance, leur patience, leur disponibilité et leur exigence. Enfin, je les remercie pour le temps passé sur la lecture de ce manuscrit et les corrections apportées.
Mes remerciements vont aussi au Dr. Bruno Constantin ui a pe is d i t g e ette équipe. Merci pour la o fia e u il a a ifest à o ga d lo s des congrès et colloques auxquels j ai pa ti ip et pou so soutie tout au long de ce chemin de recherche.
Encore une fois, Merci à eux pour cet accompagnement lors de ces trois années pleines de doutes, de uestio s, d app e tissages, de e o t es, d ha ges et d a uisitio s s ie tifi ues.
Je tiens également à exprimer toute ma gratitude au Dr. Laurence Bresson-Bepoldin et au Pr. Christophe Vandier d a oi a ept d t e les appo teu s de e a us it, ai si u au Pr. Jean-Marc Gombert et Dr. Fabrice Gouilleux d a oi a ept de fai e pa tie du ju e ualit d e a i ateu s lo s de la soutenance.
Je remercie le Pr. Frédéric Becq pour son accueil au sein du laboratoire Signalisation et Transports Ioniques Membranaires « STIM » ERL-7368.
J'adresse tous mes remerciements à Thomas Harnois et à Laëtitia Cousin pou a oi p t une oreille attentive et bienveillante sans qui ce travail de thèse n'aurait pas été le même. Merci également à Florence Domenichini pour la bo e hu eu u elle a pu appo te durant ma dernière année de thèse.
Je remercie les autres membres de l'équipe, Valérie Coronas et Patricia Arnault pour leur
présence discrète mais toujours efficace.
Au cours de ces trois années de thèse passées au sein du Pôle BioSanté, j'ai eu la chance de rencontrer et d'échanger avec de nombreuses personnes très impliquées dans leur travail. Elles ont toutes contribué de près ou de loin à ce travail. Merci pour leurs suggestions ou contributions, ou
tout simplement merci pour leurs qualités humaines.
Je remercie particulièrement Christophe Magaud pour son soutien professionnel précieux ainsi que pour toutes les discussions personnelles que l'on a pu avoir. Que ses projets se réalisent !
Merci également à Anne Cantereau-Becq et Adriana Delwail pour leur aide technique très précieuse et efficace.
Je tiens également à remercier tous les enseignants de la section CNU 65, plus particulièrement Matthieu Regnacq, Fatima Dkhissi, Thierry Bergès ainsi que Jean-Marc Muller. Me i à eu de 'a oi fait o fia e pou ejoi d e leu uipe. Ils o t pe is de découvrir avec un réel plaisir la transmission pédagogique.
Plus simplement, je tiens à remercier mes amies de l'unité Inserm 1082 : Alice, Aurélie, Lucie, Maroua, Myriam, Sara. Merci d'avoir été là pour me soutenir dans les moments les plus difficiles, d a oi p is le te ps de oute et de 'a oi toujou s do de t s o s o seils.
Un grand merci également à Nathalie qui a su accueillir, me rassurer à mon arrivée dans ce
laboratoire et accompagner mes premiers pas dans ma nouvelle vie pictavienne.
Je e e ie toutes es a ies o delaises. Mo a ie d e fa e, Clémentine, qui a toujours cru en moi depuis 25 ans qu'on se connait ! Harmonie l a e tu i e, Claire que je ne désespère pas d a ueilli su Poitie s à l o asio de so stage d i te at, et les autres qui gentiment s i t essaie t à o sujet de th se.
Sans oublier mes amis pictaves, Carole, Antoine, Chloé, Florian, Marina et Simon que je e e ie de a oi ja ais aiss les as pou e faire découvrir la vie nocturne de Poitiers. Maintenant je n'ai plus aucune excuse !
Merci à tous les doctorants du PBS pour leur présence précieuse et leur solidarité doctorale.
Bon courage à celles et ceux qui n'ont pas encore fini.
Enfin, mes plus profonds remerciements vont à mes parents et mon frère. Merci à eux de m'avoir soutenu, encouragé et aidé tout au long de ces 3 années de thèse et plus encore. Je e se ai pas là aujou d hui sa s eu et je leu dedie ette th se. Qu'ils trouvent, dans la réalisation de ce travail l'aboutissement de leurs efforts ainsi que l'expression de ma plus affectueuse gratitude. Je
remercie tendrement mon frère pour ses attentions particulières et son humour.
Que de chemin parcouru ! Que de rencontres, de soutiens précieux, d atte tio s particulières. Merci et beaucoup de reconnaissance pour la place que vous avez individuellement et
TABLE DES MATIERES
REMERCIEMENTS
...TABLE DES MATIERES
...RESUME / ABSTRACT
...LISTE DES ABREVIATIONS
...INDEX DES FIGURES ET TABLEAUX
...INTRODUCTION
...1Partie I : La Leucémie Myéloïde Chronique : Généralités cliniques et biologiqes...1
I. Définition ... 1
II. Historique ... 1
III. L'origine de la LMC ... 3
IV. Les acteurs principaux de la LMC ... 4
1. Le gène abl et sa protéine ... 5
2. Le gène bcr et sa protéine ... 8
3. Le gène bcr-abl et sa protéine ... 11
V. L'oncogenèse induite par Bcr-Abl ... 12
1. Les voies de signalisation associées à Bcr-Abl ... 12
a. La voie MAPK/ERK (Ras/Raf/MEK/ERK) ... 13
b. La voie PI3K/Akt ... 14
c. La voie JAK/STAT... 15
d. La voie des GTPases de la famille Rho ... 16
e. Les protéines du cytosquelette ... 17
f. L’i pli atio de la PKC ... 18
2. Les traitements ciblés de la LMC ... 19
a. Les traitements cytoréducteurs ... 20
b. La greffe de moelle osseuse... 20
c. Le traitement par l'Interféron-α ... 21
d. Le traitement par les inhibiteurs de tyrosine kinases ... 22
1. Les mécanismes de résistance aux ITKs ... 23
2. La cellule souche hématopoïétique ... 25
3. La cellule souche leucémique ... 27
4. La persistance de la cellule souche leucémique ... 30
Partie II : Calcium, Canaux et Physiologie...34
I. Calcium, homéostasie calcique et signal calcique ... 34
II. Les acteurs de la signalisation calcique dans les cellules non-excitables ... 35
1. Les acteurs réticulaires ... 35
2. Les acteurs de la membrane plasmique ... 39
3. Les acteurs mitochondriaux ... 41
4. Les acteurs golgiens ... 44
5. Les calciprotéines cytoplasmiques ... 44
6. Les acteurs nucléaires ... 46
III. Les influx calciques dans les cellules non-excitables ... 47
1. L'entrée constitutive de Ca2+ ... 47
2. L’e t e de Ca2+ dépendante de TRPV2 ... 47
3. Les influx SMOCs ... 48
4. Les influx SOCs ... 49
IV. Les protéines STIM et Orai ... 51
1. Structure des STIM ... 51
a. STIM1 ... 52
b. STIM2 ... 53
c. Rôles distincts entre STIM1 et STIM2 ... 54
2. Structure des Orai ... 55
a. Orai1 ... 55
b. Orai2 et Orai3 ... 58
3. Interaction entre STIM1 et Orai1 ... 59
a. Activation de STIM1 ... 59
b. Couplage entre STIM1 et Orai1 ... 60
c. Ouverture des canaux Orai1 ... 62
V. La superfamille des canaux calciques TRPs ... 64
1. La famille des TRPs ... 64
2. La structure des TRPs ... 65
4. Les TPRC dans les influx calciques ... 68
VI. Les interactions entre STIM1, Orai1 et TRPC1 ... 71
1. Activation de TRPC1 par STIM1 ... 71
2. L’a tivatio de TRPC pa STIM /O ai ... 72
VII. La modulation des SOCEs ... 75
1. L'inactivation dépendante du Ca2+ et le rôle de la mitochondrie ... 75
2. La phosphorylation ... 76
3. La modulation par les réactions d'oxydoréduction ... 78
4. Le pH ... 79
5. La température ... 79
6. Les facteurs diffusibles ... 80
7. L'ATP ... 81
8. Les protéines accessoires ... 81
VIII. La pharmacologie des SOCEs ... 85
1. L’a tivatio des SOCEs ... 85
2. L’i hi itio des SOCEs ... 86
Partie III : Calcium et Cancer...90
I. Généralités ... 90
II. Rôle de la signalisation calcique dans la prolifération des cellules tumorales ... 91
1. Le calcium et la régulation du cycle cellulaire ... 91
2. L'implication des acteurs SOCs dans la prolifération des cellules tumorales ... 95
III. Rôle de la signalisation calcique dans l'apoptose des cellules tumorales ... 98
1. Le calcium et la régulation de l'apoptose ... 98
2. L'i pli atio des a teu s SOCs da s l’apoptose des ellules tu o ales ... 100
IV. Rôle de la signalisation calcique dans la migration des cellules tumorales ... 103
1. Le calcium et la régulation de la migration ... 103
2. L'implication des acteurs SOCs dans la migration et les métastases des cellules tumorales ...106
V. LMC et SOCEs : un rôle controversé ... ..110
POSITION DU PROBLEME ET OBJECTIFS DU TRAVAIL
...112MATERIELS ET METHODES
...114Méthodes de biologie cellulaire ... 114
I. Les modèles cellulaires . ... 114
2. Entretien des lignées cellulaires ... 114
3. Congélation/ Décongélation des lignées cellulaires ... 115
II. Mesure des variations du calcium intracellulaire ... 115
1. La sonde Fura 2-AM ... 115
2. Dispositif expérimental ... 117
3. Protocole expérimental ... 118
a. La charge des cellules avec la sonde Fura 2-AM ... 118
b. Mise e pla e de l’ ha tillo ... 118
c. Mesures effectuées ... 119
d. Pharmacologie ... 120
e. Traitement des données ... 120
III. Enregistrements des mouvements cellulaires en microscopie time-lapse...120
IV. Test de prolifération cellulaire ... 121
1. Comptage cellulaire ... 122
1. Analyse du cycle cellulaire ... 122
V. Test de d te tio de l’apoptose ... 122
1. P i ipe du a uage à l’A e i e-V ... 122
2. D te tio des ellules apoptoti ues pa utilisatio o joi te d’A e i e-V/FITC et d’Iodu e de Propidium ... 123
a. Points nécessaires au réglage du cytomètre de flux ... 124
b. Préparation des cellules à analyser ... 124
c. Analyse au cytomètre de flux (FACS) ... 125
d. Traitement des données ... 125
Méthodes biochimiques ... 126
I. Préparation de lysat cellulaire ... 126
1. Extraction de protéines ... 126
2. Dosage des protéines ... 126
a. Dans le tampon RIPA ... 126
b. Dans le Laëmmli 2X ... 127
II. SDS-PAGE et Western Blot ... 127
1. Electrophorèse des protéines sur gel de polyacrylamide (SDS-PAGE) ... 127
2. Electrotransfert sur membrane de nitrocellulose ... 129
3. Détection immunologique des protéines ... 129
a. Principe ... 131
b. Protocole expérimental ... 132
III. Immunoprécipitations et co-immunoprécipitations ... 133
IV. Immunofluorescence ... 133
1. Dispositif expérimental ... 133
2. Protocole expérimental ... 134
a. Culture des cellules sur fibronectine ... 134
b. Traitements effectués ... 134
c. Fixation, Perméabilisation et Marquage ... 136
d. Analyse des images ... 137
Méthodes de biologie moléculaire ... 138
I. RT-PCR et PCR quantitative ... 138
1. Extraction des ARN totaux ... 138
2. Transcription inverse des ARN en ADNc ... 139
3. PCR après transcription inverse ou RT-PCR ... 139
a. Réaction de PCR ... 139
b. Séparation électrophorétique des produits de PCR ... 140
4. PCR quantitative ou RT-qPCR ... 141
a. Les différentes méthodes utilisées ... 141
b. Réaction de RT-qPCR ... 142
c. Analyse des données ... 144
Analyses statistiques ... 145
RESULTATS
...146Avant-propos ... 146
Partie I : Dérégulation de l'homéostasie calcique dans les cellules leucémiques exprimant Bcr-Abl.... ...147
I. Co te te de l’ tude ... .147
II. Article : Deregulation of calcium homeostasis in Bcr-Abl-dependent Chronic Myeloid Leukemia………..………148
Partie II : Etude de l'impact de l'activité tyrosine kinase de Bcr-Abl sur la signalisation calcique....181
I. Caractérisation de l’e p essio , de la lo alisatio et de l’i te a tio des a teu s al i ues majeurs des SOCs dans les lignées 32D et Ba/F3... 181
1. Expression des transcrits stim1, orai1 et trpc1/3/4/6 dans les lignées 32D et Ba/F3 ... 182
2. Expression des protéines STIM1, Orai1 et TRPC1 dans la lignée Ba/F3 ... 184
4. Interaction entre STIM1/Orai1 et STIM1/TRPC1 ... 187
II. Etude de la gulatio de la sig alisatio al i ue pa l’a tivit t osi e ki ase de B -Abl .. 188
1. Etats de phospho latio des a teu s SOCs ap s t aite e t à l’I ati i ... 188
2. Translocation nucléaire de NFAT ... 191
a. La translocation nucléaire de NFAT est diminuée dans les cellules exprimant Bcr-Abl ... 191
b. La t a slo atio u l ai e de NFAT est li e à l’a tivatio des SOCEs da s les ellules exprimant Bcr-Abl ... 195
c. La t a slo atio de NFAT est di i u e ap s t aite e t à l’I ati i ... 196
3. Prolifération in vitro des cellules 32D et Ba/F3 ... 198
a. La prolifération est dépendante des SOCEs dans les cellules exprimant Bcr-Abl ... 198
b. La p olif atio i pli ue l’a tivit t osi e ki ase de B -Abl et la signalisation calcique de manière indépendante ... 200
4. Apoptose in vitro des cellules 32D et Ba/F3 ... 203
a. Les SOCEs ne sont pas directement impliquées da s l’apoptose spo ta e des ellules exprimant p210bcr-abl ... 203
b. L’utilisatio de l’I ati i à de fai les doses se le p ot ge de l’apoptose i duite pa le YM-58483 ... 204
c. Les i hi iteu s des SOCEs e p ot ge t pas de l’apoptose i duite ... 206
DISCUSSION ET PERSPECTIVES
...207I. Alt atio de l’ho ostasie al i ue da s les ellules e p i a t B -Abl ... 207
1. Mise en évidence ... 207
2. Mécanismes impliqués dans la diminution des influx SOCEs ... 208
a. Expression des acteurs principaux de la signalisation calcique ... 208
b. Localisation et interaction des acteurs principaux de la signalisation calcique ... 210
c. Etats de phosphorylation des acteurs principaux de la signalisation calcique ... 211
3. Rôles des SOCEs dans les cellules leucémiques exprimant Bcr-Abl ... 212
a. I pa t de l’alt atio de l’ho ostasie al i ue su la t a slo atio u l ai e de NFAT .. 212
b. I pa t de l’alt atio de l’ho ostasie al i ue su la p olif atio des ellules leu i ues ...213
c. I pa t de l’alt atio de l’ho ostasie alcique sur la migration des cellules leucémiques ...214
II. Rôle de l’a tivit t osi e ki ase de B -A l da s l’ho ostasie al i ue ... 215
1. Mise en évidence ... 215
2. Mécanisme pouvant relier Bcr-Abl à l'homéostasie calcique ... 217 3. I pa t de l’a tivit t osi e ki ase su les p o essus o og i ues dépendants du Ca2+ . 218
a. Impact de l'activité tyrosine kinase sur la translocation nucléaire de NFAT ... 218
b. Impact de l'activité tyrosine kinase sur la prolifération des cellules leucémiques ... 219
c. Impact de l'activité tyrosine kinase sur l'apoptose des cellules leucémiques ... 219
CONCLUSION
...221Rôle de la signalisation calcique dans la Leucémie Myéloïde Chronique
La Leucémie Myéloïde Chronique (LMC) est une maladie clonale caractérisée par la présence du chromosome Philadelphie codant pour Bcr-Abl, une tyrosine kinase constitutivement active responsable de la leucémogenèse. Bien que très efficaces, les inhibiteurs de tyrosine kinase (ITKs) restent cependant inactifs sur les cellules souches leucémiques. Ce travail de thèse montre que la signalisation calcique, connue pour réguler de nombreux processus dans les cellules saines et cancéreuses, est importante dans la signalisation cellulaire au décours de la LMC. Le rôle des entrées calciques dépendantes des stocks (SOCEs) médiées par STIM1 (STromal Interaction Molecule 1) et les canaux Orai1 et TRPC1 ainsi que des entrées calciques induites par la thrombine a été étudié dans la leucémogenèse. Nous avons observé une diminution de ces entrées dans les cellules exprimant Bcr-A l pou a t t e e pli u e pa le ha ge e t de stœ hiométrie Orai1/STIM1. Ceci entraîne la di i utio de l a ti atio de NFAT Nuclear Factor of Activated T-cells) ainsi que des conséquences su la p olif atio et la ig atio ellulai e ais pas su l apoptose. De plus, les “OCEs so t restaurées da s les ellules a euses ap s t aite e t à l I ati i , le p i ipal ITK. Nous p oposo s alo s ue l e p essio de B -A l joue u ôle su l homéostasie calcique en entraînant une dérégulation générale des fonctions cellulaires dans les cellules leucémiques notamment via la voie PKC (Protein Kinase C). Ainsi, ces résultats montrent une dérégulation des entrées calciques dans les cellules exprimant Bcr-Abl, suggérant que la signalisation calcique puisse être une cible thérapeutique en parallèle avec les ITKs.
Mots clés : Leucémie Myéloïde Chronique, Homéostasie calcique, STIM1/Orai1/TRPC1, Leucémogenèse
Role of calcium signaling in Chronic Myeloid Leukemia
Chronic Myeloid Leukemia (CML) is a clonal disease characterized by the presence of the Philadelphia chromosome encoding for Bcr-Abl, a constitutively active tyrosine kinase responsible for leukemogenesis. Although Bcr-Abl tyrosine kinase inhibitors (TKIs) have revolutionized the therapy of Ph+ leukemia, the complete eradication of CML is limited by the emergence of resistance in hematopoietic stem cells. This thesis proposes that calcium (Ca2+) signaling pathways, known to govern a large number of functions in normal and cancer cells, may be important in CML cell signaling. Therefore, we studied the role of Store Operated-Calcium entry (SOCE) (i.e. STromal Interaction Molecule 1 (STIM1), Orai1 and TRPC1 channels) and thrombin induced Ca2+ entry in leukemogenesis. We found a decrease in both calcium entries in Bcr-Abl-expressing cells compared to normal cells. The reduced SOCE seems related to a change in stoichiometry of Orai1/STIM1. This leads to a reduction of the Nuclear Factor of Activated T-cells (NFAT) translocation and functional consequences on cell proliferation and migration but not on apoptosis. Moreover, we showed that SOCE is restored in malignant cells after treatment with Imatinib, the main TKI. We proposed that Bcr-Abl expression could impact on Ca2+ homeostasis enhancing a general disorganization of cell functions in leukemia cells notably via Protein Kinase C (PKC) pathway. Altogether this work shows a deregulation of Ca2+ entry in Bcr-Abl-expressing cells, suggesting that the Ca2+ signaling pathway could be a therapeutic target in parallel with TKIs.
LISTE DES ABREVIATIONS
-A-2-APB : 2-AminoethyldiPhenyl Borinate aa : a ides a i s
Abl : Abelson
ADN : Acide D so i oNu l i ue ADNc : A ide D so i oNu l i ue
o pl e tai e ADP : Adénosine DiPhosphate AKT (PKB): Protein Kinase b
Alox-5 : Arachidonate 5-lipoxygenase AM : A to M th leste
AMPc : Adénosine MonoPhosphate cyclique
ANT : Adenine Nucleotide Translocator Apaf-1 : Apoptotic protease activating
factor 1
ARC : Arachidonate-Regulated Calcium Channel
ARG : Abl-Related Gene ARN : A ide ‘i oNu l i ue ARNase : Acide RiboNucléase
ARNm : A ide ‘i oNu l i ue essage Asn : Asparagine
Asp : Aspartate
ATP : Ad osi e T iPhosphate AV : Annexin-V
-B-
Bad : Bcl-2-associated agonist of cell death
BAPTA : 1,2-bis (2-aminophenoxy) ethane-N,N,N',N'-tetraacetic acid Bax : Bcl-2 associated X
Bcl-2 : B cell lymphoma 2
Bcl-XL : B-cell lymphoma-extra large
Bcr : Breakpoint Cluster Region BET : B o u e d Ethidium
Bid : BH3-interacting domain death agonist
Bim (BCL2L11): Bcl-2-like protein 11 BSA : Bovin Serum Albumin
BTP : 3,5-bis(trifluoromethyl)pyrazole) -C-
Ca2+ : ion Calcium
[Ca2+]c : Concentration calcique
cytosolique
CABPs : Calcium-Binding Proteins CAD : CRAC Activation Domain CaM : CalModuline
CaMKII : Calmoduline Kinase II CaV-1 : Caveolin 1
CBD : Calcium Binding Domain CBD : cannabidiol
Cbl : Casitas B-Lineage Lymphoma CC : Coiled-Coil
Cdc42 : Cell division control protein 42 homolog
CD : Cluster of Differentiation CDI : Calcium Dependent Inactivation Cdk : Cyclin-dependent kinase cEF : canonical EF-hand CIF : Calcium Influx Factor
CIRB : calmodulin/IP3 receptor binding
c-KIT (SCFR) : Mast/Stem Cell Growth Factor Receptor
CLPs : Progéniteurs Lymphoïdes Communs
cm : centimètre
CMD : CRAC Modulatory Domain CMPs : Progéniteurs Myéloïdes
Communs CO2 : dioxygène
CPA : CycloPiazonic Acid CRAC : Calcium Release-Activated
Channels
CRACM1 : Calcium Release-Activated 1 Calcium Modulator
CRACR2A : CRAC Regulatory protein 2A CREB : cAMP-Responsive Element
Binding
CrkL : Crk-Like protein CS : Cellule Souche
CSH : Cellule Souche Hématopoïetique CSL : Cellules Souche Leucémique Ct : Cycle threshold
CTID : C-Terminal Inhibitory Domain Cu+ : ion cuivreux Cu2+ : ion cuivrique Cys : Cystéine -D- Da : Dalton DAG : DiAcylGlycerol
Dbl : Diffuse B-cell Lymphoma DH : Dbl Homology
DISC : Death-Inducing Signaling Complex
DMSO : DiMéthylSulfOxyde
dNTP : d so Nu l otideT iPhosphate DO : Densité Optique
-E-
4EBP1 : eIF4E-binding proteins EB1 : End Binding 1 protein
ECL : Enhanced ChemiLuminescence EDTA : EthyleneDiamine TetraAcetic acid EGF : Epidermal Growth Factor
EGTA : EthyleneGlycol-bis ( -amino-ethyl ether) N,N,N',N'-Tetraacetic Acid
EMRE : Essential MCU REgulator ERK : Extracellular signal-Regulated
Kinase
ERM : Ezrin/ Radixin/ Moesin
ETON : Extended TM1 Orai1 N-terminal Region
-F-
FABD : F-Actin Binding Domain FACS : Fluorescence-Activated Cell
Sorting
FAK : Focal Adhesion Kinase Fas (CD95) : First apoptosis signal FasL (CD95L) : First apoptosis signal ligand FCDI : Fast Calcium Dependent
Inactivation
FITC : Fluorescein IsoThioCyanate FKBP : FK506 Binding Protein FK506 : Tacrolimus
FOXO : Forkhead box protein O
FRET : Förster/Fluorescence resonance energy transfer
Fura-2 AM : (1-[6-amino-2-(5-carboxy-2- oxazolyl)-5-benzofuranyloxy]-2-(2-amino-5 methylphenoxy) ethane-N, N, N , N -tetraacetic acid, pentaacetoxymethyl ester)
-G-
g : force relative de la centrifugation Gab2 : Grb2-associated-binding protein2 GAP : GTPase Activating Protein GDP : Guanosine DiPhosphate GEF : Guanine nucleotide Exchanger
factor
GM-CSF : Granulocyte-Macrophage Colony-Stimulating Factor
GOK ; Gene Overexpressed in Cancer Grb2 : Growth factor receptor-bound
protein 2 GSK3β : Gl og e “ thase ki ase β GSK-5503A : 2,6-Difluoro-N-(1-(2-phenoxy-benzyl)-1H-pyrazol-3-yl) benzamide) GSK-7975A : 2,6-Difluoro-N-(1-(4-hydroxy-2- (trifluoromethyl)benzyl)-1H-pyrazol-3-yl)benzamide GTP : Guanosine TriPhosphate -H- h : heure
Hck : Hematopoietic cell kinase HDAC6 : microtubule-associated Histone
DeAcetylase 6 hEF : hidden EF-hand Hef1 : Human enhancer of
filamentation protein 1 HEK : Human Embryonic Kidney
HEPES :
N-2-HydroxyEthyl-Piperazine-N'-2-EthaneSulfonic acid H2O2 : Péroxyde d'hydrogène
Homer-1 : Homer protein homolog 1 HTRA2 : High-Temperature Requirement
Protein A2
-I-
IAP : Inhibitor of Apoptosis Proteins ICRAC : CRAC current
ICSBP : Interferon Consensus Sequence-Binding Protein IFN-α : InterFeroN-α IgG : Immunoglobuline G IκK : I B Kinase IL-3 : InterLeukine-3 IP : Iodure de Propidium IP : I u oP ipitatio iPLA2 : calcium-independent PhosphoLipase A2 IP3 : Inositol 1,4,5-trisPhosphate IP3R : Inositol 1,4,5-trisPhosphate Receptor ISOC : SOC current
ITK : Inhibiteur de Tyrosine Kinases -J-
JAK : JAnus Kinase
JNK : c-Jun N-terminal Kinase -K- K : Lysine-rich region K+ : ion potassium kb : kilobase KCl : Chlorure de potassium Kd : constante de dissociation kDa : kiloDalton KO : Knock Out
-L- La3+ : ion Lanthane
LAL : Leucémie Aigüe Lymphoblastique LAM : Leucémie Aigüe Myéloide LMC : Leucémie Myéloïde Chronique LNC : Leucémie Neutrophilique
Chronique
LRC : Leukotriene-C4-regulated Calcium channel
LRD : Lipid Rafts Domain
Lyn : Lck/Yes novel tyrosine kinase -M-
mAb : monoclonat Antibody MAPK : Mitogen-Activating Protein
Kinase
m-bcr : minor breakpoint cluster region M-bcr : Major breakpoint cluster region MCU : Mitochondrial Calcium Uniporter MCUR1 : Mitochondrial Calcium Uniporter
Regulator 1 MDM2 : E3-ubiquitine ligase MDR1 : MultiDrug-Resitance Gene MEK : Mitogen-activated ERK mHCX : mitochondrial proton-calcium
exchanger mi-ARN : micro-ARN
MICU : MItochondrial EF Hand Calcium Uniporter regulator
μ-bcr : micro breakpoint cluster region μg : microgramme μL : microLitre μ : i o t e μM : microMolaire min : minute Mg2+ : io Mag siu mL : milliLitre
MLC : Myosin Light Chain MLCK : Myosin Light Chain Kinase ML-9 : (1-(5-Chloronaphthalene-1- sulfonyl)-1H-hexahydro-1,4-diazepine hydrochloride mM : milliMolaire Mn2+ : ion Manganèse mNCX : mitochondrial sodium-calcium exchanger
MOMP : Mitochondrial Outer Membrane Permeabilization
mOsm : milliOsmol
MP : Membrane Plasmique
mTOR : mammalian/mechanistic target of rapamycin
-N- Na+ : ion sodium
NCX : Sodium-Calcium Exchanger NES : Nuclear Export Signal NFAT : Nuclear Factor of Activated
T-cells
NFB : Nuclear Factor-kappa B NHR : NFAT Homolgy Region NLS : Nuclear Localization Signal
nm : a o t e
nM : nanoMolaire
NOD/SCID : Non-Obese Diabetic/Severe Combined ImmunoDeficiency NP-40 : Nonidet P40
-O-
OAG : 1-Oleoyl-2-Acetyl-sn-Glycerol Oli : domaine d'Oligomérisation OPN : Osteopontin
-P-
p130Cas : p130Crk-associated substrate pA : picoAmpère
PBS : Phosphate Buffer Saline PDGF : Platelet-Derived Growth Factor pF : picoFarad
PFA : Pa aFo Ald h de pH : potentiel Hydrogène Ph : Chromosome Philadelphie PH : Pleckstrin Homology PH : Proline-histidine-rich region PI3K : PhosphatidylInositol-3 Kinase PIP : PhosphatidylInositol Phosphate PIP2 : PhosphatidylInositol 4,5- bisPhosphate PKA : P ot i e Ki ase A PKC : P ot i e Ki ase C PKG : P ot i e Ki ase G PLC : PhosphoLipase C
PMCA : Plasma-Membrane Calcium ATPase
PNN : PolyNucléaires Neutrophiles POST : Partner of STIM1
pS : picoSiemens PS : Phosphatidylsérine PS : Proline-serine-rich region PSA : Pe “ulfate d A o iu PTP : Permeability Transition Pore Pyk2 : Protein tyrosine kinase 2
-Q-
-R-
Rac1 : Ras-related C3 botulinum toxin substrate 1
RB1 : Retinoblastoma Protein RCPG : Récepteurs couplés aux
protéines G
RE : ‘ ti ulu E doplas i ue RhoA : Ras homolog gene family
member A
RIPA : RadioImmunoPrecipitation Assay RIP3 : ‘ epteu à l I ositol , ,
-trisPhosphate
RK : Arginine-lysine-rich region RMN : Résonnance Magnétique
Nucléaire
ROCs : Receptor-Operated Channels ROCE : Receptor-Operated Calcium entry ROS : Reactive Oxygen Species
rpm : rotation par minute
RPMI : Roswell Park Memorial Institute medium
RS : Réticulum Sarcoplasmique RTK : Récepteurs à activité tyrosine
kinase
RT-PCR : Reverse-Transcriptase Polymerase Chain Reaction RyRs : Ryanodine Receptors
-S-
SAM : Sterile Alpha Motif
SARAF : SOCE-Associated Regulatory Factor
SCDI : Slow Calcium Dependent Inactivation
SDF-1 : Stromal cell-Derived Factor 1 SDS-PAGE : SDS-PolyAcrylamide Gel
Electrophoresis sec : seconde Ser : Sérine
SERCA : Sarco-Endoplasmique Reticulum Calcium ATPase
SH1/2/3 : Src Homology region 1/2/3 SHD : STIM1 Homomerization Domain siRNA : small interfering RNA
SK3 : Small-conductance Ca2+ activated K+ channels SKF-96365 : 1-{β-[3-(4-Methoxyphenyl) propyl]-4-methoxyphenethyl}-1H-imidazole hydrochloride SKP2 : S-phase Kinase-associated Protein 2 SLC25A23 : calcium-dependent mitochondrial solute carrier SMAC : Second Mitochondria-derived
Activator of Caspases
SMOCs : Second Messenger-Operated Channels
SMOCE : Second Messenger-Operated Calcium entry
SOAR : STIM1-Orai1 Activation Region SOCs : Store-Operated Channels SOCEs : Store-Operated Calcium Entries SOCS3 : Supressor Of Cytokine Signaling 3 Sos : Son of Sevenless
SPCA : Secretory Pathway Calcium-ATPase
STAT : Signal Transducer and Activators of Transcription
STIM1/2 : STromal Interaction Molecule 1/2 SVF : “ u de Veau Fœtal
Synta 66 : (N-(2',5'-Dimethoxy[1,1'- biphenyl]-4-yl)-3-fluoro-4- pyridinecarboxamide
-T- TAE : T is A tate EDTA TBS : Tris Buffered Saline TCA : Trichloroacetic acid TEA : TetraEthyl Ammonium TEMED :
TEtraMEthyl-1-,2-Diamino-tha e
TGFβ : Transforming Growth Factor beta ∆9-THC : ∆9
-tetrahydrocannabinol Thr : Thréonine
TIRFM : Total Internal Reflection Fluorescence Microscopy TM : Domaine TransMembranaire TMEM20 : TransMEMbrane protein 20 TNF : Tumor Necrosis Factor TPEN :
N,N,N',N'-Tetrakis-(2-Pyridyl-methyl)-EthyleNediamine Trail : tumor-necrosis-factor related
apoptosis inducing ligand TRP : Transient Receptor Potential TRPA : Transient Receptor Potential
Ankyrin
TRPC : Transient Receptor Potential Canonical
TRPM : Transient Receptor Potential Melastatin
TRPML : Transient Receptor Potential MucoLipin
TRPN : Transient Receptor Potential No mechanosensitive
TRPP : Transient Receptor Potential Polycystin
TRPV : Transient Receptor Potential Vanilloid
-U-
U : U it
-V-
VDACs : Voltage-dependent anion channels
-W- WB : Western Blotting
-X-
XPB : Xeroderma Pigmentosum group B XIP : eXchanger Inhibitory Peptide
-Y- Y : tyrosine
YM-58483 : 4-methyl-4′-[3,5-bis (trifluoro- methyl)-1H-pyrazol-1-yl]-1,2,3-thiadiazole-5- carboxanilide
-Z- Zn2+ : ion zinc
INDEX DES FIGURES
Figure Titre Page
Introduction - Partie I : La leucémie Myéloïde chronique : Généralités Cliniques et Biologiques
Figure I.1 La translocation t(9 ;22)(q34 ;q11) et la génération du gène chimérique bcr-abl
2
Figure I.2 Les différentes isoformes issues de la translocation t(9 ;22)(q34 ;q11) 3 Figure I.3 Le gène c-abl localisé sur le chromosome 9q34 5 Figure I.4 Structure en domaines de la protéine p145abl 6
Figure I.5 Rep se tatio de l’ tat i a tif o fo atio fe e et de l’ tat a tif (conformation ouverte) de la kinase c-Abl
7
Figure I.6 Le gène bcr localisé sur le chromosome 22q11 9
Figure I.7 Structure en domaine de la protéine Bcr 10
Figure I.8 Structure en domaines de la protéine p210bcr-abl 11
Figure I.9 Le réseau des voies de signalisation (non exhaustif) de Bcr-Abl 13 Figure I.10 Implication des PKC dans les voies de signalisation dépendantes de Bcr-Abl 19 Figure I.11 Evolution des traitements de la LMC depuis le XIXème siècle 20
Figure I.12 Mécanisme d'action de l'Imatinib 22
Figure I.13 Schéma illustratif simplifié de la différenciation hématopoïétique 26
Figure I.14 Modèle de CSL 29
Figure I.15 Modèle de niche leucémique 30
Figure I.16 Les ITKs et la niche leucémique 32
Introduction - Partie II : Calcium, Canaux et Physiologie
Figure I.17 Dynamique de la signalisation calcique et homéostasie 35
Figure I.18 St u tu e de deu o o es d’IP3R 36
Figure I.19 St u tu e d’u e sous-unité du RYR2 37
Figure I.20 Structure de la pompe SERCA 39
Figure I.21 St u tu e de l’ ha geu Na+/Ca2+ 41
Figure I.22 Représentation schématique du complexe MCU 43
Figure I.23 La voie NFAT dépendante du Ca2+ 45
Figure I.24 Entrée non capacitive via les canaux SMOCs 49
Figure I.25 Entrée capacitive via les canaux SOCs 51
Figure I.26 Structure moléculaire de STIM1 53
Figure I.27 Structure moléculaire de STIM2 54
Figure I.28 Structure moléculaire de Orai1 56
Figure I.29 Structure cristalline de Orai1 chez la Drosophile 57 Figure I.30 Le couplage STIM1-Orai1 et l'activation des canaux CRAC 60 Figure I.31 Modèle ol ulai e et stœ hio t ie e t e les p ot i es O ai et STIM 62 Figure I.32 Mod le pou l’ouve tu e des a au O ai 63 Figure I.33 L'arbre phylogénétique de la famille des canaux ioniques TRPs 65 Figure I.34 Structure et diversités des membres de la famille TRP 66
Figure I.35 La famille des TRPC 67
Figure I.36 Fonction physiologique de Orai1 et TRPC1 dans les SOCEs 73 Figure I.37 Représentation du signalplex impliquant les différentes protéines
accessoires
82
Introduction - Partie III : Le calcium et le cancer
Figure I.38 Les principales caractéristiques du cancer 90
Figure I.40 La voie Ca2+/CaM/Calcineurine impliquée dans la régulation du cycle cellulaire
94
Figure I.41 La gulatio de l’apoptose par le Ca2 99
Figure I.42 Schéma récapitulatif du rôle de STIM1 et des canaux Orai et TRPC dans la tumorigenèse
109
Matériels et Méthodes
Figure MM.1 La sonde Fura 2 116
Figure MM.2 Dispositif expérimental de la mesure des entrées calciques 117 Figure MM.3 Rep se tatio s d’e egist e e t t pe et des pa a t es esu s pou
l’a al se des i flu al i ues
121
Figure MM.4 Fe t age t pe issu du a uage de l’apoptose pa A e i e-V/ Iodure de propidium
123
Figure MM.5 Structure de base du Phos-tagTM 131
Figure MM.6 Principe du Phos-tagTM 132
Figure MM.7 Principe de la sonde TaqMan® 144
Figure MM.8 P i ipe de l’age t i te ala t SYBR®G ee 142
Figure MM.9 Mod le d’a al se de sultat e RT-qPCR 144
Résultats
Figure R.1 Expression du transcrit orai1 182
Figure R.2 Expression du transcrit stim1 183
Figure R.3 Expression des transcrits ARNm trpc1/3/4/6 dans les lignées 32D et Ba/F3 184 Figure R.4 Co pa aiso de l’e p essio des p ot i es STIM , O ai et TRPC da s la
lignée Ba/F3.
185
Figure R.5 Co-localisation des acteurs calciques dans les cellules 32D 186 Figure R.6 Immunoprécipiation de STIM1 dans les cellules 32D 188 Figure R.7 Effet du t aite e t à l’I ati i da s les ellules D 189 Figure R.8 Etats de phosphorylation des principaux acteurs SOCs révélés par la
molécule Phos-tagTM
191
Figure R.9 Etats de phosphorylation des protéines NFAT1 et NFAT2 dans les lignées 32D
193
Figure R.10 Translocation des protéines NFAT1 et NFAT2 dans les cellules 32D 194 Figure R.11 Implication des SOCEs da s l’a tivatio de la voie NFAT da s les ellules
32D
195
Figure R.12 I pli atio de l’a tivit t soi e ki ase de B -Abl da s l’a tivatio de la voie NFAT dans les cellules 32D
196
Figure R.13 I pli atio des SOCEs et de l’a tivit t soi e ki ase de B -Abl dans l’a tivatio de la voie NFAT da s les ellules D
198
Figure R.14 Courbes effet/dose des inhibiteurs des SOCEs dans la lignée cellulaire 32D après 48h d'exposition
199
Figure R.15 Implication des SOCEs dans la prolifération des cellules exprimant Bcr-Abl 200 Figure R.16 Evolution de nombre de cellules en fonction du temps de culture en
présence de différentes doses d'Imatinib
201
Figure R.17 I pli atio de l’a tivit t osi e ki ase et des SOCEs dans les cellules exprimant Bcr-Abl
202
Figure R.18 Entrée en apoptose des cellules traitées avec les inhibiteurs des SOCEs 204 Figure R.19 Entrée en apoptose des cellules traitées avec les inhibiteurs calciques
o i s ave l’I ati i
205
INDEX DES TABLEAUX
Tableau Titre Page
Introduction - Partie II : Calcium, Canaux et Physiologie
Tableau I.1 Les propriétés des TRPC chez les vertébrés 68 Tableau I.2 Les principales protéines accessoires impliquées dans la régulation des
SOCEs
84
Tableau I.3 Pharmacologie des canaux CRAC et SOCs 89
Matériels et Méthodes
Tableau MM.1 Liste des ilieu utilis s pou les esu es d’i flu calciques 117 Tableau MM.2 Liste des p oduits pha a ologi ues utilis s da s l’ tude des p op i t s
des influx calciques
120
Tableau MM.3 Concentrations utilisées des p oduits pha a ologi ues da s l’ tude de la prolifération cellulaire
122
Tableau MM.4 Co e t atio s utilis es des p oduits pha a ologi ues da s l’ tude de l'apoptose spontanée ou induite
125
Tableau MM.5 Composition des différents gels de polyacrylamide 128 Tableau MM.6 Liste des anticorps primaires et secondaires utilisés pour la détection
immunologique des protéines
130
Tableau MM.7 Concentrations des produits pharmacologiques et drogues utilisées dans l’ tude de la t a slo atio de NFAT da s le o au ai si ue leu te ps d’i u atio
135
Tableau MM.8 Liste des anticorps primaires utilisés pour le marquage fluorescent des protéines.
136
Tableau MM.9 Liste des anticorps secondaires utilisés pour le marquage fluorescent des protéines
136
Tableau MM.10 Ta leau apitulatif des ouples d’a o es utilis es pou la RT-PCR 140 Tableau MM.11 Ta leau apitulatif des ouples d’a o es utilis es pou la méthode
SYBR® Green en RT-qPCR
143
Tableau MM.12 Ta leau apitulatif des ouples d’a o es utilis es pou la méthode Taqman® Gene Expression Assays en RT-qPCR.
1
INTRODUCTION
Partie I : La Leucémie Myéloïde Chronique : Généralités
cliniques et biologiques
I.
Définition
La leucémie myéloïde chronique (LMC) est une hémopathie maligne clonale se présentant au d ut hez les patie ts attei ts sous la fo e d u s d o e lop olif atif. Ce de ie correspond à une production clonale de cellules anormales de la lignée granuleuse, issues d u précurseur pluripotent. On constate ainsi une augmentation de la population des granulocytes, a o pag e d u ai tie de la diff e iatio . Da s plus de % des as, la aladie est diagnostiquée durant la phase « chronique » initiale. E l a se e de t aite e t, la LMC p og esse inéluctablement vers une phase dite « accélérée » puis vers une phase finale dite « blastique »
caractérisée par une perte de la capacité de différenciation des progéniteurs myéloïdes entraînant une accumulation de cellules blastiques myéloïdes ou lymphoïdes dans la moelle osseuse et dans la rate ainsi qu'un passage de ces cellules dans le sang. La mort des patients survient dans les six mois suivant le passage en phase blastique.
II.
Historique
La maladie sera décrite pour la première fois en 1845, simultanément par trois scientifiques Bennett, Craigie et Virchow (Virchow et al., 1845 ; Craigie et al., 1845 ; Bennett et al., 1845) après o se atio d a u ulatio de ellules da s le sa g a o pag e d u e spl o galie. En 1856 Virchow utilisera pou la p e i e fois le te e « leukä ie » du g e a ie υ ός « la » et αί α « sang » (Virchow, 1856. Mais e est u u si le plus tard que les observations de Nowell et Hu ge fo d o t pe is de suspe te u e o igi e g o i ue de ette pathologie e d o t a t u il e istait u e elatio de ausalit e t e la LMC, et la p se e d u h o oso e a o ale e t ou t, u ils o t ommé chromosome Philadelphie ou chromosome Ph en raison de sa découverte dans cette ville (Nowell and Hungerford, 1960). Par la suite, les travaux de Rowley démontreront que le h o oso e Ph est u h ide des h o oso es et , fo à la suite d u e t a slo atio réciproque entre les bras longs de ces deux chromosomes (t(9;22)(q34;q11.2))(Rowley, 1973)(Figure I.1).
2
Figure I.1. La translocation t(9 ;22)(q34 ;q11) et la génération du gène chimérique bcr-abl. Le chromosome de Philadelphie est le résultat de la translocation réciproque entre les bras longs des chromosomes 9 et 22, à l'origine de la LMC.
C est e ue l'origine chromosomique de la maladie sera clairement démontrée. En effet, il sera alors établi que le gène c-abl/abl1 (Abelson) du chromosome 9 est transloqué au niveau du gène bcr (Breakpoint Cluster Region) du chromosome 22 (Bartram et al., 1983; Groffen et al.,
1984; Stam et al., 1987), codant alors pour une protéine chimérique du même nom dont l'activité tyrosine kinase est dérégulée (Daley and Baltimore, 1988). Il se a ta li pa la suite ue l a ti it t osi e ki ase d A l est e uise pou la t a sfo atio ellulai e (Konopka et al., 1984; Lugo et al., 1990). En 1990, il a pu être montré par des expériences de transplantation de cellules médullaires infectées par un rétrovirus contenant le gène bcr-abl chez la souris que la présence de ce gène de
fusion, et do l e p essio de la p otéine chimérique Bcr-Abl, était nécessaire et suffisante pour déclencher un syndrome myéloprolifératif comparable à la LMC (Daley et al., 1990; Elefanty et al., 1990; Pear et al., 1998). Cepe da t, il faut ote u à la diff e e de la LMC hu ai e, la phase chronique chez la souris transgénique est quasi inexistante et seule la transformation aigüe est observée, o t a t pa o s ue t les li ites d tude de e od le et de t a spositio à l ho e. En effet, s'agissant de souris transgéniques, l'expression de bcr-abl est retrouvée dès l'embryogénèse signifiant que ce modèle est finalement peu conforme à l'apparition de la maladie chez l'homme. Ce i appo te a oi s la p eu e ue ette aladie a pou o igi e l e p essio de et o og e unique et en fait la cible thérapeutique principale dans le traitement de la LMC. De plus, cette altération chromosomique unique entraînant un phénotype leucémique a fait de la LMC, de par sa
relative simplicité, une pathologie tumorale très étudiée malgré une incidence relativement faible dans la population (environ 15% de toutes les leucémies adultes) (Jabbour and Kantarjian, 2016).
Chromosome 9
Chromosome 22
Gène abl
Gène bcr Gène bcr-abl
Chromosome de Philadelphie Translocation t(9;22)(q34;q11) Cassure chromosomique q34 q11
3
III.
L'origine de la LMC
La t a slo atio e t e le h o oso e et le h o oso e peut do e lieu à l e p essio d isoformes différentes de la protéine chimérique Bcr-Abl et ainsi être retrouvée dans différents types de leucémies, ie ue la plus f ue te soit l isofo e p bcr-abl résultant de la translocation b2a2 (Cf. Partie IV). A noter que la portion de la protéine Abl reste la même mais la longueur de la protéine Bcr peut varier dans sa partie N-te i ale. Ai si, l o og e bcr-abl est présent dans 95% des patients atteints de LMC, dans 15 à 30% des Leucémies Aigües Lymphoblastiques (LAL) de l adulte et à % des LAL de l e fa t (Bennour et al., 2012; Leoni and Biondi, 2015). De plus, il est aussi retrouvé dans 2% des cas de Leucémies Aigües Myéloïdes (LAM) (Faderl et al., 1999; Melo, 1996). L isofo e p /p bcr-abl est retrouvée chez les patients atteints de LAL (Lugo et al., 1990). Chez les patie ts attei ts de LMC, l isofo e p bcr-ablest la plus ep se t e ta dis ue l isofo e
p230bcr-abl est retrouvée dans un sous-groupe de patients développant une leucémie neutrophilique chronique à progression plus lente (Pane et al., 1996).
Figure I.2. Les différentes isoformes issues de la translocation t(9 ;22)(q34 ;q11). Alors que le point de assu e su le h o oso e se le o sta t au i eau de l e o a e de abl, plusieurs points de cassures sont identifiés le long du gène bcr sur le chromosome 22. En fonction du point de cassure impliqué, la translocation résulte en des isoformes de différentes tailles (e1a2, b2a2, b3a2, e19a2) impliqués dans différents types de leucémies. Adaptée de (Faderl et al., 1998).
4
Les l e ts p is à l o igi e de ette a o alie tog ti ue ne sont pas encore connus, la translocation chromosomique entre le chromosome 9 et le chromosome 22 aboutissant au s d o e lop olif atif. E effet, au u e e t de l e i o e e t i au u e p dispositio g ti ue au d eloppe e t de la aladie a été rapporté à ce jour. Cependant, des études épidémiologiques ont montré que l'exposition à des radiations ionisantes augmente le risque de
développer une LMC (Deininger et al., 1998; Preston et al., 1994). Des études in vitro ont d'ailleurs montré ue l e positio de lig es ellulai es à ces mêmes radiations entraîne des translocations chromosomiques dont la translocation t(9;22)(q34;q11.2) (Deininger et al., 1998). De plus, la translocation entre les gènes bcr et abl semble favorisée par leur proximité dans les cellules hématopoïétiques en interphase (Neves et al., 1999). La p se e d u e répétition segmentaire favorisant les interactions chromatiniennes (ou duplicon) à proximité des deux gènes semble également avantager la survenue de cette translocation (Saglio et al., 2002). Néanmoins, même si les e p ie es de od lisatio hez la sou is o t e t ue l o og e bcr-abl est responsable de la LMC, on peut également envisager l e iste e de fa teu s i d pe da ts de l o og e pouvant faciliter la survenue et le développement de la aladie hez l ho e. D ailleu s, il a t appo t que de nombreux sujets sains expriment bcr-abl sans pour autant développer de LMC (Biernaux et al., 1996; Bose et al., 1998), suggérant que la translocation ait lieu dans une cellule déjà engagée dans une voie de différenciation et non dans un progéniteur hématopoïétique, ou suggérant aussi l e iste e d u o t ôle de la oissa e du lo e leu i ue pa le s st e i u itai e. E fi , la présence du chromosome Ph à la fois dans la lignée myéloïde, mais aussi lymphoïde (B, rarement T) chez un même patient LMC, suggère que la translocation a lieu dans une cellule primitive commune, avec un potentiel de différenciation pour toutes les différentes lignées hématopoïétiques (Fialkow et al., 1967) . En effet, e tudia t l h t oz gotie d u g e li au h o oso e X hez des patie tes atteintes de LMC, il a été démontré le caractère clonal de la maladie et suggéré que son origine ait
lieu dans une cellule souche multipotente (Fialkow et al., 1967) (Cf. Partie VI.3).
IV.
Les acteurs principaux de la LMC
La translocation réciproque t(9 ;22)(q34 ;q11) entraîne un réarrangement des gènes situés au voisinage des points de cassure et la constitution, sur le chromosome 22, d u g e de fusio h ide comporta t la pa tie du gène bcr et la pa tie du gène abl. La conséquence moléculaire de cette translocation est la g atio de l o og e bcr-abl qui code pour une protéine chimérique Bcr-Abl (Rowley, 1973).
5
1. Le gène abl et sa protéine
L o ogène Abelson (c-abl) est localisé sur le chromosome 9 en position 9q34. Il comporte 11 exons (appelés 1a-11a), dont le premier possède deux variants (1a et 1b) séparés par un intron de 200 kb. Le locus abl occupe au total 230 kb. Ce gène est transcrit en deux ARN messagers (ARNm) de ou k selo si la t a s iptio se fait à pa ti de l e o a ou . Il code ainsi pour une protéine de 145 kDa (p145abl) dont il e iste deu a ia tes a et issues de l pissage alte atif du p e ie exon. Les deux isoformes diffèrent seulement dans leurs séquences N-terminales (Chissoe et al., 1995).
La grande majorité des points de cassure se situe da s u e zo e i t o i ue à l e t it du gène c-abl, soit e a o t du p e ie e o , soit e t e les e o s a et , soit e a al de l e o 1a (Melo, 1996) (Figure I.3). La translocation t(9 ;22)(q34 ;q11) aboutit finalement à la juxtaposition
des exons 2a à 11a du gène de c-abl à la partie du g e de bcr.
Figure I.3. Le gène c-abl localisé sur le chromosome 9q34. Le gène contient deux premiers exons alternatifs (1a et 1b) suivis par les exons 2a à 11a. Les points de cassure créant le chromosome Ph sont situés dans une région de recombinaison au niveau de l'extrémité 5' du gène soit en amont de l'exon 1b, soit entre les exons 1a et 1b ou encore e a al de l e o a.
La protéine c-Abl est un membre particulier de la famille Src des tyrosine kinases cytoplasmiques. Dans les cellules de mammifères, c-Abl est exprimée de manière ubiquitaire mais
avec différentes localisations intracellulaires dépendantes du type cellulaire. Dans les fibroblastes, la protéine est retrouvée de manière prédominante dans le noyau alors que dans les cellules primaires hématopoïétiques et les neurones, c-Abl est davantage cytoplasmique (Taagepera et al., 1998; Woodring et al., 2002; Zipfel et al., 2000; Zukerberg et al., 2000). En contraste, tous les variants Abl so t e lusi e e t toplas i ues. E fi , alo s ue l isofo e a est da a tage toplas i ue,
1b 1a 2a 3 11a 3a 4a 5a6a 7a 8a 9a10a Gène c-abl (# 9q34) Région de recombinaison ≈ k Transcrit de type 1a Transcrit de type 1b ARNm Abl
6
l isofo e est istoylé à l e t it N-terminale permettant une localisation préférentielle de cette isoforme dans le noyau (Kharbanda et al., 1997).
Au niveau structurel, c-Abl comprend plusieurs domaines distincts dont une partie commune aux kinases Src en N-terminal. En effet, elle possède des do ai es d ho ologie “H “ Ho olog : u do ai e “H , sui i d u do ai e “H et du do ai e atal ti ue “H . Le do ai e “H est le suppo t de l a ti it t osi e ki ase, constitué de deux lobes C et N, où se trouvent localisés le site de liaiso à l ATP, le site ajeu d autophospho latio et le domaine catalytique phosphotransférase. Le do ai e “H pe et l i te a tio a e des p ot i es o po ta t des sidus t osi e phosphorylés ; il est également un régulateur positif du domaine SH1. Enfin, le domaine SH2 est lui-même régulé négativement pa le do ai e “H , ui est à l o igi e d i te a tio s a e des s ue es riches en proline (pour revue : (Hantschel, 2012)). La partie C-terminale contient une séquence d e po t u l ai e NES (Nuclear Export Signal) (Taagepera et al., 1998), 3 séquences de localisation nucléaire NLS (Nuclear Localizarion Signal) (Wen et al., 1996) et enfin un domaine de liaison à l a ti e-F FABD (F-Actin Binding Domain) et un domaine de liaison à l ADN (McWhirter and Wang, 1993; Van Etten et al., 1994) (Figure I.4). Les séquences NES et NLS permettent de contrôler la localisation de la protéine c-Abl qui pourra alors faire la navette entre le noyau et le cytoplasme en fonction des signaux environnementaux (Taagepera et al., 1998).
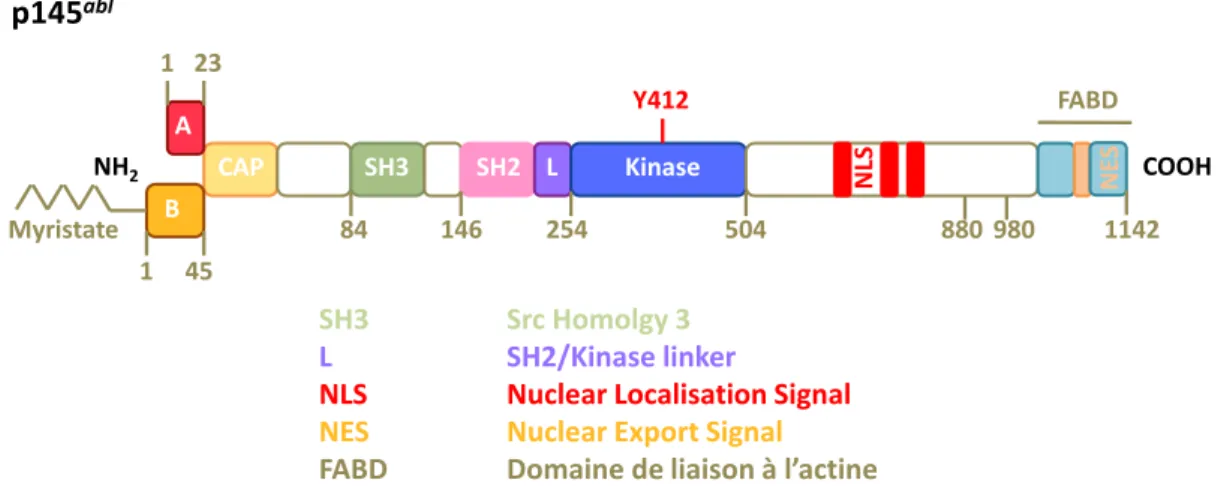
Figure I.4. La structure en domaines de la protéine p145abl. La protéine p145abl
contient plusieurs domaines structuraux importants. Deux isoformes sont générés par épissage alternatif dont l'un qui contient un site de modification de myristoylation. Les différents isoformes contiennent à l'extrémité N-terminale des séquences d'homologie aux Src (SH) et le domaine tyrosine kinase. Ces structures peuvent s'assembler en une conformation autoinhibitrice maintenant ainsi la kinase inactive. Enfin, à l'extrémité C-terminale sont retrouvés des séquences de localisation nucléaire (NLS), ou d'exportation nucléaire (NES), et un domaine de liaison à l'actine (FABD).
Kinase A B CAP SH3 SH2 L NLS NES COOH NH2 Myristate Y412 FABD 1 1 45 23 84 146 254 504 880 980 1142 SH3 Src Homolgy 3 L SH2/Kinase linker
NLS Nuclear Localisation Signal NES Nuclear Export Signal
FABD Do ai e de liaiso à l’a ti e
7
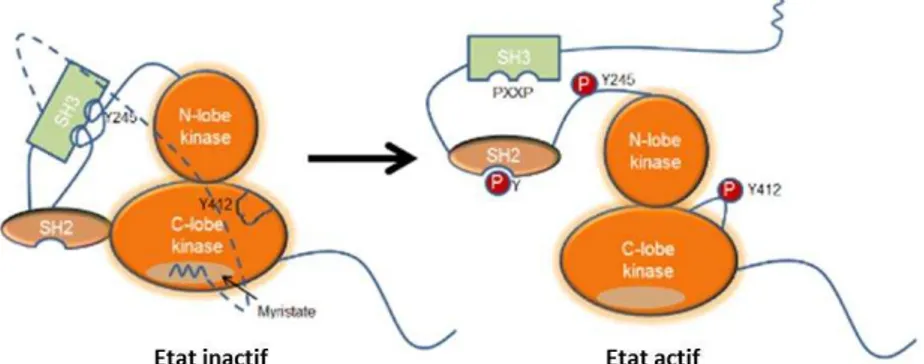
Dans sa forme normal c-Abl se trouve être dans un état fermé (inactif) qui résulte d'interactions propres à la kinase (autoinhibition) (Hantschel and Superti-Furga, 2004). De plus, la présence du groupe myristoyl au sein du lobe C du domaine kinase de c-Abl stabilise cette conformation (Hantschel et al., 2003; Nagar et al., 2003). Le passage à l tat a tif est o te u pa l'intervention d'autres protéines telles que d'autres kinases, qui s'associent à c-Abl et modifient sa
conformation. Dans la conformation fermée, la tyrosine 412 (Y412), située dans le segment d'activation dans le lobe C du domaine tyrosine kinase, n'est pas accessible aux kinases, et prévient la phosphorylation du segment d'activation en le séquestrant (Figure I.5). Ce segment d'activation lorsqu'il est non phosphorylé sur Y412 empêche l'accrochage du substrat de c-Abl et de l'ATP au niveau du domaine catalytique, grâce à cette conformation non adaptée et non reconnue par les
deux substrats (Hantschel et al., 2003; Nagar et al., 2003). Lorsque c-Abl est activée après phosphorylation sur Y412 (mais également Y245 dans le linker SH2/domaine kinase) (Brasher and Van Etten, 2000), elle acquiert son activité tyrosine kinase et peut alors phosphoryler ses substrats
Figure I.5. Rep se tatio de l’ tat i a tif conformation fermée et de l’ tat a tif (conformation ouverte) de la kinase c-Abl. Le verrouillage (état inactif) est assuré par le rapprochement des domaines SH2 et SH3 près des lobes C et N du domaine kinase. Le résidu myrisyoylé da s l e t it N-te i ale de l isofo e se lie à u e po he h d opho e da s le lo e C du do ai e ki ase, permettant la stabilisation de la conformation autoinhibée. L a ti atio de -Abl permet son déverouillage. Adaptée de : (Greuber et al., 2013)
De nombreuses protéines ont été identifiées comme étant phosphorylées par la kinase c-Abl, et la fonction de ces substrats est très diverse. Parmi ces protéines sont retrouvés des adaptateurs, d'autres kinases, des protéines du cytosquelette, des facteurs de transcription, et des protéines
impliquées dans l'organisation de la chromatine (pour revues : (Colicelli, 2010; Wang, 2014)). La kinase c-Abl est capable de phosphoryler aussi bien des protéines nucléaires que cytoplasmiques. En effet, la protéine c-A l est dot e d u e dualit st u tu ale et fonctionnelle, avec des domaines de régulation lui permettant de jouer un rôle à la fois dans le noyau et dans le cytoplasme de la cellule et de transiter entre les deux compartiments (Taagepera et al., 1998). Son action dépend alors de ette lo alisatio u l ai e ou toplas i ue. De o euses tudes fo t esso ti l a tio de c-Abl