HAL Id: tel-02089348
https://tel.archives-ouvertes.fr/tel-02089348
Submitted on 3 Apr 2019HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Protoporphyrie érythropoïétique : thérapie génique non
intégrative par oligonucléotide antisens adressé par
peptides bifonctionnels RTf1-CPP
Arienne Mirmiran
To cite this version:
Arienne Mirmiran. Protoporphyrie érythropoïétique : thérapie génique non intégrative par oligonu-cléotide antisens adressé par peptides bifonctionnels RTf1-CPP. Médecine humaine et pathologie. Université Sorbonne Paris Cité, 2017. Français. �NNT : 2017USPCC184�. �tel-02089348�
1
Thèse de doctorat
de l’Université Sorbonne Paris Cité
Préparé à l’université Paris Diderot
Ecole doctorale BioSPC (Bio Sorbonne Paris Cité)
Equipe hème, fer et pathologies inflammatoires
Titre de la thèse
Protoporphyrie Erythropoïétique :
Thérapie génique non intégrative par oligonucléotide antisens
adressé par peptides bifonctionnels RTf1-CPP
Soutenue Par
Arienne MIRMIRAN
Thèse de doctorat en génétique
Dirigée par Pr. Laurent GOUYA
Présentée et soutenue publiquement à
La faculté de médecine Xavier Bichat
Le 28 mars 2017
Président du jury : Mme Lydie DA COSTA, PU-PH, Université Paris Diderot Rapporteurs : Mr François Maillot, PU-PH, CHU de Tours
Rapporteurs : Mr Emmanuel Richard, MCU-PH, Université de Bordeaux Examinateur : Mr Dominique Germain, PU-PH, Université UVSQ
2
3
SOMMAIRE
Abréviations……….…….……...1
Liste des figures………...……….……….…....5
Liste des tableaux……….……….…9
CHAPITRE I / INTRODUCTION.………10
I/Hème………...10
1- Fonctions de l’hème……….……...…10
2- Structure et des propriétés physico-chimiques des porphyrines et de l’hème…..11
3- Métabolisme de l’hème………...12
A-Différentes étapes de biosynthèse de l’hème chez les mammifères………....12
B-Régulation de biosynthèse de l’hème……….16
1- Régulation hépatique de la biosynthèse de l’hème……….16
a-Régulation transcriptionnelle……….17
b-Régulation post-transcriptionnelle……….…...18
c-Régulation post-traductionnelle……….…....18
2- Régulation érythroïde de biosynthèse de l’hème……….……….…….….18
a-Système IRE/IRP………...20
b-Hème contrôle la transcription des gènes spécifiquement érythroïdes………..22
c-Hème régule la synthèse des protéines lors de la différentiation érythroïde…….…...23
4- Catabolisme de l’hème………....25
5- Biosynthèse de l’hème dans des conditions hypoxiques………...25
6- Trafic de l'hème et ses précurseurs à travers les membranes mitochondriales…26 a-Export de l'ALA des mitochondries………...27
b-Import mitochondrial de COPROgèneIII………...28
c-Export d’hème de la mitochondrie………...……..29
7- FECH……… ………….…...30
a-Gène FECH………..30
b-Structure de l’enzyme FECH……….…………....30
c-Substrats et les inhibiteurs de FECH……….………31
a d-Interaction avec d’autres protéines……….…..32
4
8- Complexe Transferrine/ Récepteur de la Transferrine (Tf/RTf)………….……..35
a-Structure du récepteur de la transferrine1………...36
b-Mécanisme d’internalisation de transferrine par RTf……….………....37
c-Régulation du récepteur de la transferrine1……….……39
II/ Porphyries………....……….41
A-Porphyries hépatiques aiguës………..……....42
1-Symptomatologie neurologique des porphyries hépatiques aiguës……….…...….43
2-Symptomatologie cutanée des porphyries hépatiques aiguës……….….43
3-Diagnostic……….44
4- Traitement d'une crise aiguë de porphyrie………..………44
5-Prévention des attaques aiguës récurrentes……….………….44
B-Porphyrie cutanée tardive (PCT)………..…..44
C-Porphyrie hépato-érythropoiétique……….45
D-Porphyries érythropoïétiques.………...………..45
1-Porphyrie érythropoïétique congénitale (PEC)………..……….45
2-Protoporphyrie érythropoïétique (PPE)……….…..45
A- Caractéristiques physiopathologiques et histopathologiques de PPE……..……..45
B- Caractéristiques cliniques……….….47 1-Symptômes dermatologiques………....…..47 2-Complications hépatiques……….…..47 3-Complications hématologiques……….…..48 C- Génétique de la PPE……….….….48 1-Déficit en FECH……….…………..48 D- Épidémiologie de la PPE………...50 E- Diagnostic de la PPE……….….….50 F- Traitement de la PPE………..……….…...51
1-Mesures préventives de la photosensibilité……….……...51
2-Gestion des complications hépatiques………...…….52
3-la porphyrie dominante liée à l’X (PPEDLX)……….……….53
III/Thérapie génique de la maladie PPE……….………...….54
1- Thérapie génique intégrative………...………….….54
5
2- Thérapie génique non intégrative………..………56
IV/Thérapie génique utilisant des oligonucléotides antisens………..59
1- Oligonucléotides modifiant l’épissage, SSOs………60
2- Limitation de l’utilisation des oligonucléotides antisens……….………62
3- Vectorisation d’oligonucléotides antisens……….62
4- Cell Penetrating Peptides (CPPs)……….……...…..65
A-Origine des CPPs………..………..65
B-Classification des CPPs………...………....66
i- CPPs cationiques………..………....….66
ii- CPPs amphipathiques………..……...…..68
iii- CPPs hydrophobes………..……...…...68
C-Stratégies de vectorisation des acides nucléiques par les CPPs……….……….68
i- Conjugaisons covalentes………...…....68
ii- conjugaisons non-covalentes………..…..……....70
D-Mécanisme d’internalisation des nanocomplexes CPP/acide nucléique……….……...71
E-Echappement endosomal………...…….74
F-Adressage ciblé de nanocomplexes CPP/oligonucléotide………....……….76
G-Adressage subcellulaire des acides nucléiques………...….…….78
H-Poly (oligo-D-arginine) avec le pont disulfure interne ………..…….……79
I-PepFect14………..80
J-Peptides ciblant RTf1 humain………...……….81
V/ Objectif du projet……….………...83
CHAPITRE II/ MATERIELS ET METHODES………85
1-Synthèse des peptides et les oligonucléotides antisens……….85
2-Culture cellulaire……….86
a- Lignée UT7-Epo……….…….86
b- Lignées lymphoblastoïdes (LBCL)………86
c- Fibroblastes……….….87
d- Cellules primaires de progéniteurs érythroïdes à partir de cellules CD34+ périphériques ………...…….. .88
3-Expression de RTf1 (CD71) dans différents lignées cellulaires………...………90
4-Etude de transfection in vitro des nanocomplexes ligands RTf1-CPP/ ASO-V1…...…90 c
6
a- Formation des nanocomplexes peptides/ASOs……….……90
b- Transfection des cellules par les nanocomplexes Peptide/ oligonucléotides……..91
c- Transcription reverse………....91
d- PCR qualitative………...………91
e- PCR quantitative………..………..92
5-Etude de transfection ex vivo des nanocomplexes ligands RTf1-CPP/ ASO-V1 dans des progéniteurs érythroides à partir des cellules CD34+……….……93
a- Mesure de la PPIX intracellulaire……….………93
b- Etude de marquage phénotypique……….94
c- Etude de la morphologie des cellules au cours de la différenciation érythroïde……….…94
d- Test de proliferation………...……….94
6-Détermination de la taille de nanocomplexe P1-9R / ASO-V1………94
7-Mesure de potentiel zêta de nanocomplexe P1-9R/ASO-V1………95
8-Etude de la biodisponibilité intracellulaire des peptides et des nanocomplexes par la microscopie confocale………...………..96
a- Internalisation des peptides dans des fibroblastes humains………...96
b- Internalisation de l’ASO-V1 par le peptide dans des fibroblastes humains………96
CHAPITRE III/RESULTATS……….….…...………97
1-Expression de CD71 (RTf1) dans différents lignées cellulaires……….…………..97
2-Recherche du meilleur ratio de charge entre peptides et ASO en utilisant des lignées lymphoblastoides de malades PPE………98
3-Cinétique de l’effet des complexes RTf1-CPP/ASO dans le temps……….…………..101
4-Etude de l’impact quantitatif sur l’ARN FECH WT de la transfection de l’ASO-V1 par des nanocomplexes RTf1-CPP dans des lignées lymphoblatoides de malades PPE……….…103
5- Etude de l’efficacité de la transfection de l’ASO-V1 par les peptides bi fonctionnels RTf1-CPP dans les progéniteurs érythroïdes d’un sujet porteur de l’allèle hypomorphec.31548C………..……….108
5- Etude de l’efficacité de la transfection de l’ASO-V1 par les petidesbifonctionnels RTf1-CPP précédemment sélectionnés, dans les progéniteurs érythroïdes d’un malade PPE………....111
7
i- Patient A………..……… 112
a-Impact des nanocomplexes Peptide/ASO sur la prolifération et la viabilité des cellules………....112
b-Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique……….112
c- Quantité d’ARN FECH WT………113
d- Mesure de la PPIX……….….. 114
ii- Patient B………...…115
a-Impact des nanocomplexes Peptide/ASO sur la prolifération et la viabilité des cellules……….………...116
b-Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique……….116
c- Quantité d’ARN FECH WT……….…………...116
d- Mesure de la PPIX………117
iii- Patient C………...………118
a-Impact des nanocomplexes Peptide/ASO sur la prolifération et la viabilité des cellules………118
b-Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique……….…119
c-Quantité d’ARN FECH WT, Peptide B2-9R………..…………119
d-Mesure de la PPIX, Peptide B2-9R……….……….120
e- Quantité d’ARN FECHWT, Peptide P1-9R………...………121
f- Mesure de la PPIX, Peptide P1-9R………..…122
g-Etude phénotypiques de la différenciation érythroïde, Peptide P1-9R………123
h- Morphologies des cellules, P1-9R et B2-9R………124
7-Mesure de la taille des nanocomplexes par DLS et de leur potentiel zêta………126
8-Etude de la biodisponibilité intracellulaire des peptides et nanocomplexes par la microscopie confocale………...…………128
CHAPITRE IV/DISCUSSION.………...………..….……132
CHAPITRE V/PERSPECTIVES………..……….139
Référence………...………142
e
1
Abréviations principales
ABCB10: ATP Binding Cassette Subfamily B Member 10 ACO1 : Aconitase 1ALA : Acide δ-Aminolévulinique
ALA-D : Acide delta aminolévulinique déhydratase ALAS : Acide δ-Aminolévulinique synthase
ALAS1 : Acide δ-Aminolévulinique synthase 1 ALAS2 : Acide δ-Aminolévulinique synthase 1 AntimiR: anti-miRNA
ASC : Anémie Sidéroblastique Congénitale ASO: Anti-Sense Oligonucléotide
ATPiF1: ATPase inhibitory Factor 1 CFUs: Colony-Forming Unit
CH: Coproporphyrie Héréditaire CMF: Cytomètrie en flux
COPROgène I : Coproporphyrinogène I COPROgène III : Coproporphyrinogène III CPOX : Coproporphyrinogène oxydase CPP : Cell-Penetrating Peptides
DALAD: Déficit en Acide Delta-AminolévuliniqueDéhydratase DLS : Dynamic Light Scattering
DMD : Dystrophie Musculaire de Duchenne Egr-1: Early growth response protein 1 EPO: Erythropoïétine
ERC: Endosomal Recycling Compartment FECH: Ferrochélatase
2 FOXO1: Forkhead box protein O1
FSC: Forward Scatter FXN: Frataxine
GAG: GlycosAminoGlycanes HDAC3: Histone Deacetylase 3 HIF: Hypoxia Inducible Factors HJV: Hemojuvelin
HMBS: Hydroxyméthylbilane synthase HO-1: Heme oxygenase 1
HRE: Heme Responsive Element HRI: Heme Regulated Inhibitor HRM: Heme Regulatory Motif HS: Héparanes Sulfates
KEAP1: Kelch-like ECH-associated protein 1 IRE: Iron Responsive Element
IRP: Iron Regulatory protein LONP1: Lon peptidase 1 LNA: Locked Nucleic Acid MARE: Maf Recognition Element MFRN1: Mitoferrin-1
MFS: Major Facilitator Superfamily MGG: May-Grünwald-Giemsa miRNA: microRNAs
MOE: Methoxy-ethyl
MSH: Melanocyte Stimulating Hormone NCoR : Nuclear receptor CoRepressors
3 ON: oligonucléotide
PAI: Porphyrie Aiguë Intermittente PBG : Porphobilinogène
PBGS: Porphobilinogène synthase PBGD : Porphobilinogène désaminase PC: Porphyrie Cutanée
PEC: Porphyrie Erythropoïétique Congénitale
PGC-1α: Peroxisome proliferator-activated receptor Gamma Coactivator 1-alpha PGRMC1: Progesterone Receptor Membrane Component 1
PHA : porphyries hépatiques aiguës PHE: Porphyrie Hépato-Erythropoiétique PLP: Pyridoxal 5' Phosphate
PMO: Phosporodiamidate Morpholino PNA: Peptide Nucleic Acid
PPE: Protoporphyrie Erythropoiétique
PPEDLX: Protoporphyrie Erythropoiétique Dominante Liée à l’X PPIX : Protoporphyrinogène IX
PPOX : Protopophyrinogène oxidase PROTOgène IX : Protoporphyrinogène IX PTD:Protein Transduction Domain PV: Porphyrie Variegata
Reα: orphan nuclear receptor Rev-erbα RTf : Récepteur de la transferrine RVG : Rabies Virus Glycoprotein SCARA: Scavenger Receptor Class A siRNA: Small Interfering RNAs
4 SSC: Side Scatter
SSO: Splice Switching Oligonucleotide
STAT5: Signal Transducer and Activator of Transcription 5 SUCLA2: Succinyl-CoA synthetase
URO-D : Uropophyrinogène décarboxylase UROgène I : Uroporphyrinogène I
UROgène III : Uroporphyrinogène III UROS : Uroporphyrinogène synthase III VHL: Von Hippel Lindau
5
Liste des figures
Figure 1) Structure des porphyrines et l’hème……….………...……..11
Figure 2) La première étape de la voie de biosynthèse de l'hème……….…………...13
Figure 3) Les différentes étapes enzymatiques de la voie de biosynthèse de l'hème………..15
Figure 4) Régulation hépatique de biosynthèse de l’hème………...16
Figure 5) Mécanismes de régulation transcriptionnelle d’ALAS1 par l’hème………...17
Figure 6) Régulation érythroïde de biosynthèse de l’hème……….………..20
Figure 7) Modulation post-transcriptionnelle par le fer via le système IRE/IRP.…...21
Figure 8) Modulation post-transcriptionnelle par l’hème via le système IRE/IRP……...22
Figure 9) L’implication de BACH-1 dans la régulation de la transcription des gènes tels que l’hème oxygénase et les globines………...23
Figure 10) L’hème contrôle la traduction des ARNm HBA et HBB dans les progéniteurs érythroïdes en différenciation……….……...24
Figure 11) La stabilité des protéines HIF est dépendante de la biodisponibilté du fer et l’oxygène……….………...26
Figure 12) Le trafic de l'hème et ses précurseurs à travers les membranes mitochondriales……….………..………..28
Figure 13) Structure de la FECH………..………..……31
Figure 14) La ferrochélatase, localisation et partenaires………...…………...…33
Figure 15) Modèle proposé du complexe mitochondriale de protéines impliquées dans le métabolisme de l’hème………...……….…...…..34
Figure 16) Structure du récepteur de la transferrine………..………...…...…37
Figure 17) Endocytose médié par le récepteur de la transferrine………..……...…..….38
Figure 18) Biosynthèse de l’hème : enzymes, métabolites et porphyries………...…..…41
Figure 19) Caractéristiques cliniques des porphyries………...……...….42
Figure 20) Aperçu de la physiopathologie de la PPE………...…..46
Figure 21) Les lésions chroniques, les rainurages autour des lèvres……..…………....…….47
Figure 22) Co-transmission d'une mutation FECH délétère en trans d’allèle hypomorphe c.315-48C……….……….…………...49
Figure 23) Représentation schématique de l’épissage dans l'exon 3-exon4 du gène FECH………...………..……50
6
Figure 25) Le niveau normal de ferrochélatase exprimé dans la peau protègerait la souris
contre la photosensibilité………..……….…….……..56
Figure 26) Emplacement de l’oligonucléotide antisens corrigeant l’anomalie d’épissage du gène FECH……….….………..57
Figure 27) Effet d’ASO-V1 transfecté par le lipofectamine 2000 dans des lignées lymphoblastoïdes issues de patients PPE……….…….….……….57
Figure 28) Preuve de concept dans des progéniteurs érythroïdes de patients PPE………...58
Figure 29) Mécanismes antisens……….….……….…...59
Figure 30) Mécanismes de modulation de l'expression génique induite par l'oligonucléotide……….…………..…...61
Figure 31) Les barrières membranaires limitant la distribution des acides nucléiques aux compartiments subcellulaires………...….…..…………63
Figure 32) Différents systèmes pour le transfert de gènes……….….…………..65
Figure 33) Exemple d’une conjugaison covalente de type thiol-maléimide entre le vecteur peptidique et l’oligonucléotide……….………….………...69
Figure 34) La formation du nanocomplexe entre les peptides et l’oligonucléotide par le biais des interactions électrostatiques, ou hydrophobes et/ou de l’hydrogène………..…...70
Figure 35) Mécanismes d'entrée de CPPs dans les cellules cibles………....72
Figure 36) La route de l'endocytose médiée par un récepteur………....……….……74
Figure 37) Favorisation d’effet d’éponge à proton par les polymères protonnables…...…75
Figure 38) La stratégie d’adressage d’ASO-V1 aux progéniteurs érythroïdes………..84
Figure 39) Pédigrée de la famille PPE………87
Figure 40) Le potentiel zêta représente la charge de surface d’une nanoparticule….….…..95
Figure 41) Mesure de l’expression de CD71 par cymomètre en flux……….…….………….97
Figure 42) Exemple de la mesure qualitative de l’effet d’une gamme de ratio sur l’épissage exon3-exon4 du gène FECH par des ligands RTf1 conjugués au CPP 9R……….………….99
Figure 43) Exemple de la mesure qualitative de l’effet d’une gamme de ratio sur l’épissage exon3-exon4 du gène FECH par des ligands RTf1 conjugués au PepFect14………100
Figure 44) Cinétique de l’effet sur l’épissage en lignées lymphoblastoïdes PPE……...…...102
Figure 45) Cinétique de l’effet sur l’épissage en lignée UT7-EPO……….………..…..102
Figure 46) Quantification de l’ARN FECH WT après transfection de nanocomplexes P2-9R/ASO………..………..104
7
Figure 47) Quantification de l’ARN FECH WT après transfection de nanocomplexes P1-9R/ASO………...……….105 Figure 48) Quantification de l’ARN FECH WT après transfection de nanocomplexes B2-9R/ASO……….………..………...106 Figure 49) Quantification de l’ARN FECH WT après transfection de nanocomplexes B18-PepFect14/ASO………...107 Figure 50) Redirection de l’épissage du site cryptique vers le site physiologique exon3-exon4 par les nanocomplexes RTf1-CPP/ASO-V1 en culture de progéniteurs érythroïdes d’un sujet porteur de l’allèle hypomorphe……….……...110 Figure 51) Impact des nanocomplexes Peptide/ASO sur la prolifération des cellules après la transfection de progéniteurs érythroïdes d’une malade PPE (Sujet A)………….………...112 Figure 52) Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique après transfection de progéniteur érythroïdes d’un patient PPE (Sujet A)………….….…112 Figure 53) Augmentation de la quantité d’ARN FECH WT après la redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet A)………...………113 Figure 54) Mesure de la production de la PPIX après une redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet A)……….………114 Figure 55) Impact des nanocomplexes Peptide/ASO sur la prolifération des cellules après la transfection de progéniteurs érythroïdes d’une malade PPE (Sujet B)………..…...115 Figure 56) Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique après transfection de progéniteurs érythroïdes d’un patient PPE (Sujet B)……….116 Figure 57) Augmentation de la quantité d’ARN FECH WT après la redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythoïdes d’un malade PPE (Sujet B)……….………116 Figure 58) Mesure de la PPIX après une redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet B)………...………117 Figure 59) Impact des nanocomplexes Peptide/ASO sur la prolifération des cellules après la transfection de progéniteurs érythroïdes d’un malade PPE (Sujet C)………..118 Figure 60) Redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique après transfection de progéniteurs érythroïdes d’un patient PPE (Sujet C)………119
8
Figure 61) Augmentation de la quantité d’ARN FECH WT après la redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs
érythroïdes d’un malade PPE (Sujet C)………..……….119
Figure 62) Mesure de la production de PPIX après une redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet C)………..………...120
Figure 63) La quantité d’ARN FECH WT après la redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet C)……….………..…121
Figure 64) Mesure de la production de PPIX après une redirection de l’épissage exon3-exon4 du site cryptique vers le site physiologique dans des progéniteurs érythroïdes d’un malade PPE (Sujet C)……….………122
Figure 65) Effet des nanocomplexes P1-9R/ASOs sur la différenciation érythroïde dans des progéniteurs érythroïdes d’un malade PPE(C)……….………...123
Figure 66) Effet des nanocomplexes peptide/ASOs sur la différenciation érythroïde dans des progéniteurs érythroïdes d’un malade PPE(C)………....124
Figure 67) Mesure du potentiel Zêta dans de l’eau………...………..127
Figure 68) Mesure du potentiel Zêta dans du PBS………...…………...128
Figure 69) Co-localisation entre les peptides et la Tf……….129
Figure 70) Co-localisation entre Tf Alexa 647 et ASO-V1 Alexa 594……….……….….130
9
Liste des tableaux
Tableau 1) Principales hémoprotéines humaines et leurs fonctions………..……..…10
Tableau 2) Exemple des peptides cationiques………...….66
Tableau 3) Exemple des peptides amphipathiques………...….68
Tableau 4) Exemple des peptides hydrophobes……….…....68
Tableau 5) Exemples de CPPs conjugués directement aux oligonucléotides et leurs applications………....69
Tableau 6) Exemples de nanocomplexes CPP/oligonucléotide et leurs applications…….….71
Tableau 7) Exemples des modifications favorisant l’échappement endosomal des CPPs et d’ONs……….76
Tableau 8) Peptides ciblant le RTf1 humain……….…….82
Tableau 9) Liste des peptides ligands RTf1 +/- CPP……….……86
Tableau 10) Caractéristique clinique et biologique des patients PPE et du sujet contrôl porteur de l’allèle hypomorphe………...……88
Tableau 11) Protocole de préparation du milieu de culture des CD34+ en fonction des jours de culture………...……89
10
CHAPITRE I: INTRODUCTION I/ Hème
1-Fonctions de l’hème
L’hème est le groupement prosthétique d’un ensemble de protéines appelées hémoprotéines. Elles sont impliquées dans de nombreux processus cellulaires tels que le transfert d'électrons, le transport et le stockage musculaire de l’oxygène, la détoxification cellulaire et l'apoptose (Tab.1)1. De plus, l’hème exerce un rôle régulateur essentiel. Des études récentes révèlent le rôle de l’hème dans la maturation des microARN et le contrôle du cycle circadien 2,3. L’hème inhibe les canaux dépendants du calcium (par exemple les canaux BK SLO-1) 4 et exerce tout un ensemble de régulation par sa capacité à se fixer sur des protéines ayant des motifs HRM (Heme Regulatory Motif). Ces régulations concernent aussi bien la régulation de la transcription (par exemple : les gènes CYC1, CYC2, CTT1) que la régulation de la traduction par HRI (Heme-Regulated Inhibitor) et des régulations post-traductionnelles (ALAS1, IRP1, BACH1). Cette molécule est également un effecteur dans plusieurs voies cellulaires telles que la différenciation des tissus neuronaux, adipeux et érythroïde. Bien que l'hème soit synthétisé dans chaque cellule humaine pour les respirations cellulaires et des réactions d’oxydoréduction, il est très majoritairement produit dans les cellules érythropoïétiques pour la synthèse de l’hémoglobine et dans les cellules parenchymateuses hépatiques pour la synthèse des cytochromes P450. L’ensemble de ces fonctions rend la molécule d’hème indispensable à la vie. Le contrôle de la production de l'hème diffère entre ces deux tissus, principalement en raison de la différence dans les taux de synthèse de l'acide 5-aminolévulinique. Les macrophages de la rate et du foie dégradent l’hème et recyclent le fer après l’erythrophagocytose de l’hème par l’hème oxygénase-1 5.
Hémoprotéines Rôle principal
Hémoglobine Transport O2 Myoglobine Stockage O2 HRI (Heme Regulator Inhibitor) Régulation traductionnelle
Cytochromes mitochondriaux Transfert d’électrons Péroxydase Oxydation
Catalase Dismutation H2O2 Tryptophane dioxygénase Dégradation du tryptophane
Cytochromes Transfert d’électrons, oxydation Cyclooxygénase Synthèse de prostaglandines Guanylate synthase Transport du NO Oxyde nitrique synthase Synthèse du NO
11
2-Structure et des propriétés physico-chimiques des porphyrines et de l’hème
Les porphyrines, molécules précurseurs de l’hème, sont des pigments rouges, fluorescents, de structure cyclique, aromatique et tétrapyrrolique. Les noyaux pyrroles A, B, C, D sont liés entre eux par des ponts méthènes (-CH=) entre deux carbones α (Fig.1). Le suffixe «inogène» indique un état réduit des ponts méthènes tandis que le suffixe «ine» représente l’état oxydé. Seuls les dérivés oxydés sont colorés et fluorescents. La détection et la quantification des porphyrines par spectrofluoromètrie dans les milieux biologiques est possible car les porphyrines libres ou complexées à certains métaux (ex : Zinc) émettent une fluorescence rouge intense quand elles sont exposées à la lumière violette (longueur d’onde d’excitation : 405nm, dite bande de Soret). A B
Figure 1) Structure des porphyrines et l’hème. A) Les porphyrines sont des pigments aromatiques (Rouge) et tétrapyrroliques (Bleu). Les noyaux pyrroles A, B, C, D sont liés entre eux par des ponts méthènes (-CH=) entre deux carbones α. B) L’atome de fer (Violet) se fixe au centre de la porohyrine par les atomes d’azote (Vert) pour former l’hème.
Les complexes métalliques de porphyrine jouent un rôle important dans les activités biologiques comme par exemple des complexes de fer dans les hémoprotéines, les complexes de magnésium dans les chlorophylles, et un complexe de cobalt dans la vitamine B12. L’hème est un cofacteur constitué d'un anneau organique hétérocyclique de porphyrine, qui contient un ion central de fer à l’état ferreux (Fe2+).
12 3-Métabolisme de l’hème
La biosynthèse de l’hème est un processus complexe qui se produit dans toutes les cellules grâce à huit réactions enzymatiques réparties entre les mitochondries et le cytosol. La première ainsi que les trois dernières prennent place dans la mitochondrie tandis que les étapes intermédiaires sont cytoplasmiques (Fig.3).
A-Différentes étapes de biosynthèse de l’hème chez les mammifères
• Etape I
La première étape de la voie de biosynthèse de l'hème est la condensation du succinyl-CoA et de la glycine pour former l'acide δ-aminolévulinique (ALA) dans la matrice mitochondriale (Fig.2). Cette réaction est catalysée par l’enzyme ALA synthase (ALAS; EC 2.3.1.37). L’ALA est le précurseur général pour tous les tétrapyrroles naturels et représente la seule source de carbone et les atomes d'azote nécessaires pour la formation de l'hème. Le premier rapport sur l'incorporation des atomes d'azote provenant de la glycine en hème a été publié en 1945, par Shemin et Rittenberg 6. Après l'identification de l'ALA comme un précurseur potentiel de la biosynthèse de l'hème, l’activité de l’ALA synthase a été simultanément décrite en 1958 par les groupes de Shemin et Neubergeren 7 et Laver WG et Neuberger A 8. Il existe deux isoformes d’ALAS : ALAS1 et ALAS2, codées par deux gènes. Leur homologie de séquence est de 75 % au niveau du domaine catalytique. Les isoformes d’ALAS sont les seuls dans cette voie qui sont codées par deux gènes différents. Le gène AlAS1 est situé sur le chromosome 3 et est exprimé de façon ubiquitaire. Ce gène contient 12 exons et code pour une protéine fonctionnelle de 640 acides aminés. Le gène ALAS2 est situé sur le chromosome X et contient 11 exons. Ce gène code pour une protéine fonctionnelle de 587 acides aminés et est exprimé uniquement dans les cellules érythroïdes 9.
13
Figure 2) La première étape de la voie de biosynthèse de l'hème. La première étape de la voie de biosynthèse de l’hème se déroule dans la matrice mitochondriale. Elle consiste à la condensation du succinyl-CoA et de la glycine pour former l'acide δ-aminolévulinique (ALA). Cette réaction requiert le PLP (Pyridoxal 5'-Phosphate) qui est une coenzyme dérivée d’une vitamine, la Pyridoxine.
• Etape II
La deuxième étape de la biosynthèse de l’hème se déroule dans le cytosol. Elle consiste en la condensation de deux molécules d’ALA par l’acide delta aminolévulinique déhydratase (ALA-D, E.C.4.3.1.8) pour former la molécule monopyrrolique de porphobilinogène (PBG). La protéine delta aminolévulinate déhydratase nommée aussi sous le nom de porphobilinogène synthase (PBGS) est composée de 8 sous unités identiques et présente 4 sites catalytiques. Le gène ALA-D est localisé sur le chromosome 9 et contient 2 exons alternatifs non codants, 1A et 1B, et 11 exons codant, 2 à 12. Les exons 1A et 1B sont épissés à l’exon 2 de façon alternative. Les exons ont 2 promoteurs distincts qui sont activés suivant les tissus: ubiquitaire pour 1A et érythroïde pour 1B 10. L'activité de l’ALA-D est activée par des groupements thiols et du zinc. L’intoxication par le plomb entraîne une inhibition de l’ALA-D en raison du remplacement des ions de zinc par un autre cation divalent le Pb2+11.
• Etape III
Dans la troisième étape de cette voie, l’hydroxyméthylbilane est généré par des liaisons «tête à queue» de quatre molécules PBG par l'hydroxyméthylbilane synthase (HMBS) ou la porphobilinogène désaminase (PBGD, E.C.4.3.1) 12. Le gène PBGD, situé dans le chromosome 11, est composé de 15 exons qui génèrent deux ARNm épissés de façon alternative codant pour deux isoformes de l'enzyme 13. La forme érythroïde de PBGD possède un promoteur
14
spécifiquement érythroïde situé dans l’intron 1, qui aboutit après transcription à un enchainement exon2-exon3 alors que dans la forme ubiquitaire, la maturation de l’ARN pré-messager aboutit à un enchainement exon1-exon3. Le site d’initiation de la traduction spécifiquement érythroïde est situé dans l'exon 3 14. La protéine fonctionnelle de PBGD possède 361 acides aminés.
• Etape IV
La conversion d’hydroxyméthylbilane en uroporphyrinogène III (UROgène III) nécessite l'action de l’enzyme uroporphyrinogène synthase III (UROS, E.C.4.2.1.75). Cette réaction, catalysée sans cofacteur, est une inversion du noyau pyrrole D d’hydroxyméthylbilane, suivi d’une cyclisation pour donner UROgène III. Le gène uroporphyrinogène synthase III est situé sur le chromosome 10 et est composé de 16 exons codant pour une protéine de 265 acides aminés. En absence de l’enzyme HMBS, l'hydroxyméthylbilane se cyclise spontanément en uroporphyrinogène I (UROgène I) qui se transforme en coproporphyrinogène I (COPROgène I) par une décarboxylation. Le COPROgène I est une impasse métabolique de la synthèse de l'hème du fait de son incapacité à être transporté.
• Etape V
Dans le cytosol, les chaînes latérales du macrocycle UROgène III sont décarboxylées séquentiellement par l’uropophyrinogène décarboxylase (URO-D, E.C.4.1.1.37) pour former le coproporphyrinogène III (COPROgène III). Le gène UROD est situé sur le chromosome 1 et contient 10 exons qui génèrent deux ARNm épissés de façon alternative. Un seul ARNm code pour l'enzyme fonctionnelle de 367 acides aminés.
• Etape VI
Toutes les étapes restantes de la biosynthèse de l'hème prennent place à l'intérieur des mitochondries. Le COPROgène III est importée dans l'espace inter-membranaire mitochondrial, où il est transformé en protoporphyrinogène IX (PROTOgène IX) par la coproporphyrinogène oxydase (CPOX, E.C.1.3.3.3). Le gène CPOX est localisé sur le chromosome 3 et est constitué de 8 exons codant pour une protéine de 454 acides aminés. Ce gène possède un promoteur unique qui est régulé différemment dans les tissus érythroïdes et non érythroïdes. Il exprime deux ARNm distincts grâce à l’utilisation de deux signaux de polyadénylation alternatifs.
• Etape VII
L’avant dernière étape de la biosynthèse de l’hème est l'oxydation du PROTOgène IX en protoporphyrine IX (PPIX) par la protopophyrinogène oxidase (PPOX E.C.1.3.3.4). Cette réaction nécessite trois molécules d’oxygène et entraîne la perte de six protons et six électrons, ce qui génère trois molécules de peroxyde d’hydrogène. La PPOX est une flavoprotéine synthétisée
15
dans le cytoplasme et est transloquée vers la mitochondrie où sa localisation exacte reste soumise à débat. Le gène PPOX est situé sur le chromosome 1, et est composé de 14 exons codant pour une protéine de 477 acides aminés.
+ ALA synthétase Glycine Hème Me Me Pr Pr Me Me NH NH HN NH Vi Vi Fe2+ Protoporphyrine IX PROTOgène oxydase PROTOgène IX Ferrochélatase COPROgène oxydase COPROgène III Me Pr Pr Pr Pr Me Me Me NH NH HN NH Me Pr Pr Pr Pr Me Me Me NH NH HN NH COPROgène I Ac Pr Pr Pr Pr Ac Ac Ac NH NH NH HN UROgène III Pr Ac UROgène I Pr Ac Ac Pr Pr Ac NH NH NH HN NH 2 CH -COOH CH2-NH2 ALA PBG UROgène décarboxylase PBG désaminase Hydroxyméthylbilane Pr Me Me Pr Me Me NH Vi Vi N N HN 2 COOH ' CH2 NH SCoA SuccinylCoA -2 CH2 HOOC CH CO Me Pr Pr Me Me Me NH HN Vi Vi N N N N N N Fe2+N Ac Pr Ac Pr NH NH NH NH HO Ac Pr Ac Pr 2 CH HOOC-2 CH -C CH2-NH2 HOOC-2 CH 2 CH O UROgène III synthétase ALA déshydrase CYTOSOL MITOCHONDRIE
Figure 3) Les différentes étapes enzymatiques de la voie de biosynthèse de l'hème.
• Etape VIII
Enfin, le fer ferreux est incorporé dans la PPIX par la dernière enzyme de la voie de biosynthèse de l’hème, la ferrochélatase (FECH), pour former l’hème dans la matrice mitochondriale. Le
16
premier rapport décrivant l’activité de ferrochélatase dans les érythrocytes aviaires a été publiée en 1956 par Ashenbrucker et al 15. Cependant, il a fallu attendre 25 ans de plus jusqu'à la première purification de la ferrochélatase à partir de mitochondries de foie de rat pour caractériser l’activité FECH. Au début des années 1990, les gènes codant pour la ferrochélatase venant de plusieurs organismes eucaryotes ont été clonés et séquencés. De nos jours, la ferrochélatase est l’une des enzymes de la voie de biosynthèse de l'hème la mieux caractérisée (voir à la fin du chapitre 1).
B) Régulation de biosynthèse de l’hème
1-La régulation hépatique de la biosynthèse de l’hème
La première étape de la biosynthèse de l'hème catalysée par ALAS est l’étape régulée. Dans les cellules non-érythrocytaires, en particulier dans les hépatocytes, l’hème ou son produit oxydé, l’hémine, contrôlent l’activité enzymatique d’ALAS1 par un rétrocontrôle négatif à de multiples niveaux : transcriptionnel, post-transcriptionnel, post-traductionnel en régulant la translocation de l’ALA synthase du cytosol vers la mitochondrie et finalement par une inhibition protéolytique de l’enzyme (Fig.4).
17 a- Régulation transcriptionnelle
Le premier mécanisme implique PGC-1α (Peroxisome proliferator-activated receptor Gamma Coactivator 1-alpha) qui est un coactivateur transcriptionnel du gène ALAS1 16. PGC-1α est régulé soit par le statut nutritionnel via la voie AKT (phosphorylation de FOX-1) et la voie cAMP (phosphorylation de CREB), et par la quantité d’hème libre. Il a été montré que Reα (orphan nuclear receptor Rev-erbα) agit comme un senseur de l’hème dans l’organisme 17. La fixation de l’hème sur Reα entraine le recrutement du complexe corépresseur HDAC3 (NCoR/histone deacetylase 3), ce qui réprime la transcription de PGC-1α 18 et par conséquent la transcription du gène ALAS1 19. Le deuxième mécanisme alternatif de régulation transcriptionnel d’ALAS1 médié par l’hème est basé sur l’identification d’un HRE (Heme Responsive Element) dans la région proximale du promoteur d’ALAS1 chez la souris, ainsi que sur la liaison combinatoire d’Egr-1 (Early growth response protein 1) et ses corépresseurs NBA1 et NBA 2 à HRE 20. L’interaction entre Egr-1 et le corépresseur est renforcée par l’augmentation de la concentration de l’hème intracellulaire, ce qui conduit à la répression de la transcription d’ALAS1. Ces deux mécanismes peuvent réguler de manière synergique la transcription du gène ALAS1, médié par l’hème.
Figure 5) Mécanismes de régulation transcriptionnelle d’ALAS1 par l’hème, modifié d’après Gloria C Ferreira, 2013 21. L’hème libre se lie à Reα et entraine le recrutement du complexe corépresseur NCoR/HDAC3 sur la région «enhancer» du gène PGC-1α. L’hème libre se lie également à Egr-1 et provoque l’interaction entre Egr-1 et ses corépresseurs NBA1 et NBA2 sur l’HRE dans la région proximale du gène ALAS1. La cascade de signalisation de l’insuline diminue la transcription d’ALAS1 par l’inactivation de FOXO1 et CREB, qui sont des régulateurs positifs de la transcription d’ALAS1.
18 b- Régulation post-transcriptionnelle
Chez l'homme, l'épissage alternatif de l’ARN, dans les cellules hépatiques, génère deux transcrits ALAS1 avec des régions non traduites distinctes à l’extrémité 5' (5'-UTR). Dans la forme majeure d’ARNm ALAS1, l’exon 1B est manquant alors que cet exon est conservé dans la forme mineure d’ARNm ALAS1. L’Hème diminue la stabilité de la forme majeure au niveau post-transcriptionnel par l'augmentation de la dégradation de transcrit, alors que la forme mineure est résistant au dommage médié par l’hème 22.
c- Régulation post-traductionnelle
L’hème peut également affecter l’ALAS1 au niveau post-traductionnel. Il existe trois motifs de liaison à l’hème (HRM) du côté N-terminal de la séquence de ciblage mitochondrial de protéine pre-ALAS1. La liaison de l’hème à ce motif entraine une diminution de transport de pré-ALAS1 vers les mitochondries, où la forme mature de protéine est produite après le clivage du signal de translocation 23.
Il a été récemment démontré que l’hème augmente également la dégradation de la forme mature mitochondriale d’ALAS1 via la protéolyse médiée par LONP1, une protéase dépendant de l’ATP qui contrôle le renouvellement sélectif des protéines de la matrice mitochondriale 24. L’autre protéase mitochondriale dépendante de l'ATP qui dégrade l’ALAS1 au niveau post-traductionnel est ClpXP. Ce dernier est un hétéro-multimères de CLPX et CLPP. Il est encore difficile de savoir si ClpXP dégrade directement l’ALAS1, mais il a été suggéré que CLPX reconnaît et déplie d’abord les structures tertiaires des protéines cibles, et CLPP dégrade ensuite la cible dépliée 25. L’ALAS1 subit un changement conformationnel suite à l’association de l’hème à son motif de liaison à l’hème. Ce changement conformationnel renforce la formation d’un complexe entre ALAS1 et la protéase ClpXP dans la mitochondrie, et accélère ainsi la dégradation de la protéine ALAS1 afin de maintenir le niveau de l'hème intracellulaire approprié. Il est possible que LONP1 et ClpXP régulent de manière coopérative la dégradation d’ALAS1 dans les mitochondries des cellules de mammifères 26.
2-Régulation érythroïde de biosynthèse de l’hème
La biosynthèse érythroïde de l'hème est absolument dépendante de l'absorption du fer par des érythroblastes en maturation, car le fer est non seulement nécessaire pour l’étape finale de la production de l’hème, l'incorporation du fer2+ dans le PPIX, mais il contrôle également l'expression d’ALAS2 et l’activité de la FECH. Le fer est acquis par les progéniteurs érythroïdes en différenciation via l’endocytose du RTf1 (Récepteur de la Transferrine 1) et est transféré par la Mitoferrin vers les mitochondries pour la synthèse de l'hème 27. Cependant, lorsque le fer est
19
biodisponible, il stimule la synthèse d’ALAS2 pour fabriquer l’hème afin de produire l’hémoglobine, un tétramère de chaines de globines 2α, 2β, et l’hème. L’ALAS2 est le premier facteur limitant de la voie de biosynthèse de l’hème. L’expression d’ALAS2 est régulée par des facteurs de transcription spécifiquement érythroïdes tels que GATA1. En dépit de son rôle sur la production de l’hème, le fer intra-mitochondrial participe également à la formation des clusters fer-souffre. La FECH ainsi que la protéine régulatrice IRP1 possèdent respectivement un centre [2Fe/2S] et [4Fe/4S]. La FECH est le deuxième facteur limitant de la voie de biosynthèse de l’hème car son expression est augmentée au cours de la différenciation érythroïde et est contrôlée par des facteurs de transcription Sp1, NF-E2 et des éléments de GATA. Au niveau post-transcriptionnel, l'expression de FECH est contrôlée par la disponibilité des clusters fer-soufre néoformés dont la biogenèse dépend du fer, ainsi que des machines d'assemblage de cluster soufre. Le pH mitochondrial joue un rôle important sur l’assemblage et la stabilité du cluster fer-souffre et subséquemment l’activité de ferrochélatase. L’activité FECH serait donc régulée par le facteur inhibiteur ATPiF1 (ATPase inhibitory factor 1) qui module le pH mitochondrial et le potentiel redox. ATPiF1 est un régulateur de l’ATP synthase (ou ATP phosphohydrolase (H+-transporting)). ATP synthase est une protéine à double fonction qui soit synthétise l’ATP en utilisant un gradient H + généré à travers de la membrane mitochondriale ou hydrolyse l’ATP dans une réaction inverse (activité ATPase) en cas d’absence du potentiel de membrane ou du gradient du pH. Campanella M et al ont démontré qu’ATPiF1 stimule l’activité ATP synthase en inhibant son activité ATPase 28. D’après les expériences effectuées dans les modèles de poisson zèbre et les cellules murines portant le gène muté ATPiF1, il a été démontré que l’augmentation du pH au niveau de la matrice mitochondriale a entrainée la diminution de l’activité FECH et ainsi l’hémoglobinisation 29, 30. La régulation négative de FECH pendant une érythropoïèse en carence en fer a été observée chez les souris Irp2-/-, pendant la différenciation érythroïdes des cellules MEL limitée en fer, et enfin dans des conditions d’une biogénèse déficiente de cluster fer-soufre 31.
L’hème, de son côté, joue également des rôles régulateurs multiples au cours de la différenciation des précurseurs érythroïdes étant donné qu'il commande sa propre synthèse et régule l'expression du gène spécifiquement érythroïde majeur de la voie de biosynthèse de l’hème, l’ALAS2 (Fig.6). Un équilibre très précis entre la synthèse de l’hème et des globines est nécessaire car l’hème libre est extrêmement toxique pour l’organisme. La régulation du pool de l’hème libre dans le tissu érythroïde se réalise par trois voies principales :
• Le niveau de biosynthèse de l’hème par l’ALAS2 et la FECH
20
• Export de l’hème par FLVCR1
Figure 6) Régulation érythroïde de biosynthèse de l’hème.
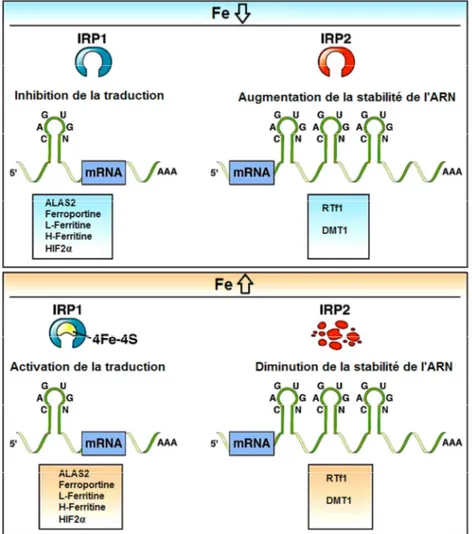
a- Le système IRE/IRP
Les protéines régulatrices IRP1 et IRP2 (Iron Regulatory Protein) sont capables de se fixer sur une séquence spécifique d'ARN messager ayant une structure tertiaire tige boucle nommée IRE (Iron Responsive Element). La liaison d’IRP à un IRE en 5' empêche l’initiation de la traduction tandis que sa liaison à un IRE en 3' protège l'ARNm de la dégradation par des ribonucléases intracellulaires. En situation de biodisponibilité normale du fer, les IRPs ne se fixent pas aux IREs, car IRP1 se complexe avec un cluster fer-soufre [4Fe-4S] pour devenir l'aconitase (ACO1) dont la fonction est actuellement inconnue. Contrairement à IRP1, IRP2 ne se complexe pas avec le cluster fer-souffre et est dégradé par le protéasome (Fig.7). Durant l’érythropoïèse, IRP2 contrôle l’absorption du fer et la biosynthèse de l’hème. Il a été démontré que les souris IRP2 -/-développe une anémie microcytaire hypochrome avec une disponibilité du fer réduite pour l'érythropoïèse. Le niveau d’expression du RTf1 chez les souris IRP2-/- est très faible. Le transcrit du RTf1 contient une séquence IRE en 3' et perd sa stabilité en cas d’absence d’IRP2. Par conséquent, les cellules ne parviennent pas à acquérir des quantités suffisantes du fer pour l’érythropoïèse en dépit de l'approvisionnement du fer physiologique. Ces souris développent également la protoporphyrie érythropoïétique due à l’accumulation élevée du PPIX libre ou Zn-PPIX (~ 200-fois plus de Zn-PPIX libre). Le transcrit d’ALAS2 contient un IRE en 5' et est également soumis à cette régulation post-transcriptionnelle. Lorsqu’Irp2 est invalidé, la traduction d’ALAS2
21
n’est pas inhibée par le mécanisme IRP/IRE et ainsi la biosynthèse de l’hème est suractivée. Cependant, l’absence du fer à un niveau approprié pour la production de l’hème, entraine l’accumulation du PPIX libre ou Zn-PPIX 32,33,34. L'expression de plusieurs autres gènes impliqués dans le métabolisme du fer est aussi régulée par le système IRE/IRP. Les transcrits des H-ferritine, L-ferritine, mAconitase et ferroportine contienne un IRE en 5' tandis que le transcrit de DMT1 possède un IRE en 3' 35. Au total, le système IRE/IRP permet une régulation balancée entre stockage/utilisation/export du fer d’une part et son apport cellulaire et mitochondrial d’autre part.
Figure 7) Modulation post-transcriptionnelle par le fer via le système IRE/IRP, modifié d’après Shah YM et Xie L, 2014 36. Lorsque la concentration du fer est faible, la liaison entre IRP1 et IRP2 avec les ARNm ayant des séquences IREs est renforcée. La liaison d’IRP aux transcrits qui contienne un IRE en 5' tels que L-ferritine, H- ferritine, Ferroportine, ALAS2 réprime la traduction. Alors que la liaison d’IRP aux transcrits qui possèdent un IRE en 3' tels que RTf1 et DMT1, stabilise l’ARNm de la dégradation par des ribonucléases intracellulaires.
Les données suggèrent que l'hème pourrait également favoriser sa propre synthèse au cours de la différenciation érythroïde par la régulation de l’expression d’ALAS2 via le système IRE/IRP. L’hème interagit avec la séquence consensus HRM d’IRP2 et entraine l'oxydation d’IRP2 qui
22
déclenche son ubiquitination et sa dégradation 37 (Fig.8). L’augmentation du niveau de l'hème dans les précurseurs érythroïdes stimule donc la production de protéine ALAS2 due à la perte d’IRP2. En conclusion, l'hème participe au contrôle de l'homéostasie du fer dans les progéniteurs érythroïdes en contrôlant l’oxydation/dégradation d’IRP2.
Figure 8) Modulation post-transcriptionnelle par l’hème via le système IRE/IRP. L’hème interagit avec la séquence consensus d’HRM d’IRP2 et entraine l'oxydation d’IRP2 qui déclenche son ubiquitination et sa dégradation.
b- L’hème contrôle la transcription des gènes spécifiquement érythroïdes
L’hème est impliqué dans la régulation transcriptionnelle de plusieurs gènes tels que l’hème oxygénase-1 (HO-1) et les gènes de globines par l’intermédiaire d’un répresseur de la transcription : BACH-1 38. Ce dernier possède cinq motifs HRM et est spécifique des séquences trans-activatrices MARE (Maf Recognition Element) au niveau des promoteurs. Lorsque l’hème se lie au domaine C-terminal du BACH-1, il inhibe son activité de liaison à l'ADN et induit son dissociation des protéines MAFs, déclenche son exportation du noyau et induit son ubiquitination et la dégradation 39,40,41. Dans des conditions normales, la protéine KEAP1 (Kelch-like ECH-associated protein 1) séquestre NrF2 dans le cytoplasme 42 et par conséquent BACH-1, qui est en compétition avec NrF2, se complexe avec les protéines MAFs. De ce fait, L’expression des gènes est réprimée par la fixation d’hétéro-dimère de BACH-1 et MAF sur la séquence MARE. Au contraire, le stress oxydatif induit par un excès d'hème provoque la dissociation du complexe KEAP1/NrF2 et l'accumulation nucléaire de NrF2 (Nuclear factor erythroïd 2-related Factor-2) 43
. L’hétéro-dimère de NrF2 et MAF se fixent ensemble sur la séquence MARE et stimulent la transcription des gènes (Fig.9).
23
Figure 9) L’implication de BACH-1 dans la régulation de la transcription des gènes tels que l’hème oxygénase et les globines, modifié d’après Yuji Naito, 2011 44. Dans des conditions normales, BACH-1 reconnait des séquences enhancers MARE et réprime la transcription des gènes tels que HO-1. L’augmentation de l’hème inhibe la liaison de BACH-1 à l'ADN et induit sa dissociation des protéines MAFs, déclenche son exportation du noyau et induit son ubiquitination et la dégradation. De ce fait, la séquence MARE est disponible pour les complexes activateurs de transcription y compris Maf/Nrf2.
De plus, l'hème contrôle également la transcription d'autres gènes exprimés de manière ubiquitaire tels que les protéines de stockage du fer, L-ferritin et l’exporteur du fer, la ferroportine par ce mécanisme 45. Ce mécanisme de régulation pourrait contribuer à adapter le métabolisme du fer dans des érythroblastes en fonction de leur stade afin d’assurer une biodisponibilité suffisante du fer pour la synthèse d’hème.
c- L’hème régule la synthèse des protéines lors de la différenciation érythroïde
La synthèse des protéines dans les progéniteurs érythroïdes dépend principalement du niveau de l'hème qui est détectée par HRI (Heme Regulated Inhibitor, eIF2α kinase), un membre de la famille des protéines kinases capables de phosphoryler la sous-unité α du facteur d'initiation de la traduction chez les eucaryotes (eIF2α). Une fois phosphorylée, eIF2α entraine l'inhibition de la synthèse protéique 46. HRI est exprimé de manière prédominante dans le compartiment érythroïde et son activité de kinase est directement régulée par le niveau de l’hème. L’hème inhibe l’activité de phosphorylation en se liant au domaine kinase d’HRI via un motif HRM. Par conséquent, la
24
phosphorylation d’eIF2α est diminuée et le taux de la traduction est augmenté, principalement des ARNm HBA et HBB. D'autre part, lors de la carence en hème, l'activation d’HRI et la phosphorylation ultérieure d’eIF2α provoquent l'inhibition de la synthèse protéique. Ainsi, seulement après l'initiation de la biosynthèse de l'hème dans les progéniteurs érythroïdes en différenciation, l’inhibition de l'activité HRI permet la synthèse des globines α et ß. De cette manière, HRI est fondamental pour coordonner la synthèse d’hème et de chaines de globine évitant ainsi une accumulation d’hème pro-oxydant et des chaines de globine seule (Fig.10). Les souris Hri-/- montrent une anémie macrocytaire hyperchrome légère avec une réponse adaptative altérée à une carence en fer. La réponse adaptative normale à une carence en fer est d'arrêter la synthèse d'hémoglobine, ce qui entraine une anémie microcytaire hypochrome. Les souris Hri-/- soumises à un régime de carence en fer développent une anémie hyperchrome avec une destruction accrue des précurseurs de globules rouges en phase tardive et une hyperplasie érythroïde compensatoire au niveau médullaire. L’excès des chaines de globine, après un déficit en HRI, précipite et forment des inclusions protéiques toxiques, responsable de la mort des progéniteurs érythroïdes. Ces données démontrent clairement le rôle essentiel des HRI pour arrêter la synthèse d’α- et β-globine dans les progéniteurs érythroïdes lorsque le fer est en carence, ce qui évite la précipitation toxique des chaînes de globine 47.
Figure 10) L’hème contrôle la traduction des ARNm HBA et HBB dans les progéniteurs érythroïdes en différenciation, modifié après D.Chiabrando, 2014 48. La traduction des ARNm de globines est régulée par l’hème via la protéine kinase HRI. Ce dernier est inactivé lorsqu’il se lie à l’hème. Par conséquent, le facteur d’initiation de traduction eIF2α n’est pas phosphorylé et entraine l’initiation de synthèse de protéines.
25 4-Catabolisme de l’hème
L’hème libre devrait rapidement être dégradé par l’hème oxygénase (HO), en monoxyde de carbone, biliverdine et fer. La biliverdine est réduite par la biliverdine réductase en bilirubine. La bilirubine est ensuite transportée vers le foie où elle est conjuguée avec un sucre solubilisant appelé l’acide glucuronique. Cette forme plus soluble de bilirubine (conjuguée) est excrétée dans la bile. Le fer est recyclé. Il existe deux isoenzymes catalytiquement actifs pour HO, 1, HO-2. Les gènes humains codant pour HO-1 et HO-2 sont localisés respectivement sur le chromosome 22 et du chromosome 16. HO-1 est une isoenzyme inductible, et son expression est régulée positivement par son substrat "hème" et le stresse oxydatif (voir paragraphe 2b).
5-La biosynthèse de l’hème dans des conditions hypoxiques
L’érythropoïétine (EPO) est une glycoprotéine de 34 kDa, composée de 165 acides aminés sous sa forme circulante. Son rôle principal est la prévention de l'apoptose dans les cellules érythroïdes dépendantes de l'EPO, les CFUs (colony-forming unit) et des érythroblastes qui n’ont pas encore commencé la synthèse de l'hémoglobine. L'hypoxie est l’inducteur physiologique primaire pour la production d'EPO, qui, en fonction des conditions hypoxiques, augmente les taux sériques d'EPO jusqu'à plusieurs centaines de fois par rapport au taux basal. EPO est produit à 90% par les fibroblastes péri-tubulaires du cortex rénal et à 10% par le foie, principalement les hépatocytes et les cellules d’Ito situées autour des sinus dans les espaces de Disse 49. L’EPO-R est son récepteur qui est également inductible par l’hypoxie. Il est exprimé au niveau des progéniteurs érythropoïétiques et exerce un effet par de multiples voies 50. Au niveau des tissus érythropoïétiques, l’hypoxie entraîne une prolifération des précurseurs érythroïdes via l'EPO. La réponse cellulaire à l’hypoxie est contrôlée par une famille de facteurs de transcription : HIFs (Hypoxia-Inducible Factors). HIF est un hétéro-dimère composé de deux sous unités, HIF-α et HIF-β. En conditions normoxiques, la sous unité HIF-α est hydroxylée par des prolyl hydroxylases PHD et est dégradée par le protéasome. La forme hydroxylée de HIF-α se fixe à VHL (Von Hippel Lindau) et le complexe HIF-α/VHL facilite la dégradation de HIF-α via le protéasome. Contrairement à HIF-α, la sous unité HIF-β est stable en conditions normoxiques. En situation d’hypoxie, les prolyl hydroxylases sont inactivées et par conséquent, HIF-α se dimérise avec HIF-β. Ce nouveau complexe se lie à de courtes séquences d’ADN qui contiennent les HREs et qui régulent la transcription oxygéno-dépendante des gènes cibles de HIF dont celui de l’EPO (Fig.11). Il existe trois sous unités régulatrices HIF- α : HIF1, HIF2 et HIF3. La sous unité HIF-1α est exprimé de façon ubiquitaire et est le facteur majeur de réponse à l'hypoxie. Zhang F.L et al ont démontré que l'expression d’ALAS2 est régulée par HIF-1α dans des conditions hypoxiques. Ils ont émis l’hypothèse que l'augmentation du niveau d'expression d’ALAS2
26
contribue à l’hémoglobinisation des cellules érythroïdes dans des conditions hypoxiques, et qu’ALAS2 coopère avec l'EPO pour augmenter l'érythropoïèse pendant l'hypoxie 51,52.
Figure 11) La stabilité des protéines HIF est dépendante de la biodisponibilité du fer et l’oxygène, modifié d’après Yatrik M. Shah and Liwei Xie, 2015 53. Le fer est majoritairement utilisé pour la synthèse des globules rouges; la carence en fer réduit le nombre de globules rouges et le transport d'oxygène, conduisant à une hypoxie tissulaire. Une diminution de l'oxygène cellulaire ou la biodisponibilité du fer inhibe l’hydroxylation de la sous unité HIF-α par des prolyl hydroxylases PHD. Les espèces réactives de l’oxygène dans les mitochondries (mtROS) sont également nécessaires pour la stabilisation de HIF. HIF-α stabilisé se complexe avec la sous unité HIF-β. Ce nouveau complexe se lie à de courtes séquences d’ADN qui contiennent les HRE et qui régulent la transcription oxygéno-dépendante des gènes cibles de HIF dont celui de l’EPO.
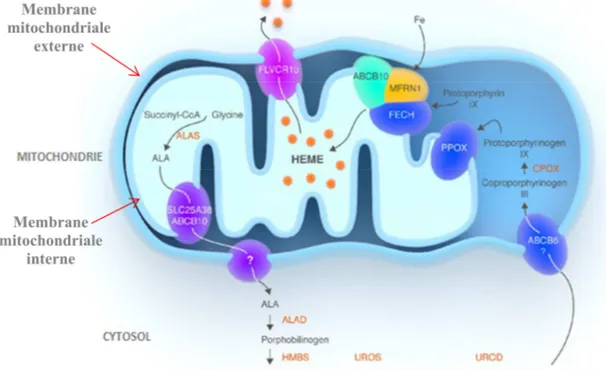
6-Le trafic de l'hème et ses précurseurs à travers les membranes mitochondriales
Alors que toutes les étapes enzymatiques conduisant à la production de l’hème sont bien caractérisées, les mécanismes de transport des métabolites entre le cytosol et la mitochondrie et inversement sont très mal connus. Comment l’ALA est-il exporté à travers les deux membranes mitochondriales? Comment le COPROgèneIII est-il importé dans l'espace inter-membranaire de la mitochondrie? Comment l’hème est-il exporté hors des mitochondries? C’est seulement dans la
27
dernière décennie que certaines de ces questions ont commencé à trouver des réponses et plusieurs nouveaux acteurs du métabolisme de l’hème ont été identifiés.
a- Export de l'ALA des mitochondries
Les transporteurs SLC25A38 et l’ABCB10 ont été proposés pour exporter l’ALA des mitochondries (Fig.12). SLC25A38 est un membre de la famille des transporteurs SLC25 de la membrane interne de la mitochondrie qui facilite la production d’ALA par l’import de glycine dans les mitochondries et échangerait la glycine contre l’ALA à travers la membrane mitochondriale interne 54, 55. SLC25A38 est fortement et préférentiellement exprimé dans les cellules érythroïdes et est importante pour la biosynthèse de l'hème chez les eucaryotes.
Un seul gène a été connu pendant longtemps pour entrainer l'anémie sidéroblastique congénitale (ASC) lorsqu'il est muté: le gène spécifiquement érythroïde d’ALAS2. Cependant, l’équipe Guernsey a trouvé des nouvelles mutations de type "perte de fonction" dans le gène SLC25A38 qui sont responsables de l’apparition de la maladie chez les patients atteints de la forme autosomique récessive d’ASC. Afin de confirmer le rôle de SLC25A38 dans l'érythropoïèse, Guernsey et al ont réalisé des expériences utilisant dans des poissons zèbre des oligonucléotides antisens de type morpholino inhibant la production d’ARNm. La diminution de l’expression de SLC25A38 chez les poissons zèbre a provoqué une anémie semblable à un déficit d’Alas2. Le manque d’YDL119c, l'orthologue de SLC25A38, chez les levures présente également un défaut de biosynthèse d’ALA 55.
ABCB10 est un membre de la famille "ATP binding cassette sub-family", qui utilisent l'énergie de l’hydrolyse de l'ATP pour le transport de divers substrats à travers les membranes cellulaires. ABCB10 est une protéine de la membrane mitochondriale interne qui s’homodimèrise pour former un transporteur fonctionnel 56. En plus de son rôle dans la stabilisation de MFRN1 (Mitoferrin-1) qui est l’importeur essentiel du fer dans la mitochondrie pour la biosynthèse de l'hème 57, 58, ABCB10 joue un rôle important au cours de la différenciation érythroïde. Les souris Abcb10 -/- meurent in utero en raison d’une anémie sévère 59. Il a d'abord été proposé qu’ABCB10 puisse exporter l’hème des mitochondries 60. Cependant, l’extinction d’ABCB10 provoque une diminution des niveaux cellulaire et mitochondrial de l’hème associée à la réduction de l’activité de plusieurs enzymes ayant un groupement prosthétique héminique. L'administration d'ALA restaure complètement le niveau de l'hème dans les cellules sous exprimées pour ABCB10 alors que la surexpression d’ALAS2 ne parvient pas à le faire. Ainsi, il a été proposé qu’ABCB10 puisse faciliter la synthèse mitochondriale d'ALA ou son exportation à partir de la mitochondrie. SLC25A38 et ABCB10 sont situées sur la membrane mitochondriale
28
interne. Il reste à comprendre comment l'ALA est transporté à travers la membrane mitochondriale externe 48.
Figure 12) Le trafic de l'hème et ses précurseurs à travers les membranes mitochondriales, modifié après D.Chiabrando et al, 2014 48.
b- L’import mitochondrial de COPROgèneIII
ABCB6 a été proposé comme étant le transporteur de COPROgèneIII du cytoplasme vers l'espace inter-membranaire mitochondrial (Fig.12). ABCB6 est situé dans la membrane mitochondriale externe et son expression est régulée positivement après la stimulation de la différenciation érythroïde des cellules MEL et G1ER ainsi que par le niveau d’hème. ABCB6 a été trouvé aussi sur la membrane plasmique, dans le compartiment de Golgi et dans les lysosomes. Cependant certaines études ne parviennent pas à détecter ABCB6 dans les mitochondries 61, 62,63. ABCB6 se lie aux porphyrines, y compris l’hème, et les essais de compétition suggèrent que le COPROgèneIII soit le substrat principal 61. Cependant, la localisation mitochondriale d’ABCB6 ainsi que le rôle d’ABCB6 dans la translocation des porphyrines dans les mitochondries restent controversés. En outre, ABCB6 a été associée à d'autres fonctions non liées à l'homéostasie des porphyrines. Elle code pour un nouveau système de groupe sanguin Langereis (Lan). L’antigène du groupe sanguin Lan est exprimé à la membrane des globules rouges ainsi que des cellules du carcinome hépatocellulaire 64. La manifestation clinique, chez différents individus atteints de porphyrie ayant les mêmes mutations, est très variable. Fukuda et alont démontré très récemment