THÈSE
Pour l'obtention du grade de
DOCTEUR DE L'UNIVERSITÉ DE POITIERS UFR des sciences fondamentales et appliquées Institut de physiologie et biologie cellulaires - IPBC (Poitiers)
(Diplôme National - Arrêté du 7 août 2006) École doctorale : Biologie-santé - Bio-santé (Limoges)
Secteur de recherche : Aspects moléculaire et cellulaire de la biologie
Présentée par :
Mahmoud El Maassarani
Identification de gènes cibles d'ErbB380kDa et
caractérisation de leur implication au cours de la progression du cancer de la prostate
Directeur(s) de Thèse : Paule Séité
Soutenue le 28 août 2014 devant le jury Jury :
Président Jean-Marc Muller Professeur des Universités, Université de Poitiers
Rapporteur Edith Bonnelye Chargé de recherche CNRS, INSERM de Lyon
Rapporteur Maxime Lehmann Professeur des Universités, Université de Strasbourg
Membre Paule Séité Professeur des Universités, Université de Poitiers
Membre Gérard Benoit Chargé de recherche CNRS, Université de Lyon 1
Pour citer cette thèse :
Mahmoud El Maassarani. Identification de gènes cibles d'ErbB380kDa et caractérisation de leur implication au
cours de la progression du cancer de la prostate [En ligne]. Thèse Aspects moléculaire et cellulaire de la biologie.
THESE DE DOCTORAT
En vue de l’obtention duGRADE DE DOCTEUR DE L’UNIVERSITE DE POITIERS
Spécialité : Biologie moléculaire
Présentée et soutenue par Mahmoud EL MAASSARANI Le 28 Aout 2014
Identification de gènes cibles d’ErbB3
80kDaet caractérisation de
leur implication au cours de la progression du cancer de la
prostate
JURY
Président : Pr. Jean-Marc Muller Equipe 2RCT, Université de Poitiers. Rapporteurs : Dr. Edith BONNELYE CR1 CNRS, Inserm U1033, Lyon
Pr. Maxime LEHMANN UMR 7213 CNRS, Université de Strasbourg Examinateurs : Dr. Gérard BENOIT CR1 UMR 5534 CNRS, Université de Lyon 1
Pr. Paule SEITE Equipe 2RCT, Université de Poitiers.
Thèse dirigée par le Pr. Paule SEITE Université de Poitiers
Faculté des sciences fondamentales et appliquées Ecole doctorale Bio-santé n°524
I
Résumé
Pour croître et proliférer, les cellules cancéreuses de la prostate activent des voies de signalisation dépendantes des androgènes. L’intervention thérapeutique en première ligne du cancer de la prostate (CaP) s'appuie donc d’abord sur le blocage de l’axe androgènes-récepteur aux androgènes (RA) mais rapidement, les patients développent des tumeurs résistantes (CRPC, Castration Resistant Prostate Cancer).
Les récepteurs à activité tyrosine kinase de la famille ErbB semblent jouer un rôle dans cette résistance, en particulier le récepteur ErbB3. En effet, l’inactivation des voies en aval d’ErbB1 et ErbB2, en association avec les anti-androgéniques n’empêche pas la progression vers l’hormono-indépendance, et une accumulation nucléaire d’ErbB3 est observée dans les CRPC en même temps que la voie PI3K-Akt est réactivée.
Dans ce contexte, nous avons validé l’expression d’une isoforme nucléaire ErbB380kDa chez les patients et dans des lignées hormono-sensible (LNCaP) et hormono-résistante (PC3). Par ChIP-on-chip, nous avons isolé 353 promoteurs cibles communs aux deux lignées, 245 spécifiques à la lignée LNCaP et 925 à la lignée PC3, et montré qu’ErbB380kDa est un co-régulateur transcriptionnel des gènes étudiés, parmi lesquels GATA2. L’analyse in silico de ces promoteurs révèle des sites de liaison pour les facteurs de transcription GATA2 et MZF1 au niveau des régions liant ErbB380kDa. Un complexe nucléaire GATA2-MZF1-ErbB380kDa est retrouvé dans les cellules LNCaP et PC3.
Des travaux récents montrent que GATA2 s’associe au RA pour réguler l’expression de gènes et qu’il pourrait être participé à la dissémination métastatique dans le CaP.
Nos résultats suggèrent qu’ErbB380kDa pourrait jouer un rôle régulateur, en amont de GATA2, dans les processus de résistance et l’apparition de métastases. Cette isoforme nucléaire insensible aux traitements actuels apparaît donc comme une cible privilégiée pour le ciblage thérapeutique.
II
Abstract
Prostate cancer (PCa) is dependent on androgens and functional androgen-receptor (AR) for growth and proliferation. Androgen-directed therapy is used at the first stages of the disease but cancer cells frequently become resistant (CRPC) by inappropriate reactivation of AR activity. As ErbB receptors are expressed in PCa cells, therapies aiming at inactivate the pathways downstream have been tested in advanced prostate cancers alongside hormone-based therapy. Still, a significant proportion of CRPC treated by ErbB1/2 inhibitors resist to treatment. ErbB3 could be responsible for this failure through both its unexpected nuclear localization and the reactivation of the PI3K-Akt pathway in those advanced tumors.
We have described a nuclear ErbB380kDa isoform, expressed in hormone-sensitive (LNCaP) and hormone-resistant (PC3) PCa cell lines that accumulates in the nucleus of tumor cells during cancer progression. ChIP-on-chip experiments led us to characterize 353 target promoters binding ErbB380kDa in both cell lines; 245 promoters specific to LNCaP and 925 specific to PC3 cells, among which the promoter of GATA2. We show that ErbB380kDa functions as a transcriptional co-regulator for the studied genes, potentially through its interaction with transcription factors. In silico analysis revealed binding sites for GATA2 and MZF1 transcription factors on the target promoters, and a complex GATA2-MZF1-ErbB380kDa has been found in LNCaP and PC3 cells. Recent publications have reported a role for GATA2 in the regulation of RA responsive-genes and in metastatic spreading. We propose that ErbB380kDa could act, upstream of GATA2, to induce resistance mechanisms and facilitate cancer progression. Thus, ErbB380kDa emerges as a putative target for the development of new therapies in prostate cancer.
III
Remerciements
Je n’aurais pas pu soutenir cette thèse sans la lecture attentive et les remarques précieuses du jury. Je remercie donc chaleureusement Dr. Edith Bonnelye et Pr. Maxime Lehmann, pour avoir accepté de rapporter ma thèse et le Dr. Gérard Benoit pour avoir bien voulu en être examinateur. Je tiens à remercier Pr. Muller Jean-Marc d’avoir accepté d’être président du jury.
Je souhaite remercier sincèrement ma directrice de thèse Paule Séité pour la confiance qu’elle m’a témoigné du début jusqu’à la fin du travail, pour sa disponibilité, sa compréhension et sa communication. C’est grâce à elle que j’ai appris beaucoup de choses dont certaines me seront fort utile non pas seulement au niveau professionnel mais aussi au niveau personnel. Je n’oublierai pas son aide précieuse dans la relecture et la correction de ma thèse. Paule, ces quelques lignes sont peu de choses par rapport à tout ce que tu m'as apporté, je t’exprime ma très profonde gratitude et je suis tout à fait d’accord avec Dominique, que tu es le meilleur chef qu’un thésard peut avoir.
Je tiens à remercier tous les membres de l’équipe 2RCT: Corinne, Agnès, Alice pour son organisation et ses aides en enseignement, et Brigitte pour son aide précieuse en tout ce qui concerne la bio-informatique et l’apport de sa collaboration avec Ahmed Moussa de l’université de Tanger- Maroc, que je remercie profondément. Je remercie également toute l’équipe du Pr. Alain Kitzis en particulier Sylvie, Barbara et Fréderic. Je remercie le Dr. Gaëlle Fromont et Julie Godet de service d’anatomopathologie de CHU de Poitiers, pour l’accès aux tissus des patients. Je remercie les membres de l’équipe de Pr. Marc Mesnil avec qui j’ai travaillé ces quatre ans de thèse, en particulier Annie-Claire, Arnaud et Laurent. Et enfin, je remercie toute l’équipe d’enseignement en biochimie et particulièrement le Franck Morel et le Jean François Jegou.
Je remercie tous les thésards du laboratoire Sylvain, Aurélie, Madryssa, Souheyla, Amandine, Renaud et mon ami de galère ‘rédaction et préparation des TP les weekends’ Hanitriniaina, merci pour ton soutien, tes conseils pour la bibliographie, je n’oublierai jamais nos discussions et les poses café ‘foumar’ en terrasse.
Mes plus profonds remerciements vont à mes chers parents qui tiennent une place immense dans mon cœur et qui ont fait de leur mieux pour que je puisse compléter mes études et réaliser mon projet. Joumana, Nazir, ce sont vos valeurs inestimables qui m’ont conduit jusque-là. Grand merci pour mes beaux-parents, d’abord de m’avoir confié le plus cher et le
IV plus beau diamant du monde ‘ma princesse Aïda’, et ensuite de m’avoir toujours soutenu motivé.
A ma princesse Aïda,
‘Aujourd’hui, j’ai envie de te dire un énorme MERCI, pour tout ce que tu me donnes, tout ce que tu m’apportes de bon depuis dix ans. Pour ta présence à mes côtés dans le meilleur comme dans le pire, pour ton amour et ton soutien pendant ces années de thèse. MERCI car c’est grâce à toi que j’ai enfin réalisé ce projet, grâce à ton aide précieuse, à tes conseils et à ta patience. MERCI car aujourd’hui et depuis plus de sept mois tu portes notre petit fils Zein, qui sera parmi nous très bientôt et qui sera notre plus beau cadeau. Aïda et Zein, je vous aime tous les deux’
V
Table des matières
INTRODUCTION 1
Première Partie : Les récepteurs trans-membranaires de la famille ErbB et leur
signalisation 2
I. Les récepteurs de la famille ErbB 2
A. Structure des récepteurs ErbB 2
1. Le domaine extracellulaire et ses propriétés ... 2 2. Le domaine transmembranaire ... 3 3. Le domaine intracytoplasmique ... 4
B. Activation des récepteurs ErbB 4
1. Les ligands ... 4 2. Les mécanismes de dimérisation ... 6 3. Les mécanismes d’activation hétérologue ... 7
C. Dégradation et recyclage des récepteurs ErbB 10
1. Endocytose clathrine-dépendante ... 11 2. Endocytose clathrine-indépendante ... 13 3. Devenir des récepteurs internalisés ... 14
II. Les voies de signalisation cytoplasmiques en aval des récepteurs ErbB 16
A. La voie MAPK 17
B. La voie PI3K 18
C. La voie STAT 19
III. Les voies de signalisation nucléaires des récepteurs ErbB 19 A. Mécanisme de translocation et fonctions nucléaires d’ErbB1 19
1. Mécanisme de translocation nucléaire du récepteur ErbB1 ... 20 2. Fonctions nucléaires du récepteur ErbB1 ... 21
B. Mécanisme de translocation et fonctions nucléaires d’ErbB2 24
1. Mécanisme de translocation nucléaire du récepteur ErbB2 ... 24 2. Fonctions nucléaires du récepteur ErbB2 ... 24
C. Mécanisme de translocation et fonctions nucléaires d’ErbB4 25
1. Mécanisme de translocation nucléaire du récepteur ErbB4 ... 26 2. Fonctions nucléaires du récepteur ErbB4 ... 27
VI
D. Localisation et fonctions nucléaires d’ErbB3 28
1. Les séquences NLS du récepteur ErbB3 ... 29
2. La localisation nucléaire d’ErbB3185kDa ... 29
3. La localisation nucléaire des variants d’ErbB3 ... 29
4. Les variants d’ErbB3 ... 30
5. Les fonctions nucléaires du récepteur ErbB3. ... 31
IV. Physiologie des récepteurs ErbB et leurs rôles dans les cancers 33 A. Physiologie des récepteurs ErbB au cours du développement 33 B. Physiopathologie des récepteurs ErbB 34 1. Altérations du gène ErbB1 dans le cancer ... 35
2. Altérations du gène ErbB2 dans le cancer ... 35
V. Thérapies anti-tumorales ciblant les récepteurs ErbB 36 A. Les thérapies qui ciblent le récepteur ErbB1 36 1. Les anticorps monoclonaux thérapeutiques (mAb) ... 36
2. Les inhibiteurs d’activité tyrosine kinase (TKIs) ... 37
3. ErbB1 et résistance aux thérapies ... 38
B. Les thérapies qui ciblent le récepteur ErbB2 39 1. Les anticorps monoclonaux thérapeutiques (mAb) ... 39
1. Les inhibiteurs d’activité kinase (TKIs) ... 41
2. ErbB2 et résistance aux thérapies ... 41
C. Les thérapies qui ciblent le récepteur ErbB3 42 1. ErbB3, acteur majeur de la voie PI3K-AKT ... 42
2. ErbB3, un récepteur clé de l’échappement aux thérapies ciblées ... 42
3. Les anticorps monoclonaux thérapeutiques (mAb) ... 43
Deuxième partie : Le cancer de la prostate 46 I. La prostate normale 46 A. Anatomie de la prostate 46 B. Les androgènes dans la prostate normale 48 1. L’axe hypothalamo-hypophysaire ... 48
2. Le récepteur aux androgènes (RA) ... 49
3. Les voies non génomiques dépendantes du RA ... 52
II. Le cancer de la prostate 53
VII
1. Epidémiologie ... 53
2. Etiologie ... 53
3. Classification des tumeurs ... 54
4. Les traitements ... 56
B. De l’androgéno-dépendance à la résistance 58 1. L’axe androgènes-récepteur aux androgènes ... 58
2. Prolifération et apoptose dans les CRPC ... 59
3. Les mécanismes moléculaires de l’androgéno-indépendance, dépendants du RA ... 61
4. Les mécanismes moléculaires de l’androgéno-indépendance, indépendants du RA ... 67
Troisième partie : Le récepteur ErbB3 dans le cancer de la prostate 70 I. Voies dépendantes du récepteur ErbB3 associées au cancer de la prostate 70 A. Amplification du récepteur ErbB3 70 B. Activation de la voie PI3K/AKT par les dimères B2/B3 et B1/B3 71 1. Dimère ErbB2/ErbB3 ... 71
2. Dimère ErbB1/ErbB3 ... 73
C. Les isoformes ErbB3 et leur rôle dans le cancer de la prostate 73 1. Les formes sécrétées d’ErbB3 ... 73
2. Les formes nucléaires d’ErbB3 ... 73
II. Rôle d’ErB3 dans l’échappement aux thérapies 74 III. Problématique 75 RESULTATS 78 Première partie :Publication 1 et résultats supplémentaires 79
A. Découverte du variant ErbB380kDa, une isoforme nucléaire du récepteur ErbB3185kDa 79 B. Caractéristiques du variant ErbB380kDa 80 C. Validation de la fonction nucléaire d’ErbB380kDa dans la lignée H358 80 D. Conclusion 80 Synthèse I 81 Deuxième partie :Publication 2 et résultats supplémentaires 83
A. De la pertinence d’ErbB380kDa dans le cancer de la prostate 83 1. Recherche de l’expression d’ErbB380kDa dans les tumeurs de prostate. ... 84 2. Caractérisation des lignées cellulaires de prostate pour l’expression d’ErbB3 89
VIII
B. Recherche de cibles génomiques du variant ErbB380kDa 94 1. Mise au point et validation de l’immunoprécipitation de chromatine sur les lignées de prostate ... 94 2. ChIP-on-chip ... 96
C. Les cibles génomiques d’ErbB380kDa dans les lignées tumorales 100 1. Validation des cibles par ChIP-qPCR ... 100 2. Mise en évidence de l’activité transcriptionnelle d’ErbB380kDa sur les cibles 105
D. Identification de facteurs de transcription au niveau des gènes cibles
d’ErbB380kDa 108
1. Facteurs de transcription répertoriés sur les gènes cibles d’ErbB380kDa ... 108 2. Analyse in silico des séquences cibles sur les promoteurs validés par ChIP-qPCR ... 110 3. Mise en évidence d’un complexe ErbB3/GATA2/MZF1 dans le noyau des lignées LNCaP et PC3 ... 112
Synthèse II 115
DISCUSSION 118
ANNEXES 135
IX
Table des figures
Figure 1 : Structure des récepteurs ErbB. 3
Figure 2 : Structure de l’ARNm et de la protéine EGF. 5
Figure 3 : Mécanismes d’activation et de dimérisation des récepteurs ErbB après fixation du
ligand. 7
Figure 4 : Structure du dimère ErbB1 activé par l’EGF. 8
Figure 5 : Activation hétérologue par « triple-membrane-passing-signalling ». 9
Figure 6 : Activation hétérologue en réponse à un stress oxydatif. 9
Figure 7 : Modèle d’endocytose clathrine- dépendante. 12
Figure 8 : Séquences et modifications post-traductionnelles associées à l’endocytose
clathrine-dépendante des récepteurs ErbB. 13
Figure 9 : Devenir des récepteurs internalisés. 15
Figure 10 : Protéines liant les phosphotyrosines des domaines C-ter des récepteurs ErbB. 17 Figure 11 : Schéma simplifié des voies de signalisation cytoplasmiques en aval du récepteur
ErbB1. 18
Figure 12 : Transport intracellulaire du récepteur vers le noyau. 21
Figure 13 : Fonctions nucléaires du récepteur ErbB1. 23
Figure 14 : Translocation nucléaire du récepteur ErbB4. 27
Figure 15 : Les variants du récepteur ErbB3. 32
Figure 16 : Les anticorps et les inhibiteurs TKIs ciblant les récepteurs ErbB. 40
Figure 17 : Anatomie de l’appareil génital masculin et coupe histologique de la glande
prostatique. 46
Figure 18 : Processus de développement de la prostate. 48
Figure 19 : Structure du locus RA et de la protéine chez l’homme. 50
Figure 20 : Voies génomiques dépendantes du RA. 51
Figure 21 : Voies non génomiques dépendantes du RA. 52
Figure 22 : Les voies conventionnelles de l’androgéno-indépendance, dépendantes du RA. 64 Figure 23 : Les voies non conventionnelles de l’androgéno-indépendance, dépendantes du
RA. 66
Figure 24 : Voies dépendantes du récepteur ErbB3 dans le cancer de la prostate. 71
Figure 25 : Expression et localisation d’ErbB3 dans les tissus prostatiques non tumoraux et
tumoraux. 86
Figure 26 : Expression et localisation d’ErbB3 et Ebp1 dans des tissus prostatiques non
tumoraux et tumoraux. 88
Figure 27 : Recherche des isoformes des transcrits d’ErbB3 dans les lignées cellulaires de
prostate. 90
Figure 28 : Expression endogène de la protéine ErbB3 dans les lignées de prostate. 92
Figure 29 : Expression des isoformes d’ErbB3 dans les lignées cellulaires. 93
Figure 30 : Immunoprécipitation de la protéine ErbB3 endogène dans les lignées de prostate
et validation de la ChIP. 96
Figure 31 : Contrôle qualité du ChIP-on-chip. 98
Figure 32 : Diagramme de Venn de la répartition des cibles d’ErbB3 dans les lignées
X
Figure 33: Choix des amorces pour la validation ChIP-qPCR. 103
Figure 34 : Validation des 21 promoteurs cibles par ChIP-qPCR. 104
Figure 35 : Validation fonctionnelle de la spécificité et de l’efficacité des si-ErbB3. 106
Figure 36 : Expression transcriptionnelle des gènes cibles en réponse à l’inhibition de
l’expression d’ErbB3 par des ARN interférents. 108
Figure 37 : Position des sites de liaison pour les facteurs de transcription GATA2 et MZF1 et
des sondes positives en ChIP, sur les promoteurs des gènes GATA2 et MAP3K14. 112
Figure 38 : Co-Immunoprécipitation d’ErbB380kDa, GATA2 et MZF1. 113
Figure 39 : Représentation schématique des interactions potentielles entre la signalisation
nucléaire d’ErbB380kDa et les processus métastatiques dans l’os. 131
Figure 40 : Schéma récapitulatif des fonctions potentielles d’ErbB3 dans la progression du
cancer de la prostate. 134
Figure 41 : Localisation subcellulaire des protéines ErbB380kDa, ErbB3185kDa GATA2 et
MZF1 dans la lignée LNCaP. 138
Figure 42 : Localisation subcellulaire des protéines ErbB380kDa, ErbB3185kDa GATA2 et
MZF1 dans la lignée PC3. 139
Figure 43 : Séquence NM_001982.3 montrant les positions des amorces PCR et qPCR
(Figure 27) et les si ARN utilisés (Figure 35). 140
Figure 44 : Expression des protéines ErbB3, GATA2 et MZF1 dans des tissus tumoraux à
XI
Tableaux
Tableau 1 : Localisation nucléaire des récepteurs de la famille ErbB dans les cellules
tumorales. 28
Tableau 2 : Anticorps utilisés en immunocytochimie. 85
Tableau 3 : Les lignées cellulaires utilisées. 89
Tableau 4 : Etude ontologique des gènes cibles d’ErbB3 dans les trois lignées. 100
Tableau 5 : Ontologie des gènes cibles sélectionnés. 101
Tableau 6 : Facteurs de transcription susceptibles de fixer les cibles d’ErbB3 nucléaire. 110 Tableau 7 : Sites potentiels de fixation de GATA2 et MZF1 dans la séquence promotrices des
cibles d’ErbB380kDa validées. 111
Tableau 8 : Amorces utilisées pour la validation des cibles en ChIP-qPCR. 136
Tableau 9 : Amorces utilisées pour la RT-qPCR. 137
XII
Abréviations
ABC : ATP-Binding Cassette transportersADAM : A Disintegrin And Metalloprotease domain ADCC : Antibody-Dependent Cell-mediated Cytotoxicity
ADNc : Acide DésoxyriboNucléique complémentaire ADT : Thérapie Anti-androgénique
AP : Adaptator Protein
ATM : Ataxia Telangiectasia Mutated ARE: Androgen Response Element Bcl-2 : B-cell lymphoma 2
BCRP : Breast Cancer Resistant Protein BTC : Betacellulin
CAB : Complete Androgen Blockade CAF : CBP-associated Factor CaP : Cancer de la Prostate Cav-1 : Cavéoline-1
CBP : CREB-Binding Protein CCND1 : Cycline D1
CDK : Cyclin-dependent kinase ChIP : Chromatin ImmunoPrecipitation CIE : Clathrin Independent Endocytosis CME : Clathrin Mediated Endocytosis COPI : Coat Protein Complex I COX-2 : Cyclo-OXygénase 2
CRPC : Castration-Resistant Prostate Cancer CTD : Carboxyl Tail Domain
C-ter : C-terminal
DBD : DNA-Binding Domain
DDX-10 : DEAD (Asp-Glu-Ala-Asp) box polypeptide
10
DHT : Dihydrotestostérone Ebp1 : ErbB3 Binding Protein 1 EEA1 : Early Endosome Antigen 1 E2F1 : E2 Transcription Factor 1 EGF : Epithelial Growth Factor
EGFR : Epithelial Growth Factor Receptor EP : Endosome Primaire
Erk : Extracellular signal Regulated Kinases FDA : Food and Drug Administration FGF : Fibroblast Frowth Factors FSH : Hormone Folliculo-stimulante GAPDH : GlycerAldehyde-3-Phosphate
DeHydrogenase
GnRH :Gonadotropin Releasing Hormone GPCR : Récepteur Couplé aux Protéines G GTPase : Guanosine TriPhosphate hydrolase Grb : Growth factor receptor-bound protein GS3K : Glycogène Synthase 3 Kinase
HB-EGF : Heparin-Binding Epidermal Growth Factor HBP : Hyperlasie Bénigne de la Prostate
HDAC : Histone Désacétylase
HDM2 : Human double minute 2, E3 ubiquitin
protein ligase
HP1 : Hetero-chromatin binding Protein 1
HNSCC : Head and Neck Cancers are Squamous Cell
carcinomas
HRG : Heréguline HS : Hormono-Sensible HSP : Heat Shock Protein ICD : intracellular domain IGF : Insulin-like Growth Factor IgG : immunoglobuline
IL : Interleukine
ILV : Intraluminal Vesicles IN : INput
iNOS : inducible Nitric Oxide Synthase IP : Immunoprécipitation
JAK : Janus Kinase JNK : Jun Kinase kb : kilo base kDa : kilo Dalton
KGF : Keratinocyte Growth Factor KO : KnockOut
LBD : Ligand-Binding Domain LH : Luteinizing Hormone
LNCaP : Lymph Node Carcinoma of the Prostate cell
line
Lys : Lysosome
MAPK : Mitogen-Activated Protein Kinase MMP : Matrix MetalloProteinases
miARN : micro-ARN
mTOR: Mammalian Target Of Rapamycin Myc : Myelocytomatosis oncogene NCor : Nuclear receptor Corepressor NE : Neuro-Endocrine
Nedd4 : Neural precursor cell expressed developmentally down-regulated protein 4
NF-KB : Nuclear Factor-Kappa B NGF : Neural Growth Factor NIP : Néoplasie Intra-épithéliale NK : Natural Killer
NLS : Nuclear Localization Signal NO : Nitric Oxide
NR4A3 : nuclear receptor subfamily 4, group A,
member 3
Nrdp1 : Neuregulin receptor degradation protein-1 NRG : Neuregulin
NSCLC : Non-Small-Cell Lung Carcinoma NTD : N-terminal Domain
N-ter : N-terminal Nup : Nucléoporine
XII
ORF : Open Reading Frame
PAK1 : p21 Protein (Cdc42/Rac)-Activated Kinase 1 PBS : Phosphate Buffered Saline
PC3 : Prostatic Carcinoma 3 cell line PCNA : Proliferating Cell Nuclear Antigen PI3K : PhosphatIdylinostol-3 Kinase PIP3 : Plasma membrane Intrinsic Protein 3 PKC : Protéine Kinase C
PLC : Phospholipase C
PML : Promyelocytic Leukemia Protein PMT : Poids Moléculaire Théorique PRCA1 : PRostate CAncer 1 PSA : Prostate-Specific Antigen
PTEN : Phosphatase and TENsin homolog PY : Tyrosine Phosphorylée
Pyk2: Pyruvate Kinase 2
qRT-PCR : quantitative Reverse
Transcription-Polymerase Chain Reaction
RA : Récepteur aux Androgènes
RAccr : Récepteur aux Androgènes muté Cortisol and
Cortisone Responsive
Raf : Rapidly accelerated fibrosarcoma
RE : Réticulum Endoplasmique RP : Récepteur à la Progestérone RTK : Récepteur Tyrosine-Kinase SDS-PAGE: Sodium Dodecyl Sulfate
PolyAcrylamide Gel Electrophoresis
SFK : Src Family Kinase Shc : Src homology
sh-RNA : short-hairpin RNA si-ARN : small interfering ARN
SIN3A : SIN3 transcription regulator family member
A
SMRT : Silencing Mediator for Retinoid an Thyroid
hormone receptor
STAT: Signal Transducer and Activator of Transcription
SRC1 : Steroid Receptor Coactivator 1
TACE : Tumor necrosis factor α-Converting Enzyme TGF : Transforming Growth Factor
TK : domaine Tyrosine Kinase TKI : tyrosine kinase inhibator TM : Transmembrane Domain
v-Myc : Myelocytomatosis viral oncogene
1
2
Première Partie : Les récepteurs trans-membranaires de
la famille ErbB et leur signalisation
Pour survivre, se multiplier et se différencier, les cellules s'appuient sur des signaux de leur environnement. Plus précisément, les cellules ont besoin d'établir des moyens de communication qui reposent sur la transmission des signaux de l'extérieur de la cellule vers l'intérieur. Les systèmes de signalisation transmembranaires comprennent des récepteurs de surface et des ligands qui se lient à ces récepteurs. Les ligands, comme les récepteurs, sont généralement de nature protéique et la liaison entre les deux déclenche un signal intracellulaire induisant à son tour une cascade de signalisation.
I. Les récepteurs de la famille ErbB
Les récepteurs de la famille ErbB (erythroblastic leukemia viral oncogene homologue) constituent une famille de récepteurs transmembranaires à activité tyrosine-kinase (RTK), qui sont à l’origine d’un large éventail de réponses cellulaires. Ils régulent la croissance, la différenciation, la progression du cycle cellulaire, l’adhérence, la migration, l’apoptose...
La famille ErbB est composée de quatre membres EGFR/ErbB1/HER1, ErbB2/HER2, ErbB3/HER3 et ErbB4/HER4, qui sont exprimés de manière ubiquitaire dans les cellules épithéliales, mésenchymateuses ou nerveuses.
A. Structure des récepteurs ErbB
Prenant pour référence la structure primaire d’ErbB1/HER1 [Ulrich et al, 1984], ces récepteurs sont caractérisés par la présence d’un domaine extracellulaire fortement glycosylé destiné à recevoir un ligand spécifique, d’un domaine transmembranaire unique et d’un domaine intracellulaire qui contient le domaine kinase (Figure 1).
1. Le domaine extracellulaire et ses propriétés
Le domaine extracellulaire se caractérise par 4 régions distinctes désignées I à IV de l’extrémité N-terminale vers le domaine transmembranaire (TM). Les domaines I et III, riches en résidus leucine, interviennent pour la liaison avec le ligand. Les domaines II et IV, riches en résidus cystéine, établissent entre eux des ponts disulfures pour maintenir la molécule en
3 conformation inactive en absence de ligand. Le domaine II est également impliqué dans l’homo ou l’hétéro-dimérisation qui se produit entre les membres de la famille ErbB après activation par le ligand.
En l’absence de certains acides aminés critiques dans les domaines II et IV du récepteur ErbB2/ HER2, ce dernier est incapable de fixer les ligands des récepteurs ErbB. Ce récepteur orphelin possède cependant une activité catalytique et fonctionne par hétéro-dimérisation avec d’autres ErbB, avec une affinité particulière pour ErbB3.
2. Le domaine transmembranaire
Le domaine extracellulaire est prolongé par un domaine transmembranaire (TM) unique constitué de 23 à 25 résidus hydrophobes, structurés en hélice alpha (Bocharov et al., 2008). La séquence varie selon le récepteur, mais des motifs communs dits GG4, constitués de 2 résidus Glycine distants de 4 acides aminés (G-XXX-G) sont retrouvés en N-ter et en C-ter du domaine TM chez tous les membres de la famille, à l’exception du récepteur ErbB3 qui est dépourvu du motif GG4 en C-ter. L’interaction entre motifs N-ter est associée à la conformation active du récepteur, alors que l’interaction entre motifs C-ter correspond à une
Figure 1 : Structure des récepteurs ErbB.
Les récepteurs sont schématisés et leurs facteurs de croissance spécifiques sont indiqués. Les chiffres le long des récepteurs indiquent la position des acides aminés délimitant chaque domaine ou sous-domaine. Les domaines extracellulaires sont constitués de quatre segments I à IV. Les domaines I et III d’ErbB2 ne fixent aucun ligand connu. Dans la région intracytoplasmique, le domaine kinase (PKD) d’ErbB3 est non fonctionnel. EGF : Epithelial Growth Factor, EPG : Epigen, TGFα: Transforming Growth Factor-alpha, AR : Amphiregulin, BTC : Betacellulin, HB-EGF : heparin-binding epidermal growth-factor like growth factor, EPR : epiregulin, Nrg-1/2/3/4 : neuregulin-1/2/3/4.
4 conformation inactive. Ces résidus permettent l’ancrage de la protéine et préviennent tout mouvement vertical de la protéine dans la membrane (Escher et al., 2009).
3. Le domaine intracytoplasmique
La partie intracellulaire des récepteurs ErbB contient un petit segment juxta-membranaire, un domaine à activité tyrosine kinase et une queue C-terminale, riche en résidus tyrosine.
Les études réalisées sur ErbB1 montrent que le segment juxtamembranaire participe à l’activation du domaine catalytique du récepteur lors de la dimérisation (Jura et al., 2009; Red Brewer et al., 2009).
Le domaine à activité kinase est séparé en deux sous-domaines : un lobe N-terminal de petite taille qui contient un important domaine régulateur de l’activité catalytique, le domaine aC, et un grand lobe C-terminal au niveau duquel se trouve le domaine d’activation proprement dit (Taylor et al., 2012). Si cette structure est bien conservée entre les 4 membres de la famille, il est important de noter que des mutations retrouvées au sein des motifs conservés du domaine kinase d’ErbB3 (His759 → Gln759, Asn834→ Asp834) rendent ce récepteur incapable d’autophosphorylation (Guy et al., 1994). Cependant, une fois qu’ErbB3 a fixé son ligand, le lobe C de son domaine kinase inactif est capable d’interagir avec un lobe N d’un autre récepteur ErbB pour initier l’activation du dimère (Citri et al., 2003; Linggi and Carpenter, 2006).
La queue C-terminale du domaine intracytoplasmique est une région riche en résidus tyrosines (Wilson et al., 2009). Le profil de phosphorylation de ces tyrosines génère une grande variété de sites de liaison pour des protéines adaptatrices qui orientent la spécificité du signal intracellulaire transduit. Les principales voies activées par ces récepteurs sont les voies MAPK (mitogen-activated protein kinase), PI3K (phosphatidylinositol-3 kinase) et STAT (signal transducer and activator of Transcription) que nous décrirons plus loin.
B. Activation des récepteurs ErbB
1.
Les ligandsLa première découverte de facteurs de croissance remonte aux années 1950, avec la caractérisation du NGF (Neural Growth Factor) par Rita Montalcini (Cohen and
Levi-5 Montalcini, 1957). En 1962, est découvert l’EGF (Epidermal Growth Factor), rapidement reconnu par la suite comme le chef de file d’une famille de facteurs de croissance. Ce n’est qu’en 1975 que Graham Carpenter et ses collègues trouveront la protéine capable de lier l’EGF, le récepteur EGFR/ErbB1 (Epithelial Growth Factor Receptor) (Carpenter et al., 1975).
Les facteurs de croissance de type EGF sont caractérisés par la présence d'une ou de plusieurs unités structurelles de type EGF ou EGF-like (Figure 2). Chaque unité est constituée d’une séquence de 35–40 acides aminés incluant six résidus cystéine conservés dans un motif CX7CX3–5CX10–12CXCX5GXRC (C-Cys; G-Gly; R-Arg; X, autres aa). Les trois ponts disulfures qui relient les cystéines deux à deux (C1/C3, C2/C4 et C5/C6) confèrent au ligand sa capacité de liaison au récepteur (Savage et al., 1973). Ces facteurs de croissance sont synthétisés sous forme de précurseurs puis exportés vers la membrane plasmique à travers la voie de biosynthèse des protéines membranaires. Le domaine de croissance, contenant les unités EGF, correspond à la région extracellulaire, suivie par une région hydrophobe (domaine transmembranaire) et une région cytoplasmique (Figure 2). Ces précurseurs sont ensuite clivés par des métalloprotéinases (ADAM) et métalloprotéinases de matrice (MMPs), pour libérer un domaine N-terminal soluble (Singh and Harris, 2005). La production des
b) a)
c)
Figure 2 : Structure de l’ARNm et de la protéine EGF. a) Schéma de l’ARNm EGF
faisant apparaître de 5’ vers 3’ : les séquences EGF-like (1-8) en jaune, la séquence EGF en vert et la séquence TM qui codera le domaine trans-membranaire du précurseur de l’EGF. b) Le précurseur transmembranaire de l’EGF comprend : un peptide signal (20 aa) et la séquence EGF proprement dite (53 aa) encadrée par 2 régions de 950 aa en N-ter et 184 aa en C-ter. Cette dernière séquence inclut le domaine transmembranaire, constitué d’acides aminés hydrophobes. c) L’EGF mature, soluble, obtenu par clivage protéolytique est constitué de 53 aa, dont 6 cystéines conservées associées par des ponts disulfures (Zeng and Harris, 2014).
6 ligands EGF solubles par clivage de l’ectodomaine des précurseurs nécessite l’activation des métalloprotéinases par des récepteurs couplés aux protéines G (GPCRs) (Gschwind et al., 2001).
Les ligands de la famille EGF sont divisés en trois groupes:
- le premier comprend l’EGF, le TGF-α (Transforming growth factor-α) et l’amphiréguline (RA) qui lient spécifiquement ErbB1.
- Le second groupe comprend la bétacelluline (BTC), HB-EGF (Heparin-Binding EGF) et l’épiréguline (EPR), qui montrent une double spécificité de liaison pour ErbB1 et ErbB4.
- Le troisième groupe enfin, est composé des neurégulines (NRGs) : NRG1 et NRG2 fixent ErbB3 et ErbB4, alors que NRG3 et 4 sont spécifiques d’ErbB4 (Hynes and Lane, 2005).
Comme nous l’avons déjà indiqué précédemment, ErbB2 est un récepteur orphelin dont la structure ne permet pas la liaison de ligands de type EGF mais cette caractéristique en fait le partenaire privilégié d’hétérodimérisation des autres récepteurs.
2. Les mécanismes de dimérisation
La liaison du ligand au domaine extracellulaire des récepteurs ErbB, déclenche la dimérisation du récepteur ou la stabilisation des dimères inactifs préexistants. Ces changements intermoléculaires sont liés à des changements conformationnels des récepteurs eux-mêmes (changement intramoléculaire) et aboutissent enfin à la phosphorylation des résidus tyrosine au niveau de domaine intracellulaire.
En l’absence de ligand et en situation physiologique, les récepteurs ErbB sont le plus souvent présents à la membrane cellulaire sous forme de monomères inactifs en raison de l’interaction entre les segments II et IV, riches en cystéines, du domaine extracellulaire (Figure 3). Ce repliement contraint de la molécule empêche ainsi toute activation constitutive des voies en aval des récepteurs (Garrett et al., 2003).
La fixation du ligand sur les segments I et III, riches en leucine, de ce même domaine extra-cellulaire, entraîne alors la séparation des segments II et IV du monomère (Figure 3). Ces changements conformationnels augmentent l’affinité du récepteur pour son ligand et
7 favorisent l’homo ou l’hétérodimérisation via l’interaction entre les domaines II de chaque co-récepteur. Selon la spécificité du ligand et la nature des récepteurs présents à la membrane, 28 combinaisons théoriques sont possibles si l’on prend en compte les isoformes du récepteur ErbB4. Cependant, les combinaisons ErbB2/ErbB2 et ErbB3/ErbB3 n’existent pas en raison de l’absence de ligand capable de lier et d’activer ErbB2 d’une part, et de l’absence d’activité intrinsèque du domaine kinase d’ErbB3 d’autre part (Roskoski, 2014).
Nous avons vu précédemment que la structure des domaines kinases est divisée en deux lobes, appelés N et C, qui coopèrent pour former le site actif. Au cours de la dimérisation, le lobe C de la kinase activatrice (récepteur ayant fixé le ligand), active le domaine kinase de son co-récepteur (receveur) par contact direct avec le lobe N, formant ainsi une boucle qui permet l’activation et la phosphorylation des tyrosines de ce récepteur (Figure 3 et 4). Cette boucle sert ensuite d’ancrage à diverses molécules adaptatrices comme la PLC (Phospholipase C), Grb (Growth factor receptor-bound) et Shc (Src homology), impliquées dans les voies de signalisation, que nous décrirons plus loin.
3. Les mécanismes d’activation hétérologue
L’idée selon laquelle la fixation du ligand induit la dimérisation/activation des récepteurs ErbB a évolué suite à la mise en évidence de mutations somatiques d’ErbB1 qui affectent la conformation du récepteur et rendent ces mutants actifs indépendamment de tout ligand (Jura et al., 2009; Kumar et al., 2008; Paez et al., 2004; Yun et al., 2007; Zhou et al., 2009).
Figure 3 : Mécanismes d’activation et de dimérisation des récepteurs ErbB après fixation du ligand. Dans la
majorité des cas, l’interaction entre les domaines II (vert) et IV (rouge) des récepteurs induit une conformation contrainte, qui ne permet pas la liaison du ligand. Ces monomères sous forme inactive (A). La fixation du ligand sur les domaines I (bleu) et III (jaune) stabilise le récepteur (B) et rend le domaine II accessible pour la dimérisation (C). En gris sont figurés les domaines tyrosine kinase (TK). Les tyrosines phosphorylées en C-terminal du dimère sont indiquées en orange.
8 Une phosphorylation anormale d’ErbB1 peut aussi être induite par l’action de cytokines, de molécules d’adhésion, où résulter d’une transactivation par des récepteurs couplés aux protéines G. GPCR et RTK sont co-exprimés dans les cellules et peuvent fonctionner de manière croisée pour répondre à des stimuli extracellulaires dans des contextes physiologiques complexes selon un modèle appelé « triple membrane passing signalling »
(Figure 6).Il a été montré que divers ligands de GPCR (acide lysophosphatidique, thrombine, endothéline…) sont capables d’induire la phosphorylation d’ErbB1 par deux types de mécanismes. Le premier consiste en l’activation de métalloprotéinases de la matrice extracellulaire qui vont cliver en retour les pro-ligands du récepteur ErbB1 et aboutir au mécanisme classique d’activation ligand-dépendant (Carpenter, 1999; Prenzel et al., 1999).
Figure 4 : Structure du dimère ErbB1 activé par l’EGF. Le monomère activateur est figuré en
bleu gris et le monomère receveur en violet. Les différents domaines (I-IV) de la région extracellulaire (EC) sont indiqués. Les domaines II sont réunis par des bras de dimérisation. Dans le cytoplasme, le lobe N du domaine TK receveur est activé par le lobe C du domaine TK activateur et trans-phosphorylé.
9 Le second mécanisme n’implique pas les métalloprotéinases mais repose sur l’activation de médiateurs tels que des seconds messagers ou les tyrosine-kinases Pyk2 (Pyruvate kinase 2) ou Src qui phosphoryleraient alors directement le domaine intracytoplasmique du récepteur (Wetzker and Böhmer, 2003). Ces mécanismes sont susceptibles de jouer un rôle au niveau de microdomaines membranaires où se concentrent des GRPC et des récepteurs ErbB.
Plus récemment, il a été montré qu’un stress oxydatif pouvait activer le récepteur ErbB1(Khan et al., 2008). En réponse à ces signaux, une modification de la répartition des lipides intramembranaires à la fois par destruction des radeaux lipidiques (Lambert et al., 2006) et par l’expression accrue de céramides (Filosto et al., 2011), serait capable d’activer ErbB1 sous forme monomérique sans induire la dimérisation du récepteur (Filosto et al., 2011) (Figure 6).
Figure 5 : Activation hétérologue par « triple-membrane-passing-signalling ».Le signal initial induit par les ligands des GRPC (outside-in) est transformé par les médiateurs cytoplasmiques en un signal autocrine-paracrine (inside-out) par le biais des métalloprotéinases. Ces enzymes libèrent le ligand qui active alors les voies cytoplasmiques en aval des RTK (outside-in) (Wetzker and Böhmer, 2003).
Figure 6 : Activation hétérologue en réponse à un stress oxydatif.
Selon ce modèle, le stress oxydatif induirait une augmentation des céramides avec pour conséquence une modification de la répartition du cholestérol membranaire. Les monomères EGFR/ErbB1, activés par Src, seraient stabilisés. Par interaction avec la protéine cavéoline-1 (Cav-1) ils seraient alors transportés sous forme active vers une région péri-nucléaire encore mal caractérisée (Filosto et al., 2011).
10
C. Dégradation et recyclage des récepteurs ErbB
La signalisation des récepteurs ErbB est cruciale pour le développement et le maintien de l’homéostasie dans différents tissus adultes. Si la vision traditionnelle de l’activation constituée par la séquence ligand-recepteur-dimérisation, a évolué fortement, il n’en reste pas moins que quel que soit le mécanisme mis en jeu, le niveau de cette signalisation doit être régulé. De la même manière qu’il est possible de contrôler les signalisations sous-jacentes des récepteurs ErbB, les cellules utilisent diverses voies pour réguler l’abondance et la distribution spatiale des récepteurs ErbB, ainsi que la durée et l'intensité des signalisations.
Comme toutes les protéines transmembranaires, les récepteurs ErbB sont synthétisés dans le réticulum endoplasmique, transportés ensuite vers l’appareil de Golgi où les domaines extracellulaires sont glycosylés, avant d’être délivrés à la membrane plasmique (Todderud and Carpenter, 1989). Les récepteurs ErbB nouvellement synthétisés sont principalement dirigés vers les membranes baso-latérales des cellules épithéliales. Des études portant principalement sur le positionnement des récepteurs ErbB1 au niveau de la membrane plasmique ont révélé une répartition hétérogène et cellule-dépendante des dimères formés après activation, ainsi que la présence de regroupements plus importants (clusters) à travers la membrane (Abulrob et al., 2010; Nagy et al., 2010). Bien que peu d'informations existent sur le regroupement des autres membres de la famille, la distribution d'ErbB1, ErbB2 et ErbB3 d’un type cellulaire à l’autre peut être nettement différente, suggérant l’importance du contrôle spatial de cette famille de récepteurs (Kaufmann et al., 2011).
Le principal mécanisme mis en jeu dans le contrôle spatial ainsi que dans le contrôle quantitatif des récepteurs ErbB est l’endocytose, qui a lieu le plus souvent après stimulation des récepteurs par le ligand. L’endocytose d’ErbB1 est le modèle de référence pour tous les récepteurs de cette famille, mais aussi d’une manière plus générale pour tous les RTKs. Pour cette raison, la plupart des données citées dans ce chapitre se rapporteront donc à ErbB1 (Grant and Donaldson, 2009).
Plusieurs paramètres, parmi lesquels la présence de motifs d’acides aminés particuliers, la spécificité des résidus phosphorylés et la nature des protéines intracytoplasmiques recrutées par le domaine C-terminal, interviennent pour orienter et
11 réguler l’internalisation des récepteurs à travers deux grandes voies d’endocytose décrites ci-après.
1. Endocytose clathrine-dépendante
Pour ce type d’endocytose, l’internalisation des récepteurs est initiée par une invagination qui se produit au niveau de domaines membranaires caractérisés, d’une part par la distribution différentielle de protéines membranaires et sous-membranaires appelées clathrines, d’autre part par l’interaction de ces structures avec le cytosquelette sous-jacent (Casaletto and McClatchey, 2012; Grecco et al., 2011; McNiven, 2006).
L’utilisation d’inhibiteurs chimiques de la clathrine ou l’expression d’ARN interférents dirigés contre le gène codant la chaîne lourde de cette protéine, bloque très fortement l’internalisation des récepteurs ErbB1, suggérant que l’endocytose clathrine-dépendante CME (Clathrin Mediated Endocytosis) est la principale voie d’internalisation des complexes ErbB1-ligand dans les conditions physiologiques. C’est aussi la voie la plus rapide (Huang et al., 2004; von Kleist et al., 2011; Lampugnani et al., 2006; Zheng et al., 2008).
Les mécanismes conventionnels d’endocytose clathrine-dépendante font intervenir deux motifs d’acides aminés qui permettent le recrutement du complexe AP-2 (clathrin Adaptator Protein Complex 2) par le domaine intracytoplasmique de récepteurs membranaires (Huang et al., 2003). Le motif Y974RAL recrute la sous-unité m2 du complexe AP-2 et le motif di- leucine L1010 /L1011 recrute la sous-unité b2 d’AP-2 dont elle phosphoryle la tyrosine en position 6 (Figure 7 A) (Huang et al., 2003).Cependant, la réelle implication de ces motifs dans la CME d’ErbB1 reste posée en raison des résultats contradictoires obtenus après déplétion d’AP-2 par des ARN interférents : certaines équipes montrent qu’AP-2 est indispensable à ce processus ((Huang et al., 2004; Johannessen et al., 2006) alors que d’autres ne voient pas d’effet inhibiteur de ces siARN sur l’endocytose (Motley et al., 2003).
12
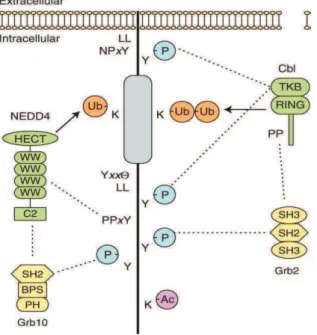
Un autre mécanisme fait intervenir des processus d’ubiquitinylation (Figure 7 B). Sur le domaine intracytoplasmique des récepteurs ErbB des résidus tyrosine phosphorylés servent de sites d’ancrage pour des ubiquitine-ligases spécifiques (Acconcia et al., 2009). ErbB1 est ubiquitinylé par l’E3-ubiquitine ligase Cbl (Levkowitz et al., 1999). Cbl peut interagir directement avec ErbB1 grâce au résidu tyrosine Y1045 phosphorylé, mais cette interaction est insuffisante pour provoquer l’endocytose. En revanche, une interaction indirecte via la protéine adaptatrice Grb2 (Growth factor receptor-bound protein 2), qui fixe simultanément les résidus Y1068-Y1086 phosphorylés ou via des protéines intracytoplasmiques à motif UIM (Ubiquitin Interacting Motif) et les molécules de clathrines, concentrerait ErbB1 dans les puits à clathrines et déclencherait ensuite la CME (Goh and Sorkin, 2013; Liu et al., 2009; Waterman et al., 2002).
Comme ErbB1, le récepteur ErbB2 recrute Cbl via la tyrosine phosphorylée Y1045, mais son domaine C-ter est incapable de recruter Grb2. Ceci limite donc la localisation spatiale d’ErbB2 au niveau des puits à clathrine (moins de 1/10 000). En conséquence, ce récepteur est internalisé par un autre mécanisme, indépendant des clathrines, qui sera décrit ci-après (Chan et al., 2004; Hommelgaard et al., 2004; Klapper et al., 2000). L’ubiquitinylation d’ErbB3 est préférentiellement assurée par Nrdp1 (Neuregulin receptor degradation protein-1) alors que celle d’ErbB4 est attribuée à Nedd4 (neural precursor cell expressed developmentally down-regulated protein 4) (Chen et al., 2010).
Figure 7 : Modèle d’endocytose clathrine- dépendante. En réponse à la fixation du ligand, le domaine
intra-cytoplasmique du récepteur RTK va s’associer aux clathrines sous-membranaires par 2 voies: A) à travers une interaction directe avec des motifs présents sur le récepteur ou par l’intermédiaire de protéines se liant aux phosphotyrosines (PTB). B) après ubiquitinylation du récepteur par une E3-ubiquitine ligase (Cbl) des protéines
cytoplasmiques possédant des domaines SH (Src homology) comme Grb, ou possédant un motif d’interaction avec les ubiquitines (UIM) sont recrutées et se lient ensuite aux molécules de clathrine. D’après Goh and Sorkin, 2013 (Goh and Sorkin, 2013).
13 Le schéma suivant résume les principaux motifs et modifications post-traductionnelles des récepteurs ErbB impliqués dans l’endocytose clathrine-dépendante (Figure 8).
D’autres modifications post-traductionnelles que nous ne détaillerons pas ici participent à ce mécanisme d’endocytose et à sa régulation. Ainsi, la phosphorylation de la thréonine Thr654 d’ErbB1 par la protéine kinase C (PKC) exerce un effet négatif sur la CME, alors que la phosphorylation des sérines 1046 et 1047 la stimule (Countaway et al., 1992).
2. Endocytose clathrine-indépendante
L’endocytose dépendante des clathrines est un processus rapide, mais dont la capacité d’internalisation est limitée. Il peut donc être saturé en cas de surexpression du ligand (EGF 20-100 ng/ml) ou des récepteurs. Quand les conditions ne sont plus physiologiques, la voie CME peut être relayée par une voie plus lente appelée CIE pour Clathrin Independant Endocytosis (Wiley, 1988) (Figure 9). Plusieurs mécanismes sont associés à ce type d’endocytose. Le premier est la macropinocytose, une endocytose induite et transitoire qui implique des réarrangements membranaires formant de grosse vésicules hétérogènes. Ce processus a été mis en évidence dans des cellules de carcinome épidermoïde exprimant des taux élevés du récepteur ErbB1 activé (Haigler et al., 1979). Plus récemment, l’utilisation
Figure 8 : Séquences et modifications post-traductionnelles associées à l’endocytose clathrine-dépendante des récepteurs ErbB. Les
résidus tyrosine phosphorylés (PY) des récepteurs lient les domaines SH2 de la protéine Grb2, dont les domaines SH3 s’associent aux motifs riches en proline de l’E3-ubiquitine ligase Cbl (à droite). Les ubiquitine ligases de la famille NEDD se lient aux motifs PPxY des récepteurs ErbB et recrutent Grb10, dont le domaine SH2 s’associe aux tyrosines phosphorylées du récepteur (à gauche) (Goh and Sorkin, 2013).
14 d’amiloride, un inhibiteur spécifique de la macropinocytose a confirmé l’implication de ce type d’endocytose dans le trafic intracellulaire d’ErbB3 (Koumakpayi et al., 2011).
Le second mécanisme fait intervenir des domaines membranaires riches en lipides, cholesterol et sphingomyeline, et en cavéoline, l’ensemble formant des radeaux lipidiques (lipid rafts) (Figure 8) (Lajoie and Nabi, 2010; Lajoie et al., 2009; Salani et al., 2010). Dans ces deux types de mécanismes, la plupart des complexes récepteurs-ligands sont internalisés avec une vitesse lente, favorisant ainsi l’exposition des récepteurs actifs à la surface cellulaire (Ke CIE=0,05-0,1min-1 vs Ke CME= 0,2-0,4 min-1).
3. Devenir des récepteurs internalisés
Les vésicules d’endocytose formées qu’elles soient ou non clathrine-dépendantes, vont ensuite fusionner avec des endosomes primaires (EE).
Trois voies sont alors possibles :
- les récepteurs sont recyclés à la membrane plasmique par un processus rapide de rétroendocytose (2 à 5mn) ou après fusion avec des vésicules de recyclage péricentriolaires (VRP) (30 à 60mn).
- L’endosome primaire (EP) mature pour former un endosome de tri ou endosome multivésiculaire (SE/MVB) dont le pH acide peut induire la dissociation des complexes ligand-récepteurs. Une partie des récepteurs de ces endosomes de tri vont pouvoir être recyclés alors que d’autres seront dégradés après fusion avec les lysosomes primaires (Lys) (Figure 9).
- Enfin, les endosomes contenant les récepteurs peuvent être dirigés vers le noyau. Cette voie, sera traitée dans le chapitre spécifique sur les fonctions nucléaires des récepteurs ErbB.
15 Après activation par l’EGF, ErbB1 se retrouve donc au niveau des endosomes primaires puis des endosomes de tri. Des prolongements tubulaires formés à partir de ces derniers vont permettre le ré-adressage de certains récepteurs à la membrane (Figure 9). Mais la majorité des récepteurs se retrouve au niveau de vésicules intra-luminales (ILV) qui se forment par bourgeonnement à partir de la membrane de l’endosome. La stabilité du complexe EGF-ErbB1 favorise la translocation des récepteurs dans ces vésicules et permet par la suite la fusion avec les lysosomes où les récepteurs seront dégradés (Roepstorff et al., 2008). Si le rôle de l’ubiquitinylation dans l’internalisation est encore controversé, il est acquis en revanche que Cbl joue un rôle majeur dans cette voie de dégradation lysosomale : le processus est en effet sévèrement altéré si l’on mute les résidus lysines cibles de l’ubiquitinylation (Figure 8) ou les tyrosines phosphorylées liant l’ubiquitine-ligase Cbl (Grøvdal et al., 2004; Huang et al., 2006; Jiang and Sorkin, 2003). Il semble que la nature du ligand influence le devenir des récepteurs internalisés. Il a été montré que les complexes TGFa-ErbB1 sont beaucoup moins stables que les complexes EGF-ErbB1 dans l’environnement acide des endosomes. Ils se dissocient rapidement ce qui entraîne une déphosphorylation et une désubiquitinylation du récepteur et favorise alors la voie du recyclage (Longva et al., 2002).
Le récepteur ErbB2, moins susceptible d’être internalisé qu’ErbB1, est généralement recyclé à la membrane plasmique (Austin et al., 2004), ce qui n’est pas le cas d’ErbB3 et ErbB4 dont l’ubiquitinylation induit préférentiellement la dégradation (Carraway, 2010; Omerovic et al., 2007).
Figure 9 : Devenir des récepteurs internalisés. Les récepteurs internalisés
après fixation du ligand se retrouvent au niveau des endosomes primaires (EP). Certains récepteurs seront recyclés rapidement à la membrane (2-5 min) alors que d’autres seront dirigés vers les endosomes matures (SE/MVB). En fonction de différents paramètres tels que la nature et la stabilité du complexe ligand-récepteur, l’ubiquitinylation du récepteur, ils seront orientés soit vers un recyclage tardif (30-60 min) soit vers une dégradation lysosomale. D’après Goh and Sorkin, 2013 (Goh and Sorkin, 2013)
16 L’avenir de ces récepteurs dépend donc de la nature des dimères formés, de la nature du ligand et de la dissociation ou non du complexe récepteur-ligand au sein des vésicules d’endocytose. Par ailleurs, ces processus d’internalisation ne réduisent pas seulement le taux des récepteurs à la surface cellulaire et l’intensité de signalisation intracellulaire de ces récepteurs, mais aussi la concentration des facteurs de croissance dans le milieu extracellulaire (Sorkin and Von Zastrow, 2002).
II. Les voies de signalisation cytoplasmiques en aval des
récepteurs ErbB
Comme nous venons de le voir, la spécificité et la puissance des signaux intracellulaires médiés par le réseau ErbB sont déterminées par la nature et la quantité des récepteurs à la membrane, l'identité et la quantité du ligand, la composition des oligomères mais également les résidus tyrosines présents en C-ter des récepteurs et les protéines adaptatrices qui s’y lient (Yarden and Sliwkowski, 2001). Les différences de séquences au niveau des régions régulatrices en C-ter créent en effet après phosphorylation une multitude de sites de liaisons pour des protéines aussi variées que la PLC, Grb, Shc ou des kinases comme PI3K (p85) (Figure 10). Les récepteurs ErbB initient également des voies de signalisation qui impliquent la PLCγ, des GTPases (Guanosine TriPhosphate hydrolases) comme Rho et Rac, ou des isoformes de STAT (Olayioye et al., 2000).
En fonction du profil de phosphorylation et par le biais de multiples médiateurs, les récepteurs ErbB activent les voies de signalisation Ras/Raf/MAPK (mitogen-activated protein kinase) ou PI3K/AKT (phosphatidylinositol-3 kinase), ou encore activent directement des facteurs de transcription comme les STAT (Signal Transducer and Activator of Transcription) (Burgess et al., 2003; Holbro and Hynes, 2004).
17
A. La voie MAPK
La voie MAPK régule l’activité de facteurs de transcription impliqués dans la progression du cycle cellulaire, la migration et l’invasion (Raman et al., 2007; Yarden and Sliwkowski, 2001). Tous les récepteurs ErbB sont capables d’initier cette voie de signalisation après activation. La fixation du ligand sur le récepteur ErbB transmet le signal à la cellule par l’intermédiaire de la GTPase Ras, présente à la face interne de la membrane plasmique et recrutée par l’intermédiaire de Grb2 ou Shc (Figure 11, A). Sous forme activée liée au GTP, Ras recrute et active la serine-thréonine kinases Raf (MAP kinase kinase kinase). Raf activée phosphoryle à son tour MEK (MAP kinase kinase kinase) qui en réponse phosphoryle Erk1 et Erk2 (Extracellular Signal regulated kinases, MAPKs). L’activation des Erks conduit à leur translocation dans le noyau où elle régule l’expression des gènes en phosphorylant des facteurs de transcription. Selon le même mécanisme, en recrutant et activant la kinase MEKK1, Rac permet la translocation nucléaire de la kinase JNK (Jun kinase). Ces facteurs se lient ensuite aux régions régulatrices de gènes de prolifération et de migration dont ils activent la transcription, proto-oncogènes comme Fos, Jun et Myc mais aussi des facteurs à doigts de Zinc comme Sp1, Egr1 et certains membres de la famille Ets.
Figure 10 : Protéines liant les phosphotyrosines des domaines C-ter des récepteurs ErbB. Les différentes
protéines sont présentées pour chaque récepteur. Elles se lient via leurs domaines SH2 (Src homology domain 2) ou les domaines de reconnaissance de résidus phosphotyrosine. Les résidus phosphorylés sont signalés par leur position dans la protéine. Le récepteur ErbB3 présente principalement des sites de liaisons de la sous unité p85 de la PI3K. Ralt, PSD-95, Pick1 et Erbin sont des partenaires des récepteurs ErbB mais ils se lient indépendamment de la phosphorylation (Olayioye, 2001).
18
B. La voie PI3K
La voie PI3K est impliquée dans la résistance à l’apoptose, la prolifération, la migration et l’angiogenèse (Scaltriti and Baselga, 2006; Vivanco and Sawyers, 2002). L’activation de cette voie par les récepteurs ErbB nécessite l’association de la sous-unité adaptatrice p85 de PI3K au récepteur, par l’intermédiaire de ses domaines SH2. Ceci entraîne alors l’activation allostérique de la sous unité catalytique p110, la production du second messager PIP3 (Plasma membrane Intrinsic Protein 3) et le recrutement d’Akt. Phosphorylée, Akt peut réguler la survie cellulaire soit en inactivant des facteurs pro-apoptotiques telles que la caspase 9 ou Bad (Leterrier and Constantin, 1999; Song et al., 2005) ou en bloquant l’expression de gènes pro-apototiques comme TRAIL - TNF-related apoptosis inducing ligand), Fas ligand, TRADD (TNFRSF1A-associated via death domain) (Datta et al., 1999) soit, à l’inverse, en activant la voie NF-KB (Nuclear Factor-Kappa B) anti-apoptotique (Barkett and Gilmore, 1999; Lauder et al., 2001). Akt intervient également dans la croissance cellulaire et la prolifération à travers l’activation de la serine/thréonine kinase mTOR (Mammalian Target Of Rapamycin) ou l’inhibition de GS3K (Glycogène synthase 3 kinase) qui stabilise la cycline D1 (Inoki et al., 2002; Kozma and Thomas, 2002).
Le potentiel et la cinétique d’activation de la voie PI3K par les récepteurs ErbB sont hétérogènes car dépendant des ligands impliqués et des dimères formés. C’est la principale
19 voie activée en aval d’ErbB3 en raison du nombre élevé de sites de liaison pour la sous unité p85 de PI3K, alors qu’ErbB1 ou ErbB2 l’activent indirectement après hétérodimérisation avec ErbB3 ou ErbB4.
C.
La voie STAT
La voie STAT (Signal Transducer and Activator of Transcription) est impliquée dans la régulation du cycle cellulaire, de l’apoptose et de la différenciation (Calò et al., 2003). La liaison du ligand à son récepteur induit l’activation des kinases intracellulaires JAK (Janus Kinase) ou Src, qui phosphorylent les protéines cytoplasmiques STAT. Les STAT ainsi activées forment des dimères, par interaction entre le domaine SH2 d’un partenaire et les tyrosines phosphorylées de l’autre, puis sont transloquées dans le noyau où elles régulent l’expression de gènes de prolifération (Aaronson and Horvath, 2002).
III. Les voies de signalisation nucléaires des récepteurs ErbB
Un nombre croissant de publications fait état de la présence des récepteurs de la famille ErbB dans les noyaux de cellules normales ou tumorales et de fonctions associées à cette translocation. Selon le récepteur, cette localisation nucléaire peut concerner la molécule dans sa totalité (Bueter et al., 2006; Dittmann et al., 2005; Marti et al., 1991; Offterdinger et al., 2002; Thompson et al., 2007; Xie and Hung, 1994; Xu et al., 2009), ou seule la région intracytoplasmique (Adilakshmi et al., 2011; Andrique et al., 2012; Ni et al., 2001). Les récepteurs ErbB nucléaires pourraient ainsi participer à des mécanismes de régulation transcriptionnelle, prolifération, réplication… et se retrouver associés, dans les cellules tumorales, aux mécanismes de résistance aux thérapies (Carpenter and Liao, 2009; Schlessinger, 2004; Wang and Hung, 2009; Wang et al., 2010b).
A. Mécanisme de translocation et fonctions nucléaires d’ErbB1
C’est en 1991 qu’a été rapportée pour la première fois la présence du récepteur ErbB1 dans les noyaux des hépatocytes au cours de la régénération du foie chez le rat (Marti et al., 1991). La forme entière d’ErbB1 (170kDa) a par la suite été retrouvée dans les noyaux de cellules de cancer du sein (Wang et al., 2010b), de l’ovaire (Xia et al., 2009), du pancréas (Jaganathan et al., 2011; Merlin et al., 2011), de la tête et du cou (Li et al., 2010), des poumons (Das et al., 2007) et de la peau (Xu et al., 2009). Deux isoformes du récepteur
20 ErbB1 de 145 kDa et 45 kDa ont été décrites dans les noyaux de glioblastome (de la Iglesia et al., 2008; Lo, 2010; Piccione et al., 2012) et dans le cancer de la prostate (Edwards et al., 2006). La présence de ce récepteur dans les noyaux des cellules tumorales, suggère qu’il joue un rôle important dans la tumorigenèse (Tableau 1).
La présence d’ErbB1 nucléaire dans les cancers du sein (Hadzisejdić et al., 2010), de l’ovaire (Xia et al., 2009), de l’oropharynx (Psyrri et al., 2008), du poumon (Traynor et al., 2013) est inversement corrélée avec la survie des patients et constitue donc un facteur de mauvais pronostic.
1. Mécanisme de translocation nucléaire du récepteur ErbB1
L’endocytose clathrine-dépendante et -indépendante du récepteur ErbB1 est cruciale pour la régulation négative, la dégradation et le recyclage du récepteur vers la membrane comme nous l’avons vu précédemment. Suite à l’activation par l’EGF, l’endocytose représente aussi la première étape de translocation du récepteur vers les compartiments intracellulaires comme l’appareil de golgi et le réticulum endoplasmique (Wang et al., 2010b). C’est l’état de phosphorylation du récepteur ErbB1 qui détermine sa destination après endocytose. Il est dirigé vers le noyau lorsque le résidu sérine 229 est phosphorylé par AKT (Huang et al., 2011). L’endosome précoce contenant ErbB1, fusionne avec l’appareil de golgi, puis le récepteur ErbB1 s’achemine vers le réticulum endoplasmique grâce au complexe COPI (Coat Protein Complex I), par un processus appelé transport rétrograde. En induisant la dissociation de ce complexe au niveau du Golgi, par la brefeldine A ou par l’utilisation d’un mutant dominant négatif de la GTPase ARF1, on observe une inhibition du transport d’ErbB1 vers le RE (réticulum endoplasmique) et le noyau (Wang et al., 2010b). Une fois dans le réticulum endoplasmique, le récepteur ErbB1 est dirigé vers le noyau par un mécanisme importine-α/β-dépendant.
La translocation nucléaire implique le signal de localisation nucléaire (NLS) constitué de treize acides aminés en position 645-657 du domaine transmembranaire du récepteur ErbB1. Cette séquence peptidique contient trois groupements d’acides aminés basiques RRRHIVRKRTLRR capables de fixer les groupements latéraux acides de l’importine-α et l’importine-β (Giri et al., 2005; Lo et al., 2006). L’importine-α/β permet à ErbB1 de traverser la membrane nucléaire externe pour atteindre la membrane nucléaire interne, où se trouve la protéine Sec61β. A son tour, Sec61β internalise le récepteur ErbB1 dans le nucléoplasme (Wang and Hung, 2012; Wang et al., 2010c).