THESE DE DOCTORAT
Pour l’obtention du grade de
DOCTEUR DE L’UNIVERSITÉ DE POITIERS
Faculté de Médecine et de Pharmacie Diplôme National – Arrêté du 07 août 2006
Ecole Doctorale BioSanté n°524
Secteur de recherche : Aspects Moléculaires et Cellulaires de la Biologie
Présentée par
Dominique FAUVIN
Caractérisation de nouvelles fonctions du suppresseur
de tumeurs p14
ARFà travers deux exemples :
Le récepteur à activité tyrosine kinase ErbB3 et
le facteur de transcription UBF1.
Thèse dirigée par le Dr. Paule SÉITÉ
Soutenue publiquement le 14 décembre 2009 Devant la Commission d’Examen
JURY
Président : Pr. Omar BENZAKOUR Université Poitiers, CNRS UMR 6187 Rapporteurs :
Pr. Franck TOLEDO Université Paris VI, CNRS UMR 3244 Dr. Ivan BIECHE Université Paris V, INSERM U735 Examinateur :
THESE DE DOCTORAT
Faculté de Médecine et de Pharmacie Ecole Doctorale BioSanté n°524
Secteur de recherche : Aspects Moléculaires et Cellulaires de la Biologie
Dominique FAUVIN
Caractérisation de nouvelles fonctions du suppresseur
de tumeurs p14
ARFà travers deux exemples :
Le récepteur à activité tyrosine kinase ErbB3 et
le facteur de transcription UBF1.
Institut de Physiologie et Biologie Cellulaire
Equipe Pathologies Moléculaires de l’Adressage et de la Signalisation
UMR 6187, CNRS
Directeur Pr. Frédéric Becq.
Thèse dirigée par le Dr. Paule SÉITÉ
Adresse : Pôle Biologie Santé,
UMR6187, PMAS,
40 avenue du Recteur Pineau,
86 022 Poitiers Cedex
Mail :
dominique.fauvin@univ-poitiers.fr
paule.seite@univ-poitiers.fr
RÉSUMÉ
La protéine ARF (p14ARF chez l’Homme et p19ARF chez la Souris) est une protéine nucléolaire qui contrôle négativement le cycle cellulaire par le relais de p53. Elle peut également arrêter la prolifération dans des modèles cellulaires déficients en p53, ce qui suggère l’existence de voies de régulation alternatives à la voie ARF-MDM2-p53, impliquant d’autres cibles. Ce travail de thèse a été initié autour des rôles de la protéine ARF à travers la caractérisation de nouveaux partenaires, dans un contexte indépendant de p53.
1ère partie : Nous avons mis en évidence une diminution d’expression du transcrit ErbB3/HER3 en réponse à la surexpression de p14ARF, dans une lignée tumorale déficiente en p53 (H358, p53-/-). Ce transcrit code un récepteur membranaire à activité Tyrosine-Kinase de la famille EGF-R, le récepteur ErbB3. L’activation de ces récepteurs induit l’expression de gènes nécessaires à des mécanismes aussi variés que la prolifération, la migration cellulaire, la différenciation, l’angiogénèse, à travers les voies de signalisation MAPK et PI3K. De récentes études montrent également que certains récepteurs de cette même famille sont parfois transloqués dans le noyau pour y jouer un rôle direct d’activateur de la transcription de gènes cibles.
Nous avons mis en évidence :
- la présence dans le noyau d’une protéine ErbB3 100 kDa correspondant à l’extrémité intracytoplasmique du récepteur.
- ErbB3 100 kDa se lie et régule positivement le promoteur de la Cycline D1, induisant la prolifération des cellules H358.
- Cet effet ne nécessite pas l’activation du récepteur par son ligand et est indépendant des voies de signalisation cytoplasmiques.
-Enfin, la protéine ARF, lorsqu’elle est surexprimée, séquestre ErbB3 100 kDA au sein du nucléole, inhibant son interaction activatrice avec le promoteur de la cycline D1 et régulant négativement la prolifération cellulaire.
2ème partie : Nous avons précédemment décrit une association spécifique entre p14ARF et le promoteur de l’ADN ribosomique (ADNr), dans cette même lignée cellulaire H358.
La transcription du précurseur ARNr 47S fait intervenir le facteur de transcription UBF1, dont la phosphorylation est médiée par les complexes Cycline D/CDK4-6 de la phase G1. Dans notre modèle cellulaire, l’expression d’ARF et sa liaison au promoteur ADNr, s’accompagne d’une hypophosphorylation d’UBF1 et d’un ralentissement du processus de transcription du 47S in vitro et in vivo. Cet effet paraît médié par la diminution d’expression de la cycline D1 réduisant la formation des complexes cycline D/CDK4/6.
Ce travail dessine une nouvelle voie de régulation de la prolifération et de la croissance cellulaire, par laquelle p14ARF inhiberait la prolifération cellulaire en délocalisant ErbB3 des noyaux vers les nucléoles, diminuerait ainsi la transcription de la cycline D1, et la transcription de l’ADNr.
ABSTRACT
The nucleolar ARF protein (p14ARF in human and p19ARF in mouse) negatively regulates the cell cycle through the well known ARF-MDM2-p53 pathway. However, ARF can also exert its tumor suppressor functions independently of p53, suggesting the existence of alternative pathways.
The aim of this work consisted in the characterization of new p14ARF functions, involving two partners, the ErbB3 membrane receptor and the ribosomal transcription factor UBF1, in p53 deficient cell lines.
1st part: By studying the expression of ErbB3 in H358 lung adenocarcinoma cells (p53-/-) we have discovered that:
- independently of ligand activation, the 185 kDa ErbB3 receptor was translocated into the nucleus as a short 100kDa protein corresponding to the intracytoplasmic domain of the receptor.
- the nuclear ErbB3 protein was found to interact with and negatively regulate the CyclinD1 promoter, activating cell proliferation.
- in response to p14ARF overexpression, the 100 kDa ErbB3 was down-regulated and partially delocalized in the nucleolus, thus inhibiting CyclinD1 promoter activation.
2nd part : We have previously described a specific interaction between p14ARF and the ribosomal DNA promoter in the H358 cell line. UBF1 is a major transcription factor for rDNA transcription whose phosphorylation is mediated by CyclinD/CDK4-6 complexes. We show that, in response to p14ARF overexpression, UBF1 is hypo-phosphorylated and the transcription of the 47S precursor is reduced both in vitro and in vivo. This suggests that ARF can exert a negative transcriptional control on ribosome biogenesis. This regulation is associated with reduced Cyclin D1 expression.
REMERCIEMENTS
Je n’aurais pas pu soutenir cette thèse sans la relecture attentive et l’analyse précise de mon travail.
Je remercie donc chaleureusement les Pr. Franck Toledo et Dr. Ivan Bièche pour avoir accepté de rapporter ma thèse.
Je remercie le Pr. Omar Benzakour d’avoir bien voulu en être examinateur. Merci à tous les trois d’avoir consacré du temps pour juger ce travail.
Je tiens également à remercier le Pr. Frédéric BECQ de m’avoir accueillie dans l’Institut de Physiologie et Biologie Cellulaire, UMR6187.
Je tiens évidemment à remercier le Pr. Alain KITZIS de m’avoir permis d’intégrer son équipe « Pathologies Moléculaires de l’Adressage et de la Signalisation ». Vous m’avez montré à plusieurs reprises que je pourrai toujours compter sur vous.
Je souhaite exprimer toute ma gratitude au Docteur Paule SÉITÉ pour m’avoir accueillie au sein de son laboratoire. Je suis consciente d’avoir eu beaucoup de chance d’avoir été encadrée par une personne comme toi.
En plus de tes qualités scientifiques, tu es enjouée et sympathique, honnête et franche. Autant de qualités qui me font dire que tu es la meilleure chef qu’un thésard puisse avoir. Tu m’as laissé beaucoup d’autonomie, mais étais toujours là quand je n’y arrivais plus! Merci de m’avoir soutenue durant mes moments de doutes !
La formation que j’ai eue au sein de ton labo a été excellente, tant au niveau scientifique qu’humain.
Tu es de plus très impliquée pour tes thésards, trop certainement, ce qui ne te facilite pas le quotidien… ;-p J’espère n’avoir jamais trop tiré sur la corde durant ces 3 ans, mais si c’est le cas, j’en suis vraiment désolée.
Un VRAI Grand MERCI !!!!
MERCI !
Je remercie tout particulièrement les étudiants du laboratoire avec qui j’ai eu plaisir à travailler :
- Olivier, Laetitia et Nissrine pour la formation, tant intellectuelle que technique, qu’ils m’ont donnée.
- Hélène pour cette petite dose de bonne humeur qu’elle a insufflée durant ma dernière année de thèse. Bien que tes « chef, oui chef » et bavardages me sortent par les yeux, j’ai eu beaucoup de plaisir à travailler avec toi. Bonne continuation !!!
- Les étudiants de Master et Licence et surtout Emilien mon chouchou…
Je remercie sincèrement tous les membres de l’équipe PMAS/LGCM, pour les bons moments passés à vos côtés, dans la salle café, au RU, en réunions, au hasard du couloir, lors des apéritifs ou repas d’équipe… Marie-Claude, Marlène, Ayman,
Véronique, Barbara, Valérie, Nathalie, Patricia, Fred, Vincent, Sylvie, Agnès et Isabelle merci pour tous ces bons moments passés ensemble.
Je remercie tous les « jeunes chercheurs » du laboratoire avec qui j’ai toujours eu un grand plaisir à travailler. Mais surtout, soyons honnêtes, pour toutes les soirées passées ensemble : Hélène, Romain, Fleur, Tristan, Thomas H, Thomas D, Jacques…
Pour leur aide technique, je remercie chaleureusement Sylvie Patri pour tous ses conseils de PCR-quantitative, Jean-Marc Gombert pour sa « bonne humeur ;-p » lorsqu’il m’aidait dans mes expériences de cytométrie en Flux et Anne Cantereau pour son aide précieuse en microscopie confocale.
Cette thèse aurait eu un autre goût si je n’avais pas croisé les thésards, étudiants et post-doc des autres laboratoires et de l’IBMIG, les membres de l’ADBEP et du comité d’organisation de BIOTechno ! Merci pour toutes ces réunions, soirées tarot, poker ou jeu de rôle, repas et fiesta !
Merci à mes amis, tout particulièrement aux anciens du primaire et collège (Anaïs, Griet et Caro pour nos « Camps »), du lycée (Milie, Anne et Pascale pour nos week-ends de folie) et de la fac (Angie, Delphes, Olivier, Reinhard…). Je vous adore et ai vraiment beaucoup de chance d’avoir des supers amis comme vous. J’espère ne jamais vous perdre de vue !
Je remercie toute ma famille pour m’avoir soutenue, avoir toujours cru en moi et avoir toujours été là dans mes moments de doute ! Vous êtes vraiment géniaux : mes parents chéris, mes sœurettes que j’aime, mes supers beaux-frères, et ma pucinette Louise que j’aime tellement !!!
Et en remerciant ma famille, je pense aussi à mamie et papi, pour leur grand confiance en moi, leurs supers mails, lettres et câlins dès que j’en avais besoin ! Yves et Babettes, François et Françoise, Roland et Anne-lise, et tous mes cousins-cousines ! Nous avons la chance d’être une famille très unie, et ceci m’a beaucoup aidée durant ces 3 dernières années. Ces retrouvailles familiales, bien que trop souvent pluvieuses, étaient des havres de réconfort. Merci d’être vous !!!
Merci à tous ceux qui m’ont permis d’avancer dans ma vie, au hasard d’une discussion, d’une rencontre, d’un bout de chemin passé ensemble…
TABLE DES MATIÈRES
ABRÉVIATIONS ... 1
INTRODUCTION ... 4
Chapitre 1 : La protéine suppresseur de tumeurs ARF ... ………...5
I. Le locus INK4a/ARF ... 5
1) Structure et expression ... 5
2) Importance du locus dans la tumorigenèse. Eléments de régulation.. ... 6
2a) p16INK4a ... 6
2b) p14ARF ... 8
2c) Régulations conjointes 11
II. La protéine ARF ... 12
1) Caractéristiques biochimiques et structurales. ... 12
1a) Composition en acides aminés ... 12
1b) Localisation intracellulaire ... 13
2) Expression et régulation de la protéine ARF ... 15
2a) Régulation transcriptionnelle ... 16
L’oncogène Ras ... 16
c-Myc ... 17
La famille E2F ... 17
2b) Régulation par interaction protéine-protéine ... 18
Pex19p ... 18
Dégradation par la voie du protéasome ... 19
III. Fonctions de ARF ... 21
1) La voie ARF-MDM2-p53. ... 21
1a) Découverte du rôle p53 dépendant de la protéine ARF. ... 21
1b) La protéine MDM2 ... 22
1c) La protéine p53 ... 23
1d) MDM2 régulateur majeur de la réponse p53 ... 25
1e) Régulation par la protéine suppresseur de tumeurs ARF. ... 26
2a) Mise en évidences de voies alternatives ... 30
2b) Régulation en G1/S ... 30
ARF et le complexe DP1/E2F1 ... 31
ARF et Myc ... 31
2c) Régulation en G2/M ... 33
2d) ARF et le contrôle de la biogenèse des ribosomes. ... Rappel sur la biogenèse des ribosomes ... 34
ARF régule la maturation du transcrit 47S ... 36
Chapitre 2 : Les récepteurs membranaires ErbB………... .39
I. Les récepteurs EGF-R/ErbB/HER à activité tyrosine kinase ... 39
1) Les membres de la famille ErbB ... 39
2) Activation des récepteurs ErbB ... 44
2a) Mécanisme de dimérisation des récepteurs ... 44
2b) Les ligands des récepteurs EGF-R ... 45
2c) Activation hétérologue ... 46
3) Voies de signalisation en aval des récepteurs EGF-R ... 47
3a) Phosphorylation des récepteurs ... 47
3b) Voies de signalisation activées ... 48
4) Inactivation/Dégradation des récepteurs ErbB ... 50
II. Fonctions des récepteurs de la famille EGF-R ... 52
1) Fonctions physiologiques des récepteurs EGF-R. ... 52
2) Implication des récepteurs EGF-R dans les processus tumorigènes. ... 53
3) Thérapies ciblées sur les récepteurs EGF-R 55
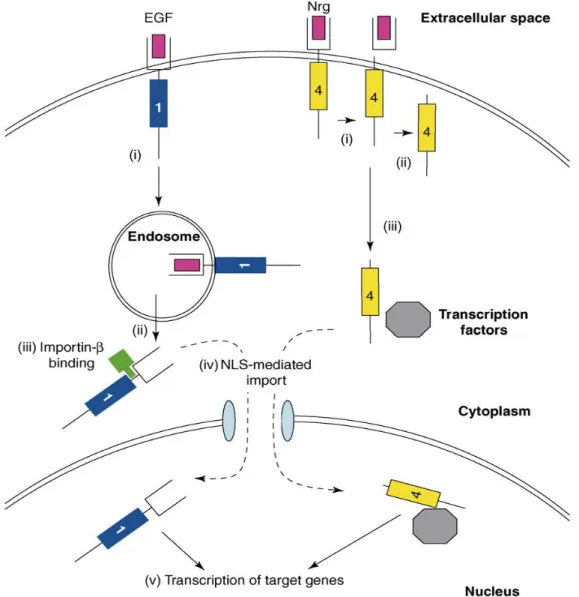
III. Expression nucléaire des récepteurs EGF-R : Vers de nouvelles fonctions ? ... 56
1) Import et localisation nucléaire ... 57
1a) Mécanisme de localisation nucléaire des récepteurs EGFR et ErbB2 ... 57
1b) Localisation nucléaire du récepteur ErbB3 ... 59
1c) Mécanisme de localisation nucléaire du récepteur ErbB4 ... 59
2) Fonctions nucléaires des récepteurs de la famille EGF-R ... 60
2a) Gènes cibles régulés par EGFR ... 60
2b) Gènes cibles régulés par ErbB2 ... 62
Chapitre 3 : Transcription de l'ADN ribosomique……… ... …63
I. Le nucléole : site majeur de synthèse des ARN ribosomiques ... 63
II. La transcription de l’ADNr ... 65
1) Le promoteur ADNr ... 65
2 Complexe d’initiation de la transcription ... 66
2a) SL1 : ... 66
2b) hRRN3 : ... 67
2c) UBF1 : ... 68
3) Mise en place du complexe d’initiation sur le promoteur. ... 69
III. Régulation de la transcription du 47S ... 70
1) Un mécanisme hautement contrôlé ... 70
2) Nombre de copies du gène de l’ADNr ... 71
3) Régulation des facteurs du complexe transcriptionnel ... 71
3a) Le facteur de transcription UBF1 ... 72
3b) Le facteur de transcription SL1 ... 73
4) Régulation de la transcription par des oncogènes et des suppresseurs de tumeurs ... 74
4a) RB inhibe la transcription de l’ADNr. ... 74
4b) p53 inhibe la transcription de l’ADNr. ... 76
4c) Myc régule la transcription de l’ADNr. ... 76
4d) ARF inhibe la transcription de l’ADNr. ... 77
RÉSULTATS ... 78
Chapitre 1 : Régulation de l’expression et de la fonction du récepteur membranaire ErbB3 par le suppresseur de tumeur p14ARF……… ... 79
I. p14ARF régule la transcription du gène ErbB3 ... 80
1) Analyse par Differential Display et test d’activité luciférase ... 80
1a) Résultats du Differential Display ... 80
1b) Tests d’activité luciférase ... 81
1c) Analyse du cycle cellulaire par cytométrie en flux ... 82
2) p14ARF se lie au promoteur du gène ErbB3. ... 84
2a) Validation de l’anticorps anti-p14ARF ... 85
II. p14 modifie l’expression et la localisation intracellulaire de la protéine ErbB3 ... 87
1) Le niveau d’expression d’ErbB3 est réduit en réponse à la surexpression de p14ARF. ... 87
2) L’expression de p14ARF ne régule pas l’expression des autres EGF-R. ... 88
3) Mesure de l’expression membranaire d’ErbB3 par cytométrie en flux. ... 89
4) Etude par immunofluorescence de la localisation subcellulaire d’ErbB3. ... 90
III. Caractérisation de la protéine ErbB3 nucléaire ... 92
1) Analyse de l’expression du récepteur ErbB3 après fractionnement cellulaire. ... 92
2) Vérification de la spécificité de l’anticorps C-17 anti-ErbB3. ... 94
3) Caractérisation de la protéine 100 kDa. ... 96
3a) Expériences d’immunoprécipitation ... 96
Immunoprécipitation avec l’anticorps C-17 ... 97
Immunoprécipitation avec l’anticorps Ab-5 ... 97
3b) Immunofluorescence ... 98
IV. Etude des fonctions de la protéine ErbB3 dans le noyau ... 99
1) Signalisation dépendante du récepteur ErbB3 dans la lignée H358. ... 99
2) La protéine nucléaire ErbB3 régule la transcription du gène cycline D1. ... 101
2a) La surexpression d’ErbB3 active le promoteur cycline D1. ... 102
2b) Construction d’un mutant de délétion N-ErbB3 et validation... 103
Construction du vecteur pEGFP- N-ErbB3 ... 103
Expression de la protéine de fusion N-GFP- N-ErbB3-C. ... 104
Localisation cellulaire de la protéine de fusion N-GFP- N-ErbB3-C. ... 105
2c) La surexpression du mutant N-GFP- N-ErbB3-C active le promoteur du gène cycline D1. ... 106
2d) ErbB3 100 kDa se lie au promoteur du gène cycline D1 ... 107
3) Conséquences fonctionnelles de la liaison d’ErbB3 100 kDa sur le promoteur de la cycline D1. ... 108
3a) Conséquences sur l’expression de la protéine cycline D1. ... 108
3b) Conséquences sur la prolifération cellulaire. ... 109
V. Et p14ARF dans tout ça ? ... 111
1) Interaction physique entre p14ARF et la protéine ErbB3 ... 112
1a) Expériences de GST-Pull-down ... 112
2) p14 régule la fonction d’ErbB3 sur le promoteur cycline D1 ... 114
2a) L’expression de p14ARF inhibe la fixation d’ErbB3 au promoteur cycline D1. .... 114
2b) L’expression de p14ARF régule négativement la transcription du gène cycline D1. ... 116
2c) p14ARF ne se lie pas au promoteur cycline D1. ... 117
2d) Le contrôle exercé par p14ARF sur le promoteur de la cycline D1 dépend d’ErbB3 100 kDa. ... 118
3) Conséquences fonctionnelles de cette régulation. ... 119
3a) Conséquences sur l’expression de la protéine cycline D1. ... 119
3b) Conséquences sur la prolifération cellulaire. ... 120
Chapitre 2 : Le suppresseur de tumeurs p14ARF est un régulateur transcriptionnel de l'ADNr……… ... 123
I. p14ARF régule la transcription des gènes ADNr. ... 123
1) La surexpression de p14ARF induit une diminution de l’activité du promoteur ADNr. ... 123
1a) Conséquences de la surexpression de p14ARF par transfection dans la lignée H358wt. ... 123
1b) Conséquences de la surexpression de p14ARF par induction dans la lignée H358/Tet-On/p14ARF . ... 125
1c) Conséquences de la surexpression de p14ARF par un signal oncogénique. ... 126
2) Conséquences de la surexpression de p14ARF sur la synthèse du précurseur ARNr 47S. ... 127
II. p14ARF interagit avec le complexe d’initiation de la transcription. ... 129
1) p14ARF délocalise UBF1 de la zone fibrillaire vers la zone granulaire du nucléole. ... 129
2) p14ARF interagit physiquement avec UBF1 ... 130
3) Compétition entre p14ARF et UBF1 pour la liaison au promoteur ADNr. ... 131
3a) Dynamique de liaison des protéines p14ARF, UBF1 et RPA194 au promoteur. ... 131
3b) Conséquences de cette compétition sur l’activation du promoteur. ... 133
III. p14ARF modifie l’activation post-traductionnelle d’UBF1. ... 134
2) Etude de la phosphorylation d’UBF1 au cours du cycle cellulaire en fonction de l’expression de p14ARF
. ... 135
2a) Analyse du cycle ... 135
2b) Synchronisation des cellules ... 136
2c) p14ARF contrôle la phosphorylation d’UBF1 à la transition G1/S ... 138
DISCUSSION ... 142
MATÉRIELS ET MÉTHODES ... 159
I. Techniques de Biologie Cellulaire ... 160
1) Lignées cellulaires ... 160
Traitement des cellules H358wt à l’héréguline. ... 160
2) Transfection des cellules eucaryotes ... 161
ARN interférents ... 161
3) Test d’activité luciférase ... 161
3a) Conditions de transfection ... 161
3b) Mesure de l’activité luciférase ... 162
4) Fractionnement cellulaire ... 162
4a) Séparation des fractions protéiques cytoplasmique et nucléaire ... 162
4b) Purification des nucléoles ... 163
5) Analyse du cycle cellulaire par Cytométrie en Flux (FACS) ... 164
6) Immunofluorescence indirecte et analyse confocale ... 165
7) Test de prolifération ... 166
8) Immunoprécipitation de Chromatine (ChIP) ... 166
8a) Crosslinking et Immunoprécipitation de Chromatine ... 167
8b) PCR semi-quantitative ... 168
II. Techniques de Biochimie ... 170
1) Extraction protéique ... 170
2) Dosage des protéines ... 170
2a) Méthode utilisant l’acide bicinchoninique (BCA) ... 170
2b) Méthode de Bradford ... 171
3) GST-pulldown ... 171
5) Electrophorèse en gel de polyacrylamide dénaturant (SDS-PAGE) : ... 172
5a) SDS-PAGE et transfert sur membrane ... 172
5b) Immunodétection ... 173
III. Techniques de Biologie Moléculaire ... 176
1) Préparation des Bactéries DH5α compétentes ... 176
2) Transformation de bactéries ... 176
3) Extraction plasmidique ... 177
4) Electrophorèse en gel d’agarose ... 177
5) Plasmides ... 177
5a) Vecteurs d’expression ... 177
5b) Vecteurs luciférase ... 178
6) Pulse-chase ... 179
ANNEXES ... 180
Annexe 1 : Séquence nucléotidique du promoteur ErbB3…… ... 181
Annexe 2 : Séquence nucléotidique d'ErbB3……… ... ….182
Annexe 3 : Séquence protéique d'ErbB3………. ... 184
Annexe 4 : Séquence nucléotidique du promoteur Cycline D1………… ... ……….185
TABLE DES FIGURES
1 : Organisation du locus INK4a/ARF 6
2 : Les altérations de la bande chromosomique 9p21 dans la tumorigenèse 10 3 : Comparaison des séquences d’acides aminées humaine et murine d’ARF 13 4 : Mécanisme de dégradation des protéines par la voie ubiquitine-protéasome 20 5 : Représentation schématique de la structure de la protéine hdm2 23 6 : Représentation schématique de la structure de la protéine p53 24 7 : La voie ARF-MDM2-p53. Une boucle de rétrocontrôle négative contrôle le
niveau de p53 27
8 : Schéma simplifié de la biogenèse des ribosomes montrant les différentes étapes
de maturation du transcrit 47S 36
9 : Interaction p19ARF-NPM et biogenèse des ribosomes 38
10 : Structure type des récepteurs de la famille ErbB 40
11 : Sites de phosphorylation d’ErbB3 et protéines contenant un domaine SH2 (Src
Homology 2) recrutables 42
12 : Structures des 4 membres de la famille ErbB 44
13 : Conceptualisation du changement conformationnel du récepteur lors de la liaison
du ligand 45
14 : Modèles d’activation hétérologue des EGF-R. 47
15 : Sites d’autophosphorylation des récepteurs ErbB et protéines impliquées dans
les voies de signalisation associées 48
16 : Schéma d’activation des voies de signalisations MAPK et PI3K par le dimère
ErbB2/ErbB3 50
17 : Trafic et dégradation du récepteur EGFR activé 52 49
18 : Mécanismes de translocation nucléaire des récepteurs EGFR et ErbB4 58 19 : Modes d’action associés à l’EGFR : mécanismes de signalisation cytoplasmique (A) et nouveaux mécanismes associés à une localisation nucléaire (B) 61
20 : Structure du nucléole 64
21 : Organisation schématique des gènes de l’ADNr de mammifères 66 22 : SL1 est un complexe d’environ 300 kDa comprenant la TATA-binding protein
23 : Monomère UBF1 68 24 : Complexe d’initiation de la transcription de l’ARN polymérase 1 70 25 : Le facteur de transcription UBF chez l’homme : ses domaines d’interaction et
ses différents sites de phosphorylation 72
26 : Modifications post-traductionnelles de SL1 73
27 : Résumé des voies de signalisation, oncogènes et suppresseurs de tumeurs qui
régulent la transcription de l’ADNr 74
28 : Régulation de la transcription de l’ADNr par Rb 76
29 : L’ADNc ErbB3 est sous-exprimé en réponse à la surexpression de p14ARF dans
les cellules H358wt 80
30 : Activité du promoteur ErbB3 en fonction de la surexpression de p14ARF 81 31 : Analyse par cytométrie en flux de la ploïdie des cellules induites ou non pour
p14ARF après marquage à l’iodure de propidium. 83
32 : p14ARF se lie au promoteur ErbB3 86
33 : Expression du récepteur membranaire ErbB3 en fonction de l’expression de
p14ARF dans 3 lignées cellulaires 88
34 : Expression des récepteurs membranaires de la famille EGF-R dans la lignée H358/Tet-On/p14ARF en fonction de l’expression de p14ARF 89 35 : Analyse par cytométrie en flux du marquage membranaire d’ErbB3 en fonction de l’expression de p14ARF
90 36 : Analyse confocale de la localisation subcellulaire d’ErbB3 en fonction de l’expression de p14ARF
91 37 : Analyse de l’expression du récepteur ErbB3 sur des fractions protéiques
cytoplasmique, nucléaire et nucléolaire 93
38 : Spécificité de l’anticorps 95
39 : Immunoprécipitations d’ErbB3 dans des extraits nucléaires et cytoplasmiques de
cellules H358wt 97
40 : Analyse confocale de la localisation cellulaire de la protéine de fusion N
-GFP-ErbB3-C en fonction de l’expression de p14ARF 99
41 : Statut des voies de signalisation PI3K et MAPK dans la lignée H358 101 42 : Activité du promoteur Cycline D1 en fonction de la surexpression d’ErbB3 102 43 : Séquence de localisation nucléaire (NLS) du récepteur ErbB4 : homologie avec une séquence localisée juste après le segment transmembranaire chez ErbB3 104
44 : Analyse par Western Blot de l’expression du mutant N-GFP- N-ErbB3-C 104
45 : Expression intracellulaire de la protéine GFP- N-ErbB3 105 46 : Activité du promoteur Cycline D1 en fonction de la surexpression d’ErbB3 ou
de la protéine mutante N-GFP- N-ErbB3-C 106
47 : ErbB3 interagit spécifiquement avec le promoteur Cycline D1 108 48 : Effets de la surexpression des protéines GFP-ErbB3 et GFP- N-ErbB3 sur le
niveau d’expression de la protéine Cycline D1 109
49 : Test de prolifération au BrdU 110
50 : Recherche de complexes p14ARF-ErbB3 par GST-Pull-down et SDS-PAGE 113 51 : Recherche de complexes p14ARF-ErbB3 par co-immunoprécipitation 114 52 : p14ARF contrecarre l’interaction d’ErbB3 avec le promoteur Cycline D1 115 53 : Activité luciférase du promoteur Cycline D1 en fonction du taux d’expression
de p14ARF 116
54 : p14ARF ne s’associe pas au promoteur Cycline D1 117 55 : Mesure de l’activité du promoteur Cycline D1 en fonction de l’expression d’ErbB3 et de p14ARF
118 56 : La surexpression de p14ARF induit une diminution d’expression des protéines
ErbB3 (185 et 100 kDa) et Cycline D1 120
57 : p14ARF régule la prolifération des cellules dès 60 heures de surexpression 121
58 : La voie p14ARF/ErbB3/Cycline D1. 122
59 : La surexpression de p14ARF inhibe l’activité du promoteur de l’ADNr dans les
cellules H358wt 124
60 : L’induction de p14ARF dans les cellules H358/Tet-On/p14ARF inhibe l’activité du
promoteur de l’ADNr 125
61 : En réponse à la surexpression d’E2F1, l’induction de p14ARF réduit l’activité du
promoteur ADNr 126
62 : La néosynthèse d’ARNr 47S diminue dans les cellules surexprimant p14ARF par
rapport aux cellules témoins 128
63 : p14ARF colocalise avec UBF1 dans la zone granulaire du nucléole 129
64 : UBF1 est un partenaire de p14ARF 131
65 : Evaluation quantitative de la liaison des protéines p14ARF, UBF1 et Pol1 au
66 : L’expression ectopique d’UBF1 corrige l’effet inhibiteur de p14ARF sur le
promoteur 133
67 : Phosphorylation d’UBF1 au cours de l’induction de p14ARF dans la lignée
H358/Tet-On/p14ARF 135
68 : Cycle de référence des cellules H358/Tet-On/p14ARF 136 69 : Suivi du cycle cellulaire après synchronisation à l’hydroxyurée 137 70 : Phosphorylation d’UBF1 au cours du cycle cellulaire, en fonction du niveau d’expression de p14ARF
138 71 : La diminution d’expression de Cycline D1 induite par p14ARF est corrélée à
l’hypo-phosphorylation d’UBF1 140
72 : La voie p14ARF/UBF1 dans le contrôle de la transcription de l’ADNr 141 73 : Les récepteurs ErbB, une cible de choix pour des thérapies anti-cancéreuses 152 74 : Schéma récapitulatif des voies p14ARF/ErbB3/Cycline D1 et p14ARF/Cycline
D1/UBF1 158
75 : Principe de la technique d’Immunoprécipitation de Chromatine (ChIP) 167
TABLEAUX
Tableau 1 : Régulateurs d’ARF 16
Tableau 2 : Partenaires d’ARF 29
Tableau 3 : Anticorps primaires 174
1
ABRÉVIATIONS
aa Acide aminé
ADNr Acide DésoxyriboNucléique Ribosomique AKT Sérine/thréonine kinase/ PKB
AR Amphiréguline
ARF Alternative Reading Frame
ARNr Acide RiboNucléique Ribosomique ATM Ataxia Telangiectasia Mutated ATP/ADP/AMP Adénosine Tri-/Di-/Mono-Phosphate ATR Ataxia Telangiectasia Related
ATRS AT-Rich consensus Site
BCA Bicinchoninic Acid
BET Bromure d’éthidium
Bmi1 B lymphoma Mo-MLV insertion region 1
BrDU BromoDéoxyUridine
BSA Bovine Serum Albumin
BTC BeTaCelluline
CDK Cyclin Dependent Kinase
CDKi Cyclin Dependent Kinase inhibitor CDK1 Cyclin Dependant Kinase 1 / p34cdc2
CF Centre Fibrillaire
CFD Composant Fibrillaire Dense
CG Composant Granulaire
ChIP Chromatin ImmunoPrecipitation
CK2 Caséine Kinase 2
COX-2 CycloOXygénase 2
CPE Core Promoter Element
DD Differential Display
DKO Double KO
DMEM Dulbecco's Modified Eagle Medium
DO Densité Optique
Dox Doxycycline
DP1 DNA binding Protein 1
DTT DiThioThreitol
EDTA Ethylene Diamine Tetraacetic Acid
EGF Epidermal Growth-Factor
EGF-R Epidermal Growth-Factor Receptor EGTA Ethylene Glycol Tetraacetic Acid
EPR EPiRéguline
ErbB v-Erb-B2 erythroblastic leukemia viral oncogene homolog
ET EndoThéline
2 FACS Fluorescent-activated cell sorting
FANCC Fanconi ANemia Complementation group C GAPDH GlycerAldehyde 3-Phosphate DeHydrogenase GFP Green Fluorescent Protein
GPCRs G protein-coupled receptors
Grb2 Growth factor receptor-bound protein 2 Grb7 Growth factor receptor-bound protein 7 GST Glutathion S Transférase
HB-EGF Heparin-Binding EGF-like factor
HDAC1 Histone DéACétylase 1
HDM2 Human Double Minut 2
HEPES HydroxyEthyl Piperazine Ethane Sulfonic acid
HER Human Epidermal Receptor
HMG High Mobility Group
HPV E6 Human PapillomaVirus E6 oncoprotein hrDNA Human ribosomal DesoxyriboNucleic Acid
HRG HéRéGuline/NeuRéGuline (NRG)
HU HydroxyUrée
IGS InterGenic Spacer
INK4 INhibitor of cyclin-dependent Kinase 4
IP Iodure de Propidium
IP Immunoprécipitation
IPTG IsoPropyl-beta-D-ThioGalactopyranoside IRES Internal Ribosome Entry Site
ITS Internal Transcribed Spacer
kb Kilobase
kDa Kilo-Dalton
KO Knock-Out
LB Lysogeny broth
LPA LysoPhosphatidic Acid
MAPK Mitogen-Activated Protein Kinase
MCS Multiples Cloning Site
MDM2 Murin Double Minute 2
MEF Mouse Embryo Fibroblast
MMPs Membrane MetalloProtease
MOPS 3-(N-MOrpholino) Propanesulfonic acid
mTOR mammalian Target Of Rapamycin
MTS1 Multiple Tumor Suppressor 1
NES Nuclear Export Signal
NLS Nuclear Localisation Signal
NO Nitric Oxyde
NoLS Nucleolar Localisation Signal NOR Nucleolar Organizer Region
3 NP40 Tergitol-type NP-40/ nonyl phenoxylpolyethoxylethanol
NPC NasoPharyngial Carcinoma
NPM NucleoPhosMine / B23 / NO38 / Numatrin
NR4A Nuclear Receptor 4A
ns Non significatif
NS Non Synchronisé
P3CA Calcineurine
p85 Sous-unité p85 de la PI3K
pb Paires de bases
PBS Phosphate Buffered Saline
P/CAF p300/CBP-Associated Factor
PCR Polymerase Chain Reaction
PI3K Phosphatidyl-Inositol 3 Kinase
PIC Pre-Initiation Complex
PIC Protease Inhibitor Cocktail
PFA ParaFormAldéhyde
PLC Phospholipase C
PMSF PhenylMethaneSulphonylFluoride
Pol1 ARN polymérase 1
pRb Protein Retinoblastoma
PTB PhosphoTyrosine Binding
RNAse A RiboNucléAse A
RPMI Roswell Park Memorial Institute Medium RTK Récepteur à activité Tyrosine Kinase
S6K S6 Kinase-1
SDS Sodium Dodecyl Sulfate
SDS-PAGE Sodium Dodecyl Sulfate PolyAcrylamide Gel Electrophoresis
SH2 Src Homology 2
SHC SH2 Domain Containing protein
SiRNA Small Interferent RNA
SL1 Selectivity Factor 1/Transcription Initiation Factor (TIF-1B)
SN Sérum Normal
SV40 Simian Vacuolating Virus 40
SVF Sérum de Veau Fœtal
TACE Tumor necrosis factor-Alpha-Converting Enzyme
TAF TBP-Associated Factor
TBP TATA Binding Protein
TGFα Transforming Growth Factor α
TKO Triple KO
Topo1 Topoisomérase 1
TPA 12-O-TetradecanoylPhorbol-13-Acetate
Ub Ubiquitine
UBF Upstream Binding Factor
UPE Upstream Promoter Element/ Upstream Control Element (UCE)
4
INTRODUCTION
Le développement d’un cancer est un processus multi-étape impliquant des mutations « gain de fonctions », qui activent à travers les oncogènes les voies de signalisation impliquées dans la progression du cycle cellulaire (McCormick, 1999), et des mutations « perte de fonctions » sur des gènes suppresseur de tumeurs (Sherr & McCormick, 2002).
La cellule altérée par ces mutations susceptibles d’entraîner une perte de contrôle de la prolifération, met en jeu des processus de réparation et/ou d’apoptose. Un faible pourcentage de cellules, endommagées au niveau de ces mêmes systèmes de réparation, ne pourra pas mettre en place ces contrôles et permettra l’émergence d’un clone tumoral.
Au cours de ma thèse, je me suis efforcée de comprendre certains des mécanismes moléculaires mis en jeu dans la cellule par la protéine suppresseur de tumeurs p14ARF, acteur clé du contrôle de la prolifération chez l’homme. Produite par et pour la cellule, p14ARF protège efficacement du développement tumoral par son action de stabilisation de la protéine p53. Depuis quelques années, il a cependant été montré que p14ARF exerce également des fonctions régulatrices du cycle cellulaire dans un contexte déficient en p53, conduisant à la recherche de nouveaux partenaires impliqués dans de nouvelles voies.
5
Chapitre 1 :
La protéine suppresseur de tumeurs
ARF
I. Le locus INK4a/ARF
1) Structure et expression
Le locus INK4a/ARF localisé sur le bras court du chromosome 9 (9p21) chez l’homme est fréquemment remanié dans les tumeurs solides (adénocarcinomes pancréatiques, mélanomes, cancers du poumon) et hémopathies malignes (leucémies aiguës lymphoblastiques) (Ruas & Peters, 1998). Cette constatation a logiquement conduit à suspecter l’existence d’un gène suppresseur de tumeurs dans cette région chromosomique, mais la réalité s’est révélée beaucoup plus complexe dans la mesure où ce locus contient 2 gènes imbriqués partageant 2 exons (Figure 1) :
- le gène INK4a, le premier à avoir été découvert, possède 3 exons initialement appelés exons 1, 2 et 3. Le transcrit correspondant code la protéine p16INK4a dont l’existence a été rapportée pour la première fois en 1993 par Xiong et collaborateurs (Xiong et al., 1993). La protéine p16INK4a se lie aux complexes cycline D/CDK4/6 responsables de la phosphorylation de pRb (protéine du Rétinoblastome), empêchant ainsi l’activation du facteur de transcription E2F1 et bloquant la progression du cycle cellulaire en G1/S.
- le gène ARF a été découvert par la suite, par la mise en évidence d’un exon supplémentaire localisé en amont de l’exon 1 du gène p16INK4a. A partir d’un promoteur
6 spécifique appelé P (le promoteur du gène INK4a devenant alors P ), ce gène donne naissance à un transcrit issu des exons 1 (spécifique), 2 et 3 (communs avec p16INK4a). Par épissage alternatif et décalage du cadre de lecture au niveau de l’exon 2, la protéine codée par ce transcrit ne présente aucune homologie avec p16INK4a (Duro et al., 1995). Cette protéine appelée ARF1 pour Alternative Reading Frame, a été mise en évidence initialement chez la souris (p19ARF) (Quelle et al., 1995). La protéine humaine p14ARF (14 kDa), plus courte, a été identifiée ultérieurement (Della Valle et al., 1997).
Figure 1 : Organisation du locus INK4a/ARF et expression des protéines p14ARF et 16INK4a
Pβ: promoteur du gène ARF Pα: promoteur du gène INK4a
2) Importance du locus dans la tumorigenèse. Eléments de
régulation.
2a) p16INK4a
Chez la souris, l’inactivation spécifique de p16INK4a
(souris KO, Knock Out) a confirmé le rôle de la protéine p16INK4a dans l’apparition de cancers (Serrano et al., 1996).
1
Dans ce manuscrit, la protéine sera généralement désignée par l’acronyme ARF, à l’exception des données spécifiques à l’une ou l’autre des espèces humaine et murine qui seront alors désignées par p14ARF et p19ARF respectivement.
7 Dans les cancers humains, l’expression de p16INK4a est très souvent altérée avec une fréquence de 25 à 70% selon le type de cancer (Ruas & Peters, 1998).
Il existe au moins trois mécanismes d’inactivation :
- délétion des deux allèles, dans les leucémies lymphoblastiques (Hebert et al., 1994).
- délétion d’un allèle et mutation au sein de l’allèle résiduel, notamment dans les mélanomes (Liu et al., 1995). Dans les mélanomes familiaux, des mutations germinales sont retrouvées à travers la région codante d’INK4a y compris dans l’exon 1 qui n’est pas partagé avec p14ARF mais également dans les régions non codantes. Il est important de noter que ces mutations altèrent généralement la capacité de p16INK4A à interagir avec sa principale cible CDK4, dont le gène est également considéré comme gène de susceptibilité au mélanome.
- délétion d’un allèle et méthylation de l’allèle résiduel, dans un certain nombre de cancers, notamment du poumon (Tanaka et al., 2005) et du foie (Ishikawa et al., 2004). Cette inactivation a été observée dans les cancers de la tête et du cou, de l’œsophage, du poumon, de la vessie, du colon, du sein, et également dans les leucémies, les lymphomes et les glioblastomes (Foulkes et al., 1997; Ruas & Peters, 1998). L’inactivation la plus fréquente est observée dans le cancer de la prostate (98% des cas) (Schutte et al., 1997), ainsi que dans les adénocarcinomes pancréatiques (Hu et al., 1997).
D’une manière générale, l’inactivation de p16INK4a
s’exerce au niveau transcriptionnel à travers l’expression de facteurs répresseurs de transcription tels que Id1, Id3 ou Bmi-1 (Ohtani et al., 2001)(Alani et al., 2001)(Jacobs et al., 1999a). Au niveau
post-8 transcriptionnel, le facteur ASF/SF2 pourrait influencer négativement l’épissage de l’ARNm, de même que les protéines hnRNP A1, hnRNP A2 et AUF semblent altérer la stabilité du transcrit (Zhu et al., 2002)(Wang et al., 2005).
Plus récemment, a été rapportée l’existence d’un micro RNA (mi-R24) qui inhibe à la fois l’initiation et l’élongation de p16, supprimant ainsi l’expression de la protéine (Lal et al., 2008)
2b) p14
ARFEn raison du statut particulier du locus, et en particulier de l’imbrication des deux gènes INK4a et ARF, s'est immédiatement posée la question de la participation de l'un et/ou l'autre gène dans les processus malins humains.
Chez la souris, les premières expériences d’invalidation du locus (souris KO) ont été effectuées par mutation et/ou délétion des exons 2 et 3 (Serrano et al., 1996). Or, si la perte de l'exon 3 pouvait en principe être sans conséquence, l'exon 2 est codant pour les deux protéines dans un cadre de lecture différent. Pour cette raison, il était impossible de définir avec certitude la part qui revenait à chaque protéine dans les événements de la transformation maligne.
Les expériences d’invalidations touchant le seul exon 1 ont permis de résoudre cette ambiguïté. Des fibroblastes embryonnaires murins (MEF) spécifiquement p19ARF-/- ont été obtenus par l’équipe de C. Sherr à Memphis. Ces cellules expriment la protéine
p16INK4a sous sa forme sauvage, mais pas la protéine p19ARF, confirmant que l’altération
de l’exon 1 ne gène pas la transcription de INK4a à partir du promoteur P (Kamijo et al., 1997) L’inactivation de p19ARF dans ces MEFs est suffisante pour qu’ils échappent à la sénescence réplicative, dernière barrière avant l’immortalisation et la transformation
9 maligne (Serrano et al., 1997). Par ailleurs, ces cellules ARF- /- peuvent être immortalisées par la seule expression d’une protéine Ha-ras oncogénique alors que la transformation cellulaire nécessite habituellement une coopération entre l’activation de Ras et l’inactivation d’un suppresseur de tumeurs. Ces données ont constitué les premiers arguments en faveur d’un rôle suppresseur de tumeurs de la protéine p19ARF
. In vivo, les souris p19ARF-/- développent un éventail varié de tumeurs dans les 18 mois qui suivent leur naissance, et ce de manière sensiblement identique à ce que l’on observe chez des animaux déficients pour p53. La tumeur la plus fréquemment observée est le lymphome lymphocytique, mais on trouve également des sarcomes et des carcinomes, particulièrement des carcinomes pulmonaires (Kamijo et al., 1999).
L’inactivation du locus ARF dans les tumeurs humaines est le plus souvent la conséquence d’une délétion qui touche également le gène INK4a (p16INK4a
) et parfois même le gène INK4b situé en 5’ (la protéine p15INK4b codée par ce gène est également un suppresseur de tumeurs) (Figure 2).
Les mutations ponctuelles ou la méthylation du promoteur, qui sont les événements les plus fréquents à l’origine de l’inactivation du gène INK4a, sont en revanche plus rares pour ARF. Des cas de mutations ponctuelles ainsi que la méthylation du promoteur P dans des lignées tumorales et des tumeurs ont cependant été rapportés (Robertson & Jones, 1998)(Sato et al., 2002)(Hsu et al., 2004)(Ishida et al., 2005)(Kawamoto et al., 2006)(Lee et al., 2006)(Badal et al., 2008).
10
Figure 2 : Les altérations de la bande chromosomique 9p21 dans la tumorigenèse humaine.
Les tableaux décrivent les altérations génétiques (délétions et mutations ponctuelles
↓
) et épigénétiques (méthylation du promoteur☼
) de ce locus retrouvées dans des tumeurs humaines. NPC: carcinome nasopharyngial. (Ortega et al., 2002).Une étude réalisée sur 53 cas de mélanomes a montré que seul un patient présentait une mutation germinale hétérozygote au niveau de l’exon 1 du gène ARF, en l’absence de toute mutation germinale sur les exons 1α, 2, et 3 de CDKN2A et les exons 1 et 2 de CDKN2B (Laud et al., 2006). Une délétion germinale de ARF n’altérant pas INK4a a également été rapportée dans un cas de mélanome associé à des tumeurs du système nerveux (NST) (Randerson-Moor et al., 2001).
Plus récemment, une analyse génétique et épigénétique, réalisée sur 58 cas de mélanomes métastasés ainsi que sur 9 lignées tumorales a montré, dans 30% des
11 mélanomes métastatiques, une altération du gène ARF sans modification de INK4a, faisant apparaître aujourd’hui ARF comme un probable gène de susceptibilité au mélanome (Freedberg et al., 2008).
2c) Régulations conjointes
.Les éléments de régulation décrits ci-dessus concernent le gène INK4a ou le gène ARF. Pour compléter ce tableau, il est important de signaler l’existence de délétions de grande ampleur impliquant la totalité du locus INK4/ARF altérant l’expression conjointe de p14ARF
, p15INK4b, p16INK4a.
De tels événements sont par exemple décrits dans les gliomes. Chez les modèles murins, la délétion du seul locus n’induit pas spontanément l’apparition de tumeurs. En revanche, l’activation de H-Ras dans ce contexte augmente fortement l’incidence des tumeurs (Uhrbom et al., 2002)(Solomon et al., 2008). De même, une délétion germinale du locus INK4/ARF a été observée dans un cas de mélanome familial associé à des tumeurs du système nerveux (NST). Cette délétion de 403 kb emporte les régions INK4b, INK4a et ARF empêchant l’expression des gènes correspondants mais également celle d’un transcrit anti-sens (ANRIL) dont le premier exon est situé dans la région promotrice du gène ARF. La régulation transcriptionnelle des gènes ARF et ANRIL, mais aussi de INK4b et INK4a, pourrait être coordonnée tant au niveau physiologique que pathologique. Le mécanisme d’action de ce transcrit reste à élucider (Pasmant et al., 2007).
12
II. La protéine ARF
1) Caractéristiques biochimiques et structurales.
1a) Composition en acides aminés
La protéine p19ARF est constituée de 169 acides aminées (aa) qui lui confèrent un poids moléculaire de 19238 Da alors que la protéine humaine contient 132 aa (13902 Da) (Figure 3). Un examen des séquences entières montre qu’il n’existe que 50% d’homologie entre p14ARF
et p19ARF. De plus, les 37 derniers résidus de la protéine murine sont absents de la protéine humaine. Cette différence structurelle reste globalement inexpliquée, seule la protéine Pex19p est connue pour s’associer spécifiquement à cette région C-terminale chez la souris (Wadhwa et al., 2002).
Les exons 1 chez l’homme et la souris présentent 61% d’homologie au niveau nucléotidique suggérant une conservation parmi les espèces. En revanche, l’identité n’est plus que de 44% au niveau protéique. C’est pourtant cette région N-terminale qui est la mieux conservée de la protéine sur le plan fonctionnel, et en particulier les 14 premiers acides aminés (11 résidus identique chez l’homme et la souris), qui permettent à ARF d’assurer sa fonction suppresseur de tumeurs (Figure 3).
Une des caractéristiques biochimiques principales d’ARF réside dans sa richesse en résidus arginine (22% des aa) et en aa hydrophobes. Les aa basiques confèrent à la protéine un point isoélectrique extrêmement élevé (12,4 chez l’homme). De ce fait, ARF est non structurée aussi bien in vivo qu’in vitro, et doit probablement former des complexes avec d’autres molécules (acides) pour à la fois être repliée correctement et pour neutraliser sa charge à pH physiologique.
13
Figure 3 : Comparaison des séquences d’acides aminées humaine et murine d’ARF.
Les résidus conservés dans les deux espèces sont surlignés en gris. Les motifs fonctionnels, incluant les séquences de localisation nucléolaire, sont aussi spécifiés. NoLs = Signal de Localisation Nucléolaire. D’après (Gallagher et al., 2006).
1b) Localisation intracellulaire
S’il n’y a pas de motif structurel reconnaissable dans la protéine ARF, il faut signaler en revanche la présence de séquences particulières de localisation intracellulaire :
- Pour la protéine murine p19ARF, la situation est assez simple. Au niveau de l’exon 1 il existe un motif reconnu comme signal de localisation nucléolaire (NoLS, acides aminés 26-37) avec une signature caractéristique, la séquence RRPR (KFVRSRRPRTAS). De fait, la délétion des résidus 26 à 37 de l’exon 1 chez la souris altère la localisation nucléolaire de p19ARF qui se retrouve alors essentiellement nucléaire. De manière surprenante, la région 26-37 concernée n’est pas conservée au sein des espèces et en particulier chez l’homme (Weber et al., 1999).
- Pour la protéine humaine, la principale séquence de localisation nucléolaire est retrouvée au niveau de l’exon 2 (résidus 83-101) avec un domaine RRPR en position 88-91(GAQLRRPRHSHPTRARRCP). La délétion de cette région au niveau de la protéine entraîne une perte partielle de localisation nucléolaire (Zhang & Xiong, 1999).
14 D’autres séquences semblent donc intervenir chez l’homme dans la localisation de la protéine :
- Un travail mené au laboratoire a montré qu’un mutant de délétion dépourvu des aa 85 à 132 présente une localisation nucléoplasmique mais aussi nucléolaire suggérant un rôle possible de la région 65 à 85 (exon 2) dans la localisation de la protéine (Ayrault et al., 2003).
- Par la suite, une étude a rapporté l’existence d’un signal de localisation nucléolaire dans l’exon 1 correspondant aux 22 premiers aa de p14ARF
(Lohrum et al., 2000). Ces résultats ont été confirmés et précisés par l’équipe d’Hélène Rizos, qui a délimité ce domaine aux aa 1 à 13 (MVRRFLVTLRIRR) (Rizos et al., 2000).
L’ensemble de ces résultats va dans le sens d’une participation des exons 1 et 2 à la localisation nucléolaire chez l’homme. Mais le caractère extrêmement basique de la protéine ARF complique les choses en raison de sa propension à contracter des liaisons électrostatiques qui pourraient aboutir à une localisation non spécifique.
Les travaux de Lindström et collaborateurs ont montré qu’après surexpression de p14ARF obtenue soit de manière ectopique soit par activation oncogénique, la protéine était retrouvée dans la partie granulaire périphérique du nucléole (Lindstrom et al., 2000). Après traitement par une ribonucléase (ARNase A), cette localisation granulaire est perdue au profit d’une translocation rapide de la protéine vers le nucléoplasme. Ces résultats indiquent que la localisation nucléolaire de p14ARF est probablement dépendante de l’interaction physique (directe ou indirecte) de la protéine avec des ARNs présents dans le nucléole, site de synthèse d’ARNs ribosomiques et de leur assemblage en sous-unités ribonucléoprotéiques. Cette hypothèse a été confirmée par la
15 suite par des travaux qui montrent que la protéine ARF intervient dans la régulation de la biogenèse des ribosomes au niveau de la transcription et de la maturation des ARN ribosomiques (Ayrault et al., 2006a; Bertwistle et al., 2004; Itahana et al., 2003).
2) Expression et régulation de la protéine ARF
ARF est une protéine ubiquitaire qui se démarque de ce fait des autres suppresseurs de tumeurs, le plus souvent induits en réponse à des stress cellulaires comme les dommages à l’ADN, des conditions d’hypoxie ou la privation de nutriments. En effet, l’expression de la protéine ARF est induite par des signaux oncogéniques provenant de la surexpression de protéines prolifératives ou de la perte d’expression de régulateurs négatifs du cycle cellulaire.
En faisant la différence entre signaux de croissance prolongés et activation oncogénique, les cellules doivent être capables d’exprimer rapidement les protéines nécessaires à l’arrêt du cycle afin d’éviter leur prolifération anarchique, mais également d’arrêter leur production quand leurs effets ne sont plus requis. En conséquence, la transcription et le niveau d’expression de la protéine ARF sont placés sous un contrôle strict qui peut s’exercer soit de manière directe, par liaison de facteurs de transcription sur le promoteur de ARF, soit de façon indirecte par interaction de la protéine ARF avec une protéine partenaire.
Les paragraphes qui suivent présentent des exemples de ces différents modes de
régulation. Une liste non exhaustive des différents facteurs impliqués est présentée sous
16
2a) Régulation transcriptionnelle
L’oncogène Ras
L’expression de l’oncogène Ha-RasG12V (mutation activatrice de la glycine 12 en valine = Ras*) dans des cultures primaires de fibroblastes embryonnaires normaux, induit un arrêt de prolifération lié à l’activation de p53 (Serrano et al., 1997). Cet effet est associé à une augmentation significative du transcrit p19ARF (5 à 10 fois) dans les MEF normaux. La même expérience réalisée sur des MEF ARF- /- montre que dans ces cellules le niveau de p53 reste inchangé en présence de Ras*. Chez la souris, la protéine p19ARF est donc essentielle pour l’activation de p53 en réponse au signal oncogénique Ras* (Palmero et al., 1998). Il est important de noter que l’induction d’ARF par Ras mutée est propre au modèle murin.
17 c-Myc
La surexpression de c-Myc par infection rétrovirale de MEFs primaires induit l’expression spécifique du transcrit ARF et la surexpression de la protéine p19ARF
sans modifier l’expression du transcrit INK4a ni de la protéine p16INK4a
. Par ailleurs, dans ces mêmes cellules, on observe une augmentation du taux de transcrit p53, une accumulation de protéines codées par les gènes-cibles de p53 (MDM2 et p21) et la mort des cellules par apoptose (Zindy et al., 1998).
En revanche, les cellules déficientes pour ARF ou p53 (MEFs ARF-/- ou p53 -/-) deviennent résistantes à la mort cellulaire. Chez la souris, la protéine p19ARF est donc induite et impliquée dans le déclenchement de l’apoptose en réponse à la surexpression de Myc. Myc active également l’expression de Bmi1, autre facteur de transcription, capable lui d’inhiber ARF. Ces deux facteurs de transcription collaboreraient, Myc en activant p19ARF et Bmi1 en l'inhibant, afin d’obtenir une réponse modulée et appropriée aux signaux perçus par les cellules (Eischen et al., 1999; Jacobs et al., 1999b).
La famille E2F
E2F1 appartient à la famille des facteurs de transcription hétérodimériques E2F dont la fonction est la coordination de l’expression de gènes régulant la progression cellulaire. Cette famille est constituée de facteurs répresseurs ou activateurs de transcription selon la croissance cellulaire ou le stade du cycle. Chez l’homme, la surexpression de E2F1-2 et 3 est associée à une augmentation massive de l’expression du transcrit ARF dans des cellules quiescentes restimulées par du sérum (DeGregori et al., 1997; Dimri et al., 2000). Cette augmentation pourrait passer par la liaison directe
18 le promoteur du gène ARF ou par le recrutement d’E2F1-3 sur le promoteur par l’intermédiaire de facteurs Sp1 (Parisi et al., 2002).
On retrouve également des facteurs E2F associés à la régulation négative de la transcription d’ARF. En particulier, Aslanian et collaborateurs ont montré que E2F3 était requis pour la répression transcriptionnelle du gène p19ARF sous des conditions normales de prolifération (Aslanian et al., 2004). Les MEFs dérivés de souris E2F3-/- prolifèrent peu et ne répondent pas aux signaux mitogéniques (facteurs sériques) (Humbert et al., 2000). Dans ces mêmes cellules, on observe une expression de p19ARF associée à l’activation de p53.
2b) Régulation par interaction protéine-protéine
Nous venons de voir qu’il existe de nombreux régulateurs transcriptionnels du
gène ARF. Ce processus de régulation est efficace puisqu’il met en jeu la régulation
directe du gène concerné. Pourtant, il existe d’autres moyens empruntés par la cellule, afin de moduler l’action de la protéine ARF sans modifier son expression. En effet, il est
possible de réguler les fonctions d’ARF par interaction directe avec d’autres protéines.
Pex19p
Pex19p est une protéine cytosolique impliquée dans la biogenèse des peroxysomes, l’assemblage et la stabilité des structures membranaires (Gotte et al., 1998; Lambkin & Rachubinski, 2001; Matsuzono et al., 1999). Il a été montré que Pex19p interagit physiquement avec la protéine p19ARF et la maintient au sein du cytosol. De cette manière, ARF ne peut plus exercer sa fonction de stabilisation de la protéine p53, alors soumise au processus de dégradation induit par MDM2. La transfection cellulaire d’un ARN interférent (siRNA) dirigé contre Pex19p permet de
19 restaurer la localisation nucléaire de p19ARF (Sugihara et al., 2001). Ce complexe protéique est exclusivement observé chez la souris, car Pex19p interagit avec les 41 acides aminés de la partie C-terminale de p19ARF, absents dans la structure de la protéine humaine p14ARF (Figure 3) (Wadhwa et al., 2002).
Dégradation par la voie du protéasome
Pour rappel, la voie ubiquitine-protéasome est le mécanisme principal de dégradation des protéines chez les eucaryotes. Ce mécanisme nécessite l’intervention successive de différentes enzymes (Figure 4) : L’enzyme E1 est une enzyme dite d’activation de l’ubiquitine. En présence d’ATP, elle va former une liaison Thiol-ester entre un résidu cystéine et une molécule d’ubiquitine. Cette molécule d’ubiquitine est ensuite transférée sur une enzyme E2 dite enzyme de conjugaison qui va la fixer sur le substrat cible. Cette fixation se fait par l’intermédiaire d’une E3-ubiquitine ligase (enzyme de reconnaissance) qui s’intercale entre l’ubiquitine et le substrat. L’ubiquitine est alors fixée à la protéine cible par une liaison covalente entre l’un des résidus glycine de sa partie C-terminale et un groupement NH2 d’une lysine de la protéine cible. Par la suite, d’autres molécules d’ubiquitine vont être ajoutées permettant la reconnaissance du substrat par le protéasome 26S et sa dégradation sous forme de peptides inactifs de 7 à 9 acides aminés.
Il semble que la dégradation de p19ARF (demi-vie d’environ 6 heures) implique le protéasome (Kuo et al., 2004a). Ces données sont surprenantes puisque la protéine, bien que de nature extrêmement basique, ne possède qu’un seul résidu Lysine (Lys 26), qui plus est non conservé au cours de l’évolution et absent de la protéine humaine p14ARF
20
Figure 4 : Mécanisme de dégradation des protéines par la voie ubiquitine-protéasome.
Ub : ubiquitine ATP/ADP/AMP : adénosine tri-/di-/mono-phosphate (Cell Signaling).
Pourtant, il a été montré que le traitement des cellules par un inhibiteur du protéasome (MG132) induit une accumulation nucléolaire de p19ARF. Par ailleurs, la dégradation protéasomale de p19ARF ne dépend pas de son interaction avec son partenaire MDM2 qui possède pourtant une activité E3-ubiquitine ligase (Kuo et al., 2004a). La poly-ubiquitination de p19ARF concerne l’extrémité N-terminale de la protéine et dépend d’une E1-ubiquitine activating enzyme fonctionnelle (Kuo et al., 2004b).
Lorsqu’elle est impliquée dans des complexes protéiques au sein du nucléole (NPM/B23, biogenèse des ribosomes), p19ARF semble protégée de ce système de dégradation (Korgaonkar et al., 2005).
21
III. Fonctions de ARF
1) La voie ARF-MDM2-p53.
1a) Découverte du rôle p53 dépendant de la protéine ARF.
Le produit alternatif du locus INK4a ne possède pas d’homologie avec les autres membres de la famille INK4 ni séquences remarquables. Cependant, ARF présente des similitudes avec la protéine p16INK4a, au niveau de son organisation génomique (Figure 1) mais également sur le plan fonctionnel. La protéine ARF, contrairement à p16INK4a, n’est pas un inhibiteur de CDKs, mais son expression ectopique induit aussi l’arrêt du cycle cellulaire. Toutefois, l’arrêt induit par p14ARF diffère de celui effectué par
p16INK4a car il ne dépend pas du statut de pRb dans la cellule, mais de la protéine p53
comme l’ont montré les observations suivantes :
- En premier lieu, les tumeurs observées chez des souris déficientes pour le locus ARF/INK4a présentent rarement des mutations ou des délétions de p53. Ainsi, un modèle de souris utilisant un gène H-ras muté (G12V) sur un fond génétique INK4a-déficient montre l’apparition de mélanomes malins avec un temps de latence court et une forte pénétrance (Chin et al., 1997). Pourtant, chez ces souris, p53 n’est pas mutée et reste fonctionnelle. La même observation est faite dans les mélanomes malins humains.
- De plus, l’expression ectopique de p19ARF est capable d’inhiber l’entrée en phase S de MEFs normaux mais pas de MEFs issus de souris KO pour le gène p53 (MEFs p53-/-) (Kamijo et al., 1997).
- Dans des cellules primaires, la neutralisation de p53 (oncoprotéine virale E6 du virus HPV, ou mutant dominant négatif) inhibe également la capacité d’ARF à exercer cette fonction suppresseur de tumeurs (Pomerantz et al., 1998).
22 - Enfin, la surexpression de p14ARF induit dans les cellules humaines une stabilisation de la protéine p53 (Argentini et al., 2001; Barak et al., 1993; Ganguli & Wasylyk, 2003) avec pour conséquence l’activation de deux gènes cibles de p53 : p21CIP et MDM2 (Dulic et al., 1994; el-Deiry et al., 1993).
1b) La protéine MDM2
Le gène MDM2 a été découvert en 1987 dans une lignée de souris BALB/c transformée (Cahilly-Snyder et al., 1987; Fakharzadeh et al., 1991). Dans cette lignée, ce gène était amplifié sur des structures extra-chromosomiques, appelés Double Minute, d’où son nom « MDM2 » chez la souris, « HDM2 » chez l’homme. La surexpression de la protéine MDM2 (491 acides aminés, 90 kDa) induit la transformation des cellules et confère au gène MDM2 un statut d’oncogène (Fakharzadeh et al., 1991). Cette protéine présente des domaines fonctionnels importants (Figure 5) :
- un signal de localisation nucléaire (NLS) qui confère à la protéine une localisation principalement nucléoplasmique,
- un signal d’export nucléaire (NES) qui permet à cette protéine d’effectuer des navettes noyau-cytoplasme en relation avec sa fonction E3 ubiquitine ligase,
- un domaine en doigts de zinc et un domaine RING finger d’interaction avec l’ADN (Iwakuma & Lozano, 2003).
MDM2, comme HDM22 chez l’homme, présente des domaines d’interactions avec des protéines partenaires, p300/CBP, la protéine ribosomale L5, MDMX, p53 et ARF.
2
En raison de la conservation de la structure et des fonctions entre les protéines humaine et murine, l’acronyme Mdm2 sera utilisé pour désigner cette protéine dans le reste du texte.
23
Figure 5 : Représentation schématique de la structure de la protéine HDM2.
Les sites d’interaction de HDM2 avec d’autres partenaires sont indiqués sur la partie supérieure de la figure, les chiffres entre parenthèses correspondent à la position des acides aminés intervenant dans ces interactions. (NLS : Signal de localisation nucléaire ; NES : Signal d’export nucléaire). D’Après (Lozano & Montes de Oca Luna, 1998).
1c) La protéine p53
L'analyse des séquences d'ADNc de p53 de diverses espèces a permis d'analyser des caractéristiques structurales et évolutives de cette protéine (Figure 6). Cinq régions sont conservées au cours de l’évolution, délimitant les domaines I à V (Soussi & May, 1996).
La région centrale correspondant aux domaines II à V concentre 80 à 90 % des mutations trouvées dans les cancers : l’incidence des mutations de p53 est élevée dans les cancers humains en accord avec la fonction suppresseur de tumeurs de cette protéine (Poumon 70%, colon, ovaire, vessie environ 60%).
Les fonctions portées par les parties N- et C-terminales ont également été identifiées. Le domaine N-terminal, acide et riche en proline, contient le domaine de transactivation qui interagit avec la machinerie transcriptionnelle de base et forme des complexes avec des facteurs de transcription spécifiques.
24
Figure 6 : Représentation schématique de la structure de la protéine p53.
Les chiffres entre parenthèses indiquent la position des acides aminés des domaines particuliers. Les chiffres romains indiquent les régions conservées au cours de l’évolution. NLS : Signal de localisation nucléaire ; NES : Signal d’export nucléaire. D’Après (May & May, 1999).
Parmi les nombreuses cibles transcriptionnelles de p53, nous ne citerons ici que celles impliquées dans l’arrêt du cycle cellulaire :
- p21Cip1, un inhibiteur de kinases dépendantes des cyclines (CDKi), qui peut activer l’arrêt du cycle cellulaire à la fois au point de restriction de la phase G1 et en
phase G2 (Agarwal et al., 1995; Bates et al., 1998b; Harper et al., 1993).
- la protéine 14-3-3 le dans la transduction de signaux et le maintien de l’arrêt du cycle cellulaire en phase G2, par liaison et séquestration de
protéines CDK dans le cytoplasme (Hermeking et al., 1997; Laronga et al., 2000; Muslin & Xing, 2000).
D’autres cibles sont impliquées dans l’apoptose, telles les protéines pro-apoptotiques Bax (Miyashita & Reed, 1995), Noxa (Oda et al., 2000a), PUMA (Nakano & Vousden, 2001; Yu et al., 2001) et p53AIPI (Oda et al., 2000b). Ces protéines mitochondriales induisent une perte de potentiel de la membrane mitochondriale et le relargage du Cytochrome C, activant ainsi la cascade apoptotique Apaf-1/caspase-9 (Bossy-Wetzel & Green, 1999).