Dynamique de la formation des coprs multilamellaires et
de l'enrobage de bactéries par différents protozoaires
Mémoire
Alicia Durocher
Maîtrise en microbiologie - avec mémoire
Maître ès sciences (M. Sc.)
Dynamique de la formation des corps
multilamellaires et de l’enrobage de bactéries par
différents protozoaires
Mémoire
Alicia Durocher
Sous la direction de :
Résumé
Plusieurs protozoaires, des eucaryotes unicellulaires ubiquitaires, sont des prédateurs de bactéries. Cependant, les relations bactéries-protozoaires sont complexes et peuvent donner lieu à des interactions particulières. Certains protozoaires, dont l’amibe sociale Dictyostelium discoideum, peuvent produire des corps multilamellaires (CML) lorsqu’ils digèrent des bactéries. Ces structures avaient initialement été identifiées comme des déchets métaboliques, mais il a été suggéré qu’elles pourraient avoir des rôles supplémentaires. Ce ne sont pas toutes les bactéries qui sont digérées par tous les protozoaires, et certaines bactéries résistantes à la digestion peuvent être enrobées dans les corps fécaux de ces protozoaires. Les corps fécaux partagent des similitudes avec les CML. Ce projet de maîtrise visait à éclairer certains aspects des interactions bactéries-protozoaires, en caractérisant la voie phagocytique d’amibes sociales environnementales afin d’examiner leur production potentielle de CML, et en analysant l’impact de différentes caractéristiques bactériennes sur la morphologie de l’enrobage par des ciliés. Ces objectifs furent atteints en cultivant les protozoaires d’intérêt en présence de bactéries digestibles (pour la production de CML) ou non (pour l’enrobage) et en faisant appel à diverses méthodes de microscopie. Nos résultats montrent que les quatre isolats d’amibes sociales environnementales, qui appartiennent tous au genre Dictyostelium, mais qui présentent des caractéristiques différentes, peuvent produire des CML lorsque cultivés sur bactéries digestibles. Pour l’étude de la morphologie de l’enrobage de bactéries, les résultats suggèrent que l’hydrophobicité de surface et la taille de l’espèce bactérienne seraient les caractéristiques ayant le plus fort impact sur la morphologie des corps fécaux. Toutefois, il n’est pas exclu que d’autres facteurs interviennent également, incluant des facteurs qui n’étaient pas à l’étude dans ce projet. Ces résultats approfondissent notre compréhension des relations bactéries-protozoaires, mais de nombreuses autres questions sont toujours sans réponse et le développement de méthodes d’analyse plus raffinées sera primordial pour répondre à ces questions.
Abstract
Many protozoa, ubiquitous unicellular eukaryotes, are predators of bacteria. However, bacteria-protozoa relationships are complex and can lead to some particular interactions. Some protozoa, including the social amoeba Dictyostelium discoideum, can produce multilamellar bodies (MLBs) upon digesting bacteria. These structures were initially identified as metabolic waste, but it has been suggested that they could have additional roles. Not all bacteria can be digested by all protozoa, and some digestion-resisting bacteria can be packaged into the fecal bodies produced by protozoa. These fecal bodies share similarities with MLBs. This master’s project was meant to shed a light on some aspects of bacteria-protozoa interactions, by characterizing the phagocytic pathway of some environmental social amoebae and by analyzing the impact of bacterial characteristics on the morphology of bacteria packaging. These objectives were met by cultivating the protozoa species of interest with either digestible bacteria (for MLB production) or undigestible bacteria (for packaging) and using diverse microscopy methods. Our results show that the four environmental isolates of social amoebae, belonging to the Dictyostelium genus but presenting distinct characteristics, can produce MLBs upon growth on digestible bacteria. As for the study of bacteria packaging morphology, results suggest that a bacteria’s surface hydrophobicity and cell size are the characteristics impacting packaging morphology the most. However, it is not excluded that other factors may intervene as well, including some not considered in this project. These results bring new understanding to bacteria-protozoa relationships, but many questions remain. The development of more refined analysis method will be paramount to answering these.
Table des matières
Résumé... ii
Abstract ... iii
Table des matières ... iv
Liste des figures... vi
Liste des tableaux ...vii
Liste des abréviations ... viii
Liste des sigles ... ix
Liste des acronymes... x
Remerciements... xiii
Avant-propos ... xv
Introduction ... 1
Hypothèse et objectifs de recherche ... 11
Chapitre 1: Various dictyostelids from the environment can produce multilamellar bodies 13 1.1 Résumé ... 13
1.2 Abstract ... 13
1.3 Introduction ... 14
1.4 Materials and methods ... 15
1.5 Results ... 18
1.6 Discussion ... 25
1.7 Conclusion ... 28
1.8 Bibliography ... 28
Chapitre 2 Non-pathogenic bacteria packaged by Tetrahymena: study of fecal pellet morphology and bacterial factors affecting it ... 31
2.1 Résumé ... 31
2.2 Abstract ... 31
2.3 Introduction ... 32
2.4 Materials and methods ... 33
2.5 Results and discussion ... 34
2.6 Limitations ... 41
2.7 References ... 41
2.8 Additional file ... 43
Discussion du chapitre 1 ... 49
Discussion du chapitre 2 ... 51
Conclusion ... 55
Bibliographie ... 58
Liste des figures
Figure I.1 : La progression de bactéries digestibles ou non au sein de la voie phagocytique
des protozoaires...1
Figure I.2 : Les compartiments de la voie endocytique de D. discoideum...2
Figure I.3 : Le cycle de vie des amibes sociales comporte une phase de croissance unicellulaire et une phase de développement multicellulaire...4
Figure I.4 : Des CML produits par l’amibe sociale D. discoideum...6
Figure I.5 : Différentes morphologies peuvent être observées lorsque des bactéries sont enrobées...8
Figure 1.1 : Growth of the amoeboid isolates on K. aerogenes...19
Figure 1.2 : Multicellular development of the amoeboid isolates...20
Figure 1.3 : Average cell size (µm2) of the amoeboid isolates…...21
Figure 1.4 : H36 staining is positive for all of the amoeboid isolates...22
Figure 1.5 : The H36 antibody displays different protein band patterns in the amoeboid isolates...23
Figure 1.6 : MLBs produced by amoeboid isolates were visible both inside the cells and in the medium...24
Figure 2.1 : DIC and DAPI fluorescence microscopy of pellets produced by T. pyriformis...36
Figure 2.2 : TEM images of bacteria packaging by Tetrahymena...37
Figure 2.3 : Bacterial cells are viable in pellets produced by Tetrahymena...40
Figure S1 : The material surrounding M. luteus pellets in formation is thick and of undefined nature...47
Figure S2 : Des inclusions de nature inconnue sont visibles dans le cytoplasme de Cupriavidus sp...64
Liste des tableaux
Tableau 1.1 : Sequence identities in 18S rDNA of the isolates and closest relative...18 Tableau S1 : Amoeba-resistant bacteria packaged by ciliates...45 Tableau S2 : Characteristics of the four bacteria studied...46
Liste des abréviations
et al.: et alii (et les autres personnes) OD595: densité optique à 595 nm
Liste des sigles
ADN/DNA : acide désoxyribonucléique/deoxyribonucleic acid AMPc : adénosine monophosphate cyclique
ARB : amoeba-resisting bacteria (bactérie résistante aux amibes) ARN/RNA : acide ribonucléique/ribonucleic acid
ARNr/rRNA : acide ribonucléique ribosomal/ribosomal ribonucleic acid ATCC: American Type Culture Collection
BMC : BioMed Central
CML/MLB : corps multilamellaire/multilamellar body
DIC : differential interference contrast (contraste interférentiel) FRQNT: Fonds de recherche du Québec – Nature et technologie IBIS : Institut de biologie intégrative et des systèmes
PBS : phosphate-buffered saline (tampon phosphate salin) PCB : plate-count broth (bouillon de dénombrement)
PCR : polymerase chain reaction (réaction de polymérase en chaîne) PFA : paraformaldéhyde
PP : protéose peptone
PRB : protozoa-resisting bacteria (bactérie résistante aux protozoaires) rpm : révolutions par minute
SEM : scanning electron microscopy (microscopie électronique à balayage) SM : standard medium (milieu standard)
SPP : sucre protéose peptone
TEM : transmission electron microscopy (microscopie électronique à transmission) TSA : trypticase soy agar (gélose trypticase soya)
TSB: trypticase soy broth (bouillon trypticase soya) UV : ultra-violet
Liste des acronymes
CRI : Centre de recherche en infectiologie DAPI : 4',6-diamidino-2-phénylindole
À mes parents, pour leur support
constant.
Time flows in a strange
way on a Sunday.
Remerciements
Merci à mes parents d’avoir toujours cru en moi et d’avoir travaillé fort pour me permettre de poursuivre mon éducation comme je le désirais. Votre support a été crucial à ma réussite et vous m’avez toujours poussée à creuser plus loin dans mes apprentissages. Je remercie également ma sœur Florence, qui a toujours su me remonter le moral dans les moments plus difficiles. J’aimerais également remercier mon parrain Gilles et ma marraine Jackie pour leur soutien et leurs encouragements.
Merci à Steve d’être un directeur de maîtrise incomparable. Je me considère très chanceuse d’avoir un chercheur aussi compréhensif, éclairé et ouvert comme directeur. Je suis très heureuse d’avoir pu reprendre le flambeau du projet enrobage/CML et je te suis très reconnaissante de m’avoir sélectionnée pour ça.
Je remercie les membres de mon comité d’encadrement, Caroline Duchaine et Sébastien Faucher, pour leurs judicieux conseils tout au long de mon parcours de maîtrise. Je suis très honorée que vous ayez accepté de m’accorder votre temps et j’espère bien que ce présent document vous satisfera!
Un gros merci à tous les membres du labo Charette que j’ai côtoyés au cours de mon baccalauréat et de ma maîtrise : Katherine, Marie-Stéphanie, Antony, Sabrina, Alex, Perrine, Marie-Ange, Nava, Pierre-Étienne, Gabrielle, William et tous les autres. Le labo Charette a une ambiance de travail incroyable et c’est grâce à vous tous. Un merci tout spécial à mes co-autrices : Alix, qui m’a montré pas mal tout ce que je sais faire au labo, Cynthia, qui est toujours d’une grande aide ainsi qu’une voisine de bureau très agréable, et Valérie, qui répond patiemment à mes mille et une questions pas toujours pertinentes, qui supporte mes questionnements existentiels et qui m’a offert son soutien constant tout au long de ma maîtrise.
Merci au FRQNT de m’avoir accordé une bourse de maîtrise et d’avoir permis le financement de ce projet.
Merci à la plateforme de microscopie de l’IBIS pour leur assistance, particulièrement en microscopie électronique. Merci également à la plateforme de microscopie du CRI pour leur aide généreuse!
Finalement, un énorme merci à mon amoureux, François, de m’avoir endurée tout au long de ma maîtrise et d’avoir su gérer mon stress quand j’en étais incapable. Merci pour toutes les fois où tu es venu m’ouvrir la porte des tunnels la fin de semaine et pour tous les allongés que tu m’as faits. Merci d’avoir écouté toutes mes présentations et de m’avoir convaincue que j’étais capable de parler en public. Merci d’avoir vu mon potentiel quand je ne le pouvais pas. Je t’adore et ma maîtrise aurait clairement été beaucoup plus laborieuse sans ta présence à mes côtés.
Avant-propos
Le chapitre 1 est constitué de l’article « Various dictyostelids from the environment can produce multilamellar bodies », publié en ligne le 31 juillet 2020 au Canadian Journal of Microbiology (https://doi.org/10.1139/cjm-2020-0187). Cet article a pour auteurs Alicia F. Durocher, Cynthia Gagné-Thivierge et Steve J. Charette. J’ai participé à cette publication à titre de première auteure. J’ai rédigé l’article, créé les figures, sauf les figures 1 et 3 créées par Cynthia Gagné-Thivierge et la figure 5 créée par Steve Charette, et réalisé toutes les manipulations présentées à l’exception de l’amplification PCR et du séquençage du gène de l’ARNr 18S des amibes environnementales, réalisés par Cynthia Gagné-Thivierge, et de l’immunobuvardage au H36, réalisé par Cynthia Gagné-Thivierge et Steve Charette. J’ai participé à la conception des expérimentations et à l’analyse des résultats avec les autres coauteurs. Les numéros des figures et tableaux ont été ajustés par rapport à l’article original pour leur intégration à ce mémoire.
Le chapitre 2 est constitué de l’article « Non-pathogenic bacteria packaged by Tetrahymena: study of fecal pellet morphology and bacterial factors affecting it », soumis sous cette forme le 2 mars 2020 à BMC Research Notes. Puisque la publication de l’article n’a pas été approuvée sous cette forme, il sera soumis après édition dans un autre journal en temps opportun. Cet article a pour auteurs Alicia F. Durocher, Alix M. Denoncourt, Valérie E. Paquet et Steve J. Charette. J’ai participé à cette publication à titre de première auteure. J’ai rédigé l’article, créé les figures et réalisé toutes les manipulations présentées. J’ai participé à la conception des expérimentations et à l’analyse des résultats avec les autres coauteurs. Les numéros des figures ont été ajustés par rapport à l’article original pour leur intégration à ce mémoire.
Introduction
Les protozoaires sont des organismes eucaryotes unicellulaires ubiquitaires et retrouvés dans des environnements variés, tant naturels que créés par l’Humain (Barker et Brown, 1994). Une grande part d’entre eux sont des prédateurs des bactéries, mais d’autres groupes trophiques sont possibles. Le groupe des protozoaires est plutôt fonctionnel que réellement valable d’un point de vue évolutif ou phylogénétique, puisque les organismes regroupés sous l’appellation « protozoaires » forment un groupe hétéroclite comprenant une grande diversité de morphologies, de tailles, d’habitats et de modes de vie (Fenchel, 1987). Ils sont grossièrement classés en quatre groupes selon leur mode de locomotion, soient les amibes, qui utilisent des pseudopodes; les ciliés, qui se déplacent à l’aide de cils motiles; les flagellés, qui utilisent des flagelles; et les apicomplexés, qui sont des parasites obligatoires non motiles. Dans le cadre de mon projet de maîtrise, deux groupes m’étaient d’un intérêt particulier, les amibes sociales, qui font partie du groupe des amibes, et les ciliés.
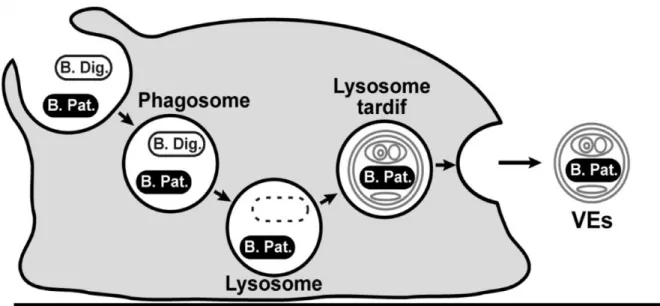
Figure I.1 : La progression de bactéries digestibles ou non au sein de la voie phagocytique des protozoaires. Lorsque des bactéries digestibles (B. Dig,) ou pathogènes/non-digestibles (B. Pat.) sont ingérées, elles se retrouvent d’abord dans les phagosomes du protozoaire, qui fusionnent ensuite avec les lysosomes afin de digérer les particules phagocytées. Le phagosome/lysosome tardif contient de potentiels restes non digérés, qui seront ensuite exocytosés sous forme de vésicule (VEs), comme des CML ou des bactéries enrobées. Image fournie par et reproduite avec la permission de Steve Charette.
La phagocytose est l’ingestion de particules, par exemple des bactéries, par une cellule eucaryote. Outre les cellules immunitaires des organismes multicellulaires, certains protozoaires, dont les amibes et les ciliés, sont également capables de phagocytose et peuvent s’en servir pour se nourrir. La voie phagocytique des protozoaires peut mener à la digestion de la particule ingérée ou à son exocytose si non digestible (Fenchel, 1987). Les vacuoles digestives progressent dans la voie phagocytique en traversant différents stades : la coupe phagocytique se referme après la captation d’une particule pour former un phagosome, les phagosomes initiaux rencontrent des lysosomes, responsables de l’acidification de la vacuole, pour former des phagolysosomes, qui deviennent ensuite des phagosomes tardifs, contenant les restes indigestes qui seront éjectés (Kessin, 2001). La figure I.1 illustre la progression de bactéries digestibles ou non dans la voie phagocytique d’un protozoaire. Plusieurs protéines sont impliquées dans la voie phagocytique et l’utilisation d’anticorps produits contre celles-ci permet l’étude de la phagocytose, de la digestion et de phénomènes connexes comme la production de corps multilamellaires ou l’enrobage de bactéries (Mercanti et al., 2006). La figure I.2 présente certaines des protéines de l’amibe Dictyostelium discoideum détectées par des anticorps, dont l’anticorps H36 qui a été utilisé dans cette étude.
Figure I.2 (page précédente). Les compartiments de la voie endocytique de D.
discoideum. Des cellules ont été cultivées en milieu liquide avant d’être traitées en immunofluorescence et observées en microscopie confocale. A) Protéine associée à l’anticorps H36 (rouge), protéine p80 (vert) et protéine Rh-50 (bleu). B) Les protéines H+-ATPase (rouge), p80 (vert) et Rh-50 (bleu). Dans le panneau A, on peut distinguer la membrane plasmique en rouge, les lysosomes et post-lysosomes en vert et la vacuole contractile en bleu. Dans le panneau B, les post-lysosomes (astérisques) peuvent être distingués des lysosomes grâce à l’absence de H+-ATPase. Cette dernière est aussi présente dans la vacuole contractile. Échelle: 5 µm. Image fournie par et reproduite avec la permission de Steve Charette.
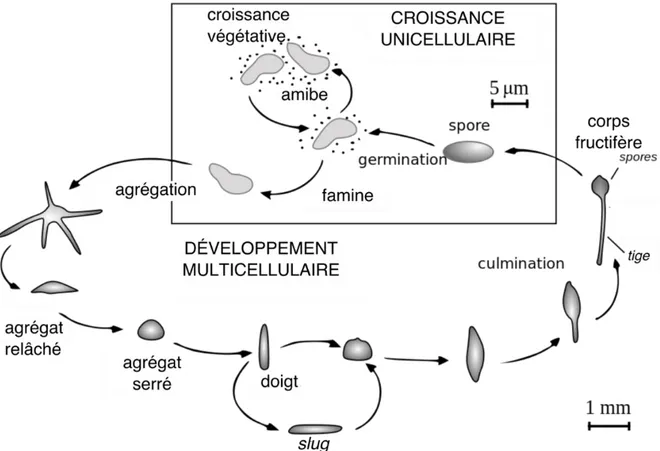
Les amibes sociales, parmi lesquelles on retrouve le genre Dictyostelium qui est particulièrement étudié, se déplacent à l’aide de pseudopodes et se nourrissent principalement de bactéries dans la nature, malgré que certaines souches de laboratoire soient capables de croissance axénique. Ces organismes sont, entre autres, retrouvés sur le sol forestier, dans des endroits humides. La distinction principale des amibes sociales par rapport au reste des amibes est leur cycle de vie particulier, marqué par une phase de développement multicellulaire. En conditions de croissance avantageuses, les amibes sociales croissent de façon unicellulaire. Lors de périodes de famine, les cellules s’agrègent en amas multicellulaire pour éventuellement former une structure appelée corps fructifère, tel que montré dans la figure I.3, composée d’une tige constituée de cellules s’étant sacrifiées et d’une tête contenant des spores prêtes à être dispersées par l’eau ou des animaux afin de rejoindre un environnement plus favorable (Bonner, 1982; Smith et al., 2014). 20% des cellules de la population formeront la tige du corps fructifère, tandis que les cellules restantes deviendront des spores. Chez Dictyostelium, l’agrégation a lieu grâce à l’AMPc, qui contrôle la chimiotaxie (Kessin, 2001). La formation de corps fructifères illustre bien la complexité du développement multicellulaire chez les amibes sociales, car il demande un haut niveau de coopération entre les cellules et peut mener à des phénomènes inattendus, comme l’altruisme, car 20% des cellules se sacrifient pour former la tige du corps fructifère; la tricherie, où certaines cellules destinées à mourir pour former la tige rejoignent plutôt les cellules de la spore; ou même de l’agriculture primitive, observée lorsque des amibes sociales emportent certaines souches bactériennes avec elles dans la spore (Brock et al., 2011; Strassmann et al., 2000). Des stratégies de résistance aux tricheurs sont même apparues (Khare et al., 2009). Il transparaît donc que les interactions entre amibes sociales
sont vastes et que plusieurs aspects de ces interactions sont encore potentiellement inconnus.
Figure I.3 : Le cycle de vie des amibes sociales comporte une phase de croissance unicellulaire et une phase de développement multicellulaire. Une fois agrégée, les cellules d’amibes sociales vont progressivement culminer pour former un corps fructifère permettant la dispersion des spores. Cette figure a été modifiée (traduction) à partir de celle produite par Tijmen Stam, IIVQ (conversion SVG) et user:Hideshi (version
originale) - en:Image:Dicty Life Cycle H01.png, CC BY-SA 3.0,
https://commons.wikimedia.org/w/index.php?curid=1782175.
Contrairement aux amibes qui doivent se déformer pour bouger, les ciliés se déplacent à l’aide de courts cils et ont une structure cellulaire fixe, incluant une ouverture analogue à une bouche désignée « cytostome », permettant l’ingestion de particules et de liquide. La phagocytose a exclusivement lieu à cet endroit, alors que les amibes peuvent phagocyter une particule à partir de n’importe quel endroit de la membrane cellulaire (Fenchel, 1987). Les ciliés sont sensibles à la concentration de bactéries dans leur environnement et ne peuvent se nourrir de bactéries que dans des environnements où celles-ci sont concentrées (Fenchel, 1980; Iriberri et al., 1994). Parmi ce groupe, on retrouve le genre Tetrahymena,
qui est capable d’un haut taux d’ingestion, soit près de 100% de son volume par heure, comme d’autres ciliés (Fenchel, 1987; Iriberri et al., 1995). Ce genre est donc particulièrement intéressant pour étudier le processus de phagocytose. Tetrahymena s’alimente par filtration (filter-feeding) et ses vacuoles alimentaires transitent au travers de la cellule assez rapidement, en 25 minutes chez T. pyriformis (Thurman et al., 2010). Des mutants axéniques se nourrissant par macropinocytose sont également disponibles pour la recherche (Cassidy-Hanley, 2012; Curds et Cockburn, 1968). Dans la nature, le genre Tetrahymena est associé aux écosystèmes d’eau douce (Fenchel 1987).
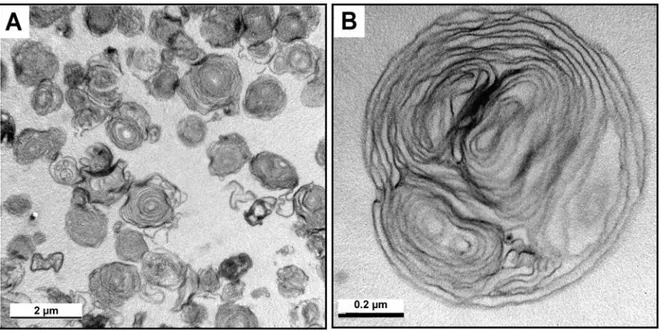
Les corps multilamellaires (CML) sont des structures produites par de nombreux types de cellules eucaryotes, qui serviraient au stockage et à la sécrétion de lipides (Schmitz et Müller, 1991). Ils sont semblables à des sphères concentriques de membrane, à l’image des couches d’un oignon, et sont habituellement de forme ronde, tel que montré à la figure I.4. Chez les protozoaires, leur production a été observée chez des amibes libres et des ciliés (Berk et al., 2008; Chekabab et al., 2012), ainsi que chez plusieurs souches de l’amibe sociale Dictyostelium discoideum, principalement chez des souches de laboratoire axéniques (Barondes et al., 1985; Cooper et al., 1986; Emslie et al., 1998; Fukuzawa et Ochiai, 1993; Gezelius, 1959; Gezelius, 1961; Hohl, 1965; Marchetti et al., 2004; Mercer et Shaffer, 1960).
Des structures similaires aux CML produits par D. discoideum sont présentes chez d’autres organismes, dont les humains. La fonction normale des CML chez l’humain est le transport de lipides spécialisés vers des sites spécifiques. Par exemple, ils permettent le maintien d’un film de dipalmytoilphosphatidylcholine à la surface des poumons, assurant leur bon fonctionnement. À l’inverse, ils peuvent aussi être impliqués dans certaines pathologies, comme le syndrome de détresse respiratoire, causé par des pneumocytes incapables de sécréter des corps multilamellaires et qui représente une cause importante de mortalité chez les prématurés; le psoriasis, où la sécrétion de corps lamellaires est restreinte chez les cellules de la peau en hyperprolifération; et la maladie de Niemann-Pick, où une accumulation de corps multilamellaires résulte en l’accumulation de sphingomyéline dans les lysosomes (Schmitz et Müller, 1991).
Figure I.4 : Des CML produits par l’amibe sociale D. discoideum. Les CML ont une apparence caractéristique, montrant qu’ils sont composés de multiples couches de membranes concentriques. Image adaptée de Paquet et al., 2013.
Les CML ont une composition particulière en protéines et en lipides qui fournit certains renseignements quant à leur origine et leur possible fonction. Chez D. discoideum, une comparaison du profil lipidique des CML, qui sont riches en lipides neutres et en phospholipides, principalement la phosphatidyléthanolamine et la phosphatidylcholine, avec ceux de l’amibe les ayant produits et de la bactérie que l’amibe avait consommée a montré que les CML avaient un profil lipidique plus proche de celui des amibes (Paquet et al., 2013). Cela suggère que les CML seraient directement/principalement issus du métabolisme de l’amibe les produisant, plutôt que d’être composés des restes des bactéries qui auraient été digérées. Diverses protéines ont également été identifiées comme étant associées aux CML. On retrouve, entre autres, la discoïdine, une lectine endogène impliquée dans l’adhésion aux substrats extracellulaires; PhoPQ, une protéine hypothétique similaire à AprA, un répresseur de prolifération sécrété par les cellules de D. discoideum lorsqu’elles sont en croissance; et SctA, une protéine sécrétée associée aux pycnosomes, des structures membraneuses produites par D. discoideum (Barondes et al., 1985; Brock et Gomer, 2005; Denoncourt et al., 2016; Sabra et al., 2016).
Bien qu’initialement identifiés comme des déchets métaboliques (Gezelius, 1959; Hohl, 1965), il est de plus en plus considéré que la formation de CML représente une trop grande dépense énergétique pour être un simple mode d’évacuation des restes de la phagocytose.
Des rôles alternatifs ont été proposés pour les CML, dont celui de réserve de nourriture ou encore de mode de communication intercellulaire. Il a été observé que lorsque des amibes naïves D. discoideum DH1-10 étaient exposées aux CML produits par des amibes nourries avec Klebsiella aerogenes, les amibes naïves ingèrent les CML et ceux-ci semblent être dégradés (Denoncourt et al., 2017a) suggérant au minimum un rôle de source d’énergie pour les CML des amibes sociales. Les protéines associées aux CML évoquent aussi une implication de ces structures dans la communication intercellulaire. La protéine PhoPQ est particulièrement intéressante pour cette théorie, étant l’analogue de AprA. Chez D. discoideum, AprA est sécrétée par les cellules en croissance et réprime la prolifération; cependant, seules les cellules végétatives sont sensibles à l’action chimiorépulsive d’AprA : la protéine n’a pas d’effet sur les cellules affamées (starved) (Brock et Gomer, 2005; Phillips et Gomer, 2012). Ainsi, les CML pourraient être impliqués dans la réponse à la famine chez Dictyostelium et permettre la coordination de l’agrégation des cellules pour la formation des corps fructifères.
Certaines bactéries sont capables de résister à la phagocytose par les protozoaires. Certaines vont user de mécanismes pour éviter d’être ingérées, comme une taille augmentée, une haute vitesse de déplacement ou le camouflage de leurs traits de surface, entre autres, tandis que d’autres vont être ingérées, mais résisteront à la digestion, seront capables de se multiplier dans le protozoaire ou produiront des toxines intracellulaires (Matz et Kjelleberg, 2005). Certaines espèces pathogènes vont infecter la cellule et causer sa mort, leur permettant de s’échapper. D’autres vont coloniser la cellule, comme Legionella pneumophila, une bactérie pathogène intracellulaire qui utilise les amibes comme habitat, menant à sa propagation et parfois à des infections chez les humains (Abu Kwaik et al., 1998).
La phagocytose pose un stress sur les communautés bactériennes et a donc une influence sur les caractéristiques de ces communautés (Jürgens et Matz, 2002). Différentes méthodes peuvent être utilisées par les bactéries pour résister à la phagocytose. La taille d’une bactérie joue un rôle important dans l’ingestion, car certains protozoaires, par exemple les ciliés, sont sensibles à la taille des particules ingérées (Fenchel, 1980). Ainsi, certaines bactéries vont avoir tendance à produire des cellules filamenteuses lorsqu’elles croissent en présence de protozoaires (Matz et Kjelleberg, 2005). L’agrégation de cellules bactériennes en microcolonies ou en biofilms peut également être avantageuse pour les bactéries soumises au stress de prédation.
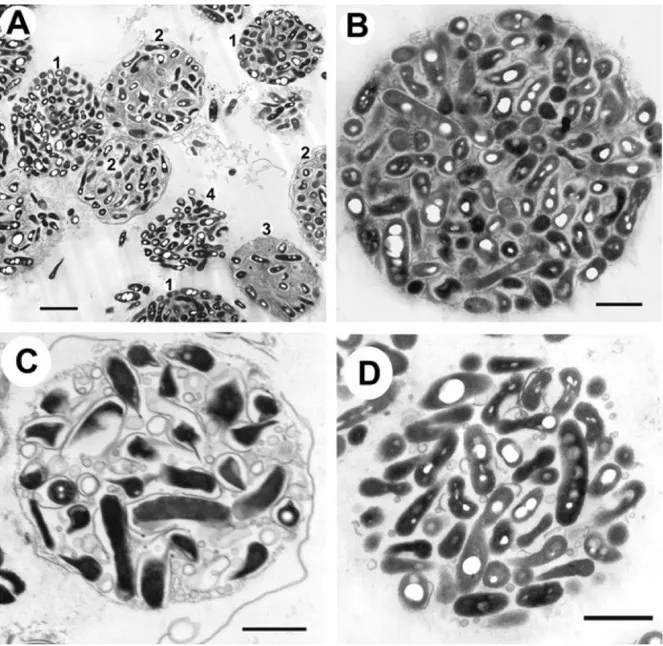
Figure I.5 : Différentes morphologies peuvent être observées lorsque des bactéries sont enrobées. Lorsque la bactérie L. pneumophila est enrobée par le cilié
Tetrahymena tropicalis, les vésicules contenant les bactéries présentent diverses
morphologies (A). Celles-ci peuvent contenir plus (B) ou moins (C) de membrane entre les cellules bactériennes, voire très peu (D). Figure tirée de Berk et al., 2008.
L’enrobage de bactéries est un phénomène observé lorsque des bactéries ayant résisté à la digestion suite à leur phagocytose par un protozoaire sont expulsées dans les corps fécaux produits par cet eucaryote (Denoncourt et al., 2014). La structure formée est constituée d’un agrégat de cellules bactériennes au centre, entouré d’une ou plusieurs couches de membrane, tel que montré à la figure I.5. Ces couches de membranes retiennent les cellules bactériennes ensemble, mais offrent également une protection très
avantageuse pour celles-ci. Éventuellement, lorsque les bactéries recommencent à se diviser, la membrane est rompue et les bactéries retrouvent leur forme libre (Gourabathini et al., 2008).
Les avantages de l’enrobage pour les bactéries sont nombreux. Il a été observé que l’enrobage peut procurer aux bactéries une résistance à la gentamicine, à certains produits chlorés, à un pH faible, à des cycles de gel-dégel, à la sonication, à la dessiccation et aux rayons UV (Berk et al., 1998; Berk et al., 2008; Brandl et al., 2005; Espinoza-Vergara et al., 2019; Gourabathini et al., 2008; Koubar et al., 2011; Raghu Nadhanan et Thomas, 2014). De plus, l’enrobage peut favoriser la survie à long terme en conditions de famine, dans certains cas jusqu’à 6 mois (Espinoza-Vergara et al., 2019; Koubar et al., 2011). Certaines espèces pathogènes sont également plus virulentes après avoir été éjectées de l’enrobage (Espinoza-Vergara et al., 2019; Faulkner et al., 2008).
Parmi les bactéries ayant déjà été identifiées comme capables d’être enrobées, on retrouve principalement des espèces pathogènes, incluant L. pneumophila, Vibrio cholerae, Listeria monocytogenes, Salmonella enterica, Helicobacter pylori, Campylobacter jejuni ainsi que Escherichia coli O157:H7 (Berk et al., 1998; Berk et al., 2008; Brandl et al., 2005; Espinoza-Vergara et al., 2019; Gourabathini et al., 2008; Marciano-Cabral et Cabral, 2003; Raghu Nadhanan et Thomas, 2014; Smith et al., 2012; Trigui et al., 2016). Pour certaines de ces bactéries, les mécanismes spécifiques nécessaires à l’enrobage ont été identifiés. Chez L. pneumophila, le système Dot/Icm est essentiel pour la résistance à la digestion et l’enrobage, tandis que chez V. cholerae, le gène ompU codant pour une protéine de la membrane externe est indispensable pour survivre à la digestion (Berk et al., 2008; Espinoza-Vergara et al., 2019). Chez E. coli, aucun facteur précis permettant l’enrobage n’a été identifié, mais l’enrobage semble être lié à la virulence, étant donné que des souches non virulentes d’E. coli sont facilement digérées par Tetrahymena, tandis que des souches pathogènes diarrhéagéniques sont enrobées par ce cilié (Berk et al., 2008; Smith et al., 2012). Cependant, même si peu d’études se sont penchées sur l’enrobage chez les bactéries non pathogènes, certaines d’entre elles ont pu être identifiées comme pouvant être enrobées. Parmi celles-ci, on retrouve Mycobacterium smegmatis, une mycobactérie faisant partie de la flore humaine normale et généralement considérée non virulente, qui peut être enrobée par T. pyriformis (Denoncourt et al., 2017b). Il a également été montré que l’amibe sociale D. discoideum pouvait enrober des souches appartenant aux genres Microcococcus, Cupriavidus, Rathayibacter et Microbacterium, toutes non pathogènes
(Paquet et Charette, 2016). Malgré ces développements, aucune étude extensive de l’enrobage de bactéries non pathogènes par des ciliés n’a été conduite jusqu’à maintenant. L’enrobage représente également des avantages potentiels dans un contexte d’infections, particulièrement pour les infections respiratoires et gastro-intestinales. En effet, une protection contre divers types de stress est procurée aux bactéries par l’enrobage, dont certains types associés à la propagation des bactéries pathogènes. Une résistance contre la dessiccation et les rayons UV pourrait avantager des bactéries en contexte d’aérosolisation, qui est une voie de propagation possible pour certains agents pathogènes respiratoires, par exemple Mycobacterium tuberculosis et L. pneumophila (Muder et al., 1986; Riley, 1957). De plus, l’enrobage réunit plusieurs cellules bactériennes en une seule particule, habituellement d’un diamètre qualifié de respirable (<10 µm), ce qui pourrait également faciliter la propagation d’infections respiratoires (Berk et al., 1998; Macher, 1999; Nicas et Sun, 2006). Il a également été souligné qu’une résistance accrue aux pH faibles pourrait protéger les bactéries enrobées contre l’acide gastrique et permettre l’apparition d’infections gastro-intestinales plus aisément (Espinoza-Vergara et al., 2019). Cependant, aucun cas d’infection humaine associé à l’enrobage de bactéries n’a été reporté jusqu’à présent, mais il serait difficile d’identifier une telle infection avec les moyens disponibles pour le moment.
Différentes morphologies d’enrobage sont possibles. Les bactéries enrobées peuvent être entourées de plusieurs couches de membrane, d’une seule couche, de fragments ou même ne pas être recouvertes de membrane dans certains cas (figure I.5) (Berk et al., 2008). L’analyse de l’enrobage en microscopie électronique à balayage (SEM) a révélé dans certains cas que le matériel membraneux retenant les cellules était similaire à un filet ou une toile(Denoncourt et al., 2017b; Smith et al., 2012). Il n’a pas encore été déterminé quels facteurs influencent cette morphologie, mais il a été observé que des différences étaient présentes selon le protozoaire et la bactérie impliqués dans l’enrobage. Par exemple, la même bactérie enrobée par deux protozoaires différents ne produira pas nécessairement la même morphologie d’enrobage. Si on compare l’enrobage de L. pneumophila par une amibe comme Acanthamoeba castellanii à celui produit par un cilié comme Tetrahymena tropicalis, on observe que l’enrobage par A. castellanii est abondant en membranes et que seules quelques bactéries sont enrobées, tandis qu’avec T. tropicalis il y a beaucoup moins de membrane, mais beaucoup plus de bactéries (Berk et al., 1998; Berk et al., 2008). À l’inverse, le même protozoaire ne produira pas forcément la même morphologie d’enrobage
avec toutes les bactéries qu’il enrobe. Quand Campylobacter jejuni est enrobé par T. pyriformis, une bonne quantité de membrane est présente autour de l’amas de cellules; avec Mycobacterium smegmatis, l’enrobage ne produit qu’une mince couche de membrane (Denoncourt et al., 2017b; Trigui et al., 2016).
L’enrobage de bactéries illustre la complexité des relations bactéries-protozoaires, car c’est une interaction extrêmement spécifique. Différentes souches de la même espèce ne seront pas nécessairement toutes enrobées au même degré, tel que montré par Espinoza-Vergara et collaborateurs avec quatre souches de Vibrio cholerae (Espinoza-Vergara et al., 2019). Des souches de la même espèce ne sont même pas toujours toutes enrobées, comme c’est le cas pour E. coli, dont les souches pathogènes sont souvent enrobées, contrairement aux souches non pathogènes qui sont digérées plus efficacement (Berk et al., 2008; Smith et al., 2012). Également, une bactérie enrobée par un protozoaire en particulier ne sera pas nécessairement enrobée par tous les protozoaires, pas même par ceux similaires à celui qui l’a enrobé (Espinoza-Vergara et al., 2019).
Hypothèse et objectifs de recherche
L’hypothèse de recherche guidant ce projet de maîtrise était que la production de CML et de bactéries enrobées serait un phénomène répandu chez les protozoaires, possible avec différentes bactéries. La recherche sur la production de CML chez les amibes sociales s’est jusqu’à maintenant limitée à l’espèce D. discoideum et généralement à des souches de laboratoire capables de croissance axénique. Bien que des hypothèses intéressantes aient été émises quant au rôle des CML de Dictyostelium, leur production pourrait être un artéfact de laboratoire. Il serait important de montrer que la production de ces structures est possible avec d’autres espèces d’amibes sociales pour solidifier ces hypothèses. L’enrobage de bactéries a jusqu’à maintenant été principalement étudié avec des souches bactériennes pathogènes. Cependant, il a été montré que la pathogénicité n’est pas nécessaire à la résistance à la digestion et que les lacunes de la voie phagocytique permettraient à certaines bactéries d’éviter la digestion (Thurman et al., 2010). De plus, l’enrobage de bactéries non pathogènes a été démontré possible avec D. discoideum (Paquet et Charette, 2016), mais aucune étude ne s’est penchée sur la question de façon exhaustive avec les ciliés.
Pour répondre à cette hypothèse de recherche, deux objectifs spécifiques étaient visés. Ceux-ci étaient :
A. Caractériser la voie phagocytique d’amibes sociales isolées de l’environnement. B. Observer la morphologie des bactéries enrobées produites par deux protozoaires de
types ciliés du genre Tetrahymena lorsque mis en co-culture avec 4 bactéries aux caractéristiques différentes (forme et taille de la cellule, résultat du test de Gram, hydrophobicité de surface).
L’objectif A vise à comparer les caractéristiques de la voie phagocytique d’amibes sociales issues de l’environnement, non axéniques, à celle de D. discoideum DH1-10, une souche axénique bien caractérisée et déjà identifiée comme étant productrice de CML (Paquet et al., 2013). Il était attendu que certaines de ces souches produisent également des CML, ce qui aiderait à solidifier les théories selon lesquelles les CML auraient un rôle particulier chez les amibes sociales, tel que décrit par Denoncourt et al., 2017a.
L’objectif B vise à éclaircir les facteurs contrôlant la morphologie de l’enrobage de bactéries. Les caractéristiques à l’étude ont été sélectionnées pour leur potentiel à influencer l’enrobage : la taille et la forme de la cellule sont soupçonnées de déterminer de quelle façon les cellules peuvent être disposées dans la vacuole alimentaire des ciliés et éventuellement dans la structure excrétée; la composition de la paroi bactérienne pourrait influencer la quantité de membrane présente autour des cellules, car l’origine de cette membrane est pour le moment inconnue, bien qu’il ait été proposé que des cellules déjà mortes avant l’ingestion pourraient être la source de cette membrane (Berk et al., 2008); et l’hydrophobicité a déjà été identifiée comme importante pour la résistance à la digestion en général, mais l’effet de cette caractéristique sur l’enrobage est toujours inconnu (Siegmund et al., 2018).
Chapitre 1: Various dictyostelids from the environment
can produce multilamellar bodies
Alicia F. Durocher1,2,3, Cynthia Gagné-Thivierge1,2,3 and Steve J. Charette1,2,3*
1. Institut de Biologie Intégrative et des Systèmes, Pavillon Charles-Eugène-Marchand, Université Laval, Québec, QC, Canada
2. Centre de recherche de l’Institut universitaire de cardiologie et de pneumologie de Québec, Université Laval, Québec, QC, Canada
3. Département de biochimie, de microbiologie et de bio-informatique, Faculté des sciences et de génie, Université Laval, Québec, QC, Canada
1.1 Résumé
Les corps multilamellaires (CML), des structures composées de couches membranaires concentriques, sont produits par divers protozoaires incluant certains ciliés, amibes libres et amibes sociales. Il fut initialement supposé que les CML de l’amibe sociale Dictyostelium discoideum soient des déchets, mais des rôles en communication intercellulaire ou comme réserves de nourriture ont été proposés. Parmi les dictyostélides, ce phénomène a seulement été observé chez D. discoideum, principalement avec des souches de laboratoire, habituellement en culture axénique. Il fut supposé que d’autres amibes sociales puissent produire des CML. Quatre isolats d’amibes sociales environnementales ont été caractérisés. Toutes les souches, appartenant au genre Dictyostelium, produisaient des CML similaires à ceux d’une souche de laboratoire de D. dictyostelium, selon nos analyses en microscopie (épifluorescence et TEM). Conséquemment, des CML pourraient être produits par des dictyostélides variés, semblablement aux souches de laboratoire, supportant un rôle des CML dans le cycle de vie de ces amibes.
1.2 Abstract
Multilamellar bodies (MLBs), structures composed of concentric membrane layers, are known to be produced by different protozoa including species of ciliates, free-living amoebae and Dictyostelium discoideum social amoebae. Initially believed to be metabolic waste, potential roles like cell communication and food storage have been suggested for D. discoideum MLBs, which could be useful for the multicellular development of social amoebae and as a food source. However, among dictyostelids, this phenomenon has only
been observed with D. discoideum, and mainly with laboratory strains usually grown in axenic conditions. It was thought that other social amoebae may also be able to produce MLBs. Four environmental social amoeba isolates were characterized. All strains belong to the Dictyostelium genus including some likely to be Dictyostelium giganteum. They have distinctive phenotypes comprising their growth rate on Klebsiella aerogenes lawns and the morphology of their fruiting bodies. They all produce MLBs similar to those produced by a D. discoideum laboratory strain when grown on K. aerogenes lawns, as revealed by analysis using the H36 antibody in epifluorescence microscopy as well as by transmission electron microscopy. Consequently, this study shows that MLBs are produced by various dictyostelid species, which further supports a role for MLBs in the lifestyle of amoebae.
1.3 Introduction
Multilamellar bodies (MLBs) are composed of concentric lipid membrane layers. They are involved in lipid storage and secretion in many eukaryotic cells (Schmitz and Müller, 1991). MLBs can be produced as part of the phagocytosis process and secreted by D. discoideum social amoebae upon feeding on digestible bacteria (Paquet et al., 2013).
In starvation conditions, such as when preys become inaccessible, D. discoideum enters a multicellular phase beginning with aggregation of the cells to form a migrating slug, leading to the creation of spore-containing fruiting bodies to withstand long-term starvation (Kessin, 2001). MLB production is mostly observed during the unicellular vegetative state, although MLBs can also be produced by aggregating cells (Hohl, 1965). Different MLB morphologies are possible depending, among others, on the density of membrane layers or the type of bacteria phagocytosed by D. discoideum cells (Denoncourt et al., 2014; Hohl, 1965; Paquet et al., 2013).
While MLBs were originally considered to be metabolic waste, it was noted that the formation of MLBs represents a cost for the cell that seems too large for simple waste evacuation. Biochemical analyses of both lipids and proteins found in these structures as well as functional analyses of D. discoideum MLBs were performed using, among others, the H36 antibody which binds to proteins on the MLBs. These analyses suggested additional roles for MLBs, including extracellular nutrient storage and a potential intermediary in communication between cells (Denoncourt et al., 2016; Denoncourt et al., 2017a; Paquet et al., 2013). Lipid analysis in particular showed that MLB lipids were mostly of amoeboid origin,
suggesting that although bacteria are necessary for MLB production, the process could be controlled by amoebae.
MLB production has been observed in a few protozoa so far, including free-living amoeba such as Acanthamoeba and ciliates including Tetrahymena (Berk et al., 2008; Chekabab et al., 2012). In the case of the dictyostelid group, MLBs have only been observed with strains of the D. discoideum species (Barondes et al., 1985; Cooper et al., 1986; Emslie et al., 1998; Fukuzawa and Ochiai, 1993; Gezelius, 1959; Gezelius, 1961; Hohl, 1965; Marchetti et al., 2004; Mercer and Shaffer, 1960). Considering that some of these strains were severely modified at the phenotypic level to stand axenic growth, it cannot be excluded that MLB production by dictyostelids could be a laboratory artifact without the observation of the same phenomenon with amoebae retrieved from the environment and from species other than D. discoideum. Observing MLB production with different environmental strains would strengthen current theories about their additional roles.
It was hypothesized that environmental dictyostelids can produce MLBs as well as D. discoideum laboratory strains, as the possible roles suggested for these structures would be useful to wild strains of social amoebae (Denoncourt et al., 2016; Denoncourt et al., 2017a). Intercellular communication is essential to coordinating multicellular development, and a role as food storage could be beneficial in starvation periods. This study aimed to characterize the phagocytic pathway of environmental social amoebae and compare it to that of D. discoideum DH1-10, a common laboratory strain (Cornillon et al., 2000). All environmental isolates, from various dictyostelid species, were found to produce MLBs similar to the laboratory strain.
1.4 Materials and methods
Isolation of social amoebae. Samples of soil from temperate deciduous forest ground were taken from different places in Quebec City, Canada. Soil samples were serially diluted in water (1/10, 1/100 and 1/1000). A volume of 150 µl of each dilution was mixed with a volume of 150 µl of Klebsiella aerogoenes bacteria grown overnight and plated on 10-cm diameter Petri dish containing SM medium (10 g/L bacteriological peptone, 1 g/L yeast extract, 2.2 g/L KH2PO4, 1 g/L K2HPO4, 1 g/L MgSO4, 20 g/L agar, 1% glucose), followed by incubation
at room temperature. K. aerogenes bacteria were generously gifted by Pierre Cosson (Benghezal et al., 2006). After 4 to 5 days of incubation, a part of the biomass from the border of phagocytic plaques displaying fruiting bodies was sampled using a micropipette
tip and inoculated on the center of an SM medium Petri dish containing fresh bacterial lawns. 12 isolates were obtained using this method.
Amoeba strains. 4 strains (JLJV2, E1, E2 and 3.2-2) displaying various phenotypes of fruiting bodies and speed of growth on bacterial lawn were selected from the 12 initially isolated upon confirming that they could be serially cultivated on bacterial lawns and kept frozen at -70°C.
18S identification. PCR amplification of 18S rRNA gene was conducted using primers Euk-4/18-F CTGGTTGATYCTGCCAGT-3’) (Hendriks et al., 1989) and EukR (5’-TGATCCTTCTGCAGGTTCACCTAC-3’) (Medlin et al., 1988). For JLJV2 and 3.2-2, Accustart II polymerase (Quantabio, USA) was used for PCR amplification while E1 and E2 were amplified with GoTaq polymerase (Promega, USA). Hybridization was conducted at 55 °C or 60 °C. PCR products were sequenced using Sanger technology (ABI3730xl DNA analyser). Sequences obtained were analyzed with BLAST to allow identification of the amoeba strains, along with comparison of the percent identity between isolates (Altschul et al., 1990).
Characteristics of environmental amoebae growth on bacterial lawn. For each amoeba strain, 40 cells suspended in HL5 medium were plated on a 15-cm Petri dish of SM agar along with 300 µl of a K. aerogenes suspension at an optical density at 595 nm of 2. Two plates were prepared per isolate and were incubated in the dark at room temperature. The growth rate of 10 phagocytic plaques of each isolate was measured over the course of a week at least once a day, or more as necessary, judging from the rate of growth of the plaques. Appearance of fruiting bodies was also evaluated at the end of the incubationusing a Motic SMZ-168 series stereomicroscope equipped with a Moticam Pro 252A camera (Motic, Canada). Additional pictures were taken with an iPhone 6S through the stereomicroscope eyepiece.
H36 immunofluorescence. Cells were harvested from the periphery of a phagocytic plaque, suspended in HL5 medium and cell density was adjusted to 500 000 cells/ml. 500 000 cells of each strain were placed on a sterile coverslip and left to adhere for 2 to 3 hours. Once the cells had adhered to the coverslip, H36 immunofluorescence was conducted as previously described (Paquet et al., 2013). The H36 antibody was detected with an Alexa 568-coupled anti-mouse IgG secondary antibody (Invitrogen, Burlington, Ontario, Canada). Samples were also stained with 4′,6-diamidino-2-phenylindole (DAPI) to facilitate observations and
discrimination between cells and cellular debris. Samples were observed with a Zeiss Axio Observer Z1 microscope equipped with an Axiocam camera (Carl Zeiss, Canada), in differential interference contrast (DIC) and epifluorescence microscopy at 630X magnification. H36 immunofluorescence was reproduced at least 3 times for each amoeboid isolate. Average cell size for all isolates was also determined based on the measure of 30 cells per isolates from immunofluorescence picture using Fiji (Schindelin et al., 2012; Schindelin et al., 2015). To verify H36 staining of MLBs, the cell suspension was centrifuged at 200 x g for 5 minutes, to concentrate most of the amoeboid cells in the pellet and recover mostly MLBs in the supernatant fraction. The supernatant was collected and placed on the coverslip and processed for immunofluorescence in the same way as the samples in which no amoeboid cells were removed.
Western blot of H36. Cells were harvested from the border of phagocytic plaques using micropipette tips. The biomass was resuspended in 4 ml of HL5 in a 15 ml Falcon tube and centrifuged at 200 x g for 5 minutes. The supernatant containing bacteria was discarded. Amoebae were washed an additional time using same volume of HL5 and centrifugation force to remove the remaining bacteria. The amoebae were resuspended in 4 ml of HL5 and an aliquot was used to count the amoebae with a hemocytometer. The cells were centrifuged again and resuspended to a concentration of 300 000 cells/15 µl in HL5 mixed with one-half volume of 3x TEX loading buffer (0.22 M Tris, pH 6,8, 23.5% glycerol, 9% SDS and traces of Bromophenol blue). A control lysate was also prepared at 300 000 cells/15 µl with axenic D. discoideum DH1-10 cells grown in HL5.
The solubilized proteins were separated on 12% SDS-PAGE in reducing conditions (5% (v/v) 2-mercaptoethanol added to the samples loaded on the gel). Proteins in the gels were electrotransferred to a nitrocellulose membrane and then stained with Ponceau S to confirm the quantity of proteins on the membrane. The latter was then immersed in 50 ml TBS (10 mM Tris, pH 7.4, and 150 mM NaCl) for 5 min and after that incubated with TBSM (TBS with 7% skim milk) 2 h at room temperature to block non-specific binding. The membrane was washed five times for 5 min with TBST (TBS with 0.1% Tween 20) and was incubated for 90 min at room temperature with H36 (ascite diluted 1:10,000 in TBST). The membrane was then washed three times for 5 min in TBST and was incubated for 1 h at room temperature with goat anti-mouse IgG IRDye 680RD (Li-cor, USA) according to the manufacturer’s instructions. The membrane was washed six times for 5 min in TBST. The protein bands were acquired with the Odyssey Fc Imaging System (Li-cor, USA).
Transmission electron microscopy (TEM). Samples from amoeboid cultures on SM medium supplemented with a K. aerogenes lawn were collected from the periphery of phagocytosis plaques and resuspended in 1X glutaraldehyde, then processed as previously described (Paquet et al. 2013). Observations were made using a transmission electron microscope (either JEOL 1230 with E1 and E2 or FEI Tecnai Spirit G2 with JLJV2 and 3.2-2) at 80 kV. TEM micrographs were used to measure average MLB size (n= 38).
1.5 Results
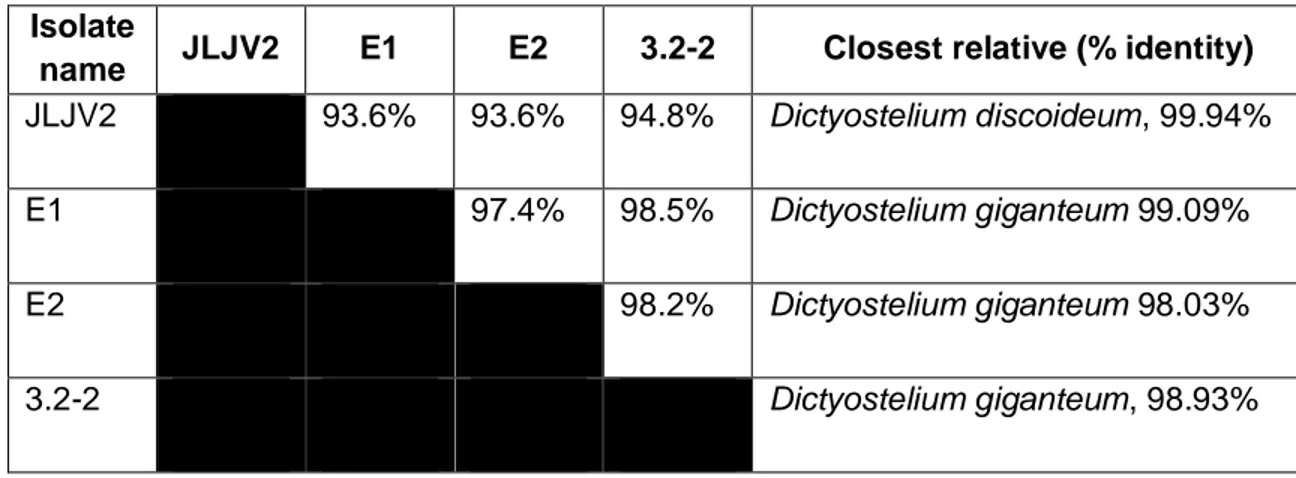
The amoeba used in this study were isolated from forest soil in Quebec City, QC, Canada. A precise 18S identification was only possible for isolate JLJV2, which was identified as Dictyostelium discoideum (Table 1.1). For the other isolates, a partial identification was attained. All strains were found to be Dictyostelium species, similar to D. giganteum, D. bruneum, D. gargantuum and D. purpureum, but closest to D. giganteum. Percent identity was high between all isolates, with JLJV2 being the most distinct from the other strains. However, it should be noted that only a 797 bp forward sequence could be obtained for isolate E2. Thus, only forward sequences were compared to obtain the percent identity between the isolates. For identification of the isolates, forward and reverse sequences were used with JLJV2, E1 and 3.2-2.
Table 1.1. Sequence identities in 18S rDNA of the isolates and closest relative. Isolate
name JLJV2 E1 E2 3.2-2 Closest relative (% identity)
JLJV2 93.6% 93.6% 94.8% Dictyostelium discoideum, 99.94%
E1 97.4% 98.5% Dictyostelium giganteum 99.09%
E2 98.2% Dictyostelium giganteum 98.03%
3.2-2 Dictyostelium giganteum, 98.93%
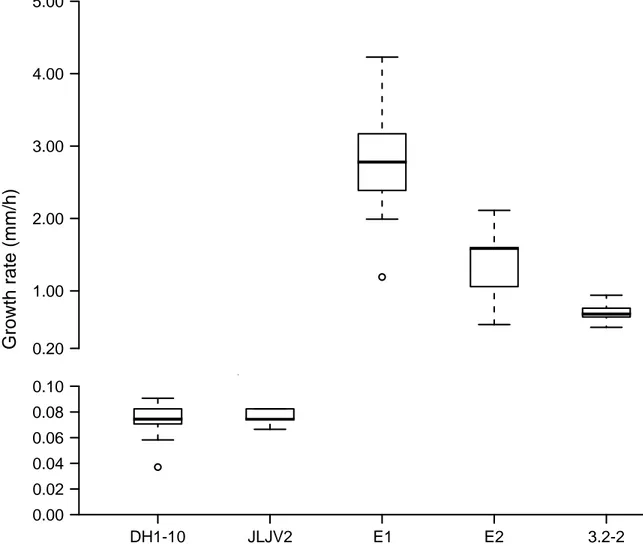
Strains E1, E2 and 3.2-2 had a much higher growth rate than D. discoideum DH1-10, used in this study for comparison, as measured by phagocytic plaque growth rate on K. aerogenes. JLJV2 grew at a similar rate than DH1-10 (Figure 1.1). Growth rates were statistically different between all strains except DH1-10 and JLJV2, as shown by a p-value under 0.05 following analysis with the Mann-Whitney U test or Student’s t-test.
Figure 1.1. Growth of the amoeboid isolates on K. aerogenes. The graph presents the growth rate of phagocytic plaques (mm/h) for the various isolates and DH1-10 on
K. aerogenes. The mean for the growth of 20 phagocytic plaques per condition is shown with
the standard deviation. Asterisks (*) indicate p < 0.05 between two values.
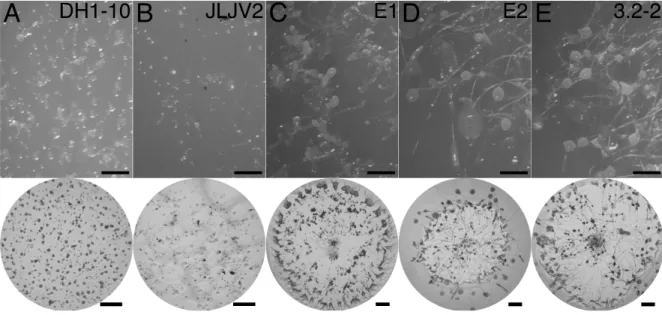
Typical multicellular development of the amoeboid isolates is presented in Figure 1.2. Strains E2 and 3.2-2 produced similar abundant fruiting bodies. These structures were quite large and tall compared to those produced by DH1-10. The strain JLJV2 produced short fruiting bodies, similar to the ones of DH1-10 but with a lower abundance. E1 had the most distinctive multicellular development, as this strain was defective for the culmination step of fruiting body development (Figure 1.2C). Only slugs, mounds and occasional finger formations could be observed for this isolate.
0.00 0.02 0.04 0.06 0.08 0.10 0.20 1.00 2.00 3.00 4.00 5.00 DH1-10 JLJV2 E1 E2 3.2-2
G
ro
w
th
r
a
te
(
m
m
/h
)
Figure 1.2. Multicellular development of the amoeboid isolates. Typical images of fruiting bodies and phagocytosis plaques produced by each strain are presented. Top row shows stereomicroscope images taken with a Motic Camera (scale: 1 mm, 100x) while the bottom row shows images taken through the eyepiece of the stereomicroscope (scale: 2 mm, A and B: 100x, C to E: 64x).
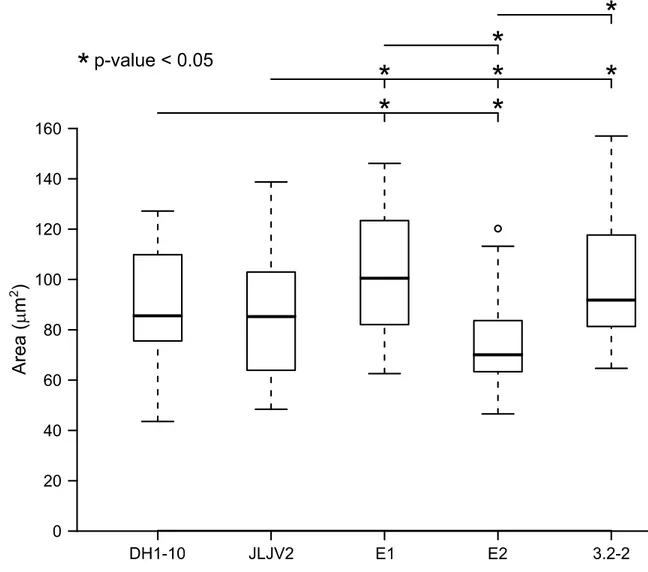
JLJV2 and DH1-10 cells were of a similar size, while E1 and 3.2-2 were larger and E2 was the strain with the smallest cells (Figure 1.3). Cell sizes were significantly different from one another, as indicated by a p-value under 0.05 following analysis of variance using either the Mann-Whitney U test or Student’s t-test, except for these pairs: 10 and JLJV2, DH1-10 and 3.2-2, and E1 and 3.2-2.
Figure 1.3. Average cell size (µm2) of the amoeboid isolates. Asterisks (*) indicate
p < 0.05 between two values.
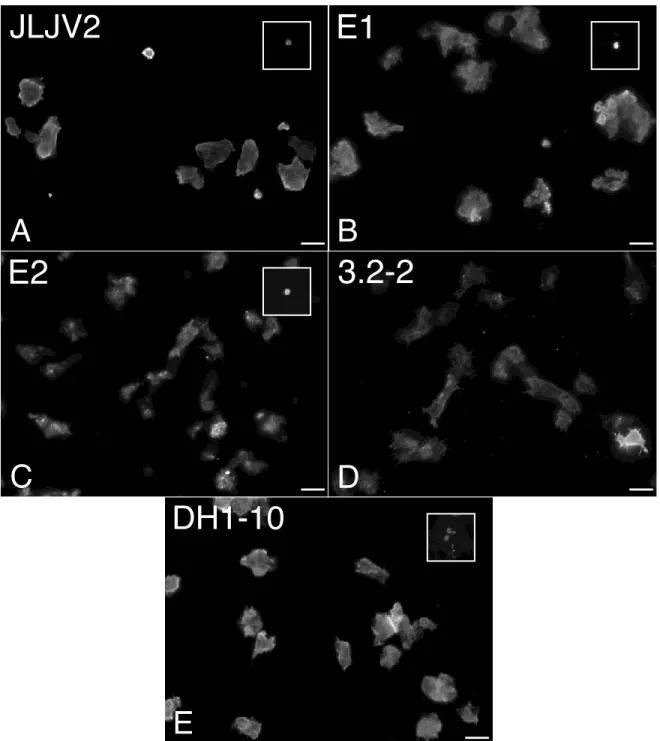
In D. discoideum grown axenically in HL5, the proteins recognized the by H36 antibody are mostly present in membranes but are excluded from phagocytic cups and phagosomes (Denoncourt et al., 2016; Mercanti et al., 2006). MLB staining has also been reported with this antibody (Denoncourt et al., 2016; Paquet et al., 2013). Figure 1.4 shows fluorescence images of H36 staining which was positive for cells of all wild isolates and DH1-10. Staining is concentrated at the plasma membrane for all strains, as is typically observed with axenic DH1-10 (Mercanti et al., 2006), with some cytoplasmic signal observed in some strains. Inserts for JLJV2, E1, E2 and DH1-10 show characteristic staining of MLBs, with staining for JLJV2, E2 and DH1-10 forming a ring around the MLB (Paquet et al., 2013). H36 staining of MLBs was not observed with isolate 3.2-2.
Figure 1.4. H36 staining is positive for all of the amoeboid isolates. JLJV2 (A), E1(B), E2 (C) and 3.2-2 (D) cells show a staining pattern similar to that observed with DH1-10 (E) upon immunofluorescent staining with the H36 antibody (red), as well as DAPI staining (blue). Scale bars equivalent to 10 µm. The H36 antibody labels several structures inside the cells, so it is not possible to be sure which intracellular elements marked by the antibody correspond to MLBs (Denoncourt et al. 2017). All pictures taken at 630X magnification. Inserts show H36 staining on MLBs produced by the associated isolate, except for 3.2-2 where no MLB could be conclusively targeted with H36 staining.
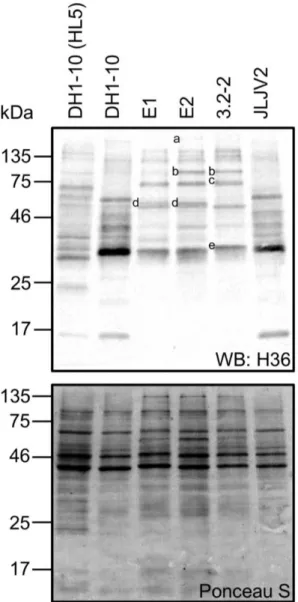
The H36 antibody recognizes N-acetylglucosamine-1-phosphate, a post-translational modification that can be found on many proteins (Denoncourt et al., 2016). A Western blot was performed to determine the profile of bands detected by H36 with the amoeboid isolates (Figure 1.5). The first interesting observation is that the band pattern obtained for DH1-10 is different between cells cultured axenically in HL5 and cells grown in the presence of bacteria. An intense band at around 30 kDa and other additional bands are visible for cells grown in the presence of bacteria compared to axenic cells. JLJV2 has a band profile equivalent to that of DH1-10. For their part, E1, E2 and 3.2-2 have a different band pattern compared to DH1-10. In addition, even if their band pattern is similar, E1, E2 and 3.2-2 show some differences with a few bands common to two out of three isolates again suggesting that they are distinct strains.
Figure 1.5. The H36 antibody displays different protein band patterns in the amoeboid isolates. Proteins from cell extracts were separated on a 12% SDS-PAGE and transferred on a nitrocellulose membrane. Before performing the western blot (WB) with the H36 antibody, the proteins on the membrane were stained with Ponceau S to confirm equivalent amount of proteins in each lane. On the image of the western blot, letters correspond to bands specific to E1, E2 and 3.2-2 individually or in pairs.
Figure 1.6 (page précédente). MLBs produced by amoeboid isolates were visible both inside the cells and in the medium. MLBs present inside (A) and outside (B) JLJV2 cells, inside (C) and outside (D) E1 cells, inside (E) and outside (F) E2 cells, and inside (G) and outside (H) 3.2-2 cells. Magnification: A, 13500X; B, 18500X; C, 2500X; D, 4000X; E, 2500X; F, 6000X; G, 4800X; H, 18500X.
Figure 1.6 presents TEM images of MLBs produced by each amoeboid isolate, both in formation inside the cells and secreted in the medium. The MLBs produced by JLJV2, E1, E2 and 3.2-2 all had a similar appearance whether inside or outside the cells. Some MLBs produced by JLJV2 and E1 contained tighter membrane layers. This morphology was observed both inside and outside of the cell. All strains also produced looser MLBs, including some that were not entirely round.Some MLBs presented “U-shaped” layers of membrane, such as in Figure 1.6F and H. MLBs may also include amorphous material as previously described (Denoncourt et al. 2016). The size of MLBs was on average 1.1 0.3 by 1.3 0.3 µm, a size close to that of a bacterium, which is in accordance with MLB sizes that have been reported previously, ranging from 0.1 to about 2 µm (Schmitz and Müller, 1991).
1.6 Discussion
The production of MLBs by all isolates studied suggests that these structures could be produced by dictyostelids in general. These results support theories proposing a supplementary role for MLBs beyond being a metabolic byproduct by demonstrating that MLB production is not an artefact resulting, among other reasons, of the axenization of the laboratory strains (Denoncourt et al., 2016; Denoncourt et al., 2017a).
Identification of the species based on 18S sequencing was not entirely conclusive, which was expected as there is a high level of homogeneity within social amoebae. This is particularly the case for the species D. giganteum and D. purpureum, which seem to exist in multi-clonal groups with different genotypes (Sathe et al., 2010). Percent identity between the isolates is also high, as expected from the identification obtained. JLJV2 is the only strain fairly identified as D. discoideum and its sequence was consequently not as similar to the other isolates. However, because the four isolates displayed very divergent phenotypes, especially for the growth rates and fruiting body characteristics as seen in Figures 1.1, 1.2 and 1.3, it is plausible that this study has analyzed the production of MLB from four distinct dictyostelid species. Strain E1 was particularly interesting due to its culmination defect. Culmination of fruiting bodies is controlled by many genes (Kessin, 2001; Loomis, 2015),
and since a full genome sequencing of E1 was not conducted, the exact reasons for this defect are unknown. It was, however, interesting to compare this likely mutant strain to culminating strains, and it should be noted that while being defective for culmination, the strain still produces MLBs similar to other strains. It should therefore be considered in future studies on the role of MLBs that if they are indeed involved in intercellular communication, they do not seem to be involved in culmination, or that at least a culmination defect does not hinder their production.
In a previous study, analysis of the lipid composition of the membranes constituting MLBs showed that they were of amoeboid origin, rather than being formed from bacterial remains, as was suggested earlier (Hohl, 1965; Paquet et al., 2013). Subsequently, some proteins associated with MLBs, such as discoidin, a lectin involved in adhesion, or SctA, a constitutively secreted protein, suggest that these bodies could play a role in intercellular communication (Barondes et al., 1985; Denoncourt et al., 2016). PhoPQ, a hypothetical protein similar to AprA, has been detected in MLBs. AprA, which regulates proliferation in Dictyostelium discoideum, can act as a chemorepellent for vegetative cells, but the effect is not observed on starved cells (Brock and Gomer, 2005; Phillips and Gomer, 2012). As starvation triggers multicellular development in social amoebae, the presence of PhoPQ in MLBs is particularly interesting for a possible role in intercellular communication. A study has also shown that naïve D. discoideum cells exposed to MLBs produced by another D. discoideum population will ingest and degrade these MLBs, which could indicate a role as food storage (Denoncourt et al., 2017a). MLBs could therefore be more than a byproduct of bacteria digestion.
These results demonstrate that other Dictyostelium species are able to produce MLBs, opening the door to new research questions. For example, it can be asked if MLBs produced by one species can be recognized by another species, have an effect on it or can be phagocytosed and digested by this second Dictyostelium species. As a first step to answer these questions, a biochemical characterization of MLBs from these isolates would be mandatory to see if they are similar to those produced by D. discoideum (Barondes et al., 1985; Denoncourt et al., 2016; Paquet et al., 2013) and, for example, see if cell signaling molecules are found in these MLBs. However, a limitation for studies of this kind is the fact that these environmental isolates are not axenic, compared to laboratory strains, and that axenic cultures can be useful for some experiments (Denoncourt et al., 2017a; Paquet et al., 2013). Axenization was attempted for these strains using HL5 medium, which is routinely
used for D. discoideum culture. It was partly conclusive for isolate JLJV2, which was the only isolate to grow in axenic medium. However, axenic JLJV2 cells appeared damaged when observed in TEM (data not shown).
Although we used environmental isolates, the production of MLBs was only observed in a laboratory setting. It was, however, shown here that H36 staining could be used to detect MLBs from JLJV2, E1 and E2. This finding could be used in further research to detect MLBs directly from environmental samples. However, since H36 is also detected in amoeboid cells, another marker could be used. This alternative marker would be especially useful as the H36 antibody detects a post-translational protein modification rather than a specific protein (Denoncourt et al., 2016), which likely explains the difference in the signal observed depending on the amoeboid strain, as shown by our results with 3.2-2. Although 3.2-2 is similar to E1 and E2 based on sequence identity, no H36 signal could be detected from MLBs. The post-translational modification recognized by H36 may not be present in 3.2-2 MLB proteins as suggested by the difference seen in the band patterns recognized by H36 in western blot for E1, E2 and 3.2-2, especially the d band specific to E1 and E2 (Figure 1.5).
Another candidate for antibody detection of MLBs would be SctA, a protein found in pycnosomes, which could be embedded into MLBs. SctA could be an interesting target for detection of dictyostelid MLBs in the environment, as the protein is constitutively secreted and is not found in significant quantities inside cells (Denoncourt et al., 2016; Sabra et al., 2016). SctA staining of MLBs produces a less intense fluorescent signal than what is observed with H36, but the specificity to MLBs could allow an easier detection. In fact, detection of SctA combined with the use of H36 to label MLBs would be an interesting approach for analysis of environmental samples. SctA staining was not attempted here as the sctA gene has not been detected in the genome of D. giganteum. However, it has been identified in other Dictyostelium species, such as D. discoideum and D. purpureum, as determined using BLAST (Altschul et al., 1990). Additional antibodies could be developed to facilitate detection of MLBs from complex samples. Identification of MLBs directly in environmental samples would be crucial to support theories of a supplementary role for MLBs.