© Nicolas Malenfant, 2021
L’endopeptidase Yps1 participe à la dégradation des
protéines GPI sans ancrage dans le contexte
métabolique d’opi3
Mémoire
Nicolas Malenfant
Maîtrise en biochimie - avec mémoire
Maître ès sciences (M. Sc.)
L’endopeptidase Yps1 participe à la dégradation des
protéines GPI sans ancrage dans le contexte
métabolique d’opi3Δ
Mémoire
Nicolas Malenfant
Sous la direction de :
Yves Bourbonnais, directeur de recherche
Christian Landry, codirecteur de recherche
iii
Résumé
Les protéines à ancrage glycosylphosphatidylinositol (GPI-APs) s’associent à des microdomaines membranaires riches en stérols et sphingolipides appelés radeaux lipidiques. Afin de pouvoir s’y associer correctement, la protéine doit s’attacher à l’ancrage GPI via une liaison éthanolamine. Cette liaison est possible grâce au complexe transamidase qui orchestre le clivage de la séquence en C-terminal de la protéine. Certains défauts génétiques qui ont été préalablement identifiés dans la biosynthèse des ancrages GPI favorisent une perte abondante de GPI-APs sans ancrage. Ceci peut être le résultat d’une protéolyse catalysée par le complexe transamidase ou principalement par leur dégradation par le protéasome. Il semblerait qu’un autre mécanisme non-identifié soit également impliqué à de plus faibles niveaux. De récents travaux ont identifié un mutant de levure (opi3Δ) pour la perte de GPI-APs sans ancrage. Ce mutant est défectueux dans la voie de méthylation responsable de la synthèse de phosphatidylcholine. Cette perte résulte de l’interférence métabolique avec les étapes de biosynthèse du GPI qui utilise de la phosphatidyléthanolamine (PE). L’étude contenue dans ce mémoire montre que la surcharge en glycoprotéines d’opi3Δ nécessite l’endopeptidase de levure Yps1 pour libérer les cellules de ces GPI-APs sans ancrage. L’inactivation de YPS1 dans ces conditions mène à leur dégradation et réduit le taux spécifique de croissance. Dans ce modèle de défaut métabolique, nos résultats suggèrent que la surcharge en glycoprotéines, associée à un profil de O-glycosylation modifié, sont nécessaires pour mettre les sites de clivage de Yps1 en évidence. Ces conditions sont rencontrées lors d’infections virales. Étant donné que plusieurs GPI-APs chez les mammifères sont impliquées dans l’immunité, les résultats obtenus dans cette étude permettent de discuter des impacts possibles de cette interférence métabolique sur la sévérité de la maladie causée par SARS-CoV2.
iv
Table des matières
Résumé ... iii
Liste des tableaux ... vi
Liste des figures ... vii
Liste des abréviations ... ix
Remerciements ... x
Avant-propos ... xi
Introduction ... 1
Organisme modèle : Saccharomyces cerevisiae ... 1
Protéines à ancrage GPI et radeaux lipidiques ... 1
Les radeaux lipidiques ... 1
Les protéines à ancrage GPI en général... 2
Biosynthèse des protéines à ancrage GPI ... 4
Remodelage de l’ancrage GPI après son attachement à la protéine ... 5
Transport et association à la surface cellulaire ... 6
Contrôle qualité des protéines mal repliées ... 7
ERAD ... 7
UPR ... 8
La microlipophagie ... 8
Le contrôle qualité des protéines à ancrage GPI et le mécanisme RESET ... 9
Défauts dans la biosynthèse du groupement GPI ... 9
Mutations causant la sécrétion des protéines à ancrage GPI chez la levure ... 11
Criblage des mutations non-essentielles de la levure ... 11
Classification des mutants sécrétant des quantités massives de Yps1 ... 12
Le mutant opi3Δ ... 12
Rôle du gène OPI3 ... 12
L’inactivation du gène OPI3 interfère avec plusieurs étapes de synthèse du groupement GPI . 13 Problématique ... 14
Hypothèses et objectifs ... 15
Chapitre 1 ... 17
1.1 Titre de l’article inséré ... 17
1.2 Auteurs ... 17
1.3 Résumé ... 17
1.4 Abstract ... 18
1.5 Introduction ... 18
1.6 Results ... 21
Native Yps1p is required for the shedding of plasmid-encoded model GPI-APs in the opi3Δ mutant ... 21
v
Requirement for native Yps1p in the shedding of anchor-less GPI-APs in the opi3Δ mutant is
conditional ... 25
An altered O-glycosylation profile in anchor-less GPI-APs is associated with their Yps1p-dependent shedding in the opi3Δ mutant ... 29
1.7 Discussion ... 32
1.8 Methods ... 40
Strains and media ... 40
DNA manipulations and transformations ... 41
Cellular extraction, supernatant concentration and immunodetection ... 42
Growth assays ... 43 Statistical analysis ... 43 1.9 Acknowledgements ... 43 1.10 Author Contributions ... 43 1.11 Additional Information ... 44 1.12 References... 44 1.13 Supplementary information ... 52 Figure S1 ... 52 Figure S2 ... 53 Table S1 ... 54 Table S2 ... 55 Additional references ... 56 Chapitre 2 ... 59 2 Discussion générale ... 59 Conclusion ... 64 Références ... 65 Annexe ... 72 Figure A1 ... 72 Figure A2 ... 73
vi
Liste des tableaux
Chapitre 2
Table I : Yeast strains used in this study………..……….……….. p.41
Table II : Oligonucleotide primers used in this study……….………….……….. p.41
Table III : Plasmids used in this study………..………. p.41 Table SI : Predicted furin-type cleavage in Yps1DLp………. p.54
Table SII : Reported COVID-19 clinical manifestation and their association with inherited GPI
vii
Liste des figures
Introduction
Figure 1 : Association des protéines à ancrage GPI avec un microdomaine structuré nommé
radeau lipidique à la surface cellulaire ………...……… p.2
Figure 2 : Structure générale d’une protéine à ancrage GPI.……….……….………. p.3
Figure 3 : Étapes nécessaires à la synthèse d’un ancrage GPI et son attachement à une
protéine……...……….……….. p.4
Figure 4 : Différentes étapes de remodelage de l’ancrage GPI nécessaires à la sortie de la
protéine à ancrage GPI du RE……….……… p.6
Figure 5 : Schématisation du transport des protéines à ancrage GPI à partir du réticulum
endoplasmique (ER) jusqu’à sa destination finale……….………. p.7
Figure 6 : Schématisation des différentes actions possibles menant à la perte de protéine
à ancrage GPI dans le milieu extracellulaire.……….……….. p.10
Figure 7 : Schématisation de l’expérimentation liée au criblage des mutations de levures
non-essentielles.……….……….………. p.11
Figure 8 : Cette figure présente le rôle de la protéine Opi3 au sein de la voie de synthèse
de phosphatidylcholine (PC) cytosolique…….………. p.12
Figure 9 : Schématisation de la compétition pour le PE resultant en une perte de protéine
à ancrage GPI.……….………. p.14
Figure 10 : Schématisation de l’hypothèse suggérant comment la peptidase de levure Yps1
peut être impliquée dans le clivage des protéines à ancrage GPI non-conformes chez le
mutant opi3Δ……….……….……….………... p.16
Chapitre 2
Figure 1 : Fate of GPI-Aps from biosynthetic mutants involved in GPI anchor assembly…… p.19
Figure 2 : Yps1 variants and antisera used in this study………... p.22
Figure 3 : Loss of genomic YPS1 in the opi3Δ mutant abolishes secretion of model GPI-APs
and promotes their degradation………. p.24
Figure 4 : Loss of genomic YPS1 in the mcd4ts mutant does not hamper secretion of model
GPI-APs.………. p.26
Figure 5 : Yps1-catalyzed secretion of GPI-APs at the ER membrane is promoted by
environmental change caused by an overload of mannoproteins………. p.28
Figure 6 : Yps1-catalyzed release of GPI-APs upon mannoprotein overload in the opi3Δ
mutant is associated with higher heterogeneity in the O-glycosylated profile………. p.30
Figure 7 : Graphical summary (a) and possible implications of the model for thrombosis (b)
and viral propagation (c) upon SARS-CoV2 infection………. p.34
Figure S1 : Shedding of the Yps1p variants in the opi3Δ mutant causes the release of a >
32 kDa degradation product……….………... p.52
Figure S2 : Mean growth curves and specific growth rates of (a) WT (BY4741) and (b) the
viii
Annexe
Figure A1 : Résultats montrant que la sécrétion des protéines GPI dans le mutant opi3Δ
résulte du défaut d’attachement entre la protéine et son ancrage GPI……… p.72
Figure A2 : L’inactivation de certains gènes du sentier de la synthèse du
ix
Liste des abréviations
GPI: Gycosylphosphatidylinositol
GPI-APs : Protéines à ancrage GPI; GPI-anchored proteins
RE : Réticulum endoplasmique; ER endoplamic reticulum
EtN : Phosphoéthanolamine
COPII : Protéine de recouvrement du complexe II; Coat protein complex II
ERES : Sites de sortie associés au RE; ER exit sites
AD : Maladie d’Alzheimer; Alzheimer’s disease
PD : Maladie de Parkinson; Parkinson’s disease
ERAD : sentier de dégradation protéique associé au RE;
Endoplasmic-reticulum-associated protein degradation
UPS : système d’ubiquitination du protéasome; ubiquitin-proteasome system
UPR : réponse face à des protéines mal repliées; unfolded protein response
LD : Gouttelettes lipidiques; Lipid Droplets
ESCRT : complexes endosomal de triage protéique requis pour le transport ; endosomal
sorting protein complexes required for transport
RESET : export rapide du RE induit par un stress; rapid ER stress-induced export
PC : Phosphatidylcholine
PE : Phosphatidyléthanolamine
MMPE : Monométhylphosphatidyléthanolamine
IGD : Inherited GPI deficiencies
HPMR : Hyperphosphatasia with mental retardation GPI-T : GPI transamidase
Man : Mannose
MSR : Membrane stress response
WT : Wild type
N-ent loop : N-terminal entrance loop
S/T : Serine/Threonine
EtN-P : Ethanolamine phosphate Dol-P-Man : Dolichol-phosphate mannose T2D : Diabète de type II; type II diabetes
SARS : Syndrome respiratoire aigu sévère; severe acute respiratory syndrome
PNH : Paroxysmal nocturnal hemoglobinuria MAC : Membrane attack complex
ARDS : Acute respiratory distress syndrome
mGPD : Mitochondrial glycerol-phosphate dehydrogenase AGEs : Advanced glycation end-products
x
Remerciements
Je veux tout d’abord remercier mon directeur de recherche Dr Yves Bourbonnais, qui m’a permis de réaliser ce projet de grande envergure au sein de son laboratoire. Ses conseils judicieux et son écoute m’ont permis de réaliser cette aventure avec plus de facilité et d’outils qui me serviront dans mes futurs projets. J’aimerais également remercier mon codirecteur de recherche, Dr Christian Landry, pour ses précieux conseils ainsi que pour le temps consacré à mon travail lors des rencontres de laboratoire. Par le fait même, je dois également remercier les membres de mon comité d’encadrement, Dre. Marie Filteau ainsi que Dr Simon Hardy pour toute l’attention portée à mon égard.
J’ai aussi eu la chance de travailler avec d’excellents collègues lors de ce passage aux études graduées. Ils ont su répondre à de nombreux questionnements grâce à leur expérience. Alexandre Dubé et Isabelle Gagnon-Arsenault, je vous remercie. De plus, j’ai pu partager mes expériences de laboratoire avec différents stagiaires : Florence Ladonne, maintenant aux études graduées, Frédéric Bégin ainsi que Nicolas Méthais. Merci pour votre aide et pour les discussions que nous avons partagées.
J’ai réussi à passer au travers cette épreuve avec le soutien de mes amis et ma famille. À mes parents, merci pour toute l’écoute lors de ce parcours. Finalement, j’aimerais remercier ma conjointe Amélie, pour la patience qu’elle a eue dans les moins bons moments et de m’avoir aidé à persévérer dans cette aventure, je t’aime.
xi
Avant-propos
Lors de mon passage à la maîtrise, Dr Bourbonnais m’a permis de participer à la conception d’un projet qui a mené à la rédaction d’un article scientifique. Avec l’aide de précédents travaux de notre laboratoire, j’ai pu participer à la conception du projet, à la réalisation de la majorité des travaux ainsi qu’à la rédaction du brouillon complet pour cet article.Mon directeur de maîtrise ainsi qu’une autre étudiante à la maîtrise dans le laboratoire Bourbonnais, Florence Ladonne, sont également coauteurs. La contribution de chacun est expliquée au chapitre 1 à la section 1.10.
L’article que j’intègre ici est intitulé « Glycosylation status, a molecular rheostat regulating
protease-dependent shedding of glycosylphosphatidylinositol-anchored » pour lequel je suis premier auteur. Il
sera soumis dans le journal scientifique « Scientific reports ». Comme la date de soumission demeure inconnue pour le moment, j’ai mis l’intégralité du manuscrit dans ce mémoire, incluant le matériel supplémentaire.
1
Introduction
Organisme modèle : Saccharomyces cerevisiae
Saccharomyces cerevisiae est une espèce de levure principalement connu pour son utilisation
alimentaire dans la fabrication du pain, du vin et de la bière depuis déjà plusieurs millénaires. Avec le développement de la génétique, cette levure fut bien caractérisée. En effet, son génome entier est maintenant séquencé (Goffeau et al. 1996) et est donc un modèle de choix pour les chercheurs dans les domaines de la génétique classique et moléculaire ainsi que pour la biologie des systèmes. S.
cerevisiae nous permet de faire des transformations par l’ADN (ajout ou délétion de gènes) avec une
grande efficacité et facilité, lui conférant ainsi un grand avantage lors du processus du choix de l’organisme modèle. De plus, les cellules de levures sont petites, facile à conserver et cultiver en plus de posséder une vitesse de division cellulaire plutôt rapide (environ 1.5 heures) nous permettant de réaliser plusieurs expériences variées dans un court délai. Sur les 6275 gènes de cet organisme, il est estimé que 31% d’entre eux ont des orthologues dans le génome humain, ce qui lui confère un autre avantage (Botstein, Chervitz, and Cherry 1997). De ce fait, lors de l’étude de pathologies, l’utilisation de la levure comme modèle peut permettre de caractériser et comprendre le mécanisme de développement de certaines maladies. En raison de tous ces avantages, l’organisme modèle
Saccharomyces cerevisiae est celui choisi pour les études effectuées dans le laboratoire Bourbonnais.
Protéines à ancrage GPI et radeaux lipidiques
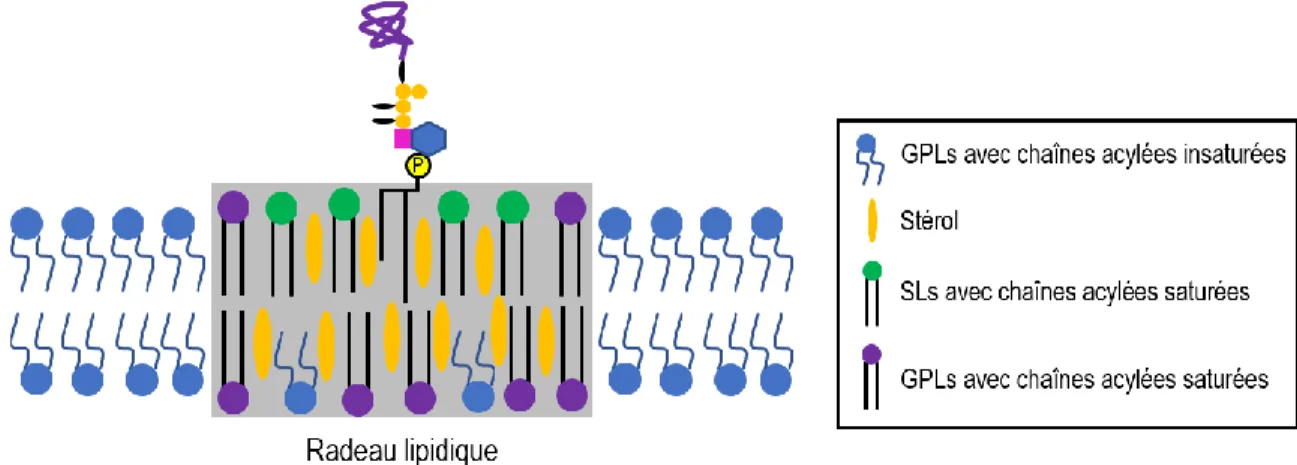
Les radeaux lipidiquesLes protéines à ancrage glycosylphosphatidylinositol (GPI-APs) sont associées à des microdomaines structurés appelés radeaux lipidiques (Sharom and Lehto 2002; Waheed and Freed 2010). Ces structures, schématisées à la figure 1, sont riches en stérols, sphingolipides ainsi qu’en glycérophospholipides. Chacune de ces composantes ont leur importance. En effet, les stérols s’associent principalement avec les chaînes acylées saturés hydrophobes des sphingolipides et des glycérolipides (Korade and Kenworthy 2008). C’est d’ailleurs cette association qui permet d’avoir un structure ordonnée (Pike 2009). Bien que leurs fonctions ne soient pas toutes découvertes, ces domaines membranaires sont impliqués dans certaines fonctions cellulaires clés. En effet, ils permettent de réguler l’association de certaines protéines avec leur substrat (Levental et al. 2010). Ils sont également connus pour jouer un rôle dans la transduction du signal à la membrane plasmique. Ils
2
participent activement dans les processus de signalisation de l’immunoglobuline E (Field, Holowka, and Baird 1995), du récepteur antigénique des cellules T (Zhang, Trible, and Samelson 1998) et B (Gupta and DeFranco 2007) et du récepteur de croissance épidermal chez les mammifères (Kovács et al. 2017). Leur importance chez la levure est aussi grande. En effet, ces microdomaines semblent critiques pour le bon fonctionnement protéique des activités essentielles à la croissance, la survie et la mort de la cellule (Mollinedo 2012). Ainsi, bien que l’on retrouve la plupart des GPI-APs ultimement liées à la paroi cellulaire chez la levure, leur présence dans ces microdomaines membranaires semble constituer une étape obligatoire.
Figure 1 : Association des protéines à ancrage GPI avec un microdomaine structuré nommé radeau lipidique à la surface cellulaire. Figure inspirée de Waheed and Freed (Waheed and Freed 2010)
Les protéines à ancrage GPI en général
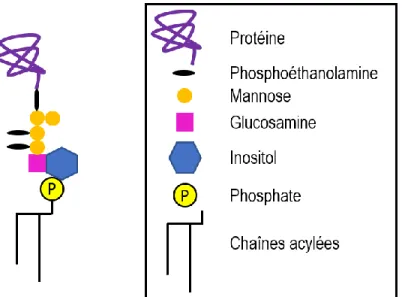
Les protéines à ancrage GPI sont présentes chez tous les eucaryotes (Nosjean, Briolay, and Roux 1997). En effet, elles sont obtenues à la suite d’une modification post-traductionnelle qui ajoute le groupement GPI à l’extrémité C-terminale de la protéine (Pittet and Conzelmann 2007). Chez la levure, elles peuvent être associées soit au feuillet externe de la membrane plasmique ou bien à la paroi cellulaire (Caro et al. 1997). La structure générale du groupement GPI est complexe et est présentée à la figure 2. Il s’agit d’un glycolipide composé d’un phosphatidylinositol et de résidus glucidiques, soit d’une molécule de glucosamine et de mannoses. Bien que le cœur de l’ancrage GPI demeure commun, il y a des différences dans la composition finale des ancrages GPI entre les espèces et les protéines GPI, ce qui pourrait conférer certaines propriétés particulières aux GPI-APs (Ferguson, Hart, and Kinoshita 2017). Chez la levure, les fonctions connues de la plupart de ces protéines sont liées à l’assemblage ou encore au remodelage de la paroi cellulaire (Pittet and Conzelmann 2007).
3
Figure 2 : Structure générale d’une protéine à ancrage GPI. Figure inspirée de Kinoshita (Kinoshita and Fujita 2016).
Noter que le schéma n’est pas à l’échelle, la protéine occupant dans l’espace une place considérablement plus grande que le groupement GPI. Ce ratio sera d’ailleurs utilisé dans toutes les figures subséquentes.
Bien qu’il y ait à ce jour peu de protéines GPI identifiées (~60 chez les levures et ~150 chez l’Homme), il y a pratiquement autant de gènes qui sont responsables de leur régulation. De ceux-ci, environ 26 gènes, la plupart d’entre eux étant essentiels, sont connus pour leur implication dans la synthèse et le remodelage de l’ancrage GPI (Kinoshita and Fujita 2016). Chez les mammifères, plusieurs de ces protéines sont requises dans de nombreux processus biologiques (Paulick and Bertozzi 2008), comme le développement (McKean and Niswander 2012), la neurogénèse (Park et al. 2013) et l’immunité (Takeda et al. 1993). Un défaut dans la synthèse des groupements GPI entraîne d’importantes pathologies chez les humains comme l’hémoglobinurie paroxystique nocturne (Krawitz et al. 2013), une anémie rare, ainsi que l’hyperphosphatasie alcaline avec un retard mental, une maladie génétique (Chiyonobu et al. 2014). Chez la levure, aucune protéine GPI n’est essentielle. La synthèse et le remodelage de l’ancrage GPI le sont cependant, suggérant ainsi un rôle collectif qui est critique. Le maintien de l’intégrité des radeaux lipidiques en fait partie puisque les GPI-APs sont invariablement associées à ces structures (Sharom and Lehto 2002). L’association se faisant avec le groupement GPI, un défaut de synthèse de ces groupements pourrait donc avoir un impact majeur sur les radeaux lipidiques. Or, plusieurs protéines transmembranaires fonctionnent normalement dans ces microdomaines membranaires.
4
Biosynthèse des protéines à ancrage GPI
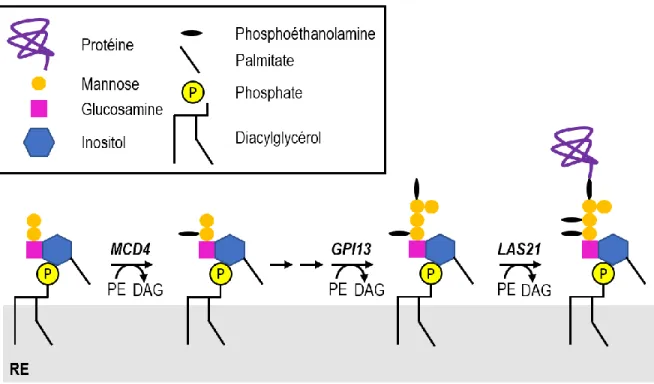
La biosynthèse des protéines à ancrage GPI a lieu en trois étapes. En premier lieu, le groupement GPI est assemblé dans la membrane du réticulum endoplasmique (RE). Les différentes parties du corps de l’ancrage, tel que décrit à la section précédente, sont assemblées (Ferguson, Hart, and Kinoshita 2017). En effet, la chaîne acylée sur l’inositol (palmitate), la glucosamine et les mannoses sont ajoutés à un phosphatidylinositol. Il y a par la suite en premier lieu l’addition de phosphoéthanolamine (EtN) sur les mannoses. Ces ajouts sont essentiels pour l’attachement de la protéine à l’ancrage GPI. Les enzymes responsables de catalyser ces trois étapes sont Mcd4, Gpi13 et Las21, respectivement (Gaynor et al. 1999; Flury, Benachour, and Conzelmann 2000; Benachour et al. 1999). Comme vous le constaterez à la figure 3, toutes les enzymes et les étapes doivent se synchroniser afin de compléter la synthèse du groupement GPI.
Figure 3 : Étapes nécessaires à la synthèse d’un ancrage GPI et son attachement à une protéine. Figure inspirée de
Kinoshita (Kinoshita and Fujita 2016)
En second lieu, l’ancrage GPI s’attache aux protéines nouvellement synthétisées à un acide aminé précis appelé le site ω (Thomas, Dwek, and Rademacher 1990). Cette étape a lieu dans la lumière du RE et doit se faire en collaboration avec le clivage du peptide signal d’addition de groupement GPI en C-terminal de la protéine. En effet, les précurseurs de protéines GPI comportent des séquences
5
signales à chacune de leurs extrémités. En N-terminale, elles permettent l’entrée dans le RE tandis que les résidus hydrophobes en C-terminalservent à réguler l’attachement avec la protéine(Thomas, Dwek, and Rademacher 1990). Lorsque ces étapes sont complétées, l’ancrage GPI attachera la protéine via le groupement EtN ajouté par Gpi13 à l’aide du complexe transamidase GPI. Ce dernier clivera la séquence en C-terminal de la protéine et l’attachera par la suite au groupement GPI. En troisième lieu, le groupement GPI est remodelé afin de permettre à la protéine à ancrage GPI de quitter le RE et de pouvoir se rendre dans l’appareil de Golgi. À cet endroit, les protéines subissent d’autres modifications post-traductionnelles (Pittet and Conzelmann 2007). Finalement, elles seront transportées à la membrane plasmique ou à la paroi cellulaire où elles pourront être fonctionnelles (Kinoshita and Fujita 2016). Le remodelage de l’ancrage GPI et le transport des protéines seront abordés plus en détails dans les sections suivantes.
Remodelage de l’ancrage GPI après son attachement à la protéine
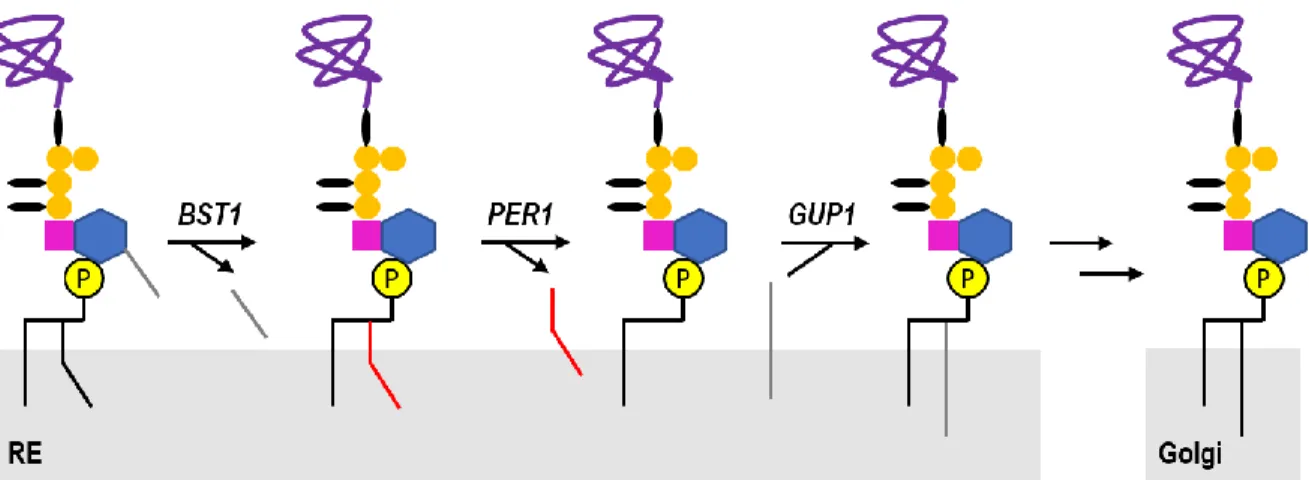
Après l’attachement de la protéine à l’ancrage GPI, ce dernier est remodelé, c’est-à-dire que ses constituants sont modifiés afin de rendre l’ancrage fonctionnel. Bien que ces modifications ne soient pas essentielles à la viabilité cellulaire, elles le sont toutefois pour assurer le transport des protéines à ancrage GPI du RE à la surface cellulaire. La modification commune est catalysée par l’enzyme Bst1 qui enlève un groupement acyl (palmitate) de l’inositol, étape critique impliquée dans le contrôle qualité pour la sortie de la protéine du RE. Plusieurs autres modifications peuvent prendre place selon la protéine, telle que la nature de l’ancrage (diacylglycérol ou céramide) avec des chaînes acylées de longueur et insaturation variées (Kinoshita and Fujita 2016). Ceci explique la très grande diversité qui existe chez ce type de protéine. Ces modifications ne touchent cependant pas le cœur de l’ancrage qui lui, reste commun entre les protéines à ancrage GPI des diverses espèces. Plusieurs des gènes impliqués dans le remodelage sont bien connus, tel que présenté à la figure 4. Comme on le voit, après l’étape de Bst1, la protéine Per1 est responsable d’enlever le groupement acyl en position sn-2 du phosphatidylinositol, le transformant ainsi en lyso-phosphatidylinositol (Fujita et al. 2006). Finalement, Gup1 fera l’addition en sn-2 d’une chaîne acylé de 26 carbones afin de compléter le remodelage (Bosson, Jaquenoud, and Conzelmann 2006). Dans le cas des ancrage GPI contenant un céramide, d’autres étapes peuvent être nécessaires (Sipos et al. 1997).
6
Figure 4 : Différentes étapes de remodelage de l’ancrage GPI nécessaires à la sortie de la protéine à ancrage GPI du RE. Figure inspirée de Kinoshita (Kinoshita and Fujita 2016)
Transport et association à la surface cellulaire
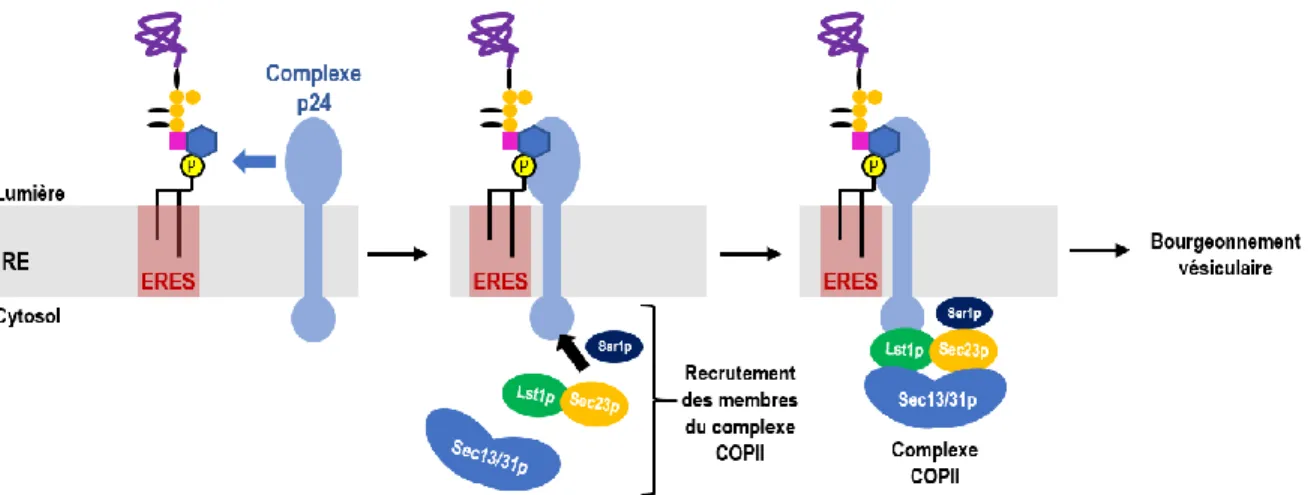
Tel que mentionné plus tôt, une fois que les protéines à ancrage GPI sont matures, elles quittent le RE vers l’appareil de Golgi où d’autres modifications post-traductionnelles auront lieu, après quoi elles seront acheminées vers la surface cellulaire. Chez la levure, il a été démontré que les GPI-APs sont transportées dans leur propre vésicule indépendamment des autres protéines qui sont elles aussi destinées à la voie de sécrétion (Mayor and Riezman 2004). Outre une bonne maturation des protéines, d’autres critères semblent liés à leur transport. Chez la levure, il a été montré que la sortie des GPI-APs du RE requiert leur association avec les sphingolipides et/ou céramides établissant ainsi la formation des microdomaines membranaires très tôt dans la voie de sécrétion (Watanabe et al. 2002). Tout comme leur biosynthèse, le transport de ces protéines est hautement régulé. Les GPI-APs correctement repliées et assemblées seront transportées du RE à l’appareil de Golgi via des vésicules de transport ayant une membrane recouverte de protéines. Ces vésicules sont formées à partir de la polymérisation de la protéine de recouvrement cytosolique du complexe II (COPII; Coat protein
complex II) qui se lient au groupement GPI correctement remodelé par l’intermédiaire d’un adaptateur
composé de plusieurs protéines appelé complexe p24 (figure 5) (Muñiz and Riezman 2016). Ceci a lieu à des domaines spécifiques du RE nommés « sites de sortie associés au RE » (ERESs; ER exit
sites). Afin d’avoir un transport efficace via le sentier de sécrétion, les protéines se concentreront aux
ERESs avant d’être capturées de façon directe ou indirecte dans les vésicules COPII (Doering and Schekman 1996; Castillon et al. 2011).
7
Figure 5 : Schématisation du transport des protéines à ancrage GPI à partir du réticulum endoplasmique (ER) jusqu’à sa destination finale. Figure inspirée de Muniz et al. (Muñiz and Riezman 2016).
Contrôle qualité des protéines mal repliées
Chaque cellule doit s’assurer de faire la synthèse de macromolécules conformes. Le contrôle qualité des protéines est donc un élément clé pour leur survie. Si une altération se produit et mène à l’accumulation de protéines mal repliées, il y aura un défaut de fonction dans la cellule. Un tel phénomène se doit d’être pris en charge rapidement afin d’éliminer les protéines défectueuses de la cellule. Les irrégularités dans la réponse peuvent mener à différentes maladies chez l’homme tel des cancers (Dai et al. 2007), la fibrose kystique (Koulov et al. 2010), des maladies lysosomales (Sawkar, D'Haeze, and Kelly 2006) ainsi que des troubles neurodégénératifs comme la maladie d’Alzheimer (AD) (Caughey and Lansbury 2003), de Parkinson (PD) (Cohen and Kelly 2003) et de Huntington (Morimoto 2008). Au fil des ans, différents processus de réponse à l’accumulation de protéines mal repliées ont été découverts et les principaux seront décrits dans les prochaines sections.
ERAD
Le sentier de dégradation « Endoplasmic-reticulum-associated protein degradation » (ERAD) cible les protéines mal repliées du réticulum endoplasmique en faisant l’ubiquitination de ces dernières (Lemus and Goder 2014; Berner, Reutter, and Wolf 2018). Cette modification post-traductionnelle fixe des protéines d’ubiquitine de façon covalente sur les lysines, cystéines ou sérines/thréonines du substrat. Ce processus se produit lorsque le sentier repère certaines structures spécifiques aux protéines non conformes, tel des glycanes immatures, des régions hydrophobes exposées ou encore des résidus cystéines non-associés (Shimizu, Okuda-Shimizu, and Hendershot 2010). Ces protéines sont transportées du réticulum endoplasmique au cytosol pour se faire ubiquitiner par le «
ubiquitin-8
proteasome system » (UPS). Les protéines marquées sont ensuite dégradées par le protéasome 26S,
un complexe de dégradation cellulaire (Biederer, Volkwein, and Sommer 1996). La perte de fonctions de certaines composantes du sentier ERAD est associée au développement de certaines maladies comme par exemple la maladie de Parkinson et la fibrose kystique (Sato et al. 2009).
UPR
Lorsque la dégradation via ERAD n’est pas suffisante pour réduire le stress engendré par l’accumulation des protéines non-repliées ou mal-repliées dans la lumière du RE, une seconde réponse cellulaire, appelé « unfolded protein response » (UPR), s’active augmentant la transcription de plusieurs gènes (Hetz and Papa 2018). L’UPR est d’ailleurs conservée chez les mammifères et les levures (Kimata and Kohno 2011). Ce processus remplit principalement trois rôles. Il doit restaurer le fonctionnement cellulaire en stoppant la traduction de protéines, dégrader les protéines mal-repliées et augmenter la production de chaperonnes impliquées dans le repliement de protéines (Wu, Ng, and Thibault 2014). Si toutefois ces fonctions ne sont pas atteintes dans le temps requis, l’UPR signalera à la cellule d’entrer en apoptose (Shore, Papa, and Oakes 2011). Une suractivation de l’UPR est associée à la maladie à prion ainsi qu’à certaines maladies neurodégénératives comme la Chorée de Huntington, la maladie d’Alzheimer, de Parkinson et de Creutzfeldt-Jakob (Hetz and Saxena 2017). Des pistes de traitements ciblent d’ailleurs l’inhibition de l’UPR (Moreno et al. 2013).
La microlipophagie
Bien que le prochain type de contrôle qualité décrit dans cette section ne soit pas directement lié aux protéines mal-repliées, ce mécanisme est impliqué en support à l’UPR lors de conditions précises. En effet, les cellules ayant des niveaux de phospholipides altérés créent des gouttelettes lipidiques (LD;
lipid droplets) pour tenter de répondre à ce stress (Fei et al. 2011). Ces gouttelettes sont produites à
la membrane du RE. Comme ce stress déclenche également l’UPR, un groupe de recherche a montré que les LD contiennent également des protéines ubiquitinées de même que des chaperonnes du RE (Vevea et al. 2015; Wu, Ng, and Thibault 2014). Ces LD sont ensuite dégradées par la vacuole par un processus ressemblant à la microautophagie. Ce processus est appelé la microlipophagie et est requis pour diminuer le stress cellulaire et ramener ou conserver les niveaux de phospholipides à ceux du type sauvage lorsqu’il n’y a pas de stress. Contrairement à l’autophagie induite par l’absence de source d’azote, la microlipophagie ne requiert pas ATG7, médiateur de l’assemblage de l’autophagosome, mais plutôt le complexe ESCRT (endosomal sorting protein complexes required for transport) ainsi que
9
les protéines vacuolaires de classe E localisées au RE. Ce mécanisme montre le lien entre la synthèse de LD et la microlipophagie pour la réponse adaptative du défaut lipidique et des protéines endommagées au RE.
Le contrôle qualité des protéines à ancrage GPI et le mécanisme RESET
Les protéines à ancrage GPI sont soumises au même contrôle qualité que décrit précédemment. Toutefois, le signal de sortie du RE contenu dans le groupement GPI, après remodelage de celui-ci, compétitionne avec la rétro-translocation dans le cytosol des protéines GPI mal-repliées pour leur dégradation par le système ERAD (Sikorska et al. 2016). Ainsi, une très petite proportion des GPI-APs mal repliées semblent emprunter la voie ERAD, du moins chez la levure. Même mal-repliées, les GPI-APs quittent le RE pour être acheminées à la surface cellulaire. Leur séjour à cet endroit est cependant transitoire puisque ces protéines mal-repliées sont en effet endocytosées pour être dégradées par le lysosome selon un mécanisme appelé RESET (export rapide du RE induit par un stress; rapid ER
stress-induced export) (Satpute-Krishnan et al. 2014). En empêchant la sortie des protéines GPI
mal-repliées du RE, suite à la délétion d’un gène codant pour une sous-unité du complexe p24 (tmp21Δ), il y a agrégation de la protéine GPI dans le RE. Dans la maladie d’Alzheimer, plusieurs évidences pointent vers une mauvaise fonction de la protéine Tmp21 (Qiu et al. 2019).
Défauts dans la biosynthèse du groupement GPI
Au contraire des protéines transmembranaires où les mécanismes de contrôle de qualité reposent essentiellement sur la structure correcte de la chaîne polypeptidique, d'autres mécanismes doivent être mis en place pour les protéines GPI dans les cas où il y a perturbation de la synthèse, du remodelage ou de l'attachement du groupement GPI à la protéine. Dans ce cas, la protéine peut en principe être correctement repliée mais son association aux radeaux lipidiques est perdue.
Bien que la plupart des gènes impliqués dans la biosynthèse du groupement GPI soient essentiels pour la viabilité (Kawagoe et al. 1996; Leidich, Drapp, and Orlean 1994), des mutants dits hypomorphiques (c. à d. l’expression et/ou l’activité de la protéine est réduite mais non complètement abolie) ont été identifiés pour presque toutes les étapes de la biosynthèse ou du remodelage des groupements GPI (Kinoshita 2020). Chez l’Homme, l'un des symptômes de ces mutations est un retard développemental (Pagnamenta et al. 2017). Plusieurs des mutants, mais pas tous, sont également associés à une hyperphostatasie qui résulte de la sécrétion de la phosphatase alcaline non-spécifique
10
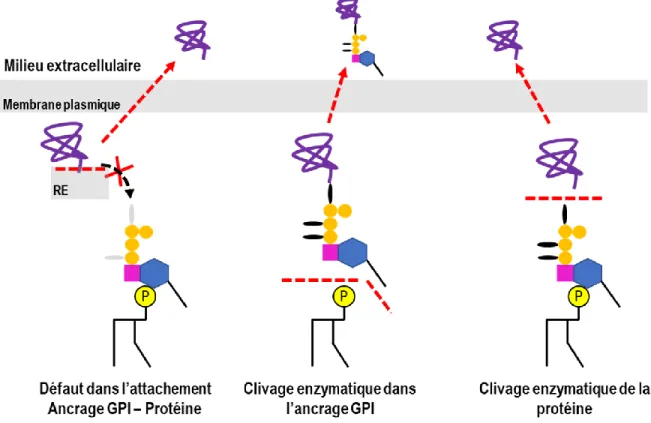
au tissu, une protéine GPI (Murakami et al. 2012). Le relâchement de formes solubles de la phosphatase alcaline peut être le résultat de divers processus comme le montre la figure 6. Tout d’abord, les étapes de la biosynthèse du GPI empêchant le transfert du groupement sur la protéine peuvent conduire soit à sa dégradation par la voie ERAD (Ashok and Hegde 2008; Sikorska et al. 2016) ou à sa sécrétion dans le milieu extracellulaire (figure 6, gauche). Dans ce dernier cas, puisque le peptide signal en C-terminal spécifiant l'attachement du groupement GPI est hydrophobe et est inséré dans la membrane du RE, la sécrétion de la protéine implique une activité protéolytique. Si par contre les étapes de la voie de biosynthèse interfèrent avec le remodelage des chaînes acylées du groupement GPI, affectant ainsi l'association de la protéine avec les radeaux lipidiques, deux mécanismes différents peuvent conduire à la sécrétion de la protéine. Soit le groupement GPI non mature est hydrolysé par une activité de type phospholipase (figure 6, centre) ou alors une peptidase opère le clivage de la protéine la libérant ainsi de son ancrage GPI (figure 6, droite).
Figure 6 : Schématisation des hypothèses des différentes actions possibles menant à la perte de protéine à ancrage GPI dans le milieu extracellulaire.
Les défauts génétiques dans la biosynthèse du groupement GPI n'expliquent qu'environ 0,15% des cas présentant un retard développemental (Pagnamenta et al. 2017), et plusieurs précurseurs essentiels à la biosynthèse du groupement GPI sont partagés dans les différentes voies métaboliques
11
de la cellule. Notre équipe a donc émis l'hypothèse que des mutations ou perturbations du métabolisme, non spécifiques à la voie de synthèse du GPI, pourraient conduire soit à une mauvaise association des protéines GPI aux radeaux lipidiques ou encore à une interférence avec le transfert du groupement GPI sur les protéines. Dans les deux cas, ceci serait associé à une sécrétion accrue de ce type de protéines à l'extérieur de la cellule. Dans la section suivante, les travaux ayant conduit à l'élaboration de mon projet de maîtrise, et auxquels j'ai participé, seront brièvement décrits.
Mutations causant la sécrétion des protéines à ancrage GPI chez la levure
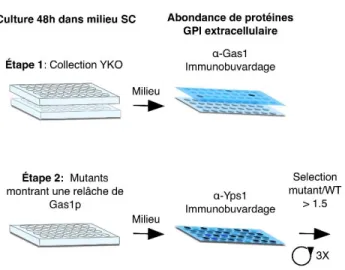
Criblage des mutations non-essentielles de la levureAfin d’identifier les gènes dont la fonction est nécessaire pour assurer la stabilité des protéines GPI dans les membranes, notre laboratoire a réalisé une expérience de criblage. En utilisant une banque de mutations non-essentielles de levure, c’est-à-dire des mutations viables pour notre modèle dans les conditions standard de croissance, nous avons mesurer la perte des GPI-APs dans le milieu extracellulaire pour cibler le gènes nécessaires à cette stabilité membranaire (figure 7) (Mémoire de maîtrise de Alexandre Dubé, 2011). À la suite de l’analyse des résultats, 30 mutants ont été retenus. Les principaux phénotypes qu’ils présentent sont un stress du RE, de paroi cellulaire et/ou un défaut vacuolaire/lysosomal.
Figure 7 : Schématisation de l’expérimentation liée au criblage des mutations de levures non-essentielles. Le type
de criblage contenu dans cette figure se nomme immunobuvardage par point. Lors de cette analyse, le surnageant de la culture en multi-plaque est passé à travers une membrane de PVDF et les protéine sont détecter à l’aide d’anticorps. Étape
1 : Présélection de mutants perdant la GPI-APs modèle Gas1 (n=1). Étape 2 : Les mutants sélectionnés à l’étape 1 sont
maintenant testés pour la perte de la GPI-APs modèle Yps1 (n=3). Les mutants qui ont été considérés pour leur perte de GPI-APs abondante sont ceux ayant une perte supérieure à 1.5 fois par rapport au type sauvage.
12
Classification des mutants sécrétant des quantités massives de Yps1
Une analyse d’enrichissement sur les 30 mutants a été faite afin de détecter s’ils avaient des processus biologiques en commun. Il y a eu détection d’enrichissement pour les processus suivants : maintien de la localisation des protéines et des compartiments cellulaires (annotations GO : appareil de Golgi et système endomembranaire) dans la cellule.Aucune fonction moléculaire particulière n’était toutefois associée à ces mutants.La comparaison des formes relâchées de Yps1 dans le milieu de culture par ces mutants a révélé que cinq d’entre eux montraient une forme de plus haut poids moléculaire comparé aux autres mutants. De plus, trois de ces mutants touchent des gènes (BST1, PER1 et GUP1) qui sont directement impliqués dans le remodelage de l’ancrage GPI tel que décrit précédemment (Kinoshita and Fujita 2016). Un autre gène, FYV6 est impliqué dans la régulation de la réparation des cassures d’ADN doubles brins via la ligation d’extrémités non-homologue et est essentiel pour la survie de la cellule en présence de la toxine K1 (Wilson 2002; Pagé et al. 2003). Le dernier de ces cinq gènes,
OPI3, est impliqué dans la synthèse de phosphatidylcholine (PC) (Kodaki and Yamashita 1987), le phospholipide le plus abondant dans la composition des membranes de la cellule. Ce mutant sera décrit en détail dans la section suivante puisqu’il sera le modèle de défaut métabolique menant à une sécrétion abondante de GPI-APs utilisé lors de ce projet de maîtrise.
Le mutant opi3Δ
Rôle du gène OPI3Le gène non-essentiel de levure OPI3 code pour une PE méthyl-transférase qui convertit la phosphatidyléthanolamine (PE) en PC (figure 8). Il catalyse les deux dernières étapes de méthylation de cette voie métabolique alors que l’enzyme Cho2 est responsable de la première méthylation (Kodaki and Yamashita 1987).
Figure 8 : Cette figure présente le rôle de la protéine Opi3 au sein de la voie de synthèse de phosphatidylcholine (PC) cytosolique. Les métabolites sont en noir ; PS : Phosphatidylsérine, PE : phosphatidyléthanolamine, MMPE :
monométhylphosphatidyléthanolamine, DMPE : diméthylphosphatidyléthanolamine et PC : Phosphatidylcholine. Les gènes condant pour les enzymes sont en rouge.
13
L’inactivation du gène OPI3 interfère avec plusieurs étapes de synthèse du groupement GPI
Ce gène a été découvert il y a près de 40 ans chez la levure sur la base d’une excrétion accrue d’inositol dans le milieu « overproducer of inositol » associée à son inactivation. C’est d’ailleurs d’où ce gène détient son nom (Greenberg, Reiner, and Henry 1982). Sa délétion empêche la synthèse de PC via le sentier CDP-DAG (McGraw and Henry 1989), obligeant la cellule à synthétiser le PC via le sentier de Kennedy qui utilise la choline exogène et le diacylglycérol comme précurseurs (Kennedy and Weiss 1956). En absence de choline dans le milieu, ce qui est le cas en milieu synthétique complet, les niveaux de PC de la cellule sont extrêmement faibles, mais la cellule est viable. L’intérêt pour ce gène, et du gène CHO2, s’est récemment renouvelé avec la découverte qu’une inactivation de l’un ou l’autre causait des gouttelettes lipidiques hypertrophiées (supersized lipid droplets) lorsque la souche est cultivée en milieu synthétique (Fei et al. 2011). De plus, un stress du RE est associé au mutant opi3Δ et sa viabilité en milieu synthétique dépend d’une réorganisation extensive de l’expression de son génome appelé la « membrane stress response » qui inclut le déclenchement de l’UPR (Thibault et al. 2012).
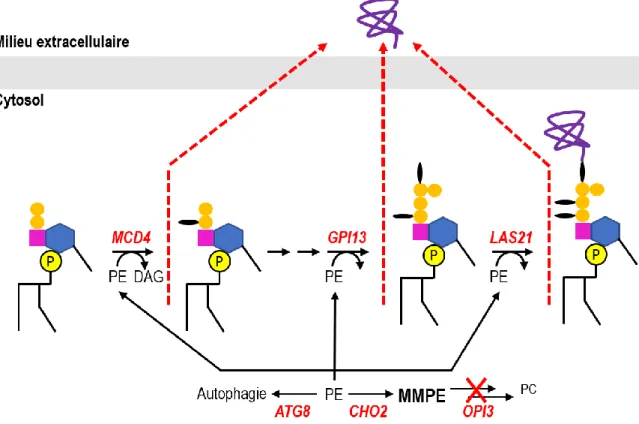
Notre laboratoire a mis en évidence que le mécanisme par lequel les protéines GPI sont sécrétées dans le milieu extracellulaire chez opi3Δ est dû à la déplétion du PE. Le PE est le donneur du groupement éthanolamine qui est impliqué dans trois étapes clés de la biosynthèse du GPI (figure 9; Mcd4, Gpi13 et Las21). Les premières données expérimentales à l’effet que le même pool de PE participait à la biosynthèse du GPI et du PC ont été apportées par Wilson-Zbinden et al. (Wilson-Zbinden et al. 2015). Ceux-ci ont montré que le mutant conditionnel mcd4-174ts peut croître à 37°C, mais seulement en absence du gène CHO2. Nous avons étendu ces travaux (Dubé et al. 2021) en montrant que; 1) le mutant cho2Δ ne sécrète pas de protéines GPI même s'il partage plusieurs phénotypes avec opi3Δ; 2) les doubles mutants gpi13-3ts cho2Δ et gpi13-5ts cho2Δ peuvent également pousser à 37°C; 3) le double mutant mcd4-174ts opi3Δ pousse moins bien à la température permissive (30°C) et finalement; 4) les niveaux de monométhylphosphatidyléthanolamine (MMPE) résultant de la première étape de méthylation du PE par Cho2, et qui sont extrêmement élevés dans le simple mutant opi3Δ (Thibault et al. 2012), sont pratiquement nuls dans le double mutant opi3Δ
gut2Δ (voir figure A1 en Annexe). Le double mutant opi3Δ gut2Δ est un modificateur positif de opi3Δ
caractérisé par l'absence de protéines GPI dans le milieu extracellulaire (voir figure A2 en Annexe).
14
catalyse l’oxydation du glycérol-3-phosphate en dihydroxyacétone phosphate (DHAP) (Grauslund and Rønnow 2000). Deux des trois enzymes étant essentiels pour la synthèse du GPI (Mcd4 et Gpi13), et donc pour son transfert sur les protéines, la sécrétion des protéines GPI modèles Yps1 et Gas1 dans le mutant opi3Δ repose donc essentiellement sur une activité protéolytique au niveau du RE tel que présenté précédemment (figure 6, gauche).
Figure 9 : Schématisation de la compétition pour le PE resultant en une perte de protéine à ancrage GPI. Figure
inspirée de plusieurs travaux (Birner et al. 2001; Wilson-Zbinden et al. 2015; Ferguson, Hart, and Kinoshita 2017).
Problématique
Murakami et al. (Murakami et al. 2012) ont récemment proposé deux mécanismes expliquant la sécrétion des protéines GPI lorsque le groupement GPI ne peut être transféré sur la protéine dans le modèle animal des cellulesCHO (Chinese Hamster Ovary; (Kim, Kim, and Lee 2012)). Pour les étapes de la biosynthèse du GPI interrompues après l'addition du premier résidu mannose, c'est le complexe transamidase normalement impliqué dans le clivage du peptide signal C-terminal couplé au transfert du groupement GPI qui serait responsable de la protéolyse, la relâche et la sécrétion de la protéine GPI. Pour les étapes antérieures à l'addition du premier résidu mannose au groupement GPI, la majorité des protéines GPI serait dégradée, mais une quantité non-négligeable de celles-ci serait
15
sécrétée par l'action potentielle d'une peptidase. La nature de cette peptidase est toutefois inconnue, aussi bien dans les cellules animales que chez la levure.
Hypothèses et objectifs
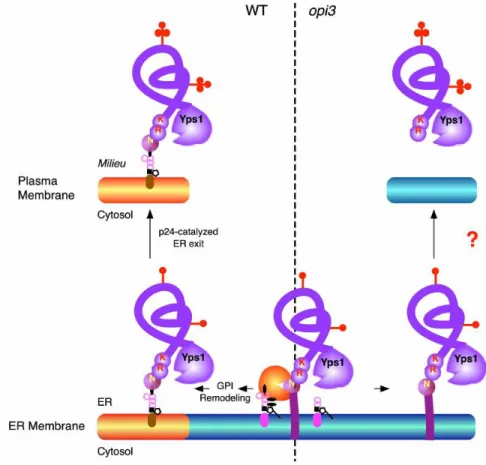
La peptidase Yps1, initialement nommée Yap3, est l’une des premières protéines à ancrage GPI à avoir été identifiée chez la levure (Ash et al. 1995). Bien qu’aucune fonction physiologique précise ne soit encore associée à cette enzyme, des GPI-APs telles que Gas1, Yps1 elle-même et Utr2 ont été décrites comme des substrats naturels de cette peptidase (Gagnon-Arsenault et al. 2008; Miller, DiDone, and Krysan 2010). Dans tous ces cas, l’action de la peptidase mène à la sécrétion des substrats de la peptidase Yps1 dans le milieu extracellulaire. D’ailleurs, Miller et al. (Miller, DiDone, and Krysan 2010) montre avec leurs travaux que la peptidase Yps1 permet de sécréter l’excès de GPI-APs en agissant de concert avec les mécanismes UPR et ERAD. Dans le contexte du mutant opi3Δ, les niveaux d’expression de cette protéine sont parmi les plus fortement augmentés dans la cellule en comparaison avec ceux du type sauvage (Thibault et al. 2012). Ceci suggère donc que l’endopeptidase Yps1joue un rôle dans la réponse au stress, malgré le fait qu’elle se retrouve en très grande quantité dans le milieu de culture.
Hypothèse 1 : Nous avons posé l’hypothèse que la protéine Yps1 puisse être impliquée dans la sécrétion des protéines GPI lorsque la synthèse des groupements GPI est avortée chez le mutant
opi3Δ (figure 10).
Objectif 1 : Déterminer si l’activité de Yps1 est requise pour la sécrétion de protéines GPI modèles
chez le mutant opi3Δ.
Objectif 2 : Si impliquée dans la sécrétion des protéines GPI dans le mutant opi3Δ (objectif 1),
déterminer si la protéine Yps1 est également nécessaire lorsque la synthèse des groupements GPI est bloquée dans le mutant mcd4-174ts. Le site d’action de Mcd4 étant l’étape la plus précoce et susceptible d’être perturbée par la mutation opi3Δ (voir figure 3).
Hypothèse 2 : Puisque le clivage d'une protéine GPI au niveau du RE serait en compétition directe
avec l'attachement du groupement GPI à cette même protéine dans un contexte normal, un mécanisme de régulation doit s'opérer pour limiter la protéolyse dans des contextes très précis.
16
Objectif 3 : Déterminer le(s) mécanisme(s) par lequel la protéolyse des protéines GPI par Yps1 est
régulée.
Figure 10 : Schématisation de l’hypothèse suggérant comment la peptidase de levure Yps1 peut être impliquée dans le clivage des protéines à ancrage GPI non-conformes chez le mutant opi3Δ. Gauche : Dans le type sauvage,
la biosynthèse normale de la protéine et de son ancrage permet à la GPI-APs d’être acheminée de RE à la membrane plasmique à l’aide du complexe p24. Droite : Une déficience dans la biosynthèse de l’ancrage GPI ne permet pas à la protéine de s’attacher à l’ancrage. Toutefois, Yps1 est détectée dans le milieu de culture, suggérant que son activité enztmatique est impliquée pour son clivage.
17
Chapitre 1
1.1
Titre de l’article inséré
Glycosylation status, a molecular rheostat regulating protease-dependent shedding of glycosylphosphatidylinositol-anchored proteins.
1.2
Auteurs
Nicolas Malenfant 1,2, Florence Ladonne 1,3 and Yves Bourbonnais 1,4,5
1 Institut de biologie intégrative et des systèmes, Département de biochimie, microbiologie &
bio-informatique, Université Laval, 1030 avenue de la médecine, Québec, Canada, G1V 0A6.
2 nicolas.malenfant.1@ulaval.ca,
3 florence.ladonne.1@ulaval.ca,
4 yves.bourbonnais@bcm.ulaval.ca, 418-656-2131 ext.: 407069
5 To whom all correspondence and material request should be addressed
1.3
Résumé
Des défauts génétiques dans la biosynthèse des ancrages GPI favorisent une sécrétion abondante de GPI-APs sans ancrage. Cette sécrétion peut être le résultat d’une protéolyse catalysée par une transamidase ou principalement par une dégradation protéosomale et un faible niveau de sécrétion via un mécanisme inconnu. Récemment, un mutant de levure (opi3Δ) défectueux dans la synthèse de la phosphatidylcholine par la voie de méthylation s'est avéré libérer des GPI-APs sans ancrage. Dans cette étude, nous démontrons que lorsqu’il y a surcharge en glycoprotéine dans le mutant opi3Δ, l’endopeptidase de levure Yps1 est essentielle pour le clivage et la libération de ces GPI-APs sans ancrage. Chez ce mutant, nous proposons que l’association entre la surcharge en glycoprotéine et un profil de O-glycosylation modifié est nécessaire pour dévoiler les sites de clivage de Yps1p. De telles conditions peuvent être rencontrées lors d’infections virales.
18
1.4
Abstract
Glycosylphosphatidylinositol-anchored proteins (GPI-APs) are associated with membrane microdomains rich in sphingolipids and sterols. Attachment of the anchor to GPI-AP by an ethanolamine linkage is carried out by a transamidase complex that orchestrates cleavage of the GPI-AP C-terminal signal sequence with anchor addition. Previously identified genetic defects in GPI anchor biosynthesis either promote an abundant secretion of anchor-less GPI-APs by a transamidase-catalyzed proteolysis or mostly proteasomal degradation and a low level of secretion by an unknown mechanism. Recently, a yeast mutant (opi3Δ) defective in phosphatidylcholine synthesis by the methylation pathway was found to release anchor-less GPI-APs. This results from metabolic interference with the GPI biosynthetic steps requiring phosphatidylethanolamine. Here we demonstrate that upon glycoprotein overload in the opi3Δ mutant, the yeast Yps1 endopeptidase is essential for the cleavage and release of anchor-less GPI-APs. Loss of YPS1 under these conditions leads to their degradation and is deleterious for growth. In this model of metabolic disorder, glycoprotein overload is associated with an altered O-glycosylation profile that we propose is needed to unmask Yps1p cleavage sites. Such conditions might be encountered upon viral infections. As several mammalian GPI-APs are involved in immunity, possible impacts of this metabolic interference on disease severity caused by SARS-CoV2 are discussed.
1.5
Introduction
Glycosylphosphatidylinositol (GPI)-anchored proteins (GPI-APs) are invariably associated with membrane microdomains known as "membrane rafts", rich in sterols and sphingolipids, throughout the eukaryotic kingdom 1,2. Involved in many diverse molecular functions ranging from enzymes, surface receptors, adhesion molecules, and complement regulators, this special class of proteins is not abundant with ~150 and ~60 proteins in human and in yeast, respectively 3. While yeast GPI-APs are mainly constituents and/or implicated in the cell wall construction with no orthologs in metazoa 4, structure of the GPI anchor and the cell machinery involved in its synthesis and remodeling have been largely conserved throughout evolution 5,6. The importance of this modification is underscored by the large number of genes (~26) involved in its synthesis and/or remodeling3, and because defects in GPI anchor assembly cause embryonic lethality in mammals 7 and is essential for yeast growth 8. In higher eukaryotes, GPI-APs play important roles in diverse biological processes including brain development 9, neurogenesis 10 and immunity 11.
19
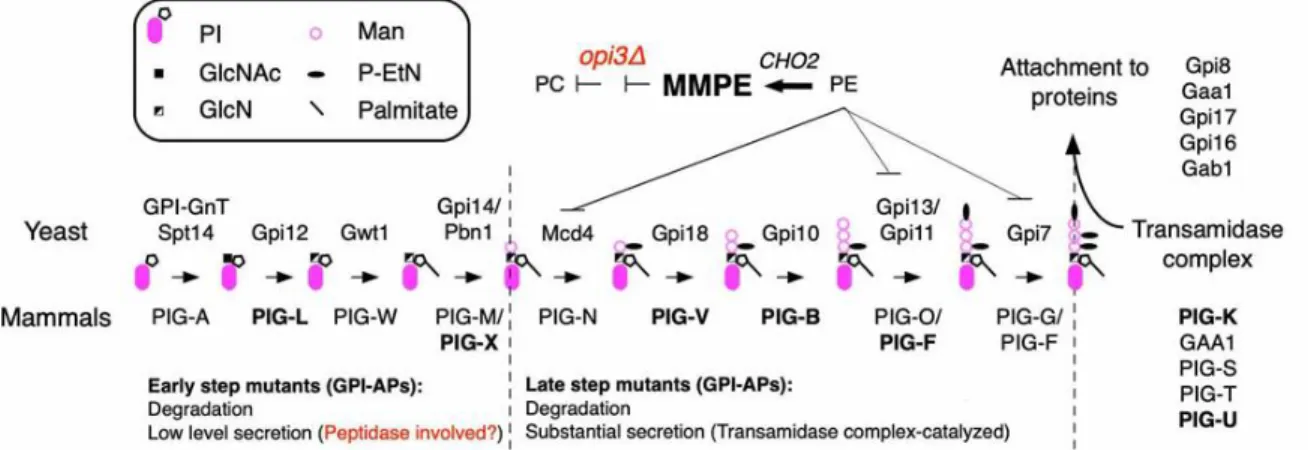
In the past decade, thanks to exome and genome sequencing 12, inherited GPI deficiencies (IGD) were identified for almost every step (21 genes) in the biosynthesis and maturation of the GPI anchor 13. While the most common symptoms associated with IGD are seizures, developmental delay and intellectual disability, mutations affecting specific steps of the pathway cause hyperphosphatasia with mental retardation (HPMR), also known as Mabry syndrome. First described in 1970 14, this syndrome results from the secretion of tissue non-specific alkaline phosphatase, a GPI-AP. Mutations causing HPMR were first described for the human PIG-V gene 15. Since then, six additional genes involved in GPI biosynthesis were added to the list 13,16. As schematized in Fig. 1, Murakami et al.17 proposed a mechanism explaining the secretion of tissue non-specific alkaline phosphatase, and other GPI-APs, from mutants accumulating immature GPI anchors that could not be attached to GPI-APs. Using various GPI biosynthetic mutants established in CHO cells, they found that the GPI transamidase (GPI-T) complex, which usually coordinates the cleavage of the hydrophobic C-terminal signal peptide with GPI anchor attachment, can also cleave GPI-APs at this site in the absence of a functional anchor. Transamidase-catalyzed cleavage of the GPI-APs was proposed to proceed if the GPI anchor intermediate contains at least one mannose (Man) residues (Fig.1 "Late step mutants"). In early steps mutants, before addition of the first Man by PIG-X, the bulk of GPI-APs were degraded. Yet, a low level of GPI-AP secretion was observed in these mutants suggesting the implication of a currently unknown peptidase.
Figure 1. Fate of GPI-Aps from biosynthetic mutants involved in GPI anchor assembly.
Anchor assembly steps presented with orthologous enzymes from mammals (bottom) and yeast (top). They are classified as ‘’Early’’ or ‘’Late’’ step mutants according to the fate (secretion vs degradation) of GPI-Aps in CHO mutants (bold)17.
Steps requiring phosphatidylethanolamine (PE) are indicated. These are affected in the yeast opi3Δ mutant due to the stimulated conversion of PE into monomethyl-PE (MMPE) by Cho2 enzyme18. PC; phosphatidylcholine.
20
In a recent yeast genome-wide screen to uncover loss of function mutations affecting membrane raft integrity, as monitored by the secretion of model GPI-APs (Gas1p and Yps1p), the opi3Δ mutant was identified 18. The OPI3 gene, an ortholog of mammalian PEMT 19, encodes the phospholipid methyltransferase catalyzing the last two methylation steps that converts phosphatidylethanolamine (PE) into phosphatidylcholine (PC) 20. Like many mutants affecting PC homeostasis, such as the cho2Δ mutant, opi3Δ mutant cells accumulate supersized lipid droplets when grown in choline limiting conditions 21. However, unlike the quantitative release in the medium of GPI-APs in the opi3Δ mutant, GPI-APs are stably maintained at the cell surface in cho2Δ yeast 18. In the opi3Δ mutant, the Cho2-catalyzed methylation step of PE is favored causing a vast accumulation of monomethyl-PE (MMPE)
18,21. Importantly, this competition for PE between Cho2p and the enzymes involved in GPI anchor
synthesis (Mcd4, Gpi13 and Las21/Gpi7; Fig. 1) provided an explanation for the secretion of GPI-APs in the opi3Δ mutant; incomplete or aborted GPI anchor synthesis and cleavage of anchor-less GPI-APs 18.
To cope with this severe anchoring deficit of GPI-APs the opi3Δ yeast mutant has elaborated a sophisticated response referred to as the "Membrane Stress Response" (MSR) 22, characterized by an extensive rewiring of gene expression. An essential component of this response is the activation of the
Unfolded Protein Response (UPR) which is evoked by cells to stimulate the ER-associated degradation
(ERAD) pathway by the proteasome 23,24. Apart from the canonical UPR targets involved in ERAD, phospholipid biosynthesis and ER chaperones, the GPI-AP Yps1 peptidase was found to be substantially overexpressed in the opi3Δ mutant (mean log2 = 3.02 vs wild type (WT) yeast) 22, hence pointing for Yps1-catalyzed proteolysis as a component of MSR. This GPI-anchored aspartic peptidase is a member of the yeast yapsin family 25. It is initially synthesized as a single chain protein that is matured into a heterodimer produced by an internal proteolytic cleavage within an N-terminal entrance loop (N-ent loop) 26. Yps1p was previously shown to be implicated in ectodomain shedding of various surface proteins, including the transmembrane protein Msb2 27 and the GPI-APs Gas1p and Utr2p as well as its own shedding 28,29. The study with Utr2p also suggested that Yps1p is part of a new protein quality control mechanism, functioning in parallel to proteasomal-mediated degradation, to release misfolded and/or excess GPI-APs.
21
The aim of the present study was to investigate whether the yeast Yps1 enzyme is implicated in the release of GPI-APs in the opi3Δ mutant. Globally, our data indicate that Yps1p catalyzes the shedding of anchor-less GPI-APs accumulating in this mutant, but only upon overexpression of glycoproteins. Our results suggest that metabolic interference between the glycosylation pathways and mannose addition steps in GPI anchor synthesis causes an altered glycosylation profile necessary to unmask Yps1 cleavage sites in GPI-APs.
1.6
Results
Native Yps1p is required for the shedding of plasmid-encoded model GPI-APs in the opi3Δ mutant
To monitor the secretion of GPI-APs, and the possible function of the yeast Yps1 peptidase in this process, the previously developed single-chain Yps1p variants 30 with a functional (Yps1DLp) or disrupted catalytic site (Yps1DL2p) were expressed from plasmids in different genetic backgrounds (Fig. 2a). These variants are lacking the N-ent loop (amino acids 118-185 of Yps1p) and are poorly detected by the rabbit antiserum 294 (α-294; Fig. 2a) raised against amino acids 35-246 of Yps1p 31. Given the overexpression scheme and different sizes, single-chain Yps1DL/2p can be distinguished from native Yps1p by western blotting with antiserum 268 (α-268; Fig. 2a) derived from the C-terminal part of Yps1p (amino acids 245- 532 of Yps1p 30). A soluble, secreted version of the inactive peptidase (Yps1ssDL2p; Fig. 2a) was also used as a control to assess the integrity of the secretory pathway. When expressed in wild type (WT) yeast cultured at pH 6.0, Yps1DLp was found to promote its self-shedding from the cell surface and accumulated a ~50 kDa form, after Endo Hf treatment, into the culture medium 30. This treatment was used to remove N-glycosylated chains that in Yps1p, because of heterogeneity, caused poorly resolved species migrating as a smear on SDS-PAGE. This Yps1p species results from self-catalyzed cleavage at sites internal to the pro-peptide (P) 30 and at a site (K498) 28 preceding the serine/threonine rich domain (S/T) (Fig. 2a; first and last black arrowheads, respectively). In contrast, when expressed in the opi3Δ mutant, very little of this molecular species was secreted and instead, a larger isoform (~60 kDa) was secreted 18. Because the opi3Δ mutant interferes with the addition of ethanolamine phosphate (EtN-P) in GPI biosynthesis (Fig. 1; 18), this secreted molecular species must arise from proteolytic cleavage of the anchor-less precursor at the ER membrane (Fig. 2b). By analogy with metazoans 17, this may be catalyzed by the yeast GPI-T complex.
22
However, contiguous to the ω site used by GPI-T complex (N; Fig. 2b), a pair basic site (KR; Fig. 2b) is also present, which is a preferred cleavage site for Yps1p in vitro 32. This arrangement around the ω site is a hallmark of yeast GPI-APs maintained at the plasma membrane as opposed to cell wall-associated GPI-APs 33. It therefore prompted us to investigate whether Yps1p could, in some conditions, shed this class of anchor-less GPI-APs at these sites (red arrow; Fig. 2a & b).
Figure 2. Yps1 variants and antisera used in this study. (a) Native Yps1p (top) in α/β heterodimer maintained by disulfide
bridge (SS) that results from a self-catalyzed proteolysis within an N-terminal entrance loop (L). All variants derived from Yps1p are single chain peptidases where the loop (L) was replaced by short analogous peptide found in Yps3p (aa96-102).
Some variants (Yps1DLp and Yps1DL2p) retained the GPI anchor signal (AS) or missed both the AS and the serine/threonine (S/T)-rich domain (Yps1ssDL2p). The Yps1DL2p and Yps1ssDL2p variants are also catalytically inactive (D101E). P; Pro-peptide. Yps1 antiserum α-294 preferentially detects native Yps1p, because of epitopes present within the
loop, whereas the α-268 antiserum detects all Yps1 proteins. (b) Hypothetical Yps1p self-catalyzed cleavage at the ER membrane at a basic pair site (KR) located next to the GPI attachment site (N; ω site) to release the ectodomain. GPI-T complex; GPI transamidase complex.
As shown in Fig. 3a (left panel) a substantial amount of Yps1DLp was secreted as a major band of ~50 kDa upon expression in wild type (WT) yeast hence confirming its self-shedding activity of at an internal site (K498) 30. However, its catalytically inactive isoform (D101E; Yps1DL2p) was not. All immunoreactive Yps1DL2p being cell-associated, this indicates that the native Yps1 peptidase cannot proteolytically process membrane-bound Yps1DL2p under these conditions. In contrast, both Yps1DLp and Yps1DL2p were secreted when expressed in the opi3Δ single mutant. However, compared to the total Yps1 immunoreactive material observed upon Yps1DL2 expression in WT yeast, when expressed in the opi3Δ mutant the percentage of immunoreactive material recovered with Yps1DLp or Yps1DL2p was significantly lower (Fig.3a; histogram left panel). The main secreted species migrated slightly
23
above the 58 kDa protein marker. Based on its predicted molecular weight (MW = 48.36 kDa), and considering extensive O-glycosylation, this species is likely the full-length protein without the C-terminal anchor sequence (see schematic representations in Fig. 3a). A smaller, and somewhat less abundant, species of > 32 kDa was also secreted. Although this minor species was not always retained on the gel after SDS-PAGE, absent from the blot presented in Fig. 3a (left panel) but observed on other blots (Fig. S1), it was also detected as a degradation product upon expression of the soluble Yps1ssDL2p protein (Fig. 3a; right panel). Based on its size (predicted MW of 29.27 kDa), this species is thought to correspond to a peptide fragment resulting from cleavage after R201 and R478 (▽; see below). Using an algorithm (ProP 1.0 Server; http://www.cbs.dtu.dk/services/ProP/) to detect putative mammalian furin cleavage sites (an ortholog of yeast Kex2p), R201 and R478 gave scores close to other known utilized sites in Yps1DLp (Table S1). Cleavage at these sites splits apart the two catalytic aspartate residues (D101 and D371) of Yps1DLp (or native Yps1p) and would consequently inactivate the peptidase. Plasmid expression of the soluble form Yps1ssDL2p in WT and the opi3Δ mutant, used as an internal control for the secretory apparatus, resulted in efficient secretion (Fig. 3a; left panel). Yet, compared to WT yeast, a trend toward a lower recovery of total immunoreactive material was observed when expressed in the opi3Δ mutant (Fig.3a; histogram left panel). In addition to the full- length His-tagged protein (predicted MW of 45.58 kDa), other species were observed in the medium of both strains. These include the >32 kDa species also detected with the Yps1DLp variant.
When the model GPI-APs Yps1-DLp and Yps1-DL2p were expressed in the opi3Δ yps1Δ double mutant, the Yps1-immunoreactive signal could barely be detected, either intracellularly or in the medium (Fig. 3a; right panel). Furthermore, a sharp reduction in the secretion of the soluble Yps1ssDL2 protein was noticed when expressed in the opi3Δ yps1Δ double mutant compared to that observed for the single yps1Δ mutant. Overall, these data support a role for native Yps1p in the release of anchor-less GPI-APs in the opi3Δ mutant and suggest the working model presented in Fig. 3b. In contrast to WT yeast, synthesis of GPI in opi3Δ is partially blocked at multiple steps resulting in the accumulation of GPI intermediates and anchor-less GPI-APs at the ER membrane.
24
Figure 3. Loss of genomic YPS1 in the opi3Δ mutant abolishes secretion of model GPI-Aps and promotes their degradation. (a) Representative Western blots (n = 3 biological replicates, except for Yps1ssDL2; n = 2) and corresponding
histograms comparing the total intracellular (I) and extracellular (E) Yps1-immunoreactive material (Yps1-antiserum 268-6; α-268) from the indicated yeast strains and plasmids. Anti-actin (α-act) was used as the loading control. Note that the extracellular material was 10 X concentrated compared to cell extracts. Postulated secreted Yps1 fragments derived from Yps1DLp and Yps1ssDL2p, and the postulated cleavage sites used (arrowheads), are schematized on the right-end side. The percentage of immunoreactivity in each fraction was determined relative to the total immunoreactive material found in YPS1DL2 and YPS1ssDL2, both set to 100% for the GPI and soluble forms, respectively. Error bars in the histogram are SD associated with the intra- (under the bars) and extracellular (above the bars) material. *** = p < 0.001; ** p < 0.01; * p <
0.05. (b) Cartoon illustrating the major conclusions drawn from (a). Native Yps1p is implicated in the shedding of
anchor-less GPI-Aps at the ER (left panel), its absence causes degradation of these species (center panel) as well as a sharp reduction in the amount of secreted soluble species (right panel).