APRIN(PDS5B) et PALB2, deux protéines
impliquées dans la réparation de l’ADN par
recombinaison homologue et l’apparition de
cancers
Thèse
Anthony Couturier
Doctorat en Biologie cellulaire et moléculaire
Philosophiæ Doctor (Ph.D.)
Québec, Canada
APRIN(PDS5B) et PALB2, deux protéines
impliquées dans la réparation de l’ADN par
recombinaison homologue et l’apparition de
cancers
Thèse
Anthony Couturier
Sous la direction de :
iii
Au Canada, en 2015, il était estimé que 78 000 personnes allaient mourir d’un cancer, représentant 30 % de tous les décès et faisant de celui-ci la première cause de mortalité. De plus, 196 900 nouveaux cas de cancers seraient découverts au cours de cette même année (Canadian
Cancer Society’s Advisory Committee on Cancer Statistics. Canadian Cancer Statistics 2015. Toronto, ON : Canadian Cancer Society; 2015).
L’intégrité du génome est chaque jour menacée par des conditions environnementales qui endommagent l’ADN (ultraviolets, produits chimiques divers, etc.). Parmi les différents types de lésions, l’un des plus délétères et pouvant mener au cancer est la cassure double-brin (CDB). Celle-ci peut être réparée suivant deux mécanismes majeurs : la jonction des extrémités non homologues (Non-Homologous End-Joining ou NHEJ) ou la Recombinaison Homologue (RH). Cette dernière, prépondérante pendant les phases S/G2, consiste en la réparation d’une CDB grâce à l’utilisation d’une chromatide sœur comme modèle, permettant une réparation fidèle du dommage. La RH est sous la dépendance de diverses protéines, dont RAD51, PALB2 et BRCA2. Ces deux dernières sont connues pour être mutées dans les cancers du sein et des ovaires. Ainsi, la compréhension de l’implication de chaque acteur dans la RH est un objectif fondamental dans la lutte contre le cancer et constitue l’objectif général de cette thèse.
En 2012, une étude a montré qu’une nouvelle protéine, APRIN (Androgen-induced
PRoliferation INhibitor), appartenant au complexe cohésine, interagissait avec BRCA2 et jouait un
rôle dans la RH. Les rôles précis d’APRIN dans ce mécanisme restaient toutefois à être définis. Le projet principal de cette thèse repose sur la caractérisation fonctionnelle d’APRIN dans la réparation par RH. Nous révélons qu’APRIN aurait un rôle spécifique et indépendant de celui de la cohésine dans la RH, et pourrait agir à diverses étapes cruciales de ce mécanisme. De plus, nos données montrent que le niveau d’expression d’APRIN pourrait être un marqueur de prédiction dans le cancer ovarien.
Étant donné qu’APRIN interagit aussi avec PALB2, autre partenaire essentiel de BRCA2, nous avons également étudié et caractérisé les fonctions de divers mutants de PALB2. Nous faisons ainsi la découverte inattendue d’un nouveau phénotype induit par une troncation de cette protéine associée à certains cancers agressifs.
Ainsi, cette thèse apporte des informations supplémentaires et indispensables à la compréhension de la réparation de l’ADN par RH et de la survenue de certains cancers.
iv
Cancer is the leading cause of death in Canada (30% of all deaths). By 2015, it was estimated that 78,000 Canadians would die of cancer. Moreover, 196,900 new cases of cancer were discovered in that same year (Canadian Cancer Society’s Advisory Committee on Cancer Statistics.
Canadian Cancer Statistics 2015. Toronto, ON: Canadian Cancer Society; 2015).
Each day, genome integrity is threatened by environmental conditions that can induce DNA damages (UV, chemicals, etc.). Amongst different types of lesions, double-strand breaks (DSB) are one of the most deleterious and can lead to cancer development. DSB can be repaired following two different and major pathways: Non-Homologous End-Joining (NHEJ) and Homologous Recombination (HR). HR mainly takes place during S/G2 phases and uses the intact sister chromatid as a template for repair. This renders HR to be a faithful mechanism, which depends on three important proteins: RAD51, PALB2 and BRCA2. The last two proteins are frequently found mutated in both breast and ovarian cancers. Understanding how each HR actor is specifically implicated in this pathway is important to develop strategies to fight cancer and constitutes the general aim of this thesis.
In 2012, APRIN (Androgen-induced PRoliferation INhibitor), known as a regulator of the cohesin complex, was described as a new interacting partner of BRCA2 and an actor of HR. Nevertheless, how APRIN was specifically implicated in HR remained unclear. Hence, the major objective of this thesis was to functionally characterize APRIN in HR. We show that APRIN possesses a specific and independent role from the cohesin complex in HR and could be implicated in different steps during this repair pathway. Interestingly, APRIN expression levels in ovarian cancer tumours may be used as a prognostic marker.
Moreover, to achieve the general objective of this thesis, we focused on another BRCA2 partner : PALB2. We studied the impact of different truncating mutations found in cancers on PALB2 functions. Surprisingly, we discovered that the consequence of an aggressive cancer-causing mutation could stem from an aberrant localization of PALB2.
Consequently, this thesis provides important new information on the repair of DSB by HR and on the occurrence of cancers.
v
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des tableaux ... ix
Liste des figures ... x
Liste des figures supplémentaires ... xii
Liste des abréviations ... xiii
Remerciements ... xx
Avant-propos ... xxii
Chapitre 1 : Introduction ... 1
1.1 Les dommages de l’ADN ... 1
1.2 Le cycle cellulaire ... 4
1.3 La détection d’une lésion de l’ADN ... 6
1.4 Les premières étapes de détection d’une cassure double-brin de l’ADN ... 8
1.5 Les cassures double-brin de l’ADN ... 10
1.5.1
Bref aperçu ... 10
1.5.2
Les cassures double-brin bénéfiques ... 10
1.6 La réparation des cassures double-brin de l’ADN ... 13
1.7 Le NHEJ : la jonction des extrémités non-homologues ... 14
1.7.1
Le C-NHEJ : la voie classique ... 15
1.7.2
Le Alt-NHEJ : la voie alternative ... 16
1.8 La réparation homologue ... 17
1.8.1
L’étape présynaptique : de la résection à la formation du filament RAD51 ... 17
1.8.2
L’étape synaptique ... 21
1.8.3
L’étape postsynaptique ... 23
1.8.4
Le SDSA ... 24
1.8.5
Le BIR ... 25
1.8.6
Le SSA ... 26
1.9 Les protéines BRCA1-PALB2-BRCA2 et RAD51 au cœur de la RH ... 27
1.9.1
La protéine RAD51 ... 28
1.9.2
La protéine BRCA1 ... 32
1.9.3
La protéine BRCA2 ... 33
1.9.4
La protéine PALB2 ... 37
1.10 La régulation du choix de la voie de réparation de la cassure double-brin ... 42
1.11 Cohésine et caractéristique ... 44
1.11.1
Bref historique ... 44
1.11.2
Structure et composition du complexe cohésine ... 45
1.11.3
La cohésion des chromatides sœurs ... 46
1.11.4
Les protéines du complexe cohésine ... 47
1.11.5
La protéine PDS5 ... 50
1.11.6
La cohésine et la réparation de l’ADN ... 55
1.11.7
Défauts de cohésine et destin clinique ... 57
1.12 Le Cancer ... 59
1.12.1
Brève introduction ... 59
vi
Chapitre 2 : DNA Damage Signalling and Repair Inhibitors : The
Long-Sought-After Achilles’ Heel of Cancer ... 65
2.1 Avant-propos ... 66
2.2 Résumé ... 67
2.3 Abstract ... 68
2.4 Introduction ... 69
2.5 The ATM and ATR Kinases ... 71
2.5.1
Dual ATM/ATR Inhibitors ... 72
2.5.2
Selective ATM Inhibitors ... 72
2.5.3
Selective ATR Inhibitors ... 74
2.6 The Chk1 Kinase ... 78
2.6.1
Roles of Chk1 in the DNA Damage Response ... 78
2.6.2
Phosphorylation of Chk1 ... 78
2.6.3
Targeting Chk1 as a Novel Strategy in Cancer Therapy ... 79
2.6.4
Inhibitors of Chk1 ... 79
2.7 The Chk2 Kinase ... 82
2.8 The Poly (ADP) Ribose Polymerase ... 85
2.8.1
Talazoparib (BMN 673; BioMarin Pharmaceutical Inc., Novato, CA, USA) ... 87
2.8.2
Veliparib (ABT-888; AbbVie Inc., North Chicago, IL, USA) ... 87
2.8.3
Rucaparib (AG-014699; Clovis Oncology, Boulder, CO, USA) ... 88
2.8.4
Niraparib (MK-4827 ; Tesaro Inc., Waltham, MA, USA) ... 88
2.8.5
INO-1001 (Selleck Chemicals, Euromedex, France) ... 89
2.8.6
E7016 (Eisai Co., Ltd; Tokyo, Japan) ... 89
2.8.7
CEP-9722 (Cephalon, Inc.; Frazer, PA, USA) ... 89
2.8.8
PARP Trapping: Mechanism of Action of PARP Inhibitors ... 89
2.9 The MRE11-RAD50-NBS1 Complex ... 93
2.9.1
Mirin ... 95
2.9.2
PFM39, PFM01 and PFM03 ... 95
2.10 The RAD51 Protein, a Central HR Protein ... 96
2.10.1
DIDS ... 96
2.10.2
Halenaquinone/Xestoquinone ... 97
2.10.3
B02 and Derivatives ... 97
2.10.4
RI-1 and RI-2 ... 98
2.10.5
IBR2 and IBR120 ... 99
2.11 The RAD52 Protein ... 101
2.12 The Non-Homologous End-Joining (NHEJ) Proteins ... 102
2.12.1
Selective DNA-PK Inhibitors ... 103
2.12.2
Nucleotide-Based Inhibitors of DNA-PK ... 107
2.12.3
Ku70/Ku80 Inhibitors ... 108
2.12.4
DNA Ligase IV Inhibitors ... 108
2.12.5
XRCC4 Inhibitors ... 110
2.12.6
Alt-NHEJ Inhibitors ... 110
2.13 Conclusions ... 111
vii
3.1 Avant-propos ... 122
3.2 Résumé ... 123
3.3 Abstract ... 124
3.4 Introduction ... 125
3.5 PALB2 : a key player for genome stability ... 126
3.5.1
Brief overview of BRCA2 functions ... 126
3.5.2
Contributions of PALB2 to genome maintenance and HR ... 127
3.5.3
MRG15 : another key interactor of PALB2 ... 131
3.5.4
PALB2 is an FA gene ... 132
3.6 PALB2 : an important tumour suppressor ... 133
3.6.1
Mutations of PALB2 in female and male breast cancer ... 133
3.6.2
Mutations of PALB2 in ovarian cancer ... 138
3.6.3
PALB2 mutations in pancreatic cancer ... 139
3.6.4
Insights from animal models ... 140
3.6.5
Functional studies of disease-related mutations ... 142
3.6.6
Using PALB2 defect as a therapeutic strategy ... 143
Chapitre 4 : Investigating cancer-causing mutations in the tumour
suppressor PALB2 reveals a novel cancer mechanism
using hidden nuclear export signal in the WD40
repeat motif ... 145
4.1 Avant-propos ... 146
4.2 Résumé ... 147
4.3 Abstract ... 148
4.4 Introduction ... 149
4.5 Results ... 151
4.5.1
PALB2 DNA binding is required to stimulate RAD51 activity ... 151
4.5.2
A mutation leading to a truncation in the WD40 domain induces a cellular mislocalization ... 155
4.5.3
The Q988X cancer-causing mutation also causes cytoplasmic accumulation ... 157
4.5.4
PALB2 WD40 masks a nuclear export sequence ... 157
4.5.5
Unfolding the WD40 repeat of other proteins ... 162
4.6 Discussion ... 164
4.7 Material and Methods ... 166
4.8 Supplementary figures ... 169
viii
5.1 Avant-propos ... 182
5.2 Résumé ... 183
5.3 Abstract ... 184
5.4 Introduction ... 185
5.5 Results ... 187
5.5.1
APRIN is recruited to laser-induced DNA damage sites, colocalizes with RAD51 and interacts directly with RAD51 ... 187
5.5.2
Purified APRIN binds different DNA intermediates of HR ... 189
5.5.3
APRIN binds DNA via two distinct domains and is phosphorylated at its C-terminal domain ... 191
5.5.4
Purified APRIN stimulates RAD51-mediated D-loop formation ... 191
5.5.5
APRIN enhances the annealing of homologous single-strands ... 192
5.5.6
APRIN enhances homologous-strand annealing in synergy with BRCA2 ... 194
5.5.7
APRIN does not play a role in NHEJ or CSR unlike cohesins ... 195
5.5.8
Depletion of APRIN leads to the sensitization of cells to PARP inhibition in zebrafish. ... 197
5.5.9
Low levels of APRIN correlate with a better survival of ovarian cancer patients ... 198
5.6 Discussion ... 200
5.7 Material and methods ... 202
5.8 Supplementary figures ... 210
5.9 Tableaux ... 218
Chapitre 6 : Discussion ... 221
6.1 APRIN une nouvelle protéine impliquée dans la RH ... 221
6.1.1
APRIN est recrutée au niveau des dommages de l’ADN ... 221
6.1.2
APRIN est un médiateur de la recombinaison homologue ... 221
6.1.3
Le rôle d’APRIN est indépendant de celui de la cohésine et pourrait être régulé par sa phosphorylation ... 222
6.1.4
APRIN un marqueur de prédiction pour le cancer ovarien ? ... 224
6.2 Une mutation tronquante de PALB2 a une conséquence inattendue sur sa
fonction ... 226
6.2.1
Les fonctions biochimiques de PALB2 sont altérées différemment suivant la troncation ... 226
6.2.2
La mutation W1038X révèle un NES dans la partie C-terminale de PALB2 ... 227
6.2.3
La présence d’un NES caché au sein d’un domaine WD40 : un phénomène retrouvé dans une autre protéine ... 229
6.2.4
Utiliser le NES de PALB2 comme une cible thérapeutique ... 230
Chapitre 7 : Conclusion et perspectives ... 233
ix
Chapitre 1 : Introduction
Tableau 1.1 : Rôle des protéines Pds5 au sein de différents organismes ... 51
Tableau 1.2 : Différents stades dans le cancer ovarien ... 62
Tableau 1.3 : Différents grades dans le cancer ovarien ... 62
Chapitre 3 : Exploring the roles of PALB2 at the crossroads of DNA
repair and cancer
Tableau 3.1 : PALB2 truncating mutations found in cancer and Fanconi’s anemia patients ... 133Chapitre 4 : Hidden NES in the WD40 repeat motif of PALB2
Tableau 4.1 : Oligonucleotides used in this study ... 177Chapitre 5 : Functions for APRIN in HR and in ovarian cancer prediction
Tableau 5.1 : Clinicopathological characteristics of the RMA sample set (n=208) ... 218Tableau 5.2 : Primers used for DNA probes and their sequences ... 218
Tableau 5.3 : Primers used for His/Flag TAP constructs and their sequences ... 219
Tableau 5.4 : siRNA and shRNA sequences ... 220
Chapitre 6 : Discussion
Tableau 6.1 : Différents patrons de phosphorylation pour PDS5A et APRIN à la suite d’un dommage à l’ADN ... 224x
Chapitre 1 : Introduction
Figure 1.1 : Les sources de dommages à l’ADN ... 2
Figure 1.2 : Le cycle cellulaire et les points de contrôle ... 4
Figure 1.3 : La réponse aux dommages l’ADN ... 7
Figure 1.4 : Les premières étapes de détection d’une cassure double-brin de l’ADN ... 9
Figure 1.5 : Le mécanisme de commutation de classe ... 11
Figure 1.6 : Les différents types de réparation des cassures double-brin de l’ADN ... 13
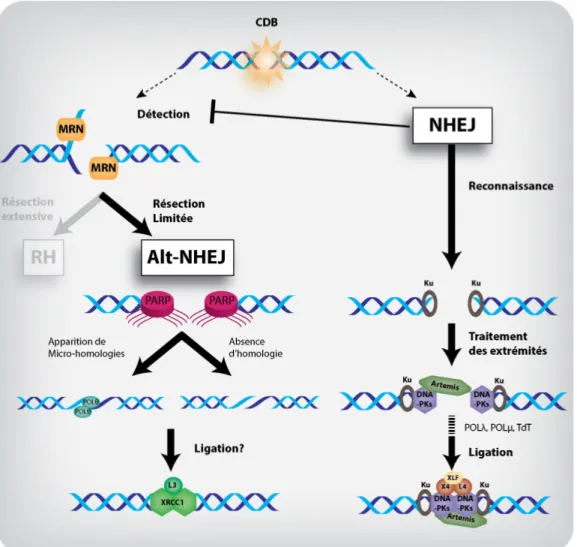
Figure 1.7 : La réparation par NHEJ versus Alt-NHEJ ... 14
Figure 1.8 : L’étape présynaptique de la réparation homologue ... 19
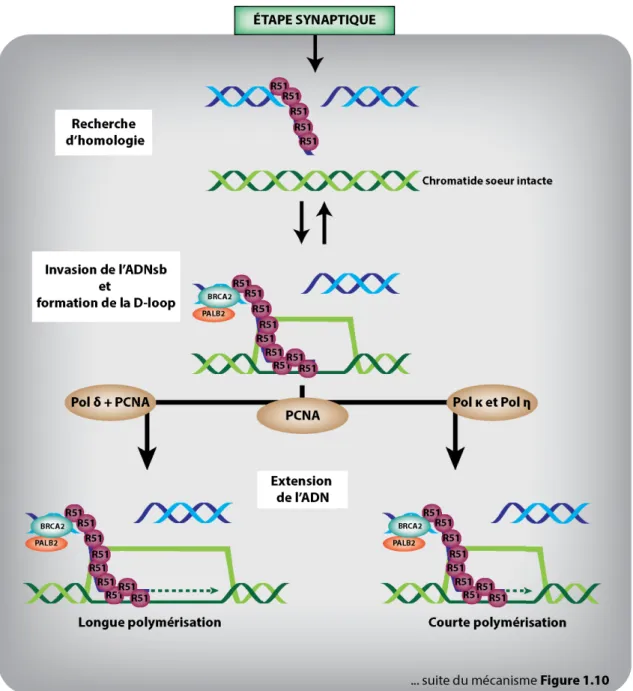
Figure 1.9 : L’étape synaptique de la recombinaison homologue ... 21
Figure 1.10 : L’étape postsynaptique de la recombinaison homologue composée de trois différents mécanismes de réparation ... 25
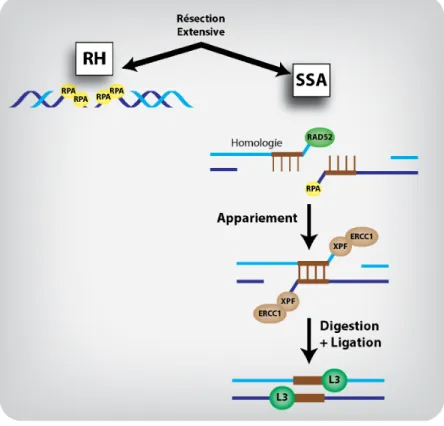
Figure 1.11 : L’appariement d’ADN simple-brin (SSA) ... 26
Figure 1.12 : Les domaines fonctionnels de BRCA1, PALB2, BRCA2 et RAD51 ... 27
Figure 1.13 : Les régulateurs négatifs de la protéine RAD51 ... 31
Figure 1.14 : Les différents rôles de BRCA2 ... 36
Figure 1.15 : La protéine PALB2 : domaines et partenaires importants ... 37
Figure 1.16 : Le choix du type de réparation des cassures double-brin est finement régulé ... 43
Figure 1.17 : Le complexe cohésine ... 45
Figure 1.18 : Le cycle de la cohésine ... 47
Figure 1.19 : La protéine SMC ... 48
Figure 1.20 : La localisation du gène PDS5B (APRIN) sur le chromosome 13 ... 52
Figure 1.21 : Les domaines fonctionnels de PDS5A et PDS5B ... 53
Figure 1.22 : Schéma d’un ovaire ... 61
Chapitre 2 : DNA damage signalling and repair inhibitors
Figure 2.1 : The DNA damage response pathways and its inhibitors ... 70Figure 2.2 : Schematic representation of DNA repair by homologous recombination ... 94
Figure 2.3 : Schematic representation of DNA repair by non-homologous end-joining (NHEJ): classical NHEJ (C-NHEJ) or alternative NHEJ (alt-NHEJ) ... 103
Figure 2.4 : The synthetic lethality pathway model based on a deficiency in Alt-NHEJ and the double-strand break repair pathway ... 112
Chapitre 3 : Exploring the roles of PALB2 at the crossroads of DNA
repair and cancer
Figure 3.1 : Roles of PALB2 in HR ... 127Figure 3.2 : Schematic representation of PALB2 mutations in exons or assigned domains of PALB2 ... 129
xi
Figure 4.1 : PALB2 wild-type and mutant proteins ... 152
Figure 4.2 : Competition EMSAs with five different DNA substrates and PALB2 mutant proteins ... 154
Figure 4.3 : Cellular localization of PALB2 mutant proteins ... 156
Figure 4.4 : Cellular localization of WD40-mutant PALB2 and WD40-truncated variant proteins 158
Figure 4.5 : A sequence contained in PALB2 WD40-domain acts as a CRM1-dependent nuclear export signal ... 159
Figure 4.6 : Effect of PALB2 W1038X mutant proteins on RAD51 foci formation ... 161
Figure 4.7 : RBBP4, another WD40 domain containing protein, becomes cytoplasmic due to a cancer causing mutation ... 163
Chapitre 5 : Functions for APRIN in HR and in ovarian cancer prediction
Figure 5.1 : APRIN is recruited to laser-induced DNA damage sites and interacts directly with RAD51 ... 188
Figure 5.2 : APRIN binds DNA and preferentially single-strand DNA and D-loop structures and enhances RAD51 mediated D-loop formation ... 190
Figure 5.3 : APRIN is proficient in single-strand annealing ... 193
Figure 5.4 : APRIN enhances the homologous-strand annealing in synergy with BRCA2 ... 194
Figure 5.5 : APRIN is not implicated in Non-Homologous End-Joining (NHEJ) nor Switch Class Recombination (CSR) ... 196
Figure 5.6 : Zebrafish xenografted with APRIN-depleted Hela cells are sensitive to Olaparib ... 197
Figure 5.7 : Low APRIN expression levels are correlated with better survival of ovarian cancer patients ... 199
Chapitre 6 : Discussion
xii
Chapitre 2 : DNA damage signalling and repair inhibitors
Supplementary figure 2.1 : The ATM/ATR inhibitors ... 114
Supplementary figure 2.2 : The Chk1/Chek2 inhibitors inhibitors ... 115
Supplementary figure 2.3 : The PARP inhibitors that have reached clinical evaluation ... 116
Supplementary figure 2.4 : The MRN (MRE11-RAD50-NBS1) complex inhibitors ... 117
Supplementary figure 2.5 : The RAD51 inhibitors ... 118
Supplementary figure 2.6 : The DNA-PK inhibitors ... 119
Supplementary figure 2.7 : The DNA ligase IV and XRCC4 inhibitors ... 120
Chapitre 4 : Hidden NES in the WD40 repeat motif of PALB2
Supplementary figure 4.1 : DNA binding of PALB2 mutant proteins ... 169Supplementary figure 4.2 : Stimulation of RAD51-mediated D-loop formation by PALB2 mutant proteins ... 170
Supplementary figure 4.3 : Localization of PALB2 mutant proteins to double-strand breaks induced by neocarzinostatin in HEK293T cells ... 171
Supplementary figure 4.4 : Cellular localization of PALB2 mutant proteins in the absence or presence of DNA damage ... 172
Supplementary figure 4.5 : Results of NES prediction by ValidNESs and NetNES 1.1 server on PALB2 ... 173
Supplementary figure 4.6 : Conservation of the PALB2 NES, throughout different species ... 174
Supplementary figure 4.7 : The cellular mislocalization of PALB2 W1038X is almost completely abrogated after theatment with the CRM1 inhibitor KPT-330 ... 175
Supplementary figure 4.8 : Cellular localization of W1038X and W1038XmutNES ... 176
Chapitre 5 : Functions for APRIN in HR and in ovarian cancer prediction
Supplementary figure 5.1 : Two-step affinity purification of APRIN ... 210Supplementary figure 5.2 : APRIN scheme and nuclear property ... 211
Supplementary figure 5.3 : APRIN binds differents types of DNA structures ... 212
Supplementary figure 5.4 : Purification of APRIN fragments ... 213
Supplementary figure 5.5 : APRIN is phosphorylated on in its C-terminal domain ... 214
Supplementary figure 5.6 : APRIN C-terminal DNA binding properties ... 215
Supplementary figure 5.7 : AnnexinV staining and cell viability ... 216
xiii
293 T Cellules immortalisées de rein embryonnaire humain exprimant l’antigène T du
virus SV40
53BP1 de l’anglais « p53-Binding Protein 1 »
ABC de l’anglais « ATP-binding cassette »
Abraxas de l’anglais « BRCA1-A complex subunit Abraxas (From the Greek god and the
gene Abra1) »
ADN Acide DésoxyriboNucléique
ADNdb ADN double-brin
ADNsb ADN simple-brin
ADP Adénosine DiPhosphate
AF Anémie de Fanconi
AID de l’anglais « Activation-Induced cytidine Deaminase »
Alt-NHEJ de l’anglais « Alternative-NHEJ »
ANOVA de l’anglais « ANalysis Of Variance »
APRIN de l’anglais « Androgen-induced proliferation inhibitor »
AR de l’anglais « Androgen Receptor »
ARN Acide RiboNucléique
ATM de l’anglais « Ataxia Telangiectasia Mutated »
ATP Adénosine TriPhosphate
ATR de l’anglais « ATM and RAD3 related »
ATRIP de l’anglais « ATR-Interacting Protein »
BARD1 de l’anglais « BRCA1 associated RING domain protein 1 »
BER Réparation par excision de bases ( de l’anglais « Base Excision Repair »)
BHQ de l’anglais « Black Hole Quencher »
BIR de l’anglais « Break Induced Replication »
BLM de l’anglais « BLoom syndrome Mutated protein »
BMN de l’anglais « BioMarin Pharmaceutical Inc., Novato, CA, USA »
BRC de l’anglais « Breast cancer (repeats) »
BRCA1/2 de l’anglais « BReast CAncer 1/2, early onset »
BRCT de l’anglais « BRCA1 C Terminus domain »
BSA de l’anglais « Bovine Serum Albumin »
CBP de l’anglais « CREB-binding protein »
CDB Cassure Double-Brin
CDC25 de l’anglais « Cell Division Cycle 25 »
CDDP Cisplatine
CDK de l’anglais « Cyclin-Dependent Kinase »
xiv
CIP de l’anglais « Calf-Intestinal Phosphatase »
COE Cancer Ovarien Épithélial
COSMIC de l’anglais « Catalogue of Somatic Mutations In Cancer »
CPT Camptothécine
CRISPR de l’anglais « Clustered Regularly Interspaced Short Palindromic Repeats »
CRM1 de l’anglais « Chromosome Region Maintenance 1 » (ou Exportine 1/XPO1)
CSB Cassure simple-brin
CSR de l’anglais « Class switch recombination »
CtIP de l’anglais « C-terminal binding protein Interacting Protein »
CTL de l’anglais « Control »
D-loop de l’anglais « Displacement loop »
DAPI 4', 6 - diamidino -2-phenylindole
DBD de l’anglais « DNA binding domain »
DDR de l’anglais « DNA damage response »
DEB DiEpoxyButane
DMSO DiMéthylSulfOxyde
DNA de l’anglais « Deoxyribonucleic acid »
DNA-PKcs de l’anglais « DNA-dependent Protein Kinase catalytic subunit »
DNA2 de l’anglais « DNA replication ATP-dependent helicase/nuclease DNA2 »
DSB de l’anglais « Double-strand break »
DSBR de l’anglais « Double-Strand Break Repair »
DSS1 de l’anglais « 6S proteasome complex subunit DSS1 »
DTT DiThioThréitole
EDTA de l’anglais « Ethylene Diamine Tetraacetic Acid »
eGFP de l’anglais « enhanced Green Fluorescent Protein »
EGTA de l’anglais « Ethylene Glycol Tetraacetic Acid »
EME1 de l’anglais « Essential Meiotic Endonuclease 1 »
EMSA de l’anglais « Electrophoretic Mobility Shift Assay »
EMSY Description des auteurs: « since the word ‘SISTER’ appears in the first line of
the protein sequence, we named this protein after the first author’s sister, who is a breast cancer nurse »
EOC de l’anglais « Epithelial Ovarian Cancer »
ERCC1 de l’anglais « DNA excision repair protein Excision Repair
Cross-Complementation group 1 »
EXO1 EXOnucléase 1
FA de l’anglais « Fanconi Anemia »
xv
J/M/N/O
FBH1 de l’anglais « F-box only protein 18 »
FBS de l’anglais « Fetal Bovine Serum »
FIGO de l’anglais « Federation International of Gynecology and Obstetrics »
FPC de l’anglais « Familial Pancreatic Cancer »
GAPDH glycéraldéhyde -3 - phosphate déshydrogénase
GDP Guanosine-5’-Diphosphate
GEN1 de l’anglais « Flap endonuclease GEN homolog 1 »
GFP de l’anglais « Green Fluorescent Protein »
GST Glutathion S-transférase
GTP Guanosine-5’-Triphosphate
H2AX de l’anglais « H2A Histone Family, Member X »
HAT de l’anglais « Histone AcetylTransferase »
HD de l’anglais « Helical Domain »
HDAC de l’anglais « Histone DeACetylase »
HEAT de l’anglais « Huntingtin, Elongation factor 3 (EF3), protein phosphatase 2A
(PP2A), and the yeast kinase TOR1 » HEK de l’anglais « Human Embryonic Kidney »
HeLa cellules provenant d’un carcinome humain du col de l’utérus d’une patiente
nommée Henrietta Lacks
HELB de l’anglais « Human DNA helicase B »
HEPES acide 4 - (2-hydroxyéthyl)-1 -pipérazine éthane sulfonique
HGS de l’anglais « High Grade Serous »
HIS Histidine
HJ de l’anglais « Holliday Junction »
HR de l’anglais « Homologous Recombination »
HTH de l’anglais « Helix-Turn-Helix »
HU Hydroxyurée
ICL de l’anglais « Interstrand crosslink »
IF Immunofluorescence
IL de l’anglais « interleukin »
IP Immunoprécipitation
IPTG IsoPropyl β-D-1-ThioGalactopyranoside
KCL de l’anglais « potassium chloride »
kDa kiloDalton
KPT Karyopharm
xvi
MBP de l’anglais « Maltose-binding protein »
MDC1 de l’anglais « Mediator of DNA Damage Checkpoint protein 1 »
MFI de l’anglais « Mean Fluorescent Intensity »
MMC Mitomycine C
MMEJ de l’anglais « Microhomology-Mediated End-Joining »
MMR de l’anglais « Mismatch repair »
MMS de l’anglais « Methyl Methane Sulfonate »
MOPS de l’anglais « 3-(N-morpholino)propanesulfonic acid »
MRE11 de l’anglais « Meiotic REcombination protein 11 »
MRG15 de l’anglais « (Mortality factor)-related gene on chromosome 15 »
MRN MRE11-RAD50-NBS1
MS de l’anglais « Mass Spectrometry »
MUS81 de l’anglais « MMS and UV Sensitive protein 81 »
MW de l’anglais « Molecular Weight »
NBS1 de l’anglais « Nijmegen Breakage Syndrome protein 1 »
NCS NéoCarzinoStatine
NER de l’anglais « Nucleotide Excision Repair »
NES de l’anglais « Nuclear Export Signal/Sequence »
NF de l’anglais « Nuclear Fraction »
NHEJ de l’anglais « Non homologous end joining »
NIPBL de l’anglais « Nipped-B-like »
NLS de l’anglais « Nuclear Localization Signal »
NMCII de l’anglais « Non-muscle Myosin IIC »
NMD de l’anglais « Nonsense-mediated mRNA decay »
NPC de l’anglais « Nuclear Pore Complex »
OB de l’anglais « Oligonucleotide/Oligosaccharide Binding-fold domain »
P/CAF de l’anglais « P300/CBP-associated factor »
p21 de l’anglais « Cyclin-dependent kinase inhibitor 1 »
P300 de l’anglais « E1A binding protein p300 (E1A = adenovirus early region 1A) »
p53 de l’anglais « cellular tumor antigen p53 or Tumor protein p53 »
PALB2 de l’anglais « Partner and Localizer of BRCA2 »
PARI de l’anglais « PCNA-associated recombination inhibitor »
PARP de l’anglais « Poly (ADP-Ribose) Polymerase »
PAXX paralogue de XRCC4 et XLF
PBS de l’anglais « phosphate buffered saline »
xvii
PI3K de l’anglais « Phosphoinositide 3-kinase »
PIKK de l’anglais « Phospho-Inositide 3-Kinase related Kinase »
PMSF de l’anglais « PhenylMethylSulfonyl Fluoride »
POL Polymérase
POL Q/X Polymérase Q/X
RAD de l’anglais « Radiation sensitive »
RAD50 de l’anglais « DNA repair protein RAD50 »
RAD51 de l’anglais « DNA repair protein 51 homolog »
RAD52 de l’anglais « DNA repair protein 52 homolog »
RAG de l’anglais « recombination-activating genes »
RecA/Q de l’anglais « Recombination protein A/Q »
RECQ5 de l’anglais « ATP-dependent DNA helicase Q5 »
RH Recombinaison Homologue
RI Rayonnements Ionisants
RING de l’anglais « Really Interesting New Gene »
RMI1 de l’anglais « RecQ-mediated genome instability protein 1 »
RNA de l’anglais « Ribonucleotidic acid »
RNF 138/168/8 de l’anglais « RiNg Finger protein 138/168/8 » ROS de l’anglais « Reactive oxygene species »
RPA de l’anglais « Replication protein A »
RT de l’anglais « Room temperature »
SA de l’anglais « Splayed Arm »
SAC de l’anglais « Spindle Assembly Checkpoint »
SDS DodécylSulfate de Sodium
SDSA de l’anglais « Synthesis-Dependent Strand Annealing »
SEM de l’anglais « Standard Error of the Mean »
SFR1 de l’anglais « Swi5-dependent recombination DNA repair protein 1 homolog »
shRNA de l’anglais « small hairpin RNA »
SINE de l’anglais « Small Inhibitors of Nuclear Export »
siRNA de l’anglais « small interfering RNA »
SLX1/SLX4 de l’anglais « Synthetic Lethal of unknown [X] function protein 1/4 »
SMC de l’anglais « Structure Maintenance of Chromosomes »
SS de l’anglais « Single-Strand »
SSA de l’anglais « Single-Strand Annealing »
SSBR de l’anglais « Single-Strand Break Repair »
xviii
TAP de l’anglais « Tandem Affinity Purification »
TBE Tris, Borate, EDTA
TBS de l’anglais « Tris-Buffered Salin »
TEV de l’anglais « Tobacco Etch Virus »
TGF de l’anglais « Transforming Growth Factor »
TLS de l’anglais « TransLesion Synthesis »
TMA de l’anglais « Tumor Tissue MicroArray »
TOPBP1 de l’anglais « Topoisomerase II-Binding Protein 1 »
TOP I/II/III de l’anglais « DNA topoisomerase 1/2/3 »
TREX de l’anglais « TRanscription/Export »
UBE2D de l’anglais « Ubiquitin conjugating enzyme E2 D2 »
UV UltraViolet
V(D)J de l’anglais « Variable, Diversity and Joining »
WAPL de l’anglais « Wings Apart-Like »
WB de l’anglais « Western Blot »
WD40 Répétition d’environ 40 acides aminés souvent terminée par le dipeptide
Tryptophane-Aspartate (W-D)
XLF de l’anglais « XRCC4-like factor »
XPF de l’anglais « Xeroderma Pigmentosum Group F-Complementing Protein »
XRCC 2/3/4 de l’anglais « X-ray Repair Cross-Complementing protein group 2/3/4 »
γ-H2AX H2AX phosphorylée sur la sérine 139
°C Degré Celcius
xix
Nobody gets justice. People only get good luck or bad luck. Orson Well The will to win, the desire to succeed, the urge to reach your full potential ... these are the keys that will unlock the door to personal excellence.
xx
C’est par ces quelques mots que nous avons l’occasion de remercier et célébrer ceux qui ont contribué à notre réussite. Je suis de ceux qui pensent qu’il faut être reconnaissant envers tous, car tous nous enseignent. Nous apprenons tous, tant des situations positives que négatives dont se compose notre vie. Je remercie donc par avance toutes les personnes qui de près comme de loin ont contribué à ma réussite actuelle.
Mon arrivée au Québec, mon évolution et mon épanouissement scientifique n’auraient pu être possibles sans la confiance du Dr Jean-Yves Masson. J’ai toujours été conscient que mon acceptation dans son laboratoire fut un pari risqué et c’est pour cette raison que j’ai essayé d’être (et espère avoir été) à la hauteur de ses attentes. Son esprit « canadien », convivial et chaleureux à la fois, compétiteur et travailleur aussi, m’a aidé à me façonner en tant que scientifique, mais également en tant qu’être humain. J’y ai appris une pléthore d’informations concernant la stabilité du génome, le cancer et la réparation de l’ADN, mais aussi concernant l’organisation, la prise de décision et l’avancée d’un projet. J’ai aussi pu mettre à profit mes compétences informatiques. En somme, le Dr Jean-Yves Masson a changé l’avenir qui m’était destiné pour un avenir dont certains envieraient. Je ne crois pas qu’il y ait assez de mots pour exprimer mes remerciements et ma gratitude au Dr Jean-Yves Masson. Puisse-t-il aussi être un exemple pour les chefs, chercheurs et autres, à accepter et donner leur chance à toutes les personnes, qui comme moi, se sentent parfois rejetées.
Mon acclimatation dans le laboratoire fut grandement facilitée par la gentillesse et la disponibilité de tous les membres du laboratoire Masson passés et présents. Je pense sans discernement aux pièces maitresses du laboratoire : Marie-Christine, Yan et Amélie. À Hélène présente à mes débuts, qui fut aussi d’une aide précieuse. Je pense aux étudiantes à la maitrise : Stéphanie qui fut d’une exemplarité incroyable ; Mariline, Anne-Marie et Maripier à qui je souhaite plein de courage et de bonne chance pour la suite de l’Aventure. Je pense au postdoctorant, Hemanta, compatriote de paillasse dont le calme est parfois contagieux. Je pense aux doctorants passés : Rémi, qui fut pour moi l’exemple à suivre tant sa capacité à travailler était infinie ; Ranjan et Jana, deux êtres travailleurs et bourrés de bonne volonté ; Marie-Michelle, une force de la nature ; Joris, Émilie et Denis, trois français totalement différents et à l’opposé de certains clichés préétablis. Finalement, je pense au doctorant qui a commencé l’aventure folle de thésards avec moi : Niraj. Bien que nos cultures soient à des années-lumière l’une de l’autre, j’y ai trouvé en sa personne le frère que je n’ai jamais eu : réconfortant et virevoltant, compétiteur et amical, il aura été un pilier durant tout mon doctorat. Nos discussions parfois tumultueuses ont été d’une aide
xxi
fois sincères et honnêtes.
Je souhaite remercier les différents membres du jury qui ont accepté de superviser et évaluer mon travail. Je remercie le centre de recherche en cancérologie qui m’a octroyé une année durant la prestigieuse bourse Luc Bélanger. Je remercie également Isabelle Brodeur pour les corrections apportées à mon manuscrit. Je remercie également toutes les personnes du centre de recherche qui grâce à leurs différentes implications ont su redonner un brin de soleil à mes journées.
En dehors des heures scientifiques, j’ai partagé quelques bons moments avec différentes personnes dont je remercie particulièrement pour m’avoir accepté dans leurs cercles d’amis respectifs. J’ai encore en mémoire certaines excursions américaines, certaines parties de tarot, mais aussi le partage de la culture et gastronomie québécoise. Je pense que vous vous reconnaitrez. Également et grâce à certains d’entre vous, je m’y suis découvert comme étant un fin cuisinier capable de faire une pièce montée, une bouillabaisse et diverses autres choses que je n’aurai jamais imaginées. Dans le même contexte, je souhaite remercier Niraj qui m’a permis de découvrir la culture indienne, la perception de la musique et bien d’autres. J’ai partagé énormément de choses et de moments avec lui et j’espère que nos chemins se croiseront à nouveau. Merci, mon Ami, et bon courage pour la suite ! Aussi, j’ai pris beaucoup de plaisir à jouer dans l’équipe de football/soccer du centre de recherche durant les 4 années de mon doctorat. Ce fut parfois le moment clé de ma semaine, celui qui permettait de relâcher la pression. J’aimerai aussi remercier ceux qui ont rendu cela possible lorsque je me trouvais encore en France : Benoît, Olivier, Josselin et Wilhelm. Merci pour votre aide précieuse, vos discussions et tout ce que vous m’avait apporté qui a toujours été important et parfois au détriment de vos vies respectives.
De façon plus intime, j’aimerais honorer ma famille : mes parents Dominique et Marie et mes deux sœurs Alicia et Mélanie. Sans eux, rien de tout et plus encore n’aurait été possible. Je vous dois tout, c’est grâce à vous que je suis ici aujourd’hui. Je n’oublie pas non plus tous les autres membres de ma famille ici ou ailleurs maintenant. Un indéfinissable Merci !
Finalement, je voudrais remercier celle avec qui tout débuta lors d’un congrès scientifique et avec qui je partage tout mon temps depuis maintenant presque trois ans : Véronique. Elle est et aura été cette personne qui lorsque vous la rencontrez plus rien n’a le même gout, tout est vécu différemment et devient possible.
Je vous souhaite une agréable lecture de cette thèse. Ma culture française vous conseillerait même de l’apprécier avec un bon verre de vin rouge.
xxii
Le laboratoire du Dr Jean-Yves Masson s’intéresse à la réparation de l’ADN et spécifiquement à mieux comprendre la réparation des cassures double-brin de l’ADN. C’est dans le but d’atteindre cet objectif principal que mes travaux de thèse se sont concentrés. La compréhension du rôle de chaque acteur au sein même de ce processus est primordiale. Cette compréhension est indispensable à la conception de traitements spécifiques ciblant les acteurs impliqués dans la réponse aux dommages de l’ADN. Ainsi, lors de mon arrivée au laboratoire il y a 4 années, j’ai eu pour objectif principal de caractériser spécifiquement les rôles d’APRIN, une nouvelle protéine interagissant avec BRCA2 et appartenant à un complexe impliqué dans la cohésion des chromatides sœurs, au sein de la recombinaison homologue. Le long voyage du doctorat m’a permis d’être impliqué dans l’écriture de différentes revues de littérature. J’ai finalement participé à un projet concernant une mutation spécifique de PALB2, un autre partenaire de BRCA2.
Ainsi, le Chapitre 1 de ce manuscrit constitue une introduction sur la réparation de l’ADN, la cohésine et l’impact d’une dérégulation des protéines de la réparation.
Le Chapitre 2 intitulé : « DNA Damage Signalling and Repair Inhibitors : The
Long-Sought-After Achilles’ Heel of Cancer » (Denis Velic*, Anthony M. Couturier*, Maria Tedim Ferreira,
Amélie Rodrigue, Guy G. Poirier, Fabrice Fleury et Jean-Yves Masson) correspond à une revue de littérature publiée dans le journal Biomolecules sur les inhibiteurs importants de la réponse aux dommages de l’ADN pouvant être utilisés pour lutter contre le cancer. Cette revue est intégrée dans cette thèse puisqu’elle complète parfaitement l’introduction de ce manuscrit. (* co- auteurs)
Parmi les partenaires de BRCA2, PALB2 est de loin de l’un des plus importants. Durant mon doctorat, nous avions décidé de faire une revue de littérature des mutations de PALB2, publiée dans le journal Biochemical Journal. Ce travail intitulé « Exploring the roles of PALB2 at the
crossroads of DNA repair and cancer » (Joris Pauty, Amélie Rodrigue, Anthony Couturier, Rémi
Buisson et Jean-Yves Masson) est présenté dans le Chapitre 3 de ce manuscrit. Quelques temps après la parution de cette revue, le gène PALB2 fut caractérisé comme un nouveau gène de susceptibilité au cancer du sein. L’intégration de cette revue est aussi un moyen d’introduire le chapitre 4 portant sur l’étude fonctionnelle de mutations spécifiques de PALB2.
xxiii
novel cancer mechanism using hidden nuclear export signal in the WD40 repeat motif » (Joris
Pauty*, Anthony M. Couturier*, Amélie Rodrigue, Marie-Christine Caron, Yan Coulombe, et Jean-Yves Masson). Ceux-ci initiés par Dr Joris Pauty, concerne la caractérisation biochimique de divers mutants de PALB2 et sont en révision dans le journal Nucleic Acids Research. L’intégration de ce
chapitre permet de faire un lien entre BRCA2, PALB2, la recombinaison homologue et APRIN. (*
co- auteurs).
Le Chapitre 5 présente le projet principal de mon doctorat. Ces travaux intitulés « Functions
for APRIN(PDS5B) in homologous recombination and in ovarian cancer prediction » (Anthony
M. Couturier, Hubert Fleury, Anne-Marie Patenaude, Victoria L. Bentley, Yan Coulombe, Amélie Rodrigue, Joshi Niraj, Joris Pauty, Graham Dellaire, Javier Di Noia, Anne-Marie Mes-Masson et Jean-Yves Masson) sont en cours de révision dans le journal Nucleic Acids Research.
Finalement, j’ai eu l’occasion de participer à divers autres projets au cours de mon doctorat, mais la présentation de ceux-ci dans ce manuscrit dévierait du but principal de ma thèse.
– J’ai participé à d’autres travaux sur la protéine PALB2, en collaboration avec le Dr Detlev Schindler, à travers lesquels j’ai caractérisé l’impact d’une mutation sur un des rôles de PALB2 dans la réparation de l’ADN. Néanmoins, la rédaction du manuscrit n’est pas encore terminée rendant la présentation de ces résultats prématurée.
– J’ai aussi pris part aux travaux de Niraj Joshi sur la protéine FANCD2 et dans lesquels j’y ai effectué des expériences biochimiques.
– Finalement, j’ai eu la chance de travailler en collaboration avec le Dr Stéphane Richard sur l’impact de la méthylation de la nucléase FEN1 par SET7. Dans ce projet, j’ai étudié la conséquence de cette modification sur l’activité in vitro de la protéine. Ces travaux ont nécessité la purification d’une dizaine de protéines.
1
Chapitre 1 : Introduction
Mes travaux de doctorat portent spécifiquement sur la cassure double-brin de l’ADN et sa réparation par recombinaison homologue (RH), et notamment sur des protéines impliquées dans cette réparation, dont une possède un rôle dans la cohésion des chromatides sœurs. Ainsi, cette introduction a d’abord pour but d’établir un lien entre le complexe cohésine, le cycle cellulaire et la réparation des CDB.
1.1
Les dommages de l’ADN
Au cours de la vie, les cellules transmettent leur information génétique via leur acide désoxyribonucléique (ADN). Ce dernier doit être pertinemment dupliqué pendant la phase S, et protégé pendant la phase G2 du cycle cellulaire. Cette duplication va donner naissance aux chromatides sœurs qui, durant la mitose (M), seront séparées et réparties dans chaque cellule fille. Le bon fonctionnement de ce processus repose, avant tout, sur un point crucial : la cohésion des chromatides sœurs. Un des acteurs principaux impliqués dans la cohésion des chromatides est le complexe cohésine. Cette capacité qu’ont les cellules à pouvoir « attraper » et garder à proximité deux molécules d’ADN ayant la même composition est aussi utilisée pour lutter contre l’instabilité génomique. Cette instabilité est souvent la conséquence de dommages de l’ADN dont les causes sont toutes aussi riches que variées [1, 2].
Notre ADN est la cible constante de divers agents et réactions qui vont changer sa composition chimique. Ces dommages peuvent être spontanés ou engendrés par le métabolisme cellulaire, par exemple par une mauvaise incorporation d’un nucléotide durant la réplication, une modification erronée d’une base, soit par une déamination, dépurination, alkylation ou par les espèces oxydatives de l’oxygène. Il existe aussi plusieurs facteurs environnementaux qui peuvent contribuer à endommager l’ADN. C’est le cas des ultraviolets du soleil qui vont promouvoir les liens entre deux bases pyrimidines covalentes, formant ce qu’on appelle un dimère de thymines.
Ces défauts, si non réparés, peuvent conduire à l’apparition de substitutions ou délétions après la réplication. Certains, comme les dimères de thymines, peuvent même bloquer la machinerie réplicative. D’ailleurs, dans un autre contexte, la machinerie réplicative bien que réputée pour sa grande efficacité peut elle même introduire des erreurs, via, par exemple, l’incorporation de mauvais nucléotides. D’autres causes peuvent être très dangereuses, comme les radiations ionisantes et les agents chimiques génotoxiques qui vont produire une variété de lésions de l’ADN passant par la modification de bases, la formation de pontages inter/intrabrins, les cassures
simple-2
brin (CSB) et double-brin de l’ADN (CDB). Ainsi, il existe un très grand nombre de causes pouvant altérer l’ADN. Ces causes et dommages de l’ADN sont détaillés de façon schématique dans la
Figure 1.1.
Il a été estimé que chaque jour une cellule pourrait subir environ 100 000 lésions de son ADN provoquées par des causes endogènes ou exogènes [3], et que 50 CDB pouvaient avoir lieu par cycle cellulaire [4].
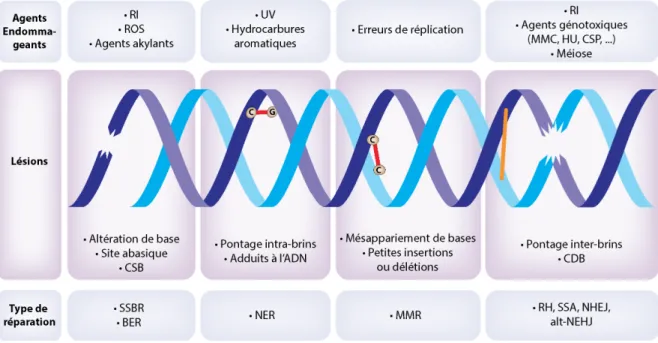
Figure 1.1 : Les sources de dommages à l’ADN
Divers types de dommages de l’ADN peuvent se produire dans une cellule. Ces lésions sont la conséquence d’agents endommageant spécifiques. Ces dommages vont être détectés et réparés suivant des voies de réparation différentes et spécifiques. RI : Rayonnements ionisants ; ROS : Espèce réactive de l’oxygène ; CSB : Cassure brin, SSBR : Réparation des cassures simple-brin ; BER : Réparation par excision de bases ; UV : Ultraviolet ; ADN : Acide désoxyribonucléique ; NER : Réparation par excision de nucléotides ; MMR : Réparation de mésappariement de bases ; MMC : Mitomycine C ; HU : Hydroxyurée; CDDP : Cisplatine ; CDB : Cassure double-brin ; RH : Recombinaison homologue ; SSA : Appariement simple-brin ; NHEJ : jonction classique des extrémités non homologues ; Alt-NHEJ : jonction alternative des extrémités non homologues. Illustration par Anthony M. Couturier.
3
Suivant le type de dommage engendré et/ou la phase du cycle cellulaire, la cellule va mettre en place un système de réparation spécifique. De façon générale, les CSB seront réparées par la voie de réparation des CSB (SSBR, « Single-Strand Break Repair »). Les petites modifications de bases seront corrigées par la voie de l’excision de bases (BER, « Base Excision Repair ») alors que les altérations plus complexes le seront par la voie d’excision de nucléotides (NER, « Nucleotide
Excision Repair ») (Figure 1.1). Les mésappariements de bases seront corrigés par la voie des
« mauvais assortiments » (« MisMatch Repair », MMR).
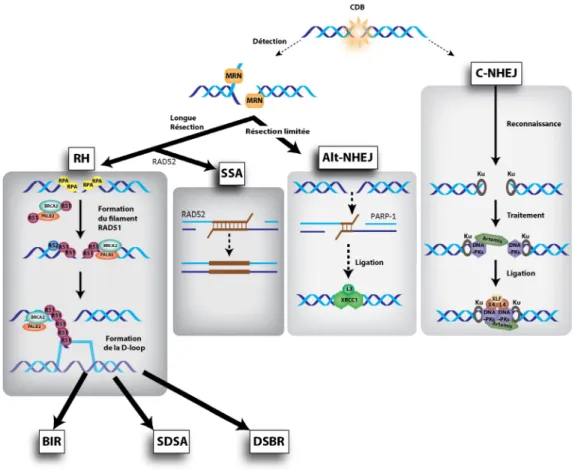
La CDB est l’une des lésions les plus délétères et doit être précisément réparée. Au moins quatre voies différentes peuvent réparer cette lésion : (i) la RH ; (ii) la jonction des extrémités non homologues (communément appelée NHEJ, « Non-homologous End-Joining ») ; (iii) le NHEJ alternatif (ou Alt-NHEJ) et (iv) l’appariement des extrémités simple-brin d’ADN (communément appelé SSA, « Single-Strand Annealing ») [2].
Durant la réparation des CDB, la cohésion et les protéines importantes à son bon
fonctionnement sont mises en jeu. Lorsque cette cassure est non ou mal réparée, elle peut avoir des conséquences dévastatrices pour la cellule. Par exemple, la présence de grandes délétions ou de réarrangements géniques qui peuvent mener à l’apparition de cancers [2]. La plus grande partie de notre savoir sur cette réponse aux dommages de l’ADN (DDR, « DNA Damage Response ») provient de l’étude des maladies causées par des défauts dans les voies de signalisation et de réparation des dommages à l’ADN. Ainsi, la caractérisation de tous les acteurs mis en jeu permet une meilleure compréhension des mécanismes de réparation, mais aussi de l’apparition de certaines maladies et leurs soins.
4
1.2
Le cycle cellulaire
Les cellules vivantes ont la capacité de se dupliquer. Cet évènement qui consiste en la séparation d’une cellule en deux suit des étapes précises, regroupées sous le terme de cycle cellulaire. Ce dernier est divisé en quatre phases (Figure 1.2). La réplication du matériel génétique se déroule pendant la phase S (Synthèse) et sa ségrégation en deux molécules d’ADN a lieu pendant la phase M (Mitotique). Les phases S et M sont temporairement séparées par deux intervalles, ou deux phases G (Gap) : les phases G1 et G2. Durant ces phases G1 et G2, la cellule croît, se prépare et arrive à maturité respectivement pendant les phases S et M [5]. Durant chaque cycle cellulaire, les chromosomes doivent donc être copiés de façon parfaite et en un seul exemplaire pour être équitablement distribués à chaque cellule fille. Ce processus repose en grande partie sur la cohésion des chromatides sœurs, contrôlée par le complexe cohésine. Le dépôt de la cohésine et son état de cohésion sont finement régulés, spatialement et temporellement, durant le cycle cellulaire.
Figure 1.2 : Le cycle cellulaire et les points de contrôle
Le cycle cellulaire est composé de 4 phases spécifiques et 4 points de contrôle importants. G1 : Gap 1 ; G2 : Gap 2 ; S : Synthèse ; M : Mitose. Illustration par Anthony M. Couturier.
Chez les eucaryotes, le cycle cellulaire est contrôlé par un réseau protéique de régulation. Ce processus consiste en la vérification du bon déroulement d’évènements spécifiques et dans un ordre bien défini. Ce réseau est composé principalement de cyclines et de kinases dépendantes des cyclines (CDK « cycline dependant kinase »). Comme la cellule est une entité plus perspicace qu’on ne le croit, elle a aussi mis en place un réseau de protéines permettant de contrôler cette régulation. Il existe deux aspects importants dans cette régulation : les points de contrôle (l’anglicisme « checkpoint » est souvent utilisé) des structures de l’ADN, qui peuvent arrêter le cycle cellulaire suite à un dommage de l’ADN ou une réplication incomplète ; et un point appelé le « point de restriction » qui est défini comme le point après lequel une cellule devient apte à entrer dans le cycle cellulaire et y progresser indépendamment des signaux extérieurs. L’importance de ces
5
checkpoints et du point de restriction pour le bon déroulement du cycle cellulaire est mise en avant
par la très grande fréquence de mutations touchant les acteurs de cette régulation lors de cancers. Ces mutations étant bien souvent le fruit d’une mauvaise gestion de ces mêmes checkpoints ou autres protéines régulatrices.
La réplication de l’ADN et sa réparation sont finement régulées durant le cycle cellulaire grâce aux points de contrôle. Il existe 4 points de contrôle (Figure 1.2) : le point de contrôle G1/S ou point de restriction, qui permet aux cellules de contrôler si les toutes les conditions sont remplies pour entamer la réplication ; le point de contrôle intra-S, qui permet d’arrêter la réplication si un dommage est détecté et ainsi permet à la cellule de se protéger contre des agressions extérieures ou provenant de la cellule elle-même ; le point de contrôle G2/M, qui permet de s’assurer que l’ADN a été dument répliqué ; et le point de contrôle M ou point de contrôle de l’assemblage du fuseau, qui arrête le cycle cellulaire si le fuseau mitotique n’est pas convenablement assemblé [6, 7].
6
1.3
La détection d’une lésion de l’ADN
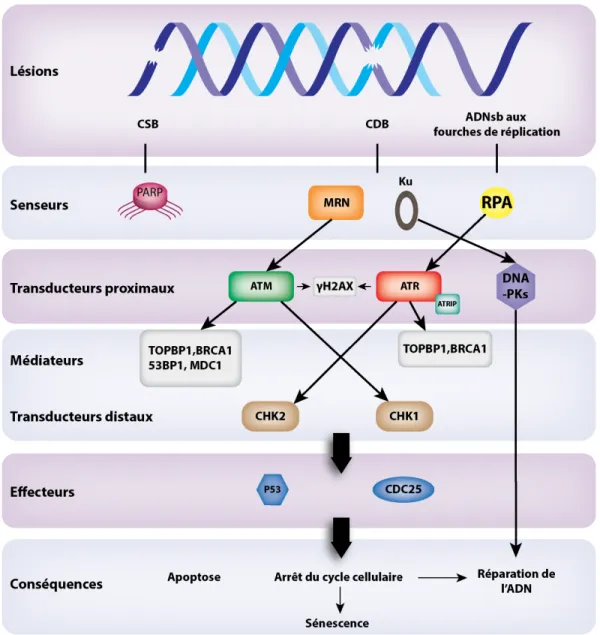
Lors d’une lésion de l’ADN, une réponse va être mise en place impliquant trois types d’acteurs différents : les senseurs, les transducteurs et les effecteurs-médiateurs [8] (Figure 1.3).
§ Les senseurs :
Dans une première étape, la cellule va détecter la lésion de l’ADN grâce à différents senseurs liant l’ADN [9]. Le complexe MRN (Mre11/Rad50/NBS1) va permettre la détection des cassures double-brin de l’ADN [10]. Ce complexe est parfois en compétition avec le complexe Ku70/80 pour la détection d’une CDB. La protéine PARP (« Poly (ADP-Ribose) Polymerase ») agit en tant que senseur principal des CSB. Finalement, RPA (« Replication Protein A ») va lier les régions d’ADN simple-brin (ADNsb) qui sont exposées au niveau des fourches de réplication bloquées ou après la résection [11]. Ces senseurs ont généralement pour rôle de commencer la signalisation spécifique, mais aussi de diffuser le signal.
§ Les transducteurs :
Les transducteurs sont des kinases spécifiques. On retrouve des kinases apicales (ou proximales) qui transmettent le signal des senseurs et des kinases distales qui transmettent le signal des kinases apicales.
Les kinases apicales sont les protéines ATM (« Ataxia Telangiectasia Mutated ») et ATR
(« ATM and Rad3 related ») appartenant à la famille des PIKK (« Phospho-Inositide 3-Kinase
related Kinases »), impliquées dans la régulation des points de contrôle [12] et permettant la
réparation. ATR est recrutée par son partenaire ATRIP (« ATR-Interacting Protein ») qui reconnait RPA couplé à l’ADN. Finalement, DNA-PK (« DNA-dependent Protein Kinase ») est aussi considérée comme une protéine kinase apicale qui va transmettre le signal du senseur Ku.
Les kinases distales sont CHK1 et CHK2 (« Checkpoint kinase 1 and 2 ») [8]. On considère
que CHK1 est plus souvent phosphorylée par ATR alors que CHK2 par ATM. L’activation de ces kinases va permettre la mise en place des différents points de contrôle G1/S, intra-S et G2/M. Ces kinases sont capables d’initier les mécanismes de réparation de l’ADN, mais aussi, dans le cas d’une activation prolongée du point de contrôle, d’initier l’apoptose.
D’autres protéines transductrices majeures caractérisées sous le terme de médiateurs, comme 53BP1 (« p53-Binding Protein 1 »), MDC1 (« Mediator of DNA Damage Checkpoint protein 1 »), TOPBP1 (« Topoisomerase II-Binding Protein 1 »), BRCA1 (« BReast CAncer 1, early onset »), participent dans le choix des voies distinctes de réparation [8].
7 § Les effecteurs :
L’activation des kinases distales peut amener l’activation d’effecteurs nécessaire pour engendrer la réponse cellulaire aux dommages, comme p53 et la phosphatase CDC25 (« Cell
Division Cycle 25 »), pouvant entrainer dans le cas ou la lésion n’est pas réparée soit la mort
cellulaire par apoptose ou la sénescence [13].
Figure 1.3 : La réponse aux dommages l’ADN
Cette réponse va mettre en jeu divers acteurs importants. La lésion de l’ADN va être détectée par des senseurs. Ces senseurs vont recruter des transducteurs proximaux responsables de la diffusion du signal. Ces derniers vont être responsables de la phosphorylation de l’histone H2A. Le signal va ensuite être transmis à des médiateurs responsables du choix de réparation, mais aussi à des transducteurs distaux responsables de la transmission du signal à divers effecteurs impliqués dans la mort cellulaire par apoptose, l’arrêt du cycle cellulaire ou la sénescence. CSB : Cassure simple-brin ; CDB : Cassure double-simple-brin ; ADNsb : ADN simple-simple-brin ; Ku : Ku70/80. Illustration par Anthony M. Couturier.
8
1.4
Les premières étapes de détection d’une cassure
double-brin de l’ADN
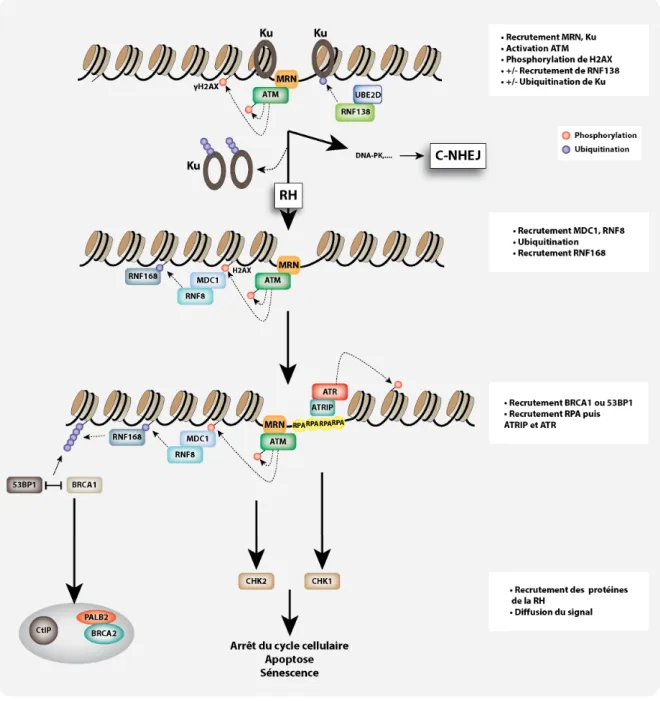
La détection et la réponse à une CDB sont en grande partie dues à l’action de la protéine ATM. Cette protéine kinase de la famille des PI3K est activée aux sites de dommages par son interaction avec le complexe MRN, généralement un des premiers acteurs à y être recruté [14].
Comme précédemment dit, la CDB va être détectée par les complexes Ku ou MRN. En réalité, il semblerait que ces complexes soient recrutés au même moment, mais qu’une compétition finement régulée soit mise en jeu afin de se diriger vers un mécanisme de réparation précis (RH ou NHEJ). Récemment, deux études ont montré qu’une E3 ubiquitine ligase, RNF138 (« RiNg Finger
protein 138 »), était également recrutée au niveau d’une CDB où elle ubiquitine des facteurs clés
permettant ainsi de promouvoir la RH [15, 16]. En présence d’un facteur spécifique (UBE2D, « Ubiquitin conjugating enzyme E2 D2 »), RNF138 ubiquitine le complexe Ku permettant possiblement son éviction de la CDB entrainant ainsi la réparation par RH (Figure 1.4).
Lorsque MRN est recruté au niveau d’une CDB, il va permettre l’activation de la protéine ATM. Ensuite, une des premières cibles de la kinase ATM est le variant d’histone H2AX (« H2A
Histone Family, Member X ») qui va être phosphorylé sur sa sérine 139 et donner naissance à
γ-H2AX. Dans un même élan, cette protéine est reconnue par MDC1 (« Mediator of DNA damage
checkpoint protein 1 ») [17] (Figure 1.4). Cette dernière possède deux rôles importants : le
recrutement de molécules supplémentaires ATM et MRN pour permettre la diffusion du signal aux abords de la CDB ; et le recrutement de RNF8, une E3 ubiquitine ligase qui reconnait la forme MDC1 phosphorylée par ATM [18]. RNF8 est indispensable au recrutement d’une autre E3 ubiquitine ligase, RNF168, qui va reconnaitre les produits d’ubiquitination de RNF8. Ces deux protéines amplifient l’ubiquitination des histones et permettent le recrutement de plusieurs autres facteurs [19]. Parmi ces facteurs, les protéines 53BP1 et BRCA1-RAP80 impliquées chacune dans des voies de réparation des CDB différentes [20] (voir plus loin pour plus de renseignements). La kinase ATR est, quant à elle, recrutée lorsque l’ADN simple-brin formé au cours de la réparation est recouvert par RPA et détecté par le partenaire de ATR, ATRIP. Finalement, ATM et ATR vont ensuite phosphoryler diverses cibles permettant l’arrêt du cycle cellulaire, l’entrée en sénescence, voire l’apoptose [21] (Figure 1.4).
9
Figure 1.4 : Les premières étapes de détection d’une cassure double-brin de l’ADN
La détection de la cassure double-brin met en jeu diverses protéines entrainant une cascade de réactions (plus de détails sont fournis dans le texte). Ku : Ku70/80. Illustration par Anthony M. Couturier.
10
1.5
Les cassures double-brin de l’ADN
1.5.1 Bref aperçu
Une cassure double-brin est définie comme la cassure simultanée deux brins complémentaires de l’ADN. Ceci en fait une des formes de lésions de l’ADN les plus cytotoxiques. Les sources des CDB sont nombreuses et on en distingue de types endogènes et exogènes. Les erreurs dans le métabolisme de l’ADN (réplication lors d’une cassure simple-brin, fourche de réplication bloquée), les nucléases endogènes, les réarrangements géniques programmés, les forces physiques ou les dérivés réactifs de l’oxygène (ROS, « Reactive oxygene species ») produits lors de la respiration oxydative sont autant de sources endogènes pouvant provoquer la formation de CDB (Figure 1.1). Les sources de types exogènes, quant à elles, peuvent être naturelles : les rayons cosmiques (provenant de l’univers et du soleil et bombardant continuellement la terre), les radiations naturelles terrestres (provenant de différents dépôts naturels d’uranium, potassium et thorium) ou même un virus [22]. D’autres sources exogènes peuvent aussi être produites par l’homme. C’est le cas des armes de destruction massive, de la radiographie, de la radiothérapie (radiations ionisantes), des drogues génotoxiques ou radiomimétiques (thérapie anticancer). L’homme peut aussi induire la production de CDB grâce à l’utilisation de diverses stratégies (I-SceI, CRISPR-Cas9…). Toutes ces causes engendrent des CDB non programmées qui peuvent être très néfastes pour la cellule si elles restent non réparées. La cellule peut elle aussi provoquer des CDB volontairement durant divers mécanismes. Ces cassures, souvent bénéfiques et très importantes pour le fonctionnement
cellulaire, seront discutées dans la section 1.5.2.
La réparation des CDB met en jeu l’implication de cohésion et de protéines importantes à son fonctionnement. La plus grande partie de notre savoir sur cette réponse aux dommages de l’ADN (DDR, « DNA Damage Response ») provient de l’étude des maladies causées par des défauts dans les voies de signalisation et de réparation des dommages à l’ADN. Ainsi, la caractérisation de tous les acteurs mis en jeu dans cette réponse permet une meilleure compréhension des mécanismes, mais aussi de l’apparition de certaines maladies et leur guérison.
1.5.2 Les cassures double-brin bénéfiques
Les CDB bénéfiques sont celles dont la cellule se sert pour son propre fonctionnement. Un exemple de CDB bénéfique est la CDB méiotique, entrainant l’apparition d’enjambements («
cross-overs ») et cruciale pour la ségrégation des chromosomes homologues provenant de chaque parent
11
acteurs majeurs dans l’apparition des CDB pendant la méiose. SPO11 est aussi impliquée dans l’apparition de CDB dans les neurones de souris, ce rôle reste néanmoins à être mieux étudié [25].
Durant le cycle cellulaire, des CDB peuvent se créer grâce à l’action des topoisomérases (TOPI et TOPII chez les eucaryotes). Ces enzymes ubiquitaires permettent de renverser un état de superhélice de l’ADN (hélice positive ou négative chez les eucaryotes). Cette action joue un rôle prépondérant dans la réplication, la recombinaison, la ségrégation des chromosomes et leur transcription.
Des CDB sont aussi produites pour initier la recombinaison V(D)J nécessaire à l’assemblage des gènes produisant les divers récepteurs immunoglobulines et les récepteurs des cellules T [26]. Les réarrangements géniques des immunoglobulines sont indispensables pour créer une diversité de récepteurs antigéniques et ne sont possibles que grâce à l’utilisation de CDB [27]. Les extrémités d’ADN produites vont être liées entre elles grâce à la réparation par NHEJ (qui sera décrite à la
section 1.7).
Finalement, la commutation de classe (CSR, « Class Switch Recombination ») est un type de réarrangement génique se produisant seulement dans les cellules B matures. Ce processus permet d’échanger le fragment d’ADN codant pour la forme IgM avec d’autres régions présentent sur le même gène et codant pour d’autres classes d’anticorps (IgG, IgE, IgA) [28] (Figure 1.5). Les cellules murines B de lymphome (CH12F3) sont souvent utilisées pour étudier ce processus, car elles présentent de hauts taux de recombinaison pour l’IgA [29].
Figure 1.5 : Le mécanisme de commutation de classe
Schéma représentant la commutation de classe (CSR). Plus de détails sont fournis dans le texte. Ss : région de changement, AID : Activation-induced cytidine deaminase. Ig : Immunoglobuline. Illustration par Anthony M. Couturier.