Effets de l’isolement des tourbières sur les variations
génétiques et phénotypiques entre les populations de
l’orchidée Platanthera blephariglottis.
Mémoire
Laurent De Vriendt
Maîtrise en biologie végétale
Maître ès sciences (M.Sc.)
Résumé
Cette étude visait à caractériser la connectivité des tourbières du Québec méridional en analysant la variabilité phénotypique et génétique des populations de l’orchidée Platanthera blephariglottis. Quinze traits phénotypiques furent mesurés sur 24 individus dans 20 populations et des tests de germination ont été réalisés. Les analyses génétiques ont été effectuées suivant un protocole de « Genotyping-By-Sequencing ». L’isolement des populations fut évalué via la distance à la population la plus proche et le nombre de populations présentes dans différents rayons autour des sites. Des différences entre les populations ont été trouvées pour tous les traits phénotypiques étudiés et pour les taux de germination. La variation de plusieurs traits phénotypiques et une baisse des taux de germination étaient corrélées à l’isolement. La structure génétique des populations présentait un patron d’isolement par distance, signifiant la présence d’échanges entre populations rapprochées. Les tourbières constituent ainsi un réseau dynamique d’écosystèmes interconnectés pour P. blephariglottis.
Abstract
The aim of this study was to characterize the connectivity of peatlands in southern Quebec using phenotypic and genetic variability among populations of the orchid Platanthera blephariglottis. Fifteen phenotypic traits were measured on 24 individuals in 20 populations and germination trials were conducted. Genetic analyses were realised through a Genotyping-By-Sequencing protocol. Isolation of populations was evaluated by the distance to the nearest population and by the number of populations occurring within different buffers around studied sites. Differences were found between populations for all phenotypic traits and several of these differences were correlated with isolation. A diminished germination was also correlated to increased isolation. The genetic structure of populations followed a pattern of Isolation-By-Distance (IBD), meaning that exchanges of propagules occur between close populations. We can thus consider peatlands as a dynamic network of interconnected patches for P. blephariglottis.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux ... ix
Liste des figures ... xi
Remerciements ... xv
Avant-propos ... xvii
Chapitre 1 Introduction générale ... 1
1.0 Problématique ... 1
1.1 Conséquences de la fragmentation ... 1
1.2 Espèces rares et écosystèmes naturellement fragmentés ... 3
1.3 Les tourbières ... 5
1.4 La platanthère à gorge frangée ... 7
1.5 Objectifs et hypothèses ... 8
Chapitre 2 How connected are populations of the peatland orchid Platanthera blephariglottis, as revealed by genetic and phenotypic traits variation. ... 11
2.1 Résumé ... 11 2.2 Abstract ... 12 2.3 Introduction ... 12 2.4 Methods ... 16 2.4.1 Study area ... 16 2.4.2 Sampling ... 16 2.4.3 Germination tests ... 17 2.4.4 Isolation measures ... 18
2.4.5 DNA extraction, libraries preparation and sequencing ... 19
2.4.6 Processing of raw sequence read data ... 19
2.4.7 Statistical analysis ... 20
2.4.8 Genetic analyses ... 20
2.5 Results ... 21
2.5.1 Differences among populations: phenotype and germination ... 21
2.5.2 Sequencing results ... 22
2.5.3 Differences among populations: genetic grouping ... 23
2.5.4 Effect of population isolation: phenotype, germination and genetic ... 26
2.6 Discussion ... 28
viii
2.6.2 Genetic evidences of connectivity ... 30
2.6.3 Further considerations ... 31
Chapitre 3 Conclusion générale ... 33
Références ... 37
Liste des tableaux
Tableau 1 Liste des traits phénotypiques étudiés et résultats des analyses en composantes
principales (ACP). ... 23 Tableau 2 Résultats des analyses Mixtes de l’effet de l’isolement sur les variations
phénotypiques et sur la différentiation génétique des populations. ... 27 Tableau 3 Résultats des tests de Mantel entre les différentiations génétiques et phénotypiques
Liste des figures
Figure 1 Carte de la zone étudiée incluant la localisation des populations échantillonnées, des populations non-échantillonnées et des regroupements génétiques des populations. ... 17 Figure 2 Réseau phylogénétique et phénogramme (UPGMA-clustering) montrant les
À tous ceux et celles pour qui la nature n’est pas un acquis mais une responsabilité
Remerciements
Mes premiers remerciements vont à ma directrice, Monique Poulin, pour son inestimable aide, pour ses précieux conseils et suggestions, ses encouragements soutenus, sa compréhension lors des moments difficiles et pour m’avoir toujours supporté lors des prises de décisions. Merci également à mes co-directeurs, Stéphanie Pellerin et Simon Joly, pour votre aide générale dans le projet, de sa planification à sa rédaction, mais aussi pour votre enthousiasme et votre disponibilité tout au long du projet. À vous trois, vous avez vraiment été une merveilleuse équipe de directeurs! Merci!
Je dois ensuite remercier François Belzile sans qui l’utilisation de technologies dernier cri en génétique n’aurait pas été possible. Je suis vraiment content d’avoir pu travailler dans votre laboratoire. Merci également à Martine Jean qui m’a supervisé lors de mes travaux en laboratoire de génétique et qui m’a montré que la bioinformatique ce n’est pas si pire! Merci à Sébastien Bélanger de m’avoir montré de A à Z les secrets de l’extraction d’ADN chez les plantes. Vraiment, j’ai été choyé d’être si bien entouré lors de mon bref séjour dans le labo Belzile.
Je remercie également Marc-André Lemay, pour son travail acharné lors de l’échantillonnage. On en a marché des tourbières au gros soleil! Merci d’avoir été toujours si enthousiaste et de m’avoir fait part de tes idées et de tes connaissances sur les plantes. Sans oublier tes expériences sur la germination des graines d’orchidée, félicitations et un gros merci!
Je remercie tous les gens du labo Poulin, ceux et celles qui étaient là à mon arrivée tout comme ceux et celles qui m’accompagnent dans la fin de ma rédaction. Ce fut un plaisir de partager avec vous tous ces moments sérieux et moins sérieux. Un merci spécial à Audrey Comtois pour son coup de pouce sur le terrain!
Je dois bien sûr remercier ma famille et mes amis qui m’ont poussé à faire un retour aux études et qui m’ont soutenu du début du bac jusqu’à la fin de la maîtrise. Merci à mes parents, à mon frère et mes sœurs, de perpétuellement croire en moi, savoir m’aider et me
xvi
faire voir le positif même durant les moments les plus durs et de partager avec moi les joies de la réussite et des bons coups. Merci à tous mes amis et amies de m’avoir fait décrocher quand j’en avais besoin et m’avoir changé les idées. C’est vraiment rafraichissant de vous savoir avec moi et ce fut indispensable à la réussite de ce projet!
Merci à tous mes partenaires financiers, sans qui rien de tout ceci n’aurait pu être possible, soit le Jardin Botanique de Montréal et le Centre de la Science de la Biodiversité du Québec (CSBQ). J’ai également bénéficié d’une bourse d’études du programme de Bourses en Milieu de Pratique (BMP Innovation) du Fonds de recherche du Québec sur la nature et les technologies (FRQNT) et du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG). Finalement, merci à Hélène Crépeau pour son assistance et ses conseils en analyses statistiques et merci à tous ceux et celles qui ont contribué de près ou loin à la réussite de ce projet.
Avant-propos
Ce projet d’étude de la connectivité des tourbières du Québec méridional a initialement été pensé et développé par Monique Poulin, Stéphanie Pellerin ainsi que par l’auteur de ce mémoire. Les données récentes de présence des populations de l’espèce étudiée ainsi que de nombreuses données sur les composantes environnementales entourant ces populations furent fournies par Stéphanie Pellerin et son ancien étudiant Vincent Laroche. Simon Joly a rapidement été incorporé au projet afin d’y ajouter son expertise en génétique des populations, laquelle a entre autres aidé à la sélection des sites d’études ainsi qu’à l’analyse et l’interprétation des résultats. François Belzile et Martine Jean ont rendu le volet génétique possible grâce à leur expertise en séquençage « Genotyping-by-Sequencing » et leurs installations. Marc-André Lemay a réalisé les expériences de germination, nous permettant d’avoir des données relatives au fitness des populations. Hélène Crépeau a apporté son expertise en statistiques, autant pour les analyses que pour la rédaction de la méthodologie. Le développement général du projet, la planification et la réalisation de l’échantillonnage, la caractérisation de l’isolement des sites, les extractions d’ADN et la préparation des « pools » génétiques, l’analyse statistique de l’ensemble des données, l’interprétation des résultats et la rédaction du présent mémoire, incluant tous les tableaux et figures, ont été exécutés par l’auteur du présent mémoire.
Le deuxième chapitre de ce mémoire a été rédigé sous la forme d’un article scientifique. Cet article a été principalement rédigé par l’auteur de ce mémoire avec l’aide et la collaboration de Monique Poulin, Stéphanie Pellerin, Simon Joly, François Belzile, Martine Jean et Marc-André Lemay. Cet article a été écrit en anglais afin d’être publié dans une revue scientifique de calibre international.
Chapitre 1 Introduction générale
1.0 Problématique
La baisse de la biodiversité à l’échelle planétaire est un des grands enjeux de notre temps. Le rythme de disparition des espèces s’accroît si rapidement que l’idée d’une sixième extinction de masse est de plus en plus acceptée (Barnosky et al. 2011; Dirzo et al. 2014). Bien que de nombreuses causes soient impliquées, la destruction des habitats naturels demeure l’une des principales (Krauss et al. 2010; Pimm and Raven 2000; Sala et al. 2000). Dans la plupart des cas, cette destruction se présente sous la forme d’une fragmentation, soit la transformation d’une grande surface d’habitat en plusieurs petites aires dont la somme est inférieure à l’aire d’origine et dont la connectivité varie selon la distance géographique entre les aires résiduelles et la nature de la matrice environnante. Pour les plantes, il est généralement admis que la fragmentation implique une diminution directe de la taille effective des populations ainsi qu’une augmentation de l’isolement des fragments restants (Fahrig 2003). Les conséquences génétiques de l’isolement sont multiples et peuvent conduire à l’extinction des populations (Aguilar et al. 2008; Frankham 2005; Newman et al. 2013). Néanmoins, certaines espèces prospèrent au sein d’écosystèmes naturellement fragmentés, tels que les tourbières. Afin de protéger convenablement ces milieux, il convient de savoir si ceux-ci représentent un archipel d’écosystèmes isolés les uns des autres ou, à l’opposé, un réseau dynamique où il y a des échanges de propagules entre les sites. En effet, dans le cas où des échanges seraient présents entre les populations végétales de tourbières, la destruction de certains sites pourrait accroître les distances à franchir et réduire les échanges fructueux, menant ainsi aux conséquences néfastes de l’isolement chez les populations restantes.
1.1 Conséquences de la fragmentation
La fragmentation de l’habitat ou sa dégradation peuvent mener à l’isolement des populations végétales et à une diminution de leur abondance, autant dans le paysage que localement (Fahrig 2003). Un des effets immédiats suivant cette perte d’individus est souvent un goulot d’étranglement génétique et donc, une réduction de la variation génétique au sein des populations (Ellstrand and Elam 1993; Nei et al. 1975; Young et al.
2
1996). La dépression endogamique et la dérive génétique réduiront davantage la diversité génétique, conduisant éventuellement à la fixation d’allèles récessifs. Ceux-ci diminueront la fécondité des individus, augmenteront la mortalité des graines et des jeunes plants et diminueront le recrutement de nouveau individus dans la population, réduisant ainsi le potentiel évolutif des populations et augmentant leur risque d’extinction (Aguilar et al. 2006; Aguilar et al. 2008; Ellstrand and Elam 1993; Frankham 2005; Leimu et al. 2006; Lowe et al. 2005; Newman et al. 2013; Young et al. 1996). Cet enchainement de conséquences dues à l’isolation d’une population et menant à son extinction fut baptisé « extinction vortex » par Soulé and Mills (1998). Les effets de la fragmentation sont toutefois complexes et l’érosion génétique des populations n’est pas toujours observée suite à la fragmentation (Lowe et al. 2005; Young et al. 1996).
La connectivité entre les fragments restants peut permettre de contrer les conséquences négatives de l’isolement (Brudvig et al. 2009; Martensen et al. 2008; Neel 2008; Staddon et al. 2010). Chez les plantes, cette connectivité, permettant les échanges génétiques entre populations, est principalement réalisée via le transport du pollen et des graines. Conséquemment, l’impact de la fragmentation sur la connectivité est lié à l’impact sur les vecteurs de dispersion. Dans le cas d’un mutualisme plante-pollinisateur, la matrice environnante résultant de la fragmentation a souvent un effet négatif sur la reproduction des plantes, réduisant les taux de pollinisation et de production de fruits (Aizen and Feinsinger 1994; Cunningham 2000). La transformation de la matrice du paysage peut cependant engendrer simultanément des effets négatifs et positifs. Par exemple, l’expansion des terres agricoles peut nuire au déplacement des pollinisateurs entre les fragments (Krewenka et al. 2011; Llorens et al. 2012) tout en augmentant la diversité totale de pollinisateurs, bien que celle-ci soit souvent dominée par des espèces généralistes (Winfree et al. 2007). Dans le cas d’une espèce dont le pollen est dispersé par le vent, les aires ouvertes favorisent généralement la connectivité (Culley et al. 2002), tel que démontré chez Acer saccharum Marsh., pour qui les champs agricoles augmentent la connectivité entre individus comparativement à la forêt continue (Young et al. 1993). De manière générale, les espèces dont le pollen est dispersé par des animaux ou dont les graines sont transportées par des animaux sont considérées plus vulnérables à la fragmentation que les espèces dépendantes
des vecteurs abiotiques pour la dispersion de propagules (Aguilar et al. 2006; Garcia et al. 2007; Nathan and Muller-Landau 2000).
La sensibilité d’une espèce végétale à la fragmentation peut également dépendre de son degré de spécialisation et de dépendance envers ses pollinisateurs (Aguilar et al. 2006; Ashworth et al. 2004; Bond 1994). Les espèces spécialistes, n’ayant qu’un ou quelques pollinisateurs, sont considérées plus vulnérables à la fragmentation que les espèces généralistes (Bond 1994; Waser et al. 1996). Le mode de reproduction peut aussi influencer l’impact de l’isolement. La diversité génétique des populations d’espèces autofécondes ou « autogames », serait ainsi moins affectée par une diminution de la taille de la population que les espèces à reproduction croisée obligatoire ou « allogames » (Aguilar et al. 2008; Honnay and Jacquemyn 2007). La reproduction végétative, ou reproduction clonale, permettrait également d’augmenter le temps de génération et d’ainsi protéger les populations des effets génétiquement néfastes de la fragmentation (Honnay and Bossuyt 2005). D’ailleurs, les espèces ayant un mutualisme plante-pollinisateur très spécialisé auraient généralement développé une utilisation accrue de la reproduction végétative comme mécanisme alternatif aux baisses d’abondance de pollinisateurs (Bond 1994).
1.2 Espèces rares et écosystèmes naturellement fragmentés
Bien qu’il soit relativement aisé de concevoir les impacts de la fragmentation et de l’isolement subséquent sur les écosystèmes continus, tels qu’une forêt, certaines espèces sont naturellement rares et persistent malgré une abondance faible et éparse dans le paysage (Barrett et al. 1991). Afin de survivre dans ces conditions, ces espèces possèdent généralement diverses adaptations ou alternatives telles que la reproduction végétative (Pauw and Bond 2011; Wolf et al. 2000b) et l’autogamie (Goodwillie 2001; Moeller and Gebre 2005) qui peuvent assurer un certain recrutement même chez les petites populations ou en absence de pollinisateurs. La production d’une banque de graines dormantes dans le sol peut aussi augmenter la persistance des populations (Kalisz and McPeek 1993) en réduisant l’impact des variations environnementales (Menges 2000; Nathan and Muller-Landau 2000). De plus, une banque de graines permet de préserver la diversité génétique
4
même en présence de fluctuations importantes de la taille des populations (Del Castillo 1994; Tansley and Brown 2000). La dormance à l’état de corme, de bulbe ou de rhizome se rencontre chez de nombreux végétaux dont les orchidées (Wells and Willems 1991) chez qui elle peut durer jusqu’à 12 ans (Tamm 1972). Cette dormance permettrait d’obtenir sensiblement les mêmes résultats qu’une banque de graines et d’ainsi diminuer l’impact d’évènements stochastiques sur les petites populations (Wallace 2002). Malgré ces diverses adaptations et alternatives, les espèces rares sont néanmoins théoriquement considérées comme ayant une diversité génétique moindre que celle des espèces communes et abondantes (Ellstrand and Elam 1993; Hamrick et al. 1990; Karron 1987), bien que certaines études tendent à prouver le contraire (Gitzendanner and Soltis 2000).
Une partie de la confusion concernant la susceptibilité à la fragmentation des espèces rares et des espèces abondantes viendrait du fait que certaines espèces rares sont naturellement rares alors que d’autres espèces ne sont rares que depuis peu de temps, soit sur une échelle de temps humaine (Aguilar et al. 2008). Une espèce autrefois commune et maintenant rare devrait montrer une empreinte génétique de la fragmentation plus prononcée qu’une espèce naturellement rare (Aguilar et al. 2008). Les espèces abondantes et les espèces rares depuis peu de temps pourraient ainsi être plus vulnérables à la fragmentation que les espèces naturellement rares (Aguilar et al. 2008; Honnay and Jacquemyn 2007).
De la même manière qu’on trouve naturellement certaines espèces en petites populations isolées, certains écosystèmes forment des archipels dans le paysage. Les espèces végétales endémiques à ces écosystèmes naturellement en archipel ont possiblement évolué de manière semblable aux espèces naturellement rares afin de pallier les conséquences néfastes de l’isolement. Elles pourraient ainsi utiliser davantage la reproduction végétative, l’autogamie ou la production d’une banque de graines et donc être plus résilientes à la fragmentation comparativement aux espèces dont l’habitat n’a été que récemment fragmenté (Wolf et al. 2000a). Ces écosystèmes naturellement fragmentés pourraient agir comme un réseau dynamique avec des échanges entre les communautés végétales ou, à l’inverse, comme un ensemble de fragments isolés les uns des autres,
indépendants et autosuffisants. Cela dépend évidemment des espèces en cause et de leurs caractéristiques morphologiques et reproductrices. Les populations de certaines espèces inféodées à ces écosystèmes en archipel pourraient en effet bénéficier d’échanges de propagules entre les parcelles et ainsi représenter un réseau dynamique. Pour ces espèces, la perte d’habitat accentuant l’isolement des parcelles pourrait éventuellement réduire la connectivité entre les populations et affecter leur fonctionnement et persistance (Bossuyt 2007).
1.3 Les tourbières
Les tourbières sont des milieux humides qui se trouvent naturellement sous la forme d’un archipel dans le paysage. Il s’agit d’un des types de milieux humides les plus abondants au monde (Parish et al. 2008), dont plus de 40% se trouvent en Amérique du Nord (Lappalainen 1996). Les tourbières sont caractérisées par leur sol saturé en eau, lequel permet la dominance des sphaignes et ralentit la décomposition de la matière organique du sol, permettant ainsi l’accumulation de la tourbe. Cette particularité en fait d’excellents puits de carbone : bien que les tourbières n’occupent que 3% de la surface terrestre, on y retrouve 30% du carbone des sols (Parish et al. 2008). Certains types de tourbières remplissent également des services hydrologiques tels que la filtration de l’eau et l’atténuation des risques d’inondations (Barkham 1993; Erwin 2009; Zedler and Kercher 2005). Il existe deux principaux types de tourbières : les tourbières ombrotrophes ou « bogs » et les tourbières minérotrophes ou « fens ». Les bogs se différencient des fens par l’absence d’enrichissement en minéraux causé par le ruissèlement provenant des écosystèmes adjacents (Batzer and Baldwin 2012). L’approvisionnement en eau des bogs est donc principalement dû aux précipitations. Par conséquent, les bogs sont plus pauvres en éléments minéraux et ont un pH généralement plus faible que les fens (Batzer and Baldwin 2012; Wells 1981). Les deux types de tourbières sont toutefois des milieux anoxiques, parfois acides, pauvres en nutriments et donc considérés comme stressants et difficiles pour les plantes, ce qui a conduit à l’installation et à l’adaptation d’une flore et d’une biodiversité unique (Barkham 1993).
6
Bien que les tourbières soient généralement dans un état naturel en régions éloignées, surtout dans les latitudes boréales, leur situation est préoccupante en régions habitées où les pressions anthropiques, dont le drainage pour l’expansion urbaine, l’agriculture ou pour l’exploitation de la tourbe, menacent leur intégrité (Joosten 2009; Pellerin and Poulin 2013; Poulin et al. 2004; Raeymaekers et al. 1999; Wilson and Provan 2003). Jusqu’à maintenant, la protection et la conservation des tourbières se sont principalement appuyées sur la sélection des sites en fonction de caractères spécifiques à chaque site dont la taille, la diversité de micro-habitats et l’intégrité, menant entre autres à leur nomination en tant que site Ramsar (Bragg et al. 2003; Poulin et al. 2006), mais délaissant ainsi l’implication possible de la connectivité au sein de l’archipel. Très peu d’études se sont intéressées aux impacts de la fragmentation et de l’isolation sur les plantes de tourbières. Une étude sur la mousse Sphagnum affine Ren. & Card. n’a pas observé une diversité génétique moindre dans les sites fragmentés par les activités humaines (Thingsgaard 2001). La perte de diversité génétique dans les sites nordiques non fragmentés fut plutôt associée à l’effet fondateur de la recolonisation post-glacière et à la dérive génétique qui s’en suivit (Thingsgaard 2001). Toutefois, deux autres études ont observé un effet négatif de la fragmentation et de l’isolement sur des plantes de tourbières. Dans un premier temps, l’exploitation de la tourbe a réduit la diversité génétique des sous-populations de Polytrichum commune Hedw. en plus d’augmenter leur différenciation, et ce, malgré le fait que P. commune possède un mécanisme de dispersion des spores efficace (Wilson and Provan 2003) et que les mousses se reproduisent généralement de manière végétative. Deuxièmement, la fragmentation a réduit la diversité génétique et le fitness des populations de Swertia perennis L., une espèce commune de fens en Suisse (Lienert et al. 2002). Les plantes de tourbières pourraient ainsi vraisemblablement être affectées par la perte d’habitat et l’isolement, bien qu’évoluant dans un écosystème déjà naturellement réparti en parcelles isolées. Globalement, les conséquences de la fragmentation sur les populations de plantes ne sont pas encore assez connues pour permettre l’établissement de directives générales de conservation appropriées (Heinken and Weber 2013). Il convient donc d’approfondir la question de l’impact de la fragmentation sur les espèces trouvées en tourbières : les tourbières forment-elles un réseau dynamique d’écosystèmes connectés les uns les autres ou, à l’opposé, un ensemble de sites isolés, indépendants?
1.4 La platanthère à gorge frangée
La platanthère à gorge frangée, Platanthera blephariglottis var. blephariglottis (Willd.) Lindl., est une orchidée endémique des tourbières du nord-est de l’Amérique du Nord qui croît sur des substrats acides ayant un pH de 3.8 à 4.7 (Brown and Scott 1997). On la trouve donc fréquemment dans les bogs, mais également dans certaines autres tourbières légèrement minérotrophes, comme les fens pauvres (Laroche et al. 2012). Sa présence est indicatrice de l’intégrité écologique des tourbières ombrotrophes au Québec (Laroche et al. 2012). Conséquemment, l’analyse de la connectivité des populations de cette espèce pourrait non seulement permettre la sauvegarde d’une espèce emblématique, mais également mener à la conservation d’un ensemble de tourbières écologiquement intègres.
Les populations de P. blephariglottis peuvent atteindre plusieurs milliers d’individus (pers. obs.) dont les inflorescences supportent de 2 à 40+ fleurs (Cole and Firmage 1984)(photos en annexes 1 et 2). Il est déjà connu que P. blephariglottis possède des variations morphologiques entre populations (Argue 2012; Brown and Scott 1997; Hardin 1961). Certaines de ces variations peuvent être dues à l’environnement, tel que rapporté par Cole and Firmage (1984) qui ont observé que les inflorescences entourées d’arbres et d’arbustes étaient plus grandes que celles que l’on retrouve dans un environnement dégagé. Selon une étude menée à Terre-Neuve par Brown and Scott (1997), l’aire des sépales latéraux et des pétales latéraux varient d’une population à l’autre, alors que Hardin (1961) observe une diminution de la taille générale en plus d’une variation de la forme des feuilles et de la longueur du labelle et de l’éperon chez les populations nordiques. Ainsi, plusieurs variations phénotypiques sont connues chez P. blephariglottis. Toutefois, bien que l’environnement puisse être un facteur important, certaines de ces variations morphologiques pourraient également être dues à l’isolement géographique ou génétique des populations.
Au moins onze pollinisateurs, neuf espèces de Lépidoptères et deux espèces d’hyménoptères, sont connus pour P. blephariglottis (Cole and Firmage 1984). Il ne s’agit donc pas d’un mutualisme plante-pollinisateur très spécialisé. Les lépidoptères ou
8
« papillons » seraient les principaux pollinisateurs et incluent notamment le genre Hemaris, famille des Sphingidae, lesquels sont connus pour leurs habitudes nomades et vagabondes (Tartaglia 2013). Bien que peu de données soient disponibles sur les distances de vols chez les papillons, certaines espèces pourraient voler jusqu’à 10 km entre deux sources de nectar (Janzen 1984), ce qui pourrait promouvoir la connectivité entre populations végétales rapprochées. Bien que l’allogamie semble fortement favorisée par le mécanisme de pollinisation, l’autogamie peut également se produire (Cole and Firmage 1984). P. blephariglottis est diploïde à 2n=40 (Argue 2012) et à notre connaissance, une seule étude génétique sur les allozymes fut faite sur P. blephariglottis (Cowden 1998).
1.5 Objectifs et hypothèses
L’objectif général de ce mémoire est d’évaluer l’impact de l’isolement des tourbières sur les variations phénotypiques et génétiques des populations de P. blephariglottis afin d’approfondir nos connaissances sur la connectivité des tourbières et d’ainsi permettre la formulation de mesures de conservation adaptées à ces milieux qui sont souvent naturellement présents sous la forme d’un archipel.
Les objectifs spécifiques du mémoire sont donc :
(1) Estimer les variations phénotypiques et génétiques des populations de l’orchidée P. blephariglottis.
(2) Quantifier l’isolement de chacune des populations étudiées et déterminer si les variations phénotypiques et génétiques sont associées à l’isolement des populations.
(3) Évaluer si certaines variations phénotypiques sont corrélées aux variations génétiques. Nous nous attendons à trouver des différences phénotypiques entre les populations de P. blephariglottis, d’une part parce que plusieurs études ont déjà démontré que certaines différences phénotypiques existaient entre les populations (Argue 2012; Brown and Scott
1997; Hardin 1961), mais également parce que le genre Platanthera est reconnu pour son évolution morphologique rapide (Efimov 2011). L’utilisation d’une des dernières technologies de séquençage, soit le Genotyping-By-Sequencing (GBS) sur plateforme Illumina, nous permettra d’obtenir une grande quantité de marqueurs génétiques Single-Nucleotide-Polymorphism (SNP) afin de comparer les fréquences alléliques des populations et de caractériser leur différentiation. L’accès aux données de présences récentes et de données sur l’habitat des populations de P. blephariglottis (CDPNQ 2013; Laroche et al. 2012) et de communautés végétales des tourbières de cette région (Poulin et al. 2002) nous permettra d’établir le type de tourbières propices à P. blephariglottis. Les présences historiques pourront ainsi être revisitées et les tourbières candidates seront explorées afin de caractériser avec confiance l’isolement dans le paysage des populations échantillonnées. Les hypothèses suivantes ont été émises :
(1) Il y a des différences phénotypiques et génétiques entre les populations et celles-ci peuvent être expliquées, du moins en partie, par l’isolement.
Chapitre 2 How connected are populations of the
peatland orchid Platanthera blephariglottis, as revealed
by genetic and phenotypic traits variation.
12.1 Résumé
La connectivité de l’habitat est une composante importante en conservation des végétaux puisque l’isolement d’une population peut engendrer une diminution de son potentiel d’adaptation ainsi qu’une augmentation de son risque d’extinction via la dépression endogamique et la dérive génétique. Les tourbières sont présentes sous la forme d’un écosystème naturellement fragmenté dans le Québec méridional, mais leur connectivité reste méconnue. Nous avons étudié l’orchidée Platanthera blephariglottis afin de savoir si les tourbières représentent un réseau dynamique d’écosystèmes ou, à l’opposé, un ensemble d’habitats isolés les uns des autres. Quinze traits phénotypiques ont été mesurés chez 24 individus, et ce, dans 20 populations différentes. Des tests de germination ont été réalisés pour les mêmes populations. Des échantillons de feuilles ont été récoltés dans les 20 mêmes populations et six populations additionnelles pour effectuer une analyse génétique via un protocole de Genotyping-By-Sequencing (GBS) sur plateforme Illumina. L’isolement des populations fut évalué en fonction de la distance à la population la plus proche et du nombre de populations présentes dans différents rayons autour des sites. Des différences entre les populations ont été trouvées pour tous les traits phénotypiques. Nos résultats indiquent également que la structure génétique des populations suivait un patron d’isolement par distance (Isolation-By-Distance), signifiant ainsi la présence d’échanges de graines et/ou de pollen entre les populations rapprochées. La variation de plusieurs traits phénotypiques ainsi qu’une baisse des taux de germination étaient corrélées à l’isolement des populations. Considérant ces informations, il est clair que, bien que les tourbières soient présentes sous forme d’archipel dans le sud du Québec, elles constituent un réseau dynamique d’écosystèmes interconnectés pour P. blephariglottis.
12
2.2 Abstract
Habitat connectivity is important in plant conservation since the isolation of a population can diminish its adaptation potential and increase its risk of extinction though inbreeding depression and genetic drift. Peatlands occur as a naturally patchy ecosystem in southern Quebec, but their connectivity remains unknown. We studied the orchid Platanthera blephariglottis to assess whether peatlands represent a dynamic network or, at the opposite, a group of patches isolated from each other. Fifteen phenotypic traits were measured on 24 individuals in 20 populations. Germination trials were conducted for the same populations. Leaf sample were taken in the 20 populations and in six additional populations to conduct genetic analyses through a Genotyping-By-Sequencing (GBS) protocol on an Illumina platform. Isolation of populations was evaluated by the distance to the nearest population and by the number of populations occurring in different buffer widths around studied sites. Differences were found between populations for all phenotypic traits. Our results also indicated that the genetic structure of populations followed a pattern of Isolation-By-Distance (IBD), meaning that seeds and/or pollen exchanges occur between close populations. Variations of several phenotypic traits, as well as a diminished germination rate, were correlated with the isolation of populations. Considering this information, it is clear that, even if peatlands are naturally occurring in the form of an archipelago in southern Québec, they consist of a dynamic network of interconnected patches for P. blephariglottis.
2.3 Introduction
Degradation and destruction of habitats are recognized as some of the principal threats to terrestrial biodiversity across the planet (Krauss et al. 2010; Sala et al. 2000). The increasing pressure for land use change leads to habitats that are smaller, fragmented and isolated within a human-modified matrix, which in turn makes remnant populations more prone to extinction (Lande 1988; Young et al. 1996). Isolation, or the loss of connectivity among fragments, and reduction of population sizes may trigger genetic loss through random genetic drift and increased inbreeding and selfing, possibly leading to a lower
fitness, a diminished capacity of adaptation and eventually a higher risk of extinction (Aguilar et al. 2008; Frankham 2005; Newman et al. 2013).
The response of plant species to fragmentation depends on their life-history traits (Ewers and Didham 2006; Henle et al. 2004). Stronger effects are expected to occur for adult generation of short-lived species compared to long-lived species (Young et al. 1996). As well, species dependent on seeds for reproduction, especially short-lived seeds, are less resistant to fragmentation effects than plants reproducing clonally via vegetative spread (Honnay et al. 2005) or those producing a persistent seed bank (Henle et al. 2004). Outcrossing plants are also more susceptible to suffer from reduced effective population size than are selfing plants (Aguilar et al. 2008; Honnay and Jacquemyn 2007). On the other hand, connectivity among isolated patches can be maintained via key processes such as pollination and seed dispersal. In the specific case of plant-pollinator mutualism, the matrix resulting from fragmentation could influence pollination rates by reducing or increasing both pollinators movement between undisturbed patches (Krewenka et al. 2011; Llorens et al. 2012) and pollinator diversity (Winfree et al. 2007). For example, agricultural landscapes could be favourable to wind-pollinated species, which generally disperse pollen through relatively open areas (Culley et al. 2002). It remains that the role of life history traits interacts with attributes of pollinators and seed dispersers as well as with parameters of the landscape context to define population connectivity and determine the response of plant populations to fragmentation (Aparicio et al. 2012; Kolb and Diekmann 2005). Using proxies such as genetic diversity or structure in the remaining populations may simplify the task of assessing how they are connected in a fragmented landscape.
Most studies on the consequences of fragmentation on plants have focused on threatened and rare species or were conducted in few habitat types making difficult to draw accurate conclusions and guidelines for conservation (Cruzan 2001; Gonzales and Hamrick 2005; Heinken and Weber 2013; Wallace 2002). Rare species are generally associated with higher vulnerability to fragmentation (Ellstrand and Elam 1993; Leimu et al. 2006), but in their meta-analysis of 52 species, Honnay and Jacquemyn (2007) reported that common species could be just as vulnerable as rare species. Furthermore, Aguilar et al. (2008) distinguished
14
species that are “historically or naturally rare” from species that became recently rare as a consequence of anthropogenic habitat loss and warned that fragmentation could affect both differently. Once common and now rare species should show higher decrease in genetic diversity than naturally rare species after habitat loss. Despite these important considerations, most studies have mainly focused on anthropogenically fragmented ecosystems, like forests and grasslands, while few studies have been conducted on naturally patchy ecosystems, which could respond differently to fragmentation (Wolf et al. 2000a). Peatlands are naturally isolated ecosystems that generally occur as archipelagos in the landscape in southern Québec. Their conservation has mainly focussed on the exceptional character of specific sites, notably through their nomination as Ramsar sites (Bragg et al. 2003), therefore ignoring any form of connectivity within the archipelago. Yet, only few studies have investigated fragmentation and isolation effects on peatland plant populations and results were mixed. For instance, Thingsgaard (2001) found lower genetic diversity in the peatland moss Sphagnum affine Ren. & Card. in unfragmented than in anthropogenically fragmented areas. On the other hand, commercial peat extraction increased subpopulations differentiation of Polytrichum commune Hedw., despite the fact that P. commune possesses effective spore dispersal mechanisms (Wilson and Provan 2003) and that mosses generally rely on vegetative reproduction. Finally, fragmentation was found to reduce genetic diversity and plant fitness for the common fen species Swertia perennis L., regardless of the naturally patchy distribution of fens (Lienert et al. 2002). It thus appears that peatland plant species can respond to habitat loss and remain sensitive to connectivity, even if they are associated to an ecosystem that naturally occurs as isolated patches in the landscape.
The White Fringed Orchid, Platanthera blephariglottis var. blephariglottis (Willd.) Lindl. is a species commonly found in ombrotrophic peatlands (bogs), and occasionally in poor minerotrophic peatlands (fens) or peaty meadows in northeastern North America (Brown and Scott 1997; Laroche et al. 2012). Inflorescences have two to more than 40 white flowers and populations encompass tens to several thousands of individuals. It has been shown to be a strong indicator of peatland’s integrity (Laroche et al. 2012) since its
presence and abundance are positively linked to the Floristic Quality Assessment Index, FQAI (Swink and Wilhelm 1994), suggesting increased abundance in less disturbed sites. Increasing our knowledge on the sensitivity of this species to peatland’s connectivity could help implementing conservation networks that both efficiently protect one of the most charismatic species associated to peatlands and are composed of sites of high integrity. Platanthera blephariglottis is already known to have morphological variations between populations (Argue 2012; Brown and Scott 1997; Hardin 1961). Some of these variations might be due to environmental conditions, such as reported by Cole and Firmage (1984) who found that inflorescences under trees or shrub thickets were taller than the ones in open field. However, it remains unknown if some of these phenotypic differences are also related to geographic or genetic isolation. At least 11 pollinators are known for P. blephariglottis (Cole and Firmage 1984), including butterflies and hawkmoths (Hemaris sp.). Travel distances are generally unknown for butterflies, but moths can fly up to 10 km (Janzen 1984) and hawkmoths are considered strong flyers with vagrant habit (Tartaglia 2013), which should favour connectivity among close populations. Finally, P. blephariglottis is diploid with 2n=40 (Argue 2012) which should facilitate genetic assessments. To our knowledge, only allozyme analysis have been conducted so far on this species (Cowden 1998).
The objective of this study was to assess the connectivity of peatland through the genetic structure and phenotypic differences between populations of P. blephariglottis. Increasing our knowledge on whether naturally patchy ecosystems work as dynamic networks is of conservation concern as ignoring connectivity among patches in the implementation of conservation network could affect the persistence of populations or metapopulations (Bossuyt 2007; Vandewoestijne et al. 2008). We hypothesised that connectivity among close populations should foster genetic and phenotypic similarities while isolated and more distant populations would show increased distinctiveness. We based our study on a Genotyping-By-Sequencing (GBS) protocol in which within-population DNA was pooled across all sampled individuals, a promising approach for ecological studies at reduced costs (Futschik and Schlötterer 2010; Gautier et al. 2013; Sham et al. 2002). The assessment of
16
phenotypic variability was carried over fifteen morphological traits as well as germination rates, which can be associated to the fitness of populations.
2.4 Methods
2.4.1 Study areaThe study area is located in southern Quebec, Canada, alongside the southern ridge of the St. Laurent River. Forests cover approximately 45% of the region, agricultural lands 40%, and peatlands 4% (Poulin et al. 2002). Numerous peatlands are scattered throughout the landscape with more than 50 peatlands bigger than 40 ha and inter-peatland distances ranging from a few hundred meters to more than five kilometers.
2.4.2 Sampling
A population of P. blephariglottis was defined as a peatland containing the orchid, regardless of the peatland area and population size. Prior to this study, there were 24 known populations within the study area (CDPNQ 2013; Laroche et al. 2012). These 24 populations were sampled for genetic analysis while 20 populations were selected for phenotypic analysis (Fig. 1). The later were those for which data on vegetation communities was available (Laroche et al. 2012). Additionally, two distant populations, one about 208 km north of the study zone and another one about 132 km south, were sampled for genetic analysis to root phylogenetic trees and to better estimate the significance of genetic relations between populations within the study area. A voucher of P. blephariglotis was collected from all populations and deposited at the Marie-Victorin Herbarium [MT]. In the 20 populations studied for phenotypic traits, we sampled 24 randomly chosen individuals from the zone with the highest density of inflorescences. On each plant, three random fully developed flowers were selected. We measured a total of 15 traits using electronic sliding callipers (Table 1; Annexe 3). Traits were selected based on known variations (Argue 2012) and achievable measurement accuracy. The basal leave of many plants, which is always the longest, were grazed in many plants. Statistical adjustments were made to compensate for missing data. Measurements were done from July 21 to
July 31. This short period should not have induced any bias since blooming of inflorescences for P. blephariglottis can last up to 46 days and each flower remains receptive for up to 10 days (Cole and Firmage 1984; Smith and Snow 1976).
In the 26 populations selected for genetic analyses, leaf material from 24 random individuals was collected and immediately stored in anhydrous calcium sulphate (Drierite, 8 mesh). Dried samples were kept at room temperature until analysis.
Figure 1 Map of the studied zone. Groups of genetically more similar populations are framed. More genetically distinct populations are pointed with arrows.
2.4.3 Germination tests
We conducted germination tests to obtain a broad estimate of population fitness. In each of the 20 populations studied for phenotypic traits, we collected four mature but closed capsules on 10 randomly selected individuals. Sphagnum mosses were collected from each site and then thoroughly mixed together to serve as a germination substrate (Diez 2007;
18
Jacquemyn et al. 2007). Seeds from the same individual were pooled and germination trials on Sphagnum substrate were conducted, using an “Ex situ germination” protocol (Lemay et al. 2015). A nylon filter with a pore size of 100 μm (catalogue #NY1H09000, EMD Millipore, Billerica, Massachusetts, USA) was deposited on 1 cm of the mixed Sphagnum mosses in polystyrene petri dishes. Small squares of 7 x 7 mm of nitrocellulose filters (pore size of 0.45 µm, catalogue #HAWP04700, EMD Millipore) were subsequently put on the nylon filter and moistened with distilled water. About 100 seeds per individuals were deposited on a cluster of three squares, with a total of 5 clusters (5 individuals) per petri dish. To prevent drying and light exposure, petri dishes were sealed with Parafilm (Bemis Flexible Packaging, Neenah, Wisconsin, USA) and wrapped with aluminum foil. To break potential seed dormancy, plates were kept in a refrigerator at 4°C for 11 weeks before being incubated at 20°C in growth chambers in darkness for nine weeks. Using a dissecting microscope, swollen seeds with or without rhizoids were scored as germinated, based on germination criteria of other orchid species (Kauth et al. 2006; Zettler and McInnis Jr 1994).
2.4.4 Isolation measures
To quantify isolation among populations, we searched for undetected presences in all surrounding bogs bigger than 20 ha with at least 10 ha of suitable habitat for P. blephariglottis, based on data from Poulin et al. (2002) and Laroche et al. (2012), who observed that P. blephariglottis was present in herbaceous dominated habitats and absent in dense ericaceous shrubs. Fifty-seven additional peatlands met habitat criteria and field visits revealed that P. blephariglottis was present in 46 of these peatlands. To evaluate the degree of isolation of each population, we used the software ArcGis 10.0 (ESRI, Redlands, CA) with data on peatlands location and geometry (Poulin et al. 2002) to calculate the linear distances between each studied populations as well as the distance to the nearest population, furthermore called Nearest Neighbour (NN), whether sampled or not. We also calculated the area of the peatland where each studied population occurred. Adjusted buffers of 2, 5 and 10 kilometers width were generated for each studied populations and we counted the number of populations within each buffer (Annexe 4).
2.4.5 DNA extraction, libraries preparation and sequencing
Individual DNA extractions were conducted with Qiagen DNeasy 96 plant kit (Qiagen, cat. no. 69181) using 15 mg of dried material. DNA was quantified using Thermo Scientific Nanodrop 8000 spectrophotometer instrument (Fisher Scientific) and DNA quality was checked on 1% agarose gels. Population pools were prepared to a concentration of 20 ng/ul, ensuring equal contribution from each individual extract. Pool replicates were made from the same individual DNA extracts for four populations to test the reliability of the pooling method. A PstI/MspI enzymes combination was used for library preparation and all pools were sequenced as a multiplex on one lane of an Illumina HiSeq2000 (at the McGill University-Génome Québec Innovation Center in Montreal, QC, Canada).
To validate our approach of DNA-pooling at the population level, we genotyped 15 individuals from two populations and also sequenced a DNA pool for these two populations. This was important to make sure that the allelic frequencies estimate obtained with the DNA-pooling approach concurred with that obtained by an individual level genotyping. We found that the average allelic frequencies obtained with the individual genotyping of the 15 individual were highly correlated with the allelic frequencies of the pool for both populations (r=0.96 and r=0.93), confirming the validity of our DNA pooling approach.
2.4.6 Processing of raw sequence read data
Since a reference genome was not available for P. blephariglottis, we used the UNEAK pipeline (Lu et al. 2013) from TASSEL (Bradbury et al. 2007) to assemble contigs and call SNPs. We used a minimum minor allele frequency (mnMAF) of 0.05 and minimum call rate (mnC) of 0.80. Since the UNEAK pipeline cannot operate a filtration based on depth coverage and because the maximum number of reads stored per allele is capped at 127, we manually performed additional filtrations with EXCEL. Because our pools consisted of 24 diploid individuals, we removed positions that had a mean depth coverage of 48 reads or less among pools as well as every SNP that had a score of 127 for both alleles in one or more pools. We used pairwise comparisons of allelic frequencies between the four pools and replicate pairs to validate the efficiency of every filtration steps.
20
2.4.7 Statistical analysis
To synthesize phenotypic traits in fewer variables, we conducted two Principal Component Analyses (PCA): one on flower traits and the second on vegetative traits (Table 1). Previously, imputation of missing data was made after confirming with Chi-Square Test and Markov’s chains (Little 1988) that the high percentage (28%) of missing data due to grazing regarding the length of the longest leaf were randomly distributed throughout the data set (p=0.239). To test for morphological and germination differences among populations, an ANOVA was performed using the MIXED procedure of SAS which allowed to account for data heterogeneity using the group option of the repeated statement (SAS Institute Inc., 2011). To assess the influence of environmental parameters (buffers, NN, area) on the PCA vectors and germination, a regression model with populations as random factor was conducted in the MIXED procedure of SAS.
Plant communities were analysed to evaluate their possible influence on the phenotype of orchids. More precisely, the influence of plant species composition on populations’ estimates of the PCA vectors as well as on germination was assessed with a Redundancy Analysis (RDA) followed by 1000 permutations to test RDA axes significance, using R software (Team 2014). Species cover data were previously Hellinger transformed to account for double absence (Legendre and Gallagher 2001). For all phenotype and germination variables, there was no significant effect of local plant communities (RDA results: Flower Size r2 adj = -0.015 p=0.78; Flower Shape r2 adj = 0.022 p= 0.101; Plant Size r2 adj = 0.018 p= 0.14; Plant Foliage r2 adj = -0.016 p= 0.84; Germination r2 adj = 0.013 p= 0.21). Therefore, differences between populations would not be considered to arise from environmental differences between sampled sites as communities reflect local factors such as water table level and minerotrophy levels.
2.4.8 Genetic analyses
To evaluate the genetic distinctiveness of populations, allelic frequencies were used to generate a distance matrix between populations based on Nei’s genetic distance (Nei 1972) using the package “ade4” (Dray and Dufour 2007) in R. A phenogram, using UPGMA clustering, was then built from the resulting distance matrix using the “cluster” package
(Maechler et al. 2014). This phenogram was converted into a tree using “ape” package (Paradis et al. 2004) and Evolutionary Distinctiveness (ED) values were then estimated for each population using the “picante” package (Kembel et al. 2010). The ED is a metric used by the Zoological Society of London in their Edge of Existence program (http://www.edgeofexistence.org) and gives an estimate of a species or a population uniqueness based on the branch lengths in an evolutionary tree. We also used the SplitsTree4 software (Huson and Bryant 2006) to build a split network, that was then used to calculate a Shapley metric (SH) value (Volkmann et al. 2014). The SH metric is an independently derived approach based on Game Theory (Haake et al. 2008) that explicitly considers the species (or population) individual contribution to the future diversity. It is essentially the equivalent of the ED, but values are calculated based on phylogenetic networks, which likely better represent relationships between populations than a bifurcating tree. They are expected to provide more accurate estimates of the populations genetic distinctiveness than ED values which are based on a dichotomous tree (Volkmann et al. 2014). To test whether or not the isolation of a population was linked to its genetic distinctiveness, the relationships between ED and SH values and the environmental parameters (buffers, NN, area) were investigated by looking for significant correlations using the CORR procedure in SAS program. To determine if there was connectivity between close sites, we tested for an Isolation-by-distance pattern by performing a Mantel test with 9999 permutations between Nei’s pairwise genetic distances and pairwise geographical distances. We also used Mantel tests to explore links between genetic distances and phenotypic differences in populations using Euclidian distance matrixes generated from the populations’ estimates of the phenotypic PCA vectors and from germination results.
2.5 Results
2.5.1 Differences among populations: phenotype and germination
The first two axes of the two PCA synthesized 56% and 68% of the variability of plants and flowers, respectively. For flowers, all traits were positively correlated along the first eigenvector (Table 1), meaning that when one trait was bigger, the others traits tended to be
22
bigger as well. This is often associated to a size effect, a common result for biological data, which means that populations with a higher value for this vector had bigger flowers than those with a lower value. Along the second eigenvector, all length traits were correlated together and opposed to all width traits (Table 1), clearly representing a shape effect. Consequently, populations with higher value for this vector had stocky flowers, while populations with a lower value had elongated flowers. For plant traits, the first eigenvector also revealed a size effect as all traits were positively correlated (Table 1). Therefore, populations with a higher value had bigger plants with more flowers than those with a lower value. Along the second eigenvector, the number of leaves was opposed to the length of the longest leaf, which for P. blephariglottis is always at the base of the plant with decreasing leaves along the stem. Consequently, we associated this vector with a foliage effect: populations with a higher value had bushier plants with many short leaves whereas populations with a lower value showed individuals with fewer but longer leaves. For all four selected eigenvectors, populations significantly differed in terms of flower size (F = 12.20 p<0.0001), flower shape (F = 13.36 p<0.0001), plant size (F = 5.30 p<0.0001), and plant foliage (F = 9.95 p<0.0001). A total of 19050 seeds were assessed during germination trials and average rates of germination were calculated for each population. Results ranged from 37% to 83%, with significant differences between populations (F = 3.64; p<0.0001).
2.5.2 Sequencing results
A total of 175 235 842 reads were sequenced on the lane and the UNEAK pipeline identified 113 163 SNPs. The number of reads per pool ranged from 2,9 to 8,5 millions. After filtration steps, we obtained 2841 SNPs with 0,05% of missing data and a mean coverage of 105,9 reads/SNP. Pair-wise comparisons of allelic frequencies for the four populations for which a replicate was made showed very high correlations (r≥ 0,96 in the 4 cases). This is also very close to what we found between pools and the average of 15 individuals analysed separately, for the 2 populations in preliminary tests (r=0.96 for pop1, r=0.93 for pop2). This shows that our pooling method gave results similar to what we would have obtained through individual sequencing and therefore provides reliable results when repeated on the same individual extracts.
Tableau 1 List of studied phenotypic traits and principal component analysis (PCA) on flowers and on plants. Flower traits were conducted on 3 random flowers per plant. A total of 24 individuals were measured in 20 populations, totalizing 480 plants. For flowers, Component 1 is associated with a “Size” effect and Component 2 with a “Shape” effect. For plants, Component 1 is associated with a “Size” effect and Component 2 with a “Foliage” effect.
Component Component
Flower 1 2 Plant 1 2
Size Shape Size Foliage
Variability explained (%) 40.77 15.07 Variability explained (%) 51.23 17.27
Central sepal length 0.45 -0.16 Inflorescence height 0.39 -0.17
Central sepal width 0.41 0.23 Inflorescence size 0.47 -0.12
Lateral petal length 0.42 -0.23 Number of flowers 0.49 0.03
Lateral petal width 0.26 0.60 Number of leaves 0.01 0.68
Column width 0.34 0.19 Longest leaf length 0.18 -0.59
Labellum length 0.39 -0.33 Longest leaf width 0.39 0.32
Labellum width 0.22 0.44 Stem diameter 0.45 0.19
Spur length 0.27 -0.42
2.5.3 Differences among populations: genetic grouping
The phenogram resulting from the UPGMA clustering shows that the four replicates (GPB2, S2PB2, NDL2 and HER2) got paired with their corresponding population (Annexe 5) and that in all cases, the similarity in allelic frequencies with replicates was greater then the similarity between different populations.
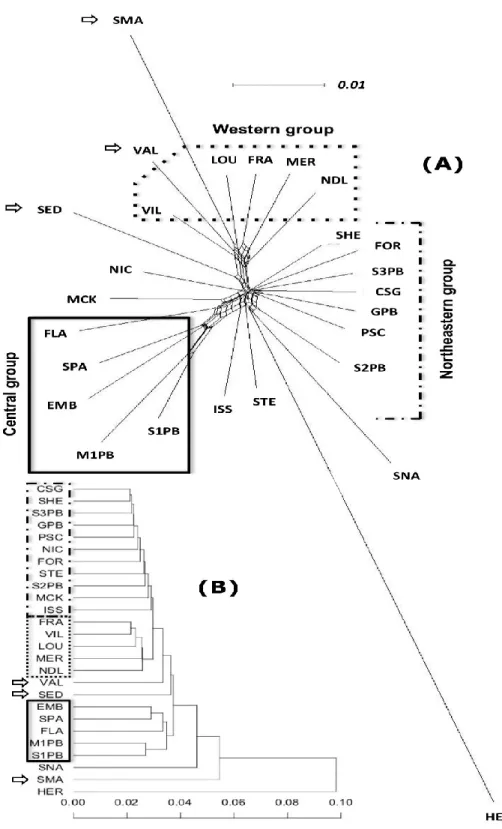
Three groups of populations could be identified in the phenogram constructed from genetic distances, while some populations appeared genetically more isolated (Fig. 2B). These groups (Western, Central, Northeastern) corresponded to the spatial distribution of populations across the study areas (Fig. 1). As expected, the two geographically outsider populations (SNA, 208 km north; HER, 132 km south) were isolated in the phenogram, suggesting a large genetic distance with the populations of the studied area. Surprisingly though, the population of SMA from the studied area was located between the two outsider populations (SNA, HER). Yet, this SMA population, with two others (VAL and SED), were the three most geographically isolated populations (Fig. 1), corresponding to the results of the phenogram.
24
The population clustering following the phylogenetic network approach was similar than the one resulting from the phenogram approach (Fig. 2A). Again, the two outsider populations SNA and HER were closely located on long branches. Western populations were again grouped together in the network as well as the Central group. The Northeastern group was also recognized but was scattered in the center of the network as some populations (ISS, MCK) lodged closer to the Central group. This is in accordance with the geography since these two populations were spatially close to the Central group. Overall, groups were similar in both clustering analyses. The three geographically isolated populations (SMA, VAL and SED) also remained more distinct from the others.
Figure 2 Phylogenetic network (A), using SplitsTree4 version 4.13.1, and phenogram (B), using UPGMA clustering, based on Nei’s genetic distance calculated on allelic frequencies. Groups of more similar populations are framed and more distinct populations are pointed with arrows. SNA and HER stand for the 208 km north population and the 132 km south population, respectively.
26
2.5.4 Effect of population isolation: phenotype, germination and genetic
Differences between populations in phenotypic traits and germination were not linked to the area of the peatlands (Table 2). Germination rate and the type of foliage were negatively correlated with the distance to the nearest neighbour (NN), meaning that the shorter the distance between a peatland and his closest neighbour was, the higher was the germination rate and the bushier were the plants. The shape of flowers, the size of plants and the rate of germination were all significantly influenced by the amount of populations within the landscape. More precisely, the shape of flowers was positively correlated to the amount of populations in the landscape regardless of the scale considered (2, 5 or 10 km buffer zone). The germination rate was positively correlated with the amount of populations in the landscape, but only at the two larger scales. Finally, the plant size was negatively correlated with the amount of populations in the landscape at the two smaller scales.
The evolutionary distinctiveness (ED), calculated from UPGMA clustering, was significantly and positively correlated to the NN (Table 2), meaning that the greater the distance between a population and its nearest neighbour was, the more genetically distinct the population was. The ED was also negatively correlated to the number of populations within the 2 km and 5 km buffers; populations with more neighbours in a 2 and 5 km radius were less distinct genetically from all other studied populations. However, these results were only significant when using ED values. When using the SH values, which result from the network approach, no correlation was found significant (Table 2) but the coefficients of correlation (r) were all above 0.30 and the relation was very close to be significant for the 5 km buffer (p=0.055). Additionally, all correlations with SH followed the same trend as the ones with ED: positive correlation with NN and negative correlations with all buffers. These results suggest that genetic distinctiveness increases with the NN distance and diminishes with an increasing numbers of neighbouring populations.
Tableau 2 Results of the MIXED procedure linking genetic and phenotypic differences to isolation parameters. Area refers to the peatlands areas. NN stands for Nearest Neighbour and Buffer X km stands for the number of populations of P. blephariglottis within a radius of X km. ED is the Evolutionary Distinctiveness and SH is Shapley value. P-values below 0.05 are accompanied by * symbol.
Area NN Buffer 2 km Buffer 5 km Buffer 10 km
n r p-value r p-value r p-value r p-value r p-value Flower size 1440 -0.12 0.160 0.10 0.225* -0.01 0.877* -0.02 0.8295* 0.11 0.1979* Flower shape 1440 0.03 0.727 -0.16 0.0594* 0.24 0.0006* 0.28 <.0001* 0.19 0.0146* Plant size 480 -0.01 0.934 0.15 0.1375* -0.26 0.0052* -0.22 0.0211* -0.11 0.2867* Plant foliage 480 -0.08 0.532 -0.36 0.0001* 0.20 0.0883* 0.19 0.1185* 0.21 0.0664* Germination rate 200 -0.06 0.645 -0.25 0.0279* 0.22 0.0606* 0.30 0.0039* 0.32 0.0016* ED 24 -0.05 0.845 0.46 0.0425* -0.46 0.0424* -0.52 0.0179* -0.40 0.084* SH 24 0.06 0.808 0.37 0.1053* -0.31 0.1768* -0.44 0.0552* -0.39 0.0922*
28
We found a positive correlation between pairwise Nei’s genetic distances between populations and pairwise geographical distances, thus indicating a pattern of Isolation By Distance (IBD) (Mantel test; r= 0.313; p= 0.0233). A significant positive correlation was also observed between the genetic distance and plant size (Table 3).
Tableau 3 Results of Mantel Tests between phenotypic traits and genetic distances. P-values below 0.05 are accompanied by * symbol.
Nei's genetic distance r p-value Flower Size 0.07 0.276* Flower Shape 0.06 0.248* Plant Size 0.40 0.031* Plant Foliage 0.09 0.219* Germination 0.36 0.053*
2.6 Discussion
The objective of this study was to investigate peatland’s connectivity of populations of P. blephariglottis, hypothesizing that connectivity among close populations should foster genetic and phenotypic similarities while isolated and more distant populations would exhibit increased distinctiveness. We found that, although occurring in naturally patchy ecosystems, populations of P. blephariglottis showed some level of connectivity among close peatlands and increased isolation was correlated to both phenotypic and genetic differentiations.
2.6.1 The footprint of isolation on phenotype and germination
Isolation seemed to be affecting populations since significant differences between populations were found for phenotypes as well as for germination. Differences can generally be expected in studies covering great distances, such as Hardin (1961) who found a cline in P. blephariglottis from a small northern form to large southern form, along the east coast of the United States. However, our study was restricted to a much smaller area with 122 km between the two most distant populations and it was thus surprising to found so many differences over that area. Plasticity could hardly be responsible of local