Le récepteur IOS1 d’Arabidopsis thalisana - modulateur de l’homéostasie protéique du réticulum endoplasmique en réponse au stress biotique
Texte intégral
(2) THÈSE DE DOCTORAT Le récepteur IOS1 d’Arabidopsis thaliana : modulateur de l’homéostasie protéique du réticulum endoplasmique en réponse au stress biotique. Laïla GIORDANO Institut Sophia Agrobiotech ISA. Présentée en vue de l’obtention du grade Devant le jury, composé de : Directrice de de docteur en Science de la Vie et de la Sylvie German-Retana, Recherche INRA, Laboratoire de Biologie du Santé de l’Université Côte d’Azur Fruit et Pathologie de Bordeaux, France Responsable Aurélien Boisson-Dernier, Dirigée par : Harald Keller d’équipe, Institut de Botanique de l’Université de Cologne, Allemagne Soutenue le : 16 décembre 2019 Benoit Lefebvre, Chargé de recherche INRA, Laboratoire des Interactions Plantes-Microbes, Toulouse, France Béatrice Bailly-Maitre, Chargée de recherche INSERM, Centre Méditerranéen de Médecine Moléculaire, Nice, France.
(3)
(4) Le récepteur IOS1 d’Arabidopsis thaliana modulateur de l’homéostasie protéique du réticulum endoplasmique en réponse au stress biotique. Présidente du jury Béatrice Bailly-Maitre, Chargée de recherche INSERM au Centre Méditerranéen de Médecine Moléculaire, Nice, France Rapporteurs Sylvie German-Retana, Directrice de Recherche INRA au Laboratoire de Biologie du Fruit et Pathologie de Bordeaux, France Aurélien Boisson-Dernier, Responsable d’équipe à l’Institut de Botanique de l’Université de Cologne, Allemagne Examinateur Benoit Lefebvre, Chargé de recherche INRA au Laboratoire des Interactions PlantesMicrobes, Toulouse, France.
(5)
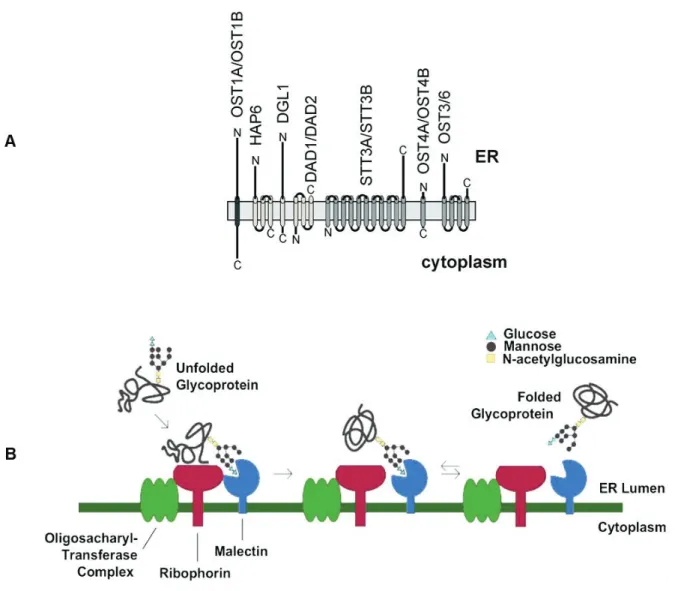
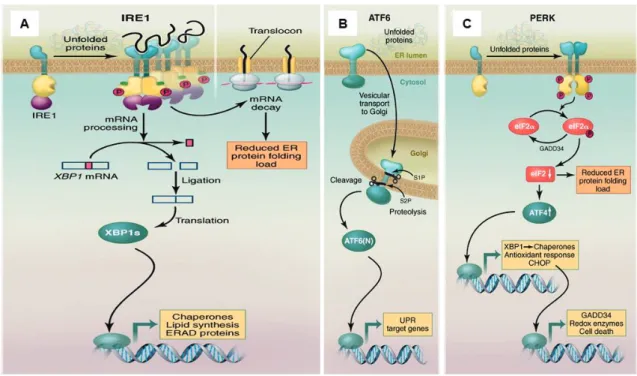
(6) Le récepteur IOS1 d’Arabidopsis thaliana : modulateur de l’homéostasie protéique du réticulum endoplasmique en réponse au stress biotique Les cellules végétales possèdent un nombre éminent de récepteurs localisés à la membrane plasmique. Ils sont spécialisés dans la détection des changements environnementaux et permettent à la plante de s'adapter en conséquence. Environ 200 de ces récepteurs sont composés d'un domaine extracellulaire avec des répétitions riches en leucine (LRR) et d’un domaine kinase intracellulaire. Nous avons préalablement identifié un membre de cette famille de récepteurs chez Arabidopsis, qui contribue au succès d’infection par des agents pathogènes filamenteux biotrophes, comme l'oomycète Hyaloperonospora arabidopsidis (Hpa). Une plante mutante pour le gène du récepteur perd sa sensibilité à l'infection et d'après ce phénotype le récepteur a été nommé "Impaired Oomycete Susceptibility 1" (IOS1). IOS1 régule négativement la voie de signalisation à l'hormone acide abscissique (ABA) au cours de l’infection. De plus, il a été démontré que le récepteur fait partie d'un complexe de récepteurs du plasmalemme qui détecte les infections bactériennes et déclenche les réactions de l’immunité innée. La région extracellulaire de l'IOS1 contient un domaine supplémentaire appelé « Malectin-Like Domain » (MLD). Le MLD présente de fortes similitudes structurelles avec la malectine animale qui réside dans le réticulum endoplasmique (RE), où elle interagit avec les ribophorines du complexe oligosaccharyltransférase (OST). Les protéines de ce complexe assurent la maturation post-traductionnelle des protéines en ajoutant des N-glycosylations. Les ribophorines surveillent. le. repliement. correct. des. glycoprotéines. néo-synthétisées.. Les. changements environnementaux modifient fréquemment le taux de protéines produites qui nécessitent une maturation. Si le système de surveillance n'est pas efficace, des protéines néo-synthétisées s'accumulent dans le RE et génèrent l’« Unfolded Protein Response » (UPR). Le mécanisme de contrôle de la maturation des glycoprotéines et les mécanismes de l'UPR existent également chez les cellules végétales. Afin de caractériser les fonctions du domaine extracellulaire (ED) de l'IOS1, nous montrons par microscopie confocale à balayage laser que le MLD est le médiateur de la rétention du récepteur dans la RE. Ici, le MLD du récepteur atténue l'UPR déclenché par l'infection avec l'oomycète. Nous avons identifié la ribophorine végétale HAP6 et l'atténuateur de mort cellulaire Bax-Inhibitor-1 (BI-1) comme.
(7) protéines résidant dans la RE qui interagissent avec l’ED de l'IOS1. Dans des expériences de complémentation fonctionnelle impliquant le mutant ios1-1 transformé avec des domaines IOS1 individuels, nous avons évalué le rôle de IOS1 et du MLD dans les réponses d’Arabidopsis à la signalisation de stress. Nous montrons que le MLD atténue l'UPR pendant l'interaction plante-oomycète, favorisant ainsi une infection réussie. Nous montrons également que la signalisation ABA est en corrélation positive avec l'UPR, indiquant que l’interférence de IOS1 avec la signalisation hormonale est une conséquence de l’interférence avec l'UPR. Ensemble, nos données suggèrent que les différents domaines du récepteur IOS1 ciblent des fonctions distinctes dans différents compartiments subcellulaires. Mots-clés : Interaction Plante-Agent Pathogène, Récepteur LRR-RLK, Domaine Malectin-Like, Stress du Réticulum Endoplasmique, UPR, Ribophorine, Bax Inhibitor1, Signalisation ABA..
(8) The IOS1 Arabidopsis thaliana receptor: modulator of endoplasmic reticulum protein homeostasis in response to biotic stress Plant cells have a diversifying number of plasma membrane-localized receptors, which are specialized in detecting environmental changes and allow the plant to adapt accordingly. About 200 of these receptors are composed of an extracellular domain with leucine-rich repeats (LRR) and an intracellular kinase domain. We have previously identified a member of this receptor family in Arabidopsis, which contributes to the infection success by biotrophic filamentous pathogens, such as the oomycete Hyaloperonospora arabidopsidis (Hpa). The plant mutant for the receptor gene loses its susceptibility to infection, and according to this phenotype the receptor has been named "Impaired Oomycete Susceptibility 1" (IOS1). IOS1 negatively regulates the abscisic acid (ABA) hormone signaling pathway upon infection. In addition, the receptor has been shown to be part of a plasma membrane receptor complex that detects bacterial infections and triggers innate immunity. The extracellular region of IOS1 harbors an additional so-called Malectin-Like Domain (MLD), which has strong structural similarities to animal malectin. Animal malectin resides in the endoplasmic reticulum (ER), where it interacts with ribophorins from the oligosaccharyltransferase (OST) complex. Proteins from this complex ensure post-translational protein maturation by adding N-glycosylations. Ribophorins monitor the correct folding of neosynthetized glycoproteins. Environmental changes frequently alter the rate of proteins that are produced and require maturation. If monitoring system is not efficient, neosynthetized proteins accumulate in the ER and generate the "Unfolded Protein Response" (UPR). The mechanism for controlling glycoprotein maturation and the UPR also exist in plant cells. In order to characterize the functions of the extracellular domain (ED) of IOS1, we show by confocal laser-scanning microscopy that the MLD mediates a retention of the receptor in the ER. Here, the MLD of the receptor attenuates the UPR, which is triggered by the oomycete infection. We identified the plant ribophorin HAP6 and the cell death attenuator Bax-Inhibitor-1 (BI-1) as ERresiding proteins that interact with the ED of IOS1. In functional complementation experiments involving the ios1-1 mutant transformed with individual IOS1 domains, we further evaluated the role of IOS1 and the MLD in the plant responses to ER and ABA stress signaling. We show that the MLD attenuates the UPR during the plant-oomycete.
(9) interaction, thus promoting successful infection. We also show that ABA signaling correlates positively with the UPR, indicating that the observed IOS1-mediated regulation of hormone signaling is a consequence of interference with the UPR. Taken together, our data suggest that individual domains of the IOS1 receptor target distinct functions in different subcellular compartments. Keywords : Plant-Pathogen Interaction, LRR-RLK Receptor, Malectin-Like Domain, Endoplasmic Reticulum Stress, Unfolded Protein Response, Ribophorin, Bax Inhibitor1, ABA signaling.
(10) Remerciements Je tiens à remercier les membres du jury d’avoir accepté d’évaluer mes 3 ans de travail, Sylvie German-Retana et Aurélien Boisson-Dernier en tant que rapporteurs, Béatrice Bailly-Maître et Benoit Lefebvre en tant qu’examinateurs. Un remercîment particulier pour Sylvie qui m’a accompagnée et encouragée dans la voie de la recherche depuis le master, pour son implication dans la conclusion de mon cursus de docteur, je suis très heureuse qu’elle soit rapportrice de ma thèse. Je suis très reconnaissante à Harald qui était toujours attentif et à mon écoute, ainsi que de m’avoir encadré et guidé durant ces trois années. Il m’a transmis de bon cœur toutes les connaissances et les compétences qui m’ont permis de réaliser et réussir mon projet de recherche. Je remercie également Valérie pour sa bienveillance, ses conseils précieux, son aide, son implication et son grand intérêt à la réalisation de ma thèse. Un grand merci à Franck pour son accueil dans l’équipe IPO et sa sympathie quotidienne. Je remercie aussi les membres de mes comités de thèse, Benoit Lefebvre, Claire Veneault-Fourrey et Bruno Favery qui ont su m’aiguiller les premières années sur les directions à prendre pour l’avancement des recherches. Merci à toutes les personnes qui ont contribué à la réalisation de ce travail, à Olivier pour son aide et ses conseils de microscopie, Maëlle pour ses astuces de biochimie, Joffrey pour ses recommandations pour les expériences, et bien-sûr Laurent et Benoit toujours prêts à m’aider. Un grand merci à mes amis Danila, Lucie, Joffrey, Marine et tous les doctorants avec qui j’ai pu passer de bons moments. Les Remerciements spéciaux vont à mes parents qui m’ont soutenu durant tout mon cursus, à mon frère, à ma sœur et aux membres de ma grande famille qui étaient toujours là pour moi. Un merci infini à Issam, mon amour, pour sa bienveillance, sa patience, son soutien, ses encouragements et sa quiétude qui m’étaient indispensables pour finir ce doctorat avec sérénité. Merci d’être là, merci de colorer ma vie, merci pour tout..
(11)
(12) Table des matières Introduction............................................................................................................... 7 I Interaction Plante Oomycète ............................................................................................ 9 1.1 Impact sur l’agriculture mondiale .............................................................................. 9 1.2 Les oomycètes, modèles d’étude ............................................................................12 1.3 L’état des lieux sur le récepteur Impaired Oomycete Susceptibility 1 (IOS1) ...........15 x. Identification du gène IOS1 .................................................................................15. x. Implication de IOS1 dans l’interaction plante-microbe .........................................15. x. Implication de IOS1 dans la signalisation à l’acide abscicique ............................18. II Dialogue moléculaire lors de l’interaction plante-microbe ...............................................20 2.1 Défenses constitutives .............................................................................................20 2.2 Défenses induites ....................................................................................................21 x. L’immunité déclenchée par un agent pathogène .................................................21. x. L’immunité déclenchée par un effecteur .............................................................22. x. Activation de la PTI par une bactérie pathogène .................................................22. 2.3 Les récepteurs RLKs comportant un domaine MLD.................................................26 x. FERONIA, récepteur MLD-RLK ..........................................................................27. x. ANXUR, récepteur MLD-RLK..............................................................................30. x. SYMRK, récepteur MLD-LRR-RLK .....................................................................30. III Le Réticulum Endoplasmique .......................................................................................33 3.1 Structure et fonctionnement.....................................................................................33 3.2 Le complexe OligoSaccharyl Transferase ...............................................................35 3.3 L’UPR : Unfolded Protein Response ........................................................................38 x. L’UPR chez les animaux .....................................................................................39. x. L’UPR chez les plantes .......................................................................................43. 3.4 ERAD, le système de dégradation associé au RE et l’autophagie ...........................52. Objectifs de la thèse ....................................................................................................55 Résultats ................................................................................................................. 57 I Caractérisation du récepteur IOS1 ..................................................................................59 1.1 IOS1 localise à la membrane plasmique et est retenu au RE par son MLD .............63 1.2 L’ED de IOS1 s’associe aux membranes et est glycosylé. .......................................67 1.3 L’expression de IOS1 chez Arabidopsis dépend de l’interaction avec l’oomycète ....70 II À la recherche des fonctions de IOS1 ............................................................................73 1.
(13) 2.1 Rôle du MLD en réponse à l’infection pathogénique ................................................73 2.2 Les marqueurs des voies de signalisation UPR .......................................................75 2.3 Suite à l’infection par Hpa, les plantes activent les voies UPR .................................78 III À la recherche des interactants de IOS1 .......................................................................83 3.1 L’ED de IOS1 interagit avec la ribophorine HAP6 ....................................................85 x. La proximité entre ED et HAP6 est indiquée en levure........................................85. x. ED co-localise et immunoprécipite avec HAP6 in planta .....................................86. 3.2 HAP6 influe le développement de l’oomycète en régulant l’UPR .............................89 x. HAP6 altère le développement de Hpa ...............................................................90. x. HAP6 active les voies UPR bZIP28 et bZIP60 en condition d’infection ...............91. 3.3 BI-1 régulateur de la mort cellulaire, acteur dans l’interaction plante-oomycete .......94 x. L’ED de IOS1 est physiquement proche de BI-1 .................................................94. x. BI-1 est impliqué dans le contrôle du développement d’Hpa ...............................96. IV Le MLD de IOS1 est impliqué dans la signalisation à l’ABA ..........................................98 4.1 L’ED de IOS1 réduit la sensibilité à l’ABA de ios1-1 ................................................98 4.2 Le MLD régule négativement la signalisation à l’ABA après infection ....................100 4.3 Le stress du RE régule positivement la signalisation à l’ABA .................................102. Discussion et Perspectives ................................................................................. 103 I Le double rôle de IOS1 .................................................................................................105 II Le clivage de IOS1 expliquerait sa modularité .............................................................108 III IOS1 acteur de la signalisation UPR en collaboration avec l’OST ...............................110 IV La signalisation à l’ABA est activée par l’UPR ............................................................114. Matériels et Méthodes .......................................................................................... 117 I Matériel biologique et conditions de culture ..................................................................119 II Méthodes .....................................................................................................................121 III Lignées, plasmides et amorces ...................................................................................129 Bibliographie .................................................................................................................135. Article .................................................................................................................... 147. 2.
(14) Table des figures et tableaux Figure 1. Symptômes des maladies du mildiou ........................................................ 10 Figure 2. Infection d’Arabidopsis thaliana par Hyaloperonospora arabidopsidis....... 13 Figure 3. Réponses des mutants ios1 aux infections pathogènes ............................ 17 Figure 4. Réponses de IOS1 à l’acide abscicique (ABA) .......................................... 19 Figure 5. Le modèle en zigzag illustre le système immunitaire des plantes ............. 23 Figure 6. BAK1 le récepteur kinase au centre de l’immunité .................................... 25 Figure 7. Arbre phylogénétique des protéines LRR-RLK d’Arabidopsis thaliana ...... 26 Figure 8. Alignement des séquences des récepteurs MLD-RLK .............................. 29 Figure 9. L’activité oligosaccharyltransférase est associée à un complexe protéique composé de ribophorines ......................................................................................... 37 Figure 10. Les trois voies de la signalisation UPR animale ...................................... 40 Figure 11. Les deux voies de la signalisation UPR végétale. ................................... 45 Figure 12. Organisation des domaines de IOS1 et prédiction topologique ............... 60 Figure 13. Prédiction de la structure de IOS1 et de son MLD................................... 62 Figure 14. Localisation subcellulaire de IOS1........................................................... 64 Figure 15. Localisation subcellulaire des domaines de IOS1 ................................... 66 Figure 16. L’ED de IOS1 est fortement glycosylé ..................................................... 68 Figure 17. Arabidopsis exprime le MLD en condition d’inoculation ........................... 71 Figure 18. Schéma de la mutation ios1-1 ................................................................. 72 Figure 19. Le MLD favorise la sensibilité à Hpa ....................................................... 74 Figure 20. Les gènes marqueurs sélectionnés sont représentatifs des deux voies de signalisation UPR chez les plantes ........................................................................... 76 Figure 21. Réponse des gènes marqueurs de l’UPR au traitement tunicamycine chez les plantes ................................................................................................................ 77 Figure 22. IOS1 régule négativement le stress du RE lors de l’infection par Hpa .... 79 Figure 23. Les mutants ios1-1 infectés par Hpa accumulent l'ARNm bZIP60 épissé 80 Figure 24. Le MLD de IOS1 attenue le stress du RE induit par Hpa ........................ 80 Figure 25. La mutation ios1-2 n'affecte ni l'interaction avec Hpa, ni les réponses au stress du RE provoqué par un agent pathogène ...................................................... 82 Figure 26. HAP6 est le gène codant la ribophorine II le plus exprimé en condition d’inoculation.............................................................................................................. 84 Figure 27. L’ED de IOS1 interagit avec la ribophorine HAP6 en levure .................... 86 Figure 28. L’ED de IOS1 s’associe avec la ribophorine HAP6 ................................. 87 Figure 29. Chez les mutants hap6 l'accumulation du transcrit HAP6 est réduite, mais pas supprimée .......................................................................................................... 89 3.
(15) Figure 30. Les mutants knock-down hap6 présentent un phénotype de sensibilité à l’infection par Hpa légèrement augmenté. ................................................................ 90 Figure 31. Les mutants knock-down hap6 n’activent pas de réponse UPR en condition d’infection .................................................................................................. 91 Figure 32. IOS1 et HAP6 montrent des profils d’expression opposés en condition de stress ........................................................................................................................ 92 Figure 33. Le domaine ED de IOS1 interagit avec BI-1 ............................................ 94 Figure 34. Le domaine ED de IOS1, HAP6 et BI-1 formeraient un complexe au RE 95 Figure 35. Le mutant bi-1-2 est moins sensible à l’infection oomycète, la lignée surexpresseur complémente ce phénotype. .................................................................. 96 Figure 36. Le MLD rétablie le seuil de sensibilité basal à l’ABA ............................... 99 Figure 37. Le MLD régule négativement la signalisation à l’ABA lors d'infection. ... 101 Figure 38. Le MLD régule négativement la signalisation à l’ABA lors du stress du RE ............................................................................................................................... 102 Figure 39. Rôles des différents domaines de IOS1 lors d’interactions avec des bactéries ou des biotrophes filmenteux. ................................................................. 107 Figure 40. IOS1 ∆ GDPC localise au RE ................................................................ 109 Figure 41. Le complexe entre HAP6, BI-1 et le MLD permet la répression de l’UPR ............................................................................................................................... 113 Figure supplémentaire 1. Séquence en acides aminés de IOS1 annotée .............. 125 Tableau 1. Lignées d’Arabidopsis mutantes ........................................................... 129 Tableau 2. Lignées d’Arabidopsis transformées ..................................................... 129 Tableau 3. Constructions pour les protéines de fusion ........................................... 129 Figure supplémentaire 2 : Schémas des plasmides utilisés ................................... 130 Tableau 4. Liste des amorces................................................................................. 131. 4.
(16) Abréviations. Lexique. Gènes. ABA. Acide Abscissique. 6K2. 6 KDa. AD. Alzheimer's disease - Maladie d'Alzheimer. ANX. Anxur. ADN. Acide Désoxyriboucléique. ATF6. Activating Transcription Factor 6. ADN-T. ADN de Transfert. BAK. Bcl-2 Homologous Antagonist Killer. AG. Appareil de Golgi. BAK1. BRI1-Associated Receptor Kinase 1. ARN. Acide Riboucléique. BAX. Bcl-2-Associated X Protein. BABA. Beta-aminobutyric acid. BcL2. B-Cell Lymphoma 2. BiFC. Bimolecular Fluorescence Complementation. BI1. Bax Inhibitor 1. Col. Columbia. BiP. Luminal Binding Protein. DTT. bZIP. Basic Leucine-Zipper. COPII. Coat Protein. Cub. C-Terminal Ubiquitin. FAO. Dithiothréitol Endoplasmic Reticulum Associated Degradation - Dégradation associée au RE Endoplasmic Reticulum Quality Control - Contrôle qualité du RE Effector-triggered immunity - Immunité déclanchée par les effecteurs Food and Agriculture Organization of the United Nations. DGL1. Defective Glycosylation 1. GDPC. Glycine Acide aspartique Proline Cystéine. EFR. EF-Tu Receptor. GFP. Green Fluorescence Protein. elF2. Elongation Factor 2. GlcNac. N-Acétylglucosamine. FER. Feronia. Hpa. Hyaloperonospora arabidopsidis. FLG22. Flagellin 22. HR. Hypersensitive Response - Réponse hypersensible. FLS2. Flagellin-Sensitive 2. J/Hpi. Jour/Heure post-inoculation. FRK1. Flg22-Induced Receptor-Like Kinase 1. KO. Knockout. HAC1. Homologous To Atf/Creb1. Ler. Landsberg erecta. HAP6. Hapless 6. LRR. Leucin Rich Repeat - répetition riche en leucine. HSP. Heat Shock Protein. mbSUS. membrane based Split Ubiquitin System. IOS1. Impaired Oomycete Susceptibility 1. MLD. Malectin-Like Domain. IRE1. Inositol-Requiring Enzyme 1. MP. Membrane Plasmique. Nub. N-Terminal Ubiquitin. OST. PDI. Protein Disulfide Isomerase. PTI. Oligosaccharyltransferase Pathogen-associated molecular pattern - Motif moléculaire associé aux agents pathogènes Peptide N-glycosidase Pattern Recognition Receptor - Récepteur à reconnaissance de motif PAMP triggered immunity - Immunité déclanchée par les PAMP. PVX. Potato Virux X. RC. ERAD ERCQ ETI. PAMP. COR15A Cold-Regulated Gene. PERK. Protein Kinase R (Pkr)-Like Endoplasmic Reticulum Kinase. Ppat5. Hyaloperonospora parasitica Isolate Maks9 Ppat5. PPIASE. Phosphatase. PR1. Pathogenesis-Related Gene 1. RALF. Rapid Alkalinization Factor. Replication Complex - Complexes de réplication. RD29A. Resistance to Desiccation. RE. Réticulum Endoplasmique. RPN. Ribophorin. RER. Réticulum Endoplasmique Rugueux. S1P. Site-1 Protease. RFP. Red Florescence Protein. STT3. Staurosporin and Temperature Sensitive 3. RLK. Receptor Like Kinase. SubAB. Subtilase Cytotoxin. ROS. Reactive Oxygen Species - espèces réactives d'oxygène. SWP1. Suppressor of Wbp1. SER. SYMRK. Symbiose Receptor Kinase. TBSV. Smooth ER - Réticulum endoplasmique lisse Signal Recognition Particle - Particule de reconnaissance du signal Tomato Bushy Stunt Virus. TuMV. Turnip Mosaïc Virus. UPR. Unfolded Protein Response - Réponse aux protéines non-pliées. VIGS. Virus-Induced Gene Silencing. Y2H. Yeast 2 Hybrid. PNGase PRR. SRP. TGB3. Triple Gene Block Protein. WBP1. Wheat Germ AgglutininǦBinding Protein 1. XBP1. X-Box Binding Protein 1. 5.
(17) 6.
(18) Introduction. 7.
(19) 8.
(20) I Interaction Plante Oomycète 1.1 Impact sur l’agriculture mondiale La population mondiale est estimée à de 9,8 milliards d’humains sur Terre pour 2050, cette croissance démographique, et la demande alimentaire liée, doit être soutenue par une production alimentaire globale nécessitant de progresser de 70% entre 2010 et 2050. Environ 1,5 milliards d’hectares de terres arables sont aujourd’hui cultivés dans le monde. Selon la FAO (Food and Agriculture Organization of the United Nations) cette surface suffirait pour nourrir la future population mondiale, mais elle est concentrée dans quelques pays. Il serait nécessaire d’augmenter cette surface dans les pays en développement, principalement en Afrique subsaharienne et en Amérique latine (FAO 2009). L’augmentation de la production alimentaire peut être obtenue à 90% par l’amélioration des rendements, grâce à la recherche et au développement de nouvelle technologies, techniques agricoles et variétés culturales (Bruinsma, 2017). La production agricole mondiale doit être optimisée afin de subvenir à nos besoins, et malheureusement de nombreuses pertes de production sont causées par des facteurs climatiques comme le réchauffement planétaire induisant la sécheresse ; ou des facteurs biologiques comme la dévastation des cultures par des agents pathogènes, incluant virus, bactéries, champignons, oomycètes, et des insectes ravageurs (Bélanger and Pilling, 2019). On estime que les pertes causées par des organismes nuisibles peuvent représenter jusqu’à 20% de la totalité de la production (Savary et al., 2019). Les pertes causées par les agents pathogènes représentent près de 220 milliards de dollars (Bélanger and Pilling, 2019), et celles causées par les insectes dévastateurs autours de 70 milliards de dollars (Bradshaw et al., 2016). Parmi ces menaces biotiques, ce sont les champignons et les oomycètes qui représentent le plus grand risque pour la sécurité alimentaire mondiale (Fisher et al., 2012). Les épidémies phytopathogènes causées par les champignons et les oomycètes ont modifié le cours de l’histoire humaine. Au milieu du XIXe siècle le Mildiou a entrainé la grande famine de la pomme de terre en Irlande qui en 10 ans, fit plus d’un million de victimes et trois millions d’émigrants à destination des Etats-Unis, de la Grande Bretagne ou encore du Canada et de l’Australie. Cette épidémie due à l’agent pathogène Phytophthora infestans a détruit les cultures en s’attaquant aux feuilles et aux tubercules. Aujourd’hui encore, les oomycètes provoquent des maladies dévastatrices dans l’agriculture mondiale, représentés majoritairement par le genre Phytophthora attaquant les 9.
(21) pommes de terre, les tomates, les cacaoyers, les cocotiers et d’autres plantes tropicales, les Plasmopara sur les vignes et les tournesols, ou encore les Peronospora sur le soja et des plantes ornementales (Figure 1), causant des pertes de plus de 6 millions de dollars annuels (Fisher et al., 2012). Les stratégies actuelles de lutte contre ces maladies comprennent la lutte chimique et le déploiement de cultivars résistants. Suite au Grenelle de l’Environnement de 2008, le plan Ecophyto vise à réduire de 50% l'utilisation des produits phytopharmaceutiques en zones agricoles et non agricoles en France, à l’horizon 2025. Dans le cas des oomycètes phytopathogènes, les fongicides sont souvent inopérants, car les oomycètes et les champignons montrent des similitudes dans leur mode d’infection et de développement, mais se différencient par leurs caractéristiques physiologiques. La plupart des fongicides utilisés en agriculture conventionnelle ciblent des métabolismes spécifiques aux champignons dans la synthèse des ergostérols ou de la chitine, ce qui est inefficace contre les oomycètes. En effet, les oomycètes ne synthétisent pas d’ergostérols et leurs parois sont essentiellement composées de β-1-3glucane et β-1-6glucane, ainsi que de cellulose (β-1-4glucane), contrairement aux champignons qui ont une paroi composée de chitine.. Figure 1. Symptômes des maladies du mildiou (A) Phytophthora infestans sur la pomme de terre (B) Phytophthora infestans sur la tomate (C) Plasmopara viticola sur la vigne (D) Peronospora sparsa sur le rosier.. 10.
(22) Dans le contexte de réduction des intrants, l’étude des interactions entre les plantes et leurs bioagresseurs est indispensable pour comprendre les mécanismes de défense mis en place par les plantes, et les systèmes de contournements des résistances mis en place par les agents pathogènes. La résistance aux produits agrochimiques est courante en raison de l'adaptabilité de l’agent pathogène. Les molécules utilisées comme fongicides ont un mode d’action à large spectre, ce qui permet aux souches de Phytophthora de développer des traits de résistance. Un exemple peut être donné par la molécule fongicide anti-oomycète Phenylamide Mefenoxam, utilisée depuis plus de 30 ans. Cette molécule est inhibitrice de la synthèse d’ARN, mais certaines populations de P. infestans en sont insensibles. L’étude des mécanismes de résistance a permis de mettre en avant un polymorphisme mononucléotidique (SNP) dans des isolats insensibles. Cet allèle insensible code pour une sous-unité de l’ARN polymérase II rendant l’oomycète résistant à ces molécules (Randall et al., 2014). L’utilisation d’oomycides a donc des limites dans son efficacité, et des répercutions nocives sur les écosystèmes. L’utilisation de variétés résistantes est une alternative efficace, mais montre ses limites, il arrive régulièrement que des isolats contournent le fonctionnement du gène de résistance introgressé dans la plante. Les sélectionneurs cherchent donc à accumuler des facteurs génétiques résistants dans les nouvelles variétés. De nouvelles méthodes de luttes durables et respectueuses de l’environnement doivent être développées afin d’éviter toutes les pertes agricoles.. 11.
(23) 1.2 Les oomycètes, modèles d’étude Les oomycètes sont des microorganismes eucaryotes filamenteux non photosynthétiques, classés parmi les hétérokontes, ils sont phylogénétiquement proches des algues brunes et des diatomées. Les oomycètes phytopathogènes se développent au sein des tissus végétaux sous forme de mycélium, formant des hyphes filamenteuses non cloisonées, composées de cellules multi-nucléées. Ils sont majoritairement polyphages, capables d’infecter un grand nombre d’espèces dans les écosystèmes naturels, et également des espèces d’intérêt agronomique, comme vu précédemment. Ces microorganismes peuvent être nécrotrophes, comme ceux représentés par le genre Pythium, il existe des espèces parasites hémibiotrophes, avec une phase biotrophe puis nécrotrophe, comme les Phytophthora, ou bien encore des espèces biotrophes comme les espèces des genres Peronospora et Plasmopara. La plupart du temps, les oomycètes se multiplient de façon asexuée par multiplication clonale, produisant des milliers de spores à chaque génération. Lorsque les conditions environnementales sont défavorables, un cycle sexué peut avoir lieu, conduisant à la formation d’oospores, ayant un rôle plus important pour la survie de l’organisme, que pour sa dispersion. Dans l’objectif de mieux comprendre les interactions entre les plantes et les oomycètes via leur dialogue moléculaire, le pathosystème composé de la plante modèle Arabidopsis thaliana et de l’oomycète Hyaloperonospora arabidopsidis (Hpa) est étudié (Figure 2A). Hpa est un parasite biotrophe obligatoire du genre Peronospora, spécialisé sur Arabidopsis. Au laboratoire en conditions contrôlées, cette espèce se développe en grande partie à l’intérieur de l’hôte afin d’augmenter sa biomasse avant de se reproduire par multiplication clonale (Figure 2B). Son cycle de vie asexué est responsable de la dissémination de la maladie et s’accomplit en 7 jours post inoculation (jpi) (Koch and Slusarenko, 1990). Au contact de la surface foliaire, la spore forme un tube germinatif se développant en un appressorium permettant la pénétration des tissus végétaux. L’oomycète forme ensuite des hyphes qui cheminent par voie intercellulaire et infectent chacune des cellules rencontrées, par la formation d’une structure nourricière intracellulaire appelée haustorie. L’haustorie se trouve dans un environnement appelé « matrice extrahaustoriale », espace par lequel transitent des nutriments, divers métabolites et des protéines, sécrétés par le microbe et par la plante (Hardham, 2007) (Figure 2C). 12.
(24) Figure 2. Infection d’Arabidopsis thaliana par Hyaloperonospora arabidopsidis (A) Sporulation (B) Développement de l’hyphe infectieux (C) Schéma d’une haustorie. Photographie de Hans van Pelt, adapté de (Fawke et al., 2015).. 13.
(25) Cette interface d’échange délimitée par les membranes des deux organismes, permet la nutrition du parasite aux dépens de la plante. Ainsi, l’oomycète n’est jamais en contact direct avec le cytoplasme de la cellule hôte mais, met tout en œuvre pour déréguler. le. métabolisme.. La. manipulation. des. fonctions. végétales. par. l’affaiblissement des défenses permet de favoriser son installation dans le site nourricier (Hardham, 2007). Pour se défendre, les cellules végétales déclenchent des mécanismes de l’immunité pouvant conduire jusqu’à la mort des cellules voisines du point d’infection, c’est la réponse hypersensible (HR) (Ponchet et al., 1999). Ce processus régule l’étendue de l’agent pathogène dans les tissus et peut bloquer son développement. Si le microbe est capable de détourner cette défense, alors le réseau mycélien envahit les tissus foliaires en quelques jours. En condition climatique humide favorable, les hyphes ressortent par les stomates et se différencient en conidiophores porteurs des conidiospores. Nous pouvons alors voir apparaitre à l’œil nu un duvet blanc (Figure 2A), comprenant les spores de dissémination à la surface des feuilles infectées. Ces spores se disperseront sur d’autres tissus afin de perpétuer le cycle de vie des oomycètes. Afin de proposer de nouvelles méthodes de lutte contre les oomycètes, il est essentiel d’étudier le dialogue moléculaire mis en place entre les plantes et les microorganismes. L’un des objectifs de recherche de l’équipe IPO est d’identifier des fonctions végétales manipulées par le parasite dans le but d’accomplir son cycle de vie. Le pathosystème A. thaliana et Hpa est un bon modèle pour étudier les interactions biotrophes et la reprogrammation génétique des cellules infectées.. 14.
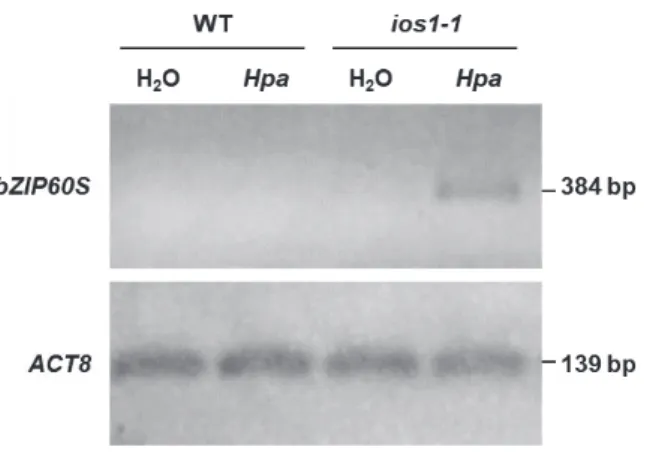
(26) 1.3 L’état des lieux sur le récepteur Impaired Oomycete Susceptibility 1 (IOS1) x. Identification du gène IOS1. D’après l’analyse du transcriptome d’A. thaliana infecté par Hpa, il a été montré que 21% du transcriptome est différentiellement exprimé durant le processus d’infection, à des stades précoces (< 1 jpi) correspondant à l’initiation de l’infection, et tardifs (> 4 jpi) correspondant au développement invasif et à l’initiation de la sporulation de l’oomycète (Hok et al., 2011). Parmi les gènes dont l’expression est induite chez A. thaliana au cours de l’infection de Hpa, certains sont impliqués dans les processus de défense et de réponse au stress, visant à empêcher le développement de la maladie. Les microbes ont une capacité d’adaptation et de détournement des défenses très efficaces, ce qui leur permet de s’établir tout de même au sein des tissus végétaux. Les analyses qui ont suivi se sont donc focalisées sur la recherche de facteurs de sensibilité de la plante permettant de favoriser le développement du parasite. Une surexpression du gène IOS1 (At1g51800), a été mise en évidence lors des stades précoces de l’infection et persistant jusqu’aux stades tardifs (Hok et al., 2011). x. Implication de IOS1 dans l’interaction plante-microbe. Des tests d’infection menés sur les mutants Knock-Out de ce gène (Figure 3A), ont montré que seulement le mutant allélique ios1-1 (fond génétique Landsberg erecta) présentait une résistance accrue à l’infection de Hpa. En effet ce mutant comporte un nombre de conidiospores par mg de feuille inférieur par rapport à celui du type sauvage (Hok et al., 2014). Les lignées mutantes ios1-1 complémentées avec le gène IOS1 complet, retrouvent un phénotype sauvage sensible après infection. IOS1 est donc un facteur de sensibilité à l’infection par les oomycètes, ce qui lui a valu son nom Impaired Oomycete Susceptibility (IOS1). Les tests d’infection réalisés sur les autres mutants d’insertion (ios1-2 et ios1-3) de fond génétique Columbia, ont montré un phénotype comparable au type sauvage (Figure 3A) (Hok et al., 2014). Le mutant ios1-1 possède une insertion d’un T-DNA au début de la séquence du gène, dans le domaine MLD. Les mutants ios1-2 et ios1-3 possèdent des insertions dans la séquence du domaine kinase. Il a été montré qu’en condition d’infection, les transcrits de IOS1 étaient de taille différente (Figure 3B) chez le sauvage et chez les mutants, laissant penser que ios1-1 n’exprimerait qu’une petite partie de la protéine, alors que les ios1-2 et ios1-3 produiraient un transcrit dont la traduction donnerait une protéine 15.
(27) avec un domaine kinase tronqué (Hok et al., 2014). Il a été suggéré que cette dissemblance génère des réponses différentes face à l’infection et expliquerait la différence de sensibilité entre ios1-1, ios1-2 et ios1-3 (Hok et al., 2014). D’autres chercheurs proposent une autre explication, l’impact de l’interaction Arabidopsis /Hpa dépendrait des génotypes Ler ou Col, respectivement ios1-1 et ios1-2/3, expliquant les différences de sporulation (Ried et al., 2019). Des tests de sensibilité ont été effectués sur les mutants ios1 avec d’autres organismes phytopatogènes (Figure 3C). D’une part avec des microbes filamenteux comme l’oomycète hemibiotrophe Phytophthora parasitica et le champignon biotrophe Erysiphe cruciferarum (Oïdium), et d’autre part avec des microbes bactériens comme Ralstonia solanacearum (Hok et al., 2014) et l’hémibiotrophe Pseudomonas syringae (Yeh et al., 2016). Suite à l’infection par les agents filamenteux, les phénotypes sont identiques au phénotype observé en interaction avec Hpa, ios1-1 présente moins de sporulation comparé à la lignée sauvage et aux mutants ios1-2 et ios1-3. Pour l’inoculation avec R. solanacearum, les plantes sauvages et mutantes montrent une sensibilité identique après inoculation des racines, le développement de cette bactérie se produit indépendamment de l’expression de IOS1 (Figure 3C). A l’inverse, les 3 versions des mutants ios1 sont hypersensibles à la bactérie P. syringae et les plantes sur-expresseurs de IOS1 sont plus résistantes à cette bactérie que les sauvages (Yeh et al., 2016). IOS1 serait nécessaire à l’établissement des agents pathogènes (hémi)biotrophes filamenteux, et jouerait un rôle différent dans le développement des bactéries.. 16.
(28) Figure 3. Réponses des mutants ios1 aux infections pathogènes (A) ios1-1 est moins sensible à l’attaque d’Hpa que la lignée sauvage (WT). Taux de sporulation d’Hpa après 7 jours d’inoculation des lignées d’Arabidopsis sauvages (WT Ler et Col) et chez les mutants alléliques ios1-1, ios1-2, et ios1-3. Le graphique représente les moyennes du nombre de conidiospores par mg de plantules fraiches, à partir de 3 réplicats biologiques de 20 échantillons individuels chacun. (B) Localisation des sites d’insertions (triangles noirs) pour les mutants ios1-1, ios1-2, et ios1-3. Les flèches et nombres indiques les amorces utilisées pour obtenir les amplicons des transcrits présentés au dessous. ios1-1 exprime faiblement le transcrit du couple 1-3, les mutants ios1-2 et ios13 produisent des transcrits qui pourront potentiellement se traduire en un récepteur quasiment complet, pouvant expliquer les résultats du graphique A. (C) ios1-1 présente une de perte de sensibilité face au champignon biotrophe Erysiphe cruciferarum (Hok et al., 2014). Le phénotype inverse est observé suite à l’infection bactérienne par Pseudomonas syringae. Pour l’infection par la bactérie Ralstonia solanacearum, ios1-1 ne présente pas de phénotype différent comparé au WT. Les astérisques indiquent les différences statistiquement significatives d’après un test de Student (*, p<0.05; ***, p<0.001). Adapté de (Hok et al., 2011, 2014; Yeh et al., 2016). 17.
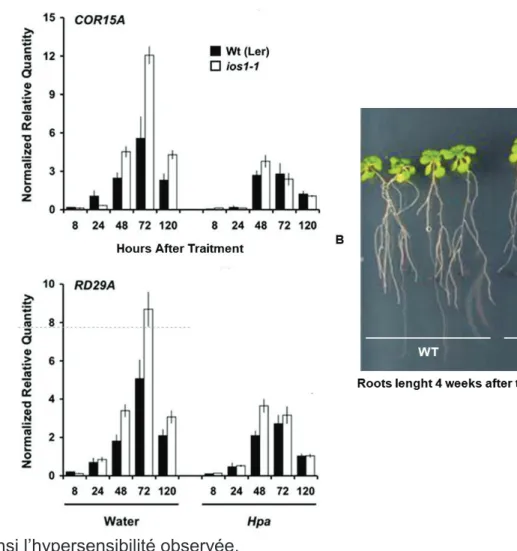
(29) x. Implication de IOS1 dans la signalisation à l’acide abscicique. En condition d’infection, IOS1 est sur-exprimé dans les cellules voisines des hyphes infectieuses ainsi que les cellules hébergeant les haustories de Hpa, suggérant un rôle de IOS1 dans le contact direct entre les cellules végétales et l’agent pathogène (Hok et al., 2014). Le phénotype de résistance à Hpa observé est indépendant de la mise en place des mécanismes de défense étant donné qu’aucune sur-induction de gènes codant pour des protéines de résistance (comme PR1) n’a été constaté chez le mutant ios1-1 (Hok et al., 2014). Ces résultats ont amené à penser que la protéine IOS1 jouerait un rôle dans d’autres voies de signalisation. Lors de la caractérisation de IOS1, l’analyse du profil d’expression au cours du développement de la plante nous a montré que le promoteur IOS1 est activé dans des tissus spécifiques tels que le cortex et la zone d’élongation racinaire, et au niveau de la zone d’abscission d’organes aériens comme les stipules, le stigmate, les sépales, les pétales et les fruits (Hok et al., 2014). L’expression de ce gène dans les zones d’abscission et de croissance ont supposé une implication de IOS1 dans la voie de l’acide abscissique (ABA), actives dans ces mêmes sites. L’ABA est une phytohormone de la famille des terpénoïdes, active dans le développement végétatif. L’ABA a un effet inhibiteur général de la croissance cellulaire, elle inhibe l’allongement des tiges et des racines primaires, et prolonge la dormance des bourgeons et des graines en cas de conditions climatiques défavorables. Des études ont montré que l’ABA est également impliquée dans les réponses de défense au contact d’agent pathogène (Ton et al., 2009). L’induction de la fermeture des stomates par l’ABA est une des premières réponses observées suite à la perception de bioagresseurs. En effet, les stomates constituent des ouvertures naturelles pour les microbes et facilitent leur passage au travers des tissus végétaux. Les oomycètes, tels que Hpa, utilisent ces voies de sortie pour y former un conidiophore afin de sporuler à l’extérieur du tissu infecté et se disséminer. La fermeture des stomates induites par l’ABA constitue donc un obstacle à la sporulation de l’oomycète. Il a été montré que IOS1 régule de façon négative l’expression de gènes marqueurs des voies de signalisation de l’ABA (COR15A Cold Regulated 15A et RD29A Resistance to Desiccation 29A) (Hok et al., 2014) (Figure 4A). En présence d’ABA exogène, le mutant ios1-montre une hypersensibilité à la phytohormone qui se manifeste par une élongation racinaire réduite et un taux de germination 18.
(30) significativement plus faible par rapport au type sauvage (Hok et al., 2014) (Figure 4B). L’induction de l’expression de IOS1 chez Arabidopsis dans le cadre de l’infection par Hpa semble alors être bénéfique à l’oomycète, qui réprime activement les voies de signalisation de l’ABA en faveur de l’infection et de sa sporulation. L’absence de IOS1 conduit donc à une sur-activation des voies de signalisation de l’ABA, ce qui explique. ainsi l’hypersensibilité observée.. Figure 4. Réponses de IOS1 à l’acide abscicique (ABA) (A) La signalisation ABA interférant avec la maladie est atténuée par IOS1. Expériences de RTqPCR pour déterminer l'évolution dans le temps des niveaux de transcription des gènes marqueurs COR15A et RD29A contrôlés par l'ABA dans des plantes de type sauvage (Wt) et ios1-1 infectées ou non par Hpa. (B) Le mutant ios1-1 est hypersensible à l’ABA. Les racines du mutant sont plus courtes que celle du WT après traitement ABA, il est plus sensible à l’inhibition de l’élongation racinaire induite par l’ABA. Adapté de (Hok et al., 2014). 19.
(31) II Dialogue moléculaire lors de l’interaction plante-microbe Le succès de l’infection microbienne est dû à la manipulation des défenses et du métabolisme par l’agent pathogène, au-delà des barrières mises en place par la plante pour favoriser son installation. 2.1 Défenses constitutives La première phase de défense est dite constitutive, et empêche la plupart des infections. Elle peut être une caractéristique physique, comme la cuticule enrobant les feuilles et les tiges, limitant l’entrée des agents pathogènes foliaires (Hématy et al., 2009) ou une caractéristique chimique. En effet, les cellules végétales produisent aussi des. métabolites. secondaires. toxiques,. appelés. phytoanticipines,. qui. sont. généralement des composés azotés (dont des alcaloïdes), des composés terpénoïdes et des composés phénoliques (tanins), qui peuvent altérer le développement des prédateurs herbivores et microbiens (Wittstock and Gershenzon, 2002). Afin de détourner ces défenses basales, les agents pathogènes ont dû s’adapter au cours de leur évolution et s’orienter vers la création de structures spécialisées. Par exemple, certains filamenteux sont capables d’exercer une force supérieure à 8 MPa dans leur appressorium afin de percer mécaniquement la cuticule et d’atteindre les cellules du parenchyme (Howard et al., 1991). Les microorganismes phytopathogènes produisent eux aussi des toxines pouvant altérer l’intégrité de la cuticule, ou sécrètent des enzymes cutinases ou cellulases qui leur permettent d’entrer en interaction directe avec les cellules végétales (Quoc and Bao Chau, 2017). Les agents pathogènes hémibiotrophes dans leur phase nécrotrophes, et nécrotrophes produisent des toxines qui contribuent à la virulence, et induisent la mort des tissus infectés. Les micro-organismes pathogènes ont développé des stratégies d’attaque afin d’infecter efficacement les cellules végétales, notamment par la sécrétion de facteurs de pathogénicité. Au cours de l’évolution, afin de répondre à ces attaques, les plantes ont étendu leur système de défense en créant des mécanismes de perception des microbes afin de réguler les tentatives d’infection. Des réactions spécifiques de défense sont alors mises en place, et s’apparentent d’ailleurs à un système immunitaire.. 20.
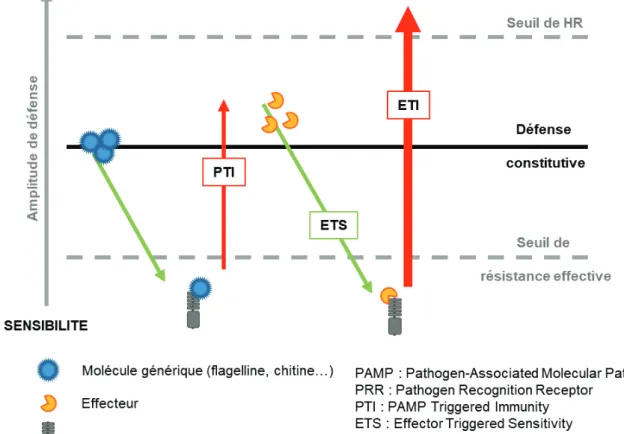
(32) 2.2 Défenses induites x. L’immunité déclenchée par un agent pathogène. La mise en place des défenses immunitaires est possible seulement si la plante détecte la présence d’un agent pathogène. Les molécules microbiennes reconnues peuvent être des métabolites, des oligossacharides ou des protéines, et sont nommées PAMPs pour Pathogen-Associated Molecular Patterns. Le terme PAMP est d’abord apparu dans le système animal pour décrire les motifs moléculaires conservées pour la reconnaissance du « non –soi » (Janeway, 1989). Chez les plantes, le développement du principe de la reconnaissance du non-soi et de la mise en place d’une immunité basale ou innée, similaire à celle des animaux est apparu plus tard (Nürnberger and Brunner, 2002). Les PAMPs sont généralement des molécules nécessaires à l’accomplissement du cycle de vie du microbe. Leur détection se fait par des récepteurs situés à la surface des cellules, les PRR (PatternRecognition Receptor) (Zipfel, 2014). Ces récepteurs sont spécialisés dans la reconnaissance des séquences conservées de PAMPs et peuvent être dotés d’un domaine kinase intracellulaire, assurant la signalisation, et/ou être en lien avec des corecepteurs et des cascades de MAPK (Mitogen-Activated Protein Kinase) (Boutrot and Zipfel, 2017). Cette signalisation entraine le déclanchement de l’immunité basale, aussi appelée Pathogen-Triggered Immunity (PTI), afin de limiter l’invasion microbienne (Figure 5). Les premières défenses induites sont un dépôt de callose et un renforcement de la paroi afin de solidifier les barrières physiques, la production de phytotoxines ou d’enzymes litiques pour digérer l’agent pathogène ou encore la fermeture des stomates pour empêcher l’entrée et la sortie des microbes. Toutes ces signalisations sont régulées par les phytohormones. Au cours de l’évolution, une fois que les plantes ont déployé un système immunitaire permettant la détection de PAMPs, certains agents pathogènes ont développé la capacité d’éviter ce type de résistance en supprimant les défenses de la plante.. 21.
(33) x. L’immunité déclenchée par un effecteur. Lorsque l’agent pathogène interagit avec les cellules végétales, il sécrète des molécules effectrices pour moduler les voies de défense et assurer l’infection (Jones and Dangl, 2006). D’autres protéines réceptrices intracellulaires reconnaissent ces effecteurs, ce sont des protéines NLRs, comportant des domaines Nucleotide-binding et Leucine-rich repeat (Jones et al., 2016). La reconnaissance des effecteurs ou de leurs activités par des récepteurs spécifiques déclenchent les réponses ETI (Effector Triggered Immmunity). Les premières réponses de défense sont la libération d’espèces réactives d’oxygène (ROS), ce burst oxydatif entraine la HR, et la mort des tissus infectés (Levine et al., 1994). Le modèle en zigzag illustre les défenses de la plantes mises en place au cours de l’interaction avec des agents pathogènes (Jones and Dangl, 2006) (Figure 5). x. Activation de la PTI par une bactérie pathogène. La perception de fragments microbiens comme la flagelline lors d’attaque de bactéries ou la chitine lors d’attaque de champignons, apparait comme un système conservé chez beaucoup d’espèces de plantes (Zipfel, 2009). Dans l’étude des processus de réponse à l’infection microbienne, le cas de l’interaction plante-bactérie est le plus courant. La plupart des bactéries phytopathogènes sont dotées de flagelles constitués d’une protéine appelée flagelline (flg). Celle-ci présente une séquence peptidique conservée de 22 acides aminés en N-terminal. Le peptide flg22 est l’éliciteur bactérien le plus étudié, sa présence est détectable par les cellules végétales et induit les réponses de défense de la plante (Felix et al., 1999). Chez Arabidopsis thaliana, le PRR responsable de la reconnaissance de flg22 est un récepteur de type Leucin-Rich Repeat Receptor Like Kinase (LRR RLK), la FLAGELLIN-SENSING 2 (FLS2), qui a été identifié par criblage d’une banque de mutants pour leur insensibilité à la flagelline (Gómez-Gómez and Boller, 2000). L’activation de la voie de signalisation des défenses nécessite une association hétérodimérique entre le ligand flg22, FLS2 et le co-récepteur BAK1 (Brassinosteroid insensitive Associated Receptor Kinase 1) (Albert et al., 2013), ces deux récepteurs n’interagissent qu’en présence du ligand flg22. BAK1 est un co-récepteur LRR-RLK multifonctionnel identifié initialement en interaction avec le récepteur LRR-RLK BRI1 (Brassinosteroid Insensitive 1) (Nam and Li, 2002), pour la perception de brassinostéroïdes, phytohormones impliquées dans le 22.
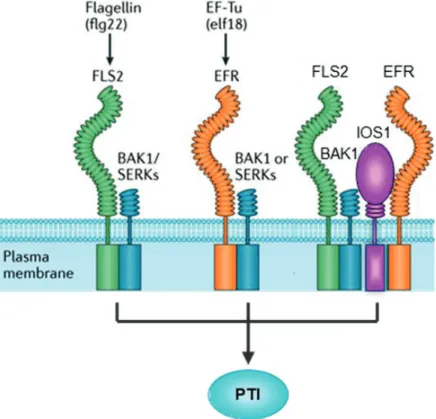
(34) développement et lors des réponses à l’environnement. Lors de l’interaction entre Arabidopsis et la bactérie P. syringae, FLS2 et son co-récepteur BAK1 perçoivent l’épitope flg22, et activent l’immunité (Figure 6). Les plantes mutantes bak1 déclenchent des réponses anormales après traitement à la flagelline, indiquant que BAK1 agit positivement sur la signalisation dépendante des PRR, initiant l’immunité (Chinchilla et al., 2007).. Figure 5. Le modèle en zigzag illustre le système immunitaire des plantes Des défenses d’amplitudes différentes sont mises en place durant l’interaction plante-microbe. La plante possède des barrières constitutives que l’agent pathogène doit contourner. Les premières molécules du non-soi à être reconnues sont des molécules génériques (phase 1). Les plantes détectent les motifs moléculaires associés aux agents pathogènes (PAMPs) via des PRR pour activer l’immunité déclenchée par les PAMPs (PTI). Si les agents pathogènes résistent à la PTI, la phase 2 est mise en place, avec la libération d’effecteurs interférant avec la PTI, entrainant une sensibilité déclenchée par les effecteurs (ETS). Là, un effecteur peut être reconnu par des récepteurs NB-LRR, ce qui active l’immunité déclenchée par un effecteur (ETI), c’est une version amplifiée du PTI qui peut dépasser le seuil d’induction de la réponse hypersensible (HR). Une troisième phase d’attaque du pathogène peut être effective avec la libération de nouveaux effecteurs. Adapté de (Jones and Dangl, 2006).. 23.
(35) Un autre exemple très étudié de reconnaissance de PAMPs est le recepteur EFR (EF-Tu Receptor), également de la famille des LRR-RLK, pour la détection du facteur d’élongation bactérien EF-Tu ou de peptides dérivés elf18 et elf26 (Kunze et al., 2004). Suite à la liaison entre EFR et EF-Tu, des réponses aux PAMPs sont activées, entrainant une induction de la résistance basale (PTI) (Zipfel et al., 2006). Le récepteur EFR détecte les peptides bactériens et s’associe rapidement avec BAK1 en présence du ligand, initiant la phosphorylation du récepteur BAK1 (Schulze et al., 2010) (Figure 6). BAK1 est un récepteur indispensable à la signalisation de la défense, et l’identification de son répertoire d’interactants nous permettrait d’avoir une vision globale de son mode d’action pour la transduction des signaux de défense. Il a été montré que le récepteur IOS1 était un acteur essentiel de la PTI contre P. syringae (Yeh et al., 2016).En recherchant les gènes dont l’expression est sensible à l’acide ß-aminobutirique (BABA), molécule utilisée comme activatrice de la résistance induite, les mutants ios1 se présentent comme candidats. Les plantules d’Arabidopsis traitées avec les peptides flg22 ou elf18 montrent une accumulation des transcrits d’IOS1 dès une heure après application. Les mutants ios1 présentent des réponses au PTI défectueuses, en observant le gène marqueur FLG22-Induced Receptor-Like Kinase1 (FRK1) après le traitement aux peptides précédents. Celui-ci est significativement moins exprimé chez le mutant que chez la plante sauvage. De plus, IOS1 est requis pour l’activation complète des voies de signalisation MAPK, après la perception de PAMPs. Il a également été montré que IOS1 immuno-précipitait avec les récepteurs FLS2 et EFR, après production en bactérie E. coli. Dans des expériences de BiFC (Bimolecular Fluoresecnce Complementation) IOS1 interagit avec FLS2, EFR et BAK1 en protoplastes d’Arabidopsis co-infectés, après traitement à la flg22 ou au peptide elf18 (Figure 6). IOS1 est associé avec BAK1 de manière indépendante du ligand proposé, et régule positivement la formation du complexe FLS2/BAK1 lors du traitement aux PAMPs. Cette équipe a conclu que IOS1 est une protéine régulatrice des voies de signalisation médiées par FLS2 et EFR vers une activation de la PTI en réponse à P. syringae.. 24.
(36) La grande famille protéique des LRR-RLK comprend probablement d’autres récepteurs membranaires pour la détection de PAMPs. Ces protéines ont la particularité de former des complexes multiprotéiques dynamiques lors de la liaison avec une molécule microbienne, et ces associations sont nécessaires à la transmission de la signalisation de la PTI (Figure 6).. Figure 6. BAK1 le récepteur kinase au centre de l’immunité Les motifs bactériens flg22 ou EF-Tu sont respectivement reconnus par les PRR FLS2 et EFR. Le corécepteur BAK1 recrute spécifiquement ces récepteurs et permet la transduction du signal. FLS2, EFR et BAK1 forment un complexe avec IOS1. Adapté de (Couto and Zipfel, 2016; Yeh et al., 2016). 25.
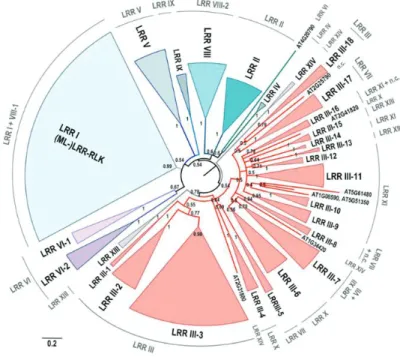
(37) 2.3 Les récepteurs RLKs comportant un domaine MLD Chez Arabidopsis, la famille des LRR-RLK comprend 215 membres (Hok et al., 2011) (Figure 7). Les LRR-RLKs sont impliqués dans les voies de signalisation du développement, en réponse à des hormones, à des changements environnementaux ainsi que lors de l’interaction avec des organismes microbiens (Liu et al., 2017). Les LRR-RLKs sont constitués d’un domaine kinase cytoplasmique suivi d’un domaine transmembranaire servant d’ancrage à la membrane. Une caractéristique de la famille de ces récepteurs est qu’ils détiennent un domaine extracellulaire pourvu de répétitions riches en leucine (motifs LRR) impliquées dans des interactions protéiques (Kobe and Kajava, 2001). IOS1 fait partie d’un cluster de 11 gènes issus de duplications d’un gène ancestral (At1g51790) qui sont subdivisées en deux sousgroupes résultant de deux autres séries indépendantes de duplications (Hok et al., 2011). Ces gènes codent tous pour des LRR-RLKs et ont la particularité de posséder un domaine extracellulaire « Malectin-Like » (MLD). Le MLD est nommé ainsi d’après sa ressemblance structurelle à la malectine identifiée chez le Xénope (Schallus et al., 2008). Le MLD de IOS1 est composé de deux sous-unités malectine. Des domaines MLD similaires à celui de IOS1 sont retrouvés dans 20% des LRR-RLK chez A. thaliana, bien que leur fonction soit peu étudiée (Hok et al., 2011) (Figure 7).. Figure 7. Arbre phylogénétique des protéines LRR-RLK d’Arabidopsis thaliana La famille des LRR-RLK est composée de 11 groupes. Le groupe LRR I contient les récepteurs arborant un domaine malectin-like (Hok et al., 2011).. 26.
(38) x. FERONIA, récepteur MLD-RLK. FERONIA (FER) est un récepteur similaire à IOS1 possédant une MLD, un domaine transmembranaire, une kinase cytoplasmique mais pas de LRR (Figure 8). Le MLD de FER est composé de deux sous-unités (tout comme IOS1) (Li et al., 2016). Cette protéine a la particularité d’avoir des fonctions multiples durant le développement, lors de la reproduction et dans la perception de l’environnement. Elle a été identifiée dans un premier temps comme un régulateur clé de la fertilité chez Arabidopsis. FER contrôle l’interaction pollen-ovule permettant la fécondation (Huck et al., 2003), dans les conditions normales, le tube pollinique germe en réponse à des molécules attractives du gamétophyte femelle, et se rapproche au plus près des sacs embryonnaires, afin de féconder les ovules. Chez les mutants fer perte de fonction, le processus de réception du tube pollinique est défaillant, ce qui entraine une incapacité de ces derniers à se rompre afin de libérer les gamètes, on parle d’infertilité féminine. Les mutants fer ont aussi des phénotypes sévères lors du développement des plantules, les plantes sont insensibles aux stimuli auxiniques pour l’élongation des poils absorbants racinaires (Duan et al., 2010), et sont hypersensibles à l’inhibition de l’élongation racinaire provoquée par l’ABA (Yu et al., 2012). FER est aussi nécessaire à la croissance des racines après un stress salin du sol important. En cas de stress les cellules sont abimées et FER permet le signalement des dommages via des canaux calciques. Tous ces phénotypes suggèrent que FERONIA recoupe plusieurs voies de signalisation dans la régulation du développement des plantules. FER participe aussi à la signalisation des réponses face aux agents pathogènes. Chez le mutant KO fer-4, les gènes marqueurs de la réponse immunitaire frk1, wrky53 et pdf1.2 sont régulés positivement de manière constitutive (Masachis et al., 2016). Afin d’expliquer son rôle dans la signalisation à la défense, il est nécessaire de trouver quel est le ligand de ce récepteur. Ce récepteur se lie à un peptide de la famille des RALF (Rapid Alcalinization Factor). En interaction avec RALF1, FER déclenche une voie de signalisation conduisant à une alcalinisation apoplastique et à une inhibition de l'élongation cellulaire (Haruta et al., 2014). Les RALFs ont été découverts chez Nicotiana tabacum pour leur fonction dans le développement des racines (Pearce et al., 2001), et sont capables d’ajuster le pH du milieu dans lequel la plante croit. Le génome d’Arabidopsis, code une famille d’au moins 35 peptides RALFs différents (Murphy and De Smet, 2014). Les microbes pathogènes fongiques utilisent des 27.
Figure

Documents relatifs
Consequently, better regenerator efficiency is obtained (distance improvement ratio of 1.4 to 1.6 compared to 1.2 at 10 dBm). The greater the fibre launched power, the stronger
Cambridge-London-New York-New Rochelle-Melbourne- Sydney, Cambridge University Press, 1984, xv+189p., bibl., index (“Cambridge Studies in Cultural Systems
In the proposed GBS algorithm, the incident field on or near the first reflecting surface is expanded into a superposition of frame windows, each of which radiates in the form of
The second principle is that any given physical sensor node can execute (locally) a task for a given application deployed in the overlay. Any given sensor node may
We show how an interactive graph visualization method based on maximal modularity clustering can be used to explore a large epidemic network.. The visual representation is used
A Spectral Database for the Recognition of Urban Objects in Kaunas City: Performance and Morphometric Issues. Sébastien Gadal 12 , Gintautas Mozgeris 3 , Donatas Jonikavicius 3