HAL Id: tel-01828072
https://tel.archives-ouvertes.fr/tel-01828072
Submitted on 3 Jul 2018
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
CARACTERISATION BIOCHIMIQUE ET
STRUCTURALE DE BACTERIOCINES CIBLANT LE
METABOLISME DU PEPTIDOGLYCANE
BACTERIEN, ALTERNATIVE POTENTIELLE AUX
ANTIBIOTIQUES.
Dimitri Cherier
To cite this version:
Dimitri Cherier. CARACTERISATION BIOCHIMIQUE ET STRUCTURALE DE
BACTERI-OCINES CIBLANT LE METABOLISME DU PEPTIDOGLYCANE BACTERIEN, ALTERNATIVE POTENTIELLE AUX ANTIBIOTIQUES.. Biochimie, Biologie Moléculaire. Université Paris-Saclay, 2017. Français. �NNT : 2017SACLS529�. �tel-01828072�
N N T : 2 0 17 S A C LS 5 29
Caractérisation biochimique et structurale de
bactériocines ciblant le métabolisme du
peptidoglycane bactérien, alternative
potentielle aux antibiotiques.
Thèse de doctorat de l'Université Paris-Saclay
Préparée à l’Université Paris-Sud
École doctorale n°569 : Innovation thérapeutique :
du fondamental à l'appliqué (ITFA)
Spécialité de doctorat : Biochimie et Biologie structuraleThèse présentée et soutenue à Orsay, le 14 Décembre 2017, par
Dimitri Chérier
Composition du Jury : Nicolas Bayan
Professeur, Université Paris-Sud
(–UMR9198-I2BC) Président
Ivo Gomperts Boneca
Directeur de recherche INSERM, Institut Pasteur Rapporteur
Jean-Emmanuel Hugonnet
Maître de conférences, Université Pierre et Marie Curie
(–UMRS 1138) Rapporteur
Didier Hilaire
Expert confirmé en biologie, Direction Générale de l’Armement
(–DGA Maîtrise NRBC) Examinateur
Sylvie Rebuffat
Professeur, Muséum National d’Histoire Naturelle
(– UMR 7245 CNRS-MNHN) Examinatrice
Hélène Barreteau
Maître de conférences, Université Paris-Sud
(– UMR9198-I2BC) Directrice de thèse
Dominique Mengin-Lecreulx Directeur de recherche CNRS
2
REMERCIEMENTS
Je tiens tout d’abord à remercier Monsieur Ivo Gomperts Boneca et Monsieur Jean-Emmanuel Hugonnet d’avoir accepté d’être rapporteur de ce travail. Je remercie également Madame Sylvie Rebuffat et Messieurs Nicolas Bayan et Didier Hilaire d’avoir accepté de faire partie de mon jury de thèse et d’avoir accepté d’évaluer ce travail.
Je tiens également à adresser ces remerciements à la Direction Générale de l’Armement, qui m’a permis de réaliser ce doctorat en m’accordant un financement de 3 ans.
Je tiens ensuite à remercier vivement le Dr. Dominique Mengin-Lecreulx de m’avoir accueilli au sein de son équipe de recherche Enveloppes Bactériennes et Antibiotiques. « Merci Chef » comme j’ai eu si souvent l’occasion de te le dire au cours de ma thèse. Merci pour l’ensemble des conseils que tu as pu me prodiguer, que ce soit au niveau scientifique ou autre (en mécanique notamment !). Merci pour ta disponibilité et pour le soutien que tu m’as pu m’apporter, plus particulièrement durant cette longue période qu’a constitué la rédaction de ce manuscrit. J’espère que nos chemins se recroiseront à l’avenir, pourquoi pas à La ferme du bout des prés autour d’un cochon grillé pour commencer ? Ou bien encore au bord d’un terrain de rugby ?
Je tiens également à remercier le Dr. Hélène Barreteau. Hélène, un immense merci pour tout ! Sans toi la réalisation de cette thèse n’aurait pas été possible. C’est à toi qu’est revenue la lourde tâche de m’encadrer, de m’aiguiller et de me conseiller durant ces trois années, ce que tu as fait à la perfection ! Merci de m’avoir offert la possibilité de réaliser cette expérience qu’est la thèse ! Merci pour tous les moments partagés (pizzas, sushis, bières, concours de sudoku… Tu veux un moshi ?). Merci de ne m’avoir jamais fait de remarques le lundi matin quand j’arrivais un peu trop fatigué suite à victoire sur le pré et la troisième mi-temps qui l’avait accompagnée. Merci pour ta joie de vive et ta bonne humeur permanentes ! Merci pour tout ! Le petit scarabée, comme tu m’as surnommé, te remercie du fond du cœur.
Je tiens également à remercier tous les autres membres de l’équipe. Merci mon Rodolpho pour les moments de franche rigolade, merci pour ta gentillesse et ta bienveillance. Ton caractère de cochon va me manquer, mais je suis sûr que nous nous reverrons très prochainement autour d’une bière avec Kanello ! Je tiens également à remercier Delphine Patin, ma voisine de bureau. Merci pour l’aide que tu as pu m’apporter, pour tes conseils et pour les manips que tu as réalisées pour moi. Tes « ça me gave, ça me gave, ça me gave » resteront gravé dans ma mémoire ! Je remercie également Thierry Touzé. Merci pour tes suggestions, tes idées de manips qui m’ont permis d’avancer dans ce travail. Merci également pour nos débats enflammés qui ont animés de nombreuses pauses café ! Je tiens également à remercier Xudong Tian. Merci Xudong pour ta gentillesse, ta sympathie et tout le reste, parce qu’avec toi « y’a pas de problème ». Cette chambrée commune au Portugal nous aura permis de mieux nous connaître ! Ne change pas ! Je te souhaite le meilleur pour la fin de ta thèse ; J’assisterai avec un vif plaisir à ta soutenance ! Je tiens également à remercier le Dr. Didier Blanot. Tes passages hebdomadaires au laboratoire ont toujours été très enrichissants. Merci pour ton expertise et ta gentillesse. Merci pour la relecture que tu as faite de ce manuscrit et pour les corrections que tu as pu y apporter. Je tiens également à saluer Victorien Decoin avec qui j’ai partagé de très bons moments lors son passage au laboratoire en tant que « post-doc ». Merci d’être venu me voir à Marcoussis lors des Championnats de France de 7 malgré une chaleur accablante et une absence totale d’endroits ombragés !
Je tiens également à remercier particulièrement Inès Gallay ainsi que Stéphane Plancqueel pour leur gentillesse et leur disponibilité lors de la réalisation des expériences de cristallographie. Merci à vous deux, maintenant nous n’avons plus qu’à croiser les doigts pour avoir une structure exploitable prochainement.
Je tiens également à remercier Magalie Aumont-Nicaise pour son aide, ses conseils et par-dessus tout sa bonne humeur ! Merci de m’avoir permis de réaliser les tests d’interactions sur cette belle plateforme !
Je remercie tout particulièrement Marie-Hélène Sarda et Martine Denis pour leur gentillesse et le travail formidable qu’elles effectuent.
Je tiens également à remercier mes collègues enseignants, Moussa, Fred, Boris, Marianne…
Je tiens à également à remercier toutes les personnes avec qui j’ai pu échanger au sein de l’institut : Agathe, Marielle, Agnès, Dominique, Michel (merci pour le dépannage !), Olivier, Corentin, Sébastien, Jean, Ahmed, Herman, Sylvie, Noureddine…
3 Je tiens à remercier chaleureusement Margarida Gomes et Adeline Pichard-Kostuch. Un immense merci à vous deux ! Merci de me supporter depuis le Master, ça ne doit pas être facile tous les jours ! Merci d’avoir été là pour moi dans les bons comme dans les mauvais moments ! Merci pour ces nombreux jeudis soirs que l’on a passé ensemble, en compagnie de Clément Madru, que je salue également. Merci ma petite Maggie, Beijinhos ! Merci Petit Faron, merci de m’avoir épaulé dans la dernière ligne droite de la rédaction, notamment de m’avoir évité de perdre un temps fou dans les méandres d’ADUM ! Petit faron « you’re eating my food’s food and i don’t appreciate that ».
Je tiens à adresser un hommage tout particulier à Monsieur Thomas Ducrocq aka « Sustanon » qui figure dans ces remerciements en partie grâce à un « pour combien » mais également parce que t’es un chic type ! Ce défi de te faire apparaître dans mes remerciements, tu me l’as lancé pendant une de nos fameuses séances de bibis avec Bruno et je tenais à le coucher sur le papier pour me le remémorer dans quelques années !
Merci également à la famille du CA.ORSAY.RC. J’ai passé des saisons formidables à vos côtés et jouer en votre compagnie m’a permis de garder un certain équilibre essentiel pour pouvoir mener à bien ce travail de thèse.
Je tiens également à remercier Romaric Jumetz, mon copain de toujours. Je n’ai pas besoin d’en dire beaucoup plus. Merci pour ton soutien, et dans quelques semaines, je pourrai de nouveau servir de co-pilote et rattraper le temps perdu ! Par la même occasion, je tiens à remercier Ludovic Sotoca, qui m’a encouragé depuis l’autre bout du globe, en Nouvelle-Zélande. Merci mon Lulu et ne t’en fais pas pour ta calvitie, la science progresse à grands pas !
Merci à mes nouveaux colocs : Léo, La Bapt, Alexis et Alexis ! Merci de m’avoir fait à manger quand je rentrais tard le soir après une longue journée de rédaction. Merci de m’avoir accepté aux Lulus !
Je tiens également à remercier mes parents du fond du cœur ! Merci de m’avoir toujours poussé à continuer mes études. Je crois que je ne peux pas aller beaucoup plus loin maintenant ! Merci Maman, merci papa, merci Garf !
Enfin, je tiens à remercier Charlène Devilleger d’avoir eu la patience et le courage de me supporter, surtout pendant la période de rédaction ! Merci beaucoup, car sans toi ça aurait été beaucoup plus dur ! Je remercie également tes parents et ton frère pour leur accueil chaleureux à chaque fois que je suis venu chez vous.
4
Table des matières
Table des matières ... 4
Liste des Figures ... 8
Liste des tableaux ... 12
Liste des abréviations ... 13
Introduction Bibliographique ... 15
I. Le Peptidoglycane : ... 16
1. Structure et fonction du peptidoglycane :... 17
2. Biosynthèse du peptidoglycane : ... 19
Les étapes cytoplasmiques : ... 19
Les étapes membranaires : ... 26
Polymérisation du peptidoglycane : ... 30
3. Recyclage du peptidoglycane : ... 33
4. Variabilité du peptidoglycane : ... 34
Variabilité des chaînes de glycanes : ... 34
Variabilité de la partie peptidique : ... 35
II. Les antibiotiques :... 38
1. Les différentes cibles des antibiotiques : ... 38
2. Les antibiotiques qui ciblent la biosynthèse de la paroi bactérienne : ... 39
Exemple de la fosfomycine qui cible une étape cytoplasmique de la biosynthèse du peptidoglycane : ... 40
Exemple de la bacitracine qui cible une étape du métabolisme de l’undécaprényl phosphate : ... 40
Les antibiotiques ciblant les étapes de polymérisation du peptidoglycane : ... 40
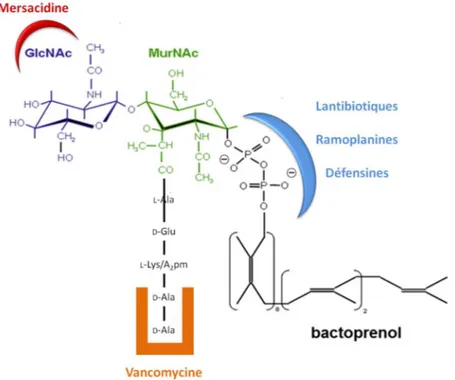
Les molécules antimicrobiennes ciblant le lipide II : ... 41
3. La résistance aux antibiotiques : ... 44
III. Les bactériocines : ... 48
1. Définition : ... 48
2. Les bactériocines des bactéries à Gram positif : ... 48
3. Les bactériocines des bactéries à Gram négatif : ... 49
Les bactériocines de faible masse moléculaire des bactéries à Gram négatif : ... 50
Les bactériocines de haute masse moléculaire des bactéries à Gram négatif : ... 52
IV. Les colicines : ... 53
1. Classification des colicines : ... 53
2. Organisation génétique des colicines : ... 54
3. Organisation structurale des colicines :... 55
4. Mode d’action des colicines : ... 57
Libération des colicines : ... 57
Liaison à la cellule cible : ... 58
5
Maturation des colicines : ... 68
Activité cytotoxique des colicines : ... 69
5. La protéine d’immunité : ... 73
Protéines d’immunité des colicines ionophoriques : ... 73
Protéines d’immunité des colicines nucléasiques : ... 73
V. La colicine M et ses homologues : ... 75
1. La colicine M : ... 75
Caractéristiques de la colicine M : ... 75
Structure tridimensionnelle de la colicine M :... 75
Mode d’action de la colicine M : ... 77
2. Les homologues de la colicine M : ... 88
Les homologues issus de Pseudomonas spp. : ... 88
Les homologues issus de Burkholderia spp. : ... 94
Les homologues issus de Pectobacterium carotovorum : ... 95
VI. Objectifs de l’étude : ... 99
Matériels et Méthodes ... 102
1. Produits et réactifs : ... 103
2. Souches bactériennes, plasmides et conditions de culture :... 103
Souches : ... 103
Milieux de culture : ... 104
Plasmides : ... 104
3. Techniques de biologie moléculaire : ... 106
Oligonucléotides : ... 106
PCR : ... 107
Extraction d’ADN plasmidique : ... 107
Restriction et ligation des fragments d’ADN : ... 108
Construction des plasmides : ... 108
Mutagenèse dirigée : ... 109
Préparation des cellules compétentes et transformation : ... 109
4. Surproduction, extraction et purification des protéines recombinantes : ... 110
Surproduction : ... 110
Extraction :... 111
Purification par chromatographie d’affinité : ... 111
Purification par chromatographie d’exclusion stérique : ... 111
Purification par chromatographie échangeuse de cations : ... 112
Purification des protéines produites en corps d’inclusion : ... 113
5. Dosage des protéines : ... 113
Détermination de la concentration protéique par spectrophotométrie microvolume :114 Détermination de la concentration protéique par analyse en acides aminés : ... 114
6. Protéolyse ménagée : ... 114
7. Synthèse de substrats lipidiques et production d’un analogue du produit de réaction : .... 114
Synthèse et purification de C55-PP-MurNAc(-pentapeptide)-[14C]GlcNAc : ... 115
Synthèse et purification de lipides II solubles : ... 115
Préparation et purification du 1-PP-MurNAc-pentapeptide : ... 116
8. Spectrométrie de masse MALDI-TOF : ... 117
Analyses du lipide II en C15 et du 1-PP-MurNAc-pentapeptide ... 117
Analyse de la protéine PcaM1 : ... 118
6
Evaluation de l’activité enzymatique : ... 118
Recherche de ligand de la protéine PaeM : ... 119
Evaluation de l’activité cytotoxique : ... 119
10. Analyses physiologiques : ... 120
Suivi d’incorporation de l’A2pm dans le peptidoglycane : ... 120
Analyse de la distribution cellulaire du meso-A2pm : ... 120
Quantification des pools de prénols dans les membranes : ... 120
Observations microscopiques : ... 121
11. Cristallogenèse : ... 121
Principe : ... 121
Criblage automatisé : ... 123
Optimisation des conditions de cristallisation : ... 123
Micro-ensemencement : ... 123
Essais de co-cristallisation et trempage des cristaux :... 124
Collecte des données de diffraction et résolution de structure : ... 124
12. Microcalorimétrie : ... 124
Titration par microcalorimétrie isotherme (ITC) : ... 125
Calorimétrie différentielle par balayage (DSC) : ... 127
Résultats et Discussion ... 128
I. Synthèse de substrats solubles :... 129
1. Synthèse de lipides II solubles : ... 129
2. Détermination de l’activité catalytique des protéines ColM, PaeM et PcaM1 vis-à-vis des lipides II solubles : ... 132
3. Optimisation des conditions de synthèse du lipide II en C15 : ... 135
4. Purification et confirmation de l’identité du lipide II en C15 : ... 136
5. Synthèse, purification et confirmation de l’identité du 1-PP-MurNAc-pentapeptide : ... 138
II. Etudes structurales et biochimiques de la protéine PaeM et de deux variants : . 140 1. Production et purification de la PaeM :... 140
2. Identification d’un ligand potentiel de la PaeM : ... 141
Par le test de détection d’activité enzymatique : ... 141
Par microcalorimétrie : ... 142
3. Essais de co-cristallisation de la PaeM avec le C15-PP : ... 144
4. Essais de cristallogenèse de la protéine PaeM D241A : ... 148
Production et purification de la PaeM D241A : ... 148
Cristallogenèse de la protéine PaeM D241A : ... 150
5. Etude structurale d’une forme tronquée en N-terminal de la PaeM : ... 157
Genèse d’une forme tronquée par protéolyse ménagée : ... 158
Construction de la protéine PaeM Δ1-30 : ... 159
Production et purification de la PaeM Δ1-30 : ... 159
Surproduction et purification de la PaeM Δ1-30 : ... 161
Détermination de l’activité enzymatique de la PaeM Δ1-30 : ... 163
Cristallogenèse de la PaeM Δ1-30 : ... 163
6. Mise en évidence d’une certaine cytotoxicité de la PaeM vis-à-vis d’E. coli : ... 169
Confirmation de la cytotoxicité réelle de la PaeM : ... 169
Dépendance de la cytotoxicité de la PaeM vis-à-vis de partenaires protéiques : ... 171
Recherche d’une cytotoxicité identique de la part des autres homologues de la ColM : ... ... 174
7
III. Etude de la cytotoxicité de la PcaM1 vis-à-vis d’E. coli : ... 175
1. Contexte de l’étude : ... 175
2. Article : ... 176
3. Résumé des principaux résultats obtenus : ... 199
4. Expériences complémentaires :... 200
Etude de la cytotoxicité de la PcaM1 D222N vis-à-vis d’E. coli :... 200
Etude de la cytotoxicité des homologues de la ColM issus de Pseudomonas spp. vis-à-vis d’E. coli par le biais de l’expression périplasmique : ... 203
IV. Création de colicines chimères ciblant spécifiquement E. coli : ... 206
1. Conception des colicines chimères : ... 206
2. Construction des vecteurs permettant l’expression des colicines chimères : ... 208
3. Détermination des conditions d’expression et purification des colicines chimères : ... 209
4. Purification des colicines chimères à partir des corps d’inclusion : ... 212
5. Evaluation de l’activité in vitro et in vivo des colicines chimères : ... 214
6. Evaluation de la cytotoxicité des colicines chimères vis-à-vis d’E. coli par le biais d’une expression périplasmique : ... 217
Construction des vecteurs d’expression périplasmique : ... 217
Expression périplasmique des colicines chimères : ... 218
Analyse du contenu en prénols des membranes des cellules traitées par les colicines chimères : ... 219
Conclusions et Perspectives ... 223
8
Liste des Figures
Figure 1 : Représentation schématique des différents types de parois bactériennes ... 17
Figure 2 : Structure du peptidoglycane chez Escherichia coli. ... 18
Figure 3 : Schéma de la voie de biosynthèse du peptidoglycane chez E. coli ... 19
Figure 4 : Formation de l’UDP-N-acétylglucosamine. ... 20
Figure 5 : Structure cristallographique des protéines GlmS, GlmM et GlmU ... 21
Figure 6 : Synthèse de l’acide UDP-N-acétylmuramique. ... 22
Figure 7 : Réaction catalysée par les Mur ligases (d’après Barreteau et al., 2008). ... 24
Figure 8 : Structures tridimensionnelles des Mur ligases ... 25
Figure 9 : Topologie membranaire et structure cristallographique de MraY d’Aquifex aeolicus ... 27
Figure 10 : Structure du lipide II d’une bactérie à Gram négatif... 28
Figure 11 : Structure cristallographique de MurJ de Thermosipho africanus et mécanisme putatif de la translocation du lipide II. ... 30
Figure 12 : Polymérisation du peptidoglycane ... 31
Figure 13 : Voies de transpeptidation. ... 32
Figure 14 : Schéma de la biosynthèse et du recyclage du peptidoglycane.. ... 33
Figure 15 : Variabilité des chaînes de glycanes. ... 35
Figure 16 : Variabilité de la partie peptidique ... 36
Figure 17 : Cibles cellulaires des antibiotiques ... 39
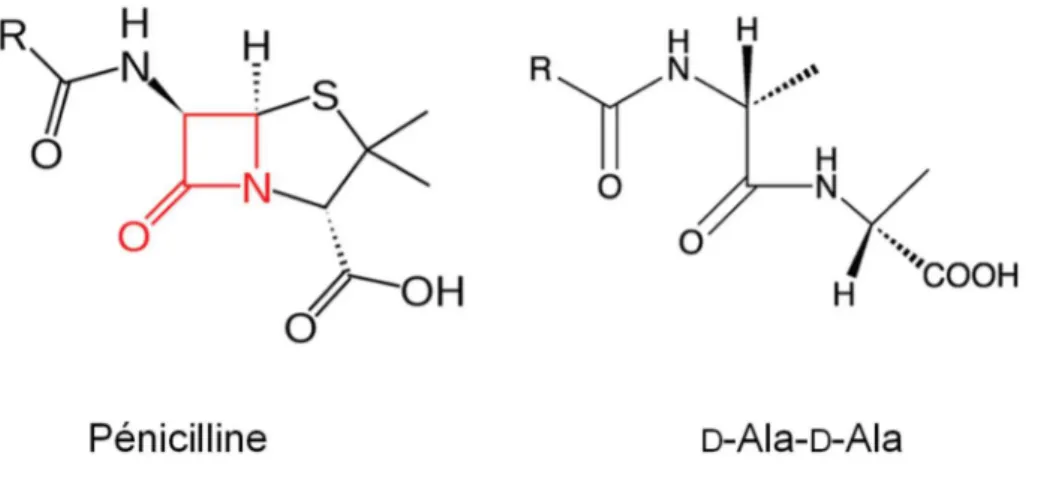
Figure 18 : Homologie structurale entre la pénicilline et le dipeptide terminal D-Ala-D-Ala ... 41
Figure 19 : Structure primaire de la nisine ... 42
Figure 20 : Structure tridimensionnelle de la nisine en complexe avec le lipide II ... 42
Figure 21 : Le lipide II, cible de nombreux antibactériens ... 44
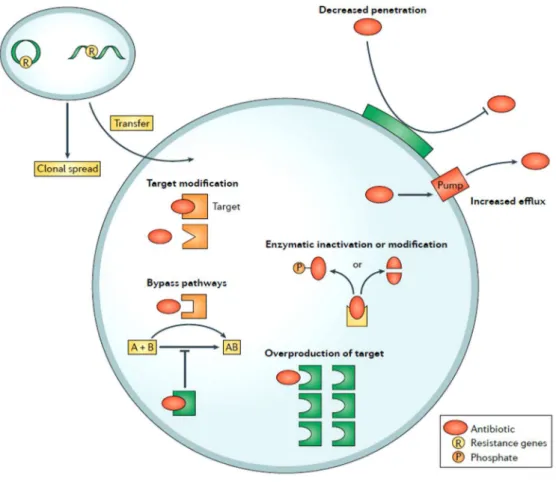
Figure 22 : Illustration de l’acquisition d’une résistance à un antibiotique au sein d’une culture bactérienne. ... 45
Figure 23 : Mécanismes de résistance aux antibiotiques ... 46
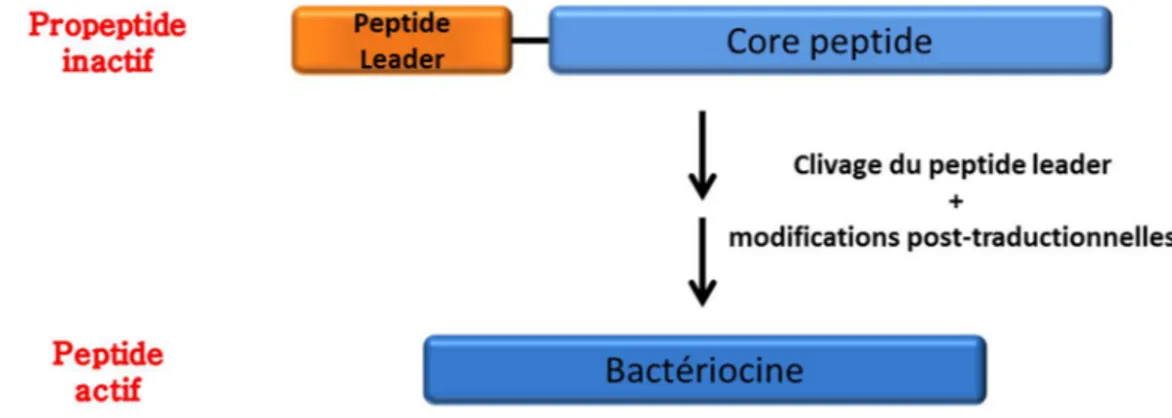
Figure 24 : Schéma général de maturation des bactériocines de faible masse moléculaire ... 51
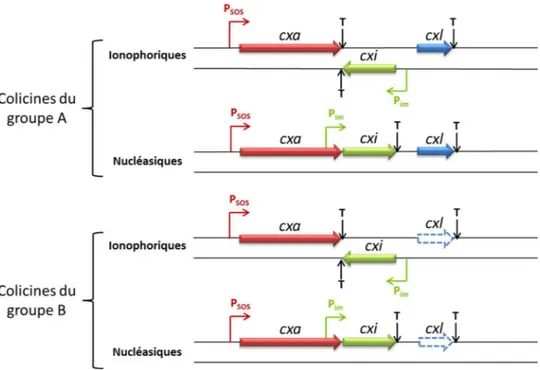
Figure 25 : Classification des colicines ... 53
Figure 26 : Organisation génétique des colicines ... 55
Figure 27 : Schéma général de l’organisation modulaire en trois domaines des colicines ... 56
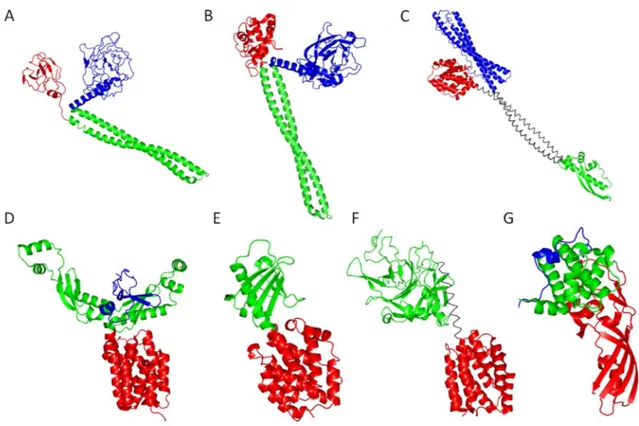
Figure 28 : Exemples de structures tridimensionnelles de colicines ... 57
9
Figure 30 : Homologies entre les systèmes de translocation TonB et Tol ... 61
Figure 31 : Superposition des structures tridimensionnelles des domaines C-terminaux de TolA et TonB ... 62
Figure 32 : Représentation schématique du translocon formé par la ColE9 avec les protéines BtuB et OmpF ... 63
Figure 33 : Modèle de translocation pour les colicines du groupe A ... 64
Figure 34 : Présentation des deux modèles de translocation proposés pour la colicine Ia à travers le récepteur Cir ... 66
Figure 35 : Modèle de translocation de la colicine B à travers son récepteur FepA. ... 67
Figure 36 : Modèle général de translocation pour les colicines du groupe B. ... 68
Figure 37 : Modèle de translocation des colicines nucléasiques ... 69
Figure 38 : Structures tridimensionnelles des domaines ionophoriques isolés des colicines A, N, B, Ia et E1 ... 70
Figure 39 : Représentation schématique des deux modèles d’insertion d’un domaine ionophorique au sein de la membrane interne ... 71
Figure 40 : Modèle putatif de formation du pore pour la ColA ... 71
Figure 41 : Topologie membranaire des protéines d’immunité de types A et E1 associées aux colicines ionophoriques. ... 73
Figure 42 : Structures tridimensionnelles de domaines nucléasiques de colicines en complexe avec leurs protéines d’immunité ... 74
Figure 43 : Organisation génétique des colicines B et M. ... 75
Figure 44 : Structure tridimensionnelle de la colicine M ... 76
Figure 45 : Structure du complexe formé entre FhuA et TonB ... 78
Figure 46 : Structure tridimensionnelle de FkpA sous sa forme dimérique ... 80
Figure 47 : Morphologie cellulaire d’une souche d’E. coli K-12 ROW/V/22.1 traitée par la colicine M. ... 82
Figure 48 : Mode d’action de la colicine M ... 83
Figure 49 : Localisation des résidus du site actif à la surface de la ColM ... 84
Figure 50 : Structure tridimensionnelle de la Cmi. ... 86
Figure 51 : Alignement de séquence de la ColM avec ses homologues ... 88
Figure 52 : Structure tridimensionnelle de la PsyM ... 90
Figure 53 : Structure tridimensionnelle de la PaeM ... 91
Figure 54 : Comparaison de l’orientation des chaînes latérales des résidus essentiels à l’activité de ColM et PaeM ... 92
10 Figure 55 : Topologie membranaire des différentes protéines d’immunité associées aux homologues
de la ColM ... 93
Figure 56 : Structure tridimensionnelle du récepteur de membrane externe FusA. ... 96
Figure 57 : Structure tridimensionnelle de la PcaM2 ... 97
Figure 58 : Modèle de liaison de la PcaM2 à la surface de FusA ... 98
Figure 59 : Oxydation de l’UDP-MurNAc-pentapeptide par le métapériodate de sodium pour l’obtention de l’analogue de produit de réaction 1-PP-MurNAc-pentapeptide ... 117
Figure 60 : Diagramme de phase ... 122
Figure 61 : Schéma d’un appareil d’ITC ... 126
Figure 62 : Schéma d’une expérience d’ITC. ... 126
Figure 63 : Essai de synthèse de C10-lipide II avec les enzymes MraYBs et MurGEc. ... 129
Figure 64 : Suivi de synthèse du lipide II en C10... 130
Figure 65 : Suivi de synthèse du lipide II en C15... 131
Figure 66 : Isomérie du C10-P, du C15-P et du C55-P... 132
Figure 67 : Dégradation in vitro des lipides II en C10 et C15 par les enzymes ColM, PaeM et PcaM1 .. 133
Figure 68 : Synthèse de lipide II en C15 ... 135
Figure 69 : Synthèse de lipide II en C15 en présence d’un excès de C15-PP ... 136
Figure 70 : Spectre de masse du lipide II en C15 purifié ... 137
Figure 71 : Spectre de masse du 1-PP-MurNAc-L-Ala-γ-D-Glu-L-Lys-D-Ala-D-Ala purifié.. ... 138
Figure 72 : Profil de purification de la protéine PaeM. ... 140
Figure 73 : Thermogramme de la PaeM seule ... 143
Figure 74 : Thermogramme de la PaeM en absence ou en présence de 1-PP-MurNAc-pentapeptide. ... 144
Figure 75 : Cinétique d’apparition des cristaux au sein de la condition H04 du kit « Classics » en présence (A) ou en absence (B) de C15-PP. ... 147
Figure 76 : Profil de purification de la protéine PaeM D241A ... 149
Figure 77 : Images des cristaux de la PaeM D241A ... 152
Figure 78 : Structure tridimensionnelle du mutant catalytique PaeM D241A ... 152
Figure 79 : Comparaison des sites actifs des protéines PaeM et PaeM D241A ... 154
Figure 80 : Site actif de la PaeM ... 155
Figure 81 : Mécanisme putatif de coupure du lipide II en deux étapes par la PaeM. ... 156
Figure 82 : Profils de digestion de la PaeM par les protéases trypsine, chymotrypsine et protéinase K. ... 158
Figure 83 : Profil de purification de la PaeM à l’issue de la protéolyse ménagée par la protéinase K.. ... 159
11
Figure 84 : Profils de purification de la protéine PaeM Δ1-30 (étiquette histidine en C-terminal) .... 160
Figure 85 : Profils de purification de la protéine PaeM Δ1-30 possédant l’étiquette His6 en N-terminal ... 161
Figure 86 : Profil de purification de la protéine PaeM Δ1-30. ... 162
Figure 87 : Dégradation du lipide II in vitro par la PaeM Δ1-30 ... 163
Figure 88 : Image de cristaux de PaeM Δ1-30 ... 165
Figure 89 : Délimitation des différents domaines de la PaeM ... 167
Figure 90 : Evaluation de la cytotoxicité de la PaeM vis-à-vis d’une souche d’E. coli BW25113 ... 169
Figure 91 : Evaluation de l’activité cytotoxique de différentes préparations de PaeM purifiées sur une souche d’E. coli (BW25113) ... 170
Figure 92 : Détermination de la quantité minimale de protéine PaeM nécessaire pour induire la lyse d’E. coli ... 170
Figure 93 : Evaluation de l’activité cytotoxique de la PaeM D241A sur une souche d’E. coli BW25113. ... 171
Figure 94 : Dépendance de la cytotoxicité de la PaeM sur E. coli vis-à-vis des protéines FhuA, TonB et FkpA ... 171
Figure 95 : Inhibition de l’activité cytotoxique de la PaeM par la protéine Cmi ... 173
Figure 96 : Evaluation de l’activité cytotoxique des protéines PflM, PsyM et PcaM1 sur une souche d’E. coli BW25113 ... 174
Figure 97 : Effet de l’expression périplasmique de la PcaM1 D222N sur la croissance d’E. coli ... 201
Figure 98 : Effet de l’expression périplasmique des homologues issus de Pseudomonas spp. sur la croissance d’E. coli ... 204
Figure 99 : Analyses par HPLC du contenu en prénols des membranes des cellules d’E. coli traitées par les différents homologues issus de Pseudomonas spp. ... 205
Figure 100 : Représentation schématique des différentes colicines chimères construites ... 208
Figure 101 : Profil de purification des chimères CCA1, CCA3 et de la ColM 121QM122 :: 121EF122 ... 210
Figure 102 : Cytotoxicité de la ColM 121QM122 :: 121EF122 vis-à-vis d’une souche d’E. coli BW25113 ... 211
Figure 103 : Profils de purification des chimères CCA1 et CCC2 renaturées à partir des corps d’inclusion ... 213
Figure 104 : Profils électrophorétiques des différentes chimères renaturées et purifiées. ... 214
Figure 105 : Dégradation du lipide II in vitro par les colicines chimères ... 215
Figure 106 : Effet de l’expression périplasmique des différentes colicines chimères sur la croissance d’E. coli ... 218
Figure 107 : Analyse par HPLC du contenu en prénols des membranes des cellules traitées par les différentes colicines chimères ... 220
12
Liste des tableaux
Tableau 1 : Classification des bactériocines ... 48
Tableau 2 : Activité in vitro et in vivo de différents mutants de la colicine M ... 84
Tableau 3 : Activité de la ColM vis-à-vis des lipides II ramifiés ... 85
Tableau 4 : Activités spécifiques de la ColM et de ses homologues ... 89
Tableau 5 : Liste des souches d’E. coli utilisées. ... 103
Tableau 6 : Liste des plasmides ... 104
Tableau 7 : Oligonucléotides utilisés ... 106
Tableau 8 : Identités, masses moléculaires et temps de rétention des composés utilisées pour la calibration de la colonne Hi-Load 16/600 Superdex S200 ... 112
Tableau 9 : Activité résiduelle (en pourcentage) de la PaeM en présence des différentes molécules testées ... 141
Tableau 10 : Température de demi-dénaturation et enthalpies de dénaturation de la PaeM en absence ou en présence de 1-PP-MurNAc-L-Ala-γ-D-Glu-L-Lys-D-Ala-D-Ala. ... 143
Tableau 11 : Conditions de cristallisation obtenues pour la PaeM seule. ... 145
Tableau 12 : Conditions de cristallisation de la PaeM en présence de C15-PP ... 146
Tableau 13 : Conditions de cristallisation de la PaeM D241A... 150
Tableau 14 : Liste des conditions de cristallisation de la PaeM Δ1-30 ... 163
Tableau 15 : Optimisation des conditions de cristallisation de la PaeM Δ1-30. ... 166
Tableau 16 : Conditions d’expression optimales des cinq colicines chimères et de la ColM 121QM122 :: 121EF122. ... 210
13
Liste des abréviations
- 2YT : Milieu yeast-tryptone - M63 : Milieu minimum
- ADN : Acide désoxyribonucléique - AHT : Anhydrotétracyline
- Amp : Ampicilline
- ARN : Acide ribonucléique - ATP : Adénosine triphosphate - C10-P : Néryl phosphate - C15-P : Farnésyl phosphate - C15-PP : Farnésyl pyrophosphate - C55-OH : Undécaprénol - C55-P : Undécaprényl phosphate - C55-PP : Undécaprényl pyrophosphate - Cam : Chloramphénicol - CCA : Chimère coli-aeruginosa - CCC : Chimère coli-carotovorum
- Cmi : Protéine d’immunité associée à la colicine M - ColM : Colicine M
- Da : Dalton
- DDM : n-dodecyl-β-D-maltopyranoside
- DO : Densité optique
- DSC : Differential scanning calorimetry / Calorimétrie différentielle par balayage - E : Fraction d’élution
- EB : Extrait brut
- FT : Flow through / Fraction non retenue - GlcNAc : N-acétylglucosamine
- ITC : Isothermal titration calorimetry / Titration par calorimétrie isotherme - Kan : Kanamycine
- MALDI-TOF : Matrix assisted laser desorption ionisation – Time of flight - meso-A2pm : acide meso-diaminopimélique
- MraYAa : MraY d’Aquifex aeolicus
- MraYBs : MraY de Bacillus subtilis
- MurGEc : MurG d’Escherichia coli
- MurNAc : acide N-acétylmuramique - Ni2+-NTA : nickel-nitrilotriacétate
- PaeM : homologue de la colicine M issu de Pseudomonas aeruginosa - PcaM : homologue de la colicine M issu de Pectobacterium carotovorum - PflM : homologue de la colicine M issu de Pseudomonas fluorescens - Pi : Phosphate inorganique
14 - RMSD : Root mean square deviation
- SDS-PAGE : Sodium dodecyl sulfate – polyacrylamide gel electrophoresis - TCA : acide trichloroacétique
- TLC : Thin layer chromatography / Chromatographie sur couche mince - UDP : Uridine diphosphate
15
16
I. Le Peptidoglycane :
Le peptidoglycane (ou muréine) est une macromolécule biologique essentielle et spécifique de la paroi de la plupart des eubactéries (Neidhardt, 1996; Rogers et al., 1980; Vollmer et al., 2008a). Il s’agit d’un polymère continu localisé à la périphérie de la membrane plasmique. Sa principale fonction est de protéger la bactérie contre les effets de la pression osmotique interne exercée par le cytoplasme sur la membrane cytoplasmique. Le peptidoglycane est également responsable du maintien d’une forme cellulaire définie. Il est aussi associé aux processus de croissance et de division cellulaire (Nanninga, 1998; Vollmer et al., 2008b). L’absence de peptidoglycane dans un milieu hypotonique conduit à la mort de la bactérie suite à la rupture de sa membrane cytoplasmique. Dans certaines conditions, des bactéries dépourvues de peptidoglycane, appelées « formes L », peuvent survivre sous forme de protoplastes ou sphéroplastes. Cependant, la forme caractéristique de la bactérie est perdue et la division cellulaire est fortement altérée (Smith, 1968; Errington, 2017). Le peptidoglycane sert également de point d’ancrage pour d’autres constituants majeurs de la paroi bactérienne tels que les acides teichoïques (Baddiley, 1972) ou la lipoprotéine de Braun (Braun, 1975).
Chez les bactéries à Gram positif, la paroi est caractérisée par la présence d’une épaisse couche de peptidoglycane qui entoure la membrane cytoplasmique et qui se retrouve en contact direct avec le milieu extérieur. Cette couche atteint une épaisseur comprise entre 20 et 50 nm et peut représenter jusqu’à 50% de la paroi (Figure 1A) (Shockman et Barrett, 1983). Chez les bactéries à Gram négatif, la couche de peptidoglycane est beaucoup plus fine, avec une épaisseur comprise entre 3 et 6 nm (Gan et al., 2008). Chez ces bactéries, le peptidoglycane représente 10 à 20% de la paroi bactérienne et est situé dans l’espace périplasmique, entre la membrane cytoplasmique et la membrane externe. Cette dernière est une membrane asymétrique dont la face extérieure est composée exclusivement de lipopolysaccharides dont la longueur de la chaîne glucidique varie en fonction des espèces bactériennes (Kamio et Nikaido, 1976; Op den Kamp, 1979) (Figure 1B).
Les corynébactéries et les mycobactéries présentent une paroi atypique dont certains aspects sont communs à la fois aux bactéries à Gram positif et aux bactéries à Gram négatif. Ainsi, leur membrane cytoplasmique est entourée par une couche de peptidoglycane liée de façon covalente par des liaisons phosphodiester à un polymère d’arabinogalactane. Cette couche d’arabinogalactane est elle-même estérifiée par des acides mycoliques. Cette structure constitue le complexe « mycolyl-arabinogalactan-peptidoglycan » (mAGP complex) (Brennan, 2003). Au-dessus de ce complexe repose une deuxième couche de lipides de nature diverse. Ces lipides sont dits « libres » car ils sont liés de façon non covalente à la couche d’acides mycoliques. Cet ensemble lipidique forme une pseudo-bicouche lipidique appelée mycomembrane et est traversé par des porines. Enfin, la couche la plus externe de cette paroi particulière est constituée majoritairement de polysaccharides et de protéines. Cette partie est appelée capsule chez les espèces pathogènes (Zuber et al., 2008).
17
Figure 1 : Représentation schématique des différents types de parois bactériennes. A : Représentation de la paroi des bactéries à Gram négatif. B : Représentation de la paroi des bactéries à Gram positif. C : Représentation de la paroi des mycobactéries. (D’après (Brown et al., 2015).
1. Structure et fonction du peptidoglycane :
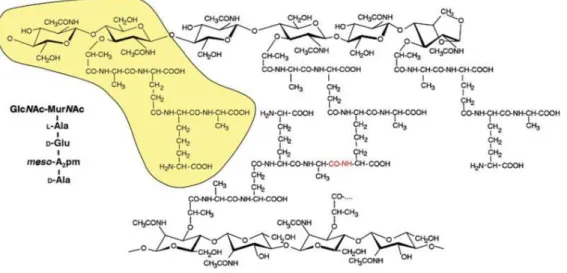
Le peptidoglycane est un hétéropolymère constitué de longues chaînes de sucres reliées entre elles par de courts peptides (Schleifer et Kandler, 1972; van Heijenoort, 2001; Vollmer et al., 2008a). La partie osidique est composée d’une alternance d’acide acétylmuramique (MurNAc) et de N-acétylglucosamine (GlcNAc) reliés par des liaisons β-(1→4). La fonc on acide carboxylique du motif lactoyl en C3 de chaque MurNAc est substituée par un pentapeptide. Au sein du peptidoglycane naissant, la séquence de ce pentapeptide est dans la majorité des cas L-alanyl-γ-D
-glutamyl-meso-diaminopimelyl(ou L-lysyl)-D-alanyl-D-alanine (Figure 2). Dans le peptidoglycane mature, la partie
peptidique est délestée de l’un voire des deux résidus de D-alanine terminaux. La connexion entre deux
chaînes osidiques est assurée par ces sous-unités peptidiques. En effet, la liaison entre deux chaînes de glycanes s’établit généralement entre la fonction amine libre de l’acide diaminé en position 3 d’une sous-unité accepteuse et la fonction carboxylique de la D-alanine en position 4 d’une sous-unité
donneuse appartenant à une chaîne voisine. Cette condensation est responsable de la perte du cinquième résidu (D-alanine) de la sous-unité donneuse. La réticulation entre deux chaînes osidiques
peut se faire soit de façon directe, comme mentionné précédemment, soit de façon indirecte par l’intervention d’un polypeptide court de composition variable, comme par exemple un penta-glycine chez Staphylococcus aureus (Vollmer et al., 2008a).
18
Figure 2 : Structure du peptidoglycane chez Escherichia coli. La composition de l’unité monomérique au sein du peptidoglycane mature est représentée en gras. La structure de cette même unité est représentée sur fond jaune. La liaison peptidique entre deux chaînes voisines est représentée en rouge (d’après Mengin-Lecreulx et Lemaitre, 2005).
Le peptidoglycane est une structure essentielle qui est retrouvée chez presque toutes les bactéries. Il est le produit d’une voie de biosynthèse complexe faisant intervenir un grand nombre d’enzymes. Cette voie, très conservée au sein du monde bactérien, se décompose toujours en trois étapes principales qui se déroulent successivement au niveau du cytoplasme, de la membrane interne et enfin du périplasme (Barreteau et al., 2008; Bouhss et al., 2008; Sauvage et al., 2008).
19
2. Biosynthèse du peptidoglycane :
Figure 3 : Schéma de la voie de biosynthèse du peptidoglycane chez E. coli. La biosynthèse du peptidoglycane se déroule au niveau de trois compartiments cellulaires. Les premières étapes de synthèse se déroulent au niveau du cytoplasme et aboutissent à la formation des deux principaux précurseurs cytoplasmiques : GlcNAc (hexagone orange) et l’UDP-MurNAc (hexagone bleu) porteur de sa chaîne pentapeptidique. La biosynthèse se poursuit ensuite au niveau de la membrane interne. Le motif phospho-MurNAc-pentapeptide est tout d’abord ajouté sur l’undécaprényl phosphate pour former le lipide I. Le motif GlcNAc est par la suite ajouté sur le lipide I pour former le lipide II. Ce dernier est ensuite transloqué du côté périplasmique par une flippase. Il est alors pris en charge par les « penicillin-binding proteins » (PBPs) qui sont responsables de l’ajout de l’unité disaccharide-pentapeptide au sein du peptidoglycane mature. Le point d’interrogation désigne des enzymes encore non identifiées.
Les étapes cytoplasmiques :
La synthèse du peptidoglycane débute toujours au niveau du cytoplasme par la formation de deux principaux précurseurs nucléotidiques : l’N-acétylglucosamine (GlcNAc) et l’acide UDP-N-acétylmuramoyl-pentapeptide (UDP-MurNAc-pentapeptide). La formation de ces deux précurseurs peut se diviser en quatre étapes : (i). formation de l’UDP-GlcNAc à partir du fructose-6-phosphate, (ii). formation de l’UDP-MurNAc à partir de l’UDP-GlcNAc, (iii). assemblage de la sous-unité peptidique pour former l’UDP-MurNAc-pentapeptide, (iv). synthèses annexes permettant la formation de l’acide
20
Synthèse de l’UDP-N-acétylglucosamine :
L’UDP-GlcNAc est synthétisé à partir du fructose-6-phosphate en quatre étapes faisant intervenir trois enzymes différentes (Figure 4) : GlmS, GlmM et GlmU.
Figure 4 : Formation de l’UDP-N-acétylglucosamine.
La première étape est responsable de la transformation du fructose-6-phosphate en glucosamine-6-phosphate sous l’action de la glucosamine-6-phosphate synthase (GlmS). La L
-glutamine est utilisée comme source d’azote par GlmS (Badet et al., 1987). Cette réaction suit un mécanisme réactionnel de type « bi-bi » ordonné au cours duquel la fixation du fructose-6-phosphate est toujours suivie par celle de la L-glutamine (Badet et al., 1988; Mouilleron et al., 2011). Il s’agit d’une
enzyme dimérique où chaque monomère est composé de deux domaines indépendants à la fois au niveau structural et au niveau fonctionnel (Teplyakov et al., 2001 ; Mouilleron et al., 2011) (Figure 5A). En effet, le domaine N-terminal de GlmS est responsable de la fixation et de l’hydrolyse de la glutamine en glutamate et en ammoniaque. Le domaine C-terminal est quant à lui responsable de la transformation du fructose-6-phosphate en glucosamine-6-phosphate. C’est l’ammoniac produit au niveau du domaine N-terminal qui est utilisé comme source d’azote pour l’amination du sucre. Il est transporté du domaine N-terminal au domaine C-terminal par un système de « channelling » faisant intervenir des résidus hydrophobes. D’autre part, la dimérisation de la protéine GlmS est essentielle pour l’orientation des domaines N-terminal et C-terminal l’un par rapport à l’autre, ce qui permet la fermeture du domaine de liaison du fructose-6-phosphate ainsi que la formation du système de « channelling » nécessaire à l’activité (Floquet et al., 2007).
Dans la deuxième étape, la glucosamine-6-phosphate formée par GlmS est transformée en glucosamine-1-phosphate sous l’action de la phosphoglucosamine mutase (GlmM). Cette protéine appartient à la famille des hexose-phosphate mutases. Elle possède en effet au niveau de sa structure
primaire un motif consensus typique de cette famille d’enzymes :
(GA)(LIVM)X(LIVM)(ST)(PGA)S*HXPX4(GN) (Mengin-Lecreulx et van Heijenoort, 1996). Cette séquence caractéristique contient la sérine catalytique (S*) dont la phosphorylation est nécessaire pour l’activité
21 enzymatique. Il a été montré qu’il s’agissait de la sérine 102 pour GlmM d’E. coli (Jolly et al., 1999). La transformation de la glucosamine-6-phosphate en glucosamine-1-phosphate se fait selon un mécanisme ping-pong nécessitant l’intervention d’un intermédiaire catalytique, la glucosamine-1-6-diphosphate, qui est à la fois le premier produit et le second substrat de la réaction catalysée par cette enzyme (Jolly et al., 1999). Le transfert du groupement phosphate sur la glucosamine-1-phosphate pour former la glucosamine-1-6-diphosphate nécessite que la sérine catalytique soit phosphorylée. GlmM est capable de s’autophosphoryler in vitro en présence d’ATP ou d’un autre nucléotide triphosphate pour pouvoir effectuer cette réaction de transfert (Jolly et al., 2000). Pour réaliser cette réaction, GlmM doit être sous forme dimérique. La structure tridimensionnelle de l’homodimère a par ailleurs été résolue (Figure 5B) (Mehra-Chaudhary et al., 2011a, 2011b).
Figure 5 : Structure cristallographique des protéines GlmS, GlmM et GlmU. A : Structure tridimensionnelle de GlmS sous forme monomérique en présence de fructose-6-phosphate (magenta). Les domaines glutaminase et isomérase sont représentés respectivement en vert et bleu. Les points rouges représentent le trajet emprunté par l’ammoniac (code PDB : 1JXA) (D’après Barreteau et al., 2008). B : Structure tridimensionnelle de GlmM sous forme dimérique (code PDB : 3PDK). C : Structure tridimensionnelle de GlmU sous forme trimérique en présence de CoA et d’UDP-GlcNAc (en jaune) ainsi qu’en présence d’ions cobalt (en rose) (code PDB : 1HV9) (Olsen et Roderick, 2001).
La dernière étape aboutissant à la formation de l’UDP-GlcNAc à partir de la glucosamine-1-phosphate est catalysée par la N-acétylglucosamine-1-glucosamine-1-phosphate uridyltransférase ou GlmU (Mengin-Lecreulx et van Heijenoort, 1993). GlmU est une enzyme trimérique et bifonctionnelle responsable de l’acétylation et de l’uridylation de la glucosamine-1-phosphate (Mengin-Lecreulx et van Heijenoort, 1994). Ainsi, GlmU catalyse au niveau de son domaine C-terminal le transfert du groupement acétyl provenant de l’acétyl-CoA sur la glucosamine-1-phosphate (Glc1-P) pour former la N-acétylglucosamine-1-P (GlcNAc-1-P). Enfin, l’uridylation de la GlcNAc-1-P, permettant la formation de l’UDP-GlcNAc à partir d’UTP, se fait au niveau du domaine N-terminal. Ces deux réactions se font de manière ordonnée, l’acétylation précédant l’uridylation, même si une certaine capacité du domaine N-terminal à pouvoir transférer directement l’UMP sur la GlcN-1-P a été démontrée in vitro et dans certains contextes particuliers in vivo (Pompeo et al., 2001). Il a par ailleurs été établi que le transfert du GlcNAc-1-P du domaine C-terminal vers le domaine N-terminal ne faisait pas intervenir de système de « channelling ». Ce produit intermédiaire est en effet relargué dans le milieu puis capté par le domaine N-terminal (Gehring et al., 1996). La résolution de la structure tridimensionnelle de GlmU en présence de CoA et de GlcN-1-P a permis de mettre en évidence la nécessité de la trimérisation pour
22 la réaction d’acétylation. En effet, le site catalytique responsable de l’addition du groupement acétyl sur la GlcN-1-P se situe à l’interface de deux monomères. De plus, l’extrémité C-terminale du dernier monomère est impliquée dans la liaison de la partie 3’-5’ ADP du CoA. La protéine GlmU sous sa forme trimérique contient donc trois sites d’acétylation indépendants (Olsen et Roderick, 2001; Olsen et al., 2007) (Figure 5C). En revanche, la trimérisation n’est pas essentielle pour la réaction d’uridylation (Pompeo et al., 2001).
L’UDP-GlcNAc est donc produit à partir du fructose-6-phosphate suite aux actions successives des enzymes GlmS, GlmM et GlmU. Il est ensuite utilisé comme substrat par deux enzymes impliquées dans la biosynthèse du peptidoglycane : MurA, au niveau cytoplasmique, pour permettre in fine la formation d’UDP-MurNAc, et MurG, au niveau de la membrane cytoplasmique, pour former le lipide II, dernier précurseur de la voie de biosynthèse du peptidoglycane (Barreteau et al., 2008; Bouhss et al., 2008). Il est important de noter que l’UDP-GlcNAc intervient également dans la synthèse d’autres macromolécules bactériennes telles que les lipopolysaccharides (LPS), les acides teichoïques ou l’antigène commun aux entérobactéries (ECA). Il intervient également dans la synthèse du poly-β-(1-6)-N-acétyl-D-glucosamine (PNAG), polymère impliqué dans la formation de biofilms chez E. coli (Konopka, 2012).
Synthèse de l’acide UDP-N-acétylmuramique :
La formation de l’acide UDP-N-acétylmuramique (UDP-MurNAc) à partir de l’UDP-GlcNAc se fait en deux étapes et nécessite l’intervention de deux enzymes distinctes : MurA et MurB (Figure 6).
Figure 6 : Synthèse de l’acide UDP-N-acétylmuramique.
Lors de la première étape, catalysée par MurA, un groupement énolpyruvate provenant du phosphoénolpyruvate (PEP) est transféré sur le groupement hydroxyle en position 3 de l’UDP-GlcNAc. Cela aboutit à la formation de l’UDP-GlcNAc-énolpyruvate (Brown et al., 1994). Cette réaction enzymatique est la première directement impliquée dans la voie de biosynthèse du peptidoglycane. MurA a fait l’objet de nombreuses études biochimiques et structurales qui ont permis de proposer un mécanisme catalytique d’addition-élimination faisant intervenir un intermédiaire tétraédrique (Skarzynski et al., 1998). La résolution de structures tridimensionnelles de MurA seule ou en présence de différents ligands (UDP-GlcNAc, PEP ou analogues de PEP) a permis de mettre en évidence les différents changements conformationnels que subit la protéine au cours de son cycle réactionnel. En effet, la liaison des substrats permet le passage de l’enzyme d’une forme ouverte à une forme fermée,
23 favorable au transfert du groupement énolpyruvate (Schönbrunn et al., 1998). Le type de réaction catalysée par MurA est un processus biochimique rare qui est uniquement retrouvée pour la protéine AroA intervenant dans le métabolisme de l’acide shikimique (Byczynski et al., 2003).
La seconde étape permettant la formation de l’UDP-MurNAc est catalysée par l’enzyme MurB qui est responsable de la réduction du groupement énolpyruvate de l’UDP-GlcNAc-énolpyruvate. MurB est une flavoprotéine qui adopte un mécanisme catalytique de type ping-pong bi-bi mettant en jeu deux demi-réactions successives. Elle catalyse dans un premier temps la réduction du FAD en FADH2 à
l’aide des ions hydrures du NADPH. Le FADH2 ainsi formé est ensuite utilisé lors de la deuxième
demi-réaction pour réduire l’UDP-GlcNAc-énolpyruvate afin de former l’UDP-MurNAc (Benson et al., 1993). Cette dernière étape de réduction conduit à la formation d’un groupement lactoyl sur lequel viendra se fixer le pentapeptide. Les études structurales de protéines MurB provenant de différentes espèces ont permis de mettre en évidence une structure commune en trois domaines. Les domaines 1 et 2 sont responsables de la fixation du FAD alors que le domaine 3 permet quant à lui la fixation du NADPH et de l’UDP-GlcNAc-énolpyruvate (Benson et al., 1996 ; Kim et al., 2007).
Les protéines MurA et MurB permettent donc, par leurs actions successives, de produire à partir de l’UDP-GlcNAc, l’UDP-MurNAc sur lequel va être fixé le pentapeptide qui permettra la réticulation du peptidoglycane.
Synthèse de l’UDP-MurNAc-pentapeptide :
L’UDP-MurNAc-pentapeptide est synthétisé à partir de l’UDP-MurNAc suite aux actions consécutives de quatre enzymes hautement spécifiques appelées Mur ligases ou Mur synthétases : MurC, MurD, MurE et MurF (Figure 3) (van Heijenoort, 2001; Barreteau et al., 2008). Ces quatre enzymes permettent les ajouts successifs de la L-Ala, du D-Glu, d’un acide diaminé tel que le
meso-A2pm - essentiellement retrouvé chez les bactéries à Gram négatif - ou la L-Lys - essentiellement
retrouvée chez les bactéries à Gram positif - et enfin d’un dipeptide dont la séquence est en général
D-Ala-D-Ala. Ce pentapeptide est greffé au niveau du groupement D-lactoyl de l’UDP-MurNAc. Chez E.
coli, les gènes codant pour ces quatre protéines sont situés au sein d’un même opéron : l’opéron mra (« murein region A ») qui contient à la fois des gènes responsables de la biosynthèse du peptidoglycane, mais aussi des gènes impliqués dans la division cellulaire (Mengin-Lecreulx et al., 1998). Ces enzymes ont été très étudiées chez E. coli et leurs gènes (murC, murD, murE et murF) ont été clonés, séquencés et caractérisés comme étant uniques et essentiels à la survie cellulaire. Il existe d’autre part chez E. coli une autre Mur ligase, Mpl, enzyme non essentielle qui catalyse l’addition en bloc du tripeptide L-Ala-γ-D-Glu-meso-A2pm sur l’UDP-MurNAc lors du processus de recyclage du
peptidoglycane (Mengin-Lecreulx et al., 1996).
Les Mur ligases sont des enzymes à trois substrats : (i). l’ATP, (ii). l’MurNAc ou un UDP-MurNAc-acide aminé ou peptide, (iii). un acide aminé ou un dipeptide. Ces enzymes réalisent toutes le même type de réaction aboutissant à la formation d’une liaison amide ou peptidique avec hydrolyse concomitante de l’ATP en ADP et Pi. La formation de cette liaison passe en premier lieu par l’activation
du groupement carboxyle présent au niveau du substrat nucléotidique par l’ATP. L’acyl-phosphate formé subit alors une attaque nucléophile de la fonction amine de l’acide aminé ou du dipeptide. Cela conduit à la formation d’un intermédiaire tétraédrique hautement énergétique. Il y alors élimination du groupement phosphate et formation de la liaison peptidique (Figure 7) (Bouhss et al., 2002).
24
Figure 7 : Réaction catalysée par les Mur ligases (d’après Barreteau et al., 2008).
L’alignement des séquences de différentes Mur ligases a permis d’identifier 6 résidus invariants ainsi qu’une séquence consensus de fixation de l’ATP (Bouhss et al., 1997; Eveland et al., 1997). Ces éléments ont permis de définir la superfamille des Mur ligases. D’autres enzymes non impliquées dans la biosynthèse du peptidoglycane, mais catalysant le même type de réaction, ont été rattachées à la famille des Mur ligases du fait de la présence de ces mêmes acides aminés invariants. Il s’agit de la protéine FolC, impliquée dans le métabolisme du folate (Sheng et al., 2000), de la protéine CapB impliquée dans la synthèse de la capsule de Bacillus anthracis (Candela et Fouet, 2006) et de la région C-terminale de la cyanophicine synthétase (Ziegler et al., 1998).
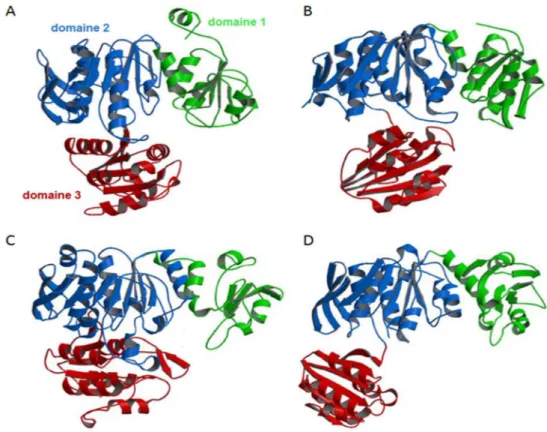
Plusieurs structures tridimensionnelles de Mur ligases ont été obtenues en présence ou absence de différents ligands. L’analyse de ces différentes structures a permis de mettre en évidence la présence d’une architecture commune en trois domaines (Figure 8) (Smith, 2006). Le domaine N-terminal est responsable de la fixation du substrat nucléotidique. Sa structure, de type « Rossman-fold », varie légèrement entre chaque Mur ligase afin de pouvoir accommoder un substrat dont la partie peptidique est de plus en plus longue. Le domaine central qui permet de lier l’ATP possède quant à lui une structure caractéristique des protéines capables de fixer des mononucléotides telles que les ATPases ou les GTPases. Le domaine C-terminal, qui possède comme le domaine N-terminal un repliement de type « Rossman-fold », est quant à lui responsable de la fixation de l’acide aminé ou du dipeptide. Les six résidus invariants caractéristiques de cette famille d’enzymes sont situés à l’interface des trois domaines. L’analyse des différentes structures en présence et en absence de substrats a permis de montrer que ces enzymes existaient à la fois sous une forme ouverte et une forme fermée. Ce passage de la forme ouverte à la forme fermée semble être provoqué par la liaison des différents substrats (Smith, 2006; Šink et al., 2016).
25
Figure 8 : Structures tridimensionnelles des Mur ligases. Les structures de MurC (A), MurD (B), MurE (C) et MurF (D) sont représentées en « cartoon ». Les différents domaines sont indiqués pour MurC et le code couleur utilisé est identique pour l’ensemble des Mur ligases. Le domaine N-terminal (domaine 1) est coloré en vert, le domaine central (domaine 2) en bleu et le domaine C-terminal (domaine 3) en rouge. Les enzymes MurC, MurD et MurE sont dans une conformation fermée alors que MurF est dans une conformation ouverte (D’après Smith, 2006).
Voies annexes :
On retrouve dans la composition du peptidoglycane des acides aminés de la série D. Ce type
d’acide aminé n’est pas fréquemment rencontré et ils sont uniquement synthétisés pour intégrer la voie de biosynthèse du peptidoglycane.
2.1.4.1. Formation de la D-alanine :
La D-alanine est produite, chez E. coli et d’autres espèces bactériennes, à partir de la L-Ala sous l’action d’une alanine racémase. En fonction des espèces, on retrouve une ou deux alanine racémases. Chez E. coli, deux ont été identifiées, codées respectivement par les gènes alr et dadX. L’expression de la protéine Alr est constitutive alors que celle de DadX est induite uniquement lorsque la L-Ala est
utilisée comme source de carbone et d’énergie (Barreteau et al., 2008).
2.1.4.2. Formation du dipeptide D-Ala-D-Ala :
Le dipeptide D-Ala-D-Ala résulte de la condensation de deux molécules de D-Ala. Cette réaction est catalysée par une ligase et nécessite la consommation d’une molécule d’ATP. Selon les espèces bactériennes, un ou deux gènes codent pour cette fonction (ddlA et ddlB chez E. coli). Le mécanisme réactionnel met en jeu la formation d’un acyl-phosphate ainsi que la formation d’un intermédiaire tétraédrique (Mullins et al., 1990; Healy et al., 2000). Bien que ce mécanisme réactionnel soit proche de celui des Mur ligases, les D-Ala:D-Ala ligases (Ddl) ont été identifiées comme appartenant à la famille
26
2.1.4.3. Formation du D-glutamate :
Le D-glutamate rentre dans la composition du peptidoglycane mais il est également un
composant de la capsule chez certaines bactéries (Foster et Popham, 2002). Sa formation peut être catalysée par une D-Ala:D-Glu transaminase ou par une glutamate racémase MurI (Doublet et al., 1993;
Fotheringham et al., 1998).
Les étapes membranaires :
Lors des étapes membranaires de la biosynthèse du peptidoglycane, le motif phospho-MurNAc-pentapeptide du précurseur nucléotidique est dans un premier temps ajouté au transporteur lipidique undécaprényl phosphate (C55-P) pour former le lipide I. Le motif GlcNAc de l’UDP-GlcNAc est
ensuite fixé sur ce lipide I pour former le lipide II.
Métabolisme de l’undécaprényl phosphate :
L’undécaprényl phosphate (C55-P), également appelé bactoprénol, est un lipide essentiel
impliqué dans la biosynthèse du peptidoglycane mais également dans celle d’autres constituants de la paroi bactérienne comme les LPS, les acides teichoïques ou bien l’antigène commun aux entérobactéries (ECA) (Bouhss et al., 2008; Manat et al., 2014; Barreteau et al., 2017). Le C55-P est
généré à partir de l’undécaprenyl pyrophosphate (C55-PP) suite à une étape de déphosphorylation. Ce
C55-PP peut être soit néo-synthétisé, soit issu d’une étape de recyclage. Le C55-PP néo-synthétisé sur la
face interne de la membrane plasmique est le produit de l’undécaprényl pyrophosphate synthase (UppS), suite à la condensation d’une molécule de farnésyl pyrophosphate (C15-PP) et de huit
molécules d’isopentényl pyrophosphate (C5-PP) (Figure 3) (Ogura et Koyama, 1998). Le C55-PP recyclé
est, quant à lui, libéré au niveau de la face externe de la membrane plasmique suite aux étapes de polymérisation du peptidoglycane. La déphosphorylation du C55-PP permettant de former du C55-P est
catalysée par des undécaprényl pyrophosphate phosphatases (UppP). Chez E. coli, cette activité essentielle est portée par la protéine BacA, ainsi que par les protéines PgpB, YbjG et LpxT appartenant à la superfamille des PAP2 (Phosphatidic Acid Phosphatases of type 2) (El Ghachi et al., 2004, 2005). Ces quatre phosphatases sont des protéines intégrales de membrane dont le site actif est localisé du côté périplasmique (Tatar et al., 2007; Touzé et al., 2008; Fan et al., 2014; Manat et al., 2015). Cette orientation commune suggère donc que ces protéines sont impliquées uniquement dans la déphosphorylation du C55-PP localisé sur la face externe de la membrane plasmique. En revanche, le
mécanisme de déphosphorylation du C55-PP néo-synthétisé reste flou et les acteurs impliqués dans
cette étape n’ont pour l’heure pas été identifiés. Deux hypothèses concernant la déphosphorylation du C55-PP néo-synthétisé sont avancées : (i). l’implication d’une phosphatase spécifique active du côté
cytoplasmique ; (ii). l’export de ce lipide du côté périplasmique par une flippase où il serait alors déphosphorylé par les phosphatases BacA, PgpB, YbjG ou LpxT, puis réimporté par une autre flippase (Figure 3).
Formation du lipide I :
L’enzyme MraY (phospho-N-acétylmuramoyl-pentapeptide transférase) est responsable du transfert du motif phospho-MurNAc-pentapeptide de l’UDP-MurNAc-pentapeptide sur l’undécaprényl phosphate, permettant ainsi la formation du lipide I (C55-PP-MurNAc-pentapeptide) avec libération
27 1998). Comme les gènes mur codant pour les Mur ligases, ce gène est situé au sein du cluster mra (Ikeda et al., 1991). MraY est une protéine intégrale de membrane, qui contient dix segments transmembranaires, quatre boucles périplasmiques et cinq boucles cytoplasmiques. Les séquences correspondant à trois des cinq boucles cytoplasmiques sont relativement conservées et permettent de rattacher MraY à la famille des polyisoprényl-phosphate N-acétylhexosamine-1-phosphate transférases (Bouhss et al., 2008). Cette topologie semble conservée pour l’ensemble des eubactéries (Bouhss et al., 1999). La structure tridimensionnelle de MraY issue d’Aquifex aeolicus a récemment été résolue et confirme la topologie membranaire précédemment établie (Figure 9) (Chung et al., 2013). Le pool de lipide I a été estimé chez E. coli à environ 700 molécules par cellule (van Heijenoort et al., 1992). Cette faible quantité s’explique par le fait que le lipide I est un intermédiaire transitoire dont la synthèse est efficacement couplée avec les réactions situées en aval dans la voie de biosynthèse du peptidoglycane, en particulier avec la synthèse de lipide II. En comparaison, le nombre de molécules de C55-P a été estimé à environ 40 000 par cellule chez E. coli (Barreteau et al., 2009a).
Figure 9 : Topologie membranaire et structure cristallographique de MraY d’Aquifex aeolicus. A : Topologie membranaire de MraYAa. Les chiffres représentent les numéros des hélices transmembranaires et les lettres les boucles cytoplasmiques. B : Structure cristallographique de MraYAa. Les mêmes couleurs sont utilisées pour identifier les hélices entre la topologie membranaire et la structure. (code PDB : 4J72) (d’après Chung et al., 2013).
Formation du lipide II :
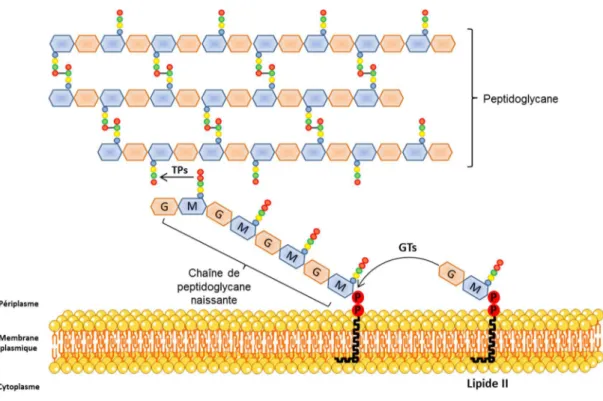
Le lipide II (C55-PP-MurNAc(-pentapeptide)-GlcNAc) constitue le dernier précurseur dans la
voie de biosynthèse du peptidoglycane. Il est le résultat de la condensation entre le motif GlcNAc, issu de l’UDP-GlcNAc, avec une molécule de lipide I. Cette condensation a lieu au niveau du groupement hydroxyle en position 4 du MurNAc et du groupement hydroxyle en position 1 de la glucosamine, aboutissant ainsi à la formation d’une liaison β-(1→4) entre ces résidus (Figure 10).
28
Figure 10 : Structure du lipide II d’une bactérie à Gram négatif.
Cette réaction est catalysée par l’enzyme MurG (également appelée translocase II) et permet ainsi la formation du motif disaccharide-pentapeptide, monomère de base du peptidoglycane, porté par le transporteur lipidique (Figure 3). Le gène codant pour la protéine MurG est également localisé au sein du cluster mra. Il est unique et essentiel pour la viabilité cellulaire chez E. coli, tout comme le gène codant pour MraY (Salmond et al., 1980; Mengin-Lecreulx et al., 1991). MurG est décrite comme une enzyme soluble associée à la face interne de la membrane plasmique. Le fait que MurG soit située du côté cytoplasmique a permis de conclure que le motif disaccharide-pentapeptide était assemblé en amont de la translocation à travers la membrane plasmique (Bupp et van Heijenoort, 1993). La protéine MurG issue d’E. coli a été la première à être purifiée (Crouvoisier et al., 1999). Elle est composée de deux domaines séparés par une crevasse au sein de laquelle est situé le site actif (Ha et al., 2000; Hu et al., 2003). Ce dernier est constitué de 13 résidus invariants importants à la fois pour la liaison des différents substrats et pour la catalyse (Crouvoisier et al., 2007).
Le lipide II est synthétisé par MurG sur la face cytoplasmique de la membrane plasmique. Cependant, la polymérisation du peptidoglycane a lieu au sein du périplasme. Le motif disaccharide-pentapeptide a donc besoin d’être transporté à travers la membrane cytoplasmique pour être pris en charge par les PBPs.
Translocation du lipide II à travers la membrane plasmique :
La translocation du lipide II à travers la membrane plasmique est une étape essentielle dans la voie de biosynthèse du peptidoglycane. A l’heure actuelle, l’identité du ou des différents acteurs impliqués dans ce mécanisme n’a pas été clairement établie.
Il a d’abord été proposé que le lipide II puisse diffuser de manière spontanée à travers la membrane pour atteindre le périplasme (Bugg et Brandish, 1994). Cependant, dans une étude réalisée en 2007, van Dam et ses collaborateurs ont réfuté cette hypothèse en montrant à l’aide d’un lipide II fluorescent qu’une translocation non assistée à travers la membrane n’était pas assez rapide pour subvenir aux besoins de la synthèse du peptidoglycane dans le périplasme. Ils ont également montré que la translocation du lipide II à travers la membrane plasmique faisait intervenir une machinerie
29 protéique spécialisée ne nécessitant aucune source d’énergie (ATP ou force proton-motrice) (van Dam et al., 2007). C’est dans ce contexte que plusieurs protéines intégrales de membranes ont été proposées comme candidats potentiels au transport du lipide II.
La protéine FtsW ainsi que ses homologues (RodA retrouvée chez E. coli et SpoVE chez B. subtilis) appartiennent à la famille SEDS (Shape, Elongation, Division and Sporulation) et sont depuis longtemps considérés comme des acteurs de ce processus métabolique pour diverses raisons : (i). ces enzymes sont essentielles dans la voie de biosynthèse du peptidoglycane ; (ii). elles sont impliquées dans des complexes multiprotéiques intervenant dans les mécanismes d’élongation et de division (Boyle et al., 1997; Matsuhashi et al., 1990) ; (iii). ce sont toutes des protéines intégrales de membrane. Ainsi, FtsW et RodA ont été les premières protéines à avoir été évoquées comme étant potentiellement impliquées dans le transport du lipide II (Höltje, 1998). Par ailleurs, il a été récemment montré que la protéine FtsW d’E. coli possédait in vitro une activité flippase pour le lipide II au sein de protéoliposomes reconstitués, mais également au sein de vésicules membranaires issues d’une souche d’E. coli surexprimant cette même protéine (Mohammadi et al., 2011). Cependant, un autre candidat a également été proposé pour accomplir cette fonction. Il s’agit de la protéine MurJ (ou MviN), qui est une protéine intégrale de membrane appartenant à la superfamille des protéines exportatrices MOP (Multidrug/Oligosaccharidyl-lipid/Polysaccharide) et qui est essentielle à la synthèse du peptidoglycane chez E. coli (Inoue et al., 2008; Ruiz, 2008). On retrouve également dans cette superfamille des MOP la protéine Wzx qui est responsable du transport à travers la membrane de différents intermédiaires portés par le C55-PP intervenant dans les voies de biosynthèse de l’antigène
O, des LPS ou bien de l’ECA. Cela a donc renforcé l’idée que MurJ puisse être la flippase du lipide II, étant donné que le motif disaccharide-peptide est fixé sur ce même transporteur lipidique. Une étude récente, mettant en jeu un test d’accessibilité du lipide II à la colicine M au sein d’une souche d’E. coli ΔmurJ, a permis de montrer in vivo l’implication de la protéine MurJ dans la translocation du lipide II à travers la membrane plasmique. Ce test a permis de mettre en évidence une absence de dégradation du lipide II par la colicine M suite à l’inactivation de la protéine MurJ, mettant ainsi en avant son implication dans la translocation de ce lipide (Sham et al., 2014). Cependant, l’orthologue de MurJ chez B. subtilis n’est pas essentiel à la croissance, suggérant ainsi que cette protéine ne joue pas le rôle de flippase du lipide II chez cette espèce (Fay et Dworkin, 2009). Il a par contre été montré que l’inactivation simultanée des gènes codant pour les protéines MurJ et AmJ chez B. subtilis est létale. Par ailleurs, l’expression de la protéine AmJ permet de restaurer la croissance au sein d’une souche d’E. coli ΔmurJ. Ces deux informations suggèrent que la protéine AmJ posséde également une activité flippase pour le lipide II et que les protéines MurJ et AmJ ont donc une fonction redondante (Meeske et al., 2015).
La structure cristallographique de MurJ a récemment été résolue. Elle est constituée de 14 hélices transmembranaires (TMs) qui sont séparées en deux domaines principalement. Les TMs 1 à 6 constituent le domaine N-terminal et les TMs 7 à 12 constituent le domaine C-terminal. Ces deux lobes sont reliés par un axe de pseudo-symétrie d’ordre 2. Les TMs 13 et 14 sont situées à la périphérie du domaine C-terminal. Ces deux hélices permettent la formation d’un sillon hydrophobe qui donne sur une cavité chargée positivement à l’une de ses extrémités, située à l’interface des domaines N-terminal et C-terminal. Une expérience de docking avec du lipide II permet de supposer raisonnablement que le sillon hydrophobe accommoderait la chaîne hydrophobe et que la cavité chargée positivement lierait quant à elle le groupement pyrophosphate ainsi que les sucres. Le pentapeptide se logerait à l’extrémité opposée de la cavité. MurJ existe sous deux états, une forme avec la cavité donnant sur le
30 cytoplasme et une forme où la cavité centrale donne sur le périplasme. Ces deux états sont en accord avec le transport du lipide II à travers la membrane (Figure 11) (Kuk et al., 2017).
Figure 11 : Structure cristallographique de MurJ de Thermosipho africanus et mécanisme putatif de la translocation du lipide II. A : Structure cristallographique de MurJTa. Le lobe N-terminal constitué des TMs 1 à 6 est représenté en bleu. Le lobe C-terminal constitué des TMs 7 à 12 est représenté en vert. Les TMs 13 et 14 sont représentés en marron. B : mécanisme putatif de translocation du lipide II (d’après Kuk et al., 2017).
Il est important de noter qu’à l’heure actuelle, tous ces résultats ne permettent pas de conclure de façon certaine quant à l’identité de la flippase responsable de la translocation du lipide II. En effet, aucune activité in vitro de transport du lipide II n’a pu être mise en évidence pour la protéine MurJ (Mohammadi et al., 2011) et aucune activité in vivo n’a pu être mise en évidence pour la protéine FtsW (Sham et al., 2014). Il a récemment été montré que la protéine RodA, orthologue de FtsW, possédait une activité glycosyltransférase, suggérant ainsi un rôle similaire pour FtsW (Meeske et al., 2016). D’autre part, la protéine AmJ n’est pas présente chez E. coli. L’identité de la protéine responsable du transport du lipide II risque donc de faire débat encore quelque temps.
Polymérisation du peptidoglycane :
Une fois transloqué du côté périplasmique, le lipide II sert de substrat pour les étapes finales de biosynthèse du peptidoglycane. Ces étapes de polymérisation et de réticulation sont assurées par les « penicillin-binding proteins » (PBPs) qui sont ainsi nommées ainsi car elles sont la cible de cette famille d’antibiotiques. Les PBPs catalysent les réactions de transglycosylation et de transpeptidation nécessaires à l’incorporation du motif disaccharide-peptide au sein du peptidoglycane préexistant (Scheffers et Pinho, 2005). Les glycosyltransférases (GTs) catalysent dans un premier temps la synthèse des nouvelles chaînes de glycanes. Cette réaction est suivie par la transpeptidation s’effectuant entre deux motifs peptidiques situés sur deux chaînes de glycanes adjacentes (une existante et une néo-synthétisée) (Figure 12) (Sauvage et al., 2008).