HAL Id: hal-02797974
https://hal.inrae.fr/hal-02797974
Submitted on 5 Jun 2020
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Signatures génomiques de l’histoire évolutive des races
caprines françaises
Claire Oget
To cite this version:
Claire Oget. Signatures génomiques de l’histoire évolutive des races caprines françaises. Sciences du Vivant [q-bio]. 2016. �hal-02797974�
École Nationale Supérieure Agronomique de Toulouse (ENSAT) Institut National de la Recherche Agronomique (INRA)
SIGNATURES GÉNOMIQUES DE L’HISTOIRE
ÉVOLUTIVE DES RACES CAPRINES FRANÇAISES
Claire OGET
Rapport de stage de fin d’études présenté en vue de l’obtention du diplôme d’ingénieur agronome, spécialisation Systèmes et Produits de l’Elevage (SysPEl)
Tuteurs INRA : Isabelle PALHIÈRE et Bertrand SERVIN Tutrices ENSAT : Zulma VITEZICA et Cécile BONNEFONT
Castanet-Tolosan (31) Du 29 février au 26 août 2016 Ch èv re d es F o ss és ( S o u rc e : Ca p g en es)
TABLE DES MATIÈRES
TABLE DES MATIÈRES... ii
LISTE DES ANNEXES ... iv
TABLE DES ILLUSTRATIONS ... v
REMERCIEMENTS ... 1
RÉSUMÉ ... 2
ABSTRACT ... 3
INTRODUCTION ... 4
1 Histoire de la domestication de la chèvre ... 5
2 Les races caprines françaises ... 6
3 Etudes de diversité génétique déjà réalisées sur les races caprines françaises ... 11
4 Objectifs du stage ... 12
ANALYSE DE LA DIVERSITÉ GÉNÉTIQUE CAPRINE ... 14
1 Matériel et méthodes ... 14
1.1 Données génétiques ... 14
1.1.1 Le projet ADAPTmap ... 14
1.1.2 Particularités de la puce caprine 50K ... 14
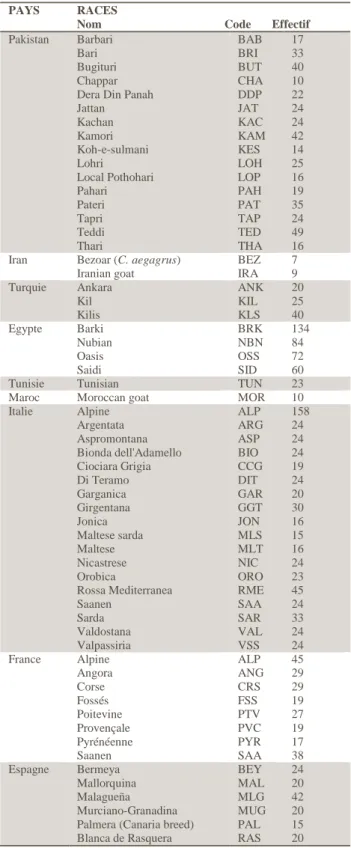
1.1.3 Les différents jeux de données étudiés ... 15
1.2 Méthodes utilisées ... 17
1.2.1 Variabilité intra-population : coefficients de consanguinité et de parenté ... 17
1.2.2 Analyse en Composantes Principales ... 17
1.2.3 Admixture : recherche de la structure des populations ... 18
1.2.4 Les arbres phylogénétiques ... 19
2 Résultats et discussion des analyses effectuées sur les races mondiales ... 20
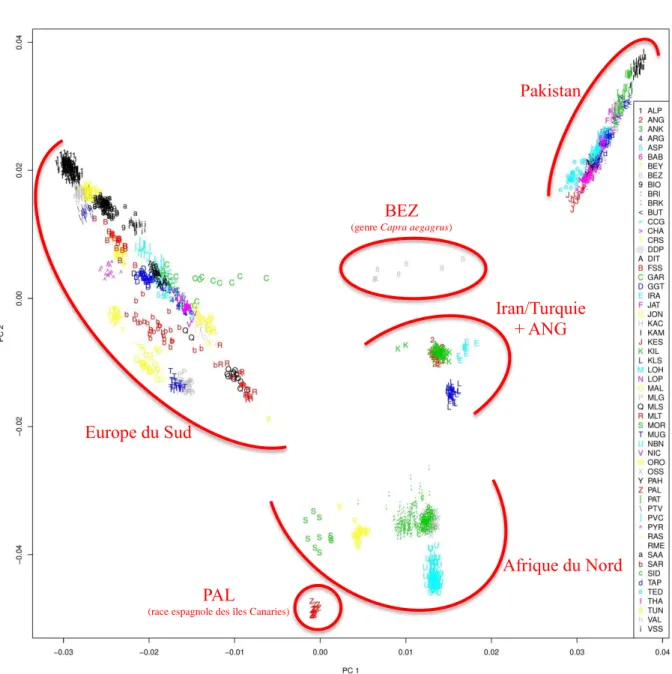
2.1 Migration via la mer Méditerranée ... 20
2.1.1 ACP ... 20
2.1.2 Structure de la population ... 20
2.1.3 Arbres phylogénétiques ... 21
2.1.4 Schéma d’interprétation... 22
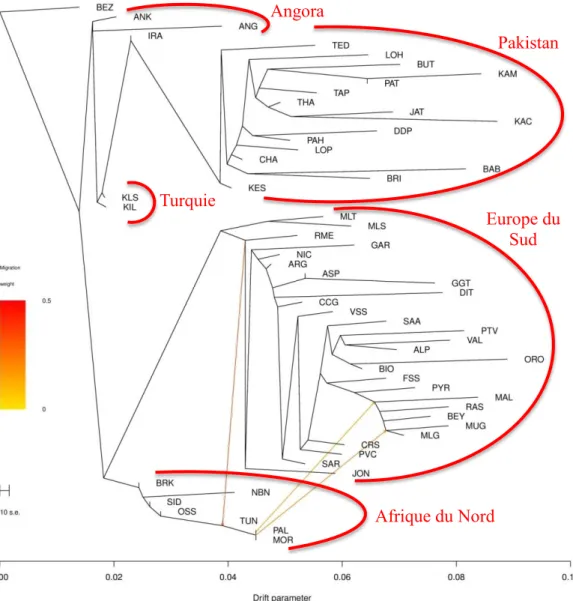
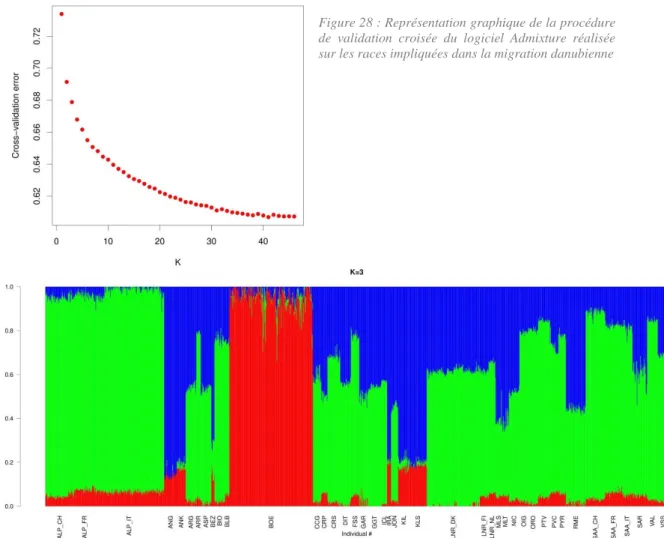
2.2 Migration via le Danube ... 23
2.2.1 ACP ... 23
2.2.2 Structure de la population ... 23
2.2.3 Arbres phylogénétiques ... 24
3 Résultats et discussion des analyses effectuées sur les races françaises ... 25
3.1 Variabilité intra-population ... 25
3.2 ACP ... 26
3.3 Structure de la population ... 27
3.4 Arbres phylogénétiques ... 28
3.5 Etude du cas particulier de la race Provençale ... 28
DÉTECTION DE TRACES DE SÉLECTION ... 31
1 Matériel et méthodes ... 31
1.1 Notion de balayage sélectif ... 31
1.2 Tests FLK et hapFLK ... 32
1.3 Recherche de gènes candidats et de races impliquées ... 33
1.4 Données génétiques étudiées ... 33
2 Résultats et discussion ... 34
2.1 Approche globale : régions significatives ... 34
2.2 Approche détaillée : recherche de gènes candidats ... 34
2.2.1 Le chromosome 5 et le gène ADAMTS20 ... 34
2.2.2 Le chromosome 6 et la caséine αS1 ... 36
2.2.3 Le chromosome 11 ... 38
2.2.4 Le chromosome 13 et ses gènes de couleur ... 39
DISCUSSION ET PERSPECTIVES ... 42
CONCLUSION ... 46
RÉFÉRENCES BIBLIOGRAPHIQUES ... 47
LISTE DES ANNEXES
Annexe 1 : Arbre phylogénétique (a) avec ses résidus (b) obtenus grâce au logiciel TreeMix sur les races impliquées dans la migration méditerranéenne ... 51 Annexe 2 : Arbre phylogénétique (a) avec ses résidus (b) obtenus grâce au logiciel TreeMix sur les races impliquées dans la migration danubienne ... 52 Annexe 3 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Alpine, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 53 Annexe 4 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Saanen, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 53 Annexe 5 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Provençale, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 54 Annexe 6 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Angora, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 54 Annexe 7 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Poitevine, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 55 Annexe 8 : Représentation graphique des relations entre coefficients de parenté (a) et de consanguinité (b) pedigree et génomiques pour les individus de la race Fossés, avant tri sur les apparentements. La droite rouge correspond à la régression linéaire, et la noire à la droite y = x. Fhat2 est un des estimateurs de la consanguinité calculé par le logiciel PLINK. ... 55 Annexe 9 : Arbre phylogénétique (a) avec ses résidus (b) obtenus grâce au logiciel TreeMix sur les races françaises ... 56 Annexe 10 : Liste des gènes candidats présents au sein des régions significatives (Source : d'après NCBI 2016) ... 57 Annexe 11 : Nuage de points des p-valeurs des tests hapFLK du chromosome 5. Le trait horizontal noir correspond au seuil de significativité de 10-3 choisi. ... 58 Annexe 12 : Fréquences haplotypiques (nombre d’haplotype fixé à 15)) estimées par le logiciel hapFLK par race dans la région 30-40 Mb du chromosome 5. Les droites noires verticales délimitent la région significative ... 58 Annexe 13 : Nuage de points des p-valeurs des tests hapFLK du chromosome 6. Le trait horizontal noir correspond au seuil de significativité de 10-3 choisi. ... 59 Annexe 14 : Nuage de points des p-valeurs des tests hapFLK du chromosome 11. Le trait horizontal noir correspond au seuil de significativité de 10-3 choisi. ... 59 Annexe 15 : Nuage de points des p-valeurs des tests hapFLK du chromosome 13. Le trait horizontal noir correspond au seuil de significativité de 10-3 choisi. ... 59
TABLE DES ILLUSTRATIONS
Figure 1 : Photographie d'une chèvre Alpine (Source : Capgenes)...7
Figure 2 : Photographie d'une chèvre Saanen (Source : Capgenes)...7
Figure 3 : Photographie d'une chèvre du Rove (Source : Capgenes)...7
Figure 4 : Photographie d'une chèvre Angora (Source : Capgenes)...8
Figure 5 : Photographie d'une chèvre des Pyrénées (Source : Capgenes)...8
Figure 6 : Photographie d'une chèvre Poitevine (Source : Capgenes)...8
Figure 7 : Photographie de chèvres Provençales (Source : Capgenes)...9
Figure 8 : Photographie de chèvres des Fossés (Source : Capgenes)...9
Figure 9 : Photographie de chèvres du Massif Central (Source : Capgenes)...9
Figure 10 : Photographie d'une chèvre de Lorraine (Source : Capgenes)...10
Figure 11 : Photographie d'une chèvre Corse (Source : Capgenes)...10
Figure 12 : Photographie de chèvres Créoles (Source : Capgenes)...11
Figure 13 : Photographie d'une chèvre Boer (Source : Capgenes)...11
Figure 14 : Photographie de chèvres Péi (Source : Capgenes)...12
Figure 15 : Arbre phylogénétique établi à partir des distances de Reynolds entre races ou groupes de races (Source : Lenstra et al. 2016). Les couleurs indiquent le regroupement régional des populations ou des races génétiquement proches (bleu pour les races du Nord de l’Europe, vert pour celles du Centre, vert kaki pour celles du Sud-Ouest, orange pour celles du Sud-Est, et rouge pour celles d’Asie du Sud-Ouest). Les populations en noir n’appartiennent à aucun de ces regroupements...13
Figure 16 : Spectre des MAF de 50 000 SNPs tirés aléatoirement parmi les données de séquence publiques de chèvres marocaines issues du projet Nextgen (Source : ftp://ftp.ebi.ac.uk/pub/databases/nextgen/) ...15
Figure 17 : Spectre des MAF des marqueurs de la puce 50K caprine sur les chèvres marocaines séquencées au sein du projet Nextgen (Source : ftp://ftp.ebi.ac.uk/pub/databases/nextgen/) ...15
Figure 18 : Représentation schématique d'un arbre phylogénétique...19
Figure 19 : Éboulis des valeurs propres de l'ACP réalisée sur les races impliquées dans la migration méditerranéenne ...20
Figure 20 : Représentation selon les deux premiers axes de l'ACP réalisée sur les races impliquées dans la migration méditerranéenne...20
Figure 21 : Représentation graphique de la procédure de validation croisée du logiciel Admixture réalisée sur les races impliquées dans la migration méditerranéenne...20
Figure 22 : Représentation graphique pour K=3 de la structuration des populations obtenue avec le logiciel ADMIXTURE sur les races impliquées dans la migration méditerranéenne...21
Figure 23 : Arbre phylogénétique obtenu grâce au logiciel hapFLK sur les races impliquées dans la migration méditerranéenne...21
Figure 24 : Arbre phylogénétique obtenu grâce au logiciel TreeMix sur les races impliquées dans la migration méditerranéenne avec trois évènements de migration...22
Figure 25 : Schéma d'interprétation des arbres phylogénétiques obtenus dans l'étude de la migration via la Méditerranée de la chèvre domestique (Source : Google Maps). Les traits rouges représentent la migration proposée par les arbres obtenus avec hapFLK et TreeMix, et les traits pleins jaunes/oranges, les évènements de migration proposés par TreeMix...22
Figure 26 : Éboulis des valeurs propres de l'ACP réalisée sur les races impliquées dans la migration danubienne ...23
Figure 27 : Représentation selon les axes 1-2 (a) et 1-3 (b) de l'ACP réalisée sur les races impliquées dans la migration danubienne...23
Figure 28 : Représentation graphique de la procédure de validation croisée du logiciel Admixture réalisée sur les races impliquées dans la migration danubienne...23 Figure 29 : Représentation graphique pour K=3 de la structuration des populations obtenue avec le logiciel ADMIXTURE sur les races impliquées dans la migration danubienne...23 Figure 30 : Arbre phylogénétique obtenu grâce au logiciel hapFLK sur les races impliquées dans la migration danubienne...24 Figure 31 : Arbre phylogénétique obtenu grâce au logiciel TreeMix sur les races impliquées dans la migration danubienne avec trois évènements de migration...24 Figure 32 : Éboulis des valeurs propres de l'ACP réalisée sur les races françaises...26 Figure 33 : Représentation selon les axes 1-2 (a), 3-4 (b), 5-6 (c) et 7-8 (d) de l'ACP réalisée sur les races françaises...26 Figure 34 : Représentation graphique de la procédure de validation croisée du logiciel Admixture réalisée sur les races françaises...27 Figure 35 : Représentation graphique pour K=6 (a), K=7 (b) et K= 8 (c) de la structuration des populations obtenue avec le logiciel ADMIXTURE sur les races françaises...27 Figure 36 : Arbre phylogénétique obtenu grâce au logiciel hapFLK sur les races françaises...28 Figure 37 : Arbre phylogénétique (a) avec ses résidus (b) obtenus grâce au logiciel TreeMix sur les races françaises avec deux évènements de migration...28 Figure 38 : Arbre phylogénétique obtenu grâce au logiciel TreeMix sur un jeu de données comprenant les races françaises et la italienne Maltaise issue de Sicile (MLT) et Sardaigne (MLS)...29 Figure 39 : Représentation schématique du devenir des allèles d'un locus au sein d'une population (Vekemans 2015)...31 Figure 40 : Représentation schématique du phénomène de balayage sélectif "complet" (hard sweep) (a) et "doux" (soft sweep) (b)...31 Figure 41 : Exemple d'arbre phylogénétique sur lequel la matrice de parenté est établie (Source : Bonhomme et al. 2010)...32 Figure 42 : Nuage de points des pvaleurs des tests FLK sur le génome entier (les chromosomes sont délimités par le changement de couleur)...34 Figure 43 : Nuage de points des pvaleurs des tests hapFLK sur le génome entier (les chromosomes sont délimités par le changement de couleur). Le trait horizontal noir correspond au seuil de significativité de 10-3choisi...34 Figure 44 : Schéma récapitulatif des conséquences de mutations du gène ADAMTS20 chez la souris à ceinture blanche (Source : d’après Silver et al.)...35 Figure 45 : Fréquences alléliques (a) et haplotypiques (nombre d’haplotypes fixé à 40) (b) respectivement calculées et estimées par le logiciel hapFLK par race dans la région 30-40 Mb du chromosome 5. Les droites noires verticales délimitent la région significative...35 Figure 46 : Arbres global (a) réalisé sur le génome entier, et locaux testés avec une approche SNP par SNP (b) et une approche haplotypique (c), construits avec le logiciel hapFLK sur la région significative du chromosome 5...36 Figure 47 : Photos de chèvres des Fossés (a) et des Pyrénées (b) présentant une robe avec une ceinture blanche au niveau de la région lombaire (Source : www.naturablog.com et www.midipyrenees.fr) ...36 Figure 48 : Arbre phylogénétique des allèles de la caséine alpha s1 caprine (Source : Grosclaude et al. 1994) ...37 Figure 49 : Evolution des fréquences alléliques du gène CSN1S1 chez tous les individus génotypés par Capgènes de race Alpine (a1) avec les effectifs associés (a2) et de race Saanen (b1) avec les effectifs associés (b2) nés entre 1984 et 2015...37 Figure 50 : Fréquences alléliques (a) et haplotypiques (nombre d’haplotypes fixé à 40) (b) respectivement calculées et estimées par le logiciel hapFLK par race dans la région 75-85 Mb du
chromosome 6. Les droites noires verticales délimitent la région significative...38
Figure 51 : Arbres global (a) réalisé sur le génome entier, et locaux testés avec une approche SNP par SNP (b) et une approche haplotypique (c), construits avec le logiciel hapFLK sur la région significative du chromosome 6...38
Figure 52 : Fréquences alléliques (a) et haplotypiques (nombre d’haplotypes fixé à 40) (b) respectivement calculées et estimées par le logiciel hapFLK par race dans la région 40-50 Mb du chromosome 11. Les droites noires verticales délimitent la région significative...39
Figure 53 : Arbres global (a) réalisé sur le génome entier, et locaux testés avec une approche SNP par SNP (b) et une approche haplotypique (c), construits avec le logiciel hapFLK sur la région significative du chromosome 11...39
Figure 54 : Fréquences alléliques (a) et haplotypiques (nombre d’haplotypes fixé à 40) (b) respectivement calculées et estimées par le logiciel hapFLK par race dans la région 45-70 Mb du chromosome 13. Les droites noires verticales délimitent la région significative...40
Figure 55 : Arbres global (a) réalisé sur le génome entier, et locaux testés avec une approche SNP par SNP (b) et une approche haplotypique (c), construits avec le logiciel hapFLK sur le début (49-51 Mb) de la région significative du chromosome 13...41
Figure 56 : Arbres global (a) réalisé sur le génome entier, et locaux testés avec une approche SNP par SNP (b) et une approche haplotypique (c), construits avec le logiciel hapFLK sur la fin (60-65 Mb) de la région significative du chromosome 13...41
Figure 57 : Photographies des races Saanen (a), Fossés (b) et Poitevine (c) (Sources : www.capgenes.com, www.ouest-france.fr et www.chevre-poitevine.org) ...41
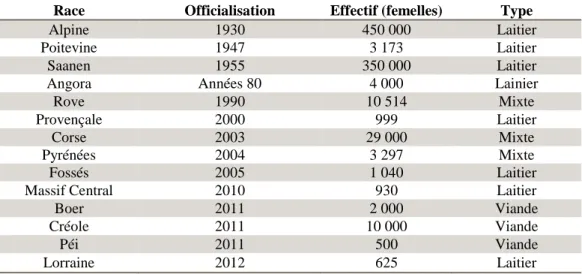
--- Tableau 1 : Les races caprines françaises officielles et leurs effectifs (Sources : Capgenes et Markey et al. 2014)...6
Tableau 2 : Résultats d'une étude de variabilité génétique sur 4 races caprines (Source : d’après Institut de l'Elevage 2011)...11
Tableau 3 : Races impliquées dans la migration de la chèvre domestique via la mer Méditerranée....15
Tableau 4 : Races impliquées dans la migration de la chèvre domestique via le Danube...16
Tableau 5 : Détail des effectifs des races françaises étudiées...16
Tableau 6 : Caractéristiques des races françaises étudiées *valeur calculée sur les individus ayant une ascendance connue **valeur calculée sur les individus ayant une ascendance connue du jeu de données initial...25
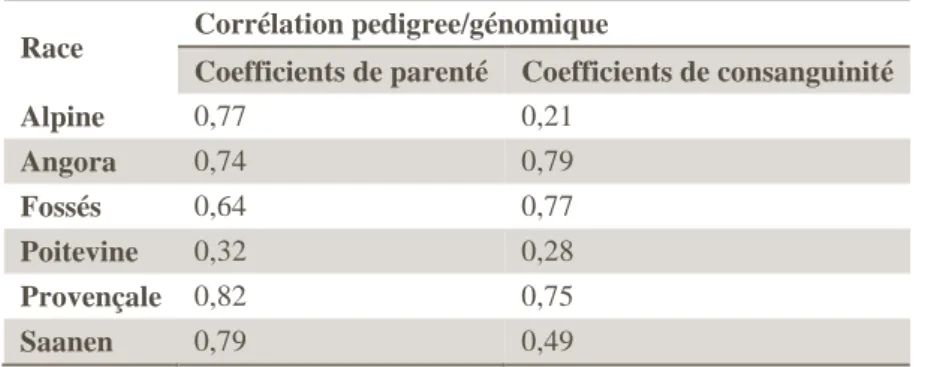
Tableau 7 : Corrélations entre les données pedigree et les données génomiques...26
Tableau 8 : Tableau récapitulatif des caractéristiques des régions significatives...34
REMERCIEMENTS
Je souhaiterais remercier Mme Isabelle Palhière et M. Bertrand Servin, mes encadrants au sein de l’INRA, pour m’avoir accompagnée et guidée au cours de mon stage grâce à leur grande écoute, leur disponibilité, et leurs conseils. Ils m’ont fait découvrir et apprécier le monde de la recherche dans lequel j’envisage aujourd’hui de continuer à évoluer. Je remercie aussi leurs collègues Mme Rachel Rupp et Mme Gwenola Tosser-Klopp pour m’avoir aidée lors de mon étude concernant la détection de traces de sélection, et avec lesquelles je vais à présent élaborer mes futures années professionnelles au cours d’un projet de thèse.
Merci également à Mme Delphine Duclos, Mme Coralie Danchin et M. Xavier Rognon de m’avoir écoutée et conseillée au cours des visioconférences que l’on a pu réaliser, ainsi que pour tous les documents bibliographiques et les données de pedigree qu’ils m’ont fournies durant ce stage.
Un grand merci à toute l’équipe d’Isabelle, l’équipe GeSPR (Génétique et Sélection des Petits Ruminants) pour m’avoir intégrée et suivi l’évolution de mon stage au cours de leurs réunions hebdomadaires, ainsi qu’à toute l’équipe de Bertrand, l’équipe DynaGen (Dynamique des Génomes et des Populations) qui m’a fourni de précieux conseils lors d’une présentation de mes résultats au cours du stage. Je remercie aussi toute l’unité de recherche GenPhySE (Génétique Physiologie et Systèmes d’Elevage) qui m’a accueillie et permis de réaliser ce stage, et plus particulièrement M. Dominique François et M. Dominique Hazard, mes collègues de bureau durant ces 6 mois, toujours disponibles pour répondre à mes questions.
Je tiens également à remercier Mme Zulma Vitezica, Mme Cécile Bonnefont et la scolarité de l’ENSAT pour m’avoir aidée à réaliser ce stage, ainsi que toute l’équipe pédagogique pour la préparation théorique et pratique.
Enfin, je souhaiterais remercier mon compagnon, mes parents ainsi que mes proches pour leur soutien qui m’a permis depuis toujours d’être épanouie, d’avancer dans mes études, et d’envisager mes futurs personnel et professionnel.
RÉSUMÉ
Ce rapport présente les études effectuées au cours des 6 mois de mon stage de fin d’études à l’INRA. Durant ce stage, j’ai eu l’opportunité d’étudier un grand jeu de données génomiques sur l’espèce caprine (puce caprine 50K SNPs), intégré à l’initiative internationale ADAPTMap d’étude de la diversité caprine mondiale. Cette espèce, domestiquée il y a 11 000 ans en Asie Mineure, a suivi la migration de l’Homme en Europe et en Asie au cours du Néolithique, a été introduite dans toute l’Afrique, et se retrouve donc aujourd’hui dans le monde entier. En France, 14 races caprines sont officiellement reconnues par le Ministère de l’Agriculture. En exploitant une partie de ces données mondiales, j’ai étudié si la diversité génétique actuelle pouvait aider à caractériser ou apporter des éléments quant à la migration de la chèvre via la mer Méditerranée (57 races internationales) et via le Danube (36 races internationales). Je me suis ensuite concentrée sur l’analyse des 8 races françaises génotypées (223 individus) pour mieux comprendre leur origine dans un contexte mondial. Les résultats obtenus à partir de différentes approches (ACP avec le logiciel PLINK, structure des populations avec Admixture, arbres phylogénétiques avec hapFLK et TreeMix) permettent de mettre en évidence une diversité génétique bien structurée au sein des races présentes entre le berceau de domestication et l’Ouest de l’Afrique du Nord et l’Europe du Sud, confirmant ainsi l’existence d’une voie de migration méditerranéenne. Ces résultats révèlent également la proximité génétique des races françaises. Puis, j’ai identifié des signatures de sélection au sein de 6 races françaises (Alpine, Corse, Fossés, Poitevine, Provençale, Pyrénées, Saanen), à partir d’une approche de différenciation entre les races (logiciel hapFLK). Quatre régions ont été détectées sur les chromosomes 5, 6, 11 et 13, mettant en lumière des gènes candidats (ADAMTS20, CSN1S1, ASIP et ATRN) sélectionnés au cours de l’histoire évolutive de ces races. Ces résultats majeurs constituent la première étude qui caractérise les races caprines françaises dans leur ensemble et devraient être utiles dans divers domaines tels que la gestion des races, les recherches futures sur les gènes sélectionnés, ou encore la conservation de la biodiversité.
ABSTRACT
This report presents the studies realized during my six month end of study internship at INRA. During this internship, I had the opportunity to analyze a large genomic data set on the caprine species (GoatSNP50 BeadChip), integrated into the international initiative ADAPTmap studying global caprine diversity. This species was domesticated 11 000 years ago in Asia Minor and followed human migration to Europe and to Asia through Neolithic. It has been introduced all across Africa and it is now raised all over the world. 14 breeds are officially recognized by the Ministry of Agriculture in France. By exploiting a part of these international data, I studied if the present genetic diversity could help to characterize or help to understand the migration of goat via the Mediterranean Sea (57 international breeds) and via the Danube (36 international breeds). I then focused on the analysis of 8 genotyped French breeds (223 individuals) to better understand their origin in a global context. The results obtained from different approaches (PCA using PLINK software, populations structure using Admixture, phylogenetic trees using hapFLK and TreeMix) highlight a well-structured genetic diversity within breeds located between the domestication cradle and West of North Africa and Southern Europe, which confirm the existence of a Mediterranean migration route. These results also reveal the genetic proximity of French breeds. Then, I identified with a differentiation between breeds approach (hapFLK software) selection signatures in 6 French breeds (Alpine, Corse, Fossés, Poitevine, Provençale, Pyrénées, Saanen). Four regions were detected on chromosomes 5, 6, 11 and 13, bringing to light candidate genes (ADAMTS20, CSN1S1, ASIP and ATRN) which have been selected over evolution history of these breeds. These major results represent the first study that characterizes French caprine breeds as a whole. They should be useful in various fields such as breeds’ management, future researches on selected genes, or biodiversity conservation.
INTRODUCTION
La conservation de la biodiversité est aujourd’hui une question centrale dans le monde dans un contexte de changement climatique. Que ce soit en effet les végétaux ou les animaux, la modification de l’environnement dans lequel évoluent les êtres vivants provoque des variations de l’équilibre écologique, causant l’expansion de certaines espèces et la disparition d’autres. Depuis la sédentarisation de l’Homme et le développement de l’agriculture au Néolithique (environ 9 000 ans av. J.-C.), ces modifications ont pris de l’ampleur, de manière directe (espèces domestiquées) ou indirectes (espèces dont le milieu de vie a été dégradé), principalement dans les zones où l’Homme s’est implanté. Les espèces animales actuelles, élevées pour nourrir la planète, telles que la volaille, les porcins, les bovins, les ovins, ou encore les caprins, sont le fruit de la sélection de l’Homme au cours des siècles. Cette sélection a toujours favorisé au sein d’une espèce les races les plus productrices ou possédant des qualités recherchées par l’Homme, comme par exemple des caractéristiques morphologiques (taille, forme des cornes, etc.), ou des caractéristiques comportementales (adaptabilité, maniabilité, etc.). Les races délaissées par l’Homme sont alors bien souvent amenées à disparaître, emmenant avec elles une certaine diversité.
Dans le cadre de mon stage de fin d’études, je me suis intéressée à l’espèce caprine Capra hircus ou chèvre domestique. Cette espèce, appartenant au règne animal, s’insère dans la sous-famille des caprinés (chamois, chèvres, moutons, etc.) au sein de la famille des bovidés (antilopes, vaches, etc.). Parmi le genre Capra, les espèces les plus proches de la chèvre domestique sont la chèvre sauvage ou bézoard (Capra aegagrus), l’ibex (Capra ibex), ou encore les markhors (Capra falconeri) et les bouquetins (Capra nubiana, Capra pyrenaica, etc.). Des études génétiques ont montré que le plus proche parent de la chèvre domestique est le bézoard, qui serait même son ancêtre (Takada et al. 1997, Manceau et al. 1999).
Étudier les différences entre races d’une même espèce a toujours été réalisé par l’Homme et on retrouve quelques traces écrites de ces observations faites sur l’espèce caprine dans la littérature (Vigne 2008, Denis 2008). Elles étaient basées sur des différences morphologiques telles que la forme des os, la couleur de la robe, etc. Grâce à l’avancée des techniques et des connaissances en génétique, les scientifiques peuvent aujourd’hui fonder leurs études sur le génome des individus. En génétique animale, de nombreuses études ont été effectuées sur la diversité des espèces domestiques à l’aide de différents marqueurs moléculaires (Lenstra et al. 2005). Un marqueur génétique est une séquence d'ADN variable selon les individus, dont les différentes formes (allèles) peuvent être déterminées (génotypage). Le but de ces études est d’analyser le polymorphisme de l’ADN afin de connaître la structuration des populations et leur histoire évolutive depuis la domestication. On retrouve dans ces études les marqueurs suivants : microsatellites, AFLP (Amplification Fragment Length Polymorphism, ou polymorphisme de longueur des fragments d'amplification), ADN mitochondrial, ADN du chromosome Y, SNPs (Single-Nucleotide Polymorphism ou polymorphisme nucléotidique). Les marqueurs les plus utilisés sont les microsatellites et plus récemment les SNPs qui sont moins onéreux à obtenir et plus enclins à se développer en termes de densité dans les années à venir, permettant ainsi de capter plus d’information génétique qu’ils ne procurent à présent.
La thématique de la diversité génétique chez la chèvre domestique a donc déjà été de nombreuses fois abordée par les scientifiques à travers le monde (Cañón et al. 2006, Kijas et al. 2012, Nicoloso et al. 2015, Radhika et al. 2015, Lenstra et al. 2016). Cependant, les races caprines françaises ont été peu étudiées, malgré la diversité assez conséquente avec 14 races officiellement reconnues par le Ministère de l’Agriculture. L’étude que je vais aborder dans ce rapport est l’analyse
de la diversité génétique des races caprines françaises afin de comprendre leur évolution au cours de l’Histoire, ainsi que les signatures de sélection que l’on peut détecter, preuves des évènements passés.
1 Histoire de la domestication de la chèvre
La diversité génétique au sein d’une espèce est le fruit des évènements génétiques et démographiques qui ont eu lieu conjointement au cours de l’histoire évolutive, c’est-à-dire la conséquence des « pressions évolutives » (mutation, sélection, dérive génétique, et migration). La structure des populations d’une même espèce révèle en effet d’une part les évènements aléatoires de mutation génétique, mais également les effets d’une sélection faite soit de manière naturelle, en conséquence d’une meilleure adaptabilité au milieu de vie, soit réalisée par la main de l’Homme, souvent dans un but de production. L’Homme a donc joué un rôle important dans la conservation ou la disparition de la diversité génétique depuis la domestication des espèces de rente occidentales actuelles.
D’après de nombreuses traces archéologiques (Vigne 1988) et des études génétiques de plus en plus précises (Colli et al. 2015), on sait à présent que la chèvre a été domestiquée il y a 11 000 ans dans le Sud-Est de l’Anatolie (Turquie actuelle) et sur les Monts Zagros (Iran central), ce qui coïncide avec la zone biogéographique du « Croissant fertile ». Ce petit ruminant, facilement transportable et possédant de bonnes capacités d’adaptation aux conditions environnementales, a ensuite suivi la migration de l’Homme au Néolithique vers l’Europe occidentale, qui a débuté il y a 7 500 ans. Cette migration a été assez peu étudiée, contrairement aux autres espèces de bovidés plus productives, mais deux voies hypothétiques sont mises en avant dans la littérature (Vigne 1988) : une voie de migration méditerranéenne, et une voie de migration dite « Danubienne ». Concernant, les races caprines françaises, elles proviendraient de la voie de migration méditeranéenne qui a amené la chèvre dans un premier temps dans les pays européens du Sud tels que la Grèce et l’Italie, et par la suite en métropole française.
Il existe très peu de preuves retraçant ensuite l’évolution de la chèvre domestique en France en dehors des preuves archéologiques. Ces dernières apportent des éléments quant aux conduites d’élevage, par exemple grâce aux proportions des débris osseux qui révèlent l’abattage des jeunes mâles plutôt que des mâles adultes en Ardèche en 5 500 av. J.-C. (Lacroix 2008). En effet, la chèvre a très peu intéressé les auteurs anciens, car jusqu’au XXème siècle, ce petit ruminant est considéré en France comme la « vache du pauvre » (Denis 2008). Cette réputation, bien que positive dans le sens où c’est une espèce capable de s’adapter aux conditions les plus difficiles, a influencé le désintérêt de la population française dans un contexte de modernisation de l’agriculture au cours du XIXème siècle, siècle du commencement de la création des races domestiques actuelles. De plus, les effectifs par exploitation sont faibles en raison de la non spécialisation des élevages qui possèdent des chèvres en complémentarité d’autres animaux, ce qui ne permet pas de dégager des agriculteurs « leaders de race », qui sont à l’origine de la reconnaissance de race, comme cela a été le cas dans d’autres espèces.
Les auteurs de la fin du XVIIIème et du XIXème siècles se basent sur des critères morphologiques pour distinguer les individus (Denis 1988). Dans le cas de la chèvre, cela correspond à la couleur de la robe, ou bien au gabarit, ou encore à la forme des cornes. La chèvre étant un animal qui a très peu intéressé la population française, il n’y a pas eu de réelle standardisation de la morphologie avant la création des races à la fin du XXème siècle. C’est pourquoi, il n’y a pas de distinction dans la littérature entre les races en France aux XVIIIème et XIXème siècles, à l’exception des races Angora et Cachemire, races importées d’Asie, élevées pour la laine qu’elles produisent et morphologiquement très différentes de la chèvre « commune » française. Ce n’est qu’en 1886 qu’un
Rapport de stage de fin d’études Claire OGET
Tableau 1 : Les races caprines françaises officielles et leurs effectifs (Sources : Capgenes et Markey et al. 2014)
Race Officialisation Effectif (femelles) Type
Alpine 1930 450 000 Laitier
Poitevine 1947 3 173 Laitier
Saanen 1955 350 000 Laitier
Angora Années 80 4 000 Lainier
Rove 1990 10 514 Mixte
Provençale 2000 999 Laitier
Corse 2003 29 000 Mixte
Pyrénées 2004 3 297 Mixte
Fossés 2005 1 040 Laitier
Massif Central 2010 930 Laitier
Boer 2011 2 000 Viande
Créole 2011 10 000 Viande
Péi 2011 500 Viande
auteur, Sanson, commence à s’intéresser à la classification des chèvres. Il distingue trois grands groupes de chèvres : les chèvres d’Europe, les chèvres d’Asie, et les chèvres d’Afrique. Au sein des chèvres d’Europe, il fait ensuite la description des différences morphologiques entre le groupe alpin, le groupe pyrénéen, le groupe du Massif Central, et enfin, la population Corse.
Les principales régions « berceaux » de l’élevage caprin en France sont les régions Poitou-Charentes, Rhône-Alpes, et Centre. Les chèvres sont principalement élevées pour leur lait dans un but de fabrication fromagère. On dénombre en 1900 1,8 million de chèvres (Spindler 1988, Le Jaouen 2002). Jusqu’en 1973, le cheptel caprin français va décroître, atteignant le nombre de 880 000 individus. Les causes de ce déclin sont multiples : désintérêt des éleveurs, épidémies successives de fièvre aphteuse dans les années 20, guerres mondiales, etc. C’est grâce à un accompagnement technique et scientifique des éleveurs, permis grâce à l’action conjointe de l’Unité Nationale de Sélection et de Promotion de Race (UPRA) caprine, créée en 1966 et devenue Capgenes dans les années 2000, qui représentait les trois races laitières alors reconnues (Alpine, Saanen, Poitevine), de l’Institut Technique de l’Elevage Ovin et Caprin (ITOVIC), créé en 1967, qui deviendra l’Institut de l’Elevage (Idele) en 1991, et de l’Institut National de la Recherche Agronomique (INRA) qui a développé depuis les années 50 un pôle de recherches caprines, que le cheptel caprin va retrouver une croissance. En 2010, le recensement agricole dénombre alors 955 000 chèvres.
2 Les races caprines françaises
La chèvre « commune » de France a su s’adapter au fil des siècles aux conditions dans lesquelles elle était élevée par l’Homme. Les éleveurs ont consciemment ou non sélectionné des caractères tels que la productivité, la résistance aux maladies, etc., et ont permis l’acquisition de phénotypes particuliers et reconnaissables, créant ainsi des races locales dans de nombreuses régions françaises. Les races caprines locales au cours du XXème siècle sont progressivement remplacées par des races très productives et mieux adaptées à la production intensive laitière (meilleure conformation des mamelles, meilleures capacités de production laitière, poils moins longs, etc.), introduites en France dans les années 20 et originaires de Suisse et de l’Est de la France. Ces races locales ont, dans les années 60-70, quasi disparu. Les associations d’éleveurs des races locales en déclin vont alors commencer à rechercher la reconnaissance de l’Etat afin d’obtenir des aides et conserver le patrimoine génétique, et donc la diversité génétique des races caprines françaises. Des programmes de conservation, soutenus par l’Institut de l’Elevage, vont être mis en place à partir des années 80 et vont aboutir à l’officialisation des races locales.
Aujourd’hui, 14 races caprines sont officiellement reconnues par le Ministère de l’Agriculture. Parmi elles, deux races originaires de l’Est de la France et de Suisse sont grandement majoritaires car elles représentent plus de 90% des effectifs nationaux et 97,1% du total des lactations recensées au Contrôle Laitier Officiel en 2015 (Institut de l’Elevage 2016a): l’Alpine et la Saanen. 11 autres races sont des races locales, dont 8 sont présentes en métropole. Parmi ces races locales, 5 (Provençale, Fossés, Massif Central, Péi et Lorraine) sont des races à très petits effectifs (< 1000 chèvres) qui survivent grâce à des associations d’éleveurs dans le cadre de programmes de conservation, 3 autres (Poitevine, Rove et Pyrénées) ont été sauvées de l’extinction et sont maintenues à des effectifs de quelques milliers de chèvres, les 3 dernières (Corse, Boer et Créole) ont des effectifs intermédiaires stables dus à l’isolation géographique de leurs milieux d’élevage. La dernière, la race Angora serait originaire d’Asie et a la particularité d’être élevée pour sa production de laine. Le Tableau 1 présente les effectifs des races officielles, classées par ordre chronologique d’officialisation. Toutes ces races sont fédérées et gérées par Capgenes.
Rapport de stage de fin d’études Claire OGET
Figure 2 : Photographie d'une chèvre Saanen (Source : Capgenes)
Figure 3 : Photographie d'une chèvre du Rove (Source : Capgenes)
Figure 1 : Photographie d'une chèvre Alpine (Source : Capgenes)
Nous allons voir à présent brièvement les différentes caractéristiques de ces races, dans l’ordre décroissant des effectifs en commençant par les races présentes en métropole, afin de mettre en lumière la diversité phénotypique des chèvres françaises.
Chèvre Alpine (chamoisée)
Originaire des Alpes françaises et suisses, la chèvre Alpine (Figure 1) est la chèvre la plus répandue en France avec environ 450 000 chèvres dont 150 072 au Contrôle Laitier Officiel en 2015, soit 57,1% des lactations quantifiées (Institut de l’Elevage 2016a). Elle a été introduite en France dans les années 20 et s’est installée dans tout le Centre et l’Ouest de la métropole. La création du Livre Généalogique en 1930 a abouti à l’officialisation de la race. Cette chèvre a remplacé les races locales lors de son expansion et plus particulièrement la chèvre de Savoie présente dans les Alpes françaises, aux patrons de couleurs plus variés, qui n’est pas officialisée aujourd’hui. Reconnaissable grâce à sa robe dite « chamoisée » (de couleur jaune foncé à brune claire) au poil ras et de gabarit moyen (la femelle pèse entre 50 et 70 kg, le bouc entre 80 et 100 kg), la chèvre Alpine s’adapte à tous les types d’élevage (stabulation, pâturage). Elle possède aujourd’hui une grande capacité de production laitière avec en moyenne 909 kg de lait par lactation en 296 jours (Institut de l’Elevage 2016a) grâce au développement du schéma de sélection depuis les années 70. Ce schéma de sélection a eu pour objectif depuis sa création l’amélioration des qualités laitières (quantités de lait, de matière protéique et de matière grasse, et taux protéique et butyreux) et prend également en compte aujourd’hui la morphologie (plus particulièrement des mamelles) et le nombre de cellules somatiques dans le lait (Danchin-Burge 2012). Ainsi, elle possède au dernier Contrôle Laitier Officiel en 2015 de bons taux avec en moyenne un taux protéique (TP) de 33,3 g/kg et un taux butyreux (TB) de 37,6 g/kg (Institut de l’Elevage 2016a).
Chèvre Saanen
Originaire de la vallée de la Saane dans le canton de Berne en Suisse, la chèvre Saanen (Figure 2) est la plus répandue à travers le monde, et la deuxième race caprine française en termes d’effectifs, avec environ 350 000 chèvres dont 105 088 au Contrôle Laitier Officiel en 2015, soit 40,0% des lactations quantifiées (Institut de l’Elevage 2016a). Implantée plutôt dans le Sud de la France depuis les années 50, la chèvre Saanen s’est rapidement répandue sur tout le territoire français après la création du Livre Généalogique en 1955. Cette race, de robe uniformément blanche au poil ras et de grand gabarit (la femelle pèse entre 50 et 90 kg, le bouc entre 80 et 120 kg), s’adapte très bien à la production laitière intensive. Soumise au même schéma de sélection que la race Alpine, elle se distingue de cette dernière par ses très grandes capacités laitières avec en moyenne 954 kg de lait par lactation en 308 jours et ses taux un peu plus faibles avec en moyenne un TP de 32,0 g/kg et un TB de 35,5 g/kg (Institut de l’Elevage 2016a).
Chèvre du Rove
La chèvre du Rove (Figure 3) est originaire de l’arrière-pays méditerranéen (village du Rove). Présente au XIXème siècle au sein des grands troupeaux ovins qui transhumaient, cette race possède des qualités laitières exceptionnelles avec en moyenne un TP de 31,2 g/kg et un TB de 41,0 g/kg, malgré une quantité de lait produite faible (en moyenne 273 kg de lait par lactation en 179 jours) (Institut de l’Elevage 2016a), qui sont à l’origine du fromage la brousse du Rove. Après avoir frôlé l’extinction en 1979 (effectif estimé à 500 individus) à la suite de l’arrêt de la transhumance à pied et la mise en place de la prophylaxie pour l’éradication de la brucellose, cette race a survécu grâce à l’Association de Défense des Caprins du Rove (ADCR). Connue pour sa couleur rouge avec parfois quelques petites mouchetures blanches, et surtout grâce à sa cornure très développée, la chèvre du Rove est capable de
Rapport de stage de fin d’études Claire OGET
Figure 6 : Photographie d'une chèvre Poitevine (Source : Capgenes)
Figure 5 : Photographie d'une chèvre des Pyrénées (Source : Capgenes)
Figure 4 : Photographie d'une chèvre Angora (Source : Capgenes)
valoriser les terrains très pauvres. Cette race est aujourd’hui sauvée de l’extinction avec plus de 10 000 chèvres présentes dans environ 150 élevages dont un au Contrôle Laitier Officiel, et est inscrite dans un programme de conservation bien géré par l’ADCR et soutenu par l’Institut de l’Elevage. Race mixte (à la fois destinée à la production de viande et de lait), 90% de son effectif est présent dans le grand Sud-Est (Danchin-Burge 2008).
Chèvre Angora
Présente au Tibet en 2000 av. J.-C., la chèvre Angora (Figure 4) a été importée en Turquie dans la province d’Angora (aujourd’hui Ankara) au XIème
s. Cette chèvre produit une fibre de grande qualité, la fibre Mohair, transformée en vêtements importés en France de Turquie dès le XVIème s. La race est importée en France au XIXème s. pour le développement de l’industrie du Mohair. Cette première population disparaît au cours du XXème s. et c’est à partir des années 80 que de nouvelles importations sont réalisées depuis le Canada, le Texas, l’Afrique du Sud, l’Australie et la Nouvelle-Zélande. Ces individus sont les souches de la race actuelle puisqu’aucune importation n’a été réalisée depuis, établissant ainsi une variabilité assez restreinte (Institut de l’Elevage 2011). Aujourd’hui, un programme de sélection est mis en place par Capgenes, en collaboration avec l’INRA et l’Institut de l’Elevage avec 2 500 chèvres (35 élevages) dans le noyau de sélection, qui permettent la diffusion du progrès génétique vers les 4 000 chèvres de la race. Ce schéma a pour objectif l’obtention d’animaux de 18 mois, produisant des fibres d’un diamètre inférieur à 30 microns (Danchin-Burge 2012). Les producteurs valorisent leurs produits grâce à la charte qualité « Le Mohair des Fermes de France ».
Chèvre des Pyrénées
Originaire de toute la chaîne pyrénéenne de l’Ariège à l’Atlantique, la chèvre des Pyrénées (Figure 5) était considérée au début des années 90 comme quasi disparue en raison de la désertification et de la concurrence des races sélectionnées. Cette race mixte, à l’origine présente dans les troupeaux ovins destinés à la production de viande, est de format moyen à poils longs et de robe souvent noire mais pouvant être « décolorée par le soleil ». C’est grâce à des actions réalisées par le Conservatoire du Patrimoine Biologique Régional (CPBR) de Midi-Pyrénées et le Conservatoire des Races d’Aquitaine, qui ont permis la création de l’Association Chèvre des Pyrénées, que la race a pu être sauvée. Elle comptabilise aujourd’hui environ 3 300 chèvres. La majorité des élevages sont allaitants avec une spécialisation dans la production de chevreaux (20% des élevages sont fromagers, Ricordeau et al. 1999). Les 3 élevages laitiers contrôlés (57 chèvres) montrent une production laitière modeste (en moyenne 312 kg de lait par lactation en 214 jours) avec des taux faibles (TP de 29,0 g/kg et TB de 30,3 g/kg) (Institut de l’Elevage 2016a). Cette race est également connue pour avoir une forte structuration par vallée (Ricordeau et al. 1999) et un taux de consanguinité élevée (Cañón et al. 2006).
Chèvre Poitevine
Originaire de Poitou-Charentes, cette race locale (Figure 6) de couleur brun foncé avec ventre et face intérieure des membres blancs, aux poils longs, comptait au début du XXème s. plus de 40 000 individus qui alimentaient les laiteries de la région. Dans les années 20, une succession d’épidémies de fièvre aphteuse a décimé les troupeaux qui ont été reconstitués grâce à des croisements avec les races Alpine et Saanen qui se développaient dans cette région. En 1947, les fromageries coopératives des Deux-Sèvres et de la Vienne créent le Livre Généalogique Caprin Poitevin. Cette race, officialisée assez tôt par rapport aux autres races locales, n’a finalement pas suivi le mouvement d’amélioration génétique à la suite de l’union des organismes de sélection au sein de l’UPRA caprine en 1966. Les effectifs diminuent face aux performances plus faibles de la race (520 kg de lait par lactation en moyenne en 259 jours, Institut de l’Elevage 2016a). En 1986, l’Association pour la Défense et le
Rapport de stage de fin d’études Claire OGET
Figure 8 : Photographie de chèvres des Fossés (Source : Capgenes)
Figure 9 : Photographie de chèvres du Massif Central (Source : Capgenes)
Figure 7 : Photographie de chèvres Provençales (Source : Capgenes)
Développement de la Chèvre Poitevine (ADDCP) est créée et redynamise la race. Aujourd’hui, la race compte plus de 3 000 chèvres dont 483 sont au Contrôle Laitier Officiel. Les taux historiquement forts, sont aujourd’hui plus faibles que ceux des races sélectionnées (TP de 30,9 g/kg et TB de 34,0 g/kg) (Institut de l’Elevage 2016a) malgré une particularité connue de cette race qui est la fréquence importante (environ 40%) de l’allèle fort originel B1 de la caséine αs1 qui confère au lait une bonne capacité fromagère (Ricordeau et al. 1996).
Chèvre Provençale
La chèvre Provençale (Figure 7) est la chèvre locale de tout l’arrière-pays provençal. A
l’inverse de la chèvre du Rove, cette race ne transhumait pas et restait sur l’exploitation. Race locale avec un bon potentiel laitier (en moyenne sur 34 chèvres : 691 kg de lait par lactation en 272 jours d’après les résultats de Contrôle Laitier Officiel de 2009), elle a frôlé l’extinction au début des années 90 avec environ 200 individus. Sa redynamisation a été effectuée à partir de 1999 grâce à une poignée d’éleveurs regroupés au sein de l’Association de sauvegarde et de développement de la chèvre Commune Provençale. La chèvre Provençale est aujourd’hui inscrite dans un programme de conservation géré par l’Institut de l’Elevage. Cette race n’a jamais subi d’uniformisation au niveau de la couleur de sa robe. On peut la reconnaître grâce à son poil mi-long, sa grande carrure et ses oreilles longues et pendantes. On évoque chez les auteurs anciens (Buffon 1755 et Mauron 1947) une origine syrienne à cette race dont l’héritage principal, malgré les nombreux croisements avec des chèvres européennes, reste aujourd’hui ces oreilles plutôt pendantes.
Chèvre des Fossés
Le berceau d’origine de la chèvre des Fossés (Figure 8) est le grand Ouest de la France (Bretagne, Pays de la Loire, Basse Normandie). Cette race relique, qui compte actuellement 1 040 chèvres, a été sauvée par les actions réalisées par l’Institut de l’Elevage, accompagné d’opérateurs régionaux tels que l’Ecomusée du Pays de Rennes, dès le milieu des années 90. En 2007 est créée l’Association de Sauvegarde et de Promotion (ASP) de la Chèvre des Fossés qui permet aujourd’hui d’assurer la survie de la race au sein d’un programme de conservation soutenu par l’Institut de l’Elevage. Particulièrement bien adaptée au climat humide, cette race de petit format possède des poils de longueur variable. Sa capacité laitière est faible d’après les résultats de Contrôle Laitier Officiel de 2012 avec 260 kg de lait par lactation en 216 jours (moyenne sur 60 chèvres). La couleur de la robe n’est pas uniformisée mais les éleveurs préfèrent les robes pie. Aujourd’hui élevée au sein de toutes petites exploitations, cette race semble avoir été particulièrement bien conservée en raison d’un berceau d’origine non localisé dans une région caprine, mais aussi en raison de la présence de plusieurs souches bien variables qui annonce une structuration forte de la population ainsi que de la dynamique de reproduction (toutes les femelles sont mises à la reproduction et le nombre de femelles mises au même bouc est généralement faible).
Chèvre du Massif Central
Comme beaucoup d’autres races locales françaises, la chèvre du Massif Central (Figure 9), originaire du Centre de la France (Massif Central, Limousin, Berry, Vallée du Rhône) avait quasiment disparu lors de l’introduction des races sélectionnées. Ce n’est qu’en 1994 que la Fédération pour promouvoir l’Elevage des Races domestiques Menacées (FERME) lance une opération de sauvegarde qui aboutit à la création de l’Association pour le Renouveau de la Chèvre du Massif Central (ARCMC) en 1996. La race qui compte aujourd’hui 930 chèvres, inscrite dans un programme de conservation soutenu par l’Institut de l’Elevage, n’est pas standardisée sur sa couleur de robe. En 2015, 2/3 des chèvres contrôlées au contrôle laitier officiel et simplifié (sur 57 individus) produisaient
Rapport de stage de fin d’études Claire OGET
Figure 11 : Photographie d'une chèvre Corse (Source : Capgenes)
Figure 12 : Photographie de chèvres Créoles (Source : Capgenes)
Figure 13 : Photographie d'une chèvre Boer (Source : Capgenes)
Figure 10 : Photographie d'une chèvre de Lorraine (Source : Capgenes)
entre 176 et 368 kg de lait par lactation sur une durée variant de 152 à 249 jours, soit une production modeste, avec des taux compris entre 25,7 et 30,4 g/kg pour le TP et 25,1 et 35,2 g/kg pour le TB, soit un lait plutôt pauvre (Institut de l’Elevage 2016b). D’après la littérature, cette population serait hétérogène car elle engloberait les populations des Cévennes, du Mont D’Or, d’Auvergne et du Falgoux.
Chèvre de Lorraine
La chèvre de Lorraine (Figure 10) est la dernière race en date à avoir été reconnue officiellement en 2012. Originaire du pays lorrain, cette race locale laitière, d’une robe herminée grise, est le fruit de croisements entre la chèvre commune et les chèvres introduites par la Société Régionale d'Acclimatation du Nord-Est pendant la guerre. Après avoir frôlé l’extinction en 2006 avec seulement 78 individus recensés, l’Association des « Amis de la Chèvre de Lorraine » a permis la redynamisation de la race. C’est aujourd’hui la race caprine française présente en métropole avec le plus petit nombre d’animaux (625).
Chèvre Corse
Race locale, la chèvre Corse (Figure 11) n’a jamais été en voie d’extinction grâce à
l’insularité de son berceau d’origine. En effet, les races sélectionnées introduites depuis la Suisse ne sont pas arrivées jusqu’en Corse, permettant à la race locale de survivre sans avoir subi de chute d’effectif ou d’épisodes de croisement. Cette race de petit gabarit (la femelle pèse entre 30 et 40 kg, le bouc entre 45 et 60 kg), à la couleur non standardisée, possède des poils longs qui sont le fruit de l’adaptation aux parcours épineux de son environnement. Race mixte, on élève le « cabri de Noël » sous la mère, et on transforme le lait en de nombreux fromages locaux dont le bruccio. Aujourd’hui, on compte 29 000 chèvres dont 3 684sont au contrôle laitier non officiel. La capacité laitière de cette race est assez modeste avec les 2/3 des chèvres produisant entre 121 et 330kg de lait par lactation sur une durée variant de 144 à 258 jours mais riche avec un TP compris entre 28,6 et 35,6 g/kg et un TB compris entre 35,3 et 51,8g/kg (Institut de l’Elevage 2016b).
Chèvre Créole
La chèvre Créole (Figure 12), élevée en Guadeloupe, département français d’outre-mer, est
une population caprine locale qui s’est développée suite à de nombreuses importations (Afrique, Inde, Europe). Cette appellation de race est également celles d’autres races des îles des Caraïbes et d’Amérique du Sud. Le génotype de cette race serait proche des races africaines. La chèvre Créole possède une très bonne adaptabilité aux contraintes climatiques du milieu tropical ainsi qu’une reproduction aisée, associée à des taux de fertilité et de prolificité élevés, et de bonnes qualités maternelles. On remarque peu d’homogénéité de couleur de robe chez cette race à viande, avec cependant une fréquence élevée de la couleur noire. Elevée pour sa production de cabris, cette race, qui compte environ 10 000 chèvres, possède un schéma de sélection grâce à la coopérative Cabricoop en collaboration avec la Chambre d’Agriculture de la Guadeloupe et l’INRA, avec 300 mères dans la base de sélection. Ce schéma de sélection a pour objectif l’amélioration des caractères de production (croissance, rendement carcasse), mais aussi des qualités maternelles et de l’adaptation au milieu.
Chèvre Boer
La race Boer (Figure 13), présente aujourd’hui sur l’île de la Réunion, département français
d’outre-mer, est originaire d’Afrique du Sud, issue d’un croisement entre les races locales et les races laitières européennes. Cette race à viande a ensuite été introduite sur l’île de la Réunion en 1976 où
Rapport de stage de fin d’études Claire OGET
Tableau 2 : Résultats d'une étude de variabilité génétique sur 4 races caprines (Source : d’après Institut de l'Elevage 2011).
Population de référence Nombre moyen de générations connues Nombre efficace d’ancêtres Consanguinité moyenne en 2009 Poitevine 1 989 5,23 60 3,4 % Saanen 125 797 6,34 51 2,4 % Alpine 183 611 7,81 46 2,5 % Angora 1 229 5,69 34 2,1 %
Figure 14 : Photographie de chèvres Péi (Source : Capgenes)
elle a été croisée à nouveau aux races locales. Reconnue officiellement en 2011, la chèvre Boer est principalement de couleur blanche avec une tête rousse. Elle compte aujourd’hui environ 2 000 chèvres. Cette race est soutenue par l’Association Réunionnaise de Sélectionneurs de Cabri (ARSEC) qui souhaite mettre en place un schéma de sélection dans les années à venir, avec l’appui de Capgenes.
Chèvre Péi
Race à viande locale de l’île de la Réunion, la chèvre Péi (Figure 14) fait partie intégrante du patrimoine culturel et historique réunionnais. Absorbée par la race Boer au cours du XXème siècle, cette race possède une diversité de phénotypes. Ses particularités sont d’une part, une très bonne capacité de reproduction avec de bonnes qualités maternelles, et d’autre part une forte résistance aux pathologies. Cette race rustique, reconnue officiellement en 2011, et qui compte actuellement environ 500 chèvres, va certainement bénéficier dans les années à venir d’actions d’associations locales afin de préserver ces qualités.
3 Etudes de diversité génétique déjà réalisées sur les races caprines
françaises
Quelques-unes des races caprines françaises sont présentes dans la littérature dans des analyses de diversité. En effet, une étude de variabilité génétique sur la base des pedigrees de 9 races de petits ruminants a été réalisée en 2011 (Institut de l’Elevage 2011). Parmi ces races, on retrouve les 4 races caprines les plus étudiées jusqu’à présent et les plus renseignées concernant leur généalogie, grâce à leur schéma de sélection : l’Alpine, la Saanen, l’Angora, et la Poitevine.
Le Tableau 2 résume les valeurs intéressantes pour notre étude. La population de référence est le nombre d’individus pris en compte dans l’étude et correspond aux femelles nées de deux parents connus entre 2006 et 2009. Les races Alpine et Saanen possèdent une population de référence presque 100 fois supérieure aux races Poitevine et Angora, ce qui est dû aux effectifs de ces deux grandes races. Le nombre moyen de générations connues varie également puisqu’elle est de l’ordre de 5 pour les races Poitevine et Angora et de 6 et 7 respectivement pour les races Saanen et Alpine. Cela montre bien l’efficacité des schémas de sélection concernant l’ascendance des individus. Ces valeurs constituent donc des bons indicateurs pour les calculs de diversité sur la base des pedigrees. Le nombre efficace d’ancêtres correspond au « nombre d’ancêtres, qui en ayant tous des contributions parfaitement égales, engendreraient une population du même niveau de diversité génétique que la population de référence ». Ce nombre nous indique donc le degré de diversité génétique au sein d’une race. On constate que la Poitevine semble être la race avec le plus de variabilité avec 60 ancêtres efficaces, tandis que l’Alpine et la Saanen ont des valeurs similaires et plus réduites (46 et 51), lié à l’utilisation de l’insémination artificielle et au schéma de sélection qui conduit à réduire le nombre de souches utilisées. En revanche, l’Angora apparaît comme étant la race la moins variable (34 ancêtres). Cela peut s’expliquer par le nombre limité de souches importées en France et par l’application d’une sélection efficace. Les coefficients moyens de consanguinité calculés dans cette étude sont de l’ordre de 2% pour les races sélectionnées dont l’ascendance est bien connue (consanguinité maximale), et de 3% pour la Poitevine. Par conséquent, l’Alpine et la Saanen ont un niveau de consanguinité vraiment plus faible que l’Angora et la Poitevine. Ceci est lié à une volonté de gérer la variabilité génétique dans le cadre du schéma depuis des années.
Des études plus élargies d’un point de vue géographique et fondées sur l’analyse de marqueurs moléculaires ont été réalisées. Ces études s’intéressent aux races traditionnelles non sélectionnées qui semblent être les témoins de la diversité génétique d’une région. En 2006 est réalisée une étude de
Rapport de stage de fin d’études Claire OGET
Figure 15 : Arbre phylogénétique établi à partir des distances de Reynolds entre races ou groupes de races (Source : Lenstra et al. 2016). Les couleurs indiquent le regroupement régional des populations ou des races génétiquement proches (bleu pour les races du Nord de l’Europe, vert pour celles du Centre, vert kaki pour celles du Ouest, orange pour celles du Est, et rouge pour celles d’Asie du Sud-Ouest). Les populations en noir n’appartiennent à aucun de ces regroupements.
diversité génétique caprine à partir de 30 microsatellites (un microsatellite est une succession de motifs (très courtes séquences) d'ADN, répétés à l'identique et composés chacun de deux à cinq bases) sur des races d’Europe et du Moyen-Orient (Cañón et al. 2006). Parmi les races étudiées, on trouve la race du Rove, la chèvre Corse, la chèvre des Pyrénées, et l’Alpine (environ 30 d’individus par race). Les indices de fixation de Wright, FIS et FST sont calculés. Le FIS est un indicateur de la consanguinité
moyenne d’une race, et le FST nous indique la variabilité génétique due à l’effet race, soit la
différenciation entre races.
Les résultats de cette étude nous révèlent que la chèvre Pyrénéenne est la race la plus consanguine des races étudiées (FIS = 0,293), tandis que les autres races françaises ont des valeurs de
l’ordre de 0,1. Une analyse de structure du jeu de données global de cette étude inscrit ensuite 3 races françaises (Rove, Pyrénées et Corse) parmi les races de l’Ouest de l’Europe tandis que l’Alpine est rattachée aux races du Centre-Nord de l’Europe.
Une autre étude permet de la même manière de situer certaines races françaises au sein de la diversité des populations européennes (Lenstra et al. 2016). Cette étude, également réalisée à partir de 27 microsatellites, s’intéresse aux mêmes races : Rove, Pyrénées et Corse. Les résultats nous confirment la forte consanguinité de la chèvre des Pyrénées (FIS = 0,28) ainsi que l’appartenance au
groupe du Sud-Ouest de l’Europe des races Corse et Pyrénées comme on peut le voir sur la Figure 15. En revanche, la chèvre du Rove semble être ici plus originale car elle n’appartient à aucun des groupes présents.
4 Objectifs du stage
Comme nous venons de le voir, certaines races caprines françaises ont déjà été étudiées dans la littérature d’un point de vue de leur diversité génétique, que ce soit sur la base de leur pedigree, ou bien à partir de données de microsatellites. Ces deux approches sont limitées respectivement par le degré de connaissance du pedigree de la race étudiée, et par le degré de représentativité du génome des marqueurs génétiques utilisés, qui est faible (les deux analyses décrites précédemment sont réalisées à partir d’une trentaine de marqueurs). De plus, aucune étude ne se concentre sur les races françaises dans leur ensemble, on retrouve d’un côté soit les races dont le pedigree est assez bien connu (« grandes » races françaises telles que l’Alpine, la Saanen, l’Angora et la Poitevine), soit quelques races locales (Pyrénées, Corse et Rove) qui semblent être caractéristiques de la génétique caprine française pour les études à une plus grande échelle.
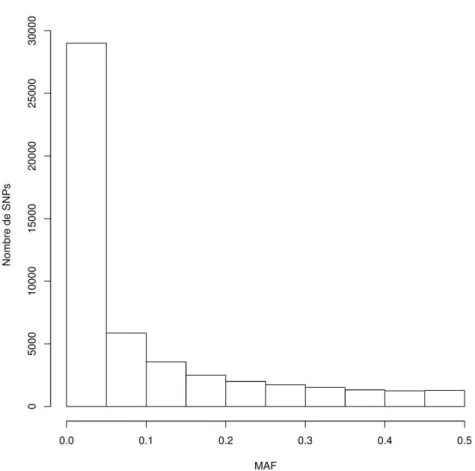
En 2011, une puce 50K SNPs a été mise au point pour l’espèce caprine (Tosser-Klopp et al. 2014). Un SNP est un marqueur moléculaire constitué d’une seule paire de bases ADN à un locus connu du génome. Cette nouvelle technologie permet d’étudier le génome d’individus avec une densité moyenne (50 000 marqueurs ici), qui ouvre de grandes opportunités pour les analyses de diversité génétique. Par ailleurs, cette densification des marqueurs permet également d’interroger spécifiquement les régions du génome pour rechercher des signatures de sélection.
Les objectifs de ce stage sont de réaliser, sur des données génétiques obtenues à partir de cette puce caprine de moyenne densité nouvellement disponible, des analyses de diversité génétique sur les races françaises échantillonnées, et de détecter des signatures de sélection qui seraient caractéristiques de certaines races.
Nous verrons dans un premier temps dans ce rapport quelques éléments bibliographiques qui retracent l’Histoire de la domestication de la chèvre, les races françaises actuelles, ainsi que le projet
dans lequel s’inscrit ce stage. Ensuite, je présenterai l’étude réalisée sur la diversité des races. Cette étude portera sur trois jeux de données différents : un jeu comprenant des races qui témoignent de la migration de la chèvre domestique depuis le berceau de domestication vers l’Ouest via la mer Méditerranée, un jeu comprenant des races qui témoignent de la migration de la chèvre domestique depuis le berceau de domestication vers le Nord-Ouest via le Danube, et un jeu comprenant uniquement les races françaises. Cette étude de diversité a pour but d’analyser les variabilités génétiques intra et inter populations afin de préciser les relations de proximité qui existent entre les races de chèvres domestiques actuelles. Puis, j’analyserai les traces de sélection que l’on peut détecter sur le jeu de données des races françaises disponibles. Cette analyse a pour objectif de mettre en lumière des parties du génome qui auraient été sélectionnées chez certaines races au cours de leur histoire évolutive. Enfin, nous terminerons par une discussion sur les résultats obtenus et sur les perspectives des recherches effectuées durant ce stage.
Rapport de stage de fin d’études Claire OGET
Figure 17 : Spectre des MAF des marqueurs de la puce 50K caprine sur les chèvres marocaines séquencées au sein du projet Nextgen (Source :
ftp://ftp.ebi.ac.uk/pub/databases/nextgen/)
Figure 16 : Spectre des MAF de 50 000 SNPs tirés aléatoirement parmi les données de séquence publiques de chèvres marocaines issues du projet Nextgen (Source :