H
es
·so(jfJ
/h?()
VALAIS
WALLIS
Haute Ecole d'Ingénierie
n
Hochschule für Ingenieurwissenschaften lIiIDip
l
ôme
20] 3
Qualitative
an
d
QU(J[lf
l
i
ti
t
ative
Amino
Acid
Analysis
~
~
Assay Development a
n
d
M
e
t
hod implementation
11'" Professeur
Marc Mathieu
Expert
Alain Razaname
Date de la remise du rapport
16 août 2013
n r.:Il!!!!!In HES-SO Valais-Wallis' rtedu Rawyl 47 •c.P.·1950 Sion 2
Ce rapport est l'original remis par l'étudiant.
HES-SO Valais
lSiITVl
c::x::I:K:J
Données du travail de diplôme
Daten der Diplomarbeit
FO 1.2.02.07.AB pof/31 10112009
D
FSI Année académique /Studienjahr NoTD/Nr. DA[g] FTV 2012/2013 ca/2013/85
Mandant /Auftraggeber Etudiant /Student Lieu d'exécution /Ausführungsort [g] HES-SO Valais-Wallis Megane Schweickhardt [g]
HES-SO Valais-Wallis 0 Industrie
0 Industrie 0 Ecole hôte
0 Ecole hôte Professeur /Dozent Expert /Experte (données complètes)
Marc Mathieu Alain Razaname
Travail confidentiel /vertrauliche Arbeit Debio Recherche Pharmaceutique 0 oui / ja1 [g] non / nein Rue du Levant 14611920 Martigny
Qualitiative and Quantitative Amino Acid Analysis: Assay Development and Method implementation Titre /Titel
Par sa signature, /'étudiant-e s'engage àrespecter strictement la directive et le caractère confidentiel du travail de diplôme qui lui est confié et des informations mises àsa disposition.
Durch seine Unterschrift verpflichtet sich der Student, die Richtlinie einzuhalten sowie die Vertraulichkeit der Diplomarbeit und der dafür zur Verfügung gestellten Informationen zu wahren.
Megane Schweickhardt
Description et Objectifs /Beschreibung und Ziele
This work aims to set-up and a peptide hydrolysis system followed by data generation using a UPLC chromatography system. Optimization of experimental and analytical parameters using model peptides and SOP writing are envisaged.
Stages:
1. Current litterature review 2. Set-up of instrument
3. Synthesis ofdemo peptide-sequences 4. Analysis of samples
5. Optimisation of experimental parameters
Délais1 Termine
Attribution du thème1Ausgabe des Auftrags:
29.04.2013
Exposition publique1Ausstellung Diplomarbeiten:
30.08.2013
Fin des travaux de diplôme dans les laboratoires de l'école 1
Ende der Diplomarbeiten in den Labors der Schule:
12.07.2013 1 16h15
Remise du rapport1Abgabe des Schlussberichts:
16.08.2013 1 12h00
Défense orale1 Mündliche Verteidigung:
dès la semaine 35 1 ab Woche 35
Signature ou visa1 Unterschrift oder Visum
Responsable de la filière ~~./' 1
Leiterdes
r=:
»:
:
~
EtudianUStudent:....
\\
..
...
$
L
h
.
weÀJ:lli
.
~
Objectif du projet
Mise en place d’un appareillage et développement d’une méthode d’analyse d’acides aminés par chromatographie UPLC et détection UV. Identification et dosage des acides aminés présents dans une séquence peptidique modèle, après hydrolyse et dérivatisation.
Méthodes | E xpérie nces | Résultats
Les acides aminés sont les composants de base des protéines. L’analyse d’acides aminés est un outil de vérification de présence de ceux-ci. Elle permet également la détermination de leur concentration et donc d’en extrapoler celle de la protéine.
Initialement la synthèse en phase solide d’un peptide modèle contenant une sélection d’acides aminés dans des proportions différentes a été réalisée. Puis, après montage de l’appareillage, une procédure d’hydrolyse standard a été optimisée. Différentes concentrations en acide chlorhydrique et de durées d’hydrolyse ont été évalués. Pour la partie de dérivatisation des acides aminés, un kit commercial utilisant du 6-aminoquinolyl-N-hydroxysuccinimidyl carbamate a été utilisé. Finalement une méthode analytique de chromatographie UPLC a été mise au point afin d’identifier et de quantifier les acides aminés présents dans une séquence peptidique.
Les résultats obtenus avec les analyses par UPLC ont permis à la fois de retrouver la séquence du peptide modèle synthétisé mais aussi de déterminer la quantité d’acide aminé contenue dans la poudre isolée après purification. La méthode a de plus été vérifiée sur d'autres peptides test.
Qualitative and quantitative amino acid analysis :
assay development and method implementation
Dip lômante Megane Schweickhardt
Trava il de dip lôme | é d i t i o n 2 0 1 3 | Filière Technologies du vivant Domaine d’application Chimie Analytique Professeur responsable Dr. Marc Mathieu Marc.Mathieu@hevs.ch
Station d’hydrolyse de peptides Séparation du standard d’acide amine obtenue par chromatographie UPLC et détection UV.
Remerciements
Je tiens à adresser mes remerciements sincères aux personnes qui m’ont offert la possibilité de réaliser ce travail de Bachelor. Je souhaite remercier particulièrement : Le Dr. Marc Mathieu pour sa disponibilité et ses précieux conseils.
Vanessa Gaillard, laborantine au laboratoire peptides, pour ses coups de mains bénéfiques.
Yann Dini, mon cousin, enseignant et responsable du groupe EIL à Monthey, pour sa relecture et ses pistes suggérées.
Mes proches pour leur soutien au quotidien. Plus spécialement ma maman pour ses corrections orthographiques, mon papa pour son aide logistique et Anthony pour sa motivation.
Et enfin tous ceux qui, de près ou de loin, ont contribué au bon déroulement de ce travail.
Abréviations
Le tableau A ci-dessous liste de manière exhaustive les abréviations utilisées dans ce rapport.
Tableau A : Liste exhaustive des abréviations utilisées dans le rapport
Abréviation Nom Structure
AMQ 6-aminoquinoline AQC 6-aminoquinolyl-N-hydroxysuccinimidyl carbamate Boc tert-butoxycarbonyle DCM dichlorométhane DIEA N,N-diisopropyléthylamine DMF N,N-diméthylformamide Fmoc 9-fluorenylméthoxycarbonyle
HPLC chromatographie liquide haute performance -
HTBU O-Benzotriazole-N,N,N’,N’-tetraméthyl-uronium-hexafluoro-phosphate
NMP 1-méthyl-2-pyrrolidinone
PmC 2,2,5,7,8-pentaméthylchroman-6-sulfonyle
SPPS synthèse peptidique en phase solide -
tBu tert-butyle
TFA acide trifluoroacétique
Tis triisopropylsilane
-Acides aminés
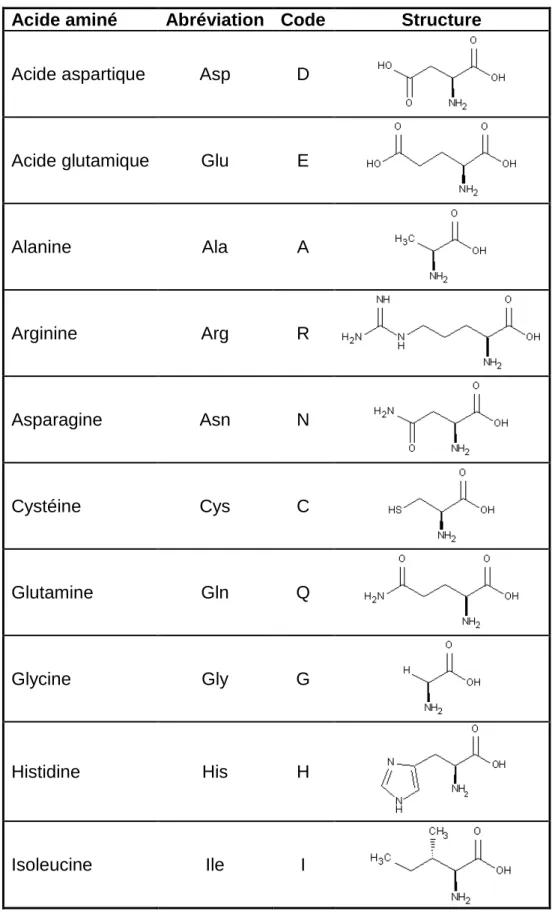
Le tableau B ci-dessous récapitule les acides aminés existants, ainsi que leurs abréviations, leurs codes et leurs structures.
Tableau B : Récapitulatif des acides aminés
Acide aminé Abréviation Code Structure
Acide aspartique Asp D
Acide glutamique Glu E
Alanine Ala A Arginine Arg R Asparagine Asn N Cystéine Cys C Glutamine Gln Q Glycine Gly G Histidine His H Isoleucine Ile I
Leucine Leu L Lysine Lys K Méthionine Met M Phénylalanine Phe F Proline Pro P Sérine Ser S Thréonine Thr T Tryptophane Trp W Tyrosine Tyr Y Valine Val V
Table des matières
1. Introduction ... 2 2. Partie théorique ... 3 2.1 Synthèse peptidique ... 3 2.2 Hydrolyse ... 5 2.3 Dérivatisation ... 62.4 Analyse par chromatographie UPLC ... 8
3. Matériel et méthodes ... 9
3.1 Réactifs, appareillage et matériel ... 9
3.2 Méthodes ... 11
4. Résultats ... 12
4.1 Synthèse peptidique ... 12
4.2 Hydrolyse ... 14
4.3 Analyse par UPLC ... 19
5. Discussion ... 26
5.1 Synthèse peptidique ... 26
5.2 Hydrolyse ... 26
5.3 Analyse par chromatographie UPLC ... 27
6. Conclusion et perspectives ... 28
1. Introduction
Les protéines sont des macromolécules essentielles dans la vie de la cellule. [1] Leur poids moléculaire avoisine les 10'000 daltons pour les plus petites, et plusieurs centaines de milliers pour les plus grosses. [2] Elles peuvent jouer un rôle structurel, un rôle dans la mobilité, un rôle catalytique, un rôle de régulation ou encore un rôle d’expression des gènes. Leur structure est complexe et influe sur la fonction qu’elles jouent dans la vie de la cellule. [1] Les protéines sont des chaines linéaires d’acides aminés liés entre eux par des liaisons peptidiques. Un peptide est une petite protéine composée d’une courte chaine allant de deux à une dizaine d’acides aminés. Lorsque le nombre d’acides aminés composant la chaine linéaire dépasse une dizaine d’acides aminés, on parle de polypeptide. Quant aux protéines, il s’agit de polypeptides fonctionnels ou de combinaisons fonctionnelles de polypeptides. [3] Les protéines du vivant sont composées presque exclusivement de vingt acides aminés. Le carbone alpha des ces derniers est à la fois lié à un groupe amine (–NH2) et à un groupe acide (–COOH). La nature utilise ces deux groupes pour assembler entre eux les acides aminés dans les protéines, en formant des liaisons peptidiques. Les acides aminés comportent également une chaine latérale, qui identifie l’acide aminé et dont la nature va déterminer ses propriétés physico-chimiques. [3]
L’analyse d’acides aminés est un outil nécessaire dans de nombreux domaines de la recherche et également dans diverses activités d’analyse de produits. L’analyse qualitative et quantitative de peptides ou de protéines hydrolysés permet la détermination de leur composition en acides aminés, ainsi que de mesurer directement la concentration de ceux-ci. [4] L’analyse d’acides aminés d’échantillons d’urine ou de plasma fournit également des informations essentielles permettant la détermination de l’état métabolique et nutritionnel d’un patient. Il s’agit donc d’un puissant outil en ce qui concerne le diagnostic des maladies. [5]
Le but de ce travail de diplôme a été de mettre en place et de développer une méthode d’analyse d’acides aminés par chromatographie UPLC et détection UV. Tout d’abord, un peptide modèle a été synthétisé en phase solide. Celui-ci se compose de plusieurs acides aminés présents dans des proportions différentes. Ensuite, une méthode d’hydrolyse a été optimisée, après montage de l’appareillage. Puis, une dérivatisation a été effectuée. Finalement, une méthode analytique de chromatographie UPLC a été mise au point, dans le but d’identifier et de quantifier les acides aminés présents dans des séquences peptidiques modèles.
2. Partie théorique
2.1 Synthèse peptidique
La synthèse peptidique permet la production de peptides. Elle offre un large panel de techniques et de procédures permettant aussi bien la formation de petits peptides que celle de protéines de grande taille. [6] Robert Bruce Merrifield est le pionnier de la synthèse peptidique en phase solide (SPPS). Il a reçu pour son travail le prix Nobel de chimie, en 1984. [6] [7] La SPPS a révolutionné la manière de produire des peptides, en simplifiant les processus pointilleux et fastidieux qui existaient alors. De plus, cette méthode a également rendu possible l’automatisation de la synthèse, grâce à de nouveaux instruments robotisés. La SPPS est donc aujourd’hui une méthode de choix pour produire des peptides. [6]
La figure 1 illustre l’appareillage permettant la synthèse peptidique manuelle. Il se compose d’un réacteur muni d’un robinet et d’une fritte (1), d’une fiole à filtrer (2) ainsi que d’une trompe à eau permettant de mettre l’installation sous vide.
Figure 1 : Appareillage pour la synthèse peptidique manuelle
Lors d’une synthèse peptidique une résine doit tout d’abord être placée dans le réacteur, la structure de celle utilisée dans le cadre de ce travail est illustrée par la figure 2. La résine se compose de petites billes qui contiennent des « linkers » sur lesquels le premier acide aminé va pouvoir se lier de manière covalente, du côté de son groupe carboxylique. Le côté du groupe aminé est quant à lui protégé par un groupe protecteur temporaire (ex. Fmoc). Avant de coupler un second acide aminé, cette protection doit être otée. Cela est réalisé avec des conditions douces, de manière à préserver le reste du peptide. Après déprotection, un second acide aminé peut être fixé, toujours du côté de son groupe carboxylique. Les deux acides aminés sont liés entre eux par une liaison peptidique. La synthèse peptidique se poursuit ensuite en une série de cycles répétitifs de déprotection – lavage – couplage – lavage, jusqu’à ce que la séquence désirée soit obtenue. [6] [7] [8]
1
Figure 2 : Schéma de la résine utilisée (Rink Amide NovaGelTM)
Certains acides aminés contiennent des groupes fonctionnels sur leur chaine latérale. Ceux-ci doivent être protégés par un groupe protecteur permanent (ex. Boc, pmc, …) faute de quoi, ils pourraient interagir avec des extrémités libres ou d’autres groupes de chaines latérales. Cela aurait pour conséquence de réduire le rendement de la synthèse et également la pureté du peptide synthétisé. [8]
A la fin de la synthèse, le peptide est séparé de la résine grâce à une dernière étape de déprotection. Les groupes protecteurs des chaines latérales sont quant à eux éliminés par une étape de clivage.
La figure 3 représente un schéma global de la synthèse peptidique manuelle.
L’HBTU et le DIEA sont tous deux des catalyseurs qui permettent de transformer le groupe hydroxyle, du côté du groupe carboxylique, en groupe partant. De cette manière, l’attaque de l’azote sur le carbonyle peut avoir lieu.
Il est important de comprendre que les acides aminés sont crochés du côté de l’acide carboxylique. Pour cette raison, la synthèse peptidique doit s’effectuer dans le sens contraire de la notation normale qui prévoit que l’on donne la séquence d’un peptide du N-terminus au C-terminus. Par exemple, si la séquence du peptide est AKR, le premier acide aminé qui doit être fixé sur la résine est l’arginine (R), suivi de la lysine (K) et enfin l’alanine (A). [6] [8]
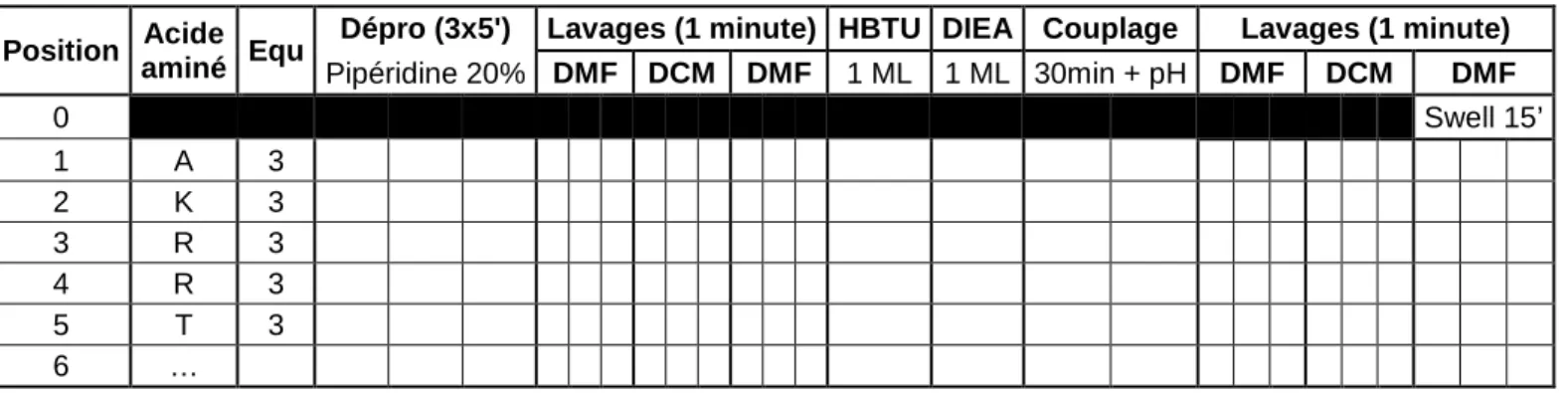
Le tableau 1 permet de suivre l’évolution de la synthèse avec ses nombreux cycles. Il permet également à l’opérateur de savoir où il en est exactement. Pour cela, chaque fois qu’une étape est réalisée, une coche est faite dans la case correspondante.
Tableau 1 : Tableau permettant le suivi de la synthèse peptidique
Position Acide aminé Equ
Dépro (3x5') Lavages (1 minute) HBTU DIEA Couplage Lavages (1 minute)
Pipéridine 20% DMF DCM DMF 1 ML 1 ML 30min + pH DMF DCM DMF 0 - - - Swell 15’ 1 A 3 2 K 3 3 R 3 4 R 3 5 T 3 6 …
2.2 Hydrolyse
Les acides aminés composant un peptide sont liés entre eux par des liaisons peptidiques. Ces dernières sont des liaisons covalentes qui se forment entre un acide carboxylique et un groupement amine, provoquant la perte d’une molécule d’eau. La formation d’une telle liaison peut aisément être inversée par addition d’eau. Ce processus est appelé hydrolyse. La figure 4 illustre ces deux phénomènes. [9]
L’hydrolyse en phase gazeuse sous argon ou sous vide, l’hydrolyse en phase liquide en conditions acides ou basiques ainsi que l’hydrolyse par irradiation avec des micro-ondes constituent les différents types d’hydrolyse existants. Chaque méthode présente des avantages et des inconvénients. De plus, les conditions de chacune d’elles sont plus ou moins drastiques. La méthode en phase liquide sous conditions acides est la plus répandue. [10]
Dans le cadre de ce travail, une hydrolyse aqueuse avec des conditions acides a été réalisée car c’est celle qui était proposée avec l’appareillage de la maison Eldex® à disposition. L’acide convertit l’asparagine et la glutamine respectivement en acide aspartique et acide glutamique. Plus la durée de l’hydrolyse augmente, et plus la thréonine, la sérine et, dans une moindre mesure la tyrosine, sont peu à peu détruites. La quantification de ces acides aminés est donc difficile ; il est nécessaire de réaliser plusieurs hydrolyses de 24, 48 et 72 h et d’extrapoler au temps zéro. La cystéine et la méthionine sont également partiellement détruites. Quant au tryptophane, il est totalement détruit. En revanche, une hydrolyse en milieu alcalin préserve le tryptophane, qui peut de ce fait être quantifié. En ce qui concerne l’alanine, l’isoleucine et la valine, un temps d’hydrolyse plus long permet d’obtenir des résultats plus précis. En effet, il s’agit d’acides aminés aliphatiques, et leur hydrolyse n’est pas toujours terminée après 24 h. [1]
2.3 Dérivatisation
La dérivatisation est un procédé qui permet de transformer un composé chimique en un produit de même structure chimique, appelé un dérivé. [12] En chromatographie liquide, des dérivés sont quasiment systématiquement préparés. Cela permet d’améliorer la réponse du détecteur à la substance analysée. La dérivatisation peut se faire soit avant la séparation (dérivatisation pré-colonne) soit après (dérivatisation post-colonne). Les deux techniques les plus utilisées sont les dérivatisations pré-colonne externes et post-pré-colonne en continu. [13]
La dérivatisation pré-colonne externe ne nécessite aucune modification du matériel. Elle est moins restrictive du point de vue des conditions de réaction, par rapport à la technique post-colonne. Le désavantage majeur provient de la présence du réactif en excès de sous-produit qui risque d’interférer lors de la séparation. Par ailleurs, il est également possible que le groupe introduit dans les molécules modifie les propriétés chromatographiques de l’échantillon. [13]
La dérivatisation post-colonne en continu s’effectue dans un réacteur placé entre la colonne et le détecteur. Il n’est pas nécessaire que la réaction soit totale, pour autant que la réaction soit reproductible. Néanmoins, il est nécessaire que la réaction soit rapide et qu’un excès de réactif ne donne pas de réponse au détecteur. Le principal avantage vient du fait que le processus de séparation et celui de détection peuvent être optimisés. En revanche, les éluants les mieux adaptés aux séparations chromatographiques constituent rarement le milieu réactionnel idéal pour la dérivatisation, ce qui peut donc poser problème. [13]
Dans le cadre de ce travail, une dérivatisation pré-colonne externe a été réalisée. Il s’agit d’une dérivatisation proposée en kit par Waters®. Le réactif de dérivatisation est le 6-aminoquinolyl-N-hydroxysuccinimidyl carbamate (AQC). Sa structure est illustrée dans la figure 5.
Figure 5 : Structure du 6-aminoquinolyl-N-hydroxysuccinimidyl carbamate
La figure 6 décrit les différentes réactions qui se déroulent lors de la dérivatisation. Les deux premières réactions correspondent à la réaction entre l’AQC et un acide aminé primaire et secondaire. Comme illustré par la troisième réaction, si l’AQC réagit avec de l’eau, il se transforme en 6-aminoquinoline (AMQ). Ce produit ne gène cependant en rien l’analyse ultérieure. L’AMQ généré peut réagir avec l’AQC pour former du bis-aminoquinoline urée. Ce dernier a une solubilité limitée dans l’acétonitrile. C’est pourquoi lors d’un stockage prolongé, il est possible qu’un précipité se forme, si de l’humidité est entrée en contact avec le réactif. Cependant, la faible concentration de l’urée n’a pas d’effet sur l’analyse car son temps de rétention est plus long que n’importe quel dérivé d’acide aminé et a une fluorescence très faible. Si la solution est conservée au frigo ou au congélateur, la durée de sa conservation est augmentée. [14] [15]
Lors de la préparation des échantillons, il y a une étape de reconstitution durant laquelle de l’HCl 20 mmol/l est ajouté. Cela permet de mieux dérivatiser les acides aminés acides, telles l’arginine, l’histidine et la lysine. [15]
La préparation du standard mais aussi celle des échantillons nécessite dix minutes de chauffage. Cette étape permet de transformer la chaine phénolique latérale de la tyrosine en phénol libre. Cela donne un produit labellisé sur un seul groupe amino. [15]
La stabilité des dérivés avec l’AQC est de loin supérieure à celle obtenue avec beaucoup d'autres réactifs d'acides aminés, tels que le phénylisothiocyanate ou l’o-phtalaldéhyde. Avec l’AQC, seule une infime dégradation est observable à température ambiante, en une semaine. [14]
2.4 Analyse par chromatographie UPLC
La chromatographie en phase liquide à haute performance (HPLC) est la méthode de choix pour l’analyse et la purification d’une large gamme de molécules. En particulier, l’HPLC avec ses différents modes d’analyses (chromatographie en phase inverse, d’affinité, échangeuse d’ions, …) est devenue la technique de référence pour la caractérisation des peptides et des protéines. C’est pourquoi, les progrès dans les sciences biologiques et biomédicales, durant ces dix dernières, années ont été rapides. [16]
La chromatographie en phase liquide à ultra haute performance (UPLC) a progressivement été adoptée dans les laboratoires industriels, grâce à sa haute résolution, sa haute vitesse et son économie de solvants. Les colonnes utilisées avec les méthodes UPLC ont une taille de particules d’environ 2 µm. Cela permet de réduire à la fois le temps d'analyse de près de 80% et d'économiser la consommation des éluants d’au moins 80% par rapport à une méthode HPLC, utilisant des colonnes conventionnelles avec une taille de particules d’environ 3.5 µm. Ces économies sont accomplies sans sacrifier les performances de séparation. En outre, la durée des analyses beaucoup plus courte permet de réduire significativement le temps de développement de méthodes UPLC. Les principes du développement de méthodes HPLC peuvent s’appliquer au développement de méthodes UPLC. Par ailleurs, de nombreuses méthodes HPLC existantes et utilisées dans l'industrie peuvent être converties en méthodes UPLC. [17]
Les mélanges de peptides et de protéines sont élués systématiquement par un gradient de concentration croissant de solvant organique. Cela en raison de la large gamme d’hydrophobicité des chaines latérales d’acides aminés. [14] [16]
L’ordre d'élution est fortement dépendant du pH. Lorsque les éluants ont un pH faible, l’élution d’AMQ avant les acides aminés polaires est favorisée. A un pH plus élevé, l’AMQ élue dans la région d'acides aminés polaires. Une élution à un pH faible simplifie donc le développement de séparation, puisque le pic n’interfère pas avec les pics d’acides aminés. [14]
3. Matériel et méthodes
3.1 Réactifs, appareillage et matériel
3.1.1. Réactifs
Le tableau 2 liste les différents réactifs employés durant ce travail de diplôme ainsi que leur provenance et leur sécurité.
Tableau 2: Réactifs employés dans le cadre de ce travail
Réactif Fournisseur N° N° CAS Sécurité
1-méthyl-2-pyrrolidinone Acros Organics 127630250 872-50-4 2-propanol Fluka 34965 67-63-0 AccQ∙Tag Ultra Eluent A
Concentrate Waters
® WAT052885 -
AccQ∙Tag Ultra Eluent B Waters® WAT052890 75-05-8 AccQ·Fluor Borate Buffer Waters® WAT052880 1303-96-4 AccQ·Fluor reagent diluent Waters® WAT052880 75-05-8
AccQ·Fluor reagent powder Waters® WAT052880 148757-94-2 - Acétonitrile Lab Scan
PLG02C11X-066 75-05-8 Acide chlorhydrique 32% Acros Organics 389310025 7647-01-0 Acide chlorhydrique 6 M
(ampoule) Sigma Aldrich 84429 7647-01-0 Acide trifluoroacétique Biosolve 20234135 76-05-1 Amino Acid Hydrolysate
Standard Ampoule Waters
® WAT088122 -
Azote 4.5 Pangas - 7727-37-9 Azote liquide Pangas - 7727-37-9
Dichlorométhane Laboratory
Reagent Grade D/1850/15 75-09-2
Eau miliQ HES-SO - - - Ethanol absolu Lavanchy TEC 11825 64-17-5
Ether CoChimy - 60-29-7
Fmoc-Ala-OH Aapptec® AFA101 35661-39-3 - Fmoc-Arg(PmC)-OH Novabiochem® 04-12-1073 119831-72-0 - Fmoc-Leu-OH Aapptec® AFL101 35661-60-0 - Fmoc-Lys(Boc)-OH Aapptec® AFK105 71989-26-9 - Fmoc-Thr(tBu)-OH Bachem B-1245 71989-35-0 - L-Alanine Sigma Aldrich A7627 56-41-7 - L-Arginine Fluka 11010 74-79-3 - L-Asparagine monohydraté Fluka 11160 579413-8 - L-Glutamine Fluka 49419 56-85-9 - L-Glycine Fluka 50046 56-40-6 - L-Histidine Sigma Aldrich H8000 71-00-1 - L-Isoleucine Orgamol - 73-32-5 - L-Leucine Sigma Aldrich L8000 61-90-5 - L-Lysine Sigma Aldrich L5501 56-87-1 - L-Méthionine - - 63-68-3 - L-Phénylalanine Fluka 78020 63-91-2 - L-Proline Fluka 81710 147-85-3 - L-Serine Fluka 84960 56-45-1 - L-Thréonine Sigma Aldrich T8625 72-19-5 - L-Tryptophane Sigma Aldrich 93659 73-22-3 - L-Tyrosine Fluka 93829 60-18-4
L-Valine Acros Organics 140811000 72-18-4 - N,N-diisopropyléthylamine Acros Organics 115225000 7087-68-5
N,N-diméthylformamide Brenntag 81388-156 68-12-2
N-Acétyl-L-cystéine Acros Organics 160280250 616-91-1 -
O-Benzotriazole-N,N,N’,N’- tetraméthyl-uronium-hexafluoro-phosphate
Phénol Fluka 77610 108-95-2 Pipéridine Acros Organics 147180010 110-89-4
Rink Amide NovagelTM Novabiochem® 855031 - - Triéthylamine Acros Organics 157910010 121-44-8
Triisopropylsilane Acros Organics 214922500 6485-79-6
3.1.2. Appareillage et matériel
Les appareillages ainsi que le matériel employés dans le cadre de ce travail de diplôme sont listés exhaustivement dans les méthodes dans les annexes A à D.
3.2 Méthodes
3.2.1. Synthèse peptidique
Les marches à suivre détaillées pour le bon déroulement de la synthèse peptidique, la déprotection et le clivage, la vérification par UPLC-MS de même que la purification sont décrites dans l’annexe A.
3.2.2. Hydrolyse
La marche à suivre détaillée pour le bon déroulement de l’hydrolyse est décrite dans l’annexe B.
3.2.3. Analyse par chromatographie UPLC
Avant d’être analysé par chromatographie UPLC, les échantillons de peptide doivent tout d’abord être dérivatisés. La marche à suivre détaillée pour le bon déroulement de la dérivatisation est décrite dans l’annexe C.
La préparation des éluants nécessaires pour les analyses par chromatographie UPLC ainsi que celle des standards d’acides aminés est décrite dans l'annexe D. La méthode UPLC utilisée y est également décrite.
4. Résultats
4.1 Synthèse peptidique
Un peptide modèle contenant une sélection d’acides aminés dans des proportions diverses a été synthétisé selon la méthode SPPS. Le choix des acides aminés composants le peptide a été réalisé sur la base du chromatogramme de la figure 7. Pour être sélectionnés, les acides aminés devaient être bien séparés les uns des autres. La séquence choisie est la suivante : AKRRTALRL. Le choix s’est fait arbitrairement, d’autres acides aminés auraient très bien pu être choisis.
Figure 7 : Séparation théorique du standard d'acides aminés selon la méthode fournie par Waters® Une analyse par chromatographie UPLC-MS a ensuite été réalisée afin de certifier que le bon peptide ait été synthétisé. Le principe de la MS est de séparer des molécules chargées (ions) en fonction de leur rapport masse/charge (m/z). Par exemple, si un peptide a une masse théorique de 1’000 g/mol. Un pic avec rapport m/z = 1’000 sera présent s’il est chargé une fois mais, s’il est chargé deux fois, un pic avec un rapport m/z = 500 sera présent. Les deux pics correspondent à la même molécule mais chargée différemment.
Dans le cadre de ce travail, le peptide synthétisé a une masse théorique de 1083.3 g/mol. Comme le montre la figure 8, à 2.24 minutes un pic avec un rapport m/z = 542.6 est présent. Cela signifie que le bon peptide a été synthétisé, et qu’il est chargé deux fois (542.6 · 2 = 1083.3 g/mol).
Figure 8 : Chromatogrammes de l’analyse par UPLC-MS du peptide et spectre de masse à 2.24 minutes.
Le chromatogramme illustre également la présence d’une impureté, à 2.10 minutes. La figure 9 montre que cette impureté a un rapport m/z = 615.3. Si la molécule est aussi chargée deux fois, sa masse est donc de 1228.6 g/mol. Il y a donc 145.3 g/mol en trop pour que la masse de l’impureté corresponde à la masse du peptide synthétisé.
Le peptide synthétisé a ensuite été purifié par UPLC préparative. Grâce à cette méthode, de multiples fractions ont été récoltées et c’est ainsi que le peptide et l’impureté ont pu être séparés. Les chromatogrammes ainsi que les spectres MS des fractions récoltées se trouvent en annexe E. Après purification, les fractions semblables ont été rassemblées. Deux grandes fractions ont été obtenues : celle du peptide et celle de l’impureté. Chacune d’elle a finalement été lyophilisées.
4.2
Hydrolyse
Une procédure d'hydrolyse de base était fournie avec la station d'hydrolyse Eldex®. Cependant, plusieurs paramètres ont été testés et modifiés afin d’optimiser au mieux la méthode.
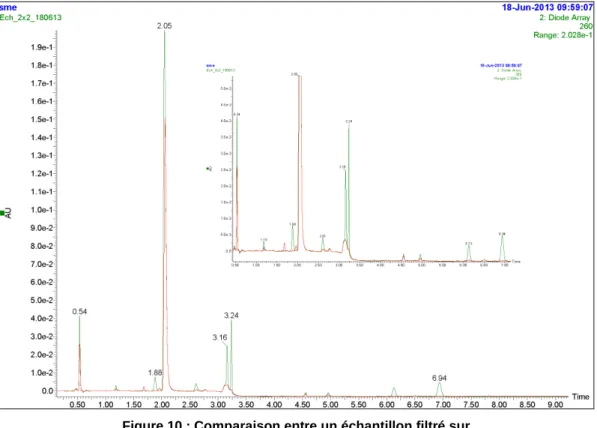
Tout d’abord, la nécessité de filtrer les échantillons de peptide sur 0.45 µm avant hydrolyse a été testée. La figure 10 compare deux échantillons : le premier a été préalablement filtré (vert) tandis que le second a été hydrolysé tel quel (rouge). Mieux vaut filtrer les échantillons. En effet, cela permet d’obtenir une ligne de base plus nette, des pics plus intenses et plus fins ainsi que d’éliminer certaines impuretés.
Figure 10 : Comparaison entre un échantillon filtré sur 0.45 µm (vert) et un échantillon non filtré (rouge)
La procédure fournie par Eldex® recommande de laisser le peptide s’hydrolyser soit 24 h à 105 °C (rouge), soit 1 h à 150 °C (vert). Les deux méthodes ont été testées afin de voir si une des deux étaient préférables à une autre. Les résultats sont illustrés par la figure 11. Ils montrent que l’hydrolyse de 24 h est préférable à celle de 1 h. En effet, la ligne de base est meilleure et les pics des analytes plus intenses et plus fins. En outre, de nombreuses impuretés présentes au début de l’hydrolyse de 1 h sont éliminées dans l’hydrolyse de 24 h.
Figure 11 : Comparaison entre un échantillon hydrolysé 1 h à 150°C (vert) et un échantillon hydrolysé 24 h à 105 °C (rouge)
Comme vu précédemment, l’hydrolyse de 24 h à 105 °C est la meilleure. Cependant, un temps de réaction de 24 h est long et contraignant. C’est pourquoi, plusieurs échantillons hydrolysés 1, 2, 4, 5 et 6 heures ont été prélevés, afin d'observer l’évolution de l’hydrolyse au cours du temps. Les résultats sont illustrés dans les figures 12 (1, 2 et 4 h) et 13 (4, 5 et 6 h). Une variation de la ligne de base est observable entre 3.50 min et 4.50 min, ainsi qu’un changement dans l’intensité des pics.
Figure 12 : Comparaison entre des échantillons hydrolysés à 105 °C durant 1 h (rouge), 2 h (vert) et 4 h (violet)
Figure 13 : Comparaison entre des échantillons hydrolysés à 105 °C durant 4 h (rouge), 5 h (vert) et 6 h (violet)
Les illustrations de la figure 14 ci-dessous représentent de manière agrandie certaines parties du chromatogramme de la figure 13. Elles permettent de mieux distinguer les variations de la ligne de base ainsi que de l’intensité des pics, dues aux différents temps d'hydrolyse.
Figure 14 : Agrandissement de différentes parties du chromatogramme de la figure 9
Une comparaison est également faite entre un échantillon d’une durée d’hydrolyse de 6 (vert) et de 24 h (violet). La figure 15 illustre ces résultats. A nouveau, un changement dans la ligne de base ainsi que dans l’intensité des pics est observable.
Un dernier test a été réalisé concernant l’utilisation d’HCl. La procédure de base préconise l’utilisation d’HCl 6 M en ampoule contenant 1% de phénol (V:V). L’emploi d’HCl concentré serait assurément plus commode et moins onéreux. De plus, le phénol étant un composé extrêmement toxique, mieux vaudrait le remplacer par un réactif moins dangereux voir même, si possible, ne pas l’utiliser du tout. Les résultats obtenus avec ces deux méthodes sont illustrés dans la figure 16. Ceux-ci montrent que de meilleurs résultats sont obtenus avec l’HCl 32% (rouge), plutôt qu’avec l’HCl 6 M contenant 1% de phénol (vert). Dans le premier cas, la ligne de base est plus plate et les pics plus intenses et plus fins.
Figure 16 : Comparaison entre des échantillons hydrolysés 24 h à 105 °C avec de l’HCl 6 M contenant 1% de phénol (vert) et de l’HCl 32% (rouge)
4.3 Analyse par UPLC
4.3.1. Développement de méthode
La compagnie Waters® propose une méthode toute faite pour l'analyse des acides aminés. Le tableau 3 présente le gradient d’élution fourni par la méthode de Waters®.
Tableau 3 : Gradient d'élution de la méthode fournie par Waters®
Temps [min] Eluant A [%] Eluant B [%] Curve 0 99.9 0.1 0.54 99.9 0.1 6 5.74 90.9 9.1 7 7.74 78.8 21.2 6 8.04 40.4 59.6 6 8.64 40.4 59.6 6 8.73 99.9 0.1 6 9.50 99.9 0.1 6
Lors de l'analyse du standard d'acides aminés, le résultat attendu est celui illustré par la figure 17.
Figure 17 : Séparation théorique du standard d'acides
aminés selon la méthode fournie par Waters®
En utilisant le bon appareillage, la même colonne, les mêmes éluants et les mêmes conditions d’analyse, des résultats identiques devraient donc être obtenus. Cependant, les résultats acquis ne correspondent pas, comme le montre la figure 18.
Figure 18 : Séparation pratique du standard d'acides
aminés selon la méthode fournie par Waters®
La méthode a donc dû être modifiée, dans le but d’obtenir une meilleure séparation. Les seuls paramètres qui ont été changés sont le gradient d’éluants ainsi que le temps d’analyse. Après plusieurs essais, le résultat est celui visible sur la figure 19. Le gradient utilisé est décrit dans l’annexe D.
Figure 19 : Séparation pratique du standard d'acides aminés
4.3.2. Identification des pics
Le standard d’acide aminé contient dix-sept acides aminés. Suite aux modifications apportées à la méthode de base, l’ordre d’élution de ceux-ci a peut-être été modifié. Pour cette raison, des solutions de chaque acide aminé présent dans le standard ont été préparées et analysées avec la méthode « AccQ_Tag ». Sur les dix-sept acides aminés présents dans le standard, quinze ont pu être identifiés, comme illustré dans la figure 20. Seules la glutamine et la glycine, même en analysant des standards plus concentrés, n’ont pu être reconnues. Par ailleurs, l’acide glutamique, l’asparagine ainsi que le tryptophane sont tous trois des acides aminés qui ne peuvent pas être analysés selon cette méthode analytique, puisqu’ils ne font pas partie du standard.
Figure 20 : Identification des pics du standard d’acides aminés
4.3.3. Analyses qualitatives de peptides modèles
En premier lieu, le peptide synthétisé, dont la séquence est AKRRTALRL, a été analysé. Grâce au standard d’acides aminé, les cinq acides aminés présents dans la séquence ont été identifiés sur le chromatogramme. Cela est visible dans la figure 21.
Figure 21 : Identification des pics du peptide synthétisé (AKRRTALRL)
L’impureté qui a été isolée lors de l’étape de purification a également été analysée. La séquence de celle-ci est inconnue. La figure 22 montre que quatre acides aminés ont été identifiés au moyen du standard : l’alanine, l’arginine, l’isoleucine et la thréonine.
Un second peptide modèle a été analysé. Sa séquence est LSQEQLEHRERSLQ. Ni l’acide glutamique ni la glutamine ne peuvent être identifiés. En effet, l’acide glutamique ne fait pas partie du standard et la glutamine n’a pas pu être reconnue dans le standard. En ce qui concerne les quatre autres acides aminés, l’histidine et la leucine n’ont pas pu être identifiées. L’arginine et la serine ont quant à elles pu être identifiées. Enfin, la petite pointe vers 3.25 minutes, laisse supposer la présence de thréonine, comme le montre la figure 23.
Figure 23 : Identification des pics du second peptide modèle (LSQEQLEHRERSLQ)
Un troisième peptide modèle a été analysé. Sa séquence est RRRYYFVIHK. Sur les sept acides aminés présents dans le peptide, seuls trois sont identifiés : l’arginine, l’histidine et l’isoleucine. La lysine, la phénylalanine, la tyrosine et la valine quant à elles restent introuvables. Bien que l’alanine et la thréonine ne fassent pas partie de la séquence du peptide modèle, elles se retrouvent sur le chromatogramme, comme l’illustre la figure 24.
Figure 24 : Identification des pics du troisième peptide modèle (RRRYYFVIHK)
Le pic très intense de 2.06 minutes, présent sur chaque chromatogramme, correspond à l’AMQ. Quant au pic de 0.54 minute, il s’agit probablement du pic de solvant (l’éthanol). Une analyse d’un blanc d’éthanol permettrait de confirmer cette hypothèse. Sur les chromatogrammes, de petits pics sont visibles, il s’agit vraisemblablement d’impuretés présentes dans les peptides, peut être des résidus de la synthèse peptidique.
4.3.4. Analyses quantitatives de peptides modèles
Chaque acide aminé est présent dans le standard d’acides aminés à une concentration de 2.5 mmol/l, à l’exception de la cystéine, présente à 1.25 mmol/l. A la suite de l’intégration des pics d’acides aminés du standard, il est possible de quantifier les acides aminés présents dans les peptides modèles.
Les calculs peuvent se faire soit avec l’aire des pics soit avec la hauteur, cela n’influence pas le résultat. Les calculs présentés dans le tableau 4 ont été effectués selon l’aire des pics, ils permettent de déterminer la concentration de certains acides aminés dans les peptides modèles analysés. Les calculs sont détaillés à l’annexe F.
Tableau 4 : Dosage des acides aminés dans les peptides modèles Aire Conc [mg/ml] Conc [mmol/l] Proportion Peptide : AKRRTALRL Leucine 1252,06 1,41 10.8 2 Lysine 851,94 0,72 5.0 1 Impureté : ??? Isoleucine 201.69 0.22 1.68 - Peptide : LSQEQLEHRERSLQ Serine 109,91 0,07 0,7 - Peptide : RRRYYFVIHK Histidine 61,84 0,06 0,4 (1) Isoleucine 218,52 0,24 1,8 (5)
Certains pics n’étant pas séparés de manière optimale, il n’a par conséquent pas été possible des les doser. Cela est le cas de l’alanine, l’arginine et la thréonine.
Les proportions entre la leucine et la lysine dans le peptide synthétisé (AKRRTALRL) correspondent. En revanche pour le troisième peptide modèle (RRRYYFVIHK), alors que l’histidine et l’isoleucine sont toutes les deux présentes une fois, le rapport ne concorde pas. Pour ce qui est de l’impureté et du second peptide modèle (LSQEQLEHRERSLQ), aucune relation ne peut être établie puisqu’un seul acide aminé est dosé.
Grâce à la concentration en acide aminé calculée et en connaissant la masse molaire du peptide, il devrait être possible, par extrapolation, de déterminer la concentration en peptide. Cependant, lors de la préparation des échantillons pour l’hydrolyse, seuls quelques milligrammes de peptides ont été pesés. Ces quantités infimes induisent une trop grosse erreur pour permettre de faire ce calcul de la concentration en peptide.
5. Discussion
5.1 Synthèse peptidique
Le protocole appliqué pour la synthèse peptidique est celui utilisé par le laboratoire peptidique. Il a déjà été développé et optimisé. Grâce à cela, aucun problème n’a été rencontré et la synthèse s’est déroulée avec succès. En effet, comme le montre le spectre de masse de la figure 8, la masse attendue est trouvée.
5.2 Hydrolyse
Suite aux résultats obtenus avec les tests effectués sur l’hydrolyse, la méthode de base a pu être améliorée. Tout d’abord, il est nécessaire de filtrer les échantillons avant de les hydrolyser : cela permet d’éliminer des impuretés indésirables. Ensuite, laisser l’hydrolyse s’effectuer durant 24 h à 105 °C (plutôt qu’une heure à 150 °C) permet d’obtenir de meilleurs résultats. [11] Enfin, l’utilisation d’HCl 32% est plus efficace que celle d’HCl 6 M contenant 1% de phénol. En effet, il s’agit d’HCl plus concentré ce qui permet une hydrolyse plus efficace. Cependant, comme vu dans la partie théorique au point 2.2, certains acides aminés supportent mal les conditions de l’hydrolyse et sont partiellement détruits (cystéine, méthionine, sérine, thréonine, tryptophane et tyrosine). Le fait d’utiliser de l’HCl encore plus concentré peut donc contribuer à accentuer cette dégradation. En revanche, pour les acides aminés qui nécessiteraient un temps d’hydrolyse plus long (alanine, isoleucine et valine), cela améliore certainement leur hydrolysation. Idéalement, le choix de l’utilisation d’un HCl plutôt que d’un autre devrait se faire en fonction des acides aminés présents ou non dans les peptides analysés. Cela permettrait d’obtenir les meilleurs résultats qui soient pour l’hydrolyse. Enfin, si le tryptophane doit être dosé, une hydrolyse alcaline devrait être réalisée puisque celui-ci est complètement détruit lors d’une hydrolyse acide.
Une fois l’hydrolyse terminée, les tubes d’échantillon doivent être lavés. Cependant, cette tâche n’est pas aisée, car les tubes sont très petits et leur embouchure très fine. Par conséquent, il est difficile de nettoyer leur intérieur correctement, surtout si des résidus restent collés contre les parois. Or, pour réaliser des analyses qui soient le plus sensible possible, il est nécessaire d’avoir des tubes d’échantillon qui soient très propres. Le traitement des tubes d’échantillon par un acide ou une base ne fonctionnant pas parfaitement, une pyrolyse de ceux-ci à 500 °C semble donc être la meilleure solution. [15]
Si plusieurs tubes d’échantillon sont hydrolysés en même temps, il est indispensable des les marquer afin de les reconnaître facilement. Certaines méthodes ne fonctionnent absolument pas : le stylo indélébile s’efface, les bouchons des tubes RMN fondent et le scotch se décolle et noircit (impossible de retrouver la couleur d’origine). La meilleure méthode trouvée consiste à enrouler du fil de fer autour des tubes (un fil de fer sur un premier tube, deux sur un second, etc). Une seconde méthode envisageable consiste à graver des numéros dans le verre des tubes d’échantillon au moyen d’un stylo à pointe de diamant.
5.3 Analyse par chromatographie UPLC
Le développement de la méthode chromatographique par UPLC a permis d’obtenir une séparation satisfaisante permettant une identification qualitative des pics d’acides aminés. Sur les dix-sept acides aminés présents dans le standard, seules la glutamine et la glycine n’ont pas pu être identifiées. Sur le chromatogramme du standard, deux pics n’ont pas été attribués. Un de ces derniers correspond certainement à un pic de NH3.
La partie identification pourrait être améliorée en concentrant les échantillons. Cela permettrait d’obtenir un signal des pics d’acides aminés plus intense. De cette manière, certains acides aminés n’ayant pas été identifiés (telles l’histidine et la leucine dans le peptide LSQEQLEHRERSLQ) pourraient manifestement l’être.
La partie quantification est une partie à améliorer. Tout d’abord, lors de la préparation de l’échantillon à hydrolyser, il faut peser une beaucoup plus grande quantité de peptide que ce qui a été fait ici, à savoir, quelques milligrammes. Cela permettrait d’être précis et de pouvoir calculer de manière plus exacte la concentration en peptide. Si nécessaire, préparer d’abord une solution de peptide concentrée puis, la diluer (lors de l’hydrolyse il faut introduire entre 5 et 10 µg de peptide dans un tube d’échantillon). De plus, une amélioration de la séparation de certains pics est à envisager (notamment l’alanine, l’arginine et la thréonine), afin de permettre le dosage d’un plus grand nombre d’acide aminé. Cela permettrait également une meilleure précision de la moyenne arithmétique. L’amélioration de cette séparation pourrait se faire en variant le pH des éluants puisque comme vu dans la partie théorique, au point 2.4, l’élution est fortement dépendante du pH. Un des points essentiels lors de l’analyse d’acides aminés est d’éviter toute contamination. Cela aurait pour effet d’augmenter le bruit de fond et pourrait également créer des pics indésirables qui pourraient être confondus avec des pics d’acides aminés. Pour cette raison, il est nécessaire de travailler le plus proprement possible, d’employer des ustensiles impeccables, de filtrer les échantillons et également d’utiliser des réactifs de grande qualité. [15]
Comme le détecteur UV n’est pas sélectif, le pic AMQ est très large et peut compromettre l'analyse de l’histidine. Le détecteur UV pourrait être remplacé par un détecteur de fluorescence. La limite de détection serait, de ce fait, beaucoup plus élevée. [15] La détection se ferait avec une longueur d’onde à 248 nm pour l’excitation et à 395 nm pour l’émission. [14]
La méthode n’est pas reproductible puisque entre les résultats obtenus dans le cadre de ce travail et ceux qui étaient attendus selon la méthode fournie par Waters®, une énorme différence existe. Faire des analyses sur un second appareillage selon la méthode de Waters® serait intéressant. Si les résultats obtenus sont les mêmes que ceux obtenus avec le premier appareillage, cela signifie peut être qu’une erreur systématique a été commise. En revanche, si les résultats correspondent à ceux attendus, cela peut vouloir dire qu’un problème avec le premier appareillage est survenu. Enfin, si encore d’autres résultats sont obtenus, cela indique que la méthode n’est vraiment pas reproductible.
6. Conclusion et perspectives
Un système d’analyse d’acides aminés par chromatographie UPLC et détection UV a commencé à être mis en place. Néanmoins, des améliorations doivent encore être réalisées pour que l’analyse fonctionne correctement.
Un peptide modèle a été synthétisé avec succès et a servi pour la suite du travail. Le système d’hydrolyse de peptide affecté est opérationnel. Différents tests ont été menés sur l’appareillage et ont permis de rendre l’hydrolyse la plus efficace possible. Suite au développement de la méthode chromatographique, une grande partie des acides aminés a pu être identifiée dans des peptides modèles. La quantification de certains d’entre eux a également été rendue possible. Cependant, l’extrapolation de la concentration d’acide aminé en celle de peptide n’a pas été possible, car la pesée de peptide initiale était trop faible pour permettre un résultat fiable.
Des pistes pour l’amélioration de la méthode sont proposées ci-après.
Tout d’abord, une analyse d’un peptide modèle du début à la fin est à refaire. Lors de la préparation de la solution peptidique initiale, une plus grande quantité de peptide doit impérativement être pesée, idéalement au minimum 50 mg. Cette solution devra ensuite être diluée puisque, la solution à hydrolyser doit contenir entre 5 et 10 µg de peptide, faute de quoi, le temps d’hydrolyse sera fortement augmenté.
Ensuite, une mise au point de la méthode UPLC permettrait une meilleure séparation de certains acides aminés et ainsi, un dosage de ces derniers (notamment la zone de l’alanine, l’arginine et la thréonine).
De plus, des solutions d’acide glutamique et de glutamine plus concentrées que celles déjà analysées pourraient éventuellement être préparées pour être vraiment certain qu’aucune identification de ces deux acides aminés ne soit possible.
En outre, comme le montre les chromatogrammes des échantillons analysés, les pics d’acides aminés sont de faibles intensités. C’est peut être pour cette raison que certains acides aminés ne sont pas visibles. Lors de la préparation des échantillons pour l’analyse sur UPLC, des échantillons plus concentrés devraient être préparés. Lors de la dérivatisation, il faudrait, par exemple, diviser par deux la quantité des réactifs ajoutés aux échantillons hydrolysés.
D’autre part, une fois que la méthode est suffisamment développée et que des résultats satisfaisants sont obtenus, des tests statistiques pourraient être réalisés dans le but de voir si la méthode est précise, exacte, robuste et linéaire.
Par ailleurs, l’emploi d’un standard interne, une substance dont on ajoute la même quantité à tous les échantillons, aux blancs et aux standards, permettrait de compenser certains types d’erreurs aléatoires et systématiques. Pour cela, le standard interne doit être adroitement choisi, afin que les signaux de l’analyte et du standard soient tous les deux proportionnels aux fluctuations de la méthode. Ainsi, le rapport des réponses sera indépendant de ces variations. La principale difficulté dans l’utilisation d’un standard interne provient de la nécessité d’introduire ce dernier de manière reproductible aussi bien dans les échantillons que dans le standard. Il doit produire un signal qui est à la fois semblable à celui de l’analyte mais cependant assez différent pour être différencié par l’appareil. Il faut également qu’il soit absent de la matrice de l’échantillon, dans le but que sa seule et unique source soit la quantité ajoutée. Dans le cadre de ce travail, le standard interne idéal serait un acide aminé non présent dans la séquence du peptide à analyser. Par exemple, avec le peptide AKRRTALRL, la phénylalanine pourrait servir comme standard interne. [18] Enfin, les éluants utilisé dans le cadre de ce travail, sont des éluants déjà tout prêt provenant de chez Waters®. Le problème est qu’ils sont onéreux. Il serait donc intéressant et avantageux de les fabriquer soi-même. En se fiant à la fiche de sécurité des éluants, il est possible de connaitre leur composition. L’éluant A se compose d’acétate de sodium trihydraté, d’acide phosphorique et de triéthylamine. L’éluant B se compose d’acétonitrile et d’eau. Une adaptation serait cependant nécessaire car les proportions exactes ne sont pas connues. Une publication propose également d’autres éluants : 10% d’acétonitrile, 6% d’acide formique et de 84% de formate d’ammonium dans de l’eau pour l’éluant A et 100% d’acétonitrile pour l’éluant B. [19]
7. Bibliographie
[1] Les protéines, http://fr.wikipedia.org/wiki/Prot%C3%A9ine (05.08.13)
[2] Nicolay L., Les principaux nutriments, Script sur la nutrition humaine, HES-SO / Valais-Wallis, Sion (2012)
[3] Segura J.M., Les protéines, Script sur la chimie des produits naturels, HES-SO / Valais-Wallis, Sion (2011)
[4] Herat T., Grumbach E., Mazzeo J., UPLC Amino Acids Analysis Solution – Application Note, Waters Corporation, Milford
[5] Sherma J., Amino Acid Analyzers – A field Guide to Instrumentation, Journal of AOAC International, Vol. 86, N° 6 (2003)
[6] Howl J., Peptide Synthesis and Applications, Methods in Molecular Biology (Vol. 298), HumanA Press, New Jersey (2005) 3-24
[7] Synthèse de Merrifield, http://de.wikipedia.org/wiki/Merrifield-Synthese (10.06.13) [8] Synthèse peptidique, http://en.wikipedia.org/wiki/Peptide_synthesis (10.06.13) [9] Petsko G.A., Ringe D., Structure et fonction des protéines, De Boeck, Bruxelles (2008, 1st ed.) 8
[10] Smith B.J., Protein Sequencing Protocols, Methods in Molecular Biology (Vol. 211), Humana Press, New Jersey (2003, 2nd ed.) 110-122
[11] Walker J.M., Basic Protein and Peptide Protocols, Methods in Molecular Biology (Vol. 32), Humana Press, New Jersey (1994, 2nd ed.) 262
[12] Dérivatisation, http://en.wikipedia.org/wiki/Derivatization (30.07.13)
[13] Mendham J., Denney R.C., Barnes J.D., Thomas M.J.K, Analyse chimique quantitative de Vogel, De Boeck, Bruxelles (2005, 1st ed.) 303 / 310-311
[14] Molnár-Perl I., Quantitation Of Amino Acids And Amines by Chromatography – Methods and Protocols, Journal of Chromatography Library (Vol. 70), Elsevier, Amsterdam (2005, 1st ed.) 242-267
[15] Cooper C., Packer N., Williams K., Amino Acid Analysis Protocols, Methods in Molecular Biology (Vol. 159), Humana Press, New Jersey (2001) 39-47
[16] Aguilar M.I., HPLC of Peptides and Proteins – Methods and Protocols, Methods in Molecular Biology (Vol. 251), Humana Press, New Jersey (2004) 3-5
[17] Quanyun A.X., Ultra-High Performane Liquid Chromatography and its Applications, Wiley, New Jersey (2013) 1
[18] Skoog D.A., Holler F.J., Nieman T.A., Principes d’Analyses Instrumentale, De Boeck, Bruxelles (2003, 1st ed.) 18
[19] Salazar C., Armenta J., Shulaev V., An UPLC-ESI-MS/MS Assay Using 6-aminoquinolyl-N-Hydroxysuccinimidyl Carbamate Derivatization for Targeted Amino Acid Analysis : Application to Screening of Arabidopsis, Metabolites,2, 398-428 (2012)
Table des annexes
Annexe A : Marche à suivre pour la synthèse peptidique ... 2
A.1 Synthèse peptidique ... 2 A.2 Déprotection et clivage ... 4 A.3 Vérification par UPLC-MS ... 5 A.4 Purification par UPLC préparative ... 7
Annexe B : Marche à suivre pour l’hydrolyse ... 8
Annexe C : Marche à suivre pour la dérivatisation ... 13
Annexe D : Marche à suivre pour les analyses par chromatographie UPLC . 15
Annexe E : Chromatogramme de la purification par UPLC-MS ... 18
Annexe A : Marche à suivre pour la synthèse peptidique
A.1 Synthèse peptidique Réactifs DCM DIEA DMF HBTU NMP Pipéridine Appareillage et matériel Agitateur plat, Stuart®, SSM5 Bain à ultrasons, Elmasonic, S30H
Balance analytique, Sartorius, TE64-0CE Centrifugeuse, Eppendorf Centrifuge, 5810 Chronomètre
Embouts de micropipette bleus de 200 – 1000 µl, Ratiolab®, 2100610 Embouts de micropipette jaunes de 1 – 200 µl, Axygen®, T-200-Y Fiole à filtrer
Micropipettes, Pipetman® Neo, P20, P200 N et P1’000 N Papier pH-Fix 0-14, Macherey-Nagel, 921-10
Réacteur avec frite et robinet Trompe à eau
Tuyaux pour le vide
Seringues de 5 ml, ONCE, 62-5202
Marche à suivre
1) Faire le montage pour la synthèse peptidique manuelle comme illustré sur la figure précédente.
2) Préparer les solutions suivantes :
a. Solution HBTU 0.5 M : 47.4 g d'HBTU / 250 ml de DMF. Attention, il se dissout mal, peser dans un cylindre gradué, ajouter la bonne quantité de DMF, bien dissoudre sous agitation, couvrir avec un papier d’alu.
b. Solution DIEA 2M : 85 ml de DIEA / 165 ml de NMP.
c. Solution pipéridine à 20% (V:V) dans du DMF : 40 ml de pipéridine / 160 ml de DMF.
3) Préparer et imprimer le fichier Excel prévu à cet effet (cf. Annexe A).
4) Peser tous les acides aminés (a.a) à l’avance, les conserver au congélateur. 5) Peser la résine au fond du tube.
6) Ajouter du DMF et laisser la résine gonfler durant 15’ (cela permet d’ouvrir les pores de la résine).
7) Déprotéger avec la pipéridine à 20% (3 x 5 min).
8) Laver avec du DMF (3 x 1 min), du DCM (3 x 1 min) et du DMF (3 x 1 min). 9) Couplage :
a. Solubiliser l’a.a dans 1 ml d’HBTU et 1 ml de DIEA. Si besoin, passer aux ultrasons. Faire cette étape n’importe quand dans la ligne.
b. Ajouter l’a.a dans le tube.
c. Vérifier le pH (celui-ci doit être basique) à l’aide d’un papier pH. d. Laisser coupler durant 30 min (45 minutes pour l'arginine).
10) Laver avec du DMF (3 x 1 min), du DCM (3 x 1 min) et du DMF (3 x 1 min). 11) Reprendre au point 7) jusqu’à ce que la synthèse soit terminée.
Remarques
• A chaque étape de la synthèse, bien brasser avec une petite spatule.
• A la fin de la synthèse, le peptide est analysé par UPLC-MS afin de s’assurer que le bon peptide a été synthétisé. Si synthèse d’un long peptide, faire des contrôles au cours de la synthèse pour s’assurer qu’aucune erreur n’a été commise.
• Ne s’arrêter qu’à la fin d’une ligne.
• Durant une pause de midi, laisser le DMF du dernier lavage.
• A la fin de la journée, enlever le DMF du dernier lavage, fermer le tube et le mettre au congélateur.
• Lorsque la synthèse doit être effectuée sur plusieurs jours, faire 2 – 3 lavages avec du DMF avant de poursuivre la synthèse (la résine a pu s'asséchée pendant la nuit).
• Certains a.a sont plus difficiles à coupler que d’autres (p. ex. l’arginine). Un temps de couplage plus long est donc conseillé (non pas 30 mais 45 min).
• Bien cocher sur les cases de l’annexe A chaque fois qu’une étape est réalisée. Une seule erreur et tout est à refaire !!
A.2 Déprotection et clivage Réactifs Eau miliQ Ether TFA Tis Appareillage et matériel Agitateur plat, Stuart®, SSM5 Bain à ultrasons, Elmasonic, S30H
Balance analytique, Sartorius, TE64-0CE Centrifugeuse, Eppendorf Centrifuge, 5810 Chronomètre
Embouts de micropipette bleus de 200 – 1000 µl, Ratiolab®, 2100610 Embouts de micropipette jaunes de 1 – 200 µl, Axygen®, T-200-Y Fiole à filtrer
Micropipettes, Pipetman® Neo, P20, P200 N et P1’000 N Papier pH-Fix 0-14, Macherey-Nagel, 921-10
Réacteur avec frite et robinet Trompe à eau
Tuyaux pour le vide
Seringues de 5 ml, ONCE, 62-5202
Tubes centrifuge de 15 ml, Corning® CentriStar™, 430791
Marche à suivre
Les étapes décrites ci-après s’effectuent lorsque la synthèse peptidique est terminée. Attention, s’il y a des a.a soufrés la procédure est un peu différente !!
1) Déprotection
Enlever le groupe Fmoc sur le dernier a.a.
• Déprotéger avec la pipéridine à 20% (3 x 5 min). • Laver avec du DMF (3 x 1 min)
• Laver avec du DCM (3 x 1 min). 2) Cliver
Enlever la résine et les groupes protecteurs sur les côtés (Boc, pmc, tBu). • Préparer un mélange TFA / Tis / H2O miliQ (95% : 2.5% : 2.5%).
• Transférer le peptide dans un tube falcon de 15 ml.
• Ajouter 1 ml du mélange TFA / Tis / H2O miliQ (si besoin ajouter un peu plus de mélange).
3) Filtrer
• Mettre un petit filtre au fond d’un tube d’une seringue de 5 ml.
• Placer le tube de la seringue (sans le poussoir) au dessus d’un tube falcon (15 ml).
• Transvaser la solution (peptide clivé) dans le tube de la seringue.
• Appliquer de la pression dans le tube de la seringue avec une petite poire prévue à cet effet. Attention à ne pas relâcher la pression tant que la poire est sur le tube sinon tout remonte ! Faire également attention à ce que le tube de la seringue n’aille pas dans le tube falcon sinon l’écriture du tube part et va dans le filtrat (à cause des vapeurs du TFA) !
• Rincer le tube falcon avec 300 µl de TFA (utiliser un embout de micropipette avec un filtre !), transvaser dans le tube de la seringue, appliquer à nouveau de la pression.
• Pipeter 300 µl de TFA (utiliser un embout de micropipette avec un filtre !) directement dans le tube de la seringue, appliquer la pression.
• Appliquer cette fois de la pression avec le poussoir de la seringue pour être certain de récupérer tout le peptide.
4) Concentrer
• Placer le tube falcon sous courant d’azote.
• Laisser buller durant ~ 10 minutes, jusqu’à ce qu’il ne reste plus qu’environ 0.5 ml.
5) Précipiter
• Compléter la solution concentrée à 10 ml avec de l’éther glacé. • Laisser au congélateur pendant 30 minutes.
• Centrifuger 4 minutes à 4'000 rpm.
• Enlever le surnageant (aux déchets organiques). Taper le tube pour que le précipité se décolle du fond du tube.
• Répéter les quatre dernières étapes une deuxième fois.
• Laisser l’éther s’évaporer sous chapelle. Une poudre est obtenue. A.3 Vérification par UPLC-MS
Réactifs Acétonitrile Eau miliQ TFA
Appareillage et matériel
Bouchons pour vials, VWR, 548-0373
Colonne : ACQUITY UPLC® BEH C18, 1.7 µm, 1.0x50 mm, Part No. 186002344
Insert de 0.1 ml, BGB®, 110502 UPLC-MS :
• Module 2487 Dual λ Absorbance Detector • Module Binary Solvent Manager
• Module Sample Manager
• Module TUV Detector
• Micromass® Quattro microTM API
Tubes centrifuge de 15 ml, Corning® CentriStar™, 430791 Vials de 1.1 ml, VWR, 548-0081
Marche à suivre
Quelques grains de peptide sont dissouts dans de l’H2O contenant 0.1% de TFA. Si ça ne se dissout pas, ajouter un minimum d’ACN contenant 0.1% de TFA.
La solution est transférée dans un vial et analysée par UPLC-MS par la méthode suivante :
Nom de la méthode : Analytical002
Colonne : ACQUITY UPLC® BEH C18, 1.7 µm, 1.0x50
mm
Eluant A : H2O avec 0.1% de TFA
Eluant B : ACN avec 0.1% de TFA
Débit : 0.14 ml/min Gradient : Temps [min] Eluant A [%] Eluant B [%] Curve 0.00 98.0 2.0 8.00 10.0 90.0 6 9.00 0.0 100.0 6 11.00 98.0 2.0 1 Volume d’injection : 1.0 µl
Mode d’injection : Partial Loop Needle Overfill
Solvant de nettoyage faible : 600 µl
Solvant de nettoyage fort : 500 µl
Température des échantillons : 4.0 °C
Température de la colonne : 30 °C
Détection : UV à 214 et 280 nm
Durée de la méthode : 11.00 min
Mode d’ionisation : positive
Range de masses : 100.0 – 1'000.0
A.4 Purification par UPLC préparative
Réactifs Acétonitrile Eau miliQ TFA
Appareillage et matériel
Bouchons pour vials, VWR, 548-0373 Eprouvettes en verre
Insert de 0.1 ml, BGB®, 110502
Lyophilisateur, Aapptec®, Sharp Freeze-110 UPLC/MS :
• Micromass® Quattro microTM API
• Module 2487 Dual λ Absorbance Detector
• Module 2777C Sample Manager
• Module 515 HPLC Pump
Tubes centrifuge de 15 ml, Corning® CentriStar™, 430791 Vials de 1.1 ml, VWR, 548-0081
Marche à suivre
La méthode utilisée pour la purification est la suivante : Débit de 30 ml/min
Gradient de 1 à 35% d’acétonitrile contenant 0.1% de TFA en 25 minutes
Certaines des fractions récoltées on ensuite été analysée par UPLC-MS avec la méthode décrite plus haute. Les résultats se trouvent en annexe 5. Les fractions 19 à 25 sont réunies, elles correspondent à l’impureté. Les fractions 29 à 43 sont rassemblées, elles correspondent au peptide.
Les fractions réunies sont maintenant lyophilisées, de manière à obtenir le peptide sous forme de poudre. L’eau est ainsi éliminée par sublimation. La procédure est la suivante :
Congeler les fractions dans de l’azote liquide durant une dizaine de minutes. Mettre les fractions dans lyophilisateur.
Laisser jusqu’à ce qu’il ne reste que de la poudre (pour un volume d’environ 15 ml compter 1 jour, pour un volume d’environ 30 ml compter 2 à 3 jours).
Annexe B : Marche à suivre pour l’hydrolyse
Réactifs Azote en bouteille Eau miliQ Ethanol HCl 32% NaCl Triéthylamine AppareillageEldex® Hydrolysis/Derivatization Station
Embouts de micropipette jaunes de 1 – 200 µl, Axygen®, T-200-Y Filtres pour seringue de 0.45 µm, ExapureTM, PTFE
Huile résistante aux acides pour la pompe Alcatel HV-0304 Manomètre
Micropipettes, Pipetman® Neo, P20 N, P200
Microtubes centrifuges de 1.7 ml, Axygen®, MCT-175-C Pince en Téflon
Pompe à vide Alcatel HV-0304 Seringues de 2 ml, ONCE, 62-2201
Tube de réaction en verre, Eldex®, P/N 1163
Tubes pour échantillons en verre (6 x 50 mm), Eldex®, P/N 3002
Schéma de l’installation
1) Mise en marche / arrêt de la station 2) Mise en marche / arrêt du four 3) Connections pour le vide et l’azote 4) Vial de réaction
5) Vanne du vide
6) Vanne de l’azote
7) Molette de réglage de la température
8) Réglage de la température
9) Affichage de la température 10) Jauge à vide
Remarque :
Tests préalables
Lors de la première prise en main de la station, plusieurs manipulations sont à effectuer, afin de s’assurer du bon fonctionnement de la station d’hydrolyse. Ces tests sont décrits ci-après.
Contrôle des tuyaux
• Vérifier que les tuyaux pour le vide soient en bon état. Si ce n’est pas le cas, les changer.