INFORMATION TO USERS
This manuscript has been reproduced from the microfilm master. UMI films the text directly from the original or copy submitted. Thus, some thesis and dissertation copies are in typewriter face, while others may be from any type of computer printer.
The quality of this reproduction is dependent upon the quality of the copy submitted. Broken or indistind print, colored or poor quality illustrations and photographs, print bleedthrough, substandard margins, and improper alignment can adversely affect reproduction.
ln the unlikely event that the author did not send UMI a complete manuscript and there are missing pages, these will be noted. Also, if unauthorized copyright material had to be removed, a note will indicate the deletion.
Oversize materials (e.g., maps, drawings, charts) are reproduced by sectioning the original, beginning at the upper left-hand corner and continuing from left to right in equal sections with small overlaps.
ProQuest Information and Leaming
300 North Zeeb Road, Ann Arbor, Ml 48106-1346 USA 800-521-0600
NOTE TO USERS
This reproduction is the best copy available.
Université de Sherbrooke
La réplication des télomères et la réplication conventionnelle: deux mécanismes concertés
Par Isabelle Dionne
Thèse présentée à la Faculté de Médecine En vue de 1 ·obtention du grade de
philosophae doctor (Ph.D.) en microbiologie
l+I
National Libraryof Canada Bibliothèque nationale du Canada
Acquisitions and Acquisitions et
Bibliographie SeNices services bibliographiques
315 Welllnglon SlrNt 315, rue Wellngton 011awa ON K1A ON4 on.a-ON K1A ON4
c.'8da c.wda
The author bas
granted
a
non-exclusive licence allowing the
National
Ltbrary
of Qmada to
reproduce, loan, distnbute or
sen
copies of
this
thesis
in
microform,
paper
or electronic formats.
The author retains ownersbip of the
copyright in
this
thesis. Neither the
thesis nor substantial
extracts
from
it
may
be
printed or otherwise
reproduced without the author's
permission.
0-612-74229-6
L'auteur a accordé
unelicence non
exclusive permettant
à la
Bibliothèque nationale du Canada de
reproduire, prêter, distribuer ou
vendre des
copies
de
cette thèse sous
la forme de microfiche/film, de
reproduction sur papier ou sur format
électronique.
L'auteur conserve
la
propriété du
droit
d'autem
qui protège
cettethèse.
Ni la thèse ni des
extraits
substantiels
de
celle-ci ne doivent être imprimés
ou autrement reproduits sans son
autorisation.
TABLE DES MATIÈRES
Table des matières ... .!
Liste des figures ... .111
Liste des abréviations ... .IV Résumé ... VI Introduction ... 1
La réplication de l'ADN ... l Les ADN polymérases ... 2
Les autres composantes de la machinerie de répl ication ... .5
Les télomères ... 9
L ·ADN télomérique ... 10
La répression télomérique et le télosome ... 15
Les composantes protéiques de la chromatine télomérique ... 17
La réplication des télomères ... 21
Les plasmides linéaires chez S. cerevisiae ... 26
L'objectif du projet ... 27 Article l ... 29 Préambule ... 29 Article 11 ... 36 Préambule ... 36 Article lli ... 44 Préambule ... 44
Nouvelle étape lors de la réplication des télomères ... 57
Couplage de la réplication des télomères avec la réplication conventionnelle ... 60
Coordination de la synthèse du brin C-riche et du brin G-riche ... 63
Conclusion ... 70
Remerciements ... 72
LISTE DES FIGURES
Figure 1. Schématisation de la réplication de l 'ADN ... 2
Figure 2. Modèle de la fourche de réplication de I' ADN ... 8
Figure 3. La structure du t-loop ... 12
Figure 4. Modèle de la formation de l'hétérochromatine télomérique chez S. cerevisiae .. 13
Figure 5. Structure du télomère de S. cerevisiae ... 14
Figure 6. Modèle d'élongation par la télomérase chez Tetrahymena ... 22
Figure 7. Modèle de la réplication des télomères chez la levure S. cerevisiae ... 25
Figure 8. Modèle de la coordination de la réplication du brin G-riche et du brin C-riche des télomères ... 67
Figure 9. Modèle de la réplication des télomères ... 71
LISTE DES ABRÉVIATIONS
ALT
ARN pol IIARS
FEN-1 HML HMR nt pb PCNA PENT pol-a pol-ô pol-E rDNA RF-CRPA
SV40TAS
t-loop TPE.. alternative lengthening of telomeres" ARN polymérase II
.. autonomously replicating sequence" .. flap endonuclease ou 5' -exonuclease 1"
··mating type cassette-left" .. mating type cassette-right"
nucléotides paires de base
.. proliferating cell nuclear antigen" "primer extension/nick translation"
ADN polymérase alpha ADN polymérase delta ADN polymérase epsilon ADN ribosomal
"replication factor C" .. replication protein A" "simian virus 40"
"telomere-associated sequences" "telomere-loop"
RÉSUMÉ
Les télomères sont constitués de répétitions de courtes séquences dont un des brins est riche en guanine (G) et l'autre riche en cytosine (C). Les télomères de
Saccharomyces cerevisiae sont répliqués par la télomérase qui ajoute des répétitions
nouvelles au brin G-riche. Lors de la réplication des télomères de levure, les
extrémités des chromosomes acquièrent de longues extensions simple-brin 3' riche en G. Ces extensions 3' sont générées à la fin de la phase S. Le candidat idéal pour la génération de ces queues G-riche était la télomérase. Toutefois, dans une souche de levure dépourvue de la télomérase, nous avons aussi retrouvé des extensions 3' télomériques durant la phase S. Ces résultats démontrent l'existence d'une activité indépendante de la télomérase pour la génération de ces queues riche en G. Cette activité pourrait être une exonucléase 5' -3' nécessaire pour générer une structure uniforme à tous les télomères puisque suite à la réplication semi-conservative, une extrémité du chromosome va comporter une courte extension 3' et l'autre extrémité, un bout franc. Pour déterminer si cette activité de dégradation du brin C-riche était dépendante du passage de la machinerie de réplication, nous avons utilisé un plasmide
linéaire comportant des télomères à chacune de ses extrémités et qui ne peut se répliquer. Dans un tel cas, les télomères n'acquièrent pas d'extensions simple-brin riche en G contrairement à un plasmide correspondant comportant une origine de réplication. Ceci nous permettait de proposer un lien entre la machinerie de réplication conventionnelle et le maintien des télomères.
Pour confirmer le lien pouvant exister entre la réplication des télomères et la machinerie de réplication conventionnelle, nous avons examiné les télomères d'une souche de levure possédant une mutation dans la sous-unité catalytique de I' ADN polymérase a. En plus de posséder des télomères allongés par rapport à une souche de type sauvage, un tel mutant démontre une augmentation de la quantité de simple-brin télomérique G-riche. Cette augmentation du simple-brin terminal est probablement causée par une synthèse inefficace du brin C-riche par la machinerie du brin retardé puisque ce simple-brin télomérique est généré durant la phase S. L'augmentation de la taille des télomères étant dépendante de la télomérase, nous proposons donc un
couplage de la synthèse du brin G-riche par la télomérase avec la synthèse du brin C-riche par la machinerie de réplication conventionnelle pour permettre une régulation de la taille des télomères.
INTRODUCTION
La réplication de I' ADN
Dans une cellule eucaryote en prolifération, la duplication du matériel génétique survient à toutes les phases S du cycle cellulaire. Ce processus doit se produire une seule fois par cycle cellulaire et doit être complété de façon très précise. En général, la régulation et l'intégration de la réplication au cycle cellulaire surviennent au niveau de l'initiation via l'interaction avec d'autres processus qui contrôlent la progression du cycle cellulaire. La réplication de l'ADN chez les eucaryotes est semi-conservative c'est-à-dire que chaque molécule fille d' ADN bicaténaire contient un brin d'origine parental et un autre nouvellement synthétisé (Revue dans Brush et Kelly 1996). Chaque brin d'ADN parental sert de matrice à la synthèse d'un brin fille complémentaire. Par conséquent, il doit y avoir ouverture du duplex d' ADN par des hélicases pour permettre l'instauration d'une fourche de réplication. Ce sont les ADN polymérases qui synthétisent le brin d'ADN fille dans la direction 5'-3' en utilisant une amorce puisqu'elles sont incapables de faire la synthèse d'ADN
de novo.
Pour les cellules eucaryotes, la synthèse d'ADN est généralement initiée par une courte amorce d'ARN qui fournit un 3 '-OH libre aux ADN polymérases. Étant donné la polarité inversée (5'-3' et 3'-5') des deux brins du duplex d'ADN, un seul brin peut être formé en continu par une synthèse 5'-3', c'est le brin avancé (Figure 1). Le brin retardé est le résultat de la synthèse discontinue d'ADN qui est en sens inverse de la progression de la fourche de réplication. La synthèse discontinue d' ADN va générer de courts fragments d'ADN, chacun débutant par une amorce d' ARN, appelés3' 5'
- : amorce d' ARN
- a : fragment d 'Okazaki 3' 5' Figure 1: Schématisation de la réplication de I' ADN au brin avancé (synthèse continue) et au brin retardé (synthèse discontinue).
biochimiquement à l'aide d'un système de réplication in vitro utilisant l'origine du virus simien
40 (SV40), un virus d'ADN double-brin circulaire, et génétiquement à l'aide de la levure
Saccharomyces cerevisiae. Étant donné que mon
système d'étude est S. cerevisiae, je vais me
concentrer sur les mécanismes et le mode d'action des facteurs impliqués dans cet organisme. Bien que la régulation de l'initiation de la réplication soit très importante, je ne vais toutefois pas en discuter puisqu'on ne retrouve pas d'événements d'initiation aux télomères (Wellinger et al. 1993a). Par contre, la machinerie de réplication atteignant les télomères, je vais donc présenter les différents facteurs retrouvés à la fourche de réplication.
Les ADN polymérases
Les ADN polymérases sont nécessaires pour la réplication de l' ADN mais aussi pour la réparation et la recombinaison de l' ADN. Chez la levure, il existe 5 ADN polymérases nucléaires et une ADN polymérase mitochondriale (Revue dans Hubscher et al. 2000). Il y a trois ADN polymérases nécessaires pour la réplication du génome et qui sont essentielles à la survie des cellules: la polymérase a (pol-a), la polymérase 8 (pol-o) et la polymérase E (pol-E) (Campbell 1993). La pol-a est composée de 4 sous-unités (167-kD, 86-(167-kD, 48-kD et 58-kD) qui sont encodées par les gènes POLJICDCJ 7, POLI 2, PRIJ
et PR/2 respectivement. L'activité catalytique pour la synthèse de l' ADN est accomplie par la sous-unité Pollp (Budd et Campbell 1987). Des mutants thermosensibles de
pol l !cdc 17 ont des télomères allongés, arrêtent à la fin de la phase S ou au début de G2 dû à un arrêt de la synthèse d' ADN, démontrent une augmentation de la recombinaison mitotique entre des gènes répétés et dans les régions terminales des chromosomes et une augmentation de la perte des chromosomes (Carson et Hartwell 1985; Aguilera et Klein 1988; Lucchini et al. 1990; Adams et Holm 1996). Le complexe de la pol-a comprend aussi une activité primase qui est formée d'un hétérodimère constitué des sous-unités Pri l p et Pri2p (Plevani et al. 1985; Plevani et al. 1988). Par contre, Pri l p est suffisante pour l'activité primase in vitro et Pri2p médierait ou stabiliserait la liaison de Pri l p à
Pol 1 p (Longhese et al. 1993; Santocanale et al. 1993 ). Le rôle de Pol l 2p n'est pas très bien connu mais cette protéine serait impliquée au niveau de l'initiation de la réplication de l' ADN (Foiani et al. 1994).
La pol-ô est composée de 3 sous-unités (125-kD, 58-kD et 55-kD) qui sont encodées par les gènes POL3/CDC2, POL31/HYS2 et POL32 respectivement (Burgers 1998; Gerik et al. 1998; Hashimoto et al. 1998). POL3 et POL3 l sont essentiels à la survie cellulaire (Boulet et al. 1989; Sitney et al. 1989; Sugimoto et al. 1995). Pol3p est la sous-unité catalytique tandis que le rôle des deux autres sous-sous-unités est inconnu . La pol-ô contient une activité exonucléolytique 3'-5' qui agit comme fonction d'édition durant la synthèse del' ADN (Morrison et al. 1991; Simon et al. 1991).
Pour sa part, la pol-& contient une sous-unité catalytique de 256-kD encodée par le gène POL2 ainsi que 4 sous-unités de 80-kD, 34-kD (et 31-kD) et 29-kD encodées par les
Ohya et al. 2000). Les gènes POL2 et DP82 sont essentiels à la survie cellulaire et les deux sont nécessaires pour la réplication de l' ADN des chromosomes (Morrison et al. 1990; Araki et al. 1991a; Araki et al. 1992; Budd et Campbell 1993). Une souche de levure
dpb3tl. est viable mais démontre une augmentation du taux de mutation spontanée
suggérant que DP 83 est requis pour la fidélité de la réplication (Araki et al. 1991 b ). DP 84
joue un rôle dans le maintien de la structure du complexe de la pol-e (Ohya et al. 2000). La pol-e, tout comme la pol-ô, a une activité exonucléolytique 3 '-5' (Morrison et Sugino 1994). La sous-unité catalytique de pol-e aurait aussi un rôle au niveau des mécanismes de surveillance de la qualité de l' ADN (checkpoints) qui préviendrait l'entrée en mitose lorsqu'il y a des dommages à l' ADN ou lorsque l' ADN n'est pas répliqué complètement (Navas et al. 1995; Navas et al. 1996).
La pol-ô et la pol-e sont différentes de la pol-a du fait qu'elles ont une activité exonucléase 3'-5' d'autocorrection, elles sont processives et elles n'ont pas d'activité primase. En plus de leur rôle dans la réplication du génome, la pol-ô et la pol-e auraient un rôle à jouer dans la réparation de l'ADN (Revue dans Burgers 1998). Jusqu'à tout récemment, aucun rôle n'avait été suggéré pour la pol-a au niveau de la réparation de l 'ADN chez la levure. Par contre, de récentes découvertes suggèrent que toutes les ADN polymérases réplicatives, a, ô et E, soient nécessaires pour la réparation des cassures
double-brin de l' ADN par recombinaison homologue et ainsi, la pol-a initierait la réplication de l' ADN sur le brin retardé pour produire un événement appelé "hreak induced replication" (BIR) (Holmes et Haber 1999).
Les autres composantes de la machinerie de réplication
En plus des ADN polymérases, il y a d'autres facteurs qui entrent en jeu pour la duplication complète des chromosomes. La protéine de réplication A (RP A; replication arotein A) est un hétérotrimère qui lie l' ADN simple-brin (Brill et Stillman 1989). Chacun des gènes qui encodent les trois sous-unités, RF A 1, RF A2 et RF A3, sont essentiels chez S.
cerevisiae (Brill et Stillman 1991 ). La fonction de RP A est conservée chez tous les
eucaryotes dans différents aspects du métabolisme de l' ADN (Revue dans Wold 1997) incluant la réplication, la réparation et la recombinaison (Longhese et al. 1994; Firmenich et al. 1995; Smith et Rothstein l 995).
Le complexe RF-C (replication factor
g
est composé de 5 sous-unités, encodées par les gènes RFC 1 à RFC5, qui sont conservés chez tous les eucaryotes (Revue dans Hübscher et al. 1996). Chacun des 5 gènes chez S. cerevisiae est essentiel à la survie cellulaire (Howell et al. 1994; Li et Burgers l 994b; Li et Burgers l 994a; Noskov et al. 1994; Cullmann et al. 1995). Le rôle principal de RF-C est de charger le facteur de processivité, PCNA (voir plus bas), sur l' ADN à une jonction amorce-matrice (Tsurimoto et Stillman 1991). RF-C joue aussi un rôle dans la réparation et les mécanismes de surveillance (Revue dans Budd et Campbell 2000a).PCNA (Qroliferating ç_ell nuclear ~tigen) est un facteur de processivité pour la
pol-ô et la pol-e et est encodé par le gène essentiel POL30 (Sauer et Burgers 1990; Burgers 1991; Lee et al. 1991 ). PCNA est un homotrimère qui forme un anneau fermé dont le trou central est assez grand pour contenir un duplex d'ADN (Krishna et al. 1994). PCNA a aussi un rôle dans la réparation del' ADN (Revue dans Budd et Campbell 2000a).
Rad27p est une exo/endonucléase 5' -3' homologue à FEN-1 (flap endonucléase ou 5'-exonucléase 1) chez les mammifères (Harrington et Lieber 1994b; Harrington et Lieber 1994a). Comme son nom l'indique, FEN-1 coupe préférentiellement une structure '"flap" c'est-à-dire un fragment 5' d' ADN ou d' ARN qui n'est pas apparié à la matrice d' ADN (Harrington et Lieber 1994a). La délétion du gène RAD27 n'est pas létale pour les cellules mais démontre plusieurs anomalies au niveau de la stabilité génomique: un taux élevé de mutations spontanées (Tishkoff et al. l 997b ), l'expansion et la contraction des séquences micro- et mini-satellites (Johnson et al. 1995; Freudenreich et al. 1998; Kokoska et al. 1998; Schweitzer et Livingston 1998; Spiro et al. 1999), une altération de la structure des télomères (Parenteau et Wellinger 1999), une augmentation de la recombinaison spontanée (Sommers et al. 1995; Vallen et Cross 1995; Tishkoff et al. l 997b; Freudenreich et al. 1998; Lobachev et al. 2000) et une augmentation des bris de chromosomes (Vallen et Cross 1995; Freudenreich et al. 1998). Les fonctions biologiques de FEN-1 et Rad27p semblent avoir été conservées durant l'évolution puisque l'expression de FENJ humain
dans un mutant de levure rad27â complémente pour la croissance à température restrictive et pour la sensibilité aux agents alkylants (Frank et al. 1998; Greene et al. 1999; Hansen et al. 2000).
Dna2p est une hélicase nucléase essentielle pour la réplication et la réparation de l' ADN (Budd et al. 1995; Fiorentino et Crabtree 1997; Bae et al. 1998; Formosa et Nittis 1999; Budd et Campbell 2000b ). Les activités hélicase et endonucléase de Dna2p sont essentielles pour la viabilité cellulaire (Budd et al. 1995; Budd et al. 2000; Lee et al. 2000). Il a été démontré que Dna2p et Rad27p interagissent physiquement et génétiquement suggérant qu'il y a collaboration entre ces deu.x protéines pour la
maturation des fragments d'Okazaki (Budd et Campbell 1997; Parenteau et Wellinger 1999; Bae et Seo 2000).
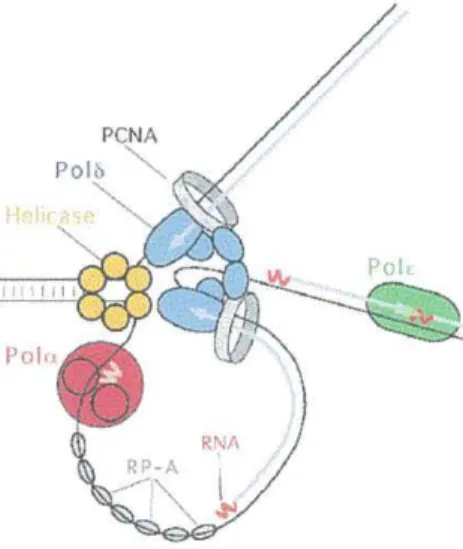
La coordination des différents facteurs retrouvés à la fourche de réplication a surtout été étudiée à l'aide de réactions de réplication reconstituées de l'origine de SV40 (Revue dans Waga et Stillman 1998). L'activité primase intrinsèque de la pol-a est requise pour initier la synthèse d'ADN de novo du brin avancé et du brin retardé. Une courte amorce d' ARN est synthétisée pour ensuite être allongée par l'activité ADN polymérase de la pol-a générant ainsi un hybride ARN-ADN d'environ 40nt de long. Le RF-C, qui est le facteur de chargement (loading clamp), lie l'hybride ARN-ADN pour charger le facteur de processivité, PCNA, ainsi que la pol-ô à la fourche de réplication délogeant par le fait même la pol-a de la matrice d' ADN. Ce changement de polymérase survient probablement à tous les événements d'initiation du brin avancé ou du brin retardé. Les études de la réplication de SV 40 indiquent que ce virus peut se répliquer en absence de la pol-E. Toutefois, il y a plusieurs évidences qui suggèrent un rôle de la pol-E au niveau de la réplication dans les cellules eucaryotes. Le gène qui encode la sous-unité catalytique de la pol-E est essentiel pour la croissance cellulaire chez S. cerevisiae et chez Schizosaccharomyces pombe et des mutants thermosensibles de ces gènes démontrent un rôle de la pol-& au niveau de la réplication (Morrison et al. 1990; Budd et Campbell 1993; D'Urso et Nurse 1997). De plus, des études de "crosslinking" ont montré que la pol-& de mammifère est associée avec de l' ADN en réplication et que la pol-E de S. cerevisiae est localisée à la fourche de réplication ou du moins tout près (Zlotkin et al. 1996; Aparicio et al. 1997). Pour l'instant, on ne connait pas exactement le rôle de chacune des polymérases
le brin retardé puisqu'il y a des résultats divergents (Revue dans Burgers 1998). Un premier modèle propose que la pol-8 et la pol-a répliquerait chacune un brin distinct ceci étant basé sur des résultats de mutants de la pol-8
(pol3exo) et de la pol-a (pol2exo-) comportant des
mutations dans leur domaine exonucléase d' autocorrection (proofreading) (Shcherbakova et Pavlov 1996). Le deuxième modèle (Figure 2) suggère plutôt un rôle de la pol-8 pour
Figure 2: Modèle de la fourche de réplication de !'ADN comprenant un dimère de la pol-ô. La pol-E accomplit la maturation des fragments d'Okazaki. Il y a synthèse simultanée du brin avancé et du brin retardé. Tiré de Burgers 1998.
l'élongation au brin avancé et au brin retardé puisque la pol-8 forme un dimère (Burgers et Gerik 1998) et un rôle de la pol-a au niveau de la maturation des fragments d'Okazaki puisqu'un mutant pol2exo- montre un taux de mutations spontanées moins élevé qu'un
mutantpol3exo- (Morrison et al. 1991; Simon et al. 1991; Morrison et Sugino 1994).
Des protéines additionnelles sont nécessaires pour la maturation des fragments d'Okazaki en un brin d' ADN continu. Il y a trois étapes requises pour la maturation des fragments d'Okazaki: la dégradation des amorces d'ARN, le remplissage des trous laissés par cette dégradation et la ligation de l' ADN. Pour le modèle présentement discuté chez les eucaryotes c'est la RNase HI et FENl qui vont accomplir la première étape (Ishimi et al. 1988; Goulian et al. 1990; Waga et Stillman 1994). La RNase HI a une activité endonucléolytique qui permet de cliver l'ARN qui est attaché en 5' d'un fragment d'ADN comme c'est le cas pour un fragment d'Okazaki. Par contre, la RNase HI est incapable
1994; Turchi et al. 1994; Rumbaugh et al. 1997). C'est FENl, l'endo/exonucléase 5'-3', qui enlèverait le dernier ribonucléotide. Pour les mammifères, la pol-o, le RPA, le PCNA, le RFC, la RNase HI, FENl et l' ADN ligase 1 sont nécessaires et suffisants pour reconstituer la synthèse du brin retardé in vitro (Waga et Stillman 1994). Par contre, chez la levure c'est plutôt un modèle impliquant Dna2p et Rad27p qui a été proposé puisque ces deux protéines interagissent physiquement et génétiquement suggérant une collaboration entre ces deux protéines pour la maturation des fragments d'Okazaki (Budd et Campbell 1997; Parenteau et Wellinger 1999). Dna2p déplacerait la portion ARN du fragment d'Okazaki générant ainsi une structure .. flap" (un duplex d' ADN qui a une partie non-appariée en 5') pouvant être clivée par Rad27p. Chez la levure, il y a des homologues de la RNase HI de mammifère, RNHL et RNH35. Pour l'instant, il y a peu d'évidences in vivo indiquant un rôle de RNHI ou de RNH35 dans la maturation des fragments d'Okazaki. Les études chez SV40 ont montré qu'une fois que les amorces d' ARN ont été dégradées, il y a remplissage des trous laissés par la dégradation par une ADN polymérase et c'est l' ADN ligase I qui est responsable de liguer les fragments d' ADN adjacents pour former une molécule d' ADN continue (Turchi et al. l 994; Waga et al. l 994).
Les télomères
Les télomères ont été définis dans les années 1930 comme étant une composante essentielle qui stabilise les extrémités des chromosomes. Hermann J. Muller a utilisé les rayons X pour produire des réarrangements de chromosomes chez la Drosophile mais il n'a pas réussi à trouver de délétions terminales du chromosome. Il a donc proposé que les extrémités des chromosomes étaient des structures spécialisées qu'il a nommé télomères
(Muller 1938). Les travaux de Barbara McClintock sur le maïs ont pour leur part démontré que les chromosomes endommagés étaient réactifs et fusionnaient les uns aux autres tandis que les extrémités naturelles des chromosomes étaient stables (McClintock 1939; McClintock 1941 ). Aujourd'hui, on sait que les télomères sont des structures nucléoprotéiques essentielles aux extrémités des chromosomes eucaryotes. Les télomères accomplissent plusieurs fonctions dont deux qui sont essentielles pour l'intégrité du génome: la protection des extrémités des chromosomes contre la dégradation et la fusion ainsi que la réplication complète des chromosomes (Revue dans Dandjinou et al. 1999).
L 'ADN télomérique
Les premières séquences télomériques à avoir été identifiées sont celles de ciliés dont font partie Tetrahymena et Oxytricha (Blackburn et Gall 1978; Klobutcher et al. 1981 ). L'isolation et le séquençage de l' ADN télomérique a été facilité par l'abondance des télomères dans le macronoyau de ces protozoaires ciliés. L' ADN télomérique des ciliés est constitué de répétitions de courtes séquences d' ADN. Pour Tetrahymena et
Oxytricha, les séquences télomériques sont C4A2/T2G4 et C4~/T4G4 respectivement.
Aujourd'hui, les séquences télomériques d'une grande variété d'organismes sont connues et malgré que les séquences entre elles soient différentes, il y a certains aspects qui sont conservés. Les répétitions sont toujours riches en résidus guanine (G) et l'orientation des séquences télomériques est conservée: le brin riche en G se dirige toujours de
5'
à 3' vers la fin du chromosome (Tableau l et revue dans Wellinger et Sen 1997). Le nombre de répétitions télomériques varie d'une espèce à l'autre, et même pour chacun des télomèresTableau l: Séquence des répétitions télomériques de différents organismes
Organismes Séquences répétées Références
Protozoaires
Tetrahymena T2G4 Blackburn et Gall 1978
Oxytricha T4G4 Klobutcher et al. 1981
Eup lotes T4G4 Klobutcher et al. 1981
Trypanosoma T2AG3 Blackburn et Challoner 1984;
Champignons Van der Ploeg et al. 1984
Saccharomyces (TG) 1.6TG2_3 Shampay et al. 1984 Schiwsacc/zaromyces T 1-2ACA0.1Co.1G1-6 Sugawara et Szostak 1986
Plantes
Arabidopsis T3AG3 Rk~hards et Ausubel 1988
Venébrés
Homo Sapiens T2AG3 Moyzis et al. 1988
La plupart des autres vertébrés T2AG3 Meyne et al. 1989
La séquence du brin qui va des· à 3 • vers l'extrémité du chromosome est représentée.
d'une même espèce. La longueur des télomères varie de 38 pb chez les ciliés Oxytricha et
Euplotes jusqu'à des dizaines de kilobases pour les cellules de mammifères et chacun des
organismes a une taille de télomères qui se situe autour d'une moyenne. Chez la levure S.
cerevisiae, la séquence télomérique est C2.3A(CA)1-~(TG)1-6TG2.3 (qu'on abbrévie
c,_
3A!fG1.3) et chacun des télomères comporte 300±75pb de ces séquences (Shampay et al. 1984; Wang et Zakian 1990).
La structure physique terminale exacte d'un chromosome est seulement connue pour certains protozoaires et elle consiste en une extension 3' riche en G de 12-16 bases (Klobutcher et al. 1981; Pluta et al. 1982; Henderson et Blackburn 1989). Chez S.
cerevisiae, des extensions de 50-1 OOb riches en G sont présentes à la fin de la phase S
mais les techniques utilisées ne permettent pas d'affirmer que des queues plus courtes soient présentes à toutes les phases du cycle cellulaire (voir plus loin) (Wellinger et al. 1993a; Wellinger et al. 1993b). Pour les cellules humaines, il a été montré que les
cellulaire et même en absence de télomérase (Makarov et al. 1997; McElligott et Wellinger 1997; Wright et al. 1997). On retrouve aussi de telles extensions riches en G aux télomères de poulets, de souris, de plantes et de Trypanosomes (Venkatesan et Price 1998; Hemann et Greider 1999; Riha et al. 2000; Munoz-Jordan et al. 2001). Il y a présentement une controverse quant à la présence de ces queues riches en G des deux côtés d'un chromosome. En utilisant un essai de "primer extension/nick translation" (PENT), Makarov et al. ont observé qu'il y avait des queues G-riches sur plus de 80% des extrémités des chromosomes humains (Makarov et al. 1997). De leur côté, Wright et al. ont utilisé une purification par colonne d'affinité qui leur a permis de démontrer que les chromosomes humains étaient asymétriques, possédant une longue extension d'un côté et une très courte extension (<12 nt) sur le côté
opposé (Wright et al. 1997). Des résultats A. The classical view semblables ont été observés chez la plante
puisque les queues G-riches ne sont détectées
que sur la moitié des extrémités
Telomenc DNA
5
!
Duplex DNA0 0 0 0
binding protcin.. (eg. R.1plp, TRFI) 5' \ 363
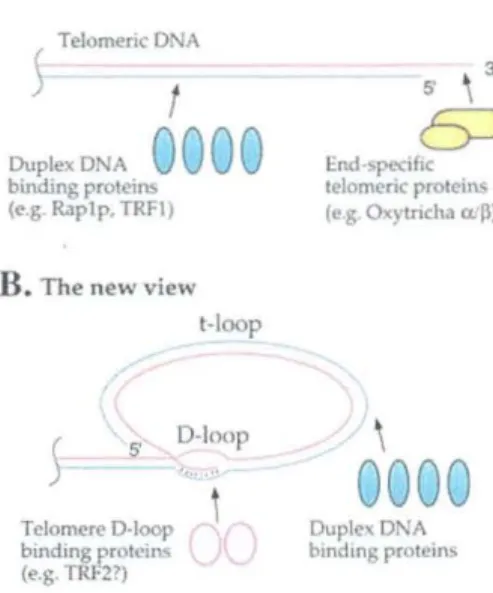
lnd-,pt.><:1f1c telomeric protcins {e S O<ytncha a.!})chromosomiques et cette fois en utilisant l'essai B. The new view PENT (Riha et al. 2000). Malgré cette
divergence de résultats, on savait qu'il y avait une queue G-riche sur au moins une fraction des télomères mais on ne savait pas si les télomères adoptaient une structure particulière. Récemment, il a été démontré par microscopie électronique que, in vivo, l' ADN télomérique
l·loop ) s
~ '~"'
Tclomcrc D·loopoc
bind1ng prot~in' (e.g TRF2?)\
0000
Duplex DNA bmdmg prote1rl.'Figure 3: La structure du t-loop. La nouvelle vision de la structure du télomère
comparativement à l'ancienne.
L' ADN télomérique se replie sur lui-même et forme une structure en lasso. L'extension 3' G-riche s'introduit dans la portion double-brin pour former un D-loop (displacement loop). Tiré de Greider 1999.
de mammifères forme une grande boucle que l'on nomme t-loop (telomere-loop) (Griffith et al. 1999). Cette structure est formée par le repliement du télomère sur lui-même qui crée une structure en lasso et par l'invasion de l'extrémité simple-brin terminale dans la portion télomérique double-brin (Figure 3). Malgré que l'on ne connaisse pas le mécanisme par lequel les t-loops sont formés, on sait que TRF2, une protéine qui lie le double brin télomérique, aurait un rôle à jouer dans la formation ou la stabilisation de ces structures (Griffith et al. 1999). Les t-loops ont aussi été observés chez Oxytricha fallax et
Trypanosoma brucei (Murti et Prescott 1999; Munoz-Jordan et al. 2001). Par conséquent,
les t-loops semblent être une structure conservée par les eucaryotes au cours de l'évolution. Par contre, on ne sait pas si les t-loops sont présents chez S. cerevisiae malgré
qu'il a déjà été proposé que les télomères se repliaient sur eux-mêmes selon différentes études (Figure 4; Grunstein 1997; de Bruin et al. 2000; de Bruin et al. 2001). Les limites de la technique ne permettent pas l'observation directe des t-loops, mais différentes observations ne supportent pas la formation de telles structures chez S. cerevisiae. De
longues extensions simple-brin semblent importantes pour la formation des t-loops et chez la levure, on ne retrouve de telles structures qu'à la fin de la phase S (Wellinger et al. 1993b). De plus, il ne semble pas y avoir d'homologue de TRF2 chez la levure, la protéine qui est nécessaire pour la formation des t-loops chez les mammifères (Li et al. 2000).
Chez la plupart des organismes, on retrouve une région juxtaposée aux télomères qu'on nomme
r
SIR3•' ., "•'If If
.... -- If . :,-:.il.
,.-RAP1
SIR2 SIR4
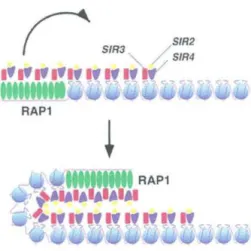
Figure 4: Modèle de la formation de l'hétérochromatine télomérique chez S.
cerevisiae. Rapl se lie aux répétitions
télomériques C1_3A. Les cercles sont des nucléosomes. Tiré de Grunstein 1998
X
Y'(o-4) 5.2 ou 6.7 kb Xhol-300bp
. (TG1.3)f3' -5' TRF: 1.3kb~Figure 5: Structure du télomère de S. cerevisiae. Chacune des extrémités des chromosomes comporte 300 ± 75 pb de répétitions C1•3A/TG1•3• Les télomères comportent aussi des éléments sous-télomériques, X et Y'. Voir la description dans le texte. La figure n'est pas à l'échelle.
TAS (telomere-associated sequences) et qui est composée d'éléments répétés (Henderson 1995). Ces séquences sont très dynamiques et varient beaucoup pour une même espèce et pour les espèces entre elles. Les T ASs qui sont le mieux caractérisées sont celles de S.
cerevisiae (Louis 1995). Il y a l'élément Y' que l'on retrouve sur 75% des télomères (0-4
copies par télomère) et qui est un élément conservé de 6.7 kb (Y' long) ou 5.2 kb (Y' court) (Figure 5; Chan et Tye 1983a; Chan et Tye 1983b; Walmsley et al. 1984; Louis et Haber 1992). À chacun des télomères, il y a un élément X qui a une taille hétérogène variant de 0.3 kb à 3.75 kb et qui se retrouve du côté centromérique de l'élément Y' (ou du télomère s'il n'y a pas d'élément Y') (Figure 5; Louis 1995). La seule région qui est partagée par tous les télomères est un petit élément de 473 pb qu'on appelle le "noyau X" et qui contient une origine de réplication et un site de liaison pour Abfl p sur 31 télomères des 32 (Louis et al. 1994; Pryde et al. 1995). Le rôle des TASs reste encore à élucider. Puisqu'on ne retrouve pas d'éléments Y' à tous les télomères, ces séquences ne sont certes pas essentielles pour la stabilité du chromosome. Les séquences Y' pourraient avoir un rôle structural pour le maintien des télomères par un mécanisme dépendant de la recombinaison ou bien comme zone tampon pour protéger contre la répression
transcriptionnelle due aux télomères (voir section suivante) (Louis et Haber 1992; Lundblad et Blackburn 1993; Louis et al. 1994 ). L'élément X est retrouvé à tous les télomères ce qui laisse croire qu'il y aurait une fonction associée à cet élément sous-télomérique mais qui reste encore non-identifiée.
La répression télomérique et le télosome
La répression transcriptionnelle chez S. cerevisiae est causée par une structure
spécialisée de la chromatine qui réprime l'expression des gènes. Une telle répression transcriptionnelle est retrouvée aux loci HML et HMR de ''mating-type", au locus de rDNA et aux télomères (Gottschling et al. 1990; Loo et Rine 1995; Bryk et al. 1997; Fritze et al. 1997; Smith et Boeke 1997). Ce phénomène a aussi été observé chez la Drosophile,
chez S. pombe et chez Trypanosoma (Levis et al. 1985; Nimmo et al. 1994; Horn et Cross
1995; Rudenko et al. 1995). Chez la levure, la répression transcriptionnelle télomérique ou TPE (!elomere 12osition ~ffect) a été observée lorsque des gènes transcrits par l' ARN Pol II
ont été insérés de façon artificielle près d'un télomère (Gottschling et al. 1990). L'état de répression observé est épigénétique puisque la transcription peut être réprimée dans certaines cellules et active dans d'autres cellules et ce phénomène est réversible. L'efficacité de la répression transcriptionnelle est inversement proportionnelle à la distance du gène rapporteur par rapport au télomère (Gottschling et al. 1990; Renauld et al. 1993). Le TPE est observé lorsque le promoteur du gène rapporteur est situé de 1-3 kb des répétitions télomériques C1_3A/TG1_3 et à des distances plus grandes que 3.5 kb, le TPE est
aboli (Renauld et al. 1993). Le TPE se produit dans toutes les souches de type sauvage, à tous les télomères et pour tous les gènes testés (Gottschling et al. 1990). Le TPE est aussi
influencé par les régions sous-télomériques qui peuvent être activatrices ou inhibitrices selon le contexte (Fourel et al. 1999; Pryde et Louis 1999). On retrouve deux exemples de TPE dans un contexte naturel: le rétrotransposon TyS qui est à 1.8 kb du télomère du chromosome III-L est réprimé transcriptionnellement ainsi que l'ORF YFR057W dont la fonction est inconnue qui se trouve au chromosome VI-R (Palas et al. 1997; Vega-Palas et al. 2000).
Les patrons de digestion à la nucléase micrococcale et à la DNase 1 ont permis d'affirmer que les séquences télomériques C1.3A/TG1.3 aux extrémités des chromosomes sont organisées dans une structure non-nucléosomale que l'on nomme télosome (Wright et al. 1992). Les télosomes peuvent être distingués des nucléosomes à différents points de vue. Les télosomes contiennent plus d' ADN que les nucléosomes (Wright et al. 1992). Les interactions ADN-protéine du télosome sont stables à des concentrations de bromure d'éthidium qui, normalement, défont les nucléosomes mais elles sont moins stables que les interactions histone-ADN dans des solutions salines (Wright et Zakian 1995). Contrairement aux répétitions C1.3A/TG1.3, les éléments sous-télomériques X et Y' ainsi que les gènes placés artificiellement près d'un télomère sont assemblés en nucléosomes (Wright et al. 1992). Il y a tout de même des différences entre les nucléosomes sous-télomériques et les nucléosomes qui sont ailleurs dans le génome. Les sites de reconnaissance pour la dam méthylase dans le gène URA3 sont beaucoup moins accessibles lorsque URA3 est près d'un télomère que lorsqu'il est à sa position naturelle sur le chromosome V (Gottschling 1992). De plus, les histones se trouvant dans les régions sous-télomériques sont hypoacétylées par rapport aux histones des régions euchromatiques (Braunstein et al. 1993). Cet état d'hypoacétylation des histones est une caractéristique
importante pour la répression transcriptionnelle et pour la formation de l'hétérochromatine aux télomères ou aux loci HML et HMR (Revue dans Shore 1995).
Les composantes protéiques de la chromatine télomérique
Une des fonctions des télomères est d'empêcher la dégradation et aussi les fusions entre les extrémités des chromosomes. Pour accomplir cette fonction, les télomères doivent être reconnus comme étant des structures distinctes c'est-à-dire ne pas être reconnus comme des cassures double-brin de l' ADN et ainsi être fusionnés. On croit que les facteurs qui se lient aux télomères auraient un rôle à jouer dans cette fonction de protection. Malgré que les télomères soient des structures qui ont été conservées au cours de l'évolution, les protéines retrouvées aux télomères de S. cerevisiae ne sont pas nécessairement homologues à celles retrouvées chez les mammifères par exemple. Les différentes protéines qui se lient aux télomères chez S. cerevisiae méritent donc une attention particulière.
Raplp
RAPl est un gène essentiel qui encode une protéine de 827 aa requise pour la répression et l'activation de plusieurs gènes (Revue dans Shore 1994; Shore 1997). C'est le domaine de liaison à l' ADN de Raplp qui est essentiel pour la survie cellulaire (Graham et al. 1999). Rap 1 p lie
r
ADN double-brin qui correspond à la séquence consensus suivante: (A/G)(A/C)ACCCANNCA(C/T)(C/T) (Buchman et al. 1988). Les séquences télomériques de la levure peuvent être liées par Raplp à toutes les 18 pb in vitro (Gilson et al. 1993; Strahl-Bolsinger et al. 1997). Il a aussi été démontré que Rap 1 p lie l' ADNtélomérique in vivo (Conrad et al. 1990; Wright et al. 1992; Strahl-Bolsinger et al. 1997). Rap 1 p semble impliquée dans un mécanisme de décompte de protéines (protein-counting mechanism) qui régulerait la taille des télomères (Marcand et al. 1997). Il y a différents mutants de rapl qui affectent soit la taille des télomères, soit la répression transcriptionnelle aux télomères (Lustig et al. 1990; Sussel et Shore 1991; Kyrion et al. 1992; Kyrion et al. 1993; Liu et al. 1994). La surexpression de Raplp produit une augmentation de la taille des télomères, une croissance lente ainsi qu'un haut taux de perte de chromosomes (Conrad et al. 1990).
Riflp et Rif2p
Rifl p et Rif2p ont été identifiées comme étant des protéines qui peuvent interagir avec le domaine C-terminal de Raplp par double-hybride (Hardy et al. 1992; Wotton et Shore 1997). RIF 1 et RIF2 sont des gènes non-essentiels qui encodent des protéines de 1916 aa et 395 aa respectivement (Hardy et al. 1992; Wotton et Shore 1997). Riflp et Rif2p interagissent une avec l'autre (Wotton et Shore 1997). Un mutant rifl~ ou rifl~ a
des télomères qui sont plus longs d'environ 100-300 ph comparativement à des cellules de type sauvage, ils ont un niveau de TPE augmenté ainsi qu'une augmentation de la fréquence de perte de chromosomes (Hardy et al. 1992; Kyrion et al. 1993; Wotton et Shore 1997). Dans un double mutant rifl ~ rifl~, chacun des phénotypes est augmenté
suggérant que les mutations agissent en synergie (Wotton et Shore 1997).
Sirl, Sir3, Sir4
Les gènes SIRI, SIR2, SIR3 et SIR4 C§.ilent information regulator) ont été identifiés dans un criblage qui recherchait des mutants incapables de maintenir l'état de répression aux loci HML et HMR (Rine et Herskowitz 1987). La répression transcriptionnelle aux
télomères nécessite seulement les protéines Sir2p, Sir3p et Sir4p (Aparicio et al. 1991), tandis qu'au locus de rDNA, c'est seulement Sir2p qui est nécessaire (Bryk et al. 1997; Fritze et al. 1997; Smith et Boeke 1997). SIR2 encode une protéine de 562 aa qui est conservée des bactéries jusqu'à l'homme (Brachmann et al. 1995). Sir2p est une histone déacétylase qui est responsable de la déacétylation des histones H3 et H4 (lmai et al. 2000). S/R3 et S/R4 encodent des protéines de 973 et 1358 aa respectivement. Des mutants sir3~ et sir-1~ ont des télomères plus courts que des cellules de type sauvage d'environ 50 pb et l 00-150 pb respectivement.
Ku70 et Ku80
YKU70 et YKU80 encodent des protéines de 70-kD et 80-kD respectivement
formant un complexe qui est impliqué dans la réparation non-homologue des cassures double-brin de l' ADN (Featherstone et Jackson 1999). Le complexe Ku a la capacité de lier des extrémités d' ADN et joue un rôle au niveau du maintien des télomères. En effet, l'absence de l'une ou l'autre des sous-unités du complexe Ku résulte en la diminution de la taille des télomères (Boulton et Jackson 1996; Porter et al. 1996). De plus, pour un mutant
yku70~ ou yku80~, il y a présence de longues extensions riches en G au niveau des
télomères que l'on retrouve tout au long du cycle cellulaire (Grave! et al. 1998; Polotnianka et al. 1998). La répression transcriptionnelle des gènes situés près des télomères est altérée dans des cellules dépourvues du complexe Ku (Boulton et Jackson 1998; Gravel et al. 1998; Laroche et al. 1998; Nugent et al. 1998). Le rôle de Ku serait d'établir une structure normale de la chromatine télomérique en agissant comme facteur pouvant lier les extrémités chromosomiques. Le complexe Ku est aussi important pour la localisation des télomères à la périphérie du noyau (Laroche et al. 1998). Pour des cellules
diploïdes de type sauvage, les 64 télomères sont regroupés dans environ 6 à 8 foci à la périphérie nucléaire (Gotta et al. 1996). Dans un mutant yku70& ou yku80&, plusieurs de ces foci sont perdus et le signal correspondant à l' ADN télomérique est dispersé dans le noyau (Laroche et al. 1998). Le complexe Ku permet probablement l'encrage à la membrane nucléaire par son interaction avec Mlp2p, une composante du pore nucléaire (Galy et al. 2000). Également, lors de dommages à l'ADN, le complexe Ku ainsi que les protéines Sir et Rap 1 p sont relâchés des télomères et sont recrutés aux sites de dommage sous le contrôle des gènes de '"checkpoint" RAD9 et MEC l (Martin et al. 1999; McAinsh et al. 1999; Mills et al. 1999). Ceci laisse supposer que les télomères seraient un entrepôt pour la machinerie de réparation qui peut être mobilisée lors des dommages à l' ADN. Il a aussi été proposé que le complexe Ku aiderait au recrutement ou à l'activation de la télomérase par une association avec TLC l, l' ARN de la télomérase (Peterson et al. 2001 ).
Cdcl3p
CDC I 3 est un gène essentiel qui encode une protéine de 924 aa requise pour la protection des télomères (Garvik et al. 1995). Cdc 13p lie l' ADN simple-brin TG1•3 in vitro et interagit avec les télomères in vivo (Lin et Zakian 1996; Nugent et al. 1996; Boums et al. 1998). En absence de Cdc 13p, il y a une dégradation du brin C-riche au niveau des télomères qui s'étend jusque dans la région sous-télomérique ce qui cause un arrêt du cycle cellulaire en G2/M dépendant de RAD9 (Garvik et al. 1995). Cdc l 3p interagit avec Stnlp et Tenlp par essai double hybride et la surexpression de STNJ et TENI supprime la létalité de l'allèle cdcl 3-1 à la température restrictive (Grandin et al. 1997; Grandin et al. 2001). Les phénotypes associés à des mutants stnl et tenl sont très similaires à un mutant
télomères (Grandin et al. 1997; Grandin et al. 2001 ). En plus de son rôle de protection des télomères, Cdc 13p a aussi un rôle au niveau de la réplication des télomères. Le mutant
cdcl 3-2esr subit une perte de ses séquences télomériques ce qui mène à la sénescence
cellulaire (Nugent et al. 1996) (voir prochaine section). Ce mutant serait incapable de recruter la télomérase aux télomères et ainsi de permettre leur réplication par la télomérase.
La réplication des télomères
La réplication complète des chromosomes linéaires eucaryotes présente un problème qui a été postulé pour la première fois par Watson et par Olovnikov (Watson 1972; Olovnikov 1973). En effet, les ADN polymérases nécessitent une amorce d' ARN pour initier la réplication et synthétisent I' ADN dans la direction 5' -3'. La réplication conventionnelle génère ainsi un trou à l'extrémité 5' du nouveau brin synthétisé dû à l'enlèvement de la dernière amorce d' ARN. Si aucun mécanisme n'est prévu pour contrer cette perte de séquences, les chromosomes vont raccourcir à chacune des divisions cellulaires. Pour la plupart des eucaryotes, le problème de réplication complète des chromosomes est résolu par l'enzyme télomérase (Revue dans Greider 1995). La télomérase est une ribonucléoprotéine qui contient des protéines essentielles et une composante ARN essentielle. La composante ARN comprend -1.5 répétions du brin C qui est utilisée comme matrice pour l'ajout de nucléotides au brin G-riche du télomère faisant de la télomérase une transcriptase inverse (Figure 6). In vitro, le substrat de la télomérase est un ADN simple-brin G-riche sur lequel est ajouté en 3' des répétitions du brin G-riche (Lingner et Cech 1996).
Chez S. cerevisiae, 5 gènes ont été identifiés comme étant essentiels pour le maintien des séquences télomériques in vivo par la voie de la télomérase: TLCJ (telomerase ~omponent 1), ESTJ,
EST2, EST3 (~ver âhorter !elomeres) et CDCJ 3/EST4
(Lundblad et Szostak 1989; Singer et Gottschling 1994; Lendvay et al. 1996). La délétion des gènes
TLCJ, ESTJ, EST2 et EST3 ainsi que l'allèle
cdc13-2est, résulte en une perte progressive des séquences
télomériques qm éventuellement mène à la sénescence après environ 50 à 100 générations. On estime à environ 3 pb par génération la perte des
Élongation
l
Translocationl
{---ccccAACCCCAACCC r---aaGGITGGGGTIGGGGTfGGGGTI
Élongation
l
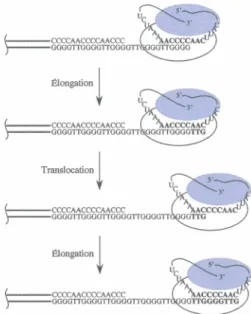
Figure 6: Modèle d'élongation par la télomérase chez Tetrahymena. Premièrement, il y a
alignement de la matrice d' ARN de la télomérase avec I 'ADN du brin G-riche. Par la suite, la télomérase utilise sa matrice pour allonger l'extension 3' du télomère. Finalement, il y a translocation de la télomérase pour un allongement subséquent. Adapté de Greider et Blackburn 1989.
séquences télomériques (Lundblad et Szostak 1989; Singer et Gottschling 1994; Lendvay et al. 1996). Par contre, seules des mutations dans les gènes TLCJ, qui encode la composante ARN, et EST2, qui encode la composante catalytique, abolissent l'activité de la télomérase in vitro (Cohn et Blackburn 1995; Counter et al. 1997; Lingner et al. 1997). Estlp est associé avec l'ARN de la télomérase in vivo et lie de l'ADN simple-brin TG1_3 possédant une extrémité 3' in vitro (Lin et Zakian 1995; Steiner et al. 1996; Virta-Pearlman et al. 1996; Zhou et al. 2000). Étant donné que ESTJ n'est pas nécessaire pour l'activité de la télomérase in vitro, son rôle pourrait être d'amener la télomérase aux télomères en liant l' ADN télomérique. En plus de son rôle de protection des télomères contre les nucléases (voir plus haut), Cdc13p pourrait aussi recruter la télomérase aux télomères via Estlp puisqu'il a été montré qu'ils interagissaient ensemble (Qi et Zakian
2000). Le rôle de Cdc l 3p dans le recrutement de la télomérase est supporté par la démonstration que l'expression d'une protéine de fusion Cdcl3p-Est2p ne requiert plus Estlp pour le maintien des télomères (Evans et Lundblad 1999). Est3p est une sous-unité de la télomérase qui se lie à TLCJ et son interaction avec TLC/ nécessite la présence de
Est2p c'est-à-dire d'un noyau catalytique intact (Hughes et al. 2000).
Chez la levure, il y a aussi des mécanismes indépendants de la télomérase pour le maintien des télomères. Les cellules dépourvues de TLC l ou des gènes EST peuvent se diviser -50-l OO fois avant de mourir (Lundblad et Blackburn 1993; Singer et Gottschling 1994; Lendvay et al. 1996). Par contre, on peut tout de même retrouver des cellules qui vont réussir à échapper à la mort et qui vont croître à la même vitesse que des cellules de type sauvage. On appelle ces cellules des survivantes et elles ont surtout été caractérisées pour la mutation estll! malgré qu'on retrouve aussi ces survivantes dans les souches est2!!,
est3, cdc/Jest et tic/!! (Lundblad et Blackburn 1993; Singer et Gottschling 1994; Lendvay
et al. 1996). La majorité des survivantes ont des blocs télomériques C1_3A!fG1_3 très courts en plus d'une amplification des éléments Y' (survivants de type I) (Lundblad et Blackburn 1993). Les survivantes de type II qui surviennent dans environ 10% des cas ont des télomères longs et très hétérogènes pouvant atteindre 12 kb (Lundblad et Blackburn 1993; Teng et Zakian 1999; Teng et al. 2000). Les survivantes de type I surviennent par un mécanisme dépendant de RAD51 et les survivantes de type II sont dépendantes de RA.D50 tandis que les deux mécanismes sont dus à la recombinaison homologue dépendante de
RAD52 (Lundblad et Blackburn 1993; Le et al. 1999; Teng et al. 2000). Rad5lp est une
protéine d'échange de brin (strand-exchange protein) qui est homologue à RecA de
dans la recombinaison homologue et la réparation non-homologue des cassures double-brin del' ADN et la mutation d'une ou l'autre des sous-unités produit des télomères courts (Kironmai et Muniyappa 1997; Boulton et Jackson 1998).
Certaines lignées cellulaires humaines immortalisées et certaines tumeurs maintiennent leurs télomères en absence de télomérase par un mécanisme alternatif d'allongement des télomères @ltemative !engthening of !elomeres; ALT) (Bryan et al.
l 995; Bryan et al. 1997). Les télomères des lignées cellulaires et des tumeurs ALT sont très longs et hétérogènes comme pour les survivants de type II chez la levure. Le groupe de Reddel a montré que dans une lignée cellulaire humaine ALT, le mécanisme alternatif pour le maintien des télomères était de la recombinaison homologue (Dunham et al. 2000).
Chez la levure, l' ADN télomérique est répliqué tardivement durant la phase S du cycle cellulaire et au même moment va acquérir une longue extension 3' du brin G-riche (McCarroll et Fangman 1988; Reynolds et al. 1989; Ferguson et al. 1991; Wellinger et al. l 993a; Wellinger et al. l 993b). Cette extension G-riche est transitoire puisqu'elle disparaît lorsque les cellules entrent en G2/M. Ces extensions G-riches sont d'au moins 30 nt et on les retrouve à chacune des extrémités d'un plasmide linéaire (Wellinger et al. 1996). La technique de détection utilisée ne permet pas d'affirmer s'il y a de courtes extensions (<30 nt) dans les autres phases du cycle cellulaire. La télomérase est le meilleur candidat étant responsable de ces extensions G-riches mais ce n'est certainement pas le seul. En effet, la réplication bidirectionnelle d'un chromosome de levure génère une molécule d' ADN comportant une extrémité franche et une extrémité avec une courte extension 3' (Lingner et al. 1995). ln vitro, la télomérase ne peut pas utiliser de l' ADN double-brin comme substrat (Lingner et Cech 1996), suggérant que ce n'est pas la télomérase qui est
(?) (?) 5' l' l' 5'
i
Réplication seml·con1erva1he·'
3' 5' 3' + 5·~~•---t~---t~~---+ ~, 3' 3' 5'i
(exonucliue brln-spiclnque?) 5' 3' 3' 5' + 5' 3' 3' 5' 5'i
1dlonpmenl par lélomél'WIC
et rempllssa!elréparalion pur ADN polym rDSe
3'
l' s•
+
5' 3'
l' 5'
Figure 7: Modèle de réplication des télomères chez S. cerevisiae. Suite à la réplication conventionnelle, il y aurait une exonucléase qui dégraderait le brin C-riche à chacune des extrémités du chromosome. Ces extrémités 3' simple-brin peuvent servir de substrat pour la télomérase. Finalement, il y a maturation du long simple-brin G-riche pour générer de courtes extensions 3 '. Chez la levure, on ne connait pas la structure terminale exacte (indiqué par(?)).
responsable de créer le long simple-brin G-riche sur au moins un des bouts du chromosome. De plus, il a été démontré que ces extensions G-riches sont générées même en absence de télomérase (Wellinger et al. 1996). Ceci implique un mécanisme indépendant de la télomérase, probablement une exonucléase qui dégraderait une partie du brin C-riche (Figure 7). Le fait de créer des queues G-riches fournit un substrat pour la télomérase et permet la génération de courtes extension du brin 3' ( <30 nt) qui sont probablement nécessaires pour la liaison des protéines liant spécifiquement les extrémités. En effet, chez Oxytricha et d'autres ciliés reliés, il y a des protéines qui lient spécifiquement l'extension G-riche (Gottschling et Zakian 1986; Price 1990). Ceci
pourrait être une caractéristique universelle de tous les eucaryotes (Henderson et Blackburn 1989; Lingner et al. 1995).
Une fois que la télomérase et/ou l'exonucléase a agi aux télomères, le brin C-riche doit être resynthétisé et le mécanisme le plus plausible pour accomplir cette fonction est la machinerie de réplication conventionnelle (Revue dans Price 1997). Due à la polarité des brins, c'est toujours le brin retardé qui synthétise le brin C-riche. D'ailleurs, certaines évidences chez la levure laissent croire que la machinerie de réplication du brin retardé serait responsable de la synthèse du brin C-riche. Carson et Hartwell ont été les premiers à démontrer que des mutants de la pol-a. avaient des télomères plus longs que les cellules de type sauvage (Carson et Hartwell 1985). De plus, différents allèles de la pol-a. et de RF-C produisent aussi un allongement des télomèrcs et un mutant rad2 7 ô produit une augmentation et une hétérogénéité du simple brin G-riche (Adams et Holm 1996; Parenteau et Wellinger 1999). Durant la synthèse de télomères de novo chez Euplotes, l'ajout d'aphidicoline qui inhibe la pol-a. et la pol-8 mais pas la télomérase, amène un changement dans la longueur du brin C et cause une augmentation de la longueur et de l'hétérogénéité du brin G (Fan et Price 1997). Ces évidences suggèrent une interaction entre la réplication des télomères et la machinerie de réplication conventionnelle.
Les plasmides linéaires chez S.
cerevisiae
Chez la levure S. cerevisiae, on peut introduire des plasmides linéaires qui permettent, entre autre, d'étudier différents aspects moléculaires de la réplication. Pour qu'un plasmide linéaire puisse se propager d'une cellule mère à une cellule fille, il doit
(Stinchcomb et al. 1979; Szostak et Blackburn 1982). L'origine de réplication chez la levure est communément appelée ARS pour "autonomously replicating sequence" puisqu'elles ont originalement été identifiées comme séquences qui permettaient à un plasmide de se répliquer de façon autonome (Revue dans Newton 1996). Les séquences télomériques à chacune des extrémités du plasmide sont essentielles pour que le plasmide ne soit pas dégradé. Chez S. cerevisiae, chacun des blocs télomériques est d'environ 300 pb. Originalement, les plasmides linéaires introduits chez la levure comportaient des séquences télomériques de Tetrahymena ou d'Oxytricha et ces plasmides étaient maintenus grâce à l'ajout d'environ 300 pb de séquences télomériques TG1•3/C1•3A de
levure (Szostak et Blackburn 1982; Pluta et al. 1984; Shampay et al. 1984; Wang et Zakian 1990). Les séquences sous-télomériques X et Y' ne sont pas nécessaires pour le maintien du plasmide dans les cellules (Wellinger et Zakian 1989). On peut aussi ajouter un centromère sur un plasmide linéaire ce qui a pour effet de limiter le nombre de plasmides par cellule (7 à 40) (Murray et Szostak 1983; Wellinger et Zakian 1989; Runge et al. 1991). Un plasmide linéaire est beaucoup moins stable qu'un plasmide circulaire correspondant qui lui est retrouvé en l ou 2 copies par cellule (Clarke et Carbon 1980; Stinchcomb et al. 1982; Dani et Zakian 1983) . L'absence d'un centromère permettra la présence d'un nombre encore plus élevé de plasmides par cellule facilitant ainsi leur détection (Murray et Szostak 1983).
L'objectif du projet
Initialement, les seules informations que nous avions concernant la réplication des télomères étaient l'acquisition par les télomères d'une extension simple-brin riche en G
après que la réplication conventionnelle soit terminée. Malgré ces données nous ne savions pas si la réplication des télomères était couplée avec la machinerie de réplication conventionnelle ou bien si la télomérase agissait indépendamment. Le but de mon travail a été d'éclaircir cet aspect de la biologie des télomères en répondant aux questions suivantes:
1) Est-ce que la génération des extensions 3' G-riches est dépendante de la télomérase? 2) Est-ce que l'activité exonucléase 5'-3' subit une régulation dépendante du cycle
cellulaire?
3) Le passage de la machinerie de réplication est-il nécessaire pour la génération des queues G-riches à la fin de la phase S?
4) Est-ce que des mutants de la machinerie de réplication conventionnelle ont une altération de la structure de leurs télomères?
ARTICLEI
Dionne, 1. et Wellinger, R.J. (1996) Cell cycle-regulated generation of single-stranded G-rich DNA in the absence of telomerase. Proc. Nat/. Acad. Sei. USA 93, 13902-13907.
Préambule
Ce manuscrit rapporte l'existence d'une activité indépendante de la télomérase pour la génération des longues extensions 3' G-riche durant la phase S. Cette activité subit une régulation durant le cycle cellulaire. La présence du simple-brin télomérique riche en Ga été détecté à l'aide d'une nouvelle technique native d'hybridation directement dans le gel. J'ai réalisé toutes les expériences présentées dans cet article.
Proc. Natl . .1lcad. Sei. USA
Vol. 93, pp. 13902-13907, November 1996 Gene tics
Cell cycle-regulated generation of single-stranded G-rich DNA in
the absence of telomerase
(lelomere replication/tclomerase RNA/ycast) ISABELLE DIONNE AND RAYMUND J. WELLINGER*
Facult.! de Mc!decinc:, Dc!purtemc:nt de: Micrubiulugic et lnfcctiulngic, Univc:rsit.! de Sherbrookc. 31Xll 12 cmc Avenue Nord. Sherbrooke. OC J 1 H SN-1, Canada
Communicared by Da1•id M. Prescotr, University of Colorado, 8011/der, CO. Septr:mber 16, 1996 (received for rr:view, May 6, 1996)
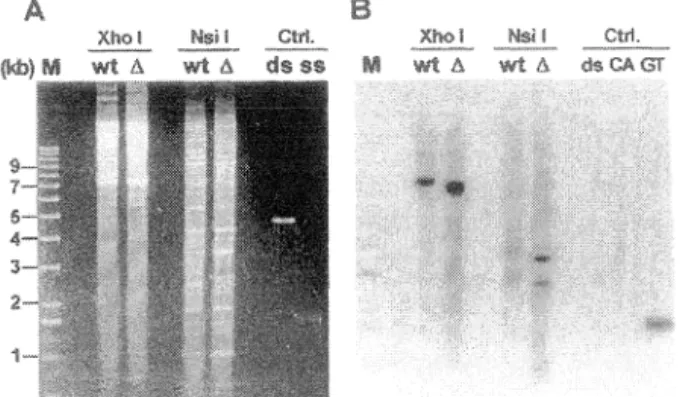
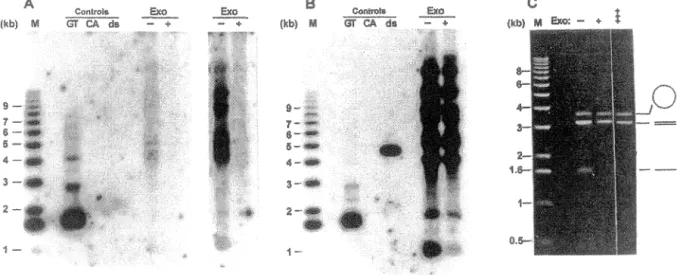
ABSTRACT Current models or telomere replication pre-dict that due to the properties of the polymerases impllcated in semiconservallve replicatlon or linear DNA, the two daugh· ter molecules have one end that is blunt and one end with a short 3' overhang. Telomerase is thought to extend the short 3' overhang to produce long single-stranded overhangs. Re-cently, such overhangs, or TG1_3 tails, were shown to occur on both telomeres or replicated linear plasmlds in yeast. More-over, indirect evidence suggested that the TG1_3 tails also occurred in a yeast strain lacking telomerase. We report herein a novel in-gel hybridization technique to probe telo-meres für single-stranded DNA. Using this method, il is shown directly that in yeast strains lacking the TLCI gene encoding the yeast telomerase RNA, TG1.J single-stranded DNA was generated on chromosomal and plasmid telomeres. The single-stranded DNA only appeared in S phase and was sensitive to digestion with a single-strand-specific exonuclease. These data demonstrate that during replication or telomeres, TGi-J tails can be generated in a way that is independent or telomerase-mediated strand elongation. ln wlld-type strains, these TG,_3 tails could subsequently serve as substrates für telomerase and telomere binding proteins on ail telomeres. Telomeric DNA of most eukaryoticorganisms consists of short tandemly repeated sequences (1, 2). Due to the nature of the repeats, one strand is usually rich in guanines (G-rich strand) and this strand always runs 5' - 3' toward the end of the chromosomes (2). The actual amount of double-stranded DNA made up of these repeats varies considerably between different organisms and between different telomeres in the same organism. For instance, in the yeast Saccharomyces cerevisiae the telomeric rcpeats can be described as C1_3A/ TG1_3 sequences and there are 250-350 bp of these repeats on each telomere (3). The physical structure of the very ends of the chromosomes is only known for some protozoans and there consists in a short 10-16 base overhang of the G-rich strand
( 4-6). Pro teins that bind specifically to this terminal structure have been identified in Oxytriclza and related ciliates (7-9). A similar activity has been detected in extracts of Xenopus eggs (10). Based on these results, it has been proposed that a short overhang of the G-rich strand is a general feature of eukaryotic telomeres (6) and that this structure is bound by terminus-and structure-specific telomere-binding proteins (2, 11).
Telomeres are essential for chromosome integrity in at least two ways: they function as a protection against random fusion events and degradation (12. 13) and they are involved in the completion of the duplication of chromosomal DNA (14). Ali conventional DNA polymerases require a primer and a tem-plate and synthesize DNA in the 5' _. 3' direction. Given these properties, conventional replication is expected to leave short primer-sized gaps on the 5' end of the strands that were generated by lagging-strand synthesis (2, 11). Upon successive The publication costs of this article were defrayed in part page charge
divisions and in the absence of compensating mechanisms, chromosomal termini are cxpectcd to gradually losc terminal sequences until telomere functions arc lost, chromosomes become unstable, and the cclls die (see, for example, refs. 15-17). However, an enzymatic activity called telomerase can elongate the short single-stranded 3' overhangs in a DNA-template-independent manner (ref. 18, and for a review, see ref. 19). The enzyme uses a sequence within its associated RNA as the template for telomeric repear addition (16, 20). Such long, single-stranded, and G-rich tails (hereafter called TG1_3 tails) are present on yeast telomeres in late S phase, when conventional replication has essentially been completed (21, 22). Fill-in synthesis on TGi-3 tails would complete the replication of telomeres, preventing the loss of sequences and reestablishing short 3' overhangs.
The lelomeres on which the newly synthesized strand is made by leading-strand synthesis are thought to becomc blunt-ended after conventional replication is completc (2. 11 ). However, since the terminal structure of the telomeres is expectcd to be uniform and terminus binding proteins may require a short 3' overhang, this blunt end would have to be processed into an end with a short 3' overhang ( 11 ). At lcast in vitro, telomerase requires a single-stranded 3' end for strand elongation ( 11, 23). lt is not clear whethcr in vivo, a blunt-ended double-stranded molecule cou Id serve as a substrate for a telomerase holoenzyme and whether this enzyme could generate TG1_3 tails on ends replicated by leading-strand
synthesis (24 ). As alternatives, this conversion could be achieved by tclomerase-independent activities such as exo-nucleases or helicases (24, 25).
Reccntly, the gene encoding the yeast telomerase RNA TLC I was cloned ( 16). Cc lis lacking this RNA progrcssively lose tclomeric sequcnces from thcir telomeres and die after 80-100 generations ( 16). Introduction of a restriction site by in vitro mutagenesis of the tcmplating rcgion in the TLCI gene resulted in the incorporation of the restriction site into newly synthesized chromosomal telomeres ( 16). Moreover, extracts prepared from c/cl::LEU2 yeast strains lack detectable telom-erase activity (26). These results demonstrate that the TLC/-encoded RNA is an essential component for yeast telomerasc-mediated TG1_3 synthesis in vivo.
The existence of yeast strains lacking telomerase activity provides a unique opportunity to address of the rote of telomerase in the formation of telomere replication interme-diates in vivo. One of the major predictions of the current models of telomere replication is that the long TQ1_3 tails observed at the end of S phase are generated by the yeast telomerase. Using a nove( nondenaturing in-gel hybridization technique, we report hcrein that TG1_3 single-stranded DNA was detected on plasmid and chromosomal telomeres of cells that lacked the TLCJ gene. This single-stranded DNA was sensitive to digestion with a single-strand-specific exonuclease and, therefore, constituted terminal TG1_3 tails. As reported