Structure et diversité de la communauté de protistes de
la lagune de Havre-aux-Maisons, Canada
Thèse
Bérangère Péquin
Doctorat interuniversitaire en Océanographie
Philosophiae Doctor (Ph.D.)
Québec, Canada
Résumé
La lagune de Havre-aux-Maisons aux Îles-de-la-Madeleine dans le Golfe du Saint-Laurent est un système semi-fermé, avec un temps de renouvellement de l’eau jusqu’à 45 jours. Une activité mytilicole modérée a lieu dans la lagune; les moules (Mytilus edulis L.) sont principalement suspendues sous la surface de l’eau. Les moules dépendent de la matière en suspension pour leur nourriture, incluant les micro-organismes eucaryotes planctoniques (protistes et phytoplancton). La répartition spatiale et temporelle des espèces microbiennes marines eucaryotes est déterminée par les conditions physiques, biologiques et chimiques. L’objectif principal de cette thèse était de caractériser la communauté microbienne eucaryote par des méthodes moléculaires (séquençage à haut-débit), à court et moyen terme. Les échantillons ont été prélevés à partir d’un site unique de Juin à Octobre 2009, et pour deux semaines en Août 2010 dans des zones avec ou sans ferme de moules. Les acides gras de la lagune et ceux assimilés et rejetés par M.edulis ont aussi été examinés en 2009 pour déterminer le lien entre profile lipidique planctonique et celui des moules. Dans l’ensemble la vitesse moyenne de vent avait un effet sur la composition en espèces planctoniques en 2009, il y avait une alternance entre les dinoflagellés et les petites cellules photosynthétiques. Ce changement de communauté a peu d’effet sur la nutrition des moules, qui ont ajusté leurs profils d’acides gras indépendamment de la source de nourriture disponible dans l’échelle de temps de l’étude. En 2010, l’échantillonnage aux deux jours pendant deux semaines, la communauté planctonique était similaire dans et hors la ferme de moules, mais différait à chaque échantillonnage. Au niveau de l’espèce, peu d’entre elles semblaient impacter par la présence des moules, ce qui suggére une forte homogénéisation, verticale et horizontale, de l’eau de la lagune. En date, il s’agit de l’étude la plus exhaustive caractérisant la communauté microbienne eucaryotes concernant sa diversité et sa strucuture dans une lagune des Îles de la Madeleine, où la communauté est sensible aux événements météorologiques extérieurs.
Abstract
The Havre-aux-Maisons lagoon in the Magdalen Islands in Gulf of St-Lawrence is a semi-enclosed system with water residence time of up to 45 days. Limited myticulture is carried out in the lagune; mostly suspended mussels farming, (Mytilus edulis L.). The mussels depend on suspended material, including planktonic microbial eukaryotes (protists and phytoplankton) for food. The spatial and temporal distribution of marine eukaryotic microbial species is thought to be determined by physical, chemical and biological conditions. The main goal of this thesis was to characterize the microbial eukaryotic community uising molecular methods (high throughput sequencing) over medium and short terms. The samples were collected from a single site from June to October 2009 and for two weeks in August 2010 in areas away from and within a mussel farm. Fatty acids from the lagoon plankton and those assimilated and rejected by M. edulis were also examined in 2009 to determine any links between planktonic and mussel lipid profiles. Overall average wind field had the strongest effect on planktonic species composition and in 2009 there was an alternation between dinoflagellates and small photosynthetic cells. The community had little effect on the nutrition of mussels, which adjusted their fatty acid profiles independent of the source of food available over the time scale investigated. In 2010, when sampling every 2 days for two weeks, the plankton community was similar in both farm and off farm sites, but differed on each sampling occasion. At the level of species, very few species were impacted by the presence of mussels suggesting strong horizontal as well as vertical mixing in the lagoon. This is the most exhaustive study to date on the diversity and community structure of microbial eukaryotics in a Magdalen Island lagoon, where we found that the community was most sensitive to external weather events.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ...vii
Liste des tableaux...xi
Liste des figures...xiii
Liste des abréviations et des sigles ...xv
Remerciements ...xix
Avant-‐propos ...xxi
Chapitre 1 : Introduction générale... 1
1.1 Les lagunes côtières ...1
1.2 Les protistes...3
1.2.1 Diversité des protistes ... 3
1.2.2 Structure de la communauté de protistes... 5
1.2.3 Méthodologie employée ... 5
1.3 Mytilus edulis (L.) ...8
1.3.1 Description de l’espèce... 8
1.3.2 Les acides gras ... 9
1.3.3 Méthodologie employée ... 9
1.4 Site d’étude...10
1.5 Structure de la thèse ...10
Chapitre 2 : Wind drives microbial eukaryote communities in a temperate closed lagoon
... 13
Résumé...13
Abstract...15
2.1 Introduction...17
2.2 Materials and Methods ...18
2.2.1 Sample collection... 18
2.2.2 Sample processing ... 19
2.2.3 DNA amplification and sequencing... 20
2.2.4 Bioinformatics analysis ... 20
2.2.5 Environmental data, statistical and model analysis ... 21
2.3 Results ...22
2.3.1 Environmental conditions... 22
viii
2.3.3 Community composition...25
2.3.4 Drivers of the community changes over time...29
2.3.5 Potential predator prey interactions. ...31
2.4. Discussion... 32
2.4.1 Picophytoplankton, wind and nutrients ...32
2.4.2 Picophytoplankton species over the season...33
2.4.3 Role of Dinoflagellates ...34
2.4.4 Species succession and potential for Harmful Algal Blooms...35
2.4.5 Conclusions ...36
2.5 Acknowledgments ... 36
Chapitre 3 : Fatty acids in a closed lagoon in the Magadalen Islands, Canada: recent
assimilation and metabolic mobility by cultured mussels. ... 37
Résumé ... 37
Abstract... 39
3.1 Introduction... 41
3.2 Material & Methods ... 42
3.2.1 Sample collection...42
3.2.2 Fatty acids ...44
3.2.3 Statistical analyses ...44
3.3 Results... 46
3.3.1 Mussels and environmental conditions ...46
3.3.2 Flow cytometry ...46
3.3.3 Fatty acids composition ...49
3.3.4 Correlations ...53
3.4 Discussion... 55
3.5 Acknowledgments ... 59
Chapitre 4 : Short term temporal changes in rRNA inferred microbial eukaryotic
communities in a shallow lagoon with mariculture activities... 61
Résumé ... 61
Abstract... 63
4.1 Introduction... 65
4.2 Materiels & Methods... 66
4.2.1 Sample collection...66
4.2.2 RNA collection and extraction ...68
4.2.3 Pyrosequencing...69
4.2.4 Read analysis...69
4.2.5 Statistical and UniFrac analysis ...70
4.3 Results... 70
4.3.1 General environmental setting ...70
4.3.2 Community composition over the 2 weeks ...71
4.5 Acknowledgments...84
Chapitre 5 : Conclusion générale ... 85
5.1 Synthèse ...85
5.2 Commentaires méthodologiques ...87
5.2.1 Apport de méthodes... 87
5.2.2 Biais méthodologiques ... 88
5.3 Perspectives...90
Bibliographie... 91
Liste des tableaux
Table 2.1: Physical and environmental variables in the HAM Lagoon 2009 at depth 2.5m... 23
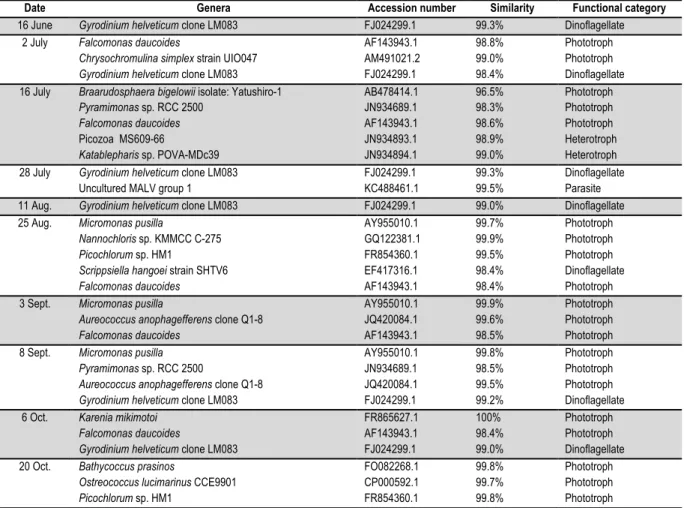
Table 2.2: Most abundant ribotypes (>100 reads) on each date over sampling period... 27
Table 2.3: Spearman rank correlation rho values comparing biological and environmental variables ... 31
Table 3.1: Physical and environmental variables in the HAM Lagoon 2009... 47
Table 3.2: Percentage of different fatty acids recovered by class of lipids SFA, MUFA, PUFA and EFA (20:4n6 + 20:5n3 + 22 :6n3) in three type of samples seston, digestive gland (DG) and feces. ... 49
Table 3.3: Spearman’s rank correlation (p-value < 0.05) between environmental variables and the major lipids classes... 53
Table 3.4: Percentage of selected PUFA that contributed to clustering, for the three types of samples seston, digestive gland (DG) and feces... 54
Table 3.5: Correlation between differents sample types for a subset of selected PUFA fatty acids... 55
Table 4.1: Sites coordinates and sequencing effort for small and large fraction with the number of sequencing samples by site. ... 67
Table 4.2: Abundant genus (> 300 reads by sample) in large and small fraction... 77
Table 4.3: Spatial two-way ANOVA for 11 August fo small fraction... 80
Liste des figures
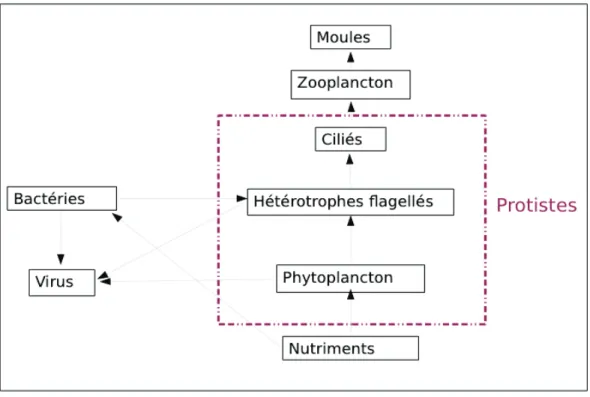
Figure 1.1: Schéma du réseau trophique aquatique. Le sens des flèches indique un transfert d'énergie. ... 2
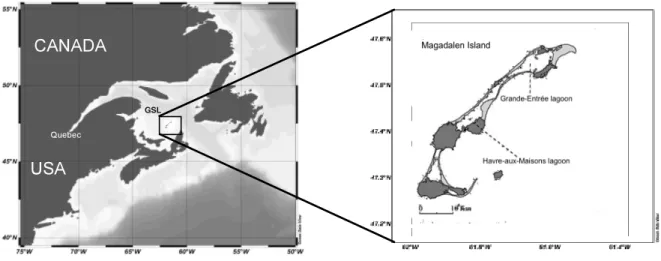
Figure 1.2 : Carte des Îles de la Madeleine. ... 3
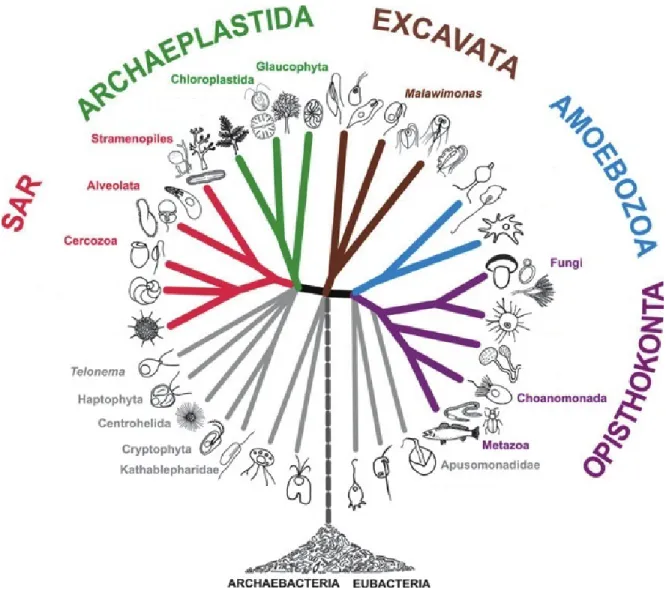
Figure 1.3 : Arbre phylogénétique des eucaryotes modifié de Adl et al. (2012). Les groupes indiqués sont ceux dans lesquels les protistes sont retrouvés... 4
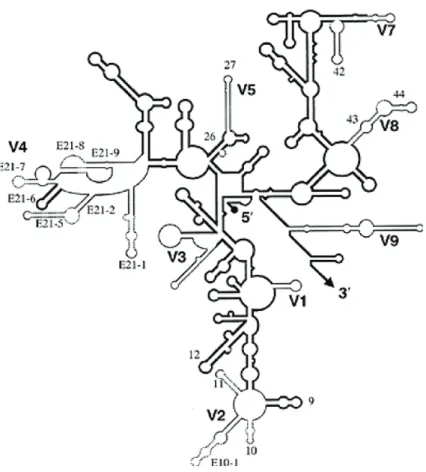
Figure 1.4 : Structure secondaire de la petite sous-unité ribosomique des eukaryotes, 18S, Ueda-Nishimura and Mikata (2000)... 7
Figure 2.1: Map of Magdalen Islands, Gulf of St. Lawrence (GSL). ... 19
Figure 2.2: Meterological data, from the the airport at Havre-aux-Maisons from 1 June to 20 October 2009... 24
Figure 2.3: Finer level taxonomic assignation normalized reads classified as phototrophs (panel A),
heterotrophs (panel B) and dinoflagellates (panel C). ... 26
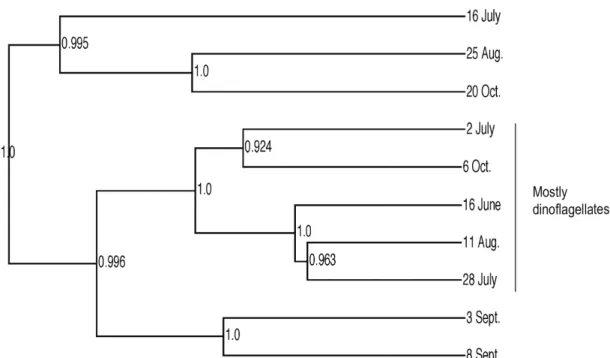
Figure 2.4: Weighted UniFrac analysis of community similarity with jackknife coefficients shown at the nodes. Based on 1000 jackknife replicats on 1700 reads per sample... 29
Figure 2.5: Biplot representing dbRDA with abundant ribotypes and environmental variabes selected by forward selection; Abbreviations as in Table 2.1. ... 30
Figure 3.1: Temporal trends from June to October 2009. The results of one-way ANOVA are given at the top right of each graph. The letters over the points indicate significant differences among the dates based on a Tukey test (A) SPM concentrations (mg l-1), (B) mussel length (mm), (C) mussel weight (g), (D) concentration of total lipids in the digestive glands (dg) (mg g-1) wet mass. ... 48
Figure 3.2 : Distance based redundancy Analysis (dbRDA) based on fatty acids contributed to dissimilarity.. 50
Figure 3.3 : Non-metric multidimensional scaling (nMDS) of fatty acids. The different groups of fatty acids by month were determined by Simper analysis with fatty acids that contributed significantly to dissimilarity in seston (A), digestive gland (B) and feces (C). ... 51
Figure 4.1: Map of sampling sites in Havre-aux-Maisons lagoon ... 67
Figure 4.2: Daily wind and rain over from 9 August and the following 2 weeks of sampling (top panel). Nutrient concentration SRP, Silicate and Nitrate over the same period (bottom panel)... 71
Figure 4.3: Percentage of reads classified by major taxonomic group for small fraction (a) and large fraction (B). ... 73
Figure 4.4 : Unweighted UniFrac community clustering, by date (day and month) site 2 control (2C) or site 2 farm (2F) and small or large fractions... 74
Figure 4.5 : Weighted UniFrac community clustering by date (day and month) site 2 control (2C) or site 2 farm (2F) and small or large fractions. ... 75
Figure 4.6 : Weighted UniFrac for small fraction temporal (day and month) community of the sites 2 farm (F) and 2 control (C). ... 78
Figure 4.7 : Weighted UniFrac for large fraction temporal (day and month) community of the sites 2 farm (F) and 2 control (C). ... 79
Liste des abréviations et des sigles
18S : petite sous-unité ribosomiqueAA : acide arachidonique ou arachidonic acid ADN ou DNA : acide désoxyribonucléique
ADNc ou cDNA : acide désoxyribonucléique complémentaire ARN ou RNA : acide riboxynucléique
ARNr ou rRNA : acide riboxynucléique ribosomique Blast : basic local alignment search tool
Chl a : chlorophylle a
dbRDA : analyse de redondance basée sur la distance ou distance based redondancy analysis DHA : acide docosahexaenoïque ou docosahexaenoic acid
EDTA : acide éthylène-diamine-tétraacétique EFA : acides gras essentiels ou essential fatty acid EPA : acide eicosapentaenoïque ou eicosapentaenoic acid FCM : cytométrie en flux ou flow cytometry
GEL : lagune de la Grande-Entrée
HAB : bloom d'algues toxique ou harmful alga bloom HAM : Havre-aux-Maisons
MID : multiplex identifier
MUFA : acide gras monoinsaturé ou monoinsaturated fatty acid NCBI : national center for biotechnologie information
xvi
nMDS : non-metric multidimensional scaling
OTU : unité taxonomique opérationnelle ou operational taxonomic unit PCR : polymerase chain reaction
PCoA : principal coordinates analysis
POM : matière organique paticulaire ou particulate organic mater PUFA : acide gras polyinsaturé ou polyinsaturated fatty acid SDA : acide stéaridonique ou stearidonic acid
SFA : acide gras saturé ou saturated fatty acid Si : silicate
SPM : matière particulaire en suspension ou suspended particulate mater SRP : soluble reactive phosphorus
À Timothée
À Eliott
Remerciements
Tout d’abord, je tiens à remercier ma directrice de thèse Dr Connie Lovejoy de m’avoir accueillie pour faire mon doctorat dans son laboratoire. Sa passion, sa patience, son perfectionnisme, sa bonne humeur et son soutien sont toujours au rendez-vous que ce soit par beau temps ou mauvais temps. Mon co-directeur Dr Rejean Tremblay avec qui j’ai eu un autre très bon point de vue dans la manière de faire et tout aussi enrichissant. Pour nos échanges toujours très productifs malgré le « nuage noir » qui a longtemps compromis nos rencontres. Également les membres de mon comité le Dr Steve Charette pour son encouragement et ses bonnes idées. Le Dr Ladd E Johnson pour sa remise en question de méthodes utilisées et aux combiens intéressants. Je tiens surtout à vous remercier profondément pour la confiance en moi que j’ai pu développer tout au long de ces années. Il y en a eu du chemin… Au Dr Guillaume Massé qui a rejoint mon comité pour la dernière ligne droite. Merci à Tove Gabrielsen d’avoir accepté d’être mon examinatrice externe.
Tous les gens du projet AquaMeN qui ont permis ce doctorat et surtout mon autre AquaWomen du labo Vani Mohit à qui je dois un spécial grand et gros merci, avec qui j’ai énormément partagé, tous les hauts et les bas que nous avons traversé ensemble, heureusement que tu étais là. Merci à Maurice Gaudet et François Bourque du MAPAQ et de Merinov de m'avoir donné des informations relatives aux moules et à la lagune de Havre-aux-Maisons. Thomas Guyondet avec qui j'ai communiqué brièvement pour les mêmes raisons. En parallèle à cela tous les gens que j’ai côtoyé plus ou moins tous les jours. Tout d’abord les « vieilles du labo » Sophie Charvet, Cindy Dasilva, Emmanuelle Medrinal, Mary Thaler avec qui j’ai pu partager tous nos moments de doute, de stress et de joie reliés à nos doctorats et le côté personnel tout aussi important. Il y a aussi ceux qui sont partis vers d’autres horizons : Marie Lionard et André Comeau avec qui j’ai fait du très bon travaille et aussi pas mal de papotage. Puis il y a les « plus jeunes » du labo avec qui le quotidien est toujours agréable Sophie Crevcoeur, Nathalie Joli, Deo Onda, Jérôme Comte, Adam Monier, Ramon Terrado, Annabelle Baya, Robyn Edgar, Vincent Carrier. Enfin deux formidables personnes Marianne Potvin et Marie-Josée Martineau, uniques et primordiales aux labos. Je n’oublie pas non plus la gang du labo Vincent.
La fin des mes remerciements est plus intime, merci à toutes les merveilleuses personnes qui j’ai croisées sur ma route, parties plus ou moins loin. Merci à ma famille et belle-famille pour leur soutien malgré les kilomètres qui nous séparent. Merci à Clémence ma cop’s avec qui j’ai pu échanger nos différents points de vue sur le doctorat, déverser tout mon trop-plein d’émotion et parler de tout et de rien pour le plus grand bien de notre morale. Merci à Timothée pour son amour inconditionnel et son soutien inépuisable pour traverser cette aventure si importante qu’est un doctorat. Finalement, merci à Eliott notre fils, d’avoir pointé le bout de son nez durant ce doctorat et d’avoir remis nos vies en perspective.
Avant-propos
Cette thèse de doctorat interuniversitaire en Océanographie inclut cinq chapitres, l'introduction générale, trois chapitres de recherches scientifiques et une conclusion générale. Elle a été réalisé dans le cadre du programme AquaMeN (Aquaculture, Moules et Nutriments) financé par le CRSNG (Conseil de Recherche en Sciences Naturelles et Génie du Canada). Ce projet visait à intégrer différentes échelles trophiques et le recyclage des nutriments appliqué à la gestion de l'aquaculture. Tous les échantillons ont été récoltés dans la lagune de Havre-aux-Maisons (HAM) aux Îles de la Madeleine se situant dans le golfe du Saint-Laurent au Canada. J'ai été responsable de la partie sur les microorganismes marins eucaryotes. L'échantillonnage a été effectué pour le chapitre 2 et le chapitre 3 par Vani Mohit, Nicolas Toupoint et Lisandre Gilmore Solomon. L'échantillonnage du chapitre 4 a été planifié et réalisé par moi-même avec l'aide de Vani Mohit. J'ai réalisé toutes les analyses en laboratoire ainsi que celles relevant de la bio-informatique. J'ai rédigé cette thèse en intégralité.
Chapitre 1 - Introduction générale
Chapitre 2 – Péquin B, Mohit V, Poisot T, Tremblay R, Lovejoy C. Wind drives microbial eukaryote
communities in a temperate closed lagoon. Soumis à Estuarine Coastal and Shelf Science
Chapitre 3 – Péquin B, Lovejoy C, Gilmore Solomon L, Myrand B, Tremblay R. Fatty acidsin a closed lagoon
in the Magadalen Islands, Canada : recent assimilation and metabolic mobility by cultured mussels. Soumis à Aquaculture.
Chapitre 4 – Péquin B, Mohit V, Tremblay R, Lovejoy C. Short term temporal changes in rRNA inferred
microbial eukaryotic communities in a shallow lagoon with mariculture activities. En préparation, en vue d'une soumission prochaine.
Chapitre 5 - Conclusion générale
Ces travaux ont été présentés lors de différentes présentations orales et par affiches lors de conférences et séminaires. Ils ont été présentés aux assemblées générales de Québec-Océan de 2009, 2011, 2012. Entre autres :
B Péquin, R Tremblay, C Lovejoy « Biodiversity over a season in a temperate lagoon » Affiche présentée à la Gordon Research Conference in Marine Microbes, Tilton (USA), 4-9 juillet 2010.
xxii
B Péquin « Étude de la diversité de la communauté de protistes aux Iles de la Madeleine dans une ferme de moules et sous influence environnementale » Séminaire présenté à l’interlab de l’Institut de Biologie Intégrative et des Systèmes, Québec (Canada), 19 décembre 2012.
En parallèle à cette thèse, j'ai contribué à deux autres articles :
Lionard M, Péquin B, Lovejoy C, Vincent WF, 2012. Benthic cyanobacterial mats in the high Arctic : multi-layer strucuture and fluoresence responses to osmotic stress. Frontiers in Microbiology 3, 1-10.
Poisot T*, Péquin B*, Gravel D*, 2013. High-throughput sequencing : a roadmap toward community ecology. Ecology and Evolution 3, 1125-1139 (* contributions égales).
Mes données de pyroséquençage du chapitre 2 et 4 ont également été utilisées pour le papier suivant : Comeau AM, Vincent WF, Bernier L, Lovejoy C, 201X. Novel chytrid lineages dominate fungal communities in diverse marine, freshwater and ice habitats. ISME Journal, submitted
.
Finalement, j’ai participé à la préparation des cultures de microalgues en vue du séquençage génomique et transcriptomique de celles-ci ainsi que les tests de détermination de la taille du génome. Publiées dans le papier de Terrado R, Monier A, Edgar R, Lovejoy C, 2015. Diversity of nitrogen assimilation pathways among microbial photosynthetic eukaryotes. Journal of Phycology 51,490-506.
Chapitre 1 : Introduction générale
1.1 Les lagunes côtières
La plupart des lagunes sont des enclaves d’eau salée qui communiquent avec la mer ou l’océan par des ouvertures plus ou moins larges. Le temps de résidence de l'eau est assez long, et la circulation des courants marins plus ou moins restreinte (Kennish and Paerl 2010). La caractéristique fermée des lagunes permet d’étudier les processus intrinsèques qui sont les processus biologiques structurant la communauté tel la prédation ou la compétition pour les ressources. À l’opposé les processus extrinsèques à la communauté peuvent influencer sa structure c’est le cas des marées, des apports de nutriments, du vent, qui sont davantage physiques.
Les lagunes côtières sont parmis les écosystèmes les plus productifs de la planète. La faible profondeur permet la productivité primaire de la surface jusqu’au fond. Celle-ci est la formation de matière organique végétale issue de la photosynthèse. Les producteurs primaires sont des phototrophes, c’est le cas des micro ou macro-algues, des bactéries photosynthétiques ou encore des zostères retrouvés sur le fond des lagunes. La productivité de l'écosystème est alors possible sur toute la hauteur de la colonne d'eau ce qui n'est pas le cas pour la majorité des systèmes océaniques. Les nutriments présents pour la production primaire viennent soient d’apports extérieurs (rives, exploitations agricoles, pluie, vent, etc.) que l’on nomme apports allochtones; soient intérieurs, apports autochtones, provenant du recyclage des nutriments par les organismes présents. Lorsque l’apport allochtone est faible les nutriments sont abondaments recyclés, la chaine trophique est soutenu par le picophytoplancton. L'excrétion de la matière organique dissoute (DOM) par le picophytoplancton vivant ou mort est transférée dans la boucle microbienne (Azam et al. 1983). Celle-ci est l’utilisation du carbone et des nutriments par les bactéries comme source d’énergie et permettant la production de biomasse. Ce système contraste avec un réseau trophique soutenu par des diatomées, comme dans les estuaires, où l’énergie est transférée aux macroorganismes qui broutent les diatomées (Gilbert et al. 2010). Les producteurs primaires sont ensuite consommés par les hétérotrophes microflagellés (dinoflagellés, rhizaria, telonemia, picozoa), eux-mêmes servant de proies aux ciliés. Au niveau supérieur, la prédation se fait par le zooplancton et les moules ainsi que les autres invertébrés marins, puis finalement par des plus gros organismes. Les bactéries de la boucle microbienne vont servir de proies aux bactérivores et de « compartiment » détritivore. Les virus par leur infection sur les producteurs primaires et les hétérotrophes microflagellés vont apporter une source supplémentaire de matière organique dissoute à la boucle microbienne (Figure 1.1).
2
Figure 1.1: Schéma du réseau trophique aquatique. Le sens des flèches indique un transfert d'énergie. Les lagunes ont un très fort potentiel récréatif, touristique et économique de par leur faible profondeur et leurs particularités physiques. De plus, elles soutiennent un grand nombre de services écosystémiques. Les services écosystémiques (Millenium Ecosystem Assessment 2005) sont une grande fonction d’un écosystème dont l'être humain peut bénéficier. Le service qui est à la base du fonctionnement des écosystèmes est le service « porteur ». Il se définit comme les processus biotiques et abiotiques d'un écosystème. On distingue la productivité, la décomposition et le flux de nutriments (Loreau et al. 2001) comme vu précédemment. Le service « porteur » soutient le service « approvisionnement » tel que la nourriture. Dans le cas des lagunes, le service approvisionnement est la culture de moules, de pétoncles, d’huîtres et autres bivalves. Les installations sont facilement accessibles et déplaçables. La mariculture entraîne en revanche une perte de la valeur esthétique, une gêne pour la navigation, des problèmes d'allocation d'espace entre les divers usagers, la potentielle contamination par la terre provenant des cultures et enfin la déplétion en nutriments et/ou phytoplancton (Stevens et al. 2008). Celle-ci peut influencer le réseau trophique de la lagune. De ce point de vue, la mariculture structure la chaîne trophique par le haut (top-down); c'est à dire, les niveaux trophiques inférieurs sont soumis à la pression des prédateurs supérieurs (ici les bivalves). Le cas contraire est une structuration de la chaîne trophique par le bas (bottom-up); c'est-à-dire que les prédateurs supérieurs sont dépendants des fluctuations en nutriments et en producteurs primaires.
La lagune de Havre-aux-Maisons aux Îles de la Madeleine, dans le golfe du St-Laurent au Canada est l’exacte description d’une lagune côtière présentant des services écosystémiques. La mytiliculture est une activité économique importante dans les deux principales lagunes (Figure 1.2). L'industrie maricole y est présente depuis 1984. La production de moules est d'environ 193 tonnes/an (moyenne pour les 7 dernières années). Le réseau trophique de la lagune de Havre-aux-Maisons n’est pas totalement connu. Plusieurs études ont été réalisées sur les bivalves, les macro-organismes, les flux de nutriments cependant la partie micorscopique été peu connue. Récemment, Mohit et al. (2014) ont permis de déterminer la population de bactérie. Le compartiment phytoplanctonique a été déterminé dans cette thèse. C’est ainsi que les objectifs principaux de cette thèse sont :
- Déterminer la diversité et la structure de la communauté de protistes - Etudier l’interaction entre la communauté de protistes et Mytilus edulis
Figure 1.2 : Carte des Îles de la Madeleine.
1.2 Les protistes
1.2.1 Diversité des protistes
Un protiste est un eucaryote unicellulaire. On en distingue trois types : (i) les protistes autotrophes font de la photosynthèse. Ce sont les producteurs primaires, qui utilisent l’énergie lumineuse pour fixer le carbone inorganique et le transformer en biomolécule grâce à l’utilisation de photosystèmes. (ii) Les protistes hétérotrophes brouteurs de bactéries et/ou d'autotrophes. (iii) Finalement, les mixotrophes combinent l'autotrophie et l'hétérotrophie en fonction des conditions environnementales. Selon les ressources nutritives
4
dont ils ont besoin et l’énergie dépensée à récupérer cette ressource, ils alternent tour à tour la photosynthèse (dans le but de synthétiser eux-mêmes leur source carbonée) ou la prédation pour récupérer une source carbonée déjà existante. La diversité des protistes est très grande aussi bien au niveau taxonomique que écologique. Il existe 10 phyla majeurs. Nous pouvons citer les alvéolés dans lesquels on retrouve principalement les dinoflagellés et les ciliés; les chlorophytes où l'on retrouve les algues vertes et la lignée des végétaux; les cryptophytes, les haptophytes et les picozoa, qui sont largement étudiés; et les straménopiles dans lesquels on retrouve les diatomées (Figure 1.3). Il est courant de catégoriser les protistes par leur taille. Nous distinguons le picoplancton entre 0,2 et 3 μm, le nanoplancton entre 3 et 20 μm et enfin le microplancton entre 20 et 200 μm (Sieburth et al. 1978).
Figure 1.3 : Arbre phylogénétique des eucaryotes modifié de Adl et al. (2012). Les groupes indiqués sont ceux dans lesquels les protistes sont retrouvés.
Le groupe des Alveolates contient principalement les ciliés, les dinoflagellés et les Marines Alveolates (MALV). Parmis eux, les dinoflagellés ont une morphologie carcatéristiques et une très grande diversité d’espèce. Ils ont une grande diversité fonctionnelles phototrophes, hétérotrophes et mixotrophes. Leur taille varie entre 10 et 100µm. Certaines espèces de dinoflagellés peuvent être toxiques pour l’homme comme Alexandrium tamarense qui forme des « marées rouges » et consommés par les bivalves. Le groupe des Straménopiles est très étudié, il posséde entre autres la phylum des diatomées très connu. Il y a également le groupe des Straménopiles Marins (MAST) qui n’ont aucun représentant en culture mais que l’on retrouve de façon cosmopolite sur la planète. Les cercozoa sont un groupes très peu étudiés pour cause ils sont souvent nommés petits flagellés c’est à dire un groupe dont on ne sait pas grand chose. Ils sont hétérotrophes et leur taille est estimée entre 3 et 50 µm de diamètre. Les haptophytes possèdent une grande diversité morphologique. Ils sont protégés ou non de plaques de carbonate de calcium nommés coccolithes. Ils sont mixotrophes et font entre 3 et 20 µm. C’est un groupe d’intérêt en aquaculture, par exemple l’epèce Isochrysis galbana est utilisée pour nourrire les larves de moules. Le groupe des chloroplastida est fait des chlorophytes et des plantes terrestres. Les chlorophytes ont une grande diversité morphologique et phototrophes faisant entre 1µm et 2mm.
1.2.2 Structure de la communauté de protistes
Les conditions environnementales influencent les producteurs primaires de plusieurs façons. Entre autre, elles peuvent impacter les réserves et les apports de matière organique. C'est le cas du vent qui, par exemple, entraîne les poussières continentales chargées en Fer vers le milieu aquatique, ce qui bénéficiera directement aux organismes photosynthétiques. Les conditions environnementales entraînent une dynamique de succession des protistes tout au long de l’année (Margalef 1958, 1962). Au printemps, l’apport de nutriments provenant du ruissellement hivernal et la lumière en grande quantité favorisent les efflorescences de diatomées. La transition vers l’été est marquée par la présence d’haptophytes. L’été en lui-même est caractérisé par les dinoflagellés, lorsque l'abondance des proies est importante. Enfin en automne, la lumière plus faible et le retour de la matière organique apportés par le vent et la pluie favorisent les efflorescences de petites cellules (certains chlorophytes). La spatialité peut être définie sur deux plans (horizontal et vertical). De manière horizontale, les protistes peuvent présenter différents assemblages de communauté selon qu'ils se trouvent proche de la côte ou au large (Dasilva et al. 2014). De manière verticale, la stratification des masses d'eau peut influencer la répartition de la communauté de protistes. Les masses d'eaux se distinguent les unes entre les autres par leur densité, leur température, leur salinité et leur provenance.
1.2.3 Méthodologie employée
Nous utiliserons les techniques moléculaires pour obtenir une bonne résolution de la diversité taxonomique et fonctionnelle des protistes. L’apport des méthodes moléculaires est indéniable pour étudier les protistes. Elles
6
permettent de connaître davantage les différents genres présents en plus d'apporter une information sur leur rôle fonctionnel. La biologie moléculaire a permis de mettre en évidence certains taxons non détectés en microscopie et non cultivés. C'est le cas des Straménopiles Marins (MAST) ou encore des picozoa. Les techniques moléculaires ont aussi permis d'approfondir les connaissances sur la diversité phylogénétique du picoplancton. Auparavant, ils n'étaient pas détectés en microscopie en raison de leur trop petite taille. L'avènement du séquençage haut-débit, et la génération de milliers de séquences par échantillon, a permis de mettre en évidence la biosphère rare, c'est à dire les organismes qui sont présents à très faibles abondances. La principale technique utilisée pour décrire la structure des communautés est le séquençage à haut-débit par la méthode du pyroséquençage 454.
Le gène codant la petite sous unité ribosomique des eucaryotes (18S) est retrouvé chez tous les eucaryotes. Il possède une partie très conservée parmi toutes les espèces, et d'autres parties plus variables qui permettent l'assignation phylogénétique des espèces. L’identification taxonomique des protistes de cette thèse se fait grâce au séquençage de la partie V4 du 18S (Figure 1.4) et un recoupement avec les bases de données taxonomique existante. La partie V4 du 18S est une des parties les plus variables flanquées de régions conservées. Cette configuration nous permet d’amplifier les 400 paires de bases de la région V4. Les amorces utilisées pour l’amplifcation sont universelles. Le séquençage de cette zone est donc très reproductible quel que soit l’environnement étudié. Les bases de données taxonomiques concernant cette région sont bien renseignées. Dans un premier temps la communauté planctonique est identifiée en séquençant le gène codant le 18S, la forme ADN. De cette manière tous les organismes eucaryotes présents dans l’eau sont identifiés. Il peut s'agir d'organismes morts ou vivants (Chapitre 2). Dans un second temps les protistes sont identifiés par l’intermédiaire de la forme ARN du18S. Cet ARN est spécifique aux organismes vivants. En effet seuls ceux métaboliquement actifs transcrivent l'ADN en ARN. Pour étudier l’ARN, nous l'extrayons des échantillons et le transformons en ADN complémentaire que nous séquençons toujours en ciblant le 18S (Chapitre 4).
Figure 1.4 : Structure secondaire de la petite sous-unité ribosomique des eukaryotes, 18S, Ueda-Nishimura and Mikata (2000).
Sur le terrain l'eau est filtrée et fractionnée au moyen de trois filtres, dans le but d'isoler différentes classes de tailles d’organismes. Le premier filtre (pores de 50 μm de diamètre) permet d’exclure les organismes de très grande taille, principalement le zooplancton. Le deuxième filtre (pores de 3 μm – grande fraction) permet de récupérer les organismes entre 3 μm et 50 μm, le nanoplancton. Enfin, la petite fraction, le picoplancton, est récupérée grâce à un filtre de 0.2 μm. De retour au laboratoire, l'ADN est extrait par la méthode d'extraction au sel. Le gène codant la petite sous-unité de l'ARN ribosomique des eucaryotes (ADN 18S) est amplifié par la réaction de polymérase en chaîne (PCR). L'ADN ainsi amplifié est récupéré et purifié au moyen d’un kit. Les échantillons sont ensuite séquencés. La première étape fixe une séquence d’ADN issue de la PCR sur une bille pour l’amplifier à nouveau. La seconde étape est le séquençage individuel de la bille et donc de l’ADN fixé sur cette bille. Ainsi, chaque séquence présente dans la PCR d’origine est susceptible d’être séquencée. Le jeu de données ainsi obtenu contient des milliers de séquences pour chaque échantillon. La communauté sera alors décrite avec une très fine résolution, puisque les groupes taxonomiques les plus rares seront représentés. Les séquences sont ensuite nettoyées via un pipeline bioinformatique avec le logiciel Mothur (Schloss et al. 2009). Toutes les séquences provenant des différents échantillons sont regroupées lors du traitement informatique. La première chose consiste à enlever les séquences contenant des bases ambiguës,
8
inférieures à 150 nucléotides, supérieures à 600 nucléotides ou contenant une amorce forward inattendue. Ensuite, les séquences se rapportant à des métazoaires sont éliminées ainsi que les chimères. Par la suite, les séquences sont alignées. Celles mal alignées sont supprimées. Une fois l'alignement propre, un regroupement basé sur un algorithme permet de former des Unités Taxonomiques Opérationnelles (OTU). On regroupe des séquences dans un OTU si leur pourcentage de similarité est au moins de 98%. Toutes les séquences qui ne forment pas des OTU, c'est-à-dire les séquences uniques (singletons) sont éliminées du jeu de données. Enfin, nous avons choisi de re-séparer les échantillons et de les ré-échantillonner au même nombre de séquences pour chaque échantillon. Nous pourrons ainsi les comparer entre eux.
1.3 Mytilus edulis (L.)
1.3.1 Description de l’espèce
L’espèce de moules étudiée ici, la moule bleue Mytilus edulis (L.), est une des plus importantes en terme d'abondance de la côte Est du Canada. Le phytoplancton constitue la majeure partie de leur diète (Kreger and Newell 1996). Cependant, elles peuvent aussi consommer des bactéries et du zooplancton. La larviphagie a aussi été décrite dans les fermes de moules. Famme et Kofoed (1983) ont montré que la moule bleue adapte son alimentation aux taxons présents dans son environnement. Les particules absorbées, mesurant entre 1 et 35 µm, sont retenues par les branchies pour ensuite passer dans le tractus intestinal (Stoheimer et al. 2012). La rétention est possible mais moindre pour les particules entre 35 et 200 µm. Selon la quantité de nourriture disponible, les moules peuvent réaliser une première sélection juste après le passage par les branchies. Une partie rejoint le tractus digestif, la seconde partie est alors rejetée sous forme de pseudofèces. Suite à la digestion, les moules rejettent des fèces. Les pseudofèces sont produites lorsque la concentration de la matière particulaire en suspension (SPM) est supérieure à 4.5 – 5 mg L-1 (Widdows et al. 1979).
La saisonnalité a aussi son importance pour les moules. Nous avons vu ci-dessus la dynamique de succession saisonnière des protistes. Ceux-ci étant la principale source de nourriture des moules, l’alimentation des bivalves va par conséquent changer au cours de l'année (Ezgeta-Balić et al. 2012). L’état physiologique des moules (par exemple en période de gamétogénèse) est un autre paramètre à prendre en compte. Lorsque les moules produisent des gamètes, la demande en énergie est très élevée. Une fois la ponte effectuée la demande en énergie diminue. L’arrivée de l’automne permet de stocker les nutriments pour passer l’hiver. La demande en énergie est à ce moment-là importante, mais plus lente, car elle dure plusieurs mois.
Le sujet principal de cette thèse est l’interaction entre les moules et la communauté microbienne. En effet, il est connu que les moules peuvent stimuler une efflorescence phytoplanctonique par le recyclage des nutriments lors de la resuspension des sédiments (Asmus and Asmus 1991; Prins et al. 1997). Elles peuvent
aussi entraîner une perte de biomasse de la communauté phytoplanctonique (Stevens et al. 2008), une perte de productivité primaire (Riemann et al. 1990), ou encore un changement dans l'identité des espèces de phytoplancton présentes (Baker et al. 1998). Ces déséquilibres se produisent aux niveaux biotique, abiotique et local (extinction d’espèces, cycles biogéochimiques, pH etc.) (Hooper et al. 2005), entraînant une rétroaction directe ou indirecte sur la production de moules. En effet, si les espèces planctoniques, leur principale source de nourriture, venaient à manquer, les moules ne pourraient plus croître. Si le déséquilibre se fait sur la matière organique, principale source d'énergie de la communauté planctonique, de manière indirecte cela pourra affecter la croissance des moules.
1.3.2 Les acides gras
Une des principales sources d'énergie des moules est l'utilisation métabolique des acides gras (AG). Ceux-ci sont retrouvés dans les lipides qui sont des constituants essentiels pour les organismes.
Les lipides sont des constituants indispensables aux organismes vivants. Ce sont des molécules amphiphiles c’est à dire ayant un côté hydrophyile et l’autre hydrophobes. Ils sont classés par leurs srucutres. Ils sont retrouvés dans de nombreux constituants cellulaires. Par exemple la classe des phospholipides est la classe de lipides qui constitue les membranes cellulaires. La classe des stéroles retrouvés dans les hormaones entre autre. Un dernier exemple comme les triglycérides source d’énergie. Ceux-ci sont constitués de trois molécules de glycérol rattaché chacune à un acide gras.
Les acides gras sont des chaînes carbonées possédant entre 13 et 28 atomes de carbone. Elles sont entrecoupées par une ou plusieurs doubles liaisons ou insaturations. Le nombre d'insaturations permet de diviser les acides gras en trois catégories : les acides gras saturés (SFA), les acides gras monoinsaturés (MUFA) qui possèdent une seule double liaison et les acides gras polyinsaturés (PUFA) avec plusieurs doubles liaisons. Les acides gras ont la particularité de pouvoir être utilisé directement, sans phase de transformation, lors du passage d’un compartiment trophique à l’autre. Le proverbe «nous sommes ce que nous mangeons» est alors approprié.
1.3.3 Méthodologie employée
Les acides gras issus des lipides permettent de caractériser les sources nutritives des moules, en déterminant ceux présents dans la lagune, ceux ingérés par les moules et enfin ceux rejetés dans les fèces. Sur le terrain, de l’eau est filtrée pour identifier les acides gras de la lagune. Après la récolte sur le terrain, ls moules sont misent en stabulation, c’est à dire dans de l’eau pure pour récupérer au bout de 24h la glande digestive et les fèces afin de déterminer la compostion en acides gras. Pour récupérer les acides gras, les lipides sont extraits selon la méthode de Folch modifié par Parrish (1999) puis une trans-estérification totale est réalisée sur les
10
lipides afin de ne garder que les acides gras (AG). Deux acides gras standards (le C19:0 et le C23:0) sont utilisés lors de l’extraction et de l’estérification. Les échantillons sont ensuite analysés en chromatographie en phase gazeuse (CPG), couplée à un spectromètre de masse. Ils permettent de distinguer les acides gras selon leur temps de rétention à travers la colonne et leur masse. Les acides gras standards absents des matrices biologiques nous permettent d’avoir une calibration lors du passage en chromatographie puisque leur temps de rétention et leur masse sont connus. Les chromatogrammes issus de cette analyse sont comparés à des banques d'acides gras de référence. Également, pour valider l'identification sur notre propre colonne, nous passons un standard contenant 37 acides gras les plus connus. On peut ainsi identifier la composition de nos échantillons.
1.4 Site d’étude
Les îles de la Madeleine sont reliées entre elles par des dunes de sables, entraînant la formation de lagunes. Ce sont celles de la Grande-Entrée au Nord-Est, Havre-aux-Maisons au centre et le bassin de Havre-Aubert au Sud-Est. La lagune de Havre-aux-Maisons est un exemple de lagune côtière. Selon Bird (1982), la largeur des entrées à marées hautes est inférieure à 20% de la longueur totale de la barrière de la lagune. La lagune de Havre-aux-Maisons présente de faibles concentrations moyennes en chlorophylle a. Il n'y a pas d'apport fluvial par les rives. La décharge des eaux de pluie est faible en matière organique dû à l’absence d’arbre sur les rives à cause du vent trop important. Le temps de résidence de l'eau varie entre 25 et 45 jours. Vingt-cinq jours correspondent à l'effet seul des marées et quarante-cinq jours à l'effet simultané des marées et de la direction du vent (Koutitonsky 2002, Guyondet et Koutitonsky 2008). La profondeur maximale est de 6 mètres. Les moules bleues Mytilus edulis sont commercialisées après 2 ans de vie passés dans lagune. La culture se fait en boudins suspendus dans la colonne d’eau. Les moules pondent en général au mois de mai, puis entament ensuite un repos gamétogénique, et reforment leurs réserves pour passer l'hiver. Les moules des îles ne semblent pas relarguer de pseudofèces. Cela est peut-être dû à la trop faible concentration en SPM de la lagune. Pour le chapitre 2 et le chapitre 3, les échantillons sont pris dans le site d’élevage expérimental du Ministère de l'Agriculture, des Pêcheries et de l'Alimentation du Québec (MAPAQ). Il n'y a pas de récolte commerciale à cet endroit. Pour le chapitre 4, la ferme de moules utilisée est celle de Mr. Michel Fournier.
1.5 Structure de la thèse
Cette thèse a pour objectifs (i) l’étude de la diversité et la détermination de la structure de la commuauté de prostistes de la lagune de Havre-aux-Maisons; (ii) l'étude de l'interaction entre les moules et la communauté de protistes. Pour répondre à ces objectifs, trois grands axes sont abordés dans cette thèse: la caractérisation de la communauté de protistes, la nutrition des moules et enfin l'influence des moules sur la communauté de protistes. Chaque chapitre adressera un de ces axes.
Le chapitre 2, «Le vent détermine la communauté microbienne eucaryote dans une lagune fermée tempérée», décrit la communauté taxonomique et fonctionnelle des protistes présents dans la lagune de Havre-aux-Maisons. Les changements temporels observés sont dus, à l'influence du vent. Pour cela, un site dans une ferme de moules a été échantillonné dix fois sur une période allant de juin à octobre 2009. Les facteurs environnementaux, la taxonomie, le rôle fonctionnel des espèces ainsi que leurs interactions ont été déterminés afin d’obtenir une image, la plus complète possible, de la succession dans la communauté de protistes de la lagune de Havre-aux-Maisons. L’hypothèse de ce chapitre est
- Les changements d’espèces eucaryotes sont le résultat de processus intrinsèques.
Le chapitre 3, «Acides gras dans une lagune fermée aux Îles de la Madeleine, Canada : récente assimilation et mobilité métabolique par les moules cultivées», détermine les variations du régime alimentaire des moules de la lagune. Pour cela, un site dans une ferme de moules a été échantillonné dix fois sur une période s'étalant de juin à octobre 2009. Les profils en acides gras de l'eau de la lagune, de la glande digestive et des fèces des moules sont déterminés. Les abondances des cellules photosynthétiques et bactériennes sont incluses dans ce chapitre. L’hypothèse est
- La compostition en acides gras retrouvés chez les moules est le reflet de ceux retrouvés dans la lagune.
Le chapitre 4 intitulé « Changements temporels à court terme de la communauté microbienne eucaryotes inférés par l’ARNr dans une lagune peu profonde ayant des activités maricoles » tente de déterminer l'impact de la ferme de moules sur la communauté taxonomique de protistes. Le but est d'identifier si la pression de prédation des moules influence la communauté des protistes. Pour cela, trois sites dans la ferme de moules et trois sites hors de la ferme de moules ont été échantillonnés tous les deux jours durant quinze jours pour un total de 48 échantillons. L'abondance, la biomasse et l'identification taxonomique de la communauté de protistes ont été comparées entre les sites avec ou sans moules. L’hypothèse de ce chapitre est
- La communauté de protiste change à court terme (aux 2 jours) - La ferme de moule impacte la communauté de protistes
Une image assez complète de la caractérisation et de la dynamique de la communauté de protistes est ainsi réalisée. Plusieurs niveaux trophiques sont pris en compte de manière à intégrer nos connaissances sur les protistes de la lagune de Havre-aux-Maisons ainsi que les interactions entre les organismes de ces différents niveaux trophiques.
Chapitre 2 : Wind drives microbial eukaryote
communities in a temperate closed lagoon
Résumé
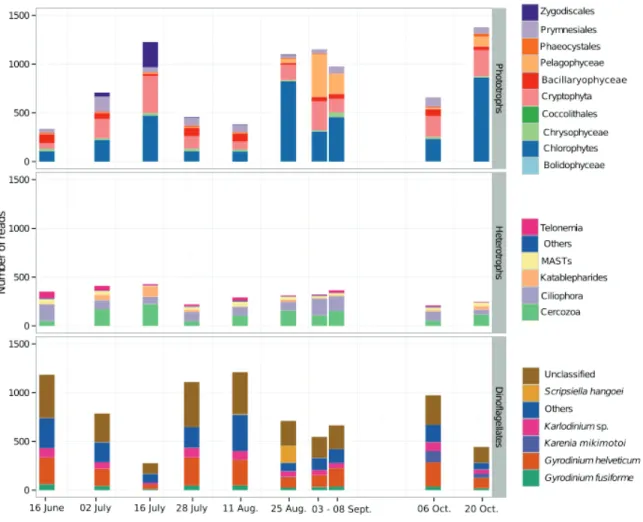
Les micro-organismes eucaryotes pélagiques sont fonctionnellement et phylogénétiquement divers et sont des composants importants des écosystèmes côtiers, mais il y a peu de compréhension des facteurs intrinsèques et extrinsèques qui influence ces communautés. À cette fin nous avons échantillonnés les eucaryotes microbiens marins dans une lagune tempérée aux Îles de la Madeleine dans le Golfe du Saint-Laurent durant 10 occasions entre juin et octobre 2009. Nous estimions que la communauté estivale serait influencée par des processus successifs intrinsèques, par l’absence d’apport fluviaux et du faible échange avec le milieu marin avoisinant. Les concentrations en picophytoplancton et le nanophytoplancton ont été estimées par la cytométrie de flux (FCM) et la composition taxonomique de la communauté a été investiguée par séquençage haut débit d’amplicons de la région V4 du gène codant l’ARN ribosomique 18S. Les concentrations cellulaires en picophytoplancton fluctuaient sensiblement mais il n’y avait pas d’évidence d’un déclin de la taille cellulaire du picophytoplancton durant l’été. Les taxons dominants de la communauté variaient également et l’analyse UniFrac prenant en compte l’abondance divisait la communauté en trois groupes. Le plus grand groupe (5 dates) était dominé par les dinoflagellés, les deux autres groupes étaient caractérisés par différents taxons de pico et nanophytoplancton. L’alternance entre les dinoflagellés et les petits phytoplanctons était corrélée avec la vitesse du vent jusqu’à cinq jours avant l’échantillonnage et les grands événements occasionnels de vent. Les petits phytoplanctons augmentaient suivant les grands événements de vent et les périodes de vent ayant une moyenne élevée. Au contraire, les dinoflagellés dominaient durant les périodes de faible vent.
Abstract
Pelagic microbial eukaryotes are functionally and phylogenetically diverse and are ubiquitous in coastal ecosystems, but there is little understanding of the intrinsic and extrinsic factors that influence these communities. To this end we sampled the marine microbial eukaryotes in a temperate lagoon in the Magdalen Islands of the Gulf of St. Lawrence on 10 occasions between June and October 2009. The summer community was expected to be mostly influenced by intrinsic successional processes because of a lack of riverine inputs and little exchange with the surrounding Gulf of St. Lawrence. Picophytoplankton and nanophytoplankton concentrations were estimated from flow cytometry (FCM) and the taxonomic composition of the communities was investigated by way of high throughput amplicon sequencing of the V4 region of the 18S rRNA gene. Picophytoplankton cell concentrations fluctuated markedly but there was no evidence of a decline in phytoplankton average cell size over the summer. The taxonomic dominants of the communities also varied, with a weighted UniFrac analysis placing the communities into three major clusters. The largest cluster (5 dates) was dominated by dinoflagellates, while the two other clusters were characterized by different pico and nanophytoplankton taxa. The alternation between the dinoflagellates and the small phytoplankton, was correlated with average wind speed over the preceding five days and occasional high wind events. Smaller phytoplankton increased following high wind events and over periods of greater average wind speeds. In contrast dinoflagellates dominated during periods with lower average wind speeds.
2.1 Introduction
Summer production in temperate coastal regions is supported by either external nutrient inputs (Guadayol et al. 2009) or efficient nutrient recycling by microbial communities (Canton et al. 2012). Groundwater and rivers contribute much of the external nutrient input into estuaries, while upwelling and tidal mixing are a source of nutrients to the euphotic zone in open coastal systems. These mechanisms for external nutrient input do not always occur in closed lagoons that nonetheless can support a high level of secondary production and even aquiculture activities (Roy et al. 1991) and nutrient recycling is thought to sustain microbial food webs over the summer growing season.
Diverse microbial eukaryotes are key components of these microbial food webs and the timing and occurrence of particular clades and genera with differing trophic strategies may point to predominant processes in the ecosystem over time and space (Caron et al. 2012). Larger primarly heterotrophic marine microbial eukaryotes referred to as protist microzooplankton (Revelante and Gilmartin 1983; Dupuy et al. 1999) are a primary food source for mesozooplankton and shellfish (Sherr and Sherr 1994, 2002; Trottet et al. 2007) and secondary productivity may be directly dependent on microzooplankton success. Microzooplankton graze on each other, nanophytoplankton (2-20 µm diameter) and picophytoplankton (0.2-2µm diameter). However, much of what is known about the in situ interactions among these groups is based on bulk properties and some microscopy observations (Sanders et al. 2000; Sherr and Sherr 2002) and in particular a broad understanding of factors driving relative abundance of individual taxa is lacking.
The complex hydrography of most coastal regions also confounds interpretation of species occurrences since communities can be rapidly replaced by advection and upwelling at a given site. In contrast, closed lagoons have limited biotic exchange with the open ocean making them suitable for investigating processes that select for microbial eukaryotic species and potential species interactions. The closed lagoons in the Magdalen Islands Archipelago are relatively isolated from extrinsic drivers: they have little exchange with the adjacent Gulf of St. Lawrence, have weak mean currents (between 1 to 5 cm s-1), and relatively long hydraulic residence times (>20 days). In addition, with no rivers or streams there is little surface runoff and they have small catchments (Koutitonsky et al. 2002; Guyondet and Koutitonsky 2008). Compared to other coastal systems, the lagoons typically have low standing stocks of inorganic nutrients (Robert et al. 2013) and photosynthetic biomass estimated from chlorophyll a (Chl a) over the summer season is between 0.8 – 3 µg l-1 (Trottet et al. 2007). By way of contrast, Chl a concentrations are typically >6 µg l-1 in other coastal systems (Gilmartin and Revelante 1978; Pennock 1985; Pérez-Ruzafa et al. 2005).
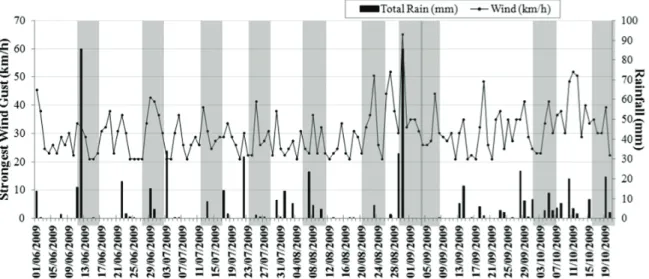
In the Magdalen Island lagoons, precipitation is a major source of external nitrate, but most precipitation is over winter (Souchu and Mayzaud 1991; Guyondet and Koutitonsky 2008), and the influence of occasional
18
summer rain events on phytoplankton has not been investigated. Smaller cells, especially picophytoplankton are favored under low nutrient conditions (Raven 1998) and it could be expected that in the absence of external nutrient inputs, the phytoplankton in the Magdalen Island lagoons would be made up of increasingly smaller sized cells as the summer progressed. Such changes would be manifest as a shift from nanoplankton to picophytoplankton. In addition, since nano and picophytoplankton are phylogenetically diverse (Vaulot et al. 2008) species changes would be expected to occur along with size differences. In this study, we test the hypothesis that change in microbial eukaryotic species composition is a result of intrinsic processes (Record et al. 2010), where nutrients become increasingly limiting and species better able to scavenge nutrients will become the dominant phototrophs that support microbial eukaryote grazers. Other intrinsic processes could include ecological interactions (e.g. synergy, competition or predator prey interactions) among species or functional clades in the lagoon. An alternative scenario would have species changes occurring as a result of external events such as storms, which has been reported for bacterial communities (Yeo et al. 2013).
In this study we enumerated photosynthetic (chlorophyll containing) eukaryotes and cyanobacteria using flow cytometry (FCM) to detect any changes in the cell size of the community over time. We then applied high throughput multiplex tag sequencing of the V4 region of the 18S rRNA gene to taxonomically identify and functionally classify the microbial eukaryotes from the same samples. Using these data we then addressed the question of whether community taxonomic composition may have been influenced by extrinsic factors especially wind and rain events. The use of high throughput amplicon sequence data also enabled us to follow specific taxa over time. With the goal of separating intrinsic and extrinsic processes operating on the communities, we applied multivariate statistics to link specific taxa with environmental variables.
2.2 Materials and Methods
2.2.1 Sample collection
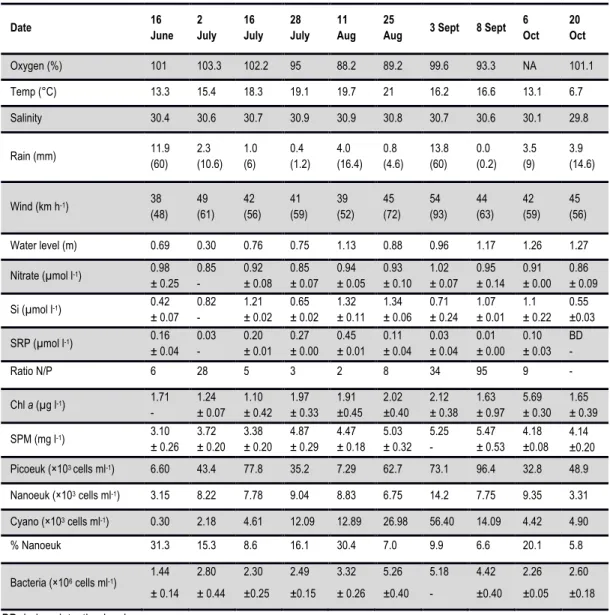
The Havre-aux-Maisons lagoon (HAM: Canada, Québec, Gulf of Saint Lawrence, Magdalen Islands) is relatively pristine with development limited to mussel farming operations covering less than 5 % of the superficial area (Myrand and Gaudreault, 1996; Moisan et al. 2012). A single site (47° 25.730ʹ N: 61° 48.832ʹ W: Figure 2.1) was visited approximately every two weeks from 16 June to 8 September and again on 6 and 20 October 2009. All sampling was done between 9:00 and 11:00 am. Temperature and salinity were recorded down the water column with a Yellow Springs Instruments™ YSI 30/25 FT probe and oxygen saturation values with a YSI 550A dissolved oxygen probe. Water samples were collected into cleaned carboys from 2.5 m below the surface using a submersible electric Rule 3700 bilge pump (Kings Pumps™) and transported to a shore-based laboratory for filtration and processing. Duplicate water samples were taken for nutrient analysis. The samples were pre-filtered through a 25 mm 0.45 µm Millipore™ LCR membrane
syringe filter into acid cleaned and sample rinsed (3 times) 15 mL polycarbonate microtubes. The nutrient samples were then frozen and stored at -20°C until analysis. Duplicate samples for chlorophyll a (Chl a) were filtered onto 25 mm diameter Whatman™ GF/F glass fiber filters and the filters kept at -20°C until analysis. Duplicate samples for total suspended particulate material (SPM) was collected by filtering 2 L of water onto pre-combusted, pre-weighed Whatman™ GF/C filters; the filters with material were rinsed with a solution of ammonium formate to remove salts and then stored to -80°C until analysis. Following pre-filtration through a 40 µm mesh, samples for FCM were collected into 1.5 mL cryotubes and preserved by the addition of 25 % glutaraldehyde (final concentration of 0.1 % v/v), left at room temperature in the dark for 15 min and then transferred to -80°C. For DNA collection, two liters of water were prefiltered through a 50 µm net and then sequentially through a 3 µm pore size 47 mm diameter polycarbonate Millipore™ filter and a 0.2 µm pore size Sterivex™ Unit (Millipore) using a peristaltic pump system. The 3 µm filters (large fraction) were placed into cryovials with 1.8 mL of buffer (40 mM EDTA; 50 mM Tris pH=8.3; 0.75 M sucrose). The same buffer was added to the Sterivex units (small fraction, 0.2-3.0 µm) and both fractions were immediately placed into -80°C freezer until DNA extraction.
Figure 2.1: Map of Magdalen Islands, Gulf of St. Lawrence (GSL). The dark grey areas are regions of solidrock whereas the light grey regions surrounding the lagoons are sand bars.
2.2.2 Sample processing
Nutrient concentrations; soluble reactive phosphate (SRP), soluble silicate (Si), and nitrite+nitrate (Nitrate), were determined using standard protocols (Grasshoff et al. 2009) adapted for the AA3 Autoanalyzer (SEAL Analytical Inc) with a detection limit of 0.01 µmol l- 1 for each nutrient. The filters for Chl a determination were extracted in 70°C ethanol, which allows for a rapid extraction without degradation of the Chl a (Arvola 1981; Wright et al. 2005) and concentrations determined spectrophotometrically using a Cary 300 Bio UV-Visible spectrophotometer following an acidification step (Wright et al. 2005). The filters for SPM were oven dried at
20
65 ºC for 48 hours and reweighed. Concentrations of heterotrophic bacteria, cyanobacteria, nano- and picophytoplankton, were determined using a Beckmann Coulter Epics Altra™ flow cytometer as described by Belzile et al. (2008). DNA was extracted from the 3 µm filters and Sterivex units based on a salt extraction protocol (Aljanabi and Martinez 1997) with modifications as described by Harding et al. (2011). The extracted DNA was resuspended in TE buffer and stored at -80 °C, final concentrations averaged 35 ng DNA µl–1 (±16 ng µl–1).
2.2.3 DNA amplification and sequencing
Genomic DNA was amplified using primers specific for the V4 region of the 18S rRNA gene, using the forward (E572F) and reverse primers (E1009R) described by Comeau et al. (2011). The V4 region is consistently better at discriminating lower level taxa compared to other variable regions of the 18S rRNA gene in marine microbial eukaryotes (Luddington et al. 2012; Hugerth et al. 2014). To decrease PCR bias, for each size fraction (> 3 µm and 0.2- 3µm) amplicons from three separate reactions, using 0.5 µL, 1 µL, 2 µL of template, were pooled and purified using QIAGEN QIAquick™ PCR purification kit. The concentrations of the final pooled DNA amplicons were quantified spectrophotometrically with the Nanodrop™ ND-1000. Separate multiplex identifier oligonucleotide tags (MIDs from Roche Scientific) were used for each of the 10 samples, with amplicons from large and small fractions mixed based on the proportions of picophytoplankton and nanophytoplankton estimated from flow cytometry (Table 2.1). The pooled tagged amplicons were sequenced at the IBIS/Université Laval Plate-forme d’Analyses Génomiques on 1/8th plate on GS-FLX, using Titanium Chemistry (Roche Scientific). All reads from this study have been deposited in the NCBI Short Read Archive (SRA) under accession SRP043016.
2.2.4 Bioinformatics analysis
Raw reads (nucleotide sequences) were processed to remove unresolved nucleotides (Ns), reads shorter than 150 Ns, and sequences with one or more 7-mer or longer homopolymers identified using mothur (v 1.27.0 on Mac OSX, Schloss et al. 2009). The 10 MID tags were merged, metazoan reads identified and removed, reads were then chimera checked via UCHIME (Edgar et al. 2011) run in mothur. After removal of chimeras, reads were aligned against the eukaryote Silva Database (http://www.arb-silva.de) based on the secondary structure of the SSU rRNA gene. After this alignment step, poorly aligned reads were removed. We then used the furthest neighbor clustering option in mothur to generate Operational Taxonomic Units (OTUs) at a similarity level of 98%. OTUs that occurred only once in the entire dataset (singletons), were removed, leaving only what we termed ‘high quality reads’ to facilitate comparative and statistical tests.
Reads from the pooled MID tags, were then separated and randomly re-sampled to ensure the same number of reads from each of 10 original samples, based on the sample with the fewest ‘high quality reads’, resulting
in 2278 reads from each sample. Phylogenetic similarities among eukaryotic microbial communities were assessed using weighted and normalized UniFrac distances implemented in QIIME v1.6 (Caporaso et al. 2010). UniFrac distances were inferred from the OTU approximate maximum-likelihood phylogeny reconstructed using FastTree v2.1 (Price et al. 2010). Clustering of UniFrac distances was computed using UPGMA, and statistical support was based on 1000 jackknife replicates; each re-sampling 1700 reads per sample, which corresponded to 75% of the reads per sample. Taxonomic identities of all reads were assigned based on a curated marine eukaryote reference database (Comeau et al. 2011), which was derived from the NCBI taxonomy (http://www.ncbi.nlm.nih.gov/taxonomy) and Silva databases (based on the SSU Ref database v. 102). This reference database includes representative aligned full length environmental sequences from the North Atlantic, Scotia Shelf, (Dasilva et al. 2014). This same database was annotated, by adding approximate size and what is known about trophic function of the genera level taxa, following the approach of Lovejoy et al. (2002), which provides a means of comparing communities where the same classification criteria are applied. The reads were assigned to 196 genera level taxa. Of these 121 valid taxonomic genera were then binned by likely functional group where sufficient information existed. The pico and nanoflagellates were binned as phototrophic, heterotrophic or parasitic; potential mixotrophs were considered phototrophic to facilitate comparisons with the FCM data, which detects all chlorophyll containing cells. The dinoflagellates were binned separately since the trophic status of many dinoflagellates is not easily determined by phylogeny; they may be heterotrophic, mixotrophic or phototrophic (Taylor 1976; Saldarriaga et al. 2004). Reads classified to the same final species level taxon (99% similarity excluding indels) were considered ribotypes. This approach ignores artifacts generated by 454 pyrosequencing errors that typically occur as indels and not substitutions (Balzer et al. 2010). Ribotypes with more than 100 representatives in a single sample, hereafter referred to as “abundant ribotypes”, were subjected to a separate Basic Local Alignment Search Tool (BLAST) search of the nr/nt database at the National Center for Biotechnology Information (NCBI) using the NCBI parsers from BioPython (Cock et al. 2009) and an in-house python (v2.7) script (https://gist.github.com/tpoisot/5613748). When using 98% similarity to determine OTUs, in mothur, there were few OTUs with more than 40 reads. However once the reads were aligned, indels ignored and the reads classed as ribotypes, we found that distinct ribotypes dominated the reads in each sample. Spearman’s Rank correlation tests were carried out in R (http:// www.R-project.org, version 3.1.2) and SigmaPlot (version 11) to determine significant associations among the functional categories, environmental variables and FCM data.
2.2.5 Environmental data, statistical and model analysis
As an indication of tidal level at the time of sampling, the water level above chart datum was obtained from Fisheries and Oceans Canada, (http://www.dfo-mpo.gc.ca). Meteorological data was collected from the Havre-aux-Maisons Airport, which is 4 km from the sampling site (Environment Canada; www.climat.meteo.gc.ca).